Область техники
Изобретение относится к биотехнологии, иммунологии и вирусологии. Оно касается рекомбинантных векторов, которые могут быть использованы в фармацевтической промышленности для разработки вакцин.
Уровень техники
Вакцинация считается одним из наиболее эффективных способов борьбы с инфекционными заболеваниями. Принимая во внимание, что широкая вакцинопрофилактика имеет не столь длительную историю применения, уже к настоящему моменту, считается доказанным факт, что она спасла больше жизней, чем любое другое вмешательство в области общественного здравоохранения. Согласно исследованию, опубликованному в журнале The Lancet, иммунизация населения только против 10 патогенов в 98 странах позволила в период 2000-2019 гг. спасти жизни около 37 млн людей.
Вирусные векторы представляют собой относительно новую вакцинную платформу, основанную на использовании рекомбинантных вирусов для доставки генетического материала, кодирующего антиген, в клетки млекопитающих. В настоящее время шесть вакцин на основе вирусных векторов уже разрешены к применению и еще более 100 находятся на разных стадиях доклинических и клинических исследований.
Одним из наиболее востребованных вирусных векторов является вектор на основе рекомбинантного аденовируса. К преимуществам данного типа векторов можно отнести высокую безопасность, широкий диапазон тканевого тропизма, хорошо охарактеризованный геном, высокую пакующую емкость, возможность получения препаратов с высокими титрами и т.д. После введения в организм млекопитающих аденовирусы индуцируют устойчивый Т-клеточный и гуморальный иммунный ответ к вакцинному антигену. Кроме того, данные векторы приводят к активации врожденного иммунитета и не требуют добавления адъюванта в вакцинную композицию.
Среди аденовирусных векторов наиболее изученным является вектор на основе аденовируса человека 5 серотипа.
Известно решение (CN1276777C), в котором предложена вакцина против тяжелого острого респираторного синдрома на основе рекомбинантного аденовируса человека 5 серотипа, содержащего последовательность S белка вируса SARS-CoV.
Известно решение по заявке на изобретение US20080267992A1, где описана вакцина против тяжелого острого респираторного синдрома на основе рекомбинантного аденовируса человека 5 серотипа, содержащего последовательность полного протективного антигена S вируса SARS-CoV, или последовательность, которая включает домен S1 антигена S вируса SARS-CoV или домен S2 антигена S вируса SARS-CoV, или оба домена. Кроме того, данный рекомбинантный аденовирус в составе экспрессионной кассеты содержит промотор цитомегаловируса человека (CMV-промотор) и сигнал полиаденилирования бычьего гормона роста (polyА BGH).
Известно решение CN111218459, в котором разработан экспрессионный вектор на основе аденовируса человека 5 серотипа, в котором были удалены Е1, Е3 области, содержащий ген S белка. Данный вектор применяется для создания вакцины против COVID-19.
Однако ограничением к широкому применению векторов на основе аденовируса человека 5 серотипа является наличие у части населения предсуществующего иммунного ответа. В связи с этим актуальным направлением является разработка различных векторов, отличающихся генетически, в том числе на основе аденовирусов других серотипов. Однако из более 80 известных серотипов аденовирусов человека в настоящий момент в качестве экспрессионных векторов используется меньше 10 серотипов.
Таким образом, в области техники существует потребность в разработке новых экспрессионных векторов на основе других серотипов аденовируса.
Осуществление изобретения
Технической задачей заявленной группы изобретений является расширение арсенала экспрессионных векторов на основе аденовирусов человека.
Технический результат заключается в создании экспрессионного вектора на основе аденовируса человека 19 серотипа, который способен индуцировать иммунный ответ к вакцинному антигену.
Указанный технический результат достигается тем, что создан экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий: константную последовательность с делецией Е1 и Е3областей, внутри которой находится экспрессионная кассета с целевым геном; вариабельный участок на правом конце генома размером до 1000 п.о.
В частном варианте исполнения экспрессионного вектора константная последовательность до экспрессионной кассеты имеет последовательность SEQ ID NO:1, а после экспрессионной кассеты имеет последовательность SEQ ID NO:2.
Также в частном варианте исполнения экспрессионного вектора экспрессионная кассета в качестве гена вакцинного антигена содержит ген S белка вируса SARS-CoV2 SEQ ID NO:3.
В другом варианте исполнения экспрессионного вектора экспрессионная кассета в качестве гена вакцинного антигена содержит ген гемагглютинина вируса гриппа А H1N1 SEQ ID NO:4.
Кроме того, представлен вариант исполнения экспрессионного вектора, в котором экспрессионная кассета в качестве гена вакцинного антигена содержит ген гемагглютинина вируса гриппа А H3N2 SEQ ID NO:5.
Кроме того, представлен вариант исполнения экспрессионного вектора, в котором экспрессионная кассета в качестве гена вакцинного антигена содержит ген гемагглютинина вируса гриппа B/Yamagata SEQ ID NO:6.
Кроме того, представлен вариант исполнения экспрессионного вектора, в котором экспрессионная кассета в качестве гена вакцинного антигена содержит ген гемагглютинина вируса гриппа B/Victoria SEQIDNO:7.
Указанный технический результат также достигается тем, что разработан способ получения экспрессионного вектора, отличающийся тем, что в нем используется челночная плазмида, содержащая последовательность, которая гомологична участку генома аденовируса, серотип которого отличается от 19.
Также технический результат достигается тем, что разработан способ применения экспрессионного вектора для индукции иммунного ответа к вакцинному антигену.
Краткое описание фигур
На фиг. 1
представлено схематичное изображение экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего константную последовательность с делецией Е1 и Е3областей,вариабельный участок на правом конце генома размером до 1000 п.о. и экспрессионную кассету с целевым геном.
На фиг.2 представлена схема получения плазмиды pAd19-dlE1.
На фиг.3 представлена схема получения плазмиды pAd19-dlE1-dlE3.
На фиг.4 представлена схема получения плазмиды pAd19-dlE1-dlE3-S-nCov.
На фиг. 5 представлена электрофореграмма результатов ПЦР-анализа полученного экспрессионного вектора Ad19-S-nCov с праймерами, фланкирующими участок гена гексона Ad19 и праймерами, фланкирующими ген Sбелка SARS-CoV2.
1 - Маркер молекулярного веса;
2 -Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-S-nCov.
На фиг. 6 представлена электрофореграмма результатов ПЦР-анализа полученных вариантов экспрессионного вектора Ad19-HA-H1, Ad19-HA-H3, Ad19-HA-B(Victoria), Ad19-HA-B(Yamagata) с праймерами, фланкирующими участок гена гексона Ad19.
1 - Маркер молекулярного веса;
2 - Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-HA-H1;
6 - Ad19-HA-H3;
7 - Ad19-HA-B(Victoria);
8 -Ad19-HA-B(Yamagata).
На фиг. 7 представлена электрофореграмма результатов ПЦР-анализа экспрессионного вектора Ad19-HA-H1 с праймерами, фланкирующими участок гена гемагглютинина вируса гриппа А H1N1.
1 - Маркер молекулярного веса;
2 - Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-HA-H1.
На фиг. 8 представлена электрофореграмма результатов ПЦР-анализа экспрессионного вектора Ad19-HA-H3 с праймерами, фланкирующими участок гена гемагглютинина вируса гриппа А H3N2.
1 - Маркер молекулярного веса;
2 - Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-HA-H3.
На фиг. 9 представлена электрофореграмма результатов ПЦР-анализа экспрессионного вектора Ad19-HA-B(Yamagata) с праймерами, фланкирующими участок гена гемагглютинина вируса гриппа BYamagata.
1 - Маркер молекулярного веса;
2 - Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-HA-B(Yamagata).
На фиг. 10 представлена электрофореграмма результатов ПЦР-анализа экспрессионного вектора Ad19-HA-B(Victoria) с праймерами, фланкирующими участок гена гемагглютинина вируса гриппа BVictoria.
1 - Маркер молекулярного веса;
2 - Отрицательный контроль;
3 - Положительный контроль;
4 - Контроль выделения;
5 - Ad19-HA-B(Victoria).
Краткое описание нуклеотидных последовательностей:
SEQIDNO:1 последовательность константной последовательности разработанного экспрессионного вектора до экспрессионной кассеты.
SEQIDNO:2 последовательность константной последовательности разработанного экспрессионного вектора после экспрессионной кассеты.
SEQIDNO:3 последовательность гена S белка вируса SARS-CoV-2;
SEQIDNO:4 последовательность гена гемагглютинина вируса гриппа А H1N1;
SEQIDNO:5 последовательность гена гемагглютинина вируса гриппа А H3N2;
SEQIDNO:6 последовательность гена гемагглютинина вируса гриппа BYamagata;
SEQIDNO:7 последовательность гена гемагглютинина вируса гриппа BVictoria;
Реализация изобретения
Авторы изобретения на основании проведенных исследований определили серотипы аденовирусных векторов, которые настолько отличаются генетически от аденовируса человека 5 серотипа, чтобы при последовательной иммунизации не влиять на генерацию антиген-специфических иммунных ответов против вакцинного антигена. Для дальнейшей работы был выбран аденовирус человека 19 серотипа. Затем был отобран вирусный клон, отличающийся повышенными ростовыми свойствами, который в дальнейшем был использован для получения экспрессионного вектора.
Способ получения данного экспрессионного вектора основан на способе получения аденовирусных векторов, описанном в патенте RU 2 731 356 C9, и включает следующие этапы: получение экспрессионной кассеты с целевым геном; получение челночной плазмиды, содержащей экспрессионную кассету с целевым геном; получение плазмиды, содержащей аденовирусный геном и экспрессионную кассету с целевым геном; получение рекомбинантного аденовируса. Главной отличительной особенностью нового метода является использование челночной плазмиды, содержащей области (плечи гомологии), которые только частично гомологичны геному аденовируса человека 19 серотипа. Во время гомологичной рекомбинации между челночной плазмидой и плазмидой, содержащей геном аденовируса человека 19 серотипа с делецией Е1 и Е3 областей, образуется новая плазмида. При этом в последовательностьна правом конце генома аденовируса может попасть последовательность (разного размера) из челночной плазмиды, т.к. гомология в данной области ниже 100%. У всех вариантов разработанного экспрессионного вектора данная область немного отличается, поэтому она была названа «вариабельный участок». Схематичное изображение разработанного экспрессионного вектора представлено на фиг.1.
Использование челночной плазмиды с плечами гомологии аденовируса человека 26 серотипа для получения экспрессионного вектора на основе аденовируса человека 19 дает преимущество в скорости получения экспрессионного вектора и снижает его себестоимость.
В приведенных ниже примерах проиллюстрировано получение вариантов экспрессионного вектора, в котором экспрессионная кассета в качестве целевого гена содержит генS белка вируса SARS-CoV-2 или гемагглютинин вируса гриппа. Результаты проведенных экспериментов показывают, что при введении данных вариантов экспрессионного вектора в организм млекопитающих формируется иммунный ответ против соответствующего антигена. Поэтому различные варианты экспрессионного вектора могут быть использованы в составе иммунобиологических средств для профилактики инфекционных заболеваний.
Осуществление изобретения подтверждается следующими примерами.
Пример 1. Получение плазмиды, содержащей геном аденовируса человека 19 серотипа с делецией Е1 и Е3 областей.
В работе использовали аденовирус человека 19-го серотипа из Государственной коллекции вирусов ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России. Данный аденовирус добавляли в культуру клеток почки человеческого эмбриона(НЕК293). Далее клетки культивировали в инкубаторе при температуре 37°С, 5% СО2 до наступления цитопатического действия (определяли путем микроскопирования). Затем аденовирус выделяли из клеток и концентрировали методом ультрацентрифугирования в градиенте хлористого цезия, после чего выделяли вирусную ДНК из вирионов методом фенол-хлороформной экстракции. Нуклеотидная последовательность аденовируса человека 19-го серотипа была определена методом полногеномного секвенирования.
ДНК, выделенную из вирионов аденовируса человека 19-го серотипа, использовали в реакции гомологичной рекомбинации с плазмидой pAd26-Ends (получена в ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России), линеаризованной с помощью эндонуклеазы рестрикции MluI. Плазмида pAd26-Ends содержит два участка, гомологичных геному аденовируса человека 26 серотипа (два плеча гомологии). Одно плечо гомологии представляет собой начало генома аденовируса человека 26-го серотипа (от левого инвертированного концевого повтора до Е1-области) и последовательность вирусного генома, включающую pIX белок. Второе плечо гомологии содержит последовательность нуклеотидов после ORF3 Е4 области до конца генома. Далее проводили трансформацию клеток E.coli штамма BJ5183полученной смесью. В результате была получена плазмида pAd19-dlE1.
На следующем этапе с помощью стандартных генно-инженерных методов была получена плазмида р19-dlE3, которая представляет собой челночный вектор, содержащий два плеча гомологии, фланкирующих Е3 область. Далее проводили гомологичную рекомбинацию плазмиды р19-dlE3 и плазмиды pAd19-dlE1, полученной на предыдущем этапе. Для этого обе плазмиды были линеаризованы по уникальным сайтам рестрикции и смешаны. После чего проводили трансформацию клеток E.coli штамма BJ5183полученной смесью.
В результате проведенной работы была получена pAd19-dlE1-dlE3, содержащая геном аденовируса человека 19 серотипа с делецией Е1 и Е3 областей.
Пример 2. Способ получения экспрессионного вектора на основе генома аденовируса человека 19 серотипа.
Способ получения экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего константную последовательность с делецией Е1 и Е3областей,внутри которой находится экспрессионная кассета с геном вакцинного антигена, и вариабельный участок на правом конце генома размером до 1000 п.о. состоит из следующих этапов.
Этап 1. Получение экспрессионной кассеты.
На первом этапе необходимо было разработать дизайн экспрессионной кассеты, которая содержит промотор, целевой ген и сигнал полиаденилирования. Кроме того, в состав экспрессионной кассеты может входить полилинкерная последовательность, необходимая для генно-инженерных манипуляций. В качестве промотора авторы использовали промотор цитомегаловируса человека (CMV). Однако, специалисту среднего уровня очевидно, что в качестве промотора в данной экспрессионной кассете может быть использован любой из известных промоторов, обеспечивающих экспрессию целевого гена.
В качестве целевого гена может быть использована последовательность любого вакцинного антигена, в том числе модифицированная. В данном примере авторы использовали оптимизированные последовательности гена S белка вируса SARS-CoV-2 (SEQIDNO:3), гена гемагглютинина вируса гриппа А H1N1(SEQIDNO:4), гена гемагглютинина вируса гриппа А H3N2(SEQIDNO:5), гена гемагглютинина вируса гриппа B/Yamagata(SEQIDNO:6) и гена гемагглютинина вируса гриппа B/Victoria(SEQIDNO:7).
В качестве сигнала полиаденилирования в данных примерах был использован сигнал позднего полиаденилирования SV40.
Фрагменты экспрессионной кассеты были синтезированы ЗАО «Евроген» и собраны в ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» с помощью стандартных генно-инженерных методик. Таким образом, были получены следующие экспрессионные кассеты:
1) Экспрессионная кассета, которая в качестве целевого гена содержит генS белка вируса SARS-CoV-2 SEQIDNO:3.
2) Экспрессионная кассета, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа А H1N1 SEQIDNO:4.
3) Экспрессионная кассета, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа А H3N2 SEQIDNO:5.
4) Экспрессионная кассета, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа B/Yamagata SEQIDNO:6.
5) Экспрессионная кассета, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа B/Victoria SEQIDNO:7.
Этап 2. Получение челночной плазмиды, содержащей экспрессионную кассету.
В работе использовали плазмиду pAd26-Ends (получена в ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России),которая содержит участки, гомологичные геному аденовируса человека 26 серотипа.
На основе плазмиды pAd26-Ends генно-инженерным методом были получены конструкции, содержащие экспрессионные кассеты, полученные на предыдущем этапе, а также несущие участки, гомологичные геному аденовируса человека 26 серотипа. Таким образом, были получены плазмиды:
1) pArms-26-S-nCoVс экспрессионной кассетой, которая в качестве целевого гена содержит генS белка вируса SARS-CoV-2 SEQIDNO:3.
2) pArms-26-HA-H1с экспрессионной кассетой, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа А H1N1 SEQIDNO:4.
3) pArms-26-HA-H3с экспрессионной кассетой, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа А H3N2 SEQIDNO:5.
4) pArms-26-HA-B(Yamagata)с экспрессионной кассетой, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа B/Yamagata SEQIDNO:6.
5) pArms-26-HA-B(Victoria)с экспрессионной кассетой, которая в качестве целевого гена содержит ген гемагглютинина вируса гриппа B/Victoria SEQIDNO:7.
Этап 3. Получение плазмиды, содержащей геном аденовируса 19 серотипа и экспрессионную кассету с целевым геном.
Конструкцииp Arms-26-S-nCoV, pArms-26-HA-H1, pArms-26-HA-H3, pArms-26-HA-B(Yamagata), pArms-26-HA-B(Victoria), полученные на предыдущем этапе, линеаризовали по уникальному сайту гидролиза между плечами гомологии. Затем каждую плазмиду смешивали с лианеризованной плазмидой pAd19-dlE1-dlE3. В результате гомологичной рекомбинации были получены плазмиды pAd19-dlE1-dlE3-S-nCoV, pAd19-dlE1-dlE3-HA-H1, pAd19-dlE1-dlE3-HA-H3, pAd19-dlE1-dlE3-HA-B(Yamagata),pAd19-dlE1-dlE3-HA-B(Victoria). Данные плазмиды помимо экспрессионной кассеты несут геном аденовируса человека 19 серотипа с делецией E1 и Е3-областей. Однако за счет того, что рекомбинация каждый раз происходила между двумя плазмидами, содержащими плечи гомологии от аденовирусов разных серотипов, образовалась вариабельная область. В данную область может попасть последовательность (разного размера) из челночной плазмиды, т.к. гомология в данной области ниже 100%.При получении каждого экспрессионного вектора с новым целевым геном данная область может меняться.
Этап 4. Получение рекомбинантного аденовируса.
На последнем этапе плазмиды pAd19-dlE1-dlE3-S-nCoV, pAd19-dlE1-dlE3-HA-H1, pAd19-dlE1-dlE3-HA-H3, pAd19-dlE1-dlE3-HA-B(Yamagata),pAd19-dlE1-dlE3-HA-B(Victoria) гидролизовали специфическими эндонуклеазами рестрикции для удаления векторной части. Полученными препаратами ДНК трансфицировали клетки культуры НЕК293.
В результате был получен экспрессионный вектор, на основе генома аденовируса человека 19 серотипа, содержащий константную последовательность с делецией Е1 и Е3 областей (SEQIDNO:1), вариабельный участок на правом конце генома размером до 1000 п.о. и экспрессионную кассету с целевым геном S белка вируса SARS-CoV-2 SEQ ID NO:3 (Ad19-S-nCoV), или геном гемагглютинина вируса гриппа А H1N1 SEQ ID NO:4 (Ad19-HA-H1), или геном гемагглютинина вируса гриппа А H3N2 SEQ ID NO:5 (Ad19-HA-H3), или геном гемагглютинина вируса гриппа B/Yamagata SEQ ID NO:6 (Ad19-HA-B(Yamagata)), или геном гемагглютинина вируса гриппа B/Victoria SEQ ID NO:7 (Ad19-HA-B(Victoria)).
Пример 3. Оценка подлинности полученных вариантов разработанного экспрессионного вектора.
Для проверки подлинности полученных вариантов разработанного экспрессионного вектора использовали метод ПЦР. Для каждого образца вируса использовали праймеры, комплементарные фрагменту генома аденовируса, и уникальные праймеры на целевой ген:
1) образец ДНК: Ad19-S-nCoV
праймеры, фланкирующие участок векторной части экспрессионного вектора:
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
праймеры, фланкирующие участок гена Sбелка SARS-CoV-2 и сигнала полиаденилированияSV40:
Nov-S-F AAGAATTTCACCACCGCTCCA
SV40-R GTGGTATGGCTGATTATGATC
Результаты ПЦР анализа представлены на фиг.5.
2) образец ДНК: Ad19-HA-H1
праймеры, фланкирующие участок векторной части экспрессионного вектора:
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
праймеры, фланкирующие участок гена гемагглютинина вируса гриппа гемагглютинина вируса гриппа H1N1:
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
H1-прямой GGGCATTCACCATCCTCC
H1-обратный CAGGTTTCCAGTTGCTTCG
Результаты ПЦР анализа представлены на фиг.6-7.
3) Образец ДНК: Ad19-HA-H3
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
H3-прямой: TCCCGAGAAACAGACCCG
H3-обратный: GCCGATTCAGCTTCCCAT
Результаты ПЦР анализа представлены на фиг.6-8.
4) Образец ДНК: Ad19-HA-B(Yamagata)
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
B-PH-прямой: CTTGGACAGGATTGCTGCA
B-PH-обратный: TCTGCTCACCATGTAGACG
Результаты ПЦР анализа представлены на фиг.6-9.
5) Ad19-HA-B(Victoria)
hexon19-прямой: AGAAACAGCCGATAATCCTCC
hexon19-обратный: ATCGATATTGTCACCAGTTTTAAG
B-aus-прямой: TCTGCGGGCTGATACAATAAG
B-aus-обратный: GCTAAATTCTCCTGCATCAAAG
Результаты ПЦР анализа представлены на фиг.5.
Для проведения ПЦР использовали набор 5X Screen Mix-HS (Евроген, Россия). Готовили реакционную смесь из расчета для 1 пробы: 5-кратная смесь (Евроген, Россия) 4 мкл, по 0,1 мкл каждого праймера, 14,8 мкл деионизированной воды, 1 мкл образца ДНК. ПЦР проводили в амплификаторе Bio-Rad T100 Thermal Cycler (Bio-Rad Laboratories, США). Продукты амплификации анализировали в горизонтальном гель электрофорезе с помощью системы детектирования гелей Gel-Doc EZ Imager (Bio-Rad, США). Для определения молекулярного веса фрагментов ДНК использовали маркер молекулярного веса 1 kb DNA Ladder (Евроген, Россия).
Как видно из полученных данных в каждом экспериментальном образце были обнаружена ДНК аденовируса человека 19 серотипа и целевой ген.
Таким образом, результаты эксперимента подтверждают получение экспрессионных векторов на основе генома аденовируса человека 19 серотипа, содержащего константную последовательность с делецией Е1 и Е3 областей, вариабельный участок на правом конце генома размером до 1000 п.о. и экспрессионную кассету с целевым геном.
Пример 4. Оценка иммуногенности разработанного экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном S белка вируса SARS;вариабельный участок на правом конце генома размером до 1000 п.о.
Целью данного эксперимента являлось определение способности разработанного вектора на основе аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном S белка вируса SARS; вариабельный участок на правом конце генома размером до 1000 п.о. (Ad19-S-nCov), индуцировать развитие гуморального иммунного ответа у млекопитающих.
В примере представлены данные, касающиеся изменения титра антител против Sбелка вируса SARS через 21 день после однократной внутримышечной иммунизации животных Ad19-S-nCov мышей линии C57BL/6, самки 18г. Все животные были разделены на 7 групп по 6 животных, которым внутримышечно вводили:
1) Ad19-S-nCov108БОЕ/мышь
2) Ad19-S-nCov 109БОЕ/мышь
3) Ad19-S-nCov 1010БОЕ/мышь
4) Ad19-null 108БОЕ/мышь
5) Ad19-null 109БОЕ/мышь
6) Ad19-null 1010БОЕ/мышь
7) фосфатно-солевой буфер
Через три недели у животных отбирали кровь из хвостовой вены и выделяли сыворотку крови. Титр антител определяли методом иммуноферментного анализа (ИФА) по следующему протоколу:
1) Белок (S) адсорбировали на лунках 96-луночного планшета для ИФА в течение 16 часов при температуре +4°С.
2) Далее для избавления от неспецифического связывания осуществилась "забивка" планшета 5% молоком, растворенном в TPBS в объеме 100 мкл на лунку. Инкубировали на шейкере при температуре 37°С на протяжении часа.
3) Методом 2-кратных разведений разводили образцы сыворотки иммунизированных мышей. Всего было приготовлено 12 разведений каждого образца.
4) Добавляли по 50 мкл каждого разведенного образца сыворотки в лунки планшета.
5) Далее проводили инкубацию в течение 1 часа при 37°С.
6) После инкубации проводилась трехкратная промывка лунок фосфатным буфером.
7) Затем добавляли вторичные антитела против иммуноглобулинов мыши, конъюгированные с пероксидазой хрена.
8) Далее проводили инкубацию в течение 1 часа при 37°С.
9) После инкубации проводилась трехкратная промывка лунок фосфатным буфером
10) Затем добавили раствор тетраметилбензидина (ТМВ), который является субстратом пероксидазы хрена и в результате реакции превращается в окрашенное соединение. Реакцию останавливали через 15 минут добавлением серной кислоты. Далее с помощью спектрофотометра измеряли оптическую плотность раствора (OD) в каждой лунке при длине волны 450 нм.
Титр антител определяли, как последнее разведение, в котором оптическая плотность раствора была достоверно выше, чем в группе отрицательного контроля. Полученные результаты (среднее геометрическое значение) представлены в таблице 1.
Таблица 1 - Титр антител к белку S в сыворотке крови мышей (среднее геометрическое значение титра антител).
Таблица 1.
Результаты эксперимента показали, что разработанный экспрессионный вектор, на основе генома аденовируса человека 19 серотипа, содержащего константную последовательность с делецией Е1 и Е3 областей, вариабельный участок на правом конце генома размером до 1000 п.о. и экспрессионную кассету с геном S белка вируса SARS-CoV2, введенный в организм млекопитающего, индуцирует гуморальный иммунный ответ к Sбелку SARS-CoV-2 во всем диапазоне выбранных доз. При этом, очевидно, что увеличение доз будет приводить к увеличению титра антител в крови млекопитающих до наступления токсического эффекта.
Пример 5. Оценка иммуногенности разработанного экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа А H1N1; вариабельный участок на правом конце генома размером до 1000 п.о.
Целью данного эксперимента являлось определение способности разработанного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа А H1N1; вариабельный участок на правом конце генома размером до 1000 п.о.(Ad19-HA-H1), индуцировать развитие гуморального иммунного ответа у млекопитающих.
В примере представлены данные, касающиеся изменения титра антител против гемагглютинина вируса гриппа А H1N1 через 21 день после однократной внутримышечной иммунизации животных Ad19-HA-H1мышей линии C57BL/6, самки 18г. Все животные были разделены на 7 групп по 6 животных, которым внутримышечно вводили:
1) Ad19-HA-H1,108БОЕ/мышь
2) Ad19-HA-H1, 109БОЕ/мышь
3) Ad19-HA-H1, 1010БОЕ/мышь
4) Ad19-null, 108БОЕ/мышь
5) Ad19-null, 109БОЕ/мышь
6) Ad19-null, 1010БОЕ/мышь
7) фосфатно-солевой буфер
Через три недели у животных отбирали кровь из хвостовой вены и выделяли сыворотку крови. Титр антител определяли методом иммуноферментного анализа (ИФА) по следующему протоколу:
1) Гемагглютинин вируса гриппа А H1N1 адсорбировали на лунках 96-луночного планшета для ИФА в течение 16 часов при температуре +4°С.
2) Далее для избавления от неспецифического связывания осуществилась "забивка" планшета 5% молоком, растворенном в TPBS в объеме 100 мкл на лунку. Инкубировали на шейкере при температуре 37°С на протяжении часа.
3) Методом 2-кратных разведений разводили образцы сыворотки иммунизированных мышей. Всего было приготовлено 12 разведений каждого образца.
4) Добавляли по 50 мкл каждого разведенного образца сыворотки в лунки планшета.
5) Далее проводили инкубацию в течение 1 часа при 37°С.
6) После инкубации проводилась трехкратная промывка лунок фосфатным буфером.
7) Затем добавляли вторичные антитела против иммуноглобулинов мыши, конъюгированные с пероксидазой хрена.
8) Далее проводили инкубацию в течение 1 часа при 37°С.
9) После инкубации проводилась трехкратная промывка лунок фосфатным буфером
10) Затем добавили раствор тетраметилбензидина (ТМВ), который является субстратом пероксидазы хрена и в результате реакции превращается в окрашенное соединение. Реакцию останавливали через 15 минут добавлением серной кислоты. Далее с помощью спектрофотометра измеряли оптическую плотность раствора (OD) в каждой лунке при длине волны 450 нм.
Титр антител определяли, как последнее разведение, в котором оптическая плотность раствора была достоверно выше, чем в группе отрицательного контроля. Полученные результаты (среднее геометрическое значение) представлены в таблице 2.
Таблица 2 - Титр антител кгемагглютинину вируса гриппа А H1N1 в сыворотке крови мышей (среднее геометрическое значение титра антител).
Таблица 2.
Результаты эксперимента показали, что разработанный экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа А H1N1, и вариабельный участок на правом конце генома размером до 1000 п.о., введенный в организм млекопитающего, индуцирует гуморальный иммунный ответ к гемагглютинину вируса гриппа А H1N1 во всем диапазоне выбранных доз. При этом очевидно, что увеличение доз будет приводить к увеличению титра антител в крови млекопитающих до наступления токсического эффекта.
Пример 6. Оценка иммуногенности разработанного экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа А H3N2; вариабельный участок на правом конце генома размером до 1000 п.о.
Целью данного эксперимента являлось определение способности разработанного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа А H3N2; вариабельный участок на правом конце генома размером до 1000 п.о.(Ad19-HA-H3), индуцировать развитие гуморального иммунного ответа у млекопитающих.
В примере представлены данные, касающиеся изменения титра антител против гемагглютинина вируса гриппа А H3N2 через 21 день после однократной внутримышечной иммунизации животных экспрессионным вектором Ad19-HA-H3мышей линии C57BL/6, самки 18г. Все животные были разделены на 7 групп по 5 животных, которым внутримышечно вводили:
1) Ad19-HA-H3,108БОЕ/мышь
2) Ad19-HA-H3,109БОЕ/мышь
3) Ad19-HA-H3, 1010БОЕ/мышь
4) Ad19-null, 108БОЕ/мышь
5) Ad19-null, 109БОЕ/мышь
6) Ad19-null, 1010БОЕ/мышь
7) фосфатно-солевой буфер
Через три недели у животных отбирали кровь из хвостовой вены и выделяли сыворотку крови. Титр антител определяли методом иммуноферментного анализа (ИФА) по следующему протоколу:
1) Гемагглютинин вируса гриппа А H3N2 адсорбировали на лунках 96-луночного планшета для ИФА в течение 16 часов при температуре +4°С.
2) Далее для избавления от неспецифического связывания осуществилась "забивка" планшета 5% молоком, растворенном в TPBS в объеме 100 мкл на лунку. Инкубировали на шейкере при температуре 37°С на протяжении часа.
3) Методом 2-хкратных разведений разводили образцы сыворотки иммунизированных мышей. Всего было приготовлено 12 разведений каждого образца.
4) Добавляли по 50 мкл каждого разведенного образца сыворотки в лунки планшета.
5) Далее проводили инкубацию в течение 1 часа при 37°С.
6) После инкубации проводилась трехкратная промывка лунок фосфатным буфером.
7) Затем добавляли вторичные антитела против иммуноглобулинов мыши, конъюгированные с пероксидазой хрена.
8) Далее проводили инкубацию в течение 1 часа при 37°С.
9) После инкубации проводилась трехкратная промывка лунок фосфатным буфером
10) Затем добавили раствор тетраметилбензидина (ТМВ), который является субстратом пероксидазы хрена и в результате реакции превращается в окрашенное соединение. Реакцию останавливали через 15 минут добавлением серной кислоты. Далее с помощью спектрофотометра измеряли оптическую плотность раствора (OD) в каждой лунке при длине волны 450 нм.
Титр антител определяли, как последнее разведение, в котором оптическая плотность раствора была достоверно выше, чем в группе отрицательного контроля. Полученные результаты (среднее геометрическое значение) представлены в таблице 3.
Таблица 3 - Титр антител к гемагглютинину вируса гриппа А H3N2 в сыворотке крови мышей (среднее геометрическое значение титра антител).
Таблица 3.
Результаты эксперимента показали, что разработанный экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий константную последовательность с делецией Е1 и Е3областей, внутри которой находится экспрессионная кассетас геном гемагглютинина вируса гриппа А H3N2, и вариабельный участок на правом конце генома размером до 1000 п.о., введенный в организм млекопитающего, индуцирует гуморальный иммунный ответ к гемагглютинину вируса гриппа А H3N2 во всем диапазоне выбранных доз. При этом очевидно, что увеличение доз будет приводить к увеличению титра антител в крови млекопитающих до наступления токсического эффекта.
Пример 7. Оценка иммуногенности разработанного экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BYamagata; вариабельный участок на правом конце генома размером до 1000 п.о.
Целью данного эксперимента являлось определение способности разработанного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BYamagata; вариабельный участок на правом конце генома размером до 1000 п.о. (Ad19-HA-B(Yamagata)), индуцировать развитие гуморального иммунного ответа у млекопитающих.
В примере представлены данные, касающиеся изменения титра антител против гемагглютинина вируса гриппа B/Yamagataчерез 21 день после однократной внутримышечной иммунизации животных экспрессионным вектором Ad19-HA-B(Yamagata)мышей линии C57BL/6, самки 18г. Все животные были разделены на 7 групп по 6 животных, которым внутримышечно вводили:
1) Ad19-HA-B(Yamagata),108БОЕ/мышь
2) Ad19-HA-B(Yamagata),109БОЕ/мышь
3) Ad19-HA-B(Yamagata), 1010БОЕ/мышь
4) Ad19-null, 108БОЕ/мышь
5) Ad19-null, 109БОЕ/мышь
6) Ad19-null, 1010БОЕ/мышь
7) фосфатно-солевой буфер
Через три недели у животных отбирали кровь из хвостовой вены и выделяли сыворотку крови. Титр антител определяли методом иммуноферментного анализа (ИФА) по следующему протоколу:
1) Гемагглютинин вируса гриппа B/Yamagata адсорбировали на лунках 96-луночного планшета для ИФА в течение 16 часов при температуре +4°С.
2) Далее для избавления от неспецифического связывания осуществилась "забивка" планшета 5% молоком, растворенном в TPBS в объеме 100 мкл на лунку. Инкубировали на шейкере при температуре 37°С на протяжении часа.
3) Методом 2-хкратных разведений разводили образцы сыворотки иммунизированных мышей. Всего было приготовлено 12 разведений каждого образца.
4) Добавляли по 50 мкл каждого разведенного образца сыворотки в лунки планшета.
5) Далее проводили инкубацию в течение 1 часа при 37°С.
6) После инкубации проводилась трехкратная промывка лунок фосфатным буфером.
7) Затем добавляли вторичные антитела против иммуноглобулинов мыши, конъюгированные с пероксидазой хрена.
8) Далее проводили инкубацию в течение 1 часа при 37°С.
9) После инкубации проводилась трехкратная промывка лунок фосфатным буфером
10) Затем добавили раствор тетраметилбензидина (ТМВ), который является субстратом пероксидазы хрена и в результате реакции превращается в окрашенное соединение. Реакцию останавливали через 15 минут добавлением серной кислоты. Далее с помощью спектрофотометра измеряли оптическую плотность раствора (OD) в каждой лунке при длине волны 450 нм.
Титр антител определяли, как последнее разведение, в котором оптическая плотность раствора была достоверно выше, чем в группе отрицательного контроля. Полученные результаты (среднее геометрическое значение) представлены в таблице 4.
Таблица 4 - Титр антител к гемагглютинину вируса гриппа B/Yamagataв сыворотке крови мышей (среднее геометрическое значение титра антител).
Таблица 4.
Результаты эксперимента показали, что разработанный экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BYamagata, и вариабельный участок на правом конце генома размером до 1000 п.о., введенный в организм млекопитающего, индуцирует гуморальный иммунный ответ к гемагглютинину вируса гриппа BYamagata во всем диапазоне выбранных доз. При этом очевидно, что увеличение доз будет приводить к увеличению титра антител в крови млекопитающих до наступления токсического эффекта.
Пример 8. Оценка иммуногенности разработанного экспрессионного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BVictoria; вариабельный участок на правом конце генома размером до 1000 п.о.
Целью данного эксперимента являлось определение способности разработанного вектора на основе генома аденовируса человека 19 серотипа, содержащего: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BVictoria; вариабельный участок на правом конце генома размером до 1000 п.о. (Ad19-HA-B(Victoria)), индуцировать развитие гуморального иммунного ответа у млекопитающих.
В примере представлены данные, касающиеся изменения титра антител против гемагглютинина вируса гриппа B/Victoria через 21 день после однократной внутримышечной иммунизации животных экспрессионным вектором Ad19-HA-B (Victoria) мышей линии C57BL/6, самки 18г. Все животные были разделены на 7 групп по 6 животных, которым внутримышечно вводили:
1) Ad19-HA-B(Victoria),108БОЕ/мышь
2) Ad19-HA-B(Victoria),109БОЕ/мышь
3) Ad19-HA-B(Victoria), 1010БОЕ/мышь
4) Ad19-null, 108БОЕ/мышь
5) Ad19-null, 109БОЕ/мышь
6) Ad19-null, 1010БОЕ/мышь
7) фосфатно-солевой буфер
Через три недели у животных отбирали кровь из хвостовой вены и выделяли сыворотку крови. Титр антител определяли методом иммуноферментного анализа (ИФА) по следующему протоколу:
1) Гемагглютинин вируса гриппа B/Victoriaадсорбировали на лунках 96-луночного планшета для ИФА в течение 16 часов при температуре +4°С.
2) Далее для избавления от неспецифического связывания осуществилась "забивка" планшета 5% молоком, растворенном в TPBS в объеме 100 мкл на лунку. Инкубировали на шейкере при температуре 37°С на протяжении часа.
3) Методом 2-хкратных разведений разводили образцы сыворотки иммунизированных мышей. Всего было приготовлено 12 разведений каждого образца.
4) Добавляли по 50 мкл каждого разведенного образца сыворотки в лунки планшета.
5) Далее проводили инкубацию в течение 1 часа при 37°С.
6) После инкубации проводилась трехкратная промывка лунок фосфатным буфером.
7) Затем добавляли вторичные антитела против иммуноглобулинов мыши, конъюгированные с пероксидазой хрена.
8) Далее проводили инкубацию в течение 1 часа при 37°С.
9) После инкубации проводилась трехкратная промывка лунок фосфатным буфером
10) Затем добавили раствор тетраметилбензидина (ТМВ), который является субстратом пероксидазы хрена и в результате реакции превращается в окрашенное соединение. Реакцию останавливали через 15 минут добавлением серной кислоты. Далее с помощью спектрофотометра измеряли оптическую плотность раствора (OD) в каждой лунке при длине волны 450 нм.
Титр антител определяли, как последнее разведение, в котором оптическая плотность раствора была достоверно выше, чем в группе отрицательного контроля. Полученные результаты (среднее геометрическое значение) представлены в таблице 5.
Таблица 5 - Титр антител к гемагглютинину вируса гриппа B/Victoriaв сыворотке крови мышей (среднее геометрическое значение титра антител).
Таблица 5.
Результаты эксперимента показали, что разработанный экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с геном гемагглютинина вируса гриппа BVictoria, и вариабельный участок на правом конце генома размером до 1000 п.о., введенный в организм млекопитающего, индуцирует гуморальный иммунный ответ к гемагглютинину вируса гриппа BVictoria во всем диапазоне выбранных доз. При этом очевидно, что увеличение доз будет приводить к увеличению титра антител в крови млекопитающих до наступления токсического эффекта.
Пример 9. Проверка безопасности разработанных вариантов экспрессионного вектора на основе аденовируса человека 19 серотипа.
Целью данного эксперимента являлась проверка токсичности разработанного экспрессионного вектора на основе аденовируса человека 19 серотипа при однократном внутримышечном введении (острая токсичность) на мышах.
В исследовании были использованы аутбредные мыши, обоих полов, массой 18-20 грамм, возрастом 6-8 недель.
Минимальной дозой для токсикологических экспериментов была выбрана доза для мыши 108в.ч., как наиболее близкая к терапевтической. Для пересчета доз не использовался коэффициент межвидового пересчета, дозы получены путем прямого пересчета на массу тела, согласно рекомендация ВОЗ для вакцинных препаратов.
В результате, для введения мышам в данном эксперименте выбрали следующие дозы средства:
109в.ч. - увеличенная эффективная доза (ЭД) для мышей в 20 раз;
1010в.ч. - увеличенная ЭД для мышей в 200 раз;
1011в.ч. - увеличенная ЭД для мышей в 2000 раз;
Таким образом, были получены следующие экспериментальные группы животных:
1) Ad19-S-nCov,109в.ч./мышь, 20 мышей;
2) Ad19-S-nCov,1010в.ч./мышь, 20 мышей;
3) Ad19-S-nCov,1011в.ч./мышь, 20 мышей;
4) Ad19-HA-H1,109в.ч./мышь, 20 мышей;
5) Ad19-HA-H1,1010в.ч./мышь, 20 мышей;
6) Ad19-HA-H1,1011в.ч./мышь, 20 мышей;
7) Ad19-HA-H3,109в.ч./мышь, 20 мышей;
8) Ad19-HA-H3,1010в.ч./мышь, 20 мышей;
9) Ad19-HA-H3,1011в.ч./мышь, 20 мышей;
10) Ad19-HA-B(Yamagata),109в.ч./мышь, 20 мышей;
11) Ad19-HA-B(Yamagata),1010в.ч./мышь, 20 мышей;
12) Ad19-HA-B(Yamagata),1011в.ч./мышь, 20 мышей;
13) Ad19-HA-B(Victoria),109в.ч./мышь, 20 мышей;
14) Ad19-HA-B(Victoria),1010в.ч./мышь, 20 мышей;
15) Ad19-HA-B(Victoria),1011в.ч./мышь, 20 мышей;
16) Фосфатно-солевой буферный раствор, 20 мышей.
Клинический осмотр каждого животного проводили ежедневно в течение 14 дней, регистрируя признаки интоксикации и число павших животных.
Фиксировали следующие параметры функционального состояния лабораторных животных: активность, передвижение, внешний вид, состояние шерсти, глаз, ушей, зубов, конечностей. Физиологические функции: дыхание, слюноотделение, слюна, моча, экскрет. На протяжении эксперимента все животные оставались живы. Во всех группах животные выглядели здоровыми, активно поедали корм, адекватно реагировали на раздражители, проявляли исследовательский интерес. Шерстный покров густой, ровный и блестящий, плотно прилегал к поверхности тела, выпадения или ломкости шерсти не выявлено. Мышечный тонус не отличался повышенной возбудимостью. Ушные раковины без корок, не воспалены, подергиваний не замечено. Зубы обычного цвета, без поломок. Мыши были средней упитанности, истощением не страдали. Область живота в объеме не увеличена. Дыхание ровное, незатрудненное. Слюноотделение в норме. Частота мочеиспускания, цвет мочи, желудочно-кишечные показатели, мышечный тонус, рефлексы соответствовали физиологической норме. Поведение опытных животных не отличалось от контрольных.
На 14 сутки от начала эксперимента, осуществляли запланированную эвтаназию мышей методом дислокации шейных позвонков. В ходе проведения исследования животные в тяжелом состоянии с признаками неминуемой смерти не наблюдались, гибели животных не было.
Проводили полную некропсию тел всех животных. При некропсии исследовали внешнее состояние тела, внутренние поверхности и проходы, полость черепа, грудную, брюшную и тазовую полости с находящимися в них органами и тканями, шею с органами и тканями и скелетно-мышечную систему.
При макроскопическом исследовании не обнаружено влияния средства на состояние внутренних органов мышей, различий между контрольными и опытными группами не найдено. Cтатистически достоверных различий в массе органов между опытными и контрольной группами не обнаружено. Набор массы животных в опытных и контрольных группах не отличался.
Таким образом, в ходе проведенной работы определяли безопасность разработанных средств путем оценки острой токсичности. Исходя из полученных данных, можно заключить, что все исследованные варианты экспрессионного вектора на основе аденовируса человека 19 серотипа безопасны.
Промышленная применимость
Все приведенные примеры подтверждают способность разработанного экспрессионного вектора на основе аденовируса человека 19 серотипа индуцировать гуморальный ответ против целевого антигена и его промышленную применимость для создания вакцин против различных инфекционных заболеваний.
--->
Список последовательностей
<ST26SequenceListing dtdVersion="V1_3" fileName="Ad19.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2023-04-15">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023-04-14</ApplicationNumberText>
<FilingDate>2023-04-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>2023-04-14</ApplicantFileReference>
<ApplicantName languageCode="ru">федеральное государственное
бюджетное учреждение "Национальный исследовательский центр
эпидемиологии и микробиологии имени почетного академика Н.Ф.Гамалеи"
Министерства здравоохранения Российской Федерации</ApplicantName>
<ApplicantNameLatin>The National Research Center for Epidemiology and
Microbiology named after Honorary Academician N.F. Gamaleya of the
Ministry of Health of the Russian Federation </ApplicantNameLatin>
<InventionTitle languageCode="ru">Экспрессионный вектор на основе
аденовируса человека 19 серотипа и способ его
применения</InventionTitle>
<SequenceTotalQuantity>7</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>536</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..536</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catcatcaataatataccccacaaagtaaacaaaagttaatatgcaaatgag
cttttgaattttaacggttttggggcggagccaacgctgattggacgagaaacggtgatgcaaatgacgt
cacgacgcacggctaacggtcgccgcggaggcgtggcctagcccggaagcaagtcgcggggctgatgacg
tataaaaaagcggactttagacccggaaacggccgattttcccgcggccacgcccggatatgaggtaatt
ctgggcggatgcaagtgaaattaggtcattttggcgcgaaaactgaatgaggaagtgaaaagcgaaaaat
accggtccctcccagggcggaatatttaccgagggccgagagactttgaccgattacgtgggggtttcga
ttgcggtgtttttttcgcgaatttccgcgtccgtgtcaaagtccggtgtttatgtcacagatcagctggt
ttaaacactgttaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>26612</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26612</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggaaacgtatacctataaaggcgggtgtcttacgagggtctttttgcttt
tctgcagacatcatgaacgggactggcggggccttcgaaggggggctttttagcccttatttgacaaccc
gcctgccgggatgggccggagttcgtcagaatgtgatgggatcgacggtggatgggcgcccagtgcttcc
agcaaattcctcgaccatgacctacgcgaccgtggggaactcgtcgctcgacagcaccgccgcagccgcg
gcagccgcagccgccatgacagcgacgagactggcctcgagctacatgcccagcagcggtagtagcccct
ctgtgcccagttccatcatcgccgaggagaaactgctggccctgctggccgagctggaagccctgagccg
ccagctggccgccctgacccagcaggtgtccgagctccgcgaacagcagcagcagcaaaataaatgattc
aataaacacagattctgattcaaacagcaaagcatctttattatttattttttcgcgcgcggtaggccct
ggtccacctctcccgatcattgagagtgcggtggattttttccaggacccggtagaggtgggattggatg
ttgaggtacatgggcatgagcccgtcccggggatggaggtagcaccactgcatggcctcgtgctctgggg
tcgtgttgtagatgatccagtcatagcaggggcgctgggcgtggtgctggatgatgtccttgaggaggag
actgatggccacggggagccccttggtgtaggtgttggcaaagcggttgagctgggagggatgcatgcga
ggggagatgatgtgcagtttggcctggatcttgaggttggcgatgttgccgcccagatcccgcctggggt
tcatgttgtgcaggaccaccaggacggtgtagcccgtgcacttggggaacttatcatgcaacttggaagg
gaatgcgtggaagaatttggagacgcccttgtgcccgcccaggttttccatgcactcatccatgatgatg
gcgatgggcccgtgggctgcggctttggcaaagacgtttctggggtcagagacatcataattatgctcct
gggtgagatcatcataagacattttaatgaattttgggcggagggtgccagattgggggacgatggttcc
ctcgggccccggggcgaagttcccctcgcagatctgcatctcccaggctttcatctcggagggggggatc
atgtccacctgcggggcgatgaaaaaaacggtttccggggcgggggtgatgagctgcgaggagagcaggt
ttctcaacagctgggacttgccgcacccggtcgggccgtagatgaccccgatgacgggttgcaggtggta
gttcaaggacatgcagctgccgtcgtcccggaggaggggggccacctcgttgagcatgtctctaacttgg
aggttttcccggacgagctcgccgaggaggcggtccccgcccagcgagaggagctcttgcagggaagcaa
agtttttcaggggcttgagtccgtcggccatgggcatcttggcgagggtctgcgagaggagttcgagacg
gtcccagagctcggtgacgtgctctacggcatctcgatccagcagacttcctcgtttcgggggttgggac
gactgcgactgtagggcacgagacgatgggcgtccagcgcggccagcgtcatgtccttccagggtctcag
ggtccgcgtgagggtggtctccgtcacggtgaaggggtgggcccctggctgggcgcttgcaagggtgcgc
ttgagacttatcctgctggtgctgaaacgggcacggtcttcgccctgcgcgtcggcgagatagcagttga
ccatgagctcgtagttgagggcctcggcggcgtggcccttggcgcggagcttgcccttggaagagcgtcc
gcaggcgggacagaggagggattgcagggcgtagagcttgggcgcaagaaagaccgactcgggagcgaaa
gcgtccgctccgcagtgggcgcagacggtctcgcactcgacgagccaggtgagctcgggctgctcggggt
caaaaaccagttttcccccgttctttttgatgcgcttcttacctcgcgtctccatgagtctgtgtccgcg
ctcggtgacaaacaggctgtcggtgtccccgtagacggacttgattggcctgtcctgcaggggcgtcccg
cggtcctcctcgtagagaaactcggaccactctgagacaaaggcgcgcgtccacgccaagacaaaggagg
ccacgtgcgaggggtagcggtcgttgtccaccagggggtccaccttttccaccgtgtgcagacacatgtc
cccctcctccgcatccaagaaggtgattggcttgtaggtgtaggccacgtgaccgggggtccccgacggg
ggggtataaaagggggcgggtctgtgctcgtcctcactctcttccgcgtcgctgtccacgagcgccagct
gttggggtaggtattccctctcgagagcgggcatgacctcggcactcaggttgtcagtttctagaaacga
ggaggatttgatgttggcttgccctgccgcgatgctttttaggagactttcatccatctggtcagaaaag
actatttttttattgtcaagcttggtggcaaaggagccatagagggcgttggagagaagcttggcgatgg
atctcatggtctgatttttgtcacggtcggcgcgctccttggccgcgatgttgagctggacatactcgcg
cgcgacacacttccattcggggaagacggtggtgcgctcgtcaggcacgatcctgacgcgccagccgcgg
ttatgcagggtgaccaggtccacgctggtggccacctcgccgcgcaggggctcgttggtccagcagagtc
tgccgcccttgcgcgagcagaacggtggcagcacatcaagcagatgctcgtcaggggggtccgcatcgat
ggtgaagatgccgggacagagttccttgtcaaaatagtctatttttgaggatgcatcatccaaggccatc
tgccactcgcgggcggccattgctcgctcgtaggggttgaggggcggaccccagggcatgggatgcgtga
gggcggaggcgtacatgccgcaaatgtcgtagacatagatgggctccgagaggatgccgatgtaggtggg
ataacagcgccccccgcggatgctggcgcgcacgtagtcatacaactcgtgcgagggggccaagaaggcg
gggccgagattggtgcgctggggctgctcggcgcggaagacaatctggcgaaagatggcgtgcgagttgg
aggagatggtgggccgttggaagatgttaaagtgggcgtggggcaagcggaccgagtcgcggatgaagtg
cgcgtaggagtcttgcagcttggcgacgagctcggcggtgacgaggacgtccatggcgcagtagtccagc
gtttcgcggatgatgtcataacccgcctctcctttcttctcccacagctcgcggttgagggcgtattcct
cgtcatccttccagtactcccggagcgggaatcctagatcgtccgcacggtaagagcccagcatgtagaa
atggttcacggccttgtagggacagcagcccttctccacggggagggcgtaagcttgtgcggccttgcgg
agcgaggtgtgcgtcagggcgaaggtgtccctgaccatgactttcaagaactggtacttgaaatccgagt
cgtcgcagccgccgtgctcccatagctcgaaatcggtgcgcttcttcgagagggggttaggcagagcgaa
agtgacgtcattgaagagaatcttgcctgctcgcggcatgaaattgcgggtgatgcggaaagggcccggg
acggaggctcggttgttgatgacctgggcggcgaggacgatctcgtcgaagccgttgatgttgtgcccga
cgatgtagagttccatgaatcgcgggcggcctttgatgtgcggcagctttttgagctcctcgtaggtgag
gtcctcggggcattgcaggccgtgctgctcgagcgcccattcctggagatgtgggttggcttgcatgaag
gaagcccagagctcgcgggccatgagggtctggagctcgtcgcgaaagaggcggaactgctggcccacgg
ccatcttttcgggtgtgacgcagtagaaggtgagggggtcccgctcccagcgatcccagcgtaagcgcgc
ggctagatcgcgagcaagggcgaccagctctgggtcccccgagaatttcatgaccagcatgaaggggacg
agctgcttgccgaaggaccccatccaggtgtaggtttctacatcgtaggtgacaaagagccgctccgtgc
gaggatgagagccgattgggaagaactggatttcctgccaccagttggacgagtggctgttgatgtgatg
aaagtagaaatcccgccggcgaaccgagcactcgtgctgatgcttgtaaaagcgtccgcagtactcgcag
cgctgcacgggctgtacctcatccacgagatacacagcgcgtcccttgaggaggaacttcaggagtggcg
gccctggctggtggttttcatgttcgcctgcgtgggactcaccctggggctcctcgaggacggagaggct
gacgagcccgcgcgggagccaggtccagatctcggcgcggcgggggcggagagcgaagacgagggcgcgc
agttgggagctgtccatggtgtcgcggagatccaggtccgggggcagggttctgaggttgacctcgtaga
ggcgggtgagggcgtgcttgagatgcagatggtacttgatttctacgggtgagttggtggccgtgtccac
gcattgcatgagcccgtagctgcgcggggccacgaccgtgccgcggtgcgcttttagaagcggtgtcgcg
gacgcgctcccggcggcagcggcggttccggccccgcgggcaggggcggcagaggcacgtcggcgtggcg
ctcgggcaggtcccggtgttgcgccctgagagcgctggcgtgcgcgacgacgcggcggttgacatcctgg
atctgccgcctctgcgtgaagaccactggccccgtgactttgaacctgaaagacagttcaacagaatcaa
tctcggcgtcattgacggcggcctgacgcaggatctcttgcacgtcgcccgagttgtcctggtaggcgat
ctcggacatgaactgctcgatctcctcctcctggagatcgccgcgacccgcgcgctccacggtggcggcg
aggtcattcgagatgcgacccatgagctgcgagaaggcgcccaggccgctctcgttccagacgcggctgt
agaccacgtccccgtcggcgtcgcgcgcgcgcatgaccacctgcgcgaggttgagctccacgtgccgcgt
gaagacggcgtagttgcgcaggcgctggaagaggtagttgagggtggtggcgatgtgctcggtgacgaag
aagtacatgatccagcggcgcaggggcatctcgctgatgtcgccaatggcctccagcctttccatggcct
cgtagaaatccacggcgaagttgaaaaactgggcgttgcgggccgagaccgtgagctcgtcttccaggag
ccgaatgagctcggcgatggtggcgcgcacctcgcgctcgaaatccccgggggcctcctcctcctcttcc
tcttcttccatgacgacctcttcttctatttcttcctctgggggcggtggtggtggcggggcccgacgac
gacggcgacgcaccgggagacggtcgacgaagcgctcgatcatctccccgcggcggcgacgcatggtttc
ggtgacggcgcgaccccgttcgcgaggacgcagcgtgaagacgccgccggtcatctcccggtaatggggt
gggtccccgttgggcagcgatagggcgctgacgatgcatcttatcaattgcggtgtaggggacgtgagcg
cgtcgagatcgaccggatcggagaatctttcgaggaaagcgtctagccaatcgcagtcgcaaggtaagct
caaacacgtagcagccctgtggacgctgttagaattgcggttgctgatgatgtaattgaagtaggcgttt
ttgaggcggcggatggtggcgaggaggaccaggtccttgggtcccgcttgctggatgcggagccgctcgg
ccatgccccaggcctggccctgacaccggctcaggttcttgtagtagtcatgcatgagcctctcaatgtc
atcactggcggaggtggagtcttccatgcgggtgaccccgacgcccctgagcggctgcacgagcgccagg
tcggcgacgacgcgctcggcgaggatggcctgttgcacgcgggtgagggtgtcctggaagtcgtccatgt
cgacgaagcggtggtaggccccggtattgatggtgtaggtgcagttggccatgagcgaccagttgacggt
ctgcaggccgggctgcacgacctcggagtacctgagccgcgagaaggcgcgcgagtcgaagacgtagtcg
ttgcaggtgcgcacgaggtactggtagccgactaggaagtgcggcggcggctggcggtagagcggccagc
gctgggtggccggcgcgcccggggccaggtcctcgagcatgaggcggtggtagccgtagaggtagcggga
catccaggtgatgccggcggcggtggtggaggcgcgcgggaactcgcggacgcggttccagatgttgcgc
agcggcaggaaatagtccatggtcggcacggtctggccggtgagacgcgcgcagtcattgacgctctaga
ggcaaaaacgaaagcggttgagcgggctcttcctccgtagcctggcggaacgcaaacgggttaggtcgcg
tgtgtaccccggttcgagtcccctcgaatcaggctggagccgcgactaacgtggtattggcactcccgtc
tcgacccgagcccgatagccgccaggatacggcggagagccctttttgccggccgcggggagtcgctaga
cttgaaagcggccgaaaaccctgccgggtagtggctcgcgcccgtagtctggagaagcatcgccagggtt
gagtcgcggcagaacccggttcgaggacggccgcggcgagcgggacttggtcaccccgccgatttaaaga
cccacagccagccgacttctccagttacgggagcgagcccccttttttctttttgccagatgcatcccgt
cctgcgccaaatgcgtcccacccccccggcgaccaccgcgaccgcggccgtagcaggcgccggcgctagc
cagccaccacagacagagatggacttggaagagggcgaagggctggcgagactgggggcgccgtccccgg
agcgacatccccgcgtgcagctgcagaaggacgtgcgcccggcgtacgtgcctgcgcagaacctgttcag
ggaccgcagcggggaggagcccgaggagatgcgcgactgccggtttcgggcgggcagggagctgcgcgag
ggcctggaccgccagcgcgtgctgcgcgacgaggatttcgagccgaacgagcagacggggatcagccccg
cgcgcgctcacgtggcggcggccaacctggtgacggcctacgagcagacggtgaagcaggagcgcaactt
ccaaaagagtttcaacaaccatgtgcgcacgctgatcgcgcgcgaggaggtggccctgggcctgatgcac
ctgtgggacctggcggaggccatcgtgcagaacccggacagcaagcctctgacggcgcagctgttcctgg
tggtgcagcacagcagggacaacgaggcgttcagggaggcgctgctgaacattgccgagcccgagggtcg
ctggctgctggagctgatcaacatcttgcagagcatcgtagtgcaggagcgcagcctgagcctggccgag
aaggtggcggcgatcaactactcggtgctgagcctgggcaagttttacgcgcgcaagatttacaagacgc
cgtacgtgcccatagacaaggaggtgaagatagacagcttttacatgcgtatggcgctcaaggtgttgac
gctgagcgacgacctgggcgtgtatcgcaacgaccgcatccacaaggccgtgagcacgagccggcggcgc
gagctgagcgaccgtgagctgatgctgagtctgcgccgggcgctggtagggggcgccgccggcggcgagg
agtcctacttcgacatgggggcggacctgcattggcagccgagccggcgcgccttggaggccgcctacgg
tccagaggacttggatgaggaagaggaagaggaggaggatgcacccgctgcggggtactgacgcctccgt
gatgtgtttttagatgtcccagcaagccccggaccccgccataagggcggcgctgcaaagccagccgtcc
ggtctagcatcggacgactgggaggccgcgatgcaacgcatcatggccctgacgacccgcaaccccgagt
cctttagacaacagccgcaggccaacagactctcggccattctggaggcggtggtcccctctcggaccaa
ccccacgcacgagaaggtgctggcgatcgtgaacgcgctggcggagaacaaggccatccgtcccgacgag
gccgggctggtgtacaacgccctgctggagcgcgtgggccgctacaacagcacgaacgtgcagtccaacc
tggaccggctggtgacggacgtgcgcgaggccgtggcgcagcgcgagcggttcaagaacgagggcctggg
ctcgctggtggcgctgaacgccttcctggcgacgcagccggcgaacgtgccgcgcgggcaggacgattac
accaactttatcagcgcgctgcggctgatggtgactgaggtgccccagagcgaggtgtaccagtcgggcc
cggactactttttccagacgagccggcagggcttgcagacggtgaacctgagtcaggctttcaagaacct
gcgcgggttgtggggcgtgcaggcgcccgtgggcgaccggtcgacggtgagcagcttgctgacgcccaac
tcgcggctgttgctgctgcttatcgcgcccttcaccgacagcggcagcgtgaaccgcaactcgtacctgg
gccacctgctgacgctgtaccgcgaggccataggccaggcgcaggtggacgagcagaccttccaggagat
cacgagcgtgagtcgcgcgctgggccaaaacgacaccgacagtctgagggccaccctgaacttcttgctg
accaatagacagcagaagatcccggcgcagtacgcgctgtcggccgaggaggaaaggatcctgagatatg
tgcagcagagcgtagggcttttcctgatgcaggagggcgccacccccagcgccgcgctggacatgaccgc
gcgcaacatggaacctagcatgtacgccgccaaccggccgttcatcaataagctgatggactacctgcac
cgcgcggcgtccatgaactcggactactttacaaacgccatcctgaacccgcactggctcccgccgccgg
ggttctacacgggcgagtacgacatgcccgaccccaacgacgggttcctgtgggacgacgtggacagcgc
ggtgttctccccgaccttgcaaaagcgccaggaggcggtacgcacgcccgcgagcgagggcgcggtgggt
cggagcccctttcctagcttagggagtttgcatagcttgccgggctcggtgaacagcggcagggtgagcc
ggccgcgcttgctgggcgaggacgagtacctgaacgactcgctgctgcagccgccacgggtcaagaacgc
catggccaataacgggatagagagtctggtggacaaactgaaccgctggaagacctacgctcaggaccat
agggacgcgcccgcgccgcggcgacagcgccacgaccggcagcggggcctggtgtgggacgacgaggact
cggccgacgatagcagcgtgttggacttgggcgggagcggtggggccaacccgttcgcgcatctgcagcc
caaactggggcggcggatgttttgaaatgcaaaataaaactcaccaaggccatagcgtgcgttctcttcc
ttgttagagatgaggcgcgcggtggtgtcttcctctcctcctccctcgtacgagagcgtgatggcgcagg
cgaccctggaggttccgtttgtgcctccgcggtatatggctcctacggagggcagaaacagcattcgtta
ctcggagctggctccgcagtacgacaccactcgcgtgtacttggtggacaacaagtcggcggacatcgct
tccctgaactaccaaaacgaccacagcaacttcctgaccacggtggtgcagaacaacgatttcacccccg
ccgaggccagcacgcagacgataaattttgacgagcggtcgcggtggggcggtgatctgaagaccattct
gcacaccaacatgcccaatgtgaacgagtacatgttcaccagcaagtttaaggcgcgggtgatggtgtct
aggaagcggccagagggggcgacagatgcaagtcaggatattttaaagtatgagtggtttgagtttaccc
tgcccgagggcaacttttccgagaccatgaccatagacctgatgaacaacgccatcttggaaaactactt
gcaagtggggcggcagaatggcgtgctggagagcgatatcggagtcaagtttgacagcaggaatttcaag
ctgggctgggacccggtgaccaagctggtgatgccaggggtctacacctacgaagccttccacccggacg
tggtgctgctgccgggctgcggggtggacttcacagagagccgcctgagcaacctcctgggcattcgcaa
gaagcaacctttccaagagggcttcagaatcatgtatgaggatctagaaggcggtaatatccccgctctc
ctggataccaaaaaatatctggatagcaagaaagaaattgaagatgcaaaacagaaagcggcccagccag
gtggcgagatcagaggagacagtgctgatactaaagctgcagaacaggcggctgaaaaagagctggttat
tgagcccatcgagcaagatgaaagcaagagaagctataatgtgatcaagggtacccatgacaccctgtac
cgaagttggtacctgtcctatacctacggggaccccgagaagggggtgcagtcgtggacgctgctcacca
ccccggacgtcacctgcggcgcggagcaagtctactggtcgctgccggacctcatgcaagaccccgtcac
cttccgctctacccagcaagtcagcaactaccctgtggtcggcgccgagctcatgcccttccgcgccaag
agcttttacaacgacctcgccgtctactcccagctcatccgcagctacacctccctcacccacgtcttca
accgcttccccgacaaccagatcctctgccgcccgcccgcgcccaccatcaccaccgtcagtgaaaacgt
gcctgctctcacagatcacgggacgcttccgctgcgcagcagtatccgcggagtccagcgagtgaccgtc
actgacgcccgtcgccgcacctgtccctacgtctacaaggccctgggcatagtcgcgccgcgcgtgctct
ccagtcgcaccttctaaaaaatgtctattctcatctcgcccagcaataacaccggctggggtcttactag
gcccagcaccatgtacggaggagccaagaagcgctcccagcagcaccccgtccgcgtacgcggccacttc
cgcgctccctggggcgcttacaagcgcgggcggactgccaccgccgccgccgtgcgcaccaccgtcgacg
acgtcatcgactcggtggtcgccgacgcgcgcaactatacccccgccccttcgaccgtggacgcggtcat
cgacagcgtggtggccgacgcgcgcgactatgccagacgcaagagccggcggcgacgaatcgccaggcgc
caccggagcacgcccgccatgcgcgccgctcgggctctgctgcgccgcgccagacgcacgggccgccggg
ccatgatgcgagccgcgcgtcgcgccgccgccgcgcccacccccgcaggcaggactcgcagacgagcggc
cgccgccgccgccgcggccatctctagcatgaccagacccaggcgcggaaacgtgtactgggtgcgcgac
tccgtcacgggcgtgcgcgtgcccgtgcgcacccgtcctcctcgtccctgatctaatgcttgtgtcctcc
cccgcaagcgacgatgtcaaagcgcaaaatcaaggaggagatgctccaggtcgtcgccccggagatttac
ggaccaccccaggcggaccagaaaccccgcaaaatcaagcgggttaaaaaaaaggatgaggtggacgagg
gggcagtagagtttgtgcgcgagttcgctccgcggcggcgcgtaaattggaaggggcgcagggtgcagcg
cgtgttgcggcccggcacggcggtggtgttcacgcccggcgagcggtcctcggtcaggagcaagcgtagc
tatgacgaggtgtacggcgacgacgacatcctggaccaggcggcggagcgggcgggcgagttcgcctacg
ggaagcggtcgcgcgaagaggagctgatctcgctgccgctggacgagagcaaccccacgccgagcctgaa
gcccgtgaccctgcagcaggtgctgccccaggcggtgctgctgccgagccgcggggtcaagcgcgagggc
gagagcatgtacccgaccatgcagatcatggtgcccaagcgccggcgcgtggaggacgtgctggacaccg
tgaaaatggatgtggagcccgaggtcaaggtgcgccccatcaagcaggtggcgccgggcctgggcgtgca
aaccgtggacattcagatccccaccgacatggatgtcgacaaaaaaccctcgaccagcatcgaggtgcaa
accgacccctggctcccagcctccaccgctaccacgtccacttctaccgccgccacggctaccgagcctc
ccaggaggcgaagatggggcgccgccagccggctgatgcccaactacgtgttgcatccttccattatccc
gacgccgggctaccgcggcacccggtactacgccagccgcaggcgcccagccagcaaacgccgccgccgc
accgccacccgccgccgtctgccccccgcccgcgtgcgccgcgtaaccacgcgccggggccgctcgctcg
ttctgcccaccgtgcgctaccaccccagcatcctttaatccgtgtgctgtgatactgttgcagagagatg
gctctcacttgccgcctgcgcatccccgttccgaattaccgaggaagatcccgccgcaggagaggcatgg
caggcagcggcctgaaccgccgccggcggcgggccatgcgcaggcgcctgagtggcggctttctgcccgc
gctcatccccataatcgcggcggccatcggcacgatcccgggcatagcttccgttgcgctgcaggcgtcg
cagcgccgttgatgtgcgaataaagcctctttagactctgacacacctggtcctgtatatttttagaatg
gaagacatcaattttgcgtccctggctccgcggcacggcacgcggccgttcatgggcacctggaacgaga
tcggcaccagccagctgaacgggggcgccttcaattggagcagtgtctggagcgggcttaaaaatttcgg
ctcgacgctccggacctatgggaacaaggcctggaatagtagcacggggcagttgttgagggaaaagctc
aaagaccagaacttccagcagaaggtggtggacggcctggcctcgggcattaacggggtggtggacatcg
cgaaccaggcagtgcagcgcgagataaacagccgtctggacccgcggccgcccacggtggtggagatgga
agatgcaactcttccgccgtcgaagggcgagaagcggccgcggccagatgcggaggagacgatcctgcag
gtggacgagccgccctcgtacgaggaggccgtgaaggccggcatgcccaccacgcgcatcatcgcgccac
tggccacgggtgtaatgaaacccgccacccttgacctgcctccaccacccacgcccgctccaccgaaggc
agctccggttgtgcagccccctccggtggcgaccgccgtgcgccgcgtccccgcccgccgccaggcccag
aactggcagagcacgctgcacagtattgtgggcctgggagtgaaaagtctgaagcgccgccgatgctatt
gagagagaggaaggaggacactaaagggagagcttaacttgtatgtgccttaccgccagagaacgcgcga
agatggccaccccctcgatgatgccgcagtgggcgtacatgcacatcgccgggcaggacgcctcggagta
cctgagcccgggtctggtgcagtttgcccgcgccaccgacacgtacttcagcctgggcaacaagtttagg
aaccccacggtggccccgacccacgatgtgaccacggaccggtcccagcgtctgacgctgcgcttcgtgc
ccgtggatcgcgaggacaccacgtactcgtacaaggcgcgcttcactctggccgtgggcgacaaccgggt
gctagacatggccagcacttactttgacatccgcggcgtcctggaccgcggtcccagcttcaaaccctac
tcgggcacggcttacaacagcctggcccccaagggcgcccccaactccagtcagtgggatgctcaagaaa
aaaatggacaaggaggaaatgacatggttaccaaaactcacacatttggtgtggctgctatgggaggaac
aaatattacaaaccagggtttgttaattggaactgaagaaacagccgataatcctccaaaggaaatcttt
gcagacaaattattccagccagaacctcaagtaggagaggaaaactggcaagacaccaatgcattctatg
gaggcagggctcttaagaaggaaactaaaatgaaaccatgctatggatcttatgctagaccaacaaacac
aagtggcggacaggctaagcttaaaactggtgacaatatcgatcctaccaaggatttcgacatagatctt
gctttcttcgatactcctggcggaaatcctccagcaggtggtagtggaacggaagaatacaaagcagaca
ttgttatgtacactgaaaatgtcaaccttgaaacacctgacactcatgtggtgtacaaaccaggcaaaga
ggatgaaagttctgaggccaacttggttcagcagtccatgcccaacagacccaactacattggcttcaga
gacaattttgtggggctcatgtattacaacagcactggcaacatgggtgtgctggctggtcaggcctctc
agttgaatgctgtggtggacttgcaagacagaaacacagagctgtcttaccagctcttgctagattctct
gggcgacagaaccaggtactttagcatgtggaactctgcggtggacagctatgatcccgacgtcaggatc
attgaaaatcacggtgtggaagatgaacttccaaactattgctttccattggatggctctggtaccaatg
ctgcctaccaaggtgtaaaggttcaagatggtgaagacggggataaagaaactgaatgggaaaaagatac
caaagtcgcagatcgtaaccaactgtgcaagggtaacatctttgccatggagatcaacctccaggccaac
ctgtggaagagttttctgtactcgaacgtggccttgtacctgcccgactcctacaagtacacgccggcca
acgtcacgctgcccgccaacaccaacacctacgagtacatgaacggccgcgtggtagccccctcgctggt
ggacgcatacgtcaacatcggtgcgcgctggtcgctggaccccatggacaacgtcaaccccttcaaccac
caccgcaacgctggcctgcgctaccgctccatgcttctgggcaacggccgctacgtgcccttccacatcc
aagtgccccaaaagttctttgccatcaagaacctgctcctgctccccggctcctacacctacgagtggaa
cttccgcaaggatgtcaacatgatcctgcagagttccctcggaaacgacctgcgcgttgacggcgcctcc
gtgcgctttgacagcgttaacctctacgccacattcttccccatggcgcacaacaccgcctccaccctgg
aagccatgctgcgcaacgacaccaacgaccagtccttcaacgactacctctcggccgccaacatgctcta
ccccatcccggccaaggccaccaacgtgcccatctccatcccctcgcgcaactgggccgccttccgaggc
tggagttttacccggctcaagaccaaggaaactccctccctcggctcgggtttcgacccctactttgtat
actctggctccatcccctatctcgacgggaccttctacctcaaccacaccttcaagaaggtctccatcat
gttcgactcctcggtcagctggcccggcaacgaccggctgctcacgccgaacgagttcgagatcaagcgc
agcgtcgacggggagggctacaatgtggcccaatgcaacatgaccaaggactggttcctcgtccagatgc
tctcccactacaacatcggctaccagggcttccatgtgcccgagggctacaaggaccgcatgtactcctt
cttccgcaacttccagcccatgagcaggcaggtggtcgatgagatcaactacaaggactacaaggccgtc
accctgcccttccagcacaacaactcgggcttcaccggctacctagcacccaccatgcgtcaggggcagc
cctaccccgccaacttcccctacccgctcattggctccaccgcagtgccctccgtcacccagaaaaagtt
cctctgcgacagggtcatgtggcgcatccccttctccagcaacttcatgtccatgggcgccctcaccgac
ctgggtcagaacatgctctacgccaactcggcccacgcgctcgacatgaccttcgaggtggaccccatgg
atgagcccaccctcctctatcttctcttcgaagttttcgacgtggtcagagtgcaccagccgcaccgcgg
cgtcatcgaggccgtctacctgcgcacgcccttctccgccggcaacgccaccacataagcatgagcggct
ccagcgaaagagagctcgcggccatcgtgcgcgacctgggctgcgggccctactttttgggcacccacga
caagcgcttcccgggcttcctcgccggcgacaagctggcctgcgccatcgtcaacacggccggccgcgag
accgggggcgtgcactggctcgccttcggctggaacccgcgctcgcgcacctgctacatgttcgacccat
ttgggttctcggaccgccggctcaaacagatttacagcttcgagtacgaggccatgctgcgccgcagcgc
cctggcctcctcgcccgaccgctgtctcagcctcgagcagtccacccagaccgtgcaggggcccgactcc
gccgcctgcggacttttctgttgcatgttcttgcatgctttcgtgcactggcccgaccaacccatggacg
ggaaccccaccatgaacttgctgacgggggtgcccaacggcatgctacagtcgccacaggtgctgcccac
cctcaggcgcaaccaggaggagctctaccgcttcctcgcgcgccactccccttactttcgctcccaccgc
gccgccatcgaacacgccaccgcttttgacaaaatgaaacaactgcgtgtatctcaataaacagcacttt
tattttacatgcactggagtatatgcaagttatttaaaagtcgaaggggttatcccgcttgtcgttgtgc
gccgcgctggggagggccacgttgcggtactggaacttgggctgccacttgaactcggggatcaccagtt
tgggaacagcaatctcggggaaggtctcgctccacatgcgccggctcatctgcagggcgcccagcatgtc
aggcgcggagatcttgaaatcacagttggggccggtgctctgcgcgcgcgagttgcggtacacggggttg
cagcactggaacaccatcagactggggtacttcacactggccagcacgctcttgtcgctgatctgatcct
tgtccaggtcctcggcattgcttaggccaaacggggtcatcttgcacagctggcggcccaagaagggcac
gctctgaggcttgtggttacactcgcagtgcacgggcatcagcatcatccccgcgccgcgctgcatattc
gggtagagggccttgacaaaagccgagatctgcttgaaagcttgctgggccttggccccctcgctgaaaa
acagaccgcagctcttcccgctgaactggttattcccgcatccggcatcctgcacgcagcagcgcgcgtc
atggctggtcagttgcaccacgcttcttccccagcggttctgggtcaccttggccttgctgggttgctcc
ttcagcgcgcgctgtccgttctcgctggtcacatccatctccaccacgtggtccttgtggatcatcaccg
ttccatgcagacacttgagctggccttccacctcggtgcagccgtgatcccacaggacgcatccggtgca
ctcccagttcttgtgcgcgatcccgctgtgactgaagatgtaaccttgcaacatgcggcccatcacggtg
ctaaatgatttactggtgctgaaggtcagttgcaggccgcgggcctcctcgttcatccaggtctgacaca
tcttctggaagatctcggtctgctcgggcatgagcttgtaggcatcgcgcaggccgctgtcgacgcggta
gcgttccatcagcacgttcatggtatccatgcccttctcccaggacgagaccagaggcaaactcaggggg
ttgcgcacgttcaggataccgggggtcgcgggctcgacgatgcgttttccgtccttgccttccttcaaca
gaaccggaggctggctgaatcccactcccacgatcacggcttcttcctggggcatttcttcgtcggggtc
cgtcttggtcacatgcttggtctttctggcttgcttcttttttggagggctgtccacggggaccacgtcc
tcctcggaagacccggagcccacccgctgatactttcggcgcttggtgggcagaggaggtggcggcgagg
ggctcctctcctgctccggcggatagcgcgccgacccgtggccccggggcggagtggcctctcgctccat
gaaccggcgcacgtcctgactgccgccggccattgtttcctaggggaagatggaggagcagccgcgtaag
caggagcaggaggaggacttaaccacccacaagcaacccaaaatcgagcaggacctgggcttcgaagagc
cggctcgtctagaacccccacaggatgaacaggagcacgagcaagacgcaggccaggaggagaccgacgc
tgggctcaagcatggctacctgggaggagaggaggatgtgctgctgaaacacctgcagcgccagtccctc
atcctccgggacgccctggccgaccggagcgaaacccccctcagcgtcgaggagctaagtagggcctacg
agctcaaccttttctcgccgcgcgtgccccccaaacgccagcccaacggcacatgcgagcccaacccgcg
tctcaacttctatcccgttttcgcggtccccgaggcccttgccacctatcacatctttttcaagaaccaa
aagatccccgtctcctgccgcgccaaccgcacccgcgccgacgcgctcctcgctctggggcccggcgcgc
gcatacctgatatcgcttccctggaagaggtgcccaagatcttcgaagggctcggtcgggacgagacgcg
cgcggcgaacgctctgaaagaaacagcagaggaagagggtcacactagcgccttggtagagttggaaggc
gacaacgccaggctggccgtgctcaagcgcagcgtcgagctcacccacttcgcctaccccgccgtcaacc
tcccgcccaaggtcatgcgtcgcatcatggatcagctcatcatgccccacatcgaggccctcgatgaaag
tcaggagcagcggcccgaggacgcccagcccgtggtcagcgacgagcagcttgcgcgctggctcgggaac
cgcgacccccaggccctggagcagcggcgcaagctcatgctggccgtggtcctggtcaccctcgagctgg
aatgcatgcgccgcttcttcaccgaccccgacaccctgcgcaaggtggaggagaccctgcactacacttt
cagacacgggttcgtcaggcaggcctgcaagatctccaacgtggagctgaccaacctggtttcctgcctg
gggatcctgcacgagaaccgcctggggcagaccgtgctccactctaccctcaagggcgaggcgcgtcggg
actatgtccgcgactgcgtctttctttttctctgtcacacatggcaagcggccatgggcgtgtggcagca
gtgtctcgaggacgagaacctgaaggagctggacaagcttcttgctagaaacctcaaaaagctgtggacg
ggcttcgacgagcgcaccgtcgcctcggacctggccgagatcgtcttccccgagcgcctgaggcagacgc
tgaaaggcgggctgcccgacttcatgagccaaagcatgttgcaaaactaccgcactttcattctcgagcg
atcggggatcctgcccgccacctgcaacgctttcccctccgactttgttccgctgagctaccgcgagtgt
cccccgccgctgtggagccactgctacctcttgcaactggccaactacatcgcccaccactcggatgtga
tcgaggacgtgagcggcgaggggctgctagagtgccactgccgctgcaacctgtgctccccgcaccgctc
cctggtctgcaacccccagctcctcagcgagacccaggtcatcggtaccttcgagctgcaaggtccgcag
gagtccaccgctccgctgaaacttacgccggggttgtggacttccgcgtacctgcgcaaatttgtacccg
aggactaccacgcccatgagataaagttcttcgaggaccaatcgcggccgcagcacgcggatctcacggc
ctgcgtcatcacccagggcgcgatcctcgcccaattgcacgccatccaaaaatcccgccaagagtttctt
ctgaaaaagggtagaggggtatacctggacccccagacgggcgaggtgctcaacccgggtcttccccagc
atgccgaggaagaagcaggagccgctagtggaggagatggaagaagaatgggacagccaggcagaggagg
acgaatgggaggaggagacagaggaggaagaattggaagaggtggaagaggagcaggcaacagagcagcc
cgtcgccgcaccatccgcgccggcagccccggcggtcacggatacaacctccgcagctccggccaagcct
cctcgtagatgggatcgagtgaagggtgacggtaagcacgagcggcagggctaccgatcatggagggccc
acaaagccgcgatcatcgcctgcttgcaagactgcggggggaacatcgctttcgcccgccgctacctgct
cttccaccgcggggtgaacatcccccgcaacgtgttgcattactaccgtcaccttcacagctaagaaaaa
gcaagtcagaggagtcgccggaggaggaggaggcctgagaatcgcggcgaacgagccctcgaccaccagg
gagctgaggaaccggatcttccccactctttatgccatttttcagcagagtcgaggtcagcagcaagagc
tcaaagtaaaaaaccggtctctgcgctcgctcacccgcagttgcttgtaccacaaaaacgaagatcagct
gcagcgcactctcgaagacgccgaggctctgttccacaagtactgcgcgctcactcttaaagactaaggc
gcgcccacccggaaaaaaggcgggaattacctcatcgccaccatgagcaaggagattcccaccccttaca
tgtggagctatcagccccagatgggcctggccgcgggcgcctcccaggactactccacccgcatgaactg
gcttagtgccggcccctcgatgatctcacgggtcaacggggtccgtaaccatcgaaaccagatattgttg
gagcaggcggcggtcacctccacgcccagggcaaagctcaacccacgtaattggccctccaccctggtgt
atcaggaaatccccgggccgactaccgtactacttccgcgtgacgcactggccgaagtccgcatgactaa
ctcaggtgtccagctggccggcggcgcttcccggtgcccgctccgcccacaatcgggtataaaaaccctg
gtgatccgaggcagaggcacacagctcaacgacgagttggtgagctcttcgatcggtcttcgaccggacg
gagtgttccaactagccggagccgggagatcgtccttcactcccaaccaggcctacctgaccttgcagag
cagctcttctgagcctcgctccggaggcatcggaaccctccagttcgtggaggagtttgtgccctcagtc
tacttcaaccccttctcgggatcgccaggcctctacccggacgagttcataccgaacttcgacgcagtga
gagaagcggtggacggctacgactgaatgtcccatggtgactcggctgagctcgctcggttgaggcatct
ggaccactgccgccgcctgcgctgcttcgcccgggagagctgcggactcatctactttgagtttcccgag
gagcaccccaacggccctgcgcacggagtgcggatcaccgtagagggtaccaccgagtctcacctggtca
ggttcttcacccagcaacccttcctggtcgaacgggaccggggcaccaccacctacaccgtctactgcat
ctgtcctaccccgaagttgcatgagaatttttgctgtactctttgtggtgagtttaataaaagctgaact
agtggcgcgatggcgctggaatacgccgattaaaagcccaaaaaccaatcagacccttcctcatttcccc
atccccaatactcataagaataaatcattggaattaatcattcaataaagatcacttacttgaaatctga
aagtatgtctctggtgtagttgttcagcaacacctcggtaccctcttcccagctctggtactccagtccc
cggcgggcggcgaacttcctccacaccttgaaagggatgtcaaattcctggtccacaattttcattgtct
tccctctcagatgtcaaagaggctccgggtggaagatgacttcaaccccgtctacccctatggctacgcg
cggaatcagaatatccccttcctcactcccccctttgtctcctccgatggattcaaaaacttcccccctg
gggtactgtcactcaaactggctgatccaatcaccattaccaatggggatgtatccctcaaggtgggagg
tggtctcactttgcaagatggaagcctaactgtaaaccctaaggctccactgcaagttactactgataaa
aaacttgagcttgcatatgataatccatttgaatgtagtgctaataaatttagtttaaaagtaggacatg
gattaaaagtattagatgaaaaaagtgctgcggggttaaaagatttaattggcaaacttgtggttttaac
aggaaaaggaataggcactgaaaatttagaaaatacagatggtagcagcagaggaattggtataaatgta
agagcaagagaagggttgacatttgacaatgatggatacttggtagcatggaacccaaagtatgacacgc
gcacactttggacaacaccagacacatctccaaactgcacaattgctcaggataaggactctaaactcac
tttggtacttacaaagtgtggaagtcaaatattagctaatgtgtctttgattgtggtcgcaggaaagtac
cacatcataaataataagacaaatccagaaataaaaagttttactattaaactgttatttaataagaacg
gagtgcttttagacaactcaaatcttggaaaagcttattggaactttagaagtggaaattccaatgtttc
gacagcttatgaaaaagcaattggttttatgcctaatttagtagcgtatccaaaacccagtaattctaaa
aaatatgcaagagacatagtttatggaactatatatcttggtggaaaacctgatcagccagcagtcatta
aaactacctttaaccaagaaactggatgtgaatactctatcacatttgactttagttggtccaaaaccta
tgaaaatgttgaatttgaaaccacctcttttaccttctcctatattgcccaagaatgaaagaccaataaa
cgtgtttttcatttcaaattttcatgtatctttattgatttttacaccagcacgggtagtcagtctccca
ccaccagcccatttcacagtgtacacggttctctcagcacgggtggccttaaataggggaatgttctgat
tagtgcgggaactcaacttggggtctataatccacacagtttcctggcgagccaaacgggggtcggtgat
tgagatgaagccgtcctctgaaaagtcatccaagcgggcctcacagtccaaggtcacagtctggtggaat
gagaagaacgcacagattcatactcggaaaacaggatgggtctgtgcctctccatcagcgccctcagcag
tctctgccgccggggctcggtgcggctgctgcagatgggatcgggatcacaagtctctctgactatgatc
ccaacagccttcagcatcagtctcctggtgcgtcgggcacagcaccgcatcctgatctctgccatgttct
cacagtaagtgcagcacataatcaccatgttattcagcagcccataattcagggcgctccagccaaagct
catgttggggatgatggaacccacgtgaccatcgtaccagatgcggcagtatatcaggtgcctgcccctc
ataaacacactgcccatatacatcatctctttgggcatgtttctgttcacaatctgacggtaccagggga
agcgctggttgaacatgcacccgtaaatgactctcctgaaccacacggcaagcagggtgcctcccgcccg
acactgcagggagcccggggatgaacagtggcaatgcagtatccagcgctcgtacccgctcaccatctga
gctctcaccaagtccagggtagcggggcacaggcacactgacatacatctttttaaaatttttatttcct
ctggggtcaggatcatatcccagggtactggaaattcttggagcagtgtaaagccagcagcacatggtaa
tccacggacagaacttacattatgataatctgcatgatcacaatcgggcaacaaagggtgttgttcagtc
agagaggccctggtctcctcatcagatcgtggtaaacgggccctgcgatatggatgatggcggagcaagc
tcgactgatcctcggtttgcattgtagtgaattctcttgcgtaccttgtcgtacttctgccagcagaaat
gggcccttgaacagcagatacctctccttctcctgtctttccgctgctgacgctcagtcatccaactgaa
gtacagccattcccgcaggttctcgagcagctcctcagcatctgatgaaacaaaagtcctgtccatgcgg
attccccttaatacatcagccaggacattgtaggccatcccaatccagttaatgcagcctggtctatcat
tcagaggaggtgggggaagaactggaagaaccatttttttattccaagcggtctcgaaggacgataaagt
gtaagtcacgcaggtgacagcgttcccctccgctgtgctggtggaaacagacagccaggtcaaaacctac
tctattttcaaggtgctcgactgtagcttcgagcagtggctctacgcgtacatccagcatcaaaatcaca
ttaaaggctggccctccatcgatttcatcaatcatcaggttacactcgttcaccatccccaggtaattct
catttttccagccttggattatttctacaaattgttggtgtaagtccactccgcacatgtggtaaagctc
ccacagcgccccctccactttcataatcaggcagaccttcataatagaaacagatcctgctgctccacca
cctgcagcgtgttcaaaacaacaagattcaataaggttctgccctccgccctcagctcgcgcctcaatgt
cagctgcaaaaagtcacttaaatcctgggccactacagctgacaattcagagccagggctaagcgtggga
ctggcaagcgtgagggaaaactttaatgatccaaagctagcacccaaaaactgcatgctggaataagctc
tctttgtgtctccggtgatgccttccaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>3819</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3819</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtttgtgttccttgtgttattgccactagtctctagtcagtgtgtgaacc
tgaccacaagaacccagctgcctccagcctacaccaacagctttaccagaggcgtgtactaccccgacaa
ggtgttcagatccagcgtgctgcactctacccaggacctgttcctgcctttcttcagcaacgtgacctgg
ttccacgccatccacgtgtccggcaccaatggcaccaagagattcgacaaccccgtgctgcccttcaacg
acggggtgtactttgccagcaccgagaagtccaacatcatcagaggctggatcttcggcaccacactgga
cagcaagacccagagcctgctgatcgtgaacaacgccaccaacgtggtcatcaaagtgtgcgagttccag
ttctgcaacgaccccttcctgggcgtctactatcacaagaacaacaagagctggatggaaagcgagttcc
gggtgtacagcagcgccaacaactgcaccttcgagtacgtgtcccagcctttcctgatggacctggaagg
caagcagggcaacttcaagaacctgcgcgagttcgtgttcaagaacatcgacggctacttcaagatctac
agcaagcacacccctatcaacctcgtgcgggatctgcctcagggcttctctgctctggaacccctggtgg
atctgcccatcggcatcaacatcacccggtttcagacactgctggccctgcacagaagctacctgacacc
tggcgatagcagcagcggatggacagctggtgccgccgcttactatgtgggctacctgcagcctagaacc
ttcctgctgaagtacaacgagaacggcaccatcaccgacgccgtggattgtgctctggatcctctgagcg
agacaaagtgcaccctgaagtccttcaccgtggaaaagggcatctaccagaccagcaacttccgggtgca
gcccaccgaatccatcgtgcggttccccaatatcaccaatctgtgccccttcggcgaggtgttcaatgcc
accagattcgcctctgtgtacgcctggaaccggaagcggatcagcaattgcgtggccgactactccgtgc
tgtacaactccgccagcttcagcaccttcaagtgctacggcgtgtcccctaccaagctgaacgacctgtg
cttcacaaacgtgtacgccgacagcttcgtgatccggggagatgaagtgcggcagattgcccctggacag
acaggcaagatcgccgactacaactacaagctgcccgacgacttcaccggctgtgtgattgcctggaaca
gcaacaacctggactccaaagtcggcggcaactacaattacctgtaccggctgttccggaagtccaatct
gaagcccttcgagcgggacatctccaccgagatctatcaggccggcagcaccccttgtaacggcgtggaa
ggcttcaactgctacttcccactgcagtcctacggctttcagcccacaaatggcgtgggctatcagccct
acagagtggtggtgctgagcttcgaactgctgcatgcccctgccacagtgtgcggccctaagaaaagcac
caatctcgtgaagaacaaatgcgtgaacttcaacttcaacggcctgaccggcaccggcgtgctgacagag
agcaacaagaagttcctgccattccagcagtttggccgggatattgccgataccacagacgccgtacgag
atccccagacactggaaatcctggacatcaccccttgcagcttcggcggagtgtctgtgatcacccctgg
caccaacaccagcaatcaggtggcagtgctgtaccaggacgtgaactgtaccgaagtgcccgtggccatt
cacgccgatcagctgacacctacatggcgggtgtactccaccggcagcaatgtgtttcagaccagagccg
gctgtctgatcggagccgagcacgtgaacaatagctacgagtgcgacatccccatcggcgctggcatctg
tgccagctaccagacacagacaaacagccccagacgggccagatctgtggccagccagagcatcattgcc
tacacaatgtctctgggcgccgagaacagcgtggcctactccaacaactctatcgctatccccaccaact
tcaccatcagcgtgaccacagagatcctgcctgtgtccatgaccaagaccagcgtggactgcaccatgta
catctgcggcgattccaccgagtgctccaacctgctgctgcagtacggcagcttctgcacccagctgaat
agagccctgacagggatcgccgtggaacaggacaagaacacccaagaggtgttcgcccaagtgaagcaga
tctacaagacccctcctatcaaggacttcggcggcttcaatttcagccagattctgcccgatcctagcaa
gcccagcaagcggagcttcatcgaggacctgctgttcaacaaagtgacactggccgacgccggcttcatc
aagcagtatggcgattgtctgggcgacattgccgccagggatctgatttgcgcccagaagtttaacggac
tgacagtgctgccaccactgctgaccgatgagatgatcgcccagtacacatctgccctgctggccggcac
aatcacaagcggctggacatttggagctggcgccgctctgcagatcccctttgctatgcagatggcctac
cggttcaacggcatcggagtgacccagaatgtgctgtacgagaaccagaagctgatcgccaaccagttca
acagcgccatcggcaagatccaggacagcctgagcagcacagcaagcgccctgggaaagctgcaggacgt
ggtcaaccagaatgcccaggcactgaacaccctggtcaagcagctgtcctccaacttcggcgccatcagc
tctgtgctgaacgacatcctgagcagactggacaaggtggaagccgaggtgcagatcgacagactgatca
ccggaaggctgcagtccctgcagacctacgttacccagcagctgatcagagccgccgagattagagcctc
tgccaatctggccgccaccaagatgtctgagtgtgtgctgggccagagcaagagagtggacttttgcggc
aagggctaccacctgatgagcttccctcagtctgcccctcacggcgtggtgtttctgcacgtgacatacg
tgcccgctcaagagaagaatttcaccaccgctccagccatctgccacgacggcaaagcccactttcctag
agaaggcgtgttcgtgtccaacggcacccattggttcgtgacccagcggaacttctacgagccccagatc
atcaccaccgacaacaccttcgtgtctggcaactgcgacgtcgtgatcggcattgtgaacaataccgtgt
acgaccctctgcagcccgagctggacagcttcaaagaggaactggataagtactttaagaaccacacaag
ccccgacgtggacctgggcgacatcagcggaatcaatgccagcgtcgtgaacatccagaaagagatcgac
cggctgaacgaggtggccaagaatctgaacgagagcctgatcgacctgcaagaactggggaagtacgagc
agtacatcaagtggccctggtacatctggctgggctttatcgccggactgattgccatcgtgatggtcac
aatcatgctgtgttgcatgaccagctgctgtagctgcctgaagggctgttgtagctgtggcagctgctgc
aagttcgacgaggacgattctgagcccgtgctcaaaggagtcaaattacattacaca</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>1698</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1698</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaggcgatactcgtggttatgctgtacacatttaccaccgcaaatgcag
acacgttatgtatcggttatcatgcgaacaattcaaccgacactgtggacacagttcttgagaagaatgt
caccgtgacacactctgtcaatcttctggaagacaagcataacgggaaactatgcaagctccgcggggta
gcacctttgcatcttggcaaatgtaacattgctggctggatcctgggaaatccagagtgcgaatccctct
ccacagctcgaagttggtcctacattgtggagacatcgaattcagacaacggcacgtgctacccaggaga
tttcatcaattatgaggagctgcgggagcagcttagctccgtgtcatcctttgaaaggtttgaaatcttc
cccaagacaagtagctggcctaatcacgactcggacaatggcgtcacggcagcatgtcctcacgctggag
ccaagagcttctacaagaacctgatctggctggttaagaaaggaaagtcttacccaaagatcaaccaaac
ctacattaatgataaagggaaagaggtcctcgtgctgtggggcattcaccatcctcctactattgctgac
caacaaagtctctatcagaatgcagatgcctatgtgtttgtggggacaagtcgttacagcaagaagttca
agccagagatagcaacccggcccaaggtgcgggatcaggaaggcagaatgaactattactggacactcgt
ggaaccgggagacaaaataaccttcgaagcaactggaaacctggtggcgcccagatatgcctttaccatg
gagcgcgatgctggctctggtattatcatttcagatacgcccgtccacgattgcaataccacttgtcaga
cgcccgagggtgctataaacaccagcctgccctttcagaatgtgcatccaatcaccattggcaaatgtcc
aaagtacgtgaaaagcacaaaattgcgcctggccaccggattgaggaatgtcccttctatccagtctcgt
ggcctgttcggggccattgctggcttcatcgagggtggctggacagggatggtagatggatggtacggtt
atcaccatcagaatgagcaagggagcggctatgccgccgacctgaagagcactcagaatgccattgataa
gattactaacaaagtaaactctgttatcgaaaagatgaatacacagtttaccgccgttggtaaggagttc
aaccaccttgagaaacggatcgagaatctaaacaagaaggttgatgatggcttcctggacatttggactt
acaatgccgaactgttggttctactggaaaacgaacgaactcttgactatcacgattccaacgtgaagaa
cttgtatgagaaagtcaggaaccagttaaagaacaacgccaaggagattggaaacggctgctttgaattc
taccacaaatgcgacaacacctgcatggaaagtgtcaagaacgggacttatgactacccgaaatactcag
aggaagctaaattaaacagagagaaaatcgatggagtgaagctggactccaccaggatctaccagatact
cgcgatctattccactgtggccagttcattggtgctggtagtctccctgggtgctatcagcttctggatg
tgctctaatggttctcttcagtgtagaatatgtatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>1698</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1698</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaagactatcattgctctctctaacattctctgtctggtgttcgctcaga
aaattcctggcaatgacaatagcacagccacgctgtgcctcgggcaccatgcagtcccaaacggaaccat
cgtgaagacaatcacaaatgaccgaattgaagtgaccaatgctactgagcttgtccagaactcctccata
ggtgaaatatgcggctcgcctcatcagatccttgatggagggaactgcacactaatagatgctctattgg
gtgaccctcagtgtgacggctttcagaataaggagtgggacctctttgttgaaaggtcccgggccaacag
caactgttacccttatgatgtgccagattatgcctcccttaggtccctagttgcctcatccggcacactg
gagtttaaaaatgagagcttcaattggactggcgtcaagcaaaacggaacaagttctgcgtgcataaggg
gctctagtagtagcttctttagtcggttaaattggctgaccagcttaaacaacatatatccagcacagaa
cgtgactatgccaaacaaggaacaatttgacaagttgtacatttggggggtgcaccacccggatacggac
aagaaccaaatctccctgtttgctcagtcatcaggacgtatcacagtatctaccaagcgcagccagcagg
ccgtcatcccaaatatcggctctaggcccagaatccgcggtatccctagtagaatctccatctattggac
aattgtaaaacccggagacattctgttgattaacagcaccgggaatctcattgctcctcgcggttacttc
aaaatccgaagcgggaaatcttccataatgcggtcagatgcacccattggcaaatgtaagtctgaatgca
tcactccaaatgggagcattcccaatgacaagccgttccaaaatgtaaaccgcatcacctacggggcctg
tcccagatatgttaagcaaagcaccctgaaattggcaacaggcatgcgaaatgttcccgagaaacagacc
cgtggcatctttggcgccatagcgggtttcatagagaatggatgggagggaatggtggatggttggtacg
gtttcaggcaccagaactctgagggaagaggacaggcggcagatttgaagagcactcaggccgcaatcga
tcaaatcaatgggaagctgaatcggctcatcgggaaaaccaacgagaaattccatcagattgagaaggaa
ttctcagaggtcgaaggccgggtgcaggaccttgagaaatatgtggaggacactaagatcgatttatggt
catacaacgcggagcttctggtggccctggagaaccagcatacgattgacctcactgactcggaaatgaa
caaactgtttgagaaaaccaagaagcagctgagggaaaatgctgaggatatgggcaatggttgtttcaag
atttaccacaaatgcgacaatgcctgcatcggctcaataagaaacgaaacctatgaccacaatgtgtacc
gcgatgaagcactgaacaaccggttccagatcaagggagtcgagctgaagtctgggtacaaagattggat
cctctggatttccttcgccatgagttgtttcctgctgtgtattgctctgctcgggttcatcatgtgggcc
tgccagaagggcaacattagatgcaacatttgcatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>1752</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1752</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaggccatcattgtgctgctcatggttgtgacaagcaatgctgaccgga
tctgcactgggatcacctcttccaactcacctcacgtggtcaagacagctactcaaggagaggtgaatgt
cactggtgtgattccactgaccacaactcccaccaagagctacttcgccaacctcaaaggcaccaggact
agagggaaactgtgcccagactgtctcaactgcaccgatctggacgtggcccttggcaggcctatgtgcg
tcggaactacacccagtgctaaggcatccatcctgcacgaagttagaccagtgacctctgggtgcttccc
tatcatgcatgacaggaccaagattaggcagcttcctaatctgttgagaggatatgagaaaatcaggctg
agcactcagaacgtgattgacgcagaaaaggctccaggtggaccctacagacttggaacctctggctcct
gtcctaacgccactagcaagatcggcttctttgccaccatggcttgggcagtcccaaaggacaactacaa
gaatgctactaacccactgacagtggaggttccctacatctgtaccgaaggagaggaccagatcaccgtc
tggggattccactctgataacaagacccaaatgaagagcctctatggagactccaatcctcagaagttca
ctagctctgccaatggagtgacaactcactatgtctcccagattggtgactttccagatcaaaccgaaga
cggaggcctgcctcagagtggcagaatcgtcgtggattacatgatgcagaaaccagggaagaccggaact
atcgtgtaccaaagaggtgtcttgctgcctcagaaggtgtggtgtgcctctggcaggagcaaggtgatca
aaggatccctgcccttgattggtgaggcagactgcctccatgaggagtacggtggattgaacaagagcaa
accttactatacaggtaagcatgcaaaagccattggcaactgtcctatctgggtgaagactccactgaaa
ctcgccaatgggaccaagtacagaccacctgctaaactgttgaaggagagaggcttctttggagccattg
ctgggttcctcgagggaggctgggaaggaatgatcgctggttggcacggctacactagccacggagctca
tggtgtggcagttgctgccgacctgaagtccacccaagaagccatcaacaagattactaagaatctcaac
agcttgagtgagctggaagtcaagaatcttcagaggctgtctggagccatggatgagctccacaacgaaa
tcctggagcttgatgagaaagtggatgacctcagagctgacactatatcctctcagatcgagcttgctgt
cttgctgagcaacgaaggaatcattaacagtgaggacgagcacctcttggcactggagcggaaactcaag
aaaatgctgggtccctccgccgtggacatcggaaatggatgctttgaaactaaacacaagtgcaaccaga
cctgcttggacaggattgctgcaggcacattcaatgctggcgagttctcacttcccacttttgattcctt
gaacatcacagctgccagcttgaatgacgatggcctggacaaccacaccattctgctctattactccact
gctgcctctagcttggctgtgaccctgatgcttgccatcttcatcgtctacatggtgagcagagacaacg
tttcctgcagcatctgtctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>1746</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1746</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaggccataattgtgctgctcatggtggtaacgtccaatgcagaccgaa
tctgcactgggatcacgtcgtcaaactcaccacatgtcgtcaaaactgctactcagggtgaggtcaacgt
gactggtgtaatcccactgaccacgacacccaccaaatcgcattttgcgaatctcaaagggacagaaacc
cgcgggaagctatgccctaaatgcttaaactgcacagacttagatgtagccttgggccggccaaagtgca
ctgggaaaatcccctctgccagggtgtctatcctccacgaagtcaggcctgttacctctgggtgctttcc
tatcatgcacgatcggacaaagattagacagctgcctaaccttctccgaggatatgagcatgtcaggctt
agcactcacaacgttatcaatacagaggacgcaccaggagggccctacgagatcggcacctccgggagct
gtctgaacattaccaatggcaagggattcttcgccacaatggcttgggccgtgccaaagaacaaaacagc
caccaatccgttaacaatagaggtaccgtatatttgtactgaggaggaagaccagattaccgtttggggg
ttccactccgacgacgagacacaaatggcaaggctctatggggattcaaagccccagaagttcacctcct
ctgccaacggcgtgaccacacactacgtctcacagattggtggctttccaaatcagaccgaggacggcgg
gctaccgcagagtggcagaattgtggttgactacatggtgcaaaaatctggcaaaacgggcaccattacc
tatcaaagaggtattctgctgcctcaaaaggtgtggtgcgcaagtggcaagagcaaggtgatcaagggat
ccttgcccttaattggcgaagcggattgcctccatgagaaatacggtggactgaacaaaagcaagcctta
ttacacaggggagcacgccaaggccatcggaaattgtccaatatgggtgaaaactcccttgaagctggcc
aatggcaccaaatatcgtcctcctgccaaactgctgaaggaacgcggtttcttcggcgccattgctggtt
tcttagagggcggatgggaaggaatgattgccggttggcacggctacacatcccatggggcacatggagt
ggcggtggcagcggaccttaagagcactcaggaggccatcaacaagatcacaaagaatctcaacagcttg
agtgagctggaagtgaagaatcttcagcgtctaagcggcgccatggacgaactccacaacgaaatactcg
aactagatgagaaagtggatgatctgcgggctgatacaataagctcacagatcgaactcgcagtcctgct
ttccaacgagggcatcataaacagtgaggatgaacatctcttggcgcttgagagaaagctgaagaaaatg
ctggggccctctgctgtcgagataggaaatggatgttttgaaaccaaacacaagtgtaaccagacctgtc
tggaccgcatcgctgctggtacctttgatgcaggagaatttagcctgcccacctttgatagcctgaacat
tactgctgcatctctgaatgacgatggcttggacaatcacactatcctgctttactactcaactgctgcc
tccagtttggctgtgacgctgatgatcgctatcttcgtggtttatatggtcagtcgggacaatgtttctt
gctccatttgtctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Иммунобиологическое средство и способ его использования для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2 (варианты) | 2020 |
|
RU2720614C1 |
| Иммунобиологическое средство для индукции иммунного ответа против вируса Ласса и способ его применения | 2024 |
|
RU2823965C1 |
| Вакцина против гриппа типа А, гриппа типа B и COVID-19 | 2021 |
|
RU2751485C1 |
| Средство для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2 в жидкой форме (варианты) | 2021 |
|
RU2743963C1 |
| Иммунобиологическое средство и способ его использования для индукции специфического иммунитета к вирусу ближневосточного респираторного синдрома (варианты) | 2018 |
|
RU2709659C1 |
| Экспрессионный вектор на основе аденовируса человека 5 серотипа, индуцирующий кросс-протективный иммунитет к вирусам гриппа А субтипа Н1, и фармацевтическая композиция на его основе | 2023 |
|
RU2802753C1 |
| Применение средства для индукции специфического иммунитета против вируса тяжелого острого респираторного синдрома SARS-CoV-2 у детей | 2021 |
|
RU2761904C1 |
| Иммунобиологическое средство и способ его использования для индукции специфического иммунитета против вирусов SARS-CoV-2 вариант B.1.617.2 (Delta) и SARS-CoV-2 вариант B.1.1.529 (Omicron) (варианты) | 2022 |
|
RU2779634C1 |
| Иммунобиологическое средство для индукции иммунного ответа против вируса гриппа и его применение | 2024 |
|
RU2838904C1 |
| Экспрессионный вектор на основе аденовируса человека 5 серотипа индуцирующий кросс-протективный иммунитет к вирусам гриппа А субтипа H3 и фармацевтическая композиция на его основе. | 2023 |
|
RU2814189C1 |



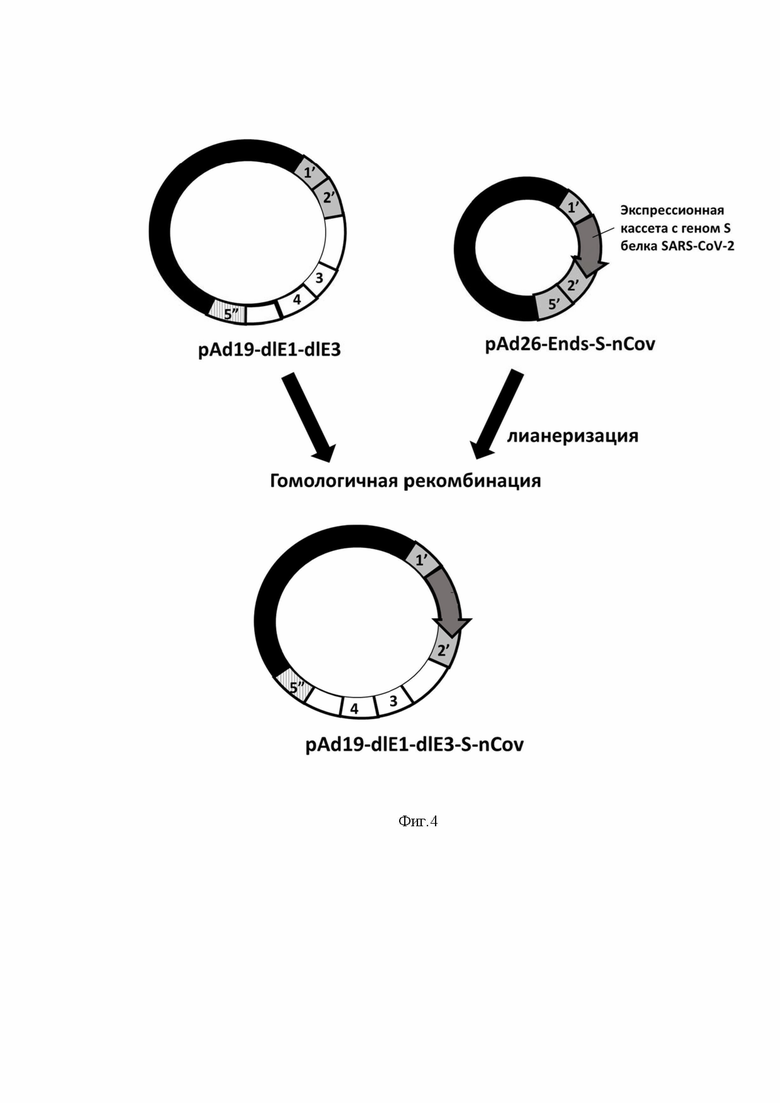




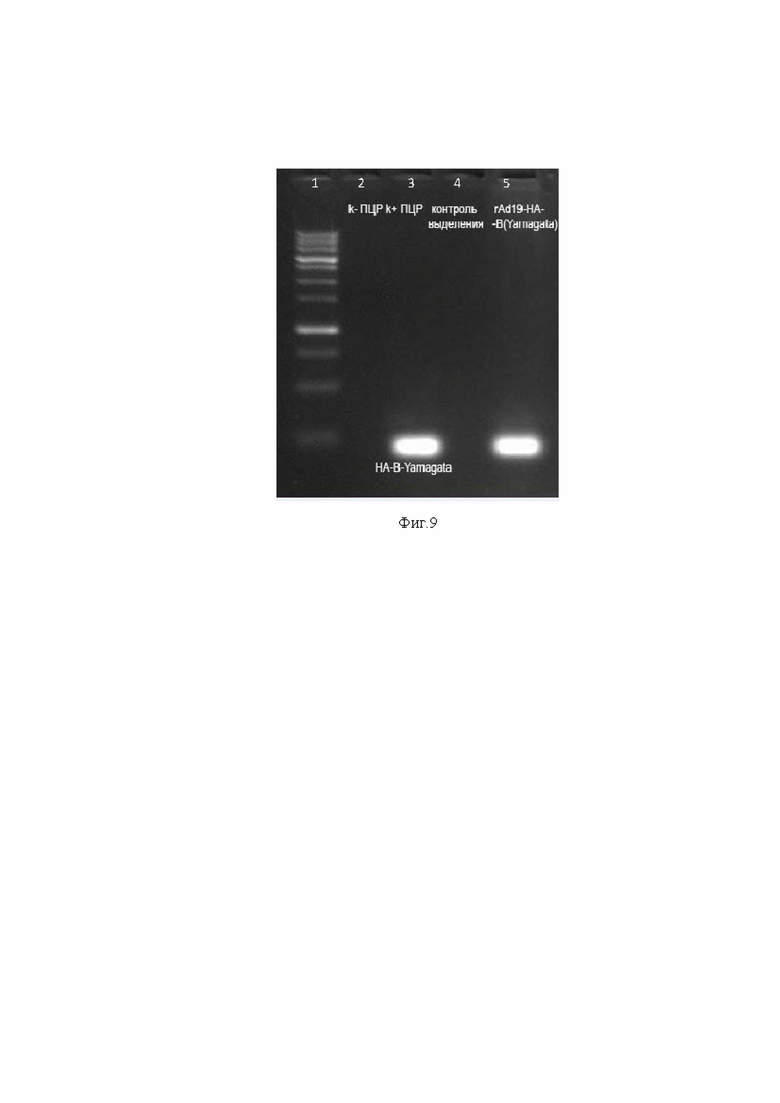

Группа изобретений относится к биотехнологии, иммунологии и вирусологии. Создан экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с целевым геном; вариабельный участок на правом конце генома размером до 1000 п.о. В частном варианте исполнения экспрессионного вектора константная последовательность до экспрессионной кассеты имеет последовательность SEQ ID NO:1, а после экспрессионной кассеты имеет последовательность SEQ ID NO:2. Экспрессионная кассета в качестве гена вакцинного антигена может содержать SEQ ID NO:3, или SEQ ID NO:4, или SEQ ID NO:5, или SEQ ID NO:6, или SEQ ID NO:7. Также описан способ получения экспрессионного вектора, отличающийся тем, что в нем используется челночная плазмида, содержащая последовательность, которая гомологична участку генома аденовируса, серотип которого отличается от 19. Группа изобретений обеспечивает создание экспрессионного вектора на основе аденовируса человека 19 серотипа, который способен индуцировать иммунный ответ к вакцинному антигену. 3 н. и 6 з.п. ф-лы, 10 ил., 5 табл., 9 пр.
1. Экспрессионный вектор на основе генома аденовируса человека 19 серотипа, содержащий: константную последовательность с делецией Е1 и Е3 областей, внутри которой находится экспрессионная кассета с целевым геном; вариабельный участок на правом конце генома размером до 1000 п.о.
2. Экспрессионный вектор по п.1, в котором константная последовательность имеет последовательность SEQIDNO:1 до экспрессионной кассеты и последовательность SEQIDNO:2 после экспрессионной кассеты.
3. Экспрессионный вектор по п.1, в котором экспрессионная кассета в качестве целевого гена содержит ген S белка вируса SARS-CoV-2 SEQIDNO:3.
4. Экспрессионный вектор по п.1, в котором экспрессионная кассета в качестве целевого гена содержит генгемагглютинина вируса гриппа А H1N1 SEQIDNO:4.
5. Экспрессионный вектор по п.1, в котором экспрессионная кассета в качестве целевого гена содержит генгемагглютинина вируса гриппа А H3N2 SEQIDNO:5.
6. Экспрессионный вектор по п.1, в котором экспрессионная кассета в качестве целевого гена содержит генгемагглютинина вируса гриппа B/Yamagata SEQIDNO:6.
7. Экспрессионный вектор по п.1, в котором экспрессионная кассета в качестве целевого гена содержит генгемагглютинина вируса гриппа B/Victoria SEQIDNO:7.
8. Способ получения экспрессионного вектора по п.1, отличающийся тем, что в нем используется челночная плазмида, содержащая последовательность, которая гомологична участку генома аденовируса, серотип которого отличается от 19.
9. Применение экспрессионного вектора по п.1 для индукции иммунного ответа к целевому антигену.
| Dennis Lapuente, Jana Fuchs, Protective mucosal immunity against SARS-CoV-2 after heterologous systemic prime-mucosal boost immunization, Nature Communications, 26 November 2021, найдено в интернете 31.08.2023 https://www.nature.com/articles/s41467-021-27063-4 | |||
| WO 2020041361 A1, 27.02.2020 | |||
| C | |||
| Thirion, Hanns Lochmuller, Z | |||
| Ruzsics et al |
Авторы
Даты
2024-01-17—Публикация
2023-04-16—Подача