ОБЛАСТЬ ТЕХНИКИ
Изобретение относится к персонализированной медицине для онкологических заболеваний, а именно к системе обнаружения химерных продуктов генов во всех типах опухолевых образцов, основанной на анализе данных РНК-секвенирования, а именно детекции дисбаланса покрытия экзонов гена.
УРОВЕНЬ ТЕХНИКИ
Химерные гены образуются в результате структурных изменений хромосом и являются характерной чертой генома раковых клеток. Такие изменения генома могут служить драйверными мутациями канцерогенеза, если в образовании химерного продукта принимает участие протоонкоген или ген-супрессор опухолевого роста. Особый интерес представляют химерные продукты, одним из партнеров которых являются гены рецепторных тирозинкиназ, в частности, гены ALK, ROS1, MET, RET, NTRK1, NTRK2, NTRK3, FGFR1, FGFR3, FLT1, FLT4. Так, транслокации генов ALK, ROS1, RET, NTRK1, NTRK2, NTRK3, FGFR1, FGFR3, FLT1, FLT4 - клинически значимые биомаркеры, важные для назначения противораковой терапии (Hallberg, Palmer, 2013, doi: 10.1038/nrc3580; Sorokin et al., 2022, doi: 10.1177/17588359221144108).
При этом химерные гены состоят из двух частей (названных здесь и далее 5’- u 3’-частями согласно их положению в составе химерного гена). В подавляющем большинстве случаев, функционально значимые химерные онкогены содержат фрагмент гена рецепторной тирозинкиназы в составе 3’-части. При этом такая 3’-часть химерного онкогена содержит последовательность полного тирозинкиназного домена. Это обеспечивает активацию сигнальных путей, усиливающих пролиферативный потенциал опухолевых клеток. С другой стороны, 5’- часть химерного онкогена может быть представлена фрагментами самых разных генов, которые, как правило, обладают более высоким уровнем экспрессии, чем ген тирозинкиназного рецептора, в норме экспрессирующийся на слабом уровне. В результате геномной перестройки 5’-часть сильно экспрессирующегося гена оказывается связана с 3’- частью слабо экспрессирующегося гена рецепторной тирозинкиназы, содержащей последовательность тирозинкиназного домена. В результате существенно усиливается связанная с этим тирозинкиназная активность.
Например, описано более тридцати 5’-генов-партнеров, которые объединяются с 3’-частью гена ALK, содержащей последовательность тирозинкиназного домена (Hallberg, Palmer, 2013, doi: 10.1038/nrc3580; Lovly et al., 2014, doi: 10.1038/nm.3667). К опухолям, где детектированы химерные гены ALK, относятся крупноклеточная лимфома, диффузная крупноклеточная В-клеточная лимфома, глиома, немелкоклеточный рак легкого, колоректальный рак, рак молочной железы, яичников и пищевода (Hallberg, Palmer, 2013, doi: 10.1038/nrc3580). Транслокации ALK не являются редкостью и обнаруживаются примерно в 8% случаев немелкоклеточного рака легкого (Kris et al., 2014, doi: 10.1001/jama.2014.3741).
Кризотиниб, церитиниб, алектиниб, бригатиниб и лоратиниб одобрены FDA США для лечения немелкоклеточного рака легкого с химерным геном ALK. Ответ на терапию, ингибирующую ALK, также описан в исследованиях почечноклеточного рака и рака толстой кишки (Drilon et al., 2017, doi: 10.1158/2159-8290).
Кроме того, разработаны также препараты, специфично блокирующие активность химерных онкогенов, содержащих части генов ROS1, RET, NTRK1, NTRK2, NTRK3, FGFR1, FGFR3, FLT1, FLT4. Так, для лечения пациентов, в опухолях которых обнаруживаются химеры с геном ROS1, применяют препараты кризотиниб, церитиниб, бригатиниб, энтректиниб; при наличии химер с геном RET - препараты пралсетиниб и селперкатиниб; при наличии химер с генами NTRK1, NTRK2, NTRK3 - препараты ларотректиниб и энтректиниб; при наличии химер с генами FGFR1, FGFR2, FGFR3 - препарат пемигатиниб; при наличии химер с генами FLT1, FLT4 - препарат селперкатиниб (Sorokin et al., 2022, doi: 10.1177/17588359221144108).
Ввиду высокой значимости наличия транслокаций вышеперечисленных генов для эффективного подбора противораковой терапии, разработан ряд тестов для обнаружения данного типа биомаркеров. Такие тесты используют как «классические» подходы, основанные на методах ПЦР, флуоресцентной in situ гибридизации (FISH) или на иммуногистихимическом исследовании (ИГХ), а также инновационные подходы, основанные на секвенировании нового поколения (NGS): на РНК-секвенировании и на геномном секвенировании опухолей (Sorokin et al., 2022, doi: 10.1177/17588359221144108).
Например, для гена ALK предложен ряд способов анализа транслокаций с часто встречающимся геном-партнером EML4 с использованием специфических наборов праймеров, позволяющих амплифицировать исследуемый фрагмент и анализировать результаты с помощью полимеразной цепной реакции (ПЦР) в реальном времени, капиллярного или обычного гель-электрофореза, а также детекции методом FISH (патентные заявки WO 2011087709 опубл.2011-07-21, WO 2011095894 опубл. 2011-08-11, RU 2509153 C1 опубл. 2014-03-10). Также предложен метод детекции продуктов амплификации кДНК EML4-ALK с использованием флуоресцентной гибридизации с биочипами (патентная заявка WO 2010132888 опубл. 2010-11-18). Существенное ограничение всех предложенных методик - детекция ограничена наборами праймеров, то есть методы применимы для анализа только одного клинически значимого биомаркера: химерные гены ALK с геном EML4.
Антитела для обнаружения транслокаций ALK Ventana ALK (D5F3) CDx Assay (Ventana Medical Systems, Tucson, AZ) методом ИГХ одобрены FDA США и зарегистрированы в РФ РУ № РЗН 2020/9786 от 25.03.2020. Стоит отметить, ИГХ детектирует уровень экспрессии белков тирозинкиназных рецепторов, что служит лишь косвенным признаком наличия транслокации. В связи с чем нередко результаты анализа ИГХ не согласуются с данными, полученными методами ПЦР или FISH (Mok T, Peters S, Camidge DR, et al. 2021).
Другие примеры стандартных методов детекции химер приведены ниже. Для детеции химер генов NTRK1-3 используются наборы VENTANA pan-TRK (Roche), для детекции химер генов FGFR1-3 используют наборы Cytotest: FGFR1 Break Apart FISH Probe Kit u FGFR3 Break Apart FISH Probe Kit. Для детекции химер ROS1 используют набор VENTANA BenchMark ULTRA anti-ROS1 (Roche). Для детекции перестроек RET используют набор Cytotest RET Break Apart FISH Probe Kit.
Кроме того, эти и другие перестройки в генах рецепторных тирозинкиназ также детектируются с помощью методов NGS - либо непосредственно в виде химерного РНК-транскрипта, либо на уровне геномной ДНК опухоли (Sorokin et al., 2022, doi: 10.1177/17588359221144108).
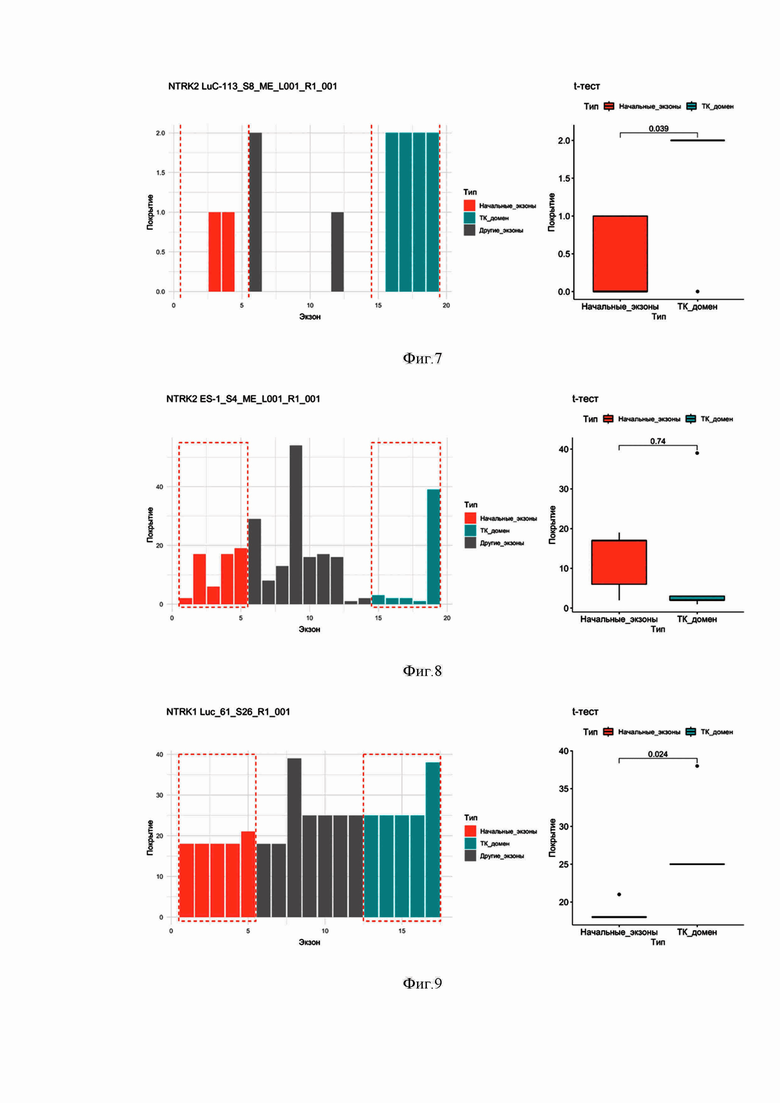
Наиболее близким к заявляемому подходу является способ детекции химерных генов, основанный на детекции внутреннего дисбаланса экспресссии генов методом амплификации в реальном времени 5’ и 3’-частей гена (US11021758B2, опубл. 2021-06-01). При образовании химерных продуктов 5’ и 3’-части гена оказываются под контролем различных регуляторных элементов, что может приводить к повышению уровня экспрессии части гена, оказавшейся слитой с более сильным промотором. В частности, клинической значимостью при диагностике и выборе терапии онкологических заболеваний обладают химерные гены, образованные в результате слияния 3’-части потенциального протоонкогена с более сильным промотором. Поэтому авторы предлагают амплифицировать в реальном времени 5’ и 3’-части гена (нетранслируемые области, UTR, untranslated region) набором разработанных праймеров и затем рассчитывать разницу экспрессии (IDE, Intragenic Differential Expression) по формуле:
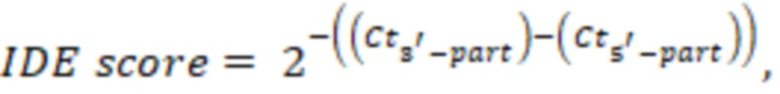
Ct - пороговый цикл амплификации.
Превышение IDE порогового значения, определенного экспериментально, свидетельствует о значительной разнице в уровнях экспрессии 5’ и 3’-частей гена и, следовательно, об образовании его химерных генов.
Описанный в патенте US11021758B2 метод обладает возможностью детектировать химерные продукты исследуемого гена независимо от его 5’-гена партнера и точного расположения точки разрыва/химеризации. Однако позволяет анализировать дисбаланс экспрессии только ограниченного числа генов, заданного набором праймеров. Кроме того, генетический полиморфизм в участках посадки ПЦР-праймеров способен привести к получению ложноположительных или ложноотрицательных результатов и, тем самым, снизить информативность теста.
Мы предлагаем подход для детекции внутреннего дисбаланса экспрессии экзонов генов рецепторных тирозинкиназ, основанный на анализе данных глубокого секвенирования тотальной мРНК опухолевого образца. РНК-секвенирование не ограничено набором специфических праймеров и потенциально позволяет детектировать дисбаланс всех экспрессирующихся в образце генов. Также применение РНК-секвенирования позволяет объединить детекцию химерных генов с определением других клинически значимых биомаркеров в одной экспериментальной процедуре.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Задачей, решаемой настоящим изобретением, является создание эффективного подхода к детекции структурных изменений генов тирозинкиназных рецепторов в раковых опухолях. Описанный в настоящем изобретении подход заключается в анализе характера экспрессии генов с целью детектировать дисбаланс экспрессии 5’ и 3’-частей гена. При этом особое внимание уделяется фрагментам, кодирующим тирозинкиназный домен. Описанный в изобретении подход решает задачу детекции клинически значимого биомаркера в различных типах опухолевых образцов, в том числе и в фиксированных в формалине парафинизированных образцах.
В качестве исходных данных используются результаты транскриптомных исследований (РНК-секвенирования) образцов клеток и тканей индивидуальных онкобольных. В качестве стартового биоматериала для исследования используются свежие, парафинизированные или законсервированные иным путем образцы опухолевой ткани. Исследованием, предоставляющими исходные данные для алгоритма, может быть омиксное генетическое профилирование: высокопроизводительное профилирование экспрессии генов с использованием глубокого секвенирования тотальной мРНК (РНК-секвенирование).
На основе результатов профилирования генной экспрессии детектируется дисбаланс экспрессии 5’ и 3’-частей гена рецепторной тирозинкиназы. При этом наличие дисбаланса в экспрессии 5’ и 3’-частей свидетельствует о наличии в исследуемом образце химерного продукта анализируемого гена.
Указанная задача решается путем оценки характера покрытия экзонов гена прочтениями РНК-секвенирования и состоит по меньшей мере из следующих этапов:
(а) получают образец ткани пациента, имеющий опухолевых онкопролиферативный фенотип;
(б) получают данные по экспрессии тотальной мРНК или РНК выбранной панели генов, включающей гены тирозинкиназных рецепторов;
(в) картируют прочтения РНК-секвенирования на экзоны исследуемого гена и нормализуют полученное число прочтений тем или иным способом, например - нормализуют полученное число прочтений путем деления числа на длину соответствующего экзона и суммарное число прочтений в образце;
(г) сравнивают уровень покрытия экзонов 5’ и 3’-частей гена тирозинкиназного рецептора, где 3’-часть соответствует тирозинкиназному домену; детекция значимой разницы в покрытии - признак наличия химерного продукта анализируемого гена в образце. При этом для гена тирозинкиназного рецептора анализируют покрытие экзонов, кодирующих тирозинкиназный домен, и сравнивают их покрытие с экзонами того же гена, расположенными на его 5’-конце; например, сравнивают покрытие экзонов, кодирующих тирозинкиназный рецептор, с покрытием такого же числа экзонов на 5’-конце гена, начиная с первого экзона включительно.
При осуществлении изобретения достигается следующий технический результат: разработан новый, эффективный способ детекции клинически значимых химерных онкогенов тирозинкиназных рецепторов ALK, ROS1, RET, NTRK1, NTRK2, NTRK3, FGFR1, FGFR3, FLT1, FLT4 в различных типах опухолевых образцов, включая фиксированные в формалине парафинизированные ткани. Данный способ может быть автоматизирован, что предотвращает потенциальные ошибки, связанные с ручным расчетом, и позволяет потенциально обнаружить химерные продукты всех экспрессируемых в образце генов тирозинкиназных рецепторов, характерные для конкретной опухоли исследуемого пациента.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
Сущность изобретения поясняется рисунками, на которых;
Фиг. 1. Диаграмма покрытия экзонов гена ALK прочтениями РНК-секвенирования в парафинизированном образце ткани рака поджелудочной железы РС-10 с химерным транскриптом, содержащим последовательность тирозинкиназного домена ALK.
Фиг. 2. Диаграмма покрытия экзонов гена ALK прочтениями РНК-секвенирования в парафинизированном образце ткани глиобластомы ВТ-23 без химерного транскрипта, содержащего последовательность тирозинкиназного домена ALK.
Фиг. 3. Диаграмма покрытия экзонов гена ROS1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-46 с химерным транскриптом, содержащим последовательность тирозинкиназного домена ROS1.
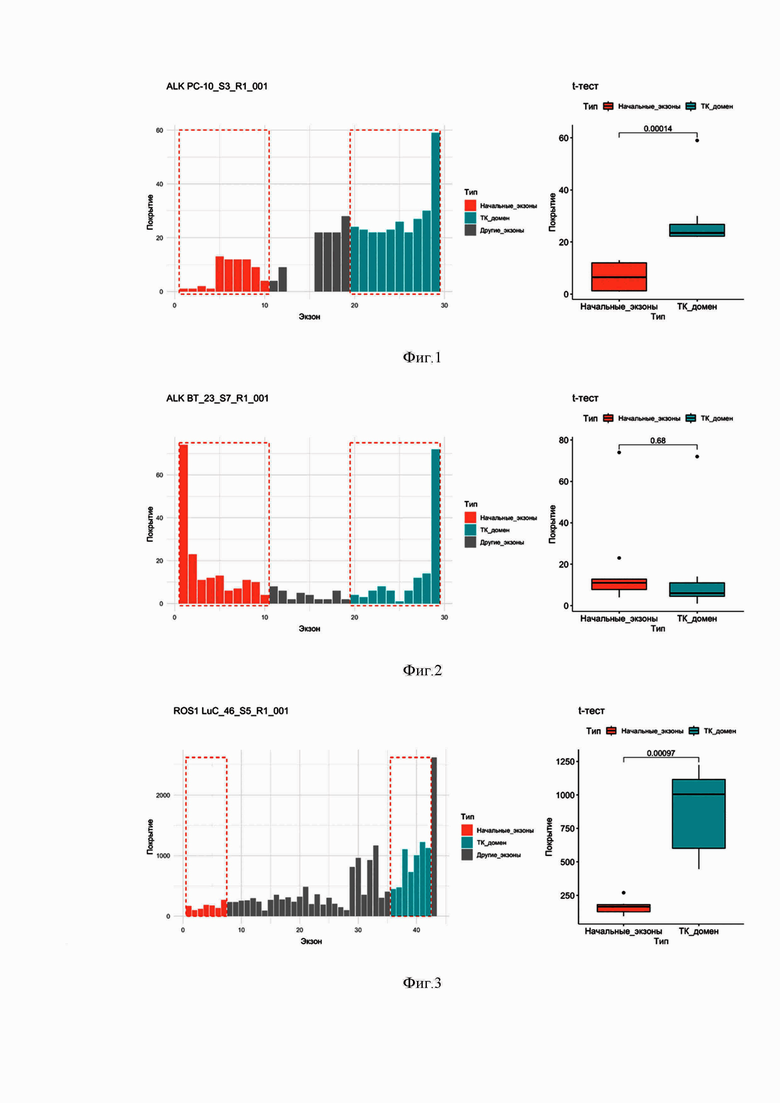
Фиг. 4. Диаграмма покрытия экзонов гена ROS1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака почки КС-108 без химерного транскрипта, содержащего последовательность тирозинкиназного домена ROS1.
Фиг. 5. Диаграмма покрытия экзонов гена RET прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-100 с химерным транскриптом, содержащим последовательность тирозинкиназного домена RET.
Фиг. 6. Диаграмма покрытия экзонов гена RET прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-53 без химерного транскрипта, содержащего последовательность тирозинкиназного домена RET.
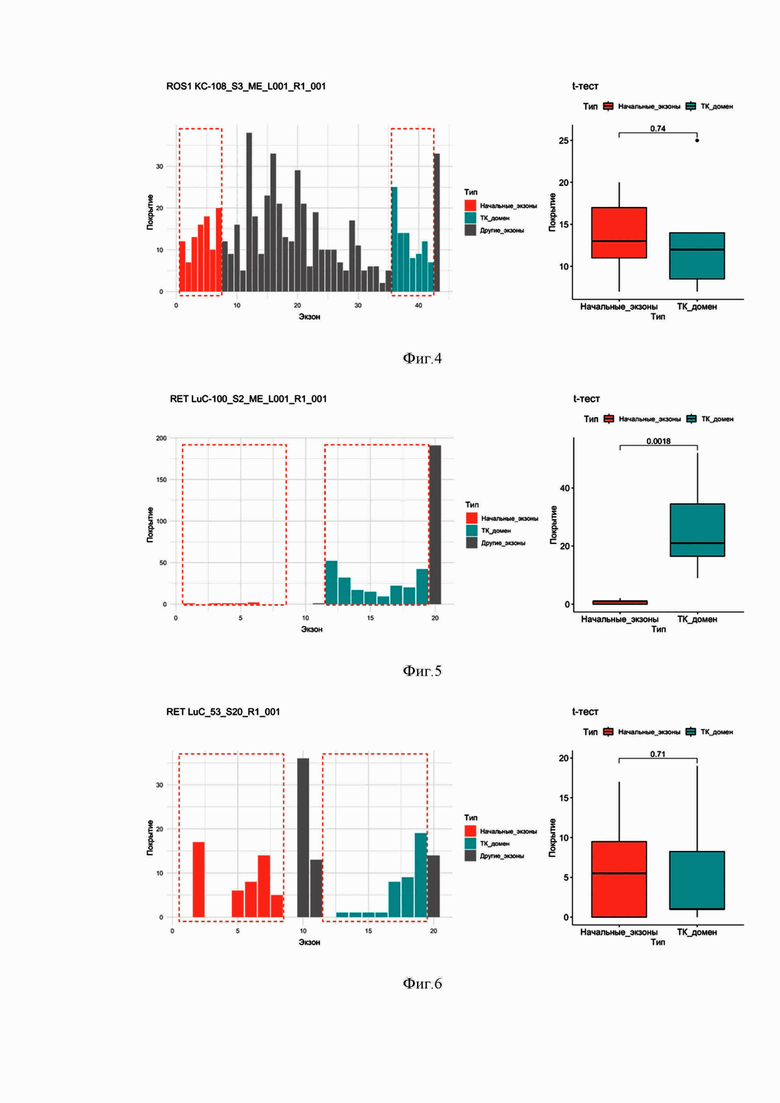
Фиг. 7. Диаграмма покрытия экзонов гена NTRK2 прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-113 с химерным транскриптом, содержащим последовательность тирозинкиназного домена NTRK2.
Фиг. 8. Диаграмма покрытия экзонов гена NTRK2 прочтениями РНК-секвенирования в парафинизированном образце ткани рака пищевода ЕС-1 без химерного транскрипта, содержащего последовательность тирозинкиназного домена NTRK2.
Фиг. 9. Диаграмма покрытия экзонов гена NTRK1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-61 с химерным транскриптом, содержащим последовательность тирозинкиназного домена NTRK1.
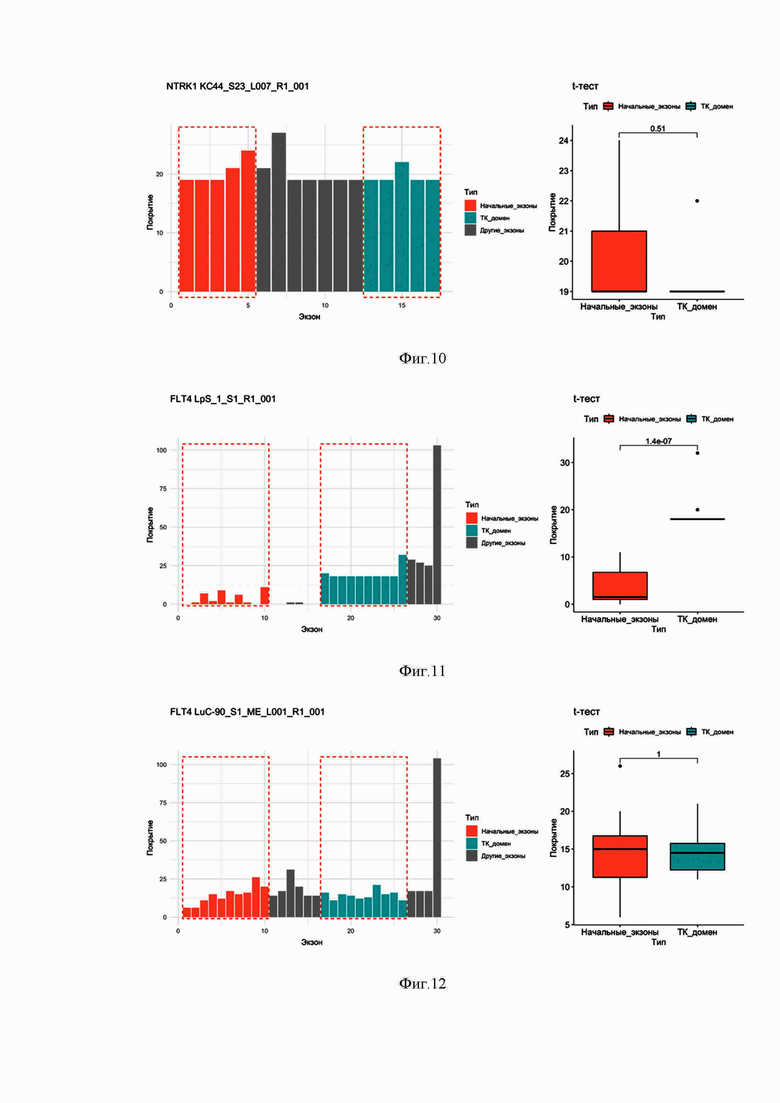
Фиг. 10. Диаграмма покрытия экзонов гена NTRK1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака почки КС-44 без химерного транскрипта, содержащего последовательность тирозинкиназного домена NTRK1.
Фиг. 11. Диаграмма покрытия экзонов гена FLT4 прочтениями РНК-секвенирования в парафинизированном образце ткани липосаркомы LpS-1 с химерным транскриптом, содержащим последовательность тирозинкиназного домена FLT4.
Фиг. 12. Диаграмма покрытия экзонов гена FLT4 прочтениями РНК-секвенирования в парафинизированном образце ткани рака легкого LuC-90 без химерного транскрипта, содержащего последовательность тирозинкиназного домена FLT4.
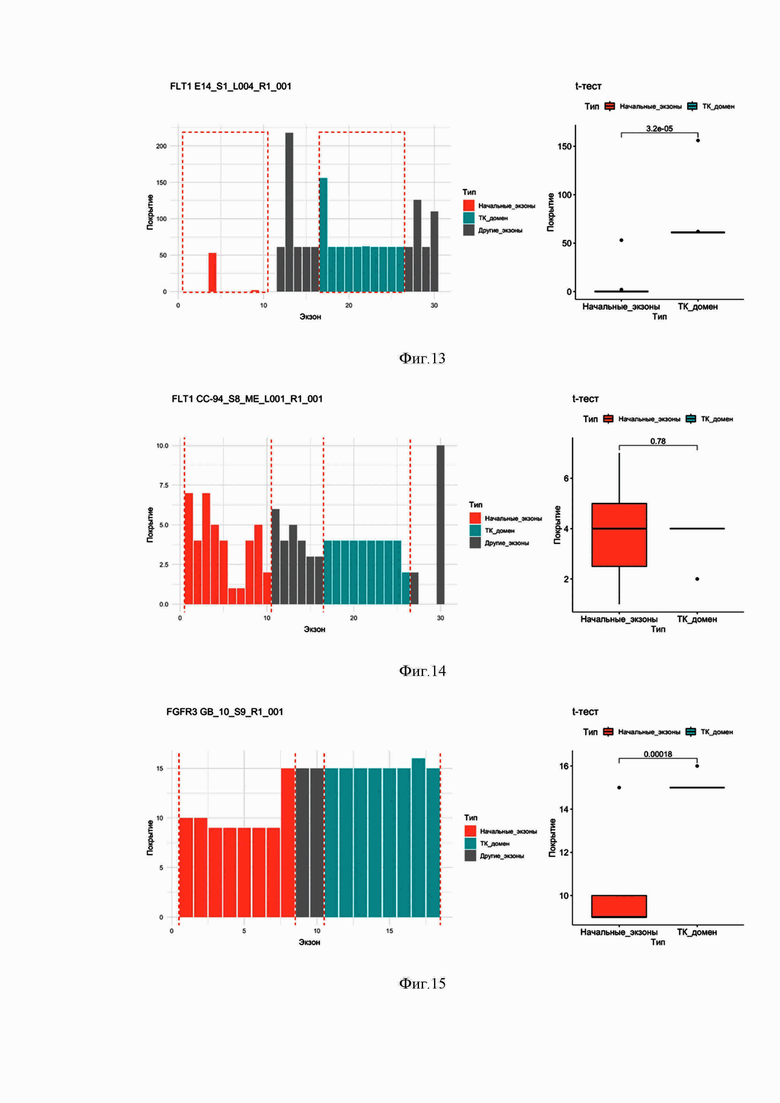
Фиг. 13. Диаграмма покрытия экзонов гена FLT1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака пищевода Е-14 с химерным транскриптом, содержащим последовательность тирозинкиназного домена FLT1.
Фиг. 14. Диаграмма покрытия экзонов гена FLT1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака толстой кишки СС-94 без химерного транскрипта, содержащего последовательность тирозинкиназного домена FLT1.
Фиг. 15. Диаграмма покрытия экзонов гена FGFR3 прочтениями РНК-секвенирования в парафинизированном образце ткани глиобластомы Gb-10 с химерным транскриптом, содержащим последовательность тирозинкиназного домена FGFR3.
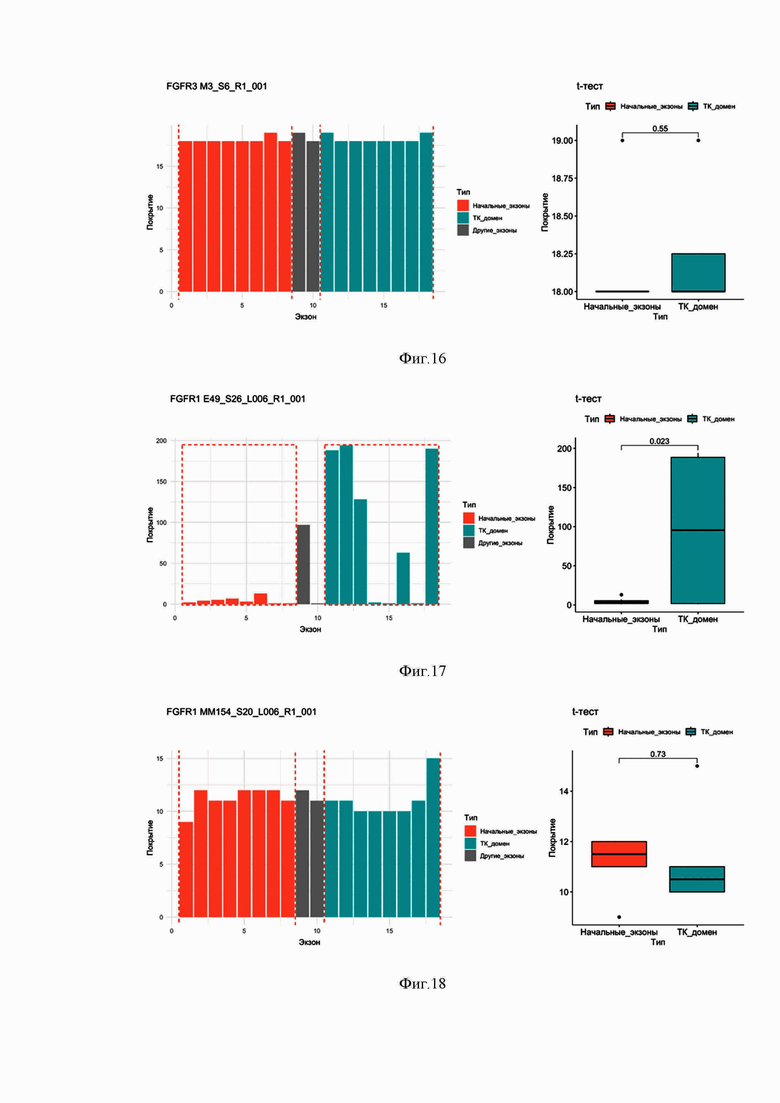
Фиг. 16. Диаграмма покрытия экзонов гена FGFR3 прочтениями РНК-секвенирования в парафинизированном образце ткани меланомы М-3_S4 без химерного транскрипта, содержащего последовательность тирозинкиназного домена FGFR3.
Фиг. 17. Диаграмма покрытия экзонов гена FGFR1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака пищевода Е-49 с химерным транскриптом, содержащим последовательность тирозинкиназного домена FGFR1.
Фиг. 18. Диаграмма покрытия экзонов гена FGFR1 прочтениями РНК-секвенирования в парафинизированном образце ткани рака щитовидной железы ММ-154 без химерного транскрипта, содержащего последовательность тирозинкиназного домена FGFR1.
Эти рисунки не охватывают и, кроме того, не ограничивают весь объем вариантов реализации данного технического решения, а представляют собой только иллюстративный материал частного случая его реализации.
ВАРИАНТ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из».
Под «пациентом», следует понимать человека (предпочтительно) или другое млекопитающее. Под «тканью» пациента или субъекта следует понимать значение, принятое в медицинской литературе - это система клеток и межклеточного вещества, объединённых общим происхождением, строением и выполняемыми функциями. В описании данного изобретения это может быть кровь, твердая ткань различного происхождения (например, эпителиальная, соединительная, нервная или мышечная) или часть любого органа пациента или субъекта.
Под образцом ткани пациента, имеющим пролиферативный фенотип, понимают образец опухолевой ткани, клетки которой обладают способностью к неконтролируемому делению в результате патологического изменения (обычно в результате изменения генетического аппарата клеток). Такой образец может быть частью доброкачественной или злокачественной опухоли.
Под пролиферативным или онкологическим заболеванием понимают заболевание, характеризуемое патологическим изменением генетического аппарата клеток, приводящее к неконтролируемому делению определенной популяции клеток. Примерами пролиферативных заболеваний могут служить миелопролиферативные заболевания, лимфопролиферативные заболевания, пролиферативные заболевания соединительной ткани и другие заболевания.
Под данными по экспрессии тотальной мРНК следует понимать данные, показывающие абсолютное или относительное количество всех или более 300 видов молекул мРНК в образце.
Под данными по экспрессии РНК выбранной панели генов, включающей гены тирозинкиназных рецепторов, следует понимать данные, показывающие абсолютное или относительное количество прочтений РНК-секвенирования, покрывающих экзоны не менее одного гена тирозинкиназного рецептора.
Под “картированием прочтений” стоит понимать биоинформатический метод анализа результатов секвенирования, состоящий в определении позиций в референсном геноме, откуда с наибольшей вероятностью могло быть получено каждое конкретное прочтение.
Под “покрытием экзона” стоит понимать количество прочтений, которые частично или полностью картируются на какой-то участок экзона исследуемого гена.
Под “5’-частью” стоит понимать часть полинуклеотида (гена или транскрипта), расположенную ближе к 5’-концу полинуклеотида относительно 3’-части. Применительно к химерным продуктам и транслокациям “5’-часть” означает область, расположенную в направлении 5’, то есть до точки химеризации/точки разрыва.
Под “3’-частью” стоит понимать часть полинуклеотида (гена или транскрипта), расположенную ближе к 3’-концу полинуклеотида относительно 5’-части. Применительно к химерным продуктам и транслокациям “3’-часть” означает область, расположенную в направлении 3’, то есть после точки химеризации/точки разрыва.
При описании известных химерных генов и их транскриптов “5’-часть” и “3’-часть” обозначают, как правило, регионы, расположенные по разные стороны от точки разрыва и являющиеся частями разных генов.
Если не определено отдельно, технические и научные термины в данном описании имеют стандартные значения, общепринятые в научной и технической литературе.
Представленный подход решает задачу обнаружения химерных продуктов генов рецепторных тирозинкиназ в различных типах образцов пролиферативной ткани для более эффективного назначения противораковой терапии при помощи технологии, основанной на анализе дисбаланса покрытия экзонов генов прочтениями РНК-секвенирования.
В качестве образцов патологической ткани могут браться образцы свежей, парафинизированной или законсервированной иным способом ткани. В предпочтительном варианте осуществления изобретения анализ ткани производится у пациента с пролиферативным или онкологическим заболеванием ткани. Также для патологической ткани предпочтительно проводить анализ участка ткани пациента, имеющей пролиферативный фенотип и содержащей не менее 10% опухолевых клеток. Для этого возможно проводить дополнительную очистку ткани, имеющей пролиферативный фенотип, от других окружающих ее тканей с помощью методов, известных специалистам.
Алгоритм эффективен для анализа данных по экспрессии мРНК генов в различных типах образцов.
Первой решаемой технической задачей (1) является картирование прочтений РНК-секвенирования на экзоны исследуемого гена или исследуемых генов исходя из данных тотального РНК-секвенирования или анализа РНК-секвенирования панели генов.
При этом первая техническая задача разделяется на ряд подзадач:
1.1. Получение транскриптомных данных для анализируемого образца.
1.2. Картирование прочтений на экзоны исследуемого гена или группы исследуемых генов и их нормализация.
Второй решаемой технической задачей (2) является анализ характера покрытия экзонов 5’- и 3’-частей гена для детекции дисбаланса экспрессии.
Вторая техническая задача разделяется на следующие подзадачи:
2.1. Сравнение уровня покрытия экзонов, кодирующих тирозинкиназный домен, и экзонов, расположенных на 5’-части гена, с помощью статистических методов, например, применяя t-test.
2.2. Вывод о наличии превышения уровня покрытия экзонов, кодирующих тирозинкиназный домен, над уровнем покрытия экзонов, расположенных на 5’-части гена.
Особенности технологического решения
В сравнении с существующим наиболее близким техническим решением, представленный способ обладает рядом преимуществ. Во-первых, применение РНК-секвенирования позволяет анализировать характер экспрессии всех генов в образце, то есть потенциально возможно детектировать химерные продукты всех известных генов. В то время как анализ на основе ПЦР ограничен набором праймеров, комплиментарных к конкретному гену или ограниченному набору генов. Во-вторых, в отличие от методов на основе ПЦР, данный способ не чувствителен к наличию полиморфных позиций в сайтах посадки ПЦР-праймеров, что может искажать результаты ПЦР-анализа. В-третьих, метод РНК-секвенирования позволяет комбинировать определение различных биомаркеров в одной экспериментальной процедуре, что сокращает время подбора терапии. В-четвертых, в отличие от методов, основанных на поиске химерных прочтений РНК-секвенирования, представленный метод адаптирован для анализа в том числе парафинизированных материалов, степень деградации нуклеиновых кислот в которых повышена.
Анализ дисбаланса покрытия экзонов
Для детекции значимой разницы уровней экспрессии 5’- и 3’-частей гена предложен следующий подход:
Применение статистических тестов, например t-теста, для сравнения уровня покрытия экзонов, составляющих 5’- и 3’-части гена. Детекция в результате применения статистического критерия статистически значимого превышения уровня покрытия экзонов 3’-части над 5’-частью свидетельствует о дисбалансе экспрессии соответствующего гена и, следовательно, о наличии продуктов его перестройки в образце (фиг. 1); напротив, отсутствие дисбаланса говорит об отсутствии продуктов перестройки гена (фиг. 2). Например, в качестве критерия статистической значимости может быть задано значение p-value для t-теста 0.05. Данный подход не требует дополнительной информации о характере покрытия экзонов соответствующих генов в образцах какой-либо контрольной группы, что позволяет применять его непосредственно для анализа любых наборов данных РНК-секвенирования, содержащих информацию по генам рецепторных тирозинкиназ. Для применения данного подхода не требуется предварительной информации о положении точки разрыва/химеризации для корректного разделения экзонов анализируемого гена на группы 5’ и 3’-частей, то есть расположенных до и после точки химеризации. Это достигается тем, что за 3’-часть гена принимается группа экзонов, кодирующих тирозинкиназных домен, а за 5’-часть гена принимается группа из стольких же экзонов, следующих подряд друг за другом начиная с первого экзона включительно.
Нижеследующие примеры работы системы приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Детекция химерной онкогенной перестройки гена ALK в образцах рака поджелудочной железы и глиобластомы.
Фиксированные в формалине парафинизированные образцы рака поджелудочной железы РС-10 и глиобластомы ВТ-23 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена ALK ENST00000389048.8, входящего в проект MANE (Matched Annotation between NCBI and EBI). Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов РС-10 (фиг. 1) и ВТ-23 (фиг. 2). В качестве критерия статистической значимости испоьзовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена ALK в образце РС-10 (фиг. 1), которая подтверждается статистическим тестом: t-тест, p-value = 0,00014. В то же время, в образце ВТ-23 (фиг. 2) статистически достоверной разницы между покрытием 5’- и 3’-частей гена ALK не выявлено (p-value = 0,68).
В результате анализа делается вывод о наличии химерного онкогена ALK в образце РС-10 и об отсутствии химерного онкогена ALK в образце ВТ-23.
Проведена экспериментальная проверка сделанных выводов с использованием иммуногистохимической (ИГХ) системы детекции химерных онкогенов ALK Ventana ALK (D5F3) CDx Assay (Ventana Medical Systems), зарегистрированной в РФ РУ № РЗН 2020/9786 от 25.03.2020. С использованием данной системы было подтверждено наличие химерного онкогена в образце РС-10 и отсутствие химерного онкогена в образце ВТ-23.
Пример 2. Детекция химерной онкогенной перестройки гена ROS1 в образцах рака легкого и рака почки.
Фиксированные в формалине парафинизированные образцы рака легкого LuC-46 и рака почки KC-108 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена ROS1. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов LuC-46 (фиг. 3) и KC-108 (фиг. 4). В качестве критерия статистической значимости испоьзовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена ROS1 в образце LuC-46 (фиг. 3), которая подтверждается статистическим тестом: t-тест, p-value = 0,00097. В то же время, в образце КС-108 (фиг. 4) статистически достоверной разницы между покрытием 5’- и 3’-частей гена ROS1 не выявлено (p-value = 0,74).
В результате анализа делается вывод о наличии химерного онкогена ROS1 в образце LuC-46 и об отсутствии химерного онкогена ROS1 в образце КС-108.
Проведена экспериментальная проверка сделанных выводов с использованием иммуногистохимической (ИГХ) системы детекции химерных онкогенов ROS1 VENTANA BenchMark ULTRA anti-ROS1 (Roche). С использованием данной системы было подтверждено наличие химерного онкогена в образце LuC-46 и отсутствие химерного онкогена ROS1 в образце КС-108.
Пример 3. Детекция химерной онкогенной перестройки гена RET в образцах рака легкого.
Фиксированные в формалине парафинизированные образцы рака легкого LuC-100 и LuC-53 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена RET. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов LuC-100 (фиг. 5) и LuC-53 (фиг. 6). В качестве критерия статистической значимости испоьзовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена RET в образце LuC-100 (фиг. 5), которая подтверждается статистическим тестом: t-тест, p-value = 0,0018. В то же время, в образце LuC-53 (фиг. 6) статистически достоверной разницы между покрытием 5’- и 3’-частей гена RET не выявлено (p-value = 0,71).
В результате анализа делается вывод о наличии химерного онкогена RET в образце LuC-100 и об отсутствии химерного онкогена RET в образце LuC-53.
Проведена экспериментальная проверка сделанных выводов с использованием основанной на флуоресцентной ин ситу гибридизации системы детекции химерных онкогенов RET Cytotest RET Break Apart FISH Probe Kit. С использованием данной системы было подтверждено наличие химерного онкогена в образце LuC-100 и отсутствие химерного онкогена RET в образце LuC-53.
Пример 4. Детекция химерной онкогенной перестройки гена NTRK2 в образцах рака легкого и рака пищевода.
Фиксированные в формалине парафинизированные образцы рака легкого LuC-113 и рака пищевода ЕС-1 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена NTRK2. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов LuC-113 (фиг. 7) и ЕС-1 (фиг. 8). В качестве критерия статистической значимости испоьзовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена NTRK2 в образце LuC-113 (фиг. 7), которая подтверждается статистическим тестом: t-тест, p-value = 0,039. В то же время, в образце ЕС-1 (фиг. 8) статистически достоверной разницы между покрытием 5’- и 3’-частей гена NTRK2 не выявлено (p-value = 0,74).
В результате анализа делается вывод о наличии химерного онкогена NTRK2 в образце LuC-113 и об отсутствии химерного онкогена NTRK2 в образце ЕС-1.
Проведена экспериментальная проверка сделанных выводов с использованием основанной на ИГХ системы детекции химерных онкогенов VENTANA pan-TRK (Roche). С использованием данной системы было подтверждено наличие химерного онкогена NTRK2 в образце LuC-113 и отсутствие химерного онкогена NTRK2 в образце ЕС-1.
Пример 5. Детекция химерной онкогенной перестройки гена NTRK1 в образцах рака легкого и рака почки.
Фиксированные в формалине парафинизированные образцы рака легкого LuC-61 и рака почки КС-44 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена NTRK1. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов LuC-61 (фиг. 9) и КС-44 (фиг. 10). В качестве критерия статистической значимости испоьзовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена NTRK1 в образце LuC-61 (фиг. 9), которая подтверждается статистическим тестом: t-тест, p-value = 0,024. В то же время, в образце КС-44 (фиг. 10) статистически достоверной разницы между покрытием 5’- и 3’-частей гена NTRK1 не выявлено (p-value = 0,51).
В результате анализа делается вывод о наличии химерного онкогена NTRK1 в образце LuC-61 и об отсутствии химерного онкогена NTRK1 в образце КС-44.
Проведена экспериментальная проверка сделанных выводов с использованием основанной на ИГХ системы детекции химерных онкогенов VENTANA pan-TRK (Roche). С использованием данной системы было подтверждено наличие химерного онкогена NTRK1 в образце LuC-61 и отсутствие химерного онкогена NTRK1 в образце КС-44.
Пример 6. Детекция химерной онкогенной перестройки гена FLT4 в образцах рака легкого и липосаркомы.
Фиксированные в формалине парафинизированные образцы липосаркомы LpS-1 и рака легкого LuC-90 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена FLT4. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов LpS-1 (фиг. 11) и LuC-90 (фиг. 12). В качестве критерия статистической значимости использовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена FLT4 в образце LpS-1 (фиг. 11), которая подтверждается статистическим тестом: t-тест, p-value = 1,4е-07. В то же время, в образце LuC-90 (фиг. 12) статистически достоверной разницы между покрытием 5’- и 3’-частей гена FLT4 не выявлено (p-value = 1).
В результате анализа делается вывод о наличии химерного онкогена FLT4 в образце LpS-1 и об отсутствии химерного онкогена FLT4 в образце LuC-90.
Проведена экспериментальная проверка сделанных выводов с использованием глубокого РНК-секвенирования с покрытием 100 миллионов прочтений на библиотеку и прямой детекции химерных онкогенов. С использованием данной системы было подтверждено наличие химерного онкогена FLT4 в образце LpS-1 и отсутствие химерного онкогена FLT4 в образце LuC-90.
Пример 7. Детекция химерной онкогенной перестройки гена FLT1 в образцах рака пищевода и рака толстой кишки.
Фиксированные в формалине парафинизированные образцы рака пищевода Е-14 и рака толстой кишки СC-94 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена FLT1. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов Е-14 (фиг. 13) и СC-94 (фиг. 14). В качестве критерия статистической значимости использовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена FLT1 в образце Е-14 (фиг. 13), которая подтверждается статистическим тестом: t-тест, p-value = 3,2е-05. В то же время, в образце СС-94 (фиг. 14) статистически достоверной разницы между покрытием 5’- и 3’-частей гена FLT1 не выявлено (p-value = 0,78).
В результате анализа делается вывод о наличии химерного онкогена FLT1 в образце Е-14 и об отсутствии химерного онкогена FLT1 в образце СС-94.
Проведена экспериментальная проверка сделанных выводов с использованием глубокого РНК-секвенирования с покрытием 100 миллионов прочтений на библиотеку и прямой детекции химерных онкогенов. С использованием данной системы было подтверждено наличие химерного онкогена FLT1 в образце Е-14 и отсутствие химерного онкогена FLT1 в образце СС-94.
Пример 8. Детекция химерной онкогенной перестройки гена FGFR3 в образцах глиобластомы и меланомы.
Фиксированные в формалине парафинизированные образцы глиобластомы GB-10 и меланомы М-3 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена FGFR3. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов GB-10 (фиг. 15) и M-3 (фиг. 16). В качестве критерия статистической значимости использовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена FGFR3 в образце GB-10 (фиг. 15), которая подтверждается статистическим тестом: t-тест, p-value = 0,00018. В то же время, в образце M-3 (фиг. 16) статистически достоверной разницы между покрытием 5’- и 3’-частей гена FGFR3 не выявлено (p-value = 0,55).
В результате анализа делается вывод о наличии химерного онкогена FGFR3 в образце GB-10 и об отсутствии химерного онкогена FGFR3 в образце M-3.
Проведена экспериментальная проверка сделанных выводов с использованием основанной на флуоресцентной ин ситу гибридизации системы FGFR3 Break Apart FISH Probe Kit. С использованием данной системы было подтверждено наличие химерного онкогена FGFR3 в образце GB-10 и отсутствие химерного онкогена FGFR3 в образце M-3.
Пример 9. Детекция химерной онкогенной перестройки гена FGFR1 в образцах рака пищевода и рака щитовидной железы.
Фиксированные в формалине парафинизированные образцы глиобластомы рака пищевода Е-49 и рака щитовидной железы ММ-154 были получены для исследования представленного метода.
Сначала была выделена тотальная РНК и проведено полнотраскриптомное секвенирование. Для удаления рРНК и приготовления библиотек использовали KAPA RNA HyperPrep Kit with RiboErase (HMR) (Roche Sequencing, США). Различные адаптеры KAPA UDI Primer Mix (Roche Sequencing, США) использовались для объединения образцов в одном запуске. Секвенирование проводили на приборе Illumina NextSeq 550 в режиме прочтения с одного конца, длина прочтения 50 пар оснований, расчетная глубина 30 миллионов «сырых» прочтений на образец. Качество запуска анализировалось программным обеспечением Illumina SAV. Для демультиплексирования использовали программу Illumina Bcl2fastq2 v2.17.
Для расчета уровня покрытия экзонов использовали пакет программного обеспечения BEDtools multicov с параметрами «по умолчанию». В качестве входных файлов были BAM (Binary Alignment/Map) файлы результатов РНК-секвенирования отдельно каждого соответствующего образца и BED (Browser Extensible Data) файл, содержащий координаты экзонов транскрипта гена FGFR1. Полученные значения числа прочтений нормировали на длину соответствующего экзона и глубину секвенирования образца.
Получили картину распределения прочтений для образцов Е-49 (фиг. 17) и MМ-154 (фиг. 18). В качестве критерия статистической значимости использовали значение p-value для t-теста 0.05. Очевидна значимая разница уровней покрытия 5’ и 3’-частей гена FGFR1 в образце Е-49 (фиг. 17), которая подтверждается статистическим тестом: t-тест, p-value = 0,023. В то же время, в образце MМ-154 (фиг. 18) статистически достоверной разницы между покрытием 5’- и 3’-частей гена FGFR1 не выявлено (p-value = 0,73).
В результате анализа делается вывод о наличии химерного онкогена FGFR1 в образце Е-49 и об отсутствии химерного онкогена FGFR1 в образце MМ-154.
Проведена экспериментальная проверка сделанных выводов с использованием основанной на флуоресцентной ин ситу гибридизации системы FGFR1 Break Apart FISH Probe Kit. С использованием данной системы было подтверждено наличие химерного онкогена FGFR1 в образце Е-49 и отсутствие химерного онкогена FGFR1 в образце МM-154.
Таким образом, применение предложенного способа позволяет детектировать химерные онкогены в опухолевых образцах без предварительного знания о втором гене-партнере химерного продукта или точном положении точки химеризации / позиции перестройки внутри гена рецепторной тирозинкциназы.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОЦЕНКИ ДЕТЕКЦИИ ТРАНСЛОКАЦИЙ ГЕНА ALK В ПАРАФИНИЗИРОВАННЫХ ОПУХОЛЕВЫХ ОБРАЗЦАХ С ПОМОЩЬЮ РНК-СЕКВЕНИРОВАНИЯ | 2023 |
|
RU2812346C1 |
| Набор высокоспецифичных олигонуклеотидных праймеров для детекции генетических вариантов методом высокопроизводительного секвенирования ДНК | 2024 |
|
RU2833429C1 |
| Тест-классификатор клинического ответа на лечение сорафенибом индивидуальных пациентов с раком почки | 2018 |
|
RU2747746C2 |
| ЭКСПРЕССИЯ FGFR И ЧУВСТВИТЕЛЬНОСТЬ К ИНГИБИТОРУ FGFR | 2016 |
|
RU2728674C2 |
| СПОСОБ ОЦЕНКИ СТАТУСА 14-ГО ЭКЗОНА ГЕНА MET ПО ДАННЫМ РНК СЕКВЕНИРОВАНИЯ | 2023 |
|
RU2817869C1 |
| Способ создания таргетной панели для исследования геномных регионов для выявления терапевтических биомаркеров ингибиторов иммунных контрольных точек (ИКТ) | 2023 |
|
RU2818360C1 |
| КРИСТАЛЛИЧЕСКАЯ ФОРМА (S)-N-(5-((R)-2-(2,5-ДИФТОРФЕНИЛ)-ПИРРОЛИДИН-1-ИЛ)-ПИРАЗОЛО[1,5-A]ПИРИМИДИН-3-ИЛ)-3-ГИДРОКСИПИРРОЛИДИН-1-КАРБОКСАМИДА ГИДРОСУЛЬФАТА | 2015 |
|
RU2723990C2 |
| СПОСОБ ВЫЯВЛЕНИЯ МУТАЦИЙ В СЛОЖНЫХ СМЕСЯХ ДНК | 2014 |
|
RU2613489C2 |
| ИНДИВИДУАЛИЗИРОВАННЫЕ ПРОТИВООПУХОЛЕВЫЕ ВАКЦИНЫ | 2012 |
|
RU2670745C9 |
| ИНДИВИДУАЛИЗИРОВАННЫЕ ПРОТИВООПУХОЛЕВЫЕ ВАКЦИНЫ | 2012 |
|
RU2779946C2 |
Изобретение относится к области биотехнологии, в частности к способу, позволяющему детектировать продукты экспрессии химерных генов рецепторных тирозинкиназ в широком спектре образцов патологических тканей пациентов на основе данных об экспрессии РНК. Способ основан на анализе характера экспрессии отдельных генов и поиске дисбаланса покрытия экзонов, свидетельствующем о наличии структурной перестройки гена. Подход основан на анализе транскриптомных данных, что потенциально позволяет обнаружить химерные продукты всех экспрессируемых в образце генов. Кроме того, данный способ может быть автоматизирован, что предотвращает потенциальные ошибки, связанные с ручным расчетом. Применение данного способа позволит эффективно и точно детектировать химерные транскрипты во всех типах опухолевых образцов, включая фиксированные в формалине парафинизированные образцы, степень деградации нуклеиновых кислот в которых повышена. Изобретение позволяет повысить доступность персонализированной медицины и эффективность подбора противораковой терапии. 18 ил., 9 пр.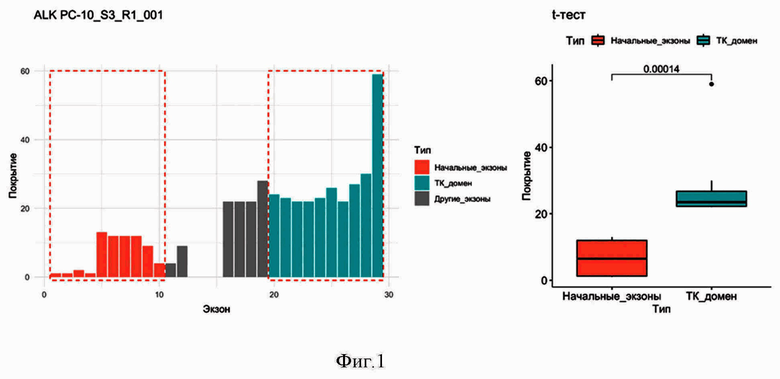
Способ детекции транслокаций генов тирозинкиназных рецепторов в образцах опухолевой ткани с использованием данных РНК-секвенирования, включающий детекцию химерных генов рецепторных тирозинкиназ при анализе данных транскриптомного секвенирования путем оценки характера покрытия гена прочтениями РНК-секвенирования, который состоит по меньшей мере из следующих этапов:
(а) получают образец ткани пациента, имеющий пролиферативный фенотип;
(б) получают данные по секвенированию тотальной мРНК или РНК выбранной панели генов, включающей гены тирозинкиназных рецепторов;
(в) картируют прочтения РНК-секвенирования на экзоны исследуемых генов тирозинкиназных рецепторов и нормализуют полученное число;
(г) сравнивают уровень покрытия экзонов 5’- и 3’-частей гена с использованием статистического теста либо группы статистических тестов, при этом детекция значимой разницы – признак наличия химерного продукта анализируемого гена в образце.
| СПОСОБ АНАЛИЗА ТРАНСЛОКАЦИЙ EML4-ALK, АССОЦИИРОВАННЫХ С ЧУВСТВИТЕЛЬНОСТЬЮ РАКА ЛЕГКОГО К ПРОТИВООПУХОЛЕВОЙ ТАРГЕТНОЙ ТЕРАПИИ | 2012 |
|
RU2509153C1 |
| US 20120115144 A1, 10.05.2012 | |||
| EP 4119675 A1, 18.01.2023 | |||
| Разработка критериев для верификации химерных транскриптов, обнаруженных в ходе анализа данных РНК-секвенирования злокачественных опухолей / Е | |||
| Н | |||
| Рабушко, М | |||
| И | |||
| Сорокин, М | |||
| В | |||
| Сунцова, А | |||
| А | |||
| Буздин // XXXV зимняя молодёжная научная школа "Перспективные | |||
Авторы
Даты
2024-03-04—Публикация
2023-12-03—Подача