Область техники, к которой относится изобретение
Группа изобретений относится к области биотехнологии и генетической инженерии, а именно к плазмиде, применяемой для геномного редактирования бактерий рода Bacillus и способу внесения модификаций в геном бактерий рода Bacillus.
Уровень техники
Бактерии рода Bacillus - востребованный объект промышленной биотехнологии, что связано с их способностью к продукции ряда метаболитов, таких как витамины [1], пуриновые нуклеозиды [2], поли-гамма-глутаминовой кислоты [3], инсектициды, антимикробных соединений (бактериоцины, пептидные антибиотики). Также бактерии рода Bacillus способны синтезировать и секретировать большие количества белка ферментов (амилазы, протеазы) во внеклеточную среду [4,5]. Эти особенности позволяют рассматривать бактерии рода Bacillus в качестве эффективных штаммов-продуцентов, что требует эффективных методов манипуляции с геномом бактерий.
Существует ряд инструментов для манипуляции с геномом бактерий рода Bacillus [6,7], чаще всего они основаны на использовании селективных маркеров и гомологичной рекомбинации или системы Cre/lox [8].
Также существуют методы внесения модификации в геном бактерий рода Bacillus с помощью систем CRISPR/Cas. Данная система представляет собой вектор, обеспечивающий экспрессию Cas нуклеазы и направляющей ее гидовой РНК (гидРНК или crРНК), которые образуют комплекс и связываются со специфической последовательностью (протоспейсером) ДНК для ее разрезания. При этом фрагменты, кодирующие Cas нуклеазу и гидРНК могут находиться на разных плазмидах (двухплазмидная система) или на одной плазмиде (одноплазмидная система). Чаще всего в качестве нуклеазы в подобных системах используется Cas9 нуклеаза или ее модификации, реже используется нуклеаза Cpf1 (другое название Cas12a) или ее модификации.
Cas нуклеаза и направляющая ее гидРНК образуют комплекс Cas/гидРНК, который связывается со специфической последовательностью ДНК и вносит в нее двунитевой разрыв, что приводит, к гибели клонов дикого типа и выживанию мутантных клонов. Репарация двунитевого разрыва ДНК может происходить по пути гомологичной рекомбинации в присутствии плечей гомологии или по пути негомологичного соединения концов (non-homologous end joining, NHEJ). В результате репарации происходит делеция, инсерция или другая мутация в области ДНК (протоспейсер), которая ранее распознавалась комплексом Cas/гидРНК. Мутация в специфичной последовательности протоспейсера (или в ПАМ элементе) не позволяет комплексу Cas/гидРНК произвести разрыв ДНК, что и приводит к выживанию мутантов.
В клетках эукариот негомологичное соединение концов является основным способом репарации двуцепочечных разрывов ДНК. В то же время в клетках бактерий негомологичное соединение концов встречается редко и происходит с низкой частотой [9-11]. Вместе с тем, этот метод позволяет быстро и эффективно вводить делеции в требуемый сайт в отсутствие плечей гомологии. Кроме того, данный способ позволяет вносить делеции в штаммах, дефектных по системе гомологичной рекомбинации (штаммы recA-), что необходимо для целей конструирования.
Известны системы негомологичного соединения концов у микобактерий (Mycobacterium tuberculosis или Mycobacterium smegmatis) [12,13]. Показано, что эти системы из Mycobacterium tuberculosis или Mycobacterium smegmatis способны работать в клетках Leptospira [14], Escherichia coli [15,16] и Bacillus subtilis [17]. Гены, кодирующие элементы системы негомологичного соединения концов идентифицированы и в других бактериях, включая Bacillus subtilis [9], однако их функциональность не доказана.
Предложенный авторами изобретения способ позволяет с помощью плазмиды, кодирующей Cas/гидРНК вносить делеции неконтролируемого размера в геномы бактерий рода Bacillus с использованием собственной системы негомологичного соединения концов. Кроме того, этот способ позволяет отбирать случайные мутации в области ПАМ-элемента и протоспейсера ДНК мишени.
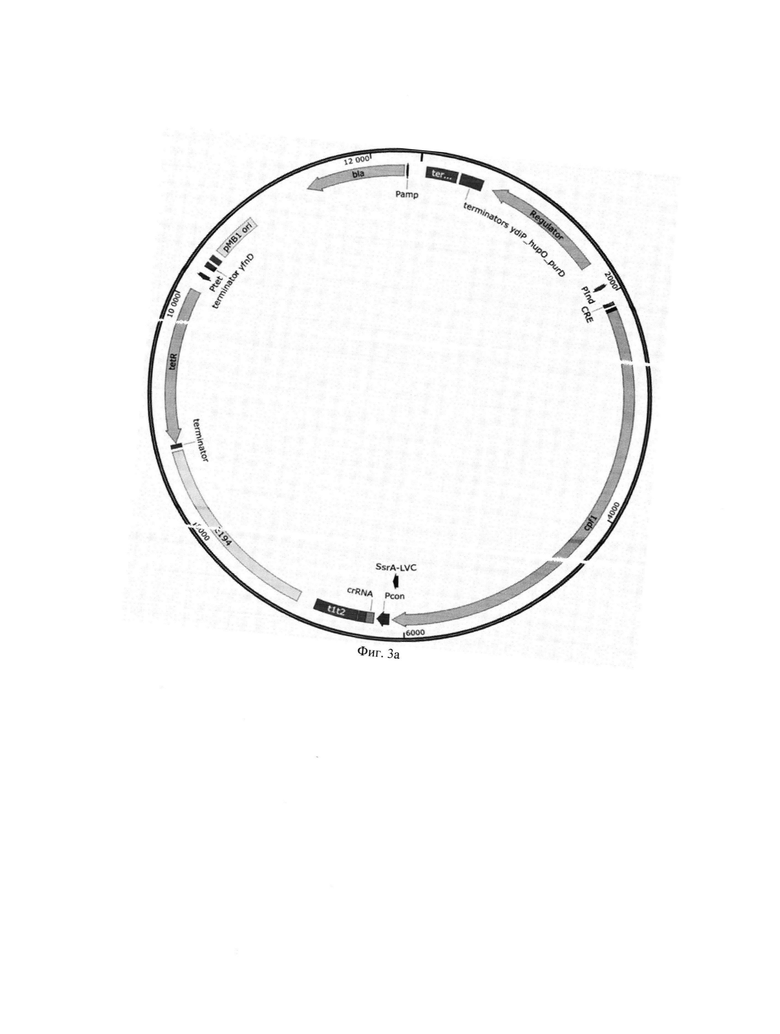
Впервые предложена плазмида для внесения модификаций в геном бактерий рода Bacillus с использованием системы репарации негомологичного соединения концов, содержащая ген cpƒ1 под контролем индуцируемого промотора, ген, кодирующий репрессор для соответствующего индуцируемого промотора, операторную последовательностью cre, область для встраивания последовательности направляющей гидРНК (crРНК), включающей спейсер к ДНК мишени, расположенную под контролем сильного конститутивного промотора (Фиг. 3а).
Идуцируемый промотор выбирают из группы, включающей промоторы Pxy1A, Pgrac, Pspac, PmanP, PtetA, Pmt1A или другой индуцируемый промотор [18-22].
Сильный конститутивный промотор выбирают из группы, включающей промоторы Pveg, Р43, PymdA, PserA или другой конститутивный промотор [23,24].
В одном из вариантов в качестве индуцируемого промотора плазмида содержит промоторы Pxy1A или Pgrac.
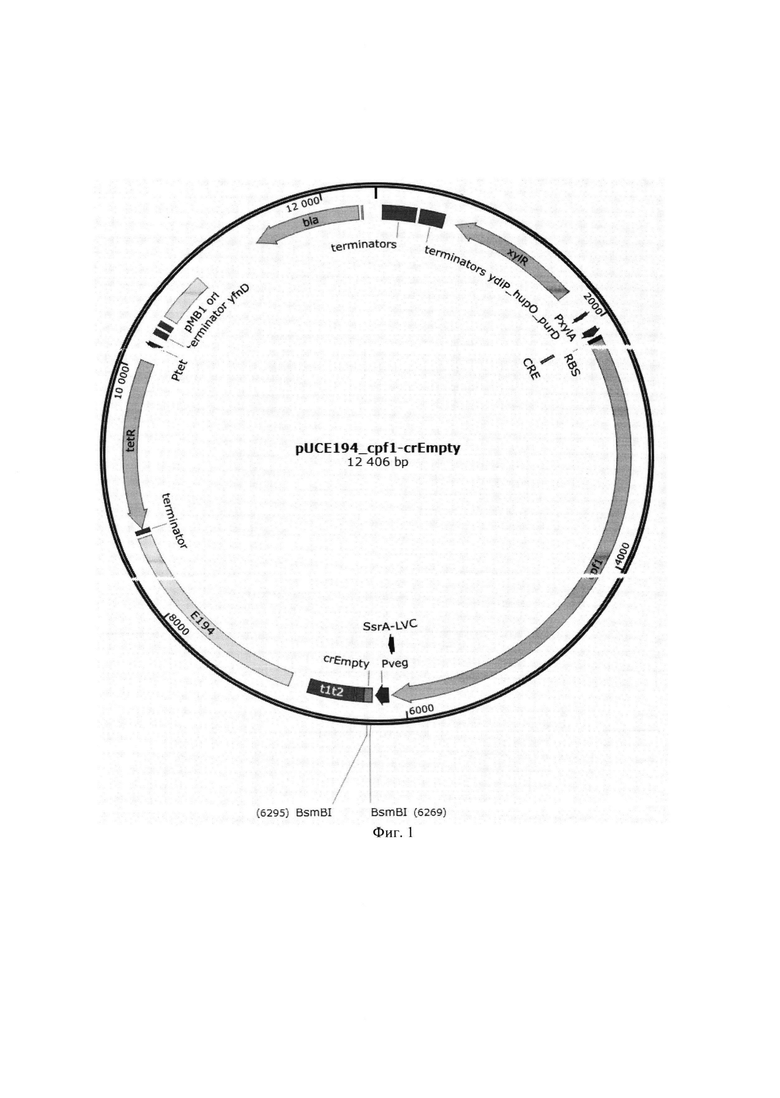
В одном из вариантов плазмида представляет плазмиду pUCE194_cpf1-crSpcR, содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор Xy1R для промотора Pxy1A, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Pveg. Плазмида pUCE194_cpf1-crSpcR депонирована в БРЦ ВКПМ под номером В-14351 (Фиг. 3).
В еще одном варианте плазмида представляет плазмиду pUCE194_Pgrac-cpf1_crSpcR содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора Pgrac, ген, кодирующий репрессор для промотора Pgrac, операторную последовательность cre для репрессии гена нуклеазы Cpf1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Р43 (Фиг. 4).
В еще одном варианте плазмида представляет плазмиду pUCE194_cpf1-crAmyS содержащую ген cpƒ1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор для промотора PxylA, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену термостабильной альфа-амилазы amyS, расположенную под контролем сильного конститутивного промотора Pveg (Фиг. 5).
В еще одном варианте плазмида представляет плазмиду pUCE194_cpf1-crAprE содержащую ген cpƒ1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор для промотора PxylA, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену щелочной протеазы aprE, расположенную под контролем сильного конститутивного промотора Pveg (Фиг. 6).
Авторы неожиданно обнаружили, что полное подавление синтеза нуклеазы cpƒ1 позволяет создать систему редактирования генома бактерий рода Bacillus с использованием собственной системы негомологичного соединения концов в геноме бактерий без обеспечения плечей для гомологичной рекомбинации, которая при этом не оказывает токсичного эффекта на клетки в состоянии без индукции промотора. Полное подавление синтеза нуклеазы cpƒ1 достигается двойной репрессией транкрипции гена cpƒ1 - посредством репрессора для индуцируемого промотора и репрессора СсрА.
Следовательно, другим аспектом изобретения является способ модификации генома бактерий рода Bacillus путем внесения мутаций с помощью репарации по пути негомологичного соединения концов, предусматривающий трансформацию бактерий плазмидой согласно изобретению, содержащей Cpf1/гидРНК, выращивание культуры клеток до титра 106 - 109 и индукцию промотора для синтеза нуклеазы Cpf1 с последующим отбором мутаций. Образование эффекторного комплекса Cpf1/гидРНК вносит двунитевой разрыв в ДНК бактерий с последующей репарацией. Использование строго контролируемого индуцируемого промотора позволяет выращивать культуру клеток до высокого титра без токсичного влияния на клетку, что позволяет обойти низкую эффективность репарации без использования плечей для гомологичной рекомбинации, что является преимуществом представленной системы редактирования над известной системой «All-in-One» [25].
После внесения модификаций в геном, плазмида с системой редактирования гидРНК/Cpf1, легко элиминируется из клеток при повышенной температуре (37-43°С) без селективного давления антибиотика, что является дополнительным преимуществом представленной системы редактирования над известной системой «All-in-One» [25].
В одном из вариантов предложенного способа клетки бактерий В. subtilis трансформируют плазмидой pUCE194_cpf1-crSpcR.
В другом варианте способа клетки бактерий В. subtilis трансформируют плазмидой pUCE194_Pgrac-cpf1_crSpcR.
В еще одном варианте способа клетки бактерий В. licheniformis трансформируют плазмидой pUCE194_cpf1-crAmyS.
В еще одном варианте способа клетки бактерий В. amyloliquefaciens трансформируют плазмидой pUCE194_cpf1-crAprE.
Раскрытие сущности изобретения
Техническим результатом является то, что предложена плазмида для внесения модификаций в геном бактерий рода Bacillus с использованием системы репарации негомологичного соединения концов, содержащая ген cpƒ1 под контролем индуцируемого промотора, ген, кодирующий репрессор для соответствующего индуцируемого промотора, операторную последовательностью cre для дополнительной репрессии гена cpƒ1 репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающую спейсер к ДНК мишени, расположенную под контролем сильного конститутивного промотора (Фиг. 3а).
Индуцируемый промотор выбирают из группы, включающей промоторы Pxy1A, Pgrac, Pspac, PmanP, PtetA, Pmt1A или другой индуцируемый промотор.
Сильный конститутивный промотор выбирают из группы, включающей промоторы Pveg, Р43, PymdA, PserA или другой конститутивный промотор.
В одном из вариантов плазмида представляет плазмиду pUCE194_cpf1-crSpcR, содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор Xy1R для промотора Pxy1A, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Pveg, депонированную в БРЦ ВКПМ под номером В-14351 (Фиг. 3). Конструирование плазмиды описано в примере 3. Последовательность спейсера представлена в табл. 2.

В еще одном варианте плазмида представляет плазмиду pUCE194_Pgrac-cpf1_crSpcR, содержащую ген cpƒ1 под контролем индуцируемого промотора Pgrac, ген, кодирующий репрессор для промотора Pgrac, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Р43 (Фиг. 4). Конструирование плазмиды описано в примере 4. Последовательность спейсера указана в таблице 2.
В еще одном варианте плазмида представляет плазмиду pUCE194_cpf1-crAmyS содержащую ген cpƒ1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор для промотора Pxy1A, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену термостабильной альфа-амилазы, расположенную под контролем сильного конститутивного промотора Pveg (Фиг. 5). Конструирование плазмиды описано в примере 5. Последовательность спейсера указана в таблице 2.
В еще одном варианте плазмида представляет плазмиду pUCE194_cpf1-crAprE содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора Pxy1A, ген, кодирующий репрессор для промотора Pxy1A, операторную последовательность cre для репрессии гена cpƒ1 белком репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену щелочной протеазы aprE, расположенную под контролем сильного конститутивного промотора Pveg (Фиг. 6). Конструирование плазмиды описано в примере 6. Последовательность спейсера указана в таблице 2.
Праймеры, используемые при конструировании плазмид, приведены в таблице 1.


Одним из преимуществ использования предложенной плазмиды относительно известных систем редактирования является полное подавление синтеза нуклеазы Cpf1 в отсутствии индуктора, что позволяет совместить на одной плазмиде ген нуклеазы, последовательность кодирующую гидРНК, и производить разрыв мишени ДНК в строго определенное время путем добавления индуктора. В качестве индуктора в зависимости от соответствующего индуцируемого промотора могут быть использованы индукторы, выбираемые из группы, включающей ксилозу, лактозу, ИПТГ, галактозу, арабинозу, маннозу, мальтозу, ангидротетрациклин, кумат и другие.
Предложенная согласно изобретению плазмида для редактирования генома бактерий рода Bacillus, обеспечивая полное подавление синтеза нуклеазы cpƒ1 посредством репрессора для индуцируемого промотора и репрессора СсрА, при этом не оказывает токсичного эффекта на клетки в состоянии без индукции промотора, позволяя выращивать клетки до титра 106 - 109 с последующей индукцией промотора, что приводит к синтезу Cpf1 нуклеазы и образованию эффекторного комплекса Cpf1/гидРНК, который вносит двунитевой разрыв в ДНК бактерий, и позволяет эффективно отбирать редкие события, такие как модификации с использованием системы репарации - негомологичного соединения концов или спонтанные мутации в области протоспейсера или ПАМ-элемента ДНК мишени, осуществляя негативную селекцию путем разрушения ДНК клеток дикого типа и оставляя в живых мутанты. После внесения модификации плазмиду при необходимости элиминируют при культивировании при повышенной температуре без селективного давления (без использования маркеров устойчивости к антибиотикам).
Следовательно, еще одним аспектом изобретения является способ внесения модификаций в геном бактерий рода Bacillus с использованием негомологичного соединения концов, предусматривающий трансформацию бактерий плазмидой согласно изобретению, содержащей Cpf1/гидРНК, выращивание культуры клеток до титра 106 - 109 и индукцию промотора для экспрессии гена нуклеазы Cpf1 с последующим отбором мутантов. При необходимости, последующая элиминация плазмиды Cpf1/гидРНК из культуры клеток осуществляется при повышенной температуре без селективного давления.
Предложенный способ позволяет отбирать редкие события, такие как модификации с использованием системы репарации - негомологичного соединения концов или спонтанные мутации в области протоспейсера или ПАМ-элемента ДНК мишени, осуществляя негативную селекцию путем разрушения ДНК клеток дикого типа и оставляя в живых мутанты (Примеры 7-9).
Модификации может быть подвержен любой штамм В. subtilis или другой штамм бактерий рода Bacillus, чувствительный к тетрациклину и совместимый с репликоном Е194. Для модификации, в качестве мишени может использоваться любая нуклеотидная последовательность ДНК на хромосоме бактерий (в области протоспейсера), граничащая с последовательностью ПАМ-элемента (протоспейсер ассоциированный мотив), который представлен тремя нуклеотидами 5'-TTN-3'.
В одном из вариантов предложенного способа клетки бактерий В. subtilis трансформируют плазмидой pUCE194_cpf1-crSpcR (пример 7). Для отбора (тестирования) модификаций использовали штамм Bacillus subtilis ΔprmA-rsmE-rimO::spcR, который получен на основе дикого штамма Bacillus subtilis 168 [26] (депонирован в БРЦ ВКПМ под номером В-1727 (АТСС 23857)) в результате введения в этот штамм направленных генетических модификаций: инактивации трех генов prmA, rsmE, rimO встраиванием в эту область гена spcR, сообщающего устойчивость клеток В. subtilis к спектиномицину.
Для внесения модификаций в геном штамма В. subtilis ΔprmA-rsmE-rimO::spcR плазмида pUCE194cpf1-crSpcR содержит спейсер, комплементарный протоспейсеру в последовательности гена spcR, таким образом гидРНК нацеливает нуклеазу cpƒ1 на последовательность гена spcR.
В другом варианте предложенного способа клетки штамма Bacillus subtilis ΔprmA-rsmE-rimO::spcR бактерий В. subtilis трансформируют плазмидой pUCE194_Pgrac-cpf1_crSpcR (пример 7).
В другом варианте осуществления способа для получения мутантов использовали штамм Bacillus licheniformis АТСС 14580 (депонирован в БРЦ ВКПМ под номером В-8054 [27]. Для получения мутантов по гену альфа-амилазы использовалась плазмида pUCE194_cpf1-crAmyS, содержащая спейсер, комплементарный протоспейсеру в последовательности гена термостабильной альфа-амилазы amyS, таким образом гидРНК нацеливает нуклеазу cpƒ1 на последовательность гена альфа-амилазы amyS (пример 8).
В еще одном варианте осуществления способа клетки штамма Bacillus amyloliquefaciens HK1 [28] трансформируют плазмидой pUCE194_cpf1-crAprE и отбирают мутации в гене щелочной протеазы aprE.
В соответствии с изобретением после индукции двунитевого разрыва ДНК происходит его репарация с использованием системы негомологичного соединения концов, или отбираются спонтанные мутанты, с мутациями в области протоспейсера или ПАМ-элемента, которые были подвержены разрыву эффекторным комплексом Cpf1/гидРНК. В результате, выживают мутантные клоны, в которых изменена последовательность протоспейсера (или ПАМ-элемента). Клоны дикого типа, в протоспейсере (или ПАМ-элементе) которых не произошла модификация, погибают. Из полученных клонов выделяется ДНК, и проверяется на наличие внесенных модификаций методом ПЦР [29] и секвенированием (Фиг. 7-12, Примеры 7-9).
Определения
CRISPR - (от англ. clustered regularly interspaced short palindromic repeats - короткие палиндромные повторы, регулярно расположенные группами) - особые локусы бактерий и архей, состоящие из прямых повторяющихся последовательностей, которые разделены уникальными последовательностями (спейсерами). Спейсеры заимствуются из чужеродных генетических элементов, с которыми сталкивалась клетка (бактериофагов, плазмид). РНК, транскрибирующиеся с локусов CRISPR, процессируется в короткие зрелые гидРНК длиной 42-44 нуклеотида совместно с ассоциированными белками Cas обеспечивают адаптивный иммунитет за счет комплементарного связывания РНК с нуклеиновыми кислотами чужеродных элементов и последующего разрушения их Cas нуклеазами.
гидРНК (или crРНК) - последовательность РНК, состоящая из консервативной части и вариабельной части, называемой спейсером длиной 23-25 нуклеотидов.
Спейсер - последовательность ДНК, комплементарная последовательности протоспейсера. Спейсер заимствуются из чужеродных генетических элементов, с которыми сталкивалась клетка и интегрируется в последовательность CRISPR.
Протоспейсер - последовательность ДНК, в которой происходит двунитевой разрыв комплексом Cas/гидРНК при наличии ПАМ элемента.
ПАМ элемент - (от англ. protospacer adjacent motif - протоспейсер-ассоциированный мотив) - для нуклеазы Cas12a (Cpf1) это последовательность 5'-TTN-3', прилегающая к последовательности протоспейсера, в присутствии ПАМ элемента комплекс Cpf1/гидРНК вносит двунитевой разрыв ДНК в области протоспейсера, с образованием липких концов.
Cpf1 (альтернативное название Cas12a) - CRISPR associated protein 12а, РНК-зависимая ДНК-эндонуклеаза, направляемая гидРНК, последовательность гена амплифицирована из генома бактерии Francisella novicida U112.
Комплекс Cpf1/гидРНК - эффекторный комплекс, белка нуклеазы Cpf1 (Cas12a) и гидРНК образует нуклеопротеиновый комплекс, который распознает и разрезает таргетируемую ДНК в области протоспейсера.
Челночная плазмида - в данном изобретении означает, что плазмида способна к репликации в клетках Е. coli и В. subtilis.
pE194ts - термочувствительный репликон, позволяющий реплицироваться плазмиде pUCE194_cpf1_crEmpty в клетках В. subtilis при температуре 30°С. При повышеной температуре репликация плазмиды не происходит. Репликон получен из плазмиды pXen5 [30].
рМВ1 - репликон, позволяющий реплицироваться плазмиде в клетках Е. coli, получен из плазмиды pUC18 (Thermo scientific).
tetR - ген устойчивости клеток Е. coli и бактерий В. subtilis к тетрациклину, получен из плазмиды pHY300PLK [31].
bla - ген β-лактамазы, сообщающий устойчивость клеток Е. coli к ампициллину, получен из плазмиды pUC18.
spcR - ген сообщающий устойчивость клеток В. subtilis к спектиномицину.
Pxy1A-cre - индуцируемый ксилозой промотор, репрессируется в отсутствии ксилозы собственным репрессором Xy1R. В присутствии глюкозы обеспечивается катаболитная репрессия посредством белка СсрА, связывающегося с операторной последовательностью cre-элементом. Промотор клонирован из плазмиды pPxy1Acre_MCS [32].
NHEJ (англ. non-homologous end joining) - негомологичное соединение концов, путь репарации ДНК для внесения модификаций в геном Bacillus.
Краткое описание чертежей
На Фиг. 1 изображена схема плазмиды pUCE194_cpf1_crEmpty
На Фиг. 2 представлена схема плазмиды pUCE194_Pgrac-cpf1-crEmpty
На Фиг. 3а представлена схема плазмиды для редактирования генома бактерий рода Bacillus с использованием системы репарации негомологичного соединения концов, кодирующая Cpf1/гидРНК
На Фиг. 3 представлена схема плазмиды pUCE194_cpf1-crSpcR
На Фиг. 4 представлена схема плазмиды pUCE194_Pgrac-cpf1-crSpcR
На фиг. 5 представлена схема плазмиды pUCE194_cpf1-crAmyS
На Фиг. 6 представлена схема плазмиды pUCE194_cpf1-crAprE
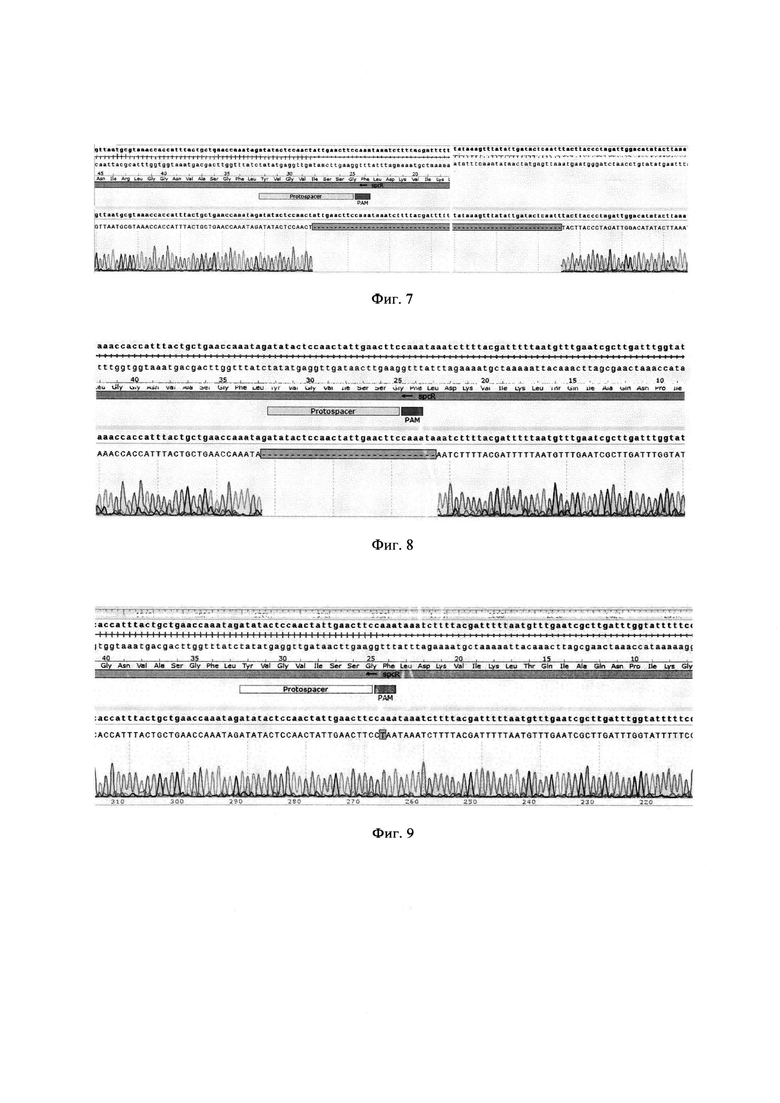
На Фиг. 7 изображена делеция 134 п.н. в геноме В. subtilis ΔprmA-rsmE-rimO::spcR в последовательности гена spcR с использованием негомологичного соединения концов.
На Фиг. 8 изображена делеция 30 п.н. в геноме В. subtilis ΔprmA-rsmE-rimO::spcR в последовательности гена spcR с использованием негомологичного соединения концов.
На Фиг. 9 изображена замена А->Т в ПАМ элементе, в геноме В. subtilis ΔprmA-rsmE-rimO::spcR в последовательности гена spcR с использованием негомологичного соединения концов.
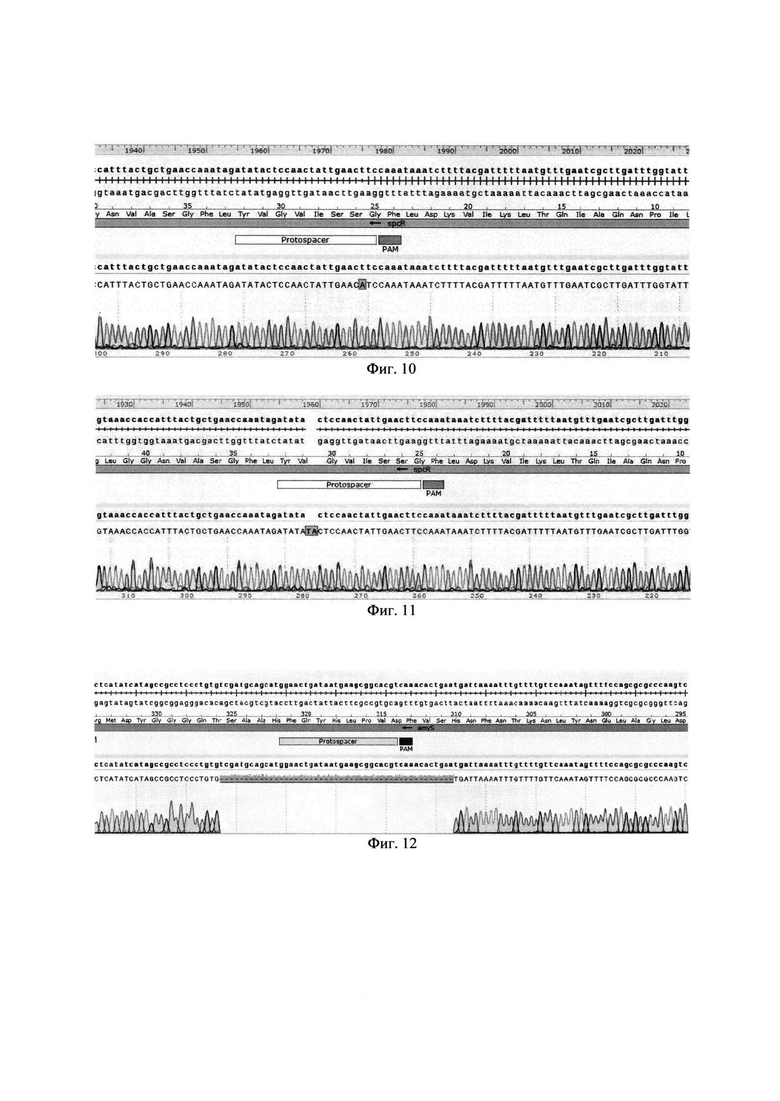
На Фиг. 10 изображена замена Т->А в протоспейсере, в геноме В. subtilis ΔprmA-rsmE-rimO::spcR в последовательности гена spcR с использованием негомологичного соединения концов.
На Фиг. 11 изображена вставка двух нуклеотидов AT в протоспейсере, в геноме В. subtilis AprmA-rsmE-rimO::spcR в последовательности гена spcR с использованием негомологичного соединения концов.
На Фиг. 12 изображена делеция 47 п.н. в геноме В. licheniformis АТСС 14580 в последовательности гена amyS с использованием негомологичного соединения концов.
Примеры осуществления настоящего изобретения
Пример 1. Конструирование рекомбинантной плазмиды pUCE194_cpf1_crEmpty с нуклеотидной последовательностью Seq ID N1.
Плазмида pUCE194_cpf1_crEmpty представляет челночную плазмиду, основанную на репликоне рМВ1 для Е. coli и репликоне pE194ts для В. subtilis, содержащую ген нуклеазы Cpf1 под индуцируемым промотором Pxy1A-cre [32], область для клонирования гидРНК под сильным конститутивным промотором Pveg, маркеры устойчивости к ампицилину и тетрациклину для Е. coli и В. subtilis соответственно.
Термочувствительная область начала репликации (ori) E194, обеспечивающая репликацию плазмиды в В. subtilis, амплифицирована методом ПЦР с помощью пары праймеров Р1/Р2 с плазмиды pXen5, используемой в качестве матрицы. Полученный линейный фрагмент ДНК размером 1719 п.н. собирают методом Гибсона с фрагментом 2240 п.н. линерезованной плазмиды pUC18 по сайтам рестрикции NdeI/PvuII. В результате получают челночную плазмиду pUCE194.
С помощью пар праймеров Р3/Р4 и Р5/Р6 из плазмиды pHY300PLK, используемой в качестве матрицы, методом ПЦР амплифицируют два линейных фрагмента ДНК, кодирующие ген устойчивости к антибиотику тетрациклину с заменами в сайтах рестрикции BsmBI и AarI, добавляют сайт рестрикции SmaI перед промотором гена устойчивости к тетрациклину. Фрагменты ДНК, используя сборку Гибсона, встраивают в плазмиду pUCE194 по сайту ClaI. В результате получают плазмиду pUCE194_tet. Используя пару праймеров Р7/Р8 и плазмиду pPxy1Acre_MCS в качестве матрицы, получают линейный фрагмент ДНК размером 2109 п.н. Данный фрагмент ДНК кодирует репрессор xy1R, промотор Pxy1A-cre, сайт множественного клонирования, и терминатор t1t2 гена rrnB. Фрагмент ДНК размером 2109 п.н. элюируется из геля и встраивается методом Гибсона в плазмиду pUCE194_tet по сайту PstI. В результате получают плазмиду pUCE194_Pxy1A_MCS.
Плазмиду pUCE194_Pxy1A_MCS обрабатывают рестриктазами AatII/XbaI, в полученный фрагмент размером 7470 п.н. встраивают кассету из терминаторов сборкой Гибсона с использованием праймеров Р9/Р10. Кассету терминаторов, используемую в качестве матрицы ДНК, синтезировали ранее коммерческой фирмой. В результате проведенных манипуляций получают плазмиду pUCE194_Pxy1A_MCS_T.
В плазмиду pUCE194_Pxy1A_MCS_T по сайтам NotI/ApaI сборкой Гибсона встраивают амплифицированный с использованием праймеров Р11/Р12 фрагмент ДНК, кодирующий ген cpƒ1, сайт посадки рибосом, ssrA-tag - метку ускоренного протеолиза. В качестве матрицы для ПЦР реакции используют геномную ДНК Francisella novicida U112. В результате проведенных манипуляций получают плазмиду с названием pUCE194_tet_Pxy1A_MCS_T_cpf1.
В плазмиду pUCE194_tet_Pxy1A_MCS_T_cpf1 по сайту BamHI встраивают последовательность ДНК, кодирующую сильный конститутивный промотор Pveg, сайты рестрикции, терминатор транскрипции Teno. Данную последовательность амплифицируют с использованием праймеров Р13/Р14. В качестве матрицы используют ДНК, синтезированную ранее коммерческой фирмой. В результате проведенных манипуляций получают плазмиду с названием pUCE194_tet_Pxy1A_MCS_T_cpf1_Pveg.
В плазмиду pUCE194_tet_Pxy1A_MCS_T_cpf1_Pveg перед промотором гена устойчивости к тетрациклину по сайту SmaI сборкой Гибсона встраивают два терминатора, синтезированные двумя парами Р15/Р16, Р17/Р18 перекрывающихся праймеров; последовательности терминаторов разделены сайтом рестрикции SmaI. В результате проведенной манипуляции получена плазмида pUCE194_tet_Pxy1A_MCS_T_cpf1_Pveg_T-SmaI-T.
В плазмиду pUCE194_tet_Pxy1A_MCS_T_cpf1_Pveg_T-SmaI-T, под промотор Pveg, сборкой Гибсона по сайтам BamHI/ApaI встраивают фрагмент ДНК, амплифицированный с использованием праймеров Р19/20. Праймеры перекрываются в 3'-области и выступают в качестве матрицы для ПЦР. Встроенный фрагмент ДНК содержит два прямых палиндромных повтора, между которыми встроены два сайта рестрикции BsmBI, необходимые для встраивания последовательности спейсера. В результате проведенных манипуляций получают плазмиду с названием pUCE194_cpf1_crEmpty, схема данной плазмиды представлена на Фигуре 1, последовательность представлена в SEQ ID NO: 1. Данная плазмида депонирована в БРЦ ВКПМ под номером В-14352.
Пример 2. Получение плазмиды pUCE194_Pgrac-cpf1_crEmpty.
В плазмиду pUCE194_tet (из примера 1) по сайту PstI из плазмиды pHT01 клонируют индуцируемый промотор Pgrac и ген репрессора lacI (с убранными последовательностями сайтов рестрикции BsmBI), получив в результате плазмиду pUCE194_tet_Pgrac. Следующие этапы конструирования плазмиды pUCE194_Pgrac-cpf1_crEmpty соответствуют этапам из примера 1, за исключением встраивания констиутивного промотора, где в плазмиду pUCE194_tet_Pgrac_MCS_T_cpf1 по сайту BamHI встраивают последовательность ДНК, кодирующую сильный конститутивный промотор Р43, сайты рестрикции, терминатор транскрипции Teno, в результате проведенных манипуляций получают плазмиду с названием pUCE194_tet_Pgrac_MCS_T_cpf1_P43.
В плазмиду pUCE194_tet_Pgrac_MCS_T_cpf1_P43 аналогично примеру 1, перед промотором гена устойчивости к тетрациклину по сайту SmaI сборкой Гибсона встраивают два терминатора, последовательности терминаторов разделены сайтом рестрикции SmaI. В результате проведенной манипуляции получена плазмида pUCE194_tet_Pgrac_MCS_T_cpf1_P43_T-SmaI-T.
В плазмиду pUCE194_tet_Pgrac_MCS_T_cpf1_P43_T-SmaI-T, аналогично примеру 1, под промотор Р43, сборкой Гибсона по сайтам BamHI/ApaI встраивают фрагмент ДНК содержащий два прямых палиндромных повтора, между которыми встроены два сайта рестрикции BsmBI, необходимые для встраивания последовательности спейсера. В результате получают плазмиду pUCE194_Pgrac-cpf1_crEmpty, схема данной плазмиды представлена на Фигуре 2.
Пример 3. Получение рекомбинантной плазмиды pUCE194_cpf1-crSpcR для внесения модификаций в геном В. subtilis ΔprmA-rsmE-rimO::spcR с использованием негомологичного соединения концов.
К плазмиде pUCE194_cpf1_crEmpty (пример 1), обработанной эндонуклеазой рестрикции BsmBI, добавляют олигонуклеотиды Р21/Р22 и проводят реакцию лигирования, встроив таким образом последовательность спейсера к гену устойчивости к спектиномицину, между прямыми палиндромными повторами и получив в результате плазмиду pUCE194_cpf1-crSpcR, используемую для модификации генома В. subtilis ΔprmA-rsmE-rimO::spcR, схема данной плазмиды представлена на Фигуре 3. Последовательность спейсера указана в таблице 2.
Пример 4. Получение рекомбинантной плазмиды pUCE194_Pgrac-cpf1_crSpcR для внесения модификаций в геном В. subtilis ΔprmA-rsmE-rimO::spcR с использованием негомологичного соединения концов.
К плазмиде pUCE194_Pgrac-cpf1_crEmpty (пример 2), обработанной эндонуклеазой рестрикции BsmBI, добавляют олигонуклеотиды Р21/Р22 и проводят реакцию лигирования, встроив таким образом последовательность спейсера к гену устойчивости к спектиномицину, между прямыми палиндромными повторами и получив в результате плазмиду pUCE194_Pgrac-cpf1_crSpcR, используемую для модификации генома В. subtilis ΔprmA-rsmE-rimO::spcR, схема данной плазмиды представлена на Фигуре 4. Последовательность спейсера указана в таблице 2.
Пример 5. Получение рекомбинантной плазмиды pUCE194_cpf1-crAmyS для внесения модификаций в геном Bacillus licheniformis АТСС 14580 с использованием негомологичного соединения концов.
В плазмиду pUCE194_cpf1_crEmpty (пример 1) аналогично, примеру 3, по сайтам BsmBI встраивают последовательность спейсера к гену термостабильной альфа-амилазы amyS, получив в результате плазмиду pUCE194_cpfl-crAmyS для внесения делеций в ген термостабильной альфа-амилазы amyS на хромосоме В. licheniformis АТСС 14580, схема данной плазмиды представлена на Фигуре 5. Последовательность спейсера указана в таблице 2.
Пример 6. Получение рекомбинантной плазмиды pUCE194_cpf1-crAprE для внесения модификаций в геном Bacillus amyloliquefaciens с использованием негомологичного соединения концов.
В плазмиду pUCE194_cpf1_crEmpty (пример 1) Аналогично, примеру 3, по сайтам BsmBI встраивают последовательность спейсера к гену щелочной протеазы aprE, получив в результате плазмиду pUCE194_cpf1-crAprE для внесения делеций в ген щелочной протеазы aprE на хромосоме Bacillus amyloliquefaciens HK1 схема данной плазмиды представлена на Фигуре 6. Последовательность спейсера указана в таблице 2.
Пример 7. Внесение модификаций в геном В. subtilis с использованием негомологичного соединения концов.
Штамм В. subtilis ΔprmA-rsmE-rimO:.spcR трансформируют плазмидой полученной по примерам 3 или 4 (pUCE194_cpf1-crSpcR или pUCE194_Pgrac-cpfl_crSpcR). Клоны, содержащие данную плазмиду, инокулируют в жидкую среду LB с тетрациклином и выращивают в течение ночи на 30°С при постоянной аэрации (200 об/мин.). К ночной культуре клеток добавляют индуктор ксилозу или ИПТГ. Индукция длится в течении двух часов на 30°С при постоянной аэрации (200 об/мин). При титре культуры клеток 109 после индукции клетки титруют до разведения 10-3 и высевают на агаризованную среду LB с соответствующим индуктором (ксилозой или ИПТГ) и 20 мкг/мл тетрациклина и инкубируют на 30°С в течении 1-2 суток. После инкубации на чашках с разведением 10-3 вырастает порядка 20-50 клонов, которые пересевают на агаризованную LB среду с соответствующим индуктором и 20 мкг/мл тетрациклина. Из клонов выделяют хромасомальную ДНК методом [33], ставят ПЦР с праймерами Р27/Р28 и отправляют на секвенирование. Результаты четырех полученных модификаций показывают делеции разного размера, замены и вставки, расположенные в гене spcR, представленью на Фиг. 7-11.
При необходимости избавления от плазмиды, клетки Bacillus выращивают в LB среде без тетрациклина при 37-43°С при постоянной аэрации (200 об/мин) в течении ночи, ночную культуру рассевают до отдельных колоний на агаризованную среду LB без тетрациклина и выращивают на 37-43°С. Отдельные колонии проверяют на чувствительность к тетрациклину. Отсутствие роста на среде с тетрациклином свидетельствует о потере плазмиды.
Пример 8 Внесение модификаций в геном В. licheniformis с использованием негомологичного соединения концов.
Штамм В. licheniformis АТСС 14580 трансформируют плазмидой полученной по примеру 5 (pUCE194_cpf1-crAmyS). Клоны, содержащие данную плазмиду, инокулируют в жидкую среду LB с тетрациклином и выращивают в течение ночи на 30°С при постоянной аэрации (200 об/мин). К ночной культуре клеток добавляют ксилозу. Индукция длится в течении двух часов на 30°С при постоянной аэрации (200 об/мин.). После индукции, при титре культуры клеток 109 клетки титруют до разведения 10-3 и высевают на агаризованную среду LB с индуктором ксилозой и 20 мкг/мл тетрациклина и инкубируют на 30°С в течении 1-2 суток. После инкубации на чашках с разведением 10-3 вырастает порядка 20-70 клонов, которые пересевают на агаризованную LB среду с ксилозой и 20 мкг/мл тетрациклина и инкубируют в течении суток на 30°С. Из клонов выделяют хромасомальную ДНК методом [33], ставят ПЦР с соответствующими праймерами и отправляют на секвенирование. В результате было проверено 10 случайных мутантных клонов, среди которых были получены модификации в виде делеций разного размера, замены и вставки, расположенные в гене термостабильной альфа-амилазы, одна из мутаций представлена на Фигуре 12.
При необходимости избавления от плазмиды, клетки Bacillus выращивают в LB без тетрациклина на 37-43°С при постоянной аэрации (200 об/мин) в течении ночи, ночную культуру рассевают до отдельных колоний на агаризованную среду LB без тетрациклина и выращивают на 37-43°С. Отдельные колонии проверяют на чувствительность к тетрациклину. Отсутствие роста на среде с тетрациклином свидетельствует о потере плазмиды.
Пример 9. Внесение модификаций в геном Bacillus amyloliquefaciens с использованием негомологичного соединения концов.
Штамм Bacillus amyloliquefaciens HK1 трансформируют плазмидой, полученной по примеру 6 (pUCE194_cpf1-crAprE). Клоны, содержащие данную плазмиду, инокулируют в жидкую среду LB с тетрациклином и выращивают в течение ночи на 30°С при постоянной аэрации (200 об/мин.). К ночной культуре клеток добавляют индуктор ксилозу. Индукция длится в течении двух часов на 30°С при постоянной аэрации (200 об/мин).
После индукции, при титре культуры клеток 109 клетки титруют до разведения 10-3 и высевают на агаризованную среду LB с индуктором ксилозой и 20 мкг/мл тетрациклина и инкубируют на 30°С в течении 1-2 суток. После инкубации на чашках с разведением 10-3 вырастает порядка 20-50 клонов, которые пересевают на агаризованную LB среду с ксилозой и 20 мкг/мл тетрациклина и инкубируют в течение суток на 30°С. Из клонов выделяют хромосомальную ДНК методом [33], ставят ПЦР с соответствующими праймерами и отправляют на секвенирование. В результате было проверено 10 случайных мутантных клонов, среди которых были получены модификации в виде делеций разного размера, замены и вставки, расположенные в гене щелочной протеазы aprE (данные не представлены).
При необходимости избавления от плазмиды, клетки Bacillus выращивают в LB среде без тетрациклина на 37-43°С при постоянной аэрации (200 об/мин.) в течении ночи, ночную культуру рассевают до отдельных колоний на агаризованную среду LB без тетрациклина и выращивают на 37-43°С. Отдельные колонии проверяют на чувствительность к тетрациклину. Отсутствие роста на среде с тетрациклином свидетельствует о потере плазмиды.
Источники информации
1. RU 2261273 C2 - Способ получения рибофлавина, штамм bacillus subtilis - продуцент рибофлавина (варианты) - Google Patents [Electronic resource]. URL: https://patents.google.com/patent/RU2261273C2/ru (accessed: 01.11.2022).
2. RU 2405833 C2 - Method for microbiologial synthesis of purine nucleoside of 5'-aminoimidazole-4-carboxamide riboside (aicar) and bacillus subtilis strain - producer of aicar - Google Patents [Electronic resource]. URL:
https://patents.google.com/patent/RU2405833C2/en (accessed: 01.11.2022).
3. RU 2313578 C2 - Method for preparing poly-gamma-glutamic acid and microorganism used in this method - Google Patents [Electronic resource]. URL: https://patents.google.com/patent/RU2313578C2/en (accessed: 01.11.2022).
4. Pandey A. et al. Advances in microbial amylases // Biotechnol. Appl. Biochem. Wiley, 2000. Vol. 31, №2. P. 135.
5. Zukowski M M. Production of comercially valuable products. 1992. P. 311-337.
6. Fabret C., Ehrlich S.D., Noirot P. A new mutation delivery system for genome-scale approaches in Bacillus subtilis // Mol. Microbiol. Mol Microbiol, 2002. Vol. 46, №1. P. 25-36.
7. Zhang X.Z. et al. mazF, a novel counter-selectable marker for unmarked chromosomal manipulation in Bacillus subtilis // Nucleic Acids Res. Nucleic Acids Res, 2006. Vol. 34, №9.
8. Yan X. et al. Cre/lox system and PCR-based genome engineering in Bacillus subtilis // Appl. Environ. Microbiol. Appl Environ Microbiol, 2008. Vol. 74, №17. P. 5556-5562.
9. Weller G.R. et al. Identification of a DNA nonhomologous end-joining complex in bacteria// Science (80-.). Science, 2002. Vol. 297, №5587. P. 1686-1689.
10. Shuman S., Glickman M.S. Bacterial DNA repair by non-homologous end joining // Nature Reviews Microbiology. Nat Rev Microbiol, 2007. Vol. 5, №11. P. 852-861.
11. Gupta R. et al. Mycobacteria exploit three genetically distinct DNA double-strand break repair pathways // Mol. Microbiol. Mol Microbiol, 2011. Vol. 79, №2. P. 316-330.
12. Sun B. et al. A CRISPR-Cpf1-Assisted Non-Homologous End Joining Genome Editing System of Mycobacterium smegmatis // Biotechnol. J. Wiley-VCH Verlag, 2018. Vol. 13, №9.
13. Yan M.-Y. et al. A CRISPR-Assisted Nonhomologous End-Joining Strategy for Efficient Genome Editing in Mycobacterium tuberculosis // MBio / ed. Hatfull G.F. mBio, 2020. Vol. 11, №1.
14. Fernandes L.G. V., Nascimento A.L.T.O. A Novel Breakthrough in Leptospira spp.Mutagenesis: Knockout by Combination of CRISPR/Cas9 and Non-homologous End-Joining Systems // Front. Microbiol. Frontiers Media SA, 2022. Vol. 13.
15. Su T. et al. A CRISPR-Cas9 assisted non-homologous end-joining strategy for one-step engineering of bacterial genome // Sci. Rep.Nature Publishing Group, 2016. Vol. 6, №1. P. 1-11.
16. Zheng X. et al. An efficient system for deletion of large DNA fragments in Escherichia coli via introduction of both Cas9 and the non-homologous end joining system from Mycobacterium smegmatis // Biochem. Biophys. Res. Commun. Elsevier B.V., 2017. Vol. 485, №4. P. 768-774.
17. Tian J. et al. Efficient Large-Scale and Scarless Genome Engineering Enables the Construction and Screening of Bacillus subtilis Biofuel Overproducers // Int. J. Mol. Sci. MDPI, 2022. Vol. 23, №9.
18. Bhavsar A.P., Zhao X., Brown E.D. Development and Characterization of a Xylose-Dependent System for Expression of Cloned Genes in Bacillus subtilis: Conditional Complementation of a Teichoic Acid Mutant // Appl. Environ. Microbiol. 2001. Vol. 67, №1. P. 403-410.
19. Phan T.T.P., Nguyen H.D., Schumann W. Novel plasmid-based expression vectors for intra- and extracellular production of recombinant proteins in Bacillus subtilis // Protein Expr. Purif. Protein Expr Purif, 2006. Vol. 46, №2. P. 189-195.
20. Yansura D.G., Henner D.J. Use of the Escherichia coli lac repressor and operator to control gene expression in Bacillus subtilis // Proc. Natl. Acad. Sci. U.S.A. National Academy of Sciences, 1984. Vol. 81, №2 I. P. 439-443.
21. Sun Т., Altenbuchner J. Characterization of a mannose utilization system in bacillus subtilis // J. Bacteriol. American Society for Microbiology (ASM), 2010. Vol. 192, №8. P. 2128-2139.
22. Falkenberg K.B. et al. The ProUSER2.0 Toolbox: Genetic Parts and Highly Customizable Plasmids for Synthetic Biology in Bacillus subtilis // ACS Synth. Biol. American Chemical Society, 2021. Vol. 10, №12. P. 3278-3289.
23. Guiziou S. et al. A part toolbox to tune genetic expression in Bacillus subtilis. // Nucleic Acids Res. 2016. Vol. 44, №15. P. 7495-7508.
24. Song Y. et al. Promoter Screening from Bacillus subtilis in Various Conditions Hunting for Synthetic Biology and Industrial Applications // PLoS One / ed. Isalan M. Public Library of Science, 2016. Vol. 11, №7. P. e0158447.
25. Hao W. et al. Design and Construction of Portable CRISPR-Cpf1-Mediated Genome Editing in Bacillus subtilis 168 Oriented Toward Multiple Utilities // Front. Bioeng. Biotechnol. Frontiers Media S.A., 2020. Vol. 8.
26. Kunst F. et al. The complete genome sequence of the gram-positive bacterium Bacillus subtilis // Nature. Nature Publishing Group, 1997. Vol. 390, №6657. P. 249-256.
27. Rey M.W. et al. Complete genome sequence of the industrial bacterium Bacillus licheniformis and comparisons with closely related Bacillus species // Genome Biol. Genome Biol. 2004. Vol. 5, №10. P. r77.
28. Bacillus amyloliquefaciens strain:HK1 (ID 358264) - BioProject - NCBI [Electronic resource]. URL: https://www.ncbi.nlm.nih.gov/bioproject/PRJNA358264 (accessed: 01.11.2022).
29. Saiki R.K. et al. Enzymatic amplification of β-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia // Science (80-.). Science, 1985. Vol. 230, №4732. P. 1350-1354.
30. Francis K.P. et al. Visualizing pneumococcal infections in the lungs of live mice using bioluminescent Streptococcus pneumoniae transformed with a novel gram-positive lux transposon. // Infect. Immun. 2001. Vol.69, №5. P. 3350-3358.
31. Ishiwa H., Shibahara H. New shuttle vectors for Escherichia coli and Bacillus subtilis. II. Plasmid pHY300PLK, a multipurpose cloning vector with a polylinker, derived from pHY460 // Japanese J. Genet. 1985. Vol. 60, №3. P. 235-243.
32. Gnuchikh E.Y., Manukhov I.V., Zavilgelsky G.B. Biosensors to Assess the Activity of Promoters and Chaperones in Bacillus subtilis Cells // Appl. Biochem. Microbiol. Pleiades journals, 2021. Vol. 57, №8. P. 877-885.
33. Sidoruk K.V. et al. A Method of DNA Extraction from a Wide Range of Objects via Treatment with Ammonium Salts // Appl. Biochem. Microbiol. Pleiades journals, 2021. Vol. 57, №8. P. 899-906.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Плазмида для редактирования генома бактерий рода Bacillus и
способ внесения модификаций в геном бактерий рода Bacillus.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2022-12-13">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">НИЦ «Курчатовский
институт»</ApplicantName>
<ApplicantNameLatin>NRC Kurchatov Institute</ApplicantNameLatin>
<InventionTitle languageCode="ru">Плазмида для редактирования генома
бактерий рода Bacillus и способ внесения модификаций в геном бактерий
рода Bacillus</InventionTitle>
<SequenceTotalQuantity>1</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>12406</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12406</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus sp.</INSDQualifier_value>
<NonEnglishQualifier_value>Bacillus
sp.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccactttatccaattttcgtttgttgaactaatgggtgctttagttga
agaataaaagaccacattaaaaaatgtggtcttttgtgtttttttaaaggatttgagcgtagcgaaaaat
ccttttctttcttatcttgataataagggtaactattgccgtcgtccattccgacagcatcgccagtcac
tatggcgtgctgctagaattgtatactgctgtcccatgtcatttcctcctttttctttttcatgtagtat
gcgcttgtatggaggaaaatattaaaaagccgctgaatgaggttcagcggcttttttgttaatcctgttt
gtaaagaagagctttcagggctcgttgttgtttctcatatacatgtaataatacggactaaaaagtatcg
gagctggataaaaccagctccgttttttatctttaattttttttgcgtttaaaagaagaaagctgctcgc
atagcgagcagctcttttttatgccaaaaataagtgaggaaaacccgcagaatagctgcgggttttttgt
tatcaaaaaagcctagagagaagggagaaggtgccatgtcactattgcttcagaaatactcctagaataa
aaaaactcatctttaaagatgagctgtccattccataaaaaattacattgtaatcatgtccagaaaatga
tcaatcacaatggaggacattcctaatgccggtgcattctgtcctaaggaagatggcaataattcatagc
tattgcctaattgggaataaacccttgatgatacttcacttctcattgaatttaaaaccataggatgcga
ttcaattatgctatttcttaaaattacggcttgtgggttgaaagtatttagaatattggtaaggcctatt
cctaaatagaatccaaaattttgtaatgcatttaaggttccgatatcattcagatgggcgaggtttatga
tatcttgataggacagttttttctctttggtctgaagagattttaataaagccttctctgaagcatacaa
ttcccagcatcctcggtttccgcaactgcatttaggaccattaaagtctattgtcatatgtcccatttct
ccagagaagccgcttactcctctatataaatgattgttgataataacaccgatccctattcctgtgctga
tacttacgtaaataatgttatcgtgattttttgcagctccaaatactttttctccatatgcgccagcatt
tgcctcattttcaataaaaacaggcacattgtacttctcttgtatcgaagattttaagtcaatatctctc
cagttggagttcggagtgaaaacaattttttgatctttatcaatgagtccaggcacgcaaatacctatac
caataagcccgtacggagattggggcatttgcgtaataaagtgatgaatcatatcaatcaaaatgtcttt
cgttatttctggagaattggattccaaatggcggtattgatcaagaacgattgttccttcaaggtctgtt
aaaatgccattaatataatccacaccaacatctattccaacggagtatcctgcctttttattaaaaacaa
gcatgacaggtcttcttccgccacttgattgtccttgacctatttcaaataccatactttctttcattaa
cgtgtttacctgtgatgagacagttgatttatttaatccagtcatttcagataattttgctcttgaaata
ggtgaatttttaaggatttcttttaataataacttttgatttacttttttgacaaaggtttgatcagcga
tatccacttcatccactccatttgtttaatctttaaattaagtatcaacatagtacatagcgaatcttcc
ctttattatatctaatgtgttcataaaaaactaaaaaaaatattgaaaatactgacgaggttatataaga
tgaaaataagttagtttgtttaaacaacaaactaataggtgatgtacttactatatgaaataaaatgcat
ctgtatttgaatgaatttatttttaagggggaaatcacatggctcaatctcattccagttcaatcaacta
ttttgtaagcgttaacaaagtggtttaagagctcggtaccgcggccgcatttgattaactaataagaggt
gcaaacatgtcaatttatcaagaatttgttaataaatatagtttaagtaaaactctaagatttgagttaa
tcccacagggtaaaacacttgaaaacataaaagcaagaggtttgattttagatgatgagaaaagagctaa
agactacaaaaaggctaaacaaataattgataaatatcatcagttttttatagaggagatattaagttcg
gtttgtattagcgaagatttattacaaaactattctgatgtttattttaaacttaaaaagagtgatgatg
ataatctacaaaaagattttaaaagtgcaaaagatacgataaagaaacaaatatctgaatatataaagga
ctcagagaaatttaagaatttgtttaatcaaaaccttatcgatgctaaaaaagggcaagagtcagattta
attctatggctaaagcaatctaaggataatggtatagaactatttaaagccaatagtgatatcacagata
tagatgaggcgttagaaataatcaaatcttttaaaggttggacaacttattttaagggttttcatgaaaa
tagaaaaaatgtttatagtagcaatgatattcctacatctattatttataggatagtagatgataatttg
cctaaatttctagaaaataaagctaagtatgagagtttaaaagacaaagctccagaagctataaactatg
aacaaattaaaaaagatttggcagaagagctaacctttgatattgactacaaaacatctgaagttaatca
aagagttttttcacttgatgaagtttttgagatagcaaactttaataattatctaaatcaaagtggtatt
actaaatttaatactattattggtggtaaatttgtaaatggtgaaaatacaaagagaaaaggtataaatg
aatatataaatctatactcacagcaaataaatgataaaacactcaaaaaatataaaatgagtgttttatt
taagcaaattttaagtgatacagaatctaaatcttttgtaattgataagttagaagatgatagtgatgta
gttacaacgatgcaaagtttttatgagcaaatagcagcttttaaaacagtagaagaaaaatctattaaag
aaacactatctttattatttgatgatttaaaagctcaaaaacttgatttgagtaaaatttattttaaaaa
tgataaatctcttactgatctatcacaacaagtttttgatgattatagtgttattggtacagcggtacta
gaatatataactcaacaaatagcacctaaaaatcttgataaccctagtaagaaagagcaagaattaatag
ccaaaaaaactgaaaaagcaaaatacttatctctagaaactataaagcttgccttagaagaatttaataa
gcatagagatatagataaacagtgtaggtttgaagaaatacttgcaaactttgcggctattccgatgata
tttgatgaaatagctcaaaacaaagacaatttggcacagatatctatcaaatatcaaaatcaaggtaaaa
aagacctacttcaagctagtgcggaagatgatgttaaagctatcaaggatcttttagatcaaactaataa
tctcttacataaactaaaaatatttcatattagtcagtcagaagataaggcaaatattttagacaaggat
gagcatttttatctagtatttgaggagtgctactttgagctagcgaatatagtgcctctttataacaaaa
ttagaaactatataactcaaaagccatatagtgatgagaaatttaagctcaattttgagaactcgacttt
ggctaatggttgggataaaaataaagagcctgacaatacggcaattttatttatcaaagatgataaatat
tatctgggtgtgatgaataagaaaaataacaaaatatttgatgataaagctatcaaagaaaataaaggcg
agggttataaaaaaattgtttataaacttttacctggcgcaaataaaatgttacctaaggttttcttttc
tgctaaatctataaaattttataatcctagtgaagatatacttagaataagaaatcattccacacataca
aaaaatggtagtcctcaaaaaggatatgaaaaatttgagtttaatattgaagattgccgaaaatttatag
atttttataaacagtctataagtaagcatccggagtggaaagattttggatttagattttctgatactca
aagatataattctatagatgaattttatagagaagttgaaaatcaaggctacaaactaacttttgaaaat
atatcagagagctatattgatagcgtagttaatcagggtaaattgtacctattccaaatctataataaag
atttttcagcttatagcaaagggcgaccaaatctacatactttatattggaaagcgctgtttgatgagag
aaatcttcaagatgtggtttataagctaaatggtgaggcagagcttttttatcgtaaacaatcaatacct
aaaaaaatcactcacccagctaaagaggcaatagctaataaaaacaaagataatcctaaaaaagagagtg
tttttgaatatgatttaatcaaagataaacgctttactgaagataagtttttctttcactgtcctattac
aatcaattttaaatctagtggagctaataagtttaatgatgaaatcaatttattgctaaaagaaaaagca
aatgatgttcatatattaagtatagatagaggtgaaagacatttagcttactatactttggtagatggta
aaggcaatatcatcaaacaagatactttcaacatcattggtaatgatagaatgaaaacaaactaccatga
taagcttgctgcaatagagaaagatagggattcagctaggaaagactggaaaaagataaataacatcaaa
gagatgaaagagggctatctatctcaggtagttcatgaaatagctaagctagttatagagtataatgcta
ttgtggtttttgaggatttaaattttggatttaaaagagggcgtttcaaggtagagaagcaggtctatca
aaagttagaaaaaatgctaattgagaaactaaactatctagttttcaaagataatgagtttgataaaact
gggggagtgcttagagcttatcagctaacagcaccttttgagacttttaaaaagatgggtaaacaaacag
gtattatctactatgtaccagctggttttacttcaaaaatttgtcctgtaactggttttgtaaatcagtt
atatcctaagtatgaaagtgtcagcaaatctcaagagttctttagtaagtttgacaagatttgttataac
cttgataagggctattttgagtttagttttgattataaaaactttggtgacaaggctgccaaaggcaagt
ggactatagctagctttgggagtagattgattaactttagaaattcagataaaaatcataattgggatac
tcgagaagtttatccaactaaagagttggagaaattgctaaaagattattctatcgaatatgggcatggc
gaatgtatcaaagcagctatttgcggtgagagcgacaaaaagttttttgctaagctaactagtgtcctaa
atactatcttacaaatgcgtaactcaaaaacaggtactgagttagattatctaatttcaccagtagcaga
tgtaaatggcaatttctttgattcgcgacaggcgccaaaaaatatgcctcaagatgctgatgccaatggt
gcttatcatattgggctaaaaggtctgatgctactaggtaggatcaaaaataatcaagagggcaaaaaac
tcaatttggttatcaaaaatgaagagtattttgagttcgtgcagaataggaataacgcaggcaaaacaaa
ttcatttaatcaaaatgttgctcttgtttgttaataattcattcaagggggccactgcagatagaatttt
gtcaaaataattttattgacaacgtcttattaacgttgatataatttaaattttatttgacaaaaatggg
ctcgtgttgtacaataaatgtaggggcccgtctaagaactttaaataatttctactgttgtagatgagac
ggaagatatcgtctcagtctaagaactttaaatggcgcgccggatccacttaaacaagtaataaaaagac
cgccgggattttctctcggcggcttttttatgctttctttagagagatccccatgccgaactcagaagtg
aaacgccgtagcgccgatggtagtgtggggtctccccatgcgagagtagggaactgccaggcatcaaata
aaacgaaaggctcagtcgaaagactgggcctttcgttttatctgttgtttgtcggtgaacgctctcctga
gtaggacaaatccgccgggagcggatttgaacgttgcgaagcaacggcccggagggtggcgggcaggacg
cccgccataaactgccaggcatcaaattaagcagaaggccatcctgacggatggcctttttgcgtttcta
caaactcttcctgtcgtcatatctacaagccatccccccacagatacggtaaactagcctcgtttttgca
tcagctcgattctcctcctctttctatattagtacttttgaaaattttagcatttttacatgctacctat
cattatagcactatcgaatctcatctataaacttatattggtgaatataaataaaaagattgatgcgaat
tctcacatcgaacggattgttgatgattacgaaaatattaagagcacagactattacacagaaaatcaag
aattaaaaaaacgtagagagagtttgaaagaagtagtgaatacatggaaagaggggtatcacgaaaaaag
taaagaggttaataaattaaagcgagagaatgatagtttgaatgagcagttgaatgtatcagagaaattt
caagctagtacagtgactttatatcgtgctgcgagggcgaatttccctgggtttgagaaagggtttaata
ggcttaaagagaaattctttaatgattccaaatttgagcgtgtgggacagtttatggatgttgtacagga
taatgtccagaaggtcgatagaaagcgtgagaaacagcgtacagacgatttagagatgtagaggtacttt
tatgccgagaaaactttttgcgtgtgacagtccttaaaatatacttagagcgtaagcgaaagtagtagcg
acagctattaactttcggttgcaaagctctaggatttttaatggacgcagcgcatcacacgcaaaaagga
aattggaataaatgcgaaatttgagatgttaattaaagacctttttgaggtctttttttcttagattttt
ggggttatttaggggagaaaacataggggggtactacgacctcccccctaggtgtccattgtccattgtc
caaacaaataaataaatattgggtttttaatgttaaaaggttgttttttatgttaaagtgaaaaaaacag
atgttgggaggtacagtgatggttgtagatagaaaagaagagaaaaaagttgctgttactttaagactta
caacagaagaaaatgagatattaaatagaatcaaagaaaaatataatattagcaaatcagatgcaaccgg
tattctaataaaaaaatatgcaaaggaggaatacggtgcattttaaacaaaaaaagatagacagcactgg
catgctgcctatctatgactaaattttgttaagtgtattagcaccgttattatatcatgagcgaaaatgt
aataaaagaaactgaaaacaagaaaaattcaagaggacgtaattggacatttgttttatatccagaatca
gcaaaagccgagtggttagagtatttaaaagagttacacattcaatttgtagtgtctccattacatgata
gggatactgatacagaaggtaggatgaaaaaagagcattatcatattctagtgatgtatgagggtaataa
atcttatgaacagataaaaataattaacagaagaattgaatgcgactattccgcagattgcaggaagtgt
gaaaggtcttgtgagatatatgcttcacatggacgatcctaataaatttaaatatcaaaaagaagatatg
atagtttatggcggtgtagatgttgatgaattattaaagaaaacaacaacagatagatataaattaatta
aagaaatgattgagtttattgatgaacaaggaatcgtagaatttaagagtttaatggattatgcaatgaa
gtttaaatttgatgattggttcccgcttttatgtgataactcggcgtatgttattcaagaatatataaaa
tcaaatcggtataaatctgaccgatagattttgaatttaggtgtcacaagacactcttttttcgcaccag
cgaaaactggtttaagccgactgcgcaaaagacataatccgggaattcctgttataaaaaaaggatcaat
tttgaactctctcccaaagttgatcccttaacgatttagaaatccctttgagaatgtttatatacattca
aggtaaccagccaactaatgacaatgattcctgaaaaaagtaataacaaattactatacagataagttga
ctgatcaacttccataggtaacaacctttgatcaagtaagggtatggataataaaccacctacaattgca
atacctgttccctctgataaaaagctggtaaagttaagcaaactcattccagcaccagcttcctgctgtt
tcaagctacttgaaacaattgttgatataactgttttggtgaacgaaagcccacctaaaacaaatacgat
tataattgtcatgaaccatgatgttgtttctaaaagaaaggaagcagttaaaaagctaacagaaagaaat
gtaactccgatgtttaacacgtataaaggacctcttctatcaacaagtatcccaccaatgtagccgaaaa
taatgacactcattgttccagggaaaataattacacttccgatttcggcagtacttagctggtgaacatc
tttcatcatataaggaaccatagagacaaaccctgctactgttccaaatataattcccccacaaagaact
ccaatcataaaaggtatatttttccctaatccgggatcaacaaaaggatctgttactttcctgatatgtt
ttacaaatatcaggaatgacagcacgctaacgataagaaaagaaatgctatatgatgttgtaaacaacat
aaaaaatacaatgcctacagacattagtataattcctttgatatcaaaatgaccttttatccttacttct
ttctttaataatttcataagaaacggaacagtgataattgttatcataggaatgagtagaagataggacc
aatgaatataatgggctatcattccaccaatcgctggaccgactccttctcccatggctactatcgatcc
aataagaccaaatgctttacccctattttcctttggaatatagcgcgcaactacaaccattacgagtgct
ggaaatgcagctgcaccagccccttgaataaaacgagccataataagtaaggaaaagaaagaatggccaa
caaacccaattaccgacccgaaacaatttattataattccaaataggagtaaccttttgatgcctaattg
atcagatagctttccatatacagctgttccaatggaaaaggttaacataaaggctgtgttcacccagttt
gtactcgccggtggtttattaaaatcatttgcaatatcaggtaatgacacgttcaaaaccatttcattta
atacgctaaaaaaagataaaatgcaaagccaaattaaaatttggttgtgtcgtaaattcgattgtgaata
ggatgtattcacatttcaccctccaataatgagggcagacgtagtttatagggttaatgatacgcttccc
tcttttaattgaaccctgttacattcattattcattacacttcataattaattcctcctaaacttgatta
aaacattttaccacatataaactaagttttaaattcagtatttcatcacttatacaacaatatggcccgt
tcgattcactgcattaatgaatcgcccaatgcattaagctgaaaaaacccctgccaggcggcagggggtt
ttttaatccagtaactattacccgggtactaatgtagtaagaaaaaagccggcccattacaggccggctt
tttttacgctttaatggcggggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttc
ctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggta
atacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggcca
ggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaa
tcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagc
tccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaa
gcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctggg
ctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaac
ccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtag
gcggtgctacagagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctg
cgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgct
ggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctt
tgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagatt
atcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatat
gagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttc
gttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccc
cagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagcaataaaccagccagcc
ggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccggg
aagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggt
gtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcc
cccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcag
tgttatcactcatggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttc
tgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttgctcttgcccg
gcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttctt
cggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaa
ctgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgca
aaaaagggaataagggcgacacggaaatgttgaatactcatactcttcctttttcaatattattgaagca
tttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggt
tccgcgcacatttccccgaaaagtgccacctgacgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2699523C2 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2766685C2 |
| СПОСОБ ПРОДУКЦИИ ПУРИНОВЫХ НУКЛЕОЗИДОВ И НУКЛЕОТИДОВ МЕТОДОМ ФЕРМЕНТАЦИИ С ИСПОЛЬЗОВАНИЕМ БАКТЕРИЙ, ПРИНАДЛЕЖАЩИХ К РОДУ Escherichia ИЛИ Bacillus | 2006 |
|
RU2365622C2 |
| ТАНДЕМНЫЙ ПРОМОТОР, ФУНКЦИОНИРУЮЩИЙ В БАКТЕРИИ РОДА Bacillus, ТРАНСФОРМИРОВАННАЯ БАКТЕРИЯ РОДА Bacillus - ПРОДУЦЕНТ ЦЕЛЕВОГО ПРОДУКТА, СОДЕРЖАЩАЯ УКАЗАННЫЙ ПРОМОТОР, И СПОСОБ ПОЛУЧЕНИЯ ЦЕЛЕВОГО ПРОДУКТА С ИСПОЛЬЗОВАНИЕМ УКАЗАННОЙ БАКТЕРИИ | 2019 |
|
RU2723722C1 |
| НУКЛЕАЗА CPF1 ИЗ БАКТЕРИИ Ruminococcus bromii, МОЛЕКУЛА ДНК ИЛИ РНК, КОДИРУЮЩАЯ НУКЛЕАЗУ, ВЕКТОР, СОДЕРЖАЩИЙ УКАЗАННУЮ МОЛЕКУЛУ ДНК, СИСТЕМА CRISPR/CPF1, СОДЕРЖАЩАЯ УКАЗАННУЮ НУКЛЕАЗУ И НАПРАВЛЯЮЩУЮ РНК, КЛЕТКА-ХОЗЯИН ДЛЯ ПОЛУЧЕНИЯ НУКЛЕАЗЫ CPF1, СПОСОБ ПОЛУЧЕНИЯ НУКЛЕАЗЫ CPF1 И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2816876C1 |
| БАКТЕРИЯ РОДА BACILLUS, ПРОДУЦИРУЮЩАЯ ГИАЛУРОНОВУЮ КИСЛОТУ, И СПОСОБ ПОЛУЧЕНИЯ ГИАЛУРОНОВОЙ КИСЛОТЫ С ИСПОЛЬЗОВАНИЕМ УКАЗАННОЙ БАКТЕРИИ | 2019 |
|
RU2719140C1 |
| БАКТЕРИЯ РОДА BACILLUS, ПРОДУЦИРУЮЩАЯ L-АМИНОКИСЛОТУ, И СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТЫ | 2002 |
|
RU2299907C2 |
| БАКТЕРИЯ Bacillus subtilis, ПРОДУЦИРУЮЩАЯ 5`-АМИНОИМИДАЗОЛ-4-КАРБОКСАМИДРИБОЗИД (АИКАР), И СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА АИКАР ПУТЕМ КУЛЬТИВИРОВАНИЯ ТАКОЙ БАКТЕРИИ | 2013 |
|
RU2542387C1 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2014 |
|
RU2688462C2 |
| Рекомбинантная плазмида pUSB2-AmQ, обеспечивающая синтез белка альфа-амилазы Bacillus amyloliquefaciens, и штамм Bacillus subtilis/pUSB2-AmQ - продуцент белка альфа-амилазы Bacillus amyloliquefaciens | 2020 |
|
RU2747627C1 |





Группа изобретений относится к биотехнологии и генетической инженерии. Предложена плазмида для внесения модификаций в геном бактерий рода Bacillus с использованием системы репарации негомологичного соединения концов, содержащая ген cpf1, под контролем индуцируемого промотора, ген, кодирующий репрессор для соответствующего индуцируемого промотора, операторную последовательность cre для дополнительной репрессии гена cpfl репрессором СсрА, область для встраивания последовательности, направляющей гидРНК, включающей спейсер к ДНК-мишени, расположенную под контролем сильного конститутивного промотора. Также предложен способ модификации генома бактерий рода Bacillus по пути негомологичного соединения концов с использованием указанной плазмиды. Группа изобретений обеспечивает контролируемую экспрессию нуклеазы Cpf1 в бактериях рода Bacillus и позволяет вносить направленные модификации в геном бактерий рода Bacillus без использования маркеров устойчивости к антибиотикам. 2 н. и 12 з.п. ф-лы, 12 ил., 2 табл., 9 пр.
1. Плазмида для внесения модификаций в геном бактерий рода Bacillus с использованием системы репарации негомологичного соединения концов, содержащая ген cpf1, под контролем индуцируемого промотора, ген, кодирующий репрессор для соответствующего индуцируемого промотора, операторную последовательность cre для дополнительной репрессии гена cpfl репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к ДНК-мишени, расположенную под контролем сильного конститутивного промотора.
2. Плазмида по п. 1, отличающаяся тем, что индуцируемый промотор выбирают из группы, включающей промоторы PxylA, Pgrac, Pspac, PmanP, PtetA, PmtlA.
3. Плазмида по п. 1, отличающаяся тем, что сильный конститутивный промотор выбирают из группы, включающей промоторы Pveg, Р43, PymdA, PserA.
4. Плазмида по п. 1, отличающаяся тем, что индуцируемый промотор выбирают из PxylA или Pgrac.
5. Плазмида по п. 1, отличающаяся тем, что представляет плазмиду pUCE194_cpf1-crSpcR, содержащую ген cpf1 под контролем индуцируемого промотора PxylA, ген, кодирующий репрессор XylR для промотора PxylA, операторную последовательность cre для репрессии гена cpf1 белком-репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Pveg, депонированная в БРЦ ВКПМ под номером В-14351.
6. Плазмида по п. 1, отличающаяся тем, что представляет плазмиду pUCEl94_Pgrac-cpf1_crSpcR, содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора Pgrac, ген, кодирующий репрессор для промотора Pgrac, операторную последовательность cre для репрессии гена cpf1 белком-репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену устойчивости к спектиномицину, расположенную под контролем сильного конститутивного промотора Р43.
7. Плазмида по п. 1, отличающаяся тем, что представляет плазмиду pUCE194_cpf1-crAmyS, содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора PxylA, ген, кодирующий репрессор для промотора PxylA, операторную последовательность cre для репрессии гена cpf1 белком-репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену термостабильной альфа-амилазы, расположенную под контролем сильного конститутивного промотора Pveg.
8. Плазмида по п. 1, отличающаяся тем, что представляет плазмиду pUCE194_cpf1-crAprE, содержащую ген нуклеазы Cpf1 под контролем индуцируемого промотора PxylA, ген, кодирующий репрессор для промотора PxylA, операторную последовательность cre для репрессии гена cpf1 белком-репрессором СсрА, область для встраивания последовательности направляющей гидРНК, включающей спейсер к гену щелочной протеазы aprE, расположенную под контролем сильного конститутивного промотора Pveg.
9. Способ модификации генома бактерий рода Bacillus по пути негомологичного соединения концов, предусматривающий трансформацию бактерий плазмидой по п. 1, выращивание культуры клеток до титра 106-109 и индукцию промотора для гена нуклеазы Cpf1 с последующим отбором мутаций.
10. Способ модификации генома бактерий рода Bacillus по п. 9, отличающийся тем, что дополнительно плазмиду элиминируют из культуры клеток при температуре 37-43°С без селективного давления антибиотика.
11. Способ по п. 9, отличающийся тем, что клетки бактерий представляют клетки В. subtilis, и для трансформации используют плазмиду pUCE194_cpf1-crSpcR.
12. Способ по п. 9, отличающийся тем, что клетки бактерий представляют клетки В. subtilis, и для трансформации используют плазмиду pUCE194_Pgrac-cpf1_crSpcR.
13. Способ по п. 9, отличающийся тем, что клетки бактерий представляют клетки В. licheniformis, и для трансформации используют плазмиду pUCE194_cpf1-crAmyS.
14. Способ по п. 9, отличающийся тем, что клетки бактерий представляют клетки В. amyloliquefaciens, и для трансформации используют плазмиду pUCE194_cpf1-crAprE.
| HAO W | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Front Bioeng Biotechnol | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| CN 110951741 A, 03.04.2020 | |||
| WO 2021175756 A1, 10.09.2021 | |||
| WU Y | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2024-03-22—Публикация
2022-12-27—Подача