Область техники
Изобретение относится к генетической конструкции (плазмиде) и может быть использовано для получения рекомбинантного белка щелочной протеазы Alkalihalobacillus clausii. Получаемый белок может быть использован в пищевой промышленности для получения пищевых гидролизатов (из рыбы, молока и сои), а также в различных детергентах (стиральные порошки, моющие средства для посуды, растворы для обработки кожи).
Уровень техники
Щелочная протеаза - это гидролитический фермент, который расщепляет белки до более мелких пептидов и аминокислот. Щелочные протеазы активны в диапазоне рН от нейтрального до щелочного. Эти ферменты широко используются в производстве детергентов, пищевых продуктов и в других областях (Sharma et al., 2019). Известны коммерческие протеазы такие как, BIOTOUCH® ROC 250 LC, Lavergy™ Pro, PRO-IE0025.
Широким спектром действия обладает щелочная протеаза Alkalihalobacillus clausii, расщепляющая различные субстраты, включая, окисленную В-цепь инсулина, казеин, гемоглобин и склеропротеины, такие как кератин, альфа-кератин и эластин (Yadav et al., 2019). Фермент стабилен и совместим с сильными анионными поверхностно-активными веществами (SDS) и окислителями (перекись водорода и перборат натрия (Joo et al., 2003). Комплекс физико-химических свойств протеазы Alkalihalobacillus clausii делает ее привлекательной как для научных целей, так и для промышленности.
Увеличение спроса на щелочные протеазы, особенно широкого спектра действия, создает потребность в методах увеличения коммерческого производства. Эффективность синтеза и секреции рекомбинантных белков в настоящее время может быть изменена в значительной степени за счет генно-инженерных манипуляций, конструирования экспрессионной кассеты, включающей последовательность промотера и сигнального пептида, подбором подходящего продуцента. На данный момент большинство протеаз синтезируется в штаммах-продуцентах, относящихся к разным видам рода Bacillus. Среди них выделяется Bacillus subtilis. Достоинствами использования системы Bacillus subtilis являются безопасность (Bacillus subtilis как платформа для промышленного использования имеет статус GRASj, синтез и секреция рекомбинантных белков непосредственно в культуральную среду, легкость масштабирования.
Известен патент «Protease enzyme variants and uses thereof)) (EP 3636735 A1, https://patents.google.com/patent/EP3636735A1/en), в котором описывается генетическая конструкция, кодирующая сериновую протеазу грибного происхождения для моющих веществ. Фермент активен в широком диапозоне рН и стабильно функционирует при низких и средних температурах. Сериновая протеаза грибного происхождения, согласно патенту, может быть получена в более высокопродуктивных грибах-продуцентах.
Также известен патент «Subtilase Variants)) (US 20090163400 A1, https://patents.google.com/patent/US20090163400A1/en), где описываются различные генетические конструкции, кодирующие ряд мутантных вариантов протеазы бациллярного происхождения с аминокислотными заменами, позволяющих увеличить стабильность протеаз в моющих веществах. В патенте точно не указаны штаммы-продуценты, которые будут использованы для получения фермента.
Наиболее близким к заявляемому изобретению является патент «Способ получения алкалофильного штамма бацилл с пониженным уровнем внеклеточной щелочной протеазы, способ получения высокощелочной протеазы и мутантная форма высокощелочной протеазы» (RU 2060276 C1, https://patents.google.com/patent/RU2060276C1/ru). В патенте описывается получение рекомбинантной плазмиды, содержащей ген внеклеточной щелочной протеазы и ее мутантных форм, а также получение штамма продуцента Bacillus. К недостаткам данного способа следует отнести низкий уровень синтеза целевого белка, получение продуцента при интеграции целевой последовательности в геном. Известно, что продуценты на основе интеграции могут обладать нестабильностью с исключением генетических участков после нескольких циклов культивирования. Раскрытие изобретения
Техническим результатом заявляемого изобретения является генетическая конструкция (плазмида), обеспечивающая синтез и секрецию белка щелочной протеазы Alkalihalobacillus clausii в системе Bacillus subtilis.
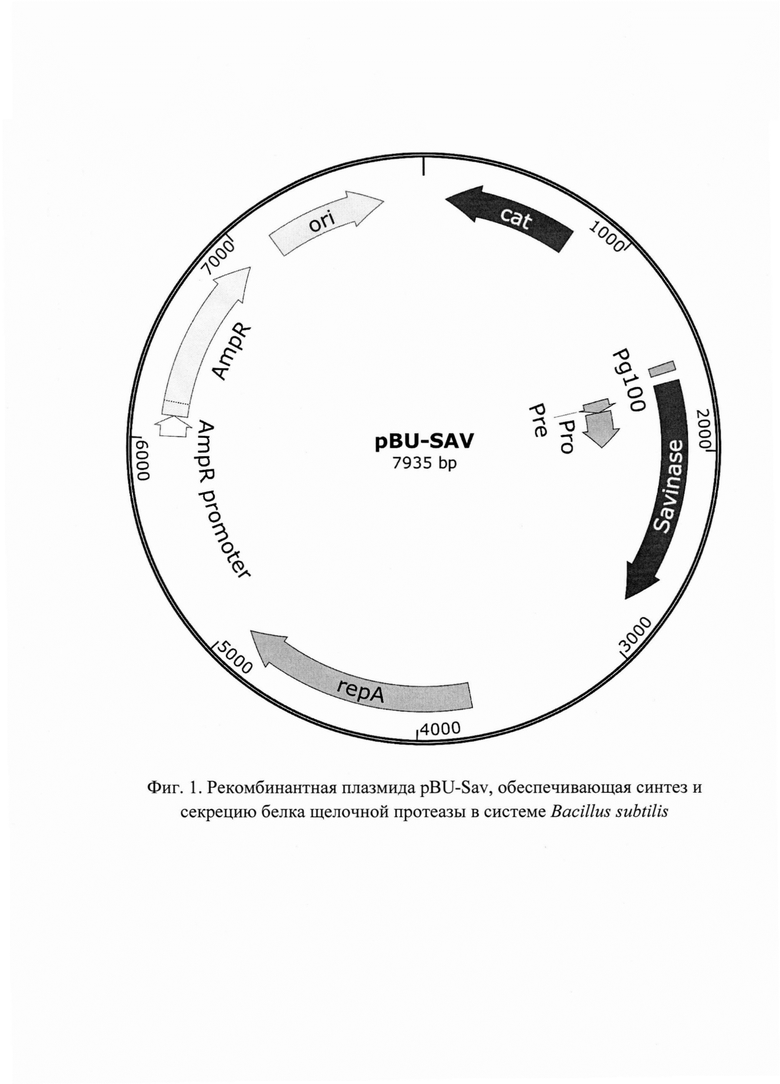
Указанный технический результат достигается получением получением генетической конструкции pBU-Sav (SEQ ID NO: 2), содержащей кодон-оптимизированную под Bacillus subtilis нуклеотидную последовательность (SEQ ID NO: 1) и обеспечивающей внеклеточную экспрессию щелочной протеазы в системе Bacillus subtilis. Генетическая конструкция pBU-Sav содержит в соответствии с физической и генетической картой, представленной на Фиг. 1, следующие элементы:
нуклеотидная последовательность, кодирующая устойчивость к хлорамфениколу (cat) (113-763 п. н.);
нуклеотидная последовательность сильного синтетического промотора Pg100 (1562-1605 п. н.);
нуклеотидная последовательность, кодирующая препептид, обеспечивающий экспорт белка из клетки (Pre) (1655-1735 п. н.);
нуклеотидная последовательность, кодирующая пропептид (Pro) (1736-1987 п. н.);
нуклеотидная последовательность, кодирующая ген щелочной протеазы (SEQ ID NO: 1) (1655-2797 п. н.);
нуклеотидная последовательность начала репликации в Bacillus subtilis (repA) (3737-4929 п. н.);
ген устойчивости к антибиотику ампициллин AmpR (6117-6977 п. н.) и бактериальный промотор гена устойчивости к ампициллину (6012-6116 п. н.), позволяющие проводить препаративную наработку плазмиды в E.coli;
- участок начала репликации ori (7148-7736 п. н.).
Генетическая конструкция pBU-Sav не содержит последовательности, кодирующей белок-репрессор LacI. Наличие белка репрессора LacI в составе экспрессионной кассеты обеспечивает контроль синтеза мРНК, однако в случае постоянного синтеза при интеграции он оказывает отрицательное влияние.
Указанный технический результат достигается разработкой способа получения белка щелочной протеазы с аминокислотной последовательностью SEQ ID NO: 3, который включает культивирование штамма Bacillus subtilis, внеклеточную экспрессию целевого белка и его скрининг.
Изобретение имеет ряд преимуществ по сравнению с близкими по сущности аналогами. Щелочная протеаза Alkalihalobacillus clausii стабильна с сильными анионными поверхностно-активными веществами, активна при температуре до 60°С и при рН=11. Конструкция pBU-Sav структурно и сегрегационно стабильна, имеет индуцибельный промотер Pg100, который обеспечивает высокоэффективное внеклеточное производство рекомбинантных белков. Экспрессия белка в системе Bacillus subtilis позволяет получать рекомбинантный фермент в секретируемой форме, за счет чего снижаются расходы на выделение и очистку конечного продукта.
Изобретение иллюстрируется следующими графическими фигурами. На фиг.1 представлена физическая и генетическая карта генетической конструкции pBU-Sav. На фиг.2 представлена электрофореграмма, подтверждающая секрецию белка щелочной протеазы: 1 - белковый маркер (Thermo Scientific Unstained Protein Molecular Weight Marker), 2-1 клон до наработки белка, 3-1 клон после 16 часов культивирования, 4-2 клон до наработки белка, 5-2 клон после 16 часов культивирования, 6-3 клон до наработки белка, 7-3 клон после 16 часов культивирования,
Для лучшего понимания сущности предлагаемого изобретения ниже приведены примеры (1-4) его осуществления.
Пример 1. Конструирование рекомбинатной плазмиды pBU-Sav для синтеза щелочной протеазы Alkalihalobacillus clausii в системе Bacillus subtilis
Нуклеотидная последовательность, кодирующая щелочную протеазу Alkalihalobacillus clausii, была заимствована из базы данных GenBank (WP_094423791).
Оптимизацию кодонного состава под систему экспрессии Bacillus subtilis проводили на сервере Thermo Fisher [https://www.thermofisher.com/]. Полученная последовательность представлена в SEQ ID NO: 1. После оптимизации был проведен анализ различия частот использования кодонов, а также негативных элементов с помощью серверов GenScript [https://www.genscript.com/tools/rare-codon-analysis] и EFMCalculator [http://barricklab.org/django/efm/].
Нуклеотидную последовательность промотера Pg100 и сайты рестрикции BamHI и AatII вводили в последовательность Pre-Pro-Sav одновременно при помощи праймеров Sub-prom-F, Sub-prom-R (таблица 1). Амплификацию нуклеотидной последовательности, кодирующей щелочную протеазу, проводили по стандартному протоколу. Реакционная смесь, объемом 50 мкл содержала 10×буфер Pfu, 20 нг плазмидной ДНК pSav2, кодирующей щелочную протеазу, 25 мМ dNTP (смесь дезоксинуклеотидтрифосфатов), 10 пмоль каждого олигонуклеотидного праймера (таблица 1), 0,25 ед. Pfu-полимеразы. Реакцию проводили в амплификаторе Techne TC-PLUS (ТС-5000 Plus) PCR Thermal Cycler.

Температурно-временной профиль ПНР: 95°С - 5 мин. 1 цикл.
30 циклов:
95°С - 30 сек.
58°С - 30 сек.
72°С - 30 сек.
ПЦР-продукт (Pre-Pro-Sav) наносили на 1% агарозный гель, выделяли из геля с использованием набора для очистки ДНК из агарозного геля и реакционных смесей Cleanup Standard («Евроген», Москва) в соответствии с протоколом и рекомендациями производителя.
ПЦР-продукт (Pre-Pro-Sav) и вектор pBU обрабатывали эндонуклеазами рестрикции BamHI и AatII («СибЭнзим», г. Новосибирск). Реакцию ферментативного гидролиза проводили в условиях, рекомендованных производителем.
Далее проводили лигирование ПЦР-продукта Pre-Pro-Sav и вектора pBU лигазой фага Т4 («СибЭнзим», г. Новосибирск) в соответствии с рекомендациями производителя.
Продуктами лигазной реакции трансформировали клетки Е. coli, штамм Stbl3 методом химической трансформации с помощью CaCl2 по общепринятой методике.
Клетки Е. coli штамм Stbl3, трансформированные экспрессионной плазмидой pBU-Sav, селективно культивировали в 5 мл жидкой питательной среды LB с добавлением антибиотика в концентрации 25 мкг/мл. Выделяли полученную плазмидную ДНК pBU-Sav с помощью набора Plasmid Miniprep («Евроген», Москва) в соответствии с инструкцией производителя.
Секвенирование плазмидной генетической конструкции pBU-Sav проводили по методу Сэнгера. Для секвенирования использовали набор CEQ2000Dye Terminator Cycle Sequencing Kit и 16-капилярный автоматический секвенатор ABI 3130x1.
Пример 2. Получение рекомбинантного штамма Bacillus subtilis BDV-1/pBU-Sav, обеспечивающего синтез щелочной протеазы Alkalihalobacillus clausii
Полученной рекомбинантной плазмидой pBU-Sav были электропорированы клетки Bacillus subtilis, штамм BDV-1. Культуру клеток штамма Bacillus subtilis подращивали до ODλ600 = 0,2. о.е, в количестве 50 мл. Затем в культуру добавили 0,5 гр. трионина, 1 гр. глицил-глицина, 0,05 гр. триптофана и 15 мкл TWEEN 80 и инкубировали в течение часа при 30°С. После культуру клеток инкубировали 20 мин на льду. 50 мл культуры центрифугировали 10 мин 5000g при 40С на центрифуге Eppendorf Centrifuge 5810 R. Осажденную биомассу дважды промывали в 5 мл ЕР Buffer (0,5 М трегалоза, 0,5 М сорбитол, 0,5 М маннитол, 0,7 мМ MgCl2, 0,5 мМ K2HPO4, 0,5 мМ KH2PO4, рН = 7,4). После промывки ресуспендировали в 0,5 мл EPBuffer. 100 мкл суспензии клеток перенесли в 2 мл холодные кюветы для электропорации и добавили ДНК с расчетом 25 нг/мкл. Произвели электропорацию при 2,5 kV на электропораторе Eppendorf Eporator®. К электропорированным клеткам добавляли питательную среду YTx2 с 0,5 М сорбитолом и 0,38 М маннитолом. Инкубировали 3 часа при 37°С. 50 и 100 мкл культуры высевали на чашки с агаризованной средой YTx2, содержащие 5 мкг/мл хлорамфеникола. Чашки помещали в термостат 30°С и инкубировали в течение ночи. На следующий день были идентифицированы отдельные колонии, содержащие плазмиду pBU-Sav.
Пример 3. Получение рекомбинатного белка щелочной протеазы Полученные клоны В.subtilis, штамм BDV-1 после электропорации рекомбинантной плазмидой pBU-Sav культивировали в качалочных колбах объемом 750 мл в течение16 часов при 30°С, 170 об/мин. Биомассу бактериальных клеток отделяли от культуральной среды центрифугированием (7000 об/мин), в течение 10 мин, 4°С. Наличие целевого белка щелочной протеазы (молекулярный вес 26,7 kDa) в культуральной среде анализировали с помощью 12% ПААГ (фиг.2).
Пример 4. Измерение активности щелочной протеазы
Активность определяют по общепринятой методике по гидролизу О-(4-итрофенилфосфорил)холина (NPPC) [J. Biochem - 1968 - N.63 - Р.681-683]. За единицу активности принимают количество фермента, необходимое для образования 1 мкмоля нитрофенола в минуту.
Активность фермента щелочной протеазы, полученной с помощью конструкции pBU-Sav и рекомбинантного штамма BDV-1/pBU-Sav, может достигать до 200 ЕД/мл культуральной жидкости.
Список литературы
1. Sharma М., Gat Y., Агуа S., Kumar V., Panghal A., Kumar A. A review on microbial alkaline protease: an essential tool for various industrial approaches. Industrial Biotechnology, 15(2), 69-78. A review on microbial alkaline protease: An essential tool for various industrial approaches // Industrial Biotechnology, 2019. Vol.15, №2. P. 69-78.
2. Yadav V.K., Singh V., Mishra V. Alkaline protease: A tool to manage solid waste and its utility in detergent industry // Microbial Genomics in Sustainable Agroecosystems. Springer, Singapore, 2019. P. 231-254.
3. Joo H.S., Kumar C.G., Park G.C., Paik S.R., Chang C.S. Oxidant and SDS-stable alkaline protease from Bacillus clausii 1-52: production and some properties // Journal of applied microbiology 2003, Vol 95. №2. P. 267-272.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="RU-3-pBU-Sav.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2022-12-16">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>RU-3-pBU-Sav</ApplicationNumberText>
<FilingDate>2022-12-16</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>RU-3-pBU-Sav</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>RU-3-pBU-Sav</ApplicationNumberText>
<FilingDate>2022-12-16</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБОУ ВО
«Алтайский государстРIенный
СѓРЅРёРIерситет»</ApplicantName>
<ApplicantNameLatin>Altai State University</ApplicantNameLatin>
<InventionTitle languageCode="ru">Рекомбинантная
плаЕмида pBU-Sav, обеспечиРIающая синтеЕ
Рё секрецию белка щелочной протеаЕы РI
системе Bacillus subtilis</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1143</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1143</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaagaaaccgttggggaaaattgtcgcaagcaccgcactactcattt
ctgttgcttttagttcatcgatcgcatcggctgctgaagaagcaaaagaaaaatatttaattggctttaa
tgagcaggaagctgtcagtgagtttgtagaacaagtagaggcaaatgacgaggtcgccattctctctgag
gaagaggaagtcgaaattgaattgcttcatgaatttgaaacgattcctgttttatccgttgagttaagcc
cagaagatgtggacgcgcttgaactcgatccagcgatttcttatattgaagaggatgcagaagtaacgac
aatggcgcaatcagtgccatggggaattagccgtgtgcaagccccagctgcccataaccgtggattgaca
ggttctggtgtaaaagttgctgtcctcgatacaggtatttccactcatccagacttaaatattcgtggtg
gcgctagctttgtaccaggggaaccatccactcaagatgggaatgggcatggcacgcatgtggccgggac
gattgctgctttaaacaattcgattggcgttcttggcgtagcgccgagcgcggaactatacgctgttaaa
gtattaggggcgagcggttcaggttcggtcagctcgattgcccaaggattggaatgggcagggaacaatg
gcatgcacgttgctaatttgagtttaggaagcccttcgccaagtgccacacttgagcaagctgttaatag
cgcgacttctagaggcgttcttgttgtagcggcatctgggaattcaggtgcaggctcaatcagctatccg
gcccgttatgcgaacgcaatggcagtcggagctactgaccaaaacaacaaccgcgccagcttttcacagt
atggcgcagggcttgacattgtcgcaccaggtgtaaacgtgcagagcacatacccaggttcaacgtatgc
cagcttaaacggtacatcgatggctactcctcatgttgcaggtgcagcagcccttgttaaacaaaagaac
ccatcttggtccaatgtacaaatccgcaatcatctaaagaatacggcaacgagcttaggaagcacgaact
tgtatggaagcggacttgtcaatgcagaagcggcaacacgctaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>7935</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7935</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttaagttattggtatgactggttttaagcgcaaaaaaagttgctttttc
gtacctattaatgtatcgttttagaaaaccgactgtaaaaagtacagtcggcattatctcatattataaa
agccagtcattaggcctatctgacaattcctgaatagagttcataaacaatcctgcatgataaccatcac
aaacagaatgatgtacctgtaaagatagcggtaaatatattgaattacctttattaatgaattttcctgc
tgtaataatgggtagaaggtaattactattattattgatatttaagttaaacccagtaaatgaagtccat
ggaataatagaaagagaaaaagcattttcaggtataggtgttttgggaaacaatttccccgaaccattat
atttctctacatcagaaaggtataaatcataaaactctttgaagtcattctttacaggagtccaaatacc
agagaatgttttagatacaccatcaaaaattgtataaagtggctctaacttatcccaataacctaactct
ccgtcgctattgtaaccagttctaaaagctgtatttgagtttatcacccttgtcactaagaaaataaatg
cagggtaaaatttatatccttcttgttttatgtttcggtataaaacactaatatcaatttctgtggttat
actaaaagtcgtttgttggttcaaataatgattaaatatctcttttctcttccaattgtctaaatcaatt
ttattaaagttcatttgatatgcctcctaaatttttatctaaagtgaatttaggaggcttacttgtctgc
tttcttcattagaatcaatccttttttaaaagtcaatattactgtaacataaatatatattttaaaaata
tcccactttatccaattttcgtttgttgaactaatgggtgctttagttgaagaataaagaccacattaaa
aaatgtggtcttttgtgtttttttaaaggatttgagcgtagcgaaaaatccttttctttcttatcttgat
aataagggtaactattgccgatcgtccattccgacagcatcgccagtcactatggcgtgctgctagcgcc
attcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagct
ggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacgttgt
aaaacgacggccagtgaattcgagctcaggccttaactcacattaattgcgttgcgctcactgcccgctt
tccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcg
tattgggcgtacggtacgatctttcagccgactcaaacatcaaatcttacaaatgtagtctttgaaagta
ttacatatgtaagatttaaatgcaaccgttttttcggaaggaaatgatgacctcgtttccaccggaatta
gcttggtaccaaaggaggtaaggatcactagaaaattttttaaaaaatctcttgacattggaagggagat
atgttattataagaattgcggaattgtgagcggataacaattcccatataaaggaggaaggatccatgaa
gaaaccgttggggaaaattgtcgcaagcaccgcactactcatttctgttgcttttagttcatcgatcgca
tcggctgctgaagaagcaaaagaaaaatatttaattggctttaatgagcaggaagctgtcagtgagtttg
tagaacaagtagaggcaaatgacgaggtcgccattctctctgaggaagaggaagtcgaaattgaattgct
tcatgaatttgaaacgattcctgttttatccgttgagttaagcccagaagatgtggacgcgcttgaactc
gatccagcgatttcttatattgaagaggatgcagaagtaacgacaatggcgcaatcagtgccatggggaa
ttagccgtgtgcaagccccagctgcccataaccgtggattgacaggttctggtgtaaaagttgctgtcct
cgatacaggtatttccactcatccagacttaaatattcgtggtggcgctagctttgtaccaggggaacca
tccactcaagatgggaatgggcatggcacgcatgtggccgggacgattgctgctttaaacaattcgattg
gcgttcttggcgtagcgccgagcgcggaactatacgctgttaaagtattaggggcgagcggttcaggttc
ggtcagctcgattgcccaaggattggaatgggcagggaacaatggcatgcacgttgctaatttgagttta
ggaagcccttcgccaagtgccacacttgagcaagctgttaatagcgcgacttctagaggcgttcttgttg
tagcggcatctgggaattcaggtgcaggctcaatcagctatccggcccgttatgcgaacgcaatggcagt
cggagctactgaccaaaacaacaaccgcgccagcttttcacagtatggcgcagggcttgacattgtcgca
ccaggtgtaaacgtgcagagcacatacccaggttcaacgtatgccagcttaaacggtacatcgatggcta
ctcctcatgttgcaggtgcagcagcccttgttaaacaaaagaacccatcttggtccaatgtacaaatccg
caatcatctaaagaatacggcaacgagcttaggaagcacgaacttgtatggaagcggacttgtcaatgca
gaagcggcaacacgctaagacgtccccggggcagcccgcctaatgagcgggcttttttcacgtcacgcgt
ccatggagatctttgtctgcaactgaaaagtttataccttacctggaacaaatggttgaaacatacgagg
ctaatatcggcttattaggaatagtccctgtactaataaaatcaggtggatcagttgatcagtatatttt
ggacgaagctcggaaagaatttggagatgacttgcttaattccacaattaaattaagggaaagaataaag
cgatttgatgttcaaggaatcacggaagaagatactcatgataaagaagctctaaaactattcaataacc
ttacaatggaattgatcgaaagggtggaaggttaatggtacgaaaattaggggatctacctagaaagcca
caaggcgataggtcaagcttaaagaacccttacatggatcttacagattctgaaagtaaagaaacaacag
aggttaaacaaacagaaccaaaaagaaaaaaagcattgttgaaaacaatgaaagttgatgtttcaatcca
taataagattaaatcgctgcacgaaattctggcagcatccgaagggaattcatattacttagaggatact
attgagagagctattgataagatggttgagacattacctgagagccaaaaaactttttatgaatatgaat
taaaaaaaagaaccaacaaaggctgagacagactccaaacgagtctgtttttttaaaaaaaatattagga
gcattgaatatatattagagaattaagaaagacatgggaataaaaatattttaaatccagtaaaaatatg
ataagattatttcagaatatgaagaactctgtttgtttttgatgaaaaaacaaacaaaaaaaatccacct
aacggaatctcaatttaactaacagcggccaaactgagaagttaaatttgagaaggggaaaaggcggatt
tatacttgtatttaactatctccattttaacattttattaaaccccatacaagtgaaaatcctcttttac
actgttcctttaggtgatcgcggagggacattatgagtgaagtaaacctaaaaggaaatacagatgaatt
agtgtattatcgacagcaaaccactggaaataaaatcgccaggaagagaatcaaaaaagggaaagaagaa
gtttattatgttgctgaaacggaagagaagatatggacagaagagcaaataaaaaacttttctttagaca
aatttggtacgcatataccttacatagaaggtcattatacaatcttaaataattacttctttgatttttg
gggctattttttaggtgctgaaggaattgcgctctatgctcacctaactcgttatgcatacggcagcaaa
gacttttgctttcctagtctacaaacaatcgctaaaaaaatggacaagactcctgttacagttagaggct
acttgaaactgcttgaaaggtacggttttatttggaaggtaaacgtccgtaataaaaccaaggataacac
agaggaatccccgatttttaagattagacgtaaggttcctttgctttcagaagaacttttaaatggaaac
cctaatattgaaattccagatgacgaggaagcacatgtaaagaaggctttaaaaaaggaaaaagagggtc
ttccaaaggttttgaaaaaagagcacgatgaatttgttaaaaaaatgatggatgagtcagaaacaattaa
tattccagaggccttacaatatgacacaatgtatgaagatatactcagtaaaggagaaattcgaaaagaa
atcaaaaaacaaatacctaatcctacaacatcttttgagagtatatcaatgacaactgaagaggaaaaag
tcgacagtactttaaaaagcgaaatgcaaaatcgtgtctctaagccttcttttgatacctggtttaaaaa
cactaagatcaaaattgaaaataaaaattgtttattacttgtaccgagtgaatttgcatttgaatggatt
aagaaaagatatttagaaacaattaaaacagtccttgaagaagctggatatgttttcgaaaaaatcgaac
taagaaaagtgcaataaactgctgaagtatttcagcagttttttttatttagaaatagtgaaaaaaatat
aatcagggaggtatcaatatttaatgagtactgatttaaatttatttagactggaattaataattaacac
gtagactaattaaaatttaatgagggataaagaggatacaaaaatattaatttcaatccctattaaattt
taacaagggggggattaaaatttaattagaggtttatccacaagaaaagaccctaataaaatttttacta
gggttataacactgattaatttcttaatgggggagggattaaaatttaatgacaaagaaaacaatctttt
aagaaaagcttttaaaagataataataaaaagagctttgcgattaagcaaaactctttactttttcattg
acattatcaaattcatcgatttcaaattgttgttgtatcataaagttaattctgttttgcacaacctttt
caggaatataaaacacatctgaggcttgttttataaactcagggtcgctaaagtcaatgtaacgtagcat
atgatatggtatagcttccacccaagttagcctttctgcttcttctgaatgtttttcatatacttccatg
ggtatctctaaatgattttcctcatgtagcaaggtatgagcaaaaagtttatggaattgatagttcctct
ctttttcttcaacttttttatctaaaacaaacactttaacatctgagtcaatgtaagcataagatgtttt
tccagtcataatttcaatcccaaatcttttagacagaaattctggacgtaaatcttttggtgaaagaatt
tttttatgtagcaatatatccgatacagcaccttctaaaagcgttggtgaatagggcattttacctatct
cctctcattttgtggaataaaaatagtcatattcgtccatctacctatcctattatcgaacagttgaact
ttttaatcaaggatcagtcctttttttcattattcttaaactgtgctcttaactttaacaactcgatttg
tttttccagatctcgagggtaactagcctcgccgatcccgcaagaggcccggcagtcaggtggcactttt
cggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatga
gacaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgt
cgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagta
aaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatcc
ttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggt
attatcccgtattgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggtt
gagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgcca
taaccatgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgc
ttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccata
ccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcg
aactacttactctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccact
tctgcgctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgc
ggtatcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtc
aggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaact
gtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctag
gtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcag
accccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaac
aaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggta
actggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttca
agaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcga
taagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacg
gggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagc
tatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaac
aggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccac
ctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacg
cggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctga
ttctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgc
agcgagtcagtgagcgaggaagcggaagagcgcccaatacgcatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>269</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..269</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AQSVPWGISRVQAPAAHNRGLTGSGVKVAVLDTGISTHPDLNIRGGASF
VPGEPSTQDGNGHGTHVAGTIAALNNSIGVLGVAPSAELYAVKVLGASGSGSVSSIAQGLEWAGNNGMHV
ANLSLGSPSPSATLEQAVNSATSRGVLVVAASGNSGAGSISYPARYANAMAVGATDQNNNRASFSQYGAG
LDIVAPGVNVQSTYPGSTYASLNGTSMATPHVAGAAALVKQKNPSWSNVQIRNHLKNTATSLGSTNLYGS
GLVNAEAATR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантная плазмида pBU-LipA, обеспечивающая синтез белка липазы А штамма Bacillus natto IAN | 2023 |
|
RU2808501C1 |
| УЛУЧШЕННАЯ ПРОДУКЦИЯ БЕЛКА В BACILLUS | 2008 |
|
RU2515112C2 |
| СПОСОБ ПОЛУЧЕНИЯ ГИАЛУРОНАНА В РЕКОМБИНАНТНОЙ КЛЕТКЕ-ХОЗЯИНЕ | 2002 |
|
RU2346049C2 |
| Рекомбинантная плазмида pUSB2-AmQ, обеспечивающая синтез белка альфа-амилазы Bacillus amyloliquefaciens, и штамм Bacillus subtilis/pUSB2-AmQ - продуцент белка альфа-амилазы Bacillus amyloliquefaciens | 2020 |
|
RU2747627C1 |
| ТАНДЕМНЫЙ ПРОМОТОР, ФУНКЦИОНИРУЮЩИЙ В БАКТЕРИИ РОДА Bacillus, ТРАНСФОРМИРОВАННАЯ БАКТЕРИЯ РОДА Bacillus - ПРОДУЦЕНТ ЦЕЛЕВОГО ПРОДУКТА, СОДЕРЖАЩАЯ УКАЗАННЫЙ ПРОМОТОР, И СПОСОБ ПОЛУЧЕНИЯ ЦЕЛЕВОГО ПРОДУКТА С ИСПОЛЬЗОВАНИЕМ УКАЗАННОЙ БАКТЕРИИ | 2019 |
|
RU2723722C1 |
| СПОСОБ АМПЛИФИЦИРОВАНИЯ ЛОКУСОВ В БАКТЕРИАЛЬНОЙ КЛЕТКЕ | 2009 |
|
RU2577985C2 |
| СПОСОБ ЭКСПРЕССИИ | 2013 |
|
RU2644199C2 |
| ЭКСПРЕССИОННЫЕ ВЕКТОРЫ ДЛЯ УЛУЧШЕННОЙ СЕКРЕЦИИ БЕЛКА | 2012 |
|
RU2661790C2 |
| ПРОТИВОМИКРОБНЫЕ ПОЛИПЕПТИДЫ ИЗ PSEUDOPLECTANIA NIGRELLA | 2002 |
|
RU2336278C2 |
| СПОСОБ ЭКСПРЕССИИ | 2012 |
|
RU2642324C2 |

Изобретение относится к биотехнологии и генной инженерии, в частности к генетической конструкции, и может быть использовано для получения рекомбинантного белка щелочной протеазы Alkalihalobacillus clausii. Получена генетическая конструкция pBU-Sav SEQ ID NO: 2, содержащая кодон-оптимизированную под Bacillus subtilis нуклеотидную последовательность SEQ ID NO: 1 и обеспечивающую внеклеточную экспрессию щелочной протеазы в системе Bacillus subtilis, имеющей элементы в соответствии с физической и генетической картой, представленной на Фиг. 1. Изобретение позволяет обеспечивать эффективный синтез и секрецию белка щелочной протеазы Alkalihalobacillus clausii в системе Bacillus subtilis. 2 ил., 1 табл., 4 пр.
Генетическая конструкция pBU-Sav SEQ ID NO: 2, содержащая кодон-оптимизированную под Bacillus subtilis нуклеотидную последовательность SEQ ID NO: 1, обеспечивающая внеклеточную экспрессию щелочной протеазы Alkalihalobacillus clausii в системе Bacillus subtilis, имеющая в соответствии с физической и генетической картой, представленной на Фиг. 1, следующие элементы:
- нуклеотидная последовательность, кодирующая устойчивость к хлорамфениколу (cat) 113-763 п. н.;
- нуклеотидная последовательность сильного синтетического промотора Pg100 1562-1605 п. н.;
- нуклеотидная последовательность, кодирующая препептид, обеспечивающий экспорт белка из клетки (Pre) 1655-1735 п. н.;
- нуклеотидная последовательность, кодирующая пропептид (Pro) 1736-1987 п. н.;
- нуклеотидная последовательность, кодирующая ген щелочной протеазы SEQ ID NO: 1 1655-2797 п. н.;
- нуклеотидная последовательность начала репликации в Bacillus subtilis (repA) 3737-4929 п. н.;
- ген устойчивости к антибиотику ампициллин AmpR 6117-6977 п. н. и бактериальный промотор гена устойчивости к ампициллину 6012-6116 п. н., позволяющие проводить препаративную наработку плазмиды в E.coli;
- участок начала репликации ori 7148-7736 п. н.
| RU 2060276 C1, 20.05.1996 | |||
| ЭКСПРЕССИОННЫЕ ВЕКТОРЫ ДЛЯ УЛУЧШЕННОЙ СЕКРЕЦИИ БЕЛКА | 2012 |
|
RU2661790C2 |
| EP 3636735 A1, 15.04.2020. | |||
Авторы
Даты
2024-04-01—Публикация
2023-02-15—Подача