Область техники
Изобретение относится к генетической конструкции (плазмиде), обеспечивающей синтез рекомбинантного белка - липазы A Bacillus natto IAN. Способ включает дизайн генетической конструкции и методы трансформации.
Уровень техники
Липазы - ферменты, осуществляющие гидролиз триацилглицеридов с образованием жирных кислот и глицерина. Основными коммерчески-значимыми характеристиками, которыми должны обладать липазы, являются термостабильность, устойчивость к щелочным условиям, органическим растворителям (RU 2508402 С1).
Источником липолитических ферментов являются грибы и бактерии, в частности многие бациллы (RU 2508402 С1). В изобретениях RU 2500812 C1, RU 2475532 C1, RU 2451075 C1, RU 2575804 C1 (Journal of Biotechnology. - 1997. - V. 56. - Р. 89-102), (Protein Expression and Purification. - 2003. - V. 28. -N. 1. - P. 102-1), RU 93042492 A, RU 2508402 C1 приводятся методы получения термостабильных липаз в продуцентах Bacillus licheniformis, Yarrowia lipolytica, Candida parapsilosis, Geobacillus thermocalenulatus, Pichia pastoris, Rhizopus orizae, Escherichia coli. К недостаткам изобретений можно отнести невысокую активность продуцентов, либо трудозатратный способ выделения и очистки ферментного препарата.
Липаза A (LipA) является небольшим белком с массой 19,3 кДа, продуцируемым Bacillus subtilis. В отличие от большинства липаз, LipA не имеет lid домена, закрывающего активный участок фермента, и не проявляет межфазной активации (Borrelli G. М., Trono D., 2015). Оптимальный рН для работы LipA 10,0, температурный интервал 35-40°С. LipA проявляет активность в отношении сложных эфиров глицерина и жирных кислот, длина цепи которых составляет от 6 до 18 атомов углерода, максимальная активность показана для эфиров жирных кислот с длиной цепи С8-С14 (Eggert T. et al. 2002; R. Jing Ma et al. 2017). На практике LipA широко используется при синтезе фармацевтических препаратов, в частности для получения энантиомеров лекарственных веществ (Безбородов А.М., Загустина Н.А., 2014).
Получение LipA проводят в культурах Е. coli и В. subtilis. По сравнению с Е. coli В. subtilis имеет ряд преимуществ: 1) В. subtilis непатогенна; 2) продуцент В. subtilis способен обеспечить синтез и секрецию рекомбинантных белков непосредственно в культуральную среду; 3) культура В. subtilis легко масштабируется, возможно культивирование в больших ферментерах (R. Jing Ma et al. 2017). Однако, несмотря на достоинства, продукция LipA в рекомбинантной В. subtilis все еще ограничена из-за таких проблем, как низкий уровень транскрипции, блокирование секреторного аппарата, деградация внеклеточной протеазы и так далее. Поэтому поиск и конструирование векторов, позволяющих оптимизировать регуляторные элементы экспрессии В. subtilis, являются актуальными научными задачами.
Раскрытие изобретения
Задачей настоящего изобретения является разработка экспрессионной системы и структуры плазмиды для получения липазы А, продуцируемой штаммом Bacillus natto IAN.
Техническим результатом изобретения является экспрессионная генетическая конструкция pBU-LipA, содержащая нуклеотидную последовательность белка липазы А, обеспечивающая экспрессию белка липазы А штамма Bacillus natto IAN в культуральную среду.
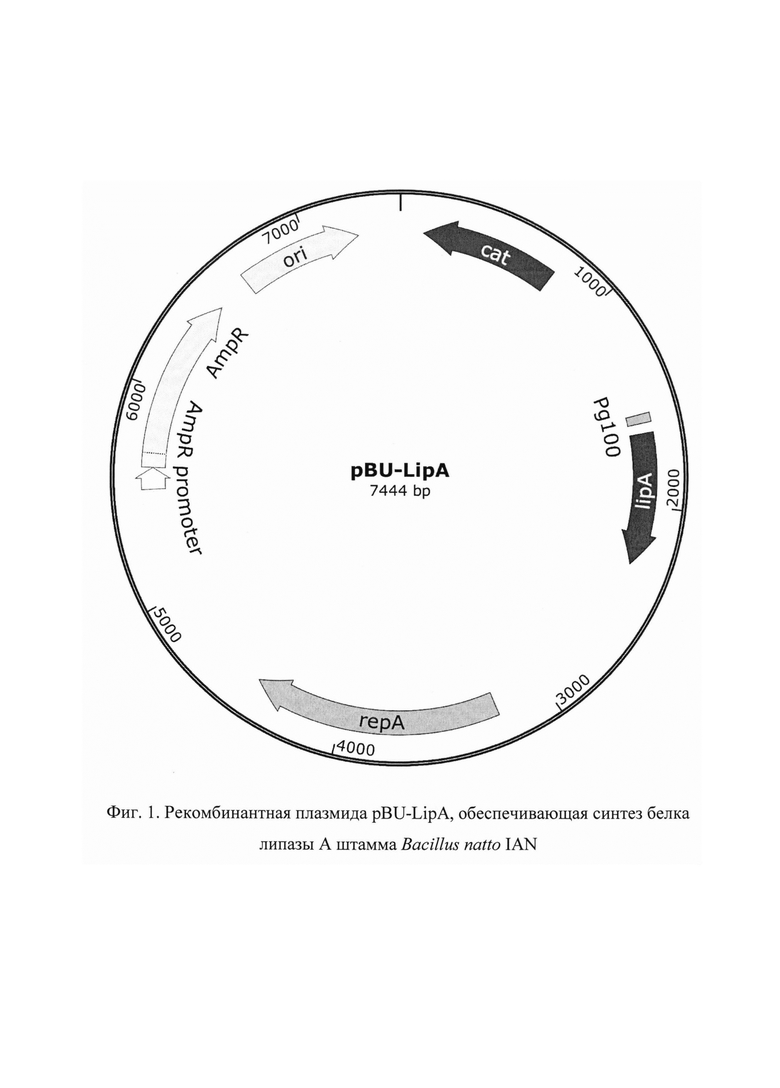
Указанный технический результат достигается получением рекомбинантной плазмиды pBU-LipA, размером 7444 пар нуклеотидов (п.н.), характеризующейся физической и генетической картой, представленной на фиг. 1, имеющей нуклеотидную последовательность SEQ ID NO: 1, обеспечивающей синтез рекомбинантного белка липазы А штамма Bacillus natto IAN массой 19,3 кДа в клетках Bacillus subtilis. Рекомбинантный ген белка липазы А, имеющей нуклеотидную последовательность SEQ ID NO: 2 от нуклеотида в положении 1650 п. о. до нуклеотида в положении 2285 п. о., кодирующий целевой белок липазы A Bacillus natto полной аминокислотной последовательностью SEQ ID NO: 3, встроен по сайтам рестрикции BamHI и AatII в область полилинкера вектора pBU, содержащего сильный промотор, что обеспечивает эффективную наработку рекомбинантного белка и его экспорт в культуральную среду.
Изобретение иллюстрируется следующими графическими фигурами. На фиг. 1 представлена физическая и генетическая карта рекомбинантной плазмиды pBU-LipA, предназначенной для синтеза липазы A Bacillus natto в клетках Bacillus subtilis, имеющей следующие основные структурно-функциональные регионы:
- нуклеотидная последовательность, кодирующая устойчивость к хлорамфениколу (cat) (113-763 п. н.);
- нуклеотидная последовательность синтетического промотора Pg100 (1557-1600 п. н.);
- нуклеотидная последовательность, кодирующая ген липазы А (SEQ ID NO: 1) (1650-2285 п. н.);
- нуклеотидная последовательность начала репликации в Bacillus subtilis (repA) (3246-4438 п. н.);
- ген устойчивости к антибиотику ампициллин AmpR (5626-6486 п. н.) и бактериальный промотор гена устойчивости к ампициллину (5521-5625 п. н.), обеспечивающие репликацию плазмиды в E.coli;
- участок начала репликации ori (6657-7245 п. н.).
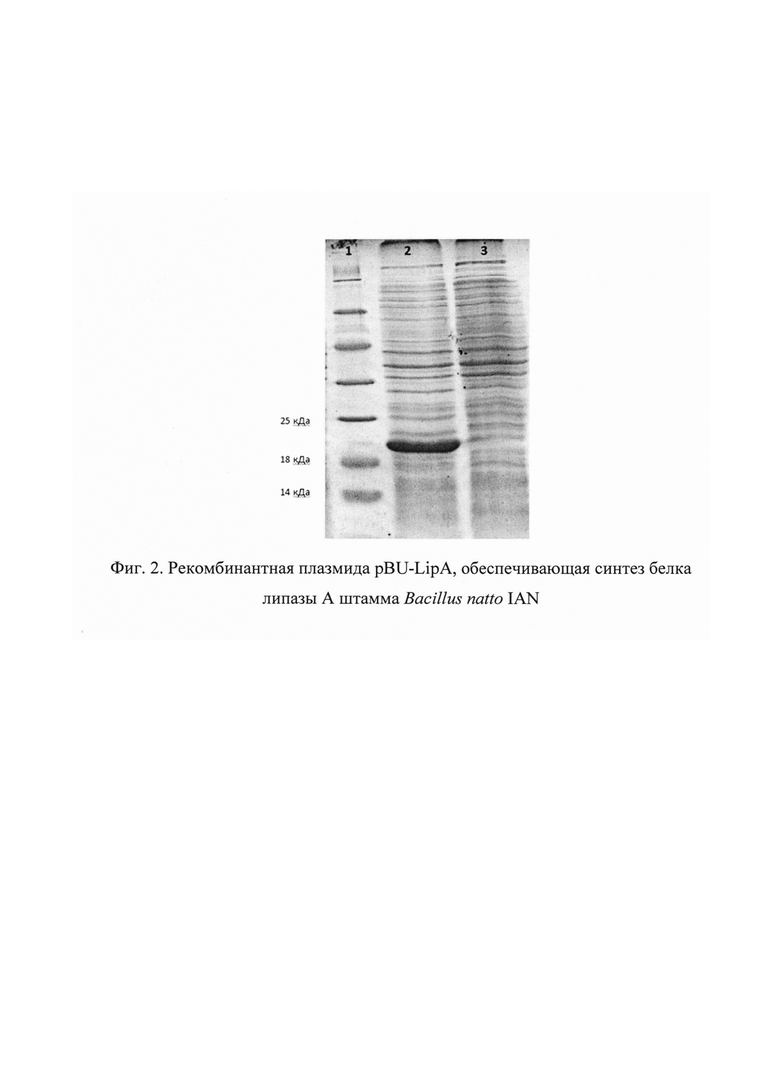
На фиг. 2. представлена электрофореграмма, иллюстрирующая наработку липазы A Bacillus natto IAN в клетках Bacillus subtilis: 1 - маркер молекулярного веса (Thermo Scientific Unstained Protein Molecular Weight Marker); 2 - культуральная жидкость, содержащая рекомбинантный белок липазы A Bacillus natto IAN; 3 - культуральная жидкость, полученная при культивировании нетрансформированной Bacillus subtilis (отрицательный контроль).
Для лучшего понимания сущности предлагаемого изобретения ниже приведены примеры (1-3) его осуществления.
Пример 1. Конструирование рекомбинантной плазмиды pBU-LipA для синтеза липазы A Bacillus natto IAN в системе Bacillus subtilis
Последовательность гена липазы А была амплифицирована из колонии дикого штамма Bacillus natto из коллекции инжинирингового центра «Промбиотех». Для амплификации использовались праймеры LipA-F и LipA-R (таблица 1). ПЦР-продукт гена липазы А размером 636 п.н. в составе реакционной смеси был нанесен на агарозный гель, выделен из геля с помощью набора «Евроген» Cleanup Standard (Россия) в соответствии с рекомендациями производителя, встроен в состав вектора pJET.2.1, входящего в набор CloneJET PCR Cloning Kit (Thermo Scientific™), в соответствии с инструкциями производителя. Полученной плазмидой pJET-LipA трансформировали культуру Е. coli штамм Stbl3 по общепринятой методике с помощью CaCl2. Далее выделяли плазмидную ДНК pJET-LipA с помощью набора Plasmid Miniprep («Евроген», Москва) в соответствии с инструкцией производителя и проверяли секвенированием по методу Сэнгера с помощью набора CEQ2000Dye Terminator Cycle Sequencing Kit в соответствии с инструкциями производителя. 
Далее в нуклеотидную последовательность липазы А вводили сайты рестрикции BamHI и AatII с помощью ПЦР с использованием праймеров LpA-F и LpA-R (таблица 1), в качестве матрицы для амплификации была использована плазмида pJET-LipA.
На основе вектора pBU, оптимизированного под штамм Bacillus subtilis, получали конструкцию pBU-LipA (фиг. 1), содержащую нуклеотидную последовательность LipA. Для этого проводили ферментативный гидролиз вектора pBU и ПЦР-продукта нуклеотидной последовательности, кодирующей липазу А, эндонуклеазами рестрикции BamHI и AatII. Далее проводили лигирование гидролизованных ПЦР-продукта и вектора pBU и последующую трансформацию по общепринятой методике с помощью CaCl2 культуры E.coli штамм Stbl3 полученной плазмидой. Выделяли полученную плазмидную ДНК pBU-LipA с помощью набора Plasmid Miniprep («Евроген», Москва) в соответствии с инструкцией производителя. Идентичность плазмиды pBU-LipA спроектированной подтверждали секвенированием по методу Сэнгера с помощью набора CEQ2000Dye Terminator Cycle Sequencing Kit в соответствии с инструкциями производителя.
Пример 2. Получение рекомбинантного штамма Bacillus subtilis, обеспечивающего синтез липазы A Bacillus natto IAN
Полученной рекомбинантной плазмидой pBU-LipA была проведена трансформация методом электропорации клеток В. subtilis WB800N. Культуру клеток в количестве 50 мл подращивали до ODλ600=0.2 о.е. Затем в культуру добавили 0,5 гр. трионина, 1 гр. глицил-глицина, 0,05 гр. триптофана и 15 мкл TWEEN 80 и инкубировали в течение часа при 30°С. После культуру клеток инкубировали 20 мин на льду. 50 мл культуры центрифугировали 10 мин 5000g при 4°С на центрифуге AvantiJ-301. Осажденную биомассу дважды промыли в 5 мл ЕР Buffer (0,5 М трегалоза, 0,5 М сорбитол, 0,5 М маннитол, 0,7 мМ MgCl, 0,5 мМ K2HPO4, 0,5 мМ KH2PO4, рН=7,4). После промывки ресуспендировали в 0,5 мл ЕР Buffer. 100 мкл суспензии клеток перенесли в 2 мл холодные кюветы для электропорации и добавили ДНК с расчетом 25 нг/мкл. Произвели электропорацию при 2,5 kV на электропораторе MicroPulser. К про электропорированным клеткам добавили питательную среду YTx2 с 0,5 М сорбитолом и 0,38 М маннитолом. Инкубировали 3 часа при 37°С. 50 и 200 мкл культуры высевали на чашки с агаризованной средой YTx2, содержащие 5 мкг/мл хлорамфеникола. Чашки помещали в термостат 30°С и инкубировали в течение ночи. На следующий день были идентифицированы отдельные колонии, содержащие плазмиду pBU-LipA.
Пример 3. Получение рекомбинантного белка липазы A Bacillus natto IAN
Полученные клоны В. subtilis, штамм SVB-1 после электропорации рекомбинантной плазмидой pBU-LipA культивировали в качалочных колбах объемом 750 мл в течение 16 часов при 30°С, 170 об/мин. Биомассу бактериальных клеток отделяли от культуральной среды центрифугированием (7000 об/мин), в течение 10 мин, 4°С. Наличие целевого белка липазы А Bacillus natto IAN (молекулярный вес 19,3 kDa) в культуральной среде анализировали с помощью 12% ПААГ (фиг. 2).
Список использованных источников
1. RU 2508402 С1. Термостабильная липаза из бактерии Thermosyntropha lipolytica, активная в щелочной среде. Гумеров В.М. (RU), Марданов А.В. (RU), Равин Н.В. (RU). Патентообладатель(и): Федеральное государственное бюджетное учреждение науки Центр "Биоинженерия" Российской академии наук (RU). Заявка: 2012141676/10. Дата подачи заявки: 02.10.2012. Опубликовано: 27.02.2014. Бюл. №6.
2. Borrelli G. М., Trono D. Recombinant lipases and phospholipases and their use as biocatalysts for industrial applications // International journal of molecular sciences. 2015. Vol. 16. №9. P. 20774-20840.
3. T. Eggert, G. van Pouderoyen, G. Pencreac'h, I. Douchet, R. Verger, B. W. Dijkstra, K.-E. Jaeger Biochemical properties and three-dimensional structures of two extracellular lipolytic enzymes from Bacillus subtilis Colloids and Surfaces B: Biointerfaces 26 (2002) 37-46.
4. R. Jing Ma, Y. H. Wang, L. Liu, L. L. Bai, R. Ban Production enhancement of the extracellular lipase LipA in Bacillus subtilis: Effects of regulatory elements and Sec pathway components. Protein Expression and Purification (2017), doi: 10.1016/j.pep.2017.09.011.
5. Безбородов A.M., Загустина H.А. Липазы в реакциях катализа в органическом синтезе (обзор). Прикладная биохимия и микробиология, 2014, том 50, №4, с. 347-373.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="RU-pBU-LipA.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2022-12-16">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>RU-1-pBU-LipA</ApplicationNumberText>
<FilingDate>2022-12-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>RU-1-pBU-LipA</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>RU-1-pBU-LipA</ApplicationNumberText>
<FilingDate>2022-12-14</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБОУ ВО
«Алтайский государстРIенный
СѓРЅРёРIерситет»</ApplicantName>
<ApplicantNameLatin>Altai State University</ApplicantNameLatin>
<InventionTitle languageCode="ru">Рекомбинантная
плаЕмида pBU-LipA, обеспечиРIающая синтеЕ
белка липаЕы А штамма Bacillus natto
IAN</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>7444</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7444</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttaagttattggtatgactggttttaagcgcaaaaaaagttgctttttc
gtacctattaatgtatcgttttagaaaaccgactgtaaaaagtacagtcggcattatctcatattataaa
agccagtcattaggcctatctgacaattcctgaatagagttcataaacaatcctgcatgataaccatcac
aaacagaatgatgtacctgtaaagatagcggtaaatatattgaattacctttattaatgaattttcctgc
tgtaataatgggtagaaggtaattactattattattgatatttaagttaaacccagtaaatgaagtccat
ggaataatagaaagagaaaaagcattttcaggtataggtgttttgggaaacaatttccccgaaccattat
atttctctacatcagaaaggtataaatcataaaactctttgaagtcattctttacaggagtccaaatacc
agagaatgttttagatacaccatcaaaaattgtataaagtggctctaacttatcccaataacctaactct
ccgtcgctattgtaaccagttctaaaagctgtatttgagtttatcacccttgtcactaagaaaataaatg
cagggtaaaatttatatccttcttgttttatgtttcggtataaaacactaatatcaatttctgtggttat
actaaaagtcgtttgttggttcaaataatgattaaatatctcttttctcttccaattgtctaaatcaatt
ttattaaagttcatttgatatgcctcctaaatttttatctaaagtgaatttaggaggcttacttgtctgc
tttcttcattagaatcaatccttttttaaaagtcaatattactgtaacataaatatatattttaaaaata
tcccactttatccaattttcgtttgttgaactaatgggtgctttagttgaagaataaagaccacattaaa
aaatgtggtcttttgtgtttttttaaaggatttgagcgtagcgaaaaatccttttctttcttatcttgat
aataagggtaactattgccgatcgtccattccgacagcatcgccagtcactatggcgtgctgctagcgcc
attcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagct
ggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacgttgt
aaaacgacggccagtgaattcgagctcaggccttaactcacattaattgcgttgcgctcactgcccgctt
tccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcg
tattgggcgtacgatctttcagccgactcaaacatcaaatcttacaaatgtagtctttgaaagtattaca
tatgtaagatttaaatgcaaccgttttttcggaaggaaatgatgacctcgtttccaccggaattagcttg
gtaccaaaggaggtaaggatcactagaaaattttttaaaaaatctcttgacattggaagggagatatgtt
attataagaattgcggaattgtgagcggataacaattcccatataaaggaggaaggatccatgaaatttg
taaaaagaaggatcattgcacttgtaacaattttgatgctgtctgttacatcgctgtttgcgttgcagcc
gtcagtaaaagccgctgaacacaatccagtcgttatggttcacggcattggaggggcatcattcaatttt
gcgggaattaagagctatctcgtatctcagggctggtcgcgggacaagctgtatgcagttgatttttggg
acaagacaggcacaaattataacaatggcccggtattgtcacgatttgtgcaaaaggttttagatgaaac
gggtgcgaaaaaagtggatattgtcgctcacagtatggggggcgcgaacacactttactacataaaaaat
ctggacggcggaaataaagttgaaaacgtcgtgacgcttggcggcgccaaccgtttgacgacaggcaagg
cgcttccgggaacagatccaaatcaaaagattttatacacatccatttacagcagtgccgatatgattgt
catgaattatttatcaagattagacggtgctagaaacgttcaaatccatggcgttggacacatcggcctt
ctgtacagcagccaagtcaacagcctgattaaagaagggctgaacggcgggggccagaatacaaaccatc
atcaccatcaccactaagacgtccccggggcagcccgcctaatgagcgggcttttttcacgtcacgcgtc
catggagatctttgtctgcaactgaaaagtttataccttacctggaacaaatggttgaaacatacgaggc
taatatcggcttattaggaatagtccctgtactaataaaatcaggtggatcagttgatcagtatattttg
gacgaagctcggaaagaatttggagatgacttgcttaattccacaattaaattaagggaaagaataaagc
gatttgatgttcaaggaatcacggaagaagatactcatgataaagaagctctaaaactattcaataacct
tacaatggaattgatcgaaagggtggaaggttaatggtacgaaaattaggggatctacctagaaagccac
aaggcgataggtcaagcttaaagaacccttacatggatcttacagattctgaaagtaaagaaacaacaga
ggttaaacaaacagaaccaaaaagaaaaaaagcattgttgaaaacaatgaaagttgatgtttcaatccat
aataagattaaatcgctgcacgaaattctggcagcatccgaagggaattcatattacttagaggatacta
ttgagagagctattgataagatggttgagacattacctgagagccaaaaaactttttatgaatatgaatt
aaaaaaaagaaccaacaaaggctgagacagactccaaacgagtctgtttttttaaaaaaaatattaggag
cattgaatatatattagagaattaagaaagacatgggaataaaaatattttaaatccagtaaaaatatga
taagattatttcagaatatgaagaactctgtttgtttttgatgaaaaaacaaacaaaaaaaatccaccta
acggaatctcaatttaactaacagcggccaaactgagaagttaaatttgagaaggggaaaaggcggattt
atacttgtatttaactatctccattttaacattttattaaaccccatacaagtgaaaatcctcttttaca
ctgttcctttaggtgatcgcggagggacattatgagtgaagtaaacctaaaaggaaatacagatgaatta
gtgtattatcgacagcaaaccactggaaataaaatcgccaggaagagaatcaaaaaagggaaagaagaag
tttattatgttgctgaaacggaagagaagatatggacagaagagcaaataaaaaacttttctttagacaa
atttggtacgcatataccttacatagaaggtcattatacaatcttaaataattacttctttgatttttgg
ggctattttttaggtgctgaaggaattgcgctctatgctcacctaactcgttatgcatacggcagcaaag
acttttgctttcctagtctacaaacaatcgctaaaaaaatggacaagactcctgttacagttagaggcta
cttgaaactgcttgaaaggtacggttttatttggaaggtaaacgtccgtaataaaaccaaggataacaca
gaggaatccccgatttttaagattagacgtaaggttcctttgctttcagaagaacttttaaatggaaacc
ctaatattgaaattccagatgacgaggaagcacatgtaaagaaggctttaaaaaaggaaaaagagggtct
tccaaaggttttgaaaaaagagcacgatgaatttgttaaaaaaatgatggatgagtcagaaacaattaat
attccagaggccttacaatatgacacaatgtatgaagatatactcagtaaaggagaaattcgaaaagaaa
tcaaaaaacaaatacctaatcctacaacatcttttgagagtatatcaatgacaactgaagaggaaaaagt
cgacagtactttaaaaagcgaaatgcaaaatcgtgtctctaagccttcttttgatacctggtttaaaaac
actaagatcaaaattgaaaataaaaattgtttattacttgtaccgagtgaatttgcatttgaatggatta
agaaaagatatttagaaacaattaaaacagtccttgaagaagctggatatgttttcgaaaaaatcgaact
aagaaaagtgcaataaactgctgaagtatttcagcagttttttttatttagaaatagtgaaaaaaatata
atcagggaggtatcaatatttaatgagtactgatttaaatttatttagactggaattaataattaacacg
tagactaattaaaatttaatgagggataaagaggatacaaaaatattaatttcaatccctattaaatttt
aacaagggggggattaaaatttaattagaggtttatccacaagaaaagaccctaataaaatttttactag
ggttataacactgattaatttcttaatgggggagggattaaaatttaatgacaaagaaaacaatctttta
agaaaagcttttaaaagataataataaaaagagctttgcgattaagcaaaactctttactttttcattga
cattatcaaattcatcgatttcaaattgttgttgtatcataaagttaattctgttttgcacaaccttttc
aggaatataaaacacatctgaggcttgttttataaactcagggtcgctaaagtcaatgtaacgtagcata
tgatatggtatagcttccacccaagttagcctttctgcttcttctgaatgtttttcatatacttccatgg
gtatctctaaatgattttcctcatgtagcaaggtatgagcaaaaagtttatggaattgatagttcctctc
tttttcttcaacttttttatctaaaacaaacactttaacatctgagtcaatgtaagcataagatgttttt
ccagtcataatttcaatcccaaatcttttagacagaaattctggacgtaaatcttttggtgaaagaattt
ttttatgtagcaatatatccgatacagcaccttctaaaagcgttggtgaatagggcattttacctatctc
ctctcattttgtggaataaaaatagtcatattcgtccatctacctatcctattatcgaacagttgaactt
tttaatcaaggatcagtcctttttttcattattcttaaactgtgctcttaactttaacaactcgatttgt
ttttccagatctcgagggtaactagcctcgccgatcccgcaagaggcccggcagtcaggtggcacttttc
ggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgag
acaataaccctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtc
gcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaa
aagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatcct
tgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggta
ttatcccgtattgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttg
agtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccat
aaccatgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgct
tttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccatac
caaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcga
actacttactctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccactt
ctgcgctcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtctcgcg
gtatcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtca
ggcaactatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaactg
tcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctagg
tgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcaga
ccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaaca
aaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaa
ctggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaa
gaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgat
aagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacgg
ggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagct
atgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaaca
ggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacc
tctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgc
ggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgat
tctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgca
gcgagtcagtgagcgaggaagcggaagagcgcccaatacgcatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>636</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..636</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaatttgtaaaaagaaggatcattgcacttgtaacaattttgatgc
tgtctgttacatcgctgtttgcgttgcagccgtcagtaaaagccgctgaacacaatccagtcgttatggt
tcacggcattggaggggcatcattcaattttgcgggaattaagagctatctcgtatctcagggctggtcg
cgggacaagctgtatgcagttgatttttgggacaagacaggcacaaattataacaatggcccggtattgt
cacgatttgtgcaaaaggttttagatgaaacgggtgcgaaaaaagtggatattgtcgctcacagtatggg
gggcgcgaacacactttactacataaaaaatctggacggcggaaataaagttgaaaacgtcgtgacgctt
ggcggcgccaaccgtttgacgacaggcaaggcgcttccgggaacagatccaaatcaaaagattttataca
catccatttacagcagtgccgatatgattgtcatgaattatttatcaagattagacggtgctagaaacgt
tcaaatccatggcgttggacacatcggccttctgtacagcagccaagtcaacagcctgattaaagaaggg
ctgaacggcgggggccagaatacaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>212</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..212</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MKFVKRRIIALVTILMLSVTSLFALQPSVKAAEHNPVVMVHGIGGASFN
FAGIKSYLVSQGWSRDKLYAVDFWDKTGTNYNNGPVLSRFVQKVLDETGAKKVDIVAHSMGGANTLYYIK
NLDGGNKVENVVTLGGANRLTTGKALPGTDPNQKILYTSIYSSADMIVMNYLSRLDGARNVQIHGVGHIG
LLYSSQVNSLIKEGLNGGGQNTN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантная плазмида pBU-Sav, обеспечивающая синтез и секрецию белка щелочной протеазы в системе Bacillus subtilis | 2023 |
|
RU2816513C1 |
| Рекомбинантная плазмида pUSB2-AmQ, обеспечивающая синтез белка альфа-амилазы Bacillus amyloliquefaciens, и штамм Bacillus subtilis/pUSB2-AmQ - продуцент белка альфа-амилазы Bacillus amyloliquefaciens | 2020 |
|
RU2747627C1 |
| ФЕРМЕНТАТИВНОЕ ПОЛУЧЕНИЕ 1-БУТАНОЛА | 2006 |
|
RU2429295C2 |
| БАКТЕРИЯ РОДА BACILLUS, ПРОДУЦИРУЮЩАЯ ГИАЛУРОНОВУЮ КИСЛОТУ, И СПОСОБ ПОЛУЧЕНИЯ ГИАЛУРОНОВОЙ КИСЛОТЫ С ИСПОЛЬЗОВАНИЕМ УКАЗАННОЙ БАКТЕРИИ | 2019 |
|
RU2719140C1 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК ДЛЯ ОБНАРУЖЕНИЯ АГЕНТОВ, ПОВРЕЖДАЮЩИХ ГЕНЕТИЧЕСКИЙ АППАРАТ КЛЕТКИ (ВАРИАНТЫ) | 2005 |
|
RU2311459C2 |
| ФЕРМЕНТАТИВНОЕ ПОЛУЧЕНИЕ ЧЕТЫРЕХУГЛЕРОДНЫХ СПИРТОВ | 2006 |
|
RU2394913C2 |
| Плазмида для редактирования генома бактерий рода Bacillus и способ внесения модификаций в геном бактерий рода Bacillus | 2022 |
|
RU2815835C1 |
| БАКТЕРИЯ - ПРОДУЦЕНТ ПРОДУКТА РЕАКЦИИ, КАТАЛИЗИРУЕМОЙ БЕЛКОМ, ОБЛАДАЮЩИМ АКТИВНОСТЬЮ 2-ОКСОГЛУТАРАТ-ЗАВИСИМОГО ФЕРМЕНТА, И СПОСОБ ПРОДУКЦИИ УКАЗАННОГО ПРОДУКТА | 2008 |
|
RU2444568C2 |
| СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА ПУРИНОВОГО НУКЛЕОЗИДА 5'-АМИНОИМИДАЗОЛ-4-КАРБОКСАМИДРИБОЗИДА (АИКАР) И ШТАММ БАКТЕРИЙ BACILLUS SUBTILIS - ПРОДУЦЕНТ АИКАР | 2008 |
|
RU2405833C2 |
| СПОСОБ ПРОДУКЦИИ 2-АМИНО-3-МЕТИЛ-4-КЕТОПЕНТАНОАТА | 2007 |
|
RU2392323C2 |
Изобретение относится к области биотехнологии. Предложена рекомбинантная плазмида pBU-LipA, обеспечивающая синтез рекомбинантного белка липазы А штамма Bacillus natto IAN массой 19,3 кДа в клетках Bacillus subtilis. Размер плазмиды 7444 п. н. Плазмида характеризуется физической и генетической картой, представленной на фиг. 1. Рекомбинантный ген, кодирующий целевой белок липазы A Bacillus natto, встроен по сайтам рестрикции BamHI и AatII в область полилинкера вектора pBU, содержащего сильный промотор и сигнальную последовательность. Изобретение обеспечивает эффективную наработку рекомбинантного белка липазы A и его экспорт в культуральную среду. 2 ил., 1 табл., 3 пр.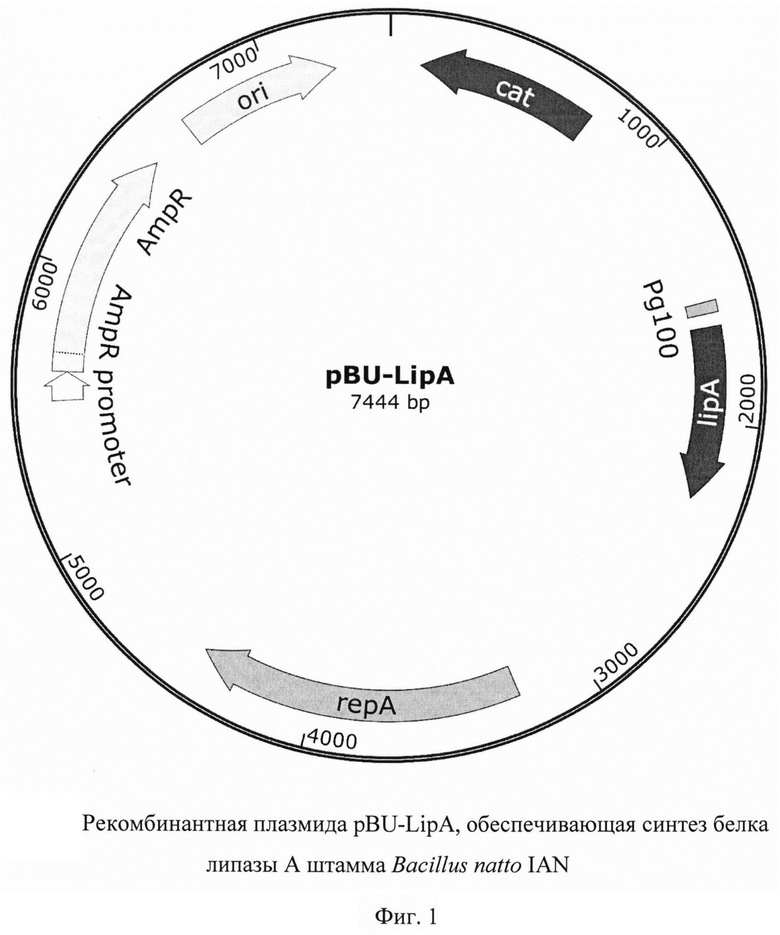
Рекомбинантная плазмида pBU-LipA, обеспечивающая синтез рекомбинантного белка липазы А штамма Bacillus natto IAN массой 19,3 кДа в клетках Bacillus subtilis, имеющая нуклеотидную последовательность SEQ ID NO: 1 размером 7444 п. н. и содержащая в соответствии с физической и генетической картой следующие структурно-функциональные регионы:
- нуклеотидная последовательность, кодирующая устойчивость к хлорамфениколу (cat) (113-763 п. н.);
- нуклеотидная последовательность синтетического промотора Pg100 (1557-1600 п. н.);
- ген липазы А с нуклеотидной последовательностью SEQ ID NO: 2 от нуклеотида в положении 1650 по 2285 п. н., кодирующий целевой белок липазы A Bacillus natto полной аминокислотной последовательностью SEQ ID NO: 3 и встроенный по сайтам рестрикции BamHI и AatII в область полилинкера вектора pBU, содержащего сильный промотор и сигнальную последовательность, что обеспечивает эффективную наработку рекомбинантного белка и его экспорт в культуральную среду;
- нуклеотидная последовательность начала репликации в Bacillus subtilis (repA) (3246-4438 п. н.);
- ген устойчивости к антибиотику ампициллину AmpR (5626-6486 п. н.) и бактериальный промотор гена устойчивости к ампициллину (5521-5625 п. н.), позволяющие проводить препаративную наработку плазмиды в Е. coli;
- участок начала репликации ori (6657-7245 п. н.).
| РЕКОМБИНАНТНЫЙ ШТАММ БАКТЕРИЙ BACILLUS LICHENIFORMIS - ПРОДУЦЕНТ ТЕРМОСТАБИЛЬНОЙ ЛИПАЗЫ | 2012 |
|
RU2500812C1 |
| ШТАММ БАКТЕРИИ Escerichia coli XL1-blue/pQS-G3, ПРОДУЦЕНТ ТЕРМОСТАБИЛЬНОЙ ЛИПАЗЫ БАКТЕРИИ Geobacillus stearothermophilus G3 | 2013 |
|
RU2540873C1 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pYP2, КОДИРУЮЩАЯ ФЕРМЕНТАТИВНО-АКТИВНУЮ ЧАСТЬ ПОЛИПЕПТИДА ЛИПАЗЫ, БЕЛКА ПИЩЕВАРИТЕЛЬНОГО ТРАКТА ПЧЕЛИНОЙ ОГНЕВКИ | 2012 |
|
RU2507261C1 |
| WO 2009094084 A1, 30.07.2009 | |||
| JISHENG MA et al., Overexpression and characterization of a lipase from Bacillus subtilis, Protein Expression and Purification, 2006, Volume 45, Issue 1, pp.22-29. | |||
Авторы
Даты
2023-11-28—Публикация
2023-02-15—Подача