Изобретение относится к медицине, клинической лабораторной диагностике, в частности к проблеме изучения инфекционных вирусных заболеваний, и касается поиска новых направлений в специфической диагностике хронического вирусного гепатита С.
По данным Всемирной организации общественного здравоохранения, более 71 млн человек в мире страдают хроническим гепатитом С (ХГС). В РФ ежегодно регистрируется более 40 тыс.случаев впервые выявленного хронического вирусного гепатита С (ХВГС).
Источником инфекции являются лица, инфицированные ВГС. Механизм передачи инфекции - гемоконтактный, который осуществляется за счет крови и ее компонентов, в меньшей степени других физиологических жидкостей.
Развитие инфекции наступает через 2-12 недель после заражения (в среднем 7 недель) и длится от 2 до 12 недель. У большинства пациентов острая стадия протекает бессимптомно либо выражается общими клиническими проявлениями - слабость, недомогание, тяжесть в правом подреберье при физических нагрузках, миалгия, субфебрилитет и тд. Превышение аланинаминотрансферазы (АЛТ) так же не специфично.
Различают семь основных генотипов ВГС и более 60 субтипов. Относительно нуклеотидных последовательностей, генетическое разнообразие между генотипами ВГС составляет около 30%, а различие между субтипами -15%.
Генетическая рекомбинация является механизмом, участвующим в генетической гетерогенности ВГС. Рекомбинация создает генетическую изменчивость за счет перестройки геномных молекул во время элонгации РНК, когда полимераза переключается с донорских на акцепторные молекулы, в результате чего образуется РНК с признаками обоих родительских вирусов. Для возникновения рекомбинации требуется коинфекция или суперинфекция в одной и той же клетке двумя родительскими вирусами. Рекомбинация ВГС имеет важные клинические и эпидемиологические последствия, влияющие на молекулярную эпидемиологию, надзор за возникающими новыми линиями и штаммами с лекарственной устойчивостью.
Помимо прямого цитопатического воздействия на ткань печени, ХВГС характеризуется хроническим воспалительным процессом, что приводит к повышению уровня аминотрансфераз, развитию цирроза в 10-20% в течение 20-30 лет. У пациентов, живущих с ХВГС в течение 30 лет и более, частота цирроза оценивается в 41%, что почти в 3 раза выше, чем частота, прогнозируемая через 20 лет.
Появление точечных мутаций из-за отсутствия корректирующей способности у РНК-зависимой РНК-полимеразы является основным элементом, способствующим высокой генетической изменчивости ВГС. Частота мутаций ВГС in vivo составляет около 2,5×10-5 на нуклеотид на репликацию генома.
Внешнее селективное давление, такое как иммунный ответ хозяина и противовирусное лечение определяют накопление мутаций в субгеномных регионах.
По сравнению с терапией интерфероном, благодаря появлению препаратов прямого противовирусного действия (ПГШД), значительно увеличилась частота достижения устойчивого вирусологического ответа (более 90%), сократилась продолжительность курса (до 12 недель), снизилось количество побочных явлений и режим приема препарата стал более удобен для пациентов. Также использование новых лекарственных средств позволило обеспечить лечение пациентам с циррозом печени и другими осложнениями и достигать высоких значений устойчивого вирусологического ответа, что при предшествующей схеме терапии было проблематично.
На данный момент выделяют 4 группы препаратов прямого противовирусного действия, нацеленных на 3 вирусных неструктурных белка-мишени: ингибиторы NS3-протеазы, ингибиторы NSSA-репликативного комплекса, нуклеотдиные и ненуклеотидные ингибиторы NS5B РНК-полимеразы.
Тем не менее, для доли пациентов (около 5%) противовирусная терапия оказывается неэффективной. Неудачи могут быть связаны с феноменом лекарственной устойчивости вируса к противовирусным препаратам. По некоторым данным, в 83% случаев неудачного исхода терапии обнаруживались одна или несколько мутаций резистентности в участках генома, кодирующих белки-мишени для действия ПППД.
Возникновение устойчивости вируса связано с наличием нуклеотидных замен, появляющихся спонтанно в генах NS3, NS5A, NS5B ВГС при каждом цикле репликации. Большинство подобных замен в геноме нарушают фитнес (способность вируса выживать и размножаться в определенных условиях) дикого вируса в обычных условиях, но в условиях приема ПППД мутантные штаммы имеют преимущество перед диким типом вируса.
Увеличение числа случаев рекомбинантных вариантов ВГС может повлиять на выбор оптимальной терапии против ВГС, поскольку пангенотипные схемы недоступны для многих пациентов из-за их высокой стоимости. Лечение пациентов с рекомбинантным ВГС затруднено по сравнению с другими генотипами. В таких случаях необходимо контролировать все этапы терапии для предотвращения неудач из-за различного действия противовирусных препаратов на разные генотипы.
Известен метод, предложенный в работе коллег, посвященный обнаружению рекомбинантных форм ВГС, представляющий собой генотипирование и определение точки рекомбмнации ВГС посредством секвенирования фрагментов белков core, NS5B и NS2 [Чуб Е.В., Сиволобова Г.Ф., Нетесов С.В., Кочнева Г.В. Рекомбинантные варианты вируса гепатита С в Сибири. Молекулярная генетика, микробиология и вирусология. 2019;37(2):64-75. https://doi.org/10.17116/molgen20193702164]. Данный метод позволяет определить рекомбинантные формы ВГС генотипов 1b, 3a, 2a, 2c, 2k. 2a, 2c, 2k. Недостаток метода заключается в ограниченном количестве определяемых генотипов и не позволяет определить мутации ЛУ в других регионах вируса.
Наиболее близким по сущности к заявляемому изобретению и выбранным за прототип является метод, посвященный обнаружению мутаций лекарственной устойчивости ВГС к ПППД, представляющий собой секвенирование фрагментов белков NS3, NS5A, NS5B [Dietz J, et al Patterns of Resistance-Associated Substitutions in Patients With Chronic HCV Infection Following Treatment With Direct-Acting Antivirals. Gastroenterology. 2018 Mar;154(4):976-988.e4. doi: 10.1053/j.gastro.2017.11.007.]. Данный метод позволяет определить мутации лекарственной устойчивости к ингибиторам NS3, NS5A, NS5B ВГС генотипов 1-6 методом гнездовой ПЦР. Недостаток метода заключается в отсутствии возможности определения рекомбинантой формы вируса.
Общим недостатком всех вышеуказанных методов является отсутствие возможности выявления рекомбинантных форм ВГС на ряду с определением мутаций лекарственной устойчивости к ПППД.
Авторами предложен метод одновременного выявления мутаций лекарственной устойчивости ВГС в регионах NS3, NS5A, NS5B и рекомбинантных штаммов ВГС с помощью определения и дальнейшего филогенетического анализа регионов Core и NS5B с последующим определением точки рекомбинации, расположенной в гене NS2.
Технический результат - определение мутаций лекарственной устойчивости ВГС к ПППД на ряду с возможным определением рекомбинантной формы вируса. Этот результат достигается за счет получения нуклеотидных последовательностей пяти регионов (Core, NS2, NS3, NS5A, NS5B) и сравнения их с референсными последовательностями.
Сущность метода заключается в том, что на первом этапе проводят амплификация РНК вируса с использованием олигонуклеотидных праймеров, специфичных для каждого генотипа, комплементарных областям наибольшего сходства геномов различных изолятов ВГС, получая в качестве результата пять продуктов амплификации (Core, NS2, NS3, NS5A, NS5B). Для повышения чувствительности проводят вторую полимеразную цепную реакцию с использованием продукта амплификации первой реакции, внутренних (вложенных, гнездовых) праймеров для ВГС, комплементарных участкам амплифицируемых на втором этапе фрагментов.
На первом этапе используются следующие олигонуклеотидные праймеры:
Регион Core для всех генотипов:
Core-fl 5'-GCAACAGGGAACCTTCCTGGTTGCTC-3'
Core-rl 5'-CGTAGGGGACCAGTTCATCATCAT-3'
Регион NS2 для генотипа 2:
NS2-fl 5'-TAGTGTTTGACATAACCAAGTGGC-3'
NS2-rl 5'-CCCCTTCGGGCGGAGACG-3'
Регион NS3 генотип 1а:
NS3-fl-la 5'-CCRRATGGAGACCAAGMTCATYACGT-3'
NS3-rl-la 5'-TCNGTDGARTGGCACTCRTCACA-3'
Регион NS3 генотип lb:
NS3-fl-lb 5'-GAYATGGAGAYYAAGRTCATYACCTGG-3'
NS3-rl-lb 5'-TCCAGCCGTCTCCGCTTGG-3'
Регион NS3 генотип 2:
NS3-fl-2 5'-CTCACMCCTATGTCGGAYTGGGC-3'
NS3-rl-2 5'-CCATCGGCGAGRAATTTGCCATAYGTGGA-3'
Регион NS3 генотип 3а:
NS3-fl-3a 5'-GAYCACCTAGCRCCAATGCAACA-3'
NS3-rl-3a5'-TAGGTGGARTAGGTCAGTTTRGCACCAGTTG-3'
Регион NS5A генотип la:
NS5A-fl-la 5'-GTTGGCCCGGGCGAGG-3'
NS5A-rl-la 5'-CGCCCATCTCCTGCCTCCA-3'
Регион NS5A генотип lb:
NS5A-fl-lb 5'-TGGATGAACCGGCTGATAG-3'
NS5A-rl-lb 5'-CCACAGGAGRTTGGCCTC-3'
Регион NS5A генотип 2:
NS5A-fl-2 5'-GCGGTCCAGTGGATGAACAG-3'
NS5A-rl-2 5'-ACTCRATCCGGGTCACRTC-3'
Регион NS5A генотип 3а:
NS5A-fl-3a 5'-TGGATGAACAGGCTCATYGC-3'
NS5A-rl-3a 5'-ATGTTRCTGCCCATCTCTTG-3'
Регион NS5B генотипы 1,2,3:
NS5B-lf 5'-GTSTGGIARGACYTICTGGAAGAC-3'
NS5B-lr 5'-RGIGCRGARTACCTRGTCATAGCCT-3'
Состав амплификационной смеси на первом этапе:
4 пмоль/л прямого праймера, 30 пмоль/л обратного праймера, по 0,8 ммоль/л всех нуклеозидтрифосфатов, 8,2 ммоль/л MgCb, 1 ед. рекомбинантной Taq ДНК-полимеразы (Fermentas), буфер для Taq ДНК-полимеразы (750 ммоль/л Трис-HCl, (рН 8,8), 200 ммоль/л (NH4)2SO4, 0,1% (v/v) твин 20), глицерин 5% от конечного объема, 10 мкл раствора ДНК, вода без нуклеаз до конечного объема 50 мкл. Условия проведения ГЩР представлены в таблице 1.
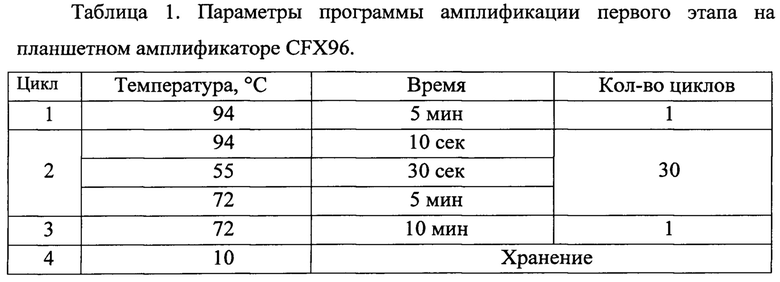
На втором этапе используют продукт амплификации первого этапа, а также следующие олигонуклеотиды, в одной пробирке:
Core-f2 5'-AACCTTCCTGGTTGCTCTTTCTCTAT-3'
Core-r2 5'-GTTCATCATCATATCCCATGCCAT-3'
Регион NS2 для генотипа 2:
NS2-f2 5'-TAGTGTTTGACATAACCAAGTGGC-3'
NS2-r2 5'-CCCCTTCGGGCGGAGACG-3'
Регион NS3 генотип 1 a:
NS3-f2-la 5' GCRTGYGGBGACATCATYAACGG-3'
NS3-r2-la 5'- CAVCCRCCRTCRGCAAGGAACTTG-3'
Регион NS3 генотип lb:
NS3-f2-lb 5'-GCRGCRTGYGGGGACATCAT-3'
NS3-r2-lb 5'-CCARGACYGTGCCRATGCCCA-3'
Регион NS3 генотип 2:
NS3-f2-2 5'-ATGGAGAAGAAGGTCATCGTCTGGGG-3'
NS3-r2-2 5'-GTCCTAATGTTGGGATTGATGCCATGTGC-3'
Регион NS3 генотип 3а:
NS3-f2-3a 5'-GCCACTGAACCTGTAATATTTAGTCCCATGG-3'
NS3-r2-3a 5'- GTGCGGTTCCCAGTGCGGA-3'
Регион NS5A генотип la:
NS5A-f2-la 5'-TCCCCCACGCACTAYGTG-3'
NS5A-r2-la 5'-GTGCAAGTTGCCTTGAGAG-3'
Регион NS5A генотип lb:
NS5A-f2-lb 5'-TCCCCCACGCACTAYGTG-3'
NS5A-r2-lb 5'-CTRGCYGARGAGCTGGCC-3'
Регион NS5A генотип 2:
NS5a-f2-2 5'-CACTACGTGRCRGAGTCTG-3'
NS5a-r2-2 5'-AGCTGGCTYGCTGARGAGC-3'
Регион NS5A генотип 3а:
NS5A-f2-3a 5'-GTYCCCGAGAGCGATGCYGC-3'
NS5A-r2-3a 5'-AGTTGGCTGGRGAYGAGCTYGC-3'
Регион NS5B генотипы 1,2,3:
NS5B-f2 5'-IACYATCATGGCIAARARYGAGGT-3'
NS5B-r2 5'-ACCTRGTCATAGCCTCCGTGAA-3'
Состав амплификационной смеси на втором этапе:
20 пмоль/л каждого праймера, по 0,8 ммоль/л всех нуклеозидтрифосфатов, 7,5 ммоль/л MgCb, 1 ед. рекомбинантной Taq ДНК-полимеразы (Fermentas), буфер для Taq ДНК-полимеразы (750 ммоль/л Трис-НС1, (рН 8,8), 200 ммоль/л (NH4)2S04, 0,1% (v/v) твин 20), DMSO 8% от конечного объема, формамид 4% от конечного объема, 10 мкл продукта амплификации первого этапа, вода без нуклеаз до конечного объема 50 мкл. Условия проведения ПЦР представлены в таблице 2.
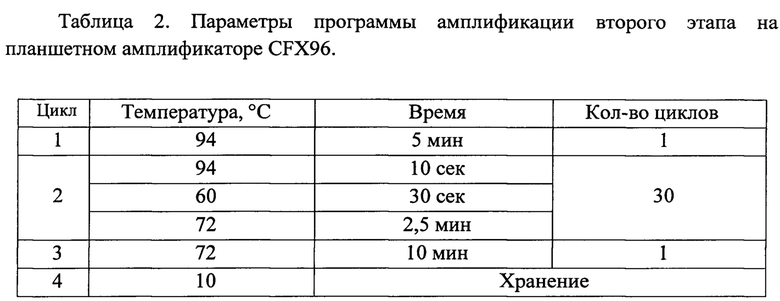
Далее следует этап очистки продуктов амплификации методом спиртового осаждения в присутствии ацетата натрия, для чего смесь из 2 мкл 3М ацетата натрия, 2 мкл 0,125М EDTA и 1 мкл гликогена вносили в 20 мкл продукта амплификации и инкубировали при комнатной температуре в присутствии охлажденного 96% этилового спирта 15 минут. Центрифугировали при 14000 об/мин, 4°С 15 мин супернатан удаляли и дважды промывали осадок охлажденным 70% этиловым спиртом, повторяя процедуру центрифугирования на холоде. Промытый осадок сушили.
Для анализа качества очищения продуктов амплификации осадок растворяли в 30 мкл ТЕ-буфера и визуализировали в агарозном геле.
Концентрацию НК измеряли на флюориметре Qubit 2.0 по стандартной методике, рекомендованной производителем.
Очищенный фрагмент с концентрацией 50-100 нг в зависимости от нуклеотидного состава анализируемого участка использовали для постановки секвенирующих реакций с прямого и обратного праймеров для каждой пары праймеров второго этапа ГШР для каждого образца.
При анализе продукта секвенирующей реакции в генетическом анализаторе ABI PRISM 3500 (Applied Biosystems, США) использовали набор реагентов ABI PRISM BigDye Terminator v3.1. (Applied Biosystems, США), согласно инструкции производителя. Для этого готовили смесь для секвенирующей реакции следующего состава: матрица 0,2-8,0 мкл, праймер для секвенирования 3,0 мкл (концентрация 1,6 пмол/мкл), ABI PRISM BigDye Terminator v3.1 Kit 8 мкл, деионизованная вода до конечного объема смеси 20 мкл. Секвенирующую реакцию проводили при условиях: устанавливали 25 циклов амплификации в режиме: 96°С - 10 сек, 50°С - 5 сек, 60°С - 4 мин. Продукты секвенирующей реакции очищали с использованием вышеприведенных методов, очищенный осадок растворяли в SLS-буфере и помещали в генетический анализатор.
Для определения принадлежности изолятов ВГС к генотипам и субгенотипам, а также определения рекомбинантных вариантов вируса были выбраны фрагменты генома, включающий регионы Core и NS5B.
Первичный анализ полученных в ходе секвенирования фрагментов проводили с помощью программы NCBI Blast в сравнении с представленными в международной базе данных GenBank нуклеотидными последовательностями референсных образцов. Выравнивание нуклеотидных последовательностей проводили в программе MEGA версия 5, используя алгоритм ClustalW. Поскольку для выбранных для секвенирования регионов показана высокая скорость эволюции, для построения филогенетических деревьев и последующего филогенетического анализа использовали метод присоединения соседей (Neighbor-joining), позволяющий оптимизацию дерева в соответствии с критерием «сбалансированной минимальной эволюции». Визуализацию и оформление филогенетических деревьев осуществляли в программе MEGA версия 5. Для оценки достоверности реконструированной топологии построенных филогенетических деревьев проводили бутстреп (bootstrap) анализ (500 повторностей).
Для определения рекомбинантного вида ВГС последовательности регионов Core и NS5B с помощью программы NCBI Blast сравнивались с представленными в международной базе данных GenBank нуклеотидными последовательностями референсных образцов. При выявлении принадлежности данных последовательностей к отличным генотипам, проводился анализ наличия точки рекомбинации, находящейся внутри целевого продукта NS2 (между нуклеотидами 3175 и 3176) посредством сравнения полученной последовательности с референсами.
Для определения наличия мутаций лекарственной устойчивости ВГС к ПППД была использована программа Geno2pheno HCV resistance.
Аналитическую чувствительность метода проверяли путем поэтапного разведения. Были определены 10 образцов плазмы крови, содержащие различные концентрации ВГС. Вирусную нагрузку предварительно определяли с помощью коммерческого набора для количественного определения РНК ВГС в клиническом материале методом полимеразной цепной реакции (ПЦР) с гибридизационно-флуоресцентной детекцией в режиме «реального времени» с помощью набора «АмплиСенс® HCV-Mohhtop-FL» (ФБУН ЦНИИЭ, Москва).
Каждый образец поэтапно разводили предварительно проанализированной плазмой крови без ВГС (чистой плазмой). Аликвоту образца объемом 100 мкл вносили в микропробирку Eppendorf объемом 1,5 мл, добавляли 100 мкл чистой плазмы, тщательно пипетировали и переносили 100 мкл полученного пула в новую микропробирку, куда снова добавляли 100 мкл чистой плазмы, пипетировали и 100 мкл нового пула переносили в третью пробирку и тд. до десятикратного последовательного разведения (таблица 3).

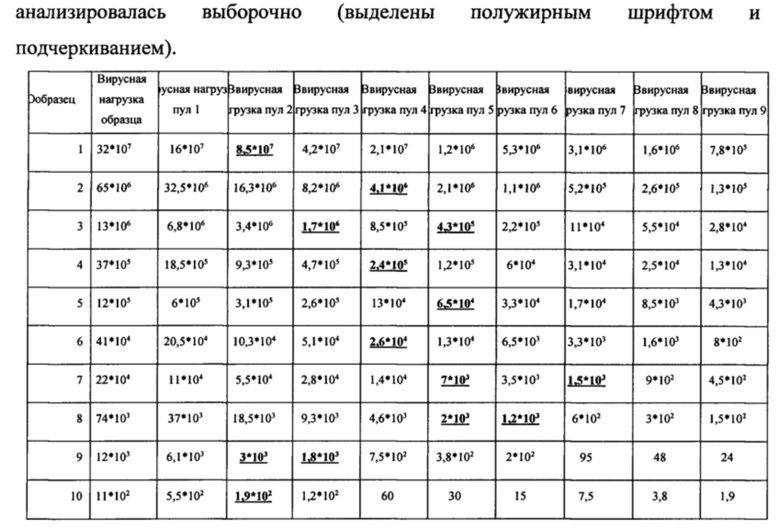
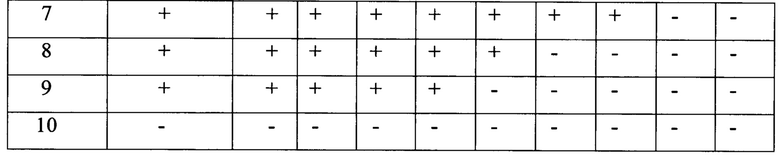
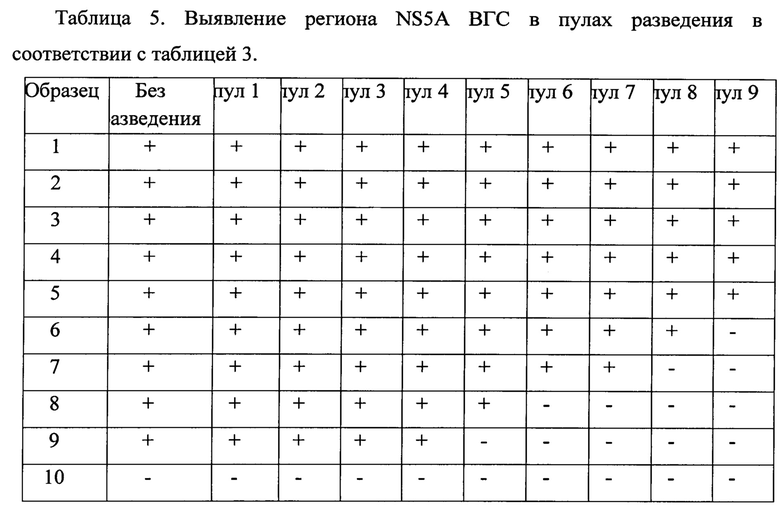
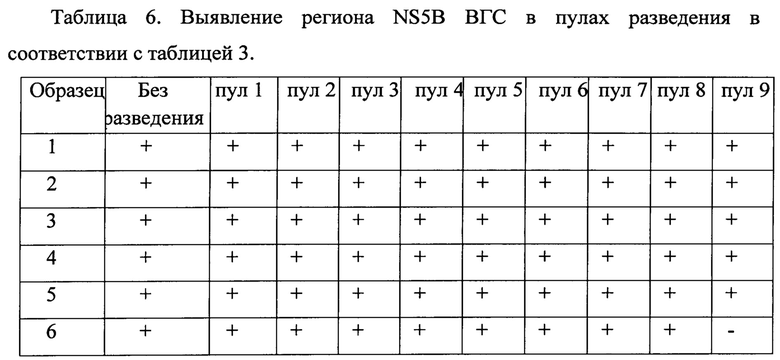
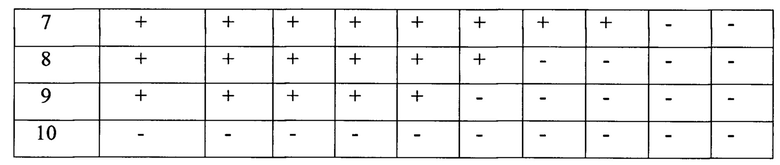
После разведения осуществляли экстракцию РНК из каждого пула разведения каждого образца с помощью комплекта реагентов для выделения РНК/ДНК из клинического материала «РИБО-преп» (ФБУН ЦНИИЭ, Москва). Полученные образцы РНК амплифицировали, согласно предложенному методу. Результаты выявления вируса в пулах разведения представлены в таблицах 4-8.
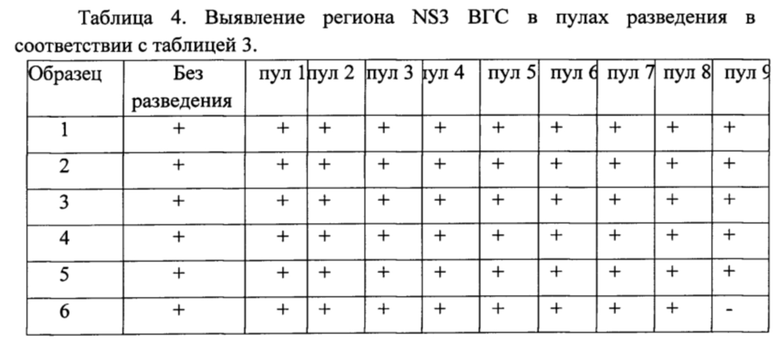
с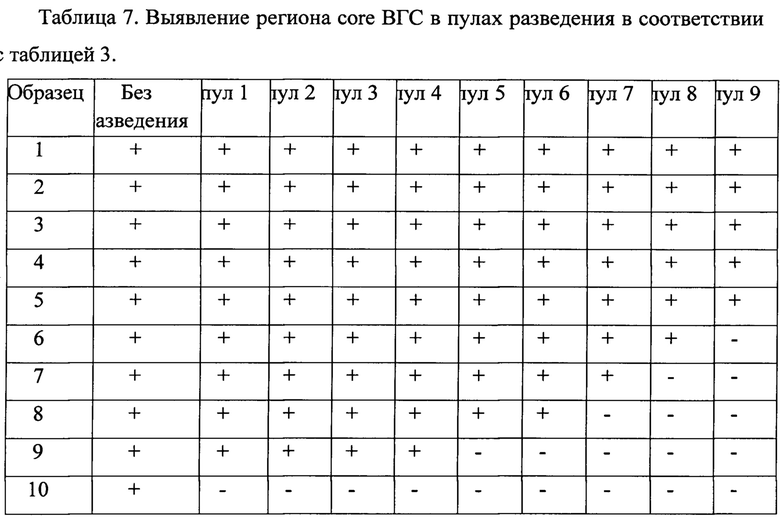
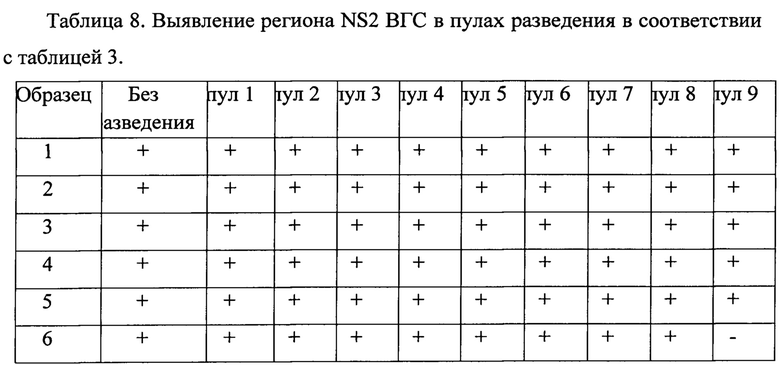
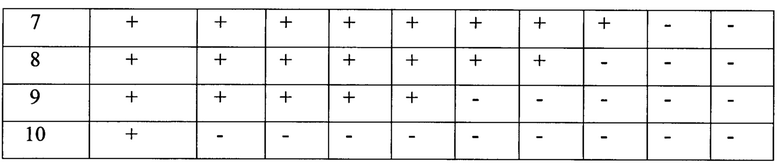
На основании анализа данных выявлено, что необходимая вирусная нагрузка для проведения секвенирующей реакции и получения нуклеотидной последовательности искомых фрагментов генома для определения генотипа вируса и мутаций ЛУ составляет 1,5×103МЕ/мл и более.
Аналитическую специфичность метода проверяли путем получения продуктов амплификации четырех регионов (core, NS3, NS5A, NS5B) наиболее распространенных в РФ генотипов ВГС (1, 2, 3). Для получения продукта амплификации региона NS2 использовали образцы плазмы с ВГС генотипа 2 т.к определение точки рекомбинации актуально для межгенотипных рекомбинантов, имеющих на 3'-конце нуклеотидную последовательность, относящуюся к генотипу 2, а на 5'-конце нуклеотидную последовательность, относящуюся к другому генотипу.
Были определены 10 образцов плазмы крови разных генотипов ВГС удовлетворительной вирусной нагрузки для дальнейшего секвенирования (1,5×103 МЕ/мл и более).
Вирусную нагрузку предварительно определяли с помощью коммерческого набора для количественного определения РНК ВГС в клиническом материале методом полимеразной цепной реакции (ГЩР) с гибридизационно-флуоресцентной детекцией в режиме «реального времени» с помощью набора «АмплиСенс® HCV-Mohhtop-FL» (ФБУН ЦНИИЭ, Москва).
Генотипирование вируса предварительно определяли с помощью коммерческого набора для определения генотипа ВГС в клиническом материале методом полимеразной цепной реакции (ГЩР) с гибридизационно-флуоресцентной детекцией в режиме «реального времени» с помощью набора «АмплиСенс® НСУ-генотип-И,» (ФБУН ЦНИИЭ, Москва). Список образцов представлен в таблице 9.
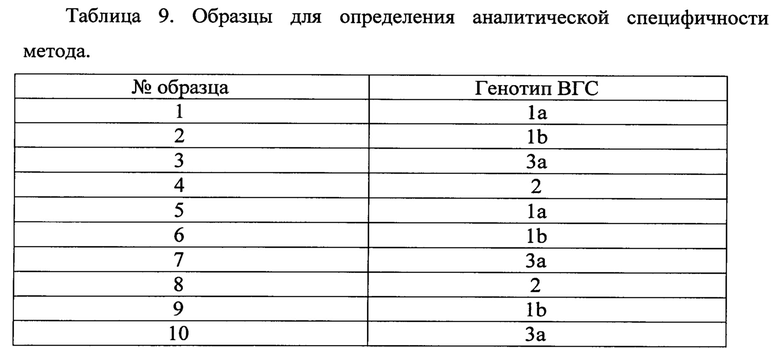
Полученные образцы РНК амплифицировали, согласно предложенному методу. Результаты специфичности олигонуклеотидных праймеров в зависимости от генотипа ВГС представлены в таблице 10-13. 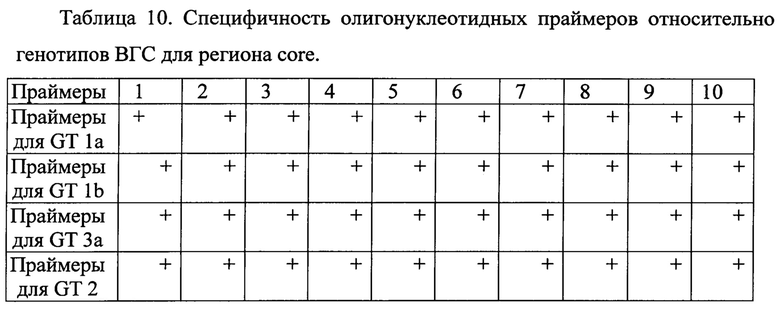
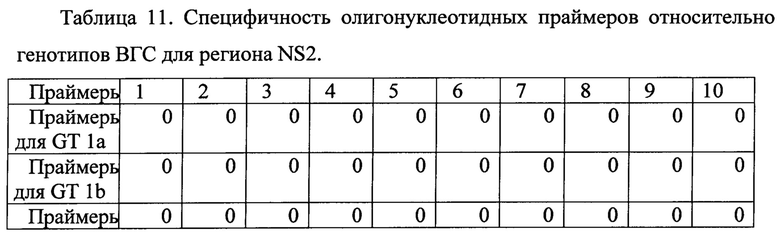
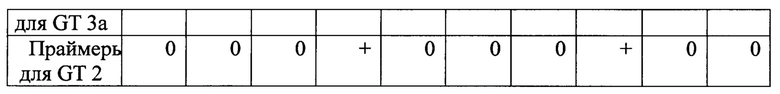
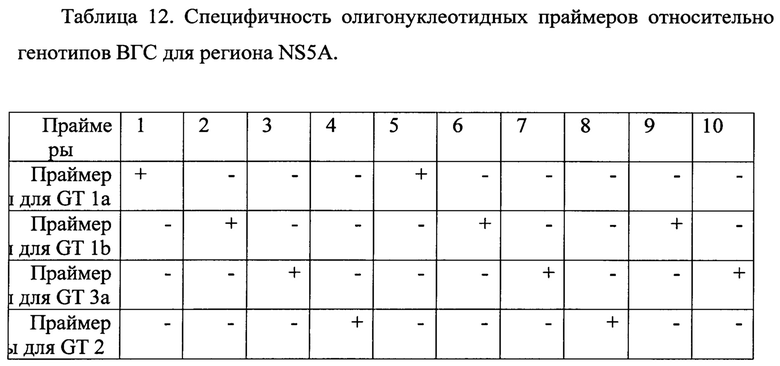
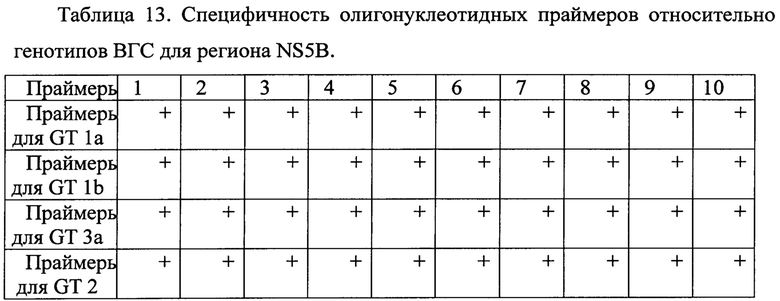
На основании анализа полученных данных выявлено, что для определения нуклеотидных последовательностей регионов core, NS5B достаточно двух неспецифических для всех генотипов пар олигонуклеотидных праймеров. Для определения нуклеотидных последовательностей регионов NS3, NS5A разных генотипов, напротив, необходимо использование праймеров, специфичных для каждого отдельного генотипа.
Для определения нуклеотидной последовательности региона NS2 ВГС генотипа 2 так же достаточно двух пар специфических праймеров.
Сущность изобретения поясняется чертежами, где на фиг.1-5 представлен электрофорез продуктов амплификации второго этапа (регионы Core, NS2, NS3, NS5A, NS5B соответственно) последовательных разведений образца №7, вирусная нагрузка 22*104 МЕ/мл.
Продукты амплификации седьмого разведения (предположительно около 1500 МЕ/мл) видны слабо, однако концентрации продукта достаточно для проведения секвенирующей реакции и получения нуклеотидных последовательностей искомых фрагментов генома. Ниже приведены примеры конкретного использования предложенного метода.
Пример 1.
Больные ХВГС с вирусологическим прорывом на терапии ПППД - 31 пациентов. Распределение генотипов в данной группе было следующим: 1а-26%(n=8), 1b-29%(n=9), 3а-45%(n=14). Возраст пациентов варьировал от 33 до 62 и составил в среднем 45.8±8.38 лет. Результаты определения вирусной нагрузки варьировали от 3,1×103 до 4,2×107 МЕ/мл. Нуклеотидная последовательность участков NS3, NS5A, NS5B определена во всех образцах. Мутации, ассоциированные с лекарственной устойчивостью, были обнаружены в 87% (n=27). Отсутствие мутаций резистентности в 4 случаях предположительно может быть связано с отсутствием приверженности пациентов к терапии, назначение неверной схемы лечения либо реинфицированием другим штаммом вируса.
Значимые нуклеотидные замены в исследуемой группе представлены в таблице 14.
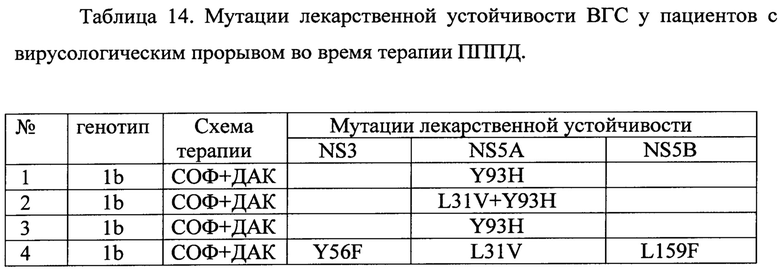
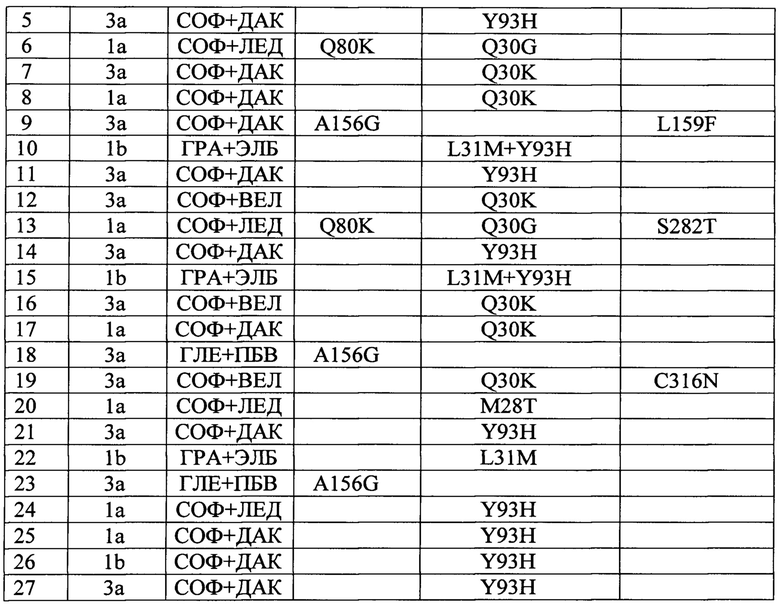
При анализе данной группы пациентов рекомбинантных форм вируса обнаружено не было.
Пример 2
Из 153 образцов сывороток пациентов с установленным диагнозом ХВГС без предшествующей терапии, повторно определить РНК ВГС удалось в 83,6% (n=128) образцах. Причиной неудачи амплификации в 25 образцах, по-видимому, стала деградация РНК в результате повторного размораживания для проведения предыдущих экспериментов и длительное хранения сывороток. Распределение генотипов ВГС в данной группе было следующим: 1а-5,5%(n=7), 1b-58,5% (n=75), 3а-36% (n=46). Возраст пациентов варьировал от 24 до 81 и составил в среднем 46.8±11.78 лет.
Результаты определения вирусной нагрузки варьировали от неопределяемой (менее 300 МЕ/мл) до 1,8×108 МЕ/мл. Как упоминалось выше, из 153 пациентов только у 128 удалось определить вирусную нагрузку и продолжить дальнейшее исследование. Нуклеотидная последовательность всех трех участков NS3, NS5A, NS5B определена в 94 образцах. Неудачи амплификации, по-видимому, были обусловлены более высоким уровнем полиморфизма последовательностей, кодирующих целевые белки, по сравнению с 5'-НТО областью, использовавшейся для детекции инфекции и участком NS5B, использовавшимся для определения генотипа ВГС. Таким образом, у 12 образцов удалось амплифицировать и получить нуклеотидные последовательности двух регионов и у 22 образцов - одного региона.
Мутации, ассоциированные с устойчивостью ВГС к ПППД были обнаружены в 9% случаев (n=11). В регионе NS3-5 значимых нуклеотидных замен, в регионах NS5A и NS5B 4 и 2 значимых нуклеотидных замен соответственно. Все значимые мутации ВГС представлены в таблице 15.
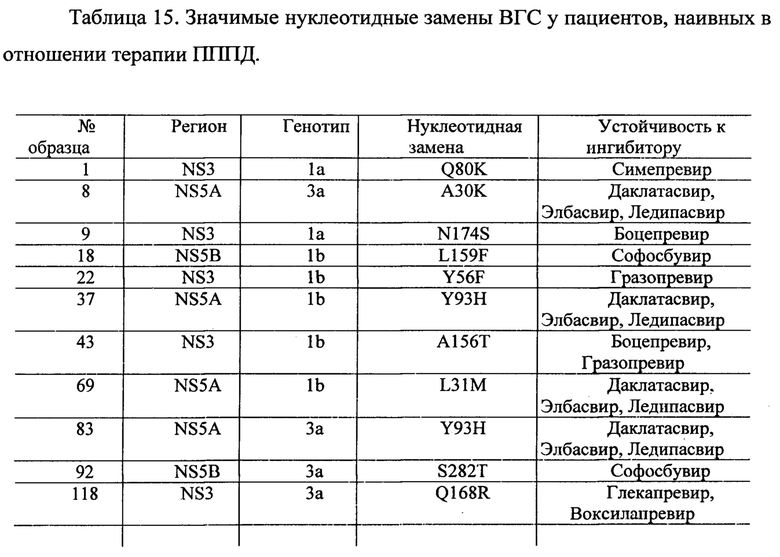
Рекомбинантных форм при анализе группы пациентов выявлено не было, таким образом, необходимости в определении точки рекомбинации в регионе NS2 не было.
Пример 3.
Из 278 образцов сывороток пациентов с впервые выявленной ВИЧ инфекцией определить РНК ВГС удалось 105 образцах. Распределение генотипов ВГС в данной группе было следующим: 1а-12% (n=13), 1b-54%(n=57), 2а-3%(n=2), 2k-1%(n=1), 3а-30%(n=32). Возраст пациентов составил от 18 до 65 и составил в среднем 38.7±9.48 лет.
Результаты определения вирусной нагрузки варьировали от неопределяемой (менее 300 МЕ/мл) до 2,3×107 МЕ/мл. Из-за низкой вирусной нагрузки (менее 300 МЕ/мл) в 18 образцах, дальнейшее исследование удалось продолжить для 87 образцов. Нуклеотидная последовательность всех трех участков NS3, NS5A, NS5B определена в 64 образцах. Неудачи амплификации, по-видимому, были обусловлены более высоким уровнем полиморфизма последовательностей, кодирующих целевые белки, по сравнению с 5'-НТО областью, использовавшейся для детекции инфекции и участком NS5B, использовавшимся для определения генотипа ВГС. Таким образом, у 9 образцов удалось амплифицировать и получить нуклеотидные последовательности двух регионов и у 14 образцов - одного региона.
Мутации, ассоциированные с устойчивостью ВГС к ПППД были обнаружены в 13% случаев (n=11). В регионе NS3-3 значимых нуклеотидных замен, в регионах NS5A и NS5B 3 и 8 значимых нуклеотидных замен соответственно.
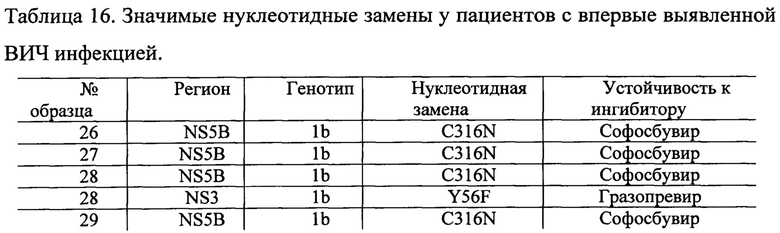
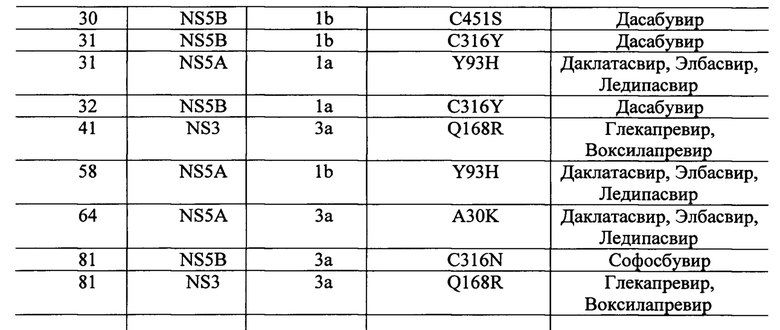
При анализе группы пациентов у одного пациента была выявлена рекомбинантная форма вируса 2k/lb. По результатам генотипирования были выявлены несоответствия между кластеризацией при филогеннетическом анализе регионов core и NS5B- 2k и 1b соответственно. При анализе региона NS2 была выявлена точка перекреста в нуклеотидной позиции 3175 генома ВГС (согласно нумерации нуклеотидов изолята pJ6CF субтипа 2а, AF177036 в базе данных GenBank).
Пример 4.
Из 148 образцов сывороток пациентов с вирусологической неэффективностью АРВТ определить РНК ВГС удалось 97 образцах. Распределение генотипов ВГС в данной группе было следующим: 1a-7% (n=7), 1b-53% (n=52), 2-2% (n=2), 3а-38% (n=37).
Возраст пациентов варьировал от 18 до 65 и составил в среднем 34.3±7.27 лет. Количество мужчин в группе преобладало по сравнению с женщинами-71% и 28% соответственно.
Результаты определения вирусной нагрузки варьировали от 5,3×103 до 2,3×108 МЕ/мл. Нуклеотидная последовательность всех трех участков NS3, NS5A, NS5B определена в 73 образцах. Неудачи амплификации, по-видимому, были обусловлены более высоким уровнем полиморфизма последовательностей, кодирующих целевые белки, по сравнению с 5'-НТО областью, использовавшейся для детекции инфекции и участком NS5B, использовавшимся для определения генотипа ВГС.Таким образом у 9 образцов удалось амплифицировать и получить нуклеотидные последовательности двух регионов и у 15 образцов- одного региона.
Мутации, ассоциированные с устойчивостью ВГС к ПППД были обнаружены в 27% случаев (n=26). В регионе NS3-6 значимых нуклеотидных замен, в регионах NS5A и NS5B 15 и 10 значимых нуклеотидных замен соответственно.
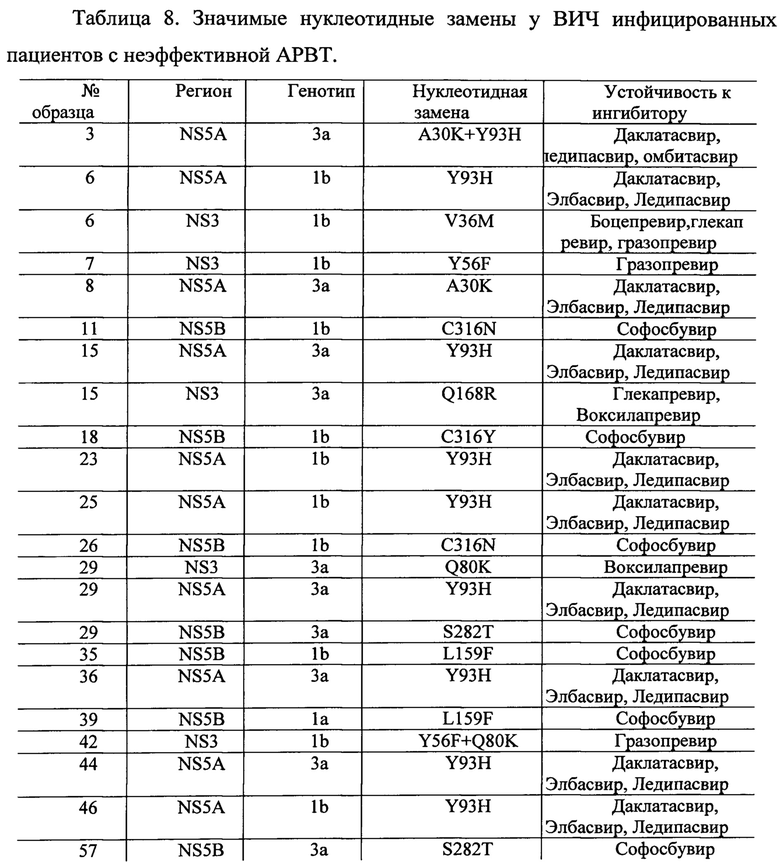
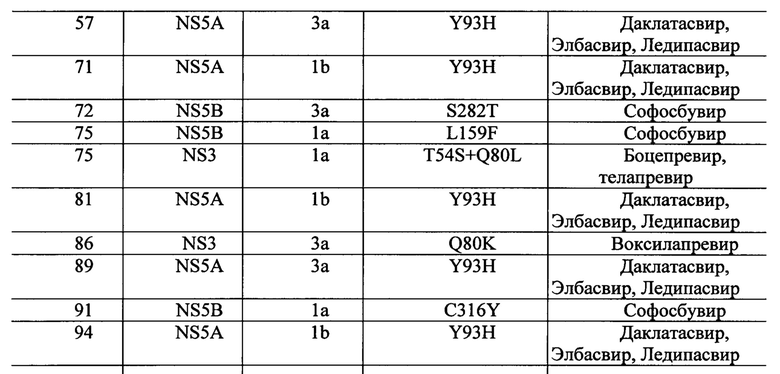
Рекомбинантных форм при анализе групп пациентов выявлено не было, таким образом, необходимости в определении точки рекомбинации в регионе NS2 не было.
Предложенный метод отличается от прототипа тем, что наряду с определением мутаций лекарственной устойчивости возможно определение рекомбинантной формы вируса, что важно при назначении/изменении схемы лечения у пациентов с ХВГС.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ выявления
рекомбинантных форм вируса гепатита С .xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2023-07-05">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>нет </ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия человека»
</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur
Institute</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ выявления рекомбинантных
форм вируса гепатита С и мутаций лекарственной устойчивости вируса
гепатита С к препаратам прямого противовирусного действия методом
полимеразной цепной реакции с последующим
секвенированием</InventionTitle>
<SequenceTotalQuantity>42</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaacagggaaccttcctggttgctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverce PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaggggaccagttcatcatcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtgtttgacataaccaagtggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccccttcgggcggagacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccrratggagaccaagmtcatyacgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcngtdgartggcactcrtcaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gayatggagayyaagrtcatyacctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccagccgtctccgcttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcacmcctatgtcggaytgggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatcggcgagraatttgccataygtgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaycacctagcrccaatgcaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taggtggartaggtcagtttrgcaccagttg</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggcccgggcgagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcccatctcctgcctcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggatgaaccggctgatag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccacaggagrttggcctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcggtccagtggatgaacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actcratccgggtcacrtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q57">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggatgaacaggctcatygc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgttrctgcccatctcttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtstggnargacytnctggaagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>rgngcrgartacctrgtcatagcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q67">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaccttcctggttgctctttctctat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q71">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttcatcatcatatcccatgccat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q73">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtgtttgacataaccaagtggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q77">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccccttcgggcggagacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q79">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcrtgyggbgacatcatyaacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q83">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cavccrccrtcrgcaaggaacttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q85">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcrgcrtgyggggacatcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q89">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccargacygtgccratgccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q91">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccactgaacctgtaatatttagtcccatgg</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q95">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcggttcccagtgcgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q97">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcccccacgcactaygtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q101">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcaagttgccttgagag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q103">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcccccacgcactaygtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q107">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctrgcygargagctggcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q109">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactacgtgrcrgagtctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q113">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctggctygctgargagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q115">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtycccgagagcgatgcygc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q119">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttggctggrgaygagctygc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q121">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>nacyatcatggcnaararygaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q125">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctrgtcatagcctccgtgaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к области вирусологии. Описан способ выявления рекомбинантных форм вируса гепатита С и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия на основе двухэтапной полимеразной цепной реакции для целевых генов с последующим их секвенированием. Технический результат заключается в определении мутаций лекарственной устойчивости ВГС к ПППД наряду с возможным определением рекомбинантной формы вируса. 1 з.п. ф-лы, 5 ил., 17 табл., 4 пр.
1. Способ выявления рекомбинантных форм вируса гепатита С и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия на основе двухэтапной полимеразной цепной реакции для целевых генов с последующим их секвенированием, отличающийся тем, что на первом этапе применяют асимметричную ПЦР с протяженными олигонуклеотидными праймерами, а именно: регион NS3 генотип la: прямой праймер 5'-ccrratggagaccaagmtcatyacgt-3', обратный праймер 5'-tcngtdgartggcactcrtcaca-3'; регион NS3 генотип lb: прямой праймер 5'-gayatggagayyaagrtcatyacctgg-3', обратный праймер 5'-tccagccgtctccgcttgg-3'; регион NS3 генотип 2: прямой праймер 5'-ctcacmcctatgtcggaytgggc-3', обратный праймер 5'-ccatcggcgagraatttgccataygtgga-3'; регион NS3 генотип 3а: прямой праймер 5'-gaycacctagcrccaatgcaaca-3', обратный праймер 5'-taggtggartaggtcagtttrgcaccagttg-3'; регион NS5A генотип la: прямой праймер 5'-gttggcccgggcgagg-3', обратный праймер 5'-cgcccatctcctgcctcca-3'; регион NS5A генотип lb: прямой праймер 5'-tggatgaaccggctgatag-3', обратный праймер 5'-ccacaggagrttggcctc-3'; регион NS5A генотип 2: прямой праймер 5'-gcggtccagtggatgaacag-3', обратный праймер 5'-actcratccgggtcacrtc-3'; регион NS5A генотип 3а: прямой праймер 5'-tggatgaacaggctcatygc-3', обратный праймер 5'-atgttrctgcccatctcttg-3; регион NS5B генотипы 1, 2, 3: прямой праймер 5'-gtstggiargacytictggaagac-3', обратный праймер 5'-rgigcrgartacctrgtcatagcct-3'; полученные продукты на первом этапе используют для второго этапа амплификации со следующими парами вложенных праймеров: регион NS3 генотип la: прямой праймер 5'-gcrtgyggbgacatcatyaacgg-3', обратный праймер 5'-cavccrccrtcrgcaaggaacttg-3'; регион NS3 генотип lb: прямой праймер 5'-gcrgcrtgyggggacatcat-3', обратный праймер 5'-ccargacygtgccratgccca-3'; регион NS3 генотип 2: прямой праймер 5'-atggagaagaaggtcatcgtctgggg-3', обратный праймер 5'-gtcctaatgttgggattgatgccatgtgc-3'; регион NS3 генотип 3а: прямой праймер 5'-gccactgaacctgtaatatttagtcccatgg-3', обратный праймер 5'-gtgcggttcccagtgcgga-3'; регион NS5A генотип la: прямой праймер 5'-tcccccacgcactaygtg-3', обратный праймер 5'-gtgcaagttgccttgagag-3'; регион NS5A генотип lb: прямой праймер 5'-tcccccacgcactaygtg-3', обратный праймер 5'-ctrgcygargagctggcc-3'; регион NS5A генотип 2: прямой праймер 5'-cactacgtgrcrgagtctg-3', обратный праймер 5'-agctggctygctgargagc-3'; регион NS5A генотип 3а: прямой праймер 5'-gtycccgagagcgatgcygc-3', обратный праймер 5'-agttggctggrgaygagctygc-3'; регион NS5B генотипы 1, 2, 3: прямой праймер 5'-iacyatcatggciaararygaggt-3', обратный праймер 5'-acctrgtcatagcctccgtgaa-3', для определения наличия мутаций лекарственной устойчивости ВГС к препаратам прямого противовирусного действия последовательности регионов NS3, NS5A, NS5B сравнивают с представленными в международной базе данных GenBank нуклеотидными последовательностями референсных образцов, а для определения рекомбинантного вида ВГС последовательности регионов Core и NS5B сравнивают с представленными в международной базе данных GenBank нуклеотидными последовательностями референсных образцов, при выявлении принадлежности данных последовательностей к отличным генотипам проводят анализ наличия точки рекомбинации, находящейся внутри целевого продукта NS2, посредством сравнения полученной последовательности с референсами.
2. Способ по п. 1, отличающийся тем, что наряду с определением мутаций лекарственной устойчивости вируса к препаратам прямого противовирусного действия в регионах NS3, NS5A, NS5B проводят одновременное определение рекомбинантных форм ВГС, применяя асимметричную ПЦР с протяженными олигонуклеотидными праймерами, а именно: регион Core для всех генотипов: прямой праймер 5'-gcaacagggaaccttcctggttgctc-3', обратный праймер 5'-cgtaggggaccagttcatcatcat-3'; регион NS2 для генотипа 2: прямой праймер 5'-tagtgtttgacataaccaagtggc-3', обратный праймер 5'-ccccttcgggcggagacg-3'; полученные продукты на первом этапе используют для второго этапа амплификации со следующими парами вложенных праймеров: регион Core для всех генотипов: прямой праймер 5'-aaccttcctggttgctctttctctat-3', обратный праймер 5'-gttcatcatcatatcccatgccat-3'; регион NS2 для генотипа 2: прямой праймер 5'-tagtgtttgacataaccaagtggc-3', обратный праймер 5'-ccccttcgggcggagacg-3.
| Dietz J | |||
| et al | |||
| Patterns of Resistance-Associated Substitutions in Patients With Chronic HCV Infection Following Treatment With Direct-Acting Antivirals | |||
| Gastroenterology | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Serre SB | |||
| et al | |||
| Identification of alpha interferon-induced envelope mutations of hepatitis C virus in vitro associated with increased viral fitness | |||
Авторы
Даты
2024-08-12—Публикация
2023-07-27—Подача