Группа изобретений относится к биотехнологии и медицине, а именно к инфекционным болезням и медицинской генетике, в частности к проблеме прогностической оценки развития гепатотоксических побочных эффектов у ВИЧ-инфицированных лиц при терапии антиретровирусными препаратами. Диагностику проводят с использованием ДНК, выделенной из цельной крови или сухих пятен крови (сухой капли крови).
Низкая приверженность лечению при антиретровирусной терапии (APT) ВИЧ - наиболее частая причина прекращения APT - зависит от многих факторов, в том числе медицинских: токсичность используемых лекарств, приводящая к побочным реакциям при приеме препаратов и, соответственно, плохой переносимости лечения из-за проявлений гепатотоксичности [Мусатов В.Б., Яковлев А.А., Чайка Н.А., Келли Д., Амирханян Ю.А. Основные причины и современные методы коррекции низкой приверженности к антиретровирусной терапии у трудных пациентов. ВИЧ-инфекция и иммуносупрессии. 2018. т. 10 (4). стр. 37-56. DOI: 10.22328/2077-9828-2018-10-4-37-56]. Гепатотоксичность представляет собой повреждение печени, вызванное чужеродными веществами (ксенобиотиками), в том числе лекарственными препаратами. Гепатотоксичность и ее выраженность зависят от токсического потенциала лекарственных препаратов, модифицируемых факторов риска и генетических особенностей пациента. В среднем, только у 25-30% ВИЧ-инфицированных лиц гепатотоксичность сопровождается клинически выраженными симптомами, однако значимое поражение печени может привести к тяжелым последствиям и летальному исходу [Журавлева М.В., Прокофьев А.Б., Подымова А.С., Бердникова Н.Г., Сереброва С.Ю., Демченкова Е.Ю. Контроль за нежелательными реакциями при проведении антиретровирусной терапии ВИЧ/СПИД при применении взаимозаменяемых лекарственных препаратов. Безопасность и риск фармакотерапии. 2017; 5(3): 126-132.]. Следует отметить, что понимание значимости гепатотоксичности при APT увеличивается с каждым годом в связи с распространенностью среди ВИЧ-инфицированных лиц хронических вирусных гепатитов В (ХГВ) и С (ХГС) [Останкова Ю. В., Щемелев А.Н., Зуева Е.Б., Чурина М.А., Валутите Д.Э., Семенов А.В. Молекулярная эпидемиология и фармакорезистентность ВИЧ у пациентов с вирусологической неэффективностью антиретровирусной терапии в Архангельской области. ВИЧ инфекция и иммуносупрессии. 2019. Том 11. №4. с. 65-72. http://dx.doi.org/10.22328/2077-9828-2019-11-4-79-90. Семенов А.В., Останкова Ю. В., Серикова Е.Н., Зуева Е.Б., Тотолян Арег А. Оптимизация алгоритма диагностики маркеров хронического гепатита В у пациентов с впервые выявленной ВИЧ-инфекцией. Клиническая лабораторная диагностика. 2020. 65(9). 574-579. doi: 10.18821/0869-2084-2020-65-9-574-579.], так как у таких больных на фоне терапии чаще развиваются гепатотоксические эффекты.
Согласно литературным данным, определение нулевых/делеционных мутаций генов GSTM1, GSTT1, CYP2D6 у ВИЧ-инфицированных лиц дает ценную прогностическую информацию в отношении возможного развития гепатотоксического эффекта при APT [Абдрашитов Р.Х., Гильдеева Г.Н., Раменская Г.В., Смирнов В.В. Обзор существующих методик оценки активности CYP2D6 с применением экзогенных и эндогенных маркеров. Фармакокинетика и Фармакодинамика. 2015; (1):4-11; Seidegard J, Vorachek WR, Pero RW, Pearson WR. Hereditary differences in the expression of the human glutathione transferase active on trans-stilbene oxide are due to a gene deletion. Proc Natl Acad Sci. 1988. 85:7293-7. doi: 10.1073/pnas.85.19.7293; Nefic H. The Genetic Variation of CYP2D6 Gene in the Bosnian Population. Med Arch. 2018 Dec;72(6):396-400. doi: 10.5455/medarh.2018.72.396-400; Sophonnithiprasert T, Saelee P, Pongtheerat T. GSTM1 and GSTT1 copy number variants and the risk to Thai females of hepatocellular carcinoma. J Gastrointest Oncol. 2019 Apr;10(2):324-329. doi: 10.21037/jgo.2018.09.14; Ivanov A.V., Valuev-Elliston V.T., Ivanova O.N., Kochetkov S.N., Starodubova E.S., Bartosch В., Isaguliants M.G. Oxidative Stress during HIV Infection: Mechanisms and Consequences. Oxid. Med. Cell. Longev. 2016; 2016:8910396. doi: 10.1155/2016/8910396; Singh H.O., Lata S., Angadi M., Bapat S., Pawar J., Nema V., Ghate M.V., Sahay S., Gangakhedkar R.R. Impact of GSTM1, GSTT1 and GSTP1 gene polymorphism and risk of ARV-associated hepatotoxicity in HIV-infected individuals and its modulation. Pharmacogenomics J. 2017. 17(l):53-60. doi: 10.1038/tpj.2015.88; Zhu Y, Yang JH, Hu JP, Qiao M. Association of glutathione S-transferases (GSTT1, GSTM1 and GSTP1) genes polymorphisms with nonalcoholic fatty liver disease susceptibility: A PRISMA-compliant systematic review and meta-analysis. Medicine (Baltimore). 2022. 101(38):e30803. doi:10.1097/MD.0000000000030803]. Это позволяет выявлять пациентов для углубленного обследования, своевременного назначения адекватной поддерживающей терапии, а также проведения комплекса профилактических мероприятий.
Известен способ определения делеционных мутаций генов GSTM1 и GSTT1 у ВИЧ-инфицированных лиц для определения риска развития гепатотоксичности при APT [Singh Н.О., Lata S., Angadi M., Bapat S., Pawar J., Nema V., Ghate M.V., Sahay S., Gangakhedkar R.R. Impact of GSTM1, GSTT1 and GSTP1 gene polymorphism and risk of ARV-associated hepatotoxicity in HIV-infected individuals and its modulation. Pharmacogenomics J. 2017. 17(1):53-60. doi: 10.1038/tpj.2015.88.]. Известен «Набор реагентов для выявления генетических полиморфизмов в генах GSTT1 и GSTM1 человека методом полимеразной цепной реакции (ПЦР) с электрофоретической детекцией продуктов амплификации в агарозном геле "АмплиСенс® GSTT1 / GSTMl-EPh"». Однако, методы, выявляющие только гомозиготные нулевые генотипы очевидно ограничены так как не позволяют определить гетерозиготы GSTM1+/0 и GSTT1+/0, ферментативная активность которых наличествует, но имеет сравнительно более низкий уровень, чем при гомозиготах GSTM1+/+и GSTT1+/+. Кроме того, ограничением является необходимость использования агарозного гель-электрофореза для детекции результатов. Известен способ определения мутаций гена CYP2D6, включая делеционную мутацию методом ПЦР с последующим секвенированием [Kim EY, Lee SS, Jung HJ, Jung HE, Yeo CW, Shon JH, Shin JG. Robust CYP2D6 genotype assay including copy number variation using multiplex single-base extension for Asian populations. Clin Chim Acta. 2010 Dec 14; 411(23-24):2043-8. doi: 10.1016/j.cca.2010.08.042.]. Очевидным ограничением метода является необходимость прямого секвенирования фрагмента генома, что редко возможно в рамках рутинной лабораторной диагностики.
Наиболее близким по сущности к заявляемому изобретению и выбранным за прототип является метод, предложенный в работе I. Girault с соавторами [Girault I, Lidereau R, Biche I. Trimodal GSTT1 and GSTM1 genotyping assay by real-time PCR. Int J Biol Markers. 2005 Apr-Jun; 20(2):81-86. doi: 10.5301/JBM.2008.2569.]. Согласно методу, выделяют ДНК, выделенную из лейкоцитов крови. Затем производят одновременную амплификацию участков генов GSTM1, GSTT1 и нормировочного гена, кодирующего альбумин (ALB) с использованием набора праймеров и зондов TaqMan, а также с использованием в качестве калибратора образцов гомозиготных по аллелям GSTM1 и GSTT1 дикого типа. Регистрируют полученные результаты посредством гибридизационно-флуоресцентной детекции и определяют в режиме реального времени относительное количество копий генов GSTM1, GSTT1 с использованием сравнительного метода СТ (ΔΔСТ), позволяющего измерять разницу СТ (ΔCT) между целевым и эталонным геном, а затем сравнивать значения ΔCT анализируемых образцов с образцом-калибратором, имеющим две копии целевой последовательности. Число копий мишени рассчитывается как удвоенное относительное количество. По результатам оценки относительного количества копий целевых генов делают вывод о наличии делеционного полиморфизма для каждого гена в гомо- или гетерозиготном состоянии. Недостатком способа является выявление делеционных мутаций только генов II фазы биотрансформации ксенобиотиков, но отсутствие выявления делеционной мутации генов I фазы. Недостатком способа является выбор нуклеотидной последовательности зонда GSTM1, имеющего сайты, за счет которых зонд может отжигаться на себя, тем самым снижая эффективность ПЦР. Недостатком способа является выбор комплекта олигодезоксирибонуклеотидных праймеров с различными температурами отжига, в результате чего эффективность ПЦР целевых и нормировочного гена отличаются.
Задачей, на решение которой направлено заявляемое изобретение, является расширение арсенала способов, предназначенных для оценки на доклиническом этапе генетической предрасположенности человека к развитию гепатотоксических побочных эффектов при терапии с использованием антиретровирусных препаратов.
Технический результат - создание способа определения наследственной предрасположенности человека к проявлению гепатотоксичности антиретровирусных препаратов и расширение арсенала средств, используемых для прогноза гепатотоксичности при антиретровирусной терапии.
Авторами предложен способ, согласно которому ДНК выделяют из биологического материала, например, цельной крови или из сухой капли крови, а затем проводят мультиплексную постановку ПЦР участков генов GSTM1, GSTT1, CYP2D6 и эталонного нормировочного гена RPP30 с использованием следующих олигодезоксирибонуклеотидных праймеров и флуоресцентно меченых зондов:

Состав амплификационной смеси представляет собой буферный раствор, содержащий Трис-HCl рН 8,8 (при 25°С), КО, 6-7 мМ MgCl2, дезоксинуклеозидтрифосфаты, глицерол, Tween 20, SynTaq ДНК-полимеразу с ингибирующими активность фермента антителами (или 1 ед. рекомбинантной Taq ДНК-полимеразы, или Hot-start Taq ДНК-полимеразы).
ПЦР и детекцию результатов проводят при следующих условиях:
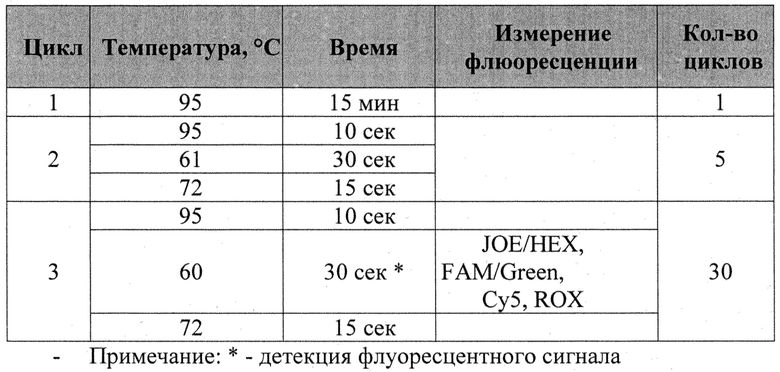
Параллельно с амплификацией анализируемых образцов проводят амплификацию контрольного образца, представляющего собой ранее охарактеризованный образец ДНК, в котором все три целевых гена дикого типа находятся в гомозиготном состоянии.
Первичный анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени».
Анализируют кривые накопления флуоресцентного сигнала по четырем каналам:
по каналу для флуорофора FAM регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента гена GSTM1;
по каналу для флуорофора HEX регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента гена GSTT1;
по каналу для флуорофора Су5 регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента гена CYP2D6;
по каналу для флуорофора ROX регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента ДНК RPP30.
Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (устанавливается в середине линейного участка прироста флуоресценции положительного контроля в логарифмической шкале), что определяет наличие (или отсутствие) для данной пробы значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат амплификации по каналу считается положительным, если кривая однократно пересекается с пороговой линией в области достоверного прироста флуоресценции, отрицательным в случае отсутствия пересечения кривой с пороговой линией (нет значения Ct), сомнительным во всех других случаях.
Анализ GSTM1, GSTT1, CYP2D6 проводят с нормализацией на эталонный ген RPP30, относительное число копий целевых генов определяют с использованием метода AΔCT [Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001 Dec;25(4):402-8. doi: 10.1006/meth.2001.1262.] в сравнении с охарактеризованным образцом-калибратором, имеющим две копии каждой из целевых последовательностей, на основании чего делают выводы о наличии аллеля дикого типа или делеционного полиморфизма в гетеро- или гомозиготном состоянии для каждого анализируемого гена и функциональной состоятельности соответствующих ферментов. При выявлении сочетания генотипов GSTM1 0/0 GSTT1 0/0, GSTM1 0/+ GSTT1 0/0, GSTM1 0/+ GSTT1 0/+, GSTM1 0/+ GSTT1 0/0 в присутствии CYP2D6 0/0 или CYP2D6 0/+делают вывод о наличии у обследуемого генетической предрасположенности к развитию гепатотоксического эффекта при антиретровирусной терапии.
Предложенный способ отличается от прототипа выявлением делеционных мутаций трех целевых генов, использованием нормировочного гена RPP30, набором олигодезоксирибонуклеотидных праймеров и зондов.
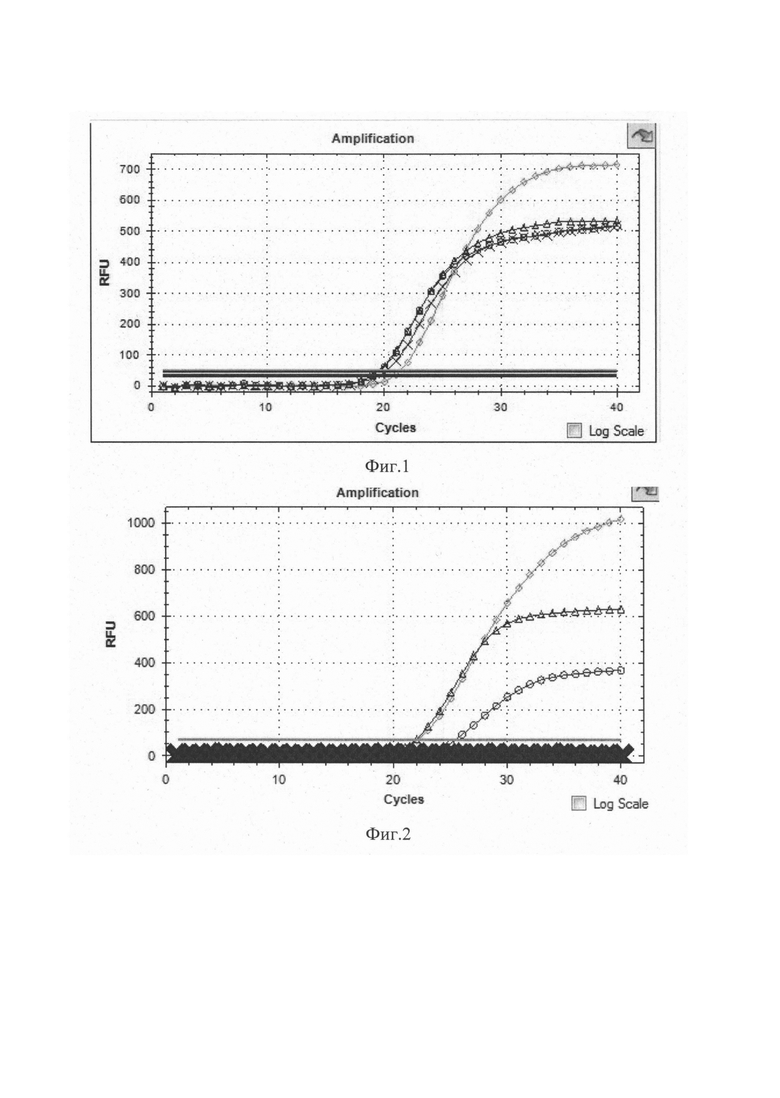
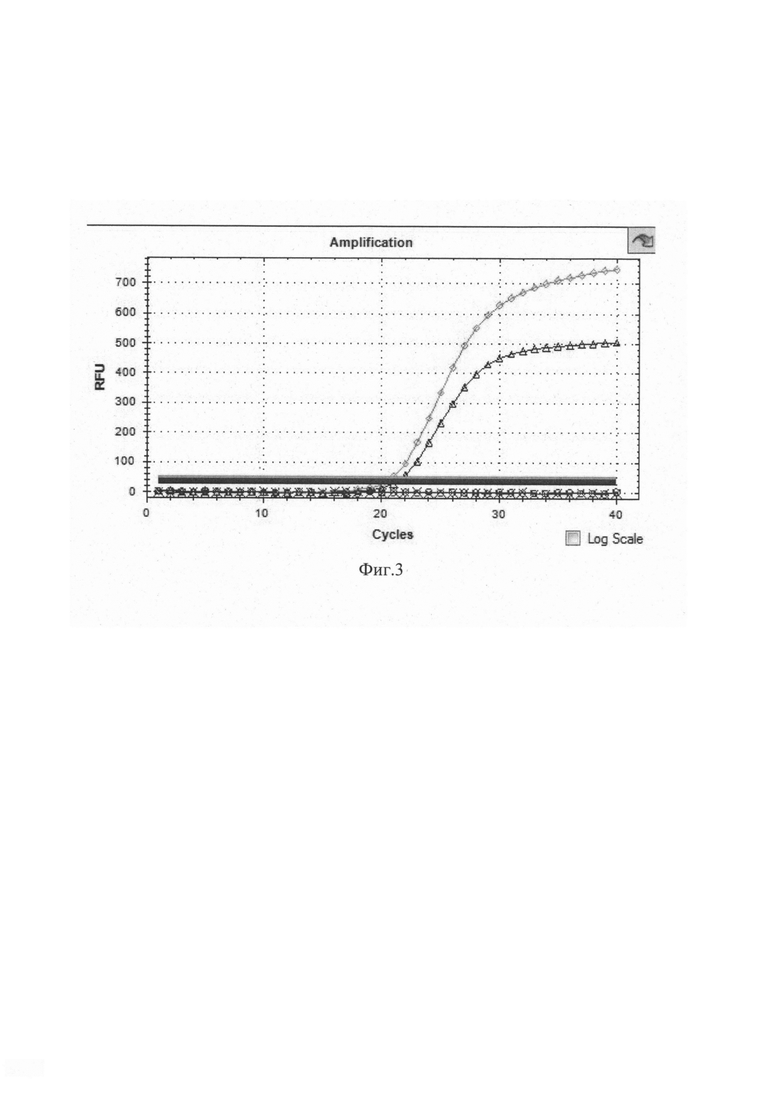
Сущность изобретения поясняется чертежом, где на фиг. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции в ходе амплификации всех анализируемых генов, включая три целевых гена и один нормировочный, при «диком» генотипе, то есть, каждый целевой ген гомозиготен по «дикой» аллели. А также чертежами, где на фиг. 2 представлен двухкопийный нормировочный ген RPP30 (ROX - ромбы), двухкопийный целевой ген GSTT1 (HEX - треугольники), однокопийный (0/+) целевой ген GSTM1 (FAM - кружок), целевой ген CYP2D6 с гомозиготной делеционной мутацией (Су5 - крест); где на фиг. З представлен двухкопийный нормировочный ген RPP30 (ROX - ромбы), двухкопийный целевой ген GSTT1 (HEX - треугольники), целевые гены GSTM1 (FAM -кружок) и CYP2D6 (Су5 - крест) с гомозиготной делеционной мутацией.
Ниже приведены примеры конкретного использования предложенного метода, являющиеся одним из возможных способов его реализации. Пример 1.
Были обследованы образцы крови 176 ВИЧ-инфицированных пациентов, проживающих в Северо-Западном федеральном округе, получавших две и более схем антиретровирусной терапии (APT), направленных в 2014-2016 гг.на определение лекарственной устойчивости вируса в связи с вирусологической неэффективностью APT; 112 образцов крови от лиц с впервые выявленной ВИЧ-инфекцией; 123 пациента без ВИЧ, вирусных гепатитов и клинических проявлений хронических и/или острых заболеваний, включая гепатотоксические проявления. Частоты гомозиготных делеций GSTM1 и GSTT1 у пациентов с неэффективной APT составили 41,47% и 44,88%, соответственно, сочетанный генотип GSTM1 0/0+ GSTT1 0/0 - 22,15% от группы. В контрольной группе сочетанный генотип GSTM1 0/0+ GSTT1 0/0 - 12,19% от группы. При анализе распределения генотипов: GSTM1 0/0+ GSTT1+/-, GSTM1+/-+ GSTT1 0/0, GSTM1 0/0+ GSTT1 0/0, GSTM1+/-+ GSTT1+/- выявлены достоверные отличия между контрольной группой и ВИЧ-инфицированными лицами с неэффективной APT -%2=18,103, df3, р=0,0004. Распределение генотипов генов GST в группе пациентов с впервые выявленной инфекцией ВИЧ не отличалось от такового в контрольной группе. Показан риск неэффективности APT при сочетанном генотипе GSTM1 0/0+ GSTT1 0/0 - OR=2,05, р<0,05. Риск вирусологической неэффективности APT при GSTM1 0/- GSTT1 0/- CYP2D6 0/- более чем в пять раз выше, чем у лиц с генотипом GSTM1+/+ GSTT1+/+CYP2D6+/+-OR=5,7, р<0,05.
Пример 2.
Большая часть ВИЧ-инфицированных больных с низкой приверженностью APT сообщали о гепатотоксических проявлениях лечения антиретровирусными препаратами. Среди 400 ВИЧ-инфицированных лиц с низкой приверженностью терапии у 332 человек, что составило 83% обследованных, был представлен по крайней мере один из целевых генов GSTM1, GSTT1, CYP2D6 с делеционной мутацией в гомозиготном состоянии. У 278 человек (69,5%) одновременно выявляли делеционную мутацию другого целевого гена в гомо- или гетерозиготном состоянии. У 161 (40,25%) ВИЧ-инфицированных больных с низкой приверженностью APT были выявлены сочетания генотипов GSTM1 0/0 GSTT1 0/0, GSTM1 0/+ GSTT1 0/0, GSTM1 0/+ GSTT1 0/+, GSTM1 0/+ GSTT1 0/0 в присутствии CYP2D6 0/0 или CYP2D6 0/+.
Пример 3.
Обследованы 297 ВИЧ-инфицированных лиц на антиретровирусной терапии с гепатотоксическими проявлениями различной степени тяжести. Показано, что в 87,88% случаев у пациентов с гепатотоксическими проявлениями был представлен по крайней мере один из целевых генов GSTM1, GSTT1, CYP2D6 с делеционной мутацией в гомозиготном состоянии. У 92,22% лиц с тяжелыми гепатотоксическими проявлениями выявляли следующие сочетания целевых генов: GSTM1 0/0 GSTT1 0/0 CYP2D6 0/0, GSTM1 0/0 GSTT1 0/0 CYP2D6 0/+, GSTM1 0/+ GSTT1 0/0 CYP2D6 0/0, GSTM1 0/0 GSTT1 0/+CYP2D6 0/0, GSTM1 0/+ GSTT1 0/0 CYP2D6 0/+, GSTM1 0/0 GSTT1 0/+CYP2D6 0/+. При оценке риска развития тяжелых гепатотоксических эффектов показано, что OR=16,21, р<0,05 при сочетанном генотипе GSTM1 0/0 GSTT1 0/0 CYP2D6 0/0.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="1.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-02-20">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022130994/20(067648)</ApplicationNumberText>
<FilingDate>2022-11-28</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека»</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur
Institute</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ПРОГНОСТИЧЕСКОЙ ОЦЕНКИ
ГЕПАТОТОКСИЧНОСТИ У ВИЧ-ИНФИЦИРОВАННЫХ ЛИЦ ПРИ АНТИРЕТРОВИРУСНОЙ
ТЕРАПИИ НА ОСНОВЕ ОПРЕДЕЛЕНИЯ ДЕЛЕЦИОННОГО ПОЛИМОРФИЗМА ГЕНОВ
БИОТРАНСФОРМАЦИИ КСЕНОБИОТИКОВ GSTM1, GSTT1, CYP2D6 ЧЕЛОВЕКА И НАБОР
ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО МЕЧЕНЫХ
ЗОНДОВ</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacctgcattcgttcatgtgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcccagagcacctcacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagcccatctgggaaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttggacctgcgagcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagcaagagcagagaggagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgtgcatcattctcattgtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtgtcccagcaaagttcatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcggctgtctccacaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a FAM fluorophore at the 5' end and a dark quencher
RTQ1 or BHQ1 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccatgagcaggcacagtgagtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a HEX fluorophore at the 5' end and a dark quencher
RTQ1 or BHQ1 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caccatccccaccctgtcttcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a Cy5 fluorophore at the 5' end and a dark quencher
RTQ2 or BHQ2 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagtgcaggggccgagggag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a ROX fluorophore at the 5' end and a dark quencher
RTQ2 or BHQ2 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttctgacctgaaggctctgcgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ прогностической оценки развития гепатоцеллюлярной карциномы на основе определения полиморфизма гена человека IFNAR-1 и набор олигодезоксирибонуклеотидных праймеров | 2022 |
|
RU2811763C1 |
| Способ выявления рекомбинантных форм вируса гепатита C и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия методом полимеразной цепной реакции с последующим секвенированием | 2023 |
|
RU2824565C1 |
| Способ выявления вируса Nipah методом, основанным на применении дезоксирибозима 10-23 | 2023 |
|
RU2816271C1 |
| Способ определения источника ВИЧ-инфекции при молекулярно-эпидемиологических расследованиях с использованием набора олигодезоксирибонуклеотидных праймеров и разработанной региональной базы данных | 2023 |
|
RU2830591C1 |
| Способ выявления вируса Guanarito методом, основанным на применении дезоксирибозима 10-23 | 2024 |
|
RU2827922C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления вируса Henipavirus hendraense методом DETECTR с изотермической амплификацией | 2024 |
|
RU2834907C1 |
| Способ выявления вируса Mammarenavirus machupoense методом DETECTR с изотермической амплификацией | 2024 |
|
RU2832917C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ВИРУСА ОБЕЗЬЯНЬЕЙ ОСПЫ ВИДА MONKEYPOX МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ (MPX AMP PS) | 2023 |
|
RU2803898C1 |
| Способ детекции и генотипирования патогенных лептоспир в клинических образцах | 2024 |
|
RU2840144C1 |
Группа изобретений относится к биотехнологии. Описан способ прогностической оценки гепатотоксичности у ВИЧ-инфицированных лиц при антиретровирусной терапии. Способ основан на определении делеционного полиморфизма генов биотрансформации ксенобиотиков человека. Он предусматривает экстракцию ДНК из биологического материала с последующим проведением полимеразной цепной реакции для целевых генов и эталонного нормировочного гена с использованием набора олигодезоксирибонуклеотидных праймеров и соответствующих флуоресцентно меченых зондов, комплементарных участкам выявляемых фрагментов. Также описан набор олигодезоксирибонуклеотидных праймеров и флуоресцентно меченых зондов для проведения амплификации целевых генов GSTM1, GSTT1, CYP2D6 и эталонного нормировочного гена RPP30. Технический результат заявленного изобретения заключается в создании способа определения наследственной предрасположенности человека к проявлению гепатотоксичности антиретровирусных препаратов. 2 н. и 2 з.п. ф-лы, 3 ил., 2 табл., 3 пр.
1. Способ прогностической оценки гепатотоксичности у ВИЧ-инфицированных лиц при антиретровирусной терапии на основе определения делеционного полиморфизма генов биотрансформации ксенобиотиков человека, предусматривающий экстракцию ДНК из биологического материала с последующим проведением полимеразной цепной реакции для целевых генов и эталонного нормировочного гена с использованием набора олигодезоксирибонуклеотидных праймеров и соответствующих флуоресцентно меченых зондов, комплементарных участкам выявляемых фрагментов, отличающийся тем, что используют: GSTM1F-cacctgcattcgttcatgtgac, GSTM1R-aagcaagagcagagaggagac, GSTM1zondFAM-FAM-gccatgagcaggcacagtgagtgc-RTQ1/BHQ1, GSTT1F-gtcccagagcacctcacc, GSTT1R-gtgtgcatcattctcattgtgg, GSTT1zondHEX-HEX-caccatccccaccctgtcttcca-BHQ1, CYP2D6*5F-tgagcccatctgggaaaca, CYP2D6*5R-ggtgtcccagcaaagttcatg, CYP2D6*5zondCy5-Cy5-cagtgcaggggccgagggag-RTQ2/BHQ2, RPP30F-tttggacctgcgagcg, RPP30R-gagcggctgtctccacaagt, RPP30-zondROX-ROX-ttctgacctgaaggctctgcgcg-RTQ2/BHQ2, при этом анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени», анализ целевых генов проводят с нормализацией на эталонный ген, относительное число копий целевых генов определяют с использованием метода ΔΔСТ в сравнении с охарактеризованным образцом-калибратором, имеющим две копии каждой из целевых последовательностей, на основании чего делают выводы о наличии аллеля дикого типа или делеционного полиморфизма в гетеро- или гомозиготном состоянии для каждого анализируемого гена и функциональной состоятельности соответствующих ферментов.
2. Способ по п. 1, отличающийся тем, что проводят одновременную амплификацию в одной емкости участков трех целевых генов GSTM1, GSTT1, CYP2D6 и эталонного нормировочного гена, тем самым выявляя среди анализируемых образцов образцы с гетеро- и гомозиготными делеционными мутациями целевых генов.
3. Способ по п. 1, отличающийся тем, что в качестве нормировочного гена используют ген белковой субъединицы р30 рибонуклеазы Р (RPP30), имеющий постоянный уровень в цельной крови и в сухой капле крови, зависящий только от количества клеток во всех образцах вне зависимости от индивидуальных особенностей пациентов.
4. Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно меченых зондов для проведения амплификации целевых генов GSTM1, GSTT1, CYP2D6 и эталонного нормировочного гена RPP30: GSTM1F-cacctgcattcgttcatgtgac, GSTM1R-aagcaagagcagagaggagac, GSTM1zondFAM-FAM-gccatgagcaggcacagtgagtgc-RTQ1/BHQ1, GSTT1F-gtcccagagcacctcacc, GSTT1R-gtgtgcatcattctcattgtgg, GSTT1zondHEX-HEX-caccatccccaccctgtcttcca-BHQ1, CYP2D6*5F-tgagcccatctgggaaaca, CYP2D6*5R-ggtgtcccagcaaagttcatg, CYP2D6*5zondCy5-Cy5-cagtgcaggggccgagggag-RTQ2/BHQ2, RPP30F-tttggacctgcgagcg, RPP30R-gagcggctgtctccacaagt, RPP30-zondROX-ROX-ttctgacctgaaggctctgcgcg-RTQ2/BHQ2.
Авторы
Даты
2023-11-16—Публикация
2022-11-28—Подача