Группа изобретений относится к биотехнологии и медицине, а именно к инфекционным болезням и медицинской генетике, в частности к проблеме прогностической оценки развития гепатоцеллюлярной карциномы при хронических вирусных гепатитах. Диагностику проводят с использованием ДНК, выделенной из цельной крови или сухих пятен крови (сухой капли крови).
Вирусные гепатиты (ВГ), предпосылкой которых являются вирусы гепатита В (ВГВ), С (ВГС), D (BID), относятся к наиболее значимым причинам заболеваемости и смертности во всем мире. Несмотря на наличие прививок от вируса гепатита В (ВГВ), эффективность которых составляет от 98% до 100%, именно этот патоген стал причиной примерно 820000 смертей в 2019 году. Причем смерти были связаны преимущественно с последствиями цирроза печени и гепатоцеллюлярной карциномой (ГЦК) [Robinson A, Wong R, Gish RG. Chronic Hepatitis В Virus and Hepatitis D Virus: New Developments. Clin Liver Dis. 2023 Feb;27(1):17-25. doi: 10.1016/j.cld.2022.08.001].
Гепатоцеллюлярная карцинома является четвертой по значимости причиной смертности от рака во всем мире и ведущей причиной смерти при циррозе печени. Прогноз при ГЦК неблагоприятный, во всем мире смертность приближается к показателям заболеваемости [Ganesan Р, Kulik LM. Hepatocellular Carcinoma: New Developments. Clin Liver Dis. 2023 Feb;27(1):85-102. doi: 10.1016/j.cld.2022.08.004]. Ранняя диагностика с постоянным наблюдением демонстрировала лучшие результаты, чем диагностика на основе симптомов, с соответствующими объединенными показателями 3-летней выживаемости 50,8% и 21,9%. Однако большинство случаев ГЦК диагностируется на поздних стадиях, что приводит к росту и метастазированию злокачественной опухоли и высокой смертности [Li Q, Chen К, Huang W, Ma H, Zhao X, Zhang J, Zhang Y, Fang C, Nie L. Minimally invasive photothermal ablation assisted by laparoscopy as an effective preoperative neoadjuvant treatment for orthotopic hepatocellular carcinoma. Cancer Lett. 2021 Jan 1;496:169-178. doi: 10.1016/j.canlet.2020.09.024]. Рентгеновская компьютерная томография (KT), магнитно-резонансная томография (МРТ) и ультразвуковое исследование (УЗИ) позволяют визуализировать солидные опухоли глубоко в тканях, однако их чувствительности недостаточно для обнаружения очагов ГЦК размером около миллиметра. Оптическая визуализация ГЦК представляет огромный интерес из-за биобезопасности, превосходной чувствительности и возможности молекулярной визуализации, но она не может обеспечить высокое разрешение в глубоких тканях. Таким образом, ранняя диагностика ГЦК по-прежнему представляет собой клиническую проблему [Qi S, Zhang Y, Liu G, Chen J, Li X, Zhu Q, Yang Y, Wang F, Shi J, Lee CS, Zhu G, Lai P, Wang L, Fang C. Plasmonic-doped melanin-mimic for CXCR4-targeted NIR-II photoacoustic computed tomography-guided photothermal ablation of orthotopic hepatocellular carcinoma. Acta Biomater. 2021 Jul 15;129:245-257. doi: 10.1016/j.actbio.2021.05.034].
Диагностика и наблюдение за ГЦК развиваются с появлением новых биомаркеров и разработкой рекомендаций по персонализированному скринингу, основанных на причине и факторах риска заболевания. К таким факторам относятся как инфицирование гепатотропными патогенами, прежде всего вирусами гепатита В, С, D, так и нарушение обмена веществ и/или особенности метаболизма индивидуума. Неинвазивное прогнозирование долгосрочного риска ГЦК при прогрессирующем фиброзе печени, инфекции вирусными гепатитами и других факторах риска крайне необходимо для скрининга ГЦК [Fujiwara N, Kobayashi М, Fobar AJ, Hoshida A, Marquez С A, Koneru B, Panda G, Taguri M, Qian T, Raman I, Li QZ, Hoshida H, Sezaki H, Kumada H, Tateishi R, Yokoo T, Yopp AC, Chung RT, Fuchs ВС, Baumert TF, Marrero JA, Parikh ND, Zhu S, Singal AG, Hoshida Y. A blood-based prognostic liver secretome signature and long-term hepatocellular carcinoma risk in advanced liver fibrosis. Med (N Y). 2021 Jul 9;2(7):836-850.e10. doi: 10.1016/j.medj.2021.03.017].
К биомаркерам, способным дать прогностические сведения о риске развития ГЦК несомненно следует отнести мутации ряда генов человека. В том числе генов, потенциально влияющих на конверсию острой инфекции ВГ в хроническую, развитие цирроза печени и ГЦК. Особенно важным это становится в связи с тем, что элиминация вируса, особенно на поздних стадиях заболевания, не гарантирует окончательного заживления повреждения печени и не устраняет риск декомпенсации печени или развития ГЦК [Wirth ТС, Manns MP. The impact of the revolution in hepatitis С treatment on hepatocellular carcinoma. Ann Oncol. 2016;27: 1467-1474. doi: 10.1093/annonc/mdw219]. Изменчивость различных генов иммунной системы может объяснить различия в исходе инфекции. Например, известна связь семи однонуклеотидных замен в генах IL-28B (rs12979860), JAK1 (rs11576173 и rs1497056), TYK2 (rs280519), OAS1 (rs2057778), SOCS1 (rs33932899) и RNASEL (rs3738579) с тяжелой степенью некровоспалительной активности печени у пациентов с ХГС [Lopez-Rodriguez R, Hernandez-Bartolome A, Borque MJ, Rodriguez-Munoz Y, Martin-Vilchez S, Garcia-Buey L, Gonzalez-Moreno L, Real-Martinez Y, Munoz de Rueda P, Salmeron J, Vidal-Castineira JR, Lopez-Larrea C, Rodrigo L, Moreno-Otero R, Sanz-Cameno P. Interferon-related genetic markers of necroinflammatory activity in chronic hepatitis C. PLoS One. 2017 Jul 12;12(7):e0180927. doi: 10.1371/journal.pone.0180927].
Интерфероны (IFN)-α/β представляют собой цитокины, участвующие как в врожденном, так и в адаптивном иммунном ответе. Интерферон типа 1 (IFN1) представляет собой первую линию иммунной защиты хозяина, играет значимую роль во врожденном иммунном ответе и является важным цитокином, опосредующим противовирусный ответ хозяина. Индуцированные интерфероном пути передачи сигнала представляют собой точно настроенную сеть взаимодействий, которые запускаются при связывании IFN с его рецептором (IFNR). Поскольку IFN являются противоопухолевыми и противовирусными цитокинами, ожидается, что генетические профили генов, участвующих в этих путях передачи сигнала (например, IFNR), будут влиять на восприимчивость пациентов к раку и, при вовлечении ВГВ, к хроническому вирусному гепатиту В и связанной с ВГВ ГЦК [Karamitros Т, Papatheodoridis G, Paraskevis D, Hatzakis A, Mbisa JL, Georgopoulou U, Klenerman P, Magiorkinis G. Impact of Interferon-α Receptor-1 Promoter Polymorphisms on the Transcriptome of the Hepatitis В Virus-Associated Hepatocellular Carcinoma. Front Immunol. 2018 Apr 16;9:777. doi: 10.3389/fimmu.2018.00777].
Известно, что полиморфные варианты IFNAR-1 в промоторной области гена (-568, -408, -77 и -3) и в кодирующих областях (C/G в интроне 3 и C/G в экзоне 4) связаны с предрасположенностью к хронизации вирусного гепатита и его прогрессированию [Rehman SU, Rauf М, Abbas Z, Hamed MH, Qadri I. Role of Some Predominant Host Immunomodulators' Single Nucleotide Polymorphisms in Severity of Hepatitis В Virus and Hepatitis С Virus Infection. Viral Immunol. 2016 Dec;29(10):536-545. doi: 10.1089/vim.2016.0062]. Известно, что IFNAR-1 является одним из генов, чья экспрессия связана с канцерогенезом печени [Bellodi-Privato М, Kubrusly MS, Stefano JT, Soares 1С, Wakamatsu A, Oliveira AC, Alves VA, Bacchella T, Machado MC, D'Albuquerque LA. Differential gene expression profiles of hepatocellular carcinomas associated or not with viral infection. Braz J Med Biol Res. 2009 Dec;42(12):119-1127. doi: 10.1590/s0100-879x2009005000037; Damdinsuren B, Nagano H, Wada H, Noda T, Natsag J, Marubashi S, Miyamoto A, Takeda Y, Umeshita K, Doki Y, Dono K, Monden M. Interferon alpha receptors are important for antiproliferative effect of interferon-alpha against human hepatocellular carcinoma cells. Hepatol Res. 2007 Jan;37(1):77-83. doi: 10.1111/j.l872-034X.2007.00007.x].
Таким образом, определение мутаций гена IFNAR-1 и уровня его экспрессии у инфицированных ВГВ, ВГС, BTD лиц дает ценную прогностическую информацию в отношении возможного развития гепатоцеллюлярной карциномы, что позволяет выявлять пациентов для углубленного обследования, своевременного назначения адекватной терапии, а также проведения комплекса профилактических мероприятий.
Известен способ определения уровня экспрессии гена IFNAR-1 в цельной крови [Lalle Е, Bordi L, Caglioti С, Garbuglia AR, Castilletti С, Taibi С, Cristofari F, Capobianchi MR. IFN-Alpha receptor-1 upregulation in PBMC from HCV naive patients carrying cc genotype, possible role of IFN-lambda. PLoS One. 2014 Apr 1;9(4):e93434. doi: 10.1371/journal.pone.0093434]. Недостатком метода является необходимость для работы предварительного выделения мононуклеаров из периферической крови, а также использование только одного нормировочного гена, кроме того, не проводится определение мутаций гена IFNAR-1 в рамках этого способа, в то время как есть мутации, не влияющие на уровень экспрессии основного транскрипта IFNAR-1, однако играющие регулирующую роль в экспрессии вторичного, укороченного, специфичного для ГЦК транскрипта, что, в свою очередь, совпадает с нарушениями сигнальных путей, связанных с раком, и модификациями экспрессии FN-1 [Karamitros Т, Papatheodoridis G, Paraskevis D, Hatzakis A, Mbisa JL, Georgopoulou U, Klenerman P, Magiorkinis G. Impact of Interferon-a Receptor-1 Promoter Polymorphisms on the Transcriptome of the Hepatitis В Virus-Associated Hepatocellular Carcinoma. Front Immunol. 2018 Apr 16;9:777. doi: 10.33 89/fimmu.2018.00777].
Известен способ определения потенциально патогенетически значимых мутаций гена IFNAR-1 при оценке повышения риска развития хронического вирусного гепатита В [Zhang G, deWeerd NA, Stifter SA, Liu L, Zhou B, Wang W, Zhou Y, Ying В, Ни X, Matthews AY, Ellis M, Triccas JA, Hertzog PJ, Britton WJ, Chen X, Feng CG. A proline deletion in IFNAR1 impairs IFN-signaling and underlies increased resistance to tuberculosis in humans. Nat Commun. 2018 Jan 8;9(1):85. doi: 10.1038/s41467-017-02611-z]. Недостатком способа является необходимость для работы предварительного выделения мононуклеаров из периферической цельной крови, а также определение ряда мутаций без возможности анализа полиморфизма всех экзонов гена IFNAR-1, кроме того, не осуществляется одновременной оценки уровня экспрессии IFNAR-1, в то время как он может быть связан не только с собственными мутациями, но и с мутациями иных генов, участвующих в общих с IFNAR-1 сигнальных путях [Chen J, Xu W, Chen Y, Xie X, Zhang Y, Ma C, Yang Q, Han Y, Zhu C, Xiong Y, Wu K, Liu F, Liu Y, Wu J. Matrix Metalloproteinase 9 Facilitates Hepatitis В Virus Replication through Binding with Type I Interferon (IFN) Receptor 1 To Repress IFN/JAK/STAT Signaling. J Virol. 2017 Mar 29;91(8):e01824-16. doi: 10.1128/JVI.01824-16], а также с наличием вирусной инфекции [Sedefio-Monge V, Santos-Lopez G, Rocha-Gracia RC, Melendez-Mena D, Ramirez-Mata A, Vallejo-Ruiz V, Reyes-Leyva J. Quantitative analysis of interferon alpha receptor subunit 1 and suppressor of cytokine signaling 1 gene transcription in blood cells of patients with chronic hepatitis C. Virol J. 2010 Sep 18;7:243. doi: 10.1186/1743-422X-7-243].
Наиболее близким по сущности к заявляемому изобретению и выбранным за прототип является метод, предложенный в работе Mohammadarian Akbari с соавторами [Akbari М, Akhavan-Bahabadi М, Shafigh N, Taheriazam A, Hussen ВМ, Sayad A, Fathi М, Taheri М, Ghafouri-Fard S, Fathi M. Expression analysis of IFNAR1 and TYK2 transcripts in COVID-19 patients. Cytokine. 2022 May; 153:155849. doi: 10.1016/j.cyto.2022.155849]. Согласно методу, тотальную РНК выделяют из цельной крови, затем осуществляют обратную транскрипцию, получая кДНК, и проводят амплификацию целевого гена одновременно с эталонным нормировочным геном HPRT, уровень экспрессии оценивают по значениям Ct. Недостатком способа является использование одного нормировочного гена для нормирования данных. Недостатком способа нормирования данных с использованием одного эталонного гена является вариабельность результатов анализа, так как не существует идеального нормировочного гена, постоянного в независимости от ткани и состояния клеток в анализируемом образце. В связи с этим выбор эталонного гена является одним из самых ответственных этапов при проведении анализа. Наиболее оптимальным можно считать подход, при котором анализируется одновременно несколько эталонных генов. Кроме того, недостатком является то, что не проводится определение мутаций гена IFNAR-1, в то время как есть мутации, не влияющие на уровень экспрессии основного транскрипта IFNAR-1, однако играющие регулирующую роль в экспрессии вторичного, укороченного, специфичного для ГЦК транскрипта, как уже было упомянуто выше.
Задачей, на решение которой направлено заявляемое изобретение, является расширение арсенала способов, предназначенных для оценки на доклиническом этапе генетической предрасположенности человека к развитию гепатоцеллюлярной карциномы при вирусном гепатите.
Технический результат - повышение чувствительности и достоверности диагностики, снижение ложноположительных (низкие уровни экспрессии гена IFNAR-1) результатов, а значит и расширение возможностей метода, расширение арсенала средств, используемых для прогноза развития гепатоцеллюлярной карциномы при вирусном гепатите.
Авторами предложен способ, согласно которому в образце кДНК, полученном при выделении РНК в присутствии ДНКазы из биологического материала (цельной крови или сухой капли крови) с последующей обратной транскрипцией, определяют уровень экспрессии целевого гена IFNAR-1 и двух эталонных нормировочных генов HPRT и RPP30. Регистрируют полученные результаты посредством гибридизационно-флуоресцентной детекции в режиме реального времени. Уровень экспрессии целевого гена IFNAR-1 определяют путем пересчета полученных в ходе работы значений на калибровочных графиках с использованием в качестве калибраторов серии последовательных разведений контрольного образца, представляющего собой ранее охарактеризованный образец кДНК, в котором представлен ген IFNAR-1 дикого типа. Параллельно осуществляют ПНР для получения протяженных продуктов амплификации нуклеотидной последовательности транскрипта целевого гена.
Сущность метода заключается в том, что экстрагируют РНК из цельной крови или сухих пятен крови (сухой капли крови), осуществляют обратную транскрипцию, получая кДНК, после чего используют полученный образец кДНК для одновременной амплификации в одной емкости участков целевого гена IFNAR-1 и двух эталонных нормировочных генов HPRT и RPP30, а также для амплификации в других емкостях фрагментов транскрипта гена IFNAR-1 с последующим секвенированием по Сэнгеру.
Состав амплификационной смеси представляет собой буферный раствор, содержащий Трис-HCl рН 8,8 (при 25°С), KCl, 6-7 мМ MgCl2, дезоксинуклеозидтрифосфаты, глицерол, Tween 20, Phusion-полимераза.
При определении нуклеотидной последовательности полного транскрипта гена IFNAR-1 для амплификации и последующей секвенирующей реакции используют набор олигодезоксирибонуклеотидных праймеров, комплементарных участкам выявляемых фрагментов, при этом в одной емкости для ПНР используют пару праймеров IFNAR-1F1/R1, фланкирующую фрагмент протяженностью 5944 нуклеотидов, область 1-5944 нт., согласно представленной в международной базе данных GenBank референтной последовательности (NM_001384504.1), в другой емкости используют пару праймеров IFNAR-1F2/R2, фланкирующую фрагмент протяженностью 4607 нуклеотидов, область 1551-6157 нт., для секвенирующих реакций используют уже упомянутые праймеры, а также праймеры IFNAR-1F3/R3, IFNAR-1F4/R4, IFNAR-1F5/R5:


ПЦР и детекцию результатов проводят при следующих условиях: после денатурации при 95°С в течение 15 минут устанавливают 45 циклов амплификации в режиме: 95°С - 30 сек, 52°С - 30 сек, 72°С - 6 мин 30 сек; после 15-го и 30-го циклов элонгация при 72°С - 8 мин; затем финальная элонгация при 72°С - 10 мин.
При определении уровня экспрессии для амплификации используют набор олигодезоксирибонуклеотидных праймеров и соответствующих флуоресцентно меченых зондов, комплементарных участкам выявляемых фрагментов:

ПЦР и детекцию результатов проводят при следующих условиях:
Первичный анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени».
Анализируют кривые накопления флуоресцентного сигнала по четырем каналам:
- по каналу для флуорофора HEX регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента гена IFNAR-1;
- по каналу для флуорофора Су5 регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента гена HPRT;
- по каналу для флуорофора ROX регистрируется сигнал, свидетельствующий о накоплении продукта амплификации фрагмента ДНК RPP30.
Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (устанавливается в середине линейного участка прироста флуоресценции положительного контроля в логарифмической шкале), что определяет наличие (или отсутствие) для данной пробы значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат амплификации по каналу считается положительным, если кривая однократно пересекается с пороговой линией в области достоверного прироста флуоресценции, отрицательным в случае отсутствия пересечения кривой с пороговой линией (нет значения Ct), сомнительным во всех других случаях.
Анализ экспрессии IFNAR-1 проводят с нормализацией на эталонные гены, при этом вводится «нормировочный коэффициент», вычисляемый как среднее геометрическое или среднее арифметическое от значения всех эталонных нормировочных генов. Относительный уровень экспрессии IFNAR-1 определяют с использованием метода ΔΔCT [Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001 Dec;25(4):402-8. doi: 10.1006/meth.2001.1262] в сравнении с последовательной серией разведений кДНК охарактеризованного образца-калибратора, в котором представлен ген IFNAR-1 дикого типа, на основании чего делают выводы о нормальном или сниженном уровне экспрессии целевого гена.
При выявлении сниженной экспрессии IFNAR-1 и/или мутаций нуклеотидной последовательности транскрипта, приводящей к изменениям аминокислотной последовательности продукта гена, делают вывод о наличии у обследуемого предрасположенности к развитию ГЦК.
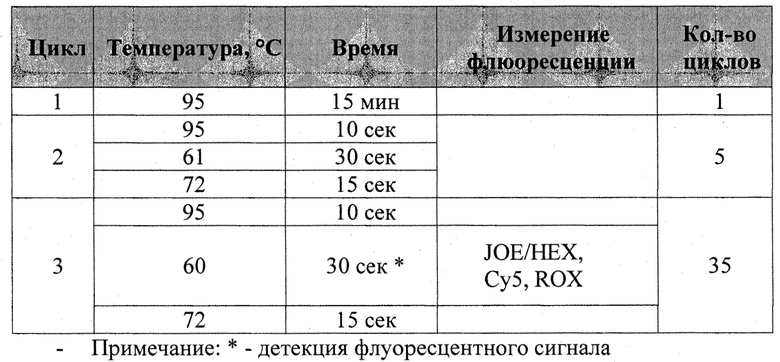
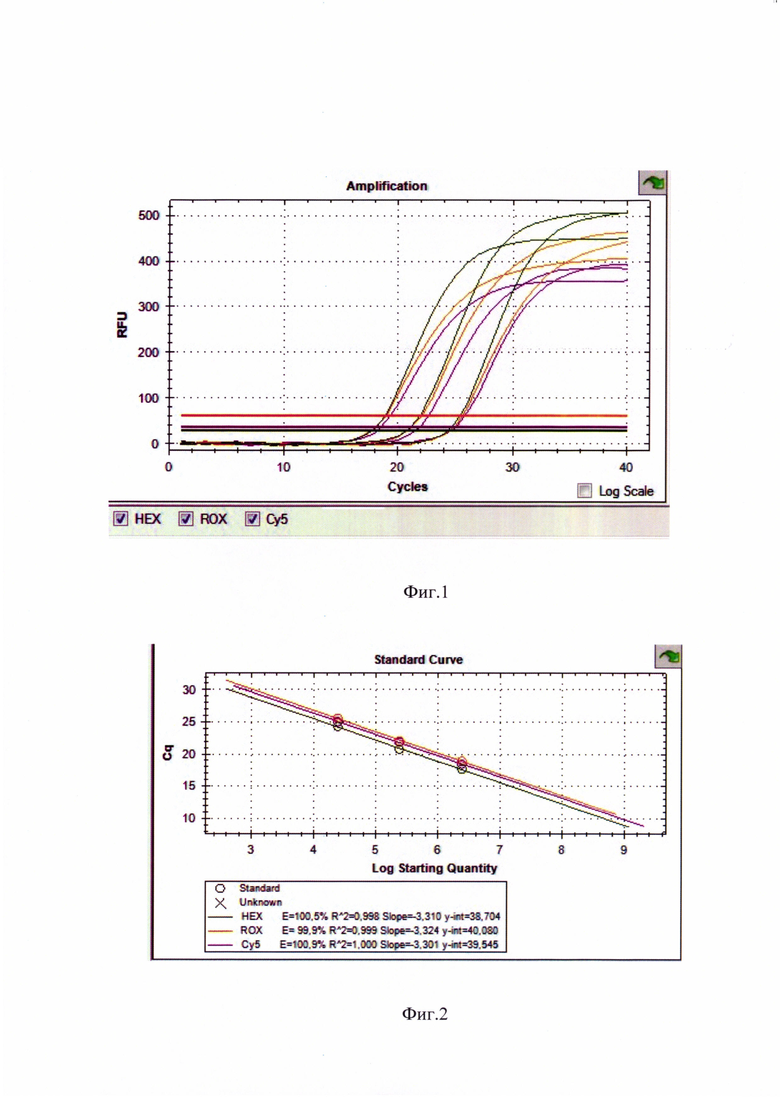
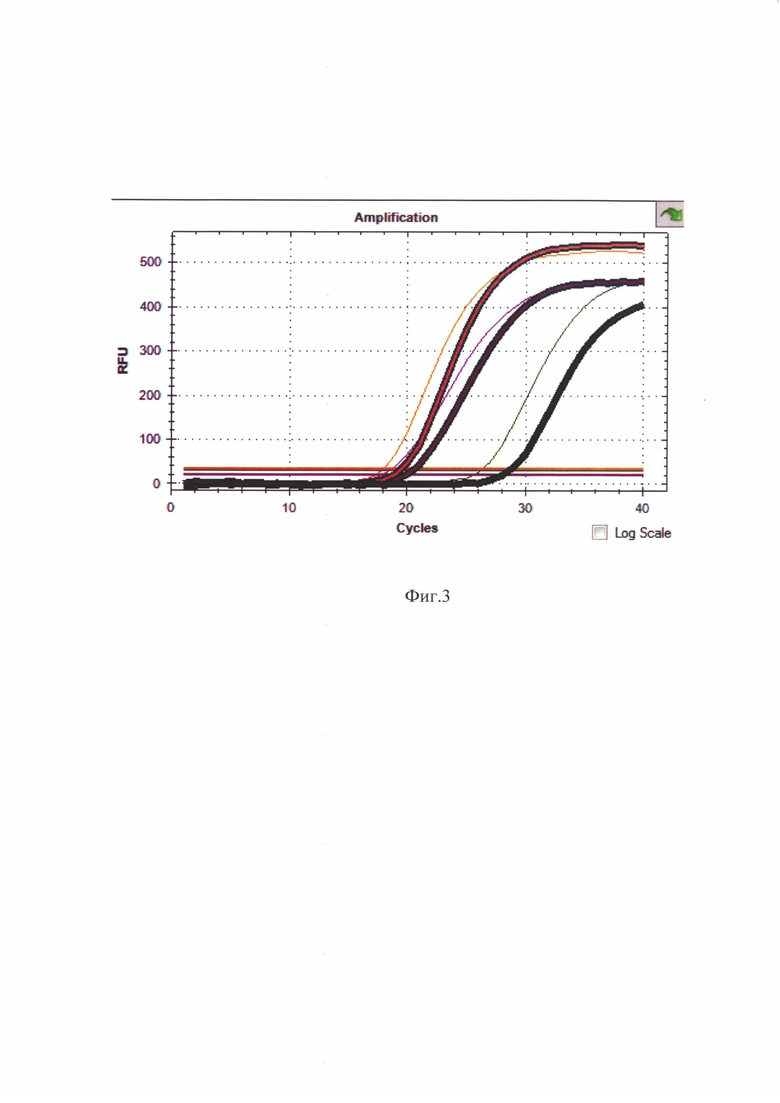
Сущность изобретения поясняется чертежами, где на фиг.1 представлены кривые флуоресценции трех последовательных разведений кДНК контрольного образца-калибратора, в котором представлен ген IFNAR-1 дикого типа, отражающие динамику образования продукта реакции в ходе амплификации всех анализируемых генов, включая один целевой ген IFNAR-1 (HEX - зеленый) и два нормировочных HPRT (Су5 - фиолетовый), RPP30 (ROX - оранжевый) при «диком» генотипе; на фиг.2 представлены калибровочные графики, демонстрирующие эффективность ПЦР целевого и нормировочных генов. А также фиг.3 где представлены кривые флуоресценции, отражающие динамику образования продукта реакции в ходе амплификации всех анализируемых генов, включая один целевой ген IFNAR-1 (HEX - зеленый) и два нормировочных HPRT (Су5 - фиолетовый), RPP30 (ROX - оранжевый) при сниженном уровне экспрессии IFNAR-1.
Таким образом, предложенный способ отличается от прототипа использованием двух эталонных нормировочных генов HPRT и RPP30 при оценке уровня экспрессии целевого гена IFNAR-1, оценкой не только экспрессии гена IFNAR-1, но и нуклеотидной последовательности его транскрипта для выявления патогенетически значимых мутаций, набором олигодезоксирибонуклеотидных праймеров.
Ниже приведены примеры конкретного использования предложенного метода, являющиеся одним из возможных способов его реализации.
Пример 1
Исследованы биоптаты печени и образцы цельной крови, полученные от 298 пациентов с хроническим вирусным гепатитом В с различной тяжестью течения и прогрессированием заболевания. Показана ассоциация полиморфизма L168V-C/G в 4 экзоне (rs2257167) с острой печеночной недостаточностью, развившейся на фоне хронической, и ГЦК: χ2=4,287 OR=3,7, df=1,95% ДИ: 1,185 - 1,913%, р<0,0384.
Пример 2
Обследованы 78 больных хроническим вирусным гепатитом В с установленной ГЦК, а также 134 больных без тяжелой патологии печени и 200 условно здоровых лиц. Показано значительное снижение уровня экспрессии IFNAR-1 у пациентов с ГЦК по сравнению с больными ХГВ без осложнений и по сравнению со здоровыми людьми - χ2=8,472, р<0,05. Показан высокий риск развития ГЦК при снижении уровня экспрессии IFNAR-1: OR=3,8 df=1,95% ДИ: 1,610 - 8,970%, р=0,0024.
Пример 3
Обследовали жителей стран Средней Азии, в том числе 119 условно здоровых лиц и 149 больных ХГВ с ГЦК или ОХПН. Показана ассоциация полиморфизма I499T-T/C в 11 экзоне (rs113181057) с ОХПН: χ2=6,44 OR=3,008, df=1,95% ДИ: 1,352 - 6,694%, р=0,0111, а также с ГЦК: χ2=13,492 OR=5,207, df=1,95% ДИ: 2,194 - 12,355%, р<0,0002.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="СПОСОБ
ПРОГНОСТИЧЕСКОЙ ОЦЕНКИ РАЗВИТИЯ ГЕПАТОЦЕЛЛЮЛЯРНОЙ КАРЦИНОМЫ НА ОСНОВЕ
ОПРЕДЕЛЕНИЯ ПОЛИМОРФИЗМА ГЕНА ЧЕЛОВЕКА IFNAR-1 И НАБОР
ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ.xml" softwareName="WIPO
Sequence" softwareVersion="2.2.0" productionDate="2023-02-20">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022131541/20(068845)</ApplicationNumberText>
<FilingDate>2022-12-01</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия человека»
(ФБУН НИИ эпидемиологии и микробиологии имени Пастера), ул. Мира, 14
, Санкт-Петербург, 197101, Российская Федерация</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur Institute
</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ прогностической оценки
развития гепатоцеллюлярной карциномы на основе определения
полиморфизма гена человека IFNAR-1 и набор
олигодезоксирибонуклеотидных праймеров</InventionTitle>
<SequenceTotalQuantity>19</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcacctgatggcctatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggagcatgaagaactggatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a HEX fluorophore at the 5' end and a dark quencher
RTQ1 or BHQ1 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggccagcaagcgcatctccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgctcgagatgtgatgaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcaggtcagcaaagaatttatag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a Cy5 fluorophore at the 5' end and a dark quencher
RTQ2 or BHQ2 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcacattgtagccctctgtgtgctcaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttggacctgcgagcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcggctgtctccacaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Oligonucleotide fluorescent probe
carrying a ROX fluorophore at the 5' end and a dark quencher
RTQ2 or BHQ2 at the 3' end</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttctgacctgaaggctctgcgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctctcgctctgcacacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaagagcctccttggaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagatgcatcaattatgtcttctttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaagttactggtattttgttattac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctgtgttcctgtccatcactg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccttcaaccttttcttccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggaagaaaaggttgaaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtggaatgcaactgcctttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Forward PCR primer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttcagtgaacaagcgctgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q57">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Reverse primer PCR</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcctccttggaacgaactaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Группа изобретений относится к биотехнологии и медицине. Диагностику проводят с использованием олигодезоксирибонуклеотидных праймеров и соответствующего одной из пар праймеров флуоресцентно меченого зонда, комплементарных участкам выявляемых фрагментов, а также с использованием стандартных образцов, представляющих собой последовательную серию разведений кДНК охарактеризованного образца-калибратора. Экстрагируют РНК из цельной крови или сухих пятен крови (сухой капли крови), осуществляют обратную транскрипцию, получая кДНК, после чего используют полученный образец кДНК для одновременной амплификации в одной емкости участков целевого гена IFNAR-1 и двух эталонных нормировочных генов HPRT и RPP30, а также для амплификации в других емкостях фрагментов транскрипта гена IFNAR-1 с последующим секвенированием по Сэнгеру. При определении уровня экспрессии для амплификации регистрируют полученные результаты с помощью программного обеспечения используемого прибора для проведения ПНР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Анализ экспрессии IFNAR-1 проводят с нормализацией на эталонные гены, при этом вводится «нормировочный коэффициент», вычисляемый как среднее геометрическое или среднее арифметическое от значения всех эталонных нормировочных генов. Относительный уровень экспрессии IFNAR-1 определяют с использованием метода ΔΔСТ в сравнении с последовательной серией разведений кДНК охарактеризованного образца-калибратора, в котором представлен ген IFNAR-1 дикого типа, на основании чего делают выводы о нормальном или сниженном уровне экспрессии целевого гена. При определении нуклеотидной последовательности полного транскрипта гена IFNAR-1 осуществляют амплификацию двух перекрывающихся фрагментов транскрипта: протяженностью 5944 нуклеотидов, область 1-5944 нт., и протяженностью 4607 нуклеотидов, область 1551-6157 нт., согласно представленной в международной базе данных GenBank референтной последовательности (NM_0013 84504.1). Затем осуществляют прямое секвенирование по Сэнгеру полученных фрагментов транскрипта гена IFNAR-1 и делают выводы о наличии или отсутствии потенциально патогенетически значимых мутаций. При выявлении сниженной экспрессии IFNAR-1 и/или мутаций нуклеотидной последовательности транскрипта, приводящей к изменениям аминокислотной последовательности продукта гена, делают вывод о наличии у обследуемого предрасположенности к развитию ГЦК. Обеспечивается повышение чувствительности и достоверности диагностики, снижение ложноположительных (низкие уровни экспрессии гена IFNAR-1) результатов, а значит и расширение возможностей метода, расширение арсенала средств, используемых для прогноза развития гепатоцеллюлярной карциномы при вирусном гепатите. 2 н. и 2 з.п. ф-лы, 3 ил., 3 табл., 3 пр.
1. Способ прогностической оценки развития гепатоцеллюлярной карциномы на основе определения полиморфизма гена человека IFNAR-1, предусматривающий экстракцию РНК из биологического материала с последующей обратной транскрипцией и проведением полимеразной цепной реакции, при этом в одной из емкостей осуществляют амплификацию фрагмента целевого генов и эталонных нормировочных генов с использованием набора олигодезоксирибонуклеотидных праймеров и соответствующих флуоресцентно меченых зондов, комплементарных участкам выявляемых фрагментов, отличающийся тем, что используют IFNAR-1F-cagcacctgatggcctatcac, IFNAR-1R-tggagcatgaagaactggatg, IFNAR-1zondFffiX-HEX-ggccagcaagcgcatctccag-BHQ1, FIPRTF-cttgctcgagatgtgatgaagg, FIPRTR-cagcaggtcagcaaagaatttatag, HPRT-zondCy5-Cy5-atcacattgtagccctctgtgtgctcaagg-RTQ2/BHQ2, RPP30F-tttggacctgcgagcg, RPP30R-gagcggctgtctccacaagt, RPP30-zondROX-ROX-ttctgacctgaaggctctgcgcg-RTQ2/BHQ2, при этом анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени, анализ целевых генов проводят с нормализацией на эталонные гены, при этом используют нормировочный коэффициент, вычисляемый как среднее геометрическое или среднее арифметическое от значения всех эталонных нормировочных генов, относительный уровень экспрессии IFNAR-1 определяют с использованием метода ААСТ в сравнении с последовательной серией разведений кДНК охарактеризованного образца-калибратора, в котором представлен ген IFNAR-1 дикого типа, на основании чего делают выводы о нормальном или сниженном уровне экспрессии целевого гена, одновременно в двух других емкостях для определения нуклеотидной последовательности транскрипта гена IFNAR-1 осуществляют амплификацию фрагментов 1-5944 нт. и 1551-6157 нт. (NM_001384504.1) с использованием следующих праймеров, комплементарных участкам выявляемых фрагментов: IFNAR-1F1-gctctcgctctgcacacag, IFNAR-1R1-tgaagagcctccttggaacg, IFNAR-1F2-gagatgcatcaattatgtcttctttc, IFNAR-1R2-tggaagttactggtattttgttattac, продукты амплификации затем используют для секвенирующей реакции, применяя вышеуказанные олигонуклеотидные праймеры, а также следующие дополнительные: IFNAR-1F3-tctgtgttcctgtccatcactg, IFNAR-1R3-gccttcaaccttttcttccacc, IFNAR-1F4-ggtggaagaaaaggttgaaggc, IFNAR-1R4-gagtggaatgcaactgcctttt, IFNAR-1F5-ttttcagtgaacaagcgctgag, IFNAR-1R5-gagcctccttggaacgaactaa.
2. Способ по п. 1, отличающийся тем, что проводят одновременную амплификацию в одной емкости участков целевого гена IFNAR-1 и двух эталонных нормировочных генов HPRT и RPP30.
3. Способ по п. 1, отличающийся тем, что одновременно с оценкой уровня экспрессии целевого гена осуществляют также определение нуклеотидной последовательности его транскрипта.
4. Набор олигодезоксирибонуклеотидных праймеров для проведения амплификации целевого гена IFNAR-1 и эталонных нормировочных генов HPRT и RPP30: IFNAR-1F- cagcacctgatggcctatcac, IFNAR-1R-tggagcatgaagaactggatg, IFNAR-1zondHEX-HEX-ggccagcaagcgcatctccag-BHQ1, HPRTF-cttgctcgagatgtgatgaagg, HPRTR-cagcaggtcagcaaagaatttatag, HPRT-zondCy5-Cy5-atcacattgtagccctctgtgtgctcaagg-RTQ2/BHQ2, RPP30F-tttggacctgcgagcg, RPP30R-gagcggctgtctccacaagt, RPP30-zondROX-ROX-ttctgacctgaaggctctgcgcg-RTQ2/BHQ2, IFNAR-1F1-gctctcgctctgcacacag, IFNAR-1R1-tgaagagcctccttggaacg, IFNAR-1F2-gagatgcatcaattatgtcttctttc, IFNAR-1R2-tggaagttactggtattttgttattac, IFNAR-1F3-tctgtgttcctgtccatcactg, IFNAR-1R3-gccttcaaccttttcttccacc, IFNAR-1F4-ggtggaagaaaaggttgaaggc, IFNAR-1R4-gagtggaatgcaactgcctttt, IFNAR-1F5-ttttcagtgaacaagcgctgag, IFNAR-1R5-gagcctccttggaacgaactaa.
| CN 109504763 A, 22.03.2019 | |||
| РАЗРЫВНАЯ ЗАСТЕЖКА | 1930 |
|
SU38600A1 |
| СПОСОБЫ АМПЛИФИКАЦИИ ДНК ДЛЯ СОХРАНЕНИЯ СТАТУСА МЕТИЛИРОВАНИЯ | 2018 |
|
RU2754038C2 |
Авторы
Даты
2024-01-17—Публикация
2022-12-01—Подача