Предлагаемое изобретение относится к областям медицинской микробиологии, биотехнологии и молекулярной биологии, а именно к молекулярной диагностике и может быть использовано для лабораторной диагностики холеры, как в практическом здравоохранении, так и в научно-исследовательских учреждениях и лабораториях.
В настоящее время остается актуальной проблема распространения острой кишечной инфекции-холеры, которая характерна особой тяжестью течения, высокой летальностью и тенденцией к эпидемическому, а иногда и пандемическому распространению, поэтому отнесена ВОЗ к особо опасным карантинным инфекциям (1, 2).
Наиболее часто эпидемические вспышки холеры регистрируются в странах Африки, Латинской Америки, Юго-Восточной Азии. По оценкам ВОЗ, ежегодно регистрируется 3-5 млн. случаев заболевания холерой, из которых 100-120 тыс. заканчиваются смертельно (3). В связи с этим, холера на сегодняшний день остается глобальной проблемой мирового здравоохранения, что определяет необходимость совершенствования лабораторной диагностики данной инфекции.
Несмотря на многолетней опыт и имеющиеся подробные рекомендации по лабораторной диагностике, идентификация холерного вибриона традиционными методами (культуральные, биохимические и иммунологические) требует значительных затрат и времени. Новый и высокоспецифичный метод петлевой изотермической амплификации (LAMP) из-за его простоты, быстроты и пригодности для крупно масштабного скрининга на месте является альтернативным многообещающим инструментом для чувствительного и быстрого обнаружения холерных вибрионов, в том числе, содержащих ген холерного токсина СТХ (4). Известны различные коммерческие тест-системы для видовой идентификации, а также позволяющие определять наличие различных факторов патогенности холерных вибрионов. Они как правило основаны на методе ПЦР (5). Современный уровень развития лабораторной диагностики характеризуется появлением новых ускоренных способов альтернативных ПЦР. Изотермические методы амплификации нуклеиновых кислот становятся перспективной альтернативой ПЦР и значительно упрощают реализацию способов амплификации.
Одним из таких способов является технология петлевой амплификации (LAMP), которая была разработана Tsugunori Notomi и его коллегами (Япония) в 2000 г. (6), заключается в использовании четырех или шести олигонуклеотидных праймеров, что позволяет значительно повысить специфичность реакции, и фермента Bst-полимеразы, которая отличается сильной вытесняющей активностью.
Данный способ позволяет провести то же исследование, что и с помощью реакции ПЦР, но значительно быстрее, специфичнее, не требует дорогого приборного оснащения и квалифицированного персонала.
Однако, в связи с высокой стоимостью реактивов зарубежного производства, одним из этапов предлагаемой работы, в целях повышения экономической эффективности способа, была проведена оценка возможности применения реагентов для LAMP отечественного производства (ООО «Биолабмикс»).
За прототип выбран способ идентификации холерного вибриона на основе метода LAMP разработанный С. Srisuk с соавторами (7), заключающийся в том, что подобран набор из пяти праймеров, которые специфически распознают ген ompW V. cholerae. Оптимальные временные и температурные условия для анализа составляют 75 минут при 65°С. Специфичность и чувствительность разработанных праймеров была проверена на 16 штаммах V. cholerae, а также штаммах других близкородственных видов.
Отличие данного способа от разработанного нами является использование пяти, а не шести праймеров (отсутствует один петлевой праймер), в связи с чем и времени на реакцию требуется больше. При этом следует отметить маленькую выборку штаммов холерных вибрионов, на которых проверена специфичность праймеров.
Отсутствие отечественных наборов, предназначенных для выявления V. cholerae методом петлевой изотермической амплификации, поставило перед разработчиками задачу сконструировать собственный набор олигонуклеотидных праймеров.
При этом был разработан способ детекции видоспецифичного для Vibrio cholerae гена ompW и гена ctxA, кодирующего холерный токсин СТХ, с помощью реакции петлевой изотермической амплификации (LAMP). Предлагаемое изобретение позволяет быстро в течение 30-60 минут, с высокой специфичностью и чувствительностью 1×104 м.кл./мл. выявлять присутствие штаммов Vibrio cholerae, в том числе, продуцирующих холерный токсин.
Технической задачей предлагаемого изобретения является разработка новых высокоспецифичных олигонуклеотидных праймеров для быстрого, специфичного и чувствительного выявления штаммов Vibrio cholerae в пробах чистых культур, основанного на петлевой изотермической амплификации фрагмента гена-мишени.
Поставленная задача достигается тем, что в известном способе идентификации и определения патогенности штаммов Vibrio cholerae методом петлевой изотермической амплификации (LAMP), включающем выделение ДНК из исследуемого материала, проведение петлевой изометрической амплификации и учет результатов, отличие состоит в том, что амплификацию проводят с помощью набора олигонуклеотидных праймеров к гену ompW и набора праймеров к гену ctxA, следующего состава:
к гену ompW

к гену ctxA
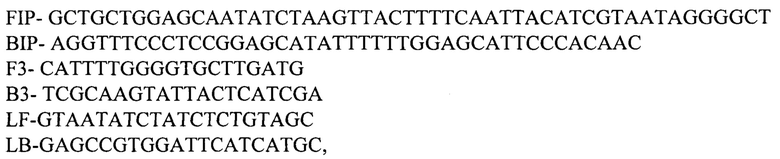
при этом реакцию петлевой изометрической амплификации проводят в два этапа, первоначально к гену ompW, кодирующему белок внешней мембраны холеры, для определения вида Vibrio cholerae, а затем к гену ctxA, кодирующему холерный токсин, для определения вирулентности, учет результатов осуществляют с помощью амплификатора с программой 65°С - 60 сек и снятием сигнала при каждом цикле и длительностью 60 циклов, а детекцию результатов амплификации осуществляют по каналам флуорофора FAM, где положительным считают образец, при анализе которого наблюдают рост флуоресцентного сигнала на цветовом канале амплификатора.
При этом для проведения предворительно готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP- праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл, при этом положительный контроль включает в себя все компоненты реакции и препарат ДНК Vibrio cholerae (ctx+),a в случае отрицательного контроля в реакционную смесь вместо исследуемого препарата ДНК вносят соответствующее количество деионизованной воды, не содержащей исследуемой ДНК.
Кроме того амплификацию проводят в объеме 25 мкл и реакционная смесь содержит: 10хбуфер для Bst ДНК-полимеразы - 2.5 мкл; MgSO4 (100 mM) - 2 мкл; смесь dNTP - (25 mM) - 1,5 мкл; смесь праймеров к гену ompW - 2,5 мкл; Bst ДНК-полимераза - 2 мкл; флуоресцентный краситель EvaGreen (20Х водный раствор) - 1,25 мкл; образец ДНК - 2 мкл; вода, свободная от нуклеаз - до 25 мкл.
Техническим результатом, достигаемым при осуществлении данного изобретения, являются новые высокоспецифичные олигонуклеотидные праймеры для быстрого, специфичного и чувствительного выявления штаммов V. Cholera в пробах чистых культур методом петлевой изотермической амплификации (LAMP).
Обоснование выбора праймеров и ДНК-мишени
Для конструирования праймеров был использован интернет-сервис Primer Explorer 5 (LAMP primer designing software: http://primerexplorer.jp/e/), BLAST NCBI.
В качестве целевой мишени взяты ген ompW, кодирующий белок внешней мембраны и ген ctxA, кодирующий холерный токсин СТХ.
OmpW является членом основного семейства белков, которые локализуются на внешней мембране бактерий и участвуют в транспорте небольших гидрофобных молекул и железа (8). Ген ompW присутствует в штаммах V. cholerae и широко используется в качестве специфической мишени для обнаружения и идентификации V. cholerae (9). Ген ctxA кодируют холерный токсин СТХ (белковый экзотоксин). Холерный токсин секретируется вирулентными штаммами после попадания бактерии V. cholerae в организм человека.
Последовательности для подбора праймеров к генам ompW и ctxA были получены из базы GenBank NCBI:
к гену ompW

к гену ctxA
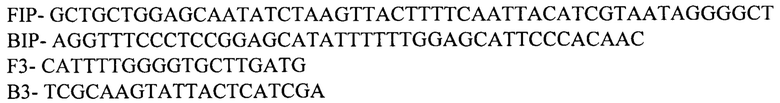
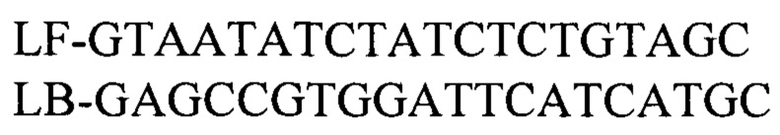
Способ осуществляется следующим образом.
Этапы:
1) подготовка смесей праймеров для проведения LAMP, исследуемых и контрольных образцов (положительный и отрицательный контроли);
2) выделение нуклеиновых кислот из исследуемых и контрольных образцов;
3) проведение петлевой изотермической амплификации (LAMP):
а) - определение принадлежности исследуемых культур к виду Vibrio cholerae - с праймерами к гену ompW;
б) - определение вирулентности штаммов Vibrio cholerae - с праймерами к гену ctxA;
4) учет и интерпретация результатов анализа.
Из полученной исследуемой чистой культуры готовят суспензию в 2 мл 0,9% раствора натрия хлорида в концентрации 1×109 м.кл./мл по стандарту мутности бактериальных взвесей ОСО 42-28-86-2018 (ФГБУ «НЦЭСМП» Минздрава России).
Исследуемые бактериальные суспензии обеззараживают в соответствии с требованиями МУ 1.3.2569-09 и СанПиН 3.3686-21 (10,11).
По первому этапу готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP- праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл.
Положительный контроль включает в себя все компоненты реакции и препарат ДНК Vibrio cholerae (ctx+). В случае отрицательного контроля в реакционную смесь вместо исследуемого препарата ДНК вносится соответствующее количество деионизованной воды, не содержащей исследуемой ДНК.
По второму этапу выделяют ДНК V.cholerae из исследуемого материала с помощью зарегистрированных в установленном порядке наборов реагентов, в соответствии с инструкцией производителя.
Третий этап - с полученными пробами ДНК проводят реакцию петлевой изотермической амплификации (LAMP).
а) При этом на первом этапе с целью определения принадлежности к виду Vibrio cholerae реакцию ставят с праймерами к гену ompW.
Реакцию проводят в объеме 25 мкл и реакционная смесь содержит:
10хбуфер для Bst ДНК-полимеразы - 2.5 мкл; MgSO4 (100 mM) - 2 мкл; смесь dNTP - (25 mM) - 1,5 мкл; смесь праймеров к гену ompW - 2,5 мкл; Bst ДНК-полимераза - 2 мкл; флуоресцентный краситель EvaGreen (20Х водный раствор) - 1,25 мкл; образец ДНК - 2 мкл; вода, свободная от нуклеаз - до 25 мкл.
Для мониторинга флуоресценции окрашенной реакционной смеси в режиме реального времени используют соответствующий амплификатор с программой: 65°С - 60 сек и снятием сигнала при каждом цикле и длительностью 60 циклов.
Четвертый этап-детекцию результатов амплификации осуществляют по каналу FAM. Положительным считается образец, при анализе которого наблюдают рост флуоресцентного сигнала на цветовом канале амплификатора, используемом для учета.
Результат считается достоверным только в случае прохождения положительного контроля реакции и отсутствия амплификации специфического продукта для отрицательного контроля реакции.
б) На втором этапе для определения вирулентности, идентифицированных на первом этапе как Vibrio cholerae штаммов, проводят реакцию LAMP с праймерами к гену ctxA по той же схеме.
При использовании готовых наборов для проведения петлевой изотермической амплификации в режиме реального времени с использованием флуоресцентных красителей или с визуальным колориметрическим способом детекции реакцию проводят согласно инструкции производителя реакционной смеси.
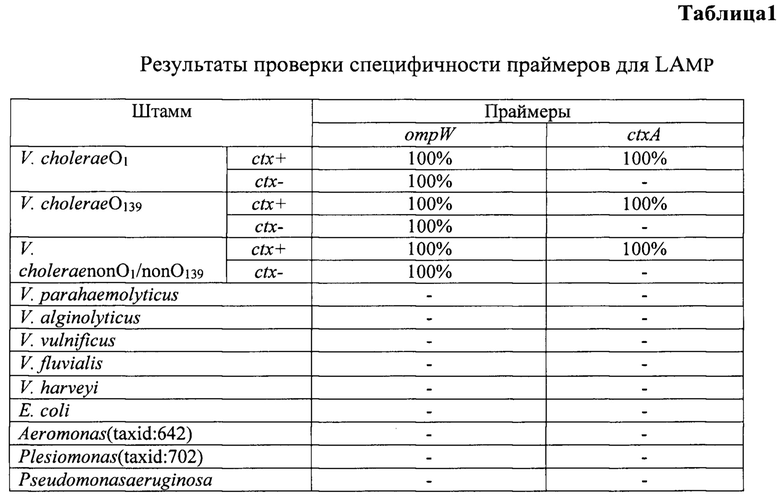
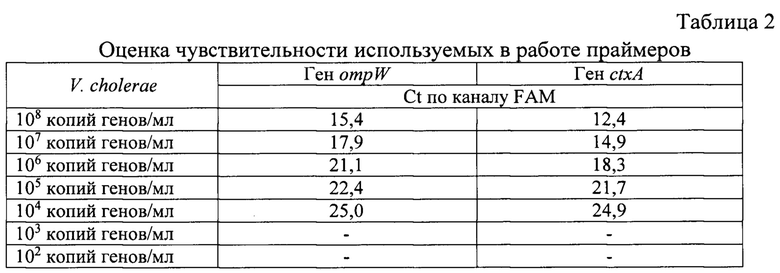
Апробацию праймеров осуществляли на наборе коллекционных и свежевыделенных штаммов V. cholerae, взятых из коллекции ФКУЗ Ростовского-на-Дону противочумного института Роспотребнадзора. Специфичность отобранных праймеров подтвердили путем исследования культур близкородственных видов и родов в количестве 57 штаммов. Чувствительность реакции амплификации с праймерами оценивают при исследовании проб ДНК, выделенных из десятикратных разведений штаммов холерных вибрионов. В результате проведенной оценки установлено, что данные праймеры обладают 100% специфичностью и чувствительностью 1×104 м.кл./мл. (см. таблицу 1, 2).
Сущность изобретения поясняется примерами
Пример 1.
Определение принадлежности культуры к виду V. cholerae в реакции LAMP с помощью праймеров к гену ompW.
Штаммы V. cholerae O1 №№20000, V. cholerae O139 №16064, V. cholerae non O1/non O139 №19874, Vibrio parahaemolyticus №19005 с точно установленной видовой принадлежностью взяты из коллекции ФКУЗ Ростовского-на-Дону противочумного института Роспотребнадзора. В качестве положительного контроля взят штамм V. cholerae O1 Р-15415. В качестве отрицательного контрольного образца используют деионизованную стерильную воду, свободную от ДНКаз.
Бактериальные взвеси клеток суточных культур вибрионов, выросших на питательной среде, готовят в 2 мл 0,9% стерильного раствора натрия хлористого по стандартному образцу мутности 5 единиц (ОСО 42-28-86-2018), что соответствует 1×109 микробных клеток в 1 мл для холерного вибриона. Обеззараживание подготовленных бактериальные взвесей проводят в соответствии с МУ 1.3.2569-09.
Из обеззараженных проб выделяют ДНК с помощью набора реагентов «ДНК-сорб В» («Интерлабсервис», Москва). Работу проводят в соответствии с инструкцией производителя.
Затем проводят постановку петлевой изотермической амплификации (LAMP) с выделенной ДНК с использованием предложенных праймеров к гену ompW по вышеизложенной технологии.
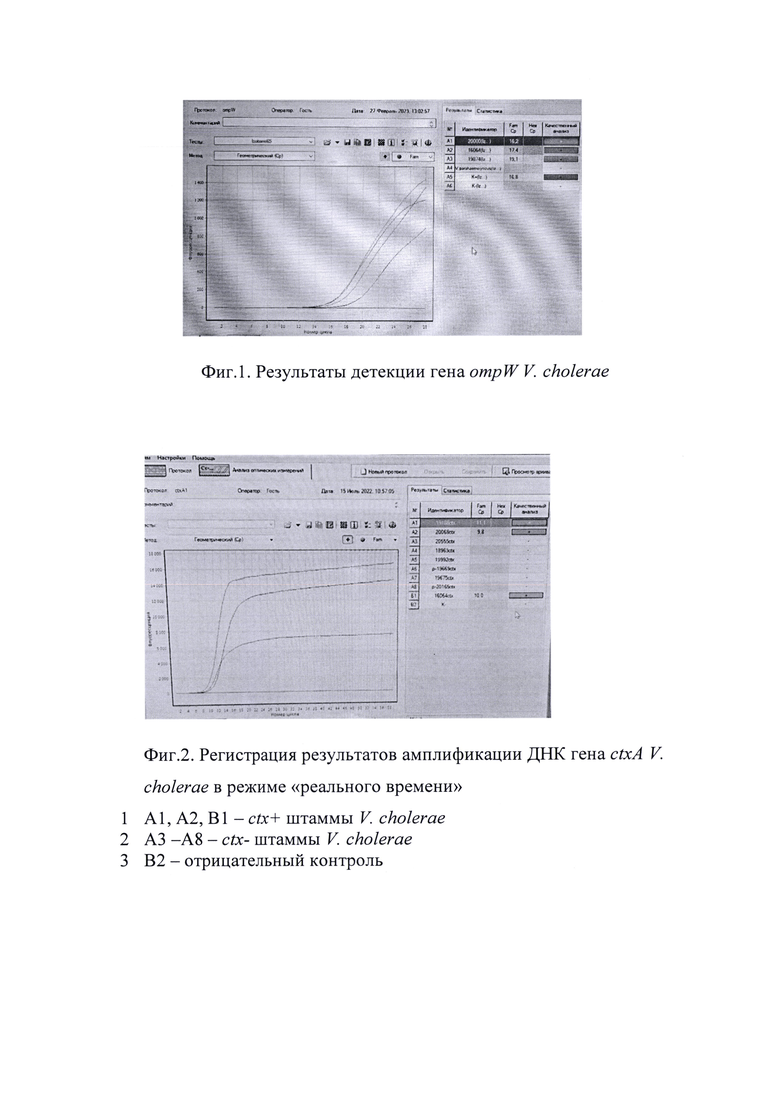
Учет результатов осуществляют с помощью реал-тайм амплификатора ДТ-Lite (ДНК-технология, Россия) с программой: 65°С - 60 сек и снятием сигнала при каждом цикле и длительностью 60 циклов. Результаты учитывают в реальном времени по наличию флуоресцентного сигнала характерного для флуорофора FAM (фиг. 1).
На фигуре 1 результаты детекции гена ompW V. cholerae с помощью предложенных праймеров, где А1-А3-штаммы V. cholerae №№20000, 16064, 19874; А4 - штамм Vibrio parahaemolyticus №19005; А5 - положительный контроль; А6 - отрицательный контроль.
Таким образом - график показывает, что кривые штаммов А1-А3 и А5 имеют флуоресцентный сигнал, что подтверждает их принадлежность к виду V. cholerae.
Пример 2.
Идентификация патогенных штаммов Vibrio cholerae (по наличию фактора вирулентности - гена ctxA)
Штаммы V. cholerae O1 19188 (ctx+), 20068 (ctx+), 20555 (ctx-), 18963 (ctx-), 19992 (ctx-), V. cholerae non O1/non P-19669 (ctx-), 19675 (ctx-), P-20165 (ctx-), V. cholerae O139 16064 (ctx+) с точно установленной видовой принадлежностью и с заведомо известными данными по наличию гена ctxA взяты из коллекции ФКУЗ Ростовского-на-Дону противочумного института Роспотребнадзора. В качестве отрицательного контроля был взят штамм Vibrio vulnificus №15828.
Исследование проводят как в примере 1. Петлевую изотермическую амплификацию проводят с применением праймеров к гену ctx А. Технология и условия проведения реакции сходные. Детектирующее флуоресцентное свечение молекулы флуорофора FAM свидетельствуют о наличии гена ctxA у штаммов V. cholerae (и как следствие о продукции холерного токсина) (фиг. 2), где кривые штаммов А1, А2, В1 - штаммы V. cholerae, содержащие ген холерного токсина; A3- А8 - атоксигенные ctx- штаммы V. cholerae; В2 - отрицательный контроль штамм Vibrio vulnificus №15828.
Использование предлагаемого изобретения позволяет за счет набора новых высокоспецифичных праймеров быстро в течение 30-60 минут, достоверно, и эффективно проводить идентификацию представителей вида V. cholerae, а также определять патогенность штаммов, дифференцируя токсигенные штаммы от нетоксигенных методом изотермической петлевой амплификации.
Кроме того, простота в исполнении анализа, низкая себестоимость, высокая чувствительность 1×104 м.кл./мл и специфичность данных праймеров дает возможность применять их в практике бактериологических лабораторий, центров гигиены и эпидемиологии, лечебно-профилактических, противочумных и других учреждений.
Источники информации:
1. Шувалова Е.П., Белозеров Е.С., Беляева Т.В., Змушко Е.И. Инфекционные болезни. Спб. СпецЛит. 2016: 783 с.
2. Andrew S. Azman, Kara Е. Rudolph, Derek A.T. Cummings, Justin Lessler. The incubation period of cholera: A systematic review. Infection. 2013;. 66 (5): 432-438. doi: 10.1016/j.jinf.2012.11.013.
3. Всемирная организация здравоохранения (16 декабря 2022 г.). Новости о вспышках болезней. Холера - ситуация в мире. См. по адресу: https://www.who.int/ru/emergencies/disease-outbreak-news/item/2022-DON426.
4. Notomi Т., Okayama Н., Masubuchi Н. et al. Loop-mediated isothermal amplification of DNA // Nucleic Acids Res. - 2000. - Vol. 28. - e63-e63. DOI: 10.1093/nar/28.12.e63.
5. МУК 4.2.3745-22. Методы лабораторной диагностики холеры. Методические указания. Москва 2022.
6. Notomi Т., Okayama Н., Masubuchi Н., Yonekawa Т., Watanabe K., Amino N., Hase Т. Loop-mediated isothermal amplification of DNA. Nucleic Acids. Res., 2000, vol. 28, no. 12, e63 (7 p.). DOI: 10.1093/nar/28.12.e63.
7. Srisuk C., Chaivisuthangkura P., Rukpratanporn S., Longyant S., Sridulyakul P., Sithigorngul P. Rapid and sensitive detection of Vibrio cholerae by loop-mediated isothermal amplification targeted to the gene of outer membrane protein ompW. Lett Appl. Microbiol. 2010; 50(1): 36-42. DOI: 10.1111/j.1472-765X.2009.02749.x.
8. Gil F., Ipinza F., Fuente J., Fumeron R., Villarreal J.M., Aspee A. et al. The ompW (porin) gene mediates methyl viologen (paraquat) efflux in Salmonella enterica serovar typhimurium. RES. Microbiol. 2007; 158: 529-36. DOI: 10.1016/j.resmic.2007.05.004.
9. Nandi В., Nandy R.K., Mukhopadhyay S., Nair G.В., Shimada Т., Ghose A.C. et al. Rapid method for species-specific identification of Vibrio cholerae using primers targeted to the gene of outer membrane protein OmpW. J Clin Microbiol. 2000; 38: 4145-51. DOI: 10.1128/JCM.38.11.4145-4151.2000.
10. СанПиН 3.3686-21. Санитарно-эпидемиологические требования по профилактике инфекционных болезней.
11. Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности: методические указания МУ 1.3.2569-09. - М.; 2009.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ
идентификации и определения патогенности штаммов Vibrio cholerae
методом петлевой изотермической амплификации (LAMP)..xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-03-15">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>185</ApplicationNumberText>
<FilingDate>2023-03-15</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>185</ApplicantFileReference>
<ApplicantName languageCode="ru">Ростовский- на –Дону противочумный
институт Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>Rostov-on-Don Plague Control Research Institute
of the Rospotrebnadzor</ApplicantNameLatin>
<InventorName languageCode="ru">Чемисова Ольга
Сергеевна</InventorName>
<InventorNameLatin>Chemisova Olga Sergeevna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ идентификации и определения
патогенности штаммов Vibrio cholerae методом петлевой
изотермической амплификации (LAMP).</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggggacttgctgctaacgttttcgttgaggaaccagctatc</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>53</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..53</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcactcaatccagcattagtaccttttgaattacaccactttctttg
atga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcataccacacagaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtccatatgttggtgcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttggcctttgattatatgctcaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaccattaaagctttcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgctggagcaatatctaagttacttttcaattacatcgtaatagggg
ct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggtttccctccggagcatattttttggagcattcccacaac</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattttggggtgcttgatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgcaagtattactcatcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaatatctatctctgtagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagccgtggattcatcatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления гена холодового шока csh1 у штаммов Vibrio cholerae O1 и неО1/неО139 методом петлевой изотермической амплификации (LAMP) | 2023 |
|
RU2819101C1 |
| Способ оценки внутривидовой конкуренции токсигенных и нетоксигенных штаммов Vibrio cholerae О1 и О139 серогрупп с помощью ПЦР в режиме реального времени | 2023 |
|
RU2806564C1 |
| Способ выявления гена tcpA Classical V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2024 |
|
RU2837089C1 |
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| Способ обнаружения жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor в окружающей среде с помощью бактериофага M3 методом количественной ПЦР | 2023 |
|
RU2808577C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
| Набор реагентов и способ детекции возбудителя малярии Plasmodium falciparum | 2024 |
|
RU2839159C1 |
| СИНТЕТИЧЕСКИЕ ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И СПОСОБ ВЫСОКОЧУВСТВИТЕЛЬНОГО ЭКСПРЕСС-ВЫЯВЛЕНИЯ ДНК ВИРУСА АФРИКАНСКОЙ ЧУМЫ СВИНЕЙ МЕТОДОМ ПЕТЛЕВОЙ ИЗОТЕРМИЧЕСКОЙ АМПЛИФИКАЦИИ В ПРИСУТСТВИИ ДНК ВНУТРЕННЕГО КОНТРОЛЬНОГО ОБРАЗЦА | 2022 |
|
RU2799410C1 |
| Набор реагентов для обнаружения возбудителей клещевых нейроинфекций | 2022 |
|
RU2800261C1 |
| Способ идентификации токсигенных генетических вариантов возбудителя холеры Эль Тор с набором мутаций в генах вирулентости и генах острова пандемичности VSPII методом мультиплексной полимеразной цепной реакции | 2022 |
|
RU2815711C2 |
Изобретение относится к области биотехнологии. Описан способ идентификации и определения патогенности штаммов Vibrio cholerae методом петлевой изотермической амплификации (LAMP). Способ включает выделение ДНК из исследуемого материала, проведение петлевой изометрической амплификации и учет результатов. Он отличается тем, что амплификацию проводят с помощью разработанного набора праймеров к гену ompW и набора праймеров к гену ctxA. Техническим результатом, достигаемым при осуществлении данного изобретения, являются новые высокоспецифичные олигонуклеотидные праймеры для быстрого, специфичного и чувствительного выявления штаммов V. cholerae в пробах чистых культур методом петлевой изотермической амплификации (LAMP). 2 з.п. ф-лы, 2 ил., 2 табл., 2 пр.
1. Способ идентификации и определения патогенности штаммов Vibrio cholerae методом петлевой изотермической амплификации (LAMP), включающий выделение ДНК из исследуемого материала, проведение петлевой изометрической амплификации и учет результатов, отличающийся тем, что амплификацию проводят с помощью набора праймеров к гену ompW и набора праймеров к гену ctxA, следующего состава:
к гену ompW
FIP- ATGGGGACTTGCTGCTAACGTTTTCGTTGAGGAACCAGCTATC
BIP- AATCACTCAATCCAGCATTAGTACCTTTTGAATTACACCACTTTCTTTGATGA
F3-TGGCATACCACACAGAAG
В3-GTCCATATGTTGGTGCGG
LoopF-TTGGCCTTTGATTATATGCTCAAT
LoopB-GTACCATTAAAGCTTTCATC
к гену ctxA
FIP- GCTGCTGGAGCAATATCTAAGTTACTTTTCAATTACATCGTAATAGGGGCT
BIP- AGGTTTCCCTCCGGAGCATATTTTTTGGAGCATTCCCACAAC
F3- CATTTTGGGGTGCTTGATG
B3- TCGCAAGTATTACTCATCGA
LF-GTAATATCTATCTCTGTAGC
LB-GAGCCGTGGATTCATCATGC,
при этом реакцию петлевой изометрической амплификации проводят в два этапа, первоначально к гену ompW, кодирующему белок внешней мембраны холеры, для определения вида Vibrio cholerae, а затем к гену ctxA, кодирующему холерный токсин, для определения вирулентности, учет результатов осуществляют с помощью амплификатора с программой 65°С - 60 сек и снятием сигнала при каждом цикле и длительностью 60 циклов, а детекцию результатов амплификации осуществляют по каналам флуорофора FAM, где относящимся к виду Vibrio cholerae и патогенным считают тот образец, при анализе которого наблюдают рост флуоресцентных сигналов на цветовых каналах амплификатора.
2. Способ по п.1, отличающийся тем, что для проведения LAMP предварительно готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP- праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл, при этом положительный контроль включает в себя все компоненты реакции и препарат ДНК Vibrio cholerae ctx+, a в случае отрицательного контроля в реакционную смесь вместо исследуемого препарата ДНК вносят соответствующее количество деионизованной воды, не содержащей исследуемой ДНК.
3. Способ по п. 1, отличающийся тем, что амплификацию проводят в объеме 25 мкл и реакционная смесь содержит: 10хбуфер для Bst ДНК-полимеразы -2.5 мкл; MgSO4 100 mM - 2 мкл; смесь dNTP - 25 mM - 1,5 мкл; смесь праймеров к гену ompW - 2,5 мкл; Bst ДНК-полимераза - 2 мкл; флуоресцентный краситель EvaGreen 20Х водный раствор - 1,25 мкл; образец ДНК - 2 мкл; вода, свободная от нуклеаз - до 25 мкл.
| СПОСОБ ВЫЯВЛЕНИЯ ТОКСИГЕННЫХ ШТАММОВ 01 VIBRIO CHOLERAE "ПОСТГАИТЯНСКОЙ" ЛИНИИ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2021 |
|
RU2766192C1 |
| CN 105648054 A, 08.06.2016 | |||
| CN 103160606 A, 19.06.2013 | |||
| DE 102015012691 A1, 30.03.2017. | |||
Авторы
Даты
2024-05-29—Публикация
2023-03-27—Подача