УРОВЕНЬ ТЕХНИКИ ДЛЯ ИЗОБРЕТЕНИЯ
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
[0001] Настоящее изобретение относится, главным образом, к модифицированным, неприродным доменам α1-α2 лигандов NKG2D с присоединенными полипептидами, имеющими свойства связывания специфической мишени, например, антителами или фрагментами антител, избирательно доставляемым к химерным рецепторам антигенов (CAR), содержащим модифицированные, неприродные рецепторы NKG2D на модифицированных клетках млекопитающих.
ИНФОРМАЦИЯ ОБ УРОВНЕ ТЕХНИКИ ДЛЯ ИЗОБРЕТЕНИЯ
[0002] Антитело (Ab), фигура 1, также известное как иммуноглобулин (Ig), у многих млекопитающих, включая человека, представляет собой большой, Y-образный белок, используемый иммунной системой для идентификации и нейтрализации чужеродных объектов, таких как бактерии и вирусы (Charles Janeway (2001). Immunobiology. (5th ed.), Chapter 3. Garland Publishing. ISBN 0-8153-3642-X. (полный электронный текст на NCBI Bookshelf). Антитело узнает уникальную часть чужеродной мишени, называемой антигеном. Каждая верхушка двух плеч «Y» антитела содержит антигенсвязывающий участок или паратоп, (структурный аналог замка), который является специфическим для одного конкретного эпитопа (структурного аналога ключа) антигена, позволяя точное связывание этих структур. С использованием этого механизма связывания, антитело может метить микроорганизм или инфицированную клетку для атаки другими частями иммунной системы или может нейтрализовать свою мишень напрямую, например, посредством блокирования части микроорганизма, которая является необходимой для его проникновения и выживаемости. Продукция антител является главной функцией гуморальной или «адаптивной», иммунной системы. Антитела секретируются плазматическими клетками. Антитела в природе могут возникать в двух физических формах, растворимой форме, секретируемой из клетки, и связанной с мембраной форме, присоединенной к поверхности B-клетки через «стебель» Y.
[0003] Антитела представляют собой гликопротеины, принадлежащие к суперсемейству иммуноглобулинов и, как правило, состоят из основных структурных единиц - каждая с двумя большими тяжелыми цепями и двумя малыми легкими цепями. Существует несколько различных типов тяжелых цепей антитела, и несколько различных видов антител, сгруппированных в различные изотипы, на основании того, какой тяжелой цепью они обладают. Пять различных изотипов антител известны у млекопитающих (Market E, Papavasiliou FN (October 2003). «V(D)J recombination and the evolution of the adaptive immune system». PLoS Biol. 1 (1): E16. doi:10.1371/journal.pbio.0000016. PMC 212695. PMID 14551913). Хотя общая структура всех антител является очень сходной, небольшая область на верхушке каждого плеча Y-образного белка является необычайно вариабельной, позволяя существование миллионов антител с немного отличающимися структурами верхушек, или антигенсвязывающих участков. Эта область известна как гипервариабельная или вариабельная область. Каждый из этих природных вариантов может связываться с отличным антигеном. Это необычайное разнообразие антител позволяет иммунной системе адаптироваться и узнавать эквивалентное широкое множество антигенов (Hozumi N, Tonegawa S (1976). «Evidence for somatic rearrangement of immunoglobulin genes coding for variable and constant regions». Proc. Natl. Acad. Sci. U.S.A. 73 (10): 3628-3632. doi:10.1073/pnas.73.10.3628. PMC 431171. PMID 824647.)
[0004] Природная «Y»-образная молекула Ig состоит из четырех полипептидных цепей; двух идентичных тяжелых цепей и двух идентичных легких цепей, соединенных посредством дисульфидных связей. Каждая тяжелая цепь имеет две главных области, константную область (CH) и вариабельную область (VH). Константная область является по существу идентичной во всех антителах одного и того же изотипа, но отличается в антителах различных изотипов. Легкая цепь также имеет два последовательных домена: меньшую константную область (CL) и вариабельную область (VL) (Woof J, Burton D (2004). «Human antibody-Fc receptor interactions illuminated by crystal structures». Nat Rev Immunol 4 (2): 89-99. doi:10.1038/nri1266. PMID 15040582).
[0005] Некоторые части антитела имеют одинаковые функции. Каждое из двух плеч Y, например, содержит участки, которые могут связываться с антигенами и, таким образом, узнавать специфические чужеродные объекты. Эту область антитела называют Fv (фрагментом, вариабельной) областью. Она состоит из одного вариабельного домена из тяжелой цепи (VH) и одной вариабельной области из легкой цепи (VL) антитела (Hochman J, Inbar D, Givol D (1973). An active antibody fragment (Fv) composed of the variable portions of heavy and light chains. Biochemistry 12 (6): 1130-1135. doi:10.1021/bi00730a018. PMID 4569769). Паратоп сформирован на одном конце Fv и представляет собой область для связывания с антигенами. Он состоит из вариабельных петель из β-цепей, трех на каждой VL и VH, и является ответственным за связывание с антигеном. Эти 6 петель обозначены как определяющие комплементарность области (CDR) (North B, Lehmann A, Dunbrack RL (2010). «A new clustering of antibody CDR loop conformations». J Mol Biol 406 (2): 228-256. doi:10.1016/j.jmb.2010.10.030. PMC 3065967. PMID 21035459).
[0006] Полипептиды, которые можно использовать, имеющие специфическую функцию связывания антигена, могут происходить из CDR вариабельных областей антител. Эти два вариабельных домена антитела, один из легкой цепи (VL) и один из тяжелой цепи (VH), каждый с 3 CDR, могут являться слитыми в тандеме, в любом порядке, с использованием одного, короткого линкерного пептида из от 10 до приблизительно 25 аминокислот для получения полипептида линейного одноцепочечного вариабельного фрагмента (scFv), содержащего один из вариабельных доменов каждой из тяжелой и легкой цепи (Bird, R. E., Hardman, K. D., Jacobson, J. W., Johnson, S., Kaufman, B. M., Lee, S. M., Lee, T., Pope, S. H., Riordan, G. S., and Whitlow, M. (1988) Single-chain antigen-binding proteins, Science 242, 423-426; Huston, J. S., Levinson, D, Mudgett-Hunter, M, Tai, M-S, Novotny, J, Margolies, M.N., Ridge, R., Bruccoleri, RE., Haber, E., Crea, R., and Opperman, H. (1988). Protein engineering of antibody binding sites: Recovery of specific activity in an anti-digoxin single-chain Fv analogue produced in Escherichia coli. PNAS 85: 5879-5883).
[0007] Линкер обычно является богатым глицином для гибкости, так же как серином, треонином или заряженными аминокислотами для растворимости, и может соединять либо N-конец VH с C-концом VL, либо наоборот. Этот белок сохраняет специфичность исходного иммуноглобулина, несмотря на удаление константных областей и введение одного линкера. Этот формат позволяет специалисту в области технологии рекомбинантной ДНК генетически сливать линейный scFv с N- или C-концом исходного белка, чтобы придать исходному белку свойства связывания антигена scFv. Существует множество других предложенных или полученных аранжировок поливалентных и тандемных областей scFv, однако, что важно, как описано ниже, все имеют по меньшей мере два пространственно отдаленных конца, (Le Gall, F.; Kipriyanov, SM; Moldenhauer, G; Little, M (1999). «Di-, tri- and tetrameric single chain Fv antibody fragments against human CD19: effect of valency on cell binding». FEBS Letters 453 (1): 164-168. doi:10.1016/S0014-5793(99)00713-9. PMID 10403395).
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0008] Настоящее изобретение относится к модифицированным доменам α1-α2 лигандов NKG2D, присоединенным к гетерологичным полипептидам, в некоторых вариантах осуществления, антителам или фрагментам антител. Модифицированные лиганды избирательно связываются с родственными неприродными рецепторами NKG2D, которые, в свою очередь, связываются избирательно со своими родственными модифицированными лигандами. Неприродные рецепторы NKG2D могут экспрессироваться на поверхностях клеток иммунной системы и образовывать химерный рецептор на поверхности этой эффекторной клетки. Гетерологичная молекула, присоединенная к лиганду, может также связывать специфическую молекулу на поверхности клетки-мишени, таким образом, доставлять иммуноэффекторную клетку к клетке-мишени. Такие эффекторные клетки включают лимфоциты, B-клетки, плазматические клетки, моноциты, макрофаги и дендритные клетки.
[0009] В некоторых вариантах осуществления, настоящее изобретение относится к модифицированному, неприродному лиганду для модифицированного, неприродного рецептора NKG2D, где лиганд имеет присоединенную гетерологичную молекулу, которая избирательно связывает белок HIV, присутствующий на поверхности клетки, инфицированной HIV, где модифицированный лиганд со своей гетерологичной молекулой может избирательно связываться с модифицированным, неприродным рецептором NKG2D CAR-клетки и вызывает разрушение инфицированной HIV клетки.
[0010] В следующих вариантах осуществления, белок HIV, с которым избирательно связывается гетерологичная молекула, представляет собой белок оболочки HIV.
[0011] В следующих вариантах осуществления, эпитоп белка оболочки, с которым гетерологичная молекула избирательно связывается, содержит SEQ ID NO: 169 или SEQ ID NO: 170.
[0012] В некоторых вариантах осуществления настоящего изобретения, модифицированный, неприродный лиганд содержит SEQ ID NO: 68, 69, 70, 71 или 72.
[0013] В следующих вариантах осуществления, модифицированный, неприродный рецептор NKG2D содержит SEQ ID NO: 54 или 154.
[0014] В некоторых вариантах осуществления настоящего изобретения, белок HIV экспрессирован на инфицированной HIV клетке, подвергнутой шоку или активации посредством механизма или средства, или реактивирующего после латентности средства, как известно, провоцирующих экспрессию белка HIV на латентной инфицированной HIV клетке.
[0015] В некоторых вариантах осуществления настоящего изобретения, CAR-клетка имеет множество связанных модифицированных, неприродных лигандов с различными, отдельными гетерологичными молекулами, связывающими различные эпитопы, белки или другие молекулы на поверхности инфицированной HIV клетки.
[0016] В некоторых вариантах осуществления настоящего изобретения, модифицированный, неприродный рецептор NKG2D, содержащий SEQ ID NO: 54 или 154, присутствует на CAR-клетке, где модифицированный рецептор NKG2D связывает модифицированный, неприродный лиганд, состоящий из SEQ ID NO: 68, 69, 70, 71 или 72, к которому присоединены гетерологичные молекула или атом, которые не связывают белок HIV.
[0017] В следующих вариантах осуществления, гетерологичные молекула или атом модулируют функцию CAR-клетки. В следующих вариантах осуществления, клеточная функция включает пролиферацию, дифференцировку, абляцию, визуализацию, антагонизм иммуносупрессии, хоминг или цитолиз клетки, не инфицированной HIV.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[0018] Файл патента или заявки содержит по меньшей мере один чертеж, выполненный в цвете. Копии этой публикации патента или патентной заявки с цветными чертежом(чертежами) могут быть представлены офисом после запроса и оплаты необходимого взноса.
[0019] Фигура 1. Рисунок типичного антитела млекопитающего, показывающий его Y-образную структуру и структурные компоненты.
[0020] Фигура 2. Строение типичного CAR (Gill & June, 2015, в цитируемом документе).
[0021] Фигура 3. Направленный на структуру мутагенез домена α1-α2 MICA для усиления аффинности для NKG2D. Структура домена α1-α2 MICA (PDB 1HYR) с его связывающей NKG2D поверхностью, окрашенной темно-серым, где 57 специфических аминокислотных участков подвергали обширному мутагенезу.
[0022] Фигура 4. Остатки тирозина Y152 и Y199 внутри природного гомодимера NKG2D.
[0023] Фигура 5. Выравнивание белковой последовательности доменов α1-α2 из MICA и ULBP 1-6. Аминокислоты, выделенные серым, выбраны для мутагенеза NNK в ULBP2 (60 аминокислот) и ULBP3 (36 аминокислот). Остатки, выделенные черным, идентифицированы как ключевые положения для выбора и идентифицированы как мутации, модулирующие аффинность связывания с NKG2D (таблицы 6 и 7).
[0024] Фигура 6. Результаты ELISA для слитых с антителом R3 белков с неприродными доменами α1-α2, отобранными по связыванию с Y152A NKG2D-Fc. (A) слитый с антителом R3 HC25 белок не является избирательным для Y152A NKG2D. (B) слитый с антителом R3 HC25.17 (SEQ ID NO.: 73) белок является избирательным для Y152A NKG2D, более, чем для природного NKG2D-Fc. (C) слитый с антителом R3 HC.U2RW белок не является избирательным для Y152A NKG2D более, чем для природного NKG2D-Fc. (D) слитый с антителом R3 HC.U2S3 (SEQ ID NO.: 74) белок является избирательным для Y152A NKG2D более, чем для природного NKG2D-Fc.
[0025] Фигура 7. Оценка соотношений клеток эффектор:мишень (E:T) для уничтожения инфицированных HIV первичных CD4 T-клеток посредством CAR-T-клеток с использованием различных концентраций специфических нацеленных на HIV MicAbodies. Один миллион первичных происходящих из миндалин клеток, инфицированных вирусом Bal-GFP R5 (~10% инфекция; 1X104 инфицированных клеток), инкубировали с 1X105 нетрансдуцированных CD8 (0:1) или с 1X104 (1:1) или 2X105 (20:1) CAR-T-клеток в присутствии различных концентраций четырех различных MicAbodies с широким спектром нейтрализации HIV. Клетки окрашивали через 24 час и оценивали посредством проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- клеткам, либо экспрессирующим, либо не экспрессирующим GFP. Показаны результаты, усредненные для 3 исследований.
[0026] Фигура 8. Специфическое уничтожение инфицированных вирусом R5 первичных CD4 клеток посредством CAR-T в комбинации со специфическим для HIV MicAbody. Один миллион первичных происходящих из миндалин клеток, инфицированных вирусом Bal-GFP R5 (~1X104 инфицированных клеток), инкубировали с 1X105 CAR-T-клеток в присутствии различных концентраций специфических для HIV MicAbodies или специфического для B-клетки нацеливающего на CD20 MicAbody, или нацеливающего на HER2 MicAbody (Her2). Клетки окрашивали через 24 час и анализировали посредством проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- и либо GFP+, либо GFP-. Показаны результаты, усредненные для 4 исследований.
[0027] Фигура 9. Специфическое уничтожение инфицированных перенесенным вирусом/вирусом-основателем F4 первичных CD4 клеток посредством CAR-T в комбинации со специфическим для HIV MicAbody. Один миллион первичных происходящих из миндалин клеток, инфицированных вирусом F4-GFP (T/F) (~1X104 инфицированных клеток) инкубировали с использованием 1X105 конвертируемый CAR-T-клеток в присутствии различных концентраций 4 различных специфических для HIV MicAbodies, нацеливающего на CD20 MicAbody (Ritux) или нацеливающего на HER2 MicAbody (Her2). Клетки окрашивали через 24 час и продолжали с использованием проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- и либо GFP+, либо GFP-.
[0028] Фигура 10. Уничтожение посредством CAR-T и MicAbody реактивированных латентно инфицированных клеток-резервуаров от пациентов с авиремией, хронически инфицированных HIV и подвергаемых ART. CD4+ T-клетки выделяли посредством бесконтактного отрицательного отбора из PBMC, собранных от известных инфицированных HIV пациентов, подвергаемых ART, и реактивировали в течение 72 час с использованием 100 нМ форбол-миристат-ацетата (PMA)+1 мкМ иономицина. Затем клетки промывали дважды и инкубировали в течение 48 часов с конвертируемый CAR-T-клетками или нетрансдуцированными CD8 T-клетками в присутствии 0,1 или 1 нМ смеси равных концентраций MicAbodies на основе bNAb HIV (3BNC60, 3BNC117, PGT121 и 10-1074), обозначенной MIX. Затем клетки центрифугировали, и РНК выделяли из осадков клеток. Ассоциированную с клетками РНК HIV измеряли посредством цкПЦР.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0029] Клетки естественные киллеры (NK), клетки линий моноцитов-макрофагов, и конкретные (CD8+ αβ и γδ) T-клетки иммунной системы играют важные роли у человека и других млекопитающих в качестве врожденной защиты первой линии против неопластических и инфицированных вирусом клеток (Cerwenka, A., and L.L. Lanier. 2001. NK cells, viruses and cancer. Nat. Rev. Immunol. 1:41-49). Клетки NK и конкретные T-клетки экспонируют на своих поверхностях NKG2D, выступающий, гомодимерный, поверхностный иммунорецептор, ответственный за узнавание клетки-мишени и активацию врожденной защиты против патологической клетки (Lanier, LL, 1998. NK cell receptors. Ann. Rev. Immunol. 16: 359-393; Houchins JP et al. 1991. DNA sequence analysis of NKG2, a family of related cDNA clones encoding type II integral membrane proteins on human NK cells. J. Exp. Med. 173: 1017-1020; Bauer, S et al., 1999. Activation of NK cells and T cells by NKG2D, a receptor for stress-inducible MICA. Science 285: 727-730). Молекула NKG2D человека имеет подобный лектину C-типа внеклеточный домен, который связывается со своими родственными лигандами, на 84% идентичными или гомологичными по последовательности мономерным MICA и MICB, полиморфным аналогам родственных цепям класса I главного комплекса гистосовместимости (MHC) гликопротеинов (MIC) (Weis et al. 1998. The C-type lectin superfamily of the immune system. Immunol. Rev. 163: 19-34; Bahram et al. 1994. A second lineage of mammalian MHC class I genes. PNAS 91:6259-6263; Bahram et al. 1996a. Nucleotide sequence of the human MHC class I MICA gene. Immunogenetics 44: 80-81; Bahram and Spies TA. 1996. Nucleotide sequence of human MHC class I MICB cDNA. Immunogenetics 43: 230-233). Непатологическая экспрессия белков MIC, как правило, ограничена эпителием кишечника, кератиноцитами, эндотелиальными клетками и моноцитами, однако, измененная поверхностная экспрессия этих белков MIC возникает в ответ на множество типов клеточного стресса, таких как пролиферация, окисление, вирусная инфекция и тепловой шок, и помечает клетку как патологическую (Groh et al. 1996. Cell stress-regulated human MHC class I gene expressed in GI epithelium. PNAS 93: 12445-12450; Groh et al. 1998. Recognition of stress-induced MHC molecules by intestinal γδT cells. Science 279: 1737-1740; Zwirner et al. 1999. Differential expression of MICA by endothelial cells, fibroblasts, keratinocytes and monocytes. Human Immunol. 60: 323-330). Патологическая экспрессия белков MIC также, по-видимому, вовлечена в некоторые аутоиммунные заболевания (Ravetch, JV and Lanier LL. 2000. Immune Inhibitory Receptors. Science 290: 84-89; Burgess, SJ. 2008. Immunol. Res. 40: 18-34). Дифференциальная регуляция лигандов NKG2D, таких как полиморфные MICA и MICB, является важной для предоставления иммунной системе средств для идентификации и ответа на широкий диапазон признаков критической ситуации, в то же время все еще с защитой здоровых клеток от нежелательной атаки (Stephens HA, (2001) MICA and MICB genes: can the enigma of their polymorphism be resolved? Trends Immunol. 22: 378-85; Spies, T. 2008. Regulation of NKG2D ligands: a purposeful but delicate affair. Nature Immunol. 9: 1013-1015).
[0030] Вирусная инфекция является обычным индуктором экспрессии белка MIC и идентифицирует инфицированную вирусом клетку для атаки NK или T-клетки (Groh et al. 1998; Groh et al. 2001. Co-stimulation of CD8+ αβT-cells by NKG2D via engagement by MIC induced on virus-infected cells. Nat. Immunol. 2: 255-260; Cerwenka, A., and L.L. Lanier. 2001). Фактически, чтобы избегать такой атаки на его клетку-хозяина, цитомегаловирус и другие вирусы имеют эволюционировавшие механизмы, предотвращающие экспрессию белков MIC на поверхности клетки, которую они инфицируют, чтобы избежать возбуждения врожденной иммунной системы (Lodoen, M., K. Ogasawara, J.A. Hamerman, H. Arase, J.P. Houchins, E.S. Mocarski, and L.L. Lanier. 2003. NKG2D-mediated NK cell protection against cytomegalovirus is impaired by gp40 modulation of RAE-1 molecules. J. Exp. Med. 197:1245-1253; Stern-Ginossar et al., (2007) Host immune system gene targeting by viral miRNA. Science 317: 376-381; Stern-Ginossar et al., (2008) Human microRNAs regulate stress-induced immune responses mediated by the receptor NKG2D. Nature Immunology 9: 1065-73; Slavuljica, I A Busche, M Babic, M Mitrovic, I Gašparovic, Đ Cekinovic, E Markova Car, EP Pugel, A Cikovic, VJ Lisnic, WJ Britt, U Koszinowski, M Messerle, A Krmpotic and S Jonjic. 2010. Recombinant mouse cytomegalovirus expressing a ligand for the NKG2D receptor is attenuated and has improved vaccine properties. J. Clin. Invest. 120: 4532-4545).
[0031] Несмотря на их стресс, множество злокачественных клеток, таких как клетки злокачественной опухоли легкого и злокачественной опухоли мозга глиобластомы, также избегают экспрессии белков MIC и в результате, могут являться особенно агрессивными, поскольку они тоже избегают врожденной иммунной системы (Busche, A et al. 2006, NK cell mediated rejection of experimental human lung cancer by genetic over expression of MHC class I chain-related gene A. Human Gene Therapy 17: 135-146; Doubrovina, ES, MM Doubrovin, E Vider, RB Sisson, RJ O’Reilly, B Dupont, and YM Vyas, 2003. Evasion from NK Cell Immunity by MHC Class I Chain-Related Molecules Expressing Colon Adenocarcinoma (2003) J. Immunology 6891-99; Friese, M. et al. 2003. MICA/NKG2D-mediated immunogene therapy of experimental gliomas. Cancer Research 63: 8996-9006; Fuertes, MB, MV Girart, LL Molinero, CI Domaica, LE Rossi, MM Barrio, J Mordoh, GA Rabinovich and NW Zwirner. (2008) Intracellular Retention of the NKG2D Ligand MHC Class I Chain-Related Gene A in Human Melanomas Confers Immune Privilege and Prevents NK Cell-Mediated Cytotoxicity. J. Immunology, 180: 4606 -4614).
[0032] Структура с высоким разрешением MICA человека, связанного с NKG2D, разрешена и показывает, что домен α3 MICA не имеет прямого взаимодействия с NKG2D (Li et al. 2001. Complex structure of the activating immunoreceptor NKG2D and its MHC class I-like ligand MICA. Nature Immunol. 2: 443-451; код доступа 1HYR в банке данных белков). Домен α3 MICA, подобно домену MICB, соединен с платформенным доменом α1-α2 посредством короткого, гибкого линкерного пептида, и сам расположен естественным образом как «спейсер» между платформой и поверхностью экспрессирующей MIC клетки. 3-мерные структуры доменов α3 MICA и MICB человека являются почти идентичными (среднеквадратичное расстояние <1 Å на 94 C-αα) и функционально взаимозаменяемыми (Holmes et al. 2001. Structural Studies of Allelic Diversity of the MHC Class I Homolog MICB, a Stress-Inducible Ligand for the Activating Immunoreceptor NKG2D. J Immunol. 169: 1395-1400).
[0033] Описаны конкретные неприродные домены α1-α2 лигандов NKG2D, модифицированные для связывания природных рецепторов NKG2D человека с более высокой аффинностью, чем природные домены α1-α2 (Candice S. E. Lengyel, Lindsey J. Willis, Patrick Mann, David Baker, Tanja Kortemme, Roland K. Strong and Benjamin J. McFarland. Mutations Designed to Destabilize the Receptor-Bound Conformation Increase MICA-NKG2D Association Rate and Affinity. Journal of Biological Chemistry Vol. 282, no. 42, pp. 30658-30666, 2007; Samuel H. Henager, Melissa A. Hale, Nicholas J. Maurice, Erin C. Dunnington, Carter J. Swanson, Megan J. Peterson, Joseph J. Ban, David J. Culpepper, Luke D. Davies, Lisa K. Sanders, and Benjamin J. McFarland. Combining different design strategies for rational affinity maturation of the MICA-NKG2D interface. Protein Science 2012 VOL 21:1396-1402. В настоящем описании авторы настоящего изобретения описывают неприродные домены α1-α2 лигандов NKG2D, модифицированные для связывания неприродных рецепторов NKG2D, самих подвергнутых мутациям в участках, которые впоследствии приводят к нарушению или потере связывания с природными доменами α1-α2 лигандов NKG2D (David J. Culpepper, Michael K. Maddox, Andrew B. Caldwell, and Benjamin J. McFarland. Systematic mutation and thermodynamic analysis of central tyrosine pairs in polyspecific NKG2D receptor interactions. Mol Immunol. 2011 January; 48(4): 516-523; Патентная заявка USPTO 14/562534; Предварительная патентная заявка USPTO 62/088456)). По настоящему изобретению получают биспецифические молекулы, состоящие из специфически модифицированных неприродных доменов α1-α2 и специфических нацеливающих гетерологичных молекул, включая, но без ограничения, гетерологичные пептиды или полипептиды, которые связывают химерные рецепторы антигенов (CAR), где рецептор CAR состоит из эктодомена неприродного рецептора NKG2D, который связывает модифицированные домены α1-α2 с большей аффинностью, чем он связывает природные домены α1-α2. Генетические модифицированные клетки иммунной системы, например, B-клетки, T-клетки, NK-клетки и макрофаги, содержащие такие CAR, могут затем преодолевать многие недостатки, включая известные виды тяжелой системной токсичности и ускользание антигена, современных терапевтических средств на основе CAR-T- и CAR-NK-клеток, как описано ниже (Kalos M, Levine, BL, Porter, DL, Katz, S, Grupp, SA, Bagg, A and June, C.. T Cells with chimeric antigen receptors have potent antitumor effects and can establish memory in patients with advanced leukemia. Sci Transl Med 2011;3:95ra73; Morgan RA, Yang JC, Kitano M, Dudley ME, Laurencot CM, Rosenberg SA. Case report of a serious adverse event following the administration of T cells transduced with a chimeric antigen receptor recognizing ERBB2. Mol Ther 2010, 18:843-851; Gill and June 2015).
[0034] T-клетки, NK-клетки и макрофаги можно модифицировать с использованием технологий переноса генов для прямой и стабильной экспрессии на их поверхности связывающих доменов антитела, придающих новые антигенные специфичности (Saar Gill & Carl H. June. Going viral: chimeric antigen receptor T-cell therapy for hematological malignancies. Immunological Reviews 2015. Vol. 263: 68-89; Wolfgang Glienke, Ruth Esser, Christoph Priesner, Julia D. Suerth, Axel Schambach, Winfried S. Wels, Manuel Grez, Stephan Kloess, Lubomir Arseniev and Ulrike Koehl. 2015. Advantages and applications of CAR-expressing natural killer cells. Front. Pharmacol. doi: 10.3389/fphar.2015.00021). CAR-T-клетки представляют собой применения этого способа, который комбинирует домен узнавания антигена специфического антитела с внутриклеточным доменом цепи CD3-ζ, который является первичным передатчиком сигналов от эндогенных T-клеточных рецепторов (TCR), в один химерный белок вместе с костимулирующей молекулой, такой как CD27, CD28, ICOS, 4-1BB или OX40, фигура 2. Таким образом сконструированные CAR могут запускать активацию T-клетки при связывании целевого антигена, способом, сходным с эндогенным T-клеточным рецептором, но не зависимым от главного комплекса гистосовместимости (MHC).
[0035] В рамках изобретения, «растворимый белок MIC», «растворимый MICA» и «растворимый MICB» относятся к белку MIC, содержащему домен α1-α2 в присутствии или в отсутствие доменов α3 белка MIC, но без мотива прикрепления к мембране, трансмембранных или внутриклеточных доменов. Лиганды NKG2D, ULBP1-6, естественным образом не имеют домена α3 (Cerwenka A, Lanier LL. 2004. NKG2D ligands: unconventional MHC class I-like molecules exploited by viruses and cancer. Tissue Antigens 61 (5): 335-43. doi:10.1034/j.1399-0039.2003.00070.x. PMID 12753652). «Домен α1-α2» лиганда NKG2D относится к белковому домену лиганда, который связывает рецептор NKG2D.
[0036] В некоторых вариантах осуществления, домены α1-α2 белков неприродного лиганда NKG2D по изобретению являются по меньшей мере на 80% идентичными или гомологичными нативному или природному домену α1-α2 лиганда NKG2D, SEQ ID NO: 1-19. В других вариантах осуществления, модифицированный домен α1-α2 является на 85% идентичным нативному или природному домену α1-α2 лиганда NKG2D. В других вариантах осуществления, модифицированный домен α1-α2 является на 90% идентичным нативному или природному домену α1-α2 природного белка лиганда NKG2D и связывает неприродный NKG2D.
[0037] Платформенный домен α1-α2 растворимого белка MIC является диффундируемым в межклеточном или внутрисосудистом пространстве у млекопитающего. Предпочтительно, платформенные домены α1-α2 неприродных белков MIC по изобретению являются по меньшей мере на 80% идентичными или гомологичными нативному или природному домену α1-α2 белка MICA или MICB человека и связывают природный NKG2D или, в конкретных примерах, связывают модифицированные, неприродные рецепторы NKG2D. В некоторых вариантах осуществления, платформенный домен α1-α2 является на 85% идентичным нативному или природному платформенному домену α1-α2 белка MICA человека, MICB человека или ULBP1-6 человека и связывает природный NKG2D или модифицированный, неприродный NKG2D. В других вариантах осуществления, платформенный домен α1-α2 является на 90%, 95%, 96%, 97%, 98% или 99% идентичным нативному или природному платформенному домену α1-α2 белка MICA человека, MICB человека или ULBP1-6 человека и связывает природный рецептор NKG2D или модифицированный, неприродный рецептор NKG2D.
[0038] В некоторых вариантах осуществления, гетерологичную пептидную метку можно сливать с N-концом или C-концом домена α1-α2 или другого растворимого белка MIC, чтобы способствовать очистке растворимого белка MIC. Последовательности метки включают такие пептиды, как полигистидин, myc-пептид или метка FLAG. Такие метки можно удалять после выделения молекулы MIC способами, известными специалисту в данной области.
[0039] В других вариантах осуществления изобретения, специфические мутации в доменах α1-α2 лигандов NKG2D можно осуществлять для получения неприродных доменов α1-α2, которые связывают неприродные рецепторы NKG2D, сами сконструированные таким образом, чтобы иметь уменьшенную аффинность для природных лигандов NKG2D. Это можно осуществлять, например, посредством генной инженерии. Неприродный рецептор NKG2D, модифицированный таким образом, можно использовать для получения на поверхности NK-клеток, T-клеток, макрофагов или стволовых клеток иммунной системы химерного рецептора антигена (CAR) на основе неприродного NKG2D, который может предпочтительно связываться и подвергаться активации молекулами, состоящими из изобретательских неприродных доменов α1-α2. Эти пары неприродных рецепторов NKG2D и их изобретательских родственных неприродных лигандов NKG2D могут обеспечивать важные преимущества безопасности, эффективности и изготовления для лечения злокачественной опухоли и вирусных инфекций, по сравнению с современными CAR-T-клетками и CAR-клетками NK, как описано ниже.
[0040] Модифицированные T-клетки с CAR возникли в качестве многообещающего способа адоптивной T-клеточной терапии злокачественной опухоли, и CAR, нацеливающие на множество различных молекул, тестировали в CAR-T-клетках а качестве терапевтических средств против злокачественных новообразований (Porter DL, Levine BL, Kalos M, Bagg A, June CH. Chimeric antigen receptor-modified T cells in chronic lymphoid leukemia. N Engl J Med. 365:725-733.). В то время как заметную клиническую эффективность наблюдали у сотен пациентов, подвергаемых адоптивному переносу T-клеток, экспрессирующих специфические для CD19 химерные рецепторы антигенов, способы конструирования на заказ CAR для нацеливания на специфический антиген, выделения аутологичных T-клеток от пациента, генетической модификации аутологичных T-клеток для экспрессии индивидуализированного CAR, размножения модифицированных клеток in vitro и контроля качества их получения все были трудоемкими и дорогостоящими. В настоящее время это является практически осуществимым только в контексте больших академических центров с большим опытом и ресурсами (Gill & June, 2015).
[0041] Как только аутологичные CAR-T-клетки вводят посредством инфузии обратно пациенту-донору, их размножение in vivo невозможно контролировать---«терапия живыми клетками», и не существует взаимосвязи зависимости ответа от дозы с эффективностью (Gill & June, 2015). Кроме того, ускользание от CAR-T-клетки может происходить посредством ускользания из-за потери антигена (Stephan A. Grupp, M.D., Ph.D., Michael Kalos, Ph.D., David Barrett, M.D., Ph.D., Richard Aplenc, M.D., Ph.D., David L. Porter, M.D., Susan R. Rheingold, M.D., David T. Teachey, M.D., Anne Chew, Ph.D., Bernd Hauck, Ph.D., J. Fraser Wright, Ph.D., Michael C. Milone, M.D., Ph.D., Bruce L. Levine, Ph.D., and Carl H. June, M.D. Chimeric Antigen Receptor-Modified T Cells for Acute Lymphoid Leukemia. N Engl J Med 2013;368:1509-1518), и этот путь ускользания можно наиболее быстро подвергать нацеливанию посредством последовательной терапии с использованием дифференциально нацеленной CAR-T-клетки или посредством первоначальной инфузии продукта T-клетки, содержащего несколько CAR двух или более специфичностей, что дополнительно усложняет производственные процессы и контроль качества.
[0042] В дополнение к CAR-T-клеткам, нацеливающим на опухоли с использованием одноцепочечных связывающих доменов антитела (scFv), CAR-T-клетки с использованием связывающего лиганд домена рецептора NKG2D исследовали у животных и недавно у человека (Sentman CL, Meehan KR. NKG2D CARs as cell therapy for cancer. Cancer J. 2014 Mar-Apr;20(2):156-9. doi: 10.1097/PPO.0000000000000029; Manfred Lehner, Gabriel Götz, Julia Proff, Niels Schaft, Jan Dörrie, Florian Full, Armin Ensser, Yves A. Muller, Adelheid Cerwenka, Hinrich Abken, Ornella Parolini, Peter F. Ambros, Heinrich Kovar, Wolfgang Holter. Redirecting T Cells to Ewing's Sarcoma Family of Tumors by a Chimeric NKG2D Receptor Expressed by Lentiviral Transduction or mRNA Transfection Research Article | published 15 Feb 2012 | PLOS ONE 10.1371/journal.pone.0031210; www.clinicaltrials.gov NCT02203825). Поскольку экспрессия лиганда NKG2D увеличена на поверхности подвергнутых стрессу клеток, таких как клетки опухолей и инфицированные вирусом клетки, это семейство природных лигандов NKG2D представляет значительный интерес в качестве мишеней для видов иммунотерапии против вирусных инфекций и злокачественных опухолей (Spear P, Wu MR, Sentman ML, Sentman CL. NKG2D ligands as therapeutic targets. Cancer Immun. 2013 May 1;13:8.; Song DG, Ye Q, Santoro S, Fang C, Best A, Powell DJ Jr., Chimeric NKG2D CAR-expressing T cell-mediated attack of human ovarian cancer is enhanced by histone deacetylase inhibition. Hum Gene Ther. 2013 Mar;24(3):295-305). Один NKG2D-CAR представлял собой слитый белок из полноразмерного рецептора NKG2D и CD3ζ (NKG2Dζ); другой представлял собой только эктодомен NKG2D, слитый в обратной ориентации с каркасом CAR второго поколения, состоящим из трансмембранного и внутриклеточного доменов из CD28 и передающего сигналы домена из CD3ζ (NKG2D28ζ). Поскольку активация NKG2D зависит от присутствия DAP10, сконструировали также CAR-T-клетку, где DAP10 совместно экспрессировался с NKG2Dζ (NKG2Dζ10). T-клетки, экспрессирующие любой из вышеуказанных NKG2D-CAR, продуцировали IFNɣ и TNFα в ответ на стимуляцию лигандом NKG2D, и in vitro эффективно уничтожали опухоли-мишени, экспрессирующие лиганды NKG2D (Heather VanSeggelen, Joanne A. Hammill, Anna Dvorkin-Gheva, Daniela G.M. Tantalo, Jacek M. Kwiecien, Galina F. Denisova, Brian Rabinovich, Yonghong Wan, Jonathan L. Bramson, T cells engineered with chimeric antigen receptors targeting NKG2D ligands display lethal toxicity in mice, Molecular Therapy accepted article preview online 30 June 2015; doi:10.1038/mt.2015.119). Цитотоксический потенциал клеток NK против широкого спектра подтипов опухолей также можно было заметно увеличивать посредством экспрессии CAR на основе NKG2D-DAP10-CD3ζ (Yu-Hsiang Chang, John Connolly, Noriko Shimasaki, Kousaku Mimura, Koji Kono, and Dario Campana. Chimeric Receptor with NKG2D Specificity Enhances Natural Killer Cell Activation and Killing of Tumor Cells. Cancer Res; 73(6) March 15, 2013).
[0043] Однако после инфузии сингенным мышиным хозяевам возникала значительная токсичность при использовании этих конструкций CAR-T, которые связываются и подвергаются активации посредством природных лигандов природного рецептора NKG2D. Признаки токсичности, включая плохое физическое состояние, сгорбленную позу, затрудненное дыхание и уменьшенную центральную температуру тела, наблюдали у несущих опухоль и свободных от опухоли мышей, подвергнутых лечению основанный на NKG2D-CAR-T-клетками по сравнению с необработанными контрольными мышами. Тяжесть токсичности NKG2D-CAR-T-клеток менялась, где NKG2Dζ10 являлся тяжело токсичным, для NKG2D28ζ показана промежуточная токсичность, и NKG2Dζ являлся переносимым. Клинические симптомы токсичности и частоты смертности обострялись, когда мышей подвергали химиотерапии до адоптивного переноса T-клеток, экспрессирующих любой из NKG2D-CAR (VanSeggelen et al. 2015). Известно, что химиотерапия и радиоактивное излучение индуцируют лиганды NKG2D на здоровых в ином отношении тканях (Xiulong Xu, Geetha S Rao, Veronika Groh, Thomas Spies, Paolo Gattuso, Howard L Kaufman, Janet Plate and Richard A Prinz. Major histocompatibility complex class I-related chain A/B (MICA/B) expression in tumor tissue and serum of pancreatic cancer: Role of uric acid accumulation in gemcitabine-induced MICA/B expression. BMC Cancer 2011, 11:194 doi:10.1186/1471-2407-11-194; Gannagé M, Buzyn A, Bogiatzi SI, Lambert M, Soumelis V, Dal Cortivo L, Cavazzana-Calvo M, Brousse N, Caillat-Zucman Induction of NKG2D ligands by gamma radiation and tumor necrosis factor-alpha may participate in the tissue damage during acute graft-versus-host disease. Transplantation. 2008 Mar 27;85(6):911-5. doi: 10.1097/TP.0b013e31816691ef.). Дополнительная характеризация выявила, что токсичность соответствовала системному цитокиновому шторму и летальным уровням воспаления в легких. Эти данные предупреждают, что нужно соблюдать необычайную осторожность при использовании природных лигандов NKG2D для целевой иммунотерапии, и показывают, что усиление экспрессии T-клетками сильно активирующих CAR может являться неблагоприятным in vivo (VanSeggelen et al. 2015).
[0044] CAR-T-клетки, CAR-клетки NK и макрофаги, содержащие эктодомены неприродных рецепторов NKG2D, которые не связывают или только слабо связывают природные лиганды NKG2D, не подвергаются вышеуказанной форме активации и таким образом, не могут являться настолько токсикогенными, как клетка, экспрессирующая CAR на основе природного рецептора NKG2D. Кроме того, эктодомены неприродных рецепторов NKG2D на клетках не подвергаются понижающей регуляции посредством природных лигандов NKG2D в растворимом формате или на супрессорных клетках миелоидного происхождения (MDSC) (Deng W, Gowen BG, Zhang L, Wang L, Lau S, Iannello A, Xu J, Rovis TL, Xiong N, Raulet DH, 2015. Antitumor immunity. A shed NKG2D ligand that promotes natural killer cell activation and tumor rejection. Science. 2015 Apr 3;348(6230):136-9. doi: 10.1126/science.1258867. Epub 2015 Mar 5). Однако, когда такие CAR-клетки, несущие эктодомены неприродных рецепторов NKG2D, привлекаются посредством биспецифических молекул с родственными неприродными доменами α1-α2 по настоящему изобретению и его гетерологичного нацеливающего мотива, который нашел и связал предназначенную для него мишень, CAR активируется, и эффекторные функции CAR-клетки проявляются. Эффекторные функции CAR-T-клетки, CAR-клетки NK и CAR-клетки макрофага могут прекращать или нарушать жизнеспособность или функцию целевой клетки. Целевая клетка может включать злокачественную клетку, иммуносупрессивную клетку опухоли, клетку, вносящую вклад в аутоиммунное заболевание, клетку, инфицированную вирусом, например, но без ограничения, HIV, вирусом гепатита, HTLV-1, CMV, EBV и другими вирусами герпеса.
[0045] Поскольку CAR-T-клетки или CAR-клетки NK, содержащие эктодомены неприродного рецептора NKG2D, не активируются, за исключением присутствия привлеченной биспецифической молекулы, содержащей родственные неприродные домены α1-α2, их активацию можно контролировать посредством введения биспецифических молекул, которые, в качестве биофармацевтических средств, могут проявлять фармакокинетику и фармакодинамику, хорошо известные в данной области. В случае, когда развивается неблагоприятное событие, терапевт может просто модифицировать режим дозирования вводимой биспецифической молекулы, вместо того, чтобы быть вынужденным разворачивать индуцированный механизм самоубийства для разрушения введенных посредством инфузии CAR-клеток, как делают в настоящее время (Monica Casucci and Attilio Bondanza. Suicide Gene Therapy to Increase the Safety of Chimeric Antigen Receptor-Redirected T Lymphocytes. J Cancer. 2011; 2: 378-382). Кроме того, такие биспецифические молекулы с различными специфическими нацеливающими мотивами можно вводить одновременно или последовательно, чтобы способствовать нацеливанию на возникновение устойчивости и ускользания клетки опухоли или инфицированной вирусом клетки в результате утраты антигена-мишени без необходимости получения, размножения и инфузии множества различных аутологичных CAR-клеток (Gill & June, 2015). Поскольку все конструкции CAR могут являться идентичными для всех CAR-клеток, и специфичность нацеливания определяется просто посредством нацеливающего мотива полученной биспецифической молекулы по настоящему изобретению, производственные процессы могут являться упрощенными и менее дорогими.
[0046] В процессе эволюции образовалось множество вирусов с механизмами для избегания уничтожения их клетки-хозяина посредством природной системы иммунного надзора, особенно зависимыми от NKG2D компонентами. Например, аденовирус, цитомегаловирус (CMV), вирусы герпеса, HIV, вирус-1 T-клеточной лимфомы человека (HTLV-1) и вирусы папилломы все имеют один или несколько механизмов. Такие вирусы могут экспрессировать на поверхности инфицированных ими клеток-хозяев вирусные антигены, эпитопы которых могут служить в качестве специфических для вируса молекулярных мишеней для связывания антителами, фрагментами антител или другими молекулярными нацеливающими мотивами. Эти экспонированные на клеточной поверхности молекулярные мишени являются привлекательными в качестве мишеней для антител или видов адоптивной клеточной терапии (ACT) для предотвращения распространения вирусной инфекции или лечения вирусной инфекции посредством уничтожения инфицированных вирусом клеток.
[0047] Латентность HIV-1 образуется на ранних стадиях в ходе острой инфекции и первоначально обнаружена в CD4+ T-клетках памяти. Этот резервуар, хоть и является почти транскрипционно молчащим, является полностью способным к образованию инфекционного вируса, когда клетку-хозяина реактивируют посредством стимуляции антигеном или цитокином, или когда прерывают антиретровирусную терапию (ART). Резервуар латентного HIV в первую очередь обнаружен в лимфоидных тканях, где находится >98% CD4+ T-клеток. Несмотря на то, что ART является способной супрессировать вирусную репликацию, она не способна уничтожать латентные резервуары (Ruelas, D.S. and W.C. Greene, An integrated overview of HIV-1 latency. Cell, 2013. 155(3): p. 519-29.). Попытки очистки от латентного HIV-1 были первоначально сфокусированы на реактивации латентных провирусов с использованием цитокинов или активирующих T-клеточный рецептор средств. Однако, эти способы приводили к тяжелым побочным эффектам и имели низкую эффективность. Так называемый способ «шока и уничтожения», вместо этого, включает реактивацию транскрипционно молчащих провирусов посредством введения обращающих латентность средств (LRA), представляющих собой химические соединения, способные индуцировать транскрипцию HIV-1 (Cary, D.C., K. Fujinaga, and B.M. Peterlin, Molecular mechanisms of HIV latency. J Clin Invest, 2016. 126(2): p. 448-54.). После реактивации латентных вирусов, гликопротеин оболочки HIV, gp160, экспрессируется на поверхности активированной клетки, процессированным до gp120 и gp41. Домены V1, V2, V3, C1, C2 и N-сегмент gp120 предоставляют привлекательные мишени для атаки на инфицированные HIV клетки с использованием нейтрализующих антител и, как описано по настоящему изобретению, CAR-T-клеток.
[0048] После реактивации латентно инфицированных клеток, прогноз состоял в том, что эти клетки могут продуцировать вирусы (что можно остановить посредством проведения ART), и что эти клетки могут умирать посредством апоптоза из-за вирусных цитопатических эффектов, таким образом, уменьшая размер латентного резервуара. Тестирование этой гипотезы показало, что реактивированные клетки не умирают, и размер латентного резервуара не уменьшается (Shan L, Deng K, Shroff NS, Durand CM, Rabi SA, Yang HC, Zhang H, Margolick JB, Blankson JN, Siliciano RF, Stimulation of HIV-1-specific cytolytic T lymphocytes facilitates elimination of latent viral reservoir after virus reactivation. Immunity. 2012;36(3) p.491-501.). Две главные проблемы все еще присутствуют после реактивации резервуара. Первая относится к появлению вирусов, которые являются устойчивыми к уничтожению посредством CTL (Deng K, Pertea M, Rongvaux A, Wang L, Durand CM, Ghiaur G, Lai J, McHugh HL, Hao H, Zhang H, JB, Gurer C, Murphy AJ, Valenzuela DM, Yancopoulos GD, Deeks SG, Strowig T, Kumar P, Siliciano JD, Salzberg SL, Flavell RA, Shan L, Siliciano RF Broad CTL response is required to clear latent HIV-1 due to dominance of escape mutations. Nature. 2015 Jan 15;517(7534) p. 381-5). Это представляет собой общераспространенную проблему у хронически инфицированных индивидуумов, которых не подвергали лечению с использованием ART в ходе первых 6 месяцев инфекции (большинства хронически инфицированных индивидуумов). Вторая проблема вытекает из воздействия на CTL связанного с HIV хронического воспаления, приводящего к истощению CTL (Cella M, Presti R, Vermi W, Lavender K, Turnbull E, Ochsenbauer-Jambor C, Kappes JC, Ferrari G, Kessels L, Williams I; CHAVI Clinical Core B, McMichael AJ, Haynes BF, Borrow P, Colonna M; NIAID Center for HIV/AIDS Vaccine Immunology. Loss of DNAM-1 contributes to CD8+ T-cell exhaustion in chronic HIV-1 infection. Eur J Immunol. 2010 Apr;40(4):p. 949-54). Кажется вероятным, что являются необходимыми новые способы уничтожения реактивированных клеток-резервуаров, исключающие проблемы вирусной устойчивости и клеточного истощения. Авторы настоящего изобретения предложили конструирование конвертируемый CAR-T-клеток, принимающих преимущество нейтрализующих HIV антител широкого спектра для нацеливания CTL для уничтожения реактивированного резервуара клеток.
[0049] Таким образом, настоящее изобретение умножает разнообразие и практичность этого примечательного, очень многообещающего иммунологического способа для управления течением злокачественной опухоли или вирусных инфекций с использованием CAR-T-клеток, CAR-клеток NK и/или макрофагов, с преодолением в то же время множества из этих современных, известных сложностей ACT.
[0050] В рамках изобретения, «пептид», «полипептид» и «белок» использованы взаимозаменяемо; и «гетерологичная молекула», «гетерологичный пептид», «гетерологичная последовательность» или «гетерологичный атом» представляет собой молекулу, пептид, нуклеиновую кислоту или аминокислотную последовательность или атом, соответственно, которая не является природной или в норме не обнаружена в физическом объединении с рассматриваемой молекулой. В рамках изобретения, «неприродный» и «модифицированный» использованы взаимозаменяемо. В рамках изобретения, «природный» и «нативный» использованы взаимозаменяемо, и «NKG2D» и «рецептор NKG2D» использованы взаимозаменяемо. Термин «антитело» в настоящем описании использован в самом широком смысле и конкретно охватывает моноклональные антитела, мультиспецифические антитела (например, биспецифические антитела) и фрагменты антител, при условии, что они имеют желательную биологическую активность. «Фрагменты антител» содержат часть интактного антитела, предпочтительно, содержащую его антигенсвязывающую область. Примеры фрагментов антител включают фрагменты Fab, Fab', F(ab')2 и Fv; диатела; линейные антитела; молекулы одноцепочечных антител; и мультиспецифические антитела, сформированные из фрагмента(фрагментов) антител.
[0051] Термин «содержащий», который использован взаимозаменяемо с «включающий», «вмещающий» или «характеризующийся», представляет собой включительное или неограничивающее выражение и не исключает дополнительных, не перечисленных элементов или стадий способа. Фраза «состоящий из» исключает любой элемент, стадию или ингредиент, не указанный в пункте формулы изобретения. Фраза «состоящий в основном из» ограничивает объем пункта формулы изобретения до указанных материалов или стадий и материалов или стадий, которые существенно не влияют на основные и новые характеристики заявленного изобретения. Настоящее описание включает варианты осуществления по изобретению композиций и способов, соответствующие объему каждой из этих фраз. Таким образом, композиция или способ, содержащие перечисленные элементы или стадии, включает конкретные варианты осуществления, в которых композиция или способ в основном состоит из или состоит из этих элементов или стадий.
[0052] Полное содержание всех ссылок, процитированных в настоящем описании, таким образом, приведено в качестве ссылки, вне зависимости от того, включено ли оно предварительно конкретно или нет. В рамках изобретения, каждый из терминов единственного числа предназначен для включения форм как единственного, так и множественного числа.
[0053] При наличии в настоящее время полностью описанного изобретения, специалисту в данной области понятно, что его можно осуществлять в пределах широкого диапазона эквивалентных параметров, концентраций и условий, без отклонения от содержания и объема изобретения, и без излишнего экспериментирования. В то время как это изобретение описано в связи с конкретными вариантами его осуществления, понятно, что возможны его дополнительные модификации. Это описание предназначено, чтобы охватывать любые варианты, применения или адаптации изобретения, следуя, в общем, принципам изобретения и включая такие отклонения от настоящего описания, которые находятся в пределах известной или общепринятой практики в области, к которой относится изобретение, и могут быть применены к существенным признакам, указанным в настоящем описании ранее.
[0054] Пример 1 (Модифицированные домены α1- α2 лигандов NKG2D.) Существуют примеры прикрепления полипептидов к NKG2DL, которые были модифицированы для значительного усиления аффинности их связывания с рецептором NKG2D человека. Домен α1-α2 белков MIC представляет собой NKG2DL для рецептора NKG2D. Эта аффинность является достаточной для физиологической активации клеток NK и стимуляции лизиса клеток, экспрессирующих нативные полноразмерные белки MIC, необратимо привязанные к двумерной поверхности плазматической мембраны «клетки-мишени» (Bauer S, Groh V, Wu J, Steinle A, Phillips JH, Lanier LL, Spies T., Science. 1999 Jul 30;285(5428):727-9.). Однако, поскольку сконструированные растворимые белки MIC по настоящему изобретению обратимо связывают специфические антигены-мишени на поверхности клеток-мишеней, аффинность связывания сконструированного растворимого белка MIC с NKG2D может непосредственно влиять на стабильность растворимого зависимого от MIC комплекса, сформированного между клетками NK и клетками, экспрессирующими антигены-мишени. Особенно, если аффинность между sMICA и NKG2D является увеличенной посредством значительно более медленной скорости диссоциации или скорости отсоединения модифицированного sMICA от NKG2D, ожидают, что уничтожение на основе клеток NK будет больше при более низких плотностях молекул растворимого MIC, связанных с клеткой-мишенью. До настоящего изобретения не было идентифицировано никаких мутаций α1-α2, которые изменяют активность уничтожения растворимых белков MIC или значительно уменьшают скорость выключения связывания для увеличения аффинности белков MIC для NKG2D. Попытки вычислительного дизайна показали, что три мутации в домене α1-α2 MICA дикого типа: N69W, K152E и K154D (WED-MICA) в комбинации могут умеренно влиять на аффинность связывания NKG2D посредством влияния на стабильность несвязанного MICA и таким образом, скорость его ассоциации или скорость включения связывания с NKG2D (Lengyel CS, Willis LJ, Mann P, Baker D, Kortemme T, Strong RK, McFarland BJ.J Biol Chem. 2007 Oct 19;282(42):30658-66. Epub 2007 Aug 8). В последующей обширной работе по вычислительному дизайну в той же группе, сканируя посредством повторяющихся вычислений 22 положения аминокислот MICA, теоретически находящихся в контакте с NKG2D, в соответствии с опубликованными структурными описаниями (Li P, Morris DL, Willcox BE, Steinle A, Spies T, Strong RK., Nat Immunol. 2001 May;2(5):443-451), показали экспериментально, что при комбинации с ранее сконструированными 3 изменениями, дополнительный рациональный, повторяющийся вычислительный дизайн MICA количественно изменял его аффинность для NKG2D от слабой (Kd ~2,5 мкМ) до умеренно тесной (Kd=51 нМ) с использованием всего семи комбинированных мутаций (Henager, Samuel H., Melissa A. Hale, Nicholas J. Maurice, Erin C. Dunnington, Carter J. Swanson, Megan J. Peterson, Joseph J. Ban, David J. Culpepper, Luke D. Davies, Lisa K. Sanders, and Benjamin J. McFarland, 2102, Combining different design strategies for rational affinity maturation of the MICA-NKG2D interface. Protein Science 21:1396-1402). В отличие от этого, в экспериментальном способе по настоящему изобретению экспериментально отбирали аминокислотные модификации MICA, которые замедляли скорость диссоциации между доменом α1-α2 MICA и NKG2D, начиная с MICA, стабилизированного посредством 3 замен WED из Lengyel et al (Lengyel CS, Willis LJ, Mann P, Baker D, Kortemme T, Strong RK, McFarland BJ., J Biol Chem. 2007 Oct 19;282(42):30658-66. Epub 2007 Aug 8).
[0055] Этот пример относится к модификации аффинности связывания NKG2D для растворимых белков MIC посредством конструирования специфических мутаций в выбранных положениях аминокислот внутри домена α1-α2, которые влияют на кинетику скорости выключения связывания и таким образом, изменяют опосредованную клеткой NK активность уничтожения изобретательских неприродных, целевых молекул MIC.
[0056] Для конструирования растворимых неприродных доменов α1-α2 с измененной аффинностью для NKG2D, 57 остатков в домене α1-α2 выбраны для обширного мутагенеза (фигура 12). Библиотеки синтетической ДНК, кодирующие домен α1-α2 и содержащие мутагенные кодоны NNK в каждом из 57 положений аминокислот, синтезировали, индивидуально клонировали в форме слитых белков с минорным белком оболочки pIII фага M13, и частицы фага, экспонирующие подвергнутые мутагенезу варианты α1-α2, продуцировали в клетках E.coli SS320, в соответствии со стандартными способами (Andris-Widhopf, J., Steinberger, P., Fuller, R., Rader, C., and Barbas, C. F., 3rd. (2011) Generation of human Fab antibody libraries: PCR amplification and assembly of light- and heavy-chain coding sequences, Cold Spring Harbor protocols 2011). Библиотеки фагов α1-α2 сортировали по увеличенной аффинности связывания с использованием рекомбинантного биотинилированного NKG2D в качестве антигена-мишени и подвергали циклическим повторяющимся раундам целенаправленно продленного связывания, продленной отмывки и элюции клонов фагов, для отбора высоко аффинных вариантов, обогащенных по медленным скоростям диссоциации или выключения связывания. Группа специфических аминокислотных мутаций встречались с высокой частотой в 6 положениях в α1-α2 и отобраны в качестве предпочтительных аминокислотных замен с усиленной аффинностью связывания NKG2D (фигура 3, таблица 1).
[0057] Таблица 1. Отобранные мутации аффинности в указанных 6 положениях аминокислот домена α1-α2 MIC. Аминокислоты из SEQ ID NO.: 20 в каждом из 6 положений показаны жирным шрифтом в первой строке таблицы. Идентифицированные мутации аффинности перечислены с уменьшающейся частотой сверху вниз. Все аминокислоты представлены однобуквенными сокращенными наименованиями IUPAC.
[0058] Авторы настоящего изобретения синтезировали ДНК-полинуклеотиды (SEQ ID NO. 21-24), кодирующие домены α1-α2 из 4 репрезентативных вариантов 15, 16, 17, 18, содержащих различные комбинации специфических открытых мутаций (таблица 2).
[0059] Таблица 2. Последовательности специфических вариантов домена α1-α2. Специфические аминокислотные замены для вариантов 15, 16, 17 и 18 (SEQ ID NO.: 25-28, соответственно) перечислены относительно аминокислот из SEQ ID NO.:20, выделенных жирным шрифтом. Все аминокислоты представлены однобуквенными сокращенными наименованиями IUPAC.
[0060] К NKG2DL в вышеуказанном примере, авторы настоящего изобретения напрямую присоединяли гетерологичные молекулы, такие как полипептид, к каждому из этих 4 модифицированных α1-α2 NKG2DL с использованием линкерного пептида. Четыре меченных His белка (SEQ ID NO.: 25-28), состоящих из модифицированных NKG2DL с присоединенными гетерологичными молекулами, экспрессировали в клетках насекомых и очищали для характеризации их аффинности связывания NKG2D и кинетических параметров связывания. С использованием конкурентного ELISA связывания, авторы настоящего изобретения определили относительную аффинность связывания NKG2D 4 модифицированных вариантов α1-α2. Растворимым NKG2DL дикого типа (WT), белком sMICA, покрывали все лунки планшета maxisorp для ELISA для предоставления партнера по связыванию для реагента NKG2D-Fc человека. Растворы четырех вариантов α1-α2, так же как домены α1-α2 WT и WED (SEQ ID NO.: 20), титровали в лунках для ELISA и позволяли конкурентно ингибировать связывание 2нМ NKG2D-Fc человека с WT sMICA, покрывающим планшет. Уровень NKG2D-Fc человека, связанного с WT NKG2DL на планшете, детектировали с использованием антитела против Fc-HRP. На фигуре 13, панель A, показаны варианты 16, 17 и 18, имеющие значения IC50 0,7, 0,6, 0,5 нМ, в то время как вариант 15 имел значение IC50 1,7 нМ, все имели значительно лучшее связывание с NKG2D, в 27, 32, 38 и 11 раз лучше, чем NKG2DL WT, соответственно, так же как значительно лучшее, чем WED-MICA (таблица 3).
[0061] Таблица 3. Параметры равновесия и кинетики связывания для вариантов α1-α2. Значения IC50 выводили из 4-параметрического подбора кривых для титрований конкурентного связывания (фигура 12), и кинетические параметры связывания выводили из однократного экспоненциального подбора кривых для кинетики связывания (фигура 13). Равновесные константы связывания (Kd) выводили из кинетических параметров связывания с использованием уравнения Kd=kOFF/kON.
[0062] Важно, что относительные различия IC50 также приводили к лучшему связыванию с мышиным NKG2D-Fc и демонстрировали способность улучшать связывание растворимых, модифицированных доменов α1-α2 среди человеческих и не относящихся к человеку рецепторов NKG2D, важное свойство для доклинической разработки лекарственного средства.
[0063] Для понимания кинетической основы измененной аффинности, как скорости связывания, так и скорости диссоциации для связывания вариантов α1-α2 NKG2DL с поверхностью, покрытой биотинилированным NKG2D человека, измеряли с использованием интерферометрии биослоев (Octet) при 100 нМ каждого из модифицированных белков α1-α2. В соответствии с результатами IC50 в ELISA, для каждого из вариантов 16, 17 и 18 показаны значительные уменьшения скорости диссоциации (в 18 раз относительно WT), что в большой степени отвечает за увеличение аффинности (в ~30 раз относительно a1-a2 WT; таблица 3). Хотя для варианта 15 показана сходная медленная скорость диссоциации, как и для 16, 17 и 18, его скорость связывания была уменьшена, что приводило к аффинности, более сильной, чем для WT, но слабее, чем для вариантов 16, 17 и 18. Поскольку единственным различием между вариантом 15 (SEQ ID NO.:25) и 16 (SEQ ID NO.:26) являлось K125N против K125L, мутация в положении 125 явно изменяла скорость связывания, в то время как уменьшенную скорость диссоциации приписывали мутации H161R. Таким образом, в то время как отобранную группу мутаций NKG2DL (таблица 1) использовали доля увеличения аффинности α1-α2 для NKG2D посредством значительного уменьшения скорости диссоциации, конкретные замены изменяли также скорость связывания, что приводило к диапазону постепенных увеличений аффинности, которые, как показали авторы настоящего изобретения, имеют дифференциальную активность в анализах опосредованного клетками NK уничтожения, как описано ниже.
[0064] Способность аффинных вариантов α1-α2 перенацеливать опосредованный клетками NK лизис экспрессирующих FGFR3 клеток-мишеней, показана in vitro в анализе высвобождения кальцеина. Линию клеток естественных киллеров (NK) человека, NKL, совместно культивировали с нагруженными кальцеином клетками-мишенями P815, эктопически экспрессирующими FGFR3, и титровали с использованием растворимых модифицированных белков MIC. Результаты на фигуре 15 показали, что активность уничтожения специфических для FGFR3 растворимых вариантов MIC коррелировала с аффинностью их сконструированных α1-α2. Конкретно, для вариантов 16, 17 и 18 показано в ~15 раз большее уничтожение, чем для WT, при 0,78 нМ. WED-MICA (SEQ ID NO.:20) являлся только немного лучшим, чем WT. Таким образом, по настоящему изобретению описаны аминокислотные замены в домене α1-α2, которые увеличивали аффинность связывания NKG2D посредством уменьшения скорости выключения связывания растворимого белка MIC с NKG2D человека и следовательно, приводили к предсказуемо увеличенной активности уничтожения. Для WED-MICA, для которого показана несколько большая аффинность, чем у WT MICA, для NKG2D, из-за увеличения скорости связывания, а не уменьшения скорости диссоциации, не показано существенного улучшения уничтожения клетки-мишени. Кроме того, для WED-MICA показано существенно более слабое связывание с мышиным NKG2D, даже чем для WT MICA, в то время как для каждого из вариантов 15, 16, 17 и 18 показана большая аффинность как для человеческого, так и для мышиного NKG2D.
[0065] Эти аффинные варианты α1-α2 NKG2DL 15, 16, 17 и 18 усиливали аффинность связывания присоединенного полипептида с рецептором NKG2D и таким образом, усиливали опосредованный клетками NK лизис целевых клеток.
[0066] Пример 2. (Неприродные домены α1-α2 лигандов NKG2D и родственные неприродные рецепторы NKG2D, с которыми они связываются)
[0067] Домен α1-α2 MICA и другие лиганды NKG2D связывают рецептор NKG2D в известном специфическом участке (Li et al 2001; Benjamin J. McFarland, Tanja Kortemme, Shuyuarn F. Yu, David Baker, and Roland K. Strong. Symmetry Recognizing Asymmetry: Analysis of the Interactions between the C-Type Lectin-like Immunoreceptor NKG2D and MHC Class I-like Ligands. Structure, Vol. 11, 411-422, April, 2003) и управляют активацией несущего рецептор NKG2D иммуноцита, который, как следствие, уничтожает клетки-мишени, экспонирующие MICA или другие лиганды. Авторы настоящего изобретения использовали фаговый дисплей для конструирования неприродных доменов α1-α2 MICA посредством обширного мутагенеза в 57 специфических участках, вероятно, вовлеченных в связывание с NKG2D (фигура 16). Библиотеки синтетической ДНК, кодирующие домен α1-α2 и содержащие мутагенные кодоны NNK в каждом их 57 положений аминокислот, синтезировали, индивидуально клонировали в форме слитых белков с минорным белком оболочки pIII фага M13, и частицы фага, экспонирующие подвергнутые мутагенезу варианты α1-α2, продуцировали в клетках E.coli SS320, в соответствии со стандартными способами (Andris-Widhopf, J., Steinberger, P., Fuller, R., Rader, C., and Barbas, C. F., 3rd, 2011. Generation of human Fab antibody libraries: PCR amplification and assembly of light- and heavy-chain coding sequences, Cold Spring Harbor protocols 2011). Библиотеки фагов α1-α2 сортировали по увеличенной аффинности связывания с использованием рекомбинантного биотинилированного NKG2D в качестве антигена-мишени и подвергали циклическим повторяющимся раундам целенаправленно продленного связывания, продленной отмывки и элюции клонов фагов, для отбора высоко аффинных вариантов, обогащенных по медленным скоростям диссоциации или выключения связывания. Группа специфических аминокислотных мутаций в 9 положениях в домене α1-α2 отобраны в качестве предпочтительных участков аминокислотных замен с усиленной аффинностью связывания NKG2D. Авторы настоящего изобретения синтезировали ДНК-полинуклеотиды, кодирующие домены α1-α2 из 8 репрезентативных вариантов (SEQ ID NO: 29-36), содержащих различные комбинации специфических мутаций (таблица 4).
[0068] Таблица 4. Варианты неприродного домена α1-α2, отобранные по увеличенной аффинности для природного рецептора NKG2D, и вариант MICwed, описанный ранее (McFarland et al., 2003). Положения указанных аминокислотных замен даны со ссылкой на положения остатков в SEQ ID NO.: 7, и представлены общепринятые наименования вариантов и их SEQ ID NO.
[0069] ДНК-полинуклеотиды, кодирующие 8 вариантов доменов α1-α2, амплифицировали с использованием праймеров для ПЦР (SEQ ID NO.: 37-38). С использованием рестрикционных ферментов Blp1 и Sap1, каждый субклонировали в конструкцию для экспрессии слитого белка меченного His α1-α2-α3-Fv (SEQ ID NO.:39) для замены последовательности, кодирующей природные (wt) последовательности α1-α2, на мутантные последовательности α1-α2. 9 слитых белков (SEQ ID NO.: 40-48) экспрессировали в клетках 293 (Система экспрессии Expi293™, Life Technologies, Thermo Fisher, Inc.) и подвергали аффинной очистке с использованием Ni-аффинной хроматографии (HisTrap HP, GE Healthcare Life Sciences).
[0070] Для конструирования белков рецептора NKG2D, авторы настоящего изобретения синтезировали ДНК, кодирующую внеклеточный домен («эктодомен») рецептора дикого типа (SEQ ID No.:49) и использовали праймеры для ПЦР (SEQ ID NO.: 50-51) и участки XbaI и BamHI для клонирования синтетической ДНК в экспрессирующий вектор с N-концевой меткой His-avitag (SEQ ID NO.: 78). His-avitag-природный NKG2D (SEQ ID NO.:52) временно экспрессировали в клетках 293 и очищали с использованием Ni-аффинной хроматографии. После очистки, белки NKG2D подвергали сайт-специфическому биотинилированию с использованием BirA для присоединения группы биотина к последовательности avitag (стандартный реакционный набор с биотин-протеин-лигазой BirA, Avidity, LLC, Aurora, CO.).
[0071] Для характеризации и сравнения кинетических параметров связывания природного и 8 вариантов доменов α1-α2 с природным NKG2D, авторы настоящего изобретения измеряли их связывание с поверхностью, покрытой биотинилированным природным эктодоменом NKG2D, с использованием интерферометрии биослоев (Octet) при 100 нМ каждого из слитых белков α1-α2-α3-Fv. Результаты показаны в таблице 5.
[0072] Таблица 5: Кинетические параметры связывания слитых белков α3-Fv с доменом дикого типа (wt или природным) и 8 вариантами домена α1-α2 с природным NKG2D. MICwed-Fv в данном случае исследовали в 2 отдельных анализах Octet, одном, сравнивающем со слитым белком α3-Fv с доменом α1-α2 wt, и другом, сравнивающем со слитыми белками α3-Fv с 7 другими неприродными доменами α1-α2. Общепринятые наименования каждого из вариантов доменов α1-α2 и SEQ ID NO. слитых с ними белков α3-Fv представлены вместе с их значениями аффинности (Kd) в молях (М), скоростей связывания (kon) в обратных молях-секундах (1/Мс), и скоростей диссоциации или выключения связывания (kdis) в обратных секундах.
[0073] Как показано в таблице 5, отобранные мутации домена α1-α2 в качестве слитых белков с гетерологичными полипептидами α3-Fv из SEQ ID NO.: 42-48 увеличивали аффинность домена α1-α2 для природного NKG2D посредством значительного уменьшения скорости диссоциации. Скорость диссоциации лежала в диапазоне в от 20 до более чем 100 раз медленнее, чем для wt (SEQ ID NO.:40) и ранее описанного варианта MICwed домена α1-α2 (SEQ ID NO.:41).
[0074] В этом примере по настоящему изобретению, авторы настоящего изобретения дополнительно показали, как описано ниже, что неприродный домен α1-α2 (DSM25, SEQ ID NO.:31, таблица 4), который в форме слитого белка α1-α2-α3-Fv имел высокую аффинность для и очень медленную скорость диссоциации от природного NKG2D (таблица 2; SEQ ID NO.:43), имел тесную аффинность связывания с неприродным рецептором NKG2D, содержащим специфическую мутацию, прекращающую его связывание с природными лигандами NKG2D. Другими авторами показано, что мутации тирозина 152 и тирозина 199 в NKG2D человека, эквиваленте положений 73 и 120 эктодомена NKG2D (SEQ ID NO.:49 и фигура 4), прекращают связывание с природным лигандом, MICA (David J. Culpepper, Michael K. Maddox1, Andrew B. Caldwell, and Benjamin J. McFarland. Systematic mutation and thermodynamic analysis of central tyrosine pairs in polyspecific NKG2D receptor interactions. Mol Immunol. 2011 January ; 48(4): 516-523).
[0075] Для конструирования белков неприродного рецептора NKG2D, авторы настоящего изобретения использовали праймеры для ПЦР (SEQ ID NO.:50-51) для клонирования ДНК, кодирующей природный эктодомен NKG2D (SEQ ID NO.:49), и ее вставки в экспрессирующий вектор с N-концевой меткой His-avitag SEQ ID NO.:52 для получения His-avitag-NKG2D (SEQ ID NO.:53). Сайт-направленный мутагенез проводили для конструкции ДНК природного эктодомена NKG2D для введения мутаций Y152A, Y199A или Y152A плюс Y199A, и получили три неприродных варианта NKG2D человека (SEQ ID NO.: 54-56, соответственно). Природный NKG2D и 3 неприродных мутанта NKG2D с метками His-avitag временно экспрессировали в клетках 293 и очищали с использованием Ni- аффинной хроматографии. После очистки, белки NKG2D подвергали сайт-специфическому биотинилированию с использованием BirA для присоединения группы биотина к последовательности avitag (стандартный реакционный набор с биотин-протеин-лигазой BirA, Avidity, LLC, Aurora, CO.).
[0076] Для получения слитых белков гетерологичных полипептидов α3-Fc с доменом α1-α2 MICwed (SEQ ID NO.:29) и доменом α1-α2 DSM25 (SEQ ID NO.: 31), ДНК-полинуклеотиды кодирующие домены α1-α2, амплифицировали с использованием праймеров для ПЦР (SEQ ID NO.: 37-38). С использованием рестрикционных ферментов XbaI и NcoI, каждый субклонировали в конструкцию для экспрессии слитого белка α1-α2-α3-Fc (SEQ ID NO.:57) для замены последовательности, кодирующей природные (wt) последовательности (wt) α1-α2, на мутантные последовательности α1-α2. 3 слитых белка, MICA-Fc (SEQ ID NO.: 58), MICwed-Fc (SEQ ID NO.: 59), и MICv25-Fc (SEQ ID NO.: 60) экспрессировали в клетках 293 (Система экспрессии Expi293™ Expression System, Life Technologies, Thermo Fisher, Inc.) и подвергали аффинной очистке с использованием аффинной хроматографии с белком A (кат. no. 20334, Pierce Biotechnology, Rockford, IL).
[0077] В дополнение к очистке вышеуказанных 3 слитых с Fc белков лиганд NKG2D-Fc, слитые белки MICB-Fc, ULBP1-Fc, ULBP2-Fc, ULBP3-Fc и ULBP4-Fc закупали из R&D Systems, Inc. (Minneapolis, MN). Связывание различных слитых белков домен α1-α2-Fc с белками как с природным, так и с неприродным эктодоменом NKG2D, анализировали с использованием способа ELISA на основе планшетов. Всеми слитыми белками α1-α2-Fc с природными и неприродным доменами покрывали в течение ночи при 4°C отдельные лунки 96-луночных планшетов Maxisorp с использованием покрывающей концентрации 2 мкг/мл в фосфатно-солевом буфере (PBS). Планшеты промывали 3 раза в PBS/0,05% Tween20 при 20-22°C и блокировали с использованием 0,5% бычьего сывороточного альбумина в течение 2 часов. Биотинилированные природные и неприродные рецепторные белки NKG2D титровали против связанных с планшетом лигандов NKG2D в течение 2 часов при 20-22°C, промывали 3 раза с использованием PBS/0,05% Tween20 при 20-22°C, и связанные белки NKG2D впоследствии детектировали с использованием стадии вторичной детекции с стрептавидином-HRP и проявляли с использованием 1-Step Ultra TMB Elisa. Природная форма эктодомена NKG2D (SEQ ID NO.:49) являлась способной связывать все тестированные слитые белки домен α1-α2-Fc. Лиганд с неприродным доменом α1-α2 MIC-v25 связывался с наивысшей аффинностью (EC50=14 нМ), которая была в 8 раз лучше, чем для MICwed, и более чем в 100 раз лучше, чем для всех тестированных лигандов с природным доменом α1-α2. Все тестированные лиганды, как с природными, так и с неприродными доменами α1-α2, теряли связывание с мутантным рецептором NKG2D Y199A (SEQ ID NO.:55; фигура 18, панель B) и с двойным мутантным рецептором Y152A плюс Y199A (SEQ ID NO.:56). Однако, из всех тестированных лигандов с природными и с неприродными доменами α1-α2, только неприродный домен α1-α2 (SEQ ID NO.:31) MICv25-Fc (SEQ ID NO.:60) сохранял связывание с мутантом Y152A эктодомена NKG2D (SEQ ID NO.:54) с EC50 50 нМ.
[0078] В то время как для специфичности связывания природного NKG2D показана предпочтительность высокоаффинных неприродных лигандов, его сильное связывание с природными лигандами NKG2D, которые присутствуют на конкретных здоровых тканях и многих подвергнутых стрессу тканях, создает необычайный риск токсичности при использовании современных способов с NKG2D CAR (VanSeggelen et al. 2015). Неприродный рецептор NKG2D Y152A специфически связывался с единственным белком, состоящим из высокоаффинного неприродного домена α1-α2, сконструированного для заметно уменьшенной скорости диссоциации. Этот прототипический пример подчеркивает способность неприродных доменов α1-α2 связывать неприродные рецепторы NKG2D, таким образом, обеспечивая избирательный контроль неприродных NKG2D-CAR с использованием биспецифических белков, содержащих изобретательский неприродный домен α1-α2 лигандов NKG2D.
[0079] Пример 3 (Модифицированные домены α1-α2 лигандов NKG2D).
[0080] Этот вариант осуществления относится к дополнительным аффинным вариантам α1-α2 NKG2DL, полученным посредством конструирования доменов α1-α2 белков ULBP. Белки ULBP содержат домены α1-α2, которые являются лигандами NKG2D, способными связываться с рецептором NKG2D (Cerwenka A, Lanier LL (2004). NKG2D ligands: unconventional MHC class I-like molecules exploited by viruses and cancer. Tissue Antigens 61 (5): 335-43. doi:10.1034/j.1399-0039.2003.00070.x. PMID 12753652). Эта аффинность связывания NKG2D является достаточной для физиологической активации клеток NK и стимуляции лизиса клеток, экспрессирующих нативные полноразмерные белки ULBP, естественным образом и необратимо привязанные к двумерным поверхностям плазматической мембраны «клетки-мишени» (Cerwenka A, Lanier LL (2004). NKG2D ligands: unconventional MHC class I-like molecules exploited by viruses and cancer. Tissue Antigens 61 (5): 335-43. doi:10.1034/j.1399-0039.2003.00070.x. PMID 12753652). Однако, поскольку сконструированные растворимые домены α1-α2, слитые с гетерологичными полипептидами, в конкретных вариантах осуществления настоящего изобретения, обратимо связывают специфические антигены-мишени на поверхности клетки-мишени, аффинность связывания сконструированных доменов α1-α2 ULBP с NKG2D может напрямую влиять на стабильность искусственного синапса, сформированного между клетками NK и клетками, экспрессирующими антигены-мишени, как уже показано посредством сконструированных растворимых белков MIC (примеры 21-2). Для внесения разнообразия в репертуар сконструированных неприродных доменов α1-α2 в качестве лигандов NKG2D, белки ULBP использовали в качестве субстрата или исходной точки для модификации на основе фагового дисплея их аффинности связывания с NKG2D. Несмотря на структурную гомологию, наблюдаемую между ULBP и MICA (Radaev, S., Rostro, B., Brooks, AG., Colonna, M., Sun, PD. (2001) Conformational plasticity revealed by the cocrystal structure of NKG2D and its class I MHC-like Ligand ULBP3. Immunity 15, 1039-49.), гомология последовательности составляет <50% для доменов α1-α2 ULBP, относительно MICA. Таким образом, авторы настоящего изобретения искали идентичность положений кодонов в доменах α1-α2 ULBP, которые улучшают аффинность связывания NKG2D.
[0081] Для конструирования растворимых, неприродных доменов α1-α2, из белков ULBP, ULBP2 и ULBP3 выбраны для фагового дисплея и отбора мутантов с высокоаффинным связыванием NKG2D. Шестьдесят положений аминокислот в домене α1-α2 ULBP2 (SEQ ID NO: 61) и тридцать шесть положений аминокислот в домене α1-α2 ULBP3 (SEQ ID NO: 62) выбраны для обширного мутагенеза. Кроме того, мутации консервативного цистеина до серина выполнены в C8S в ULBP2 (SEQ ID NO: 61) и C103S в ULBP3 (SEQ ID NO: 62), уничтожающие неспаренные свободные остатки цистеина для увеличения стабильности и функционирования лигандов NKG2D с присоединенными полипептидами, так же как для улучшения процессов пэннинга фагов. Библиотеки синтетической ДНК, кодирующие эти домены α1-α2 с модификацией цистеина до серина, и содержащие мутагенные кодоны NNK в каждом из выбранных положений аминокислот, синтезировали, индивидуально; клонировали в форме слитых белков с минорным белком оболочки pIII фага M13; и частицы фага, экспонирующие подвергнутые мутагенезу варианты α1-α2 ULBP2 или ULBP3, продуцировали в клетках E.coli SS320, в соответствии со стандартными способами (Andris-Widhopf, J., Steinberger, P., Fuller, R., Rader, C., and Barbas, C. F., 3rd. (2011). Generation of human Fab antibody libraries: PCR amplification and assembly of light- and heavy-chain coding sequences, Cold Spring Harbor protocols 2011). Библиотеки фагового дисплея α1-α2 сортировали по увеличенной аффинности связывания с NKG2D с использованием NKG2D-Fc человека в качестве белка-мишени, и подвергали циклическим повторяющимся раундам целенаправленно продленного связывания, продленной отмывки и элюции клонов фагов, для отбора высоко аффинных вариантов, обогащенных по медленным скоростям диссоциации или выключения связывания. Для ULBP2, специфические аминокислотные мутации обнаружены с высокими частотами в положениях R80, V151, V152 и A153 в α1-α2, и идентифицированы в качестве предпочтительных аминокислотных замен с усиленной аффинностью связывания NKG2D (фигура 19, панель A; и таблица 6).
[0082] Таблица 6. Отобранные мутации аффинности в указанных 4 положениях аминокислот домена α1-α2 ULBP2. Аминокислоты из SEQ ID NO: 61 в каждом из 4 положений показаны жирным шрифтом в первой строке таблицы. Идентифицированные мутации аффинности перечислены с уменьшающейся частотой сверху вниз. Все аминокислоты представлены однобуквенными сокращенными наименованиями IUPAC.
[0083] Для ULBP3, специфические аминокислотные мутации обнаружены в различных локализациях, относительно ULBP2. Положения R162 и K165 в домене α1-α2 ULBP3 содержали специфические мутации, которые идентифицированы в качестве предпочтительных аминокислотных замен с усиленной аффинностью связывания NKG2D (таблица 7). Эти модифицированные неприродные домены α1-α2, происходящие из ULBP2 и ULBP3, можно использовать для усиления связывания NKG2D во множестве терапевтических форматов, в форме одиночных белков или слитых белков с гетерологичными пептидами или полипептидами.
[0084] Таблица 7. Отобранные мутации аффинности в указанных 2 положениях аминокислот домена α1-α2 ULBP3. Аминокислоты из SEQ ID NO: 62 в каждом из 2 положений показаны жирным шрифтом в первой строке таблицы. Идентифицированные мутации аффинности перечислены с уменьшающейся частотой сверху вниз. Все аминокислоты представлены однобуквенными сокращенными наименованиями IUPAC.
[0085] Пример 4 (Связывание и цитолиз посредством модифицированных доменов α1-α2 ULBP, слитых с пептидами антител)
[0086] Следующий пример относится к присоединению полипептидов антител к NKG2DL, которые были модифицированы для значительного усиления их аффинности связывания с человеческим и мышиным рецептором NKG2D. Домен α1-α2 каждого белка ULBP представляет собой природный лиганд для рецептора NKG2D, т.е. NKG2DL. Антитела представляют собой высокостабильные гликопротеины, состоящие из двух больших тяжелых цепей и двух небольших легких цепей (фигура 1). В данной области не существовало формата антитела IgG, которое может напрямую активировать иммуноциты с использованием неприродных доменов α1-α2 ULBP, которые связываются более тесно, чем нативные домены ULBP, с рецептором NKG2D. Кроме того, домены α1-α2 ULBP обеспечивают альтернативные NKG2DL для конструирования слитых с антителом белков, которые могут иметь дифференциальные свойства in vivo, относительно доменов α1-α2 MICA. Например, ответ in vivo антитела против лекарственного средства на домены α1-α2 MICA внутри слитого с антителом белка, вероятно, не будет являться реакционноспособным по отношению к или мешать модифицированным доменам α1-α2 ULBP, из-за низкой гомологии последовательности между доменами α1-α2 ULBP и MICA (фигура 5). Этот пример показывает, что слитые белки между сконструированными лигандами NKG2D ULBP α1-α2 (таблица 6 и 7) и тяжелой цепью молекулы IgG имеют усиленное связывание NKG2D и уничтожение клетки-мишени, относительно природных лигандов NKG2D ULBP α1-α2. Это дополнительно показывает полезность слитых белков модифицированных доменов α1-α2 с гетерологичными белками или пептидами.
[0087] Для получения сконструированных слитых белков домена α1-α2 с антителами, последовательности ДНК, кодирующие модифицированные домены C8S α1-α2 ULBP2 (SEQ ID NO.: 61), варианты R80W и V151D (SEQ ID NO.: 63 и 64, соответственно) и модифицированный домен C103S α1-α2 ULBP3 (SEQ ID NO.: 62), вариант R162G (SEQ ID NO.: 65), синтезировали и клонировали в форме слитых с C-конца белков с последовательностью тяжелой цепи из специфического для Her2 антитела (Carter, P., Presta, L., Gorman, CM., Ridgway, JB., Henner, D., Wong, WL., Rowland, AM., Kotts, C., Carver, ME., Shepard, HM. (1992) Proc Natl Acad Sci 15, 4285-9.). Полученные слитые белки клонировали в экспрессирующий вектор для млекопитающих pD2509 и экспрессировали с легкой цепью исходного антитела в форме спаренных полноразмерных антител IgG. Временную экспрессию осуществляли в клетках HEK293 с использованием системы экспрессии Expi293, в соответствии с протоколом производителя (Life Technologies), и очищали с использованием стандартной аффинной хроматографии с белком A. ELISA связывания, проведенные для слитых белков α1-α2 ULBP2 и ULBP3 с тяжелой цепью антитела, показали, что модифицированные слитые с ULBP2 белки (HC_R80W и HC_V151D) и слитый с UBLP3 белок (HC_R162G) связывались с более высокой аффинностью с NKG2D человека, относительно их соответствующих природных доменов α1-α2, слитых с такой же тяжелой цепью.
[0088] Для характеризации свойств уничтожения клетки-мишени слитых белков модифицированного ULBP с антителом, линию клеток естественных киллеров (NK) человека, NKL, совместно культивировали с нагруженными кальцеином клетками-мишенями SKBR3, экспрессирующими Her2, и титровали с использованием сконструированных слитых с антителом белков. Результаты показали, что усиленная цитолитическая активность (уничтожения) специфических для Her2 слитых белков неприродные ULBP2 и неприродные ULBP3 α1-α2-антитело отражала увеличенную аффинность их сконструированных доменов α1-α2 для NKG2D. Конкретно, варианты слитых с ULBP2 белков HC_R80W и HC_V151D, и вариант слитого с ULBP3 белка HC_R162G, уничтожали клетки SKBR3 более эффективно, чем слитые с антителом белки, содержащие любой нативный домен α1-α2. Эти данные дополнительно показали, что слитые белки модифицированный α1-α2 вариант-антитело представляют собой универсальную платформу для обеспечения тесного связывания молекул IgG с NKG2D и для управления антигенспецифическим лизисом клеток.
[0089] Пример 5 (Конструирование ортогональных неприродных доменов α1-α2 с избирательным связыванием с неприродным Y152A NKG2D)
[0090] Средства для избирательного контроля видов CAR-T-клеточной терапии очень востребованы для ослабления токсичности и улучшения эффективности против опухолей (Gill and June, в цитируемом документе). Предшествующие попытки предпринимали для разработки CAR с использованием эктодомена CD16, который затем можно привлекать посредством домена Fc терапевтических моноклональных антител, обеспечивая основанный на антителах контроль нацеливания CAR-T (Chang et al., в цитируемом документе). Однако, основанный на CD16-CAR-T-клетки могут узнавать все эндогенные молекулы антител в крови и тканях, и терапевтические антитела, используемые для контроля этих клеток, могут встречать помехи из-за эндогенных рецепторов CD16 на клетках NK. Оба из этих признаков создают проблемы с не специфической для опухоли токсичностью и плохой фармакокинетикой, соответственно.
[0091] Для нацеливания на эти проблемы, авторы настоящего изобретения сконструировали неприродный NKG2D-CAR-T-клетки, которые лишены связывания со всеми природными лигандами NKG2D и могу поддаваться контролю посредством связывания высокоаффинных неприродных доменов α1-α2, как описано в примере 2. Дополнительным требованием для неприродных доменов α1-α2 является сохранение высокой аффинности для неприродного NKG2D, и исключение связывания с природными доменами NKG2D. Таким образом, сконструированные домены α1-α2, имеющие сильную избирательность для неприродных рецепторов NKG2D, по сравнению с природным NKG2D, представляют собой идеальную систему для избирательного контроля рецепторов неприродный NKG2D-CAR, или любого рецептора или белка, слитого с неприродными эктодоменами NKG2D, которые могут подвергаться избирательному привлечению неприродными доменами α1-α2.
[0092] Авторы настоящего изобретения использовали фаговый дисплей для конструирования ортогональных неприродных доменов α1-α2, демонстрирующих избирательное связывание с рецептором NKG2D Y152A. В качестве исходной точки, три неприродных домена α1-α2 с высокой аффинностью для природного NKG2D отбирали в качестве исходных доменов для дальнейшего мутагенеза и скрининга посредством фагового дисплея. Библиотеки синтетической ДНК получали для индивидуальных вариантов домена α1-α2 DSM25, ULBP2 R80W и ULBP3 R162G (SEQ ID NO.: 31, 63 и 65), в результате чего кодоны аминокислотных остатков, которые в связанном состоянии расположены в тесной близости к положению Y152 на рецепторе NKG2D, заменяли на кодоны NNK. Библиотеки DSM 25 состояли из положений NNK в остатках 71-75 и 155-159, библиотек ULBP2 R80W с кодонами NNK в положениях 154-159, и библиотек ULBP3 R162G с кодонами NNK в положениях 155 -159. Библиотеки клонировали в форме слитых белков с минорным белком оболочки pIII фага M13; и частицы фага, экспонирующие подвергнутые мутагенезу варианты домена α1-α2, продуцировали в клетках E.coli SS320, в соответствии со стандартными способами (Andris-Widhopf, J., Steinberger, P., Fuller, R., Rader, C., and Barbas, C. F., 3rd. (2011). Generation of human Fab antibody libraries: PCR amplification and assembly of light- and heavy-chain coding sequences, Cold Spring Harbor protocols 2011). Библиотеки фагового дисплея α1-α2 сортировали по высокоаффинному связыванию с неприродным рецептором NKG2D Y152A посредством избирательного захвата клонов фагов, связанных с биотинилированным белком Y152A NKG2D-Fc в присутствии небиотинилированного природного конкурентного белка NKG2D-Fc. Избирательные клоны обогащали посредством подвергания множеству циклических раундов конкурентного отбора с использованием увеличивающихся концентраций небиотинилированного природного NKG2D-Fc.
[0093] После четырех раундов отбора, клоны фагов секвенировали для идентификации специфических мутаций в областях мутагенеза NNK. В таблицах 8, 9 и 10 показаны отобранные аминокислотные остатки, как обнаружено, преобладающие для каждого домена α1-α2, полученного в результате избирательного скрининга Y152A NKG2D.
[0094] Таблица 8. Отобранные мутации в DSM25, приведшие к получению специфических для Y152A клонов фагов.
[0095] Таблица 9. Отобранные мутации в ULBP2 R80W, приведшие к получению специфических для Y152A клонов фагов.
[0096] Таблица 10. Отобранные мутации в ULBP3 R162G, приведшие к получению специфических для Y152A клонов фагов.
[0097] Для подтверждения клонов фагов, имеющих надлежащее избирательное связывание, фаги продуцировали для индивидуальных клонов: MICA25.17, MICA25.18, ULBP2.S1, ULBP2.S2, ULBP2.S3, ULBP3.S1 и ULBP3.S2 (SEQ ID NO: 66, 67, 68, 69, 70, 71 и 72, соответственно) и титровали против Y152A или природного NKG2D в ELISA связывания. На фигуре 23, панели A-C, показано, что все 7 клонов фагов имели более чем 10-кратное избирательное связывание с неприродным Y152A NKG2D, по сравнению с природным или относящимся к дикому типу NKG2D.
[0098] Для подтверждения того, что избирательные для Y152A варианты домена α1-α2 сохраняют свойства специфического связывания в контексте слитых с антителом белков, авторы настоящего изобретения клонировали MICA25.17 и ULBP2.S3 в форме слитых с C-конца белков с тяжелой цепью специфического для FGFR3 антитела, описанного ранее (Qing et al, 2009. в цитируемом документе; SEQ ID NO.: 73 и 74, соответственно). Полученные слитые белки клонировали в экспрессирующий вектор для млекопитающих pD2509 и совместно экспрессировали с легкой цепью исходного антитела в форме спаренных полноразмерных антител IgG (R3 HC25.17 и R3 HC.U2S3). Временную экспрессию осуществляли в клетках HEK293 с использованием системы экспрессии Expi293 в соответствии с протоколом производителя (Life Technologies), и очищали с использованием стандартной аффинной хроматографии с белком A. ELISA, измеряющие связывание слитых с тяжелой цепью антитела белков R3 HC25.17 и R3 HC.U2S3 α1-α2 с неприродным Y152A NKG2D и с природным NKG2D, показали их значительно большую аффинность связывания для Y152A NKG2D, относительно природного NKG2D (фигура 6, панели B и D). В отличие от этого, слитые белки антитела с DSM25 и ULBP2 R80W имели предпочтительное связывание с природным NKG2D-Fc (фигура 6, панели A и C). Совместно, эти данные показывают изобретение неприродных ортогональных доменов α1-α2, имеющих высокоаффинное связывание с неприродными рецепторами NKG2D и значительно уменьшенную аффинность связывания с природным рецептором NKG2D. Кроме того, слитые ортогональные домены α1-α2 с полипептидами антитела сохраняли свои свойства избирательного связывания и могли быть использованы для перенацеливания неприродных рецепторов NKG2D на новые антигены, например, в контексте CAR-T-клеток.
[0099] Пример 6 (Нацеливание и активность уничтожения CAR-T-клеток с использованием неприродного эктодомена NKG2D контролировали с использованием ортогональных доменов α1-α2, слитых с нацеливающими антителами).
[0100] Чтобы показать избирательный контроль CAR-T-клеток, сконструированных с химерным рецептором с внедренным неприродным эктодоменом NKG2D, авторы настоящего изобретения конструировали CAR либо с природным NKG2D, либо с неприродным Y152A эктодоменами NKG2D, на основании предшествующей работы с использованием конструкций 4-1BB/CD3-дзета CAR (Патент Campana 8399645), сливая соответствующие эктодомены NKG2D с шарнирной областью CD8 (фигура 2) CAR. Эти конструкции клонировали в лентивирусный вектор и экспрессировали в первичных положительных по CD8 T-клетках человека с использованием лентивирусной трансдукции. Полученные природный NKG2D-CAR-T-клетки имели специфическую активность уничтожения клеток in vitro, в соответствии с узнаванием природного лиганда MICA, экспрессированного на клетках-мишенях. Конкретно, результаты показали, что хотя природный NKG2D-CAR-T-клетки уничтожали клетки P1, экспрессирующие природные лиганды MICA, неприродный Y152A NKG2D-CAR-T-клетки являлись значительно инактивированными и имели намного уменьшенное уничтожение экспрессирующих MICA клеток P1. Кроме того, ортогональные слитые белки α1-α2 с тяжелой цепью антитела, R3 HC25.17 и R3 HC.U2S3, избирательно активировали неприродный Y152A CAR-T-клетки для уничтожения экспрессирующих FGFR3 клеток-мишеней P1, но являлись неспособными перенацеливать активность уничтожения природный NKG2D-CAR-T-клеток. Это отличалось от слитых белков R3 HC25 и R3 HC.U2R80W α1-α2 с тяжелой цепью антитела, которые не являлись избирательными для неприродного Y152A NKG2D и активировали как природный, так и неприродный CAR-T-клетки для уничтожения клеток-мишеней P1. Эти данные показали, что неприродные ортогональные домены α1-α2, сконструированные для избирательного связывания неприродного Y152A NKG2D, специфически активировали неприродный Y152A NKG2D-CAR-T-клетки, в то же время избегая природных рецепторов NKG2D.
[0101] Пример 7 (Конструирование ортогональных неприродных доменов α1-α2 с избирательным связыванием с неприродным Y152A/Y199F NKG2D)
[0102] Другими авторами показано, что мутации тирозина 152 или тирозина 199 в NKG2D человека, эквивалентные положениям 73 и 120 эктодомена NKG2D (SEQ ID NO.:49), могут сильно уменьшать связывание с природным лигандом, MICA (David J. Culpepper, Michael K. Maddox, Andrew B. Caldwell, and Benjamin J. McFarland. Systematic mutation and thermodynamic analysis of central tyrosine pairs in polyspecific NKG2D receptor interactions. Mol Immunol. 2011 January; 48(4): 516-523). Авторы настоящего изобретения заключили, что в то время как мутация любого остатка тирозина сильно влияла на способность NKG2D связывать его природные лиганды, одновременная мутация обоих тирозина 152 (Y152) и тирозина 199 (Y199) может фактически уничтожать способность рецептора к привлечению всеми нативными лигандами. Авторы настоящего изобретения, таким образом, решили исследовать индивидуальные и комбинированные замены Y152 и Y199 и охарактеризовать их применительно к их биохимическому поведению с целью идентификации вариантов как с одиночной, так и с двойной мутацией, неспособных привлекать никакие природные лиганды. Эти варианты, которые также хорошо экспрессировались и собирались, представляли собой особенный интерес, поскольку они олицетворяли инертные лиганды, которые можно было более просто получать для анализа.
[0103] Природный (дикого типа) эктодомен NKG2D (NKG2D.wt, SEQ ID NO: 49) и варианты-кандидаты неприродных эктодоменов NKG2D (SEQ ID NO: 75-92) - также названные «сконструированными NKG2D» или «eNKG2D», клонировали в форме слитых белков с C-концом Fc IgG1 человека (домены без Fab), посредством короткого узнаваемого фактором Xa линкера Ile-Glu-Gly-Arg (SEQ ID NO: 93) и взаимозаменяемо обозначали как Fc-NKG2D.wt или NKG2D.wt и Fc-eNKG2D или eNKG2D (SEQ ID NO: 94-112). Фрагменты ДНК gBlocks® (Integrated ДНК Technologies, San Diego, CA), соответствующие сигнальной последовательности MHCI (SEQ ID NO: 113 и 114), Fc IgG1 человека с линкером (SEQ ID NO: 115) и варианты эктодомена NKG2D (SEQ ID NO: 116-124) синтезировали и вставляли в pD2610-V12 (ATUM, Newark, CA). Конструкции ДНК для исследования замен в Y152, Y199 или комбинации мутаций Y152/Y199 (таблица 1) временно экспрессировали в клетках Expi293TM (ThermoFisher Scientific, Waltham, MA), и секретированный белок очищали посредством аффинной хроматографии с белком A (кат. no. 20334, Pierce Biotechnology, Rockford, IL). Элюированный материал характеризовали посредством эксклюзионной хроматографии (SEC) на колонках Akta Pur Superdex, и корректно собранный, подходящий по размеру материал фракционировали и отделяли от пиков агрегатов до включения в анализы.
[0104] Характеризация посредством SEC очищенного слитого белка NKG2D.Y199A-Fc выявила состав преобладающего агрегированного материала (фигура 2). В отличие от этого, материал как слитого белка природный Fc-NKG2D, так и слитого белка Fc-NKG2D.Y152A, поддавались разделению посредством дискретного, неагрегированного пика, который легко отличался от более быстро мигрирующего агрегата. Эффект мутации Y199A на агрегацию также являлся очевидным для варианта слитого белка двойной мутант Y152A/Y199A Fc-NKG2D, показывая, что она оказывала преобладающее влияние на неправильное сворачивание белка (фигура 2). Этот аспект включения Y199A с любой комбинацией мутаций Y152 в варианты NKG2D, таким образом, представлял проблему для получения материала, необходимого для дальнейших попыток конструирования, и вызывал опасения относительно сборки и представления на поверхности клетки. Следовательно, предприняты попытки исследования других замен в Y152 и Y199, которые можно комбинировать для получения более надежной молекулы. Кандидаты с комбинированными мутациями Y152 и Y199 eNKG2D исследовали в форме слитых с Fc белков и подробно описывали (таблица 1). Кроме того, для всех очищенных и экспрессированных кандидатов слитых белков Fc-eNKG2D получали профили посредством SEC, и их хроматограммы выявили различные уровни формирования агрегатов (фигуры 2 и 3, таблица 1). Из одиночных исследованных аминокислотных замен в остатке 152, аланин, серин, треонин и валин все не влияли на сборку молекулы Fc-NKG2D, хотя Y152-лейцин (Y152L) приводила к получению высоко агрегированного материала. Подобно аланину, ни глутамат, ни аспартат не являлись допустимыми в положении 199, хотя фенилаланин только умеренно увеличивал формирование агрегатов. Из исследованных комбинаций мутаций, Y152A/Y199F, Y152S/Y199F, Y152T/Y199F и Y152F/Y199F не оказывали отрицательного влияния на формирование желательного димера, в то время как другие комбинации приводили к увеличенной агрегации.
[0105] Пример 8: (Получение биспецифических молекул на основе антител, «MicAbodies», с использованием вариантов неприродного лиганда NKG2D)
[0106] Для получения вариантов неприродного MicA, слитого с IgG1 человека, ДНК-полинуклеотиды, кодирующие домены α1-α2, например, из MICwed (SEQ ID NO: 7) и MIC25 (SEQ ID NO: 31), амплифицировали посредством ПЦР с использованием праймеров, которые вводили также полинуклеотид, кодирующий либо линкер APTSSSGGGGS для слияния с C-концевой легкой цепью каппа (SEQ ID NO: 135), либо линкер GGGS для слияния с C-концевой тяжелой цепью IgG1 человека (SEQ ID NO: 136). Кроме того, две мутации вводили в домен CH2 тяжелой цепи - D265A/N297A (нумерация Kabat) - которые уменьшают связывание со всеми рецепторами FcγR, таким образом, прекращая функцию антителозависимой клеточной цитотоксичности (ADCC) (Shields et al., 2001 JBC, 276:6591-6604]. Полинуклеотид, кодирующий домен α1-α2 ULBP2 дикого типа (ULBP2.wt) без его GPI-связи (SEQ ID NO: 61), сходным образом клонировали и сливали с ДНК-полинуклеотидами, кодирующими линкеры и тяжелую цепь или легкую цепь IgG1. Эти биспецифические антитела - названные «MicAbodyTM» в единственном числе, «MicAbodies» во множественном числе - являются двухвалентными для слитого домена α1-α2. Примеры антител, используемых для получения MicAbodies для целей исследования конструирования eNKG2D, включают, но без ограничения, трастузумаб (SEQ ID NO: 137 и 138) и ритуксимаб (SEQ ID NO: 139 и 140) и следовательно, названы «трастузумаб-MicAbody» и «ритуксимаб-MicAbody», соответственно. Слитые конструкции вставляли индивидуально в pD2610-V12 (ATUM, Newark, CA) посредством клонирования способом Гибсона (New England Biolabs Inc., Ipswich, MA). Для данного антитела, узнающего специфический антиген, плазмиду, кодирующую тяжелую цепь, и плазмиду, кодирующую легкую цепь, слитые либо с природным, либо с неприродным лигандом NKG2D, совместно трансфицировали для временной экспрессии в клетках Expi293TM (ThermoFisher Scientific, Waltham, MA). Альтернативно, плазмиду, кодирующую тяжелую цепь, слитую либо с природным, либо с неприродным лигандом NKG2D, и плазмиду для легкой цепи совместно трансфицировали. Секретированные биспецифические антитела очищали посредством аффинной хроматографии с белком A (кат. no. 20334, Pierce Biotechnology, Rockford, IL), элюированный материал характеризовали посредством эксклюзионной хроматографии (SEC) на колонках Akta Pur Superdex, и фракционирование проводили по необходимости. Кроме того, анализ SDS-PAGE проводили для очищенных образцов для подтверждения ожидаемых молекулярных масс молекул, слитых с тяжелой цепью и слитых с легкой цепью.
[0107] Пример 9: (Идентификация модифицированных вариантов NK2GD, неспособных связываться либо с природными связывающими NKG2D лигандами, либо с неприродными лигандами, имеющих усиленное связывание с NKG2D дикого типа)
[0108] Аффинность связывания вариантов α1-α2 с внеклеточными доменами природного (дикого типа) NKG2D и неприродных белков eNKG2D анализировали с использованием способа ELISA на основе планшетов. Каждым из фракционированных посредством SEC слитых белков с природным Fc-NKG2D и неприродным Fc-eNKG2D покрывали в течение ночи при 4°C отдельные лунки 96-луночных планшетов Nunc Maxisorp (Thermo Fisher Scientific, Waltham, MA) с использованием концентрации покрытия 1 мкг/мл в фосфатно-солевом буфере (PBS). Планшеты промывали три раза в PBS/0,05% Tween-20 (PBS-T) at 20-22°C, и блокировали с использованием 0,5% бычьего сывороточного альбумина в PBS (PBS-B) в течение 2 часов при 20-22°C. MicAbodies титровали против связанных с планшетом природных или неприродных слитых белков Fc-NKG2D в течение 60 минут при 20-22°C в PBS/0,5% бычьем сывороточном альбумине (BSA)/0,05% Tween-20 (PBS-BT), промывали 3 раза с использованием PBS-T при 20-22°C, и связанные биспецифические белки детектировали с использованием конъюгированного с HRP антитела против каппа человека в PBS-BT (Abcam, Cambridge MA) и проявляли с использованием раствора субстрата 1-StepTM Ultra TMB ELISA (Thermo Fisher Scientific, Waltham, MA). Связывание ULBP2.wt ритуксимаб-MicAbody (SEQ ID NO: 139 и 141) выявляло различия между NKG2D дикого типа и вариантами eNKG2D с уменьшенным связыванием с последним, и варианты лигандов - MICwed (SEQ ID NO: 20 и 78) и MIC25 (SEQ ID NO: 138 и 80) - являлись более строгими при идентификации вариантов eNKG2D с прекращенным связыванием лиганда. Поведение связывания для каждого варианта eNKG2D против всех трех биспецифических лигандов выявило комбинации модификаций NKG2D, которые приводили к наибольшему уменьшению связывания лигандов дикого типа и вариантов лигандов, и позволяло отбор лидирующих инертных вариантов NKG2D.
[0109] Дополнительный биофизический анализ связывания вариантов eNKG2D с лигандами также проводили с использованием интерферометрии биослоев (BLI) с использованием системы Octet FortéBio (все FortéBio LLC, Fremont, CA). Для этих экспериментов лиганды человеческие NKG2D MICA-Fc, MICB-Fc, ULBP1-Fc, ULBP2-Fc, ULBP3-Fc и ULBP4-Fc закупали из R&D Systems, Inc. (Minneapolis, MN). Лиганды в формате MicAbody связывали на наконечниках биосенсора для связывания против Fc IgG человека (AHC). После установки фоновых значений, наконечники подвергали воздействию серий титрования слитых белков Fc-eNKG2D в диапазоне от 300 нМ до 0,41 нМ, и мониторировали кинетику связывания/диссоциации, где все стадии проводили в PBS-BT. Затем, слитые белки Fc-eNKG2D связывали на наконечниках AHC, и MicAbodies титровали для характеризации кинетики связывания.
[0110] Для определения максимального ответа, как определено посредством связывания природного NKG2D либо с MICwed, либо с MIC25, природные слитые белки Fc-NKG2D связывали на биосенсорах AHC, и 20 нМ трастузумаб-MICwed или 20 нМ трастузумаб-MIC25 MicAbodies инкубировали в течение двух минут, и затем кинетику диссоциации наблюдали в течение 30 секунд. Затем анализ связывания проводили в таких же условиях с использованием слитых рецепторов Fc-eNKG2D в качестве связывающего средства, и уровень связывания для каждого eNKG2D ранжировали как процент от максимального ответа связывания, полученного посредством Fc-NKG2D.wt (таблица 2). Для MICwed, ответы для всех вариантов Fc-eNKG2D с одиночной мутацией, за исключением Y199F, уменьшались до 50%. Y199F сохранял 100% ответ связывания. Однако, все варианты Fc-eNKG2D с двойной мутацией имели полностью прекращенное связывание с MICwed. Для MIC25, все варианты Fc-eNKG2D с одиночной мутацией и Y152V/Y199F сохраняли 100% ответ связывания, относительно связывания Fc-NKG2D дикого типа. Однако, связывание уменьшалось до 50% для нескольких из вариантов Fc-eNKG2D с двойной мутацией, включая Y152A/Y199F, Y152S/Y199F и Y152T/Y199F.
[0111] Анализы ELISA с использованием слитых белков Fc-eNKG2D в качестве связывающих средств проводили для ULBP2.wt, MICwed, MIC25 MicAbodies, титрованных, начиная с 300 нМ. Значения EC50 рассчитывали, когда возможно, с использованием GraphPad Prism (таблица 11).
Таблица 11: значения EC50 (нМ) для ELISA Fc-eNKG2D. nt=не тестировали; nb=отсутствие связывания или очень низкое связывание даже при 300 нМ, таким образом, значение EC50 не рассчитывали
[0112] Природный NKG2D связывался с MicAbodies на основе ULBP2, MICwed и MIC25 с аффинностями, рассчитанными как значения Kd 1,4, 0,007 и 0,005 нМ, соответственно. В то время как аффинность уменьшалась для MicAbodies ULBP2 и MICwed, при использовании всех кандидатов eNKG2D с одиночной мутацией, связывание MIC25 с кандидатами eNKG2D сохранялось. Однако все кандидаты eNKG2D с двойной мутацией имели прекращенное или значительно уменьшенное связывание со всеми тремя лигандами - ULBP2, MICwed и MIC25 - в форматах Micabody.
[0113] Варианты eNKG2D eNKG2D5 (Y152A/Y199F), eNKG2D7 (Y152S/Y199F), eNKG2D8 (Y152T/Y199F) и eNKG2D9 (Y152V/Y199F) имели уменьшенное или прекращенное связывание с MicAbodies на основе ULBP2, MICwed и MIC25 как по анализу Octet, так и по ELISA (таблица 2 и 3). Кроме того, eNKG2D 5, 7 и 8 имели наименьший уровень агрегации, что позволяет предполагать более надежную сборку белка при экспрессии в 293T (таблица 1). eNKG2D5 (SEQ ID NO: 102) исследовали более подробно по связыванию с лигандами дикого типа, в форме MicAbodies, связанных на наконечниках Octet AHC. Fc-NKG2D.Y152A с одиночной мутацией (SEQ ID NO: 95) имел уменьшенное связывание со всеми природными лигандами, относительно природного (SEQ ID NO: 94) NKG2D (фигура 5). Кривые ответа для связывания eNKG2D5 (Y152A/Y199F) были уменьшены даже дополнительно, относительно Y152A eNKG2D. eNKG2D5 (Y152A/Y199F, в дальнейшем обозначенный как «AF» или «NKG2D.AF»), выбран в качестве лидирующего варианта NKG2D, для которого конструировали родственные избирательные, ортогональные, неприродные лиганды.
[0114] Пример 10: (Конструирование ортогональных неприродных доменов α1-α2 с избирательным связыванием с неприродным эктодоменом NKG2D.AF)
[0115] Авторы настоящего изобретения использовали фаговый дисплей для конструирования ортогональных неприродных доменов α1-α2, имеющих избирательное связывание с рецептором NKG2D.AF (SEQ ID NO: 102). В качестве исходной точки, неприродный домен α1-α2 ULBP2.R80W (фигура 1B; SEQ ID NO: 142) с высокой аффинностью для природного, дикого типа эктодомена NKG2D (NKG2D.wt) выбран в качестве исходного домена для дальнейшего мутагенеза и скрининга посредством фагового дисплея. Библиотеки синтетической ДНК получали для домена α1-α2 ULBP2.R80W (SEQ ID NO: 108), который, кроме того, имеет мутацию C8S для исключения потенциала для дисульфидных связей. Кодоны аминокислотных остатков лиганда, которые в связанном состоянии расположены в тесной близости с положениями Y152 и Y199 на природном рецепторе NKG2D, заменяли на кодоны NNK; библиотеки состояли из кодонов NNK в положениях 154-159. Библиотеки клонировали в форме слитых белков с минорным белком оболочки pIII фага M13, и частицы фага, экспонирующие подвергнутые мутагенезу варианты домена α1-α2, продуцировали в клетках E.coli SS320, в соответствии со стандартными способами (Andris-Widhopf, J., Steinberger, P., Fuller, R., Rader, C., and Barbas, C. F., 3rd ed. (2011). Эти библиотеки фагового дисплея α1-α2 сортировали по высокоаффинному связыванию с неприродным рецептором NKG2D.AF посредством избирательного захвата клонов фагов, связанных с биотинилированным белком Fc-NKG2D.AF в присутствии небиотинилированного природного конкурентного белка Fc-NKG2D.wt. Избирательные клоны обогащали посредством подвергания множеству циклических раундов конкурентного отбора с использованием увеличивающихся концентраций небиотинилированного природного Fc-NKG2D.
[0116] После четырех раундов отбора, получали массив индивидуальных клонов в 96-луночном формате, проводили спот-ELISA для подтверждения предпочтительного дифференциального связывания для связанных с планшетом неприродных NKG2D.AF против NKG2D.wt. Связанные фаги детектировали с использованием биотинилированного моноклонального антитела E1 против белка M13 оболочки фага (ThermoFisher Scientific, Waltham, MA), детекции посредством стрептавидина-HRP (R&D Systems, Minneapolis, MN) и проявления посредством 1-Step Ultra TMB ELISA (ThermoFisher Scientific, Waltham, MA). Сигнал спот-ELISA для каждого клона выражали как соотношение связывания фага с NKG2D.AF со связыванием фага с NKG2D.wt. Фаги с соотношением, большим или равным 14, секвенировали для идентификации специфических мутаций внутри подвергнутых мутагенезу посредством NNK областей. В случаях, когда идентифицировали множество клонов, представляющих одинаковую последовательность, соотношение сигналов ELISA наносили на график, и согласованность клонов фагов подтверждали посредством кластеризации точек данных (данные не представлены).
[0117] Тридцать вариантов, идентифицированные в ELISA, размножали в индивидуальных монокультурах для получения микропартий фага с высоким титром. Концентрации очищенного фага нормализовали до OD268=0,5, затем подвергали серийным разведениям 1:3 против связанных с планшетом Fc-NKG2D.AF или Fc-NKG2D.wt с детекцией фага и проявлением ELISA, проведенными, как описано выше. Для всех тридцати вариантов, анализированных этих способом, постоянно демонстрировали избирательное связывание с NKG2D.AF, со связыванием от небольшого до отсутствующего с NKG2D.wt, даже при наивысших анализированных концентрациях фага. Для отобранных фагов также показан сдвиг на два или более log концентрации фага для достижения половины максимального связывания между NKG2D.AF и NKG2D.wt.
[0118] Для подтверждения того, что избирательные для NKG2D.AF варианты домена α1-α2 сохраняли свойства специфического связывания в контексте слитых с антителом белков, 21 вариант (таблица 5; например, SEQ ID NO: 143-150) клонировали в форме слитых с C-конца белков с линкером APTSSSGGGGS с легкой цепью антитела ритуксимаба. Полученные слитые белки клонировали в экспрессирующий вектор для млекопитающих pD2610-V12 (ATUM, Newark, CA) посредством клонирования способом Гибсона (New England Biolabs Inc., Ipswich, MA) и совместно экспрессировали с тяжелой цепью исходного антитела в форме спаренных полноразмерных антител IgG. Временную экспрессию осуществляли в клетках Expi293TM (ThermoFisher Scientific, Waltham, MA) в соответствии с протоколом производителя, и очищали с использованием стандартной аффинной хроматографии с белком A (кат. no. 20334, Pierce Biotechnology, Rockford, IL). ELISA, измеряющие связывание каждого варианта слитых с антителом белков ULBP2 α1-α2 с неприродными Fc-NKG2D.AF и с природным Fc-NKG2D.wt, показали их значительно большую аффинность связывания для NKG2D.AF, относительно природного NKG2D.wt (таблица 12).
Таблица 12: Специфичность отобранных на NKG2D.AF вариантов ULBP2 в формате ритуксимаб-MicAbody сохраняли свое связывание с NKG2D.AF по количественному ELISA. Показаны специфические аминокислотные модификации каждого варианта ULBP2, так же как соотношения их связывания со слитым белком Fc-NKG2D.wt против слитого белка Fc-NKG2D.AF.
[0119]
[0120]
[0121] Совместно, эти данные показали изобретение неприродных, ортогональных доменов α1-α2, имевших высокоаффинное связывание с неприродным рецептором NKG2D.AF и значительно уменьшенную аффинность связывания с природным рецептором NKG2D. Кроме того, слитые белки этих ортогональных доменов α1-α2 с полипептидами антител сохраняли свои свойства избирательного связывания и были использованы, например, в контексте T-клеток с химерным рецептором антигена (CAR), для перенацеливания неприродных рецепторов NKG2D.AF на специфические антигены.
[0122] Пример 11: (Идентификация неприродных лигандов NKG2D, которые могут устанавливать различия между вариантами неприродного рецептора NKG2D посредством избирательного связывания одного или другого)
[0123] Фаговый дисплей для конструирования ортогональных неприродных доменов α1-α2 с избирательным связыванием с рецептором NKG2D.Y152A (в дальнейшем обозначенным как NKG2D.YA, проводили с использованием неприродного домена α1-α2 ULBP2.R80W (SEQ ID NO: 142) в качестве исходной точки, как описано выше. Библиотеки фагового дисплея α1-α2 подвергали пэннингу по высокоаффинному связыванию с неприродным рецептором Fc-NKG2D.YA посредством избирательного захвата клонов фагов, связанных с биотинилированным белком Fc-NKG2D.YA (SEQ ID NO: 95) в присутствии конкурентного белка небиотинилированного природного Fc-NKG2D.wt (SEQ ID NO: 94). Дополнительная работа по подтверждению клонов фагов привела к идентификации вариантов с предпочтительным связыванием с Fc-NKG2D.YA против Fc-NKG2D.wt (таблица 13).
[0124] Таблица 13: Отобранные мутации в указанных локализациях аминокислот ULBP2.R80W (SEQ ID NO: 132) , приведшие к получению специфических для Y152A клонов фагов.
[0125] Для ULBP2.S3 (SEQ ID NO: 151), например, постоянно показывали избирательное связывание посредством ELISA и анализа Octet (оба в формате мономерного меченного His и биспецифического слитого антитела) с неприродным NKG2D.YA, относительно природного NKG2D.wt. Это представляло собой отдельную форму по изобретению неприродных ортогональных доменов α1-α2, имеющих высокоаффинное связывание с неприродными рецепторами NKG2D (в этом случае, NKG2D.YA, в отличие от NKG2D.AF, как в примере 2). Кроме того, слитые белки ортогональных доменов α1-α2 с полипептидами антител сохраняли свои свойства избирательного связывания и были использованы для избирательного перенацеливания неприродных рецепторов NKG2D на специфические молекулы, определенного посредством слитых гетерологичных пептидов, таких как антитела.
[0126] Для определения того, могут ли неприродный домен α1-α2 с избирательным связыванием с NKG2D.YA (ULBP2.S3, SEQ ID NO: 151) и неприродные домены α1-α2 с избирательным связыванием с NKG2D.AF устанавливать различия между этими двумя вариантами неприродного рецептора, проводили титрующие ELISA. Все 21 из отобранных вариантов α1-α2, которые связывали NKG2D.AF, напрямую сравнивали по связыванию с NKG2D.AF против NKG2D.YA. Из них, для четырех показаны свойства неспособности связывать NKG2D.wt, сильную аффинность для NKG2D.AF, и сильно уменьшенное (в 15-20 раз) или прекращенное связывание с NKG2D.YA, относительно NKG2D.AF. Эти четыре неприродных варианта α1-α2 ULBP2 - ULBP2.C, ULBP2.R, ULBP2.AA и ULBP2.AB (SEQ ID NO: 143, 145, 147 и 149) - также проверяли по изменениям прогнозированного профиля иммуногенности, относительно пептидной последовательности ULBP2 дикого типа (SEQ ID NO: 61), с использованием сервера NetMHC4.0 (для запроса связывания пептида-MHC класса I против всех представителей супертипа HLA с использованием анализа 9-членных пептидов; http://www.cbs.dtu.dk/services/NetMHC/) и сервера NetMHCII 2.3 (для запроса связывания пептида-MHC класса II против гаплотипов HLA-DR, HLA-DQ, HLA-DP с использованием анализа 15-членных пептидов; http://www.cbs.dtu.dk/services/NetMHCII/), оба алгоритма разработаны в Technical University of Denmark (http://www.bioinformatics.dtu.dk/; Andreatta M and Nielsen M, Gapped sequence alignment using artificial neural networks: application to the MHC class I system, 2016 Bioinformatics, 32:511, PMID: 26515819; Jensen KK, Andreatta M, Marcatili P, Buus S, Greenbaum JA, Yan Z, Sette A, Peters B, and Nielsen M, Improved methods for predicting peptide binding affinity to MHC class I molecules, 2018 Immunology, PMID: 29315598). Мутации, включенные в ULBP2.C, ULBP2.R и ULBP2.AB, не увеличивали прогнозированную иммуногенность, в то время как иммуногенность ULPB2.AA была слабо увеличена для нескольких гаплотипов (фигуры 8 и 9). Как следствие специфичности ULBP2.R для NKG2D.AF и отсутствия у него прогнозированной иммуногенности, ULBP2.R был отобран для дополнительного анализа ELISA для непосредственного сравнения его поведения связывания с поведением ULBP2.S3 (отобранного посредством NKG2D.YA, неприродного, ортогонального лиганда), ULBP2.R80W (неприродного лиганда с увеличенной аффинностью для NKG2D дикого типа) и ULBP2 дикого типа (ULBP2.wt). Связывание четырех реагентов ритуксимаб-MicAbody (SEQ ID NO: 139 и 151, 139 и 152, 153 и 140, и 139 и 141 в качестве тяжелой цепи и легкой цепи для ULBP2.R, ULBP2.S3, ULBP2.R80W и ULBP2.wt, соответственно) анализировали против NKG2D дикого типа (NKG2D.wt) и двух инертных, неприродных вариантов NKG2D.YA и NKG2D.AF. Данные показали, что отобранный посредством NKG2D.YA вариант ULBP2.S3 в форме MicAbody связывался с высокой аффинностью с NKG2D.YA, но не привлекал NKG2D.AF или природный NKG2D. Кроме того, отобранный посредством NKG2D.AF вариант ULBP2.R в формате MicAbody связывался с высокой аффинностью с NKG2D.AF, но не привлекал NKG2D.YA или природный NKG2D. Эти результаты показали огромный потенциал исследования оси NKG2D-лиганд MIC и разработки уникальных пар новых, избирательных неприродных рецепторов NKG2D и их соответствующих, родственных неприродных партнеров по связыванию лигандов MIC.
[0127] Пример 12: (Активность нацеливания и уничтожения CAR-T-клеток, экспрессирующих неприродный эктодомен NKG2D.AF, контролируют посредством ортогональных доменов α1-α2, слитых с гетерологичными нацеливающими полипептидами)
[0128] Средства для избирательного контроля видов CAR-T-клеточной терапии очень востребованы для ослабления токсичности и улучшения эффективности против опухолей (Gill and June, в цитируемом документе). Предшествующие попытки предпринимали для разработки CAR с использованием эктодомена CD16, который можно было затем привлекать посредством домена Fc терапевтических моноклональных антител, позволяя основанный на антителах контроль нацеливания CAR-T (Chang et al., в цитируемом документе). Однако, основанный на CD16-CAR-T-клетки могут узнавать почти все эндогенные молекулы антитела в крови и тканях, и терапевтические антитела, используемые для контроля этих клеток, могут сталкиваться с конкуренцией со стороны эндогенных рецепторов CD16 на клетках NK, PMN, моноцитах и макрофагах. Оба из этих признаков вносят вклад в проблемы не специфической для опухолей токсичности и плохой фармакокинетики, соответственно.
[0129] Природные лиганды NKG2D присутствуют на конкретных здоровых тканях и многих подверженных стрессу тканей, создавая необычайный риск токсичности при использовании современных способов NKG2D-CAR (VanSeggelen et al., 2015). Неприродный рецептор NKG2D Y152A специфически связывался с неприродным доменом α1-α2 лигандов NKG2D, составляя пример средств, посредством которых активность неприродный NKG2D-CAR можно избирательно контролировать с использованием биспецифических белков, содержащих изобретательский неприродный домен α1-α2 лигандов NKG2D.
[0130] Авторы настоящего изобретения сконструировали CAR-T-клетки с рецептором, содержащим модифицированный эктодомен Y152A/Y199F («AF») NKG2D, лишенный связывания со всеми природными лигандами NKG2D, или описанные ранее неприродные домены α1-α2, ортогональные и родственные модифицированному NKG2D Y152A (NKG2D.YA). Изобретательские родственные неприродные домены α1-α2 связывали с высокой аффинностью неприродный эктодомен NKG2D.AF и избегали связывания с природными эктодоменами NKG2D и с эктодоменом NKG2D.YA. Таким образом, сконструированные домены α1-α2, имеющие сильную избирательность для неприродного эктодомена NKG2D.AF, по сравнению с природным NKG2D и неприродным NKG2D.YA, представляют идеальную систему для избирательного контроля рецепторов неприродный NKG2D-CAR, или любого рецептора или белка, слитого с неприродными эктодоменами NKG2D, который может избирательно привлекаться посредством неприродных доменов α1-α2 по настоящему изобретению. Настоящее изобретение, кроме того, обеспечивает возможность отдельных клеток, экспрессирующих два отдельных CAR - один, содержащий NKG2D.YA, и другой NKG2D.AF - каждый передающий сигналы с использованием совершенно различных внутриклеточных доменов. Эти различные CAR могут иметь независимый, двойной контроль активности клеток посредством внеклеточного воздействия соответствующего, родственного ортогонального MicAbody или другого слитого не с антителом полипептида.
[0131] Чтобы продемонстрировать избирательный контроль CAR-T-клеток, модифицированных с использованием химерного рецептора с внедренным неприродным эктодоменом NKG2D.AF, авторы настоящего изобретения сконструировали CAR с природным NKG2D.wt (SEQ ID NO: 49), неприродным NKG2D.YA (SEQ ID NO: 54) или неприродным NKG2D.AF (SEQ ID NO: 154) эктодоменами но основании предшествующей работы с использованием конструкций 4-1BB/CD3-дзета-CAR (Campana, Патент 8399645), сливающих соответствующие эктодомены NKG2D с шарнирной областью CD8 из CAR (SEQ ID NO: 155, 157, 159). Эти конструкции (SEQ ID NO: 156, 158, 160) клонировали в лентивирусный вектор и экспрессировали в первичных положительных по CD8 T-клетках человека с использованием лентивирусной трансдукции. Клетки HeLa имеют подвергнутые повышающей регуляции уровни лигандов MIC на своей поверхности, включая MICA, MICB, ULBP3 и ULBP2/5/6 (антитело, используемое для установления этого, не может устанавливать различия между этими тремя ULBP; антитело против ULBP-2/5/6 человека, R&D Systems, Minneapolis, MN). Клетки HeLa трансфицировали, чтобы также экспрессировать либо природный ULBP1, либо отобранный посредством NKG2D.AF вариант ULBP2.R на их поверхности, и эти клетки использовали в качестве мишени для анализов уничтожения in vitro. Клетки-мишени HeLa предварительно нагружали кальцеином и подвергали воздействию NKG2D.wt-CAR-, NKG2D.YA-CAR- или NKG2D.AF-CAR-CD8 клеток при увеличивающихся соотношениях эффектора к мишени (E:T) в течение пяти часов, после чего количество кальцеина, высвобожденное в супернатант, количественно определяли и нормализовали по общему количеству кальцеина, высвобождаемого при обработке детергентом. Из-за увеличенных уровней лигандов MIC, естественным образом экспрессированных на поверхности клеток HeLa, CD8 клетки, экспрессирующие природный NKG2D (NKG2D.wt) в качестве CAR, привлекали клетки HeLa посредством этого сверхэкспрессированного природного лиганда и осуществляли цитолиз. Однако, для трансдуцированных с использованием как NKG2D.YA-, так и NKG2D.AF-CAR-CD8 клеток показан очень небольшой лизис природных клеток HeLa даже при высоких соотношениях E:T, с уровнем активности, на равных с нетрансдуцированными CD8 T-клетками. Когда ULBP1 сверхэкспрессировали на поверхности клеток HeLa, только NKG2D.wt-CAR-CD8 T-клетки значительно лизировали их. Присутствует некоторое дополнительное уничтожение при высоком соотношении E:T, с использованием NKG2D.YA-CAR-клеток, но этого не существует при использовании NKG2D.AF-CAR-клеток, что показывает, что двойная мутация Y152A/Y199F делает NKG2D даже более инертным, чем одиночная мутация Y152A. Для клеток HeLa, сверхэкспрессирующих избирательный по отношению к NKG2D.AF неприродный ULBP2.R, NKG2D.wt-CAR-клетки вызывают лизис (из-за узнавания эндогенных лигандов MIC), в то время как NKG2D.AF-CAR-клетки вызывают значительные уровни лизиса, в соответствии с привлечением рецептора и избирательного для него лиганда.
[0132] Чтобы показать, что лизис либо NKG2D.YA-, либо NKG2D.AF-CAR-клеток можно вызывать только посредством соответствующего, родственного нацеливающего MicAbody, клетки Ramos использовали в качестве мишени для цитолиза в комбинации с MicAbodies на основе ритуксимаба, связанных с неприродными ULBP2.S3 или ULBP2.R ортогональными лигандами. MicAbody ритуксимаб-ULBP2.S3 могло управлять активностью уничтожения клеток для NKG2D.YA-CAR-CD8 клеток, но не для NKG2D.AF-CAR-клеток, в то время как MicAbody ритуксимаб-ULBP2.R могло управлять активностью NKG2D.AF-CAR-, но не NKG2D.YA-CAR-клеток. Это дополнительно показывает избирательность двух неприродных вариантов ULBP2 для родственных им неприродных вариантов NKG2D, для которых они были сконструированы в качестве предпочтительных партнеров. Чтобы показать специфичность части антитела из MicAbody, проводили анализы уничтожения in vitro с использованием NKG2D.AF-CAR-CD8 клеток, которые предварительно вооружали посредством инкубации с ритуксимаб-ULBP2.R, трастузумаб-ULPB2.R (SEQ ID NO: 95 и 133, тяжелая и легкая цепь, соответственно), или эквимолярной комбинации этих двух в насыщающей общей концентрации MicAbody. После удаления несвязанного MicAbody посредством промывки, CD8 клетки вводили либо к клеткам Ramos (экспрессирующим CD20, мишень ритуксимаба), либо к CT26-Her2 (линии клеток мыши, трансфицированной для экспрессии Her2 человека), предварительно нагруженным кальцеином. После двух часов инкубации в двух различных соотношениях E:T, количество высвобожденного кальцеина оценивали количественно. Когда клетки предварительно вооружали с использованием ритуксимаб-MicAbody, только клетки Ramos подвергались лизису, в то время как трастузумаб-MicAbody направляло цитолитическую активность только против CT26-Her2 клеток. Однако, когда NKG2D.AF-CAR-CD8 клетки одновременно предварительно вооружали с использованием обоих MicAbodies ритуксимаб- и трастузумаб-ULBP2.R, обе линии клеток-мишеней подвергались лизису, что показывает, что эти CAR-клетки - посредством избирательного, привилегированного партнерства, сконструированного между рецептором и лигандом - легко становились мультиплексными и таким образом, нацеленными для привлечения к различным мишеням опухолей одновременно.
[0133] Пример 13: (Уничтожение тонзиллярных CD4 T-клеток человека, продуктивно инфицированных HIV) CD8+ T-клетки выделяли из PBMC здоровых доноров, активировали посредством бусин против CD3/CD28, и трансдуцировали с использованием CAR, состоящего из инертного NKG2D, шарнира и трансмембранного домена CD8, костимулирующего домена 4-1BB и CD3ξ. Эти CAR-T-клетки обозначены как конвертируемый CAR-клетки. Эти конвертируемый CAR-T-клетки являлись способными только к непрямому связыванию с антителами с широким спектром нейтрализации HIV, слитыми с модифицированным, неприродным лигандом, родственным инертному рецептору NKG2D конвертируемого CAR. Нетрансдуцированные CD8 T-клетки от того же донора, также получали параллельно в качестве отрицательного контроля. Получали четыре специфических для HIV MicAbodies на основании последовательности нейтрализующих антител широкого спектра 3BNC60, 3BNC117, PGT121 и 10-1074 (SEQ ID NO. 161 и 162 (3BNC60), тяжелая и легкая цепи MicAbody соответственно; 163 и 164 (3BNC117) тяжелая и легкая цепи MicAbody, соответственно; 165 и 166 (PGT121) тяжелая и легкая цепи MicAbody, соответственно; 167 и 168 (10-1074) тяжелая и легкая цепи MicAbody, соответственно). Эти MicAbodies связываются со специфическими эпитопами молекул gp160 оболочки HIV. Целевой эпитоп, связываемый 3BNC60 и 3BNC117, представляет собой SEQ ID NO.:169; посредством PGF12 и 10-1074 представляет собой SEQ ID NO.: 170; (Deng K, Pertea M, Rongvaux A, Wang L, Durand CM, Ghiaur G, Lai J, McHugh HL, Hao H, Zhang H, JB, Gurer C, Murphy AJ, Valenzuela DM, Yancopoulos GD, Deeks SG, Strowig T, Kumar P, Siliciano JD, Salzberg SL, Flavell RA, Shan L, Siliciano RF Broad CTL response is required to clear latent HIV-1 due to dominance of escape mutations. Nature. 2015 Jan 15;517(7534) p. 381-5.). MicAbodies, нацеливающие на CD20 или HER2, также задействовали в качестве отрицательного контроля.
[0134] Тонзиллярные клетки человека от 4 здоровых доноров перерабатывали для получения культуры лимфоидных агрегатов человека (HLAC). Клетки HLAC наслаивали на клетки 293T, предварительно трансфицированные ДНК, соответствующей R5-тропному HIV-1 и репортерному гену GFP. Через 24 час, клетки HLAC удаляли, и позволяли продолжение распространения инфекции HIV в течение еще 4 суток. Положительные по GFP инфицированные HLAC клетки затем подвергали воздействию нетрансдуцированных CD8 T-клеток или конвертируемый CAR-T-клеток, вооруженных указанными MicAbodies, и культивировали в течение 48 часов в присутствии 5 мкМ саквинавира для предотвращения вирусного распространения. Затем клетки собирали посредством центрифугирования, промывали и окрашивали для оценки жизнеспособности инфицированных и неинфицированных клеток с использованием проточного цитометра LSRII.
[0135] Оценка соотношений клеток эффектор:мишень (E:T) для уничтожения инфицированных HIV первичных CD4 T-клеток посредством CAR-T-клеток с использованием различных концентраций специфических нацеленных на HIV MicAbodies. Как описано выше, один миллион первичных происходящих из миндалин клеток, инфицированных вирусом Bal-GFP R5 (~10% инфекция; 1X104 инфицированных клеток) инкубировали с использованием 1X105 нетрансдуцированных CD8 (0:1) или с использовать 1X104 (1:1) или 2X105 (20:1) CAR-T-клеток в присутствии различных концентраций четырех различных MicAbodies с широким спектром нейтрализации HIV. Клетки окрашивали через 24 час и оценивали посредством проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- клеткам, либо экспрессирующим, либо не экспрессирующим GFP. Результаты, усредненные для 3 исследований, показаны на фигуре 7. В этих исследованиях, комбинация специфических для HIV MicAbodies и конвертируемый CAR-T-клеток приводило к специфическому уничтожению тонзиллярных клеток, инфицированных вирусом R5 HIV. Оптимальное соотношение эффектор:мишень для уничтожения лежало в диапазоне между 1:1 и 10:1, без уменьшения жизнеспособности неинфицированных клеток. Уничтожение являлось сильно ограниченным инфицированными клетками, т.е. клетками, экспрессирующим GFP. Для GFP-клеток, присутствующих в той же культуре, показано небольшое уменьшение или отсутствие уменьшения количества клеток (фигуры B и C; GFP+ против GFP-). Кроме того, не происходило уничтожения неинфицированных клеток, и не происходило уничтожения инфицированных клеток, когда использовали совпадающие по донору нетрансдуцированные CD8 T-клетки или не нацеливающие на HIV- MicAbodies (например, нацеливающее на CD20 MicAbody или нацеливающее на Her2 MicAbody)
[0136] Специфическое уничтожение инфицированных вирусом R5 первичных CD4 клеток посредством CAR-T в комбинации со специфическим для HIV MicAbody. Один миллион первичных происходящих из миндалин клеток, инфицированных Bal-GFP вирусом R5 (~1X104 инфицированных клеток), инкубировали с использованием 1X105 CAR-T-клеток в присутствии различных концентраций специфических для HIV MicAbodies или специфического для B-клеток нацеливающего на CD20 MicAbody, или нацеливающего на HER2 MicAbody (Her2). Клетки окрашивали через 24 час и анализировали посредством проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- и либо GFP+, либо GFP-. Результаты, усредненные для 4 исследований, показаны на фигуре 8.
[0137] Специфическое уничтожение инфицированных перенесенным вирусом/вирусом-основателем F4 первичных CD4 клеток посредством CAR-T в комбинации со специфическим для HIV MicAbody. Один миллион первичных происходящих из миндалин клеток, инфицированных вирусом F4-GFP (T/F) (~1X104 инфицированных клеток) инкубировали с 1X105 конвертируемый CAR-T-клеток в присутствии различных концентраций 4 различных специфических для HIV MicAbodies, нацеливающего на CD20 MicAbody (Ritux) или нацеливающего на HER2 MicAbody (Her2). Клетки окрашивали через 24 час и затем подвергали проточной цитометрии. Клетки отбирали по отдельным клеткам/живым/CD3+/CD8- и либо GFP+, либо GFP-. Результаты показаны на фигуре 9. Эффективное уничтожение наблюдали, когда клетки инфицировали вирусом R5 или инфицировали перенесенным вирусом/вирусом-основателем F4 HIV, представляющим собой вирусный штамм, который успешно передается горизонтально от одного индивидуума к другому.
[0138] Пример 14 (Уничтожение посредством CAR-T и MicAbody реактивированных латентно инфицированных клеток-резервуаров от пациентов с авиремией, хронически инфицированных HIV и подвергаемых ART)
Мононуклеарные клетки периферической крови (PBMC) от 6 положительных по HIV индивидуумов с авиремией, подвергаемых ART, получали посредством непрерывного проточного центрифугирования лейкафереза, с последующим центрифугированием клеток в градиентов плотности фиколла-гипака. Затем покоящиеся CD4+ T-лимфоциты выделяли посредством «бесконтактного» отрицательного отбора посредством антител. Клетки культивировали в среде RPMI, дополненной 10% эмбриональной бычьей сывороткой и пенициллином/стрептомицином. 10 миллион покоящихся CD4+ лимфоцитов стимулировали с использованием 80 нМ PMA+1 мкМ иономицина в течение 72 часов. После реактивации, клетки инкубировали в течение 48 час с CAR-T- или совпадающими по донору нетрансдуцированными CD8 клетками с различными MicAbodies в присутствии 5 мкМ саквинавира. Клетки собирали центрифугированием при 300 g в течение 10 минут. Осадки клеток затем лизировали, и РНК выделяли с использованием набора RNeasy (Qiagen). Систему для одностадийной RT-ПЦР Superscript III использовали для получения кДНК и одновременной предварительной амплификации вирусной мРНК (т.е. 10 циклов предварительной амплификации) перед анализом и количественной оценкой посредством цифровой капельной ПЦР (цкПЦР). CD4+ T-клетки, выделенные посредством бесконтактного отрицательного отбора из PBMC, собранных от известных инфицированных HIV пациентов, подвергаемых ART и реактивированных в течение 72 час с использованием 100нМ форбол-миристат-ацетата (PMA) + 1 мкМ иономицина. Затем клетки промывали дважды и инкубировали в течение 48 часов с использованием конвертируемый CAR-T-клеток или нетрансдуцированных CD8 T-клеток в присутствии 0,1 или 1 нМ смеси равных концентраций MicAbodies на основе bNAb HIV (3BNC60, 3BNC117, PGT121 и 10-1074), обозначенной MIX на фигуре. Клетки затем центрифугировали, и РНК выделяли из осадков клеток. Ассоциированную с клетками РНК HIV измеряли посредством цкПЦР. Результаты показаны на фигуре 10.
В исследованиях из этого примера реактивации латентных клеток-резервуаров (3 суток с PMA+иономицин) от инфицированных индивидуумов с авиремией, подвергаемых ART (n=6), авторы настоящего изобретения наблюдали, что CAR-T-клетки являлись способными эффективно уменьшать количество этих реактивированных клеток-резервуаров приблизительно на 50%, по сравнению с совпадающими по донору нетрансдуцированными CD8 T-клетками+смесь MicAbodies. Размер индуцируемого резервуара оценивали посредством количественной оценки ассоциированной с клетками РНК HIV в присутствии и в отсутствие индуктора и эффекторных клеток с использованием цкПЦР.
Эти обнаружения из примеров 13 и 14 совместно обеспечивают ex vivo доказательство концепции, что конвертируемый CAR-T-клетки плюс родственные MicAbodies, конструированные с использованием нейтрализующих антител IgG1 человека широкого спектра, можно использовать в качестве нового, эффективного и высоко избирательного способа уничтожения для уничтожения успешно реактивированных инфицированных HIV клеток внутри латентного резервуара HIV-1.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> XYPHOS BIOSCIENCES INC.
<120> МОДИФИЦИРОВАННЫЕ НЕПРИРОДНЫЕ ЛИГАНДЫ NKG2D, КОТОРЫЕ ИЗБИРАТЕЛЬНО
ДОСТАВЛЯЮТ ПРИСОЕДИНЕННЫЕ ГЕТЕРОЛОГИЧНЫЕ МОЛЕКУЛЫ К НЕПРИРОДНЫМ
РЕЦЕПТОРАМ NKG2D НА CAR-КЛЕТКАХ
<130> F252863
<150> US 62/797644
<151> 2019-01-28
<160> 170
<170> PatentIn версии 3.5
<210> 1
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 1
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr Leu Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 2
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 2
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Ile Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 3
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 3
Glu Pro His Ser Leu Pro Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 4
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 4
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Glu Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 5
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 5
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 6
<211> 274
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 6
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Glu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser
<210> 7
<211> 276
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA
<400> 7
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Cys Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser Gly Lys
275
<210> 8
<211> 306
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 8
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Ala Lys Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Leu Pro Pro Met Val Asn Val
165 170 175
Ile Cys Ser Glu Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala
180 185 190
Ser Ser Phe Tyr Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly
195 200 205
Val Ser Leu Ser His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp
210 215 220
Gly Asn Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly
225 230 235 240
Glu Glu Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly
245 250 255
Thr His Pro Val Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg
260 265 270
Thr Asp Phe Pro Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile
275 280 285
Ile Ile Leu Cys Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu
290 295 300
Gly Pro
305
<210> 9
<211> 318
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 9
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Ala Glu Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Met His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asn Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Arg Tyr Leu Lys Ser Gly Val
165 170 175
Ala Ile Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Cys Ser Glu
180 185 190
Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg Thr Asp Phe Pro
275 280 285
Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile Ile Ile Leu Cys
290 295 300
Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu Gly Pro
305 310 315
<210> 10
<211> 318
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 10
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Ala Lys Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Arg Tyr Leu Lys Ser Gly Val
165 170 175
Ala Ile Arg Arg Thr Val Pro Pro Met Val Asn Val Ile Cys Ser Glu
180 185 190
Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg Thr Asp Phe Pro
275 280 285
Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile Ile Ile Leu Cys
290 295 300
Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu Gly Pro
305 310 315
<210> 11
<211> 318
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 11
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asn Val Leu Gly Ala Lys Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Arg Tyr Leu Lys Ser Gly Val
165 170 175
Ala Ile Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Cys Ser Glu
180 185 190
Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg Thr Asp Phe Pro
275 280 285
Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile Ile Ile Leu Cys
290 295 300
Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu Gly Pro
305 310 315
<210> 12
<211> 318
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 12
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Ala Glu Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asn Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Arg Tyr Leu Lys Ser Gly Val
165 170 175
Ala Ile Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Cys Ser Glu
180 185 190
Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly Glu Glu Gln Lys
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg Thr Asp Phe Pro
275 280 285
Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile Ile Ile Leu Cys
290 295 300
Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu Gly Pro
305 310 315
<210> 13
<211> 318
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICB
<400> 13
Glu Pro His Ser Leu Arg Tyr Asn Leu Met Val Leu Ser Gln Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Ala Glu Gly His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Tyr Asp Arg Gln Lys Arg Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Ala Glu Thr Trp Asp Thr Glu Thr Glu
50 55 60
Asp Leu Thr Glu Asn Gly Gln Asp Leu Arg Arg Thr Leu Thr His Ile
65 70 75 80
Lys Asp Gln Lys Gly Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Ser Ser Thr Arg Gly Ser Arg His Phe Tyr Tyr
100 105 110
Asn Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Gln Glu Ser Thr
115 120 125
Val Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Thr Asn
130 135 140
Phe Trp Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr Arg Ala Met
145 150 155 160
Gln Ala Asp Cys Leu Gln Lys Leu Gln Arg Tyr Leu Lys Ser Gly Val
165 170 175
Ala Ile Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Cys Ser Glu
180 185 190
Val Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Ser Phe Tyr
195 200 205
Pro Arg Asn Ile Thr Leu Thr Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asn Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Arg Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Gly Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ala Leu Val Leu Gln Ser Gln Arg Thr Asp Phe Pro
275 280 285
Tyr Val Ser Ala Ala Met Pro Cys Phe Val Ile Ile Ile Ile Leu Cys
290 295 300
Val Pro Cys Cys Lys Lys Lys Thr Ser Ala Ala Glu Gly Pro
305 310 315
<210> 14
<211> 244
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-1 (NO ДОСТУПА Q9BZM6)
<400> 14
Met Ala Ala Ala Ala Ser Pro Ala Phe Leu Leu Cys Leu Pro Leu Leu
1 5 10 15
His Leu Leu Ser Gly Trp Ser Arg Ala Gly Trp Val Asp Thr His Cys
20 25 30
Leu Cys Tyr Asp Phe Ile Ile Thr Pro Lys Ser Arg Pro Glu Pro Gln
35 40 45
Trp Cys Glu Val Gln Gly Leu Val Asp Glu Arg Pro Phe Leu His Tyr
50 55 60
Asp Cys Val Asn His Lys Ala Lys Ala Phe Ala Ser Leu Gly Lys Lys
65 70 75 80
Val Asn Val Thr Lys Thr Trp Glu Glu Gln Thr Glu Thr Leu Arg Asp
85 90 95
Val Val Asp Phe Leu Lys Gly Gln Leu Leu Asp Ile Gln Val Glu Asn
100 105 110
Leu Ile Pro Ile Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu
115 120 125
His Glu Ala His Gly His Gly Arg Gly Ser Trp Gln Phe Leu Phe Asn
130 135 140
Gly Gln Lys Phe Leu Leu Phe Asp Ser Asn Asn Arg Lys Trp Thr Ala
145 150 155 160
Leu His Pro Gly Ala Lys Lys Met Thr Glu Lys Trp Glu Lys Asn Arg
165 170 175
Asp Val Thr Met Phe Phe Gln Lys Ile Ser Leu Gly Asp Cys Lys Met
180 185 190
Trp Leu Glu Glu Phe Leu Met Tyr Trp Glu Gln Met Leu Asp Pro Thr
195 200 205
Lys Pro Pro Ser Leu Ala Pro Gly Thr Thr Gln Pro Lys Ala Met Ala
210 215 220
Thr Thr Leu Ser Pro Trp Ser Leu Leu Ile Ile Phe Leu Cys Phe Ile
225 230 235 240
Leu Ala Gly Arg
<210> 15
<211> 246
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-2 (NO ДОСТУПА Q9BZM5)
<400> 15
Met Ala Ala Ala Ala Ala Thr Lys Ile Leu Leu Cys Leu Pro Leu Leu
1 5 10 15
Leu Leu Leu Ser Gly Trp Ser Arg Ala Gly Arg Ala Asp Pro His Ser
20 25 30
Leu Cys Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg Pro Gly Pro Arg
35 40 45
Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr
50 55 60
Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys
65 70 75 80
Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu
85 90 95
Val Val Asp Ile Leu Thr Glu Gln Leu Arg Asp Ile Gln Leu Glu Asn
100 105 110
Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu
115 120 125
Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln Phe Ser Phe Asp
130 135 140
Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg Met Trp Thr Thr
145 150 155 160
Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys
165 170 175
Val Val Ala Met Ser Phe His Tyr Phe Ser Met Gly Asp Cys Ile Gly
180 185 190
Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser
195 200 205
Ala Gly Ala Pro Leu Ala Met Ser Ser Gly Thr Thr Gln Leu Arg Ala
210 215 220
Thr Ala Thr Thr Leu Ile Leu Cys Cys Leu Leu Ile Ile Leu Pro Cys
225 230 235 240
Phe Ile Leu Pro Gly Ile
245
<210> 16
<211> 244
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-3 (NO ДОСТУПА NP_078794)
<400> 16
Met Ala Ala Ala Ala Ser Pro Ala Ile Leu Pro Arg Leu Ala Ile Leu
1 5 10 15
Pro Tyr Leu Leu Phe Asp Trp Ser Gly Thr Gly Arg Ala Asp Ala His
20 25 30
Ser Leu Trp Tyr Asn Phe Thr Ile Ile His Leu Pro Arg His Gly Gln
35 40 45
Gln Trp Cys Glu Val Gln Ser Gln Val Asp Gln Lys Asn Phe Leu Ser
50 55 60
Tyr Asp Cys Gly Ser Asp Lys Val Leu Ser Met Gly His Leu Glu Glu
65 70 75 80
Gln Leu Tyr Ala Thr Asp Ala Trp Gly Lys Gln Leu Glu Met Leu Arg
85 90 95
Glu Val Gly Gln Arg Leu Arg Leu Glu Leu Ala Asp Thr Glu Leu Glu
100 105 110
Asp Phe Thr Pro Ser Gly Pro Leu Thr Leu Gln Val Arg Met Ser Cys
115 120 125
Glu Cys Glu Ala Asp Gly Tyr Ile Arg Gly Ser Trp Gln Phe Ser Phe
130 135 140
Asp Gly Arg Lys Phe Leu Leu Phe Asp Ser Asn Asn Arg Lys Trp Thr
145 150 155 160
Val Val His Ala Gly Ala Arg Arg Met Lys Glu Lys Trp Glu Lys Asp
165 170 175
Ser Gly Leu Thr Thr Phe Phe Lys Met Val Ser Met Arg Asp Cys Lys
180 185 190
Ser Trp Leu Arg Asp Phe Leu Met His Arg Lys Lys Arg Leu Glu Pro
195 200 205
Thr Ala Pro Pro Thr Met Ala Pro Gly Leu Ala Gln Pro Lys Ala Ile
210 215 220
Ala Thr Thr Leu Ser Pro Trp Ser Phe Leu Ile Ile Leu Cys Phe Ile
225 230 235 240
Leu Pro Gly Ile
<210> 17
<211> 263
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-4/RAET1E (NO ДОСТУПА Q8TD07)
<400> 17
Met Arg Arg Ile Ser Leu Thr Ser Ser Pro Val Arg Leu Leu Leu Phe
1 5 10 15
Leu Leu Leu Leu Leu Ile Ala Leu Glu Ile Met Val Gly Gly His Ser
20 25 30
Leu Cys Phe Asn Phe Thr Ile Lys Ser Leu Ser Arg Pro Gly Gln Pro
35 40 45
Trp Cys Glu Ala Gln Val Phe Leu Asn Lys Asn Leu Phe Leu Gln Tyr
50 55 60
Asn Ser Asp Asn Asn Met Val Lys Pro Leu Gly Leu Leu Gly Lys Lys
65 70 75 80
Val Tyr Ala Thr Ser Thr Trp Gly Glu Leu Thr Gln Thr Leu Gly Glu
85 90 95
Val Gly Arg Asp Leu Arg Met Leu Leu Cys Asp Ile Lys Pro Gln Ile
100 105 110
Lys Thr Ser Asp Pro Ser Thr Leu Gln Val Glu Met Phe Cys Gln Arg
115 120 125
Glu Ala Glu Arg Cys Thr Gly Ala Ser Trp Gln Phe Ala Thr Asn Gly
130 135 140
Glu Lys Ser Leu Leu Phe Asp Ala Met Asn Met Thr Trp Thr Val Ile
145 150 155 160
Asn His Glu Ala Ser Lys Ile Lys Glu Thr Trp Lys Lys Asp Arg Gly
165 170 175
Leu Glu Lys Tyr Phe Arg Lys Leu Ser Lys Gly Asp Cys Asp His Trp
180 185 190
Leu Arg Glu Phe Leu Gly His Trp Glu Ala Met Pro Glu Pro Thr Val
195 200 205
Ser Pro Val Asn Ala Ser Asp Ile His Trp Ser Ser Ser Ser Leu Pro
210 215 220
Asp Arg Trp Ile Ile Leu Gly Ala Phe Ile Leu Leu Val Leu Met Gly
225 230 235 240
Ile Val Leu Ile Cys Val Trp Trp Gln Asn Gly Glu Trp Gln Ala Gly
245 250 255
Leu Trp Pro Leu Arg Thr Ser
260
<210> 18
<211> 332
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-5 (NO ДОСТУПА Q6H3X3)
<400> 18
Met Ala Ala Ala Ala Ser Pro Ala Phe Leu Leu Arg Leu Pro Leu Leu
1 5 10 15
Leu Leu Leu Ser Ser Trp Cys Arg Thr Gly Leu Ala Asp Pro His Ser
20 25 30
Leu Cys Tyr Asp Ile Thr Val Pro Lys Phe Arg Pro Gly Pro Arg Trp
35 40 45
Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr Asp
50 55 60
Cys Gly Ser Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys Leu
65 70 75 80
Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu Val
85 90 95
Val Asp Leu Thr Glu Gln Leu Leu Asp Ile Gln Leu Glu Asn Tyr Ile
100 105 110
Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu Gln Lys
115 120 125
Ala Glu Gly His Gly Ser Gly Ser Trp Gln Leu Ser Phe Asp Gly Gln
130 135 140
Ile Phe Leu Leu Phe Asp Ser Glu Asn Arg Met Trp Thr Thr Val His
145 150 155 160
Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys Asp Met
165 170 175
Thr Met Ser Phe His Tyr Ile Ser Met Gly Asp Cys Thr Gly Trp Leu
180 185 190
Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser Ala Gly
195 200 205
Ala Pro Pro Thr Met Ser Ser Gly Thr Ala Gln Pro Arg Ala Thr Ala
210 215 220
Thr Thr Leu Ile Leu Cys Cys Leu Leu Ile Met Cys Leu Leu Ile Cys
225 230 235 240
Ser Arg His Ser Leu Thr Gln Ser His Gly His His Pro Gln Ser Leu
245 250 255
Gln Pro Pro Pro His Pro Pro Leu Leu His Pro Thr Trp Leu Leu Arg
260 265 270
Arg Val Leu Trp Ser Asp Ser Tyr Gln Ile Ala Lys Arg Pro Leu Ser
275 280 285
Gly Gly His Val Thr Arg Val Thr Leu Pro Ile Ile Gly Asp Asp Ser
290 295 300
His Ser Leu Pro Cys Pro Leu Ala Leu Tyr Thr Ile Asn Asn Gly Ala
305 310 315 320
Ala Arg Tyr Ser Glu Pro Leu Gln Val Ser Ile Ser
325 330
<210> 19
<211> 246
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP-6 (NO ДОСТУПА NP_570970)
<400> 19
Met Ala Ala Ala Ala Ile Pro Ala Leu Leu Leu Cys Leu Pro Leu Leu
1 5 10 15
Phe Leu Leu Phe Gly Trp Ser Arg Ala Arg Arg Asp Asp Pro His Ser
20 25 30
Leu Cys Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg Pro Gly Pro Arg
35 40 45
Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr
50 55 60
Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys
65 70 75 80
Leu Asn Val Thr Met Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu
85 90 95
Val Val Asp Ile Leu Thr Glu Gln Leu Leu Asp Ile Gln Leu Glu Asn
100 105 110
Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu
115 120 125
Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln Phe Ser Ile Asp
130 135 140
Gly Gln Thr Phe Leu Leu Phe Asp Ser Glu Lys Arg Met Trp Thr Thr
145 150 155 160
Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys
165 170 175
Asp Val Ala Met Ser Phe His Tyr Ile Ser Met Gly Asp Cys Ile Gly
180 185 190
Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser
195 200 205
Ala Gly Ala Pro Leu Ala Met Ser Ser Gly Thr Thr Gln Leu Arg Ala
210 215 220
Thr Ala Thr Thr Leu Ile Leu Cys Cys Leu Leu Ile Ile Leu Pro Cys
225 230 235 240
Phe Ile Leu Pro Gly Ile
245
<210> 20
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA-WED
<400> 20
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 21
<211> 507
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий вариант 15 альфа1-альфа2
<400> 21
gctgctgagc cacacagtct ccgctacaac cttacggtgt tgagctggga cggctctgtc 60
cagagtggct ttctgactga ggtacatctc gatggtcagc ccttcctccg atgcgacaga 120
caaaagtgca gggccaagcc acagggccaa tgggccgaag atgtacttgg caataagact 180
tgggacagag aaaccagaga tctgactggc tggggtaagg acttacgcat gactctcgca 240
cacattaaag accagaagga aggtcttcat tcgctccagg aaattagagt ctgtgaaatc 300
catgaagaca acagcacaag aagttcccaa catttctact acgacggcga gctgttctta 360
tcacagaatt tagagaccaa cgagtggaca atgccccaaa gctcgagggc ccagaccctc 420
gctatgaatg tgaggaattt ccttaaggag gacgctatgg aaactgacac ccactaccat 480
gcgatgcgcg ccgattgcct gcaggaa 507
<210> 22
<211> 507
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий вариант 16 альфа1-альфа2
<400> 22
gctgctgagc cacacagtct ccgctacaac cttacggtgt tgagctggga cggctctgtc 60
cagagtggct ttctgactga ggtacatctc gatggtcagc ccttcctccg atgcgacaga 120
caaaagtgca gggccaagcc acagggccaa tgggccgaag atgtacttgg caataagact 180
tgggacagag aaaccagaga tctgactggc tggggtaagg acttacgcat gactctcgca 240
cacattaaag accagaagga aggtcttcat tcgctccagg aaattagagt ctgtgaaatc 300
catgaagaca acagcacaag aagttcccaa catttctact acgacggcga gctgttctta 360
tcacagaatt tagagaccct cgagtggaca atgccccaaa gctcgagggc ccagaccctc 420
gctatgaatg tgaggaattt ccttaaggag gacgctatgg aaactgacac ccactaccat 480
gcgatgcgcg ccgattgcct gcaggaa 507
<210> 23
<211> 507
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий вариант 17 альфа1-альфа2
<400> 23
gctgctgagc cacacagtct ccgctacaac cttacggtgt tgagctggga cggctctgtc 60
cagagtggct ttctgactga ggtacatctc gatggtcagc ccttcctccg atgcgacaga 120
caaaagtgca gggccaagcc acagggccaa tgggccgaag atgtacttgg caataagact 180
tgggacagag aaaccagaga tctgactctc tggggtaagg acttacgcat gactctcgca 240
cacattaaag accagaagga aggtcttcat tcgctccagg aaattagagt ctgtgaaatc 300
catgaagaca acagcacaag aagttcccaa catttctact acgacggcga gctgttctta 360
tcacagaatt tagagaccct cgagtggaca atgccccaaa gctcgagggc ccagaccctc 420
gctatgaatg tgaggaattt ccttaaggag gacgctatgg aaactgacac ccactaccat 480
gcgatgcgcg ccgattgcct gcaggaa 507
<210> 24
<211> 507
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий вариант 18 альфа1-альфа2
<400> 24
gctgctgagc cacacagtct ccgctacaac cttacggtgt tgagctggga cggctctgtc 60
cagcccggct ttctgactga ggtacatctc gatggtcagc ccttcctccg atgcgacaga 120
caaaagtgca gggccaagcc acagggccaa tgggccgaag atgtacttgg caataagact 180
tgggacagag aaaccagaga tctgactctc tggggtaagg acttacgcat gactctcgca 240
cacattaaag accagaagga aggtcttcat tcgctccagg aaattagagt ctgtgaaatc 300
catgaagaca acagcacaag aagttcccaa catttctact acgacggcga gctgttctta 360
tcacagaatt tagagaccct cgagtggaca atgccccaaa gctcgagggc ccagaccctc 420
gctatgaatg tgaggaattt ccttaaggag gacgctatgg aaactgacac ccactaccat 480
gcgatgcgcg ccgattgcct gcaggaa 507
<210> 25
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA, вариант 15 альфа1-альфа2
<400> 25
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Asn Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 26
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA, вариант 16 альфа1-альфа2
<400> 26
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 27
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA, вариант 17 альфа1-альфа2
<400> 27
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Leu Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 28
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICA, вариант 18 альфа1-альфа2
<400> 28
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Pro Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Leu Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 29
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICwed альфа1-альфа2
<400> 29
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 30
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM20 альфа1-альфа2
<400> 30
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Ala Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Gln Thr Asp Thr His Tyr Arg Ala Met
145 150 155 160
His Ala Asp Cys Leu Phe Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 31
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM25 альфа1-альфа2
<400> 31
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 32
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM27 альфа1-альфа2
<400> 32
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 33
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM28 альфа1-альфа2
<400> 33
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 34
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM42 альфа1-альфа2
<400> 34
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 35
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM48 альфа1-альфа2
<400> 35
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Ala Thr Asp Thr His Tyr Ile Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ala Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 36
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид DSM49 альфа1-альфа2
<400> 36
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Thr Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gln Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Phe Thr Asp Thr His Tyr Arg Ala Met
145 150 155 160
Thr Ala Asp Cys Leu Thr Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 37
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический олигонуклеотидный праймер
<400> 37
atctataatg ctgagcccca cagtcttcg 29
<210> 38
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический олигонуклеотидный праймер
<400> 38
cttgctcttc agatatcgcc gtagttc 27
<210> 39
<211> 7556
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция ДНК, экспрессирующая wt домен a1a2-Fv
<400> 39
ggagagacca cacccaagct gtctagagcc gccacgatgg ggctgggccc ggtcttcctg 60
cttctggctg gcatcttccc ttttgcacct ccgggagctg ctgctgagcc ccacagtctt 120
cgttataacc tcacggtgct gtcctgggat ggatctgtgc agtcagggtt tctcactgag 180
gtacatctgg atggtcagcc cttcctgcgc tgtgacaggc agaaatgcag ggcaaagccc 240
cagggacagt gggcagaaga tgtcctggga aataagacat gggacagaga gaccagagac 300
ttgacaggga atggaaagga cctcaggatg accctggctc atatcaagga ccagaaagaa 360
ggcttgcatt ccctccagga gattagggtc tgtgagatcc atgaagacaa cagcaccagg 420
agctcccagc atttctacta cgatggggag ctctttctct cccaaaacct ggagactaag 480
gaatggacaa tgccccagtc ctccagagct cagaccttgg ccatgaacgt caggaatttc 540
ttgaaggaag atgcaatgaa gaccaagaca cactatcacg ctatgcatgc agactgcctg 600
caggaactac ggcgatatct aaaatccggc gtagtcctga ggagaacagt gccccccatg 660
gtgcaggtga ctcgctctga ggcctctggc ggatctgggg accgtgtgac aatcacctgc 720
agagcctccc aggacgtctc cactgccgtg gcgtggtacc aacagaagcc cgggaaggca 780
cccaaactgc tcatttacag cgcatccttt ctctactctg gcgtgccgtc tcgctttagc 840
gggtccggca gcggtacaga ctttactctg accatctcct ctctgcaacc ggaggatttt 900
gcaacctatt attgccagca atcctacaca acccccccca cctttggcca gggcaccaag 960
gtggagatca agggaggttc tagccgctcc agcagctctg gaggtggagg ctctggcgga 1020
ggaggcgagg tgcaactggt ggagtctggg ggcggcctgg tccagcccgg cggaagcttg 1080
cgcctgagct gtgccgcctc cggttttacc ttcaccagca ctggaatctc ctgggtgcgc 1140
caagctcccg gcaaagggct cgaatgggtg ggccgtatct accccaccaa cggaagcacc 1200
aactatgcag acagcgtgaa ggggcgcttc actatctccg ccgacaccag caaaaacacc 1260
gcgtacctgc agatgaattc tttgagggca gaggatactg ccgtgtacta ctgcgcgagg 1320
acatacggca tttacgatct gtatgtggat tacaccgaat acgtgatgga ctattggggc 1380
cagggcactc tggtcacagt gtctagcggt ggcagctccc gcagctccag cagcggtggt 1440
ggcggtagcg gaggcggagg cgatatccag atgactcaga gtccctcttc tctgagtgct 1500
tctggcggaa gtgggcagat caccgtcaca tgtcgcgcaa gcggctttta tccttggaac 1560
atcaccctga gctggcggca ggacggcgtc agcctgtccc atgataccca acagtgggga 1620
gatgtgctcc cggacggtca gggaacttac cagacctggg ttgcaactcg catctcccag 1680
ggggaggagc agcgtttcac atgttatatg gagcactctg gccagcacag cactcatccg 1740
gtgccgtccg gaaagggatc tcatcaccat caccaccact aggatccgtt gaggtctcta 1800
aaagcgtctt cctgttctca tcacatcata tcaaggttat ataccatcaa tattgccaca 1860
gatgttactt agccttttaa tatttctcta atttagtgta tatgcaatga tagttctctg 1920
atttctgaga ttgagtttct catgtgtaat gattatttag agtttctctt tcatctgttc 1980
aaatttttgt ctagttttat tttttactga tttgtaagac ttctttttat aatctgcata 2040
ttacaattct ctttactggg gtgttgcaaa tattttctgt cattctatgg cctgactttt 2100
cttaatggtt ttttaatttt aaaaataagt cttaatattc atgcaatcta attaacaatc 2160
ttttctttgt ggttaggact ttgagtcata agaaattttt ctctacactg aagtcatgat 2220
ggcatgcttc tatattattt tctaaaagat ttaaagtttt gccttctcca tttagactta 2280
taattcactg gaattttttt gtgtgtatgg tatgacatat gggttccctt ttatttttta 2340
catataaata tatttccctg tttttctaaa aaagaaaaag atcatcattt tcccattgta 2400
aaatgccata tttttttcat aggtcactta catatatcaa tgggtctgtt tctgagctct 2460
actctatttt atcagcctca ctgtctatcc ccacacatct catgctttgc tctaaatctt 2520
gatatttagt ggaacattct ttcccatttt gttctacaag aatatttttg ttattgtctt 2580
tgggctttct atatacattt tgaaatgagg ttgacaagtt aataatcaac ctctggatta 2640
caaaatttgt gaaagattga ctggtattct taactatgtt gctcctttta cgctatgtgg 2700
atacgctgct ttaatgcctt tgtatcatgc tattgcttcc cgtatggctt tcattttctc 2760
ctccttgtat aaatcctggt tgctgtctct ttatgaggag ttgtggcccg ttgtcaggca 2820
acgtggcgtg gtgtgcactg tgtttgctga cgcaaccccc actggttggg gcattgccac 2880
cacctgtcag ctcctttccg ggactttcgc tttccccctc cctattgcca cggcggaact 2940
catcgccgcc tgccttgccc gctgctggac aggggctcgg ctgttgggca ctgacaattc 3000
cgtggtgttg tcggggaaat catcgtcctt tccttggctg ctcgcctgtg ttgccacctg 3060
gattctgcgc gggacgtcct tctgctacgt cccttcggcc ctcaatccag cggaccttcc 3120
ttcccgcggc ctgctgccgg ctctgcggcc tcttccgcct cttcgccttc gccctcagac 3180
gagtcggatc tccctttggg ccgcctcccc gcatctgtgc cttctagttg ccagccatct 3240
gttgtttgcc cctcccccgt gccttccttg accctggaag gtgccactcc cactgtcctt 3300
tcctaataaa atgaggaaat tgcatcgcat tgtctgagta ggtgtcattc tattctgggg 3360
ggtggggtgg ggcaggacag caagggggag gattggcaag acaatagcag gctttgcatt 3420
tttagacatt tagaagccta tatcttgtta cagaattgga attacacaaa aattctacca 3480
tattttgaaa gcttaggttg ttctgaaaaa aacaatatat tgttttcctg ggtaaactaa 3540
aagtcccctc gaggaaaggc ccctaaagtg aaacagtgca aaacgttcaa aaactgtctg 3600
gcaatacaag ttccactttg accaaaacgg ctggcagtaa aagggttaag aagactgtca 3660
gccttgagcg gtatcagctc actcaaaggc ggtaatacgg ttatccacag aatcagggga 3720
taacgcagga aagaacatgt gagcaaaagg ccagcaaaag gccaggaacc gtaaaaaggc 3780
cgcgttgctg gcgtttttcc ataggctccg cccccctgac gagcatcaca aaaatcgacg 3840
ctcaagtcag aggtggcgaa acccgacagg actataaaga taccaggcgt ttccccctgg 3900
aagctccctc gtgcgctctc ctgttccgac cctgccgctt accggatacc tgtccgcctt 3960
tctcccttcg ggaagcgtgg cgctttctca tagctcacgc tgtaggtatc tcagttcggt 4020
gtaggtcgtt cgctccaagc tgggctgtgt gcacgaaccc cccgttcagc ccgaccgctg 4080
cgccttatcc ggtaactatc gtcttgagtc caacccggta agacacgact tatcgccact 4140
ggcagcagcc actggtaaca ggattagcag agcgaggtat gtaggcggtg ctacagagtt 4200
cttgaagtgg tgggctaact acggctacac tagaagaaca gtatttggta tctgcgctct 4260
gctgaagcca gttaccttcg gaaaaagagt tggtagctct tgatccggca aacaaaccac 4320
cgctggtagc ggtggttttt ttgtttgcaa gcagcagatt acgcgcagaa aaaaaggatc 4380
tcaagaagat cctttgatct tttctacggg gtctgacgct cagtggaacg acgcgcgcgt 4440
aactcacgtt aagggatttt ggtcatgagt tagaaaaact catcgagcat caaatgaaac 4500
tgcaatttat tcatatcagg attatcaata ccatattttt gaaaaagccg tttctgtaat 4560
gaaggagaaa actcaccgag gcagttccat aggatggcaa gatcctggta tcggtctgcg 4620
attccgactc gtccaacatc aatacaacct attaatttcc cctcgtcaaa aataaggtta 4680
tcaagtgaga aatcaccatg agtgacgact gaatccggtg agaatggcaa aagtttatgc 4740
atttctttcc agacttgttc aacaggccag ccattacgct cgtcatcaaa atcactcgca 4800
tcaaccaaac cgttattcat tcgtgattgc gcctgagcga ggcgaaatac gcgatcgctg 4860
ttaaaaggac aattacaaac aggaatcgag tgcaaccggc gcaggaacac tgccagcgca 4920
tcaacaatat tttcacctga atcaggatat tcttctaata cctggaacgc tgtttttccg 4980
gggatcgcag tggtgagtaa ccatgcatca tcaggagtac ggataaaatg cttgatggtc 5040
ggaagtggca taaattccgt cagccagttt agtctgacca tctcatctgt aacatcattg 5100
gcaacgctac ctttgccatg tttcagaaac aactctggcg catcgggctt cccatacaag 5160
cgatagattg tcgcacctga ttgcccgaca ttatcgcgag cccatttata cccatataaa 5220
tcagcatcca tgttggaatt taatcgcggc ctcgacgttt cccgttggat atggctcatt 5280
ttttacttcc tcaccttgtc gtattatact atgccgatat actatgccga tgattaattg 5340
tcgacactgc gggggctctg tgtggtaagc aggtcttaac ctttttactg ccaatgacgc 5400
atgggatacg tcgtggcagt aaaagggctt aaatgccaac gacgcgtccc atacgttgtt 5460
ggcattttaa ttcttctctc tgcagcggca gcatgtgccg ccgctgcaga gagtttctag 5520
cgatgacagc ccctctgggc aacgagccgg gggggctgtc tttctttatg ttttaaatgc 5580
actgacctcc cacattccct ttttagtaaa atattcagaa ataatttaaa tacatcattg 5640
caatgaaaat aaatgttttt tattaggcag aatccagatg ctcaaggccc ttcataatat 5700
cccccagttt agtagttgga cttagggaac aaaggaacct ttaatagaaa ttggacagca 5760
agaaagcgag tcaggcaccg ggcttgcggg tcatgcacca ggtgcgcggt ccttcgggca 5820
cctcgacgtc ggcggtgacg gtgaagccga gccgctcgta gaaggggagg ttgcggggcg 5880
cggatgtctc caggaaggcg ggcaccccgg cgcgctcggc cgcctccact ccggggagca 5940
cgacggcgct gcccagaccc ttgccctggt ggtcgggcga cacgccgacg gtggccagga 6000
accacgcggg ctccttgggc cggtgcggcg ccaggaggcc ttccatctgt tgctgcgcgg 6060
ccagccggga accgctcaac tcggccatgc gcgggccgat ctcggcgaac accgcccccg 6120
cttcgacgct ctccggcgtg gtccagaccg ccaccgcggc gccgtcgtcc gcgacccaca 6180
ccttgccgat gtcgagcccg acgcgcgtga ggaagagttc ttgcagctcg gtgacccgct 6240
cgatgtggcg gtccggatcg acggtgtggc gcgtggcggg gtagtcggcg aacgcggcgg 6300
cgagggtgcg tacggccctg gggacgtcgt cgcgggtggc gaggcgcacc gtgggcttgt 6360
actcggtcat ggtggcggac gaaaggcccg gagatgagga agaggagaac agcgcggcag 6420
acgtgcgctt ttgaagcgtg cagaatgccg ggcctccgga ggaccttcgg gcgcccgccc 6480
cgcccctgag cccgcccctg agcccgcccc cggacccacc ccttcccagc ctctgagccc 6540
agaaagcgaa ggagcaaagc tgctattggc cgctgcccca aaggcctacc cgcttccatt 6600
gctcagcggt gctgtccatc tgcacgagac tagtgagtcg tgctacttcc atttgtcacg 6660
tcctgcacga cgcgagctgc ggggcggggg ggaacttcct gactagggga ggagtagaag 6720
gtggcgcgaa ggggccacca aagaacggag ccggttggcg cctaccggtg gatgtggaat 6780
gtgtgcgagg ccagaggcca cttgtgtagc gccaagtgcc cagcggggct gctaaagcgc 6840
atgctccaga ctgccttggg aaaagcgcct cccctacccg gtagagaaac ttgatctgtc 6900
gccgcaattc aaacttcgtg aggctccggt gcccgtcagt gacctgctat actctggaga 6960
cgacttacgg taaatggccc gcctggctga ccgcccaacg acccccgccc attgacgtca 7020
ataatgacgt atgttcccat agtaacgcca atagggactt tccattgacg tcaatgggtg 7080
gagtatttac ggtaaactgc ccacttggca gtacatcaag tgtatcatat gccaagtccg 7140
ccccctattg acgtcaatga cggtaaatgg cccgcctggc attatgccca gtacatgacc 7200
ttacgggact ttcctacttg gcagtacatc tacgtattag tcatcgctat taccatgctg 7260
atgcggtttt ggcagtacac caatgggcgt ggatagcggt ttgactcacg gggatttcca 7320
agtctccacc ccattgacgt caatgggagt ttgttttggc accaaaatca acgggacttt 7380
ccaaaatgtc gtaataaccc cgccccgttg acgcaaatgg gcggtaggcg tgtacggtgg 7440
gaggtctata taagcagagc tcgtttagtg aaccgtcaga tcgcctggag aggccatcca 7500
cgctgttttg acctccatag tggacaccgg gaccgatcca gcctccgcgt ctcagg 7556
<210> 40
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид wt MICA-Fv
<400> 40
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 41
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICwed-Fv
<400> 41
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 42
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv20-Fv
<400> 42
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Ala Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Gln Thr Asp Thr His Tyr Arg Ala Met
145 150 155 160
His Ala Asp Cys Leu Phe Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 43
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv25-Fv
<400> 43
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 44
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv27-Fv
<400> 44
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 45
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv28-Fv
<400> 45
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 46
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv42-Fv
<400> 46
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 47
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv48-Fc
<400> 47
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Ala Thr Asp Thr His Tyr Ile Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ala Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 48
<211> 558
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид MICv49-Fv
<400> 48
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Thr Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gln Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Phe Thr Asp Thr His Tyr Arg Ala Met
145 150 155 160
Thr Ala Asp Cys Leu Thr Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Gln Val Thr Arg Ser Glu
180 185 190
Ala Ser Gly Gly Ser Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
195 200 205
Gln Asp Val Ser Thr Ala Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
210 215 220
Ala Pro Lys Leu Leu Ile Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val
225 230 235 240
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
245 250 255
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
260 265 270
Ser Tyr Thr Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
275 280 285
Lys Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser Gly
290 295 300
Gly Gly Gly Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
305 310 315 320
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
325 330 335
Thr Ser Thr Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
340 345 350
Glu Trp Val Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala
355 360 365
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn
370 375 380
Thr Ala Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
385 390 395 400
Tyr Tyr Cys Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr
405 410 415
Thr Glu Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
420 425 430
Ser Ser Gly Gly Ser Ser Arg Ser Ser Ser Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
450 455 460
Ala Ser Gly Gly Ser Gly Gln Ile Thr Val Thr Cys Arg Ala Ser Gly
465 470 475 480
Phe Tyr Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser
485 490 495
Leu Ser His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Gln
500 505 510
Gly Thr Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu
515 520 525
Gln Arg Phe Thr Cys Tyr Met Glu His Ser Gly Gln His Ser Thr His
530 535 540
Pro Val Pro Ser Gly Lys Gly Ser His His His His His His
545 550 555
<210> 49
<211> 137
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический эктодомен NKG2D
<400> 49
Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr
1 5 10 15
Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr
20 25 30
Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys
35 40 45
Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln
50 55 60
Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His
65 70 75 80
Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser
85 90 95
Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu
100 105 110
Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn
115 120 125
Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 50
<211> 40
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический олигонуклеотид
<400> 50
ctgtctagag ccgccaacat ggggctgggc ccggtcttcc 40
<210> 51
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический олигонуклеотид
<400> 51
aacggatcct acacagtcct ttgcatgcag 30
<210> 52
<211> 6362
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, pD2509-CMV-Avi-His-природный
эктодомен NKG2D
<400> 52
ggagagacca cacccaagct gtctagagcc gccaacatgg ggctgggccc ggtcttcctg 60
cttctggctg gcatcttccc ttttgcacct ccgggagctg ctgctgagcc ccaccatcat 120
caccaccatg gccttaacga catcttcgaa gctcaaaaga tcgaatggca tgaaaactca 180
ttattcaacc aagaagttca aattcccttg accgaaagtt actgtggccc atgtcctaaa 240
aactggatat gttacaaaaa taactgctac caattttttg atgagagtaa aaactggtat 300
gagagccagg cttcttgtat gtctcaaaat gccagccttc tgaaagtata cagcaaagag 360
gaccaggatt tacttaaact ggtgaagtca tatcattgga tgggactagt acacattcca 420
acaaatggat cttggcagtg ggaagatggc tccattctct cacccaacct actaacaata 480
attgaaatgc agaagggaga ctgtgcactc tatgcctcga gctttaaagg ctatatagaa 540
aactgttcaa ctccaaatac atacatctgc atgcaaagga ctgtgtagga tccgttgagg 600
tctctaaaag cgtcttcctg ttctcatcac atcatatcaa ggttatatac catcaatatt 660
gccacagatg ttacttagcc ttttaatatt tctctaattt agtgtatatg caatgatagt 720
tctctgattt ctgagattga gtttctcatg tgtaatgatt atttagagtt tctctttcat 780
ctgttcaaat ttttgtctag ttttattttt tactgatttg taagacttct ttttataatc 840
tgcatattac aattctcttt actggggtgt tgcaaatatt ttctgtcatt ctatggcctg 900
acttttctta atggtttttt aattttaaaa ataagtctta atattcatgc aatctaatta 960
acaatctttt ctttgtggtt aggactttga gtcataagaa atttttctct acactgaagt 1020
catgatggca tgcttctata ttattttcta aaagatttaa agttttgcct tctccattta 1080
gacttataat tcactggaat ttttttgtgt gtatggtatg acatatgggt tcccttttat 1140
tttttacata taaatatatt tccctgtttt tctaaaaaag aaaaagatca tcattttccc 1200
attgtaaaat gccatatttt tttcataggt cacttacata tatcaatggg tctgtttctg 1260
agctctactc tattttatca gcctcactgt ctatccccac acatctcatg ctttgctcta 1320
aatcttgata tttagtggaa cattctttcc cattttgttc tacaagaata tttttgttat 1380
tgtctttggg ctttctatat acattttgaa atgaggttga caagttaata atcaacctct 1440
ggattacaaa atttgtgaaa gattgactgg tattcttaac tatgttgctc cttttacgct 1500
atgtggatac gctgctttaa tgcctttgta tcatgctatt gcttcccgta tggctttcat 1560
tttctcctcc ttgtataaat cctggttgct gtctctttat gaggagttgt ggcccgttgt 1620
caggcaacgt ggcgtggtgt gcactgtgtt tgctgacgca acccccactg gttggggcat 1680
tgccaccacc tgtcagctcc tttccgggac tttcgctttc cccctcccta ttgccacggc 1740
ggaactcatc gccgcctgcc ttgcccgctg ctggacaggg gctcggctgt tgggcactga 1800
caattccgtg gtgttgtcgg ggaaatcatc gtcctttcct tggctgctcg cctgtgttgc 1860
cacctggatt ctgcgcggga cgtccttctg ctacgtccct tcggccctca atccagcgga 1920
ccttccttcc cgcggcctgc tgccggctct gcggcctctt ccgcctcttc gccttcgccc 1980
tcagacgagt cggatctccc tttgggccgc ctccccgcat ctgtgccttc tagttgccag 2040
ccatctgttg tttgcccctc ccccgtgcct tccttgaccc tggaaggtgc cactcccact 2100
gtcctttcct aataaaatga ggaaattgca tcgcattgtc tgagtaggtg tcattctatt 2160
ctggggggtg gggtggggca ggacagcaag ggggaggatt ggcaagacaa tagcaggctt 2220
tgcattttta gacatttaga agcctatatc ttgttacaga attggaatta cacaaaaatt 2280
ctaccatatt ttgaaagctt aggttgttct gaaaaaaaca atatattgtt ttcctgggta 2340
aactaaaagt cccctcgagg aaaggcccct aaagtgaaac agtgcaaaac gttcaaaaac 2400
tgtctggcaa tacaagttcc actttgacca aaacggctgg cagtaaaagg gttaagaaga 2460
ctgtcagcct tgagcggtat cagctcactc aaaggcggta atacggttat ccacagaatc 2520
aggggataac gcaggaaaga acatgtgagc aaaaggccag caaaaggcca ggaaccgtaa 2580
aaaggccgcg ttgctggcgt ttttccatag gctccgcccc cctgacgagc atcacaaaaa 2640
tcgacgctca agtcagaggt ggcgaaaccc gacaggacta taaagatacc aggcgtttcc 2700
ccctggaagc tccctcgtgc gctctcctgt tccgaccctg ccgcttaccg gatacctgtc 2760
cgcctttctc ccttcgggaa gcgtggcgct ttctcatagc tcacgctgta ggtatctcag 2820
ttcggtgtag gtcgttcgct ccaagctggg ctgtgtgcac gaaccccccg ttcagcccga 2880
ccgctgcgcc ttatccggta actatcgtct tgagtccaac ccggtaagac acgacttatc 2940
gccactggca gcagccactg gtaacaggat tagcagagcg aggtatgtag gcggtgctac 3000
agagttcttg aagtggtggg ctaactacgg ctacactaga agaacagtat ttggtatctg 3060
cgctctgctg aagccagtta ccttcggaaa aagagttggt agctcttgat ccggcaaaca 3120
aaccaccgct ggtagcggtg gtttttttgt ttgcaagcag cagattacgc gcagaaaaaa 3180
aggatctcaa gaagatcctt tgatcttttc tacggggtct gacgctcagt ggaacgacgc 3240
gcgcgtaact cacgttaagg gattttggtc atgagttaga aaaactcatc gagcatcaaa 3300
tgaaactgca atttattcat atcaggatta tcaataccat atttttgaaa aagccgtttc 3360
tgtaatgaag gagaaaactc accgaggcag ttccatagga tggcaagatc ctggtatcgg 3420
tctgcgattc cgactcgtcc aacatcaata caacctatta atttcccctc gtcaaaaata 3480
aggttatcaa gtgagaaatc accatgagtg acgactgaat ccggtgagaa tggcaaaagt 3540
ttatgcattt ctttccagac ttgttcaaca ggccagccat tacgctcgtc atcaaaatca 3600
ctcgcatcaa ccaaaccgtt attcattcgt gattgcgcct gagcgaggcg aaatacgcga 3660
tcgctgttaa aaggacaatt acaaacagga atcgagtgca accggcgcag gaacactgcc 3720
agcgcatcaa caatattttc acctgaatca ggatattctt ctaatacctg gaacgctgtt 3780
tttccgggga tcgcagtggt gagtaaccat gcatcatcag gagtacggat aaaatgcttg 3840
atggtcggaa gtggcataaa ttccgtcagc cagtttagtc tgaccatctc atctgtaaca 3900
tcattggcaa cgctaccttt gccatgtttc agaaacaact ctggcgcatc gggcttccca 3960
tacaagcgat agattgtcgc acctgattgc ccgacattat cgcgagccca tttataccca 4020
tataaatcag catccatgtt ggaatttaat cgcggcctcg acgtttcccg ttggatatgg 4080
ctcatttttt acttcctcac cttgtcgtat tatactatgc cgatatacta tgccgatgat 4140
taattgtcga cactgcgggg gctctgtgtg gtaagcaggt cttaaccttt ttactgccaa 4200
tgacgcatgg gatacgtcgt ggcagtaaaa gggcttaaat gccaacgacg cgtcccatac 4260
gttgttggca ttttaattct tctctctgca gcggcagcat gtgccgccgc tgcagagagt 4320
ttctagcgat gacagcccct ctgggcaacg agccgggggg gctgtctttc tttatgtttt 4380
aaatgcactg acctcccaca ttcccttttt agtaaaatat tcagaaataa tttaaataca 4440
tcattgcaat gaaaataaat gttttttatt aggcagaatc cagatgctca aggcccttca 4500
taatatcccc cagtttagta gttggactta gggaacaaag gaacctttaa tagaaattgg 4560
acagcaagaa agcgagtcag gcaccgggct tgcgggtcat gcaccaggtg cgcggtcctt 4620
cgggcacctc gacgtcggcg gtgacggtga agccgagccg ctcgtagaag gggaggttgc 4680
ggggcgcgga tgtctccagg aaggcgggca ccccggcgcg ctcggccgcc tccactccgg 4740
ggagcacgac ggcgctgccc agacccttgc cctggtggtc gggcgacacg ccgacggtgg 4800
ccaggaacca cgcgggctcc ttgggccggt gcggcgccag gaggccttcc atctgttgct 4860
gcgcggccag ccgggaaccg ctcaactcgg ccatgcgcgg gccgatctcg gcgaacaccg 4920
cccccgcttc gacgctctcc ggcgtggtcc agaccgccac cgcggcgccg tcgtccgcga 4980
cccacacctt gccgatgtcg agcccgacgc gcgtgaggaa gagttcttgc agctcggtga 5040
cccgctcgat gtggcggtcc ggatcgacgg tgtggcgcgt ggcggggtag tcggcgaacg 5100
cggcggcgag ggtgcgtacg gccctgggga cgtcgtcgcg ggtggcgagg cgcaccgtgg 5160
gcttgtactc ggtcatggtg gcggacgaaa ggcccggaga tgaggaagag gagaacagcg 5220
cggcagacgt gcgcttttga agcgtgcaga atgccgggcc tccggaggac cttcgggcgc 5280
ccgccccgcc cctgagcccg cccctgagcc cgcccccgga cccacccctt cccagcctct 5340
gagcccagaa agcgaaggag caaagctgct attggccgct gccccaaagg cctacccgct 5400
tccattgctc agcggtgctg tccatctgca cgagactagt gagtcgtgct acttccattt 5460
gtcacgtcct gcacgacgcg agctgcgggg cgggggggaa cttcctgact aggggaggag 5520
tagaaggtgg cgcgaagggg ccaccaaaga acggagccgg ttggcgccta ccggtggatg 5580
tggaatgtgt gcgaggccag aggccacttg tgtagcgcca agtgcccagc ggggctgcta 5640
aagcgcatgc tccagactgc cttgggaaaa gcgcctcccc tacccggtag agaaacttga 5700
tctgtcgccg caattcaaac ttcgtgaggc tccggtgccc gtcagtgacc tgctatactc 5760
tggagacgac ttacggtaaa tggcccgcct ggctgaccgc ccaacgaccc ccgcccattg 5820
acgtcaataa tgacgtatgt tcccatagta acgccaatag ggactttcca ttgacgtcaa 5880
tgggtggagt atttacggta aactgcccac ttggcagtac atcaagtgta tcatatgcca 5940
agtccgcccc ctattgacgt caatgacggt aaatggcccg cctggcatta tgcccagtac 6000
atgaccttac gggactttcc tacttggcag tacatctacg tattagtcat cgctattacc 6060
atgctgatgc ggttttggca gtacaccaat gggcgtggat agcggtttga ctcacgggga 6120
tttccaagtc tccaccccat tgacgtcaat gggagtttgt tttggcacca aaatcaacgg 6180
gactttccaa aatgtcgtaa taaccccgcc ccgttgacgc aaatgggcgg taggcgtgta 6240
cggtgggagg tctatataag cagagctcgt ttagtgaacc gtcagatcgc ctggagaggc 6300
catccacgct gttttgacct ccatagtgga caccgggacc gatccagcct ccgcgtctca 6360
gg 6362
<210> 53
<211> 160
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, His-avitag-природный эктодомен NKG2D
<400> 53
Glu Pro His His His His His His Gly Leu Asn Asp Ile Phe Glu Ala
1 5 10 15
Gln Lys Ile Glu Trp His Glu Asn Ser Leu Phe Asn Gln Glu Val Gln
20 25 30
Ile Pro Leu Thr Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile
35 40 45
Cys Tyr Lys Asn Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp
50 55 60
Tyr Glu Ser Gln Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys
65 70 75 80
Val Tyr Ser Lys Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr
85 90 95
His Trp Met Gly Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp
100 105 110
Glu Asp Gly Ser Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met
115 120 125
Gln Lys Gly Asp Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile
130 135 140
Glu Asn Cys Ser Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
145 150 155 160
<210> 54
<211> 160
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, His-avitag-неприродный эктодомен NKG2D Y152A
<400> 54
Glu Pro His His His His His His Gly Leu Asn Asp Ile Phe Glu Ala
1 5 10 15
Gln Lys Ile Glu Trp His Glu Asn Ser Leu Phe Asn Gln Glu Val Gln
20 25 30
Ile Pro Leu Thr Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile
35 40 45
Cys Tyr Lys Asn Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp
50 55 60
Tyr Glu Ser Gln Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys
65 70 75 80
Val Tyr Ser Lys Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala
85 90 95
His Trp Met Gly Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp
100 105 110
Glu Asp Gly Ser Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met
115 120 125
Gln Lys Gly Asp Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile
130 135 140
Glu Asn Cys Ser Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
145 150 155 160
<210> 55
<211> 160
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, His-avitag-неприродный эктодомен NKG2D Y199A
<400> 55
Glu Pro His His His His His His Gly Leu Asn Asp Ile Phe Glu Ala
1 5 10 15
Gln Lys Ile Glu Trp His Glu Asn Ser Leu Phe Asn Gln Glu Val Gln
20 25 30
Ile Pro Leu Thr Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile
35 40 45
Cys Tyr Lys Asn Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp
50 55 60
Tyr Glu Ser Gln Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys
65 70 75 80
Val Tyr Ser Lys Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr
85 90 95
His Trp Met Gly Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp
100 105 110
Glu Asp Gly Ser Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met
115 120 125
Gln Lys Gly Asp Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Ala Ile
130 135 140
Glu Asn Cys Ser Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
145 150 155 160
<210> 56
<211> 160
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, His-avitag-неприродный эктодомен NKG2D
Y152A +Y199A
<400> 56
Glu Pro His His His His His His Gly Leu Asn Asp Ile Phe Glu Ala
1 5 10 15
Gln Lys Ile Glu Trp His Glu Asn Ser Leu Phe Asn Gln Glu Val Gln
20 25 30
Ile Pro Leu Thr Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile
35 40 45
Cys Tyr Lys Asn Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp
50 55 60
Tyr Glu Ser Gln Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys
65 70 75 80
Val Tyr Ser Lys Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala
85 90 95
His Trp Met Gly Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp
100 105 110
Glu Asp Gly Ser Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met
115 120 125
Gln Lys Gly Asp Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Ala Ile
130 135 140
Glu Asn Cys Ser Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
145 150 155 160
<210> 57
<211> 7640
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, экспрессирующий вектор wt MIC-Fc
<400> 57
ggagagacca cacccaagct gtctagagcc gccaacatgg ggctgggccc ggtcttcctg 60
cttctggctg gcatcttccc ttttgcacct ccgggagctg ctgctgagcc ccacagtctt 120
cgttataacc tcacggtgct gtcctgggat ggatctgtgc agtcagggtt tctcactgag 180
gtacatctgg atggtcagcc cttcctgcgc tgtgacaggc agaaatgcag ggcaaagccc 240
cagggacagt gggcagaaga tgtcctggga aataagacat gggacagaga gaccagagac 300
ttgacagggt ggggaaagga cctcaggatg accctggctc atatcaagga ccagaaagaa 360
ggcttgcatt ccctccagga gattagggtc tgtgagatcc atgaagacaa cagcaccagg 420
agctcccagc atttctacta cgatggggag ctctttctct cccaaaacct ggagactaag 480
gaatggacaa tgccccagtc ctccagagct cagaccttgg ccatgaacgt caggaatttc 540
ttgaaggaag atgcaatgga gaccgataca cactatcacg ctatgcatgc agactgcctg 600
caggaactac ggcgatatct aaaatccggc gtagtcctga ggagaacagt gccccccatg 660
gtgaatgtca cccgcagcga ggcctcagag ggcaacatta ccgtgacatg cagggcttct 720
ggcttctatc cctggaatat cacactgagc tggcgtcagg atggggtatc tttgagccac 780
gacacccagc agtgggggga tgtcctgcct gatgggaatg gaacctacca gacctgggtg 840
gccaccagga tttgccaagg agaggagcag aggttcacct gctacatgga acacagcggg 900
aatcacagca ctcaccctgt gccctctggg aaaatcgaag gacgcatgga cccaaagagt 960
tgcgacaaaa ctcacacatg cccaccgtgc ccaggtaagc cagcccaggc ctcgccctcc 1020
agctcaaggc gggacaggtg ccctagagta gcctgcatcc agggacaggc cccagccggg 1080
tgctgacacg tccacctcca tctcttcctc agcacctgaa ctcctggggg gaccgtcagt 1140
cttcctcttc cccccaaaac ccaaggacac cctcatgatc tcccggaccc ctgaggtcac 1200
atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc aagttcaact ggtacgtgga 1260
cggcgtggag gtgcataatg ccaagacaaa gccgcgggag gagcagtaca acagcacgta 1320
ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg ctgaatggca aggagtacaa 1380
gtgcaaggtc tccaacaaag ccctcccagc ccccatcgag aaaaccatct ccaaagccaa 1440
aggtgggacc cgtggggtgc gagggccaca tggacagagg ccggctcggc ccaccctctg 1500
ccctgagagt gactgctgta ccaacctctg tccctacagg gcagccccga gaaccacagg 1560
tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc ctgacctgcc 1620
tggtcaaagg cttctatccc agcgacatcg ccgtggagtg ggagagcaat gggcagccgg 1680
agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc ttcctctaca 1740
gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca tgctccgtga 1800
tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct ccgggtaaat 1860
gataggatcc ggttgaggtc tctaaaagcg tcttcctgtt ctcatcacat catatcaagg 1920
ttatatacca tcaatattgc cacagatgtt acttagcctt ttaatatttc tctaatttag 1980
tgtatatgca atgatagttc tctgatttct gagattgagt ttctcatgtg taatgattat 2040
ttagagtttc tctttcatct gttcaaattt ttgtctagtt ttatttttta ctgatttgta 2100
agacttcttt ttataatctg catattacaa ttctctttac tggggtgttg caaatatttt 2160
ctgtcattct atggcctgac ttttcttaat ggttttttaa ttttaaaaat aagtcttaat 2220
attcatgcaa tctaattaac aatcttttct ttgtggttag gactttgagt cataagaaat 2280
ttttctctac actgaagtca tgatggcatg cttctatatt attttctaaa agatttaaag 2340
ttttgccttc tccatttaga cttataattc actggaattt ttttgtgtgt atggtatgac 2400
atatgggttc ccttttattt tttacatata aatatatttc cctgtttttc taaaaaagaa 2460
aaagatcatc attttcccat tgtaaaatgc catatttttt tcataggtca cttacatata 2520
tcaatgggtc tgtttctgag ctctactcta ttttatcagc ctcactgtct atccccacac 2580
atctcatgct ttgctctaaa tcttgatatt tagtggaaca ttctttccca ttttgttcta 2640
caagaatatt tttgttattg tctttgggct ttctatatac attttgaaat gaggttgaca 2700
agttaataat caacctctgg attacaaaat ttgtgaaaga ttgactggta ttcttaacta 2760
tgttgctcct tttacgctat gtggatacgc tgctttaatg cctttgtatc atgctattgc 2820
ttcccgtatg gctttcattt tctcctcctt gtataaatcc tggttgctgt ctctttatga 2880
ggagttgtgg cccgttgtca ggcaacgtgg cgtggtgtgc actgtgtttg ctgacgcaac 2940
ccccactggt tggggcattg ccaccacctg tcagctcctt tccgggactt tcgctttccc 3000
cctccctatt gccacggcgg aactcatcgc cgcctgcctt gcccgctgct ggacaggggc 3060
tcggctgttg ggcactgaca attccgtggt gttgtcgggg aaatcatcgt cctttccttg 3120
gctgctcgcc tgtgttgcca cctggattct gcgcgggacg tccttctgct acgtcccttc 3180
ggccctcaat ccagcggacc ttccttcccg cggcctgctg ccggctctgc ggcctcttcc 3240
gcctcttcgc cttcgccctc agacgagtcg gatctccctt tgggccgcct ccccgcatct 3300
gtgccttcta gttgccagcc atctgttgtt tgcccctccc ccgtgccttc cttgaccctg 3360
gaaggtgcca ctcccactgt cctttcctaa taaaatgagg aaattgcatc gcattgtctg 3420
agtaggtgtc attctattct ggggggtggg gtggggcagg acagcaaggg ggaggattgg 3480
caagacaata gcaggctttg catttttaga catttagaag cctatatctt gttacagaat 3540
tggaattaca caaaaattct accatatttt gaaagcttag gttgttctga aaaaaacaat 3600
atattgtttt cctgggtaaa ctaaaagtcc cctcgaggaa aggcccctaa agtgaaacag 3660
tgcaaaacgt tcaaaaactg tctggcaata caagttccac tttgaccaaa acggctggca 3720
gtaaaagggt taagaagact gtcagccttg agcggtatca gctcactcaa aggcggtaat 3780
acggttatcc acagaatcag gggataacgc aggaaagaac atgtgagcaa aaggccagca 3840
aaaggccagg aaccgtaaaa aggccgcgtt gctggcgttt ttccataggc tccgcccccc 3900
tgacgagcat cacaaaaatc gacgctcaag tcagaggtgg cgaaacccga caggactata 3960
aagataccag gcgtttcccc ctggaagctc cctcgtgcgc tctcctgttc cgaccctgcc 4020
gcttaccgga tacctgtccg cctttctccc ttcgggaagc gtggcgcttt ctcatagctc 4080
acgctgtagg tatctcagtt cggtgtaggt cgttcgctcc aagctgggct gtgtgcacga 4140
accccccgtt cagcccgacc gctgcgcctt atccggtaac tatcgtcttg agtccaaccc 4200
ggtaagacac gacttatcgc cactggcagc agccactggt aacaggatta gcagagcgag 4260
gtatgtaggc ggtgctacag agttcttgaa gtggtgggct aactacggct acactagaag 4320
aacagtattt ggtatctgcg ctctgctgaa gccagttacc ttcggaaaaa gagttggtag 4380
ctcttgatcc ggcaaacaaa ccaccgctgg tagcggtggt ttttttgttt gcaagcagca 4440
gattacgcgc agaaaaaaag gatctcaaga agatcctttg atcttttcta cggggtctga 4500
cgctcagtgg aacgacgcgc gcgtaactca cgttaaggga ttttggtcat gagttagaaa 4560
aactcatcga gcatcaaatg aaactgcaat ttattcatat caggattatc aataccatat 4620
ttttgaaaaa gccgtttctg taatgaagga gaaaactcac cgaggcagtt ccataggatg 4680
gcaagatcct ggtatcggtc tgcgattccg actcgtccaa catcaataca acctattaat 4740
ttcccctcgt caaaaataag gttatcaagt gagaaatcac catgagtgac gactgaatcc 4800
ggtgagaatg gcaaaagttt atgcatttct ttccagactt gttcaacagg ccagccatta 4860
cgctcgtcat caaaatcact cgcatcaacc aaaccgttat tcattcgtga ttgcgcctga 4920
gcgaggcgaa atacgcgatc gctgttaaaa ggacaattac aaacaggaat cgagtgcaac 4980
cggcgcagga acactgccag cgcatcaaca atattttcac ctgaatcagg atattcttct 5040
aatacctgga acgctgtttt tccggggatc gcagtggtga gtaaccatgc atcatcagga 5100
gtacggataa aatgcttgat ggtcggaagt ggcataaatt ccgtcagcca gtttagtctg 5160
accatctcat ctgtaacatc attggcaacg ctacctttgc catgtttcag aaacaactct 5220
ggcgcatcgg gcttcccata caagcgatag attgtcgcac ctgattgccc gacattatcg 5280
cgagcccatt tatacccata taaatcagca tccatgttgg aatttaatcg cggcctcgac 5340
gtttcccgtt ggatatggct cattttttac ttcctcacct tgtcgtatta tactatgccg 5400
atatactatg ccgatgatta attgtcgaca ctgcgggggc tctgtgtggt aagcaggtct 5460
taaccttttt actgccaatg acgcatggga tacgtcgtgg cagtaaaagg gcttaaatgc 5520
caacgacgcg tcccatacgt tgttggcatt ttaattcttc tctctgcagc ggcagcatgt 5580
gccgccgctg cagagagttt ctagcgatga cagcccctct gggcaacgag ccgggggggc 5640
tgtctttctt tatgttttaa atgcactgac ctcccacatt ccctttttag taaaatattc 5700
agaaataatt taaatacatc attgcaatga aaataaatgt tttttattag gcagaatcca 5760
gatgctcaag gcccttcata atatccccca gtttagtagt tggacttagg gaacaaagga 5820
acctttaata gaaattggac agcaagaaag cgagtcaggc accgggcttg cgggtcatgc 5880
accaggtgcg cggtccttcg ggcacctcga cgtcggcggt gacggtgaag ccgagccgct 5940
cgtagaaggg gaggttgcgg ggcgcggatg tctccaggaa ggcgggcacc ccggcgcgct 6000
cggccgcctc cactccgggg agcacgacgg cgctgcccag acccttgccc tggtggtcgg 6060
gcgacacgcc gacggtggcc aggaaccacg cgggctcctt gggccggtgc ggcgccagga 6120
ggccttccat ctgttgctgc gcggccagcc gggaaccgct caactcggcc atgcgcgggc 6180
cgatctcggc gaacaccgcc cccgcttcga cgctctccgg cgtggtccag accgccaccg 6240
cggcgccgtc gtccgcgacc cacaccttgc cgatgtcgag cccgacgcgc gtgaggaaga 6300
gttcttgcag ctcggtgacc cgctcgatgt ggcggtccgg atcgacggtg tggcgcgtgg 6360
cggggtagtc ggcgaacgcg gcggcgaggg tgcgtacggc cctggggacg tcgtcgcggg 6420
tggcgaggcg caccgtgggc ttgtactcgg tcatggtggc ggacgaaagg cccggagatg 6480
aggaagagga gaacagcgcg gcagacgtgc gcttttgaag cgtgcagaat gccgggcctc 6540
cggaggacct tcgggcgccc gccccgcccc tgagcccgcc cctgagcccg cccccggacc 6600
caccccttcc cagcctctga gcccagaaag cgaaggagca aagctgctat tggccgctgc 6660
cccaaaggcc tacccgcttc cattgctcag cggtgctgtc catctgcacg agactagtga 6720
gtcgtgctac ttccatttgt cacgtcctgc acgacgcgag ctgcggggcg ggggggaact 6780
tcctgactag gggaggagta gaaggtggcg cgaaggggcc accaaagaac ggagccggtt 6840
ggcgcctacc ggtggatgtg gaatgtgtgc gaggccagag gccacttgtg tagcgccaag 6900
tgcccagcgg ggctgctaaa gcgcatgctc cagactgcct tgggaaaagc gcctccccta 6960
cccggtagag aaacttgatc tgtcgccgca attcaaactt cgtgaggctc cggtgcccgt 7020
cagtgacctg ctatactctg gagacgactt acggtaaatg gcccgcctgg ctgaccgccc 7080
aacgaccccc gcccattgac gtcaataatg acgtatgttc ccatagtaac gccaataggg 7140
actttccatt gacgtcaatg ggtggagtat ttacggtaaa ctgcccactt ggcagtacat 7200
caagtgtatc atatgccaag tccgccccct attgacgtca atgacggtaa atggcccgcc 7260
tggcattatg cccagtacat gaccttacgg gactttccta cttggcagta catctacgta 7320
ttagtcatcg ctattaccat gctgatgcgg ttttggcagt acaccaatgg gcgtggatag 7380
cggtttgact cacggggatt tccaagtctc caccccattg acgtcaatgg gagtttgttt 7440
tggcaccaaa atcaacggga ctttccaaaa tgtcgtaata accccgcccc gttgacgcaa 7500
atgggcggta ggcgtgtacg gtgggaggtc tatataagca gagctcgttt agtgaaccgt 7560
cagatcgcct ggagaggcca tccacgctgt tttgacctcc atagtggaca ccgggaccga 7620
tccagcctcc gcgtctcagg 7640
<210> 58
<211> 438
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, MICA-Fc
<400> 58
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Asn Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Lys Thr Lys Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ile Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys
275 280 285
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
290 295 300
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
305 310 315 320
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
325 330 335
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
340 345 350
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
355 360 365
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
370 375 380
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
385 390 395 400
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
405 410 415
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
420 425 430
Cys Leu Val Lys Gly Phe
435
<210> 59
<211> 513
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, MICwed-Fc
<400> 59
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Lys Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
His Ala Asp Cys Leu Gln Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ile Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys
275 280 285
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
290 295 300
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
305 310 315 320
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
325 330 335
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
340 345 350
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
355 360 365
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
370 375 380
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
385 390 395 400
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
405 410 415
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
420 425 430
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
435 440 445
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
450 455 460
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
465 470 475 480
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
485 490 495
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
500 505 510
Lys
<210> 60
<211> 513
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, MICv25-Fc
<400> 60
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Lys Asp Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Thr His Tyr His Ala Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val Pro Pro Met Val Asn Val Thr Arg Ser Glu
180 185 190
Ala Ser Glu Gly Asn Ile Thr Val Thr Cys Arg Ala Ser Gly Phe Tyr
195 200 205
Pro Trp Asn Ile Thr Leu Ser Trp Arg Gln Asp Gly Val Ser Leu Ser
210 215 220
His Asp Thr Gln Gln Trp Gly Asp Val Leu Pro Asp Gly Asn Gly Thr
225 230 235 240
Tyr Gln Thr Trp Val Ala Thr Arg Ile Ser Gln Gly Glu Glu Gln Arg
245 250 255
Phe Thr Cys Tyr Met Glu His Ser Gly Asn His Ser Thr His Pro Val
260 265 270
Pro Ser Gly Lys Ile Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys
275 280 285
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
290 295 300
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
305 310 315 320
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
325 330 335
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
340 345 350
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
355 360 365
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
370 375 380
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
385 390 395 400
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
405 410 415
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
420 425 430
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
435 440 445
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
450 455 460
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
465 470 475 480
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
485 490 495
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
500 505 510
Lys
<210> 61
<211> 189
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP2 альфа1-альфа2
<400> 61
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Arg
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Val Val Ala Met Ser Phe His Tyr Phe Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro Pro Met Val
180 185
<210> 62
<211> 187
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP3 альфа1-альфа2
<400> 62
Ala Ala Glu Pro His Ser Leu Trp Tyr Asn Phe Thr Ile Ile His Leu
1 5 10 15
Pro Arg His Gly Gln Gln Trp Cys Glu Val Gln Ser Gln Val Asp Gln
20 25 30
Lys Asn Phe Leu Ser Tyr Asp Cys Gly Ser Asp Lys Val Leu Ser Met
35 40 45
Gly His Leu Glu Glu Gln Leu Tyr Ala Thr Asp Ala Trp Gly Lys Gln
50 55 60
Leu Glu Met Leu Arg Glu Val Gly Gln Arg Leu Arg Leu Glu Leu Ala
65 70 75 80
Asp Thr Glu Leu Glu Asp Phe Thr Pro Ser Gly Pro Leu Thr Leu Gln
85 90 95
Val Arg Met Ser Cys Glu Ser Glu Ala Asp Gly Tyr Ile Arg Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Arg Lys Phe Leu Leu Phe Asp Ser Asn
115 120 125
Asn Arg Lys Trp Thr Val Val His Ala Gly Ala Arg Arg Met Lys Glu
130 135 140
Lys Trp Glu Lys Asp Ser Gly Leu Thr Thr Phe Phe Lys Met Val Ser
145 150 155 160
Met Arg Asp Cys Lys Ser Trp Leu Arg Asp Phe Leu Met His Arg Lys
165 170 175
Lys Arg Leu Glu Pro Thr Ala Pro Pro Met Val
180 185
<210> 63
<211> 186
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 R80W
<400> 63
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Val Val Ala Met Ser Phe His Tyr Phe Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro
180 185
<210> 64
<211> 186
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 V151D
<400> 64
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Arg
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Asp Val Ala Met Ser Phe His Tyr Phe Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro
180 185
<210> 65
<211> 184
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP3 R162G
<400> 65
Ala Ala Glu Pro His Ser Leu Trp Tyr Asn Phe Thr Ile Ile His Leu
1 5 10 15
Pro Arg His Gly Gln Gln Trp Cys Glu Val Gln Ser Gln Val Asp Gln
20 25 30
Lys Asn Phe Leu Ser Tyr Asp Cys Gly Ser Asp Lys Val Leu Ser Met
35 40 45
Gly His Leu Glu Glu Gln Leu Tyr Ala Thr Asp Ala Trp Gly Lys Gln
50 55 60
Leu Glu Met Leu Arg Glu Val Gly Gln Arg Leu Arg Leu Glu Leu Ala
65 70 75 80
Asp Thr Glu Leu Glu Asp Phe Thr Pro Ser Gly Pro Leu Thr Leu Gln
85 90 95
Val Arg Met Ser Cys Glu Ser Glu Ala Asp Gly Tyr Ile Arg Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Arg Lys Phe Leu Leu Phe Asp Ser Asn
115 120 125
Asn Arg Lys Trp Thr Val Val His Ala Gly Ala Arg Arg Met Lys Glu
130 135 140
Lys Trp Glu Lys Asp Ser Gly Leu Thr Thr Phe Phe Lys Met Val Ser
145 150 155 160
Met Gly Asp Cys Lys Ser Trp Leu Arg Asp Phe Leu Met His Arg Lys
165 170 175
Lys Arg Leu Glu Pro Thr Ala Pro
180
<210> 66
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, MICA25.17
<400> 66
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Thr Thr Leu Leu Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Ile Gly Tyr Arg Leu Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 67
<211> 182
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, MICA25.18
<400> 67
Glu Pro His Ser Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly
1 5 10 15
Ser Val Gln Ser Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro
20 25 30
Phe Leu Arg Cys Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln
35 40 45
Trp Ala Glu Asp Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg
50 55 60
Asp Leu Thr Gly Trp Gly Thr Phe Leu Arg Met Thr Leu Ala His Ile
65 70 75 80
Lys Asp Gln Lys Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys
85 90 95
Glu Ile His Glu Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr
100 105 110
Asp Gly Glu Leu Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr
115 120 125
Met Pro Gln Ser Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn
130 135 140
Phe Leu Lys Glu Asp Ala Met Glu Thr Asp Arg Ser Gly Leu Leu Met
145 150 155 160
Arg Ala Asp Cys Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val
165 170 175
Val Leu Arg Arg Thr Val
180
<210> 68
<211> 186
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2.S1
<400> 68
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Val Val Ala Thr Thr Leu Tyr Thr Trp Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro
180 185
<210> 69
<211> 186
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2.S2
<400> 69
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Val Val Ala Thr Leu Met Arg Ile Trp Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro
180 185
<210> 70
<211> 186
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2.S3
<400> 70
Ala Ala Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys
1 5 10 15
Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu
20 25 30
Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val
35 40 45
Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln
50 55 60
Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp
65 70 75 80
Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln
85 90 95
Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu
115 120 125
Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu
130 135 140
Lys Trp Glu Asn Asp Lys Val Val Ala Thr Lys Leu Tyr Leu Trp Ser
145 150 155 160
Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp
165 170 175
Ser Thr Leu Glu Pro Ser Ala Gly Ala Pro
180 185
<210> 71
<211> 184
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP3.S1
<400> 71
Ala Ala Glu Pro His Ser Leu Trp Tyr Asn Phe Thr Ile Ile His Leu
1 5 10 15
Pro Arg His Gly Gln Gln Trp Cys Glu Val Gln Ser Gln Val Asp Gln
20 25 30
Lys Asn Phe Leu Ser Tyr Asp Cys Gly Ser Asp Lys Val Leu Ser Met
35 40 45
Gly His Leu Glu Glu Gln Leu Tyr Ala Thr Asp Ala Trp Gly Lys Gln
50 55 60
Leu Glu Met Leu Arg Glu Val Gly Gln Arg Leu Arg Leu Glu Leu Ala
65 70 75 80
Asp Thr Glu Leu Glu Asp Phe Thr Pro Ser Gly Pro Leu Thr Leu Gln
85 90 95
Val Arg Met Ser Cys Glu Ser Glu Ala Asp Gly Tyr Ile Arg Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Arg Lys Phe Leu Leu Phe Asp Ser Asn
115 120 125
Asn Arg Lys Trp Thr Val Val His Ala Gly Ala Arg Arg Met Lys Glu
130 135 140
Lys Trp Glu Lys Asp Ser Gly Leu Thr Thr Asp Leu Ile Arg Arg Ser
145 150 155 160
Met Gly Asp Cys Lys Ser Trp Leu Arg Asp Phe Leu Met His Arg Lys
165 170 175
Lys Arg Leu Glu Pro Thr Ala Pro
180
<210> 72
<211> 184
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULBP3.S2
<400> 72
Ala Ala Glu Pro His Ser Leu Trp Tyr Asn Phe Thr Ile Ile His Leu
1 5 10 15
Pro Arg His Gly Gln Gln Trp Cys Glu Val Gln Ser Gln Val Asp Gln
20 25 30
Lys Asn Phe Leu Ser Tyr Asp Cys Gly Ser Asp Lys Val Leu Ser Met
35 40 45
Gly His Leu Glu Glu Gln Leu Tyr Ala Thr Asp Ala Trp Gly Lys Gln
50 55 60
Leu Glu Met Leu Arg Glu Val Gly Gln Arg Leu Arg Leu Glu Leu Ala
65 70 75 80
Asp Thr Glu Leu Glu Asp Phe Thr Pro Ser Gly Pro Leu Thr Leu Gln
85 90 95
Val Arg Met Ser Cys Glu Ser Glu Ala Asp Gly Tyr Ile Arg Gly Ser
100 105 110
Trp Gln Phe Ser Phe Asp Gly Arg Lys Phe Leu Leu Phe Asp Ser Asn
115 120 125
Asn Arg Lys Trp Thr Val Val His Ala Gly Ala Arg Arg Met Lys Glu
130 135 140
Lys Trp Glu Lys Asp Ser Gly Leu Thr Thr Tyr Phe Tyr Leu Arg Ser
145 150 155 160
Met Gly Asp Cys Lys Ser Trp Leu Arg Asp Phe Leu Met His Arg Lys
165 170 175
Lys Arg Leu Glu Pro Thr Ala Pro
180
<210> 73
<211> 657
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, R3 HC25.17
<400> 73
Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu Ala
1 5 10 15
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
20 25 30
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Thr
35 40 45
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
50 55 60
Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala Asp Ser Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
85 90 95
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr Thr Glu Tyr
115 120 125
Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
130 135 140
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
145 150 155 160
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
165 170 175
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
180 185 190
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
195 200 205
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
210 215 220
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
225 230 235 240
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Ser Glu Pro His Ser
465 470 475 480
Leu Arg Tyr Asn Leu Thr Val Leu Ser Trp Asp Gly Ser Val Gln Ser
485 490 495
Gly Phe Leu Thr Glu Val His Leu Asp Gly Gln Pro Phe Leu Arg Cys
500 505 510
Asp Arg Gln Lys Cys Arg Ala Lys Pro Gln Gly Gln Trp Ala Glu Asp
515 520 525
Val Leu Gly Asn Lys Thr Trp Asp Arg Glu Thr Arg Asp Leu Thr Gly
530 535 540
Trp Gly Thr Thr Leu Leu Met Thr Leu Ala His Ile Lys Asp Gln Lys
545 550 555 560
Glu Gly Leu His Ser Leu Gln Glu Ile Arg Val Cys Glu Ile His Glu
565 570 575
Asp Asn Ser Thr Arg Ser Ser Gln His Phe Tyr Tyr Asp Gly Glu Leu
580 585 590
Phe Leu Ser Gln Asn Leu Glu Thr Leu Glu Trp Thr Met Pro Gln Ser
595 600 605
Ser Arg Ala Gln Thr Leu Ala Met Asn Val Arg Asn Phe Leu Lys Glu
610 615 620
Asp Ala Met Glu Thr Asp Ile Gly Tyr Arg Leu Met Arg Ala Asp Cys
625 630 635 640
Leu Ser Glu Leu Arg Arg Tyr Leu Lys Ser Gly Val Val Leu Arg Arg
645 650 655
Thr
<210> 74
<211> 656
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, R3 HC.U2S3
<400> 74
Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu Ala
1 5 10 15
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
20 25 30
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Ser Thr
35 40 45
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
50 55 60
Gly Arg Ile Tyr Pro Thr Asn Gly Ser Thr Asn Tyr Ala Asp Ser Val
65 70 75 80
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
85 90 95
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Arg Thr Tyr Gly Ile Tyr Asp Leu Tyr Val Asp Tyr Thr Glu Tyr
115 120 125
Val Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
130 135 140
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
145 150 155 160
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
165 170 175
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
180 185 190
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
195 200 205
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
210 215 220
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
225 230 235 240
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly Ser Glu Pro His Ser
465 470 475 480
Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg Pro Gly Pro Arg
485 490 495
Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr
500 505 510
Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys
515 520 525
Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu
530 535 540
Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile Gln Leu Glu Asn
545 550 555 560
Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu
565 570 575
Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln Phe Ser Phe Asp
580 585 590
Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg Met Trp Thr Thr
595 600 605
Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys
610 615 620
Val Val Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly Asp Cys Ile Gly
625 630 635 640
Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser
645 650 655
<210> 75
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152A
<400> 75
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 76
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y199A
<400> 76
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Ala Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 77
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152A/Y199A
<400> 77
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Ala Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 78
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y199F eNKG2D1
<400> 78
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 79
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152S eNKG2D2
<400> 79
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ser His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 80
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152T eNKG2D3
<400> 80
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Thr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 81
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152V eNKG2D4
<400> 81
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Val His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 82
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152A/Y199F eNKG2D5
<400> 82
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 83
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152L/Y199F eNKG2D6
<400> 83
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Leu His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 84
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152S/Y199F eNKG2D7
<400> 84
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ser His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 85
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152T/Y199F eNKG2D8
<400> 85
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Thr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 86
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152V/Y199F eNKG2D9
<400> 86
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Val His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 87
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y199D eNKG2D10
<400> 87
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Asp Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 88
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y199E eNKG2D11
<400> 88
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Glu Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 89
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152D/Y199D eNKG2D12
<400> 89
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Asp His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Asp Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 90
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152E/Y199E eNKG2D13
<400> 90
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Glu His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Glu Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 91
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D Y152L eNKG2D14
<400> 91
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Leu His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 92
<211> 139
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид неприродный эктодомен NKG2D
Y152F/Y199F eNKG2D15
<400> 92
Phe Leu Asn Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu
1 5 10 15
Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn
20 25 30
Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala
35 40 45
Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu
50 55 60
Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Phe His Trp Met Gly Leu
65 70 75 80
Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile
85 90 95
Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys
100 105 110
Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr
115 120 125
Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
130 135
<210> 93
<211> 237
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид Fc IgG1 человека с линкером IEGR
<400> 93
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg
225 230 235
<210> 94
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
<400> 94
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 95
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D Y152A
<400> 95
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 96
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид Fc IgG1 человека-эктодомен NKG2D Y199A
<400> 96
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Ala Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 97
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид Fc IgG1 человека-эктодомен NKG2D Y152A/Y199A
<400> 97
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Ala Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 98
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y199F eNKG2D1
<400> 98
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 99
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y152S eNKG2D2
<400> 99
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 100
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y152T eNKG2D3
<400> 100
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Thr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 101
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y152V eNKG2D4
<400> 101
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Val His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 102
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152A/Y199F eNKG2D5
<400> 102
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 103
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152L/Y199F eNKG2D6
<400> 103
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Leu His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 104
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152S/Y199F eNKG2D7
<400> 104
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Ser His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 105
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152T/Y199F eNKG2D8
<400> 105
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Thr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 106
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152V/Y199F eNKG2D9
<400> 106
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Val His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 107
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y199D eNKG2D10
<400> 107
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Asp Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 108
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y199E eNKG2D11
<400> 108
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Glu Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 109
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152D/Y199D eNKG2D12
<400> 109
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Asp His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Asp Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 110
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152E/Y199E eNKG2D13
<400> 110
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Glu His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Glu Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 111
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1 человека-NKG2D
Y152L eNKG2D14
<400> 111
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Leu His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 112
<211> 376
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид слитый белок Fc IgG1
человека-NKG2D Y152F/Y199F eNKG2D15
<400> 112
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
20 25 30
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
35 40 45
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
50 55 60
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
65 70 75 80
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
85 90 95
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
100 105 110
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
115 120 125
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
130 135 140
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
145 150 155 160
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
165 170 175
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
180 185 190
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
195 200 205
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
210 215 220
Lys Ser Leu Ser Leu Ser Pro Gly Lys Ile Glu Gly Arg Phe Leu Asn
225 230 235 240
Ser Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Cys
245 250 255
Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn Asn Cys Tyr Gln
260 265 270
Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln Ala Ser Cys Met
275 280 285
Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys Glu Asp Gln Asp
290 295 300
Leu Leu Lys Leu Val Lys Ser Phe His Trp Met Gly Leu Val His Ile
305 310 315 320
Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser Ile Leu Ser Pro
325 330 335
Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp Cys Ala Leu Tyr
340 345 350
Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser Thr Pro Asn Thr
355 360 365
Tyr Ile Cys Met Gln Arg Thr Val
370 375
<210> 113
<211> 23
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид сигнальная последовательность MHCI
<400> 113
Met Gly Leu Gly Pro Val Phe Leu Leu Leu Ala Gly Ile Phe Pro Phe
1 5 10 15
Ala Pro Pro Gly Ala Ala Ala
20
<210> 114
<211> 69
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий
сигнальную последовательность MHCI
<400> 114
atgggccttg gcccagtgtt tctgctgttg gcaggcattt tcccttttgc tccgcccggc 60
gccgcagcc 69
<210> 115
<211> 711
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий Fc IgG1 человека с
линкером IEGR
<400> 115
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg c 711
<210> 116
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок
Fc IgG1 человека-NKG2D
<400> 116
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcatatcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggctatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 117
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок
Fc IgG1 человека-NKG2D Y152A
<400> 117
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagctcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggctatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 118
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий Fc IgG1 человека-эктодомен
NKG2D Y199A
<400> 118
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcataccatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgctata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 119
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий Fc IgG1 человека-эктодомен
NKG2D Y152A/Y199A
<400> 119
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagctcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgctata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 120
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y199F eNKG2D1
<400> 120
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcataccatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 121
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152S eNKG2D2
<400> 121
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcataccatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 122
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152T eNKG2D3
<400> 122
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcaactcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggctatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 123
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152V eNKG2D4
<400> 123
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagtgcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggctatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 124
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152A/Y199F eNKG2D5
<400> 124
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagctcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 125
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152L/Y199F eNKG2D6
<400> 125
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcactgcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 126
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152S/Y199F eNKG2D7
<400> 126
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcaagtcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 127
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152T/Y199F eNKG2D8
<400> 127
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcaactcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 128
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152V/Y199F eNKG2D9
<400> 128
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagtgcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 129
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y199D eNKG2D10
<400> 129
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcatatcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 130
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y199E eNKG2D11
<400> 130
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcataccatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgagata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 131
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152D/Y199D eNKG2D12
<400> 131
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagatcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 132
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152E/Y199E eNKG2D13
<400> 132
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcagagcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcgagata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 133
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152L eNKG2D14
<400> 133
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcactgcatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggctatata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 134
<211> 1128
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий слитый белок Fc IgG1
человека-NKG2D Y152F/Y199F eNKG2D15
<400> 134
atggacccga aaagctgcga caagactcac acttgtccgc cgtgccccgc ccccgaactg 60
ctgggtggcc cctccgtgtt cctgttcccg cctaagccta aggacaccct tatgatcagc 120
cgcacccctg aagtgacctg tgtcgtcgtg gatgtgtcac acgaggaccc ggaggtcaag 180
ttcaattggt acgtggacgg cgtggaagtg cataacgcaa agaccaagcc tcgggaggaa 240
cagtacaact cgacctaccg cgtggtgtca gtcctgactg tgctgcacca ggactggctg 300
aacgggaagg agtacaagtg caaagtgtcg aacaaggccc tgccggctcc aattgaaaag 360
accatcagca aggccaaggg ccagccaagg gaaccacagg tgtacaccct ccctccttcc 420
cgggacgagc tgaccaaaaa ccaagtgtcc ctgacttgcc ttgtgaaggg gttctaccct 480
tctgacattg ccgtcgaatg ggaatcgaac ggacagcctg aaaacaacta taagactacc 540
ccgcccgtgc tggattccga cggaagcttc ttcctgtact ccaagctgac cgtggacaag 600
tcgagatggc agcagggaaa tgtgttcagc tgctccgtga tgcatgaggc gctgcacaac 660
cactacaccc agaagtcact gagcctctcc cccggaaaga tcgaaggacg cttcttaaac 720
tcattattca accaagaagt tcaaattccc ttgaccgaaa gttactgtgg cccatgtcct 780
aaaaactgga tatgttacaa aaataactgc taccaatttt ttgatgagag taaaaactgg 840
tatgagagcc aggcttcttg tatgtctcaa aatgccagcc ttctgaaagt atacagcaaa 900
gaggaccagg atttacttaa actggtgaag tcattccatt ggatgggact agtacacatt 960
ccaacaaatg gatcttggca gtgggaagat ggctccattc tctcacccaa cctactaaca 1020
ataattgaaa tgcagaaggg agactgtgca ctctatgcct cgagctttaa aggcttcata 1080
gaaaactgtt caactccaaa tacatacatc tgcatgcaaa ggactgtg 1128
<210> 135
<211> 107
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид легкая цепь каппа человека
<400> 135
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 136
<211> 330
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид CH1-CH2-CH3 тяжелой цепи IgG1 человека
D265A/N297A
<400> 136
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Ala Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 137
<211> 450
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид тяжелая цепь трастузумаба
<400> 137
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Tyr Pro Thr Asn Gly Tyr Thr Arg Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ser Arg Trp Gly Gly Asp Gly Phe Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 138
<211> 213
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид легкая цепь трастузумаба
<400> 138
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 139
<211> 451
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид тяжелая цепь ритуксимаба
<400> 139
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Arg Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Tyr Gly Gly Asp Trp Tyr Phe Asn Val Trp Gly
100 105 110
Ala Gly Thr Thr Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 140
<211> 212
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид легкая цепь ритуксимаба
<400> 140
Gln Ile Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Ser Tyr Ile
20 25 30
His Trp Phe Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Val Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Thr Ser Asn Pro Pro Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Thr Val Ala Ala Pro Ser
100 105 110
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
115 120 125
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
130 135 140
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
145 150 155 160
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
165 170 175
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
180 185 190
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
195 200 205
Arg Gly Glu Cys
210
<210> 141
<211> 404
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ритуксимаб LC_ULBP2.wt
<400> 141
Gln Ile Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Ser Tyr Ile
20 25 30
His Trp Phe Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Val Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Thr Ser Asn Pro Pro Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys Ala Pro Thr Ser Ser Ser Gly Gly Gly Gly Ser
210 215 220
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
225 230 235 240
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
245 250 255
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
260 265 270
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
275 280 285
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Arg Asp Ile
290 295 300
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
305 310 315 320
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
325 330 335
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
340 345 350
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
355 360 365
Glu Asn Asp Lys Val Val Ala Met Ser Phe His Tyr Phe Ser Met Gly
370 375 380
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
385 390 395 400
Leu Glu Pro Ser
<210> 142
<211> 187
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ULPB2 альфа1-альфа2 вариант R80W
<400> 142
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Met Ser Phe His Tyr Phe Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser Ala Gly Ala Pro Pro Met Val
180 185
<210> 143
<211> 180
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 альфа1-альфа2 вариант ULBP2.C
<400> 143
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Thr Ile Leu Trp Gln Thr Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser
180
<210> 144
<211> 540
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий ULBP2 альфа1-альфа2
вариант ULBP2.C
<400> 144
gagccccata gtctgagcta cgacatcaca gttattccca agttcaggcc cggaccgcgc 60
tggtgtgccg tgcaaggaca agtcgacgaa aaaacctttc ttcattacga ttgcggaaat 120
aagactgtaa cgccagtctc tcctttaggt aagaagttaa acgtcactac ggcgtggaag 180
gcacaaaacc ccgtcctgcg cgaggtcgtc gacatcctga ctgaacaatt gtgggacatc 240
cagctcgaga attacactcc aaaggagcct cttaccctgc aggctagaat gtcttgcgag 300
caaaaggcag agggccactc ctccggcagc tggcagttca gtttcgacgg acaaatcttt 360
ctgttattcg attcagagaa gagaatgtgg actacagttc accccggtgc ccgtaaaatg 420
aaggagaagt gggaaaacga caaagtggtg gcgactattc tgtggcagac ttcgatggga 480
gactgcatcg gttggctgga agatttcctc atgggtatgg actccacttt ggagccatcg 540
<210> 145
<211> 180
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 альфа1-альфа2 вариант ULBP2.R
<400> 145
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Thr Leu Leu Trp Gly Trp Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser
180
<210> 146
<211> 540
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий ULBP2 альфа1-альфа2
вариант ULBP2.R
<400> 146
gagccccata gtctgagcta cgacatcaca gttattccca agttcaggcc cggaccgcgc 60
tggtgtgccg tgcaaggaca agtcgacgaa aaaacctttc ttcattacga ttgcggaaat 120
aagactgtaa cgccagtctc tcctttaggt aagaagttaa acgtcactac ggcgtggaag 180
gcacaaaacc ccgtcctgcg cgaggtcgtc gacatcctga ctgaacaatt gtgggacatc 240
cagctcgaga attacactcc aaaggagcct cttaccctgc aggctagaat gtcttgcgag 300
caaaaggcag agggccactc ctccggcagc tggcagttca gtttcgacgg acaaatcttt 360
ctgttattcg attcagagaa gagaatgtgg actacagttc accccggtgc ccgtaaaatg 420
aaggagaagt gggaaaacga caaagtggtg gcgactttgt tgtgggggtg gtcgatggga 480
gactgcatcg gttggctgga agatttcctc atgggtatgg actccacttt ggagccatcg 540
<210> 147
<211> 180
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 альфа1-альфа2 вариант ULBP2.AA
<400> 147
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Thr Met Phe Trp Ser Trp Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser
180
<210> 148
<211> 540
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий ULBP2 альфа1-альфа2
вариант ULBP2.AA
<400> 148
gagccccata gtctgagcta cgacatcaca gttattccca agttcaggcc cggaccgcgc 60
tggtgtgccg tgcaaggaca agtcgacgaa aaaacctttc ttcattacga ttgcggaaat 120
aagactgtaa cgccagtctc tcctttaggt aagaagttaa acgtcactac ggcgtggaag 180
gcacaaaacc ccgtcctgcg cgaggtcgtc gacatcctga ctgaacaatt gtgggacatc 240
cagctcgaga attacactcc aaaggagcct cttaccctgc aggctagaat gtcttgcgag 300
caaaaggcag agggccactc ctccggcagc tggcagttca gtttcgacgg acaaatcttt 360
ctgttattcg attcagagaa gagaatgtgg actacagttc accccggtgc ccgtaaaatg 420
aaggagaagt gggaaaacga caaagtggtg gcgactatgt tttggagttg gtcgatggga 480
gactgcatcg gttggctgga agatttcctc atgggtatgg actccacttt ggagccatcg 540
<210> 149
<211> 180
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 альфа1-альфа2 вариант ULBP2.AB
<400> 149
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Thr Leu Met Trp Gln Trp Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser
180
<210> 150
<211> 540
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий ULBP2 альфа1-альфа2
вариант ULBP2.AB
<400> 150
gagccccata gtctgagcta cgacatcaca gttattccca agttcaggcc cggaccgcgc 60
tggtgtgccg tgcaaggaca agtcgacgaa aaaacctttc ttcattacga ttgcggaaat 120
aagactgtaa cgccagtctc tcctttaggt aagaagttaa acgtcactac ggcgtggaag 180
gcacaaaacc ccgtcctgcg cgaggtcgtc gacatcctga ctgaacaatt gtgggacatc 240
cagctcgaga attacactcc aaaggagcct cttaccctgc aggctagaat gtcttgcgag 300
caaaaggcag agggccactc ctccggcagc tggcagttca gtttcgacgg acaaatcttt 360
ctgttattcg attcagagaa gagaatgtgg actacagttc accccggtgc ccgtaaaatg 420
aaggagaagt gggaaaacga caaagtggtg gcgactctta tgtggcagtg gtcgatggga 480
gactgcatcg gttggctgga agatttcctc atgggtatgg actccacttt ggagccatcg 540
<210> 151
<211> 180
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид, ULBP2 альфа1-альфа2 вариант ULBP2.S3
<400> 151
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
1 5 10 15
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
20 25 30
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
35 40 45
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
50 55 60
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
65 70 75 80
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
85 90 95
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
100 105 110
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
115 120 125
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
130 135 140
Glu Asn Asp Lys Val Val Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly
145 150 155 160
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
165 170 175
Leu Glu Pro Ser
180
<210> 152
<211> 404
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ритуксимаб LC_ULBP2.S3
<400> 152
Gln Ile Val Leu Ser Gln Ser Pro Ala Ile Leu Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Ser Tyr Ile
20 25 30
His Trp Phe Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Tyr
35 40 45
Ala Thr Ser Asn Leu Ala Ser Gly Val Pro Val Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Thr Ser Asn Pro Pro Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys Ala Pro Thr Ser Ser Ser Gly Gly Gly Gly Ser
210 215 220
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
225 230 235 240
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
245 250 255
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
260 265 270
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
275 280 285
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
290 295 300
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
305 310 315 320
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
325 330 335
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
340 345 350
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
355 360 365
Glu Asn Asp Lys Val Val Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly
370 375 380
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
385 390 395 400
Leu Glu Pro Ser
<210> 153
<211> 634
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид ритуксимаб HC_ULBP2.R80W
<400> 153
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Asn Met His Trp Val Lys Gln Thr Pro Gly Arg Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Tyr Pro Gly Asn Gly Asp Thr Ser Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Tyr Tyr Gly Gly Asp Trp Tyr Phe Asn Val Trp Gly
100 105 110
Ala Gly Thr Thr Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ala Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Gly Gly Gly Ser Glu Pro His Ser Leu Ser Tyr Asp Ile Thr
450 455 460
Val Ile Pro Lys Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly
465 470 475 480
Gln Val Asp Glu Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr
485 490 495
Val Thr Pro Val Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala
500 505 510
Trp Lys Ala Gln Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr
515 520 525
Glu Gln Leu Trp Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro
530 535 540
Leu Thr Leu Gln Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His
545 550 555 560
Ser Ser Gly Ser Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu
565 570 575
Phe Asp Ser Glu Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg
580 585 590
Lys Met Lys Glu Lys Trp Glu Asn Asp Lys Val Val Ala Met Ser Phe
595 600 605
His Tyr Phe Ser Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu
610 615 620
Met Gly Met Asp Ser Thr Leu Glu Pro Ser
625 630
<210> 154
<211> 156
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид эктодомен NKG2D.AF
<400> 154
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr
20 25 30
Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn
35 40 45
Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln
50 55 60
Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys
65 70 75 80
Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly
85 90 95
Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser
100 105 110
Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp
115 120 125
Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser
130 135 140
Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val
145 150 155
<210> 155
<211> 629
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид полный химерный рецептор
антигена NKG2D.wt_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 155
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr
20 25 30
Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn
35 40 45
Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln
50 55 60
Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys
65 70 75 80
Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Tyr His Trp Met Gly
85 90 95
Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser
100 105 110
Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp
115 120 125
Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser
130 135 140
Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val Thr Thr Thr Pro
145 150 155 160
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
165 170 175
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
180 185 190
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
195 200 205
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
210 215 220
Cys Ser Leu Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
225 230 235 240
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
245 250 255
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
260 265 270
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln
275 280 285
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
290 295 300
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
305 310 315 320
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
325 330 335
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
340 345 350
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
355 360 365
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ser Gly Ser
370 375 380
Gly Ser Gly Ser Gly Ser Met Val Ser Lys Gly Glu Glu Leu Phe Thr
385 390 395 400
Gly Val Val Pro Ile Leu Val Glu Leu Asp Gly Asp Val Asn Gly His
405 410 415
Lys Phe Ser Val Ser Gly Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys
420 425 430
Leu Thr Leu Lys Phe Ile Cys Thr Thr Gly Lys Leu Pro Val Pro Trp
435 440 445
Pro Thr Leu Val Thr Thr Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg
450 455 460
Tyr Pro Asp His Met Lys Gln His Asp Phe Phe Lys Ser Ala Met Pro
465 470 475 480
Glu Gly Tyr Val Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn
485 490 495
Tyr Lys Thr Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn
500 505 510
Arg Ile Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu
515 520 525
Gly His Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met
530 535 540
Ala Asp Lys Gln Lys Asn Gly Ile Lys Ala Asn Phe Lys Ile Arg His
545 550 555 560
Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln Gln Asn
565 570 575
Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn His Tyr Leu
580 585 590
Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu Lys Arg Asp His
595 600 605
Met Val Leu Leu Glu Phe Val Thr Ala Ala Gly Ile Thr Leu Gly Met
610 615 620
Asp Glu Leu Tyr Lys
625
<210> 156
<211> 1887
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий полный химерный рецептор
антигена NKG2D.wt_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 156
atggcattgc ctgttacagc tctgctgctg cccctggctc tgcttctgca tgctgccaga 60
cctctgttca atcaagaggt gcagatccct ctgaccgaga gctactgtgg cccctgtcct 120
aagaactgga tctgctacaa gaacaactgc taccagttct tcgacgagag caagaattgg 180
tacgagagcc aggccagctg catgagccag aatgccagcc tgctgaaggt gtacagcaaa 240
gaggaccagg atctgctgaa gctggtcaag agctaccact ggatgggact cgtgcacatc 300
cctacaaacg gcagctggca gtgggaggac ggctctatcc tgtctcctaa cctgctgacc 360
atcatcgaga tgcagaaggg cgactgcgcc ctgtacgcca gcagctttaa gggctacatc 420
gagaactgca gcacccctaa cacctacatc tgtatgcagc ggaccgtgac caccacacca 480
gctcctagac ctccaactcc tgctcctaca atcgccagcc agcctctgtc tctgaggcca 540
gaagcttgta gacctgctgc aggcggagcc gtgcatacaa gaggactgga tttcgcctgc 600
gacatctaca tctgggcccc tctggctgga acatgtggcg tgctgctgct gagcctggtc 660
atcaccctgt actgcagcct gaagcggggc agaaagaagc tgctgtacat ctttaagcag 720
cccttcatgc ggcccgtgca gaccacacaa gaggaagatg gctgctcctg cagattcccc 780
gaggaagaag aaggcggctg cgagctgaga gtgaagttca gccgttctgc cgacgctccc 840
gcctataagc agggacagaa ccagctgtac aacgagctga acctggggag aagagaagag 900
tacgacgtgc tggacaagcg gagaggcaga gatcctgaga tgggcggcaa gcccagacgg 960
aagaatcctc aagagggcct gtataatgag ctgcagaaag acaagatggc cgaggcctac 1020
agcgagatcg gaatgaaggg cgagcgcaga agaggcaagg gacacgatgg actgtaccag 1080
ggcctgagca ccgccaccaa ggatacctat gatgccctgc acatgcaggc cctgcctcca 1140
agatcaggct ctggttctgg cagcggcagc atggtgtcta aaggcgagga actgttcacc 1200
ggcgtggtgc ccattctggt ggaactggac ggggatgtga acggccacaa gtttagcgtt 1260
agcggcgaag gcgaagggga tgccacatac ggaaagctga ccctgaagtt catctgcacc 1320
accggcaagc tgcctgtgcc ttggcctaca ctggtcacca cactgacata cggcgtgcag 1380
tgctttagca gataccccga ccatatgaag cagcacgact tcttcaagtc cgccatgcct 1440
gagggctacg tgcaagagcg gaccatcttc tttaaggacg acggcaacta caagaccagg 1500
gccgaagtga agtttgaggg cgacaccctg gtcaaccgga tcgagctgaa gggcatcgac 1560
ttcaaagagg atggcaacat cctgggccac aagctcgagt acaactacaa cagccacaac 1620
gtgtacatca tggccgacaa gcagaagaac ggcatcaagg ccaacttcaa gatccggcac 1680
aacatcgagg acggcagcgt tcagctggcc gatcactacc agcagaacac ccctatcgga 1740
gatggccctg tgctgctccc cgacaatcac tacctgagca cacagagcgc cctgagcaag 1800
gaccccaacg agaagaggga tcacatggtg ctgctggaat ttgtgaccgc cgcaggcatc 1860
accctcggca tggacgaact gtacaaa 1887
<210> 157
<211> 629
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид полный химерный рецептор антигена
NKG2D.YA_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 157
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr
20 25 30
Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn
35 40 45
Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln
50 55 60
Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys
65 70 75 80
Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly
85 90 95
Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser
100 105 110
Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp
115 120 125
Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Tyr Ile Glu Asn Cys Ser
130 135 140
Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val Thr Thr Thr Pro
145 150 155 160
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
165 170 175
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
180 185 190
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
195 200 205
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
210 215 220
Cys Ser Leu Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
225 230 235 240
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
245 250 255
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
260 265 270
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln
275 280 285
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
290 295 300
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
305 310 315 320
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
325 330 335
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
340 345 350
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
355 360 365
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ser Gly Ser
370 375 380
Gly Ser Gly Ser Gly Ser Met Val Ser Lys Gly Glu Glu Leu Phe Thr
385 390 395 400
Gly Val Val Pro Ile Leu Val Glu Leu Asp Gly Asp Val Asn Gly His
405 410 415
Lys Phe Ser Val Ser Gly Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys
420 425 430
Leu Thr Leu Lys Phe Ile Cys Thr Thr Gly Lys Leu Pro Val Pro Trp
435 440 445
Pro Thr Leu Val Thr Thr Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg
450 455 460
Tyr Pro Asp His Met Lys Gln His Asp Phe Phe Lys Ser Ala Met Pro
465 470 475 480
Glu Gly Tyr Val Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn
485 490 495
Tyr Lys Thr Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn
500 505 510
Arg Ile Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu
515 520 525
Gly His Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met
530 535 540
Ala Asp Lys Gln Lys Asn Gly Ile Lys Ala Asn Phe Lys Ile Arg His
545 550 555 560
Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln Gln Asn
565 570 575
Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn His Tyr Leu
580 585 590
Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu Lys Arg Asp His
595 600 605
Met Val Leu Leu Glu Phe Val Thr Ala Ala Gly Ile Thr Leu Gly Met
610 615 620
Asp Glu Leu Tyr Lys
625
<210> 158
<211> 1887
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий полный химерный
рецептор антигена NKG2D.YA_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 158
atggcattgc ctgttacagc tctgctgctg cccctggctc tgcttctgca tgctgccaga 60
cctctgttca atcaagaggt gcagatccct ctgaccgaga gctactgtgg cccctgtcct 120
aagaactgga tctgctacaa gaacaactgc taccagttct tcgacgagag caagaattgg 180
tacgagagcc aggccagctg catgagccag aatgccagcc tgctgaaggt gtacagcaaa 240
gaggaccagg atctgctgaa gctggtcaag agcgcccact ggatgggact cgtgcacatc 300
cctacaaacg gcagctggca gtgggaggac ggctctatcc tgtctcctaa cctgctgacc 360
atcatcgaga tgcagaaggg cgactgcgcc ctgtacgcca gcagctttaa gggctacatc 420
gagaactgca gcacccctaa cacctacatc tgtatgcagc ggaccgtgac caccacacca 480
gctcctagac ctccaactcc tgctcctaca atcgccagcc agcctctgtc tctgaggcca 540
gaagcttgta gacctgctgc aggcggagcc gtgcatacaa gaggactgga tttcgcctgc 600
gacatctaca tctgggcccc tctggctgga acatgtggcg tgctgctgct gagcctggtc 660
atcaccctgt actgcagcct gaagcggggc agaaagaagc tgctgtacat ctttaagcag 720
cccttcatgc ggcccgtgca gaccacacaa gaggaagatg gctgctcctg cagattcccc 780
gaggaagaag aaggcggctg cgagctgaga gtgaagttca gccgttctgc cgacgctccc 840
gcctataagc agggacagaa ccagctgtac aacgagctga acctggggag aagagaagag 900
tacgacgtgc tggacaagcg gagaggcaga gatcctgaga tgggcggcaa gcccagacgg 960
aagaatcctc aagagggcct gtataatgag ctgcagaaag acaagatggc cgaggcctac 1020
agcgagatcg gaatgaaggg cgagcgcaga agaggcaagg gacacgatgg actgtaccag 1080
ggcctgagca ccgccaccaa ggatacctat gatgccctgc acatgcaggc cctgcctcca 1140
agatcaggct ctggttctgg cagcggcagc atggtgtcta aaggcgagga actgttcacc 1200
ggcgtggtgc ccattctggt ggaactggac ggggatgtga acggccacaa gtttagcgtt 1260
agcggcgaag gcgaagggga tgccacatac ggaaagctga ccctgaagtt catctgcacc 1320
accggcaagc tgcctgtgcc ttggcctaca ctggtcacca cactgacata cggcgtgcag 1380
tgctttagca gataccccga ccatatgaag cagcacgact tcttcaagtc cgccatgcct 1440
gagggctacg tgcaagagcg gaccatcttc tttaaggacg acggcaacta caagaccagg 1500
gccgaagtga agtttgaggg cgacaccctg gtcaaccgga tcgagctgaa gggcatcgac 1560
ttcaaagagg atggcaacat cctgggccac aagctcgagt acaactacaa cagccacaac 1620
gtgtacatca tggccgacaa gcagaagaac ggcatcaagg ccaacttcaa gatccggcac 1680
aacatcgagg acggcagcgt tcagctggcc gatcactacc agcagaacac ccctatcgga 1740
gatggccctg tgctgctccc cgacaatcac tacctgagca cacagagcgc cctgagcaag 1800
gaccccaacg agaagaggga tcacatggtg ctgctggaat ttgtgaccgc cgcaggcatc 1860
accctcggca tggacgaact gtacaaa 1887
<210> 159
<211> 629
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический пептид полный химерный рецептор антигена
NKG2D.AF_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 159
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Leu Phe Asn Gln Glu Val Gln Ile Pro Leu Thr
20 25 30
Glu Ser Tyr Cys Gly Pro Cys Pro Lys Asn Trp Ile Cys Tyr Lys Asn
35 40 45
Asn Cys Tyr Gln Phe Phe Asp Glu Ser Lys Asn Trp Tyr Glu Ser Gln
50 55 60
Ala Ser Cys Met Ser Gln Asn Ala Ser Leu Leu Lys Val Tyr Ser Lys
65 70 75 80
Glu Asp Gln Asp Leu Leu Lys Leu Val Lys Ser Ala His Trp Met Gly
85 90 95
Leu Val His Ile Pro Thr Asn Gly Ser Trp Gln Trp Glu Asp Gly Ser
100 105 110
Ile Leu Ser Pro Asn Leu Leu Thr Ile Ile Glu Met Gln Lys Gly Asp
115 120 125
Cys Ala Leu Tyr Ala Ser Ser Phe Lys Gly Phe Ile Glu Asn Cys Ser
130 135 140
Thr Pro Asn Thr Tyr Ile Cys Met Gln Arg Thr Val Thr Thr Thr Pro
145 150 155 160
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
165 170 175
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
180 185 190
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
195 200 205
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
210 215 220
Cys Ser Leu Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln
225 230 235 240
Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser
245 250 255
Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys
260 265 270
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln
275 280 285
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
290 295 300
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
305 310 315 320
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
325 330 335
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
340 345 350
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
355 360 365
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Ser Gly Ser
370 375 380
Gly Ser Gly Ser Gly Ser Met Val Ser Lys Gly Glu Glu Leu Phe Thr
385 390 395 400
Gly Val Val Pro Ile Leu Val Glu Leu Asp Gly Asp Val Asn Gly His
405 410 415
Lys Phe Ser Val Ser Gly Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys
420 425 430
Leu Thr Leu Lys Phe Ile Cys Thr Thr Gly Lys Leu Pro Val Pro Trp
435 440 445
Pro Thr Leu Val Thr Thr Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg
450 455 460
Tyr Pro Asp His Met Lys Gln His Asp Phe Phe Lys Ser Ala Met Pro
465 470 475 480
Glu Gly Tyr Val Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn
485 490 495
Tyr Lys Thr Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn
500 505 510
Arg Ile Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu
515 520 525
Gly His Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met
530 535 540
Ala Asp Lys Gln Lys Asn Gly Ile Lys Ala Asn Phe Lys Ile Arg His
545 550 555 560
Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln Gln Asn
565 570 575
Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn His Tyr Leu
580 585 590
Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu Lys Arg Asp His
595 600 605
Met Val Leu Leu Glu Phe Val Thr Ala Ala Gly Ile Thr Leu Gly Met
610 615 620
Asp Glu Leu Tyr Lys
625
<210> 160
<211> 1887
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический полинуклеотид, кодирующий полный химерный рецептор
антигена NKG2D.AF_CD8шарнирTM_4-1BB_CD3-дзета_EGFP
<400> 160
atggcattgc ctgttacagc tctgctgctg cccctggctc tgcttctgca tgctgccaga 60
cctctgttca atcaagaggt gcagatccct ctgaccgaga gctactgtgg cccctgtcct 120
aagaactgga tctgctacaa gaacaactgc taccagttct tcgacgagag caagaattgg 180
tacgagagcc aggccagctg catgagccag aatgccagcc tgctgaaggt gtacagcaaa 240
gaggaccagg atctgctgaa gctggtcaag agcgcccact ggatgggact cgtgcacatc 300
cctacaaacg gcagctggca gtgggaggac ggctctatcc tgtctcctaa cctgctgacc 360
atcatcgaga tgcagaaggg cgactgcgcc ctgtacgcca gcagctttaa gggcttcatc 420
gagaactgca gcacccctaa cacctacatc tgtatgcagc ggaccgtgac caccacacca 480
gctcctagac ctccaactcc tgctcctaca atcgccagcc agcctctgtc tctgaggcca 540
gaagcttgta gacctgctgc aggcggagcc gtgcatacaa gaggactgga tttcgcctgc 600
gacatctaca tctgggcccc tctggctgga acatgtggcg tgctgctgct gagcctggtc 660
atcaccctgt actgcagcct gaagcggggc agaaagaagc tgctgtacat ctttaagcag 720
cccttcatgc ggcccgtgca gaccacacaa gaggaagatg gctgctcctg cagattcccc 780
gaggaagaag aaggcggctg cgagctgaga gtgaagttca gccgttctgc cgacgctccc 840
gcctataagc agggacagaa ccagctgtac aacgagctga acctggggag aagagaagag 900
tacgacgtgc tggacaagcg gagaggcaga gatcctgaga tgggcggcaa gcccagacgg 960
aagaatcctc aagagggcct gtataatgag ctgcagaaag acaagatggc cgaggcctac 1020
agcgagatcg gaatgaaggg cgagcgcaga agaggcaagg gacacgatgg actgtaccag 1080
ggcctgagca ccgccaccaa ggatacctat gatgccctgc acatgcaggc cctgcctcca 1140
agatcaggct ctggttctgg cagcggcagc atggtgtcta aaggcgagga actgttcacc 1200
ggcgtggtgc ccattctggt ggaactggac ggggatgtga acggccacaa gtttagcgtt 1260
agcggcgaag gcgaagggga tgccacatac ggaaagctga ccctgaagtt catctgcacc 1320
accggcaagc tgcctgtgcc ttggcctaca ctggtcacca cactgacata cggcgtgcag 1380
tgctttagca gataccccga ccatatgaag cagcacgact tcttcaagtc cgccatgcct 1440
gagggctacg tgcaagagcg gaccatcttc tttaaggacg acggcaacta caagaccagg 1500
gccgaagtga agtttgaggg cgacaccctg gtcaaccgga tcgagctgaa gggcatcgac 1560
ttcaaagagg atggcaacat cctgggccac aagctcgagt acaactacaa cagccacaac 1620
gtgtacatca tggccgacaa gcagaagaac ggcatcaagg ccaacttcaa gatccggcac 1680
aacatcgagg acggcagcgt tcagctggcc gatcactacc agcagaacac ccctatcgga 1740
gatggccctg tgctgctccc cgacaatcac tacctgagca cacagagcgc cctgagcaag 1800
gaccccaacg agaagaggga tcacatggtg ctgctggaat ttgtgaccgc cgcaggcatc 1860
accctcggca tggacgaact gtacaaa 1887
<210> 161
<211> 470
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, H-цепь 3BNC60 (Fc IgG1 с
мутациями D265A/N297A (нумерация Kabat))
<400> 161
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Gln Val His Leu Ser Gln Ser Gly Ala Ala Val Thr Lys Pro Gly
20 25 30
Ala Ser Val Arg Val Ser Cys Glu Ala Ser Gly Tyr Lys Ile Ser Asp
35 40 45
His Phe Ile His Trp Trp Arg Gln Ala Pro Gly Gln Gly Leu Gln Trp
50 55 60
Val Gly Trp Ile Asn Pro Lys Thr Gly Gln Pro Asn Asn Pro Arg Gln
65 70 75 80
Phe Gln Gly Arg Val Ser Leu Thr Arg Gln Ala Ser Trp Asp Phe Asp
85 90 95
Thr Tyr Ser Phe Tyr Met Asp Leu Lys Ala Val Arg Ser Asp Asp Thr
100 105 110
Ala Ile Tyr Phe Cys Ala Arg Gln Arg Ser Asp Phe Trp Asp Phe Asp
115 120 125
Val Trp Gly Ser Gly Thr Gln Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ala
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 162
<211> 414
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, L-цепь 3BNC60 (с
добавленным ULBP2.S3)
<400> 162
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Arg Val
20 25 30
Gly Asp Thr Val Thr Ile Thr Cys Gln Ala Asn Gly Tyr Leu Asn Trp
35 40 45
Tyr Gln Gln Arg Arg Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp Gly
50 55 60
Ser Lys Leu Glu Arg Gly Val Pro Ala Arg Phe Ser Gly Arg Arg Trp
65 70 75 80
Gly Gln Glu Tyr Asn Leu Thr Ile Asn Asn Leu Gln Pro Glu Asp Val
85 90 95
Ala Thr Tyr Phe Cys Gln Val Tyr Glu Phe Ile Val Pro Gly Thr Arg
100 105 110
Leu Asp Leu Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro
115 120 125
Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu
130 135 140
Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp
145 150 155 160
Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp
165 170 175
Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys
180 185 190
Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln
195 200 205
Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Ala
210 215 220
Pro Thr Ser Ser Ser Gly Gly Gly Gly Ser Glu Pro His Ser Leu Ser
225 230 235 240
Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg Pro Gly Pro Arg Trp Cys
245 250 255
Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr Asp Cys
260 265 270
Gly Asn Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys Leu Asn
275 280 285
Val Thr Thr Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu Val Val
290 295 300
Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile Gln Leu Glu Asn Tyr Thr
305 310 315 320
Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu Gln Lys
325 330 335
Ala Glu Gly His Ser Ser Gly Ser Trp Gln Phe Ser Phe Asp Gly Gln
340 345 350
Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg Met Trp Thr Thr Val His
355 360 365
Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys Val Val
370 375 380
Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly Asp Cys Ile Gly Trp Leu
385 390 395 400
Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser
405 410
<210> 163
<211> 470
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, H-цепь 3BNC117 (Fc IgG1
с мутациями D265A/N297A (нумерация Kabat))
<400> 163
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Gln Val Gln Leu Leu Gln Ser Gly Ala Ala Val Thr Lys Pro Gly
20 25 30
Ala Ser Val Arg Val Ser Cys Glu Ala Ser Gly Tyr Asn Ile Arg Asp
35 40 45
Tyr Phe Ile His Trp Trp Arg Gln Ala Pro Gly Gln Gly Leu Gln Trp
50 55 60
Val Gly Trp Ile Asn Pro Lys Thr Gly Gln Pro Asn Asn Pro Arg Gln
65 70 75 80
Phe Gln Gly Arg Val Ser Leu Thr Arg His Ala Ser Trp Asp Phe Asp
85 90 95
Thr Tyr Ser Phe Tyr Met Asp Leu Lys Ala Leu Arg Ser Asp Asp Thr
100 105 110
Ala Val Tyr Phe Cys Ala Arg Gln Arg Ser Asp Tyr Trp Asp Phe Asp
115 120 125
Val Trp Gly Ser Gly Thr Gln Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
245 250 255
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
260 265 270
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Ala
275 280 285
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
290 295 300
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala
305 310 315 320
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
325 330 335
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
340 345 350
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
355 360 365
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
370 375 380
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
385 390 395 400
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
405 410 415
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
420 425 430
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
435 440 445
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
450 455 460
Ser Leu Ser Pro Gly Lys
465 470
<210> 164
<211> 414
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, L-цепь 3BNC117 (с
добавленным ULBP2.S3)
<400> 164
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
20 25 30
Gly Asp Thr Val Thr Ile Thr Cys Gln Ala Asn Gly Tyr Leu Asn Trp
35 40 45
Tyr Gln Gln Arg Arg Gly Lys Ala Pro Lys Leu Leu Ile Tyr Asp Gly
50 55 60
Ser Lys Leu Glu Arg Gly Val Pro Ser Arg Phe Ser Gly Arg Arg Trp
65 70 75 80
Gly Gln Glu Tyr Asn Leu Thr Ile Asn Asn Leu Gln Pro Glu Asp Ile
85 90 95
Ala Thr Tyr Phe Cys Gln Val Tyr Glu Phe Val Val Pro Gly Thr Arg
100 105 110
Leu Asp Leu Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro
115 120 125
Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu
130 135 140
Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp
145 150 155 160
Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp
165 170 175
Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys
180 185 190
Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln
195 200 205
Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Ala
210 215 220
Pro Thr Ser Ser Ser Gly Gly Gly Gly Ser Glu Pro His Ser Leu Ser
225 230 235 240
Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg Pro Gly Pro Arg Trp Cys
245 250 255
Ala Val Gln Gly Gln Val Asp Glu Lys Thr Phe Leu His Tyr Asp Cys
260 265 270
Gly Asn Lys Thr Val Thr Pro Val Ser Pro Leu Gly Lys Lys Leu Asn
275 280 285
Val Thr Thr Ala Trp Lys Ala Gln Asn Pro Val Leu Arg Glu Val Val
290 295 300
Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile Gln Leu Glu Asn Tyr Thr
305 310 315 320
Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg Met Ser Cys Glu Gln Lys
325 330 335
Ala Glu Gly His Ser Ser Gly Ser Trp Gln Phe Ser Phe Asp Gly Gln
340 345 350
Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg Met Trp Thr Thr Val His
355 360 365
Pro Gly Ala Arg Lys Met Lys Glu Lys Trp Glu Asn Asp Lys Val Val
370 375 380
Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly Asp Cys Ile Gly Trp Leu
385 390 395 400
Glu Asp Phe Leu Met Gly Met Asp Ser Thr Leu Glu Pro Ser
405 410
<210> 165
<211> 479
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 165
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Gln Met Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser
20 25 30
Glu Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Ala Ser Ile Ser Asp
35 40 45
Ser Tyr Trp Ser Trp Ile Arg Arg Ser Pro Gly Lys Gly Leu Glu Trp
50 55 60
Ile Gly Tyr Val His Lys Ser Gly Asp Thr Asn Tyr Ser Pro Ser Leu
65 70 75 80
Lys Ser Arg Val Asn Leu Ser Leu Asp Thr Ser Lys Asn Gln Val Ser
85 90 95
Leu Ser Leu Val Ala Ala Thr Ala Ala Asp Ser Gly Lys Tyr Tyr Cys
100 105 110
Ala Arg Thr Leu His Gly Arg Arg Ile Tyr Gly Ile Val Ala Phe Asn
115 120 125
Glu Trp Phe Thr Tyr Phe Tyr Met Asp Val Trp Gly Asn Gly Thr Gln
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Ala Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 166
<211> 420
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, L-цепь PGT121 (с
добавленным ULBP2.S3)
<400> 166
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Ser Asp Ile Ser Val Ala Pro Gly Glu Thr Ala Arg Ile Ser Cys
20 25 30
Gly Glu Lys Ser Leu Gly Ser Arg Ala Val Gln Trp Tyr Gln His Arg
35 40 45
Ala Gly Gln Ala Pro Ser Leu Ile Ile Tyr Asn Asn Gln Asp Arg Pro
50 55 60
Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser Pro Asp Ser Pro Phe Gly
65 70 75 80
Thr Thr Ala Thr Leu Thr Ile Thr Ser Val Glu Ala Gly Asp Glu Ala
85 90 95
Asp Tyr Tyr Cys His Ile Trp Asp Ser Arg Val Pro Thr Lys Trp Val
100 105 110
Phe Gly Gly Gly Thr Thr Leu Thr Val Leu Arg Thr Val Ala Ala Pro
115 120 125
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
130 135 140
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
145 150 155 160
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
165 170 175
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
180 185 190
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
195 200 205
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
210 215 220
Asn Arg Gly Glu Cys Ala Pro Thr Ser Ser Ser Gly Gly Gly Gly Ser
225 230 235 240
Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro Lys Phe Arg
245 250 255
Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp Glu Lys Thr
260 265 270
Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro Val Ser Pro
275 280 285
Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala Gln Asn Pro
290 295 300
Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu Trp Asp Ile
305 310 315 320
Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu Gln Ala Arg
325 330 335
Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly Ser Trp Gln
340 345 350
Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser Glu Lys Arg
355 360 365
Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys Glu Lys Trp
370 375 380
Glu Asn Asp Lys Val Val Ala Thr Lys Leu Tyr Leu Trp Ser Met Gly
385 390 395 400
Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met Asp Ser Thr
405 410 415
Leu Glu Pro Ser
420
<210> 167
<211> 479
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, H-цепь 10-1074 (Fc IgG1
с мутациями D265A/N297A (нумерация Kabat))
<400> 167
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser
20 25 30
Glu Thr Leu Ser Val Thr Cys Ser Val Ser Gly Asp Ser Met Asn Asn
35 40 45
Tyr Tyr Trp Thr Trp Ile Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp
50 55 60
Ile Gly Tyr Ile Ser Asp Arg Glu Ser Ala Thr Tyr Asn Pro Ser Leu
65 70 75 80
Asn Ser Arg Val Val Ile Ser Arg Asp Thr Ser Lys Asn Gln Leu Ser
85 90 95
Leu Lys Leu Asn Ser Val Thr Pro Ala Asp Thr Ala Val Tyr Tyr Cys
100 105 110
Ala Thr Ala Arg Arg Gly Gln Arg Ile Tyr Gly Val Val Ser Phe Gly
115 120 125
Glu Phe Phe Tyr Tyr Tyr Ser Met Asp Val Trp Gly Lys Gly Thr Thr
130 135 140
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
145 150 155 160
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
165 170 175
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
180 185 190
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
195 200 205
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
210 215 220
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
225 230 235 240
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
245 250 255
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Ala Val Ser His Glu Asp Pro Glu
290 295 300
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
465 470 475
<210> 168
<211> 428
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, L-цепь 10-1074 (с
добавленным ULBP2.S3)
<400> 168
Met Met Arg Pro Ile Val Leu Val Leu Leu Phe Ala Thr Ser Ala Leu
1 5 10 15
Ala Ser Tyr Val Arg Pro Leu Ser Val Ala Leu Gly Glu Thr Ala Arg
20 25 30
Ile Ser Cys Gly Arg Gln Ala Leu Gly Ser Arg Ala Val Gln Trp Tyr
35 40 45
Gln His Arg Pro Gly Gln Ala Pro Ile Leu Leu Ile Tyr Asn Asn Gln
50 55 60
Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Thr Pro Asp Ile
65 70 75 80
Asn Phe Gly Thr Arg Ala Thr Leu Thr Ile Ser Gly Val Glu Ala Gly
85 90 95
Asp Glu Ala Asp Tyr Tyr Cys His Met Trp Asp Ser Arg Ser Gly Phe
100 105 110
Ser Trp Ser Phe Gly Gly Ala Thr Arg Leu Thr Val Leu Arg Thr Val
115 120 125
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
130 135 140
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
145 150 155 160
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
165 170 175
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
180 185 190
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
195 200 205
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
210 215 220
Lys Ser Phe Asn Arg Gly Glu Cys Ala Pro Thr Ser Ser Ser Gly Gly
225 230 235 240
Gly Gly Ser Glu Pro His Ser Leu Ser Tyr Asp Ile Thr Val Ile Pro
245 250 255
Lys Phe Arg Pro Gly Pro Arg Trp Cys Ala Val Gln Gly Gln Val Asp
260 265 270
Glu Lys Thr Phe Leu His Tyr Asp Cys Gly Asn Lys Thr Val Thr Pro
275 280 285
Val Ser Pro Leu Gly Lys Lys Leu Asn Val Thr Thr Ala Trp Lys Ala
290 295 300
Gln Asn Pro Val Leu Arg Glu Val Val Asp Ile Leu Thr Glu Gln Leu
305 310 315 320
Trp Asp Ile Gln Leu Glu Asn Tyr Thr Pro Lys Glu Pro Leu Thr Leu
325 330 335
Gln Ala Arg Met Ser Cys Glu Gln Lys Ala Glu Gly His Ser Ser Gly
340 345 350
Ser Trp Gln Phe Ser Phe Asp Gly Gln Ile Phe Leu Leu Phe Asp Ser
355 360 365
Glu Lys Arg Met Trp Thr Thr Val His Pro Gly Ala Arg Lys Met Lys
370 375 380
Glu Lys Trp Glu Asn Asp Lys Val Val Ala Thr Lys Leu Tyr Leu Trp
385 390 395 400
Ser Met Gly Asp Cys Ile Gly Trp Leu Glu Asp Phe Leu Met Gly Met
405 410 415
Asp Ser Thr Leu Glu Pro Ser Leu Ile Ser Gly Arg
420 425
<210> 169
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, связывающий эпитоп 3BNC60
и 3BNC117
<400> 169
Ser Ser Gly Gly Asp Pro Glu Ile Val Thr His
1 5 10
<210> 170
<211> 36
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность, связывающий эпитоп PGF12 и
10-1074
<400> 170
Cys Thr Arg Pro Asn Asn Asn Thr Arg Lys Arg Ile Arg Ile Gln Arg
1 5 10 15
Gly Pro Gly Arg Ala Phe Val Thr Ile Gly Lys Ile Gly Asn Met Arg
20 25 30
Gln Ala His Cys
35
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОДИФИЦИРОВАННЫЕ ДОМЕНЫ α1-α2 НЕПРИРОДНЫХ ЛИГАНДОВ NKG2D, КОТОРЫЕ СВЯЗЫВАЮТСЯ С НЕПРИРОДНЫМИ РЕЦЕПТОРАМИ NKG2D | 2019 |
|
RU2815278C2 |
| НЕПРИРОДНЫЕ РЕЦЕПТОРЫ NKG2D, КОТОРЫЕ НЕПОСРЕДСТВЕННО НЕ ПЕРЕДАЮТ СИГНАЛ КЛЕТКАМ, С КОТОРЫМИ ОНИ СВЯЗАНЫ | 2019 |
|
RU2832538C2 |
| Белки, связывающие NKG2D, CD16 и опухолеассоциированный антиген | 2018 |
|
RU2816716C2 |
| БЕЛОК, СВЯЗЫВАЮЩИЙСЯ С NKG2D, CD16 И С ОПУХОЛЕСПЕЦИФИЧЕСКИМ АНТИГЕНОМ | 2018 |
|
RU2788531C2 |
| БЕЛКИ, СВЯЗЫВАЮЩИЕ CD33, NKG2D И CD16 | 2018 |
|
RU2820603C2 |
| БЕЛКИ, СВЯЗЫВАЮЩИЕ ВСМА, NKG2D И CD16 | 2018 |
|
RU2805254C2 |
| СПОСОБЫ ПОЛУЧЕНИЯ ЭКСПРЕССИРУЮЩИХ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР КЛЕТОК | 2015 |
|
RU2751362C2 |
| ПОЛИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ, НАЦЕЛЕННЫЕ НА NKG2D, CD16 И TROP2 | 2018 |
|
RU2820629C2 |
| КОМПОЗИЦИИ НА ОСНОВЕ НЕПРИРОДНЫХ ПАР ОСНОВАНИЙ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2799441C2 |
| БЕЛОК, СВЯЗЫВАЮЩИЙСЯ С NKG2D, CD16 И С ОПУХОЛЕСПЕЦИФИЧЕСКИМ АНТИГЕНОМ | 2018 |
|
RU2826991C2 |
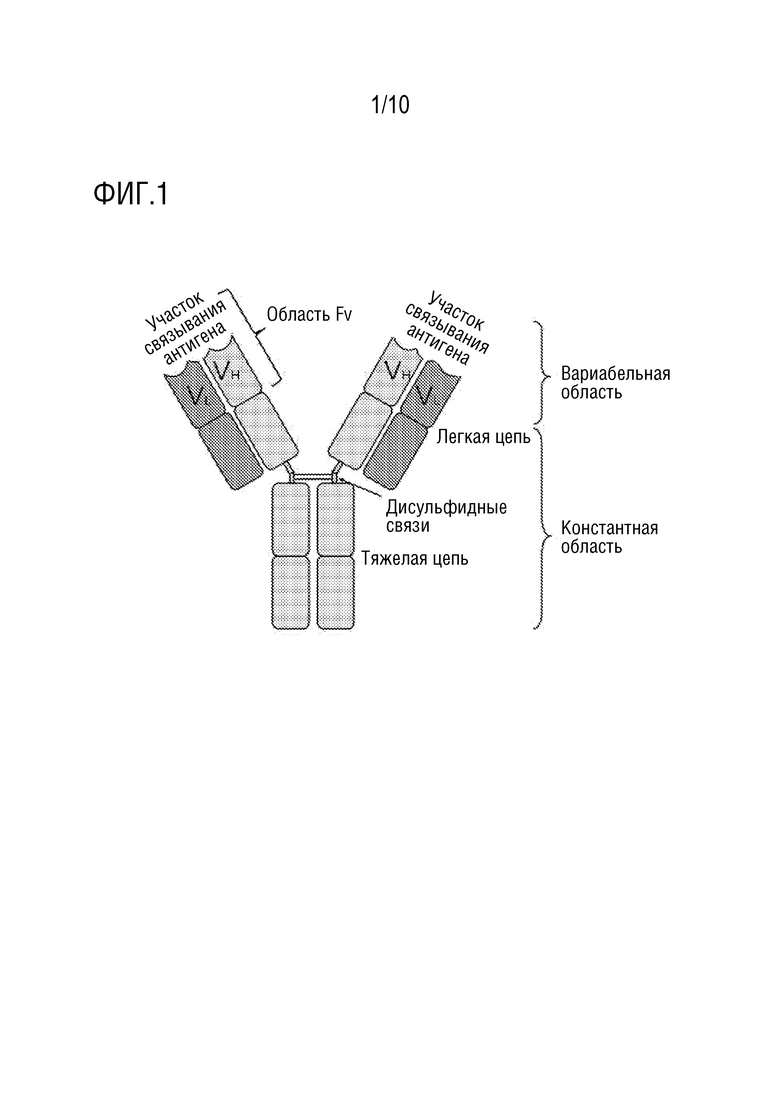
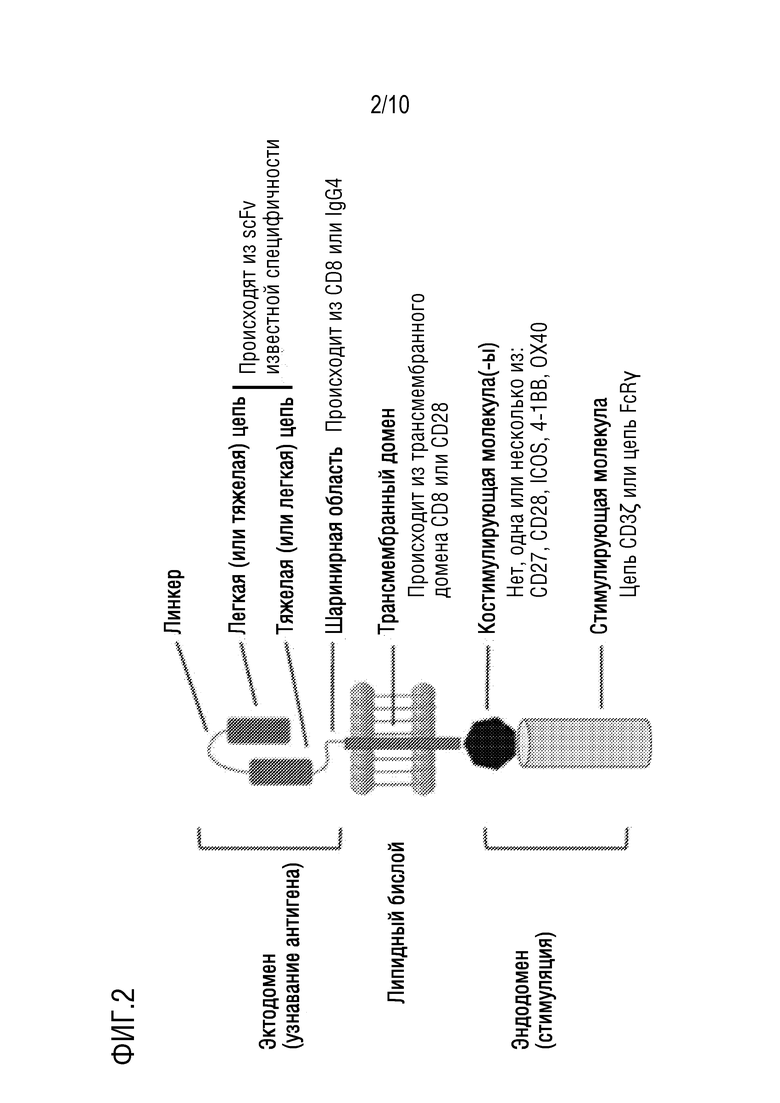
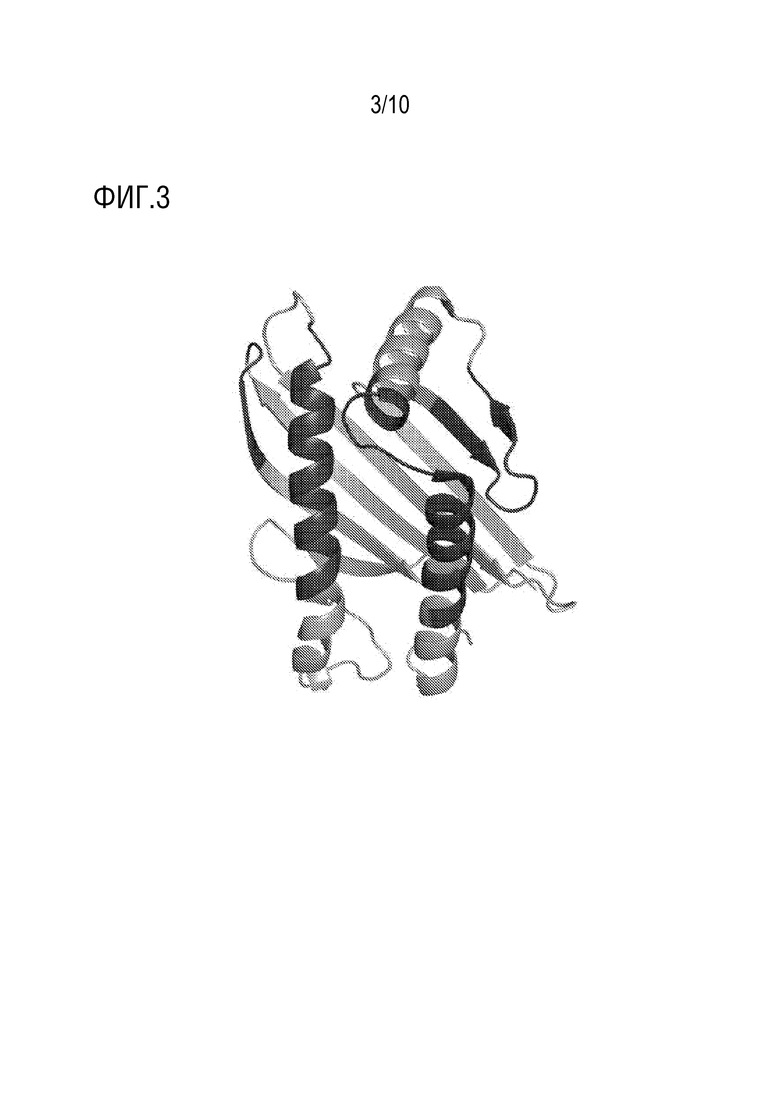


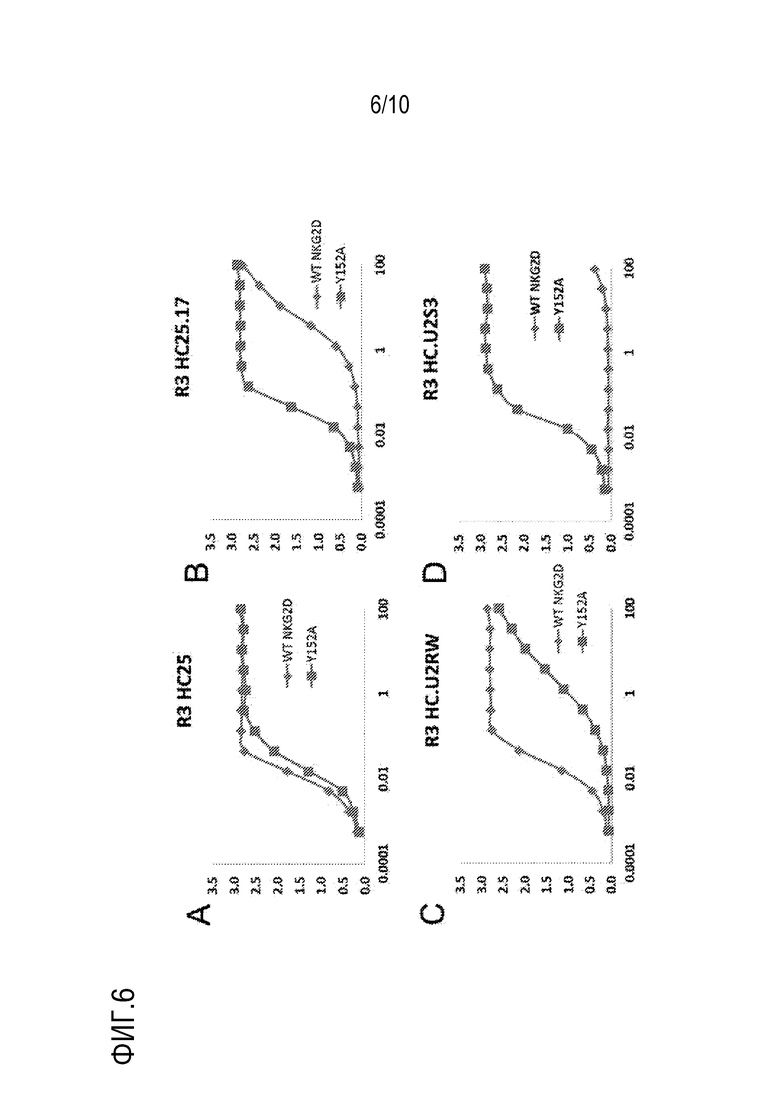
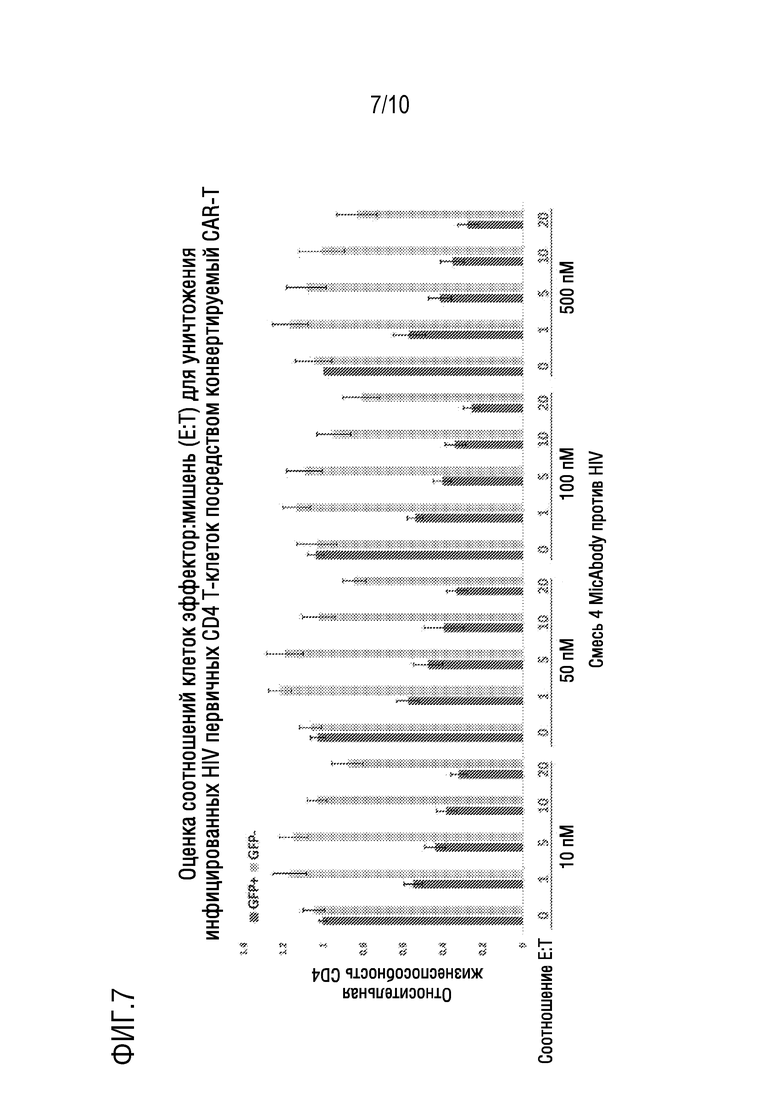

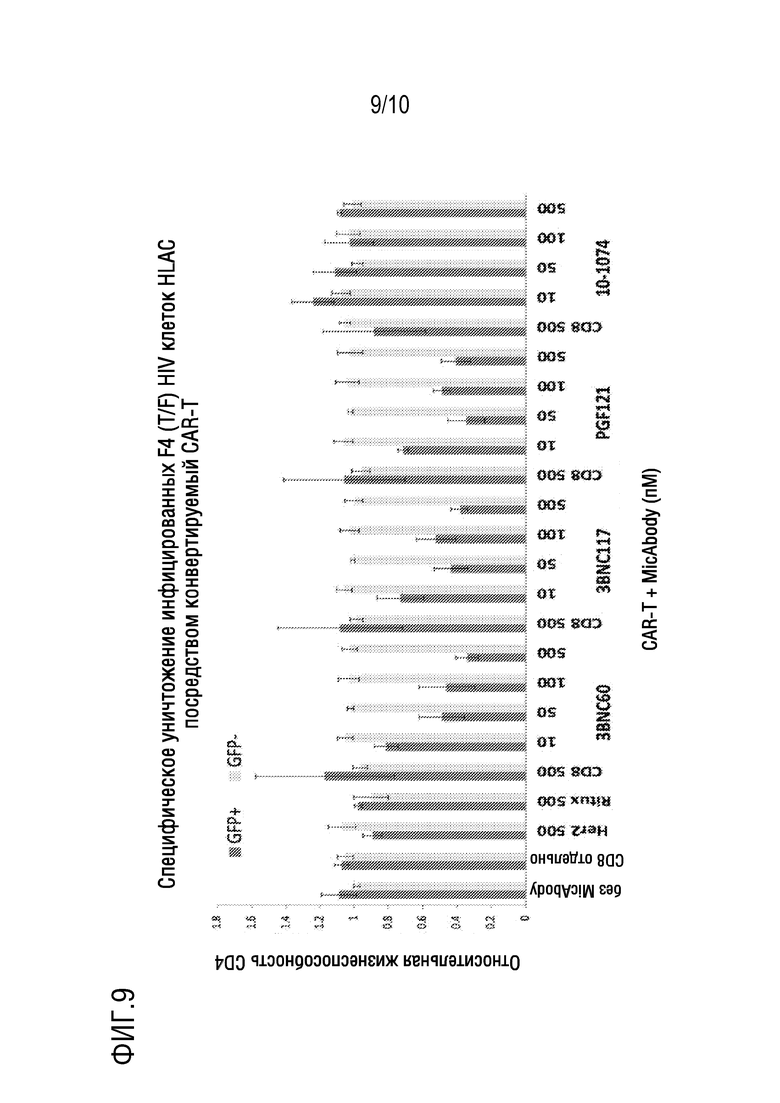
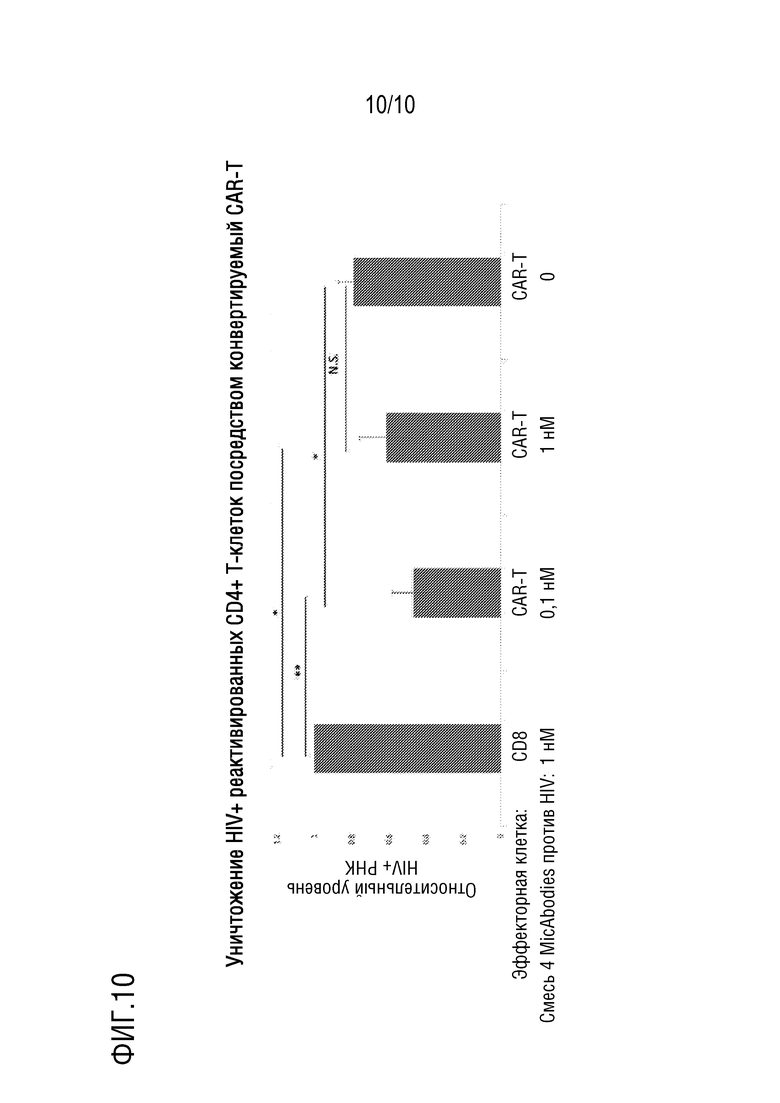
Настоящее изобретение относится к области биотехнологии. Изобретение раскрывает новые модифицированные лиганды NKG2D с присоединенными полипептидами, имеющими свойства связывания специфической мишени, например, антителами или вариабельными фрагментами антител, избирательно доставляемых к химерным рецепторам антигенов (CAR), содержащим модифицированные, неприродные рецепторы NKG2D на модифицированных клетках млекопитающих. Изобретение может быть применимо при получении CAR-клеток, способных к уничтожению инфицированных HIV клеток. 2 н. и 8 з.п. ф-лы, 14 пр., 13 табл., 10 ил.
1. Модифицированный неприродный лиганд для модифицированного неприродного рецептора NKG2D для доставки клетки, экспонирующей модифицированный неприродный рецептор NKG2D, к клетке, инфицированной HIV, где лиганд
(a) содержит
(i) SEQ ID NO: 63 или его мутант, имеющий T в положении 154; M, K, W, L или T в положении 155; L или M в положении 156; E, T, S, Q, Y или R в положении 157; L, V, I или T в положении 158; и/или W или I в положении 159; или
(ii) SEQ ID NO: 65 или его мутант, имеющий D, W, R, Y или L в положении 155; L или M в положении 156; I, Y, V или L в положении 157; R, L или T в положении 158; и/или R, I, W или K в положении 159; и
(b) имеет присоединенную гетерологичную молекулу, которая избирательно связывает белок HIV, представленный на поверхности клетки, инфицированной HIV, где указанная гетерологичная молекула избирательно связывается с последовательностью эпитопа, которая включает аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 169 и SEQ ID NO: 170, и
где модифицированный лиганд с его присоединенной гетерологичной молекулой может избирательно связываться с модифицированным неприродным рецептором NKG2D CAR-клетки и вызывать разрушение инфицированной HIV клетки.
2. Модифицированный неприродный лиганд по п.1, отличающийся тем, что указанный модифицированный неприродный лиганд модифицированного неприродного рецептора NKG2D содержит неприродный домен α1-α2 лиганда NKG2D.
3. Модифицированный неприродный лиганд по п.2, отличающийся тем, что лиганд NKG2D выбран из группы, состоящей из MICA, MICB и ULBP 1-6.
4. Модифицированный неприродный лиганд по п.2 или 3, отличающийся тем, что домен α1-α2 неприродного лиганда NKG2D по меньшей мере на 80% идентичен нативному или природному домену α1-α2 лиганда NKG2D, выбранному из группы, состоящей из SEQ ID NO: 1-19.
5. Модифицированный неприродный лиганд по п.1, отличающийся тем, что указанный модифицированный неприродный лиганд содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 68-72.
6. Модифицированный неприродный лиганд по п.1, отличающийся тем, что указанный модифицированный неприродный лиганд связывается с неприродным рецептором NKG2D, содержащим аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 54 и SEQ ID NO: 154.
7. CAR-клетка для уничтожения инфицированных HIV клеток, причем CAR-клетка имеет связанный с ней модифицированный неприродный лиганд по п.1, где указанная CAR-клетка связана со множеством модифицированных неприродных лигандов с разными, отдельными гетерологичными молекулами, связывающими разные эпитопы, белки или другие молекулы на поверхности инфицированной HIV клетки, и где CAR-клетка представляет собой CAR-Т-клетку или CAR-NK-клетку.
8. CAR-клетка по п.7, отличающаяся тем, что указанная CAR-клетка содержит модифицированный неприродный рецептор NKG2D, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 54 и SEQ ID NO: 154, и,
где модифицированный неприродный лиганд, который содержит аминокислотную последовательность, выбранную из группы, состоящий из SEQ ID NO: 68-72, к которому присоединена гетерологичная молекула или атом, которые не связывают белок HIV, дополнительно связаны с указанной CAR-клеткой.
9. CAR-клетка по п.7 или 8, в где указанная гетерологичная молекула или атом, которые не связывают белок HIV, модулируют функцию CAR-клетки.
10. CAR-клетка по п.9, где указанная функция представляет собой функцию, выбранную из группы, состоящей из пролиферации, дифференцировки, абляции, визуализации, антагонизма иммуносупрессии, хоминга и цитолиза клетки, не инфицированной HIV.
| WO 2017024131 A1, 09.02.2017 | |||
| US 20180134765 A1, 17.05.2018 | |||
| US 20100324034 A1, 23.12.2010 | |||
| US 5804440 A, 08.09.1998 | |||
| US 5817316 A, 06.10.1998 | |||
| RU 2016139643 A 13.04.2018. |
Авторы
Даты
2024-07-29—Публикация
2020-01-28—Подача