ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к рекомбинантным белкам, имеющим последовательность аминокислот, соответствующую или родственную домену гомологии к повторам тромбоспондина I типа (домен III) представителя семейства белков CCN, и их применению, в том числе, в частности, таким белкам, который усечены и/или содержат определенные модификации аминокислот. Кроме того, настоящее изобретение относится к слитым белкам, содержащим последовательность аминокислот, соответствующую или родственную домену гомологии к повторам тромбоспондина I типа представителя семейства белков CCN, скомбинированную с партнером для слияния, необязательно посредством линкерного пептида. В частности, партнер для слияния представляет собой мономерный белок, и указанные слитые белки являются мономерными.
Также в настоящей заявке раскрыты новые устойчивые к протеазам Fc-фрагменты.
УРОВЕНЬ ТЕХНИКИ
Белки CCN представляют собой семейство секретируемых гликопротеинов. Изначально для обозначения CCN была выбрана аббревиатура, происходящая от первых трех идентифицированных представителей указанного семейства генов; Cyr61, CTGF и NOV. Однако недавно смысл указанной аббревиатура был адаптирован и теперь она представляет собой сокращение от «факторов сети клеточной коммуникации» («Cellular Communication Network»), ратифицированное Комитетом по номенклатуре генов HUGO (Perbal В, Tweedie S and Bruford E, J Cell Commun Signal. 2019 Sep; 13(3):435). Указанные белки часто классифицируют как матриклеточные белки, ассоциированные с внеклеточным матриксом (ВКМ). Белки CCN не участвуют в выполнении функций скаффолда, организующих клетки в ткани, а считаются сигнальными белками и могут функционировать в качестве независимых аутокринных или паракринных сигнальных факторов, или в качестве модификаторов других внеклеточных сигнальных белков. Наряду с группой из трех белков Wnt-индуцируемого сигнального пути (WISP1/CCN4, WISP2/CCN5 и WISP3/CCN6) они включают семейство из шести гомологичных богатых цистеином белков у млекопитающих, которые были переименованы в CCN1-6.
Исходные представители семейства CCN отличает общая модульная структура, где за N-концевым сигнальным пептидом для секреции следуют четыре консервативных домена. Первый домен демонстрирует гомологию последовательностей относительно связывающих инсулиноподобный фактор роста белков (IGFBP) и, соответственно, известен как гомологичный IGF-связывающим белкам домен, хотя он и имеет пренебрежимо малую аффинность к IGF. Второй домен известен как домен гомологии с повторами фактора фон Виллебранда типа С (VWC), часто встречающийся в белках внеклеточного матрикса (ВКМ). Третий домен известен как домен гомологии с повторами тромбоспондина типа I (TSP-1), который может быть вовлечен в прикрепление белков CCN к интегринам. Четвертый домен представляет собой богатый цистеином С-концевой повтор или домен гомологии к цистиновому узлу, домен, который, как сообщалось, связывает гепарин. Пятый представитель семейства белков CCN, WISP2 (белок Wnt1-индуцируемого сигнального пути 2), также известный как CCN5, атипичен, поскольку не содержит карбокси-концевого домена цистинового узла (домена 4). Домены гомологии с TSP-1 семейства белков CCN характеризуются 34% идентичности последовательностей аминокислот и 25% сходства последовательностей (по результатам анализа ClustalOmega, см. источник ниже). Четырехдоменные белки CCN содержат 38 консервативных остатка цистеина по длине первичной последовательности, за исключением CCN6, где 4 цистеина в домене гомологии к VWC не сохраняются относительно других представителей семейства. Также, в CCN5, где отсутствует карбоксиконцевой домен гомологии к цистиновому узлу, все цистеины доменов гомологии к IGFBP, VWC и TSP-1 консервативны относительно других представителей семейства CCN.
Неконсервативная чувствительная к протеазам центральная область, часто называемая шарнирной областью, рассекает белки на две половины. Экспрессия белков CCN регулируется на транскрипционном, посттранскрипционном и трансляционном уровнях в ответ на изменения стимулов среды.
Информацию относительно доменной организации семейства белков CCN можно найти, например, в источнике: Liu et al., 2017, Journal of Diabetes, 9, pp. 462-474.
На клеточном уровне белки CCN могут играть разнообразные регуляторные роли на границе внеклеточного матрикса и поверхности клеток. Белки CCN могут регулировать клеточную адгезию, миграцию, пролиферацию, дифференцировку, апоптоз, выживание, старение и генную экспрессию. Модулируя один или более аспектов указанных клеточных функций специфичным для типа клеток способом, CCN координируют сложные биологические процессы, в том числе развитие сердечно-сосудистой и скелетной систем во время эмбриогенеза, а также воспаление, заживление ран, и повреждение и восстановление тканей у взрослых. В общем случае, 4-доменные CCN 1-4 и CCN6 (в частности, CCN1, CCN2 и CCN4) могут проявлять профибротическую активность, тогда как CCN5, который содержит только 3 домена I-III, обладает антифибротической активностью.
Белки CCN также вовлечены в разнообразные патологические состояния, такие как недостаточность органов в результате прогрессирующего фиброза, например, фиброза печени и идиопатического фиброза легких, и в инвазию и метастазирование рака. Соответствующую информацию можно найти в источнике: Jun and Lau, 2011, Nat. Rev. Drug. Discovery, 10(12), pp. 945-963. Также было показано, что, в общем случае, 4-доменные белки CCN, в частности, CCN2, вовлечены в механизмы различных фиброзных заболеваний, тогда как в доклинических моделях таких заболеваний, напротив, было показано, что повышенные уровни CCN5 могут быть благоприятными.
В источнике:  et al., J. Biol. Chem, 293:46, pp. 17953-17970, сообщается, что фактор роста соединительной ткани (CTGF), также известный как CCN2, синтезируется и секретируется в виде неактивного препробелка, который требует протеолитического расщепления для высвобождения биологически активного CCN2, и что гомодимер С-концевого фрагмента, состоящий из доменов III-IV, представляет собой биологически полностью активную форму CCN2, и, наконец, что все основные описанные виды активности CCN2 могут быть воспроизведены гомодимером С-концевых фрагментов доменов III-IV. Анализы активности, описанные
et al., J. Biol. Chem, 293:46, pp. 17953-17970, сообщается, что фактор роста соединительной ткани (CTGF), также известный как CCN2, синтезируется и секретируется в виде неактивного препробелка, который требует протеолитического расщепления для высвобождения биологически активного CCN2, и что гомодимер С-концевого фрагмента, состоящий из доменов III-IV, представляет собой биологически полностью активную форму CCN2, и, наконец, что все основные описанные виды активности CCN2 могут быть воспроизведены гомодимером С-концевых фрагментов доменов III-IV. Анализы активности, описанные  с соавторами, показывают, что ни непроцессированный полноразмерный CCN2, ни N-концевой фрагмент, состоящий из доменов I-II, не были биологически активными. Кроме того, было обнаружено, что протеолитический процессинг полноразмерного CCN2 в результате активности матриксной металл о протеиназы (ММР) высвобождал его латентную активность. В совокупности, данные, полученные
с соавторами, показывают, что ни непроцессированный полноразмерный CCN2, ни N-концевой фрагмент, состоящий из доменов I-II, не были биологически активными. Кроме того, было обнаружено, что протеолитический процессинг полноразмерного CCN2 в результате активности матриксной металл о протеиназы (ММР) высвобождал его латентную активность. В совокупности, данные, полученные  с соавторами, подразумевают, что препро-CCN2 аутоингибируют N-концевые домены I и II. Было также обнаружено, что C-концевых доменов III и IV фрагмента CCN1 и CCN3 было достаточно для активации быстрой клеточной сигнализации и стимуляции физиологических ответов клеток. Однако неизвестно, в какой степени эндопептидазное расщепление шарнирной области CCN1 и/или CCN3, или любого другого представителя семейства белков CCN, необходимо для высвобождения биологической активности.
с соавторами, подразумевают, что препро-CCN2 аутоингибируют N-концевые домены I и II. Было также обнаружено, что C-концевых доменов III и IV фрагмента CCN1 и CCN3 было достаточно для активации быстрой клеточной сигнализации и стимуляции физиологических ответов клеток. Однако неизвестно, в какой степени эндопептидазное расщепление шарнирной области CCN1 и/или CCN3, или любого другого представителя семейства белков CCN, необходимо для высвобождения биологической активности.
Известно, что CCN2 экспрессируется на высоком уровне в ходе развития, при различных патологических состояниях, включающих усиленный фиброгенез и фиброз тканей, и при некоторых видах рака (Jun and Lau, 2011, выше). Тот факт, что белки CCN вовлечены в широкий спектр патологических состояний, представляют собой внеклеточные белки, механистически вовлеченные в развитие фиброза, и демонстрируют ограниченную экспрессию в здоровых организмах, делает их привлекательными терапевтическими мишенями.
В источнике: Jeong et al., 2016, J. American College of Cardiology, 67: 13, pp. 1557-1568, описано исследование, направленное на изучение роли опосредованного аденоассоциированным вирусом переноса генов CCN5 в сердца мышей после экспериментально индуцированной перегрузки сердца давлением. В исследовании сделан вывод, что CCN5 может обращать вспять развившийся фиброз сердца за счет ингибирования образования и повышения апоптоза миофибробластов в миокарде, что позволяет предположить, что CCN5 может служить платформой для разработки средств терапии фиброза сердца.
В US 2008/0207489 раскрыт способ лечения расстройства, основанного на пролиферации гладких мышц, включающий экспрессию CCN5 или введение белка CCN5 в гладкомышечные клетки.
В ЕР 2556839 предложена композиция, содержащая генетический носитель, содержащий последовательность нуклеотидов, кодирующую полноразмерный CCN5 или CCN2ACT, и высказано предположение о его роли в лечении сердечной недостаточности. CCN2ACT в ЕР 2556839 определен как последовательность аминокислот CCN2, усеченная после K251 (нумерация Uniprot).
Хотя было описано, что избыточная экспрессия CCN5 в некоторых экспериментальных системах приводит к фенотипу, противоположному наблюдаемому при избыточной экспрессии CCN2 (Jeong et al. выше, Yoon et al., J Mol Cell Cardiol, 49 (2), 294-303 Aug 2010), о прямом антагонизме CCN5 с четырехдоменными белками CCN5, насколько известно авторам настоящего изобретения, сообщений не было. В частности, структурная основа CCN5/WISP2-опосредованного антагонизма других представителей семейства CCN была неизвестна до исследования, представленного в настоящей заявке.
Авторы настоящего изобретения на более ранней стадии показали, что полноразмерный CCN2 (FL-CCN2) представляет собой препробелок, неактивный предшественник, и что фрагмент, содержащий домены III и IV, по-видимому, осуществляет все биологически релевантные виды активности CCN2. Остается неизвестным, в какой степени белки CCN, в целом, секретируются как неактивные препробелки, которые требуют протеолитической активации. Тем не менее чувствительность полноразмерных белков CCN к нескольким протеазам, как продемонстрировано авторами настоящего изобретения ( et al., J. Biol. Chem. (2018) 293(46) 17953-17970) и другими авторами (Butler, G.S. et al. Matrix Biol 59, 23-38 (2017) и Guillon-Munos, A. et al. J Biol Chem 286, 25505- 25518 (2011)), подразумевает, что немодифицированные полноразмерные белки CCN были бы крайне неподходящими для применения в качестве лекарственных средств по причинам стабильности, как в ходе производства рекомбинантного белка, так и после введения in vivo. Указанная непригодность для применения немодифицированных полноразмерных белков CCN в качестве терапевтических белков также относится к слитым белкам полноразмерных белков CCN, например, согласно описанию для полноразмерного CCN1 (Schutze, N. et al. (2005) Protein Expr Purif 42, 219-225) и полноразмерного CCN6 (Schutze, N. et al. (2007) BMC Cell Biol 8, 45). В области техники, относящейся к белкам CCN, хорошо известно, что чувствительность указанных белков к протеолизу является одной из причин, объясняющих значительную сложность получения рекомбинантных белков CCN. Кроме того, на основании новых данных, полученных
et al., J. Biol. Chem. (2018) 293(46) 17953-17970) и другими авторами (Butler, G.S. et al. Matrix Biol 59, 23-38 (2017) и Guillon-Munos, A. et al. J Biol Chem 286, 25505- 25518 (2011)), подразумевает, что немодифицированные полноразмерные белки CCN были бы крайне неподходящими для применения в качестве лекарственных средств по причинам стабильности, как в ходе производства рекомбинантного белка, так и после введения in vivo. Указанная непригодность для применения немодифицированных полноразмерных белков CCN в качестве терапевтических белков также относится к слитым белкам полноразмерных белков CCN, например, согласно описанию для полноразмерного CCN1 (Schutze, N. et al. (2005) Protein Expr Purif 42, 219-225) и полноразмерного CCN6 (Schutze, N. et al. (2007) BMC Cell Biol 8, 45). В области техники, относящейся к белкам CCN, хорошо известно, что чувствительность указанных белков к протеолизу является одной из причин, объясняющих значительную сложность получения рекомбинантных белков CCN. Кроме того, на основании новых данных, полученных  с соавторами (
с соавторами ( et al. J Biol Chem 2018; 293(46):17953-17970), рекомбинантные полноразмерные белки CCN могут быть далеко не идеальными биологическими лекарственными средствами, поскольку их активность может зависеть от предшествующего протеолитического процессинга, что делает фармакокинетику и фармакодинамику непредсказуемыми. Кроме того, в случае слитых белков с Fc наряду с протеолитической чувствительностью компонентов, например, пептидного линкера, CCN-фрагмента и Fc-фрагмента, была показана также важность расположения компонентов для эффективности и мощности рекомбинантных слитых белков. Один из подтверждающих это примеров описан в работе, опубликованной авторами настоящего изобретения (
et al. J Biol Chem 2018; 293(46):17953-17970), рекомбинантные полноразмерные белки CCN могут быть далеко не идеальными биологическими лекарственными средствами, поскольку их активность может зависеть от предшествующего протеолитического процессинга, что делает фармакокинетику и фармакодинамику непредсказуемыми. Кроме того, в случае слитых белков с Fc наряду с протеолитической чувствительностью компонентов, например, пептидного линкера, CCN-фрагмента и Fc-фрагмента, была показана также важность расположения компонентов для эффективности и мощности рекомбинантных слитых белков. Один из подтверждающих это примеров описан в работе, опубликованной авторами настоящего изобретения ( et al. (2018)), где было обнаружено, что варианты слитых белков с Fc, содержащих домены III-IV CCN2, имеют активность, варьирующую в широком диапазоне и не являющуюся заранее легко предсказуемой.
et al. (2018)), где было обнаружено, что варианты слитых белков с Fc, содержащих домены III-IV CCN2, имеют активность, варьирующую в широком диапазоне и не являющуюся заранее легко предсказуемой.
Была описана чувствительность действий белков CCN к антагонизирующим эффектам высоких концентраций синтетических пептидов, происходящих из первичных последовательностей белков CCN. Одним из примеров является ингибирование фосфорилирования АКТ, стимулированного рекомбинантным CCN2 в фибробластах Rat2, пептидами, происходящими из домена I, домена гомологии к IGFBP, и в меньшей степени пептидами, происходящими из домена III, домена гомологии к повторам TSP-1, CCN2 (Мое et al., J. Cell Commun. Signal. (2013) 7:31-47). Другим примером является ингибирование стимулированной CCN2 (домен IV) адгезии жиронакапливающих клеток печени пептидом, происходящим из домена IV, домена гомологии к цистиновому узлу, из CCN2 (Gao R and Brigstock DR., J Biol Chem. 2004 Mar 5; 279(10):8848-55). Кроме того, сообщалось, что пептиды из домена III CCN1 (Leu et al. J. Biol. Chem, 2003, Vol. 278, NO: 36, Issue of September 5, pp. 33801-33808, 2003) и домена III CCN1, CCN2, CCN3, CCN5 и CCN6 (Karagiannis EG and Popel, The International Journal of Biochemistry & Cell Biology 39 (2007) 2314 2323) оказывают некоторые анти-ангиогенные эффекты в анализах in vitro на клетках HUVEC (Leu et al. J. Biol. Chem, 2003, и Karagiannis EG and Popel, Int J Biochem Cell Biol 39 (2007)) и анти-адгезионные эффекты на 1064SK фибробласты крайней плоти человека (Leu et al. J. Biol. Chem, 2003), указанные пептиды содержат только один (Leu et al. J. Biol. Chem, 2003) или два (Karagiannis EG and Popel, Int J Biochem Cell Biol 39, 2007) из консервативных цистеинов, которые играют центральную роль в изобретении, описанном в настоящей заявке. Цистеины в домене III белков CCN, как известно, образуют дисульфидные мостики, как продемонстрировано для CCN2, эндогенно экспрессируемого клетками HUVEC (Lu, S et al. (2015) Nat methods 12, 329-331), и очищенного рекомбинантного CCN2 ( et al., J. Biol. Chem. 2018). Дисульфидные мостики, продемонстрированные для CCN2, захватывающие С199-С228 (нумерация Uniprot), обеспечивают комплексную трехмерную структуру, в которой аминокислотная цепь сворачивается с образованием складки. Это подразумевает, что нельзя ожидать воспроизведения полного домена III белка CCN короткими пептидами, которые структурно не ограничены дисульфидными мостиками между цистеинами, как в полном домене III белков CCN, продуцируемых в эукариотических системах. Кроме того, ингибирование активности CCN2 пептидами, происходящими из первичных последовательностей доменов I, III и IV, иллюстрирует отсутствие знаний в данной области относительно того, могут ли пептиды, происходящие из конкретного домена CCN2, обеспечивать способность к ингибированию четырехдоменным белкам CCN.
et al., J. Biol. Chem. 2018). Дисульфидные мостики, продемонстрированные для CCN2, захватывающие С199-С228 (нумерация Uniprot), обеспечивают комплексную трехмерную структуру, в которой аминокислотная цепь сворачивается с образованием складки. Это подразумевает, что нельзя ожидать воспроизведения полного домена III белка CCN короткими пептидами, которые структурно не ограничены дисульфидными мостиками между цистеинами, как в полном домене III белков CCN, продуцируемых в эукариотических системах. Кроме того, ингибирование активности CCN2 пептидами, происходящими из первичных последовательностей доменов I, III и IV, иллюстрирует отсутствие знаний в данной области относительно того, могут ли пептиды, происходящие из конкретного домена CCN2, обеспечивать способность к ингибированию четырехдоменным белкам CCN.
В настоящей работе авторы настоящего изобретения на основании структурно-функционального анализа белков семейства CCN и наблюдения, что CCN2 нуждается в протеолитическом процессинге, чтобы стать биологически активным, обнаружили, что биологически активная часть белка CCN5 представлена доменом III, доменом гомологии к повторам тромбоспондина типа I. Эти новые данные привели к получению биоактивных структур на основе домена III CCN5, а также домена III других представителей семейства белков CCN.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Обнаруженная авторами настоящего изобретения взаимосвязь структуры и активности CCN5 и других белков CCN позволила получить новые биологически активные рекомбинантные белки, которые воспроизводят клеточную сигнализацию и физиологические функции клеток, приписываемые CCN-сигнализации, а также способные противодействовать другим четырехдоменным белкам CCN (Cyr61 (также известен как CCN1), CTGF (также известен как CCN2), NOV (также известен как CCN3), WISP1 (также известен как CCN4) и WISP3 (также известен как CCN6)). Другими словами, предложены белки, в том числе в форме слитых белков, на основе домена III, домена гомологии с TSP-1, белка CCN, которые воспроизводят или имеют биологическую активность CCN5, и способны антагонизировать или ингибировать эффекты 4-доменных белков CCN, CCN1-4 или CCN6. В частности, белки согласно настоящей заявке имеют антифибротическую активность и могут также осуществлять прямую противораковую активность.
Как отмечалось выше, было неожиданно обнаружено, что домен III (домен гомологии с TSP-1) других белков CCN, а именно, 4-доменных белков CCN, когда он представлен отдельным доменом в отсутствие других доменов CCN, достаточен для воспроизведения описанных видов активности CCN5. Соответственно, другими словами, домен III 4-доменных белков CCN, когда он представлен отдельным доменом в отсутствие других доменов CCN (т.е. в виде выделенного домена), имеет такую же активность, как и CCN5, или, говоря иначе, как домен III/домен гомологии с TSP-1 CNN5 (то есть активность, противоположную активности полноразмерных 4-доменных белков CCN). Соответственно, из экспериментов, раскрытых в указанной заявке, ясно, что выделенный домен гомологии с TSP-1 любого белка CCN может осуществлять ту же активность, что и домен гомологии с TSP-1 CCN5. За исключением CCN5, указанные виды активности могут быть иными, чем осуществляемые полноразмерным белком CCN.
Было обнаружено, что мономерные слитые белки, отличающиеся тем, что домен III белка CCN слит с мономерным партнером для слияния, обладают особенными преимуществами и полезностью в соответствии с настоящим изобретением и описанием в настоящей заявке.
В соответствии с первым аспектом в настоящем изобретении предложен мономерный слитый белок, содержащий:
(i) полипептид, соответствующий по меньшей мере части домена гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN;
(ii) мономерный партнер для слияния, слитый на N- или С-конце с последовательностью аминокислот по (i); и
(iii) необязательно, пептидный линкер между полипептидом по (i) и мономерным партнером для слияния по (ii),
где полипептид по (i) имеет длину от 40 до 60 аминокислот и содержит последовательность аминокислот, выбранную из SEQ ID NO: 37 или 2 6, или последовательность, по меньшей мере на 80% идентичную последовательности, выбранной из SEQ ID NO: 37 или 2-6, причем все остатки цистеина в указанной последовательности, выбранной из SEQ ID NO: 37 или 2-6, сохранены, а мономерный партнер для слияния по (ii) и пептидный линкер по (iii) не представляют собой или не содержат домен гомологии со связывающим IGF белком, домен гомологии с повторами фактора фон Виллебранда типа С или домен цистинового узла белка семейства CCN.
Как описано более подробно ниже, SEQ ID NO: 37 и 2-6 представляют усеченные фрагменты из 44 аминокислот домена III CCN5, CCN3, CCN2, CCN1, CCN4 и CCN6, соответственно, которые содержат 6 консервативных остатков цистеина указанного домена. В частности, указанные фрагменты фланкированы первым и последним остатками цистеина указанного домена. Было обнаружено, что такие фрагменты, в частности, эффективны и устойчивы к протеолитическому разложению.
Согласно варианту реализации полипептид по (i) содержит или состоит из:
(a) последовательности аминокислот, выбранной из SEQ ID NO: 1 или 8-12; или
(b) последовательности аминокислот, по меньшей мере на 80% идентичной последовательности, выбранной из SEQ ID NO: 1 или 8-12; или
(c) части последовательности аминокислот по (а) или (b), отличающейся тем, что указанная часть содержит по меньшей мере последовательность из 44 аминокислот из SEQ ID NO: 37, 6, 2, 3, 4 или 5, соответственно, или последовательность, по меньшей мере на 80% идентичную последовательности, выбранной из SEQ ID NO: 37, 6, 2, 3, 4 или 5, соответственно.
SEQ ID NO: 1 и 8-12 представляют незначительно более длинные усеченные по N-концу фрагменты домена III CCN5, CCN6, CCN3, CCN2, CCN1 и CCN4, соответственно. Указанные фрагменты содержат соответствующие последовательности SEQ ID NO: 37, 6, 2, 3, 4 и 5, соответственно, с какой-либо дополнительной C-концевой последовательностью из соответствующего домена III.
Согласно дополнительному варианту реализации полипептид по (i) содержит остаток аланина в положении, соответствующем положению 2 указанной последовательности, выбранной из SEQ ID NO: 37 или 2-6, или SEQ ID NO: 1 или 8-12. Согласно некоторым вариантам реализации последовательность аминокислот по (i) содержит последовательность аминокислот, выбранную из SEQ ID NO: 38 или 42 46, или последовательность аминокислот, по меньшей мере на 80% идентичную указанной. Согласно другому варианту реализации последовательность аминокислот по (i) содержит последовательность аминокислот, выбранную из SEQ ID NO: 7, или 47-51, или последовательность аминокислот, по меньшей мере на 80% идентичную указанной. В этом отношении было обнаружено, что замена указанного остатка в положении 2 полезна для повышения стабильности белка.
В соответствии с дополнительным аспектом настоящего изобретения мономерный слитый белок имеет последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 84, 85, 88, 89, 97, 98, 102, 103, 106, 107, ПО, 111, или последовательность аминокислот, на 80% идентичную указанной.
Во вариантах реализации указанных аспектов мономерный партнер для слияния выбирают из сывороточного альбумина, трансферрина и мономерных Fc-фрагментов, в частности, мономерных Fc-фрагментов IgG, более конкретно - IgG человека.
Как отмечалось выше, замена положения 2 во фрагментах домена III согласно настоящему изобретению улучшает стабильность фрагмента, в частности, устойчивость к разложению протеазами. Считается и предполагается в настоящей заявке, что такие варианты фрагментов домена III с модифицированной последовательностью сами по себе являются полезными белками, без связывания с партнером для слияния.
Соответственно, согласно другому аспекту настоящего изобретения также предложен белок длиной от 40 до 60 аминокислот, который содержит последовательность, или белок, который состоит из последовательности аминокислот, представленной в SEQ ID NO: 7, 38, 42 46, или 47-51, или последовательности, по меньшей мере на 80% идентичной указанной, причем указанный белок содержит остаток аланина в положении, соответствующем положению 2 указанной последовательности из SEQ ID NO: 7, 38, 42-16, 47-51, и все остатки цистеина в указанной последовательности сохранены.
Другие белки и слитые белки также предложены в качестве дополнительных аспектов настоящих изобретений, согласно подробному описанию ниже.
В соответствии с дополнительным аспектом настоящего изобретению предложен рекомбинантный белок, состоящий из формулы (I)
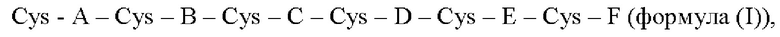
где
А представляет собой пептид формулы II
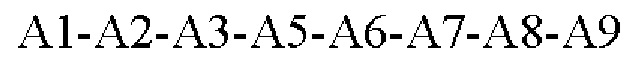
где А1 выбран из группы, состоящей из Р, А, V, I и L; А2 выбран из группы, состоящей из Е, D, A, I, L, и V; A3 выбран из группы, состоящей из G, Q, Y, S, N, W, F; А4 выбран из группы, состоящей из A, I, L, V, S, Т; А5 представляет собой аминокислоту, выбранную из группы, состоящей из Т, Y, N, G, Q и S; А6 представляет собой аминокислоту, выбранную из группы, состоящей из А, V, I, L, Р, S, Е, D, К, R и Н; А7 представляет собой W; А8 выбран из группы, состоящей из G, Т, S, Q, Y, N, Р, А, V, I и L; А9 представляет собой аминокислоту, выбранную из группы, состоящей из А, Р, L, I, V, Q; и
В представляет собой пептид формулы III 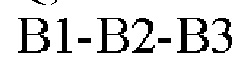
где В1 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y и Т; В2 представляет собой аминокислоту, выбранную из группы, состоящей из, Т, S, N, F, Q, Н, R и K; В3 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y, Т; при этом один из В1-В3 отсутствует; и
С представляет собой пептид формулы IV
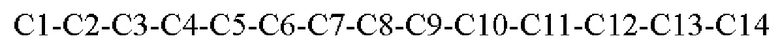
где С1 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y и Т; С2 представляет собой аминокислоту, выбранную из группы, состоящей из K, R, Н, М, Т, S, A, L, I и V; С3 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y и Т; С4 представляет собой аминокислоту, выбранную из группы, состоящей из М, F, A, I, L, V и W; С5 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Т, Y, A, I, L и V; С6 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S и Т; С7 представляет собой аминокислоту, выбранную из группы, состоящей из Н, R и L; С8 представляет собой аминокислоту, выбранную из группы, состоящей из A, L, I, и V; С9 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Т и Y; С10 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Т, Y (предпочтительно N); С11 представляет собой аминокислоту, выбранную из группы, состоящей из V, Р, A, I, L, G, Q, N, S, Т, Y, R, K, D и Е; С12 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y и Т; С13 представляет собой аминокислоту, выбранную из группы, состоящей из Н, K, R, A, L, I, V, Р, G, Q, N, S, Y и Т; С14 представляет собой аминокислоту, выбранную из группы, состоящей из F, Р, W, G, Q, N, S, Y, Т, Е и D; и
D представляет собой пептид формулы V 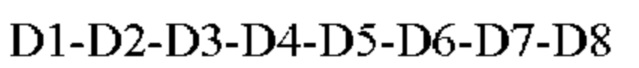
где D1 представляет собой аминокислоту, выбранную из группы, состоящей из R, K, Н, D, Е, W, Р; D2 представляет собой аминокислоту, выбранную из группы, состоящей из Р, А, L, I, V, М, W, D и Е; D3 представляет собой аминокислоту, выбранную из группы, состоящей из D, Е, A, L, I, V, R, K и Н; D4 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, S, Y, Т, R, L, K и Н; D5 представляет собой аминокислоту, выбранную из группы, состоящей из G, Q, N, S, Y, Т, D и Е; D6 представляет собой аминокислоту, выбранную из группы, состоящей из Н, R; K, G, Q, N, S, Y и Т; D7 представляет собой аминокислоту, выбранную из группы, состоящей из L, Н и R; D8 представляет собой аминокислоту, выбранную из группы, состоящей из A, L, I и V; и
Е представляет собой пептид формулы VI 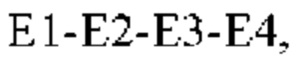
отличающийся тем, что Е1 представляет собой аминокислоту, выбранную из группы, состоящей из Р, A, L, I, V, М, W, G, Q, N, S, Т, Y, D и Е; Е2 представляет собой аминокислоту, выбранную из группы, состоящей из; Р, A, L, I, V, М, W, G, Q, N, S, Т, Y; Е3 представляет собой аминокислоту, выбранную из группы, состоящей из, R, K, Н, G, Q, N, S, Т и Y; Е4 представляет собой аминокислоту, выбранную из группы, состоящей из Р, A, L, I и V; F отсутствует или представляет собой последовательность аминокислот длиной до приблизительно 13 аминокислот,
где рекомбинантный белок содержит от 40 до 60 аминокислот.
В соответствии с одним вариантом реализации вышеописанного аспекта предложен рекомбинантный белок формулы (I), отличающийся тем, что
А1 выбран из группы, состоящей из Р, I и L; А2 выбран из группы, состоящей из Е, V, и А; A3 выбран из группы, состоящей из W, Q, и Y; А4 выбран из группы, состоящей из S, Т, и А; А5 представляет собой аминокислоту, выбранную из группы, состоящей из Т и S; А6 представляет собой аминокислоту, выбранную из группы, состоящей из А, Е, Р, S и K; А7 представляет собой W; А8 выбран из группы, состоящей из G, S и Т; А9 представляет собой аминокислоту, выбранную из группы, состоящей из Р, Q и А; и
В1 представляет собой серии (S); В2 представляет собой аминокислоту, выбранную из группы, состоящей из Т, K и R; В3 представляет собой аминокислоту, выбранную из группы, состоящей из Т и S; и
С1 представляет собой аминокислоту G; С2 представляет собой аминокислоту, выбранную из группы, состоящей из Т, L и М; С3 представляет собой G; С4 представляет собой аминокислоту, выбранную из группы, состоящей из М, F, I и V; С5 представляет собой аминокислоту, выбранную из группы, состоящей из S и А; С6 представляет собой аминокислоту, выбранную из группы, состоящей из Т и N; С7 представляет собой R; С8 представляет собой аминокислоту, выбранную из группы, состоящей из V, и I; С9 представляет собой аминокислоту, выбранную из группы, состоящей из S и Т; С10 представляет собой аспарагин N; С11 представляет собой аминокислоту, выбранную из группы, состоящей из Q, R, D, V и Е; С12 представляет собой аспарагин N; С13 представляет собой аминокислоту, выбранную из группы, состоящей из R, А, Р, и S; С14 представляет собой аминокислоту, выбранную из группы, состоящей из F, Q, S, Е и N; и
D1 представляет собой аминокислоту, выбранную из группы, состоящей из R, Е и W; D2 представляет собой аминокислоту, выбранную из группы, состоящей из L, М и Р; D3 представляет собой аминокислоту, выбранную из группы, состоящей из Е, L, V и R; D4 представляет собой аминокислоту, выбранную из группы, состоящей из Т, K и Q; D5 представляет собой аминокислоту, выбранную из группы, состоящей из Q и Е; D6 представляет собой аминокислоту, выбранную из группы, состоящей из R, Т, S и K; D7 представляет собой аргинин (R); D8 представляет собой аминокислоту, выбранную из группы, состоящей из L и I; и
Е1 представляет собой аминокислоту, выбранную из группы, состоящей из L, М, Е, N и Y; Е2 представляет собой аминокислоту, выбранную из группы, состоящей из; S, V, L и I; Е3 представляет собой аминокислоту, выбранную из группы, состоящей из, Q и R; Е4 представляет собой Р; F отсутствует или представляет собой пептид длиной до 13 аминокислот и содержащий последовательность аминокислот, выбранную из группы, состоящей из PPSRGRSPQNSAF, GQPVYSSL, EADLEEN, EQEPEQPTD, DVDIHTLI и DSNILKTIKIP,
где рекомбинантный белок содержит в общей сложности от 44 до 57 аминокислот.
В соответствии с еще одним вариантом реализации вышеописанного аспекта предложен рекомбинантный белок формулы I, где F полностью отсутствует, частично отсутствует, или представляет собой пептид длиной приблизительно 13 аминокислот, содержащий последовательность аминокислот PPSRGRSPQNSAF.
Более конкретно, предложен рекомбинантный белок, отличающийся тем, что указанный белок содержит последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38; и ее фрагменты или варианты с более чем 50% идентичностью последовательностей последовательностям аминокислот SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38.
В соответствии с другим аспектом настоящего изобретения предложены рекомбинантные белки согласно определению выше, отличающиеся тем, что указанный белок пегилирован.
Согласно другому аспекту настоящего изобретения предложен слитый белок, содержащий
(i) домен гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN;
(ii) партнер для слияния слит на N- или С-конце с доменом гомологии к повторам TSP-1 по (i), при этом указанный партнер для слияния выбран из группы, состоящей из сывороточного альбумина, трансферрина и Fc-фрагмента IgG человека;
(iii) необязательно, пептидный линкер между доменом гомологии к повторам TSP-1 и Fc-фрагментом (слитый на N- или С-конце с доменом гомологии к повторам TSP-1 по (i)).
В соответствии с вариантом реализации вышеописанного аспекта предложены слитые белки, содержащие рекомбинантный белок в соответствии с настоящим изобретением согласно описанию выше, в качестве дополнительного аспекта настоящего изобретения.
Партнер для слияния слитого белка согласно настоящему изобретению соответствует одному из вариантов реализации, выбранных из группы, состоящей из Fc-фрагментом IgG1, IgG2 или IgG4, сывороточного альбумина и трансферрина.
В соответствии с дополнительным вариантом реализации предложен слитый белок, отличающийся тем, что партнер для слияния (ii) представляет собой Fc-фрагмент IgG1, IgG2 или IgG4, содержащий стабилизирующий дисульфидный мостик. Такие мутации могут повышать термическую стабильность белка. Стабилизирующие мутации известны и были описаны в данной области техники.
В соответствии с еще одним дополнительным вариантом реализации предложен слитый белок, отличающийся тем, что партнер для слияния (ii) представляет собой Fc-фрагмент IgG1, IgG2 или IgG4, содержащий одну или более мутаций, выбранных из группы, состоящей из S228P (относится к IgG4), Е233Р (относится к IgG1 и IgG4), F234A (относится к IgG4), L234A (относится к IgG1), L234V (относится к IgG1), F234V (относится к IgG4), L235A (относится к IgG1 и IgG4), AG236 (относится к IgG1 и IgG4) и ΔK447 (относится к IgG1, IgG2 и IgG4).
Согласно другому варианту реализации слитый белок может содержать Fc-фрагмент, выбранный из группы, состоящей из SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18 и SEQ ID NO: 19.
Согласно другому варианту реализации слитый белок содержит линкер, выбранный из группы, состоящей из SEQ ID NO: 20; SEQ ID NO: 21; SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25 и SEQ ID NO: 39.
В соответствии с одним вариантом реализации указанный линкер содержит последовательность аминокислот (EAAAK)n, где n равен по меньшей мере 4, предпочтительно n равен 8.
Согласно другому варианту реализации указанный слитый белок содержит последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 26, SEQ ID NO: 27; SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 31, SEQ ID NO: 40 и SEQ ID NO: 41.
Согласно другому варианту реализации указанный партнер для слияния по (ii) представляет собой сывороточный альбумин.
В соответствии с другим вариантом реализации второго аспекта настоящего изобретения партнер для слияния по (ii) представляет собой трансферрин.
Также согласно настоящему изобретению, в соответствии с еще одним дополнительным аспектом, предложена молекула нуклеиновой кислоты (например, ДНК), кодирующая рекомбинантный белок, белок или слитый белок в соответствии с настоящим изобретением.
В соответствии с одним вариантом реализации указанного аспекта предложена последовательность ДНК, содержащая последовательность нуклеиновой кислоты, представленную в SEQ ID NO: 34, SEQ ID NO: 35, SEQ ID NO: 36 или SEQ ID NO: 86, 87, 90, 91, 99, 100, 104, 105, 108, 109, 112 или 113, и последовательностях нуклеиновых кислот, приблизительно на 80% идентичных SEQ ID NO: 34, SEQ ID NO: 35, SEQ ID NO: 36 или SEQ ID NO: 86, 87, 90, 91, 99, 100, 104, 105, 108, 109, 112 или 113.
Кроме того, в соответствии с другим аспектом настоящего изобретения предложен экспрессионный вектор, содержащий последовательность ДНК в соответствии с настоящим изобретением. Также предложена клетка-хозяин, содержащая экспрессионный вектор в соответствии с настоящим изобретением.
Наконец, предложен домен гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN, белок и слитый белок согласно определению выше для применения в качестве медикамента для лечения или предотвращения расстройств путем ингибирования или противодействия клеточной сигнализации и физиологических функций клетки, приписываемым четырехдоменным белкам семейства CCN.
Согласно одному аспекту предложен слитый белок или белок согласно определению в настоящей заявке для применения в терапии.
Указанный слитый белок или белок может быть предназначен для применения при лечении состояния, ассоциированного с активностью 4-доменного белка CCN, в частности, нежелательной или аберрантной активности 4-доменного белка CCN. Указанная активность может быть ассоциирована с фибротическим эффектом. Указанная активность может представлять собой профибротическую активность.
Согласно другому аспекту предложен слитый белок или белок согласно определению в настоящей заявке для применения при лечении или предотвращении фиброза, или любого состояния, при котором наблюдается фиброз (т.е. фибротического состояния или заболевания). Согласно дополнительному аспекту предложен слитый белок или белок согласно определению в настоящей заявке для применения при лечении рака. Также предложен слитый белок или белок согласно определению в настоящей заявке для применения при лечении воспалительных или аутоиммунных заболеваний, или метаболических заболеваний.
Также в соответствии с такими аспектами настоящего изобретения предложено применение белка или слитого белка согласно определению в настоящей заявке для изготовления медикамента для лечения или предотвращения состояния или заболевания согласно определению или описанию в настоящей заявке.
Такие аспекты также включают композицию (например, фармацевтическую композицию), содержащую белок или слитый белок согласно определению в настоящей заявке для применения при лечении или предотвращении состояния или заболевания согласно определению или описанию в настоящей заявке.
Такие аспекты также включают способ лечения или предотвращения состояния или заболевания согласно определению или описанию в настоящей заявке, отличающийся тем, что указанный способ включает введение нуждающемуся в этом субъекту белка или слитого белка согласно определению в настоящей заявке, в частности, эффективного количества указанного белка или слитого белка.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
На Фиг. 1 показаны клеточные физиологические процессы и клеточная сигнализация CCN5(dIII)-Fcv2 (вариант реализации настоящего изобретения согласно определенному в последовательности SEQ ID NO: 28).
A) показано, что слитый белок CCN5(dIII)-Fcv2 из SEQ ID NO: 28 вызывает зависимое от концентрации ингибирование фосфорилирования АКТ (серин-473) в клетках рака легкого А549.
B) показано, что слитый белок CCN5(dIII)-Fcv2 из SEQ ID. NO: 28 ингибирует пролиферацию линии клеток фибробластов легкого человека, IMR90.
C) показано, что слитый белок CCN5(dIII)-Fcv2 из SEQ ID NO: 28 ингибирует сферообразующую способность (независимый от подложки рост) линии положительных по рецептору эстрогена рака молочной железы клеток MCF-7 и линии клеток рака молочной железы с тройным негативным фенотипом MDA-MB-231.
D) показано, что слитый белок CCN5(dIII)-Fcv2 из SEQ ID NO: 28 дозозависимо ингибирует индуцированную ТФР-β SMAD репортерную активность (белки SMAD представляют собой канонические регулируемые ТФР-β транскрипционные факторы).
Все планки погрешностей отражают SD. Статистическая значимость вычислена с использованием однофакторного дисперсионного анализа ANOVA с апостериорным тестом Даннета (р<0,05 отмечено символом *).
На Фиг. 2 показан эффект разных вариантов шарнирной области Fc-фрагмента на чувствительность к протеазе вариантов реализации настоящего изобретения, где CCN5(dIII) слит с Fc-фрагментом IgG, когда тестируемый слитый белок содержит последовательность, представленную в SEQ ID NO: 28; SEQ ID NO: 29 и SEQ ID NO: 30, соответственно, см. Пример 6 ниже.
На Фиг. 3 показана склонность к агрегации варианта реализации настоящего изобретения в зависимости от структуры пептидного линкера, соединяющего CCN5(dIII) с Fc-фрагментом IgG, когда тестируемый слитый белок содержит последовательность, представленную в SEQ ID NO: 30 и SEQ ID NO: 31.
На Фиг. 4 показан слитый белок в соответствии с настоящим изобретением, содержащий домен гомологии к повторам TSP-1, который соединен на С-конце с пептидным линкером, и соединен посредством Fc-шарнира с Fc-фрагментом.
На Фиг. 5 показана сниженная чувствительность к расщеплению эндопептидазой в том случае, когда вариант реализации настоящего изобретения содержит мутацию пролин-195 домена гомологии к повторам TSP-1 CCN5, представленного в SEQ ID NO: 7 (Fc-HLn8-CCN5(dIII)-P195A, SEQ ID NO: 41), относительно варианта P195 дикого типа домена гомологии к повторам TSP-1 CCN5 (Fc-HLn8-CCN5(dIII), SEQ ID NO: 40).
На Фиг. 6 показано получение белка, соответствующего SEQ ID NO: 58, очищенного путем хроматографии с захватом на белке А. Видно, что в отсутствие восстанавливающего агента бета-меркаптоэтанола присутствует димер ((-) дорожка). Однако в присутствии бета-меркаптоэтанола ((+) дорожка), как можно видеть, первичный продукт представляет собой фрагмент после расщепления, состоящий только из Fc-фрагмента, а не интактный слитый белок, содержащий все части, кодируемые SEQ ID NO: 58 (домен гомологии с TSP-1 фрагмент, пептидный линкер и Fc-фрагмент).
На Фиг. 7 показано получение белка, соответствующего SEQ ID NO: 27, имеющего усеченную C-концевую часть, очищенного путем хроматографии с захватом на белке А. Видно, что указанный белок значимо более устойчив к разложению протеазой, чем белок, соответствующий SEQ ID NO: 58, который включает C-концевую часть.
На Фиг. 8 показано получение белка, соответствующего SEQ ID NO: 73, аналогичного белку, соответствующему SEQ ID NO: 27, очищенного путем хроматографии с захватом на белке А. В этом случае также, как можно видеть, в присутствии бета-меркаптоэтанола ((+) дорожка) указанный белок более устойчив к разложению протеазой, чем белок, соответствующий SEQ ID NO: 58.
На Фиг. 9 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белка, соответствующего SEQ ID NO: 41, продуцируемого в стабильно трансфицированных клетках. Видно, что указанный белок не демонстрирует ингибирования фосфорилирования AKT.
На Фиг. 10 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белка, соответствующего SEQ ID NO: 80, продуцируемого в стабильно трансфицированных клетках. Видно, что указанный белок не демонстрирует значимого ингибирования фосфорилирования AKT и, фактически, даже может приводить к повышению уровня фосфо-AKT при более высокой концентрации.
На Фиг. 11 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белка, соответствующего SEQ ID NO: 80, продуцируемого в транзиентно трансфицированных клетках. Можно видеть, что при продуцировании в транзиентно трансфицированных клетках указанный белок характеризуется зависимой от концентрации ингибиторной активностью в отношении фосфорилирования AKT.
На Фиг. 12 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белков, соответствующих SEQ ID NO: 84, 94 и 106. Видно, что каждый из указанных белков характеризуется зависимой от концентрации ингибиторной активностью в отношении фосфорилирования AKT.
На Фиг. 13 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белка, соответствующего SEQ ID NO: 88. Видно, что указанный белок способен ингибировать фосфорилирование АКТ в концентрациях выше 10 мкг/мл.
На Фиг. 14 представлены результаты анализа для измерения уровней фосфо-AKT (Ser-473) в клетках рака легкого А549 после введения варьирующих концентраций белков, соответствующих SEQ ID NO: 102 и 97. Видно, что оба белка характеризуются зависимой от концентрации ингибиторной активностью в отношении фосфорилирования AKT.
На Фиг. 15 представлены результаты анализа для измерения уровней фосфо-AKT в клетках рака легкого А549 после введения варьирующих концентраций белка, соответствующего SEQ ID NO: 110. Видно, что указанный белок характеризуется зависимой от концентрации ингибиторной активностью в отношении фосфорилирования AKT.
На Фиг. 16 представлены результаты ряда экспериментов, включающих белок, соответствующий SEQ ID NO: 106.
A) показывает, что указанный белок ингибирует миграцию фибробластов легких человека, индуцированную как ТФР-бета, так и CCN2.
B) показывает, что указанный белок ингибирует зарастание царапины, индуцированное как ТФР-бета, так и CCN2.
C) показывает, что указанный белок обеспечивает частичное ингибирование индукции ТФР-бета экспрессии гена COL1A1, который, как известно, является профибротическим.
D) показывает, что указанный белок обеспечивает частичное ингибирование индукции ТФР-бета экспрессии гена FN1, который, как известно, является профибротическим.
E) показывает, что указанный белок обеспечивает частичное ингибирование индукции ТФР-бета экспрессии гена АСТА2, который, как известно, является профибротическим.
F) показывает, что указанный белок обеспечивает частичное ингибирование индукции ТФР-бета экспрессии гена CCN2, который, как известно, является профибротическим.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение, как уже упоминалось, основано на неожиданном открытии того, что домен гомологии к повторам тромбоспондина типа 1 (TSP-1) CCN5 представляет собой полностью активную структуру, обеспечивающую функции клеточной сигнализации CCN5/WISP2. На основании указанных новых данных относительно активности домена гомологии к повторам TSP-1 CCN5 авторы настоящего изобретения предлагают белки, рекомбинантные белки и слитые белки в соответствии с настоящим изобретением, которые могут быть использованы для ингибирования или противодействия клеточной сигнализации и физиологическим функциям клетки, приписываемым четырехдоменным белкам CCN, т.е. CCN1, CCN2, CCN3, CCN4 и CCN6. Указанные новые данные, как считается, предположительно, критически важны для обеспечения формирования стабильной, гомогенной лекарствоподобной молекулы на основе CCN5, поскольку ранее не было установлено, какая часть полноразмерного CCN5 необходима для воспроизведения активности CCN5, и поскольку полноразмерные молекулы CCN очень восприимчивы к протеолизу и их трудно получить в активном гомогенном виде. Кроме того, эти данные, подразумевающие, что конкретная часть CCN5 может быть достаточной для воспроизводства активности, наблюдаемой, например, при транзиентной сверхэкспрессии полноразмерного белка, также противоречат преобладающему в данной области мнению, что белки CCN функционируют как матриклеточные белки. Преобладающее мнение о механизме действия белков CCN и матриклеточных белков в целом заключается в том, что разные сегменты белков CCN взаимодействует с различными другими белками ВКМ и рецепторами клеточной поверхности, таким образом, модулируя их активность, а не работают как прямые модуляторы клеточной сигнализации. Новая информация об активности домена гомологии к повторам TSP-1 CCN5, а также информация о структурной близости с другими представителями семейства белков CCN предполагает, что и домены гомологии к повторам TSP-1 других белков семейства CCN могут также быть использованы для ингибирования функций клеточной сигнализации четырехдоменных белков семейства CCN. Согласно одному аспекту рекомбинантные белки и слитые белки согласно настоящему изобретению ингибируют фосфорилирование АКТ (Ser473) в клетках А549.
Ингибирование указанной клеточной сигнализации имеет значение при лечении различных расстройств. CCN2, например, вовлечен в ряд заболеваний, в частности, заболеваний, при которых усиленный фиброгенез и тканевой фиброз представляют собой характерный патофизиологический признак.
Например, было показано, что избыточная экспрессия CCN2 сама по себе достаточна для индуцирования фиброза в легком (см. Sonnylal et el., Arthritis Rheum 62, 1523-1532 (2010)). Также было обнаружено, что CCN2 необходим для индуцированного блеомицином фиброза легких (Bonniaud, P. et al. Am J Respir Cell Mol Biol 31, 510-516 (2004)), радиационно-индуцированного фиброза легких (Bickelhaupt, S. et al. J Natl Cancer Inst 109 (2017) и фиброза легких в результате утраты экспрессии PTEN (гомолога фосфатазы и тензина) (Parapuram, S.K. et al. Matrix Biol 43, 35-41 (2015)). Кроме того, было обнаружено, что в отсутствие других стимулирующих агентов CCN2 индуцирует фиброз легких при экспрессии и секреции легочными клетками Клара (Wu, S. et al. Am J Respir Cell Mol Biol 42, 552-563 (2010)), альвеолярными эпителиальными клетками II типа (Chen, S. et al. Am J Physiol Lung Cell Mol Physiol 300, L330-340), при экспрессии со специфичного для фибробластов промотора (Sonnylal et al (2010), выше, Sonnylal, S. et al., J Cell Sci 126, 2164-2175 (2013)) или доставке аденовирусом (Bonniaud, P. et al., Am J Respir Crit Care Med 168, 770-778 (2003)). Соответственно, все имеющиеся сообщения подтверждают вывод о том, что CCN2 не только достаточен для стимуляции фиброза в коже или легком, однако также необходим для полного фиброзного фенотипа в нескольких моделях заболеваний. Фиброз легких представляет собой признак заболевания человека идиопатического фиброза легких (ИФЛ), однако это также возникает при хронической обструктивной болезни легких (ХОБЛ) (Jang, J.H. et al., COPD 14, 228-237 (2017)) и системном склерозе. На самом деле фиброз легких регистрируется как первичная причина смерти до 40% пациентов с системным склерозом (Tyndall AJ et al., Ann Rheum Dis. 2010 Oct; 69(10):1809-15). Показано, что CCN2 и другие белки CCN, такие как WISP1, также вовлечены в патофизиологию ИФЛ (Konigshoff, М. et al., J Clin Invest 119, 772-787 (2009) и ХОБЛ (Jang et al, выше) у пациентов-людей.
Другой пример представлен неопластическими расстройствами. Например, в условиях рака молочной железы CCN2, как было показано, вносит вклад в метастазирование в кости в модели рака молочной железы с тройным негативным фенотипом (MDA-MB-231) (Kang, Y. et al., Cancer Cell 3, 537-549 (2003)). Кроме того, нокдаун CCN2 в клетках рака молочной железы с тройным негативным фенотипом (MDA-MB-231), линии клеток, которая экспрессирует высокие уровни CCN2 (Chen, P.S. et al., J Cell Sci 120, 2053-2065 (2007)), снижал способность указанных клеток к миграции, тогда как избыточная экспрессия CCN2 в положительных по рецепторам гормона клетках линии рака молочной железы MCF-7, при низкой эндогенной экспрессии CCN2 (Chen et al., выше), повышала способность последних к миграции (Chen et al., выше, Chien, W. et al., Int J Oncol 38, 1741-1747 (2011)). В более позднем отчете также было описано, что избыточная экспрессия CCN2 в клетках MCF-7 увеличивает хеморезистентность, тогда как нокдаун CCN2 в клетках MDA-MB-231 снижает хеморезистентность (Wang, M.Y. et al., Cancer Res 69, 3482-3491 (2009)). Сообщалось также об увеличении хеморезистентности, вызываемой CCN2, других клеток рака молочной железы (Lai, D et al., Cancer Res 71, 2728-2738 (2011)). Кроме того, с помощью исследований сверхэкспрессии или нокдауна было также показано, что CCN2 вносит вклад в эпителиально-мезенхимальный переход (ЕМТ) и увеличивает способность к независимому от подложки росту (образованию маммосфер) клеток рака молочной железы (Chen et al., выше, Zhu, X. et al., Oncotarget 6, 25320-25338 (2015)). Обнаружение как повышенной хеморезистентности, так и усиления ЕМТ, индуцируемых CCN2, согласуется со связью, установленной между ЕМТ и хеморезистентностью и при других типах рака (Fischer, K.R. et al., Nature 527, 472-476 (2015), Zheng, X. et al., Nature 527, 525-530 (2015)).
Согласно конкретному аспекту настоящего изобретения предложен мономерный слитый белок согласно определению выше, содержащий полипептид, соответствующий по меньшей мере части домена гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN, причем последовательность указанного домен гомологии с TSP-1 может быть усечена и/или модифицирована, однако при этом остатки цистеина указанного домена сохранены. Указанный полипептид для удобства может быть назван в настоящей заявке «полипептидом TSP-1», и указанный термин, соответственно, следует понимать как не обуславливающий или не подразумевающий каких-либо ограничений до исключительно конкретной природной последовательности домена гомологии с TSP-1. Термин «полипептид TSP-1» может использоваться как синоним или взаимозаменяемо с «белком домена TSP-1» или «последовательностью домена TSP-1».
Как продемонстрировано в приведенных ниже примерах, неожиданным образом было обнаружено, что мономерные партнеры для слияния предпочтительнее для получения активных и стабильных белков по сравнению с димерными партнерами для слияния, такими как Fc-фрагменты, происходящие из IgG-белков, которые формируют димерные слитые белки. Мономерные слитые белки сохраняют активность полипептида домена TSP-1, который они содержат. Кроме того, указанные белки стабильны, в том числе применительно к протеолитическому разложению. Как подробнее описано ниже, устойчивость к протеолитическому разложению может быть повышена путем введения модификаций в последовательность аминокислот полипептида TSP-1, в том числе, в частности, замены Ala, упомянутой выше.
Соответственно, полипептид компонента (i) слитого белка может содержать инсерции, делеции, замены, мутации или любую их комбинацию относительно указанной последовательности SEQ ID NO: 37 или 2-6, или последовательности, выбранной из SEQ ID NO: 1 или 8 12, при условии, что полипептид сохраняет по меньшей мере 80% идентичности последовательностей указанной последовательности, и все остатки цистеина в указанной последовательности сохранены.
Согласно другому аспекту настоящего изобретения предложен белок (например, рекомбинантный белок), который состоит из полипептида или содержит полипептид, соответствующий по меньшей мере части домена гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN, но не в контексте слитого белка, где последовательность домена TSP-1 может быть усечена и/или модифицирована и содержит замену Ala в положении, соответствующем положению 2 SEQ ID NO: 37 или 2-6, или SEQ ID NO: 1 или 8-12, однако при этом остатки цистеина указанного домена сохранены. Иными словами, белок домена TSP-1 может быть предоставлен без другого компонента, такого как партнер для слияния, или независимо от него. Соответственно, белок домена TSP-1 не сливают или не соединяют с другим белковым доменом или компонентом, или другой функциональной или структурной последовательностью белка. Для удобства такие белки могут называться «белками с заменой на Ala».
В настоящей заявке термин «консервативный» означает, что остаток в определенной последовательности не делетирован или не заменен. Иными словами, термин «консервативный» используется как синоним (и взаимозаменяемо) с термином «сохраненный». Он просто означает, что остатки цистеина не удалены из последовательности. Соответственно, в описанном выше контексте это означает, что остатки цистеина в последовательности, выбранной из SEQ ID NO: 37 или 2-6 или 1 или 8-12, не делетированы и не удалены. Отметим, что инсерция дополнительных остатков между консервативными остатками (например, между консервативными остатками цистеина) или делеция неконсервативных остатков (например, остатков, не являющихся цистеином) может изменять положение консервативных остатков в последовательности полипептида относительно их положения в исходной референсной последовательности (например, последовательности, выбранной из SEQ ID NO: 37 или 2-6). Однако такие остатки все же считают «консервативными» согласно определению в настоящей заявке. Соответственно, термин «консервативный» не подразумевает какого-либо сужения или ограничения положения (или, более конкретно, номера положения) остатков цистеина.
Согласно некоторым вариантам реализации полипептид по (i) содержит или состоит из:
(а) последовательности аминокислот, выбранной из SEQ ID NO: 1 или 8-12; или
b) последовательности аминокислот, по меньшей мере на 80% идентичной последовательности, выбранной из SEQ ID NO: 1 или 8-12; или
c) части последовательности аминокислот по (а) или (b), отличающейся тем, что указанная часть содержит по меньшей мере последовательность из 44 аминокислот из SEQ ID NO: 37, 6, 2, 3, 4 или 5, соответственно, или последовательность, по меньшей мере на 80% идентичную последовательности, выбранной из SEQ ID NO: 37, 6, 2, 3, 4 или 5, соответственно.
Как отмечалось выше, мономерный партнер для слияния по (ii) и пептидный линкер по (iii) не представляют собой домен или не содержат домена гомологии со связывающим IGF белком, домена гомологии с повторами фактора фон Виллебранда типа С или домена цистинового узла белка семейства CCN. Иными словами, единственный домен белка семейства CCN, который может присутствовать в слитом белке согласно настоящему изобретению, представляет собой домен гомологии с TSP-1.
Сходным и аналогичным образом, в контексте белков с заменой на Ala, которые не являются слитыми белками, указанный белок не содержит каких-либо других доменов CCN (помимо белка домена TSP-1).
Согласно некоторым вариантам реализации полипептид по (i) или белок с заменой на Ala, может содержать только часть домена гомологии с TSP-1 согласно определению выше. Авторы настоящего изобретения обнаружили, что минимальный требуемый фрагмент домена TSP-1 представлен последовательностью из 44 аминокислот из SEQ ID NO: 37, 6, 2, 3, 4 или 5. Соответственно, согласно некоторым вариантам реализации длина полипептида по (i) составляет по меньшей мере 44 аминокислот. Согласно некоторым вариантам реализации полипептид по (i) имеет длину от 44 до 57 аминокислот. Однако, как отмечалось выше, в минимальном фрагменте из 44 аминокислот может присутствовать одна или более делеций аминокислот, которые расположены между остатками цистеина. Соответственно, согласно некоторым вариантам реализации длина полипептида TSP-1 может составлять менее 44 остатков, т.е. 40-43 остатка.
Согласно некоторым вариантам реализации полипептид по (i) состоит из последовательности аминокислот, выбранной из SEQ ID NO: 37 или 2-6, или последовательность, по меньшей мере на 80% идентичную последовательности, выбранной из SEQ ID NO: 37 или 2-6.
Согласно описанию выше, белки согласно настоящему изобретению, в том числе слитые белки, проявляют (или, другими словами, демонстрируют или имеют) активность CCN5, более конкретно - биологическую активность CCN5. Согласно варианту реализации указанные белки могут сохранять, или проявлять, или иметь активность домена гомологии с TSP-1 CCN5. Как вариант, указанные белки могут быть определены как проявляющие (или демонстрирующие, или имеющие) активность, в частности, биологическую активность выделенного домена гомологии с TSP-1 белка CCN. Сказанное выше может относиться к любой активности указанного домена, а также к конкретным видам активности, отражающим антифибротический эффект указанного домена гомологии с TSP-1. Такая активность может быть проанализирована (или протестирована, или детектирована) с применением любого удобного анализа или способа на основе любого конкретного биологического эффекта указанного домена.
Отметим, что активность определенного белка может удобным образом быть оценена путем анализа эффекта белка на фосфорилирование AKT. В частности, определенный белок может быть проанализирован на способность ингибировать фосфорилирование AKT (Ser-473) в клетках рака легкого человека А549, согласно описанию в Примере 2. Специалисту будет понятно, что другие аналогичные анализы могут быть разработаны для оценки этой же активности или для оценки других родственных видов антифибротической активности.
Как отмечалось выше, согласно другим аспектам настоящего изобретения предложены рекомбинантные белки, которые ингибируют или противодействуют клеточной сигнализации и физиологическим функциям клетки, приписываемым четырехдоменным белкам CCN, содержащие последовательность аминокислот в соответствии с приведенной выше формулой I.
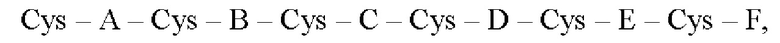
где А, В, С, D, Е и F соответствуют определению выше и в прилагаемой формуле изобретения.
Формула I представляет собой результат выравнивания домена гомологии к повторам TSP-1 структурно родственных белков семейства CCN (CCN 1 - CCN6), все из которых содержат 6 консервативных остатков цистеина, с учетом того, что аминокислоты могут быть заменены так, чтобы это не влияло на активность указанного белка (консервативные замены, как подробнее обсуждается ниже). Положение первого консервативного остатка цистеина домена гомологии к повторам TSP1 других белков CCN определяют как положение №1 рекомбинантного белка формулы I.
Пять сегментов между консервативными цистеинами обозначены как А, В, С, D и Е, соответственно.
Первый сегмент А определяет формула 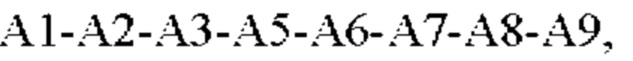 где А1-А9 соответствует определению выше. Аминокислота в положении №7 (А7) сегмента А представляет собой триптофан (W) у всех представителей семейства белков CCN и считается консервативной.
где А1-А9 соответствует определению выше. Аминокислота в положении №7 (А7) сегмента А представляет собой триптофан (W) у всех представителей семейства белков CCN и считается консервативной.
Второй сегмент В определяет формула В1-В2-В3, где В1-В3 соответствует определению выше. В соответствии с одним вариантом реализации В1 и В3 представляет собой либо серии, либо треонин.
Третий сегмент С определяет формула 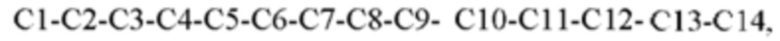 где аминокислоты С1-С14 соответствуют определению выше. В соответствии с одним вариантом реализации аминокислоты С1 и С3 представляют собой глицин (G).
где аминокислоты С1-С14 соответствуют определению выше. В соответствии с одним вариантом реализации аминокислоты С1 и С3 представляют собой глицин (G).
Согласно другому варианту реализации С7 представляет собой аргинин (R), оба из С10 и С12 представляют собой аспарагин (N).
Четвертый сегмент D определяет формула 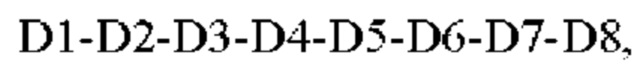 где аминокислоты D1-D8 соответствуют определению выше. В соответствии с одним вариантом реализации D7 представляет собой аргинин (R).
где аминокислоты D1-D8 соответствуют определению выше. В соответствии с одним вариантом реализации D7 представляет собой аргинин (R).
Пятый сегмент Е определяет формула 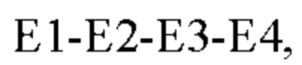 где аминокислоты Е1-Е4 соответствуют определению выше. В соответствии с одним вариантом реализации настоящего изобретения Е4 представляет собой пролин.
где аминокислоты Е1-Е4 соответствуют определению выше. В соответствии с одним вариантом реализации настоящего изобретения Е4 представляет собой пролин.
После последнего остатка цистеина расположен карбоксиконцевой пептидный сегмент вариабельной длины (F), содержащий от 0 до 13 аминокислот.
F может быть делетирован или укорочен по сравнению с последовательностями аминокислот домена гомологии к повторам TSP-1 семейства белков CCN. В соответствии с одним вариантом реализации F отсутствует. Согласно другому варианту реализации F состоит из пептида, выбранного из группы, состоящей из PPSRGRSPQNSAF, GQPVYSSL, EADLEEN, EQEPEQPTD, DVDIHTLI и DSNILKTIKIP. В соответствии с одним аспектом указанного варианта реализации рекомбинантные белки могут принимать форму последовательности аминокислот, представленную в SEQ ID NO: 8-12.
Согласно другому аспекту настоящего изобретения предложены рекомбинантные белки, содержащие последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38; и их фрагментов или вариантов по меньшей мере с 50% идентичностью последовательностей последовательностям аминокислот SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38.
Согласно одному аспекту предложен рекомбинантный белок, состоящий из последовательности аминокислот, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38; и их фрагментов или вариантов с более чем 50% идентичностью последовательностей последовательностям аминокислот SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, и SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37, SEQ ID NO: 38.
«Рекомбинантные белки» в настоящей заявке представляют собой белки, кодируемые рекомбинантными нуклеиновыми кислотами. Они экспрессируются с рекомбинантных нуклеиновых кислот в клетке-хозяине как подробнее описано ниже.
«Рекомбинантная нуклеиновая кислота» в настоящей заявке описывает молекулу нуклеиновой кислоты, которая, ввиду происхождения или в результате манипуляций, не связана с полным полинуклеотидом или с частью полинуклеотида, с которым связана в природе, и/или соединена не с тем полинуклеотидом, с которым соединена в природе, как подробнее описано ниже.
Специалисту будет понятно, что может быть введена модификация последовательности аминокислот рекомбинантных белков и слитых белков в соответствии с настоящим изобретением без изменения активности указанного белка. Аминокислоты обычно классифицируют как гидрофобные или гидрофильные и/или имеющие полярные или неполярные боковые цепи. Замены одной аминокислоты на другую, имеющую те же биохимические характеристики, общеизвестны как консервативные замены.
Консервативные замены аминокислот включают взаимные замены аминокислот внутри следующих групп:
• MILV
• FYW
• KRH
• AG
• ST
• QN
• ED
Обычно «консервативная замена аминокислоты» относится к замене аминокислоты, которая не изменяет характеристик относительного заряда или размера белка, в котором осуществляют замену аминокислоты, и, соответственно, редко изменяет структуру белка, поэтому биологическая активность также значимо не изменяется.
Специалисту будет понятно, что биологическая активность белка также может быть сохранена, если одна или несколько аминокислот делетированы, инсертированы или добавлены к последовательности аминокислот, при условии сохранения структурных и физико-химических свойств.
Символ «Δ» в настоящей заявке перед аминокислотой относится к делеции указанной аминокислоты, например, ΔК447 следует понимать как белок, в котором отсутствует К447. Также в настоящей заявке делецию конкретной аминокислоты, как вариант, обозначают символом «-», например, К447- также следует понимать как белок, в котором отсутствует К447.
Соответственно, следует понимать, что настоящим изобретением охвачены рекомбинантные белки и слитые белки согласно описанию в прилагаемой формуле изобретения, в которые могут быть введены такие модификации согласно описанию выше (замены, делеции, инсерции и добавления аминокислот) по существу без изменения их биологической активности, т.е. способности ингибировать или противодействовать клеточной сигнализации и физиологическим функциям клетки, приписываемым четырехдоменным белкам семейства CCN; CCN1, CCN2, CCN3, CCN4 и CCN6.
В настоящем описании упоминаются последовательности аминокислот. В настоящем описании применительно к последовательностям аминокислот иногда упоминается модификация рассматриваемой последовательности аминокислот или белка со ссылкой на «нумерацию Uniprot» или Eu-нумерацию. Нумерация Uniprot относится к нумерации, используемой в базе данных Uniprot (Uniprot Consortium, Nucleic Acids Res. 2019 Jan 8;47(D1):D506-D515). Нумерацию Uniprot используют для нумерации аминокислот белков CCN. Eu-нумерация относится к нумерации антитела Eu (Edelman et al., 1969, Proc Natl Acad Sci USA 63:78-85), и ее используют в отношении аминокислот в Fc-фрагментах подклассов IgG человека с мутациями или без мутаций, или химерах, отличных от дикого типа. Система нумерации Ей доступна, например, в международной информационной системе iMMunoGeneTics (IMGT) ресурса IMGT Scientific chart. Система IMGT описана в источнике: Lefranc М-Р, Biomolecules. 2014 Dec; 4(4): 1102- 1139.
В контексте настоящего описания, когда речь идет об «идентичности последовательности», последовательность по меньшей мере с х% идентичностью второй последовательности означает, что х% соответствует числу аминокислот в первой последовательности, идентичных соответствующим им аминокислотам второй последовательности при проведении оптимального выравнивания последовательностей путем глобального выравнивания по всей длине второй последовательности аминокислот. Оптимальное выравнивание обеих последовательностей выполнено, если х максимален. Выравнивание и определение процента идентичности может проводиться вручную или автоматически.
Выравнивание с целью определения процента идентичности аминокислот может быть достигнуто различными путями в пределах компетенции специалиста в данной области техники, например, с использованием открытого компьютерного обеспечения, такого как ClustalOmega (Sievers F, Higgins DG (2018) Protein Sci 27:135-145), Protein BLAST (от Национального центра биотехнологической информации (NCBI), США) или коммерческого программного обеспечения, такого как программное обеспечение Megalign (DNASTAR). Специалисты в данной области техники могут определить подходящие показатели для измерения выравнивания, в том числе любые алгоритмы, необходимые для достижения максимального выравнивания на протяжении полной длины сравниваемых последовательностей. NCBI BLAST представляет собой другой пример программного обеспечения для определения идентичности последовательности аминокислот (MacWilliam et al., Nucleic Acids Res.2013 Jul; 41(Web Server issue): W597-W600).
В соответствии с одним аспектом настоящего изобретения предложен рекомбинантный белок, содержащий последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38; и ее фрагменты или варианты по меньшей мере с 50% идентичностью последовательностей последовательностям аминокислот SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38.
Согласно другому аспекту предложен рекомбинантный белок, содержащий последовательность аминокислот по меньшей мере с 60%, 70%, 80%, 90%, или 95% идентичностью последовательностей последовательности аминокислот, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38.
Биологически активные белки и пептиды играют важную роль в клиническом лечении заболеваний человека. Однако со многими белками и пептидами возникают затруднения из-за их далеко не идеальных фармакокинетических свойств, поскольку они элиминируются в результате фильтрации почками из-за малого размера и/или протеолитического метаболизма. Такие факторы могут налагать ограничения или приводить к затруднениям при введении лекарства субъектному нуждающемуся в лечении, например, необходимость проведения постоянных инфузий или частых подкожных введений для поддержания циркулирующих концентраций белка или пептида на эффективном терапевтическом уровне. Необходимость постоянного или очень частого введения лекарственного средства клинически нежелательна из-за очевидных проблем и неудобства как для пациента, так и для лечащего врача.
Одна из стратегий увеличения времени полужизни биологически активного пептида или белка состоит в том, чтобы связать группу полиэтиленгликоля (ПЭГ) с пептидом или белком, представляющим интерес, с помощью процесса, называемого пегилированием (см., например, Dozie et al. (2015), Int. J. Mot Sci, 16(10) 25831-25864). Общая стратегия пегилирования белков состоит в проведении реакции функциональной группы на белке с комплементарной группой на молекуле ПЭГ с образованием конъюгата белка и ПЭГ. ПЭГ-фрагмент обеспечивает ряд преимуществ для увеличения стабильности белка и времени полужизни в кровотоке благодаря его гибкости, гидрофильности, вариабельному размеру и низкой токсичности.
Согласно одному варианту реализации настоящего изобретения, соответственно, предложен рекомбинантный белок согласно описанию выше, отличающийся тем, что указанный указанный белок пегилирован. Слитые белки в соответствии с настоящим изобретением могут также быть пегилированными.
Слитые белки
Другой способ избежать затруднений, связанных с медицинским применением пептидов и белков, заключается в продлении времени полужизни биоактивного белка или пептида путем получения слитых белков, (см. например Valeria et al. (2017), «А New Approach to Drag Therapy: Fc-Fusion Technology), Prim Health Care, 7:255, doi: 10.4172/2167-1079.1000255). Путем ковалентного слияния белка или пептида с белком-носителем посредством генетической рекомбинации можно увеличить молекулярную массу белка, представляющего интерес, до приблизительно 60-70 кДа, что соответствует порогу для фильтрации почками.
В настоящем изобретении предложен слитый белок, содержащий
(i) Домен гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN;
(ii) партнер для слияния, слитый на N- или С-конце с доменом гомологии к повторам TSP-1 по (i), причем указанный партнер для слияния выбран из группы, состоящей из сывороточного альбумина, трансферрина и Fc-фрагмента иммуноглобулина.
(iii) необязательно, пептидный линкер между доменом гомологии к повторам TSP-1 и Fc-фрагментом (слитым на N- или С-конце с доменом гомологии к повторам TSP-1) по (i).
В настоящем описании домен гомологии к повторам TSP-I также может называться доменом III и относиться к домену III, то есть третьему домену белков семейства CCN.
В одном предпочтительном аспекте партнер для слияния представляет собой мономерный партнер для слияния и приводит к получению мономерного слитого белка. Такие слитые белки и, в частности, их домены TSP-1, определены выше и подробнее описаны ниже.
Однако настоящее изобретение также включает другие варианты реализации, как применительно к компоненту белку домена TSP-1, так и к компоненту партнеру для слияния.
В соответствии с одним таким вариантом реализации домен гомологии к повторам TSP-1 представляет собой рекомбинантный белок формулы I согласно определению выше.
Домен гомологии к повторам TSP-1 в соответствии с другим вариантом реализации представляет собой рекомбинантный белок, имеющий последовательность аминокислот согласно определению в любой из последовательностей, представленных в SEQ ID NO: 1-12, 37 и 38, или рекомбинантный белок формулы I согласно определению выше.
В соответствии с одним вариантом реализации домен гомологии к повторам TSP-1 представляет собой рекомбинантный белок, содержащий последовательность аминокислот по меньшей мере с 50%, 60%, 70%, 80%, 90% или 95% идентичностью последовательностей последовательности аминокислот, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38.
Белки по своей природе чувствительны к разложению протеазами. Для предотвращения разложения протеазами рекомбинантных белков и слитых белков в соответствии с настоящим изобретением в последовательность аминокислот могут быть введены модификации, например, с помощью направленного мутагенеза, чтобы получить устойчивые к протеазам рекомбинантные белки и слитые белки. Например, точечная мутация может быть введена в домен гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN согласно определению в SEQ ID NO: 1-12, 37 или 38, или, более конкретно, в белок согласно определению в любой из SEQ ID NO: 1-6, 8-12 или 37, слитый белок или рекомбинантный белок. В соответствии с одним вариантом реализации вводят точечную мутацию, снижающую чувствительность к протеолитическому разложению. Неограничивающий пример точечной мутации, которая приводит к меньшему протеолизу рекомбинантных белков и слитых белков согласно настоящему изобретению, заключается во введении точечной мутации, соответствующей замене пролина на аланин в положении 195 (Р195А) домена III CCN5, такой, как показана в SEQ ID NO: 7. Аналогичная мутация может также быть введена в последовательности аминокислот, происходящие из домена III других представителей семейства CCN. SEQ ID NO: 38 соответствует усеченной последовательности из 44 аминокислот домена TSP-1 CCN5, содержащей замену на Ala. SEQ ID NO: 42-46 соответствуют усеченным последовательностям из 44 аминокислот доменов гомологии с TSP-1 CCN1, 2, 3, 4 и 6, соответственно, содержащим замену на Ala. SEQ ID NO: 47-51 соответствуют более длинным последовательностям гомологии с TSP-1 CCN1, 2, 3, 4 и 6, соответственно, содержащим замену Ala. Любая такая последовательность, или последовательность, по меньшей мере на 80% ей идентичная, может применяться в соответствии с настоящим изобретением.
Как отмечалось выше, согласно предпочтительному варианту реализации указанный партнер для слияния (ii) слитого белка в соответствии с настоящим изобретением является мономерным. Может применяться любой мономерный партнер для слияния. Соответственно, партнер для слияния может представлять собой любой белок или его часть (например, домен белка), который возникает и остается в мономерной форме при слиянии с компонентом белком с доменом гомологии с TSP-1. Соответственно, слитый белок, содержащий мономерный партнер для слияния и белок с доменом гомологии с TSP-1, остается мономером. Это означает, что он не димеризуется и не образует сам с собой мультимеры более высоких порядков.
Известны различные белки, подходящие в качестве возможные партнеров для слияния, они могут включать природные белки, или их фрагменты или варианты с модифицированной последовательностью аминокислот, а также синтетические белки или гомополимеры аминокислот. Такие белки включают, в частности, Fc-фрагменты IgG, сывороточный альбумин или трансферрин.
Партнер для слияния в настоящей заявке определен в широком смысле как второй полипептид (или вторая последовательность аминокислот), который не присутствует в комбинации (например, не расположен смежно, или не связан, прямо или непрямо) с первым полипептидом CCN с гомологией TSP-1 в природе, и соединен с первым полипептидом CCN с гомологией TSP-1 в синтетической или искусственной комбинации. Соответственно, слитый белок содержит не встречающуюся в природе комбинацию по меньшей мере двух последовательностей аминокислот или полипептидов, соединенных или слитых вместе.
Партнером для слияния может быть последовательность аминокислот длиной по меньшей мере 6, 8, 9, 10, 15, 20, 25, 30, 40 или 50 или более аминокислот. Как правило, партнер для слияния представляет собой функциональный полипептид, или, другими словами, он представляет собой полипептид, который обеспечивает функцию или свойство слитого белка, например, стабилизирует слитый белок (делая первый полипептид более стабильным) или увеличивает время его полужизни в сыворотке. Соответственно, партнер для слияния может представлять собой структурный белок или иметь структурную функцию, или он может обеспечивать активность или свойство слитого белка, например, связывающую активность (например, партнер для слияния может быть членом связывающейся пары, или может быть партнером для аффинного связывания, и т.п.). В репрезентативных примерах партнером для слияния может быть альбумин (в частности, сывороточный альбумин), фибриноген, глутатион-S-трансфераза, трансферрин, стрептавидин или стрептавидино-подобный белок, или иммуноглобулин, или его часть, в частности, Fc-часть иммуноглобулина (например IgG1, IgG2, IgG3 или IgG4), или ее часть или модифицированный вариант. Подходящие сывороточные альбумины включают бычий сывороточный альбумин (БСА), сывороточный альбумин мыши (МСА) и, в особенности, сывороточный альбумин человека (ЧСА). Другие возможные партнеры для слияния включают полипептиды, которые могут действовать, улучшая фармакокинетические свойства слитого белка, например, синтетические полипептиды, такие как гомополимер аминокислоты, пролин-аланин-сериновый полимер или эластиноподобный пептид, например, согласно описанию в источнике: Strohl, 2015, BioDrugs 29, 215-239. Может применяться любой партнер для слияния для применения с терапевтическими белками, известный в данной области техники.
Согласно варианту реализации партнером для слияния (ii) слитого белка в соответствии с настоящим изобретением может быть Fc-фрагмент IgG (любого подкласса или химеры любых подклассов), сывороточный альбумин или трансферрин.
Партнер для слияния может быть сопряжен на N- или С-конце с компонентом - белком с доменом гомологии с TSP-1 слитого белка, например, с доменом гомологии к повторам TSP-1 CCN5 или любых других белков CCN согласно определению в настоящей заявке. Он может быть присоединен прямо или непрямо, посредством линкера, как подробнее описано ниже.
Fc-фрагменты склонны образовывать димеры, и при использовании в слитых белках конструкция слитого белка проявляет склонность к включению двух копий слитого белка. Однако в данной области техники известно, что могут быть получены мономерные Fc-фрагменты и содержащие их мономерные слитые белки.
Соответственно, если партнер для слияния представляет собой Fc-фрагмент, он предпочтительно представляет собой мономерный Fc-фрагмент, такой как мономерный Fc-фрагмент IgG человека любого класса. Предусмотрены химерные Fc-фрагменты, содержащие части Fc-областей из разных классов, а также Fc-фрагменты с модифицированными последовательностями.
Слитые с Fc белки представляют растущий класс белковых терапевтических средств на основе химерных белков, состоящих из эффекторного домена, сопряженного с Fc-фрагментом IgG-изотипа. Типичным примером биофармацевтического продукта является этанерцепт (рецептор ФНО-α, связанный с Fc-фрагментом), используемый при лечении, например, ревматоидного артрита. Другим примером биофармацевтического белка слияния Fc является афлиберцепт. Афлиберцепт, слитый с Fc белок рецептора ФРЭС, используется в лечении влажной формы макулярной дегенерации и метастатического рака ободочной и прямой кишки. Основным обоснованием для получения слитых белков с Fc-фрагментом является продление времени полужизни за счет увеличения молекулярной массы, достаточного для исключения почечной экскреции и усиления почечной проксимальной канальцевой реабсорбции через неонатальный Fc-рецептор. Также рН-зависимое связывание слитых с Fc белков с неонатальным рецептором Fc (FcRn) на эндотелиальных клетках позволяет слитым белкам на основе Fc, которые в противном случае подверглись бы эндоцитозу и последующему лизосомному разложению, проходить рециклинг и попадать обратно в кровоток.
В соответствии с одним вариантом реализации предложен слитый белок, отличающийся тем, что партнер для слияния (ii) представляет собой Fc-фрагмент из IgG человека (иммуноглобулин G, также известный как иммуноглобулин γ), включая любые подклассы IgG человека. В соответствии с еще одним вариантом реализации настоящего изобретения предложен слитый белок, отличающийся тем, что партнер для слияния представляет собой Fc-фрагмент IgG1, IgG2 или IgG4. Предпочтительно, Fc-фрагмент IgG человека относится к подклассу IgG4 (SEQ ID. NO 13) или IgG2 (SEQ ID. NO 14).
IgG1, IgG2 и IgG4 часто предпочтительнее, чем IgG3, ввиду более длительного времени полужизни, составляющего приблизительно 3 недели. Специалисту будет понятно, что выбор изотипа IgG конкретного подкласса в качестве Fc-партнера для слияния зависит от требуемого продления времени полужизни и уровня цитотоксической активности итогового соединения. Терапевтические антитела, предназначенные для лечения рака или аутоиммунных заболеваний, принадлежат, по большей части, к подклассу IgG1 ввиду высокой аффинности к Fc-рецепторам и мощной способности стимулировать иммунные эффекторные функции. IgG2 и IgG4, с другой стороны, являются предпочтительными подклассами IgG для применения в качестве остова кандидатного терапевтического средства, когда требуется отсутствие иммунных эффекторных функций, поскольку иммунные эффекторные функции могут быть причиной нежелательных явлений. Склонность Fc-фрагмента к активации иммунных эффекторных функций зависит от изотипа и подкласса Ig и варьирует для разных иммунных эффекторных функций. Помимо выбора Fc-фрагмента подходящего подкласса IgG последовательность аминокислот указанного подкласса IgG может быть модифицирована, например, путем направленного мутагенеза, для снижения способности Fc-фрагментов активировать иммунные эффекторные функции.
Могут быть выбраны Fc-фрагменты, которые образуют мономеры, или, точнее, которые сохраняют или имеют мономерную форму или могут быть модифицированы путем введения мутаций, которые позволяют или облегчают получение мономерной структуры. Такие мутации в настоящей заявке называются «мутациями, обеспечивающими образование мономеров» Примеры Fc-фрагментов, которые содержат мутации, обеспечивающие образование мономеров, представлены SEQ ID NO: 54 и 55. Специалист в данной области техники знает, как ввести такие мутации и выбрать мономерные Fc-мутанты.
Избегание функции активации иммунных эффекторов Fc-фрагментами
Например, в биофармацевтический слитый белок дулаглутид (Трулисити, Trulicity™), слитый белок с агонистом GLP-1 и Fc-фрагментом, используемый один раз в неделю при лечении типа 2 диабета, вводят хорошо охарактеризованные мутации F234A и L235A в шарнирную область Fc-фрагмента IgG4 для снижения его способности активировать иммунные эффекторные функции.
В соответствии с одним вариантом реализации настоящего изобретения Fc-партнер для слияния представляет собой Fc-фрагмент IgG4, отличающийся тем, что указанный IgG4 Fc-фрагмент модифицирован так, чтобы избежать иммунных эффекторных функций, например, он содержит вышеуказанные мутации F234A и L235A.
Fc-фрагменты, устойчивые к протеазе
Другой фактор, который может уменьшить и выход в процессе изготовления, и биологический период полужизни - это расщепление слитого белка эндопептидазой. Для снижения или элиминации риска протеолитического разложения в последовательности аминокислот Fc-фрагмента могут быть введены модификации, в частности, путем введения мутации в сайты, чувствительные к протеолитическому расщеплению. В заявке на патент ЕР ЕР2654780 В1 Fc-домен константной области IgG1 был модифицирован путем замены E233-L234-L235-G236 на P233-V234-A235 (делеция G236) (нумерация ЕС) для придания итоговому модифицированному Fc-содержащему белку устойчивости к протеолитическому разложению.
Включение модификации аминокислот, раскрытой в ЕР2654780 В1, в Fc-фрагмент IgG4, сопряженный с доменом III CCN5, было признано неспособным обеспечить достаточную устойчивость к эндопептидазам. Однако улучшение устойчивости к протеазам было достигнуто путем дополнительных модификаций подтипа IgG, используемого в качестве партнер для слияния в соответствии с настоящим изобретением.
Более конкретно, было обнаружено, что слитые белки, содержащие полную шарнирную область IgG2 и константные тяжелые цепи 2 и 3 IgG4, проявляют превосходную протеолитическую устойчивость.
В источнике: Mueller JP et al., Mol. Immunol. (1997), 34(6), pp.441-452, раскрыто применение химер IgG2/IG4 в антителах IgG. Другое биофармацевтическое моноклональное антитело, экулизумаб, который используют в лечении ночной пароксизмальной гемоглобинурии и атипичного гемолитического уремического синдрома, как было показано, может подходить для применения, например, чтобы устранить способность IgG4 активировать FcγR-зависимый иммунный эффекторный ответ. Кроме того, показано, что константные домены 2 и 3 IgG4 такого химерного Fc-фрагмента также устраняют способность IgG2 активировать комплемент-зависимые иммунные эффекторные функции. Информация на эту тему приведена в отчетах Rother et al., (2007), см. Nat. Biotechnol., 25(11), pp. 1256-1264 и Mueller JP et al., выше.
Также в источнике: Borrok et al. (2017), J. Pharm. Sci. 106; 1008-1017 описано введение модификаций в Fc-фрагмент для исследования его эффекта на иммунные эффекторные функции антител (мутации FQQ-YTE). В WO 2017158426A1 раскрыты модификации антител путем введения мутаций в Fc-фрагмент для продления времени полужизни антител. В частности, раскрыты модификации в одном или более положениях 311, 434, 428, 438 и 435 в Fc-области иммуноглобулина.
Кроме того, Kinder et al. J Biol Chem. 2013 Oct 25; 288(43):30843-54 сообщают, что мутации в нижнем шарнире IgGl (т.е. Е233Р, L234V, L235A, G236-, Eu-нумерация) приводили к получению устойчивых к протеазам антител IgG1.
В соответствии с одним вариантом реализации Fc-фрагмент слитого белка в соответствии с настоящим изобретением состоит из Fc-фрагмента подкласса IgG4, включающего следующие мутации: S228P, F234A, L235A, К447-, Eu-нумерация, см. SEQ ID NO:15.
В источнике: Jacobsen et al. J Biol Chem. 2017 Feb 3;292(5): 1865-1875 сообщается, что мутация Asn297 приводит к получению агликозилированного Fc-фрагмента, что в дальнейшем приводит к отсутствию эффекторных функций IgG. Jacobsen также обнаружил, что некоторые варианты (N297G) приводили к получению антител с лучшей стабильностью и пригодностью для разработки по сравнению с другими вариантами (N297Q или N297A). Также вводили дополнительные модификации (дисульфидные мостики), что приводило к лучшей стабильности, чем у исходного IgG1.
В соответствии с настоящим изобретением, когда партнер для слияния представляет собой Fc-фрагмент, он может быть агликозилирован, без стабилизирующего дисульфидного мостика или со стабилизирующим дисульфидным мостиком, как, например, в SEQ ID NO: 16.
Насколько известно авторам настоящего изобретения, Fc-фрагмент, состоящий из полной шарнирной области IgG2 и константных тяжелых цепей 2 и 3 IgG4, ранее не использовался для получения слитых белков путем соединения указанного Fc-фрагмента с эффекторным белком.
В соответствии с одним вариантом реализации партнер для слияния предложенного слитого белка представляет собой Fc-фрагмент IgG1, который агликозилирован и стабилизирован дисульфидным мостиком, и отличается тем, что в нижний шарнир введены следующие мутации: Е233Р, L234V, L235A, G236- (Eu-нумерация) (SEQ ID NO: 17).
В соответствии с одним вариантом реализации партнер для слияния слитого белка, содержащего домен гомологии к повторам TSP-1 белка семейства CCN, представляет собой Fc-фрагмент IgG4, и отличается тем, что в нижний шарнир введены следующие мутации: Е233Р, L234V, L235A, G236- (Eu-нумерация) наряду с мутациями S228P и К477- (SEQ ID NO: 18).
Согласно одному предпочтительному варианту реализации указанный Fc-фрагмент представляет собой химеру шарнирной области IgG2 (216 ERKCCVECPPCPAPPVA-GP 238, Eu-нумерация) и любого другого из подклассов IgG. Наиболее предпочтительно Fc-фрагмент представляет собой химеру шарнирной области IgG2 и константных доменов тяжелой цепи 2 и 3 IgG4 с делецией карбоксиконцевого К477 (Eu-нумерация), представленную в SEQ ID. NO: 19. Указанный вариант реализации настоящего изобретения, как было показано, имеет улучшенные характеристики устойчивости к протеазам (см. Пример 6).
Согласно одному варианту реализации партнер для слияния мономерного слитого белка согласно настоящему изобретению представляет собой Fc-фрагмент IgGl, стабилизированный дисульфидным мостиком (R292C, V302C), агликозилированный (N297G) и содержащий мутации, обеспечивающие образование мономеров (C220Q, C226Q, C229Q, T366R, L368H, Р395К, К409Т, M428L), Eu-нумерация), как представлено в SEQ ID NO: 54.
Согласно дополнительному варианту реализации партнер для слияния мономерного слитого белка согласно настоящему изобретению представляет собой Fc-фрагмент, представляющий собой химеру шарнирной области IgG2 и константных доменов тяжелой цепи 2 и 3 IgG4 с делецией карбоксиконцевого К477- и с мутациями, обеспечивающими образование мономеров (C219Q, C220Q, C226Q, C229Q, L351F, T366R, Р395К, F405R, Y407E), и продлевающими время полужизни мутациями (M252Y, S254T, Т256Е) (Eu-нумерация), как представлено в SEQ ID NO: 55.
Хотя в примере слитого белка в соответствии с настоящим изобретением использован Fc-фрагмент, состоящий из полной шарнирной области IgG2, константных тяжелых цепей 2 и 3 IgG4, и домена III представителя семейства белков CCN, считается, что благоприятная устойчивость к протеазам также достигается, когда такой химерный Fc-фрагмент сопряжен с другими эффекторными молекулами, например, такими как VEGFR, FGF- 21 или GLP1. Эффекторная молекула является частью слитого с Fc белка, которая обеспечивает требуемые фармакодинамические свойства, тогда как Fc-фрагмент вносит вклад в фармакокинетические свойства.
Сывороточный альбумин в качестве партнера для слияния
Альтернативная стратегия для продления времени полужизни пептидов и белков заключается в использовании сывороточного альбумина (СА) в качестве партнера для слияния. IgG и СА имеют продолжительное время полужизни, равное приблизительно 19 дней, по сравнению с несколькими днями или менее для большинства других циркулирующих белков. СА также имеет аффинность к неонатальному рецептору Fc (FcRn) и избегает внутриклеточного разложения (см. Andersen et al. (2014), J Biol Chem, 289(19); pp. 13492- 13502).
Согласно одному варианту реализации настоящего изобретения предложен слитый белок согласно описанию выше, отличающийся тем, что партнером для слияния является сывороточный альбумин, предпочтительно сывороточный альбумин человека.
Согласно одному варианту реализации предложен мономерный слитый белок согласно описанию выше, отличающийся тем, что указанный слитый белок содержит аминокислоты 25 606 сывороточного альбумина человека, как представлено в SEQ ID NO: 101.
Согласно дополнительному варианту реализации настоящего изобретения указанный альбумин, например, сывороточный альбумин человека, модифицированный, например, для увеличения или уменьшения времени полужизни за счет изменения его аффинности к FcRn, с зависимостью или без зависимости от рН, что приводит к увеличению или уменьшению периода полужизни.
Трансферрин как партнер для слияния
Также альтернативной стратегией продления времени полужизни пептидов и белков является применение трансферрина в качестве партнера для слияния с использованием естественного длительного времени полужизни трансферрина (Strohl W. FJioDrugs. 2015; 29(4): 215 239). Трансферрин может быть использован в гликозилированной или негликозилированной форме.
Согласно одному варианту реализации настоящего изобретения предложен слитый белок согласно описанию выше, отличающийся тем, что партнером для слияния является трансферрин, предпочтительно трансферрин человека.
Согласно одному варианту реализации предложен мономерный слитый белок согласно описанию выше, отличающийся тем, что указанный слитый белок содержит аминокислоты 20-698 трансферрина человека, как представлено в SEQ ID NO: 53.
Линкер
Согласно другому варианту реализации слитые белки в соответствии с настоящим изобретением могут необязательно содержать пептидный линкер между партнером для слияния и эффекторной молекулой, т.е. линкер слит на N- или С-конце с доменом гомологии к повторам TSP-1 белка CCN (белок/полипептид с доменом TSP-1).
Может применяться любой пептидный линкер (при условии, что он не является последовательностью белка CCN), многие из которых известны и описаны в данной области техники. Линкер может представлять собой последовательность гибкого линкера (которая может включать повторы мотива последовательности гибкого линкера). Типичные линкеры, известные в данной области техники, богаты небольшими неполярными остатками (например, глицина) или полярными остатками (например, серина или треонина), и обычно состоят из отрезков остатков глицина и серина (GS), или остатков других аминокислот, таких как аланин, лизин и/или глутамат (А, K, и/или Е), или, фактически, любых аминокислот. Часто используемым линкером является линкер (GGGGS) (SEQ ID NO: 121), который может быть предоставлен в виде повторяющихся единиц линкера (как (GGGGS) n, где количество копий n может быть скорректировано, например от 1-10, 1-6, 1-4 и т.п.). Длина линкера может составлять 1-50, 1-45, 1-40, 1-30, 1-25, 1-20, 1-15, 1-12, 1-10, например, 1-8, 1-6, 1-5, или 1-4 аминокислоты. Различные отличающиеся линкеры описаны и использованы в примерах, представленных ниже, и любые из них могут применяться в любых слитых белках согласно настоящему изобретению.
Согласно некоторым вариантам реализации указанный линкер содержит не более 50 аминокислот.
Свойства пептидного линкера могут дополнительно улучшать сохранение эффекторных функций. Однако пептидные линкеры могут быть восприимчивы к расщеплению эндопептидазой и элиминации слитого белка. Пептидные линкеры с глицином, с остатками или без остатков серина часто используются, однако указанный дизайн не всегда дает слитые белки с требуемыми видами активности и устойчивости к эндопептидазе. В US20180273603, раскрывающем нейротрофин-связывающий белок-Fс-слитый белок, предлагается применение а-спиральных линкеров, предусматривающих повторы последовательности А (ЕАААК) A (SEQ ID NO: 14 в указанном источнике). Кроме того, в US2018/0127478 раскрыто применение аминокислотного линкера, состоящего из одного-трех повторов последовательности ЕАААK, в слитом белке с Fc.
В соответствии с настоящим изобретением линкер, состоящий из пептидной последовательности ЕАААK (SEQ ID NO: 21 в настоящей заявке), может также быть вставлен между доменом гомологии с TSP-1 и партнером для слияния (Fc-фрагментом). Более предпочтительно линкер состоит из повторяющейся последовательности аминокислот ЕАААK.
Если линкер включен в слитый белок согласно настоящему изобретению, указанный линкер находится между партнером для слияния и эффекторной молекулой, т.е. доменом III белка CCN. Линкер может быть введен на С-конце или N-конце домена III белка CCN.
Кроме того, спиральный линкер был устойчив к расщеплению эндопептидазой после экспрессии рекомбинантного белка в суспензии клеток СНО. Это важно как для целей изготовления, так и для in vivo эффективности. Кроме того, показано, что включение α-спирального линкера между Fc-фрагментом и эффекторным доменом в слитом с Fc белке снижает склонность к агрегации указанного слитого с Fc белка.
Хотя указанные результаты обнаружены для слитого белка, содержащего домен III белка CCN в качестве эффекторного белка, считается, что благоприятная пониженная склонность к агрегации и эффекты устойчивости к протеазам также достигаются, если объединить другие эффекторные молекулы с Fc-фрагментом α-спиральным линкером в соответствии с настоящим изобретением.
Согласно настоящему изобретению, соответственно, предложен слитый с Fc белок, содержащий Fc-фрагмент, который содержит последовательность пептидного линкера формулы АK1-АK2-(ЕАААK)n-АK3-АK4-АK5, где n≥4, между Fc-фрагментом и эффекторной молекулой, и где аминокислоты АK1, АK2, АK3, АK4, АK5 независимым образом отсутствуют или представляют собой аминокислоту. Линкер может быть размещен на N-конце или С-конце Fc-фрагмента. В соответствии с одним вариантом реализации n равен 8. Согласно другому варианту реализации АK1 представляет собой треонин (Т), АK1, АK2, АK3, АK4 и АK5 представляют собой Ala (А). В соответствии с одним вариантом реализации линкер для вышеуказанного Fc-слитого белка выбран из группы, состоящей из SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24 и SEQ ID NO: 25. В частности, было показано, что применение слитого белка в соответствии с настоящим изобретением, содержащего линкер, состоящий из (ЕАААK)-повтора, т.е. такой как (ЕАААK)n, где n равен 8, благоприятным образом приводит к меньшей агрегации.
Альтернативный линкер, который можно применять в соответствии с настоящим изобретением, представляет собой линкер с последовательностью аминокислот, представленной в SEQ ID NO: 20 (TEGRMD).
Согласно одному варианту реализации настоящее изобретение может, соответственно, предусматривать включение линкерного пептида между партнером для слияния и доменом III на CCN5 (например, SEQ ID NO: 1 - SEQ ID NO: 12, 37 или 38). Неограничивающие примеры слитых белков, содержащих линкер из SEQ ID NO: 20, представлены в SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30, соответственно.
В том случае, когда согласно изобретению используют вариант реализации домена III CCN5, генетически слитый на N-конце с пептидным линкером (как в SEQ ID NO 20) и Fc-фрагментом IgG подтипа IgG4, включающим следующие мутации (S228P, F234A, L235A, K447-, Eu-нумерация) (как в SEQ ID NO 15), полная последовательность соответствует SEQ ID NO: 28, также обозначаемой CCN5(dIII)-Fcv2.
В том случае, когда согласно изобретению используют вариант реализации домена III CCN5, генетически слитый на N-конце с пептидным линкером (как в SEQ ID NO: 20) и Fc-фрагментом IgG подтипа IgG4, включающим следующие мутации (S228P, Е233Р, F234V, L235A, G236-, K447-, Eu-нумерация), представленным в SEQ ID NO: 18, итоговая последовательность соответствует SEQ ID NO: 29, также обозначаемой CCN5(dIII)-Fcv2.1.
В том случае, когда согласно изобретению используют вариант реализации домена III CCN5, генетически слитый на N-конце с пептидным линкером (как в SEQ ID NO: 20) и химерным Fc-фрагментом подтипа IgG IgG2/4, представленным в SEQ ID NO: 19, итоговая последовательность соответствует SEQ ID NO: 30, также обозначаемой CCN5(dIII)-Fcv2.3.
В том случае, когда согласно изобретению используют вариант реализации домена III CCN5 (как в SEQ ID NO: 1), генетически слитый на N-конце с пептидным линкером (как в SEQ ID NO: 25) и химерным Fc-фрагментом подтипа IgG IgG2/4, представленным в SEQ ID NO: 19, итоговая последовательность соответствует SEQ ID NO: 31, также обозначаемой CCN5(dIII)-HLn8-Fcv2.3.
В соответствии с предпочтительным вариантом реализации настоящего изобретения предложен слитый белок в соответствии с настоящим изобретением, содержащий:
1) точечную мутацию в домене III белка семейства CCN, в частности, CCN5 (см. SEQ ID NO: 7, которая приводит к пониженной протеолитической чувствительности указанного домена III;
2) сконструированную химеру Fc-фрагмента IgG4 человека и IgG2 человека (SEQ ID NO: 19, которая уменьшает протеолитическую чувствительность относительно ранее описанных остовов на основе Fc-фрагмента, используемых в слитых с Fc белках; и
3) содержащий оптимизированную композицию пептидного линкера (см. SEQ ID NO: 21-25), который уменьшает протеолитическую чувствительность, усиливает биологическую активность слитого белка и уменьшает склонность к агрегации слитого белка.
Согласно некоторым вариантам реализации пептидный линкер между последовательностью аминокислот по (i) и мономерным партнером для слияния имеет последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 20-25 или 39, или последовательность аминокислот, на 80% идентичную указанной.
Альтернативные линкерные последовательности, которые могут применяться в соответствии с настоящим изобретением, представлены в SEQ ID NO: 57, 63, 65, 67 и 121.
Рекомбинантная экспрессия
Рекомбинантные белки и слитые белки в соответствии с настоящим изобретением могут быть получены путем культивирования клетки-хозяина, позволяющей экспрессию последовательностей нуклеотидов, кодирующих указанные белки. Специалист хорошо знаком с различными доступными биотехнологическими методиками, обеспечивающими экспрессию выделенных последовательностей нуклеиновых кислот для получения рекомбинантных белков путем гетерологичной экспрессии в различных системах клеток-хозяев с применением общедоступных методик генетического конструирования и систем экспрессии рекомбинантной ДНК, см. например, источники: «Recombinant Gene Expression Protocols, Methods in Molecular Biology, 1997, Ed. Rocky S Tuan, Human Press (ISSN 1064-3745) или Sambrook et al., Molecular Cloning: A laboratory Manual (third edition), 2001, CSHL Press, (ISBN 978-087969577-4). Например, последовательности нуклеиновых кислот, кодирующие рекомбинантные белки в соответствии с настоящим изобретением, могут быть инсертированы в подходящие экспрессионные векторы, содержащие все необходимые транскрипционные и трансляционные регуляторные последовательности, специально адаптированные для направления экспрессии кодирующей последовательности нуклеиновой кислоты требуемого белка в подходящей клетке-хозяине. Подходящие экспрессионные векторы представляют собой, например, плазмиды, космиды, вирусы или искусственные дрожжевые хромосомы (YAC).
Последовательности ДНК, кодирующие рекомбинантные белки согласно настоящему изобретению, могут быть синтезированы с применением способов, хорошо известных специалисту, или синтезированы коммерческими поставщиками, хорошо известными специалисту, например, Genscript, Thermo Fisher Scientific и т.п.
В соответствии с одним вариантом реализации указанного аспекта предложена молекула ДНК, содержащая последовательность нуклеиновой кислоты, представленную в SEQ ID NO: 86, 87, 90, 91, 99, 100, 104, 105, 108, 109, 112 или 113, или последовательность, по меньшей мере на 80% идентичную любой вышеупомянутой последовательности. Также предложены экспрессионные векторы, содержащие такие молекулы ДНК. В соответствии с другим вариантом реализации указанного аспекта также предложены клетки-хозяева, содержащие такие векторы.
Последовательности ДНК для экспрессии и применения для получения рекомбинантных белков могут быть инсертированы в векторы, общеизвестные как входящие векторы, с применением системы клонирования Gateway (Esposito et al, 2009, "Gateway Cloning for Protein Expression", Methods in Molecular Biology, 498, pp.31-54). Гены, клонированные во входящий вектор, могут легко быть введены в различные экспрессионные векторы путем рекомбинации. Например, синтезированная последовательность, кодирующая рекомбинантный белок или слитый белок в соответствии с настоящим изобретением, может быть рекомбинирована путем рекомбиназного клонирования BP Gateway с получением входящего вектора, который может быть использован для размножения плазмид в подходящей клетке-хозяине, например, клетках Е. coli. Согласно предпочтительному варианту реализации используют клетки E.coli, мутированные таким образом, чтобы они позволяли эффективное размножение плазмид, такие как, например, клетки One Shot Top 10™.
В соответствии с одним вариантом реализации настоящего изобретения получают экспрессионный вектор, содержащий последовательность ДНК, кодирующую рекомбинантный белок или слитый белок в соответствии с настоящим изобретением, функционально связанный с промотором. Специалисту будет понятно, что «промотор» в настоящей заявке относится к области ДНК, расположенной выше (в направлении 5') относительно кодирующей последовательности ДНК, которая контролирует и инициирует транскрипцию конкретного гена. Промотор контролирует распознавание и связывание РНК-полимеразы и других белков для инициации транскрипции. «Функционально связанный» относится к функциональной связи между промотором и второй последовательностью, когда последовательность промотора инициирует и опосредует транскрипцию последовательности ДНК, соответствующих второй последовательности. В общем случае «функционально связанный» означает, что связанные последовательности нуклеиновых кислот расположены непрерывно.
Входящий вектор, а также экспрессионный вектор, такой как полученный из принимающего вектора, упоминаемого ниже, может быть выделен с применением стандартных методик выделения плазмид, хорошо известных специалисту, например, с применением набора QIAprep™ Spin Miniprep от Qiagen™ или набора QIAGEN™ Plasmid Plus Maxi Kit.
Если используют входящий вектор, содержащий последовательность ДНК, кодирующую рекомбинантный белок или слитый белок в соответствии с настоящим изобретением, указанный входящий вектор может быть дополнительно рекомбинирован с принимающим вектором с применением рекомбиназы LR Gateway для получения экспрессионного вектора. Экспрессионный вектор может затем быть использован для экспрессии кодирующей белок последовательности ДНК в подходящей клетке-хозяине. Неограничивающий пример применимого принимающего вектора представлен, например, pUCOE-DHFR-DEST согласно описанию в источнике:  et al., J. Biol. Chem, 293:46, pp.17953-17970.
et al., J. Biol. Chem, 293:46, pp.17953-17970.
Также итоговый экспрессионный вектор может быть верифицирован с использованием стандартного анализа с расщеплением рестрикционными ферментами и гель-электрофореза
ДНК.
В соответствии с одним аспектом настоящего изобретения предложен экспрессионный вектор, содержащий последовательность нуклеиновой кислоты, кодирующую рекомбинантный белок формулы (I). В соответствии с еще одним аспектом настоящего изобретения предложен экспрессионный вектор, содержащий последовательность нуклеиновой кислоты, кодирующую белок, содержащий последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7 и SEQ ID NO: 8-12, 37, 38, 84, 85, 88, 89, 97, 98, 102, 103, 106, 107, 110 и 111; и их фрагментов или вариантов по меньшей мере с 50% идентичностью последовательностей последовательностям аминокислот SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, и SEQ ID NO: 7, SEQ ID NO:8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38; и SEQ ID NO: 84,85, 88, 89, 97,98, 102, 103, 106, 107, 110 и 111.
Согласно другому аспекту предложен экспрессионный вектор, кодирующий рекомбинантный белок, содержащий последовательность аминокислот по меньшей мере с 60%, 70%, 80%, 90% или 95% идентичностью последовательностей последовательности аминокислот, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 37 и SEQ ID NO: 38, и SEQ ID NO: 84, 85, 88, 89, 97, 98, 102, 103, 106, 107, ПО и 111.
В соответствии с вариантом реализации настоящего изобретения предложены экспрессионные векторы, кодирующие слитый белок в соответствии с настоящим изобретением.
Специалисту хорошо известно о вырожденности генетического кода, и предпочтительном использовании конкретных кодонов в различных организмах. Соответственно, в зависимости от выбора клетки-хозяина, последовательность нуклеиновой кислоты, кодирующая рекомбинантный белок и слитые белки согласно настоящему изобретению, может быть адаптирована так, чтобы содержать предпочтительные для клетки-хозяина кодоны. Соответственно, аминокислоты белков согласно настоящему изобретению может кодировать любая комбинация кодонов из представленных в таблице ниже:
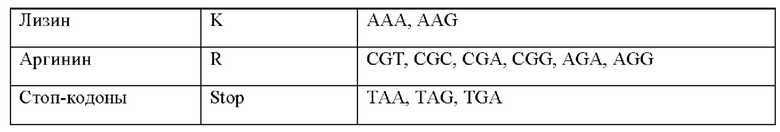
Предпочтительно указанные кодоны дополнительно оптимизированы для высоких уровней экспрессии в соответствии с выбранной клеткой-хозяином.
Для экспрессии белков с помощью технологии рекомбинантной ДНК, в дополнение к конкретному варианту реализации настоящего изобретения к N-концу последовательности белка предпочтительно добавляют последовательность ДНК, кодирующую сигнальный пептид. Сигнальный пептид может служить для направленной локализации слитого белка во время и/или после синтеза в клетке-хозяине. Он может, соответственно, представлять собой последовательность, направляющую секрецию слитого белка. Применение таких последовательностей сигнальных пептидов хорошо известно в данной области техники. Сигнальный пептид может быть представлен любой формой, например, может представлять собой сигнальный пептид IgGk-цепи, или может представлять собой сигнальный пептид из сывороточного альбумина человека (SEQ ID. NO 32).
В том случае, когда сигнальным пептидом из сывороточного альбумина человека (SEQ ID №32) дополнен N-конец SEQ ID NO: 28, последовательность белка для экспрессии может соответствовать SEQ ID NO: 33 или SEQ ID NO: 85, 89, 98, 103, 107 или 111.
Кроме того, для экспрессии белка с помощью технологии рекомбинантной ДНК, в соответствии с одним конкретным вариантом реализации настоящего изобретения, белком, имеющим последовательность аминокислот, представленную в SEQ ID NO: 33, может применяться последовательность нуклеотидов, представленная в SEQ ID NO: 34 или SEQ ID NO: 86, 90, 99, 104, 108 или 112, где 3'-конец кодирующей последовательности дополнен трансляционным стоп-кодоном.
В том случае, когда вариантом реализации изобретения является последовательность нуклеотидов из SEQ ID. NO: 34, указанная последовательность нуклеотидов предпочтительно дополнена непосредственно на 5'-конце кодирующей последовательности последовательностью KOZAK, например, GCCACC, как в SEQ ID NO: 35 или SEQ ID NO: 86, 90, 99, 104, 108 или 112. Последовательность ДНК может дополнительно быть фланкирована ДНК-элементами, позволяющими субклонирование, например, такими как рекомбиназные сайты attB Gateway. Однако для получения последовательности ДНК и облегчения субклонирования в экспрессионный вектор могут быть использована любая стратегия клонирования или синтеза. В том случае, когда последовательность ДНК включает рекомбиназные сайты Gateway, позволяющие субклонирование, последовательность нуклеотидов может соответствовать представленной в SEQ ID NO 36 или SEQ ID NO: 87, 91, 100, 105, 109 или 113.
Полученный экспрессионный вектор, включающий последовательность нуклеиновой кислоты, кодирующую рекомбинантный белок или слитый белок согласно настоящему изобретению, может быть введен в подходящие клетки-хозяева для продуцирования требуемого белка. Могут применяться различные коммерчески доступные или самостоятельно полученные клетки-хозяева. Например, экспрессионный вектор может быть перенесен в эукариотические клетки-хозяева, такие как клетки СНО, например, адаптированные к суспензионной культуре клетки СНО DG44 DHFR (дигидрофолатредуктаза-/-). Трансфекция клеток-хозяев экспрессионным вектором может быть выполнена с применением способов, хорошо известных специалисту, например, с применением электропорации.
При культивировании клеток-хозяев в подходящей культуральной среде будут продуцироваться рекомбинантные белки или слитые белки в соответствии с настоящим изобретением, кодируемые экспрессионным вектором в клетка-хозяине, и итоговый белок может быть собран и очищенный с применением способов, хорошо известных специалисту.
Экспрессионный вектор может включать сигнальные последовательности, общеизвестные как «сигнальный пептид», для секреции экспрессированного белка или слитого белка в культуральную среду.
Для выделения и очищения секретированного рекомбинантного белка из клеточной культуральной среды обычно проводят один или более этапов предварительной обработки или очищения, в первую очередь для удаления больших частиц и биомассы. Неограничивающие примеры применимых этапов предварительной обработки представлены, например, обратным осмосом, центрифугированием, методами фильтрации и диафильтрации, или их комбинацией. Затем полученный белок обычно очищают с помощью одного или более из различных хроматографических методов, хорошо известных специалисту, например, аффинной хроматографии, ионообменной хроматографии, хроматографии со смешанным режимом, хроматографии гидрофобного взаимодействия, эксклюзионной хроматографии или других хроматографических техник, или их комбинации.
Например, рекомбинантный белок или слитый белок, экспрессируемый подходящей клеткой-хозяином, может быть очищен с применением метода аффинной хроматографии, например, с применением сред MabSelect™ SuRe™, например, на колонке на 5 мл HiTrap MabSelect™ SuRe™, установленной в систему для высокопроизводительной жидкостной хроматографии, например, систему BioRad NGC Discover™ 10 Pro, оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. После загрузки образца, содержащего белок для очищения, колонку обычно промывают один или более раз одним или более применимыми промывочными буферами, после чего белок элюируют с использованием подходящего элюирующего буфера. Полученный белок может быть дополнительно очищен одним или более из перечисленных выше хроматографических способов.
Следует понимать, что различные модификации могут быть введены в последовательности нуклеиновых кислот, кодирующие рекомбинантные белки согласно настоящему изобретению, с использованием методик, хорошо известных специалисту, например, для облегчения экспрессии. Путем применения направленного мутагенеза можно ввести модификацию для адаптации кодирующей последовательности к нужному хозяину, используемому для экспрессии последовательности и, соответственно, продуцирования рекомбинантного белка. Специалисту хорошо известно о существовании специфичных для хозяев кодонов, и о том, что адаптация гетерологичной последовательности нуклеиновой кислоты с включением специфичных для хозяев кодонов повышает эффективность экспрессии, как упоминалось выше. Могут также быть введены другие модификации, например, для облегчения выделения и очищения, т.е. путем добавления последовательности, кодирующей пептид или белок, подходящие для таких целей. Также к последовательностям нуклеиновых кислот согласно настоящему изобретению могут быть дополнительно присоединены последовательности нуклеиновых кислот, кодирующие сигнальный пептид, обеспечивающий секрецию требуемого рекомбинантного белка из клетки-хозяина.
Согласно настоящему изобретению также предложена клетка-хозяин, подходящая для продуцирования рекомбинантного белка или слитого белка в соответствии с настоящим изобретением. Могут применяться различные коммерчески доступные клетки-хозяева, конкретным образом адаптированные для продуцирования рекомбинантных белков, как прокариотические клетки-хозяева, так и эукариотические клетки-хозяева. Неограничивающими примерами подходящих клеток-хозяев являются, например, клетки СНО, клетки НЕК293, клетки Pichia pastoris, клетки NS0 или клетки Е. coli.
Наконец, настоящее изобретение также относится к домену гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN, и к слитому белку, содержащему указанный домен гомологии к повторам TSP-1 для применения в качестве медикамента для лечения или предотвращения расстройств путем ингибирования или противодействия клеточной сигнализации и физиологическим функциям клетки, приписываемым белкам семейства CCN.
Согласно одному аспекту настоящего изобретения предложен белок, например, слитый белок согласно определению в настоящей заявке, для применения в терапии.
Согласно некоторым аспектам указанный белок, например, слитый белок, может быть предназначен для применения при лечении или предотвращении фиброза, или любого состояния, при котором наблюдается фиброз (т.е. любого фибротического состояния или расстройства). Фиброз может влиять на любую ткань или орган, в том числе, например, на легкие, глаз, сердце, скелетные мышцы, брюшину, почки, печень, поджелудочную железу, желчные протоки, кожу, кровеносные сосуды или более глобальные системы. В частности, состояние, при котором проявляется фиброз, может быть выбрано из фиброза легких, который может быть любой этиологии, в том числе идиопатического фиброза легких, бронхолегочной дисплазии, фиброза сетчатки, диабетической ретинопатии, возрастной макулярной дегенерации, отслоения сетчатки, индуцированной кислородом ретинопатии, глаукомы, сердечного фиброза, фиброза трансплантата после трансплантации, ассоциированного с кардиомиопатией фиброза, мышечного фиброза, мышечной дистрофии Дюшенна, перитонеального фиброза, диабетической нефропатии, хронической болезни почек (фиброза почек), острого повреждения почек, тубулоинтерстициального фиброза, хронической нефропатии аллотрансплантата, фиброза печени, неалкогольного стеатогепатита, жировой болезни печени, хронического панкреатита, билиарного фиброза, келоидов, рубцов, системного склероза, атеросклероза, эпидурального фиброза.
В контексте сердечного фиброза состояния, подлежащие лечению или предотвращению, могут включать гипертрофию сердца и сердечную недостаточность с сохраненной фракцией выброса или без нее.
Согласно дополнительному аспекту настоящего изобретения предложен белок, например, слитый белок согласно определению в настоящей заявке, для применения в лечении воспалительного или аутоиммунного заболевания. Согласно некоторым вариантам реализации указанное воспалительное заболевание выбрано из ревматоидного артрита, амиотрофического бокового склероза (ALS), воспалительного заболевания кишечника, язвенного колита, болезни Крона.
Согласно дополнительному аспекту настоящего изобретения предложен белок, например, слитый белок согласно определению в настоящей заявке, для применения при лечении рака. В этом отношении известно, что 4-доменные белки CCN могут как вызывать онкогенные ответы в выделенных раковых клетках, так и способствовать метастазированию, хеморезистентности и устойчивости к иммунотерапии, воздействуя прямо на раковые клетки или на строму опухоли. Активность белков согласно настоящему изобретению, относящаяся к ингибированию эффекта или активности 4-доменного белка CCN, соответственно, является обоснованием для их применения в лечении рака. Рак может представлять собой любое злокачественное или предзлокачественное неопластическое состояние. Это может быть рак любой ткани или любого органа. В одном из вариантов реализации рак может проявляться в виде солидных опухолей. Согласно другому варианту реализации рак может представлять собой рак системы кроветворения или находиться в системе кроветворения. Он может представлять собой первичный рак или вторичный рак, или метастазы. Рак может, соответственно, представлять собой рак поджелудочной железы, молочной железы, предстательной железы, шейки матки, яичников, печени, мочевого пузыря, мозга, крови, костей, кожи, легких или желудка. Согласно некоторым вариантам реализации рак выбран из рака поджелудочной железы, аденокарциномы протоков поджелудочной железы, рака молочной железы, рака предстательной железы, рака шейки матки, рака яичника, рака печени, гепатоцеллюлярной карциномы, уротелиального рака мочевого пузыря, рака головного мозга, глиобластомы, острого лимфобластного лейкоза, остеосаркомы, меланомы, мезотелиомы, рака желудка, плоскоклеточной карциномы полости рта, рака пищевода, рака ободочной и прямой кишки, рака легкого.
Согласно дополнительному аспекту настоящего изобретения предложен белок, например, слитый белок согласно определению в настоящей заявке, для применения при лечении метаболического заболевания. Указанное метаболическое заболевание может представлять собой или может быть ассоциировано с инсулинорезистентностью или нарушением толерантности к глюкозе. Согласно некоторым вариантам реализации указанное метаболическое заболевание выбрано из диабета 2 типа и метаболического синдрома.
Слитый белок согласно настоящему изобретению может также применяться согласно описанным выше способам лечения состояний. Аналогичным образом, слитый белок согласно настоящему изобретению может применяться в способах изготовления медикамента для применения в лечении состояний, описанных выше.
ПРИМЕРЫ
Пример 1
Экспрессия слитого белка в соответствии с настоящим изобретением
В этом примере описано получение слитого белка, содержащего аминокислоты 194-246 CCN5 (SEQ ID NO: 1), слитого на N-конце с пептидным линкером (SEQ ID NO: 20) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2) (т.е. слитого белка в соответствии с SEQ ID NO: 28). Указанный слитый белок также был дополнен N-концевой сигнальной последовательностью, происходящей из альбумина из SEQ ID NO: 32, и экспрессирован в клетках млекопитающих согласно описанию ниже.
Последовательность ДНК, представленную в последовательности SEQ ID NO: 36, синтезировал и верифицировал коммерческий поставщик. Синтезированная последовательность была рекомбинирована с pDonrZeo путем рекомбиназного клонирования BP Gateway для создания входящего вектора. После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Тор10™), выделяли входящий вектор с помощью стандартных методик выделения плазмид с применением набора QIAprep™ Spin Miniprep от Qiagen™. После выделения плазмиды входящий вектор верифицировали путем расщепления рестрикционными ферментами с последующим гель-электрофорезом ДНК в соответствии со стандартными методиками, хорошо известными специалисту.
Затем входящий вектор, содержащий последовательность SEQ ID NO: 35, рекомбинировали с принимающим вектором с применением рекомбиназы LR Gateway. Использовали принимающий вектор pUCOE-DHFR-DEST согласно описанию Kaasb0ll и соавторов, 2018, см. выше.
После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top 10™), экспрессионный вектор выделяли с помощью стандартных методик выделения плазмид с применением набора QIAGEN™ Plasmid Plus Maxi Kit. Итоговый экспрессионный вектор верифицировали с помощью стандартного расщепления рестрикционными ферментами и гель-электрофореза ДНК в соответствии со стандартными методиками, хорошо известными специалисту. Итоговый экспрессионный вектор затем переносили в адаптированные к суспензионной культуре клетки СНО ExpiCHO в соответствии с протоколом «Мах Titer» от изготовителя набора для трансфекции СНО Expifectamine™ (Gibco кат. №: А29129) и кратким описанием  и соавторов, 2018, выше. Клетки осаждали через 6 дней после трансфекции центрифугированием при 4750g в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 1 мМ и 0,5М ЭДТК до концентрации 2 мМ. Затем добавляли 96% этанол до конечной концентрации приблизительно 3%. Перед хроматографическим очищением добавляли 1М TrisHCl с рН 7,4 до конечной концентрации 25 мМ.
и соавторов, 2018, выше. Клетки осаждали через 6 дней после трансфекции центрифугированием при 4750g в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 1 мМ и 0,5М ЭДТК до концентрации 2 мМ. Затем добавляли 96% этанол до конечной концентрации приблизительно 3%. Перед хроматографическим очищением добавляли 1М TrisHCl с рН 7,4 до конечной концентрации 25 мМ.
Этап захвата при очищении выполняли путем аффинной хроматографии на хроматографической среде с белком А. В этом эксперименте использовали среду rProtein А FF (GE Healthcare). Колонку HiTrap ™ rProtein A FF на 5 мл (GE Healthcare) использовали для очищения экспрессированного рекомбинантного белка из 60 мл клеточной культуральной среды, собранной и дополненной согласно описанию, см. выше. Колонку HiTrap ™ rProtein А FF устанавливали в систему скоростной высокопроизводительной жидкостной хроматографии (систему BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм, и уравновешивали буфером, содержащим 25 мМ TrisHCl, рН 7,4, 25 мМ NaCl и 3% этанола. Собранную клеточную культуральную среду, содержащую рекомбинантный белок, загружали с помощью пробоотборного насоса со скоростью 2,5 мл/мин, с последующим промыванием промывочным буфером в количестве 6 объемов колонки (25 мМ TrisHCl рН 7,4, 25 мМ NaCl и 3% этанола)) до элюирования 0,1М цитратом Na, рН 3,0, в 3% этаноле. Элюат с показателем поглощения УФ на 280 нм, превышающим 100 мЕОП, собирали фракциями по 3 мл в пробирки с низким связыванием белков, предварительно заполненные 1 мл 1М TrisHCl, рН 9,0. Фракцию, содержащую пик поглощения УФ, концентрировали до 500 мкл с использованием концентратора Vivaspin® 20 мл, с номинальным отсечением по молекулярной массе 30 кДа. После концентрирования образец загружали в контур для загрузки образцов системы скоростной высокопроизводительной жидкостной хроматографии (FPLC) (система BioRad NGC Discover™ 10 Pro). Система FPLC-хроматографии была оснащена колонкой Superdex® 200 Increase 10/300 GL (GE Healthcare), которую уравновешивали 50 мМ NaCl, 20 мМ HEPES, рН 7,0. Вводили образец и колонку перфузировали буфером для предварительного уравновешивания (50 мМ NaCl, 20 мМ HEPES, рН 7,0) со скоростью потока 0,25 мл/мин. Было обнаружено, что основной пик поглощения УФ на 280 нм содержит очищенный рекомбинантный белок (CCN5(dIII)-Fcv2, SEQ ID NO: 28). Образцы собранных фракций объемом 10 мкл подвергали ДСН-ПААГ, используя готовые гели Mini-PROTEAN® TGX Stain-Free™ и выделенные рекомбинантные белки визуализировали с использованием системы визуализации ChemiDoc™ (BioRad).
Специалистам хорошо известно, что рекомбинантные белки могут быть получены в различных экспрессионных системах и очищены с помощью различных хроматографических способов с аналогичными результатами.
Пример 2
Последовательность ДНК, кодирующую слитый белок, содержащий аминокислоты 194 246 CCN5 (SEQ ID NO: 1), слитый на N-конце с пептидным линкером (SEQ ID NO: 20) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2) экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 28.
Полученный белок тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 в AKT) в клетках рака легкого человека А549 (Фиг. 1А). Обработанные для культивирования тканей 96-луночные стерильные полистироловые планшеты Corning Incorporated Costar® покрывали фибронектином (Sigma, кат. № F1141, разведен до 10 мкг/мл забуференным фосфатом солевым раствором по Дульбекко BioWhittaker® (Lonza, кат. №17-512F, в дальнейшем называемый ФСБ)). Раствор для покрытия, содержащий фибронектин, распределяли по лункам в объеме 100 мкл/лунку, инкубировали в течение 1 часа при комнатной температуре, затем декантировали раствор для покрытия, распределяли в покрытые фибронектином лунки по 100 мкл ФСБ, и также декантировали. Клетки А549, субкультивированные для поддержания плотности с максимальной конфлюентностью 80%, разделяли путем обработки ферментом (Аккутаза®, кат. № L0950-100 от Biowest®), разведенным в модифицированной по Дульбекко среде Игла с высоким содержанием глюкозы (Gibco кат. №: 41965-039), добавляли 10% инактивированную нагреванием фетальную бычью сыворотку (ФБС) (флаконы на 500 мл с ФБС (кат. №16000-044 от Gibco), уравновешивали до комнатной температуры, инкубировали в воде с температурой 60°С при перемешивании в течение 30 минут) и с 50 мкг/мл генсумицина (Sanofi)) до концентрации 110000 клеток/мл, и по 100 мкл раствора клеток распределяли в покрытые фибронектином лунки. Все инкубации клеток проводили в инкубаторах для клеточных культур при поддержании температуры 37°С, во влажной атмосфере с комнатным воздухом и 5% СО2. После инкубации в течение ночи клетки А549 двукратно промывали ФСБ и 90 мкл модифицированной по Дульбекко среды Игла с высоким содержанием глюкозы (DMEM, Gibco кат. №: 41965-039), и в лунки распределяли по 50 мкг/мл генсумицина (Sanofi) без ФБС. Через 18 часов инкубации в среде без ФБС клетки стимулировали 10 мкл раствора рассматриваемого рекомбинантного белка. После стимуляции в течение 60 среду декантировали и клетки собирали путем добавления 50 мкл буфера для лизиса с блокирующим реагентом, в соответствии с набором для фосфо-AKT от Cisbio (Ser473) (Cisbio Inc, кат. №: 64AKSPEG). После добавления буфера для лизиса с блокирующим реагентом 96-луночный планшет инкубируют в течение 60 минут на планшетном шейкере PST-60HL Plus (ThermoFisher) при 500 об/мин. После перемешивания лизированные образцы растирали, после чего переносили по 16 мкл из каждой лунки в белые 96-луночные малообъемные HTRF-планшеты (Cisbio Inc., кат. №: 66PL96025). Для анализа количества фосфорилированной AKT (Ser473) в каждую лунку добавляли 4 мкл смеси меченых антител (50/50 об/об смесь антител, AT к фосфо-AKT d2 и меченое криптатом AT к фосфо-AKT от Cisbio bic, кат. №: 64AKSPEG) (в отрицательные контрольные лунки добавляли только антитело с криптатом), планшеты запечатывали клейкой полимерной пленкой и инкубировали при 4°С в течение ночи, после чего считывали на планшет-ридере PolarStar Omega (BMG Labtech, Германия), оснащенном регистратором TR-FRET и эмиссионным фильтром на 337 нм и фильтрами возбуждения на 615 нм и 665 нм. Соотношение между зарегистрированными показателями возбуждения на 665 нм и 615 нм корректировали по холостым пробам, и значения для рекомбинантного белка из стимулированных лунок выражали через процент относительно стимулированных носителем лунок.
Пример 3
Последовательность ДНК, кодирующую слитый белок, содержащий аминокислоты 194-246 CCN5 (SEQ ID. NO: 1), слитый на N-конце с пептидным линкером (SEQ ID NO: 20) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2), экспрессировали для получения рекомбинантного белка в соответствии с SEQ ID NO: 28.
Полученный белок тестировали на способность ингибировать профибротическую стимулированную ТФР-β транскрипцию (со связывающих SMAD2/3 цис-элементов) в фибробластах легких человека IMR90 (Фиг. 1D). Анализ проводили в техническом отношении согласно описанию Kaasbell et al. (2018) выше, за исключением использования 2500 фибробластов легких IMR90 /лунку вместо клеток Rat2. Для стимуляции использовали белки, указанные на Фиг. 1D. Клетки IMR90 перед применением субкультивировали согласно описанию для клеток А549, см. выше. Клетки IMR90 использовали до 20 пересева, т.е. до достижения репликативного старения.
Пример 4
Последовательность ДНК, кодирующую слитый белок, содержащий аминокислоты 194-246 CCN5 (SEQ ID. NO: 1), слитый на N-конце с пептидным линкером (SEQ ID NO: 20) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2), экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 28.
Полученный белок тестировали на способность ингибировать пролиферацию клеток линии фибробластов легких человека IMR90 (Фиг. 1В). Клетки IMR90 субкультивировали согласно описанию для клеток А549 перед применением, выше. Клетки IMR90 использовали до пересева 20, т.е. до достижения репликативного старения. Для экспериментов клетки IMR90 собирали согласно описанию для клеток А549, выше, промывали ФСБ, разводили DMEM с 1% ФБС и генсумицином согласно описанию для эксперимента 2, выше, и высевали в планшеты для измерения импеданса xCELLigence с плотностью 12000 кл./лунку. Через 2 часа клетки стимулировали 10 мкл раствора рассматриваемого рекомбинантного белка или ФБС и инкубировали в течение еще 72 часов, после чего собирали данные с использованием CellTiter-Glo® (Promega Inc.) согласно описанию в  и соавторов, (2018), выше.
и соавторов, (2018), выше.
Пример 5
Последовательность ДНК, кодирующую слитый белок, содержащий аминокислоты 194-246 CCN5 (SEQ ID. NO: 1), слитый на N-конце с пептидным линкером (SEQ ID NO: 20) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2), экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 28.
Полученный белок тестировали на способность ингибировать сферообразующую способность (независимый от подложки рост) клеток положительной по рецептору эстрогена линии рака молочной железы MCF-7 и клеток линии рака молочной железы с тройным негативным фенотипом MDA-MB-231 (Фиг. 1С) согласно описанию  и соавторов, выше. Клетки MDA-MB-231 обрабатывали согласно описанию для линии клеток MCF-7
и соавторов, выше. Клетки MDA-MB-231 обрабатывали согласно описанию для линии клеток MCF-7  и соавторов, выше. Клетки линий MCF-7 и MDA-MB-231 субкультивировали согласно описанию для линии клеток А549, выше.
и соавторов, выше. Клетки линий MCF-7 и MDA-MB-231 субкультивировали согласно описанию для линии клеток А549, выше.
Пример 6
Последовательности ДНК, кодирующие слитый белок, содержащий аминокислоты 194 246 CCN5 (SEQ ID. NO: 1), слитый на N-конце с пептидным линкером (SEQ ID NO: 20) и либо Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, К447-, Eu-нумерация) (CCN5(dIII)-Fcv2), либо Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 18 (S228P, E233P, F234V, L235A, G236-, K447-, Eu-нумерация) (CCN5(dIII)-Fcv2.1), либо химерным Fc-фрагментом подклассов IgG2/4 (SEQ ID NO: 19) (CCN5(dIII)-Fcv2.3), экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 28 (CCN5(dIII)-Fcv2), SEQ ID NO: 29 (CCN5(dIII)- Fcv2.1) и SEQ ID NO: 30 (CCN5(dIII)-Fcv2.3).
В частности, экспрессионными векторами для экспрессии SEQ ID NO: 28 (CCN5(dIII)-Fcv2), SEQ ID NO: 29 (CCN5(dIII)-Fcv2.1) и SEQ ID NO: 30 (CCN5(dIII)-Fcv2.3) трансфицировали адаптированные к суспензионной культуре клетки СНО ExpiCHO в соответствии с протоколом «Мах Titer» от изготовителя набора для трансфекции СНО Expifectamine™ (Gibco, кат. №: A29129) и кратким описанием  и соавторов, выше. Клетки осаждали через 6 дней после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранных супернатантов клеточной культуральной среды разделяли в ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™, и визуализировали рекомбинантные белки с использованием системы визуализации ChemiDoc™ (BioRad). Разделенные белки затем переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad) для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad) для визуализации.
и соавторов, выше. Клетки осаждали через 6 дней после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранных супернатантов клеточной культуральной среды разделяли в ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™, и визуализировали рекомбинантные белки с использованием системы визуализации ChemiDoc™ (BioRad). Разделенные белки затем переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad) для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad) для визуализации.
На Фиг. 2 приведены данные, демонстрирующие улучшенную устойчивость к протеазам остова на основе Fc-фрагмента, состоящего из химеры IgG2/4 (показан на SEQ ID NO: 19).
CCN5/WISP2 (домен III) слитый с Fc-фрагментом IgG4 либо с помощью шарнира IgG4 с сайленсингом иммунных эффекторных функций (согласно определению в SEQ ID NO: 28); CCN5 (домен III)-Fcv2, где тот же остов IgG4 включает мутации на основе IgG2 (согласно определению в SEQ ID. NO: 29); CCN5 (домен III)-Fcv2.1, или тот же остов IgG4 с полной шарнирной областью IgG2 (согласно определению в SEQ ID NO: 30); CCN5 (домен III)-Fcv2.3 экспрессировали в системе ExpiCHO и собирали кондиционированную среду (СМ) через 6 дней. Вестерн-блоттинг и окрашивание на тотальный белок гелей ДСН-ПААГ показали, что вариант CCN5 (домен III)-Fcv2.3 наименее чувствителен к протеазам, присутствующим при культивировании. Обратим внимание, что иммунореактивность антитела против IgG4 по отношению к Fc-фрагменту частично утрачивается при замене на последовательности IgG2, и, соответственно, оценка уровня указанного белка на основании окрашивания общего белка занижена.
Пример 7
Последовательности ДНК, кодирующие слитый белок, содержащий аминокислоты 194 246 CCN5 (SEQ ID NO: 1), слитые на N-конце с пептидным линкером, описанным в SEQ ID NO: 20, и химерным Fc-фрагментом подклассов IgG2/4 (SEQ ID NO: 19) (CCN5(dIII)-Fcv2.3), или с пептидным линкером, описанным в SEQ ID NO: 25 и химерным Fc-фрагментом подклассов IgG2/4 (SEQ ID NO: 19) (CCN5(dIII)-HLn8-Fcv2.3), экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 30 (CCN5(dIII)-Fcv2.3) и SEQ ID NO: 31 (CCN5(dIII)-HLn8-Fcv2.3).
В частности, экспрессионными векторами для экспрессии SEQ ID NO: 30 (CCN5(dIII)-Fcv2.3) и SEQ ID NO: 31 (CCN5(dIII)-HLn8-Fcv2.3) трансфицировали адаптированные к суспензионной культуре клетки СНО ExpiCHO в соответствии с протоколом «Мах Titer» от изготовителя набора для трансфекции СНО Expifectamine™ (Gibco, кат. №: А29129) и кратким описанием  и соавторов, выше. Клетки осаждали через 4 дня после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранного супернатанта клеточной культуральной среды разделяли с помощью ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™. Разделенные белки переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad) для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали для визуализации в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad).
и соавторов, выше. Клетки осаждали через 4 дня после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранного супернатанта клеточной культуральной среды разделяли с помощью ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™. Разделенные белки переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad) для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали для визуализации в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad).
На Фиг. 3 приведены данные, отражающие сниженную склонность к агрегации в тех случаях, когда вариант реализации настоящего изобретения включает пептидный линкер, представленный в SEQ ID NO: 25.
Невосстанавливающий ДСН-ПААГ СМ из транзиентно трансфицированных суспензионных клеток СНО, экспрессирующих CCN5 (домен III), слитый с аминоконцом химерного Fc-фрагмента IgG2/4 посредством различных пептидных линкеров. Вестерн-блоттинг показывает, что слитый белок с последовательностью аминокислот, показанной в SEQ ID NO: 31; (dIII)-HLn8-Fcv2.3, имеет меньшую склонность к агрегации, чем слитый белок согласно настоящему изобретению, имеющий последовательность аминокислот, представленную в SEQ ID NO: 30; CCN5 (домен III)-Fcv2.3. Этот результат показывает, что пептидный линкер согласно определению в последовательности SEQ ID NO: 25, обеспечивает меньшую склонность к агрегации слитого белка по сравнению со слитым белком, содержащим пептидный линкер согласно определению в последовательности SEQ ID NO: 20.
Пример 8
Последовательности ДНК, кодирующие слитый белок, содержащий либо аминокислоты 194-246 CCN5 (SEQ ID. NO: 1), либо аминокислоты 194-246 CCN5 (SEQ ID. NO: 7), где аминокислота в положении 195 (пролин) заменена на аланин, слитый на С-конце с пептидным линкером (SEQ ID NO: 39) и Fc-фрагментом IgG подтипа IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация), экспрессировали для продуцирования рекомбинантного белка в соответствии с SEQ ID NO: 40 (Fc-HLn8-CCN5(dIII)) или SEQ ID NO:: 41 (Fc-HLn8-CCN5(dIII)-P195A).
В частности, экспрессионными векторами для экспрессии SEQ ID NO: 40 (Fc-HLn8-CCN5(dIII)) и SEQ ID NO: 41 (Fc-HLn8-CCN5(dIII)-P195A) трансфицировали адаптированные к суспензионной культуре клетки СНО ExpiCHO в соответствии с протоколом «Мах Titer» от изготовителя набора для трансфекции СНО Expifectamine™ (Gibco, кат. №: А29129) и кратким описанием  и соавторов, выше. Клетки осаждали через 3 дня после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранного супернатанта клеточной культуральной среды разделяли с помощью ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™. Разделенные белки переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad), для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad) для визуализации.
и соавторов, выше. Клетки осаждали через 3 дня после трансфекции центрифугированием при 13000 об/мин в настольной центрифуге Heraeus Biofuge Pico в течение 5 минут и собирали супернатант клеточной культуральной среды. Образцы собранного супернатанта клеточной культуральной среды разделяли с помощью ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™. Разделенные белки переносили на ПВДФ-мембраны с применением Trans-Blot Turbo, системы полусухого блоттинга (Bio-Rad), для анализа методом вестерн-блоттинга. Блот зондировали антителом против IgG4 человека, конъюгированным с пероксидазой хрена (Invitrogen, кат. №: А10654), которое использовали в сочетании с субстратом SuperSignal™ West Femto Maximum Sensitivity Substrate (ThermoFisherScientific) и системой визуализации ChemiDoc™ (BioRad) для визуализации.
На Фиг. 5 приведены данные, отражающие пониженную чувствительность к расщеплению эндопептидазой в тех случаях, когда вариант реализации настоящего изобретения включает мутацию пролин-195 в домене гомологии к повторам TSP-1 CCN5, представленную в SEQ ID NO: 7.
Восстанавливающий ДСН-ПААГ СМ из транзиентно трансфицированных суспензионных клеток СНО, экспрессирующих CCN5(домен III), слитый с карбоксильным концом Fc-фрагмента IgG4 согласно описанию в SEQ ID NO: 15, либо включающий мутацию Р195А (Fc-HLn8-CCN5(dIII)-P195A), либо экспрессирующих вариант дикого типа Р195 домена гомологии к повторам TSP-1 CCN5 (Fc-HLn8-CCN5(dIII)). Указанный блот демонстрирует, что мутация Р195А обеспечивает протеолитическую устойчивость домена гомологии к повторам TSP-1 CCN5.
Пример 9
Раскрыт слитый белок, содержащий аминокислоты 194-250 CCN5 человека (SEQ ID NO: 56), слитый на N-конце с пептидным линкером (SEQ ID NO: 57) и Fc-фрагментом IgG человека, подкласса IgG4, из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация), с получением последовательности белка, соответствующего SEQ ID NO: 58 (CCN5(dIII)-SL-Fcv0). К указанному слитому белку была дополнительно добавлена N-концевая сигнальная последовательность для секреции, происходящая из альбумина из SEQ ID NO: 32, для получения слитого белка, соответствующего SEQ ID NO: 59, и он был экспрессирован в клетках млекопитающих, как описано ниже.
Последовательность ДНК, кодирующую слитый белок из SEQ ID NO: 59, кодон-оптимизировали для экспрессии белка в клетках хомяка (используя алгоритм коммерческого поставщика), присоединяли последовательность KOZAK для трансляции на 5'-конце и вводили стоп-кодон на 3'-конце, получая последовательность ДНК из SEQ ID NO: 60. Указанную последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 61. Последовательность из SEQ ID NO: 61 синтезировал и верифицировал коммерческий поставщик. Синтезированную последовательность рекомбинировали с pDonrZeo путем рекомбиназного клонирования BP Gateway для получения входящего вектора. После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Тор10™), входящий вектор выделяли с помощью стандартных методик выделения плазмид с использованием набора QIAprep™ Spin Miniprep от Qiagen™. После выделения плазмиды входящий вектор верифицировали путем расщепления рестрикционными ферментами с последующим гель-электрофорезом ДНК в соответствии со стандартными методиками, хорошо известными специалисту.
Входящий вектор, содержащий последовательность SEQ ID NO: 60, дополнительно рекомбинировали с принимающим вектором с применением рекомбиназы LR Gateway. Использовали принимающий вектор pUCOE-DHFR-DEST согласно описанию в источнике:  et al., 2018, J. Biol. Chem, 293:46, pp. 17953-17970.
et al., 2018, J. Biol. Chem, 293:46, pp. 17953-17970.
После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top10™), экспрессионный вектор выделяли с помощью стандартных методик выделения плазмид с применением набора QIAGEN™ Plasmid Plus Maxi Kit. Итоговый экспрессионный вектор верифицировали с помощью стандартного расщепления рестрикционными ферментами и гель-электрофореза ДНК в соответствии со стандартными методиками, хорошо известными специалисту. Итоговый экспрессионным вектором затем трансфицировали адаптированные для суспензионной культуры клетки ExpiCHO в соответствии с протоколом «Мах Titer» от изготовителя набора для трансфекции СНО Expifectamine™ (Gibco, кат. №: А29129) и кратким описанием  и соавторов, 2018, выше. Клетки осаждали через 4 дня после трансфекции центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли ОДМ ПМСФ в 100% изопропаноле до концентрации 1 мМ и добавляли 0,5М ЭДТК до концентрации 2 мМ. Затем добавляли 96% этанол до конечной концентрации приблизительно 3%. Добавляли 1М TrisHCl рН 7,4 до конечной концентрации 25 мМ перед хроматографическим очищением.
и соавторов, 2018, выше. Клетки осаждали через 4 дня после трансфекции центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли ОДМ ПМСФ в 100% изопропаноле до концентрации 1 мМ и добавляли 0,5М ЭДТК до концентрации 2 мМ. Затем добавляли 96% этанол до конечной концентрации приблизительно 3%. Добавляли 1М TrisHCl рН 7,4 до конечной концентрации 25 мМ перед хроматографическим очищением.
Белок очищали аффинной хроматографией с использованием хроматографической среды с белком А. В этом эксперименте использовали хроматографическую среду rProtein А FF (GE Healthcare). Использовали колонку HiTrap ™ rProtein A FF (GE Healthcare) на 5 мл для очищения экспрессированного рекомбинантного белка из 120 мл клеточной культуральной среды, собранной и дополненной согласно описанию, см. выше. Колонку HiTrap ™ rProtein А FF устанавливали в систему скоростной высокопроизводительной жидкостной хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм и уравновешивали буфером, содержащим 25 мМ TrisHCl рН 7,4, 25 мМ NaCl и 3% этанола. Собранную клеточную культуральную среду, содержащую рекомбинантный белок, загружали с помощью пробоотборного насоса со скоростью 2,5 мл/мин, с последующим промыванием 10 объемами колонки промывочного буфера (25 мМ TrisHCl рН 7,4, 25 мМ NaCl и 3% этанола)) до элюирования ОДМ цитратом Na, рН 3,0, в 3% этаноле. Элюированные фракции объемом 3 мл собирали в пробирки с низким связыванием белков, предварительно наполненные 1 мл 1М TrisHCl, рН 9,0. Элюирование белка отслеживали по поглощению УФ на 280 нм, и образцы объединенных фракций объемом 10 мкл, содержащих пик поглощения УФ на 280 нм, подвергали ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™ в присутствии или в отсутствие восстанавливающего агента β-меркаптоэтанола, и выделенные рекомбинантные белки визуализировали с использованием системы визуализации ChemiDoc™ (BioRad).
Специалисту в данной области техники хорошо известно, что рекомбинантные белки могут быть получены в различных экспрессионных системах и очищены различными хроматографическими способами с получением аналогичных результатов.
На Фиг. 6 показана, что экспрессия и очищение белка, соответствующего SEQ ID NO: 58, действительно приводят к получению белка, мигрирующего выше ожидаемого уровня в отсутствие восстанавливающего агента β-меркаптоэтанола, что, соответственно, указывает на формирование димеров. Однако, как можно видеть по дорожке, которая содержит очищенный белок, в присутствии восстанавливающего агента β-меркаптоэтанола экспрессия и очищение белка, соответствующего SEQ ID NO: 58, приводит в первую очередь к получению расщепленных фрагментов, а не интактного белка.
Пример 10
Получали несколько вариантов последовательности SEQ ID NO: 58 с целью повысить протеолитическую устойчивость белка, соответствующего SEQ ID NO: 58. Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием для получения плазмид согласно описанию в Примере 9, и экспрессировали белки согласно описанию в Примере 9. Указанные варианты включают белки с модификациями, представленные ниже:
1) N-концевая сигнальная последовательность, происходящая из альбумина из SEQ ID NO: 32, со стороны аминоконца фрагмента CCN5, состоящего из аминокислот 194-249, включающего мутацию (P245L), соответствующего SEQ ID NO: 62, в комбинации с усеченным пептидным линкером, соответствующим SEQ ID NO: 63, и Fc-фрагментом SEQ ID NO: 15, с получением последовательности, соответствующей SEQ ID NO: 64,
2) N-концевая сигнальная последовательность, происходящая из альбумина из SEQ ID NO: 32, со стороны аминоконца фрагмента CCN5, состоящего из аминокислот 194-246, соответствующего SEQ ID NO: 1, в комбинации с вариантом пептидного линкера, соответствующим SEQ ID NO: 65, и Fc-фрагментом из SEQ ID NO: 15, с получением последовательности, соответствующей SEQ ID NO: 66,
3) N-концевая сигнальная последовательность, происходящая из альбумина из SEQ ID NO: 32, со стороны аминоконца фрагмента CCN5, состоящего из аминокислот 194-246, соответствующего SEQ ID NO: 1, в комбинации с вариантом пептидного линкера, соответствующим SEQ ID NO: 67, и Fc-фрагментом из SEQ ID NO: 15, с получением последовательности, соответствующей SEQ ID NO: 68,
4) N-концевая сигнальная последовательность, происходящая из альбумина из SEQ ID NO: 32, со стороны аминоконца фрагмента CCN5, состоящего из аминокислот 194-246, соответствующего SEQ ID NO: 1, в комбинации с вариантом пептидного линкера, соответствующим SEQ ID NO: 65, и Fc-фрагментом из SEQ ID NO: 19, с получением последовательности, соответствующей SEQ ID NO: 69.
Указанные версии (1-4, выше) белка, раскрытого в Примере 12, действительно показали некоторое повышение устойчивости к протеолитическому расщеплению во время экспрессии в системе ExpiCHO, проведенной согласно описанию в Примере 9. Однако при экспрессии белков, соответствующих SEQ ID NO: 64, SEQ ID. NO 66, SEQ ID NO: 68 и SEQ ID 69, обнаружилось, что степень протеолитической устойчивости все же была недостаточной, чтобы обеспечить получение интактных очищенных белков.
Пример 11
Получали слитый белок, содержащий аминокислоты 194-237 CCN5, где аминокислота в положении 195 (пролин) заменена на аланин (SEQ ID NO: 38), слитый на N-конце с пептидным линкером (SEQ ID NO: 21) и химерным Fc-фрагментом IgG подтипа IgG2/4 с делецией карбоксиконцевого K477- (Eu-нумерация) (SEQ ID NO: 19), с получением SEQ ID NO: 27. К слитому белку дополнительно добавляли N-концевую сигнальную последовательность для секреции, происходящую из альбумина SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 70. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 70, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 71. Указанную последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 72.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и белок, соответствующий SEQ ID NO: 70 экспрессировали путем транзиентной трансфекции клеток ExpiCHO согласно описанию в Примере 9.
Клетки осаждали через 6 дней после трансфекции центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ. Добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap ™ MabSelectSuRe™ 1 мл (GE Healthcare) непосредственно за которым следовало высаливание на колонке BioScale™ Mini Bio-Gel® Р-6 10 мл (BioRad). Колонки устанавливали в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Колонку MabSelectSuRe™ устанавливали на первый клапан для переключения колонки и уравновешивали буфером, состоящим из 30 мМ цитрата Na, рН 5,5, а колонку Bio-Gel® устанавливали на второй клапан для переключения колонки и уравновешивали буфером А2 (100 мМ NaH2PO4/Na2HPO4 рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку Bio-Gel®, 140 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на колонку MabSelectSuRe™ с помощью пробоотборного насоса со скоростью 2,0 мл/мин, с последующим промыванием 5 объемами колонки промывочного буфера А1 (30 мМ цитрат Na, рН 5,5), а затем 5 объемами колонки промывочного буфера A3 (30 мМ цитрат Na, 0,5М NaCl, рН 5,5), и затем 3 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (30 мМ лимонная кислота, рН 3,4) колонку Bio-Gel®, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 2 мл элюирующего буфера колонку MabSelectSuRe™ отключали от проточного канала и очищенный белок элюировали с колонки Bio-Gel® буфером А2. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 100 мЕОП. Собранные фракции объединяли и образец 10 мкл подвергали ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™ в присутствии восстанавливающего агента β-меркаптоэтанола, и выделенные рекомбинантные белки визуализировали с использованием системы визуализации ChemiDoc™ (BioRad).
На Фиг. 7 показано, что экспрессия и очищение белка, соответствующего SEQ ID NO: 27, где карбоксиконцевой фрагмент CCN5 усеченный, по существу более протеолитически устойчивы, чем варианты, где присутствуют все карбоксиконцевые аминокислоты CCN5 (как в SEQ ID NO: 58, 64, 66, 68 и 69), даже несмотря на то, что клеточную культуральную среду собирали позже на 2 дня после субкультивирования по сравнению с Примером 9 (Фиг. 6).
Пример 12
Получали слитый белок, содержащий аминокислоты 206-249 CCN3, где аминокислота в положении 207 (изолейцин) заменена на аланин (SEQ ID NO: 44), слитый на N-конце с пептидным линкером (SEQ ID NO: 21) и химерным Fc-фрагментом IgG подтипа IgG2/4 с делецией карбоксиконцевого K477- (Eu-нумерация) (SEQ ID NO: 19), с получением слитого белка из SEQ ID NO: 73. К слитому белку дополнительно добавляли N-концевую сигнальную последовательность для секреции, происходящую из альбумина SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 74. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 74, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 75. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 76.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и белок, соответствующий SEQ ID NO: 74, экспрессировали путем транзиентной трансфекции клеток ExpiCHO согласно описанию в Примере 9.
Клетки осаждали через 5 дней после трансфекции центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ. Добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelectSuRe™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке 53 мл HiPrep™ 26/10 (GE Healthcare). Колонки устанавливали в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Колонку MabSelectSuRe™ устанавливали на первый клапан для переключения колонки и уравновешивали буфером, состоящим из 30 мМ цитрата Na, рН 5,5, а колонку HiPrep™ устанавливали на второй клапан для переключения колонки и уравновешивали буфером А2 (100 мМ NaH2PO4/Na2HPO4 рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку HiPrep™, 260 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на колонку MabSelectSuRe™ с помощью пробоотборного насоса со скоростью 3,5 мл/мин, с последующим промыванием 5 объемами колонки промывочного буфера А1 (30 мМ цитрат Na, рН 5,5), затем 5 объемами колонки промывочного буфера A3 (30 мМ цитрат Na, 0,5М NaCl, рН 5,5), и затем 2 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (30 мМ лимонная кислота, рН 3,4) колонку HiPrep™, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 10 мл элюирующего буфера колонку MabSelectSuRe™ отключали от проточного канала, и очищенный белок элюировали с колонки HiPrep™ буфером А2. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 100 мЕОП. Собранные фракции объединяли и образец объемом 10 мкл подвергали ДСН-ПААГ с использованием готовых гелей Mini-PROTEAN® TGX Stain-Free™ в присутствии или в отсутствие восстанавливающего агента β-меркаптоэтанола и выделенные рекомбинантные белки визуализировали с использованием системы визуализации ChemiDoc™ (BioRad).
На Фиг. 8 можно видеть, что слитый белок, содержащий аминокислоты, происходящие из CCN3/NOv (домен III/домен гомологии с TSP-1), согласно описанию в SEQ ID NO: 73, аналогичный слитому белку, содержащему аминокислоты, происходящие из гомологичного CCN5 (домен III/домен гомологии с TSP-1), согласно описанию в SEQ ID NO: 27, обладает аналогичной или лучшей устойчивостью к протеолизу, чем слитый белок, содержащий аминокислоты, происходящие из CCN5, согласно описанию в Примере 11 и на Фиг. 7.
Пример 13
Слитый белок, содержащий аминокислоты 194-246 CCN5, где аминокислота в положении 195 (пролин) заменена на аланин (SEQ ID NO: 7), слитый на С-конце с пептидным линкером (SEQ ID NO: 39) и Fc-фрагментом IgG подкласса IgG4 из SEQ ID NO: 15 (S228P, F234A, L235A, K447-, Eu-нумерация), с получением последовательности белка, соответствующего SEQ ID NO: 41, дополняли N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 77. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 77, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 78. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 79.
Последовательность из SEQ ID NO: 79 синтезировал и верифицировал коммерческий поставщик. Синтезированную последовательность рекомбинировали с pDonrZeo путем рекомбиназного клонирования BP Gateway для получения входящего вектора. После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top10™), входящий вектор выделяли с помощью стандартных методик выделения плазмид с использованием набора QIAprep™ Spin Miniprep от Qiagen™. После выделения плазмиды входящий вектор верифицировали путем расщепления рестрикционными ферментами с последующим гель-электрофорезом ДНК в соответствии со стандартными методиками, хорошо известными специалисту.
Входящий вектор, содержащий последовательность SEQ ID NO: 78, дополнительно рекомбинировали с принимающим вектором с применением рекомбиназы LR Gateway. Использовали принимающий вектор pUCOE-DHFR-DEST, согласно описанию в источнике:  et al., 2018, J. Biol. Chem, 293:46, pp. 17953 - 17970.
et al., 2018, J. Biol. Chem, 293:46, pp. 17953 - 17970.
После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top10™), экспрессионный вектор выделяли с помощью стандартных методик выделения плазмид с применением набора QIAGEN™ Plasmid Plus Maxi Kit. Итоговый экспрессионный вектор верифицировали с помощью стандартного расщепления рестрикционными ферментами и гель-электрофореза ДНК в соответствии со стандартными методиками, хорошо известными специалисту. Затем итоговый экспрессионный вектор переносили в адаптированные к суспензионной культуре клетки СНО DG44 путем электропорации с использованием системы трансфекции Neon (ThermoFisherScientilic).
Клетки поддерживали в вентилируемых колбах Эрленмейера в инкубаторах для клеточных культур при 37°С с 8% CO2 на шейкерной платформе (согласно описанию Kaasbøll и соавторов, выше). Трансфицированные клетки выдерживали в течение ночи в среде CD для клеток DG44 (Gibco, кат. №12610-010) до перенесения в среду HyClone ™ ActiPro™ (без гипоксантина и тимидина, GE Healthcare), и субкультивировали до 80% жизнеспособности, в этот момент в среду добавляли 0,1 мкМ метотрексат. После добавления 0,1 мкМ метотрексата клетки субкультивировали до повторного достижения 80% жизнеспособности, в этот момент в среду добавляли 1 мкМ метотрексат. Клетки снова субкультивировали до превышения 98% жизнеспособности и снижения времени удвоения до менее чем 26 часов, после чего пул клеток считали стабильно трансфицированным. После получения стабильного пула клеток объем клеточной культуры увеличивали, чтобы обеспечить заселение стабильно трансфицированными клетками для продуцирования с плотностью, составляющей 1*106 клеток/мл. В культуры клеток добавляли 4/0,4% по объему HyClone™ Cell Boost™ 7a/7b ежедневно, начиная с 3 дня после субкультивирования. Через 10 дней клетки осаждали центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1 М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ. Добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelectSuRe™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare), согласно описанию в Примере 15. Очищенный белковый состав (без наблюдаемых признаков протеолитического процессинга) затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 9, очищенный белок, соответствующий SEQ ID NO: 41, продуцированный стабильно трансфицированным пулом суспензионных клеток СНО, неожиданным образом не показал признаков способности ингибировать фосфорилирование AKT (серии 473).
Пример 14
Получали слитый белок, содержащий аминокислоты 206-249 CCN3 (SEQ ID NO: 44), где аминокислота в положении 207 (изолейцин) заменена на аланин, слитый на N-конце с пептидным линкером (SEQ ID NO: 22) и химерным Fc-фрагментом IgG подтипа IgG2/4 (SEQ ID NO: 19), с получением последовательности белка, соответствующего SEQ ID NO: 80, которую дополняли N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 81. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 81, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 82. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 83.
Последовательность из SEQ ID NO: 83 синтезировал и верифицировал коммерческий поставщик. Синтезированную последовательность рекомбинировали с pDonrZeo путем рекомбиназного клонирования BP Gateway для получения входящего вектора. После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top10™), входящий вектор выделяли с помощью стандартных методик выделения плазмид с использованием набора QIAprep™ Spin Miniprep от Qiagen™. После выделения плазмиды входящий вектор верифицировали путем расщепления рестрикционными ферментами с последующим гель-электрофорезом ДНК в соответствии со стандартными методиками, хорошо известными специалисту.
Входящий вектор, содержащий последовательность SEQ ID NO: 82, дополнительно рекомбинировали с принимающим вектором с применением рекомбиназы LR Gateway. Использовали принимающий вектор pUCOE-DHFR-DEST, согласно описанию в источнике:  et al., 2018, J. Biol. Chem, 293:46, pp.17953-17970.
et al., 2018, J. Biol. Chem, 293:46, pp.17953-17970.
После трансфекции компетентных E.coli, мутированных так, чтобы позволять эффективное размножение плазмид (клетки One Shot Top10™), экспрессионный вектор выделяли с помощью стандартных методик выделения плазмид с применением набора QIAGEN™ Plasmid Plus Maxi Kit. Итоговый экспрессионный вектор верифицировали с помощью стандартного расщепления рестрикционными ферментами и гель-электрофореза ДНК в соответствии со стандартными методиками, хорошо известными специалисту. Затем итоговый экспрессионный вектор переносили в адаптированные к суспензионной культуре клетки СНО ExpiCHO в соответствии с протоколом «Создания и масштабирования стабильной клеточной линии с использованием продуктов ExpiCHO™» от изготовителя среды для продуцирования ExpiCHO™ Stable Production Medium (Gibco, кат. №:A3711001). Клетки поддерживали в вентилируемых колбах Эрленмейера в инкубаторах для клеточных культур при 37°С с 8% CO2 на шейкерной платформе (согласно описанию Kaasbøll и соавторов, выше). Трансфицированные клетки выдерживали в течение ночи в среде для экспрессии в клетках ExpiCHO™ до перенесения на среду для экспрессии в клетках ExpiCHO™ с добавлением 0,1 мкМ метотрексата. Затем клетки субкультивировали до повторного достижения 80% жизнеспособности, в этот момент в среду добавляли 1 мкМ метотрексат. Клетки снова субкультивировали до превышения 95% жизнеспособности и снижения времени удвоения до менее чем 20 часов, после чего пул клеток считали стабильно трансфицированным. После получения стабильного пула клеток объем клеточной культуры увеличивали, чтобы обеспечить заселение стабильно трансфицированными клетками для продуцирования с плотностью, составляющей 1*10∧6 клеток/мл. Через 5 дней клетки осаждали центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ. Добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ и добавляли 2М L-аргинин, рН 4,0 до конечной концентрации 100 мМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelect PrismA™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare), с использованием протокола согласно описанию в Примере 12, за исключением добавления 100 мМ L-Аргинина в буфер A1, А2, A3 и В1. Затем очищенный белковый состав (не проявляющий признаков протеолитического процессинга) тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 10, очищенный белок, соответствующий SEQ ID NO: 80, продуцированный стабильно трансфицированным пулом суспензионных клеток СНО, неожиданным образом не показал признаков способности ингибировать фосфорилирование AKT (серина 473) демонстрируя, что ни один из составов интактных димерных слитых с Fc белков, содержащих аминокислоты, происходящие из CCN5 (SEQ ID NO: 41, Пример 13, Фиг. 9) или аминокислоты, происходящие из CCN3 (SEQ ID NO: 80) не является биологически активным.
Пример 15
Экспрессионную плазмиду, описанную в Примере 14, содержащую SEQ ID NO: 82, кодирующую слитый белок, содержащий аминокислоты 206-249 CCN3 (SEQ ID NO: 44), где аминокислота в положении 207 (изолейцин) заменена на аланин, слитый на N-конце с пептидным линкером (SEQ ID NO: 22) и химерным Fc-фрагментом IgG подтипа IgG2/4 (SEQ ID NO: 19) с получением последовательности белка, соответствующего SEQ ID NO: 80, также дополненной N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32 и соответствующей SEQ ID NO: 81, экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 9. Клетки осаждали, согласно описанию в Примере 14, через 6 дней после трансфекции, и в среду вносили добавки согласно описанию в Примере 14. Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelect PrismA™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare), согласно описанию в Примере 14.
Очищенный белковый состав (который был частично протеолитически процессирован) затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 11, очищенный белок, соответствующий SEQ ID NO: 80, продуцированный транзиентно трансфицированными клетками ExpiCHO™, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что экспрессионная система, используемая для получения слитого белка, соответствующего SEQ ID NO: 80, и, следовательно, степень наблюдаемого протеолитического процессинга существенно влияют на активность или отсутствие активности итогового белкового состава.
Пример 16
Слитый белок, содержащий аминокислоты 206-249 CCN3 (SEQ ID NO: 44), где аминокислота в положении 207 (изолейцин) заменена на аланин, слитый на N-конце с пептидным линкером (SEQ ID NO: 21) и Fc-фрагментом с индуцирующими образование мономеров и продлевающими время полужизни мутациями (SEQ ID NO: 55), с получением последовательности белка, соответствующего SEQ ID NO: 84, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 85. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 85, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 86. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 86. Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и их экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 9. Клетки осаждали, согласно описанию в Примере 14, через 5 дней после трансфекции и в среду вносили добавки согласно описанию в Примере 14. Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelect PrismA™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare) согласно описанию в Примере 14.
Очищенный белковый состав, который демонстрировал ожидаемую мономерную форму, затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 12, очищенный белок, соответствующий SEQ ID NO: 84, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домена гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 17
Слитый белок, содержащий аминокислоты 206-249 CCN3 (SEQ ID. NO: 44), где аминокислота в положении 207 (изолейцин) заменена на аланин, слитый на N-конце с пептидным линкером (SEQ ID NO: 21) и Fc-фрагментом с индуцирующими образование мономеров и стабильность мутациями (SEQ ID NO: 54), с получением последовательности белка, соответствующего SEQ ID NO: 88, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32 с получением слитого белка, соответствующего SEQ ID NO: 89. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 89, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 90. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 91. Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9 и их экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 9. Клетки осаждали, согласно описанию в Примере 14, через 6 дней после трансфекции и в среду вносили добавки согласно описанию в Примере 14. Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ MabSelect PrismA™ 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare), согласно описанию в Примере 14.
Очищенный белковый состав, который в основном демонстрировал ожидаемую мономерную форму, затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 13, очищенный белок, соответствующий SEQ ID NO: 88, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домен гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 18
Слитый белок, содержащий аминокислоты 206-249 CCN3 (SEQ ID NO: 44), где аминокислота в положении 207 (изолейцин) заменена на аланин, слитый на N-конце с пептидным линкером (SEQ ID NO: 93) и многофункциональной меткой, включающей метку 6xHis, HaloTag и элементы Sumo* (SEQ ID NO: 92), с получением последовательности белка, соответствующего SEQ ID NO: 94, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 114. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 114, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 95. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 96. Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и их экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 15. Клетки осаждали, согласно описанию в Примере 14, через 5 дней после трансфекции. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ, добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ и 2М L-аргинин, рН 4,0 до конечной концентрации 0,1М; и добавляли имидазол до конечной концентрации 5 мМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке HiTrap™ HisTrap™ Excel 5 мл (GE Healthcare), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare). Колонки устанавливали в систему FPLC-хроматографии (BioRad NGC Discover™ 10 Pro система), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Колонку HisTrap™ устанавливали на первый клапан для переключения колонки и уравновешивали буфером А1, состоящим из 5 мМ имидазола, 50 мМ NaCl, 100 мМ L-аргинина, а колонку HiPrep™ устанавливали на второй клапан для переключения колонки и уравновешивали буфером А2 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинин, рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку HiPrep™, 250 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на колонку HisTrap™ с помощью пробоотборного насоса со скоростью 3,5 мл/мин, с последующим промыванием 5 объемами колонки промывочного буфера А1, затем 5 объемами колонки промывочного буфера A3 (5 мМ Имидазол, 0,5М NaCl, 100 мМ L-Аргинин), и затем 2 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (250 мМ Имидазол, 50 мМ NaCl, 100 мМ L-Аргинин) колонку HiPrep™, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 10 мл элюирующего буфера колонку HisTrap™ отключали от проточного канала и очищенный белок элюировали с колонки HiPrep™ буфером А2. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 60 мЕОП.
Очищенный белковый состав, который демонстрировал ожидаемую мономерную форму, затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 12, очищенный белок, соответствующий SEQ ID NO: 94, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домена гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 19
Слитый белок, содержащий аминокислоты 194-237 CCN5, где аминокислота в положении 195 (пролин) заменена на аланин (SEQ ID NO: 38), слитый на N-конце с пептидным линкером (SEQ ID NO: 21), и аминокислоты 25-609 сывороточного альбумина человека (SEQ ID NO: 52), с получением SEQ ID NO: 97, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 98. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 98, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 99. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 100.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, а суспензионные клетки СНО DG44, сконструированные для экспрессии конститутивно активной формы AKT, использовали для получения стабильного пула суспензионных клеток СНО, экспрессирующих белок из SEQ ID NO: 98 согласно описанию в Примере 13. После получения стабильного пула клеток объем клеточной культуры увеличивали, чтобы обеспечить заселение стабильно трансфицированными клетками для продуцирования с плотностью, составляющей 1*106 клеток/мл. Через 6 дней клетки осаждали центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды, добавляли ОДМ ПМСФ в 100% изопропаноле до концентрации 0,1 мМ, добавляли 0,5М ЭДТК до конечной концентрации 2 мМ, добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ и добавляли 2М L-аргинин, рН 4,0 до конечной концентрации ОДМ перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке Tricorn (GE Healthcare) заполненная 3 мл матрицы для очищения альбумина человека Capture Select™ Human Albumin Affinity Matrix (ThermoFisherScientific), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare). Колонки устанавливали в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Содержащую CaptureSelect™ колонку устанавливали на первый клапан для переключения колонки и уравновешивали буфером А1, состоящим из 100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинина, рН 6,5, а колонку HiPrep™ устанавливали на второй клапан для переключения колонки и уравновешивали буфером А1 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинина, рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку HiPrep™, 500 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на содержащую CaptureSelect™ колонку с помощью пробоотборного насоса со скоростью 2,0 мл/мин, с последующим промыванием 5 объемами колонки промывочного буфера А1, затем 5 объемами колонки промывочного буфера А2 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-Аргинин, 0,25М NaCl, рН 6,5), и затем 5 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (30 мМ лимонная кислота, рН 3,5+0,5М L-аргинин) колонку HiPrep™, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 10 мл элюирующего буфера содержащую CaptureSelect™ колонку отключали от проточного канала и очищенный белок элюировали с колонки HiPrep™ буфером А1. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 100 мЕОП.
Очищенный белковый состав, который демонстрировал ожидаемую мономерную форму, затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 14, очищенный белок, соответствующий SEQ ID NO: 97, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473) демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домена гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 20
Слитый белок сывороточного альбумина человека (аминокислоты 25-606, SEQ ID NO: 101) сливали на С-конце с пептидным линкером (SEQ ID NO: 22), соединенным с аминокислотами 194-246 CCN5 человека, где аминокислота в положении 195 (пролин) заменена на аланин (SEQ ID NO: 7), с получением SEQ ID NO: 103. Слитый белок, соответствующий SEQ ID NO: 102, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 103. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 103, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 104. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 105.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9 и DG44 суспензионные клетки СНО, сконструированные для экспрессии конститутивно активной формы AKT, использовали для получения стабильного пула суспензионных клеток СНО, экспрессирующих указанный белок из SEQ ID NO: 104 согласно описанию в Примере 13. После получения стабильного пула клеток объем клеточной культуры увеличивали, чтобы обеспечить заселение стабильно трансфицированными клетками для продуцирования с плотностью, составляющей 1*106 клеток/мл. Через 6 дней клетки осаждали центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ, добавляли 0,5М ЭДТК до конечной концентрации 2 мМ, добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ и добавляли 2М L-аргинин, рН 4,0 до конечной концентрации 0,1М перед хроматографическим очищением.
Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке Tricorn (GE Healthcare), заполненной 3 мл матрицы CaptureSelect™ Human Albumin Affinity Matrix (ThermoFisherScientific), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare) согласно описанию в Примере 19, за исключением того, что скорость загрузки образцов составляла 0,37 мл/мин вместо 2,0 мл/мин.
Очищенный белковый состав, который демонстрировал ожидаемую мономерную форму, затем тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 14, очищенный белок, соответствующий SEQ ID NO: 102, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473) демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домен гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 21
Слитый белок, содержащий аминокислоты 206-249 CCN3, где аминокислота в положении 207 (изолейцин) заменена на аланин (SEQ ID NO: 44), слитый на N-конце с пептидным линкером (SEQ ID NO: 21) и аминокислотами 25 609 сывороточного альбумина человека (SEQ ID NO: 52) с получением SEQ ID NO: 106, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 107. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 107, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 108. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 109.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и их экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 9. Клетки осаждали, согласно описанию в Примере 14, через 6 дней после трансфекции, и в среду вносили добавки согласно описанию в Примере 19. Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке Tricorn (GE Healthcare), заполненной 10 мл матрицы CaptureSelect™ Human Albumin Affinity Matrix (ThermoFisherScientific), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare). Колонки устанавливали в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Содержащую CaptureSelect™ колонку устанавливали на первый клапан для переключения колонки и уравновешивали буфером А1, состоящим из 100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинина, рН 6,5, а колонку HiPrep™ устанавливали на второй клапан для переключения колонки и уравновешивали буфером А1 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинин, рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку HiPrep™, 500 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на содержащую CaptureSelect™ колонку с помощью пробоотборного насоса со скоростью 1,0 мл/мин, с последующим промыванием 3 объемами колонки промывочного буфера А1, затем 2 объемами колонки промывочного буфера А2 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-Аргинин, 0,25М NaCl, рН 6,5), и затем 3 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (30 мМ лимонная кислота, рН 3,5+0,1М L-аргинин) колонку HiPrep™, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 15 мл элюирующего буфера содержащую Capture Select™ колонку отключали от проточного канала и очищенный белок элюировали с колонки HiPrep™ буфером А1. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 100 мЕОП.
Затем очищенный белковый состав, который содержал ожидаемую мономерную форму, тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 12, очищенный белок, соответствующий SEQ ID NO: 106, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домена гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 22
Слитый белок, содержащий аминокислоты 206-249 CCN3, где аминокислота в положении 207 (изолейцин) заменена на аланин (SEQ ID NO: 44), слитый на N-конце с пептидным линкером (SEQ ID NO: 22) и аминокислот 25-609 сывороточного альбумина человека (SEQ ID NO: 52) с получением SEQ ID NO: 110, был дополнен N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32, с получением слитого белка, соответствующего SEQ ID NO: 111. Последовательность ДНК, кодирующая слитый белок из SEQ ID NO: 111, была кодон-оптимизирована для экспрессии белка в клетках хомяка (с использованием алгоритма коммерческого поставщика), последовательность KOZAK для трансляции присоединяли на 5'-конце, а стоп-кодон вводили на 3'-конце, получая последовательность ДНК из SEQ ID NO: 112. Последовательность ДНК также дополняли на обоих концах сайтами attB Gateway, получая последовательность ДНК из SEQ ID NO: 113.
Последовательности ДНК синтезировал и верифицировал коммерческий поставщик перед субклонированием с получением плазмид согласно описанию в Примере 9, и их экспрессировали путем транзиентной трансфекции клеток ExpiCHO™ согласно описанию в Примере 9. Клетки осаждали, согласно описанию в Примере 14, через 6 дней после трансфекции, и в среду вносили добавки согласно описанию в Примере 19. Белок очищали тандемной хроматографией, состоящей из этапа захвата на колонке Tricorn (GE Healthcare), заполненной 10 мл матрицы CaptureSelect™ Human Albumin Affinity Matrix (ThermoFisherScientific), непосредственно за которым следовало высаливание на высаливающей колонке HiPrep™ 26/10 53 мл (GE Healthcare). Колонки устанавливали в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм. Содержащую CaptureSelect™ колонку устанавливали на первый клапан для переключения колонки и уравновешивали буфером А1, состоящим из 100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинина, рН 6,5, а колонку HiPrep™ устанавливали на второй клапан для переключения колонки и уравновешивали буфером А1 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинин, рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонку HiPrep™, 300 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на содержащую CaptureSelect™ колонку с помощью пробоотборного насоса со скоростью 1,0 мл/мин, с последующим промыванием 3 объемами колонки промывочного буфера А1, затем 2 объемами колонки промывочного буфера А2 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-Аргинин, 0,25М NaCl, рН 6,5), и затем 3 объемами колонки промывочного буфера А1. До элюирования элюирующим буфером (30 мМ лимонная кислота, рН 3,5+0,5М L-аргинин) колонку HiPrep™, установленную на второй клапан для переключения колонки, устанавливали в положение для подключения к проточному каналу. После элюирования 15 мл элюирующего буфера содержащую CaptureSelect™ колонку отключали от проточного канала и очищенный белок элюировали с колонки HiPrep™ буфером А1. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 100 мЕОП.
Затем очищенный белковый состав, который содержал ожидаемую мономерную форму, тестировали на способность ингибировать способствующую выживанию сигнализацию (фосфорилирование серина-473 AKT) в клетках рака легкого человека А549 согласно описанию в Примере 2. Как можно видеть на Фиг. 15, очищенный белок, соответствующий SEQ ID NO: 110, проявлял зависимую от концентрации способность ингибировать фосфорилирование AKT (серии 473), демонстрируя, что другой мономерный слитый белок, содержащий аминокислоты из домена III/домена гомологии с TSP-1 белка CCN, обладал способностью ингибировать фосфорилирование AKT (серин-473) в клетках рака легкого человека А549.
Пример 23
Экспрессионная плазмида, описанная в Примере 21, содержащая SEQ ID NO: 108, кодирующую слитый белок, содержащий аминокислоты 206-249 CCN3, где аминокислота в положении 207 (изолейцин) заменена на аланин (SEQ ID NO: 44), слитый на N-конце с пептидным линкером (SEQ ID NO: 21), и аминокислоты 25-609 сывороточного альбумина человека (SEQ ID NO: 52), также дополненный N-концевой сигнальной последовательностью для секреции, происходящей из альбумина из SEQ ID NO: 32 и соответствующей SEQ ID NO: 107, использовали для получения пула стабильно трансфицированных клеток ExpiCHO™ согласно описанию в Примере 14. Для получения партии кондиционированной среды, содержащей секретируемый белок, соответствующий SEQ ID NO: 106, пул стабильно трансфицированных клеток размножали, чтобы обеспечить заселение объема 250 мл стабильно трансфицированными клетками с плотностью 1*106 клеток/мл. В культуры клеток добавляли 5% по объему 2Х EfficientFeed™ С+(Gibco™) через день, начиная с 2 дня после субкультивирования, 3% глюкозы (10% масса/объем) на 2 день после субкультивирования и 5% глюкозы (10% масса/объем) на 6 день после субкультивирования. Через 9 дней клетки осаждали центрифугированием при 4750xg в течение 20 минут при 4°С и собирали супернатант клеточной культуральной среды. Добавляли 0,1М ПМСФ в 100% изопропаноле до концентрации 0,1 мМ. Добавляли 1М цитрат Na, рН 5,5 до конечной концентрации 30 мМ, и 2М L-аргинин, рН 4,0 до конечной концентрации 100 мМ перед хроматографическим очищением.
Белок очищали 20-хроматографией, состоящей из этапа захвата на колонке Tricorn (GE Healthcare), заполненной 10 мл матрицы CaptureSelect™ Human Albumin Affinity Matrix (ThermoFisherScientific), непосредственно за которым следовала эксклюзионная хроматография на двух последовательно соединенных колонках Superdex 200 Increase 10/300 GL (GE Healthcare). Колонки были установлены в систему FPLC-хроматографии (система BioRad NGC Discover™ 10 Pro), оснащенную проточной кюветой для УФ-детекции с длиной оптического пути 5 мм и выходным клапаном, соединенным с контуром для образцов на 5 мл. Содержащую CaptureSelect™ колонку устанавливали на первый клапан для переключения колонки и уравновешивали буфером А1, состоящим из 100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинина, рН 6,5, а колонки Superdex 200 Increase устанавливали на второй клапан для переключения колонки и уравновешивали буфером А1 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-аргинин, рН 6,5). При установленном в положение для обхода втором клапане для переключения колонки, содержащем колонки Superdex 200 Increase, 120 мл собранной клеточной культуральной среды, содержащей рекомбинантный белок, загружали на содержащую CaptureSelect™ колонку с помощью пробоотборного насоса со скоростью 3,9 мл/мин. После загрузки собранную клеточную культуральную среду, содержащую рекомбинантный белок, на содержащую CaptureSelect™ колонку, ее промывали 3 объемами колонки буфера А1, затем 2 объемами колонки буфера А2 (100 мМ NaH2PO4/Na2HPO4, 100 мМ L-Аргинин, 0,25М NaCl, рН 6,5), и затем 3 объемами колонки буфера А1. Проводили элюирование содержащей CaptureSelect™ колонки 15 мл буфера В1 (30 мМ лимонная кислота, 0,5М L-аргинин, рН 3,5), при этом которого система была настроена на сбор элюата с показателями поглощения выше 1200 мЕОП, в контур для образцов. После элюирования содержащей CaptureSelect™ колонки, соединенной с первым клапаном для переключения колонки, ее отключали от проточного канала, а второй клапан для переключения колонки устанавливали в положение для подключения колонок, содержащих Superdex 200 Increase, к проточному каналу. Затем элюат с содержащей CaptureSelect™ колонки, содержащий элюировавший белок, загружали на колонки, содержащие Superdex 200 Increase, с буфером А1 со скоростью 0,5 мл/мин. Элюирование белка отслеживали по поглощению УФ при 280 нм и запускали сбор, когда показатель поглощения превышал 200 мЕОП. Затем очищенный белковый состав, который содержал ожидаемую мономерную форму, тестировали на способность ингибировать индуцированные ТФР-β и активным CCN2 виды активности нормальных фибробластов легких человека (NHLF) (Lonza Bioscience, Кат. №: СС-2512). NHLF субкультивировали в полной ростовой среде (Lonza Bioscience BulletKit (Кат. №: СС-3132) со всеми добавками (2% фетальной бычьей сыворотки, инсулин, hFGF-B, гентамицин/амфотерицин-В)) для поддержания плотности с максимальной конфлюентностью 80% в соответствии с инструкциями продавца (Lonza Bioscience). Активный CCN2 состоял из доменов 3-4 CCN2, и был продуцирован и очищен согласно описанию у  и соавторов, 2018, выше.
и соавторов, 2018, выше.
Для тестирования эффекта белка, соответствующего SEQ ID NO: 106, на индуцированную активным CCN2 и ТФР-β миграцию клеток NHLF (анализ в системе Transwell/модифицированный анализ в камере Бойдена), клетки сначала рассоединяли трипсином/ЭДТК, нейтрализовали реагентами для нейтрализации трипсина (Lonza Bioscience, Кат. № СС-5034) и ресуспендировали в базальных ростовых средах (Базальная среда для фибробластов (LonzaBioscience Кат. №: СС-3131, без каких-либо добавок, кроме генсумицина (50 мкг/мл))), после чего высевали 30000 клеток в объеме 100 мкл на лунку с верхней стороны вкладышей Transwell с размером пор 5 мкм (24-луночный планшет, Corning® Transwell®, Кат. № CLS3402-48EA от SigmaAldrich (Merck KGaA)). Нижняя камера лунок содержала тестируемые вещества или контрольный носитель, растворенные в 500 мкл базальной ростовой среды без каких-либо добавок, кроме генсумицина. Через 20 часов инкубации вкладыши убирали из лунок, дважды промывали, погружая в забуференный фосфатом солевой раствор (ФСБ, Lonza Bioscience, Кат. №: 17-512F), после чего фиксировали в 4% формальдегиде (Solveco, Швеция, Кат. №: 621092) в течение 15 минут при 37°С. Клетки пермеабилизировали обработкой 0,1% Triton Х-100 в ФСБ в течение 10 минут, после чего дважды промывали ФСБ. Немигрировавшие клетки на верхней стороне вкладышей удаляли, соскабливая ватным тампоном, после чего мембрану оставляли для высыхания. Ядра мигрировавших клеток на обратной стороне вкладышей окрашивали раствором Hoechst 33342 20 мМ (1:5000 в ФСБ, ThermoFisherScientific, Кат. №: 62249) в течение 15 минут в темноте, после чего дважды промывали, погружая в ФСБ. Мембрану вырезали из вкладыша Transwell и монтировали на предметных стеклах, стороной с мигрировавшими клетками к стеклу, покрытому одной каплей Pro Long™ Gold Antifade (ThermoFisherScientific, Кат. №: P36934), закрывали стеклянным покровным стеклом, и по 5-10 изображений каждой лунки захватывали с помощью системы визуализации Zeiss Axio Observer Z.1. Изображения анализировали полуавтоматически с использованием программного обеспечения ImageJ v1.51k, Rasband, WS, ImageJ, от Национальных институтов здравоохранения США, Бетесда, Мэриленд, США, https://imagej.nih.gov/ij/, 1997-2018 гг..). Как можно видеть на Фиг. 16А, белок, соответствующий SEQ ID NO: 106, ингибирует миграцию, индуцированную как ТФР-β, так и активным CCN2.
Для тестирования эффекта белка, соответствующего SEQ ID NO: 106, на индуцированные активным CCN2 и ТФР-β результаты анализа зарастания царапины, NHLF рассоединяли трипсином/ЭДТК, нейтрализовали реагентами для нейтрализации трипсина (Lonza Bioscience, Кат. № СС-5034), после чего высевали 100000 клеток в объеме 1 мл в обработанные для культивирования тканей 12-луночные планшеты (Corning Costar®, Кат. № 3513). На следующий день после высевания клетки двукратно промывали 0,9% NaCl и полную ростовую среду заменяли на базальную ростовую среду. После инкубации в базальной ростовой среде в течение 16-20 часов на монослой клеток наносили царапину стерильным наконечником для пипетки на 12,5 мкл (ThermoFisherScientific, Кат. №: 94420053), клетки однократно промывали ФСБ, после чего инкубировали в 1 мл базальной ростовой среды совместно с тестируемыми веществами или носителем. Клетки инкубировали в течение дополнительных 24 часов до троекратного промывания ФСБ до фиксации в течение 15 минут при 37°С в 4% формальдегиде. После фиксации клетки снова промывали в течение 3x3 минут в ФСБ, осторожно встряхивая, пермеабилизировали 0,1% Triton Х-100 в ФСБ в течение 10 минут, осторожно встряхивая. Ядра клеток окрашивали 20 мМ раствором Hoechst 33342 (1:5000, разведенный в ФСБ), ThermoFisherScientific, Кат. №: 62249) в течение 15 минут в темноте, после чего промывали 3x5 минут в ФСБ, осторожно встряхивая. Наносили 1 каплю Pro Long™ Gold Antifade (ThermoFisherScientific, Кат. №: P36934) до монтировки препаратов и захватывали по 5 изображений, с центром на остающемся зазоре в каждой лунке, с помощью системы визуализации Zeiss Axio Observer Z.1. Изображения анализировали путем измерения размера оставшегося зазора после нанесения царапины через 3 фиксированных интервала вдоль всей длины царапины. Вычисляли среднее для всех измерений на всех изображениях из каждой лунки и засчитывали как один биологический репликат. Как можно видеть на Фиг. 16 В, белок, соответствующий SEQ ID NO: 106, ингибирует зарастание царапины, индуцированное как ТФР-β, так и активным CCN2.
Для тестирования эффекта белка, соответствующего SEQ ID NO: 106, на индуцированную ТФР-β регуляцию генов, NHLF рассоединяли трипсином/ЭДТК, нейтрализовали реагентами для нейтрализации трипсина (Lonza Bioscience, Кат. № СС-5034), после чего высевали 100000 клеток в объеме 1 мл в обработанные для культивирования тканей 12-луночные планшеты (Corning Costar®, Кат. № 3513). На следующий день после высевания клетки двукратно промывали 0,9% NaCl и полную ростовую среду заменяли на базальную ростовую среду с добавлением 0,1% инактивированной нагреванием фетальной бычьей сыворотки (Кат. № 16000-044 от Gibco™, термоинактивацию проводили согласно описанию в Примере 2). После инкубации в базальной ростовой среде с 0,1% фетальной бычьей сыворотки в течение 6 часов добавляли в лунки тестируемые вещества или контрольный носитель. Через 96 часов лунки двукратно промывали в ФСБ и экстрагировали РНК с использованием набора для экстракции РНК Qiagen RNeasy (Кат. № 74106) в соответствии с протоколом изготовителя. Концентрации РНК количественно определяли с помощью спектрофотометра NanoDrop® ND-1000 (NanoDrop Technologies, США), разводили водой без нуклеаз до конечной концентрации РНК 50 нг/мкл перед использованием 200 нг РНК из каждого репликата для получения кДНК с помощью набора для обратной транскрипции TaqMan™ (Кат. № N8080234) в соответствии с протоколом производителя. Дифференциальную генную экспрессию анализировали в полученных образцах кДНК с применением соответствующих анализов TaqMan™ и мастер-микса TaqMan Fast Advanced Master Mix (ThermoFisherScientific Кат. № 4444557). (ThermoFisherScientific Кат. № 4444557). Реакции ПЦР в реальном времени TaqMan™ проводили с получением технических трипликатов для каждого образца, используя систему ПЦР в реальном времени Applied Biosystems StepOnePlus Real Time PCR в соответствии с протоколами изготовителя. Относительные количества разных транскриптов вычисляли по стандартной кривой до усреднения данных технических трипликатов для получения единственного значения для каждого образца. Все результаты генной экспрессии сопоставляли с уровнями мРНК ГАФДГ (ThermoFisherScientific, Кат. № Hs02786624_g1) и нормировали, выражая в виде показателя кратности относительно среднего значения для лунок, стимулированных контролем-носителем. Как можно видеть на Фиг. 19C-F, белок, соответствующий SEQ ID NO: 107, обеспечивает частичное ингибирование индуцированных ТФР-β генов; COL1A1 («коллаген типа 1 α-1», ThermoFisherScientific, Кат. № Hs00164004_m1), FN1 («фибронектин 1», ThermoFisherScientific, Кат. № Hs01549976_m1), АСТА2 («гладкомышечный актин α-2», ThermoFisherScientific, Кат. № Hs00426835_g1) и CCN2 (ThermoFisherScientific, Кат. № Hs00170014_m1), которые обычно рассматривают как профибротические гены.

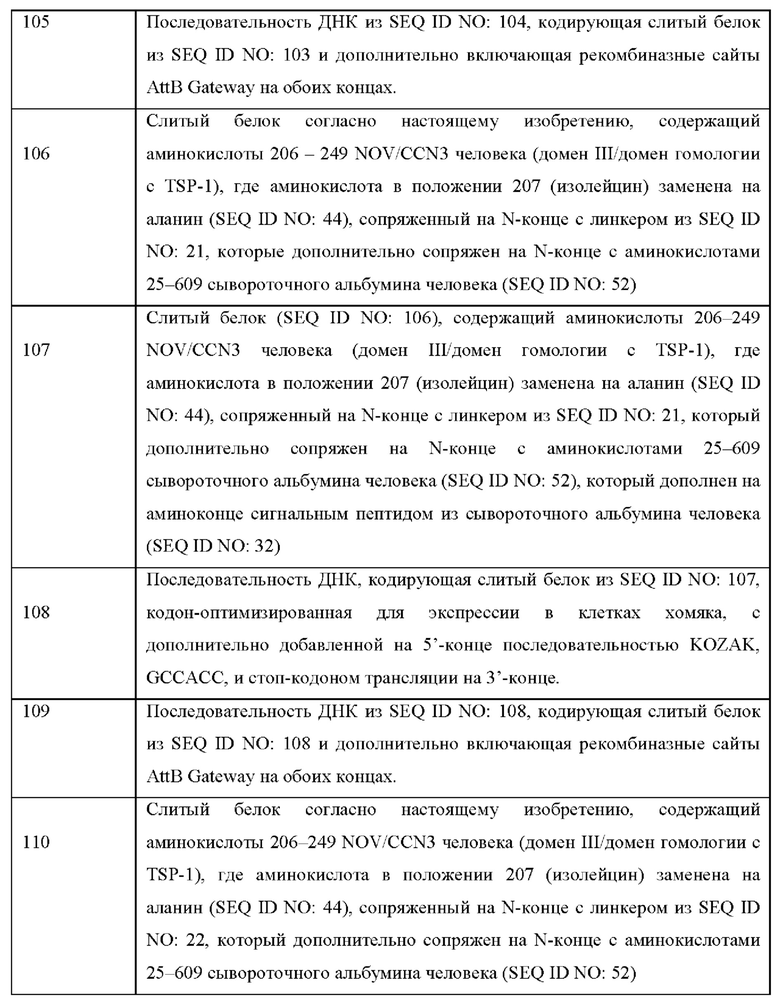
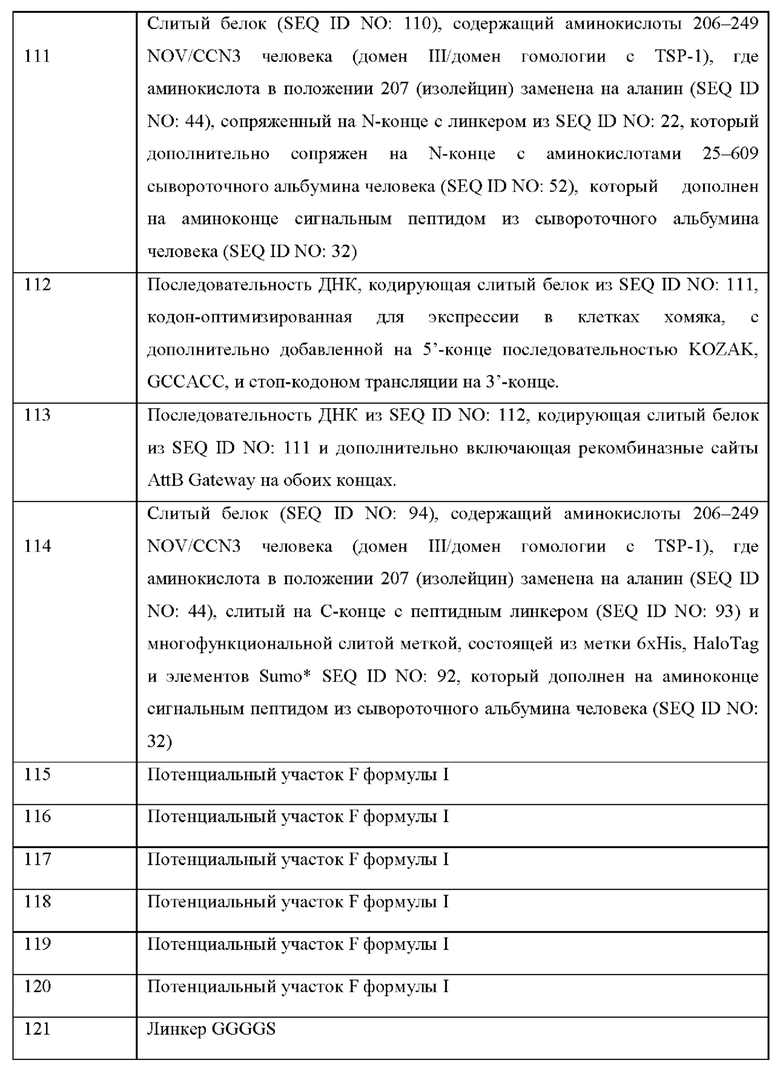
Нумерация белков CCN: в соответствии с базой данных Uniprot, согласно описанию в разделе «Подробное описание изобретения», выше. Нумерация Fc-фрагментов: в соответствии с системой нумерации Ей согласно описанию в разделе «Подробное описание изобретения», выше.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Oslo Universitetssykehus HF/ Осло Университетссикехус ХФ
<120> Recombinant CCN domain proteins and fusion proteins/
РЕКОМБИНАНТНЫЕ БЕЛКИ И СЛИТЫЕ БЕЛКИ С ДОМЕНАМИ CCN
<130> 27.11.145331/01
<150> EP 19163970.7
<151> 2019-03-20
<160> 121
<170> PatentIn, версия 3.5
<210> 1
<211> 53
<212> PRT/Белок
<213> Homo sapiens
<400> 1
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln
50
<210> 2
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 2
Cys Ile Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys
35 40
<210> 3
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 3
Cys Leu Val Gln Thr Thr Glu Trp Ser Ala Cys Ser Lys Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Ala Ser Cys Arg Leu
20 25 30
Glu Lys Gln Ser Arg Leu Cys Met Val Arg Pro Cys
35 40
<210> 4
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 4
Cys Ile Val Gln Thr Thr Ser Trp Ser Gln Cys Ser Lys Thr Cys Gly
1 5 10 15
Thr Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Pro Glu Cys Arg Leu
20 25 30
Val Lys Glu Thr Arg Ile Cys Glu Val Arg Pro Cys
35 40
<210> 5
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 5
Cys Ile Ala Tyr Thr Ser Pro Trp Ser Pro Cys Ser Thr Ser Cys Gly
1 5 10 15
Leu Gly Val Ser Thr Arg Ile Ser Asn Val Asn Ala Gln Cys Trp Pro
20 25 30
Glu Gln Glu Ser Arg Leu Cys Asn Leu Arg Pro Cys
35 40
<210> 6
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 6
Cys Leu Val Gln Ala Thr Lys Trp Thr Pro Cys Ser Arg Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Asn Arg Val Thr Asn Glu Asn Ser Asn Cys Glu Met
20 25 30
Arg Lys Glu Lys Arg Leu Cys Tyr Ile Gln Pro Cys
35 40
<210> 7
<211> 53
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN5
<400> 7
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln
50
<210> 8
<211> 55
<212> PRT/Белок
<213> Homo sapiens
<400> 8
Cys Leu Val Gln Ala Thr Lys Trp Thr Pro Cys Ser Arg Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Asn Arg Val Thr Asn Glu Asn Ser Asn Cys Glu Met
20 25 30
Arg Lys Glu Lys Arg Leu Cys Tyr Ile Gln Pro Cys Asp Ser Asn Ile
35 40 45
Leu Lys Thr Ile Lys Ile Pro
50 55
<210> 9
<211> 53
<212> PRT/Белок
<213> Homo sapiens
<400> 9
Cys Ile Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Gln Glu Pro
35 40 45
Glu Gln Pro Thr Asp
50
<210> 10
<211> 52
<212> PRT/Белок
<213> Homo sapiens
<400> 10
Cys Leu Val Gln Thr Thr Glu Trp Ser Ala Cys Ser Lys Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Ala Ser Cys Arg Leu
20 25 30
Glu Lys Gln Ser Arg Leu Cys Met Val Arg Pro Cys Glu Ala Asp Leu
35 40 45
Glu Glu Asn Ile
50
<210> 11
<211> 52
<212> PRT/Белок
<213> Homo sapiens
<400> 11
Cys Ile Val Gln Thr Thr Ser Trp Ser Gln Cys Ser Lys Thr Cys Gly
1 5 10 15
Thr Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Pro Glu Cys Arg Leu
20 25 30
Val Lys Glu Thr Arg Ile Cys Glu Val Arg Pro Cys Gly Gln Pro Val
35 40 45
Tyr Ser Ser Leu
50
<210> 12
<211> 52
<212> PRT/Белок
<213> Homo sapiens
<400> 12
Cys Ile Ala Tyr Thr Ser Pro Trp Ser Pro Cys Ser Thr Ser Cys Gly
1 5 10 15
Leu Gly Val Ser Thr Arg Ile Ser Asn Val Asn Ala Gln Cys Trp Pro
20 25 30
Glu Gln Glu Ser Arg Leu Cys Asn Leu Arg Pro Cys Asp Val Asp Ile
35 40 45
His Thr Leu Ile
50
<210> 13
<211> 229
<212> PRT/Белок
<213> Homo sapiens
<400> 13
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 14
<211> 228
<212> PRT/Белок
<213> Homo sapiens
<400> 14
Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val
1 5 10 15
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
20 25 30
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
35 40 45
His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
50 55 60
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
65 70 75 80
Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn
85 90 95
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro
100 105 110
Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln
115 120 125
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
130 135 140
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ser Val
145 150 155 160
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
165 170 175
Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
180 185 190
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
195 200 205
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
210 215 220
Ser Pro Gly Lys
225
<210> 15
<211> 228
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 15
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala
1 5 10 15
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly
225
<210> 16
<211> 232
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 16
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Cys Glu Glu Gln
65 70 75 80
Tyr Gly Ser Thr Tyr Arg Cys Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 17
<211> 231
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 17
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
20 25 30
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
35 40 45
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
50 55 60
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Cys Glu Glu Gln Tyr
65 70 75 80
Gly Ser Thr Tyr Arg Cys Val Ser Val Leu Thr Val Leu His Gln Asp
85 90 95
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
100 105 110
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
115 120 125
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
130 135 140
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
145 150 155 160
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
165 170 175
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
180 185 190
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
195 200 205
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
210 215 220
Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 18
<211> 227
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 18
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Pro Val
1 5 10 15
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
20 25 30
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
35 40 45
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
50 55 60
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
65 70 75 80
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
85 90 95
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser
100 105 110
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
115 120 125
Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
130 135 140
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
145 150 155 160
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
165 170 175
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr
180 185 190
Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
195 200 205
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
210 215 220
Ser Leu Gly
225
<210> 19
<211> 227
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 19
Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val
1 5 10 15
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
20 25 30
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
35 40 45
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
50 55 60
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
65 70 75 80
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
85 90 95
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser
100 105 110
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
115 120 125
Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
130 135 140
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
145 150 155 160
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
165 170 175
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr
180 185 190
Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
195 200 205
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
210 215 220
Ser Leu Gly
225
<210> 20
<211> 6
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 20
Thr Glu Gly Arg Met Asp
1 5
<210> 21
<211> 5
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 21
Glu Ala Ala Ala Lys
1 5
<210> 22
<211> 40
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 22
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
1 5 10 15
Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala
20 25 30
Ala Ala Lys Glu Ala Ala Ala Lys
35 40
<210> 23
<211> 42
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 23
Thr Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala
1 5 10 15
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
20 25 30
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
35 40
<210> 24
<211> 43
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 24
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
1 5 10 15
Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala
20 25 30
Ala Ala Lys Glu Ala Ala Ala Lys Ala Ala Ala
35 40
<210> 25
<211> 45
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 25
Thr Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala
1 5 10 15
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
20 25 30
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala Ala Ala
35 40 45
<210> 26
<211> 276
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 26
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro
50 55 60
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
65 70 75 80
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
85 90 95
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
100 105 110
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
115 120 125
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
130 135 140
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
145 150 155 160
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
165 170 175
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
180 185 190
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
195 200 205
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
210 215 220
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
225 230 235 240
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
245 250 255
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
260 265 270
Leu Ser Leu Gly
275
<210> 27
<211> 276
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 27
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro
50 55 60
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
65 70 75 80
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
85 90 95
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
100 105 110
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
115 120 125
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
130 135 140
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
145 150 155 160
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
165 170 175
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
180 185 190
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
195 200 205
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
210 215 220
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
225 230 235 240
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
245 250 255
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
260 265 270
Leu Ser Leu Gly
275
<210> 28
<211> 287
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 28
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Ser Lys Tyr Gly
50 55 60
Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
65 70 75 80
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
85 90 95
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
100 105 110
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
115 120 125
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
130 135 140
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
145 150 155 160
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
165 170 175
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
180 185 190
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
195 200 205
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
210 215 220
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
225 230 235 240
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
245 250 255
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
260 265 270
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
275 280 285
<210> 29
<211> 286
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 29
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Ser Lys Tyr Gly
50 55 60
Pro Pro Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
65 70 75 80
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
85 90 95
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
100 105 110
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
115 120 125
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
130 135 140
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
145 150 155 160
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
165 170 175
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
180 185 190
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
195 200 205
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
210 215 220
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
225 230 235 240
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
245 250 255
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
260 265 270
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
275 280 285
<210> 30
<211> 286
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 30
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Arg Lys Cys Cys
50 55 60
Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val
65 70 75 80
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
85 90 95
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
100 105 110
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
115 120 125
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
130 135 140
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
145 150 155 160
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
165 170 175
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
180 185 190
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
195 200 205
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
210 215 220
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
225 230 235 240
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
245 250 255
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
260 265 270
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
275 280 285
<210> 31
<211> 325
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 31
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Thr Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala
50 55 60
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
65 70 75 80
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala
85 90 95
Ala Ala Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Leu Gly
325
<210> 32
<211> 18
<212> PRT/Белок
<213> Homo sapiens
<400> 32
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser
<210> 33
<211> 305
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 33
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Ser Lys
65 70 75 80
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
85 90 95
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
100 105 110
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
115 120 125
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
130 135 140
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
145 150 155 160
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
165 170 175
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
180 185 190
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
195 200 205
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
210 215 220
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
225 230 235 240
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
245 250 255
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
260 265 270
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
275 280 285
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
290 295 300
Gly
305
<210> 34
<211> 915
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 34
atgaaatggg tcacctttat ctccctgctg ttcctgttct cctccgccta ctcttgccct 60
gagtggtcta cagcttgggg cccttgctct accacctgtg gactcggcat ggccaccaga 120
gtgtctaacc agaacagatt ctgccggctg gaaacccagc ggagactgtg cctgtctaga 180
ccctgtcctc ctagcagagg cagatcccct cagaccgagg gcagaatgga cgagtctaag 240
tacggccctc cttgtcctcc atgtcctgct ccagaagctg ctggcggccc ttccgtgttt 300
ctgttccctc caaagcctaa ggacaccctg atgatctctc ggacccctga agtgacctgc 360
gtggtggtgg atgtgtccca agaggatccc gaggtgcagt tcaattggta cgtggacggc 420
gtggaagtgc acaacgccaa gaccaagcct agagaggaac agttcaactc cacctacaga 480
gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga gtacaagtgc 540
aaggtgtcca acaagggcct gccttccagc atcgaaaaga ccatctccaa ggccaagggc 600
cagcctaggg aaccccaggt ttacaccctg cctccaagcc aagaggaaat gaccaagaac 660
caggtgtccc tgacctgcct ggtcaagggc ttctaccctt ccgatatcgc cgtggaatgg 720
gagagcaatg gccagcctga gaacaactac aagaccacac ctcctgtgct ggactccgac 780
ggctccttct ttctgtactc ccgcctgacc gtggacaagt ccagatggca agagggcaac 840
gtgttctcct gctccgtgat gcacgaggcc ctgcacaatc actacaccca gaagtccctg 900
tctctgtccc tgggc 915
<210> 35
<211> 924
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 35
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgccctgagt ggtctacagc ttggggccct tgctctacca cctgtggact cggcatggcc 120
accagagtgt ctaaccagaa cagattctgc cggctggaaa cccagcggag actgtgcctg 180
tctagaccct gtcctcctag cagaggcaga tcccctcaga ccgagggcag aatggacgag 240
tctaagtacg gccctccttg tcctccatgt cctgctccag aagctgctgg cggcccttcc 300
gtgtttctgt tccctccaaa gcctaaggac accctgatga tctctcggac ccctgaagtg 360
acctgcgtgg tggtggatgt gtcccaagag gatcccgagg tgcagttcaa ttggtacgtg 420
gacggcgtgg aagtgcacaa cgccaagacc aagcctagag aggaacagtt caactccacc 480
tacagagtgg tgtccgtgct gaccgtgctg caccaggatt ggctgaacgg caaagagtac 540
aagtgcaagg tgtccaacaa gggcctgcct tccagcatcg aaaagaccat ctccaaggcc 600
aagggccagc ctagggaacc ccaggtttac accctgcctc caagccaaga ggaaatgacc 660
aagaaccagg tgtccctgac ctgcctggtc aagggcttct acccttccga tatcgccgtg 720
gaatgggaga gcaatggcca gcctgagaac aactacaaga ccacacctcc tgtgctggac 780
tccgacggct ccttctttct gtactcccgc ctgaccgtgg acaagtccag atggcaagag 840
ggcaacgtgt tctcctgctc cgtgatgcac gaggccctgc acaatcacta cacccagaag 900
tccctgtctc tgtccctggg ctaa 924
<210> 36
<211> 997
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 36
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgc cctgagtggt ctacagcttg 120
gggcccttgc tctaccacct gtggactcgg catggccacc agagtgtcta accagaacag 180
attctgccgg ctggaaaccc agcggagact gtgcctgtct agaccctgtc ctcctagcag 240
aggcagatcc cctcagaccg agggcagaat ggacgagtct aagtacggcc ctccttgtcc 300
tccatgtcct gctccagaag ctgctggcgg cccttccgtg tttctgttcc ctccaaagcc 360
taaggacacc ctgatgatct ctcggacccc tgaagtgacc tgcgtggtgg tggatgtgtc 420
ccaagaggat cccgaggtgc agttcaattg gtacgtggac ggcgtggaag tgcacaacgc 480
caagaccaag cctagagagg aacagttcaa ctccacctac agagtggtgt ccgtgctgac 540
cgtgctgcac caggattggc tgaacggcaa agagtacaag tgcaaggtgt ccaacaaggg 600
cctgccttcc agcatcgaaa agaccatctc caaggccaag ggccagccta gggaacccca 660
ggtttacacc ctgcctccaa gccaagagga aatgaccaag aaccaggtgt ccctgacctg 720
cctggtcaag ggcttctacc cttccgatat cgccgtggaa tgggagagca atggccagcc 780
tgagaacaac tacaagacca cacctcctgt gctggactcc gacggctcct tctttctgta 840
ctcccgcctg accgtggaca agtccagatg gcaagagggc aacgtgttct cctgctccgt 900
gatgcacgag gccctgcaca atcactacac ccagaagtcc ctgtctctgt ccctgggcta 960
atctagaaac ccagctttct tgtacaaagt ggtcccc 997
<210> 37
<211> 44
<212> PRT/Белок
<213> Homo sapiens
<400> 37
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys
35 40
<210> 38
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN5
<400> 38
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys
35 40
<210> 39
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 39
Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
1 5 10 15
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
20 25 30
Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala Ala Ala
35 40
<210> 40
<211> 325
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 40
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala
1 5 10 15
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
225 230 235 240
Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala
245 250 255
Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala Ala Ala
260 265 270
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
275 280 285
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
290 295 300
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
305 310 315 320
Gly Arg Ser Pro Gln
325
<210> 41
<211> 325
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 41
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala
1 5 10 15
Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
225 230 235 240
Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala
245 250 255
Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala Ala Ala
260 265 270
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
275 280 285
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
290 295 300
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
305 310 315 320
Gly Arg Ser Pro Gln
325
<210> 42
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN1
<400> 42
Cys Ala Val Gln Thr Thr Ser Trp Ser Gln Cys Ser Lys Thr Cys Gly
1 5 10 15
Thr Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Pro Glu Cys Arg Leu
20 25 30
Val Lys Glu Thr Arg Ile Cys Glu Val Arg Pro Cys
35 40
<210> 43
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN2
<400> 43
Cys Ala Val Gln Thr Thr Glu Trp Ser Ala Cys Ser Lys Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Ala Ser Cys Arg Leu
20 25 30
Glu Lys Gln Ser Arg Leu Cys Met Val Arg Pro Cys
35 40
<210> 44
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN3
<400> 44
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys
35 40
<210> 45
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN4
<400> 45
Cys Ala Ala Tyr Thr Ser Pro Trp Ser Pro Cys Ser Thr Ser Cys Gly
1 5 10 15
Leu Gly Val Ser Thr Arg Ile Ser Asn Val Asn Ala Gln Cys Trp Pro
20 25 30
Glu Gln Glu Ser Arg Leu Cys Asn Leu Arg Pro Cys
35 40
<210> 46
<211> 44
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN6
<400> 46
Cys Ala Val Gln Ala Thr Lys Trp Thr Pro Cys Ser Arg Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Asn Arg Val Thr Asn Glu Asn Ser Asn Cys Glu Met
20 25 30
Arg Lys Glu Lys Arg Leu Cys Tyr Ile Gln Pro Cys
35 40
<210> 47
<211> 52
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN1
<400> 47
Cys Ala Val Gln Thr Thr Ser Trp Ser Gln Cys Ser Lys Thr Cys Gly
1 5 10 15
Thr Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Pro Glu Cys Arg Leu
20 25 30
Val Lys Glu Thr Arg Ile Cys Glu Val Arg Pro Cys Gly Gln Pro Val
35 40 45
Tyr Ser Ser Leu
50
<210> 48
<211> 52
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN2
<400> 48
Cys Ala Val Gln Thr Thr Glu Trp Ser Ala Cys Ser Lys Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Thr Arg Val Thr Asn Asp Asn Ala Ser Cys Arg Leu
20 25 30
Glu Lys Gln Ser Arg Leu Cys Met Val Arg Pro Cys Glu Ala Asp Leu
35 40 45
Glu Glu Asn Ile
50
<210> 49
<211> 53
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN3
<400> 49
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Gln Glu Pro
35 40 45
Glu Gln Pro Thr Asp
50
<210> 50
<211> 52
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN4
<400> 50
Cys Ala Ala Tyr Thr Ser Pro Trp Ser Pro Cys Ser Thr Ser Cys Gly
1 5 10 15
Leu Gly Val Ser Thr Arg Ile Ser Asn Val Asn Ala Gln Cys Trp Pro
20 25 30
Glu Gln Glu Ser Arg Leu Cys Asn Leu Arg Pro Cys Asp Val Asp Ile
35 40 45
His Thr Leu Ile
50
<210> 51
<211> 55
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN6
<400> 51
Cys Ala Val Gln Ala Thr Lys Trp Thr Pro Cys Ser Arg Thr Cys Gly
1 5 10 15
Met Gly Ile Ser Asn Arg Val Thr Asn Glu Asn Ser Asn Cys Glu Met
20 25 30
Arg Lys Glu Lys Arg Leu Cys Tyr Ile Gln Pro Cys Asp Ser Asn Ile
35 40 45
Leu Lys Thr Ile Lys Ile Pro
50 55
<210> 52
<211> 585
<212> PRT/Белок
<213> Homo sapiens
<400> 52
Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly Glu
1 5 10 15
Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu Gln
20 25 30
Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr Glu
35 40 45
Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp Lys
50 55 60
Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr Leu
65 70 75 80
Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu Pro
85 90 95
Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn Leu
100 105 110
Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe His
115 120 125
Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala Arg
130 135 140
Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys Arg
145 150 155 160
Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala Ala
165 170 175
Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala Ser
180 185 190
Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly Glu
195 200 205
Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe Pro
210 215 220
Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr Lys
225 230 235 240
Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp Asp
245 250 255
Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile Ser
260 265 270
Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser His
275 280 285
Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro Ser
290 295 300
Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr Ala
305 310 315 320
Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala Arg
325 330 335
Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys Thr
340 345 350
Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His Glu
355 360 365
Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu Pro
370 375 380
Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly Glu
385 390 395 400
Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val Pro
405 410 415
Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly Lys
420 425 430
Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro Cys
435 440 445
Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu His
450 455 460
Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu Ser
465 470 475 480
Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu Thr
485 490 495
Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala Asp
500 505 510
Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr Ala
515 520 525
Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln Leu
530 535 540
Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys Lys
545 550 555 560
Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu Val
565 570 575
Ala Ala Ser Gln Ala Ala Leu Gly Leu
580 585
<210> 53
<211> 679
<212> PRT/Белок
<213> Homo sapiens
<400> 53
Val Pro Asp Lys Thr Val Arg Trp Cys Ala Val Ser Glu His Glu Ala
1 5 10 15
Thr Lys Cys Gln Ser Phe Arg Asp His Met Lys Ser Val Ile Pro Ser
20 25 30
Asp Gly Pro Ser Val Ala Cys Val Lys Lys Ala Ser Tyr Leu Asp Cys
35 40 45
Ile Arg Ala Ile Ala Ala Asn Glu Ala Asp Ala Val Thr Leu Asp Ala
50 55 60
Gly Leu Val Tyr Asp Ala Tyr Leu Ala Pro Asn Asn Leu Lys Pro Val
65 70 75 80
Val Ala Glu Phe Tyr Gly Ser Lys Glu Asp Pro Gln Thr Phe Tyr Tyr
85 90 95
Ala Val Ala Val Val Lys Lys Asp Ser Gly Phe Gln Met Asn Gln Leu
100 105 110
Arg Gly Lys Lys Ser Cys His Thr Gly Leu Gly Arg Ser Ala Gly Trp
115 120 125
Asn Ile Pro Ile Gly Leu Leu Tyr Cys Asp Leu Pro Glu Pro Arg Lys
130 135 140
Pro Leu Glu Lys Ala Val Ala Asn Phe Phe Ser Gly Ser Cys Ala Pro
145 150 155 160
Cys Ala Asp Gly Thr Asp Phe Pro Gln Leu Cys Gln Leu Cys Pro Gly
165 170 175
Cys Gly Cys Ser Thr Leu Asn Gln Tyr Phe Gly Tyr Ser Gly Ala Phe
180 185 190
Lys Cys Leu Lys Asp Gly Ala Gly Asp Val Ala Phe Val Lys His Ser
195 200 205
Thr Ile Phe Glu Asn Leu Ala Asn Lys Ala Asp Arg Asp Gln Tyr Glu
210 215 220
Leu Leu Cys Leu Asp Asn Thr Arg Lys Pro Val Asp Glu Tyr Lys Asp
225 230 235 240
Cys His Leu Ala Gln Val Pro Ser His Thr Val Val Ala Arg Ser Met
245 250 255
Gly Gly Lys Glu Asp Leu Ile Trp Glu Leu Leu Asn Gln Ala Gln Glu
260 265 270
His Phe Gly Lys Asp Lys Ser Lys Glu Phe Gln Leu Phe Ser Ser Pro
275 280 285
His Gly Lys Asp Leu Leu Phe Lys Asp Ser Ala His Gly Phe Leu Lys
290 295 300
Val Pro Pro Arg Met Asp Ala Lys Met Tyr Leu Gly Tyr Glu Tyr Val
305 310 315 320
Thr Ala Ile Arg Asn Leu Arg Glu Gly Thr Cys Pro Glu Ala Pro Thr
325 330 335
Asp Glu Cys Lys Pro Val Lys Trp Cys Ala Leu Ser His His Glu Arg
340 345 350
Leu Lys Cys Asp Glu Trp Ser Val Asn Ser Val Gly Lys Ile Glu Cys
355 360 365
Val Ser Ala Glu Thr Thr Glu Asp Cys Ile Ala Lys Ile Met Asn Gly
370 375 380
Glu Ala Asp Ala Met Ser Leu Asp Gly Gly Phe Val Tyr Ile Ala Gly
385 390 395 400
Lys Cys Gly Leu Val Pro Val Leu Ala Glu Asn Tyr Asn Lys Ser Asp
405 410 415
Asn Cys Glu Asp Thr Pro Glu Ala Gly Tyr Phe Ala Ile Ala Val Val
420 425 430
Lys Lys Ser Ala Ser Asp Leu Thr Trp Asp Asn Leu Lys Gly Lys Lys
435 440 445
Ser Cys His Thr Ala Val Gly Arg Thr Ala Gly Trp Asn Ile Pro Met
450 455 460
Gly Leu Leu Tyr Asn Lys Ile Asn His Cys Arg Phe Asp Glu Phe Phe
465 470 475 480
Ser Glu Gly Cys Ala Pro Gly Ser Lys Lys Asp Ser Ser Leu Cys Lys
485 490 495
Leu Cys Met Gly Ser Gly Leu Asn Leu Cys Glu Pro Asn Asn Lys Glu
500 505 510
Gly Tyr Tyr Gly Tyr Thr Gly Ala Phe Arg Cys Leu Val Glu Lys Gly
515 520 525
Asp Val Ala Phe Val Lys His Gln Thr Val Pro Gln Asn Thr Gly Gly
530 535 540
Lys Asn Pro Asp Pro Trp Ala Lys Asn Leu Asn Glu Lys Asp Tyr Glu
545 550 555 560
Leu Leu Cys Leu Asp Gly Thr Arg Lys Pro Val Glu Glu Tyr Ala Asn
565 570 575
Cys His Leu Ala Arg Ala Pro Asn His Ala Val Val Thr Arg Lys Asp
580 585 590
Lys Glu Ala Cys Val His Lys Ile Leu Arg Gln Gln Gln His Leu Phe
595 600 605
Gly Ser Asn Val Thr Asp Cys Ser Gly Asn Phe Cys Leu Phe Arg Ser
610 615 620
Glu Thr Lys Asp Leu Leu Phe Arg Asp Asp Thr Val Cys Leu Ala Lys
625 630 635 640
Leu His Asp Arg Asn Thr Tyr Glu Lys Tyr Leu Gly Glu Glu Tyr Val
645 650 655
Lys Ala Val Gly Asn Leu Arg Lys Cys Ser Thr Ser Ser Leu Leu Glu
660 665 670
Ala Cys Thr Phe Arg Arg Pro
675
<210> 54
<211> 232
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 54
Glu Pro Lys Ser Gln Asp Lys Thr His Thr Gln Pro Pro Gln Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Cys Glu Glu Gln
65 70 75 80
Tyr Gly Ser Thr Tyr Arg Cys Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Arg Cys His Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Lys Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Thr Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 55
<211> 227
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированный Fc-фрагмент
<400> 55
Glu Arg Lys Gln Gln Val Glu Gln Pro Pro Gln Pro Ala Pro Pro Val
1 5 10 15
Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
20 25 30
Tyr Ile Thr Arg Glu Pro Glu Val Thr Cys Val Val Val Asp Val Ser
35 40 45
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
50 55 60
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
65 70 75 80
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
85 90 95
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser
100 105 110
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
115 120 125
Val Tyr Thr Phe Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
130 135 140
Ser Leu Arg Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
145 150 155 160
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Lys
165 170 175
Pro Val Leu Asp Ser Asp Gly Ser Phe Arg Leu Glu Ser Arg Leu Thr
180 185 190
Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
195 200 205
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
210 215 220
Ser Leu Gly
225
<210> 56
<211> 57
<212> PRT/Белок
<213> Homo sapiens
<400> 56
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Asn Ser Ala Phe
50 55
<210> 57
<211> 6
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 57
Ile Glu Gly Arg Met Asp
1 5
<210> 58
<211> 291
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 58
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Pro Gln Asn Ser Ala Phe Ile Glu Gly Arg Met Asp Glu
50 55 60
Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
65 70 75 80
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
85 90 95
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
100 105 110
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
115 120 125
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
130 135 140
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
145 150 155 160
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser
165 170 175
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
180 185 190
Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
195 200 205
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
210 215 220
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
225 230 235 240
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr
245 250 255
Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
260 265 270
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
275 280 285
Ser Leu Gly
290
<210> 59
<211> 309
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 59
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Pro Gln Asn Ser Ala Phe Ile Glu Gly Arg Met
65 70 75 80
Asp Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu
85 90 95
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
100 105 110
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
115 120 125
Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
130 135 140
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
145 150 155 160
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
165 170 175
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
180 185 190
Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
195 200 205
Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn
210 215 220
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
225 230 235 240
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
245 250 255
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg
260 265 270
Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys
275 280 285
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
290 295 300
Ser Leu Ser Leu Gly
305
<210> 60
<211> 936
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 60
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgccctgagt ggtctacagc ttggggccct tgctctacca cctgtggact cggcatggcc 120
accagagtgt ctaaccagaa cagattctgc cggctggaaa cccagcggag actgtgtctg 180
tccagacctt gtcctcctag ccggggcaga tcccctcaga actctgcctt tatcgagggc 240
agaatggacg agtctaagta cggccctcct tgtccaccat gtcctgctcc agaagctgct 300
ggcggccctt ccgtgtttct gttccctcca aagcctaagg acaccctgat gatctctcgg 360
acccctgaag tgacctgcgt ggtggtggat gtgtcccaag aggatcccga ggtgcagttc 420
aattggtacg tggacggcgt ggaagtgcac aacgccaaga ccaagcctag agaggaacag 480
ttcaactcca cctacagagt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 540
ggcaaagagt acaagtgcaa ggtgtccaac aagggcctgc cttccagcat cgaaaagacc 600
atctccaagg ccaagggcca gcctagggaa ccccaggttt acaccctgcc tccaagccaa 660
gaggaaatga ccaagaacca ggtgtccctg acctgcctgg tcaagggctt ctacccttcc 720
gatatcgccg tggaatggga gagcaatggc cagcctgaga acaactacaa gaccacacct 780
cctgtgctgg actccgacgg ctccttcttt ctgtactccc gcctgaccgt ggacaagtcc 840
agatggcaag agggcaacgt gttctcctgc tccgtgatgc acgaggccct gcacaatcac 900
tacacccaga agtccctgtc tctgtccctg ggctaa 936
<210> 61
<211> 1009
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 61
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgc cctgagtggt ctacagcttg 120
gggcccttgc tctaccacct gtggactcgg catggccacc agagtgtcta accagaacag 180
attctgccgg ctggaaaccc agcggagact gtgtctgtcc agaccttgtc ctcctagccg 240
gggcagatcc cctcagaact ctgcctttat cgagggcaga atggacgagt ctaagtacgg 300
ccctccttgt ccaccatgtc ctgctccaga agctgctggc ggcccttccg tgtttctgtt 360
ccctccaaag cctaaggaca ccctgatgat ctctcggacc cctgaagtga cctgcgtggt 420
ggtggatgtg tcccaagagg atcccgaggt gcagttcaat tggtacgtgg acggcgtgga 480
agtgcacaac gccaagacca agcctagaga ggaacagttc aactccacct acagagtggt 540
gtccgtgctg accgtgctgc accaggattg gctgaacggc aaagagtaca agtgcaaggt 600
gtccaacaag ggcctgcctt ccagcatcga aaagaccatc tccaaggcca agggccagcc 660
tagggaaccc caggtttaca ccctgcctcc aagccaagag gaaatgacca agaaccaggt 720
gtccctgacc tgcctggtca agggcttcta cccttccgat atcgccgtgg aatgggagag 780
caatggccag cctgagaaca actacaagac cacacctcct gtgctggact ccgacggctc 840
cttctttctg tactcccgcc tgaccgtgga caagtccaga tggcaagagg gcaacgtgtt 900
ctcctgctcc gtgatgcacg aggccctgca caatcactac acccagaagt ccctgtctct 960
gtccctgggc taatctagaa acccagcttt cttgtacaaa gtggtcccc 1009
<210> 62
<211> 56
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Модифицированная последовательность CCN5
<400> 62
Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
35 40 45
Gly Arg Ser Leu Gln Asn Ser Ala
50 55
<210> 63
<211> 4
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 63
Gly Arg Met Asp
1
<210> 64
<211> 306
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 64
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Leu Gln Asn Ser Ala Gly Arg Met Asp Glu Ser
65 70 75 80
Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
85 90 95
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
100 105 110
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln
115 120 125
Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val
130 135 140
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr
145 150 155 160
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
165 170 175
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile
180 185 190
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
195 200 205
Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser
210 215 220
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
225 230 235 240
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
245 250 255
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val
260 265 270
Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met
275 280 285
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
290 295 300
Leu Gly
305
<210> 65
<211> 6
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 65
Thr Glu Gly Arg Met Asp
1 5
<210> 66
<211> 305
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 66
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Ser Lys
65 70 75 80
Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
85 90 95
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
100 105 110
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
115 120 125
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
130 135 140
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
145 150 155 160
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
165 170 175
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
180 185 190
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
195 200 205
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
210 215 220
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
225 230 235 240
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
245 250 255
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
260 265 270
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
275 280 285
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
290 295 300
Gly
305
<210> 67
<211> 8
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 67
Thr Ala Glu Ala Ala Ala Lys Ala
1 5
<210> 68
<211> 307
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 68
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Pro Gln Thr Ala Glu Ala Ala Ala Lys Ala Glu
65 70 75 80
Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
85 90 95
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
100 105 110
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
115 120 125
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
130 135 140
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
145 150 155 160
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
165 170 175
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser
180 185 190
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
195 200 205
Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
210 215 220
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
225 230 235 240
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
245 250 255
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr
260 265 270
Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
275 280 285
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
290 295 300
Ser Leu Gly
305
<210> 69
<211> 304
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 69
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Pro Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
50 55 60
Ser Arg Gly Arg Ser Pro Gln Thr Glu Gly Arg Met Asp Glu Arg Lys
65 70 75 80
Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro
85 90 95
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
100 105 110
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp
115 120 125
Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
130 135 140
Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val
145 150 155 160
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
165 170 175
Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys
180 185 190
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
195 200 205
Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
210 215 220
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
225 230 235 240
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
245 250 255
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys
260 265 270
Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu
275 280 285
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
290 295 300
<210> 70
<211> 294
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 70
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
65 70 75 80
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
85 90 95
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
100 105 110
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
115 120 125
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
130 135 140
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
145 150 155 160
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
165 170 175
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
180 185 190
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
195 200 205
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
210 215 220
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
225 230 235 240
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
245 250 255
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
260 265 270
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
275 280 285
Leu Ser Leu Ser Leu Gly
290
<210> 71
<211> 891
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 71
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgcgccgagt ggtctacagc ttggggccct tgttctacca cctgtggcct cggcatggcc 120
accagagtgt ccaaccagaa cagattctgc cggctggaaa cccagcggag actgtgtttg 180
tccagacctt gcgaggccgc tgccaaagaa agaaagtgct gcgtggaatg ccctccttgt 240
cctgctcctc ctgtggctgg cccttccgtg tttctgttcc ctccaaagcc taaggacacc 300
ctgatgatct ctcggacccc tgaagtgacc tgcgtggtgg tggatgtgtc ccaagaggat 360
cccgaggtgc agttcaattg gtacgtggac ggcgtggaag tgcacaacgc caagaccaag 420
cctagagagg aacagttcaa ctccacctac agagtggtgt ccgtgctgac cgtgctgcac 480
caggattggc tgaacggcaa agagtacaag tgcaaggtgt ccaacaaggg actgccctcc 540
agcatcgaaa agaccatctc caaggccaag ggacagccca gagaacccca ggtgtacaca 600
ctgcctccaa gccaagagga aatgaccaag aaccaggtgt ccctgacctg cctggtcaag 660
ggcttctacc cttccgatat cgccgtggaa tgggagtcca atggccagcc tgagaacaac 720
tacaagacca cacctccagt gctggactcc gacggctcct tctttctgta ctcccgcctg 780
accgtggaca agtccagatg gcaagagggc aacgtgttct cctgctccgt gatgcacgag 840
gccctgcaca atcactacac ccagaagtcc ctgtctctgt ccctgggcta a 891
<210> 72
<211> 964
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 72
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgc gccgagtggt ctacagcttg 120
gggcccttgt tctaccacct gtggcctcgg catggccacc agagtgtcca accagaacag 180
attctgccgg ctggaaaccc agcggagact gtgtttgtcc agaccttgcg aggccgctgc 240
caaagaaaga aagtgctgcg tggaatgccc tccttgtcct gctcctcctg tggctggccc 300
ttccgtgttt ctgttccctc caaagcctaa ggacaccctg atgatctctc ggacccctga 360
agtgacctgc gtggtggtgg atgtgtccca agaggatccc gaggtgcagt tcaattggta 420
cgtggacggc gtggaagtgc acaacgccaa gaccaagcct agagaggaac agttcaactc 480
cacctacaga gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga 540
gtacaagtgc aaggtgtcca acaagggact gccctccagc atcgaaaaga ccatctccaa 600
ggccaaggga cagcccagag aaccccaggt gtacacactg cctccaagcc aagaggaaat 660
gaccaagaac caggtgtccc tgacctgcct ggtcaagggc ttctaccctt ccgatatcgc 720
cgtggaatgg gagtccaatg gccagcctga gaacaactac aagaccacac ctccagtgct 780
ggactccgac ggctccttct ttctgtactc ccgcctgacc gtggacaagt ccagatggca 840
agagggcaac gtgttctcct gctccgtgat gcacgaggcc ctgcacaatc actacaccca 900
gaagtccctg tctctgtccc tgggctaatc tagaaaccca gctttcttgt acaaagtggt 960
cccc 964
<210> 73
<211> 276
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 73
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro
50 55 60
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
65 70 75 80
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
85 90 95
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
100 105 110
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
115 120 125
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
130 135 140
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
145 150 155 160
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
165 170 175
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
180 185 190
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
195 200 205
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
210 215 220
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
225 230 235 240
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
245 250 255
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
260 265 270
Leu Ser Leu Gly
275
<210> 74
<211> 294
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 74
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala
65 70 75 80
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
85 90 95
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
100 105 110
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
115 120 125
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
130 135 140
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
145 150 155 160
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
165 170 175
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
180 185 190
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
195 200 205
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
210 215 220
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
225 230 235 240
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
245 250 255
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
260 265 270
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
275 280 285
Leu Ser Leu Ser Leu Gly
290
<210> 75
<211> 891
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 75
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaagaa agaaagtgct gcgtggaatg ccctccttgt 240
cctgctcctc ctgtggctgg cccttccgtg tttctgttcc ctccaaagcc taaggacacc 300
ctgatgatct ctcggacccc tgaagtgacc tgcgtggtgg tggatgtgtc ccaagaggat 360
cccgaggtgc agttcaattg gtacgtggac ggcgtggaag tgcacaacgc caagaccaag 420
cctagagagg aacagttcaa ctccacctac agagtggtgt ccgtgctgac cgtgctgcac 480
caggattggc tgaacggcaa agagtacaag tgcaaggtgt ccaacaaggg cctgccttcc 540
agcatcgaaa agaccatctc caaggccaag ggacagccca gagaacccca ggtgtacaca 600
ctgcctccaa gccaagagga aatgaccaag aaccaggtgt ccctgacctg cctggtcaag 660
ggcttctacc cttccgatat cgccgtggaa tgggagtcca atggccagcc tgagaacaac 720
tacaagacca cacctccagt gctggactcc gacggctcct tctttctgta ctcccgcctg 780
accgtggaca agtccagatg gcaagagggc aacgtgttct cctgctccgt gatgcacgag 840
gccctgcaca atcactacac ccagaagtcc ctgtctctgt ccctgggcta a 891
<210> 76
<211> 964
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 76
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaagaaaga aagtgctgcg tggaatgccc tccttgtcct gctcctcctg tggctggccc 300
ttccgtgttt ctgttccctc caaagcctaa ggacaccctg atgatctctc ggacccctga 360
agtgacctgc gtggtggtgg atgtgtccca agaggatccc gaggtgcagt tcaattggta 420
cgtggacggc gtggaagtgc acaacgccaa gaccaagcct agagaggaac agttcaactc 480
cacctacaga gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga 540
gtacaagtgc aaggtgtcca acaagggcct gccttccagc atcgaaaaga ccatctccaa 600
ggccaaggga cagcccagag aaccccaggt gtacacactg cctccaagcc aagaggaaat 660
gaccaagaac caggtgtccc tgacctgcct ggtcaagggc ttctaccctt ccgatatcgc 720
cgtggaatgg gagtccaatg gccagcctga gaacaactac aagaccacac ctccagtgct 780
ggactccgac ggctccttct ttctgtactc ccgcctgacc gtggacaagt ccagatggca 840
agagggcaac gtgttctcct gctccgtgat gcacgaggcc ctgcacaatc actacaccca 900
gaagtccctg tctctgtccc tgggctaatc tagaaaccca gctttcttgt acaaagtggt 960
cccc 964
<210> 77
<211> 343
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 77
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
20 25 30
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
35 40 45
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
50 55 60
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
65 70 75 80
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
85 90 95
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
100 105 110
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
115 120 125
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
130 135 140
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
145 150 155 160
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
165 170 175
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
180 185 190
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
195 200 205
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
210 215 220
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
225 230 235 240
Leu Ser Leu Ser Leu Gly Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala
245 250 255
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
260 265 270
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala
275 280 285
Ala Ala Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
290 295 300
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
305 310 315 320
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro
325 330 335
Ser Arg Gly Arg Ser Pro Gln
340
<210> 78
<211> 1038
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 78
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctccag cgcctactcc 60
gagtctaagt acggccctcc ttgtcctcca tgtcctgctc cagaagctgc tggcggccct 120
tccgtgtttc tgttccctcc aaagcctaag gacaccctga tgatctctcg gacccctgaa 180
gtgacctgcg tggtggtgga tgtgtcccaa gaggatcccg aggtgcagtt caattggtac 240
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gttcaactcc 300
acctacagag tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 360
tacaagtgca aggtgtccaa caagggcctg ccttccagca tcgaaaagac catctccaag 420
gccaagggcc agcctaggga accccaggtt tacaccctgc ctccaagcca agaggaaatg 480
accaagaacc aggtgtccct gacctgcctg gtcaagggct tctacccttc cgatatcgcc 540
gtggaatggg agagcaatgg ccagcctgag aacaactaca agaccacacc tcctgtgctg 600
gactccgacg gctccttctt tctgtactcc cgcctgaccg tggacaagtc cagatggcaa 660
gagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaatca ctacacccag 720
aagtccctgt ctctgtccct gggagctgag gccgctgcta aagaagctgc cgctaaagag 780
gccgcagcca aagaggcagc cgccaaagaa gccgctgcaa aagaggctgc tgcaaaagaa 840
gcagcagcta aagaagctgc tgccaaggcc gctgcttgtg ccgaatggtc tacagcttgg 900
ggcccttgct ctaccacctg tggactcggc atggccacca gagtgtctaa ccagaacaga 960
ttctgccggc tggaaaccca gcggagactg tgcctgtcta gaccctgtcc tcctagcaga 1020
ggcagatccc ctcagtga 1038
<210> 79
<211> 1111
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 79
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctccagcgc ctactccgag tctaagtacg gccctccttg 120
tcctccatgt cctgctccag aagctgctgg cggcccttcc gtgtttctgt tccctccaaa 180
gcctaaggac accctgatga tctctcggac ccctgaagtg acctgcgtgg tggtggatgt 240
gtcccaagag gatcccgagg tgcagttcaa ttggtacgtg gacggcgtgg aagtgcacaa 300
cgccaagacc aagcctagag aggaacagtt caactccacc tacagagtgg tgtccgtgct 360
gaccgtgctg caccaggatt ggctgaacgg caaagagtac aagtgcaagg tgtccaacaa 420
gggcctgcct tccagcatcg aaaagaccat ctccaaggcc aagggccagc ctagggaacc 480
ccaggtttac accctgcctc caagccaaga ggaaatgacc aagaaccagg tgtccctgac 540
ctgcctggtc aagggcttct acccttccga tatcgccgtg gaatgggaga gcaatggcca 600
gcctgagaac aactacaaga ccacacctcc tgtgctggac tccgacggct ccttctttct 660
gtactcccgc ctgaccgtgg acaagtccag atggcaagag ggcaacgtgt tctcctgctc 720
cgtgatgcac gaggccctgc acaatcacta cacccagaag tccctgtctc tgtccctggg 780
agctgaggcc gctgctaaag aagctgccgc taaagaggcc gcagccaaag aggcagccgc 840
caaagaagcc gctgcaaaag aggctgctgc aaaagaagca gcagctaaag aagctgctgc 900
caaggccgct gcttgtgccg aatggtctac agcttggggc ccttgctcta ccacctgtgg 960
actcggcatg gccaccagag tgtctaacca gaacagattc tgccggctgg aaacccagcg 1020
gagactgtgc ctgtctagac cctgtcctcc tagcagaggc agatcccctc agtgatctag 1080
aaacccagct ttcttgtaca aagtggtccc c 1111
<210> 80
<211> 311
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 80
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
50 55 60
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
65 70 75 80
Ala Ala Ala Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro
85 90 95
Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
100 105 110
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
115 120 125
Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
130 135 140
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
145 150 155 160
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
165 170 175
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
180 185 190
Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
195 200 205
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr
210 215 220
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
225 230 235 240
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
245 250 255
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
260 265 270
Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe
275 280 285
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
290 295 300
Ser Leu Ser Leu Ser Leu Gly
305 310
<210> 81
<211> 329
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 81
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala
65 70 75 80
Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala
85 90 95
Lys Glu Ala Ala Ala Lys Glu Arg Lys Cys Cys Val Glu Cys Pro Pro
100 105 110
Cys Pro Ala Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu
225 230 235 240
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Leu Gly
325
<210> 82
<211> 996
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 82
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaagag gctgctgcta aagaagccgc cgcaaaagag 240
gcagcagcaa aagaggctgc cgccaaagag gccgcagcca aagaagcagc agctaaagag 300
gccgctgcaa aagaacggaa gtgctgcgtg gaatgccctc cttgtcctgc tcctcctgtg 360
gctggccctt ccgtgtttct gttccctcca aagcctaagg acaccctgat gatctctcgg 420
acccctgaag tgacctgcgt ggtggtggat gtgtcccaag aggatcccga ggtgcagttc 480
aattggtacg tggacggcgt ggaagtgcac aacgccaaga ccaagcctag agaggaacag 540
ttcaactcca cctacagagt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 600
ggcaaagagt acaagtgcaa ggtgtccaac aagggcctgc cttccagcat cgaaaagacc 660
atctccaagg ccaagggaca gcccagagaa ccccaggtgt acacactgcc tccaagccaa 720
gaggaaatga ccaagaacca ggtgtccctg acctgcctgg tcaagggctt ctacccttcc 780
gatatcgccg tggaatggga gtccaatggc cagcctgaga acaactacaa gaccacacct 840
ccagtgctgg actccgacgg ctccttcttt ctgtactccc gcctgaccgt ggacaagtcc 900
agatggcaag agggcaacgt gttctcctgc tccgtgatgc acgaggccct gcacaatcac 960
tacacccaga agtccctgtc tctgtccctg ggctaa 996
<210> 83
<211> 1069
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 83
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaagaggct gctgctaaag aagccgccgc aaaagaggca gcagcaaaag aggctgccgc 300
caaagaggcc gcagccaaag aagcagcagc taaagaggcc gctgcaaaag aacggaagtg 360
ctgcgtggaa tgccctcctt gtcctgctcc tcctgtggct ggcccttccg tgtttctgtt 420
ccctccaaag cctaaggaca ccctgatgat ctctcggacc cctgaagtga cctgcgtggt 480
ggtggatgtg tcccaagagg atcccgaggt gcagttcaat tggtacgtgg acggcgtgga 540
agtgcacaac gccaagacca agcctagaga ggaacagttc aactccacct acagagtggt 600
gtccgtgctg accgtgctgc accaggattg gctgaacggc aaagagtaca agtgcaaggt 660
gtccaacaag ggcctgcctt ccagcatcga aaagaccatc tccaaggcca agggacagcc 720
cagagaaccc caggtgtaca cactgcctcc aagccaagag gaaatgacca agaaccaggt 780
gtccctgacc tgcctggtca agggcttcta cccttccgat atcgccgtgg aatgggagtc 840
caatggccag cctgagaaca actacaagac cacacctcca gtgctggact ccgacggctc 900
cttctttctg tactcccgcc tgaccgtgga caagtccaga tggcaagagg gcaacgtgtt 960
ctcctgctcc gtgatgcacg aggccctgca caatcactac acccagaagt ccctgtctct 1020
gtccctgggc taatctagaa acccagcttt cttgtacaaa gtggtcccc 1069
<210> 84
<211> 276
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 84
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Arg Lys Gln Gln Val Glu Gln Pro Pro Gln Pro Ala Pro Pro
50 55 60
Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
65 70 75 80
Leu Tyr Ile Thr Arg Glu Pro Glu Val Thr Cys Val Val Val Asp Val
85 90 95
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
100 105 110
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
115 120 125
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
130 135 140
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
145 150 155 160
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
165 170 175
Gln Val Tyr Thr Phe Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
180 185 190
Val Ser Leu Arg Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
195 200 205
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
210 215 220
Lys Pro Val Leu Asp Ser Asp Gly Ser Phe Arg Leu Glu Ser Arg Leu
225 230 235 240
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
245 250 255
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
260 265 270
Leu Ser Leu Gly
275
<210> 85
<211> 294
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 85
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Arg Lys Gln Gln Val Glu Gln Pro Pro Gln Pro Ala
65 70 75 80
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
85 90 95
Asp Thr Leu Tyr Ile Thr Arg Glu Pro Glu Val Thr Cys Val Val Val
100 105 110
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
115 120 125
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
130 135 140
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
145 150 155 160
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
165 170 175
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
180 185 190
Glu Pro Gln Val Tyr Thr Phe Pro Pro Ser Gln Glu Glu Met Thr Lys
195 200 205
Asn Gln Val Ser Leu Arg Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
210 215 220
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
225 230 235 240
Thr Thr Lys Pro Val Leu Asp Ser Asp Gly Ser Phe Arg Leu Glu Ser
245 250 255
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
260 265 270
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
275 280 285
Leu Ser Leu Ser Leu Gly
290
<210> 86
<211> 891
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 86
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaagaa agaaagcagc aggtcgagca gcctcctcag 240
cctgctcctc ctgttgctgg cccttccgtg tttctgttcc ctccaaagcc taaggacacc 300
ctgtacatca cccgcgagcc tgaagtgacc tgcgtggtgg tggatgtgtc ccaagaggat 360
cccgaggtgc agttcaattg gtacgtggac ggcgtggaag tgcacaacgc caagaccaag 420
cctagagagg aacagttcaa ctccacctac agagtggtgt ccgtgctgac cgtgctgcac 480
caggattggc tgaacggcaa agagtacaag tgcaaggtgt ccaacaaggg cctgccttcc 540
agcatcgaaa agaccatctc caaggccaag ggacagccca gagaacccca ggtgtacaca 600
ttccctccat ctcaagagga aatgaccaag aaccaggtgt ccctgcggtg cctggtcaag 660
ggcttctacc cttctgatat cgccgtggaa tgggagtcca acggccagcc tgagaacaac 720
tacaagacca ccaagcctgt gctggactcc gacggctcct tccggcttga atctagactg 780
accgtggaca agtcccggtg gcaagagggc aacgtgttct cctgctctgt gatgcacgag 840
gccctgcaca accactacac ccagaagtcc ctgtctctgt ccctgggcta a 891
<210> 87
<211> 964
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 87
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaagaaaga aagcagcagg tcgagcagcc tcctcagcct gctcctcctg ttgctggccc 300
ttccgtgttt ctgttccctc caaagcctaa ggacaccctg tacatcaccc gcgagcctga 360
agtgacctgc gtggtggtgg atgtgtccca agaggatccc gaggtgcagt tcaattggta 420
cgtggacggc gtggaagtgc acaacgccaa gaccaagcct agagaggaac agttcaactc 480
cacctacaga gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga 540
gtacaagtgc aaggtgtcca acaagggcct gccttccagc atcgaaaaga ccatctccaa 600
ggccaaggga cagcccagag aaccccaggt gtacacattc cctccatctc aagaggaaat 660
gaccaagaac caggtgtccc tgcggtgcct ggtcaagggc ttctaccctt ctgatatcgc 720
cgtggaatgg gagtccaacg gccagcctga gaacaactac aagaccacca agcctgtgct 780
ggactccgac ggctccttcc ggcttgaatc tagactgacc gtggacaagt cccggtggca 840
agagggcaac gtgttctcct gctctgtgat gcacgaggcc ctgcacaacc actacaccca 900
gaagtccctg tctctgtccc tgggctaatc tagaaaccca gctttcttgt acaaagtggt 960
cccc 964
<210> 88
<211> 281
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 88
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Pro Lys Ser Gln Asp Lys Thr His Thr Gln Pro Pro Gln Pro
50 55 60
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
65 70 75 80
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
85 90 95
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
100 105 110
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Cys Glu Glu
115 120 125
Gln Tyr Gly Ser Thr Tyr Arg Cys Val Ser Val Leu Thr Val Leu His
130 135 140
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
145 150 155 160
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
165 170 175
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
180 185 190
Thr Lys Asn Gln Val Ser Leu Arg Cys His Val Lys Gly Phe Tyr Pro
195 200 205
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
210 215 220
Tyr Lys Thr Thr Lys Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
225 230 235 240
Tyr Ser Thr Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
245 250 255
Phe Ser Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln
260 265 270
Lys Ser Leu Ser Leu Ser Pro Gly Lys
275 280
<210> 89
<211> 299
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 89
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Pro Lys Ser Gln Asp Lys Thr His Thr Gln Pro Pro
65 70 75 80
Gln Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
85 90 95
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
100 105 110
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
115 120 125
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Cys
130 135 140
Glu Glu Gln Tyr Gly Ser Thr Tyr Arg Cys Val Ser Val Leu Thr Val
145 150 155 160
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
165 170 175
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
180 185 190
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
195 200 205
Glu Leu Thr Lys Asn Gln Val Ser Leu Arg Cys His Val Lys Gly Phe
210 215 220
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
225 230 235 240
Asn Asn Tyr Lys Thr Thr Lys Pro Val Leu Asp Ser Asp Gly Ser Phe
245 250 255
Phe Leu Tyr Ser Thr Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
260 265 270
Asn Val Phe Ser Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr
275 280 285
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
290 295
<210> 90
<211> 906
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 90
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaagag cctaagagcc aggacaagac ccacacacag 240
cctccacagc ctgctccaga attgctcgga ggcccttccg tgtttctgtt ccctccaaag 300
cctaaggaca ccctgatgat ctctcggacc cctgaagtga cctgcgtggt ggtggatgtg 360
tctcacgagg atcccgaagt gaagttcaat tggtacgtgg acggcgtgga agtgcacaac 420
gccaagacaa agccctgcga ggaacagtac ggctccacct acagatgcgt gtccgtgctg 480
acagtgctgc accaggattg gctgaacggc aaagagtaca agtgcaaggt gtccaacaag 540
gccctgcctg ctcctatcga aaagaccatc tccaaggcca agggccagcc tagagaaccc 600
caggtgtaca cactgccacc ttctagggac gagctgacca agaaccaggt gtccctgaga 660
tgccacgtga agggcttcta cccctccgat atcgccgtgg aatgggagtc taatggacag 720
cccgagaaca actacaagac caccaagcct gtgctggact ccgacggctc cttcttcctg 780
tactctaccc tgaccgtgga caagtccaga tggcagcagg gcaacgtgtt ctcctgctct 840
gtgctgcacg aggccctgca caatcactac acccagaagt ccctgtctct gtcccctggc 900
aagtga 906
<210> 91
<211> 979
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 91
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaagagcct aagagccagg acaagaccca cacacagcct ccacagcctg ctccagaatt 300
gctcggaggc ccttccgtgt ttctgttccc tccaaagcct aaggacaccc tgatgatctc 360
tcggacccct gaagtgacct gcgtggtggt ggatgtgtct cacgaggatc ccgaagtgaa 420
gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc aagacaaagc cctgcgagga 480
acagtacggc tccacctaca gatgcgtgtc cgtgctgaca gtgctgcacc aggattggct 540
gaacggcaaa gagtacaagt gcaaggtgtc caacaaggcc ctgcctgctc ctatcgaaaa 600
gaccatctcc aaggccaagg gccagcctag agaaccccag gtgtacacac tgccaccttc 660
tagggacgag ctgaccaaga accaggtgtc cctgagatgc cacgtgaagg gcttctaccc 720
ctccgatatc gccgtggaat gggagtctaa tggacagccc gagaacaact acaagaccac 780
caagcctgtg ctggactccg acggctcctt cttcctgtac tctaccctga ccgtggacaa 840
gtccagatgg cagcagggca acgtgttctc ctgctctgtg ctgcacgagg ccctgcacaa 900
tcactacacc cagaagtccc tgtctctgtc ccctggcaag tgatctagaa acccagcttt 960
cttgtacaaa gtggtcccc 979
<210> 92
<211> 402
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитая метка
<400> 92
Gly His His His His His His Gly Ser Glu Ile Gly Thr Gly Phe Pro
1 5 10 15
Phe Asp Pro His Tyr Val Glu Val Leu Gly Glu Arg Met His Tyr Val
20 25 30
Asp Val Gly Pro Arg Asp Gly Thr Pro Val Leu Phe Leu His Gly Asn
35 40 45
Pro Thr Ser Ser Tyr Val Trp Arg Asn Ile Ile Pro His Val Ala Pro
50 55 60
Thr His Arg Cys Ile Ala Pro Asp Leu Ile Gly Met Gly Lys Ser Asp
65 70 75 80
Lys Pro Asp Leu Gly Tyr Phe Phe Asp Asp His Val Arg Phe Met Asp
85 90 95
Ala Phe Ile Glu Ala Leu Gly Leu Glu Glu Val Val Leu Val Ile His
100 105 110
Asp Trp Gly Ser Ala Leu Gly Phe His Trp Ala Lys Arg Asn Pro Glu
115 120 125
Arg Val Lys Gly Ile Ala Phe Met Glu Phe Ile Arg Pro Ile Pro Thr
130 135 140
Trp Asp Glu Trp Pro Glu Phe Ala Arg Glu Thr Phe Gln Ala Phe Arg
145 150 155 160
Thr Thr Asp Val Gly Arg Lys Leu Ile Ile Asp Gln Asn Val Phe Ile
165 170 175
Glu Gly Thr Leu Pro Met Gly Val Val Arg Pro Leu Thr Glu Val Glu
180 185 190
Met Asp His Tyr Arg Glu Pro Phe Leu Asn Pro Val Asp Arg Glu Pro
195 200 205
Leu Trp Arg Phe Pro Asn Glu Leu Pro Ile Ala Gly Glu Pro Ala Asn
210 215 220
Ile Val Ala Leu Val Glu Glu Tyr Met Asp Trp Leu His Gln Ser Pro
225 230 235 240
Val Pro Lys Leu Leu Phe Trp Gly Thr Pro Gly Val Leu Ile Pro Pro
245 250 255
Ala Glu Ala Ala Arg Leu Ala Lys Ser Leu Pro Asn Cys Lys Ala Val
260 265 270
Asp Ile Gly Pro Gly Leu Asn Leu Leu Gln Glu Asp Asn Pro Asp Leu
275 280 285
Ile Gly Ser Glu Ile Ala Arg Trp Leu Ser Thr Leu Glu Ile Ser Gly
290 295 300
Leu Gln Asp Ser Glu Val Asn Gln Glu Ala Lys Pro Glu Val Lys Pro
305 310 315 320
Glu Val Lys Pro Glu Thr His Ile Asn Leu Lys Val Ser Asp Gly Ser
325 330 335
Ser Glu Ile Phe Phe Lys Ile Lys Lys Thr Thr Pro Leu Arg Arg Leu
340 345 350
Met Glu Ala Phe Ala Lys Arg Gln Gly Lys Glu Met Asp Ser Leu Thr
355 360 365
Phe Leu Tyr Asp Gly Ile Glu Ile Gln Ala Asp Gln Thr Pro Glu Asp
370 375 380
Leu Asp Met Glu Asp Asn Asp Ile Ile Glu Ala His Arg Glu Gln Ile
385 390 395 400
Gly Gly
<210> 93
<211> 20
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 93
Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Ser Gly Gly
1 5 10 15
Gly Ser Ser Gly
20
<210> 94
<211> 466
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 94
Gly His His His His His His Gly Ser Glu Ile Gly Thr Gly Phe Pro
1 5 10 15
Phe Asp Pro His Tyr Val Glu Val Leu Gly Glu Arg Met His Tyr Val
20 25 30
Asp Val Gly Pro Arg Asp Gly Thr Pro Val Leu Phe Leu His Gly Asn
35 40 45
Pro Thr Ser Ser Tyr Val Trp Arg Asn Ile Ile Pro His Val Ala Pro
50 55 60
Thr His Arg Cys Ile Ala Pro Asp Leu Ile Gly Met Gly Lys Ser Asp
65 70 75 80
Lys Pro Asp Leu Gly Tyr Phe Phe Asp Asp His Val Arg Phe Met Asp
85 90 95
Ala Phe Ile Glu Ala Leu Gly Leu Glu Glu Val Val Leu Val Ile His
100 105 110
Asp Trp Gly Ser Ala Leu Gly Phe His Trp Ala Lys Arg Asn Pro Glu
115 120 125
Arg Val Lys Gly Ile Ala Phe Met Glu Phe Ile Arg Pro Ile Pro Thr
130 135 140
Trp Asp Glu Trp Pro Glu Phe Ala Arg Glu Thr Phe Gln Ala Phe Arg
145 150 155 160
Thr Thr Asp Val Gly Arg Lys Leu Ile Ile Asp Gln Asn Val Phe Ile
165 170 175
Glu Gly Thr Leu Pro Met Gly Val Val Arg Pro Leu Thr Glu Val Glu
180 185 190
Met Asp His Tyr Arg Glu Pro Phe Leu Asn Pro Val Asp Arg Glu Pro
195 200 205
Leu Trp Arg Phe Pro Asn Glu Leu Pro Ile Ala Gly Glu Pro Ala Asn
210 215 220
Ile Val Ala Leu Val Glu Glu Tyr Met Asp Trp Leu His Gln Ser Pro
225 230 235 240
Val Pro Lys Leu Leu Phe Trp Gly Thr Pro Gly Val Leu Ile Pro Pro
245 250 255
Ala Glu Ala Ala Arg Leu Ala Lys Ser Leu Pro Asn Cys Lys Ala Val
260 265 270
Asp Ile Gly Pro Gly Leu Asn Leu Leu Gln Glu Asp Asn Pro Asp Leu
275 280 285
Ile Gly Ser Glu Ile Ala Arg Trp Leu Ser Thr Leu Glu Ile Ser Gly
290 295 300
Leu Gln Asp Ser Glu Val Asn Gln Glu Ala Lys Pro Glu Val Lys Pro
305 310 315 320
Glu Val Lys Pro Glu Thr His Ile Asn Leu Lys Val Ser Asp Gly Ser
325 330 335
Ser Glu Ile Phe Phe Lys Ile Lys Lys Thr Thr Pro Leu Arg Arg Leu
340 345 350
Met Glu Ala Phe Ala Lys Arg Gln Gly Lys Glu Met Asp Ser Leu Thr
355 360 365
Phe Leu Tyr Asp Gly Ile Glu Ile Gln Ala Asp Gln Thr Pro Glu Asp
370 375 380
Leu Asp Met Glu Asp Asn Asp Ile Ile Glu Ala His Arg Glu Gln Ile
385 390 395 400
Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Ser
405 410 415
Gly Gly Gly Ser Ser Gly Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala
420 425 430
Cys Ser Lys Ser Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg
435 440 445
Asn Arg Gln Cys Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg
450 455 460
Pro Cys
465
<210> 95
<211> 1461
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 95
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
ggccaccacc atcaccatca cggctccgag atcggaaccg gctttccttt cgaccctcac 120
tacgtggaag tgctgggcga gagaatgcac tatgtggacg tgggccccag agatggaacc 180
cctgtgctgt ttctgcacgg caaccctacc tccagctacg tgtggcggaa catcatccct 240
cacgtggccc ctacacacag atgtatcgcc cctgacctga tcggcatggg caagtctgac 300
aagcctgacc tgggctactt cttcgacgac cacgtgcggt tcatggacgc ctttatcgag 360
gctctgggcc tcgaagaggt ggtgctggtc atccatgatt ggggctctgc cctgggcttt 420
cactgggcca agagaaaccc cgagagagtg aagggaatcg ccttcatgga attcatccgg 480
cctattccta cctgggacga gtggcctgag ttcgccagag agacattcca ggccttcaga 540
accaccgacg tgggcagaaa gctgatcatc gaccagaacg tgttcatcga gggcaccctg 600
cctatgggag tcgtcagacc tctgaccgag gtggaaatgg accactacag agagcccttt 660
ctgaaccccg tggaccggga acctctttgg agattcccta acgagctgcc tatcgctggc 720
gagcctgcca atattgtggc cctggtggaa gagtacatgg actggctgca tcagagcccc 780
gtgcctaagc tgctgttttg gggaacaccc ggcgtgctga ttcctcctgc tgaagctgct 840
agactggcca agagcctgcc taactgcaag gccgtggata tcggccctgg cctgaatctg 900
ctgcaagagg acaaccccga tctgatcgga tctgagatcg cccggtggct gagcaccctg 960
gaaatcagtg gactgcagga ctccgaagtg aatcaagagg ccaagcctga agtgaagccc 1020
gaagtcaagc ctgagacaca catcaacctg aaggtgtccg acggctccag cgagatcttc 1080
ttcaagatca agaaaaccac acctctgcgg cggctgatgg aagcctttgc caagagacag 1140
ggcaaagaga tggactccct gaccttcctg tacgacggca tcgagatcca ggccgatcag 1200
acccctgagg acctggacat ggaagataac gacatcattg aggcccacag agagcagatc 1260
ggcggctctg gtggtagcgg aggttctggt ggatctggtg gttcttctgg cggcggatct 1320
tctggctgtg ctgagcagac aaccgagtgg accgcctgct ctaagtcttg tggcatgggc 1380
ttctccacca gagtgaccaa ccggaacaga cagtgcgaga tgctgaagca gacccggctg 1440
tgtatggtcc gaccttgcta a 1461
<210> 96
<211> 1552
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 96
ggggacaagt ttgtacaaaa aagcaggcta taagcttgct gccaccatga aatgggtcac 60
ctttatctcc ctgctgttcc tgttctcctc cgcctactct ggccaccacc atcaccatca 120
cggctccgag atcggaaccg gctttccttt cgaccctcac tacgtggaag tgctgggcga 180
gagaatgcac tatgtggacg tgggccccag agatggaacc cctgtgctgt ttctgcacgg 240
caaccctacc tccagctacg tgtggcggaa catcatccct cacgtggccc ctacacacag 300
atgtatcgcc cctgacctga tcggcatggg caagtctgac aagcctgacc tgggctactt 360
cttcgacgac cacgtgcggt tcatggacgc ctttatcgag gctctgggcc tcgaagaggt 420
ggtgctggtc atccatgatt ggggctctgc cctgggcttt cactgggcca agagaaaccc 480
cgagagagtg aagggaatcg ccttcatgga attcatccgg cctattccta cctgggacga 540
gtggcctgag ttcgccagag agacattcca ggccttcaga accaccgacg tgggcagaaa 600
gctgatcatc gaccagaacg tgttcatcga gggcaccctg cctatgggag tcgtcagacc 660
tctgaccgag gtggaaatgg accactacag agagcccttt ctgaaccccg tggaccggga 720
acctctttgg agattcccta acgagctgcc tatcgctggc gagcctgcca atattgtggc 780
cctggtggaa gagtacatgg actggctgca tcagagcccc gtgcctaagc tgctgttttg 840
gggaacaccc ggcgtgctga ttcctcctgc tgaagctgct agactggcca agagcctgcc 900
taactgcaag gccgtggata tcggccctgg cctgaatctg ctgcaagagg acaaccccga 960
tctgatcgga tctgagatcg cccggtggct gagcaccctg gaaatcagtg gactgcagga 1020
ctccgaagtg aatcaagagg ccaagcctga agtgaagccc gaagtcaagc ctgagacaca 1080
catcaacctg aaggtgtccg acggctccag cgagatcttc ttcaagatca agaaaaccac 1140
acctctgcgg cggctgatgg aagcctttgc caagagacag ggcaaagaga tggactccct 1200
gaccttcctg tacgacggca tcgagatcca ggccgatcag acccctgagg acctggacat 1260
ggaagataac gacatcattg aggcccacag agagcagatc ggcggctctg gtggtagcgg 1320
aggttctggt ggatctggtg gttcttctgg cggcggatct tctggctgtg ctgagcagac 1380
aaccgagtgg accgcctgct ctaagtcttg tggcatgggc ttctccacca gagtgaccaa 1440
ccggaacaga cagtgcgaga tgctgaagca gacccggctg tgtatggtcc gaccttgcta 1500
aatctagagc ggccgcggta ccaacccagc tttcttgtac aaagtggtcc cc 1552
<210> 97
<211> 634
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 97
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
1 5 10 15
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
20 25 30
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly
50 55 60
Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu
65 70 75 80
Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr
85 90 95
Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp
100 105 110
Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr
115 120 125
Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu
130 135 140
Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn
145 150 155 160
Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe
165 170 175
His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala
180 185 190
Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys
195 200 205
Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala
210 215 220
Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala
225 230 235 240
Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly
245 250 255
Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe
260 265 270
Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr
275 280 285
Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp
290 295 300
Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile
305 310 315 320
Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser
325 330 335
His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro
340 345 350
Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr
355 360 365
Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala
370 375 380
Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys
385 390 395 400
Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His
405 410 415
Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu
420 425 430
Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly
435 440 445
Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val
450 455 460
Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly
465 470 475 480
Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro
485 490 495
Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu
500 505 510
His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu
515 520 525
Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu
530 535 540
Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala
545 550 555 560
Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr
565 570 575
Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln
580 585 590
Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys
595 600 605
Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu
610 615 620
Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
625 630
<210> 98
<211> 652
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 98
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr
20 25 30
Cys Gly Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys
35 40 45
Arg Leu Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp
65 70 75 80
Leu Gly Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln
85 90 95
Tyr Leu Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu
100 105 110
Val Thr Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn
115 120 125
Cys Asp Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val
130 135 140
Ala Thr Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys
145 150 155 160
Gln Glu Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn
165 170 175
Pro Asn Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr
180 185 190
Ala Phe His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu
195 200 205
Ile Ala Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe
210 215 220
Ala Lys Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp
225 230 235 240
Lys Ala Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly
245 250 255
Lys Ala Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys
260 265 270
Phe Gly Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln
275 280 285
Arg Phe Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp
290 295 300
Leu Thr Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys
305 310 315 320
Ala Asp Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp
325 330 335
Ser Ile Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu
340 345 350
Lys Ser His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp
355 360 365
Leu Pro Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys
370 375 380
Asn Tyr Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu
385 390 395 400
Tyr Ala Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu
405 410 415
Ala Lys Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp
420 425 430
Pro His Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val
435 440 445
Glu Glu Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln
450 455 460
Leu Gly Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys
465 470 475 480
Lys Val Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn
485 490 495
Leu Gly Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg
500 505 510
Met Pro Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys
515 520 525
Val Leu His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys
530 535 540
Thr Glu Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val
545 550 555 560
Asp Glu Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe
565 570 575
His Ala Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys
580 585 590
Gln Thr Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys
595 600 605
Glu Gln Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys
610 615 620
Cys Cys Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys
625 630 635 640
Lys Leu Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
645 650
<210> 99
<211> 1965
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 99
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgcgccgagt ggtctacagc ttggggccct tgttctacca cctgtggcct cggcatggcc 120
accagagtgt ccaaccagaa cagattctgc cggctggaaa cccagcggag actgtgcttg 180
tctagacctt gcgaggccgc tgccaaggac gctcataagt ctgaggtggc ccaccggttc 240
aaggacctgg gcgaagagaa cttcaaggcc ctggtgctga tcgccttcgc tcagtacttg 300
cagcagtgcc ccttcgagga ccacgtgaag ctggtcaacg aagtgaccga gttcgccaag 360
acctgcgtgg ccgatgagtc tgccgagaac tgcgacaagt ctctgcacac cctgttcggc 420
gacaagctgt gtaccgtggc taccctgaga gaaacctacg gcgagatggc cgactgctgc 480
gctaagcaag agcccgagag aaacgagtgc ttcctgcagc acaaggacga caaccctaac 540
ctgcctagac tcgtgcggcc tgaggtggac gtgatgtgta ccgccttcca cgacaacgag 600
gaaaccttcc tgaagaagta cctgtacgag atcgccagac ggcaccccta cttttacgcc 660
cctgagctgc tgttcttcgc caagcggtac aaggccgcct tcaccgagtg ttgtcaggcc 720
gctgataagg ccgcttgcct gctgcctaaa ctggacgagc tgagagatga aggcaaggcc 780
tccagcgcca agcagagact gaagtgtgcc agcctgcaga agttcggcga gagagccttt 840
aaggcctggg ccgtcgctag actgtcccag agatttccca aggccgagtt tgccgaggtg 900
tccaagctgg ttaccgacct gaccaaggtg cacaccgaat gctgtcacgg cgacctgctg 960
gaatgcgccg atgatagagc cgatctggcc aagtacatct gcgagaacca ggactccatc 1020
tcctccaagc tgaaagagtg ctgcgagaag cctctgctgg aaaagtccca ctgtatcgcc 1080
gaggtggaaa acgacgagat gcctgccgat ctgccttctc tggccgccga cttcgtggaa 1140
tctaaggacg tgtgcaagaa ctacgccgag gctaaggatg tgttcctggg catgtttctg 1200
tacgagtacg ctcggcggca ccccgactat tctgttgtgc tgctgctgag actggctaag 1260
acctacgaga caaccctcga gaagtgctgt gccgccgctg atcctcacga gtgttacgcc 1320
aaggtgttcg acgagttcaa gccactggtg gaagaacccc agaacctgat caagcagaat 1380
tgcgagctgt tcgagcagct gggcgagtac aagttccaga acgccctgct cgtgcggtac 1440
accaagaaag tgccccaggt gtccacacct acactggttg aggtgtcccg gaacctgggc 1500
aaagtgggct ctaagtgctg caagcacccc gaggccaaga gaatgccttg tgccgaggac 1560
tacctgtccg tggtgctgaa ccagctgtgc gtgctgcacg aaaagacccc tgtgtccgac 1620
agagtgacca agtgctgtac cgagagcctg gtcaacagac ggccttgctt ctctgccctg 1680
gaagtggacg agacatacgt gcccaaagag ttcaacgccg agacattcac cttccacgcc 1740
gacatctgca ccctgtccga gaaagagcgg cagatcaaga aacagaccgc tctggtggaa 1800
ctggtcaagc acaagcccaa ggccaccaaa gaacagctga aggccgtgat ggacgacttc 1860
gccgcctttg tggaaaagtg ttgcaaggcc gacgacaaag agacatgctt cgccgaagag 1920
ggcaagaaac tggtggccgc ttctcaggct gctctgggac tttaa 1965
<210> 100
<211> 2038
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 100
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgc gccgagtggt ctacagcttg 120
gggcccttgt tctaccacct gtggcctcgg catggccacc agagtgtcca accagaacag 180
attctgccgg ctggaaaccc agcggagact gtgcttgtct agaccttgcg aggccgctgc 240
caaggacgct cataagtctg aggtggccca ccggttcaag gacctgggcg aagagaactt 300
caaggccctg gtgctgatcg ccttcgctca gtacttgcag cagtgcccct tcgaggacca 360
cgtgaagctg gtcaacgaag tgaccgagtt cgccaagacc tgcgtggccg atgagtctgc 420
cgagaactgc gacaagtctc tgcacaccct gttcggcgac aagctgtgta ccgtggctac 480
cctgagagaa acctacggcg agatggccga ctgctgcgct aagcaagagc ccgagagaaa 540
cgagtgcttc ctgcagcaca aggacgacaa ccctaacctg cctagactcg tgcggcctga 600
ggtggacgtg atgtgtaccg ccttccacga caacgaggaa accttcctga agaagtacct 660
gtacgagatc gccagacggc acccctactt ttacgcccct gagctgctgt tcttcgccaa 720
gcggtacaag gccgccttca ccgagtgttg tcaggccgct gataaggccg cttgcctgct 780
gcctaaactg gacgagctga gagatgaagg caaggcctcc agcgccaagc agagactgaa 840
gtgtgccagc ctgcagaagt tcggcgagag agcctttaag gcctgggccg tcgctagact 900
gtcccagaga tttcccaagg ccgagtttgc cgaggtgtcc aagctggtta ccgacctgac 960
caaggtgcac accgaatgct gtcacggcga cctgctggaa tgcgccgatg atagagccga 1020
tctggccaag tacatctgcg agaaccagga ctccatctcc tccaagctga aagagtgctg 1080
cgagaagcct ctgctggaaa agtcccactg tatcgccgag gtggaaaacg acgagatgcc 1140
tgccgatctg ccttctctgg ccgccgactt cgtggaatct aaggacgtgt gcaagaacta 1200
cgccgaggct aaggatgtgt tcctgggcat gtttctgtac gagtacgctc ggcggcaccc 1260
cgactattct gttgtgctgc tgctgagact ggctaagacc tacgagacaa ccctcgagaa 1320
gtgctgtgcc gccgctgatc ctcacgagtg ttacgccaag gtgttcgacg agttcaagcc 1380
actggtggaa gaaccccaga acctgatcaa gcagaattgc gagctgttcg agcagctggg 1440
cgagtacaag ttccagaacg ccctgctcgt gcggtacacc aagaaagtgc cccaggtgtc 1500
cacacctaca ctggttgagg tgtcccggaa cctgggcaaa gtgggctcta agtgctgcaa 1560
gcaccccgag gccaagagaa tgccttgtgc cgaggactac ctgtccgtgg tgctgaacca 1620
gctgtgcgtg ctgcacgaaa agacccctgt gtccgacaga gtgaccaagt gctgtaccga 1680
gagcctggtc aacagacggc cttgcttctc tgccctggaa gtggacgaga catacgtgcc 1740
caaagagttc aacgccgaga cattcacctt ccacgccgac atctgcaccc tgtccgagaa 1800
agagcggcag atcaagaaac agaccgctct ggtggaactg gtcaagcaca agcccaaggc 1860
caccaaagaa cagctgaagg ccgtgatgga cgacttcgcc gcctttgtgg aaaagtgttg 1920
caaggccgac gacaaagaga catgcttcgc cgaagagggc aagaaactgg tggccgcttc 1980
tcaggctgct ctgggacttt aatctagaaa cccagctttc ttgtacaaag tggtcccc 2038
<210> 101
<211> 582
<212> PRT/Белок
<213> Homo sapiens
<400> 101
Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly Glu
1 5 10 15
Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu Gln
20 25 30
Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr Glu
35 40 45
Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp Lys
50 55 60
Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr Leu
65 70 75 80
Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu Pro
85 90 95
Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn Leu
100 105 110
Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe His
115 120 125
Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala Arg
130 135 140
Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys Arg
145 150 155 160
Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala Ala
165 170 175
Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala Ser
180 185 190
Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly Glu
195 200 205
Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe Pro
210 215 220
Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr Lys
225 230 235 240
Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp Asp
245 250 255
Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile Ser
260 265 270
Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser His
275 280 285
Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro Ser
290 295 300
Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr Ala
305 310 315 320
Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala Arg
325 330 335
Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys Thr
340 345 350
Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His Glu
355 360 365
Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu Pro
370 375 380
Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly Glu
385 390 395 400
Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val Pro
405 410 415
Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly Lys
420 425 430
Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro Cys
435 440 445
Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu His
450 455 460
Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu Ser
465 470 475 480
Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu Thr
485 490 495
Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala Asp
500 505 510
Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr Ala
515 520 525
Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln Leu
530 535 540
Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys Lys
545 550 555 560
Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu Val
565 570 575
Ala Ala Ser Gln Ala Ala
580
<210> 102
<211> 675
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 102
Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly Glu
1 5 10 15
Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu Gln
20 25 30
Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr Glu
35 40 45
Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp Lys
50 55 60
Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr Leu
65 70 75 80
Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu Pro
85 90 95
Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn Leu
100 105 110
Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe His
115 120 125
Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala Arg
130 135 140
Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys Arg
145 150 155 160
Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala Ala
165 170 175
Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala Ser
180 185 190
Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly Glu
195 200 205
Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe Pro
210 215 220
Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr Lys
225 230 235 240
Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp Asp
245 250 255
Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile Ser
260 265 270
Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser His
275 280 285
Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro Ser
290 295 300
Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr Ala
305 310 315 320
Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala Arg
325 330 335
Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys Thr
340 345 350
Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His Glu
355 360 365
Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu Pro
370 375 380
Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly Glu
385 390 395 400
Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val Pro
405 410 415
Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly Lys
420 425 430
Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro Cys
435 440 445
Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu His
450 455 460
Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu Ser
465 470 475 480
Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu Thr
485 490 495
Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala Asp
500 505 510
Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr Ala
515 520 525
Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln Leu
530 535 540
Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys Lys
545 550 555 560
Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu Val
565 570 575
Ala Ala Ser Gln Ala Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
580 585 590
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
595 600 605
Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Cys Ala
610 615 620
Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly Leu Gly
625 630 635 640
Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu Glu Thr
645 650 655
Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg Gly Arg
660 665 670
Ser Pro Gln
675
<210> 103
<211> 693
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 103
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu
20 25 30
Gly Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr
35 40 45
Leu Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val
50 55 60
Thr Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys
65 70 75 80
Asp Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala
85 90 95
Thr Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln
100 105 110
Glu Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro
115 120 125
Asn Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala
130 135 140
Phe His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile
145 150 155 160
Ala Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala
165 170 175
Lys Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys
180 185 190
Ala Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys
195 200 205
Ala Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe
210 215 220
Gly Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg
225 230 235 240
Phe Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu
245 250 255
Thr Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala
260 265 270
Asp Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser
275 280 285
Ile Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys
290 295 300
Ser His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu
305 310 315 320
Pro Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn
325 330 335
Tyr Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr
340 345 350
Ala Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala
355 360 365
Lys Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro
370 375 380
His Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu
385 390 395 400
Glu Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu
405 410 415
Gly Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys
420 425 430
Val Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu
435 440 445
Gly Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met
450 455 460
Pro Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val
465 470 475 480
Leu His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr
485 490 495
Glu Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp
500 505 510
Glu Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His
515 520 525
Ala Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln
530 535 540
Thr Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu
545 550 555 560
Gln Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys
565 570 575
Cys Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys
580 585 590
Leu Val Ala Ala Ser Gln Ala Ala Glu Ala Ala Ala Lys Glu Ala Ala
595 600 605
Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala
610 615 620
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
625 630 635 640
Cys Ala Glu Trp Ser Thr Ala Trp Gly Pro Cys Ser Thr Thr Cys Gly
645 650 655
Leu Gly Met Ala Thr Arg Val Ser Asn Gln Asn Arg Phe Cys Arg Leu
660 665 670
Glu Thr Gln Arg Arg Leu Cys Leu Ser Arg Pro Cys Pro Pro Ser Arg
675 680 685
Gly Arg Ser Pro Gln
690
<210> 104
<211> 2088
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 104
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
gacgcccaca agtctgaggt ggcccacaga ttcaaggacc tgggcgaaga gaacttcaag 120
gccctggtgc tgatcgcctt cgctcagtac ttgcagcagt gccccttcga ggaccacgtg 180
aagctggtca acgaagtgac cgagttcgcc aagacctgcg tggccgatga gtctgccgag 240
aactgcgaca agtctctgca caccctgttc ggcgacaagc tgtgtaccgt ggctaccctg 300
agagaaacct acggcgagat ggccgactgc tgcgctaagc aagagcccga gagaaacgag 360
tgcttcctgc agcacaagga cgacaaccct aacctgccta gactcgtgcg gcctgaggtg 420
gacgtgatgt gtaccgcctt ccacgacaac gaggaaacct tcctgaagaa gtacctgtac 480
gagatcgcca gacggcaccc ctacttttac gcccctgagc tgctgttctt cgccaagcgg 540
tacaaggccg ccttcaccga gtgttgtcag gccgctgata aggccgcttg cctgctgcct 600
aaactggacg agctgagaga tgaaggcaag gcctccagcg ccaagcagag actgaagtgt 660
gccagcctgc agaagttcgg cgagagagcc tttaaggcct gggccgtcgc tagactgtcc 720
cagagatttc ccaaggccga gtttgccgag gtgtccaagc tggttaccga cctgaccaag 780
gtgcacaccg aatgctgtca cggcgacctg ctggaatgcg ccgatgatag agccgatctg 840
gccaagtaca tctgcgagaa ccaggactcc atctcctcca agctgaaaga gtgctgcgag 900
aagcctctgc tggaaaagtc ccactgtatc gccgaggtgg aaaacgacga gatgcctgcc 960
gatctgcctt ctctggccgc cgacttcgtg gaatctaagg acgtgtgcaa gaactacgcc 1020
gaggccaagg atgtgttcct gggcatgttt ctgtacgagt acgctcggcg gcaccccgac 1080
tattctgttg tgctgctgct gagactggct aagacctacg agacaaccct cgagaagtgc 1140
tgtgccgccg ctgatcctca cgagtgttac gccaaggtgt tcgacgagtt caagccactg 1200
gtggaagaac cccagaacct gatcaagcag aattgcgagc tgttcgagca gctgggcgag 1260
tacaagttcc agaacgccct gctcgtgcgg tacaccaaga aagtgcccca ggtgtccaca 1320
cctacactgg ttgaggtgtc ccggaacctg ggcaaagtgg gctctaagtg ctgcaagcac 1380
cctgaggcca agagaatgcc ttgcgccgag gactacctgt ccgtggtgct gaatcagctg 1440
tgcgtgctgc acgaaaagac ccctgtgtcc gacagagtga ccaagtgctg taccgagagc 1500
ctggtcaaca gacggccttg cttctctgcc ctggaagtgg acgagacata cgtgcccaaa 1560
gagttcaacg ccgagacatt caccttccac gccgacatct gcaccctgtc cgagaaagag 1620
cggcagatca agaaacagac cgctctggtg gaactggtca agcacaagcc caaggccacc 1680
aaagaacagc tgaaggccgt gatggacgac ttcgccgcct ttgtggaaaa gtgttgcaag 1740
gccgacgaca aagagacatg cttcgccgaa gagggcaaga aactggtggc cgcttctcag 1800
gctgctgagg ccgctgctaa agaggctgcc gctaaagaag ccgcagccaa agaggcagct 1860
gcaaaagaag ctgctgcaaa agaggcagcc gccaaagagg ccgctgctaa agaagcagcc 1920
gccaagtgtg ctgagtggtc tacagcttgg ggcccctgct ctacaacctg tggactcggc 1980
atggccacca gagtgtctaa ccagaacaga ttctgccggc tggaaaccca gcggagactg 2040
tgcctgtcta gaccctgtcc tcctagcaga ggcagatccc ctcagtga 2088
<210> 105
<211> 2161
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 105
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactctgac gcccacaagt ctgaggtggc 120
ccacagattc aaggacctgg gcgaagagaa cttcaaggcc ctggtgctga tcgccttcgc 180
tcagtacttg cagcagtgcc ccttcgagga ccacgtgaag ctggtcaacg aagtgaccga 240
gttcgccaag acctgcgtgg ccgatgagtc tgccgagaac tgcgacaagt ctctgcacac 300
cctgttcggc gacaagctgt gtaccgtggc taccctgaga gaaacctacg gcgagatggc 360
cgactgctgc gctaagcaag agcccgagag aaacgagtgc ttcctgcagc acaaggacga 420
caaccctaac ctgcctagac tcgtgcggcc tgaggtggac gtgatgtgta ccgccttcca 480
cgacaacgag gaaaccttcc tgaagaagta cctgtacgag atcgccagac ggcaccccta 540
cttttacgcc cctgagctgc tgttcttcgc caagcggtac aaggccgcct tcaccgagtg 600
ttgtcaggcc gctgataagg ccgcttgcct gctgcctaaa ctggacgagc tgagagatga 660
aggcaaggcc tccagcgcca agcagagact gaagtgtgcc agcctgcaga agttcggcga 720
gagagccttt aaggcctggg ccgtcgctag actgtcccag agatttccca aggccgagtt 780
tgccgaggtg tccaagctgg ttaccgacct gaccaaggtg cacaccgaat gctgtcacgg 840
cgacctgctg gaatgcgccg atgatagagc cgatctggcc aagtacatct gcgagaacca 900
ggactccatc tcctccaagc tgaaagagtg ctgcgagaag cctctgctgg aaaagtccca 960
ctgtatcgcc gaggtggaaa acgacgagat gcctgccgat ctgccttctc tggccgccga 1020
cttcgtggaa tctaaggacg tgtgcaagaa ctacgccgag gccaaggatg tgttcctggg 1080
catgtttctg tacgagtacg ctcggcggca ccccgactat tctgttgtgc tgctgctgag 1140
actggctaag acctacgaga caaccctcga gaagtgctgt gccgccgctg atcctcacga 1200
gtgttacgcc aaggtgttcg acgagttcaa gccactggtg gaagaacccc agaacctgat 1260
caagcagaat tgcgagctgt tcgagcagct gggcgagtac aagttccaga acgccctgct 1320
cgtgcggtac accaagaaag tgccccaggt gtccacacct acactggttg aggtgtcccg 1380
gaacctgggc aaagtgggct ctaagtgctg caagcaccct gaggccaaga gaatgccttg 1440
cgccgaggac tacctgtccg tggtgctgaa tcagctgtgc gtgctgcacg aaaagacccc 1500
tgtgtccgac agagtgacca agtgctgtac cgagagcctg gtcaacagac ggccttgctt 1560
ctctgccctg gaagtggacg agacatacgt gcccaaagag ttcaacgccg agacattcac 1620
cttccacgcc gacatctgca ccctgtccga gaaagagcgg cagatcaaga aacagaccgc 1680
tctggtggaa ctggtcaagc acaagcccaa ggccaccaaa gaacagctga aggccgtgat 1740
ggacgacttc gccgcctttg tggaaaagtg ttgcaaggcc gacgacaaag agacatgctt 1800
cgccgaagag ggcaagaaac tggtggccgc ttctcaggct gctgaggccg ctgctaaaga 1860
ggctgccgct aaagaagccg cagccaaaga ggcagctgca aaagaagctg ctgcaaaaga 1920
ggcagccgcc aaagaggccg ctgctaaaga agcagccgcc aagtgtgctg agtggtctac 1980
agcttggggc ccctgctcta caacctgtgg actcggcatg gccaccagag tgtctaacca 2040
gaacagattc tgccggctgg aaacccagcg gagactgtgc ctgtctagac cctgtcctcc 2100
tagcagaggc agatcccctc agtgatctag aaacccagct ttcttgtaca aagtggtccc 2160
c 2161
<210> 106
<211> 634
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 106
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp Leu Gly
50 55 60
Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln Tyr Leu
65 70 75 80
Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu Val Thr
85 90 95
Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn Cys Asp
100 105 110
Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val Ala Thr
115 120 125
Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys Gln Glu
130 135 140
Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn Pro Asn
145 150 155 160
Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr Ala Phe
165 170 175
His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu Ile Ala
180 185 190
Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe Ala Lys
195 200 205
Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp Lys Ala
210 215 220
Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly Lys Ala
225 230 235 240
Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys Phe Gly
245 250 255
Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln Arg Phe
260 265 270
Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp Leu Thr
275 280 285
Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys Ala Asp
290 295 300
Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp Ser Ile
305 310 315 320
Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu Lys Ser
325 330 335
His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp Leu Pro
340 345 350
Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys Asn Tyr
355 360 365
Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu Tyr Ala
370 375 380
Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu Ala Lys
385 390 395 400
Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp Pro His
405 410 415
Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val Glu Glu
420 425 430
Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln Leu Gly
435 440 445
Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys Lys Val
450 455 460
Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn Leu Gly
465 470 475 480
Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg Met Pro
485 490 495
Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys Val Leu
500 505 510
His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys Thr Glu
515 520 525
Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val Asp Glu
530 535 540
Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe His Ala
545 550 555 560
Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys Gln Thr
565 570 575
Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys Glu Gln
580 585 590
Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys Cys Cys
595 600 605
Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys Lys Leu
610 615 620
Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
625 630
<210> 107
<211> 652
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 107
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys Asp
65 70 75 80
Leu Gly Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala Gln
85 90 95
Tyr Leu Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn Glu
100 105 110
Val Thr Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu Asn
115 120 125
Cys Asp Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr Val
130 135 140
Ala Thr Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala Lys
145 150 155 160
Gln Glu Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp Asn
165 170 175
Pro Asn Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys Thr
180 185 190
Ala Phe His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr Glu
195 200 205
Ile Ala Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe Phe
210 215 220
Ala Lys Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala Asp
225 230 235 240
Lys Ala Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu Gly
245 250 255
Lys Ala Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln Lys
260 265 270
Phe Gly Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser Gln
275 280 285
Arg Phe Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr Asp
290 295 300
Leu Thr Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu Cys
305 310 315 320
Ala Asp Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln Asp
325 330 335
Ser Ile Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu Glu
340 345 350
Lys Ser His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala Asp
355 360 365
Leu Pro Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys Lys
370 375 380
Asn Tyr Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr Glu
385 390 395 400
Tyr Ala Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg Leu
405 410 415
Ala Lys Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala Asp
420 425 430
Pro His Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu Val
435 440 445
Glu Glu Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu Gln
450 455 460
Leu Gly Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr Lys
465 470 475 480
Lys Val Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg Asn
485 490 495
Leu Gly Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys Arg
500 505 510
Met Pro Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu Cys
515 520 525
Val Leu His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys Cys
530 535 540
Thr Glu Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu Val
545 550 555 560
Asp Glu Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr Phe
565 570 575
His Ala Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys Lys
580 585 590
Gln Thr Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr Lys
595 600 605
Glu Gln Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu Lys
610 615 620
Cys Cys Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly Lys
625 630 635 640
Lys Leu Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
645 650
<210> 108
<211> 1965
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 108
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaggat gctcataagt ctgaggtggc ccaccggttc 240
aaggacctgg gcgaagagaa cttcaaggcc ctggtgctga tcgccttcgc tcagtacttg 300
cagcagtgcc ccttcgagga ccacgtgaag ctggtcaacg aagtgaccga gttcgccaag 360
acctgcgtgg ccgatgagtc tgccgagaac tgcgacaagt ctctgcacac cctgttcggc 420
gacaagctgt gtaccgtggc taccctgaga gaaacctacg gcgagatggc cgactgctgc 480
gctaagcaag agcccgagag aaacgagtgc ttcctgcagc acaaggacga caaccctaac 540
ctgcctagac tcgtgcggcc tgaggtggac gtgatgtgta ccgccttcca cgacaacgag 600
gaaaccttcc tgaagaagta cctgtacgag atcgccagac ggcaccccta cttttacgcc 660
cctgagctgc tgttcttcgc caagcggtac aaggccgcct tcaccgagtg ttgtcaggcc 720
gctgataagg ccgcttgcct gctgcctaaa ctggacgagc tgagagatga aggcaaggcc 780
tccagcgcca agcagagact gaagtgtgcc agcctgcaga agttcggcga gagagccttt 840
aaggcctggg ccgtcgctag actgtcccag agatttccca aggccgagtt tgccgaggtg 900
tccaagctgg ttaccgacct gaccaaggtg cacaccgaat gctgtcacgg cgacctgctg 960
gaatgcgccg atgatagagc cgatctggcc aagtacatct gcgagaacca ggactccatc 1020
tcctccaagc tgaaagagtg ctgcgagaag cctctgctgg aaaagtccca ctgtatcgcc 1080
gaggtggaaa acgacgagat gcctgccgat ctgccttctc tggccgccga cttcgtggaa 1140
tctaaggacg tgtgcaagaa ctacgccgag gctaaggatg tgttcctggg catgtttctg 1200
tacgagtacg ctcggcggca ccccgattat agtgtggtgc tgctgctgag actggctaag 1260
acctacgaga caaccctcga gaagtgctgt gccgccgctg atcctcacga gtgttacgcc 1320
aaggtgttcg acgagttcaa gccactggtg gaagaacccc agaacctgat caagcagaat 1380
tgcgagctgt tcgagcagct gggcgagtac aagttccaga acgccctgct cgtgcggtac 1440
accaagaaag tgccccaggt gtccacacct acactggttg aggtgtcccg gaacctgggc 1500
aaagtgggct ctaagtgctg caagcacccc gaggccaaga gaatgccttg cgccgaggat 1560
tacctgtccg tggtgctgaa ccagctgtgc gtgctgcacg aaaagacccc tgtgtccgac 1620
cgcgtgacca agtgctgtac agagtccctg gtcaacagac ggccctgctt ctctgccctg 1680
gaagtggacg agacatacgt gcccaaagag ttcaacgccg agacattcac cttccacgcc 1740
gacatctgca ccctgtccga gaaagagcgg cagatcaaga aacagaccgc tctggtcgaa 1800
ctggtcaagc acaagcccaa ggccaccaaa gaacagctga aggccgtgat ggacgacttc 1860
gccgcctttg tggaaaagtg ttgcaaggcc gacgacaaag agacatgctt cgccgaagag 1920
ggcaagaaac tggtggccgc ttctcaggct gctctgggac tttaa 1965
<210> 109
<211> 2038
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 109
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaggatgct cataagtctg aggtggccca ccggttcaag gacctgggcg aagagaactt 300
caaggccctg gtgctgatcg ccttcgctca gtacttgcag cagtgcccct tcgaggacca 360
cgtgaagctg gtcaacgaag tgaccgagtt cgccaagacc tgcgtggccg atgagtctgc 420
cgagaactgc gacaagtctc tgcacaccct gttcggcgac aagctgtgta ccgtggctac 480
cctgagagaa acctacggcg agatggccga ctgctgcgct aagcaagagc ccgagagaaa 540
cgagtgcttc ctgcagcaca aggacgacaa ccctaacctg cctagactcg tgcggcctga 600
ggtggacgtg atgtgtaccg ccttccacga caacgaggaa accttcctga agaagtacct 660
gtacgagatc gccagacggc acccctactt ttacgcccct gagctgctgt tcttcgccaa 720
gcggtacaag gccgccttca ccgagtgttg tcaggccgct gataaggccg cttgcctgct 780
gcctaaactg gacgagctga gagatgaagg caaggcctcc agcgccaagc agagactgaa 840
gtgtgccagc ctgcagaagt tcggcgagag agcctttaag gcctgggccg tcgctagact 900
gtcccagaga tttcccaagg ccgagtttgc cgaggtgtcc aagctggtta ccgacctgac 960
caaggtgcac accgaatgct gtcacggcga cctgctggaa tgcgccgatg atagagccga 1020
tctggccaag tacatctgcg agaaccagga ctccatctcc tccaagctga aagagtgctg 1080
cgagaagcct ctgctggaaa agtcccactg tatcgccgag gtggaaaacg acgagatgcc 1140
tgccgatctg ccttctctgg ccgccgactt cgtggaatct aaggacgtgt gcaagaacta 1200
cgccgaggct aaggatgtgt tcctgggcat gtttctgtac gagtacgctc ggcggcaccc 1260
cgattatagt gtggtgctgc tgctgagact ggctaagacc tacgagacaa ccctcgagaa 1320
gtgctgtgcc gccgctgatc ctcacgagtg ttacgccaag gtgttcgacg agttcaagcc 1380
actggtggaa gaaccccaga acctgatcaa gcagaattgc gagctgttcg agcagctggg 1440
cgagtacaag ttccagaacg ccctgctcgt gcggtacacc aagaaagtgc cccaggtgtc 1500
cacacctaca ctggttgagg tgtcccggaa cctgggcaaa gtgggctcta agtgctgcaa 1560
gcaccccgag gccaagagaa tgccttgcgc cgaggattac ctgtccgtgg tgctgaacca 1620
gctgtgcgtg ctgcacgaaa agacccctgt gtccgaccgc gtgaccaagt gctgtacaga 1680
gtccctggtc aacagacggc cctgcttctc tgccctggaa gtggacgaga catacgtgcc 1740
caaagagttc aacgccgaga cattcacctt ccacgccgac atctgcaccc tgtccgagaa 1800
agagcggcag atcaagaaac agaccgctct ggtcgaactg gtcaagcaca agcccaaggc 1860
caccaaagaa cagctgaagg ccgtgatgga cgacttcgcc gcctttgtgg aaaagtgttg 1920
caaggccgac gacaaagaga catgcttcgc cgaagagggc aagaaactgg tggccgcttc 1980
tcaggctgct ctgggacttt aatctagaaa cccagctttc ttgtacaaag tggtcccc 2038
<210> 110
<211> 669
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 110
Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser Cys Gly
1 5 10 15
Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys Glu Met
20 25 30
Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala Ala Ala
35 40 45
Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
50 55 60
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu
65 70 75 80
Ala Ala Ala Lys Asp Ala His Lys Ser Glu Val Ala His Arg Phe Lys
85 90 95
Asp Leu Gly Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala Phe Ala
100 105 110
Gln Tyr Leu Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu Val Asn
115 120 125
Glu Val Thr Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser Ala Glu
130 135 140
Asn Cys Asp Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu Cys Thr
145 150 155 160
Val Ala Thr Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys Cys Ala
165 170 175
Lys Gln Glu Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys Asp Asp
180 185 190
Asn Pro Asn Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val Met Cys
195 200 205
Thr Ala Phe His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr Leu Tyr
210 215 220
Glu Ile Ala Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu Leu Phe
225 230 235 240
Phe Ala Lys Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln Ala Ala
245 250 255
Asp Lys Ala Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg Asp Glu
260 265 270
Gly Lys Ala Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser Leu Gln
275 280 285
Lys Phe Gly Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg Leu Ser
290 295 300
Gln Arg Phe Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu Val Thr
305 310 315 320
Asp Leu Thr Lys Val His Thr Glu Cys Cys His Gly Asp Leu Leu Glu
325 330 335
Cys Ala Asp Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu Asn Gln
340 345 350
Asp Ser Ile Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro Leu Leu
355 360 365
Glu Lys Ser His Cys Ile Ala Glu Val Glu Asn Asp Glu Met Pro Ala
370 375 380
Asp Leu Pro Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp Val Cys
385 390 395 400
Lys Asn Tyr Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe Leu Tyr
405 410 415
Glu Tyr Ala Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu Leu Arg
420 425 430
Leu Ala Lys Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala Ala Ala
435 440 445
Asp Pro His Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys Pro Leu
450 455 460
Val Glu Glu Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu Phe Glu
465 470 475 480
Gln Leu Gly Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg Tyr Thr
485 490 495
Lys Lys Val Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val Ser Arg
500 505 510
Asn Leu Gly Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu Ala Lys
515 520 525
Arg Met Pro Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn Gln Leu
530 535 540
Cys Val Leu His Glu Lys Thr Pro Val Ser Asp Arg Val Thr Lys Cys
545 550 555 560
Cys Thr Glu Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala Leu Glu
565 570 575
Val Asp Glu Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr Phe Thr
580 585 590
Phe His Ala Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln Ile Lys
595 600 605
Lys Gln Thr Ala Leu Val Glu Leu Val Lys His Lys Pro Lys Ala Thr
610 615 620
Lys Glu Gln Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe Val Glu
625 630 635 640
Lys Cys Cys Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu Glu Gly
645 650 655
Lys Lys Leu Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
660 665
<210> 111
<211> 687
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 111
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Cys Ala Glu Gln Thr Thr Glu Trp Thr Ala Cys Ser Lys Ser
20 25 30
Cys Gly Met Gly Phe Ser Thr Arg Val Thr Asn Arg Asn Arg Gln Cys
35 40 45
Glu Met Leu Lys Gln Thr Arg Leu Cys Met Val Arg Pro Cys Glu Ala
50 55 60
Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala
65 70 75 80
Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala
85 90 95
Lys Glu Ala Ala Ala Lys Asp Ala His Lys Ser Glu Val Ala His Arg
100 105 110
Phe Lys Asp Leu Gly Glu Glu Asn Phe Lys Ala Leu Val Leu Ile Ala
115 120 125
Phe Ala Gln Tyr Leu Gln Gln Cys Pro Phe Glu Asp His Val Lys Leu
130 135 140
Val Asn Glu Val Thr Glu Phe Ala Lys Thr Cys Val Ala Asp Glu Ser
145 150 155 160
Ala Glu Asn Cys Asp Lys Ser Leu His Thr Leu Phe Gly Asp Lys Leu
165 170 175
Cys Thr Val Ala Thr Leu Arg Glu Thr Tyr Gly Glu Met Ala Asp Cys
180 185 190
Cys Ala Lys Gln Glu Pro Glu Arg Asn Glu Cys Phe Leu Gln His Lys
195 200 205
Asp Asp Asn Pro Asn Leu Pro Arg Leu Val Arg Pro Glu Val Asp Val
210 215 220
Met Cys Thr Ala Phe His Asp Asn Glu Glu Thr Phe Leu Lys Lys Tyr
225 230 235 240
Leu Tyr Glu Ile Ala Arg Arg His Pro Tyr Phe Tyr Ala Pro Glu Leu
245 250 255
Leu Phe Phe Ala Lys Arg Tyr Lys Ala Ala Phe Thr Glu Cys Cys Gln
260 265 270
Ala Ala Asp Lys Ala Ala Cys Leu Leu Pro Lys Leu Asp Glu Leu Arg
275 280 285
Asp Glu Gly Lys Ala Ser Ser Ala Lys Gln Arg Leu Lys Cys Ala Ser
290 295 300
Leu Gln Lys Phe Gly Glu Arg Ala Phe Lys Ala Trp Ala Val Ala Arg
305 310 315 320
Leu Ser Gln Arg Phe Pro Lys Ala Glu Phe Ala Glu Val Ser Lys Leu
325 330 335
Val Thr Asp Leu Thr Lys Val His Thr Glu Cys Cys His Gly Asp Leu
340 345 350
Leu Glu Cys Ala Asp Asp Arg Ala Asp Leu Ala Lys Tyr Ile Cys Glu
355 360 365
Asn Gln Asp Ser Ile Ser Ser Lys Leu Lys Glu Cys Cys Glu Lys Pro
370 375 380
Leu Leu Glu Lys Ser His Cys Ile Ala Glu Val Glu Asn Asp Glu Met
385 390 395 400
Pro Ala Asp Leu Pro Ser Leu Ala Ala Asp Phe Val Glu Ser Lys Asp
405 410 415
Val Cys Lys Asn Tyr Ala Glu Ala Lys Asp Val Phe Leu Gly Met Phe
420 425 430
Leu Tyr Glu Tyr Ala Arg Arg His Pro Asp Tyr Ser Val Val Leu Leu
435 440 445
Leu Arg Leu Ala Lys Thr Tyr Glu Thr Thr Leu Glu Lys Cys Cys Ala
450 455 460
Ala Ala Asp Pro His Glu Cys Tyr Ala Lys Val Phe Asp Glu Phe Lys
465 470 475 480
Pro Leu Val Glu Glu Pro Gln Asn Leu Ile Lys Gln Asn Cys Glu Leu
485 490 495
Phe Glu Gln Leu Gly Glu Tyr Lys Phe Gln Asn Ala Leu Leu Val Arg
500 505 510
Tyr Thr Lys Lys Val Pro Gln Val Ser Thr Pro Thr Leu Val Glu Val
515 520 525
Ser Arg Asn Leu Gly Lys Val Gly Ser Lys Cys Cys Lys His Pro Glu
530 535 540
Ala Lys Arg Met Pro Cys Ala Glu Asp Tyr Leu Ser Val Val Leu Asn
545 550 555 560
Gln Leu Cys Val Leu His Glu Lys Thr Pro Val Ser Asp Arg Val Thr
565 570 575
Lys Cys Cys Thr Glu Ser Leu Val Asn Arg Arg Pro Cys Phe Ser Ala
580 585 590
Leu Glu Val Asp Glu Thr Tyr Val Pro Lys Glu Phe Asn Ala Glu Thr
595 600 605
Phe Thr Phe His Ala Asp Ile Cys Thr Leu Ser Glu Lys Glu Arg Gln
610 615 620
Ile Lys Lys Gln Thr Ala Leu Val Glu Leu Val Lys His Lys Pro Lys
625 630 635 640
Ala Thr Lys Glu Gln Leu Lys Ala Val Met Asp Asp Phe Ala Ala Phe
645 650 655
Val Glu Lys Cys Cys Lys Ala Asp Asp Lys Glu Thr Cys Phe Ala Glu
660 665 670
Glu Gly Lys Lys Leu Val Ala Ala Ser Gln Ala Ala Leu Gly Leu
675 680 685
<210> 112
<211> 2070
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 112
gccaccatga aatgggtcac ctttatctcc ctgctgttcc tgttctcctc cgcctactct 60
tgtgccgagc agaccacaga gtggaccgcc tgctctaagt cttgcggcat gggcttctcc 120
accagagtga ccaaccggaa cagacagtgc gagatgctga agcagacccg gctgtgtatg 180
gttcgacctt gcgaggccgc tgccaaagag gctgctgcta aagaagccgc cgcaaaagag 240
gcagcagcaa aagaggctgc cgccaaagag gccgcagcca aagaagcagc agctaaagag 300
gccgctgcta aggacgccca caagtctgaa gtggcccacc ggtttaagga cctgggcgaa 360
gagaacttca aggccctggt gctgatcgcc ttcgctcagt acttgcagca gtgccccttc 420
gaggaccacg tgaagctggt caacgaagtg accgagttcg ccaagacctg cgtggccgat 480
gagtctgccg agaactgcga caagtctctg cacaccctgt tcggcgacaa gctgtgtacc 540
gtggctaccc tgagagaaac ctacggcgag atggccgact gctgcgctaa gcaagagccc 600
gagagaaacg agtgcttcct gcagcacaag gacgacaacc ctaacctgcc tagactcgtg 660
cggcctgagg tggacgtgat gtgtaccgcc ttccacgaca acgaggaaac cttcctgaag 720
aagtacctgt acgagatcgc cagacggcac ccctactttt acgcccctga gctgctgttt 780
ttcgccaagc ggtacaaggc cgccttcacc gagtgttgtc aggccgccga taaggccgct 840
tgtctgctgc ctaaactgga cgagctgcgc gacgaaggca aggcctcttc tgctaagcag 900
cggctgaagt gcgccagcct gcagaagttt ggcgagagag ccttcaaggc ttgggccgtc 960
gctagactgt cccagagatt tcccaaggcc gagtttgccg aggtgtccaa gctggttacc 1020
gacctgacca aggtgcacac cgaatgctgt cacggcgacc tgctggaatg cgccgatgat 1080
agagccgatc tggccaagta catctgcgag aaccaggact ccatctcctc caagctgaaa 1140
gagtgctgcg agaagcctct gctggaaaag tcccactgta tcgccgaggt ggaaaacgac 1200
gagatgcctg ccgatctgcc ttctctggcc gccgacttcg tggaatctaa ggacgtgtgc 1260
aagaactacg ccgaggccaa ggatgtgttc ctgggcatgt ttctgtacga gtacgctcgg 1320
cggcaccccg attatagtgt ggtgctgctg ctgagactgg ctaagaccta cgagacaacc 1380
ctcgagaagt gctgtgccgc cgctgatcct cacgagtgtt acgccaaggt gttcgacgag 1440
ttcaagccac tggtggaaga accccagaac ctgatcaagc agaattgcga gctgttcgag 1500
cagctgggcg agtacaagtt ccagaacgcc ctgctcgtgc ggtacaccaa gaaagtgccc 1560
caggtgtcca cacctacact ggttgaggtg tcccggaacc tgggcaaagt gggctctaag 1620
tgctgcaagc accctgaggc caagagaatg ccttgcgccg aggactacct gtccgtggtg 1680
ctgaatcagc tgtgcgtgct gcacgaaaag acccctgtgt ccgaccgcgt gaccaagtgc 1740
tgtacagagt ccctggtcaa cagacggccc tgcttctctg ccctggaagt ggacgagaca 1800
tacgtgccca aagagttcaa cgccgagaca ttcaccttcc acgccgacat ctgcaccctg 1860
tccgagaaag agcggcagat caagaaacag accgctctgg tcgagctggt taagcacaag 1920
cccaaggcca ccaaagaaca gctgaaggcc gtgatggacg acttcgccgc ctttgtggaa 1980
aagtgttgca aggccgacga caaagagaca tgcttcgccg aagagggcaa gaaactggtg 2040
gccgcttctc aggctgctct gggactttaa 2070
<210> 113
<211> 2143
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 113
ggggacaagt ttgtacaaaa aagcaggcta tggtaccgcc accatgaaat gggtcacctt 60
tatctccctg ctgttcctgt tctcctccgc ctactcttgt gccgagcaga ccacagagtg 120
gaccgcctgc tctaagtctt gcggcatggg cttctccacc agagtgacca accggaacag 180
acagtgcgag atgctgaagc agacccggct gtgtatggtt cgaccttgcg aggccgctgc 240
caaagaggct gctgctaaag aagccgccgc aaaagaggca gcagcaaaag aggctgccgc 300
caaagaggcc gcagccaaag aagcagcagc taaagaggcc gctgctaagg acgcccacaa 360
gtctgaagtg gcccaccggt ttaaggacct gggcgaagag aacttcaagg ccctggtgct 420
gatcgccttc gctcagtact tgcagcagtg ccccttcgag gaccacgtga agctggtcaa 480
cgaagtgacc gagttcgcca agacctgcgt ggccgatgag tctgccgaga actgcgacaa 540
gtctctgcac accctgttcg gcgacaagct gtgtaccgtg gctaccctga gagaaaccta 600
cggcgagatg gccgactgct gcgctaagca agagcccgag agaaacgagt gcttcctgca 660
gcacaaggac gacaacccta acctgcctag actcgtgcgg cctgaggtgg acgtgatgtg 720
taccgccttc cacgacaacg aggaaacctt cctgaagaag tacctgtacg agatcgccag 780
acggcacccc tacttttacg cccctgagct gctgtttttc gccaagcggt acaaggccgc 840
cttcaccgag tgttgtcagg ccgccgataa ggccgcttgt ctgctgccta aactggacga 900
gctgcgcgac gaaggcaagg cctcttctgc taagcagcgg ctgaagtgcg ccagcctgca 960
gaagtttggc gagagagcct tcaaggcttg ggccgtcgct agactgtccc agagatttcc 1020
caaggccgag tttgccgagg tgtccaagct ggttaccgac ctgaccaagg tgcacaccga 1080
atgctgtcac ggcgacctgc tggaatgcgc cgatgataga gccgatctgg ccaagtacat 1140
ctgcgagaac caggactcca tctcctccaa gctgaaagag tgctgcgaga agcctctgct 1200
ggaaaagtcc cactgtatcg ccgaggtgga aaacgacgag atgcctgccg atctgccttc 1260
tctggccgcc gacttcgtgg aatctaagga cgtgtgcaag aactacgccg aggccaagga 1320
tgtgttcctg ggcatgtttc tgtacgagta cgctcggcgg caccccgatt atagtgtggt 1380
gctgctgctg agactggcta agacctacga gacaaccctc gagaagtgct gtgccgccgc 1440
tgatcctcac gagtgttacg ccaaggtgtt cgacgagttc aagccactgg tggaagaacc 1500
ccagaacctg atcaagcaga attgcgagct gttcgagcag ctgggcgagt acaagttcca 1560
gaacgccctg ctcgtgcggt acaccaagaa agtgccccag gtgtccacac ctacactggt 1620
tgaggtgtcc cggaacctgg gcaaagtggg ctctaagtgc tgcaagcacc ctgaggccaa 1680
gagaatgcct tgcgccgagg actacctgtc cgtggtgctg aatcagctgt gcgtgctgca 1740
cgaaaagacc cctgtgtccg accgcgtgac caagtgctgt acagagtccc tggtcaacag 1800
acggccctgc ttctctgccc tggaagtgga cgagacatac gtgcccaaag agttcaacgc 1860
cgagacattc accttccacg ccgacatctg caccctgtcc gagaaagagc ggcagatcaa 1920
gaaacagacc gctctggtcg agctggttaa gcacaagccc aaggccacca aagaacagct 1980
gaaggccgtg atggacgact tcgccgcctt tgtggaaaag tgttgcaagg ccgacgacaa 2040
agagacatgc ttcgccgaag agggcaagaa actggtggcc gcttctcagg ctgctctggg 2100
actttaatct agaaacccag ctttcttgta caaagtggtc ccc 2143
<210> 114
<211> 484
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок
<400> 114
Met Lys Trp Val Thr Phe Ile Ser Leu Leu Phe Leu Phe Ser Ser Ala
1 5 10 15
Tyr Ser Gly His His His His His His Gly Ser Glu Ile Gly Thr Gly
20 25 30
Phe Pro Phe Asp Pro His Tyr Val Glu Val Leu Gly Glu Arg Met His
35 40 45
Tyr Val Asp Val Gly Pro Arg Asp Gly Thr Pro Val Leu Phe Leu His
50 55 60
Gly Asn Pro Thr Ser Ser Tyr Val Trp Arg Asn Ile Ile Pro His Val
65 70 75 80
Ala Pro Thr His Arg Cys Ile Ala Pro Asp Leu Ile Gly Met Gly Lys
85 90 95
Ser Asp Lys Pro Asp Leu Gly Tyr Phe Phe Asp Asp His Val Arg Phe
100 105 110
Met Asp Ala Phe Ile Glu Ala Leu Gly Leu Glu Glu Val Val Leu Val
115 120 125
Ile His Asp Trp Gly Ser Ala Leu Gly Phe His Trp Ala Lys Arg Asn
130 135 140
Pro Glu Arg Val Lys Gly Ile Ala Phe Met Glu Phe Ile Arg Pro Ile
145 150 155 160
Pro Thr Trp Asp Glu Trp Pro Glu Phe Ala Arg Glu Thr Phe Gln Ala
165 170 175
Phe Arg Thr Thr Asp Val Gly Arg Lys Leu Ile Ile Asp Gln Asn Val
180 185 190
Phe Ile Glu Gly Thr Leu Pro Met Gly Val Val Arg Pro Leu Thr Glu
195 200 205
Val Glu Met Asp His Tyr Arg Glu Pro Phe Leu Asn Pro Val Asp Arg
210 215 220
Glu Pro Leu Trp Arg Phe Pro Asn Glu Leu Pro Ile Ala Gly Glu Pro
225 230 235 240
Ala Asn Ile Val Ala Leu Val Glu Glu Tyr Met Asp Trp Leu His Gln
245 250 255
Ser Pro Val Pro Lys Leu Leu Phe Trp Gly Thr Pro Gly Val Leu Ile
260 265 270
Pro Pro Ala Glu Ala Ala Arg Leu Ala Lys Ser Leu Pro Asn Cys Lys
275 280 285
Ala Val Asp Ile Gly Pro Gly Leu Asn Leu Leu Gln Glu Asp Asn Pro
290 295 300
Asp Leu Ile Gly Ser Glu Ile Ala Arg Trp Leu Ser Thr Leu Glu Ile
305 310 315 320
Ser Gly Leu Gln Asp Ser Glu Val Asn Gln Glu Ala Lys Pro Glu Val
325 330 335
Lys Pro Glu Val Lys Pro Glu Thr His Ile Asn Leu Lys Val Ser Asp
340 345 350
Gly Ser Ser Glu Ile Phe Phe Lys Ile Lys Lys Thr Thr Pro Leu Arg
355 360 365
Arg Leu Met Glu Ala Phe Ala Lys Arg Gln Gly Lys Glu Met Asp Ser
370 375 380
Leu Thr Phe Leu Tyr Asp Gly Ile Glu Ile Gln Ala Asp Gln Thr Pro
385 390 395 400
Glu Asp Leu Asp Met Glu Asp Asn Asp Ile Ile Glu Ala His Arg Glu
405 410 415
Gln Ile Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly
420 425 430
Ser Ser Gly Gly Gly Ser Ser Gly Cys Ala Glu Gln Thr Thr Glu Trp
435 440 445
Thr Ala Cys Ser Lys Ser Cys Gly Met Gly Phe Ser Thr Arg Val Thr
450 455 460
Asn Arg Asn Arg Gln Cys Glu Met Leu Lys Gln Thr Arg Leu Cys Met
465 470 475 480
Val Arg Pro Cys
<210> 115
<211> 13
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 115
Pro Pro Ser Arg Gly Arg Ser Pro Gln Asn Ser Ala Phe
1 5 10
<210> 116
<211> 8
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 116
Gly Gln Pro Val Tyr Ser Ser Leu
1 5
<210> 117
<211> 7
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 117
Glu Ala Asp Leu Glu Glu Asn
1 5
<210> 118
<211> 9
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 118
Glu Gln Gln Pro Glu Gln Pro Thr Asp
1 5
<210> 119
<211> 8
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 119
Asp Val Asp Ile His Thr Leu Ile
1 5
<210> 120
<211> 11
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> часть рекомбинантного белка
<400> 120
Asp Ser Asn Ile Leu Lys Thr Ile Lys Ile Pro
1 5 10
<210> 121
<211> 5
<212> PRT/Белок
<213> Искусственная последовательность
<220>
<223> Линкер
<400> 121
Gly Gly Gly Gly Ser
1 5
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СЛИТЫЙ БЕЛОК ИЗ БЕЛКА DCTN1 С БЕЛКОМ RET | 2018 |
|
RU2813996C2 |
| ПОЛИВАЛЕТНЫЕ И ПОЛИСПЕЦИФИЧНЫЕ GITR-СВЯЗЫВАЮЩИЕ СЛИТЫЕ БЕЛКИ | 2016 |
|
RU2753439C2 |
| IL-12 ГЕТЕРОДИМЕРНЫЕ СЛИТЫЕ БЕЛКИ FC | 2019 |
|
RU2819097C2 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
| ПОЛИВАЛЕНТНЫЕ И ПОЛИСПЕЦИФИЧЕСКИЕ ОХ40-СВЯЗЫВАЮЩИЕ СЛИТЫЕ БЕЛКИ | 2017 |
|
RU2773052C2 |
| ТЕРАПЕВТИЧЕСКИЕ СЛИТЫЕ БЕЛКИ | 2020 |
|
RU2825292C1 |
| НОВЫЕ СЛИТЫЕ БЕЛКИ, СПЕЦИФИЧЕСКИЕ В ОТНОШЕНИИ CD137 И GPC3 | 2020 |
|
RU2814653C2 |
| ГЛИКОЗИЛИРОВАННЫЕ СЛИТЫЕ БЕЛКИ VWF С УЛУЧШЕННОЙ ФАРМАКОКИНЕТИКОЙ | 2017 |
|
RU2782212C2 |
| ТЕРАПЕВТИЧЕСКИЕ СЛИТЫЕ БЕЛКИ | 2020 |
|
RU2837545C1 |
| МНОГОФУНКЦИОНАЛЬНЫЕ СЛИТЫЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2815388C2 |
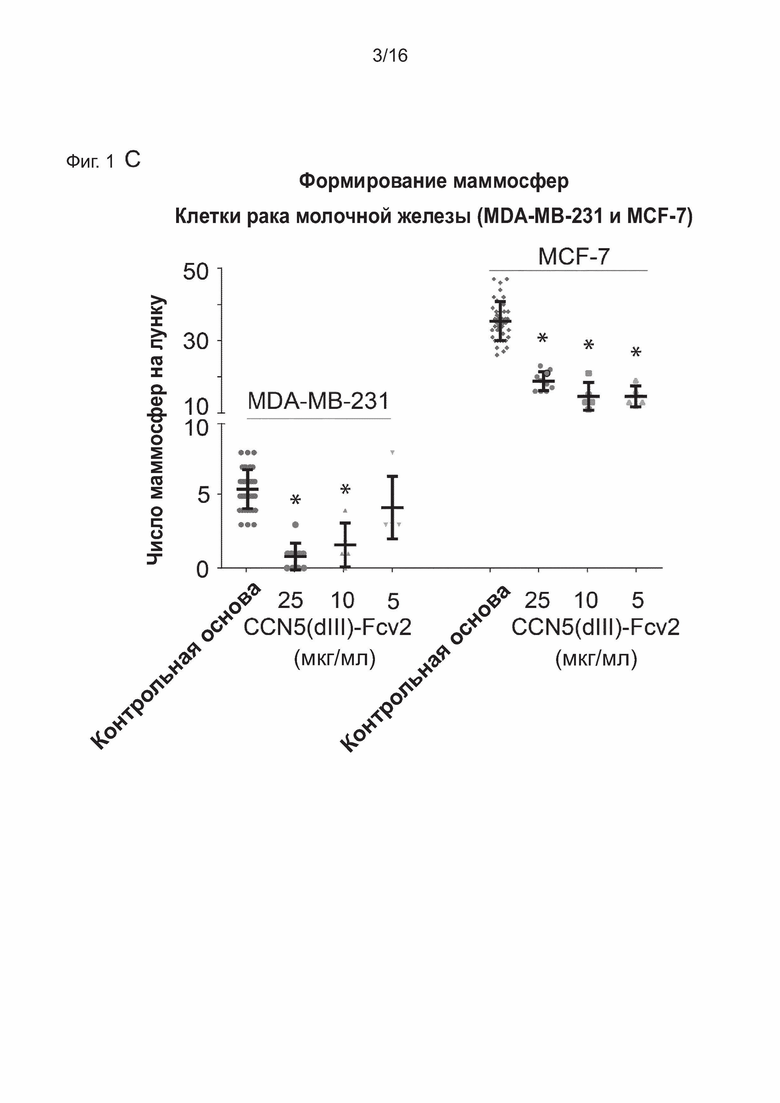
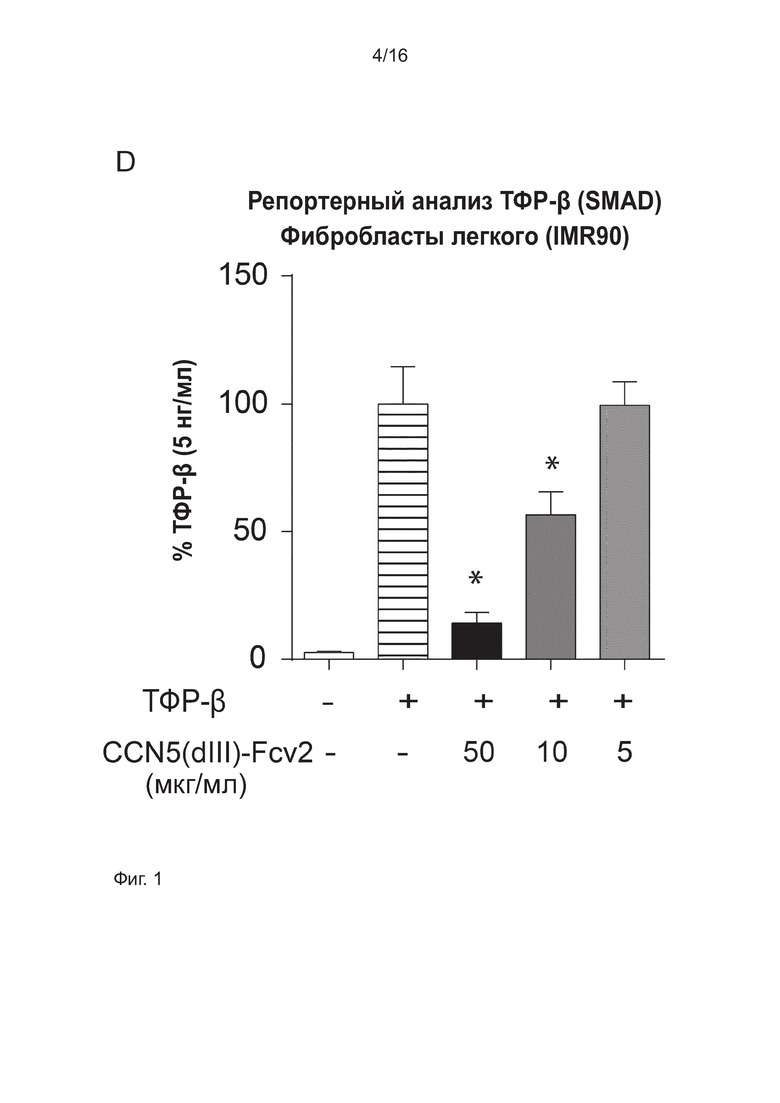
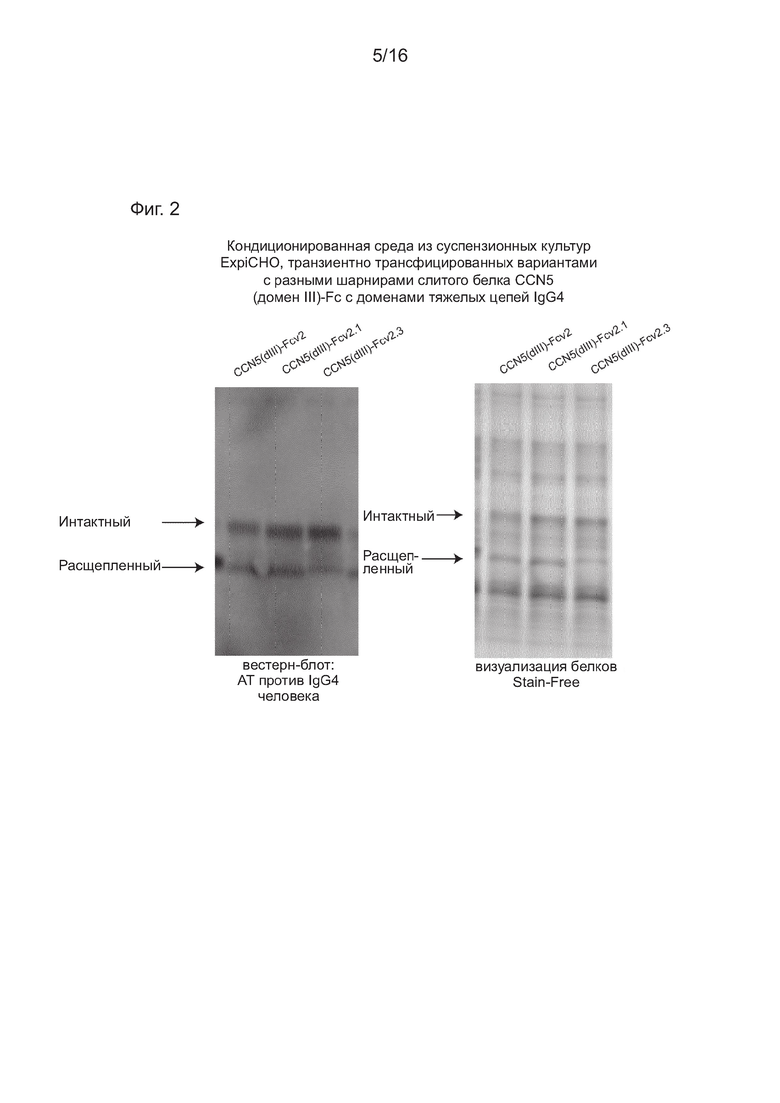

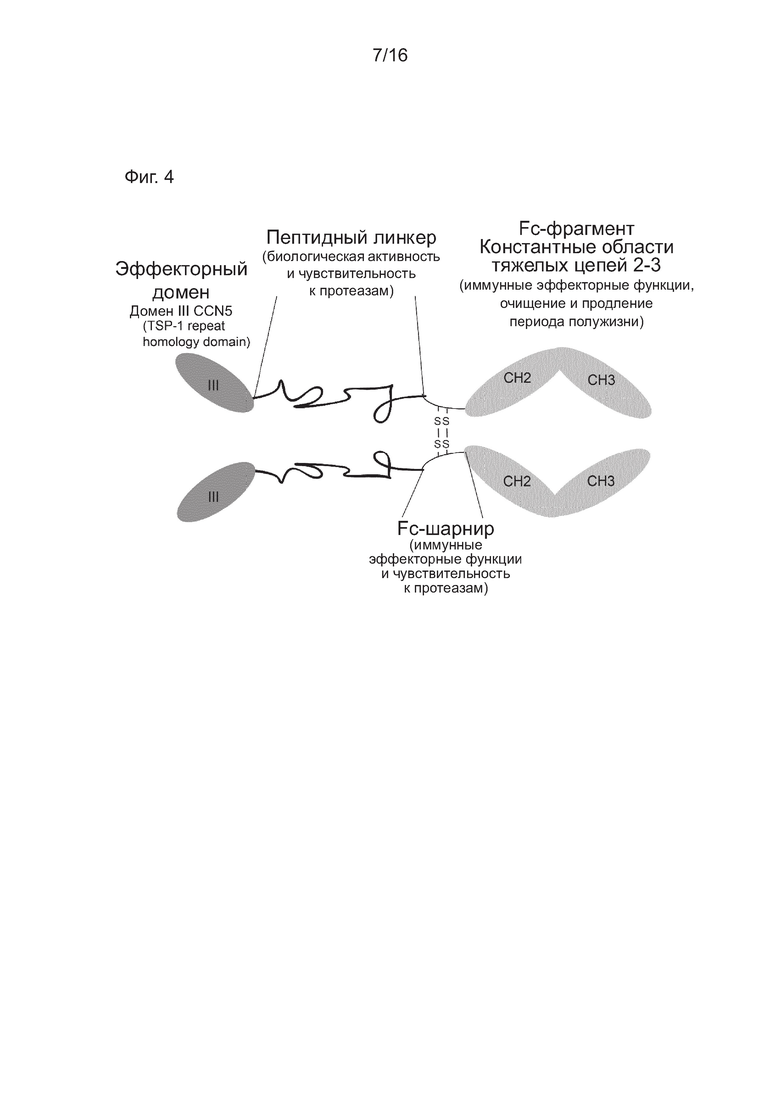
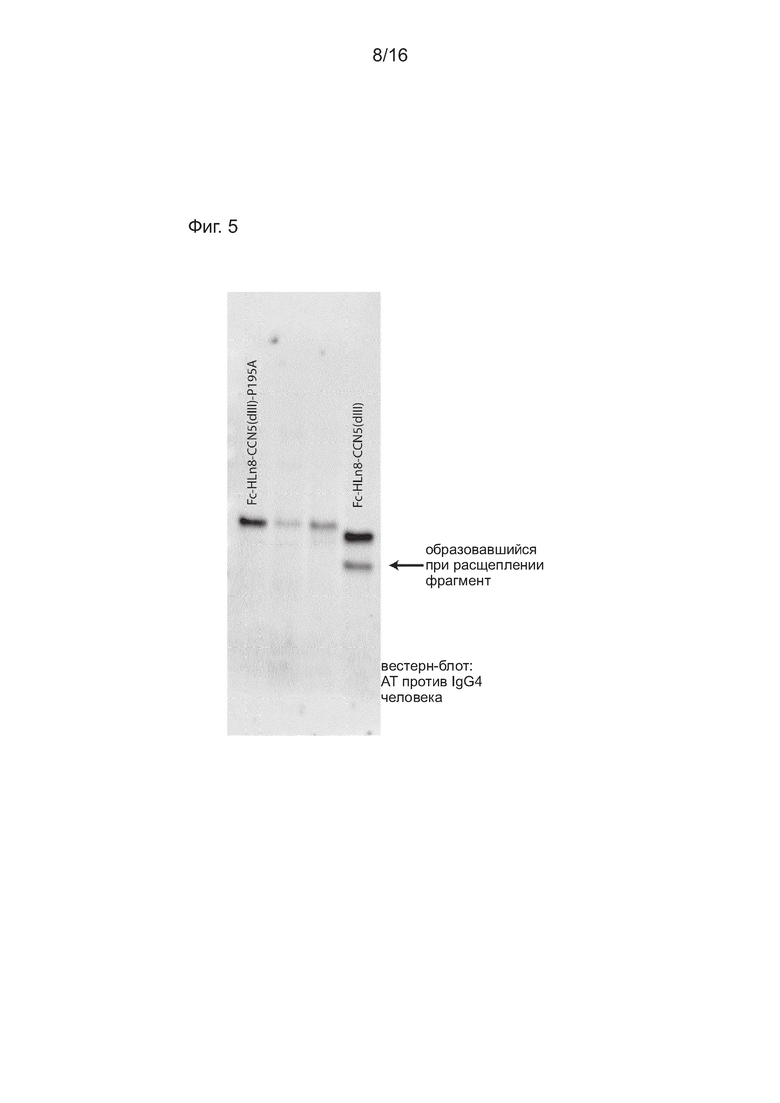
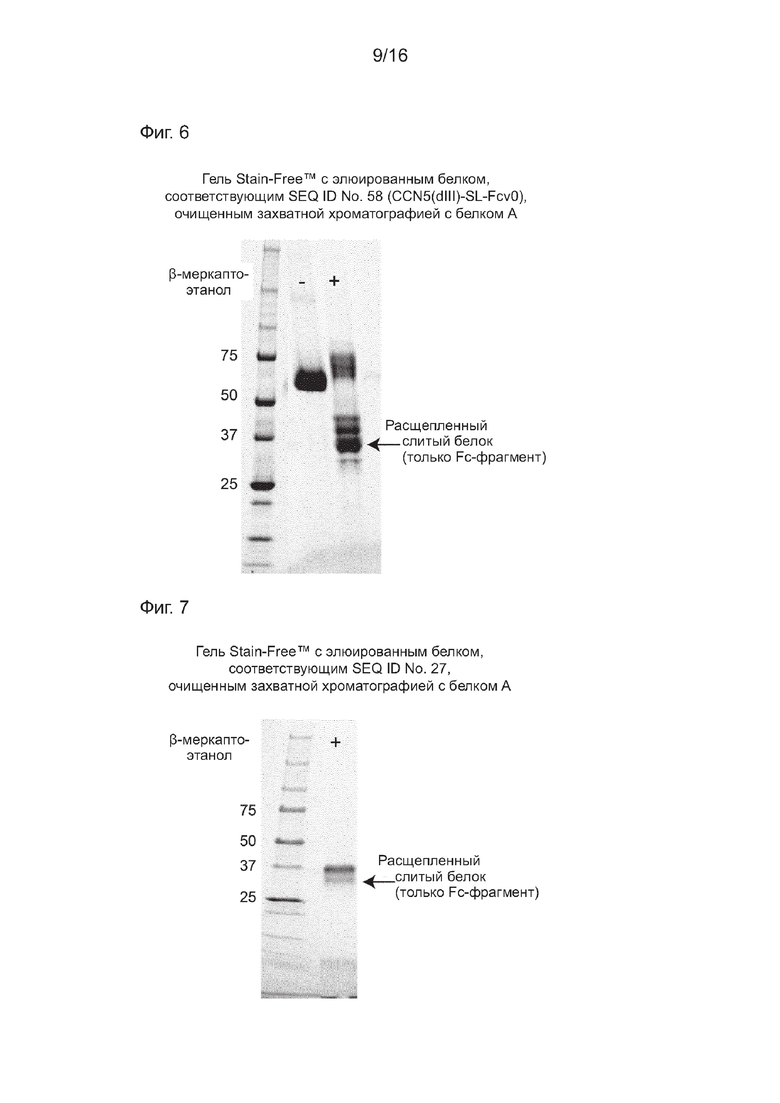
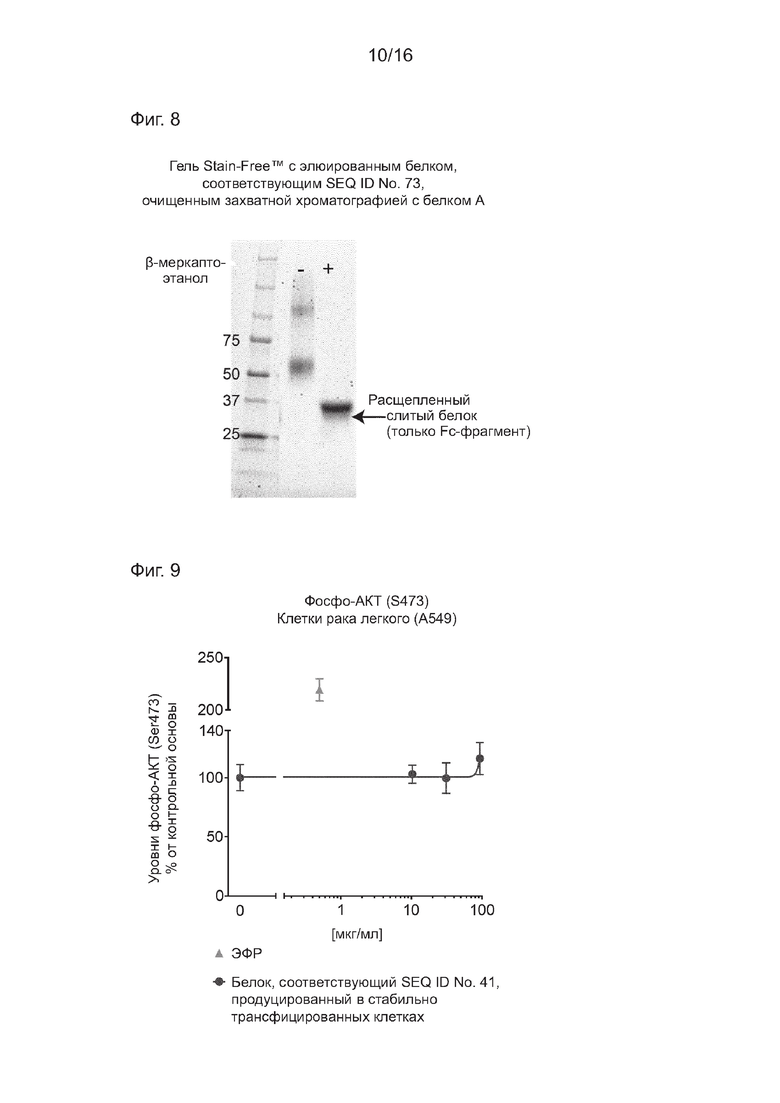
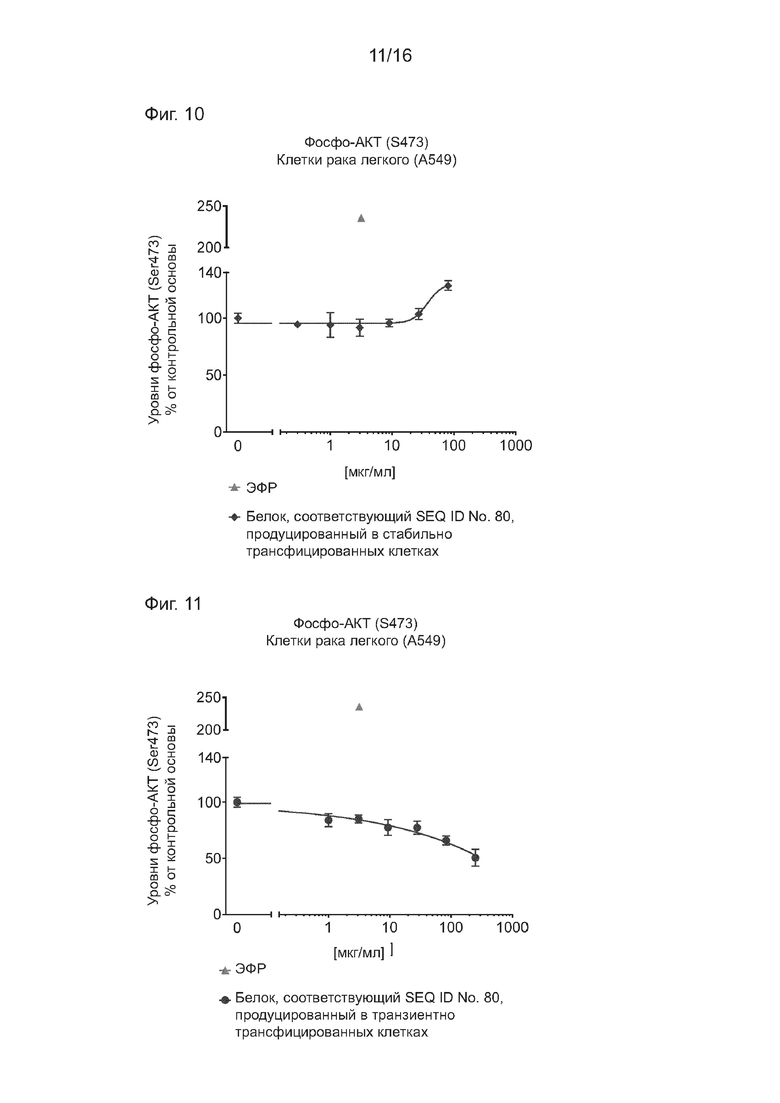
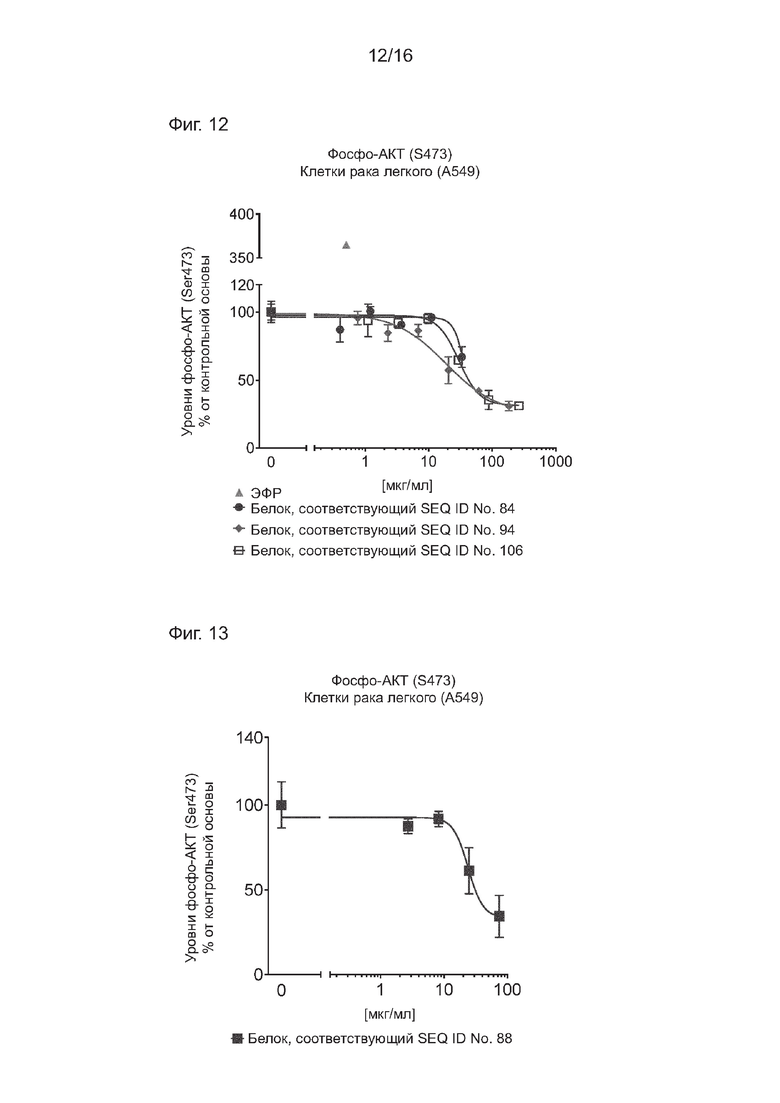
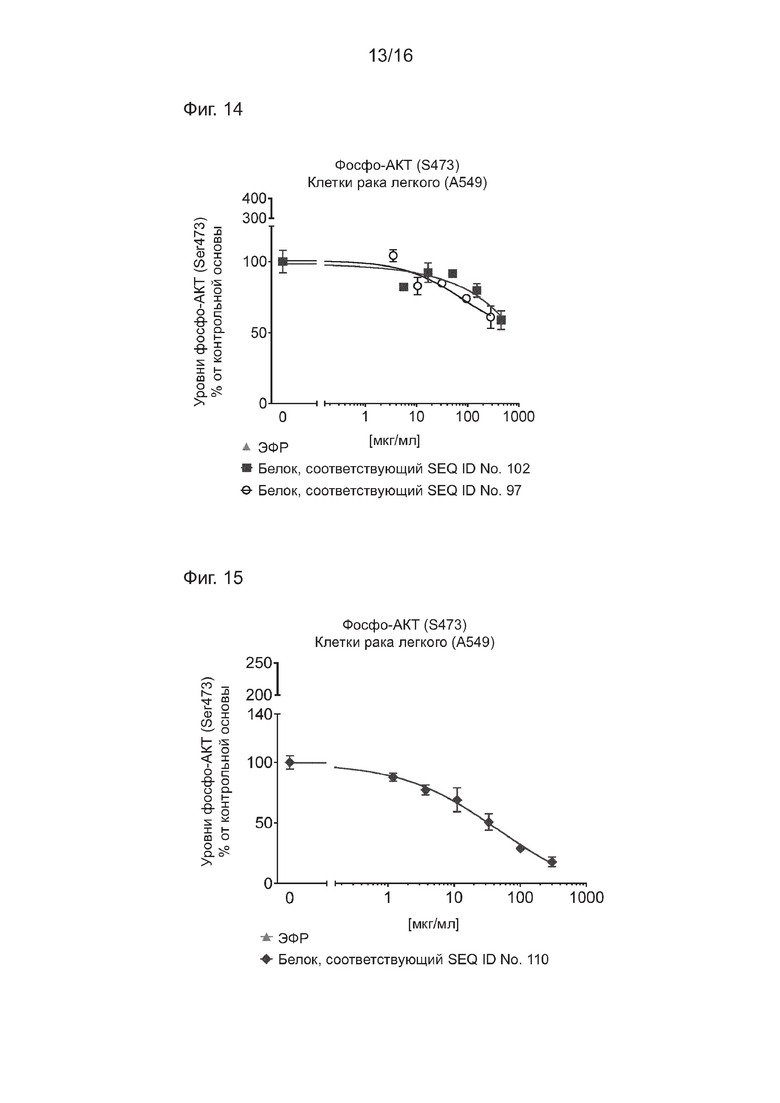
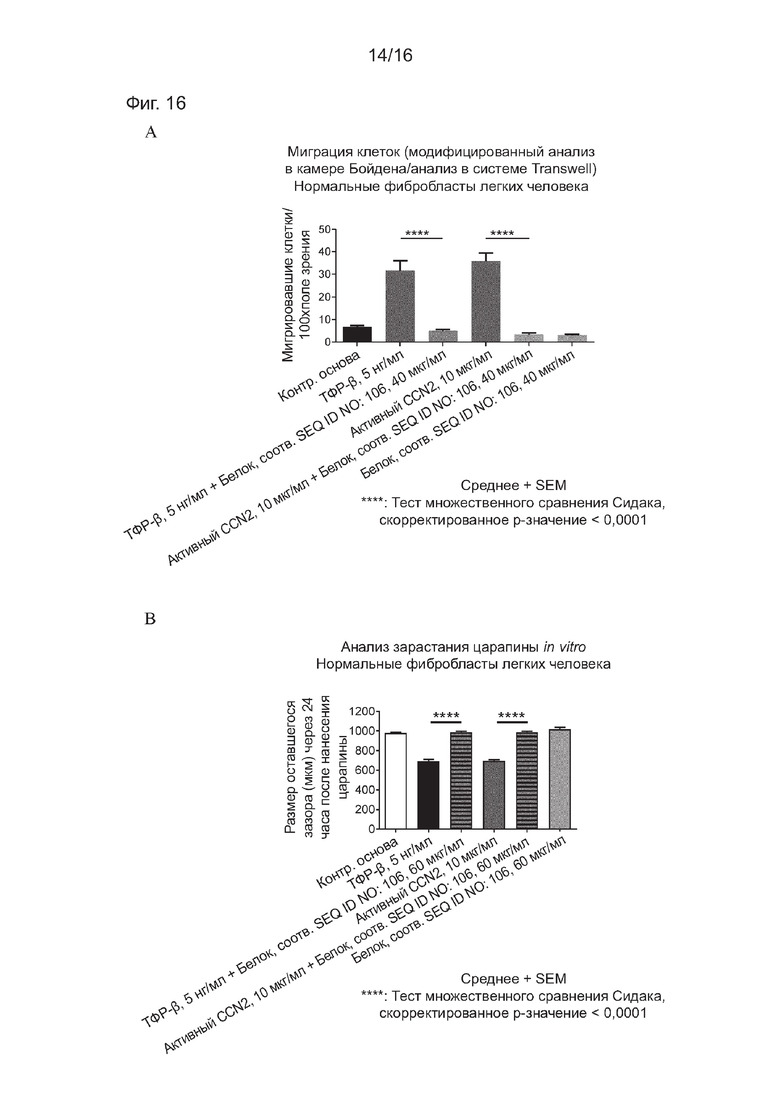
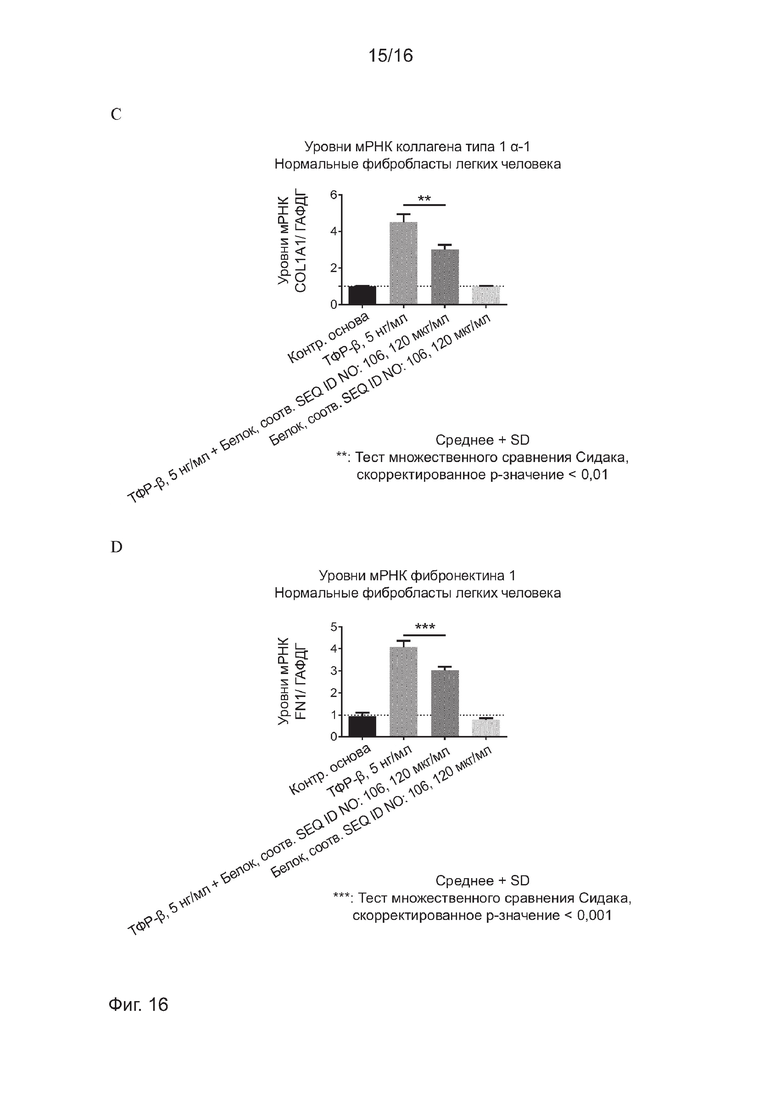
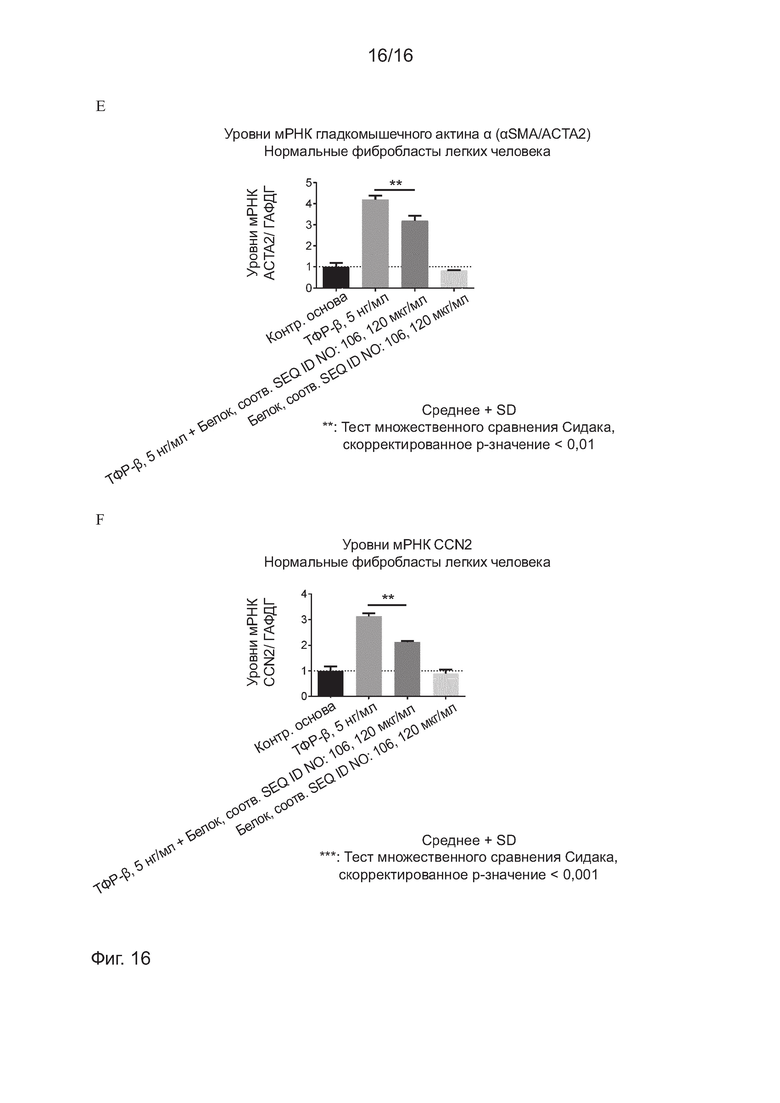
Изобретение относится к области биотехнологии, конкретно к рекомбинантным белкам тромбоспондина I типа представителя семейства белков CCN, и может быть использовано в медицине для лечения рака или фиброза. Предложен мономерный слитый белок для ингибирования эффекта или активности 4-доменного белка CCN, содержащий полипептид, соответствующий по меньшей мере части домена гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN и слитый с ним на N- или C-конце мономерный партнер для слияния, который содержит по меньшей мере 6 аминокислот и увеличивает период полувыведения из сыворотки указанного слитого белка. Изобретение обеспечивает получение слитых белков на основе усеченных фрагментов домена III белка семейства CCN, которые воспроизводят или имеют биологическую активность CCN5, способны антагонизировать или ингибировать эффекты 4-доменных белков CCN и устойчивы к протеолитическому разложению. 6 н. и 15 з.п. ф-лы, 16 ил., 1 табл., 23 пр.
1. Мономерный слитый белок для ингибирования эффекта или активности 4-доменного белка CCN, содержащий:
(i) полипептид, соответствующий по меньшей мере части домена гомологии к повторам тромбоспондина типа 1 (TSP-1) белка семейства CCN;
(ii) мономерный партнер для слияния, слитый на N- или C-конце с последовательностью аминокислот по (i), причем указанный мономерный партнер для слияния содержит по меньшей мере 6 аминокислот и увеличивает период полувыведения из сыворотки указанного слитого белка; и
(iii) необязательно, пептидный линкер между полипептидом по (i) и мономерным партнером для слияния по (ii),
при этом указанный полипептид согласно (i) (a) содержит последовательность аминокислот, выбранную из SEQ ID NO: 37 или 2, и имеет длину от 44 до 60 аминокислот, или (b) содержит последовательность, которая имеет по меньшей мере 80% идентичности с последовательностью, выбранной из SEQ ID NO: 37 или 2, и имеет длину от 40 до 60 аминокислот, причем все остатки цистеина в указанной последовательности, выбранной из SEQ ID NO: 37 или 2, сохранены, и причем указанный полипептид согласно (i) осуществляет ту же активность, что и домен гомологии с TSP-1 CCN5,
и при этом указанный мономерный партнер для слияния по (ii) и указанный пептидный линкер по (iii) не представляют собой или не содержат домен гомологии со связывающим IGF белком, домен гомологии с повторами фактора фон Виллебранда типа C или домен цистинового узла белка семейства CCN.
2. Слитый белок по п. 1, отличающийся тем, что указанный полипептид по (i) имеет длину от 44 до 57 аминокислот.
3. Слитый белок по п. 1 или 2, отличающийся тем, что указанный полипептид по (i) содержит или состоит из:
(a) последовательности аминокислот, выбранной из SEQ ID NO: 1 или 9; или
(b) последовательности аминокислот, которая имеет по меньшей мере 80% идентичности с последовательностью, выбранной из SEQ ID NO: 1 или 9; или
(c) части последовательности аминокислот по (a) или (b), при том что указанная часть содержит по меньшей мере последовательность из 44 аминокислот из SEQ ID NO: 37 или 2, соответственно, или последовательность, которая имеет по меньшей мере 80% идентичности с последовательностью, выбранной из SEQ ID NO: 37 или 2, соответственно.
4. Слитый белок по любому из пп. 1–3, отличающийся тем, что указанный полипептид состоит из последовательности аминокислот, выбранной из SEQ ID NO: 37 или 2, или последовательности, которая имеет по меньшей мере 80% идентичности с последовательностью, выбранной из SEQ ID NO: 37 или 2.
5. Слитый белок по любому из пп. 1–4, отличающийся тем, что пептидный линкер по (iii) содержит не более 50 аминокислот.
6. Слитый белок по любому из пп. 1–5, отличающийся тем, что указанный полипептид по (i) содержит остаток аланина в положении, соответствующем положению 2 указанной последовательности, выбранной из SEQ ID NO: 37 или 2, или SEQ ID NO: 1 или 9.
7. Слитый белок по любому из пп. 1–6, отличающийся тем, что последовательность аминокислот по (i) содержит последовательность аминокислот, выбранную из SEQ ID NO: 7, 38, 44 или 49, или последовательность, которая имеет по меньшей мере 80% идентичности с указанной последовательностью, причем указанный белок содержит остаток аланина в положении, соответствующем положению 2 указанной последовательности SEQ ID NO: 7, 38, 44 или 49.
8. Слитый белок по любому из пп. 1–7, отличающийся тем, что указанный мономерный партнер для слияния выбран из группы, состоящей из сывороточного альбумина, трансферрина и мономерного Fc-фрагмента IgG человека.
9. Слитый белок по п. 8, отличающийся тем, что указанный мономерный Fc-фрагмент IgG человека представляет собой мономерный Fc-фрагмент IgG1, IgG2 или IgG4.
10. Слитый белок по п. 8 или 9, отличающийся тем, что указанный мономерный Fc-фрагмент агликозилирован.
11. Слитый белок по любому из пп. 8–10, отличающийся тем, что указанный мономерный Fc-фрагмент содержит стабилизирующий дисульфидный мостик и/или мутацию, стабилизирующую против действия протеазы.
12. Слитый белок по любому из пп. 8–11, отличающийся тем, что указанный мономерный Fc-фрагмент не обладает иммунной эффекторной функцией.
13. Слитый белок по любому из пп. 1–12, отличающийся тем, что указанный пептидный линкер между последовательностью аминокислот по (i) и указанным мономерным партнером для слияния имеет последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 20–25, 39, 57, 63, 65 или 67, или последовательность аминокислот, которая имеет 80% идентичности с указанной последовательностью.
14. Слитый белок по любому из пп. 1–8, отличающийся тем, что указанный слитый белок имеет последовательность аминокислот, выбранную из группы, состоящей из SEQ ID NO: 84, 85, 88, 89, 97, 98, 102, 103, 106, 107, 110 и 111, или последовательность аминокислот, которая имеет 80% идентичности с указанной последовательностью.
15. Молекула ДНК, кодирующая мономерный слитый белок, как определено в любом из пп. 1–14.
16. Молекула ДНК по п. 15, отличающаяся тем, что указанная молекула дополнительно содержит последовательность нуклеотидов, кодирующую сигнальную последовательность.
17. Молекула ДНК по п. 15 или 16, отличающаяся тем, что указанная молекула содержит последовательность нуклеотидов согласно SEQ ID NO: 34, 35, 36, 86, 87, 90, 91, 99, 100, 104, 105, 108, 109, 112 или 113, или последовательность нуклеотидов, которая имеет по меньшей мере 80% идентичности с любой вышеуказанной последовательностью.
18. Экспрессионный вектор для обеспечения экспрессии в клетке млекопитающего, содержащий молекулу ДНК, как определено в любом из пп. 15–17.
19. Клетка-хозяин млекопитающего для экспрессии мономерного слитого белка по любому из пп. 1-14, содержащая вектор, как определено в п. 18.
20. Применение слитого белка по любому из пп. 1–14 при лечении или предотвращении фиброза, или любого состояния, при котором проявляется фиброз.
21. Применение слитого белка по любому из пп. 1–14 при лечении рака.
| FR 2858234 A1, 04.02.2005 | |||
| US 20020165185 A1, 07.11.2002 | |||
| CN 105396136 A, 16.03.2016 | |||
| WO 2007066823 A1, 14.06.2007 | |||
| KR 20180099537 A, 05.09.2018 | |||
| КУЗНЕЦОВА С.А | |||
| и др., ИММУНОРЕГУЛЯТОРНЫЕ СВОЙСТВА ТРОМБОСПОНДИНА-1, КОМПОНЕНТА ВНЕКЛЕТОЧНОГО МАТРИКСА И ИНГИБИТОРА АНГИОГЕНЕЗА, Медицинская Иммунология, 2008, т | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| Складная пожарная (штурмовая) лестница | 1923 |
|
SU499A1 |
| KONTERMANN R.E | |||
| et | |||
Авторы
Даты
2024-08-20—Публикация
2020-03-20—Подача