Область техники
[0001]
Настоящее изобретение относится к полипептиду, представляющему собой слитый белок из белка DCTN1 и белка RET; к полинуклеотиду, кодирующему полипептид; к способу детекции полипептида или полинуклеотида; к соединению, нацеленному на полипептид или полинуклеотид; и к способу скрининга соединения.
Уровень техники
[0002]
Злокачественная опухоль является основной причиной смерти в Японии, и ее лечение необходимо улучшать. Несмотря на то, что количество индивидуумов, пораженных раком щитовидной железы, увеличивается, соответствующее лечение на начальной стадии приводит к высокому уровню выживаемости, благодаря медленному прогрессированию заболевания в большинстве случаев. Заболевание, однако, почти не имеет субъективных симптомов, и ранняя диагностика является необходимой для адекватного лечения.
[0003]
Рак щитовидной железы разделяют по гистологическим типам на папиллярную злокачественную опухоль, фолликулярную злокачественную опухоль, медуллярную злокачественную опухоль, анапластическую злокачественную опухоль и злокачественную лимфому. Папиллярная злокачественная опухоль насчитывает приблизительно 80% рака щитовидной железы, и анапластическая злокачественная опухоль, которая, несмотря на то, что имеет более низкую встречаемость, как известно, имеет очень плохой прогноз (непатентный литературный источник 1).
[0004]
Известно, что папиллярная злокачественная опухоль развивается в большой степени из-за активации онкогенов, и выявлены взаимоисключающие генетические аномалии, такие как мутация в гене BRAF (50-60%), мутация в гене RAS (10-20%) и слитый ген RET (5-10%). Опубликованы также исследования, что при немелкоклеточном рак легкого, слитый ген RET присутствует с частотой 1-2%, взаимоисключающим образом с управляющими мутациями в других генах, такими как мутация в гене EGFR (непатентные литературные источники 2-5).
[0005]
Лечение лекарственными средствами является преобладающим способом лечения рака щитовидной железы на поздних стадиях, и апробировано множество ингибиторов множества киназ. Однако лекарственные средства, проявляющие эффект, специфический для управляющих мутаций в генах, остаются не одобренными. Опубликованы результаты исследований, что для положительных по слитому гену RET пациентов с раком легкого показано преимущество от ингибирования RET (непатентный литературный источник 6); также при раке щитовидной железы необходимо идентифицировать генные аномалии, такие как мутации в генах или слитые гены, которые могут являться показателем эффекта лекарственных средств.
[0006]
Очень желательна идентификация мутаций в генах (мутаций в белках), слитых генов (слитых белков) и т.д., которые могут управлять злокачественной опухолью; поскольку такая идентификация может прояснять природу злокачественной опухоли и вносить значительный вклад в разработку новых лекарственных средств для лечения злокачественных опухолей или способов тестирования, направленных на эти мутации в генах или слитые гены. Однако мутации в генах, слитые гены и т.д., которые могут управлять развитием злокачественных опухолей, еще предстоит полностью прояснить, и идентификация генных аномалий, которые могут быть ассоциированы с терапевтическими эффектами лекарственных средств, могут являться очень важными.
Список литературы
Непатентные литературные источники
[0007]
Непатентный литературный источник 1: Cancer, 115 (16), pp. 3801-7 (2009)
Непатентный литературный источник 2: Oncogene, 22 (29), pp. 4578-80 (2003)
Непатентный литературный источник 3: Cell, 159 (3), pp. 676-90 (2014)
Непатентный литературный источник 4: Cancer Discov., 3 (6), pp. 630-5 (2013)
Непатентный литературный источник 5: Nature, 511 (7511), pp. 543-50 (2014)
Непатентный литературный источник 6: Lancet Respir Med., 5 (1), pp. 42-50 (2017).
Сущность изобретения
Техническая задача
[0008]
Целью настоящего изобретения является разработка нового полипептида, представляющего собой слитый белок, включающий по меньшей мере часть белка RET; полинуклеотида, кодирующего полипептид; способа детекции полипептида или полинуклеотида; соединения, нацеленного на полипептид или полинуклеотид; и способа скрининга соединения.
Решение задачи
[0009]
Авторы настоящего изобретения провели интенсивное исследование для достижения этой цели и идентифицировали новый полипептид, в котором часть белка DCTN1 слита с частью белка RET, и полинуклеотид, кодирующий полипептид, в клетках, полученных от пациентов с раком щитовидной железы. Авторы изобретения обнаружили также способ детекции полинуклеотида или полипептида по настоящему изобретению в клетках злокачественных опухолей, и способ скрининга соединения, ингибирующего экспрессию полинуклеотида, или экспрессию и/или активность полипептида. Это является новым обнаружением и не может быть предсказано из предшествующего уровня техники, что, среди широкого диапазона белков, слитый белок, содержащий комбинацию N-концевой части белка DCTN1 и C-концевой части белка RET, естественным образом встречается внутри клеток; и что, поскольку слитый ген из DCTN1 и RET функционирует как управляющий злокачественной опухолью, слитый белок можно использовать в диагностике злокачественных опухолей. Они, кроме того, обнаружили фармацевтическую композицию, которая содержит соединение, ингибирующее RET, в качестве активного ингредиента, и которая предназначена для использования в лечении пациентов с злокачественной опухолью, имеющей экспрессию полипептида и/или полинуклеотида, и завершили настоящее изобретение.
[0010]
Конкретно, настоящее изобретение относится к следующему объекту.
[0011]
Пункт 1.
Полипептид, где N-концевая часть белка DCTN1 слита с C-концевой частью белка RET.
[0012]
Пункт 2.
Полипептид по пункту 1, выбранный из следующих (a)-(c):
[0013]
(a) полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24;
[0014]
(b) полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24; и
[0015]
(c) полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0016]
Пункт 3.
Полинуклеотид, кодирующий полипептид по пункту 1 или 2.
[0017]
Пункт 4.
Полинуклеотид по пункту 3, выбранный из следующих (d) - (f):
[0018]
(d) полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24;
[0019]
(e) полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24; и
[0020]
(f) полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0021]
Пункт 5.
Полинуклеотид по пункту 3, выбранный из следующих (g)-(i):
[0022]
(g) полинуклеотид, содержащий последовательность оснований, представленную SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23;
[0023]
(h) полинуклеотид, гибридизующийся в строгих условиях с полинуклеотидом, содержащим последовательность оснований, комплементарную последовательности оснований, представленной SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23; и
[0024]
(i) полинуклеотид, имеющий по меньшей мере 90% идентичность с последовательностью оснований, представленной SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23.
[0025]
Пункт 6.
Экспрессирующий вектор, содержащий полинуклеотид по любому из пунктов 3-5.
[0026]
Пункт 7.
Клетка, трансфицированная полинуклеотидом по любому из пунктов 3-5.
[0027]
Пункт 8.
Антитело, специфически связывающееcя с полипептидом по пункту 1 или 2.
[0028]
Пункт 9.
Способ детекции присутствия полипептида по пункту 1 или 2 в образце.
[0029]
Пункт 10.
Праймер или зонд для детекции присутствия полинуклеотида по любому из пунктов 3-5 в образце, представляющий собой полинуклеотид, выбранный из следующих (j) - (l):
[0030]
(j) полинуклеотид, представляющий собой по меньшей мере один зонд, выбранный из группы, состоящей из зондов, гибридизующихся с полинуклеотидом, кодирующим белок DCTN1, и зондов, гибридизующихся с полинуклеотидом, кодирующим белок RET;
[0031]
(k) полинуклеотид, представляющий собой зонд, гибридизующийся с точкой слияния между полинуклеотидом, кодирующим белок DCTN1, и полинуклеотидом, кодирующим белок RET; и
[0032]
(l) полинуклеотид, представляющий собой набор из смыслового праймера и антисмыслового праймера, разработанных для обрамления точки слияния между полинуклеотидом, кодирующим белок DCTN1, и полинуклеотидом, кодирующим белок RET.
[0033]
Пункт 11.
Способ детекции присутствия полинуклеотида по любому из пунктов 3-5 в образце.
[0034]
Пункт 12.
Способ диагностики злокачественной опухоли у пациента, когда присутствие полипептида по пункту 1 или 2 или полинуклеотида по любому из пунктов 3-5 детектируют в образце, полученном от пациента, посредством способа детекции по пункту 9 или 11.
[0035]
Пункт 13.
Фармацевтическая композиция для лечения злокачественной опухоли, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET, где композиция содержит соединение, ингибирующее RET, в качестве активного ингредиента.
[0036]
Пункт 14.
Способ скрининга соединения, ингибирующего экспрессию и/или активность полипептида по пункту 1 или 2, или экспрессию полинуклеотида по любому из пунктов 3-5, включающий следующие стадии (1) и (2):
[0037]
(1) стадию приведения полипептида по пункту 1 или 2, клетки, экспрессирующей полипептид по пункту 1 или 2, или полинуклеотида по любому из пунктов 3-5, или клетки по пункту 7 в контакт с тестируемым соединением; и
[0038]
(2) стадию измерения того, ингибирована ли экспрессия и/или активность полипептида по пункту 1 или 2, или экспрессия полинуклеотида по любому из пунктов 3-5 на стадии (1), или стадию измерения того, ингибирован ли рост клетки на стадии (1).
[0039]
Пункт 15.
Способ с использованием полипептида по пункту 1 или 2 или полинуклеотида по любому из пунктов 3-5 в качестве индикатора для определения того, является ли эффективной химиотерапия с использованием соединения, ингибирующего RET,
включающий определение того, что химиотерапия с использованием соединения, ингибирующего RET, является эффективной, когда полипептид по пункту 1 или 2 детектируют в образце посредством способа детекции по пункту 9, и/или когда присутствие полинуклеотида по любому из пунктов 3-5 детектируют в образце посредством способа детекции по пункту 11.
[0040]
Пункт 16.
Биомаркер для детекции злокачественной опухоли, содержащий по меньшей мере один член, выбранный из группы, состоящей из полипептидов, где N-концевая часть белка DCTN1 слита с C-концевой частью белка RET, и полинуклеотидов, кодирующих полипептиды.
[0041]
Пункт 17.
Способ лечения злокачественной опухоли, включающий
проведение химиотерапии с использованием соединения, ингибирующего RET, для пациента с злокачественной опухолью, положительного по гену, слитому из гена DCTN1 и гена RET, и/или положительного по белку, слитому из белка DCTN1 и белка RET.
[0042] Пункт 18.
Способ лечения злокачественной опухоли, включающий
детекцию присутствия полипептида по пункту 1 или 2, и/или присутствия полинуклеотида по любому из пунктов 3-5 в образце, полученном от тестируемого субъекта, и
проведение химиотерапии с использованием соединения, ингибирующего RET, для тестируемого субъекта, когда детектировано присутствие полипептида по пункту 1 или 2, и/или детектировано присутствие полинуклеотида по любому из пунктов 3-5.
[0043]
Пункт 19.
Соединение, ингибирующее RET, для применения в лечении пациента со злокачественной опухолью, положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET.
[0044]
Пункт 20.
Применение соединения, ингибирующего RET, для получения фармацевтической композиции для лечения злокачественной опухоли для лечения пациента со злокачественной опухолью, положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET.
[0045]
Пункт 21.
Способ получения реагента для определения того, является ли химиотерапия с использованием соединения, ингибирующего RET, эффективной, с использованием средств для детекции присутствия полипептида по пункту 1 или 2 в образце, и/или средств для детекции присутствия полинуклеотида по любому из пунктов 3-5 в образце.
[0046]
Пункт 22.
Комбинация антитела к DCTN1 и антитела к RET для детекции присутствия полинуклеотида по любому из пунктов 3-5.
[0047]
Пункт 23.
Применение антитела по пункту 8, комбинации антител по пункту 22 или праймера или зонда по пункту 10 для получения детектирующего реагента для детекции присутствия полипептида по пункту 1 или 2 или присутствия полинуклеотида по любому из пунктов 3-5.
Выгодные эффекты изобретения
[0048]
В настоящем изобретении показано, что полинуклеотид и/или полипептид по настоящему изобретению специфически экспрессируются в клетке злокачественной опухоли. Полинуклеотид, полипептид и клетку, экспрессирующую полинуклеотид и/или полипептид по настоящему изобретению, можно использовать в способе скрининга соединения, ингибирующего экспрессию полинуклеотида, или экспрессию и/или активность полипептида по настоящему изобретению. Использование присутствия полинуклеотида и/или полипептида по настоящему изобретению в качестве индикатора позволяет детекцию мишени, положительной по гену, слитому из гена DCTN1 и гена RET, и/или мишени, положительной по белку, слитому из белка DCTN1 и белка RET. Соединение, ингибирующее RET, можно использовать в качестве фармацевтической композиции для лечения злокачественной опухоли, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET.
Краткое описание чертежей
[0049]
Фиг. 1: Подтверждение экспрессии слитого гена DCTN1-RET и GAPDH в ткани злокачественной опухоли щитовидной железы с использованием цифровой капельной ПЦР (цкПЦР).
Фиг. 2: Подтверждение экспрессии слитого гена DCTN1-RET и GAPDH в нормальной ткани щитовидной железы с использованием цифровой капельной ПЦР (цкПЦР).
Фиг. 3: Подтверждение экспрессии полноразмерного слитого гена DCTN1-RET в нормальной ткани щитовидной железы и в ткани злокачественной опухоли щитовидной железы.
Фиг. 4: Подтверждение экспрессии слитого белка DCTN1-RET в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3:
a) Детекция слитого белка DCTN1-RET с использованием антитела к фосфорилированного RET;
b) Детекция слитого белка DCTN1-RET с использованием антитела к RET; и
c) Детекция слитого белка DCTN1-RET с использованием антитела к DCTN1.
Фиг. 5: Подтверждение роста клеток NIH/3T3, экспрессирующих слитый ген DCTN1-RET, в 3-мерной культуре.
N=3, среднее+SD.
Фиг. 6: Подтверждение онкогенности клеток NIH/3T3, экспрессирующих слитый ген DCTN1-RET, in vivo.
Фиг. 7: Подтверждение ингибирования экспрессии фосфорилированного RET посредством миРНК RET в клетках NIH/3T3, экспрессирующих слитый ген DCTN1-RET.
Фиг. 8: Подтверждение эффекта ингибирования роста на клетки NIH/3T3, экспрессирующие слитый ген DCTN1-RET, посредством миРНК RET.
Фиг. 9: Подтверждение ингибирования экспрессии фосфорилированного RET в клетках NIH/3T3, экспрессирующих слитый ген DCTN1-RET, посредством соединений, ингибирующих RET.
Описание вариантов осуществления
[0050]
Настоящее изобретение относится к новому полинуклеотиду или полипептиду; к способу детекции полинуклеотида или полипептида; к соединению, нацеленному на полинуклеотид или полипептид; и к способу скрининга соединения.
[0051]
Настоящее изобретение относится к полипептиду, в котором N-концевая часть белка DCTN1 слита с C-концевой частью белка RET (который может быть далее в настоящем описании обозначен как «полипептид по настоящему изобретению»). Настоящее изобретение также относится к полинуклеотиду, кодирующему полипептид (который может быть далее в настоящем описании обозначен как «полинуклеотид по настоящему изобретению»).
[0052]
«Белок DCTN1 (субъединицу 1 динактина)» по настоящему изобретению называют также белком ассоциированного с динеином полипептида 150 кДа или белком DAP-150, и он включает белок DCTN1 человека или не относящегося к человеку млекопитающего, где белок DCTN1 человека является предпочтительным. Белок DCTN1 кодирован геном, локализованным на 2p13.1 у человека. По настоящему изобретению, «белок DCTN1» включает изоформы (его варианты сплайсинга), и примеры белка DCTN1 человеческого происхождения включают полипептиды, содержащие аминокислотную последовательность, представленную номером доступа в GenPept NP_004073, NP_075408, NP_001128512, NP_001128513, NP_001177765 или NP_001177766. Более конкретно, примеры включают полипептиды, содержащие аминокислотную последовательность, представленную SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29 или SEQ ID NO: 30. Кроме того, «N-концевая часть белка DCTN1» по настоящему изобретению относится к полипептиду, содержащему часть или весь суперспиральный домен, который находится на N-концевой стороне белка DCTN1, и предпочтительно, к полипептиду, содержащему весь суперспиральный домен, который находится на N-концевой стороне белка DCTN1.
[0053]
«Белок RET» по настоящему изобретению называют также протоонкогенным белком RET, белком рецепторной тирозинкиназы RET или белком, подвергаемым реаранжировке в ходе трансфекции; и включает белок RET человека или не относящегося к человеку млекопитающего, где белок RET человека является предпочтительным. Белок RET кодирован геном, локализованным на 10q11.2 у человека. По настоящему изобретению, «белок RET» включает изоформы (его варианты сплайсинга), и примеры белка RET человеческого происхождения включают полипептиды, содержащие аминокислотную последовательность, представленную номером доступа в GenPept NP_066124 или NP_065681. Более конкретно, примеры включают полипептиды, содержащие аминокислотную последовательность, представленную SEQ ID NO: 31 или SEQ ID NO: 32. Кроме того, «C-концевая часть белка RET» по настоящему изобретению относится к полипептиду, содержащему киназный домен, который находится на C-концевой стороне белка RET.
[0054]
«Полипептид, в котором N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET», по настоящему изобретению, представляет собой полипептид, в котором полипептид, содержащий часть или весь суперспиральный домен, который находится на N-концевой стороне белка DCTN1, является слитым с полипептидом, содержащим киназный домен, который находится на C-концевой стороне белка RET, предпочтительно, полипептид, в котором полипептид, содержащий весь суперспиральный домен, который находится на N-концевой стороне белка DCTN1, является слитым с полипептидом, содержащим киназный домен, который находится на C-концевой стороне белка RET, и более предпочтительно, с полипептидом, выбранным из следующих (a)-(c). Эти полипептиды, предпочтительно, имеют активность киназы и/или эффект вызова пролиферации клеток.
[0055]
(a) Полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0056]
(b) Полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0057]
(c) Полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0058]
Более предпочтительно, полипептид выбран из следующих (a)-(c). Эти полипептиды, предпочтительно, имеют активность киназы или эффект вызова пролиферации клеток.
[0059]
(a) Полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 18.
[0060]
(b) Полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 18.
[0061]
(c) Полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 18.
[0062]
«Полипептид, в котором N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET», по настоящему изобретению включает полипептиды, содержащие аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24 (пункт (b) выше). Примеры полипептидов, содержащих такую аминокислотную последовательность, где N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET, включают изоформы полипептидов, содержащих аминокислотную последовательность, представленную SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24, где N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET. Эти полипептиды, предпочтительно, имеют активность киназы или эффект вызова пролиферации клеток. В рамках изобретения, «несколько аминокислот, которые заменены, делетированы или добавлены», обозначает, например, предпочтительно, 1-10 аминокислот, и более предпочтительно, 1-5 аминокислот. «Добавление» включает добавление от одной до нескольких аминокислот к N-концу или C-концу, или добавление от одной до нескольких аминокислот к обоим концам.
[0063]
Примеры полипептида, где одна или несколько аминокислот заменены, включают полипептиды, в которых валин в положении 804 (в положении 1325 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1191 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1300 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1186 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1283 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1318 в SEQ ID NO: 22 и SEQ ID NO: 24), который находится в участке-привратнике белка RET, содержащем аминокислотную последовательность, представленную номером доступа в GenPept: NP_066124 (SEQ ID NO: 31) или NP_065681 (SEQ ID NO: 32), заменен на лейцин, метионин или глутаминовую кислоту; и полипептиды, в которых тирозин в положении 806 (в положении 1327 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1193 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1302 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1188 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1285 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1320 в SEQ ID NO: 22 и SEQ ID NO: 24) заменен на цистеин, глутаминовую кислоту, серин, гистидин или аспарагин.
[0064]
Примеры также включают аминокислоты, локализованные в положении, отличном от участка-привратника, но без ограничения, полипептиды, в которых глутаминовая кислота в положении 768 (в положении 1289 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1155 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1264 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1150 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1247 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1282 в SEQ ID NO: 22 и SEQ ID NO: 24) заменен на аспарагиновую кислоту; полипептиды, в которых аланин в положении 883 (в положении 1404 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1270 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1379 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1265 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1362 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1397 в SEQ ID NO: 22 и SEQ ID NO: 24) заменен на фенилаланин или серин; полипептиды, в которых глутаминовая кислота в положении 884 (в положении 1405 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1271 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1380 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1266 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1363 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1398 в SEQ ID NO: 22 и SEQ ID NO: 24) заменена на валин; полипептиды, в которых серин в положении 891 (в положении 1412 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1278 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1387 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1273 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1370 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1405 в SEQ ID NO: 22 и SEQ ID NO: 24) заменен на аланин или лейцин; и полипептиды, в которых метионин в положении 918 (в положении 1439 в SEQ ID NO: 2 и SEQ ID NO: 4, в положении 1305 в SEQ ID NO: 6 и SEQ ID NO: 8, в положении 1414 в SEQ ID NO: 10 и SEQ ID NO: 12, в положении 1300 в SEQ ID NO: 14 и SEQ ID NO: 16, в положении 1397 в SEQ ID NO: 18 и SEQ ID NO: 20, в положении 1432 в SEQ ID NO: 22 и SEQ ID NO: 24) заменен на треонин.
[0065]
Полипептид, в котором N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET, по настоящему изобретению включает полипептиды, содержащие аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной любой из SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24, при их соответствующем выравнивании (пункт (c) выше). Эти полипептиды, предпочтительно, имеют активность киназы или эффект вызова пролиферации клеток.
[0066]
Идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24, составляет предпочтительно по меньшей мере 90%, более предпочтительно по меньшей мере 95%, и еще более предпочтительно, по меньшей мере 98%. Идентичность аминокислотной последовательности можно рассчитывать общепринятым способом.
[0067]
Полипептид по настоящему изобретению может содержать аминокислоты, составляющие белковую метку, в дополнение к аминокислотной последовательности, составляющей полипептид по настоящему изобретению. Примеры полезных меток включают метки, хорошо известные специалисту в данной области; например, полезные метки включают метки для улучшения эффективности экспрессии и метки для улучшения эффективности очистки, такие как метка His, метка Myc и метка FLAG.
[0068]
Полинуклеотид по настоящему изобретению кодирует полипептид, в котором N-концевая часть белка DCTN1 является слитой с C-концевой частью белка RET, и предпочтительно, представляет собой полинуклеотид, выбранный из следующих (d)-(i). Эти полинуклеотиды, предпочтительно, кодируют полипептиды, которые имеют активность киназы или эффект вызова пролиферации клеток.
[0069]
(d) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0070]
(e) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0071]
(f) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 12, SEQ ID NO: 14, SEQ ID NO: 16, SEQ ID NO: 18, SEQ ID NO: 20, SEQ ID NO: 22 или SEQ ID NO: 24.
[0072]
(g) Полинуклеотид, содержащий последовательность оснований, представленную SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23.
[0073]
(h) Полинуклеотид, гибридизующийся в строгих условиях с полинуклеотидом, содержащим последовательность оснований, комплементарную последовательности оснований, представленной SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23.
[0074]
(i) Полинуклеотид, имеющий по меньшей мере 90% идентичность с последовательностью оснований, представленной SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23.
[0075]
Более предпочтительно, полинуклеотид по настоящему изобретению выбран из следующих (d)-(i). Эти полинуклеотиды предпочтительно кодируют полипептиды, которые имеют активность киназы или эффект вызова пролиферации клеток.
[0076]
(d) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 18.
[0077]
(e) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, где одна или несколько аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 18.
[0078]
(f) Полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, имеющую по меньшей мере 90% идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 18.
[0079]
(g) Полинуклеотид, содержащий последовательность оснований, представленную SEQ ID NO: 17.
[0080]
(h) Полинуклеотид, гибридизующийся в строгих условиях с полинуклеотидом, содержащим последовательность оснований, комплементарную последовательности оснований, представленной SEQ ID NO: 17.
[0081]
(i) Полинуклеотид, имеющий по меньшей мере 90% идентичность с последовательностью оснований, представленной SEQ ID NO: 17.
[0082]
Полинуклеотид по настоящему изобретению включает не только его двухцепочечную ДНК, но также различные типы одноцепочечной ДНК и РНК, составляющие двухцепочечную ДНК, такие как смысловые цепи и антисмысловые цепи. Антисмысловые цепи можно использовать в качестве зондов и т.п. ДНК включает виды ДНК, полученные посредством клонирования, химического синтеза или их комбинации, такие как кДНК и геномная ДНК. Кроме того, последовательности оснований, такие как последовательности нетранслируемой области (UTR), можно добавлять к полинуклеотиду по настоящему изобретению, в дополнение к последовательности оснований для кодирования полипептида по настоящему изобретению.
[0083]
Строгие условия, в рамках изобретения включают, например, условия, описанные в Molecular Cloning: A Laboratory Manual (Second Edition, J. Sambrook et al., 1989). Конкретно, строгие условия включают такие условия, что раствор, содержащий 6×SSC (состав 1×SSC: 0,15 M хлорид натрия, 0,015 M цитрат натрия, pH 7,0), 0,5% SDS, 5×Denhalt и 100 мг/мл ДНК спермы сельди, подвергают изотермической обработке вместе с зондом при 65°C в течение 8-16 часов для проведения гибридизации.
[0084]
Идентичность с последовательностью оснований, представленной SEQ ID NO: 1, SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 11, SEQ ID NO: 13, SEQ ID NO: 15, SEQ ID NO: 17, SEQ ID NO: 19, SEQ ID NO: 21 или SEQ ID NO: 23, предпочтительно, составляет по меньшей мере 90%, более предпочтительно по меньшей мере 95%, и еще более предпочтительно, по меньшей мере 98%. Идентичность последовательности оснований можно рассчитывать общепринятым способом.
[0085]
В этом описании, фраза «имеют активность киназы» в «имеют активность киназы или эффект вызова пролиферации клеток» означает наличие ферментной активности для фосфорилирования тирозина. Кроме того, фраза «имеют эффект вызова пролиферации клеток» в «имеют активность киназы или эффект вызова пролиферации клеток» означает, что трансфекция полинуклеотида и/или полипептида по настоящему изобретению в клетку улучшает способность клетки к пролиферации, по сравнению с клеткой, в которую не трансфицируют полинуклеотид и/или полипептид. Этот эффект можно подтверждать, например, следующим образом: полинуклеотидом и/или полипептидом трансфицируют линию клеток, пролиферирующую в зависимости от цитокинов, и если линия клеток пролиферирует независимо от цитокинов, полинуклеотид и/или полипептид имеет эффект вызова пролиферации клеток.
[0086]
Полинуклеотид по настоящему изобретению можно выделять, например, с использованием библиотеки кДНК или библиотеки геномной ДНК, полученной из злокачественной опухоли щитовидной железы и т.д., сохраняющей ген, слитый из гена DCTN1 и гена RET, с использованием праймера, который специфически гибридизуется с частью последовательности оснований полинуклеотида по настоящему изобретению. Для этого праймера, можно использовать любой праймер любой последовательности и любой длины, при условии, что праймер специфически гибридизуется по меньшей мере с частью полинуклеотида по настоящему изобретению или его антисмысловой цепью. Можно использовать также способ искусственного синтеза полинуклеотидов (Nat. Methods, 11: 499-507, 2014).
[0087]
Экспрессирующий вектор по настоящему изобретению не является конкретно ограниченным, при условии, что экспрессирующий вектор содержит полинуклеотид по настоящему изобретению, и позволяет экспрессию полипептида по настоящему изобретению. Примеры включают экспрессирующие векторы, полученные посредством вставки полинуклеотида по настоящему изобретению в известный экспрессирующий вектор, подходящим образом выбранный в соответствии с хозяином для использования.
[0088]
Хозяин не является конкретно ограниченным, при условии, что хозяин представляет собой живую клетку, которая может подвергаться трансформации, и примеры включают бактерии, такие как E. coli и Bacillus subtilis; настоящие грибы, такие как дрожжи и мицелиальные грибы; клетки насекомых, такие как клетки Sf9; насекомых, таких как шелкопряд; клетки животных; и растения или клетки растительного происхождения.
[0089]
Вектор для вставки полинуклеотида по настоящему изобретению не является конкретно ограниченным, при условии, что вектор является способным к репликации в хозяине. Вектор можно подходящим образом выбирать в соответствии, например, с типом хозяина, подвергаемого введению, и со способом введения. Примеры включают плазмидную ДНК, фаговую ДНК и вирусные векторы. Для векторной ДНК для использования в конструировании экспрессирующего вектора, можно использовать широко распространенную и легко доступную векторную ДНК. Примеры включают pUC19 (Takara Bio Inc.), pTV118N (Takara Bio Inc.), pMAMneo (Clontech Laboratories, Inc.), pGEX (GE Healthcare), pET160 (Invitrogen), pDEST (Invitrogen), pIEx (Merck Millipore) и pBacPAK (Clontech Laboratories, Inc.). Примеры вирусных векторов включают ДНК-вирусы и РНК-вирусы, такие как бакуловирусные векторы, ретровирусные векторы, лентивирусные векторы (например, вирус иммунодефицита человека или HIV), аденовирусные векторы, аденоассоциированные вирусные векторы (вектор AAV), вирус герпеса, вирус осповакцины, поксвирус, вирус полиомиелита, вирус синдбис, вирус сендай и вирус обезьян-40 (SV-40).
[0090]
Трансформацию хозяина с использованием экспрессирующего вектора можно проводить, например, способом протопластов, способом компетентных клеток или способом электропорации. Полученный трансформант можно культивировать в подходящих условиях в среде, содержащей источник углерода, источник азота, соли металлов, витамины и т.д., которые может использовать хозяин.
[0091]
Примеры клетки, трансфицированной полинуклеотидом в соответствии с настоящим изобретением, включают клетки, трансформированные экспрессирующим вектором по настоящему изобретению, и клетки, в которые полинуклеотид по настоящему изобретению введен посредством редактирования генома. Клетка для использования включает клетки-хозяева, перечисленные выше. Примеры способа подтверждения того, была ли клетка трансформирована экспрессирующим вектором, включают способ детекции присутствия полипептида по настоящему изобретению и способ детекции присутствия полинуклеотида по настоящему изобретению.
[0092]
«Клетки, в которые полинуклеотид по настоящему изобретению введен посредством редактирования генома», предпочтительно, представляют собой клетки, имеющие ген, полученный посредством слияния независимо присутствующего гена DCTN1 и гена RET посредством редактирования генома, и более предпочтительно, клетки, имеющие ген, полученный посредством слияния экзона 27 DCTN1 и экзона 12 RET в соответствующих независимо присутствующих гене DCTN1 и гене RET посредством редактирования генома. Эти клетки можно получать общепринятым способом, и примеры включают способ, описанный в Cell Rep., 9(4), pp. 1219-1227 (2014), Nat. Commun., 5, 3728 (2014). Примеры способа подтверждения того, представляет ли клетка собой клетку, в которую полинуклеотид по настоящему изобретению введен посредством редактирования генома, включают способ детекции присутствия полипептида по настоящему изобретению и способ детекции присутствия полинуклеотида по настоящему изобретению.
[0093]
Полипептид по настоящему изобретению можно получать посредством получения культурального раствора и/или клеток посредством культивирования клеток, трансформированных экспрессирующим вектором по настоящему изобретению, в среде, подходящей для культивирования клеток, в подходящих условиях, и затем сбора и очистки белка из культурального раствора и/или клеток типичным способом. Полипептид по настоящему изобретению можно также получать посредством введения экспрессирующего вектора, содержащего полинуклеотид по настоящему изобретению, или РНК-матрицы или ДНК-матрицы, кодирующей полинуклеотид по настоящему изобретению, в бесклеточную систему синтеза белка (например, экстракт, полученный из линии клеток человека, экстракт ретикулоцитов кролика, экстракт зародышей пшеницы и экстракт E. coli); инкубацию полученной смеси в подходящих условиях; и сбор и очистку белка из полученного реакционного раствора типичным способом.
[0094]
По настоящему изобретению, антитело, специфически связывающееcя с полипептидом по настоящему изобретению, включает антитело, которое специфически связывается с точкой слияния N-концевой части белка DCTN1 и C-концевой части белка RET. Антитело относится к антителу, которое специфически связывается с точкой слияния N-концевой части белка DCTN1 и C-концевой части белка RET, но которое не связывается ни с одним из белка DCTN1 дикого типа и RET дикого типа.
[0095]
По настоящему изобретению, фраза «точка слияния» в «точка слияния N-концевой части белка DCTN1 и C-концевой части белка RET» относится к точке, в которой полипептид, происходящий из N-концевой части белка DCTN1, слит с полипептидом, происходящим из C-концевой части белка RET. Точка слияния в SEQ ID NO: 2 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1233 из SEQ ID NO: 2, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1234-1635 из SEQ ID NO: 2, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 4 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1233 из SEQ ID NO: 4, происходящий из N-концевой части DCTN 1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1234-1593 из SEQ ID NO: 4, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 6 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1099 из SEQ ID NO: 6, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1100-1501 из SEQ ID NO: 6, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 8 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1099 из SEQ ID NO: 8, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1100-1459 из SEQ ID NO: 8, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 10 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1208 из SEQ ID NO: 10, происходящий из N-концевой части of DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1209-1610 из SEQ ID NO: 10, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 12 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1208 из SEQ ID NO: 12, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1209-1568 из SEQ ID NO: 12, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 14 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1094 из SEQ ID NO: 14, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1095-1496 из SEQ ID NO: 14, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 16 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1094 из SEQ ID NO: 16, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1095-1454 из SEQ ID NO: 16, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 18 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1191 из SEQ ID NO: 18, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1192-1593 из SEQ ID NO: 18, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 20 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1191 из SEQ ID NO: 20, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1192-1551 из SEQ ID NO: 20, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 22 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1226 из SEQ ID NO: 22, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1227-1628 из SEQ ID NO: 22, происходящим из C-концевой части RET. Точка слияния в SEQ ID NO: 24 представляет собой точку, в которой полипептид, имеющий аминокислотную последовательность из положений 1-1226 из SEQ ID NO: 24, происходящий из N-концевой части DCTN1, слит с полипептидом, имеющим аминокислотную последовательность из положений 1227-1586 из SEQ ID NO: 24, происходящим из C-концевой части RET.
[0096]
Примеры антитела включают иммуноглобулины (например, IgA, IgD, IgE, IgG, IgM и IgY), фрагменты Fab, фрагменты F(ab')2, одноцепочечные фрагменты антител (scFv), однодоменные антитела и диатела (Nat. Rev. Immunol., 6: 343-357, 2006). Они включают, но без ограничения, моноклональные антитела и поликлональные антитела, например, из человеческих антител, гуманизированных антител, химерных антител, мышиных антител, антител ламы и антител курицы.
[0097]
Антитело можно получать различными известными способами, и способ получения не является конкретно ограниченным. Известные способы включают способ, в котором полипептид по настоящему изобретению, фрагмент полипептида, содержащий точку слияния N-концевой части белка DCTN1 и C-концевой части белка RET, или т.п., инокулируют иммунизированному животному для активации иммунной системы животного, и сыворотку животного собирают для получения поликлонального антитела; и способ получения моноклонального антитела, например, способом гибридомы и способом фагового дисплея.
[0098]
Способ скрининга соединения, ингибирующего экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, можно осуществлять способом, включающим следующие стадии (1) и (2).
[0099]
Конкретно, способ скрининга по настоящему изобретению осуществляют способом, включающим:
(1) стадию приведения полипептида по настоящему изобретению, или клетки, экспрессирующей полипептид и/или полинуклеотид по настоящему изобретению, в контакт с тестируемым соединением; и
[0100]
(2) стадию измерения того, ингибированы ли экспрессия и/или активность полипептида по настоящему изобретению, или экспрессия полинуклеотида по настоящему изобретению на стадии (1), или стадию измерения того, ингибирован ли рост клетки на стадии (1).
[0101]
Более предпочтительно, способ скрининга по настоящему изобретению представляет собой способ, включающий следующие стадии (1) и (2).
[0102]
(1) Стадия приведения клетки, экспрессирующей полипептид и/или полинуклеотид по настоящему изобретению, в контакт с тестируемым соединением.
[0103]
(2) Стадия измерения того, ингибирован ли рост клетки на стадии (1).
[0104]
Способ скрининга соединения, ингибирующего экспрессию и/или активность полипептида по настоящему изобретению или экспрессию полинуклеотида по настоящему изобретению, можно осуществлять способом, включающим следующие стадии (1)-(3).
[0105]
Конкретно, способ скрининга по настоящему изобретению осуществляют способом, включающим:
(1) стадию приведения полипептида по настоящему изобретению или клетки, экспрессирующей полипептид и/или полинуклеотид по настоящему изобретению, в контакт с тестируемым соединением;
[0106]
(2) стадию измерения того, ингибирована ли экспрессия и/или активность полипептида по настоящему изобретению, или экспрессия полинуклеотида по настоящему изобретению на стадии (1), или стадию измерения того, ингибирован ли рост клетки на стадии (1); и
[0107]
(3) стадию определения того, что тестируемое соединение ингибирует экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, когда экспрессия и/или активность полипептида по настоящему изобретению, или экспрессия полинуклеотида по настоящему изобретению ингибирована на стадии (2), или когда рост клетки на стадии (1) ингибирован на стадии (2).
[0108]
Более предпочтительно, способ скрининга по настоящему изобретению представляет собой способ, включающий следующие стадии (1) - (3).
[0109]
(1) Стадия приведения клетки, экспрессирующей полипептид и/или полинуклеотид по настоящему изобретению, в контакт с тестируемым соединением.
[0110]
(2) Стадия измерения того, ингибирован ли рост клетки на стадии (1).
[0111]
(3) Стадия определения того, что тестируемое соединение ингибирует экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, когда рост клетки на стадии (1) ингибирован на стадии (2).
[0112]
«Клетка, экспрессирующая полипептид и/или полинуклеотид по настоящему изобретению», включает клетки, трансформированные экспрессирующим вектором по настоящему изобретению, клетки, в которые введен полинуклеотид по настоящему изобретению посредством редактирования генома, первичную культуру клеток, экспрессирующих полипептид и/или полинуклеотид по настоящему изобретению, линии клеток, экспрессирующие полипептид и/или полинуклеотид по настоящему изобретению, и происходящие от пациента с злокачественной опухолью клетки, экспрессирующие полипептид и/или полинуклеотид по настоящему изобретению. Примеры способа подтверждения того, экспрессирует ли клетка полипептид и/или полинуклеотид по настоящему изобретению, включают способ детекции присутствия полипептида по настоящему изобретению и способ детекции присутствия полинуклеотида по настоящему изобретению.
[0113]
По настоящему изобретению, «экспрессия полипептида по настоящему изобретению или экспрессия полинуклеотида по настоящему изобретению ингибирована» в фразе «экспрессия и/или активность полипептида по настоящему изобретению или экспрессия полинуклеотида по настоящему изобретению ингибирована» означает, например, следующее. Клетку, экспрессирующую полипептид и/или полинуклеотид по настоящему изобретению, приводят в контакт с тестируемым соединением, и уровень экспрессии полипептида или полинуклеотида по настоящему изобретению в клетке оценивают с использованием способа детекции присутствия полипептида или полинуклеотида по настоящему изобретению. Когда для клетки, приведенной в контакт с тестируемым соединением, показан статистически значимо уменьшенный уровень экспрессии полипептида или полинуклеотида по настоящему изобретению, по сравнению с клеткой, не приводимой в контакт с тестируемым соединением, определяют, что экспрессия полипептида или полинуклеотида по настоящему изобретению ингибирована.
[0114]
Фраза «активность полипептида по настоящему изобретению ингибирована» в фразе «экспрессия и/или активность полипептида по настоящему изобретению, или экспрессия полинуклеотида по настоящему изобретению ингибирована» означает, например, следующее. Когда для полипептида по настоящему изобретению или клетки, экспрессирующей полипептид по настоящему изобретению, приведенных в контакт с тестируемым соединением, получают статистически значимо меньший процент фосфорилирования тирозина, чем для полипептида или клетки, не приводимых в контакт с тестируемым соединением, определяют, что активность полипептида по настоящему изобретению ингибирована.
[0115]
Когда для клетки, экспрессирующей полипептид по настоящему изобретению, приведенной в контакт с тестируемым соединением, показан статистически значимо ингибированный рост клетки, по сравнению с клеткой, не приводимой в контакт с тестируемым соединением, определяют, что активность полипептида по настоящему изобретению ингибирована.
[0116]
По настоящему изобретению, «фосфорилирование тирозина» включает не только фосфорилирование тирозина в белке RET (включая белок RET, слитый с другим белком), но также фосфорилирование тирозина в белке передачи сигналов ниже RET. Примеры белков передачи сигналов ниже RET включают STAT, AKT и ERK. Фосфорилирование тирозина, предпочтительно, представляет собой фосфорилирование тирозина в белке RET (включая белок RET, слитый с другим белком).
[0117]
«Тирозин процент фосфорилирования» можно измерять с использованием, например, специфического для фосфорилированного RET антитела, посредством Вестерн-блоттинга, иммунопреципитации, иммуногистохимии, ELISA или проточной цитометрии.
[0118]
По настоящему изобретению, «образец» включает не только биологические образцы (например, клетки, ткани, органы, жидкости организма (например, кровь и лимфу), желудочный сок и мочу), но также экстракты нуклеиновой кислоты (например, экстракты геномной ДНК, экстракты мРНК и препараты кДНК, и препараты кРНК, полученные из экстрактов мРНК) и экстракты белка, полученные из этих биологических образцов. Образцы могут представлять собой образцы, подвергнутые формалиновой фиксации, спиртовой фиксации, обработке замораживанием или погружению в парафин. Биологический образец для использования может представлять собой образец, собранный из живого организма, и, предпочтительно, представляет собой образец, полученный от пациента с злокачественной опухолью, и более предпочтительно, образец, содержащий клетки опухолей. Способ сбора биологического образца можно подходящим образом выбирать, в зависимости от типа биологического образца.
[0119]
Настоящее изобретение относится к способу детекции присутствия полипептида по настоящему изобретению в образце.
[0120]
По настоящему изобретению, способ детекции присутствия полипептида по настоящему изобретению в образце включает способы детекции в соответствии с общепринятыми способами, такими как ELISA, Вестерн-блоттинг или иммуногистохимическое окрашивание, с использованием антитела, специфически связывающего полипептид по настоящему изобретению; и FRET (резонансный перенос энергии флуоресценции) с использованием антитела, специфически связывающего белок DCTN1, и антитела, специфически связывающего белок RET. Способ детекции, предпочтительно, представляет собой ELISA, Вестерн-блоттинг или иммуногистохимическое окрашивание с использованием антитела, специфически связывающего полипептид по настоящему изобретению.
[0121]
Антитело, специфически связывающее белок DCTN1, и антитело, специфически связывающее белок RET, предпочтительно, представляет собой антитело, которое связывает N-концевую часть от точки слияния в белке DCTN1, и антитело, которое связывает C-концевую часть от точки слияния в белке RET. Эти антитела могут представлять собой коммерчески доступные продукты или могут быть получены типичным известным способом.
[0122]
По настоящему изобретению, способ детекции присутствия полипептида по настоящему изобретению в образце предпочтительно включает стадию детекции полипептида по настоящему изобретению с использованием антитела, специфически связывающего полипептид по настоящему изобретению, или антитела, специфически связывающего белок DCTN1, и антитела, специфически связывающего белок RET; и более предпочтительно, включает стадию детекции полипептида по настоящему изобретению с использованием антитела, специфически связывающего полипептид по настоящему изобретению. Средства для детекции присутствия полипептида по настоящему изобретению не являются конкретно ограниченными, и их примеры включают комбинацию антитела, специфически связывающего белок DCTN1, и антитела, специфически связывающего белок RET; и антитела, специфически связывающего полипептид по настоящему изобретению.
[0123]
Настоящее изобретение относится к праймеру или зонду для детекции присутствия полинуклеотида по настоящему изобретению в образце. По настоящему изобретению, средства для детекции присутствия полипептида по настоящему изобретению не являются конкретно ограниченными, и их примеры включают праймер или зонд для детекции присутствия полинуклеотида по настоящему изобретению.
[0124]
Праймер или зонд включает полинуклеотиды, выбранные из следующих (j)-(l):
(j) полинуклеотид, представляющий собой по меньшей мере один зонд, выбранный из группы, состоящей из зондов, гибридизующихся с полинуклеотидом, кодирующим белок DCTN1, и зондов, гибридизующихся с полинуклеотидом, кодирующим белок RET;
[0125]
(k) полинуклеотид, представляющий собой зонд, гибридизующийся с точкой слияния между полинуклеотидом, кодирующим белок DCTN1, и полинуклеотидом, кодирующим белок RET; и
[0126]
(l) полинуклеотид, представляющий собой набор из смыслового праймера и антисмыслового праймера, разработанных для обрамления точки слияния между полинуклеотидом, кодирующим белок DCTN1, и полинуклеотидом, кодирующим белок RET.
[0127]
По настоящему изобретению, «точка слияния» в фразе «точка слияния между полинуклеотидом, кодирующим белок DCTN1, и полинуклеотидом, кодирующим белок RET», относится к точке, в которой полинуклеотид, кодирующий белок DCTN1, слит с полинуклеотидом, кодирующим белок RET. Точка слияния в SEQ ID NO: 1 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3699 из SEQ ID NO: 1, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3700-4905 из SEQ ID NO: 1, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 3 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3699 из SEQ ID NO: 3, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3700-4779 из SEQ ID NO: 3, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 5 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3297 из SEQ ID NO: 5, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3298-4503 из SEQ ID NO: 5, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 7 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3297 из SEQ ID NO: 7, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3298-4377 из SEQ ID NO: 7, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 9 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3624 из SEQ ID NO: 9, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3625-4830 из SEQ ID NO: 9, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 11 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3624 из SEQ ID NO: 11, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3625-4704 из SEQ ID NO: 11, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 13 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3282 из SEQ ID NO: 13, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3283-4488 из SEQ ID NO: 13, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 15 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3282 из SEQ ID NO: 15, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3283-4362 из SEQ ID NO: 15, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 17 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3573 из SEQ ID NO: 17, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3574-4779 из SEQ ID NO: 17, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 19 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3573 из SEQ ID NO: 19, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3574-4653 из SEQ ID NO: 19, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 21 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3678 из SEQ ID NO: 21, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3679-4884 из SEQ ID NO: 21, происходящую из полинуклеотида, кодирующего RET. Точка слияния в SEQ ID NO: 23 представляет собой точку, в которой полинуклеотид, имеющий последовательность оснований из положений 1-3678 из SEQ ID NO: 23, происходящую из полинуклеотида, кодирующего DCTN1, слит с полинуклеотидом, имеющим последовательность оснований из положений 3679-4758 из SEQ ID NO: 23, происходящую из полинуклеотида, кодирующего RET.
[0128]
По настоящему изобретению, праймер или зонд получают в форме полинуклеотида, который специфически гибридизуется с полинуклеотидом по настоящему изобретению, на основании информации о последовательности полинуклеотида по настоящему изобретению, в соответствии с типичным известным способом. Количество оснований для праймера или зонда составляет 10-50, предпочтительно, 15-50, и более предпочтительно, 18-35.
[0129]
Праймер или зонд не обязательно должен являться полностью комплементарным, при условии, что праймер или зонд специфически гибридизуется с полинуклеотидом по настоящему изобретению. Праймер или зонд представляет собой полинуклеотид, имеющий по меньшей мере 70% идентичность, предпочтительно, по меньшей мере 80% идентичность, более предпочтительно, по меньшей мере 90% идентичность, более предпочтительно, по меньшей мере 95% идентичность, и более предпочтительно, по меньшей мере 98% идентичность с соответствующей последовательность оснований.
[0130]
Праймер или зонд по настоящему изобретению, предпочтительно, представляет собой полинуклеотид, представленный (i) SEQ ID NO: 69, (ii) SEQ ID NO: 70 или (iii) SEQ ID NO: 71, более предпочтительно, полинуклеотид, представляющий собой набор из смыслового праймера и антисмыслового праймера, представленный (iv) SEQ ID NO: 69 и SEQ ID NO: 70, и более предпочтительно, полинуклеотид, представляющий собой набор из смыслового праймера, антисмыслового праймера и зонда, представленный (v) SEQ ID NO: 69, SEQ ID NO: 70 и SEQ ID NO: 71.
[0131]
Настоящее изобретение относится к способу детекции присутствия полинуклеотида по настоящему изобретению в образце.
[0132]
По настоящему изобретению, способ детекции присутствия полинуклеотида по настоящему изобретению в образце представляет собой способ детекции в соответствии с общепринятым способом детекции, таким как Нозерн-блоттинг, Саузерн-блоттинг, RT-ПЦР, ПЦР с детекцией в реальном времени, цифровая ПЦР, микромассив ДНК, гибридизация in situ и анализ последовательности.
[0133]
По настоящему изобретению, способ детекции присутствия полинуклеотида по настоящему изобретению в образце также включает способ детекции присутствия полинуклеотида слитого гена RET, включая полинуклеотид по настоящему изобретению. Способ включает способ, в котором продукт ПЦР, амплифицированный способом 5' RACE с использованием праймера, который гибридизуется с полинуклеотидом, кодирующим белок RET (например, праймера, который гибридизуется с последовательностью с 3'-стороны от киназного домена RET), подвергают анализу последовательности.
[0134]
По настоящему изобретению, способ детекции присутствия полинуклеотида по настоящему изобретению в образце, предпочтительно, включает стадию детекции полинуклеотида по настоящему изобретению с использованием праймера или зонда по настоящему изобретению.
[0135]
Настоящее изобретение относится к фармацевтической композиции для лечения злокачественной опухоли, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET, где композиция содержит соединение, ингибирующее RET, в качестве активного ингредиента.
[0136]
Более предпочтительно, настоящее изобретение относится к фармацевтической композиции для лечения злокачественной опухоли, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET, где композиция содержит соединение, ингибирующее экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, в качестве активного ингредиента.
[0137]
По настоящему изобретению, «злокачественная опухоль, которая является положительной по гену, слитому из гена DCTN1 и гена RET», в фразе «злокачественная опухоль, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET», относится к злокачественной опухоли, которая экспрессирует полинуклеотид по настоящему изобретению, и предпочтительно, к злокачественной опухоли, в которой полинуклеотид по настоящему изобретению детектирован с использованием способа детекции присутствия полинуклеотида по настоящему изобретению.
[0138]
По настоящему изобретению, «злокачественная опухоль, которая является положительной по белку, слитому из белка DCTN1 и белка RET», в фразе «злокачественная опухоль, которая является положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET», относится к злокачественной опухоли, которая экспрессирует полипептид по настоящему изобретению, и предпочтительно, к злокачественной опухоли, в которой полипептид по настоящему изобретению детектирован с использованием способа детекции присутствия полипептида по настоящему изобретению.
[0139]
Активный ингредиент фармацевтической композиции для лечения злокачественной опухоли в соответствии с настоящим изобретением представляет собой соединение, ингибирующее RET, и более предпочтительно, соединение, ингибирующее экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению. Соединение, выбранное способом скрининга по настоящему изобретению, можно также использовать в качестве активного ингредиента. Например, соединение, как известно, ингибирующее RET, можно использовать в качестве активного ингредиента фармацевтической композиции по настоящему изобретению. Соединение, ингибирующее RET, может представлять собой соединение, ингибирующее экспрессию и/или активность других тирозинкиназ, при условии, что соединение может ингибировать экспрессию и/или активность RET, и более предпочтительно, соединение, которое может ингибировать активность RET и экспрессию и/или активность других тирозинкиназ. Примеры таких соединений включают вандетаниб, сорафениб, сунитиниб, мотесаниб, кабозантиниб, ленватиниб и соединения, описанные в брошюре WO2016/127074, брошюре WO2017/043550, брошюре WO2017/011776 и брошюре WO2017/146116.
Активный ингредиент для фармацевтической композиции для лечения злокачественной опухоли, положительной по гену, слитому из гена DCTN1 и гена RET, и/или положительной по белку, слитому из белка DCTN1 и белка RET, представляет собой соединение, ингибирующее RET; более предпочтительно, вандетаниб, кабозантиниб, ленватиниб, конденсированное соединение пиримидина, представленное формулой (1), описанное в брошюре WO2017/043550, и конденсированное соединение пиримидина, представленное формулой (1), описанное в брошюре WO2017/146116; более предпочтительно, вандетаниб, кабозантиниб, ленватиниб, примеры соединений 1-90, описанные в брошюре WO2017/043550, и примеры соединений 1-207, описанные в брошюре WO2017/146116; еще более предпочтительно, вандетаниб, кабозантиниб, ленватиниб, 4-амино-1-(трет-бутил)-N-(5-метил-1H-пиразол-3-ил)-1H-пиразолo[3,4-d]пиримидин-3-карбоксамид, 4-амино-7-(трет-бутил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-7-(1-фтор-2-метилпропан-2-ил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-(5-метил-1H-пиразол-3-ил)-7-(1-метилциклопропил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-7-(2-циклопропилпропан-2-ил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-(3-морфолинопро-1-пин-1-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-((тетрагидро-2H-пиран-4-ил)этинил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, (R)-4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-((тетрагидрофуран-2-ил)метокси)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид и 4-амино-N-[4-(метоксиметил)фенил]-6-((1-метил-1H-пиразол-4-ил)этинил)-7-(1-метилциклопропил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид; и особенно предпочтительно, 4-амино-1-(трет-бутил)-N-(5-метил-1H-пиразол-3-ил)-1H-пиразолo[3,4-d]пиримидин-3-карбоксамид, 4-амино-7-(трет-бутил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-7-(1-фтор-2-метилпропан-2-ил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-(5-метил-1H-пиразол-3-ил)-7-(1-метилциклопропил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-7-(2-циклопропилпропан-2-ил)-N-(5-метил-1H-пиразол-3-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-(3-морфолинопро-1-пин-1-ил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, 4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-((тетрагидро-2H-пиран-4-ил)этинил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид, (R)-4-амино-N-[4-(метоксиметил)фенил]-7-(1-метилциклопропил)-6-((тетрагидрофуран-2-ил)метокси)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид и 4-амино-N-[4-(метоксиметил)фенил]-6-((1-метил-1H-пиразол-4-ил)этинил)-7-(1-метилциклопропил)-7H-пирроло[2,3-d]пиримидин-5-карбоксамид.
[0140]
По настоящему изобретению, «может ингибировать экспрессию RET» в фразе «соединение может ингибировать экспрессию и/или активность RET» означает, например, следующее. Клетку, экспрессирующую полипептид и/или полинуклеотид RET, приводят в контакт с тестируемым соединением, и детектируют уровень экспрессии полипептида или полинуклеотида RET в клетке. Когда для клетки, приведенной в контакт с тестируемым соединением, показывают более низкий уровень экспрессии полипептида или полинуклеотида RET, чем в клетке, которую не приводили в контакт с тестируемым соединением, определяют, что экспрессия RET ингибирована. Такие соединения включают соединения, описанные выше, миРНК, мкРНК и аптамеры нуклеиновой кислоты (ДНК, РНК). Примеры миРНК включают CACAUGUCAUCAAAUUGUATT (SEQ ID NO: 74), GGAUUGAAAACAAACUCUATT (SEQ ID NO: 75) и GCUUGUCCCGAGAUGUUUATT (SEQ ID NO: 76); и миРНК, предпочтительно, представляет собой CACAUGUCAUCAAAUUGUATT (SEQ ID NO: 74) или GGAUUGAAAACAAACUCUATT (SEQ ID NO: 75).
[0141]
Может ли соединение «ингибировать активность RET», в фразе «соединение может ингибировать экспрессию и/или активность RET», можно определять с использованием фосфорилирования тирозина в качестве индикатора. Примеры способа измерения фосфорилирования тирозина включают способ, описанный в тестовом примере 1 в брошюре WO2017/043550.
[0142]
Кроме того, соединение можно определять как способное ингибировать активность RET с использованием клетки, экспрессирующей полипептид и/или полинуклеотид RET, когда эффект ингибирования роста клетки является индикатором. Примеры способа измерения эффекта ингибирования роста клетки включают способ, описанный в тестовом примере 3 и тестовом примере 4 из брошюры WO2017/043550.
[0143]
Злокачественная опухоль, на которую нацелена фармацевтическая композиция по настоящему изобретению, не является конкретно ограниченной, при условии, что злокачественная опухоль экспрессирует полинуклеотид и/или полипептид по настоящему изобретению, и примеры включают рак головы и шеи, рак щитовидной железы, злокачественную опухоль желудочно-кишечного тракта (например, рак пищевода, рак желудка, рак двенадцатиперстной кишки, рак печени, рак желчевыводящих путей (например, рак желчного пузыря и рак желчного протока), рак поджелудочной железы, рак тонкого кишечника, рак кишечника (например, колоректальный рак, рак ободочной кишки, рак прямой кишки) и стромальную опухоль желудочно-кишечного тракта), рак легкого (немелкоклеточный рак легкого, мелкоклеточный рак легкого), рак молочной железы, рак яичника, рак матки (например, рак шейки матки, рак эндометрия), рак почки, рак мочевого пузыря, рак предстательной железы и рак кожи. Злокачественная опухоль, предпочтительно, представляет собой рак щитовидной железы или рак легкого (немелкоклеточный рак легкого, мелкоклеточный рак легкого). Злокачественная опухоль, в рамках изобретения, включает не только первичные опухоли, но также злокачественную опухоль, распространившуюся на другие органы (например, печень).
[0144]
Препарат, содержащий соединение, ингибирующее экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, в качестве активного ингредиента, можно получать в форме фармацевтической композиции, содержащей фармацевтический носитель, так чтобы она подходила для множества лекарственных форм. Примеры лекарственных форм включают пероральные средства, средства для инъекций, суппозитории, мази и пластыри. Эти лекарственные формы можно получать способами получения, известными и общепринятыми для специалиста в данной области.
[0145]
Фармацевтический носитель для использования включает различные органические или неорганические вещества-носители, общепринятые в качестве материалов для получения препаратов, и их добавляют в качестве наполнителей, связующих веществ, дезинтегрирующих средств, смазывающих средств, покрывающих средств и т.п. для твердых составов; и растворителей, солюбилизирующих средств, суспендирующих средств, средств для придания тоничности, регуляторов pH и буферов, успокаивающих средств и т.п. для жидких составов. Необязательно, полезными являются такие добавки для составов, как консерванты, антиоксиданты, окрашивающие средства, ароматизаторы и стабилизаторы.
[0146]
При получении перорального твердого состава, наполнитель, необязательно, с наполнителем, связующим, дезинтегрирующим средством, смазывающим средством, окрашивающим средством, ароматизатором и т.д., добавляют к соединению по настоящему изобретению, и затем таблетки, покрытые таблетки, гранулы, порошки, капсулы и т.п. получают в соответствии с обычным способом.
[0147]
При получении перорального жидкого состава, регулятор pH и буфер, стабилизатор, ароматизатор и т.п. добавляют к соединению по настоящему изобретению, и жидкое лекарственное средство для внутреннего применения, лекарственное средство в форме сиропа, эликсир и т.п., получают в соответствии с обычным способом.
[0148]
При получении составов для инъекций, регулятор pH и буфер, стабилизатор, средство для придания тоничности, местный анестетик, и т.п. добавляют к соединению по настоящему изобретению, и затем средство для подкожной, внутримышечной или внутривенной инъекции получают в соответствии с обычным способом.
[0149]
Настоящее изобретение относится к способу диагностики злокачественной опухоли, когда присутствие полипептида по настоящему изобретению или полинуклеотида по настоящему изобретению детектируют в образце способом детекции присутствия полипептида по настоящему изобретению, или способом детекции присутствия полинуклеотида по настоящему изобретению. Злокачественная опухоль, диагностируемая по этому изобретению, включает злокачественные опухоли, перечисленные в качестве мишеней фармацевтической композиции по настоящему изобретению. Как описано выше, использование полипептида или полинуклеотида по настоящему изобретению позволяет диагностику злокачественной опухоли. Таким образом, полипептид и полинуклеотид по настоящему изобретению можно использовать в качестве биомаркера для детекции злокачественной опухоли.
[0150]
Настоящее изобретение относится к способу применения полипептида по настоящему изобретению или полинуклеотида по настоящему изобретению в качестве индикатора для определения того, эффективна ли химиотерапия с использованием соединения, ингибирующего RET, где способ определяет, что химиотерапия с использованием соединения, ингибирующего RET, является эффективной, когда полипептид по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению, и/или когда присутствие полинуклеотида по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению.
[0151]
Более предпочтительно, настоящее изобретение относится к способу с использованием полипептида по настоящему изобретению или полинуклеотида по настоящему изобретению в качестве индикатора для определения того, эффективна ли химиотерапия с использованием соединения, ингибирующего экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, где способ определяет, что химиотерапия с использованием соединения, ингибирующего экспрессию и/или активность полипептида по настоящему изобретению, или экспрессию полинуклеотида по настоящему изобретению, является эффективной, когда полипептид по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению, и/или когда присутствие полинуклеотида по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению.
[0152]
Более предпочтительно, настоящее изобретение относится к способу с использованием полипептида по настоящему изобретению или полинуклеотида по настоящему изобретению в качестве индикатора для определения того, эффективна ли химиотерапия с использованием соединения, полученного в способе скрининга по настоящему изобретению, где способ определяет, что химиотерапия с использованием соединения, полученного в способе скрининга по настоящему изобретению, является эффективной, когда полипептид по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению, и/или когда присутствие полинуклеотида по настоящему изобретению детектируют в образце посредством способа детекции по настоящему изобретению.
[0153]
Следующие примеры подробно описывают настоящее изобретение. Однако, настоящее изобретение не является ограниченным этими примерами.
Примеры
[0154]
Пример 1: Получение гена, слитого из гена DCTN1 и гена RET (слитого гена DCTN1-RET)
1-1: Выделение РНК, происходящей из клинического образца
РНК выделяли из ткани злокачественной опухоли щитовидной железы человека, закупленной из Asterand Bioscience, с использованием набора RNeasy Mini (Qiagen) в соответствии со следующим способом. 600 мкл буфера RLT добавляли к ткани злокачественной опухоли щитовидной железы, и наносили на колонку для центрифугирования QIAshredder, с последующим центрифугированием (16000 об/мин, 2 минуты, комнатная температура), таким образом, собирая фильтрат. Эквивалентное количество 70% водного раствора этанола добавляли к собранному фильтрату. После того, как их смешивали, смесь наносили на колонку RNeasy Mini и затем центрифугировали (10000 об/мин, 15 секунд, комнатная температура). 700 мкл буфера RW1 добавляли в колонку RNeasy Mini и центрифугировали (10000 об/мин, 15 секунд, комнатная температура). 500 мкл буфера RPE дополнительно добавляли к этому и центрифугировали (10000 об/мин, 15 секунд, комнатная температура). Таким же способом, 500 мкл буфера RPE добавляли снова, и центрифугировали (10000 об/мин, 2 минуты, комнатная температура). Колонку RNeasy Mini центрифугировали снова (16000 об/мин, 1 минута, комнатная температура), и оставшийся буфер удаляли. 40 мкл свободной от РНКазы воды наносили на колонку RNeasy Mini и центрифугировали (10000 об/мин, 1 минута, комнатная температура), таким образом, собирая фильтрат как тотальную РНК.
[0155]
1-2: Получение кДНК, происходящей из клинического образца
кДНК синтезировали из тотальной РНК, полученной в разделе 1-1 выше, с использованием набора для синтеза кДНК SuperScript VILO (Invitrogen) в соответствии со следующим способом. 500 нг тотальной РНК доводили свободной от РНКазы водой для получения количества 14 мкл, и к этому добавляли 4 мкл реакционной смеси 5×VILO и 2 мкл смеси ферментов 10×SuperScript Mix, и перемешивали. Смесь поддерживали теплой при 25°C в течение 10 минут, и затем поддерживали теплой при 42°C в течение 60 минут. Для остановки реакции, смесь, наконец, инкубировали при 85°C в течение 5 минут, таким образом, получая кДНК.
[0156]
1-3: Получение и очистка клонирующего вектора
Для амплификации слитого гена DCTN1-RET, разработаны праймеры, показанные в таблице 1: праймер 1 (SEQ ID NO: 33) в качестве смыслового праймера и праймер 2 (SEQ ID NO: 34) в качестве антисмыслового праймера, так же, как праймер 3 (SEQ ID NO: 35) в качестве смыслового праймера и праймер 4 (SEQ ID NO: 36) в качестве антисмыслового праймера для использования в гнездовой ПЦР.
[0157]
Таблица 1
[0158]
Слитый ген DCTN1-RET амплифицировали с использованием кДНК, синтезированной в разделе 1-2 выше, в качестве матрицы, с использованием этих праймеров и с использованием KOD-Plus-Neo (Toyobo), в соответствии со следующим способом. 2 мкл кДНК, 5 мкл 10×буфера для ПЦР для KOD-Plus-Neo, 5 мкл 2 мМ dNTP, 3 мкл 25 мМ MgSO4, 1 мкл KOD-Plus-Neo, 1,5 мкл праймера 1 (10 мкМ), 1,5 мкл праймера 2 (10 мкМ) и 31 мкл дважды дистиллированной воды (DDW) смешивали; и проводили ПЦР. Затем полученный продукт ПЦР разводили в 100 раз, и 2 мкл разведенного продукта ПЦР, 5 мкл 10×буфера для ПЦР для KOD-Plus-Neo, 5 мкл 2 мМ dNTP, 3 мкл 25 мМ MgSO4, 1 мкл KOD-Plus-Neo, 1,5 мкл праймера 3 (10 мкМ), 1,5 мкл праймера 4 (10 мкМ) и 31 мкл DDW смешивали; и проводили гнездовую ПЦР.
[0159]
Продукт гнездовой ПЦР разделяли посредством электрофореза с использованием 1% агарозного геля (Nacalai Tesque), и продукт ПЦР очищали из геля с использованием набора QIAquick Gel Extraction (Qiagen).
[0160]
ДНК pUC18 (Takara Bio Inc.), расщепленную рестрикционным ферментом SmaI (NEB), очищенный продукт ПЦР, T4 ДНК-лигазу (NEB) и реакционный буфер для T4 ДНК-лигазы (NEB) смешивали, и смесь инкубировали при 16°C в течение ночи. Продукт лигирования обрабатывали SmaI (NEB), и трансформацию компетентных клеток проводили следующим способом. Продукт лигирования, обработанный SmaI, добавляли к 50 мкл компетентных клеток E. coli DH5α (Takara Bio Inc.), и позволяли стоять на льду в течение 30 минут. Затем клетки подвергали тепловому шоку при 42°C в течение 30 секунд, и позволяли стоять на льду в течение 2 минут. К этому добавляли среду SOC (Takara Bio Inc.), и клетки культивировали с качанием при 37°C в течение 1 час. Затем культуральный раствор наносили на содержащую ампициллин чашку с агаризованной средой LB (Unitech), и позволяли стоять при 37°C в течение ночи. Колонии E. coli суспендировали в содержащей ампициллин среде LB (InvivoGen), и культивировали с качанием при 37°C в течение ночи. Плазмидную ДНК со вставкой слитого гена DCTN1-RET очищали из пролиферирующих E. coli с использованием набора QIAquick Spin Miniprep (Qiagen), в соответствии с протоколом, предоставленным с набором.
[0161]
1-4: Определение последовательности
Реакцию секвенирования проводили с использованием плазмидной ДНК, полученной в разделе 1-3 выше, в качестве матрицы, с использованием праймеров 5-36 для секвенирования, показанных в таблице 2, и с использованием набора для секвенирования BigDye Terminator V3.1 Cycle Sequencing; и анализ последовательности проводили с использованием анализатора ДНК Applied Biosystems 3730xl. Результаты анализа последовательности выявили, что слитый ген DCTN1-RET представлял собой ген (SEQ ID NO: 17), в котором экзоны 12-20 из варианта 2 RET (номер доступа в GenBank: NM_020975) были слиты ниже 3'-конца с экзонами 1-27 из варианта 5 DCTN1 (номер доступа в GenBank: NM_001190836).
[0162]
Таблица 2
[0163]
Пример 2: Детекция слитого гена DCTN1-RET
кДНК синтезировали из РНК, происходящей из нормальной ткани щитовидной железы человека, закупленной из Asterand Bioscience, и из РНК, происходящей из ткани злокачественной опухоли щитовидной железы человека, полученной в разделе 1-1, с использованием набора для синтеза кДНК SuperScript VILO (Invitrogen), в соответствии со следующим способом. 280 нг тотальной РНК доводили свободной от РНКазы водой для получения количества 14 мкл, и к этому добавляли, соответственно, 4 мкл реакционной смеси 5×VILO и 2 мкл смеси ферментов 10×SuperScript, и перемешивали. Смесь поддерживали теплой при 25°C в течение 10 минут, и затем поддерживали теплой при 42°C в течение 60 минут. Для окончания реакции, смесь, наконец, инкубировали при 85°C в течение 5 минут.
[0164]
Для детекции слитого гена DCTN1-RET, разработаны праймеры и зонд, показанные в таблице 3: праймер 37 (SEQ ID NO: 69) в качестве смыслового праймера для детекции слитого гена DCTN1-RET, праймер 38 (SEQ ID NO: 70) в качестве антисмыслового праймера для детекции слитого гена DCTN1-RET, и праймер 39 (SEQ ID NO: 71) в качестве зонда для детекции слитого гена DCTN1-RET (зонд: TaqMan MGB; флуоресцентный краситель: FAM (Thermo Fisher Scientific)).
[0165]
Таблица 3
[0166]
Полученную кДНК разводили в 10 раз, и 1,1 мкл этого использовали в качестве матрицы. 11 мкл смеси для цкПЦР для зонда ddPCR Supermix (Bio-Rad), 2 мкл праймера 37 (10 мкМ), 2 мкл праймера 38 (10 мкМ), 0,6 мкл праймера 39 (10 мкМ) и 1,1 мкл смеси для анализа 20×HEX для детекции GAPDH (Prime PCR ddPCR Expression Probe Assay: GAPDH, Human, Bio-Rad) смешивали; и капли получали с использованием автоматизированного генератора капель (Bio-Rad). ПЦР проводили в полученных каплях, и капли, положительные по DCTN1-RET и GAPDH, подсчитывали с использованием считывателя капель (Bio-Rad). На фиг. 1 и 2 показаны результаты.
[0167]
Слитый ген DCTN1-RET амплифицировали с использованием кДНК, синтезированной выше, в качестве матрицы, с использованием KOD-Plus-Neo (Toyobo) в соответствии со следующим способом. 2 мкл кДНК, 5 мкл 10×буфера для ПЦР для KOD-Plus-Neo, 5 мкл 2 мМ dNTP, 3 мкл 25 мМ MgSO4, 1 мкл KOD-Plus-Neo, 1,5 мкл праймера 1 (10 мкМ), 1,5 мкл праймера 2 (10 мкМ) и 31 мкл DDW смешивали, и проводили ПЦР. Затем, полученный продукт ПЦР разводили в 100 раз, и 2 мкл разведенного продукта ПЦР, 5 мкл 10×буфера для ПЦР для KOD-Plus-Neo, 5 мкл 2 мМ dNTP, 3 мкл 25 мМ MgSO4, 1 мкл KOD-Plus-Neo, 1,5 мкл праймера 3 (10 мкМ), 1,5 мкл праймера 4 (10 мкМ) и 31 мкл of DDW смешивали; и проводили гнездовую ПЦР. Продукт гнездовой ПЦР разделяли посредством электрофореза с использованием 1% агарозного геля (Nacalai Tesque) и фотографировали. На фиг. 3 показаны результаты.
[0168]
Как показано на фиг. 1, фиг. 2 и фиг. 3, в то время как слитый ген DCTN1-RET детектирован в кДНК, синтезированной из РНК, происходящей из ткани злокачественной опухоли щитовидной железы человека, слитый ген DCTN1-RET не детектирован в кДНК, синтезированной из РНК, происходящей из нормальной ткани щитовидной железы человека. Эти результаты показывают, что слитый ген DCTN1-RET можно использовать в качестве биомаркера для злокачественной опухоли.
[0169]
Пример 3: Конструирование экспрессирующего вектора для слитого гена DCTN1-RET
Для конструирования экспрессирующего вектора, праймер 40 (SEQ ID NO: 72) в качестве смыслового праймера и праймер 41 (SEQ ID NO: 73) в качестве антисмыслового праймера, сконструированы, как показано в таблице 4.
[0170]
Таблица 4
[0171]
Слитый ген DCTN1-RET амплифицировали с использованием этих праймеров, кДНК, синтезированной в разделе 1-2 выше, в качестве матрицы, и ДНК-полимеразы Prime STAR Max (TaKaRa), в соответствии со способом, описанным ниже. 1 мкл кДНК, 25 мкл 2× ДНК-полимеразы Prime STAR Max, 1 мкл праймера 40 (10 мкМ), 1 мкл праймера 41 (10 мкМ) и 22 мкл дважды дистиллированной воды (DDW) смешивали для проведения ПЦР. Полученный продукт ПЦР разделяли посредством электрофореза с использованием 1% агарозного геля (Nacalai Tesque), и продукт ПЦР очищали из геля с использованием набора GFX PCR DNA and Gel Band Purification Kit (GE Healthcare). Затем очищенный продукт ПЦР вставляли в вектор Gateway pDONR221 с использованием смеси ферментов Gateway BP Clonase II (Thermo Fisher) в соответствии со способом, описанным ниже, таким образом, получая входной вектор. Конкретно, 5,0 мкл очищенного продукта ПЦР, 3,5 мкл pDONR221 (85 нг/мкл), 4,0 мкл смеси ферментов BP Clonase II и 7,5 мкл TE смешивали, и инкубировали при 25°C в течение 90 минут. После инкубации, к этому добавляли 1 мкл протеиназы K (2 мг/мл), с последующей инкубацией при 37°C в течение 10 минут, таким образом, получая входной вектор.
[0172]
Полученный входной вектор добавляли к 50 мкл компетентных клеток E. coli DH5α (Takara Bio Inc.), и позволяли стоять на льду в течение 30 минут. Затем клетки подвергали тепловому шоку при 37°C в течение 20 секунд и позволяли стоять в течение 2 минут на льду. Затем к этому добавляли среду SOC (Takara Bio Inc.), и клетки подвергали культивированию с качанием при 37°C в течение 1 часа. Затем культуральный раствор наносили на содержащую канамицин чашку с агаризованной средой LB и позволяли стоять при 37°C в течение ночи. Колонии Escherichia coli суспендировали в содержащей канамицин среде LB, и подвергали культивированию с качанием при 37°C в течение ночи. Из выращенной Escherichia coli, плазмидную ДНК с вставкой слитого гена DCTN1-RET (клон входного вектора) очищали с использованием автоматизированной системы для выделения ДНК GENE PREP STAR PI-480 (Kurabo Industries Ltd.).
[0173]
Слитый ген DCTN1-RET вставляли в вектор pJTI Fast DEST с использованием полученной плазмиды и смеси ферментов Gateway LR Clonase II (Thermo Fisher) в соответствии со способом, описанным ниже, таким образом, получая экспрессирующий вектор.
150 нг клона входного вектора, 1 мкл вектора pJTI Fast DEST (150 нг/мкл), 2 мкл смеси ферментов LR Clonase II и буфер TE смешивали для получения всего 10 мкл, и смесь инкубировали при 25°C в течение 90 минут. После инкубации, к этому добавляли 1 мкл протеиназы K (2 мг/мл), с последующей инкубацией при 37°C в течение 10 минут, таким образом, получая вектор pJTI Fast DEST со вставленным слитым геном DCTN1-RET (вектор, экспрессирующий слитый ген DCTN1-RET). Полученный вектор, экспрессирующий слитый ген DCTN1-RET, добавляли к 50 мкл компетентных клеток E. coli DH5α (Takara Bio Inc.) и позволяли стоять в течение 30 минут на льду. Затем, клетки подвергали тепловому шоку при 37°C в течение 20 секунд, и позволяли стоять в течение 2 минут на льду. Затем к этому добавляли среду SOC (Takara Bio Inc.), и клетки подвергали культивированию с качанием при 37°C в течение 1 часа. Культуральный раствор наносили на содержащую ампициллин чашку с агаризованной средой LB и позволяли стоять при 37°C в течение ночи. Колонии Escherichia coli суспендировали в содержащей ампициллин среде LB, и подвергали культивированию с качанием при 37°C в течение ночи. Из выращенной Escherichia coli, плазмидную ДНК с вставкой слитого гена DCTN1-RET (вектор, экспрессирующий слитый ген DCTN1-RET) очищали с использованием набора Plasmid Plus Maxi (QIAGEN).
[0174]
Пример 4: Получение клетки, экспрессирующей слитый ген DCTN1-RET
4-1: Получение клетки
Для клетки-хозяина для получения клетки, экспрессирующей слитый ген DCTN1-RET, выбраны клетки эмбриональные фибробласты мыши NIH/3T3 (Американская коллекция типовых культур), и клетки трансфицировали экспрессирующим вектором с вставленным слитым геном DCTN1-RET, полученным выше, таким образом, получая клетку, экспрессирующую слитый ген DCTN1-RET. Детали способа являются следующими. Клетки NIH/3T3 получали посредством культивирования в среде для типичной культуры (2-мерной культуры клеток) при 37°C в 5% CO2; и среду для использования в 2-мерной культуре клеток получали посредством добавления сыворотки новорожденного теленка (NBCS) (Gibco) в D-MEM (с высоким содержанием глюкозы) (содержащую L-глутамин, фенол красный, пируват натрия, 1500 мг/л гидрокарбоната натрия) (Wako) для получения 10%. За сутки до проведения трансфекции, клетки NIH/3T3 рассевали в 6-луночный планшет (Iwaki) при 1,5×105 клеток/2 мл и инкубировали при 37°C в 5% CO2 в течение ночи. Реагент для трансфекции ViaFect добавляли в смешанный раствор, полученный посредством смешивания 1,5 мкг вектора, экспрессирующего слитый ген DCTN1-RET, и 1,5 мкг вектора pJTI с интегразой phiC31, таким образом, чтобы количество реагента для трансфекции ViaFect в шесть раз превышало количество смешанного раствора; и затем к этому добавляли Opti-MEM для получения общего количества 300 мкл, с последующей инкубацией при комнатной температуре в течение 5 минут, таким образом, получая раствор для трансфекции. Из лунок, в которые рассевали клетки NIH/3T3, отбирали по 300 мкл среды, и 300 мкл полученного раствора для трансфекции добавляли в лунки, с последующей инкубацией при 37°C в 5% CO2 в течение ночи. На следующие сутки, среду заменяли для удаления раствора для трансфекции. При замене среды, гигромицин B (Nacalai Tesque) добавляли в новую среду для получения 500 мкг/мл. Гигромицин B удаляет клетки, не трансфицированные вектором, экспрессирующим вставленный слитый ген DCTN1-RET. После трансфекции, при замене среды приблизительно дважды в неделю, клетки культивировали, пока они не пролиферируют. Через 22 суток после трансфекции, клетки собирали с использованием трипсина, и проводили клонирование отдельных клеток, в соответствии со следующим способом. Измеряли количество собранных клеток, и к ним добавляли среду для получения 1 клетки/200 мкл. Клетки рассевали в 96-луночный планшет (Thermo Fisher) для получения 200 мкл на лунку. После рассева, клетки наблюдали ежесуточно, и отбирали клетки, выращенные из отдельных клеток; эти клетки представляли собой клетки, экспрессирующие слитый ген DCTN1-RET- (экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3).
[0175]
4-2: Подтверждение экспрессии белка-мишени
Экспрессию слитого белка DCTN1-RET в полученных экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 подтверждали посредством Вестерн-блоттинга. Конкретно, среду отбирали из культурального флакона, с последующей однократной промывкой PBS. Концентрат разбавителя 2 для образцов (R&D Systems), содержащий ингибитор фосфатаз (Roche) и ингибитор протеаз (Roche), добавляли в культуральный флакон, и лизат клеток собирали с использованием скребка. Из собранного лизата клеток, образец белка получали посредством центрифугирования. Образец белка подвергали определению белка, и концентрацию белка доводили до предписанной концентрации. Раствор буфера для образцов с восстанавливающим реагентом (6x) для SDS-PAGE (Nacalai Tesque) добавляли к образцу белка предписанной концентрации; и смесь инкубировали при 95°C в течение 5 минут для денатурации белка, таким образом, получая образец для использования в Вестерн-блоттинге. Для отрицательного контроля, образец для Вестерн-блоттинга получали с использованием клеток NIH/3T3 (исходной линии клеток), в соответствии с таким же способом. Экспрессию белка подтверждали с использованием образцов, в соответствии со способом, описанным ниже. Белок разделяли с использованием 4-15% акриламидного геля (Bio-Rad) и 1×буфера Трис/глицин/SDS посредством SDS-PAGE (в течение 30 минут при 200 В). Белок переносили на мембрану PVDF с использованием набора для переноса Trans-Blot Turbo RTA Midi PVDF Transfer Kit (Bio-Rad) и системы для переноса Trans-Blot Turbo (Bio-Rad), и мембрану PVDF погружали в блокирующий раствор Blocking One-P на 1 час. Раствор получали посредством разведения Blocking One-P с TBS-T для получения 10%, и первичное антитело (антитело против фосфо-RET (Tyr905) (CST), mAb кролика против Ret (C31B4) (CST) и антитело против Dctn1 (Atlas Antibodies)) разводили полученным раствором для получения концентрации 1/1000. Мембрану PVDF погружали в полученный раствор и инкубировали при 4°C в течение ночи. После промывки с использованием TBS-T, мембрану PVDF погружали в раствор разведенного вторичного антитела, который получали посредством разведения антитела к IgG кролика, связанного с HRP (CST), с использованием TBS-T для получения концентрации 1/2000; и инкубировали при комнатной температуре в течение 1 час. После промывки мембраны с использованием TBS-T, белок детектировали с использованием субстрата SuperSignal West Dura Extended Duration (Thermo Fisher) с использованием анализатора изображений Amersham Imager 600 lumino (GE Healthcare). Молекулярную массу детектированного белка подтверждали с использованием предварительно окрашенных белковых стандартов Precision Plus Protein Kaleidoscope (Bio-Rad). Как показано на фиг. 4a) и 4b), эндогенный RET (150 и 175 кДа) не детектировали, когда использовали антитело против pRET или антитело против RET. Однако, полосу, по-видимому, являющуюся слитым белком DCTN1-RET, подтверждали при приблизительно 175 кДа только в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3. Кроме того, как показано на фиг. 4c), эндогенный DCTN1 детектировали при приблизительно 150 кДа при использовании антитела к DCTN1. Детектировали также полосу при приблизительно 175 кДа, выше полосы эндогенного DCTN1 при приблизительно 150 кДа, только в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3. Конкретно, полосу при приблизительно 175 кДа детектировали при использовании как антитела к RET, так и антитела к DCTN1, только в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3; это явно показывает, что слитый белок из DCTN1 и RET экспрессировался в полученных экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3.
[0176]
Пример 5: Подтверждение роста экспрессирующих слитый ген DCTN1-RET клеток NIH/3T3 посредством 3-мерной культуры клеток
Клетки NIH/3T3 хорошо росли в условиях 2-мерной культуры клеток, но с трудом росли в условиях 3-мерной культуры клеток. Однако, известно также, что клетки NIH/3T3 растут в условиях 3-мерной культуры клеток, когда онкоген экспрессируется в клетках. Таким образом, является ли слитый ген DCTN1-RET онкогеном, подтверждали с использованием этой характеристики. Экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3 и клетки NIH/3T3 культивировали при 37°C в 5% CO2 в 2-мерной культуре клеток; и собирали с использованием трипсина, с последующим определением количества клеток. Для осуществления 3-мерного культивирования клеток, среду для 3-мерной культуры клеток получали с использованием набора для получения, серий FCeM (Nissan Chemical Industries, Ltd.), D-MEM (с высоким содержанием глюкозы) (содержащей L-глутамин, фенол красный, пируват натрия, и 1500 мг/л гидрокарбоната натрия) (Wako) и сыворотки новорожденного теленка (NBCS) (Gibco). Клетки суспендировали в полученной среде для 3-мерной культуры клеток для получения 1000 клеток/90 мкл и рассевали в 96-луночный прозрачный-черный микропланшет с круглодонными, сферическими лунками (Corning), 90 мкл на лунку, с последующей инкубацией при 37°C в 5% CO2. На сутки после рассева (сутки 1) и через 8 суток после рассева (сутки 8), уровень люминесценции (импульсов в секунду: cps) измеряли с использованием детектирующего реагента для люминесценции внутриклеточного АТФ (реагента CellTiter-Glo 2.0, Promega) с использованием люминометра (EnSpire, PerkinElmer), и определили, что результат является индикатором количества жизнеспособных клеток. Затем скорость роста клеток рассчитывали по результату измерений на сутки 1 и результату измерений на сутки 8 (N=3). В результате, как показано на фиг. 5, в то время как количество клеток для клеток NIH/3T3 на сутки 8 в 2,4 раза превышало количество клеток на сутки 1, количество клеток для экспрессирующих слитый ген DCTN1-RET клеток NIH/3T3 на сутки 8 в 20,9 превышало количество клеток на сутки 1. Кроме того, в то время как не обнаружено агрегатов клеток, сформированных в клетках NIH/3T3 при 3-мерном культивировании клеток, подтвердили, что агрегаты клеток сформированы в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 при 3-мерном культивировании клеток. Это явно показывает, что трансфекция слитым геном DCTN1-RET усиливала рост клеток, позволяя предполагать, что слитый ген DCTN1-RET является онкогеном.
[0177]
Пример 6: Подтверждение онкогенности в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 in vivo
Для подтверждения онкогенности в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 in vivo, проводили эксперимент трансплантации с использованием мышей. Общеизвестно, что клетки NIH/3T3, представляющие собой исходную линию клеток, не растут подкожно у мышей nude; и можно подтвердить, вносит ли слитый ген DCTN1-RET вклад в онкогенность, или является ли слитый ген DCTN1-RET онкогеном, посредством подкожной трансплантации мышей nude с использованием экспрессирующих слитый ген DCTN1-RET- клеток NIH/3T3. Мышей nude, BALB/cAJcl-nu/nu (CLEA Japan, Inc.) использовали для трансплантации. Экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3 собирали с использованием трипсина, и суспендировали в PBS, чтобы, наконец, получить 1×108 клеток/мл. К этому добавляли эквивалентное количество матрикса базальной мембраны матригеля (Corning) и доводили для получения 5×107 клеток/мл для получения суспензии клеток для трансплантации. 0,1 мл суспензии клеток для трансплантации подкожно трансплантировали в правую сторону грудной клетки каждой мыши nude (N=10) с использованием иглы для инъекций 25G и 1-мл шприца. На сутки 10, 13 и 17 после трансплантации, длинную ось и короткую ось опухоли каждой мыши измеряли с использованием цифрового штангенциркуля (Mitutoyo Corporation), и объем опухоли рассчитывали на основании следующего уравнения.
Объем опухоли (мм3)=(длинная ось: мм)×(короткая ось: мм)×(короткая ось: мм)/2
На фиг. 6 показаны результаты измерения объема опухоли. Подтвердили, что экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3, подкожно трансплантированные мышам nude, вызывали онкогенез и хорошо росли; таким образом, этот эксперимент in vivo позволяет также предполагать, что слитый ген DCTN1-RET является онкогеном.
[0178]
Пример 7: Подтверждение ингибирования слитого белка DCTN1-RET и эффекта ингибирования роста клеток посредством миРНК в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3
Проверяли эффект обработки миРНК на экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3. миРНК для использования являлась следующей: три типа миРНК RET, показанные в таблице 5 ниже, и Silencer Select Negative Control #1 siRNA (Ambion) в качестве отрицательного контроля. Мишенью для трех типов миРНК RET является RET человека; и миРНК RET1 и миРНК RET2 содержат последовательность, которая связывается с частью RET слитого гена DCTN1-RET, в то время как миРНК RET3 не содержит последовательности, которая связывается со слитым геном DCTN1-RET. Иными словами, заключили, что миРНК RET1 и миРНК RET2 ингибируют экспрессию слитого гена DCTN1-RET, и миРНК RET3 не ингибирует. Далее описан способ из эксперимента с использованием миРНК.
[0179]
Таблица 5
[0180]
Экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3 ДЛЯ использования получали посредством культивирования в среде для 2-мерной культуры клеток при 37°C в 5% CO2. За сутки до обработки миРНК, клетки рассевали в 6-луночный планшет (Iwaki), 3×105 клеток/2 мл, и инкубировали при 37°C в 5% CO2 в течение ночи. 12 мкл миРНК каждого типа, заблаговременно доведенной до 20 мкМ с использованием воды, 4 мкл реагента для трансфекции липофектамина RNAiMAX (Thermo Fisher) и 384 мкл Opti-MEM смешивали; и инкубировали при комнатной температуре в течение 15 минут, таким образом, получая растворы миРНК. 400 мкл каждого раствора миРНК добавляли в лунки, в которые рассевали клетки, и инкубацию проводили при 37°C в 5% CO2 в течение ночи. На следующие сутки, часть инкубированных клеток отбирали в качестве образца для использования в анализе экспрессии белка; и другую часть также отбирали и повторно рассевали для использования для подтверждения роста клеток. Отбор образцов для анализа экспрессии белка и анализ экспрессии белка проводили таким же образом, как в 4-2: Подтверждение экспрессии белка-мишени, описанном выше, за исключением использования антитела к фосфо-RET (Tyr905) (CST), mAb кролика против Ret (C31B4) (CST), и mAb кролика против GAPDH (D16H11) XP (CST) в качестве первичных антител. Результаты показали, как показано на фиг. 7, что экспрессия слитого белка DCTN1-RET не была ингибирована в клетках, обработанных отрицательной контрольной миРНК (NC), сравнимо с клетками без обработки миРНК (без обработки). Когда клетки обрабатывали миРНК RET1 или миРНК RET2, подтвердили, что экспрессия слитого белка DCTN1-RET была ингибирована в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3, и не была ингибирована, когда использовали миРНК RET3.
Затем, для подтверждения эффекта ингибирования роста клеток, клетки собирали с использованием трипсина из лунок без обработки и из лунок, обработанных миРНК, и подсчитывали количество клеток. Затем проводили 3-мерное культивирование клеток таким же способом, как в примере 5, и на сутки проведения рассева (сутки 0) и через 4 суток после рассева (сутки 4), количество жизнеспособных клеток подсчитывали таким же способом, как в примере 5. Скорость роста клеток рассчитывали по результату измерения на сутки 0 и результату измерения на сутки 4. Результаты показывают, как показано на фиг. 8, что для клеток без обработки миРНК (без обработки) и для клеток, обработанных отрицательной контрольной миРНК (NC), соответственно, показано количество клеток на сутки 4 в 4,9 раз и в 3,6 раз больше, чем на сутки 0; однако, для клеток, обработанных миРНК RET1 и для клеток, обработанных миРНК RET2, показан рост только приблизительно в 2,0 раза и в 2,4 раза, соответственно, в обоих случаях заметно ниже. Для клеток, обработанных миРНК RET3, показан рост в 3,7 раза, который является по существу равным росту клеток, обработанных отрицательной контрольной миРНК, редко показывая уменьшение роста. Эти результаты выявили, что рост экспрессирующих слитый ген DCTN1-RET клеток NIH/3T3 также ингибирован, когда экспрессия RET ингибирована посредством миРНК.
[0181]
Пример 8: Эффект ингибирования роста клеток в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3
Анализ роста клеток in vitro проводили на экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3. 3-мерное культивирование клеток и рассев осуществляли таким же способом, как в примере 5. После рассева, проводили инкубацию при 37°C в 5% CO2 в течение ночи (сутки 0). Кабозантиниб, вандетаниб, алектиниб, ленватиниб и конденсированные соединения пиримидина (соединения 1-9, показанные в таблице 6), которые, как опубликовано, являются способными ингибировать RET, растворяли в диметилсульфоксиде для получения концентрации 10 ммоль/л; и далее разводили с использованием среды для 3-мерной культуры клеток, таким образом, чтобы эти соединения имели конечную концентрацию 1000, 333, 111, 37,0, 12,3, 4,12, 1,37 и 0,457 нмоль/л. Затем эти разведенные соединения индивидуально добавляли в каждую лунку (0,01 мл на лунку) планшета, в который рассевали клетки (сутки 1); и клетки инкубировали при 37°C в 5% CO2 в течение 7 суток. После культивирования (сутки 8), детектирующий реагент для люминесценции внутриклеточного АТФ (реагент CellTiter-Glo 2.0, Promega) добавляли в каждую лунку, и уровень люминесценции (импульсов в секунду: cps) измеряли с использованием люминометра (EnSpire, PerkinElmer). Скорость роста от суток 1 клеток, обработанных соединениями в различных концентрациях, рассчитывали на основании следующего уравнения, в соответствии с значением Tсутки8 и Cсутки1, и определяли концентрацию каждого тестируемого соединения, при которой рост клетки был ингибирован на 50% (GI50(нМ)).
1) Когда Tсутки8 ≥ Cсутки1
Скорость роста (%)=(Tсутки8 - Cсутки1)/(Cсутки8 - Cсутки1)×100
T: cps для лунки, в которую добавляли тестируемое соединение
C: cps для лунки, в которую не добавляли тестируемое соединение
Сутки 1: сутки, на которые добавляли тестируемое соединение
Сутки 8: сутки, на которые проводили оценку
2) Когда Tсутки 8 < Cсутки1
Скорость роста (%)=(Tсутки8 - Cсутки1)/(Cсутки1)×100
T: cps для лунки, в которую добавляли тестируемое соединение
C: cps для лунки, в которую не добавляли тестируемое соединение
Сутки 1: сутки, на которые добавляли тестируемое соединение
Сутки 8: сутки, на которые проводили оценку[0182]
Таблица 6
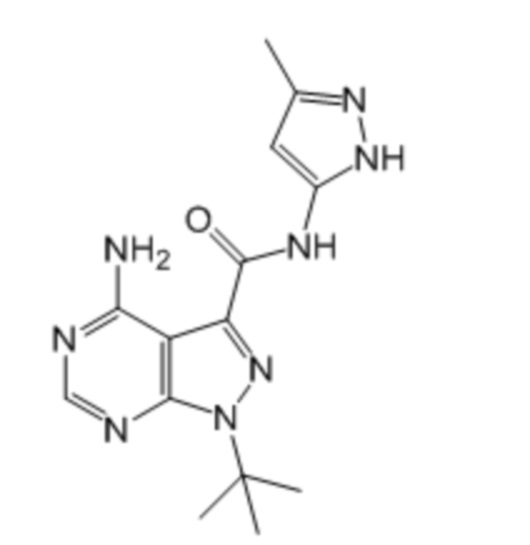
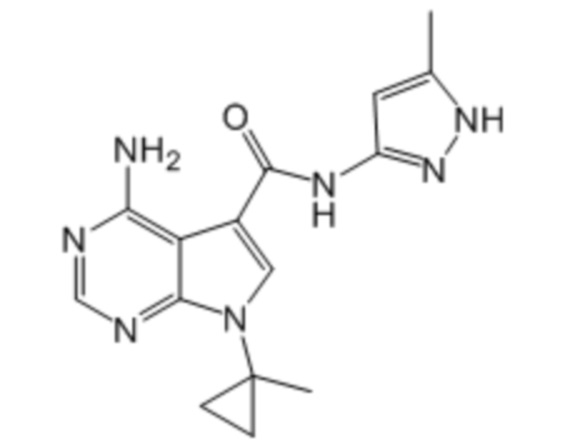
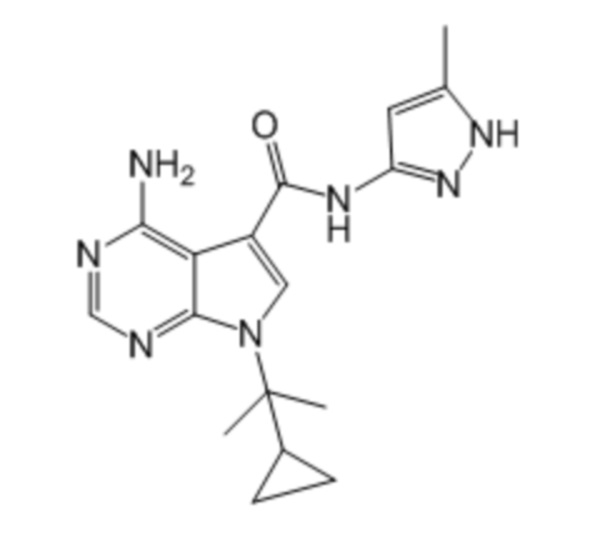
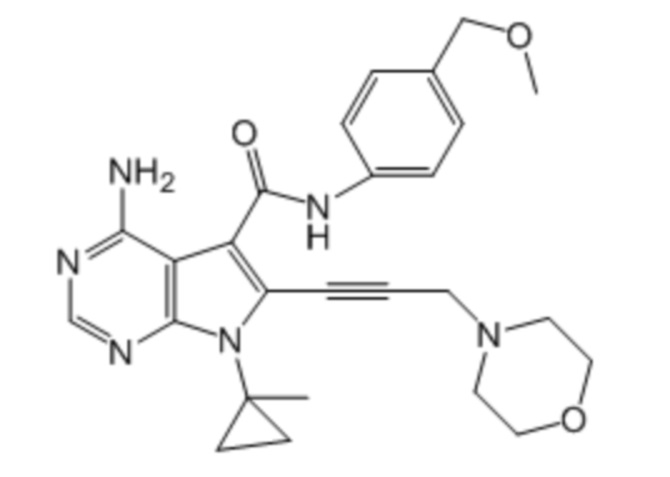
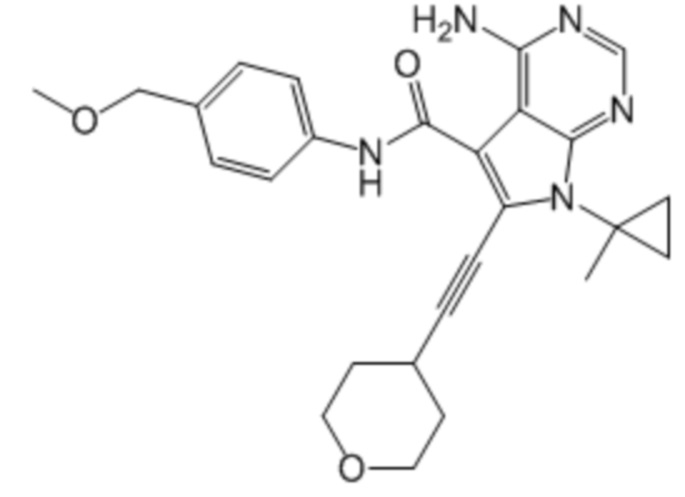
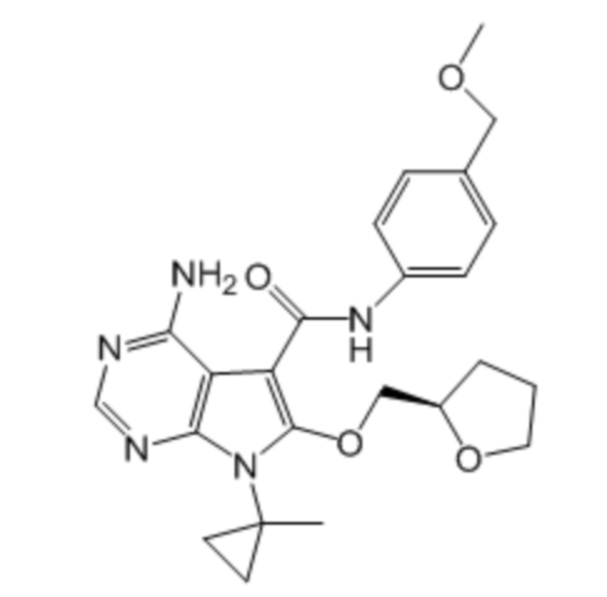
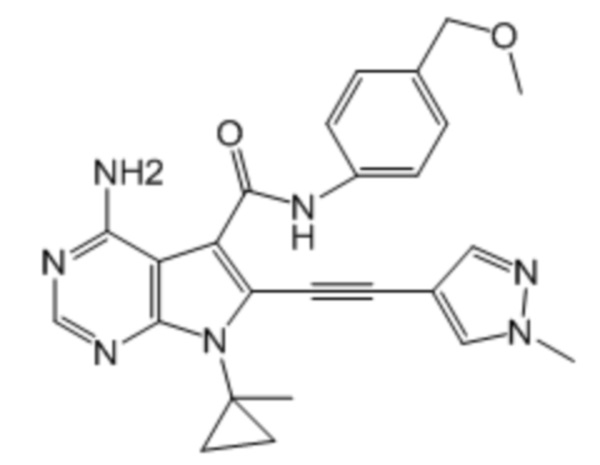
[0183]
Результаты показывают, как показано в таблице 7, что кабозантиниб, вандетаниб, ленватиниб и конденсированные соединения пиримидина (соединения 1-9) ингибировали рост экспрессирующих слитый ген DCTN1-RET клеток NIH/3T3.
[0184]
Таблица 7
Соединение 2
Соединение 3
Соединение 4
Соединение 5
Соединение 6
Соединение 7
Соединение 8
Соединение 9
Кабозантиниб
Вандетаниб
Ленватиниб
Алектиниб
5,80
5,40
5,70
4,20
3,60
11,40
17,90
4,80
91,10
68,70
50,30
>1000
[0185]
Результаты выше позволяют предполагать потенциальную полезность этих ингибиторов RET в качестве лекарственного средства против злокачественных опухолей, в которых детектирован слитый ген DCTN1-RET. Результаты позволяют предполагать также, что использование экспрессирующих слитый ген DCTN1-RET-клеток NIH/3T3 позволяет скрининг соединения, ингибирующего DCTN1-RET.
[0186]
Пример 9: Ингибирование фосфорилирования RET в экспрессирующих слитый ген DCTN1-RET клетках
Затем проверяли, ингибировано ли фосфорилирование RET в экспрессирующих слитый ген DCTN1-RET клетках посредством существующих лекарственных средств, как опубликовано, ингибирующих RET, в соответствии со способом, описанным ниже. Экспрессирующие слитый ген DCTN1-RET клетки NIH/3T3 для использования получали посредством культивирования в среде для 2-мерной культуры клеток при 37°C в 5% CO2. За сутки до обработки клеток лекарственным средством, клетки рассевали в 6-луночный планшет (Iwaki), 3×105 клеток/2 мл, и инкубировали при 37°C в 5% CO2 в течение ночи. Кабозантиниб, вандетаниб, алектиниб и ленватиниб растворяли в диметилсульфоксиде для получения концентрации 10 ммоль/л, и далее разводили с использованием PBS, таким образом, чтобы эти соединения имели конечную концентрацию 1000, 100 и 10 нмоль/л. Разведенные соединения индивидуально добавляли в каждую лунку, в которую предварительно рассевали клетки, 20 мкл на лунку (сутки 1), и инкубировали при 37°C в 5% CO2 в течение 1 час. После инкубации, образец клеток для анализа экспрессии белка отбирали таким же способом, как в примере 7, и анализировали экспрессию белка. Результаты показывают, как показано на фиг. 9, что уровни фосфорилированного RET в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 были заметно уменьшены посредством кабозантиниба и ленватиниба. Кроме того, ингибирование фосфорилирования RET посредством конденсированных соединений пиримидина также оценивали таким же способом, как выше, и подтвердили также, что фосфорилирование RET было заметно уменьшено посредством конденсированных соединений пиримидина. Эти результаты позволяют предполагать, что лекарственное средство, способное заметно уменьшать уровни фосфорилированного RET, может представлять собой соединение, которое может ингибировать рост экспрессирующих слитый ген DCTN1-RET клеток NIH/3T3, и что соединение является потенциально полезным в качестве лекарственного средства против злокачественных опухолей, в которых детектирован слитый ген DCTN1-RET. Результаты также позволяют предполагать, что использование уровней фосфорилированного RET в экспрессирующих слитый ген DCTN1-RET клетках NIH/3T3 позволяет скрининг ингибитора RET.
Список последовательностей в произвольном текстовом формате
[0187]
SEQ ID NO: 1 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из варианта 1 (v1) DCTN1 (часть из SEQ ID NO: 25) и варианта 2 (v2) RET (часть из SEQ ID NO: 31).
SEQ ID NO: 2 показывает аминокислотную последовательность слитого пептида из DCTN1 v1 и RET v2.
SEQ ID NO: 3 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v1 и варианта 4 (v4) RET (часть из SEQ ID NO: 32).
SEQ ID NO: 4 показывает аминокислотную последовательность слитого пептида из DCTN1 v1 и RET v4.
SEQ ID NO: 5 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из варианта 2 (v2) DCTN1 (часть из SEQ ID NO: 26) и RET v2.
SEQ ID NO: 6 показывает аминокислотную последовательность слитого пептида из DCTN1 v2 и RET v2.
SEQ ID NO: 7 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v2 и RET v4.
SEQ ID NO: 8 показывает аминокислотную последовательность слитого пептида из DCTN1 v2 и RET v4.
SEQ ID NO: 9 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из варианта 3 (v3) DCTN1 (часть из SEQ ID NO: 27) и RET v2.
SEQ ID NO: 10 показывает аминокислотную последовательность слитого пептида из DCTN1 v3 и RET v2.
SEQ ID NO: 11 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v3 и RET v4.
SEQ ID NO: 12 показывает аминокислотную последовательность слитого пептида из DCTN1 v3 и RET v4.
SEQ ID NO: 13 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из варианта 4 (v4) DCTN1 (часть из SEQ ID NO: 28) и RET v2.
SEQ ID NO: 14 показывает аминокислотную последовательность слитого пептида из DCTN1 v4 и RET v2.
SEQ ID NO: 15 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v4 и RET v4.
SEQ ID NO: 16 показывает аминокислотную последовательность слитого пептида из DCTN1 v4 и RET v4.
SEQ ID NO: 17 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из варианта 5 (v5) DCTN1 (часть из SEQ ID NO: 29) и RET v2.
SEQ ID NO: 18 показывает аминокислотную последовательность слитого пептида из DCTN1 v5 и RET v2.
SEQ ID NO: 19 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v5 и RET v4.
SEQ ID NO: 20 показывает аминокислотную последовательность слитого пептида из DCTN1 v5 и RET v4.
SEQ ID NO: 21 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v6 и RET v2.
SEQ ID NO: 22 показывает аминокислотную последовательность слитого пептида из DCTN1 v6 и RET v2.
SEQ ID NO: 23 показывает последовательность оснований полинуклеотида, кодирующего слитый пептид из DCTN1 v6 и RET v4.
SEQ ID NO: 24 показывает аминокислотную последовательность слитого пептида из DCTN1 v6 и RET v4.
SEQ ID NO: 33 показывает последовательность оснований праймера.
SEQ ID NO: 34 показывает последовательность оснований праймера.
SEQ ID NO: 35 показывает последовательность оснований праймера.
SEQ ID NO: 36 показывает последовательность оснований праймера.
SEQ ID NO: 37 показывает последовательность оснований праймера.
SEQ ID NO: 38 показывает последовательность оснований праймера.
SEQ ID NO: 39 показывает последовательность оснований праймера.
SEQ ID NO: 40 показывает последовательность оснований праймера.
SEQ ID NO: 41 показывает последовательность оснований праймера.
SEQ ID NO: 42 показывает последовательность оснований праймера.
SEQ ID NO: 43 показывает последовательность оснований праймера.
SEQ ID NO: 44 показывает последовательность оснований праймера.
SEQ ID NO: 45 показывает последовательность оснований праймера.
SEQ ID NO: 46 показывает последовательность оснований праймера.
SEQ ID NO: 47 показывает последовательность оснований праймера.
SEQ ID NO: 48 показывает последовательность оснований праймера.
SEQ ID NO: 49 показывает последовательность оснований праймера.
SEQ ID NO: 50 показывает последовательность оснований праймера.
SEQ ID NO: 51 показывает последовательность оснований праймера.
SEQ ID NO: 52 показывает последовательность оснований праймера.
SEQ ID NO: 53 показывает последовательность оснований праймера.
SEQ ID NO: 54 показывает последовательность оснований праймера.
SEQ ID NO: 55 показывает последовательность оснований праймера.
SEQ ID NO: 56 показывает последовательность оснований праймера.
SEQ ID NO: 57 показывает последовательность оснований праймера.
SEQ ID NO: 58 показывает последовательность оснований праймера.
SEQ ID NO: 59 показывает последовательность оснований праймера.
SEQ ID NO: 60 показывает последовательность оснований праймера.
SEQ ID NO: 61 показывает последовательность оснований праймера.
SEQ ID NO: 62 показывает последовательность оснований праймера.
SEQ ID NO: 63 показывает последовательность оснований праймера.
SEQ ID NO: 64 показывает последовательность оснований праймера.
SEQ ID NO: 65 показывает последовательность оснований праймера.
SEQ ID NO: 66 показывает последовательность оснований праймера.
SEQ ID NO: 67 показывает последовательность оснований праймера.
SEQ ID NO: 68 показывает последовательность оснований праймера.
SEQ ID NO: 69 показывает последовательность оснований праймера.
SEQ ID NO: 70 показывает последовательность оснований праймера.
SEQ ID NO: 71 показывает последовательность оснований праймера.
SEQ ID NO: 72 показывает последовательность оснований праймера.
SEQ ID NO: 73 показывает последовательность оснований праймера.
SEQ ID NO: 74 показывает последовательность оснований миРНК RET.
SEQ ID NO: 75 показывает последовательность оснований миРНК RET.
SEQ ID NO: 76 показывает последовательность оснований миРНК RET.
SEQ ID NO: 77 показывает последовательность оснований миРНК RET.
Таблица последовательностей
P17-158WOPCT_DCTN1 PROTEIN_20180820_115457_8.txt
--->
SEQUENCE LISTING
<110> ТАЙХО ФАРМАСЬЮТИКАЛ КО., ЛТД.
<120> СЛИТЫЙ БЕЛОК ИЗ БЕЛКА DCTN1 С БЕЛКОМ RET
<130> P17-158WO
<150> JP 2017-158796
<151> 2017-08-21
<160> 77
<170> PatentIn version 3.5
<210> 1
<211> 4908
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 1
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctgcggggac tgaagcctaa gaaggcaccg 420
acagcccgaa agaccacaac tcggcgaccc aagcccacgc gcccagccag tactggggtg 480
gctggggcca gtagctccct gggcccctct ggctcagcgt cagcaggtga gctgagcagc 540
agtgagccca gcaccccggc tcagactccg ctggcagcac ccatcatccc cacgccggtc 600
ctcacctctc ctggagcagt ccccccgctt ccttccccat ccaaggagga ggagggacta 660
agggctcagg tgcgggacct ggaggagaaa ctagagaccc tgagactgaa acgggcagaa 720
gacaaagcaa agctaaaaga gctggagaaa cacaaaatcc agctggagca ggtgcaggaa 780
tggaagagca aaatgcagga gcagcaggcc gacctgcagc ggcgcctcaa ggaggcgaga 840
aaggaagcca aggaggcgct ggaggcaaag gaacgctata tggaggagat ggctgatact 900
gctgatgcca ttgagatggc cactttggac aaggagatgg ctgaagagcg ggctgagtcc 960
ctgcagcagg aggtggaggc actgaaggag cgggtggacg agctcactac tgacttagag 1020
atcctcaagg ctgagattga agagaagggc tcagatggcg ctgcatccag ttatcagctc 1080
aagcagcttg aggagcagaa tgcccgcctg aaggatgccc tggtgaggat gcgggatctt 1140
tcttcctcag agaagcagga gcatgtgaag ctccagaagc tcatggaaaa gaagaaccaa 1200
gagctggaag ttgtgaggca acagcgggag cgtctgcagg aggagctaag ccaggcagag 1260
agcaccattg atgagctcaa ggagcaggtg gatgctgctc tgggtgctga ggagatggtg 1320
gagatgctga cagatcggaa cctgaatctg gaagagaaag tgcgcgagtt gagggagact 1380
gtgggagact tggaagcgat gaatgagatg aacgatgagc tgcaggagaa tgcacgtgag 1440
acagaactgg agctgcggga gcagctggac atggcaggcg cgcgggttcg tgaggcccag 1500
aagcgtgtgg aggcagccca ggagacggtt gcagactacc agcagaccat caagaagtac 1560
cgccagctga ccgcccatct acaggatgtg aatcgggaac tgacaaacca gcaggaagca 1620
tctgtggaga ggcaacagca gccacctcca gagacctttg acttcaaaat caagtttgct 1680
gagactaagg cccatgccaa ggcaattgag atggaattga ggcagatgga ggtggcccag 1740
gccaatcgac acatgtccct gctgacagcc ttcatgcctg acagcttcct tcggccaggt 1800
ggggaccatg actgcgttct ggtgctgttg ctcatgcctc gtctcatttg caaggcagag 1860
ctgatccgga agcaggccca ggagaagttt gaactaagtg agaactgttc agagcggcct 1920
gggctgcgag gagctgctgg ggagcaactc agctttgctg ctggactggt gtactcgctg 1980
agcctgctgc aggccacgct acaccgctat gagcatgccc tctctcagtg cagtgtggat 2040
gtgtataaga aagtgggcag cctgtaccct gagatgagtg cccatgagcg ctccttggat 2100
ttcctcattg aactgctgca caaggatcag ctggatgaga ctgtcaatgt ggagcctctc 2160
accaaggcca tcaagtacta tcagcatctg tacagcatcc accttgccga acagcctgag 2220
gactgtacta tgcagctggc tgaccacatt aagttcacgc agagtgctct ggactgcatg 2280
agtgtggagg taggacggct gcgtgccttc ttgcagggtg ggcaggaggc tacagatatt 2340
gccctcctgc tccgggatct ggaaacttca tgcagtgaca tccgccagtt ctgcaagaag 2400
atccgaaggc gaatgccagg gacagatgct cctgggatcc cagctgcact ggcctttgga 2460
ccacaggtat ctgacacgct cctagactgc aggaaacact tgacgtgggt cgtggctgtg 2520
ctgcaggagg tggcagctgc tgctgcccag ctcattgccc cactggcaga gaatgagggg 2580
ctacttgtgg ctgctctgga ggaactggct ttcaaagcaa gcgagcagat ctatgggacc 2640
ccctccagca gcccctatga gtgtctgcgc cagtcatgca acatcctcat cagtaccatg 2700
aacaagctgg ccacagccat gcaggagggg gagtatgatg cagagcggcc ccccagcaag 2760
cctccaccgg ttgaactgcg ggctgctgcc cttcgtgcag agatcacaga tgctgaaggc 2820
ctgggtttga agctcgaaga tcgagagaca gttattaagg agttgaagaa gtcactcaag 2880
attaagggag aggagctaag tgaggccaat gtgcggctga gcctcctgga gaagaagttg 2940
gacagtgctg ccaaggatgc agatgagcgc atcgagaaag tccagactcg gctggaggag 3000
acccaggcac tgctgcgaaa gaaggagaaa gagtttgagg agacaatgga tgcactccag 3060
gctgacatcg accagctgga ggcagagaag gcagaactaa agcagcgtct gaacagccag 3120
tccaaacgca cgattgaggg actccggggc cctcctcctt caggcattgc tactctggtc 3180
tctggcattg ctggtgaaga acagcagcga ggagccatcc ctgggcaggc tccagggtct 3240
gtgccaggcc cagggctggt gaaggactca ccactgctgc ttcagcagat ctctgccatg 3300
aggctgcaca tctcccagct ccagcatgag aacagcatcc tcaagggagc ccagatgaag 3360
gcatccttgg catccctgcc ccctctgcat gttgcaaagc tatcccatga gggccctggc 3420
agtgagttac cagctggagc gctgtatcgt aagaccagcc agctgctgga gacattgaat 3480
caattgagca cacacacgca cgtagtagac atcactcgca ccagccctgc tgccaagagc 3540
ccgtcggccc aacttatgga gcaagtggct cagcttaagt ccctgagtga caccgtcgag 3600
aagctcaagg atgaggtcct caaggagaca gtatctcagc gccctggagc cacagtaccc 3660
actgactttg ccaccttccc ttcatcagcc ttcctcaggg aggatccaaa gtgggaattc 3720
cctcggaaga acttggttct tggaaaaact ctaggagaag gcgaatttgg aaaagtggtc 3780
aaggcaacgg ccttccatct gaaaggcaga gcagggtaca ccacggtggc cgtgaagatg 3840
ctgaaagaga acgcctcccc gagtgagctt cgagacctgc tgtcagagtt caacgtcctg 3900
aagcaggtca accacccaca tgtcatcaaa ttgtatgggg cctgcagcca ggatggcccg 3960
ctcctcctca tcgtggagta cgccaaatac ggctccctgc ggggcttcct ccgcgagagc 4020
cgcaaagtgg ggcctggcta cctgggcagt ggaggcagcc gcaactccag ctccctggac 4080
cacccggatg agcgggccct caccatgggc gacctcatct catttgcctg gcagatctca 4140
caggggatgc agtatctggc cgagatgaag ctcgttcatc gggacttggc agccagaaac 4200
atcctggtag ctgaggggcg gaagatgaag atttcggatt tcggcttgtc ccgagatgtt 4260
tatgaagagg attcctacgt gaagaggagc cagggtcgga ttccagttaa atggatggca 4320
attgaatccc tttttgatca tatctacacc acgcaaagtg atgtatggtc ttttggtgtc 4380
ctgctgtggg agatcgtgac cctaggggga aacccctatc ctgggattcc tcctgagcgg 4440
ctcttcaacc ttctgaagac cggccaccgg atggagaggc cagacaactg cagcgaggag 4500
atgtaccgcc tgatgctgca atgctggaag caggagccgg acaaaaggcc ggtgtttgcg 4560
gacatcagca aagacctgga gaagatgatg gttaagagga gagactactt ggaccttgcg 4620
gcgtccactc catctgactc cctgatttat gacgacggcc tctcagagga ggagacaccg 4680
ctggtggact gtaataatgc ccccctccct cgagccctcc cttccacatg gattgaaaac 4740
aaactctatg gcatgtcaga cccgaactgg cctggagaga gtcctgtacc actcacgaga 4800
gctgatggca ctaacactgg gtttccaaga tatccaaatg atagtgtata tgctaactgg 4860
atgctttcac cctcagcggc aaaattaatg gacacgtttg atagttaa 4908
<210> 2
<211> 1635
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 2
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Arg Gly Leu Lys Pro Lys Lys Ala Pro Thr Ala Arg Lys
130 135 140
Thr Thr Thr Arg Arg Pro Lys Pro Thr Arg Pro Ala Ser Thr Gly Val
145 150 155 160
Ala Gly Ala Ser Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly
165 170 175
Glu Leu Ser Ser Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala
180 185 190
Ala Pro Ile Ile Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro
195 200 205
Pro Leu Pro Ser Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val
210 215 220
Arg Asp Leu Glu Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu
225 230 235 240
Asp Lys Ala Lys Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu
245 250 255
Gln Val Gln Glu Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu
260 265 270
Gln Arg Arg Leu Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu
275 280 285
Ala Lys Glu Arg Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile
290 295 300
Glu Met Ala Thr Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser
305 310 315 320
Leu Gln Gln Glu Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr
325 330 335
Thr Asp Leu Glu Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp
340 345 350
Gly Ala Ala Ser Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala
355 360 365
Arg Leu Lys Asp Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu
370 375 380
Lys Gln Glu His Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln
385 390 395 400
Glu Leu Glu Val Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu
405 410 415
Ser Gln Ala Glu Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala
420 425 430
Ala Leu Gly Ala Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu
435 440 445
Asn Leu Glu Glu Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu
450 455 460
Glu Ala Met Asn Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu
465 470 475 480
Thr Glu Leu Glu Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val
485 490 495
Arg Glu Ala Gln Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp
500 505 510
Tyr Gln Gln Thr Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln
515 520 525
Asp Val Asn Arg Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg
530 535 540
Gln Gln Gln Pro Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala
545 550 555 560
Glu Thr Lys Ala His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met
565 570 575
Glu Val Ala Gln Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met
580 585 590
Pro Asp Ser Phe Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val
595 600 605
Leu Leu Leu Met Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys
610 615 620
Gln Ala Gln Glu Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro
625 630 635 640
Gly Leu Arg Gly Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu
645 650 655
Val Tyr Ser Leu Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His
660 665 670
Ala Leu Ser Gln Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu
675 680 685
Tyr Pro Glu Met Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu
690 695 700
Leu Leu His Lys Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu
705 710 715 720
Thr Lys Ala Ile Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala
725 730 735
Glu Gln Pro Glu Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe
740 745 750
Thr Gln Ser Ala Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg
755 760 765
Ala Phe Leu Gln Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu
770 775 780
Arg Asp Leu Glu Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys
785 790 795 800
Ile Arg Arg Arg Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala
805 810 815
Leu Ala Phe Gly Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys
820 825 830
His Leu Thr Trp Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala
835 840 845
Ala Gln Leu Ile Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala
850 855 860
Ala Leu Glu Glu Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr
865 870 875 880
Pro Ser Ser Ser Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu
885 890 895
Ile Ser Thr Met Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr
900 905 910
Asp Ala Glu Arg Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala
915 920 925
Ala Ala Leu Arg Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys
930 935 940
Leu Glu Asp Arg Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys
945 950 955 960
Ile Lys Gly Glu Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu
965 970 975
Glu Lys Lys Leu Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu
980 985 990
Lys Val Gln Thr Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys
995 1000 1005
Glu Lys Glu Phe Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile
1010 1015 1020
Asp Gln Leu Glu Ala Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn
1025 1030 1035
Ser Gln Ser Lys Arg Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro
1040 1045 1050
Ser Gly Ile Ala Thr Leu Val Ser Gly Ile Ala Gly Glu Glu Gln
1055 1060 1065
Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
1070 1075 1080
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser
1085 1090 1095
Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile
1100 1105 1110
Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro
1115 1120 1125
Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu
1130 1135 1140
Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr
1145 1150 1155
Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr Arg
1160 1165 1170
Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln
1175 1180 1185
Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys
1190 1195 1200
Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr
1205 1210 1215
Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg
1220 1225 1230
Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu Val Leu Gly
1235 1240 1245
Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val Lys Ala Thr
1250 1255 1260
Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala Val
1265 1270 1275
Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp Leu
1280 1285 1290
Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His Pro His Val
1295 1300 1305
Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu Leu
1310 1315 1320
Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu Arg
1325 1330 1335
Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser
1340 1345 1350
Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr
1355 1360 1365
Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met
1370 1375 1380
Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala Ala
1385 1390 1395
Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp
1400 1405 1410
Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys
1415 1420 1425
Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala Ile Glu Ser
1430 1435 1440
Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser Phe
1445 1450 1455
Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly Asn Pro Tyr
1460 1465 1470
Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr Gly
1475 1480 1485
His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr Arg
1490 1495 1500
Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro Val
1505 1510 1515
Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met Val Lys Arg
1520 1525 1530
Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser Leu
1535 1540 1545
Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro Leu Val Asp
1550 1555 1560
Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp Ile
1565 1570 1575
Glu Asn Lys Leu Tyr Gly Met Ser Asp Pro Asn Trp Pro Gly Glu
1580 1585 1590
Ser Pro Val Pro Leu Thr Arg Ala Asp Gly Thr Asn Thr Gly Phe
1595 1600 1605
Pro Arg Tyr Pro Asn Asp Ser Val Tyr Ala Asn Trp Met Leu Ser
1610 1615 1620
Pro Ser Ala Ala Lys Leu Met Asp Thr Phe Asp Ser
1625 1630 1635
<210> 3
<211> 4782
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 3
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctgcggggac tgaagcctaa gaaggcaccg 420
acagcccgaa agaccacaac tcggcgaccc aagcccacgc gcccagccag tactggggtg 480
gctggggcca gtagctccct gggcccctct ggctcagcgt cagcaggtga gctgagcagc 540
agtgagccca gcaccccggc tcagactccg ctggcagcac ccatcatccc cacgccggtc 600
ctcacctctc ctggagcagt ccccccgctt ccttccccat ccaaggagga ggagggacta 660
agggctcagg tgcgggacct ggaggagaaa ctagagaccc tgagactgaa acgggcagaa 720
gacaaagcaa agctaaaaga gctggagaaa cacaaaatcc agctggagca ggtgcaggaa 780
tggaagagca aaatgcagga gcagcaggcc gacctgcagc ggcgcctcaa ggaggcgaga 840
aaggaagcca aggaggcgct ggaggcaaag gaacgctata tggaggagat ggctgatact 900
gctgatgcca ttgagatggc cactttggac aaggagatgg ctgaagagcg ggctgagtcc 960
ctgcagcagg aggtggaggc actgaaggag cgggtggacg agctcactac tgacttagag 1020
atcctcaagg ctgagattga agagaagggc tcagatggcg ctgcatccag ttatcagctc 1080
aagcagcttg aggagcagaa tgcccgcctg aaggatgccc tggtgaggat gcgggatctt 1140
tcttcctcag agaagcagga gcatgtgaag ctccagaagc tcatggaaaa gaagaaccaa 1200
gagctggaag ttgtgaggca acagcgggag cgtctgcagg aggagctaag ccaggcagag 1260
agcaccattg atgagctcaa ggagcaggtg gatgctgctc tgggtgctga ggagatggtg 1320
gagatgctga cagatcggaa cctgaatctg gaagagaaag tgcgcgagtt gagggagact 1380
gtgggagact tggaagcgat gaatgagatg aacgatgagc tgcaggagaa tgcacgtgag 1440
acagaactgg agctgcggga gcagctggac atggcaggcg cgcgggttcg tgaggcccag 1500
aagcgtgtgg aggcagccca ggagacggtt gcagactacc agcagaccat caagaagtac 1560
cgccagctga ccgcccatct acaggatgtg aatcgggaac tgacaaacca gcaggaagca 1620
tctgtggaga ggcaacagca gccacctcca gagacctttg acttcaaaat caagtttgct 1680
gagactaagg cccatgccaa ggcaattgag atggaattga ggcagatgga ggtggcccag 1740
gccaatcgac acatgtccct gctgacagcc ttcatgcctg acagcttcct tcggccaggt 1800
ggggaccatg actgcgttct ggtgctgttg ctcatgcctc gtctcatttg caaggcagag 1860
ctgatccgga agcaggccca ggagaagttt gaactaagtg agaactgttc agagcggcct 1920
gggctgcgag gagctgctgg ggagcaactc agctttgctg ctggactggt gtactcgctg 1980
agcctgctgc aggccacgct acaccgctat gagcatgccc tctctcagtg cagtgtggat 2040
gtgtataaga aagtgggcag cctgtaccct gagatgagtg cccatgagcg ctccttggat 2100
ttcctcattg aactgctgca caaggatcag ctggatgaga ctgtcaatgt ggagcctctc 2160
accaaggcca tcaagtacta tcagcatctg tacagcatcc accttgccga acagcctgag 2220
gactgtacta tgcagctggc tgaccacatt aagttcacgc agagtgctct ggactgcatg 2280
agtgtggagg taggacggct gcgtgccttc ttgcagggtg ggcaggaggc tacagatatt 2340
gccctcctgc tccgggatct ggaaacttca tgcagtgaca tccgccagtt ctgcaagaag 2400
atccgaaggc gaatgccagg gacagatgct cctgggatcc cagctgcact ggcctttgga 2460
ccacaggtat ctgacacgct cctagactgc aggaaacact tgacgtgggt cgtggctgtg 2520
ctgcaggagg tggcagctgc tgctgcccag ctcattgccc cactggcaga gaatgagggg 2580
ctacttgtgg ctgctctgga ggaactggct ttcaaagcaa gcgagcagat ctatgggacc 2640
ccctccagca gcccctatga gtgtctgcgc cagtcatgca acatcctcat cagtaccatg 2700
aacaagctgg ccacagccat gcaggagggg gagtatgatg cagagcggcc ccccagcaag 2760
cctccaccgg ttgaactgcg ggctgctgcc cttcgtgcag agatcacaga tgctgaaggc 2820
ctgggtttga agctcgaaga tcgagagaca gttattaagg agttgaagaa gtcactcaag 2880
attaagggag aggagctaag tgaggccaat gtgcggctga gcctcctgga gaagaagttg 2940
gacagtgctg ccaaggatgc agatgagcgc atcgagaaag tccagactcg gctggaggag 3000
acccaggcac tgctgcgaaa gaaggagaaa gagtttgagg agacaatgga tgcactccag 3060
gctgacatcg accagctgga ggcagagaag gcagaactaa agcagcgtct gaacagccag 3120
tccaaacgca cgattgaggg actccggggc cctcctcctt caggcattgc tactctggtc 3180
tctggcattg ctggtgaaga acagcagcga ggagccatcc ctgggcaggc tccagggtct 3240
gtgccaggcc cagggctggt gaaggactca ccactgctgc ttcagcagat ctctgccatg 3300
aggctgcaca tctcccagct ccagcatgag aacagcatcc tcaagggagc ccagatgaag 3360
gcatccttgg catccctgcc ccctctgcat gttgcaaagc tatcccatga gggccctggc 3420
agtgagttac cagctggagc gctgtatcgt aagaccagcc agctgctgga gacattgaat 3480
caattgagca cacacacgca cgtagtagac atcactcgca ccagccctgc tgccaagagc 3540
ccgtcggccc aacttatgga gcaagtggct cagcttaagt ccctgagtga caccgtcgag 3600
aagctcaagg atgaggtcct caaggagaca gtatctcagc gccctggagc cacagtaccc 3660
actgactttg ccaccttccc ttcatcagcc ttcctcaggg aggatccaaa gtgggaattc 3720
cctcggaaga acttggttct tggaaaaact ctaggagaag gcgaatttgg aaaagtggtc 3780
aaggcaacgg ccttccatct gaaaggcaga gcagggtaca ccacggtggc cgtgaagatg 3840
ctgaaagaga acgcctcccc gagtgagctt cgagacctgc tgtcagagtt caacgtcctg 3900
aagcaggtca accacccaca tgtcatcaaa ttgtatgggg cctgcagcca ggatggcccg 3960
ctcctcctca tcgtggagta cgccaaatac ggctccctgc ggggcttcct ccgcgagagc 4020
cgcaaagtgg ggcctggcta cctgggcagt ggaggcagcc gcaactccag ctccctggac 4080
cacccggatg agcgggccct caccatgggc gacctcatct catttgcctg gcagatctca 4140
caggggatgc agtatctggc cgagatgaag ctcgttcatc gggacttggc agccagaaac 4200
atcctggtag ctgaggggcg gaagatgaag atttcggatt tcggcttgtc ccgagatgtt 4260
tatgaagagg attcctacgt gaagaggagc cagggtcgga ttccagttaa atggatggca 4320
attgaatccc tttttgatca tatctacacc acgcaaagtg atgtatggtc ttttggtgtc 4380
ctgctgtggg agatcgtgac cctaggggga aacccctatc ctgggattcc tcctgagcgg 4440
ctcttcaacc ttctgaagac cggccaccgg atggagaggc cagacaactg cagcgaggag 4500
atgtaccgcc tgatgctgca atgctggaag caggagccgg acaaaaggcc ggtgtttgcg 4560
gacatcagca aagacctgga gaagatgatg gttaagagga gagactactt ggaccttgcg 4620
gcgtccactc catctgactc cctgatttat gacgacggcc tctcagagga ggagacaccg 4680
ctggtggact gtaataatgc ccccctccct cgagccctcc cttccacatg gattgaaaac 4740
aaactctatg gtagaatttc ccatgcattt actagattct ag 4782
<210> 4
<211> 1593
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 4
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Arg Gly Leu Lys Pro Lys Lys Ala Pro Thr Ala Arg Lys
130 135 140
Thr Thr Thr Arg Arg Pro Lys Pro Thr Arg Pro Ala Ser Thr Gly Val
145 150 155 160
Ala Gly Ala Ser Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly
165 170 175
Glu Leu Ser Ser Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala
180 185 190
Ala Pro Ile Ile Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro
195 200 205
Pro Leu Pro Ser Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val
210 215 220
Arg Asp Leu Glu Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu
225 230 235 240
Asp Lys Ala Lys Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu
245 250 255
Gln Val Gln Glu Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu
260 265 270
Gln Arg Arg Leu Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu
275 280 285
Ala Lys Glu Arg Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile
290 295 300
Glu Met Ala Thr Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser
305 310 315 320
Leu Gln Gln Glu Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr
325 330 335
Thr Asp Leu Glu Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp
340 345 350
Gly Ala Ala Ser Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala
355 360 365
Arg Leu Lys Asp Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu
370 375 380
Lys Gln Glu His Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln
385 390 395 400
Glu Leu Glu Val Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu
405 410 415
Ser Gln Ala Glu Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala
420 425 430
Ala Leu Gly Ala Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu
435 440 445
Asn Leu Glu Glu Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu
450 455 460
Glu Ala Met Asn Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu
465 470 475 480
Thr Glu Leu Glu Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val
485 490 495
Arg Glu Ala Gln Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp
500 505 510
Tyr Gln Gln Thr Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln
515 520 525
Asp Val Asn Arg Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg
530 535 540
Gln Gln Gln Pro Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala
545 550 555 560
Glu Thr Lys Ala His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met
565 570 575
Glu Val Ala Gln Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met
580 585 590
Pro Asp Ser Phe Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val
595 600 605
Leu Leu Leu Met Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys
610 615 620
Gln Ala Gln Glu Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro
625 630 635 640
Gly Leu Arg Gly Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu
645 650 655
Val Tyr Ser Leu Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His
660 665 670
Ala Leu Ser Gln Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu
675 680 685
Tyr Pro Glu Met Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu
690 695 700
Leu Leu His Lys Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu
705 710 715 720
Thr Lys Ala Ile Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala
725 730 735
Glu Gln Pro Glu Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe
740 745 750
Thr Gln Ser Ala Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg
755 760 765
Ala Phe Leu Gln Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu
770 775 780
Arg Asp Leu Glu Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys
785 790 795 800
Ile Arg Arg Arg Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala
805 810 815
Leu Ala Phe Gly Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys
820 825 830
His Leu Thr Trp Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala
835 840 845
Ala Gln Leu Ile Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala
850 855 860
Ala Leu Glu Glu Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr
865 870 875 880
Pro Ser Ser Ser Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu
885 890 895
Ile Ser Thr Met Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr
900 905 910
Asp Ala Glu Arg Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala
915 920 925
Ala Ala Leu Arg Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys
930 935 940
Leu Glu Asp Arg Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys
945 950 955 960
Ile Lys Gly Glu Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu
965 970 975
Glu Lys Lys Leu Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu
980 985 990
Lys Val Gln Thr Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys
995 1000 1005
Glu Lys Glu Phe Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile
1010 1015 1020
Asp Gln Leu Glu Ala Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn
1025 1030 1035
Ser Gln Ser Lys Arg Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro
1040 1045 1050
Ser Gly Ile Ala Thr Leu Val Ser Gly Ile Ala Gly Glu Glu Gln
1055 1060 1065
Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
1070 1075 1080
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser
1085 1090 1095
Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile
1100 1105 1110
Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro
1115 1120 1125
Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu
1130 1135 1140
Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr
1145 1150 1155
Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr Arg
1160 1165 1170
Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln
1175 1180 1185
Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys
1190 1195 1200
Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr
1205 1210 1215
Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg
1220 1225 1230
Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu Val Leu Gly
1235 1240 1245
Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val Lys Ala Thr
1250 1255 1260
Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala Val
1265 1270 1275
Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp Leu
1280 1285 1290
Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His Pro His Val
1295 1300 1305
Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu Leu
1310 1315 1320
Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu Arg
1325 1330 1335
Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser
1340 1345 1350
Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr
1355 1360 1365
Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met
1370 1375 1380
Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala Ala
1385 1390 1395
Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp
1400 1405 1410
Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys
1415 1420 1425
Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala Ile Glu Ser
1430 1435 1440
Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser Phe
1445 1450 1455
Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly Asn Pro Tyr
1460 1465 1470
Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr Gly
1475 1480 1485
His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr Arg
1490 1495 1500
Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro Val
1505 1510 1515
Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met Val Lys Arg
1520 1525 1530
Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser Leu
1535 1540 1545
Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro Leu Val Asp
1550 1555 1560
Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp Ile
1565 1570 1575
Glu Asn Lys Leu Tyr Gly Arg Ile Ser His Ala Phe Thr Arg Phe
1580 1585 1590
<210> 5
<211> 4506
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 5
atgatgagac aggcaccgac agcccgaaag accacaactc ggcgacccaa gcccacgcgc 60
ccagccagta ctggggtggc tggggccagt agctccctgg gcccctctgg ctcagcgtca 120
gcaggtgagc tgagcagcag tgagcccagc accccggctc agactccgct ggcagcaccc 180
atcatcccca cgccggtcct cacctctcct ggagcagtcc ccccgcttcc ttccccatcc 240
aaggaggagg agggactaag ggctcaggtg cgggacctgg aggagaaact agagaccctg 300
agactgaaac gggcagaaga caaagcaaag ctaaaagagc tggagaaaca caaaatccag 360
ctggagcagg tgcaggaatg gaagagcaaa atgcaggagc agcaggccga cctgcagcgg 420
cgcctcaagg aggcgagaaa ggaagccaag gaggcgctgg aggcaaagga acgctatatg 480
gaggagatgg ctgatactgc tgatgccatt gagatggcca ctttggacaa ggagatggct 540
gaagagcggg ctgagtccct gcagcaggag gtggaggcac tgaaggagcg ggtggacgag 600
ctcactactg acttagagat cctcaaggct gagattgaag agaagggctc agatggcgct 660
gcatccagtt atcagctcaa gcagcttgag gagcagaatg cccgcctgaa ggatgccctg 720
gtgaggatgc gggatctttc ttcctcagag aagcaggagc atgtgaagct ccagaagctc 780
atggaaaaga agaaccaaga gctggaagtt gtgaggcaac agcgggagcg tctgcaggag 840
gagctaagcc aggcagagag caccattgat gagctcaagg agcaggtgga tgctgctctg 900
ggtgctgagg agatggtgga gatgctgaca gatcggaacc tgaatctgga agagaaagtg 960
cgcgagttga gggagactgt gggagacttg gaagcgatga atgagatgaa cgatgagctg 1020
caggagaatg cacgtgagac agaactggag ctgcgggagc agctggacat ggcaggcgcg 1080
cgggttcgtg aggcccagaa gcgtgtggag gcagcccagg agacggttgc agactaccag 1140
cagaccatca agaagtaccg ccagctgacc gcccatctac aggatgtgaa tcgggaactg 1200
acaaaccagc aggaagcatc tgtggagagg caacagcagc cacctccaga gacctttgac 1260
ttcaaaatca agtttgctga gactaaggcc catgccaagg caattgagat ggaattgagg 1320
cagatggagg tggcccaggc caatcgacac atgtccctgc tgacagcctt catgcctgac 1380
agcttccttc ggccaggtgg ggaccatgac tgcgttctgg tgctgttgct catgcctcgt 1440
ctcatttgca aggcagagct gatccggaag caggcccagg agaagtttga actaagtgag 1500
aactgttcag agcggcctgg gctgcgagga gctgctgggg agcaactcag ctttgctgct 1560
ggactggtgt actcgctgag cctgctgcag gccacgctac accgctatga gcatgccctc 1620
tctcagtgca gtgtggatgt gtataagaaa gtgggcagcc tgtaccctga gatgagtgcc 1680
catgagcgct ccttggattt cctcattgaa ctgctgcaca aggatcagct ggatgagact 1740
gtcaatgtgg agcctctcac caaggccatc aagtactatc agcatctgta cagcatccac 1800
cttgccgaac agcctgagga ctgtactatg cagctggctg accacattaa gttcacgcag 1860
agtgctctgg actgcatgag tgtggaggta ggacggctgc gtgccttctt gcagggtggg 1920
caggaggcta cagatattgc cctcctgctc cgggatctgg aaacttcatg cagtgacatc 1980
cgccagttct gcaagaagat ccgaaggcga atgccaggga cagatgctcc tgggatccca 2040
gctgcactgg cctttggacc acaggtatct gacacgctcc tagactgcag gaaacacttg 2100
acgtgggtcg tggctgtgct gcaggaggtg gcagctgctg ctgcccagct cattgcccca 2160
ctggcagaga atgaggggct acttgtggct gctctggagg aactggcttt caaagcaagc 2220
gagcagatct atgggacccc ctccagcagc ccctatgagt gtctgcgcca gtcatgcaac 2280
atcctcatca gtaccatgaa caagctggcc acagccatgc aggaggggga gtatgatgca 2340
gagcggcccc ccagcaagcc tccaccggtt gaactgcggg ctgctgccct tcgtgcagag 2400
atcacagatg ctgaaggcct gggtttgaag ctcgaagatc gagagacagt tattaaggag 2460
ttgaagaagt cactcaagat taagggagag gagctaagtg aggccaatgt gcggctgagc 2520
ctcctggaga agaagttgga cagtgctgcc aaggatgcag atgagcgcat cgagaaagtc 2580
cagactcggc tggaggagac ccaggcactg ctgcgaaaga aggagaaaga gtttgaggag 2640
acaatggatg cactccaggc tgacatcgac cagctggagg cagagaaggc agaactaaag 2700
cagcgtctga acagccagtc caaacgcacg attgagggac tccggggccc tcctccttca 2760
ggcattgcta ctctggtctc tggcattgct ggtgaagaac agcagcgagg agccatccct 2820
gggcaggctc cagggtctgt gccaggccca gggctggtga aggactcacc actgctgctt 2880
cagcagatct ctgccatgag gctgcacatc tcccagctcc agcatgagaa cagcatcctc 2940
aagggagccc agatgaaggc atccttggca tccctgcccc ctctgcatgt tgcaaagcta 3000
tcccatgagg gccctggcag tgagttacca gctggagcgc tgtatcgtaa gaccagccag 3060
ctgctggaga cattgaatca attgagcaca cacacgcacg tagtagacat cactcgcacc 3120
agccctgctg ccaagagccc gtcggcccaa cttatggagc aagtggctca gcttaagtcc 3180
ctgagtgaca ccgtcgagaa gctcaaggat gaggtcctca aggagacagt atctcagcgc 3240
cctggagcca cagtacccac tgactttgcc accttccctt catcagcctt cctcagggag 3300
gatccaaagt gggaattccc tcggaagaac ttggttcttg gaaaaactct aggagaaggc 3360
gaatttggaa aagtggtcaa ggcaacggcc ttccatctga aaggcagagc agggtacacc 3420
acggtggccg tgaagatgct gaaagagaac gcctccccga gtgagcttcg agacctgctg 3480
tcagagttca acgtcctgaa gcaggtcaac cacccacatg tcatcaaatt gtatggggcc 3540
tgcagccagg atggcccgct cctcctcatc gtggagtacg ccaaatacgg ctccctgcgg 3600
ggcttcctcc gcgagagccg caaagtgggg cctggctacc tgggcagtgg aggcagccgc 3660
aactccagct ccctggacca cccggatgag cgggccctca ccatgggcga cctcatctca 3720
tttgcctggc agatctcaca ggggatgcag tatctggccg agatgaagct cgttcatcgg 3780
gacttggcag ccagaaacat cctggtagct gaggggcgga agatgaagat ttcggatttc 3840
ggcttgtccc gagatgttta tgaagaggat tcctacgtga agaggagcca gggtcggatt 3900
ccagttaaat ggatggcaat tgaatccctt tttgatcata tctacaccac gcaaagtgat 3960
gtatggtctt ttggtgtcct gctgtgggag atcgtgaccc tagggggaaa cccctatcct 4020
gggattcctc ctgagcggct cttcaacctt ctgaagaccg gccaccggat ggagaggcca 4080
gacaactgca gcgaggagat gtaccgcctg atgctgcaat gctggaagca ggagccggac 4140
aaaaggccgg tgtttgcgga catcagcaaa gacctggaga agatgatggt taagaggaga 4200
gactacttgg accttgcggc gtccactcca tctgactccc tgatttatga cgacggcctc 4260
tcagaggagg agacaccgct ggtggactgt aataatgccc ccctccctcg agccctccct 4320
tccacatgga ttgaaaacaa actctatggc atgtcagacc cgaactggcc tggagagagt 4380
cctgtaccac tcacgagagc tgatggcact aacactgggt ttccaagata tccaaatgat 4440
agtgtatatg ctaactggat gctttcaccc tcagcggcaa aattaatgga cacgtttgat 4500
agttaa 4506
<210> 6
<211> 1501
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 6
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro
930 935 940
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu
945 950 955 960
Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
965 970 975
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu
980 985 990
Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu
995 1000 1005
Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu
1010 1015 1020
Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr
1025 1030 1035
Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu
1040 1045 1050
Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu
1055 1060 1065
Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala
1070 1075 1080
Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu
1085 1090 1095
Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu Val Leu
1100 1105 1110
Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val Lys Ala
1115 1120 1125
Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala
1130 1135 1140
Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp
1145 1150 1155
Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His Pro His
1160 1165 1170
Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu
1175 1180 1185
Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu
1190 1195 1200
Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly
1205 1210 1215
Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu
1220 1225 1230
Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly
1235 1240 1245
Met Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala
1250 1255 1260
Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser
1265 1270 1275
Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val
1280 1285 1290
Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala Ile Glu
1295 1300 1305
Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser
1310 1315 1320
Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly Asn Pro
1325 1330 1335
Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr
1340 1345 1350
Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr
1355 1360 1365
Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro
1370 1375 1380
Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met Val Lys
1385 1390 1395
Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser
1400 1405 1410
Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro Leu Val
1415 1420 1425
Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp
1430 1435 1440
Ile Glu Asn Lys Leu Tyr Gly Met Ser Asp Pro Asn Trp Pro Gly
1445 1450 1455
Glu Ser Pro Val Pro Leu Thr Arg Ala Asp Gly Thr Asn Thr Gly
1460 1465 1470
Phe Pro Arg Tyr Pro Asn Asp Ser Val Tyr Ala Asn Trp Met Leu
1475 1480 1485
Ser Pro Ser Ala Ala Lys Leu Met Asp Thr Phe Asp Ser
1490 1495 1500
<210> 7
<211> 4380
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 7
atgatgagac aggcaccgac agcccgaaag accacaactc ggcgacccaa gcccacgcgc 60
ccagccagta ctggggtggc tggggccagt agctccctgg gcccctctgg ctcagcgtca 120
gcaggtgagc tgagcagcag tgagcccagc accccggctc agactccgct ggcagcaccc 180
atcatcccca cgccggtcct cacctctcct ggagcagtcc ccccgcttcc ttccccatcc 240
aaggaggagg agggactaag ggctcaggtg cgggacctgg aggagaaact agagaccctg 300
agactgaaac gggcagaaga caaagcaaag ctaaaagagc tggagaaaca caaaatccag 360
ctggagcagg tgcaggaatg gaagagcaaa atgcaggagc agcaggccga cctgcagcgg 420
cgcctcaagg aggcgagaaa ggaagccaag gaggcgctgg aggcaaagga acgctatatg 480
gaggagatgg ctgatactgc tgatgccatt gagatggcca ctttggacaa ggagatggct 540
gaagagcggg ctgagtccct gcagcaggag gtggaggcac tgaaggagcg ggtggacgag 600
ctcactactg acttagagat cctcaaggct gagattgaag agaagggctc agatggcgct 660
gcatccagtt atcagctcaa gcagcttgag gagcagaatg cccgcctgaa ggatgccctg 720
gtgaggatgc gggatctttc ttcctcagag aagcaggagc atgtgaagct ccagaagctc 780
atggaaaaga agaaccaaga gctggaagtt gtgaggcaac agcgggagcg tctgcaggag 840
gagctaagcc aggcagagag caccattgat gagctcaagg agcaggtgga tgctgctctg 900
ggtgctgagg agatggtgga gatgctgaca gatcggaacc tgaatctgga agagaaagtg 960
cgcgagttga gggagactgt gggagacttg gaagcgatga atgagatgaa cgatgagctg 1020
caggagaatg cacgtgagac agaactggag ctgcgggagc agctggacat ggcaggcgcg 1080
cgggttcgtg aggcccagaa gcgtgtggag gcagcccagg agacggttgc agactaccag 1140
cagaccatca agaagtaccg ccagctgacc gcccatctac aggatgtgaa tcgggaactg 1200
acaaaccagc aggaagcatc tgtggagagg caacagcagc cacctccaga gacctttgac 1260
ttcaaaatca agtttgctga gactaaggcc catgccaagg caattgagat ggaattgagg 1320
cagatggagg tggcccaggc caatcgacac atgtccctgc tgacagcctt catgcctgac 1380
agcttccttc ggccaggtgg ggaccatgac tgcgttctgg tgctgttgct catgcctcgt 1440
ctcatttgca aggcagagct gatccggaag caggcccagg agaagtttga actaagtgag 1500
aactgttcag agcggcctgg gctgcgagga gctgctgggg agcaactcag ctttgctgct 1560
ggactggtgt actcgctgag cctgctgcag gccacgctac accgctatga gcatgccctc 1620
tctcagtgca gtgtggatgt gtataagaaa gtgggcagcc tgtaccctga gatgagtgcc 1680
catgagcgct ccttggattt cctcattgaa ctgctgcaca aggatcagct ggatgagact 1740
gtcaatgtgg agcctctcac caaggccatc aagtactatc agcatctgta cagcatccac 1800
cttgccgaac agcctgagga ctgtactatg cagctggctg accacattaa gttcacgcag 1860
agtgctctgg actgcatgag tgtggaggta ggacggctgc gtgccttctt gcagggtggg 1920
caggaggcta cagatattgc cctcctgctc cgggatctgg aaacttcatg cagtgacatc 1980
cgccagttct gcaagaagat ccgaaggcga atgccaggga cagatgctcc tgggatccca 2040
gctgcactgg cctttggacc acaggtatct gacacgctcc tagactgcag gaaacacttg 2100
acgtgggtcg tggctgtgct gcaggaggtg gcagctgctg ctgcccagct cattgcccca 2160
ctggcagaga atgaggggct acttgtggct gctctggagg aactggcttt caaagcaagc 2220
gagcagatct atgggacccc ctccagcagc ccctatgagt gtctgcgcca gtcatgcaac 2280
atcctcatca gtaccatgaa caagctggcc acagccatgc aggaggggga gtatgatgca 2340
gagcggcccc ccagcaagcc tccaccggtt gaactgcggg ctgctgccct tcgtgcagag 2400
atcacagatg ctgaaggcct gggtttgaag ctcgaagatc gagagacagt tattaaggag 2460
ttgaagaagt cactcaagat taagggagag gagctaagtg aggccaatgt gcggctgagc 2520
ctcctggaga agaagttgga cagtgctgcc aaggatgcag atgagcgcat cgagaaagtc 2580
cagactcggc tggaggagac ccaggcactg ctgcgaaaga aggagaaaga gtttgaggag 2640
acaatggatg cactccaggc tgacatcgac cagctggagg cagagaaggc agaactaaag 2700
cagcgtctga acagccagtc caaacgcacg attgagggac tccggggccc tcctccttca 2760
ggcattgcta ctctggtctc tggcattgct ggtgaagaac agcagcgagg agccatccct 2820
gggcaggctc cagggtctgt gccaggccca gggctggtga aggactcacc actgctgctt 2880
cagcagatct ctgccatgag gctgcacatc tcccagctcc agcatgagaa cagcatcctc 2940
aagggagccc agatgaaggc atccttggca tccctgcccc ctctgcatgt tgcaaagcta 3000
tcccatgagg gccctggcag tgagttacca gctggagcgc tgtatcgtaa gaccagccag 3060
ctgctggaga cattgaatca attgagcaca cacacgcacg tagtagacat cactcgcacc 3120
agccctgctg ccaagagccc gtcggcccaa cttatggagc aagtggctca gcttaagtcc 3180
ctgagtgaca ccgtcgagaa gctcaaggat gaggtcctca aggagacagt atctcagcgc 3240
cctggagcca cagtacccac tgactttgcc accttccctt catcagcctt cctcagggag 3300
gatccaaagt gggaattccc tcggaagaac ttggttcttg gaaaaactct aggagaaggc 3360
gaatttggaa aagtggtcaa ggcaacggcc ttccatctga aaggcagagc agggtacacc 3420
acggtggccg tgaagatgct gaaagagaac gcctccccga gtgagcttcg agacctgctg 3480
tcagagttca acgtcctgaa gcaggtcaac cacccacatg tcatcaaatt gtatggggcc 3540
tgcagccagg atggcccgct cctcctcatc gtggagtacg ccaaatacgg ctccctgcgg 3600
ggcttcctcc gcgagagccg caaagtgggg cctggctacc tgggcagtgg aggcagccgc 3660
aactccagct ccctggacca cccggatgag cgggccctca ccatgggcga cctcatctca 3720
tttgcctggc agatctcaca ggggatgcag tatctggccg agatgaagct cgttcatcgg 3780
gacttggcag ccagaaacat cctggtagct gaggggcgga agatgaagat ttcggatttc 3840
ggcttgtccc gagatgttta tgaagaggat tcctacgtga agaggagcca gggtcggatt 3900
ccagttaaat ggatggcaat tgaatccctt tttgatcata tctacaccac gcaaagtgat 3960
gtatggtctt ttggtgtcct gctgtgggag atcgtgaccc tagggggaaa cccctatcct 4020
gggattcctc ctgagcggct cttcaacctt ctgaagaccg gccaccggat ggagaggcca 4080
gacaactgca gcgaggagat gtaccgcctg atgctgcaat gctggaagca ggagccggac 4140
aaaaggccgg tgtttgcgga catcagcaaa gacctggaga agatgatggt taagaggaga 4200
gactacttgg accttgcggc gtccactcca tctgactccc tgatttatga cgacggcctc 4260
tcagaggagg agacaccgct ggtggactgt aataatgccc ccctccctcg agccctccct 4320
tccacatgga ttgaaaacaa actctatggt agaatttccc atgcatttac tagattctag 4380
<210> 8
<211> 1459
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 8
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro
930 935 940
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu
945 950 955 960
Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
965 970 975
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu
980 985 990
Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu
995 1000 1005
Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu
1010 1015 1020
Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr
1025 1030 1035
Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu
1040 1045 1050
Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu
1055 1060 1065
Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala
1070 1075 1080
Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu
1085 1090 1095
Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu Val Leu
1100 1105 1110
Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val Lys Ala
1115 1120 1125
Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala
1130 1135 1140
Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp
1145 1150 1155
Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His Pro His
1160 1165 1170
Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu
1175 1180 1185
Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu
1190 1195 1200
Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly
1205 1210 1215
Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu
1220 1225 1230
Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly
1235 1240 1245
Met Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala
1250 1255 1260
Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser
1265 1270 1275
Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val
1280 1285 1290
Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala Ile Glu
1295 1300 1305
Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser
1310 1315 1320
Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly Asn Pro
1325 1330 1335
Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr
1340 1345 1350
Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr
1355 1360 1365
Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro
1370 1375 1380
Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met Val Lys
1385 1390 1395
Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser
1400 1405 1410
Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro Leu Val
1415 1420 1425
Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp
1430 1435 1440
Ile Glu Asn Lys Leu Tyr Gly Arg Ile Ser His Ala Phe Thr Arg
1445 1450 1455
Phe
<210> 9
<211> 4833
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 9
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctgcccacgc gcccagccag tactggggtg 420
gctggggcca gtagctccct gggcccctct ggctcagcgt cagcaggtga gctgagcagc 480
agtgagccca gcaccccggc tcagactccg ctggcagcac ccatcatccc cacgccggtc 540
ctcacctctc ctggagcagt ccccccgctt ccttccccat ccaaggagga ggagggacta 600
agggctcagg tgcgggacct ggaggagaaa ctagagaccc tgagactgaa acgggcagaa 660
gacaaagcaa agctaaaaga gctggagaaa cacaaaatcc agctggagca ggtgcaggaa 720
tggaagagca aaatgcagga gcagcaggcc gacctgcagc ggcgcctcaa ggaggcgaga 780
aaggaagcca aggaggcgct ggaggcaaag gaacgctata tggaggagat ggctgatact 840
gctgatgcca ttgagatggc cactttggac aaggagatgg ctgaagagcg ggctgagtcc 900
ctgcagcagg aggtggaggc actgaaggag cgggtggacg agctcactac tgacttagag 960
atcctcaagg ctgagattga agagaagggc tcagatggcg ctgcatccag ttatcagctc 1020
aagcagcttg aggagcagaa tgcccgcctg aaggatgccc tggtgaggat gcgggatctt 1080
tcttcctcag agaagcagga gcatgtgaag ctccagaagc tcatggaaaa gaagaaccaa 1140
gagctggaag ttgtgaggca acagcgggag cgtctgcagg aggagctaag ccaggcagag 1200
agcaccattg atgagctcaa ggagcaggtg gatgctgctc tgggtgctga ggagatggtg 1260
gagatgctga cagatcggaa cctgaatctg gaagagaaag tgcgcgagtt gagggagact 1320
gtgggagact tggaagcgat gaatgagatg aacgatgagc tgcaggagaa tgcacgtgag 1380
acagaactgg agctgcggga gcagctggac atggcaggcg cgcgggttcg tgaggcccag 1440
aagcgtgtgg aggcagccca ggagacggtt gcagactacc agcagaccat caagaagtac 1500
cgccagctga ccgcccatct acaggatgtg aatcgggaac tgacaaacca gcaggaagca 1560
tctgtggaga ggcaacagca gccacctcca gagacctttg acttcaaaat caagtttgct 1620
gagactaagg cccatgccaa ggcaattgag atggaattga ggcagatgga ggtggcccag 1680
gccaatcgac acatgtccct gctgacagcc ttcatgcctg acagcttcct tcggccaggt 1740
ggggaccatg actgcgttct ggtgctgttg ctcatgcctc gtctcatttg caaggcagag 1800
ctgatccgga agcaggccca ggagaagttt gaactaagtg agaactgttc agagcggcct 1860
gggctgcgag gagctgctgg ggagcaactc agctttgctg ctggactggt gtactcgctg 1920
agcctgctgc aggccacgct acaccgctat gagcatgccc tctctcagtg cagtgtggat 1980
gtgtataaga aagtgggcag cctgtaccct gagatgagtg cccatgagcg ctccttggat 2040
ttcctcattg aactgctgca caaggatcag ctggatgaga ctgtcaatgt ggagcctctc 2100
accaaggcca tcaagtacta tcagcatctg tacagcatcc accttgccga acagcctgag 2160
gactgtacta tgcagctggc tgaccacatt aagttcacgc agagtgctct ggactgcatg 2220
agtgtggagg taggacggct gcgtgccttc ttgcagggtg ggcaggaggc tacagatatt 2280
gccctcctgc tccgggatct ggaaacttca tgcagtgaca tccgccagtt ctgcaagaag 2340
atccgaaggc gaatgccagg gacagatgct cctgggatcc cagctgcact ggcctttgga 2400
ccacaggtat ctgacacgct cctagactgc aggaaacact tgacgtgggt cgtggctgtg 2460
ctgcaggagg tggcagctgc tgctgcccag ctcattgccc cactggcaga gaatgagggg 2520
ctacttgtgg ctgctctgga ggaactggct ttcaaagcaa gcgagcagat ctatgggacc 2580
ccctccagca gcccctatga gtgtctgcgc cagtcatgca acatcctcat cagtaccatg 2640
aacaagctgg ccacagccat gcaggagggg gagtatgatg cagagcggcc ccccagcaag 2700
cctccaccgg ttgaactgcg ggctgctgcc cttcgtgcag agatcacaga tgctgaaggc 2760
ctgggtttga agctcgaaga tcgagagaca gttattaagg agttgaagaa gtcactcaag 2820
attaagggag aggagctaag tgaggccaat gtgcggctga gcctcctgga gaagaagttg 2880
gacagtgctg ccaaggatgc agatgagcgc atcgagaaag tccagactcg gctggaggag 2940
acccaggcac tgctgcgaaa gaaggagaaa gagtttgagg agacaatgga tgcactccag 3000
gctgacatcg accagctgga ggcagagaag gcagaactaa agcagcgtct gaacagccag 3060
tccaaacgca cgattgaggg actccggggc cctcctcctt caggcattgc tactctggtc 3120
tctggcattg ctggtggagc catccctggg caggctccag ggtctgtgcc aggcccaggg 3180
ctggtgaagg actcaccact gctgcttcag cagatctctg ccatgaggct gcacatctcc 3240
cagctccagc atgagaacag catcctcaag ggagcccaga tgaaggcatc cttggcatcc 3300
ctgccccctc tgcatgttgc aaagctatcc catgagggcc ctggcagtga gttaccagct 3360
ggagcgctgt atcgtaagac cagccagctg ctggagacat tgaatcaatt gagcacacac 3420
acgcacgtag tagacatcac tcgcaccagc cctgctgcca agagcccgtc ggcccaactt 3480
atggagcaag tggctcagct taagtccctg agtgacaccg tcgagaagct caaggatgag 3540
gtcctcaagg agacagtatc tcagcgccct ggagccacag tacccactga ctttgccacc 3600
ttcccttcat cagccttcct cagggaggat ccaaagtggg aattccctcg gaagaacttg 3660
gttcttggaa aaactctagg agaaggcgaa tttggaaaag tggtcaaggc aacggccttc 3720
catctgaaag gcagagcagg gtacaccacg gtggccgtga agatgctgaa agagaacgcc 3780
tccccgagtg agcttcgaga cctgctgtca gagttcaacg tcctgaagca ggtcaaccac 3840
ccacatgtca tcaaattgta tggggcctgc agccaggatg gcccgctcct cctcatcgtg 3900
gagtacgcca aatacggctc cctgcggggc ttcctccgcg agagccgcaa agtggggcct 3960
ggctacctgg gcagtggagg cagccgcaac tccagctccc tggaccaccc ggatgagcgg 4020
gccctcacca tgggcgacct catctcattt gcctggcaga tctcacaggg gatgcagtat 4080
ctggccgaga tgaagctcgt tcatcgggac ttggcagcca gaaacatcct ggtagctgag 4140
gggcggaaga tgaagatttc ggatttcggc ttgtcccgag atgtttatga agaggattcc 4200
tacgtgaaga ggagccaggg tcggattcca gttaaatgga tggcaattga atcccttttt 4260
gatcatatct acaccacgca aagtgatgta tggtcttttg gtgtcctgct gtgggagatc 4320
gtgaccctag ggggaaaccc ctatcctggg attcctcctg agcggctctt caaccttctg 4380
aagaccggcc accggatgga gaggccagac aactgcagcg aggagatgta ccgcctgatg 4440
ctgcaatgct ggaagcagga gccggacaaa aggccggtgt ttgcggacat cagcaaagac 4500
ctggagaaga tgatggttaa gaggagagac tacttggacc ttgcggcgtc cactccatct 4560
gactccctga tttatgacga cggcctctca gaggaggaga caccgctggt ggactgtaat 4620
aatgcccccc tccctcgagc cctcccttcc acatggattg aaaacaaact ctatggcatg 4680
tcagacccga actggcctgg agagagtcct gtaccactca cgagagctga tggcactaac 4740
actgggtttc caagatatcc aaatgatagt gtatatgcta actggatgct ttcaccctca 4800
gcggcaaaat taatggacac gtttgatagt taa 4833
<210> 10
<211> 1610
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 10
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser
130 135 140
Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser
145 150 155 160
Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile
165 170 175
Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser
180 185 190
Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu
195 200 205
Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys
210 215 220
Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu
225 230 235 240
Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu
245 250 255
Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg
260 265 270
Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr
275 280 285
Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu
290 295 300
Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu
305 310 315 320
Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser
325 330 335
Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp
340 345 350
Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His
355 360 365
Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val
370 375 380
Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu
385 390 395 400
Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala
405 410 415
Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu
420 425 430
Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn
435 440 445
Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu
450 455 460
Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln
465 470 475 480
Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr
485 490 495
Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg
500 505 510
Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro
515 520 525
Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala
530 535 540
His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln
545 550 555 560
Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe
565 570 575
Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met
580 585 590
Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu
595 600 605
Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly
610 615 620
Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu
625 630 635 640
Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln
645 650 655
Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met
660 665 670
Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys
675 680 685
Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile
690 695 700
Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu
705 710 715 720
Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala
725 730 735
Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln
740 745 750
Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu
755 760 765
Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg
770 775 780
Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly
785 790 795 800
Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp
805 810 815
Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile
820 825 830
Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu
835 840 845
Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser
850 855 860
Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met
865 870 875 880
Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg
885 890 895
Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg
900 905 910
Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg
915 920 925
Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu
930 935 940
Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu
945 950 955 960
Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr
965 970 975
Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe
980 985 990
Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala
995 1000 1005
Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg
1010 1015 1020
Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr
1025 1030 1035
Leu Val Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro
1040 1045 1050
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu
1055 1060 1065
Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln
1070 1075 1080
His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu
1085 1090 1095
Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly
1100 1105 1110
Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser
1115 1120 1125
Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val
1130 1135 1140
Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala
1145 1150 1155
Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr
1160 1165 1170
Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln
1175 1180 1185
Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser
1190 1195 1200
Ser Ala Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys
1205 1210 1215
Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys
1220 1225 1230
Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr
1235 1240 1245
Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser
1250 1255 1260
Glu Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val
1265 1270 1275
Asn His Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp
1280 1285 1290
Gly Pro Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu
1295 1300 1305
Arg Gly Phe Leu Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu
1310 1315 1320
Gly Ser Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp
1325 1330 1335
Glu Arg Ala Leu Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln
1340 1345 1350
Ile Ser Gln Gly Met Gln Tyr Leu Ala Glu Met Lys Leu Val His
1355 1360 1365
Arg Asp Leu Ala Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys
1370 1375 1380
Met Lys Ile Ser Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu
1385 1390 1395
Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp
1400 1405 1410
Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser
1415 1420 1425
Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu
1430 1435 1440
Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn
1445 1450 1455
Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser
1460 1465 1470
Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro
1475 1480 1485
Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys
1490 1495 1500
Met Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr
1505 1510 1515
Pro Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu
1520 1525 1530
Thr Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu
1535 1540 1545
Pro Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Met Ser Asp Pro
1550 1555 1560
Asn Trp Pro Gly Glu Ser Pro Val Pro Leu Thr Arg Ala Asp Gly
1565 1570 1575
Thr Asn Thr Gly Phe Pro Arg Tyr Pro Asn Asp Ser Val Tyr Ala
1580 1585 1590
Asn Trp Met Leu Ser Pro Ser Ala Ala Lys Leu Met Asp Thr Phe
1595 1600 1605
Asp Ser
1610
<210> 11
<211> 4707
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 11
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctgcccacgc gcccagccag tactggggtg 420
gctggggcca gtagctccct gggcccctct ggctcagcgt cagcaggtga gctgagcagc 480
agtgagccca gcaccccggc tcagactccg ctggcagcac ccatcatccc cacgccggtc 540
ctcacctctc ctggagcagt ccccccgctt ccttccccat ccaaggagga ggagggacta 600
agggctcagg tgcgggacct ggaggagaaa ctagagaccc tgagactgaa acgggcagaa 660
gacaaagcaa agctaaaaga gctggagaaa cacaaaatcc agctggagca ggtgcaggaa 720
tggaagagca aaatgcagga gcagcaggcc gacctgcagc ggcgcctcaa ggaggcgaga 780
aaggaagcca aggaggcgct ggaggcaaag gaacgctata tggaggagat ggctgatact 840
gctgatgcca ttgagatggc cactttggac aaggagatgg ctgaagagcg ggctgagtcc 900
ctgcagcagg aggtggaggc actgaaggag cgggtggacg agctcactac tgacttagag 960
atcctcaagg ctgagattga agagaagggc tcagatggcg ctgcatccag ttatcagctc 1020
aagcagcttg aggagcagaa tgcccgcctg aaggatgccc tggtgaggat gcgggatctt 1080
tcttcctcag agaagcagga gcatgtgaag ctccagaagc tcatggaaaa gaagaaccaa 1140
gagctggaag ttgtgaggca acagcgggag cgtctgcagg aggagctaag ccaggcagag 1200
agcaccattg atgagctcaa ggagcaggtg gatgctgctc tgggtgctga ggagatggtg 1260
gagatgctga cagatcggaa cctgaatctg gaagagaaag tgcgcgagtt gagggagact 1320
gtgggagact tggaagcgat gaatgagatg aacgatgagc tgcaggagaa tgcacgtgag 1380
acagaactgg agctgcggga gcagctggac atggcaggcg cgcgggttcg tgaggcccag 1440
aagcgtgtgg aggcagccca ggagacggtt gcagactacc agcagaccat caagaagtac 1500
cgccagctga ccgcccatct acaggatgtg aatcgggaac tgacaaacca gcaggaagca 1560
tctgtggaga ggcaacagca gccacctcca gagacctttg acttcaaaat caagtttgct 1620
gagactaagg cccatgccaa ggcaattgag atggaattga ggcagatgga ggtggcccag 1680
gccaatcgac acatgtccct gctgacagcc ttcatgcctg acagcttcct tcggccaggt 1740
ggggaccatg actgcgttct ggtgctgttg ctcatgcctc gtctcatttg caaggcagag 1800
ctgatccgga agcaggccca ggagaagttt gaactaagtg agaactgttc agagcggcct 1860
gggctgcgag gagctgctgg ggagcaactc agctttgctg ctggactggt gtactcgctg 1920
agcctgctgc aggccacgct acaccgctat gagcatgccc tctctcagtg cagtgtggat 1980
gtgtataaga aagtgggcag cctgtaccct gagatgagtg cccatgagcg ctccttggat 2040
ttcctcattg aactgctgca caaggatcag ctggatgaga ctgtcaatgt ggagcctctc 2100
accaaggcca tcaagtacta tcagcatctg tacagcatcc accttgccga acagcctgag 2160
gactgtacta tgcagctggc tgaccacatt aagttcacgc agagtgctct ggactgcatg 2220
agtgtggagg taggacggct gcgtgccttc ttgcagggtg ggcaggaggc tacagatatt 2280
gccctcctgc tccgggatct ggaaacttca tgcagtgaca tccgccagtt ctgcaagaag 2340
atccgaaggc gaatgccagg gacagatgct cctgggatcc cagctgcact ggcctttgga 2400
ccacaggtat ctgacacgct cctagactgc aggaaacact tgacgtgggt cgtggctgtg 2460
ctgcaggagg tggcagctgc tgctgcccag ctcattgccc cactggcaga gaatgagggg 2520
ctacttgtgg ctgctctgga ggaactggct ttcaaagcaa gcgagcagat ctatgggacc 2580
ccctccagca gcccctatga gtgtctgcgc cagtcatgca acatcctcat cagtaccatg 2640
aacaagctgg ccacagccat gcaggagggg gagtatgatg cagagcggcc ccccagcaag 2700
cctccaccgg ttgaactgcg ggctgctgcc cttcgtgcag agatcacaga tgctgaaggc 2760
ctgggtttga agctcgaaga tcgagagaca gttattaagg agttgaagaa gtcactcaag 2820
attaagggag aggagctaag tgaggccaat gtgcggctga gcctcctgga gaagaagttg 2880
gacagtgctg ccaaggatgc agatgagcgc atcgagaaag tccagactcg gctggaggag 2940
acccaggcac tgctgcgaaa gaaggagaaa gagtttgagg agacaatgga tgcactccag 3000
gctgacatcg accagctgga ggcagagaag gcagaactaa agcagcgtct gaacagccag 3060
tccaaacgca cgattgaggg actccggggc cctcctcctt caggcattgc tactctggtc 3120
tctggcattg ctggtggagc catccctggg caggctccag ggtctgtgcc aggcccaggg 3180
ctggtgaagg actcaccact gctgcttcag cagatctctg ccatgaggct gcacatctcc 3240
cagctccagc atgagaacag catcctcaag ggagcccaga tgaaggcatc cttggcatcc 3300
ctgccccctc tgcatgttgc aaagctatcc catgagggcc ctggcagtga gttaccagct 3360
ggagcgctgt atcgtaagac cagccagctg ctggagacat tgaatcaatt gagcacacac 3420
acgcacgtag tagacatcac tcgcaccagc cctgctgcca agagcccgtc ggcccaactt 3480
atggagcaag tggctcagct taagtccctg agtgacaccg tcgagaagct caaggatgag 3540
gtcctcaagg agacagtatc tcagcgccct ggagccacag tacccactga ctttgccacc 3600
ttcccttcat cagccttcct cagggaggat ccaaagtggg aattccctcg gaagaacttg 3660
gttcttggaa aaactctagg agaaggcgaa tttggaaaag tggtcaaggc aacggccttc 3720
catctgaaag gcagagcagg gtacaccacg gtggccgtga agatgctgaa agagaacgcc 3780
tccccgagtg agcttcgaga cctgctgtca gagttcaacg tcctgaagca ggtcaaccac 3840
ccacatgtca tcaaattgta tggggcctgc agccaggatg gcccgctcct cctcatcgtg 3900
gagtacgcca aatacggctc cctgcggggc ttcctccgcg agagccgcaa agtggggcct 3960
ggctacctgg gcagtggagg cagccgcaac tccagctccc tggaccaccc ggatgagcgg 4020
gccctcacca tgggcgacct catctcattt gcctggcaga tctcacaggg gatgcagtat 4080
ctggccgaga tgaagctcgt tcatcgggac ttggcagcca gaaacatcct ggtagctgag 4140
gggcggaaga tgaagatttc ggatttcggc ttgtcccgag atgtttatga agaggattcc 4200
tacgtgaaga ggagccaggg tcggattcca gttaaatgga tggcaattga atcccttttt 4260
gatcatatct acaccacgca aagtgatgta tggtcttttg gtgtcctgct gtgggagatc 4320
gtgaccctag ggggaaaccc ctatcctggg attcctcctg agcggctctt caaccttctg 4380
aagaccggcc accggatgga gaggccagac aactgcagcg aggagatgta ccgcctgatg 4440
ctgcaatgct ggaagcagga gccggacaaa aggccggtgt ttgcggacat cagcaaagac 4500
ctggagaaga tgatggttaa gaggagagac tacttggacc ttgcggcgtc cactccatct 4560
gactccctga tttatgacga cggcctctca gaggaggaga caccgctggt ggactgtaat 4620
aatgcccccc tccctcgagc cctcccttcc acatggattg aaaacaaact ctatggtaga 4680
atttcccatg catttactag attctag 4707
<210> 12
<211> 1568
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 12
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser
130 135 140
Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser
145 150 155 160
Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile
165 170 175
Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser
180 185 190
Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu
195 200 205
Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys
210 215 220
Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu
225 230 235 240
Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu
245 250 255
Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg
260 265 270
Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr
275 280 285
Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu
290 295 300
Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu
305 310 315 320
Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser
325 330 335
Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp
340 345 350
Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His
355 360 365
Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val
370 375 380
Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu
385 390 395 400
Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala
405 410 415
Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu
420 425 430
Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn
435 440 445
Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu
450 455 460
Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln
465 470 475 480
Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr
485 490 495
Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg
500 505 510
Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro
515 520 525
Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala
530 535 540
His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln
545 550 555 560
Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe
565 570 575
Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met
580 585 590
Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu
595 600 605
Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly
610 615 620
Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu
625 630 635 640
Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln
645 650 655
Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met
660 665 670
Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys
675 680 685
Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile
690 695 700
Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu
705 710 715 720
Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala
725 730 735
Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln
740 745 750
Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu
755 760 765
Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg
770 775 780
Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly
785 790 795 800
Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp
805 810 815
Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile
820 825 830
Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu
835 840 845
Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser
850 855 860
Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met
865 870 875 880
Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg
885 890 895
Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg
900 905 910
Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg
915 920 925
Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu
930 935 940
Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu
945 950 955 960
Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr
965 970 975
Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe
980 985 990
Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala
995 1000 1005
Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg
1010 1015 1020
Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr
1025 1030 1035
Leu Val Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro
1040 1045 1050
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu
1055 1060 1065
Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln
1070 1075 1080
His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu
1085 1090 1095
Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly
1100 1105 1110
Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser
1115 1120 1125
Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val
1130 1135 1140
Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala
1145 1150 1155
Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr
1160 1165 1170
Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln
1175 1180 1185
Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser
1190 1195 1200
Ser Ala Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys
1205 1210 1215
Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys
1220 1225 1230
Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr
1235 1240 1245
Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser
1250 1255 1260
Glu Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val
1265 1270 1275
Asn His Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp
1280 1285 1290
Gly Pro Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu
1295 1300 1305
Arg Gly Phe Leu Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu
1310 1315 1320
Gly Ser Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp
1325 1330 1335
Glu Arg Ala Leu Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln
1340 1345 1350
Ile Ser Gln Gly Met Gln Tyr Leu Ala Glu Met Lys Leu Val His
1355 1360 1365
Arg Asp Leu Ala Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys
1370 1375 1380
Met Lys Ile Ser Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu
1385 1390 1395
Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp
1400 1405 1410
Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser
1415 1420 1425
Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu
1430 1435 1440
Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn
1445 1450 1455
Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser
1460 1465 1470
Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro
1475 1480 1485
Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys
1490 1495 1500
Met Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr
1505 1510 1515
Pro Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu
1520 1525 1530
Thr Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu
1535 1540 1545
Pro Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Arg Ile Ser His
1550 1555 1560
Ala Phe Thr Arg Phe
1565
<210> 13
<211> 4491
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 13
atgatgagac aggcaccgac agcccgaaag accacaactc ggcgacccaa gcccacgcgc 60
ccagccagta ctggggtggc tggggccagt agctccctgg gcccctctgg ctcagcgtca 120
gcaggtgagc tgagcagcag tgagcccagc accccggctc agactccgct ggcagcaccc 180
atcatcccca cgccggtcct cacctctcct ggagcagtcc ccccgcttcc ttccccatcc 240
aaggaggagg agggactaag ggctcaggtg cgggacctgg aggagaaact agagaccctg 300
agactgaaac gggcagaaga caaagcaaag ctaaaagagc tggagaaaca caaaatccag 360
ctggagcagg tgcaggaatg gaagagcaaa atgcaggagc agcaggccga cctgcagcgg 420
cgcctcaagg aggcgagaaa ggaagccaag gaggcgctgg aggcaaagga acgctatatg 480
gaggagatgg ctgatactgc tgatgccatt gagatggcca ctttggacaa ggagatggct 540
gaagagcggg ctgagtccct gcagcaggag gtggaggcac tgaaggagcg ggtggacgag 600
ctcactactg acttagagat cctcaaggct gagattgaag agaagggctc agatggcgct 660
gcatccagtt atcagctcaa gcagcttgag gagcagaatg cccgcctgaa ggatgccctg 720
gtgaggatgc gggatctttc ttcctcagag aagcaggagc atgtgaagct ccagaagctc 780
atggaaaaga agaaccaaga gctggaagtt gtgaggcaac agcgggagcg tctgcaggag 840
gagctaagcc aggcagagag caccattgat gagctcaagg agcaggtgga tgctgctctg 900
ggtgctgagg agatggtgga gatgctgaca gatcggaacc tgaatctgga agagaaagtg 960
cgcgagttga gggagactgt gggagacttg gaagcgatga atgagatgaa cgatgagctg 1020
caggagaatg cacgtgagac agaactggag ctgcgggagc agctggacat ggcaggcgcg 1080
cgggttcgtg aggcccagaa gcgtgtggag gcagcccagg agacggttgc agactaccag 1140
cagaccatca agaagtaccg ccagctgacc gcccatctac aggatgtgaa tcgggaactg 1200
acaaaccagc aggaagcatc tgtggagagg caacagcagc cacctccaga gacctttgac 1260
ttcaaaatca agtttgctga gactaaggcc catgccaagg caattgagat ggaattgagg 1320
cagatggagg tggcccaggc caatcgacac atgtccctgc tgacagcctt catgcctgac 1380
agcttccttc ggccaggtgg ggaccatgac tgcgttctgg tgctgttgct catgcctcgt 1440
ctcatttgca aggcagagct gatccggaag caggcccagg agaagtttga actaagtgag 1500
aactgttcag agcggcctgg gctgcgagga gctgctgggg agcaactcag ctttgctgct 1560
ggactggtgt actcgctgag cctgctgcag gccacgctac accgctatga gcatgccctc 1620
tctcagtgca gtgtggatgt gtataagaaa gtgggcagcc tgtaccctga gatgagtgcc 1680
catgagcgct ccttggattt cctcattgaa ctgctgcaca aggatcagct ggatgagact 1740
gtcaatgtgg agcctctcac caaggccatc aagtactatc agcatctgta cagcatccac 1800
cttgccgaac agcctgagga ctgtactatg cagctggctg accacattaa gttcacgcag 1860
agtgctctgg actgcatgag tgtggaggta ggacggctgc gtgccttctt gcagggtggg 1920
caggaggcta cagatattgc cctcctgctc cgggatctgg aaacttcatg cagtgacatc 1980
cgccagttct gcaagaagat ccgaaggcga atgccaggga cagatgctcc tgggatccca 2040
gctgcactgg cctttggacc acaggtatct gacacgctcc tagactgcag gaaacacttg 2100
acgtgggtcg tggctgtgct gcaggaggtg gcagctgctg ctgcccagct cattgcccca 2160
ctggcagaga atgaggggct acttgtggct gctctggagg aactggcttt caaagcaagc 2220
gagcagatct atgggacccc ctccagcagc ccctatgagt gtctgcgcca gtcatgcaac 2280
atcctcatca gtaccatgaa caagctggcc acagccatgc aggaggggga gtatgatgca 2340
gagcggcccc ccagcaagcc tccaccggtt gaactgcggg ctgctgccct tcgtgcagag 2400
atcacagatg ctgaaggcct gggtttgaag ctcgaagatc gagagacagt tattaaggag 2460
ttgaagaagt cactcaagat taagggagag gagctaagtg aggccaatgt gcggctgagc 2520
ctcctggaga agaagttgga cagtgctgcc aaggatgcag atgagcgcat cgagaaagtc 2580
cagactcggc tggaggagac ccaggcactg ctgcgaaaga aggagaaaga gtttgaggag 2640
acaatggatg cactccaggc tgacatcgac cagctggagg cagagaaggc agaactaaag 2700
cagcgtctga acagccagtc caaacgcacg attgagggac tccggggccc tcctccttca 2760
ggcattgcta ctctggtctc tggcattgct ggtggagcca tccctgggca ggctccaggg 2820
tctgtgccag gcccagggct ggtgaaggac tcaccactgc tgcttcagca gatctctgcc 2880
atgaggctgc acatctccca gctccagcat gagaacagca tcctcaaggg agcccagatg 2940
aaggcatcct tggcatccct gccccctctg catgttgcaa agctatccca tgagggccct 3000
ggcagtgagt taccagctgg agcgctgtat cgtaagacca gccagctgct ggagacattg 3060
aatcaattga gcacacacac gcacgtagta gacatcactc gcaccagccc tgctgccaag 3120
agcccgtcgg cccaacttat ggagcaagtg gctcagctta agtccctgag tgacaccgtc 3180
gagaagctca aggatgaggt cctcaaggag acagtatctc agcgccctgg agccacagta 3240
cccactgact ttgccacctt cccttcatca gccttcctca gggaggatcc aaagtgggaa 3300
ttccctcgga agaacttggt tcttggaaaa actctaggag aaggcgaatt tggaaaagtg 3360
gtcaaggcaa cggccttcca tctgaaaggc agagcagggt acaccacggt ggccgtgaag 3420
atgctgaaag agaacgcctc cccgagtgag cttcgagacc tgctgtcaga gttcaacgtc 3480
ctgaagcagg tcaaccaccc acatgtcatc aaattgtatg gggcctgcag ccaggatggc 3540
ccgctcctcc tcatcgtgga gtacgccaaa tacggctccc tgcggggctt cctccgcgag 3600
agccgcaaag tggggcctgg ctacctgggc agtggaggca gccgcaactc cagctccctg 3660
gaccacccgg atgagcgggc cctcaccatg ggcgacctca tctcatttgc ctggcagatc 3720
tcacagggga tgcagtatct ggccgagatg aagctcgttc atcgggactt ggcagccaga 3780
aacatcctgg tagctgaggg gcggaagatg aagatttcgg atttcggctt gtcccgagat 3840
gtttatgaag aggattccta cgtgaagagg agccagggtc ggattccagt taaatggatg 3900
gcaattgaat ccctttttga tcatatctac accacgcaaa gtgatgtatg gtcttttggt 3960
gtcctgctgt gggagatcgt gaccctaggg ggaaacccct atcctgggat tcctcctgag 4020
cggctcttca accttctgaa gaccggccac cggatggaga ggccagacaa ctgcagcgag 4080
gagatgtacc gcctgatgct gcaatgctgg aagcaggagc cggacaaaag gccggtgttt 4140
gcggacatca gcaaagacct ggagaagatg atggttaaga ggagagacta cttggacctt 4200
gcggcgtcca ctccatctga ctccctgatt tatgacgacg gcctctcaga ggaggagaca 4260
ccgctggtgg actgtaataa tgcccccctc cctcgagccc tcccttccac atggattgaa 4320
aacaaactct atggcatgtc agacccgaac tggcctggag agagtcctgt accactcacg 4380
agagctgatg gcactaacac tgggtttcca agatatccaa atgatagtgt atatgctaac 4440
tggatgcttt caccctcagc ggcaaaatta atggacacgt ttgatagtta a 4491
<210> 14
<211> 1496
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 14
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
930 935 940
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser Ala
945 950 955 960
Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile Leu Lys
965 970 975
Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val
980 985 990
Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala
995 1000 1005
Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu
1010 1015 1020
Ser Thr His Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala
1025 1030 1035
Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu
1040 1045 1050
Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu
1055 1060 1065
Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp
1070 1075 1080
Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg Glu Asp Pro Lys
1085 1090 1095
Trp Glu Phe Pro Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly
1100 1105 1110
Glu Gly Glu Phe Gly Lys Val Val Lys Ala Thr Ala Phe His Leu
1115 1120 1125
Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala Val Lys Met Leu Lys
1130 1135 1140
Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp Leu Leu Ser Glu Phe
1145 1150 1155
Asn Val Leu Lys Gln Val Asn His Pro His Val Ile Lys Leu Tyr
1160 1165 1170
Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu Leu Ile Val Glu Tyr
1175 1180 1185
Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu Arg Glu Ser Arg Lys
1190 1195 1200
Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser Arg Asn Ser Ser
1205 1210 1215
Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr Met Gly Asp Leu
1220 1225 1230
Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr Leu Ala
1235 1240 1245
Glu Met Lys Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile Leu
1250 1255 1260
Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser
1265 1270 1275
Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly
1280 1285 1290
Arg Ile Pro Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His
1295 1300 1305
Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu
1310 1315 1320
Trp Glu Ile Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro
1325 1330 1335
Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu
1340 1345 1350
Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr Arg Leu Met Leu Gln
1355 1360 1365
Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro Val Phe Ala Asp Ile
1370 1375 1380
Ser Lys Asp Leu Glu Lys Met Met Val Lys Arg Arg Asp Tyr Leu
1385 1390 1395
Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser Leu Ile Tyr Asp Asp
1400 1405 1410
Gly Leu Ser Glu Glu Glu Thr Pro Leu Val Asp Cys Asn Asn Ala
1415 1420 1425
Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp Ile Glu Asn Lys Leu
1430 1435 1440
Tyr Gly Met Ser Asp Pro Asn Trp Pro Gly Glu Ser Pro Val Pro
1445 1450 1455
Leu Thr Arg Ala Asp Gly Thr Asn Thr Gly Phe Pro Arg Tyr Pro
1460 1465 1470
Asn Asp Ser Val Tyr Ala Asn Trp Met Leu Ser Pro Ser Ala Ala
1475 1480 1485
Lys Leu Met Asp Thr Phe Asp Ser
1490 1495
<210> 15
<211> 4365
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 15
atgatgagac aggcaccgac agcccgaaag accacaactc ggcgacccaa gcccacgcgc 60
ccagccagta ctggggtggc tggggccagt agctccctgg gcccctctgg ctcagcgtca 120
gcaggtgagc tgagcagcag tgagcccagc accccggctc agactccgct ggcagcaccc 180
atcatcccca cgccggtcct cacctctcct ggagcagtcc ccccgcttcc ttccccatcc 240
aaggaggagg agggactaag ggctcaggtg cgggacctgg aggagaaact agagaccctg 300
agactgaaac gggcagaaga caaagcaaag ctaaaagagc tggagaaaca caaaatccag 360
ctggagcagg tgcaggaatg gaagagcaaa atgcaggagc agcaggccga cctgcagcgg 420
cgcctcaagg aggcgagaaa ggaagccaag gaggcgctgg aggcaaagga acgctatatg 480
gaggagatgg ctgatactgc tgatgccatt gagatggcca ctttggacaa ggagatggct 540
gaagagcggg ctgagtccct gcagcaggag gtggaggcac tgaaggagcg ggtggacgag 600
ctcactactg acttagagat cctcaaggct gagattgaag agaagggctc agatggcgct 660
gcatccagtt atcagctcaa gcagcttgag gagcagaatg cccgcctgaa ggatgccctg 720
gtgaggatgc gggatctttc ttcctcagag aagcaggagc atgtgaagct ccagaagctc 780
atggaaaaga agaaccaaga gctggaagtt gtgaggcaac agcgggagcg tctgcaggag 840
gagctaagcc aggcagagag caccattgat gagctcaagg agcaggtgga tgctgctctg 900
ggtgctgagg agatggtgga gatgctgaca gatcggaacc tgaatctgga agagaaagtg 960
cgcgagttga gggagactgt gggagacttg gaagcgatga atgagatgaa cgatgagctg 1020
caggagaatg cacgtgagac agaactggag ctgcgggagc agctggacat ggcaggcgcg 1080
cgggttcgtg aggcccagaa gcgtgtggag gcagcccagg agacggttgc agactaccag 1140
cagaccatca agaagtaccg ccagctgacc gcccatctac aggatgtgaa tcgggaactg 1200
acaaaccagc aggaagcatc tgtggagagg caacagcagc cacctccaga gacctttgac 1260
ttcaaaatca agtttgctga gactaaggcc catgccaagg caattgagat ggaattgagg 1320
cagatggagg tggcccaggc caatcgacac atgtccctgc tgacagcctt catgcctgac 1380
agcttccttc ggccaggtgg ggaccatgac tgcgttctgg tgctgttgct catgcctcgt 1440
ctcatttgca aggcagagct gatccggaag caggcccagg agaagtttga actaagtgag 1500
aactgttcag agcggcctgg gctgcgagga gctgctgggg agcaactcag ctttgctgct 1560
ggactggtgt actcgctgag cctgctgcag gccacgctac accgctatga gcatgccctc 1620
tctcagtgca gtgtggatgt gtataagaaa gtgggcagcc tgtaccctga gatgagtgcc 1680
catgagcgct ccttggattt cctcattgaa ctgctgcaca aggatcagct ggatgagact 1740
gtcaatgtgg agcctctcac caaggccatc aagtactatc agcatctgta cagcatccac 1800
cttgccgaac agcctgagga ctgtactatg cagctggctg accacattaa gttcacgcag 1860
agtgctctgg actgcatgag tgtggaggta ggacggctgc gtgccttctt gcagggtggg 1920
caggaggcta cagatattgc cctcctgctc cgggatctgg aaacttcatg cagtgacatc 1980
cgccagttct gcaagaagat ccgaaggcga atgccaggga cagatgctcc tgggatccca 2040
gctgcactgg cctttggacc acaggtatct gacacgctcc tagactgcag gaaacacttg 2100
acgtgggtcg tggctgtgct gcaggaggtg gcagctgctg ctgcccagct cattgcccca 2160
ctggcagaga atgaggggct acttgtggct gctctggagg aactggcttt caaagcaagc 2220
gagcagatct atgggacccc ctccagcagc ccctatgagt gtctgcgcca gtcatgcaac 2280
atcctcatca gtaccatgaa caagctggcc acagccatgc aggaggggga gtatgatgca 2340
gagcggcccc ccagcaagcc tccaccggtt gaactgcggg ctgctgccct tcgtgcagag 2400
atcacagatg ctgaaggcct gggtttgaag ctcgaagatc gagagacagt tattaaggag 2460
ttgaagaagt cactcaagat taagggagag gagctaagtg aggccaatgt gcggctgagc 2520
ctcctggaga agaagttgga cagtgctgcc aaggatgcag atgagcgcat cgagaaagtc 2580
cagactcggc tggaggagac ccaggcactg ctgcgaaaga aggagaaaga gtttgaggag 2640
acaatggatg cactccaggc tgacatcgac cagctggagg cagagaaggc agaactaaag 2700
cagcgtctga acagccagtc caaacgcacg attgagggac tccggggccc tcctccttca 2760
ggcattgcta ctctggtctc tggcattgct ggtggagcca tccctgggca ggctccaggg 2820
tctgtgccag gcccagggct ggtgaaggac tcaccactgc tgcttcagca gatctctgcc 2880
atgaggctgc acatctccca gctccagcat gagaacagca tcctcaaggg agcccagatg 2940
aaggcatcct tggcatccct gccccctctg catgttgcaa agctatccca tgagggccct 3000
ggcagtgagt taccagctgg agcgctgtat cgtaagacca gccagctgct ggagacattg 3060
aatcaattga gcacacacac gcacgtagta gacatcactc gcaccagccc tgctgccaag 3120
agcccgtcgg cccaacttat ggagcaagtg gctcagctta agtccctgag tgacaccgtc 3180
gagaagctca aggatgaggt cctcaaggag acagtatctc agcgccctgg agccacagta 3240
cccactgact ttgccacctt cccttcatca gccttcctca gggaggatcc aaagtgggaa 3300
ttccctcgga agaacttggt tcttggaaaa actctaggag aaggcgaatt tggaaaagtg 3360
gtcaaggcaa cggccttcca tctgaaaggc agagcagggt acaccacggt ggccgtgaag 3420
atgctgaaag agaacgcctc cccgagtgag cttcgagacc tgctgtcaga gttcaacgtc 3480
ctgaagcagg tcaaccaccc acatgtcatc aaattgtatg gggcctgcag ccaggatggc 3540
ccgctcctcc tcatcgtgga gtacgccaaa tacggctccc tgcggggctt cctccgcgag 3600
agccgcaaag tggggcctgg ctacctgggc agtggaggca gccgcaactc cagctccctg 3660
gaccacccgg atgagcgggc cctcaccatg ggcgacctca tctcatttgc ctggcagatc 3720
tcacagggga tgcagtatct ggccgagatg aagctcgttc atcgggactt ggcagccaga 3780
aacatcctgg tagctgaggg gcggaagatg aagatttcgg atttcggctt gtcccgagat 3840
gtttatgaag aggattccta cgtgaagagg agccagggtc ggattccagt taaatggatg 3900
gcaattgaat ccctttttga tcatatctac accacgcaaa gtgatgtatg gtcttttggt 3960
gtcctgctgt gggagatcgt gaccctaggg ggaaacccct atcctgggat tcctcctgag 4020
cggctcttca accttctgaa gaccggccac cggatggaga ggccagacaa ctgcagcgag 4080
gagatgtacc gcctgatgct gcaatgctgg aagcaggagc cggacaaaag gccggtgttt 4140
gcggacatca gcaaagacct ggagaagatg atggttaaga ggagagacta cttggacctt 4200
gcggcgtcca ctccatctga ctccctgatt tatgacgacg gcctctcaga ggaggagaca 4260
ccgctggtgg actgtaataa tgcccccctc cctcgagccc tcccttccac atggattgaa 4320
aacaaactct atggtagaat ttcccatgca tttactagat tctag 4365
<210> 16
<211> 1454
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 16
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
930 935 940
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser Ala
945 950 955 960
Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile Leu Lys
965 970 975
Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val
980 985 990
Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala
995 1000 1005
Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu
1010 1015 1020
Ser Thr His Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala
1025 1030 1035
Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu
1040 1045 1050
Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu
1055 1060 1065
Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp
1070 1075 1080
Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg Glu Asp Pro Lys
1085 1090 1095
Trp Glu Phe Pro Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly
1100 1105 1110
Glu Gly Glu Phe Gly Lys Val Val Lys Ala Thr Ala Phe His Leu
1115 1120 1125
Lys Gly Arg Ala Gly Tyr Thr Thr Val Ala Val Lys Met Leu Lys
1130 1135 1140
Glu Asn Ala Ser Pro Ser Glu Leu Arg Asp Leu Leu Ser Glu Phe
1145 1150 1155
Asn Val Leu Lys Gln Val Asn His Pro His Val Ile Lys Leu Tyr
1160 1165 1170
Gly Ala Cys Ser Gln Asp Gly Pro Leu Leu Leu Ile Val Glu Tyr
1175 1180 1185
Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu Arg Glu Ser Arg Lys
1190 1195 1200
Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser Arg Asn Ser Ser
1205 1210 1215
Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr Met Gly Asp Leu
1220 1225 1230
Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr Leu Ala
1235 1240 1245
Glu Met Lys Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile Leu
1250 1255 1260
Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser
1265 1270 1275
Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly
1280 1285 1290
Arg Ile Pro Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His
1295 1300 1305
Ile Tyr Thr Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu
1310 1315 1320
Trp Glu Ile Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro
1325 1330 1335
Pro Glu Arg Leu Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu
1340 1345 1350
Arg Pro Asp Asn Cys Ser Glu Glu Met Tyr Arg Leu Met Leu Gln
1355 1360 1365
Cys Trp Lys Gln Glu Pro Asp Lys Arg Pro Val Phe Ala Asp Ile
1370 1375 1380
Ser Lys Asp Leu Glu Lys Met Met Val Lys Arg Arg Asp Tyr Leu
1385 1390 1395
Asp Leu Ala Ala Ser Thr Pro Ser Asp Ser Leu Ile Tyr Asp Asp
1400 1405 1410
Gly Leu Ser Glu Glu Glu Thr Pro Leu Val Asp Cys Asn Asn Ala
1415 1420 1425
Pro Leu Pro Arg Ala Leu Pro Ser Thr Trp Ile Glu Asn Lys Leu
1430 1435 1440
Tyr Gly Arg Ile Ser His Ala Phe Thr Arg Phe
1445 1450
<210> 17
<211> 4782
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 17
atgagtgcgg aggcaagcgc ccggcctctg cgggtgggct cccgtgtaga ggtgattgga 60
aaaggccacc gaggcactgt ggcctatgtt ggagccacac tgtttgccac tggcaaatgg 120
gtaggcgtga ttctggatga agcaaagggc aaaaatgatg gaactgttca aggcaggaag 180
tacttcactt gtgatgaagg gcatggcatc tttgtgcgcc agtcccagat ccaggtattt 240
gaagatggag cagatactac ttccccagag acacctgatt cttctgcttc aaaagtcctc 300
aaaagagagg gaactgatac aactgcaaag actagcaaac tgcccacgcg cccagccagt 360
actggggtgg ctggggccag tagctccctg ggcccctctg gctcagcgtc agcaggtgag 420
ctgagcagca gtgagcccag caccccggct cagactccgc tggcagcacc catcatcccc 480
acgccggtcc tcacctctcc tggagcagtc cccccgcttc cttccccatc caaggaggag 540
gagggactaa gggctcaggt gcgggacctg gaggagaaac tagagaccct gagactgaaa 600
cgggcagaag acaaagcaaa gctaaaagag ctggagaaac acaaaatcca gctggagcag 660
gtgcaggaat ggaagagcaa aatgcaggag cagcaggccg acctgcagcg gcgcctcaag 720
gaggcgagaa aggaagccaa ggaggcgctg gaggcaaagg aacgctatat ggaggagatg 780
gctgatactg ctgatgccat tgagatggcc actttggaca aggagatggc tgaagagcgg 840
gctgagtccc tgcagcagga ggtggaggca ctgaaggagc gggtggacga gctcactact 900
gacttagaga tcctcaaggc tgagattgaa gagaagggct cagatggcgc tgcatccagt 960
tatcagctca agcagcttga ggagcagaat gcccgcctga aggatgccct ggtgaggatg 1020
cgggatcttt cttcctcaga gaagcaggag catgtgaagc tccagaagct catggaaaag 1080
aagaaccaag agctggaagt tgtgaggcaa cagcgggagc gtctgcagga ggagctaagc 1140
caggcagaga gcaccattga tgagctcaag gagcaggtgg atgctgctct gggtgctgag 1200
gagatggtgg agatgctgac agatcggaac ctgaatctgg aagagaaagt gcgcgagttg 1260
agggagactg tgggagactt ggaagcgatg aatgagatga acgatgagct gcaggagaat 1320
gcacgtgaga cagaactgga gctgcgggag cagctggaca tggcaggcgc gcgggttcgt 1380
gaggcccaga agcgtgtgga ggcagcccag gagacggttg cagactacca gcagaccatc 1440
aagaagtacc gccagctgac cgcccatcta caggatgtga atcgggaact gacaaaccag 1500
caggaagcat ctgtggagag gcaacagcag ccacctccag agacctttga cttcaaaatc 1560
aagtttgctg agactaaggc ccatgccaag gcaattgaga tggaattgag gcagatggag 1620
gtggcccagg ccaatcgaca catgtccctg ctgacagcct tcatgcctga cagcttcctt 1680
cggccaggtg gggaccatga ctgcgttctg gtgctgttgc tcatgcctcg tctcatttgc 1740
aaggcagagc tgatccggaa gcaggcccag gagaagtttg aactaagtga gaactgttca 1800
gagcggcctg ggctgcgagg agctgctggg gagcaactca gctttgctgc tggactggtg 1860
tactcgctga gcctgctgca ggccacgcta caccgctatg agcatgccct ctctcagtgc 1920
agtgtggatg tgtataagaa agtgggcagc ctgtaccctg agatgagtgc ccatgagcgc 1980
tccttggatt tcctcattga actgctgcac aaggatcagc tggatgagac tgtcaatgtg 2040
gagcctctca ccaaggccat caagtactat cagcatctgt acagcatcca ccttgccgaa 2100
cagcctgagg actgtactat gcagctggct gaccacatta agttcacgca gagtgctctg 2160
gactgcatga gtgtggaggt aggacggctg cgtgccttct tgcagggtgg gcaggaggct 2220
acagatattg ccctcctgct ccgggatctg gaaacttcat gcagtgacat ccgccagttc 2280
tgcaagaaga tccgaaggcg aatgccaggg acagatgctc ctgggatccc agctgcactg 2340
gcctttggac cacaggtatc tgacacgctc ctagactgca ggaaacactt gacgtgggtc 2400
gtggctgtgc tgcaggaggt ggcagctgct gctgcccagc tcattgcccc actggcagag 2460
aatgaggggc tacttgtggc tgctctggag gaactggctt tcaaagcaag cgagcagatc 2520
tatgggaccc cctccagcag cccctatgag tgtctgcgcc agtcatgcaa catcctcatc 2580
agtaccatga acaagctggc cacagccatg caggaggggg agtatgatgc agagcggccc 2640
cccagcaagc ctccaccggt tgaactgcgg gctgctgccc ttcgtgcaga gatcacagat 2700
gctgaaggcc tgggtttgaa gctcgaagat cgagagacag ttattaagga gttgaagaag 2760
tcactcaaga ttaagggaga ggagctaagt gaggccaatg tgcggctgag cctcctggag 2820
aagaagttgg acagtgctgc caaggatgca gatgagcgca tcgagaaagt ccagactcgg 2880
ctggaggaga cccaggcact gctgcgaaag aaggagaaag agtttgagga gacaatggat 2940
gcactccagg ctgacatcga ccagctggag gcagagaagg cagaactaaa gcagcgtctg 3000
aacagccagt ccaaacgcac gattgaggga ctccggggcc ctcctccttc aggcattgct 3060
actctggtct ctggcattgc tggtggagcc atccctgggc aggctccagg gtctgtgcca 3120
ggcccagggc tggtgaagga ctcaccactg ctgcttcagc agatctctgc catgaggctg 3180
cacatctccc agctccagca tgagaacagc atcctcaagg gagcccagat gaaggcatcc 3240
ttggcatccc tgccccctct gcatgttgca aagctatccc atgagggccc tggcagtgag 3300
ttaccagctg gagcgctgta tcgtaagacc agccagctgc tggagacatt gaatcaattg 3360
agcacacaca cgcacgtagt agacatcact cgcaccagcc ctgctgccaa gagcccgtcg 3420
gcccaactta tggagcaagt ggctcagctt aagtccctga gtgacaccgt cgagaagctc 3480
aaggatgagg tcctcaagga gacagtatct cagcgccctg gagccacagt acccactgac 3540
tttgccacct tcccttcatc agccttcctc agggaggatc caaagtggga attccctcgg 3600
aagaacttgg ttcttggaaa aactctagga gaaggcgaat ttggaaaagt ggtcaaggca 3660
acggccttcc atctgaaagg cagagcaggg tacaccacgg tggccgtgaa gatgctgaaa 3720
gagaacgcct ccccgagtga gcttcgagac ctgctgtcag agttcaacgt cctgaagcag 3780
gtcaaccacc cacatgtcat caaattgtat ggggcctgca gccaggatgg cccgctcctc 3840
ctcatcgtgg agtacgccaa atacggctcc ctgcggggct tcctccgcga gagccgcaaa 3900
gtggggcctg gctacctggg cagtggaggc agccgcaact ccagctccct ggaccacccg 3960
gatgagcggg ccctcaccat gggcgacctc atctcatttg cctggcagat ctcacagggg 4020
atgcagtatc tggccgagat gaagctcgtt catcgggact tggcagccag aaacatcctg 4080
gtagctgagg ggcggaagat gaagatttcg gatttcggct tgtcccgaga tgtttatgaa 4140
gaggattcct acgtgaagag gagccagggt cggattccag ttaaatggat ggcaattgaa 4200
tccctttttg atcatatcta caccacgcaa agtgatgtat ggtcttttgg tgtcctgctg 4260
tgggagatcg tgaccctagg gggaaacccc tatcctggga ttcctcctga gcggctcttc 4320
aaccttctga agaccggcca ccggatggag aggccagaca actgcagcga ggagatgtac 4380
cgcctgatgc tgcaatgctg gaagcaggag ccggacaaaa ggccggtgtt tgcggacatc 4440
agcaaagacc tggagaagat gatggttaag aggagagact acttggacct tgcggcgtcc 4500
actccatctg actccctgat ttatgacgac ggcctctcag aggaggagac accgctggtg 4560
gactgtaata atgcccccct ccctcgagcc ctcccttcca catggattga aaacaaactc 4620
tatggcatgt cagacccgaa ctggcctgga gagagtcctg taccactcac gagagctgat 4680
ggcactaaca ctgggtttcc aagatatcca aatgatagtg tatatgctaa ctggatgctt 4740
tcaccctcag cggcaaaatt aatggacacg tttgatagtt aa 4782
<210> 18
<211> 1593
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 18
Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg Val
1. 5 10 15
Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly Ala
20 25 30
Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu Ala
35 40 45
Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr Cys
50 55 60
Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val Phe
65 70 75 80
Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser Ala
85 90 95
Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr Ser
100 105 110
Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser
115 120 125
Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser
130 135 140
Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro
145 150 155 160
Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro
165 170 175
Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu
180 185 190
Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu
195 200 205
Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp
210 215 220
Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys
225 230 235 240
Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr
245 250 255
Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu
260 265 270
Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val
275 280 285
Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile
290 295 300
Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser
305 310 315 320
Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala
325 330 335
Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val
340 345 350
Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val
355 360 365
Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser
370 375 380
Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu
385 390 395 400
Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys
405 410 415
Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu
420 425 430
Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu
435 440 445
Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys
450 455 460
Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile
465 470 475 480
Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu
485 490 495
Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro
500 505 510
Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His
515 520 525
Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala
530 535 540
Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu
545 550 555 560
Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro
565 570 575
Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys
580 585 590
Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala
595 600 605
Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser
610 615 620
Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys
625 630 635 640
Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser
645 650 655
Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp
660 665 670
Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys
675 680 685
Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp
690 695 700
Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu
705 710 715 720
Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly
725 730 735
Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr
740 745 750
Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met
755 760 765
Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro
770 775 780
Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val
785 790 795 800
Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala
805 810 815
Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu
820 825 830
Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro
835 840 845
Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn
850 855 860
Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro
865 870 875 880
Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala
885 890 895
Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu
900 905 910
Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu
915 920 925
Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp
930 935 940
Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg
945 950 955 960
Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu
965 970 975
Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu
980 985 990
Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
995 1000 1005
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1010 1015 1020
Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser
1025 1030 1035
Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln
1040 1045 1050
Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
1055 1060 1065
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser
1070 1075 1080
Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly
1085 1090 1095
Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu
1100 1105 1110
Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp
1115 1120 1125
Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu
1130 1135 1140
Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu
1145 1150 1155
Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro
1160 1165 1170
Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala
1175 1180 1185
Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu
1190 1195 1200
Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val
1205 1210 1215
Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr
1220 1225 1230
Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu
1235 1240 1245
Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His
1250 1255 1260
Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro
1265 1270 1275
Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly
1280 1285 1290
Phe Leu Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser
1295 1300 1305
Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg
1310 1315 1320
Ala Leu Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser
1325 1330 1335
Gln Gly Met Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp
1340 1345 1350
Leu Ala Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys
1355 1360 1365
Ile Ser Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser
1370 1375 1380
Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala
1385 1390 1395
Ile Glu Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val
1400 1405 1410
Trp Ser Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly
1415 1420 1425
Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu
1430 1435 1440
Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu
1445 1450 1455
Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys
1460 1465 1470
Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met
1475 1480 1485
Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser
1490 1495 1500
Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro
1505 1510 1515
Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser
1520 1525 1530
Thr Trp Ile Glu Asn Lys Leu Tyr Gly Met Ser Asp Pro Asn Trp
1535 1540 1545
Pro Gly Glu Ser Pro Val Pro Leu Thr Arg Ala Asp Gly Thr Asn
1550 1555 1560
Thr Gly Phe Pro Arg Tyr Pro Asn Asp Ser Val Tyr Ala Asn Trp
1565 1570 1575
Met Leu Ser Pro Ser Ala Ala Lys Leu Met Asp Thr Phe Asp Ser
1580 1585 1590
<210> 19
<211> 4656
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 19
atgagtgcgg aggcaagcgc ccggcctctg cgggtgggct cccgtgtaga ggtgattgga 60
aaaggccacc gaggcactgt ggcctatgtt ggagccacac tgtttgccac tggcaaatgg 120
gtaggcgtga ttctggatga agcaaagggc aaaaatgatg gaactgttca aggcaggaag 180
tacttcactt gtgatgaagg gcatggcatc tttgtgcgcc agtcccagat ccaggtattt 240
gaagatggag cagatactac ttccccagag acacctgatt cttctgcttc aaaagtcctc 300
aaaagagagg gaactgatac aactgcaaag actagcaaac tgcccacgcg cccagccagt 360
actggggtgg ctggggccag tagctccctg ggcccctctg gctcagcgtc agcaggtgag 420
ctgagcagca gtgagcccag caccccggct cagactccgc tggcagcacc catcatcccc 480
acgccggtcc tcacctctcc tggagcagtc cccccgcttc cttccccatc caaggaggag 540
gagggactaa gggctcaggt gcgggacctg gaggagaaac tagagaccct gagactgaaa 600
cgggcagaag acaaagcaaa gctaaaagag ctggagaaac acaaaatcca gctggagcag 660
gtgcaggaat ggaagagcaa aatgcaggag cagcaggccg acctgcagcg gcgcctcaag 720
gaggcgagaa aggaagccaa ggaggcgctg gaggcaaagg aacgctatat ggaggagatg 780
gctgatactg ctgatgccat tgagatggcc actttggaca aggagatggc tgaagagcgg 840
gctgagtccc tgcagcagga ggtggaggca ctgaaggagc gggtggacga gctcactact 900
gacttagaga tcctcaaggc tgagattgaa gagaagggct cagatggcgc tgcatccagt 960
tatcagctca agcagcttga ggagcagaat gcccgcctga aggatgccct ggtgaggatg 1020
cgggatcttt cttcctcaga gaagcaggag catgtgaagc tccagaagct catggaaaag 1080
aagaaccaag agctggaagt tgtgaggcaa cagcgggagc gtctgcagga ggagctaagc 1140
caggcagaga gcaccattga tgagctcaag gagcaggtgg atgctgctct gggtgctgag 1200
gagatggtgg agatgctgac agatcggaac ctgaatctgg aagagaaagt gcgcgagttg 1260
agggagactg tgggagactt ggaagcgatg aatgagatga acgatgagct gcaggagaat 1320
gcacgtgaga cagaactgga gctgcgggag cagctggaca tggcaggcgc gcgggttcgt 1380
gaggcccaga agcgtgtgga ggcagcccag gagacggttg cagactacca gcagaccatc 1440
aagaagtacc gccagctgac cgcccatcta caggatgtga atcgggaact gacaaaccag 1500
caggaagcat ctgtggagag gcaacagcag ccacctccag agacctttga cttcaaaatc 1560
aagtttgctg agactaaggc ccatgccaag gcaattgaga tggaattgag gcagatggag 1620
gtggcccagg ccaatcgaca catgtccctg ctgacagcct tcatgcctga cagcttcctt 1680
cggccaggtg gggaccatga ctgcgttctg gtgctgttgc tcatgcctcg tctcatttgc 1740
aaggcagagc tgatccggaa gcaggcccag gagaagtttg aactaagtga gaactgttca 1800
gagcggcctg ggctgcgagg agctgctggg gagcaactca gctttgctgc tggactggtg 1860
tactcgctga gcctgctgca ggccacgcta caccgctatg agcatgccct ctctcagtgc 1920
agtgtggatg tgtataagaa agtgggcagc ctgtaccctg agatgagtgc ccatgagcgc 1980
tccttggatt tcctcattga actgctgcac aaggatcagc tggatgagac tgtcaatgtg 2040
gagcctctca ccaaggccat caagtactat cagcatctgt acagcatcca ccttgccgaa 2100
cagcctgagg actgtactat gcagctggct gaccacatta agttcacgca gagtgctctg 2160
gactgcatga gtgtggaggt aggacggctg cgtgccttct tgcagggtgg gcaggaggct 2220
acagatattg ccctcctgct ccgggatctg gaaacttcat gcagtgacat ccgccagttc 2280
tgcaagaaga tccgaaggcg aatgccaggg acagatgctc ctgggatccc agctgcactg 2340
gcctttggac cacaggtatc tgacacgctc ctagactgca ggaaacactt gacgtgggtc 2400
gtggctgtgc tgcaggaggt ggcagctgct gctgcccagc tcattgcccc actggcagag 2460
aatgaggggc tacttgtggc tgctctggag gaactggctt tcaaagcaag cgagcagatc 2520
tatgggaccc cctccagcag cccctatgag tgtctgcgcc agtcatgcaa catcctcatc 2580
agtaccatga acaagctggc cacagccatg caggaggggg agtatgatgc agagcggccc 2640
cccagcaagc ctccaccggt tgaactgcgg gctgctgccc ttcgtgcaga gatcacagat 2700
gctgaaggcc tgggtttgaa gctcgaagat cgagagacag ttattaagga gttgaagaag 2760
tcactcaaga ttaagggaga ggagctaagt gaggccaatg tgcggctgag cctcctggag 2820
aagaagttgg acagtgctgc caaggatgca gatgagcgca tcgagaaagt ccagactcgg 2880
ctggaggaga cccaggcact gctgcgaaag aaggagaaag agtttgagga gacaatggat 2940
gcactccagg ctgacatcga ccagctggag gcagagaagg cagaactaaa gcagcgtctg 3000
aacagccagt ccaaacgcac gattgaggga ctccggggcc ctcctccttc aggcattgct 3060
actctggtct ctggcattgc tggtggagcc atccctgggc aggctccagg gtctgtgcca 3120
ggcccagggc tggtgaagga ctcaccactg ctgcttcagc agatctctgc catgaggctg 3180
cacatctccc agctccagca tgagaacagc atcctcaagg gagcccagat gaaggcatcc 3240
ttggcatccc tgccccctct gcatgttgca aagctatccc atgagggccc tggcagtgag 3300
ttaccagctg gagcgctgta tcgtaagacc agccagctgc tggagacatt gaatcaattg 3360
agcacacaca cgcacgtagt agacatcact cgcaccagcc ctgctgccaa gagcccgtcg 3420
gcccaactta tggagcaagt ggctcagctt aagtccctga gtgacaccgt cgagaagctc 3480
aaggatgagg tcctcaagga gacagtatct cagcgccctg gagccacagt acccactgac 3540
tttgccacct tcccttcatc agccttcctc agggaggatc caaagtggga attccctcgg 3600
aagaacttgg ttcttggaaa aactctagga gaaggcgaat ttggaaaagt ggtcaaggca 3660
acggccttcc atctgaaagg cagagcaggg tacaccacgg tggccgtgaa gatgctgaaa 3720
gagaacgcct ccccgagtga gcttcgagac ctgctgtcag agttcaacgt cctgaagcag 3780
gtcaaccacc cacatgtcat caaattgtat ggggcctgca gccaggatgg cccgctcctc 3840
ctcatcgtgg agtacgccaa atacggctcc ctgcggggct tcctccgcga gagccgcaaa 3900
gtggggcctg gctacctggg cagtggaggc agccgcaact ccagctccct ggaccacccg 3960
gatgagcggg ccctcaccat gggcgacctc atctcatttg cctggcagat ctcacagggg 4020
atgcagtatc tggccgagat gaagctcgtt catcgggact tggcagccag aaacatcctg 4080
gtagctgagg ggcggaagat gaagatttcg gatttcggct tgtcccgaga tgtttatgaa 4140
gaggattcct acgtgaagag gagccagggt cggattccag ttaaatggat ggcaattgaa 4200
tccctttttg atcatatcta caccacgcaa agtgatgtat ggtcttttgg tgtcctgctg 4260
tgggagatcg tgaccctagg gggaaacccc tatcctggga ttcctcctga gcggctcttc 4320
aaccttctga agaccggcca ccggatggag aggccagaca actgcagcga ggagatgtac 4380
cgcctgatgc tgcaatgctg gaagcaggag ccggacaaaa ggccggtgtt tgcggacatc 4440
agcaaagacc tggagaagat gatggttaag aggagagact acttggacct tgcggcgtcc 4500
actccatctg actccctgat ttatgacgac ggcctctcag aggaggagac accgctggtg 4560
gactgtaata atgcccccct ccctcgagcc ctcccttcca catggattga aaacaaactc 4620
tatggtagaa tttcccatgc atttactaga ttctag 4656
<210> 20
<211> 1551
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 20
Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg Val
1. 5 10 15
Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly Ala
20 25 30
Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu Ala
35 40 45
Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr Cys
50 55 60
Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val Phe
65 70 75 80
Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser Ala
85 90 95
Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr Ser
100 105 110
Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser
115 120 125
Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser
130 135 140
Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro
145 150 155 160
Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro
165 170 175
Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu
180 185 190
Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu
195 200 205
Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp
210 215 220
Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys
225 230 235 240
Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr
245 250 255
Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu
260 265 270
Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val
275 280 285
Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile
290 295 300
Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser
305 310 315 320
Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala
325 330 335
Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val
340 345 350
Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val
355 360 365
Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser
370 375 380
Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu
385 390 395 400
Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys
405 410 415
Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu
420 425 430
Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu
435 440 445
Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys
450 455 460
Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile
465 470 475 480
Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu
485 490 495
Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro
500 505 510
Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His
515 520 525
Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala
530 535 540
Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu
545 550 555 560
Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro
565 570 575
Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys
580 585 590
Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala
595 600 605
Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser
610 615 620
Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys
625 630 635 640
Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser
645 650 655
Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp
660 665 670
Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys
675 680 685
Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp
690 695 700
Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu
705 710 715 720
Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly
725 730 735
Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr
740 745 750
Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met
755 760 765
Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro
770 775 780
Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val
785 790 795 800
Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala
805 810 815
Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu
820 825 830
Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro
835 840 845
Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn
850 855 860
Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro
865 870 875 880
Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala
885 890 895
Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu
900 905 910
Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu
915 920 925
Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp
930 935 940
Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg
945 950 955 960
Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu
965 970 975
Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu
980 985 990
Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
995 1000 1005
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1010 1015 1020
Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser
1025 1030 1035
Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln
1040 1045 1050
Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
1055 1060 1065
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser
1070 1075 1080
Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly
1085 1090 1095
Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu
1100 1105 1110
Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp
1115 1120 1125
Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu
1130 1135 1140
Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu
1145 1150 1155
Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro
1160 1165 1170
Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala
1175 1180 1185
Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe Pro Arg Lys Asn Leu
1190 1195 1200
Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly Lys Val Val
1205 1210 1215
Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr Thr Thr
1220 1225 1230
Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu Leu
1235 1240 1245
Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His
1250 1255 1260
Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro
1265 1270 1275
Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly
1280 1285 1290
Phe Leu Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser
1295 1300 1305
Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg
1310 1315 1320
Ala Leu Thr Met Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser
1325 1330 1335
Gln Gly Met Gln Tyr Leu Ala Glu Met Lys Leu Val His Arg Asp
1340 1345 1350
Leu Ala Ala Arg Asn Ile Leu Val Ala Glu Gly Arg Lys Met Lys
1355 1360 1365
Ile Ser Asp Phe Gly Leu Ser Arg Asp Val Tyr Glu Glu Asp Ser
1370 1375 1380
Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro Val Lys Trp Met Ala
1385 1390 1395
Ile Glu Ser Leu Phe Asp His Ile Tyr Thr Thr Gln Ser Asp Val
1400 1405 1410
Trp Ser Phe Gly Val Leu Leu Trp Glu Ile Val Thr Leu Gly Gly
1415 1420 1425
Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu Phe Asn Leu Leu
1430 1435 1440
Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys Ser Glu Glu
1445 1450 1455
Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro Asp Lys
1460 1465 1470
Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met Met
1475 1480 1485
Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro Ser
1490 1495 1500
Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr Pro
1505 1510 1515
Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro Ser
1520 1525 1530
Thr Trp Ile Glu Asn Lys Leu Tyr Gly Arg Ile Ser His Ala Phe
1535 1540 1545
Thr Arg Phe
1550
<210> 21
<211> 4887
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 21
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctggcaccga cagcccgaaa gaccacaact 420
cggcgaccca agcccacgcg cccagccagt actggggtgg ctggggccag tagctccctg 480
ggcccctctg gctcagcgtc agcaggtgag ctgagcagca gtgagcccag caccccggct 540
cagactccgc tggcagcacc catcatcccc acgccggtcc tcacctctcc tggagcagtc 600
cccccgcttc cttccccatc caaggaggag gagggactaa gggctcaggt gcgggacctg 660
gaggagaaac tagagaccct gagactgaaa cgggcagaag acaaagcaaa gctaaaagag 720
ctggagaaac acaaaatcca gctggagcag gtgcaggaat ggaagagcaa aatgcaggag 780
cagcaggccg acctgcagcg gcgcctcaag gaggcgagaa aggaagccaa ggaggcgctg 840
gaggcaaagg aacgctatat ggaggagatg gctgatactg ctgatgccat tgagatggcc 900
actttggaca aggagatggc tgaagagcgg gctgagtccc tgcagcagga ggtggaggca 960
ctgaaggagc gggtggacga gctcactact gacttagaga tcctcaaggc tgagattgaa 1020
gagaagggct cagatggcgc tgcatccagt tatcagctca agcagcttga ggagcagaat 1080
gcccgcctga aggatgccct ggtgaggatg cgggatcttt cttcctcaga gaagcaggag 1140
catgtgaagc tccagaagct catggaaaag aagaaccaag agctggaagt tgtgaggcaa 1200
cagcgggagc gtctgcagga ggagctaagc caggcagaga gcaccattga tgagctcaag 1260
gagcaggtgg atgctgctct gggtgctgag gagatggtgg agatgctgac agatcggaac 1320
ctgaatctgg aagagaaagt gcgcgagttg agggagactg tgggagactt ggaagcgatg 1380
aatgagatga acgatgagct gcaggagaat gcacgtgaga cagaactgga gctgcgggag 1440
cagctggaca tggcaggcgc gcgggttcgt gaggcccaga agcgtgtgga ggcagcccag 1500
gagacggttg cagactacca gcagaccatc aagaagtacc gccagctgac cgcccatcta 1560
caggatgtga atcgggaact gacaaaccag caggaagcat ctgtggagag gcaacagcag 1620
ccacctccag agacctttga cttcaaaatc aagtttgctg agactaaggc ccatgccaag 1680
gcaattgaga tggaattgag gcagatggag gtggcccagg ccaatcgaca catgtccctg 1740
ctgacagcct tcatgcctga cagcttcctt cggccaggtg gggaccatga ctgcgttctg 1800
gtgctgttgc tcatgcctcg tctcatttgc aaggcagagc tgatccggaa gcaggcccag 1860
gagaagtttg aactaagtga gaactgttca gagcggcctg ggctgcgagg agctgctggg 1920
gagcaactca gctttgctgc tggactggtg tactcgctga gcctgctgca ggccacgcta 1980
caccgctatg agcatgccct ctctcagtgc agtgtggatg tgtataagaa agtgggcagc 2040
ctgtaccctg agatgagtgc ccatgagcgc tccttggatt tcctcattga actgctgcac 2100
aaggatcagc tggatgagac tgtcaatgtg gagcctctca ccaaggccat caagtactat 2160
cagcatctgt acagcatcca ccttgccgaa cagcctgagg actgtactat gcagctggct 2220
gaccacatta agttcacgca gagtgctctg gactgcatga gtgtggaggt aggacggctg 2280
cgtgccttct tgcagggtgg gcaggaggct acagatattg ccctcctgct ccgggatctg 2340
gaaacttcat gcagtgacat ccgccagttc tgcaagaaga tccgaaggcg aatgccaggg 2400
acagatgctc ctgggatccc agctgcactg gcctttggac cacaggtatc tgacacgctc 2460
ctagactgca ggaaacactt gacgtgggtc gtggctgtgc tgcaggaggt ggcagctgct 2520
gctgcccagc tcattgcccc actggcagag aatgaggggc tacttgtggc tgctctggag 2580
gaactggctt tcaaagcaag cgagcagatc tatgggaccc cctccagcag cccctatgag 2640
tgtctgcgcc agtcatgcaa catcctcatc agtaccatga acaagctggc cacagccatg 2700
caggaggggg agtatgatgc agagcggccc cccagcaagc ctccaccggt tgaactgcgg 2760
gctgctgccc ttcgtgcaga gatcacagat gctgaaggcc tgggtttgaa gctcgaagat 2820
cgagagacag ttattaagga gttgaagaag tcactcaaga ttaagggaga ggagctaagt 2880
gaggccaatg tgcggctgag cctcctggag aagaagttgg acagtgctgc caaggatgca 2940
gatgagcgca tcgagaaagt ccagactcgg ctggaggaga cccaggcact gctgcgaaag 3000
aaggagaaag agtttgagga gacaatggat gcactccagg ctgacatcga ccagctggag 3060
gcagagaagg cagaactaaa gcagcgtctg aacagccagt ccaaacgcac gattgaggga 3120
ctccggggcc ctcctccttc aggcattgct actctggtct ctggcattgc tggtgaagaa 3180
cagcagcgag gagccatccc tgggcaggct ccagggtctg tgccaggccc agggctggtg 3240
aaggactcac cactgctgct tcagcagatc tctgccatga ggctgcacat ctcccagctc 3300
cagcatgaga acagcatcct caagggagcc cagatgaagg catccttggc atccctgccc 3360
cctctgcatg ttgcaaagct atcccatgag ggccctggca gtgagttacc agctggagcg 3420
ctgtatcgta agaccagcca gctgctggag acattgaatc aattgagcac acacacgcac 3480
gtagtagaca tcactcgcac cagccctgct gccaagagcc cgtcggccca acttatggag 3540
caagtggctc agcttaagtc cctgagtgac accgtcgaga agctcaagga tgaggtcctc 3600
aaggagacag tatctcagcg ccctggagcc acagtaccca ctgactttgc caccttccct 3660
tcatcagcct tcctcaggga ggatccaaag tgggaattcc ctcggaagaa cttggttctt 3720
ggaaaaactc taggagaagg cgaatttgga aaagtggtca aggcaacggc cttccatctg 3780
aaaggcagag cagggtacac cacggtggcc gtgaagatgc tgaaagagaa cgcctccccg 3840
agtgagcttc gagacctgct gtcagagttc aacgtcctga agcaggtcaa ccacccacat 3900
gtcatcaaat tgtatggggc ctgcagccag gatggcccgc tcctcctcat cgtggagtac 3960
gccaaatacg gctccctgcg gggcttcctc cgcgagagcc gcaaagtggg gcctggctac 4020
ctgggcagtg gaggcagccg caactccagc tccctggacc acccggatga gcgggccctc 4080
accatgggcg acctcatctc atttgcctgg cagatctcac aggggatgca gtatctggcc 4140
gagatgaagc tcgttcatcg ggacttggca gccagaaaca tcctggtagc tgaggggcgg 4200
aagatgaaga tttcggattt cggcttgtcc cgagatgttt atgaagagga ttcctacgtg 4260
aagaggagcc agggtcggat tccagttaaa tggatggcaa ttgaatccct ttttgatcat 4320
atctacacca cgcaaagtga tgtatggtct tttggtgtcc tgctgtggga gatcgtgacc 4380
ctagggggaa acccctatcc tgggattcct cctgagcggc tcttcaacct tctgaagacc 4440
ggccaccgga tggagaggcc agacaactgc agcgaggaga tgtaccgcct gatgctgcaa 4500
tgctggaagc aggagccgga caaaaggccg gtgtttgcgg acatcagcaa agacctggag 4560
aagatgatgg ttaagaggag agactacttg gaccttgcgg cgtccactcc atctgactcc 4620
ctgatttatg acgacggcct ctcagaggag gagacaccgc tggtggactg taataatgcc 4680
cccctccctc gagccctccc ttccacatgg attgaaaaca aactctatgg catgtcagac 4740
ccgaactggc ctggagagag tcctgtacca ctcacgagag ctgatggcac taacactggg 4800
tttccaagat atccaaatga tagtgtatat gctaactgga tgctttcacc ctcagcggca 4860
aaattaatgg acacgtttga tagttaa 4887
<210> 22
<211> 1628
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 22
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro Lys
130 135 140
Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser Leu
145 150 155 160
Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu Pro
165 170 175
Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr Pro
180 185 190
Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser Lys
195 200 205
Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys Leu
210 215 220
Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys Glu
225 230 235 240
Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys Ser
245 250 255
Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu Ala
260 265 270
Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met Glu
275 280 285
Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp Lys
290 295 300
Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu Ala
305 310 315 320
Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu Lys
325 330 335
Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr Gln
340 345 350
Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu Val
355 360 365
Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys Leu
370 375 380
Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg Gln
385 390 395 400
Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr Ile
405 410 415
Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu Met
420 425 430
Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val Arg
435 440 445
Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met Asn
450 455 460
Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg Glu
465 470 475 480
Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg Val
485 490 495
Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys Lys
500 505 510
Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu Thr
515 520 525
Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro Glu
530 535 540
Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala Lys
545 550 555 560
Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn Arg
565 570 575
His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg Pro
580 585 590
Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg Leu
595 600 605
Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe Glu
610 615 620
Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala Gly
625 630 635 640
Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu Leu
645 650 655
Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser Val
660 665 670
Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala His
675 680 685
Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln Leu
690 695 700
Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr Tyr
705 710 715 720
Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys Thr
725 730 735
Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp Cys
740 745 750
Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly Gln
755 760 765
Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser Cys
770 775 780
Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro Gly
785 790 795 800
Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln Val
805 810 815
Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val Ala
820 825 830
Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro Leu
835 840 845
Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala Phe
850 855 860
Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr Glu
865 870 875 880
Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys Leu
885 890 895
Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro Ser
900 905 910
Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu Ile
915 920 925
Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr Val
930 935 940
Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu Ser
945 950 955 960
Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser Ala
965 970 975
Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu Glu
980 985 990
Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu Thr
995 1000 1005
Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
1010 1015 1020
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
1025 1030 1035
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1040 1045 1050
Ser Gly Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly
1055 1060 1065
Gln Ala Pro Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser
1070 1075 1080
Pro Leu Leu Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser
1085 1090 1095
Gln Leu Gln His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys
1100 1105 1110
Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser
1115 1120 1125
His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg
1130 1135 1140
Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His
1145 1150 1155
Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser
1160 1165 1170
Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu
1175 1180 1185
Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr
1190 1195 1200
Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr
1205 1210 1215
Phe Pro Ser Ser Ala Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe
1220 1225 1230
Pro Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu
1235 1240 1245
Phe Gly Lys Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg
1250 1255 1260
Ala Gly Tyr Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala
1265 1270 1275
Ser Pro Ser Glu Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu
1280 1285 1290
Lys Gln Val Asn His Pro His Val Ile Lys Leu Tyr Gly Ala Cys
1295 1300 1305
Ser Gln Asp Gly Pro Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr
1310 1315 1320
Gly Ser Leu Arg Gly Phe Leu Arg Glu Ser Arg Lys Val Gly Pro
1325 1330 1335
Gly Tyr Leu Gly Ser Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp
1340 1345 1350
His Pro Asp Glu Arg Ala Leu Thr Met Gly Asp Leu Ile Ser Phe
1355 1360 1365
Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr Leu Ala Glu Met Lys
1370 1375 1380
Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile Leu Val Ala Glu
1385 1390 1395
Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser Arg Asp Val
1400 1405 1410
Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro
1415 1420 1425
Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr Thr
1430 1435 1440
Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile
1445 1450 1455
Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg
1460 1465 1470
Leu Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp
1475 1480 1485
Asn Cys Ser Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys
1490 1495 1500
Gln Glu Pro Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp
1505 1510 1515
Leu Glu Lys Met Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala
1520 1525 1530
Ala Ser Thr Pro Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser
1535 1540 1545
Glu Glu Glu Thr Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro
1550 1555 1560
Arg Ala Leu Pro Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Met
1565 1570 1575
Ser Asp Pro Asn Trp Pro Gly Glu Ser Pro Val Pro Leu Thr Arg
1580 1585 1590
Ala Asp Gly Thr Asn Thr Gly Phe Pro Arg Tyr Pro Asn Asp Ser
1595 1600 1605
Val Tyr Ala Asn Trp Met Leu Ser Pro Ser Ala Ala Lys Leu Met
1610 1615 1620
Asp Thr Phe Asp Ser
1625
<210> 23
<211> 4761
<212> ДНК
<213> Искусственная последовательность
<220>
<223> кодирующая последовательность слитого пептида
<400> 23
atggcacaga gcaagaggca cgtgtacagc cggacgccca gcggcagcag gatgagtgcg 60
gaggcaagcg cccggcctct gcgggtgggc tcccgtgtag aggtgattgg aaaaggccac 120
cgaggcactg tggcctatgt tggagccaca ctgtttgcca ctggcaaatg ggtaggcgtg 180
attctggatg aagcaaaggg caaaaatgat ggaactgttc aaggcaggaa gtacttcact 240
tgtgatgaag ggcatggcat ctttgtgcgc cagtcccaga tccaggtatt tgaagatgga 300
gcagatacta cttccccaga gacacctgat tcttctgctt caaaagtcct caaaagagag 360
ggaactgata caactgcaaa gactagcaaa ctggcaccga cagcccgaaa gaccacaact 420
cggcgaccca agcccacgcg cccagccagt actggggtgg ctggggccag tagctccctg 480
ggcccctctg gctcagcgtc agcaggtgag ctgagcagca gtgagcccag caccccggct 540
cagactccgc tggcagcacc catcatcccc acgccggtcc tcacctctcc tggagcagtc 600
cccccgcttc cttccccatc caaggaggag gagggactaa gggctcaggt gcgggacctg 660
gaggagaaac tagagaccct gagactgaaa cgggcagaag acaaagcaaa gctaaaagag 720
ctggagaaac acaaaatcca gctggagcag gtgcaggaat ggaagagcaa aatgcaggag 780
cagcaggccg acctgcagcg gcgcctcaag gaggcgagaa aggaagccaa ggaggcgctg 840
gaggcaaagg aacgctatat ggaggagatg gctgatactg ctgatgccat tgagatggcc 900
actttggaca aggagatggc tgaagagcgg gctgagtccc tgcagcagga ggtggaggca 960
ctgaaggagc gggtggacga gctcactact gacttagaga tcctcaaggc tgagattgaa 1020
gagaagggct cagatggcgc tgcatccagt tatcagctca agcagcttga ggagcagaat 1080
gcccgcctga aggatgccct ggtgaggatg cgggatcttt cttcctcaga gaagcaggag 1140
catgtgaagc tccagaagct catggaaaag aagaaccaag agctggaagt tgtgaggcaa 1200
cagcgggagc gtctgcagga ggagctaagc caggcagaga gcaccattga tgagctcaag 1260
gagcaggtgg atgctgctct gggtgctgag gagatggtgg agatgctgac agatcggaac 1320
ctgaatctgg aagagaaagt gcgcgagttg agggagactg tgggagactt ggaagcgatg 1380
aatgagatga acgatgagct gcaggagaat gcacgtgaga cagaactgga gctgcgggag 1440
cagctggaca tggcaggcgc gcgggttcgt gaggcccaga agcgtgtgga ggcagcccag 1500
gagacggttg cagactacca gcagaccatc aagaagtacc gccagctgac cgcccatcta 1560
caggatgtga atcgggaact gacaaaccag caggaagcat ctgtggagag gcaacagcag 1620
ccacctccag agacctttga cttcaaaatc aagtttgctg agactaaggc ccatgccaag 1680
gcaattgaga tggaattgag gcagatggag gtggcccagg ccaatcgaca catgtccctg 1740
ctgacagcct tcatgcctga cagcttcctt cggccaggtg gggaccatga ctgcgttctg 1800
gtgctgttgc tcatgcctcg tctcatttgc aaggcagagc tgatccggaa gcaggcccag 1860
gagaagtttg aactaagtga gaactgttca gagcggcctg ggctgcgagg agctgctggg 1920
gagcaactca gctttgctgc tggactggtg tactcgctga gcctgctgca ggccacgcta 1980
caccgctatg agcatgccct ctctcagtgc agtgtggatg tgtataagaa agtgggcagc 2040
ctgtaccctg agatgagtgc ccatgagcgc tccttggatt tcctcattga actgctgcac 2100
aaggatcagc tggatgagac tgtcaatgtg gagcctctca ccaaggccat caagtactat 2160
cagcatctgt acagcatcca ccttgccgaa cagcctgagg actgtactat gcagctggct 2220
gaccacatta agttcacgca gagtgctctg gactgcatga gtgtggaggt aggacggctg 2280
cgtgccttct tgcagggtgg gcaggaggct acagatattg ccctcctgct ccgggatctg 2340
gaaacttcat gcagtgacat ccgccagttc tgcaagaaga tccgaaggcg aatgccaggg 2400
acagatgctc ctgggatccc agctgcactg gcctttggac cacaggtatc tgacacgctc 2460
ctagactgca ggaaacactt gacgtgggtc gtggctgtgc tgcaggaggt ggcagctgct 2520
gctgcccagc tcattgcccc actggcagag aatgaggggc tacttgtggc tgctctggag 2580
gaactggctt tcaaagcaag cgagcagatc tatgggaccc cctccagcag cccctatgag 2640
tgtctgcgcc agtcatgcaa catcctcatc agtaccatga acaagctggc cacagccatg 2700
caggaggggg agtatgatgc agagcggccc cccagcaagc ctccaccggt tgaactgcgg 2760
gctgctgccc ttcgtgcaga gatcacagat gctgaaggcc tgggtttgaa gctcgaagat 2820
cgagagacag ttattaagga gttgaagaag tcactcaaga ttaagggaga ggagctaagt 2880
gaggccaatg tgcggctgag cctcctggag aagaagttgg acagtgctgc caaggatgca 2940
gatgagcgca tcgagaaagt ccagactcgg ctggaggaga cccaggcact gctgcgaaag 3000
aaggagaaag agtttgagga gacaatggat gcactccagg ctgacatcga ccagctggag 3060
gcagagaagg cagaactaaa gcagcgtctg aacagccagt ccaaacgcac gattgaggga 3120
ctccggggcc ctcctccttc aggcattgct actctggtct ctggcattgc tggtgaagaa 3180
cagcagcgag gagccatccc tgggcaggct ccagggtctg tgccaggccc agggctggtg 3240
aaggactcac cactgctgct tcagcagatc tctgccatga ggctgcacat ctcccagctc 3300
cagcatgaga acagcatcct caagggagcc cagatgaagg catccttggc atccctgccc 3360
cctctgcatg ttgcaaagct atcccatgag ggccctggca gtgagttacc agctggagcg 3420
ctgtatcgta agaccagcca gctgctggag acattgaatc aattgagcac acacacgcac 3480
gtagtagaca tcactcgcac cagccctgct gccaagagcc cgtcggccca acttatggag 3540
caagtggctc agcttaagtc cctgagtgac accgtcgaga agctcaagga tgaggtcctc 3600
aaggagacag tatctcagcg ccctggagcc acagtaccca ctgactttgc caccttccct 3660
tcatcagcct tcctcaggga ggatccaaag tgggaattcc ctcggaagaa cttggttctt 3720
ggaaaaactc taggagaagg cgaatttgga aaagtggtca aggcaacggc cttccatctg 3780
aaaggcagag cagggtacac cacggtggcc gtgaagatgc tgaaagagaa cgcctccccg 3840
agtgagcttc gagacctgct gtcagagttc aacgtcctga agcaggtcaa ccacccacat 3900
gtcatcaaat tgtatggggc ctgcagccag gatggcccgc tcctcctcat cgtggagtac 3960
gccaaatacg gctccctgcg gggcttcctc cgcgagagcc gcaaagtggg gcctggctac 4020
ctgggcagtg gaggcagccg caactccagc tccctggacc acccggatga gcgggccctc 4080
accatgggcg acctcatctc atttgcctgg cagatctcac aggggatgca gtatctggcc 4140
gagatgaagc tcgttcatcg ggacttggca gccagaaaca tcctggtagc tgaggggcgg 4200
aagatgaaga tttcggattt cggcttgtcc cgagatgttt atgaagagga ttcctacgtg 4260
aagaggagcc agggtcggat tccagttaaa tggatggcaa ttgaatccct ttttgatcat 4320
atctacacca cgcaaagtga tgtatggtct tttggtgtcc tgctgtggga gatcgtgacc 4380
ctagggggaa acccctatcc tgggattcct cctgagcggc tcttcaacct tctgaagacc 4440
ggccaccgga tggagaggcc agacaactgc agcgaggaga tgtaccgcct gatgctgcaa 4500
tgctggaagc aggagccgga caaaaggccg gtgtttgcgg acatcagcaa agacctggag 4560
aagatgatgg ttaagaggag agactacttg gaccttgcgg cgtccactcc atctgactcc 4620
ctgatttatg acgacggcct ctcagaggag gagacaccgc tggtggactg taataatgcc 4680
cccctccctc gagccctccc ttccacatgg attgaaaaca aactctatgg tagaatttcc 4740
catgcattta ctagattcta g 4761
<210> 24
<211> 1586
<212> белок
<213> Искусственная последовательность
<220>
<223> слитый пептид
<400> 24
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro Lys
130 135 140
Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser Leu
145 150 155 160
Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu Pro
165 170 175
Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr Pro
180 185 190
Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser Lys
195 200 205
Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys Leu
210 215 220
Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys Glu
225 230 235 240
Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys Ser
245 250 255
Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu Ala
260 265 270
Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met Glu
275 280 285
Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp Lys
290 295 300
Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu Ala
305 310 315 320
Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu Lys
325 330 335
Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr Gln
340 345 350
Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu Val
355 360 365
Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys Leu
370 375 380
Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg Gln
385 390 395 400
Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr Ile
405 410 415
Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu Met
420 425 430
Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val Arg
435 440 445
Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met Asn
450 455 460
Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg Glu
465 470 475 480
Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg Val
485 490 495
Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys Lys
500 505 510
Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu Thr
515 520 525
Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro Glu
530 535 540
Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala Lys
545 550 555 560
Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn Arg
565 570 575
His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg Pro
580 585 590
Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg Leu
595 600 605
Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe Glu
610 615 620
Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala Gly
625 630 635 640
Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu Leu
645 650 655
Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser Val
660 665 670
Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala His
675 680 685
Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln Leu
690 695 700
Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr Tyr
705 710 715 720
Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys Thr
725 730 735
Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp Cys
740 745 750
Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly Gln
755 760 765
Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser Cys
770 775 780
Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro Gly
785 790 795 800
Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln Val
805 810 815
Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val Ala
820 825 830
Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro Leu
835 840 845
Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala Phe
850 855 860
Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr Glu
865 870 875 880
Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys Leu
885 890 895
Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro Ser
900 905 910
Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu Ile
915 920 925
Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr Val
930 935 940
Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu Ser
945 950 955 960
Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser Ala
965 970 975
Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu Glu
980 985 990
Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu Thr
995 1000 1005
Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
1010 1015 1020
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
1025 1030 1035
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1040 1045 1050
Ser Gly Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly
1055 1060 1065
Gln Ala Pro Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser
1070 1075 1080
Pro Leu Leu Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser
1085 1090 1095
Gln Leu Gln His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys
1100 1105 1110
Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser
1115 1120 1125
His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg
1130 1135 1140
Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His
1145 1150 1155
Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser
1160 1165 1170
Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu
1175 1180 1185
Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr
1190 1195 1200
Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr
1205 1210 1215
Phe Pro Ser Ser Ala Phe Leu Arg Glu Asp Pro Lys Trp Glu Phe
1220 1225 1230
Pro Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu
1235 1240 1245
Phe Gly Lys Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg
1250 1255 1260
Ala Gly Tyr Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala
1265 1270 1275
Ser Pro Ser Glu Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu
1280 1285 1290
Lys Gln Val Asn His Pro His Val Ile Lys Leu Tyr Gly Ala Cys
1295 1300 1305
Ser Gln Asp Gly Pro Leu Leu Leu Ile Val Glu Tyr Ala Lys Tyr
1310 1315 1320
Gly Ser Leu Arg Gly Phe Leu Arg Glu Ser Arg Lys Val Gly Pro
1325 1330 1335
Gly Tyr Leu Gly Ser Gly Gly Ser Arg Asn Ser Ser Ser Leu Asp
1340 1345 1350
His Pro Asp Glu Arg Ala Leu Thr Met Gly Asp Leu Ile Ser Phe
1355 1360 1365
Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr Leu Ala Glu Met Lys
1370 1375 1380
Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile Leu Val Ala Glu
1385 1390 1395
Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser Arg Asp Val
1400 1405 1410
Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg Ile Pro
1415 1420 1425
Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr Thr
1430 1435 1440
Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile
1445 1450 1455
Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg
1460 1465 1470
Leu Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp
1475 1480 1485
Asn Cys Ser Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys
1490 1495 1500
Gln Glu Pro Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp
1505 1510 1515
Leu Glu Lys Met Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala
1520 1525 1530
Ala Ser Thr Pro Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser
1535 1540 1545
Glu Glu Glu Thr Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro
1550 1555 1560
Arg Ala Leu Pro Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Arg
1565 1570 1575
Ile Ser His Ala Phe Thr Arg Phe
1580 1585
<210> 25
<211> 1278
<212> белок
<213> Homo sapiens
<400> 25
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Arg Gly Leu Lys Pro Lys Lys Ala Pro Thr Ala Arg Lys
130 135 140
Thr Thr Thr Arg Arg Pro Lys Pro Thr Arg Pro Ala Ser Thr Gly Val
145 150 155 160
Ala Gly Ala Ser Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly
165 170 175
Glu Leu Ser Ser Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala
180 185 190
Ala Pro Ile Ile Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro
195 200 205
Pro Leu Pro Ser Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val
210 215 220
Arg Asp Leu Glu Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu
225 230 235 240
Asp Lys Ala Lys Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu
245 250 255
Gln Val Gln Glu Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu
260 265 270
Gln Arg Arg Leu Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu
275 280 285
Ala Lys Glu Arg Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile
290 295 300
Glu Met Ala Thr Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser
305 310 315 320
Leu Gln Gln Glu Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr
325 330 335
Thr Asp Leu Glu Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp
340 345 350
Gly Ala Ala Ser Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala
355 360 365
Arg Leu Lys Asp Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu
370 375 380
Lys Gln Glu His Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln
385 390 395 400
Glu Leu Glu Val Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu
405 410 415
Ser Gln Ala Glu Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala
420 425 430
Ala Leu Gly Ala Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu
435 440 445
Asn Leu Glu Glu Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu
450 455 460
Glu Ala Met Asn Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu
465 470 475 480
Thr Glu Leu Glu Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val
485 490 495
Arg Glu Ala Gln Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp
500 505 510
Tyr Gln Gln Thr Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln
515 520 525
Asp Val Asn Arg Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg
530 535 540
Gln Gln Gln Pro Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala
545 550 555 560
Glu Thr Lys Ala His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met
565 570 575
Glu Val Ala Gln Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met
580 585 590
Pro Asp Ser Phe Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val
595 600 605
Leu Leu Leu Met Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys
610 615 620
Gln Ala Gln Glu Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro
625 630 635 640
Gly Leu Arg Gly Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu
645 650 655
Val Tyr Ser Leu Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His
660 665 670
Ala Leu Ser Gln Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu
675 680 685
Tyr Pro Glu Met Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu
690 695 700
Leu Leu His Lys Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu
705 710 715 720
Thr Lys Ala Ile Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala
725 730 735
Glu Gln Pro Glu Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe
740 745 750
Thr Gln Ser Ala Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg
755 760 765
Ala Phe Leu Gln Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu
770 775 780
Arg Asp Leu Glu Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys
785 790 795 800
Ile Arg Arg Arg Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala
805 810 815
Leu Ala Phe Gly Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys
820 825 830
His Leu Thr Trp Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala
835 840 845
Ala Gln Leu Ile Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala
850 855 860
Ala Leu Glu Glu Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr
865 870 875 880
Pro Ser Ser Ser Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu
885 890 895
Ile Ser Thr Met Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr
900 905 910
Asp Ala Glu Arg Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala
915 920 925
Ala Ala Leu Arg Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys
930 935 940
Leu Glu Asp Arg Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys
945 950 955 960
Ile Lys Gly Glu Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu
965 970 975
Glu Lys Lys Leu Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu
980 985 990
Lys Val Gln Thr Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys
995 1000 1005
Glu Lys Glu Phe Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile
1010 1015 1020
Asp Gln Leu Glu Ala Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn
1025 1030 1035
Ser Gln Ser Lys Arg Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro
1040 1045 1050
Ser Gly Ile Ala Thr Leu Val Ser Gly Ile Ala Gly Glu Glu Gln
1055 1060 1065
Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
1070 1075 1080
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser
1085 1090 1095
Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile
1100 1105 1110
Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro
1115 1120 1125
Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu
1130 1135 1140
Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr
1145 1150 1155
Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr Arg
1160 1165 1170
Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln
1175 1180 1185
Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys
1190 1195 1200
Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr
1205 1210 1215
Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg
1220 1225 1230
Ala Lys Glu Glu Gln Gln Asp Asp Thr Val Tyr Met Gly Lys Val
1235 1240 1245
Thr Phe Ser Cys Ala Ala Gly Phe Gly Gln Arg His Arg Leu Val
1250 1255 1260
Leu Thr Gln Glu Gln Leu His Gln Leu His Ser Arg Leu Ile Ser
1265 1270 1275
<210> 26
<211> 1144
<212> белок
<213> Homo sapiens
<400> 26
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly Gln Ala Pro
930 935 940
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu
945 950 955 960
Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
965 970 975
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu
980 985 990
Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu
995 1000 1005
Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu
1010 1015 1020
Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp Ile Thr
1025 1030 1035
Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu Met Glu
1040 1045 1050
Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu Lys Leu
1055 1060 1065
Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro Gly Ala
1070 1075 1080
Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu
1085 1090 1095
Arg Ala Lys Glu Glu Gln Gln Asp Asp Thr Val Tyr Met Gly Lys
1100 1105 1110
Val Thr Phe Ser Cys Ala Ala Gly Phe Gly Gln Arg His Arg Leu
1115 1120 1125
Val Leu Thr Gln Glu Gln Leu His Gln Leu His Ser Arg Leu Ile
1130 1135 1140
Ser
<210> 27
<211> 1253
<212> белок
<213> Homo sapiens
<400> 27
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser
130 135 140
Ser Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser
145 150 155 160
Ser Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile
165 170 175
Pro Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser
180 185 190
Pro Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu
195 200 205
Glu Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys
210 215 220
Leu Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu
225 230 235 240
Trp Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu
245 250 255
Lys Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg
260 265 270
Tyr Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr
275 280 285
Leu Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu
290 295 300
Val Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu
305 310 315 320
Ile Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser
325 330 335
Ser Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp
340 345 350
Ala Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His
355 360 365
Val Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val
370 375 380
Val Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu
385 390 395 400
Ser Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala
405 410 415
Glu Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu
420 425 430
Lys Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn
435 440 445
Glu Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu
450 455 460
Leu Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln
465 470 475 480
Lys Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr
485 490 495
Ile Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg
500 505 510
Glu Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro
515 520 525
Pro Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala
530 535 540
His Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln
545 550 555 560
Ala Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe
565 570 575
Leu Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met
580 585 590
Pro Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu
595 600 605
Lys Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly
610 615 620
Ala Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu
625 630 635 640
Ser Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln
645 650 655
Cys Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met
660 665 670
Ser Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys
675 680 685
Asp Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile
690 695 700
Lys Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu
705 710 715 720
Asp Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala
725 730 735
Leu Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln
740 745 750
Gly Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu
755 760 765
Thr Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg
770 775 780
Met Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly
785 790 795 800
Pro Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp
805 810 815
Val Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile
820 825 830
Ala Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu
835 840 845
Leu Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser
850 855 860
Pro Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met
865 870 875 880
Asn Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg
885 890 895
Pro Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg
900 905 910
Ala Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg
915 920 925
Glu Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu
930 935 940
Glu Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu
945 950 955 960
Asp Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr
965 970 975
Arg Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe
980 985 990
Glu Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala
995 1000 1005
Glu Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg
1010 1015 1020
Thr Ile Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr
1025 1030 1035
Leu Val Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro
1040 1045 1050
Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu
1055 1060 1065
Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln
1070 1075 1080
His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu
1085 1090 1095
Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly
1100 1105 1110
Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser
1115 1120 1125
Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val
1130 1135 1140
Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala
1145 1150 1155
Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr
1160 1165 1170
Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln
1175 1180 1185
Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser
1190 1195 1200
Ser Ala Phe Leu Arg Ala Lys Glu Glu Gln Gln Asp Asp Thr Val
1205 1210 1215
Tyr Met Gly Lys Val Thr Phe Ser Cys Ala Ala Gly Phe Gly Gln
1220 1225 1230
Arg His Arg Leu Val Leu Thr Gln Glu Gln Leu His Gln Leu His
1235 1240 1245
Ser Arg Leu Ile Ser
1250
<210> 28
<211> 1139
<212> белок
<213> Homo sapiens
<400> 28
Met Met Arg Gln Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro
1. 5 10 15
Lys Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser
20 25 30
Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu
35 40 45
Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr
50 55 60
Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser
65 70 75 80
Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys
85 90 95
Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys
100 105 110
Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys
115 120 125
Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu
130 135 140
Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met
145 150 155 160
Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp
165 170 175
Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu
180 185 190
Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu
195 200 205
Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr
210 215 220
Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu
225 230 235 240
Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys
245 250 255
Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg
260 265 270
Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr
275 280 285
Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu
290 295 300
Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val
305 310 315 320
Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met
325 330 335
Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg
340 345 350
Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg
355 360 365
Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys
370 375 380
Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu
385 390 395 400
Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro
405 410 415
Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala
420 425 430
Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn
435 440 445
Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg
450 455 460
Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg
465 470 475 480
Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe
485 490 495
Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala
500 505 510
Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu
515 520 525
Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser
530 535 540
Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala
545 550 555 560
His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln
565 570 575
Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr
580 585 590
Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys
595 600 605
Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp
610 615 620
Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly
625 630 635 640
Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser
645 650 655
Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro
660 665 670
Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln
675 680 685
Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val
690 695 700
Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro
705 710 715 720
Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala
725 730 735
Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr
740 745 750
Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys
755 760 765
Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro
770 775 780
Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu
785 790 795 800
Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr
805 810 815
Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu
820 825 830
Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser
835 840 845
Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu
850 855 860
Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu
865 870 875 880
Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
885 890 895
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile Glu
900 905 910
Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val Ser Gly
915 920 925
Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser Val Pro Gly
930 935 940
Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln Gln Ile Ser Ala
945 950 955 960
Met Arg Leu His Ile Ser Gln Leu Gln His Glu Asn Ser Ile Leu Lys
965 970 975
Gly Ala Gln Met Lys Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val
980 985 990
Ala Lys Leu Ser His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala
995 1000 1005
Leu Tyr Arg Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu
1010 1015 1020
Ser Thr His Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala
1025 1030 1035
Ala Lys Ser Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu
1040 1045 1050
Lys Ser Leu Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu
1055 1060 1065
Lys Glu Thr Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp
1070 1075 1080
Phe Ala Thr Phe Pro Ser Ser Ala Phe Leu Arg Ala Lys Glu Glu
1085 1090 1095
Gln Gln Asp Asp Thr Val Tyr Met Gly Lys Val Thr Phe Ser Cys
1100 1105 1110
Ala Ala Gly Phe Gly Gln Arg His Arg Leu Val Leu Thr Gln Glu
1115 1120 1125
Gln Leu His Gln Leu His Ser Arg Leu Ile Ser
1130 1135
<210> 29
<211> 1236
<212> белок
<213> Homo sapiens
<400> 29
Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg Val
1. 5 10 15
Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly Ala
20 25 30
Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu Ala
35 40 45
Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr Cys
50 55 60
Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val Phe
65 70 75 80
Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser Ala
85 90 95
Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr Ser
100 105 110
Lys Leu Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser
115 120 125
Ser Leu Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser
130 135 140
Glu Pro Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro
145 150 155 160
Thr Pro Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro
165 170 175
Ser Lys Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu
180 185 190
Lys Leu Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu
195 200 205
Lys Glu Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp
210 215 220
Lys Ser Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys
225 230 235 240
Glu Ala Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr
245 250 255
Met Glu Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu
260 265 270
Asp Lys Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val
275 280 285
Glu Ala Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile
290 295 300
Leu Lys Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser
305 310 315 320
Tyr Gln Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala
325 330 335
Leu Val Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val
340 345 350
Lys Leu Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val
355 360 365
Arg Gln Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser
370 375 380
Thr Ile Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu
385 390 395 400
Glu Met Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys
405 410 415
Val Arg Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu
420 425 430
Met Asn Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu
435 440 445
Arg Glu Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys
450 455 460
Arg Val Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile
465 470 475 480
Lys Lys Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu
485 490 495
Leu Thr Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro
500 505 510
Pro Glu Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His
515 520 525
Ala Lys Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala
530 535 540
Asn Arg His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu
545 550 555 560
Arg Pro Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro
565 570 575
Arg Leu Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys
580 585 590
Phe Glu Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala
595 600 605
Ala Gly Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser
610 615 620
Leu Leu Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys
625 630 635 640
Ser Val Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser
645 650 655
Ala His Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp
660 665 670
Gln Leu Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys
675 680 685
Tyr Tyr Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp
690 695 700
Cys Thr Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu
705 710 715 720
Asp Cys Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly
725 730 735
Gly Gln Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr
740 745 750
Ser Cys Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met
755 760 765
Pro Gly Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro
770 775 780
Gln Val Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val
785 790 795 800
Val Ala Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala
805 810 815
Pro Leu Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu
820 825 830
Ala Phe Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro
835 840 845
Tyr Glu Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn
850 855 860
Lys Leu Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro
865 870 875 880
Pro Ser Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala
885 890 895
Glu Ile Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu
900 905 910
Thr Val Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu
915 920 925
Leu Ser Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp
930 935 940
Ser Ala Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg
945 950 955 960
Leu Glu Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu
965 970 975
Glu Thr Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu
980 985 990
Lys Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
995 1000 1005
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1010 1015 1020
Ser Gly Ile Ala Gly Gly Ala Ile Pro Gly Gln Ala Pro Gly Ser
1025 1030 1035
Val Pro Gly Pro Gly Leu Val Lys Asp Ser Pro Leu Leu Leu Gln
1040 1045 1050
Gln Ile Ser Ala Met Arg Leu His Ile Ser Gln Leu Gln His Glu
1055 1060 1065
Asn Ser Ile Leu Lys Gly Ala Gln Met Lys Ala Ser Leu Ala Ser
1070 1075 1080
Leu Pro Pro Leu His Val Ala Lys Leu Ser His Glu Gly Pro Gly
1085 1090 1095
Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg Lys Thr Ser Gln Leu
1100 1105 1110
Leu Glu Thr Leu Asn Gln Leu Ser Thr His Thr His Val Val Asp
1115 1120 1125
Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser Pro Ser Ala Gln Leu
1130 1135 1140
Met Glu Gln Val Ala Gln Leu Lys Ser Leu Ser Asp Thr Val Glu
1145 1150 1155
Lys Leu Lys Asp Glu Val Leu Lys Glu Thr Val Ser Gln Arg Pro
1160 1165 1170
Gly Ala Thr Val Pro Thr Asp Phe Ala Thr Phe Pro Ser Ser Ala
1175 1180 1185
Phe Leu Arg Ala Lys Glu Glu Gln Gln Asp Asp Thr Val Tyr Met
1190 1195 1200
Gly Lys Val Thr Phe Ser Cys Ala Ala Gly Phe Gly Gln Arg His
1205 1210 1215
Arg Leu Val Leu Thr Gln Glu Gln Leu His Gln Leu His Ser Arg
1220 1225 1230
Leu Ile Ser
1235
<210> 30
<211> 1271
<212> белок
<213> Homo sapiens
<400> 30
Met Ala Gln Ser Lys Arg His Val Tyr Ser Arg Thr Pro Ser Gly Ser
1. 5 10 15
Arg Met Ser Ala Glu Ala Ser Ala Arg Pro Leu Arg Val Gly Ser Arg
20 25 30
Val Glu Val Ile Gly Lys Gly His Arg Gly Thr Val Ala Tyr Val Gly
35 40 45
Ala Thr Leu Phe Ala Thr Gly Lys Trp Val Gly Val Ile Leu Asp Glu
50 55 60
Ala Lys Gly Lys Asn Asp Gly Thr Val Gln Gly Arg Lys Tyr Phe Thr
65 70 75 80
Cys Asp Glu Gly His Gly Ile Phe Val Arg Gln Ser Gln Ile Gln Val
85 90 95
Phe Glu Asp Gly Ala Asp Thr Thr Ser Pro Glu Thr Pro Asp Ser Ser
100 105 110
Ala Ser Lys Val Leu Lys Arg Glu Gly Thr Asp Thr Thr Ala Lys Thr
115 120 125
Ser Lys Leu Ala Pro Thr Ala Arg Lys Thr Thr Thr Arg Arg Pro Lys
130 135 140
Pro Thr Arg Pro Ala Ser Thr Gly Val Ala Gly Ala Ser Ser Ser Leu
145 150 155 160
Gly Pro Ser Gly Ser Ala Ser Ala Gly Glu Leu Ser Ser Ser Glu Pro
165 170 175
Ser Thr Pro Ala Gln Thr Pro Leu Ala Ala Pro Ile Ile Pro Thr Pro
180 185 190
Val Leu Thr Ser Pro Gly Ala Val Pro Pro Leu Pro Ser Pro Ser Lys
195 200 205
Glu Glu Glu Gly Leu Arg Ala Gln Val Arg Asp Leu Glu Glu Lys Leu
210 215 220
Glu Thr Leu Arg Leu Lys Arg Ala Glu Asp Lys Ala Lys Leu Lys Glu
225 230 235 240
Leu Glu Lys His Lys Ile Gln Leu Glu Gln Val Gln Glu Trp Lys Ser
245 250 255
Lys Met Gln Glu Gln Gln Ala Asp Leu Gln Arg Arg Leu Lys Glu Ala
260 265 270
Arg Lys Glu Ala Lys Glu Ala Leu Glu Ala Lys Glu Arg Tyr Met Glu
275 280 285
Glu Met Ala Asp Thr Ala Asp Ala Ile Glu Met Ala Thr Leu Asp Lys
290 295 300
Glu Met Ala Glu Glu Arg Ala Glu Ser Leu Gln Gln Glu Val Glu Ala
305 310 315 320
Leu Lys Glu Arg Val Asp Glu Leu Thr Thr Asp Leu Glu Ile Leu Lys
325 330 335
Ala Glu Ile Glu Glu Lys Gly Ser Asp Gly Ala Ala Ser Ser Tyr Gln
340 345 350
Leu Lys Gln Leu Glu Glu Gln Asn Ala Arg Leu Lys Asp Ala Leu Val
355 360 365
Arg Met Arg Asp Leu Ser Ser Ser Glu Lys Gln Glu His Val Lys Leu
370 375 380
Gln Lys Leu Met Glu Lys Lys Asn Gln Glu Leu Glu Val Val Arg Gln
385 390 395 400
Gln Arg Glu Arg Leu Gln Glu Glu Leu Ser Gln Ala Glu Ser Thr Ile
405 410 415
Asp Glu Leu Lys Glu Gln Val Asp Ala Ala Leu Gly Ala Glu Glu Met
420 425 430
Val Glu Met Leu Thr Asp Arg Asn Leu Asn Leu Glu Glu Lys Val Arg
435 440 445
Glu Leu Arg Glu Thr Val Gly Asp Leu Glu Ala Met Asn Glu Met Asn
450 455 460
Asp Glu Leu Gln Glu Asn Ala Arg Glu Thr Glu Leu Glu Leu Arg Glu
465 470 475 480
Gln Leu Asp Met Ala Gly Ala Arg Val Arg Glu Ala Gln Lys Arg Val
485 490 495
Glu Ala Ala Gln Glu Thr Val Ala Asp Tyr Gln Gln Thr Ile Lys Lys
500 505 510
Tyr Arg Gln Leu Thr Ala His Leu Gln Asp Val Asn Arg Glu Leu Thr
515 520 525
Asn Gln Gln Glu Ala Ser Val Glu Arg Gln Gln Gln Pro Pro Pro Glu
530 535 540
Thr Phe Asp Phe Lys Ile Lys Phe Ala Glu Thr Lys Ala His Ala Lys
545 550 555 560
Ala Ile Glu Met Glu Leu Arg Gln Met Glu Val Ala Gln Ala Asn Arg
565 570 575
His Met Ser Leu Leu Thr Ala Phe Met Pro Asp Ser Phe Leu Arg Pro
580 585 590
Gly Gly Asp His Asp Cys Val Leu Val Leu Leu Leu Met Pro Arg Leu
595 600 605
Ile Cys Lys Ala Glu Leu Ile Arg Lys Gln Ala Gln Glu Lys Phe Glu
610 615 620
Leu Ser Glu Asn Cys Ser Glu Arg Pro Gly Leu Arg Gly Ala Ala Gly
625 630 635 640
Glu Gln Leu Ser Phe Ala Ala Gly Leu Val Tyr Ser Leu Ser Leu Leu
645 650 655
Gln Ala Thr Leu His Arg Tyr Glu His Ala Leu Ser Gln Cys Ser Val
660 665 670
Asp Val Tyr Lys Lys Val Gly Ser Leu Tyr Pro Glu Met Ser Ala His
675 680 685
Glu Arg Ser Leu Asp Phe Leu Ile Glu Leu Leu His Lys Asp Gln Leu
690 695 700
Asp Glu Thr Val Asn Val Glu Pro Leu Thr Lys Ala Ile Lys Tyr Tyr
705 710 715 720
Gln His Leu Tyr Ser Ile His Leu Ala Glu Gln Pro Glu Asp Cys Thr
725 730 735
Met Gln Leu Ala Asp His Ile Lys Phe Thr Gln Ser Ala Leu Asp Cys
740 745 750
Met Ser Val Glu Val Gly Arg Leu Arg Ala Phe Leu Gln Gly Gly Gln
755 760 765
Glu Ala Thr Asp Ile Ala Leu Leu Leu Arg Asp Leu Glu Thr Ser Cys
770 775 780
Ser Asp Ile Arg Gln Phe Cys Lys Lys Ile Arg Arg Arg Met Pro Gly
785 790 795 800
Thr Asp Ala Pro Gly Ile Pro Ala Ala Leu Ala Phe Gly Pro Gln Val
805 810 815
Ser Asp Thr Leu Leu Asp Cys Arg Lys His Leu Thr Trp Val Val Ala
820 825 830
Val Leu Gln Glu Val Ala Ala Ala Ala Ala Gln Leu Ile Ala Pro Leu
835 840 845
Ala Glu Asn Glu Gly Leu Leu Val Ala Ala Leu Glu Glu Leu Ala Phe
850 855 860
Lys Ala Ser Glu Gln Ile Tyr Gly Thr Pro Ser Ser Ser Pro Tyr Glu
865 870 875 880
Cys Leu Arg Gln Ser Cys Asn Ile Leu Ile Ser Thr Met Asn Lys Leu
885 890 895
Ala Thr Ala Met Gln Glu Gly Glu Tyr Asp Ala Glu Arg Pro Pro Ser
900 905 910
Lys Pro Pro Pro Val Glu Leu Arg Ala Ala Ala Leu Arg Ala Glu Ile
915 920 925
Thr Asp Ala Glu Gly Leu Gly Leu Lys Leu Glu Asp Arg Glu Thr Val
930 935 940
Ile Lys Glu Leu Lys Lys Ser Leu Lys Ile Lys Gly Glu Glu Leu Ser
945 950 955 960
Glu Ala Asn Val Arg Leu Ser Leu Leu Glu Lys Lys Leu Asp Ser Ala
965 970 975
Ala Lys Asp Ala Asp Glu Arg Ile Glu Lys Val Gln Thr Arg Leu Glu
980 985 990
Glu Thr Gln Ala Leu Leu Arg Lys Lys Glu Lys Glu Phe Glu Glu Thr
995 1000 1005
Met Asp Ala Leu Gln Ala Asp Ile Asp Gln Leu Glu Ala Glu Lys
1010 1015 1020
Ala Glu Leu Lys Gln Arg Leu Asn Ser Gln Ser Lys Arg Thr Ile
1025 1030 1035
Glu Gly Leu Arg Gly Pro Pro Pro Ser Gly Ile Ala Thr Leu Val
1040 1045 1050
Ser Gly Ile Ala Gly Glu Glu Gln Gln Arg Gly Ala Ile Pro Gly
1055 1060 1065
Gln Ala Pro Gly Ser Val Pro Gly Pro Gly Leu Val Lys Asp Ser
1070 1075 1080
Pro Leu Leu Leu Gln Gln Ile Ser Ala Met Arg Leu His Ile Ser
1085 1090 1095
Gln Leu Gln His Glu Asn Ser Ile Leu Lys Gly Ala Gln Met Lys
1100 1105 1110
Ala Ser Leu Ala Ser Leu Pro Pro Leu His Val Ala Lys Leu Ser
1115 1120 1125
His Glu Gly Pro Gly Ser Glu Leu Pro Ala Gly Ala Leu Tyr Arg
1130 1135 1140
Lys Thr Ser Gln Leu Leu Glu Thr Leu Asn Gln Leu Ser Thr His
1145 1150 1155
Thr His Val Val Asp Ile Thr Arg Thr Ser Pro Ala Ala Lys Ser
1160 1165 1170
Pro Ser Ala Gln Leu Met Glu Gln Val Ala Gln Leu Lys Ser Leu
1175 1180 1185
Ser Asp Thr Val Glu Lys Leu Lys Asp Glu Val Leu Lys Glu Thr
1190 1195 1200
Val Ser Gln Arg Pro Gly Ala Thr Val Pro Thr Asp Phe Ala Thr
1205 1210 1215
Phe Pro Ser Ser Ala Phe Leu Arg Ala Lys Glu Glu Gln Gln Asp
1220 1225 1230
Asp Thr Val Tyr Met Gly Lys Val Thr Phe Ser Cys Ala Ala Gly
1235 1240 1245
Phe Gly Gln Arg His Arg Leu Val Leu Thr Gln Glu Gln Leu His
1250 1255 1260
Gln Leu His Ser Arg Leu Ile Ser
1265 1270
<210> 31
<211> 1114
<212> белок
<213> Homo sapiens
<400> 31
Met Ala Lys Ala Thr Ser Gly Ala Ala Gly Leu Arg Leu Leu Leu Leu
1. 5 10 15
Leu Leu Leu Pro Leu Leu Gly Lys Val Ala Leu Gly Leu Tyr Phe Ser
20 25 30
Arg Asp Ala Tyr Trp Glu Lys Leu Tyr Val Asp Gln Ala Ala Gly Thr
35 40 45
Pro Leu Leu Tyr Val His Ala Leu Arg Asp Ala Pro Glu Glu Val Pro
50 55 60
Ser Phe Arg Leu Gly Gln His Leu Tyr Gly Thr Tyr Arg Thr Arg Leu
65 70 75 80
His Glu Asn Asn Trp Ile Cys Ile Gln Glu Asp Thr Gly Leu Leu Tyr
85 90 95
Leu Asn Arg Ser Leu Asp His Ser Ser Trp Glu Lys Leu Ser Val Arg
100 105 110
Asn Arg Gly Phe Pro Leu Leu Thr Val Tyr Leu Lys Val Phe Leu Ser
115 120 125
Pro Thr Ser Leu Arg Glu Gly Glu Cys Gln Trp Pro Gly Cys Ala Arg
130 135 140
Val Tyr Phe Ser Phe Phe Asn Thr Ser Phe Pro Ala Cys Ser Ser Leu
145 150 155 160
Lys Pro Arg Glu Leu Cys Phe Pro Glu Thr Arg Pro Ser Phe Arg Ile
165 170 175
Arg Glu Asn Arg Pro Pro Gly Thr Phe His Gln Phe Arg Leu Leu Pro
180 185 190
Val Gln Phe Leu Cys Pro Asn Ile Ser Val Ala Tyr Arg Leu Leu Glu
195 200 205
Gly Glu Gly Leu Pro Phe Arg Cys Ala Pro Asp Ser Leu Glu Val Ser
210 215 220
Thr Arg Trp Ala Leu Asp Arg Glu Gln Arg Glu Lys Tyr Glu Leu Val
225 230 235 240
Ala Val Cys Thr Val His Ala Gly Ala Arg Glu Glu Val Val Met Val
245 250 255
Pro Phe Pro Val Thr Val Tyr Asp Glu Asp Asp Ser Ala Pro Thr Phe
260 265 270
Pro Ala Gly Val Asp Thr Ala Ser Ala Val Val Glu Phe Lys Arg Lys
275 280 285
Glu Asp Thr Val Val Ala Thr Leu Arg Val Phe Asp Ala Asp Val Val
290 295 300
Pro Ala Ser Gly Glu Leu Val Arg Arg Tyr Thr Ser Thr Leu Leu Pro
305 310 315 320
Gly Asp Thr Trp Ala Gln Gln Thr Phe Arg Val Glu His Trp Pro Asn
325 330 335
Glu Thr Ser Val Gln Ala Asn Gly Ser Phe Val Arg Ala Thr Val His
340 345 350
Asp Tyr Arg Leu Val Leu Asn Arg Asn Leu Ser Ile Ser Glu Asn Arg
355 360 365
Thr Met Gln Leu Ala Val Leu Val Asn Asp Ser Asp Phe Gln Gly Pro
370 375 380
Gly Ala Gly Val Leu Leu Leu His Phe Asn Val Ser Val Leu Pro Val
385 390 395 400
Ser Leu His Leu Pro Ser Thr Tyr Ser Leu Ser Val Ser Arg Arg Ala
405 410 415
Arg Arg Phe Ala Gln Ile Gly Lys Val Cys Val Glu Asn Cys Gln Ala
420 425 430
Phe Ser Gly Ile Asn Val Gln Tyr Lys Leu His Ser Ser Gly Ala Asn
435 440 445
Cys Ser Thr Leu Gly Val Val Thr Ser Ala Glu Asp Thr Ser Gly Ile
450 455 460
Leu Phe Val Asn Asp Thr Lys Ala Leu Arg Arg Pro Lys Cys Ala Glu
465 470 475 480
Leu His Tyr Met Val Val Ala Thr Asp Gln Gln Thr Ser Arg Gln Ala
485 490 495
Gln Ala Gln Leu Leu Val Thr Val Glu Gly Ser Tyr Val Ala Glu Glu
500 505 510
Ala Gly Cys Pro Leu Ser Cys Ala Val Ser Lys Arg Arg Leu Glu Cys
515 520 525
Glu Glu Cys Gly Gly Leu Gly Ser Pro Thr Gly Arg Cys Glu Trp Arg
530 535 540
Gln Gly Asp Gly Lys Gly Ile Thr Arg Asn Phe Ser Thr Cys Ser Pro
545 550 555 560
Ser Thr Lys Thr Cys Pro Asp Gly His Cys Asp Val Val Glu Thr Gln
565 570 575
Asp Ile Asn Ile Cys Pro Gln Asp Cys Leu Arg Gly Ser Ile Val Gly
580 585 590
Gly His Glu Pro Gly Glu Pro Arg Gly Ile Lys Ala Gly Tyr Gly Thr
595 600 605
Cys Asn Cys Phe Pro Glu Glu Glu Lys Cys Phe Cys Glu Pro Glu Asp
610 615 620
Ile Gln Asp Pro Leu Cys Asp Glu Leu Cys Arg Thr Val Ile Ala Ala
625 630 635 640
Ala Val Leu Phe Ser Phe Ile Val Ser Val Leu Leu Ser Ala Phe Cys
645 650 655
Ile His Cys Tyr His Lys Phe Ala His Lys Pro Pro Ile Ser Ser Ala
660 665 670
Glu Met Thr Phe Arg Arg Pro Ala Gln Ala Phe Pro Val Ser Tyr Ser
675 680 685
Ser Ser Gly Ala Arg Arg Pro Ser Leu Asp Ser Met Glu Asn Gln Val
690 695 700
Ser Val Asp Ala Phe Lys Ile Leu Glu Asp Pro Lys Trp Glu Phe Pro
705 710 715 720
Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly
725 730 735
Lys Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr
740 745 750
Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu
755 760 765
Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His
770 775 780
Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu
785 790 795 800
Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu
805 810 815
Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser
820 825 830
Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr Met
835 840 845
Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr
850 855 860
Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile
865 870 875 880
Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser
885 890 895
Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg
900 905 910
Ile Pro Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr
915 920 925
Thr Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile
930 935 940
Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu
945 950 955 960
Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys
965 970 975
Ser Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro
980 985 990
Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met
995 1000 1005
Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro
1010 1015 1020
Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr
1025 1030 1035
Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro
1040 1045 1050
Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Met Ser Asp Pro Asn
1055 1060 1065
Trp Pro Gly Glu Ser Pro Val Pro Leu Thr Arg Ala Asp Gly Thr
1070 1075 1080
Asn Thr Gly Phe Pro Arg Tyr Pro Asn Asp Ser Val Tyr Ala Asn
1085 1090 1095
Trp Met Leu Ser Pro Ser Ala Ala Lys Leu Met Asp Thr Phe Asp
1100 1105 1110
Ser
<210> 32
<211> 1072
<212> белок
<213> Homo sapiens
<400> 32
Met Ala Lys Ala Thr Ser Gly Ala Ala Gly Leu Arg Leu Leu Leu Leu
1. 5 10 15
Leu Leu Leu Pro Leu Leu Gly Lys Val Ala Leu Gly Leu Tyr Phe Ser
20 25 30
Arg Asp Ala Tyr Trp Glu Lys Leu Tyr Val Asp Gln Ala Ala Gly Thr
35 40 45
Pro Leu Leu Tyr Val His Ala Leu Arg Asp Ala Pro Glu Glu Val Pro
50 55 60
Ser Phe Arg Leu Gly Gln His Leu Tyr Gly Thr Tyr Arg Thr Arg Leu
65 70 75 80
His Glu Asn Asn Trp Ile Cys Ile Gln Glu Asp Thr Gly Leu Leu Tyr
85 90 95
Leu Asn Arg Ser Leu Asp His Ser Ser Trp Glu Lys Leu Ser Val Arg
100 105 110
Asn Arg Gly Phe Pro Leu Leu Thr Val Tyr Leu Lys Val Phe Leu Ser
115 120 125
Pro Thr Ser Leu Arg Glu Gly Glu Cys Gln Trp Pro Gly Cys Ala Arg
130 135 140
Val Tyr Phe Ser Phe Phe Asn Thr Ser Phe Pro Ala Cys Ser Ser Leu
145 150 155 160
Lys Pro Arg Glu Leu Cys Phe Pro Glu Thr Arg Pro Ser Phe Arg Ile
165 170 175
Arg Glu Asn Arg Pro Pro Gly Thr Phe His Gln Phe Arg Leu Leu Pro
180 185 190
Val Gln Phe Leu Cys Pro Asn Ile Ser Val Ala Tyr Arg Leu Leu Glu
195 200 205
Gly Glu Gly Leu Pro Phe Arg Cys Ala Pro Asp Ser Leu Glu Val Ser
210 215 220
Thr Arg Trp Ala Leu Asp Arg Glu Gln Arg Glu Lys Tyr Glu Leu Val
225 230 235 240
Ala Val Cys Thr Val His Ala Gly Ala Arg Glu Glu Val Val Met Val
245 250 255
Pro Phe Pro Val Thr Val Tyr Asp Glu Asp Asp Ser Ala Pro Thr Phe
260 265 270
Pro Ala Gly Val Asp Thr Ala Ser Ala Val Val Glu Phe Lys Arg Lys
275 280 285
Glu Asp Thr Val Val Ala Thr Leu Arg Val Phe Asp Ala Asp Val Val
290 295 300
Pro Ala Ser Gly Glu Leu Val Arg Arg Tyr Thr Ser Thr Leu Leu Pro
305 310 315 320
Gly Asp Thr Trp Ala Gln Gln Thr Phe Arg Val Glu His Trp Pro Asn
325 330 335
Glu Thr Ser Val Gln Ala Asn Gly Ser Phe Val Arg Ala Thr Val His
340 345 350
Asp Tyr Arg Leu Val Leu Asn Arg Asn Leu Ser Ile Ser Glu Asn Arg
355 360 365
Thr Met Gln Leu Ala Val Leu Val Asn Asp Ser Asp Phe Gln Gly Pro
370 375 380
Gly Ala Gly Val Leu Leu Leu His Phe Asn Val Ser Val Leu Pro Val
385 390 395 400
Ser Leu His Leu Pro Ser Thr Tyr Ser Leu Ser Val Ser Arg Arg Ala
405 410 415
Arg Arg Phe Ala Gln Ile Gly Lys Val Cys Val Glu Asn Cys Gln Ala
420 425 430
Phe Ser Gly Ile Asn Val Gln Tyr Lys Leu His Ser Ser Gly Ala Asn
435 440 445
Cys Ser Thr Leu Gly Val Val Thr Ser Ala Glu Asp Thr Ser Gly Ile
450 455 460
Leu Phe Val Asn Asp Thr Lys Ala Leu Arg Arg Pro Lys Cys Ala Glu
465 470 475 480
Leu His Tyr Met Val Val Ala Thr Asp Gln Gln Thr Ser Arg Gln Ala
485 490 495
Gln Ala Gln Leu Leu Val Thr Val Glu Gly Ser Tyr Val Ala Glu Glu
500 505 510
Ala Gly Cys Pro Leu Ser Cys Ala Val Ser Lys Arg Arg Leu Glu Cys
515 520 525
Glu Glu Cys Gly Gly Leu Gly Ser Pro Thr Gly Arg Cys Glu Trp Arg
530 535 540
Gln Gly Asp Gly Lys Gly Ile Thr Arg Asn Phe Ser Thr Cys Ser Pro
545 550 555 560
Ser Thr Lys Thr Cys Pro Asp Gly His Cys Asp Val Val Glu Thr Gln
565 570 575
Asp Ile Asn Ile Cys Pro Gln Asp Cys Leu Arg Gly Ser Ile Val Gly
580 585 590
Gly His Glu Pro Gly Glu Pro Arg Gly Ile Lys Ala Gly Tyr Gly Thr
595 600 605
Cys Asn Cys Phe Pro Glu Glu Glu Lys Cys Phe Cys Glu Pro Glu Asp
610 615 620
Ile Gln Asp Pro Leu Cys Asp Glu Leu Cys Arg Thr Val Ile Ala Ala
625 630 635 640
Ala Val Leu Phe Ser Phe Ile Val Ser Val Leu Leu Ser Ala Phe Cys
645 650 655
Ile His Cys Tyr His Lys Phe Ala His Lys Pro Pro Ile Ser Ser Ala
660 665 670
Glu Met Thr Phe Arg Arg Pro Ala Gln Ala Phe Pro Val Ser Tyr Ser
675 680 685
Ser Ser Gly Ala Arg Arg Pro Ser Leu Asp Ser Met Glu Asn Gln Val
690 695 700
Ser Val Asp Ala Phe Lys Ile Leu Glu Asp Pro Lys Trp Glu Phe Pro
705 710 715 720
Arg Lys Asn Leu Val Leu Gly Lys Thr Leu Gly Glu Gly Glu Phe Gly
725 730 735
Lys Val Val Lys Ala Thr Ala Phe His Leu Lys Gly Arg Ala Gly Tyr
740 745 750
Thr Thr Val Ala Val Lys Met Leu Lys Glu Asn Ala Ser Pro Ser Glu
755 760 765
Leu Arg Asp Leu Leu Ser Glu Phe Asn Val Leu Lys Gln Val Asn His
770 775 780
Pro His Val Ile Lys Leu Tyr Gly Ala Cys Ser Gln Asp Gly Pro Leu
785 790 795 800
Leu Leu Ile Val Glu Tyr Ala Lys Tyr Gly Ser Leu Arg Gly Phe Leu
805 810 815
Arg Glu Ser Arg Lys Val Gly Pro Gly Tyr Leu Gly Ser Gly Gly Ser
820 825 830
Arg Asn Ser Ser Ser Leu Asp His Pro Asp Glu Arg Ala Leu Thr Met
835 840 845
Gly Asp Leu Ile Ser Phe Ala Trp Gln Ile Ser Gln Gly Met Gln Tyr
850 855 860
Leu Ala Glu Met Lys Leu Val His Arg Asp Leu Ala Ala Arg Asn Ile
865 870 875 880
Leu Val Ala Glu Gly Arg Lys Met Lys Ile Ser Asp Phe Gly Leu Ser
885 890 895
Arg Asp Val Tyr Glu Glu Asp Ser Tyr Val Lys Arg Ser Gln Gly Arg
900 905 910
Ile Pro Val Lys Trp Met Ala Ile Glu Ser Leu Phe Asp His Ile Tyr
915 920 925
Thr Thr Gln Ser Asp Val Trp Ser Phe Gly Val Leu Leu Trp Glu Ile
930 935 940
Val Thr Leu Gly Gly Asn Pro Tyr Pro Gly Ile Pro Pro Glu Arg Leu
945 950 955 960
Phe Asn Leu Leu Lys Thr Gly His Arg Met Glu Arg Pro Asp Asn Cys
965 970 975
Ser Glu Glu Met Tyr Arg Leu Met Leu Gln Cys Trp Lys Gln Glu Pro
980 985 990
Asp Lys Arg Pro Val Phe Ala Asp Ile Ser Lys Asp Leu Glu Lys Met
995 1000 1005
Met Val Lys Arg Arg Asp Tyr Leu Asp Leu Ala Ala Ser Thr Pro
1010 1015 1020
Ser Asp Ser Leu Ile Tyr Asp Asp Gly Leu Ser Glu Glu Glu Thr
1025 1030 1035
Pro Leu Val Asp Cys Asn Asn Ala Pro Leu Pro Arg Ala Leu Pro
1040 1045 1050
Ser Thr Trp Ile Glu Asn Lys Leu Tyr Gly Arg Ile Ser His Ala
1055 1060 1065
Phe Thr Arg Phe
1070
<210> 33
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 33
tgtccagctt tgtgcctgat tgatgt 26
<210> 34
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 34
gctgggcact gaagagaaag gaatgc 26
<210> 35
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 35
agcaggatga gtgcggaggc aagc 24
<210> 36
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 36
ttaactatca aacgtgtcca ttaattttgc cgc 33
<210> 37
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 37
agtactgggg tggctggg 18
<210> 38
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 38
cactttggac aaggagatg 19
<210> 39
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 39
acagaactgg agctgcgg 18
<210> 40
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 40
ggactggtgt actcgctg 18
<210> 41
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 41
tcctagactg caggaaacac 20
<210> 42
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 42
catcgagaaa gtccagac 18
<210> 43
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 43
gctgctggag acattgaa 18
<210> 44
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 44
tcactgctgc tcagctca 18
<210> 45
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 45
gaggatccaa agtgggaatt 20
<210> 46
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 46
agtatctggc cgagatgaag 20
<210> 47
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 47
gcaaagacct ggagaagatg 20
<210> 48
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 48
aggacgttga actctgacag 20
<210> 49
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 49
cctttgcttc atccagaatc 20
<210> 50
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 50
gattttgtgt ttctccagct ct 22
<210> 51
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 51
cctgcttctc tgaggaagaa 20
<210> 52
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 52
gggccttagt ctcagcaaac 20
<210> 53
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 53
gagcactctg cgtgaactta 20
<210> 54
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 54
cagcttgttc atggtactga t 21
<210> 55
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 55
tggtgagtcc ttcaccag 18
<210> 56
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 56
cctagagttt ttccaagaac ca 22
<210> 57
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 57
catttaactg gaatccgacc 20
<210> 58
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 58
gactctctcc aggccagttc 20
<210> 59
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 59
ggctatcaga agtaaaacca cc 22
<210> 60
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 60
cgagagctga tggcacta 18
<210> 61
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 61
cttcatcaca agtgaagtac ttcc 24
<210> 62
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 62
cgtactccac gatgaggag 19
<210> 63
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 63
gattctggat gaagcaaagg 20
<210> 64
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 64
ggaagtactt cacttgtgat gaag 24
<210> 65
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 65
cccagccacc ccagtact 18
<210> 66
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 66
gtaaaacgac ggccagt 17
<210> 67
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 67
gttttcccag tcacgac 17
<210> 68
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 68
caggaaacag ctatgac 17
<210> 69
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 69
ctggagccac agtacccact 20
<210> 70
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 70
tccaaattcg ccttctccta 20
<210> 71
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 71
ttcatcagcc ttcctcaggg aggat 25
<210> 72
<211> 41
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 72
ggggacaagt ttgtacaaaa aagcaggctt cgccaccagc a 41
<210> 73
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> праймер
<400> 73
ggggaccact ttgtacaaga aagctgggtt ttaactatca aa 42
<210> 74
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> RET siРНК1
<220>
<221> разное
<223> n обозначает тимин
<220>
<221> разное
<222> (20)..(21)
<223> n обозначает тимин
<400> 74
cacaugucau caaauuguan n 21
<210> 75
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> RET siРНК2
<220>
<221> разное
<222> (20)..(21)
<223> n обозначает тимин
<400> 75
ggauugaaaa caaacucuan n 21
<210> 76
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> RET siРНК
<220>
<221> разное
<222> (20)..(21)
<223> n обозначает тимин
<400> 76
gcuugucccg agauguuuan n 21
<210> 77
<211> 21
<212> РНК
<213> Искусственная последовательность
<220>
<223> RET siРНК3
<220>
<221> разное
<222> (20)..(21)
<223> n обозначает тимин
<400> 77
ccacugcuac cacaaguuun n 21
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИНСЕКТИЦИДНЫЕ БЕЛКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2740312C2 |
| ГЛИКОЗИЛИРОВАННЫЕ СЛИТЫЕ БЕЛКИ VWF С УЛУЧШЕННОЙ ФАРМАКОКИНЕТИКОЙ | 2017 |
|
RU2782212C2 |
| ТЕРАПЕВТИЧЕСКИЕ СЛИТЫЕ БЕЛКИ | 2020 |
|
RU2825292C1 |
| ТРИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ, ОТНОСЯЩИЕСЯ К НИМ СПОСОБЫ И ВАРИАНТЫ ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2822200C2 |
| ТЕРАПЕВТИЧЕСКИЕ СЛИТЫЕ БЕЛКИ | 2020 |
|
RU2837545C1 |
| АНТИТЕЛА, НАПРАВЛЕННЫЕ ПРОТИВ FC-РЕЦЕПТОР-ПОДОБНОГО БЕЛКА 5, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2774158C2 |
| РЕКОМБИНАНТНЫЕ БЕЛКИ С ДОМЕНАМИ CCN И СЛИТЫЕ БЕЛКИ | 2020 |
|
RU2825102C2 |
| ТРИСПЕЦИФИЧЕСКИЕ И/ИЛИ ТРИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ПРИМЕНЕНИЕМ КРОССОВЕРНОГО ФОРМАТА С ДВОЙНЫМ ВАРИАБЕЛЬНЫМ ДОМЕНОМ (CODV) ДЛЯ ЛЕЧЕНИЯ ИНФЕКЦИИ, ВЫЗВАННОЙ HIV | 2020 |
|
RU2820164C2 |
| ДИСПЛЕЙ ИНТЕГРАЛЬНОГО МЕМБРАННОГО БЕЛКА НА ВНЕКЛЕТОЧНЫХ ОБОЛОЧЕЧНЫХ ВИРИОНАХ ПОКСВИРУСА | 2017 |
|
RU2759846C2 |
| КОМПОЗИЦИИ И СПОСОБЫ СЕЛЕКТИВНОЙ ДЕГРАДАЦИИ БЕЛКА | 2018 |
|
RU2811700C2 |
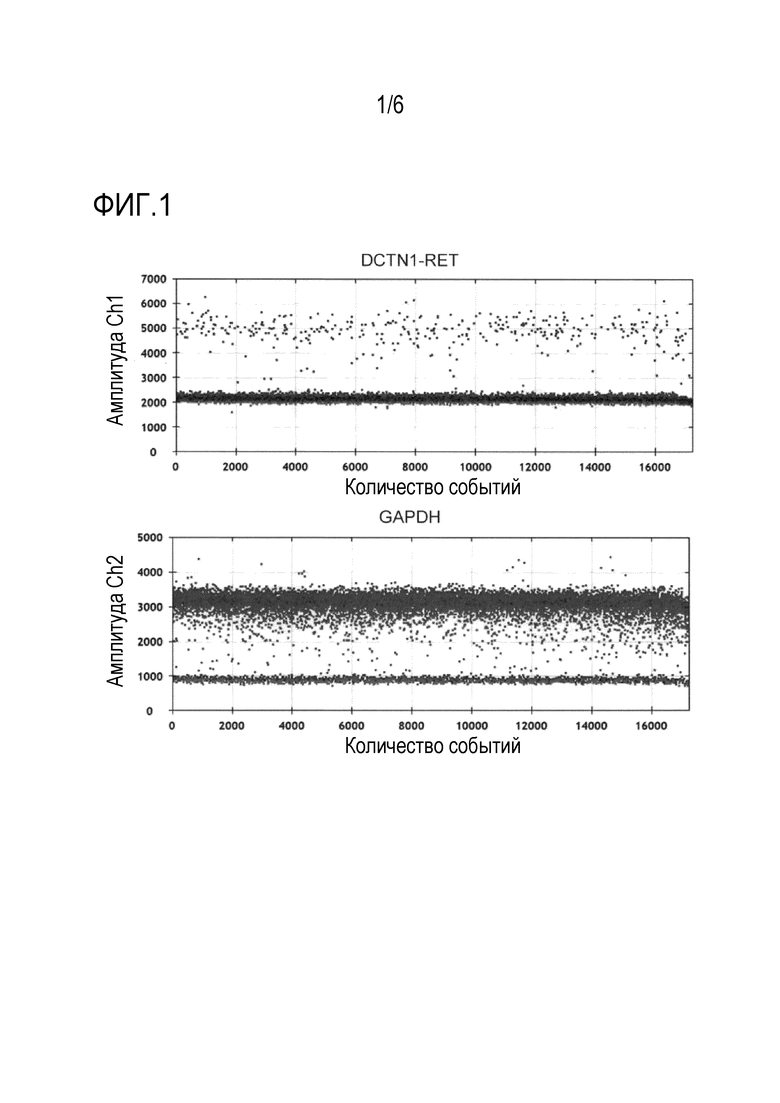

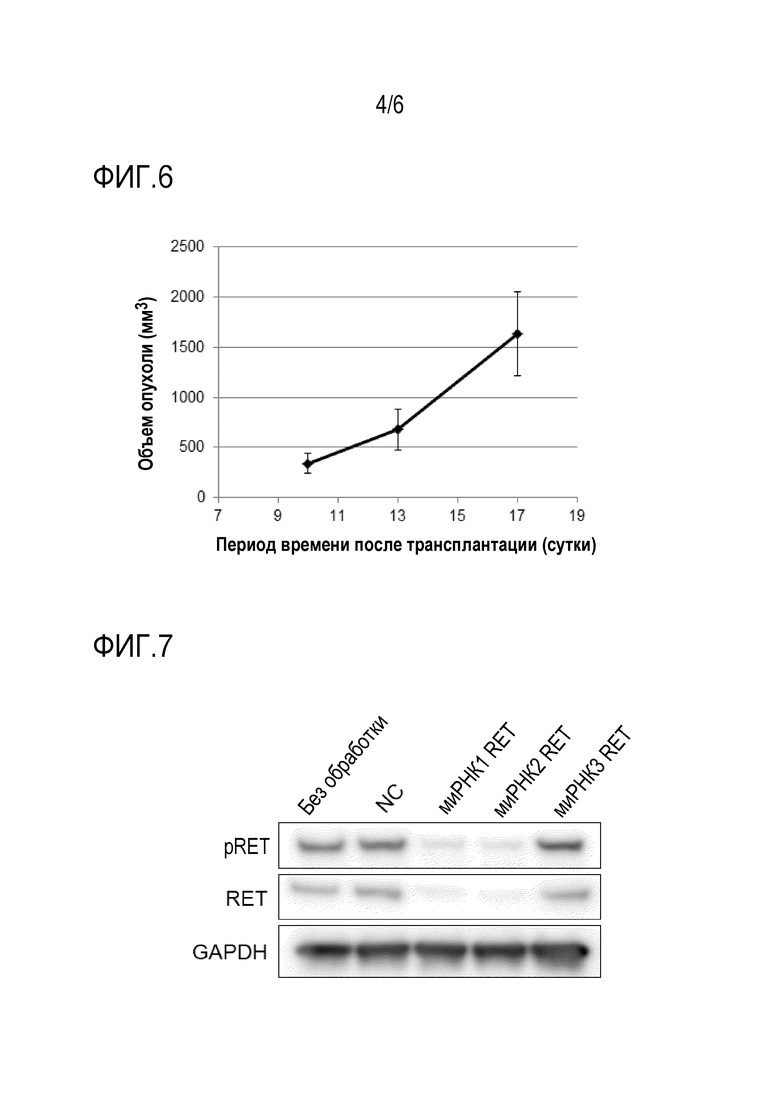
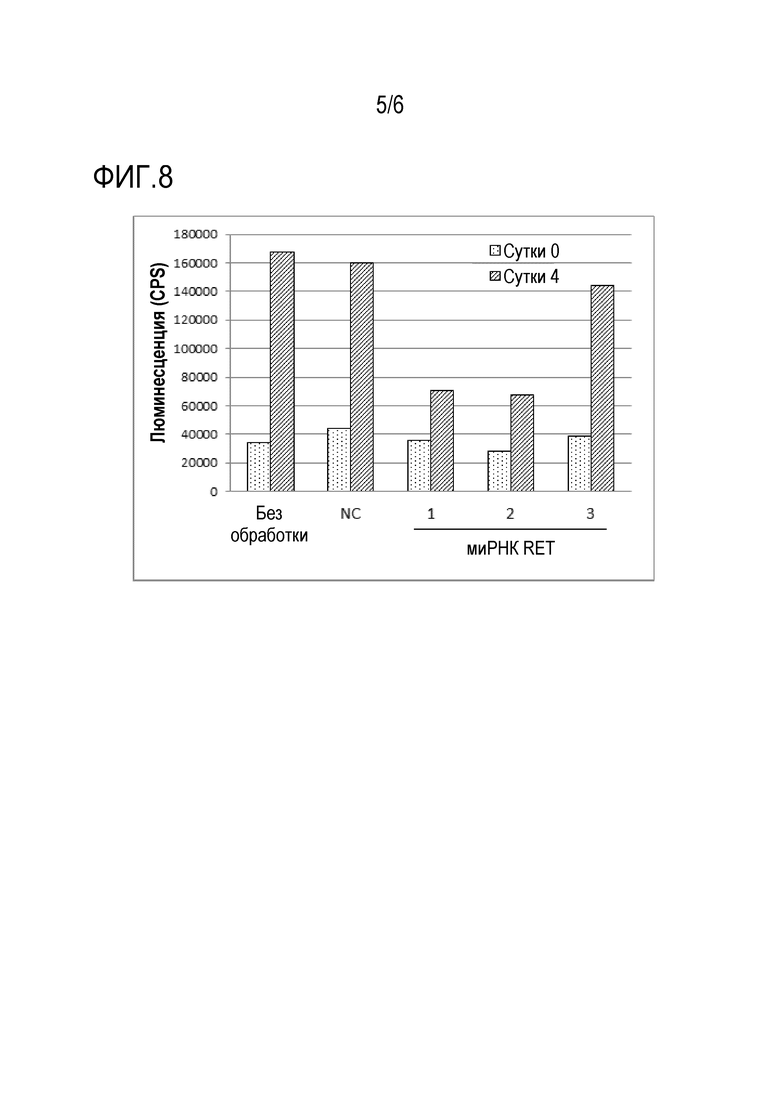
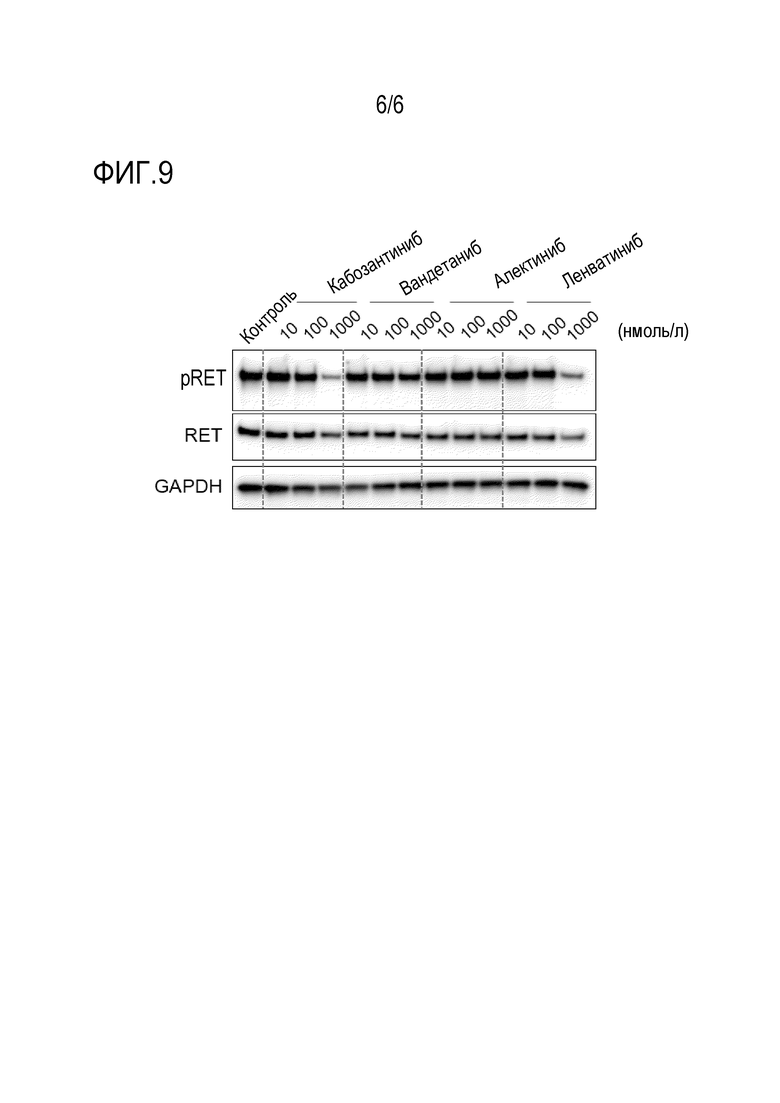
Настоящее изобретение относится к области биотехнологии, конкретно к идентификации аномалий в организме, ассоциированных с развитием злокачественных опухолей, и может быть использовано в медицине для диагностики и терапии злокачественных опухолей щитовидной железы. Идентифицирован слитый ген DCTN1-RET, состоящий из генов DCTN1 и RET, ассоциированный со злокачественной опухолью щитовидной железы. Предложены средства и способы для его детекции в организме, а также для скрининга соединений, направленных на ингибирование его экспрессии или активности кодируемого им слитого полипептида и терапию таких опухолей. Изобретение позволяет провести идентификацию и терапию злокачественной опухоли щитовидной железы, экспрессирующей слитый ген DCTN1-RET. 35 н. и 2 з.п. ф-лы, 9 ил., 7 табл., 9 пр.
1. Слитый полипептид, имеющий киназную активность и эффект вызова пролиферации клеток, в котором N-концевая часть белка DCTN1 слита с C-концевой частью белка RET, выбранный из следующих (a) или (b):
(a) полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 18; или
(b) полипептид, содержащий аминокислотную последовательность, где от 1 до 10 аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 18.
2. Полинуклеотид, кодирующий слитый полипептид по п.1.
3. Полинуклеотид по п.2, выбранный из следующих (d) и (e):
(d) полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 18;
(e) полинуклеотид, кодирующий полипептид, содержащий аминокислотную последовательность, где от 1 до 10 аминокислот заменены, делетированы или добавлены в аминокислотной последовательности, представленной SEQ ID NO: 18.
4. Полинуклеотид по п.2, выбранный из следующих (g) и (h):
(g) полинуклеотид, содержащий последовательность оснований, представленную SEQ ID NO: 17;
(h) полинуклеотид, гибридизующийся в строгих условиях с полинуклеотидом, содержащим последовательность оснований, комплементарную последовательности оснований, представленной SEQ ID NO: 17.
5. Экспрессирующий вектор, содержащий полинуклеотид по любому из пп. 2-4.
6. Клетка, трансфицированная экспрессирующим вектором по п.5, причем клетка экспрессирует полипептид по п.1.
7. Способ детекции присутствия слитого полипептида по п.1 в образце, включающий следующие стадии:
1) контактирование образца с антителом, которое специфически связывается с полипептидом, содержащим точку слияния N-концевой части белка DCTN1 и C-концевой части белка RET в слитом полипептиде по п.1;
2) детекция антитела, слитого с образцом.
8. Праймер для детекции присутствия полинуклеотида по любому из пп. 2-4 в образце, причем праймер содержит полинуклеотид, представленный (i) SEQ ID NO: 69.
9. Зонд для детекции присутствия полинуклеотида по любому из пп. 2-4 в образце, причем зонд содержит полинуклеотид, представленный (iii) SEQ ID NO: 71.
10. Способ детекции присутствия полинуклеотида по любому из пп. 2-4 в образце, включающий следующие стадии:
1) контактирование образца с праймером по п.8 или зондом по п.9;
2) детекция праймера/зонда, гибридизованного с образцом.
11. Способ диагностики злокачественной опухоли щитовидной железы у пациента, включающий детекцию слитого полипептида по п.1 в образце, полученном от пациента, посредством способа детекции по п.7, причем
когда присутствие слитого полипептида по п.1 детектируют в образце, полученном от пациента, тогда пациента диагностируют как пациента со злокачественной опухолью.
12. Способ диагностики злокачественной опухоли щитовидной железы у пациента, включающий детекцию полинуклеотида по любому из пп. 2-4 в образце, полученном от пациента, посредством способа детекции по п.10, причем
когда присутствие полинуклеотида по любому из пп. 2-4 детектируют в образце, полученном от пациента, тогда пациента диагностируют как пациента со злокачественной опухолью.
13. Способ скрининга соединения, ингибирующего экспрессию и/или активность слитого полипептида по п.1, включающий следующие стадии (1) и (2):
(1) стадию приведения слитого полипептида по п.1 в контакт с тестируемым соединением; и
(2) стадию измерения того, ингибирована ли активность слитого полипептида по п.1 на стадии (1).
14. Способ скрининга соединения, ингибирующего экспрессию и/или активность слитого полипептида по п.1, включающий следующие стадии (1) и (2):
(1) стадию приведения клетки, экспрессирующей слитый полипептид по п.1, в контакт с тестируемым соединением; и
(2) стадию измерения того, ингибирован ли рост клетки на стадии (1).
15. Способ скрининга соединения, ингибирующего экспрессию и/или активность слитого полипептида по п.1, включающий следующие стадии (1) и (2):
(1) стадию приведения клетки по п.6 в контакт с тестируемым соединением; и
(2) стадию измерения того, ингибирован ли рост клетки на стадии (1).
16. Способ скрининга соединения, ингибирующего экспрессию полинуклеотида по любому из пп. 2-4, включающий следующие стадии (1) и (2):
(1) стадию приведения клетки по п.6 в контакт с тестируемым соединением; и
(2) стадию измерения того, ингибирована ли экспрессия полинуклеотида по любому из пп. 2-4 в указанной клетке на стадии (1), или ингибирован ли рост указанной клетки на стадии (1).
17. Способ с использованием слитого полипептида по п.1 в качестве индикатора для определения того, является ли эффективной химиотерапия с использованием соединения, ингибирующего RET,
включающий определение того, что химиотерапия с использованием соединения, ингибирующего RET, является эффективной, когда слитый полипептид по п.1 детектируют в образце посредством способа детекции по п.7.
18. Способ с использованием полинуклеотида по любому из пп. 2-4 в качестве индикатора для определения того, является ли эффективной химиотерапия с использованием соединения, ингибирующего RET,
включающий определение того, что химиотерапия с использованием соединения, ингибирующего RET, является эффективной, когда присутствие полинуклеотида по любому из пп. 2-4 детектируют в образце посредством способа детекции по п.10.
19. Биомаркер для детекции злокачественной опухоли щитовидной железы, экспрессирующей слитый ген DCTN1-RET, содержащий полинуклеотид по любому из пп. 2-4.
20. Биомаркер для детекции злокачественной опухоли щитовидной железы, экспрессирующей слитый ген DCTN1-RET, содержащий слитый полипептид по п.1.
21. Способ лечения злокачественной опухоли щитовидной железы, которая является положительной в отношении экспрессии полинуклеотида по любому из пп. 2-4, включающий
проведение химиотерапии с использованием соединения, ингибирующего RET, для пациента со злокачественной опухолью, положительного в отношении экспрессии полинуклеотида по любому из пп. 2-4.
22. Способ лечения злокачественной опухоли щитовидной железы, которая является положительной в отношении слитого полипептида по п.1, включающий
проведение химиотерапии с использованием соединения, ингибирующего RET, для пациента со злокачественной опухолью, положительного в отношении экспрессии слитого полипептида по п.1.
23. Способ лечения злокачественной опухоли щитовидной железы, которая является положительной в отношении экспрессии гена полинуклеотида по любому из пп. 2-4, включающий
детекцию присутствия полинуклеотида по любому из пп. 2-4 в образце, полученном от тестируемого субъекта, и
проведение химиотерапии с использованием соединения, ингибирующего RET, для тестируемого субъекта, когда детектировано присутствие полинуклеотида по любому из пп. 2-4.
24. Способ лечения злокачественной опухоли щитовидной железы, которая является положительной в отношении экспрессии слитого полипептида по п.1, включающий
детекцию присутствия слитого полипептида по п.1 в образце, полученном от тестируемого субъекта, и
проведение химиотерапии с использованием соединения, ингибирующего RET, для тестируемого субъекта, когда детектировано присутствие слитого полипептида по п.1.
25. Применение соединения, ингибирующего RET, для лечения пациента со злокачественной опухолью щитовидной железы, положительного в отношении экспрессии гена полинуклеотида по любому из пп. 2-4.
26. Применение соединения, ингибирующего RET, для лечения пациента со злокачественной опухолью щитовидной железы, положительного в отношении экспрессии слитого полипептида по п.1.
27. Применение соединения, ингибирующего RET, для получения фармацевтической композиции для лечения злокачественной опухоли щитовидной железы для лечения пациента со злокачественной опухолью, положительного в отношении экспрессии гена полинуклеотида по любому из пп. 2-4.
28. Применение соединения, ингибирующего RET, для получения фармацевтической композиции для лечения злокачественной опухоли щитовидной железы для лечения пациента со злокачественной опухолью, положительного в отношении экспрессии слитого полипептида по п.1.
29. Способ получения реагента для определения того, является ли химиотерапия с использованием соединения, ингибирующего RET, эффективной, с использованием средств для детекции присутствия слитого полипептида по п.1 в образце, и/или средств для детекции присутствия полинуклеотида по любому из пп. 2-4 в образце.
30. Комбинация антитела к DCTN1 и антитела к RET для детекции присутствия слитого полипептида по п.1.
31. Применение комбинации антител по п.30 в получении детектирующего реагента для детекции присутствия слитого полипептида по п.1.
32. Применение праймера по п.8 или зонда по п.9 для получения детектирующего реагента для детекции присутствия полинуклеотида по любому из пп. 2-4.
33. Применение соединения, ингибирующего RET, в лечении злокачественной опухоли щитовидной железы, где лечение осуществляют способом, включающим
проведение химиотерапии с использованием соединения, ингибирующего RET, для пациента со злокачественной опухолью, положительного в отношении экспрессии гена полинуклеотида по любому из пп. 2-4.
34. Применение соединения, ингибирующего RET, в лечении злокачественной опухоли щитовидной железы, где лечение осуществляют способом, включающим
проведение химиотерапии с использованием соединения, ингибирующего RET, для пациента со злокачественной опухолью, положительного в отношении экспрессии слитого полипептида по п.1.
35. Применение соединения, ингибирующего RET, в лечении злокачественной опухоли щитовидной железы, где лечение осуществляют способом, включающим
детекцию присутствия слитого полипептида по п.1 в образце, полученном от тестируемого субъекта, и
проведение химиотерапии с использованием соединения, ингибирующего RET, для тестируемого субъекта, когда детектировано присутствие слитого полипептида по п.1.
36. Применение соединения, ингибирующего RET, в лечении злокачественной опухоли щитовидной железы, где лечение осуществляют способом, включающим
детекцию присутствия полинуклеотида по любому из пп. 2-4 в образце, полученном от тестируемого субъекта, и
проведение химиотерапии с использованием соединения, ингибирующего RET, для тестируемого субъекта, когда детектировано присутствие полинуклеотида по любому из пп. 2-4.
37. Применение полинуклеотида, представленного (i) SEQ ID NO: 69 или (ii) SEQ ID NO: 70, в получении праймера для детекции присутствия полинуклеотида по любому из пп. 2-4 в образце.
| WO 2014130975 A1, 28.08.2014, VAUGHAN K | |||
| T | |||
| et al., Cytoplasmic dynein binds dynactin through a direct interaction between the intermediate chains and p150Glued, The Journal of cell biology, 1995, v.131, n | |||
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| и др., ПЕРЕСТРОЙКИ ПРОТООНКОГЕНА C-RET В КЛЕТКАХ ПАПИЛЛЯРНОГО РАКА ЩИТОВИДНОЙ ЖЕЛЕЗЫ, Вестник Витебского | |||
Авторы
Даты
2024-02-21—Публикация
2018-08-20—Подача