Изобретение относится к области молекулярной биологии и генетическим исследованиям в селекции и пушном звероводстве.
Уровень техники
Американская норка является основным объектом клеточного пушного звероводства [Andersson, Nyman, Wallgren, 2017; Hansen, 2014], который характеризуется значительным разнообразием окрасочных форм. Среди 35 мутаций доминантной, полудоминантной и рецессивной природы, затрагивающих окраску волосяного покрова [Трапезов, Трапезова, 2009], выделяется группа мутаций, обуславливающих развитие коричневых окрасок меха. На сегодняшний день у американской норки описано 12 коричневых окрасок меха, которые формируют широкий спектр от светло-коричневой (Американский паломино (k/k)), до шоколадной (мойл (m/m)) и стандартной темно-коричневой (+/+) окрасок. Большинство коричневых окрасок меха у норки наследуются как моногенные аутосомно-рецессивные признаки, и характеризуются значительным сходством, что существенно затрудняет селекционную работу с ними [Robinson, 1975; Cirera и др., 2016], поскольку для определения статуса носительства той или иной мутации, обуславливающей развитие одной из коричневых окрасок мехового покрова, требуется проводить серии случайных скрещиваний, анализировать полученное потомство и составлять родословные животных, что является трудоемким и дорогостоящим процессом. В связи с этим целесообразным является разработка более быстрых и менее трудоемких методов, позволяющих вести отбор тех особей и популяций норки, которые будут размножаться и давать наибольшее число потомков с желаемой окраской мехового покрова.
На сегодняшний день с молекулярно-генетической точки зрения описаны по меньшей мере три мутации, приводящие к формированию коричневой окраски меха у американской норки:
- Окраска Американский паломино (k/k), характеризующаяся бледно-коричневым мехом и розовыми или красными глазами. Данная окраска наследуется как моногенный аутосомно-рецессивный признак и, как было показано ранее, обуславливается мутацией в гене TYRP1, кодирующем один из ключевых ферментов, вовлеченных в меланогенез [Cirera и др., 2016].
- Окраски мойл (m/m) и камео (mc/mc) - светло-коричневые окраски, которые наследуются как моногенный аутосомно-рецессивный признак. Согласно результатам гибридологического анализа, было описано 3 аллеля: мойл (m), камео (mc) и стандартный тип (+). Аллель камео обуславливает более темную окраску по сравнению с мойл и, видимо, доминирует на ним (+>mc>m) [Robinson, 1975]. Ранее было показано, что развитие этих окрасок мехового покрова, обуславливается мутациями в гене RAB38, кодирующем Ras-подобный белок, который обеспечивает транспорт вновь синтезированных ферментов тирозиназы и TYRP1, являющихся ключевыми ферментами для биосинтеза меланина, из эндосом в меланосомы [Manakhov и др., 2020].
В случае если генетические маркеры животных-носителей данных мутаций неизвестны, носительство определяют, используя генеалогический метод - это метод изучения родословных, с помощью которого прослеживается наследование признака в группе с указанием типа родственных связей между членами родословной [Инге-Вечтомов, 2010]. Молекулярно-генетические маркеры для животных, являющихся носителями признака пастель до настоящего времени не были известны.
Окраска пастель или королевская пастель (b/b) также наследуется как аутосомно-рецессивный признак. Особи с окраской пастель характеризуются осветленной, по сравнению со стандартной темно-коричневой, окраской меха. Пигментация носа, губ и когтей также светлее. Цвет глаз может варьироваться от светло-коричневого до немного светлее стандартного [Robinson, 1975].
Технической задачей, на решение которой направлено настоящее изобретение, является решение как минимум одной из вышеуказанных в уровне техники проблем.
Сущность изобретения
Техническим решением является использование описанного признаками в пунктах формулы изобретения.
Одной из возможных технических задач, на решение которой может быть направлено настоящее изобретение, являлось создание способа отбора особей американской норки для получения желаемой окраски меха на основе определения генотипа отдельных особей норки. Техническим результатом является более быстрый и менее трудоемкий способ отбора особей американской норки, носителей генетического маркера окраски меха пастель.
Задача решена тем, что впервые выявлен новый генетический маркер, определяющий окраску меха норок. Было обнаружено, что инсерция эндогенного ретровирусного элемента (ERV1) в первом интроне гена HPS3 (NC_058096.1:g.75137871_75137872ins[OR863243];75137865_75137871, далее HPS3ins), определяет окраску меха пастель: особи, гомозиготные по данной мутации (аллели b/b), имеют светло-коричневую окраску, в то время как особи, гетерозиготные по данной мутации (аллели +/b), характеризуются стандартной темно-коричневой окраской меха.
В одном из вариантов осуществления изобретения предложен генетический маркер окраски меха американской норки HPS3ins, в гомозиготном состоянии определяющий окраску меха пастель, представляющий собой инсерцию последовательности OR863243 (SEQ ID NO: 8) в позиции NC_058096.1:g.75137871_75137872ins[OR863243]; 75137865_75137871.
В другом варианте осуществления изобретения предложен способ выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, включающий генотипирование животного по гену HPS3, а именно выявление наличия и количества или отсутствия генетического маркера HPS3ins.
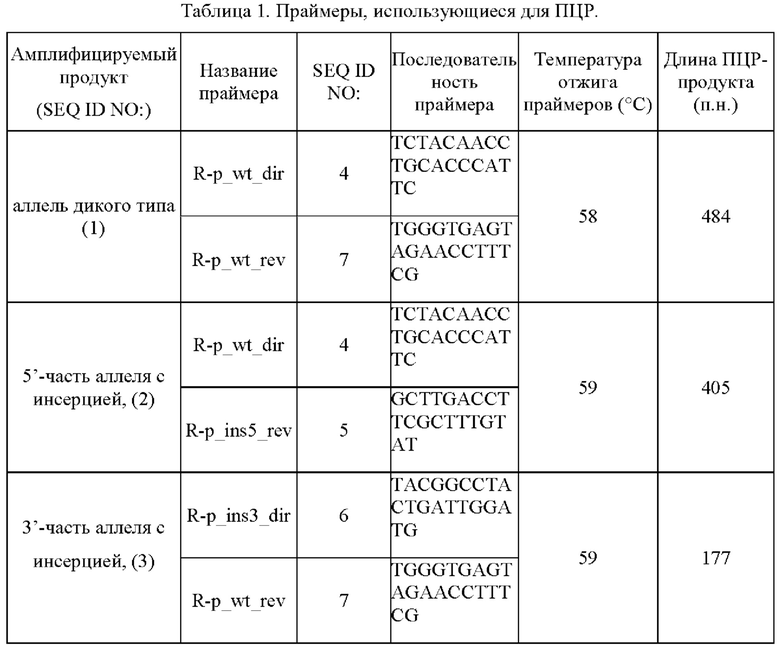
В одном из вариантов осуществления изобретения наличие и количество или отсутствие генетического маркера по п. 1 определяют путем амплификации ДНК с использованием праймеров SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7.
В одном из вариантов осуществления изобретения выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят секвенированием по методу Сенгера.
В одном из вариантов осуществления изобретения выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят секвенированием NGS.
В других вариантах осуществления изобретения выявление наличия или отсутствия однонуклеотидной замены в последовательности ДНК проводят методом ПЦР-ПДРФ, TaqMan Real-Time PCR, аллель-специфической ПЦР, гибридизации на чипах.
В одном из вариантов осуществления изобретения предлагается применение упомянутого способа выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, для отбора особей американской норки среди имеющегося поголовья фермерских норок.
В одном из вариантов осуществления изобретения предлагается упомянутое применение для отбора особей, где отобранных животных используют для получения от них потомства с заданной окраской меха.
Предложенные в настоящей группе изобретений генетический маркер и способ выявления особи норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, позволяет быстро определить особи норки, обладающие искомыми аллелями, и спрогнозировать окраску меха их потомства, не прибегая к генеалогическому методу, требующему многолетних скрещиваний.
Детальное описание изобретения
Если не указано иначе, предполагается, что все термины, обозначения и другие научные термины, используемые в данной заявке, имеют значения, которые обычно понимают специалисты в области, к которой относится настоящее изобретение. В некоторых случаях определения терминов с общепринятыми значениями приведены в данной заявке для ясности и/или для быстрой справки и понимания, и включение таких определений в настоящее описание не должно истолковываться как наличие существенного отличия значения термина от обычно подразумеваемого в данной области.
Кроме того, если по контексту не требуется иное, термины в единственном числе включают в себя термины во множественном числе, и термины во множественном числе включают в себя термины в единственном числе. Как правило, используемая классификация и методы молекулярной биологии, генетики, аналитической химии, а также гибридизации и химии белка и нуклеиновых кислот, описанные в настоящем документе, хорошо известны специалистам и широко применяются в данной области. Ферментативные реакции и способы очистки осуществляют в соответствии с инструкциями производителя, как это обычно осуществляется в данной области, или как описано в настоящем документе.
Изобретение раскрывает новый генетический маркер, а именно специфическую мутацию в гене HPS3, кодирующем HPS3 субъединицу белкового комплекса BLOC2, а также способ идентификации генов, вовлеченных в регуляцию формирования окраски меха у американской норки Neogale vison. Авторы изобретения обнаружили, что выявленный генетический маркер связан с развитием окраски меха пастель (b/b) у американской норки. Предложен способ отбора животных, являющихся носителем указанного маркера, и применение данного способа в пушном звероводстве для отбора производителей, обладающих генетическими характеристиками, необходимыми для получения потомства желаемой цветовой окраски. Возможность определения наличия или отсутствия данных маркеров в образце ДНК животного методами молекулярной биологии показана на примере их выявления путем проведения ПНР-анализа и последующего секвенирования биологических образцов американской норки. Раскрыт способ выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование желаемой цветовой вариации, включающий генотипирование животного для выявления у него маркерной полиморфной нуклеотидной вариации, ассоциированной с наличием у животного аллеля, проявляющегося в развитии определенной цветовой вариации. Предложена в качестве маркера нуклеотидная вариация в геноме, ассоциированная с изменением физиологической активности белка HPS3. Для выявления данной вариации проводят генотипирование полиморфного локуса в гене HPS3. В качестве маркера наличия в генотипе животного аллеля b используют инсерцию HPS3ins.
Изобретение относится также к мутации HPS3ins в гене HPS3, которая, предположительно приводит к нарушению сплайсинга и/или снижению уровня экспрессии гена HPS3 за счет гиперметилирования эндогенного ретровирусного элемента, распространяющегося в том числе и на область CpG-островка гена HPS3 (NC_058096.1:75137188-75137976). Авторы изобретения показали, что ген HPS3 у норки вовлечен в процессы определения окраски мехового покрова. В частности, авторы изобретения обнаружили, что инсерция HPS3ins, определяет окраску меха пастель: особи, гомозиготные по данной мутации (аллели b/b), имеют светло-коричневую окраску, в то время как особи, гетерозиготные по данной мутации (аллели +/b), характеризуются стандартной темно-коричневой окраской меха. Авторы изобретения впервые показали ассоциацию физиологической активности белка HPS3 с окраской мехового покрова американской норки, что и послужило основой создания нового способа отбора животных, являющих носителями аллелей, обеспечивающих определенную окраску, а также их число. Раскрыт новый генетический маркер HPS3ins, который может быть использован для генотипирования животных с целью выявления животных - носителей аллелей определенной цветовой вариации. Отбор таких особей является актуальной задачей для промышленного разведения американской норки, так как знание о генотипе особей позволяет подбирать их в скрещивание таким образом, чтобы в потомстве получалась максимальная доля животных с окраской меха, необходимой звероводу.
Открытие инсерции HPS3ins в первом интроне гена HPS3, позволило авторам изобретения разработать способ, позволяющий предсказывать цвет норки и ее потомства на основе различения аллелей b и + и определения генотипа отдельных особей американской норки по инсерции HPS3ins, обуславливающей развитие окраски меха пастель. В соответствии с настоящим изобретением предлагается способ отбора особей для получения желаемой окраски меха на основе определения генотипа отдельных особей норки.
Эффективность производства меха заданной окраски и разведения соответствующих линий животных может быть повышена, при возможности определения до скрещивания, является ли животное носителем мутации, влияющей на окраску мехового покрова. Используя данную информацию, селекционер может планировать скрещивания животных, увеличивая, таким образом, долю потомства с интересующей его окраской мехового покрова, что в конечном итоге приведет к уменьшению расходов звероводческой организации. В качестве инструмента для отбора животных предложено использовать мутацию в гене HPS3 (HPS3ins), которая обуславливает развитие окраски пастель:
- особи гомозиготные по данной мутации (аллели b/b) имеют светло-коричневую окраску;
- особи, гетерозиготные по данной мутации (аллели +/b) характеризуются стандартной темно-коричневой окраской меха.
Изобретение раскрывает также применение заявленного способа для селекции и разведения фермерской американской норки по желательному признаку (для получения особей с заданной окраской мехового покрова). Применение включает в себя отбор особей, генетически соответствующих желательному признаку (заданной окраске меха) среди имеющегося поголовья фермерских норок для использования в направленных селекционных программах (получение потомства с заданной окраской меха). Применение заявленного способа для отбора норок для селекции также может быть использован для защиты от генетической кражи селекционных линий американских норок определенной окраски.
Авторы изобретения провели полногеномное секвенирование и последующее сравнение геномов американских норок, характеризующихся различными типами окраски мехового покрова: стандартной темно-коричневой (дикий тип, +/+), серебристо-голубой (аллели р/р), мойл (аллели m/m), виолет (аллели а/а m/m р/р), Шедоу серебристо-голубой (аллели Sh/+р/р), а также особей из линии, характеризующейся окраской меха пастель (аллели b/b), последние происходили из двух неродственных популяций норок, содержащихся на экспериментальной звероферме ИЦиГ СО РАН и зверохозяйстве «Мермерины».
У животных с окраской меха пастель была обнаружена инсерция эндогенного ретровирусного элемента (ERV1) в первом интроне гене HPS3. Было показано, что выявленная инсерция в гене HPS3 (HPS3ins), в гомозиготном состоянии обуславливает развитие окраски меха пастель (b/b), характеризующейся светло-коричневой окраской мехового покрова.
Таким образом, настоящее изобретение относится, прежде всего, к использованию обнаруженной мутации в гене HPS3 в качестве маркера, позволяющего определить наличие или отсутствие у животного аллелей b, а также их числа. В одном из вариантов осуществления изобретения предложен генетический маркер окраски меха американской норки HPS3ins, в гомозиготном состоянии определяющий окраску меха пастель, представляющий собой инсерцию последовательности OR863243 (SEQ ID NO: 8) в позиции NC_058096.1:g.75137871_75137872ins[OR863243]; 75137865_75137871.
Изобретение относится к способу выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, включающий генотипирование животного по гену HPS3, а именно выявление наличия и количества или отсутствия генетического маркера HPS3ins.
Фрагмент ДНК исследуемой особи получается любым известным из уровня техники способом взятия биологического материала, содержащего ДНК, в том числе, но не ограничиваясь, такими способами как взятие крови, мазка, биопсии и пр. способами. Такие способы известны специалисту в данной области.
Специалисту в данной области понятно, что для осуществления генотипирования по инсерции HPS3ins может быть использован любой известный в настоящее время способ определения нуклеотидной последовательности ДНК и/или наличия определенной мутации (секвенирование по Сэнгеру, NGS, аллель-специфичная ПЦР, ПЦР-ПДРФ, TaqMan Real-Time PCR и др.).
В одном из вариантов осуществления изобретения генотипирование проводится с использованием фрагмента ДНК исследуемой особи американской норки, содержащего сайт инсерции HPS3ins, полученного путем амплификации ДНК исследуемой особи американской норки с использованием праймеров SEQ ID NO: 4 и SEQ ID NO: 7, SEQ ID NO: 4 и SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7. В результате такой амплификации могут образоваться фрагменты ДНК с последовательностями SEQ ID NO: 1 (аллель дикого типа) SEQ ID NO: 2 (5'-часть аллеля с инсерцией) и SEQ ID NO: 3 (3'-часть аллеля с инсерцией). При дальнейшем анализе продуктов амплификации можно сделать вывол, что особь американской норки, у которой амплифицируются только продукты SEQ ID NO: 2 и SEQ ID NO: 3, является носителем генетического маркера HPS3ins в каждом аллеле, а особь, у которой амплифицируются все три продукта, является носителем одного аллеля генетического маркера HPS3ins. Наличие только продукта SEQ ID NO: 1 говорит об отсутствии маркера окраски меха пастель у особи американской норки.
Изобретение также относится к применению упомянутого способа выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование желаемой цветовой вариации, для отбора особей американской норки, пригодных в племенной работе для получения желаемой окраски меха, среди имеющегося поголовья фермерских норок.
В одном из вариантов осуществления изобретения предлагается упомянутое применение для отбора особей, где отобранных животных используют для получения от них потомства с желаемой окраской меха.
В настоящее время аналогов представляемым маркерам и способу их использования для определения окраски меха пастель у американской норки не существует.
Ниже представлены примеры проведения генотипирования животных по мутациям инсерции HPS3ins (b), а также применение заявленного способа в селекции.
Пример 1. Генотипирование биологического образца
Пример 1.1. Получение ДНК
ДНК может быть получена из любой ткани животного, содержащей клеточные ядра, например, из лейкоцитов, волосяных фолликулов, ткани уха и мышечной ткани, клеток буккального эпителия и др. Пригодная для проведения ПЦР ДНК может быть выделена из образца при помощи наборов QIAamp DNA Mini Kit компании Qiagen (http://www.qiagen.com/). Однако любой метод выделения ДНК должен быть в равной степени эффективным.
Пример 1.2. Амплификация участков генома американской норки, содержащих исследуемую инсерцию
Амплификацию полученного в Примере 1.1. образца ДНК проводили с помощью наборов GenPack PCR Core компании «Лаборатория Изоген» (Россия), на приборах GeneAmp PCR System 9700 Thermal Cycler компании «Applied Biosystems» (США) no следующей программе: 94°C - 5 мин; (94°C - 30 сек; 58/59°C - 25 сек; 72°C - 20 сек) * 35 циклов; 72°C - 10 мин. Реакционная смесь объемом 20 мкл содержала: 10 мкл PCR Diluent, по 0,5 пмоль прямого и обратного праймеров (Таблица 1), 10 нг геномной ДНК.
Пример 1.3. Секвенирование полученных ПЦР продуктов
Полученные в результате ПЦР фрагменты очищали с использованием наборов Cleanup компании «Евроген» (Россия) в соответствии с инструкцией производителя.
Пробоподготовку для секвенирования проводили с помощью набора реагентов BigDye® Terminator v3.1 Cycle Sequencing Kit компании «Applied Biosystems» (США), реакционная смесь объемом 10 мкл содержала: 2 мкл BigDye™ Terminator v3.1 Ready Reaction Mix, 1 мкл BigDye™ Terminator v3.1 5X Sequencing Buffer, 0,1 пмоль прямого или обратного праймера (Таблица 1), и очищенный продукт ПЦР.
Полученные продукты реакции очищали на колонках DyeEx 2.0 Spin Kit («Qiagen», Германия) и секвенировали на приборе ABI PSISN 3730x1 компании «Applied Biosystems» (США).
Пример 1.4. Анализ полученных нуклеотидных последовательностей
Анализ проводили с использованием программ Chromas, GeneDoc и веб-сервиса Clustal Omega.
Проводили выравнивание и сравнение полученных нуклеотидных последовательностей исследуемых особей с референсными нуклеотидными последовательностями гена HPS3 (Gene ID: 122908591, NCBI) последовательностью HPS3ins (OR863243) американской норки.
Пример 1.5. Определение принадлежности исследуемых животных к линии пастель
Условные обозначения:
- Аллель «В» - отсутствует инсерция HPS3ins
- Аллель b - имеется инсерция HPS3ins
Была взята случайная выборка из 7 норок со светло-коричневой окраской мехового покрова. С целью выявления особей американской норки, являющихся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель было проведено генотипирование указных светло-коричневых животных по мутациям HPS3ins в ходе которого были получены следующие результаты (Таблица 2)
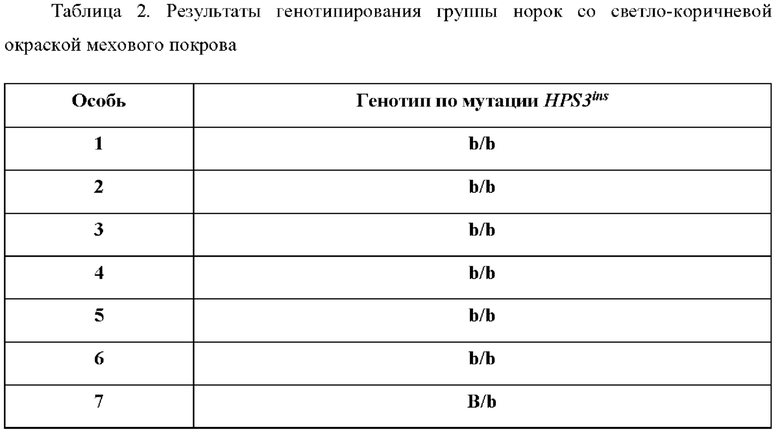
По результатам генотипирования можно сделать вывод, что образцы 1-6 действительно принадлежат к линии животных с окраской пастель, в то время как образец 7 не относится к ней и его светло-коричневая окраска обусловлена другой мутацией.
Пример 2. Применение способа для отбора особей норки в селекционной работе
Условные обозначения:
- Аллель «В» - отсутствует инсерция HPS3ins
- Аллель b - имеется инсерция HPS3ins
Предположим, имеется популяция норок со стандартной темно-коричневой окраски. В результате проведенного генотипирования по инсерции HPS3ins было выяснено, что 50% популяции гетерозиготы по аллелю с инсерцией HPS3ins (В/b), 50% популяции гомозиготы по аллелю без инсерции HPS3ins (В/В).
В случае случайного скрещивания в потомстве, в соответствии с законами Менделя [Инге-Вечтомов, 2010], ожидается следующее распределение по окраскам и генотипам:
- 6,25% животных со светло-коричневой окраской пастель (b/b)
- 93,75% животных со стандартной окраской мехового покрова (56,52% В/В; 37,50% В/b)
В случае целенаправленного скрещивания животных с генотипом В/b в потомстве от таких скрещиваний ожидается следующее распределение по окраскам и генотипам:
- 50% животных с окраской пастель (b/b)
- 50% животных со стандартной окраской мехового покрова (В/b)
Таким образом, применение предложенного способа определения окраски меха американской норки, с использованием молекулярных маркеров ДНК, может существенно увеличить долю животных с заданной окраской в потомстве. Способ позволяет определить генотип норок, который позволяет проводить скрещивание таким образом, чтобы получить максимальное количество животных заданной окраски, например, вплоть до 50% животных с окраской пастель. В случае случайного скрещивания, их доля составит 6,25% (расчеты проводились на основании частот генотипов и в соответствии с законами расщепления Менделя). Предложенный способ позволяет быстро определить особи норки, обладающие искомыми аллелями, и спрогнозировать окраску меха их потомства, не прибегая к генеалогическому методу, требующему многолетних скрещиваний.
Все публикации, патенты и заявки на патенты включены в настоящий документ посредством ссылки. Хотя в вышеприведенном описании это изобретение было описано в отношении некоторых предпочтительных вариантов его осуществления, и многие детали были изложены в целях иллюстрации, для специалистов в данной области техники будет очевидно, что изобретение допускает дополнительные варианты осуществления и что некоторые детали, описанные в данном документе, могут значительно изменяться без отклонения от сущности изобретения.
Использование терминов в единственном числе в контексте описания изобретения должно толковаться как охватывающее как единственное, так и множественное число, если иное не указано в данном документе или явно не противоречит контексту. Термины «состоящий из», «имеющий», «включающий» и «содержащий» следует толковать как неограничивающие термины, т.е. означающие «включая, но не ограничиваясь», если не указано иное. Перечисление диапазонов значений в данном документе просто предназначено для использования в качестве сокращенного способа индивидуальной ссылки на каждое отдельное значение, попадающее в этот диапазон, если здесь не указано иное, и каждое отдельное значение включено в спецификацию, как если бы оно было отдельно изложено в данном документе. Все способы, описанные в данном документе, могут выполняться в любом подходящем порядке, если иное не указано в данном документе или иным образом явно не противоречит контексту. Использование любых и всех примеров или иллюстративного языка (например, «такой как»), представленных в данном документе, предназначено просто для лучшего описания изобретения и не налагает ограничения на объем изобретения, если иное не заявлено. Никакие формулировки в описании не следует истолковывать как указывающие на какой-либо не заявленный элемент как существенный для практического применения изобретения.
Здесь описаны варианты осуществления этого изобретения, включая лучший из известных изобретателям способа осуществления изобретения. Разновидности этих вариантов осуществления могут стать очевидными для специалистов в данной области техники после прочтения предшествующего описания. Авторы ожидают, что квалифицированные специалисты будут использовать такие варианты в зависимости от обстоятельств, и авторы предполагают, что изобретение будет реализовано на практике иначе, чем конкретно описано в данном документе. Соответственно, это изобретение включает в себя все модификации и эквиваленты признаков, изложенных в прилагаемой формуле изобретения, как это разрешено действующим законодательством. Более того, любая комбинация вышеописанных признаков во всех их возможных вариациях охватывается изобретением, если иное не указано в данном документе или иным образом явно не противоречит контексту.
Заявитель просит рассмотреть представленные материалы заявки «Молекулярно-генетический маркер цветовой вариации пастель у американской норки и способ выявления особи норки, являющейся носителем аллеля, обуславливающего формирование желаемой цветовой вариации» на предмет выдачи патента на изобретение.
Список литературы
1. Инге-Вечтомов С.Г. Генетика с основами селекции: учебник для студентов высших учебных заведений. 2-е издание, перераб. и доп. // СПб.: Изд-во Н-Л, 2010. 720 с.
2. Трапезов О.В., Трапезова Л.И. Воспроизводящаяся коллекция окрасочных генотипов американской норки (Mustek vison Schreber, 1777) на экспериментальной звероферме Института цитологии и генетики СО РАН // Вестник ВОГиС. 2009. Т. 13. №3. С. 554-570.
3. Andersson А.-М., Nyman А.-K., Wallgren P. A retrospective cohort study estimating the individual Aleutian disease progress in female mink using a VP2 ELISA and its association to reproductive performance // Preventive Veterinary Medicine. 2017. T. 140. C. 60-66.
4. Cirera S. и др. A large insertion in intron 2 of the TYRP1 gene associated with American Palomino phenotype in American mink. // Mammalian genome: official journal of the International Mammalian Genome Society. 2016. T. 27. №3-4. С. 135-43.
5. Hansen H.O. The Global Fur Industry: Trends, Globalization and Specialization// Journal of Agricultural Science and Technology. 2014. T. 4. C. 543-551.
6. Manakhov A.D. и др. Genome analysis of American minks reveals link of mutations in Ras-related protein-38 gene to Moyle brown coat phenotype. // Scientific reports. 2020. T. 10. №1. C. 15876.
7. Robinson R. The American mink, mustela vison //Handbook of Genetics: Volume 4 Vertebrates of Genetic Interest. - Boston, MA: Springer US, 1975. - C. 367-398.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="2023135870_listing.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-12-28">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023135870</ApplicationNumberText>
<FilingDate>2023-12-28</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Автономная некоммерческая
образовательная организация высшего образования
"Научно-Технологический Университет
"Сириус"</ApplicantName>
<ApplicantNameLatin>Sirius University of Science and
Technology</ApplicantNameLatin>
<InventionTitle languageCode="ru">Молекулярно-генетический маркер
цветовой вариации пастель у американской норки и способ выявления
особи норки, являющейся носителем аллеля, обуславливающего
формирование желаемой цветовой вариации</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>477</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..477</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neogale vison</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctacaacctgcacccattcgggtcgcagcaggtggtgccctgcaaatt
ggagcccgagcggttctgcggcggagggcgcgacgcgctcttcgtggcggcaggctgcaaggtggaggca
ttcgccgtgaccggacaggagctgtgccagccgaggtgcgccttctccacgctcggcagggtgctgtgcc
tggcctacagcgaggccggtgagtagcagcgaggccagggggagcccgggtccccgcttggggcctcctt
cctagaggactcagcgtccgccgcggtactctgggtggggaagagactttccggaacttgctggagggtc
tcgcggggtctggctactgattactgaaggcgaggagagccgtgcggttgtcgtggcaaccacaggtgcg
cgctggcttctagggggcgggggacgctgtctgctcgctcttacctcgctcgctcgcccgaaaggttcta
ctcaccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>405</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..405</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neogale vison</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctacaacctgcacccattcgggtcgcagcaggtggtgccctgcaaatt
ggagcccgagcggttctgcggcggagggcgcgacgcgctcttcgtggcggcaggctgcaaggtggaggca
ttcgccgtgaccggacaggagctgtgccagccgaggtgcgccttctccacgctcggcagggtgctgtgcc
tggcctacagcgaggccggtgagtagcagcgaggccagggggagcccgggtccccgcttggggcctcctt
cctagaggactcagcgtccgccgcggtactctgggtggggaagagactttccggaacttgctggagggtc
tcgcggggtctggctactgattactgttagggtccgtgatcaaaggaacgagactgatacaaagcgaagg
tcaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>177</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..177</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neogale vison</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacggcctactgattggatgactccactcagtctgccccacccttacaa
ttactgaaggcgaggagagccgtgcggttgtcgtggcaaccacaggtgcgcgctggcttctagggggcgg
gggacgctgtctgctcgctcttacctcgctcgctcgcccgaaaggttctactcaccca</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctacaacctgcacccattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcttgaccttcgctttgtat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacggcctactgattggatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgggtgagtagaacctttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>11763</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11763</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neogale vison</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttagggtccgtgatcaaaggaacgagactgatacaaagcgaaggtcaag
caaagctttatttcgcgccaagcatcatgaatcgcactgaccggtcagggctttcttttataaaagatga
caactttgaccccggcgagcctactcccaatgaggttgctgacatctaggtcgtttctcactgtgtcact
gctgagggtcctggagtgcggggcagggccgctggcaccgcaggcgtcgagggaccgctgacaactggac
cgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagatgtctcaggaccgca
ggcgtcgagggaccgctgacaactggaccgcagacatcgagggaccgctgacaactggaccgcagacatc
gagggaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactgga
ccgcagacgtctcaggaccgcaggcgtcgagggaacgctgacaactggaccgcagacgtctcaggaccgc
aggcgtcgagggaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgac
aactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagatgtctca
ggaccgcaggcgtcgagggaccgctgacaactggactgcagacatcgagggaccgctgacaactggaccg
cagacatcgggggaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgggggaccgctga
caactggaccgcagacgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagacgtctc
aggaccgcaggcgtcgagggaccgctgacaactggaccgcagacgtctcaggaccgcaggcgtcgaggga
ccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccacag
acgactggactgctgggccgccggaggctgcaggctgcaggcgatggctcggggccgccgccggaggctg
gaagctgcaggcggcggtgctcggggccgccgccggaggctggaagctgcaggcggcggcgctcgaggcc
ggaggctggaagctgcaggcgacggcgctcggggccggaggctggaagctgcaggcgacggtgctcgggg
ccggaggctggaagctgcaggcgacggtgctcggggccggaggctggaagctgcaggcgacggtgctcgg
ggccgccgccggagggtgcaggcggcggcgctcggggccgccgccggagggtgcagggcggcggcgctcg
gggccgccgccggaggctgcaggcggcggcgctcggggccgccgccggagggtgcaggcggcggcgctcg
gggccgccgccggggctgcaggcggcggcgctcggggccgccgccggaggctgcaggcggcggcgctcgg
gggccgccgccggaggctgcgcaggcggcggcgctcggggcgccgccggaggctgcaggcggcggcgctc
gggccgccgccggaggctgcgggcggcggcgctcggggccgccgccggaggtgcaggcggcggcgctcgg
ggccgccgccggaggctgcaggcggcggcgctcggggccgccgcggaggctgcaggcggcggcgctcggg
gccgccgccggaggctgcaggcggcggcgctcgggccgccgccggaggctgcaggcggcggcgctcgggg
ccgccgccggaggctgcaggcggcggcgctgggccgccgccgggggtgcaggcgcggcgctcgggggcgg
cgccgccgccggaggctgcaggcggcggcgctcgggcgcgcccggagggtgggcacggcggcgctcgggg
ccgccgccgggctgcaggcggcggcgctcggggccgccgccggaggctgcaggcggcggcgctcggggcc
gccgccggaggctgcaggcggcggcgctcggggccgccgcccggaggctgcaggcggcggcgctcggggc
cgccgccggaggctgcaggcggcggcgctcggggccgccggagggtgcaggcggcggcgctcggggccgc
cgccggaggctgcgggcggcggcgctcggggccgccgccggaggctgcaggcggcggcgccggggccgcc
gccggaggctgcaggcggcggcgctcggggccgccgccggaggctgcaggcggcggcgctcggggccgcc
gccggaggctgcaggcggcggcgctcggggccgccgccgggggtgcaggcggcggcgctcggggccgccg
ccggaggctgcaggcggcggcgctcgccgccgggtgcaggcgcgccggggccgccgccggagggtgcgca
ggcggcggcgctcggggccgccgccggaggctgcaggcggcggcgctcggggccgccgccggagggtgga
ccgccaaacgcggttatagctcttacaagagctgttacagctctactatacaccgttgactcccctgctc
ttctgcgactccaccgctctccgctccacttcaccgctctccgccgacttcaccgttctccgccgacttc
accgctctccgccgacttcaccgctctccgccgacttcaccgctctccgccgacttcatccctcctgtag
cagccgctgctctcctgcaggagcccccggctctcctgcagcagcccccctcggcagcctggcatagcaa
cctttatggggtggttgagccccgcccctacacaggtggccaattgaatttcaaccttcccagcggatat
acctacggcctactgattggatgactccactcagtctgccccacccttacattcccccttttctttggag
attagtcaaatccttgacttttcagatggttctaccttatattgaacatataagactaacattttatcgc
cccaattttctttagtatagcaacaatacactcacagtttctttagtcttagtttcagtccatggtcagc
agggtcctttggctgcatttcccatttctggagggcatccttactctcagctcgtttgacatgacccgca
tgaacccaggccctgatgccttccacttttactgctgtgggggtggttaggataacagtaaatggtccct
tccaacgaggctccaaattccccacttgatagcgacgtatccaaaccaagtccccgggttggtaaggatg
tggttccaccgtctctgttccagtgtgggcaactcggattcctgtatgggcttttttaagacagattgta
atgcctgcagtgactggagtacagactgatccgtcatttctgctagggcaacctcctgtagccgaggaca
gagtggggggggtctcccaaacaatatttcaaatggggtcaccttgcttaaataaggtgtacatcgggct
ctaaggagggcaaaaggaaggagatctacccatccaccgccagtttccagaattagttttgtcaaggtct
ctttaagagtccgattcattctttctacttgccccgaactctggggcctgtatgcacaatgtaatttcca
atttgtgcccacagccttggccaatgcctgtgagactgaacttacgaatgcagggccattgtctgaccca
attgcaagaggcagcccgaacctaggaattatttcttctagtatcttttttgctactacccctgcggtct
catgcttggcaggaaaagcctccacccagcctgaaaaggtatccacaaaaaccaacatatacttgtgccc
atattttccaggtttcatttctgtaaaatctatttcccaataaattcctggttccttacctctttctctt
ttccctcttcccattttagttcttcctacattaactgcctggcattgggcacatctatcaactacatcct
gaaccatacgacctaatttagaaacctggaactgcttgcctataagctccttgagttttcgtgtccccaa
atgagtaccctgatgtatctggcctactaacttcttacctagtcttcgaggaagaattatttttcctatt
tttgtcttggcccagtccccttcatagtctaattcaacccatttctgtaaatactttaaatcctcttctg
aatagtttggtttttctggtagtgctggagccgggagtgcaactagttcttgcactttttctcgagctgc
ttgctttgcggcttgatctgctaagtagtttccttttgaaatcagatcagtccccttttgatgtcctggg
caatgcataacagccacctttttcgggtcccaaatagcctggaggagagctaatatttctcttctgtttt
taattccttttccttctgctgttaataggcccctttctttataaatagccccatgtatgtgcagtgtagc
aaaggcatacctgctgtccgtatagatgttggtggtttttcctttggccatctgcagtgccttcgtgagt
gcaattagttctgctctctgtgccgaagttccatgtggcagggctgctgtccagagtctctgctcaggag
aaaccaccgcggcccctgcatacctctttccattgactatgtagctgctcccatctgtgaacagagtcat
ctcggcatctgggaggggaatgtctctgagatccggccttattcctgtgacttgggttaagacctcccca
caatcatgtggatggtcagctagctcttctggcagcagtgtggctggatttagcactgctgggggtcgga
aggccacttttggttcattgaggagaagggcctgataccccgttactcgggcgttcgtcatccatcggtc
tggtggagtccggagaaggccctcaatcgcgtgtgtggtggttataacaagattttgcctaaagttaatt
tgctcgcatccttaaccaggagggctgttgcggcaattatgcggaaggcaaggagggaacccagctgcta
ctgggtctagtttcttactaaggtaggccactggtcttcgccaaggtcccagagtctgagttaaaactcc
tttggccactcccgatttttcatccacgtataattggaagggtttggttacatctgggatggccaatgct
ggggcactcagcaaggctgtctgtaattcccgaaatgctccctctgcttcagctgtccactcaatcctag
taagccctccttttgtcagttcatagagcggtcgggcaatctctgcaaaccgtggtatccatagcctaca
gtagcccactgttccaagaaactcccttacttgtcgggacgtggtgggacgggggtatcgggtaatcacc
tgtttcctggattcagacagtgaacgtactccctcatggaggtcaaagcctagataagtggcatagctct
gacagagctgtgctttctttgctgacacccgatacccttttgtctgcagagcctgtaagagagctctcgt
ccccacaaacacgactcatagtcctcggcggctactagcaagtcatcaacgtattgtaacagagtgactc
ctggattagaaattctgtactcacgcaaatcctcatgcagggcctcatcaaaaatggtgggcgaattctt
gaatccttggggaagtcttgtccaggtgagctggccttgaaacccctcctgtgggtcagaccactcaaaa
gcaaaaggggctgagattcccttgctataggtatacagaaaaatgcatcctttaaatctaagacagtata
tacagttcttttggggcccaggagggagagtagggtataggggttaggcactgttgggtgtatatcttca
acccgtttgttaacttcccgcaagtcctgaactggtcggtattcccagtctcggcctttttaacggggag
caaaggagtgttccatgaagatttacaaggaactaaaatcccgtctccccggaggcggttaatgtgtttc
ctgatcccttcctttgcttcctgggaaagaggatattgccggacgcgtatagccgtagctgtcgccttta
attgtaccaccactggaggttgttctttagccaagccaggggcttcccttctgcccagacctcaggaaac
tcgttctgtaggatttggaggaggctttcccctttgtcttctttggttttgagactggttcgtaaaggcg
gtattcatccactgctgccataagcacctgtacagtgggttgtctcaatgttacagtcatatctccttct
gaaaactgaatgccagctcttaacttgtggaggagatctcttcccatcaaattatatggggcattaggga
ggaccaaaatcgatgtttaactaatcccgaggctaaatttaaagtcctttctgttgtccatttatgtctc
tctgtgccatttgctccttgtacccaaacttcctctccagatagttttcccataggtttaagtaacgatg
aaaattgggcccctgtgtctaccaaaaaatcaactgatttccctccacttcaatttttacccgaggttcg
gggggggttccgaaccatggccccttcaatctctggtggcaagcattgccgtcttccttttagggcactc
cctggcccaatgtcctttttctttacagtaagcacactgattggaatctaagtcttcccgcctgccttct
ttctgcttaggtttttccttccatttactatcccctagccgtctcctccctgcctcatgtaatgtcaaaa
ccttaaccaatttttttgtctctttttcatcctcttgtgcatccctgttgttaaataccttttctgctat
tcccactaactcagtcagattttttccttcaaatccttcgattttctgaagttttctccttatatctggg
gctgactgacttgcaaaagcaagattcattgccctctgattttcaggtgcctcaggattgatcgggctat
acattttataggcttcaattagcctttctaggaaggctcctggagactcattcttaccttgagtcactgt
actaattttggataaatttatgggtctccttgctgctgcacggaggcctcccaatagagcctggcggtag
aaacgtaagctctccttaccttcatcagagtttggatcccagtcgggagattccctcgggcagtagtcct
ccatccggcggggatcgaggtccctgccaagtctctcgcttatcaccttatgggttttcgcatgatgcga
tcccgttcctccccattaaagagagccataaggagctgttgaacatctacccaggtgggttggtgagtaa
gaaaaacagtttccaagaggctaattaaatcctgagggtgatcagaaaaggatttgtgttgggttttcca
gttatagatgtcagaagaggagaaagggatatagcttaagtaaggccgccctctcccctggtagagcaac
ctcacgaaggggcagcatcggcgcagaccgtaccagtgaggtagaggaaggctgagggggataggggaaa
ggggaaggatcagggcagtctcgccgcatggtgatcccataggtggatcccgaccgtgtatgaggtggac
ttgtaaagacattttgggaccgagagtcagatttagaagacaaataaagggggcgaattccgcatcctcc
atatctgatggaggtagaacggtcttcttccctgaatcccccttatctgcttgttctggttttggtgctc
caacctgccttagagggagagaaataatttcattagcaggtggaagatgggacttgagccatgacggtgg
ctcttctatcaggtatcgccaggccacaatatatggtacttggtctcgatgtagatccaagacctcactc
agggtagaatatatctgagccaaggcaaacgttccttccggtggccaccctgtggagaaattgggccatt
ctaattgacagagtgtttttagttttcccggataaaccgtatcacctgaatgagtggctttaccttgaaa
atctttgaaatgttcaagaacaagacctaggggtgtcacaggcccttattctctgtttgtcccatttctc
caaatacaacaaacagcacaacagacaacaacaaaagaacaagacaaaaacaaacgtagacccagtgatc
cttatccagagcctgctgctgggggggctttttggtccacaaaacaccacacaatagataacaaaagaac
cagacaaagacaactgcagacccagcgattcttacccaaaatttgccacttatggcggctattttcatct
tccaaccgcctaaggggactcgaaccctcgatcgcccagggggactcgaacccctgaccgcccaggggac
tcgaaccccccaaccgcccagggggactcgaacccccgaccgcgcagggggactcgaaccccccgaccgc
ccaaggggactcgaaccccctgtcgatctatcagcagagggatggggcgtccccgcatccactccagccc
tggggagcatggctgcgtccagcttctccccggtgggctataccctggagctccaaccagacagacggat
aggatcccaatacctcggaacccaatacttcgaaacccaatgcttcgaaacccaatacctccaaacccaa
tacctccaaacccaatacctccaaacccaatacctccaaacccaatacctccaaacccaatacctccaaa
cccaatacctccaaacccaatacctccaaacccaatacctccaaacccaatacctccaaacccaatacct
ccaaacccaatacctccaaacccaatacctccgaacccaatacctccaaacccaatacctccggaggctt
ttcttaaaaattctgatgctggctcagttctcgccgtttgatcccggacgagcccccaaatgttagggtc
cgtgatcaaaggaacgagactgatacaaagcgaaggtcaagcaaagctttatttcgcgccaagcatcatg
aatcgcactgaccggtcagggctttctttataaaagatgacaactttgaccccggcgagcctactcccaa
tgaggttgctgacatctaggtcgtttctcactgtgtcactgctgagggtcctggagtgcggggcagggcc
gctggcaccgcaggcgtcgagggaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgag
gaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgc
agacatcgagggaccgctgacaactggaccgcagacatcgagggaccgctgacaactggaccgcagatgt
ctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagacgtctcaggaccgcaggcgtcgag
ggaacgctgacaactggaccgcagacgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccg
cagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagatgtctcaggaccgcagg
cgtcgagggaccgctgacaactggaccgcagatgtctcaggaccgcaggcgtcgagggaccgctgacaac
tggactgcagacatcgagggaccgctgacaactggaccgcagacatcgagggaccgctgacaactggacc
gcagatgtctcaggaccgcaggcgtcgagggaccgctgacaactggaccgcagacgtctcaggaccgcag
gcgtcgagggaccgctgacaactggaccgcagacgtctcaggaccgcaggcgtcgagggaccgctgacaa
ctggaccgcagacgtctcaggaccgcaggcgtcgggggaccgctgacaactggaccgcagatgtctcagg
accgcaggcgtcgagggaccgctgacaactggaccacagacgactggactgctgggccgccggagggctg
caggctgcaggcgatggctcggggccgccgccggaggctagaagctgcaggcggcggtgctcggggccgc
cgccggaggctggaagctgcaggcggcggcgctcgaggccggaggctggaagctgcaggcgacggcgccg
gggccggaggctggaagctgcaggcgacggtgctcggggccggaggctggaagctgcaggcgacggtgct
cggggccggaggctggaagctgcaggcgacggtgctcggggccgccgccggaggggcggcgcggcgctgg
ggccgccgcggagggtgcgggcggcggcgctcggggcgccgccgggctgcaggcggcggcgctcggggcc
gccgagggtgcaggcggcggcgctcggggccgccgccgagggctgcaggcggcggcgccgggccgccgcc
gaggctgcaggcggcggcgccggggccgccggaggctgcaggcggcggcgccgggccgccgccggaggct
gcaggcggcggcgccggggccgccgccggaggcgcaggcggcggcgctcgggccgccggggtgcaggcgg
cggcgccggggccgccgcgaggctgcaggcggcggcgctcggggccgccgagcgctgcaggcggcggcgc
cggggcgccgccgaggctgcaggcggcggcgctcgggccgccgccgaggctgcaggcggcggcgccgggc
cgcggccgaggaggcggcggcgccgggcgccgccggagggtaggcggcggcgccggggccgcgccggggg
ctgaggcggcggcgctcggggccgcgccggagggtgcaggcggcggcgctgggccgcgccgaggctgcag
gcggcggcgctcggggccgccgccggaggctgcaggcggcggcgctcggggccgccgccggggctgcagg
cgggcggcgctcggccgccgccgaggctgcaggcggcggcgctggcgccgccgggaggctgcaggcggcg
gcgccggggccgccggagggtgcaggcggcggcgccgggccgccgccggaggctgcaggcggcggcgcgc
cgccggggggtgcaggcggcggcgctcggggccgccgcggaggcgcaggcggcggcgctcggggccccgc
cgccggaggctgcaggcggcggcgctcggggccgccgccggaggtgcaggcggcggcgccggggccgccg
ccggagggtgcaggcggcggcgccggggccgccgccggaggctgcaggcggcggcgctcggggccgccgc
cggagggtggcaggcggccgctcgggccgccgccggagggtgcaggcggcggcgccggggccgccgccgg
aggctgcaggcggcggcgccggggccgccgccggagggtggaccgccaaacgcggttatagctcttacaa
gagctgttacagctctactatacaccgttgactcccctgctcttctgcgacttccaccgctctccgctcc
acttcaccgctctccgccgacttcaccgttctccgccgacttcaccgctctccgccgacttcaccgctct
ccgccgacttcaccgctctccgccgacttcatccctcctgtagcagccgctgctctcctgcaggagcccc
cggctctcctgcagcagccccctcggcagcctggcatagcaacctttatgggggtggttgagccccgccc
ctacacaggtggccaattgaatttcaaccttcccagcggatatacctacggcctactgattggatgactc
cactcagtctgccccacccttaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Молекулярно-генетические маркеры цветовых вариаций американской норки и способ выявления особей, являющихся носителем аллелей, обуславливающих формирование желаемой цветовой вариации | 2019 |
|
RU2722564C1 |
| Молекулярно-генетический маркер цветовой вариации "черный хрусталь" у американской норки и способ выявления особи норки, являющейся носителем аллеля, обуславливающего формирование желаемой цветовой вариации | 2022 |
|
RU2810183C1 |
| СПОСОБ ПОЛУЧЕНИЯ НОВЫХ ОКРАСОК МЕХА У НОРОК | 1992 |
|
RU2044480C1 |
| СПОСОБ РАЗВЕДЕНИЯ НОРОК | 1990 |
|
RU2010514C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| ГЕН И ВАРИАЦИИ, СВЯЗАННЫЕ С ФЕНОТИПОМ BM1, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2012 |
|
RU2617958C2 |
| ПРИМЕНЕНИЕ СПЕЦИФИЧЕСКИХ МАРКЕРОВ ГЕНА КОРИЧНЕВОЙ СРЕДНЕЙ ЖИЛКИ 3 (BROWN MIDRIB-3) У КУКУРУЗЫ ДЛЯ ИНТРОГРЕССИИ ПРИЗНАКОВ | 2011 |
|
RU2593958C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПОЛУЧЕНИЯ УКОРОЧЕННЫХ РАСТЕНИЙ КУКУРУЗЫ | 2016 |
|
RU2745987C2 |
| Набор ДНК-маркеров для отбора ячменя с повышенным содержанием антоцианов в перикарпе зерновки | 2022 |
|
RU2797678C1 |
| ГЕНЕТИЧЕСКИЙ ТЕСТ ДЛЯ ИДЕНТИФИКАЦИИ НОСИТЕЛЕЙ РЕЦЕССИВНОГО ГЕНА КОМПЛЕКСНЫХ ВЕРТЕБРАЛЬНЫХ МАЛЬФОРМАЦИЙ У КРУПНОГО РОГАТОГО СКОТА | 2001 |
|
RU2276690C2 |
Изобретение относится к области биотехнологии. Описан генетический маркер окраски меха американской норки, в гомозиготном состоянии определяющий окраску меха пастель, представляющий собой инсерцию эндогенного ретровирусного элемента в первом интроне гена HPS3 норки. Раскрыт способ выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, включающий генотипирование животного по гену HPS3, а именно выявление наличия и количества или отсутствия генетического маркера. Раскрыто применение способа для отбора особей американской норки, являющихся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, среди имеющегося поголовья фермерских норок. Техническим результатом является более быстрый и менее трудоемкий способ отбора особей американской норки, носителей генетического маркера окраски меха пастель. 3 н. и 8 з.п. ф-лы, 2 табл., 2 пр.
1. Генетический маркер окраски меха американской норки, в гомозиготном состоянии определяющий окраску меха пастель, представляющий собой инсерцию эндогенного ретровирусного элемента в первом интроне гена HPS3 норки.
2. Способ выявления особи американской норки, являющейся носителем по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, включающий генотипирование животного по гену HPS3, а именно выявление наличия и количества или отсутствия генетического маркера по п. 1.
3. Способ по п. 2, где наличие и количество или отсутствие генетического маркера по п. 1 определяют путём амплификации ДНК исследуемой особи американской норки с использованием праймеров SEQ ID NO: 4 и SEQ ID NO: 7, SEQ ID NO: 4 и SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7.
4. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят секвенированием по методу Сенгера.
5. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят секвенированием NGS.
6. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят методом ПЦР-ПДРФ.
7. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят методом ПЦР в реальном времени с использованием флуоресцентных зондов типа TaqMan.
8. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят методом аллель-специфичной ПЦР.
9. Способ по п. 2, где выявление наличия и количества или отсутствия генетического маркера по п. 1 проводят методом гибридизации на чипах.
10. Применение способа по любому из пп. 2 или 3 для отбора особей американской норки, являющихся носителями по меньшей мере одного аллеля, обуславливающего формирование окраски меха пастель, среди имеющегося поголовья фермерских норок.
11. Применение по п. 10, где отобранных животных используют для получения от них потомства с заданной окраской меха.
Авторы
Даты
2024-09-26—Публикация
2023-12-28—Подача