Ряд микроорганизмов обладает своего рода системой защиты от негативного воздействия конкурирующих микроорганизмов, подавляя их систему коммуникации кворум-сенсинга с помощью ферментных систем расщепляющих ацильные производные гомосерин-лактонов (АГЛ) - АГЛ-лактоназ [1]. Этот фермент может гидролизовать лактоновое кольцо и амидную связь АГЛ в неактивные молекулы и тем самым блокировать системы кворум-сенсинга и ослаблять симптомы, вызываемые патогенами. Среди бактерий рода Bacillus широко распространены гены, кодирующие АГЛ-лактоназу (aiiA), что делает данные микроорганизмы перспективным объектом для исследования [2].
У микроорганизмов, принадлежащих роду Bacillus также присутствует ген лакказы– cotA [3].
Показано, что лакказа бацилл способна деградировать молекулы афлатоксинов, содержащие лактоновое кольцо, идентичное таковому в сигнальных молекулах QS. Способность к инактивации микотоксинов и способность к ингибированию чувства кворума благодаря такому сходству нередко коррелирует [4], поэтому данный ген был также выбран как целевой.
В работе [5] изучалась афлатоксин B1-деградирующая активность штаммов Bacillus subtilis, выделенных из злаковых культур, которая оценивалась по соотношению остатков афлатоксина к его изначальной концентрации. В результате все 32 изолята обладали искомой. Было обнаружено, что афлатоксин В1 деградировал только в тесте с экстраклеточной фракцией. Такая специфическая активность внеклеточной фракции говорит о том, что в процесс включены ферментные системы, в частности, экзоферменты. Проверка на ингибирование различными веществами показала, что они ингибировались азидом натрия, дитиотреитолом, гидроксиламином. Азид натрия, дитиотреитол и гидроксиламин являются восстановителями, а азид натрия и дитиотреитол также являются предполагаемыми ингибиторами лакказы. Авторы предполагают, что лакказа может быть одним из действующих ферментов. В работах других авторах лакказа также рассматривается, как один из ключевых участников биодеградации афлатоксинов и зеараленона у грибов [10, 6], Bacillus licheniformis [7], Bacillus subtilis [8].
У Bacillus subtilis лакказа закодирована в гене cotA, наличие и экспрессия которого может быть хорошим маркером для поиска перспективных штаммов. Он содержит 1642 п.н, которые кодируют белок из 513 аминокислот.
CotA имеет высокую степень гомологии среди родственных видов Bacillus subtilis. Сам фермент описан у многих видов бацилл и других микроорганизмов, неприменительно к вопросу биотрансформации микотоксинов [9].
Цель настоящего изобретения - выявление штаммов бактерий рода Bacillus, проявляющих лактоназную активность, в которых содержатся гены aiiA и cotA.
Известен способ выявления бактериальной ДНК методом петлевой изотермической амплификации, описанный в [11]. Однако данный способ не позволяет выявить специфические гены.
Известен способ идентификации фрагмента гена TFDP1 методом ПЦР в режиме реального времени, описанном в [12]. Однако, данный способ подразумевает выделение РНК с последующим синтезом кДНК на матрице РНК, что увеличивает время проведения анализа.
Наиболее близким аналогом к заявленному способу является способ обнаружения генов устойчивости к аминогликозидам у бактерий животного происхождения, раскрытый в патенте [13].
Однако данное изобретение имеет совершенно отличный технический результат от заявленного, поскольку отличаются целевые гены для идентификации.
Техническим результатом заявленного изобретения является идентификация генов, кодирующих АГЛ-лактоназу (aiiA) и лакказу (cotA) у бактерий рода Bacillus, выделенных из кормовой добавки, либо полученных из чистых бактериальных культур. Результат достигается путем выделения из бактерий ДНК и проведением ПЦР с использованием оригинального набора праймеров для определения генов лактоназной активности с последующей визуализацией результатом путем электрофоретического разделения ампликонов в агарозном геле.
Для проведения полимеразной цепной реакции были подобраны пары праймеров, соответствующие уникальным участкам генов cotA (Bacillus subtilis subsp. subtilis str. 168), cotA (Bacillus pumilus strain WH4), aiiA (Bacillus cereus B4264), aiiA (Bacillus thuringiensis Bt407). Приведенные в таблице 1.
Таблица 1
Праймеры для идентификации генов aiiA и cotA.
(Bacillus subtilis subsp. subtilis str. 168)
(Bacillus pumilus strain WH4)
(Bacillus cereus B4264)
(Bacillus thuringiensis Bt407)
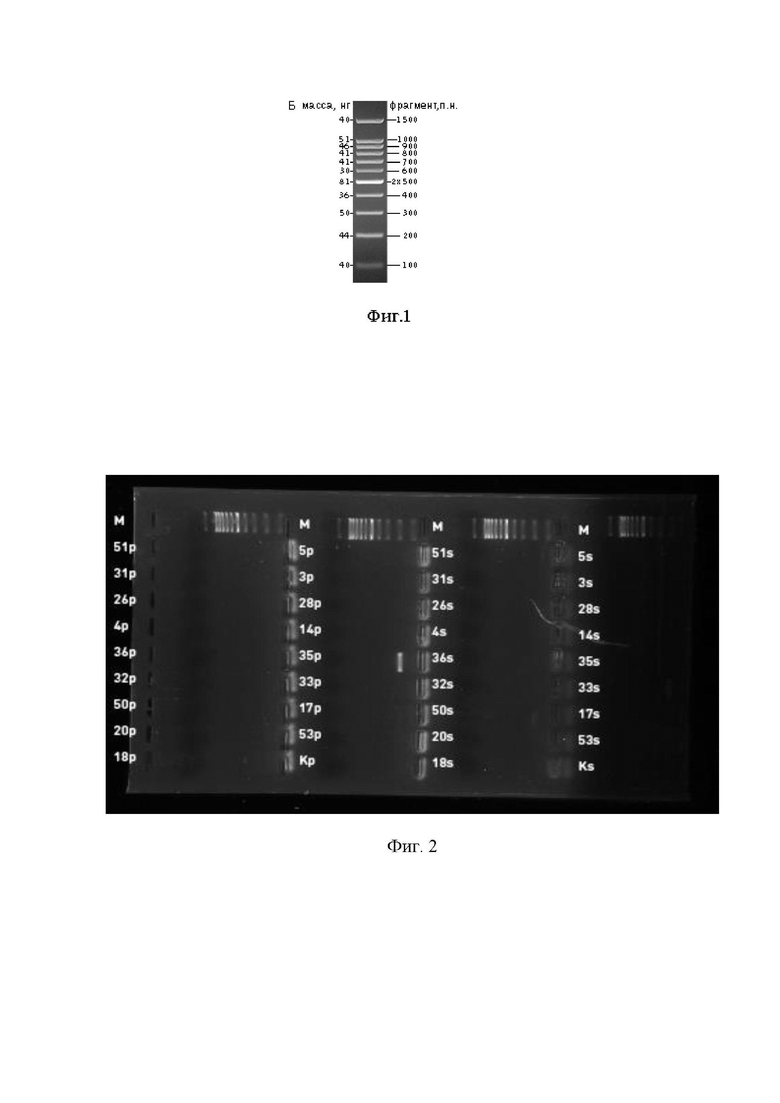
Для осуществления способа из исследуемой культуры выделяются нуклеиновые кислоты, любым способом, обеспечивающим получение ДНК, пригодной для постановки ПЦР в количестве не менее 100 нг. Проводится полимеразная реакция с указанными праймерами, где объем реакционной смеси составляет 25 мкл: растворы прямых и обратных праймеров в объеме по 1 мкл с концентрацией праймеров 0,4 мкМ, 5 мкл реактива Screen Mix (5х; Евроген, Россия), 15 мкл бидистиллированной воды и 3 мкл ДНК матрицы. Далее оцениваются результаты электрофоретического разделения продуктов амплификации. Наличие ампликонов длиной 1000п.н., характерных для подобранных пар праймеров, позволяет судить о наличии у данного штамма ферментных систем, подавляющих quorum sensing.
Пример 1
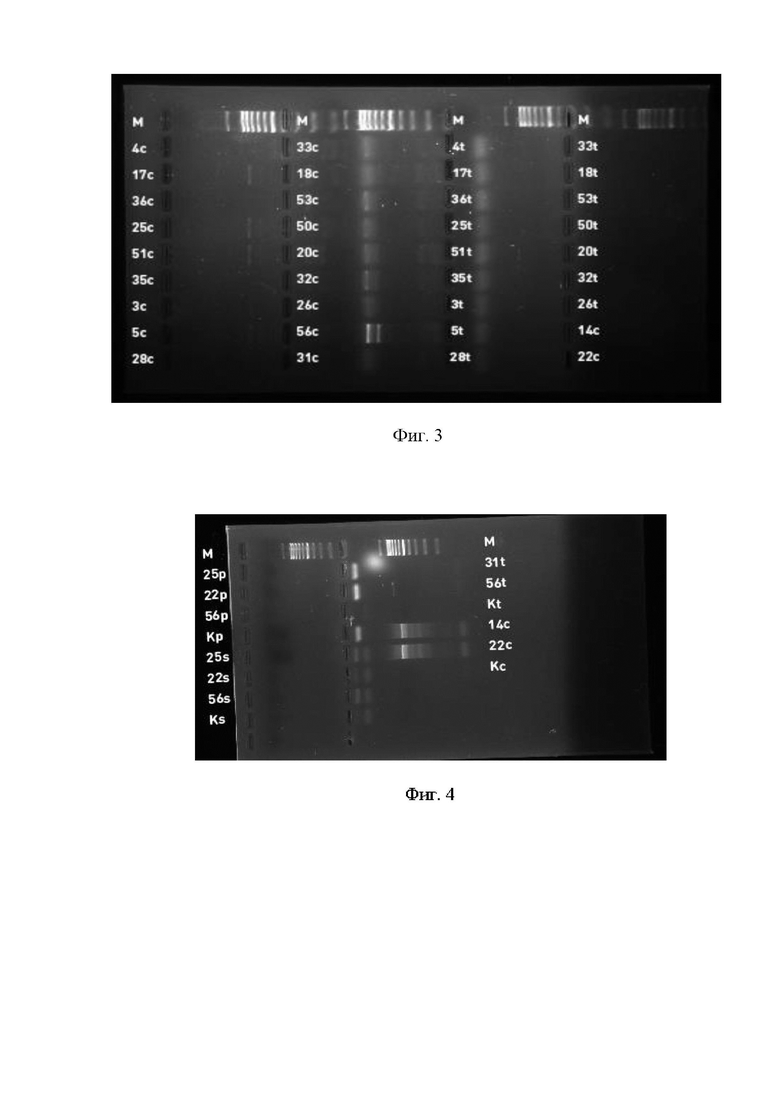
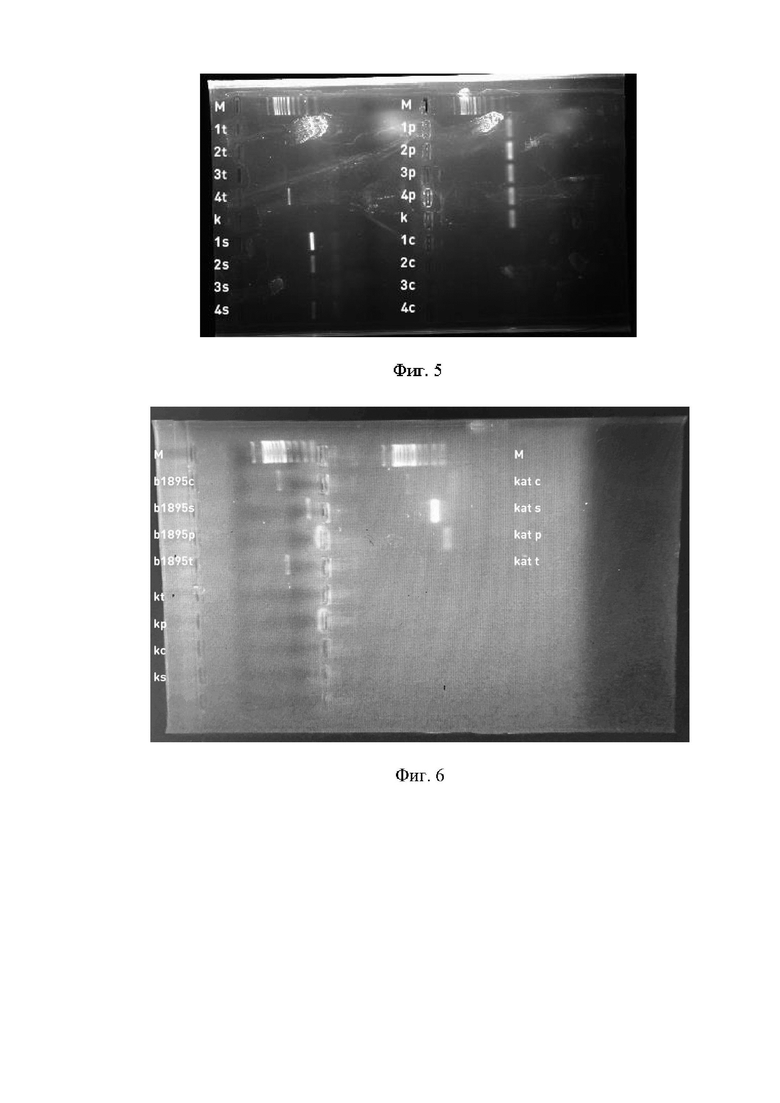
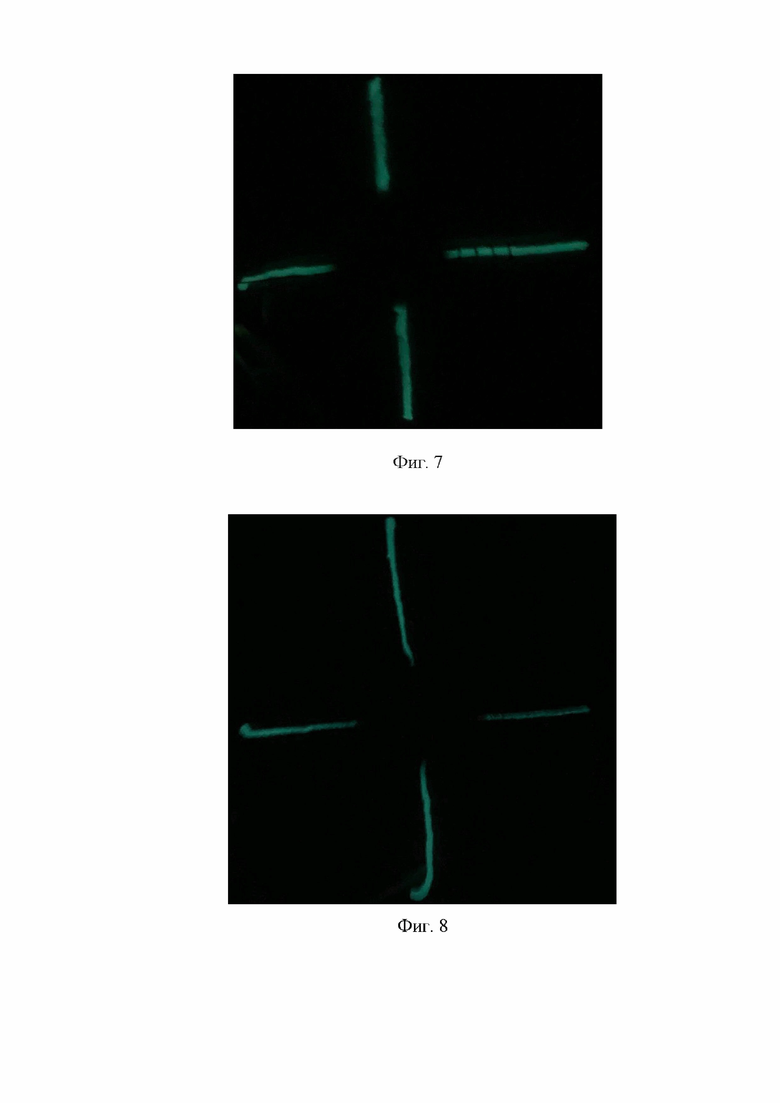
Для проверки были отобраны микроорганизмы рода Bacillus, четыре штамма выделенные из кормовой добавки для сельскохозяйственных животных «Целлобактерин», Bacillus subtilis KATMIRA и Bacillus subtilis B1895 и различные штаммы Bacillus licheniformis (№3, 4, 5, 14, 17, 18, 20, 22, 25, 26, 28, 31, 32, 33, 35, 36, 50, 51, 53, 56). Из культур отобранных штаммов была экстрагирована ДНК, с применением набора «Амплитуб-РВ» компании Синтол на магнитных частицах, по стандартному протоколу к данному набору. ПЦР осуществляли по следующей программе: удержание - 3 мин при 95°С, денатурация - 30 с при 95°С, отжиг праймеров - 30 с при 60°С, элонгация - 60 с при 72°С в течение 35 циклов. Далее проводился электрофорез в агарозном геле. Результаты представлены в Таблице 2, и на фиг. 1-6.
Для оценки результатов отбора штаммы с выявленными генами подавления quorum sensing проверили на способность подавлять обусловленную quorum sensing биолюминесценцию Aliivibrio logei AZ1. Результаты представленные на фиг. 7-12, и в таблице 3 подтверждают способность штамов, выявленных предложенным способом, подавлять quorum sensing и связанную с ним биолюминесценцию.
Таблица 2
Гены, обнаруженные у исследуемых штаммов Bacillus.
Штаммы 1c-4с – выделены из препарата Целлобактерин.
Таблица 3
Зоны подавления свечения Aliivibrio logei AZ1
Литература:
1. Watson W. T. et al. Structural basis and specificity of acyl-homoserine lactone signal production in bacterial quorum sensing //Molecular cell. – 2002. – Т. 9. – №. 3. – С. 685-694.
2. Huma N. et al. Diversity and polymorphism in AHL-lactonase gene (aiiA) of Bacillus //Journal of Microbiology and Biotechnology. – 2011. – Т. 21. – №. 10. – С. 1001-1011.
3. McKenney, Peter T.; Driks, Adam; Eichenberger, Patrick (2012). The Bacillus subtilis endospore: assembly and functions of the multilayered coat. Nature Reviews Microbiology, 11(1), 33–44. doi:10.1038/nrmicro2921
4. Prazdnova, E. V., Gorovtsov, A. V., Vasilchenko, N. G., Kulikov, M. P., Statsenko, V. N., Bogdanova, A. A., ... & Chikindas, M. L. (2022). Quorum-Sensing Inhibition by Gram-Positive Bacteria. Microorganisms, 10(2), 350.
5. Watanakij N., Visessanguan W., Petchkongkaew A. Aflatoxin B1-degrading activity from Bacillus subtilis BCC 42005 isolated from fermented cereal products //Food Additives & Contaminants: Part A. – 2020. – Vol. 37, No. 9. P. 1579-1589.
6. Zhou Z. et al. A New Laccase of Lac 2 from the White Rot Fungus Cerrena unicolor 6884 and Lac 2-Mediated Degradation of Aflatoxin B1 //Toxins. – 2020. – Vol. 12, No. 8. P. 476.
7. Guo Y. et al. CotA laccase, a novel aflatoxin oxidase from Bacillus licheniformis, transforms aflatoxin B1 to aflatoxin Q1 and epi-aflatoxin Q1 //Food chemistry. – 2020. – Vol. 325. P. 126877
8. Wang X. et al. Degradation of aflatoxin B1 and zearalenone by bacterial and fungal laccases in presence of structurally defined chemicals and complex natural mediators //Toxins. – 2019. – Vol. 11, No. 10. P. 609.
9. Reiss R., Ihssen J., Thöny-Meyer L. Bacillus pumilus laccase: a heat stable enzyme with a wide substrate spectrum //BMC biotechnology. – 2011. – Vol. 11, No. 1. P. 1-11.
10. Alberts J. F. et al. Degradation of aflatoxin B1 by fungal laccase enzymes //International journal of food microbiology. – 2009. – Vol. 135, No. 1. P. 47-52.
11. Патент РФ на изобретение № 2776163 Способ выявления ДНК бактерии Mycobacterium tuberculosis с помощью изотермической петлевой амплификации / Кечин А. А., Оскорбин И. П., Филипенко М. Л. - опубл. 14.07.2022, Бюл. № 20
12. Патент РФ на изобретение № 2463354 Способ диагностики рака мочевого пузыря с помощью онкомаркера TFDP1 (варианты) и набор для его осуществления / Заболотнева А.А., Шегай П.В., Гайфуллин Н.М., Русаков И.Г., Алексеев Б.Я., Буздин А.А. – опубл. 10.10.2012 Бюл. № 28
13. Патент РФ на изобретение № 2816522 Способ обнаружения генов устойчивости к аминогликозидам из группы aada у бактерий животного происхождения методом ПЦР с детекцией в режиме «реального времени» / Прасолова О.В., Богомазова А.Н., Путинцева А.В., Осипова Ю.А., Кирсанова Н.А., Крылова Е.В., Тимофеева И.А., Солтынская И.В. – опубл. 01.04.2024 Бюл. № 10
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="/home/k/sequence_listing.xml
" softwareName="WIPO Sequence" softwareVersion="2.3.0" productionDate="2024-
11-28">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024111011/10(024827)</ApplicationNumberT
ext>
<FilingDate>2024-11-28</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
автономное образовательное учреждение высшего образования Южный
федеральный университет</ApplicantName>
<ApplicantNameLatin>Federal State Autonomous Educational
Institution of Higher Education Southern Federal
University</ApplicantNameLatin>
<InventionTitle languageCode="ru">МЕТОД ИДЕНТИФИКАЦИИ ГЕНОВ,
КОДИРУЮЩИХ AGL-ЛАКТОНАЗУ (aiiA) И ЛАККАЗУ (CotA) В БАКТЕРИЯХ
РОДА BACILLUS</InventionTitle>
<InventionTitle languageCode="en">METHOD FOR IDENTIFICATION OF
GENES ENCODING AGL-LACTONASE (aiiA) AND LACCASE (CotA) IN BACTERIA OF THE GENUS BACILLUS</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus subtilis subspecies subtilis strain 168</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcggcacaactgaaatatggtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus subtilis subspecies subtilis strain 168</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctttccagcccttttcactt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus pumilus strain WH4</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctgcctgtcgatcatacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus pumilus strain WH4</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcttgctggtgattagggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus B4264</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtgcagcgaacggaatatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus B4264</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaatagcgactgatggcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus thuringiensis Bt407</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagccggacgaccttttat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus thuringiensis Bt407</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatggcctggagaatgacct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---


Изобретение относится к биотехнологии. Предложен способ идентификации генов, кодирующих АГЛ-лактоназу (aiiA) и лакказу (cotA) у бактерий рода Bacillus. Способ заключается в выделении нуклеиновой кислоты любым способом, обеспечивающим получение ДНК для реакционной смеси в количестве не менее 100 нг, последующем проведении полимеразной цепной реакции с использованием набора оригинальных праймеров: cotA (Bacillus subtilis subsp. subtilis str. 168): прямой TCGGCACAACTGAAATATGGTC и обратный GTCTTTCCAGCCCTTTTCACTT; cotA (Bacillus pumilus strain WH4): прямой TCCTGCCTGTCGATCATACC и обратный TGCTTGCTGGTGATTAGGGT, aiiA (Bacillus cereus B4264): прямой и обратный TGTGCAGCGAACGGAATATGTGAATAGCGACTGATGGCCT, aiiA (Bacillus thuringiensis Bt407): прямой TGAGCCGGACGACCTTTTAT и обратный GATGGCCTGGAGAATGACCT. Оценка результатов ПЦР осуществляется с помощью электрофоретического разделения продуктов амплификации по наличию целевого фрагмента длиной 1000 п.н. Изобретение позволяет идентификацию генов, кодирующих АГЛ-лактоназу (aiiA) и лакказу (cotA) у бактерий рода Bacillus, выделенных из кормовой добавки, либо полученных из чистых бактериальных культур. 12 ил., 3 табл., 1 пр.
Способ идентификации генов, кодирующих АГЛ-лактоназу (aiiA) и лакказу (cotA) у бактерий рода Bacillus, характеризующийся выделением нуклеиновой кислоты любым способом, обеспечивающим получение ДНК для реакционной смеси в количестве не менее 100 нг, проведением полимеразной цепной реакции по следующей программе: удержание - 3 мин при 95°С, денатурация - 30 с при 95°С, отжиг праймеров - 30 с при 60°С, элонгация - 60 с при 72°С в течение 35 циклов, отличающийся использованием набора оригинальных праймеров: cotA (Bacillus subtilis subsp. subtilis str. 168): прямой TCGGCACAACTGAAATATGGTC и обратный GTCTTTCCAGCCCTTTTCACTT; cotA (Bacillus pumilus strain WH4): прямой TCCTGCCTGTCGATCATACC и обратный TGCTTGCTGGTGATTAGGGT, aiiA (Bacillus cereus B4264): прямой и обратный TGTGCAGCGAACGGAATATGTGAATAGCGACTGATGGCCT, aiiA (Bacillus thuringiensis Bt407): прямой TGAGCCGGACGACCTTTTAT и обратный GATGGCCTGGAGAATGACCT в реакционной смеси объемом 25 мкл, включающей растворы прямых и обратных праймеров в объеме по 1 мкл с концентрацией праймеров 0,4 мкМ, 5 мкл реактива Screen Mix (5х; Евроген, Россия), 15 мкл бидистиллированной воды и 3 мкл ДНК матрицы, с последующей оценкой результатов ПЦР с помощью электрофоретического разделения продуктов амплификации по наличию целевого фрагмента длиной 1000 п.н.
| СПОСОБ ОБНАРУЖЕНИЯ ГЕНОВ УСТОЙЧИВОСТИ К АМИНОГЛИКОЗИДАМ ИЗ ГРУППЫ aadA У БАКТЕРИЙ ЖИВОТНОГО ПРОИСХОЖДЕНИЯ МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ «РЕАЛЬНОГО ВРЕМЕНИ» | 2023 |
|
RU2816522C1 |
| СПОСОБ ОДНОВРЕМЕННОГО ОБНАРУЖЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗНОГО КОМПЛЕКСА И ИДЕНТИФИКАЦИИ МУТАЦИЙ В ДНК МИКОБАКТЕРИЙ, ПРИВОДЯЩИХ К УСТОЙЧИВОСТИ МИКРООРГАНИЗМОВ К РИФАМПИЦИНУ И ИЗОНИАЗИДУ, НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ, НАБОР ПРАЙМЕРОВ, БИОЧИП И НАБОР ОЛИГОНУКЛЕОТИДНЫХ ЗОНДОВ, ИСПОЛЬЗУЕМЫЕ В СПОСОБЕ | 2005 |
|
RU2376387C2 |
| СПОСОБ ОБНАРУЖЕНИЯ ДНК ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА С ОДНОВРЕМЕННЫМ УСТАНОВЛЕНИЕМ ЕГО ГЕНОТИПА И ОПРЕДЕЛЕНИЕМ ГЕНЕТИЧЕСКИХ ДЕТЕРМИНАНТ МНОЖЕСТВЕННОЙ И ШИРОКОЙ ЛЕКАРСТВЕННОЙ УСТОЙЧИВОСТИ, ОЛИГОНУКЛЕОТИДНЫЙ МИКРОЧИП, НАБОР ПРАЙМЕРОВ И НАБОР ОЛИГОНУКЛЕОТИДНЫХ ЗОНДОВ, ИСПОЛЬЗУЕМЫЕ В СПОСОБЕ | 2014 |
|
RU2562866C1 |
| Сульдина Е | |||
| В | |||
| и др., Выявление генов ферментов у бактерий вида Bacillus subtilis методом rEAL-TIME PCR, Научно-теоретический журнал ВЕСТНИК Ульяновской государственной сельскохозяйственной академии, октябрь - декабрь 2021 N4 (56), с | |||
| Устройство для сортировки каменного угля | 1921 |
|
SU61A1 |
| Алина Г.В | |||
| и др., Обзор | |||
Авторы
Даты
2025-02-03—Публикация
2024-04-22—Подача