Изобретение относится к области биотехнологии, ветеринарной микробиологии и генетической инженерии и может быть использовано как способ идентификации и детекции генов генов устойчивости к аминогликозидам из группы aadA методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме «реального времени».
Аминогликозиды (АГ), относятся к старейшим классам противомикробных препаратов. АГ представляют собой бактерицидные противомикробные препараты, действие которых основано на нарушении синтеза бактериального белка за счет связывания с 30S субъединицей рибосомы. АГ должны проникнуть в бактерию, чтобы оказать свое действие, и их поглощение бактериальной клеткой является кислородозависимым процессом. Поэтому спектр активности АГ ограничивается аэробными и факультативно-анаэробными бактериями в аэробных условиях. Спектр активности АГ включает грамотрицательные бактерии, стафилококки, микобактерии, лептоспиры и некоторые простейшие. Они малоэффективны в отношении стрептококков, анаэробных бактерий и внутриклеточных бактерий. Энтерококки и стрептококки обычно проявляют определенную степень внутренней устойчивости к АГ из-за непроницаемости их клеточной стенки, хотя проникновение в бактериальную клетку может быть усилено другими противомикробными препаратами, которые ингибируют синтез клеточной стенки, такими как β-лактамные антибиотики. Поэтому АГ часто применяют в сочетании с β-лактамами. Эта комбинация также расширяет спектр действия [1].
В ветеринарии АГ используются для лечения инфекций у всех основных видов сельскохозяйственных животных и домашних животных. Всемирная организация по охране здоровья животных классифицирует их как критически важные для ветеринарии противомикробные препараты (VCIA) на основании широкого спектра и характера болезней, для лечения которых они используются у животных. АГ имеют важное значение при септицемии, болезнях пищеварительной, дыхательной и мочевыделительной систем [2].
Существуют различные методы идентификации и детекции генетических детерминант резистентности к АГ, основная часть которых основана на применении различных методов: полимеразно-цепная реакция и реакция на чипах. Однако использование таких способов, предусматривает идентификацию генов резистентности к аминогликозидам, актуальным в медицине RU 2409680 C1, RU 127074 U1, RU 2015151395 A.
Наиболее близкое по существу к изобретению является CN 101392287 (A), раскрывающий технологию обнаружения генов лекарственной устойчивости у бактерий животного происхождения к аминогликозидам (aphA3, aacC4, aadA и aacC2) методом четырехкратной ПЦР с применением электрофоретической детекции результатов амплификации. Предлагаемое изобретение предусматривает детекцию генов резистентности к аминогликозидам группы aadA в режиме «реального времени», что значительно сокращает сроки исследования.
Целью настоящего изобретения является разработка способа обнаружения генов резистентности группы aadA к аминогликозидам у бактерий животного происхождения методом ПЦР с детекцией в режиме «реального времени».
Заявленный технический результат - идентификация генов резистентности группы aadA к аминогликозидам из образцов биологического материала животных и объектов их содержания, продовольственного сырья, пищевых продуктов, без этапа выделения бактериальных изолятов, а также чистых бактериальных культур с высокой специфичностью и чувствительностью. Достигается разработкой методики выделения ДНК и использованием оригинального набора олигонуклеотидных праймеров и зондов для выявления генов группы aadA: aadA1_2, aadA1_3, aadA1b_1, aadA1_4, aadA1_5, aadA2_1, aadA2_2, aadA2b_1, aadA3_1, aadA8b_1, aadA8b_2, aadA12_1, aadA15_1, aadA17_1, aadA21_1, aadA22_1, aadA23_1, aadA24_1, aadA25 (всего 19 генов) устойчивости к аминогликозидам при проведении полимеразной цепной реакции в режиме «реального времени». Аналитическая чувствительность методики идентификации и детекции генов группы aadA составляет 2,5х104 копий/мл. Специфичность способа идентификации и детекции генов группы aadA при исследовании образцов биологического материала животных и объектов их содержания, продовольственного сырья, пищевых продуктов, без этапа выделения бактериальных изолятов, а также чистых бактериальных культур в рамках контрольной панели составляет 100%.
Для выбора мишени с целью выявления генов резистентности к аминогликозидам были проанализированы данные из базы Pathogen Detection (NCBI) [3], которая находится в открытом доступе и содержит информацию исследователей из разных стран об изолятах, выделенных из различных источников: из клинических образцов, пищевых продуктов, от животных, из окружающей среды и их характеристики (геномные последовательности, наличие генов резистентности и др.). Частота обнаружения генов резистентности к аминогликозидам aadA1 составила 23%, aadA2 - 14% от общего количества исследований ветеринарных образцов.
Для выбора и анализа ДНК последовательностей генов резистентности были использованы базы данных ResFinder, Arg-ANNOT, CARD и NCBI BARRGD, а также последовательности геномов различных грамположительных и грамотрицательных бактерий, которые были ранее получены в ФГБУ «ВГНКИ» методом полногеномного секвенирования. Подбор праймеров производили на онлайн-ресурсе «PrimerQuest Tool» (IDT), анализировалось не менее пяти наборов олигонуклеотидов для выявления каждого целевого гена. Для анализа множественного выравнивания последовательностей использовали программу Ugene и алгоритм Clustal omega. Для выбора оптимальных параметров структурных характеристик наборов праймеров использовали программы PCR Primer Stats и Oligo Analysis Tool, оценивали оптимальную температуру отжига, возможность образования димеров и «шпилек». Критерии для выбора праймеров были следующие: средняя длина - 16-25 нуклеотидов; GC состав 35-60%; температура отжига от 50 °C до 72 °C, разница в температуре отжига в паре праймеров не должна превышать 6 °C; вторичная структура (образование шпилек): по минимуму; димеры: нежелательны, необходимо избегать само- и взаимокомплементарности между 3’-концами; минимум G/C на 3’-конце праймеров (не более трех из пяти последних нуклеотидов). Специфичность праймеров оценивали при помощи онлайн-ресурса «Primer-Blast», расположенном на сайте NCBI. Выбранные в качестве мишени гены группы aadA кодируют ферменты О-аденилтрансферазы (ANT), модифицирующие аминогликозиды путем присоединения нуклеотида аденина. Модифицированные молекулы теряют способность связываться с рибосомами и подавлять синтез белка бактерий.
Сущность изобретения заключается в том, что при проведении полимеразной цепной реакции в режиме «реального времени», используют специальный оригинальный набор олигонуклеотидных праймеров и зондов для идентификации и детекции группы генов устойчивости к аминогликозидам. Выбранные нуклеотиды имеют следующую структуру (таблица 1):
Таблица 1
Характеристики праймеров и зонда для детекции генов группы aadA
°C
Оптимизированы условия амплификации: концентрации прамеров и зонда, концентрация ионов магния в реакционной смеси, температура отжига праймеров и длительность основных стадий амплификации.
Материалом для исследования служат образцы биологического материала, полученные из продовольственного сырья, пищевых продуктов, от животных (фекалии), из объектов окружающей среды (смывы с клеток, стен, оборудования; образцы фекалий, подстилка и др.), без этапа выделения бактериальных изолятов, а также чистые бактериальные культуры.
Отбор проб, выделение, идентификация бактерий и получение чистой бульонной культуры изолятов проводят по стандартным методикам в соответствии с утверждёнными нормативными документами. Отбор проб смывов с тушек, клеток, стен, оборудования и др. проводят с использованием велюр-тампонов eSwab-system. Фекалии и образцы подстилки собирают в пластиковые контейнеры для сбора биоматериала с завинчивающейся крышкой.
Для подготовки анализируемой пробы бактериального изолята отбирают дозатором 1,0 см3 жидкой бульонной культуры бактерий в предварительно промаркированную одноразовую полипропиленовую пробирку вместимостью 1,5 см3. Центрифугируют на микроцентрифуге в течение 5 мин при 8000 об/мин. Затем удаляют надосадочную жидкость. К полученному осадку добавляют 1,0 см3 той же бульонной культуры и центрифугируют в течение 5 мин при 8000 об/мин. Надосадочную жидкость удаляют. Полученный осадок анализируемой пробы в закрытых промаркированных пробирках передают на выделение ДНК.
Для подготовки анализируемой пробы смывов с тушек, клеток, стен, оборудования и др. велюр-тампон eSwab-системы с биоматериалом помещают в одноразовую полипропиленовую пробирку вместимостью 1,5 см3 с изотоническим 0,9% раствором хлорида натрия (600 мм3). Тщательно перемешивают на вортексе. Для выделения ДНК используют 100 мм3 полученного раствора.
Анализируемые пробы фекалий или пробы подстилки, отобранные в пластиковые контейнеры, готовят в виде суспензии в стерильном изотоническом 0,9% растворе хлорида натрия. В промаркированную одноразовую полипропиленовую пробирку вместимостью 1,5 см3 шпателем помещают пробу фекалий или подстилки до нижней риски, затем вносят 600 мм3 раствора натрия хлорида. Тщательно перемешивают встряхиванием. Суспензию центрифугируют при 400g в течение 2 мин. Для выделения ДНК используют 100 мм3 верхней фракции суспензии, отобранной при помощи дозатора с наконечником с фильтром.
Выделение ДНК из анализируемой пробы, осуществляют с помощью набора реагентов «АмплиПрайм ДНК-сорб-В» (ФБУН ЦНИИЭ) или «PureLink Genomic DNA» (Invitrogen), или «QIAamp DNA Mini Kit» (Qiagen) или или «PureLink Microbiome DNA Purification Kit» (Invitrogen) в соответствии с инструкцией производителя. В качестве отрицательного контроля выделения (Kв) используют 100 мм3 воды деионизованной.
Для приготовления мастермикса «ПЦР-смесь-1» в отдельных пробирках смешивают из расчета на одну реакцию следующие реагенты: растворы прямых и обратных ПЦР-праймеров и зондов в необходимой концентрации (6 пмоль специфических праймеров aadA-F и aadA-F, 3 пмоль специфического зонда aadA-Z, 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованная вода).
Смесь с полимеразой для выявления целевого гена готовили в отдельной пробирке (из расчета на каждую реакцию с учетом контролей выделения и ПЦР), вносят по 10 мм3 «ПЦР-смеси-1», 0,5 мм3 ДНК-полимеразы, 5 мм3 ПЦР-буфера с магнием. Затем вносят по 15 мм3 приготовленной смеси с полимеразой в тонкостенные пробирки и по 10 мм3 исследуемых или контрольных проб. Запускают на амплификаторе для проведения ПЦР в режиме «реального времени» RotorGene 6000 (Corbett Research, Австралия) или RotorGene Q (Qiagen, Германия) соответствующую программу термоциклирования: удержание - 15 мин при 95°С, циклирование 15 с при 95°С, 45 с при 62°С (40 циклов, детекция на втором этапе, канал Orange).
Кривые накопления флуоресцентного сигнала анализируют с помощью программного обеспечения амплификатора, при этом должны быть установлены следующие настройки: в меню основного окна «Количественный анализ» должны быть активированы кнопки «Динамич. Фон» и «Коррек. Уклона», установлено значение «Порог Фона», равное 10%, выбрана линейная шкала графического изображения результатов, выставлен «Threshold/Порог», равный 0,05.
Результаты ПЦР исследования считаются достоверными, если получены правильные результаты для положительных - ПЦР, K+ ≤ 27 и отрицательных контролей амплификации - «нет значений» и отрицательного контроля выделения ДНК - «нет значений». При идентификации мишени результаты интерпретируются в качественном формате на основании наличия значения Ct. В образце обнаружен ген устойчивости к аминогликозидам из группы aadA, если в таблице результатов по каналу Orange определено среднее значение порогового цикла Ct ≤ 33.
Пример 1. Проверка специфичности способа выявления генов группы aadA
Для подтверждения специфичности способа готовят контрольную панель, содержащую ДНК, выделенную из различных чистых бактериальных культур. В качестве контрольных используют изоляты, которые охарактеризованы полногеномно, с указанием на присутствие или отсутствие фрагмента гена aadA, а также отсутствие или присутствие других генов устойчивости к аминогликозидам, результаты представлены в таблице 2.
Таблица 2
Результаты амплификации фрагмента генов группы aadA, выделенных из чистых культур микроорганизмов контрольной панели
пп
амплификации фрагмента гена
с геном aadA1
с генами aadA2 и strA/B
с генами aadA1 и strA/B
с генами aadA1 и aac(3)
с генами aadA1 и strA/B
с генами aadA1, aadA2, aac(3)
с генами aadA1, aadA2, aph(3)
с генами aadA2, aadA22, aac(3)
с геном strA/B
к аминогликозидам
к аминогликозидам
к аминогликозидам
к аминогликозидам
В рамках предложенной панели система детекции генов резистентности показала 100% специфичность: наблюдается амплификация только фрагмента генов группы aadA. Предлагаемый способ полностью удовлетворяют критерию специфичности.
Пример 2. Оценка аналитической чувствительности способа выявления генов группы aadA
Для определения аналитической чувствительности ПЦР используют положительный контрольный образец (ПКО), представляющий собой растворы плазмидной ДНК c известной концентрацией, содержащей соответствующие клонированные фрагменты генов группы aadA. Готовят серии десятикратных разведений плазмидной ДНК в ТЕ буфере. Структуру положительного контрольного образца подтверждают методом секвенирования по Сенгеру. Секвенирование фрагментов амплификации выполняют методом "cycle sequence" с набором ABI PRISM Big Dye v. 1.1 («Applied Biosystems»,USA), согласно инструкции изготовителя с использованием капиллярного автоматического секвенатора ABI-3100 PRISM Genetic Analyzer (Applied Biosystems, США). Капиллярный электрофорез проводяти на генетическом анализаторе ABI PRISM 3130 Genetic Analyzer после очистки образцов от избытка дидезоксинуклеотидов и солей.
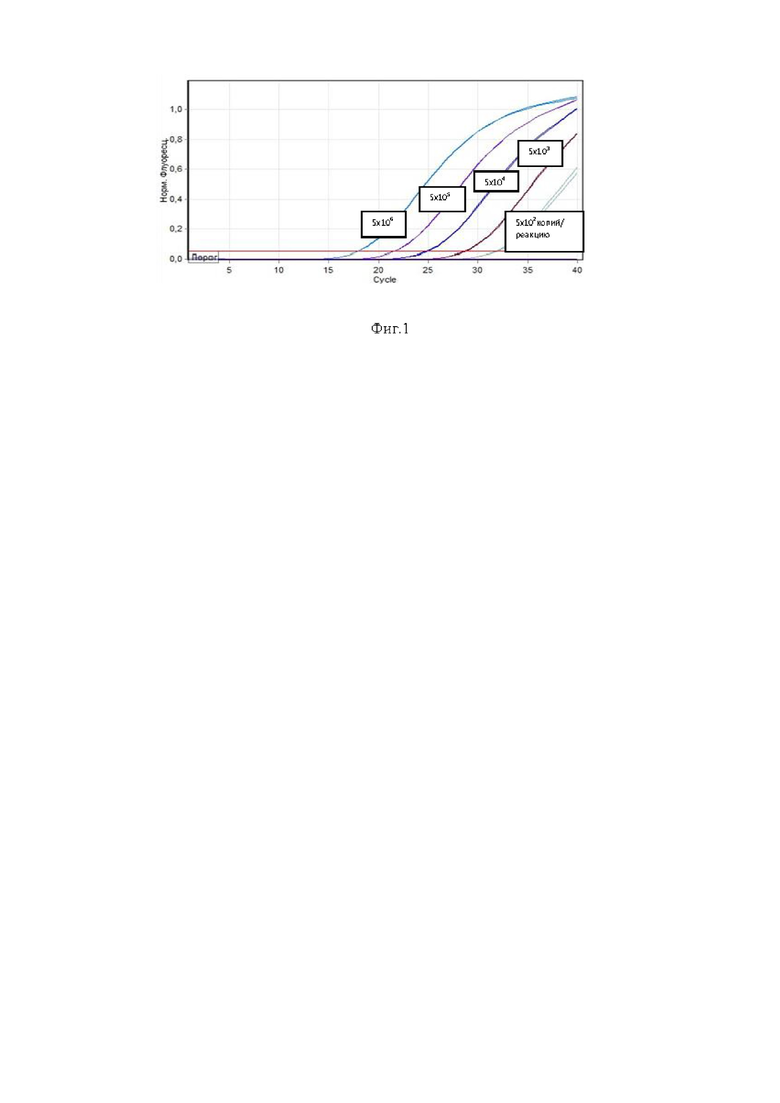
Для каждой серии разведений проводят ПЦР в двух повторах. За аналитическую чувствительность принимают наименьшую концентрацию раствора плазмидной ДНК, которая воспроизводимо дает продукт в двух повторах ПЦР. Начальная концентрация серии для генов группы aadA составляла ~5×106 копий в ПЦР (при внесении 10 мкл раствора ДНК на реакцию объёмом 25 мкл). Результаты амплификации фрагмента генов группы aadA представлены на Фиг.1 Определение чувствительности ПЦР для детекции фрагмента генов группы aadA.
Литературные источники
1. Dowling P.M. Aminoglycosides. In: Giguère S, Prescott JF, Baggot JF et al., eds. Antimicrobial Therapy in Veterinary Medicine. Ames, IA, USA: Blackwell Publishing, 2006: 207-29.
2. OIE 2021. OIE List of Antimicrobials of Veterinary Importance
3. https://www.ncbi.nlm.nih.gov/pathogens
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ обнаружения генов устойчивости к амногликозидам из
группы aadA у бактерий животного происхождения методом ПЦР с
детекцией в режиме реального времени.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2023-09-01">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023118181</ApplicationNumberText>
<FilingDate>2023-07-11</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>2023118181</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>202311881</ApplicationNumberText>
<FilingDate>2023-07-11</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="en">Федеральное государственное
бюджетное учреждение "Всероссийский государственный центр
качества и стандартизации лекарственных средств для животных и
кормов" ФГБУ "ВГНКИ", Federal state budgetary
institution "The russian state center for animal feed and drug
standardization and quality"</ApplicantName>
<ApplicantNameLatin>FGBU "VGNKI"</ApplicantNameLatin>
<InventorName languageCode="ru">Прасолова Ольга
Владимировна</InventorName>
<InventorNameLatin>Prasolova Olga Vladimirovna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ обнаружения генов
устойчивости к аминогликозидам из группы aadA у бактерий животного
происхождения методом ПЦР с детекцией в режиме "реального
времени"</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
<NonEnglishQualifier_value>unidentified</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcgatgagcgaaatgtagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttcaagtawgacgggctgata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcgcgccgaaggatgtcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ ГЕНОВ УСТОЙЧИВОСТИ К ТЕТРАЦИКЛИНАМ ИЗ ГРУППЫ tet У БАКТЕРИЙ МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ "РЕАЛЬНОГО ВРЕМЕНИ" | 2022 |
|
RU2794156C1 |
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДЛЯ ИДЕНТИФИКАЦИИ И ДЕТЕКЦИИ ГЕНОВ qnrS и qnrB, ОБЕСПЕЧИВАЮЩИХ УСТОЙЧИВОСТЬ К ФТОРХИНОЛОНАМ БАКТЕРИЙ СЕМЕЙСТВА ENTEROBACTERIACEAE, МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ «РЕАЛЬНОГО ВРЕМЕНИ» И СПОСОБ ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2810576C1 |
| Олигонуклеотидные праймеры и зонд для выявления фрагмента гена 23S рРНК бактерий семейства Enterococcaceae | 2023 |
|
RU2824466C1 |
| ШТАММ БАКТЕРИЙ SALMONELLA INFANTIS, ИСПОЛЬЗУЕМЫЙ В КАЧЕСТВЕ ПОЛОЖИТЕЛЬНОГО КОНТРОЛЯ ДЛЯ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ, А ТАКЖЕ МИКРОБИОЛОГИЧЕСКИХ ИССЛЕДОВАНИЙ, СВЯЗАННЫХ С ОПРЕДЕЛЕНИЕМ ЧУВСТВИТЕЛЬНОСТИ МИКРООРГАНИЗМОВ К АНТИБАКТЕРИАЛЬНЫМ ПРЕПАРАТАМ | 2021 |
|
RU2782213C1 |
| Способ идентификации генов, кодирующих АГЛ-лактоназу (aiiA) и лакказу (cotA) у бактерий рода Bacillus | 2024 |
|
RU2834022C1 |
| СПОСОБ ДЕТЕКЦИИ ГЕНОВ АНТИБИОТИКОУСТОЙЧИВОСТИ У ШТАММОВ ВОЗБУДИТЕЛЯ ЧУМЫ МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2011 |
|
RU2456348C1 |
| Штамм бактерий Klebsiella pneumoniae, используемый в качестве тест-культуры при детекции гена NDM методом полимеразной цепной реакции для точной диагностики и назначения антибактериальной терапии клебсиелёзных инфекций | 2022 |
|
RU2797025C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ ДНК ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННОГО АТЛАНТИЧЕСКОГО ЛОСОСЯ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2023 |
|
RU2808662C1 |
| ПОЛИРЕЗИСТЕНТНЫЙ ШТАММ БАКТЕРИЙ ESCHERICHIA COLI ДЛЯ ОПРЕДЕЛЕНИЯ БАКТЕРИЦИДНОГО ДЕЙСТВИЯ АНТИБАКТЕРИАЛЬНЫХ ПРЕПАРАТОВ В ВЕТЕРИНАРИИ | 2022 |
|
RU2797383C1 |
| ШТАММ БАКТЕРИЙ SALMONELLA ENTERICA (ВКШМ-Б-849М) В КАЧЕСТВЕ КОНТРОЛЬНОГО ШТАММА ДЛЯ МИКРОБИОЛОГИЧЕСКИХ И МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ ИССЛЕДОВАНИЙ В ОПРЕДЕЛЕНИИ АНТИБИОТИКОРЕЗИСТЕНТНОСТИ | 2020 |
|
RU2759396C1 |
Изобретение относится к области биотехнологии. Описан способ обнаружения генов устойчивости к аминогликозидам из группы aadA у бактерий животного происхождения методом ПЦР с детекцией в режиме «реального времени», включающий проведение полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени с использованием набора оригинальных олигонуклеотидов SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3. Техническим результатом заявленного изобретения является возможность идентификации генов резистентности группы aadA к аминогликозидам из образцов биологического материала животных и объектов их содержания, продовольственного сырья, пищевых продуктов, без этапа выделения бактериальных изолятов, а также чистых бактериальных культур с высокой специфичностью и чувствительностью. 1 ил., 2 табл., 2 пр.
Способ обнаружения генов устойчивости к аминогликозидам из группы aadA у бактерий животного происхождения методом ПЦР с детекцией в режиме «реального времени», включающий подготовку образца, выделение ДНК, проведение полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени с использованием набора оригинальных олигонуклеотидов SEQ ID NO 1, SEQ ID NO 2, SEQ ID NO 3, и реакционной смеси 10 мм3 «ПЦР-смеси-1», включающей растворы прямых и обратных ПЦР-праймеров и зондов в необходимой концентрации, а именно 6 пмоль специфических праймеров aadA-F и aadA-F, 3 пмоль специфического зонда aadA-Z, 2,5 мм3 раствора дНТФ с концентрацией каждого нуклеотида 1,76 ммоль/дм3, деионизованная вода, и добавлением 0,5 мм3 ДНК-полимеразы, 5 мм3 ПЦР-буфера с магнием и 10 мм3 ДНК матрицы, при этом амплификация проводится по следующей программе: удержание - 15 мин при 95°С, циклирование - 15 с при 95°С и 45 с при 62°С - 40 циклов, детекция на втором этапе, канал Orange; результаты амплификации оценивают с помощью программного обеспечения амплификатора, при этом результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на уровне 0,05 пороговой линией и устанавливают наличие в образце гена устойчивости к аминогликозидам из группы aadA при Ct≤33.
| CN 101392287 A, 25.03.2009 | |||
| CN 104212899 A, 17.12.2014 | |||
| СПОСОБ ДИАГНОСТИКИ ЧУВСТВИТЕЛЬНОСТИ ШТАММОВ MYCOBACTERIUM TUBERCULOSIS К АМИНОГЛИКОЗИДАМ | 2009 |
|
RU2409680C1 |
| Stern A.L., Van der Verren S.E., Kanchugal P.S., Näsvall J., Gutiérrez-de-Terán H., Selmer M | |||
| Structural mechanism of AadA, a dual-specificity aminoglycoside adenylyltransferase from Salmonella enterica | |||
| J Biol Chem | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Прибор для промывания газов | 1922 |
|
SU20A1 |
| doi: | |||
Авторы
Даты
2024-04-01—Публикация
2023-07-11—Подача