ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к областям молекулярной биологии и генетической инженерии, биотехнологии животных, в частности к созданию модели животного с нокаутом гена, а именно линии мышей с нокаутом гена grin3а и способу ее получения. Изобретение может быть использовано для детального изучения патогенеза и разработки терапии для нейродегенеративных заболеваний, в частности, болезни Альцгеймера. Изобретение создано при финансовой поддержке гранта СПбГУ IDPURE: 94030690.
УРОВЕНЬ ТЕХНИКИ
Болезнь Альцгеймера (БА) - одна из наиболее распространённых форм деменции, возникающая, как правило, у людей старшего возраста. Люди, страдающие от БА, сталкиваются с потерей памяти, снижением внимания, утратой речи и пространственной ориентации, нарушением базовых навыков мышления. Болезнь обычно развивается на протяжении целого ряда лет и лишает пациента способности существовать самостоятельно. В настоящее время, болезнь неизлечима и смертельна. По разным данным, заболевание поражает от 5 до 13% людей в возрасте старше 65 лет, и более 30% - в возрасте старше 85 лет (Kulichikhin KY, Fedotov SA, Rubel MS, et al. Development of molecular tools for diagnosis of Alzheimer's disease that are based on detection of amyloidogenic proteins. Prion. 2021;15(1):56-69). Большинство случаев БА являются спорадическими, и лишь небольшая часть (менее 5%) передается по наследству (Berkis LM, Yu CE, Bird TD, Tsuang DW. Genetics of Alzheimer diseas. J Geriart Psychiatry Neurol. 2010;23(4):213-227). Ассоциация по изучению болезни Альцгеймера определяет БА как деменцию, связанную с накоплением агрегированных форм пептида амилоид β и белка Tau в головном мозге (2020 Alzheimer’s Disease Facts and Figures. Alzheimers Dement. 2020;16(3):391-460). Однако причины, способствующие агрегации данных белков у большинства пациентов, остаются дискуссионными. Также остаются непонятными механизмы, приводящие к патологическим реакциям, вызывающим гибель нейронов мозга. Среди гипотез возникновения патофизиологии при БА большую экспериментальную поддержку имеет кальциевая гипотеза, основанная на том, что нарушение регуляции гомеостаза ионов Ca2+ является одним из сопутствующих факторов при БА (Zhong W, Wu A, Berglund K, et al. Pathogenesis of sporadic Alzheimer's disease by deficiency of NMDA receptor subunit GluN3A. Alzheimers Dement. 2022;18(2):222-239). При развитии БА наблюдается гиперактивность рецепторов NMDA. Рецептор NMDA является основным медиатором поступления ионов Ca2+. Гиперактивация рецепторов NMDA приводит к излишнему поступлению ионов Ca2+ в клетку, что активирует ряд ферментов, разрушающих цитозольные структуры, и в результате запускает апоптоз клетки. Есть предположение, что хроническая эксайтотоксичность также может приводить к развитию БА. Эксайтотоксичность - это патологический процесс, ведущий к повреждению и гибели нервных клеток под воздействием нейромедиатора L-глутамата, способного гиперактивировать рецептор NMDA. В мозге пациентов с БА белок GluN3A присутствует в сниженной по сравнению с нормой концентрации, поэтому он может представлять собой первичный патогенный фактор, ранний биомаркер и независимую от амилоида терапевтическую мишень. Белок Glun3A является субъединицей рецептора NMDA и кодируется геном grin3а. Структурно рецепторы семейства NMDA состоят из 4 субъединиц. Данные субъединицы разделены на три основных подсемейства: GluN1, GluN2 (A-D) и GluN3 (A-B). Процесс сборки рецептора NMDA, содержащего GluN3A, происходит в два этапа. На первом этапе субъединица GluN1 связывается в гетеродимеры или с субъединицей GluN2, или GluN3. Далее эти два гетеродимера образуют тетрамерные субъединицы (Schüler T, Mesic I, Madry C, Bartholomäus I, Laube B. Formation of NR1/NR2 and NR1/NR3 heterodimers constitutes the initial step in N-methyl-D-aspartate receptor assembly. J Biol Chem. 2008;283(1):37-46). Однако рецепторы NMDA, в состав которых входит субъединица GluN3A, не способны активироваться глутаматом, их активация осуществляется глицином. Таким образом, GluN3A является ингибирующей субъединицей рецептора NMDA. Было выдвинуто предположение, что GluN3A имеет решающее значение для устойчивого гомеостаза Ca2+, и его дефицит является патогенным для БА (Murillo A, Navarro AI, Puelles E, Zhang Y, Petros TJ, Pérez-Otaño I. Temporal Dynamics and Neuronal Specificity of Grin3a Expression in the Mouse Forebrain. Cereb Cortex. 2021;31(4):1914-1926).
Изучение моделей животных с нокаутом гена grin3а имеет большое значение для понимания патогенеза нейродегенеративных заболеваний головного мозга, в частности БА. Для поиска новых терапевтических подходов и доклинических исследований новых лекарственных препаратов для лечения БА необходимы адекватные животные модели.
Известна мышиная модель с нокаутом гена grin3a (Das S, Sasaki YF, Rothe T, et al. Increased NMDA current and spine density in mice lacking the NMDA receptor subunit NR3A. Nature. 1998;393(6683):377-381). Однако данная модель была создана методом введения культивированных эмбриональных стволовых клеток с нокаутом гена grin3a в бластоцисты мыши. Получение нокаута гена grin3a в культивированных эмбриональных стволовых клетках осуществлялось с помощью введения генетической конструкции, содержащей неомицин-позитивную селекционную кассету (Neo), путем гомологичной рекомбинации. Недостатком этой модели является то, что, как было показано, экспрессия гена Neo может вызвать изменения в экспрессии других генов, расположенных рядом с Neo-кассетой, что может исказить полученные данные при изучении патогенеза и разработки терапии БА (Jin C, Kang H, Yoo T, et al. The Neomycin Resistance Cassette in the Targeted Allele of Shank3B Knock-Out Mice Has Potential Off-Target Effects to Produce an Unusual Shank3 Isoform. Front Mol Neurosci. 2021;13:614435; Kang SS, Kurti A, Baker KE, et al. Behavioral and transcriptomic analysis of Trem2-null mice: not all knockout mice are created equal. Hum Mol Genet. 2018;27(2):211-223; Valera A, Perales JC, Hatzoglou M, Bosch F. Expression of the neomycin-resistance (neo) gene induces alterations in gene expression and metabolism. Hum Gene Ther. 1994;5(4):449-456).
В 2012-2013 годах в генной инженерии появились принципиально новые методы манипулирования генетическим материалом, основанные на применении систем CRISPR/Cas. CRISPR/Cas систему стали успешно применять для изменения генома клеток млекопитающих, что привело к созданию ряда эффективных технологий генетической инженерии животных, в том числе мышей (Williams A, Henao-Mejia J, Flavell RA. Editing the Mouse Genome Using the CRISPR-Cas9 System. Cold Spring Harb Protoc. 2016;2016(2):pdb.top087536). На данный момент система CRISPR/Cas выглядит наиболее перспективным инструментом редактирования генома, поскольку она универсальна, довольно проста в исполнении и обладает высокой сайт-специфичностью.
В данной области техники остается потребность в новых, улучшенных животных моделях БА, в том числе полученных с помощью системы CRISPR/Cas.
Настоящее изобретение преодолевает недостатки, присущие известным решениям, и решает задачу создания генетически сконструированной мышиной модели БА с точным нокаутом гена grin3а, при котором не затрагивается экспрессия других генов, подходящей для детального изучения патогенеза БА и доклинической оценки потенциальных методов лечения БА, предназначенных для использования у людей.
РАСКРЫТИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Краткое описание изобретения
В одном аспекте изобретения предложена линия генетически модифицированных мышей для моделирования болезни Альцгеймера, в которой в нуклеотидной последовательности гена grin3a делетированы нуклеотиды AC в позициях, соответствующих позициям 49844568-49844569 в последовательности, содержащейся в базе данных GenBank и имеющей идентификационный номер ID NC_000070.7, которая соответствует хромосоме 4 линии C57BL/6J Mus musculus сборки генома GRCm39. В частных воплощениях нуклеотидная последовательность гена grin3a содержит последовательность SEQ ID NO: 11.
Кроме того, предложена sgРНК (единая направляющая РНК) для получения линии мышей согласно изобретению с использованием системы CRISPR/Cas9, содержащая последовательность SEQ ID NO: 1. В частных воплощениях указанная sgРНК имеет последовательность SEQ ID NO: 2, SEQ ID NO: 3 или SEQ ID NO: 12.Также предложена комбинация crRNA/tracrRNA (CRISPR-РНК/транс-активирующая crRNA) для получения линии мышей согласно изобретению с использованием системы CRISPR/Cas9, где crRNA содержит последовательность SEQ ID NO: 1. В частном воплощении указанной комбинации crRNA имеет последовательность SEQ ID NO: 8, и tracrRNA имеет последовательность SEQ ID NO: 9. В альтернативном частном воплощении указанной комбинации crRNA имеет последовательность SEQ ID NO: 13, и tracrRNA имеет последовательность SEQ ID NO: 14.
В еще одном аспекте изобретения предложен способ получения линии мышей согласно изобретению с использованием системы CRISPR/Cas9, включающий введение компонентов указанной системы в оплодотворенную яйцеклетку мыши, причем используют sgРНК или комбинацию согласно изобретению. В частном воплощении указанного способа sgРНК или комбинацию вводят в сочетании с белком Cas9. В другом частном воплощении указанного способа sgРНК или комбинацию вводят в сочетании с мРНК Cas9. В другом частном воплощении указанного способа sgРНК или комбинацию вводят в сочетании с белком Cas9 и мРНК Cas9. В другом частном воплощении указанного способа компоненты системы CRISPR/Cas9 вводят в виде плазмиды. В предпочтительном воплощении указанного способа Cas9 представляет собой SpCas9 (Cas9 Streptococcus pyogenes).
Также предложено применение линии мышей согласно изобретению в качестве модели болезни Альцгеймера.
Кроме того, предложен набор праймеров для получения sgРНК согласно изобретению, содержащий:
прямой праймер, имеющий нуклеотидную последовательность SEQ ID NO: 4, и
обратный праймер, имеющий нуклеотидную последовательность SEQ ID NO: 5.
Также предложена матричная ДНК для транскрипции sgРНК in vitro.
Путем тщательного биоинформатического анализа последовательности гена grin3a авторы настоящего изобретения осуществили подбор направляющей РНК к первому экзону гена grin3а (обозначаемой sgРНК-grin3а) для использования с нуклеазой Cas9 и получили конструкцию CRISPR/Cas9, содержащую sgРНК-grin3а и белок Cas9, предпочтительно SpCas9. С помощью этой конструкции была получена мышиная модель БА c нокаутом гена grin3а за счет направленного введения делеции двух нуклеотидов в первом экзоне гена grin3а, приводящей к сдвигу рамки считывания и появлению стоп-кодона.
Таким образом, авторами настоящего изобретения была создана ранее не описанная модель, пригодная для детального изучения патогенеза БА и доклинической оценки потенциальных методов лечения БА, предназначенных для использования у людей.
Преимуществом настоящего изобретения является создание подходящей и легко генерируемой модели, которая может быть использована для детального изучения патогенеза БА и доклинической оценки потенциальных методов лечения БА, предназначенных для использования у людей. В мышиной модели БА согласно изобретению осуществлен точный нокаут гена grin3а, при котором не затрагивается экспрессия других генов. Полученная линия генетически модифицированных мышей была переведена на генетический фон С57BL/6, и поэтому является стабильным и удобным продуктом для исследований.
Подробное описание изобретения
Терминология
Все используемые термины имеют значения, соответствующие принятым в данной области, если более специфическое или иное толкование не определено в данном документе и не следует из контекста.
При использовании терминов в единственном числе подразумевается, там, где допускает контекст, что возможно и множественное число, и наоборот.
Термины «включающий», «содержащий», «имеющий» и т. п. означают, что помимо указанных элементов могут содержаться иные неоговоренные (несущественные) элементы. В каждом случае данные термины также охватывают значение «состоящий из», т. е. в одном из воплощений подразумевают, что описываемый объект характеризуется наличием только указанных элементов.
Термин «делеция» относится к утрате одного или нескольких оснований в нуклеотидной последовательности (в частности, хромосомы). В случае делеции длиной, не кратной трем основаниям, в кодирующей области гена нарушается порядок чтения кодонов, то есть, происходит сдвиг рамки считывания, что, как правило, приводит к преждевременной терминации трансляции и прекращению продукции соответствующего белка.
Термин «нокаут» или «KO» гена относится к методу генной инженерии, при котором из организма удаляют или делают неработоспособным заданный ген или гены.
Термин «GenBank» относится к базе данных, находящейся в открытом доступе, содержащей все аннотированные последовательности ДНК и РНК, а также последовательности закодированных в них белков.
Термин «grin3a» относится к гену grin3a, который кодирует регуляторную субъединицу ионотропного глутаматного рецептора N-метил-D-аспартата (NMDAR).
Выражение «дикий тип» или «WT» означает типичную форму организма, штамма, гена или характеристики, которые встречаются в природе в отличие от мутантных или вариантных форм.
Способы и методы биоинженерии по настоящему изобретению обычно осуществляют в соответствии с традиционными способами, хорошо известными в данной области, и как описано в различных общих и более конкретных источниках, которые цитируются и обсуждаются в настоящем описании, если не указано иное. См., например, Sambrook J. & Russell D. Molecular Cloning: A Laboratory Manual, 3rd ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. (2000); Ausubel et al., Short Protocols in Molecular Biology: A Compendium of Methods from Current Protocols in Molecular Biology, Wiley, John & Sons, Inc. (2002); Harlow and Lane Using Antibodies: A Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. (1998); и Coligan et al., Short Protocols in Protein Science, Wiley, John & Sons, Inc. (2003).
Мутация в гене grin3a, реализуемая согласно изобретению
Линия генетически модифицированных мышей согласно изобретению имеет делецию двух нуклеотидов в первом экзоне гена grin3a, который кодирует регуляторную субъединицу ионотропного глутаматного рецептора N-метил-D-аспартата (NMDAR), которая приводит к сдвигу рамки считывания и прекращению продукции белка Glun3A.
А именно, в нуклеотидной последовательности гена grin3a делетированы нуклеотиды AC в позициях, соответствующих позициям 49844568-49844569 в последовательности GenBank ID NC_000070.7. Специалистам в области генной инженерии ясно, что обозначение «GenBank ID NC_000070.7», или указание «последовательность, содержащаяся в базе данных GenBank и имеющая идентификационный номер ID NC_000070.7» представляет собой определение последовательности в базе данных GenBank, которое однозначно идентифицирует ее как хромосому 4 линии C57BL/6J Mus musculus сборки генома GRCm39, которая и содержит ген grin3a.
Альтернативно, при использовании общепринятой номенклатуры вариантов последовательностей, предложенной в den Dunnen JT, Dalgleish R, Maglott DR, et al. HGVS Recommendations for the Description of Sequence Variants: 2016 Update. Hum Mutat. 2016;37(6):564-569 (см. также https://hgvs-nomenclature.org/stable/recommendations/DNA/deletion/), линия генетически модифицированных мышей согласно изобретению может быть охарактеризована как имеющая делецию в гене grin3a, представляющую собой NC_000070.7:g.49844568_49844569del.
Альтернативно, линия генетически модифицированных мышей согласно изобретению может быть охарактеризована как имеющая фрагмент с делецией двух нуклеотидов в гене grin3a, как показано в SEQ ID NO: 11.
Специалисту ясно, что все приведенные выше варианты определения линии мышей согласно изобретению характеризуют одну и ту же мутацию гена grin3a.
При этом специалисту также ясно, что в различных линиях мышей последовательность хромосомы 4, и в том числе, последовательность гена grin3a, могут иметь различные вариации, в том числе вставки и/или делеции. Таким образом в некоторых конкретных воплощениях изобретения номера позиций, в которых осуществляется делеция, могут отличаться от 49844568-49844569 относительно последовательности хромосомы 4, имеющейся у той линии мышей, которую используют в соответствующем воплощении для получения генетически модифицированной линии по изобретению (но при этом они будут структурно и функционально соответствовать позициям 49844568-49844569 в последовательности GenBank ID NC_000070.7) и/или последовательность нуклеотидов, фланкирующих участок с делецией, может отличаться от SEQ ID NO: 11.
Система CRISPR/Cas
CRISPR/Cas (короткие палиндромные повторы, регулярно расположенные группами, и ассоциированная с ними система) - это система адаптивного приобретенного иммунитета бактерий и архей, направленная на уничтожение проникшей в клетку чужеродной ДНК, например, фагов или плазмид. К настоящему времени описано несколько различных типов систем CRISPR/Cas (см. обзор Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol. 2015;13(11):722-736). Наиболее изученной и широко используемой в генной инженерии на сегодняшний день является система CRISPR/Cas II типа, которая состоит из трех генов, кодирующих CRISPR-РНК (crRNA), транс-активирующую РНК (tracrRNA) и нуклеазу Cas9. В структуру crRNA входит спейсер длиной около 20 нуклеотидов, комплементарный целевой последовательности (также называемой протоспейсером) в геноме.
Дуплекс, образованный tracrRNA и crRNA, распознается и образует комплекс с белком Cas9, который направляется к целевой последовательности последовательностью crRNA, которая гибридизуется с целевой последовательностью. Направляемая crRNA нуклеаза Cas9 вносит двухцепочечный разрез между 3 и 4 нуклеотидами от 3’-конца протоспейсера, к которому примыкает последовательность мотива, смежного с протоспейсером (РАМ).
В контексте образования комплекса CRISPR «целевая последовательность» или «целевой фрагмент» означает последовательность, которая находится в направлении 3’ от мотива, смежного с протоспейсером (РАМ), по отношению к которой спейсер разработан так, чтобы обладать комплементарностью к ней, где гибридизация между целевой последовательностью и спейсером способствует образованию комплекса CRISPR. Полная комплементарность не обязательна при условии, что имеет место достаточная комплементарность для осуществления гибридизации и способствования образованию комплекса CRISPR.
Был создан и упрощенный вариант системы, в котором вместо crRNA и tracrRNA, транскрибируемых отдельно, используется единственная sgРНК (единая направляющая РНК), в которой зрелая crRNA соединена с частью tracrRNA с помощью синтетической структуры по типу «стебель-петля» для имитации естественного дуплекса crRNA:tracrRNA. Такая упрощенная система функционирует в виде комплекса из белка Cas9 и sgРНК.
В контексте настоящей заявки под термином «направляющая РНК» понимается РНК-компонент системы CRISPR/Cas9, который представляет собой пару crRNA/tracrRNA или sgРНК, в зависимости от типа используемой системы.
Для делеции двух нуклеотидов в первом экзоне гена grin3a и получения заявленной линии генетически модифицированных мышей может быть использована как система на основе crRNA/tracrRNA, так и упрощенная система на основе sgРНК. Выбор конкретной системы может быть осуществлен специалистом.
Подробный обзор существующих систем CRISPR/Cas приведен в статье А.В. Смирнов и др. Система CRISPR/Cas9 - универсальный инструмент геномной инженерии. Вавиловский журнал генетики и селекции. 2016;20(4):493-510.
С помощью системы CRISPR/Cas можно осуществлять все виды модификаций генома: вносить точечные мутации, встраивать в определенные места новые гены, либо осуществлять масштабные геномные мутации.
Поскольку технология CRISPR/Cas давно и интенсивно используется в генной инженерии, специалистам хорошо известно, как именно осуществляется редактирование генов с помощью этой системы. Например, руководство по использованию системы CRISPR/Cas содержится в Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 2013;8(11):2281-2308.
Оптимизация последовательности sgРНК
Тогда как последовательность, комплементарная целевому фрагменту гена (SEQ ID NO: 1), является существенной для осуществления изобретения, остальная (консервативная) часть молекулы sgРНК и, в случае использования пары crRNA/tracrRNA - молекула tracrRNA и часть crRNA, которая комплементарна tracrRNA, может иметь любую пригодную с точки зрения специалиста последовательность, каковые хорошо известны из уровня техники. Ввиду этого, настоящее изобретение не ограничивается только лишь теми последовательностями, которые непосредственно раскрыты в описании, поскольку они приведены лишь в качестве иллюстрации изобретения, а специалист может использовать раскрытую здесь идею и реализовать изобретение в виде различных дополнительных вариантов, обеспечивающих тот же технический результат.
Разумеется, специалистам известны различные варианты Cas9, которые могут использоваться в технологии CRISPR/Cas9, в том числе рекомбинантные, при условии, что они могут связываться с целевой последовательностью гена и осуществлять соответствующую нуклеазную активность. Прежде всего, это определяется тем, присутствует ли в целевой последовательности PAM, распознаваемый нуклеазой. Канонический PAM - NGG (где N - любой нуклеотид) - распознается, например, SpCas9 (Streptococcus pyogenes), которая является предпочтительным вариантом.
Вышеуказанные последовательности, которые могут варьировать, могут быть дополнительно модифицированы или оптимизированы, как известно специалистам (см. Dang Y, Jia G, Choi J, et al. Optimizing sgRNA structure to improve CRISPR-Cas9 knockout efficiency. Genome Biol. 2015;16:280; Moon SB, Kim DY, Ko JH, Kim JS, Kim YS. Improving CRISPR Genome Editing by Engineering Guide RNAs. Trends Biotechnol. 2019;37(8):870-881). Например, дуплекс повтор:антиповтор (ниже последовательности, комплементарной целевому фрагменту гена) в структуре sgРНК может варьировать по длине, от наиболее короткой, показанной в SEQ ID NO: 3 или SEQ ID NO: 12 (…GUUUUAGAGCUAGAAAUAG…) до, например, показанной в SEQ ID NO: 2 (…GUUUUAGAGCUAUGCUGUUUUGAAAAAAACAGCAUAG…) или еще длиннее (…GUUUUAGAGCUAUGCUGUUUUGGAAACAAAACAGCAUAG…); а также каждый промежуточный по длине вариант, отличающийся от указанных на 1 или более пар нуклеотидов дуплекса, а именно (…GUUUUAGAGCUAUGAAAAUAG…), (…GUUUUAGAGCUAUGGAAACAUAG…), (…GUUUUAGAGCUAUGCGAAAGCAUAG…), (…GUUUUAGAGCUAUGCUGAAAAGCAUAG…), (…GUUUUAGAGCUAUGCUGGAAACAGCAUAG…), (…GUUUUAGAGCUAUGCUGUGAAAACAGCAUAG…), (…GUUUUAGAGCUAUGCUGUUGAAAAACAGCAUAG…), (…GUUUUAGAGCUAUGCUGUUUGAAAAAACAGCAUAG…).
Все указанные выше последовательности, а также иные, известные специалистам из литературы, равным образом пригодны для реализации в настоящем изобретении.
Вне зависимости от длины дуплекса также можно внести замену U→A в полиU области (стоп-сигнал для РНК-полимеразы), следующей за последовательностью, комплементарной целевому фрагменту гена (SEQ ID NO: 1). Замена может быть произведена в любом из положений (…GAUUUA…, …GUAUUA…, …GUUAU…, …GUUUA…), предпочтительно в крайнем-3’ положении (…GUUUA…). Аналогичным образом, можно внести замену U→C или U→G.
3’-Концевая последовательность ниже дуплекса повтор:антиповтор, которая, как правило, содержит 2-3 шпильки (стебель-кольцо), линкер(ы) и негибридизованный 3’-концевой участок, также может в значительной мере варьировать. Показанные в SEQ ID NO: 2 и 3 включают в себя типичные примеры таких 3’-концевых последовательностей, но изобретение не ограничено ими, поскольку в литературе описано множество вариантов этих последовательностей, которые обеспечивают функционирование системы CRISPR/Cas9. 3’-Концевая последовательность может быть модифицирована, например, для того чтобы регулировать активность, путем обеспечения трехмерных структур, связывающих лиганд, с тем чтобы в присутствии такого лиганда редактирование не происходило. Так, описана конструкция sgРНК с богатой гуанином квадруплексной структурой на 3’-конце, обеспечивающей связь с низкомолекулярным лигандом (Liu, Xingyu et al. G-quadruplex-guided RNA engineering to modulate CRISPR-based genomic regulation. Nucleic Acids Research. 2022;50(19):11387-11400), включающая следующую последовательность на 3’-конце: …AAGUGGGCACCGGGUCGGUGGGGUUUU-3’.
Негибридизованный 3’-концевой участок может иметь различную последовательность, например UCG, полиU (UU, UUU, UUUU, UUUUU и т.д.).
В случае реализации изобретения с crRNA/tracrRNA, их последовательности по существу повторяют соответствующие части sgРНК. 3’-конец crRNA ниже последовательности, комплементарной целевому фрагменту гена, может выглядеть, соответственно, как GUUUUAGAGCUAUGCUGUUUUG-3’, а 5’-конец tracrRNA - 5’-GGAACCAUUCAAAACAGCAUAGCAAGUUAAAAU... или 5’- AAACAGCAUAGCAAGUUAAAAU... Эти последовательности 3’- и 5’-концевых участков не являются ограничивающими и могут быть изменены с учетом вышесказанного и так, как известно специалистам из литературы.
Молекула sgРНК или молекулы crRNA/tracrRNA могут быть химически модифицированы для того, чтобы, например, повысить устойчивость к экзонуклеазной активности в клетке, снизить иммунный ответ или с иной целью, как известно специалистам. Можно модифицировать входящие в нуклеозиды сахара и/или азотистые основания, а также связи между сахарами. В качестве неограничивающего примера, рибонуклеиновые основания могут быть заменены на дезоксирибонуклеиновые или иные. В частности, любой U может быть заменен на T. Арсенал доступных специалистам методов модификаций подробно раскрыт в литературе (см., например, Chen Q, Zhang Y, Yin H. Recent advances in chemical modifications of guide RNA, mRNA and donor template for CRISPR-mediated genome editing. Adv Drug Deliv Rev. 2021;168:246-258).
Методы доставки
Могут быть использованы различные методы доставки компонентов системы CRISPR/Cas9. Cas9 может быть доставлена в виде молекулы ДНК или мРНК, кодирующей ген cas9, или в виде белка, а также их комбинации. В случае доставки Cas9 в виде молекулы ДНК используют плазмиды или вирусные векторы, кодирующие белок Cas9 и молекулу sgРНК или молекулы crRNA/tracrRNA. Компоненты системы могут кодироваться в одной плазмиде/вирусном векторе или в разных плазмидах/вирусных векторах, один из которых кодирует белок Cas9, а другой - молекулу sgРНК или молекулы crRNA/tracrRNA. В случае доставки Cas9 в виде молекулы мРНК осуществляют доставку мРНК, кодирующей ген cas9, и молекулы sgРНК или молекул crRNA/tracrRNA. В случае доставки Cas9 в виде белка осуществляют доставку очищенного белка Cas9 и молекулы sgРНК или молекул crRNA/tracrRNA. Альтернативно, белок Cas9 и sgРНК или crRNA/tracrRNA объединяют в рибонуклеопротеиновый комплекс (RNP) и доставляют в виде одного элемента.
Наиболее распространенными и эффективными методами доставки являются системы доставки на основе вирусных векторов, вирусоподобных частиц (VLP) и проникающих в клетку пептидов (CPP); химические методы, в которых используются липосомы, наночастицы золота и липидные наночастицы; а также физические методы, включающие микроинъекцию, электропорацию, сонопорацию, гидродинамическую инъекцию и т.д. Системы доставки на основе вирусных векторов включают аденовирусные, аденоассоциированные вирусные и лентивирусные векторы. Дополнительные методы доставки компонентов системы CRISPR/Cas9 в клетки известны специалистам в данной области, см., например, обзор в Taha EA, Lee J, Hotta A. Delivery of CRISPR-Cas tools for in vivo genome editing therapy: Trends and challenges. J Control Release. 2022;342:345-361; Glass Z, Lee M, Li Y, Xu Q. Engineering the Delivery System for CRISPR-Based Genome Editing. Trends Biotechnol. 2018;36(2):173-185. Одним из предпочтительных способов доставки компонентов системы CRiSPR/Cas9 в форме РНК либо ДНК является микроинъекция в оплодотворенные яйцеклетки (зиготы) мышей.
В некоторых воплощениях sgРНК или комбинацию crRNA/tracrRNA вводят в сочетании с белком Cas9. В другом воплощении sgРНК или комбинацию crRNA/tracrRNA вводят в сочетании с мРНК Cas9. В другом воплощении sgРНК или комбинацию crRNA/tracrRNA вводят в сочетании с белком Cas9 и мРНК Cas9. В другом частном воплощении компоненты системы CRISPR/Cas9 вводят в виде плазмиды. Примерами такой плазмиды являются плазмиды на основе векторов pX330-U6-Chimeric_BB-CBh-hSpCas9 (Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013;339(6121):819-823) или pSpCas9(BB)-2A-GFP (PX458) (Ran, F., Hsu, P., Wright, J. et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 2013 Nov;8(11):2281-308). Эти плазмиды содержат ген SpCas9 и консервативную часть sgРНК, поэтому при клонировании в них комплементарного спейсера их также можно доставлять в зиготы с помощью микроинъекции или электропорации.
Все указанные выше методы доставки, а также иные, известные специалистам из литературы, равным образом пригодны для реализации в настоящем изобретении.
Линии мышей
В рамках изобретения с помощью предложенных авторами изобретения sgРНК или дуплекса crRNA:tracrRNA можно модифицировать любые доступные линии мышей, как инбредные (например, CBA), так и аутбредные (например, CD1), при условии, что спейсер с SEQ ID NO: 1 в молекуле sgРНК или дуплексе crRNA:tracrRNA в достаточной степени комплементарен геномной последовательности гена grin3a.
Иллюстративные инбредные линии мышей включают, не ограничиваясь перечисленным, линии CBA, BALB/c, C57BL/6, DBA/2, C3H, FVB, MRL, NZW и 129. Иллюстративные аутбредные линии мышей включают, не ограничиваясь перечисленным, линии CD-1, ICR, NMRI, CFW, SHK и мышей SCID (мыши с тяжелым комбинированным иммунодефицитом).
Одной из предпочтительных линий мышей является линия C57BL/6, которая широко используется в биомедицинских исследованиях и поэтому является особенно подходящей для получения модели заболевания человека, такого как БА. Таким образом, в предпочтительном воплощении генетически модифицированную линию мышей переводят на «генетический фон» линии C57BL/6. При этом известно, что самки инбредной линии С57BL/6 дают меньше потомства, их зиготы хуже переносят манипуляции in vitro, поэтому для гарантированного получения оплодотворенных клеток в предпочтительных воплощениях используют самок гибридов первого поколения F1(CBA×C57BL/6).
Подробный обзор различных линий мышей, подходящих для использования в лаборатории, приведен в источнике Suckow, M.A., Hashway, S., & Pritchett-Corning, K.R. (2023). The Laboratory Mouse (3rd ed.). CRC Press.
Любая из вышеперечисленных линий мышей также может быть использована в качестве реципиента для подсаживания оплодотворенных яйцеклеток в способе получения линии генетически модифицированных мышей. Особенно предпочтительной является аутбредная линия CD1. Для этой линии характерна высокая плодовитость и хорошо развитый материнский инстинкт. Более того, преимуществом использования линии CD1 является белый цвет меха, что важно при подсадке эмбрионов C57BL/6.
Способ получения линии генетически модифицированных мышей
В данном изобретении реализована линия генетически модифицированных мышей с мутацией гена grin3a со сдвигом рамки считывания и преждевременной терминацией трансляции на уровне первого экзона, приводящей к прекращению продукции белка Glun3A. Способ получения указанной линии мышей включает введение компонентов системы CRISPR/Cas9 в оплодотворенную яйцеклетку мыши, причем используют sgРНК или комбинацию crRNA/tracrRNA, описанные выше. Поскольку технология CRISPR/Cas9 давно и интенсивно используется в генной инженерии для получения животных моделей заболеваний, специалистам хорошо известно, какие именно этапы способа должны быть реализованы при редактировании генов млекопитающих (см., например, патенты RU 2764650 C1, RU 2768048 C1, RU 2791687 C1).
В частном воплощении способ включает микроинъекцию смеси sgРНК, комплементарной фрагменту первого экзона гена grin3а, способной образовывать комплекс с белком Cas9 и гибридизоваться с целевой последовательностью гена grin3а, которая находится в направлении 3’ от мотива, смежного с протоспейсером (РАМ), содержащей SEQ ID NO: 1, и белка Cas9 в оплодотворенные яйцеклетки мышей, подсадку оплодотворенных яйцеклеток после микроинъекций в матку псевдобеременной самке мыши, генотопирование рожденных детенышей и выведение линии мышей C57BL/6-grin3аKO, гомозиготных по нокауту гена. В частном воплощении sgРНК имеет последовательность SEQ ID NO: 2 или SEQ ID NO: 3. В альтернативном воплощении вместо sgРНК используют комбинацию crRNA/tracrRNA, где crRNA имеет последовательность SEQ ID NO: 8. В частном воплощении tracrRNA в указанной комбинации имеет последовательность SEQ ID NO: 9.
В предпочтительном воплощении способ характеризуется следующими стадиями:
а) осуществляют микроинъекцию конструкции, содержащей sgРНК-grin3а, содержащей SEQ ID NO: 1, и белок Cas9, в цитоплазму оплодотворенной яйцеклетки мыши гибрида F1(CBA×C57BL/6);
б) выявляют жизнеспособные зиготы;
в) пересаживают выжившие зиготы из стадии (б) псевдобеременным самкам-реципиентам, имеющим копулятивную пробку после ссаживания с вазэктомированными самцами;
г) получают новорожденных мышей поколения F0 на 21 день после пересадки зигот из стадии (в);
д) подтверждают наличие делеции двух нуклеотидов в гене grin3a, приводящей к сдвигу рамки считывания, путем амплификации целевого участка гена grin3a с последующим секвенированием фрагмента;
е) отбирают мышей с наличием делеции двух нуклеотидов в гене grin3a;
ж) проводят скрещивание мышей из стадии (е) с мышами дикого типа линии C57BL/6 для получения мышей-гетерозигот (F1) с делецией двух нуклеотидов в гене grin3a;
з) проводят скрещивание мышей из стадии (ж) для получения гомозиготной линии мышей, несущих делецию двух нуклеотидов в гене grin3a.
В частном воплощении sgРНК-grin3а имеет последовательность SEQ ID NO: 2 или SEQ ID NO: 3. В альтернативном воплощении вместо sgРНК-grin3а используют комбинацию crRNA/tracrRNA-grin3а, где crRNA имеет последовательность SEQ ID NO: 8. В частном воплощении tracrRNA в указанной комбинации имеет последовательность SEQ ID NO: 9.
Описание последовательностей
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На фиг. 1 представлено схематическое изображение введения делеции двух нуклеотидов с помощью системы CRISPR/Cas9 направленного редактирования генома согласно одному из воплощений изобретения. WT обозначает ген дикого типа; KO обозначает ген с делецией.
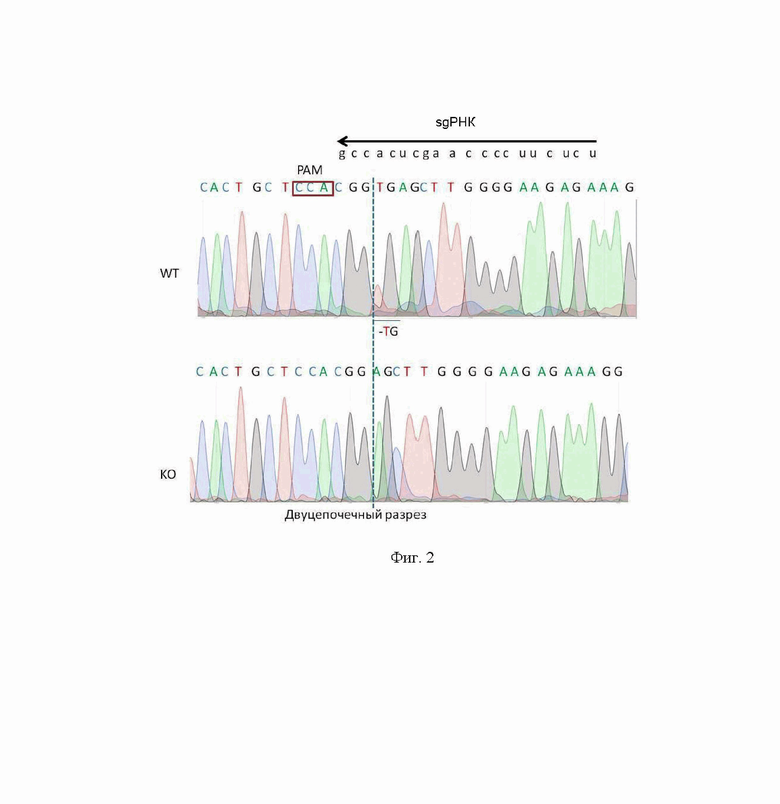
На фиг. 2 представлена хроматограмма, иллюстрирующая последовательность целевого ПЦР-фрагмента ДНК гена grin3а с делецией двух нуклеотидов согласно изобретению.
ОСУЩЕСТВЛЕНИЕ ИЗОБРЕТЕНИЯ
Пример 1
Для получения линии трансгенных мышей на первом этапе готовили генетическую конструкцию для направленного введения мутации в ген grin3a мыши, содержащую единую направляющую РНК (sgРНК-grin3a), нацеленную на данный ген, с последовательностью SEQ ID NO: 12. Подбор праймеров для получения sgРНК-grin3a для белка Cas9 проводили с помощью программы Synthego (https://www.synthego.com/products/bioinformatics/crispr-design-tool). Матричную ДНК, используемую для транскрипции sgРНК-grin3a in vitro, получали с помощью двух праймеров.
Прямой праймер с последовательностью SEQ ID NO: 4 содержал последовательность промотора T7 РНК полимеразы, последовательность спейсера и 20 нуклеотидов sgРНК.
Обратный праймер с последовательностью SEQ ID NO: 5 содержал полную последовательность sgРНК-grin3a.
Транскрипцию in vitro проводили c использованием РНК полимеразы T7 («Invitrogen», США). Для очистки sgРНК-grin3a добавляли 1 мл тризола («Thermo Fisher Scientific», США) к 100 мкл транскрипционной смеси, хорошо перемешивали, инкубировали 5 минут при комнатной температуре. Затем добавляли 200 мкл хлороформа, сильно перемешивали в течение 15 секунд и инкубировали 3 минуты при комнатной температуре. Центрифугировали смесь, 15 мин, 12000x g, при температуре +4°С. Отбирали супернатант и переносили в стерильную микропробирку типа Эппендорф. Для осаждения РНК добавляли равный объем изопропанола, инкубировали 10 минут при комнатной температуре, затем центрифугировали 10 мин, 12000х g при температуре +4°С. Осадок РНК промывали два раза добавлением 1 мл 75% этилового спирта. Высушивали на воздухе и растворяли в 30 мкл деионизированной воды. Инкубировали 10 минут при 55°С. Измеряли концентрацию РНК с помощью спектрофотометра NanoDrop 2000 («Thermo Fisher Scientific», США). Концентрация полученной мРНК составила 286 нг/мкл, А260/А280 =2,0. РНК хранили при - 80°С.
На втором этапе осуществляли получение линии мышей, нокаутных по grin3а, следующими стадиями:
1. Оплодотворенные яйцеклетки для микроинъекций мышей получали методом индукции суперовуляции. Для этого половозрелым самкам мышей линии F1(CBA×C57BL/6) (животные были получены из биоресурсной коллекции ИФАВ РАН (FFSN-2021-0005)) вводили 7 ед. гонадотропина сыворотки жеребых кобыл (ГСЖК) и через 48 час - 7 ед. хорионического гонадотропина человека (ХгЧ), после чего ссаживали с самцами линии C57BL/6 (животные были получены из биоресурсной коллекции ИФАВ РАН (FFSN-2021-0005)). Факт спаривания констатировали на следующее утро по наличию копулятивной пробки. Вымывание яйцеклеток из яичников самок проводили через 11-12 часов после спаривания с самцами. Для вымывания использовали среду ЭКО 1 "Ооклин" («ПанЭко», РФ).
2. Микроинъекции в цитоплазму оплодотворенных яйцеклеток проводили в среде ЭКО 1 "Ооклин" («ПанЭко», РФ). под инвертированным микроскопом Nikon Eclipse Ti2 («Nikon Instruments», Япония) при увеличении в 200-400 крат, используя микроманипуляторы TransferMan 4R («Eppendorf», Германия). Для изготовления игл для микроинъекций использовали пуллер микропипеток Р-97 («Sutter instrument», США), для изготовления удерживающей пипетки использовали пуллер Р-97 («Sutter instrument», США) и микрокузницу MF-900 («Narishige», Япония). После окончания микроинъекций выжившие зиготы переносили в каплю среды ЭКО ПРО «Дробление» («ПанЭко», РФ) под жидким парафиновым маслом ЭКО1 («ПанЭко», РФ) и культивировали в течение 1 часа для выявления жизнеспособных эмбрионов.
3. В качестве самок-реципиентов, которые были использованы в качестве суррогатных матерей, была выбрана аутбредная линия CD1 (животные были получены из биоресурсной коллекции ИФАВ РАН (FFSN-2021-0005). Псевдобеременных самок-рецепиентов для пересадки выживших оплодотворенных яйцеклеток после микроинъекции отбирали по наличию копулятивных пробок после ссаживания с вазэктомированными самцами линии CD1. После спаривания с вазэктомированными самцами у этих самок в период овуляции происходит гормональная перестройка аналогично беременным самкам, поэтому их репродуктивная система готова к принятию и развитию подсаженных оплодотворенных яйцеклеток.
4. Успешно перенесшие микроинъекцию оплодотворенные яйцеклетки подсаживали в яйцевод самки-реципиента спустя 1 час после микроинъекции. Подсаживание яйцеклеток осуществлялось с помощью микрохирургической операции под стереоскопическим микроскопом Nikon SMZ1270 («Nikon», Япония). Псевдобеременных самок-реципиентов в возрасте 2-3 месяца анестезировали смесью золетил/ксилазин, после чего дорсальным доступом доставали яйцевод и с помощью микрокапилляра подсаживали в него оплодотворенные яйцеклетки. Каждой самке подсаживали 5-12 оплодотворенных яйцеклеток. Мышей после операции до выхода из наркоза держали на подогреваемом до 37°С столике.
5. Вазэктомированных самцов получали путем микрохирургической операции, при которой семявыносящий проток каждого семенника разрушали раскаленным пинцетом. Самцов предварительно анестезировали смесью золетил/ксилазин, а после операции до выхода из наркоза держали на подогреваемом до 37°С столике.
6. У рожденных мышей F0 подтверждали наличие делеции, приводящей к сдвигу рамки считывания, с помощью секвенирования ДНК целевого ПЦР-фрагмента гена grin3а. Геномная ДНК была выделена из ткани рожденных детенышей, а затем целевой фрагмент был амплифицирован с помощью метода ПЦР. Подбор праймеров для амплификации и секвенирования проводили с помощью программы PerlPrimer (http://perlprimer.sourceforge.net/).
7. Выведение линейных гетерозиготных мышей из поколения F0 осуществляли путем их скрещивания с мышами линии C57BL/6 (животные были получены из биоресурсной коллекции ИФАВ РАН (FFSN-2021-0005). Данная линия мышей широко используется в биомедицинских исследованиях. Поэтому для создания генетически модифицированной линии мышей в качестве модели болезней человека важно перевести ее на «генетический фон» С57BL/6. Cамки инбредной линии С57BL/6 дают меньше потомства, их зиготы хуже переносят манипуляции in vitro, поэтому для гарантированного получения оплодотворенных клеток использовали самок гибридов первого поколения F1(CBA×C57BL/6) (п.1).
8. Для перевода на «генетический фон» С57BL/6 гетерозиготных мышей (п.7) скрещивали между собой для получения гомозиготных по делеции мышей. Наличие делеции, приводящей к сдвигу рамки считывания гена grin3а, подтверждали с помощью секвенирования ДНК целевого ПЦР-фрагмента гена grin3а (фиг. 2). Далее для освобождения от потенциальных нецелевых мутаций и вывода линии мышей на генетический фон линии C57BL/6 проводили 6 обратных скрещиваний полученных гомозиготных мышей с мышами линии C57BL/6 дикого типа. Каждое обратное скрещивание включало в себя последовательное скрещивание гомозиготных мышей с мышами C57BL/6 дикого типа и последующее скрещивание полученных гетерозигот между собой для получения гомозигот. После каждого из раундов скрещивания проверяли наличие делеции, приводящей к сдвигу рамки считывания гена grin3а, с помощью секвенирования ДНК целевого ПЦР-фрагмента гена grin3а.
Все работы с животными выполнены в соответствии с этическими нормами обращения с животными, принятыми этическим комитетом в области исследований на животных СПбГУ (Заключение No 131-03-5 от 11 октября 2022 г).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="56618-seq
(v2).xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-01-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>IN-56618/RU-NF</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное образовательное учреждение высшего образования
«САНКТ-ПЕТЕРБУРГСКИЙ ГОСУДАРСТВЕННЫЙ УНИВЕРСИТЕТ»
(СПбГУ)</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Educational Institution
of Higher Education "Saint-Petersburg State University"
(SPBU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">ЛИНИЯ ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННЫХ
МЫШЕЙ ДЛЯ МОДЕЛИРОВАНИЯ БОЛЕЗНИ АЛЬЦГЕЙМЕРА, СПОСОБ ЕЕ ПОЛУЧЕНИЯ И ЕЕ
ПРИМЕНЕНИЕ, sgРНК И КОМБИНАЦИЯ crRNA/tracrRNA, ИСПОЛЬЗУЕМЫЕ В
СПОСОБЕ, И НАБОР ПРАЙМЕРОВ</InventionTitle>
<SequenceTotalQuantity>14</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>117</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..117</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccgttttagagctatgctgttttgaaaaaaac
agcatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcttcg</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>99</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..99</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccggttttagagctagaaatagcaagttaaaa
taaggctagtaagttatcaacttgaaaaagtggcaccgagtcggtgcttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>64</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaattaatacgactcactataggtctcttccccaagctcaccggtttt
agagctagaaatagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>80</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaagcaccgactcggtgccactttttcaagttgataacggactagcct
tattttaacttgctatttctagctctaaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>124</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..124</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaattaatacgactcactataggtctcttccccaagctcaccggtttt
agagctagaaatagcaagttaaaataaggctagtaagttatcaacttgaaaaagtggcaccgagtcggtg
ctttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>128</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..128</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttaattatgctgagtgagtgatatccacgcgaccgccatgtatttcc
aaaatctcgatctttatcgttcaattttattccgatcattcaatagttgaactttttcaccgtggctcag
ccacgaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccgttttagagctatgctgttttg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>83</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..83</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaaccattcaaaacagcatagcaagttaaaataaggctagtccgttat
caacttgaaaaagtggcaccgagtcggtgcttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgcctttctcttccccaagctcaccgtggagc</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgcctttctcttccccaagctccgtggagc</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>99</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..99</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccgttttagagctagaaatagcaagttaaaat
aaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgctttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcttccccaagctcaccggttttagagctatgctgttttg</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>70</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..70</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaacagcatagcaagttaaaataaggctagtccgttatcaacttgaaaa
agtggcaccgagtcggtgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ получения генно-модифицированных кроликов с нокаутом гена LEPR с помощью системы CRISPR/Cas9 | 2023 |
|
RU2836438C1 |
| Способ получения генно-модифицированных лабораторных животных с нуль-аллелем гена P2rx3 | 2022 |
|
RU2805173C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Молекула РНК-проводника sgRNA для внесения мутаций в консервативный участок промоторной области гена PPD-D1 мягкой пшеницы с применением системы редактирования генома CRISPR/Cas9 | 2024 |
|
RU2822358C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2834229C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817384C2 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833963C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817374C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817383C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2800828C1 |

Изобретение относится к области биотехнологии, в частности к линии генетически модифицированных мышей для моделирования болезни Альцгеймера (БА). Указанная линия характеризуется тем, что в нуклеотидной последовательности гена grin3a делетированы нуклеотиды AC в позициях, соответствующих позициям 49844568-49844569 в последовательности хромосомы 4 линии C57BL/6J Mus musculus сборки генома GRCm39, которая содержит указанный ген grin3a. Изобретение также относится к способу получения указанной линии и ее применению в качестве модели болезни Альцгеймера. Настоящее изобретение обеспечивает создание генетически сконструированной мышиной модели БА с точным нокаутом гена grin3а, при котором не затрагивается экспрессия других генов, подходящей для детального изучения патогенеза БА и доклинической оценки потенциальных методов лечения БА, предназначенных для использования у людей. 3 н. и 8 з.п. ф-лы, 2 ил., 1 пр.
1. Линия генетически модифицированных мышей для моделирования болезни Альцгеймера, в которой в нуклеотидной последовательности гена grin3a делетированы нуклеотиды AC в позициях, соответствующих позициям 49844568-49844569 в последовательности хромосомы 4 линии C57BL/6J Mus musculus сборки генома GRCm39, которая содержит указанный ген grin3a.
2. Линия мышей по п. 1, где нуклеотидная последовательность гена grin3a содержит последовательность SEQ ID NO: 11.
3. Способ получения линии мышей по п. 1 или 2 с использованием системы CRISPR/Cas9, включающий введение компонентов указанной системы в оплодотворенную яйцеклетку мыши, причем используют sgРНК (единую направляющую РНК), содержащую последовательность SEQ ID NO: 1, или комбинацию crRNA/tracrRNA (CRISPR-РНК/транс-активирующая crRNA), где crRNA содержит последовательность SEQ ID NO: 1.
4. Способ по п. 3, где используют sgРНК, имеющую последовательность SEQ ID NO: 2, SEQ ID NO: 3 или SEQ ID NO: 12.
5. Способ по п. 3, где используют комбинацию crRNA/tracrRNA, в которой:
(i) crRNA имеет последовательность SEQ ID NO: 8 и tracrRNA имеет последовательность SEQ ID NO: 9; или
(ii) crRNA имеет последовательность SEQ ID NO: 13 и tracrRNA имеет последовательность SEQ ID NO: 14.
6. Способ по любому из пп. 3-5, где sgРНК или комбинацию вводят в сочетании с белком Cas9.
7. Способ по любому из пп. 3-5, где sgРНК или комбинацию вводят в сочетании с мРНК Cas9.
8. Способ по любому из пп. 3-5, где sgРНК или комбинацию вводят в сочетании с белком Cas9 и мРНК Cas9.
9. Способ по любому из пп. 3-5, где компоненты системы CRISPR/Cas9 вводят в виде плазмиды.
10. Способ по любому из пп. 3-9, где Cas9 представляет собой SpCas9 (Cas9 Streptococcus pyogenes).
11. Применение линии мышей по п. 1 или 2 в качестве модели болезни Альцгеймера.
| YOKOYAMA M et al., Mouse Models of Alzheimer’s Disease, Front | |||
| Mol | |||
| Neurosci., 2022, vol | |||
| Прибор для нагревания перетягиваемых бандажей подвижного состава | 1917 |
|
SU15A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| DE PLANO LM et al., Applications of CRISPR-Cas9 in Alzheimer's Disease and Related Disorders, International journal of molecular sciences, 2022, 23(15), 8714 | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| QING-QING XU et | |||
Авторы
Даты
2025-02-25—Публикация
2024-03-01—Подача