Область техники настоящего изобретения
Изобретение относится к областям молекулярной биологии, геномного редактирования и трансгенеза и представляет собой линию генно-модифицированных кроликов с нокаутом гена LEPR, кодирующего рецептор лептина, а также способ получения кроликов с помощью CRISPR/Cas9-опосредованного геномного редактирования, включающий получение гидовой РНК специфичной для гена LEPR кролика, микроинъекции в зиготы смеси гидовой РНК и мРНК Cas9, последующее культивирование эмбрионов in vitro до стадии развития пригодной для трансплантации, хирургическую трансплантацию эмбрионов в яйцеводы самки-реципиента и затем выявление внесенной в геном кролика генетической модификации.
Область техники
Нокаут гена - метод направленной инактивации гена в организме. Как правило, под нокаутом гена подразумевается инактивация белок-кодирующего гена, приводящая либо к полному отсутствию функционального белка, либо к продукции укороченного нефункционального белка. Нокаутные животные являются незаменимым инструментом для выявления функций генов в норме и патологии, моделирования заболеваний человека [Capecchi M.R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 2005 Jun; 6 (6): 507-12. doi:10.1038/nrg1619]. Тем не менее, нокаут гена у животного не всегда точно может выявить функцию гена у человека, а многие социально значимые заболевания человека полигенны и мультифакториальны, поэтому нокаут одного гена у животного не может привести к развитию фенотипа, моделирующего заболевание человека.
Технология получения животных с нокаутом гена была разработана на мышах и основывается на двух важнейших открытиях: выявлении того, что в клетках млекопитающих возможна гомологичная рекомбинация между геномной ДНК и экзогенным генетическим материалом [Smithies O., Gregg R.G., Boggs S.S., Koralewski M.A., Kucherlapati R.S. Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination. Nature. 1985 Sep 19-25; 317 (6034): 230-4. doi: 10.1038/317230a0], а также на получении и характеризации линий эмбриональных стволовых клеток мыши (ЭСК) [Thomas K.R., Capecchi M.R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 1987 Nov 6;51 (3): 503-12. doi:10.1016/0092-8674(87)90646-5].
Основное отличие технологии нокаута гена от методов трансгенеза, которые были разработаны примерно в тоже время в начале 80-х годов [Gordon J.W., Ruddle F.H. (1981) Integration and stable germline transmission of genes injected into mouse pronuclei. Science. 214:1244-1246] заключается в том, что при нокауте за счет гомологичной рекомбинации происходит направленная встройка генетической конструкции в целевой локус, что и приводит к конститутивной инактивации гена-мишени, тогда как при трансгенезе генетическая конструкция встраивается в геном случайным образом.
В окончательном варианте метод получения мышей с нокаутом гена был сформирован к 1988 году и включал конструирование плазмидного вектора, в котором селективный ген (как правило, ген неомицинфосфотрансферазы, дающей устойчивость к антибиотику G418) фланкирован плечам гомологии протяженность около 2 тыс. п.о., которые необходимы для гомологичной рекомбинации в локусе инактивируемого гена. Кроме того, в конструкцию включали ген тимидинкиназы вируса простого герпеса для негативной селекции. Далее полученный вектор электропорировали в ЭСК мыши и отбирали клоны с корректной рекомбинацией, после чего клетки клона имплантировали в бластоцисты мышей и получали генно-модифицированных мозаичных животных [Mansour S.L., Thomas K.R., Capecchi M.R. Disruption of the proto-oncogene int-2 in mouse embryo-derived stem cells: a general strategy for targeting mutations to non-selectable genes. Nature. 1988 Nov 24; 336 (6197): 348-52. doi:10.1038/336348a0]. Этот метод на несколько десятилетий вплоть до начала применения технологий геномного редактирования в клетках млекопитающих стал основным способом получения нокаутных животных, а авторы этого метода Smithies, Capecchi и Evans получили Нобелевскую премию в 2007 году. Однако классический подход к получению животных с нокаутом трудоемкий и занимает длительное время. В частности, необходимо конструирование достаточно сложного плазмидного вектора с плечами гомологии, для отбора клонов с корректной встройкой в силу низкой эффективности гомологичной рекомбинации необходим анализ сотен клонов ЭСК, кроме того, необходим эффективный перенос ЭСК в бластоцисты мыши, а животные первого поколения получаются мозаичными.
Появление технологий геномного редактирования, и прежде, всего CRISPR/Cas9, произвело революцию в методах получения нокаутных животных, открыв беспрецедентные возможности для модификации геномов модельных организмов и существенно упростив, и ускорив процесс получения животных с нокаутом гена [Singh P, Schimenti JC, Bolcun-Filas E. A mouse geneticist's practical guide to CRISPR applications. Genetics. 2015 Jan;199(1):1-15. doi:10.1534/genetics.114.169771].
Наиболее значимой оказалась адаптация системы CRISPR/Cas system (Clustered Regularly Interspaced Short Palindromic Repeats/ CRISPR-associated proteins) для редактирования генома в клетках млекопитающих [Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W., Marraffini L.A., Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013 Feb 15; 339 (6121): 819-23. doi: 10.1126/science.1231143], [Mali P., Yang L., Esvelt K.M., Aach J., Guell M., DiCarlo J.E., Norville J.E., Church G.M. RNA-guided human genome engineering via Cas9. Science. 2013 Feb 15; 339 (6121): 823-6. doi: 10.1126/science.1232033]. Система CRISPR/Cas представляет собой механизм приобретенного иммунитета у бактерий и архей, который используется для уничтожения чужеродного генетического материала [Horvath P., Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 2010 Jan 8; 327 (5962): 167-70. doi: 10.1126/science.1179555]. Системы CRISPR/Cas включают множество белковых и генетических компонентов и разделяются на два класса, включающие шесть типов (I - VI) и, как минимум, 29 подтипов [Makarova K.S., Wolf Y.I., Iranzo J., Shmakov S.A., Alkhnbashi O.S., Brouns S.J.J., Charpentier E., Cheng D., Haft D.H., Horvath P., Moineau S., Mojica F.J.M., Scott D., Shah S.A., Siksnys V., Terns M.P., Venclovas Č., White M.F., Yakunin A.F., Yan W., Zhang F., Garrett R.A., Backofen R., van der Oost J., Barrangou R., Koonin E.V. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol. 2020 Feb; 18 (2): 67-83. doi: 10.1038/s41579-019-0299-x]. В настоящий момент наиболее часто для геномного редактирования используют нуклеазы SpCas9 из Streptococcus pyogenes (класс II тип II) и AsCas12 из Acidaminococcus sp. (класс II тип V).
У бактерий Cas9 формирует рибонуклеопротеиновый комплекс (РНП) с двумя короткими РНК: tracrRNA и crisprRNA (crRNA), где tracrRNA выступает как скаффолд, связывающийся с Cas9, а crRNA взаимодействует с tracrRNA, и кроме того, связывается с ДНК-мишенью. Было показано, что tracrRNA и crRNA можно искусственно объединить в одну молекулу РНК, называемую single guide RNA (sgRNA), что существенно упростило использование системы CRISPR/Cas9 в клетках млекопитающих [ Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J.A., Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012 Aug 17; 337 (6096): 816-21. doi: 10.1126/science.1225829]. В отличие от SpCas9, AsCas12 и другие нуклеазы семейства Cas12 не требуют tracrRNA для процессинга CRISPR повторов, поэтому в клетках млекопитающих нуклеазе Cas12 необходимо только crRNA для эффективного редактирования генома [Zetsche B., Gootenberg J.S., Abudayyeh O.O., Slaymaker I.M., Makarova K.S., Essletzbichler P., Volz S.E., Joung J., van der Oost J., Regev A., Koonin E.V., Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 2015 Oct 22; 163 (3): 759-71. doi: 10.1016/j.cell.2015.09.038].
SpCas9 представляет собой РНК-направляемую эндонуклеазу, которая распознает специфический геномный локус за счет комплементарного взаимодействия между связанной с Cas9 гидовой РНК и нуклеотидной последовательностью в геноме длиной 20 п.о., называемой протоспейсером, за которым следуют 3 п.о. NGG, называемых protospacer adjacent motif (PAM). Для осуществления геномного редактирования sgRNA за счет комплементарности связывается с последовательностью-мишенью в геноме, направляя туда нуклеазу Cas9, которая вносит двуцепочечный разрыв в ДНК в 3 парах оснований в 5'-области от сайта PAM. Далее двуцепочечный разрыв репарируется клеточными механизмами репарации ДНК либо по механизму негомологичного соединения концов (NHEJ) либо по механизму гомологичной рекомбинации [Yamamoto Y., Gerbi S.A. Making ends meet: targeted integration of DNA fragments by genome editing. Chromosoma. 2018 Dec; 127 (4): 405-420. doi:10.1007/s00412-018-0677-6]. Репарация по механизму NHEJ приводит к формированию как правило небольших делеций или инсерций (инделов) в месте внесения разрыва, что с высокой вероятностью приводит к сдвигу рамки считывания в кодирующей последовательности гена и возникновение стоп-кодона, тем самым приводя к нокауту гена. При внесении экзогенной ДНК-матрицы для репарации одновременно с компонентами системы CRISPR/Cas9, такая матрица может быть использована клеточными механизмами репарации ДНК в качестве инструкции для внесения кодируемой матрицей нуклеотидной последовательности в геном, что позволяет осуществлять направленную модификацию геномов [Nambiar T.S., Baudrier L., Billon P., Ciccia A. CRISPR-based genome editing through the lens of DNA repair. Mol Cell. 2022 Jan 20; 82 (2): 348-388. doi:10.1016/j.molcel.2021.12.026].
Появление подобной технологии позволило получить генетически модифицированных мышей с помощью микроинъекций компонентов системы CRISRP/Cas9 непосредственно в зиготы [Wang H., Yang H., Shivalila C.S., Dawlaty M.M., Cheng A.W., Zhang F., Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 2013 May 9; 153 (4): 910-8. doi:10.1016/j.cell.2013.04.025], [Yang H., Wang H., Shivalila C.S., Cheng A.W., Shi L., Jaenisch R. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 2013 Sep 12; 154 (6): 1370-9. doi:10.1016/j.cell.2013.08.022]. Вскоре технология была перенесена на кроликов, и впервые с помощью системы CRISPR/Cas9 были получены генетически модифицированные кролики с нокаутом гена и с таргетной встройкой генетических конструкций [Yang D., Xu J., Zhu T., Fan J., Lai L., Zhang J., Chen Y.E. Effective gene targeting in rabbits using RNA-guided Cas9 nucleases. J Mol Cell Biol. 2014Feb; 6 (1): 97-9. doi: 10.1093/jmcb/mjt047], [Song J., Yang D., Xu J., Zhu T., Chen Y.E., Zhang J. RS-1 enhances CRISPR/Cas9- and TALEN-mediated knock-in efficiency. Nat Commun. 2016 Jan 28; 7: 10548. doi:10.1038/ncomms10548].
Геномное редактирование с помощью других программируемых нуклеаз, таких как TALEN (Transcription Activator-Like Effector Nuclease), также применялось для получения генетически модифицированных кроликов [Song J., Yang D., Xu J., Zhu T., Chen Y.E., Zhang J. RS-1 enhances CRISPR/Cas9- and TALEN-mediated knock-in efficiency. Nat Commun. 2016 Jan 28; 7: 10548. doi: 10.1038/ncomms10548], но в силу сложности дизайна данных нуклеаз в настоящее время они практически полностью вытеснены системой CRISPR/Cas.
Домашний кролик, начиная со времен Луи Пастера, использовавшего кроликов для разработки вакцины против бешенства [DA Mota Gomes M. Louis Pasteur and Dom Pedro II engaged in rabies vaccine development. J Prev Med Hyg. 2021 Apr 29; 62 (1): E231-E236. doi: 10.15167/2421-4248/jpmh2021.62.1.1631], и по настоящее время, остается ценной моделью в различных областях биомедицинских исследований из-за его среднего размера, высокой плодовитости, и относительно большей близости к приматам по сравнению с другими модельными объектами, такими как мыши или крысы. Технологии геномного редактирования (ZNF, TALEN и CRISPR/Cas9) нашли широкое применение для получения генно-модифицированных кроликов, прежде всего, для создания моделей для биомедицинских исследований, и в существенно меньшей степени - для изменений сельскохозяйственных характеристик [Колоскова Е.М., Каркищенко В.Н., Езерский В.А., Петрова Н.В., Максименко С.В., Матвеенко Е.Л. Трансгенные и нокаутные кролики в биомедицине и генотерапии. CRISPR/Cas9-технологии (обзор). Биомедицина. 2019; 15 (4): 12-33. https://doi.org/10.33647/2074-5982-15-4-12-33; Song J., Zhang J., Xu J., Garcia-Barrio M., Chen Y.E., Yang D. Genome engineering technologies in rabbits. J Biomed Res. 2020 Jun 12; 35 (2): 135-147. doi: 10.7555/JBR.34.20190133; Fan J., Wang Y., Chen Y.E. Genetically Modified Rabbits for Cardiovascular Research. Front Genet. 2021 Feb 2; 12:614379. doi: 10.3389/fgene.2021.614379].
Наибольшую ценность представляют кроличьи модели для исследования метаболизма липидов, атеросклероза и сердечно-сосудистых заболеваний, так как система метаболизма липидов у мышей принципиально отлична от таковой у кроликов и человека. Помимо ряда других особенностей, в отличие от человека и кроликов, у мышей в плазме холестерин преимущественно связан с липопротеинами высокой плотности, и мыши резистентны к развитию атеросклероза. Таким образом, моделирование нарушений липидного обмена, наиболее актуально именно на моделях генно-модифицированных кроликов, а не мышей [Matsuhisa F., Kitajima S., Nishijima K., Akiyoshi T., Morimoto M., Fan J. Transgenic Rabbit Models: Now and the Future. Appl. Sci. 2020, 10 (21), 7416; https://doi.org/10.3390/app10217416].
После создания в 2014 году нокаутного кролика с помощью системы CRISPR/Cas9 [Yan Q., Zhang Q., Yang H., Zou Q., Tang C., Fan N., Lai L. Generation of multi-gene knockout rabbits using the Cas9/gRNA system. Cell Regen. 2014 Sep 27; 3 (1): 12. doi: 10.1186/2045-9769-3-12] количество сообщений о нокаутных кроликах для моделей заболеваний человека резко возросло. В том числе были получены модели синдрома преждевременного старения с мышечной дистрофией и дилатационной кардиомиопатией [Sui, T.; Liu, D.; Liu, T.; Deng, J.; Chen, M.; Xu, Y.; Song, Y.; Ouyang, H.; Lai, L.; Li, Z. LMNA-mutated Rabbits: A Model of Premature Aging Syndrome with Muscular Dystrophy and Dilated Cardiomyopathy. Aging Dis. 2019, 10, 102-115], модель гиперлипидемии за счет нокаута гена APOE [Niimi, M.; Yang, D.; Kitajima, S.; Ning, B.; Wang, C.; Li, S.; Liu, E.; Zhang, J.; Eugene Chen, Y.; Fan, J. ApoE knockout rabbits: A novel model for the study of human hyperlipidemia. Atherosclerosis 2016, 245, 187-193] или нокаута аполипопротеина С-III [Yang, D.; Zhang, J.; Xu, J.; Zhu, T.; Fan, Y.; Fan, J.; Chen, Y.E. Production of apolipoprotein C-III knockout rabbits using zinc finger nucleases. J. Vis. Exp. 2013, 81, e50957]. В общей сложности было получено несколько десятков кроликов с нокаутом генов липидного обмена включая такие гены, как APOBEC1, APOB mRNA editing protein; CETP, cholesteryl ester transfer protein; CRP, C-reactive protein; EL, endothelial Lipase; HL, hepatic lipase; LCAT, lecithin: cholesterol acyltransferase; LPL, LPL, lipoprotein lipase; MMP, matrix metalloproteinase; PLTP, plasma phospholipid transfer protein и ряд других см. обзор [Fan J, Wang Y, Chen YE. Genetically Modified Rabbits for Cardiovascular Research. Front Genet. 2021 Feb 2;12:614379. doi: 10.3389/fgene.2021.614379].
Кроме того, трансгенные кролики также использовались в качестве моделей сердечной недостаточности (гипертрофическая кардиомиопатия и проаритмия), иммунных заболеваний (иммунитет к вирусной инфекции, гуморальный иммунный ответ и трансплантация) и онкогенеза из-за физиологических особенностей лабораторных кроликов.
Помимо использования в качестве моделей для биомедицинских исследований кролики представляют собой ценный сельскохозяйственный вид. Технологии геномного редактирования находят широкое применение для манипуляций с геномами животных сельскохозяйственных видов для решения классических проблем животноводства - повышение адаптивного и продуктивного потенциалов, воспроизводства животных. Редактирование генома в настоящее время используется для повышения продуктивности, разработок трансгенных пищевых продуктов, биомедицинских исследований модельных организмов и получения животных, устойчивых к болезням. К настоящему времени выполнено большое количество работ по геномному редактированию на свиньях, овцах, козах и крупном рогатом скоте, как правило, они связаны с повышением устойчивости животных к вирусным и бактериальным патогенам, и в меньшей степени - с изменениями характеристик продуктивности в желательном направлении [Jabbar A., Zulfiqar F., Mahnoor M., Mushtaq N., Zaman M.H., Din A.S.U., Khan M.A., Ahmad H.I. Advances and Perspectives in the Application of CRISPR-Cas9 in Livestock. Mol Biotechnol. 2021 Sep; 63 (9): 757-767. doi: 10.1007/s12033-021-00347-2]. Кроме того, редактирование генома в животноводстве может улучшить благополучие животных за счет уменьшения воздействия животноводства на окружающую среду [Jabbar A., Zulfiqar F., Mahnoor M., Mushtaq N., Zaman M.H., Din A.S.U, Khan M.A., Ahmad H.I. Advances and Perspectives in the Application of CRISPR-Cas9 in Livestock. Mol Biotechnol. 2021 Sep; 63 (9): 757-767. doi: 10.1007/s12033-021-00347-2; Lee K, Uh K, Farrell K. Current progress of genome editing in livestock. Theriogenology. 2020 Jul 1; 150:229-235. doi: 10.1016/j.theriogenology.2020.01.036.; Savvulidi F., Ptacek M., Savvulidi Vargova K., Stadnik L. Manipulation of spermatogonial stem cells in livestock species. J Anim Sci Biotechnol. 2019 Jun 12;10:46. doi: 10.1186/s40104-019-0355-4].
Примерами направленного изменения сельскохозяйственных признаков у кроликов могут служить изменение окраса шерсти [Xiao N., Li H., Shafique L., Zhao S., Su X., Zhang Y., Cui K., Liu Q., Shi D. A Novel Pale-Yellow Coat Color of Rabbits Generated viaMC1R Mutation With CRISPR/Cas9 System. Front Genet. 2019 Sep 18 ;10: 875. doi: 10.3389/fgene.2019.00875] или мясной продуктивности путем повреждения гена миостатина - ингибитора увеличения диаметра мышечного волокна [Zheng Y., Zhang Y., Wu L., Riaz H, Li Z., Shi D., Rehman S.U., Liu Q., Cui K. Generation of Heritable Prominent Double Muscle Buttock Rabbits via Novel Site Editing of Myostatin Gene Using CRISPR/Cas9 System. Front Vet Sci. 2022 May 20; 9: 842074. doi: 10.3389/fvets.2022.842074]. Кроме того, одна из проблем изменения характеристик продуктивности у животных сельскохозяйственных пород с помощью генетических модификаций заключается в полигенности контроля таких признаков и крайней ограниченностью числа генов, являющихся ключевыми для хозяйственно-ценных признаков. Наглядным примером может служить исследование у домашнего кролика геномных районов, контролирующих ряд хозяйственно ценных признаков [Bovo S., Schiavo G., Utzeri V.J., Ribani A., Schiavitto M., Buttazzoni L., Negrini R., Fontanesi L. A genome-wide association study for the number of teats in European rabbits (Oryctolagus cuniculus) identifies several candidate genes affecting this trait. Anim Genet. 2021 Apr; 52 (2): 237-243. doi: 10.1111/age.13036]. Оказалось, что такие характеристики, как окрас шерсти, структура шерсти и размер тела ассоциированы с генами, локализованными в 309 геномных районах, и что генетическая архитектоника этих признаков очень сложна [Ballan M., Bovo S., Schiavo G., Schiavitto M., Negrini R, Fontanesi L. Genomic diversity and signatures of selection in meat and fancy rabbit breeds based on high-density marker data. Genet Sel Evol. 2022 Jan 21; 54 (1): 3. doi: 10.1186/s12711-022-00696-9]. Это соответствует известным данным об аддитивном вкладе большинства генов, ассоциированных с проявлением хозяйственно ценных признаков [Aliloo H., Pryce J.E., González-Recio O., Cocks B.G., Hayes B.J. Validation of markers with non-additive effects on milk yield and fertility in Holstein and Jersey cows. BMC Genet. 2015 Jul 22;16:89. doi: 10.1186/s12863-015-0241-9], а также о зависимости их проявления от стадии развития, специфики кормления, влияния факторов окружающей среды [Strucken E.M., Laurenson Y.C., Brockmann G.A. Go with the flow-biology and genetics of the lactation cycle. Front Genet. 2015 Mar 26;6:118. doi: 10.3389/fgene.2015.00118].
Лептин - пептидный гормон (16 кДа), продуцируемый белой жировой тканью (относится к адипокинам) и участвующий в регуляции пищевого поведения и энергообмена, а также является плейотропным медиатором в широком диапазоне нейроэндокринных функций [Алипкина С.И., Налобин Д.С., Галиакберова А.А., Трошев Д.В., Краснов М.С., Богуславский Д.В., Глазко Т.Т., Глазко В.И., Косовский Г.Ю. Лептин и его рецептор в норме и при патологии. Успехи современной биологии. 2019. Т. 139. № 4. С. 352-364. DOI: 10.1134/S0042132419040033].
Эффекты лептина реализуются за счет его связывания с рецептором лептина, кодируемого геном LEPR. Ген LEPR кролика локализован на 13 хромосоме содержит 28 экзонов, включая альтернативные. В базе Genbank указано 14 изоформ транскрипта гена LEPR кролика, которые кодируют 5 изоформ белка. Lepr представляет собой трансмембранный рецептор.
Длинная форма рецептора состоит из трех доменов - внеклеточного, трансмембранного и внутриклеточного. Рецептор лептина относят к семейству JAK-STAT и основные эффекты лептина опосредованы активаций JAK2 протеинкиназы и транскрипционного фактора STAT3. Помимо этого лептин активирует такие сигнальные белки как SOCS3 и PTP1b, MAP-киназу, IRS (субстрат инсулинового рецептора) [Friedman J.M. Leptin and the endocrine control of energy balance. Nat Metab. 2019 Aug; 1(8): 754-764. doi: 10.1038/s42255-019-0095-y]. Также описана растворимая изоформа рецептора лептина, представляющая собой отщепленную внеклеточную часть рецептора, которая циркулирует в крови и играет роль лептин-связывающего белка. [Schaab M, Kratzsch J. The soluble leptin receptor. Best Pract Res Clin Endocrinol Metab. 2015 Oct;29(5):661-70. doi: 10.1016/j.beem.2015.08.002].
Показано, что взаимодействие лептина с его рецептором необходимо для регуляции аппетита, расхода энергии, поддержания гомеостаза глюкозы и фертильности. Для человека описано 52 клинических случая ожирения, обусловленного мутациями в гене лептинового рецептора [Berger C., Klöting N. Leptin Receptor Compound Heterozygosity in Humans and Animal Models. Int J Mol Sci. 2021 Apr 25; 22 (9): 4475. doi: 10.3390/ijms22094475. PMID: 33922961; PMCID: PMC8123313]. Созданы генетически модифицированные грызуны, нокаутные по гену лептинового рецептора, однако их фенотип не вполне совпадает с симптомами, наблюдаемыми у людей [Israel D, Chua S Jr. Leptin receptor modulation of adiposity and fertility. Trends Endocrinol Metab. 2010 Jan; 21 (1): 10-6. doi: 10.1016/j.tem.2009.07.004. Epub 2009 Oct 23. PMID: 19854659; PMCID: PMC2818174]. Важно отметить, что эффект снижения массы животных опосредуется взаимодействием лептина с лептиновым рецептором в гипоталамусе, где представлена длинная изоформа Lepr.
Были получены экспериментальные данные об участии лептина в индукции и регуляции хронических воспалительных процессов и дегенеративных заболеваний, в том числе, аутоиммунных патологий, диабета 2-го типа и рака [Iikuni N., Lam Q.L., Lu L. et al. Leptin and inflammation// Curr Immunol.Rev. 2008. - Vol. 4, N 2. - P. 70-79]. Кроме того, активно проводятся исследования направленные на изучения роли лептина не только в развитии ожирения и поддержании энергетического гомеостаза, но и в регуляции репродуктивных функций [Brewer C.J., Balen A.H. The adverse effects of obesity on conception and implantation // Reproduction. -2010. - Vol. 140, N 3. - P. 347-364]. В тоже время весь спектр влияний, связанных с активацией/ингибированием лептиновых рецепторов, изучен пока недостаточно.
В данном изобретении для создания модели морбидного ожирения и нарушений липидного и углеводного обмена у кроликов было предложено получить кроликов с нокаутом гена рецептора лептина LEPR с помощью системы CRISPR/Cas9.
Подобные работы, в том числе с применение системы CRISPR/Cas9 проводились ранее на модели грызунов (мыши и крысы). Структура гена Lepr мыши и крыс сходна с таковой у кроликов. В частности, ген Lepr мыши содержит 21 экзон, а наиболее длинный транскрипт (NM_146146) включает 19 экзонов. Мыши с генетическими нарушениями (diabetes/db) и (obese/ob), приводящими к метаболическим нарушениям, гипергликемии, ожирению и симптомам диабета, были описаны в 60-70 е годы XX века [Hummel K.P., Dickie M.M., Coleman D.L. Diabetes, a new mutation in the mouse. Science. 1966 Sep 2; 153 (3740):1127-8. doi: 10.1126/science.153.3740.1127], [Coleman D.L. Obese and diabetes: two mutant genes causing diabetes-obesity syndromes in mice. Diabetologia. 1978 Mar; 14 (3): 141-8. doi: 10.1007/BF00429772]. Однако только спустя 30 лет было установлено, что фенотип ob/ob обусловлен мутацией в гене лептина [Zhang Y., Proenca R., Maffei M., Barone M., Leopold L., Friedman J.M. Positional cloning of the mouse obese gene and its human homologue. Nature. 1994 Dec 1; 372 (6505): 425-32. doi: 10.1038/372425a0], а фенотип db/db - мутацией в гене Lepr [Chen H., Charlat O., Tartaglia L.A., Woolf E.A., Weng X., Ellis S.J., Lakey N.D., Culpepper J., Moore K.J., Breitbart R.E., Duyk G.M., Tepper R.I., Morgenstern J.P. Evidence that the diabetes gene encodes the leptin receptor: identification of a mutation in the leptin receptor gene in db/db mice. Cell. 1996 Feb9; 84(3): 491-5. doi: 10.1016/s0092-8674(00)81294-5].
Таким образом мыши линии db/db - первая модель генетических нарушений в гене Lepr, приводящих к ожирению, диабету и нарушению фертильности у животных. Было установлено, что у таких мышей транскрипт Lepr содержит инсерцию длиной 106 п.о. между 18 и 19 экзонами транскрипта, в результате чего формируется укороченный белок, лишенный почти всего внутриклеточного домена, необходимого для передачи сигнала по JAK-STAT3-сигнальному пути. Было установлено, что в геноме мышей линии db/db присутствует точечная замена G->T в 3’НТО транскрипта Lepr, которая приводит к появлению донорного сайта сплайсинга в 3’-области относительно стоп-кодона, что приводит ко включению в транскрипт 106 п.о. в виде аберрантного экзона [Chen H., Charlat O., Tartaglia L.A., Woolf E.A., Weng X., Ellis S.J., Lakey N.D., Culpepper J., Moore K.J., Breitbart R.E., Duyk G.M., Tepper R.I., Morgenstern J.P. Evidence that the diabetes gene encodes the leptin receptor: identification of a mutation in the leptin receptor gene in db/db mice. Cell. 1996 Feb9; 84 (3): 491-5. doi: 10.1016/s0092-8674(00)81294-5].
Недавно была выявлена еще одна спонтанная мутация в гене Lepr у мышей, приводящая к развитию симптомов, характерных для линии db/db. Делеция 1 п.о. в 11 экзоне привела к появлению преждевременного стоп-кодона и формированию укороченного белка Lepr длиной 530 аминокислот (вместо 1162 у дикого типа) [Berger C., Heyne H.O., Heiland T., Dommel S., Höfling C., Guiu-Jurado E., Lorenz J., Roßner S., Dannemann M., Kelso J., Kovacs P., Blüher M., Klöting N. A novel compound heterozygous leptin receptor mutation causes more severe obesity than in Lepr db/db mice. J Lipid Res. 2021; 62: 100105. doi:10.1016/j.jlr.2021.100105].
Развитие технологий получения нокаутных животных c помощью гомологичной рекомбинации в эмбриональных стволовых клетках (ЭСК) и применение рекомбиназ Cre и Flp для геномной инженерии в мышах позволило создать модель индуцибельного нокаута гена Lepr. Были получены мыши, у которых 17 экзон гена Lepr фланкирован LoxP сайтами. При этом фенотип схожий с таковым у мышей линии db/db (морбидное ожирение, диабет, нарушение фертильности) наблюдался как у мышей, несущих в геноме селективную кассету PGK-neo до ее удаления с помощью рекомбиназы Flp, так и у мышей с делецией 17 экзона за счет рекомбиназы Cre [McMinn J.E., Liu S.M., Dragatsis I., Dietrich P., Ludwig T., Eiden S., Chua S..C Jr. An allelic series for the leptin receptor gene generated by CRE and FLP recombinase. Mamm Genome. 2004 Sep;15 (9): 677-85. doi: 10.1007/s00335-004-2340-1]. Очевидно, что встройка кассеты PGK-neo в 16 интрон гена Lepr сама по себе приводит к формированию укороченного рецептора Lepr c нефункциональным внутриклеточным доменом, так как вырезание этой кассеты in vivo с помощью Flp восстанавливает нормальный фенотип мышей [McMinn J.E., Liu S.M., Dragatsis I., Dietrich P., Ludwig T., Eiden S., Chua S.C. Jr. An allelic series for the leptin receptor gene generated by CRE and FLP recombinase. Mamm Genome. 2004 Sep;15 (9): 677-85. doi: 10.1007/s00335-004-2340-1]. Наблюдаемый фенотип у мышей с гомозиготной инактивацией Lepr включал морбидное ожирение, гиперфагию, резистентность к инсулину и дефект фертильности.
Используя линию мышей с флакнированным LoxP сайтами экзоном 17 гена Lepr, в ряде работ были получены животные с тканеспецифичным нокаутом гена. Например, были получены мыши с нейрон-специфическим нокаутом Lepr за счет скрещивания с линий мышей с нейрон-специфической экспрессией Cre (CaMKIIα-Cre) [McMinn J.E., Liu S.M., Liu H., Dragatsis I., Dietrich P., Ludwig T., Boozer C.N., Chua SC Jr. Neuronal deletion of Lepr elicits diabesity in mice without affecting cold tolerance or fertility. Am J Physiol Endocrinol Metab. 2005 Sep;289(3):E403-11. doi: 10.1152/ajpendo.00535.2004].
В другой работе были описан мыши с нейрон-специфическим и с печень-специфическим нокаутом Lepr, [Cohen P., Zhao C., Cai X., Montez J.M., Rohani S.C., Feinstein P., Mombaerts P., Friedman J.M. Selective deletion of leptin receptor in neurons leads to obesity. J Clin Invest. 2001 Oct; 108 (8): 1113-21. doi: 10.1172/JCI13914]. В отличие от работы McMinn JE et al 2004, в данном случае LoxP сайты фланкировали первый экзон гена Lepr. Тканеспецифический нокаут достигался за счет скрещивания с мышами Synapsin-Cre (мозг) и альбумин-Cre (печень). В этой работе было установлено, что именно действие лептина на рецептор Lepr в гипоталамусе играет существенную роль в регуляции аппетита, так как только в случае нокаута Lepr в мозге, но не в печени, развивалось ожирение у экспериментальных животных.
Используя мышей с первым экзоном гена Lepr, фланкированным LoxP сайтами, были получены животные с нокаутом Lepr в ткани поджелудочной железы путем скрещивания с мышами, экспрессирующим Cre под контролем промотора Pdx1 [Morioka T., Asilmaz E., Hu J., Dishinger J.F., Kurpad A.J., Elias C.F., Li H., Elmquist J.K., Kennedy R.T., Kulkarni R.N. Disruption of leptin receptor expression in the pancreas directly affects beta cell growth and function in mice. J Clin Invest. 2007 Oct; 117 (10): 2860-8. doi: 10.1172/JCI30910].
В другой работе были получены мыши с кондиционным нокаутом гена Lepr в периферических тканях (печень, жировая ткань тонкий кишечник), но не в ЦНС. Это достигалось за счет использования тамоксифен-индуцибельной транслокации рекомбиназы Cre-ERT2 в ядро. При интраперитонеальном введении тамоксифена мышам он не проникает в мозг, за счет этого индуцибельный нокаут Lepr достигался лишь в периферических тканях [Guo K., McMinn J.E., Ludwig T., Yu Y.H., Yang G., Chen .L, Loh D., Li C., Chua S. Jr., Zhang Y. Disruption of peripheral leptin signaling in mice results in hyperleptinemia without associated metabolic abnormalities. Endocrinology. 2007 Aug; 148(8): 3987-97. doi: 10.1210/en.2007-0261]. Это приводило к выраженной гиперлептинемии, связанной с нарушением петли обратной связи, ингибирующей продукцию лептина в жировой ткани. Однако нарушение периферического сигналинга через Lepr не вызывало ожирения, не нарушало количество принимаемой пищи, не меняло уровень глюкозы и терморегуляцию.
Еще одна модель индуцибельного нокаута у мыши - инактивация гена Lepr в клетках эндотелия путем скрещивания с мышами со специфической экспрессией тамоксифен-индуцибельной Cre в клетках эндотелия (Tie2-Cre) [Hubert A., Bochenek M.L., Schütz E., Gogiraju R., Münzel T., Schäfer K. Selective Deletion of Leptin Signaling in Endothelial Cells Enhances Neointima Formation and Phenocopies the Vascular Effects of Diet-Induced Obesity in Mice. Arterioscler Thromb Vasc Biol. 2017 Sep; 37 (9): 1683-1697. doi:10.1161/ATVBAHA.117.309798], [Gogiraju R., Witzler C., Shahneh F., Hubert A., Renner L., Bochenek M.L., Zifkos K., Becker C., Thati M., Schäfer K. Deletion of endothelial leptin receptors in mice promotes diet-induced obesity. Sci Rep. 2023 May 22; 13 (1):8276. doi:10.1038/s41598-023-35281-7].
Важно отметить, что тканеспецифический нокаут Lepr в репродуктивной системе самок мышей приводит к нарушению фертильности за счет нарушения родовой активности. Это потенциально может влиять на поддержание и размножение линий животных с конститутивным нокаутом Lepr [Pennington K.A., Oestreich A.K., Cataldo K.H., Fogliatti C.M., Lightner C., Lydon J.P., Schulz L.C. Conditional knockout of leptin receptor in the female reproductive tract reduces fertility due to parturition defects in mice. Biol Reprod. 2022 Aug 9;107 (2): 546-556. doi: 10.1093/biolre/ioac062]. В этой работе также использовали ранее описанную линию мышей Lepr flox/flox [McMinn J.E., Liu S.M., Dragatsis I., Dietrich P., Ludwig T., Eiden S, Chua S.C. Jr. An allelic series for the leptin receptor gene generated by CRE and FLP recombinase. Mamm Genome. 2004 Sep; 1 5(9): 677-85. doi: 10.1007/s00335-004-2340-1], которую скрещивали с мышами с экспрессией Cre под контролем прогестеронового рецептора.
Отличие описанных индуцибельных моделей нокаута гена Lepr от предложенной в данном изобретении заключается в том, что нокаут, во-первых, Cre-индуцибельный, что требует скрещивания с линиями мышей, экспрессирующих Cre. Во-вторых, осуществляется делеция 17 экзона который расположен близко к С-концевой части белка, что приводит к делеции лишь внутриклеточного сигнального домена, кодируемого данным экзоном, но при этом может формироваться укороченный белок Lepr, сохраняющий свой внеклеточный домен, который может функционировать как как растворимый лептин-связывающий белок.
Крысы также представляют собой часто используемую модель морбидного ожирения, вызванного дефицитом гена Lepr. Как и в случае мышиной модели, вначале были выявлена линия крыс со спонтанно возникшей аутосомной мутацией, приводящей к ожирению. Таких крыс назвали Zucker или fatty (fa/fa) [Argilés J.M. The obese Zucker rat: a choice for fat metabolism 1968-1988: twenty years of research on the insights of the Zucker mutation. Prog Lipid Res.1989;28 (1): 53-66. doi: 10.1016/0163-7827(89)90007-6]. Впоследствии было установлено, что мутация произошла в гене Lepr, и она приводит к возникновению преждевременного стоп-кодона. В частности, замена T->A, приводит к появлению стоп-кодона в позиции 763 аминокислотного остатка, и как следствие формирования укороченного белка, лишенного трансмембранного и внутриклеточного доменов [Wu-Peng X.S., Chua S.C. Jr., Okada N., Liu S.M., Nicolson M., Leibel R.L. Phenotype of the obese Koletsky (f) rat due to Tyr763Stop mutation in the extracellular domain of the leptin receptor (Lepr): evidence for deficient plasma-to-CSF transport of leptin in both the Zucker and Koletsky obese rat. Diabetes. 1997Mar; 46 (3): 513-8. doi: 10.2337/diab.46.3.513].
Система CRISPR/Cas9 была использована для получения крыс с нокаутом Lepr. Мишень для гидовой РНК была локализована в 4 экзоне гена, а нокаутных животных получали с помощью микроинъекций смеси гидовой РНК и мРНК Cas9. В результате было получена линия крыс с делецией 298 п.о. приводящей к возникновению преждевременного стоп-кодона [Bao D., Ma Y., Zhang X., Guan F., Chen W., Gao K., Qin C., Zhang L.. Preliminary Characterization of a Leptin Receptor Knockout Rat Created by CRISPR/Cas9 System. Sci Rep. 2015 Nov 5; 5: 15942. doi: 10.1038/srep15942]. В части методов и стратегии получения нокаутных животных эта работа наиболее близка к заявленному изобретению.
Как видно из обзора литературных данных, были получены различные модели как конститутивного, так и индуцибельного тканеспецифического нокаута гена Lepr у мышей и крыс. Однако в литературе нет данных о нокауте гена LEPR у кроликов. С точки зрения локализации мутаций в гене Lepr показано, что индуцибельный нокаут Lepr возможен за счет делеции как первого, так и 17-го экзонов гена у мыши. В части методов и стратегии получения нокаутных животных наиболее близка к заявленному изобретению работа [Bao D., Ma Y., Zhang X., Guan F., Chen W., Gao K., Qin C., Zhang L. Preliminary Characterization of a Leptin Receptor Knockout Rat Created by CRISPR/Cas9 System. Sci Rep. 2015 Nov 5; 5: 15942. doi: 10.1038/srep15942] в которой использовали микроинъекции в зиготы смеси гидовой РНК и мРНК Cas9 для внесения мутации у крыс в области 4-го экзона Lepr.
В предложенном изобретении с помощью системы CRISPR/Cas9 была внесена делеция в гене LEPR кролика, затрагивающая часть 10 экзона, 10 интрон и часть 11 экзона, что отличается от области гена, делетируемой у мышей с индуцибельным нокаутом или крыс, полученных с помощью системы CRISPR/Cas9. В результате делеции в гене LEPR кролика возникает преждевременный стоп-кодон, приводящий к формированию укороченного до 450 а.о. белка. Во-первых, стоит отметить, что наличие стоп-кодона ближе к 5’-области транскрипта с большой вероятностью будет приводить к деградации такого транскрипта по механизму nonsense mediated decay. Во-вторых, даже если такой аберрантный транскрипт будет экспрессироваться на достаточно высоком уровне, то укороченный белок до 450 а.о. будет лишен трансмембранного домена и не будет заякорен в плазматической мембране. Известно, что существует растворимая форма рецептора LEPR длиной 839 а.о. у человека, получаемая за счет действия протеаз на внеклеточную часть рецептора Lepr. Таким образом, укороченный до 450 а.о. белок Lepr кролика по всей видимости не сможет выступать как растворимая форма Lepr, которая является основным белком плазмы крови, связывающим гормон лептин [Schaab M., Kratzsch J. The soluble leptin receptor. Best Pract Res Clin Endocrinol Metab. 2015 Oct;29(5):661-70. doi: 10.1016/j.beem.2015.08.002]. Таким образом, впервые была создана модель нокаута гена LEPR у кроликов, при этом в точки зрения локализации внесенного генетического дефекта данная модель существенно отличается от всех раннее описанных моделей нокаута гена у млекопитающих.
Известны способы получения генетически модифицированных кроликов. В частности, в изобретении CN114958847A (ген RHOB) описан основной метод получения нокаутных кроликов, включающий конструирование вектора для экспрессии гидовой РНК, синтез мРНК Cas9, получение и микроинъекции в оплодотворенные зиготы, культивирование и развитие оплодотворенных зигот, пересадку инъецированных зигот самкам-реципиентам. В ряде других документов также описаны похожие способы создания кроличьих моделей: CN 115125244 A (гены MAPT и PSEN1, кролик проявляет фенотип болезни Альцгеймера), CN 114958911 A (ген EIF4G1, болезнь Паркинсона), CN 116497022 A (мутация p.I151V гена SOD1).
Кроме того, найдены многочисленные патентные документы, в которых описаны кроличьи модели заболеваний человека, например, такие как модель кролика с гипопротеинемией при двойном нокауте гена альбумина (CN 116200389 A), кроличья модель β-талассемии за счет мутации гена HBB2 (WO 2022120786 A1), кроличья модель гиперлипидемии или атеросклероза за счет мутации по гену LDLR (CN 108753837 A), нокаут гена GADD45a с использованием технологии CRISPR/Cas9 (CN 107630043 A). В литературе содержится описание различных трансгенных кроликов и способов их получения, однако не было найдено ни одного похожего технического решения на предложенное изобретение по гену LEPR. Таким образом, анализ научной и патентной литературы показывает, что до настоящего момента не сообщалось о создании кроликов с нокаутом гена LEPR, тем самым подтверждаю новизну и актуальность заявленного изобретения.
Раскрытие изобретения
Задачей заявляемого изобретения является разработка способа получения генетически-модифицированных кроликов с нокаутом гена LEPR с помощью системы CRISPR/Cas9.
Поставленная задача решается подбором гидовых РНК для нуклеазы Cas9 с использованием онлайн ресурса ChopChop (https://chopchop.cbu.uib.no). Выбранные гидовые РНК имеют мишени SEQ ID NO1 и SEQ ID NO2, расположенные в 10 экзоне транскрипта гена LEPR кролика. Для выбора оптимальной гидовой РНК проводят секвенирование фрагмента генома кролика породы советская шиншилла, несущего мишени для выбранных гидовых РНК. На основе результатов секвенирования выбирают sgLepr-2, мишень для которой имеет последовательность SEQ ID NO2 и несет один однонуклеотидный полиморфизм. Далее поставленная задача решается путем создания рекомбинантной плазмиды pX330-sgLepr-2 (SEQ ID NO3), экспрессирующей нуклеазу Cas9 под контролем CMV промотора и гидовую РНК sgLepr-2 под контролем U6 промотора. Рекомбинантная плазмида используется в качестве матрицы в ПЦР для получения ДНК-матрицы для in vitro транскрипции.
Далее, с помощью транскрипции in vitro получают гидовую РНК sgLepr-2 (SEQ ID NO4), оценку ее активности и специфичности проводят с помощью теста на расщепление ДНК-субстрата in vitro с использованием рекомбинантного белка Cas9.
Поставленная задача также решается способом получения и культивирования эмбрионов кролика, включающим осеменение самок, хирургическое изъятие яичников, вымывание из них яйцеклеток и культивирование их в среде DMEM/F12 с добавлением 10%-ной фетальной сыворотки крупного рогатого скота и антибиотиков пенициллина и стрептомицина при +38.5°С, 100% влажности и 7% CO2.
Также поставленная задача решается способом проведения микроинъекций компонентов системы CRISPR/Cas9 (смеси мРНК нуклеазы Cas9 и гидовой РНК, имеющей последовательность SEQ ID NO4) в цитоплазму оплодотворенных яйцеклеток кролика и последующей хирургической трансплантации инъецированных эмбрионов кролика в яйцеводы самок-реципиентов, у которых индуцируют овуляцию за 15-24 часа до пересадки эмбрионов путем инъекции гонадотропин-рилизинг-фактора. Через 28-33 дня после трансплантации эмбрионов самкам-реципиентам происходит либо естественный окрол, либо новорожденных кроликов извлекают с помощью кесарева сечения.
Генотипирование кроликов проводят с помощью ПЦР. Для этого выделяют геномную ДНК из тканей животных дикого типа и предполагаемых генно-модифицированных кроликов. С помощью олигонуклеотидных праймеров SEQ ID NO5 и SEQ ID NO6 в ПЦР амплифицируют участок гена LEPR кролика длиной 642 п.о., несущий мишень для гидовой РНК sgLepr-2 (SEQ ID NO2). Продукты ПЦР разделяют в 8%-ном акриламидом геле и выявляют генно-модифицированного кролика с нокаутом гена LEPR по наличию в геле укороченного фрагмента ДНК, длиной 356 п.о., имеющего нуклеотидную последовательность SEQ ID NO7.
Предложенный авторами способ получения генно-модифицированных кроликов с нокаутом гена LEPR с помощью микроинъекций в зиготы кроликов компонентов системы CRISPR/Cas9 в патентной и научной литературе не описан. Способ основан на внесении с помощью системы CRISPR/Cas9 генетической модификации в области 10 экзона гена LEPR, которая приводит к возникновению преждевременного кодона терминации трансляции в транскрипте гена LEPR, тем самым приводя к формированию укороченного нефункционального рецептора лептина. Предложенным способом авторами решена задача по создания линии кроликов с нокаутом гена LEPR, которая может быть использована в качестве модели нарушений липидного обмена и ожирения, вызванных генетическими нарушениями.
Технический результат заключается во внесении генетической модификации (делеции) в геном кролика в локусе гена LEPR и достигается за счет микроинъекций в цитоплазму оплодотворенной яйцеклетки кролика смеси мРНК нуклеазы Cas9 и гидовой РНК (SEQ ID NO4), которую получают с помощью транскрипции in vitro.
Краткое описание чертежей
Фигура 1. На Фигуре 1 показаны результаты поиска гидовых РНК к гену LEPR с помощью веб-сервера ChopChop. В верхней части рисунка указано положение всех возможных гидовых РНК с NGG PAM сайтом в транскрипте гена LEPR длиной 3630 п.о. В списке ниже схемы показаны гидовые РНК в соответствии с рангом (rank) от наиболее оптимальной (номер 1) к менее оптимальной. Для дальнейшего анализа были выбраны гидовые РНК с рангом 1 и 2 (sgLepr-1 и sgLepr-2), их положение в транскрипте показано стрелками, в списке они выделены рамкой. Результат анализа показывает, что в геноме кролика присутствует одна полностью комплементарная мишень для гидовой РНК (столбец MM0 -1), и отсутствуют мишени с 1, 2 и 3 некомплементарными основаниями (MM1, MM2, MM3 - 0).
Фигура 2. На Фигуре 2 показаны результаты секвенирования геномного локуса гена LEPR кролика породы советская шиншилла. Под хроматограммами секвенирования по Сэнгеру приведена нуклеотидная последовательность мишеней для гидовых РНК. Стрелками показано положение выявленных однонуклеотидных полиморфизмов. Для гидовой РНК sgLepr-1 выявлено два полиморфизма: C/G во второй позиции протоспейсера и G/A в PAM-сайте. Для гидовой РНК sgLepr-2 выявлен полиморфизм A/T в 20-м положении протоспейсера. Для дальнейшей работы выбрана sgLepr-2.
Фигура 3. На Фигуре 3 показана генетическая карта плазмидной конструкции pX330-sgLepr-2, имеющей размер 8487 п.о. Показаны основные элементы плазмиды: “U6 promoter” - промотор U6; “sgLepr-2” - последовательность, соответствующая протоспейсеру для гидовой РНК sgLepr-2 в 10 экзоне гена LEPR кролика; “gRNA scaffold” - каркас гидовой РНК; “CMV enhancer” - немедленный ранний энхансер цитомегаловируса человека (CMV); “chicken beta-actin promoter” - промотор β-актина курицы; “hybrid intron” - гибридный интрон; “Kozak sequence” - последовательность Kozak; “3хFLAG-NLS-Cas9-NLS” - открытая рамка считывания 3хFLAG-NLS-Cas9-NLS; “bGH poly(A) signal” - сигнал полиаденилирования из бычьего гена гормона роста; “AAV2 ITR” - инвертированный обратный повтора вируса AAV2; “AmpR promoter” -промотор гена β-лактамазы; “AmpR” - открытая рамка считывания β-лактамазы; “ColE1 origin” - точка начала репликации ColE1/pMB1/pBR322/pUC; “f1 ori” - точка начала репликации бактериофага f1.
Фигура 4. На Фигуре 4А показан пример анализа в 2%-ном ТАЕ агарозном геле 1 мкл синтезированной in vitro гидовой РНК sgLepr-2 (дорожка «sgLepr-2»). Дорожка «Mw» -маркер молекулярного веса ДНК - O’GeneRuler 50bp DNA Ladder (#SM1133, Thermo Scientific). Размер некоторых полос маркера обозначен слева от рисунка. Из Фигуры 4А видно, что гидовая РНК мигрирует в виде двух полос, одна из которых соответствует по молекулярному весу ожидаемому размеру около 103 п.о, вторая полоса соответствует форме гидовой РНК со вторичными структурами, замедляющими ее подвижность в агарозном геле. Видно, что препарат не содержит примесных полос или признаков деградации РНК в виде шмера.
На Фигуре 4Б показаны результаты теста на расщепление ДНК-субстрата in vitro рибонуклеопротеиновым (РНП) комплексом белка Cas9 и гидовой РНК sgLepr-2. Дорожка «sgScr» - ДНК-субстрат длиной 642 п.о. (продукт амплификации на матрице геномной ДНК кролика породы советская шиншилла с праймерами SEQ ID NO5 и SEQ ID NO6, полученный как описано в Примере 2) инкубировали с РНП, содержащим гидовую РНК, не имеющую мишеней в геноме кролика. Дорожка «sgLepr-2» - ДНК-субстрат инкубировали с РНП, содержащим гидовую РНК sgLepr-2. Дорожка «Mw» - маркер молекулярного веса ДНК - O’GeneRuler 50bp DNA Ladder (#SM1133, Thermo Scientific). Из Фигуры 4Б видно, что sgLepr-2 вызывает расщепление субстрата с образованием фрагментов ожидаемого размера (340 и 302 п.о.), что подтверждает функциональность и специфичность полученной гидовой РНК.
Фигура 5. На Фигуре 5 показана эмбрионы кролика до проведения микроинъекций (А) и через 120 часов после микроинъекций смеси реагентов для геномного редактирования. Получали снимки эмбрионов, находящихся в чашках Петри в культуральной среде.
Фигура 6. На Фигуре 6А показаны результаты анализа продуктов ПЦР с помощью электрофореза в 8%-ном акриламидом геле. Из Фигуры 6А видно, что на матрице геномной ДНК кролика, родившегося после пересадки эмбрионов, инъецированных компонентами системы CRISPR/Cas9 для редактирования гена LEPR (дорожка «KO»), амплифицируются два продукта. Одна полоса, соответствует фрагменту длиной 642 п.о., амплифицируемому на матрице геномной ДНК кролика дикого типа (дорожка «wt»), а вторая полоса имеет размер 356 п.о. и присутствует только в образце «KO». Длины полос маркера молекулярного веса ДНК O’GeneRuler 50 bp DNA Ladder указаны справа. Положение соответствующих продуктов ПЦР указано стрелками. Продукт длиной 356 п.о. имеет нуклеотидную последовательность SEQ ID NO7.
На Фигуре 6Б показана схема геномного локуса LEPR дикого типа (wt) в области 10 и 11 экзонов, имеющих размер 291 и 118 п.о. Положение олигонуклеотидных праймеров (SEQ ID NO5 и SEQ ID NO6) показано в виде стрелок (F и R). На панели «KO» показана схема геномного локуса LEPR генно-модифицированного кролика. Видно, что в результате делеции длиной 295 п.о. утрачивается часть 10 экзона, 10 интрон и часть 11 экзона. Место внесения двуцепочечного разрыва в геноме кролика с помощью нуклеазы Cas9 и гидовой РНК sgLepr-2 показано треугольником.
На Фигуре 6В показан результат секвенирования продукта ПЦР длиной 356 п.о. с нуклеотидной последовательность SEQ ID NO7. Под нуклеотидной последовательностью указана трансляция открытой рамки считывания транскрипта XM_051856399, транскрибирующегося с геномной ДНК с указанной делецией. Трансляция с 1 по 450 аминокислоту соответствует белку Lepr дикого типа, после которых за счет инсерции случайных 9 п.о. TTTAAAGTA транслируется бессмысленный пептид FKVVPCS, после чего следует кодон терминации трансляции TGA (Фигура 6В).
На Фигуре 6Г показано фото генно-модифицированного кролика в возрасте около двух недель.
Осуществление настоящего изобретения
Пример 1. Выбор гидовой РНК для нуклеазы Cas9 для внесения инделов в ген LEPR кролика
Для выбора гидовой РНК использовали он-лайн ресурс ChopChop (https://chopchop.cbu.uib.no/). В качестве запроса вводили нуклеотидную последовательность одного из самых длинных транскриптов гена LEPR (XM_051856399, транскрипт вариант 8, кодирует белок изоформы Х1, и состоит из 21 экзона). Настройки поиска использовали следующие: Target - “Paste”, In - “ Oryctolagus cuniculus”, Using -“ CRISRP/Cas9”, For - “knock-out”, Options - настройка “Intersection”, то есть поиск мишени во всех изоформах транскриптов. На Фигуре 1 показаны результаты поиска гидовых РНК. Сверху приведено расположение всех возможных гидовых РНК с NGG PAM сайтом на транскрипте гена LEPR. Ниже приведен список гидовых РНК в соответствии с рангом (rank) от оптимальной (номер 1) к менее оптимальной. Для дальнейшего анализа были выбраны гидовые РНК с рангом 1 и 2 (выделены рамкой), которые помимо наивысшего ранга имели зеленую цветовую кодировку, указывающую на высокую специфичность. В частности, результат анализа на Фигуре 1 показывает, что в геноме кролика присутствует полностью комплементарная мишень для гидовой РНК (столбец MM0 -1), и отсутствуют мишени с 1, 2 и 3 некопмлементарными основаниями (MM1, MM2, MM3 - 0), что указывает на высокую специфичность выбранных гидовых РНК. Гидовые РНК #1 (SEQ ID NO1 GCTGACACGATCATTCACAACGG; sgLepr-1) и #2 (SEQ ID NO2, ATAATTCCGCATAGCGATGATGG; sgLepr-2) имеют мишени в 10 экзоне гена LEPR, и мишень для данных гидовых РНК присутствуют во всех изоформах транскрипта гена LEPR кролика.
Пример 2. Анализ нуклеотидной последовательности генома кролика в области 10 экзона гена LEPR
Геномы животных и человека содержат однонуклеотидные полиморфизмы (SNP, single nucleotide polymorphism), которые могут варьировать у разных пород сельскохозяйственных животных, и отсутствовать в референсной последовательности генома данного вида в базе данных Genbank. Присутствие SNP в PAM-сайте или затравочной последовательности (seed region) мишени для гидовой РНК может существенно снизить эффективность внесения разрывов в геном нуклеазой Cas9. Поэтому для выявления возможных SNP амплифицировали участок генома кролика, включающий последовательности 10 экзона гена LEPR, в которых находятся мишени для выбранных гидовых РНК. Геномную ДНК из тканей кролика породы советская шиншилла как описано в Примере 7а (см. ниже).
Участок генома кролика длиной 642 п.о. амплифицировали в реакции объёмом 25 мкл включающей:
Реакцию проводили при следующих условиях:
+94°C -3 мин; 37x циклов +94°C -30 с, +62°C -30 с, +72°C -45 с; +72°C -3 мин, +4°С - постоянно.
Далее наличие продукта ПЦР анализировали с помощью электрофореза в 1%-ном агарозном геле, очищали из реакционной смеси с помощью набора реагентов DNA Clean & Concentrator-25 (Zymo Research, D4034) и секвенировали по Сэнгеру, используя олигонуклеотидный праймер SEQ ID NO5.
Примеры хроматограмм секвенирования продукта ПЦР по Сэнгеру приведены на Фигуре 2. Было обнаружено что мишень для гидовой РНК sgLepr-1 в геноме породы кролика советская шиншилла содержит два SNP: C/G во второй позиции протоспейсера и G/A в PAM-сайте. Мишень в геноме для гидовой РНК sgLepr-2 содержит один SNP A/T в 20-м положении протоспейсера. На основании этих результатов для получения кроликов с нокаутом гена LEPR была выбрана гидовая РНК sgLepr-2 так как в геноме присутствует лишь один SNP, и значит, как минимум один аллель будет эффективно разрезаться нуклеазой Cas9.
Пример 3. Конструирование рекомбинантной плазмиды pX330-sgLepr-2
Рекомбинантную плазмиду pX330-sgLepr-2 (SEQ ID NO3), экспрессирующую гидовую РНК sgLepr-2 под контролем промотора U6, и нуклеазу Cas9 из Streptococcus pyogenes с 3xFLAG эпитопом на N-конце, последовательностью NLS вируса SV40 на N-конце и последовательностью NLS из белка нуклеоплазмина на С-конце под контролем CMV промотора, получают на основе плазмиды pX330-U6-Chimeric_BB-CBh-hSpCas9 (Addgene #42230), описанной ранее [Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W.,Marraffini L.A., Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013 Feb 15; 339 (6121): 819-23. doi: 10.1126/science.1231143. PMID: 23287718]. Для этого одноцепочечные синтетические олигонуклеотиды 5- caccgataattccgcatagcgatga-3 (SEQ ID NO8) и 5- aaactcatcgctatgcggaattatc-3 (SEQ ID NO9) гибридизуют для формирования двуцепочечных олигонуклеотидов с липкими концами и клонируют в вектор pX330-U6-Chimeric_BB-CBh-hSpCas9 по сайту рестрикции BbsI.
Полученная рекомбинантная плазмида pX330-sgLepr-2 (Фигура 3) содержит следующие элементы: промотор U6 длиной 241 п.о. в позиции 1-241 п.о.; последовательность, соответствующая протоспейсеру для гидовой РНК sgLepr-2 в 10 экзоне гена LEPR кролика, длиной 20 п.о. в позиции 251-270; каркас гидовой РНК длиной 76 п.о. в позиции 271-346 п.о.; немедленный ранний энхансер цитомегаловируса человека (CMV) длиной 286 п.о. в позиции 443-728 п.о.; промотор β-актина курицы длиной 257 п.о. в позиции 730-986 п.о.; гибридный интрон длиной 228 п.о. в позиции 987-1214 п.о.; последовательность Kozak длиной 10 п.о. в позиции 1226-1235 п.о.; открытая рамка считывания 3хFLAG-NLS-Cas9-NLS длиной 4269 п.о. в позиции 1232-5500 п.о.; сигнал полиаденилирования из бычьего гена гормона роста длиной 208 п.о. в позиции 5534-5741 п.о.; инвертированный обратный повтора вируса AAV2 длиной 141 п.о. в позиции 5750-5890 п.о.; промотор гена β-лактамазы длиной 105 п.о. в позиции 6702-6806 п.о.; открытая рамка считывания β-лактамазы длиной 861 п.о. в позиции 6807-7667 п.о.; точка начала репликации ColE1/pMB1/pBR322/pUC длиной 589 п.о. в позиции 7838-8426 п.о.; точка начала репликации бактериофага f1 в позиции 5965-6420 п.о. длиной 456 п.о.;
Пример 4. Получение гидовой РНК sgLepr-2 с помощью транскрипции in vitro
Гидовую РНК sgLepr-2 синтезировали с помощью транскрипции in vitro (IVT) с использованием набора реагентов HiScribe® T7 High Yield RNA Synthesis Kit (#02040S, New England Biolabs).
Пример 4а. Получение ДНК-матрицы для IVT с помощью ПЦР
Вначале с помощью ПЦР получают ДНК-матрицу для IVT. Для этого на матрице плазмиды pX330-sgLepr-2 амплифицируют фрагмент длиной 122 п.о., включающий последовательность гидовой РНК sgLepr-2 и каркас гидовой РНК (gRNA scaffold) с праймерами 5-tgtaatacgactcactatagggataattccgcatagcgatga-3 (SEQ ID NO10) и 5-aaaagcaccgactcggtgcc-3 (SEQ ID NO11). Прямой праймер содержит последовательность промотора T7-РНК полимеразы, с которого начинается транскрипция in vitro.
Продукт амплифицируют, используя высокоточную ДНК-полимеразу в реакции объемом 50 мкл содержащей:
Реакцию проводят при следующих условиях:
95°C -2 мин;
5х циклов: 95°С - 15 с, 72°С - 20 с;
5х циклов: 95°С - 15 с, 66°С - 10 с, 72°С - 20 с
25х циклов: 95°С - 15 с, 63°С - 10 с, 72°С - 20 с;
72°С - 10 мин, 4°С - постоянно.
Далее, наличие продукта ПЦР проверяют с помощью электрофореза в 2%-ном TAE-агарозном геле, после чего очищают продукт ПЦР используя набор реагентов MinElute® Reaction Cleanup Kit (QIAGEN, #28204). Продукт ПЦР элюируют в объем 30 мкл используя свободную от нуклеаз воду (Ambion, #AM9937). Концентрацию ПЦР-продукта определяют с помощью спектрофотометра Nanophometer ND-120 (Implen), концентрация матрицы для IVT составляет 29 нг/мкл и имеет нуклеотидную последовательность SEQ ID NO12.
Пример 4б. Реакция транскрипции гидовой РНК sgLepr-2 in vitro
Далее проводят реакцию транскрипции in vitro используя набор реагентов HiScribe® T7 High Yield RNA Synthesis Kit (#02040S, New England Biolabs) в соответствии с протоколом для коротких РНК транскриптов длиной менее 300 п.о.
Реакция транскрипции объемом 20 мкл включает:
Реакцию инкубируют при +37°С в течение 5 часов, после чего добавляют в реакцию 1 мкл фермента TURBO DNase, 2 ед./мкл (Ambion, #AM2238) для разрушения ДНК-матрицы и инкубируют при +37С в течение 20 мин.
Пример 4в. Очистка гидовой РНК sgLepr-2
Синтезированную в реакции транскрипции гидовую РНК очищают с помощью набора реагентов miRNeasy Mini Kit (Qiagen, #217004). В частности, 20 мкл реакции транскрипции переносят в микроцентрифужную пробирку объемом 1.7 мл, добавляют 700 мкл реагента QIAzol Lysis Reagent, перемешивают на вортексе и инкубируют при комнатной температуре в течение 5 минут. Затем добавляют 140 мкл хлороформа, перемешивают образец на вортексе 15 секунд и инкубируют при комнатной температуре 2-3 минуты. Центрифугируют образец 15 минут на максимальной скорости при +4°C и переносят водную фазу в чистую микроцентрифужную пробирку. Добавляют к образцу 1.5V спирта (96%), перемешивают путем пипетирования и наносят образец на колонку RNeasy mini column, центрифугируют при 16100g в течение 15 сек при комнатной температуре, после чего удаляют проскок. Далее, колонку промывают с помощью 700 мкл буфера RWT, центрифугируя 15 сек при тех же условиях. Затем колонку промывают с помощью 500 мкл буфера RPE, центрифугируя 15 сек при тех же условиях, затем повторяют промывку буфером RPE, центрифугируя 2 мин при тех же условиях для подсушивания колонки. Далее, РНК элюируют используя 100 мкл свободной от нуклеаз воды. Концентрацию гидовой РНК определяют с помощью спектрофотометра Implen Nanophotometer N120 или аналогичного. Полученную гидовую РНК хранят в аликвотах при -80°С.
Для анализа чистоты и качества полученной гидовой РНК sgLepr-2 1 мкл РНК разделяют в 2%-ном ТАЕ-агарозном геле. На Фигуре 4А показаны результаты электрофореза, из которых видно, что гидовая РНК мигрирует в виде двух полос, одна из которых соответствует по молекулярному весу ожидаемому размеру гидовой РНК около 103 п.о, вторая полоса соответствует обычно присутствующей в препарате гидовой РНК форме со вторичными структурами, замедляющими ее подвижность в агарозном геле. Видно, что препарат не содержит примесных полос или признаков деградации РНК в виде шмера. Полученная гидовая РНК имеет нуклеотидную последовательность SEQ ID NO4.
Пример 5. Оценка активности гидовой РНК sgLepr-2 с помощью in vitro теста на расщепление субстрата с рибонуклеопротеиновым комплексом (РНП)
Рекомбинантный белок Cas9 получали как описано в [Maslennikova A., Kruglova N., Kalinichenko S, Komkov D, Shepelev M, Golubev D, Siniavin A, Vzorov A, Filatov A, Mazurov D. Engineering T-Cell Resistance to HIV-1 Infection via Knock-In of Peptides from the Heptad Repeat 2 Domain of gp41. mBio. 2022 Feb 22;13(1):e0358921. doi: 10.1128/mbio.03589-21]. Для проведения in vitro теста белок Cas9 разводят в однократном буфере Orange (#BO5, Thermo Scientific) до концентрации 3 мкМ. Гидовую РНК sgLepr-2 разводят в свободной от нуклеаз воде (Ambion, #AM9937) до концентрации 3 мкМ. Общий объем реакции in vitro теста на расщепление составляет 30 мкл и включает следующие компоненты:
Вначале для сборки РНП смешивают все компоненты кроме ДНК-субстрата (продукт ПЦР длиной 642 п.о., амплифицированный на матрице геномной ДНК кролика породы советская шиншилла с праймерами SEQ ID NO5 и SEQ ID NO6 как описано в Примере 2, и несущий мишень для гидовой РНК sgLepr-2). Смесь инкубируют при комнатной температуре в течение 10 мин для формирования РНП. Далее, добавляют ДНК-субстрат и инкубируют реакцию при +37°С в течение 30 мин. Затем для разрушения гидовой РНК добавляют 1 мкл РНКазы (10 мкг/мкл) и инкубируют реакцию 10 мин при +37°С, после чего для разрушения белка Cas9 и высвобождения ДНК-субстрата из комплекса с РНП добавляют 1 мкл протеиназы К (20 мкг/мкл) и инкубируют реакцию при +56°С в течении 10 мин. К образцам добавляют 6 мкл Orange DNA Loading Dye (6X) (ThermoFisher Scientific, #R0631) и 18 мкл полученной смеси разделяют в 2%-ном агарозном геле. Результаты in vitro теста на расщепление субстрата с РНП показаны на Фигуре 4Б. Видно, что в образце с гидовой РНК sgLepr-2 ПЦР-продукт длиной 642 п.о. расщепляется с формирование двух фрагментов ожидаемого размера 302 и 340 п.о., что свидетельствует о пригодности полученной гидовой РНК для внесения двуцепочечных разрывов ДНК в гене LEPR кролика в месте, определяемом последовательностью гидовой РНК sgLepr-2.
Пример 6. Получение кроликов с нокаутом гена LEPR
Пример 6а. Получение яйцеклеток кроликов
Для получения зигот кроликов самок-доноров осеменяют любым из доступных способов (искусственное осеменение, естественная случка) за 16-18 часов до промывания яйцеводов. Репродуктивные органы (матка, яйцеводы и яичники) отбирают, либо post mortem на бойне, либо прижизненным методом с проведением хирургической операции по удалению яйцеводов - тубэктомии. Для извлечения зигот из яйцеводов готовят промывочный раствор, состоящий из среды Хенкса, с добавлением антибиотика гентамицина и 2%-ой фетальной сыворотки крупного рогатого скота. Далее в подготовленный (отпрепарированный) яйцевод со стороны рога матки вводят на 1.5-2 см иглу калибра 21G, и шприцом объемом 10 мл с промывочным раствором (t=+25-29°С) и плавным нажатием на поршень осуществляют промывание яйцеводов. Сбор промывочного раствора с зиготами осуществляют в чашку Петри. После промывания проводят поиск зигот с применением микроскопа при 150-200-х-кратном увеличении. Если промывочный раствор с зиготами мутный, содержит следы крови, жира и т.д., то для более эффективного поиска зигот его фильтруют через фильтровальную систему для эмбрионов (например, через универсальную фильтровальную систему Miniflush® Minitube, Германия). Обнаруженные зиготы (показаны на Фигуре 5А) с помощью пластикового капилляра с внутренним диаметром 300 мкм и ротовой пипетки помещают в заранее приготовленную лунку четырехлуночного планшета, заполненную средой DMEM/F12 с добавлением 10%-ой фетальной сыворотки крупного рогатого скота и антибиотиков пенициллина и стрептомицина. Полученные зиготы культивируют в течение 30 минут при +38.5°С, 100% влажности и 7% CO2, чтобы убедиться в их жизнеспособности, затем помещают в камеру для микроинъекций.
Пример 6б. Подготовка смеси CRISPR/Cas9 реагентов для микроинъекций в зиготы кролика для получения нокаута гена LEPR
Для проведения микроинъекций в зиготы кролика готовят смесь компонентов системы редактирования генома CRISPR/Cas9 объемом 50 мкл как указано ниже.
1 мкг/мкл
Смесь разделяют на аликвоты объемом 8 мкл и хранят при -80°С до проведения микроинъекций.
Пример 6в. Проведение микроинъекции в зиготы
Микроинъекции компонентов системы редактирования генома в виде смеси мРНК нуклеазы Cas9 и гидовой РНК sgLepr-2 (см. Пример 6б) осуществляют в цитоплазму полученных зигот. Процедуру проводят в среде HEPES-KSOM под контролем микроскопа Zeiss Axiovert 200М при увеличении 400Х, с помощью микроманипуляторов Narishige и инъектора Eppendorf FemtoJet (Эппендорф, Франция). Для изготовления игл для микроинъекций используют пуллер Sutter instrument Со Р-97 (США), для изготовления удерживающей пипетки используют пуллер Narishige PC-10 и микрокузницу Narishige MF-900. После окончания микроинъекций выжившие зиготы помещают в заранее приготовленную лунку четырехлуночного планшета, заполненную средой DMEM/F12 с добавлением 10%-ой фетальной сыворотки крупного рогатого скота и антибиотиков пенициллина и стрептомицина.
Пример 6г. Подготовка самок-реципиентов
Для трансплантации (пересадки) эмбрионов в качестве самок-реципиентов отбирали здоровых половозрелых самок других пород (например, белый великан или калифорнийская). Для индукции овуляции самкам-реципиентам за 15-24 часа до пересадки эмбрионов инъецировали препараты, содержащие гонадотропин-рилизинг-фактор согласно инструкции производителя.
Пример 6д. Хирургическая трансплантация микроинъецированных эмбрионов самкам-реципиентам
Процедуру трансплантации эмбрионов проводили методом введения эмбрионов в воронку яйцевода самки-реципиента. Доступ к яйцеводам обеспечивали лапаротомией. Для анестезии и наркоза использовали препараты ксилазин, золетил, пропофол согласно инструкции производителя, с этой целью также можно применять ингаляционные виды наркоза. В качестве среды для трансплантации использовали среду Spermwash (Gynotec, Канада) или Ооклин (Панэко, Россия). Для 1 процедуры трансплантации брали 8-10 эмбрионов на стадии морулы (16-32 бластомера) из расчета 4-5 морулы в каждый яйцевод. В капилляр (катетер) с внутренним диаметром 300 мкм морулы заряжали таким образом: столбик среды без эмбрионов, столбик воздуха, столбик среды с эмбрионами, что обеспечивало полный выход эмбрионов из капилляра (катетера). После извлечения яичника с яйцеводом в его воронку вводили капилляр (катетер) на 1-4 см с заряженными в него эмбрионами и осуществляли выход эмбрионов во внутреннюю полость яйцевода либо шприцом объемом 1 мл, либо с помощью ротовой пипетки.
Пример 6е. Получение новорожденных кроликов с помощью кесарева сечения
Через 28-33 суток после трансплантации эмбрионов происходил либо естественный окрол, либо проводили кесарево сечение, что часто бывает при одноплодной беременности. В случае кесарева сечения, для анестезии и наркоза использовали препараты ксилазин, золетил, пропофол согласно инструкции производителя, с этой целью также можно применять ингаляционные виды наркоза. Доступ к матке обеспечивали лапаротомией, после чего вскрывали беременный рог матки и извлекали плод. Далее проводили экстирпацию матки, убеждались, что нет кровотечения, и послойно зашивали рану.
Пример 7. Генотипирование животных
Пример 7а. Выделение геномной ДНК из тканей кролика
Выделение геномной ДНК из тканей животных проводят в соответствии с протоколом [Green M.R., Sambrook J. Preparation of Genomic DNA from Mouse Tails and Other Small Samples. Cold Spring Harb Protoc. 2017 Sep 1 ;2017(9) :pdb.prot093518. doi:10.1101/pdb.prot093518. PMID: 28864567] с небольшими модификациями. В частности, образцы тканей животных лизируют при +56°С в течение 14-16 часов в 250 мкл буфера SNET (20 мМ Tris-HCl pH=8.0, 400 мМ NaCl, 5 мМ ЭДТА pH=8.0, 1% додецилсульфат натрия), содержащем 0.8 мкг/мкл протеиназы К (Sigma, #P2308). Далее, к образцам добавляют 125 мкл смеси хлороформа:изоамилового спирта в соотношении 24:1 и 125 мкл фенола, насыщенного 1М Tris-HCl pH=8.0. Образцы встряхивают на миксере и инкубируют 10-15 мин при постоянном перемешивании. Образцы центрифугируют в течение 5 мин при 16000g при комнатной температуре. Собирают верхнюю фазу и добавляют 250 мкл изопропанола, перемешивают образцы. Для осаждения геномной ДНК образцы центрифугируют в течении 15 мин при 16000g при +4°С. Затем осадок промывают 70% этанолом, высушивают и растворяют в 400 мкл буфера 10 мМ Трис-HCl, 0,5 мМ ЭДТА pH=9.0.
Пример 7б. Генотипирование кроликов с нокаутом LEPR помощью ПЦР
Генотипирование новорожденных кроликов проводят с помощью ПЦР с праймерами (SEQ ID NO5 и SEQ ID NO6), амплифицирующими участок генома кролика длиной 642 п.о., содержащий мишень для гидовой РНК sg-Lepr-2, как описано в Примере 2. В качестве отрицательного контроля используют геномную ДНК кролика породы советская шиншилла дикого типа. Далее, продукты ПЦР анализируют с помощью электрофореза в 8%-ном акриламидом геле. Результаты анализа показаны на Фигуре 6А, из которой видно, что на матрице геномной ДНК кролика, родившегося после пересадки эмбрионов, инъецированных компонентами системы CRISPR/Cas9 для редактирования гена LEPR (дорожка «KO»), амплифицируются два продукта. Одна полоса, соответствует фрагменту длиной 642 п.о., амплифицируемому на матрице геномной ДНК кролика дикого типа (дорожка «wt»), а вторая полоса имеет размер около 350 п.о. и присутствует только в образце «KO», свидетельствуя о том, что в результате геномного редактирования с помощью системы CRISPR/Cas9 произошла делеция в геноме кролика в локусе гена LEPR. Для, подтверждения полученных результатов и определения нуклеотидной последовательности фрагмента ДНК, несущего делецию, продукты ПЦР разделяют в 2%-ном ТАЕ-агарозном геле, вырезают обе полосы из геля, очищают из геля с помощью набора реагентов QIAEX II (Qiagen, #20021) и секвенируют по Сэнгеру используя олигонуклеотидный праймер SEQ ID NO5. В результате, было обнаружено, что нуклеотидная последовательность тяжелого фрагмента ДНК длиной 642 п.о. соответствовала фрагменту генома кролика дикого типа (данные не показаны). При этом было установлено, что короткий фрагмент ПЦР в образце «KO» имеет длину 356 п.о. и нуклеотидную последовательность SEQ ID NO 7. Анализ нуклеотидной последовательности показал, что короткий фрагмент образуется в результате делеции протяженностью 295 п.о. и инсерции случайных 9 п.о. TTTAAAGTA. Таким образом было подтверждено, что с помощью системы CRISPR/Cas9 был получен генно-модифицированный кролик, несущий делецию в геноме в области гена LEPR. Схема геномного локуса LEPR дикого типа и локуса с делеций в области 10 и 11 экзонов показана на Фигуре 6Б. Дальнейший анализ нуклеотидной последовательности гена LEPR кролика показал, что в результате делеции утрачиваются 58 п.о. экзона 10, полный интрон 10 длиной 144 п.о. и часть экзона 11 длиной 93 п.о., тем самым приводя к формированию в геноме гибридного экзона между 5’-областью 10 экзона и 3’-областью 11 экзона (Фигура 6Б). Таким образом, сплайсинг такого гибридного экзона с высокой вероятностью будет идти с использованием акцепторного сайта 10-го экзона и донорного сайта 11-го экзонов гена LEPR.
Анализ нуклеотидной последовательности предполагаемого транскрипта гена LEPR (XM_051856399), который будет транскрибироваться с геномной последовательности с обнаруженной делецией, указывает на то, что с такого транскрипта будут транслироваться 450 N-концевых аминокислотных остатков белка LEPR начиная с кодона инициации трансляции ATG во втором экзоне гена LEPR, за которыми в одной рамке считывания будет транслироваться бессмысленный пептид FKVVPCS, после чего трансляция такого укороченного белка будет обрываться за счет кодона терминации трансляции TGA (Фигура 6В). Таким образом, можно заключить что поставленная задача изобретения решена и с помощью CRISPR/Cas9-опосредованного геномного редактирования получен генно-модифицированный кролик с нокаутом гена LEPR. Фотография кролика приведена на Фигуре 6Г.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Rabbits.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-12-21">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>-</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт биологии гена Российской академии
наук </ApplicantName>
<ApplicantNameLatin>Institute of Gene Biology Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ получения
генно-модифицированных кроликов с нокаутом гена LEPR с помощью
системы CRISPR/Cas9</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>21..>23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>PAM site</INSDQualifier_value>
<NonEnglishQualifier_value>CGG</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location>1..>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q4">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>Protospacer</INSDQualifier_value>
<NonEnglishQualifier_value>GCTGACACGATCATTCACAA
</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgacacgatcattcacaacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location>1..>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>Protospacer</INSDQualifier_value>
<NonEnglishQualifier_value>ATAATTCCGCATAGCGATGA
</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>21..>23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>PAM site</INSDQualifier_value>
<NonEnglishQualifier_value>TGG</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataattccgcatagcgatgatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>8487</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8487</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagggcctatttcccatgattccttcatatttgcatatacgatacaagg
ctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtag
aaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttacc
gtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccgataattccg
catagcgatgagttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaag
tggcaccgagtcggtgcttttttgttttagagctagaaatagcaagttaaaataaggctagtccgttttt
agcgcgtgcgccaattctgcagacaaatggctctagaggtacccgttacataacttacggtaaatggccc
gcctggctgaccgcccaacgacccccgcccattgacgtcaatagtaacgccaatagggactttccattga
cgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagta
cgccccctattgacgtcaatgacggtaaatggcccgcctggcattgtgcccagtacatgaccttatggga
ctttcctacttggcagtacatctacgtattagtcatcgctattaccatggtcgaggtgagccccacgttc
tgcttcactctccccatctcccccccctccccacccccaattttgtatttatttattttttaattatttt
gtgcagcgatgggggcggggggggggggggggcggggcgaggggcggggcggggcgaggcggagaggtgc
ggcggcagccaatcagagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccc
tataaaaagcgaagcgcgcggcgggcgggagtcgctgcgcgctgccttcgccccgtgccccgctccgccg
ccgcctcgcgccgcccgccccggctctgactgaccgcgttactcccacaggtgagcgggcgggacggccc
ttctcctccgggctgtaattagctgagcaagaggtaagggtttaagggatggttggttggtggggtatta
atgtttaattacctggagcacctgcctgaaatcactttttttcaggttggaccggtgccaccatggacta
taaggaccacgacggagactacaaggatcatgatattgattacaaagacgatgacgataagatggcccca
aagaagaagcggaaggtcggtatccacggagtcccagcagccgacaagaagtacagcatcggcctggaca
tcggcaccaactctgtgggctgggccgtgatcaccgacgagtacaaggtgcccagcaagaaattcaaggt
gctgggcaacaccgaccggcacagcatcaagaagaacctgatcggagccctgctgttcgacagcggcgaa
acagccgaggccacccggctgaagagaaccgccagaagaagatacaccagacggaagaaccggatctgct
atctgcaagagatcttcagcaacgagatggccaaggtggacgacagcttcttccacagactggaagagtc
cttcctggtggaagaggataagaagcacgagcggcaccccatcttcggcaacatcgtggacgaggtggcc
taccacgagaagtaccccaccatctaccacctgagaaagaaactggtggacagcaccgacaaggccgacc
tgcggctgatctatctggccctggcccacatgatcaagttccggggccacttcctgatcgagggcgacct
gaaccccgacaacagcgacgtggacaagctgttcatccagctggtgcagacctacaaccagctgttcgag
gaaaaccccatcaacgccagcggcgtggacgccaaggccatcctgtctgccagactgagcaagagcagac
ggctggaaaatctgatcgcccagctgcccggcgagaagaagaatggcctgttcggaaacctgattgccct
gagcctgggcctgacccccaacttcaagagcaacttcgacctggccgaggatgccaaactgcagctgagc
aaggacacctacgacgacgacctggacaacctgctggcccagatcggcgaccagtacgccgacctgtttc
tggccgccaagaacctgtccgacgccatcctgctgagcgacatcctgagagtgaacaccgagatcaccaa
ggcccccctgagcgcctctatgatcaagagatacgacgagcaccaccaggacctgaccctgctgaaagct
ctcgtgcggcagcagctgcctgagaagtacaaagagattttcttcgaccagagcaagaacggctacgccg
gctacattgacggcggagccagccaggaagagttctacaagttcatcaagcccatcctggaaaagatgga
cggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaagcagcggaccttcgacaac
ggcagcatcccccaccagatccacctgggagagctgcacgccattctgcggcggcaggaagatttttacc
cattcctgaaggacaaccgggaaaagatcgagaagatcctgaccttccgcatcccctactacgtgggccc
tctggccaggggaaacagcagattcgcctggatgaccagaaagagcgaggaaaccatcaccccctggaac
ttcgaggaagtggtggacaagggcgcttccgcccagagcttcatcgagcggatgaccaacttcgataaga
acctgcccaacgagaaggtgctgcccaagcacagcctgctgtacgagtacttcaccgtgtataacgagct
gaccaaagtgaaatacgtgaccgagggaatgagaaagcccgccttcctgagcggcgagcagaaaaaggcc
atcgtggacctgctgttcaagaccaaccggaaagtgaccgtgaagcagctgaaagaggactacttcaaga
aaatcgagtgcttcgactccgtggaaatctccggcgtggaagatcggttcaacgcctccctgggcacata
ccacgatctgctgaaaattatcaaggacaaggacttcctggacaatgaggaaaacgaggacattctggaa
gatatcgtgctgaccctgacactgtttgaggacagagagatgatcgaggaacggctgaaaacctatgccc
acctgttcgacgacaaagtgatgaagcagctgaagcggcggagatacaccggctggggcaggctgagccg
gaagctgatcaacggcatccgggacaagcagtccggcaagacaatcctggatttcctgaagtccgacggc
ttcgccaacagaaacttcatgcagctgatccacgacgacagcctgacctttaaagaggacatccagaaag
cccaggtgtccggccagggcgatagcctgcacgagcacattgccaatctggccggcagccccgccattaa
gaagggcatcctgcagacagtgaaggtggtggacgagctcgtgaaagtgatgggccggcacaagcccgag
aacatcgtgatcgaaatggccagagagaaccagaccacccagaagggacagaagaacagccgcgagagaa
tgaagcggatcgaagagggcatcaaagagctgggcagccagatcctgaaagaacaccccgtggaaaacac
ccagctgcagaacgagaagctgtacctgtactacctgcagaatgggcgggatatgtacgtggaccaggaa
ctggacatcaaccggctgtccgactacgatgtggaccatatcgtgcctcagagctttctgaaggacgact
ccatcgacaacaaggtgctgaccagaagcgacaagaaccggggcaagagcgacaacgtgccctccgaaga
ggtcgtgaagaagatgaagaactactggcggcagctgctgaacgccaagctgattacccagagaaagttc
gacaatctgaccaaggccgagagaggcggcctgagcgaactggataaggccggcttcatcaagagacagc
tggtggaaacccggcagatcacaaagcacgtggcacagatcctggactcccggatgaacactaagtacga
cgagaatgacaagctgatccgggaagtgaaagtgatcaccctgaagtccaagctggtgtccgatttccgg
aaggatttccagttttacaaagtgcgcgagatcaacaactaccaccacgcccacgacgcctacctgaacg
ccgtcgtgggaaccgccctgatcaaaaagtaccctaagctggaaagcgagttcgtgtacggcgactacaa
ggtgtacgacgtgcggaagatgatcgccaagagcgagcaggaaatcggcaaggctaccgccaagtacttc
ttctacagcaacatcatgaactttttcaagaccgagattaccctggccaacggcgagatccggaagcggc
ctctgatcgagacaaacggcgaaaccggggagatcgtgtgggataagggccgggattttgccaccgtgcg
gaaagtgctgagcatgccccaagtgaatatcgtgaaaaagaccgaggtgcagacaggcggcttcagcaaa
gagtctatcctgcccaagaggaacagcgataagctgatcgccagaaagaaggactgggaccctaagaagt
acggcggcttcgacagccccaccgtggcctattctgtgctggtggtggccaaagtggaaaagggcaagtc
caagaaactgaagagtgtgaaagagctgctggggatcaccatcatggaaagaagcagcttcgagaagaat
cccatcgactttctggaagccaagggctacaaagaagtgaaaaaggacctgatcatcaagctgcctaagt
actccctgttcgagctggaaaacggccggaagagaatgctggcctctgccggcgaactgcagaagggaaa
cgaactggccctgccctccaaatatgtgaacttcctgtacctggccagccactatgagaagctgaagggc
tcccccgaggataatgagcagaaacagctgtttgtggaacagcacaagcactacctggacgagatcatcg
agcagatcagcgagttctccaagagagtgatcctggccgacgctaatctggacaaagtgctgtccgccta
caacaagcaccgggataagcccatcagagagcaggccgagaatatcatccacctgtttaccctgaccaat
ctgggagcccctgccgccttcaagtactttgacaccaccatcgaccggaagaggtacaccagcaccaaag
aggtgctggacgccaccctgatccaccagagcatcaccggcctgtacgagacacggatcgacctgtctca
gctgggaggcgacaaaaggccggcggccacgaaaaaggccggccaggcaaaaaagaaaaagtaagaattc
ctagagctcgctgatcagcctcgactgtgccttctagttgccagccatctgttgtttgcccctcccccgt
gccttccttgaccctggaaggtgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcat
tgtctgagtaggtgtcattctattctggggggtggggtggggcaggacagcaagggggaggattgggaag
agaatagcaggcatgctggggagcggccgcaggaacccctagtgatggagttggccactccctctctgcg
cgctcgctcgctcactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcccgggcggcctca
gtgagcgagcgagcgcgcagctgcctgcaggggcgcctgatgcggtattttctccttacgcatctgtgcg
gtatttcacaccgcatacgtcaaagcaaccatagtacgcgccctgtagcggcgcattaagcgcggcgggt
gtggtggttacgcgcagcgtgaccgctacacttgccagcgccttagcgcccgctcctttcgctttcttcc
cttcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccg
atttagtgctttacggcacctcgaccccaaaaaacttgatttgggtgatggttcacgtagtgggccatcg
ccctgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaa
ctggaacaacactcaactctatctcgggctattcttttgatttataagggattttgccgatttcggtcta
ttggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatt
ttatggtgcactctcagtacaatctgctctgatgccgcatagttaagccagccccgacacccgccaacac
ccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgg
gagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgagacgaaagggcctcgtgatacgc
ctatttttataggttaatgtcatgataataatggtttcttagacgtcaggtggcacttttcggggaaatg
tgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataacc
ctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttatt
cccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaaaagatgctg
aagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttt
tcgccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcccgt
attgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcac
cagtcacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccatgag
tgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcac
aacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacg
agcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcgaactacttac
tctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgctcg
gcccttccggctggctggtttattgctgataaatctggagccggtgagcgtggaagccgcggtatcattg
cagcactggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaactat
ggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaa
gtttactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcc
tttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtaga
aaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaacca
ccgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggcttca
gcagagcgcagataccaaatactgttcttctagtgtagccgtagttaggccaccacttcaagaactctgt
agcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgt
cttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgt
gcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaag
cgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgc
acgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttg
agcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggccttttt
acggttcctggccttttgctggccttttgctcacatgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>103</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..103</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggataattccgcatagcgatgagttttagagctagaaatagcaagtta
aaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgctttt</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctttatctgatatcctgcttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttagctcactatacccacaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>356</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..356</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctttatctgatatcctgcttttcttccttcttcagatgtcatttact
ttccacctaaaattctgacaagtgttgggtctaacgtttcttttcactgcatctataaaaatgagcacaa
gattgtgtcctcaaaacagattgtttggtggatgaatttagctgagaaaattcctcaaagccagtatacc
gttgtgaatgatcgtgtcagcaaagttacttttcccaatttgaatgcaaccaaacctcgagggaagttta
cctacgatgcatttaaagtagtaccttgcagttgaggtatcataggtatgtgttatttttgttgtttttt
ttatttctgtgggtatagtgagctaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caccgataattccgcatagcgatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaactcatcgctatgcggaattatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtaatacgactcactatagggataattccgcatagcgatga</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaagcaccgactcggtgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>122</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..122</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtaatacgactcactatagggataattccgcatagcgatgagttttag
agctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgct
ttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ получения генно-модифицированных мышей, экспрессирующих миниген антитромбина III человека, с помощью микроинъекций TelN-линеаризованного фрагмента ДНК | 2022 |
|
RU2806568C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ конструирования минигенов млекопитающих и рекомбинантная плазмида pgC1HDR, кодирующая миниген ингибитора С1 эстеразы человека, предназначенная для получения гуманизированных по гену Serping1 мышей | 2022 |
|
RU2805177C1 |
| Способ получения генно-модифицированных лабораторных животных с нуль-аллелем гена P2rx3 | 2022 |
|
RU2805173C1 |
| Способ получения кролика с нокаутом гена CSN2 | 2023 |
|
RU2834016C1 |
| ЛИНИЯ ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННЫХ МЫШЕЙ ДЛЯ МОДЕЛИРОВАНИЯ БОЛЕЗНИ АЛЬЦГЕЙМЕРА, СПОСОБ ЕЕ ПОЛУЧЕНИЯ И ЕЕ ПРИМЕНЕНИЕ, sgPHK И КОМБИНАЦИЯ crRNA/tracrRNA, ИСПОЛЬЗУЕМЫЕ В СПОСОБЕ, И НАБОР ПРАЙМЕРОВ | 2024 |
|
RU2835497C1 |
| Технология получения мышей с гуманизированным участком Gnao1 в области однонуклеотидного полиморфизма rs587777057 для тестирования РНК-терапии для GNAO1 c.607 G>A пациентов | 2021 |
|
RU2757121C1 |
| Способ усиления направленного редактирования гена CXCR4 в CD4-лимфоцитах человека в целях создания резистентности клеток к ВИЧ-1 | 2021 |
|
RU2779300C1 |
| Способ получения нокаута гена CD209 в эмбрионах Bos taurus путем трансдукции зигот адено-ассоциированными вирусами, кодирующими saCas9 и соответствующую гидовую РНК | 2022 |
|
RU2800917C1 |
| Способ редактирования гена GJB2 для исправления патогенного варианта c.del35G в клетках человека, культивируемых in vitro | 2021 |
|
RU2780677C1 |


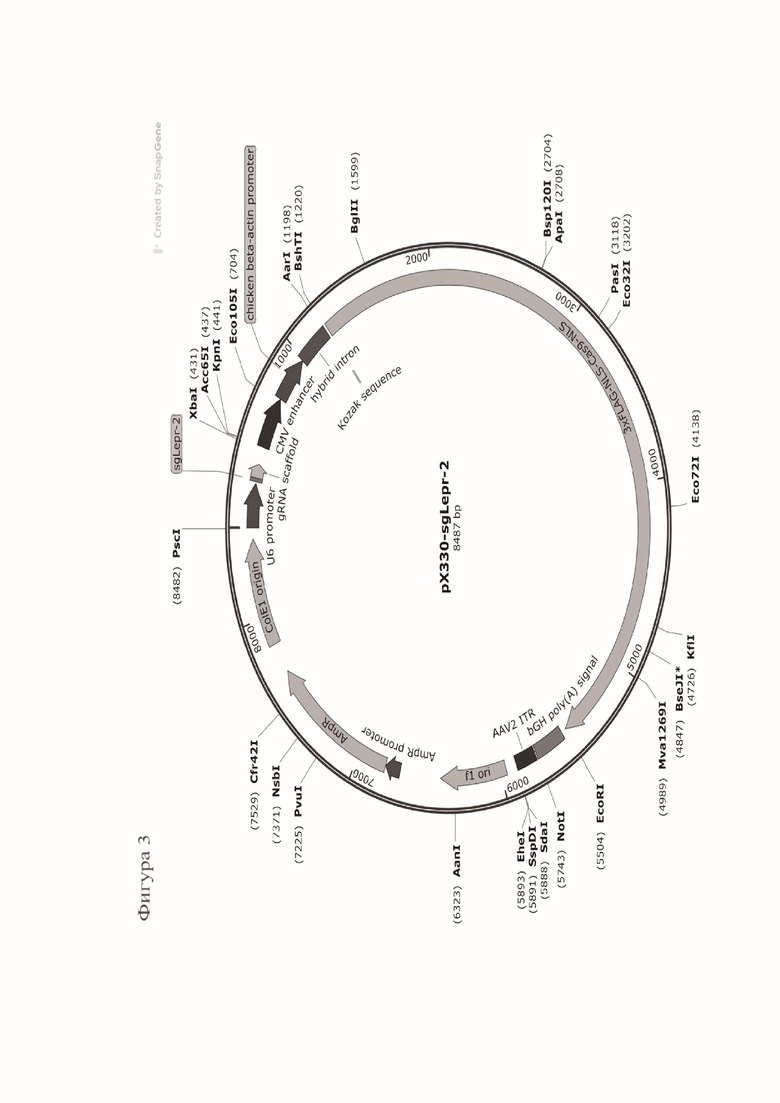
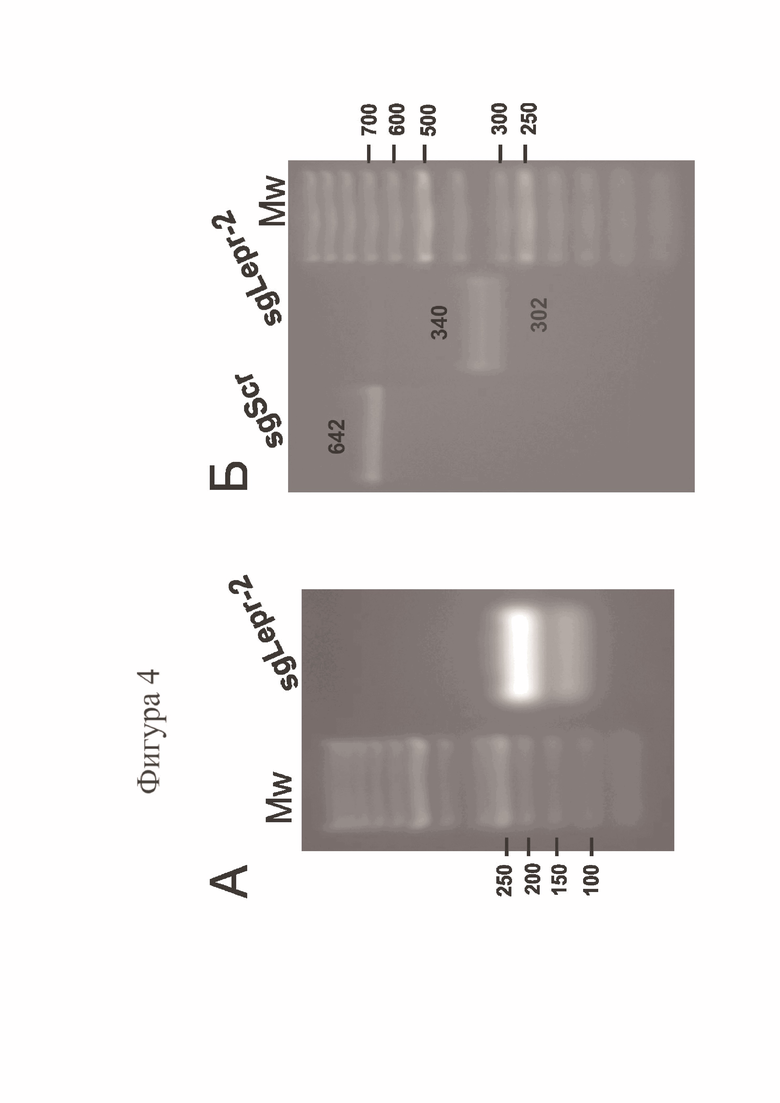

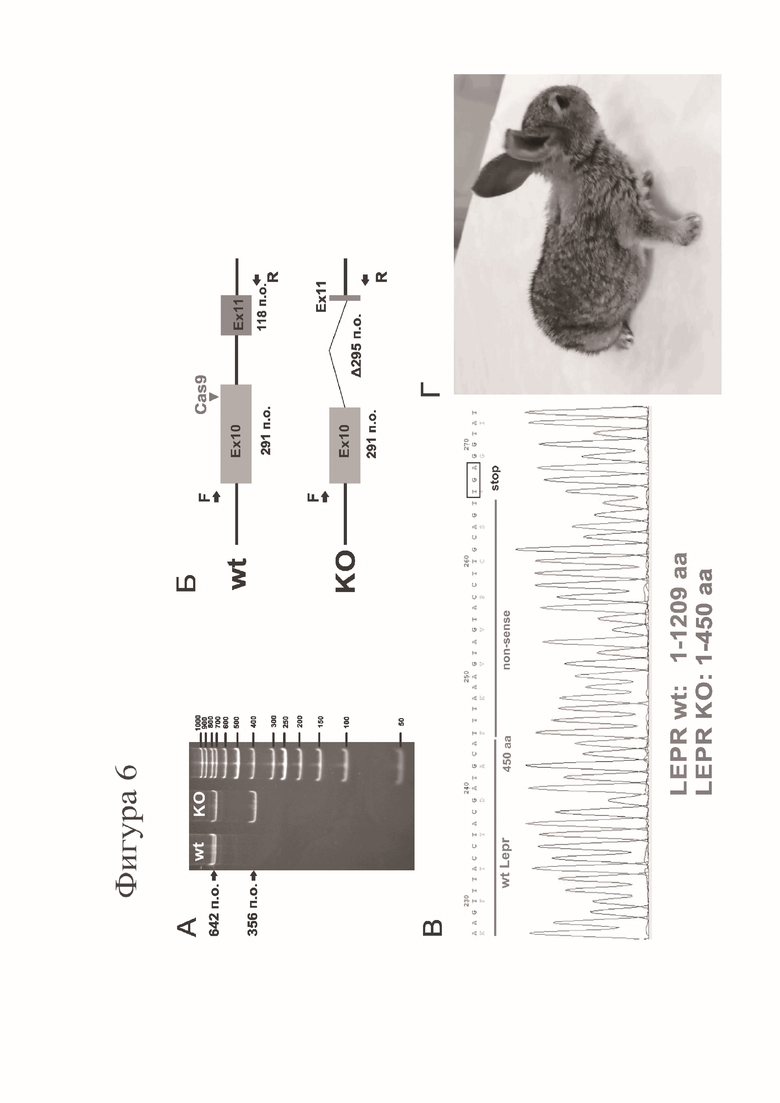
Изобретение относится к области биотехнологии, в частности к генно-модифицированному кролику с нокаутом гена LEPR. Указанный кролик предназначен для создания модели морбидного ожирения и нарушений липидного и углеводного обмена и несет в себе делецию в гене LEPR кролика, включающую часть 10 экзона, 10 интрон и часть 11 экзона. Настоящее изобретение также относится к способу получения указанного генно-модифицированного кролика с помощью системы CRISPR/Cas9. Настоящее изобретение обеспечивает генетическую модификацию (делецию) в геноме кролика в локусе гена LEPR. 2 н.п. ф-лы, 6 ил., 7 пр.
1. Генно-модифицированный кролик с нокаутом гена LEPR для создания модели морбидного ожирения и нарушений липидного и углеводного обмена, несущий делецию в гене LEPR кролика, включающую часть 10 экзона, 10 интрон и часть 11 экзона.
2. Способ получения генно-модифицированного кролика по п.1 с помощью системы CRISPR/Cas9, включающий следующие стадии:
1) получение гидовой РНК последовательностью SEQ ID NO 11, направляющей нуклеазу Cas9 к последовательности 10 экзона гена LEPR кролика, включающее следующие стадии:
1.1. амплификация фрагмента ДНК последовательностью SEQ ID NO 10 с помощью ПЦР с праймерами последовательностями SEQ ID NO 8 и SEQ ID NO 9 на матрице рекомбинантной плазмидной ДНК pX330-sgLepr-2 последовательностью SEQ ID NO 7, экспрессирующей нуклеазу Cas9 и гидовую РНК;
1.2. получение гидовой РНК последовательностью SEQ ID NO 11 с помощью транскрипции in vitro с использованием ДНК-матрицы последовательностью SEQ ID NO 10;
2) получение оплодотворенных яйцеклеток кролика, включающее осеменение самок, хирургическое изъятие яичников, вымывание из них яйцеклеток и культивирование их в среде DMEM/F12 с добавлением 10%-ной фетальной сыворотки крупного рогатого скота и антибиотиков пенициллина и стрептомицина при +38.5°С, 100% влажности и 7% CO2;
3) микроинъекции в цитоплазму оплодотворенных яйцеклеток кролика смеси мРНК нуклеазы Cas9 и гидовой РНК последовательностью SEQ ID NO 11;
4) культивирование и хирургическая трансплантация инъецированных яйцеклеток кролика в яйцеводы самок-реципиентов, у которых индуцируют овуляцию за 15-24 часа до пересадки яйцеклеток путем инъекции гонадотропин-рилизинг-фактора;
5) естественный окрол либо извлечение новорожденных кроликов с помощью кесарева сечения через 28-33 дня после трансплантации яйцеклеток самкам-реципиентам.
| BAO, D | |||
| et al | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| PEDROSO, JOAO A.B | |||
| et al | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Соломорезка | 1918 |
|
SU157A1 |
| MATSUHISA F | |||
| et al | |||
| Transgenic Rabbit | |||
Авторы
Даты
2025-03-17—Публикация
2023-12-21—Подача