Область техники
Изобретение относится к области молекулярной биотехнологии и касается метода редактирования генома, в первую очередь, получения нокаута выбранных генов, в данном случае CD209. Изобретение может быть использовано для получения животных с нокаутом гена CD209, приводящим к устойчивости животных к некоторым патогенам.
Изобретение создано при финансовой поддержке Министерства Науки и Высшего образования Российской Федерации в рамках Соглашения № 075-15-2019-1661 от 31.10.2019 на базе Центра высокоточного редактирования и генетических технологий для биомедицины (ЦВРГТБ) ИБГ РАН.
Уровень техники
Редактирование и модификация генома – это современные подходы, которые позволяют получать животных с требуемыми характеристиками: составом молока [1, 2], объемом мышц [3, 4] или устойчивостью к инфекционным заболеваниям [5, 6]. На сегодняшний день получены генетически модифицированные представители различных видов, в том числе грызуны [7, 8], приматы [9, 10] и крупный рогатый скот [6, 11, 12]. Основной способ генетической модификации и редактирования генома животных – внесение модификаций в геном эмбрионов на ранних стадиях развития. Для достижения этой цели используют различные методики: трансплантацию генетически модифицированных in vitro ЭСК в полость бластоцисты с последующим формированием мозаичного зародыша [13, 14], или доставку реагентов для редактирования генов в развивающиеся эмбрионы путем инъекции РНК или ДНК в зиготу или ооцит [15-20] или электропорации зиготы [21-23]. Основным недостатком этих методов является то, что они требуют дорогостоящего оборудования и высокой квалификации операторов.
Генетическая модификация клеток (в том числе эмбрионов) путем трансдукции - простой метод: требуется только базовое лабораторное оборудование (CO2-инкубаторы, микроскопы); нет необходимости делать микроинъекции; вирусы, используемые для трансдукции, могут производиться в другом месте и легко транспортируются. Поэтому производство трансгенных эмбрионов может осуществляться вблизи ферм (где лаборатории зачастую оснащены лишь базовым оборудованием). Таким образом, такой подход позволит выполнить все необходимые процедуры (извлечение эмбрионов, генную модификацию и перенос эмбрионов животному) в одном месте, снизив риск повреждения эмбрионов при транспортировке.
Для доставки генетического материала в зиготу можно использовать различные типы вирусов. Для этих процедур используют ретровирусы [24], особенно лентивирусы [25], аденовирусы [26] и аденоассоциированные вирусы [27].
Аденоассоциированные вирусы (ААВ) широко используются в области получения генетически модифицированных клеток и организмов. Было показано, что ААВ служат эффективным инструментом для получения трансгенных мезенхимальных стволовых клеток свиньи [28], сперматозоидов и эмбрионов [29], нейронов взрослых мышей [30], мышиных [26, 27] и бычьих эмбрионов [27].
Аденоассоциированные вирусы имеют значительно меньшие вирионы по сравнению с ретровирусами или аденовирусами. Их небольшой (около 20 нм) размер [31, 32] облегчает проникновение вириона ААВ в зародыш.
ААВ традиционно считаются безопасной вирусной векторной платформой из-за того, что вирус-помощник необходим для возникновения инфекции в естественных условиях. Аденоассоциированный вирус может инфицировать клетку, но для цикла его размножения требуется вирус-помощник, такой как аденовирус или вирус простого герпеса. Все серотипы рекомбинантных векторов ААВ, созданных в отсутствие хелперных вирусов, рассматриваются как агенты группы риска 1 (агенты, не связанные с заболеванием у здоровых взрослых людей), если трансген не кодирует для токсического или потенциально туморогенного гена. Следовательно, многие рекомбинантные векторы ААВ можно использовать без дополнительных опасений по поводу биобезопасности [33].
В рамках данного изобретения трансдукция бычьих эмбрионов с помощью ААВ была выбрана в качестве потенциального подхода к редактированию генома. Пять различных серотипов ААВ использовали для оценки их способности доставлять генетический материал в эмбрионы крупного рогатого скота.
У млекопитающих CD209 (также известный как DC-SIGN; специфичная для дендритных клеток молекула межклеточной адгезии) является одним из ключевых белков, участвующих во многих иммунологических событиях. CD209 представляет собой лектиновый рецептор С-типа, присутствующий на поверхности макрофагов и дендритных клеток [34]. Являясь частью первой линии врожденного иммунитета, CD209 довольно не специфически распознает широкий круг возбудителей: вирусы, бактерии, грибы, протисты. Во время коэволюции в системах хозяин/патоген некоторые патогены научились использовать CD209 для дальнейшего распространения (например, CD209+ DC используются для переноса вирионов ВИЧ из места контакта на слизистой оболочке в кровоток и лимфатические узлы) и, таким образом, поражать различные типы клеток. Влияние CD209 на разные стадии инфекционного процесса показано для ВИЧ [35], вируса гепатита С [36], вируса Эбола, вируса Денге, коронавирусов, микобактерий туберкулеза [37, 38], лейшманий [39] и другими инфекционными агентами (обзор в [40]).
Один из возможных механизмов, связанных с CD209, защиты патогенов от иммунной системы был выяснен для вирусов ВИЧ и гепатита С. Было показано, что даже если обычно после связывания с CD209 патогены интернализуются в протеасомы для будущей презентации антигена, при определенных обстоятельствах (таких как наличие дополнительных рецепторов) после связывания CD209 ВГС и ВИЧ частицы могут интернализоваться в специализированные, нелизосомальные, эндосомы с низким рН [36, 41, 42]. Недавно было продемонстрировано, что мыши с нокаутом CD209 менее восприимчивы к инфекции Toxoplasma gondii: инвазия и распространение паразитов происходили медленнее, а смертность снизилась [43].
Недавно было показано, что некоторые изоформы CD209 могут влиять на развитие резистентности коров к заражению паратуберкулезом [44-46]. Более того, сообщалось, что сверхэкспрессия CD209 в бычьих макрофагах in vitro приводит к усилению аденовирусной инфекции [47]. Таким образом, получение нокаута CD209 актуально для лучшего понимания механизмов инфекции при паратуберкулезе и аденовирусах. Получение такого нокаута потенциально может позволить животным стать устойчивыми к различным инфекциям.
Краткое описание изобретения
Задачей заявляемого изобретения является разработка метода получения эмбрионов Bos taurus с нокаутом гена CD209 и получение соответствующих эмбрионов.
Задача решается тем, что с использованием ААВ, кодирующих компоненты системы CRISPR/Cas9, разработан соответствующий метод и получены эмбрионы Bos taurus с нокаутом гена CD209, для чего:
1. На основе вектора px601 (SEQ ID NO: 1) была получена плазмидная конструкция px601+CD209-sg, содержащая гидовую РНК (SEQ ID NO: 2) против соответствующего участка (3 экзона гена Bos taurus) гена CD209, под контролем U6 промотора и ОРС гена saCas9 под контролем CAG промотора.
2. Был разработан способ получения эмбрионов с нуль-аллелем гена CD209, характеризующийся следующими стадиями:
а) Сборка в клетках упаковочной линии HEK293T стока ААВ, содержащего ОРС saCas9 и гидовую РНК;
б) In vitro оплодотворение яйцеклеток Bos taurus сперматозоидами Bos taurus;
в) Инфекция эмбрионов ААВ;
г) Проведение анализа наличия сдвига рамки считывания в гене CD209 в бластоцистах (г), путем амплификации целевого участка гена CD209 с праймерами cd209-check-f (SEQ ID NO: 3) и cd209-check-r (SEQ ID NO: 4) с последующим секвенированием фрагмента с использованием праймера cd209-check-r (SEQ ID NO: 4).
Технический результат изобретения: разработан способ генетического редактирования эмбрионов Bos taurus и получены эмбрионы с нуль-аллелем гена CD209.
Краткое описание чертежей
На Фиг. 1 представлена схема взаимного расположения первых экзонов гена CD209, гидовой РНК и праймеров для анализа генотипа.
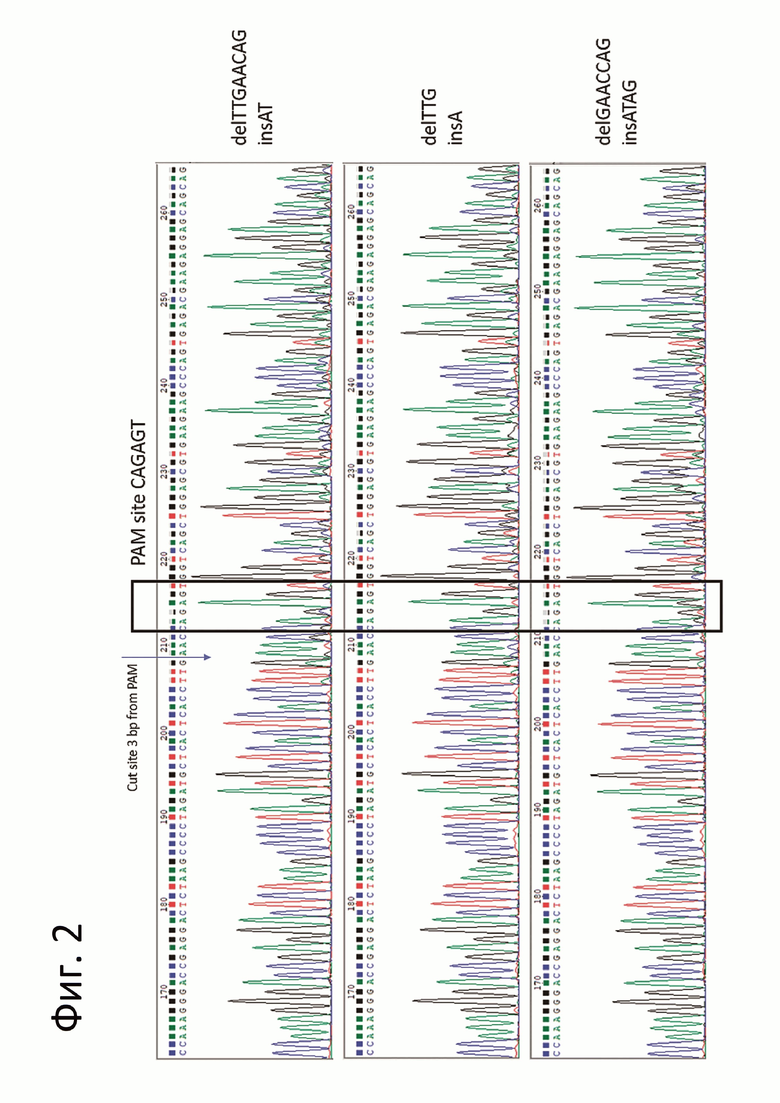
На Фиг. 2 представлены хроматограммы, полученные в результате секвенирования мозаичных эмбрионов: двоящиеся сиквенсы, соответствующий мозаичным эмбрионам в части клеток которых произошло редактирование генома.
Осуществление изобретения
Способ осуществляется следующим образом:
1. Подбор гидовой РНК для внесения разреза в ген bCD209 с целью внесения сдвина рамки считывания осуществлялся с использованием онлайн инструмента CRISPOR TEFOR (http://crispor.tefor.net/) с учетом последующего использования нуклеазы SаCas9. Была подобрана гидовая РНК (CD209-sg, последовавательности SEQ ID NO: 1) к третьему экзону гена bCD209. В результате NHEJ-репарации после их разрезания должен образовываться сдвиг рамки считывания в первом экзоне, приводящий к изменению аминокислотной последовательности белка и преждевременному образованию стоп-кодона (Фиг. 1). Выбранную сгРНК мы заклонировали в плазмидный вектор px601, предназначенный для одновременной экспрессии белка saCas9 и сгРНК. Последовательность гидовой РНК была синтезирована с использованием двух олигонуклеотидов SEQ ID NO: 5, SEQ ID NO: 6.
Разрезание вектора проводили с использованием рестриктазы BbsI-HF (New England Biolabs), дефосфорилирование вектора с помощью фермента FastAP (Thermofisher Scientific), затем очистка в агарозном геле и последующее выделение с помощью набора Monarch DNA Gel Extraction Kit.
Олигонуклеотиды фосфорилировались с использованием фермента PNK kinase (Thermofisher Scientific), отжигались друг с другом и лигировались с приготовленным ранее вектором и использованием фермента T4 DNA ligase (Thermofisher Scientific). Лигазная смесь трансформировалась в компетентные клетки штамма XL1blue (Евроген). Скрининг колоний осуществлялся с использованием пцр-наборов компании Изоген и праймеров U6-forv (SEQ ID NO: 7) и реверсного праймера, использовавшегося для клонирования. По две колонии, содержащие сгРНК, наращивались в ночной культуре и выделялись с помощью набора Monarch Plasmid Miniprep Kit. Анализ полученных плазмид проводили методом секвенирования по Сэнгеру. Сиквенс полученных плазмид осуществлялся с использованием праймера U6-forv (SEQ ID NO: 7) компанией Евроген. Анализ сиквенсов проводился с использованием программного обеспечения Chromas.
2. Протестированную конструкцию, выделенную набором Maxiprep Plasmid Purification Kit (Qiagen) вместе с плазмидами Rep2-Cap и pHelper (обе – Agilent) использовали для трансфекции клеток HEK293. Рекомбинантные ААВ получали как описано в [49]. Прикрепленные клетки HEK293Т поддерживали в среде DMEM с высоким содержанием глюкозы (Gibco, США), с добавлением 10% об./об. фетальной бычьей сыворотки (FBS, Biosera Europe, Франция) и пенициллина со стрептомицином. Трансфекцию проводили линейным ПЭИ (полиэтиленимин, масс. 25000) (Polysciences, США) Через 72 ч после трансфекции клетки лизировали 0,5% об./об. Triton X-100, 5 мкг/мл РНКазы А и 10 мкг/мл ДНКазы А (Calbiochem, США) в течение 1 ч при 37°С с встряхиванием. Затем осветленную и концентрированную вирусную суспензию загружали в модифицированную версию йодиксанола (Sigma Aldrich, США) со ступенчатым градиентом плотности (60%, 40% и 25%) [50] и подвергали ультрацентрифугированию в течение 1 ч при 350 000 g и 18°С. Фракцию, содержащую ААВ, подвергали диализу с использованием мембраны 100 кДа против буфера для хранения (PBS, 350 мМ NaCl, 0,1% Pluronic F-68 (Gibco)), а очищенную суспензию дополнительно концентрировали и стерилизовали [51]. Для определения титра использовали метод, основанный на использовании праймеров и флуоресцентного зонда, нацеленного на последовательности ITR (Inverted terminal Repeats) [52]. Отсутствие белковых примесей во время приготовления подтверждали окрашиванием серебром (ThermoFisher, США).
3. Получение яйцеклеток
Ооциты были выделены из яичников, полученных на местных скотобойнях. Полученные после забоя и разделки туши яичники транспортировали в лабораторию в течение 3-4 часов при температуре 38.5оС. Выделение ооцит-кумулюсных комплексов (содержащих ооциты и вспомогательные клетки кумулюса) из яичников проводили путём аспирации в результате пунктирования крупных фолликулов. Аспирацию производили с помощью предварительно нагретой до температуры 38.5оС среды G-MOPS (Vitrolife, Швеция) посредством инсулиновых шприцов (объёмом 1 мл) с иглой 30G.
Полученные ооциты затем были подвергнуты процедуре созревания in vitro (IVM - in vitro maturation) для достижения стадии метафазы второго деления мейоза, что необходимо для возможности оплодотворения ооцитов. Для этого ооцит-кумулюсные комплексы помещали в среду BO-IVM medium (IVF Bioscience, UK), покрытую слоем парафинового масла (Sage, USA) для предотвращения испарения и изменения осмолярности среды, и культивировали при температуре 38.5оС в увлажненной температуре в атмосфере 6.5% CO2 и 5%O2 в пластиковых чашках Петри диаметром 60 мм.
4. Подготовка сперматозоидов
Для проведения экстракорпорального оплодотворения была использована криоконсервированная сперма. Для криоконсервации свежую сперму, полученную путём донирования, помещали в 0,2 мл лунки, изготовленные во фторопластовой плате, помещенному над поверхностью жидкого азота. Плату со спермой инкубировали над жидким азотом над высотой 5 см в течение 1,5-2 минут после чего погружали непосредственно в жидкий азот. После удаления платы из жидкого азота, гранулы спермы собирали и помещали для хранения в сосуд Дьюара.
Криоконсервированную сперму размораживали при температуре 37оС в течение 1 минуты, а затем центрифугировали в градиенте плотности (80% Percoll SpermGrad (Vitrolife, Швеция)) объёмом 3 мл в течение 10 минут при 700g. После центрифугирования осадок отмывали с помощью буферного раствора SpermRinse buffer medium SpermGrad (Vitrolife, Швеция), с добавлением 5 МЕ/мл гепарина в течение 5 минут при 350g.
5. Экстракорпоральное оплодотворение
Для проведения экстракорпорального оплодотворения сперматозоиды (в концентрации ~1*106-2*106 в мл) и ооцит-кумулюсные комплексы помещали в среду BO-IVF medium (IVF Bioscience, UK) на 15 часов для оплодотворения. Затем эмбрионы трижды отмывали в среде BO-IVF medium (IVF Bioscience, UK). Процедуру жкстракорпорального оплодотворения производили в CO2-инкубаторе при температуре 38.5°C в увлажненной атмосфере, содержащей 6.5%CO2 и 5%O2.
6. Трансдукция эмбрионов и культивирование
Культивирование эмбрионов производили в CO2-инкубаторе при температуре 38.5°C в увлажненной атмосфере, содержащей 6.5%CO2, эмбрионы культивировали в среде BO-IVC medium (IVF Bioscience, UK), покрытой слоем парафинового масла (Sage, USA) для предотвращения испарения и изменения осмолярности среды. Культивирование эмбрионов осуществляли до стадии бластоцисты (170-180 часов культивирования) без смены среды.
Трансдукцию эмбрионов осуществляли с помощью аденоассоциированных вирусов. Для этого в среду для культивирования эмбрионов (в тот момент, когда эмбрионы находились на стадии зиготы) добавляли вирионы ААВ в концентрации 5*109 вирусных геномов в расчёте на 100 мкл культуральной среды.
7. Анализ генотипа эмбрионов. На 7 день после трансдукции эмбрионы отмывали от среды в TE-буфере (10 мМ Tris, 1 мМ ЭДТА), лизировали в 5 мкл буфера для лизиса бластоцист (0,1 М Tris-HCl, 0,1 M KCl, 0,02% Gelatin, 0,45% Tween-20, 0,125 mg Proteinase K) в течение 20 минут при температуре 56 градусов, а затем 10 минут при температуре 95 градусов. Используя лизат в качестве матрицы, амплифицировали целевой фрагмент с использованием 10 пМ праймеров SEQ ID NO: 3 и SEQ ID NO: 4 и Phire Tissue Direct PCR Master Mix (Thermofisher) по протоколу: денатурация 98°С - 30 сек; далее следовало 35 циклов: 98°С - 5 сек, 63°С - 5 сек; 72°С - 10 сек; и последний синтез 72°С - 1 мин. После прохождения реакции ПЦР-смесь очищали в 1,5% агарозном геле, полученный продукт выделяли набором Monarch DNA Gel Extraction Kit, секвенировали в компании Евроген и использованием праймера SEQ ID NO:4. Полученные сиквенсы можно подразделить на две категории: сиквенсы дикого типа, последовательность в которых соответствует дикому типу, и двоящиеся сиквенсы, демонстрирующие мозаицизм эмбриона (Фиг. 2).
Пример 1. Получение эмбрионов Bos taurus с редактированным геном CD209 в части клеток. По результатам секвенирования по Сэнгеру были обнаружены 3 эмбриона из 22 проверенных, содержащих инсерции и делеции в области PAM-сайта. Сиквенсы этих эмбрионов представлены на фигуре 2. Инсерции и делеции произошли на расстоянии в три нуклеотида от PAM сайта (самом вероятном месте разрезания SaCas9). В первом эмбрионе произошла делеция delTTGAACAG и инсерция insAT, во втором эмбрионе делеция delTTG и инсерция insA, в третьем эмбрионе делеция delGAACCAG и инсерция insATAG.
Список литературы:
1. Clark A.J. Genetic modification of milk proteins // Am. J. Clin. Nutr. 1996. Vol. 63, №4. P. 633-638.
2. Shepelev M.V., Kalinichenko S.V., Deykin A.V., Korobko I.V. Production of Recombinant Proteins in the Milk of Transgenic Animals: Current State and Prospects // Acta Naturae. 2018. Vol. 10, №3. P. 40-47.
3. Tabebordbar M., Zhu K., Cheng J.K.W., Chew W.L., Widrick J.J., Yan W.X., Maesner C., Wu E.Y., Xiao R., Ran F.A., Cong L., Zhang F., Vandenberghe L.H., Church G.M., Wagers A.J. In vivo gene editing in dystrophic mouse muscle and muscle stem cells // Science. 2016. Vol. 351, №6271. P. 407-411.
4. Bengtsson N.E., Hall J.K., Odom G.L., Phelps M.P., Andrus C.R., Hawkins R.D., Hauschka S.D., Chamberlain J.R., Chamberlain J.S. Muscle-specific CRISPR/Cas9 dystrophin gene editing ameliorates pathophysiology in a mouse model for Duchenne muscular dystrophy // Nat. Commun. 2017. Vol.8, №14454.
5. Xu K., Zhou Y., Mu Y., Liu Z., Hou S., Xiong Y., Fang L., Ge C., Wei Y., Zhang X., Xu C., Che J., Fan Z., Xiang G., Guo J., Shang H., Li H., Xiao S., Li J., Li K. CD163 and pAPNdouble-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance // Elife. 2020. Vol.9, №e57132.
6. Yuan M., Zhang J., Gao Y., Yuan Z., Zhu Z., Wei Y., Wu T., Han J., Zhang Y. HMEJ-based safe-harbor genome editing enables efficient generation of cattle with increased resistance to tuberculosis // J. Biol. Chem. 2021. Vol.296, №100497.
7. Anderson K.R., Haeussler M., Watanabe C., Janakiraman V., Lund J., Modrusan Z., Stinson J., Bei Q., Buechler A., Yu C., Thamminana S.R., Tam L., Sowick M.A., Alcantar T., O'Neil N., Li J., Ta L., Lima L., Roose-Girma M., Rairdan X., Durinck S., Warming S. CRISPR off-target analysis in genetically engineered rats and mice // Nat Methods. 2018. Vol.15. Р. 512–514.
8. Bruter A.V., Korshunova D.S., Kubekina M.V., Sergiev P.V., Kalinina A.A., Ilchuk L.A., Silaeva Y.Y., Korshunov E.N., Soldatov V.O., Deykin A.V. Novel transgenic mice with Cre-dependent co-expression of GFP and human ACE2: a safe tool for study of COVID-19 pathogenesis // Transgenic Res. 2021. Vol.30. Р. 289–301.
9. Chan A.W., Chong K.Y., Martinovich C., Simerly C., Schatten G. Transgenic monkeys produced by retroviral gene transfer into mature oocytes // Science. 2001. Vol.291. Р. 309–312.
10. Qiu P., Jiang J., Liu Z., Cai Y., Huang T., Wang Y., Liu Q., Nie Y., Liu F., Cheng J., Li Q., Tang Y.C., Poo M.M., Sun Q., Chang H.C. BMAL1 knockout macaque monkeys display reduced sleep and psychiatric disorders // Natl. Sci. Rev. 2019. Vol.6. Р. 87–100.
11. Sun Z., Wang M., Han S., Ma S., Zou Z., Ding F., Li X., Li L., Tang B., Wang H., Li N., Che H., Dai Y. Production of hypoallergenic milk from DNA-free beta-lactoglobulin (BLG) gene knockout cow using zinc-finger nucleases mRNA // Sci. Rep. 2018. Vol.8, №15430.
12. Proudfoot C., Carlson D.F., Huddart R., Long C.R., Pryor J.H., King T.J., Lillico S.G., Mileham A.J., McLaren D.G., Whitelaw C.B., Fahrenkrug S.C. Genome edited sheep and cattle // Transgenic Res. 2015. Vol.24. Р. 147–153.
13. Sumiyama K., Matsumoto N., Garçon-Yoshida J., Ukai H., Ueda H.R., Tanaka Y. Easy and efficient production of completely embryonic-stem-cell-derived mice using a micro-aggregation device // PloS. one. 2018. Vol.13.e0203056.
14. Tachibana M., Sparman M., Ramsey C., Ma H., Lee H.S., Penedo M.C., Mitalipov S. Generation of chimeric rhesus monkeys // Cell. 2012. Vol.148. Р. 285–295.
15. Whitworth K.M., Benne J.A., Spate L.D., Murphy S.L., Samuel M.S., Murphy C.N., Richt J.A., Walters E., Prather R.S., Wells K.D. Zygote injection of CRISPR/Cas9 RNA successfully modifies the target gene without delaying blastocyst development or altering the sex ratio in pigs // Transgenic Res. 2017. Vol.26.Р. 97–107.
16. Tan M., van Tol H., Mokry M., Stout T., Roelen B. Microinjection induces changes in the transcriptome of bovine oocytes // Sci. Rep. 2020. Vol.10, №11211.
17. Su X., Chen W., Cai Q., Liang P., Chen Y., Cong P., Huang J. Production of non-mosaic genome edited porcine embryos by injection of CRISPR/Cas9 into germinal vesicle oocytes // J. Genet. Genomics. 2019. Vol.46. Р. 335–342.
18. Brinster R.L., Chen H.Y., Warren R., Sarthy A., Palmiter R.D. Regulation of metallothionein - thymidine kinase fusion plasmids injected into mouse eggs // Nature. 1982. Vol.296. Р. 39–42.
19. Delerue F., Ittner L.M. Generation of Genetically Modified Mice through the Microinjection of Oocytes // Jo. V. E. 2017. Vol.124, №55765.
20. Palmiter R.D., Brinster R.L., Hammer R.E., Trumbauer M.E., Rosenfeld M.G., Birnberg N.C., Evans R.M. Dramatic growth of mice that develop from eggs microinjected with metallothionein-growth hormone fusion genes // Nature. 1982. Vol.300. Р. 611–615.
21. Hashimoto M., Takemoto T. Electroporation enables the efficient mRNA delivery into the mouse zygotes and facilitates CRISPR/Cas9-based genome editing // Sci. Rep. 2015. Vol.5, №11315.
22. Qin W., Dion S.L., Kutny P.M., Zhang Y., Cheng A.W., Jillette N.L., Malhotra A., Geurts A.M., Chen Y.G., Wang H. Efficient CRISPR/Cas9-Mediated Genome Editing in Mice by Zygote Electroporation of Nuclease // Genetics. 2015. Vol.200. Р. 423–430.
23. Chen S., Lee B., Lee A.Y., Modzelewski A.J., He L. Highly Efficient Mouse Genome Editing by CRISPR Ribonucleoprotein Electroporation of Zygotes // J. Biol. Chem. 2016. Vol.291. Р. 14457–14467.
24. Haskell R.E., Bowen R.A. Efficient production of transgenic cattle by retroviral infection of early embryos // Mol. Reprod. Dev. 1995. Vol.40. Р.386–390.
25. Xu Y.N., Uhm S.J., Koo B.C., Kwon M.S., Roh J.Y., Yang J.S., Choi H.Y., Heo Y.T., Cui X.S., Yoon J.H., Ko D.H., Kim T., Kim N.H. Production of transgenic Korean native cattle expressing enhanced green fluorescent protein using a FIV-based lentiviral vector injected into MII oocytes // J. Genet. Genomics. 2013. Vol.40. Р. 37–43.
26. Yoon Y., Wang D., Tai P., Riley J., Gao G., Rivera-Pérez J.A. Streamlined ex vivo and in vivo genome editing in mouse embryos using recombinant adeno-associated viruses // Nat. Commun. 2018. Vol.9, №412.
27. Mizuno N., Mizutani E., Sato H., Kasai M., Ogawa A., Suchy F., Yamaguchi T., Nakauchi, H. Intra-embryo Gene Cassette Knockin by CRISPR/Cas9-Mediated Genome Editing with Adeno-Associated Viral Vector // iScience. 2018. Vol. 9. Р. 286–297.
28. Stiehler M., Duch M., Mygind T., Li H., Ulrich-Vinther M., Modin C., Baatrup A., Lind M., Pedersen F.S., Bünger C.E. Optimizing viral and non-viral gene transfer methods for genetic modification of porcine mesenchymal stem cells // Adv. Exp. Med. Biol. 2006. Vol.585. Р. 31–48.
29. Zeng W., Tang L., Bondareva A., Honaramooz A., Tanco V., Dores C., Megee S., Modelski M., Rodriguez-Sosa J.R., Paczkowski M., Silva E., Wheeler M., Krisher R.L., Dobrinski I. Viral transduction of male germline stem cells results in transgene transmission after germ cell transplantation in pigs // Biol. Reprod. 2013. Vol. 88, №27.
30. Chan K.Y., Jang M.J., Yoo B.B., Greenbaum A., Ravi N., Wu W.L., Sánchez-Guardado L., Lois C., Mazmanian S.K., Deverman B.E., Gradinaru V. Engineered AAVs for efficient noninvasive gene delivery to the central and peripheral nervous systems // Nat. Neurosci. 2017. Vol.20. Р. 1172–1179.
31. Naso M.F., Tomkowicz B., Perry W.L. 3rd, Strohl W.R. Adeno-Associated Virus (AAV) as a Vector for Gene Therapy // BioDrugs. 2017. Vol.31. Р. 317–334.
32. Wu Z., Yang H., Colosi P. Effect of genome size on AAV vector packaging // Mol. Ther. 2010. Vol. 18. Р. 80–86.
33. Dismuke D.J., Tenenbaum L., Samulski R.J. Biosafety of recombinant adeno-associated virus vectors // Curr. Gene Ther. 2013. Vol. 13. Р. 434–452.
34. McGreal E.P., Miller J.L., Gordon S. Ligand recognition by antigen-presenting cell C-type lectin receptors // Curr. Opin. Immunol. 2005. Vol. 17. Р. 18–24.
35. Geijtenbeek T.B., Kwon D.S., Torensma R., van Vliet S.J., van Duijnhoven G.C., Middel J., Cornelissen I.L., Nottet H.S., KewalRamani V.N., Littman D.R., Figdor C.G., van Kooyk Y. DC-SIGN, a dendritic cell-specific HIV-1-binding protein that enhances trans-infection of T cells // Cell. 2000. Vol. 100. Р. 587–597.
36. Ludwig I.S., Lekkerkerker A.N., Depla E., Bosman F., Musters R.J., Depraetere S., van Kooyk Y., Geijtenbeek T.B. Hepatitis C virus targets DC-SIGN and L-SIGN to escape lysosomal degradation // J. Virol. 2004. Vol. 78. Р. 8322–8332.
37. Geijtenbeek T.B., Van Vliet S.J., Koppel E.A., Sanchez-Hernandez M., Vandenbroucke-Grauls C.M., Appelmelk B., Van Kooyk Y. Mycobacteria target DC-SIGN to suppress dendritic cell function // J. Exp. Med. 2003. Vol. 197. Р.7–17.
38. Tailleux L., Pham-Thi N., Bergeron-Lafaurie A., Herrmann J.L., Charles P., Schwartz O., Scheinmann P., Lagrange P.H., de Blic J., Tazi A., Gicquel B., Neyrolles O. DC-SIGN induction in alveolar macrophages defines privileged target host cells for mycobacteria in patients with tuberculosis // PLoS. Med. 2005.Vol. 2, №e381.
39. Caparrós E., Serrano D., Puig-Kröger A., Riol L., Lasala F., Martinez I., Vidal-Vanaclocha F., Delgado R., Rodríguez-Fernández J.L., Rivas L., Corbí A.L., Colmenares M.. Role of the C-type lectins DC-SIGN and L-SIGN in Leishmania interaction with host phagocytes // Immunobiology. 2005. Vol. 210. Р. 185–193.
40. Rahimi N. C-type Lectin CD209L/L-SIGN and CD209/DC-SIGN: Cell Adhesion Molecules Turned to Pathogen Recognition Receptors // Biology. 2020. Vol. 10, №1.
41. Kwon D.S., Gregorio G., Bitton N., Hendrickson W.A., Littman D.R. DC-SIGN-mediated internalization of HIV is required for trans-enhancement of T cell infection // Immunity. 2002. Vol.16. Р. 135–144.
42. Miyauchi K., Kim Y., Latinovic O., Morozov V., Melikyan G.B. HIV enters cells via endocytosis and dynamin-dependent fusion with endosomes // Cell. 2009. Vol. 137. P. 433–444.
43. Njiri O.A., Zhang X., Zhang Y., Wu B., Jiang L., Li Q., Liu W., Chen T. CD209 C-Type Lectins Promote Host Invasion, Dissemination, and Infection of Toxoplasma gondii // Front Immunol. 2020.Vol.11, №656.
44. Kumar S., Kumar S., Singh R.V., Chauhan A., Kumar A., Bharati J., Singh S.V. Association of genetic variability in CD209 gene with bovine paratuberculosis disease: a case-control study in the Indian cattle population // Anim. Biotechnol. 2020. Vol.28. Р.1–8.
45. Gopi B., Singh R.V., Kumar S., Kumar S., Chauhan A., Kumar A., Singh S.V. Single-nucleotide polymorphisms in CLEC7A, CD209 and TLR4 gene and their association with susceptibility to paratuberculosis in Indian cattle // J. Genet. 2020. Vol.99, №14.
46. Canive M., Casais R., Jimenez J.A., Blanco-Vazquez C., Amado J., Garrido J.M., Juste R.A., Alonso-Hearn M. Correlations between single nucleotide polymorphisms in bovine CD209, SLC11A1, SP110 and TLR2 genes and estimated breeding values for several traits in Spanish Holstein cattle // Heliyon. 2020. Vol.6, №e04254.
47. Günther P.S., Mikeler E., Hamprecht K., Schneider-Schaulies J., Jahn G., Dennehy K.M. CD209/DC-SIGN mediates efficient infection of monocyte-derived dendritic cells by clinical adenovirus 2C isolates in the presence of bovine lactoferrin // J. Gen. Virol. 2011. Vol.92. Р.1754–1759.
48. Danilov K.A., Vassilieva S.G., Polikarpova A.V., Starikova A.V., Shmidt A.A., Galkin I.I., Tsitrina A.A., Egorova T.V., Orlov S.N., Kotelevtsev Y.V. In vitro assay for the efficacy assessment of AAV vectors expressing microdystrophin // Exp. Cell Res. 2020. Vol.392, №112033.
49. Buclez P.O., Dias Florencio G., Relizani K., Beley C., Garcia L., Benchaouir R.. Rapid, scalable, and low-cost purification of recombinant adeno-associated virus produced by baculovirus expression vector system // Mol. Ther. Methods. Clin. Dev. 2016. Vol.3, №16035.
50. Zolotukhin S., Byrne B.J., Mason E., Zolotukhin I., Potter M., Chesnut K., Summerford C., Samulski R.J., Muzyczka N. Recombinant adeno-associated virus purification using novel methods improves infectious titer and yield // Gene Ther. 1999. Vol.6. Р.973–985.
51. Aurnhammer C., Haase M., Muether N., Hausl M., Rauschhuber C., Huber I., Nitschko H., Busch U., Sing A., Ehrhardt A., Baiker A. Universal real-time PCR for the detection and quantification of adeno-associated virus serotype 2-derived inverted terminal repeat sequences // Hum. Gene Ther. Methods. 2012. Vol.23. Р.18–28.
--->
Перечень последовательностей
DTD Version: V1_3
File Name: CD209 в эмбрионах Bos taurus .xml
Software Name: WIPO Sequence
Software Version: 2.1.2
Production Date: 2022-11-03
General Information:
Current application / IP Office: RU
Current application / Applicant file reference: non
Applicant name: Федеральное государственное бюджетное учреждение
науки Институт биологии гена Российской академии наук
Applicant name / Language: ru
Applicant name / Name Latin: Institute of Gene Biology Russian
Academy of Sciences
Invention title: Способ получения нокаута гена CD209 в эмбрионах Bos
taurus путем трансдукции зигот аденоассоциированными вирусами,
кодирующими saCas9 и соответствующую гидовую РНК ( ru )
Sequence Total Quantity: 7
Sequences:
Sequence Number (ID): 1
Length: 7446
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..7446
> mol_type, other DNA
> organism, synthetic construct
Residues:
cctgcaggca gctgcgcgct cgctcgctca ctgaggccgc ccgggcgtcg
ggcgaccttt 60
ggtcgcccgg cctcagtgag cgagcgagcg cgcagagagg gagtggccaa
ctccatcact 120
aggggttcct gcggcctcta gactcgaggc gttgacattg attattgact
agttattaat 180
agtaatcaat tacggggtca ttagttcata gcccatatat ggagttccgc
gttacataac 240
ttacggtaaa tggcccgcct ggctgaccgc ccaacgaccc ccgcccattg
acgtcaataa 300
tgacgtatgt tcccatagta acgccaatag ggactttcca ttgacgtcaa
tgggtggagt 360
atttacggta aactgcccac ttggcagtac atcaagtgta tcatatgcca
agtacgcccc 420
ctattgacgt caatgacggt aaatggcccg cctggcatta tgcccagtac
atgaccttat 480
gggactttcc tacttggcag tacatctacg tattagtcat cgctattacc
atggtgatgc 540
ggttttggca gtacatcaat gggcgtggat agcggtttga ctcacgggga
tttccaagtc 600
tccaccccat tgacgtcaat gggagtttgt tttggcacca aaatcaacgg
gactttccaa 660
aatgtcgtaa caactccgcc ccattgacgc aaatgggcgg taggcgtgta
cggtgggagg 720
tctatataag cagagctctc tggctaacta ccggtgccac catggcccca
aagaagaagc 780
ggaaggtcgg tatccacgga gtcccagcag ccaagcggaa ctacatcctg
ggcctggaca 840
tcggcatcac cagcgtgggc tacggcatca tcgactacga gacacgggac
gtgatcgatg 900
ccggcgtgcg gctgttcaaa gaggccaacg tggaaaacaa cgagggcagg
cggagcaaga 960
gaggcgccag aaggctgaag cggcggaggc ggcatagaat ccagagagtg
aagaagctgc 1020
tgttcgacta caacctgctg accgaccaca gcgagctgag cggcatcaac
ccctacgagg 1080
ccagagtgaa gggcctgagc cagaagctga gcgaggaaga gttctctgcc
gccctgctgc 1140
acctggccaa gagaagaggc gtgcacaacg tgaacgaggt ggaagaggac
accggcaacg 1200
agctgtccac caaagagcag atcagccgga acagcaaggc cctggaagag
aaatacgtgg 1260
ccgaactgca gctggaacgg ctgaagaaag acggcgaagt gcggggcagc
atcaacagat 1320
tcaagaccag cgactacgtg aaagaagcca aacagctgct gaaggtgcag
aaggcctacc 1380
accagctgga ccagagcttc atcgacacct acatcgacct gctggaaacc
cggcggacct 1440
actatgaggg acctggcgag ggcagcccct tcggctggaa ggacatcaaa
gaatggtacg 1500
agatgctgat gggccactgc acctacttcc ccgaggaact gcggagcgtg
aagtacgcct 1560
acaacgccga cctgtacaac gccctgaacg acctgaacaa tctcgtgatc
accagggacg 1620
agaacgagaa gctggaatat tacgagaagt tccagatcat cgagaacgtg
ttcaagcaga 1680
agaagaagcc caccctgaag cagatcgcca aagaaatcct cgtgaacgaa
gaggatatta 1740
agggctacag agtgaccagc accggcaagc ccgagttcac caacctgaag
gtgtaccacg 1800
acatcaagga cattaccgcc cggaaagaga ttattgagaa cgccgagctg
ctggatcaga 1860
ttgccaagat cctgaccatc taccagagca gcgaggacat ccaggaagaa
ctgaccaatc 1920
tgaactccga gctgacccag gaagagatcg agcagatctc taatctgaag
ggctataccg 1980
gcacccacaa cctgagcctg aaggccatca acctgatcct ggacgagctg
tggcacacca 2040
acgacaacca gatcgctatc ttcaaccggc tgaagctggt gcccaagaag
gtggacctgt 2100
cccagcagaa agagatcccc accaccctgg tggacgactt catcctgagc
cccgtcgtga 2160
agagaagctt catccagagc atcaaagtga tcaacgccat catcaagaag
tacggcctgc 2220
ccaacgacat cattatcgag ctggcccgcg agaagaactc caaggacgcc
cagaaaatga 2280
tcaacgagat gcagaagcgg aaccggcaga ccaacgagcg gatcgaggaa
atcatccgga 2340
ccaccggcaa agagaacgcc aagtacctga tcgagaagat caagctgcac
gacatgcagg 2400
aaggcaagtg cctgtacagc ctggaagcca tccctctgga agatctgctg
aacaacccct 2460
tcaactatga ggtggaccac atcatcccca gaagcgtgtc cttcgacaac
agcttcaaca 2520
acaaggtgct cgtgaagcag gaagaaaaca gcaagaaggg caaccggacc
ccattccagt 2580
acctgagcag cagcgacagc aagatcagct acgaaacctt caagaagcac
atcctgaatc 2640
tggccaaggg caagggcaga atcagcaaga ccaagaaaga gtatctgctg
gaagaacggg 2700
acatcaacag gttctccgtg cagaaagact tcatcaaccg gaacctggtg
gataccagat 2760
acgccaccag aggcctgatg aacctgctgc ggagctactt cagagtgaac
aacctggacg 2820
tgaaagtgaa gtccatcaat ggcggcttca ccagctttct gcggcggaag
tggaagttta 2880
agaaagagcg gaacaagggg tacaagcacc acgccgagga cgccctgatc
attgccaacg 2940
ccgatttcat cttcaaagag tggaagaaac tggacaaggc caaaaaagtg
atggaaaacc 3000
agatgttcga ggaaaagcag gccgagagca tgcccgagat cgaaaccgag
caggagtaca 3060
aagagatctt catcaccccc caccagatca agcacattaa ggacttcaag
gactacaagt 3120
acagccaccg ggtggacaag aagcctaata gagagctgat taacgacacc
ctgtactcca 3180
cccggaagga cgacaagggc aacaccctga tcgtgaacaa tctgaacggc
ctgtacgaca 3240
aggacaatga caagctgaaa aagctgatca acaagagccc cgaaaagctg
ctgatgtacc 3300
accacgaccc ccagacctac cagaaactga agctgattat ggaacagtac
ggcgacgaga 3360
agaatcccct gtacaagtac tacgaggaaa ccgggaacta cctgaccaag
tactccaaaa 3420
aggacaacgg ccccgtgatc aagaagatta agtattacgg caacaaactg
aacgcccatc 3480
tggacatcac cgacgactac cccaacagca gaaacaaggt cgtgaagctg
tccctgaagc 3540
cctacagatt cgacgtgtac ctggacaatg gcgtgtacaa gttcgtgacc
gtgaagaatc 3600
tggatgtgat caaaaaagaa aactactacg aagtgaatag caagtgctat
gaggaagcta 3660
agaagctgaa gaagatcagc aaccaggccg agtttatcgc ctccttctac
aacaacgatc 3720
tgatcaagat caacggcgag ctgtatagag tgatcggcgt gaacaacgac
ctgctgaacc 3780
ggatcgaagt gaacatgatc gacatcacct accgcgagta cctggaaaac
atgaacgaca 3840
agaggccccc caggatcatt aagacaatcg cctccaagac ccagagcatt
aagaagtaca 3900
gcacagacat tctgggcaac ctgtatgaag tgaaatctaa gaagcaccct
cagatcatca 3960
aaaagggcaa aaggccggcg gccacgaaaa aggccggcca ggcaaaaaag
aaaaagggat 4020
cctacccata cgatgttcca gattacgctt acccatacga tgttccagat
tacgcttacc 4080
catacgatgt tccagattac gcttaagaat tcctagagct cgctgatcag
cctcgactgt 4140
gccttctagt tgccagccat ctgttgtttg cccctccccc gtgccttcct
tgaccctgga 4200
aggtgccact cccactgtcc tttcctaata aaatgaggaa attgcatcgc
attgtctgag 4260
taggtgtcat tctattctgg ggggtggggt ggggcaggac agcaaggggg
aggattggga 4320
agagaatagc aggcatgctg gggaggtacc gagggcctat ttcccatgat
tccttcatat 4380
ttgcatatac gatacaaggc tgttagagag ataattggaa ttaatttgac
tgtaaacaca 4440
aagatattag tacaaaatac gtgacgtaga aagtaataat ttcttgggta
gtttgcagtt 4500
ttaaaattat gttttaaaat ggactatcat atgcttaccg taacttgaaa
gtatttcgat 4560
ttcttggctt tatatatctt gtggaaagga cgaaacaccg gagaccacgg
caggtctcag 4620
ttttagtact ctggaaacag aatctactaa aacaaggcaa aatgccgtgt
ttatctcgtc 4680
aacttgttgg cgagattttt gcggccgcag gaacccctag tgatggagtt
ggccactccc 4740
tctctgcgcg ctcgctcgct cactgaggcc gggcgaccaa aggtcgcccg
acgcccgggc 4800
tttgcccggg cggcctcagt gagcgagcga gcgcgcagct gcctgcaggg
gcgcctgatg 4860
cggtattttc tccttacgca tctgtgcggt atttcacacc gcatacgtca
aagcaaccat 4920
agtacgcgcc ctgtagcggc gcattaagcg cggcgggtgt ggtggttacg
cgcagcgtga 4980
ccgctacact tgccagcgcc ttagcgcccg ctcctttcgc tttcttccct
tcctttctcg 5040
ccacgttcgc cggctttccc cgtcaagctc taaatcgggg gctcccttta
gggttccgat 5100
ttagtgcttt acggcacctc gaccccaaaa aacttgattt gggtgatggt
tcacgtagtg 5160
ggccatcgcc ctgatagacg gtttttcgcc ctttgacgtt ggagtccacg
ttctttaata 5220
gtggactctt gttccaaact ggaacaacac tcaactctat ctcgggctat
tcttttgatt 5280
tataagggat tttgccgatt tcggtctatt ggttaaaaaa tgagctgatt
taacaaaaat 5340
ttaacgcgaa ttttaacaaa atattaacgt ttacaatttt atggtgcact
ctcagtacaa 5400
tctgctctga tgccgcatag ttaagccagc cccgacaccc gccaacaccc
gctgacgcgc 5460
cctgacgggc ttgtctgctc ccggcatccg cttacagaca agctgtgacc
gtctccggga 5520
gctgcatgtg tcagaggttt tcaccgtcat caccgaaacg cgcgagacga
aagggcctcg 5580
tgatacgcct atttttatag gttaatgtca tgataataat ggtttcttag
acgtcaggtg 5640
gcacttttcg gggaaatgtg cgcggaaccc ctatttgttt atttttctaa
atacattcaa 5700
atatgtatcc gctcatgaga caataaccct gataaatgct tcaataatat
tgaaaaagga 5760
agagtatgag tattcaacat ttccgtgtcg cccttattcc cttttttgcg
gcattttgcc 5820
ttcctgtttt tgctcaccca gaaacgctgg tgaaagtaaa agatgctgaa
gatcagttgg 5880
gtgcacgagt gggttacatc gaactggatc tcaacagcgg taagatcctt
gagagttttc 5940
gccccgaaga acgttttcca atgatgagca cttttaaagt tctgctatgt
ggcgcggtat 6000
tatcccgtat tgacgccggg caagagcaac tcggtcgccg catacactat
tctcagaatg 6060
acttggttga gtactcacca gtcacagaaa agcatcttac ggatggcatg
acagtaagag 6120
aattatgcag tgctgccata accatgagtg ataacactgc ggccaactta
cttctgacaa 6180
cgatcggagg accgaaggag ctaaccgctt ttttgcacaa catgggggat
catgtaactc 6240
gccttgatcg ttgggaaccg gagctgaatg aagccatacc aaacgacgag
cgtgacacca 6300
cgatgcctgt agcaatggca acaacgttgc gcaaactatt aactggcgaa
ctacttactc 6360
tagcttcccg gcaacaatta atagactgga tggaggcgga taaagttgca
ggaccacttc 6420
tgcgctcggc ccttccggct ggctggttta ttgctgataa atctggagcc
ggtgagcgtg 6480
gaagccgcgg tatcattgca gcactggggc cagatggtaa gccctcccgt
atcgtagtta 6540
tctacacgac ggggagtcag gcaactatgg atgaacgaaa tagacagatc
gctgagatag 6600
gtgcctcact gattaagcat tggtaactgt cagaccaagt ttactcatat
atactttaga 6660
ttgatttaaa acttcatttt taatttaaaa ggatctaggt gaagatcctt
tttgataatc 6720
tcatgaccaa aatcccttaa cgtgagtttt cgttccactg agcgtcagac
cccgtagaaa 6780
agatcaaagg atcttcttga gatccttttt ttctgcgcgt aatctgctgc
ttgcaaacaa 6840
aaaaaccacc gctaccagcg gtggtttgtt tgccggatca agagctacca
actctttttc 6900
cgaaggtaac tggcttcagc agagcgcaga taccaaatac tgttcttcta
gtgtagccgt 6960
agttaggcca ccacttcaag aactctgtag caccgcctac atacctcgct
ctgctaatcc 7020
tgttaccagt ggctgctgcc agtggcgata agtcgtgtct taccgggttg
gactcaagac 7080
gatagttacc ggataaggcg cagcggtcgg gctgaacggg gggttcgtgc
acacagccca 7140
gcttggagcg aacgacctac accgaactga gatacctaca gcgtgagcta
tgagaaagcg 7200
ccacgcttcc cgaagggaga aaggcggaca ggtatccggt aagcggcagg
gtcggaacag 7260
gagagcgcac gagggagctt ccagggggaa acgcctggta tctttatagt
cctgtcgggt 7320
ttcgccacct ctgacttgag cgtcgatttt tgtgatgctc gtcagggggg
cggagcctat 7380
ggaaaaacgc cagcaacgcg gcctttttac ggttcctggc cttttgctgg
ccttttgctc 7440
acatgt
7446
Sequence Number (ID): 2
Length: 26
Molecule Type: RNA
Features Location/Qualifiers:
- source, 1..26
> mol_type, other RNA
> organism, Bos taurus
Residues:
gatgctcact caccttgaac cagagt
26
Sequence Number (ID): 3
Length: 22
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..22
> mol_type, other DNA
> organism, synthetic construct
Residues:
tgaacacaag gagccagatg ac
22
Sequence Number (ID): 4
Length: 20
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..20
> mol_type, other DNA
> organism, synthetic construct
Residues:
gcactgaccc tgagccaaat
20
Sequence Number (ID): 5
Length: 25
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..25
> mol_type, other DNA
> organism, synthetic construct
Residues:
caccgcctca aactgtgtag tggtc
25
Sequence Number (ID): 6
Length: 25
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..25
> mol_type, other DNA
> organism, synthetic construct
Residues:
aaacgaccac tacacagttt gaggc
25
Sequence Number (ID): 7
Length: 21
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..21
> mol_type, other DNA
> organism, synthetic construct
Residues:
gagggcctat ttcccatgat t
21
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ получения генно-модифицированных кроликов с нокаутом гена LEPR с помощью системы CRISPR/Cas9 | 2023 |
|
RU2836438C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ усиления направленного редактирования гена CXCR4 в CD4-лимфоцитах человека в целях создания резистентности клеток к ВИЧ-1 | 2021 |
|
RU2779300C1 |
| Способ получения генно-модифицированных лабораторных животных с нуль-аллелем гена P2rx3 | 2022 |
|
RU2805173C1 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМНОЙ ДНК ЭУКАРИОТИЧЕСКОЙ КЛЕТКИ НА ОСНОВЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩЕЙ БЕЛОК SUCAS9NLS | 2022 |
|
RU2804422C1 |
| Средство разрезания ДНК на основе Cas9 белка из биотехнологически значимой бактерии Clostridium cellulolyticum | 2018 |
|
RU2712497C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Streptococcus uberis NCTC3858 | 2022 |
|
RU2788197C1 |
| Способ редактирования гена GJB2 для исправления патогенного варианта c.del35G в клетках человека, культивируемых in vitro | 2021 |
|
RU2780677C1 |
| Способ получения генно-модифицированных мышей, экспрессирующих миниген антитромбина III человека, с помощью микроинъекций TelN-линеаризованного фрагмента ДНК | 2022 |
|
RU2806568C1 |
| Средство обнаружения нуклеиновых кислот на основе ScCas12a белка из бактерии Sedimentisphaera cyanobacteriorum | 2023 |
|
RU2820345C1 |

Настоящее изобретение относится к области биотехнологии и включает в себя плазмиду, полученную на основе вектора px601 и кодирующую белок SaCas9 и гидовую РНК против фрагмента последовательности 3 экзона гена Bos taurus CD209, предназначенную для сборки аденоассоциированных вирусов, а также способ получения эмбрионов Bos taurus и их последующей трансдукции полученными аденоассоциированными вирусами для получения мозаичных нокаутов по гену CD209. 2 н.п. ф-лы, 2 ил., 1 пр.
1. Плазмидная конструкция для сборки аденоассоциированных вирусов, полученная на основе вектора px601, имеющего последовательность SEQ ID NO: 1, кодирующая ОРС гена saCas9 под контролем CAG промотора и гидовую РНК последовательностью SEQ ID NO: 2 против 3 экзона гена Bos taurus гена CD209 под контролем U6 промотора.
2. Способ получения эмбрионов Bos taurus, несущих нуль-аллель гена CD209, характеризующийся следующими стадиями:
a) трансфекция клеток HEK293, включающая сборку с помощью плазмидной конструкции по п. 1 в клетках упаковочной линии HEK293T стока аденоассоциированных вирусов, кодирующих ОРС saCas9 и гидовую РНК;
b) in vitro оплодотворение яйцеклеток Bos taurus сперматозоидами Bos taurus;
c) инфекция эмбрионов аденоассоциированными вирусами из пункта (а);
d) проведение анализа наличия сдвига рамки считывания в гене CD209 в бластоцистах, путем амплификации целевого участка гена CD209 с праймерами cd209-check-f последовательностью SEQ ID NO: 3 и cd209-check-r последовательностью SEQ ID NO: 4 с последующим секвенированием фрагмента с использованием праймера cd209-check-r последовательностью SEQ ID NO: 4.
| DEYKIN A.V | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| US 20210181215 A1, 17.06.2021 | |||
| OWEN J.R | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
Авторы
Даты
2023-08-01—Публикация
2022-11-30—Подача