В открытых базах данных не было обнаружено российских патентов, посвященных ПЦР-диагностике вируса ямчатости древесины яблони. Единственным обнаруженным патентом является RU2384045C2, предметом которого является разработка способа оценки растений яблони на вирусную инфекцию с использованием оптических характеристик листьев – степени когерентности светорассеяния и оценки изменения оптической плотности листа. Данный подход предполагает выявление заражённости ASPV, в том числе при латентном протекании инфекции. Однако, эффективность предполагаемого подхода в значительной степени зависит от квалификации персонала, а также использование специализированного оборудования.
Было обнаружено несколько патентов Китайской Народной Республики, предметом которых являются методы ПЦР-диагностики ASPV.
CN103382508A (прекратил свое действие, но может быть восстановлен). Предметом патентования являются 4 тест-системы на 3 ключевых вируса (в т.ч. ASPV) с использованием мультиплексной ПЦР с обратной транскрипцией. При этом, способом детекции в предлагаемом подходе является гель-электрофорез, использование которого сопряжено с высокими рисками контаминации рабочего пространства продуктами амплификации, приводящей, в конечном итоге к получению ложноположительных результатов. Также проблемой мультиплексного подхода является возможное конкурентное ингибирование параллельных реакций, особенно в случаях, когда генетический материал патогена находится в низкой концентрации. Кроме того, не вполне понятна чувствительность разработанных систем.
CN111057794A (прекратил свое действие, но может быть восстановлен). Предмет патентования – несколько праймерных систем для выявления ключевых вирусных патогенов яблони, в т.ч. ASPV. Однако и в данном случае детекция осуществляется исключительно электрофоретическим методом.
CN104561387A (действующий). Предмет патентования – система выявления вируса ямчатости яблони с помощью флуоресцентной ПЦР с использованием гидролизуемых (TaqMan) зондов. Предложенный подход позволяет избежать риска контаминации и увеличить скорость проведения анализа. Заявленная чувствительность тест-системы – 100 копий/реакцию.
Литературные данные свидетельствуют о том, что для диагностики ASPV используются, главным образом, иммунохимические методы (ELISA) и различные подходы, основанные на амплификации нуклеиновых кислот. Иммунохимические методы [1-3] просты в исполнении, однако не обладают достаточной чувствительностью. В ряде ранних работ, посвященных ПЦР-детекции ASPV [4-7], детекция результатов анализа осуществлялась с помощью гель-электрофореза, что влекло высокие риски контаминации рабочей зоны продуктами амплификации, и, как следствие, получения ложноположительных результатов. Помимо классических вариантов ПЦР, для диагностики ASPV используются также изотермические методы амплификации нуклеиновых кислот. Lu et al., [8], предложили способ детекции ASPV на основе петлевой изотермической амплификации (LAMP), сопряжённой с обратной транскрипцией. Согласно данным авторов, метод обладает высокой чувствительностью, на 2-3 порядка превышающей стандартную ПЦР. Однако при этом необходимо отметить, что сравнение проводилось с тест-системами, характеризующимися не самыми высокими показателями чувствительности; кроме того, проблемой изотермических методов, в т.ч. LAMP, является тот факт, что в реакции используется 6-8 праймеров. Из-за высокой полиморфности вирусов яблони, необходимость использования такого количества олигонуклеотидов может вызвать проблемы со специфичностью тест-систем.
Тест-системой с наиболее оптимальными характеристиками, и наиболее близкой к разработке, описываемой в настоящем патенте, является предложенный Nickel and Fajardo [9] предложили метод мультиплексной ПЦР с использованием гидролизуемых (TaqMan) зондов для определения трех вирусов яблони в одной пробе. Как было упомянуто выше, ограничением мультиплексной ПЦР может быть ингибирование одной или нескольких параллельных реакций амплификации. Кроме того, авторы не дают точной информации о чувствительности тест-системы: предел обнаружения составил 2 фг тотальной кДНК, однако данных по минимально детектируемому количеству геном-эквивалентов на реакцию не приводится.
Список иллюстраций
Рис. 1. Выравнивание фрагментов генов белка оболочки ASPV и близкородственных вирусов, использованных для подбора прямого (ASPVF) и обратного (ASPVR) праймеров. Последовательности праймеров подчёкнуты.
Рис. 2. Карта вектора pAL2T, использованного для клонирования положительных контролей.
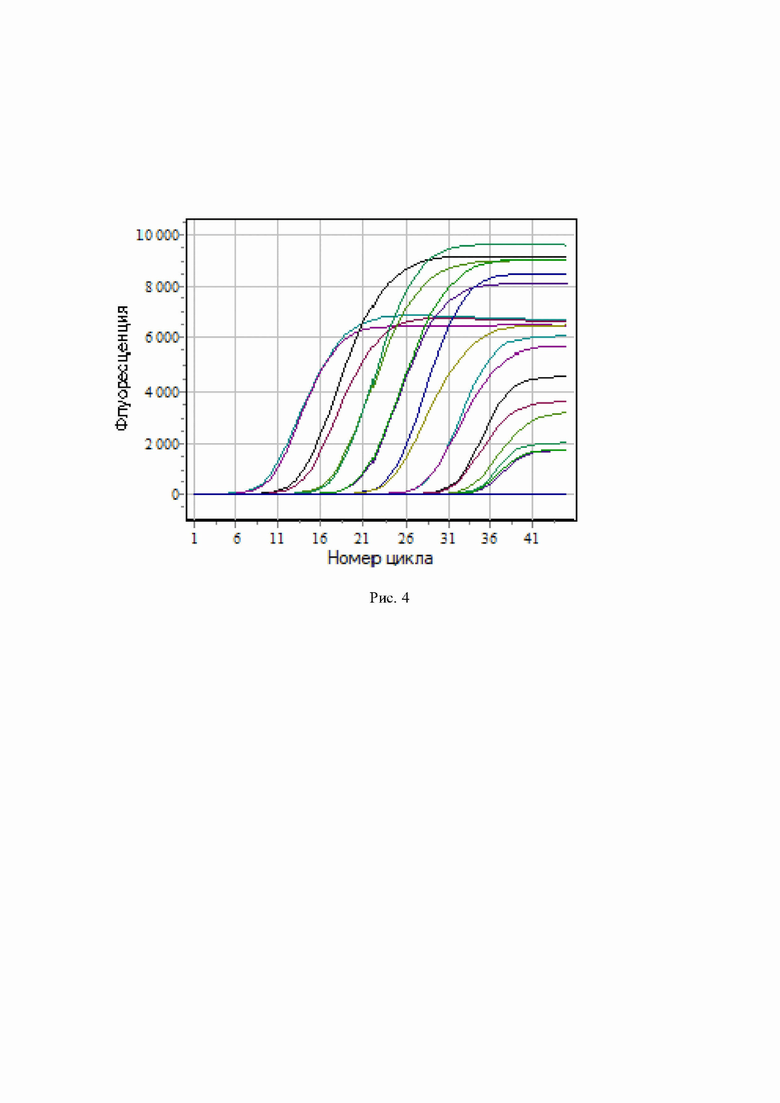
Рис. 3. Результаты ПЦР последовательных десятикратных разведений плазмиды, содержащей специфический продукт амплификации. По оси абсцисс показано логарифмические значения концентраций (геном-эквиваленты/реакцию), по оси ординат – значения соответствующих пороговых циклов.
Рис. 4. Флуоресцентный сигнал, детектируемый при ПЦР-анализе последовательных десятикратных разведений плазмид, содержащих специфический ампликон, с парой праймеров ASPVF-ASPVR и зондом ASPVT.
Техническим результатом является разработка тест-системы для проведения высокоспецифичной идентификации кДНК, с высокой чувствительностью для обнаружения вируса ямчатости древесины яблони ASPV методом ПЦР в реальном времени.
Предлагаемое техническое решение позволяет повысить чувствительность детекции, а также избежать проблем, связанных с контаминацией рабочего пространства продуктами амплификации.
Дизайн праймеров проводили с помощью выравнивания последовательностей нуклеотидов, депонированных в базе данных NCBI c использованием алгоритма ClustalW. Помимо последовательностей нуклеотидов ASPV, для выравнивания использовались последовательности нуклеотидов близкородственных видов. Наиболее вариабельным и пригодным для подбора праймеров был фрагмент гена белка оболочки. На рис. 1 приведены выравнивания фрагментов генов белка оболочки ASPV и близкородственных вирусов, использованных для подбора праймеров.
В результате проведённого биоинформатического анализа был проведён дизайн олигонуклеотидов: прямого праймера (Seq1), обратного праймера (Seq2) и флуоресцентно-меченого зонда (Seq3).
Оценку специфичности тест-системы проводили с использованием заражённых образцов листьев и коры яблони, предоставленных сотрудниками Ставропольского государственного аграрного университета. Диагностика проводилась визуально с использование контрольных (не заражённых) растений в качестве референсов.
Ниже приведены результаты ПЦР-анализа 18 образцов растений яблони.
Табл. 1. Результаты ПЦР-анализа специфичности используемых систем.
Продукты ПЦР, полученные в результате амплификации фрагмента ДНК «положительных» образцов были проклонированы в плазмидный вектор pAL2T (схема вектора приведена на рис. 2). Соответствие проклонированной последовательности фрагменту генома ASPV подтверждали с помощью секвенирования по методу Сэнгера. Для вектора, содержащего вставку специфического продукта (pAL2T-ASPV, Seq4), было проведено определение концентрации с использованием флуориметра QUBIT. Плазмида pAL2T-ASPV затем использовалась в качестве положительного контрольного образца.
Чувствительность тест-системы определяли с использованием последовательных десятикратных разведений плазмиды pAL2T-ASPV.
Табл. 2. Определение чувствительности тест-системы
Зависимость сигнала ПЦР (пороговый цикл) от концентрации ДНК в реакционной пробирке, выраженной в количестве геном-эквивалентов, приведена на рис. 3. На рис. 4 показаны уровни флуоресцентного сигнала при ПЦР последовательных десятикратных разведений положительного контрольного образца.
Тест-системы для определения РНК вируса ямчатости древесины яблони используются следующим образом.
Предварительно выделяют РНК из биологического материала любым подходящим методом. В настоящей работе использовался подход, основанный на использовании реагента TRIZOL с последующей очисткой смесью фенол:хлороформ и переосаждением этиловым спиртом.
Проводится реакция обратной транскрипции, для чего может быть использован термостат, либо ПЦР-амплификатор.
Для проведения реакции используют готовую смесь для амплификации, состоящую из буфера, дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров, флуоресцентно-меченого зонда, Taq-полимеразы. Готовую реакционную смесь смешивают с образцами кДНК, положительным контрольным образцом и отрицательным контрольным образцом. ПЦР проводили в амплификаторе ДТ96 по следующей программе амплификации с детекцией в каналах FAM и HEX: 94°C – 90 с 1 цикл; 94°C – 30 с, 60°С – 30 с – 5 циклов; 94°С – 10 с, 60°С – 30 с – 45 циклов.
Результат, для конкретного образца, считается положительным образца при превышении порового уровня флуоресценции в канале FAM выше определённого уровня. При этом. Пороговый уровень флуоресценции определяется программным обеспечением оборудования.
Для апробации разработанной тест-системы сотрудниками Ставропольского государственного аграрного университета было передано 6 образцов листьев яблони (вместе с черешками), выделенных в различных хозяйствах в разных районах Ставропольского края. Для проведения анализа на первом этапе проводилось выделение РНК. Для этого зелёную массу листьев гомогенизировали в 1 мл реагента TRIZOL (Ambion, Германия), после чего к гомогенату добавляли 0.3 мл хлороформа, центрифугировали 10 мин при 13400 об/мин, к верхней фазе добавляли 500 мкл изпропанола, инкубировали 10 мин во льду. После чего повторно центрифугировали 10 мин при 13400 об/мин, удаляли надосадочную жидкость, осадок промывали 75 % этанолом, после чего растворяли в 50 мкл деионизированной воды. Полученные РНК использовали для обратной транскрипции, которую проводили с использованием набора MMLV RT kit (Евроген, Россия) по протоколу производителя. Полученные кДНК использовали в качестве матриц в ПЦР в реальном времени с использованием разработанных специфических праймеров и зонда. ПЦР и анализ результатов проводили по протоколам, приведённым выше. В таблице 3 приведены результаты ПЦР-анализа.
Табл. 3. Результаты ПЦР-анализа образцов, переданных из Ставропольского государственного аграрного университета
Как видно из полученных данных, 2 из 6 переданных образцов заражён ASPV. Достоверность полученных данных была подтверждена секвенированием продуктов амплификации, показавшим соответствие их нуклеотидных последовательностей структурам специфического фрагмента ASPV.
По результатам проведённого исследования были предложены рекомендации по принятию мер фитосанитарного контроля. В частности, рекомендовано не использовать зараженные растения в качестве посадочного и/или прививочного материала; желательно утилизировать уже собранный материал.
Перечень используемых нуклеотидных последовательностей
Seq1
Олигонуклеотид: прямой праймер
Наименование: ASPVF
Последовательность (5’-3’): AATCAYTCWGGRAGRTCYTGTGA
В структуру входят вырожденные нуклеотиды Y=C или T; W=A или T; R=G или А
Seq2
Олигонуклеотид: обратный праймер
Наименование: ASPVR
Последовательность (5’-3’): TAYCTCATRTADGGNGCRAAYCTGC
В структуру входят вырожденные нуклеотиды: Y=C или T; R=G или А; D= A или G или T; N = A или G или C или T
Seq3
Олигонуклеотид: флуоресецнтно-меченый зонд
Наименование: ASPVT
Последовательность (5’-3’): (BHQ1) – GATTGGCCAGTKGA(FAMdT)AARGCHCTAATHTTC
В структуру входят вырожденные нуклеотиды: K=G или T; R=G или А; H=A или С или Т
В структуру входят флуорофор 6-карбоксифлуоресцеин (6-FAM) и гаситель флуоресценции Black Hole Quencher 1 (BHQ1)
Seq4
Специфический продукт амплификации, представляющий собой фрагмент гена белка РНК-зависимой РНК-полимеразы вируса ямчатости древесины яблони, проклонированный в плазмидный вектор pAL2T, длина 168 п.н.
Последовательность (5’-3’): TCATTCAGGAAGGTCTTGTGAGGATTGGCCAGTTGATAAGGCTCTAATTTTCATGAAGTCCCAACTTTGCACAAAGTTTGACAACAGATTCAGAAGTGCAAAAGCTGGTCAAACTCTGGCTTGCTTTCAACATTCAGTTCTGTGCAGATTTGCTCCTTACATGAGGTA
Список литературы
1. Paduch-Cichal E., Tomala K. Detection of Apple chlorotic leaf spot virus (ACLSV) and Apple stem grooving virus (ASGV) in different tissues of ‘mustu’ apple cultivar trees by ELISA. Phytopathologia Polonica 2007, 43, 53-59.
2. Метлицкая К.В., Упадышев М.Т., Петрова А.Д. Диагностика вредоносных вирусов на сортах яблони в Московской области. Сборник научных работ «Плодоводство и ягодоводство России», Методы и способы защиты растений от вредителей и болезней 2018, Т. 55, 271-274.
3. Кузмицкая П.В., Урбанович О.Ю., Козловская З.А. Идентификация вируса хлоротической пятнистости листьев яблони методом ОТ-ПЦР. Молекулярная и прикладная генетика 2013, 15, 26-32.
4. Kinard G.R., Scott S. W. Detection of Apple chlorotic leaf spot and Apple stem grooving viruses using RT-PCR. Plant Disease 1996, 80, 616-621.
5. Kundu J.K. The occurrence of Apple stem pitting virus and Apple stem grooving virus within field-grown apple cultivars evaluated by RT-PCR. Plant Protection Science 2003, 39, 88-92.
6. Kundu J.K. The application of RT-PCR assay for the detection of Apple stem pitting virus and Apple stem grooving virus in four apple cultivars. Plant Protection Science 2002, 38, 13-17.
7. Park H., Yoon J., Kim H., Baek K. Multiplex RT-PCR assay for the detection of Apple stem grooving virus and apple chlorotic leaf spot virus in infected Korean apple cultivars. Plant Pathology Journal 2006, 22, 168-173.
8. Lu Y., Yao B., Wang G., Hong N. The detection of ACLSV and ASPV in pear plants by RT-LAMP assays. Journal of Virological Methods 2017, 252, 80-85.
9. Nickel O., Fajardo T.V.M. Detection of viruses in apples and pears by real time RT-PCR using 5’-hydrolysis probes. Journal of Plant Pathology 2014, 96, 207-213.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd" PUBLIC "-
//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-08-13" softwareVersion="2.2.0"
softwareName="WIPO Sequence" fileName="ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА
ЯМЧАТОСТИ ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ.xml"
dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ЯМЧАТОСТИ
ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ</ApplicationNumberText>
<FilingDate/>
</ApplicationIdentification>
<ApplicantFileReference>ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ЯМЧАТОСТИ
ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное бюджетное
учреждение науки Государственный научный центр Российской Федерации Институт
биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова
Российской академии наук</ApplicantName>
<ApplicantNameLatin>Shemyakin-Ovchinnikov Institute of Bioorganic
Chemistry</ApplicantNameLatin>
<InventionTitle languageCode="ru">ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА
ЯМЧАТОСТИ ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcaytcwggragrtcytgtga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tayctcatrtadggngcraayctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gattggccagtkgaaargchctaathttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>168</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..168</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcattcaggaaggtcttgtgaggattggccagttgataaggctctaattttcatgaagt
Cccaactttgcacaaagtttgacaacagattcagaagtgcaaaagctggtcaaactctggcttgctttcaacattca
gttctgtgcagatttgctccttacatgaggta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА БОРОЗДЧАТОСТИ ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2835202C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ХЛОРОТИЧЕСКОЙ ПЯТНИСТОСТИ ЛИСТЬЕВ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2835209C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ ЧЁРНОГО РАКА ЯБЛОНИ SPHAEROPSIS MALORUM МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2840150C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ ФИТОФТОРОЗА КОРНЕВОЙ ШЕЙКИ PHYTOPHTHORA CACTORUM МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2838921C1 |
| СПОСОБ ЭКСТРАКЦИИ РНК ИЗ РАСТИТЕЛЬНЫХ ОБРАЗЦОВ | 2008 |
|
RU2389795C1 |
| Способ дифференциации генома вакцинного штамма "ВНИИЗЖ" от полевых изолятов вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков температур плавления ампликонов и применением асимметричного красителя SYBR Green | 2023 |
|
RU2822037C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией | 2023 |
|
RU2818960C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Набор олигонуклеотидных праймеров для выявления РНК вируса желтой лихорадки методом изотермической амплификации в режиме реального времени | 2024 |
|
RU2824209C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫХ ЗОНДОВ ДЛЯ СУБТИПОСПЕЦИФИЧНОЙ ИДЕНТИФИКАЦИИ РНК ВИРУСА ДЕНГЕ НА ОСНОВЕ МУЛЬТИПЛЕКСНОЙ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2012 |
|
RU2483115C1 |

Изобретение относится к области молекулярной биологии. Изобретение представляет собой тест-систему для обнаружения вируса ямчатости древесины яблони методом ПЦР в реальном времени. Тест-система включает: смесь для проведения амплификации, состоящую из реакционного буфера, дезоксинуклеотидтрифосфатов, двух олигонуклеотидных праймеров, специфичных к фрагменту гена, кодирующего РНК-зависимую РНК полимеразу, одного олигонуклеотидного зонда, меченого флуоресцентным красителем FAM, положительного контрольного образца, представляющего собой рекомбинантную плазмидную ДНК, содержащую соответствующий фрагмент генома вируса. Техническим результатом является разработка тест-системы для проведения высокоспецифичной идентификации кДНК, с высокой чувствительностью для обнаружения вируса ямчатости древесины яблони ASPV методом ПЦР в реальном времени. 4 ил., 3 табл.
Тест-система для обнаружения вируса ямчатости древесины яблони ASPV, включающая смесь для проведения амплификации, состоящую из реакционного буфера, дезоксинуклеотидтрифосфатов, двух олигонуклеотидных праймеров Seq1 и Seq2, специфичных к фрагменту гена, кодирующего белок оболочки вируса, одного олигонуклеотидного зонда Seq3, меченого флуоресцентным красителем FAM, положительного контрольного образца, представляющего собой рекомбинантную плазмидную ДНК Seq4, содержащую соответствующий фрагмент генома вируса
| Аппарат для регистрации артериального и венозного давления | 1932 |
|
SU33634A1 |
| CN 102634608 B, 08.04.2015 | |||
| СПОСОБ ОЦЕНКИ РЕАКЦИИ РАСТЕНИЙ ЯБЛОНИ НА ЛАТЕНТНУЮ ВИРУСНУЮ ИНФЕКЦИЮ | 2008 |
|
RU2384045C2 |
| Yoon J | |||
| Y | |||
| et al | |||
| Genetic diversity of a natural population of Apple stem pitting virus isolated from apple in Korea //The plant pathology journal | |||
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
| Способ обработки медных солей нафтеновых кислот | 1923 |
|
SU30A1 |
| - С | |||
| Регулятор давления для автоматических тормозов с сжатым воздухом | 1921 |
|
SU195A1 |
Авторы
Даты
2025-03-31—Публикация
2024-08-21—Подача