В открытых базах данных не обнаружено патентов, предметом которых являлась бы ПЦР-диагностика Sphaeromyces malorum. Найдены патенты Китайской Народной Республики CN 108179219A (прекратил свое действие, но может быть восстановлен) и Республики Корея KR 20230112858A (действующий), посвящённые ПЦР-диагностике близкородственного вида гриба Botryosphaeria dothidea. В обоих случаях детекция осуществляется методом гель-электрофореза.
Alves et al. [1] предложили систему видоспецифического определения S. malorum (syn. Botryosphaeria obtusa) с использованием микросателлитных маркеров. Данная система позволяет проводить дифференцировку видов, однако сложна технически и не применима для рутинной диагностики. Smith and Stanosz [2] описали систему ПЦР-диагностики близкородственных S. Malorum видов Diplodia pinea and D. scrobiculata, являющихся возбудителями болезней сосны. Детекция результатов проводилась с помощью гель-электрофореза, пределы чувствительности составили 50-100 пг ДНК на реакцию. Также описана тест-система на основе SCAR-маркеров [3], позволяющая выявлять S. Malorum с высокой чувствительностью (до 0.1 пг/реакцию), однако также использующая электрофоретический метод детекции результатов.
Наиболее близкой по своим характеристикам к предлагаемой разработке является тест-система, описанная в [4], чувствительность которой составила 0.125 пг ДНК/реакцию. Авторы использовали интеркалирующий краситель (SYBR Green), однако такой подход сопряжен с риском неспецифической детекции, связанным с тем, что данный краситель может взаимодействовать с любой двуцепочечной ДНК.
Список иллюстраций
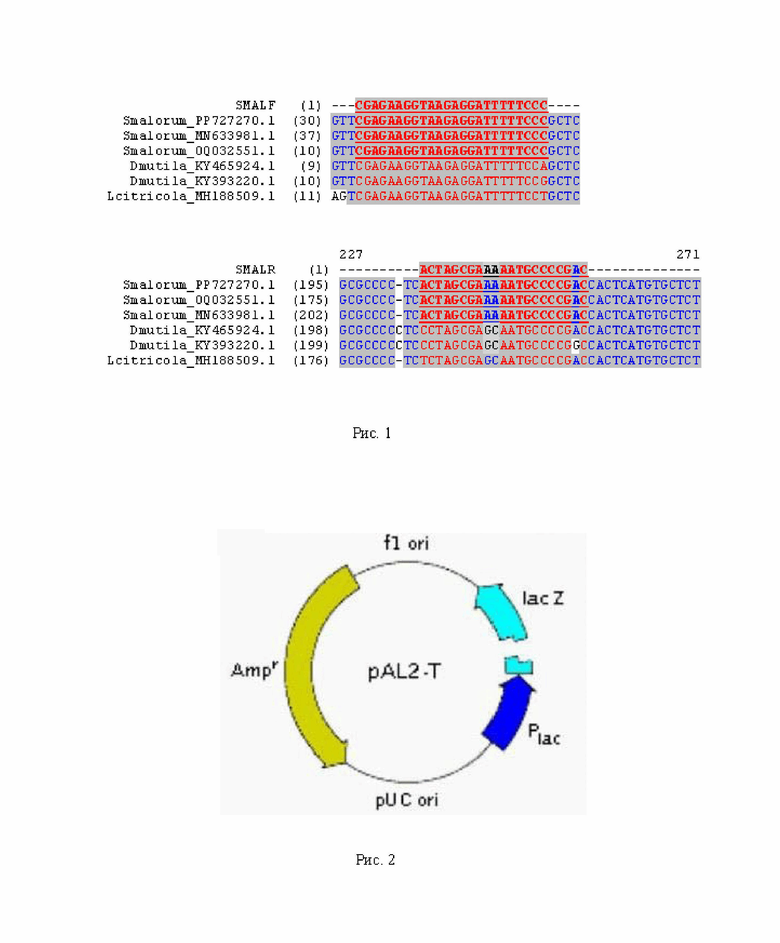
Рис. 1. Выравнивание фрагментов генов TEF1a S. malorum и близкородственных видов, использованных для подбора прямого (SMALF) и обратного (SMALR) праймеров. Последовательности праймеров подчёкнуты.
Рис. 2. Карта вектора pAL2T, использованного для клонирования положительных контролей.
Рис. 3. Результаты ПЦР последовательных десятикратных разведений плазмиды, содержащей специфический продукт амплификации. По оси абсцисс показано логарифмические значения концентраций (геном-эквиваленты/реакцию), по оси ординат – значения соответствующих пороговых циклов.
Рис. 4. Флуоресцентный сигнал, детектируемый при ПЦР-анализе последовательных десятикратных разведений плазмид, содержащих специфический ампликон, с парой праймеров SMALF-SMALR и зондом SMALT.
Техническим результатом является разработка тест-системы для проведения высокоспецифичной идентификации кДНК, с высокой чувствительностью для обнаружения вируса чёрного рака яблони Sphaeropsis malorum методом ПЦР в реальном времени.
Предлагаемое техническое решение позволяет повысить чувствительность детекции, а также избежать проблем, связанных с контаминацией рабочего пространства продуктами амплификации.
Дизайн праймеров проводили с помощью выравнивания последовательностей нуклеотидов, депонированных в базе данных NCBI c использованием алгоритма ClustalW. Помимо последовательностей нуклеотидов S. malorum, для выравнивания использовались последовательности нуклеотидов близкородственных видов. Наиболее вариабельным и пригодным для подбора праймеров был фрагмент гена фактора элонгации трансляции 1 альфа TEF1α. На рис. 1 приведены выравнивания фрагментов гена YPT1 и S. malorum близкородственных видов, использованных для подбора праймеров.
В результате проведённого биоинформатического анализа был проведён дизайн олигонуклеотидов: прямого праймера (Seq1), обратного праймера (Seq2) и флуоресцентно-меченого зонда (Seq3).
Оценку специфичности тест-системы проводили с использованием заражённых образцов древесины яблони, предоставленных сотрудниками Ставропольского государственного аграрного университета. Диагностика проводилась с помощью визуального осмотра на предмет признаков заражения фитофторозом корневой шейки.
Ниже приведены результаты ПЦР-анализа 10 образцов.
Табл. 1. Результаты ПЦР-анализа специфичности используемых систем
Продукты ПЦР, полученные в результате амплификации фрагмента ДНК «положительных» образцов, были проклонированы в плазмидный вектор pAL2T (схема вектора приведена на рис. 2). Соответствие проклонированной последовательности фрагменту генома S. malorum подтверждали с помощью секвенирования по методу Сэнгера. Для вектора, содержащего вставку специфического продукта (pAL2T-SMAL, Seq4), было проведено определение концентрации с использованием флуориметра QUBIT. Плазмида pAL2T-SMAL затем использовалась в качестве положительного контрольного образца.
Чувствительность тест-системы определяли с использованием последовательных десятикратных разведений плазмиды pAL2T-SMAL.
Табл. 2. Определение чувствительности тест-системы
Зависимость сигнала ПЦР (пороговый цикл) от концентрации ДНК в реакционной пробирке, выраженной в количестве геном-эквивалентов, приведена на рис. 3. На рис. 4 показаны уровни флуоресцентного сигнала при ПЦР последовательных десятикратных разведений положительного контрольного образца.
Тест-системы для определения ДНК возбудителя фитофтороза корневой шейки S. Malorum используются следующим образом.
Предварительно выделяют ДНК из биологического материала любым подходящим методом. В настоящей работе использовался подход, основанный на использовании нескольких буферов, содержащих цетилтриметиламмоний бромид (ЦТАБ) в качестве детергента, с последующей экстракцией смесью фенол:хлороформ и переосаждением этанолом.
Для проведения реакции используют готовую смесь для амплификации, состоящую из буфера, дезоксинуклеозидтрифосфатов, олигонуклеотидных праймеров, флуоресцентно-меченого зонда, Taq-полимеразы. Готовую реакционную смесь смешивают с образцами кДНК, положительным контрольным образцом и отрицательным контрольным образцом. ПЦР проводили в амплификаторе ДТ96 по следующей программе аплификации с детекцией в каналах FAM и HEX: 94°C – 90 сек 1 цикл; 94°C – 30 сек, 64°C – 30 сек – 5 циклов; 94°C – 10 сек, 64°C – 30 сек – 45 циклов
Результат, для конкретного образца, считается положительным образца при превышении порового уровня флуоресценции в канале FAM выше определённого уровня. При этом. Пороговый уровень флуоресценции определяется программным обеспечением оборудования.
Для апробации разработанной тест-системы сотрудниками Ставропольского государственного аграрного университета было передано 4 образца древесины яблони, из различных районов Ставропольского края. Для проведения анализа на первом этапе проводилось выделение ДНК. Для этого был адаптирован метод, основанный на использовании цетилтриметиламмоний бромида (ЦТАБ) в качестве детергента. 2 г молотой коры гомогенизировали в фарфоровой ступке с небольшим количеством буфера 2Х ЦТАБ буфер (0.5-1 мл), предварительно прогретого до температуры 65°С на водяной бане. Для более эффективной гомогенизации добавляли корунд. Полученную суспензию переносили в 50 мл пробирки и доводили объем буфером 2Х ЦТАБ буфером до 25 мл. Перемешивали и добавляли 50 мкл 2-меркаптоэтанола. Пробирки с гомогенатом инкубировали при температуре 65°С на водяной бане в течение 2 часов, при периодическом перемешивании. Затем охлаждали до комнатной температуры и инкубировали при температуре 4°С в течение ночи. В пробирки с гомогенатом добавляли равный объем смеси хлороформ:изоамиловый спирт в соотношении 24:1, перемешивали и инкубировали при комнатной температуре на 20 мин. Центрифугировали при комнатной температуре 10 мин на скорости 5000 об/мин. Верхнюю водную фазу переносили в чистые пробирки, добавляли 0.2 объёма буфера 5Х ЦТАБ буфера и 1 мкл РНКазы. Раствор перемешивали и инкубировали 20 мин при температуре 65°С на водяной бане. Добавляли равный объем смеси хлороформ:изоамиловый спирт в соотношении 24:1, перемешивали и инкубировали при комнатной температуре 10 мин. Центрифугировали 10 мин на скорости 12000 об/мин. Верхнюю водную фазу переносили в чистые пробирки (на 50 мл). К водной фазе добавляли равный объем буфера для преципитации, перемешивали и инкубировали 5 мин при комнатной температуре. Центрифугировали при комнатной температуре 15 мин на скорости 12000 об/мин. Надосадочную жидкость удаляли, осадок растворяли в 2 мл буфера HS-TE. Добавить 2V 95% этанола, перемешать и инкубировать в течение ночи при темепературе -20°С. Центрифугировать при температуре 40°С 15 мин на скорости 12000 об/мин. Надосадочную жидкость удаляли, а к осадку добавляли 400 мкл деионизированной воды и 200 мкл 3М ацетата натрия. Инкубировали во льду 20 мин. По ½ полученного объема переносили в чистые пробирки на 1.5 мл, добавляли по 2 объёма 95% этанола и инкубировали ночь при температуре -20°С. Центрифугировали при комнатной температуре 10 мин на скорости 13500 об/мин. Осадок растворяли в 50-100 мкл деионизированной воды.
Состав буферов, используемых в работе:
2Х ЦТАБ: 2% СТАВ; 0.1М Трис HCl (pH8.0); 20mM EDTA; 1.4 M NaCl
5Х ЦТАБ – 5% СТАВ; 350mM EDTA
Буфер для преципитации: 1% СТАВ; 50 mМ Трис HCl (pH8.0); 10mM EDTA
HS-TE: 10 мМ Трис HCl (pH8.0); 1mM EDTA; 1 M NaCl.
Полученные ДНК использовали в качестве матриц в ПЦР в реальном времени с использованием разработанных специфических праймеров и зонда. ПЦР и анализ результатов проводили по протоколам, приведённым выше. В таблице 3 приведены результаты ПЦР-анализа.
Табл. 3. Результаты ПЦР-анализа образцов, переданных из Ставропольского государственного аграрного университета
Как видно из полученных данных, 1 из 4 переданных образцов заражен S. malorum. Достоверность полученных данных была подтверждена секвенированием продуктов амплификации, показавшим соответствие их нуклеотидных последовательностей структурам специфического фрагмента возбудителя корневой шейки.
По результатам проведённого исследования были предложены рекомендации по принятию мер фитосанитарного контроля. В частности, рекомендовано не использовать зараженные растения в качестве посадочного и/или прививочного материала; желательно утилизировать уже собранный материал.
Список литературы
1. Alves A., Phillips A.J.L., Henriques I., Correia A. Rapid differentiation of species of Botryosphaeriaceae by PCR fingerprinting. Research in Microbiology 2007, 158, 112-121.
2. Smith D.R., Stanosz G.R. A species-specific PCR assay for detection of Diplodia pinea and D scrobiculata in dead red and Jack pines with collar rot symptoms. Plant Disease 2006, 90, 307-313.
3. Martin M.T.; Cuesta M.J., Martin L. Development of SCAR primers for PCR assay to detect Diplodia seriata. International Scholarly Research Notices 2014, 824106.
4. Pouzoulet J., Rolshausen P.E., Schiavon M., Bol S., Travadon R. et al. A method to detect and quantify Eutypa lata and Diplodia seriata-complex DNA in grapevine pruning wounds. Plant Disease 2017, 101, 1470-1480.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="ТЕСТ-СИСТЕМА ДЛЯ
ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ ЧЁРНОГО РАКА ЯБЛОНИ SPHAEROPSIS MALORUM.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2024-08-13">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ
ЧЁРНОГО РАКА ЯБЛОНИ SPHAEROPSIS MALORUM МЕТОДОМ ПЦР В РЕАЛЬНОМ
ВРЕМЕНИ</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ
ЧЁРНОГО РАКА ЯБЛОНИ SPHAEROPSIS MALORUM МЕТОДОМ ПЦР В РЕАЛЬНОМ
ВРЕМЕНИ</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Государственный научный центр Российской
Федерации Институт биоорганической химии им. академиков М.М. Шемякина
и Ю.А. Овчинникова Российской академии наук</ApplicantName>
<ApplicantNameLatin>Shemyakin-Ovchinnikov Institute of Bioorganic
Chemistry</ApplicantNameLatin>
<InventionTitle languageCode="ru">ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ
ВОЗБУДИТЕЛЯ ЧЁРНОГО РАКА ЯБЛОНИ SPHAEROPSIS MALORUM МЕТОДОМ ПЦР В
РЕАЛЬНОМ ВРЕМЕНИ</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagaaggtaagaggatttttccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcggggcatttttcgctagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggggcatttttcgtggtgggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>192</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..192</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagaaggtaagaggatttttcccgctcccgcactgcgtgcacagccca
ccttatcgctcggtgaggggcatttttcgtggtggggtttggcccgcgctaaacctcgtctgagctcggc
aaactggccgcacttggttttttgcgaccggggtctggccgatgcgcccctcactagcgaaaaatgcccc
gac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВОЗБУДИТЕЛЯ ФИТОФТОРОЗА КОРНЕВОЙ ШЕЙКИ PHYTOPHTHORA CACTORUM МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2838921C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ЯМЧАТОСТИ ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2837395C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА БОРОЗДЧАТОСТИ ДРЕВЕСИНЫ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2835202C1 |
| ТЕСТ-СИСТЕМА ДЛЯ ОБНАРУЖЕНИЯ ВИРУСА ХЛОРОТИЧЕСКОЙ ПЯТНИСТОСТИ ЛИСТЬЕВ ЯБЛОНИ МЕТОДОМ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2024 |
|
RU2835209C1 |
| Способ преимплантационного генетического тестирования болезни Гентингтона | 2024 |
|
RU2840728C1 |
| Способ преимплантационного генетического тестирования ахондроплазии | 2022 |
|
RU2795482C1 |
| Способ преимплантационного генетического тестирования наследственной зонулярной катаракты | 2022 |
|
RU2799541C1 |
| Способ преимплантационного генетического тестирования синдрома Мартина-Белл | 2022 |
|
RU2796834C1 |
| Способ преимплантационного генетического тестирования Синдрома Марфана | 2023 |
|
RU2837158C1 |
| Способ преимплантационного генетического тестирования синдрома Смита-Лемли-Опица | 2023 |
|
RU2816650C1 |
Изобретение относится к области молекулярной биологии. Описана тест-система для обнаружения вируса возбудителя чёрного рака яблони Sphaeropsis malorum, включающая смесь для проведения амплификации, состоящую из реакционного буфера, дезоксинуклеотидтрифосфатов, двух олигонуклеотидных, одного олигонуклеотидного зонда, положительного контрольного образца. Изобретение позволяет повысить чувствительность детекции Sphaeropsis malorum, а также избежать проблем, связанных с контаминацией рабочего пространства продуктами амплификации. 4 ил., 3 табл.
Тест-система для обнаружения вируса возбудителя чёрного рака яблони Sphaeropsis malorum, включающая смесь для проведения амплификации, состоящую из реакционного буфера, дезоксинуклеотидтрифосфатов, двух олигонуклеотидных праймеров Seq1 и Seq2, специфичных к фрагменту гена фактора элонгации трансляции 1 альфа, одного олигонуклеотидного зонда Seq3, меченого флуоресцентным красителем FAM, положительного контрольного образца, представляющего собой рекомбинантную плазмидную ДНК Seq4, содержащую соответствующий фрагмент генома патогена.
| Аппарат для регистрации артериального и венозного давления | 1932 |
|
SU33634A1 |
| Alves A | |||
| et al | |||
| Rapid differentiation of species of Botryosphaeriaceae by PCR fingerprinting | |||
| Research in microbiology, 2007, v | |||
| Система механической тяги | 1919 |
|
SU158A1 |
| RU 2001108572 A, 20.08.2004. | |||
Авторы
Даты
2025-05-19—Публикация
2024-08-21—Подача