Изобретение относится к области медицины, а именно к молекулярно-генетической диагностике, и может быть использовано для выявления специфических мутаций в генах с использованием аллель-специфической полимеразной цепной реакции (ПЦР). Предлагаемый способ предназначен для диагностики заболеваний в кардиологии, в частности для заболеваний, связанных с удлинением интервала QT, ассоциированных с мутацией R176W в гене KCNH2.
Внезапная сердечная смерть (ВСС) является неожиданным фатальным событием. Наиболее частой причиной ВСС является желудочковая тахиаритмия, возникающая при ишемической болезни сердца (примерно 75%-80%); дилатационная кардиомиопатия (идиопатическая и постмиокардитная, примерно 10%-15%) или редкие моногенные мутации в сердечных ионных каналах или связанных с ними белках (1%-2%).
Известно, что генетические факторы играют важную роль в патогенезе большого числа болезней, к настоящему времени подтверждены молекулярно-генетическим анализом наследственные синдромы и нозологические формы, проявляющиеся нарушением ритма сердца [Школьникова М.А. и др. Генетически детерминированные нарушения ритма сердца, 2011].
Синдром удлиненного интервала QT (LQTS) - наследственное заболевание, сопровождающееся удлинением интервала QT на ЭКГ покоя (QTc>=460 мс), синкопальными состояниями и высоким риском внезапной смерти вследствие развития полиморфной желудочковой тахикардии. Существуют две его клинические формы: синдром Романо-Уорда, с удлинением интервала QT на ЭКГ, нормальным слухом и аутосомно-доминантным типом наследования (для развития клинической картины заболевания достаточно 1 мутации) и синдром Джервелла-Ланге-Нильсена, с более тяжёлым течением, врождённой двусторонней нейросенсорной глухотой и аутосомно-рецессивным типом наследования (для развития клинической картины заболевания необходимы 2 мутации). Частота синдрома Романо - Уорда в популяции составляет 1:5000. Синдром Джервелла-Ланге-Нильсена в популяции встречается с частотой около 1.6-6:1000000, а среди лиц с врождённой двусторонней глухотой с частотой 3:1000. Идентифицированы 12 генов, ответственных за развитие заболевания. Возможна прямая ДНК-диагностика синдрома удлинённого интервала QT. Поиск мутаций проводится в генах KCNQ1, KCNH2, SCN5A, KCNE1, KCNE2, KCNJ2, CAV3 и SCN4B. При анализе кодирующей последовательности перечисленных генов удаётся выявить мутации и установить молекулярно-генетическую форму заболевания более чем у 70% пробандов. Мутации в этих генах также могут быть причиной синдрома детской внезапной смерти (около 20% этих смертей).
На сегодня в гене KCNH2 выявлено 688 уникальных мутаций, из которых 414 - патогенные. В основном это миссенс-мутации (~48 %) и синонимичные замены (~21 %). 46 патогенных мутаций KCNH2 связаны с синдромом удлиненного интервала QT и встречаются в 30-35 % случаев синдрома LQT2 [Crotti L, Celano G, Dagradi F, Schwartz PJ. Congenital long QT syndrome. Orphanet J Rare Dis. 2008 Jul 7;3:18].
Исследование, проведенное Sanguinetti et al. (1995), сосредоточилось на экспрессии гена HERG (KCNH2) в ооцитах Xenopus laevis, что позволило детально исследовать биофизические свойства калиевого канала и его реакцию на различные фармакологические препараты. Полученные данные продемонстрировали, что белки HERG формируют каналы типа I(Kr). Однако выяснилось, что для проявления некоторых специфических форм лекарственной чувствительности может потребоваться участие дополнительной субъединицы. Поскольку блокировка I(Kr) представляет собой устоявшийся механизм, ведущий к возникновению лекарственных аритмий сердца, результаты исследования предоставляют важные механистические связи между определенными наследственными и приобретенными формами синдрома удлиненного интервала QT (LQT).
Калиевые каналы KCNH2 (hERG) играют неотъемлемую роль в регуляции возбудимости гладкомышечных клеток. Некоторые пути, управляемые ангиотензином II, оксидом азота и блокатором адренергических рецепторов, участвуют в модуляции свойств калиевых каналов KCNH2. KCNQ1 и KCNH2 - два наиболее распространенных гена калиевых каналов, вызывающих синдром удлиненного интервала QT (LQTS), наследственную сердечную аритмию, характеризующуюся удлинением интервала QT и повышенным риском развития желудочковой тахикардии типа «пируэт» и внезапной смерти.
Поэтому на сегодняшний день для изучения экспрессивности заболевания многие исследования направлены на выявление мутаций и распространенных вариантов, которые могут изменять фенотип LQTS.
Показано, что генетический полиморфизм KCNH2 (1956, C>T) - новый биомаркер, связанный с реакцией на БКК и блокаторы α,β-ADR у пациентов с гибертонической болезнью. Полиморфизм KCNH2 (1956, C>T) связан с эффективностью антигипертензивных препаратов, блокаторов кальциевых каналов и блокаторов АДР, и может служить новым биомаркером для индивидуализированной терапии некоторыми антигипертензивными препаратами (Fazhong He, The KCNH2 Genetic Polymorphism (1956, C>T) Is a Novel Biomarker That Is Associated with CCB and α,β-ADR Blocker Response in EH Patients in China, 2013).
Использование молекулярно-генетических методов для диагностики, дифференциальной диагностики и выбора тактики лечения синдрома удлинённого интервала QT зачастую имеет решающее значение. Особенно актуальной ДНК-диагностика является при бессимптомном течении заболевания, когда при отсутствии клинических признаков заболевания у пациентов сохраняется высокий риск внезапной смерти. Точное установление молекулярно-генетического варианта заболевания позволяет подобрать больному адекватную лекарственную терапию с учетом нарушения функционирования конкретного типа ионного канала.
Существует метод анализа генетических вариаций, основанный на секвенировании амплифицированных фрагментов ДНК методом Сенгера [PMID: 34002542]. Однако у этого метода есть значительные недостатки, такие как высокая стоимость как оборудования, так и реактивов, что препятствует его широкому применению в научных исследованиях.
Известен способ (RU 2803796) полноэкзомного секвенирования, в панель которого входит ген KCNH2. Метод полноэкзомного секвенирования - это секвенирование всех белок-кодирующих генов человека (приблизительно 20000), а секвенирование экзома с клинической целью - это секвенирование порядка 5000 генов, про которые на настоящий момент известна ассоциация с генетическими болезнями или признаками. Экзом составляет всего ~1% от полного генома человека, но приблизительно 85% всех болезнетворных вариантов находятся именно в белок-кодирующих областях. С помощью технологии секвенирования экзома можно определить последовательность 90-95% целевых участков человека, и некоторые участки поддаются секвенированию с помощью этой методики несколько хуже. Способность метода выявить болезнетворный вариант зависит от того, в каком участке он находится (Online Mendelian Inheritance in Man, OMIM. http://www.ncbi.nlm.nih.gov/omim/). Метод предназначен для поиска однонуклеотидных замен в кодирующих участках генов человека. Некоторые другие типы генетических вариантов могут быть выявлены, но поддаются обнаружению с меньшей вероятностью, чем однонуклеотидные замены: это относится, в частности, к коротким делециям или вставкам (инделам) и изменениям числа коротких тандемных повторов (Database of Single Nucleotide Polymorphisms (dbSNP). http://www.ncbi.nlm.nih.gov/SNP/).
Поэтому данный метод имеет ряд существенных ограничений, таких как высокая стоимость проведения анализа, значительные временные затраты на получение и интерпретацию результатов, а также высокая частота ложноположительных результатов. Кроме того, использование полноэкзомного секвенирования для выявления единичных мутаций, таких как NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2, является нецелесообразным из-за избыточности получаемых данных и неоправданно высоких затрат.
В настоящее время, аллель-специфическая ПЦР - это эффективный метод выявления мутаций в геномной ДНК, широко используемый для анализа однонуклеотидных полиморфизмов (SNPs). Так как этот метод позволяет найти небольшое количество мутантных ДНК среди большого количества ДНК дикого типа, он также применяется для анализа соматических мутаций.
В основе метода лежит элонгация аллель-специфических праймеров (allele specific primer extension), строго комплементарных нуклеотидной последовательности матричной ДНК, расположенной на 3'-конце. При неполной комплементарности эффективность удлинения праймера во время ПЦР резко снижается либо отсутствует.
Известно применение метода ПЦР в режиме "реального времени" с применением аллель-специфических флуоресцентных зондов для генотипирования полиморфного локуса rs12561767 (GA) гена SERBP1 у человека (RU 2803522).
Также в диагностике заболеваний применяется метод выявления мутаций в гене ATP7B, предусматривающий выделение ДНК из образцов крови пациентов по одному из стандартных протоколов с последующим анализом при помощи аллель-специфической ПЦР и электрофореза продуктов амплификации (RU 2821573).
Преимущества метода заключаются в его специфичности, чувствительности, невысокой стоимости и быстроте получения результатов, так как при правильно подобранных условиях анализ SNPs занимает всего 3-4 часа. Он позволяет с высокой точностью различать аллели в геноме в одностадийной ПЦР без дополнительного этапа разделения продуктов реакции.
Прототипом изобретения является определение мутаций в гене KCNQ1 and KCNH2 (Xianqin Zhang et al. Protective effect of KCNH2 single nucleotide polymorphism K897T in LQTS families and identification of novel KCNQ1 and KCNH2 mutations, 2008), что расширяет мутационный и клинический спектр LQTS и дает представление о факторах, которые определяют удлинение QT, связанное с повышенным риском внезапной смерти.
Однако, существует потребность в создании быстрого, недорогого и легко воспроизводимого способа идентификации варианта мутации NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2, с доступной всем исследователям структурой праймеров и аллель-специфических зондов, который мог бы использоваться в качестве «рутинного» метода генотипирования в любой ПЦР-лаборатории.
Выдвигаемая техническая проблема, на разрешение которой направлено заявляемое изобретение, является разработка эффективного и быстрого способа обнаружения варианта NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2.
Разработан способ генотипирования полиморфного локуса rs36210422 (G>A) гена методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации, отличающийся тем, что идентификацию аллельных вариантов rs36210422 (G>A) гена KCNH2 осуществляют с использованием прямого и обратного праймера для аллеля дикого типа -SEQ ID NO 1 и SEQ ID NO 2, для мутантного SEQ ID NO 1 и SEQ ID NO 3, соответственно.
Техническим результатом заявленного изобретения является создание новой аллель-специфической ПЦР, позволяющей точно и быстро идентифицировать мутацию NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2. Заявленный способ позволяет точно идентифицировать наличие как аллелей дикого типа, так и мутантных аллелей гена KCNH2 на основе специфичности разработанных уникальных праймеров SEQ ID NO 1 - SEQ ID NO 3, что дает возможность широко использовать его в молекулярно-генетической диагностике.
В разработанном способе использует обычную амплификацию фрагментов ДНК по протоколу стандартной ПЦР с последующим анализом продуктов стандартным методом электрофореза в агарозном геле, что сильно удешевляет диагностику.
Использование данного метода значительно снижает так же и денежные затраты, что делает диагностику более экономически выгодной по сравнению с другими известными методами. Для проведения аллель-специфической ПЦР не требуется сложного оборудования или специализированных навыков, что делает метод доступным для большинства лабораторий. Метод требует минимального количества биологического материала, что особенно важно при работе с ограниченными образцами.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
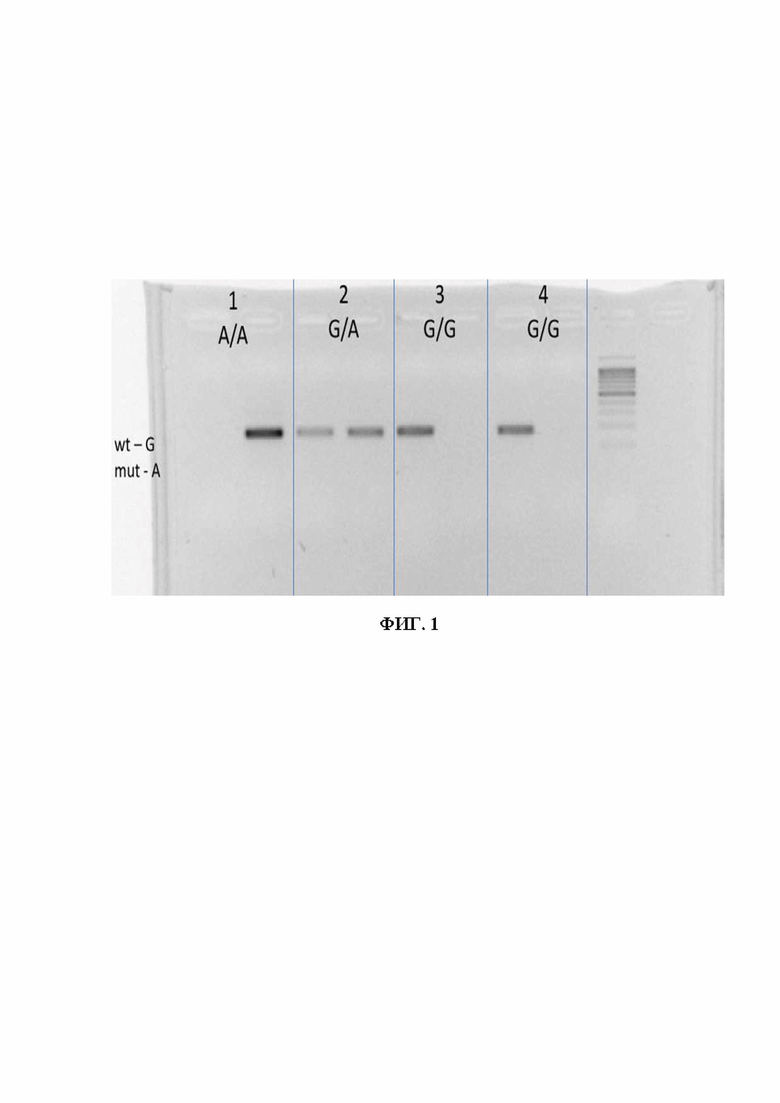
ФИГ. 1. Электрофореграмма результатов аллель-специфической ПЦР. 1, 2, 3 и 4 - условное обозначение, присваеваемое пациентам для обезличивания; A - мутантный аллель; G - нормальный аллель.
ОСУЩЕСТВЛЕНИЕ ИЗОБРЕТЕНИЯ
Сущность технического решения заключается в проведении амплификации в двух параллельных пробирках, содержащих различные пары праймеров. В первой пробирке используется пара праймеров, специфичных к участку гена KCNH2, содержащему неизмененный (дикий тип, WT) аллель. Во второй пробирке применяется пара праймеров, направленных на амплификацию участка гена, содержащего мутацию (замена R/W). Такой подход позволяет одновременно детектировать наличие мутации, а также генотип пациента.
Было обследовано 30 пациентов.
Способ осуществляют следующим образом:
1. Выделение ДНК из биологического образца (кровь, слюна, ткани и т.д.) с последующим измерением концентрации на спектрофотометре.
2. Подготовка реакционных смесей для ПЦР, включающих специфические праймеры, комплементарные участку гена KCNH2, где потенциально содержится мутация NM_000238.4:c.526C>T (p.Arg176Trp)
3. Проведение ПЦР с использованием аллель-специфических праймеров, позволяющих амплифицировать только мутантный аллель в одной смеси, только нормальный аллель во второй смеси.
4. Анализ результатов ПЦР с помощью электрофореза или других методов детекции, позволяющих определить наличие или отсутствие мутации.
Реакционная смесь для проведения аллель-специфической ПЦР включает следующие компоненты:
1. ДНК-матрица - 1мкл;
2. Праймер KCNH2 asPCR (F) - 1мкл;
3. Праймер KCNH2 asPCR R176-WT (R) (для второй смеси KCNH2 asPCR R176W (R)) - 1мкл;
4. Мастер-микс для ПЦР с ДНК-полимеразой с «горячим» стартом, содержащий два красителя - 12,5мкл;
5. H2O, nuclease-free - 8,5 мкл;
6. DMSO - 1мкл.
Экспериментально подобраны оптимальные условия для проведения генотипирования, амплификация проводится в термоциклере) с использованием следующих этапов:
1. Денатурация - 95°C - 3 мин;
2. Циклирование - 38 циклов - 95°C, 20 сек - 62°C, 12 сек - 72°C ,12 сек;
3. Финальная элонгация - 72°C - 15 сек;
4. Охлаждение - до 4-10°C.
С применением открытого ресурса University of California Santa Cruz (UCSC) [https://genome.ucsc.edu/index.html] выбран сиквенс, фланкирующий искомую однонуклеотидную замену [G>A] rs36210422 гена KCNH2, и затем с помощью доступного онлайн програмного обеспечения OligoAnalize [https://eu.idtdna.com/pages/tools/oligoanalyzer] подобрана последовательность олигонуклеотидов, используемых для проведения ПЦР-реакции (таблица 1).
Длина целевого ампликона 175 п.н.
Таблица 1.
Набор олигонуклеотидных праймеров.
Прямой праймер (KCNH2 asPCR (F) CCCTCCTTCCCTTACCCA) (SEQ ID NO 1) остается неизменным и служит для амплификации общего участка ДНК, содержащего интересующий полиморфизм.
Обратные праймеры различаются в зависимости от того, какой аллель необходимо индивидуализировать:
- для выявления мутантного аллеля используется специфичный обратный праймер KCNH2 asPCR R176W (R) GCACCGACGACTCCCAG (SEQ ID NO 3);
- для амплификации аллеля дикого типа (wild type, WT) применяется обратный праймер KCNH2 asPCR R176-WT (R) GCACCGACGACTCCCG (SEQ ID NO 2).
Сама идентификация аллелей осуществляется путем применения двух пар праймеров: SEQ ID NO 1 и SEQ ID NO 3, SEQ ID NO 1 и SEQ ID NO 2.
После проведения амплификации происходит электрофорез в 1% агарозном геле с последующим этапом анализ полученных данных. Пример получаемых результатов находится на фигуре1.
На фигуре 1 показано: в представленном образце 1 наблюдается гомозиготная мутация R176W- амплификация ДНК была успешной только в одной пробирке, содержащей специфические праймеры для данной мутации. Во втором образце фиксируется гетерозиготное состояние, при котором амплификация произошла в обеих пробирках, что указывает на наличие как мутантной, так и нормальной аллелей. Образцы 3 и 4 показывают гомозиготное состояние по норме (wild type, wt), в которых амплификация была выполнена исключительно в пробирке с праймерами, специфичными для нормальной последовательности, что подтверждает отсутствие мутантного аллеля).
Для аллеля дикого типа результат анализируется, где были задействованы праймеры SEQ ID NO 1 и SEQ ID NO 2, для мутантного, соответственно, SEQ ID NO 1 и SEQ ID NO 3. Таким образом, если мы видим на агарозе результат только амплификации первой пары, тогда пациент гомозигота норма, если обеих пар, то гетерозигота, если только второй пары, то гомозигота R176W.
ПРИМЕР.
Пациент К. 21 год, пол - мужской. Диагноз: Синдром удлиненного интервала QT. Жалобы: впервые удлинение интервала QT было выявлено в возрасте 11 лет при плановом обследовании перед посещением спортивной секции. Исследование: панель генов «Первичные формы синдрома удлиненного интервала QT».
Методом секвенирования нового поколения было проведено секвенирование белок-кодирующих генов образца ДНК и проведен биоинформатический поиск в панели ассоциированных генов. Выявлен патогенный низко-пенетрантный генетический вариант NM_000238:c.526C>T (p.Arg176Trp) в гомозиготном состоянии в последовательности 4 экзона гена KCNH2.
Обнаруженный генетический вариант был верифицирован с использованием данного изобретения, основанного на аллель-специфической полимеразной цепной реакции.
1. Выделение ДНК из крови проводили колоночным методом следующим образом. На первом этапе был отобран лейкоцитарный слой. Далее клетки были пролизированы с помощью специального буфера и протаиназы К (набор для выделения Magen). Далее добавляли спирт и переносили весь объем на колонку. После термостатирования при комнатной температуре (5 мин) производили центрифугирование в течение 1 минуты при 20000 rcf. После этого колонку чистили двухэтапно промывочными растворами с чередующим центрифугированием в течении 1 минуты при 10000 rcf. С полученной чистой колонки элюировали ДНК 60 мкл элюирующим буфером. Последний этап провели дважды для увелечения выхода продукта.
2. Подготовка образцов ДНК к генотипированию. Качество выделенной ДНК оценивали по степени чистоты и концентрации раствора на спектрофотометре NanoDrop (Thermo Fisher Scientific, США). Все анализируемые образцы ДНК были разведены деионизированной водой до концентрации 15-20 нг/мкл при А260/280=1,5-2,0.
3. Выявление мутации NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2 проводили с помощью полимеразной цепной реакции. Для генотипирования использовали две пары фланкирующих праймеров, SEQ ID NO 1 и SEQ ID NO 3, SEQ ID NO 1 и SEQ ID NO 2.
Аллель-специфическую ПЦР проводили в 25 мкл двух реакционных смесей, содержащей 2х Taq М мастер-микс («Intifica», Санкт-Петербург, Россия), 15 нг ДНК, по 1 мкМ каждого праймера (SEQ ID NO 1 и SEQ ID NO 3 - для первой смеси, SEQ ID NO 1 и SEQ ID NO 2 - для второй смеси), 4% DMSO, 8,5 мкл H2O, nuclease-free. Реакция амплификации состояла из стадии нагревания до 95°C в течение 3 минут, затем 38 циклов [95°C в течение 20 секунд и 62°C в течение 12 секунд, 72°C в течение 12 секунд], затем финальная элонгация при 72°C в течение 15 секунд, охлаждение для хранения до 4°C.
4. После проведения аллель-специфической реакции генотипирование осуществляли по данным гель-электрофореза.
ПРИМЕР 2.
Пациент Н. 14 лет, пол - женский. Исследование: поиск генетического варианта NM_000238:c.526C>T (p.Arg176Trp) в последовательности гена KCNH2, выявленного ранее у пробанда. Ранее обнаруженный у пробанда генетический вариант был проверен у пациента Н. с использованием настоящего изобретения на основе аллель-специфичных полимеразных цепных реакций.
Методика выполнения исследования как в примере 1.
Результаты исследования: генетический вариант NM_000238:c.526C>T (p.Arg176Trp) в гетерозиготном состоянии в последовательности 4 экзона гена KCNH2 выявлен у пациента Н.
Таким образом, предложенный способ идентификации генотипирования полиморфного локуса rs36210422 (G>A) гена осуществляют посредством проведения аллель-специфической ПЦР в присутствии разработанных уникальных специализированных олигонуклеотидных праймеров, соответствующих мутантным и нормальным аллелям гена KCNH2.
В разработанном способе использует обычную амплификацию фрагментов ДНК по протоколу стандартной ПЦР с последующим анализом продуктов стандартным методом электрофореза в агарозном геле, что сильно удешевляет диагностику.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="KCNH2.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2025-02-25">
<ApplicationIdentification>
<IPOfficeCode></IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2025-02-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>20250225</ApplicantFileReference>
<ApplicantName languageCode="ru">ФГБНУ РНЦХ им. акад. Б.В.
Петровского</ApplicantName>
<ApplicantNameLatin>Petrovsky National Research Centre of
Surgery</ApplicantNameLatin>
<InventorName languageCode="ru">Заклязьминская Елена
Валерьевна</InventorName>
<InventorNameLatin>Zaklyazminskaya E.V.</InventorNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования
полиморфного локуса rs36210422 (G>A) гена KCNH2</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccctccttcccttaccca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaccgacgactcccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaccgacgactcccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 | 2025 |
|
RU2837876C1 |
| Способ поиска мутаций гена ATP7B для постановки диагноза болезни Вильсона (Вильсона-Коновалова) | 2023 |
|
RU2821573C1 |
| Способ прогнозирования привычной невынашиваемости беременности по экспрессии генов микроРНК в биопсийном материале эндометрия | 2025 |
|
RU2837879C1 |
| Способ идентификации полиморфизмов Cys1079Gly и Cys1079Phe медь-транспортной АТФ-азы Вильсона | 2020 |
|
RU2756112C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ диагностики варианта SOPH c.5741G>A в гене NBAS | 2024 |
|
RU2819985C1 |
| НОВЫЕ ПРАЙМЕРЫ ДЛЯ СКРИНИРОВАНИЯ ШИЗОФРЕНИИ И СПОСОБ ЕЕ СКРИНИРОВАНИЯ | 2002 |
|
RU2338788C2 |
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНАХ EGFR, KRAS И BRAF С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2014 |
|
RU2552483C1 |
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНЕ PI3K С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2013 |
|
RU2549682C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ ПОЛИМОРФИЗМА R287Q В 8 ЭКЗОНЕ ГЕНА ЦИТОЗОЛЬНОЙ ЭПОКСИДГИДРОЛАЗЫ У ЧЕЛОВЕКА | 2006 |
|
RU2346053C2 |
Изобретение относится к области молекулярной биологии. Описан cпособ генотипирования полиморфного локуса rs36210422 (G>A) гена методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации. Идентификацию аллельных вариантов rs36210422 (G>A) гена KCNH2 осуществляют с использованием прямого и обратного праймеров: для аллеля дикого типа - SEQ ID NO: 1 и SEQ ID NO: 2, для мутантного - SEQ ID NO: 1 и SEQ ID NO: 3 соответственно. Техническим результатом заявленного изобретения является создание новой аллель-специфической ПЦР, позволяющей точно и быстро идентифицировать мутацию NM_000238.4:c.526C>T (p.Arg176Trp) в экзоне 4 гена KCNH2. 1 ил., 1 табл., 2 пр.
Способ генотипирования полиморфного локуса rs36210422 (G>A) гена методом аллель-специфической ПЦР с последующим электрофорезом продуктов амплификации, отличающийся тем, что идентификацию аллельных вариантов rs36210422 (G>A) гена KCNH2 осуществляют с использованием прямого и обратного праймеров: для аллеля дикого типа -SEQ ID NO: 1 и SEQ ID NO: 2, для мутантного - SEQ ID NO: 1 и SEQ ID NO: 3 соответственно.
| Zhang X | |||
| et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| BMC medical genetics, 2008, v | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| СПОСОБ ДИФФЕРЕНЦИРОВАННОГО ПРИМЕНЕНИЯ ДАННЫХ СЕКВЕНИРОВАНИЯ ДНК ДЛЯ ПАЦИЕНТОВ ПОСЛЕ ТРАНСПЛАНТАЦИИ ПОЧКИ | 2022 |
|
RU2803796C1 |
| WO 2016130088 A1, 18.08.2016. | |||
Авторы
Даты
2025-04-07—Публикация
2025-02-25—Подача