[0001] ОБЛАСТЬ ИЗОБРЕТЕНИЯ
[0002] Варианты осуществления настоящего изобретения относятся к секвенированию нуклеиновых кислот. В частности, варианты осуществления способов и композиций, предложенные в настоящем документе, относятся к получению индексированных библиотек одиночных ядер и одиночных клеток с использованием хеширующих олигонуклеотидов и/или нормализующих олигонуклеотидов и получению из них данных о последовательности.
[0003] ПЕРЕКРЕСТНЫЕ ССЫЛКИ НА РОДСТВЕННЫЕ ЗАЯВКИ
[0004] Настоящая заявка испрашивает преимущество по предварительной заявке США № 62/812,853, поданной 1 марта 2019 г., которая полностью включена в настоящий документ путем ссылки.
[0005] ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[0006] Настоящая заявка содержит перечень последовательностей, представленный в электронной форме посредством EFS-Web в Бюро по регистрации патентов и товарных знаков США в виде текстового файла ASCII с именем IP-1815-PCT_ST25.txt, имеющего размер 4 килобайта и созданного 28 февраля 2020 г. Информация, содержащаяся в перечне последовательностей, включена в настоящий документ путем ссылки.
[0007] ФИНАНСИРОВАНИЕ ЗА СЧЕТ ГОСУДАРСТВЕННЫХ СРЕДСТВ
[0008] Настоящее изобретение было выполнено с государственной поддержкой в рамках грантов №№ HG007811, HD088158 и R01 HG006283, предоставленных Национальными институтами здравоохранения, и гранта № DGE1258485, предоставленного Национальным научным фондом. Государство обладает определенными правами на изобретение.
[0009] ПРЕДПОСЫЛКИ СОЗДАНИЯ ИЗОБРЕТЕНИЯ
[0010] Высокопроизводительные скрининги (HTS) являются краеугольным камнем процесса поиска фармацевтических лекарственных средств (J. R. Broach, J. Thorner, Nature 384 (Suppl), 14-16 (1996), Pereira, J. A. Williams, Br. J. Pharmacol. 152, 53-61 (2007)). Однако традиционные HTS имеют по меньшей мере два основных ограничения. Во-первых, считывания большинства из них ограничены совокупными клеточными фенотипами, например пролиферацией (D. Shum et al., J. Enzyme Inhib. Med. Chem. 23, 931-945 (2008), C. Yu et al., Nat. Biotechnol. 34, 419-423 (2016)), морфологией (Z. E. Perlman et al., Science 306, 1194-1198 (2004), Y. Futamura et al., Chem. Biol. 19, 1620-1630 (2012)) или высокоспецифичным молекулярным считыванием (J. Kang et al., Nat. Biotechnol. 34, 70-77 (2016), K. L. Huss, P. E. Blonigen, R. M. Campbell, J. Biomol. Screen. 12, 578-584 (2007)). Как правило, упускаются тонкие изменения в клеточном состоянии или экспрессии генов, которые в ином случае могли бы пролить свет на механизмы или выявить нецелевые эффекты.
[0011] Во-вторых, даже при выполнении HTS в сочетании с более комплексным молекулярным фенотипированием, таким как транскрипционное профилирование (C. Ye et al., Nat. Commun. 9, 4307 (2018), E. C. Bush et al., Nat. Commun. 8, 105 (2017), A. Subramanian et al., Cell 171, 1437-1452.e17 (2017), J. Lamb et al., Science 313, 1929-1935 (2006)), ограничения массового анализа заключаются в том, что даже явно относящиеся к одному «типу» клетки могут демонстрировать гетерогенные ответы (M. B. Elowitz, A. J. Levine, E. D. Siggia, P. S. Swain, Science 297, 1183-1186 (2002), C. Trapnell, Genome Res. 25, 1491-1498 (2015)). Такая клеточная гетерогенность может быть особенно актуальна в условиях in vivo. Например, все еще по большей части неизвестно, обусловлена ли выживаемость редких субпопуляций клеток при воздействии химиотерапевтических препаратов их генетикой, эпигенетическим состоянием или каким-либо другим аспектом (S. M. Shaffer et al., Nature 546, 431-435 (2017), S. L. Spencer, S. Gaudet, J. G. Albeck, J. M. Burke, P. K. Sorger, Nature 459, 428-432 (2009)). Кроме того, разброс и уровни технического шума часто затрудняют извлечение биологически значимой информации.
[0012] В принципе, секвенирование транскриптомов одиночных клеток (scRNA-seq) представляет собой форму многопараметрического молекулярного фенотипирования, которое могло бы преодолеть оба ограничения. Однако затраты в расчете на каждый образец и на каждую клетку для большинства технологий scRNA-seq остаются высокими, что не позволяет проводить скрининги даже в умеренных масштабах. Недавно в ряде групп были разработаны способы «хеширования клеток», в которых клетки из разных образцов молекулярно маркируют и смешивают перед анализом scRNA-seq. Однако современные подходы к хешированию требуют относительно дорогих реагентов (например, антител (M. Stoeckius et al., Genome Biol. 19, 224 (2018)) или химически-модифицированных ДНК-олигонуклеотидов (J. Gehring, J. H. Park, S. Chen, M. Thomson, L. Pachter, bioRxiv 315333 [Preprint] 5 May 2018. doi.org/10.1101/315333, C. S. McGinnis et al., Nat. Methods 16, 619-626 (2019)), использования протоколов, зависящих от типа клеток (D. Shin, W. Lee, J. H. Lee, D. Bang, Sci. Adv. 5, eaav2249 (2019)), и/или использования платформ scRNA-seq с высокой стоимостью в расчете на клетку.
[0013] ИЗЛОЖЕНИЕ СУЩНОСТИ ЗАЯВКИ
[0014] Секвенирование одиночных клеток и секвенирование одиночных ядер при большом количестве клеток способами комбинаторного индексного секвенирования одиночных клеток (sci-) продемонстрировало свою эффективность в разделении популяций в клетках и сложных тканях по транскриптомам, доступности хроматина, мутационным различиям и другим различиям. В одном способе, описанном в настоящем документе, при хешировании ядер или хешировании клеток используют хеширующие олигонуклеотиды в целях повышения производительности анализа образцов и увеличения обнаружения дублетов при высоких частотах коллизий. В другом способе, описанном в настоящем документе, нормализующем хешировании, используют нормализующие олигонуклеотиды в качестве стандарта для упрощения оценки и удаления технического шума межклеточных вариаций и для повышения чувствительности и специфичности.
[0015] В настоящем документе предложены способы получения библиотеки секвенирования. В одном из вариантов осуществления библиотека включает в себя нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, и способ включает в себя получение множества клеток в первом множестве компартментов и приведение ядер, выделенных из клеток каждого компартмента, или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом для получения хешированных ядер или хешированных клеток. В одном из вариантов осуществления по меньшей мере одна копия хеширующего олигонуклеотида связывается с выделенными ядрами или клетками. В одном из вариантов осуществления хеширующий олигонуклеотид включает в себя хеширующий индекс. В одном из вариантов осуществления хеширующий индекс в каждом компартменте включает в себя индексную последовательность, которая отличается от индексных последовательностей в других компартментах. Связь между хеширующим олигонуклеотидом и выделенными ядрами или клетками может быть неспецифической, например, посредством абсорбции. Способ может дополнительно включать в себя объединение хешированных ядер или хешированных клеток разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток. В одном из вариантов осуществления способ может дополнительно включать в себя воздействие заданного условия на множество клеток каждого компартмента. Воздействие заданного условия может происходить в любой момент способа и в одном из вариантов осуществления происходит до приведения в контакт.
[0016] В одном из вариантов осуществления способ может необязательно включать в себя обработку объединенных хешированных клеток или объединенных хешированных ядер с использованием способа комбинаторного индексирования одиночных клеток для получения библиотеки секвенирования, содержащей нуклеиновые кислоты, из множества одиночных ядер. Примеры подходящих для использования способов комбинаторного индексирования одиночных клеток включают в себя, без ограничений, секвенирование транскриптомов одиночных ядер, секвенирование транскриптомов одиночных клеток и секвенирование доступного для транспозона хроматина, полногеномное секвенирование в одиночных ядрах, секвенирование доступного для транспозона хроматина в одиночных ядрах, sci-HiC, DRUG-seq, sci-CAR, sci-MET, sci-Crop, sci-perturb или sci-Crispr.
[0017] Также в настоящем описании предложен способ нормализации библиотеки секвенирования. В одном из вариантов осуществления библиотека секвенирования включает в себя нуклеиновые кислоты из множества одиночных ядер или одиночных клеток. В одном из вариантов осуществления способ включает в себя получение первого множества компартментов, включающих в себя выделенные ядра или клетки, и приведение выделенных ядер или клеток каждого компартмента в контакт с популяциями нормализующих олигонуклеотидов, причем члены каждой популяции нормализующих олигонуклеотидов связаны с выделенными ядрами или клетками. В одном из вариантов осуществления приведение в контакт происходит до распределения изолированных ядер или клеток по компартментам. Нормализующие олигонуклеотиды могут быть связаны с выделенными ядрами или клетками до компартментализации или после компартментализации. Связь между нормализующим олигонуклеотидом и выделенными ядрами или клетками может быть неспецифической, например, посредством абсорбции. Способ может дополнительно включать в себя объединение меченых ядер или меченых клеток разных компартментов для получения объединенных меченых ядер или объединенных меченых клеток. В одном из вариантов осуществления способ может дополнительно включать в себя воздействие заданного условия на множество клеток каждого компартмента. Воздействие заданного условия может происходить в любой момент способа и в одном из вариантов осуществления происходит до приведения в контакт.
[0018] Определения
[0019] Если не указано иное, следует понимать, что термины, используемые в настоящем документе, принимают свое обычное значение в соответствующей области. Ниже приведен ряд терминов, используемых в настоящем документе, и их значения.
[0020] В настоящем документе термины «организм» и «субъект» используются взаимозаменяемо и относятся к микроорганизмам (например, прокариотическим или эукариотическим), животным и растениям. Примером животного является млекопитающее, такое как человек.
[0021] В настоящем документе термин «тип клеток» предназначен для обозначения клеток на основе морфологии, фенотипа, источника развития или других известных или распознаваемых отличительных характеристик клеток. Из одного организма (или одного вида организмов) можно получить множество разных типов клеток. К примерам типов клеток относятся, без ограничений, гаметы (включая женские гаметы, например, яйцеклетки или оотиды, и мужские гаметы, например, сперма), эпителий яичников, фибробласты яичников, яичек, мочевого пузыря, эпителий поджелудочной железы, альфа-клетки поджелудочной железы, иммунные клетки, В-клетки, Т-клетки, естественные киллерные клетки, дендритные клетки, раковые клетки, эукариотические клетки, стволовые клетки, клетки крови, мышечные клетки, жировые клетки, клетки кожи, нервные клетки, костные клетки, бета-клетки поджелудочной железы, эндотелиальные клетки поджелудочной железы, В-лимфобласты костного мозга, макрофаги костного мозга, эритробласты костного мозга, дендритные клетки костного мозга, адипоциты костного мозга, остеоциты костного мозга, хондроциты костного мозга, промиелобласты, мегакариобласты костного мозга, клетки мочевого пузыря, В-лимфоциты головного мозга, глиальные клетки головного мозга, нейроны, астроциты головного мозга, нейроэктодерма, макрофаги головного мозга, микроглию головного мозга, эпителий головного мозга, кортикальные нейроны, фибробласты головного мозга, эпителий молочной железы, эпителий толстой кишки, В-лимфоциты толстой кишки, эпителий молочной железы, миоэпителий молочной железы, фибробласты молочной железы, энтероциты толстой кишки, эпителий шейки матки, эпителий протоков молочной железы, эпителия языка, дендритные клетки миндалин, B-лимфоциты миндалин, лимфобласты периферической крови, Т-лимфобласты периферической крови, кожные Т-лимфоциты периферической крови, естественные киллеры периферической крови, В-лимфобласты периферической крови, моноциты периферической крови, миелобласты периферической крови, монобласты периферической крови, промиелобласты периферической крови, макрофаги периферической крови, базофилы периферической крови, эндотелий печени, тучные клетки печени, эпителий печени, B-лимфоциты печени, эндотелий селезенки, эпителий селезенки, B-лимфоциты селезенки, гепатоциты печени, печень, фибробласты, эпителий легких, эпителий бронхов, фибробласты легких, В-лимфоциты легких, шванновские клетки легких, плоские клетки легких, макрофаги легких, остеобласты легких, нейроэндокринные клетки, альвеолярные клетки легких, эпителий желудка и фибробласты желудка.
[0022] В настоящем документе термин «ткань» обозначает набор или агрегат клеток, которые действуют совместно и выполняют одну или более конкретных функций в организме. Клетки необязательно могут быть морфологически аналогичными. К примерам тканей относятся, без ограничений, эмбриональные ткани, придаток яичка, глаз, мышцы, кожа, сухожилие, вена, артерия, кровь, сердце, селезенка, лимфатический узел, кость, костный мозг, легкое, бронхи, трахея, кишечник, тонкая кишка, толстый кишечник, ободочная кишка, прямая кишка, слюнная железа, язык, желчный пузырь, аппендикс, печень, поджелудочная железа, головной мозг, желудок, кожа, почка, мочеточник, мочевой пузырь, уретра, гонада, яичко, яичник, матка, фаллопиевы трубы, тимус, гипофиз, щитовидная железа, надпочечник или паращитовидная железа. Ткань может быть получена из любых из различных органов человека или другого организма. Ткань может быть здоровой тканью или нездоровой тканью. К примерам нездоровых тканей относятся, без ограничений, злокачественные образования репродуктивной ткани, легких, молочной железы, ободочной и прямой кишки, предстательной железы, носоглотки, желудка, яичек, кожи, нервной системы, костей, яичников, печени, гематологических тканей, поджелудочной железы, матки, почек, лимфоидных тканей и т. д. Злокачественные образования могут относиться к различным гистологическим подтипам, например, к карциноме, аденокарциноме, саркоме, фиброаденокарциноме, нейроэндокринным или недифференцированным образованиям.
[0023] В настоящем документе термин «компартмент» обозначает область или объем, который отделяет или изолирует что-либо от других объектов. К примерам компартментов относятся, без ограничений, флаконы, пробирки, лунки, капли, болюсы, гранулы, сосуды, поверхностные элементы или области или объемы, разделенные физическими силами, такими как поток текучей среды, магнетизм, электрический ток или т. п. В одном из вариантов осуществления компартмент представляет собой лунку многолуночного планшета, такого как 96-луночный или 384-луночный планшет. В настоящем документе капля может включать в себя гидрогелевую гранулу, которая представляет собой гранулу для инкапсулирования одного или более ядер или клеток, и содержит гидрогелевую композицию. В некоторых вариантах осуществления капля представляет собой гомогенную каплю гидрогелевого материала или полую каплю, имеющую полимерную гидрогелевую оболочку. Гомогенная или полая капля может быть способна инкапсулировать одно или более ядер или клеток.
[0024] В настоящем документе термин «транспосомный комплекс» относится к интегрирующему ферменту и нуклеиновой кислоте, включающей в себя сайт распознавания интеграции. «Транспосомный комплекс» представляет собой функциональный комплекс, образованный транспозазой и сайтом распознавания транспозазы, который способен катализировать реакцию транспонирования (см., например, Gunderson et al., WO 2016/130704). К примерам интегрирующих ферментов относятся, без ограничений, интеграза или транспозаза. К примерам сайтов распознавания интеграции относится, без ограничений, сайт распознавания транспозазы.
[0025] Предполагается, что в настоящем документе термин «нуклеиновая кислота» соответствует его использованию в данной области и включает в себя нуклеиновые кислоты природного происхождения или их функциональные аналоги. Особенно подходящие для использования функциональные аналоги способны гибридизоваться с нуклеиновой кислотой специфичным для последовательности образом или могут использоваться в качестве матрицы для репликации конкретной нуклеотидной последовательности. Нуклеиновые кислоты природного происхождения обычно имеют каркас, содержащий фосфодиэфирные связи. Структура аналога может иметь альтернативную каркасную связь, в том числе любую из множества известных в данной области. Нуклеиновые кислоты природного происхождения обычно содержат сахар дезоксирибозу (например, присутствующий в дезоксирибонуклеиновой кислоте (ДНК)) или сахар рибозу (например, присутствующий в рибонуклеиновой кислоте (РНК)). Нуклеиновая кислота может содержать любой из множества аналогов этих остатков сахаров, известных в данной области. Нуклеиновая кислота может включать в себя нативные или ненативные основания. В связи с этим нативная дезоксирибонуклеиновая кислота может содержать одно или более оснований, выбранных из группы, состоящей из аденина, тимина, цитозина или гуанина, и рибонуклеиновая кислота может иметь одно или более оснований, выбранных из группы, состоящей из аденина, урацила, цитозина или гуанина. Подходящие для использования ненативные основания, которые могут быть включены в нуклеиновую кислоту, известны в данной области. Примеры ненативных оснований включают в себя запертую нуклеиновую кислоту (ЗНК), мостиковую нуклеиновую кислоту (МНК) и псевдокомплементарные основания (Trilink Biotechnologies, г. Сан-Диего, штат Калифорния, США). Основания ЗНК и МНК можно встраивать в олигонуклеотид ДНК и повышать прочность и специфичность гибридизации олигонуклеотида. Основания ЗНК и МНК и варианты использования таких оснований известны специалисту в данной области и являются стандартными.
[0026] В настоящем документе термин «целевая» при использовании применительно к нуклеиновой кислоте служит семантическим идентификатором нуклеиновой кислоты в контексте способа или композиции, описанных в настоящем документе, и не обязательно ограничивает структуру или функцию нуклеиновой кислоты теми, которые явно указаны. Целевая нуклеиновая кислота может представлять собой по существу любую нуклеиновую кислоту с известной или неизвестной последовательностью. Это может быть, например, фрагмент геномной ДНК (например, хромосомная ДНК), внехромосомная ДНК, такая как плазмида, бесклеточная ДНК, РНК (например, мРНК), белки (например, белки клетки или клеточной поверхности) или кДНК. Секвенирование может приводить к определению последовательности всей целевой молекулы или ее части. Цели могут быть получены из первичного образца нуклеиновой кислоты, такого как ядро. В одном из вариантов осуществления цели можно превращать в матрицы, подходящие для амплификации, путем размещения универсальных последовательностей на конце или на концах каждого целевого фрагмента. Цели также можно получать из образца первичной РНК посредством обратной транскрипции в кДНК. В одном из вариантов осуществления термин «целевой» используется применительно к подгруппе ДНК, РНК или белков, присутствующих в клетке. Для направленного секвенирования используют отбор и выделение интересующих генов, или областей, или белков, как правило, путем ПЦР-амплификации (например, специфичных для области праймеров) или способа захвата на основе гибридизации или использования антител. Целенаправленное обогащение может происходить на различных стадиях способа. Например, целенаправленное представление РНК можно получить с использованием специфичных к цели праймеров на стадии обратной транскрипции или путем обогащения на основе гибридизации подгруппы из более сложной библиотеки. Примером является секвенирование экзома или анализ L1000 (Subramanian et al., 2017, Cell, 171;1437-1452). Целенаправленное секвенирование может включать в себя любой из процессов обогащения, известных специалисту в данной области.
[0027] В настоящем документе термин «универсальный» при использовании для описания нуклеотидной последовательности относится к области последовательности, которая является общей для двух или более молекул нуклеиновых кислот или образцов, причем молекулы также имеют области последовательности, которые отличаются друг от друга. Универсальная последовательность, присутствующая в разных членах набора молекул, может обеспечивать захват множества разных нуклеиновых кислот с использованием популяции универсальных захватывающих нуклеиновых кислот, например, захватывающих олигонуклеотидов, комплементарных участку универсальной последовательности, например, универсальной последовательности захвата. Не имеющие ограничительного характера примеры универсальных последовательностей захвата включают в себя последовательности, идентичные или комплементарные праймерам P5 и P7. Аналогичным образом, универсальная последовательность, присутствующая в разных членах набора молекул, может обеспечивать репликацию (например, секвенирование) или амплификацию множества разных нуклеиновых кислот с использованием популяции универсальных праймеров, которые комплементарны участку универсальной последовательности, например, универсальной якорной последовательности. В одном из вариантов осуществления в качестве сайта, с которым отжигается универсальный праймер (например, праймер секвенирования для чтения 1 или чтения 2) для секвенирования, используют универсальные якорные последовательности. Таким образом, захватный олигонуклеотид или универсальный праймер включает в себя последовательность, которая может специфически гибридизоваться с универсальной последовательностью.
[0028] Термины «P5» и «P7» можно использовать, когда речь идет об универсальной последовательности захвата или захватном олигонуклеотиде. Термины «P5’» («P5-штрих») и «P7’» («P7-штрих») относятся к последовательности, комплементарной P5 и P7, соответственно. Следует понимать, что в способах, предложенных в настоящем документе, можно использовать любую подходящую универсальную последовательность захвата или захватный олигонуклеотид и что использование праймеров P5 и P7 представляет собой только примеры осуществления. Способы использования захватывающих олигонуклеотидов, таких как Р5 и Р7, или комплементарных им последовательностей на проточных кюветах известны в данной области и описаны в качестве примера в публикациях WO 2007/010251, WO 2006/064199, WO 2005/065814, WO 2015/106941, WO 1998/044151 и WO 2000/018957. Например, в способах, представленных в настоящем документе, для гибридизации с комплементарной последовательностью и амплификации последовательности может использоваться любой подходящий прямой праймер для амплификации, иммобилизованный или в виде раствора. Аналогичным образом, в способах, представленных в настоящем документе, для гибридизации с комплементарной последовательностью и амплификации последовательности может использоваться любой подходящий обратный праймер для амплификации, иммобилизованный или в виде раствора. Специалистам в данной области будет понятно, как создать и использовать последовательности праймеров, которые подходят для захвата и/или амплификации нуклеиновых кислот в соответствии с описанием в настоящем документе.
[0029] В настоящем документе термин «праймер» и его производные по существу относятся к любой нуклеиновой кислоте, которая может гибридизоваться с интересующей целевой последовательностью. Как правило, праймер функционирует в качестве субстрата, на котором можно полимеризовать нуклеотиды с помощью полимеразы или с которым можно лигировать нуклеотиды; однако в некоторых вариантах осуществления праймер может встраиваться в цепь синтезированной нуклеиновой кислоты и обеспечивать сайт, с которым может гибридизоваться другой праймер для праймирования синтеза новой цепи, которая комплементарна синтезированной молекуле нуклеиновой кислоты. Праймер может включать в себя любую комбинацию нуклеотидов или их аналогов. В некоторых вариантах осуществления праймер представляет собой одноцепочечный олигонуклеотид или полинуклеотид. Термины «полинуклеотид» и «олигонуклеотид» в настоящем документе используются взаимозаменяемо для обозначения полимерной формы нуклеотидов любой длины и могут включать в себя рибонуклеотиды, дезоксирибонуклеотиды, их аналоги или их смеси. Следует понимать, что термины в качестве эквивалентов включают в себя аналоги ДНК, РНК, кДНК или антительно-олигонуклеотидные конъюгаты, полученные из нуклеотидных аналогов и применимые к одноцепочечным (таким как смысловые или антисмысловые) и двухцепочечным полинуклеотидам. Термин, используемый в настоящем документе, также охватывает кДНК, являющуюся комплементарной или являющуюся копией ДНК, полученной с РНК-матрицы, например, под действием обратной транскриптазы. Данный термин относится только к первичной структуре молекулы. Таким образом, термин включает в себя трех-, двух- и одноцепочечную дезоксирибонуклеиновую кислоту (ДНК), а также трех-, двух- и одноцепочечную рибонуклеиновую кислоту (РНК).
[0030] В настоящем документе термин «адаптер» и его производные, например, «универсальный адаптер», по существу относится к любому линейному олигонуклеотиду, который может быть присоединен к молекуле нуклеиновой кислоты изобретения. В некоторых вариантах осуществления адаптер по существу не комплементарен 3’-концу или 5’-концу любой целевой последовательности, присутствующей в образце. В некоторых вариантах осуществления подходящие длины адаптеров находятся в диапазоне около 10-100 нуклеотидов, около 12-60 нуклеотидов или около 15-50 нуклеотидов. Как правило, адаптер может включать в себя любую комбинацию нуклеотидов и/или нуклеиновых кислот. В некоторых аспектах адаптер может включать в себя одну или более расщепляемых групп в одном или более положениях. В другом аспекте адаптер может включать в себя последовательность, по существу идентичную или по существу комплементарную по меньшей мере участку праймера, например универсального праймера. В некоторых вариантах осуществления адаптер может включать в себя штрихкод (также называемый в настоящем документе меткой или индексом) для помощи в коррекции нижележащих ошибок, идентификации или секвенировании. Термины «адаптор» и «адаптер» используются на взаимозаменяемой основе.
[0031] В настоящем документе термин «каждый» применительно к набору элементов предназначен для обозначения отдельного элемента в наборе, но не обязательно относится к каждому элементу в наборе, если только иное явно не определяется контекстом.
[0032] В настоящем документе термин «транспорт» относится к движению молекулы через текучую среду. Термин может включать в себя пассивный транспорт, такой как движение молекул по градиенту их концентрации (например, пассивную диффузию). Термин также может включать в себя активный транспорт, при котором молекулы могут двигаться по их градиенту концентрации или против их градиента концентрации. Таким образом, транспорт может включать в себя подачу энергии для перемещения одной или более молекул в желаемом направлении или в желаемое место, например к сайту амплификации.
[0033] В настоящем документе термин «амплификация», «амплифицировать» или «реакция амплификации» и их производные по существу относятся к любому действию или процессу, в котором по меньшей мере участок молекулы нуклеиновой кислоты реплицируется или копируется в по меньшей мере одну дополнительную молекулу нуклеиновой кислоты. Дополнительная молекула нуклеиновой кислоты необязательно включает в себя последовательность, по существу идентичную или по существу комплементарную по меньшей мере некоторому участку матричной молекулы нуклеиновой кислоты. Матричная молекула нуклеиновой кислоты может быть одноцепочечной или двухцепочечной, а дополнительная молекула нуклеиновой кислоты может независимо быть одноцепочечной или двухцепочечной. Амплификация необязательно включает в себя линейную или экспоненциальную репликацию молекулы нуклеиновой кислоты. В некоторых вариантах осуществления такая амплификация может выполняться с использованием изотермических условий; в других вариантах осуществления такая амплификация может включать в себя термоциклирование. В некоторых вариантах осуществления амплификация представляет собой мультиплексную амплификацию, которая включает в себя одновременную амплификацию множества целевых последовательностей в одной реакции амплификации. В некоторых вариантах осуществления термин «амплификация» включает в себя амплификацию по меньшей мере некоторого участка нуклеиновых кислот на основе ДНК и РНК по отдельности или в комбинации. Реакция амплификации может включать в себя любой из процессов амплификации, известных специалисту в данной области. В некоторых вариантах осуществления реакция амплификации включает в себя полимеразную цепную реакцию (ПЦР).
[0034] В настоящем документе термин «условия амплификации» и его производные по существу относятся к условиям, подходящим для амплификации одной или более нуклеотидных последовательностей. Такая амплификация может быть линейной или экспоненциальной. В некоторых вариантах осуществления условия амплификации могут включать в себя изотермические условия или альтернативно могут включать в себя условия термоциклирования или комбинацию изотермических условий и условий термоциклирования. В некоторых вариантах осуществления условия, подходящие для амплификации одной или более нуклеотидных последовательностей, включают в себя условия полимеразной цепной реакции (ПЦР). Как правило, условия амплификации относятся к реакционной смеси, которая является достаточной для амплификации нуклеиновых кислот, например, одной или более целевых последовательностей, фланкированных универсальной последовательностью, или для амплификации амплифицированной целевой последовательности, лигированной с одним или более адаптерами. Как правило, условия амплификации включают в себя катализатор для амплификации или для синтеза нуклеиновых кислот, например полимеразу; праймер, который обладает некоторой степенью комплементарности с подлежащей амплификации нуклеиновой кислотой; и нуклеотиды, такие как дезоксирибонуклеотидтрифосфаты (дНТФ), для стимулирования удлинения праймера после гибридизации с нуклеиновой кислотой. Условия амплификации могут потребовать гибридизации или отжига праймера с нуклеиновой кислотой, удлинения праймера и стадии денатурации, на которой удлиненный праймер отделяют от нуклеотидной последовательности, подвергающейся амплификации. Как правило, но не обязательно условия амплификации могут включать в себя термоциклирование; в некоторых вариантах осуществления условия амплификации включают в себя множество циклов, на которых повторяются стадии отжига, удлинения и разделения. Как правило, условия амплификации включают в себя катионы, такие как Mg2+ или Mn2+, и также могут включать в себя различные модификаторы ионной силы.
[0035] В настоящем документе термин «реамплификация» и его производные относятся по существу к любому процессу, в котором по меньшей мере участок молекулы амплифицированной нуклеиновой кислоты дополнительно амплифицируют посредством любого подходящего процесса амплификации (называемого в некоторых вариантах осуществления «вторичной» амплификацией), в результате чего получается реамплифицированная молекула нуклеиновой кислоты. Вторичная амплификация не обязательно должна быть идентична исходному процессу амплификации, в котором была получена амплифицированная молекула нуклеиновой кислоты; не обязательно, чтобы молекула реамплифицированной нуклеиновой кислоты была полностью идентична или полностью комплементарна молекуле амплифицированной нуклеиновой кислоты; все, что необходимо, - это чтобы молекула реамплифицированной нуклеиновой кислоты включала в себя по меньшей мере участок молекулы амплифицированной нуклеиновой кислоты или комплементарную ей последовательность. Например, реамплификация может предполагать использование других условий амплификации и/или других праймеров, включая праймеры, специфичные для иной цели, чем первичная амплификация.
[0036] В настоящем документе термин «полимеразная цепная реакция» (ПЦР) относится к способу Mullis из патентов США №№ 4,683,195 и 4,683,202, который описывает способ повышения концентрации сегмента интересующего полинуклеотида в смеси геномной ДНК без клонирования или очистки. Данный процесс амплификации интересующего полинуклеотида состоит из введения большого избытка двух олигонуклеотидных праймеров в смесь ДНК, содержащую желаемый интересующий полинуклеотид, с последующей серией термоциклирования в присутствии ДНК-полимеразы. Два праймера комплементарны соответствующим цепям интересующего двухцепочечного полинуклеотида. Сначала смесь денатурируют при повышенной температуре, а затем праймеры отжигают с комплементарными последовательностями внутри интересующей молекулы полинуклеотида. После отжига праймеры удлиняют полимеразой с образованием новой пары комплементарных цепей. Стадии денатурации, отжига с праймером и достройки полимеразой можно повторять множество раз (что называется термоциклированием) для получения высокой концентрации амплифицированного сегмента желаемого интересующего полинуклеотида. Длину амплифицированного сегмента (ампликона) интересующего желаемого полинуклеотида определяют по относительным положениям праймеров относительно друг друга, и, следовательно, эта длина является контролируемым параметром. В силу повторения этого процесса способ называют ПЦР. Поскольку желаемые амплифицированные сегменты интересующего полинуклеотида становятся преобладающими нуклеотидными последовательностями (с точки зрения концентрации) в смеси, говорят, что они «ПЦР-амплифицированы». В одной из модификаций описанного выше способа целевые молекулы нуклеиновой кислоты можно амплифицировать с помощью ПЦР, используя множество разных пар праймеров, в некоторых случаях - одну или более пар праймеров, на интересующую целевую молекулу нуклеиновой кислоты, таким образом образуя мультиплексную ПЦР-реакцию.
[0037] В настоящем документе термин «мультиплексная амплификация» относится к избирательной и неслучайной амплификации двух или более целевых последовательностей в образце с использованием по меньшей мере одного специфичного для цели праймера. В некоторых вариантах осуществления мультиплексную амплификацию выполняют таким образом, чтобы некоторые или все из целевых последовательностей амплифицировались в одном реакционном сосуде. «Плексия» или «плекс» заданной мультиплексной амплификации по существу относится к числу разных специфичных для цели последовательностей, которые амплифицируются в ходе этой одной мультиплексной амплификации. В некоторых вариантах осуществления плексия может составлять около 12-плекс, 24-плекс, 48-плекс, 96-плекс, 192-плекс, 384-плекс, 768-плекс, 1536-плекс, 3072-плекс, 6144-плекс или выше. Также существует возможность обнаруживать амплифицированные целевые последовательности посредством нескольких разных методологий (например, гель-электрофорез с последующей денситометрией, количественная оценка с помощью биоанализатора или количественной ПЦР, гибридизация с меченым зондом; включение биотинилированных праймеров с последующим обнаружением конъюгата авидина с ферментом; включение 32P-меченных дезоксинуклеотидтрифосфатов в амплифицированную целевую последовательность).
[0038] В настоящем документе термин «амплифицированные целевые последовательности» и его производные относится по существу к нуклеотидной последовательности, полученной амплификацией целевых последовательностей с использованием специфичных для цели праймеров и способов, предложенных в настоящем документе. Амплифицированные целевые последовательности могут быть либо смысловыми (т. е. положительная цепь), либо антисмысловыми (т. е. отрицательная цепь) по отношению к целевым последовательностям.
[0039] В настоящем документе термины «лигирование», «лигировать» и их производные по существу относятся к способу ковалентного связывания двух или более молекул друг с другом, например ковалентного связывания двух или более молекул нуклеиновых кислот друг с другом. В некоторых вариантах осуществления лигирование включает в себя соединение одноцепочечных разрывов между соседними нуклеотидами нуклеиновых кислот. В некоторых вариантах осуществления лигирование включает в себя образование ковалентной связи между концом первой и концом второй молекул нуклеиновой кислоты. В некоторых вариантах осуществления лигирование может включать в себя образование ковалентной связи между 5'-фосфатной группой одной нуклеиновой кислоты и 3'-гидроксильной группой второй нуклеиновой кислоты с образованием таким образом лигированной молекулы нуклеиновой кислоты. Как правило, для целей настоящего описания амплифицированная целевая последовательность может быть лигирована с адаптером для получения лигированной с адаптером амплифицированной целевой последовательности.
[0040] В настоящем документе термин «лигаза» и его производные по существу относятся к любому агенту, способному катализировать лигирование двух молекул субстрата. В некоторых вариантах осуществления лигаза включает в себя фермент, способный катализировать соединение одноцепочечных разрывов между соседними нуклеотидами нуклеиновой кислоты. В некоторых вариантах осуществления лигаза включает в себя фермент, способный катализировать образование ковалентной связи между 5'-фосфатом одной молекулы нуклеиновой кислоты и 3'-гидроксилом другой молекулы нуклеиновой кислоты с образованием таким образом лигированной молекулы нуклеиновой кислоты. Подходящие лигазы могут включать в себя, без ограничений, ДНК-лигазу Т4, РНК-лигазу Т4 и ДНК-лигазу E. coli.
[0041] В настоящем документе термин «условия лигирования» и его производные по существу относятся к условиям, подходящим для лигирования двух молекул друг с другом. В некоторых вариантах осуществления условия лигирования подходят для сшивания одноцепочечных разрывов или зазоров между нуклеиновыми кислотами. В настоящем документе термин «одноцепочечный разрыв» или «зазор» соответствует использованию данного термина в данной области. Как правило, одноцепочечный разрыв или зазор может быть лигирован в присутствии фермента, такого как лигаза, при подходящих температуре и pH. В некоторых вариантах осуществления ДНК-лигаза Т4 может соединять одноцепочечный разрыв между нуклеиновыми кислотами при температуре около 70-72°C.
[0042] В настоящем документе термин «проточная кювета» относится к камере, содержащей твердую поверхность, через которую могут протекать один или более жидких реагентов. Примеры проточных кювет и связанных с ними систем для работы с жидкостями и платформ для обнаружения, которые можно легко использовать в способах настоящего изобретения, описаны, например, в публикациях Bentley et al., Nature 456:53-59 (2008), WO 04/018497; US 7,057,026; WO 91/06678; WO 07/123744; US 7,329,492; US 7,211,414; US 7,315,019; US 7,405,281 и US 2008/0108082.
[0043] В настоящем документе термин «ампликон» применительно к нуклеиновой кислоте обозначает продукт копирования нуклеиновой кислоты, причем продукт имеет нуклеотидную последовательность, которая совпадает с по меньшей мере участком нуклеотидной последовательности нуклеиновой кислоты или комплементарна ему. Ампликон можно получать любым из разнообразных способов амплификации, в которых используют нуклеиновую кислоту или ее ампликон в качестве матрицы, включая, например, полимеразное удлинение, полимеразную цепную реакцию (ПЦР), амплификацию по типу катящегося кольца (RCA), удлинение с лигированием или цепную реакцию лигирования. Ампликон может представлять собой молекулу нуклеиновой кислоты, имеющую одну копию конкретной нуклеотидной последовательности (например, продукт ПЦР) или множество копий нуклеотидной последовательности (например, конкатамерный продукт RCA). Первый ампликон целевой нуклеиновой кислоты, как правило, представляет собой комплементарную копию. Последующие ампликоны представляют собой копии, которые формируют после создания первого ампликона из целевой нуклеиновой кислоты или из первого ампликона. Последующий ампликон может иметь последовательность, которая по существу комплементарна целевой нуклеиновой кислоте или по существу идентична целевой нуклеиновой кислоте.
[0044] В настоящем документе термин «сайт амплификации» относится к сайту в матрице или на ней, где можно создать один или более ампликонов. Сайт амплификации может быть дополнительно выполнен с возможностью содержания, удержания или прикрепления по меньшей мере одного ампликона, создаваемого в сайте.
[0045] В настоящем документе термин «матрица» относится к популяции сайтов, которые можно отличать друг от друга по их относительному положению. Разные молекулы, находящиеся на разных сайтах матрицы, можно отличать друг от друга в соответствии с положениями сайтов в матрице. Отдельный сайт матрицы может содержать одну или более молекул конкретного типа. Например, сайт может включать в себя одну целевую молекулу нуклеиновой кислоты, имеющую конкретную последовательность, или сайт может включать в себя несколько молекул нуклеиновой кислоты, имеющих одинаковую последовательность (и/или комплементарную ей последовательность). Сайты матрицы могут представлять собой разные элементы, расположенные на одном и том же субстрате. Примеры элементов включают в себя, без ограничений, лунки в субстрате, гранулы (или другие частицы) в субстрате или на нем, выступы из субстрата, ребра на субстрате или каналы в субстрате. Сайты матрицы могут представлять собой отдельные субстраты, каждый из которых несет свою молекулу. Разные молекулы, прикрепленные к отдельным субстратам, можно идентифицировать по положениям субстратов на поверхности, с которой связаны субстраты, или по положениям субстратов в жидкости или геле. Примеры матриц, в которых отдельные субстраты расположены на поверхности, включают в себя, без ограничений, те, которые имеют гранулы в лунках.
[0046] В настоящем документе термин «емкость» применительно к сайту и нуклеотидному материалу обозначает максимальное количество нуклеотидного материала, которое может занимать сайт. Например, термин может относиться к общему количеству молекул нуклеиновых кислот, которые могут занимать сайт в конкретном состоянии. Можно использовать и другие меры, включая, например, общую массу нуклеотидного материала или общее количество копий конкретной нуклеотидной последовательности, которая может занимать сайт в конкретном состоянии. Как правило, емкость сайта в отношении целевой нуклеиновой кислоты будет по существу эквивалентна емкости сайта в отношении ампликонов целевой нуклеиновой кислоты.
[0047] В настоящем документе термин «захватный агент» относится к материалу, химическому веществу, молекуле или их фрагменту, способному присоединять, удерживать или связывать целевую молекулу (например, целевую нуклеиновую кислоту). Примеры захватных агентов включают в себя, без ограничений, захватную нуклеиновую кислоту (также называемую в настоящем документе захватным олигонуклеотидом), которая комплементарна по меньшей мере участку целевой нуклеиновой кислоты, член пары связывания рецептор-лиганд (например, авидин, стрептавидин, биотин, лектин, углевод, белок, связывающийся с нуклеиновой кислотой, эпитоп, антитело и т. д.), который способен связываться с целевой нуклеиновой кислотой (или с присоединенной к ней линкерной функциональной группой), или химический реагент, способный образовывать ковалентную связь с целевой нуклеиновой кислотой (или присоединенным к ней линкерной функциональной группой).
[0048] В настоящем документе термин «репортерная функциональная группа» может относиться к любой идентифицируемой метке, маркеру, индексам, штрихкодам или группе, которая позволяет определять состав, идентичность и/или источник исследуемого аналита. В некоторых вариантах осуществления репортерная функциональная группа может включать в себя антитело, которое специфически связывается с белком. В некоторых вариантах осуществления антитело может включать в себя обнаруживаемую метку. В некоторых вариантах осуществления репортер может включать в себя антитело или аффинный реагент, меченный нуклеотидной меткой. Нуклеотидная метка может обнаруживаться, например, посредством лигирования на близком расстоянии (PLA) или удлинения на близком расстоянии (PEA) либо считывания на основе секвенирования (Shahi et al. Scientific Reports volume 7, номер изделия: 44447, 2017) или CITE-seq (Stoeckius et al. Nature Methods 14:865-868, 2017).
[0049] В настоящем документе термин «клональная популяция» относится к популяции нуклеиновых кислот, которая является гомогенной по отношению к конкретной нуклеотидной последовательности. Гомогенная последовательность, как правило, имеет длину по меньшей мере 10 нуклеотидов, но может быть даже длиннее, включая, например, по меньшей мере 50, 100, 250, 500 или 1000 нуклеотидов. Клональную популяцию можно получить из одной целевой нуклеиновой кислоты или матричной нуклеиновой кислоты. Как правило, все из нуклеиновых кислот в клональной популяции будут иметь одинаковую нуклеотидную последовательность. Следует понимать, что в клональной популяции может происходить небольшое количество мутаций (например, из-за артефактов амплификации), и это не будет отклонением от клональности.
[0050] В настоящем документе термин «обеспечение» в контексте композиции, изделия, нуклеиновой кислоты или ядра обозначает получение композиции, изделия, нуклеиновой кислоты или ядра, приобретение композиции, изделия, нуклеиновой кислоты или ядра или иное получение соединения, композиции, изделия или ядра.
[0051] В настоящем документе термин «индекс» (также называемый «индексной областью», «индексным адаптером», «меткой» или «штрихкодом») относится к уникальной нуклеотидной метке, которую можно использовать для идентификации образца или источника нуклеотидного материала. Если образцы нуклеиновых кислот получены из множества источников, нуклеиновые кислоты в каждом образце нуклеиновых кислот могут быть мечены разными нуклеотидными метками, так чтобы можно было идентифицировать источник образца. Можно использовать любой подходящий индекс или набор индексов, известный в данной области и показанный на примерах в описаниях патента США № 8,053,192, публикации PCT № WO 05/068656 и патентной публикации США № 2013/0274117. В некоторых вариантах осуществления индекс может включать в себя индексную последовательность из шести оснований Index 1 (i7), последовательность из восьми оснований Index 1 (i7), последовательность из восьми оснований Index 2 (i5e), последовательность из десяти оснований Index 1 (i7) или последовательность из десяти оснований Index 2 (i5) от компании Illumina, Inc. (г. Сан-Диего, штат Калифорния, США).
[0052] В настоящем документе термин «уникальный молекулярный идентификатор», или «UMI», относится к молекулярной метке, случайной, неслучайной или полуслучайной, которая может быть присоединена к молекуле нуклеиновой кислоты. Введенный в молекулу нуклеиновой кислоты UMI можно использовать для коррекции последующей систематической ошибки амплификации путем прямого подсчета уникальных молекулярных идентификаторов (UMI), секвенированных после амплификации.
[0053] Термин «и/или» обозначает один или все из перечисленных элементов или комбинацию из любых двух или более из перечисленных элементов.
[0054] Слова «предпочтительный» и «предпочтительно» относятся к вариантам осуществления изобретения, которые могут обеспечивать определенные преимущества при определенных обстоятельствах. Однако другие варианты осуществления также могут являться предпочтительными при тех же или других обстоятельствах. Более того, приведение в настоящем документе одного или более предпочтительных вариантов осуществления не означает, что другие варианты осуществления не являются полезными, и не предполагает исключение других вариантов осуществления из объема изобретения.
[0055] Термины «содержит» и их вариации не имеют ограничительного характера при употреблении этих терминов в описании и формуле изобретения.
[0056] Следует понимать, что везде, где варианты осуществления описаны в настоящем документе с использованием формулировки «включает в себя», «включать в себя», «включающий» и т. п., также предусмотрены иные аналогичные варианты осуществления, описанные с использованием терминов «состоящий из» и/или «состоящий по существу из».
[0057] Если не указано иное, термины «один» и «по меньшей мере один» используются взаимозаменяемо и обозначают «один или более одного».
[0058] Также в настоящем документе диапазоны числовых значений, указанные по конечным точкам, включают в себя все числовые значения, содержащиеся в пределах этого диапазона (например, диапазон от 1 до 5 включает в себя значения 1, 1,5, 2, 2,75, 3, 3,80, 4, 5 и т. д.).
[0059] Для любого способа, описанного в настоящем документе, который включает в себя отдельные стадии, можно выполнять эти стадии в любом осуществимом порядке. Кроме того, в соответствующих случаях можно одновременно выполнять любую комбинацию двух или более стадий.
[0060] В настоящем описании ссылка на «один вариант осуществления», «вариант осуществления», «определенные варианты осуществления» или «некоторые варианты осуществления» и т. д. обозначает, что конкретный признак, конфигурация, композиция или характеристика, описанные в связи с вариантом осуществления, включены в по меньшей мере один вариант осуществления изобретения. Таким образом, появление таких фраз в различных местах данного описания необязательно относится к одному и тому же варианту осуществления изобретения. Более того, конкретные признаки, конфигурации, композиции или характеристики можно комбинировать любым подходящим образом в одном или более вариантах осуществления.
[0061] КРАТКОЕ ОПИСАНИЕ ФИГУР
[0062] Приведенное ниже подробное описание иллюстративных вариантов осуществления настоящего изобретения лучше всего будет понятно при чтении совместно с приведенными ниже графическими материалами.
[0063] На ФИГ. 1 представлена общая блок-схема общего иллюстративного способа для одного из вариантов осуществления хеширования ядер или клеток в соответствии с настоящим описанием.
[0064] На ФИГ. 2 представлена общая блок-схема общего иллюстративного способа для одного из вариантов осуществления нормализующего хеширования в соответствии с настоящим описанием.
[0065] На ФИГ. 3 представлена общая блок-схема общего иллюстративного способа для одного из вариантов осуществления комбинаторного индексирования одиночных клеток с хешированием ядер в соответствии с настоящим описанием.
[0066] На ФИГ. 4 представлено использование в sci-PLEX полиаденилированных одноцепочечных олигонуклеотидов для мечения ядер, что позволяет выполнять хеширование клеток и обнаружение дублетов. (A) Флуоресцентные изображения пермеабилизованных ядер после инкубации с DAPl (вверху) и одноцепочечным дигонуклеотидом, конъюгированным с Alexa Fluor-647 (внизу). (B) Общий вид sci-Plex. Клетки, соответствующие разным воздействиям, лизируют в лунке и их ядра метят специфичными для лунки «хеширующими» олигонуклеотидами с последующей фиксацией, объединением и анализом sci-RNA-seq. (C) График рассеяния, отображающий количество UMI из транскриптомов одиночных клеток, полученных из смеси хешированных человеческих клеток HEK293T и мышиных клеток NIH3T3. Точки окрашены в зависимости от присвоенного хеширующего олигонуклеотида. (D) Коробчатая диаграмма, отображающая количество UMI мРНК, выделенных в расчете на клетку для свежих человеческих и мышиных клеточных линий в сравнении с замороженными. (E) График рассеяния в эксперименте с перегрузкой; оси такие же, как в (C). Выявленные олигонуклеотидные коллизии (красные) с высокой чувствительностью идентифицируют клеточные коллизии.
[0067] На ФИГ. 5 представлено хеширование короткими полиаденилированными одноцепочечными олигонуклеотидами, обеспечивающее стабильное недорогое мечение ядер для sci-RNA-seq и последующего обнаружения дублетов. A) Изображения, полученные с помощью флуоресцентной микроскопии, показывают отсутствие окрашивания конъюгированными с красителем Alexa 647 олигонуклеотидами (справа) непермеабилизованных клеток H3-GFP+ NIH3T3 (слева). B) Создание полиаденилированных хеширующих олигонуклеотидов (вверху) и индексированного праймера, используемого для обратной транскрипции (внизу). C) Выявленное количество UMI в расчете на клетку. Клетки с менее чем 10 хеш-UMI (красная линия) исключали из дальнейшего анализа. D) Распределение коэффициентов обогащения для клеток. Коэффициенты обогащения рассчитывали как отношение количества UMI для самого распространенного хеширующего олигонуклеотида к количеству UMI для второго по распространенности хеширующего олигонуклеотида. Для различения дублетов и синглетов использовали отсечку по коэффициенту обогащения 15 (красная линия). E) Коробчатая диаграмма количества клеток, выделенных на лунку для каждой клеточной линии. F) Расположение лунок планшета для культивирования, причем цвет указывает на количество выделенных клеток, а контур указывает на клеточную линию. Следует отметить, что, хотя в расчете на лунку было выделено больше клеток NIH3T3, в лунках клеток каждого типа было извлечено аналогичное количество клеток. G) Количества UMI в логарифмическом масштабе с агрегацией по генам и нормализацией по размерному коэффициенту, полученное при sci-RNA-seq на свежих препаратах в сравнении с замороженными. Размерные коэффициенты рассчитываются как логарифмические количества, наблюдаемые в одной клетке, поделенные на геометрическое среднее логарифмических количеств по всем измеренным клеткам. Черной линией обозначено y=x. Красная линия аппроксимирована с показанной корреляцией Пирсона. H) Коробчатая диаграмма количества хеш-UMI в логарифмическом масштабе, полученных при sci-RNA-seq в клетках HEK293t (человека) или NIH3T3 (мыши) из свежих препаратов в сравнении с замороженными. I) Теоретическая (красные столбцы) и наблюдаемая (черные точки для отдельных лунок и синие столбцы для средних) частота дублетов в зависимости от количества ядер, отсортированных в конечном планшете при sci-RNA-seq. J) График эксперимента «скотный двор» с Фиг. 1E после удаления дублетов, выявленных с помощью хеширования. K) Коробчатая диаграмма в логарифмическом масштабе количества РНК UMI в синглетных и дублетных клетках, определенного на основе чистоты хеш-UMI. Следует отметить, что это «внутривидовые дублеты», т. е. человек-человек или мышь-мышь, которые плохо обнаруживаются с помощью стандартных экспериментов типа «скотный двор».
[0068] На ФИГ. 6 представлено, что sci-Plex обеспечивает мультиплексную химическую транскриптомику с разрешением до одиночных клеток. (A) Диаграмма, на которой показаны соединения и соответствующие цели, проанализированные в пилотном эксперименте sci-Plex. Клетки легочной аденокарциномы A549 обрабатывали либо несущей средой [диметилсульфоксид (ДМСО) или этанол], либо одним из четырех соединений (BMS345541, дексаметазон, нутлин-3a или SAHA). (B) Вложение UMAP для химически возмущенных клеток А549 с цветовой кодировкой по лекарственному средству. (C) Вложение UMAP для химически возмущенных клеток А549, разделенных по обработкам с цветовой кодировкой дозы. (D и E) Экспрессия канонических (D) целевых генов, активируемых (ANGPTL4) и подавляемых (GDF15) глюкокортикоидным рецептором, в зависимости от дозы дексаметазона или (E) целевых генов p53 в зависимости от дозы нутлин-3a. Оси y демонстрируют процентное содержание клеток с по меньшей мере одним чтением, соответствующим транскрипту. (F) Оценка кривой «доза-ответ» для жизнеспособности клеток A549, обработанных BMS345541, дексаметазоном, нутлин-3a и SAHA, по относительному количеству клеток, выделенных при каждой дозе.
[0069] На ФИГ. 7 представлено, что анализ sci-Plex различает транскрипционные ответы клеток A549 на четыре низкомолекулярных соединения и получает оценки «доза-ответ», аналогичные признанным анализам. A) Схема эксперимента для клеток A549 в 96-луночных планшетах. Клетки обрабатывали в течение 24 часов в двух 96-луночных планшетах с использованием 7 доз (или несущей среды), распределенных вдоль каждой колонки. Сохраняли клетки, B) содержащие более 30 UMI хеширующих олигонуклеотидов и C) имеющие коэффициент обогащения более 10. D) Сохраненные клетки имели медианное количество хеш-UMI 78 и медианное количество РНК UMI 4681. E) Вложение UMAP для химически возмущенных клеток А549, эквивалентное Фиг. 2B, но цвет клеток указывает на то, были они обработаны несущей средой или одним из четырех низкомолекулярных соединений. F) Вложение UMAP для химически возмущенных клеток А549, эквивалентное Фиг. 2B, но клетки имеют цвет в зависимости от кластера, определенного по алгоритму пика плотности в Monocle 3. G) Рисунок, показывающий, как объединение ядер со штрихкодами сохраняет относительные количества клеток. H) Оценки жизнеспособности посредством подсчета доли выделенных хешированных ядер (серый) в сравнении с CellTiter-Glo (красный, n=6). I) График рассеяния прогнозируемых количеств клеток (ось х) и оценок жизнеспособности по CellTiter-Glo (ось y) для всех протестированных обработок и доз (коэффициент корреляции Пирсона и критерий хи-квадрат).
[0070] На ФИГ. 8 продемонстрировано, что дозозависимые дифференциально экспрессируемые гены (DEG) дают ожидаемые транскрипционные модули. A) Upset-диаграмма, отображающая пересечения дозозависимых DEG между обработками (вертикальные столбцы), а также общее количество дозозависимых DEG на обработку (горизонтальные столбцы). Ген определяется как дозозависимый DEG, если псевдопуассонова регрессионная модель, соотносящая экспрессию в данной клетке с дозой полученного клеткой лекарственного средства, демонстрирует значимый эффект дозы (критерий Вальда) после коррекции Бенджамини - Хохберга (FDR < 0,05). См. главу «Способы», где приведено полное описание регрессионного моделирования. Четыре крайних левых вертикальных столбца соответствуют специфичным для конкретного лекарственного средства дозозависимым DEG, а крайний правый вертикальный столбец соответствует дозозависимым DEG, общим для всех четырех лекарственных средств. B) Анализ набора генов (GSA), выполненный с дозозависимыми DEG с использованием функции runGSA(), полученной из пакета piano, и набора генов Hallmarks, полученного из MSigDB (45). Цвет тепловой карты указывает на значение из статистики направленного обогащения GSA со значениями, оканчивающимися на уровне -10 или +10 для визуализации.
[0071] На ФИГ. 9 продемонстрировано, что sci-Plex обеспечивает глобальное определение транскрипционного профиля для тысяч химических возмущений в одном эксперименте. (A) Схема крупномасштабного эксперимента sci-Plex (sci-RNA-seq3). Всего были протестированы 188 низкомолекулярных соединений с точки зрения эффектов на человеческие клеточные линии A549, K562 и MCF7 в четырех дозах и в биологической повторности после 24 часов обработки. Позиции доз и лекарственных средств в планшетах варьировали в разных повторах, и для каждого условия получали медианное значение от 100 до 200 клеток. Цвета обозначают клеточную линию, сигнальный путь для соединения и дозу. (B) Вложения UMAP для клеток А549, К562 и MCF7 в нашем скрининге, причем цвет каждой клетки определяется сигнальным путем, на который нацелено соединение, воздействующее на данную клетку. Для упрощения визуализации значимых молекулярных фенотипов мы добавляли прозрачность клеткам, обработанным соединением или комбинациями доз, которые заметно не изменяли распределение соответствующих клеток в пространстве UMAP по сравнению с контролем - несущей средой (точный критерий Фишера, FDR < 1%). (C) Оценки жизнеспособности, полученные при подсчете ядер на основе хеша при каждой дозе выбранных соединений (бозутиниб выделен красным текстом). Строки представляют дозы соединения, увеличивающиеся сверху вниз, а столбцы представляют отдельные соединения. На панели примечаний сверху показана в широком смысле клеточная активность, на которую нацеливается каждое соединение. (D) Вложения UMAP отмечены в зависимости от обработки ингибитором MEK траметинибом (красный), ингибитором HSP90 (фиолетовый) или контролем - несущей средой (серый). (E) Уровни экспрессии HSP90AA1 в клетках, подверженных воздействию увеличивающихся доз траметиниба. Оси y указывают на процентную долю клеток с по меньшей мере одним чтением, соответствующим транскрипту.
[0072] На ФИГ. 10 представлено мечение клеток на основе хеша в крупномасштабном эксперименте sci-Plex. A) Схема хеширования для sci-Plex с использованием 188 соединений. В эксперименте использовали 52 96-луночных планшета, в которых каждая лунка была маркирована комбинацией из двух олигонуклеотидов, один из которых специфичен для одного 96-луночного культурального планшета, а другой специфичен для лунки внутри этого культурального планшета. B) Хотя это теоретически можно осуществить с использованием лишь 96 луночных хеширующих олигонуклеотидов, мы использовали 768 лунок, и это означает, что из 39 936 возможных пар планшетных и луночных хеширующих олигонуклеотидов лишь небольшое количество (12,5%) комбинаций являются ожидаемыми («легальными»), в то время как большинство являются неожидаемыми («нелегальными»). C) Наблюдаемые пары планшетных и луночных хеширующих олигонуклеотидов были сильно обогащены «легальными» комбинациями. D) Диаграмма рассеяния клеток HEK293T и NIH3T3, высеянных в одну лунку ОТ, в крупномасштабном эксперименте sci-Plex. E-H) Отсечки по хеш-UMI (панели E и G) и коэффициенту обогащения (панели F и H), использованные для луночных хеширующих олигонуклеотидов (панели E и F) и планшетных хеширующих олигонуклеотидов (панели G и H). Отсечки по коэффициенту обогащения соответствуют более чем 5-кратному обогащению. Отсечки по хеш-UMI соответствуют > 5.
[0073] На ФИГ. 11 представлены параметры контроля качества в крупномасштабном эксперименте sci-Plex. A) Коробчатая диаграмма в логарифмическом масштабе количества РНК UMI для клеток, прошедших отсечки по хеш- и РНК UMI, для каждой из трех клеточных линий. B) Корреляция нормированных по размерному фактору количеств генов между повторностями для каждой из трех клеточных линий. Черной линией обозначено y=x. Красная линия аппроксимирована с показанной корреляцией Пирсона. C) Коробчатые диаграммы, отражающие количество клеток в группе несущей среды, полученное из каждой из 8 контрольных лунок с несущей средой в каждой повторности для клеток A549, K562 и MCF7.
[0074] На ФИГ. 12 представлено, что воздействие на клетки соединений изменяет их распределение по клеточным кластерам. Тепловая карта, демонстрирующая логарифмически преобразованное соотношение клеток, обработанных конкретным лекарственным средством, по сравнению с клетками с контролем - несущей средой в каждом Лувенском сообществе. Столбцы соответствуют кластерам в пространстве PCA (см. ФИГ. 13A-C), а строки соответствуют соединениям, аннотированным по сигнальному пути и цели. Серым цветом обозначено соединение, которое не является существенно обогащенным или обедненным по сравнению с несущей средой в соответствующем кластере (точный критерий Фишера, FDR < 1%).
[0075] На ФИГ. 13 представлено, как sci-Plex идентифицирует специфичное для пути обогащение по соединениям в кластерах UMAP. A-C) Вложение UMAP с Фиг. 3 B с цветами клеток в зависимости от их отнесения к Лувенским сообществам в пространстве PCA для клеток A549 (панель A), K562 (панель B) и MCF7 (панель C). D) Вложение UMAP для клеток А549 с Фиг. 3B. Клетки, обработанные агонистом глюкокортикоидного рецептора (GR) триамцинолона ацетонидом, выделены зеленым цветом, тогда как все остальные клетки окрашены серым цветом. Эти клетки составляют подавляющее большинство (95%) клеток в кластере 18 из панели A. E) Процент клеток A549, экспрессирующих целевые гены GR ANGPTL4 и GDF15, в зависимости от увеличения дозы синтетического агониста GR триамцинолона ацетонида. F-H) Вложение UMAP для клеток А549 с цветами клеток в зависимости от обработки различными дозами эпотилона A (F), эпотилона B (G) или с цветами в зависимости от индекса пролиферации (H). На вкладках представлены увеличенные изображения отдельных фокусов, индуцированных обработкой. Обработки с наибольшим количеством клеток в каждом ограничительном блоке приведены на панели H, причем количество клеток указано в скобках.
[0076] На ФИГ. 14 показано количество дозозависимых дифференциально экспрессируемых генов, обнаруженных для каждой категории соединений. Значимые дозозависимые дифференциально экспрессируемые гены (FDR < 0,05) сгруппированы по клеточным линиям и имеют цветовую кодировку по целевому сигнальному пути.
[0077] На Фиг. 15 представлена корреляция «псевдомассового» sci-Plex с массовым RNA-seq. A) Log10 транскриптов на миллион (TPM) для кодирующих белок генов, измеренные с помощью массового RNA-seq (оси x) в зависимости от нормализованных по размерному фактору агрегированных профилей одиночных клеток для клеток, обработанных несущей средой, по sci-Plex (ось y). Результаты приведены как для клеток A549, так и для клеток K562. Черной линией показана линия y=х, а синей линией показана линейная аппроксимация с показанной корреляцией Пирсона. B) Диаграммы рассеяния для выбранных соединений со сравнением статистически значимых оценок, полученных на основе аппроксимации линейными моделями данных по одиночным клеткам (оси x) в зависимости от оценок, полученных массовым RNA-seq с использованием DESeq2 (оси y). Черной линией обозначено y=x. Синей линией показана аппроксимация с показанной корреляцией Пирсона.
[0078] На ФИГ. 16 представлено, что модерированные Z-оценки в анализе L1000 коррелируют с дозозависимыми бета-значениями по sci-Plex. A) Для выбранной комбинации соединение - клеточная линия (трихостатин A в клетках MCF7) был построен график зависимости модерированных Z-оценок из анализа L1000 с обработкой в течение 24 часов каждой из восьми доз (оси y) (11) от дозозависимых бета-значений по данным sci-Plex (оси x). Показаны все гены, входящие в анализ L1000 и значимые по дозозависимым эффектам в sci-Plex (p-значение < 0,01). Линия представляет собой аппроксимацию с показанной корреляцией Спирмена. B) Коробчатая диаграмма корреляций Спирмена между значимыми вычисленными дозозависимыми бета-значениями по sci-Plex и модерированными Z-оценками L1000 из данных LINCS L1000 для измеренных генов при самой высокой дозе в клетках MCF7. Соединения представлены группами по сигнальным путям, на которые они нацелены. Красная точка соответствует флувестранту. C) Аналогично панели A, но для флувестранта в клетках MCF7 при наивысшей дозе (10 мкМ). D) Аналогично панели B, но для клеток A549. Красная точка соответствует триамциналона ацетониду. C) Аналогично панели A, но для триамциналона ацетонида в клетках A549 при наивысшей дозе (10 мкМ).
[0079] На ФИГ. 17 представлены результаты измерений одиночных клеток, показывающие изменение статуса пролиферации в клетках, обработанных несущей средой и каждой дозой каждого лекарственного средства. A-C) UMAP-проекция для A549(A), K562 (B) и MCF7 (C) с цветовой кодировкой по индексу пролиферации. Высокий индекс пролиферации указывает на увеличение совокупной экспрессии транскриптов, которые являются маркерами фазы G1/S или фазы G2/M (43). (D-F) График плотности распределения клеток по клеточному циклу для клеток, обработанных соединением (синяя заливка), или клеток, обработанных несущей средой (красная линия). Серая линия обозначает отсечку, используемую для различения пролиферирующих клеток (больше отсечки) и непролиферирующих клеток (меньше отсечки). G-I) Соотношение процентной доли клеток, обозначенных как низкопролиферирующие для каждой дозы каждого лекарственного средства (ось x), и медианной оценочной жизнеспособности этой комбинации (ось y). Каждая черная точка соответствует клеткам, обработанным той же дозой данного лекарственного средства. Красные точки обозначают обработку несущей средой. J) Вулканная диаграмма, показывающая log2-кратность изменения для значимых (q-значение < 0,01) дифференциально экспрессируемых генов между верхней и нижней фракциями клеток, обработанных несущей средой.
[0080] На ФИГ. 18 представлено, что измерения одиночных клеток позволяют оценить состояние пролиферации и жизнеспособность при использовании разных комбинаций лекарственное средство - доза. Тепловая карта, отражающая оценки относительной скорости пролиферации, процентной доли клеток, проявляющих низкий коэффициент пролиферации, и оценочной жизнеспособности для каждой пары соединение (строка) - доза (столбец).
[0081] На ФИГ. 19 представлено, что sci-Plex позволяет разграничить популяции пролиферирующих и непролиферирующих клеток. A) Схема показывает, что изменения клеточного состояния (вверху) и изменения относительных частот субпопуляций (внизу) выглядят идентичными при анализе образца на совокупные показатели, таком как массовая RNA-seq. Адаптировано из источника (14). B, C) Корреляции Пирсона между дозозависимыми размерами эффектов, оцененными при сравнении клеток с высоким и низким коэффициентом пролиферации для каждой клеточной линии (панель B) и класса лекарственных средств (панель C). D) Величины эффектов по генам оценивали на клетках с высоким (βdh) и низким (βdl) коэффициентами пролиферации для 4 выбранных соединений. Величины эффекта выражали в виде преобразованных к log2-кратностям изменений на отрезке. Показаны четыре класса генов: гены, значимые только в клетках с высоким коэффициентом пролиферации (зеленый); гены, значимые только в клетках с низким коэффициентом пролиферации (фиолетовый); в клетках как с высоким, так и с низким коэффициентом, а также с согласованными оценочными эффектами (красный); в клетках как с высоким, так и с низким коэффициентом, но с несогласованными оценочными эффектами (синий). Лекарственное средство имело согласованные дозозависимые эффекты на ген h в клетках с высоким (βdh) и низким (βdh) показателями, если | βdh - βdl | составляет менее 10 процентов от 1/2(| βdh | + βdl |). Черной линией обозначено y=x.
[0082] На ФИГ. 20 представлено, что скрининг с помощью sci-Plex позволяет определять жизнеспособность и сигнатуры экспрессии, которые воспроизводимы с помощью валидирующих экспериментов и ортогональных наборов данных. A) Оценки жизнеспособности для клеток К562 (красный), А549 (синий) и MCF7 (зеленый), которые были подвергнуты воздействию несущей среды или растущих доз ингибитора Src/Abl бозутиниба (n=6 повторностей в культуре, критерий суммы рангов Уилкоксона). Для каждой клеточной линии значения количества клеток нормализовали к среднему значению количества клеток в контроле - несущей среде. Планки погрешностей представляют стандартную ошибку среднего, n=8. B) Значения ЕС50 для клеточных линий гематопоэтического и лимфоидного происхождения, из легких или ткани молочной железы, для которых оценки жизнеспособности можно получить из публикации Cancer Cell Line Encyclopedia (CCLE), при воздействии ингибиторов Abl AZD0530 (левая панель) или нилотиниба (правая панель). C-E) Высшие баллы согласованности (мера, обобщающая сходства между сигнатурами транскрипции, индуцированными разными лекарственными средствами (11, 12)) для ингибиторов MEK и HSP из базы данных CMAP по всем клеточным линиям (сводные данные, панель C) или для клеток A549 (панель D) и MCF7 (панель E) по отдельности. Применена отсечка по баллам согласованности +/- 90, как в (11).
[0083] На ФИГ. 21 представлена корреляция обусловленных соединениями молекулярных сигнатур для клеток А549, выявленных при скрининге sci-Plex. Тепловая карта описывает корреляцию Пирсона для бета-коэффициентов у дозозависимых дифференциально экспрессируемых генов при каждой попарной комбинации соединений во время скрининга. Для упрощения визуализации корреляции Пирсона ограничили значением 0,6.
[0084] На ФИГ. 22 представлена корреляция обусловленных соединениями молекулярных сигнатур для клеток K562, выявленных при скрининге sci-Plex. Тепловая карта описывает корреляцию Пирсона для бета-коэффициентов у дозозависимых дифференциально экспрессируемых генов при каждой попарной комбинации соединений во время скрининга. Для упрощения визуализации корреляции Пирсона ограничили значением 0,6.
[0085] На ФИГ. 23 представлена корреляция обусловленных соединениями молекулярных сигнатур для клеток MCF7, выявленных при скрининге sci-Plex. Тепловая карта описывает корреляцию Пирсона для бета-коэффициентов у дозозависимых дифференциально экспрессируемых генов при каждой попарной комбинации соединений во время скрининга. Для упрощения визуализации корреляции Пирсона ограничили значением 0,6.
[0086] На ФИГ. 24 показаны кластерограммы корреляции молекулярных сигнатур, обусловленных соединениями. Кластерограммы, демонстрирующие корреляцию Пирсона бета-коэффициентов у дозозависимых дифференциально экспрессируемых генов для каждой попарной комбинации соединений при скрининге на клетках A549 (A), K562 (B) и MCF7 (C). Названия соединений имеют цветовой код в соответствии с целевым сигнальным путем.
[0087] На ФИГ. 25 представлено вложение UMAP для лекарственных средств на основе их дозозависимых эффектов на экспрессию каждого гена. Каждое лекарственное средство представлено в UMAP как вектор оценок эффекта (см. главу «Способы») для всех генов. Форма точки соответствует типу клеток, а цвет соответствует классу соединений.
[0088] На ФИГ. 26 представлены попарные расстояния между вложениями PCA для лекарственных средств на основе их дозозависимых эффектов. A) Тепловая карта попарных расстояний между двумя типами клеток (столбцы) для данного лекарственного средства (строки) в пространстве уменьшенной размерности PCA. Иерархически кластеризованы для визуализации специфичных для типов клеток ответов на каждое лекарственное средство. B) Вставки выделенных участков тепловой карты с аннотацией сигнального пути показаны слева. Конкретные соединения, отмеченные красной стрелкой, показаны справа (C-E) в виде вложений UMAP. F) Обработанные траметинибом клеточные линии выделены для иллюстрации колокализации A549 и K562. Цветные точки соответствуют меченому соединению, а все остальные лекарственные средства показаны серым цветом. Форма кодирует клеточную линию, от которой был захвачен каждый профиль эффектов (квадраты: MCF7; треугольники; K562; круг; A549).
[0089] На ФИГ. 27 представлено, что траектория ингибитора HDAC регистрирует клеточную гетерогенность по ответу на лекарственное средство и биохимической аффинности. (A) Выравнивание методом MNN и вложения UMAP для транскрипционных профилей клеток, обработанных одним из 17 ингибиторов HDAC. Корень псевдодозы показан красной точкой. (B) Гребневые диаграммы, отражающие распределение клеток по псевдодозе в зависимости от дозы, показаны для трех ингибиторов HDAC с различными биохимическими аффинностями. (C) Соотношение между TC50 и средним значением log10(IC50) по результатам измерений in vitro. Звездочками обозначены соединения с растворимостью < 200 мМ (в ДМСО), которые не были включены в аппроксимацию.
[0090] На ФИГ. 28 представлено, что типы клеток, обработанных ингибитором HDAC, выравниваются и позволяют реконструировать соединенную траекторию псевдодозы. A) Вложение UMAP, выделяющее реконструированную траекторию псевдодозы относительно выровненных методом ближайшего соседа клеток, обработанных ингибитором HDAC, и клеток, обработанных несущей средой. В качестве корневых узлов (красные точки) были выбраны узлы на основном графе, у которых более 50% ближайших соседей аннотированы как клетки, обработанные несущей средой. B) Распределение каждой клеточной линии в пределах вложения. C) Гистограмма, отражающая долю каждой псевдодозовой группы, занятой клетками, обработанными каждой дозой. D) Гистограмма, отражающая долю каждой псевдодозовой группы, занятой клетками, обработанными каждым соединением. E) Доля в пределах каждой псевдодозовой группы, соответствующая каждой клеточной линии.
[0091] На ФИГ. 29 представлены гребневые диаграммы, отражающие распределение клеток по псевдодозе для каждой комбинации ингибитора HDAC и дозы для соединений, локализованных по траектории HDAC.
[0092] На ФИГ. 30 представлено контактное ингибирование пролиферации клеток через 72 часа после воздействия лекарственного средства. Типичные изображения светлопольной микроскопии клеток А549, обработанных несущей средой (А), или указанной дозой активатора SIRT1 SRT2104 (В) или ингибитора HDAC абексиностатом (С). Оценки жизнеспособности, определяемые по количеству выделенных клеток для каждой комбинации лекарственное средство/доза, нормализованному к количеству клеток в лунках с контролем - несущей средой.
[0093] На ФИГ. 31 представлено, что выравнивание клеток А549 через 24 и 72 часа после обработки выявляет времязависимые ответы на различные низкомолекулярные соединения. (A-C) Вложение UMAP для клеток А549 через 24 и 72 часа после обработки в отсутствие коррекции на различия в жизнеспособности и пролиферации (A) после линейного преобразования данных для учета изменений коэффициента пролиферации и жизнеспособности (B) и после выравнивания данных методом взаимных ближайших соседей после линейной трансформации (C). Клетки имеют цветовую кодировку по временной точке их сбора. (D-F) Вложения UMAP, аналогичные панелям A-C, где клетки имеют цвет в зависимости от совокупной нормализованной оценки экспрессии генов-маркеров G1/S. (G-I) Вложения UMAP, аналогичные панелям A-C, где клетки имеют цвет в зависимости от совокупной нормализованной оценки экспрессии генов-маркеров G2/M. (J-L) Вложения UMAP, аналогичные панелям A-C, где клетки имеют цвет в зависимости от коэффициента пролиферации. (M-O) Вложения UMAP, аналогичные панелям A-C, с визуализацией клеток, обработанных контролем - несущей средой. (P) Вложения UMAP с панели C, причем клетки имеют цвет в зависимости от целевого сигнального пути, на который воздействует обработка, которой они подвергались. (Q) Доля клеток в группах по целевым сигнальным путям. Следует отметить, что лишь часть из наших 188 соединений в ограниченном количестве сигнальных путей проверялась через 72 часа. (R) Доля клеток в группах по активностям, на которые нацелены обработки соединениями - эпигенетическими регуляторами. (S) Доля клеток в группах по соединению HDAC.
[0094] На ФИГ. 32 представлено, что ингибирование бромодомена, активация сиртуина и ингибирование гистондеацетилазы индуцируют характерные транскриптомные ответы. (A-D) Вложения UMAP для выровненных методом MNN клеток A549 через 24 и 72 часа после обработки универсальными ингибиторами HDAC абексиностатом (A) или белиностатом (B), ингибитором бромодомена JQ1 (C) и активатором SIRT1 SRT2104 (D). Клетки имеют цвет в зависимости от дозы, воздействию которой подвергалась каждая клетка.
[0095] На ФИГ. 33 представлено, что гетерогенный ответ на большинство ингибиторов HDAC, по-видимому, не определяется клеточной асинхронностью. A) Выровненные вложения UMAP для клеток, подвергнутых воздействию ингибиторов HDAC в течение 24 или 72 часов. Клетки окрашены в зависимости от их продвижения по псевдодозе. B) Выровненные вложения UMAP для клеток, которые подвергали воздействию несущей среды (серые клетки) или меченого ингибитора HDAC в течение 24 (красные клетки) или 72 (синие клетки) часов. C) Гребневые диаграммы, отображающие плотность клеток А549, подвергшихся воздействию ингибитора HDAC, на выровненной траектории псевдодозы. Результаты представлены для 8 ингибиторов HDAC, которые анализировали как через 24, так и через 72 часа. Серыми и цветными линиями обозначены клетки, обработанные ингибиторами в течение 24 или 72 часов соответственно.
[0096] На ФИГ. 34 представлено, что траектория транскрипции клеток, обработанных ингибитором HDAC, соответствует измерениям IC50 in vitro. A) Для каждого соединения и каждой клеточной линии кривые реакции на псевдодозу аппроксимировали, используя пакет drc R. В качестве ответа использовали среднее положение каждой дозы вдоль траектории псевдодозы. Показаны два иллюстративных примера для белиностата (вверху) и трихостатина A (TSA) (внизу). Пунктирными вертикальными линиями представлена транскрипционная EC50 (TC50) для каждого соединения в каждой клеточной линии. Затененная серая область обозначает 95%-е доверительные интервалы для каждой оценки TC50. B) График, отражающий совокупное измеренное in vitro среднее значение log10(IC50 [M]) относительно log(TC50), с цветовой кодировкой в зависимости от растворимости, указанной компанией Selleckchem Chemicals. Точки, обозначенные (*), для аппроксимаций не использовали. C) Зависимость log10(IC50 [M]) от log(TC50) для каждой изоформы HDAC. Цветовая кодировка точек в зависимости от использованного ингибитора HDAC.
[0097] На ФИГ. 35 представлено, что линейные модели идентифицируют псевдодозозависимые модули пролиферации и метаболизма. A) Гистограмма общего количества значимых дозозависимых и псевдодозозависимых DEG (FDR < 0,05). B) Upset-диаграмма, отображающая пересечения значимых псевдодозозависимых DEG между тремя типами клеток. C) Тепловая карта псевдодозы, изображающая 4308 генов, которые значительно изменялись в зависимости от псевдодозы. Каждая строка соответствует ожидаемой экспрессии гена в трех клеточных линиях, аппроксимированной моделью, описанной в разделе «Анализ дифференциальной экспрессии» в главе «Способы». Гены (строки) масштабировали и стандартизировали в пределах каждой клеточной линии, а затем соединяли три матрицы и выполняли иерархическую кластеризацию. После этого кластеры из иерархической кластеризации использовали в качестве входных данных для GSAhyper с использованием набора генов Hallmarks. Показаны выбранные гены и наборы генов, характеризующие каждый кластер (справа).
[0098] На ФИГ. 36 представлено, что обработка ингибитором HDAC индуцирует остановку клеточного цикла во всех трех клеточных линиях. A) Доля клеток, экспрессирующих РНК для AURKA и CDKN1A по псевдодозовым группам. Черными столбцами обозначается 95%-й доверительный интервал после бутстреппинга. B) Коробчатые диаграммы, отражающие долю клеток во фракции с низкой пролиферацией при данной дозе лекарственного средства по псевдодозовым группам. C) Анализ содержания ДНК в трех клеточных линиях после обработки ДМСО (вверху) или 10 мкМ абексиностата (внизу). D) Количественный анализ данных проточной цитометрии, отражающий количество клеток в каждой категории содержания ДНК.
[0099] На ФИГ. 37 представлено, что воздействие ингибитора HDAC приводит к секвестрации ацетата в виде ацетилированных лизинов. A) Количественный анализ измерений методом проточной цитометрии общего количества клеточных ацетилированных лизинов в клетках A549 (левая панель), MCF7 (средняя панель) и K562 (правая панель), обработанных 10 мкМ прациностата, 10 мкМ абексиностата или контролем - несущей средой. Планки погрешностей обозначают стандартное отклонение среднего значения (критерий суммы рангов Уилкоксона, n=3 повторности в культуре, * p < 0,05, *** p < 0,005). B) Репрезентативные гистограммы проточной цитометрии для эксперимента с количественным анализом на панели A. Синие заштрихованные области и красные линии соответствуют контролю - несущей среде ДМСО и 10 мкМ абексиностату соответственно.
[00100] На ФИГ. 38 представлен общий транскрипционный ответ на ингибиторы HDAC, указывающий на депривацию ацетил-КоА. (A) Тепловая карта центрированной по строкам и z-масштабированной экспрессии генов, демонстрирующая повышение экспрессии псевдодозозависимых генов, принимающих участие в клеточном метаболизме углерода. (B) Диаграмма ролей генов из (А) в регуляции цитоплазматического ацетил-КоА. Красными кружками обозначены ацетильные группы. Ферменты показаны серым цветом. Транспортеры показаны зеленым цветом (FA, жирная кислота; Ац-КоА, ацетил-КоА; C, цитрат).
[00101] На ФИГ. 39 представлено, что добавление предшественников ацетил-КоА снижает, а ингибирование ферментов, восполняющих пулы ацетил-КоА, усиливает продвижение по траектории псевдодозы ингибитора HDAC. A-D) Вложения UMAP для транскриптомов одиночных клеток A549 (панели A и B) и MCF7 (панели C и D) после воздействия ингибиторами HDAC прациностатом или абексиностатом в присутствии или в отсутствие предшественников ацетил-КоА или ингибиторов ферментов, восполняющих пулы ацетил-КоА. UMAP строили по клеткам из всех условий эксперимента. Клетки имеют цветовую кодировку в зависимости от псевдодозовой группы (панели A и C) или дозы (панели B и D). E) Диаграмма Венна для перекрытия дифференциально экспрессированных генов по траекториям между исходной траекторией HDACi или траекториями HDACi A549 или MCF7 из этого нового эксперимента. F, H) Коробчатые диаграммы оценок псевдодозы при выбранных условиях у клеток, которые подвергали воздействию 1 или 10 мкМ прациностата с одновременной обработкой предшественниками ацетил-КоА или без нее для клеток A549 (панель H) или MCF7 (панель L). Значения нормализовали по клеткам, обработанным несущей средой. Критерий суммы рангов Уилкоксона. G, I) Коробчатые диаграммы оценок псевдодозы при выбранных условиях у клеток, которые подвергали воздействию несущей среды и прациностата с одновременной обработкой предшественниками ацетил-КоА или без нее для клеток A549 (панель I) или MCF7 (панель M). Значения нормализовали по клеткам, обработанным несущей средой. Критерий суммы рангов Уилкоксона. J, L) Тепловые карты, отображающие долю клеток на псевдодозовую группу, для клеток, обработанных различными предшественниками ацетил-КоА, в подвергнутых воздействию прациностата клетках A549 (F) или MCF7 (J). K, M) Тепловые карты, отражающие долю клеток на псевдодозовую группу для клеток, обработанных различными ингибиторами, нацеленными на ферменты, восполняющие пулы ацетил-КоА в подвергнутых воздействию прациностата клетках A549 (панель G) и MCF7 (панель K).
[00102] На ФИГ. 40 представлена корреляция величин эффектов между дифференциально экспрессируемыми генами после ингибирования HDAC на основе исходного скрининга по сравнению с новым экспериментом. A-B) Коррекция оценок величины эффекта (бета-коэффициенты) для дифференциально экспрессированных генов между контролем - несущей средой и 10 мкМ абексиностата (панель A) или 10 мкМ прациностата (панель B) в клетках A549. C-D) Коррекция оценок величины эффекта (бета-коэффициенты) для дифференциально экспрессированных генов между контролем - несущей средой и 10 мкМ абексиностата (панель С) или 10 мкМ прациностата (панель D) в клетках MCF7. Оси X соответствуют крупномасштабному эксперименту sci-Plex. Оси Y соответствуют целенаправленному последующему эксперименту sci-Plex.
[00103] На ФИГ. 41 представлены ядерные метки (хеши) для мультиплексных анализов ATAC-seq для одиночных клеток и подход к анализу доступного хроматина с использованием комбинаторного индексирования. A. Клетки из отдельных образцов выращивают и обрабатывают в отдельных лунках. В экспериментальных лунках клетки лизируют и выделяют ядра. В каждую лунку добавляют специфичные для лунок одноцепочечные олигонуклеотидные ДНК-метки и фиксация захватывает метки внутрь ядер. Затем ядра из всех образцов объединяют для последующих стадий комбинаторного индексирования. B. Схема того, каким образом комбинаторное индексирование добавляет одинаковые молекулярные индексы к меткам и доступной ДНК внутри ядра. C. Выделенные метки правильно идентифицируют клетки из образцов, содержащих только мышиные (NIH-3T3) или только человеческие (A549) клетки в экспериментах со смесями типа «скотный двор». D. Ядра, содержащие множество меток, представляют собой дублеты. E. Распределение количества маркирующих UMI, выделенных в расчете на клетку. Красная линия представляет собой требуемую отсечку для включения клетки в дальнейший анализ. F. Распределение коэффициентов обогащения метки на клетку. Коэффициент обогащения для клетки отражает количество наиболее распространенной метки, поделенное на количество второй по распространенности метки в ядре. Клетки под красной линией (коэффициент обогащения < 5) назвали дублетами.
[00104] На ФИГ. 42 представлена стратегия маркировки, позволяющая сопоставлять профили хроматина одиночных клеток с экспериментальными группами. A. План эксперимента. B. Представление алгоритмом Uniform Manifold Approximation and Projection (UMAP) всех выделенных клеток с цветовой кодировкой по воздействию, определенному метками. Следует отметить, что расстояние между двумя клетками указывает на величину различия в картине доступного хроматина. C. Кривые «доза - ответ» для каждого лекарственного средства аппроксимировали количеством клеток, выделенных для каждой дозы воздействия. D. UMAP-представление клеток из каждой группы воздействия лекарственным средством с цветовой кодировкой в соответствии с дозой, определяемой метками. E. Дорожки обозревателя, показывающие профили псевдомассового ATAC из клеток, обработанных растущей дозировкой SAHA.
[00105] На ФИГ. 43 представлено, что лесенка хеширующих олигонуклеотидов может быть захвачена ядрами и может служить внешними стандартами в экспериментах sci-RNA-seq. (A) Экспериментальный обзор способа хеш-лесенки. Ядра выделяют из клеток, прикрепляют лесенку из хеширующих олигонуклеотидов, а затем обрабатывают sci-RNA-seq. (B) Коробчатая диаграмма количества UMI хеширующих олигонуклеотидов на клетку, причем все хеширующие олигонуклеотиды добавлялись в разных количествах. (C) Диаграмма рассеяния для ожидаемых и наблюдаемых количеств UMI хеш-лесенки UMI, демонстрирующая клетку с низкой (слева) и высокой (справа) эффективностью захвата хеша.
[00106] На ФИГ. 44 представлено, что хеш-лесенка расширяет наши возможности по обнаружению глобального снижения уровней транскриптов, вызванное флавопиридолом. (A) Обзор эксперимента. Клетки HEK293T обрабатывали флавопиридолом в течение разных периодов времени и метили лесенкой хеширующих олигонуклеотидов и дополнительным хеширующим олигонуклеотидом для мультиплексирования перед подготовкой к sci-RNA-seq. (B) Коробчатая диаграмма, демонстрирующая общее количество РНК UMI для клеток, обработанных флавопиридолом, в разные моменты времени. (C) Гистограмма, показывающая количество дифференциально экспрессируемых генов в ответ на флавопиридол с использованием традиционной и хеш-лестничной методик нормализации. (D) Скрипичный график, демонстрирующий отношение оценок величины эффекта для распространенных дифференциально экспрессируемых генов, рассчитанных с помощью хеш-лесенки, в сравнении с традиционной нормализацией.
[00107] Схематические чертежи необязательно выполнены в масштабе. Аналогичные цифровые обозначения, используемые на фигурах, относятся к аналогичным компонентам, стадиям и т. п. Однако следует понимать, что использование номера для обозначения компонента на заданной фигуре не предполагает ограничения для обозначенного тем же номером компонента на другой фигуре. Кроме того, использование разных номеров для обозначения компонентов не предусматривает указания на то, что компоненты с разными номерами не могут быть идентичными или аналогичными другим пронумерованным компонентам.
[00108] ПОДРОБНОЕ ОПИСАНИЕ
[00109] Приложения
[00110] С использованием способов, описанных в настоящем документе, можно предложить множество вариантов применения и областей использования. Например, высокопроизводительные способы для одиночных ядер и одиночных клеток можно использовать для поиска лекарственных средств. Измерения транскрипционных различий в одиночных клетках, индуцированных агентом или генетическим нарушением, можно использовать для скрининга лекарственных средств. В одном из вариантов применения для классификации вариантов генов и генома используется редактирование генома (Findlay, Nature, 2018 562(7726):217-222). В другом варианте применения способы могут использоваться для понимания здоровья и заболевания, научных, медицинских, диагностических, биомедицинских исследований, применения в клинике или выявления биомаркеров.
[00111] Воздействие заданных условий на клетки
[00112] Предложенный в настоящем документе способ можно использовать для получения библиотек секвенирования из множества одиночных клеток. В одном из вариантов осуществления способ включает в себя воздействие на клетки разными заданными условиями. Способ включает в себя воздействие на группы клеток разными заданными условиями (ФИГ. 1, блок 10). Разные условия могут включать в себя, например, разные условия культивирования (например, разные среды, разные условия окружающей среды), разные дозы агента, разные агенты или комбинации агентов. Агенты описаны в настоящем документе. Ядра или клетки каждой подгруппы клеток и/или образца или образцов метят с использованием хеширования ядер, объединяют и анализируют массовыми мультиплексными способами секвенирования одиночных ядер или одиночных клеток. Можно использовать по существу любой способ секвенирования одиночных ядер или одиночных клеток, включая, без ограничений, секвенирование транскриптома одиночных ядер (предварительная заявка на патент США № 62/680,259 и публикация Gunderson et al. (WO2016/130704)), полногеномное секвенирование одиночных ядер (опубликованная заявка на патент США № US 2018/0023119) или секвенирование в одиночных ядрах доступного для транспозона хроматина (патент США № 10,059,989), sci-HiC (Ramani et al., Nature Methods, 2017, 14:263-266), DRUG-seq (Ye et al., Nature Commun., 9, article number 4307) или любой комбинации аналитов из ДНК, РНК и белков, например, sci-CAR (Cao et al., Science, 2018, 361(6409):1380-1385). Хеширование ядер используют для демультиплексирования и идентификации отдельных клеток или ядер в разных условиях.
[00113] Клетки могут быть получены из любого организма и из клеток любого типа или любой ткани организма. Как правило, клетки распределены по первому множеству компартментов. В одном из вариантов осуществления компартмент представляет собой лунку многолуночного устройства, например, 96-луночного, 384-луночного планшета или 1536-луночного планшета, причем клетки в компартменте называются подгруппой клеток. В одном из вариантов осуществления клетки в подгруппе являются генетически гомогенными, а в другом варианте осуществления клетки в подгруппе являются генетически гетерогенными. Количество клеток может варьировать и зависеть от практических ограничений оборудования (например, размера лунок многолуночного планшета, количества индексов), используемого на других стадиях способа, как описано в настоящем документе. В одном из вариантов осуществления количество клеток в подгруппе может составлять не более чем 100 000 000, не более чем 10 000 000, не более чем 1 000 000, не более чем 100 000, не более чем 45 000, не более чем 35 000, не более чем 25 000, не более чем 15 000, не более чем 5 000, не более чем 1 000, не более чем 500 или не более чем 50.
[00114] В одном из вариантов осуществления каждая подгруппа клеток подвергается воздействию агента или возмущения. Агент может по существу представлять собой что-либо, вызывающее изменения в клетке. Например, агент может изменять транскриптом клетки, изменять структуру хроматина клетки, изменять активность белка в клетке, изменять ДНК клетки или изменять редактирование ДНК клетки. Примеры агентов включают в себя, без ограничений, соединение, такое как белок (включая антитело), нерибосомный белок, поликетид, органическую молекулу (включая органическую молекулу массой 900 дальтон или менее), неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту или их комбинацию. В одном из вариантов осуществления агент вызывает генетическое нарушение, например редактирующий ДНК белок, такой как CRISPR или Talen. В одном из вариантов осуществления агент представляет собой лекарственное средство, такое как терапевтическое лекарственное средство. В одном из вариантов осуществления клетка может представлять собой клетку дикого типа, а в другом варианте осуществления клетка может быть генетически модифицирована для обеспечения генетического нарушения, например, нокина гена, нокаута гена или сверхэкспрессии (Szlachta et al., Nat Commun., 2018, 9:4275). В одном из вариантов осуществления клетки модифицируют путем внесения генетического нарушения, воздействия агента, и любые возникшие изменения, например, в организации транскрипции генома, можно идентифицировать в одиночных клетках (Datlinger et al., Nature Methods, 2017, 14(3):297-30 [sci-Crop]; Adhemar et al., Cell, 2016, 167:1883-1896 [sci-Crispr]; and Dixit et al., Cell, 2016, 167(7):1853-1866) [sci-Perturb]. Необязательно в этих вариантах осуществления при использовании гидовой РНК для нацеливания на генетическое возмущение способ может дополнительно включать в себя идентификацию и подтверждение фактического редактирования генома.
[00115] Подгруппы клеток можно подвергать воздействию одного и того же агента, но в разных компартментах многолуночного устройства можно изменять разные переменные факторы, что позволяет тестировать множество переменных в одном эксперименте. Например, в одном планшете можно тестировать разные дозировки, разную продолжительность воздействия и разные типы клеток. В одном из вариантов осуществления клетки могут экспрессировать белок, имеющий известную активность, и можно оценивать влияние агента на активность в разных условиях. Используемое для мечения ядер хеширование ядер позволяет позднее идентифицировать нуклеиновые кислоты, происходящие из определенной подгруппы ядер или клеток, например, из одной лунки многолуночного планшета.
[00116] Хеширование клеток и ядер
[00117] При получении библиотек секвенирования из множества клеток или множества одиночных ядер клетки или ядра можно приводить в контакт с хеширующим олигонуклеотидом. Использование хеширующих олигонуклеотидов необязательно, и его можно использовать в сочетании с нормализующими олигонуклеотидами. Нормализующие олигонуклеотиды описаны в настоящем документе. В одном из вариантов осуществления приведение в контакт осуществляется после того, как клетки подверглись необязательному воздействию заданных условий. Как правило, приведение в контакт с хеширующим олигонуклеотидом происходит, когда ядра или клетки разделены на множество компартментов. В одном из вариантов осуществления ядра могут быть выделены (ФИГ. 1, блок 11) и помечены (ФИГ. 1, блок 12) хеширующим олигонуклеотидом. В другом варианте осуществления ядра подвергают воздействию хеширующего олигонуклеотида до выделения из клеток. Авторы определили, что любое разрушение клеточной мембраны позволяет пометить ядра хеширующими олигонуклеотидами. Таким образом, ядра могут быть помечены в присутствии или в отсутствие цитоплазматического материала. Ядра могут быть пермеабилизованы, и, как правило, их пермеабилизуют в процессе мечения хеширующими и/или нормализующими олигонуклеотидами, например, ядра могут быть пермеабилизованы до, во время или после мечения ядер хеширующими олигонуклеотидами и/или нормализующими олигонуклеотидами. Способы пермеабилизации мембран известны в данной области. В другом варианте осуществления клетки приводят в контакт с хеширующим олигонуклеотидом.
[00118] Выделение ядер осуществляют путем инкубации клеток в буфере для лизирования клеток в течение по меньшей мере от 1 до 20 минут, например, 5, 10 или 15 минут. Необязательно клетки можно подвергать воздействию внешней силы, способствующей лизису, например, движению через пипетку. Пример буферного раствора для лизиса клеток включает в себя 10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2, 0,1% IGEPAL CA-630 и 1% SUPERase In RNase Inhibitor. Специалисту в данной области понятно, что эти концентрации компонентов могут быть несколько изменены без снижения пригодности лизирующего клетки буфера для выделения ядер. Специалисту в данной области будет понятно, что ингибиторы РНКазы, BSA и/или поверхностно-активные вещества могут быть пригодны для использования в буферах, используемых для выделения ядер, и что в буфер могут быть введены другие добавки для других последующих применений для комбинаторного индексирования одиночных клеток.
[00119] В одном из вариантов осуществления клетки или ядра обрабатывают с использованием любого способа sci-seq для измерения аналита или аналитов в клетке, включая, без ограничений, ДНК, РНК, белка или их комбинации.
[00120] В одном из вариантов осуществления ядра выделяют из отдельных клеток, которые прикреплены или находятся в суспензии. Способы выделения ядер из отдельных клеток известны специалисту в данной области. В одном из вариантов осуществления ядра выделяют из клеток, присутствующих в ткани. Способ получения выделенных ядер, как правило, включает в себя подготовку ткани и выделение ядер из подготовленной ткани. В одном из вариантов осуществления все стадии проводятся на льду.
[00121] Подготовка ткани может включать в себя мгновенное замораживание ткани в жидком азоте, а затем режущее или тупое воздействие на ткань для измельчения ткани до фрагментов диаметром 1 мм или менее. Необязательно можно использовать активные на холоде протеазы и/или другие ферменты для разрушения межклеточных соединений. Режущее воздействие может быть выполнено при помощи лезвия, рассекающего ткань на небольшие фрагменты. Использования тупого воздействия можно достичь путем измельчения ткани молотком или аналогичным объектом, и полученный состав из раздавленной ткани называется порошком.
[00122] При традиционных методиках извлечения ядер из ткани ткань обычно инкубируют с тканеспецифическим ферментом (например, трипсином) при высокой температуре (например, 37 °C) в течение периода от 30 минут до нескольких часов, а затем клетки лизируют буферным раствором для лизирования клеток, извлекая ядра. Способ выделения ядер, описанный в настоящем документе и в предварительной заявке на патент США № 62/680,259, имеет ряд преимуществ: (1) Искусственные ферменты не вводятся, а все стадии проводятся на льду. Это снижает потенциальное нарушение клеточных состояний (например, состояние транскриптома, состояние хроматина или состояние метилирования). (2) Это было подтверждено для многих типов тканей, включая ткани головного мозга, легких, почек, селезенки, сердца, мозжечка, и патологических образцов, таких как опухолевые ткани. По сравнению с традиционными методиками выделения ядер из тканей, при которых используют разные ферменты для разных типов тканей, новая методика потенциально может снижать систематическую ошибку при сравнении состояний клеток разных тканей. (3) Способ также снижает затраты и повышает эффективность за счет исключения стадии ферментативной обработки. (4) По сравнению с другими методиками извлечения ядер (например, тканевых гомогенизаторов Даунса) эта методика более надежна для разных типов тканей (например, метод Даунса требует оптимизации циклов Даунса для разных тканей) и позволяет обрабатывать большие фрагменты образцов с высокой пропускной способностью (например, способ Даунса ограничен размером гомогенизатора).
[00123] Необязательно выделенные ядра могут не содержать нуклеосом или могут быть подвергнуты воздействию условий, при которых происходит уменьшение количества нуклеосом в ядрах, с образованием безнуклеосомных ядер (см., например, заявку на патент США № US 2018/0023119). Безнуклеосомные ядра можно использовать для полногеномного секвенирования одиночных ядер.
[00124] Лизирующий буфер может включать в себя хеширующий олигонуклеотид, используемый для хеширования ядер (ФИГ. 1, блок 12). Альтернативно хеширующий олигонуклеотид может отсутствовать в лизирующем буфере, но может присутствовать на последующей стадии, если это будет сочтено уместным специалистом в данной области.
[00125] Хеширующий олигонуклеотид включает в себя одноцепочечную или двухцепочечную нуклеотидную последовательность, которая представляет собой ДНК, РНК или их комбинацию. Хеширующий олигонуклеотид может быть устойчив к ДНКазе или устойчив к РНКазе. Хеширующий олигонуклеотид может включать в себя полинуклеотидные компоненты, такие как, без ограничений, индекс, UMI и универсальную последовательность, в любой комбинации. Хеширующий олигонуклеотид может также включать в себя другие компоненты, не относящиеся к нуклеиновым кислотам, включая белок, такой как антитело. В одном из вариантов осуществления хеширующий олигонуклеотид включает в себя 5’-область, специфичную для подгруппы индексную последовательность и 3’-концевую последовательность. 5’-область может представлять собой 5’«ПЦР-рукоятку» или универсальную последовательность, которую можно использовать на последующей стадии для амплификации и добавления конкретных нуклеотидов к хеширующиим олигонуклеотидам. 5’«ПЦР-рукоятка» может включать в себя нуклеотидную последовательность, идентичную или комплементарную универсальной последовательности захвата. 3’-концевая последовательность может представлять собой любую серию нуклеотидов, пригодную для применения на последующей стадии. Например, если последующие стадии включают в себя получение транскриптомной библиотеки, 3’-концевая последовательность может включать в себя полиаденилированную последовательность. В другом варианте осуществления хеширующий олигонуклеотид включает в себя нуклеотидную последовательность, которую можно использовать на последующей стадии лигирования, стадии амплификации, стадии достройки праймера или их комбинации, с целью добавления специфической для подгруппы индексной последовательности и других нуклеотидов, подходящих для использования на последующих стадиях способа, таких как 5’-область и/или полиаденилированный 3’-конец. Любая дополнительная манипуляция с хеширующим олигонуклеотидом для добавления специфической для подгруппы индексной последовательности и любого другого элемента может быть выполнена до стадии объединения, описанной в настоящем документе. В одном из вариантов осуществления хеширующие олигонуклеотиды можно добавлять до, во время или после стадии лизиса клеток или воздействия агента. В одном из вариантов осуществления хеширующий олигонуклеотид можно добавлять без стадии лизиса клеток.
[00126] Индексную последовательность, также называемую меткой или штрихкодом, также можно использовать в качестве маркера, характерного для компартмента, в котором находилась конкретная целевая нуклеиновая кислота. Соответственно, индекс представляет собой полинуклеотидную метку, которая присоединена к каждой из целевых нуклеиновых кислот, присутствующих в конкретном компартменте, и присутствие индекса служит показателем или используется для идентификации компартмента, в котором популяция ядер или клеток находилась на данной стадии способа. Специфичная для подгруппы индексная последовательность хеширующего олигонуклеотида указывает на то, какие нуклеиновые кислоты получены из субпопуляции клеток, подвергшихся воздействию конкретного заданного условия, и позволяют отличить эти нуклеиновые кислоты от нуклеиновых кислот, которые относятся к субпопуляциям клеток, подвергшихся воздействию других конкретных заданных условий.
[00127] Индексная последовательность (например, специфичная для подгруппы индексная последовательность хеширующего олигонуклеотида, идентифицирующая популяцию индексная последовательность нормализующего олигонуклеотида или другой индекс), используемая в настоящем документе, может представлять собой любую подходящую последовательность из любого подходящего количества нуклеотидов длиной, например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или более. Четырехнуклеотидная метка позволяет мультиплексировать 256 образцов, а шестинуклеотидная метка позволяет обрабатывать 4096 образцов. Индекс или штрихкод можно вводить множеством разных способов, включая, без ограничений, прямое включение в олигонуклеотид, лигирование, удлинение, адсорбцию и специфические или неспецифические взаимодействия олигонуклеотида или олигонуклеотидов или амплификации.
[00128] Хеширующий олигонуклеотид связывается с ядрами или клетками компартмента и необязательно фиксируется к ядрам или клеткам. Связывание может быть специфическим или неспецифическим. Хеширующий олигонуклеотид, который специфически связывается с клеткой или ядром, может включать в себя необязательный домен, опосредующий специфическое связывание. Примеры доменов включают в себя лиганд рецептора на поверхности ядра или клетки, антитело или фрагмент антитела, аптамер или специфическую олигонуклеотидную последовательность. Авторы установили, что хеширующий олигонуклеотид будет неспецифически связываться с ядром в количестве, достаточном для более последующего демультиплексирования в способе. Таким образом, в одном из вариантов осуществления хеширующий олигонуклеотид неспецифически связывается с ядрами или клетками компартмента. В одном из вариантов осуществления неспецифическое связывание осуществляется посредством абсорбции. В одном из вариантов осуществления хеширующий олигонуклеотид можно добавлять на любой стадии перед объединением. В одном из вариантов осуществления хеширующий олигонуклеотид представляет собой смесь одного или более олигонуклеотидов, причем каждый олигонуклеотид имеет уникальный штрихкод или уникальную последовательность. Хеширующий олигонуклеотид может содержать штрихкод или последовательность для введения штрихкода на более поздней стадии после связывания. В одном из вариантов осуществления комбинация хеширующих олигонуклеотидов с уникальным штрихкодом может быть сигнатурой конкретного экспериментального условия, агента, образца или воздействия.
[00129] Уникальный хеширующий олигонуклеотид, присутствующий в каждой подгруппе, фиксируют к ядрам или клеткам этой подгруппы посредством воздействия поперечно-сшивающего соединения. К подходящим для использования примерам поперечно-сшивающего соединения относятся, без ограничений, параформальдегид, формалин или метанол. Другие подходящие для использования примеры описаны в публикации Hermanson (Bioconjugate Techniques, 3rd Edition, 2013). Параформальдегид может присутствовать в концентрации от 1% до 8%, например 5%. Обработка ядер параформальдегидом может включать в себя добавление параформальдегида к суспензии ядер и инкубацию при 0 °C. В некоторых вариантах осуществления хеширующий олигонуклеотид не является поперечно-сшитым, но остается связанным.
[00130] Манипуляция с ядрами или клетками, включая стадии объединения и распределения, описанные в настоящем документе, могут включать в себя использование буфера для ядер. Пример буфера для ядер включает в себя 10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 1% SUPERase In RNase Inhibitor (20 ЕД/мкл, Ambion) и 1% BSA (20 мг/мл, NEB). Специалисту в данной области будет понятно, что эти концентрации компонентов могут быть несколько изменены без снижения пригодности этого буфера для ядер, в котором суспендируют ядра.
[00131] Выделенные фиксированные ядра или клетки можно использовать сразу же или разделить на аликвоты и быстро заморозить в жидком азоте для последующего использования. При подготовке к использованию после замораживания размороженные ядра можно подвергнуть пермеабилизации, например, с помощью 0,2% tritonX-100 в течение 3 минут на льду, и кратковременной ультразвуковой обработке для уменьшения слипания ядер.
[00132] Способ дополнительно включает в себя объединение подгрупп ядер или клеток с последующим распределением объединенных ядер или клеток во второе множество компартментов (ФИГ. 1, блок 13). Количество ядер или клеток, присутствующих в подгруппе и, следовательно, в каждом компартменте, может составлять по меньшей мере 1. Количество ядер или клеток в подгруппе не имеет ограничительного характера и может измеряться миллиардами. В одном из вариантов осуществления количество в подгруппе составляет не более чем 100 000 000, не более чем 10 000 000, не более чем 1 000 000, не более чем 100 000, не более чем 10 000, не более чем 4000, не более чем 3000, не более чем 2000 или не более чем 1000. В одном из вариантов осуществления количество ядер, присутствующих в подгруппе, может составлять от 1 до 1000, от 1000 до 10 000, от 10 000 до 100 000, или от 100 000 до 1 000 000, или от 1 000 000 до 10 000 000 или от 10 000 000 до 100 000 000. В одном из вариантов осуществления каждый компартмент может представлять собой лунку многолуночного планшета, такого как 96- или 384-луночный планшет. В одном из вариантов осуществления каждый компартмент может представлять собой каплю. Способы распределения ядер по подгруппам известны специалисту в данной области и являются стандартными. Хотя можно использовать цитометрию посредством сортировки клеток с активацией флуоресценции (FACS), также можно использовать и простое разведение. В одном из вариантов осуществления FACS-цитометрия не используется.
[00133] Количество компартментов на первой стадии распределения (ФИГ. 1, блок 13) может зависеть от используемого формата. Например, количество компартментов может составлять от 2 до 96 компартментов (при использовании 96-луночного планшета), от 2 до 384 компартментов (при использовании 384-луночного планшета). В одном из вариантов осуществления можно использовать множество планшетов. Например, можно использовать компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 96-луночных планшетов или компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 384-луночных планшетов. Если используемый тип компартмента представляет собой каплю, которая содержит два или более ядер или клеток, можно использовать любое количество капель, например по меньшей мере 10 000, по меньшей мере 100 000, по меньшей мере 1 000 000 или по меньшей мере 10 000 000 капель.
[00134] После того как ядра или клетки были помечены хеширующим олигонуклеотидом, объединены и распределены по подгруппам, можно использовать разные процедуры для окончательного получения библиотек разных нуклеиновых кислот в ядрах или клетках и последовательности нуклеиновых кислот (ФИГ. 1, блок 14). В одном из вариантов осуществления библиотеки транскриптомов одиночных ядер получают, как подробно описано в настоящем документе (см. пример 1), и в другом варианте осуществления библиотеки доступного хроматина одиночных ядер получают, как подробно описано в настоящем документе (см. пример 2); однако процедура, используемая после мечения ядер хеширующим олигонуклеотидом, не имеет ограничительного характера. Например, библиотеки, полученные с использованием способов комбинаторного индексирования одиночных клеток, например, библиотеки одиночных ядер цельных клеток, библиотеки доступного для транспозона хроматина или библиотеки одиночных ядер цельных клеток для определения статуса метилирования, могут быть получены с использованием ядер или клеток, которые были хешированы, как описано в настоящем документе.
[00135] Нормализующее хеширование
[00136] При получении библиотек секвенирования из множества клеток или множества одиночных ядер клетки или ядра можно приводить в контакт с популяциями нормализующих олигонуклеотидов. Использование нормализующих олигонуклеотидов является необязательным и может использоваться в сочетании с хешированием клеток или ядер, как описано в настоящем документе. Приведение в контакт с нормализующими олигонуклеотидами может происходить до или после того, как клетки подвергаются необязательному воздействию заданных условий. Контакт с популяциями нормализующих олигонуклеотидов может происходить, когда ядра или клетки находятся в массе, до разделения на множество компартментов. Альтернативно ядра могут быть выделены и разделены на множество компартментов (ФИГ. 2, блок 20) и помечены (ФИГ. 2, блок 22) популяциями нормализующих олигонуклеотидов. В другом варианте осуществления ядра подвергают воздействию нормализующих олигонуклеотидов до выделения из клеток. Как и в случае с хеширующими олигонуклеотидами, любое разрушение клеточной мембраны позволяет маркировать ядра нормализующими олигонуклеотидами. Таким образом, ядра могут быть помечены в присутствии или в отсутствие цитоплазматического материала. Ядра могут быть пермеабилизованы, и, как правило, их пермеабилизуют в процессе мечения нормализующими олигонуклеотидами, например, ядра могут быть пермеабилизованы до, во время или после мечения ядер нормализующими олигонуклеотидами. Способы пермеабилизации мембран известны в данной области. В другом варианте осуществления клетки приводят в контакт с нормализующими олигонуклеотидами.
[00137] Для нормализующего хеширования можно использовать способы выделения ядер, подготовки ткани, извлечения ядер и т. д., описанные в настоящем документе для хеширования клеток и ядер. Лизирующий буфер может включать в себя популяции нормализующих олигонуклеотидов, используемых для нормализующего хеширования (ФИГ. 2, блок 22). Альтернативно нормализующие олигонуклеотиды могут отсутствовать в лизирующем буфере, но могут присутствовать на последующей стадии, если специалист в данной области сочтет это уместным. Специалисту в данной области будет понятно, что при использовании как хеширующих олигонуклеотидов, так и нормализующих олигонуклеотидов, клетки или ядра можно подвергать действию хеширующих олигонуклеотидов и нормализующих олигонуклеотидов в одно и то же время или в разное время в ходе реализации способа.
[00138] Нормализующее хеширование служит иным целям, нежели хеширование клеток или ядер. Хеширующие олигонуклеотиды, как правило, используют для мечения множества подгрупп клеток или ядер, причем клетки или ядра в каждой подгруппе метят одним уникальным олигонуклеотидом. Во время секвенирования библиотеки, полученной из клетки или ядра, идентифицируют уникальный хеширующий олигонуклеотид, связанный с клеткой или ядром, что позволяет позднее идентифицировать нуклеиновые кислоты библиотеки, происходящие из конкретной подгруппы ядер или клеток, например, из одной лунки многолуночного планшета. С другой стороны, нормализующее хеширование можно использовать в качестве способа стандартизации, такого как способ стандартизации библиотеки секвенирования. При нормализующем хешировании клетки или ядра в массе или в подгруппах подвергают воздействию композиции нормализующих олигонуклеотидов, причем композиция включает в себя множество популяций нормализующих олигонуклеотидов. Во время секвенирования библиотеки, полученной из клетки или ядра, идентифицируют и подсчитывают популяции нормализующих олигонуклеотидов, связанных с клеткой или ядром, и количества используют в качестве внешнего стандарта для удаления технического шума межклеточных вариаций переменного фактора, такого как экспрессия гена. Таким образом, нормализующие олигонуклеотиды можно использовать в качестве стандарта для оценки чувствительности и количественной точности библиотеки секвенирования. Этот тип стандарта позволяет оценивать влияние технических переменных факторов, измерять эффективность биоинформатических инструментов и улучшать точный анализ образца.
[00139] Нормализующий олигонуклеотид, как правило, имеет те же характеристики, что и хеширующий олигонуклеотид. Нормализующий олигонуклеотид включает в себя одноцепочечную или двухцепочечную нуклеотидную последовательность, которая представляет собой ДНК, РНК или их комбинацию. Нормализующий олигонуклеотид может быть устойчив к ДНКазе или устойчив к РНКазе. Нормализующий олигонуклеотид может включать в себя нуклеотидные компоненты, такие как, без ограничений, индекс, UMI и универсальная последовательность, в любой комбинации. Нормализующий олигонуклеотид может также включать в себя другие компоненты, не относящиеся к нуклеиновым кислотам, включая белок, такой как антитело. В одном из вариантов осуществления нормализующий олигонуклеотид включает в себя 5’-область, специфичную для популяции индексную последовательность и 3’-концевую последовательность. 5’-область может представлять собой 5’«ПЦР-рукоятку» или универсальную последовательность, которую можно использовать на последующей стадии для амплификации и добавления конкретных нуклеотидов к нормализующим олигонуклеотидам. 5’«ПЦР-рукоятка» может включать в себя нуклеотидную последовательность, идентичную или комплементарную универсальной последовательности захвата. 3’-концевая последовательность может представлять собой любую серию нуклеотидов, пригодную для применения на последующей стадии. Например, если последующие стадии включают в себя получение транскриптомной библиотеки, 3’-концевая последовательность может включать в себя полиаденилированную последовательность. В другом варианте осуществления нормализующий олигонуклеотид включает в себя нуклеотидную последовательность, которую можно использовать на последующей стадии лигирования, стадии амплификации, стадии достройки праймера или их комбинации, с целью добавления специфической для подгруппы индексной последовательности и других нуклеотидов, подходящих для использования на последующих стадиях способа, таких как 5’-область и/или полиаденилированный 3’-конец. Любая дополнительная манипуляция с нормализующим олигонуклеотидом для добавления специфической для подгруппы индексной последовательности и любого другого элемента может быть выполнена до стадии объединения, описанной в настоящем документе. В одном из вариантов осуществления нормализующие олигонуклеотиды можно добавлять до, во время или после стадии лизиса клеток или воздействия агента. В одном из вариантов осуществления нормализующие олигонуклеотиды можно добавлять без стадии лизиса клеток.
[00140] Композиция нормализующих олигонуклеотидов, которая воздействует на множество клеток или ядер, включает в себя множество различных популяций нормализующих олигонуклеотидов. Количество различных популяций может составлять по меньшей мере 2, и нет теоретического верхнего предела по количеству отдельных популяций, которые могут присутствовать в композиции; однако практические вопросы, такие как стоимость получения множества нормализующих олигонуклеотидов и время на вычисления, необходимое для анализа множества разных нормализующих олигонуклеотидов, могут ограничивать количество отдельных популяций. Без ограничений, максимальное количество присутствующих в композиции популяций может составлять 2, 4, 8, 16, 24, 32, 40, 48, 56, 64, 72, 80, 88, 96 или 100. Например, количество популяций, присутствующих в композиции, может составлять по меньшей мере 2, по меньшей мере 4, по меньшей мере 8, по меньшей мере 16, по меньшей мере 24, по меньшей мере 32, по меньшей мере 40, по меньшей мере 48, по меньшей мере 56, по меньшей мере 64, по меньшей мере 72, по меньшей мере 80, по меньшей мере 88 или по меньшей мере 96 и не более чем 10, не более чем 96, не более чем 88, не более чем 80, не более чем 72, не более чем 64, не более чем 56, не более чем 48, не более чем 40, не более чем 32, не более чем 24, не более чем 16, не более чем 8 или не более чем 4 в любой комбинации.
[00141] В одном из вариантов осуществления каждый нормализующий олигонуклеотид в одной популяции включает в себя одну уникальную индексную последовательность, которая отсутствует в любой другой популяции композиции. В одном из вариантов осуществления нормализующие олигонуклеотиды одной популяции присутствуют в некоторой концентрации, а другие популяции присутствуют в других концентрациях, например, концентрации по меньшей мере двух популяций отличаются, и в одном из вариантов осуществления концентрации каждой популяции различаются. Таким образом, композиция нормализующих олигонуклеотидов может включать в себя множество популяций олигонуклеотидов, причем каждая популяция включает в себя отличающуюся индексную последовательность, а концентрация всех популяций одинакова; или композиция нормализующих олигонуклеотидов может включать в себя множество популяций олигонуклеотидов, причем каждая популяция включает в себя отличающуюся индексную последовательность, но концентрация по меньшей мере двух популяций отличается. В тех вариантах осуществления, в которых каждая популяция включает в себя отличающуюся индексную последовательность, но концентрация одной или более популяций отличается, известна взаимосвязь между каждой индексной последовательностью и ее концентрацией.
[00142] В другом варианте осуществления каждый нормализующий олигонуклеотид в одной популяции включает в себя по меньшей мере 2 уникальных индексных последовательности, которые отсутствуют в любой другой популяции композиции. Например, одна популяция включает в себя индексные последовательности 1-4, вторая популяция включает в себя индексные последовательности 5-8, а третья популяция включает в себя индексные последовательности 9-12. Таким образом, в одном из вариантов осуществления композиция нормализующих олигонуклеотидов может включать в себя множество популяций олигонуклеотидов, причем каждая популяция включает в себя набор разных индексных последовательностей, а концентрация всех популяций одинакова. В другом варианте осуществления композиция нормализующих олигонуклеотидов может включать в себя множество популяций олигонуклеотидов, причем каждая популяция включает в себя отличный набор индексных последовательностей, но концентрация других популяций отличается, например, концентрации по меньшей мере двух популяций различаются, и в одном варианте осуществления концентрации всех популяций различаются. В тех вариантах осуществления, в которых каждая популяция включает в себя отличный набор индексных последовательностей, но концентрация одной или более популяций отличается, известна взаимосвязь между всеми индексными последовательностями и их концентрациями.
[00143] Концентрация нормализующих олигонуклеотидов в композиции может частично зависеть от эффективности захвата нормализующих олигонуклеотидов клетками или ядрами. В целом захват нормализующих олигонуклеотидов определяется такими факторами, как, без ограничений, обработка образца и глубина секвенирования. Для клеточных линий млекопитающих авторы обнаружили, что эффективность захвата является низкой, но все же дает полезные данные (пример 3). Эмпирически было установлено, что композицию нормализующих олигонуклеотидов можно сконструировать таким образом, чтобы захватывались около 6 миллионов нормализующих олигонуклеотидов на ядро для получения медианного количества UMI, равного 1000-5000. Специалист в данной области может определить эффективность захвата для любого типа клеток и концентрации нормализующих олигонуклеотидов, необходимых в композиции для получения полезных данных нормализации. Без ограничений, используемая концентрация нормализующих олигонуклеотидов означает концентрацию, которая приводит к связыванию олигонуклеотидов с клетками в количестве, аналогичном количеству измеряемого клеточного аналита (например, ДНК, РНК, белка и т. д.). В одном из вариантов осуществления в качестве концентрации каждой популяции композиции нормализующих олигонуклеотидов можно выбрать концентрацию от по меньшей мере 0,001 зептомоль до не более чем 100 аттомоль. Например, концентрация может составлять по меньшей мере 0,001 зептомоль, по меньшей мере 0,01 зептомоль, по меньшей мере 0,1 зептомоль, по меньшей мере 1 аттомоль или по меньшей мере 10 аттомоль и не более 100 аттомоль, не более чем 10 аттомоль, не более чем 1 аттомоль, не более чем 0,1 зептомоль или не более чем 0,01 зептомоль в любой комбинации. В одном из вариантов осуществления концентрация популяции на самом низком уровне и концентрация популяции на самом высоком уровне варьируются на 1, 2, 3, 4, 5 или 6 порядков величины.
[00144] Нормализующий олигонуклеотид связывается с ядрами или клетками компартмента и необязательно фиксируется к ядрам или клеткам. Описанные в настоящем документе связывание и фиксацию хеширующего олигонуклеотида можно использовать и для нормализующего олигонуклеотида. Например, связывание нормализующего олигонуклеотида может быть специфическим или неспецифическим. В одном из вариантов осуществления неспецифическое связывание осуществляется посредством абсорбции. В одном из вариантов осуществления нормализующий олигонуклеотид, который специфически связывается с клеткой или ядром, может включать в себя необязательный домен, опосредующий специфическое связывание. Популяции нормализующих олигонуклеотидов можно фиксировать к ядрам или клеткам, как описано в настоящем документе. В некоторых вариантах осуществления нормализующий олигонуклеотид не является поперечно-сшитым, но остается связанным.
[00145] Манипулирование ядрами или клетками, связанными с хеширующими олигонуклеотидами, нормализующими олигонуклеотидами или их комбинацией, включая стадии объединения и распределения, описанные в настоящем документе, может включать в себя использование буфера для ядер. Пример буфера для ядер включает в себя 10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 1% SUPERase In RNase Inhibitor (20 ЕД/мкл, Ambion) и 1% BSA (20 мг/мл, NEB). Специалисту в данной области будет понятно, что эти концентрации компонентов могут быть несколько изменены без снижения пригодности этого буфера для ядер, в котором суспендируют ядра.
[00146] В вариантах осуществления, в которых к клеткам или ядрам в массе добавляли нормализующие олигонуклеотиды, способ может дополнительно включать в себя распределение объединенных клеток или ядер во множество компартментов. Альтернативно в вариантах осуществления, в которых нормализующие олигонуклеотиды были добавлены к клеткам или ядрам, присутствующим в подгруппах (ФИГ. 2, блок 22), способ может дополнительно включать в себя объединение подгрупп ядер или клеток с последующим распределением объединенных ядер во второе множество компартментов (ФИГ. 2, блок 24). Количество ядер или клеток, присутствующих в подгруппе и, следовательно, в каждом компартменте, может составлять по меньшей мере 1. Количество ядер или клеток в подгруппе не имеет ограничительного характера и может измеряться миллиардами. В одном из вариантов осуществления количество в подгруппе составляет не более чем 100 000 000, не более чем 10 000 000, не более чем 1 000 000, не более чем 100 000, не более чем 10 000, не более чем 4000, не более чем 3000, не более чем 2000 или не более чем 1000. В одном из вариантов осуществления количество ядер, присутствующих в подгруппе, может составлять от 1 до 1000, от 1000 до 10 000, от 10 000 до 100 000, или от 100 000 до 1 000 000, или от 1 000 000 до 10 000 000 или от 10 000 000 до 100 000 000. В одном из вариантов осуществления каждый компартмент может представлять собой лунку многолуночного планшета, такого как 96- или 384-луночный планшет. В одном из вариантов осуществления каждый компартмент может представлять собой каплю. Способы распределения ядер по подгруппам известны специалисту в данной области и являются стандартными. Хотя можно использовать цитометрию посредством сортировки клеток с активацией флуоресценции (FACS), также можно использовать и простое разведение. В одном из вариантов осуществления FACS-цитометрия не используется.
[00147] Количество компартментов на первой стадии распределения (ФИГ. 2, блок 24) может зависеть от используемого формата. Например, количество компартментов может составлять от 2 до 96 компартментов (при использовании 96-луночного планшета), от 2 до 384 компартментов (при использовании 384-луночного планшета). В одном из вариантов осуществления можно использовать множество планшетов. Например, можно использовать компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 96-луночных планшетов или компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 384-луночных планшетов. Если используемый тип компартмента представляет собой каплю, которая содержит два или более ядер или клеток, можно использовать любое количество капель, например по меньшей мере 10 000, по меньшей мере 100 000, по меньшей мере 1 000 000 или по меньшей мере 10 000 000 капель.
[00148] После того как ядра или клетки были помечены нормализующим олигонуклеотидом и, если это применимо или необходимо, распределены по подгруппам, можно использовать разные процедуры для окончательного получения библиотек разных нуклеиновых кислот в ядрах или клетках и последовательности нуклеиновых кислот (ФИГ. 2, блок 26). Процедура, используемая после мечения ядер нормализующими олигонуклеотидами, не носит ограничительного характера.
[00149] Комбинаторное индексирование транскриптомов одиночных клеток
[00150] Приведенное ниже описание способа комбинаторного секвенирования одиночных клеток ориентировано на seq-RNA и не носит ограничительного характера. В одном из вариантов осуществления способ включает в себя индексирование нуклеиновых кислот мРНК в распределенных ядрах (ФИГ. 3, блок 30). На данной стадии к имеющимся олигонуклеотидам, например, к хеширующим олигонуклеотидам, к нормализующим олигонуклеотидам или к обоим, также добавляется индекс. Этот индекс отличается от специфичного для подгруппы индекса, присутствующего в хеширующих олигонуклеотидах, и специфичного для популяции индекса, присутствующего в нормализующих олигонуклеотидах, и называется первым индексом. Соответственно, нуклеиновые кислоты, производные от молекул мРНК, после этой стадии имеют первый индекс, нуклеиновые кислоты, производные от хеширующих олигонуклеотидов, включают в себя специфичный для подгруппы индекс и первый индекс, а нуклеиновые кислоты, производные от нормализующих олигонуклеотидов, включают в себя специфичный для популяции индекс и первый индекс. В одном из вариантов осуществления генерация ядер для включения первого индекса включает в себя использование обратной транскриптазы с праймером олиго-dT для добавления индекса, случайной нуклеотидной последовательности и универсальной последовательности. Случайную последовательность используют в качестве уникального молекулярного идентификатора (UMI) для маркировки уникальных фрагментов нуклеиновых кислот. Случайную последовательность можно также использовать для помощи в удалении дубликатов при дальнейшей обработке. Универсальная последовательность служит комплементарной последовательностью для гибридизации на стадии лигирования, описанной в настоящем документе. Воздействие этими компонентами на ядра в условиях, подходящих для обратной транскрипции, приводит к получению популяции индексированных ядер, причем каждое ядро содержит две популяции индексированных фрагментов нуклеиновых кислот. Одна популяция получена в результате обратной транскрипции хеширующего олигонуклеотида или нормализующего олигонуклеотида, гибридизованного с праймером олиго-dT, а другая популяция получена в результате обратной транскрипции нуклеиновых кислот мРНК, гибридизованных с праймером олиго-dT. Индексированные фрагменты нуклеиновых кислот могут включать в себя и, как правило, включают в себя в синтезированной цепи индексную последовательность, указывающую на конкретный компартмент.
[00151] Индексированные ядра из множества компартментов можно объединять (ФИГ. 3, блок 31). Например, объединяют индексированные ядра из 2-24 компартментов, из 2-96 компартментов (если используется 96-луночный планшет) или из 2-384 компартментов (когда используется 384-луночный планшет). В одном из вариантов осуществления объединяют индексированные ядра из множества планшетов. Например, объединяют компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 96-луночных планшетов или компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 384-луночных планшетов. В одном из вариантов осуществления объединяют компартменты от четырех 386-луночных планшетов. Затем подгруппы этих комбинированных индексированных ядер, называемые в настоящем документе объединенными индексированными ядрами, распределяют в третье множество компартментов (ФИГ. 3, блок 31). Количество ядер в подгруппе и, следовательно, в каждом компартменте, частично основано на желании уменьшить коллизии индексов, т. е. попадание двух ядер, имеющих одинаковый индекс, в один и тот же компартмент на этой стадии способа. В одном из вариантов осуществления количество ядер, присутствующих в подгруппах, является приблизительно одинаковым. Способы распределения ядер по подгруппам известны специалисту в данной области и являются стандартными. К примерам относится, без ограничений, простое разведение. В одном из вариантов осуществления FACS-цитометрия не используется.
[00152] После распределения ядер по подгруппам следует встраивание в индексированные фрагменты нуклеиновых кислот в каждом компартменте второй индексной последовательности для генерации фрагментов с двойным индексированием. В результате получается дополнительное индексирование индексированных фрагментов нуклеиновых кислот (ФИГ. 3, блок 32).
[00153] В одном из вариантов осуществления встраивание второй индексной последовательности включает в себя лигирование шпилечного лигирующего дуплекса к индексированным фрагментам нуклеиновых кислот в каждом компартменте. При использовании шпилечного лигирующего дуплекса для введения универсальной последовательности, индекса или их комбинации в конец фрагмента целевой нуклеиновой кислоты, как правило, один конец дуплекса используется в качестве праймера для последующей амплификации. Напротив, в одном из вариантов осуществления используемый в настоящем документе шпилечный лигирующий дуплекс не выступает в качестве праймера. Преимуществом использования шпилечного лигирующего дуплекса, описанного в настоящем документе, является уменьшение лигирования «с собой самим», наблюдаемое для многих шпилечных лигирующих дуплексах, описанных в данной области. В одном из вариантов осуществления лигирующий дуплекс включает в себя пять элементов: 1) универсальную последовательность, которая комплементарна универсальной последовательности, присутствующей в праймере олиго-dT, 2) второй индекс, 3) идезоксиU, 4) нуклеотидную последовательность, которая может образовывать шпильку, и 5) обратный комплемент второго индекса. Вторые индексные последовательности уникальны для каждого компартмента, в котором размещены распределенные индексированные ядра (ФИГ. 3, блок 31) после добавления посредством обратной транскрипции первого индекса.
[00154] Ядра с двумя индексами из множества компартментов могут быть объединены (ФИГ. 3, блок 33). Например, объединяют ядра с дважды индексированными ядрами из 2-24 компартментов, из 2-96 компартментов (когда используется 96-луночный планшет) или из 2-384 компартментов (когда используется 384-луночный планшет). В одном из вариантов осуществления объединяют дважды индексированные ядра из множества планшетов. Например, объединяют компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 96-луночных планшетов или компартменты из по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 и т. д. 384-луночных планшетов. В одном из вариантов осуществления объединяют компартменты от четырех 386-луночных планшетов. Затем подгруппы этих комбинированных дважды индексированных ядер, называемые в настоящем документе объединенными индексированными ядрами, распределяют в четвертое множество компартментов (ФИГ. 3, блок 33). Количество ядер в подгруппе и, следовательно, в каждом компартменте частично основано на желании уменьшить коллизии индексов, т. е. попадание двух ядер, имеющих одинаковый индекс транспозазы, в один и тот же компартмент на этой стадии способа. В одном из вариантов осуществления в каждую лунку распределяют от 100 до 30 000 ядер. В одном из вариантов осуществления количество ядер в лунке составляет по меньшей мере 100, по меньшей мере 500, по меньшей мере 1000 или по меньшей мере 5000. В одном из вариантов осуществления количество ядер в лунке составляет не более чем 30 000, не более чем 25 000, не более чем 20 000 или не более чем 15 000. В одном из вариантов осуществления количество ядер, присутствующих в подгруппе, может составлять от 100 до 1000, от 1000 до 10 000, от 10 000 до 20 000 или от 20 000 до 30 000. В одном из вариантов осуществления в каждую лунку распределены 2500 ядер. В одном из вариантов осуществления количество ядер, присутствующих в подгруппах, является приблизительно одинаковым. Способы распределения ядер по подгруппам известны специалисту в данной области и являются стандартными. К примерам относится, без ограничений, простое разведение. В одном из вариантов осуществления FACS-цитометрия не используется.
[00155] За распределением ядер с двумя индексами по подгруппам может следовать синтез второй цепи ДНК (ФИГ. 3, блок 34). Альтернативно синтез второй цепи ДНК может происходить до распределения, например, в массе.
[00156] Затем ядра могут быть подвергнуты тагментации (ФИГ. 3, блок 35). Каждый компартмент, содержащий ядра с двойным индексом, включает в себя транспосомный комплекс. Транспосомный комплекс может быть добавлен в каждый компартмент до, после или одновременно с добавлением в компартмент подгруппы ядер. Транспосомный комплекс, который представляет собой транспозазу, связанную с сайтом распознавания транспозазы, может вставлять сайт распознавания транспозазы в целевую нуклеиновую кислоту в ядре в ходе процесса, который иногда называют «тагментацией». В некоторых таких событиях вставки одна цепь сайта распознавания транспозазы может переноситься в целевую нуклеиновую кислоту. Такая цепь называется «переносимой цепью». В одном из вариантов осуществления транспосомный комплекс включает в себя димерную транспозазу, имеющую две субъединицы и две несвязные транспозонные последовательности. В другом варианте осуществления транспозаза включает в себя димерную транспозазу, имеющую две субъединицы и связную транспозонную последовательность. В одном из вариантов осуществления 5’-конец одной или обеих цепей сайта распознавания транспозазы может быть фосфорилирован.
[00157] Некоторые варианты осуществления могут включать в себя использование гиперактивной транспозазы Tn5 и сайта распознавания транспозазы Tn5 (Goryshin and Reznikoff, J. Biol. Chem., 273:7367 (1998)) или транспозазу MuA и сайт распознавания транспозазы Mu, содержащий концевые последовательности R1 и R2 (Mizuuchi, K., Cell, 35: 785, 1983; Savilahti, H, et al., EMBO J., 14: 4893, 1995). Специалист в данной области также после оптимизации может использовать мозаичные концевые (ME) последовательности Tn5.
[00158] К дополнительным примерам систем транспонирования, которые можно использовать с определенными вариантами осуществления композиций и способов, предложенных в настоящем документе, относятся Staphylococcus aureus Tn552 (Colegio et al., J. Bacteriol., 183: 2384-8, 2001; Kirby C et al., Mol. Microbiol., 43: 173-86, 2002), Ty1 (Devine & Boeke, Nucleic Acids Res., 22: 3765-72, 1994 и международная публикация WO 95/23875), транспозон Tn7 (Craig, N L, Science. 271: 1512, 1996; Craig, N L, обзор в: Curr Top Microbiol Immunol., 204:27-48, 1996), Tn/O и IS10 (Kleckner N, et al., Curr Top Microbiol Immunol., 204:49-82, 1996), транспозаза Mariner (Lampe D J, et al., EMBO J., 15: 5470-9, 1996), Tc1 (Plasterk R H, Curr. Topics Microbiol. Immunol., 204: 125-43, 1996), P-элемент (Gloor, G B, Methods Mol. Biol., 260: 97-114, 2004), Tn3 (Ichikawa & Ohtsubo, J Biol. Chem. 265:18829-32, 1990), бактериальные инсерционные последовательности (Ohtsubo & Sekine, Curr. Top. Microbiol. Immunol. 204: 1-26, 1996), ретровирусы (Brown, et al., Proc Natl Acad Sci USA, 86:2525-9, 1989) и ретротранспозон дрожжей (Boeke & Corces, Annu Rev Microbiol. 43:403-34, 1989). К дополнительным примерам относятся IS5, Tn10, Tn903, IS911 и сконструированные версии ферментов семейства транспозаз (Zhang et al., (2009) PLoS Genet. 5:e1000689. Epub 2009 Oct 16; Wilson C. et al (2007) J. Microbiol. Methods 71:332-5).
[00159] К другим примерам интеграз, которые можно использовать со способами и композициями, предложенными в настоящем документе, относятся ретровирусные интегразы и сайты распознавания интеграз для таких ретровирусных интеграз, например, интеграз из ВИЧ-1, ВИЧ-2, SIV, PFV-1, RSV.
[00160] Транспозонные последовательности, подходящие для использования со способами и композициями, описанными в настоящем документе, предложены в заявках на патент США № 2012/0208705, опубликованной заявке на патент США № WO 2012/0208724 и опубликованной заявке на международный патент № WO 2012/061832. В некоторых вариантах осуществления транспозонная последовательность включает в себя первый сайт распознавания транспозазы, второй сайт распознавания транспозазы и необязательную индексную последовательность, присутствующую между двумя сайтами распознавания транспозаз.
[00161] Некоторые транспосомные комплексы, подходящие для использования в настоящем изобретении, включают в себя транспозазу, имеющую две транспозонные последовательности. В некоторых таких вариантах осуществления две транспозонные последовательности не связаны друг с другом; иными словами, транспозонные последовательности не являются связными друг с другом. Примеры таких транспосом известны в данной области (см., например, опубликованную заявку на патент США № 2010/0120098).
[00162] В некоторых вариантах осуществления транспосомный комплекс включает в себя транспозонную нуклеотидную последовательность, которая связывает две субъединицы транспозазы с образованием «петлевого комплекса» или «петлевой транспосомы». В одном примере транспосома включает в себя димерную транспозазу и транспозонную последовательность. Петлевые комплексы могут обеспечивать вставку транспозонов в целевую ДНК с сохранением информации о порядке исходной целевой ДНК и без фрагментирования целевой ДНК. Следует понимать, что петлевые структуры могут вставлять в целевую нуклеиновую кислоту желаемые нуклеотидные последовательности, такие как индексы, сохраняя при этом физическую связность целевой нуклеиновой кислоты. В некоторых вариантах осуществления транспозонная последовательность петлевого транспосомного комплекса может включать в себя сайт фрагментации, так что транспозонная последовательность может быть фрагментирована с созданием транспосомного комплекса, содержащего две транспозонных последовательности. Такие транспосомные комплексы могут использоваться для обеспечения того, чтобы соседние фрагменты целевой ДНК, в которую вставляются транспозоны, принимали кодовые комбинации, которые можно однозначно собирать на более поздней стадии анализа.
[00163] Транспосомный комплекс может необязательно включать в себя индексную последовательность, также называемую индексом транспозазы. Индексная последовательность присутствует в составе транспозонной последовательности. Использование транспосомного комплекса, имеющего индекс, позволяет получать целевые фрагменты нуклеиновых кислот, которые содержат дополнительный индекс. В одном из вариантов осуществления индексная последовательность может присутствовать на переносимой цепи, цепи сайта распознавания транспозазы, которая переносится в целевую нуклеиновую кислоту.
[00164] Таким образом, тагментацию можно использовать для получения фрагментов нуклеиновых кислот, имеющих разные типы нуклеотидных последовательностей на каждом конце. В одном из вариантов осуществления полученные фрагменты нуклеиновых кислот включают в себя разные нуклеотидные последовательности на каждом конце, такие как последовательность праймера N5 на одном конце и праймер N7 на другом конце, или разные универсальные последовательности на каждом конце. Примеры подходящих универсальных последовательностей включают в себя, например, шпилечный лигирующий дуплекс и универсальную последовательность, с которой может связываться универсальный праймер. В других вариантах осуществления тагментацию можно использовать для получения фрагментов нуклеиновых кислот, имеющих на каждом конце нуклеотидную последовательность одинакового типа. В одном из вариантов осуществления полученные фрагменты нуклеиновых кислот включают в себя на каждом конце нуклеотидную последовательность, имеющую универсальную последовательность, с которой может связываться универсальный праймер, и индекс или как универсальную последовательность, так и индекс. Универсальная последовательность может служить комплементарной последовательностью для гибридизации на описанной в настоящем документе стадии амплификации для введения третьего индекса.
[00165] За тагментацией ядер и процессингом фрагментов нуклеиновых кислот может следовать процесс очистки для повышения чистоты молекул. Можно использовать любой приемлемый процесс очистки, такой как электрофорез, эксклюзионная хроматография размеров или т. п. В некоторых вариантах осуществления для отделения желаемых молекул ДНК от, например, невстроенных праймеров, и для выбора нуклеиновых кислот в зависимости от размера можно использовать твердофазные парамагнитные микроносители с обратимой иммобилизацией. Твердофазные парамагнитные гранулы с обратимой иммобилизацией доступны в продаже в компаниях Beckman Coulter (Agencourt AMPure XP), Thermofisher (MagJet), Omega Biotek (Mag-Bind), Promega Beads (Promega) и Kapa Biosystems (Kapa Pure Beads).
[00166] Удаление идезоксиU, присутствующего в области шпильки шпилечного легирующего дуплекса, необязательно встроенного во фрагменты нуклеиновой кислоты, может происходить до, во время или после очистки. Удаление остатка урацила может быть выполнено любым доступным способом, и в одном из вариантов осуществления используется специфический для урацила вырезающий реагент (USER), предложенный компанией NEB.
[00167] За тагментацией ядер может следовать встраивание во фрагменты нуклеиновых кислот с двойным индексом в каждом компартменте третьей индексной последовательности для получения трижды индексированных фрагментов, причем третья индексная последовательность в каждом компартменте отличается от первой и второй индексных последовательностей в компартментах. Это приводит к дополнительному индексированию индексированных фрагментов нуклеиновых кислот (ФИГ. 3, блок 36) перед иммобилизацией и секвенированием. Третий индекс может быть включен посредством стадии амплификации, такой как ПЦР. В одном из вариантов осуществления универсальные последовательности, присутствующие на концах фрагментов нуклеиновых кислот с двойным индексированием (например, нуклеотидную последовательность шпилечного лигирующего дуплекса на одном конце и нуклеотидную последовательность, вставленную транспосомным комплексом, на другом конце) можно использовать для связывания с праймерами и удлинения в реакции амплификации. Как правило, используют два разных праймера. Один праймер гибридизуется с универсальными последовательностями на 3’-конце одной цепи фрагментов нуклеиновых кислот с двумя индексами, а второй праймер гибридизуется с универсальными последовательностями на 3’-конце другой цепи фрагментов нуклеиновых кислот с двумя индексами. Таким образом, якорные последовательности (например, сайт, с которым отжигают для секвенирования универсальный праймер, такой как секвенирующий праймер для чтения 1 или чтения 2) на всех праймерах могут быть разными. Каждый из подходящих праймеров может включать в себя дополнительные универсальные последовательности, такие как универсальная последовательность захвата (например, сайт, с которым гибридизуется захватный олигонуклеотид, причем захватный олигонуклеотид может быть иммобилизован на поверхности твердого субстрата). Поскольку каждый праймер включает в себя индекс, результатом данной стадии является добавление дополнительной индексной последовательности, по одной с каждого конца фрагментов нуклеиновых кислот, для получения фрагментов с тройным индексом. В одном из вариантов осуществления для добавления тройного индекса можно использовать индексированные праймеры, такие как индексированный праймер P5 и индексированный праймер P7. Тройные индексированные фрагменты объединяют, и их можно подвергнуть стадии очистки, как описано в настоящем документе.
[00168] Полученные фрагменты с тройным индексом в совокупности образуют библиотеку нуклеиновых кислот, которая может быть иммобилизована и затем секвенирована. Термин «библиотека», также называемая в настоящем документе «библиотекой секвенирования», относится к набору фрагментов нуклеиновых кислот из отдельных ядер, содержащих известные универсальные последовательности на их 3’- и 5’-концах. В настоящем варианте осуществления библиотека включает в себя нуклеиновые кислоты полного транскриптома из одного или более выделенных ядер, и она может использоваться для выполнения секвенирования полного транскриптома.
[00169] Подготовка иммобилизованных образцов для секвенирования
[00170] Для секвенирования можно приготовить множество фрагментов с множественными индексами. Например, в тех вариантах осуществления, в которых получают транскриптомные библиотеки фрагментов с тройными индексами, фрагменты с тройными индексами объединяют и очищают, обогащают, как правило, путем иммобилизации и/или амплификации, а затем секвенируют (ФИГ. 3, блок 37). Способы прикрепления индексированных фрагментов из одного или более источников к субстрату известны специалистам в данной области. В одном из вариантов осуществления индексированные фрагменты обогащают с использованием множества захватных олигонуклеотидов, обладающих специфичностью к индексированным фрагментам, и захватные олигонуклеотиды могут быть иммобилизованы на поверхности твердого субстрата. Например, захватные олигонуклеотиды могут включать в себя первый член универсальной связывающейся пары, и причем второй член связывающейся пары иммобилизован на поверхности твердого субстрата. Аналогичным образом, способы амплификации иммобилизованных фрагментов с тройным индексом включают в себя, без ограничений, мостиковую амплификацию и кинетическое исключение. Способы иммобилизации и амплификации перед секвенированием описаны, например, в публикациях Bignell et al. (US 8,053,192), Gunderson et al. (WO2016/130704), Shen et al. (US 8,895,249) и Pipenburg et al. (US 9,309,502).
[00171] Объединенный образец можно иммобилизовать при подготовке к секвенированию. Секвенирование можно выполнять на матрице из отдельных молекул или можно проводить амплификацию перед секвенированием. Амплификацию можно проводить с использованием одного или более иммобилизованных праймеров. Иммобилизованный (-ые) праймер (-ы) может (могут) представлять собой, например, сплошной слой на плоской поверхности или на пуле гранул. Пул гранул может быть выделен в эмульсию, причем одна гранула находится в каждом «компартменте» эмульсии. При концентрации всего в один темплат на «компартмент» на каждой грануле амплифицируется только один темплат.
[00172] В настоящем документе термин «твердофазная амплификация» относится к любой реакции амплификации нуклеиновых кислот, проведенной на твердой подложке или в связи с ней так, что все или участок амплифицированных продуктов иммобилизуются на твердой подложке по мере их образования. В частности, термин включает в себя твердофазную полимеразную цепную реакцию (твердофазную ПЦР) и твердофазную изотермическую амплификацию, которые представляют собой реакции, аналогичные стандартной амплификации в жидкой фазе, за исключением того, что один или оба из прямого и обратного праймеров для амплификации иммобилизованы на твердой подложке. Твердофазные ПЦР охватывают такие системы, как эмульсии, в которых один праймер заякоривают на грануле, а другой находится в свободном растворе, и образование колоний в твердофазных гелевых матрицах, в которых один праймер заякоривается на поверхности, а другой находится в свободном растворе.
[00173] В некоторых вариантах осуществления твердая подложка имеет рельефную поверхность. Термин «структурированная поверхность» относится к расположению разных областей в открытом слое твердой подложки или на нем. Например, одна или более областей могут быть элементами, в которых присутствуют один или более праймеров для амплификации. Элементы могут быть разделены промежуточными областями, в которых отсутствуют праймеры для амплификации. В некоторых вариантах осуществления рельеф может иметь координатный формат из x-y элементов, расположенных в строках и столбцах. В некоторых вариантах осуществления рельеф может иметь повторяющееся расположение элементов и/или промежуточных областей. В некоторых вариантах осуществления рельеф может иметь случайное расположение элементов и/или промежуточных областей. Примеры рельефных поверхностей, которые можно использовать в способах и композициях, описанных в настоящем документе, описаны в патентах США №№ 8,778,848, 8,778,849 и 9,079,148 и заявке на патент США № 2014/0243224.
[00174] В некоторых вариантах осуществления твердая подложка содержит матрицу из лунок или углублений на поверхности. Такую подложку можно изготовить с использованием различных методик, известных в данной области, в том числе, без ограничений, фотолитографии, методик штамповки, методик литья и методик микротравления. Специалистам в данной области будет понятно, что методика зависит от состава и формы субстрата матрицы.
[00175] Элементы на структурированной поверхности могут представлять собой лунки в матрице лунок (например, микролунки или нанолунки) на стеклянных, кремниевых, пластиковых или других подходящих твердых подложках со структурированным ковалентно связанным гелем, таким как поли(N-(5-азидоацетамидилпентил)акриламид-со-акриламид) (PAZAM, см., например, публикации США № 2013/184796, WO 2016/066586 и WO 2015/002813). Процесс позволяет получить гелевые подушечки, используемые для секвенирования, которые могут сохранять стабильность в течение прогонов секвенирования с большим количеством циклов. Ковалентное связывание полимера с лунками полезно для сохранения геля в структурированных элементах на протяжении всего срока службы структурированного субстрата при различных вариантах использования. Однако во многих вариантах осуществления гель необязательно ковалентно связан с лунками. Например, при некоторых условиях в качестве гелевого материала может использоваться не содержащий силана акриламид (SFA, см., например, патент США № 8,563,477), который не имеет ковалентной связи с какой-либо частью структурированного субстрата.
[00176] В конкретных вариантах осуществления структурированный субстрат можно получить путем структурирования материала твердой подложки с помощью лунок (например, микролунок или нанолунок), покрытия структурированной подложки гелевым материалом (например, PAZAM, SFA или их химически модифицированными вариантами, например, ацидолизированный вариант SFA (ацидо-SFA)) и полировки подложки с гелем, например, химической или механической полировки, чтобы сохранить гель в лунках и удалить или инактивировать практически весь гель в промежуточных областях на поверхности структурированного субстрата между лунками. Праймер для нуклеиновых кислот может быть прикреплен к гелевому материалу. Затем раствор фрагментов с тройным индексом можно привести в контакт с полированным субстратом, так чтобы отдельные фрагменты с тройным индексом засевали отдельные лунки посредством взаимодействий с праймерами, присоединенными к гелевому материалу; однако целевые нуклеиновые кислоты не будут занимать промежуточные области из-за отсутствия или неактивности гелевого материала. Амплификация фрагментов с тройным индексом будет ограничена лунками, так как отсутствие или неактивность геля в промежуточных областях предотвращает миграцию растущей колонии нуклеиновых кислот наружу. Процесс может быть удобен для производства, поскольку является масштабируемым и использует традиционные микро- и нанотехнологические способы.
[00177] Хотя описание включает в себя «твердофазные» способы амплификации, в которых иммобилизован только один праймер амплификации (другой праймер обычно присутствует в свободном растворе), в одном из вариантов осуществления предусмотрена твердая подложка, на которой иммобилизированы как прямой, так и обратный праймеры. На практике будут присутствовать «множество» идентичных прямых праймеров и/или «множество» идентичных обратных праймеров, иммобилизованных на твердой подложке, поскольку в процессе амплификации необходим избыток праймеров для поддержания амплификации. В настоящем документе ссылки на прямой и обратный праймеры следует интерпретировать соответственно как включающие «множества» таких праймеров, если иное не диктуется контекстом.
[00178] Как будет понятно читателю, являющемуся специалистом, для любой данной реакции амплификации требуется по меньшей мере один тип прямого праймера и по меньшей мере один тип обратного праймера, специфичный для амплифицируемого темплата. Однако в определенных вариантах осуществления прямой и обратный праймеры могут включать в себя специфичные для темплата участки идентичной последовательности и могут иметь полностью идентичную нуклеотидную последовательность и структуру (включая любые ненуклеотидные модификации). Иными словами, можно проводить твердофазную амплификацию с использованием только одного типа праймеров, и такие однопраймерные способы входят в объем настоящего описания. В других вариантах осуществления можно использовать прямой и обратный праймеры, которые содержат идентичные специфические для темплата последовательности, но которые отличаются некоторыми другими структурными особенностями. Например, один тип праймера может содержать ненуклеотидную модификацию, отсутствующую в другом.
[00179] Праймеры для твердофазной амплификации предпочтительно иммобилизуют путем одноточечного ковалентного присоединения к твердой подложке на 5’-конце праймера или вблизи него, оставляя специфичный для темплата участок праймера свободным для отжига с распознаваемым темплатом и свободную 3’-гидроксильную группу для достройки праймера. Для этой цели можно использовать любое подходящее средство ковалентного связывания, известное в данной области. Выбранная для связывания химическая реакция зависит от характера твердой подложки и любых примененных к ней производных или функционализирующих модификаций. Сам праймер может включать в себя функциональную группу, которая может представлять собой ненуклеотидную химическую модификацию для облегчения присоединения. В конкретном варианте осуществления праймер может включать в себя серосодержащий нуклеофил, такой как фосфоротиоат или тиофосфат, на 5’-конце. В случае полиакриламидных гидрогелей на твердой подложке данный нуклеофил будет связываться с бромацетамидной группой, присутствующей в гидрогеле. Более конкретным способом прикрепления праймеров и темплатов к твердой подложке является прикрепление 5’-фосфоротиоата к гидрогелю, образованному из полимеризованного акриламида и N-(5-бромацетамидилпентил)акриламида (BRAPA), как описано в международной патентной публикации № WO 05/065814.
[00180] В определенных вариантах осуществления изобретения можно использовать твердые подложки, содержащие инертный субстрат или матрикс (например, стеклянные пластины, полимерные гранулы и т. д.), которые были «функционализированы», например, посредством нанесения слоя или покрытия из промежуточного материала, содержащего реакционноспособные группы, которые обеспечивают ковалентное связывание с биомолекулами, такими как полинуклеотиды. Примеры таких субстратов включают в себя, без ограничений, полиакриламидные гидрогели, нанесенные на инертный субстрат, такой как стекло. В таких вариантах осуществления биомолекулы (например, полинуклеотиды) могут напрямую ковалентно прикрепляться к промежуточному материалу (например, гидрогелем), но промежуточный материал сам по себе может быть нековалентно прикреплен к субстрату или матриксу (например, стеклянному субстрату). Термин «ковалентное присоединение к твердой подложке» следует интерпретировать, соответственно, как охватывающий данный тип размещения.
[00181] Объединенные образцы можно амплифицировать на гранулах, причем каждая гранула содержит прямой и обратный праймеры амплификации. В конкретном варианте осуществления библиотеку фрагментов с тройным индексом используют для получения кластерных матриц с колониями нуклеиновых кислот, аналогичными описанным в патентной публикации США № 2005/0100900, патентах США №№ 7,115,400, WO 00/18957 и WO 98/44151 посредством твердофазной амплификации и, более конкретно, твердофазной изотермической амплификации. Термины «кластер» и «колония» в настоящем документе используются взаимозаменяемо и относятся к дискретному сайту на твердой подложке, содержащему множество идентичных иммобилизованных цепей нуклеиновых кислот и множество идентичных иммобилизованных комплементарных цепей нуклеиновых кислот. Термин «кластеризованная матрица» относится к матрице, образованной из таких кластеров или колоний. В этом контексте термин «матрица» не предполагает упорядоченного расположения кластеров.
[00182] Термин «твердая фаза» или «поверхность» используется для обозначения либо плоской матрицы, в которой праймеры прикреплены к плоской поверхности, например, к стеклянным, кварцевым или пластиковым стеклам микроскопа, либо к аналогичным устройствам проточных кювет; гранул, причем один или два праймера прикреплены к гранулам, и гранулы подвергают амплификации; или матрицы из гранул на поверхности после амплификации на гранулах.
[00183] Кластеризованные матрицы можно получить с использованием либо процесса термоциклирования, как описано в WO 98/44151, либо процесса, в котором температуру поддерживают на постоянном уровне, а циклы удлинения и денатурации выполняют с использованием изменений реагентов. Такие способы изотермической амплификации описаны в заявке на патент WO 02/46456 и в патентной публикации США № 2008/0009420. В связи с более низкими температурами, используемыми в изотермическом процессе, в некоторых вариантах осуществления он является особенно предпочтительным.
[00184] Следует понимать, что любую из методологий амплификации, описанных в настоящем документе или по существу известных в данной области, можно использовать с универсальными или специфичными для цели праймерами для амплификации иммобилизованных фрагментов ДНК. Подходящие способы амплификации включают в себя, без ограничений, полимеразную цепную реакцию (ПЦР), амплификацию с замещением цепей (SDA), транскрипционно-опосредованную амплификацию (ТМА) и амплификацию на основе нуклеотидной последовательности (NASBA), как описано в патенте США № 8,003,354. Вышеуказанные способы амплификации можно использовать для амплификации одной или более интересующих нуклеиновых кислот. Например, ПЦР, включая мультиплексную ПЦР, SDA, ТМА, NASBA и т. п., можно использовать для амплификации иммобилизованных фрагментов ДНК. В некоторых вариантах осуществления в реакцию амплификации включены праймеры, специфичные для интересующего полинуклеотида.
[00185] Другие подходящие способы амплификации полинуклеотидов могут включать в себя технологии удлинения и лигирования олигонуклеотидов, амплификации по типу катящегося кольца (RCA) (Lizardi et al., Nat. Genet. 19:225-232 (1998)) и лигирование олигонуклеотидных зондов (OLA) (см. по существу патенты США №№ 7,582,420, 5,185,243, 5,679,524 и 5,573,907; EP 0 320 308 B1; EP 0 336 731 B1; EP 0 439 182 B1; WO 90/01069; WO 89/12696 и WO 89/09835). Следует понимать, что эти методологии амплификации могут быть разработаны с возможностью амплификации иммобилизованных фрагментов ДНК. Например, в некоторых вариантах осуществления способ амплификации может включать в себя реакции амплификации с лигирующим зондом или лигирования олигонуклеотидных зондов (OLA), которые содержат праймеры, напрямую нацеленные на интересующую нуклеиновую кислоту. В некоторых вариантах осуществления способ амплификации может включать в себя реакцию удлинения праймеров - лигирования, которая содержит праймеры, напрямую нацеленные на интересующую нуклеиновую кислоту. В качестве не имеющего ограничительного характера примера праймеров для реакции удлинения и лигирования праймеров, которые могут быть специально выполнены с возможностью амплификации интересующей нуклеиновой кислоты, амплификация может включать в себя праймеры, используемые для анализа GoldenGate (Illumina, Inc., г. Сан-Диего, штат Калифорния, США), как показано на примерах в патенте США №№ 7,582,420 и 7,611,869.
[00186] ДНК-наносферы также можно использовать в комбинации со способами и композициями, описанными в настоящем документе. Способы создания и использования ДНК-наносфер для геномного секвенирования можно найти, например, в патентах США и публикациях патентов США №№ 7,910,354, 2009/0264299, 2009/0011943, 2009/0005252, 2009/0155781, 2009/0118488 и описаны, например, в публикации Drmanac et al. (2010, Science 327(5961): 78-81). Вкратце, после фрагментации ДНК геномной библиотеки адаптеры лигируют с фрагментами, причем лигированные адаптером фрагменты циркуляризуют посредством лигирования циркуляризационной лигазой и проводят амплификацию по типу катящегося кольца (как описано в публикации Lizardi et al., 1998. Nat. Genet. 19:225-232 и US 2007/0099208 A1). Удлиненная конкатамерная структура ампликонов способствует скручиванию, в результате чего образуются компактные ДНК-наносферы. ДНК-наносферы могут захватываться на субстраты, предпочтительно создавая упорядоченную или рельефную матрицу таким образом, чтобы сохранялось расстояние между наносферами, чтобы можно было секвенировать отдельные ДНК-наносферы. В некоторых вариантах осуществления, например, таких как используемые компанией Complete Genomics (г. Маунтин-Вью, штат Калифорния, США), перед циркуляризацией проводят последовательные раунды лигирования адаптера, амплификации и расщепления для получения конструктов с головой и хвостом, имеющих несколько фрагментов геномной ДНК, разделенных адаптерными последовательностями.
[00187] Примеры способов изотермической амплификации, которые могут использоваться в способе настоящего изобретения, включают в себя, без ограничений, амплификацию с множественным вытеснением (MDA), пример которой приведен в публикации Dean et al., Proc. Natl. Acad. Sci. USA 99:5261-66 (2002), или изотермическую амплификацию нуклеиновых кислот с замещениями цепей, пример которой приведен в патенте США № 6,214,587. Другие не основанные на ПЦР способы, которые могут использоваться в настоящем изобретении, включают в себя, например, амплификацию с замещением цепей (SDA), описанную, например, в Walker et al., Molecular Methods for Virus Detection, Academic Press, Inc., 1995; патентах США №№ 5,455,166 и 5,130,238 и публикации Walker et al., Nucl. Acids Res. 20:1691-96 (1992), или амплификацию с замещением гиперразветвленной цепи, описанную, например, в публикации Lage et al., Genome Res. 13:294-307 (2003). Можно использовать способы изотермической амплификации, например, замещающую цепь полимеразу Phi 29 или большой фрагмент ДНК-полимеразы Bst, 5’->3’ экзо- для амплификации геномной ДНК со случайными праймерами. Полимеразы позволяют использовать преимущества, которые дает их высокая процессивность и активность по замещению цепей. Высокая процессивность позволяет полимеразам производить фрагменты длиной 10-20 тыс. п. н. Как указано выше, более мелкие фрагменты могут быть получены в изотермических условиях с использованием полимераз, обладающих низкой процессивностью и активностью по замещению цепей, таких как полимераза Кленова. Дополнительное описание реакций амплификации, условий и компонентов подробно представлено в описании патента США № 7,670,810.
[00188] Другим способом амплификации полинуклеотидов, подходящим для настоящего описания, является ПЦР с метками, в которой используется популяция двухдоменных праймеров, имеющих константную 5’-область, за которой следует случайная 3’-область, как описано, например, в публикации Grothues et al. Nucleic Acids Res. 21(5):1321-2 (1993). Первые циклы амплификации выполняют для обеспечения множества инициаций на денатурированной нагреванием ДНК на основе индивидуальной гибридизации со случайно синтезированной 3’-областью. Вследствие характера 3’-области предполагается, что сайты инициации случайно распределены по всему геному. После этого несвязанные праймеры можно удалить и провести дополнительную репликацию с использованием праймеров, комплементарных константной 5’-области.
[00189] В некоторых вариантах осуществления изотермическую амплификацию можно выполнить с использованием амплификации с кинетическим исключением (KEA), также называемой эксклюзионной амплификацией (ExAmp). Библиотеку нуклеиновых кислот настоящего описания можно получить с использованием способа, который включает в себя стадию взаимодействия реагента для амплификации для получения множества сайтов амплификации, каждый из которых включает в себя по существу клональную популяцию ампликонов отдельной целевой нуклеиновой кислоты, послужившей затравкой для сайта. В некоторых вариантах осуществления реакция амплификации продолжается до тех пор, пока не будет получено достаточное количество ампликонов для заполнения емкости соответствующего сайта амплификации. Такое заполнение уже засеянного сайта до емкости подавляет посадку и амплификацию целевых нуклеиновых кислот на сайте, в результате чего на сайте получается клональная популяция ампликонов. В некоторых вариантах осуществления кажущуюся клональность можно обеспечить даже в том случае, если емкость сайта амплификации не заполнена до того, как вторая целевая нуклеиновая кислота достигнет сайта. При некоторых условиях амплификация первой целевой нуклеиновой кислоты может продолжаться до момента получения достаточного количества копий для эффективного вытеснения или преодоления продукции копий второй целевой нуклеиновой кислоты, достигающей сайта. Например, в варианте осуществления, в котором используется процесс мостиковой амплификации на круговом элементе диаметром менее 500 нм, было определено, что после 14 циклов экспоненциальной амплификации первой целевой нуклеиновой кислоты загрязнение того же сайта второй целевой нуклеиновой кислотой приведет к количеству загрязняющих ампликонов, недостаточному для того, чтобы оно неблагоприятно сказалось на анализе секвенирования методом синтеза с использованием платформы для секвенирования Illumina.
[00190] В некоторых вариантах осуществления сайты амплификации в матрице могут быть, но не обязательно должны быть полностью клональными. Вместо этого в некоторых областях применения отдельный сайт амплификации может быть преимущественно заполнен ампликонами первого фрагмента с тройным индексом и может также содержать низкий уровень загрязняющих ампликонов второй целевой нуклеиновой кислоты. Матрица может иметь один или более сайтов амплификации, содержащих низкий уровень загрязняющих ампликонов, при условии, что уровень загрязнения не оказывает неприемлемого влияния на последующее использование матрицы. Например, если матрица предназначена для использования при обнаружении, приемлемым уровнем загрязнения будет уровень, который недопустимым образом не влияет на соотношение сигнал/шум или на разрешение методики обнаружения. Соответственно, кажущаяся клональность по существу уместна для конкретного использования или применения матрицы, полученной способами, описанными в настоящем документе. Примеры уровней загрязнения, которые могут быть приемлемы для конкретных областей применения на отдельном сайте амплификации, включают в себя, без ограничений, не более 0,1%, 0,5%, 1%, 5%, 10% или 25% загрязняющих ампликонов. Матрица может включать в себя один или более сайтов амплификации, имеющих эти примеры уровней загрязняющих ампликонов. Например, до 5%, 10%, 25%, 50%, 75% или даже 100% сайтов амплификации в матрице могут содержать некоторое количество загрязняющих ампликонов. Следует понимать, что в матрице или другом наборе сайтов по меньшей мере 50%, 75%, 80%, 85%, 90%, 95% или 99% или более сайтов могут быть клональными или кажущимися клональными.
[00191] В некоторых вариантах осуществления кинетическое исключение может возникать, когда процесс происходит с достаточно высокой скоростью, чтобы эффективно исключить появление другого события или процесса. Возьмем, например, создание нуклеотидной матрицы, где сайты матрицы случайным образом засеяны из раствора фрагментами с тройным индексом, и копии фрагментов с тройным индексом создаются в процессе амплификации, заполняя каждый из засеянных сайтов до его емкости. В соответствии со способами кинетического исключения настоящего изобретения процессы посева и амплификации могут происходить одновременно в условиях, в которых скорость амплификации превышает скорость посева. Таким образом, относительно высокая скорость получения копий в сайте, засеянном первой целевой нуклеиновой кислотой, будет эффективно исключать засевание сайта амплификации второй нуклеиновой кислотой. Способы амплификации с кинетическим исключением могут выполняться так, как подробно описано в публикации заявки на патент США № 2013/0338042.
[00192] В кинетическом исключении можно использовать относительно низкую скорость инициирования амплификации (например, низкую скорость получения первой копии фрагмента с тройным индексом) относительно более высокой скорости получения последующих копий фрагмента с тройным индексом (или первой копии фрагмента с тройным индексом). В примере из предыдущего абзаца кинетическое исключение происходит из-за относительно низкой скорости посева фрагментов с тройным индексом (например, относительно медленной диффузии или транспорта) относительно высокой скорости, с которой происходит амплификация и заполнение сайта копиями фрагмента-затравки с тройным индексом. В другом примере осуществления кинетическое исключение может происходить вследствие задержки в образовании первой копии фрагмента с тройным индексом, засеявшего сайт (например, отсроченная или медленная активация) относительно высокой скорости, с которой образуются и заполняют сайт последующие копии. В этом примере конкретный сайт может быть засеян несколькими разными фрагментами с тройным индексом (например, в каждом сайте перед амплификацией могут присутствовать несколько фрагментов с тройным индексом). Однако образование первой копии любого данного фрагмента с тройным индексом может активироваться случайным образом так, чтобы средняя скорость образования первой копии была относительно низкой по сравнению со скоростью, с которой создаются последующие копии. В этом случае, хотя конкретный сайт может быть засеян несколькими разными фрагментами с тройным индексом, кинетическое исключение позволит провести амплификацию только одного из этих фрагментов с тройным индексом. Более конкретно, после активации для амплификации первого фрагмента с тройным индексом сайт быстро заполняется его копиями, таким образом предотвращая образование на сайте копий второго фрагмента с тройным индексом.
[00193] В одном из вариантов осуществления способ осуществляют с одновременной (i) транспортировкой фрагментов с тройным индексом до сайтов амплификации со средней скоростью транспортировки и (ii) амплификацией фрагментов с тройным индексом, находящихся на сайтах амплификации, со средней скоростью амплификации, причем средняя скорость амплификации превышает среднюю скорость транспортировки (патент США № 9,169,513). Соответственно, в таких вариантах осуществления кинетическое исключение может достигаться за счет использования относительно низкой скорости транспортировки. Например, может быть выбрана достаточно низкая концентрация фрагментов с тройным индексом для достижения желаемой средней скорости транспортировки, причем более низкие концентрации приводят к более медленным средним скоростям транспортировки. Альтернативно или дополнительно для снижения скорости транспортировки можно использовать раствор с высокой вязкостью и/или введение в раствор реагентов для молекулярного стеснения. Примеры подходящих для использования реагентов для молекулярного стеснения включают в себя, без ограничений, полиэтиленгликоль (ПЭГ), фиколл, декстран или поливиниловый спирт. Примеры реагентов и составов для молекулярного стеснения представлены в патенте США № 7,399,590, включенном в настоящий документ путем ссылки. Еще одним фактором, который можно корректировать для достижения желаемой скорости транспортировки, является средний размер целевых нуклеиновых кислот.
[00194] Амплификационный реагент может включать в себя дополнительные компоненты, которые способствуют образованию ампликонов и в некоторых случаях повышают скорость образования ампликонов. Примером является рекомбиназа. Рекомбиназа может способствовать образованию ампликона, обеспечивая повторяющееся внедрение/удлинение. Более конкретно рекомбиназа может облегчать внедрение фрагмента с тройным индексом в полимеразу и удлинение праймера полимеразой с использованием фрагмента с тройным индексом в качестве темплата для образования ампликона. Данный процесс можно повторять как цепную реакцию, в которой ампликоны, полученные в результате каждого цикла внедрения/удлинения, служат в качестве темплатов в последующем цикле. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, облегченная рекомбиназой амплификация может проводиться изотермически. Для облегчения амплификации по существу желательно включать АТФ или другие нуклеотиды (или, в некоторых случаях, их негидролизуемые аналоги) в реагент для облегченной рекомбиназой амплификации. Смесь рекомбиназы и связывающегося с одной цепью белка (SSB) особенно полезна, поскольку SSB может дополнительно облегчать амплификацию. Примеры составов для облегченной рекомбиназой амплификации включают в себя составы, продаваемые в виде наборов TwistAmp производства компании TwistDx (г. Кэмбридж, Великобритания). Используемые компоненты реагента для облегченной рекомбиназой амплификации и условия реакции представлены в публикациях US 5,223,414 и US 7,399,590.
[00195] Еще одним примером компонента, который можно включить в реагент для облегчения образования ампликонов и в некоторых случаях для повышения скорости образования ампликона, является геликаза. Геликаза может способствовать образованию ампликонов, обеспечивая цепную реакцию образования ампликонов. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, облегченную геликазой амплификацию можно проводить в изотермических условиях. Смесь геликазы и связывающегося с одной цепью белка (SSB) особенно полезна, поскольку SSB может дополнительно облегчать амплификацию. Примеры составов для облегченной геликазой амплификации включают в себя составы, доступные в продаже в виде наборов IsoAmp производства компании Biohelix (г. Беверли, штат Массачусетс, США). Кроме того, примеры подходящих для использования составов, включающих в себя геликазный белок, описаны в US 7,399,590 и US 7,829,284.
[00196] Еще одним примером компонента, который можно включать в амплификационный реагент для облегчения образования ампликонов и в некоторых случаях для повышения скорости образования ампликона, является белок, связывающийся с исходной молекулой.
[00197] Использование в секвенировании/способы секвенирования
[00198] После прикрепления фрагментов с тройным индексом к поверхности определяют последовательность иммобилизованных и амплифицированных фрагментов с тройным индексом. Секвенирование можно проводить с использованием любой подходящей методики секвенирования, и способы определения последовательности иммобилизованных и амплифицированных фрагментов с тройным индексом, включающие в себя повторный синтез цепи, известны в данной области и описаны, например, в Bignell et al. (US 8,053,192), Gunderson et al. (WO2016/130704), Shen et al. (US 8,895,249) и Pipenburg et al. (US 9,309,502).
[00199] Способы, описанные в настоящем документе, можно использовать в сочетании с разнообразными методиками секвенирования нуклеиновых кислот. В частности, к применимым методикам относятся методики, в которых нуклеиновые кислоты крепятся в фиксированных положениях на матрице таким образом, чтобы их относительные положения не изменялись, и в которых матрицу многократно визуализируют. Особенно подходящими являются варианты осуществления, в которых изображения получены в разных цветовых каналах, например, в соответствии с разными метками, используемыми для различения одного типа нуклеотидных оснований от другого. В некоторых вариантах осуществления процесс определения нуклеотидной последовательности фрагмента с тройным индексом может представлять собой автоматизированный процесс. Предпочтительные варианты осуществления включают в себя методики «секвенирования путем синтеза» (SBS).
[00200] Методики SBS по существу включают в себя ферментативное удлинение появляющейся полинуклеотидной цепи посредством итерационного добавления нуклеотидов в соответствии с цепью-темплатом. В традиционных способах SBS однонуклеотидный мономер можно вводить в целевой нуклеотид в присутствии полимеразы при каждой доставке. Однако в способах, описанных в настоящем документе, в целевую нуклеиновую кислоту при доставке в присутствии полимеразы можно вводить более одного типа нуклеотидного мономера.
[00201] В одном из вариантов осуществления нуклеотидный мономер включает в себя запертые нуклеиновые кислоты (ЗНК) или мостиковые нуклеиновые кислоты (МНК). Использование ЗНК или МНК в нуклеотидном мономере повышает прочность гибридизации между нуклеотидным мономером и последовательностью праймера секвенирования, присутствующей на иммобилизованном фрагменте с тройным индексом.
[00202] При SBS можно использовать нуклеотидные мономеры, имеющие терминаторную функциональную группу, или мономеры, которые не имеют терминаторных функциональных групп. Способы с использованием не имеющих терминаторов нуклеотидных мономеров включают в себя, например, пиросеквенирование и секвенирование с использованием меченных γ-фосфатом нуклеотидов, как более подробно описано в настоящем документе. В способах с использованием нуклеотидных мономеров, не содержащих терминаторов, количество добавляемых в каждом цикле нуклеотидов по существу варьирует и зависит от шаблонной последовательности и способа подачи нуклеотидов. В случае методик SBS, в которых используются нуклеотидные мономеры, имеющие терминаторную функциональную группу, терминатор может быть практически необратимым в используемых условиях секвенирования, как в случае традиционного секвенирования по Сэнгеру, в котором используются дидезоксинуклеотиды, или терминатор может быть обратимым, как в случае способов секвенирования, разработанных Solexa (в настоящее время - Illumina, Inc.).
[00203] В методиках SBS могут использоваться нуклеотидные мономеры, имеющий маркировочную функциональную группу, или мономеры, не имеющие маркировочной функциональной группы. Соответственно, события включения могут обнаруживаться на основе характеристики метки, например, флуоресценции метки; характеристики нуклеотидного мономера, например, молекулярной массы или заряда; побочного продукта встраивания нуклеотида, например, высвобождения пирофосфата; или т. п. В вариантах осуществления, в которых в реагенте для секвенирования присутствуют два или более разных нуклеотида, разные нуклеотиды могут быть отличимы друг от друга, или альтернативно две или более разные метки могут являться неотличимыми используемыми методиками детектирования. Например, разные нуклеотиды, присутствующие в реагенте для секвенирования, могут иметь разные метки, и их можно различать с помощью соответствующих оптических устройств, примеры которых представлены способами секвенирования, разработанными Solexa (в настоящее время - Illumina, Inc.).
[00204] Предпочтительные варианты осуществления включают в себя методики пиросеквенирования. Пиросеквенирование обнаруживает высвобождение неорганического пирофосфата (PPi) при встраивании конкретных нуклеотидов в формирующуюся цепь (Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M. and Nyren, P. (1996) “Real-time DNA sequencing using detection of pyrophosphate release.” Analytical Biochemistry 242(1), 84-9; Ronaghi, M. (2001) “Pyrosequencing sheds light on DNA sequencing.” Genome Res. 11(1), 3-11; Ronaghi, M., Uhlen, M. and Nyren, P. (1998) “A sequencing method based on real-time pyrophosphate.” Science 281(5375), 363; патенты США №№ 6,210,891; 6,258,568 и 6,274,320). Во время пиросеквенирования выделяющийся PPi можно обнаружить посредством ему превращения сразу в аденозинтрифосфат (АТФ) АТФ-сульфуразой, а уровень выработанного АТФ обнаруживают по продукции фотонов люциферазой. Нуклеиновые кислоты, подлежащие секвенированию, можно прикрепить к элементам матрицы, и матрицу можно визуализировать с регистрацией хемилюминесцентных сигналов, продуцируемых вследствие внедрения нуклеотидов в элементы матрицы. Изображение можно получить после обработки матрицы конкретным типом нуклеотида (например, A, T, Ц или Г). Изображения, полученные после добавления каждого типа нуклеотидов, будут отличаться в зависимости от того, какие элементы матрицы обнаруживаются. Эти различия в изображении отражают различающийся состав последовательности на элементах матрицы. Однако относительные местоположения каждого элемента на изображениях останутся неизменными. Изображения можно сохранять, обрабатывать и анализировать с помощью способов, описанных в настоящем документе. Например, изображения, полученные после обработки матрицы каждым из разных типов нуклеотидов, можно обрабатывать так, как показано в примерах настоящего документа для изображений, полученных из разных каналов обнаружения в способах секвенирования на основе обратимых терминаторов.
[00205] В другом примере типа SBS цикл секвенирования выполняют путем постадийного добавления обратимых терминаторных нуклеотидов, содержащих, например, отщепляемую или светообесцвечиваемую метку-краситель, как описано, например, в WO 04/018497 и патенте США № 7,057,026. Такой подход введен в коммерческое использование компанией Solexa (в настоящее время - Illumina Inc.), а также описан в WO 91/06678 и WO 07/123,744. Наличие флуоресцентно-меченных терминаторов, причем когда возможно как обращение терминирования, так и отщепление флуоресцентной метки, способствует эффективному секвенированию с циклической обратимой терминацией (CRT). Также можно одновременно сконструировать полимеразы, способные эффективно встраивать эти модифицированные нуклеотиды и выполнять с ними удлинение цепи.
[00206] В некоторых вариантах осуществления секвенирования на основе обратимых терминаторов метки по существу не ингибируют удлинение цепи в условиях реакции SBS. Однако детекторные метки могут быть выполнены с возможностью удаления, например, посредством отщепления или деградации. Изображения можно получить после встраивания меток в элементы матрицы с нуклеиновыми кислотами. В конкретных вариантах осуществления каждый цикл включает в себя одновременную доставку к матрице четырех разных типов нуклеотидов, и каждый тип нуклеотидов имеет спектрально отличающуюся метку. Затем можно получить четыре изображения, каждое из них с помощью канала обнаружения, селективного для одной из четырех разных меток. Альтернативно разные типы нуклеотидов могут добавляться последовательно, и изображение матрицы можно получать после каждой стадии добавления. В таких вариантах осуществления на каждом изображении будут показаны элементы с нуклеиновыми кислотами, в которые встроены нуклеотиды конкретного типа. Различные элементы будут присутствовать или отсутствовать на разных изображениях из-за разного состава последовательностей на каждом элементе. Однако относительное положение элементов на изображениях останется неизменным. Изображения, полученные такими способами SBS с обратимым терминатором, можно сохранять, обрабатывать и анализировать, как описано в настоящем документе. После стадии захвата изображения метки можно удалять, а также можно удалять обратимые терминаторные функциональные группы для последующих циклов добавления и обнаружения нуклеотидов. Удаление меток после их обнаружения в конкретном цикле и перед последующим циклом может обеспечивать преимущество, которое заключается в снижении фонового сигнала и перекрестных помех между циклами. Примеры подходящих меток и способов удаления описаны в настоящем документе.
[00207] В конкретных вариантах осуществления некоторые или все из нуклеотидных мономеров могут включать в себя обратимые терминаторы. В таких вариантах осуществления обратимые терминаторы/отщепляемые флуорофоры могут включать в себя флуорофоры, связанные с функциональной группой рибозы посредством 3'-сложноэфирной связи (Metzker, Genome Res. 15:1767-1776 (2005)). При других подходах химические реакции терминаторов отделены от отщепления флуоресцентной метки (Ruparel et al., Proc Natl Acad Sci USA 102: 5932-7 (2005)). В публикации Ruparel et al. описана разработка обратимых терминаторов, в которых для блокирования удлинения цепи использовали небольшую 3’-аллильную группу, но их можно легко разблокировать краткой обработкой палладиевым катализатором. Флуорофор присоединяли к основанию посредством фотоотщепляемого линкера, который легко расщеплялся при 30-секундном воздействии длинноволнового УФ-излучения. Таким образом, в качестве расщепляемого линкера можно использовать либо дисульфидное восстановление, либо фоторасщепление. Другим подходом к обратимой терминации является использование естественной терминации цепи, происходящей после помещения громоздкого красителя на дНТФ. Присутствие заряженного громоздкого красителя на дНТФ может выступать в качестве эффективного терминатора за счет стерического и/или электростатического затруднения. Одно событии внедрения предотвращает дальнейшее внедрение, пока краситель не будет удален. Расщепление красителя удаляет флуорофор и эффективно обращает терминацию. Примеры модифицированных нуклеотидов также описаны в патентах США №№ 7,427,673 и 7,057,026.
[00208] Дополнительные примеры систем и способов SBS, которые можно использовать со способами и системами, описанными в настоящем документе, описаны в патентных публикациях США №№ 2007/0166705, 2006/0188901, 2006/0240439, 2006/0281109, 2012/0270305 и 2013/0260372, патенте США № 7,057,026, публикации PCT № WO 05/065814, опубликованной заявке на патент США № 2005/0100900 и публикациях PCT №№ WO 06/064199 и WO 07/010,251.
[00209] В некоторых вариантах осуществления может использоваться обнаружение четырех разных нуклеотидов с использованием менее чем четырех разных меток. Например, SBS может быть выполнено с использованием способов и систем, описанных во включенных материалах патентной публикации США № 2013/0079232. В качестве первого примера, пара типов нуклеотидов могут обнаруживаться на одной и той же длине волны, но различаться по значениям интенсивности членов одной пары или по изменению одного члена пары (например, посредством химической, фотохимической или физической модификации), что приводит к появлению или исчезновению явного сигнала по сравнению с сигналом, обнаруживаемым для другого члена пары. В качестве второго примера, три из четырех разных типов нуклеотидов могут обнаруживаться в конкретных условиях, в то время как четвертый тип нуклеотидов не имеет метки, которая может быть обнаружена в этих условиях или минимально обнаруживается в этих условиях (например, минимальное обнаружение вследствие фоновой флуоресценции и т. д.). Встраивание первых трех типов нуклеотидов в нуклеиновую кислоту можно определить на основе наличия соответствующих им сигналов, а встраивание нуклеотидов четвертого типа в нуклеиновую кислоту можно определить на основе отсутствия или минимального обнаружения любого сигнала. В качестве третьего примера, один тип нуклеотидов может включать в себя метку (-и), которая (-ые) обнаруживается (-ются) в двух разных каналах, тогда как другие типы нуклеотидов обнаруживаются не более чем в одном из каналов. Вышеупомянутые три примера конфигураций не считаются взаимоисключающими и могут использоваться в различных комбинациях. Примером варианта осуществления, объединяющим все три примера, является способ SBS на основе флуоресценции, в котором используется первый тип нуклеотидов, обнаруживаемый в первом канале (например, дАТФ, имеющий метку, обнаруживаемую в первом канале при возбуждении первой длиной волны возбуждения), второй тип нуклеотидов, обнаруживаемый во втором канале (например, дЦТФ, имеющий метку, обнаруживаемую во втором канале при возбуждении второй длиной волны возбуждения), третий тип нуклеотидов, обнаруживаемый как в первом, так и во втором каналах (например, дТТФ, имеющий по меньшей мере одну метку, обнаруживаемую в обоих каналах при возбуждении первой и/или второй длинами волн возбуждения), и четвертый тип нуклеотидов, в котором отсутствует метка, которая не обнаруживается или минимально обнаруживается в любом канале (например, дГТФ без метки).
[00210] Кроме того, как описано во включенных в настоящий документ материалах публикации США № 2013/0079232, данные секвенирования могут быть получены с использованием одного канала. В таких так называемых однопигментных подходах к секвенированию первый тип нуклеотидов маркируют, но метку удаляют после создания первого изображения, а второй тип нуклеотидов маркируют только после создания первого изображения. Третий тип нуклеотидов сохраняет свою метку как на первом, так и на втором изображениях, а четвертый тип нуклеотидов остается немаркированным на обоих изображениях.
[00211] В некоторых вариантах осуществления может использоваться секвенирование посредством методик лигирования. В таких методиках используют ДНК-лигазу для встраивания олигонуклеотидов и идентифицируют встраивание таких олигонуклеотидов. Олигонуклеотиды, как правило, имеют разные метки, коррелирующие с идентичностью конкретного нуклеотида в последовательности, с которой гибридизуются олигонуклеотиды. Как и в случае с другими способами SBS, изображения можно получить после обработки матрицы из элементов с нуклеиновыми кислотами мечеными реагентами для секвенирования. На каждом изображении будут видны элементы с нуклеиновыми кислотами, имеющими внедренные метки конкретного типа. Разные элементы будут присутствовать или отсутствовать на разных изображениях из-за разного состава последовательности в каждом элементе, но относительное местоположение элементов на изображениях останется неизменным. Изображения, полученные способами секвенирования на основе лигирования, можно хранить, обрабатывать и анализировать, как описано в настоящем документе. Примеры систем и способов SBS, которые можно использовать со способами и системами, описанными в настоящем документе, описаны в патентах США №№ 6,969,488, 6,172,218 и 6,306,597.
[00212] В некоторых вариантах осуществления можно использовать секвенирование через нанопоры (Deamer, D. W. & Akeson, M. “Nanopores and nucleic acids: prospects for ultrarapid sequencing.” Trends Biotechnol. 18, 147-151 (2000); Deamer, D. and D. Branton, “Characterization of nucleic acids by nanopore analysis”, Acc. Chem. Res. 35:817-825 (2002); Li, J., M. Gershow, D. Stein, E. Brandin, and J. A. Golovchenko, “DNA molecules and configurations in a solid-state nanopore microscope” Nat. Mater. 2:611-615 (2003)). В таких вариантах осуществления фрагмент с тройным индексом проходит через нанопору. Нанопора может представлять собой синтетическую пору или биологический мембранный белок, такой как α-гемолизин. По мере прохождения фрагмента с тройным индексом через нанопору каждая пара оснований может быть идентифицирована посредством измерения флуктуаций электрической проводимости поры. (патент США № 7,001,792; Soni, G. V. & Meller, “A. Progress toward ultrafast DNA sequencing using solid-state nanopores.” Clin. Chem. 53, 1996-2001 (2007); Healy, K. “Nanopore-based single-molecule DNA analysis.” Nanomed. 2, 459-481 (2007); Cockroft, S. L., Chu, J., Amorin, M. & Ghadiri, M. R. “A single-molecule nanopore device detects DNA polymerase activity with single-nucleotide resolution.” J. Am. Chem. Soc. 130, 818-820 (2008)). Данные, полученные в результате секвенирования через нанопоры, можно хранить, обрабатывать и анализировать, как описано в настоящем документе. В частности, данные можно рассматривать как изображение в соответствии с примером обработки оптических изображений и других изображений, описанных в настоящем документе.
[00213] В некоторых вариантах осуществления могут использоваться способы, включающие в себя мониторинг активности ДНК-полимеразы в режиме реального времени. Внедрения нуклеотидов могут быть обнаружены посредством взаимодействий резонансного переноса энергии флуоресценции (FRET) между несущей флуорофор полимеразой и γ-фосфат-меченными нуклеотидами, как описано, например, в патентах США № 7,329,492 и 7,211,414, или внедрения нуклеотидов могут быть обнаружены с помощью волноводов с нулевой модой, как описано, например, в патенте США № 7,315,019, и с использованием флуоресцентных аналогов нуклеотидов и сконструированных полимераз, как описано, например, в патенте США № 7,405,281 и патентной публикации США № 2008/0108082. Освещение можно ограничить объемом порядка зептолитра вокруг связанной с поверхностью полимеразы таким образом, чтобы можно было наблюдать встраивание флуоресцентно-меченных нуклеотидов при низком фоне (Levene, M. J. et al. “Zero-mode waveguides for single-molecule analysis at high concentrations.” Science 299, 682-686 (2003); Lundquist, P. M. et al. “Parallel confocal detection of single molecules in real time.” Opt. Lett. 33, 1026-1028 (2008); Korlach, J. et al. “Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nano structures.” Proc. Natl. Acad. Sci. USA 105, 1176-1181 (2008)). Изображения, полученные такими способами, могут быть сохранены, обработаны и проанализированы, как описано в настоящем документе.
[00214] Некоторые варианты осуществления SBS включают в себя обнаружение протона, высвобождаемого при встраивании нуклеотида в продукт удлинения. Например, при секвенировании на основе обнаружения высвобожденных протонов может использоваться электрический детектор и связанные с ним методики, которые доступны в продаже от компании Ion Torrent (г. Гилфорд, штат Коннектикут, США, филиал компании Life Technologies), или способы и системы секвенирования, описанные в публикациях США №№ 2009/0026082; 2009/0127589; 2010/0137143 и 2010/0282617. Описанные в настоящем документе способы амплификации целевых нуклеиновых кислот с использованием кинетического исключения могут быть легко применены к субстратам, используемым для обнаружения протонов. Более конкретно, способы, изложенные в настоящем документе, можно использовать для получения клональных популяций ампликонов, используемых для обнаружения протонов.
[00215] Преимуществом является то, что вышеописанные способы SBS можно реализовать в мультиплексных форматах таким образом, чтобы одновременно манипулировать множеством разных фрагментов с тройным индексом. В конкретных вариантах осуществления разные фрагменты с тройным индексом можно обрабатывать в общем реакционном сосуде или на поверхности конкретного субстрата. Это позволяет удобно доставлять реагенты для секвенирования, удалять непрореагировавшие реагенты и обнаруживать события внедрения мультиплексным способом. В вариантах осуществления, в которых используются связанные с поверхностью целевые нуклеиновые кислоты, фрагменты с тройным индексом могут иметь форму матрицы. В формате матрицы фрагменты с тройным индексом, как правило, могут быть связаны с поверхностью пространственно различимым образом. Фрагменты с тройным индексом могут быть связаны посредством прямой ковалентной связи, присоединения к грануле или другой частице либо посредством связывания с полимеразой или другой молекулой, которая присоединена к поверхности. Матрица может включать в себя одну копию фрагмента с тройным индексом в каждом сайте (также называемом элементом), или же в каждом сайте или элементе может присутствовать множество копий, имеющих одинаковую последовательность. С помощью способов амплификации, таких как мостиковая амплификация или эмульсионная ПЦР, можно получать множество копий, как более подробно описано в настоящем документе.
[00216] В способах, описанных в настоящем документе, можно использовать матрицы, имеющие элементы с любым из разнообразных значений плотности, включая, например, по меньшей мере около 10 элементов/см2, 100 элементов/см2, 500 элементов/см2, 1000 элементов/см2, 5000 элементов/см2, 10 000 элементов/см2, 50 000 элементов/см2, 100 000 элементов/см2, 1 000 000 элементов/см2, 5 000 000 элементов/см2 или выше.
[00217] Преимущество способов, описанных в настоящем документе, заключается в том, что они обеспечивают быстрое и эффективное обнаружение на множестве см2 параллельно. Соответственно, в настоящем описании предложены интегрированные системы, способные подготавливать и обнаруживать нуклеиновые кислоты с использованием методик, известных в данной области, например, как в примерах, приведенных в настоящем документе. Таким образом, интегрированная система настоящего изобретения может включать в себя компоненты для работы с жидкостями, способные доставлять реагенты для амплификации и/или реагенты для секвенирования к одному или более иммобилизованным фрагментам с тройным индексом, причем система включает в себя такие компоненты, как насосы, клапаны, резервуары, магистрали для жидкости и т. п. Проточная кювета может быть выполнена и/или использована в интегрированной системе для обнаружения целевых нуклеиновых кислот. Примеры проточных кювет описаны, например, в патенте США № 8,241,573 и патенте США № 8,951,781. Как показано в примере проточных кювет, один или более компонентов для работы с жидкостями интегрированной системы можно использовать для способа амплификации и для способа обнаружения. Если взять в качестве примера вариант осуществления секвенирования нуклеиновых кислот, то один или более компонентов для работы с жидкостями интегрированной системы можно использовать для способа амплификации, описанного в настоящем документе, и для доставки реагентов для секвенирования в способе секвенирования, таком как описанный в примерах выше. Альтернативно интегрированная система может включать в себя отдельные системы для работы с жидкостями для реализации способов амплификации и для реализации способов обнаружения. Примеры интегрированных систем секвенирования, способных создавать амплифицированные нуклеиновые кислоты и определять последовательность нуклеиновых кислот, включают в себя, без ограничений, платформу MiSeqTM (Illumina, Inc., г. Сан-Диего, штат Калифорния, США) и устройства, описанные в патенте США № 8,951,781.
[00218] Композиции
[00219] В настоящем документе также предложены композиции. В процессе реализации способов, описанных в настоящем документе, на практике могут быть получены различные композиции. Например, может быть получена композиция, содержащая клетки или ядра, имеющие хеширующий олигонуклеотид, присоединенный неспецифически или специфически. Другие композиции включают в себя композиции, содержащие множество популяций нормализующих олигонуклеотидов, как описано в настоящем документе. Может быть получена композиция, включающая в себя множество компартментов, причем каждый компартмент содержит множество популяций нормализующих олигонуклеотидов, или может быть получена композиция, содержащая множество ядер или клеток, причем ядра или клетки связаны со множеством популяций нормализующих олигонуклеотидов.
[00220] ПРИМЕРЫ ОСУЩЕСТВЛЕНИЯ
[00221] Вариант осуществления 1. Способ получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение множества клеток в первом множестве компартментов;
(b) воздействие на множество клеток каждого компартмента заданным условием;
(c) приведение ядер, выделенных из клеток каждого компартмента, или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
причем с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
при этом хеширующий олигонуклеотид содержит хеширующий индекс,
причем хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток; и
(d) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток.
[00222] Вариант осуществления 2. Способ по варианту осуществления 1, дополнительно включающий воздействие на клетки или ядра поперечно-сшивающим соединением для фиксации хеширующих олигонуклеотидов к клеткам или выделенным ядрам.
[00223] Вариант осуществления 3. Способ по любому из вариантов осуществления 1-2, в котором поперечно-сшивающее соединение содержит параформальдегид, формалин или метанол.
[00224] Вариант осуществления 4. Способ по любому из вариантов осуществления 1-3, в котором заданное условие включает в себя воздействие агента.
[00225] Вариант осуществления 5. Способ по любому из вариантов осуществления 1-4, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
[00226] Вариант осуществления 6. Способ по любому из вариантов осуществления 1-5, в котором хеширующий олигонуклеотид содержит одноцепочечную нуклеиновую кислоту.
[00227] Вариант осуществления 7. Способ по любому из вариантов осуществления 1-6, в котором хеширующий олигонуклеотид состоит из одноцепочечной нуклеиновой кислоты.
[00228] Вариант осуществления 8. Способ по любому из вариантов осуществления 1-7, в котором нуклеиновая кислота хеширующего олигонуклеотида содержит ДНК, РНК или их комбинацию.
[00229] Вариант осуществления 9. Способ по любому из вариантов осуществления 1-8, в котором хеширующий олигонуклеотид содержит домен, который опосредует специфическое связывание хеширующего олигонуклеотида с поверхностью клеток или ядер.
[00230] Вариант осуществления 10. Способ по любому из вариантов осуществления 1-9, в котором домен содержит лиганд, антитело или аптамер.
[00231] Вариант осуществления 11. Способ по любому из вариантов осуществления 1-10, в котором связь между хеширующим олигонуклеотидом и клетками или выделенными ядрами является неспецифической.
[00232] Вариант осуществления 12. Способ по любому из вариантов осуществления 1-11, в котором неспецифическая связь между хеширующим олигонуклеотидом и клетками или выделенными ядрами осуществляется посредством абсорбции.
[00233] Вариант осуществления 13. Способ по любому из вариантов осуществления 1-12, дополнительно включающий обработку объединенных хешированных клеток или объединенных хешированных ядер с использованием способа комбинаторного индексирования одиночных клеток для получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер, причем нуклеиновые кислоты содержат множество индексов.
[00234] Вариант осуществления 14. Способ по любому из вариантов осуществления 1-13, в котором способ комбинаторного индексирования одиночных клеток представляет собой секвенирование транскриптомов одиночных ядер, секвенирование транскриптомов одиночных клеток и секвенирование доступного для транспозона хроматина, полногеномное секвенирование в одиночных ядрах, секвенирование доступного для транспозона хроматина в одиночных ядрах, sci-HiC, DRUG-seq, sci-CAR, sci-MET, sci-Crop, sci-perturb или sci-Crispr.
[00235] Вариант осуществления 15. Способ по любому из вариантов осуществления 1-14, дополнительно включающий
(e) распределение подгрупп объединенных хешированных клеток или хешированных ядер во второе множество компартментов и приведение каждой подгруппы в контакт с обратной транскриптазой или ДНК-полимеразой и праймером, причем праймер в каждом компартменте содержит первую индексную последовательность, которая отличается от первых индексных последовательностей в других компартментах, для получения индексированных ядер, содержащих индексированные фрагменты нуклеиновых кислот;
(f) объединение индексированных клеток или индексированных ядер для создания объединенных индексированных клеток или объединенных индексированных ядер;
(g) распределение подгрупп объединенных индексированных клеток или объединенных индексированных ядер в третье множество компартментов и введение второй индексной последовательности в индексированные фрагменты нуклеиновых кислот для создания клеток с двойным индексированием или ядер с двойным индексированием, содержащих фрагменты нуклеиновых кислот с двойным индексом, причем введение включает в себя лигирование, удлинение праймера, амплификацию или транспонирование;
(h) объединение клеток с двойным индексированием или ядер с двойным индексированием для получения объединенных ядер или клеток с двойным индексом;
(i) распределение подгрупп клеток или объединенных ядер с двойным индексированием в четвертое множество компартментов и введение третьей индексной последовательности в индексированные фрагменты нуклеиновых кислот для создания клеток с тройным индексированием или ядер с тройным индексированием, содержащих фрагменты нуклеиновых кислот с тройным индексом, причем введение включает в себя лигирование, удлинение праймера, амплификацию или транспонирование;
(j) объединение фрагментов с тройным индексом для получения таким образом библиотеки секвенирования, содержащей транскриптомные нуклеиновые кислоты из множества одиночных ядер.
[00236] Вариант осуществления 16. Способ по любому из вариантов осуществления 1-15, в котором (g) включает в себя приведение каждой подгруппы в контакт с транспосомным комплексом, причем транспосомный комплекс в каждом компартменте содержит транспозазу и вторую индексную последовательность в условиях, подходящих для лигирования второй индексной последовательности к концам индексированных фрагментов нуклеиновых кислот, содержащих первую индексную последовательность, для получения ядер с двойным индексированием, содержащих фрагменты нуклеиновых кислот с двойным индексом, при этом вторая индексная последовательность отличается от вторых индексных последовательностей в других компартментах.
[00237] Вариант осуществления 17. Способ по любому из вариантов осуществления 1-16, в котором (i) включает в себя приведение каждой подгруппы в контакт с праймером, содержащим третью индексную последовательность и универсальную последовательность праймера; причем приведение в контакт включает в себя условия, подходящие для амплификации и введения третьей индексной последовательности в концы фрагментов нуклеиновых кислот с двойным индексом, причем третья индексная последовательность отличается от третьих индексных последовательностей в других компартментах.
[00238] Вариант осуществления 18. Способ по любому из вариантов осуществления 1-17, в котором компартменты представляют собой лунку или каплю.
[00239] Вариант осуществления 19. Способ по любому из вариантов осуществления 1-18, дополнительно включающий:
получение поверхности, содержащей множество сайтов амплификации,
причем сайты амплификации содержат по меньшей мере две популяции присоединенных одноцепочечных захватных олигонуклеотидов, имеющих свободный 3’-конец, и
приведение поверхности, содержащей сайты амплификации, в контакт с фрагментами с тройным индексом в условиях, подходящих для получения множества сайтов амплификации, каждый из которых содержит клональную популяцию ампликонов из отдельного фрагмента, содержащего множество индексов.
[00240] Вариант осуществления 20. Композиция, содержащая хешированные клетки или хешированные ядра по варианту осуществления 1.
[00241] Вариант осуществления 21. Композиция, содержащая объединенные хешированные клетки или объединенные хешированные ядра по варианту осуществления 1.
[00242] Вариант осуществления 22. Многолуночный планшет, причем компартменты многолуночного планшета содержат композицию по любому из вариантов осуществления 20 или 21.
[00243] Вариант осуществления 23. Многолуночный планшет по варианту осуществления 22, причем компартмент многолуночного планшета содержит от 50 до 100 000 000 клеток или ядер.
[00244] Вариант осуществления 24. Капля, причем капля содержит композицию по любому из вариантов осуществления 20 или 21.
[00245] Вариант осуществления 25. Капля по варианту осуществления 24, причем капля содержит от 50 до 100 000 000 клеток или ядер.
[00246] Вариант осуществления 26. Способ получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение первого множества компартментов, содержащих выделенные ядра или клетки, и приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
причем с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
при этом хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
причем хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток; и
(b) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток.
[00247] Вариант осуществления 27. Способ получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение первого множества компартментов, содержащих выделенные ядра или клетки, и приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
причем с выделенными ядрами или клетками связывается по меньшей мере одна копия хеширующего олигонуклеотида посредством абсорбции,
при этом хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
причем хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток; и
(b) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток.
[00248] Вариант осуществления 28. Способ получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества ядер или клеток, включающий:
(a) получение множества компартментов, содержащих ядра или клетки, причем ядра или клетки содержат хеширующий олигонуклеотид, который содержит специфичный для компартмента индекс;
(b) объединение ядер или клеток из разных компартментов во втором компартменте для получения объединенных хешированных ядер или объединенных хешированных клеток.
[00249] Вариант осуществления 29. Способ по любому из вариантов осуществления 26-28, дополнительно включающий воздействие на клетки каждого компартмента заданного условия или воздействие на клетки каждого компартмента заданного условия с последующим выделением ядер из множества клеток перед стадией (a).
[00250] Вариант осуществления 30. Способ по любому из вариантов осуществления 28-29, в котором заданное условие включает в себя воздействие агента.
[00251] Вариант осуществления 31. Способ по любому из вариантов осуществления 28-30, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
[00252] Вариант осуществления 32. Композиция, содержащая множество популяций нормализующих олигонуклеотидов, причем композиция содержит первую популяцию нормализующих олигонуклеотидов, содержащих первую индексную последовательность, и другие популяции нормализующих олигонуклеотидов, каждая из которых содержит уникальную индексную последовательность, отличную от индексных последовательностей других популяций, и при этом концентрация каждой популяции одинакова.
[00253] Вариант осуществления 33. Композиция, содержащая множество популяций нормализующих олигонуклеотидов, причем композиция содержит первую популяцию нормализующих олигонуклеотидов, содержащих первую индексную последовательность, и другие популяции нормализующих олигонуклеотидов, каждая из которых содержит уникальную индексную последовательность, отличную от индексных последовательностей других популяций, и при этом концентрации по меньшей мере двух из популяций отличаются.
[00254] Вариант осуществления 34. Композиция, содержащая множество популяций нормализующих олигонуклеотидов, причем композиция содержит первую популяцию нормализующих олигонуклеотидов, содержащих набор первых индексных последовательностей, и другие популяции нормализующих олигонуклеотидов, каждая из которых содержит набор уникальных индексных последовательностей, отличных от набора индексных последовательностей других популяций, и при этом концентрация каждой популяции одинакова.
[00255] Вариант осуществления 35. Композиция, содержащая множество популяций нормализующих олигонуклеотидов, причем композиция содержит первую популяцию нормализующих олигонуклеотидов, содержащих набор первых индексных последовательностей, и другие популяции нормализующих олигонуклеотидов, каждая из которых содержит набор уникальных индексных последовательностей, отличных от набора индексных последовательностей других популяций, и при этом концентрации по меньшей мере двух из популяций отличаются.
[00256] Вариант осуществления 36. Композиция по любому из вариантов осуществления 32-35, причем композиция содержит от 2 до 100 популяций нормализующих олигонуклеотидов.
[00257] Вариант осуществления 37. Композиция по любому из вариантов осуществления 32-36, в которой нормализующие олигонуклеотиды содержат одноцепочечную ДНК.
[00258] Вариант осуществления 38. Композиция по любому из вариантов осуществления 32-37, в которой нормализующие олигонуклеотиды содержат уникальный молекулярный идентификатор.
[00259] Вариант осуществления 39. Композиция по любому из вариантов осуществления 32-38, в которой нормализующие олигонуклеотиды содержат универсальную последовательность.
[00260] Вариант осуществления 40. Композиция по любому из вариантов осуществления 32-39, в которой нормализующие олигонуклеотиды содержат ненуклеотидный компонент.
[00261] Вариант осуществления 41. Композиция по любому из вариантов осуществления 32-40, в которой ненуклеотидный компонент содержит белок.
[00262] Вариант осуществления 42. Композиция по любому из вариантов осуществления 32-41, в которой ненуклеотидный компонент содержит белок.
[00263] Вариант осуществления 43. Композиция по любому из вариантов осуществления 32-42, в которой первая популяция присутствует в композиции в самой низкой концентрации нормализующих олигонуклеотидов, а одна из других популяций присутствует в композиции в самой высокой концентрации нормализующих олигонуклеотидов, и причем самая низкая и самая высокая концентрации отличаются в 1-10 000 раз.
[00264] Вариант осуществления 44. Множество компартментов, причем каждый компартмент содержит композицию по любому из вариантов осуществления 32-43.
[00265] Вариант осуществления 45. Множество компартментов по варианту осуществления 44, причем компартменты содержат лунки или капли.
[00266] Вариант осуществления 46. Множество компартментов по любому из вариантов осуществления 44-45, причем каждый компартмент дополнительно содержит ядра или клетки и причем множество популяций нормализующих олигонуклеотидов связаны с ядрами или клетками.
[00267] Вариант осуществления 47. Множество компартментов по любому из вариантов осуществления 44-46, причем концентрация нормализующих олигонуклеотидов каждой популяции выбрана из диапазона от по меньшей мере 0,001 зептомоль до не более чем 100 аттомоль.
[00268] Вариант осуществления 48. Популяция ядер или клеток, причем ядра или клетки содержат композицию по любому из вариантов осуществления 32-47 и причем члены каждой популяции нормализующих олигонуклеотидов связаны с ядрами или клетками.
[00269] Вариант осуществления 49. Популяция по варианту осуществления 48, в которой связь между ядрами или клетками и нормализующими олигонуклеотидами является неспецифической.
[00270] Вариант осуществления 50. Способ нормализации библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение первого множества компартментов, содержащих выделенные ядра или клетки;
(b) приведение выделенных ядер или клеток каждого компартмента в контакт с композицией по любому из вариантов осуществления 32-35, причем члены каждой популяции нормализующих олигонуклеотидов связаны с выделенными ядрами или клетками; и
(c) объединение меченых ядер или меченых клеток разных компартментов для получения объединенных меченых ядер или объединенных меченых клеток.
[00271] Вариант осуществления 51. Способ по варианту осуществления 50, дополнительно включающий воздействие на клетки каждого компартмента заданного условия или воздействие на клетки каждого компартмента заданного условия с последующим выделением ядер из множества клеток перед стадией (a).
[00272] Вариант осуществления 52. Способ по любому из вариантов осуществления 50-51, в котором заданное условие включает в себя воздействие агента.
[00273] Вариант осуществления 53. Способ по любому из вариантов осуществления 50-52, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
[00274] Вариант осуществления 54. Способ по любому из вариантов осуществления 50-53, дополнительно включающий перед стадией (b) приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
причем с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
при этом хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
причем хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах и отличается от индексных последовательностей нормализующих олигонуклеотидов, присутствующих в компартменте, для получения меченых хешированных ядер или меченых хешированных клеток; и
объединение меченых хешированных ядер или меченых хешированных клеток разных компартментов для получения объединенных меченых хешированных ядер или объединенных меченых хешированных клеток.
[00275] Вариант осуществления 55. Способ по любому из вариантов осуществления 50-54, дополнительно включающий воздействие на клетки или ядра поперечно-сшивающим соединением для фиксации нормализующих олигонуклеотидов к клеткам или выделенным ядрам.
[00276] Вариант осуществления 56. Способ по любому из вариантов осуществления 50-55, в котором поперечно-сшивающее соединение содержит параформальдегид, формалин или метанол.
[00277] Вариант осуществления 57. Способ по любому из вариантов осуществления 50-56, в котором связь между нормализующим олигонуклеотидом и клетками или выделенными ядрами является неспецифической.
[00278] Вариант осуществления 58. Способ по любому из вариантов осуществления 50-57, в котором неспецифическая связь между нормализующим олигонуклеотидом и клетками или выделенными ядрами осуществляется посредством абсорбции.
[00279] Вариант осуществления 59. Способ по любому из вариантов осуществления 50-58, дополнительно включающий обработку объединенных меченых хешированных клеток или объединенных меченых хешированных ядер с использованием способа комбинаторного индексирования одиночных клеток для получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, причем нуклеиновые кислоты содержат множество индексов.
[00280] Вариант осуществления 60. Способ по любому из вариантов осуществления 50-59, в котором способ комбинаторного индексирования одиночных клеток представляет собой секвенирование транскриптомов одиночных ядер, секвенирование транскриптомов одиночных клеток и секвенирование доступного для транспозона хроматина, полногеномное секвенирование в одиночных ядрах, секвенирование доступного для транспозона хроматина в одиночных ядрах, sci-HiC, DRUG-seq, sci-CAR, sci-MET, sci-Crop, sci-perturb или sci-Crispr.
[00281] Вариант осуществления 61. Способ нормализации библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение выделенных ядер или клеток;
(b) приведение выделенных ядер или клеток в контакт с композицией по любому из вариантов осуществления 32-35, причем члены каждой популяции нормализующих олигонуклеотидов связаны с выделенными ядрами или клетками; и
(c) распределение подгрупп меченых ядер или меченых клеток на множество компартментов.
[00282] Вариант осуществления 62. Способ по варианту осуществления 61, дополнительно включающий воздействие на клетки заданного условия или воздействие на клетки заданного условия с последующим выделением ядер из множества клеток перед стадией (a).
[00283] Вариант осуществления 63. Способ по любому из вариантов осуществления 61-62, в котором заданное условие включает в себя воздействие агента.
[00284] Вариант осуществления 64. Способ по любому из вариантов осуществления 61-63, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
[00285] Вариант осуществления 65. Способ по любому из вариантов осуществления 61-64, дополнительно включающий после стадии (с) приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
причем с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
при этом хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
причем хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах и отличается от индексных последовательностей нормализующих олигонуклеотидов, присутствующих в компартменте, для получения меченых хешированных ядер или меченых хешированных клеток; и
объединение меченых хешированных ядер или меченых хешированных клеток разных компартментов для получения объединенных меченых хешированных ядер или объединенных меченых хешированных клеток.
[00286] Вариант осуществления 66. Способ по любому из вариантов осуществления 61-65, дополнительно включающий воздействие на клетки или ядра поперечно-сшивающим соединением для фиксации нормализующих олигонуклеотидов к клеткам или выделенным ядрам.
[00287] Вариант осуществления 67. Способ по любому из вариантов осуществления 61-66, в котором поперечно-сшивающее соединение содержит параформальдегид, формалин или метанол.
[00288] Вариант осуществления 68. Способ по любому из вариантов осуществления 61-67, в котором связь между нормализующим олигонуклеотидом и клетками или выделенными ядрами является неспецифической.
[00289] Вариант осуществления 69. Способ по любому из вариантов осуществления 61-68, в котором неспецифическая связь между нормализующим олигонуклеотидом и клетками или выделенными ядрами осуществляется посредством абсорбции.
[00290] Вариант осуществления 70. Способ по варианту осуществления любого из вариантов осуществления 61-69, дополнительно включающий обработку объединенных меченых хешированных клеток или объединенных меченых хешированных ядер с использованием способа комбинаторного индексирования одиночных клеток для получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, причем нуклеиновые кислоты содержат множество индексов.
[00291] Вариант осуществления 71. Способ по любому из вариантов осуществления 61-70, в котором способ комбинаторного индексирования одиночных клеток представляет собой секвенирование транскриптомов одиночных ядер, секвенирование транскриптомов одиночных клеток и секвенирование доступного для транспозона хроматина, полногеномное секвенирование в одиночных ядрах, секвенирование доступного для транспозона хроматина в одиночных ядрах, sci-HiC, DRUG-seq, sci-CAR, sci-MET, sci-Crop, sci-perturb или sci-Crispr.
[00292] ПРИМЕРЫ
[00293] Пример 1
[00294] Массивно-мультиплексная химическая транскриптомика с разрешением до одиночных клеток
[00295] В высокопроизводительных химических скринингах, как правило, используют грубые анализы, такие как выживание клеток, что ограничивает возможности изучения механизмов действия, нецелевых эффектов и гетерогенных ответов. В настоящем документе мы предлагаем «sci-Plex», в котором используется «хеширование ядер» для количественного определения глобальных транскрипционных ответов на тысячи независимых воздействий с разрешением до одиночных клеток. В качестве доказательства концепции мы использовали sci-Plex для скрининга трех раковых клеточных линий, подвергнутых воздействию 188 соединений. В совокупности мы создали профиль ~650 000 транскриптомов одиночных клеток по ~5000 независимым образцам в одном эксперименте. Наши результаты показывают существенную межклеточную гетерогенность ответов на конкретные соединения, общности в ответах на семейства соединений и представление о разных свойствах внутри семейств. В частности, наши результаты с ингибиторами гистондеацетилазы подтверждают представление о том, что хроматин действует как важный резервуар ацетата в раковых клетках. Этот пример также доступен в публикации Srivatsan et al., 2020, Science, 367:45-51).
[00296] Для обеспечения экономически эффективного высокопроизводительного скрининга (HTS) с фенотипированием на основе секвенирования транскриптомов одиночных клеток (scRNAseq) мы описываем новую стратегию мечения образцов (хеширования), которая основана на мечении ядер немодифицированными одноцепочечными ДНК-олигонуклеотидами. Последние усовершенствования комбинаторного индексирования одиночных клеток (sci-RNA-seq3) снизили стоимость подготовки библиотеки scRNAseq до < 0,01 долл. США на клетку, причем в каждом эксперименте (21) были профилированы миллионы клеток. В настоящем изобретении мы сочетаем хеширование ядер с sci-RNA-seq в едином рабочем процессе для мультиплексной транскриптомики под названием «sci-Plex». В качестве доказательства концепции мы используем sci-Plex для выполнения HTS на трех раковых клеточных линиях с профилированием тысяч независимых воздействий в одном эксперименте. Мы дополнительно изучили, как химическая транскриптомика с разрешением до одиночных клеток может пролить свет на механизмы действия. В частности, мы обнаружили, что связанные с регуляцией генов изменения, возникающие вследствие обработки ингибиторами гистондеацетилазы (HDAC), согласуются с моделью, в которой они препятствуют пролиферации, ограничивая способность клетки извлекать ацетат из хроматина (22, 23).
[00297] Результаты
[00298] Хеширование ядер позволяет проводить sci-RNA-seq на множестве образцов
[00299] В способах комбинаторного индексирования одиночных клеток (sci-) для специфического мечения молекулярного содержимого больших количеств одиночных клеток или ядер (24) используется мечение разделенным пулом штрихкодов. Образцы можно штрихкодировать этими же индексами, например, посредством помещения каждого образца в свою собственную лунку во время обратной транскрипции при sci-RNA-seq (21, 25), но такое ферментативное мечение в масштабе тысяч образцов практически недостижимо и экономически невозможно. Чтобы обеспечить молекулярное профилирование одиночных клеток в большом количестве независимых образцов в рамках sci-эксперимента, мы разработали недорогую процедуру мечения.
[00300] Мы отметили, что одноцепочечная ДНК (оцДНК) специфически окрашивала ядра пермеабилизованных клеток, но не интактных клеток (Фиг. 4A и Фиг. 5A). Таким образом, предполагается, что полиаденилированный оцДНК-олигонуклеотид можно использовать для мечения популяций ядер способом, совместимым со sci-RNA-seq (Фиг. 4B и Фиг. 5B). Для проверки этой концепции был проведен эксперимент типа «скотный двор». Мы по отдельности высевали человеческие (HEK293T) и мышиные (NIH3T3) клетки, использовав по 48 лунок 96-луночного культурального планшета. Затем выполняли лизирование ядер в присутствии 96 специфических для лунок полиаденилированных олигонуклеотидов оцДНК («хеширующих олигонуклеотидов») и зафиксировали полученные ядерные суспензии параформальдегидом. После мечения или «хеширования» ядер молекулярным штрихкодом мы объединяли ядра и проводили двухуровневый эксперимент sci-RNA-seq. Поскольку хеширующие олигонуклеотиды были полиаденилированы, они имели возможность комбинаторного индексирования идентично эндогенным мРНК. Как и предполагалось, мы получали чтения, соответствующие как эндогенным мРНК [медианное значение 4740 уникальных молекулярных идентификаторов (UMI) на клетку], так и хеширующим олигонуклеотидам (медианное значение 270 UMI на клетку).
[00301] Мы разработали статистическую платформу для идентификации хеширующих олигонуклеотидов, связанных с каждой клеткой, с частотой, превышающей фоновый уровень (таблица S1). Мы наблюдали 99,1% соответствие между распределением по видам на основе хеширующих олигонуклеотидов и на основе эндогенных клеточных транскриптомов (Фиг. 4 C и Фиг. 5, C-F). Кроме того, связывание хеширующих олигонуклеотидов с ядрами было устойчиво к циклу замораживания-размораживания, что указывает на возможность мечения и хранения образцов (Фиг. 4D и Фиг. 5, G и H). Эти результаты показывают, что хеширующие олигонуклеотиды устойчиво метят ядра способом, совместимым с sci-RNA-seq.
[00303] В sci-экспериментах «коллизии» представляют собой случаи, в которых две или более клеток были случайно мечены одной и той же комбинацией штрихкодов (24). Для оценки хеширования как средства обнаружения дублетов, возникших в результате коллизий, мы варьировали количество ядер, загруженных в реакционную лунку для полимеразной цепной реакции, получив диапазон прогнозируемых частот коллизий (от 7 до 23%), который хорошо соответствовал наблюдению (Фиг. 5 I). Хеширующие олигонуклеотиды облегчали идентификацию подавляющего большинства межвидовых дублетов (95,5%) и не выявляемых иными способами межвидовых дублетов (Фиг. 4E и Фиг. 5, J и K).
[00304] sci-Plex обеспечивает мультиплексную химическую транскриптомику с разрешением до одиночных клеток
[00305] Затем мы оценивали, может ли хеширование ядер позволить проводить химический скрининг посредством мечения клеток, которые подверглись определенному воздействию, с последующим анализом транскрипционного профиля одиночных клеток в виде многопараметрического фенотипического анализа. Клетки А549, представляющие собой клеточную линию легочной аденокарциномы человека, мы подвергали воздействию одного из четырех соединений: дексаметазона (агонист кортикостероидов), нутлин-3a (антагонист p53-Mdm2), BMS-345541 (ингибитор-зависимой от ядерного фактора kB транскрипции) или вориностата [субероиланилидгидроксамовая кислота (SAHA), ингибитор HDAC] в течение 24 часов в семи дозах и в трех повторностях: всего 84 комбинации «лекарственное средство - доза - повторность» и дополнительные контроли - несущие среды (Фиг. 6A и Фиг. 7A). Ядра из каждой лунки мы метили и подвергали их анализу sci-RNA-seq2 (Фиг. 7, B-D и таблица 1).
[00306] Для визуализации этих данных с использованием алгоритма Uniform Manifold Approximation and Projection (UMAP) (26) и обнаружения Лувенского сообщества с целью идентификации специфических для соединений кластеров клеток, распределенных в зависимости от дозы, мы использовали программное обеспечение Monocle 3 (21) (Фиг. 6, B и C, а также Фиг. 7, E и F). Для количественного определения «среднего по популяции» транскрипционного ответа клеток А549 на каждое из четырех лекарственных средств мы моделировали экспрессию каждого гена в зависимости дозы посредством обобщенной линейной регрессии. Всего 7561 ген был чувствителен к по меньшей мере одному лекарственному средству, а 3189 генов дифференциально экспрессировались в ответ на множество лекарственных средств (Фиг. 8A и непоказанные данные). К ним относились канонические цели дексаметазона (Фиг. 6D) и нутлина-3a (Фиг. 6E). Анализ онтологии генов с дифференциальной экспрессией генов позволил выявить задействование специфических для лекарственных средств путей (например, гормональная сигнализация для дексаметазона; p53-сигнализация для нутлина-3a; Фиг. 8B). Кроме того, мы оценивали возможность использования количества клеток, полученных для каждой концентрации, для оценки токсичности аналогично традиционным скрининговым исследованиям. После аппроксимации кривой ответа полученных количеств клеток мы логически вывели «балл жизнеспособности» по данным sci-Plex - показатель, который соответствовал измерениям, являющимся «золотым стандартом» (Фиг. 6 F и Фиг. 7, G. I).
[00307] sci-Plex имеет масштаб в тысячи образцов и позволяет проводить HTS
[00308] Для оценки того, как sci-Plex масштабируется до HTS, выполняли скрининг 188 соединений, нацеленных на широкий спектр ферментов и молекулярных путей (Фиг. 9A). Половину этой панели выбирали с нацеливанием на транскрипционные и эпигенетические регуляторы. Другую половину выбирали для оценки различных механизмов действия. Три хорошо охарактеризованные человеческие раковые клеточные линии - A549 (легочная аденокарцинома), K562 (хронический миелогенный лейкоз) и MCF7 (аденокарцинома молочной железы) - подвергали воздействию каждого из этих 188 соединений в четырех дозах (10 нМ, 100 нМ, 1 мМ и 10 мМ) в двух повторностях, рандомизируя соединения и дозы по всем положениям лунок в повторных культуральных планшетах (данные не показаны). Эти условия наряду с контролями - несущими средами в данном эксперименте обуславливали 4608 из 4992 независимо обработанных популяций клеток. После обработки проводили лизис клеток для открытия ядер, хешировали их специфической комбинацией из двух олигонуклеотидов (Фиг. 10A) и выполняли sci- RNA-seq3 (21). После секвенирования и фильтрации в зависимости от чистоты хеширования (Фиг. 10, B-F) получали транскриптомы 649 340 одиночных клеток с медианным количеством мРНК UMI, равным 1271, 1071 и 2407 для A549, K562 и MCF7 соответственно (Фиг. 11 A). Совокупные профили экспрессии для каждого типа клеток в лунках повторностей имели высокую степень согласованности (корреляция по Пирсону=0,99) (Фиг. 11B).
[00309] Визуализация профилей sci-RNA-seq отдельно для каждой клеточной линии позволяла выявить специфические для соединений транскрипционные ответы, а также паттерны, общие для множества соединений. В каждой из клеточных линий алгоритм UMAP проецировал большую часть клеток в центральную массу, фланкированную кластерами меньшего размера (Фиг. 9B). Эти более мелкие кластеры в основном состояли из клеток, обработанных соединениями только одного или двух классов (ФИГ. 12 и 13, A-C). Например, клетки А549, обработанные триамцинолона ацетонидом, синтетическим агонистом глюкокортикоидных рецепторов, были заметно обогащены одним таким малым кластером, содержащим 95% клеток [точный критерий Фишера, ожидаемая доля ложноположительных результатов (FDR) < 1%; Фиг. 13, D и E]. Хотя многие лекарственные средства ассоциировались с внешне гомогенным транскрипционным ответом, мы также выявляли случаи, когда одно и то же лекарственное средство индуцировало различные транскрипционные состояния. Например, в клетках А549 стабилизирующие микротрубочки соединения эпотилон A и эпотилон B ассоциировались с тремя такими очагами обогащения, каждый из которых состоял из клеток из обоих соединений при всех четырех дозах (Фиг. 13, F и G). Клетки в каждом очаге отличались друг от друга, но были транскрипционно аналогичны другим видам обработки: недавно выявленным дестабилизатором микротрубочек, ригосертибом (27); UNC0397, ингибитором SETD8; или необработанными пролиферирующими клетками (Фиг. 13H).
[00310] Затем оценивали влияние каждого лекарственного средства на «средний по популяции» транскриптом каждой клеточной линии. Всего в по меньшей мере одной клеточной линии дифференциально экспрессировались дозозависимым образом 6238 генов (FDR < 5%; Фиг. 14 и непоказанные данные). Массовые измерения RNA-seq, полученные для пяти соединений в четырех дозах и для несущей среды, согласовывались с усредненными значениями экспрессии генов и оценочными размерами эффектов для идентичным образом обработанных одиночных клеток, хотя корреляции между малыми размерами эффектов были снижены (Фиг. 15). Более того, дозозависимые профили эффектов sci-Plex коррелировали с результатами измерений L1000 (11) для соответствующих соединений (ФИГ. 16).
[00311] Гены, ассоциированные с клеточным циклом, обладали высокой вариабельностью в отдельных клетках, и многие лекарственные средства уменьшали долю клеток, экспрессирующих маркерные гены пролиферации (ФИГ. 17 и 18). В принципе, scRNA-seq должен обеспечивать возможность отличать изменения долей клеток, находящихся в различных транскрипционных состояниях, от изменений генной регуляции при этих состояниях. С другой стороны, массовое профилирование транскриптомов будет путать эти два сигнала (Фиг. 19A) (14). Поэтому мы провели исследование дозозависимой дифференциальной экспрессии на подгруппах клеток, соответствующих одному и тому же лекарственному средству, но экспрессирующих высокие или низкие уровни маркерных генов пролиферации (Фиг. 19B). Корреляция между дозозависимыми эффектами для этих двух фракций клеток каждого типа варьировалась по классам лекарственных средств (Фиг. 19 C) при несколько противоречивых эффектах отдельных соединений (Фиг. 19 D). Анализ жизнеспособности, выполненный так же, как в пилотном эксперименте, показал, что после воздействия лекарственного средства в наибольшей дозе лишь 52 (27%) соединений вызывали снижение жизнеспособности на 50% или более (Фиг. 9C и Фиг. 11C). Из лекарственных средств, снижающих жизнеспособность, мы наблюдали более высокую чувствительность К562 к ингибитору Src и Abl бозутинибу (Фиг. 9C), и этот результат подтверждал подсчет клеток (Фиг. 20A). Этот результат согласуется с клетками K562, несущими конститутивно активную гибридную киназу BCR-ABL (28), и с наблюдаемой повышенной чувствительностью гематопоэтических и лимфоидных раковых клеточных линий к ингибиторам Abl (29) (Фиг. 20B).
[00312] Для оценки того, вызывает ли каждое соединение аналогичные ответы в трех клеточных линиях, кластеризовали соединения с использованием размеров эффектов для дозозависимых генов в виде нагрузок в каждой клеточной линии (ФИГ. 21-24). Совместный анализ трех клеточных линий выявил общие и специфические для типа клеток ответы на разные соединения (ФИГ. 25 и 26). Например, траметиниб, ингибитор митоген-активированной протеинкиназы (MEK), индуцировал транскрипционно отличающийся ответ в клетках MCF7. Изучение UMAP-проекций позволяло выявить, что обработанные траметинибом клетки MCF7 были рассеяны среди контролей - несущих сред, что отражает ограниченные эффекты. Напротив, обработанные траметинибом клетки А549 и К562, несущие активирующие мутации KRAS и ABL (30) соответственно, были плотно кластеризованы, что согласуется с сильным специфическим транскрипционным ответом на ингибирование сигнализации MEK траметинибом (Фиг. 9D). Кроме того, мы обнаружили, что клетки A549 и K562 появлялись вблизи кластеров, обогащенных с помощью ингибиторов HSP90, ключевого шаперона сворачивания белков (Фиг. 9D). Это наблюдение подтверждалось соответствующими изменениями экспрессии HSP90AA1 в клетках, обработанных траметинибом (Фиг. 9E). Анализ карты связей (11, 12) выявил дополнительные доказательства того, что ингибиторы MEK действительно индуцируют сигнатуры экспрессии генов с высокой степенью сходства с воздействиями на HSP90 (Фиг. 20C), особенно в случае A549, но не в случае MCF7 (Фиг. 20, D и E). Эти результаты согласуются с предыдущими наблюдениями регуляции HSP90AA1 ниже от сигнализации MEK (31) и позволяют предположить, что сходство в транскриптомах одиночных клеток, обработанных различными соединениями, может показать лекарственные средства, которые воздействуют на эти целевые пересекающиеся молекулярные пути.
[00313] Анализ химических и механистических свойств ингибиторов HDAC
[00314] В каждой из трех клеточных линий наиболее заметный ответ на соединения давали клетки, обработанные одним из 17 ингибиторов HDAC (Фиг. 9 B, темно-синий, и непоказанные данные). Для оценки сходства траекторий «доза - ответ» между клеточными линиями было выполнено сопоставление обработанных HDAC клеток и обработанных несущей средой клеток всех трех клеточных линий с использованием сопоставления методом взаимных ближайших соседей (MNN) (32) для получения консенсусной траектории ингибитора HDAC, которую мы назвали «псевдодозой» [аналогично «псевдовремени» (33)] (Фиг. 27A и Фиг. 28). Мы отметили, что некоторые ингибиторы HDAC индуцировали гомогенные ответы, при которых практически все клетки локализованы в относительно узком диапазоне траектории ингибитора HDAC при каждой дозе (например, прациностат в A549), тогда как другие лекарственные средства индуцировали гораздо большую клеточную гетерогенность (Фиг. 27B и Фиг. 29).
[00315] Такую гетерогенность можно объяснить тем, что клетки исполняют определенную транскрипционную программу асинхронно, и доза лекарственного средства, которая воздействует на клетки, модулирует скорость прохождения ими этой программы. Для проверки этой гипотезы секвенировали транскриптомы клеток 64 440 клеток A549, которые были обработаны в течение 72 часов одним из 48 соединений, включая многие из ингибиторов HDAC из крупномасштабного скрининга sci-Plex. При учете эффектов зависимого от конфлюентности клеточного цикла и выравнивания MNN (ФИГ. 30 и 31) UMAP-проекция с совместными вложениями выявила новые очаговые концентрации клеток через 72 часа, которые не были очевидны в момент 24 часа, например, SRT1024 (Фиг. 32). Однако для большинства исследованных ингибиторов HDAC не наблюдали, чтобы клетки, получавшие заданную дозу, перемещались дальше вдоль выровненной траектории HDAC через 72 часа (Фиг. 33). Это позволяет предположить, что доза многих ингибиторов HDAC определяет величину клеточного ответа, но не скорость его протекания, и что не всю наблюдаемую гетерогенность можно объяснить исключительно несинхронностью (Фиг. 33).
[00316] Далее оценивали, объясняет ли аффинность данного ингибитора HDAC к цели глобальный транскрипционный ответ на соединение. Использовали дозозависимые модели для оценки медианной транскрипционно эффективной концентрации каждого соединения (TC50), т. е. концентрации, необходимой для прохождения клетки до половины псевдодозовой траектории ингибитора HDAC (Фиг. 34 A и непоказанные данные). Для сравнения производных от транскрипции показателей эффективности с биохимическими свойствами каждого соединения мы получали опубликованные значения медианной ингибирующей концентрации (IC50) для каждого соединения в анализах in vitro, выполненных на восьми очищенных изоформах HDAC (данные не показаны). За исключением двух относительно нерастворимых соединений наши расчетные значения TC50 увеличивались в зависимости от значений IC50 соединения (Фиг. 27C и Фиг. 34, B и C).
[00317] Для оценки компонентов траектории ингибитора HDAC выполняли анализ дифференциальной экспрессии с использованием псевдодозы в качестве непрерывной ковариаты. Из 4308 генов, которые имели значимую дифференциальную экспрессию по этой консенсусной траектории, 2081 (48%) отвечали зависимым от типа клеток образом, а 942 (22%) демонстрировали одинаковый характер ответа во всех трех клеточных линиях (Фиг. 35, A и B и непоказанные данные). Одним из выраженных паттернов ответа, общих для трех клеточных линий, является обогащение генами и путями, указывающими на продвижение в сторону остановки клеточного цикла (ФИГ. 35C и 36, A и B). Окрашивание ДНК и проточная цитометрия подтверждали, что ингибирование HDAC приводило к накоплению клеток, находящихся в фазе G2/M клеточного цикла (34) (Фиг. 36, C и D).
[00318] Общий ответ на ингибирование HDAC включал в себя не только остановку клеточного цикла, но и измененную экспрессию генов, вовлеченных в клеточный метаболизм (Фиг. 35C). Ацетилтрансферазы и деацетилазы гистонов регулируют доступность хроматина и активность транскрипционных факторов посредством добавления или удаления заряженных ацетильных групп (35-37). Ацетат, продукт опосредованного HDAC класса I, II и IV деацетилирования гистонов и предшественник ацетилкофермента A (ацетил-КоА), необходим для ацетилирования гистонов, но также играет важную роль в метаболическом гомеостазе (23, 38, 39). Ингибирование ядерного деацетилирования ограничивает рециркуляцию связанных с хроматином ацетильных групп как для катаболических, так и для анаболических процессов (39). Соответственно, было отмечено, что ингибирование HDAC приводило к секвестрации ацетата в виде заметно повышенных уровней ацетилированного лизина после воздействия дозы 10 мМ ингибиторов HDAC прациностата и абексиностата (Фиг. 37).
[00319] При дальнейшей проверке псевдодозозависимых генов мы обнаружили, что ферменты, критически важные для синтеза цитоплазматического ацетил-КоА из цитрата (ACLY) или ацетата (ACSS2), имели повышенную экспрессию (Фиг. 38A). Гены, участвующие в цитоплазматическом цитратном гомеостазе (GLS, IDH1 и ACO1), клеточном импорте цитрата (SLC13A3) и митохондриальной продукции и экспорте цитрата (CS, SLC25A1) также имели повышенную экспрессию. Повышение экспрессии SIRT2, деацетилирующего тубулин, также наблюдалось в ответ на ингибирование HDAC.
[00320] Наряду с увеличением количества связанного с хроматином ацетата эти транскрипционные ответы указывают на важное для метаболизма истощение резервов клеточного ацетил-КоА в клетках с ингибированной HDAC (Фиг. 38 B). Для дополнительной проверки этого факта мы попытались сместить распределение клеток по траектории ингибитора HDAC посредством модуляции концентраций ацетил-КоА в клетках. Клетки A549 и MCF7 мы обрабатывали прациностатом в присутствии и в отсутствие предшественников ацетил-КоА (ацетат, пируват или цитрат) или ингибиторов ферментов (ACLY, ACSS2 или PDH), участвующих в восполнении пулов ацетил-КоА. После обработки клетки собирали и обрабатывали с помощью sci-Plex и строили траектории для каждой клеточной линии (ФИГ. 39 и 40). Как в клетках А549, так и в клетках MCF7 введение ацетата, пирувата и цитрата оказалось способно не дать клеткам, обработанным прациностатом, достичь конца траектории ингибитора HDAC (Фиг. 39, F, J, H и L). В клетках MCF7 как ингибирование ACLY, так и ингибирование ACSS2 сдвигало клетки дальше вдоль траектории ингибитора HDAC, хотя такой сдвиг не наблюдался в случае A549 (Фиг. 39, G, K, I и M). Полученные результаты в совокупности свидетельствуют о том, что основной характеристикой ответа клеток на ингибиторы HDAC и, возможно, связанной с ними токсичности является индукция состояния недостаточности ацетил-КоА.
[00321] Обсуждение
[00322] В настоящем изобретении мы предлагаем sci-Plex - массивно-мультиплексную платформу для транскриптомики одиночных клеток. В sci-Plex используется химическая фиксация для экономичного и необратимого мечения ядер короткими немодифицированными оцДНК-олигонуклеотидами. В описанном в настоящем документе эксперименте для доказательства концепции мы применяли sci-Plex для количественного определения дозозависимых ответов раковых клеток на 188 соединений посредством анализа, который является как многопараметрическим (глобальная транскрипция), так и имеющим высокое разрешение (одиночные клетки). Посредством профилирования нескольких различных раковых клеточных линий мы различали общие и специфичные для клеточных линий молекулярные ответы на каждое соединение.
[00323] sci-Plex обеспечивает некоторые отчетливые преимуществами по сравнению с обычными HTS: он позволяет различать различные эффекты соединений на клеточные субпопуляции (включая сложные системы in vitro, такие как перепрограммирование клеток, органоиды и синтетические эмбрионы); он позволяет выявить гетерогенность клеточного ответа на воздействие; он позволяет измерить, как под действием лекарственных средств сдвигаются относительные пропорции транскрипционно отличающихся подгрупп клеток. Подчеркивая эти особенности, наше исследование дает представление о механизме действия ингибиторов HDAC. В частности, мы обнаружили, что основные транскрипционные ответы на ингибиторы HDAC включают в себя остановку клеточного цикла и заметные сдвиги в генах, связанных с метаболизмом ацетил-КоА. Для некоторых ингибиторов HDAC наблюдали четкую гетерогенность в ответах на уровне одиночных клеток. Хотя ингибирование HDAC традиционно рассматривается через механизмы, непосредственно связанные с регуляцией хроматина, наши данные подтверждают альтернативную, хотя и не взаимоисключающую модель, в которой ингибиторы HDAC нарушают рост и пролиферацию, препятствуя способности раковых клеток извлекать ацетат из хроматина (22, 23, 39). По существу вариации в ацетатных резервах клеток являются потенциальными объяснениями их гетерогенных ответов на ингибиторы HDAC.
[00324] Поскольку стоимость секвенирования одиночных клеток продолжает падать, возможности использования sci-Plex для фундаментальных и прикладных целей в биомедицине могут быть существенными. Эксперименты для доказательства концепции, описанные в настоящем документе, состоящие из приблизительно 5000 независимых видов обработки с профилированием транскрипции > 100 одиночных клеток на одну обработку, потенциально можно масштабировать в сторону создания всеобъемлющего и имеющего высокое разрешение атласа клеточных ответов на фармакологические воздействия (например, сотен клеточных линий или генетических окружений, тысяч соединений, многоканального профилирования одиночных клеток и т. д.). Этому будут способствовать простота и низкая стоимость олигонуклеотидного хеширования в сочетании с гибкостью и экспоненциальной масштабируемостью комбинаторного индексирования одиночных клеток.
[00325] Цитируемые документы
[00326] 1. J. R. Broach, J. Thorner, Nature 384 (Suppl), 14-16 (1996).
[00327] 2. D. A. Pereira, J. A. Williams, Br. J. Pharmacol. 152, 53-61 (2007).
[00328] 3. D. Shum et al., J. Enzyme Inhib. Med. Chem. 23, 931-945 (2008).
[00329] 4. C. Yu et al., Nat. Biotechnol. 34, 419-423 (2016).
[00330] 5. Z. E. Perlman et al., Science 306, 1194-1198 (2004).
[00331] 6. Y. Futamura et al., Chem. Biol. 19, 1620-1630 (2012).
[00332] 7. J. Kang et al., Nat. Biotechnol. 34, 70-77 (2016).
[00333] 8. K. L. Huss, P. E. Blonigen, R. M. Campbell, J. Biomol. Screen. 12, 578-584 (2007).
[00334] 9. C. Ye et al., Nat. Commun. 9, 4307 (2018).
[00335] 10. E. C. Bush et al., Nat. Commun. 8, 105 (2017).
[00336] 11. A. Subramanian et al., Cell 171, 1437-1452.e17 (2017).
[00337] 12. J. Lamb et al., Science 313, 1929-1935 (2006).
[00338] 13. M. B. Elowitz, A. J. Levine, E. D. Siggia, P. S. Swain, Science 297, 1183-1186 (2002).
[00339] 14. C. Trapnell, Genome Res. 25, 1491-1498 (2015).
[00340] 15. S. M. Shaffer et al., Nature 546, 431-435 (2017).
[00341] 16. S. L. Spencer, S. Gaudet, J. G. Albeck, J. M. Burke, P. K. Sorger, Nature 459, 428-432 (2009).
[00342] 17. M. Stoeckius et al., Genome Biol. 19, 224 (2018).
[00343] 18. J. Gehring, J. H. Park, S. Chen, M. Thomson, L. Pachter, Highly multiplexed single-cell RNA-seq for defining cell population and transcriptional spaces. bioRxiv 315333 [Preprint] 5 May 2018. doi.org/10.1101/315333.
[00344] 19. C. S. McGinnis et al., Nat. Methods 16, 619-626 (2019).
[00345] 20. D. Shin, W. Lee, J. H. Lee, D. Bang, Sci. Adv. 5, eaav2249 (2019).
[00346] 21. J. Cao et al., Nature 566, 496-502 (2019).
[00347] 22. M. A. McBrian et al., Mol. Cell 49, 310-321 (2013).
[00348] 23. S. A. Comerford et al., Cell 159, 1591-1602 (2014).
[00349] 24. D. A. Cusanovich et al., Science 348, 910-914 (2015).
[00350] 25. J. Cao et al., Science 357, 661-667 (2017).
[00351] 26. L. McInnes, J. Healy, UMAP: Uniform Manifold Approximation and Projection for Dimension Reduction. arXiv:1802.03426 [stat.ML] (9 February 2018).
[00352] 27. M. Jost et al., Mol. Cell 68, 210-223.e6 (2017).
[00353] 28. G. Grosveld et al., Mol. Cell. Biol. 6, 607-616 (1986).
[00354] 29. E. K. Greuber, P. Smith-Pearson, J. Wang, A. M. Pendergast, Nat. Rev. Cancer 13, 559-571 (2013).
[00355] 30. J. Barretina et al., Nature 483, 603-607 (2012).
[00356] 31. C. Dai et al., J. Clin. Invest. 122, 3742-3754 (2012).
[00357] 32. L. Haghverdi, A. T. L. Lun, M. D. Morgan, J. C. Marioni, Nat. Biotechnol. 36, 421-427 (2018).
[00358] 33. C. Trapnell et al., Nat. Biotechnol. 32, 381-386 (2014).
[00359] 34. W. Brazelle et al., PLOS ONE 5, e14335 (2010).
[00360] 35. J.-S. Roe, F. Mercan, K. Rivera, D. J. Pappin, C. R. Vakoc, Mol. Cell 58, 1028-1039 (2015).
[00361] 36. J. E. Brownell et al., Cell 84, 843-851 (1996).
[00362] 37. J. Taunton, C. A. Hassig, S. L. Schreiber, Science 272, 408-411 (1996).
[00363] 38. S. K. Kurdistani, Curr. Opin. Genet. Dev. 26, 53-58 (2014).
[00364] 39. K. E. Wellen et al., Science 324, 1076-1080 (2009).
[00365] Материалы и способы
[00366] Клеточная культура
[00367] Клетки A549 и клетки K562 были любезно предоставлены доктором Robert Bradley (UW) и доктором David Hawkins (UW) соответственно. Клетки MCF7 (кат. № HTB-22), NIH3T3 (кат. № CRL-1658) и HEK293T (кат. № CRL-11268) приобретали в ATCC. Клетки A549 и MCF7 культивировали в среде DMEM (ThermoFisher, 11995073) с добавлением 10% FBS (ThermoFisher, кат. № 26140079) и 1% пенициллина-стрептомицина (ThermoFisher, 15140122). Клетки К562 культивировали в RPMI 1640 (Fisher Scientific, кат. № 11-875-119) с добавлением 10% FBS и 1% пенициллина-стрептомицина и поддерживали в диапазоне 0,2-1×106 клеток/мл. Все клетки культивировали при 37 C в атмосфере с 5% CO2. Прикрепленные клетки рассевали по достижении конфлюэнтности 90% путем промывки DPBS (Life Technologies, кат. № 14190-250), трипсинизации с использованием TryPLE (Fisher Scientific, кат. № 12-604-039) и рассевания в соотношении 1 : 4 (MCF7) или 1 : 10 (A549, NIH3T3 и HEK293T).
[00368] Получение соединений
[00369] Дексаметазон был приобретен в компании Sigma-Aldrich и ресуспендирован в этаноле класса «для молекулярной биологии» (Fisher Scientific). BMS-345541 (S8044), вориностат (S1047) и нутлин-3a (S8059) приобретали в компании Selleck Chemicals и ресуспендировали в ДМСО (VWR Scientific, 97063-136). Специально отобранные для скрининга соединения в 96-луночном формате были приобретены в компании Selleck Chemicals и ресуспендированы до 10 мМ в ДМСО (данные не показаны). Соединения разводили в соответствующей несущей среде до 1000х от желаемой для обработки концентрации и хранили при -80°C до использования.
[00370] Обработка лекарственными средствами
[00371] Для экспериментов в 96-луночном формате прикрепленные клетки трипсинизировали, промывали PBS и высевали на обработанные для культивирования тканей 96-луночные плоскодонные планшеты (Thermo Fisher Scientific, кат. № 12-656-66) в количестве 25 000 клеток на лунку в 100 мкл среды. Суспензионные клетки промывали PBS и высевали в 96-луночные планшеты для культивирования тканей с V-образным дном (Thermo Fisher Scientific, кат. № 549935) в количестве 25 000 клеток на лунку в 100 мкл среды. Клеткам давали возможность восстановиться в течение 24 часов, а затем обрабатывали 1 мкл раствора 1 : 10 соответствующего соединения или несущей среды в PBS с поддержанием концентрации несущей среды 0,1% во всех лунках. Затем клетки подвергали воздействию низкомолекулярных соединений в указанной концентрации в течение 24 или 72 часов. Для экспериментов, в которых клетки одновременно обрабатывали ингибиторами HDAC и либо ацетатом, пируватом, цитратом, ингибитором ACSS2 (EMD Millipore Inc., кат. № 533756,), ингибитором ACLY (Cayman Chemicals, BMS-303141, кат. № 943962-47-8), либо ингибитором PDH (Cayman Chemicals, кат. № 504817), обработку клеток проводили через 24 часа после посева и собирали через 24 часа. В данной серии экспериментов все лунки содержали итоговую концентрацию 0,2% ДМСО для соответствия обработке как ингибитором HDAC, так и ингибиторами метаболических процессов.
[00372] CellTiter Glo
[00373] Клетки A549, MCF7 и K562 высевали в 96-луночные планшеты, давали им на прикрепление 24 часа и обрабатывали BMS345541, дексаметазоном, нутлином-3A, SAHA, как описано выше. Через 24 часа после обработки планшетам позволяли нагреться до комнатной температуры и оценивали жизнеспособность с помощью анализа жизнеспособности CellTiter-Glo (Promega) в соответствии с инструкциями производителя. Люминесценцию измеряли с использованием сканера для планшетов BioTek Synergy. Для каждой обработки лекарственным средством показания люминесценции нормализовали к средним значениям интенсивности люминесценции в лунках, обработанных носителем - ДМСО.
[00374] Количество клеток, которые были подвергнуты воздействию бозутиниба
[00375] Клетки A549, MCF7 и K562 высевали в 12-луночные планшеты в количестве 2,8×105 клеток на лунку. Через 24 часа, отведенных на прикрепление, клетки А549 и MCF7 в течение 24 часов подвергали воздействию 0,1, 1 и 10 мкМ бозутиниба или контроля - несущей среды ДМСО. После обработки прикрепленные клетки отделяли с помощью TrypLE или непосредственно ресуспендировали в 1 мл среды и клетки подсчитывали на автоматическом счетчике клеток Countess II FL (ThermoFisher).
[00376] Данные энциклопедии раковых клеточных линий и карты связей и анализ
[00377] Данные фармакологического профилирования загружали из портала данных Cancer cell line encyclopedia (CCLE) (доступного в Интернете по адресу portals.broadinstitute.org/ccle/data). Выделяли данные и строили график для клеточных линий, происходящих из гемопоэтической и лимфоидной ткани, легких и тканей молочной железы, подвергавшихся воздействию ингибиторов Abl - AZD0530 и нилотиниба. Данные карты связей (CMAP) загружали из управляющего приложения CUE на портале данных CMAP (доступен в Интернете по адресу clue.io/command?q = /home). Лучшие соединения и баллы связи (полученные с помощью команды /conn) экспортировали между классом пертурбагенов «ингибитор MEK», (CP_MEK_INHIBITOR) и классом пертурбагенов «ингибитор HSP» (CP_HSP_INHIBITOR) по всем клеточным линиям (сводные данные) или по отдельным клеточным линиям, перекрывающимся с нашим исследованием (A549 и MCF7). Затем результаты фильтровали для получения данных о воздействии ингибитора. Чтобы определить, каким образом изменяются связи по всем клеточным линиям в сравнении с отдельными клеточными линиями, фильтровали лучшие связи, перекрывающихся со сводными данными о связях в данных об отдельных клеточных линиях. К баллам связей применяли пороговое значение 90 как в ассоциированном исследовании CMAP (11).
[00378] Проточная цитометрия
[00379] Клетки A549 и MCF7 высевали на чашки диаметром 6 см в количестве 1,6×106 клеток на чашку. Клетки К562 высевали во флаконы Т25 см2 в количестве 1,6×106 клеток на флакон. Через 24 часа, отведенных на прикрепление, клетки А549 и MCF7 в течение 24 часов подвергали воздействию 10 мкМ абексиностата, 10 мкМ прациностата или контроля - несущей среды ДМСО. После обработки клетки собирали так, как описано выше, осажденные клетки дважды промывали в PBS, ресуспендировали в 500 мкл холодного PBS и фиксировали путем добавления 5 мл ледяного этанола при встряхивании на низкой скорости. Клетки хранили при -20°C до анализа методом проточной цитометрии. Для проведения проточной цитометрии этанол удаляли и фиксированные клетки дважды промывали PBS, содержащим 1% BSA (PBS-B), и блокировали в течение 1 часа при комнатной температуре. Затем блокирующий буфер удаляли и клетки инкубировали в PBS, содержащем 1% BSA и 0,1% Triton X-100 (PBS-BT), а также разведенное 1 : 500 мышиное антитело к ацетил-лизину (кат. № ICP0390, ImmuneChem Pharmaceuticals Inc), в течение 2 часов при комнатной температуре. После инкубации клетки дважды промывали PBS-BT и инкубировали с конъюгатом козьего антимышиного антитела с Alexa-647 в PBS-BT в течение 1 часа при комнатной температуре. Наконец, клетки дважды промывали PBS-BT, один раз PBS-B и ресуспендировали в PBS-B, содержащем 5 мкг/мл Hoechst 33258 (Life Sciences) для окрашивания ДНК. Затем уровни общего ацетилированного лизина и ДНК анализировали методом проточной цитометрии на проточном цитометре LSRII (BD Biosciences). Количественный анализ и последующий анализ проводили с помощью системы FlowJo10 (FlowJo.LLC).
[00380] Сбор клеток, выделение ядер и хеширование образцов
[00381] Для сбора прикрепленных клеток среду удаляли, а клетки промывали 100 мкл DPBS и трипсинизировали 50 мкл Tryp-LE в течение 15 минут при 37°C. После отделения клеток от планшета для культивирования реакционную смесь гасили 150 мкл ледяной DMEM, содержащей 10% FBS. Суспензии клеток получали посредством пипетирования и весь объем переносили в 96-луночный планшет с V-образным дном. Затем клетки осаждали центрифугированием при 300 x g в течение 6 минут, промывали 100 мкл ледяного DPBS и повторно осаждали при 300 x g в течение 6 минут.
[00382] Лизирование проводили в 96-луночном планшете с V-образным дном. После удаления PBS клеточные суспензии лизировали и метили, используя 50 мкл холодного лизирующего буфера (10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2, 0,1% IGEPAL CA-630) (24), с добавлением 1% Superase RNA Inhibitor и 400 фемтомоль хеширующего олигонуклеотида вида 5’- GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG-[штрихкод из 10 п. н.]- BAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA-3’ (SEQ ID NO:1) где B представляет собой G, C или T (IDT). Для скрининга большого количества соединений использовали по 500 фемтомоль дополнительного олигонуклеотида, чтобы уникальным образом проиндексировать каждый 96-луночный планшет, подлежащий обработке. После лизирования 3 циклами многоканальной пипетки клетки фиксировали путем добавления 200 мкл фиксирующего буфера (5% параформальдегида, 1,25 x PBS). Заем ядра фиксировали на льду в течение 15 минут, после чего объединяли в ванночке. Ядра объединяли путем внесения в коническую пробирку объемом 50 мл и осаждения центрифугированием при 500 x g в течение 5 минут. Затем клетки ресуспендировали в 500 мкл буфера для суспендирования ядер (NSB; (10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 1% Superase RNA Inhibitor, 1% 0,2 мг/мл сверхчистого BSA)). Наконец, ядра из всех планшетов объединяли в одну коническую пробирку и ядра осаждали центрифугированием при 500 x g в течение 5 минут. Затем ядра повторно суспендировали в 1 мл NSB и быстро замораживали в жидком азоте аликвотами по 100 мкл. После этого ядра хранили при -80°C до дальнейшей обработки с помощью sci-RNA-seq.
[00383] Получение библиотек sci-RNA-seq2
[00384] Замороженные ядра размораживали на льду и центрифугировали при 500 g в течение 5 минут. Затем клетки пермеабилизировали в буфере для пермеабилизации (NSB+0,25% Triton-X) в течение 3 минут, после чего центрифугировали. После еще одной отмывки в NSB получали двухуровневые библиотеки sci-RNA-seq, как описано ранее (25). Вкратце, ядра осаждали при 500 x g в течение 5 минут и ресуспендировали в 100 мкл NSB. Количество клеток получали путем окрашивания ядер 0,4% трипановым синим (Sigma-Aldrich) и подсчитывали с использованием гемоцитометра. Затем наносили по 5000 ядер в 2 мкл NSB и 0,25 мкл смеси 10 мМ дНТФ (Thermo Fisher Scientific, кат. № R0193) в лунки 96-луночного планшета с полуюбкой twin.tec LoBind (Fisher Scientific, кат. № 0030129512), после чего в каждую лунку добавляли 1 мкл имеющего уникальный индекс олиго-dT (25 мкМ)(25), инкубировали при 55°C в течение 5 минут и помещали на лед. После этого в каждую лунку добавляли по 1,75 мкл смеси для обратной транскрипции (1 мкл буфера Superscript IV для синтеза первой цепи, 0,25 мкл 100 мМ DTT, 0,25 мкл Superscript IV и 0,25 мкл рекомбинантного ингибитора рибонуклеазы RNAseOUT), инкубировали планшеты при 55°C в течение 10 минут и помещали на лед. Для остановки реакции в каждую лунку добавляли по 5 мкл стоп-раствора (40 мМ ЭДТА, 1 мМ спермидина и 0,5% BSA). Лунки объединяли, используя наконечники с широким каналом, ядра переносили в пробирку для проточной цитометрии через фильтровальный колпачок 0,35 мкм и добавляли DAPI до конечной концентрации 3 мкМ. Затем объединенные ядра сортировали на сортировщике клеток FACS Aria II в количестве 150 клеток на лунку в 96-луночные планшеты LoBind, содержащие по 5 мкл буфера EB (Qiagen). После сортировки в каждую лунку добавляли по 0,75 мкл смеси для второй цепи (0,5 мкл буфера для синтеза второй цепи мРНК и 0,25 мкл фермента для синтеза второй цепи мРНК, New England Biolabs), синтез второй цепи проводили при 16°C в течение 150 минут. Тагментацию выполняли путем добавления 5,75 мкл смеси для тагментации (0,01 мкл специализированного фермента TDE1 в 5,74 мкл 2x буфера Nextera TD, Illumina) и инкубировали планшеты в течение 5 минут при 55°C. Реакцию прекращали добавлением 12 мкл ДНК-связывающего буфера (Zymo) и инкубировали в течение 5 минут при комнатной температуре. В каждую лунку добавляли по 36 мкл гранул Ampure XP, очищали ДНК с использованием стандартного протокола Ampure XP (Beckman Coulter) с элюированием 17 мкл буфера EB и переносили ДНК в новый 96-луночный планшет LoBind. Для проведения ПЦР в каждую лунку добавляли по 2 мкл индексированного P5, 2 мкл индексированного P7 (25) и 20 мкл мастер-микса NEBNext High-Fidelity (New England Biolabs) и выполняли ПЦР следующим образом: 75°C в течение 3 минут, 98°C в течение 30 секунд и 18 циклов при 98°C в течение 10 секунд, 66°C в течение 30 секунд и 72°C в течение 1 минуты с последующим конечным удлинением при 72°C в течение 5 минут. После ПЦР все лунки объединяли, концентрировали с использованием набора DNA clean and concentrator kit (Zymo) и очищали посредством чистящего реагента 0,8X Ampure XP. Конечные концентрации библиотек определяли с помощью Qubit (Invitrogen), библиотеки визуализировали с использованием системы TapeStation D1000 DNA Screen tape (Agilent) и библиотеки секвенировали на Nextseq 500 (Illumina) с использованием высокопроизводительного набора из 75 циклов (чтение 1: 18 циклов, чтение 2: 52 цикла, индекс 1: 10 циклов и индекс 2: 10 циклов).
[00385] Получение библиотек sci-RNA-seq3
[00386] Замороженные ядра размораживали так, как описано ранее, и получали трехуровневые библиотеки sci-RNA-seq, как описано в (21). Ядра осаждали при 500 x g в течение 5 минут, трижды промывали NSB, и небольшую аликвоту ядер окрашивали 0,4% трипановым синим (Sigma-Aldrich) и подсчитывали ядра с помощью гемоцитометра. Затем 80 000 ядер в 22 мкл NSB и 2 мкл смеси 10 мМ дНТФ распределяли в каждую лунку 96-луночных планшетов с полуюбкой LoBind, добавляли по 2 мкл совместимых с лигированием индексированных олиго-dT праймеров, инкубировали при 55 C в течение 5 минут и помещали на лед. После этого в каждую лунку добавляли по 14 мкл смеси для обратной транскрипции (8 мкл буфера Superscript IV для синтеза первой цепи, 2 мкл 100 мМ DTT, 2 мкл Superscript IV и 2 мкл рекомбинантного ингибитора рибонуклеазы RNAseOUT), и выполняли ОТ-ПЦР в термоциклере с использованием следующей программы: 4 C в течение 2 минут, 10 C в течение 2 минут, 20 C в течение 2 минут, 30 C в течение 2 минут, 40 C в течение 2 минут, 50 в течение 2 минут и 55 C в течение 15 минут. После RT в каждую лунку добавляли по 60 мкл ядерного буфера, содержащего BSA (NBB, 10 мМ трис-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2 и 1% BSA), ядра объединяли при помощи наконечника с широким каналом, ядра осаждали центрифугированием при 500 x g в течение 10 минут и удаляли супернатант. Второй цикл комбинаторного индексирования выполняли посредством лигирования индексированных праймеров с 5’-концом индексированных ОТ кДНК. Ядра ресуспендировали в NSB и добавляли по 10 мкл в каждую лунку 96-луночных планшетов LoBind, после чего в каждую лунку добавляли по 8 мкл индексированных праймеров для лигирования вместе с 22 мкл смеси для лигирования (20 мкл буфера Quick ligase и 2 мкл Quick ligase, New England Biolabs). Затем лигирование выполняли при 25 °C в течение 10 минут. После лигирования в каждую лунку добавляли по 60 мкл NBB, ядра объединяли с помощью наконечника с широким каналом, к ядрам добавляли еще 40 мл NBB и ядра осаждали центрифугированием при 600 x g в течение 10 минут и удаляли супернатант. Затем ядра промывали однократно 5 мл NBB, ресуспендировали в 4 мл NBB, мультиплеты удаляли фильтрованием с использованием клеточного сита Flowmi с размером пор 40 мкм (Sigma-Aldrich), подсчитывали ядра и помещали по 5000 ядер в лунки 96-луночных планшет LoBind в объеме 5 мкл. Планшеты, содержащие ядра, замораживали и хранили при -80 C до дальнейшей обработки. После размораживания в каждую лунку добавляли по 5 мкл смеси для синтеза второй цепи (3 мкл буфера для элюирования, 1,33 мкл буфера для синтеза второй цепи мРНК и 0,66 мкл фермента для синтеза второй цепи мРНК) и инкубировали при 16 C в течение 3 часов. Тагментацию выполняли путем добавления 10 мкл смеси для тагментации (0,01 мкл специализированного фермента TDE1 в 9,99 мкл 2x буфера Nextera TD, Illumina) и планшеты инкубировали в течение 5 минут при 55 C. После тагментации в каждую лунку добавляли по 20 мкл буфера для связывания ДНК и планшеты инкубировали при комнатной температуре в течение 5 минут. Затем в каждую лунку добавляли по 40 мкл гранул Ampure XP и инкубировали планшеты в течение 5 минут при комнатной температуре. После выделения гранул при помощи магнитной стойки супернатант удаляли и гранулы дважды промывали 80% этанолом. Затем в каждую лунку добавляли по 10 мкл реакционной смеси USER (1 мкл 10-кратного буфера USER и 1 мкл фермента USeR в безнуклеазной воде, New England Biolabs), гранулы ресуспендировали и инкубировали при 37 C в течение 15 минут. После инкубации в каждую лунку добавляли по 7 мкл буфера для элюирования и супернатант переносили в новый 96-луночный планшет LoBind после связывания гранул на магнитной стойке. После инкубации при 85 C в течение 10 минут генерировали библиотеки с использованием 15 циклов ПЦР. После ПЦР-амплификации библиотеку секвенирования очищали, сначала концентрируя 1 мл ПЦР-библиотеки с использованием реактива для очистки 1x Ampure, а затем прогоняли полученный продукт на 2% агарозном геле, содержащем бромид этидия. Гель разрезали и выделяли 2 фрагмента - молекулы хеша (220 п. н.-250 п. н.) и библиотеку РНК (250 п. н.-1000 п. н.). После экстракции геля и дополнительной очистки реактивом 1x Ampure библиотеки РНК секвенировали на приборе NovaSeq 6000 (Illumina) (чтение 1: 34 п. н., чтение 2: 100 п. н., индекс 1: 10 п. н. и индекс 2: 10 п. н.) и хеш-библиотеки секвенировали на 75 циклах NextSeq (чтение 1: 34 п. н., чтение 2: 38 п. н., индекс 1: 10 п. н. и индекс 2: 10 п. н.)
[00387] Получение библиотек массового секвенирования РНК
[00388] Обработанные соединением клетки сначала трипсинизировали и собирали, как описано ранее. Затем клетки лизировали в планшетах с V-образным дном с использованием 26 мкл NSB. Добавляли по 2 мкл 25 мкМ индексированных праймеров ОТ-ПЦР и отжигали при 65 C в течение 5 минут. Затем проводили реакцию ОТ-ПЦР с использованием системы SuperScript IV, добавляя по 8 мкл 5X буфера SuperScript, 2 мкл SuperScript IV, 2 мкл 10 мМ смеси дНТФ, 2 мкл 100 мМ DTT и 2 мкл рекомбинантного ингибитора рибонуклеаз RNAseOUT в лунку. Реакцию проводили в течение 10 минут при 55°C, а затем останавливали путем тепловой инактивации (80°C в течение 10 минут). Затем библиотеки объединяли и избыток праймера ОТ-ПЦР удаляли посредством либо двух очисток при помощи 0,7-x SPRI, либо одной очистки 0,7-x SPRI с последующей обработкой Exo-1 и инактивацией. Двухцепочечную ДНК получали путем инкубации при 16°C в течение 3 часов со смесью для синтеза второй цепи, содержащей 0,5 мкл фермента и 2 мкл реакционного буфера для второй цепи в конечном объеме 20 мкл. После синтеза второй цепи библиотеки тагментировали при помощи 1 мкл коммерческого реагента Nextera с использованием 20,5 мкл 2x буфера TD. Реакции останавливали, используя 40 мкл буфера Zymo Clean and Concentrate, и инкубировали при комнатной температуре в течение 5 минут. Затем библиотеки очищали посредством чистящего реагента 1x SPRI и элюировали в 16 мкл элюирующего буфера. Библиотеки секвенирования генерировали посредством ПЦР с использованием 2 мкл индексированных праймеров P7 и P5 и 20 мкл 2x мастер-микса NEB Next. Наконец, библиотеки объединяли, очищали посредством чистящего реагента 1x SPRI и количественно оценивали. Библиотеки секвенировали на секвенаторе Nextseq 500 (Illumina) с использованием набора 75 высокопроизводительных циклов (чтение 1: 18 циклов, чтение 2: 52 цикла, индекс 1: 10 циклов и индекс 2: 10 циклов).
[00389] Предварительная обработка данных секвенирования
[00390] Сначала прогоны секвенирования демультиплексировали с использованием bcl2fastq v.2.18. Оставляли только штрихкоды, совпадающие с индексами обратной транскрипции в пределах дистанции редактирования 2 п. н. Для библиотек sci-RNA-seq3 сохраняли штрихкоды, которые совпадали как с предоставленными индексами обратной транскрипции, так и с индексами лигирования в пределах дистанции редактирования 2 п. н. После присвоения индексов полиА-хвосты обрезали с помощью trim-galore, и чтения картировали с транскриптомом человека (hg-38) или транскриптомом человек-мышь (hg-38 и MM-10) с помощью модуля выравнивания STAR. После выравнивания чтения фильтровали по качеству выравнивания и удаляли дубликаты. Чтения считали дубликатами, если они (1) картировались на одном и том же гене, (2) картировались на одном и том же штрихкоде клетки и (3) содержали одинаковый уникальный молекулярный идентификатор (UMI). Чтения, удовлетворяющее первым двум критериям и отличающееся дистанцией редактирования 1 от ранее наблюдавшегося UMI, также помечали как дубликаты и отбрасывали. Не являющиеся дубликатами чтения были связаны с генами с помощью bedtools (40), чтобы они пересекались с аннотированной генной моделью. Все 3’-UTR в генной модели были удлинены на 100 п. н.
[00391] для учета возможности того, что некоторые аннотации 3’ UTR-областей генов могут быть слишком короткими, что приводит к неправильному аннотированию чтений генов как межгенных. Штрихкоды клеток считали соответствующими настоящей клетке, если количество уникальных чтений, ассоциированных со штрихкодом, было больше заданного в интерактивном режиме порога на «коленном графике». Чтения из клеток, прошедших данный порог по количеству UMI, сначала агрегировали в формат разреженной матрицы, а затем загружали и сохраняли в виде объекта CDS для анализа с помощью Monocle 3.
[00392] Назначение маркировок образцов по чтениям хешей
[00393] Для идентификации чтений хешей анализировали демультиплексированные чтения, совпадающие с комбинаторными индексирующими штрихкодами. Чтения считали чтениями хешей, если они соответствовали двум критериям: (1) первые 10 п. н. чтения 2 совпадали с хеш-штрихкодом в эксперименте в пределах дистанции редактирования, равной двум, и (2) содержали последовательность полиА между парами оснований 12-16 чтения 2. Затем эти чтения дедублировали по штрихкоду клеток и свертывали по UMI для получения вектора hi числа UMI хеширующих олигонуклеотидов для каждого ядра i в эксперименте.
[00394] Чтобы назначить каждому ядру i культуральную лунку, из которой оно было взято, мы проверяем, обогащена ли библиотека sci-RNA-seq конкретным хеш-штрихкодом. Мы сравниваем UMI хеша ядра с «фоновым распределением», которое в идеальных обстоятельствах будет равномерным распределением. На практике незначительные колебания концентраций хеширующих олигонуклеотидов, добавляемых в каждую лунку с выделенными ядрами, могут потребовать эмпирической оценки фона. Для этого мы просто усредняем относительные UMI хеша из индексов клеток, для которых было собрано менее чем < UMI мРНК, предполагая, что это отражает вклад в библиотеку супернатанта в лунках ОТ-ПЦР, фрагментов мусора и т. п. Затем мы сравниваем хеш-UMI hi ядра i с этим фоном с помощью критерия хи-квадрат. После корректировки итоговых значений p для множественных тестов по Бенджамини - Хохбергу мы отбрасываем нулевую гипотезу, что hi обусловлена фоновым распределением при указанной FDR (в данном исследовании использовали 5% FDR). После этого ядра с количеством хешей, которое считается отличным от фона, оценивают на обогащение одной хеш-последовательностью. Коэффициенты обогащения рассчитывали как отношение количества UMI для самого распространенного хеширующего олигонуклеотида к количеству UMI для второго по распространенности хеширующего олигонуклеотида. Более конкретно, если количество UMI для наиболее распространенного хеша в ядре i в два раза больше, чем для второго наиболее распространенного, то i обозначается как единственный. определяли в каждом эксперименте на основе анализа распределения этих соотношений и выбора значения для разделения немеченых клеток и отдельных меченых клеток. Клетки, которые достигали нижнего увеличения численности уникального хеширующего олигонуклеотида, помечали как мультиплет или продукты распада клеток и выбрасывали.
[00395] Анализ зависимости «доза - ответ»
[00396] Анализ зависимости «доза - ответ» проводили в R с использованием пакета drc (41) посредством аппроксимации четырехпараметрической лог-логистической модели для каждого лекарственного средства к количеству клеток, выделенных в данных одноклеточной РНК секвенирования при каждой дозе. Клетки, выжившие удвоенный анализ и КК, группировали по их культуральной лунке происхождения и подсчитывали. Затем эти количества корректировали с учетом изменений в восстановлении в зависимости от типа клеток и культуральной чашки, как описано ниже. Вектор x количества клеток в лунках соответствовал модели
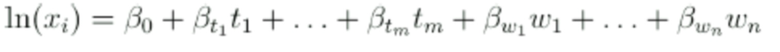 ,
,
[00397] где Ti и Wj представляют собой переменные двоичного индикатора, кодирующие тип клеток и культуральный планшет соответственно. Затем скорректированные количества клеток для данной лунки культурального планшета с клетками определенного типа рассчитываются следующим образом:
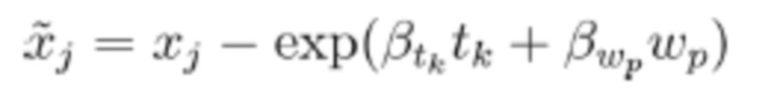 .
.
[00398] После этого скорректированные полуночные количества клеток группировали по типам и лекарственным средствам и подавали в качестве входных данных для функции drm() пакета drc с модельной формулой «количество_клеток ~ лог_доза» и функцией семейства моделей LL.4(). Эта процедура позволяет аппроксимировать модель:
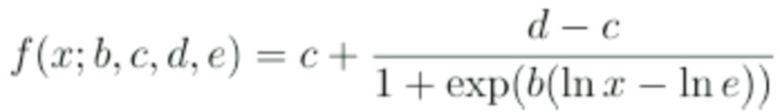 .
.
[00399] В приведенной выше модели параметры и соответствуют нижнему и верхнему асимптотическим пределам ответа соответственно. Крутизна кривой ответа отражена в b, а e представляет собой параметр, который кодирует полумаксимальную «эффективную дозу» (ED50).
[00400] Кривые «доза - ответ» позволяют аннотировать клетки в соответствии с влиянием условий их культивирования на жизнеспособность. Каждой клетке присваивали «показатель жизнеспособности», который представляет собой просто ожидаемую долю клеток из несущей среды, остающихся после воздействия данной дозы соединения. Эти количества клеток генерируются с помощью функции predict() пакета drc, а затем нормализуются относительно соответствующего контроля - несущей среды.
[00401] Уменьшение размерности и анализ траектории
[00402] Профили экспрессии генов визуализировали при помощи программного обеспечения Monocle 3, в котором используется алгоритм UMAP для их проецирования в двух- или трехмерное пространство. Вкратце, сначала Monocle 3 рассчитывает размерные коэффициенты для каждой клетки. Размерные коэффициенты рассчитывались как логарифмические количества UMI, наблюдаемые в одной клетке, поделенные на геометрическое среднее логарифмических количеств UMI по всем измеренным клеткам. После масштабирования количеств UMI для каждого ядра в соответствии с размерным коэффициентом библиотеки Monocle3 добавляет псевдоколичество 1 и выполняет логарифмическое преобразование количеств. Затем эти логарифмически преобразованные профили проецируются на первые 25 основных компонент. Эти PCA-координаты были преобразованы Monocle 3 (с применением подхода, аналогичного функции removeBatchEffect() в пакете limma (42)) в соответствии с моделью «~ log(UMI) + повторность» (Фиг. 9) или «~ log(UMI) + жизнеспособность+коэффициент пролиферации+повторность». Для инициализации UMAP используют скорректированные координаты PCA для каждой клетки. Если не указано иное, UMAP запускали со следующими параметрами: 50 ближайших соседей, min_dist=0,1, оценка межклеточного расстояния по косинусному коэффициенту. UMAP-проекцию клеток после двойного ингибирования HDAC и добавления предшественника ацетил-КоА или ингибирования фермента, образующего ацетил-КоА, выполняли так, как описано выше, за исключением того, что инициализацию PCA проводили по 1000 наиболее сильно распределенных генов. Затем в этом пространстве UMAP выполняли обнаружение Лувенских сообществ с использованием Python-пакета «louvain». После этого проводили реконструкцию траектории, как описано в (21).
[00403] Для определения того, демонстрируют ли клетки, обработанные конкретной комбинацией соединение/доза, обогащение в пространстве UMAP, мы создали таблицы сопряженности для количества клеток, обработанных соединением или несущей средой, внутри и снаружи кластеров, и использовали пакет stats R для реализации точного критерия Фишера в целях проверки обогащения. Для наглядного представления вызванного лекарственными средствами обогащения на ФИГ. 9B была добавлена непрозрачность к клеткам, получившим минимальную дозу в комбинации соединение/доза, проходящую пороговый критерий FDR < 1% и log2 коэффициента несогласия > 2,5. Клетки, прошедшие эти фильтры, использовали для получения тепловой карты доли обогащенных клеток по кластерам, представленной на Фиг. 12.
[00404] Оценка коэффициента пролиферации Для получения оценки коэффициента пролиферации для одной клетки для каждой клетки суммировали и подвергали логарифмическому преобразованию нормализованному по размерному коэффициенту экспрессию маркерных генов клеточного цикла (из таблицы S5 в (43)). Оценки были рассчитаны таким образом как для G1S, так и для G2M. Термин «коэффициент пролиферации» относится к общему пролиферативному состоянию клетки и рассчитывается как логарифмированная сумма совокупных уровней экспрессии генов G1S и G2M.
[00405] Анализ дифференциальной экспрессии
[00406] Для проверки того, экспрессируется ли ген дифференциально в клеточной линии дозозависимым образом при воздействии соединения, мы аппроксимировали его количество UMI (скорректированное по размерному коэффициенту библиотеки), зарегистрированное в каждом ядре, с помощью генерализованной линейной модели:
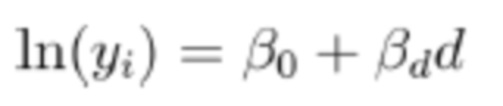 ,
,
[00407] где Yi представляет собой случайную переменную с квазипуассоновской оценкой, d представляет собой логарифмически преобразованную дозу оцениваемого соединения. Аппроксимация моделей осуществлялась с помощью программного обеспечения Monocle 3, которое использует пакет speedglm. Для аппроксимации регрессионной моделью эффекта каждого лекарственного средства на каждый ген мы сначала идентифицируем подгруппу клеток, релевантных для модели. Для определения эффектов на ген G в клетках типа C при обработке лекарственным средством D включаются все клетки типа C, которые обрабатывали любой дозой D. К ним добавляются клетки типа C, которые обрабатывали контролем - несущей средой. Затем проводили аппроксимацию указанной выше моделью, связывающей уровень экспрессии G по всем этим клеткам. Гены считаются дозозависимыми дифференциально экспрессируемыми генами (DEG), если аппроксимирующие их модели включают в себя член βd, который существенно отличается от нуля при оценке с помощью критерия Вальда (с коррекцией по Бенжамини - Хохбергу, p < 0,05). P-значения для членов βd объединяли для всех соединений и для всех генов перед внесением поправки на множественные тесты.
[00408] Для оценки гена на дифференциальную экспрессию в зависимости от «псевдодозы» yв консенсусной траектории ингибирования HDAC мы проводили аппроксимацию моделью
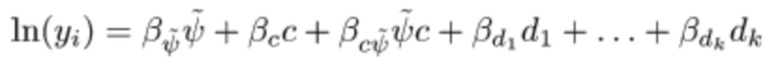 ,
,
[00409] где Yi представляет собой квазипуассоновскую переменную, фиксирующую количество UMI для гена, y кодирует значения псевдодозы, сглаженные посредством естественного сплайна, c представляет собой коэффициент, кодирующий тип клеток, и βcy отражает взаимодействие между типом клеток и псевдодозой. Член βdJdJ кодирует (логарифмически) дозозависимые эффекты соединения.
[00410] Попарная корреляция подвергнутых скринингу соединений
[00411] Для идентификации соединений, которые приводят к аналогичным дозозависимым изменениям в клеточных транскриптомах, мы рассчитывали корреляцию Пирсона между всеми попарными сочетаниями соединений. Мы создали матрицу ген х соединение для объединения дозозависимых генов по всем соединениям, где каждая запись представляет собой бета-коэффициент для определяющего дозозависимость члена βd, а затем рассчитывали корреляцию Пирсона для каждой пары лекарственных средств с помощью функции cor.test() в пакете R stats с указанием по использованию полных наблюдений. Затем полученная корреляционная матрица была иерархически кластеризована с использованием пакета pheatmap в R. Достоверность каждой попарной корреляции определяли с помощью функции corr.test() из пакета psych в R с указанием в качестве коррекции на множественные проверки гипотез метода Бенджамини - Хохберга.
[00412] Анализ обогащения наборов генов
[00413] После аппроксимации с помощью общей линейной модели гены, имеющие значимые коэффициенты (порог FDR 5%), использовали при анализе обогащения наборов генов с использованием пакета piano в R (44). Вкратце, наборы генов ранжировали по усредненному в пределах набора статистическому критерию Вальда, соответствующему члену генерализованной линейной модели, оцениваемому функцией runGSA() пакета piano. Гены рандомизировали по наборам для установления нулевого распределения для ранга каждого набора. После 10 000 преобразований функция runGSA() вычисляла p-значения с использованием «смешанной» направленной политики обогащения. Для визуализации выбирали лучшие наборы генов, соответствующие тем, которые имели статистику с наибольшей величиной обогащения.
[00414] Выравнивание клеток, обработанных ингибиторами HDAC
[00415] Для организации клеток, обработанных ингибиторами HDAC, в траекторию отбирали выборки клеток, чтобы сделать равными количества клеток, представленных в трех клеточных линиях или обработках через 24 и 72 часа. Затем совместно вычисляли координаты PCA, после чего проводили выравнивание с помощью функции mnCorrect из пакета scran (32). Эти скорректированные координаты использовали для инициализации UMAP в Monocle 3. Затем главным графиком аппроксимировали данные, используя функцию lean_graph(). Для определения начала траектории мы сопоставляли каждую клетку с ближайшим к ней узлом главного графика, а затем выбирали все узлы главного графика, у которых большинство сопоставленных клеток были обработаны несущей средой. Псевдодозы для всех остальных клеток y измеряли как геодезическое расстояние между ближайшим к ним узлом главного графика и узлом-началом.
[00416] Для количественного определения эффективности каждого ингибитора HDAC сначала группировали все клетки из каждой повторности по обработке и дозе, а затем рассчитали среднюю псевдодозу для каждой клетки. Затем аппроксимировали зависимость средних значений псевдодозы от концентрации соединения с использованием пакета для drc (41). Мы использовали четырехпараметрическую лог-логистическую модель, где максимальный ответ фиксировался на наивысшем значении псевдодозы, достигаемом для всех соединений и доз. Затем брали параметр модели e, как описано в разделе «Анализ зависимости "доза - ответ"» выше, в качестве транскрипционного EC50 (TC50) для каждого соединения.
[00417] Пример 2
[00418] Массивно-мультиплексное профилирование доступности хроматина в одиночных клетках
[00419] Клетки подвергаются изменению экспрессии генов в ответ на многочисленные связанные со средой, развитием и терапией стимулы, и специфические ответы значительно варьируют у отдельных типов клеток. Такое перепрограммирование генома формируется за счет связывающихся с ДНК факторов транскрипции (TF), которые направляют динамическую систему изменений состояния хроматина вблизи промоторов и энхансеров (Takahashi and Yamanaka 2016). Однако измерение и интерпретация такого геномного взаимодействия остается чрезвычайно сложной задачей в гетерогенных популяциях клеток или образцах ткани. Для решения этой проблемы был разработан новый подход - sciPlex-ATAC-seq, который позволяет одновременно профилировать доступный геном в тысячах отдельных клеток из практически неограниченных экспериментальных условий.
[00420] Обзор технологии
[00421] Были разработаны эффективно масштабируемые способы секвенирования транскриптомов одиночных клеток (sci-RNA-seq) и профилирования доступности хроматина посредством комбинаторного индексирования (sci-ATAC-seq), для которого не требуется физическое выделение одиночных клеток (Cusanovich et al. 2015; Cao et al. 2017). В этих способах применяется стратегия комбинаторного молекулярного индексирования, которая позволяет профилировать экспоненциально большее количество клеток при каждом цикле штрихкодирования, что значительно снижает стоимость эксперимента.
[00422] В данном случае, чтобы обеспечить одновременную обработку множества образцов, мы разработали систему мечения клеток, так чтобы при секвенировании доступного хроматина также одновременно выполнялся анализ меток внутри каждого ядра. В подходе с мечением используется склонность выделенных ядер к поглощению одноцепочечных ДНК-олигонуклеотидов (меток), которые затем могут быть захвачены посредством мягкой фиксации (Srivatsan et al. 2020). По мере того как ядра проходят множество циклов индексирования, к меткам и хроматину в каждом ядре добавляются пригодные для секвенирования штрихкоды, и в конечном итоге получается уникальная комбинация штрихкодов для материала из каждой клетки. Таким образом, все секвенированные фрагменты хроматина и метки, имеющие одинаковую комбинацию штрихкодов, получены из одной и той же клетки. При использовании этой стратегии мечения можно получать доступные профили хроматина в тысячах отдельных клеток практически из любого количества имеющих уникальные метки образцов одновременно (Фиг. 41A, 41B).
[00423] Результаты
[00424] Ядра с метками из одноцепочечных ДНК-олигонуклеотидов, обеспечивающие параллельную обработку множества образцов для определения профиля доступности хроматина в одиночных клетках
[00425] Для проверки того, может ли наш подход к мечению точно определить, к какому образцу относятся отдельные клетки, был проведен эксперимент по смешиванию видов, в котором человеческие и мышиные клеточные линии были по отдельности помечены идентифицирующими виды олигонуклеотидами (хешами), с последующим объединением и получением профилей доступности хроматина в одиночных клетках посредством комбинаторного индексирования. Клетки каждой из мышиной (NIH-3T3) и человеческой (A549) клеточных линий разделяли на три образца, и каждый из них по отдельности метили одним из шести олигонуклеотидов. Вкратце, из каждого из шести образцов выделяли ядра, кратковременно инкубировали с отдельной олигонуклеотидной меткой, а затем фиксировали 1% формальдегидом (Фиг. 41A). После фиксации все клетки объединяли и ядерные метки отжигали с общим захватным олигонуклеотидом, получая структуру, напоминающую тагментированную ДНК, что позволило проводить дальнейший совместный анализ с геномной ДНК. Затем объединенные ядра тагментировали в массе посредством Tn5 с помощью адаптеров Nextera, а 5’-концы фосфорилировали с помощью T4PNK. После этого выполняли комбинаторное индексирование как для ядерной ДНК, так и для захваченных олигонуклеотидных меток, используя следующие стадии: 1) ядра равномерно распределяли по 96 лункам, лигировали специфичным для лунки штрихкодом 1 на концах N7, 2) объединяли все лунки и снова равномерно распределяли ядра по 96 лункам, лигировали специфичным для лунок штрихкодом 2 на концах N5 и 3) объединяли все лунки и снова распределяли ядра в последний 96-луночный планшет для добавления штрихкода 3 на основе ПЦР (Фиг. 41B). Поскольку крайне маловероятно, что материал из двух ядер получит одну и ту же комбинацию штрихкодов, ожидается, что все фрагменты хроматина и молекулы меток с соответствующими штрихкодами будут получены из одной и той же клетки.
[00426] Если добавленные метки действительно остаются внутри фиксированных ядер на протяжении всей описанной подготовки библиотеки, ожидается, что удастся выделить метки, используемые на человеческих образцах, исключительно из человеческих клеток, а мышиные метки - из мышиных клеток. Действительно, виды клеток можно определить исключительно на основе молекул меток, выделенных из каждого ядра (Фиг. 411C). Более того, если обнаруживалось, что клетки имеют смесь специфических меток человека и мыши, они также имели смесь фрагментов хроматина обоих видов, что указывает на дублеты (Фиг. 41D). В этом эксперименте получили более 100 молекул меток из большинства клеток (Фиг. 41E), и наиболее распространенная метка в ядре обычно встречается по меньшей мере в 5 раз большем количестве, чем любая другая молекула метки (Фиг. 41F), и это указывает на очень малый фон, обусловленный смешением меток.
[00427] SciPlex-ATAC позволяет получить мультиплексный профиль хроматина одиночных клетках при химическом скрининге.
[00428] Для демонстрации способности к мультиплексированию множества образцов в нашем подходе мы провели химический скрининг на клетках легочной аденокарциномы (A549), чтобы изучить изменение характеристик хроматина в ответ на четыре соединения с известным влиянием на глобальную регуляцию генов. Каждую лунку 96-луночного культурального планшета обрабатывали в течение 24 часов нутлином-3A (агонистом p53), SAHA (ингибитором гистондеацетилазы широкого спектра действия), BMS345541 (ингибитором NF-kB), дексаметазоном (агонистом глюкокортикоидных рецепторов) или контролем - несущей средой. Более того, мы варьировали дозу лекарственного средства, добавленного в каждую лунку, так, чтобы эффекты каждого соединения можно было оценивать в семи разных концентрациях. Наконец, все виды обработки проводили в трех биологических повторностях (Фиг. 42A). После нанесения уникальных меток в каждую лунку клетки при всех воздействиях объединяли для параллельной подготовки к профилированию доступного хроматина в одиночных клетках.
[00429] В этом пилотном эксперименте были получены высококачественные профили хроматина из 7770 клеток, которые разделялись на дискретные популяции по организации хроматина. Важно отметить, что при использовании секвенируемых меток для идентификации условий обработки каждой клетки было очевидно, что структура популяции в значительной степени обусловлена специфичным для лекарственных средств воздействием на профили хроматина (Фиг. 42B). Более того, дозозависимые тенденции показали все большее изменение регуляторных характеристик при увеличении доз, особенно при широком ингибировании гистондеацетилаз (SAHA) (Фиг. 42C, правая нижняя панель). Интересно, что только на основе этих данных также удалось определить пороговые значения, выше которых конкретные лекарственные средства становятся токсичными для клеток (Фиг. 42D). Данный эксперимент демонстрирует возможность анализа доступности хроматина в отдельных клетках из почти 100 образцов одновременно. Ведущийся анализ этих данных направлен на изучение дифференциально доступных сайтов генома, отвечающих на обработку лекарственными средствами. Например, некодирующие сайты ближе к 5’-концу от локуса COPS7A демонстрировали дозозависимую доступность в ответ на SAHA (Фиг. 42E).
[00430] Способы
[00431] Введение хеширующих меток в ядра
[00432] Прикрепленные клетки, выращенные в 96-луночном формате, сначала готовили посредством аспирации имеющейся среды. Затем в каждую лунку добавляли по 50 мкл среды TrypLE (Termo-Fisher) и планшет инкубировали при 37 C в течение 15 минут для A549. После инкубации добавляли 150 мкл 1x DMEM (Gibco) + 10% FBS (Gibco) для гашения реакции TrypLE. Затем объем 200 мкл суспензии клеток из каждой лунки переносили в 96-луночный планшет с V-образным дном, сохраняя ориентации лунок. После этого клетки центрифугировали в течение 5 минут при 300 g для осаждения клеток перед аспирацией среды. Клеточный осадок промывали 100 мкл 1x DPBS и затем осаждали при 300 g в течение 5 минут. Для получения суспензионных клеток содержимое лунок сначала переносили в планшет с v-образным дном, а затем осаждали при 300 g в течение 5 минут. Затем клетки промывали в 200 мкл 1x DPBS, снова центрифугировали и удаляли DPBS. Далее для выделения ядер из клеток осадки ресуспендировали и осторожно пипетировали вверх и вниз несколько раз в 50 мкл холодного лизирующего буфера (10 мМ TrisHCl, 10 мМ NaCl, 3 мМ MgCl2, 0,1% Igepal, 0,1% Tween20) или лизирующего буфера OMNI (10 мМ TrisHCl, 10 мМ NaCl, 3 мМ MgCl2, 0,1% Igepal, 0,1% Tween20, 0,01% дигитонина (Promega), 1x ингибитор протеаз (Thermo Pierce Protease Inhibitor Tablets, без ЭДТА)). Затем к ядрам добавляли одноцепочечные ДНК-олигонуклеотидные метки (хеши) (ориентируясь на приблизительно 1 нмоль молекул хеша на 25 000 клеток) в лизирующем буфере и инкубировали на льду в течение 5 минут. Затем к образцам добавляли ледяной фиксирующий буфер (1,5% формальдегида, 1,25 x DPBS (gibco)) до достижения конечной концентрации формальдегида 1% и осторожно перемешивали. Фиксацию проводили в течение 15 минут на льду. В этот момент ядра из разных образцов (т. е. из разных лунок) объединяли и выполняли дополнительные стадии на этом едином пуле. Сначала фиксатор удаляли, центрифугируя пул образцов при 500 x g в течение 5 минут. Затем осадок ресуспендировали в буфере для суспендирования ядер (10 мМ TrisHCl, 10 мМ NaCl, 3 мМ MgCl2) + 0,1% Tween 20. Ядра повторно осаждали, а затем снова суспендировали в буфере для замораживания (50 мМ Tris pH 8,0, 25% глицерин, 5 мМ Mg(OAc)2, 0,1 мМ ЭДТА, 5 мМ DTT, 1x ингибитор протеазы (Thermo Pierce)) в конечной концентрации 2,5 миллиона ядер/мл (два уровня) или 5 миллионов ядер/мл (три уровня). Затем объединенные пробы быстро замораживали в жидком азоте и хранили при -80 C.
[00433] Совместный захват хеширующих олигонуклеотидов и получение профилей ATAC при двухуровневом sci-ATAC
[00434] Объединенные помеченные хешами ядра размораживали на льду, проверяли целостность ядер, подсчитывали и при необходимости дополнительно доводили до 2,5 миллиона ядер/мл. Затем по 2 мкл ядер распределяли во все лунки 96-луночного планшета с сильным связыванием. Для захвата хеш-молекул каждого ядра в каждую лунку добавляли 1 мкл 25 мкM одноцепочечных ДНК-олигонуклеотидов (5’-TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGNNNNNNNNXXXXXXXXXXTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTVN-3’ (SEQ ID NO:2)) («X» представляет собой специфичный для лунки штрихкод, а «N» представляет собой уникальный молекулярный индекс (UMI)). Затем планшет инкубировали при 55°C в течение 5 минут и немедленно возвращали на лед на 5 минут. После этого захватные олигонуклеотиды, отожженные с хеш-молекулами, удлиняли посредством добавления в каждую лунку 3 мкл мастер-микса NEBNext High-Fidelity 2X PCR Master Mix и инкубации при 55°C в течение 10 минут. После удлинения во все лунки добавляли по 12 мкл 2X буфера для тагментации (20 мМ Tris pH 7,3, 10 мМ MgCl2, 20% DMF) и 4 мкл (40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2, 0,4% NP40, 0,4% Tween20). Наконец, в каждую лунку добавляли по 1 мкл индексированной Tn5 (Cusanovich et al. 2015) и выполняли тагментацию при 55°C в течение 15 минут, после чего возвращали на лед. Тагментацию останавливали, добавив во все лунки по 25 мкл ледяной холодной 40 мМ ЭДТА+1 мМ спермидина, а затем инкубируя при 37 C в течение 15 минут. Затем все лунки объединяли и добавляли DAPI до конечной концентрации 3 мкM для проведения сортировки клеток с активацией флуоресценции (FACS). Используя сортировку с активацией флуоресценции, в каждую лунку нового 96-луночного планшета с сильным связыванием, содержащую 12 мкл буфера для поперечного сшивания (11 мкл EB (Qiagen), 0,5 мкл 1% SDS, 0,5 мкл 20 мг/мл протеиназы K (Promega), распределяли ограниченное количество клеток (варьируемое в ходе эксперимента в зависимости от желаемой ожидаемой частоты дублетов). Поперечные сшивки обращали посредством инкубации планшетов при 65°C в течение 13,5 ч на блоке ПЦР. Затем использовали ПЦР для добавления второго цикла специфичных для лунок штрихкодов как к хеш-меткам, так и к тагментированному хроматину. В каждую лунку добавляли по 3,65 мкл Tween-20, 1,25 мкл индексированного праймера Nextera P5, 1,25 мкл индексированного праймера Nextera P7 и 18,125 мкл мастер-микса NBNext High-Fidelity 2X Master Mix. Условия ПЦР были следующими: 72°C в течение 5 мин, 98°C в течение 30 с (повторить три следующих стадии 23 раза: 98°C в течение 10 с, 63°C в течение 30 с, 72°C в течение 1 мин), 72°C в течение 5 мин, удерживать при 4°C.
[00435] Затем амплифицированные библиотеки из каждой лунки объединяли и концентрировали с помощью набора реагентов для очистки и концентрирования Zymogen (с использованием 5-кратного буфера для связывания ДНК), после чего элюировали в 100 мкл EB. Для отделения хеш-библиотеки от библиотеки ATAC концентрированную объединенную библиотеку пропускали через 1% агарозный гель и очищали на геле. Библиотека хешей выглядела как полоса размером 199 п. н., а библиотека ATAC была вырезана из области ~ 200-3000 п. н. Экстракцию из геля проводили с помощью набора для ПЦР и экстракции геля Nucleospin и элюировали в 50 мкл (библиотека ATAC) или 25 мкл (библиотека хешей).
[00436] Совместный захват хешей и получение профилей ATAC при трехуровневом sci-ATAC
[00437] Меченные хешами ядра размораживали на льду, проверяли целостность ядер, подсчитывали и при необходимости дополнительно доводили до 5 миллионов ядер/мл. Затем по 10 мкл ядер распределяли в лунки 96-луночного планшета с сильным связыванием. Чтобы все ядра захватили молекулы хешей, в каждую лунку добавляли 2 мкл 25 мкМ «захватных» одноцепочечных ДНК-олигонуклеотидов (5’-TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGNNNNNNNNTT+TTT+TTT+TTT+TTT+TTT+TTT+TTT+TTT+TTT+TVN-3’) («N» обозначает уникальный молекулярный индекс (UMI), «+T» обозначает наличие запертых нуклеиновых кислот, которые повышают температуру плавления захватного олигонуклеотида, отожженного с хеширующим олигонуклеотидом). Затем планшет инкубировали при 55°C в течение 5 минут и немедленно возвращали на лед на 5 минут. Затем в каждую лунку добавляли 35,5 мкл реакционной смеси для Tn5 (25 мкл 2X буфера для тагментации, 8,25 мкл 1x DPBS, 0,5 мкл 1% дигитонина, 0,5 мкл 10% TWEEN-20, 1,25 мкл воды). Наконец, в каждую лунку добавляли по 2,5 мкл фермента Nextera Tn5 (конечный объем=50 мкл). Планшет запечатывали клейкой лентой и центрифугировали при 500 x g в течение 30 с. Затем выполняли тагментацию путем инкубации планшета при 55°C в течение 30 мин. Тагментацию останавливали, добавляя во все лунки по 50 мкл ледяного раствора 40 мМ ЭДТА+1 мМ спермидина, а затем инкубировали при 37°C в течение 15 минут. Содержимое лунок объединяли с помощью наконечников с широким каналом, и тагментированные ядра осаждали в течение 5 минут при 4°C при 500 x g и удаляли супернатант. Ядра осторожно ресуспендировали в 500 мкл 40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2+0,1% Tween-20 и снова центрифугировали при 500 x g в течение 5 минут при 4°C. Супернатант аспирировали и осадок повторно суспендировали в 110 мкл 40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2+0,1% Tween-20.
[00438] Затем 5’-концы тагментированного хроматина и захваченных хеширующих олигонуклеотидов внутри зафиксированных ядер фосфорилировали посредством реакции, опосредованной полинуклеотидкиназой (PNK). 110 мкл ресуспендированных ядер смешивали с 55 мкл 10x буфера T4PNK (NEB), 55 мкл rATP (NEB), 110 мкл безнуклеазной воды, 220 мкл T4PNK (NEB) и распределяли по 5 мкл реакционной смеси в каждую лунку 96-луночного планшета. Затем планшет запечатывали, центрифугировали при 500 x g в течение 30 секунд, а затем инкубировали при 37°C в течение 30 минут.
[00439] После киназных реакций первый уровень индексирования достигали путем специфического прикрепления индексированных олигонуклеотидов к «N7-меченной» стороне тагментированного хроматина и захваченных молекул хешей. N7-специфическое лигирование выполняли путем добавления 10 мкл 2X раствора T7-лигазного буфера, 0,18 мкл 1000 мкM N7-накладочного (шплинтового) олигонуклеотида (5'-CACGAGACGACAAGT-3' (SEQ ID NO: 3)), 1,12 мкл безнуклеазной воды, 2,5 мкл ДНК-лигазы Т7, 1,2 мкл 50 мкM N7-олигонуклеотида (5'-CAGCACGGCGAGACTNNNNNNNNNNGACTTGTC-3' (SEQ ID NO: 4), где «N» обозначают специфичный для лунки индекс) непосредственно во все лунки, содержащие киназную реакционную смесь (конечный объем лунки=20 мкл). Затем планшет запечатывали, центрифугировали при 500 x g в течение 30 с и проводили лигирование при 25°C в течение 1 ч. Лигирование останавливали путем добавления в каждую лунку 20 мкл ледяного раствора 40 мМ ЭДТА+1 мМ спермидина и инкубации при 37°C в течение 15 мин. Используя наконечники с широким каналом, содержимое лунок объединяли в коническую пробирку объемом 15 мл и увеличивали объем путем добавления трех объемов 40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2+ 0,1% Tween-20. Ядра осаждали в течение 10 мин при 500 x g и 4°C и ресуспендировали в 550 мкл 40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2+0,1% Tween-20.
[00440] Второй уровень индексирования выполняли посредством лигирования индексированных олигонуклеотидов с фосфорилированной «N5-меченной» стороны тагментированного хроматина и захваченных молекул хешей. Таким образом, по 5 мкл объединенных ресуспендированных ядер распределяли по всем лункам нового 96-луночного планшета. Затем проводили вторую реакцию лигирования, добавляя во все лунки по 10 мкл 2X раствора T7 лигазного буфера, 0,18 мкл 1000 мкM N5-накладочного (шплинтового) олигонуклеотида (5’-GCCGACGACTGATTA-3’ (SEQ ID NO:5)), 1,12 мкл безнуклеазной воды, 2,5 мкл ДНК-лигазы Т7, 1,2 мкл 50 мкM N5-олигонуклеотида (5’-CACCGCACGAGAGGTNNNNNNNNNNGTAATCAG-3’ (SEQ ID NO: 6), где «N» представляют собой специфический для лунки индекс) (конечный объем лунки=20 мкл). Затем планшет запечатывали, центрифугировали при 500 x g в течение 30 с и проводили лигирование при 25°C в течение 1 ч. Лигирование останавливали путем добавления в каждую лунку 20 мкл ледяного раствора 40 мМ ЭДТА+1 мМ спермидина и инкубации при 37°C в течение 15 мин. Используя наконечники с широким каналом, содержимое лунок объединяли в коническую пробирку объемом 15 мл и увеличивали объем путем добавления трех объемов 40 мМ TrisHCl, 40 мМ NaCl, 12 мМ MgCl2+ 0,1% Tween-20. Ядра осаждали в течение 10 мин при 500 x G и 4°C и осторожно ресуспендировали в 500 мкл буфера EB (Qiagen). Для распределения в лунки для ПЦР ядра либо окрашивали DAPI (конечная концентрация 3 мкМ), либо сортировкой вносили в лунки 96-луночного планшета (185 ядер на лунку), содержащего буфер для устранения поперечных сшивок (11 мкл буфера EB (Qiagen), 0,5 мкл протеиназы K (Roche), 0,5 мкл 1% SDS), либо подсчитывали и доводили до концентрации 1850/мл. По 10 мкл разведенных ядер распределяли по всем лункам 96-луночного планшета и добавляли по 1 мкл буфера EB (Qiagen) с 0,5 мкл протеиназы K (Qiagen) и 0,5 мкл 1% SDS для устранения поперечных сшивок. Затем планшеты запечатывали, центрифугировали при 500 x g в течение 30 секунд и поперечные сшивки удаляли инкубацией планшетов при 65°C в течение 16 часов.
[00441] Третий уровень индексирования осуществляли с помощью ПЦР. Следовательно, смесь для ПЦР содержит 2,5 мкл 25 мкМ праймера P7 (5’-CAAGCAGAAGACGGCATACGAGATNNNNNNNNNNCAGCACGGCGAGACT-3’ (SEQ ID NO:7)), 2,5 мкл 25 мкМ праймера P5 (5’-AATGATACGGCGACCACCGAGATCTACACNNNNNNNNNNCACCGCACGAGAGGT-3’ (SEQ ID NO:8), 25 мкл мастер-микса NBNext High Fidelity 2X Master Mix, 7 мкл воды, 1 мкл 20 мг/мл BSA (NEB). Важно отметить, что каждая лунка получила уникальную специфичную для лунки комбинацию праймеров P7 и P5. Условия ПЦР были следующими: 72°C в течение 5 мин, 98°C в течение 30 с (повторить три следующих стадии 20 раз: 98°C в течение 10 с, 63°C в течение 30 с, 72°C в течение 1 мин) 72°C в течение 5 мин, выдерживать при 4°C
[00442] Затем амплифицированные библиотеки из каждой лунки объединяли и концентрировали с помощью набора реагентов для очистки и концентрирования Zymogen (с использованием 5-кратного буфера для связывания ДНК), после чего элюировали в 100 мкл EB.
[00443] Процитированные источники для примера 2
[00444] Cao, Junyue, Jonathan S. Packer, Vijay Ramani, Darren A. Cusanovich, Chau Huynh, Riza Daza, Xiaojie Qiu, et al. 2017. “Comprehensive Single-Cell Transcriptional Profiling of a Multicellular Organism.” Science 357 (6352): 661-67.
[00445] Cusanovich, Darren A., Riza Daza, Andrew Adey, Hannah A. Pliner, Lena Christiansen, Kevin L. Gunderson, Frank J. Steemers, Cole Trapnell, and Jay Shendure. 2015. “Multiplex Single Cell Profiling of Chromatin Accessibility by Combinatorial Cellular Indexing.” Science 348 (6237): 910-14.
[00446] Srivatsan, Sanjay R., José L. McFaline-Figueroa, Vijay Ramani, Lauren Saunders, Junyue Cao, Jonathan Packer, Hannah A. Pliner, et al. 2020. “Massively Multiplex Chemical Transcriptomics at Single-Cell Resolution.” Science 367 (6473): 45-51.
[00447] Takahashi, Kazutoshi, and Shinya Yamanaka. 2016. “A Decade of Transcription Factor-Mediated Reprogramming to Pluripotency.” Nature Reviews. Molecular Cell Biology 17 (3): 183-93.
[00448] Пример 3
[00449] Стратегия мечения ядер обеспечивает объективный стандарт для нормализации транскриптомов одиночных клеток
[00450] Реферат
[00451] Несмотря на то что данные секвенирования РНК одиночных клеток изменили наше понимание биологии, они могут страдать от разрозненности и высокого уровня технического шума, часто препятствуя возможности извлекать биологически значимую информацию. В настоящем документе описан простой, но универсальный способ мечения отдельных ядер немодифицированными одноцепочечными ДНК-олигонуклеотидами, которые захватываются в ходе секвенирования транскриптома одиночных ядер. Когда ядра помечены «лесенкой» отдельных олигонуклеотидов, присутствующих в разных известных концентрациях, мы можем получить количества чтений каждого олигонуклеотида, пропорциональное его концентрации в лесенке, вместе с транскриптомом внутри каждого ядра. Используя лесенку количеств олигонуклеотидов, охватывающую три порядка величины, можно оценить долю выпадений транскриптов. Более того, мы показываем, что, используя количество лесенок в каждой клетке в качестве внешнего стандарта, мы можем оценить и устранить технический шум межклеточных вариаций экспрессии генов, значительно улучшая анализ дифференциальной экспрессии. Наконец, посредством химического ингибирования удлинения при транскрипции мы показываем, что нормализация количества транскриптов в каждой клетке, основанная на соответствующих количествах в олигонуклеотидных лесенках, позволяет обнаруживать глобальные направленные изменения в экспрессии генов, которые в ином случае были бы упущены.
[00452] Способы
[00453] Клеточная культура
[00454] Клетки A549 и HEK293T культивировали в среде DMEM, содержащей 10% эмбриональной бычьей сыворотки и 1% пенициллина и стрептомицина, при 37 C с 5% CO2.
[00455] Для эксперимента по изучению временной динамики при введении флавопиридола клетки HEK293T высевали на 6-луночный культуральный планшет с плотностью 4×105 клеток на лунку. Для эксперимента по временной динамике совместного введения ингибитора HDAC и дексаметазона клетки A549 высевали на 96-луночный культуральный планшет с плотностью 2,5×104 клеток на лунку.
[00456] Обработка лекарственными средствами
[00457] Клетки выращивали в течение 24 часов, после чего их высевали на планшеты для культивирования клеток. Для эксперимента по временной динамике введения флавопиридола в каждую из 6 лунок добавляли по 0,9 мкл 1 мМ раствора флавопиридола для получения конечной концентрации 300 нМ. Для экспериментов для исследования динамики при обработке HDACi и совместной обработке HDACi и дексаметазоном в каждую лунку 96-луночного планшета добавляли по 1 мкл 1 мкМ раствора абексиностата или прациностата для получения конечной концентрации 10 мкМ. За два часа до обработки HDACi добавляли 1 мкл 100 мкМ раствора дексаметазона. В качестве носителя для обработки флавопиридолом и HDACi использовали ДМСО, а в качестве носителя для обработки дексаметазоном использовали этанол.
[00458] Конструкция хеш-лесенки
[00459] Захват хеш-лесенки ядрами определяется факторами, включающими в себя, без ограничений, обработку образца и глубину секвенирования. В случае клеточных линий млекопитающих мы обнаружили, что эффективность захвата хешей очень низкая. Эмпирическим путем мы определили, что лесенка должен быть сконструирована таким образом, чтобы на в расчете ядро захватывалось около 6 миллионов молекул хешей (предполагая, что каждое ядро поглощает одинаковое количество хеш-молекул в растворе) для получения медианного количества UMI хешей, равного 1000-5000. Для пилотного эксперимента использовали хеш-лесенку, состоящую из 8 разных хеширующих олигонуклеотидов в диапазоне количества от 0,1-12,8 аттомоль на ядро. Для остальных экспериментов использовали хеш-лесенку, состоящую из 48 разных хеширующих олигонуклеотидов в диапазоне от 0,025 зептомоль до 2 аттомоль на ядро.
[00460] Сбор клеток, выделение ядер и захват хеш-лесенки
[00461] Для сбора клеток среду удаляли, а клетки промывали DPBS и отделяли от планшета с помощью trypLE. Трипсинизацию trypLE гасили равным объемом ледяной среды. Клетки осаждали центрифугированием при 500 g в течение 5 минут, промывали ледяной DPBS и ресуспендировали в ледяной DPBS. Затем клетки подсчитывали на гемоцитометре с использованием 0,4% трипанового синего. Около 2 миллионов клеток осаждали центрифугированием при 500 g в течение 5 минут и ресуспендировали в 1 мл ледяного лизирующего буфера (10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 0,1% IGEPAL CA-630) с добавлением 1% ингибитора Superase RNA Inhibitor и соответствующего количества хеш-лесенки. После лизирования при осторожном пипетировании клетки фиксировали путем добавления 4 мл фиксирующего буфера (5% параформальдегида в 1,25X PBS) на льду в течение 20 мин. После фиксации клеток их промывали с помощью 1 мл буфера для суспендирования ядер (10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2, 1% ингибитора Superase RNA Inhibitor, 1% 0,2 мг/мл NEBNext BSA) и ресуспендировали в 100 мкл NSB.
[00462] Получение библиотек sci-RNA-seq2
[00463] Затем выделенные и хешированные ядра пермеабилизировали в буферном растворе для пермеабилизации (0,25% Triton-X в NSB) в течение 3 минут на льду, а затем центрифугировали. После еще одной отмывки в NSB получали двухуровневые библиотеки sci-RNA-seq, как описано (пример 1, Cao et al., Nature, 2019, 566:496-502). Вкратце, ядра осаждали при 500 x g в течение 5 минут и ресуспендировали в 100 мкл NSB. Количество клеток получали путем окрашивания ядер 0,4% трипановым синим (Sigma-Aldrich) и подсчитывали с использованием гемоцитометра. Затем наносили по 5000 ядер в 2 мкл NSB и 0,25 мкл смеси 10 мМ дНТФ (Thermo Fisher Scientific, кат. № R0193) в лунки 96-луночного планшета с полуюбкой twin.tec LoBind (Fisher Scientific, кат. № 0030129512), после чего в каждую лунку добавляли 1 мкл имеющего уникальный индекс олиго-dT (25 мкМ), инкубировали при 55 C в течение 5 минут и помещали на лед. Затем в каждую лунку добавляли по 1,75 мкл смеси для обратной транскрипции (1 мкл буфера Superscript IV для синтеза первой цепи, 0,25 мкл 100 мМ DTT, 0,25 мкл Superscript IV и 0,25 мкл рекомбинантного ингибитора рибонуклеазы RNAseOUT), инкубировали при 55 C в течение 10 минут и помещали на лед. Для остановки реакции в каждую лунку добавляли по 5 мкл стоп-раствора (40 мМ ЭДТА и 1 мМ спермидина). Лунки объединяли, используя наконечники с широким каналом, ядра переносили в пробирку для проточной цитометрии через фильтровальный колпачок 0,35 мкм и добавляли DAPI до конечной концентрации 3 мкМ. Затем объединенные ядра сортировали на сортировщике клеток FACS Aria II в количестве 25-50 клеток на лунку в 96-луночные планшеты LoBind, содержащие по 5 мкл буфера EB (Qiagen). После сортировки в каждую лунку добавляли по 0,75 мкл смеси для второй цепи (0,5 мкл буфера для синтеза второй цепи мРНК и 0,25 мкл фермента для синтеза второй цепи мРНК, New England Biolabs), синтез второй цепи проводили при 16 C в течение 150 минут. Тагментацию выполняли путем добавления 6 мкл смеси для тагментации (0,02 мкл специализированного фермента TDE1 в 6 мкл 2x буфера Nextera TD, Illumina) и планшеты инкубировали в течение 5 минут при 55 C. Реакцию прекращали добавлением 12 мкл ДНК-связывающего буфера (Zymo) и инкубировали в течение 5 минут при комнатной температуре. В каждую лунку добавляли по 36 мкл гранул Ampure XP, очищали ДНК с использованием стандартного протокола Ampure XP (Beckman Coulter) с элюированием 17 мкл буфера EB и переносили ДНК в новый 96-луночный планшет LoBind. Для проведения ПЦР в каждую лунку добавляли по 2 мкл индексированного P5, 2 мкл индексированного P7 и 20 мкл мастер-микса NEBNext High-Fidelity (New England Biolabs) и выполняли ПЦР следующим образом. 72 C в течение 5 минут, 98 C в течение 30 секунд и 19 циклов при 98 C в течение 10 секунд, 66 C в течение 30 секунд и 72 C в течение 1 минуты с последующим конечным удлинением при 72 C в течение 5 минут. После ПЦР все лунки объединяли, концентрировали с использованием набора DNA clean and concentrator kit (Zymo) и очищали посредством чистящего реагента 0,8X Ampure XP. Конечные концентрации библиотек определяли с помощью Qubit (Invitrogen), библиотеки визуализировали с использованием системы TapeStation D1000 DNA Screen tape (Agilent) и библиотеки секвенировали на Nextseq 500 (Illumina) с использованием высокопроизводительного набора из 75 циклов (чтение 1: 18 циклов, чтение 2: 52 цикла, индекс 1: 10 циклов и индекс 2: 10 циклов).
[00464] Результаты
[00465] В экспериментах sci-RNA-seq в качестве контрольных образцов можно использовать хеш-лесенки
[00466] Основываясь на нашей технологии «хеширования» [1], задали вопрос, можно ли использовать лесенку хеширующих олигонуклеотидов, охватывающую широкий диапазон концентраций, для мечения ядер и использовать в качестве внешних контролей в экспериментах sci-RNA-seq (Фиг. 43A). Мы полагаем, что посредством мечения каждого ядра смесью различных хеширующих олигонуклеотидов с диапазонами концентраций, отражающими численность эндогенных транскриптов мРНК, мы смогли бы контролировать технический шум и использовать лесенку в качестве посредника для количественного определения абсолютной численности транскриптов в одиночных клетках.
[00467] Для проверки нашей гипотезы мы разработали лесенку, содержащую восемь разных хеширующих олигонуклеотидов с теоретическим количеством в диапазоне от 0,1 до 12,8 аттомоль на ядро, и вводили ее в клетки HEK293T на стадии лизирования в ходе получения библиотеки sci-RNA-seq. Как и ожидалось, мы получали чтения как с эндогенных мРНК, так и с хеш-лесенки. Наблюдаемое количество уникальных молекулярных идентификаторов (UMI) хеширующих олигонуклеотидов глобально отражало относительную численность каждого хеширующего олигонуклеотида в лесенке (Фиг. 43B). Если рассмотреть количество UMI хеш-лесенки в отдельных клетках, то 1806 из 1937 клеток с чтениями по меньшей мере одной хеш-молекулы (93%) имели чтения всех восьми хеш-молекул и демонстрировали сильную корреляцию между ожидаемым и наблюдаемым количеством хешей (Фиг. 43C, правый график). Мы также идентифицировали клетки с низкой эффективностью захвата хешей (Фиг. 43C, левый график), которая, вероятно, отражает вариабельность при обработке образцов в экспериментах sci-RNA-seq.
[00468] Нормализация на основе хеш-лесенки позволяет обнаруживать глобальные изменения в уровнях транскриптов в одиночных клетках.
[00469] Стандартные подходы к нормализации для данных scRNA-seq масштабируют значения экспрессии генов в клетке на размерный коэффициент, пропорциональный общему количеству РНК в клетке. Поскольку данные по экспрессии преобразуются в показатели относительной численности, они не могут зарегистрировать потенциальные глобальные сдвиги численности транскриптов, например, в ответ на подавление транскрипции. Таким образом, мы задали вопрос, можно ли использовать нашу хеш-лесенку для точного определения глобальных изменений в уровнях транскрипции, вызванных обработкой ингибитором CDK флавопиридолом.
[00470] Флавопиридол представляет собой ингибитор циклин-зависимой киназы, которая, как известно, вызывает резкое снижение глобальных уровней транскрипции [2]. Используя количества выделенных в расчете на клетку олигонуклеотидов лесенки в качестве контроля нормализации для каждой клетки, мы попытались измерить изменения в экспрессии генов, обусловленные обработкой флавопиридолом в течение увеличивающихся периодов времени (Фиг. 44А). Как и ожидалось, клетки, подвергнутые воздействию ингибитора удлинения при транскрипции в течение наибольшего времени, демонстрировали наибольшее снижение выделенной РНК в расчете на клетку (Фиг. 44B).
[00471] Затем сравнили эффекты традиционных и основанных на хеш-лесенке подходов к нормализации в анализах дифференциальной экспрессии. При традиционном подходе к нормализации, основанном на общем размерном коэффициенте РНК, количество генов с повышенной экспрессией в ответ на обработку флавопиридолом было равно количеству генов с пониженной экспрессией, хотя известно, что флавопиридол прекращает транскрипцию, и это является показателем неправильной нормализации [3] (Фиг. 44C, левый график). Однако нормализация, основанная на количестве полученных хешей лесенки, позволила успешно получить большее количество генов с подавленной экспрессией, которые, по известным данным, имеют сниженную экспрессию при обработке флавопиридолом, и уменьшить количество ложно идентифицированных генов с повышенной экспрессией [4] (Фиг. 44C, правый график). Оценки величины эффекта, рассчитанные с помощью подхода, основанного на использовании хеш-лесенки, были в среднем выше для подавленных генов и ниже для усиленных генов по сравнению с традиционным подходом, что дополнительно подчеркивает способность хеш-лесенки определять глобальные изменения в транскрипции, обусловленные флавопиридолом.
[00472] Процитированные источники для примера 3
[00473] [1] Srivatsan et al. Massively multiplex chemical transcriptomics at single-cell resolution. Science 367, 6473, 45-51 (2020)
[00474] [2] Kelland, L.R. Flavopiridol, the first cyclin-dependent kinase inhibitor to enter the clinic: current status. Expert Opin. Investig. Drugs 12, 2903-2911 (2000)
[00475] [3] Athanasiadou et al. A complete statistical model for calibration of RNA-seq counts using external spike-ins and maximum likelihood theory. PLoS Comput. Biol. 15, 3 (2019)
[00476] [4] Lü et al. Transcriptional signature of flavopiridol-induced tumor cell death. Mol. Cancer Ther. 7, 861-872 (2004).
[00477] Настоящее описание проиллюстрировано следующими примерами. Следует понимать, что конкретные примеры, материалы, количества и процедуры следует интерпретировать в широком смысле в соответствии с сущностью и объемом изложения, как описано в настоящем документе.
[00478] Полное описание всех патентов, заявок на патенты и публикаций, а также материалов, доступных в электронной форме (включая, например, публикации нуклеотидных последовательностей, например, в GenBank и RefSeq, а также публикации аминокислотных последовательностей в, например, SwissProt, PIR, PRF, PDB, а также трансляции аннотированных кодирующих областей в GenBank и RefSeq), процитированные в настоящем документе, полностью включены в настоящий документ путем ссылки. Дополнительные материалы, на которые даны ссылки в публикациях (такие как дополнительные таблицы, дополнительные фигуры, дополнительные материалы и методы и/или дополнительные экспериментальные данные), также полностью включены в настоящий документ путем ссылки. В случае несоответствия между описанием настоящей заявки и описанием (-ями) каких-либо других документов, включенных в настоящий документ путем ссылки, приоритет имеет описание настоящей заявки. Указанные выше подробное описание и примеры приведены только для обеспечения лучшего понимания. Их не следует рассматривать как наложение ненужных ограничений. Описание не ограничивается исключительно приведенными и описанными деталями, и в изложение также должны быть включены изменения, очевидные специалистам в данной области, в соответствии с пунктами формулы изобретения.
[00479] Если не указано иное, все числа, выражающие количества компонентов, молекулярных масс и т. п., используемые в описании и формуле изобретения, следует понимать как модифицированные во всех случаях термином «приблизительно» Соответственно, если не указано иное, числовые параметры, указанные в описании и формуле изобретения, являются приближенными значениями, которые могут варьироваться в зависимости от желаемых свойств, которые требуется получить посредством настоящего описания. По самой меньшей мере, но не в качестве попытки ограничить применение теории эквивалентов к объему формулы изобретения, необходимо рассматривать каждый числовой параметр по меньшей мере с учетом числа представленных значащих цифр и с применением стандартных методик округления.
[00480] Хотя числовые диапазоны и параметры, устанавливающие широкий объем объекта описания, являются приблизительными, числовые значения, указанные в конкретных примерах, представлены настолько точно, насколько это возможно. Однако все числовые значения по своей природе содержат определенные ошибки, неизбежно вытекающие из стандартного отклонения, выявленного в их соответствующих тестовых измерениях.
[00481] Все заголовки предназначены для удобства читателя и не должны использоваться для ограничения смысла текста, который следует за заголовком, если не указано иное.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Illumina, Inc.
University of Washington
Srivatsan, Sanjay
McFaline-Figueroa, Jose
Ramani, Vijay
Cao, Junyue
Booth, Gregory
Shendure, Jay
Trapnell, Cole
Steemers, Frank
<120> ВЫСОКОПРОИЗВОДИТЕЛЬНЫЕ БИБЛИОТЕКИ ИЗ ОДИНОЧНЫХ ЯДЕР И
ОДИНОЧНЫХ КЛЕТОК И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ИСПОЛЬЗОВАНИЯ
<130> IP-1815-PCT
<150> 62/812,853
<151> 2019-03-01
<160> 8
<170> PatentIn, версия 3.5
<210> 1
<211> 67
<212> ДНК
<213> искусственная
<220>
<223> хеширующий олигонуклеотид
<220>
<221> misc_feature
<222> (35)..(35)
<223> B представляет собой G, C или T
<400> 1
gtctcgtggg ctcggagatg tgtataagag acagbaaaaa aaaaaaaaaa aaaaaaaaaa 60
aaaaaaa 67
<210> 2
<211> 83
<212> ДНК
<213> искусственная
<220>
<223> одноцепочечный ДНК-олигонуклеотид
<220>
<221> misc_feature
<222> (34)..(51)
<223> n представляет собой a, c, g или t
<220>
<221> misc_feature
<222> (83)..(83)
<223> n представляет собой a, c, g или t
<400> 2
tcgtcggcag cgtcagatgt gtataagaga cagnnnnnnn nnnnnnnnnn nttttttttt 60
tttttttttt tttttttttt tvn 83
<210> 3
<211> 15
<212> ДНК
<213> искусственная
<220>
<223> шплинтовый олигонуклеотид
<400> 3
cacgagacga caagt 15
<210> 4
<211> 33
<212> ДНК
<213> искусственная
<220>
<223> Олигонуклеотид N7
<220>
<221> misc_feature
<222> (16)..(25)
<223> n представляет собой a, c, g или t
<400> 4
cagcacggcg agactnnnnn nnnnngactt gtc 33
<210> 5
<211> 15
<212> ДНК
<213> искусственная
<220>
<223> шплинтовый олигонуклеотид
<400> 5
gccgacgact gatta 15
<210> 6
<211> 33
<212> ДНК
<213> искусственная
<220>
<223> Олигонуклеотид N5
<220>
<221> misc_feature
<222> (16)..(25)
<223> n представляет собой a, c, g или t
<400> 6
caccgcacga gaggtnnnnn nnnnngtaat cag 33
<210> 7
<211> 49
<212> ДНК
<213> искусственная
<220>
<223> Праймер P7
<220>
<221> misc_feature
<222> (25)..(34)
<223> n представляет собой a, c, g или t
<400> 7
caagcagaag acggcatacg agatnnnnnn nnnncagcac ggcgagact 49
<210> 8
<211> 54
<212> ДНК
<213> искусственная
<220>
<223> Праймер P5
<220>
<221> misc_feature
<222> (30)..(39)
<223> n представляет собой a, c, g или t
<400> 8
aatgatacgg cgaccaccga gatctacacn nnnnnnnnnc accgcacgag aggt 54
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЕ БИБЛИОТЕКИ ОДИНОЧНЫХ КЛЕТОК И СПОСОБЫ ПОЛУЧЕНИЯ И ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2838946C2 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2744175C1 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2833615C2 |
| ПОЛНОГЕНОМНЫЕ БИБЛИОТЕКИ ОТДЕЛЬНЫХ КЛЕТОК ДЛЯ БИСУЛЬФИТНОГО СЕКВЕНИРОВАНИЯ | 2018 |
|
RU2770879C2 |
| КРУПНОМАСШТАБНЫЕ МОНОКЛЕТОЧНЫЕ БИБЛИОТЕКИ ТРАНСКРИПТОМОВ И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2019 |
|
RU2773318C2 |
| АНАЛИЗ МНОЖЕСТВА АНАЛИТОВ С ИСПОЛЬЗОВАНИЕМ ОДНОГО АНАЛИЗА | 2019 |
|
RU2824049C2 |
| СИСТЕМА АНАЛИЗА ДЛЯ ОРТОГОНАЛЬНОГО ДОСТУПА К БИОМОЛЕКУЛАМ И ИХ МЕЧЕНИЯ В КЛЕТОЧНЫХ КОМПАРТМЕНТАХ | 2017 |
|
RU2771892C2 |
| СПОСОБЫ ИНКАПСУЛИРОВАНИЯ ОДИНОЧНЫХ КЛЕТОК, ИНКАПСУЛИРОВАННЫЕ КЛЕТКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2793717C2 |
| СПОСОБЫ ИНКАПСУЛИРОВАНИЯ ОДИНОЧНЫХ КЛЕТОК, ИНКАПСУЛИРОВАННЫЕ КЛЕТКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2750567C2 |
| СПОСОБЫ И СРЕДСТВА ПОЛУЧЕНИЯ БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ | 2019 |
|
RU2815513C2 |


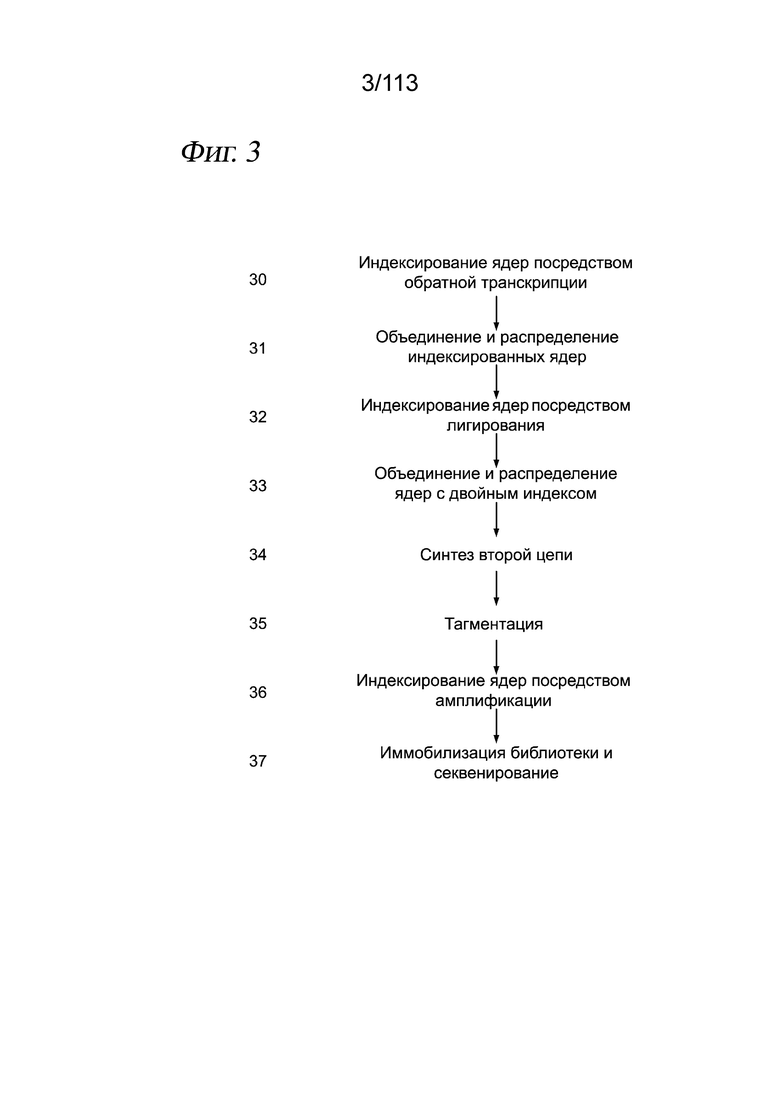

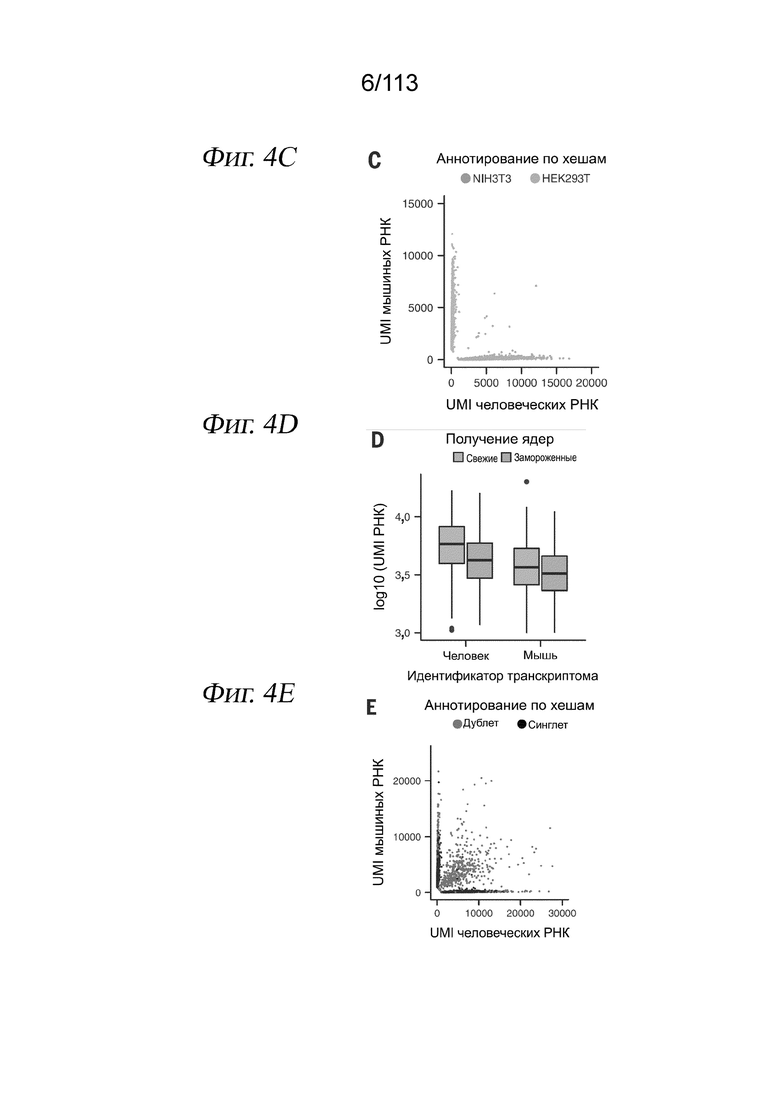
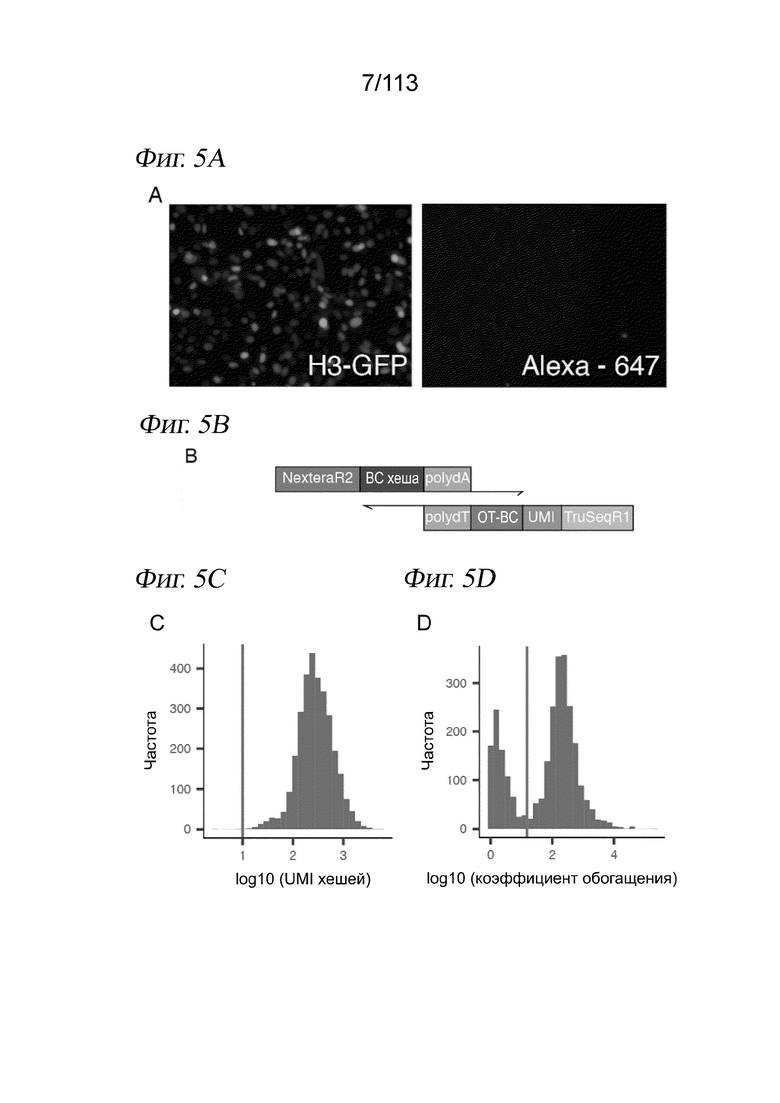
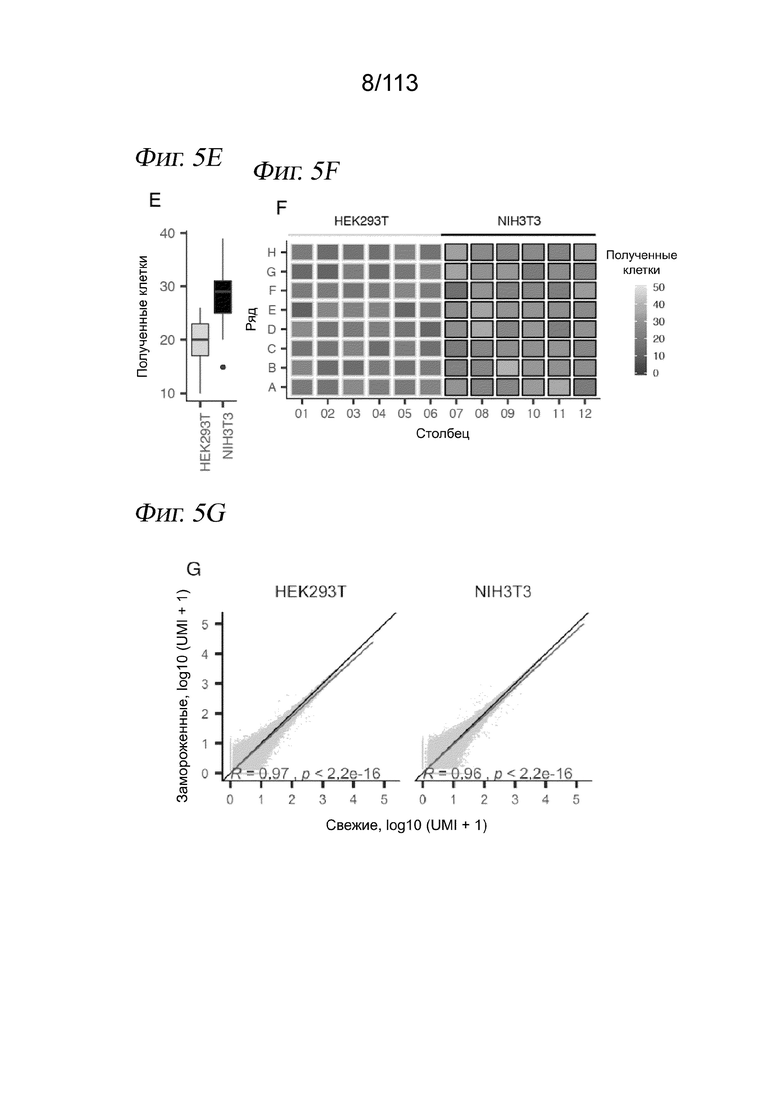
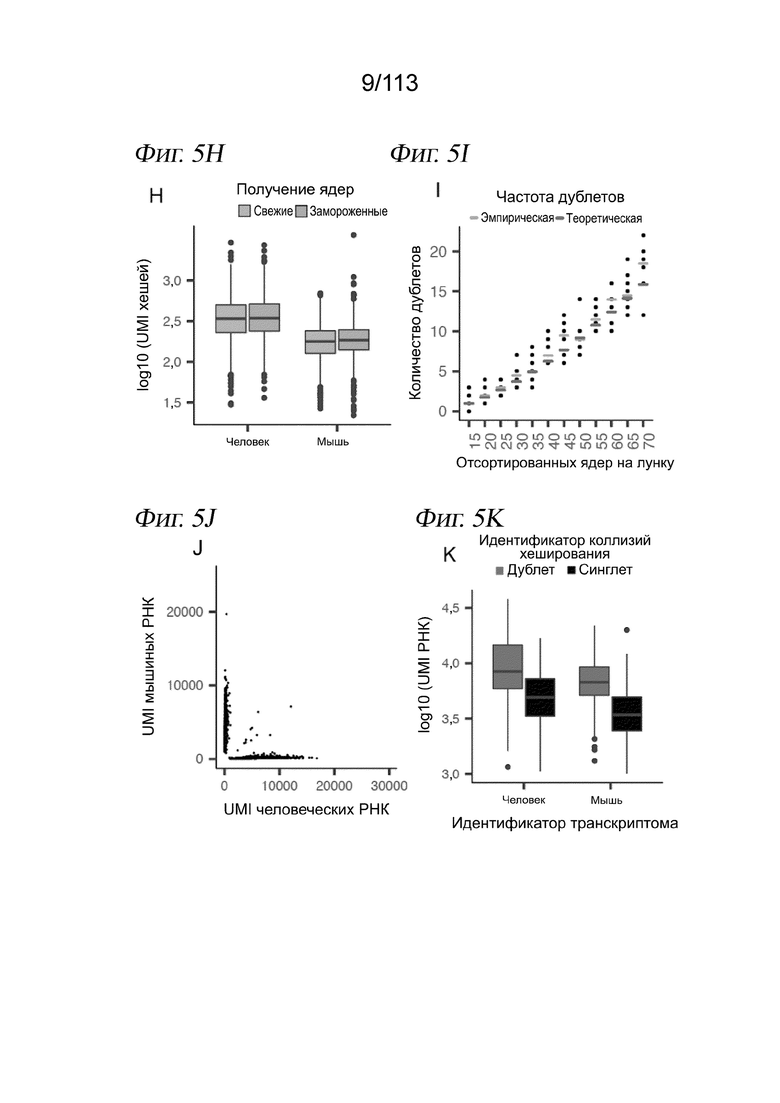
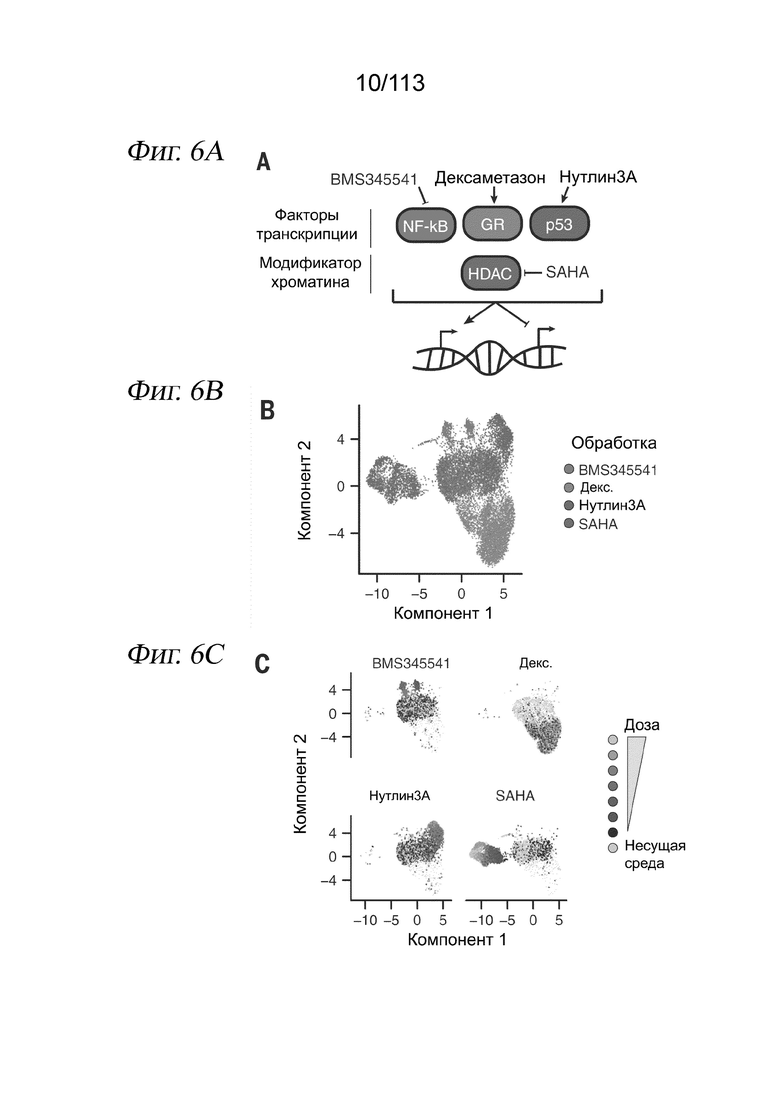
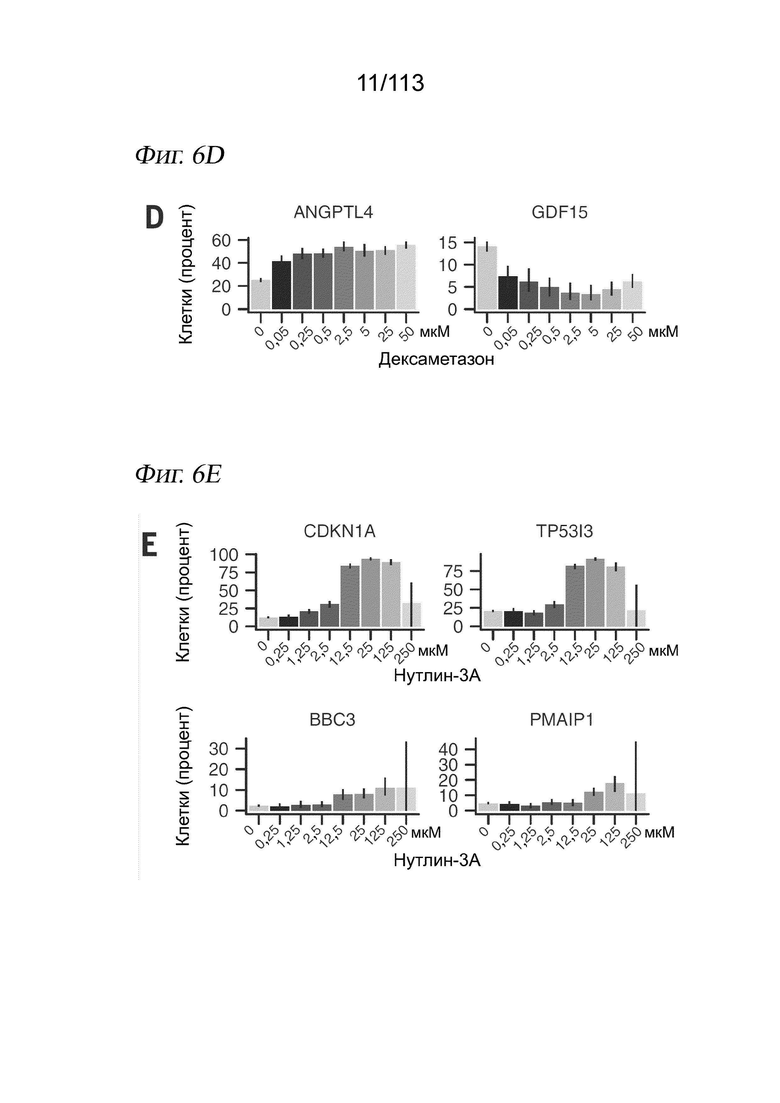
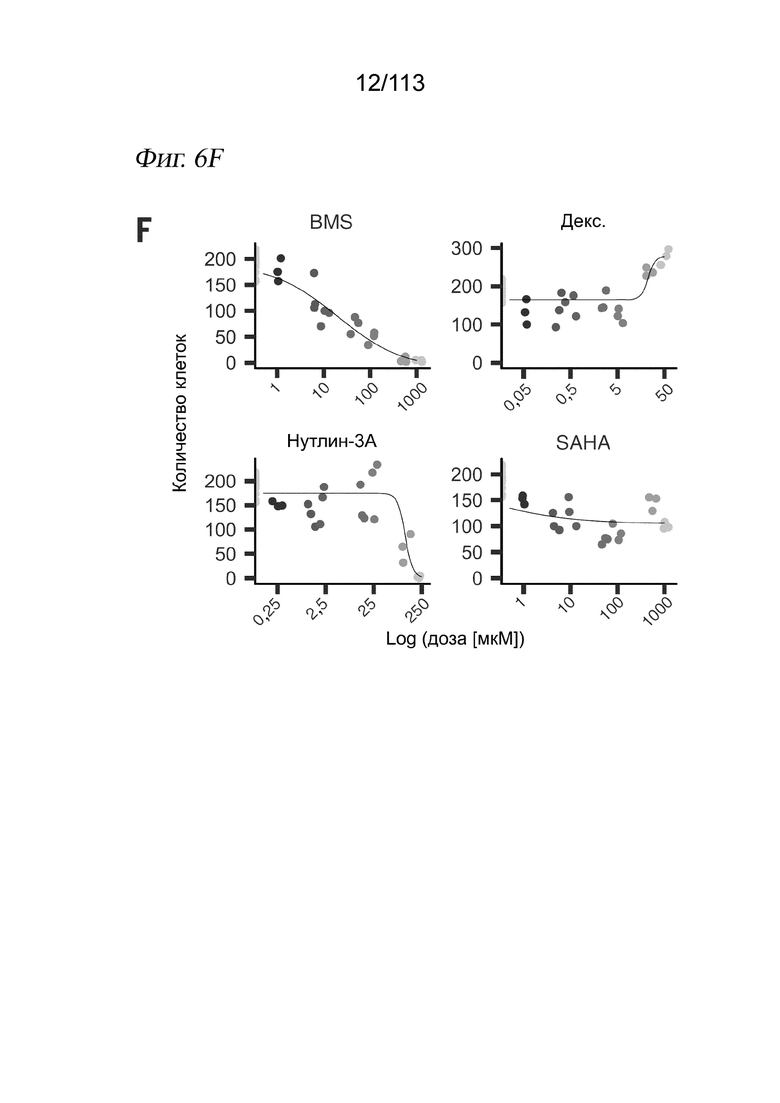
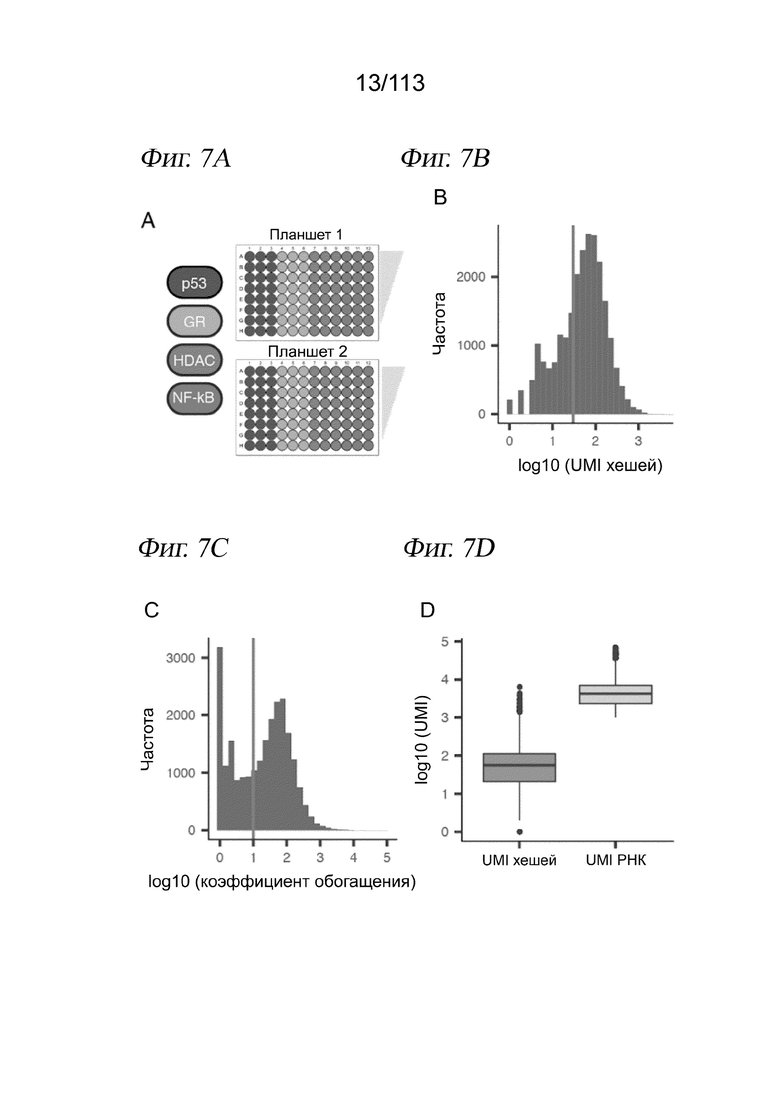
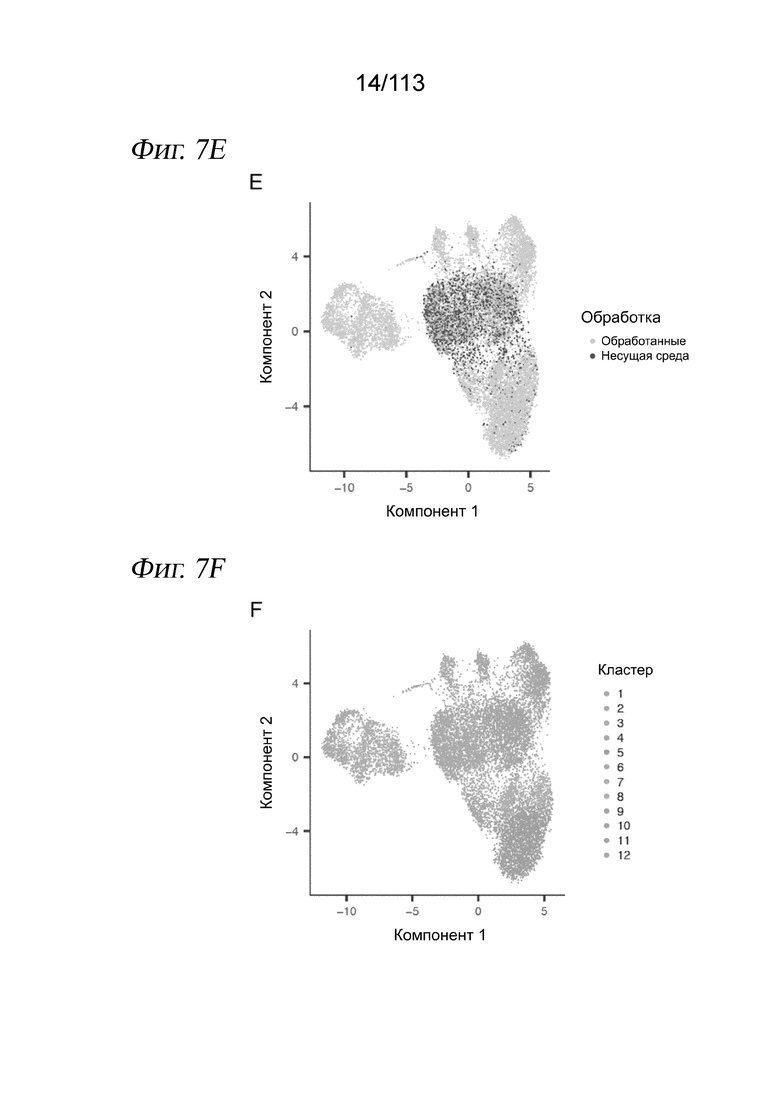
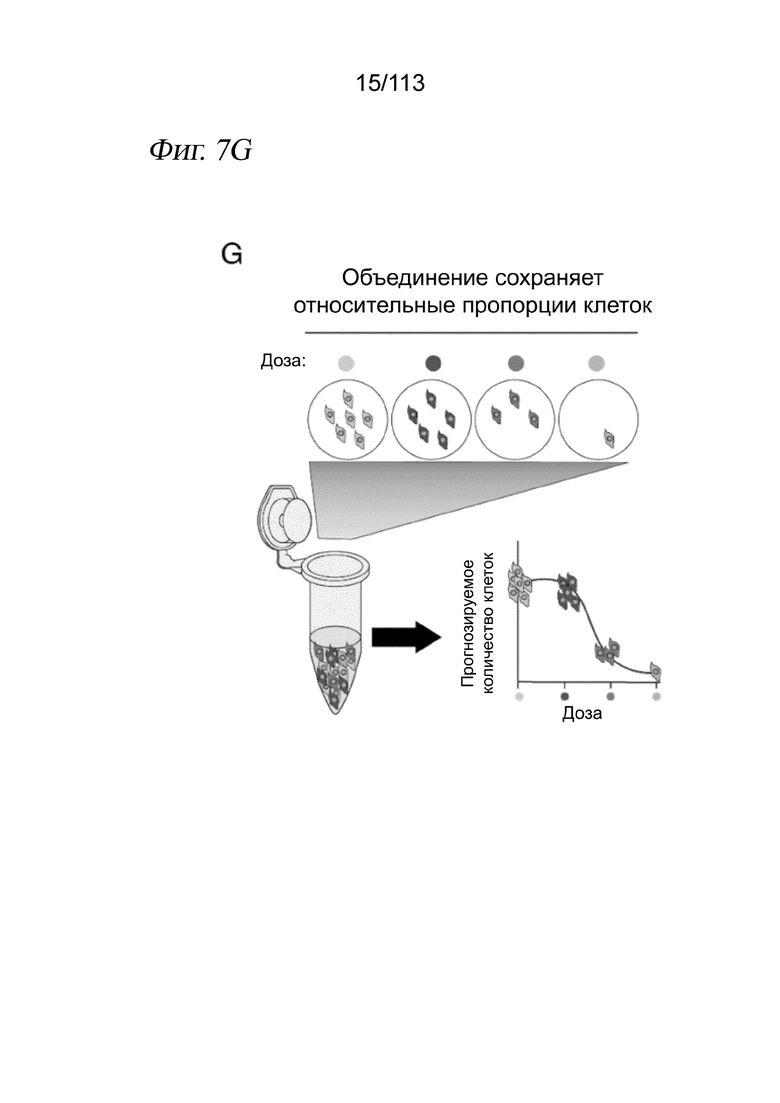
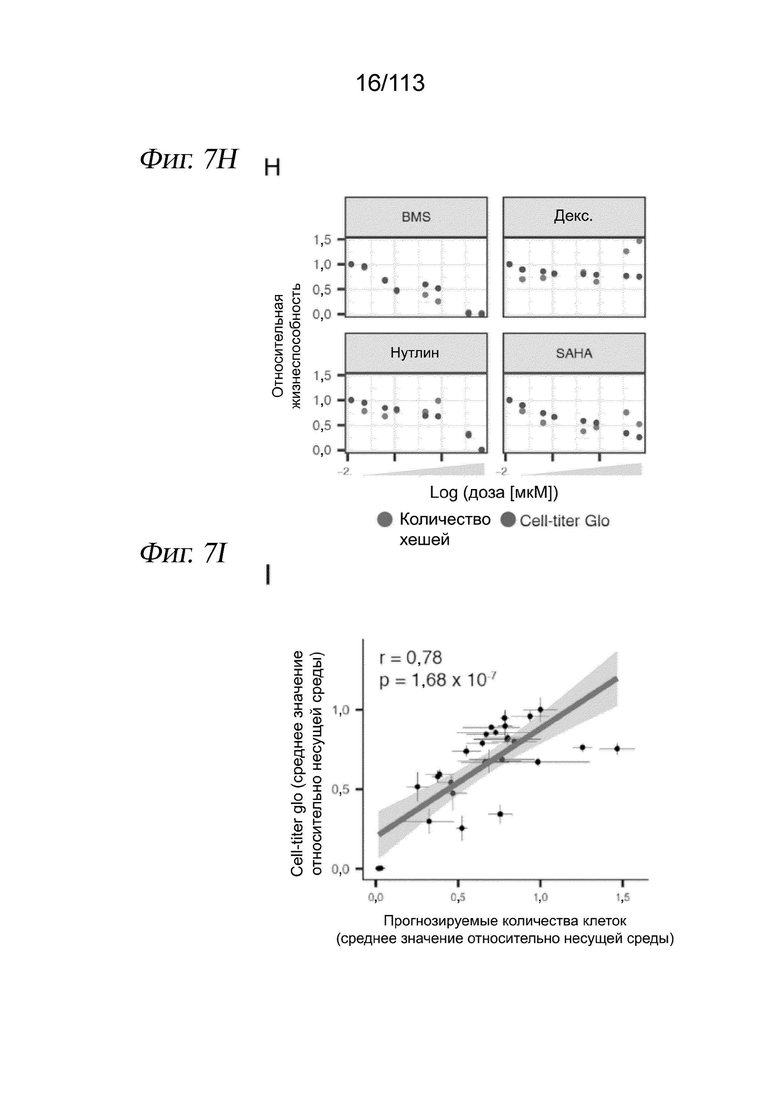
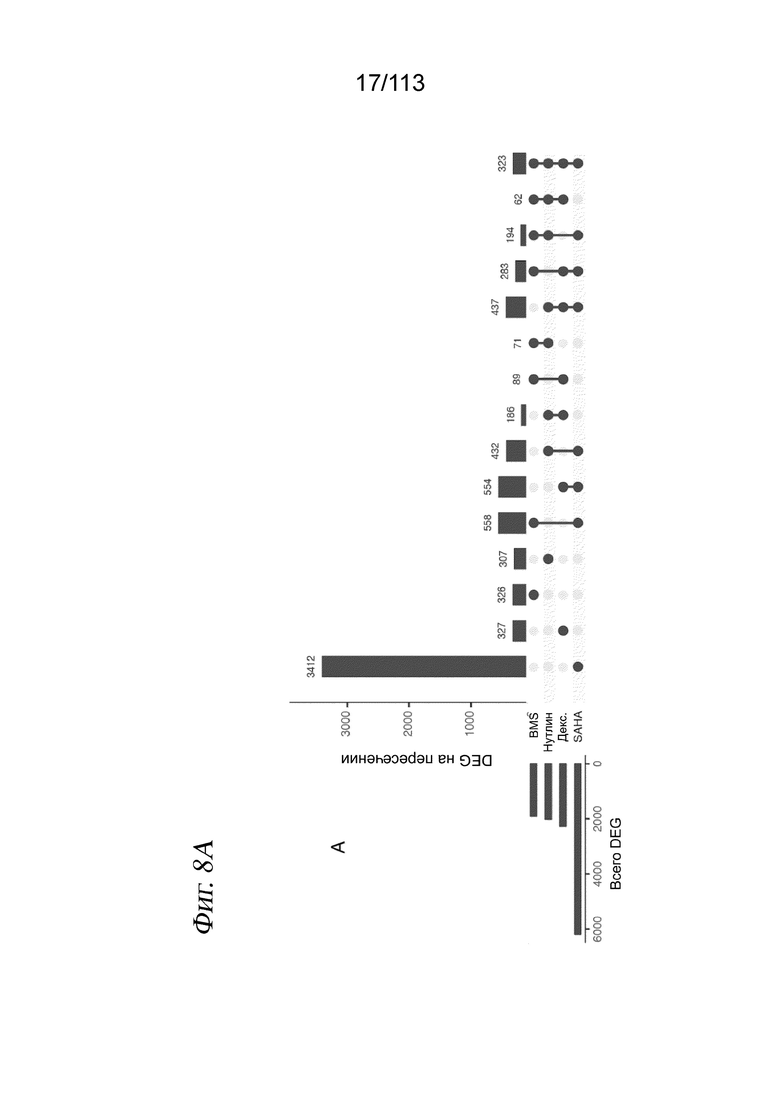
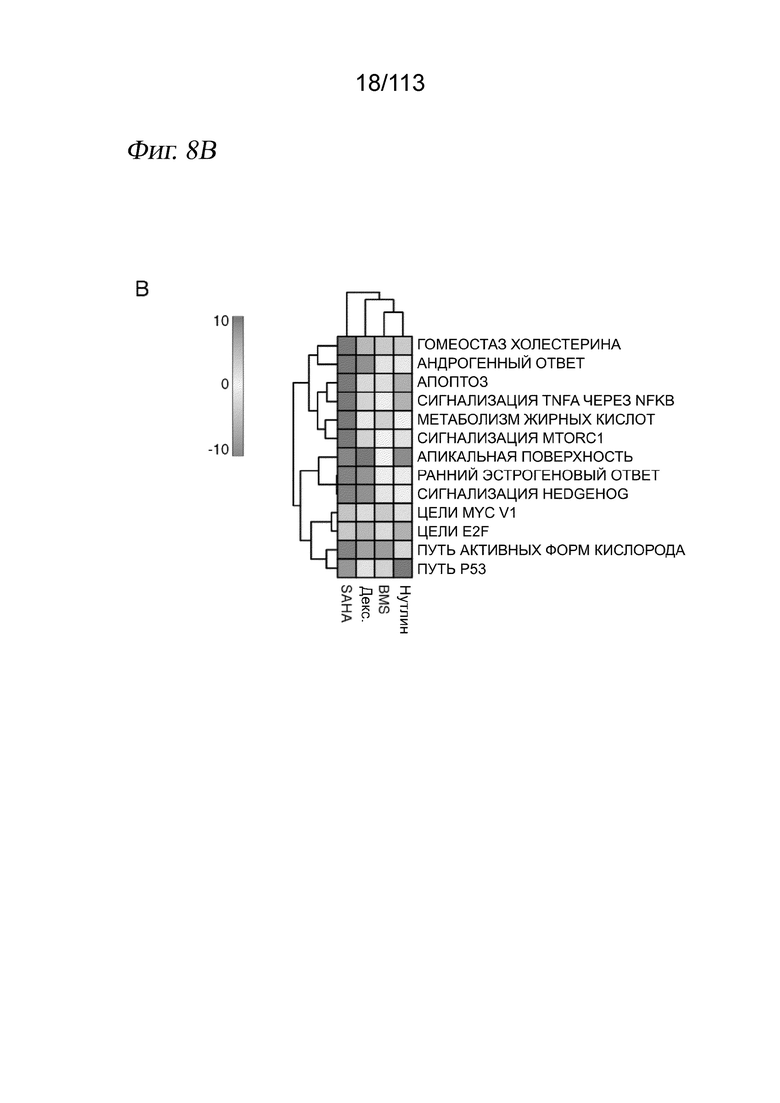
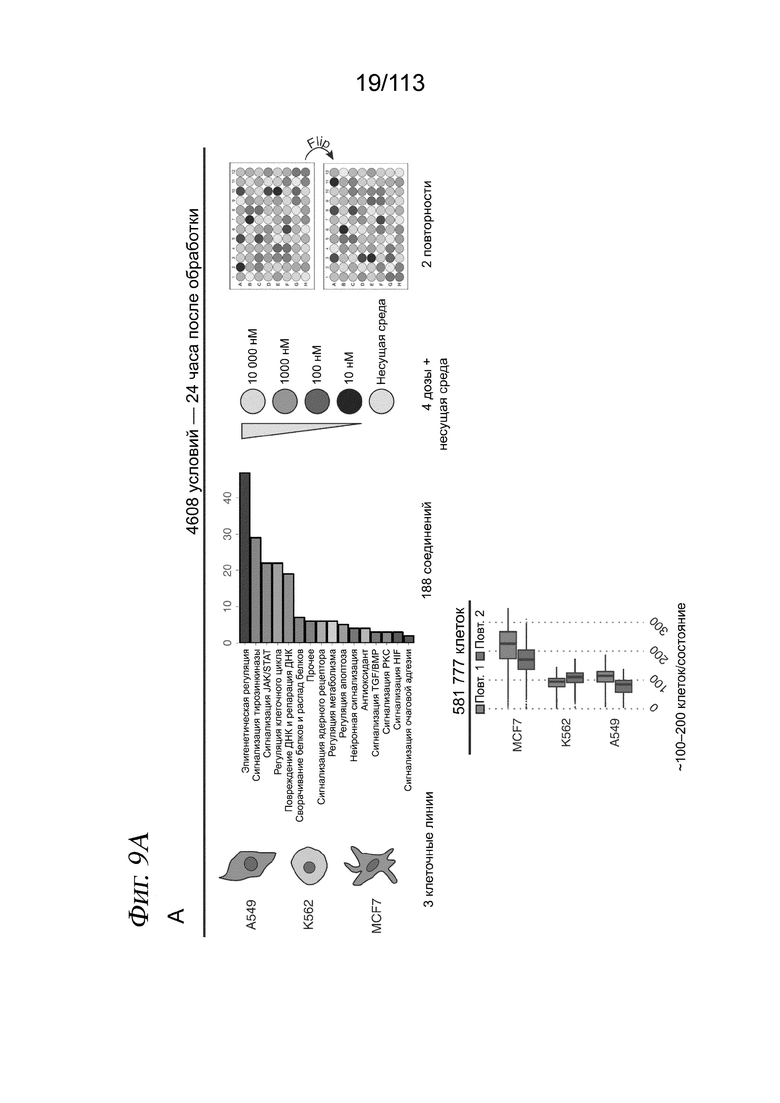
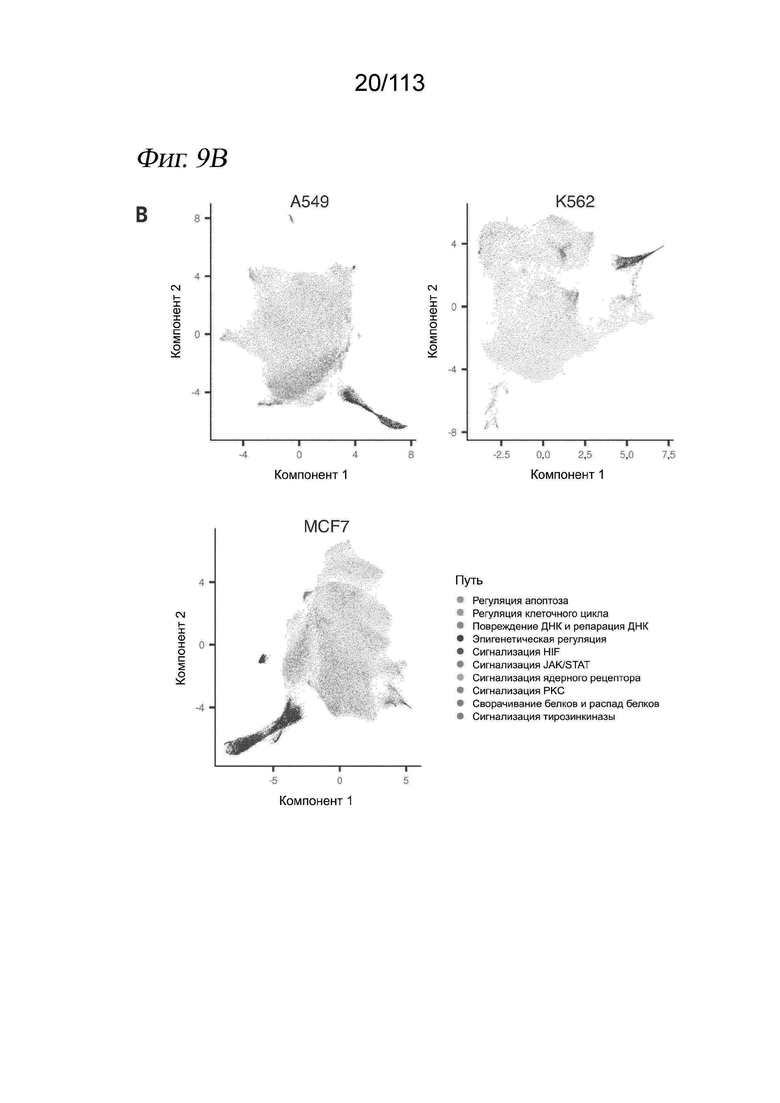

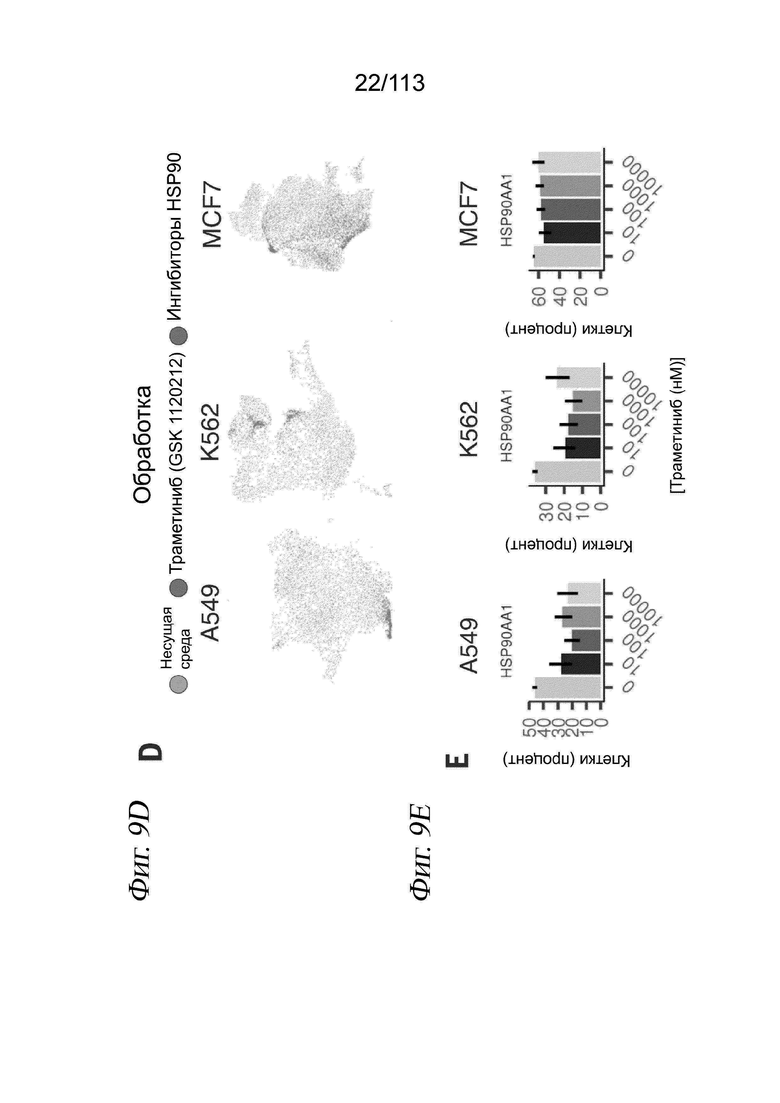

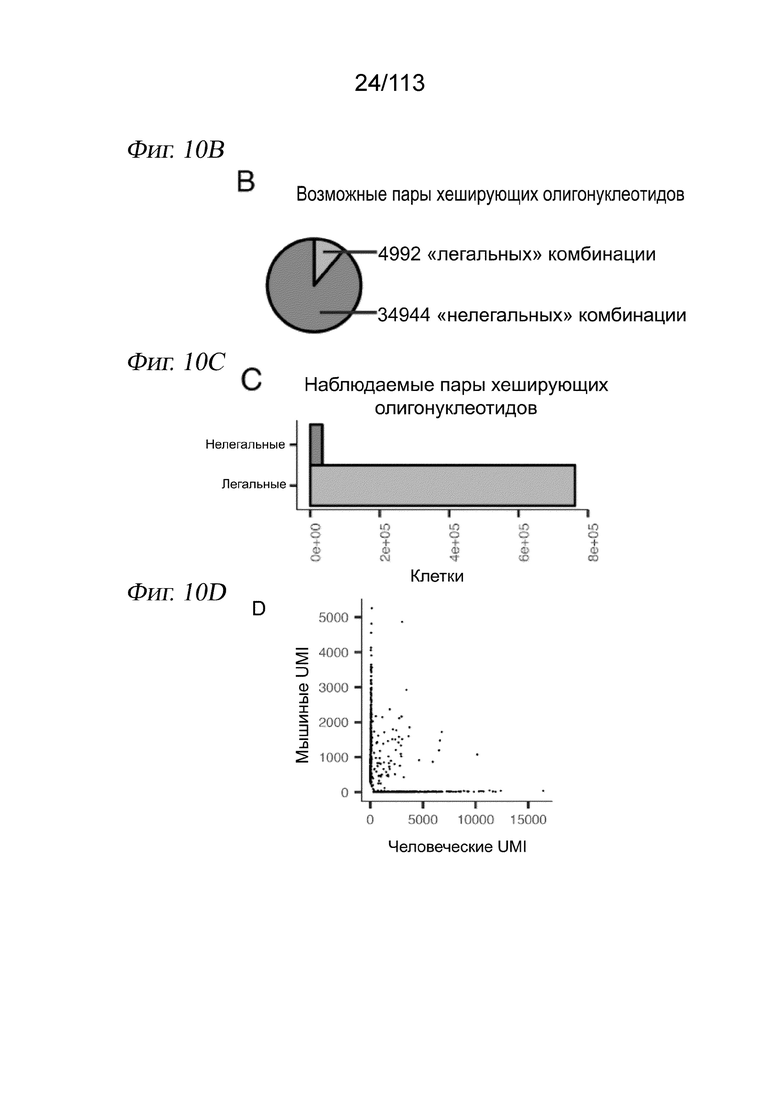
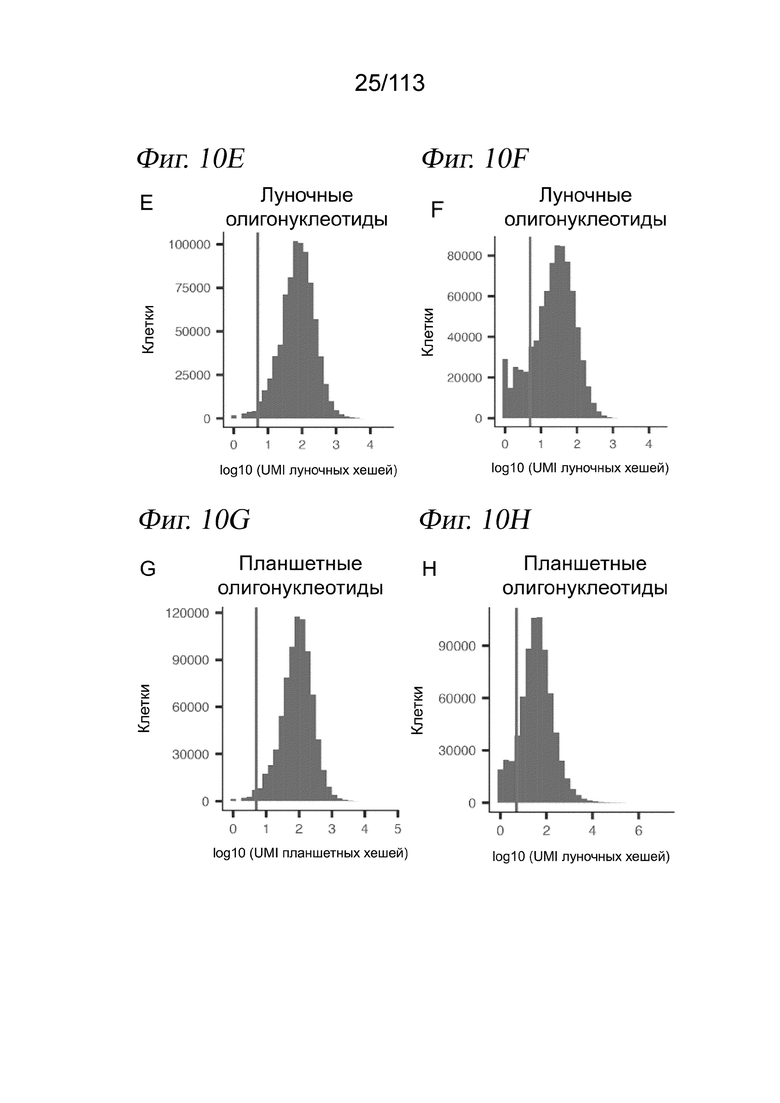
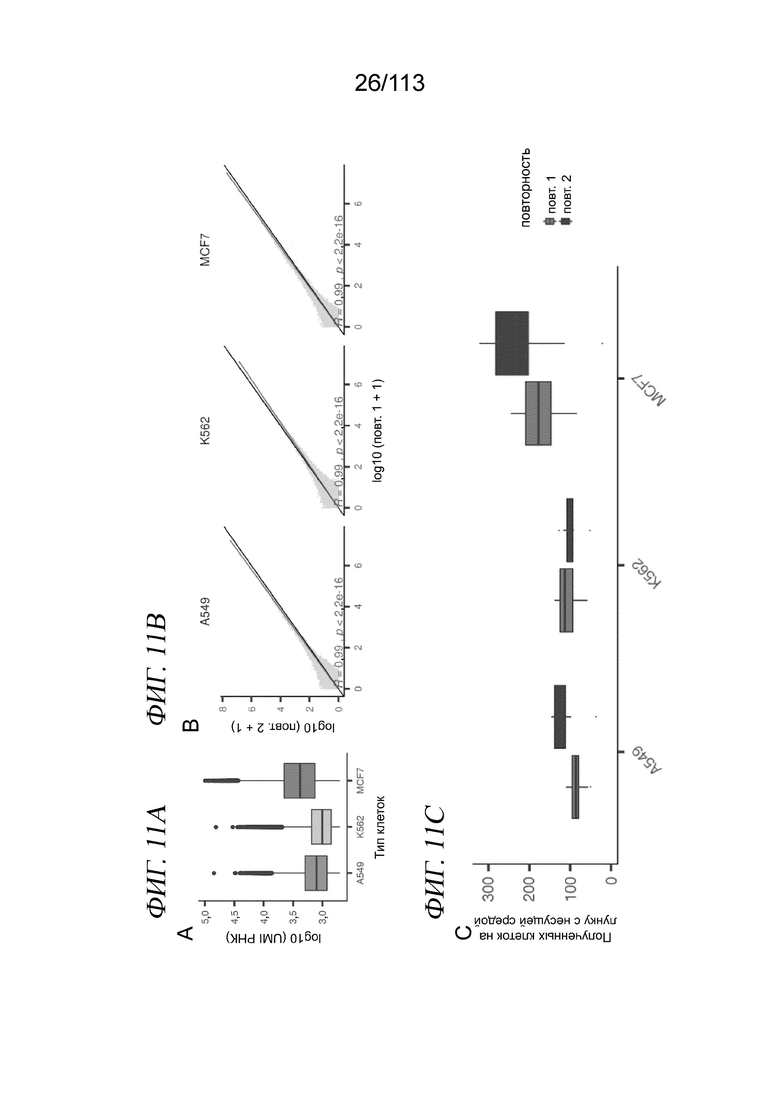




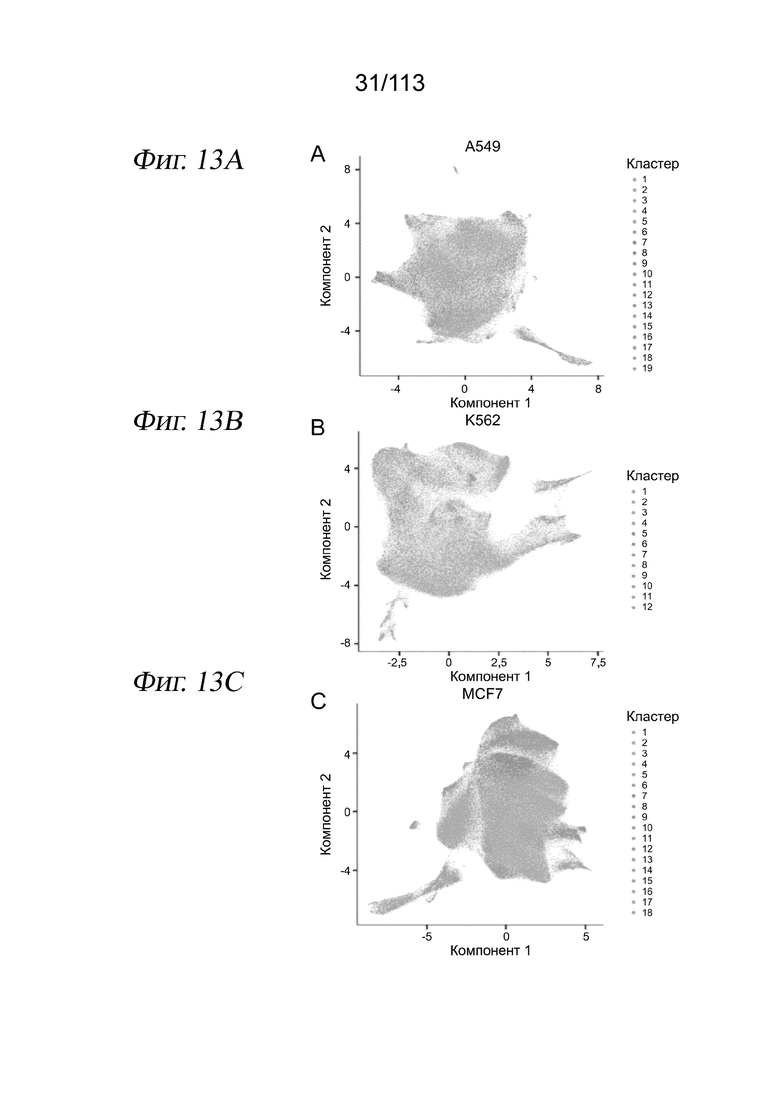
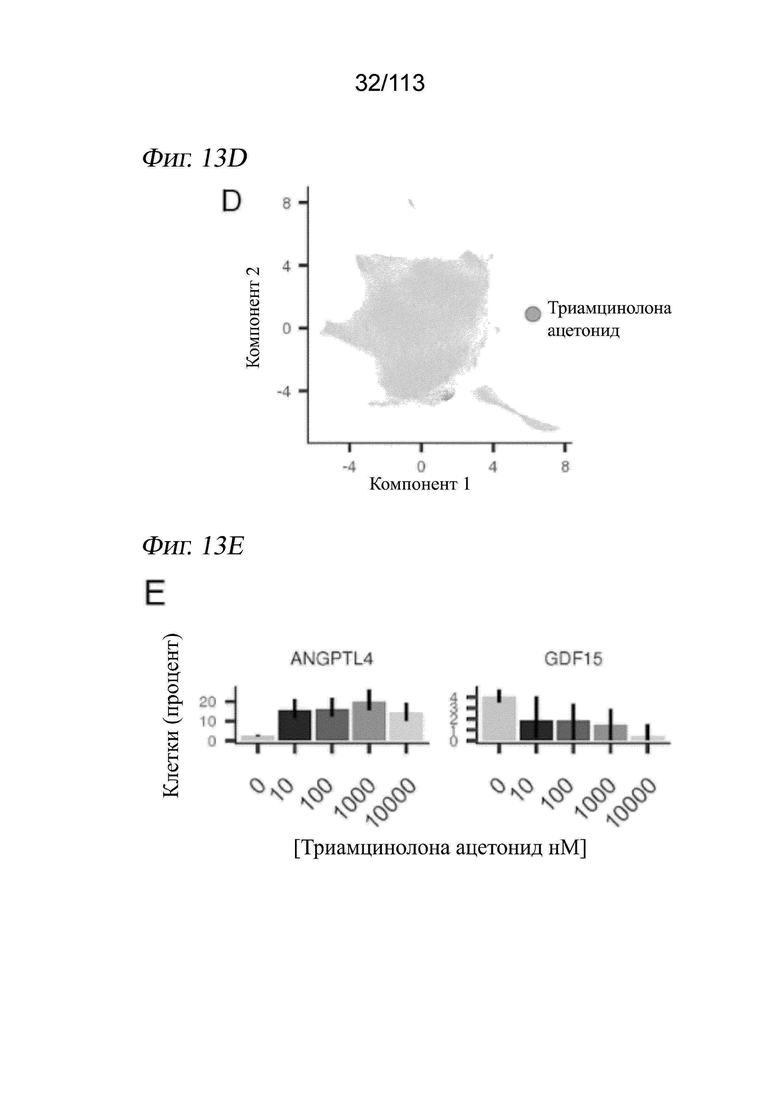
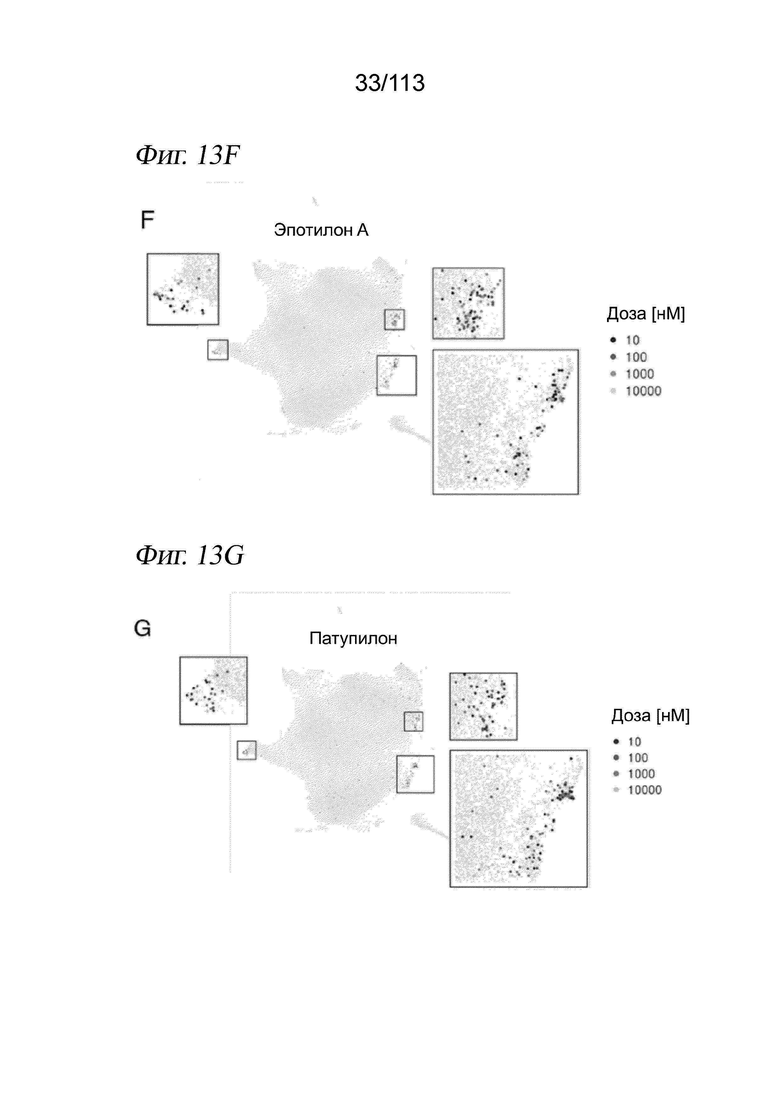
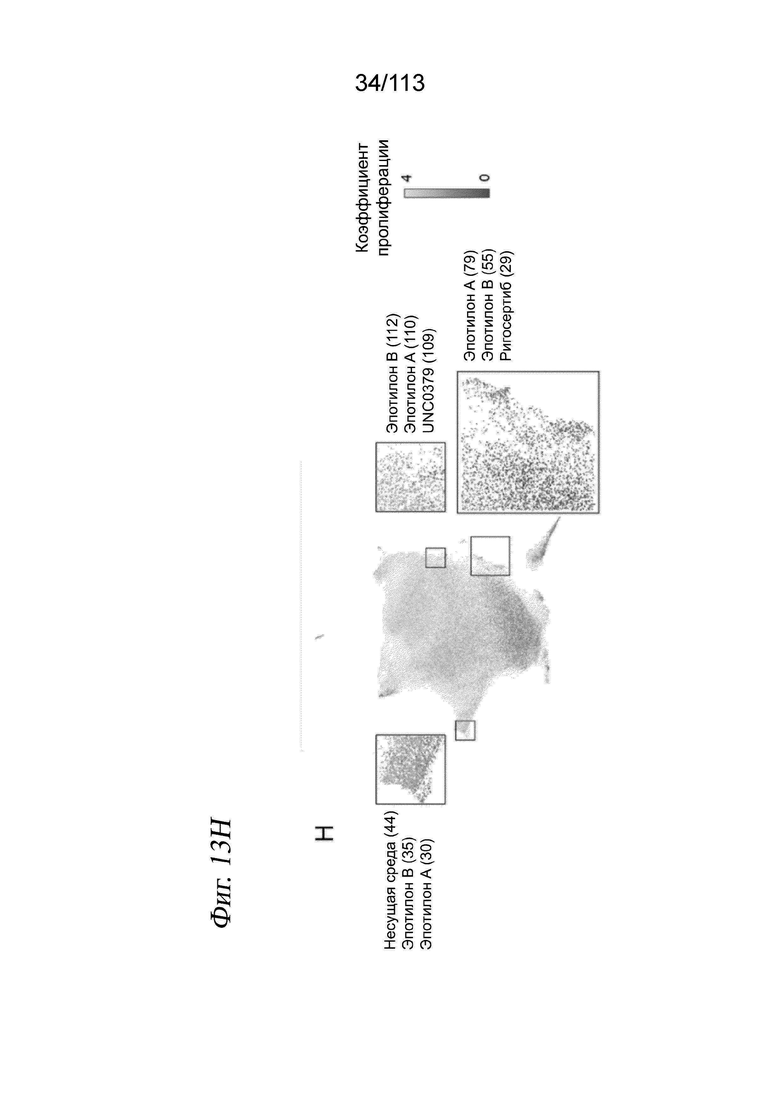
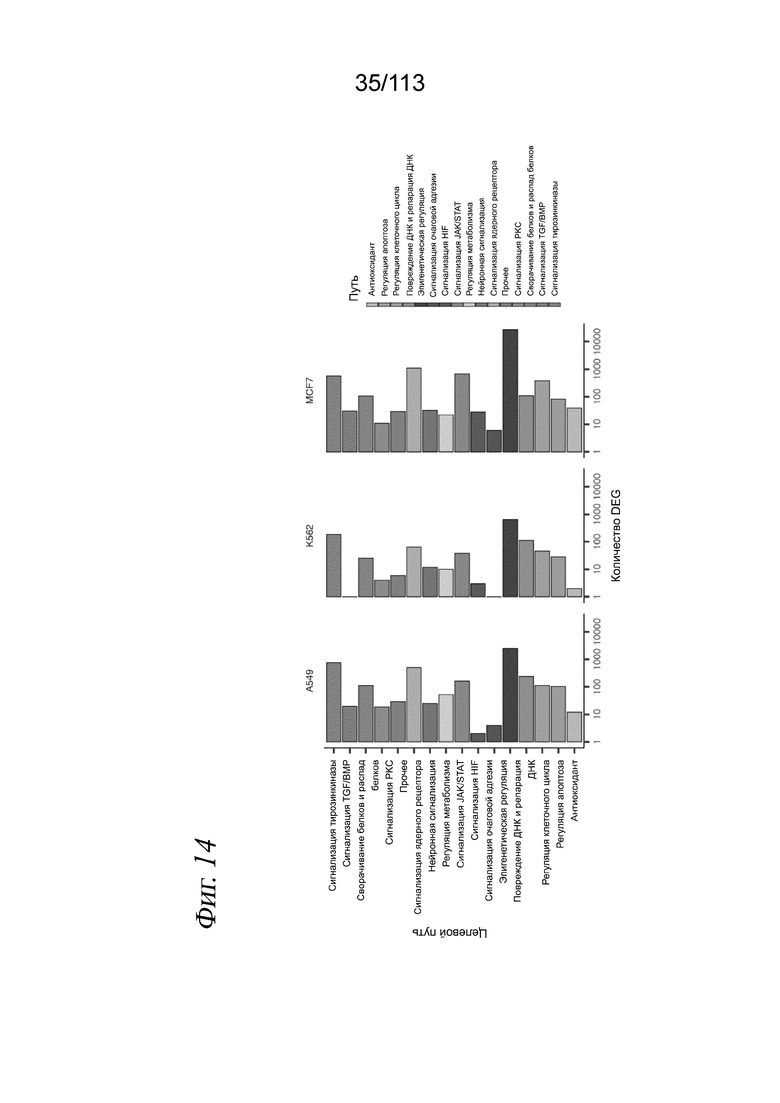
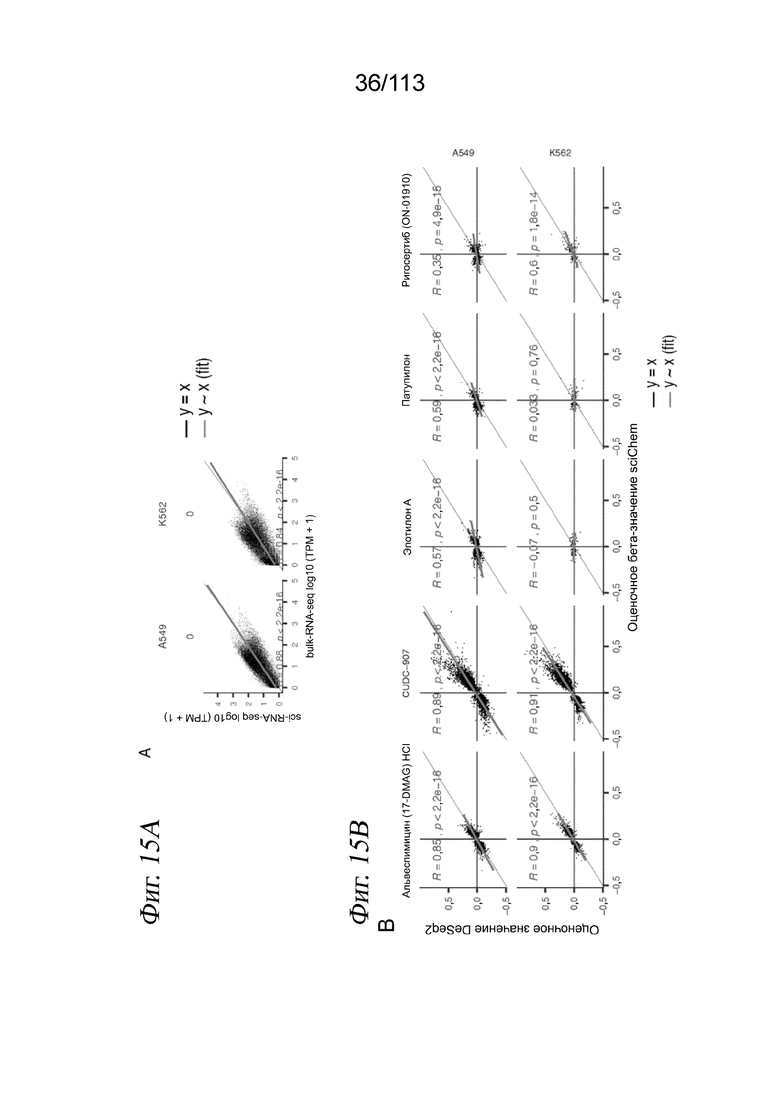
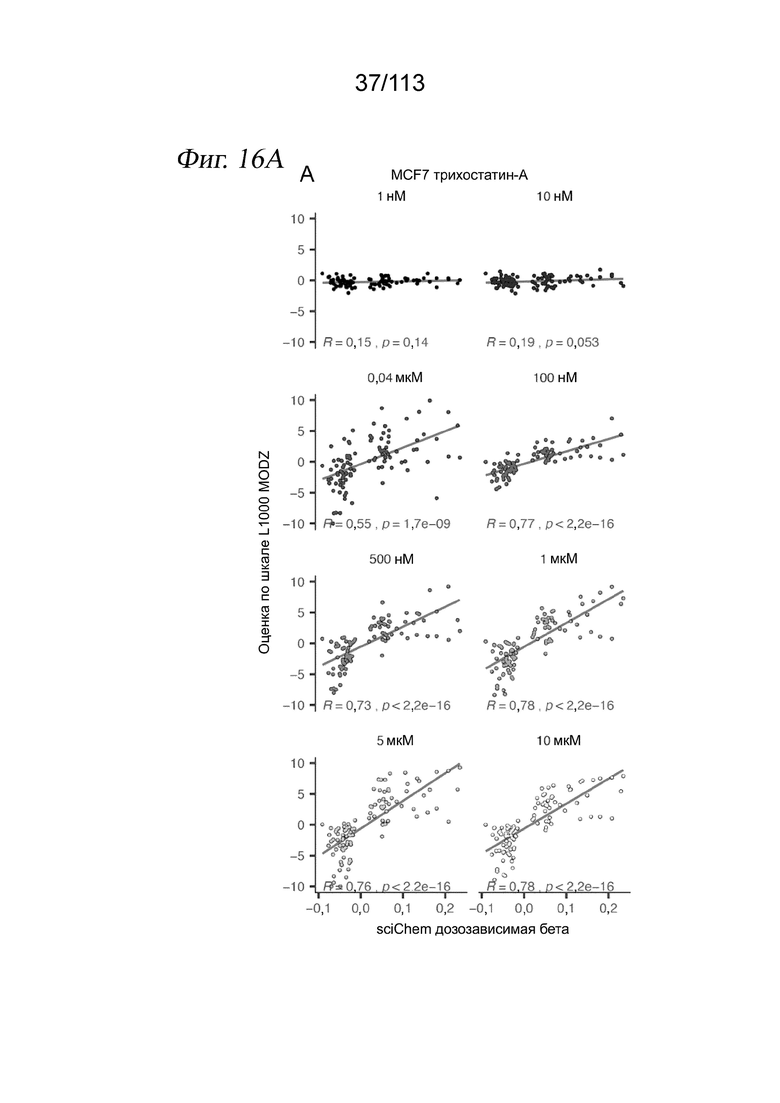
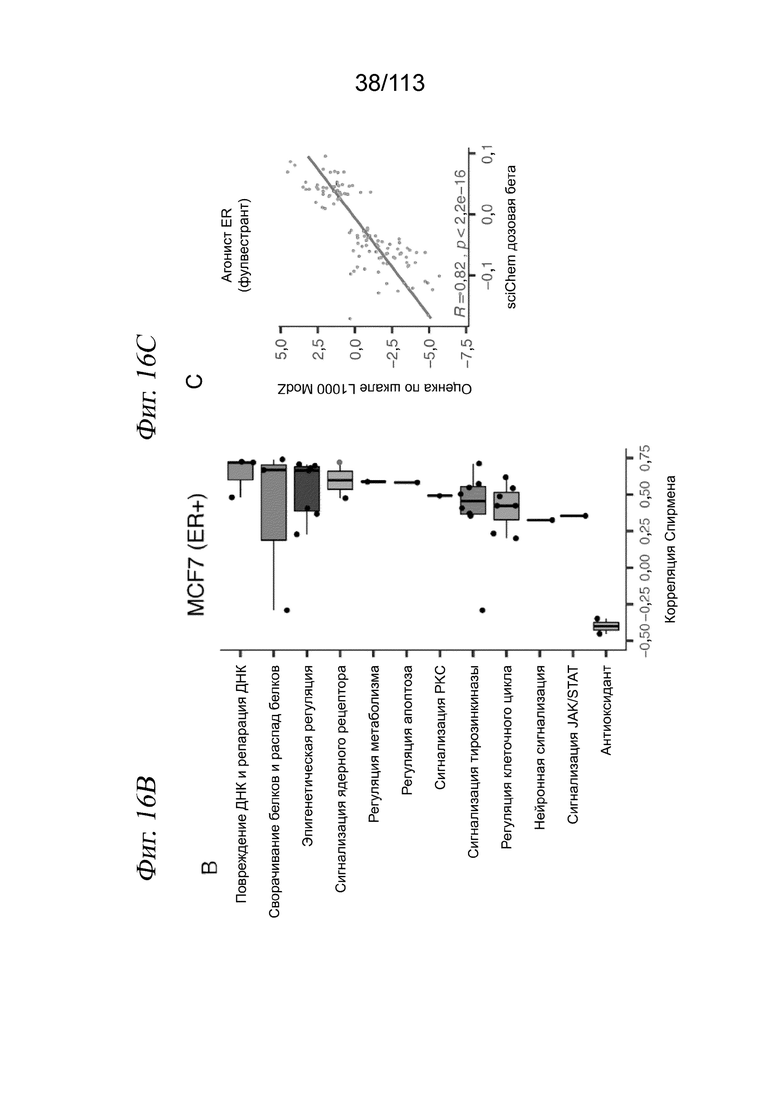
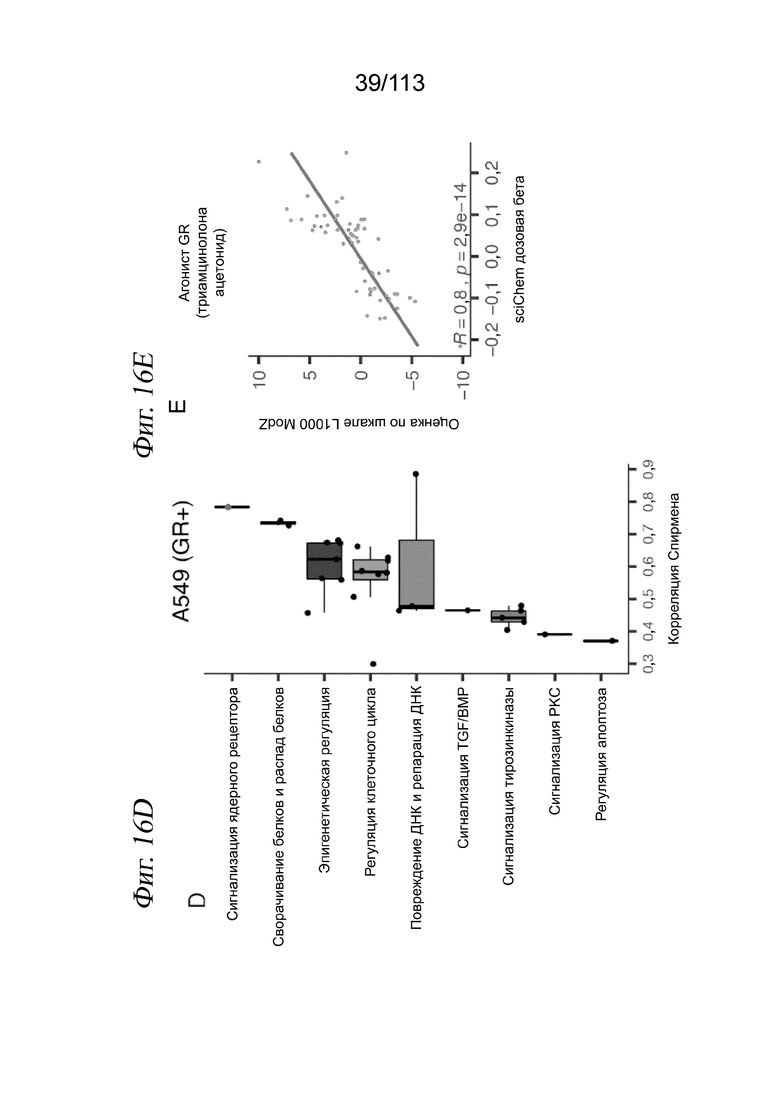
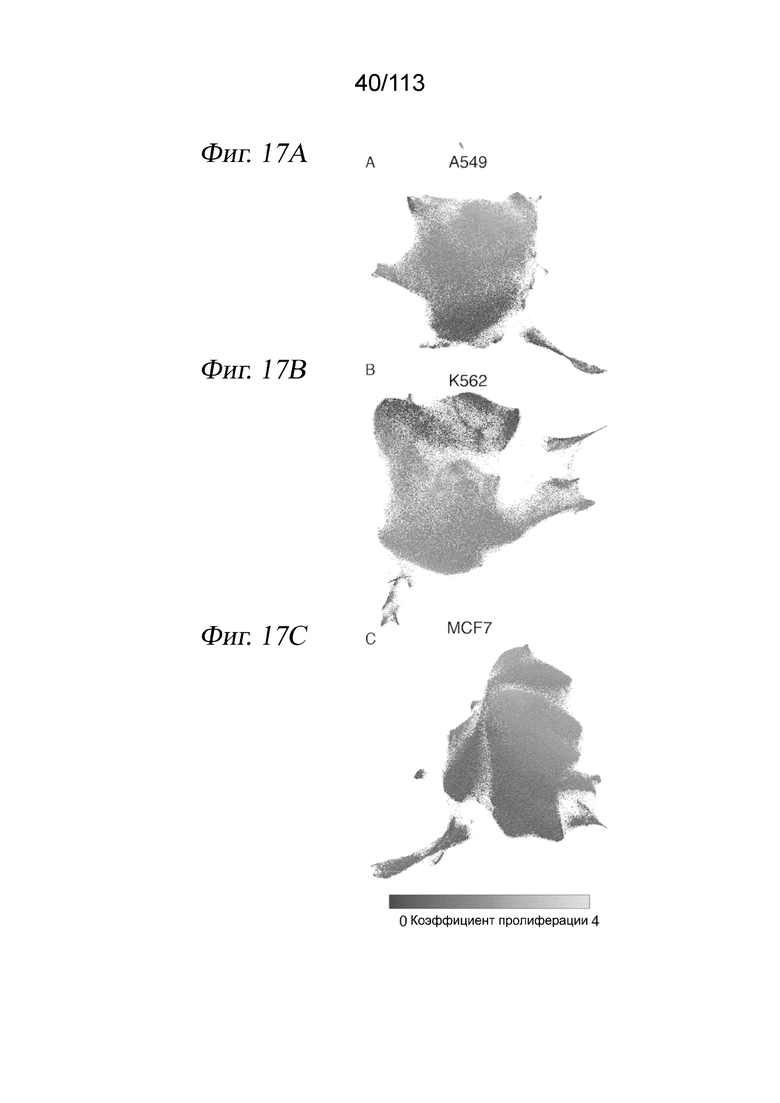
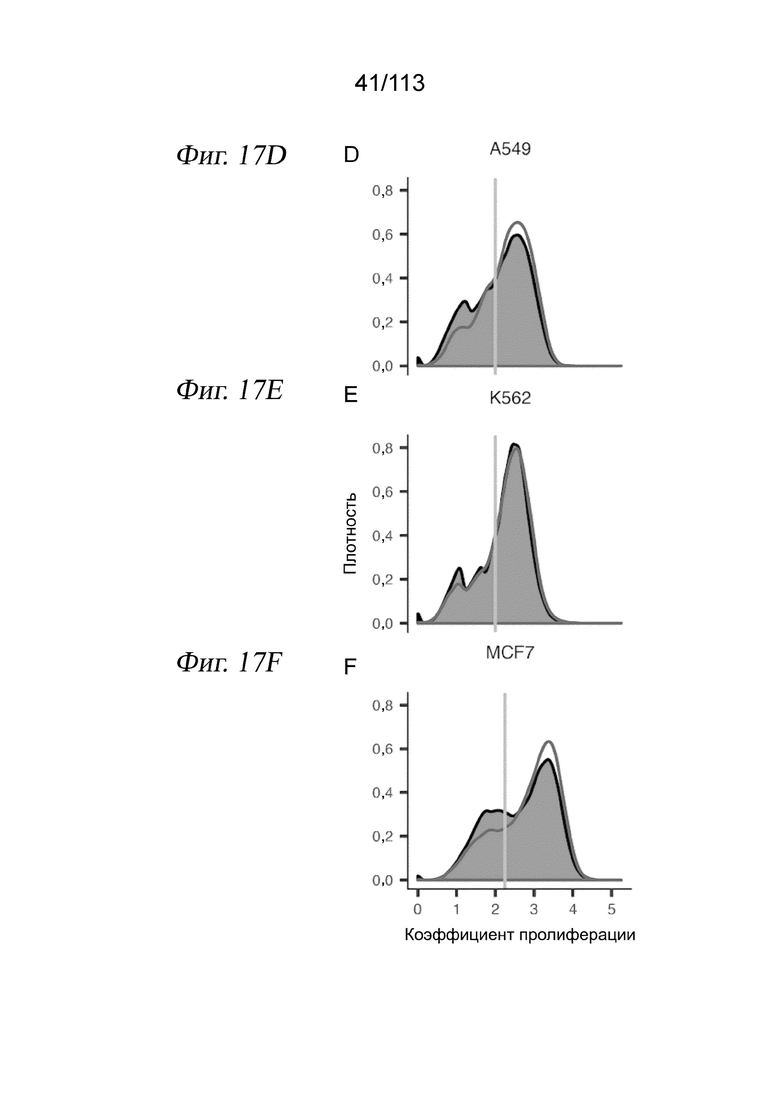
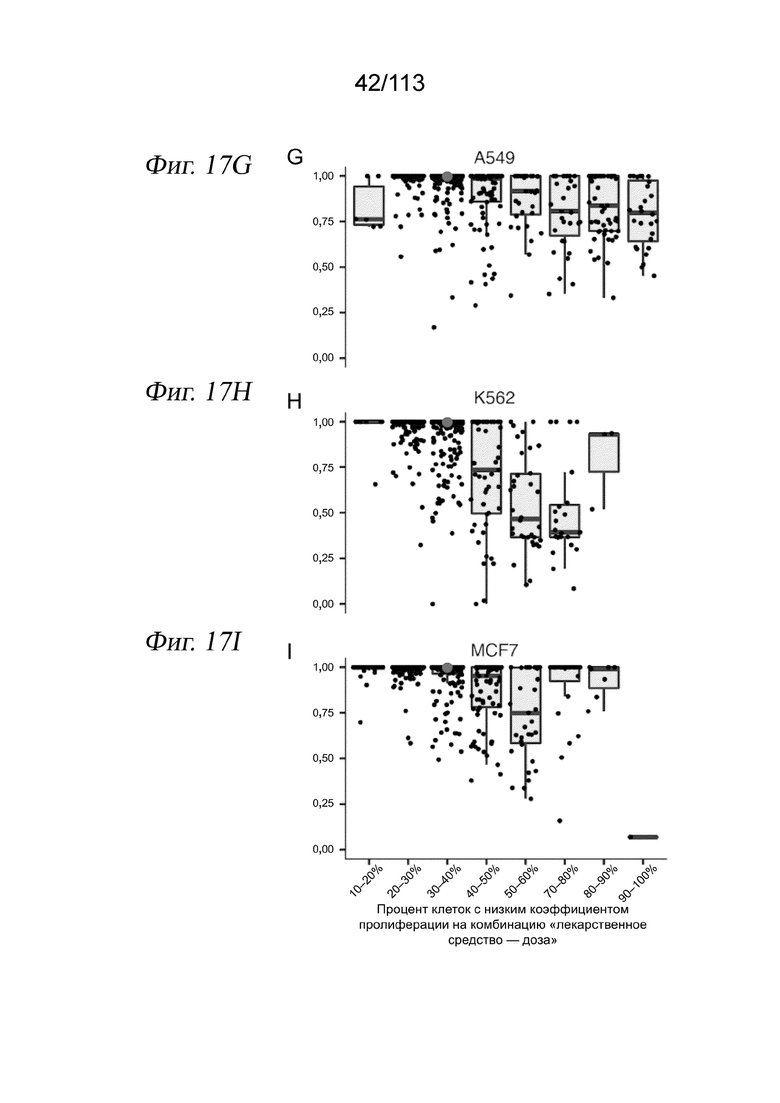
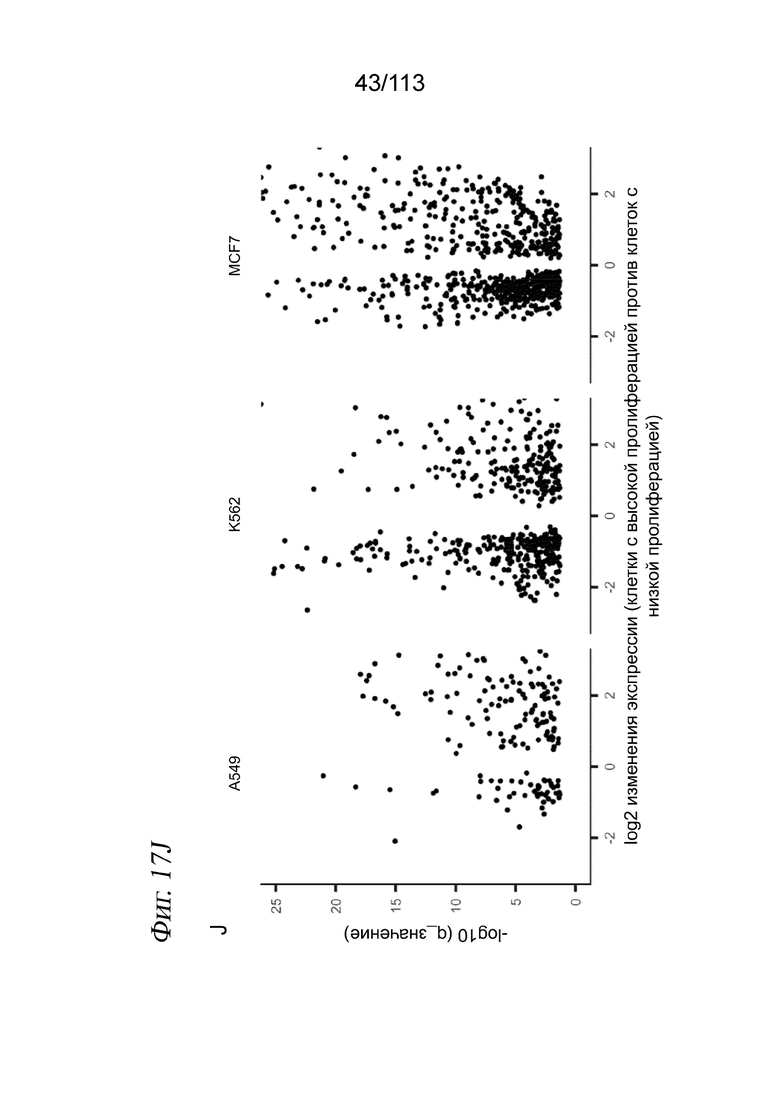


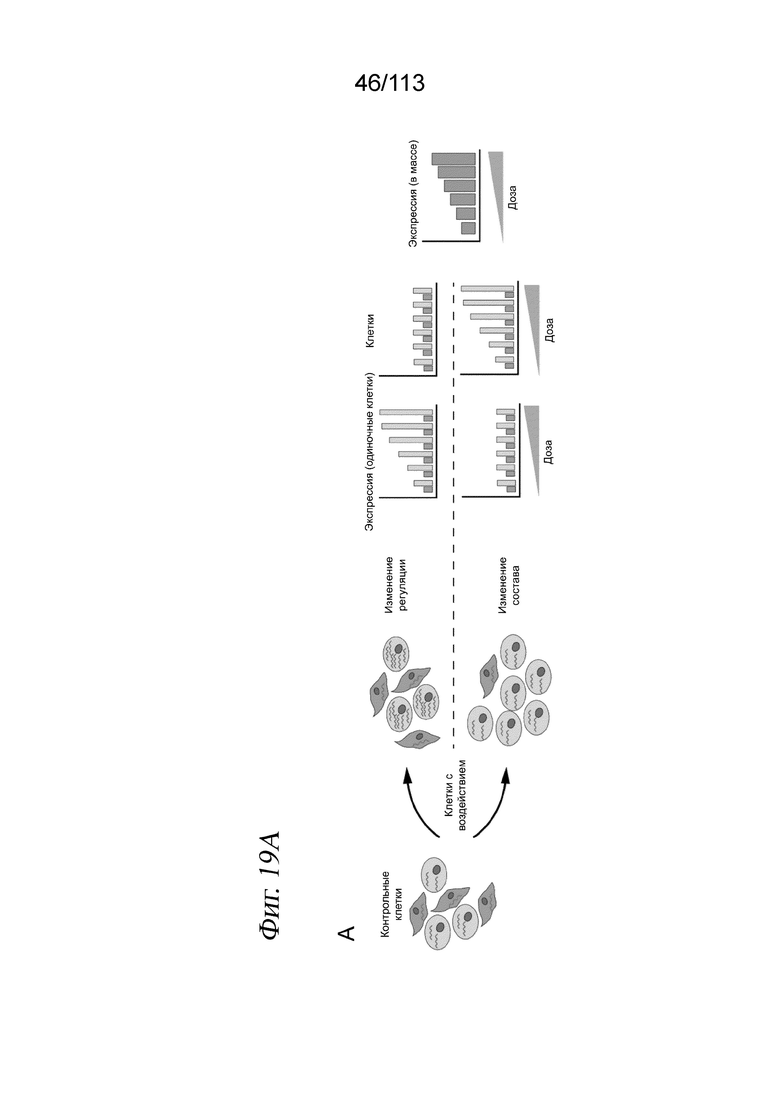
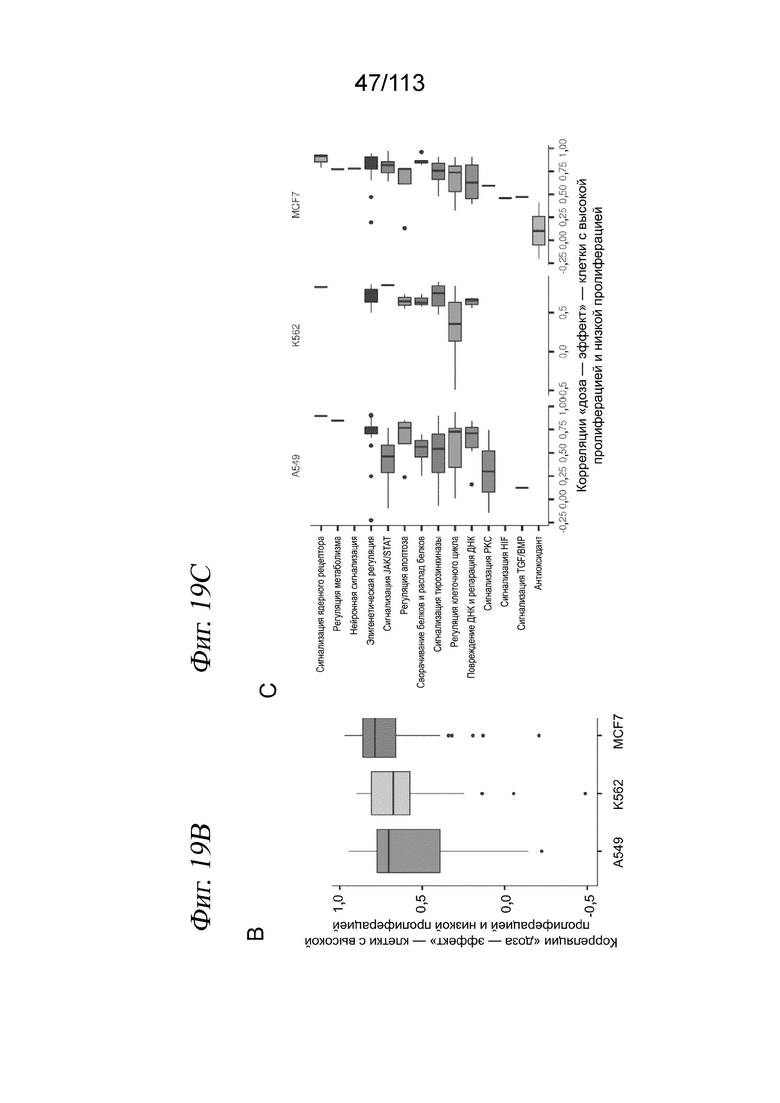
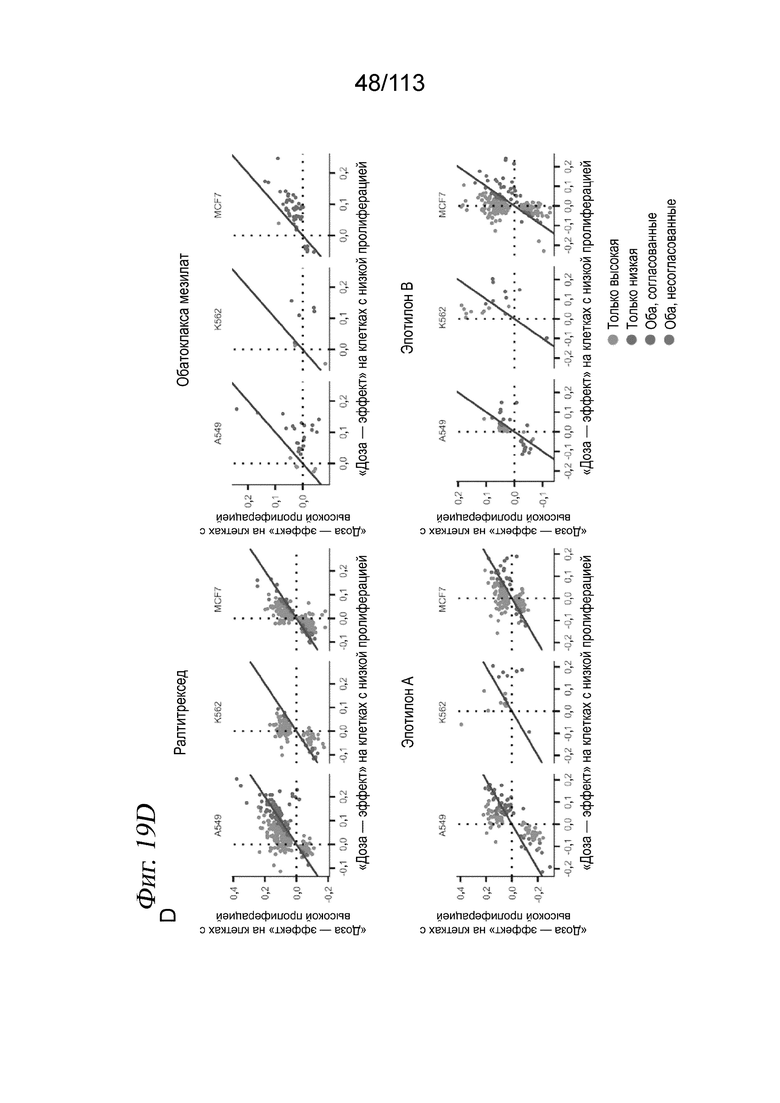
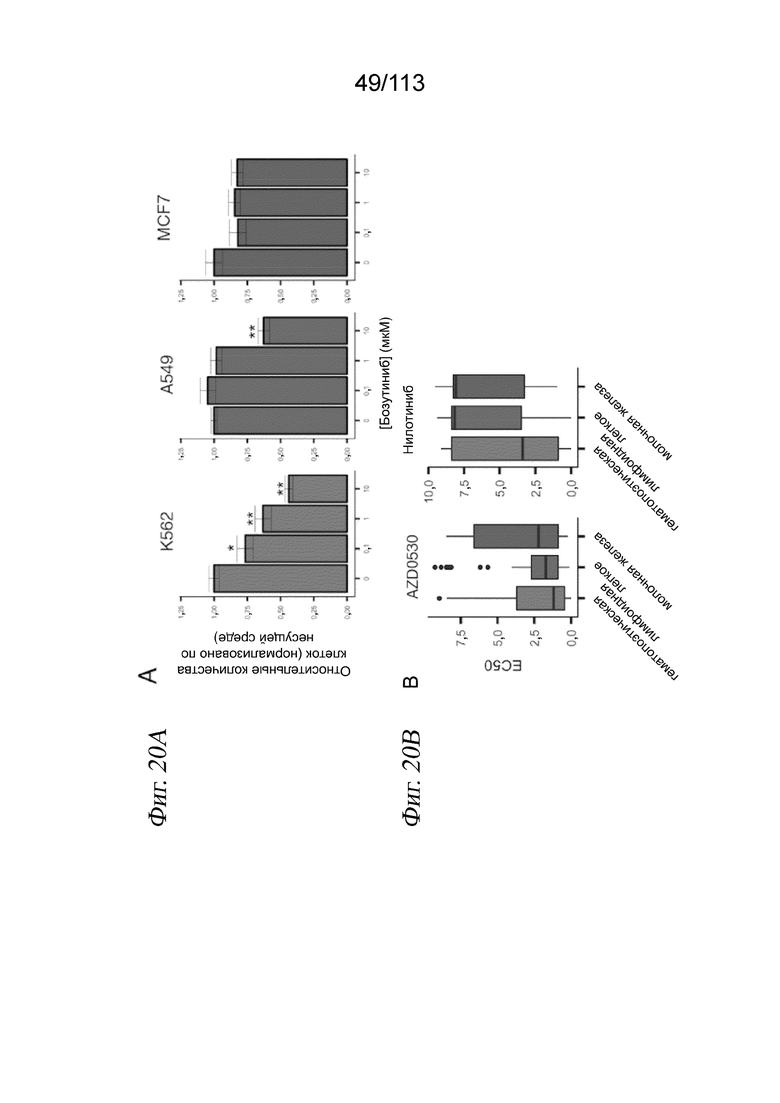
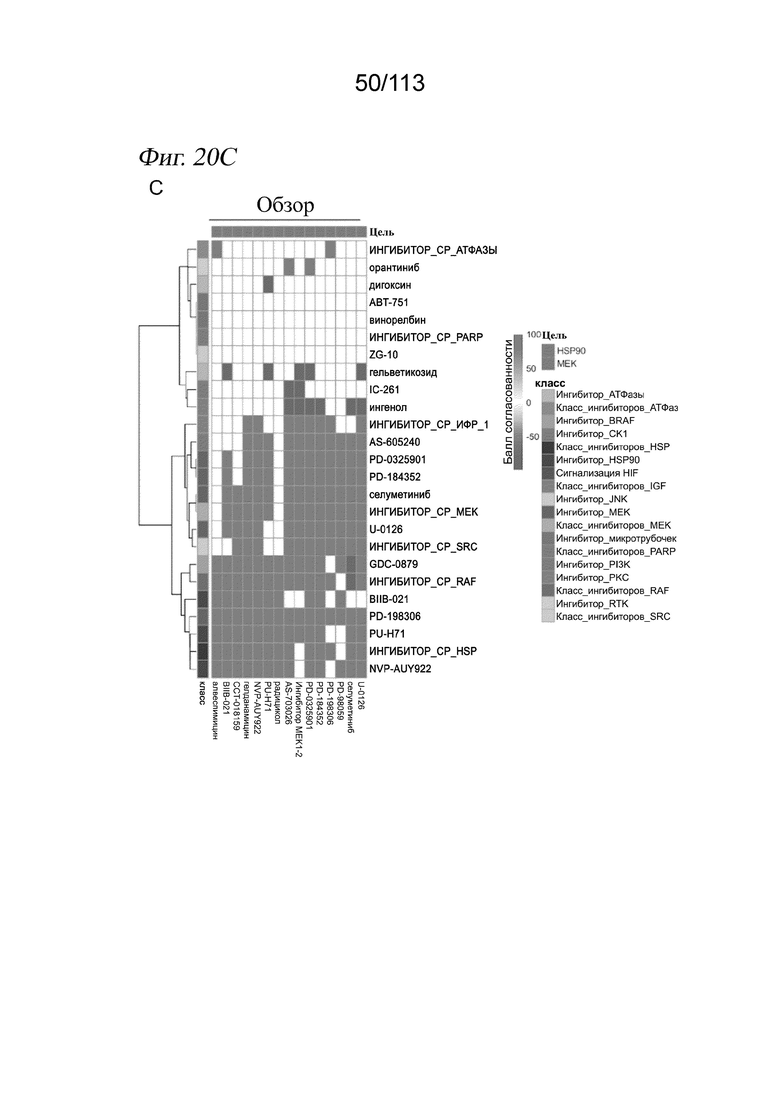

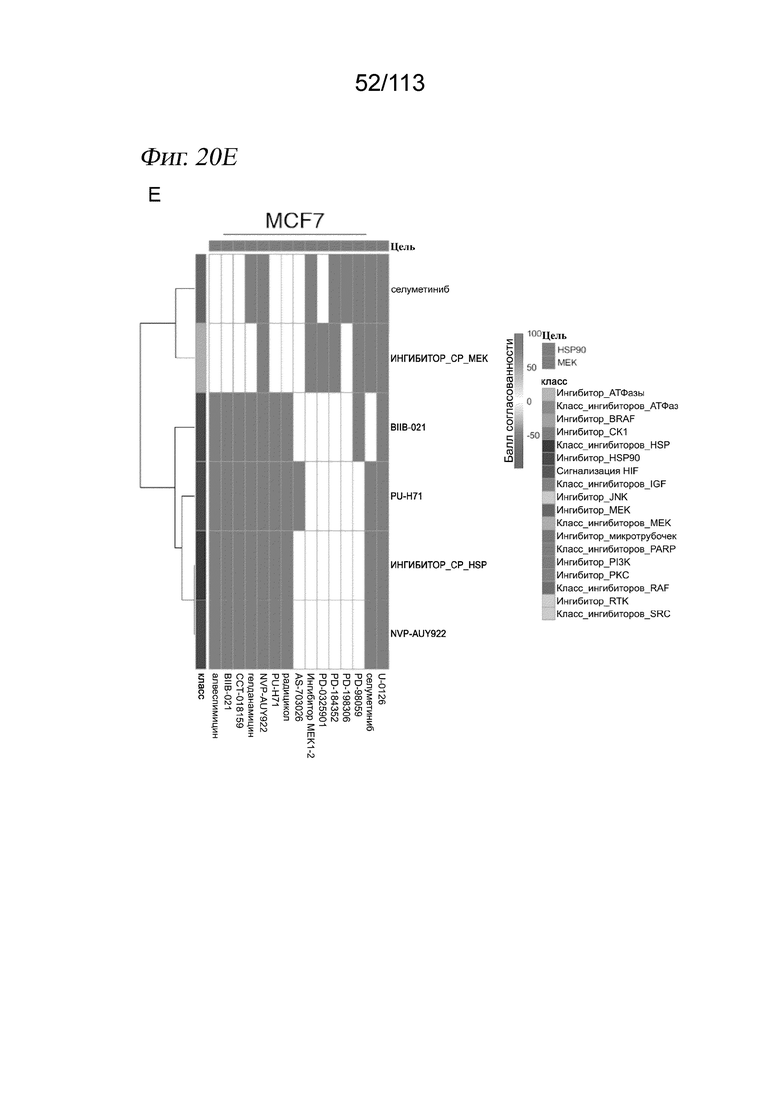


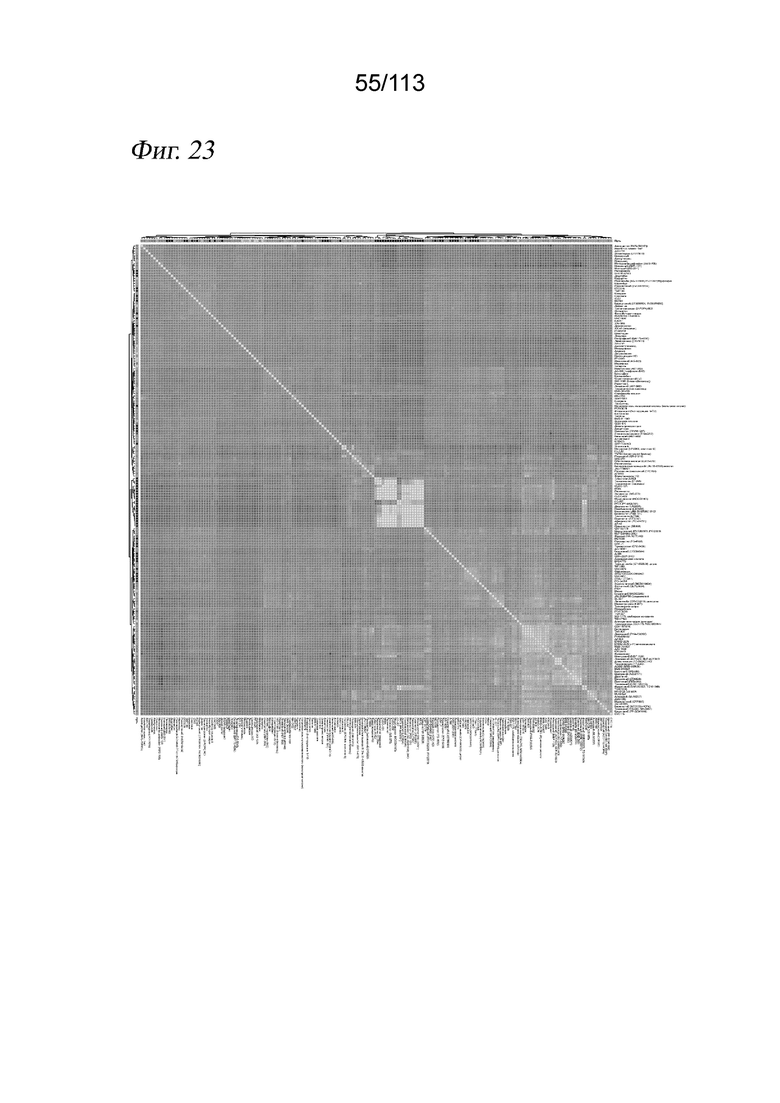
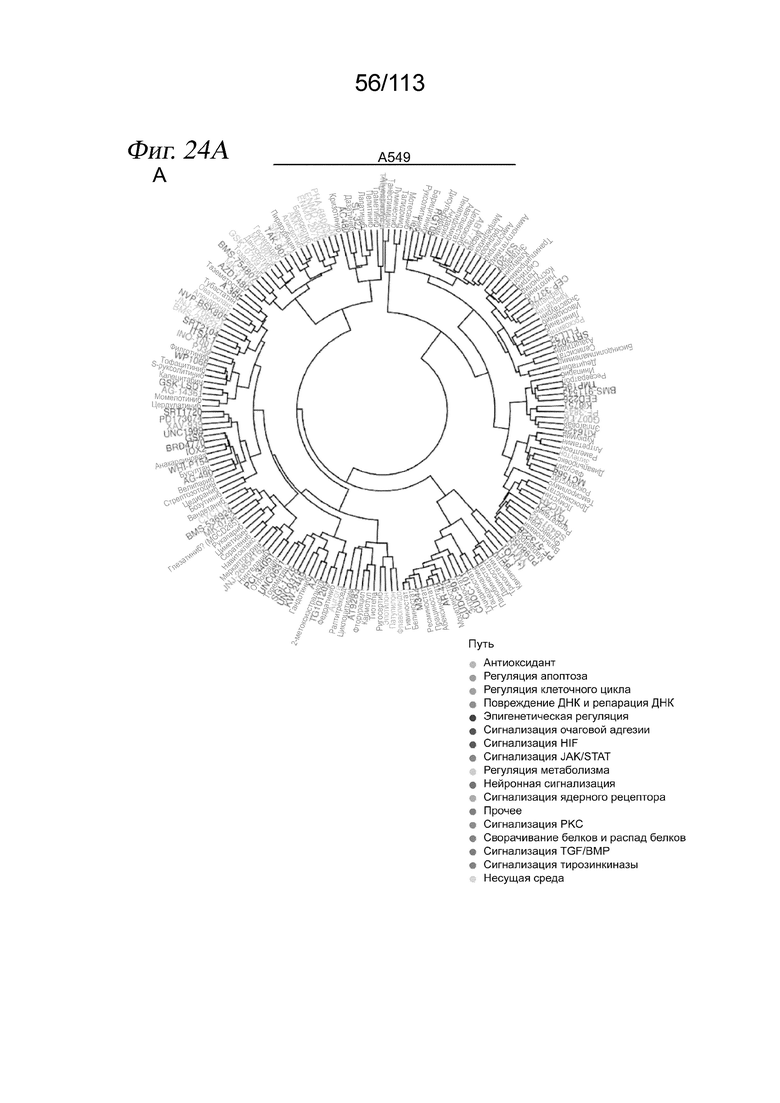
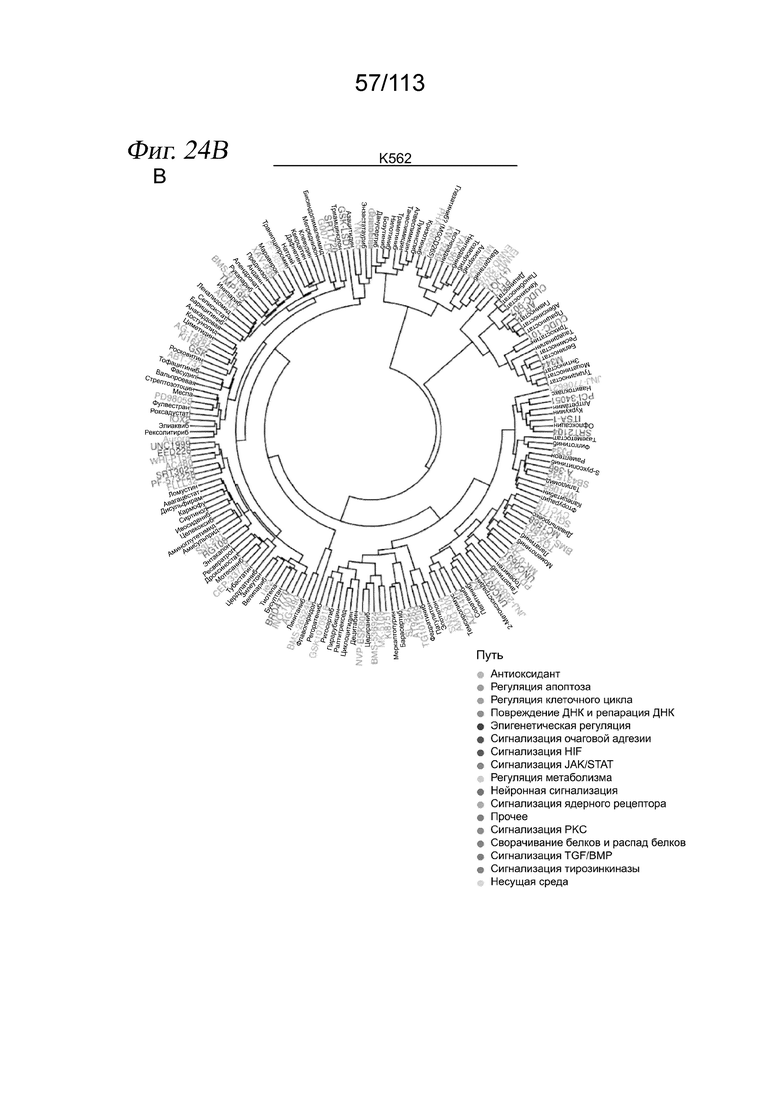
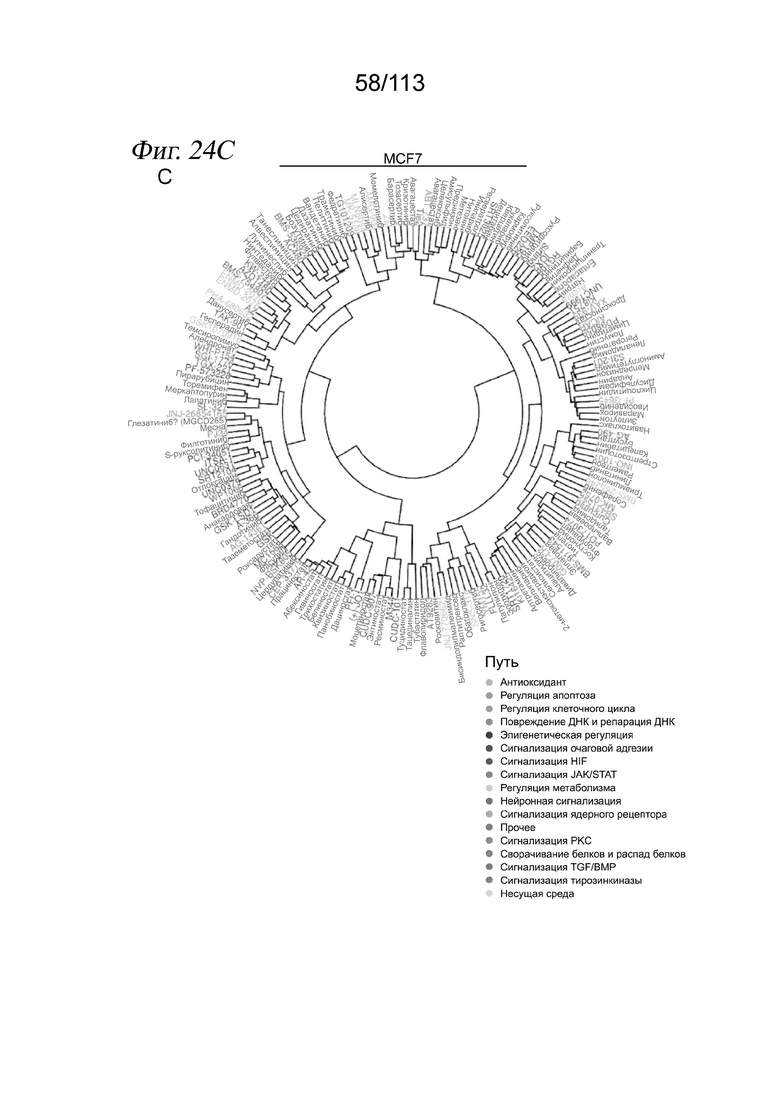
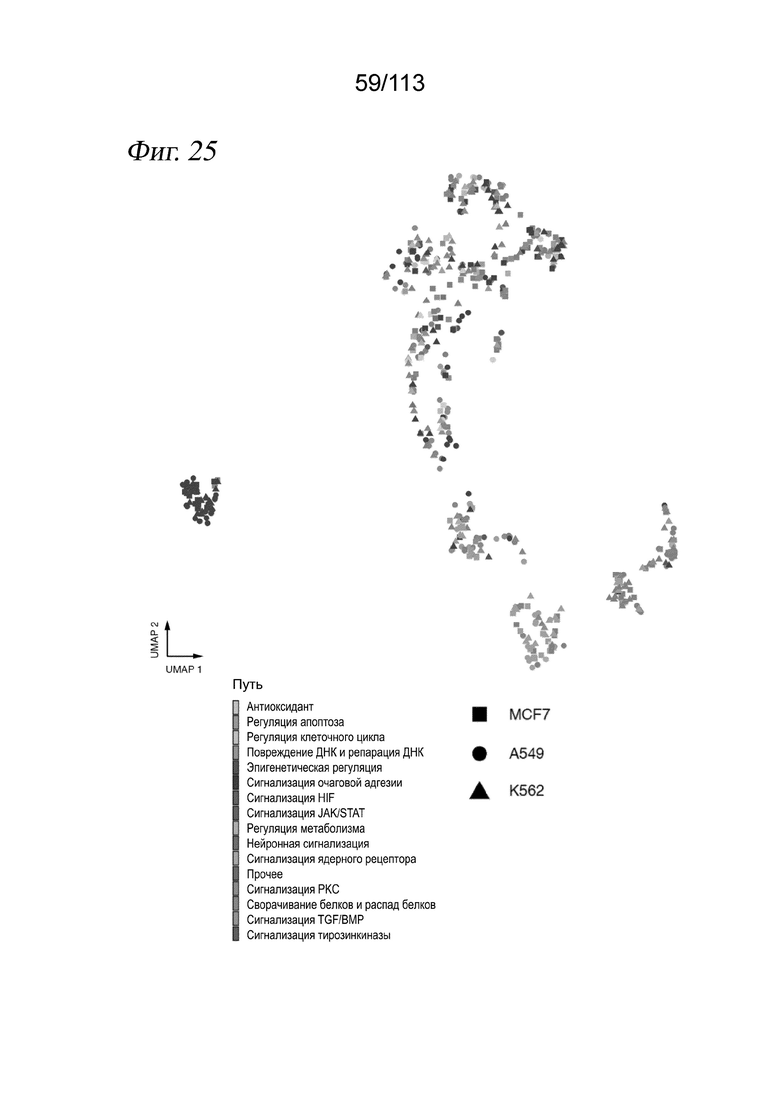

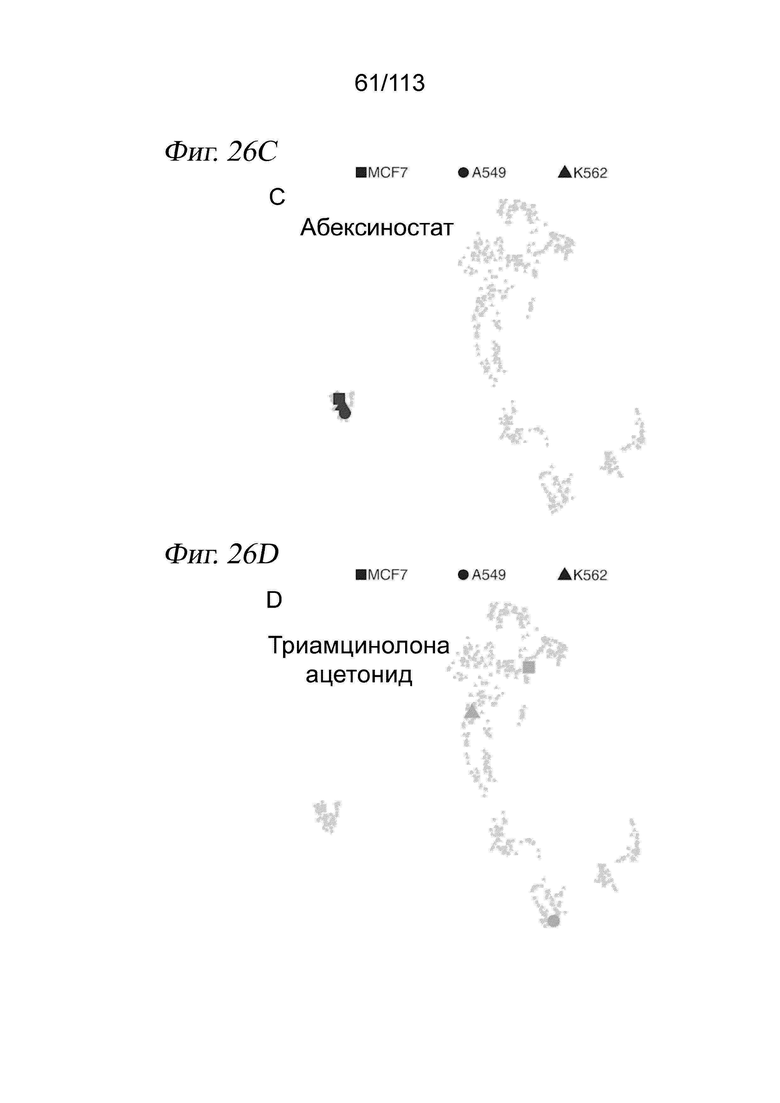
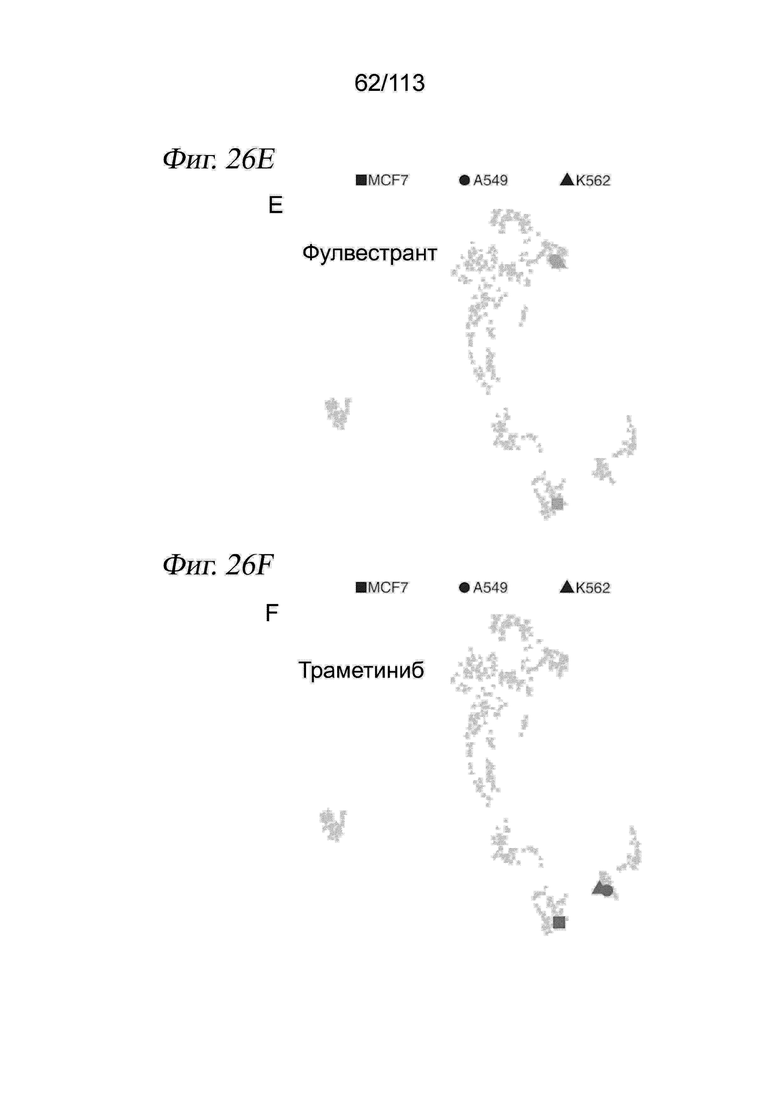
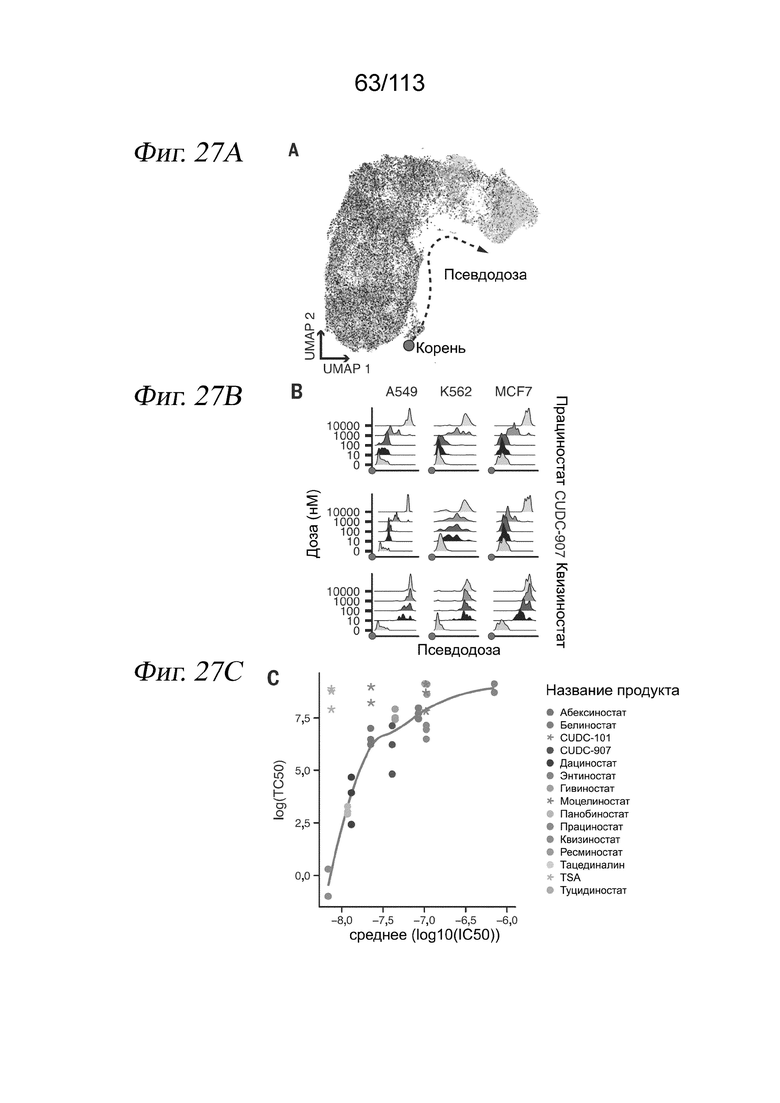
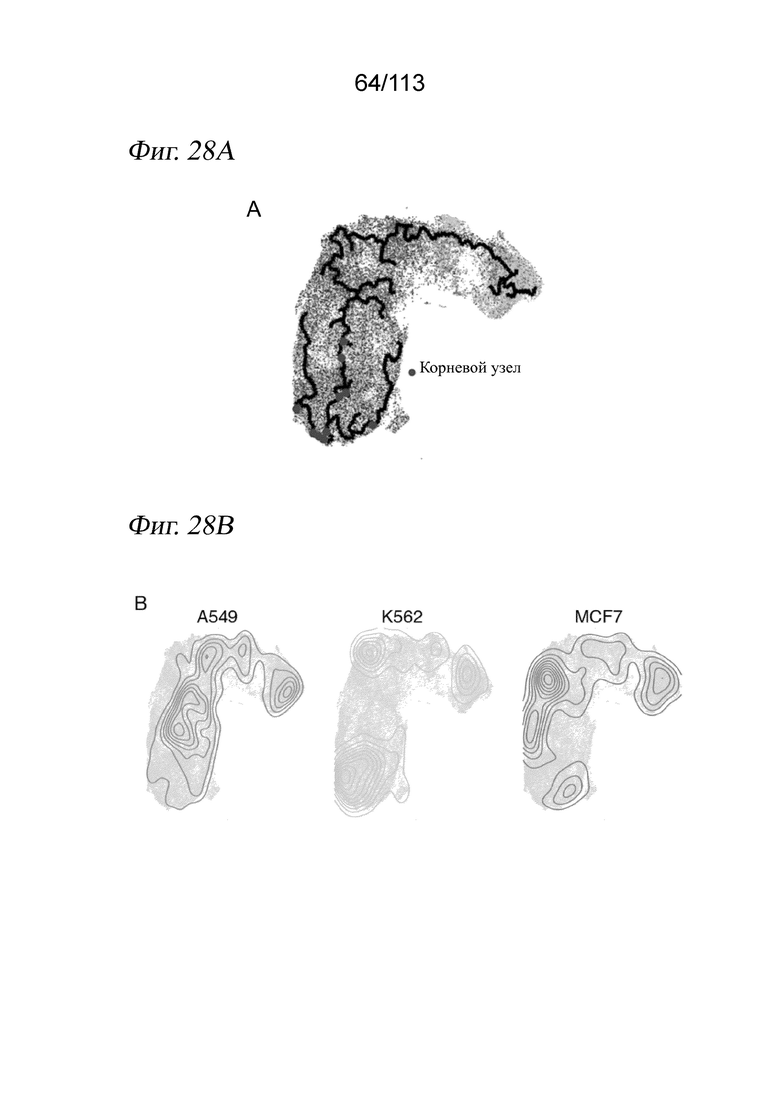
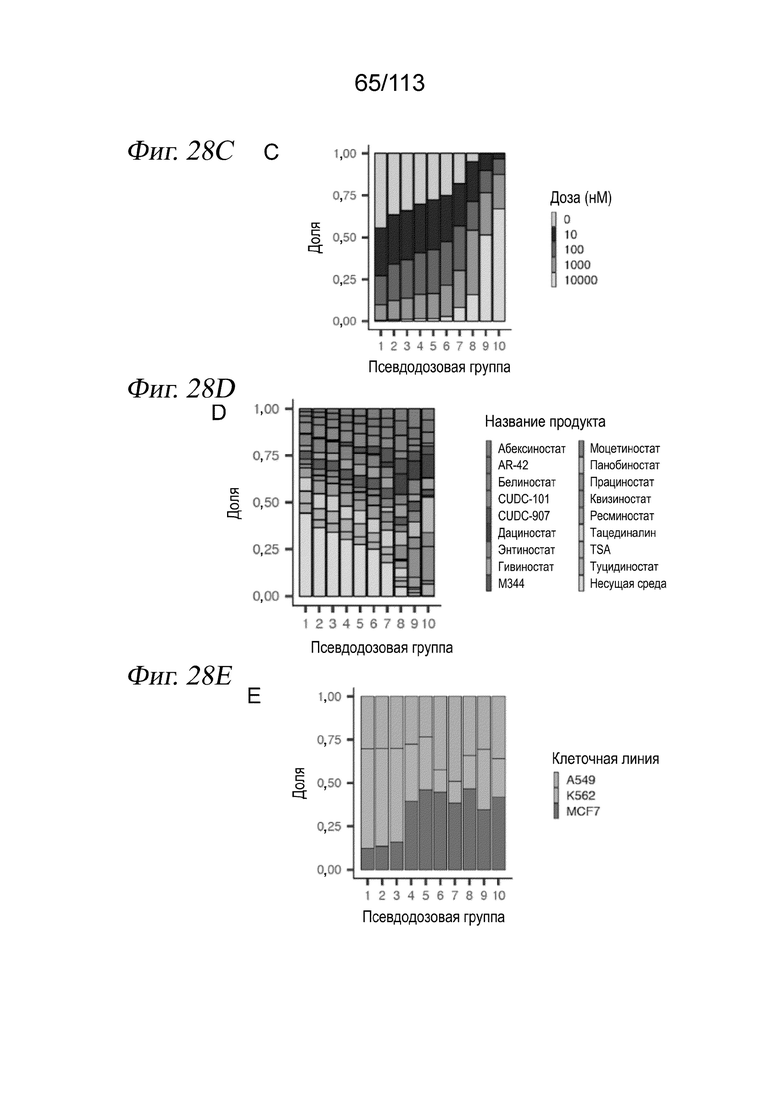



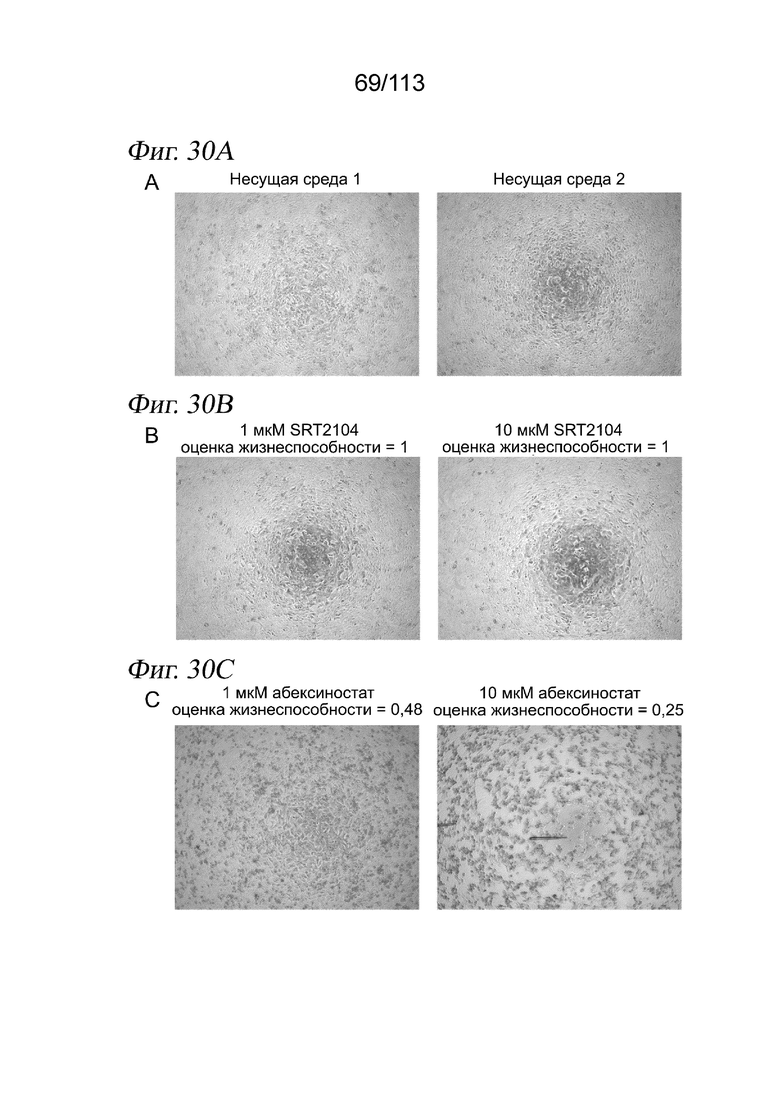
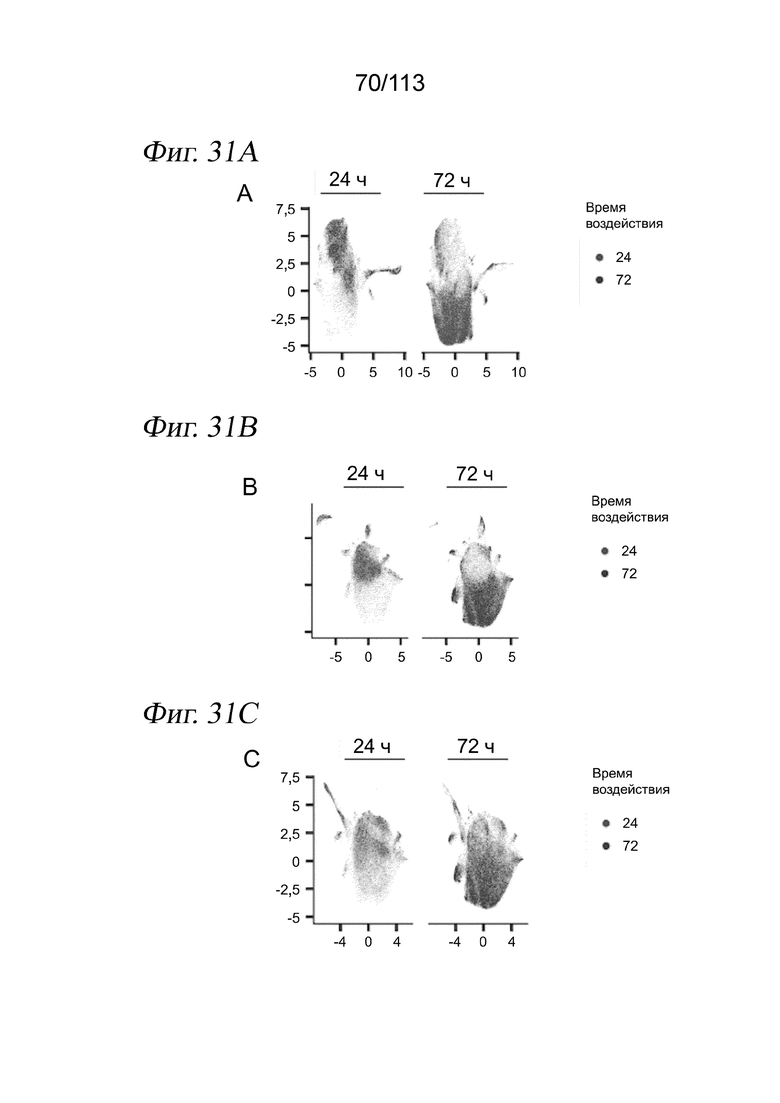
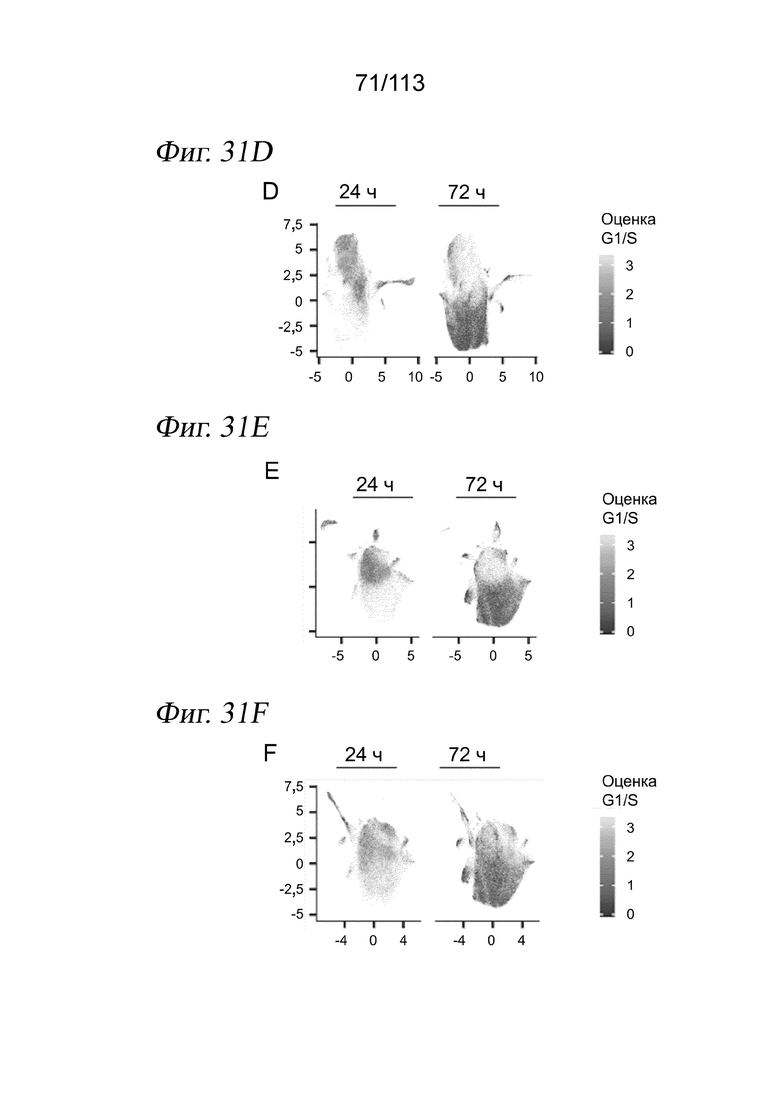
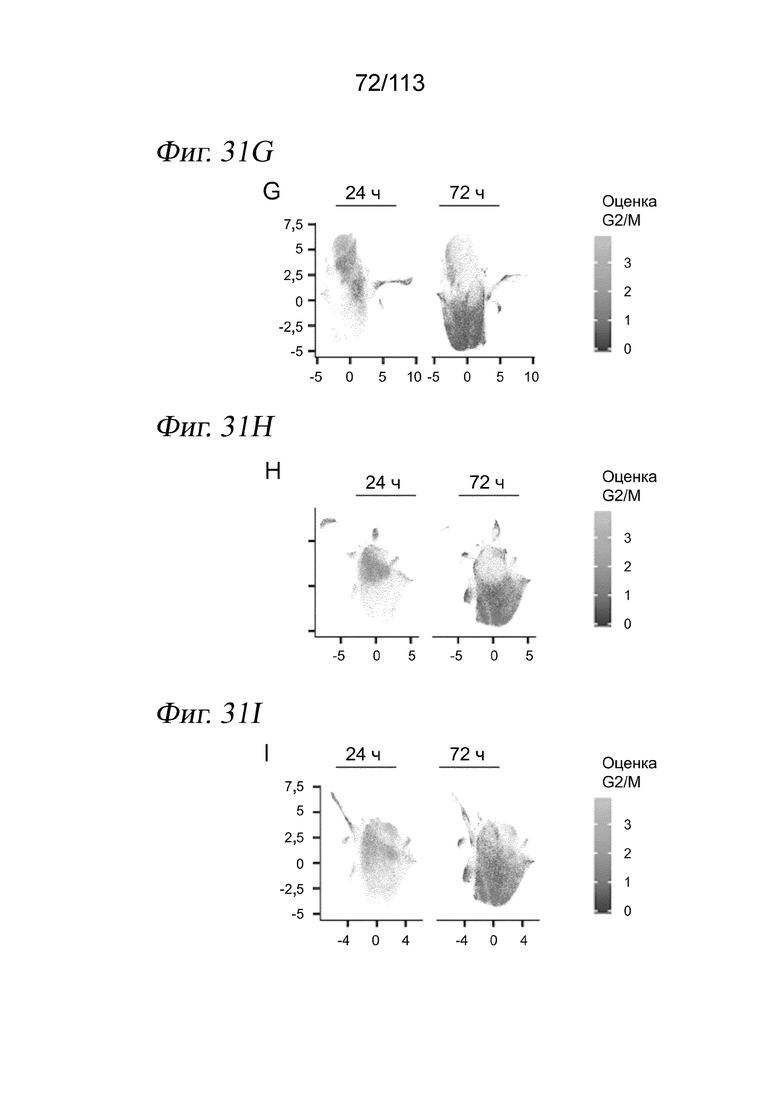
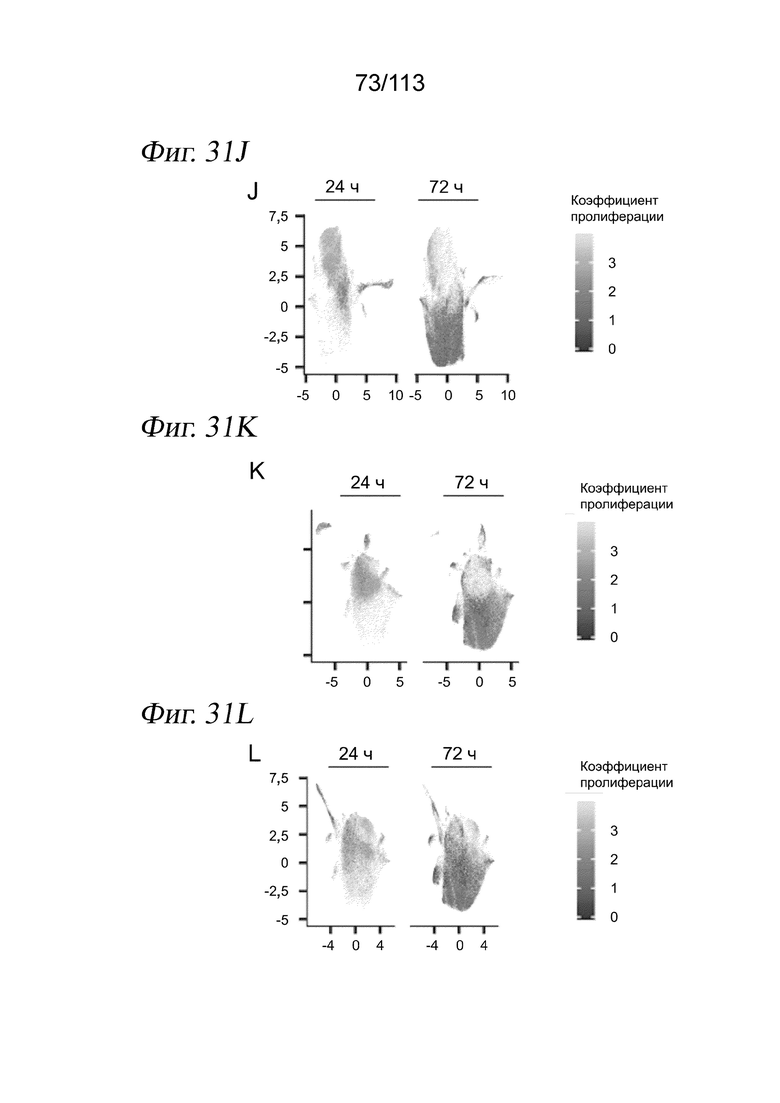
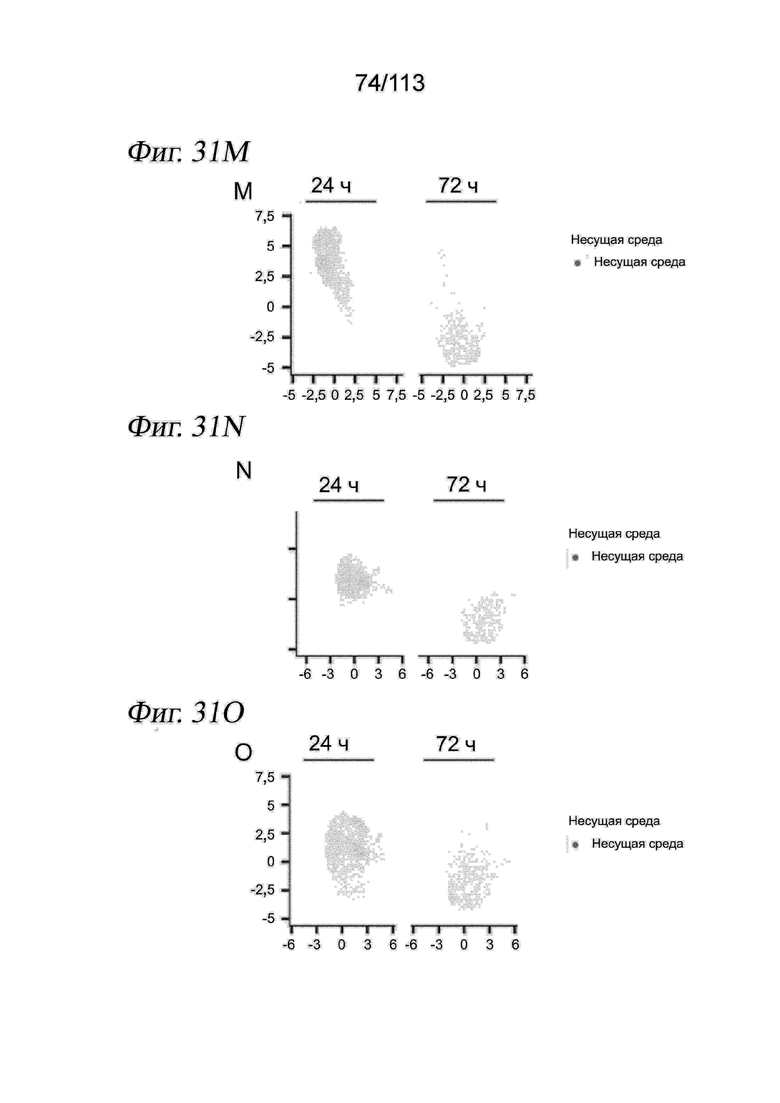
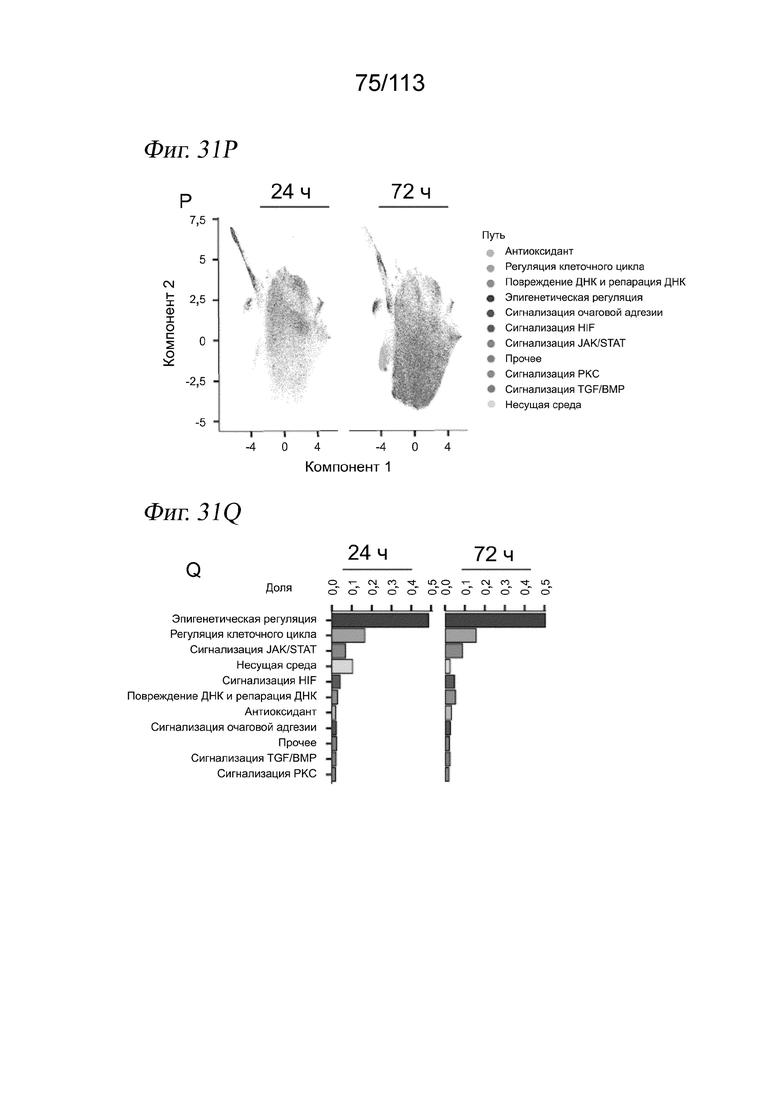
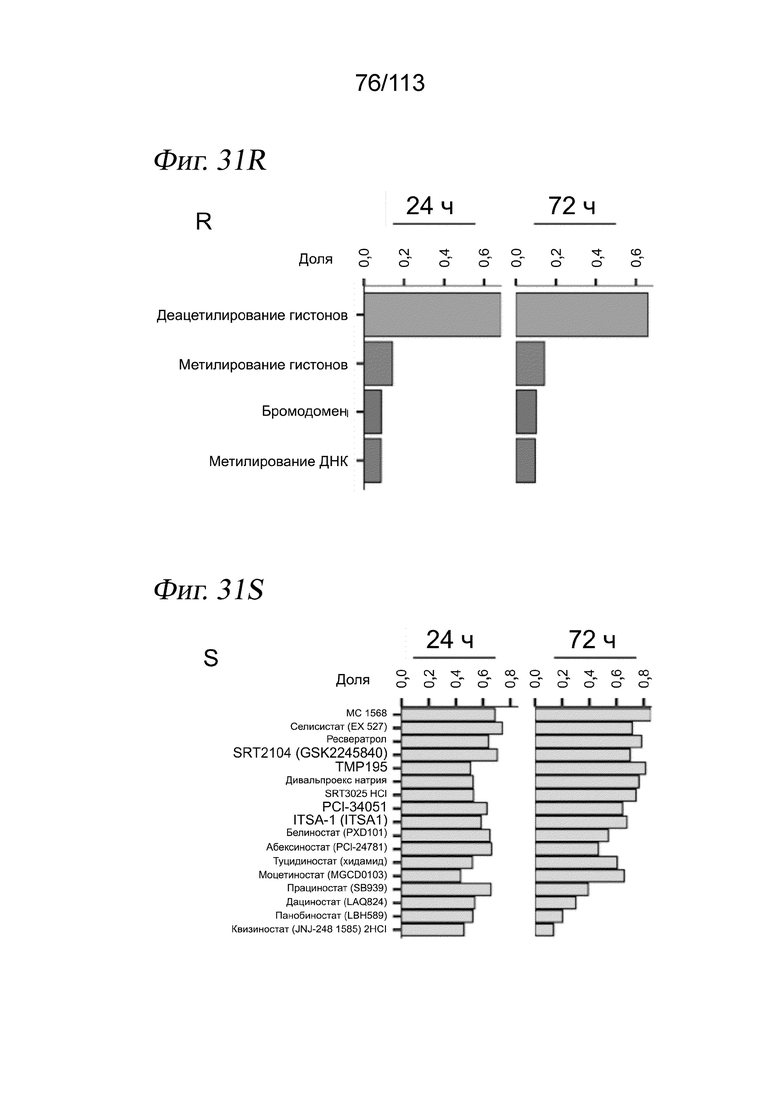
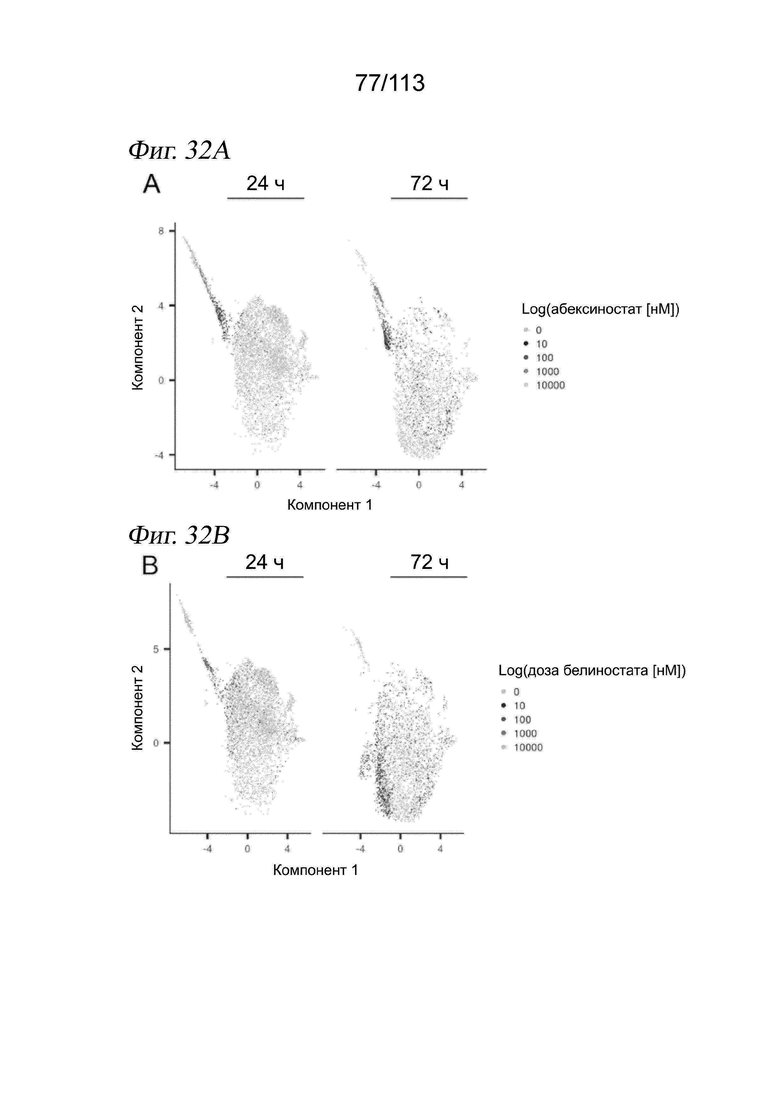
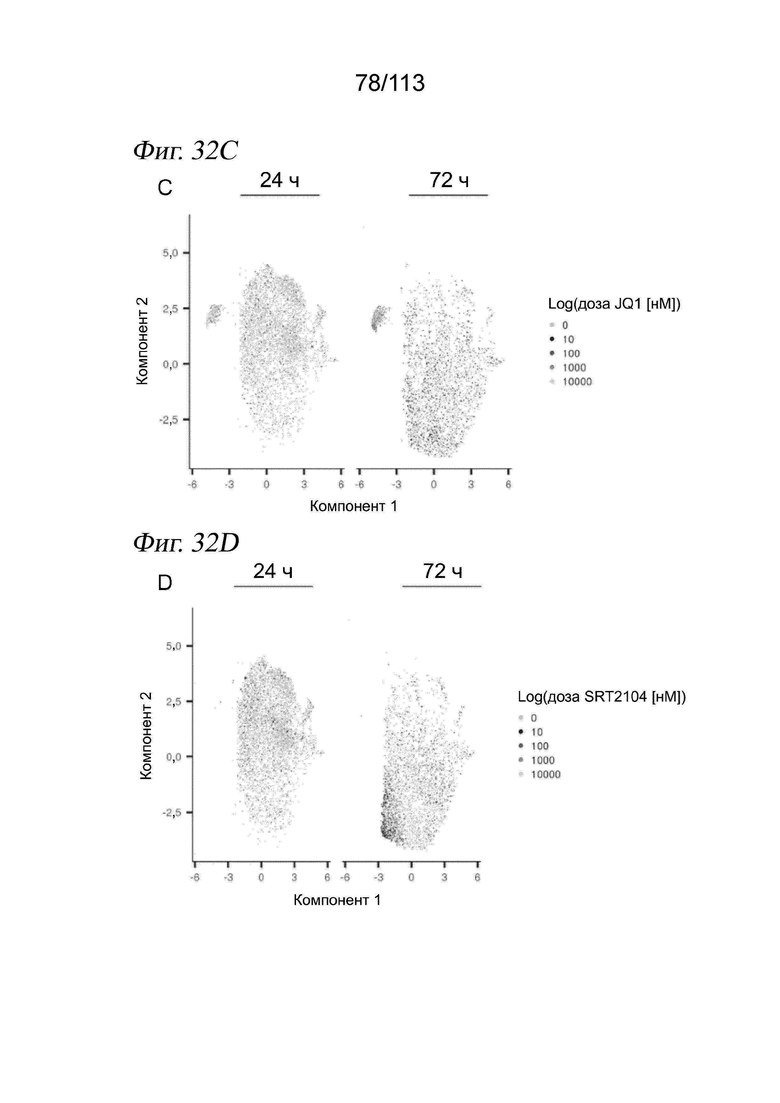
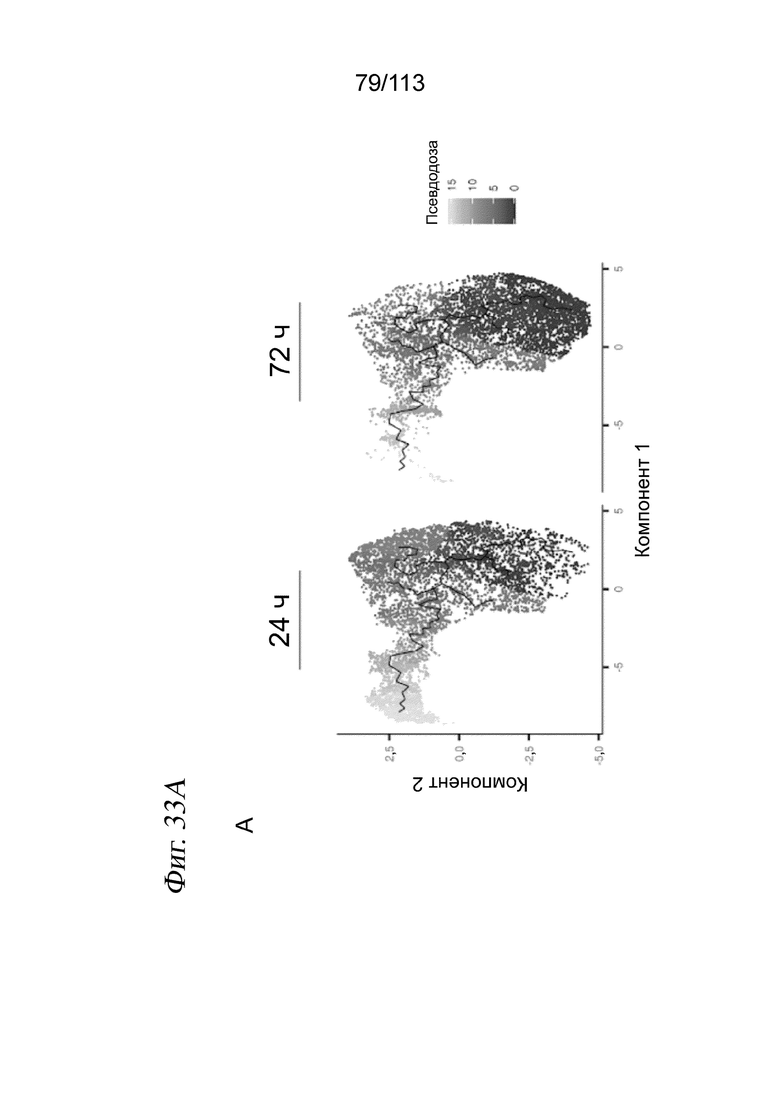
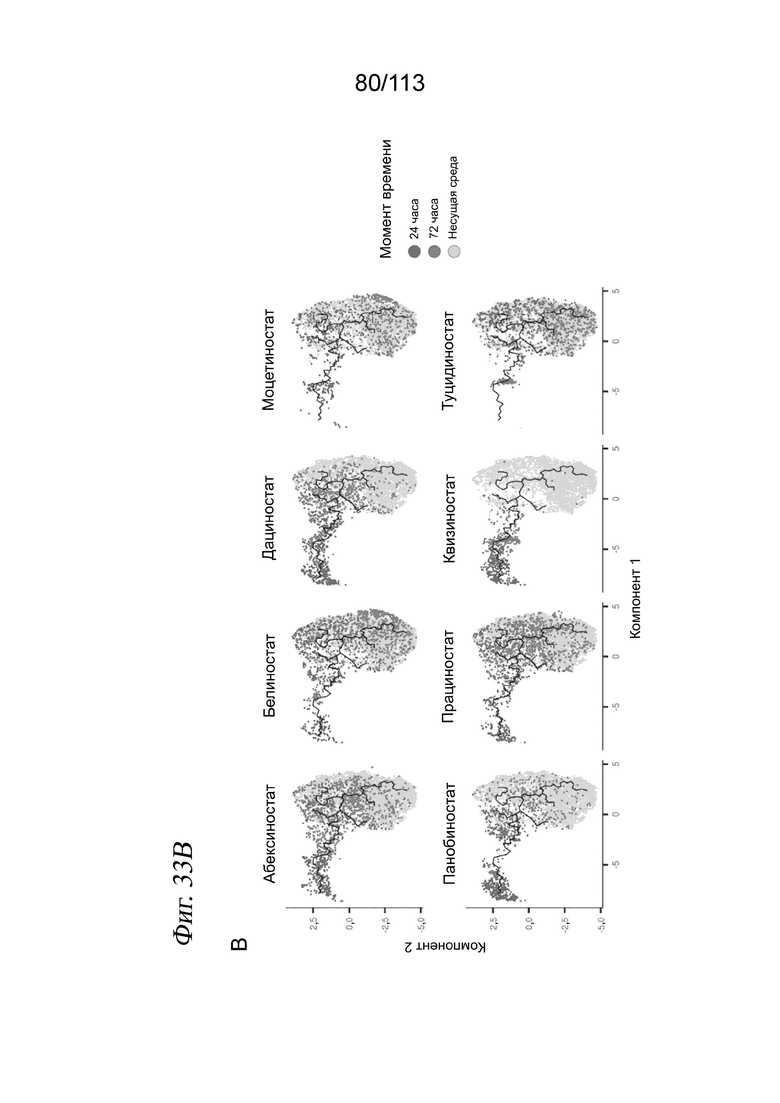
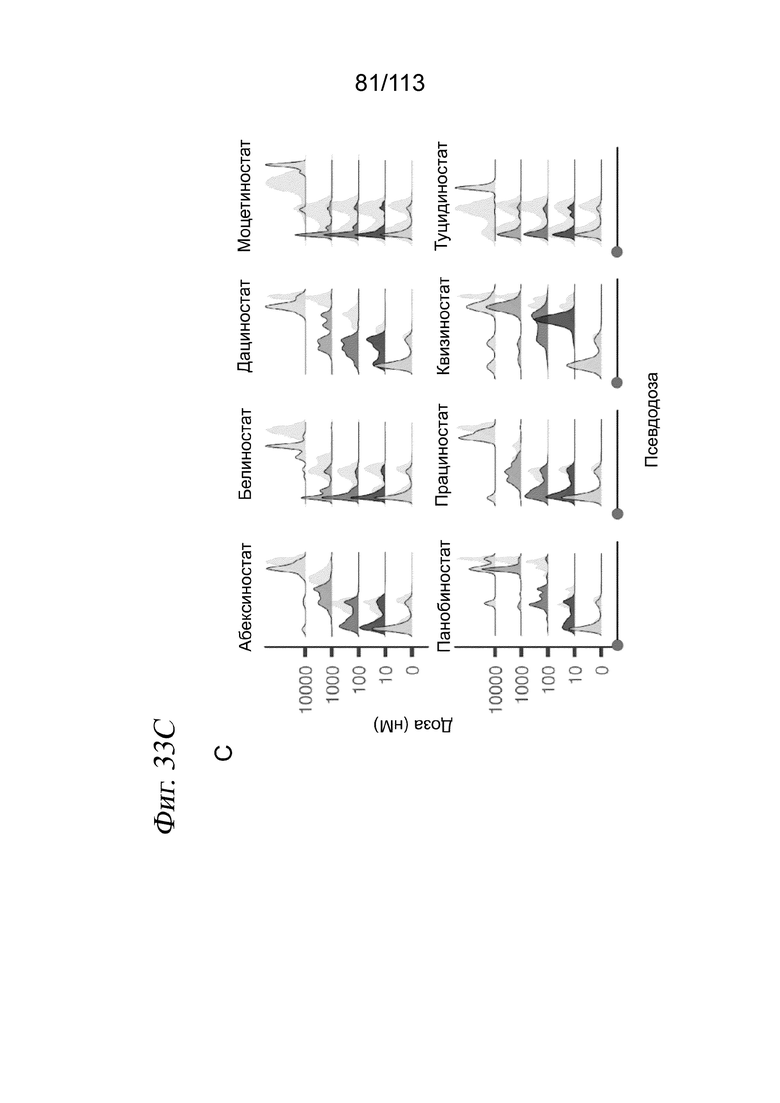
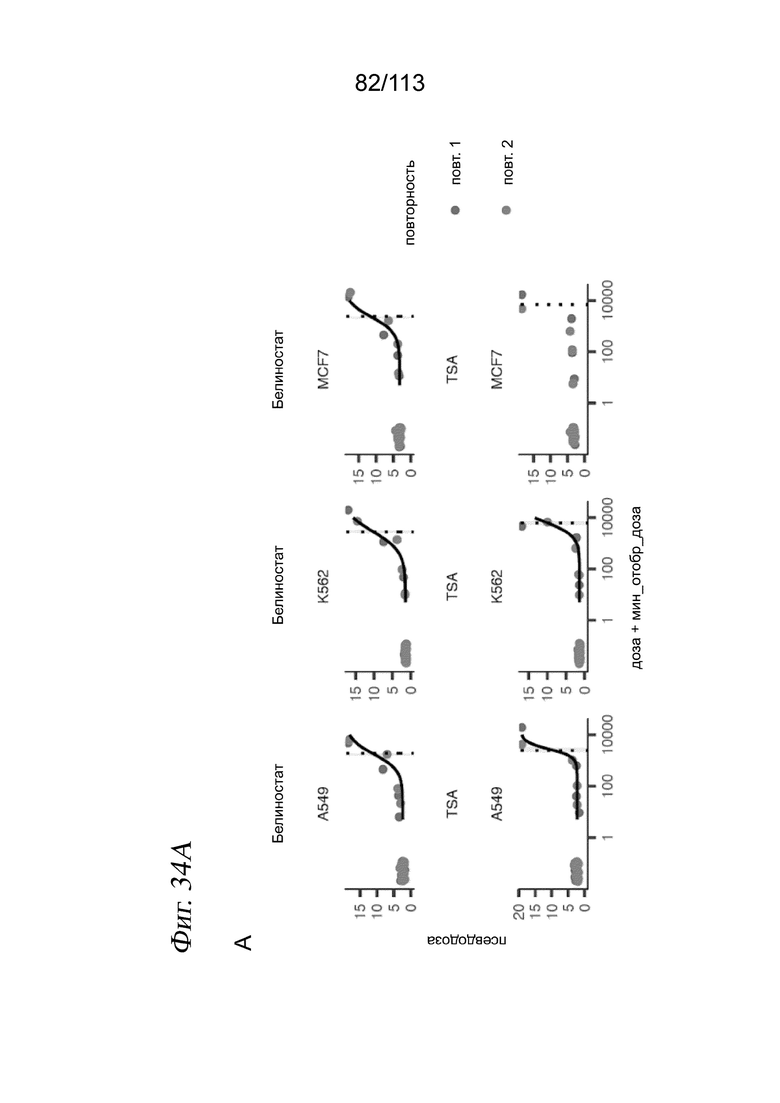
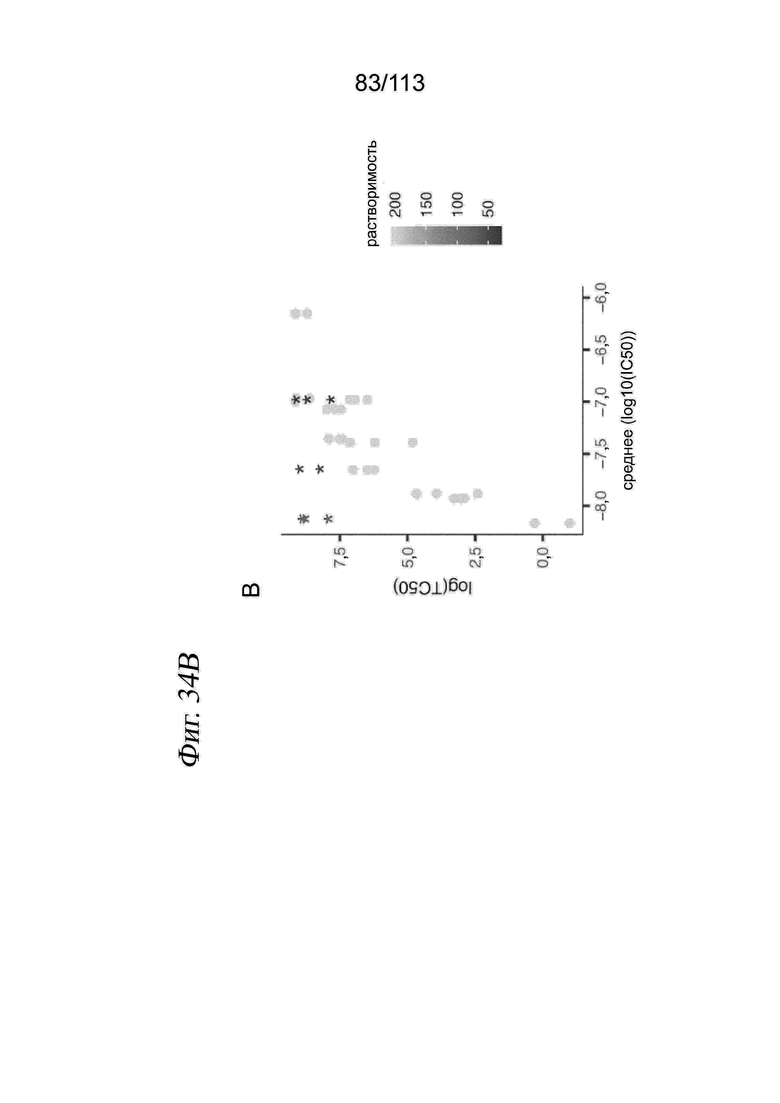
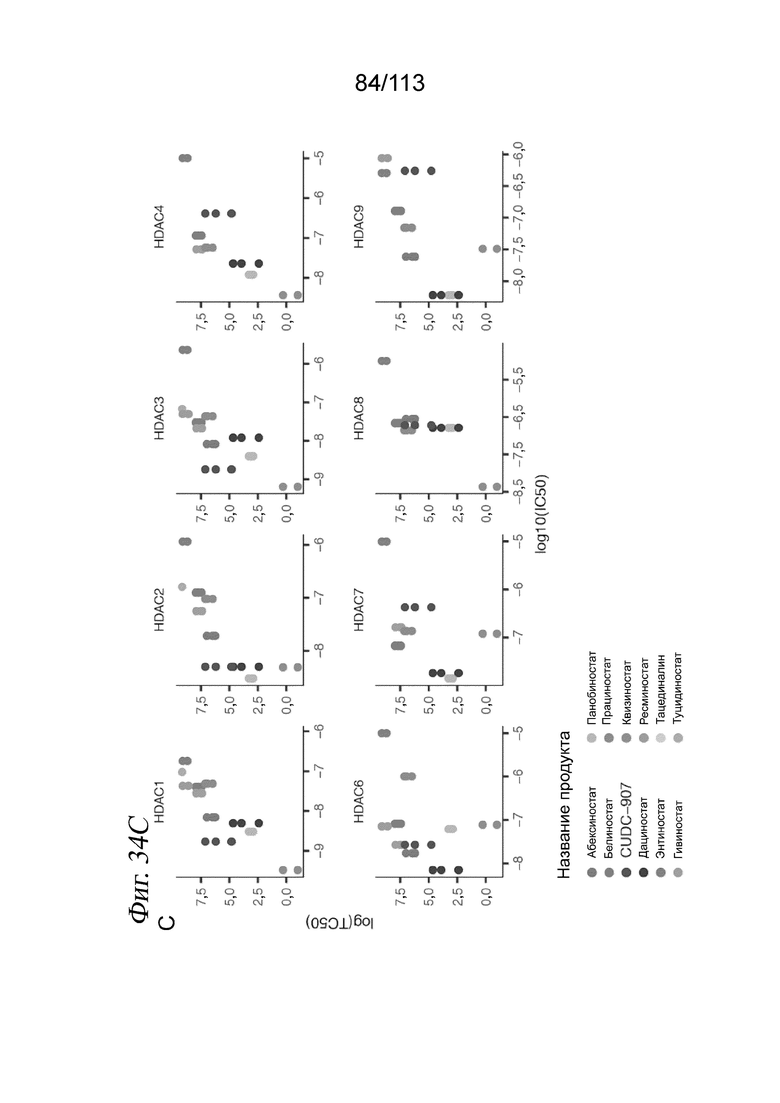
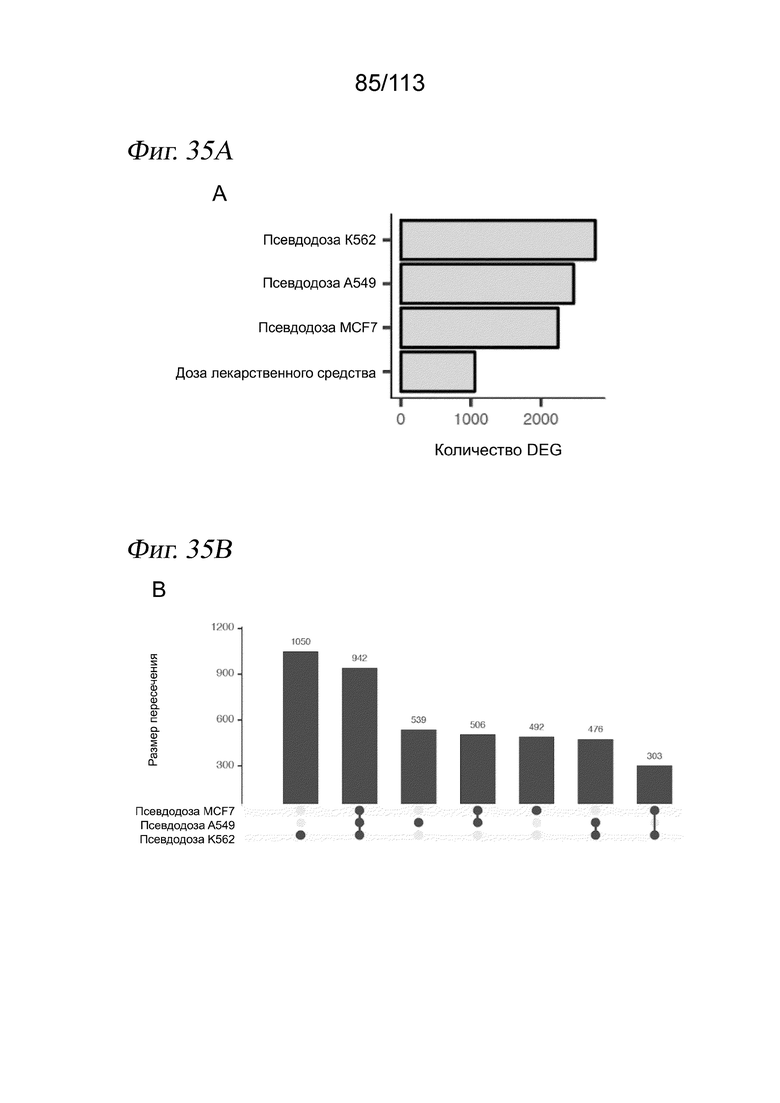

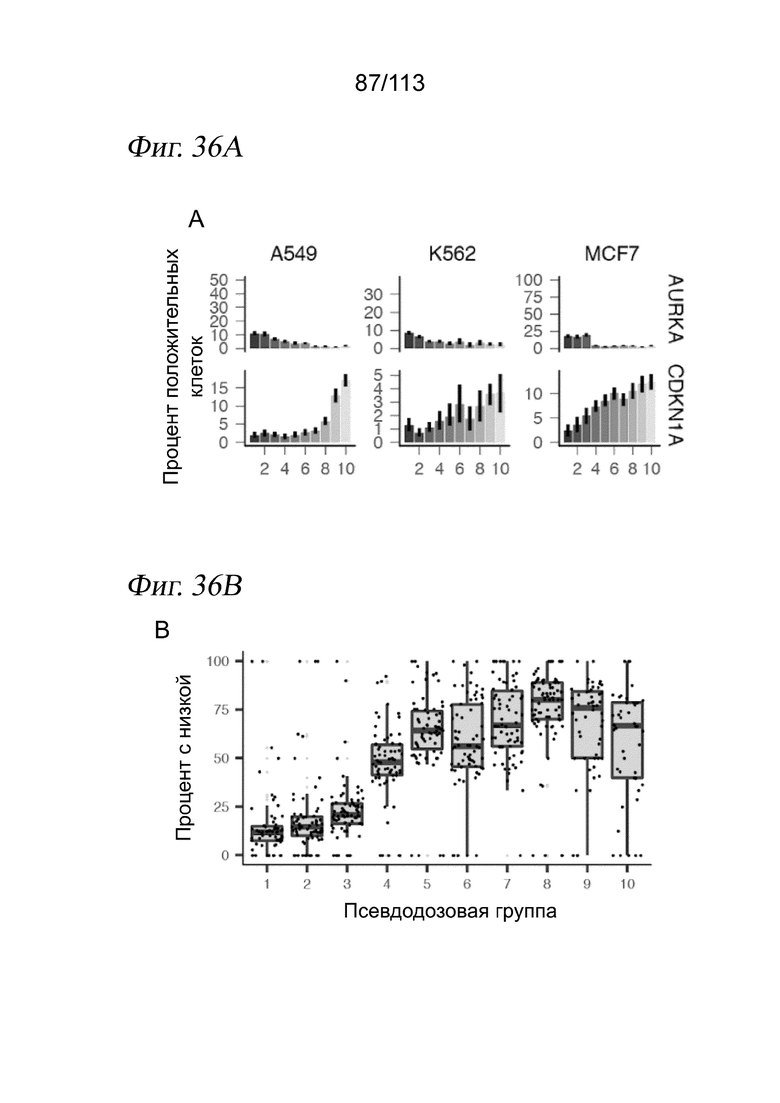
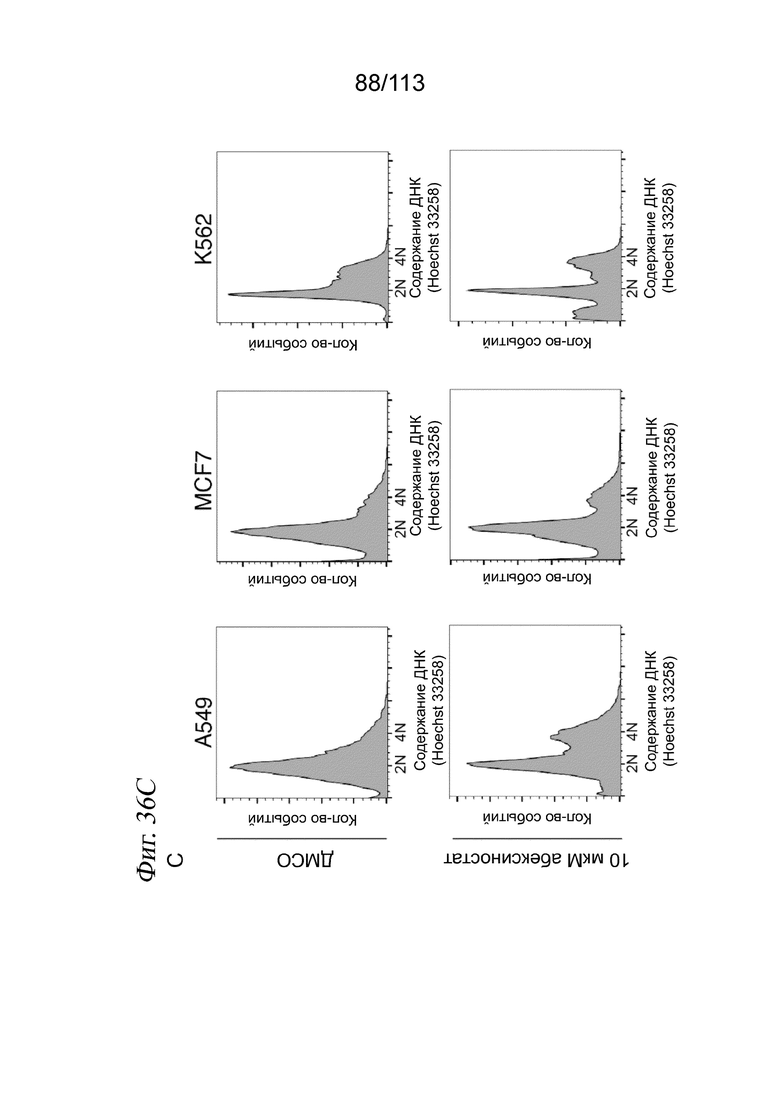
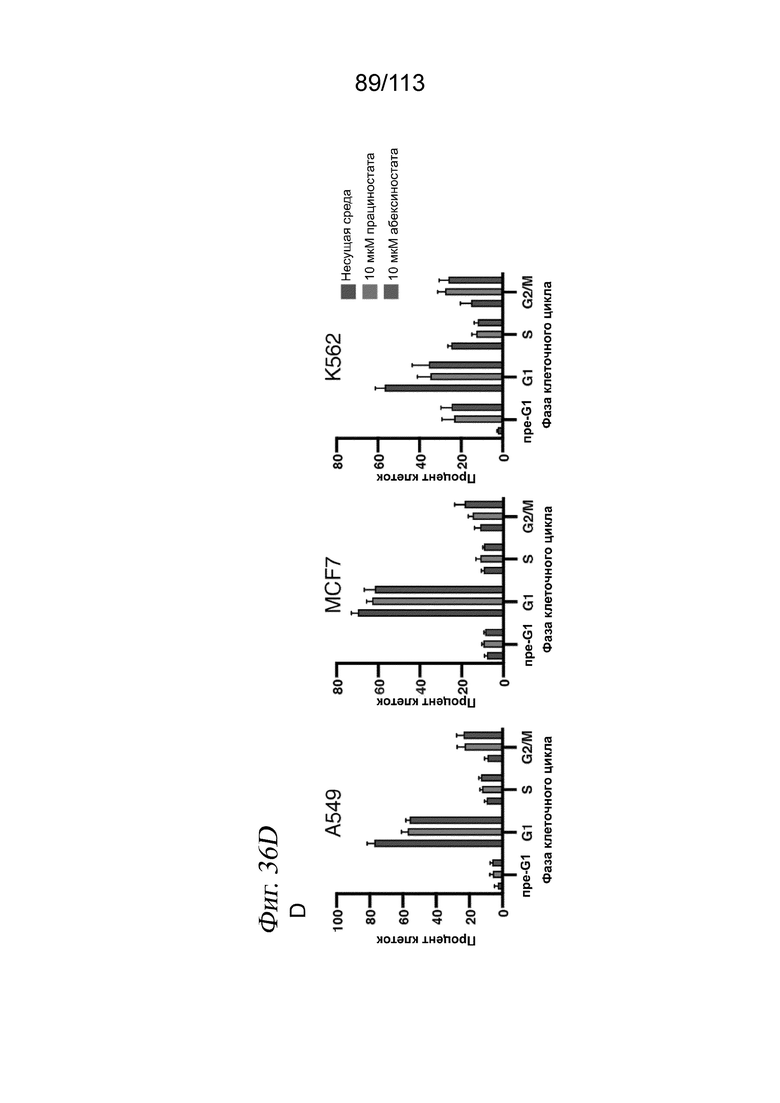
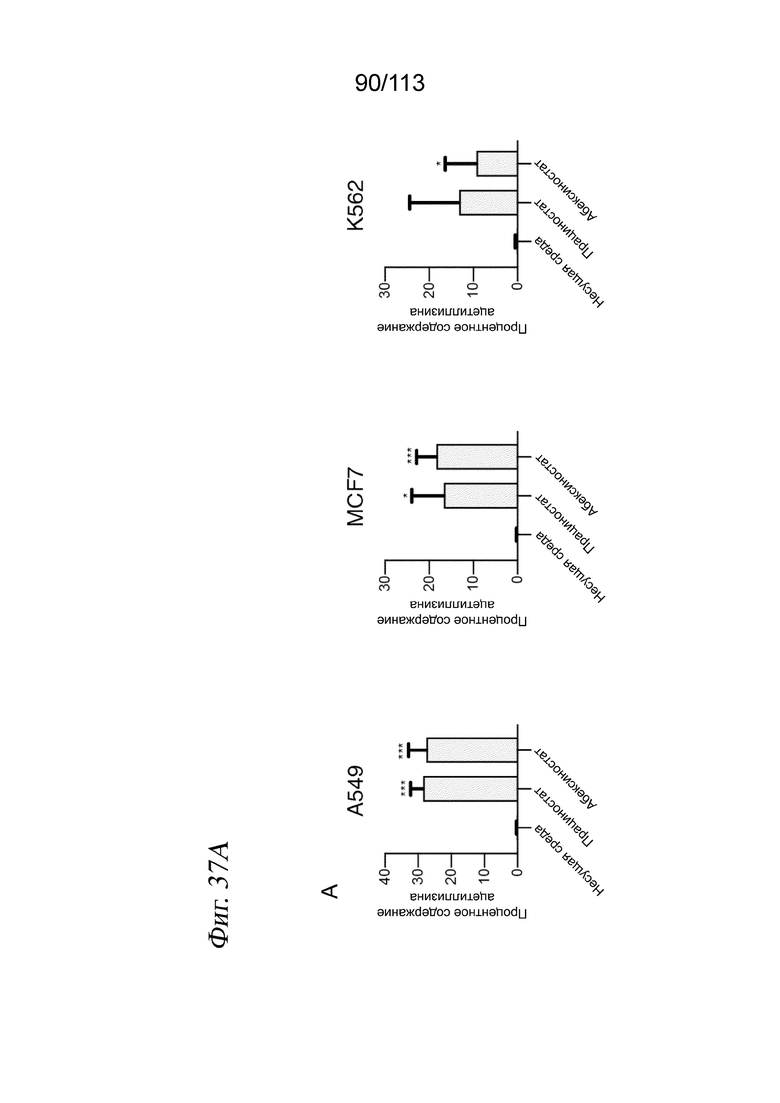
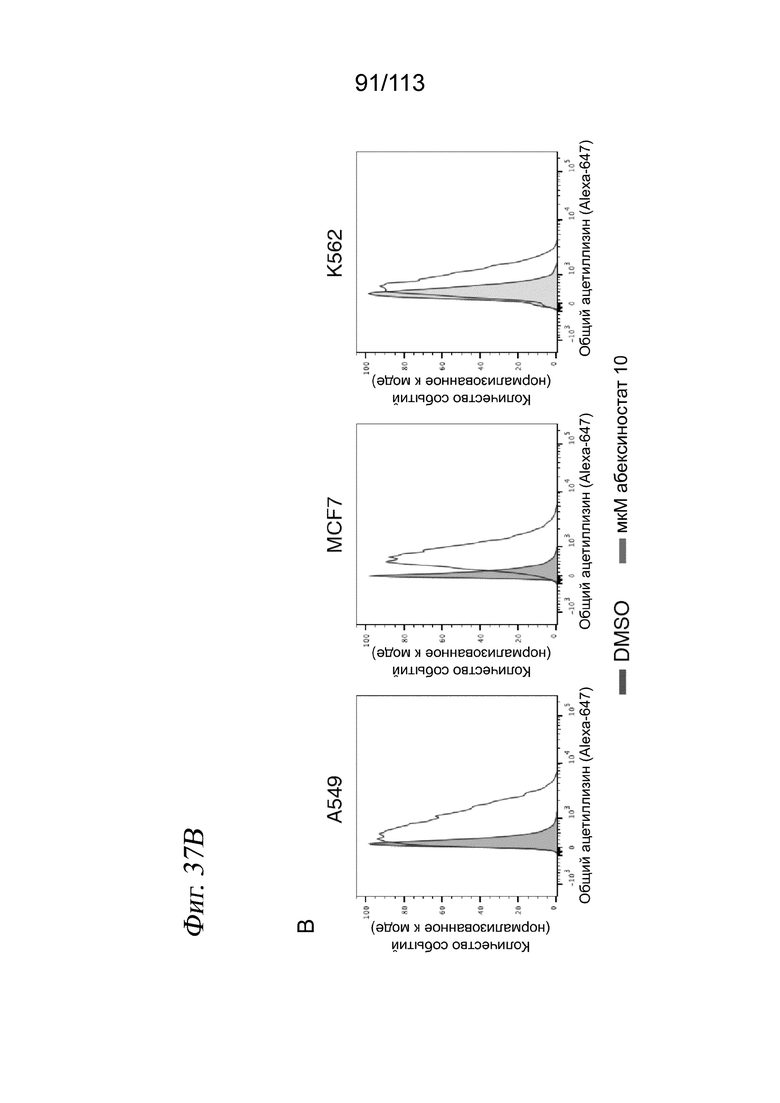
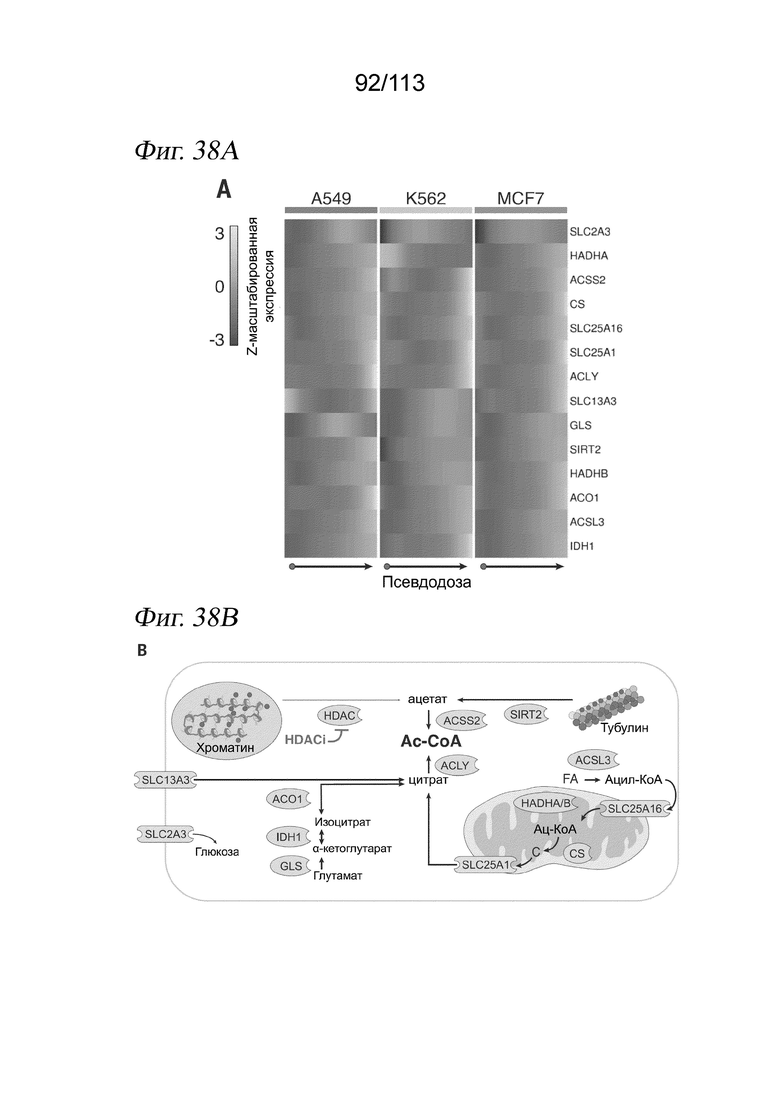
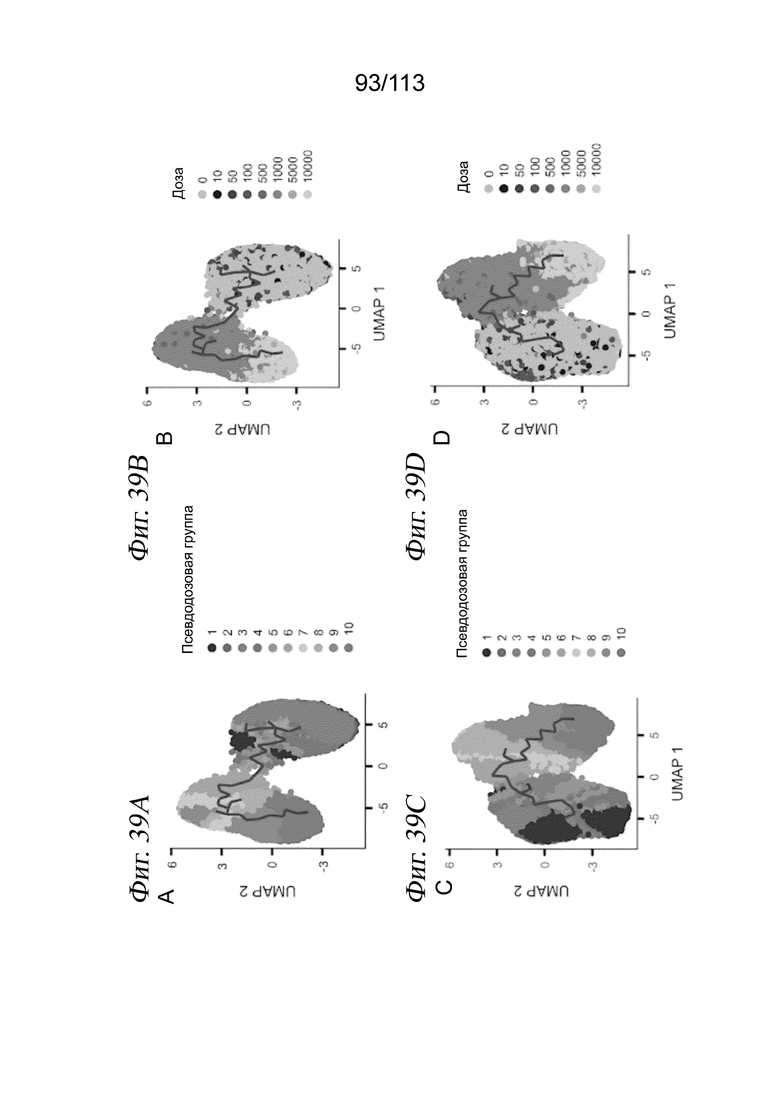
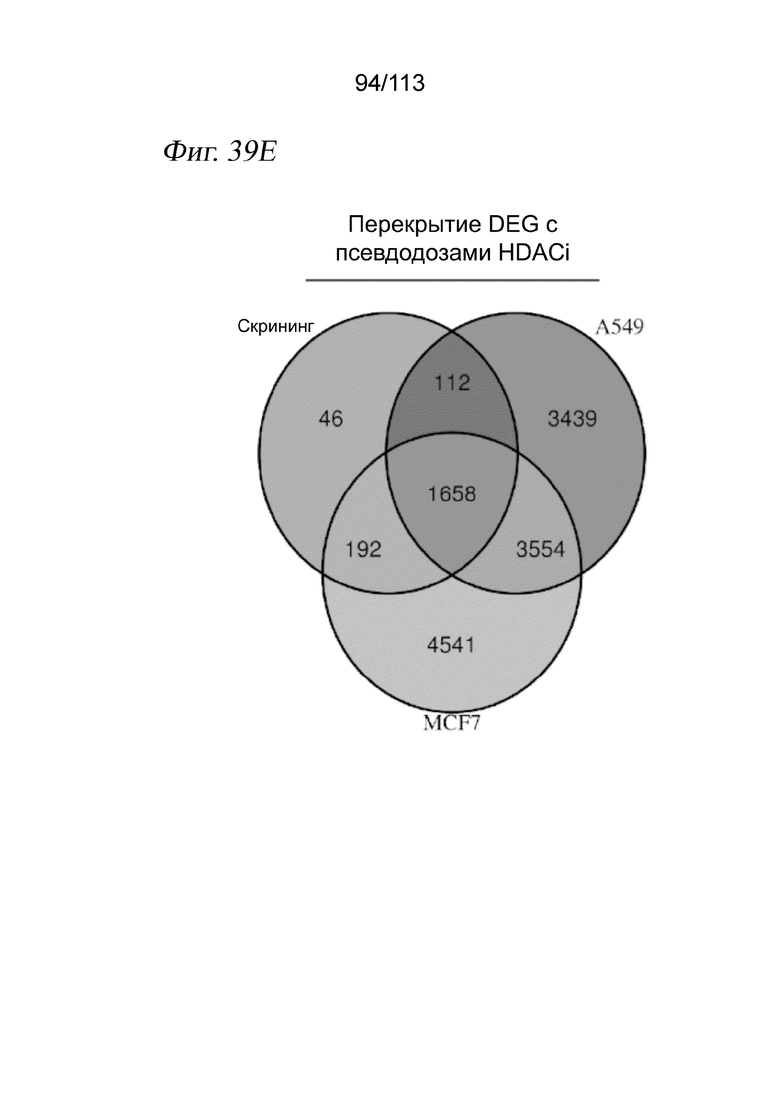
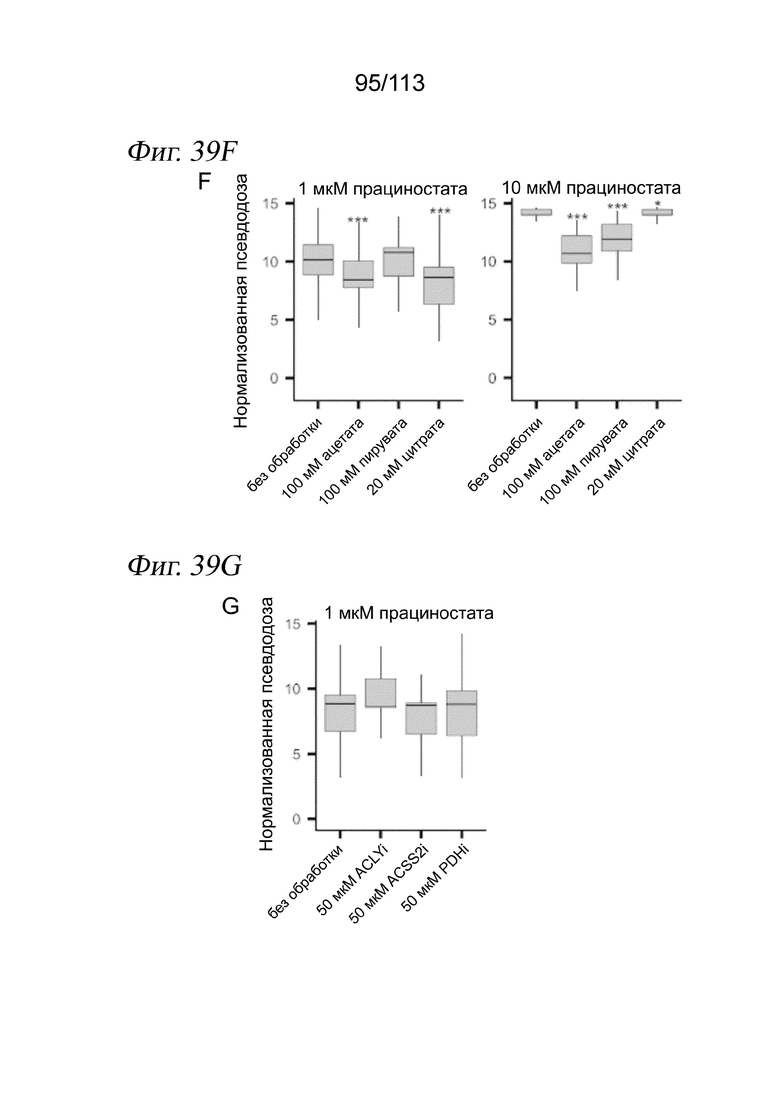
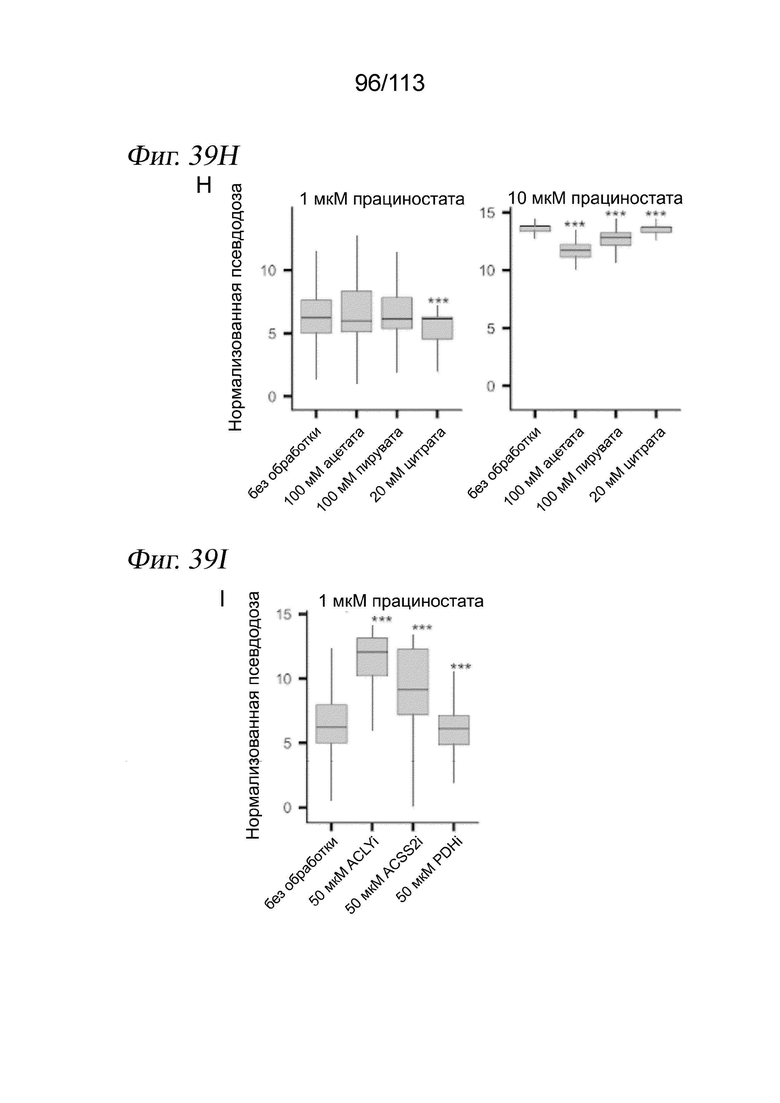


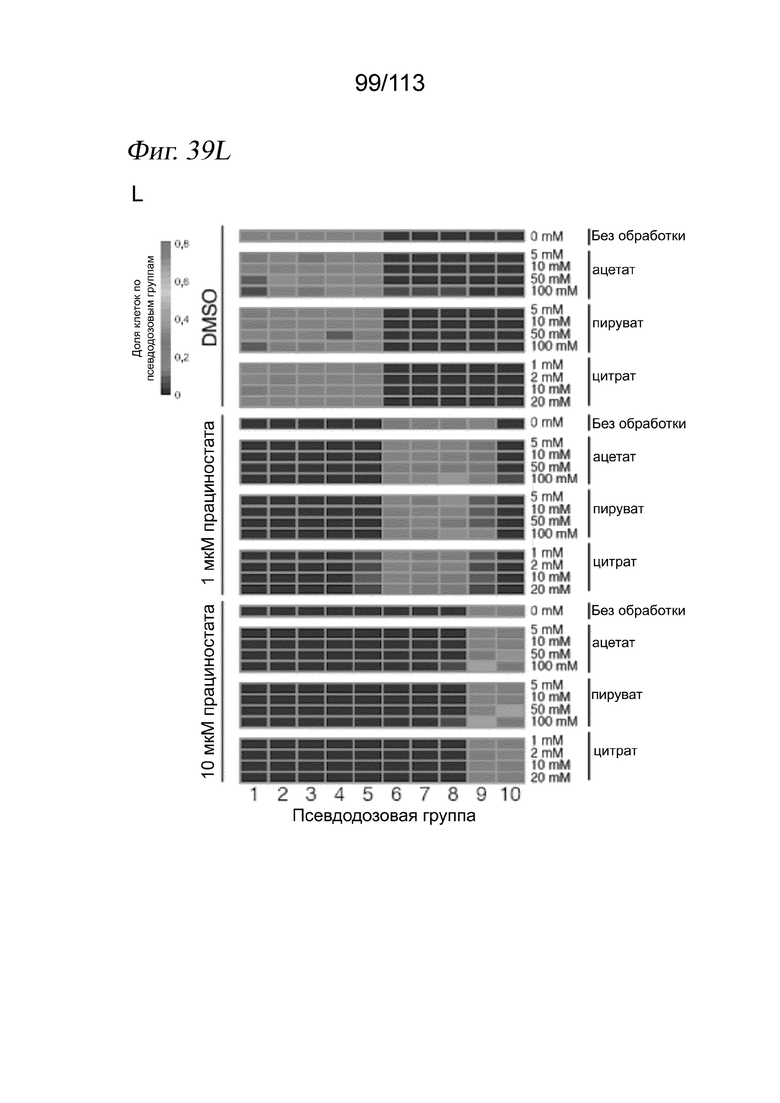

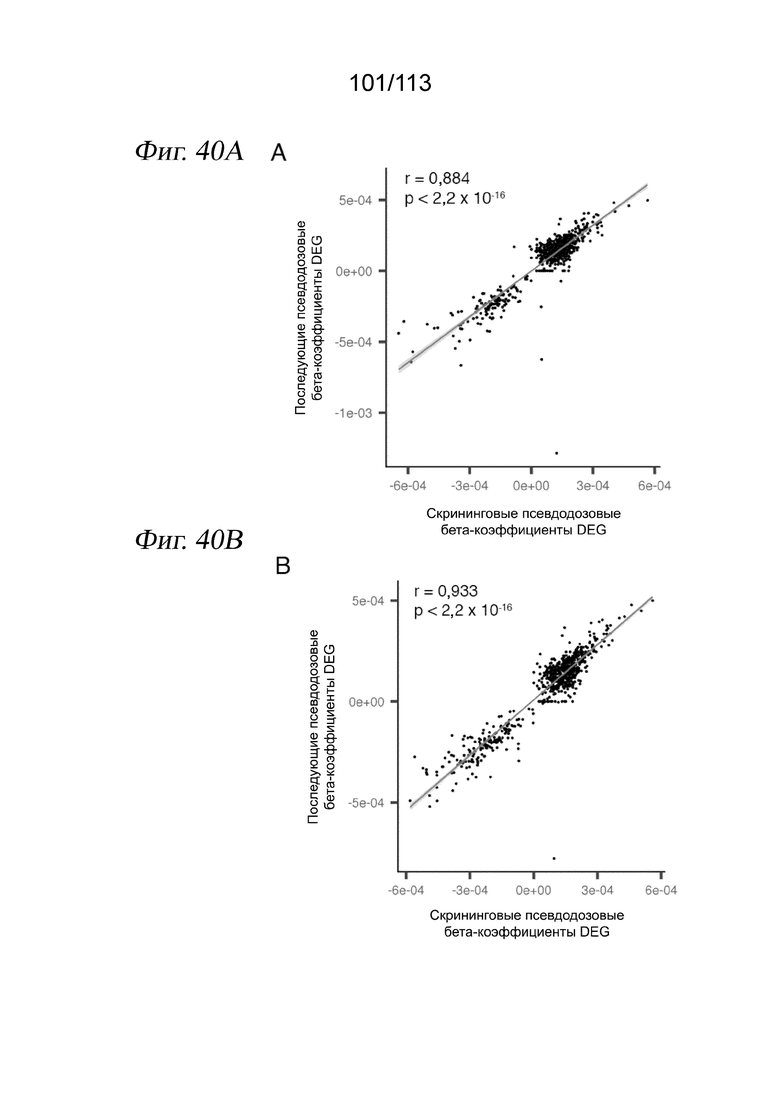
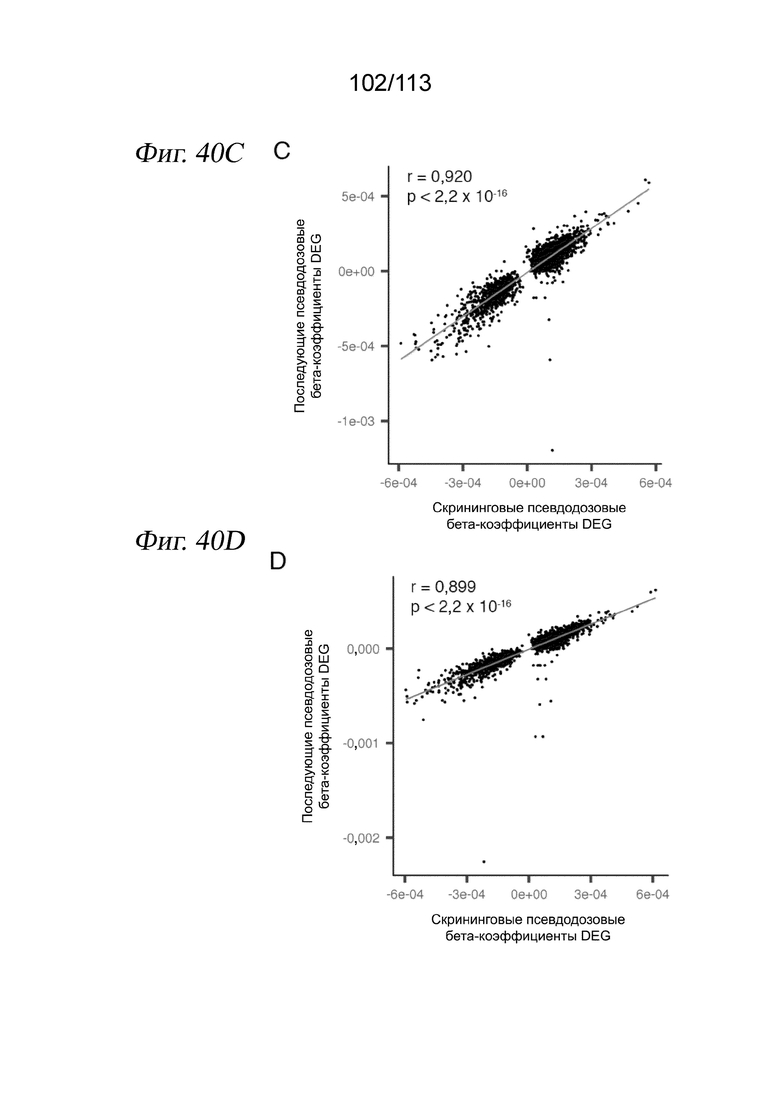
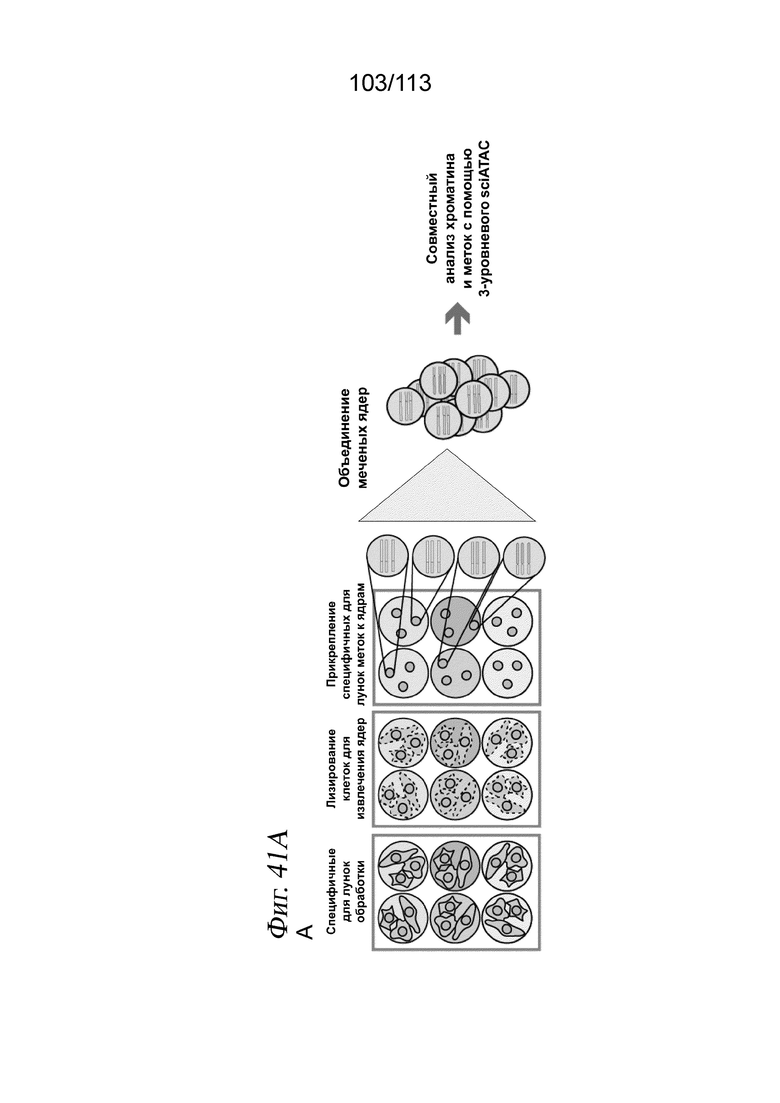
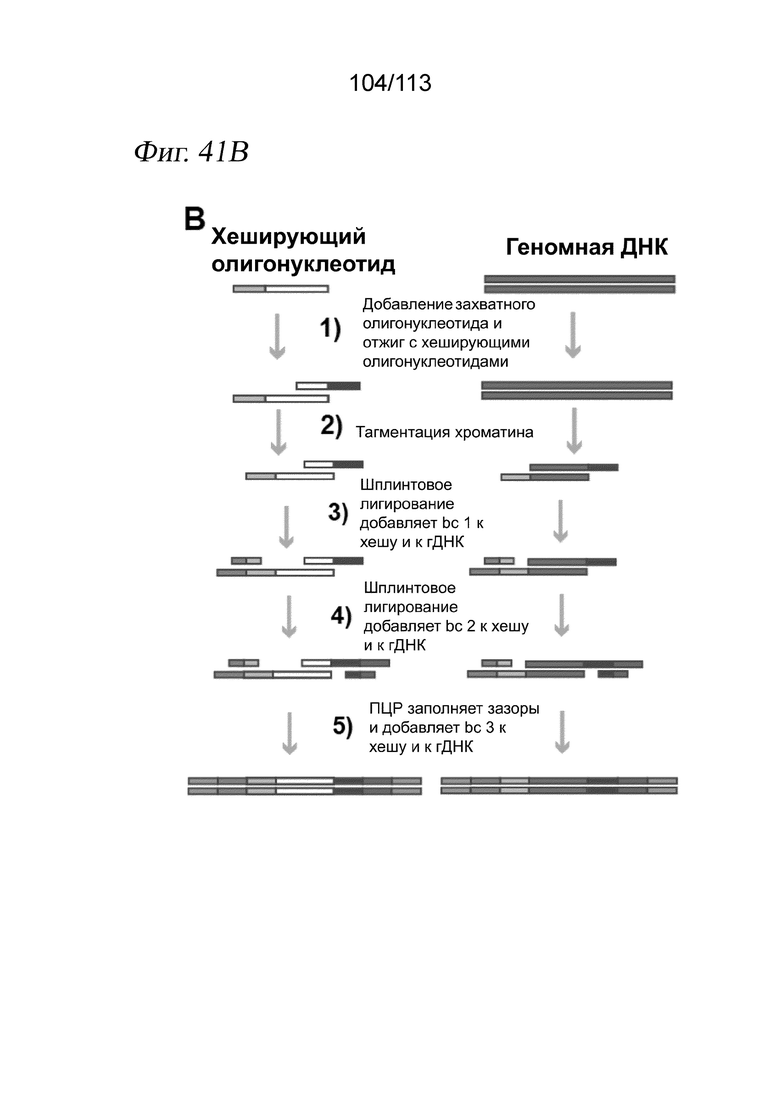
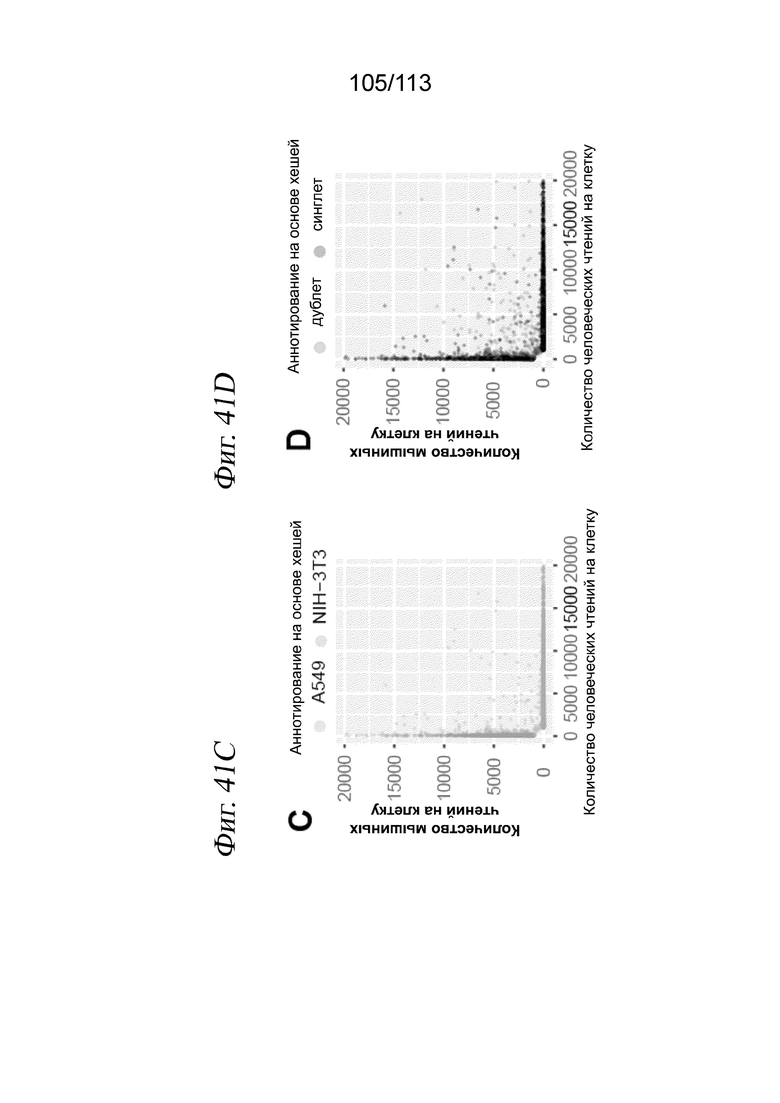
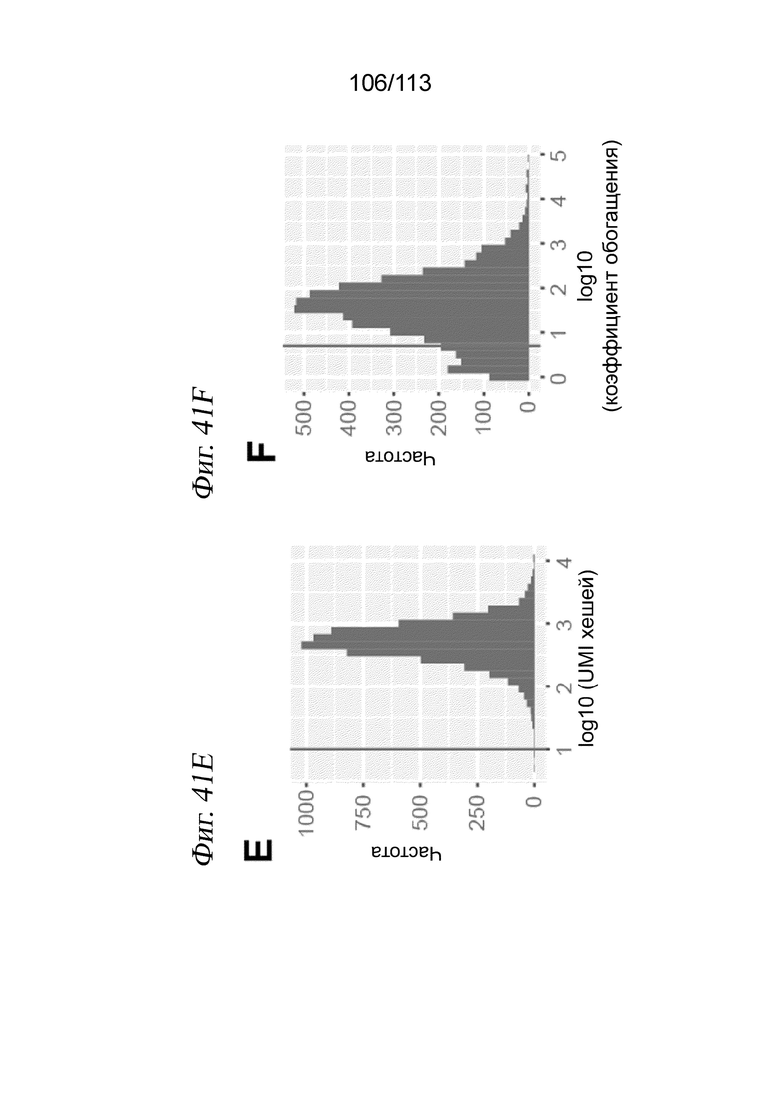
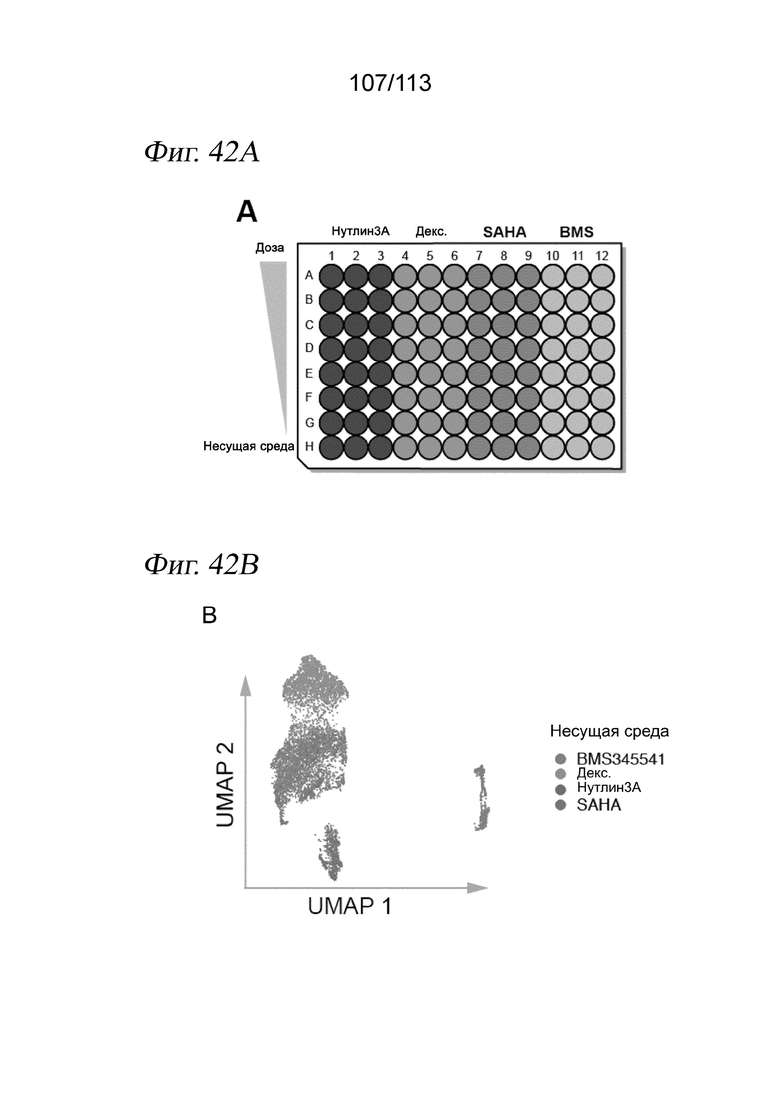
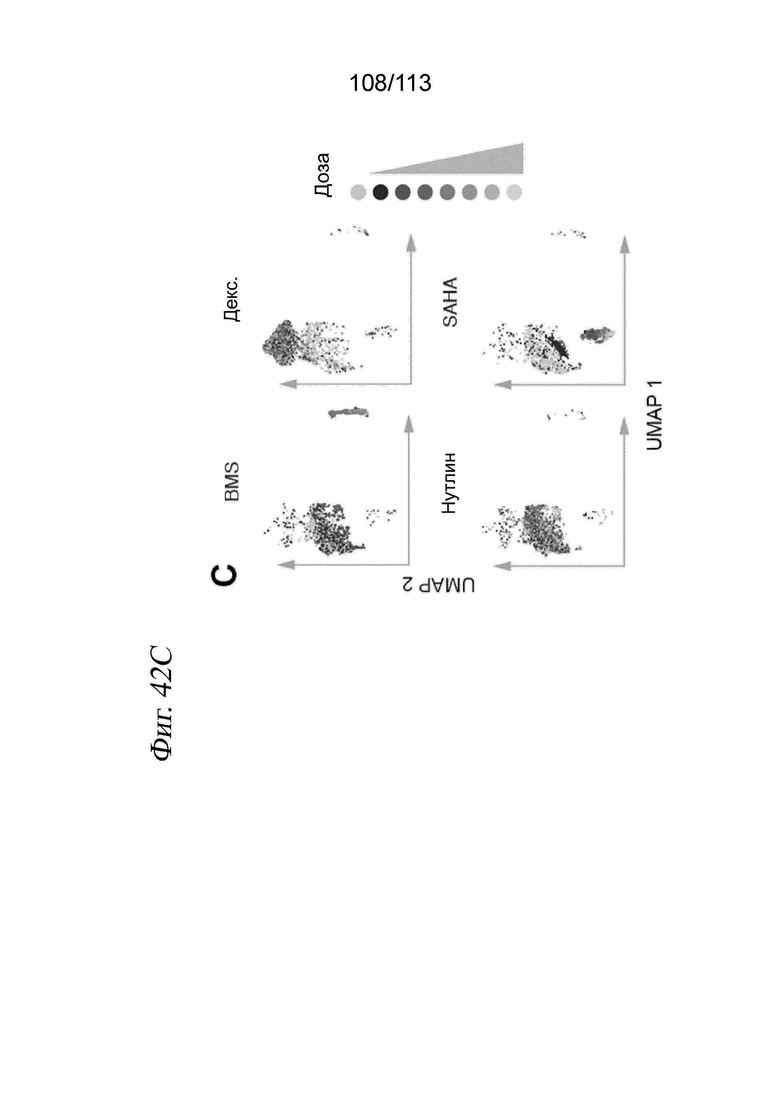
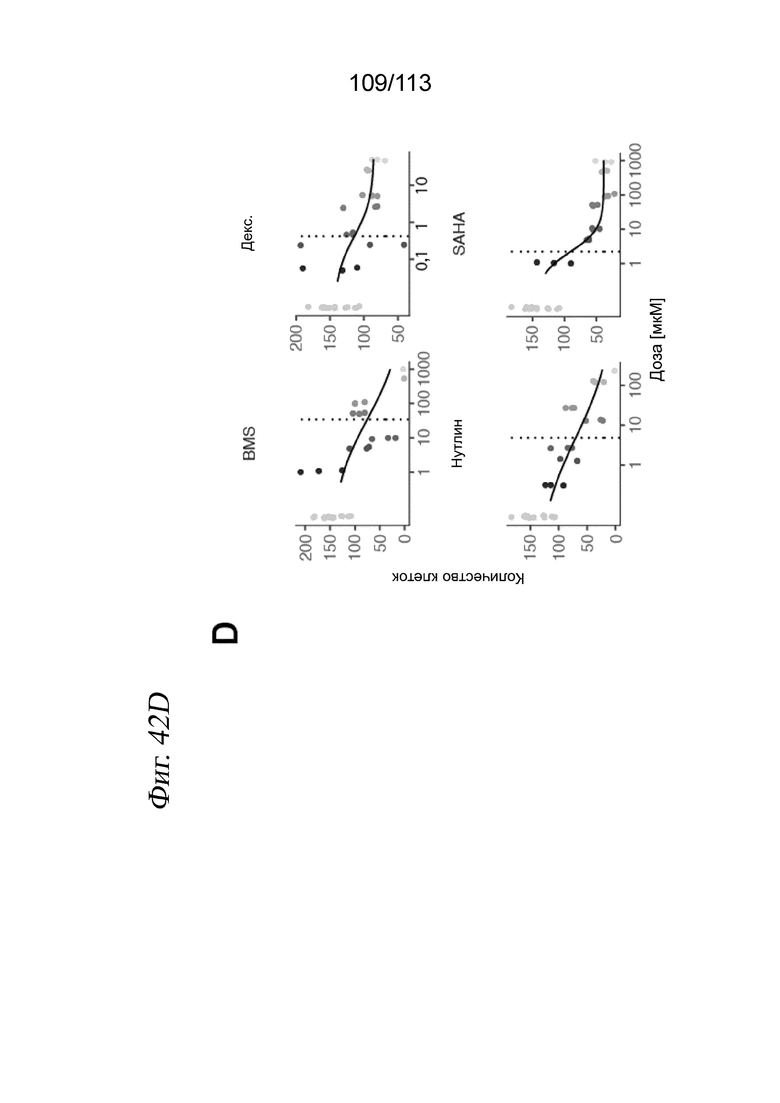

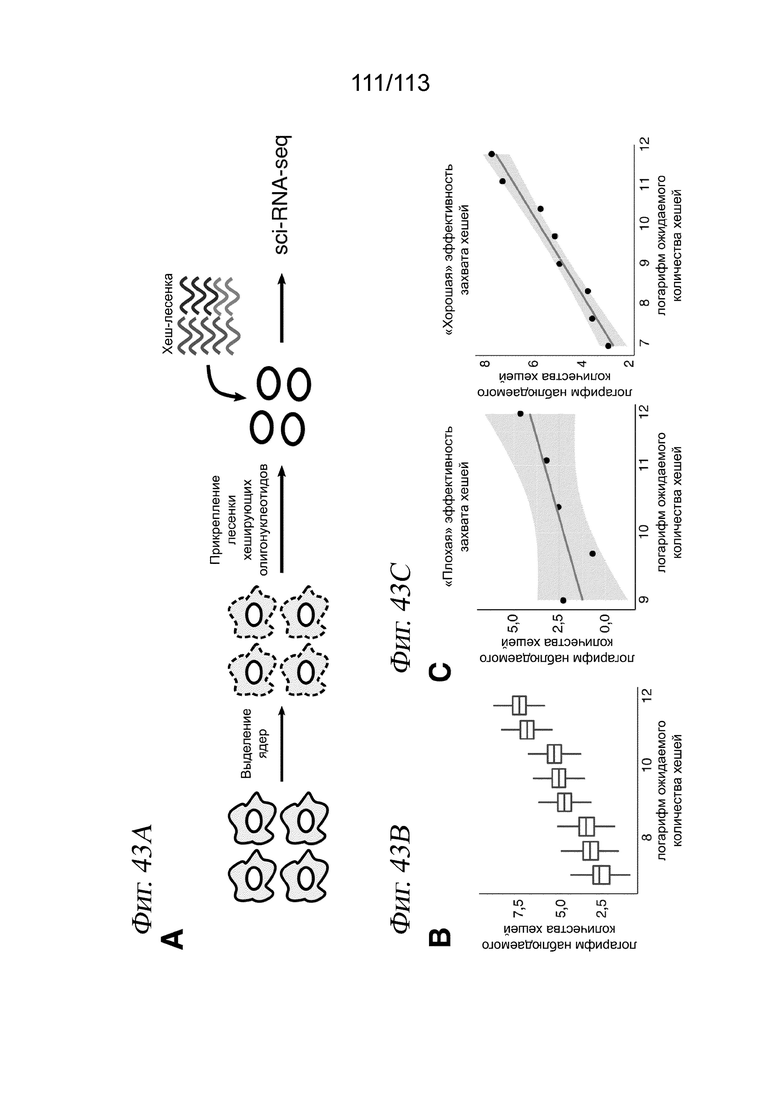
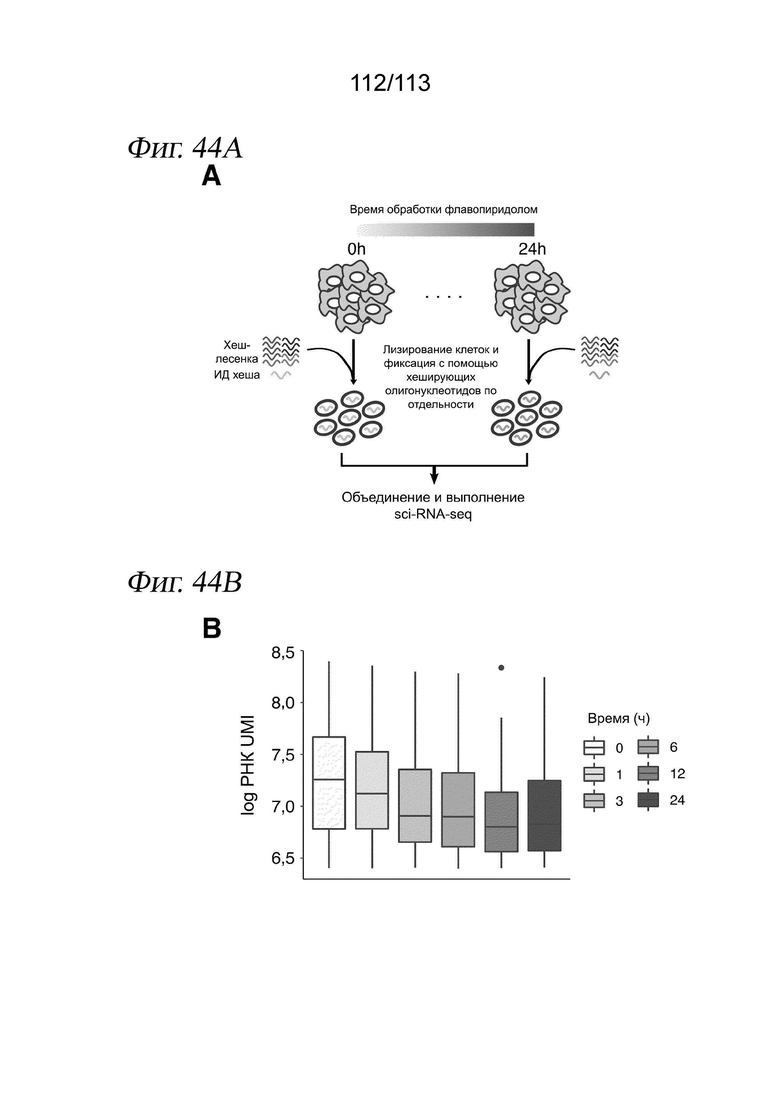
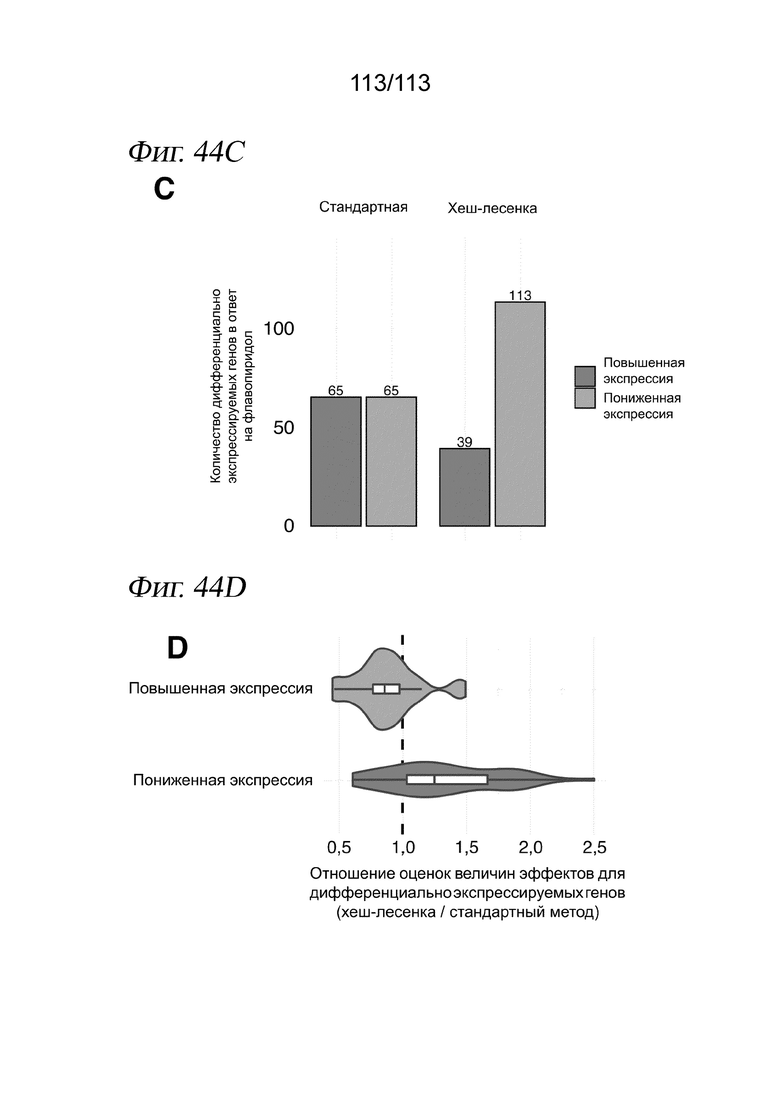
Настоящая группа изобретений относится к области биотехнологии. Раскрываются способы получения библиотек для секвенирования, включающих в себя нуклеиновые кислоты из множества одиночных клеток или ядер, меченных хеширующими олигонуклеотидами, причем связь между ними является неспецифической. Изобретение позволяет повысить производительность анализа образцов при HTS-скрининге. 6 н. и 23 з.п. ф-лы, 44 ил., 1 табл., 3 пр.
1. Способ получения библиотеки для секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение множества клеток во множестве компартментов;
(b) воздействие на множество клеток каждого компартмента заданным условием, где указанное заданное условие вызывает изменение клетки;
(c) приведение ядер, выделенных из клеток каждого компартмента, или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
где с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
где хеширующий олигонуклеотид содержит хеширующий индекс,
где хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток;
где связь между хеширующим олигонуклеотидом и выделенными ядрами или клетками является неспецифической;
(d) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток; и
(e) обработку объединенных хешированных клеток или объединенных хешированных ядер с использованием способа комбинаторного индексирования одиночных клеток с получением библиотеки для секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, где нуклеиновые кислоты содержат множество индексов.
2. Способ по п. 1, дополнительно включающий воздействие на клетки или ядра поперечно-сшивающим соединением для фиксации хеширующих олигонуклеотидов к клеткам или выделенным ядрам.
3. Способ по п. 2, в котором поперечно-сшивающее соединение содержит параформальдегид, формалин или метанол.
4. Способ по п. 1, в котором заданное условие включает в себя воздействие агента.
5. Способ по п. 1 или 4, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
6. Способ по п. 1, в котором хеширующий олигонуклеотид содержит одноцепочечную нуклеиновую кислоту.
7. Способ по п. 1, в котором хеширующий олигонуклеотид состоит из одноцепочечной нуклеиновой кислоты.
8. Способ по пп. 1, 6 или 7, в котором нуклеиновая кислота хеширующего олигонуклеотида содержит ДНК, РНК или их комбинацию.
9. Способ по п. 1, в котором хеширующий олигонуклеотид содержит домен, который опосредует специфическое связывание хеширующего олигонуклеотида с поверхностью клеток или ядер.
10. Способ по п. 9, в котором домен содержит лиганд, антитело или аптамер.
11. Способ по п. 1, в котором неспецифическая связь между хеширующим олигонуклеотидом и клетками или выделенными ядрами осуществляется посредством абсорбции.
12. Способ по п. 1, где способ комбинаторного индексирования одиночных клеток представляет собой секвенирование транскриптомов одиночных ядер, секвенирование транскриптомов одиночных клеток и секвенирование доступного для транспозона хроматина, полногеномное секвенирование в одиночных ядрах, секвенирование доступного для транспозона хроматина с в одиночных ядрах, sci-HiC, DRUG-seq, sci-CAR, sci-MET, sci-Crop, sci-perturb или sci-Crispr.
13. Способ по п. 12, дополнительно включающий:
(f) распределение подгрупп объединенных хешированных клеток или хешированных ядер во второе множество компартментов и приведение каждой подгруппы в контакт с обратной транскриптазой или ДНК-полимеразой и праймером, где праймер в каждом компартменте содержит первую индексную последовательность, которая отличается от первых индексных последовательностей в других компартментах, для получения индексированных ядер или индексированных клеток, содержащих индексированные фрагменты нуклеиновых кислот;
(g) объединение индексированных клеток или индексированных ядер для создания объединенных индексированных клеток или объединенных индексированных ядер;
(h) распределение подгрупп объединенных индексированных клеток или объединенных индексированных ядер в третье множество компартментов и введение второй индексной последовательности в индексированные фрагменты нуклеиновых кислот для создания клеток с двойным индексированием или ядер с двойным индексированием, содержащих фрагменты нуклеиновых кислот с двойным индексом, где введение включает в себя лигирование, удлинение праймера, амплификацию или транспонирование;
(i) объединение клеток с двойным индексированием или ядер с двойным индексированием для получения объединенных ядер или клеток с двойным индексом;
(j) распределение подгрупп клеток или объединенных ядер с двойным индексированием в четвертое множество компартментов и введение третьей индексной последовательности в индексированные фрагменты нуклеиновых кислот для создания клеток с тройным индексированием или ядер с тройным индексированием, содержащих фрагменты нуклеиновых кислот с тройным индексом, где введение включает в себя лигирование, удлинение праймера, амплификацию или транспонирование;
(k) объединение фрагментов с тройным индексом для получения таким образом библиотеки секвенирования, содержащей транскриптомные нуклеиновые кислоты из множества одиночных ядер.
14. Способ по п. 13, в котором (h) включает в себя приведение каждой подгруппы в контакт с транспосомным комплексом, где транспосомный комплекс в каждом компартменте содержит транспозазу и вторую индексную последовательность в условиях, подходящих для лигирования второй индексной последовательности к концам индексированных фрагментов нуклеиновых кислот, содержащих первую индексную последовательность, для получения ядер с двойным индексированием, содержащих фрагменты нуклеиновых кислот с двойным индексом, где вторая индексная последовательность отличается от вторых индексных последовательностей в других компартментах.
15. Способ по п. 13, в котором (j) включает в себя приведение каждой подгруппы в контакт с праймером, содержащим третью индексную последовательность и универсальную последовательность праймера, где приведение в контакт включает в себя условия, подходящие для амплификации и встраивания третьей индексной последовательности в концы фрагментов нуклеиновых кислот с двойным индексом, где третья индексная последовательность отличается от третьих индексных последовательностей в других компартментах.
16. Способ по п. 1, в котором компартменты содержат лунку или каплю.
17. Способ по пп. 13-15, дополнительно включающий:
получение поверхности, содержащей множество сайтов амплификации,
где сайты амплификации содержат по меньшей мере две популяции присоединенных одноцепочечных захватных олигонуклеотидов, имеющих свободный 3’-конец, и
приведение поверхности, содержащей сайты амплификации, в контакт с фрагментами с тройным индексом в условиях, подходящих для получения множества сайтов амплификации, каждый из которых содержит клональную популяцию ампликонов из отдельного фрагмента, содержащего множество индексов.
18. Композиция для применения в получении библиотеки для секвенирования, содержащая хешированные клетки или хешированные ядра,
где указанные хешированные клетки или хешированные ядра присутствуют во множестве компартментов,
где указанные хешированные клетки или хешированные ядра содержат по меньшей мере одну копию связанного хеширующего олигонуклеотида,
где указанный хеширующий олигонуклеотид содержит хеширующий индекс, и
где указанный хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах.
19. Композиция для применения в получении библиотеки для секвенирования, содержащая объединенные хешированные клетки или объединенные хешированные ядра,
где указанные объединенные хешированные клетки или объединенные хешированные ядра присутствуют во множестве компартментов,
где хешированные клетки указанных объединенных хешированных клеток или хешированные ядра указанных объединенных хешированных ядер содержат по меньшей мере одну копию связанного хеширующего олигонуклеотида,
где указанный хеширующий олигонуклеотид содержит хеширующий индекс, и
где указанный хеширующий индекс в каждом компартменте содержит смесь индексных последовательностей.
20. Композиция по п. 18 или 19, где указанное множество компартментов содержится в многолуночном планшете.
21. Композиция по п. 20, где компартмент указанного многолуночного планшета содержит от 50 до 100000000 клеток или ядер.
22. Композиция по п. 18 или 19, где указанную композицию содержит капля.
23. Композиция по п. 22, где капля содержит от 50 до 100000000 клеток или ядер.
24. Способ получения объединенных хешированных ядер или объединенных хешированных клеток, содержащих нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение множества компартментов, содержащих выделенные ядра или клетки, и приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
где с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида,
где хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
где хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток;
где связь между хеширующим олигонуклеотидом и выделенными ядрами или клетками является неспецифической; и
(b) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток.
25. Способ получения объединенных хешированных ядер или объединенных хешированных клеток, содержащих нуклеиновые кислоты из множества одиночных ядер или одиночных клеток, включающий:
(a) получение множества компартментов, содержащих выделенные ядра или клетки, и приведение выделенных ядер или клеток каждого компартмента в контакт с хеширующим олигонуклеотидом,
где с выделенными ядрами или клетками связана по меньшей мере одна копия хеширующего олигонуклеотида посредством абсорбции,
где хеширующий олигонуклеотид содержит нуклеиновую кислоту и хеширующий индекс,
где хеширующий индекс в каждом компартменте содержит индексную последовательность, которая отличается от индексных последовательностей в других компартментах, для получения хешированных ядер или хешированных клеток; и
(b) объединение хешированных ядер или хешированных клеток из разных компартментов для получения объединенных хешированных ядер или объединенных хешированных клеток.
26. Способ получения объединенных хешированных ядер или объединенных хешированных клеток, содержащих нуклеиновые кислоты из множества ядер или клеток, включающий:
(a) получение множества компартментов, содержащих ядра или клетки, где ядра или клетки содержат хеширующий олигонуклеотид, который содержит специфичный для компартмента индекс, где хеширующий олигонуклеотид связан с выделенными ядрами или клетками посредством неспецифической связи;
(b) объединение ядер или клеток из разных компартментов во втором компартменте для получения объединенных хешированных ядер или объединенных хешированных клеток.
27. Способ по п. 26, дополнительно включающий воздействие на клетки каждого компартмента заданного условия или воздействие на клетки каждого компартмента заданного условия с последующим выделением ядер из множества клеток перед стадией (a).
28. Способ по п. 27, в котором заданное условие включает в себя воздействие агента.
29. Способ по п. 28, в котором агент содержит белок, нерибосомный белок, поликетид, органическую молекулу, неорганическую молекулу, молекулу РНК или молекулу, вызывающую РНК-интерференцию, углевод, гликопротеин, нуклеиновую кислоту, лекарственное средство или их комбинацию.
| STOECKIUS M | |||
| et al | |||
| Cell Hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics, Genome Biology, 2018, v | |||
| Способ изготовления электрических сопротивлений посредством осаждения слоя проводника на поверхности изолятора | 1921 |
|
SU19A1 |
| STOECKIUS M | |||
| et al | |||
| Simultaneous epitope and transcriptome measurement in single cells, Nature Methods, 2017, v | |||
| Паровоз для отопления неспекающейся каменноугольной мелочью | 1916 |
|
SU14A1 |
| BIRNBAUM K.D | |||
| Power in Numbers: Single-Cell RNA-Seq | |||
Авторы
Даты
2025-04-21—Публикация
2020-03-02—Подача