[0001] ПЕРЕКРЕСТНЫЕ ССЫЛКИ НА РОДСТВЕННЫЕ ЗАЯВКИ
[0002] Настоящая заявка испрашивает преимущество по предварительной заявке США № 62/950,670, поданной 19 декабря 2019 г., которая полностью включена в настоящий документ путем ссылки
[0003] ФИНАНСИРОВАНИЕ ЗА СЧЕТ ГОСУДАРСТВЕННЫХ СРЕДСТВ
[0004] Настоящее изобретение выполнено при государственной поддержке, грант № T32 HL007828 Национальных институтов здравоохранения. Государство обладает определенными правами на изобретение.
[0005] ОБЛАСТЬ ИЗОБРЕТЕНИЯ
[0006] Варианты осуществления настоящего описания относятся к секвенированию нуклеиновых кислот. В частности, варианты осуществления способов и композиций, предложенных в настоящем документе, относятся к получению комбинаторных индексированных библиотек секвенирования одиночных клеток и получению из них данных о последовательности. В некоторых вариантах осуществления данные о последовательности, полученные из библиотек, являются исчерпывающими, а в других вариантах осуществления данные о последовательности, полученные из библиотек, позволяют охарактеризовать редкие события.
[0007] ПРЕДПОСЫЛКИ СОЗДАНИЯ ИЗОБРЕТЕНИЯ
[0008] Комбинаторное индексирование одиночных клеток (sci-) представляет собой методологическую базу, в которой для уникального маркирования содержимого в виде нуклеиновых кислот больших количеств одиночных клеток или ядер используют штрихкодирование с разделением пула для получения комбинаторных библиотек секвенирования одиночных клеток. Современные геномные методики одиночной клетки часто включают применение транспосомного комплекса для добавления уникальной метки на одной стадии; однако для этого требуется большое количество индивидуальных модифицированных транспозонов.
[0009] Методики определения генома одиночной клетки позволяют устранять клеточные различия, которые трудно определить при изучении основной популяции клеток. Во многих важных сферах применения, таких как онкология, иммунология и метагеномика, большой интерес и сложность представляет определение характеристик редких клеток. Современные способы секвенирования одиночных клеток позволяют параллельно охарактеризовать миллионы одиночных клеток; однако характеристика на основе комплексного секвенирования редких клеток в популяции без обогащения является дорогостоящей и сложной задачей.
[0010] ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
[0011] В настоящем документе предложены способы применения транспосомного комплекса во время комбинаторного индексирования одиночной клетки без необходимости получения индивидуальных модифицированных транспозонов.
[0012] В одном варианте осуществления в настоящем описании предложен способ получения библиотеки секвенирования, которая включает нуклеиновые кислоты из множества одиночных ядер или клеток. Способ включает обеспечение множества ядер или клеток, причем ядра или клетки включают нуклеосомы, и приведение множества ядер или клеток в контакт с транспосомным комплексом, который включает транспозазу и универсальную последовательность. В одном варианте осуществления при контакте с транспосомным комплексом множество ядер или клеток находятся в массе, а в другом варианте осуществления при контакте с транспосомным комплексом множество ядер или клеток распределено в первом множестве компартментов, причем каждый компартмент включает подмножество ядер или клеток или представляет собой образец. Приведение в контакт дополнительно включает условия, подходящие для встраивания универсальной последовательности в нуклеиновые кислоты ДНК, что приводит к образованию двухцепочечных нуклеиновых кислот ДНК, которые включают универсальную последовательность. В тех вариантах осуществления, в которых приведение в контакт происходит со множеством ядер или клеток, находящихся в массе, способ также включает распределение множества ядер или клеток в первое множество компартментов, причем каждый компартмент включает подгруппу ядер или клеток. Молекулы ДНК в каждой подгруппе ядер или клеток обрабатывают с получением индексированных ядер или клеток. Процессинг включает добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, первой индексной последовательности, специфичной для компартмента, с получением индексированных нуклеиновых кислот, присутствующих в индексированных ядрах или клетках. Процессинг может включать лигирование, достройку праймера, гибридизацию, амплификацию или их комбинацию. Индексированные ядра или клетки можно объединять для получения объединенных индексированных ядер или клеток.
[0013] В одном варианте осуществления обеспечение может включать обеспечение множества ядер или клеток во множестве компартментов, причем каждый компартмент включает подмножество ядер или клеток или представляет собой образец. Приведение в контакт может включать приведение каждого компартмента в контакт с транспосомным комплексом, и способ может дополнительно включать объединение ядер или клеток после приведения в контакт с получением объединенных ядер или клеток.
[0014] В одном варианте осуществления приведение в контакт включает приведение каждой подгруппы в контакт с двумя транспосомными комплексами, причем один транспосомный комплекс включает первую транспозазу, включающую первую универсальную последовательность, а второй транспосомный комплекс включает вторую транспозазу, включающую вторую универсальную последовательность, причем приведение в контакт дополнительно включает условия, подходящие для встраивания первой универсальной последовательности и второй универсальной последовательности в нуклеиновые кислоты ДНК, что приводит к образованию двухцепочечных нуклеиновых кислот ДНК, включающих первую и вторую универсальные последовательности.
[0015] В одном варианте осуществления способ может дополнительно включать распределение объединенных индексированных ядер или клеток, которые включают индексированные ядра или клетки, во второе множество компартментов, причем каждый компартмент включает подгруппу ядер или клеток, и процессинг молекул ДНК в каждой подгруппе ядер или клеток для получения ядер или клеток с двойным индексированием. Процессинг может включать добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, второй индексной последовательности, специфичной для компартмента, с получением нуклеиновых кислот с двойным индексированием, присутствующих в индексированных ядрах или клетках. Способ может включать объединение ядер или клеток с двойным индексированием для получения объединенных ядер или клеток с двойным индексированием.
[0016] В одном варианте осуществления способ может дополнительно включать распределение объединенных индексированных ядер или клеток, которые включают ядра или клетки с двойным индексированием, в третье множество компартментов, причем каждый компартмент включает подгруппу ядер или клеток, и процессинг молекул ДНК в каждой подгруппе ядер или клеток для получения ядер или клеток с тройным индексированием. Процессинг может включать добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, третьей индексной последовательности, специфичной для компартмента, с получением нуклеиновых кислот с тройным индексированием, присутствующих в индексированных ядрах или клетках. Способ может включать объединение ядер или клеток с тройным индексированием для получения объединенных ядер или клеток с тройным индексированием.
[0017] В одном варианте осуществления способ может дополнительно включать получение индексированных нуклеиновых кислот (например, с двойным индексированием, с тройным индексированием и т. п.) из объединенных индексированных ядер или клеток, и таким образом получают библиотеку секвенирования из множества ядер или клеток.
[0018] В настоящем документе также предложены способы определения и/или характеристики субпопуляции клеток. В одном варианте осуществления способ включает обеспечение библиотеки секвенирования, такой как комбинаторная библиотека секвенирования одиночных клеток. Необязательно библиотеку секвенирования получают из популяции клеток или ядер, наделенных свойством. Способ может включать анализ библиотеки секвенирования посредством целенаправленного секвенирования. Целенаправленное секвенирование может быть основано на биологическом признаке, который обычно присутствует в небольшой части клеток, использованных для создания библиотеки. Примеры биологического признака включают, без ограничений, нуклеотидную последовательность, указывающую на класс клеток, видовой тип или состояние заболевания. В дополнение к целенаправленному секвенированию биологического признака секвенирование также включает определение последовательности индексных последовательностей, которые присутствуют на той же модифицированной целевой нуклеиновой кислоте, что и биологический признак. Результатом является идентификация членов библиотеки секвенирования, которые происходят из тех же клеток или ядер, что и члены библиотеки, которые включают биологический признак. Способ дополнительно включает изменение библиотеки секвенирования для увеличения представления тех членов, которые происходят из тех же клеток или ядер, что и члены библиотеки, которые включают биологический признак. Изменение может включать обогащение желаемых членов библиотеки секвенирования или истощение нежелательных членов библиотеки секвенирования с получением подбиблиотеки.
[0019] Определения
[0020] Если не указано иное, следует понимать, что термины, используемые в настоящем документе, принимают свое обычное значение в соответствующей области. Ниже приведен ряд терминов, используемых в настоящем документе, и их значения.
[0021] В настоящем документе термины «организм», «субъект» используются взаимозаменяемо и относятся к микроорганизмам (например, прокариотическим или эукариотическим), животным и растениям. Примером животного является млекопитающее, такое как человек.
[0022] В настоящем документе термин «тип клеток» предназначен для обозначения клеток на основе морфологии, фенотипа, источника развития или других известных или распознаваемых отличительных характеристик клеток. Из одного организма (или одного вида организмов) можно получить множество разных типов клеток. К примерам типов клеток относятся, без ограничений, гаметы (включая женские гаметы, например яйцеклетки или оотиды, и мужские гаметы, например сперма), эпителий яичников, фибробласты яичников, яичек, мочевого пузыря, иммунные клетки, В-клетки, Т-клетки, естественные клетки-киллеры, дендритные клетки, раковые клетки, эукариотические клетки, стволовые клетки, клетки крови, мышечные клетки, жировые клетки, клетки кожи, нервные клетки, костные клетки, клетки поджелудочной железы, эндотелиальные клетки, эпителий поджелудочной железы, альфа-клетки поджелудочной железы, бета-клетки поджелудочной железы, эндотелиальные клетки поджелудочной железы, лимфобласты костного мозга, В-лимфобласты костного мозга, макрофаги костного мозга, эритробласты костного мозга, дендритные клетки костного мозга, адипоциты костного мозга, остеоциты костного мозга, хондроциты костного мозга, промиелобласты, мегакариобласты костного мозга, клетки мочевого пузыря, В-лимфоциты головного мозга, глиальные клетки головного мозга, нейроны, астроциты головного мозга, нейроэктодерма, макрофаги головного мозга, микроглия головного мозга, эпителий головного мозга, кортикальные нейроны, фибробласты головного мозга, эпителий молочной железы, эпителий толстой кишки, В-лимфоциты толстой кишки, эпителий молочной железы, миоэпителий молочной железы, фибробласт молочной железы, энтероциты толстой кишки, эпителий шейки матки, эпителий протоков молочной железы, эпителий языка, дендритные клетки миндалин, B-лимфоциты миндалин, лимфобласты периферической крови, Т-лимфобласты периферической крови, кожные Т-лимфоциты периферической крови, естественные клетки-киллеры периферической крови, В-лимфобласты периферической крови, моноциты периферической крови, миелобласты периферической крови, монобласты периферической крови, промиелобласты периферической крови, макрофаги периферической крови, базофилы периферической крови, эндотелий печени, тучные клетки печени, эпителий печени, B-лимфоциты печени, эндотелий селезенки, эпителий селезенки, B-лимфоциты селезенки, гепатоциты печени, фибробласты печени, эпителий легких, эпителий бронхов, фибробласты легких, В-лимфоциты легких, шванновские клетки легких, плоские клетки легких, макрофаги легких, остеобласты легких, нейроэндокринные клетки, альвеолярные клетки легких, эпителий желудка и фибробласты желудка. В одном варианте осуществления ряд различных типов клеток, полученных из одного организма, может включать клетки организма и другие клетки, такие как клетки симбиотических или патогенных микроорганизмов, связанных с организмом. Примеры симбиотических или патогенных микроорганизмов, связанных с организмом, включают, без ограничений, прокариотические и эукариотические микроорганизмы, присутствующие в образце микробиома из организма или присутствующие в ткани, и необязательно вызывающие заболевание.
[0023] В настоящем документе термин «ткань» обозначает набор или агрегат клеток, которые действуют совместно и выполняют одну или более конкретных функций в организме. Клетки необязательно могут быть морфологически аналогичными. К примерам тканей относятся, без ограничений, эмбриональные ткани, придаток яичка, глаз, мышцы, кожа, сухожилие, вена, артерия, кровь, сердце, селезенка, лимфатический узел, кость, костный мозг, легкое, бронхи, трахея, кишечник, тонкая кишка, толстый кишечник, ободочная кишка, прямая кишка, слюнная железа, язык, желчный пузырь, аппендикс, печень, поджелудочная железа, головной мозг, желудок, кожа, почка, мочеточник, мочевой пузырь, уретра, гонада, яичко, яичник, матка, фаллопиева труба, тимус, гипофиз, щитовидная железа, надпочечник или паращитовидная железа. Ткань может быть получена из любых из различных органов человека или другого организма. Ткань может быть здоровой тканью или нездоровой тканью. К примерам нездоровых тканей относятся, без ограничений, злокачественные образования репродуктивной ткани, легких, молочной железы, ободочной и прямой кишки, предстательной железы, носоглотки, желудка, яичек, кожи, нервной системы, костей, яичников, печени, гематологических тканей, поджелудочной железы, матки, почек, лимфоидных тканей и т. д. Злокачественные образования могут относиться к различным гистологическим подтипам, например, к карциноме, аденокарциноме, саркоме, фиброаденокарциноме, нейроэндокринным или недифференцированным образованиям.
[0024] В настоящем документе термин «образец» и его производные используются в самом широком смысле и включают любую пробу, культуру и т. п., которые, предположительно, включают целевую нуклеиновую кислоту и/или белок-мишень. В некоторых вариантах осуществления образец содержит ДНК, РНК, белок или их комбинацию. Образец может включать любую биологическую, клиническую, хирургическую, сельскохозяйственную, атмосферную или водную пробу, содержащую одну или более нуклеиновых кислот и/или один или более белков. Термин также включает любую выделенную нуклеиновую кислоту из образца, такого как геномная ДНК или транскриптом, и любой выделенный белок из образца. В некоторых вариантах осуществления образец включает набор клеток или ядер.
[0025] В настоящем документе термин «компартмент» обозначает область или объем, который отделяет или изолирует что-либо от других объектов. К примерам компартментов относятся, без ограничений, флаконы, пробирки, лунки, капли, болюсы, гранулы, сосуды, поверхностные элементы или области или объемы, разделенные физическими силами, такими как поток текучей среды, магнетизм, электрический ток или т. п. В одном из вариантов осуществления компартмент представляет собой лунку многолуночного планшета, такого как 96-луночный или 384-луночный планшет. В одном варианте осуществления компартмент представляет собой лунку (например, микролунку или нанолунку) рельефной поверхности. В настоящем документе капля может включать гидрогелевую гранулу, которая представляет собой гранулу для инкапсуляции одного или более ядер или клеток, и содержит гидрогелевую композицию. В некоторых вариантах осуществления капля представляет собой гомогенную каплю гидрогелевого материала или полую каплю, имеющую полимерную гидрогелевую оболочку. Гомогенная или полая капля может быть способна инкапсулировать одно или более ядер или клеток. В некоторых вариантах осуществления капля представляет собой каплю, стабилизированную поверхностно-активным веществом.
[0026] В настоящем документе термин «транспосомный комплекс» относится к интегрирующему ферменту и нуклеиновой кислоте, включающей сайт распознавания интеграции. «Транспосомный комплекс» представляет собой функциональный комплекс, образованный транспозазой и сайтом распознавания транспозазы, который способен катализировать реакцию транспонирования (см., например, Gunderson et al., WO 2016/130704). К примерам интегрирующих ферментов относятся, без ограничений, интеграза или транспозаза. К примерам сайтов распознавания интеграции относится, без ограничений, сайт распознавания транспозазы.
[0027] В настоящем документе термин «нуклеиновая кислота» используется взаимозаменяемо с термином «полинуклеотид» и «олигонуклеотид». «Нуклеиновая кислота» соответствует его использованию в данной области и включает нуклеиновые кислоты природного происхождения или их функциональные аналоги. Особенно подходящие для использования функциональные аналоги способны гибридизоваться с нуклеиновой кислотой специфичным для последовательности образом или могут использоваться в качестве матрицы для репликации конкретной нуклеотидной последовательности. Нуклеиновые кислоты природного происхождения обычно имеют каркас, содержащий фосфодиэфирные связи. Структура аналога может иметь альтернативную каркасную связь, в том числе любую из множества известных в данной области. Нуклеиновые кислоты природного происхождения обычно содержат сахар дезоксирибозу (например, присутствующий в дезоксирибонуклеиновой кислоте (ДНК)) или сахар рибозу (например, присутствующий в рибонуклеиновой кислоте (РНК)). Нуклеиновая кислота может содержать любой из множества аналогов этих остатков сахаров, известных в данной области. Нуклеиновая кислота может включать нативные или ненативные основания. В связи с этим нативная дезоксирибонуклеиновая кислота может содержать одно или более оснований, выбранных из группы, состоящей из аденина, тимина, цитозина или гуанина, и рибонуклеиновая кислота может иметь одно или более оснований, выбранных из группы, состоящей из аденина, урацила, цитозина или гуанина. Подходящие для использования ненативные основания, которые могут быть включены в нуклеиновую кислоту, известны в данной области. Примеры ненативных оснований включают запертую нуклеиновую кислоту (ЗНК), мостиковую нуклеиновую кислоту (МНК) и псевдокомплементарные основания (Trilink Biotechnologies, г. Сан-Диего, штат Калифорния, США). Основания ЗНК и МНК можно встраивать в олигонуклеотид ДНК и повышать прочность и специфичность гибридизации олигонуклеотида. Основания ЗНК и МНК и варианты использования таких оснований известны специалисту в данной области и являются стандартными. Если не указано иное, термин «нуклеиновая кислота» включает природную и неприродную ДНК, мРНК и некодирующую РНК, например РНК без поли-A на 3'-конце, и нуклеиновые кислоты, полученные из РНК, например кДНК. Термин «нуклеиновая кислота» относится только к первичной структуре молекулы. Таким образом, термин включает трех-, двух- и одноцепочечную дезоксирибонуклеиновую кислоту («ДНК»), а также трех-, двух- и одноцепочечную рибонуклеиновую кислоту («РНК»).
[0028] В настоящем документе термин «целевая» обозначает семантический идентификатор для молекулы, источник, функцию, идентичность и/или композицию которой исследуют. Примеры мишеней включают, без ограничений, нуклеиновую кислоту и белок. В настоящем документе термин «целевая» при использовании применительно к нуклеиновой кислоте служит семантическим идентификатором нуклеиновой кислоты в контексте способа или композиции, описанных в настоящем документе, и не обязательно ограничивает структуру или функцию нуклеиновой кислоты теми, которые явно указаны. Целевая нуклеиновая кислота может представлять собой по существу любую нуклеиновую кислоту с известной или неизвестной последовательностью. Это может быть, например, фрагмент геномной ДНК (например, хромосомная ДНК), внехромосомная ДНК, такая как плазмида, бесклеточная ДНК, РНК (например, РНК или некодирующая РНК), белки (например, белки клетки или клеточной поверхности) или кДНК. Целевая нуклеиновая кислота может представлять собой нуклеиновую кислоту, которая присоединена к соединению, такому как антитело, которое специфически связывает биомолекулу, такую как белок, гликан, протеогликан или липид (заявка на патент США Pub2018/0273933). Секвенирование может приводить к определению последовательности всей целевой молекулы или ее части. Цели могут быть получены из первичного образца нуклеиновой кислоты, такого как ядро. В одном варианте осуществления мишени можно превращать в матрицы, подходящие для амплификации, путем размещения универсальных последовательностей на одном или обоих концах каждого целевого фрагмента. Цели также можно получать из образца первичной РНК посредством обратной транскрипции в кДНК. В одном из вариантов осуществления термин «целевой» используется применительно к подгруппе ДНК, РНК или белков, присутствующих в клетке. Для направленного секвенирования используют отбор и выделение генов или областей, или белков, представляющих интерес, как правило, путем ПЦР-амплификации (например, специфичных для области праймеров) или способа захвата на основе гибридизации или использования антител. Целенаправленное обогащение может происходить на различных стадиях способа. Например, целенаправленное представление РНК можно получать с использованием специфичных к цели праймеров на стадии обратной транскрипции или путем обогащения на основе гибридизации подгруппы из более сложной библиотеки. Примером является секвенирование экзома или анализ L1000 (Subramanian et al., 2017, Cell, 171;1437-1452). Целенаправленное секвенирование может включать любой из процессов обогащения, известных специалисту в данной области. Целевая нуклеиновая кислота, имеющая универсальную последовательность на одном или обоих концах, может называться модифицированной целевой нуклеиновой кислотой. Если не указано иное, ссылка на нуклеиновую кислоту, такую как целевая нуклеиновая кислота, включает как одноцепочечные, так и двухцепочечные нуклеиновые кислоты. В одном варианте осуществления библиотеки обогащены с использованием индексной последовательности или индексных последовательностей. В некоторых вариантах осуществления обогащение вовлекает одну или более индексных последовательностей, присоединенных к той же молекуле библиотеки, например введенных посредством комбинаторного индексирования.
[0029] В настоящем документе термин «универсальный» при использовании для описания нуклеотидной последовательности относится к области последовательности, которая является общей для двух или более молекул нуклеиновых кислот, причем молекулы также имеют области последовательности, которые отличаются друг от друга. Универсальная последовательность, которая присутствует в разных членах набора молекул, например членах библиотеки секвенирования, может обеспечивать захват множества различных нуклеиновых кислот с использованием популяции универсальных последовательностей захвата. Не имеющие ограничительного характера примеры универсальных последовательностей захвата включают последовательности, идентичные или комплементарные праймерам P5 и P7. Аналогичным образом универсальная последовательность, присутствующая в разных членах набора молекул, может обеспечивать репликацию (например, секвенирование) или амплификацию множества разных нуклеиновых кислот с использованием популяции универсальных праймеров, которые комплементарны участку универсальной последовательности, например универсального сайта связывания праймера. Термины «A14» и «B15» можно использовать, когда речь идет об универсальном сайте связывания праймера. Термины «A14'» («A14-штрих») и «B15'» («B15-штрих») относятся к последовательности, комплементарной A14 и B15 соответственно. Следует понимать, что в способах, представленных в настоящем документе, можно использовать любые подходящие универсальные сайты связывания праймера, и что использование праймеров A14 и B15 представляет собой только примеры осуществления. В одном варианте осуществления в качестве сайта, с которым отжигают универсальный праймер (например, праймер секвенирования для чтения 1 или чтения 2) для секвенирования, используют универсальный сайт связывания праймера.
[0030] Термины «P5» и «P7» можно использовать при ссылке на универсальную последовательность захвата или захватный олигонуклеотид. Термины «P5’» («P5-штрих») и «P7’» («P7-штрих») относятся к последовательности, комплементарной P5 и P7 соответственно. Следует понимать, что в способах, предложенных в настоящем документе, можно использовать любую подходящую универсальную последовательность захвата или захватный олигонуклеотид и что использование праймеров P5 и P7 представляет собой только примеры осуществления. Способы использования захватных олигонуклеотидов, таких как Р5 и Р7, или комплементарных им последовательностей на проточных кюветах известны в данной области и описаны в качестве примера в публикациях WO 2007/010251, WO 2006/064199, WO 2005/065814, WO 2015/106941, WO 1998/044151 и WO 2000/018957. Например, в способах, представленных в настоящем документе, для гибридизации с комплементарной последовательностью и амплификации последовательности может использоваться любой подходящий прямой праймер для амплификации, иммобилизованный или в виде раствора. Аналогичным образом, в способах, представленных в настоящем документе, для гибридизации с комплементарной последовательностью и амплификации последовательности может использоваться любой подходящий обратный праймер для амплификации, иммобилизованный или в виде раствора. Специалистам в данной области будет понятно, как создать и использовать последовательности праймеров, которые подходят для захвата и/или амплификации нуклеиновых кислот в соответствии с описанием в настоящем документе.
[0031] В настоящем документе термин «праймер» и его производные по существу относятся к любой нуклеиновой кислоте, которая может гибридизоваться с интересующей последовательностью. Как правило, праймер функционирует в качестве субстрата, на котором можно полимеризовать нуклеотиды с помощью полимеразы или с которым можно лигировать нуклеотидную последовательность, такую как индексную; однако в некоторых вариантах осуществления праймер может встраиваться в цепь синтезированной нуклеиновой кислоты и обеспечивать сайт, с которым может гибридизоваться другой праймер для праймирования синтеза новой цепи, которая комплементарна синтезированной молекуле нуклеиновой кислоты. Праймер может включать любую комбинацию нуклеотидов или их аналогов. Праймер может представлять собой нуклеиновую кислоту, которая является одноцепочечной, двухцепочечной или включает одноцепочечную (-ые) область (-и) и двухцепочечную (-ые) область (-и), и может включать рибонуклеотиды, дезоксирибонуклеотиды, их аналоги или их смеси. Термины «полинуклеотид» и «олигонуклеотид» в настоящем документе применяются взаимозаменяемо. Следует понимать, что термины в качестве эквивалентов включают аналоги ДНК, РНК, кДНК или антитело-олигонуклеотидные конъюгаты, полученные из нуклеотидных аналогов и применимые к одноцепочечным (таким как смысловые или антисмысловые) и двухцепочечным полинуклеотидам. Термин, используемый в настоящем документе, также охватывает кДНК, являющуюся комплементарной или являющуюся копией ДНК, полученной с РНК-матрицы, например, под действием обратной транскриптазы. Данный термин относится только к первичной структуре молекулы. Таким образом, термин включает трех-, двух- и одноцепочечную дезоксирибонуклеиновую кислоту («ДНК»), а также трех-, двух- и одноцепочечную рибонуклеиновую кислоту («РНК»).
[0032] В настоящем документе термин «адаптер» и его производные, например универсальный адаптер, обычно относится к любому линейному олигонуклеотиду, который можно присоединить к молекуле нуклеиновой кислоты описания. В некоторых вариантах осуществления адаптер по существу не комплементарен 3’-концу или 5’-концу любой целевой последовательности, присутствующей в образце. В некоторых вариантах осуществления подходящие длины адаптеров находятся в диапазоне около 10-100 нуклеотидов, около 12-60 нуклеотидов или около 15-50 нуклеотидов. Как правило, адаптер может включать любую комбинацию нуклеотидов и/или нуклеиновых кислот. В некоторых аспектах адаптер может включать одну или более расщепляемых групп в одном или более положениях. В другом аспекте адаптер может включать последовательность, по существу идентичную или по существу комплементарную по меньшей мере участку праймера, например универсального праймера. В некоторых вариантах осуществления адаптер может включать штрихкод (также называемый в настоящем документе меткой или индексом) для помощи в коррекции нижележащих ошибок, идентификации или секвенировании. Термины «адаптор» и «адаптер» используются взаимозаменяемо.
[0033] В настоящем документе термин «каждый» применительно к набору элементов предназначен для определения отдельного элемента в наборе, но не обязательно относится к каждому элементу в наборе, если только иное явно не определяется контекстом.
[0034] В настоящем документе термин «транспорт» относится к движению молекулы через текучую среду. Термин может включать пассивный транспорт, такой как движение молекул по градиенту их концентрации (например, пассивную диффузию). Термин также может включать активный транспорт, при котором молекулы могут двигаться по их градиенту концентрации или против их градиента концентрации. Таким образом, транспорт может включать подачу энергии для перемещения одной или более молекул в желаемом направлении или в желаемое место, например к сайту амплификации.
[0035] В настоящем документе термин «амплификация», «амплифицировать» или «реакция амплификации» и их производные обычно относятся к любому действию или процессу, в котором по меньшей мере участок молекулы нуклеиновой кислоты реплицируется или копируется в по меньшей мере одну дополнительную молекулу нуклеиновой кислоты. Дополнительная молекула нуклеиновой кислоты необязательно включает последовательность, по существу идентичную или по существу комплементарную по меньшей мере некоторому участку матричной молекулы нуклеиновой кислоты. Матричная молекула нуклеиновой кислоты может быть одноцепочечной или двухцепочечной, а дополнительная молекула нуклеиновой кислоты может независимо быть одноцепочечной или двухцепочечной. Амплификация необязательно включает линейную или экспоненциальную репликацию молекулы нуклеиновой кислоты. В некоторых вариантах осуществления такая амплификация может выполняться с использованием изотермических условий; в других вариантах осуществления такая амплификация может включать термоциклирование. В некоторых вариантах осуществления амплификация представляет собой мультиплексную амплификацию, которая включает одновременную амплификацию множества целевых последовательностей в одной реакции амплификации. В некоторых вариантах осуществления термин «амплификация» включает амплификацию по меньшей мере некоторого участка нуклеиновых кислот на основе ДНК и РНК по отдельности или в комбинации. Реакция амплификации может включать любой из процессов амплификации, известных специалисту в данной области. В некоторых вариантах осуществления реакция амплификации включает полимеразную цепную реакцию (ПЦР).
[0036] В настоящем документе термин «условия амплификации» и его производные по существу относятся к условиям, подходящим для амплификации одной или более нуклеотидных последовательностей. Такая амплификация может быть линейной или экспоненциальной. В некоторых вариантах осуществления условия амплификации могут включать изотермические условия или альтернативно могут включать условия термоциклирования или комбинацию изотермических условий и условий термоциклирования. В некоторых вариантах осуществления условия, подходящие для амплификации одной или более нуклеотидных последовательностей, включают условия полимеразной цепной реакции (ПЦР). Как правило, условия амплификации относятся к реакционной смеси, которая является достаточной для амплификации нуклеиновых кислот, например, одной или более целевых последовательностей, фланкированных универсальной последовательностью, или для амплификации амплифицированной целевой последовательности, лигированной с одним или более адаптерами. Как правило, условия амплификации включают катализатор для амплификации или для синтеза нуклеиновых кислот, например полимеразу; праймер, который обладает некоторой степенью комплементарности с подлежащей амплификации нуклеиновой кислотой; и нуклеотиды, такие как дезоксирибонуклеотидтрифосфаты (дНТФ), для стимулирования удлинения праймера после гибридизации с нуклеиновой кислотой. Условия амплификации могут потребовать гибридизации или отжига праймера с нуклеиновой кислотой, удлинения праймера и стадии денатурации, на которой удлиненный праймер отделяют от нуклеотидной последовательности, подвергающейся амплификации. Как правило, но не обязательно условия амплификации могут включать термоциклирование; в некоторых вариантах осуществления условия амплификации включают множество циклов, на которых повторяются стадии отжига, удлинения и разделения. Как правило, условия амплификации включают катионы, такие как Mg2+ или Mn2+, и также могут включать различные модификаторы ионной силы.
[0037] В настоящем документе термин «реамплификация» и его производные обычно относятся к любому процессу, в котором по меньшей мере участок молекулы амплифицированной нуклеиновой кислоты дополнительно амплифицируют посредством любого подходящего процесса амплификации (называемого в некоторых вариантах осуществления «вторичной» амплификацией), в результате чего получается реамплифицированная молекула нуклеиновой кислоты. Вторичная амплификация не обязательно должна быть идентична исходному процессу амплификации, в котором была получена амплифицированная молекула нуклеиновой кислоты; не обязательно, чтобы молекула реамплифицированной нуклеиновой кислоты была полностью идентична или полностью комплементарна молекуле амплифицированной нуклеиновой кислоты; все, что необходимо, - это чтобы молекула реамплифицированной нуклеиновой кислоты включала по меньшей мере участок молекулы амплифицированной нуклеиновой кислоты или комплементарную ей последовательность. Например, реамплификация может предполагать использование других условий амплификации и/или других праймеров, включая праймеры, специфичные для иной цели, чем первичная амплификация.
[0038] В настоящем документе термин «полимеразная цепная реакция» («ПЦР») относится к способу Mullis из патентов США № 4,683,195 и 4,683,202, который описывает способ повышения концентрации сегмента интересующего полинуклеотида в смеси геномной ДНК без клонирования или очистки. Данный процесс амплификации интересующего полинуклеотида состоит из введения большого избытка двух олигонуклеотидных праймеров в смесь ДНК, содержащую желаемый интересующий полинуклеотид, с последующей серией термоциклирования в присутствии ДНК-полимеразы. Два праймера комплементарны соответствующим цепям интересующего двухцепочечного полинуклеотида. Сначала смесь денатурируют при повышенной температуре, а затем праймеры отжигают с комплементарными последовательностями внутри интересующей молекулы полинуклеотида. После отжига праймеры удлиняют полимеразой с образованием новой пары комплементарных цепей. Стадии денатурации, отжига с праймером и достройки полимеразой можно повторять множество раз (что называется термоциклированием) для получения высокой концентрации амплифицированного сегмента желаемого интересующего полинуклеотида. Длину амплифицированного сегмента (ампликона) интересующего желаемого полинуклеотида определяют по относительным положениям праймеров относительно друг друга, и, следовательно, эта длина является контролируемым параметром. В силу повторения этого процесса способ называют ПЦР. Поскольку желаемые амплифицированные сегменты интересующего полинуклеотида становятся преобладающими нуклеотидными последовательностями (с точки зрения концентрации) в смеси, говорят, что они «ПЦР-амплифицированы». В одной из модификаций описанного выше способа целевые молекулы нуклеиновой кислоты можно амплифицировать с помощью ПЦР, используя множество разных пар праймеров, в некоторых случаях - одну или более пар праймеров, на интересующую целевую молекулу нуклеиновой кислоты, таким образом образуя мультиплексную ПЦР-реакцию.
[0039] В настоящем документе термин «мультиплексная амплификация» относится к избирательной и неслучайной амплификации двух или более целевых последовательностей в образце с использованием по меньшей мере одного специфичного для цели праймера. В некоторых вариантах осуществления мультиплексную амплификацию выполняют таким образом, чтобы некоторые или все из целевых последовательностей амплифицировались в одном реакционном сосуде. «Плексия» или «плекс» заданной мультиплексной амплификации относится обычно к числу разных специфичных для цели последовательностей, которые амплифицируются в ходе этой одной мультиплексной амплификации. В некоторых вариантах осуществления плексия может составлять около 12-плекс, 24-плекс, 48-плекс, 96-плекс, 192-плекс, 384-плекс, 768-плекс, 1536-плекс, 3072-плекс, 6144-плекс или выше. Также существует возможность обнаруживать амплифицированные целевые последовательности посредством нескольких разных методологий (например, гель-электрофорез с последующей денситометрией, количественная оценка с помощью биоанализатора или количественной ПЦР, гибридизация с меченым зондом; включение биотинилированных праймеров с последующим обнаружением конъюгата авидина с ферментом; включение 32P-меченных дезоксинуклеотидтрифосфатов в амплифицированную целевую последовательность).
[0040] В настоящем документе термин «амплифицированные целевые последовательности» и его производные относится обычно к полинуклеотидной последовательности, полученной с помощью амплификации целевых последовательностей с использованием специфичных для цели праймеров и способов, предложенных в настоящем документе. Амплифицированные целевые последовательности могут быть либо одними и теми же смысловыми (т. е. положительная цепь), либо антисмысловыми (т. е. отрицательная цепь) по отношению к целевым последовательностям.
[0041] В настоящем документе термины «лигирование», «лигировать» и их производные обычно относятся к процессу ковалентного связывания двух или более молекул друг с другом, например ковалентного связывания двух или более молекул нуклеиновых кислот друг с другом. В некоторых вариантах осуществления лигирование включает соединение одноцепочечных разрывов между соседними нуклеотидами нуклеиновых кислот. В некоторых вариантах осуществления лигирование включает образование ковалентной связи между концом первой и концом второй молекул нуклеиновой кислоты. В некоторых вариантах осуществления лигирование может включать образование ковалентной связи между 5'-фосфатной группой одной нуклеиновой кислоты и 3'-гидроксильной группой второй нуклеиновой кислоты с образованием таким образом лигированной молекулы нуклеиновой кислоты. Как правило, для целей настоящего описания амплифицированная целевая последовательность может быть лигирована с адаптером для получения лигированной с адаптером амплифицированной целевой последовательности.
[0042] В настоящем документе термин «лигаза» и его производные обычно относятся к любому агенту, способному катализировать лигирование двух молекул субстрата. В некоторых вариантах осуществления лигаза включает фермент, способный катализировать соединение одноцепочечных разрывов между соседними нуклеотидами нуклеиновой кислоты. В некоторых вариантах осуществления лигаза включает фермент, способный катализировать образование ковалентной связи между 5'-фосфатом одной молекулы нуклеиновой кислоты и 3'-гидроксилом другой молекулы нуклеиновой кислоты с образованием таким образом лигированной молекулы нуклеиновой кислоты. Подходящие лигазы могут включать, без ограничений, ДНК-лигазу Т4, РНК-лигазу Т4 и ДНК-лигазу E. coli.
[0043] В настоящем документе термин «условия лигирования» и его производные обычно относятся к условиям, подходящим для лигирования двух молекул друг с другом. В некоторых вариантах осуществления условия лигирования подходят для сшивания одноцепочечных разрывов или зазоров между нуклеиновыми кислотами. В настоящем документе термин «одноцепочечный разрыв» или «зазор» соответствует использованию данного термина в данной области. Как правило, одноцепочечный разрыв или зазор может быть лигирован в присутствии фермента, такого как лигаза, при подходящих температуре и pH. В некоторых вариантах осуществления ДНК-лигаза Т4 может соединять одноцепочечный разрыв между нуклеиновыми кислотами при температуре около 70-72 °C.
[0044] В настоящем документе термин «проточная кювета» относится к камере, содержащей твердую поверхность, через которую могут протекать один или более жидких реагентов. Примеры проточных кювет и связанных с ними систем для работы с жидкостями и платформ для обнаружения, которые можно легко использовать в способах настоящего изобретения, описаны, например, в публикациях Bentley et al., Nature 456:53-59 (2008), WO 04/018497; US 7,057,026; WO 91/06678; WO 07/123744; US 7,329,492; US 7,211,414; US 7,315,019; US 7,405,281 и US 2008/0108082.
[0045] В настоящем документе термин «ампликон» применительно к нуклеиновой кислоте обозначает продукт копирования нуклеиновой кислоты, причем продукт имеет нуклеотидную последовательность, которая совпадает с по меньшей мере участком нуклеотидной последовательности нуклеиновой кислоты или комплементарна ему. Ампликон можно получать любым из разнообразных способов амплификации, в которых используют нуклеиновую кислоту или ее ампликон в качестве матрицы, включая, например, полимеразное удлинение, полимеразную цепную реакцию (ПЦР), амплификацию по типу катящегося кольца (RCA), удлинение с лигированием или цепную реакцию лигирования. Ампликон может представлять собой молекулу нуклеиновой кислоты, имеющую одну копию конкретной нуклеотидной последовательности (например, продукт ПЦР) или множество копий нуклеотидной последовательности (например, конкатамерный продукт RCA). Первый ампликон целевой нуклеиновой кислоты, как правило, представляет собой комплементарную копию. Последующие ампликоны представляют собой копии, которые формируют после создания первого ампликона из целевой нуклеиновой кислоты или из первого ампликона.
[0046] В настоящем документе термин «сайт амплификации» относится к сайту в матрице или на ней, где можно создать один или более ампликонов. Сайт амплификации может быть дополнительно выполнен с возможностью содержания, удержания или прикрепления по меньшей мере одного ампликона, создаваемого в сайте.
[0047] В настоящем документе термин «матрица» относится к популяции сайтов, которые можно отличать друг от друга по их относительному положению. Разные молекулы, находящиеся на разных сайтах матрицы, можно отличать друг от друга в соответствии с положениями сайтов в матрице. Отдельный сайт матрицы может содержать одну или более молекул конкретного типа. Например, сайт может включать одну целевую молекулу нуклеиновой кислоты, имеющую конкретную последовательность, или сайт может включать несколько молекул нуклеиновой кислоты, имеющих одинаковую последовательность (и/или комплементарную ей последовательность). Сайты матрицы могут представлять собой разные элементы, расположенные на одном и том же субстрате. Примеры элементов включают, без ограничений, лунки в субстрате, гранулы (или другие частицы) в субстрате или на нем, выступы из субстрата, ребра на субстрате или каналы в субстрате. Сайты матрицы могут представлять собой отдельные субстраты, каждый из которых несет свою молекулу. Разные молекулы, прикрепленные к отдельным субстратам, можно идентифицировать по положениям субстратов на поверхности, с которой связаны субстраты, или по положениям субстратов в жидкости или геле. Примеры матриц, в которых отдельные субстраты расположены на поверхности, включают, без ограничений, те, которые имеют гранулы в лунках.
[0048] В настоящем документе термин «емкость» применительно к сайту и нуклеотидному материалу обозначает максимальное количество нуклеотидного материала, которое может занимать сайт. Например, термин может относиться к общему количеству молекул нуклеиновых кислот, которые могут занимать сайт в конкретном состоянии. Можно использовать и другие меры, включая, например, общую массу нуклеотидного материала или общее количество копий конкретной нуклеотидной последовательности, которая может занимать сайт в конкретном состоянии. Как правило, емкость сайта в отношении целевой нуклеиновой кислоты будет по существу эквивалентна емкости сайта в отношении ампликонов целевой нуклеиновой кислоты.
[0049] В настоящем документе термин «захватный агент» относится к материалу, химическому веществу, молекуле или их функциональной группе, способной присоединять, удерживать или связывать целевую молекулу (например, целевую нуклеиновую кислоту). Примеры захватных агентов включают, без ограничений, захватную последовательность (также называемую в настоящем документе захватным олигонуклеотидом), которая комплементарна по меньшей мере участку целевой нуклеиновой кислоты, член пары связывания рецептор-лиганд (например, авидин, стрептавидин, биотин, лектин, углевод, белок, связывающийся с нуклеиновой кислотой, эпитоп, антитело и т. д.), способный связываться с целевой нуклеиновой кислотой (или с присоединенной к ней линкерной функциональной группой), или химический реагент, способный образовывать ковалентную связь с целевой нуклеиновой кислотой (или присоединенной к ней линкерной функциональной группой).
[0050] В настоящем документе термин «репортерная функциональная группа» может относиться к любой идентифицируемой метке, маркеру, индексу, штрихкоду или группе, которая позволяет определять состав, идентичность и/или источник исследуемой мишени. В некоторых вариантах осуществления репортерная функциональная группа может включать антитело, которое специфически связывается с белком. В некоторых вариантах осуществления антитело может включать обнаруживаемую метку. В некоторых вариантах осуществления репортер может включать антитело или аффинный реагент, меченный нуклеотидной меткой. В одном варианте осуществления нуклеиновая кислота имеет достаточную длину, чтобы выступать в качестве субстрата транспосомного комплекса. В одном варианте осуществления нуклеотидную метку может обнаруживаться, например, посредством анализа лигирования на близком расстоянии (PLA) или анализа удлинения на близком расстоянии (PEA) либо считывания на основе секвенирования (Shahi et al. Scientific Reports volume 7, Article number: 44447, 2017) или считывание на основании эпитопа, такого как CITE-seq (Stoeckius et al. Nature Methods 14:865-868, 2017).
[0051] В настоящем документе термин «клональная популяция» относится к популяции нуклеиновых кислот, которая является гомогенной по отношению к конкретной нуклеотидной последовательности. Гомогенная последовательность, как правило, имеет длину по меньшей мере 10 нуклеотидов, но может быть даже длиннее, включая, например, по меньшей мере 50, 100, 250, 500 или 1000 нуклеотидов. Клональную популяцию можно получить из одной целевой нуклеиновой кислоты или матричной нуклеиновой кислоты. Как правило, все из нуклеиновых кислот в клональной популяции будут иметь одинаковую нуклеотидную последовательность. Следует понимать, что в клональной популяции может происходить небольшое количество мутаций (например, из-за артефактов амплификации), и это не будет отклонением от клональности.
[0052] В настоящем документе термин «уникальный молекулярный идентификатор», или UMI, относится к молекулярной метке, случайной, неслучайной или полуслучайной, которая может быть присоединена к нуклеиновой кислоте. Введенный в нуклеиновую кислоту UMI можно использовать для коррекции последующей систематической ошибки амплификации путем прямого подсчета уникальных молекулярных идентификаторов (UMI), секвенированных после амплификации.
[0053] Используемый в настоящем документе термин «экзогенное» соединение, например термин «экзогенный фермент», относится к соединению, которое в норме или в природе не встречается в конкретной композиции. Например, если конкретная композиция включает клеточный лизат, экзогенный фермент представляет собой фермент, который в норме или в природе не присутствует в клеточном лизате.
[0054] В настоящем документе термин «обеспечение» в контексте, например, композиции, изделия, нуклеиновой кислоты или ядра обозначает получение композиции, изделия, нуклеиновой кислоты или ядра, приобретение композиции, изделия, нуклеиновой кислоты или ядра или иное получение соединения, композиции, изделия или ядра.
[0055] Термин «и/или» обозначает один или все из перечисленных элементов или комбинацию из любых двух или более из перечисленных элементов.
[0056] Слова «предпочтительный» и «предпочтительно» относятся к вариантам осуществления описания, которые могут обеспечивать определенные преимущества при определенных обстоятельствах. Однако другие варианты осуществления также могут являться предпочтительными при тех же или других обстоятельствах. Более того, приведение в настоящем документе одного или более предпочтительных вариантов осуществления не означает, что другие варианты осуществления не являются полезными, и не предполагает исключение других вариантов осуществления из объема изобретения.
[0057] Термины «содержит» и их вариации не имеют ограничительного характера при употреблении этих терминов в описании и формуле изобретения.
[0058] Следует понимать, что везде, где варианты осуществления описаны в настоящем документе с использованием формулировки «включать», «включает» или «включающий» и т. п., также предусмотрены иные аналогичные варианты осуществления, описанные с использованием терминов «состоящий из» и/или «состоящий по существу из».
[0059] Если не указано иное, термины «один» и «по меньшей мере один» используются взаимозаменяемо и обозначают «один или более одного».
[0060] Также в настоящем документе диапазоны числовых значений, указанные по конечным точкам, включают все числовые значения, содержащиеся в пределах этого диапазона (например, диапазон от 1 до 5 включает значения 1, 1,5, 2, 2,75, 3, 3,80, 4, 5 и т. д.).
[0061] Для любого способа, описанного в настоящем документе, который включает отдельные стадии, можно выполнять эти стадии в любом осуществимом порядке. Кроме того, в соответствующих случаях можно одновременно выполнять любую комбинацию двух или более стадий.
[0062] В настоящем описании ссылка на «один вариант осуществления», «вариант осуществления», «определенные варианты осуществления» или «некоторые варианты осуществления» и т. д. обозначает, что конкретный признак, конфигурация, композиция или характеристика, описанные в связи с вариантом осуществления, включены в по меньшей мере один вариант осуществления описания. Таким образом, появление таких фраз в различных местах данного описания необязательно относится к одному и тому же варианту осуществления изобретения. Более того, конкретные признаки, конфигурации, композиции или характеристики можно комбинировать любым подходящим образом в одном или более вариантах осуществления.
КРАТКОЕ ОПИСАНИЕ ФИГУР
[0063] Приведенное ниже подробное описание иллюстративных вариантов осуществления настоящего изобретения лучше всего будет понятно при чтении совместно с приведенными ниже графическими материалами.
[0064] На ФИГ. 1A и 1B представлены общие блок-схемы различных вариантов осуществления общего иллюстративного способа комбинаторного индексирования одиночных клеток в соответствии с настоящим описанием.
[0065] На ФИГ. 2 показан схематический рисунок способа комбинаторного индексирования одиночных клеток, как в общем показано в способе ФИГ. 1A. Для простоты показана только одна двухцепочечная целевая нуклеиновая кислота.
[0066] На ФИГ. 3 представлена общая блок-схема одного варианта осуществления общего иллюстративного способа комбинаторного индексирования одиночных клеток в соответствии с настоящим описанием.
[0067] На ФИГ. 4 представлена общая блок-схема одного варианта осуществления общего иллюстративного способа комбинаторного индексирования одиночных клеток в соответствии с настоящим описанием.
[0068] На ФИГ. 5 показан схематический рисунок способа комбинаторного индексирования одиночных клеток, как по существу показано в способе ФИГ. 1, ФИГ. 3 или ФИГ. 4. Для простоты показана только одна двухцепочечная целевая нуклеиновая кислота.
[0069] На ФИГ. 6 представлена общая блок-схема одного варианта осуществления общего иллюстративного способа метагеномного анализа с комбинаторным индексированием одиночных клеток в соответствии с настоящим описанием.
[0070] На ФИГ. 7 представлен схематический рисунок одного варианта осуществления общего иллюстративного способа получения библиотеки секвенирования со связными индексами в соответствии с настоящим описанием.
[0071] На ФИГ. 8 представлен схематический рисунок одного варианта осуществления общего иллюстративного способа для обогащения связи с целенаправленной амплификацией в соответствии с настоящим описанием.
[0072] На ФИГ. 9 представлена схема sci-ATAC-seq3. Ядра 1,6. миллиона клеток из 59 образцов плода тагментировали транспозазой Тn5 в массе. Первые два цикла индексирования проводят путем последовательного лигирования с каждым концом транспозазного комплекса Tn5, а третий цикл - с помощью ПЦР. В качестве индекса образца использовали первый цикл индексации.
[0073] На ФИГ. 10 показана структура ампликонов, полученных из sci-ATAC-seq3, описанной в примере 1.
[0074] На ФИГ. 11 показан рабочий процесс проекта, описанный в примере 2.
[0075] Схематические чертежи необязательно выполнены в масштабе. Аналогичные цифровые обозначения, используемые на фигурах, относятся к аналогичным компонентам, стадиям и т. п. Однако следует понимать, что использование номера для обозначения компонента на заданной фигуре не предполагает ограничения для обозначенного тем же номером компонента на другой фигуре. Кроме того, использование разных номеров для обозначения компонентов не предусматривает указания на то, что компоненты с разными номерами не могут быть идентичными или аналогичными другим пронумерованным компонентам.
ПОДРОБНОЕ ОПИСАНИЕ ИЛЛЮСТРАТИВНЫХ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ
[0076] Предложенный в настоящем документе способ можно использовать для получения библиотек секвенирования из множества одиночных клеток. Можно использовать по существу любой способ получения библиотеки одиночных клеток или способ секвенирования, включая, без ограничений, способы комбинаторного индексирования одиночных клеток, такие как секвенирование одиночных ядер хроматина, доступного транспозонам (sci-ATAC, патент США № 10,059,989), полногеномное секвенирование одиночных ядер (опубликованная заявка на патент США № US 2018/0023119), одноядерное секвенирование транскриптома (предварительная заявка на патент США № 62/680,259 и Gunderson et al. (WO2016/130704)), sci-HiC (Ramani et al., Nature Methods, 2017, 14:263-266), DRUG-seq (Ye et al., Nature Commun., 9, article number 4307), или любую комбинацию аналитов из ДНК и белков, например sci-CAR (Cao et al., Science, 2018, 361(6409):1380-1385), и РНК и белков, например CITE-seq (Stoeckius et al., 2017, Nature Methods. 14 (9): 865-868). В одном варианте осуществления эксперименты с атласом клеток можно проводить со считыванием, ограниченным доступной для хроматина ДНК, цельноклеточными транскриптомами, ограниченным количеством мРНК, которые являются высокоинформативными, или их комбинацией.
[0077] Обеспечение выделенных ядер или клеток
[0078] В одном варианте осуществления способ, предложенный в настоящем документе, может включать обеспечение клеток или изолированных ядер из множества клеток (например, ФИГ. 1A, блок 10, ФИГ. 3, блок 30, ФИГ. 4, блок 40, ФИГ. 6, блок 600). Клетки могут быть из любого (-ых) организма (-ов) и из клеток любого (-ых) типа или любой ткани организма (-ов). В одном варианте осуществления клетки могут быть из биоптата, такого как ткань или жидкий биоптат. В одном варианте осуществления клетки могут быть эмбриональными клетками, например клетками, полученными из эмбриона. В одном варианте осуществления клетки или ядра могут происходить из раковой или больной ткани. В одном варианте осуществления клетки или ядра могут представлять собой иммунные клетки, такие как Т-клетки или В-клетки. В одном варианте осуществления клетки могут представлять собой клетки множества различных типов, полученные из одного организма. В одном варианте осуществления множество разных типов клеток, полученных из одного организма, может включать клетки микроорганизмов, включая прокариотические и/или эукариотические клетки. В одном варианте осуществления на этой стадии не комбинируют клетки из разных источников, например разных организмов и/или разных тканей. В одном варианте осуществления на этой стадии комбинируют клетки из разных источников, например разных организмов и/или разных тканей.
[0079] В одном варианте осуществления множество клеток может представлять собой подгруппу большей популяции клеток. Подгруппу можно отделять от других клеток на основании различий в, например, размере, морфологии или присутствии на поверхности клетки идентифицируемой молекулы, такой как белок или гликан. Способы сортировки клеток известны в данной области и включают сортировку клеток с активацией флуоресценции, сортировку клеток с магнитной активацией и микрожидкостную сортировку клеток.
[0080] Способ может дополнительно включать диссоциирование клеток и/или выделение ядер. В одном варианте осуществления использованы условия, при которых поддерживается присутствие хроматина в ядрах. В одном варианте осуществления истощены присутствующие в ядрах нуклеосомы. Способы истощения нуклеосом известны специалисту в данной области (опубликованная заявка на патент США 2018/002311).
[0081] В данной области известно множество различных способов получения библиотеки одиночной клетки (Hwang et al. Experimental & Molecular Medicine, vol. 50, Article number: 96 (2018), включая, без ограничений, способы капельного секвенирования (Drop-Seq), Seq-well и способ комбинаторного индексирования одиночных клеток. Компании, обеспечивающие продукты одиночных клеток и связанные с ними технологии, включают, без ограничений, 10X Genomics, Takara biosciences, BD biosciences, Biorad, 1cellbio, IsoPlexis, Cell см., NanoCellect и Dolomite Bio. Sci-seq представляет собой методологическую базу, в которой для уникального маркирования содержимого в виде нуклеиновых кислот больших количеств одиночных клеток или ядер используют штрихкодирование с разделением пула. Как правило, количество ядер или клеток может составлять по меньшей мере два. Верхний предел зависит от практических ограничений оборудования (например, многолуночных планшетов, количества индексов), используемого на других стадиях способа, как описано в настоящем документе. Количество ядер или клеток, которое можно использовать, не имеет ограничительного характера и может измеряться миллиардами. Например, в одном варианте осуществления количество ядер или клеток может составлять не более 1 000 000 000, не более 100 000 000, не более 10 000 000, не более 1 000 000, не более 100 000, не более 10 000, не более 1000, не более 500 или не более 50. В одном варианте осуществления количество ядер или клеток может составлять по меньшей мере 50, по меньшей мере 500, по меньшей мере 1000, по меньшей мере 10 000, по меньшей мере 100 000, по меньшей мере 1 000 000, по меньшей мере 10 000 000, по меньшей мере 100 000 000 или по меньшей мере 1 000 000 000.
[0082] В тех вариантах осуществления, в которых используют изолированные ядра, ядра можно получать путем экстракции и фиксации. Необязательно и предпочтительно способ получения выделенных ядер не включает ферментативную обработку.
[0083] В одном из вариантов осуществления ядра выделяют из отдельных клеток, которые прикреплены или находятся в суспензии. Способы выделения ядер из отдельных клеток известны специалисту в данной области. Ядра обычно выделяют из клеток, присутствующих в ткани. Способ получения выделенных ядер, как правило, включает подготовку ткани, выделение ядер из подготовленной ткани и последующую фиксацию ядер. В одном из вариантов осуществления все стадии проводятся на льду.
[0084] В одном варианте осуществления подготовка ткани включает мгновенное замораживание ткани в жидком азоте с последующим уменьшением размера ткани до кусочков диаметром 1 мм или менее. Ткань можно уменьшать в размере, подвергая ткань либо режущему, либо тупому воздействию. Режущее воздействие может быть выполнено при помощи лезвия, рассекающего ткань на небольшие фрагменты. Использования тупого воздействия можно достичь путем измельчения ткани молотком или аналогичным объектом, и полученный состав из раздавленной ткани называется порошком.
[0085] Выделение ядер можно осуществлять путем инкубации частей или порошка в буфере для лизирования клеток в течение по меньшей мере от 1 до 20 минут, например 5, 10 или 15 минут. Используют те буферы, которые способствуют лизису клеток, но сохраняют целостность ядер. Пример буфера для клеточного лизиса включает 10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 0,1% IGEPAL CA-630, 1% ингибитор рибонуклеазы (РНазы) SUPERase In (20 ЕД/мкл, Ambion) и 1% альбумин бычьей сыворотки (BSA) (20 мг/мл, NEB). В стандартных способах выделения ядер для облегчения выделения часто применяют одно или более экзогенных соединений, таких как экзогенные ферменты. Примеры используемых ферментов, которые могут присутствовать в буфере для лизиса клеток, включают, без ограничений, ингибиторы протеазы, лизоцим, протеиназу K, поверхностно-активные вещества, лизостафин, зимолазу, целлюлозу, протеазу или гликаназу и т. п. (Islam et al. Micromachines (Basel), 2017, 8(3):83; www.sigmaaldrich.com/life-science/biochemicals/biochemical-products.html?TablePage=14573107). В одном варианте осуществления в лизирующем буфере для клеток отсутствуют один или более экзогенных ферментов, используемых в способе, описанном в настоящем документе. Например, экзогенный фермент (i) не добавляют к клеткам до смешивания клеток и лизирующего буферного раствора, (ii) не присутствует в лизирующем буферном растворе для клеток до смешивания его с клетками, (iii) не добавляют к смеси клеток и лизирующего буферного раствора для клеток или их комбинации. Специалисту в данной области понятно, что эти концентрации компонентов могут быть несколько изменены без снижения пригодности лизирующего клетки буфера для выделения ядер. Экстрагированные ядра затем очищают посредством одного или более циклов промывки буфером для ядер. Пример буфера для ядер включает 10 мM Tris-HCl, pH 7,4, 10 мM NaCl, 3 мM MgCl2, 1% ингибитор РНазы SUPERase In (20 ЕД/мкл, Ambion) и 1% BSA (20 мг/мл, NEB). Как и буфер для лизиса клеток, экзогенные ферменты могут также отсутствовать в буфере для ядер, применяемом в способе настоящего описания. Специалисту в данной области будет понятно, что эти концентрации компонентов могут быть несколько изменены без снижения пригодности буфера для ядер для выделения ядер. Специалисту в данной области будет понятно, что BSA и/или поверхностно-активные вещества можно использовать в буферах, используемых для выделения ядер.
[0086] Выделенные ядра можно зафиксировать путем воздействия поперечносшивающего агента. Примеры используемых поперечносшивающих агентов включают, без ограничений, параформальдегид и формальдегид. Параформальдегид может присутствовать в концентрации от 1% до 8%, например 4%. Формальдегид может присутствовать в концентрации от 30% до 45%, например 37%. Обработка ядер поперечносшивающим агентом может включать добавление агента к суспензии ядер и инкубацию при 0 °C. Другие способы фиксации включают, без ограничений, фиксацию метанолом. После фиксации необязательно и предпочтительно следует промывка в буфере для ядер.
[0087] Выделенные фиксированные ядра можно использовать сразу же или разделять на аликвоты и быстро замораживать в жидком азоте для последующего использования. При подготовке к использованию после замораживания размороженные ядра можно подвергать увеличению проницаемости мембран, например, с помощью 0,2% triton X-100 в течение 3 минут на льду, и кратковременной ультразвуковой обработке для уменьшения слипания ядер.
[0088] При традиционных методиках извлечения ядер из ткани ткань обычно инкубируют с тканеспецифическим ферментом (например, трипсином) при высокой температуре (например, 37 °C) в течение периода от 30 минут до нескольких часов, а затем клетки лизируют буферным раствором для лизирования клеток, извлекая ядра. Способ выделения ядер, описанный в настоящем документе, имеет несколько преимуществ: (1) Искусственные ферменты не вводятся, а все стадии проводятся на льду. Это снижает потенциальное нарушение клеточных состояний (например, организацию хроматина или состояние транскриптом). (2) Новый способ подтвержден для многих типов тканей, включая ткани головного мозга, легких, почек, селезенки, сердца, мозжечка, и патологических образцов, таких как опухолевые ткани. По сравнению с традиционными методиками выделения ядер из тканей, при которых используют разные ферменты для разных типов тканей, новая методика потенциально может снижать систематическую ошибку при сравнении состояний клеток разных тканей. (3) Новый способ также снижает затраты и повышает эффективность за счет исключения стадии обработки ферментами. (4) По сравнению с другими методиками извлечения ядер (например, тканевых гомогенизаторов Даунса) новая методика более надежна для разных типов тканей (например, способ Даунса требует оптимизации циклов Даунса для разных тканей) и позволяет обрабатывать большие фрагменты образцов с высокой пропускной способностью (например, способ Даунса ограничен размером гомогенизатора).
[0089] Необязательно выделенные ядра могут не содержать нуклеосом или могут быть подвергнуты воздействию условий, при которых происходит истощение нуклеосом в ядрах, с образованием безнуклеосомных ядер.
[0090] Вставка универсальных последовательностей
[0091] Способ, предложенный в настоящем документе, включает вставку одной или более универсальных последовательностей в нуклеиновые кислоты, присутствующие в ядрах или клетках. В одном варианте осуществления встраивание одной или более универсальных последовательностей происходит до распределения подгрупп (ФИГ. 1A, блок 11, ФИГ. 1B, блок 110), а в других вариантах осуществления встраивание одной или более универсальных последовательностей происходит после распределения подгрупп (ФИГ. 3, блок 32, ФИГ. 4, блок 42, блок 45). В некоторых вариантах осуществления индекс также может быть встроен с универсальной последовательностью или может быть связан с клетками или ядрами в качестве необязательной стадии, которая является отдельной от вставки одной или более универсальных последовательностей. Необязательное индексирование ядер или клеток может происходить до или после (ФИГ. 1A, блок 12) вставки универсальной последовательности. В одном варианте осуществления индекс добавляют к образцу до распределения подгрупп ядер или клеток (ФИГ. 1A, блок 13). В некоторых вариантах осуществления индекс добавляют ко множеству образцов до распределения подгрупп ядер или клеток (ФИГ. 1A, блок 13).
[0092] В одном варианте осуществления используют транспосомный комплекс. Транспосомный комплекс - это транспозаза, связанная с сайтом распознавания транспозазы, и может вставлять сайт распознавания транспозазы в целевую нуклеиновую кислоту в ядре в ходе процесса, который иногда называют «тагментацией». В некоторых таких событиях вставки одна цепь сайта распознавания транспозазы может переноситься в целевую нуклеиновую кислоту. Такая цепь называется «перенесенной цепью». В одном из вариантов осуществления транспосомный комплекс включает димерную транспозазу, имеющую две субъединицы и две несвязные транспозонные последовательности. В другом варианте осуществления транспозаза включает димерную транспозазу, имеющую две субъединицы и связную транспозонную последовательность. В одном из вариантов осуществления 5’-конец одной или обеих цепей сайта распознавания транспозазы может быть фосфорилирован.
[0093] Некоторые варианты осуществления могут включать использование гиперактивной транспозазы Tn5 и сайта распознавания транспозазы Tn5 (Goryshin and Reznikoff, J. Biol. Chem., 273:7367 (1998)) или транспозазу MuA и сайт распознавания транспозазы Mu, содержащий концевые последовательности R1 и R2 (Mizuuchi, K., Cell, 35: 785, 1983; Savilahti, H, et al., EMBO J., 14: 4893, 1995). Специалист в данной области может также использовать мозаичные концевые (ME) последовательности Tn5.
[0094] К дополнительным примерам систем транспонирования, которые можно использовать с определенными вариантами осуществления композиций и способов, предложенных в настоящем документе, относятся Staphylococcus aureus Tn552 (Colegio et al., J. Bacteriol., 183: 2384-8, 2001; Kirby C et al., Mol. Microbiol., 43: 173-86, 2002), Ty1 (Devine & Boeke, Nucleic Acids Res., 22: 3765-72, 1994 и международная публикация WO 95/23875), транспозон Tn7 (Craig, N L, Science. 271: 1512, 1996; Craig, N L, обзор в: Curr Top Microbiol Immunol., 204:27-48, 1996), Tn/O и IS10 (Kleckner N, et al., Curr Top Microbiol Immunol., 204:49-82, 1996), транспозаза Mariner (Lampe D J, et al., EMBO J., 15: 5470-9, 1996), Tc1 (Plasterk R H, Curr. Topics Microbiol. Immunol., 204: 125-43, 1996), P-элемент (Gloor, G B, Methods Mol. Biol., 260: 97-114, 2004), Tn3 (Ichikawa & Ohtsubo, J Biol. Chem. 265:18829-32, 1990), бактериальные инсерционные последовательности (Ohtsubo & Sekine, Curr. Top. Microbiol. Immunol. 204: 1-26, 1996), ретровирусы (Brown, et al., Proc Natl Acad Sci USA, 86:2525-9, 1989) и ретротранспозон дрожжей (Boeke & Corces, Annu Rev Microbiol. 43:403-34, 1989). К дополнительным примерам относятся IS5, Tn10, Tn903, IS911 и сконструированные версии ферментов семейства транспозаз (Zhang et al., (2009) PLoS Genet. 5:e1000689. Epub 2009 Oct 16; Wilson C. et al (2007) J. Microbiol. Methods 71:332-5).
[0095] К другим примерам интеграз, которые можно использовать со способами и композициями, предложенными в настоящем документе, относятся ретровирусные интегразы и сайты распознавания интеграз для таких ретровирусных интеграз, например, интеграз из ВИЧ-1, ВИЧ-2, SIV, PFV-1, RSV.
[0096] Транспозонные последовательности, используемые со способами и композициями, описанными в настоящем документе, обеспечены в опубликованной заявке на патент США № 2012/0208705, опубликованной заявке на патент США № 2012/0208724, опубликованной международной заявке на патент № WO 2012/061832. В некоторых вариантах осуществления транспозонная последовательность включает первый сайт распознавания транспозазы и второй сайт распознавания транспозазы.
[0097] Некоторые транспосомные комплексы, подходящие для использования в настоящем изобретении, включают транспозазу, имеющую две транспозонные последовательности. В некоторых таких вариантах осуществления две транспозонные последовательности не связаны друг с другом; иными словами, транспозонные последовательности не являются связными друг с другом. Примеры таких транспосом известны в данной области (см., например, опубликованную заявку на патент США № 2010/0120098).
[0098] В одном варианте осуществления тагментацию используют для получения целевых нуклеиновых кислот, которые включают различные универсальные последовательности на каждом конце (например, универсальный сайт связывания праймера, такой как А14, на одном конце и универсальный сайт связывания праймера, такой как В15, на другом конце). Этого можно достигать путем использования двух типов транспосомных комплексов, причем каждый транспосомный комплекс включает различную нуклеотидную последовательность, которая является частью переносимой цепи. Универсальная последовательность может служить множеству целей. Например и без намерения к ограничению, она может служить в качестве комплементарной последовательности для гибридизации на последующей стадии амплификации для добавления другой нуклеотидной последовательности, например индекса, она может служить сайтом, к которому отжигается универсальный праймер (например, праймер для секвенирования для чтения 1 или чтения 2) для секвенирования, или может служить в качестве «контактной площадки» на последующей стадии для отжига нуклеотидной последовательности, которую можно использовать в качестве праймера для добавления другой нуклеотидной последовательности, такой как индекс, к целевой нуклеиновой кислоте.
[0099] В некоторых вариантах осуществления транспосомный комплекс включает транспозонную последовательность нуклеиновой кислоты, которая связывает две субъединицы транспозазы с образованием «петлевого комплекса» или «петлевой транспосомы». В одном примере транспосома включает димерную транспозазу и транспозонную последовательность. Петлевые комплексы могут обеспечивать вставку транспозонов в целевую ДНК с сохранением информации о порядке исходной целевой ДНК и без фрагментирования целевой ДНК. Следует понимать, что петлевые структуры могут вставлять в целевую нуклеиновую кислоту желаемые нуклеотидные последовательности, такие как универсальные последовательности, и сохранять при этом физическую связность целевой нуклеиновой кислоты. В некоторых вариантах осуществления транспозонная последовательность петлевого транспосомного комплекса может включать сайт фрагментации, так что транспозонная последовательность может быть фрагментирована с созданием транспосомного комплекса, содержащего две транспозонных последовательности. Такие транспосомные комплексы используют для обеспечения приема соседними фрагментами целевой ДНК, в которую вставляют транспозоны, кодовых комбинаций, которые можно однозначно собирать на более поздней стадии анализа. В одном варианте осуществления комбинации индексов добавляют после вставки в целевую нуклеиновую кислоту одной или более универсальных последовательностей.
[00100] В одном варианте осуществления фрагментирование нуклеиновых кислот выполняют с использованием сайта фрагментации, присутствующего в нуклеиновых кислотах. Как правило, сайты фрагментации вводят в целевые нуклеиновые кислоты с использованием транспосомного комплекса. В одном варианте осуществления после фрагментации нуклеиновых кислот транспозаза остается присоединенной к фрагментам нуклеиновых кислот так, что фрагменты нуклеиновых кислот, полученные из одной и той же молекулы геномной ДНК, остаются физически связанными (Adey et al., 2014, Genome Res., 24:2041-2049, Amini S. et al. (2014) Nat Genet 46: 1343-1349). Например, петлевой транспосомный комплекс может включать сайт фрагментации. Сайт фрагментации можно использовать для расщепления физической связи, но не информационной связи между индексными последовательностями, которые были встроены в целевую нуклеиновую кислоту. Расщепление можно осуществлять биохимическими, химическими или иными способами. В некоторых вариантах осуществления сайт фрагментации может включать нуклеотид или нуклеотидную последовательность, которую можно фрагментировать различными способами. Примеры сайтов фрагментации включают, без ограничений, сайт эндонуклеазы рестрикции, по меньшей мере один рибонуклеотид, расщепляемый РНКазой, аналоги нуклеотидов, расщепляемые в присутствии определенного химического агента, диольную связь, расщепляемую путем обработки периодатом, дисульфидную группу, расщепляемую химическим восстанавливающим агентом, расщепляемую функциональную группу, которую можно подвергать фотохимическому расщеплению, и пептид, расщепляемый ферментом пептидазой или другими подходящими способами (см., например, публикацию заявки на патент США № 2012/0208705, опубликованной заявке на патент США № 2012/0208724 и WO 2012/061832). В одном варианте осуществления транспозаза остается присоединенной к фрагментам нуклеиновой кислоты и сохраняет физическую связь между фрагментами нуклеиновой кислоты, происходящими из одной и той же молекулы геномной ДНК, до удаления путем применения соответствующих условий, таких как добавление денатурирующего белок агента, например додецилсульфата натрия (SDS), или хелатирующего агента, например этилендиаминтетрауксусной кислоты (ЭДТК). Этот тип подхода позволяет получать информацию о непрерывности посредством захвата непрерывно связанной, транспонированной целевой нуклеиновой кислоты (заявка на патент США № 2019/0040382). Информацию о непрерывности можно сохранять с использованием транспозазы для сохранения связи соседних фрагментов темплатной нуклеиновой кислоты в целевой нуклеиновой кислоте.
[00101] В качестве альтернативы транспозиции целевые нуклеиновые кислоты можно получать посредством фрагментации. Фрагментацию первичных нуклеиновых кислот из образца можно проводить неупорядоченным образом с помощью ферментативных, химических или механических способов с последующим добавлением адаптеров к концам фрагментов. Примеры ферментативной фрагментации включают короткие палиндромные повторы, регулярно расположенные группами (CRISPR), и ферменты, подобные эффекторной нуклеазе, подобной активатору транскрипции (TALEN), и ферменты, раскручивающие ДНК (например, хеликазы), которые могут образовывать одноцепочечные области, с которыми фрагменты ДНК могут гибридизироваться, и инициировать удлинение или амплификацию. Например, можно использовать амплификацию на основе хеликазы (Vincent et al., 2004, EMBO Rep., 5(8):795-800). В одном варианте осуществления достройку или амплификацию инициируют с помощью случайно выбранного праймера. Примеры механической фрагментации включают распыление или ультразвуковую обработку.
[00102] Фрагментация первичных нуклеиновых кислот механическими средствами приводит к образованию фрагментов с гетерогенной смесью тупых и 3'- и 5'-нависающих концов. Таким образом, желательна репарация концов фрагментов с использованием способов, известных в данной области, чтобы получить концы, оптимальные для добавления адаптеров, например, в тупые сайты. В конкретном варианте осуществления концы фрагментов популяции нуклеиновых кислот являются тупоконечными. Более конкретно, концы фрагментов являются тупоконечными и фосфорилированными. Фосфатную функциональную группу можно вводить посредством ферментативной обработки, например, с использованием полинуклеотидкиназы.
[00103] В одном варианте осуществления фрагментированные нуклеиновые кислоты получают с нависающими нуклеотидами. Например, одиночные нависающие нуклеотиды можно добавлять посредством действия определенных типов ДНК-полимеразы, таких как Taq-полимераза или exo-minus-полимераза Кленова, которая обладает нетемплат-зависимой концевой трансферазной активностью, которая добавляет одиночный дезоксинуклеотид, например нуклеотид A к 3'-концам молекулы ДНК. Такие ферменты можно использовать для добавления одиночного нуклеотида А к тупоконечному 3'-концу каждой цепи фрагментов двухцепочечной нуклеиновой кислоты. Таким образом, к 3'-концу каждой репарированной на конце цепи двухцепочечных целевых фрагментов посредством реакции с Taq-полимеразой или exo-minus-полимеразой Кленова можно добавлять A, тогда как адаптер может быть T-конструкцией с совместимым T-выступом, присутствующим на 3'-конце каждой области двухцепочечной нуклеиновой кислоты универсального адаптера. В одном примере для добавления множества нуклеотидов Т можно использовать концевую дезоксинуклеотидилтрансферазу (TdT) (Swift Biosciences, Ann Arbor, MI). Такой тип концевой модификации также предотвращает самолигирование как вектора, так и мишени таким образом, что существует смещение к образованию целевых нуклеиновых кислот, имеющих один и тот же адаптер на каждом конце.
[00104] Первичная нуклеиновая кислота может быть ДНК, РНК или гибридами ДНК/РНК. В тех вариантах осуществления, в которых первичной нуклеиновой кислотой является РНК, встраивание одной или более универсальных последовательностей в нуклеиновые кислоты, присутствующие в ядрах или клетках, как правило, включает превращение РНК в ДНК. Можно использовать различные способы, и в некоторых вариантах осуществления они включают стандартные способы, используемые для получения кДНК. Например, праймер с последовательностью поли-T на 3'-конце и адаптером ближе к 5'-концу от последовательности поли-T можно отжигать к молекулам мРНК и достраивать с использованием обратной транскриптазы. Это приводит к одностадийному превращению мРНК в ДНК и необязательно универсальной последовательности - в 3'-конец. В одном варианте осуществления праймер может также включать одну или более индексных последовательностей. В одном варианте осуществления используют случайно выбранный праймер.
[00105] С использованием различных способов некодирующую РНК можно также превращать в ДНК и необязательно модифицировать для включения в нее универсальной последовательности. Например, адаптер можно добавлять с использованием первого праймера, который включает случайно выбранную последовательность, и праймера переключения темплата, причем любой праймер может включать адаптер универсальной последовательности. Можно использовать обратную транскриптазу, имеющую активность концевой трансферазы, приводящую к добавлению нетемплатных нуклеотидов к 3'-концу синтезированной цепи, а праймер для переключения темплатов включает нуклеотиды, которые отжигаются с нетемплатными нуклеотидами, добавленными обратной транскриптазой. Примером используемого фермента обратной транскриптазы является обратная транскриптаза вируса мышиного лейкоза Молони. В конкретном варианте осуществления реагент SMARTer™, поставляемый компанией Takara Bio USA, Inc. (кат. 634926) применяют для использования переключения темплатов для добавления универсальной последовательности к некодирующей РНК и, если требуется, мРНК. Необязательно праймер для переключения темплатов можно использовать с мРНК в сочетании с праймером с последовательностью поли-Т для получения в результате добавления универсальной последовательности к обоим концам целевой нуклеиновой кислоты ДНК, полученной из РНК.
[00106] Распределение подгрупп
[00107] Предложенный в настоящем документе способ включает распределение подгрупп выделенных ядер или клеток во множество компартментов (ФИГ. 1A, блок 13, ФИГ. 1B, блок 115, ФИГ. 3, блок 31, ФИГ. 4, блок 41, блок 44). Способ может включать множество стадий распределения, на которых популяцию выделенных ядер или клеток (также называемую в настоящем документе пулом) делят на подгруппы. Как правило, подгруппы изолированных ядер или клеток, например подгруппы, присутствующие во множестве компартментов, индексируют по специфичным для компартментов индексам, а затем объединяют. Соответственно, способ, как правило, включает по меньшей мере одна стадия «разделения и объединения», заключающегося в получении объединенных выделенных ядер или клеток, их распределения и добавления специфичного для компартмента индекса, причем количество стадий «разделения и объединения» может зависеть от количества различных индексов, добавляемых к целевым нуклеиновым кислотам. Каждая исходная подгруппа ядер или клеток перед индексированием может быть уникальной среди других подгрупп. Например, каждую первую подгруппу можно получать из уникального образца, такого как уникальный организм или уникальная ткань. После индексации подгруппы можно объединять, разделять на подгруппы, индексировать и снова объединять по мере необходимости до тех пор, пока к целевым нуклеиновым кислотам не будет добавлено достаточное количество индексов. Этот процесс присваивает каждой одиночной клетке или ядру уникальный индекс или комбинации индексов и приводит к комбинаторному индексированию, которое описано в настоящем документе. После завершения индексации, например после добавления одного, двух, трех или более индексов, изолированные ядра или клетки можно лизировать. В некоторых вариантах осуществления добавление индекса и лизирование могут происходить одновременно.
[00108] Количество ядер или клеток, присутствующих в подгруппе и, следовательно, в каждом компартменте, может составлять по меньшей мере 1. В одном из вариантов осуществления количество ядер или клеток в подгруппе составляет не более 100 000 000, не более 10 000 000, не более 1 000 000, не более 100 000, не более 10 000, не более 4000, не более 3000, не более 2000 или не более 1000, не более 500 или не более 50. В одном из вариантов осуществления количество ядер или клеток, присутствующих в подгруппе, может составлять от 1 до 1000, от 1000 до 10 000, от 10 000 до 100 000, от 100 000 до 1 000 000, от 1 000 000 до 10 000 000 или от 10 000 000 до 100 000 000. В одном из вариантов осуществления количество ядер или клеток, присутствующих в подгруппе, является приблизительно одинаковым. Количество ядер или клеток, присутствующих в подгруппе и, следовательно, в каждом компартменте, частично основано на желании уменьшать коллизии индексов, которые заключаются в попадании двух ядер или клеток, имеющих одинаковую комбинацию индексов, в один и тот же компартмент на этой стадии способа. Способы распределения ядер или клеток по подгруппам известны специалисту в данной области и являются стандартными. Хотя можно использовать цитометрию посредством сортировки клеток с активацией флуоресценции (FACS), в некоторых вариантах осуществления предпочтительно использование простого разведения. В одном из вариантов осуществления FACS-цитометрия не используется. Необязательно ядра различных плоидностей можно гейтировать и обогащать с помощью окрашивания, например окрашивания DAPI (4',6-диамидино-2-фенилиндолом). Окрашивание можно также использовать для различения одиночных клеток от дублетов во время сортировки.
[00109] Количество компартментов на стадиях распределения (и последующего добавления индекса) может зависеть от используемого формата. Например, количество компартментов может составлять от 2 до 96 компартментов (при использовании 96-луночного планшета), от 2 до 384 компартментов (при использовании 384-луночного планшета) или от 2 до 1536 компартментов (при использовании 1536-луночного планшета). В одном из вариантов осуществления можно использовать множество планшетов. Примеры компартментов включают, без ограничений, лунку, каплю и микрожидкостный компартмент. В одном из вариантов осуществления каждый компартмент может представлять собой каплю. Если используемый тип компартмента представляет собой каплю, которая содержит два или более ядер или клеток, можно использовать любое количество капель, например по меньшей мере 10 000, по меньшей мере 100 000, по меньшей мере 1 000 000 или по меньшей мере 10 000 000 капель. Перед объединением подгруппы выделенных ядер или клеток, как правило, индексируют в компартментах.
[00110] Комбинаторное индексирование
[00111] Предложенный в настоящем документе способ включает добавление специфичного для компартмента индекса к ядрам или клеткам, присутствующим в образце (ФИГ. 1B, блок 112), или к подгруппам выделенных ядер или клеток, распределенных в разные компартменты (например, ФИГ. 1A, блок 14, ФИГ. 3, блок 32, ФИГ. 4, блок 42 и 45, ФИГ. 6, блок 601). В некоторых вариантах осуществления с индексом можно встраивать также универсальную последовательность. Индексную последовательность, также называемую меткой или штрихкодом, также используют в качестве маркера, характерного для компартмента, в котором находилась конкретная нуклеиновая кислота. Соответственно, в некоторых вариантах осуществления индекс представляет собой метку из нуклеотидной последовательности, которая присоединена к каждой из целевых нуклеиновых кислот, присутствующих в конкретном компартменте, присутствие которых служит показателем или используется для определения компартмента, в котором популяция выделенных ядер или клеток находилась на определенной стадии способа.
[00112] В одном варианте осуществления добавляют множество индексов. Включение каждого индекса происходит в одном цикле разделения и индексирования пула. Один, два, три или более циклов разделения и штрихкодирования пула приводят к образованию целевых нуклеиновых кислот с единичной, двойной, тройной или множественной (например, четверной или более) индексацией.
[00113] Индексы можно добавлять к одному или обоим концам целевой нуклеиновой кислоты. Например, модифицированные целевые нуклеиновые кислоты, имеющие два или более индексов, могут включать различные индексы на каждом конце, пример которых показан на ФИГ. 5A. На ФИГ. 5A целевая нуклеиновая кислота 55 модифицирована так, чтобы она включала четыре отдельных индекса: два индекса (51 и 52) на одном конце и два индекса (53 и 54) на другом конце. В других вариантах осуществления модифицированная целевая нуклеиновая кислота может включать индексы, сгруппированные вместе на одном конце или на обоих концах, пример которых показан на ФИГ. 5B. На ФИГ. 5B целевую нуклеиновую кислоту 56 модифицируют так, чтобы она включала четыре отдельных индекса (51, 52, 53 и 54) на каждом конце. Набор индексов, присутствующих на одном конце целевой нуклеиновой кислоты, может называться «связным индексом». В одном варианте осуществления связные индексы не имеют нуклеотидов между каждым из индексов. В других вариантах осуществления между одним или более индексами связного индекса может быть расположено 1, 2, 3, 4 или более нуклеотидов. Как описано в настоящем документе, связный индекс можно использовать для определения членов библиотеки, имеющей определенный набор индексов. Например, связный индекс может способствовать обогащению членов библиотеки, которые происходят из одной и той же клетки.
[00114] Индексная последовательность может иметь любое подходящее количество нуклеотидов, например 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или более. Четырехнуклеотидная метка дает возможность мультиплексирования 256 образцов на одной и той же матрице, а шестинуклеотидная метка позволяет обрабатывать 4096 образцов на одной и той же матрице.
[00115] В одном варианте осуществления индекс добавляют после встраивания универсальной последовательности в нуклеиновые кислоты ДНК ядер или клеток при помощи, например, транспосомного комплекса. Для встраивания индексной последовательности можно использовать процесс, включающий одну, две или более стадий, с использованием по существу любой комбинации лигирования, достройки, гибридизации, адсорбции, специфических или неспецифических взаимодействий праймера, или амплификации. В одном варианте осуществления индекс добавляют в процессе синтеза кДНК. В одном варианте осуществления индекс добавляют посредством тагментации. Нуклеотидная последовательность, добавляемая к одному или обоим концам целевых нуклеиновых кислот, может также включать другие используемые последовательности, такие как одна или более универсальных последовательностей и/или уникальные молекулярные идентификаторы.
[00116] Для добавления индекса к нуклеиновой кислоте, которая включает универсальную последовательность, можно использовать различные способы, и способ добавления индекса не носит ограничительного характера. В одном варианте осуществления целевые нуклеиновые кислоты имеют различную универсальную последовательность на каждом конце (например, A14 на одном конце и B15 на другом конце), и специалисту в данной области будет понятно, что специфические последовательности можно добавлять к одному или обоим концам целевой нуклеиновой кислоты. Универсальные последовательности, добавляемые транспосомным комплексом, можно использовать, например, в качестве «контактной площадки» на последующей стадии для отжига нуклеотидной последовательности, которую можно использовать в качестве праймера для добавления к целевой нуклеиновой кислоте другой нуклеотидной последовательности, такой как другой индекс и/или другая универсальная последовательность. Например, в одном варианте осуществления встраивание индексной последовательности включает лигирование праймера с одним или обоими концами нуклеиновых кислот. Лигирование праймера можно облегчать присутствием универсальной последовательности на каждом конце целевых нуклеиновых кислот. Примером праймера является дуплекс лигирования со шпилькой. Дуплекс лигирования можно лигировать к одному концу или предпочтительно к обоим концам целевых нуклеиновых кислот.
[00117] В одном варианте осуществления можно использовать тупоконечное лигирование. В другом варианте осуществления целевые нуклеиновые кислоты получают с одиночными нависающими нуклеотидами посредством, например, активности определенных типов ДНК-полимеразы, таких как Taq-полимераза или exo-minus-полимераза Кленова, которая обладает нетемплат-зависимой концевой трансферазной активностью, которая добавляет к 3'-концам целевых нуклеиновых кислот один или больше дезоксинуклеотидов, например дезоксиаденозин (A). В некоторых случаях нависающий нуклеотид имеет более одного основания. Такие ферменты можно использовать для добавления одиночного нуклеотида А к тупоконечному 3'-концу каждой цепи целевых нуклеиновых кислот. Таким образом, A можно добавлять к 3'-концу каждой цепи двухцепочечных целевых фрагментов посредством реакции с Taq или exo-minus-полимеразы Кленова, тогда как дополнительные последовательности, подлежащие добавлению к каждому концу целевой нуклеиновой кислоты, могут включать совместимый T-выступ, присутствующий на 3'-конце каждой области добавляемой двухцепочечной нуклеиновой кислоты. Такая концевая модификация также предотвращает самолигирование нуклеиновых кислот таким образом, что имеет место смещение к образованию индексированных целевых нуклеиновых кислот, фланкированных последовательностями, которые добавляют в этом варианте осуществления.
[00118] В одном варианте осуществления встраивание индекса осуществляют с помощью реакции экспоненциальной амплификации, такой как ПЦР. Универсальные последовательности, присутствующие на концах целевых нуклеиновых кислот, можно использовать для отжига последовательности, которая может служить в качестве праймеров и ее можно достраивать в реакции амплификации.
[00119] Индекс и другие используемые последовательности можно добавлять за одну стадию или за множество стадий. Например, индекс и любые другие используемые последовательности можно добавлять путем лигирования или достройки, или можно использовать двухстадийный способ, который включает, например, лигирование универсальной последовательности и последующую амплификацию для дополнительной модификации универсальной последовательности с включением индекса и любых других используемых последовательностей.
[00120] В одном варианте осуществления добавление последовательностей во время стадий индексирования добавляет универсальные последовательности, которые используют для иммобилизации и/или секвенирования целевых нуклеиновых кислот. В другом варианте осуществления индексированные целевые нуклеиновые кислоты могут быть дополнительно обработаны для добавления универсальных последовательностей, которые используют для иммобилизации и секвенирования целевых нуклеиновых кислот. Специалисту в данной области будет понятно, что в вариантах осуществления, в которых компартмент представляет собой каплю, последовательности для иммобилизации фрагментов нуклеиновых кислот являются необязательными. В одном варианте осуществления встраивание универсальных последовательностей, используемых для иммобилизации и секвенирования фрагментов, включает лигирование идентичных универсальных адаптеров (также называемых «несовпадающими адаптерами», общие характеристики которых описаны в Gormley et al., US 7,741,463, и Bignell et al., US 8,053,192), с 5'- и 3'-концами индексированных фрагментов нуклеиновых кислот. В одном варианте осуществления универсальный адаптер включает все необходимые для секвенирования последовательности, включая последовательности для иммобилизации индексированных фрагментов нуклеиновых кислот на матрице.
[00121] Полученные индексированные фрагменты в совокупности образуют библиотеку нуклеиновых кислот, которую можно иммобилизовать и затем секвенировать. Термин «библиотека», также называемая в настоящем документе «библиотекой секвенирования», относится к набору целевых нуклеиновых кислот из одиночных ядер или клеток, содержащих на своих 3'- и 5'-концах известные универсальные последовательности и различные комбинации индексов. Библиотека включает нуклеиновые кислоты из, например, доступной ДНК, целого генома или целого транскриптома, нуклеиновые кислоты, указывающие на специфический белок, или их комбинацию, и ее можно применять для выполнения секвенирования.
[00122] Индексированные фрагменты нуклеиновых кислот можно подвергать воздействию условий, которые выбирают для предварительно заданного диапазона размеров, например, длиной от 150 до 400 нуклеотидов, например от 150 до 300 нуклеотидов. Полученные индексированные фрагменты нуклеиновых кислот объединяют и могут необязательно подвергать процессу очистки для улучшения чистоты молекул ДНК путем удаления по меньшей мере части невстроенных универсальных адаптеров или праймеров. Можно использовать любой приемлемый процесс очистки, такой как электрофорез, эксклюзионная хроматография размеров или т. п. В некоторых вариантах осуществления для отделения желаемых молекул ДНК от несвязанных универсальных адаптеров или праймеров, и для выбора нуклеиновых кислот в зависимости от размера можно использовать твердофазные парамагнитные гранулы с обратимой иммобилизацией. Твердофазные парамагнитные гранулы с обратимой иммобилизацией доступны в продаже в компаниях Beckman Coulter (Agencourt AMPure XP), Thermofisher (MagJet), Omega Biotek (Mag-Bind), Promega Beads (Promega) и Kapa Biosystems (Kapa Pure Beads).
[00123] Не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 1A. В этом варианте осуществления способ включает обеспечение множества ядер или клеток (ФИГ. 1A, блок 10). Множество ядер или клеток можно получать из образца или из множества образцов. Способ дополнительно включает встраивание одной или более универсальных последовательностей в нуклеиновые кислоты, присутствующие в ядрах или клетках (ФИГ. 1A, блок 11). Необязательно способ может также включать связывание индекса с ядрами или клетками (например, ядерное или клеточное хеширование, см. WO 2020/180778), и в одном варианте осуществления связывание может представлять собой добавление индекса к нуклеиновым кислотам (ФИГ. 1A, блок 12). В одном варианте осуществления добавляют две разные универсальные последовательности, чтобы в конечном итоге получить целевые нуклеиновые кислоты с разной универсальной последовательностью на каждом конце. Способ дополнительно включает распределение подгрупп ядер или клеток, которые теперь включают универсальные последовательности, встроенные в расположенные в них нуклеиновые кислоты и необязательно по меньшей мере один индекс, во множество компартментов (ФИГ. 1A, блок 13). Нуклеиновые кислоты, присутствующие в каждом компартменте, индексируют (ФИГ. 1A, блок 14), а ядра или клетки затем объединяют (ФИГ. 1A, блок 15). После добавления одиночного индекса библиотеки нуклеиновых кислот в ядрах или клетках можно дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 1A, блок 16); однако в некоторых предпочтительных вариантах осуществления желательно добавление второго, третьего или более индексов. В одном варианте осуществления добавление каждого индекса может включать стадию «разделения и объединения» с индексацией, происходящей после разделения, например распределение подгрупп ядер или клеток во множество компартментов (ФИГ. 1A, блок 13), индексацию нуклеиновых кислот, присутствующих в каждом компартменте (ФИГ. 1A, блок 14), с последующим объединением ядер или клеток (ФИГ. 1A, блок 15). Стадия «разделения и объединения» может приводить к добавлению индекса только к одному концу или к обоим концам нуклеиновых кислот, присутствующих в ядрах или клетках. После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять и дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 1A, блок 16), причем секвенирование может быть комплексным или прицельным.
[00124] Другой не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 1B. В этом варианте осуществления способ включает обеспечение множества образцов (ФИГ. 1B, блок 110), которые первоначально обрабатывают параллельно. Способ дополнительно включает встраивание одной или более универсальных последовательностей в нуклеиновые кислоты, присутствующие в ядрах или клетках (ФИГ. 1B, блок 111), с последующим добавлением к нуклеиновым кислотам (ФИГ. 1B, блок 112) индекса, причем индекс, добавленный к каждому образцу, является уникальным, и его можно использовать в качестве индекса образца для определения того, какие нуклеиновые кислоты происходят из специфического образца. В одном варианте осуществления добавляют две разные универсальные последовательности, чтобы в конечном итоге получить целевые нуклеиновые кислоты с разной универсальной последовательностью на каждом конце. Способ дополнительно включает объединение ядер или клеток (ФИГ. 1B, блок 113). В одном варианте осуществления после добавления одного индекса библиотеки нуклеиновых кислот в ядрах или клетках можно дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 1B, блок 114); однако в некоторых предпочтительных вариантах осуществления желательно добавление второго, третьего или более индексов. В одном варианте осуществления добавление каждого индекса может включать стадию «разделения и объединения» с индексацией, происходящей после разделения, например распределение подгрупп ядер или клеток во множество компартментов (ФИГ. 1B, блок 115), индексацию нуклеиновых кислот, присутствующих в каждом компартменте (ФИГ. 1B, блок 116), с последующим объединением ядер или клеток (ФИГ. 1B, блок 117). Стадия «разделения и объединения» может приводить к добавлению индекса только к одному концу или к обоим концам нуклеиновых кислот, присутствующих в ядрах или клетках. После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять и дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 1B, блок 118), причем секвенирование может быть комплексным или прицельным.
[00125] Другой не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 2. В этом варианте осуществления способ включает использование тагментации для встраивания двух универсальных последовательностей в нуклеиновые кислоты, присутствующие в ядрах или клетках, и трех последовательных циклов индексации (ФИГ. 2A). Один транспосомный комплекс 21 включает универсальную последовательность 23 (например, A14), а другой транспосомный комплекс 22 включает универсальную последовательность 24 (B15). Вставка универсальных последовательностей в нуклеиновые кислоты происходит в множестве ядер или клеток в массе. На ФИГ. 2A также показан результат вставки двух универсальных последовательностей 23 и 24 в целевую нуклеиновую кислоту 25. Множество ядер или клеток распределяют в разные компартменты и к одной стороне нуклеиновой кислоты 25 посредством лигирования добавляют полинуклеотид 26, включающий индекс, с использованием нуклеотидов, комплементарных одной универсальной последовательности (например, A14) (ФИГ. 2B). Множество ядер или клеток объединяют и затем распределяют в разные компартменты и с другой стороны нуклеиновой кислоты 25 посредством лигирования добавляют различный полинуклеотид 27, включающий второй индекс, с использованием нуклеотидов, комплементарных одной универсальной последовательности (например, B15) (ФИГ. 2C). Множество ядер или клеток, содержащих нуклеиновые кислоты с двойным индексированием, объединяют, а затем распределяют в разные компартменты, а затем подвергают реакции ПЦР-амплификации, которая добавляет полинуклеотид 28, включающий третий индекс, к одной стороне нуклеиновой кислоты 25 и добавляет полинуклеотид 29, включающий четвертый индекс, к одной стороне нуклеиновой кислоты 25 (ФИГ. 2D). После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять и дополнительно обрабатывать для подготовки к секвенированию, причем секвенирование может быть комплексным или прицельным.
[00126] Еще один не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 3. В этом варианте осуществления способ включает обеспечение множества ядер или клеток (ФИГ. 3, блок 30). Способ дополнительно включает распределение подгрупп ядер или клеток во множество компартментов (ФИГ. 3, блок 31). Нуклеиновые кислоты, присутствующие в ядрах или клетках каждого компартмента, модифицируют путем встраивания индекса и/или универсальной последовательности (ФИГ. 3, блок 32). В альтернативном варианте осуществления нуклеиновые кислоты, присутствующие в ядрах или клетках каждого компартмента, модифицируют путем встраивания одной и той же универсальной последовательности (например, тагментации с использованием транспозона с одной и той же универсальной последовательностью) с последующим добавлением индекса, специфичного для компартмента. Ядра или клетки затем объединяют (ФИГ. 3, блок 33). После добавления индекса и/или универсальной последовательности библиотеки нуклеиновых кислот в ядрах или клетках можно дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 3, блок 34); однако в некоторых предпочтительных вариантах осуществления желательно добавление второго, третьего или более индексов. Необязательно можно также добавлять универсальные последовательности. Добавление каждого индекса может включать стадию «разделения и объединения» с индексацией, происходящей после разделения, например распределение подгрупп ядер или клеток на множество компартментов (ФИГ. 3, блок 31), индексацию нуклеиновых кислот, присутствующих в каждом компартменте (ФИГ. 3, блок 32), и последующее объединение ядер или клеток (ФИГ. 3, блок 33). Стадия «разделения и объединения» может приводить к добавлению индекса только к одному концу или к обоим концам нуклеиновых кислот, присутствующих в ядрах или клетках. После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять и дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 3, блок 34), причем секвенирование может быть комплексным или прицельным.
[00127] Дополнительный не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 4. В этом варианте осуществления способ включает анализ РНК. Получают множество ядер или клеток (ФИГ. 4, блок 40), и они могут происходить из образца или множества образцов. Подгруппы ядер или клеток распределяют во множество компартментов (ФИГ. 4, блок 41). Необязательно перед распределением способ может также включать связывание индекса с ядрами или клетками (например, ядерное или клеточное хеширование, см. WO 2020/180778), или с нуклеиновыми кислотами. Нуклеиновые кислоты, присутствующие в ядрах или клетках каждого компартмента, модифицируют с использованием обратной транскриптазы для вставки индекса и/или универсальной последовательности (ФИГ. 4, блок 42), а затем ядра или клетки объединяют (ФИГ. 4, блок 43). Способ дополнительно включает распределение подгрупп ядер или клеток во множество компартментов (ФИГ. 4, блок 44). Нуклеиновые кислоты, присутствующие в ядрах или клетках каждого компартмента, модифицируют посредством вставки другого индекса и/или универсальной последовательности (ФИГ. 4, блок 45), а затем ядра или клетки объединяют (ФИГ. 4, блок 46). После добавления индекса и/или универсальной последовательности библиотеки нуклеиновых кислот в ядрах или клетках можно дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 4, блок 47); однако в некоторых предпочтительных вариантах осуществления желательно добавление третьего, четвертого или более индексов. Необязательно можно также добавлять универсальные последовательности. Добавление каждого индекса может включать стадию «разделения и объединения» с индексацией, происходящей после разделения, например распределение подгрупп ядер или клеток на множество компартментов (ФИГ. 4, блок 44), индексацию нуклеиновых кислот, присутствующих в каждом компартменте (ФИГ. 4, блок 45), и последующее объединение ядер или клеток (ФИГ. 4, блок 46). Стадия «разделения и объединения» может приводить к добавлению индекса только к одному концу или к обоим концам нуклеиновых кислот, присутствующих в ядрах или клетках. После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять и дополнительно обрабатывать для подготовки к секвенированию (ФИГ. 4, блок 47), причем секвенирование может быть комплексным или прицельным.
[00128] Подготовка иммобилизованных образцов для секвенирования
[00129] Способы прикрепления индексированных фрагментов из одного или более источников к субстрату известны специалистам в данной области. В одном из вариантов осуществления индексированные фрагменты обогащают с использованием множества захватных последовательностей, обладающих специфичностью к индексированным фрагментам, и захватные последовательности можно иммобилизовать на поверхности твердого субстрата. Например, захватные последовательности могут включать первый член связывающей пары (например, P5’), и при этом второй член связывающей пары (P5) иммобилизуют на поверхности твердого субстрата. Аналогичным образом способы амплификации иммобилизованных индексированных фрагментов включают, без ограничений, мостиковую амплификацию и кинетическое исключение. Способы иммобилизации и амплификации перед секвенированием описаны, например, в публикациях Bignell et al. (US 8,053,192), Gunderson et al. (WO2016/130704), Shen et al. (US 8,895,249) и Pipenburg et al. (US 9,309,502).
[00130] Объединенный образец можно иммобилизовать при подготовке к секвенированию. Секвенирование можно выполнять на матрице из отдельных молекул или можно проводить амплификацию перед секвенированием. Амплификацию можно проводить с использованием одного или более иммобилизованных праймеров. Иммобилизованный (-ые) праймер (-ы) может (могут) представлять собой, например, сплошной слой на плоской поверхности или на пуле гранул. Пул гранул можно выделять в эмульсию с одиночной гранулой в каждом «компартменте» эмульсии. При концентрации всего в один темплат на «компартмент» на каждой грануле амплифицируют только один темплат.
[00131] В настоящем документе термин «твердофазная амплификация» относится к любой реакции амплификации нуклеиновых кислот, проведенной на твердой подложке или в связи с ней так, что все амплифицированные продукты или их участки иммобилизуются на твердой подложке по мере их образования. В частности, термин включает твердофазную полимеразную цепную реакцию (твердофазную ПЦР) и твердофазную изотермическую амплификацию, которые представляют собой реакции, аналогичные стандартной амплификации в жидкой фазе, за исключением того, что один или оба из прямого и обратного праймеров для амплификации иммобилизованы на твердой подложке. Твердофазные ПЦР охватывают такие системы, как эмульсии, в которых один праймер заякоривают на грануле, а другой находится в свободном растворе, и образование колоний в твердофазных гелевых матрицах, в которых один праймер заякоривается на поверхности, а другой находится в свободном растворе.
[00132] В некоторых вариантах осуществления твердая подложка имеет рельефную поверхность. Термин «рельефная поверхность» относится к расположению разных областей в открытом слое твердой подложки или на нем. Например, одна или более областей могут быть элементами, в которых присутствуют один или более праймеров для амплификации. Элементы могут быть разделены промежуточными областями, в которых отсутствуют праймеры для амплификации. В некоторых вариантах осуществления рельеф может иметь координатный формат из x-y элементов, расположенных в строках и столбцах. В некоторых вариантах осуществления рельеф может иметь повторяющееся расположение элементов и/или промежуточных областей. В некоторых вариантах осуществления рельеф может иметь случайное расположение элементов и/или промежуточных областей. Примеры рельефных поверхностей, которые можно использовать в способах и композициях, описанных в настоящем документе, описаны в патентах США № 8,778,848, 8,778,849 и 9,079,148 и публикации США № 2014/0243224.
[00133] В некоторых вариантах осуществления твердая подложка содержит матрицу из лунок или углублений на поверхности. Такую подложку можно изготовить с использованием различных методик, известных в данной области, в том числе, без ограничений, фотолитографии, методик штамповки, методик литья и методик микротравления. Специалистам в данной области будет понятно, что методика зависит от состава и формы субстрата матрицы.
[00134] Элементы на структурированной поверхности могут представлять собой лунки в матрице лунок (например, микролунки или нанолунки) на стеклянных, кремниевых, пластиковых или других подходящих твердых подложках со структурированным ковалентно связанным гелем, таким как поли(N-(5-азидоацетамидилпентил)акриламид-со-акриламид) (PAZAM, см., например, публикации США № 2013/184796, WO 2016/066586 и WO 2015/002813). Процесс позволяет получить гелевые подушечки, используемые для секвенирования, которые могут сохранять стабильность в течение прогонов секвенирования с большим количеством циклов. Ковалентное связывание полимера с лунками полезно для сохранения геля в структурированных элементах на протяжении всего срока службы структурированного субстрата при различных вариантах использования. Однако во многих вариантах осуществления гель необязательно ковалентно связан с лунками. Например, при некоторых условиях в качестве гелевого материала может использоваться не содержащий силана акриламид (SFA, см., например, патент США № 8,563,477), который не имеет ковалентной связи с какой-либо частью структурированного субстрата.
[00135] В конкретных вариантах осуществления структурированный субстрат можно изготовлять путем нанесения рельефа в виде лунок (например, микролунок или нанолунок) на материал твердой подложки, покрытия структурированной подложки гелевым материалом (например, поли(N-(5-азидоацетамидилпентил)акриламид-ко-акриламид) (PAZAM), насыщенной жирной кислотой (SFA) или их химически модифицированными вариантами, например азидолизированным вариантом SFA (азидо-SFA)) и полировки покрытой гелем подложки, например химической или механической полировки, таким образом удерживая гель в лунках и удаляя или инактивируя практически весь гель с промежуточных областей на поверхности структурированного субстрата между лунками. Праймер для нуклеиновых кислот может быть прикреплен к гелевому материалу. Затем раствор индексированных фрагментов можно приводить в контакт с полированным субстратом так, чтобы отдельные индексированные фрагменты засевали отдельные лунки посредством взаимодействий с праймерами, присоединенными к гелевому материалу; однако целевые нуклеиновые кислоты не будут занимать промежуточные области из-за отсутствия или неактивности гелевого материала. Амплификация индексированных фрагментов будет ограничена лунками, так как отсутствие или неактивность геля в промежуточных областях предотвращает миграцию растущей колонии нуклеиновых кислот наружу. Процесс может быть удобен для производства, поскольку является масштабируемым и использует традиционные микро- и нанотехнологические способы.
[00136] Хотя описание включает «твердофазные» способы амплификации, в которых иммобилизован только один праймер амплификации (другой праймер обычно присутствует в свободном растворе), в одном из вариантов осуществления предусмотрена твердая подложка, на которой иммобилизованы как прямой, так и обратный праймеры. На практике будут присутствовать «множество» идентичных прямых праймеров и/или «множество» идентичных обратных праймеров, иммобилизованных на твердой подложке, поскольку в процессе амплификации необходим избыток праймеров для поддержания амплификации. В настоящем документе ссылки на прямой и обратный праймеры следует интерпретировать соответственно как включающие «множества» таких праймеров, если иное не диктуется контекстом.
[00137] Как будет понятно читателю, являющемуся специалистом, для любой данной реакции амплификации требуется по меньшей мере один тип прямого праймера и по меньшей мере один тип обратного праймера, специфичный для амплифицируемого темплата. Однако в определенных вариантах осуществления прямой и обратный праймеры могут включать специфичные для темплата участки идентичной последовательности и могут иметь полностью идентичную нуклеотидную последовательность и структуру (включая любые ненуклеотидные модификации). Иными словами, можно проводить твердофазную амплификацию с использованием только одного типа праймеров, и такие однопраймерные способы входят в объем настоящего описания. В других вариантах осуществления можно использовать прямой и обратный праймеры, которые содержат идентичные специфические для темплата последовательности, но которые отличаются некоторыми другими структурными особенностями. Например, один тип праймера может содержать ненуклеотидную модификацию, отсутствующую в другом.
[00138] Во всех вариантах осуществления изобретения праймеры для твердофазной амплификации предпочтительно иммобилизуют путем одноточечного ковалентного присоединения к твердой подложке на 5'-конце праймера или вблизи него, оставляя специфичный для темплата участок праймера свободным для отжига с его распознаваемым темплатом и свободную 3'-гидроксильную группу для достройки праймера. Для этой цели можно использовать любое подходящее средство ковалентного связывания, известное в данной области. Выбранная для связывания химическая реакция зависит от характера твердой подложки и любых примененных к ней производных или функционализирующих модификаций. Сам праймер может включать функциональную группу, которая может представлять собой ненуклеотидную химическую модификацию для облегчения присоединения. В конкретном варианте осуществления праймер может включать серосодержащий нуклеофил, такой как фосфоротиоат или тиофосфат, на 5’-конце. В случае полиакриламидных гидрогелей на твердой подложке данный нуклеофил будет связываться с бромацетамидной группой, присутствующей в гидрогеле. Более конкретным способом прикрепления праймеров и темплатов к твердой подложке является прикрепление 5'-фосфоротиоата к гидрогелю, состоящему из полимеризованного акриламида и N-(5-бромацетамидилпентил)акриламида (BRAPA), как описано в WO 05/065814.
[00139] В определенных вариантах осуществления описания можно использовать твердые подложки, которые включают инертный субстрат или матрицу (например, стеклянные пластины, полимерные гранулы и т. д.), которые были «функционализированы», например, посредством нанесения слоя или покрытия из промежуточного материала, включающего реакционноспособные группы, которые обеспечивают ковалентное прикрепление к биомолекулам, таким как полинуклеотиды. Примеры таких субстратов включают, без ограничений, полиакриламидные гидрогели, нанесенные на инертный субстрат, такой как стекло. В таких вариантах осуществления биомолекулы (например, полинуклеотиды) могут напрямую ковалентно прикрепляться к промежуточному материалу (например, гидрогелем), но промежуточный материал сам по себе может быть нековалентно прикреплен к субстрату или матриксу (например, стеклянному субстрату). Термин «ковалентное прикрепление к твердой подложке» следует интерпретировать соответственно как охватывающий данный тип размещения.
[00140] Объединенные образцы можно амплифицировать на гранулах, причем каждая гранула содержит прямой и обратный праймеры амплификации. В конкретном варианте осуществления библиотеку индексированных фрагментов используют для получения кластеризованных матриц с колониями нуклеиновых кислот, аналогичными описанным в публикации США № 2005/0100900, патентах США № 7,115,400, WO 00/18957 и WO 98/44151 посредством твердофазной амплификации и, более конкретно, твердофазной изотермической амплификации. Термины «кластер» и «колония» в настоящем документе используются взаимозаменяемо и относятся к дискретному сайту на твердой подложке, содержащему множество идентичных иммобилизованных цепей нуклеиновых кислот и множество идентичных иммобилизованных комплементарных цепей нуклеиновых кислот. Термин «кластеризованная матрица» относится к матрице, образованной из таких кластеров или колоний. В этом контексте термин «матрица» не следует понимать как требующий упорядоченного расположения кластеров.
[00141] Термин «твердая фаза» или «поверхность» использован для обозначения либо плоской матрицы, в которой праймеры прикреплены к плоской поверхности, например, к стеклянным, кварцевым или пластиковым предметным стеклам микроскопа, либо к аналогичным устройствам проточных кювет; гранул, причем один или два праймера прикреплены к гранулам, и гранулы подвергают амплификации; или матрицы из гранул на поверхности после амплификации на гранулах.
[00142] Кластеризованные матрицы можно получить с использованием либо процесса термоциклирования, как описано в WO 98/44151, либо процесса, в котором температуру поддерживают на постоянном уровне, а циклы удлинения и денатурации выполняют с использованием изменений реагентов. Такие способы изотермической амплификации описаны в заявке на патент WO 02/46456 и в патентной публикации США № 2008/0009420. В связи с более низкими температурами, используемыми в изотермическом процессе, в некоторых вариантах осуществления он является особенно предпочтительным.
[00143] Следует понимать, что любую из методологий амплификации, описанных в настоящем документе или по существу известных в данной области, можно использовать с универсальными или специфичными для цели праймерами для амплификации иммобилизованных фрагментов ДНК. Подходящие способы амплификации включают, без ограничений, полимеразную цепную реакцию (ПЦР), амплификацию с замещением цепей (SDA), транскрипционно-опосредованную амплификацию (ТМА) и амплификацию на основе нуклеотидной последовательности (NASBA), как описано в патенте США № 8,003,354. Вышеуказанные способы амплификации можно использовать для амплификации одной или более интересующих нуклеиновых кислот. Например, ПЦР, включая мультиплексную ПЦР, SDA, ТМА, NASBA и т. п., можно использовать для амплификации иммобилизованных фрагментов ДНК. В некоторых вариантах осуществления в реакцию амплификации включены праймеры, специфичные для интересующего полинуклеотида.
[00144] Другие подходящие способы амплификации полинуклеотидов могут включать технологии удлинения и лигирования олигонуклеотидов, амплификации по типу катящегося кольца (RCA) (Lizardi et al., Nat. Genet. 19:225-232 (1998)) и лигирование олигонуклеотидных зондов (OLA) (см. по существу патенты США № 7,582,420, 5,185,243, 5,679,524 и 5,573,907; EP 0 320 308 B1; EP 0 336 731 B1; EP 0 439 182 B1; WO 90/01069; WO 89/12696 и WO 89/09835). Следует понимать, что эти методологии амплификации могут быть разработаны с возможностью амплификации иммобилизованных фрагментов ДНК. Например, в некоторых вариантах осуществления способ амплификации может включать реакции амплификации с лигирующим зондом или лигирования олигонуклеотидных зондов (OLA), которые содержат праймеры, напрямую нацеленные на интересующую нуклеиновую кислоту. В некоторых вариантах осуществления способ амплификации может включать реакцию достройки праймеров - лигирования, которая содержит праймеры, напрямую нацеленные на интересующую нуклеиновую кислоту. В качестве не имеющего ограничительного характера примера праймеров для реакции достройки и лигирования праймеров, которые могут быть специально выполнены с возможностью амплификации интересующей нуклеиновой кислоты, амплификация может включать праймеры, используемые для анализа GoldenGate (Illumina, Inc., г. Сан-Диего, штат Калифорния, США), как показано на примерах в патенте США № 7,582,420 и 7,611,869.
[00145] ДНК-наносферы также можно использовать в комбинации со способами и композициями, описанными в настоящем документе. Способы создания и применения ДНК-наносфер для геномного секвенирования можно найти, например, в патентах США и публикациях патентов США № 7,910,354, 2009/0264299, 2009/0011943, 2009/0005252, 2009/0155781, 2009/0118488, и описаны, например, Drmanac et al., 2010, Science 327(5961): 78-81. Вкратце после фрагментации ДНК геномной библиотеки адаптеры лигируют с фрагментами, причем лигированные адаптером фрагменты циркуляризуют посредством лигирования циркуляризационной лигазой, и выполняют амплификацию по типу катящегося кольца (как описано у Lizardi et al., 1998. Nat. Genet. 19:225-232 и US 2007/0099208 A1). Удлиненная конкатамерная структура ампликонов способствует скручиванию, в результате чего образуются компактные ДНК-наносферы. ДНК-наносферы могут захватываться на субстраты, предпочтительно создавая упорядоченную или рельефную матрицу таким образом, чтобы сохранялось расстояние между наносферами, чтобы можно было секвенировать отдельные ДНК-наносферы. В некоторых вариантах осуществления перед циркуляризацией проводят последовательные циклы лигирования, амплификации и расщепления адаптера для получения конструктов с головой и хвостом, имеющих несколько фрагментов геномной ДНК, разделенных адаптерными последовательностями.
[00146] Примеры способов изотермической амплификации, которые могут использоваться в способе настоящего изобретения, включают, без ограничений, амплификацию с множественным вытеснением (MDA), пример которой приведен в публикации Dean et al., Proc. Natl. Acad. Sci. USA 99:5261-66 (2002), или изотермическую амплификацию нуклеиновых кислот с замещениями цепей, пример которой приведен в патенте США № 6,214,587. Другие не основанные на ПЦР способы, которые могут использоваться в настоящем изобретении, включают, например, амплификацию с замещением цепей (SDA), описанную, например, в Walker et al., Molecular Methods for Virus Detection, Academic Press, Inc., 1995; патентах США № 5,455,166 и 5,130,238 и публикации Walker et al., Nucl. Acids Res. 20:1691-96 (1992), или амплификацию с замещением гиперразветвленной цепи, описанную, например, в публикации Lage et al., Genome Res. 13:294-307 (2003). Можно использовать способы изотермической амплификации, например, замещающую цепь полимеразу Phi 29 или большой фрагмент ДНК-полимеразы Bst, 5’->3’ экзо- для амплификации геномной ДНК со случайными праймерами. Полимеразы позволяют использовать преимущества, которые дает их высокая процессивность и активность по замещению цепей. Высокая процессивность позволяет полимеразам производить фрагменты длиной 10-20 тыс. п. н. Как указано выше, более мелкие фрагменты могут быть получены в изотермических условиях с использованием полимераз, обладающих низкой процессивностью и активностью по замещению цепей, таких как полимераза Кленова. Дополнительное описание реакций амплификации, условий и компонентов подробно представлено в описании патента США № 7,670,810.
[00147] Другим способом амплификации полинуклеотидов, подходящим для настоящего описания, является ПЦР с метками, в которой используется популяция двухдоменных праймеров, имеющих константную 5’-область, за которой следует случайная 3’-область, как описано, например, в публикации Grothues et al. Nucleic Acids Res. 21(5):1321-2 (1993). Первые циклы амплификации выполняют для обеспечения множества инициаций на денатурированной нагреванием ДНК на основе индивидуальной гибридизации со случайно синтезированной 3’-областью. Вследствие характера 3’-области предполагается, что сайты инициации случайно распределены по всему геному. После этого несвязанные праймеры можно удалить и провести дополнительную репликацию с использованием праймеров, комплементарных константной 5’-области.
[00148] В некоторых вариантах осуществления изотермическую амплификацию можно выполнить с использованием амплификации с кинетическим исключением (KEA), также называемой эксклюзионной амплификацией (ExAmp). Библиотеку нуклеиновых кислот настоящего описания можно получить с использованием способа, который включает стадию взаимодействия реагента для амплификации для получения множества сайтов амплификации, каждый из которых включает по существу клональную популяцию ампликонов отдельной целевой нуклеиновой кислоты, послужившей затравкой для сайта. В некоторых вариантах осуществления реакция амплификации продолжается до тех пор, пока не будет получено достаточное количество ампликонов для заполнения емкости соответствующего сайта амплификации. Такое заполнение уже засеянного сайта до емкости подавляет посадку и амплификацию целевых нуклеиновых кислот на сайте, в результате чего на сайте получается клональная популяция ампликонов. В некоторых вариантах осуществления кажущуюся клональность можно обеспечить даже в том случае, если емкость сайта амплификации не заполнена до того, как вторая целевая нуклеиновая кислота достигнет сайта. При некоторых условиях амплификация первой целевой нуклеиновой кислоты может продолжаться до момента получения достаточного количества копий для эффективного вытеснения или преодоления продукции копий второй целевой нуклеиновой кислоты, достигающей сайта. Например, в варианте осуществления, в котором используется процесс мостиковой амплификации на круговом элементе диаметром менее 500 нм, было определено, что после 14 циклов экспоненциальной амплификации первой целевой нуклеиновой кислоты загрязнение того же сайта второй целевой нуклеиновой кислотой приведет к количеству загрязняющих ампликонов, недостаточному для того, чтобы оно неблагоприятно сказалось на анализе секвенирования методом синтеза с использованием платформы для секвенирования Illumina.
[00149] В некоторых вариантах осуществления сайты амплификации в матрице могут быть, но не обязательно должны быть полностью клональными. Вместо этого в некоторых областях применения отдельный сайт амплификации может быть преимущественно заполнен ампликонами из первого индексированного фрагмента и может также содержать низкий уровень загрязняющих ампликонов второй целевой нуклеиновой кислоты. Матрица может иметь один или более сайтов амплификации, содержащих низкий уровень загрязняющих ампликонов, при условии, что уровень загрязнения не оказывает неприемлемого влияния на последующее использование матрицы. Например, если матрица предназначена для использования при обнаружении, приемлемым уровнем загрязнения будет уровень, который недопустимым образом не влияет на соотношение сигнал/шум или на разрешение методики обнаружения. Соответственно, кажущаяся клональность по существу уместна для конкретного использования или применения матрицы, полученной способами, описанными в настоящем документе. Примеры уровней загрязнения, которые могут быть приемлемы для конкретных областей применения на отдельном сайте амплификации, включают, без ограничений, не более 0,1%, 0,5%, 1%, 5%, 10% или 25% загрязняющих ампликонов. Матрица может включать один или более сайтов амплификации, имеющих эти примеры уровней загрязняющих ампликонов. Например, до 5%, 10%, 25%, 50%, 75% или даже 100% сайтов амплификации в матрице могут содержать некоторое количество загрязняющих ампликонов. Следует понимать, что в матрице или другом наборе сайтов по меньшей мере 50%, 75%, 80%, 85%, 90%, 95% или 99% или более сайтов могут быть клональными или кажущимися клональными.
[00150] В некоторых вариантах осуществления кинетическое исключение может возникать, когда процесс происходит с достаточно высокой скоростью, чтобы эффективно исключить появление другого события или процесса. Возьмем, например, создание матрицы нуклеиновых кислот, причем сайты матрицы случайным образом засеяны из раствора индексированными фрагментами, а копии индексированных фрагментов созданы в процессе амплификации с заполнением каждого из засеянных сайтов до его емкости. В соответствии со способами кинетического исключения настоящего изобретения процессы посева и амплификации могут происходить одновременно в условиях, в которых скорость амплификации превышает скорость посева. Таким образом, относительно высокая скорость получения копий в сайте, засеянном первой целевой нуклеиновой кислотой, будет эффективно исключать засевание сайта амплификации второй нуклеиновой кислотой. Способы амплификации с кинетическим исключением можно выполнять так, как подробно описано в публикации заявки на патент № 2013/0338042.
[00151] В кинетическом исключении можно использовать относительно низкую скорость инициирования амплификации (например, низкую скорость получения первой копии индексированного фрагмента) относительно более высокой скорости получения последующих копий индексированного фрагмента (или первой копии индексированного фрагмента). В примере из предыдущего абзаца кинетическое исключение происходит вследствие относительно низкой скорости посева индексированных фрагментов (например, относительно медленной диффузии или транспорта) относительно высокой скорости, с которой происходит амплификация и заполнение сайта копиями индексированного фрагмента-затравки. В другом примере осуществления кинетическое исключение может происходить вследствие задержки в образовании первой копии индексированного фрагмента, засеявшего сайт (например, отсроченная или медленная активация), относительно высокой скорости, с которой образуются и заполняют сайт последующие копии. В этом примере конкретный сайт может быть засеян несколькими разными индексированными фрагментами (например, в каждом сайте перед амплификацией могут присутствовать несколько индексированных фрагментов). Однако образование первой копии любого данного индексированного фрагмента можно активировать случайным образом так, чтобы средняя скорость образования первой копии была относительно низкой по сравнению со скоростью, с которой создаются последующие копии. В этом случае, хотя конкретный сайт может быть засеян несколькими разными индексированными фрагментами, кинетическое исключение позволит провести амплификацию только одного из этих индексированных фрагментов. Более конкретно, после активации для амплификации первого индексированного фрагмента сайт быстро заполняется его копиями, и таким образом предотвращается образование на сайте копий второго индексированного фрагмента.
[00152] В одном из вариантов осуществления способ осуществляют с одновременной (i) транспортировкой индексированных фрагментов до сайтов амплификации со средней скоростью транспортировки и (ii) амплификацией индексированных фрагментов, находящихся на сайтах амплификации, со средней скоростью амплификации, причем средняя скорость амплификации превышает среднюю скорость транспортировки (патент США № 9,169,513). Соответственно, в таких вариантах осуществления кинетическое исключение может достигаться за счет использования относительно низкой скорости транспортировки. Например, для достижения желаемой средней скорости транспортировки можно выбирать достаточно низкую концентрацию индексированных фрагментов, более низкие концентрации приводят к более медленным средним скоростям транспортировки. Альтернативно или дополнительно для снижения скорости транспортировки можно использовать раствор с высокой вязкостью и/или введение в раствор реагентов для молекулярного стеснения. Примеры подходящих для использования реагентов для молекулярного стеснения включают, без ограничений, полиэтиленгликоль (ПЭГ), фиколл, декстран или поливиниловый спирт. Примеры реагентов и составов для молекулярного стеснения представлены в патенте США № 7,399,590, включенном в настоящий документ путем ссылки. Еще одним фактором, который можно корректировать для достижения желаемой скорости транспортировки, является средний размер целевых нуклеиновых кислот.
[00153] Амплификационный реагент может включать дополнительные компоненты, которые способствуют образованию ампликонов и в некоторых случаях повышают скорость образования ампликонов. Примером является рекомбиназа. Рекомбиназа может способствовать образованию ампликона, обеспечивая повторяющееся внедрение/удлинение. Более конкретно, рекомбиназа может облегчать внедрение полимеразы в индексированный фрагмент и достройку праймера полимеразой с использованием индексированного фрагмента качестве темплата для образования ампликона. Данный процесс можно повторять как цепную реакцию, в которой ампликоны, полученные в результате каждого цикла внедрения/удлинения, служат в качестве темплатов в последующем цикле. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, облегченная рекомбиназой амплификация может проводиться изотермически. Для облегчения амплификации по существу желательно включать АТФ или другие нуклеотиды (или, в некоторых случаях, их негидролизуемые аналоги) в реагент для облегченной рекомбиназой амплификации. Смесь рекомбиназы и связывающегося с одной цепью белка (SSB) особенно полезна, поскольку SSB может дополнительно облегчать амплификацию. Примеры составов для облегченной рекомбиназой амплификации включают составы, продаваемые в виде наборов TwistAmp производства компании TwistDx (г. Кембридж, Великобритания). Используемые компоненты реагента для облегченной рекомбиназой амплификации и условия реакции представлены в публикациях US 5,223,414 и US 7,399,590.
[00154] Еще одним примером компонента, который можно включить в реагент для облегчения образования ампликонов и в некоторых случаях для повышения скорости образования ампликона, является геликаза. Геликаза может способствовать образованию ампликонов, обеспечивая цепную реакцию образования ампликонов. Процесс может происходить быстрее, чем стандартная ПЦР, поскольку цикл денатурации (например, посредством нагревания или химической денатурации) не требуется. Таким образом, облегченную геликазой амплификацию можно проводить в изотермических условиях. Смесь геликазы и связывающегося с одной цепью белка (SSB) особенно полезна, поскольку SSB может дополнительно облегчать амплификацию. Примеры составов для облегченной геликазой амплификации включают составы, доступные в продаже в виде наборов IsoAmp производства компании Biohelix (г. Беверли, штат Массачусетс, США). Кроме того, примеры подходящих для использования составов, включающих геликазный белок, описаны в US 7,399,590 и US 7,829,284.
[00155] Еще одним примером компонента, который можно включать в амплификационный реагент для облегчения образования ампликонов и в некоторых случаях для повышения скорости образования ампликона, является белок, связывающийся с исходной молекулой.
[00156] Способы секвенирования
[00157] После прикрепления индексированных фрагментов к поверхности определяют последовательность иммобилизованных и амплифицированных индексированных фрагментов. Секвенирование может быть комплексным или прицельным. Если желательна вся последовательность каждой клетки или ядра, присутствующего в библиотеке, можно использовать комплексное секвенирование. Примеры приложений, в которых используют комплексное секвенирование, включают, без ограничений, полногеномное секвенирование, полнотранскриптомное секвенирование и секвенирование для анализа хроматина, доступного для транспозаз (ATAC). Когда желательна информация относительно биологического признака, можно использовать прицельное секвенирование. В одном варианте осуществления прицельное секвенирование можно использовать при идентификации субпопуляции клеток или ядер, или подгруппы генома, подгруппы транскриптома, подгруппы протеома или любой их комбинации, и подробно описано в настоящем документе.
[00158] Секвенирование можно проводить с использованием любой подходящей методики секвенирования, и способы определения последовательности иммобилизованных и амплифицированных индексированных фрагментов, включающие повторный синтез цепи, известны в данной области и описаны, например, Bignell et al. (US 8,053,192), Gunderson et al. (WO2016/130704), Shen et al. (US 8,895,249) и Pipenburg et al. (US 9,309,502).
[00159] Способы, описанные в настоящем документе, можно использовать в сочетании с разнообразными методиками секвенирования нуклеиновых кислот. В частности, к применимым методикам относятся методики, в которых нуклеиновые кислоты крепятся в фиксированных положениях на матрице таким образом, чтобы их относительные положения не изменялись, и в которых матрицу многократно визуализируют. Особенно подходящими являются варианты осуществления, в которых изображения получены в разных цветовых каналах, например, в соответствии с разными метками, используемыми для различения одного типа нуклеотидных оснований от другого. В некоторых вариантах осуществления процесс определения нуклеотидной последовательности индексированного фрагмента может представлять собой автоматизированный процесс. Предпочтительные варианты осуществления включают методики последовательного синтеза (SBS).
[00160] Методики SBS по существу включают ферментативное удлинение появляющейся полинуклеотидной цепи посредством итерационного добавления нуклеотидов в соответствии с цепью-темплатом. В традиционных способах SBS однонуклеотидный мономер можно вводить в целевой нуклеотид в присутствии полимеразы при каждой доставке. Однако в способах, описанных в настоящем документе, в целевую нуклеиновую кислоту при доставке в присутствии полимеразы можно вводить более одного типа нуклеотидного мономера.
[00161] В одном из вариантов осуществления нуклеотидный мономер включает запертые нуклеиновые кислоты (ЗНК) или мостиковые нуклеиновые кислоты (МНК). Использование ЗНК или МНК в нуклеотидном мономере повышает прочность гибридизации между нуклеотидным мономером и последовательностью праймера секвенирования, присутствующей на иммобилизованном индексированном фрагменте.
[00162] При SBS можно использовать нуклеотидные мономеры, имеющие терминаторную функциональную группу, или мономеры, которые не имеют терминаторных функциональных групп. Способы с использованием не имеющих терминаторов нуклеотидных мономеров включают, например, пиросеквенирование и секвенирование с использованием меченных γ-фосфатом нуклеотидов, как более подробно описано в настоящем документе. В способах с использованием нуклеотидных мономеров, не содержащих терминаторов, количество добавляемых в каждом цикле нуклеотидов по существу варьирует и зависит от шаблонной последовательности и способа подачи нуклеотидов. В случае методик SBS, в которых используют нуклеотидные мономеры, имеющие терминаторную функциональную группу, терминатор может быть практически необратимым в используемых условиях секвенирования, как в случае традиционного секвенирования по Сэнгеру, в котором используют дидезоксинуклеотиды, или терминатор может быть обратимым, как в случае способов секвенирования, разработанных Solexa (в настоящее время - Illumina, Inc.).
[00163] В методиках SBS могут использоваться нуклеотидные мономеры, имеющий маркировочную функциональную группу, или мономеры, не имеющие маркировочной функциональной группы. Соответственно, события включения могут обнаруживаться на основе характеристики метки, например флуоресценции метки; характеристики нуклеотидного мономера, например молекулярной массы или заряда; побочного продукта встраивания нуклеотида, например высвобождения пирофосфата; или т. п. В вариантах осуществления, в которых в реагенте для секвенирования присутствуют два или более разных нуклеотида, разные нуклеотиды могут быть отличимы друг от друга, или альтернативно две или более разные метки могут являться неотличимыми используемыми методиками детектирования. Например, разные нуклеотиды, присутствующие в реагенте для секвенирования, могут иметь разные метки, и их можно различать с помощью соответствующих оптических устройств, примеры которых представлены способами секвенирования, разработанными Solexa (в настоящее время - Illumina, Inc.).
[00164] Предпочтительные варианты осуществления включают методики пиросеквенирования. Пиросеквенирование обнаруживает высвобождение неорганического пирофосфата (PPi) при встраивании определенных нуклеотидов в формирующуюся цепь (Ronaghi, M., Karamohamed, S., Pettersson, B., Uhlen, M. and Nyren, P. (1996) «Real-time DNA sequencing using detection of pyrophosphate release». Analytical Biochemistry 242(1), 84-9; Ronaghi, M. (2001) «Pyrosequencing sheds light on DNA sequencing.» Genome Res. 11(1), 3-11; Ronaghi, M., Uhlen, M. and Nyren, P. (1998) «A sequencing method based on real-time pyrophosphate.» Science 281(5375), 363; патенты США № 6,210,891; 6,258,568 и 6,274,320). Во время пиросеквенирования выделяющийся PPi можно обнаружить посредством ему превращения сразу в аденозинтрифосфат (АТФ) АТФ-сульфуразой, а уровень выработанного АТФ обнаруживают по продукции фотонов люциферазой. Нуклеиновые кислоты, подлежащие секвенированию, можно прикрепить к элементам матрицы, и матрицу можно визуализировать с регистрацией хемилюминесцентных сигналов, продуцируемых вследствие внедрения нуклеотидов в элементы матрицы. Изображение можно получить после обработки матрицы конкретным типом нуклеотида (например, A, T, Ц или Г). Изображения, полученные после добавления каждого типа нуклеотидов, будут отличаться в зависимости от того, какие элементы матрицы обнаруживаются. Эти различия в изображении отражают различающийся состав последовательности на элементах матрицы. Однако относительные местоположения каждого элемента на изображениях останутся неизменными. Изображения можно сохранять, обрабатывать и анализировать с помощью способов, описанных в настоящем документе. Например, изображения, полученные после обработки матрицы каждым из разных типов нуклеотидов, можно обрабатывать так, как показано в примерах настоящего документа для изображений, полученных из разных каналов обнаружения в способах секвенирования на основе обратимых терминаторов.
[00165] В другом примере типа SBS цикл секвенирования выполняют путем постадийного добавления обратимых терминаторных нуклеотидов, содержащих, например, отщепляемую или светообесцвечиваемую метку-краситель, как описано, например, в WO 04/018497 и патенте США № 7,057,026. Такой подход введен в коммерческое использование компанией Solexa (в настоящее время - Illumina Inc.), а также описан в WO 91/06678 и WO 07/123,744. Наличие флуоресцентно-меченных терминаторов, причем когда возможно как обращение терминирования, так и отщепление флуоресцентной метки, способствует эффективному секвенированию с циклической обратимой терминацией (CRT). Также можно одновременно сконструировать полимеразы, способные эффективно встраивать эти модифицированные нуклеотиды и выполнять с ними удлинение цепи.
[00166] В некоторых вариантах осуществления секвенирования на основе обратимых терминаторов метки по существу не ингибируют удлинение цепи в условиях реакции SBS. Однако детекторные метки могут быть выполнены с возможностью удаления, например, посредством отщепления или деградации. Изображения можно получить после встраивания меток в элементы матрицы с нуклеиновыми кислотами. В конкретных вариантах осуществления каждый цикл включает одновременную доставку к матрице четырех разных типов нуклеотидов, и каждый тип нуклеотидов имеет спектрально отличающуюся метку. Затем можно получить четыре изображения, каждое из них с помощью канала обнаружения, селективного для одной из четырех разных меток. Альтернативно разные типы нуклеотидов могут добавляться последовательно, и изображение матрицы можно получать после каждой стадии добавления. В таких вариантах осуществления на каждом изображении будут показаны элементы с нуклеиновыми кислотами, в которые встроены нуклеотиды конкретного типа. Различные элементы будут присутствовать или отсутствовать на разных изображениях из-за разного состава последовательностей на каждом элементе. Однако относительное положение элементов на изображениях останется неизменным. Изображения, полученные такими способами SBS с обратимым терминатором, можно сохранять, обрабатывать и анализировать, как описано в настоящем документе. После стадии захвата изображения метки можно удалять, а также можно удалять обратимые терминаторные функциональные группы для последующих циклов добавления и обнаружения нуклеотидов. Удаление меток после их обнаружения в конкретном цикле и перед последующим циклом может обеспечивать преимущество, которое заключается в снижении фонового сигнала и перекрестных помех между циклами. Примеры подходящих меток и способов удаления описаны в настоящем документе.
[00167] В конкретных вариантах осуществления некоторые или все из нуклеотидных мономеров могут включать обратимые терминаторы. В таких вариантах осуществления обратимые терминаторы/отщепляемые флуорофоры могут включать флуорофоры, связанные с функциональной группой рибозы посредством 3'-сложноэфирной связи (Metzker, Genome Res. 15:1767-1776 (2005)). При других подходах химические реакции терминаторов отделены от отщепления флуоресцентной метки (Ruparel et al., Proc Natl Acad Sci USA 102: 5932-7 (2005)). В публикации Ruparel et al. описана разработка обратимых терминаторов, в которых для блокирования удлинения цепи использовали небольшую 3’-аллильную группу, но их можно легко разблокировать краткой обработкой палладиевым катализатором. Флуорофор присоединяли к основанию посредством фотоотщепляемого линкера, который легко расщеплялся при 30-секундном воздействии длинноволнового УФ-излучения. Таким образом, в качестве расщепляемого линкера можно использовать либо дисульфидное восстановление, либо фоторасщепление. Другим подходом к обратимой терминации является использование естественной терминации цепи, происходящей после помещения громоздкого красителя на дНТФ. Присутствие заряженного громоздкого красителя на дНТФ может выступать в качестве эффективного терминатора за счет стерического и/или электростатического затруднения. Одно событии внедрения предотвращает дальнейшее внедрение, пока краситель не будет удален. Расщепление красителя удаляет флуорофор и эффективно обращает терминацию. Примеры модифицированных нуклеотидов также описаны в патентах США № 7,427,673 и 7,057,026.
[00168] Дополнительные примеры систем и способов SBS, которые можно использовать со способами и системами, описанными в настоящем документе, описаны в публикациях США № 2007/0166705, 2006/0188901, 2006/0240439, 2006/0281109, 2012/0270305 и 2013/0260372, патенте США № 7,057,026, публикации PCT № WO 05/065814, опубликованной заявке на патент США № 2005/0100900 и публикациях PCT № WO 06/064199 и WO 07/010,251.
[00169] В некоторых вариантах осуществления может использоваться обнаружение четырех разных нуклеотидов с использованием менее четырех разных меток. Например, SBS может быть выполнено с использованием способов и систем, описанных во включенных материалах патентной публикации США № 2013/0079232. В качестве первого примера, пара типов нуклеотидов могут обнаруживаться на одной и той же длине волны, но различаться по значениям интенсивности членов одной пары или по изменению одного члена пары (например, посредством химической, фотохимической или физической модификации), что приводит к появлению или исчезновению явного сигнала по сравнению с сигналом, обнаруживаемым для другого члена пары. В качестве второго примера, три из четырех разных типов нуклеотидов могут обнаруживаться в конкретных условиях, в то время как четвертый тип нуклеотидов не имеет метки, которая может быть обнаружена в этих условиях или минимально обнаруживается в этих условиях (например, минимальное обнаружение вследствие фоновой флуоресценции и т. д.). Встраивание первых трех типов нуклеотидов в нуклеиновую кислоту можно определить на основе наличия соответствующих им сигналов, а встраивание нуклеотидов четвертого типа в нуклеиновую кислоту можно определить на основе отсутствия или минимального обнаружения любого сигнала. В качестве третьего примера, один тип нуклеотидов может включать метку (-и), которая (-ые) обнаруживается (-ются) в двух разных каналах, тогда как другие типы нуклеотидов обнаруживаются не более чем в одном из каналов. Вышеупомянутые три примера конфигураций не считаются взаимоисключающими и могут использоваться в различных комбинациях. Примером варианта осуществления, объединяющим все три примера, является способ SBS на основе флуоресценции, в котором используется первый тип нуклеотидов, обнаруживаемый в первом канале (например, дАТФ, имеющий метку, обнаруживаемую в первом канале при возбуждении первой длиной волны возбуждения), второй тип нуклеотидов, обнаруживаемый во втором канале (например, дЦТФ, имеющий метку, обнаруживаемую во втором канале при возбуждении второй длиной волны возбуждения), третий тип нуклеотидов, обнаруживаемый как в первом, так и во втором каналах (например, дТТФ, имеющий по меньшей мере одну метку, обнаруживаемую в обоих каналах при возбуждении первой и/или второй длинами волн возбуждения), и четвертый тип нуклеотидов, в котором отсутствует метка, которая не обнаруживается или минимально обнаруживается в любом канале (например, дГТФ без метки).
[00170] Кроме того, как описано во включенных в настоящий документ материалах публикации США № 2013/0079232, данные секвенирования могут быть получены с использованием одного канала. В таких так называемых однопигментных подходах к секвенированию первый тип нуклеотидов маркируют, но метку удаляют после создания первого изображения, а второй тип нуклеотидов маркируют только после создания первого изображения. Третий тип нуклеотидов сохраняет свою метку как на первом, так и на втором изображениях, а четвертый тип нуклеотидов остается немаркированным на обоих изображениях.
[00171] В некоторых вариантах осуществления может использоваться секвенирование посредством методик лигирования. В таких методиках используют ДНК-лигазу для встраивания олигонуклеотидов и идентифицируют встраивание таких олигонуклеотидов. Олигонуклеотиды, как правило, имеют разные метки, коррелирующие с идентичностью конкретного нуклеотида в последовательности, с которой гибридизуются олигонуклеотиды. Как и в случае с другими способами SBS, изображения можно получить после обработки матрицы из элементов с нуклеиновыми кислотами мечеными реагентами для секвенирования. На каждом изображении будут видны элементы с нуклеиновыми кислотами, имеющими внедренные метки конкретного типа. Разные элементы будут присутствовать или отсутствовать на разных изображениях из-за разного состава последовательности в каждом элементе, но относительное местоположение элементов на изображениях останется неизменным. Изображения, полученные способами секвенирования на основе лигирования, можно хранить, обрабатывать и анализировать, как описано в настоящем документе. Примеры систем и способов SBS, которые можно использовать со способами и системами, описанными в настоящем документе, описаны в патентах США № 6,969,488, 6,172,218 и 6,306,597.
[00172] В некоторых вариантах осуществления можно использовать секвенирование через нанопоры (Deamer, D. W. & Akeson, M. «Nanopores and nucleic acids: prospects for ultrarapid sequencing.» Trends Biotechnol. 18, 147-151 (2000); Deamer, D. and D. Branton, «Characterization of nucleic acids by nanopore analysis», Acc. Chem. Res. 35:817-825 (2002); Li, J., M. Gershow, D. Stein, E. Brandin, and J. A. Golovchenko, «DNA molecules and configurations in a solid-state nanopore microscope» Nat. Mater. 2:611-615 (2003)). В таких вариантах осуществления индексированный фрагмент проходит через нанопору. Нанопора может представлять собой синтетическую пору или биологический мембранный белок, такой как α-гемолизин. По мере прохождения индексированного фрагмента через нанопору каждую пару нуклеотидов можно определять посредством измерения флуктуаций электрической проводимости поры. (патент США № 7,001,792; Soni, G. V. & Meller, «A. Progress toward ultrafast DNA sequencing using solid-state nanopores.» Clin. Chem. 53, 1996-2001 (2007); Healy, K. «Nanopore-based single-molecule DNA analysis.» Nanomed. 2, 459-481 (2007); Cockroft, S. L., Chu, J., Amorin, M. & Ghadiri, M. R. «A single-molecule nanopore device detects DNA polymerase activity with single-nucleotide resolution.» J. Am. Chem. Soc. 130, 818-820 (2008)). Данные, полученные в результате секвенирования через нанопоры, можно хранить, обрабатывать и анализировать, как описано в настоящем документе. В частности, данные можно рассматривать как изображение в соответствии с примером обработки оптических изображений и других изображений, описанных в настоящем документе.
[00173] В некоторых вариантах осуществления могут использоваться способы, включающие мониторинг активности ДНК-полимеразы в режиме реального времени. Внедрения нуклеотидов могут быть обнаружены посредством взаимодействий резонансного переноса энергии флуоресценции (FRET) между несущей флуорофор полимеразой и γ-фосфат-меченными нуклеотидами, как описано, например, в патентах США № 7,329,492 и 7,211,414, или внедрения нуклеотидов могут быть обнаружены с помощью волноводов с нулевой модой, как описано, например, в патенте США № 7,315,019, и с использованием флуоресцентных аналогов нуклеотидов и сконструированных полимераз, как описано, например, в патенте США № 7,405,281 и патентной публикации США № 2008/0108082. Освещение можно ограничивать объемом порядка зептолитра вокруг связанной с поверхностью полимеразы таким образом, чтобы можно было наблюдать встраивание флуоресцентно-меченных нуклеотидов при низком фоне (Levene, M. J. et al. «Zero-mode waveguides for single-molecule analysis at high concentrations.» Science 299, 682-686 (2003); Lundquist, P. M. et al. «Parallel confocal detection of single molecules in real time.» Opt. Lett. 33, 1026-1028 (2008); Korlach, J. et al. «Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nano structures.» Proc. Natl. Acad. Sci. USA 105, 1176-1181 (2008)). Изображения, полученные такими способами, могут быть сохранены, обработаны и проанализированы, как описано в настоящем документе.
[00174] Некоторые варианты осуществления SBS включают обнаружение протона, высвобождаемого при встраивании нуклеотида в продукт удлинения. Например, при секвенировании на основе обнаружения высвобожденных протонов может использоваться электрический детектор и связанные с ним методики, которые доступны в продаже от компании Ion Torrent (г. Гилфорд, штат Коннектикут, США, филиал компании Life Technologies), или способы и системы секвенирования, описанные в публикациях США № 2009/0026082; 2009/0127589; 2010/0137143; и 2010/0282617. Описанные в настоящем документе способы амплификации целевых нуклеиновых кислот с использованием кинетического исключения могут быть легко применены к субстратам, используемым для обнаружения протонов. Более конкретно, способы, изложенные в настоящем документе, можно использовать для получения клональных популяций ампликонов, используемых для обнаружения протонов.
[00175] Преимуществом является то, что вышеописанные способы SBS можно реализовать в мультиплексных форматах таким образом, чтобы одновременно манипулировать множеством разных индексированных фрагментов. В конкретных вариантах осуществления разные индексированные фрагменты можно обрабатывать в общем реакционном сосуде или на поверхности конкретного субстрата. Это позволяет удобно доставлять реагенты для секвенирования, удалять непрореагировавшие реагенты и обнаруживать события внедрения мультиплексным способом. В вариантах осуществления, в которых используют связанные с поверхностью целевые нуклеиновые кислоты, индексированные фрагменты могут иметь форму матрицы. В формате матрицы индексированные фрагменты, как правило, могут быть связаны с поверхностью пространственно различимым образом. Индексированные фрагменты могут быть связаны посредством прямой ковалентной связи, присоединения к грануле или другой частице, либо посредством связывания с полимеразой или другой молекулой, которая присоединена к поверхности. Матрица может включать одиночную копию индексированного фрагмента в каждом сайте (также называемом элементом), или же в каждом сайте или элементе может присутствовать множество копий, имеющих одну и ту же последовательность. С помощью способов амплификации, таких как мостиковая амплификация или эмульсионная ПЦР, можно получать множество копий, как более подробно описано в настоящем документе.
[00176] В способах, описанных в настоящем документе, можно использовать матрицы, имеющие элементы с любым из разнообразных значений плотности, включая, например, по меньшей мере около 10 элементов/см2, 100 элементов/см2, 500 элементов/см2, 1000 элементов/см2, 5000 элементов/см2, 10 000 элементов/см2, 50 000 элементов/см2, 100 000 элементов/см2, 1 000 000 элементов/см2, 5 000 000 элементов/см2 или выше.
[00177] Преимущество способов, описанных в настоящем документе, заключается в том, что они обеспечивают быстрое и эффективное обнаружение на множестве см2 параллельно. Соответственно, в настоящем описании предложены интегрированные системы, способные подготавливать и обнаруживать нуклеиновые кислоты с использованием методик, известных в данной области, например, как в примерах, приведенных в настоящем документе. Таким образом, интегрированная система настоящего описания может включать жидкостные компоненты, способные доставлять реагенты для амплификации и/или реагенты для секвенирования к одному или более иммобилизованным индексированным фрагментам, система включает такие компоненты, как насосы, клапаны, резервуары, жидкостные магистрали и т. п. Проточная кювета может быть выполнена и/или использована в интегрированной системе для обнаружения целевых нуклеиновых кислот. Примеры проточных кювет описаны, например, в публикации США № 2010/0111768 и публ. США с сер. № 13/273,666. Как показано в примере проточных кювет, один или более компонентов для работы с жидкостями интегрированной системы можно использовать для способа амплификации и для способа обнаружения. Если взять в качестве примера вариант осуществления секвенирования нуклеиновых кислот, то один или более компонентов для работы с жидкостями интегрированной системы можно использовать для способа амплификации, описанного в настоящем документе, и для доставки реагентов для секвенирования в способе секвенирования, таком как описанный в примерах выше. Альтернативно интегрированная система может включать отдельные системы для работы с жидкостями для реализации способов амплификации и для реализации способов обнаружения. Примеры интегрированных систем секвенирования, способных создавать амплифицированные нуклеиновые кислоты и также определять последовательность нуклеиновых кислот, включают, без ограничений, платформу MiSeqTM (Illumina, Inc., Сан-Диего, штат Калифорния, США) и устройства, описанные в публ. США сер. № 13/273,666.
[00178] Обнаружение редких событий
[00179] В настоящем описании также предложены способы идентификации и/или определения характеристик редких событий. В настоящее время способы определения характеристик редких событий в популяции без обогащения являются дорогостоящими и сложными. При использовании обогащения отбор обычно основан на некоторых биологических признаках клетки, таких как размер, морфология или присутствие на поверхности клетки идентифицируемой молекулы, такой как белок или гликан. Это приводит к ограничению типов событий, которые можно определять. Способы, представленные в настоящем документе, обеспечивают существенное улучшение в способности определять и/или давать характеристику присутствия редких событий. В целом в изобретении обеспечена идентификация, обогащение и характеристика на основе последовательности подгруппы редких одиночных клеток, присутствующих в библиотеке миллионов или миллиардов клеток. Идентификацию редких одиночных клеток можно применять для создания клеточной базы данных, которую могут использовать исследователи для определения того, какие клетки можно использовать в дополнительном анализе.
[00180] Примеры редких событий включают, без ограничений, редкие клетки в большой популяции клеток. Типы редких клеток включают, без ограничений, класс клеток, видовой тип, состояние или риск заболевания. Примеры классов редких клеток включают, без ограничений, клетки от индивидуума, имеющего изменение, например, генома, транскриптома или эпигенома. Примеры редких видовых типов включают, без ограничений, прокариотические, эукариотические или грибковые клетки. Примеры редких клеток, связанных с состоянием или риском развития заболевания, включают, без ограничений, раковые клетки.
[00181] Редкое событие обычно определено присутствием биологического признака, обычно нуклеотидной последовательности, которая коррелирует с редким событием. В одном варианте осуществления биологическим признаком является биомолекула, такая как белок, гликан, протеогликан или липид. Биомолекула может быть помечена нуклеиновой кислотой, которая прикреплена к соединению, такому как антитело, которое специфически связывает биомолекулу. Биологический признак может быть известен изначально (например, известный до практического применения способа, также называемый предопределенным) или de novo (например, биологический признак идентифицирован после целевого или комплексного секвенирования, описанного в настоящем документе).
[00182] Пример биологического признака, относящегося к геному, включает, без ограничений, изменение в иммунной клетке, такое как перестройка генов. Примером биологического признака, относящегося к транскриптому, является экспрессия одного или более определенных генов, или молекул РНК, или экспрессия определенного белка. Примеры биологических признаков, относящихся к эпигеному, включают эпигенетические паттерны, такие как, без ограничений, метка метилирования, паттерн метилирования и доступная ДНК, или экспрессия определенного белка, которая коррелирует с эпигенетическим изменением. Примеры биологических признаков, которые коррелируют с редкими видовыми типами, включают 16s рРНК или рДНК, 18s рРНК или рДНК и внутренний транскрибированный спейсер (ITS) рРНК/рДНК или экспрессию определенного белка редкими видами. Примеры биологических признаков, относящихся к состоянию или риску развития заболевания, включают клетки зародышевой линии или соматические клетки, имеющие вариантную последовательность ДНК или вариантный паттерн экспрессии РНК и/или белка, которые коррелируют с заболеванием, таким как рак.
[00183] Способ может включать определение членов библиотеки секвенирования - отдельных модифицированных целевых нуклеиновых кислот, - которые содержат редкое событие. В одном варианте осуществления способ может включать опрос библиотеки секвенирования, которая, предположительно, содержит редкое событие. Анализ библиотеки секвенирования, как правило, включает определение последовательности из двух типов нуклеотидных областей, присутствующих в библиотеке; (i) биологический признак, который коррелирует с редким событием, и (ii) индексы, присутствующие на членах библиотеки. В одном варианте осуществления можно определять последовательность более одного биологического признака.
[00184] В одном варианте осуществления нуклеотидную последовательность биологического признака идентифицируют путем прицельного секвенирования. Способы прицельного секвенирования известны в данной области и могут включать использование праймера, который гибридизуется вблизи биологического признака в местоположении и ориентации, которая служит в качестве инициирующего сайта для секвенирования. Например, когда биологическим признаком является наличие определенного однонуклеотидного полиморфизма (SNP), можно разработать праймер, который будет специфически гибридизироваться с нуклеотидами вблизи SNP. В другом примере, когда биологическим признаком является белок, можно создать праймер, который специфически отжигается к нуклеотидам нуклеиновой кислоты, которая присоединена к соединению, специфически связанному с биомолекулой. Результатом являются данные о последовательности, которые позволяют квалифицированному специалисту определить, какие члены библиотеки включают интересующий биологический признак. Определение последовательности индексов, присутствующих на членах библиотеки секвенирования, является стандартной частью методик комбинаторного индексирования одиночной клетки.
[00185] Затем данные о последовательности от прицельного секвенирования биологического признака и секвенировании индексов анализируют с использованием стандартных биоинформатических способов и определяют те комбинации индексных последовательностей, которые присутствуют на одних и тех же членах библиотеки, что и биологический признак. Эта корреляция биологического признака и индексных последовательностей приводит к определению подгруппы членов библиотеки, причем каждый член включает биологический признак и уникальную группу индексных последовательностей, и созданию клеточной базы данных. Каждая уникальная группа индексных последовательностей, также называемая в настоящем документе «маркерной индексной последовательностью», аналогичным образом присутствует на других членах библиотеки, которые происходят из одной и той же клетки или ядра, например представляющих интерес индексированных библиотек. В одном варианте осуществления маркерные индексные последовательности представляют собой связные индексы, т. е. наборы из множества индексов, присутствующих на элементах библиотеки в ряду с 0, 1, 2, 3, 4 или более нуклеотидов между каждым из индексов. Как описано в настоящем документе, данные маркерные индексные последовательности можно использовать для фокусировки последующих усилий по секвенированию на тех членах библиотеки, которые происходят от клеток или ядер, обладающих биологическим признаком, и, таким образом, снижения стоимости.
[00186] Способ может дополнительно включать изменение библиотеки секвенирования для увеличения представления тех членов библиотеки, которые происходят от клеток или ядер, которые имеют биологический признак. Такое изменение может включать обогащение (например, положительный отбор тех редких членов библиотеки, которые включают требуемой маркерной индексной последовательность) или истощение (например, отрицательный отбор, такой как селективное удаление тех членов библиотеки, которые не включают требуемую маркерную индексную последовательность).
[00187] Обогащение и истощение могут включать использование маркерных индексных последовательностей. Способы обогащения и истощения известны специалистам в данной области и включают, без ограничений, способы на основе гибридизации, такие как амплификация, специфичная для маркерной индексной последовательности (например, адаптер-фиксированная ПЦР), гибридный захват и CRISPR (d)Cas9. Для способов обогащения и истощения предпочтительно использование нуклеотидной последовательности, которая специфически гибридизуется с желаемыми маркерными индексными последовательностями. Таким образом, обогащение или истощение можно выполнять в библиотеках, содержащих связные индексы, т. е. набор множества индексов, присутствующих на членах библиотеки в ряду с 0, 1, 2, 3, 4 или более нуклеотидов между каждым из индексов (см. ФИГ. 5B). Связные индексы, коррелирующие с желаемым биологическим признаком, можно положительно выбирать и сохранять, что приведет к обогащению желаемых членов библиотеки. В альтернативном варианте осуществления можно выбирать и удалять связные индексы, которые не коррелируют с желаемым биологическим признаком, что приводит к истощению элементов библиотеки, которые коррелируют с большим количеством клеток, и de facto обогащению членов библиотеки, которые коррелируют с желаемым биологическим признаком. В одном варианте осуществления обогащение можно сочетать с целенаправленной амплификацией. Например, после создания библиотеки секвенирования реакцию амплификации можно использовать для специфической амплификации членов библиотеки, содержащих интересующий биологический признак. В одном варианте осуществления специфической амплификации можно достигать с использованием праймера, специфического для биологического признака, выполненного с возможностью отжига к нуклеотидной последовательности, имеющей биологический признак, и второго праймера, который отжигается к одной стороне всех членов библиотеки. Праймер, специфичный для биологического признака, может на своем 5'-конце включать один или более индексов и/или универсальных последовательностей.
[00188] Общая длина связного индекса зависит от размера зонда, необходимого для специфической гибридизации между зондом и членами библиотеки, имеющими желаемые маркерные индексные последовательности. В некоторых вариантах осуществления общая длина связного индекса (и, следовательно, маркерной индексной последовательности) составляет по меньшей мере 40, по меньшей мере 45, по меньшей мере 50 или по меньшей мере 55 нуклеотидов и не более 80, не более 75, не более 70 или не более 65 нуклеотидов. В одном варианте осуществления общая длина связного индекса составляет 60 нуклеотидов.
[00189] Использование обогащения либо истощения приводит к получению подбиблиотеки, которая включает увеличенное представительство тех членов библиотеки, которые происходят от клеток или ядер, обладающих биологическим признаком. Комплексное секвенирование подбиблиотеки можно проводить с использованием стандартных способов, включая описанные в настоящем документе. Увеличение представительства достаточно велико, чтобы для комплексного секвенирования потребовалось значительно меньше ресурсов и, следовательно, оно было экономически эффективным. Результатом использования комплексного секвенирования подбиблиотеки может быть идентификация одного или более дополнительных неизвестных биологических признаков.
[00190] Приложения
[00191] Способы, предложенные в настоящем описании, можно легко интегрировать в по существу любой вариант применения, который включает препарат библиотеки секвенирования, такой как полный геном, транскриптом, эпигеном, доступное (например, ATAC) и конформационное состояние (например, определение конформации хромосом (HiC)). Специалисту в данной области известно множество способов библиотеки секвенирования, которые можно использовать при создании полногеномных или прицельных библиотек (см., например, Sequencing Methods Review, доступную во Всемирной сети по адресу genomics.umn.edu/downloads/sequencing-methods-review.pdf).
[00192] В тех вариантах осуществления, которые относятся к обнаружению редких событий, способы, обеспеченные в настоящем описании, можно легко интегрировать в по существу любой вариант применения с способами комбинаторного индексирования одиночных клеток (sci), включая, без ограничений, полногеномный (например, sci-WGS-seq), эпигеномный (например, sci-MET-seq), доступный (например, sci-ATAC-seq), транскриптомный (sci-RNA-seq) и конформационный (sci-HiC-seq). В некоторых вариантах осуществления вариант применения включает использование конформационного комбинаторного индексирования одиночных клеток, которое включает лигирование на близком расстоянии с методиками связанного длинного прочтения со сшиванием. В некоторых вариантах осуществления вариант применения представляет собой совместный анализ, в котором одновременно оценивают два или более различных аналитов или информацию из образца. Примеры аналитов включают, без ограничений, ДНК, РНК и белок (например, поверхностный белок). Примеры включают, без ограничений, анализы, в которых анализируют полный геном и транскриптом, или ATAC и транскриптом (Ma et al., 2020, bioRxiv, DOI: doi.org/10.1016/j.cell.2020.09.056).
[00193] В некоторых вариантах осуществления вариант применения является метагеномикой - исследованием генетического материала, происходящего непосредственно из образцов окружающей среды. Примеры сред включают те, которые присутствуют в областях, относящихся к сельскому хозяйству (например, почвы), биотопливу (например, микробные сообщества, преобразующие биомассу), биотехнологии (например, микробные сообщества, которые продуцируют биологически активные вещества) и кишечной микробиоте (например, микробные сообщества, присутствующие в микробиоме человека или животного). Генетический материал может присутствовать в прокариотических и/или эукариотических микроорганизмах (как одноклеточных, так и многоклеточных), включая клетки грибов. Способы, описанные в настоящем документе, можно использовать для определения редких клеток независимо от того, возможна ли их культивация или нет. Биологические признаки, которые можно использовать для определения редких событий в метагеномике, включают, без ограничений, 16s рРНК или рДНК, 18s рРНК или рДНК и внутренний транскрибированный спейсер (ITS) рРНК/рДНК, или белок, кодируемый микроорганизмом. После идентификации редкие клетки можно комплексно секвенировать.
[00194] В некоторых вариантах осуществления вариант применения относится к состоянию или риску заболевания. Можно определять редкие события, такие как, без ограничений, однонуклеотидные полиморфизмы (SNP) и/или биомаркеры, которые коррелируют с заболеванием или риском заболевания, а те клетки, которые имеют SNP и/или биомаркер - комплексно секвенировать. Например, жидкий биоптат циркулирующих клеток в кровотоке субъекта или клеток биоптата ткани можно анализировать на наличие редких явлений, связанных с заболеванием или риском заболевания. Редкие события, которые можно проанализировать, включают, без ограничений, соматические драйверные мутации, с помощью которых можно определить конкретный рак. Родственный вариант применения заключается в определении всех характеристик и отслеживании развития опухоли путем получения образцов от субъекта в течение интервала времени, отбора тех клеток или ядер, которые являются раковыми, и последующего полного секвенирования подгруппы опухолевых клеток.
[00195] В некоторых вариантах осуществления вариант применения относится к иммунным клеткам. Иммунные клетки подвергаются специфическим перестройкам генов, относящимся к способности приобретенной иммунной системы определять чужеродные молекулы. Примеры иммунных клеток, которые подвергаются перестройке генов, включают, без ограничений, Т-клетки (например, перестройка Т-клеточного рецептора), антигенпредставляющие клетки (например, перестройка генов, кодирующих белки главного комплекса гистосовместимости) и В-клетки (например, перестройка генов, кодирующих антитело). Биологический признак, относящийся к изменению в иммунной клетке, может представлять собой, без ограничений, специфическую перестройку или белок, полученный в результате специфической перестройки. Иммунные клетки, имеющие специфические изменения, можно полностью характеризовать и отслеживать, включая, без ограничений, характеристику и развитие набора Т-клеточных рецепторов. В другом варианте осуществления вариант применения относится к дифференцировке клеток. Например, уровни экспрессии и/или метилирования в различных областях можно использовать для оценки событий дифференцировки, таких как корреляции между доступностью и экспрессией.
[00196] Не имеющий ограничительного характера иллюстративный вариант осуществления настоящего описания показан на ФИГ. 6. В этом варианте осуществления способ идентификации и характеристики наборов Т-клеточных рецепторов может включать обеспечение множества клеток (ФИГ. 6, блок 600) и распределение подгрупп клеток во множество компартментов (ФИГ. 6, блок 601). Множество клеток можно получать, например, из образца крови или образца лимфатического узла. Нуклеиновые кислоты, присутствующие в клетках каждого компартмента, модифицируют путем вставки индекса (ФИГ. 6, блок 602), и клетки затем объединяют (ФИГ. 6, блок 603). Дополнительные индексы добавляют на стадиях «разделения и объединения», на которых повторяют распределение (ФИГ. 6, блок 601), добавление индексов (ФИГ. 6, блок 602) и объединение (ФИГ. 6, блок 603) подгрупп. В одном варианте осуществления каждый индекс добавляют на одну и ту же сторону членов библиотеки для получения связного индекса (см. ФИГ. 5B). Универсальную последовательность можно необязательно добавлять с одним или более индексов. После добавления последнего индекса библиотеки нуклеиновых кислот в ядрах или клетках можно объединять (ФИГ. 6, блок 603) и дополнительно обрабатывать для подготовки к прицельному секвенированию интересующего биологического признака, например биологического признака, который позволяет идентифицировать Т-клеточные рецепторы, которые включают специфическую нуклеотидную последовательность, такую как та, которая может связываться с биомолекулой микроорганизма или вируса, и секвенирование индексов, связанных с интересующим биологическим признаком (ФИГ. 6, блок 604). Анализ последовательности (ФИГ. 6, блок 605) используют для идентификации маркерных индексных последовательностей, т. е. уникальных групп индексных последовательностей. Идентифицированные маркерные индексные последовательности - это (i) те последовательности, которые коррелируют с биологическим признаком и, следовательно, определяют членов библиотеки, происходящих из редких клеток; или (ii) те последовательности, которые не коррелируют с биологическим признаком и, следовательно, определяют членов библиотеки, происходящих из широко распространенных клеток. На следующих стадиях данного иллюстративного варианта осуществления описано истощение широко распространенных членов библиотеки, однако способ можно изменять так, как описано в настоящем документе, для включения обогащения редких членов библиотеки. Специфические олигонуклеотиды или последовательности гидовой РНК можно получать с возможностью гибридизации с маркерными индексными последовательностями, которые коррелируют с членами библиотеки, происходящими из широко распространенных клеток (ФИГ. 6, блок 606), а затем использовать для истощения членов библиотеки секвенирования, происходящих из широко распространенных клеток (ФИГ. 6, 607), например, путем гибридизационного захвата или расщепления CRISPR. В результате измененная библиотека содержит увеличенное представительство тех членов, которые происходят из клеток, имеющих биологический признак. Члены измененной библиотеки секвенирования можно подвергать комплексному секвенированию (ФИГ. 6, блок 608). В альтернативном варианте осуществления измененную библиотеку можно подвергать дополнительным циклам обогащения и/или истощения до тех пор, пока представительство необходимых членов библиотеки не будет достаточным для соответствия критериям характеристики. Например, члены измененной библиотеки можно секвенировать второй раз, определять маркерные индексные последовательности и создавать и использовать специфические олигонуклеотиды или последовательности гидовой РНК для истощения или обогащения измененной библиотеки.
[00197] В некоторых вариантах осуществления вариант применения включает использование связных индексов. Не имеющий ограничительного характера иллюстративный вариант осуществления подхода к получению библиотеки секвенирования со связными индексами показан на ФИГ. 7. После распределения подгрупп клеток или ядер первый специфический для компартмента индекс I1 можно добавлять к молекулам 705 ДНК, присутствующим в клетках или ядрах, посредством, например, тагментации (ФИГ. 7, стадия 701). Если первичным источником нуклеиновых кислот является РНК, нуклеиновые кислоты можно преобразовывать в ДНК с использованием перед тагментацией таких способов, как синтез кДНК. Результатом является библиотека модифицированных нуклеиновых кислот, присутствующих в клетках или ядрах, причем каждая модифицированная нуклеиновая кислота 706 на каждом конце включает специфический для компартмента индекс I1. Подгруппы можно объединять и при необходимости можно репарировать концы полученных модифицированных целевых нуклеиновых кислот, например, заполнением 3’. В одном варианте осуществления 5’-концы модифицированных целевых нуклеиновых кислот можно фосфорилировать. В одном варианте осуществления следующую стадию добавления второго индекса можно облегчать путем добавления нависания, например, G, C или поли-A хвоста к 3’-концам модифицированных целевых нуклеиновых кислот. Объединенные ячейки или ядра можно распределять во второй набор компартментов, а второй специфический для компартмента индекс I2, добавлять, например, путем лигирования адаптера, имеющего 3’-конец, модифицированный соответствующим образом, например T-хвостовой 3’-конец (ФИГ. 7, стадия 702). Это приводит к получению клеток или ядер, содержащих библиотеку модифицированных нуклеиновых кислот, причем каждая модифицированная нуклеиновая кислота 707 на каждом конце включает два специфических для компартмента индекса I1 и I2. Концы модифицированных целевых нуклеиновых кислот можно изменять для облегчения добавления следующего индекса, например, путем 5’-фосфорилирования и/или модификации 3’-концов посредством наращивания поли-А или 3’-добавления G или C. Для добавления соответствующего количества индексов объединение и добавление другого специфического для компартмента индекса можно повторять по желанию. В одном варианте осуществления адаптер с универсальными последовательностями можно включать при добавлении к распределенным подгруппам клеток или ядер последнего специфического для компартмента индекса I3 (ФИГ. 7, стадия 703). Например, чтобы получить модифицированные нуклеиновые кислоты 708, к каждому концу можно добавлять несовпадающий адаптер. Примеры универсальных последовательностей включают те последовательности, которые используют для иммобилизации элементов библиотеки на матрице (P5 и P7). Несовпадающий адаптер может также включать универсальные последовательности, подходящие для секвенирования, или в некоторых вариантах осуществления модифицированные нуклеиновые кислоты 708 можно амплифицировать (ФИГ. 7, стадия 704) и добавлять универсальные последовательности, используемых для секвенирования (i5 и i7), с получением модифицированных нуклеиновых кислот 709. Модифицированные нуклеиновые кислоты 709 можно использовать в прицельном секвенировании для определения маркерных индексных последовательностей, которые коррелируют с биологическим признаком, используемым для последующего обогащения и/или делеции.
[00198] Не имеющий ограничительного характера иллюстративный вариант осуществления обогащения связи с целенаправленной амплификацией показан на ФИГ. 8. В этом варианте осуществления получена комбинаторная библиотека одиночной клетки (например, ФИГ. 3, блок 35; ФИГ. 4, блок 47; ФИГ. 6, блок 605) и полученные модифицированные нуклеиновые кислоты (например, ФИГ. 7, модифицированная нуклеиновая кислота 709) подвергают реакции амплификации, которая специфически амплифицирует членов библиотеки, содержащих интересующий биологический признак. Модифицированные нуклеиновые кислоты 802, имеющие связные индексы, приводят в контакт с праймером 803, который может включать два домена; домен 3’, выполненный с возможностью отжига к нуклеотидной последовательности, имеющей биологический признак, и домен 5’, имеющий одну или более универсальных последовательностей или их комплемент, например i7 и P7. Реакция амплификации включает второй праймер 804, который отжигается к одной стороне всех членов библиотеки. Амплификация 801 приводит к получению модифицированных нуклеиновых кислот 805, на одном конце имеющих специфические для компартмента индексы I1-3, а на другом конце - универсальные последовательности, добавленные с двухдоменным праймером, нацеленным на биологический признак. Амплифицированные модифицированные целевые нуклеиновые кислоты можно использовать в прицельном секвенировании и секвенировании для определения маркерных индексных последовательностей, коррелирующих с интересующим биологическим признаком.
[00199] В настоящем документе также обеспечены наборы. В одном варианте осуществления набор предназначен для получения библиотеки секвенирования. В одном варианте осуществления набор включает транспосомный комплекс, в котором сайт распознавания транспозона, так что в целевую нуклеиновую кислоту можно вставлять универсальную последовательность. В другом варианте осуществления набор включает два транспосомных комплекса, причем каждый комплекс включает сайт распознавания транспозона с различной универсальной последовательностью, так что в целевую нуклеиновую кислоту можно вставлять две универсальные последовательности. В другом варианте осуществления набор включает компоненты для добавления к нуклеиновым кислотам по меньшей мере одного, двух или трех индексов. Набор может также включать другие компоненты, используемые для получения библиотеки секвенирования. Например, набор может включать по меньшей мере один фермент, который опосредует лигирование, достройку праймера или амплификацию для процессинга молекул ДНК и тем самым включения индекса. Набор может включать нуклеиновые кислоты с индексными последовательностями.
[00200] Компоненты набора обычно находятся в подходящем упаковочном материале в количестве, достаточном для по меньшей мере одного анализа или использования. Можно необязательно включать другие компоненты, такие как буферы и растворы. Кроме того, обычно включают инструкции по применению упакованных компонентов. В настоящем документе фраза «упаковочный материал» относится к одной или более физическим структурам, используемым для размещения содержимого набора. Упаковочный материал изготовлен стандартными способами, как правило, чтобы обеспечивать стерильную, не содержащую загрязнений среду. Упаковочный материал может иметь этикетку, которая указывает на то, что компоненты можно использовать для получения библиотеки секвенирования. Кроме того, упаковочный материал содержит инструкции, указывающие, как использовать материалы в наборе. Используемый в настоящем документе термин «упаковка» относится к контейнеру, такому как стекло, пластик, бумага, фольга и т п., способному удерживать компоненты набора в фиксированных границах. «Инструкции по применению» обычно включают отчетливую формулировку, описывающую концентрацию реагента или по меньшей мере один параметр способа анализа, такой как относительные количества реагента и добавляемого образца, периоды времени поддержания для добавок реагента/образца, температура, буферные условия и т. п.
[00201] Композиции
[00202] Во время или после получения библиотек секвенирования можно получать ряд молекул и композиций. Например, молекула или композиция, которую можно получать, включает модифицированную целевую нуклеиновую кислоту, с одной или обеих сторон фланкированную связным индексом. Связный индекс может включать 1, 2, 3, 4, 5, 6 или более индексов подряд, причем каждый индекс отделен от другого с помощью 1, 2, 3, 4 или более нуклеотидов. В некоторых вариантах осуществления общая длина связного индекса составляет по меньшей мере 40, по меньшей мере 45, по меньшей мере 50 или по меньшей мере 55 нуклеотидов и не более 80, не более 75, не более 70 или не более 65 нуклеотидов. Возможно получение библиотеки или композиции, включающей множество таких модифицированных целевых нуклеиновых кислот. Возможно получение объединенных библиотек и композиций, которые включают объединенные библиотеки таких полинуклеотидов.
[00203] ПРИМЕРЫ ОСУЩЕСТВЛЕНИЯ
[00204] Вариант осуществления 1. Способ определения субпопуляции клеток, имеющей биологический признак, причем способ включает:
(a) обеспечение библиотеки секвенирования одиночных клеток,
причем библиотека секвенирования содержит множество модифицированных целевых нуклеиновых кислот,
при этом модифицированные целевые нуклеиновые кислоты содержат по меньшей мере одну индексную последовательность;
(b) анализ библиотеки секвенирования с помощью прицельного секвенирования для определения индексных последовательностей, которые присутствуют на той же модифицированной целевой нуклеиновой кислоте, что и биологический признак,
причем индексные последовательности, связанные с биологическим признаком, представляют собой маркерные индексные последовательности;
(c) изменение библиотеки секвенирования для получения подбиблиотеки,
при этом подбиблиотека содержит увеличенное представительство модифицированных целевых нуклеиновых кислот, содержащих маркерные индексные последовательности, по сравнению с другими модифицированными целевыми нуклеиновыми кислотами, присутствующими в библиотеке секвенирования, которые не содержат маркерную индексную последовательность;
(d) определение нуклеотидной последовательности модифицированных целевых нуклеиновых кислот, содержащих маркерную индексную последовательность.
[00205] Вариант осуществления 2. Способ по варианту осуществления 1, в котором библиотека секвенирования одиночных клеток содержит нуклеиновые кислоты из множества образцов.
[00206] Вариант осуществления 3. Способ по любому из вариантов осуществления 1-2, в котором множество образцов содержит (i) образцы одной и той же ткани, полученной из разных организмов, (ii) образцы разных тканей из одного организма или (iii) образцы разных тканей из разных организмов.
[00207] Вариант осуществления 4. Способ по любому из вариантов осуществления 1-3, в котором на стадии (b) определяют более одной маркерной индексной последовательности.
[00208] Вариант осуществления 5. Способ по любому из вариантов осуществления 1-4, в котором комбинаторная библиотека секвенирования одиночных клеток содержит целевые нуклеиновые кислоты, представляющие весь геном клеток или ядер, или подгруппы генома.
[00209] Вариант осуществления 6. Способ по любому из вариантов осуществления 1-5, в котором подгруппа генома содержит целевые нуклеиновые кислоты, представляющие транскриптом, доступный хроматин, ДНК, конформационное состояние или белки клеток или ядер.
[00210] Вариант осуществления 7. Способ по любому из вариантов осуществления 1-6, в котором изменение предусматривает обогащение модифицированных целевых нуклеиновых кислот, содержащих маркерные индексные последовательности.
[00211] Вариант осуществления 8. Способ по любому из вариантов осуществления 1-7, в котором обогащение включает способ на основе гибридизации.
[00212] Вариант осуществления 9. Способ по любому из вариантов осуществления 1-8, в котором способ на основе гибридизации, включает гибридный захват, амплификацию или CRISPR (d)Cas9.
[00213] Вариант осуществления 10. Способ по любому из вариантов осуществления 1-9, в котором изменение предусматривает истощение модифицированных целевых нуклеиновых кислот, которые не содержат маркерных индексных последовательностей.
[00214] Вариант осуществления 11. Способ по любому из вариантов осуществления 1-10, в котором истощение включает способ на основе гибридизации.
[00215] Вариант осуществления 12. Способ по любому из вариантов осуществления 1-11, в котором способ на основе гибридизации, включает гибридный захват, амплификацию или CRISPR (d)Cas9.
[00216] Вариант осуществления 13. Способ по любому из вариантов осуществления 1-12, в котором биологический признак включает нуклеотидную последовательность, указывающую на видовой тип.
[00217] Вариант осуществления 14. Способ по любому из вариантов осуществления 1-13, в котором видовой тип включает вид клетки.
[00218] Вариант осуществления 15. Способ по любому из вариантов осуществления 1-14, в котором биологический признак включает нуклеотиды субъединицы 16s, субъединицы 18s или нетранскрипционную область ITS.
[00219] Вариант осуществления 16. Способ по любому из вариантов осуществления 1-15, в котором биологический признак включает нуклеотидную последовательность, указывающую на класс клеток.
[00220] Вариант осуществления 17. Способ по любому из вариантов осуществления 1-16, в котором класс клеток содержит паттерн экспрессии, эпигенетический паттерн, рекомбинацию иммунных генов или их комбинацию.
[00221] Вариант осуществления 18. Способ по любому из вариантов осуществления 1-17, в котором эпигенетический паттерн содержит метку метилирования, паттерн метилирования, доступную ДНК или их комбинацию.
[00222] Вариант осуществления 19. Способ по любому из вариантов осуществления 1-18, в котором биологический признак включает нуклеотидную последовательность, указывающую на состояние или риск заболевания.
[00223] Вариант осуществления 20. Способ по любому из вариантов осуществления 1-19, в котором состояние или риск заболевания включает вариантную последовательность ДНК, вариантный паттерн экспрессии или вариантный эпигенетический паттерн, который коррелирует с заболеванием.
[00224] Вариант осуществления 21. Способ по любому из вариантов осуществления 1-20, в котором вариантная последовательность ДНК содержит по меньшей мере один однонуклеотидный полиморфизм.
[00225] Вариант осуществления 22. Способ по любому из вариантов осуществления 1-21, в котором вариантный паттерн экспрессии включают экспрессию биомаркера.
[00226] Вариант осуществления 23. Способ по любому из вариантов осуществления 1-22, в котором вариантный эпигенетический паттерн включают метку метилирования, паттерн метилирования.
[00227] Вариант осуществления 24. Способ по любому из вариантов осуществления 1-23, в котором модифицированные целевые нуклеиновые кислоты содержат связный индекс по меньшей мере 2 специфических для компартмента индексных последовательностей, причем между 2 индексными последовательностями находится не более 6 нуклеотидов.
[00228] Вариант осуществления 25. Способ по любому из вариантов осуществления 1-24, в котором связный индекс присутствует на каждом конце модифицированных целевых нуклеиновых кислот.
[00229] Вариант осуществления 26. Способ по любому из пп. 1-25, в котором длина связного индекса составляет по меньшей мере 55 нуклеотидов.
[00230] Вариант осуществления 27. Способ по любому из вариантов осуществления 1-26, в котором на модифицированных целевых нуклеиновых кислотах присутствует одна копия связного индекса.
[00231] Вариант осуществления 28. Способ по любому из вариантов осуществления 1-27, в котором на модифицированных целевых нуклеиновых кислотах присутствуют две копии связного индекса.
[00232] Вариант осуществления 29. Способ по любому из вариантов осуществления 1-28, в котором множество модифицированных целевых нуклеиновых кислот библиотеки секвенирования представляет по меньшей мере 100 000 различных клеток или ядер.
[00233] Вариант осуществления 30. Способ по любому из вариантов осуществления 1-29, в котором обеспечение комбинаторной библиотеки секвенирования одиночных клеток включает:
обработку образца для получения библиотеки, причем образец представляет собой метагеномический образец, полученный из организма.
[00234] Вариант осуществления 31. Способ по любому из пп. 1-30, в котором организм является млекопитающим.
[00235] Вариант осуществления 32. Способ по любому из вариантов осуществления 1-31, в котором метагеномический образец содержит ткань, предположительно содержащую симбиотический или патогенный микроорганизм.
[00236] Вариант осуществления 33. Способ по любому из вариантов осуществления 1-32, в котором микроорганизм является прокариотическим или эукариотическим.
[00237] Вариант осуществления 34. Способ по любому из вариантов осуществления 1-33, в котором метагеномический образец содержит образец микробиома.
[00238] Вариант осуществления 35. Способ по любому из вариантов осуществления 1-34, в котором обеспечение комбинаторной библиотеки секвенирования одиночных клеток включает:
обработку образца для обеспечения библиотеки, причем образец получен из организма.
[00239] Вариант осуществления 36. Способ по любому из пп. 1-35, в котором организм является млекопитающим.
[00240] Вариант осуществления 37. Способ по любому из вариантов осуществления 1-36, в котором первичный источник нуклеиновых кислот из образца содержит РНК.
[00241] Вариант осуществления 38. Способ по любому из вариантов осуществления 1-37, в котором РНК предусматривает матричную РНК (мРНК).
[00242] Вариант осуществления 39. Способ по любому из вариантов осуществления 1-38, в котором первичный источник нуклеиновых кислот из образца содержит ДНК.
[00243] Вариант осуществления 40. Способ по любому из вариантов осуществления 1-39, в котором ДНК содержит геномную ДНК всей клетки.
[00244] Вариант осуществления 41. Способ по любому из вариантов осуществления 1-40, в котором геномная ДНК всей клетки содержит нуклеосомы.
[00245] Вариант осуществления 42. Способ по любому из вариантов осуществления 1-41, в котором первичный источник нуклеиновых кислот из образца содержит бесклеточную ДНК.
[00246] Вариант осуществления 43. Способ по любому из вариантов осуществления 1-42, в котором образец содержит раковые клетки.
[00247] Вариант осуществления 44. Способ по любому из вариантов осуществления 1-43, в котором обеспечение комбинаторной библиотеки секвенирования одиночных клеток включает обеспечение библиотеки способом комбинаторного индексирования одиночных клеток, выбранным из секвенирования транскриптома одиночных ядер, секвенирования транскриптома одиночных клеток, секвенирования транскриптома одиночных клеток и хроматина, доступного транспозонам, полногеномного секвенирования одиночных ядер, секвенирования одиночных ядер хроматина, доступного транспозонам, секвенирования эпитопа одиночных клеток, sci-HiC и sci-MET.
[00248] Вариант осуществления 45. Способ по любому из вариантов осуществления 1-44, в котором обеспечение включает обеспечение из каждой клетки или ядра двух разных комбинаторных библиотек секвенирования одиночных клеток.
[00249] Вариант осуществления 46. Способ по любому из вариантов осуществления 1-45, в котором две разные комбинаторные библиотеки секвенирования одиночных клеток выбраны из способа комбинаторного индексирования одиночных клеток, выбранного из секвенирования транскриптома одиночных ядер, секвенирования транскриптома одиночных клеток, секвенирования транскриптома одиночных клеток и хроматина, доступного транспозонам, полногеномного секвенирования одиночных ядер, секвенирования одиночных ядер хроматина, доступного транспозонам, sci-HiC и sci-MET.
[00250] Вариант осуществления 47. Способ по любому из вариантов осуществления 1-46, дополнительно включающий выполнение процедуры секвенирования для определения нуклеотидных последовательностей для нуклеиновых кислот.
[00251] Вариант осуществления 48. Способ получения библиотеки секвенирования, содержащей нуклеиновые кислоты из множества одиночных ядер или клеток, причем способ включает:
(a) обеспечение множества ядер или клеток, причем ядра или клетки содержат нуклеосомы;
(b) приведение множества ядер или клеток в контакт с транспосомным комплексом, содержащим транспозазу и универсальную последовательность, причем приведение в контакт дополнительно включает условия, подходящие для встраивания универсальной последовательности в нуклеиновые кислоты ДНК, что приводит к образованию двухцепочечных нуклеиновых кислот ДНК, содержащих универсальную последовательность;
(d) распределение множества ядер или клеток в первое множество компартментов,
причем каждый компартмент содержит подгруппу ядер или клеток;
(e) процессинг молекул ДНК в каждой подгруппе ядер или клеток для создания индексированных ядер или клеток,
причем процессинг предусматривает добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, индексной последовательности, специфичной для первого компартмента, с получением индексированных нуклеиновых кислот, присутствующих в индексированных ядрах или клетках,
при этом процессинг предусматривает лигирование, достройку праймера, гибридизацию, амплификацию или их комбинацию; и
(g) объединение индексированных ядер или клеток для получения объединенных индексированных ядер или клеток.
[00252] Вариант осуществления 49. Способ по п. 48, в котором обеспечение включает обеспечение множества ядер или клеток во множестве компартментов, причем каждый компартмент содержит подгруппу ядер или клеток, причем приведение в контакт предусматривает приведение каждого компартмента в контакт с транспосомным комплексом, и при этом способ дополнительно включает объединение ядер или клеток после приведения в контакт с образованием объединенных ядер или клеток.
[00253] Вариант осуществления 50. Способ по любому из вариантов осуществления 48-49, в котором обеспечение включает подвергание ядер химической обработке с образованием ядра с истощенными нуклеосомами и сохранением при этом целостности выделенных ядер.
[00254] Вариант осуществления 51. Способ по любому из вариантов осуществления 48-50, дополнительно включающий:
распределение объединенных индексированных ядер или клеток, содержащих индексированные ядра или клетки, во второе множество компартментов,
причем каждый компартмент содержит подгруппу ядер или клеток;
процессинг молекул ДНК в каждой подгруппе ядер или клеток для получения ядер или клеток с двойным индексированием,
причем процессинг предусматривает добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, индексной последовательности, специфичной для второго компартмента, с получением нуклеиновых кислот с двойным индексированием, присутствующих в индексированных ядрах или клетках,
при этом процессинг предусматривает лигирование, достройку праймера, гибридизацию, амплификацию или их комбинацию;
объединение ядер или клеток с двойным индексированием для получения объединенных ядер или клеток с двойным индексированием.
[00255] Вариант осуществления 52. Способ по любому из вариантов осуществления 48-51, дополнительно включающий:
распределение объединенных ядер или клеток, содержащих ядра или клетки с двойным индексированием, в третье множество компартментов,
причем каждый компартмент содержит подгруппу ядер или клеток;
процессинг молекул ДНК в каждой подгруппе ядер или клеток с образованием ядер или клеток с тройным индексированием,
причем процессинг предусматривает добавление к нуклеиновым кислотам ДНК, присутствующим в каждой подгруппе ядер или клеток, индексной последовательности, специфичной для третьего компартмента, с получением нуклеиновых кислот с тройным индексированием, присутствующих в индексированных ядрах или клетках,
при этом процессинг предусматривает лигирование, достройку праймера, гибридизацию, амплификацию или их комбинацию;
объединение ядер или клеток с тройным индексированием для получения объединенных ядер или клеток с тройным индексированием.
[00256] Вариант осуществления 53. Способ по любому из вариантов осуществления 48-52, в котором стадия распределения включает разведение.
[00257] Вариант осуществления 54. Способ по любому из вариантов осуществления 48-53, в котором компартмент содержит лунку, микрожидкостный компартмент или каплю.
[00258] Вариант осуществления 55. Способ по любому из вариантов осуществления 48-54, в котором компартменты первого множества компартментов содержат от 50 до 100 000 000 ядер или клеток.
[00259] Вариант осуществления 56. Способ по любому из вариантов осуществления 48-55, в котором компартменты второго множества компартментов содержат от 50 до 100 000 000 ядер или клеток.
[00260] Вариант осуществления 57. Способ по любому из вариантов осуществления 48-56, в котором компартменты третьего множества компартментов содержат от 50 до 100 000 000 ядер или клеток.
[00261] Вариант осуществления 58. Способ по любому из вариантов осуществления 48-57, в котором приведение в контакт включает приведение каждой подгруппы в контакт с двумя транспосомными комплексами, причем один транспосомный комплекс содержит первую транспозазу, содержащую первую универсальную последовательность, а второй транспосомный комплекс содержит вторую транспозазу, содержащую вторую универсальную последовательность, при этом приведение в контакт дополнительно включает условия, подходящие для встраивания первой универсальной последовательности и второй универсальной последовательности в нуклеиновые кислоты ДНК, приводящего к образованию двухцепочечных нуклеиновых кислот ДНК, содержащих первую и вторую универсальные последовательности.
[00262] Вариант осуществления 59. Способ по любому из вариантов осуществления 48-58, в котором добавление специфичной для компартмента индексной последовательности включает двухстадийный процесс добавления к нуклеиновым кислотам нуклеотидной последовательности, содержащей универсальную последовательность, и последующее добавление к нуклеиновым кислотам индексной последовательности, специфичной для компартмента.
[00263] Вариант осуществления 60. Способ по любому из вариантов осуществления 48-59, дополнительно включающий получение индексированных нуклеиновых кислот из объединенных индексированных ядер или клеток, с образованием таким образом библиотеки секвенирования из множества ядер или клеток.
[00264] Вариант осуществления 61. Способ по любому из вариантов осуществления 48-60, дополнительно включающий получение нуклеиновых кислот с двойным индексированием из объединенных ядер или клеток с двойным индексированием, с образованием таким образом библиотеки секвенирования из множества ядер или клеток.
[00265] Вариант осуществления 62. Способ по любому из вариантов осуществления 48-61, дополнительно включающий получение нуклеиновых кислот с тройным индексированием из объединенных ядер или клеток с тройным индексированием, с образованием таким образом библиотеки секвенирования из множества ядер или клеток.
[00266] Вариант осуществления 63. Способ по любому из вариантов осуществления 48-62, дополнительно включающий:
обеспечение поверхности, содержащей множество сайтов амплификации,
причем сайты амплификации содержат по меньшей мере две популяции присоединенных одноцепочечных захватных олигонуклеотидов, имеющих свободный 3’-конец, и
приведение поверхности, содержащей сайты амплификации, в контакт с фрагментами нуклеиновой кислоты, содержащими одну, две или три индексных последовательности, в условиях, подходящих для получения множества сайтов амплификации, каждый из которых содержит клональную популяцию ампликонов из отдельного фрагмента, содержащего множество индексов.
[00267] Вариант осуществления 64. Способ получения библиотеки нуклеиновых кислот, включающий:
(a) обеспечение множества образцов, причем каждый образец содержит множество клеток или ядер, при этом множество клеток или ядер каждого образца находится в одном или более отдельных компартментах;
(b) приведение множества ядер или клеток в контакт с транспосомным комплексом, содержащим транспозазу и универсальную последовательность, и при условии, что транспосомный комплекс не содержит индексной последовательности, причем приведение в контакт дополнительно предусматривает условия, подходящие для встраивания универсальной последовательности в нуклеиновые кислоты;
(c) добавление первой индексной последовательности к нуклеиновым кислотам каждого отдельного компартмента;
(d) объединение клеток или ядер разделенных компартментов;
(e) распределение клеток или ядер во множество компартментов; и
(f) добавление второй индексной последовательности к нуклеиновым кислотам множества компартментов.
[00268] Вариант осуществления 65. Способ по варианту осуществления 64, в котором первую индексную последовательность, вторую индексную последовательность или их комбинацию добавляют путем лигирования, достройки праймера, гибридизации, амплификации или их комбинации.
[00269] Вариант осуществления 66. Способ по любому из вариантов осуществления 64-65, в котором стадии (d)-(e) повторяют для добавления третьей или более индексных последовательностей к клеткам или ядрам множества компартментов.
[00270] Вариант осуществления 67. Способ по любому из вариантов осуществления 64-66, в котором множество ядер или клеток зафиксированы.
[00271] Вариант осуществления 68. Способ по любому из вариантов осуществления 64-67, дополнительно включающий амплификацию индексированных нуклеиновых кислот после стадии (c) или стадии (f).
[00272] Вариант осуществления 69. Способ по любому из вариантов осуществления 64-68, дополнительно включающий стадию (g) объединения нуклеиновых кислот из множества компартментов и определения последовательности нуклеиновых кислот.
[00273] Вариант осуществления 70. Способ по любому из вариантов осуществления 64-69, дополнительно включающий выполнение процедуры секвенирования для определения нуклеотидных последовательностей для нуклеиновых кислот.
[00274] Вариант осуществления 71. Способ секвенирования одиночной клетки или ядра, включающий:
(a) уникальное индексирование нуклеиновых кислот каждой клетки или ядер в образце с образованием таким образом индексированной библиотеки для каждой клетки или ядра;
(b) использование биологического признака для определения одной или более интересующих индексированных библиотек со стадии (a);
(c) обогащение интересующих индексированных библиотек со стадии (b) с образованием таким образом обогащенной библиотеки; и
(d) секвенирование обогащенной библиотеки со стадии (c).
[00275] Вариант осуществления 72. Способ по варианту осуществления 71, в котором библиотеки происходят от ДНК, РНК или белка клеток или ядер.
[00276] Вариант осуществления 73. Способ по любому из вариантов осуществления 64-72, в котором биологический признак представляет собой ДНК, РНК, белок или их комбинацию.
[00277] Вариант осуществления 74. Способ по любому из вариантов осуществления 64-73, в котором уникальная индексация на стадии (а) включает связывание по меньшей мере двух различных индексов с нуклеиновыми кислотами клеток или ядер.
[00278] Вариант осуществления 75. Способ по любому из вариантов осуществления 64-74, в котором по меньшей мере два различных индекса представляют собой связный индекс.
[00279] Вариант осуществления 76. Способ по любому из вариантов осуществления 64-75, в котором обогащенная библиотека получена посредством позитивного обогащения.
[00280] Вариант осуществления 77. Способ по любому из вариантов осуществления 64-76, в котором позитивное обогащение предусматривает амплификацию.
[00281] Вариант осуществления 78. Способ по любому из вариантов осуществления 64-77, в котором позитивное обогащение предусматривает захватный агент.
[00282] Вариант осуществления 79. Способ по любому из вариантов осуществления 64-78, в котором позитивное обогащение предусматривает твердую подложку.
[00283] Вариант осуществления 80. Способ по любому из вариантов осуществления 64-79, в котором обогащенная библиотека получена посредством негативного обогащения.
[00284] Вариант осуществления 81. Способ по любому из вариантов осуществления 64-80, в котором определение интересующей индексированной библиотеки на стадии (c) включает секвенирование индексов.
[00285] Вариант осуществления 82. Способ секвенирования одиночной клетки или ядра, включающий:
(a) обеспечение образца, причем образец содержит множество ядер или клеток;
(b) связывание первого индекса на каждом ядре или клетке в образце;
(c) разделение образца на множество компартментов;
(d) связывание второго индекса на каждом ядре или клетке из множества компартментов;
(e) объединение множества компартментов;
(f) секвенирование объединенных компартментов;
(g) определение комбинации первого и второго индексов, связанных с биологическим признаком;
(h) обогащение биологического признака из объединенных компартментов с использованием идентифицированной комбинации первого и второго индексов со стадии (g).
[00286] Вариант осуществления 83. Набор, содержащий:
(a) множество транспосомных комплексов, причем каждый транспосомный комплекс содержит транспозазу и транспозонную последовательность, при этом транспозонная последовательность не индексирована;
(b) первое множество индексных олигонуклеотидов, причем первое множество индексных олигонуклеотидов содержит олигонуклеотиды, имеющие по меньшей мере две разные последовательности; и
(c) фермент лигазу для применения с индексными олигонуклеотидами.
[00287] Вариант осуществления 84. Набор по варианту осуществления 83, дополнительно содержащий второе множество индексных олигонуклеотидов, причем второе множество индексных олигонуклеотидов содержит олигонуклеотид, имеющий последовательности, отличающиеся от первого множества индексных олигонуклеотидов.
[00288] Вариант осуществления 85. Набор по варианту осуществления 83 или 84, дополнительно содержащий третье множество индексных олигонуклеотидов, причем третье множество индексных олигонуклеотидов содержит олигонуклеотид, имеющий последовательности, отличающиеся от первого множества индексных олигонуклеотидов и второго множества индексных олигонуклеотидов.
[00289] ПРИМЕРЫ
[00290] Настоящее описание проиллюстрировано следующими примерами. Следует понимать, что конкретные примеры, материалы, количества и процедуры следует интерпретировать в широком смысле в соответствии с сущностью и объемом изложения, как описано в настоящем документе.
[00291] Пример 1
[00292] Атлас доступности хроматина для клеток человека в процессе развития
[00293] Реферат
[00294] Хроматиновый ландшафт человеческого генома формирует специфические для типа клеток программы экспрессии гена. Был разработан улучшенный анализ для профилирования доступности хроматина одиночных клеток на основании трехуровневого комбинаторного индексирования (sci-ATAC-seq3) и применен к 59 образцам плода, представляющим 15 органов, совокупное профилирование которых составляет порядка одного миллиона одиночных клеток. Использовали типы клеток, определяемые экспрессией гена в одних и тех же органах, для аннотирования этих данных, для построения каталога сотен тысяч регуляторных элементов ДНК, специфических для типа клеток, и для изучения свойств специфических для генеалогической линии факторов транскрипции, а также специфических для типа клеток обогащений сложных признаков наследуемости. Наряду с сопроводительными атласами экспрессии генов человеческих клеток в процессе развития, эти данные содержат богатый ресурс для изучения биологии человека.
[00295] Основной текст
[00296] В последние годы быстро распространились способы, эксперименты и атласы одиночных клеток. Однако подавляющее большинство усилий по-прежнему сосредоточено на экспрессии генов одиночных клеток, что отражает только один аспект клеточной, относящейся к развитию и относящейся к организму биологии. Другие аспекты, включая хроматиновый ландшафт, который формирует программы экспрессии генов, столь же важны для исследования на уровне отдельных клеток, но связаны с трудностями в виде относительной нехватки масштабируемых способов.
[00297] Каркас комбинаторного индексирования одиночных клеток (sci) включает разделение и объединение клеток или ядер в лунки, в которых в каждом цикле молекулярные штрихкоды вводят in situ в интересующие виды (например, РНК или хроматин). В ходе последовательных циклов молекулярного штрихкодирования in situ виды в рамках одной и той же клетки соответствующим образом метят уникальной комбинацией штрихкодов. Анализы sci- разработаны для профилирования доступности хроматина (sci-ATAC-seq), экспрессии генов (sci-RNA-seq), ядерной архитектуры, последовательности генома, метилирования, гистоновых маркеров и других явлений, а также совместные анализы sci-, например, для совместного профилирования доступности хроматина и экспрессии генов (CoBatch, Split-seq, Paired-seq и dscATAC-seq представляют собой способы, которые также основаны на комбинаторном индексировании одиночных клеток).
[00298] Несмотря на то что ранее можно было профилировать доступность хроматина в ~ 100 000 клетках млекопитающих посредством двухуровневой системы sci-ATAC-seq, анализ имеет ряд ограничений. Например, он требует индивидуальной загрузки фермента Tn5 со штрихкодированными адаптерами, и он ограничен 104-105 клетками на один эксперимент вследствие коллизий - клеток, получающих одну и ту же комбинацию штрихкодов. Для решения этих проблем разработан улучшенный анализ для определения профиля доступности хроматина одиночных клеток на основании трех уровней комбинаторного индексирования (sci-ATAC-seq3). В отличие от предыдущих итераций sci-ATAC-seq, анализ не зависит от молекулярно штрихкодированных комплексов Tn5 (Фиг. 9; Фиг. 10). Вместо этого первых двух циклов индексирования достигают путем лигирования к каждому концу стандартного равномерно загруженного комплекса транспозазы Tn5 (стандартный Nextera), тогда как конечный цикл индексирования и далее выполняют с помощью ПЦР. Относительный к двухуровневому sci-ATAC-seq, однако подобный sci-RNA-seq3, sci-ATAC-seq3 существенно снижает затраты на получение библиотеки на каждую клетку, а также частоту коллизий. Теоретические коэффициенты коллизий для 2-уровневого (96×384 лунки) и 3-уровневого (384×384 x 384 лунки) индексирования составляли 12% и 1,3% соответственно, а наблюдаемую частоту коллизий для 3-уровневого эксперимента «смешивания видов» с использованием объединенных равных количеств клеток GM12878 и клеток CH12.LX оценивали как 4,0%, что делало возможными эксперименты в масштабе 106 клеток. Протокол больше не требует сортировки клеток, и были также оптимизированы выбор лигазы и полимеразы, концентрация киназы и конфигурации и концентрации олигонуклеотидов для максимального увеличения количества фрагментов, полученных из каждой клетки. Следует отметить, что при сохранении обогащения в доступных областях явный выбор отдается в пользу максимальной сложности за счет специфичности к доступным сайтам. Оцененные суммарные уникальные прочтения («сложность») для каждой клетки рассчитывали с использованием Picard, и для каждой клетки рассчитывали фракцию чтений на сайте инициации транскрипции (FRiTSS). Чтения в пределах 500 п. н. TSS Gencode рассматривали в пределах TSS. В частности, было обнаружено, что условия фиксации можно настраивать таким образом, чтобы регулировать чувствительность (т. е. сложность) и специфичность (т. е. обогащение доступных участков) анализа.
[00299] Относительно атласа доступности хроматина человеческих клеток применяли sci-ATAC-seq3 к 59 образцам плода, представляющим 15 органов (надпочечник, две области мозжечка, глаз, сердце, кишечник, почка, печень, легкое, мышца, поджелудочная железа, плацента, селезенка, желудок и тимус) и при этом полностью профилировали доступность хроматина в 1,6 миллиона клеток (Фиг. 1D-E). В примере 2 описывается профилирование экспрессии генов в 4-5 миллионах клеток из тех же органов на основе перекрывающегося набора образцов. Профилированные органы охватывают разнообразные системы; наиболее заметно отсутствие костного мозга, костей, гонад и кожи.
[00300] Быстрая и единообразная обработка разнородных тканей плода представляет собой серьезную проблему. Разрабатывали новый способ извлечения ядер непосредственно из криоконсервированных тканей, который хорошо работает в различных типах тканей и дает гомогенаты, подходящие как для sci-ATAC-seq3, так и для sci-RNA-seq3. Вкратце заворачивают срезы мгновенно замороженной ткани в алюминиевую фольгу и измельчаем их в порошок с помощью охлажденного молотка и на сухом льду. Порошок ткани затем разделяют на аликвоты, одну для sci-ATAC-seq3 и другую для sci-RNA-seq3.
[00301] Для sci-ATAC-seq3 получены образцы из 23 плодов с примерным гестационным возрастом в диапазоне от 89 до 125 дней. Лизировали клетки для выделения ядер с использованием известных буферов для лизиса клеток ATAC-seq и фиксировали ядра формальдегидом перед мгновенным замораживанием для последующего процессинга. Что касается ядер из каждой ткани, приблизительно 50 000 фиксированных ядер помещали в 4 лунки 96-луночного планшета и обрабатывали для тагментации. После тагментации первый индекс, который также определял образец ткани, вводили путем лигирования с одним из свободных концов асимметричного вставленного комплекса транспозазы. После объединения и разделения второй индекс вводили путем лигирования с другим свободным концом комплекса транспозазы. После еще одного цикла объединения и разделения с помощью ПЦР добавляли конечный индекс, а полученные ампликоны объединяли для секвенирования.
[00302] Были секвенированы библиотеки sci-ATAC-seq3 из 3 экспериментов в 5 прогонах Illumina NovaSeq с получением в итоге более 50 миллиардов чтений. В качестве первоначальной проверки качества были исследованы данные на уровне ткани, т. е. перед ее разделением на одиночные клетки. Были загружены и повторно составлены карты всех доступных образцов определения чувствительности к одноконечной дезоксирибонуклеазе (DNase-seq) из тканей плода из портала данных ENCODE. Затем были идентифицированы пики доступности в каждом из наших «псевдомассовых» образцов и каждом образце ENCODE, объединены эти наборы и оценена доступность каждого образца на каждом пике в главном списке. Хотя данные sci-ATAC-seq3 были несколько менее обогащены в пиках (медианные чтения в пиках: 29% для sci-ATAC-seq3; 35% для DNase-seq ENCODE), образцы из одной и той же ткани имели сравнимую корреляцию для двух анализов (медианная корреляция Spearman: 0,93 для двух образцов из одной и той же ткани для sci-ATAC-seq3; 0,91 для DNase-seq) с большей технической воспроизводимостью для sci-ATAC-seq3 (медианная корреляция Spearman: 0,95). Более того, образцы сгруппированы в соответствующие им ткани на основании этих совокупных профилей, вне зависимости от того, проводится ли анализ с применением попарных корреляций Spearman в образцах кластеров для образцов sci-ATAC-seq3 отдельно, либо образцов sci-ATAC-seq3 и DNase-seq вместе.
[00303] После разделения чтений на основании клеточных штрихкодов и применения динамического порога, как было описано ранее, определяли 1 568 018 клеток. По данным контроля по типу «скотный двор» оценивали частоту коллизий, составляющую ~ 5% для каждого из трех экспериментов. Визуализация клеток, соответствующих «сторожевой» ткани человека, с помощью приближения и проекции однородного многообразия (UMAP) не выявила никаких очевидных экспериментальных массовых эффектов. Три образца удаляли вследствие слабого образования соответствующих нуклеосомам полос распределения размеров их фрагментов; два дополнительных образца удаляли, поскольку было захвачено очень малое количество клеток. По нашим оценкам, секвенировали медиану от 91% до 99% всех уникальных фрагментов на клетку для каждого типа ткани в этих библиотеках sci-ATAC-seq3.
[00304] Определяли пики доступности по принципу очереди для каждой ткани, а затем объединяли их с образованием основного набора из 1,05 миллиона сайтов. После оценки наличия или отсутствия чтений в каждой ячейке на каждом сайте отфильтровывали клетки более низкого качества на основании общего числа уникальных чтений (минимальные специфические для образца значения в диапазоне от 1000 до 3586), доли чтений, перекрывающей основной набор доступных сайтов (минимальные специфические для образца значения в диапазоне от 0,2 до 0,4), доли чтений, попадающих рядом с TSS (+/-1 т. п. н.; минимальные специфические для образца значения в диапазоне от 0,05 до 0,15), и количества дублетов, полученного в результате адаптации алгоритма обнаружения дублетов Scrublet, первоначально разработанного для данных scRNA-seq (за исключением ~ 10% клеток с наивысшими показателями дублетов).
[00305] После этих процедур оставались 790 957 профилей доступности хроматина одиночных клеток из 54 образцов плода. Общее количество высококачественных клеток на ткань было в диапазоне от 2421 для селезенки до 211 450 для печени. Медианное количество уникальных фрагментов на клетку для этого набора составляет 6042, причем медианное значение 0,49 перекрывает основной набор доступных сайтов, а 0,19 попадает вблизи TSS (+/-1 т. п. н.).
[00306] Высококачественные клетки подвергали латентно-семантическому индексированию (LSI) по принципу очереди для каждой ткани с использованием логарифмически преобразованного компонента частоты. Хотя не наблюдали очевидных доказательств массовых эффектов для разных образцов, соответствующих одной и той же ткани, в качестве консервативной меры применяли алгоритм гармонии для выравнивания образцов в области способа главных компонентов (PCA) для каждой ткани. Затем для каждой ткани применяли кластеризацию Louvain с использованием выровненной области PCA, с получением в начале 172 кластеров во всех тканях. С помощью UMAP дополнительно снижали размерность каждого набора данных о ткани.
[00307] Аннотация типов клеток
[00308] Как показано авторами и другими исследователями, аннотацию типов клеток в наборах данных scATAC-seq можно значительно упрощать путем использования наборов данных scRNA-seq. Для частичной автоматизации аннотаций типа клеток для таких данных scATAC-seq, во-первых, аннотировали типы клеток в данных scRNA-seq для одних и тех же тканей, как описано в сопутствующей рукописи. Во-вторых, для данных scATAC-seq вычисляли показатели доступности на уровне генов, собирая вместе количество событий транспозиции, попадающих в тела генов, простирающихся на 2 т. п. н. ближе к 5'-концу от их TSS. В-третьих, для каждого типа данных в качестве входных данных для подхода с целью поиска вероятных соответствий между кластерами scRNA-seq и scATAC-seq на основе регрессии неотрицательных наименьших квадратов (NNLS) использовали матрицы «ген за клеткой», а в результате получали начальный «подъемный» набор автоматизированных аннотаций для кластеров scATAC-seq. Наконец, вручную проверяли все автоматизированные аннотации посредством изучения скоплений вокруг маркерных генов для каждого типа клеток в каждой ткани, с выполнением модификаций назначенных меток при необходимости. Типы клеток сначала аннотировали в данных sci-RNA-seq, собранных на соответствующих тканях на основании экспрессии маркерного гена. В данных ATAC для каждой ткани определяли кластеры Louvain. Затем для каждого из этих кластеров рассчитывали показатели доступности на уровне генов и сопоставляли их с кластерами РНК на основании регрессии неотрицательных наименьших квадратов (NLS), что в некоторых случаях приводило к слиянию кластеров Louvain. Эти автоматизированные аннотации первого прохода дополнительно улучшали путем ручной проверки специфического для кластера ландшафта доступности вокруг маркерных генов. Аннотированные типы клеток демонстрировали специфическую доступность вокруг TSS известных маркерных генов. Для каждого типа клеток или неаннотированного кластера суммировали доступность вблизи TSS известных маркерных генов и нормализовали шкалу для учета различий в общем количестве чтений на клетку, а также количествах клеток по типам клеток. Данные подразумевали, что некоторые неаннотированные кластеры могут представлять не новые типы клеток, а скорее технические артефакты (например, дублеты). Было отмечено, что, хотя другие подходы продемонстрировали большой потенциал для мультимодальной интеграции данных одиночных клеток, было обнаружено, что способа NNLS типа «кластер к кластеру» достаточно для настоящих целей и требуется гораздо меньше вычислительных затрат.
[00309] В целом удалось аннотировать 150 из 172 кластеров (87%) или 163 из 172 (95%) при условии включения меток с более низкой достоверностью. Некоторые кластеры получили одну и ту же аннотацию в пределах одной и той же ткани и, таким образом, были объединены, в результате чего получилось 124 аннотации во всех тканях. Из них некоторые аннотации присутствовали во множестве тканей (например, эритробласты в 4 тканях). Разрушение в тканях привело к 54 уникальным типам аннотаций клеток, которые картируются в соотношении 1 : 1 с аннотациями, сделанными в нашем наборе данных scRNA-seq (или 59 при условии включения меток с низкой достоверностью и картированиями 1 : 2). Многие из типов клеток scRNA-seq, которые не обнаружены в данных о доступности хроматина на данном уровне разрешения, представляют собой небольшие кластеры, которые не попали в образец в достаточном для обнаружения количестве, из-за меньшего числа клеток, профилированных в данном исследовании (~ 4 миллиона (РНК) по сравнению с ~ 800 тысячами высококачественных клеток (ATAC)). С другой стороны, большинство из 9 кластеров scATAC-seq, которые оставались полностью неаннотированными, по-видимому, обусловлено неотфильтрованными дублетами, поскольку они характеризуются доступностью маркерных генов для множества соседних типов клеток в представительстве UMAP.
[00310] Определение специфических для генеалогической линии факторов транскрипции (TF)
[00311] Далее пытались интегрировать и сравнивать доступность хроматина в типах клеток во всех 15 органах. Для уменьшения эффектов грубых различий в количестве клеток на орган и/или типе клеток случайным образом отбирали по 800 клеток на тип клеток на орган (или в случаях, когда в данном органе представлено менее 800 клеток заданного типа клеток, отбирали все клетки) и выполняли визуализацию UMAP. Обнадеживает то, что типы клеток, представленные во множестве органов, сгруппированы вместе, например стромальные клетки (9 органов), эндотелиальные клетки (13 органов), лимфоидные клетки (7 органов) и миелоидные клетки (10 органов), а не в партии или отдельно. Совместно размещены также типы клеток родственные по развитию и функции, например разнообразные клетки крови, секреторные клетки, нейроны периферической нервной системы (ПНС), нейроны центральной нервной системы (ЦНС).
[00312] Ключевым вопросом в биологии развития является то, какие факторы транскрипции (TF) отвечают за создание данного разнообразия типов клеток из инвариантного генома. Далее стремились использовать объем этого атласа доступности хроматина клеток человека для систематической оценки, какие мотивы TF дифференциально доступны, и, таким образом, условно назначить ключевые регуляторы клеточного предназначения в контексте развития человека in vivo.
[00313] В первом подходе использовали модель линейной регрессии для определения, какие мотивы TF, присутствующие в доступных сайтах каждой клетки, лучше всего объясняют ее принадлежность к типу клетки. Первоначально, обрабатывая каждую ткань независимо, в каждом из 124 аннотированных кластеров типов клеток определяли наиболее обогащенные мотивы/TF из базы данных JASPAR, благодаря чему выявлены как известные, так и потенциально новые регуляторы. Например, в плаценте мотив SPI1/PU.1, -установленный регулятор развития миелоидной линии, - весьма обогащен пиками миелоидных клеток; мотив TWIST-1, необходимый для формирования стромальных клеток-предшественников, обогащен пиками стромальных клеток; мотив FOS::JUN связан с доступностью хроматина во вневорсинчатых трофобластах, типе клеток, в котором была описана специфическая активность соответствующего комплекса AP1.
[00314] Интересно, что внутри плаценты неаннотированный кластер был весьма обогащен мотивами GATA1::TAL1, - установленными регуляторами эритропоэза. Эти клетки объединены в кластер с эритробластами из других тканей в общего UMAP, и после дополнительного анализа ключевые гены эритроидных маркеров демонстрировали специфическую доступность промотора. В управляемой NNLS поточной обработке кластер не аннотирован, поскольку в исследовании scRNA-seq эритробластный кластер не обнаруживали в плаценте, возможно, потому, что плацента является одной из нескольких тканей, в которых имеется больше клеток ATAC, чем РНК. Таким образом, обогащение мотива может способствовать аннотации типа клеток, если известны ключевые регуляторы типа клеток.
[00315] Этот анализ повторяли для 54 основных типов клеток, наблюдаемых во всех тканях, т. е. после разрушения типов клеток, появляющихся во множестве тканей. Как и ожидалось, ведущие мотивы по-прежнему согласовывались со специфическими для ткани анализами, а также с литературой, например SPI1/PU.1 в миелоидных клетках; CRX в пигментном эпителии сетчатки и фоторецепторных клетках; MEF2B в кардиомиоцитах и клетках скелетных мышц (31); и SRF эндокардиальных и гладкомышечных клетках. Хотя большинство мотивов обогащено в клетках только одного или двух типов, мотивы TF нейронов, включая OLIG2, NEUROG1 и POU4F1, обогащены во множестве типов нейронов. Другим важным исключением является HNF1B, традиционно связанный с развитием почек и поджелудочной железы, мотив которых обогащен в клетках 13 типов, которые охватывают ряд специализированных эпителиальных и секреторных клеток.
[00316] POU2F1 представляет собой пример TF, который ранее не был связан с конкретной ветвью развития, а был скорее предложен как исключение в семействе POU, - широко экспрессируемый и не контролирующий специфическое направление. Напротив, было обнаружено, что его мотив обогащен в нескольких типах нейронов по меньшей мере при развитии плода человека. Обеспечивая дальнейшее подтверждение, POU2F1 специфически экспрессирован в клетках тех же типов.
[00317] В рамках данного наблюдения стремились далее использовать последовательности дополняющего атласа scRNA-seq, чтобы более обобщенно узнать, экспрессируются ли TF дифференциально в паттерне, согласующемся с дифференциальной доступностью их мотивов. Например, если рассматривать все типы клеток, аннотированные в одной и той же ткани в обоих наборах данных, экспрессия миелоидного инициирующего фактора SPI1/PU.1 имеет выраженную позитивную корреляцию с увеличением его мотива в доступных сайтах. Интересно, что в данном анализе также выявлено множество TF с отрицательной корреляцией между их экспрессией и обогащением мотива. При более тщательной проверке эти TF, как правило, являются репрессорами. Например, было описано, что GFI1B действует как ключевой репрессор для развития эритробластов и мегакариоцитов посредством рекрутирования гистондеацетилазы после связывания ее мотива и индукции закрытия хроматина, например, в локусе эмбрионального гемоглобина. В соответствии с этим наблюдается, что его экспрессия отрицательно коррелирует с обогащением мотива на доступных сайтах.
[00318] При отнесении TF к категории «активаторов» или «репрессоров» на основе терминов генной онтологии (GO) было установлено, что экспрессия TF и доступность мотивов имеют тенденцию положительно коррелировать с аннотированными активаторами и отрицательно коррелировать с аннотированными репрессорами, а корреляцию обогащения и экспрессии мотива можно использовать для прогнозирования механизма действия неклассифицированных TF. Исключения в значительной степени можно объяснить отсутствием или конфликтом терминов GO, тогда как данные поиска по литературе определяют их в категорию, прогнозируемую по параметру корреляции. Соответственно, такой вид анализа может обеспечивать систематический подход к классификации TF как активаторов или репрессоров. Например, NFATc3 по существу описан как активатор, однако данный анализ указывает на репрессивный механизм действия, особенно у развивающихся Т-клеток, где он имеет высокую степень экспрессии, но с истощением его мотива в доступных сайтах. Такой репрессивный механизм действия для NFATc3 кратко упомянут в предыдущих публикациях. Помимо общей классификации можно также получать представление о состояниях типа клеток, в которых TF может переменно выступать в качестве активатора или репрессора. Например, было предложено, что TF, включая FOXO3, в своем немодифицированном состоянии действуют в качестве активаторов, однако при фосфорилировании - в качестве репрессоров, что может объяснить их более неоднозначную взаимосвязь между экспрессией и доступностью.
[00319] Описанный выше подход позволяет систематически связывать известные TF с потенциально новыми функциями, имеет преимущество, заключающееся в том, что он не основан на предварительном выборе дифференцированно доступных сайтов для каждого типа клеток, и дополнительное преимущество, которое можно предложить, заключается в том, что можно связать экспрессию TF с доступностью соответствующего ему мотива. Однако он ограничен основанием на базах данных известных мотивов TF. В другом подходе также вычисляли показатели специфичности для каждого доступного сайта, выбирали 2000 наиболее специфических пиков для каждого типа клеток и проводили поиск de novo обогащенных мотивов в этом наборе по сравнению с CpG-совместимыми фоновыми геномными последовательностями. В целом ведущие мотивы de novo для отдельных типов клеток согласуются с ведущими известными мотивами, определяемыми с помощью линейной регрессии. Интересно, что некоторые типы клеток, которые не имели точных совпадений с известными мотивами (например, эндотелиальные, стромальные, шванновские клетки), были, тем не менее, сильно связаны с мотивами de novo. В частности, для клеток эндотелия такие результаты дополнительно обсуждаются ниже.
[00320] Перекрестно-тканевые анализы клеток крови и клеток эндотелия
[00321] Характер этого набора данных позволяет исследовать специфические для органов различия в доступности хроматина в широко встречающихся типах клеток, например клетках крови и эндотелиальных клетках. При первом проходе аннотаций типа клеток для кровеносной системы удалось дифференцировать миелоидные клетки, лимфоидные клетки, эритробласты, мегакариоциты и гемопоэтические стволовые клетки. Благодаря извлечению и повторному собранию в кластеры этих линий дифференцировки крови из всех органов удалось дополнительно определить макрофаги, В-клетки, естественные киллеры (NK-клетки) / врожденные лимфоидные клетки типа 3 (ILC 3), Т-клетки и дендритные клетки при повторном применении подхода к аннотации с использованием РНК (следует отметить, что для анализа аналогичных типов клеток из множества тканей требовалась дополнительная стадия очистки дублетов; см. «Способы»). Макрофаги можно было дополнительно разделять на группы, связанные с тканью происхождения, как было отмечено ранее, также как и фагоцитарные макрофаги. Последняя группа определена, главным образом, в селезенке, с последующим определением в печени и надпочечнике. Особый интерес в рамках линий дифференцировки представляют эритробласты, вследствие пространственно-временной динамики эритропоэза в процессе развития плода. Изначально эту линию дифференцировки обнаруживали в печени, надпочечнике, сердце и плаценте; в перекрестно-тканевом анализе дополнительно определяли эритробласты в слабопрофилированной селезенке (где первоначально аннотированы только мегакариоциты и миелоидные клетки). Соотношение эритробластов в линиях дифференцировки крови ткани является наибольшим в печени, в соответствии с тем, что этот орган является основным местом эритропоэза на этой стадии развития, за ним следуют селезенка и надпочечник, где фенокопируется тенденция, наблюдаемая в данных РНК. Неожиданное наблюдение надпочечников в качестве потенциального сайта гемопоэза плода дополнительно обсуждается в примере 2.
[00322] При дальнейшем изучении эритробластов на этой стадии развития наблюдалась доступность областей, расположенных проксимально как к гену бета-глобулина взрослого, так и к гену эмбрионального гамма-глобулина, тогда как промотор гена эмбрионального эпсилон-глобулина недоступен. Эритробластный кластер можно было дополнительно разделять на пять основных кластеров Louvain с различной доступностью хроматина, включая отдельный кластер предшественников эритробластов. Доступные области в кластере предшественников эритробластов, а также смежном кластере ранних эритробластов (эритробласт_3) обогащены GATA1::TAL1, а также другими мотивами GATA. Сравнение уровней экспрессии различных факторов GATA в клетках-предшественниках эритробластов позволяет номинально определять GATA1/2 как вероятные TF, ответственные за обогащение данного мотива. Другие кластеры эритробластов, соответствующие более поздним стадиям эритропоэза, демонстрируют обогащение мотива для NFE2/NFE2L2 (эритропобласт_1) и Kruppel-подобных факторов (KLF) (эритропобласт_2/4) и особо заметно отсутствие обогащения для доступности мотива GATA. В недавно опубликованном исследовании scRNA-seq на гемопоэтической системе мыши сообщалось об индукции GATA2 на ранней стадии эритропоэза с последующим снижением экспрессии GATA2, но при этом со стабильной экспрессией GATA1. Наоборот, исследование отсортированных культивированных in vitro популяций эритроидных клеток человека в массе показало снижение экспрессии GATA1 от клеток-предшественников к дифференцированным эритробластам в соответствии с наблюдаемым в тканях плода человека, а также повышение уровней KLF1 и NFE-2 на более поздних стадиях эритробластов. Данные результаты дополнительно указывают на то, что могут существовать эпигенетически различающиеся субпопуляции дифференцированных эритробластов, в которых ландшафт доступности образован отличающимися от GATA факторами, такими как KLF1 или NFE-2. Например, дистальный регуляторный элемент ближе к 5'-концу от GYPA, который используют в качестве рецептора инвазии эритроцитов малярийным паразитом, наиболее доступен в популяции эритробластов_1 и содержит мотив, напоминающий мотив NFE-2.
[00323] Другой интересной пронизывающей ткани системой является сосудистый эндотелий. Интересно, что не существует описанного TF, который бы экспрессировался исключительно в клетках эндотелия сосудов, и можно предположить, что специфическим для эндотелия транскриптомом комбинаторно управляют несколько TF, которые имеют перекрывающуюся экспрессию в эндотелии. В соответствии с этим в данном анализе мотивов JASPAR не наблюдается никакого одиночного, значительного обогащения в эндотелиальных клетках. С другой стороны, обнаружение мотивов de novo на 2000 наиболее специфических для эндотелия пиков показало значительное обогащение фоновыми геномными последовательностями для мотивов, напоминающих ERG и SOX15. В нашем подходе с линейным моделированием эти мотивы, вероятно, не подлежали такой интенсивной оценке, поскольку они не ограничены эндотелиальными клетками (мотив ERG является более обогащенным в мегакариоцитах; а SOX15 является обогащенным в нескольких типах клеток), а экспрессия этих TF не ограничена этим типом клеток. В соответствии с этим, ERG ранее описан как основной регулятор функции эндотелия, однако он также запускает трансдифференцировку в мегакариоциты.
[00324] Эндотелиальные клетки существуют во всех органах, где они должны выполнять как конститутивные, так и высокоспециализированные функции, такие как газообмен в легких или фильтрация жидкости в почках. В нашем исследовании обнаруживали эндотелиальные клетки в 13 из 15 органов (исключениями являются более поверхностно профилированный мозжечок и глаз). Несмотря на строгие итерационные стадии фильтрации для удаления всех остаточных загрязняющих дублетов («Способы») и в отличие от линии дифференцировки эритробластов, за счет извлечения этих клеток из разных органов и повторного собрания в кластеры удалось выявить заметное разделение в соответствии с тканью происхождения. В соответствии с этим также наблюдается тканеспецифические программы экспрессии генов, как описано в примере 2. Действительно, пики доступности, наиболее близкие к этим дифференциально экспрессируемым генам, имеют более высокий показатель специфичности в соответствующей ткани по данным ATAC. Более того, эндотелиальные клетки, полученные из практически всех органов, демонстрировали специфические обогащения мотива TF. Следует отметить, что для многих обогащенных мотивов TF также дифференциально экспрессируются в соответствующей ткани по данным РНК.
[00325] В целом эти данные указывают на то, что общая программа доступности хроматина и экспрессии генов в эндотелиальных клетках (широко распространенном типе клеток, который должен выполнять как общие, так и органоспецифические функции) опосредуется комбинацией конститутивных TF, таких как ERG и SOX15, а также тканеспецифическими TF, которые обуславливают дополнительную специализацию. Эти анализы также подчеркивают преимущества комбинирования как обогащения мотива de novo в специфических пиках, так и подходов с линейной моделью в разных тканях, для обозначения ключевых регуляторов, лежащих в основе ландшафта доступности хроматина отдельных типов клеток.
[00326] Другой интересный пример включает PAEP_MECOM-позитивный тип клеток в плаценте, определенный в обоих атласах scRNA-seq и sc-ATAC-seq. Регуляторные области в данной линии дифференцировки существенно обогащены мотивом HNF1B - фактором, традиционно связанным с развитием почек и поджелудочной железы. Например, HNF1B с высокой специфичностью экспрессирован в клеточной линии дифференцирования PAEP_MECOM в плаценте. Характер данных ATAC-seq, которые захватывают некоторые геномные чтения даже в недоступных сайтах во всех хромосомах, позволяет установить пол клеток на основе Y-хромосомы по сравнению с Х-хромосомами или чтениями аутосомного происхождения. Интересно, что PAEP_MECOM и IGFBP1_DKK-положительные типы плацентарных клеток, а также в меньшей степени плацентарные миелоидные клетки, имеют существенно более низкое соотношение чтений Y-хромосомы у плодов мужского пола. В соответствии с тем, что известно о PAEP (гликоделин) и IGFBP1, эти типы клеток потенциально соответствуют эпителиальным и стромальным клеткам эндометрия матери соответственно.
[00327] CICERO
[00328] В качестве ресурса для дальнейшего исследования для каждой ткани в наборе данных были сгенерированы показатели совместной доступности Cicero и показатели генной активности Cicero. Показатели совместной доступности Cicero можно использовать для прогнозирования цис-регуляторных взаимодействий между доступными элементами. Для создания базы данных предполагаемых цис-регуляторных взаимодействий объединяли элементы, спаренные по положительным показателям совместной доступности. Эта база данных включает 80 миллионов уникальных совместно доступных пар, включая 4,5 миллиона (6%) пар «промотор-дистальный», 76 миллионов (94%) пар «дистальный-дистальный» и 128 000 (0,2%) пар «промотор-промотор». Обнаруживали в среднем по 33 миллиона совместно доступных пар на ткань. 38% пар были уникальны только для одиночной ткани, тогда как только 0,007% пар обнаружены во всех 16 тканях. Пары, обнаруженные в большем количестве тканей, с большей вероятностью оказались парами «промотор-дистальный» и «промотор-промотор». Полученные показатели совместной доступности и показатели генной активности доступны для загрузки на веб-сайте компании.
[00329] Следует отметить, что 89% из 436 206 первоначально определенных сайтов были существенно дифференциально доступными (DA) с долей ложноположительных результатов (FDR) 1% в по меньшей мере одном из этих 85 скоплений клеток относительно контрольного набора из 2040 клеток (120 клеток, случайным образом отобранных из каждого из 17 образцов; см. «Дополнительные средства»). Для определения сайтов DA, в которых доступность была ограничена конкретным (-ыми) кластером (-ами), был адаптирован показатель для количественного определения в исследованиях специфичности экспрессии генов в scRNA-seq к доступности хроматина и рассчитан для всех 436 206 сайтов во всех 85 скоплениях. 39% (167 981/436 206) доступных сайтов классифицировали как ограниченные кластером (т. е. повышенная доступность в ограниченном количестве кластеров); 55% (92 334/167 981) из них были ограничены одиночным кластером.
[00330] Причастность типов клеток к общим признакам и заболеваниям человека
[00331] Основная доля наследуемости общих признаков и заболеваний человека, измеренная посредством полногеномного поиска ассоциаций (GWAS), распределяется на дистальные регуляторные элементы, которые часто являются специфическими для типа клеток. Следовательно, большая часть усилий потрачена на пересекающиеся сигналы полногеномного поиска ассоциаций (GWAS) с данными о гиперчувствительности ДНКазы в массе (и другими эпигенетическими признаками) для систематического связывания конкретных заболеваний с дисфункцией конкретных тканей. Однако анализ таких исследований заметно ограничен гетерогенностью клеточного типа. Учитывая степень сохранения доступности хроматина у мышей по сравнению с человеком, интерес представляет, возможно ли использование этих данных для лучшего понимания специфических для типа клеток эффектов генетической вариации, лежащих в основе сложных человеческих признаков, независимо от различий между видами. Таким образом, несмотря на тот факт, что эти данные получены на тканях мышей, авторы стремились применить современные способы обнаружения специфического для типа клеток обогащения наследуемости человека.
[00332] Для этого количественно определяли обогащение наследуемости признаков человека в пределах пиков DA для каждого из этих 85 кластеров с использованием регрессии оценки неравновесия по сцеплению (LD) (LDSC). После приложения SNP человека к ортологичным координатам в геноме мыши рассчитывали обогащение наследуемости для 32 фенотипов по пикам DA, полученным для каждого из 85 кластеров. 55 из 85 типов клеток имели обогащение по меньшей мере по одному фенотипу, в то время как 28 из 32 фенотипов были обогащены по меньшей мере по одному типу клеток. В качестве общей тенденции наблюдалось сильное обогащение наследуемости для аутоиммунных заболеваний, таких как волчанка, целиакия и болезнь Крона, в кластерах, соответствующих лейкоцитам, тогда как для неврологических признаков, таких как биполярное расстройство, уровень образования и шизофрения, обогащения происходили в клетках нейронального типа. Примечательно, что большинство этих обогащений не было очевидно в пиках, полученных из основной массы тканей, что показывает ценность типов клеток, определенных данными о доступности хроматина одиночных клеток. Многие обогащения соответствовали ожиданию. Например, наиболее сильное обогащение наследуемости для холестерина липопротеинов низкой плотности (ЛПНП), холестерина липопротеинов высокой плотности (ЛПВП) и триглицеридов находится в гепатоцитах, хотя интересно, что холестерин ЛПНП также был значимым в почечном эпителии петли Генле. Аналогичным образом наибольшее обогащение наследуемости дефицита иммуноглобулина A (IgA) представлено в кластерах Т-клеток. Эти сигналы могут также привести к более четкому пониманию важности подтипов клеток. Примером такой тенденции является связь наиболее сильных обогащений с возбуждающими нейронами, хотя обогащение наследуемости по биполярному расстройству наблюдается для множества кластеров нейронов. Наоборот, наследуемость болезни Альцгеймера не обогащена ни в одном классе нейронов. Вместо этого наиболее сильное обогащение обнаружено в кластере микроглии.
[00333] Чтобы расширить данный анализ до большего набора признаков, загружали сводную статистику (nealelab.github.io/UKBB_ldsc/) GWAS для 2419 признаков у более 300 000 индивидуумов из UK Biobank. Принимая во внимание 405 признаков с эффективным размером выборки ≥ 5000 и оцененной наследуемостью ≥ 0,01, наблюдали значимое обогащение наследуемости в 273 признаках в по меньшей мере одном типе клеток, тогда как 74 из 85 типов клеток демонстрируют обогащенную наследуемость по меньшей мере по одному признаку. Хотя в настоящем документе также наблюдаются четкие тенденции, аналогичные описанным выше для аутоиммунных и неврологических признаков, гораздо большее количество признаков, измеренных с помощью UK Biobank, отображает дополнительные тенденции. Например, многие показатели размера и состава тела (например, индекс массы тела) также связаны с типами клеток в головном мозге (Фиг. 18B). Кроме того, конкретные субпопуляции Т-клеток (12.1, 12.2) в большей степени связаны с астмой и аллергическим ринитом, чем клетки других типов, включая другие скопления Т-клеток. На более детальном уровне сердечные приступы ассоциируются с эндотелиальными клетками из печени (25.3), а не из других эндотелиальных кластеров, в то время как подагра ассоциируется с клетками проксимальных канальцев почки. Систему, которая представлена в настоящем документе, можно легко применять к данным о доступности хроматина одиночной клетки, собранным от любой ткани человека или мыши, и любому наследуемому признаку.
[00334] Одним из последствий новой конфигурации является ее совместимость с 2-уровневыми («2lv2» или «2-уровневый протокол версии 2»), а также с 3-уровневыми («3lv2») конфигурациями, в результате чего обеспечивается большая гибкость дизайна исследования (Фиг. 9).
[00335] Наконец, также проанализировали различные условия для фиксации клеток или ядер формальдегидом для обеспечения долгосрочного стабильного хранения. Было обнаружено, что буфер, используемый для фиксации, и решение о выделении ядер до или после фиксации представляли собой варианты выбора между сложностью и специфичностью. В настоящем исследовании избирали протокол фиксации, который повысил сложность/чувствительность за счет специфичности, однако конечные пользователи протокола могут сами сделать этот выбор.
[00336] Материалы и способы
[00337] Клеточная культура
[00338] Клетки GM12878 культивировали и поддерживали в среде RPMI 1640 (Thermo Fisher Scientific, кат. № 11875-093) с добавлением 15% эмбриональной бычьей сыворотки (FBS) (Thermo Fisher, кат. № SH30071.03) и 1% пенициллина-стрептомицина (Thermo Fisher, кат. № 15140122). Клетки подсчитывали и разделяли при концентрации 300 000 клеток/мл три раза в неделю. Линия мышиных клеток CH12-LX была предоставлена компанией Michael Snyder Lab в Станфорде. Клетки культивировали в среде RPMI 1640 с добавлением 10% FBS, 1% пенициллина-стрептомицина и 1×105 M бета-меркаптоэптанол (B-ME). Клетки подсчитывали и поддерживали при плотности 1×105 клеток/мл, разделяя их три раза в неделю для поддержания концентрации клеток. Обе клеточные линии инкубировали при 37 °C в атмосфере с содержанием 5% CO2.
[00339] Выделение и фиксация ядер из клеточных линий
[00340] Для получения суспензии клеток берут ~ 10-100 миллионов клеток и осаждают клетки путем центрифугирования при 500 x g в течение 5 мин при комнатной температуре. Супернатант аспирируют и осадок ресуспендируют в 1 мл лизирующего буфера Omni-ATAC (10 мМ NaCl, 3 мМ MgCl2, 10 мМ Tris-HCl pH 7,4, 0,1% NP40, 0,1% Tween 20 и 0,01% дигитонин), и инкубируют на льду в течение 3 мин. Добавляют 5 мл 10 мМ NaCl, 3 мМ MgCl2, 10 мМ Tris-HCl pH 7,4 с 0,1% Tween 20 и осаждают ядра в течение 5 мин при 500 x g при 4 °C. Супернатант аспирируют и ресуспендируют ядра в 5 мл 1X физиологического раствора с сульфатным буфером Дульбекко (DPBS) (Thermo Fisher, кат. № 14190144). Для поперечного сшивания ядер одним впрыскиванием добавляют 140 мкл 37% формальдегида с метанолом (VWR, кат. № MK501602) при конечной концентрации 1%. Инкубируют фиксирующую смесь при комнатной температуре в течение 10 минут, переворачивая каждые 1-2 минуты. Для гашения реакции поперечного сшивания добавляют 250 мкл 2,5 M глицина и инкубируют при комнатной температуре в течение 5 минут, а затем на льду в течение 15 минут, чтобы полностью остановить поперечное сшивание. Для подсчета добавляют 20 мкл погашенной поперечносшитой смеси к 20 мкл трипанового синего. Поперечносшитые ядра центрифугируют при 500 x g в течение 5 минут при 4 °C и аспирируют супернатант. Фиксированные ядра ресуспендируют в соответствующем количестве буферного раствора для замораживания (50 мМ Tris при pH 8,0, 25% глицерин, 5 мМ Mg(OAc)2, 0,1 мМ этилендиаминтетрауксусная кислота (ЭДТА), 5 мМ дитиотреитол (ДТТ) (Sigma-Aldrich, кат. № 646563-10X0,5 мл), 1 × смесь ингибиторов протеазы (Sigma-Aldrich, кат. № P8340)) для получения 2 миллионов ядер на аликвоту 1 мл, мгновенно замораживают в жидком азоте и хранят при -80 °C.
[00341] Получение и хранение ткани
[00342] Интересующую ткань выделяют и промывают в 1X сбалансированном солевом растворе Хэнкса (HBSS) (с Ca и Mg), а затем промокают досуха полувлажной марлей. Высушенную ткань помещают на сверхпрочную фольгу или в криопробирку и мгновенно замораживают с помощью жидкого азота. Замороженные ткани хранят при -80 °C.
[00343] Выделение ядер и фиксация замороженных тканей плода
[00344] В день измельчения в порошок предварительно охлаждают предварительно маркированные пробирки и отбивают молотком на сухом льду с тканевым полотенцем между сухим льдом и металлом. Создают «подкладку», взяв сверхпрочную фольгу размером 18 дюймов x 18 дюймов (46 см х 46 см), складывают ее пополам дважды для получения прямоугольника. Складывают еще два раза, чтобы получить квадрат. Помещают замороженную ткань внутрь «подкладки» фольги, затем помещают ткань в подкладке из фольги внутрь предварительно охлажденного пластикового пакета 4 мм для предотвращения выпадания ткани на сухой лед в случае разрыва фольги. Охлаждают этот пакет с тканью между 2 пластинами сухого льда. Вручную измельчают ткань внутри пакета в порошок предварительно охлажденным молотком; совершают от 3 до 5 ударов без растирающего движения перед перерывом, чтобы не нагреть образец. Охлаждают молоток и при необходимости повторно измельчают в порошок до получения однородной ткани. Аликвоту измельченной в порошок ткани помещают в предварительно маркированные и предварительно охлажденные пробирки LoBind объемом 1,5 мл и безнуклеазные пробирки объемом 1,5 мл с защелкивающимися крышками (Eppendorf, кат. № 022431021). Аликвоты измельченных в порошок тканей можно хранить при -80 °C до дальнейшей обработки.
[00345] В день выделения ядер добавляют лизирующий буфер непосредственно в пробирку или выливают замороженную аликвоту в посудину размером 60 мм с буфером для лизиса клеток и дополнительно измельчают лезвием. Если аликвоту не размораживали в какой-либо момент хранения, порошкообразная аликвота ткани должна легко выскальзывать из пробирки для хранения, и при этом образец не теряется По нашей оценке количество, составляющее ~ 20 000 клеток на мг исходного веса ткани, и производительность могут варьироваться в зависимости от ткани. Измельченную в порошок ткань ресуспендируют в 1 мл лизирующего буфера Omni (ресуспендирующий буфер (RSB) + 0,1% Tween+0,1% NP-40 и 0,01% дигитонин), затем переносят в пробирку falcon объемом 15 мл. Ядра инкубируют на льду в течение 3 минут, затем добавляют 5 мл RSB+0,1% Tween 20. Центрифугируют ядра при 500 x g в течение 5 минут при 4 °C. Супернатант аспирируют и ресуспендируют в 5 мл 1X DPBS. Для удаления комков ткани пропускают ядра в 1X DPBS через клеточное сито с размером пор 100 мкм (VWR, кат. № 10199-658). В вытяжном шкафу поперечно сшивают ядра с помощью добавления 140 мкл 37% формальдегида и метанола одним впрыскиванием до конечной концентрации 1% и быстрого перемешивания посредством переворачивания пробирки несколько раз. Инкубируют при комнатной температуре в течение ровно 10 минут, слегка переворачивая пробирку каждые 1-2 минуты. Добавляют 250 мкл 2,5 M глицина (свежеприготовленного, стерилизованного с помощью фильтра) для гашения реакции поперечного сшивания, хорошо перемешивают, переворачивая пробирку несколько раз. Инкубируют в течение 5 минут при комнатной температуре, затем на льду в течение 15 минут, чтобы полностью остановить поперечное сшивание. Подсчитывают ядра с помощью гемоцитометра для определения конечного объема добавляемого буферного раствора для замораживания, цель - заморозить ~ 1-2 миллиона ядер на пробирку. Центрифугируют поперечносшитые ядра при 500 x g в течение 5 минут при 4 °C, аспирировать супернатант и ресуспендировать осадок в 1-10 мл буфера для замораживания, дополненного 1x ингибиторами протеазы и 5 мМ ДТТ. Мгновенно замораживают ядра в жидком азоте и хранят их при -80 °C.
[00346] Обработка образцов sci-ATAC-seq3 (конструирование библиотеки и контроль качества)
[00347] Извлекают замороженные фиксированные ядра из холодильника с температурой -80 °C и помещают на слой сухого льда. Размораживают ядра на водяной бане при 37 °C до оттаивания (~ 30 с-1 мин) и переносили в пробирку falcon объемом 15 мл. Осаждают ядра при 500 x g в течение 5 минут при 4 °C. Аспирируют супернатант, не нарушая осадок, и ресуспендируют осадок в 200 мкл лизирующего буфера Omni, а затем инкубируют на льду в течение 3 минут. Вымывают лизирующий буфер 1 мл ATAC-RSB, используя 0,1% Tween 20, и осторожно переворачивают пробирку 3 раза для перемешивания. Подсчитывают ядра, взяв 20 мкл ядер и 20 мкл трипанового синего. При подсчете ядра держат на льду по возможности постоянно с этого момента. Для экспериментов с 3-уровневым индексированием при 384^3 входное число ядер составляет 4,8 миллиона по 50 000 ядер на лунку на ткань или образец, распределенный на 96 реакций. Ядра осаждают и ресуспендируют в предварительно приготовленной основной смеси для реакции тагментации (буфер Nextera TD, 1X DPBS, 0,1% дигитонин, 0,1% Tween 20 и вода). Аликвотируют 47,5 мкл ядер в смесь для тагментации с использованием наконечника с широким каналом (Rainin Instrument Co. кат. № 30389249) в луночный планшет LoBind 96 (Eppendorf, кат. № 30129512). Добавляют по 2,5 мкл фермента Nextera v2 (Illumina Inc., кат. № FC-121-1031) на лунку, герметизируют планшет адгезивной лентой и центрифугируют при 500 x g в течение 30 сек. Инкубируют планшет при 55 °C в течение 30 минут для тагментации ДНК. Реакции тагментации останавливали путем добавления 50 мкл смеси для остановки реакции (40 мМ ЭДТА с 1 мМ спермидина), затем инкубировали при 37 °C в течение 15 мин. Меченые ядра объединяли с использованием наконечников с широкими каналами и осаждали при 500 x g в течение 5 минут при 4 °C, а затем промывали ATAC-RSB с 0,1% Tween 20. Ядра осаждают при 500 x g в течение 5 минут при 4 °C, аспирируют супернатант и ресуспендируют в 384 мкл ATAC-RSB с 0,1% Tween 20. Создают основную смесь для реакции полинуклеотидкиназы (PNK) (1X буфер PNK (NEB, кат. № M0201L), 1 мМ рибоаденозинтрифосфат (rATP) (кат. № P0756S), вода и полинуклеотидкиназа Т4 (NEB, кат. № M0201L) и добавляют к ядрам. Аликвотируют 5 мкл реакционной смеси PNK в четыре 96-луночных планшета LoBind, герметизируют адгезивной лентой и центрифугируют при 500 x g в течение 5 минут при 4 °C. Реакционную смесь PNK инкубировали при 37 °C в течение 30 минут. К реакционной смеси PNK непосредственно добавляют 13,8 мкл основной смеси для лигирования (1X лигазный буфер T7 (NEB, кат. № M0318L), 9 мкМ N5_splint (IDT), вода и 2,5 мкл фермента ДНК-лигазы T7 (NEB, кат. № M0318L). С помощью многоканального или 96-головочного дозатора (Liquidator, кат. № 17010335), добавляют 1,2 мкл 50 мкМ N5_oligo (IDT) в каждую лунку четырех 96-луночных планшетов. Запечатывают адгезивной лентой и центрифугируют при 500 x g в течение 30 секунд, затем инкубируют при 25 °C в течение 1 часа. После первого цикла лигирования добавляют 20 мкл 40 мМ ЭДТА с 1 мМ спермидином для остановки реакции лигирования и инкубируют при 37 °C в течение 15 минут. С помощью наконечников с широким каналом объединяют содержимое каждой лунки в лоток и переносят в пробирку falcon объемом 50 мл. Ядра осаждают при 500 x g в течение 5 минут при 4 °C, аспирируют супернатант и ресуспендируют ядра в 1 мл ATAC-RSB с 0,1% Tween 20 для промывки остатков реакционной смеси для лигирования. Ядра осаждают при 500 x g в течение 5 минут при 4 °C и аспирируют супернатант без нарушения целостности осадка. Готовят основную смесь для лигирования N7 (1X лигазный буфер T7, 9 мкM N7_splint (IDT), вода и ДНК-лигаза T7) и ресуспендируют ядра основной смесью для лигирования. Суспендированные в основной смеси ядра переносят в лоток и с помощью наконечников с широким каналом помещают аликвоты по 18,8 мкл основной смеси для лигирования в четыре 96-луночных планшета LoBind, а затем в каждую лунку четырех 96-луночных планшетов добавляют по 1,2 мкл 50 мкМ N7_oligo (IDT). Планшеты запечатывают адгезивной лентой и центрифугируют при 500 x g в течение 30 секунд, затем инкубируют при 25 °C в течение 1 часа, после чего останавливают лигирование путем добавления 20 мкл 40 мМ ЭДТА и I мМ спермидина и инкубируют при 37 °C в течение 15 минут. Содержимое лунок объединяют в лотке с помощью наконечников с широким каналом, а затем переносят в пробирку falcon объемом 50 мл. Ядра осаждают при 500 x g в течение 5 минут при 4 °C, аспирируют супернатант и ресуспендируют ядра в 2 мл буфера EB Qiagen (Qiagen, кат. № 19086). Для подсчета ядер берут 20 мкл ресуспендированных ядер и 20 мкл трипанового синего. Ядра разводят до концентрации 100-300 ядер на мкл и аликвотируют по 10 мкл на лунку в четырех 96-луночных планшетах LoBind. Для обратного поперечного сшивания ядер готовят основную смесь для обратного поперечного сшивания ядер в виде буфера EB, протеиназы k (Qiagen, кат. № 19133) и 1% додецилсульфата натрия (SDS) (по 1 мкл/0,5 мкл/0,5 мкл на лунку соответственно) и добавляют по 2 мкл в каждую лунку с ядрами. Запечатывают адгезивной лентой, центрифугируют при 500 x g в течение 30 секунд и инкубируют при 65 °C в течение 16 часов. Для определения оптимального количества циклов выполняли ПЦР-амплификацию и отслеживали реакцию с SYBR зеленым в нескольких лунках планшета. На основании результатов теста ПЦР амплифицировали остальные планшеты с обратным поперечным сшиванием с использованием по 7,5 мкл основного раствора Nextera для ПЦР (NPM), 0,5 мкл BSA (NEB, кат. № B9000S), 1,25 мкл индексированного P5_10 мкМ (IDT), 1,25 индексированного P7_10 мкМ (IDT) и воды на лунку. В зависимости от партии тканей и восстановления ядер после двух циклов лигирования обычно требовалось 11-13 циклов. Условия циклов были следующими: 72 °C - 3 мин, 98 °C - 30 с, 11-13 циклов (98 °C - 10 с, 63 °C - 30 с, 72 °C - 1 мин) и выдерживание при 10 °C. Продукт амплификации из 96-луночного планшета объединяли в лотке, и очищали с использованием Zymo Clean & Concentrate-5 (Zymo Research кат. № D4014) в соответствии с инструкциями производителя, и разделяли между 4 колонками. Каждую колонку элюировали в 25 мкл буфера EB, а затем объединили в 1 пробирку. 100 мкл гранул AMPure (Agencourt, кат. № A63882) добавляли к очищенному продукту ПЦР для дополнительного избавления от любых остаточных димеров праймеров и в соответствии со процессами очистки производителя. Элюирование конечных библиотек из гранул в 25 мкл буфера EB Qiagen. Количественно определяют конечную библиотеку с помощью системы D5000 screentape (Agilent, screentape кат. № 5067-5588, реагенты 5067-5589) Agilent 4200 Tapestation, с установлением диапазона 200-1000 пар нуклеотидов для определения концентрации нМ фрагментов, которые будут хорошо собираться в кластеры в процессе секвенирования. Пул 2 нM создавали из эквимолярного объединения и секвенировали при концентрации загрузки 1,8 пМ с использованием высокопроизводительного 150-циклового набора NextSeq (Illumina, кат. № 20024904) с индивидуальной рецептурой и праймерами.
[00348] Обработка данных для разработки способа
[00349] Обработку данных в экспериментах типа «скотный двор», проведенных для разработки sci-ATAC-seq3, выполняли так, как описано выше. Вкратце файлы BCL преобразовывали в файлы fastq с помощью bcl2fastq версии 2.16 (Illumina). Каждое чтение связывали со штрихкодом клеток, состоящим из 4 компонентов: на конце Р5 молекулы находился добавленный адрес строки для тагментации и для ПЦР, а на конце Р7 молекулы находился добавленный адрес колонки для тагментации и ПЦР. Для исправления ошибок в таких штрихкодах их разделяли на 4 составляющих части и корректировали до ближайшего штрихкода в пределах редакционного расстояния 2 до тех пор, пока такая коррекция была однозначной на требуемом редакционном расстоянии. Если ни один из четырех штрихкодов невозможно было скорректировать в соответствии с известным штрихкодом, соответствующую пару чтений удаляли. Затем считанные данные обрезали с помощью Trimmomatic, используя параметр ILLUMINACLIP:{adapters_path}:2:30:10:1:true TRAILING:3 SLIDINGWINDOW:4:10 MINLEN:20. Обрезанные чтения картировали с гибридным человеческим/мышиным геномом (hg19/mm9) с применением Bowtie2 с параметрами -X 2000 -3 1. Затем считывание результатов картирования генома в соответствующих парах с качеством по меньшей мере 10 отфильтровывали с помощью samtools с использованием параметров -f3 -F12 -q10, и для дальнейшего анализа оставляли только чтения результатов картирования аутосом или половых хромосом. Чтения дедуплицировали по штрихкоду каждой клетки с использованием специализированного сценария. Следует отметить, что в отличие от процесса подготовки для тканей (описан ниже) пары чтений не сохранялись при дедуплицировании.
[00350] Обработка данных для образцов ткани
[00351] Способы обработки данных секвенирования для образцов ткани точно соответствуют применяемым способам, хотя и с многочисленными оптимизациями для масштабирования к более крупным наборам данных, но для удобства описание включено в настоящий документ. Файлы BCL преобразовывали в файлы fastq с помощью bcl2fastq версии 2.20 (Illumina). Для каждого из образцов в таком наборе данных чтения с откорректированными штрихкодами, содержащимися в названии чтения, записывали в отдельный файл R1/R2. Следует отметить, что картирование всех несоответствий с набором известных штрихкодов вычисляли заранее (осуществимо, вследствие небольшой длины и относительно небольшого количества штрихкодов), сценарий коррекции выполняли с использованием pypy (альтернатива интерпретатору cpython, которая гораздо быстрее работает для данной конкретной задачи), и это вычисление проводили параллельно на различных дорожках прогона секвенирования, что в совокупности заметно сокращало время работы по сравнению с предыдущим способом.
[00352] Далее обрезали основания низкого качества/адаптерные последовательности с 3’-конца с помощью Trimmomatic с использованием параметров ILLUMINACLIP:{adapters_path} TRAILING:3 SLIDINGWINDOW:4:10 MINLEN:20, а затем картировали обрезанные чтения с эталонным геномом HG19 с использованием Bowtie2 с -X 2000 3 1 в качестве параметров и далее отфильтровывали пары чтений, которые не были уникально картированы с аутосомами или половыми хромосомами с качеством картирования по меньшей мере 10 с использованием Samtools -- samtools view -L {whitelist of chromosomes} -f3 -F12 -q10 -bS. Полученные файлы BAM сортировали, выравненные чтения для каждого образца объединяли с помощью sambabamba и индексировали полученные файлы BAM. По возможности этот процесс параллельно распределяли по образцам/дорожкам и одновременно обеспечивали trimmomatic/bowtie2/sambabamba со множеством цепочек на процесс для уменьшения времени работы.
[00353] Впоследствии определяли дубликаты ПЦР внутри клеток путем определения уникального набора конечных точек фрагментов внутри каждой клетки. В предыдущей работе полученный дедуплицированный файл BAM не всегда сохранял надлежащее название чтения между парами чтений, расписанными в дедуплицированном файле BAM (он случайным образом выбирал типичное чтение для R1 и R2 для каждого уникального фрагмента независимо), что привело к проблемам с совместимостью с некоторыми инструментами, такими как SnapATAC (github.com/r3fang/SnapATAC). Эту проблему исправили, а также применили написание 1) файла BED конечных точек фрагментов для каждой клетки и 2) файла, точно отражающего файл fragments.tsv.gz, предоставленного 10х Genomics для их решения scATAC.
[00354] В каждом образце использовали файл BED уникальных конечных точек фрагментов для каждой клетки для определения пиков в каждом образце посредством MACS2 -- macs2 callpeak -t {bed} -f BED -g hs --nomodel --shift -100 --extsize 200 --keep-dup all --call-summits -n {sample_name} -o {output_dir}. Полученный файл {outdir}/{sample_name}_peaks.narrowPeak сортировали, а выходные данные получали в виде файла BED. Определения пиков из всех образцов, включенных в дальнейший анализ (дополнительно за исключением наших стандартов), объединяли с помощью bedtools с образованием основного набора пиков. Следует отметить, что, как было описано ранее, использование файлов BED для определения пиков является преднамеренным и обходит поведение macs2 на входных данных BAM. MACS2, учитывая файл BAM в качестве входных данных, либо отбрасывает одну из пар чтения, которая независимо использует R1/R2 (эффективно снижая дискретизацию), либо использует всю вставку при вычислении покрытия, если явно указано, что файл BAM получен от чтений со спаренным концом (нет намерения вычислить покрытие по всей вставке, а только конечные точки). Использование файла BED позволяет использовать все данные и рассчитывать покрытие только с использованием области вокруг конечных точек молекулы.
[00355] Для каждого образца дополнительно создавали разреженные матрицы с подсчетом 1) чтений, попадающих в главный набор пиков; 2) чтений, попадающих в генные тельца, расширенные на 2 т. п. н. ближе к 5'-концу и на области генома в 5 т. п. н. Кроме того, для контроля качества дополнительно группировали общее количество чтений от каждой клетки, поступающей из аннотированных TSS (+/-1 т. п. н. вокруг каждого TSS), областей черного списка ENCODE и данного набора объединенных пиков.
[00356] Кроме того, конструировали матрицу пиков за мотивами с использованием способа, применяемого в процессе подготовки scATAC 10x genomics (см. support.10xgenomics.com/single-cell-atac/software/pipelines/latest/algorithms/overview). Вкратце в способе, полученном в 10х, рассчитывают распределение GC% пиков и групповых пиков в равных количественных диапазонах содержания гуанина-цитозина (GC) таким образом, что случаи мотивов можно обнаруживать в каждой группе отдельно. Пакет MOODS использован для определения встречаемости мотивов для мотивов в базе данных мотивов JASPAR при пороговом p-значении 1E-7 и фоновом нуклеотидном составе, совпадающем с соответствующей группой GC, для уменьшения систематической ошибки GC. Эти совпадения применяют для конструирования мотива по матрице пиков, которую можно использовать для вычисления матриц мотива по количеству клеток в последующих анализах. Эта матрица сдвоена таким образом, что на один пик может быть засчитан только один экземпляр мотива.
[00357] Клеточные штрихкоды разделяли от распределения фоновых штрихкодов с применением модифицированной версии способа, использованного в процессе подготовки scATAC 10x genomics (см. ссылку выше). Вкратце определяли зависимость смеси двух отрицательных биномов (шум по сравнению с сигналом). Вместо способа, используемого 10x для установления начального порога между этими двумя распределениями, применяют k-среднюю кластеризацию к логарифмически масштабированному распределению общего числа фрагментов и берут максимальное значение кластера, причем в качестве начального порога используют меньшие средние значения общего числа. Этот начальный порог используют для определения начальной параметризации для двух распределений с использованием оценок максимального правдоподобия и дополнительно уточняют посредством подхода максимального увеличения ожидания. Как отмечалось в 10х, такую зависимость можно улучшать путем применения к распределению подсчетов сдвига влево. В отличие от способа 10х, определяют этот сдвиг с помощью испытания нескольких сдвигов от 2 до 12 и использования смесовой модели с наилучшей точностью зависимости. Наконец, в отличие от подхода 10x, применяют этот способ к распределению общего количества фрагментов, а не к распределению количества фрагментов в пределах полученных пиков. Выбранное конечное пороговое значение представляло собой минимальное количество, которое дает отношение шансов (в пользу сигнала) 20 или выше и исключает по меньшей мере 0,5% распределения сигнала по оценке по кумулятивной функции распределения (CDF) для распределения сигнала (обнаруживали, что в противном случае этот второй критерий предотвращает зависимости с пороговыми значениями, которые в противном случае оказывались слишком слабыми).
[00358] Контроль качества на уровне клеток, снижение размерности и кластеризация
[00359] Для каждой клетки группировали общее количество уникальных чтений и общее количество уникальных чтений, попадающих вокруг сигналов TSS (+/1 т. п. н.), в пиках и в областях черного списка ENCODE, как упомянуто выше. Используя эти суммарные значения, выбирали специфические для образца отсечки для доли уникальных чтений в пиках и доли уникальных чтений, попадающих в TSS, путем визуального контроля их распределений для каждого образца и глобальной отсечки, составляющей 0,5% от уникальных чтений, поступающих из областей черного списка ENCODE. Из-за небольшого количества образцов, имеющих автоматические пороговые значения, которые были по существу ниже, чем у других образцов в наборе данных, применяли глобальный порог, составляющий 1000 уникальных чтений на клетку (или 500 уникальных фрагментов на клетку), чтобы повысить автоматические пороговые значения для соответствующих образцов. Анализировали показатели разделения нуклеозидов на полосы, которые получали ранее, но не наблюдали четкого распределения выбросов, как делали ранее при исследовании мышиных яичек и, следовательно, не использовали эти показатели в контроле качества. Пики, перекрывающие области черного списка ENCODE или попадающие на половые хромосомы, удаляли перед последующими стадиями (последнее, чтобы избежать введения потенциальных групповых эффектов между образцами различных полов). Кроме того, исключали пики за пределами двух стандартных отклонений от среднего значения логарифмически масштабированных количеств на распределение пиков для удаления пиков с очень низкими количествами в анализируемых тканях.
[00360] Все последующие стадии проводили с одной тканью за раз, объединяя проходящие клетки из всех образцов данной ткани.
[00361] После фильтрации применяли модифицированную версию алгоритма scrublet для удаления клеток, которые с наибольшей вероятностью были дублетами. Вкратце моделируют дублеты в виде сумм случайно выбранных клеток из набора данных с использованием пика по клеточной матрице. Далее выполняют LSI, как описано ниже, с использованием матрицы исходных клеток и моделируемых дублетов. Следует отметить, что на данной стадии используют термин «обратная частота документа (IDF)», полученный из исходного набора данных без смоделированных дублетов, аналогично тому, как коэффициенты масштабирования из исходного набора данных для данных scRNA-seq применяют в scrublet. В полученном 50-размерном пространстве находят ближайших соседей каждой клетки и вычисляют долю смоделированных дублетов по соседству как показатель дублетов. В каждом образце с наивысшим показателем дублетов исключают верхние 10% клеток.
[00362] Касательно снижения размерности вначале, на основании данных, собранных в данном исследовании, обнаружили, что реализация латентно-семантического индексирования (LSI; или эквивалентного латентно-семантического анализа, или LSA), которое описывали ранее, не дала хороших результатов. Авторы решили, что это, вероятно, связано с малочисленностью, и исследовали несколько альтернативных способов, включая CisTopy и SnapATAC. Каждый из этих способов, по-видимому, изначально показал лучшие результаты, чем такая реализация LSI. Изначально не было уверенности в причине, с учетом фундаментального сходства этих способов и природы данных. Обнаруживали, что простое логарифмическое масштабирование «слово-частота слова» в LSI, которое никто не выполнял ранее, приводило к результативности, очень похожей на другие исследованные инструменты. Предполагают, что это, вероятно, связано с экспоненциальным распределением общих количеств на клетку и влиянием сильных выбросов на стадии PCA LSI при отсутствии логарифмического масштабирования. Подробное описание см. здесь: andrewjohnhill.com/blog/2019/05/06/dimensionality-reduction-for-scatac-data/. Отмечается, что разница, наблюдаемая с логарифмическим масштабированием и без него, особенно заметна для немногочисленных наборов данных, причем диапазон общих количеств на клетку является широким. Отмечается также, что впоследствии другие группы подтвердили независимые выводы авторов о том, что LSI имеет преимущества при сравнении со всеми другими существующими способами снижения размерности для scATAC. Кроме того, наблюдали очень схожие характеристики при использовании пиков или областей из 5 т. п. н. генома, поэтому принимали решение использовать пики, как делали ранее в предыдущей работе.
[00363] Подводя итог, выполняли LSI в бинаризованном диапазоне по клеточной матрице из всех проходящих клеток из каждой ткани по одной ткани за один раз. Сначала взвешивали все сайты относительно отдельных клеток по логарифмическому принципу (общее количество пиков, доступных в клетке) (логарифмически масштабированная «частота слова»). Затем умножали эти взвешенные значения на log(1+обратная частота каждого сайта по всем клеткам), «обратная частота документа». Затем использовали сингулярное разложение на матрице «частота слова-обратная частота документа» (TF-IDF) с образованием меньшего пространственного представления данных (PCA) путем сохранения только измерений со 2-го по 50-е (поскольку первое измерение, как правило, сильно коррелирует с глубиной чтения). Далее выполняли нормализацию L2 на матрице PCA в попытке дополнительного учета различий в количестве уникальных фрагментов на клетку. Эту L2-нормализованную матрицу PCA использовали для всех последующих стадий.
[00364] Хотя не наблюдались никакие признаки существенных групповых эффектов между образцами, применяли алгоритм групповой коррекции Harmony в пространстве PCA для коррекции групповых эффектов между различными образцами. Выбирали Harmony главным образом в связи с тем, что он легко масштабировался для больших наборов данных и позволил использовать существующие координаты PCA.
[00365] Это скорректированное L2-нормализованное пространство PCA использовали в качестве входных данных для кластеризации Louvain и UMAP, как реализовано в Seurat версии 3.
[00366] Показатели специфичности
[00367] Перед расчетами показателя специфичности отфильтровывали любые пики, перекрывающиеся с областями черного списка ENCODE. Для каждой пары сайт/тип клетки рассчитывали показатели специфичности, как описано ранее.
[00368] Обогащение мотива
[00369] Перед расчетами обогащения мотива отфильтровывали любые пики, перекрывающиеся с областями черного списка ENCODE. Сначала получали матрицу мотива по количеству клеток путем умножения соответствующего пика на матрицу клеток (как описано выше, агрегированную по всем клеткам в подгруппе исследуемых данных), на пик на матрице мотивов. Обратите внимание, что для снижения стоимости вычислений и уменьшения чрезмерного представления очень распространенных типов клеток при вычислениях на последующих стадиях использовали уменьшение частоты дискретизации набора данных таким образом, чтобы включить максимум 800 клеток на аннотацию (например, тип клетки). Затем для каждой аннотации выполняют отрицательную биномиальную регрессию с использованием пакета speedglm, прогнозируя общее количество мотивов с использованием двух входных переменных - индикаторного столбца для аннотации в качестве основной интересующей переменной и логарифмического значения (общего количества ненулевых элементов во входной матрице пиков) для каждой клетки в качестве ковариаты. Чтобы оценить кратность изменения числа мотивов интересующей аннотации относительно клеток от всех других аннотаций: экспонента(отрезок+коэффициент_аннотации)/экспонента(отрезок), используют коэффициент для столбца индикатора аннотации и перехвата. Этот анализ выполняют для всех мотивов во всех группах, а затем корректируют р-значения с использованием процедуры Бенджамини - Хохберга.
[00370] Пример 2
[00371] Атлас экспрессии генов человеческих клеток в процессе развития
[00372] Реферат
[00373] Появление и дифференцировка типов клеток в процессе развития человека представляют фундаментальный интерес. Для профилирования экспрессии генов в одиночных клетках применяли анализ на основании трехуровневого комбинаторного индексирования (sci-RNA-seq3) к 121 ткани плода, представляющей 15 органов и в совокупности профилировали транскрипцию в 4-5 миллионах одиночных клеток. На основании этих данных определяют типы клеток и аннотируем их по отношению к маркерным генам, экспрессионным и регуляторным модулям. Исходные анализы этих данных сосредотачивают на типах клеток, которые охватывают множество систем органов, например эпителиальные, эндотелиальные клетки и клетки крови. Интересные наблюдения включают органоспецифическую эндотелиальную специализацию, потенциально новые области фетального эритропоэза и потенциально новые типы клеток. Наряду с сопроводительными атласами доступности хроматина в процессе развития человеческих клеток, эти данные являются богатым ресурсом для изучения биологии человека.
[00374] Основной текст
[00375] По нескольким причинам авторы стремились генерировать атласы как экспрессии генов, так и доступности хроматина человеческих клеток с использованием тканей, полученных в ходе развития. Во-первых, генетические расстройства, большая часть которых включает компонент развития, составляют чрезвычайно непропорциональную долю педиатрических заболеваний и смертности. К ним относятся тысячи расстройств, наследуемых по законам Менделя, а также более распространенные заболевания (например, врожденные дефекты сердца, другие врожденные дефекты, расстройства неврологического развития и т. д.), в развитии которых по существу участвуют как генетические, так и негенетические факторы. Атлас эталонных клеток, генерируемый из развивающихся тканей, может служить основой для систематических усилий для понимания определенных молекулярных и клеточных событий, которые приводят к возникновению каждого из этих педиатрических состояний.
[00376] Во-вторых, развивающиеся ткани обеспечивают гораздо лучшую возможность изучения возникновения и дифференцировки in vivo типов клеток человека, чем зрелые ткани. По сравнению с эмбриональными тканями и тканями плода, в зрелых тканях преобладают дифференцированные клетки, и, более того, многие состояния клеток просто не имеют представительства. Путем лучшего разрешения траекторий развития in vivo, атласы одиночных клеток, генерируемые из развивающихся тканей, могут существенно расширять базовое понимание человеческой биологии in vivo, а также стратегий перепрограммирования клеток и клеточной терапии.
[00377] В-третьих, несмотря на то что уже имеются сообщения о новаторских атласах клеток для многих органов взрослого человека, независимый характер этих исследований затрудняет изучение различий между типами клеток, встречающимися в разных тканях, например эпителиальных, эндотелиальных клетках и клеток крови. В частности, сравнения на основе существующих данных затруднены из-за различий в обработке образцов и технологических платформах между группами, создающими специфические для органов атласы клеток.
[00378] В отношении атласов экспрессии генов человеческих клеток применяли недавно разработанный анализ для RNA-seq одиночных клеток на основании трехуровневой комбинаторного индексирования (sci-RNA-seq3) к 121 ткани плода, представляющей 15 органов, и в целом профилировали экспрессию генов почти в 5 миллионах клеток (Фиг. 11). В примере 1 описано профилирование доступности хроматина в 1,6 миллиона клеток из одних и тех же органов на основании перекрывающегося набора образцов. Профилированные органы охватывают разнообразные системы; наиболее заметно отсутствие костного мозга, костей, гонад и кожи.
[00379] Ткани получены из 28 плодов с примерным гестационным возрастом в диапазоне от 72 до 129 дней. Вкратце их подвергали мгновенному замораживанию, измельчению и разделяли полученный порошок для различных анализов. Для sci-RNA-seq3 ядра экстрагировали непосредственно из холодного лизированного порошка, а затем фиксировали параформальдегидом. Для почек и пищеварительных органов, в которых в большом количестве содержатся РНКазы и протеазы, использовали не ядра, а клетки, фиксированные параформальдегидом, что повышало извлечение клеток и мРНК. В каждом эксперименте ядра или клетки из данной ткани помещали в разные лунки таким образом, чтобы первый индекс протокола sci-RNA-seq3 также идентифицировал источник. В качестве группового контроля для экспериментов на ядрах в одну или несколько лунок вносили смесь ядер HEK293T человека и NIH/3T3 мыши, или ядер из распространенной «сторожевой» ткани (также использовавшейся для экспериментов sci-ATAC-seq3). В качестве группового контроля для экспериментов на клетках в одну или несколько лунок вносили клетки, полученные из распространенной ткани поджелудочной железы (ядра для которой также были профилированы).
[00380] Секвенировали библиотеки sci-RNA-seq3 из 7 экспериментов в 7 прогонах Illumina NovaSeq и в целом получали 68,6 миллиарда чтений. Обрабатывая данные, как описано выше, получали 4,979,593 профиля экспрессии генов одиночных клеток (UMI > 250). Транскриптомы одиночных клеток из контрольных лунок с человеческими и мышиными клетками были преимущественно связаны с видом (~ 5% коллизий). Приближение и проекция однородного многообразия (UMAP) ядер или клеток из «сторожевых» тканей указывали на то, что различия между типами клеток преобладают в сравнении с любыми межэкспериментальными групповыми эффектами. Интегрированный анализ с использованием seurat ядер и клеток, соответствующих распространенной ткани поджелудочной железы, также привел к высоко перекрывающимся распределениям.
[00381] Профилировали медиану 72 241 клетки или ядра на орган (максимум 2 005 512 (головной мозг); минимум 12 611 (тимус)). Несмотря на относительно поверхностное секвенирование (~ 14 000 необработанных чтений на клетку) по сравнению с другими крупномасштабными атласами RNA-seq одиночных клеток, получали сопоставимое число UMI на клетку или ядро (медиана 863 UMI и 525 генов). Как и ожидалось, ядра демонстрировали более высокую долю картирования UMI к интронам, чем клетки (56% для ядер; 45% для клеток; p < 2,2e-16, двусторонний критерий суммы рангов Уилкоксона). В дальнейшем, если не указано иное, для обозначения как клеток, так и ядер, используется термин «клетки».
[00382] По экспрессии специфического для пола гена ткани легко определяли как полученные от особи мужского (n=14) или женского (n=14) пола. Каждый из 15 органов представлял собой множество образцов (медиана 8), включая по меньшей мере два от каждого пола и диапазона гестационных возрастов. Визуализация UMAP для «псевдомассовых» транскриптом каждой ткани, собрана в кластеры в зависимости от органа, а не от индивидуума или эксперимента. Около половины экспрессируемых кодирующих белок транскриптов дифференциально экспрессировались в данном наборе псевдомассовых транскриптом (11 766 из 20 033; FDR 5%).
[00383] Для обнаружения 6,4% вероятных клеток-дублетов, что соответствует предполагаемому показателю в 12,6% дублетов, включая как дублеты внутри кластера, так и между кластерами, использовали scrublet. Для удаления клеток низкого качества, обогащенных дублетами кластеров и введенных в них клеток HEK293T и NIH/3T3, затем применяли стратегию, которую ранее разработали для 2-миллионного атласа клеток органогенеза мыши (MOCA). Все описанные ниже анализы основаны на 4 062 980 профилях экспрессии генов одиночных клеток человека, полученных из 112 тканей плода, которые оставались после этой стадии фильтрации.
[00384] Определение 77 основных типов клеток
[00385] После отфильтровывания низкокачественных клеток и обогащенных дублетами кластеров 4 миллиона профилей экспрессии генов одиночных клеток подвергали визуализации UMAP и кластеризации Louvain с помощью Monocle 3 по принципу «на каждый орган». В целом на основе специфических для типа клеток маркеров из литературы изначально определяли и аннотировали 172 типа клеток. Сворачивание общих аннотаций по тканям привело к сокращению до 77 основных типов клеток, 54 из которых наблюдали только в одном органе (например, нейронах Пуркинье в мозжечке) и 23 во множестве органов (например, эндотелиальных клетках сосудов в каждом органе). Эти 77 основных типов клеток содержали медиану 4829 клеток и составляли диапазон от 1 258 818 клеток (возбуждающие нейроны в головном мозге) до только 68 клеток (LC26A4_PAEP-положительные клетки в надпочечнике). Множество индивидуумов внесли вклад в каждый тип клеток (медиана 9). Несмотря на различия в отношении вида, стадии развития и технологии получали практически все основные типы клеток, определенные в ходе предыдущих попыток по созданию атласов, направленных на одни и те же органы. Определяли медиану 12 основных типов клеток на орган в диапазоне от 5 (тимус) до 16 (глаз, сердце и желудок). Не наблюдалось корреляции между количеством профилированных клеток и количеством идентифицированных типов клеток (ρ = -0,10, p=0,74).
[00386] В среднем определяли по 11 маркерных генов на каждый основной тип клеток (мин. 0, макс. 294; определены как дифференциально экспрессируемые гены с по меньшей мере 5-кратной разницей между первым и вторым ранжированными типами клеток относительно экспрессии; FDR 5%). Обнаружено несколько типов клеток, у которых из-за сходных типов клеток в других органах на данном уровне отсутствовали маркерные гены (например, глия энтеральной нервной системы (ENS) и шванновские клетки). По этой причине также регистрируют наборы «маркерных генов в пределах ткани», определяемых с помощью той же процедуры, однако по принципу «орган-за-органом» (в среднем 147 маркеров на тип клеток; мин. 12, макс. 778).
[00387] Хотя канонические маркеры в целом наблюдались и действительно были критичными для данного процесса аннотации, по этой информации подавляющее большинство наблюдаемых маркеров является новым. Например, OLR1, SIGLEC10 и некодирующая РНК RP11-480C22.1 относятся к одним из самых устойчивых маркеров микроглии, наряду с более известными микроглиальными маркерами, такими как CCL7a, TLR7 и CCL3. С учетом того, что эти ткани проходят активное развитие, многие из 77 основных типов клеток включают состояния перехода от предшественников к одному или нескольким типам крайне дифференцированных клеток. Например, церебральные возбуждающие нейроны демонстрируют непрерывную траекторию от клеток-предшественников нейронов PAX6+ до дифференцирующихся нейронов NEUROD6+, до зрелых нейронов SLC17A7+. В печени печеночные клетки-предшественники (DLK1+, KRT8+, KRT18+) демонстрируют непрерывную траекторию до функциональных гепатобластов (SLC22A25+, ACSS2+, ASS1+). В отличие от органогенеза мыши, где созревание транскрипционной программы тесно связано со временем развития, в этих данных у человека траектории клеточного состояния непоследовательно коррелировались с оценочными гестационными возрастами. Наиболее простое объяснение заключается в заметно более динамичной экспрессии гена на более ранних стадиях развития, т. е. органогенез по сравнению с развитием плода. Однако также возможно, что данный анализ искажают неравномерное представление и неточности в оцениваемых гестационных возрастах.
[00388] Помимо данных ручных аннотаций типов клеток с использованием системы Garnett также создавали полуавтоматические классификаторы для каждого органа, а также глобальный классификатор. Классификаторы Garnett получали независимо от кластеризации с использованием маркерных генов, по отдельности скомпилированных из литературы. Классификации по Garnett были весьма согласованными с ручными классификациями, например, 88% клеток соответствовали классификации поджелудочной железы (с расширением кластера; 5% несоответствующих, 7% неклассифицированных). Используя модели Garnett, обучаемые на этом атласе человеческих клеток, также удалось точно классифицировать типы клеток по другим наборам данных одиночных клеток, включая данные по разным способам, а также по органам взрослых индивидуумов. Например, применяли классификатор Garnett для поджелудочной железы к данным RNA-seq одиночных клеток inDrop и обнаружили, что модель правильно аннотировала 82% клеток (с расширением кластера; 11% неправильных, 8% неклассифицированных). Эти модели Garnett выложены на веб-сайте авторов, и их можно широко использовать для автоматической классификации данных об одиночных клетках из различных органов.
[00389] Интеграция в ткани и исследование неожиданных типов клеток
[00390] Далее стремились интегрировать данные и сравнивать типы клеток по всем 15 органам. Для уменьшения эффектов грубых различий в количествах клеток в образце на орган и/или типе клеток случайным образом отбирали по 5000 клеток на тип клеток на орган (или в случаях, когда в данном органе представлено менее 5000 клеток заданного типа клеток, отбирали все клетки) и выполняли визуализацию UMAP на основании наиболее дифференциально экспрессируемых генов по типам клеток в каждом органе. Как и ожидалось, типы клеток, представленные в нескольких органах, обычно группировали вместе: например стромальные клетки, лимфатические эндотелиальные клетки и мезотелиальные клетки. Связанные с развитием типы клеток обычно также размещены совместно, например разнообразные клетки крови, нейроны ПНС, мезенхима.
[00391] Это общее исследование UMAP использовали, чтобы пролить свет на типы клеток, которые не поддавались четкой аннотации, или не ожидалось их присутствие в органе, в котором они изначально наблюдались. Во многих случаях совместная локализация с аннотированным типом клеток в общем исследовании UMAP пролила свет на их идентичность. Например, в легком и надпочечнике наблюдаются клетки, которые имеют выраженную корреляцию с гигантскими клетками трофобласта из плаценты (например, экспрессирующие высокие уровни плацентарного лактогена, хорионического гонадотропина и ароматазы), указывая на то, что они являются трофобластами, которые вошли в эмбриональное кровообращение (CSH1_CSH2-положительные клетки). Более неожиданно, что в плаценте и селезенке наблюдаются клетки, которые имеют выраженную корреляцию с гепатобластами (например, экспрессирующие высокие уровни сывороточного альбумина, альфа-фетопротеина и аполипопротеинов) (AFP_ALB_положительные клетки).
[00392] В сердце наблюдались три типа клеток, которые не были ожидаемыми на основании предыдущих попыток по созданию атласа. Первая из них (SATB2_LRRC7-положительные нейроны) имеет выраженную корреляцию с возбуждающими нейронами ЦНС и экспрессирует маркеры, в том числе SATB2, PTPRD и DAB1. Согласно данным это неожиданный результат. Хотя нельзя полностью исключить загрязнение из другой ткани, эти клетки наблюдаются в каждом образце сердца (n=9) в постоянной пропорции (диапазоне), и, кроме того, также не наблюдаются другие типы ЦНС-подобных клеток в сердце. Две других имеют выраженную корреляцию с кардиомиоцитами, но экспрессируют различные программы, отражающие специализированные роли. В частности, ELF3_AGBL2-положительные кардиомиоцитоподобные клетки специфически экспрессируют многие гены, ассоциированные с легочными альвеолярными клетками, секретирующими сурфактант, включая легочный секреторный белок 1 (SCGB3A2), легочный сурфактант-ассоциированный белок B (SFTPB) и легочный сурфактант-ассоциированный белок C (SFTPC), тогда как CLC_IL5RA-положительные кардиомиоцитоподобные клетки специфически экспрессируют связанные с иммунными клетками рецепторы, включая альфа-субъединицу рецептора интерлейкина 5 (IL5RA) и специфический для гемопоэтических клеток трансмембранный белок 4 (MS4A3).
[00393] Определение характеристик специфических для типа клеток генных регуляторных сетей и путей
[00394] Далее исследовали специфическую для типа клеток экспрессию генов, кодирующих поверхностные и секретируемые белки, критически важных для регуляции межклеточных взаимодействий или взаимодействий клеток с окружающей средой. Большинство поверхностных белков (4565 из 5480) и большинство секретируемых белков (2491 из 2933) дифференциально экспрессировали в основных 77 типах клеток (FDR 0,05). Например, микроглия специфически экспрессирует связывающий сиаловую кислоту иммуноглобулиноподобный лектин 8 (SIGLEC8) и рецептор эндоцитоза окисленных ЛПНП (OLR1), оба из которых связаны с болезнью Альцгеймера; клетки эндотелия специфически экспрессируют обходной направляющий рецептор 4 (ROBO4) и молекулу адгезии эндотелиальных клеток (ESAM), обе из которых участвуют в ангиогенезе и формировании сосудистого рисунка. Аналогичным образом разные нейроны были отмечены определенными переносчиками клеточной поверхности. Например, в мозжечке наблюдается специфическая экспрессия переносчика глициновых нейромедиаторов SLC6A5 в ингибиторных вставочных нейронах, переносчика возбуждающих аминокислот SLC1A6 в нейронах Пуркинье, KCNK9 калиевого канала в гранулированных нейронах и натриевого/калиевого/кальциевого антипортера SLC24A4 в SLC24A4_PEX5L-положительных ингибиторных нейронах. Существует множество аналогичных примеров специфической для типа клеток экспрессии секретируемых белков. Особенно интересным примером является неожиданный тип клеток в селезенке (STC2_TLX1-положительные клетки), который специфически экспрессирует гликопротеин STC2, а также TF TLX1 и NKX2-3, все связанные с мезенхимальными предшественниками или стволовыми клетками.
[00395] Было показано, что некодирующие РНК играют важную роль в нормальном развитии, а также при заболевании. По этим данным в 77 основных типах клеток дифференциально экспрессировались 3130 из 10 695 некодирующих РНК (FDR 0,05), например нкРНК с высокой специфичностью к микроглии (RP11-489O18.1, RP11-480C22.1, RP11-10H3.1) или эндотелиальным клеткам (AC011526.1, RP11-554D15.1, CTD-3179P9.1). Хотя биологическая значимость таких специфических для типа клеток нкРНК остается неясной, следует отметить, что их профилей экспрессии было достаточно для разделения 77 основных типов клеток на связанные с развитием группы.
[00396] Большинство факторов транскрипции (TF) также дифференциально экспрессировалось в 77 основных типах клеток (1715 из 1984; FDR 0,05). Многие из наиболее специфических TF для каждого типа клеток соответствовали ожиданиям, например, RBPJL для ацинарных клеток, OLG1 и OLG2 для олигодендроцитов и PAX7 для клеток-спутников. В других случаях специфические для типа клеток TF давали информацию о неожиданных типах клеток, например о типе стромальных клеток, наблюдаемом в поджелудочной железе и характеризующимся экспрессией лимфоидных хемокинов (CCL19_CCL21-положительные клетки), специфически экспрессирующих TF, связанных с иммунной активацией.
[00397] На основе данных экспрессии генов авторы стремились непосредственно прогнозировать взаимодействия «TF-целевой ген». Вкратце потенциальные взаимодействия определяли по ковариансе между экспрессией TF и экспрессией целевого гена во всем наборе данных. Эти взаимодействия дополнительно фильтровали с помощью анализа связывания ChIP-seq и обогащения мотива («Способы»). Осталось 56 272 потенциальные связи «TF-целевой ген», включающие 706 TF и 12 868 целевых генов. 220 из этих 706 наборов генов, связанных с TF, демонстрировали обогащение соответствующего TF (FDR 0,05) в обработанной вручную базе данных сетей TF (TRRUST) или TF-генных сетей Enrichr (например, наиболее обогащенный TF TRRUST для 330 генов, которые связывали с E2F1, - это E2F1, скорректированное p-значение=2,2e-14; наивысшим TF Enrichr для 1219 генов, которые связывают с FLI1, является FLI1, скорректированное p-значение=5,6e-122). После изменения порядка целевых генов, присвоенных этим 706 TF и повторения анализа ни один из связанных с TF наборов генов не был существенно обогащен по соответствующему TF при том же пороговом значении.
[00398] Характеристика развития линии дифференцировки крови в разных органах
[00399] Характер этого набора данных позволяет исследовать специфические для органов различия в экспрессии генов в пределах общедоступных типов клеток, например клеток крови, эндотелиальных и эпителиальных клеток. В качестве первого такого анализа повторно собирали в кластеры 103 766 клеток, полученных из всех органов, которые соответствовали типам гемопоэтических клеток. Затем выполняли кластеризацию Louvain и на основе опубликованных генных маркеров дополнительно аннотировали мелкозернистые типы иммунных клеток, определяя в некоторых случаях очень редкие типы клеток. Например, миелоидные клетки разделяются на микроглию, макрофаги и различные подтипы дендритных клеток (CD1C+, S100A9+, CLEC9A+ и плазмоцитоидные дендритные клетки (pDC)). Микроглиальный кластер в основном происходит из головного мозга и мозжечка и хорошо отделен от макрофагов, что соответствует их различному происхождению по развитию. Лимфоидные клетки кластеризировались в несколько групп, включая В-клетки, NK-клетки, клетки ILC 3 и Т-клетки (последние включают направление тимопоэза). Кроме того, также получали очень редкие типы клеток, такие как плазматические клетки (139 клеток, которые составляют 0,1% от всех клеток крови или 0,003% от полного набора данных; главным образом в плаценте) и TRAF1+АПК (189 клеток, которые составляют 0,2% от всех клеток крови или 0,005% от полного набора данных; главным образом в тимусе и сердце).
[00400] Хотя маркеры экспрессии генов для различных типов иммунных клеток были подробно изучены, они могут быть ограничены их определением посредством ограниченного набора органов или типов клеток. Действительно, в данном случае обнаруживали, что многие общепринятые маркеры иммунных клеток экспрессировались во множестве типов клеток. Например, общепринятые маркеры Т-клеток также экспрессировались в макрофагах и дендритных клетках (CD4) или NK-клетках (CD8A), что согласуется с другими исследованиями. В 14 типах клеток крови вычисляли маркеры, специфические для панорганного типа клеток всех органов. Например, Т-клетки специфически экспрессировали CD8B и CD5, как и ожидалось, но также и TENM1. Клетки ILC 3, аннотация которых основана на экспрессии RORC и KIT, были более специфически помечены SORCS1 и JMY. Эти и другие панорганно-определяемые маркеры в будущих исследованиях можно использовать для маркировки и очистки типов клеток эмбриональной крови человека.
[00401] Как и ожидалось, различные органы демонстрировали очень разные пропорции клеток крови. Например, печень содержала самую высокую долю эритробластов, что согласуется с ее ролью в качестве основного места эритропоэза плода, тогда как Т-клетки были обогащены в тимусе, а В-клетки - в селезенке. Клетки крови, выделенные из мозжечка и головного мозга, представляли собой микроглию. Собирательный анализ также позволил определить популяции редких клеток в конкретных органах. Например, редкие звездчатые клетки печени (HSC) определяли в печени, селезенке и тимусе, но также и в сердце, легком, надпочечнике и кишечнике.
[00402] Принимая во внимание эритропоэз, наблюдали непрерывное направление от HSC до клеток промежуточного типа, смещенных относительно эритроидных базофильных предшественников мегакариоцитов (EBMP), которые затем разделяли по эритроидным, базофильным и мегакариоцитарным направлениям, в соответствии с недавним исследованием в печени зародышей мышей. Это соответствие присутствовало несмотря на различия в видах (мыши по сравнению с человеком), способах (sci-RNA-seq3 по сравнению с 10х) и органах (из пробирки по сравнению с эмбриональными). При неконтролируемой кластеризации и применения терминологии из этого исследования дополнительно разделяли непрерывный спектр эритроидных состояний на три стадии: ранние эритроидные клетки-предшественники (EEP; помечены SLC16A9 и FAM178B), коммитированные эритроидные клетки-предшественники (CEP; помечены KIF18B и KIF15), а также клетки в состоянии конечной эритроидной дифференцировки (ETD; помечены TMCC2 и HBB). Кроме того, легко определяли ранние и поздние стадии мегакариоцитарных клеток. Соответствующая динамика доступности хроматина всего генома в эритроидной линии дополнительно рассмотрена в сопутствующей рукописи.
[00403] Как и ожидалось, принимая во внимание их установленную роль в эритропоэзе плода, значительная часть иммунных клеток в печени и селезенке соответствует EEP, CEP и предшественникам мегакариоцитов. Неожиданно было обнаружено, что в каждом исследованном образце также наблюдали EEP, CEP и клетки-предшественники мегакариоцитов в надпочечнике. Поскольку не наблюдается типов клеток, которые чаще встречаются в печени и селезенке, банальное загрязнение во время извлечения в надпочечнике является маловероятным объяснением. Хотя требуется подтверждение с помощью ортогонального способа, результат указывает на возможность того, что надпочечник является дополнительным местом эритропоэза плода.
[00404] Макрофаги распространены еще более широко. Далее сортировали все макрофаги вместе с микроглией головного мозга и подвергали их независимой визуализации посредством UMAP и кластеризации Louvain. Микроглия была разделена на три подкластера, один из которых, помеченный IL1B и TNFRSF10D, вероятно, представляет собой активированную микроглию, участвующую в воспалительных ответах. Другие микроглиальные кластеры отмечали по экспрессии TMEM119 и CX3CR1 (чаще всего в головном мозге) или PTPRC и CDC14B (чаще всего в мозжечке).
[00405] Макрофаги за пределами головного мозга объединяли в три основные группы: 1) антигенпредставляющие макрофаги, обнаруживаемые главным образом в органах ЖК-тракта (кишечник и желудок) и отмеченные высокой экспрессией генов антигенпредставляющих молекул (HLA-DR1, HLA-DQA1) и генов активации воспаления (AHR); 2) периваскулярные макрофаги, обнаруженные в большинстве органов со специфической экспрессией маркеров, таких как F13A1 и COLEC12, а также новых маркеров, таких как RNASE1 и LYVE1. 3) фагоцитарные макрофаги, обогащенные в печени, селезенке и надпочечнике, со специфической экспрессией таких маркеров, как CD5L, TIMD4 и VCAM1. Фагоцитарные макрофаги имеют решающее значение для эритрофагоцитоза; их наблюдение в надпочечнике согласуется с вышеупомянутой потенциальной ролью в качестве места эритропоэза плода.
[00406] Определение характеристик эндотелиальных и эпителиальных клеток в органах
[00407] В качестве второго анализа типа одиночных клеток во множестве органов повторно собирали в кластеры клетки, полученные из всех органов, которые соответствовали сосудистому эндотелию, лимфатическому эндотелию или эндокарду. Эти три группы легко отделяли друг от друга, а сосудистые эндотелиальные клетки дополнительно образовывали кластеры, по меньшей мере в некоторой степени, в соответствии с органом. Эти органоспецифические различия легче обнаружить, чем различия между артериями, капиллярами и венами, что согласуется с предыдущими атласами клеток взрослой мыши.
[00408] В анализе дифференциальной экспрессии генов определяли 700 маркеров, которые специфически экспрессируются в подгруппе эндотелиальных клеток (FDR 0,05, более чем 2-кратная разница в экспрессии между первым и вторым ранжированными кластерами). Приблизительно треть из них (236 из 700) кодировала мембранные белки, многие из которых, по-видимому, соответствуют потенциальным специализированным функциям. Например, клетки эндотелия почек специфически экспрессировали кислоточувствительный ионный канал 2 (ASIC2), - механосенсор, участвующий в миогенной констрикции и регуляции кровотока в почке. Клетки легочного эндотелия специфически экспрессировали пептидный рецептор 1 семейства релаксинов (RXFP1), который участвует в релаксации сосудов в легком, опосредованной эндогенным оксидом азота, специфически экспрессировали симпортер 1 натрий-зависимого транспортера лизофосфатидилхолина (MFSD2A), который неотъемлемо участвует в создании и функционировании гематоэнцефалического барьера. Потенциальная регуляторная основа в отношении дифференциальной экспрессии генов в подгруппах эндотелия обсуждается в сопутствующем документе.
[00409] В качестве третьего анализа широко распространенного типа клеток повторно собирали в кластеры эпителиальные клетки, полученные из всех органов, и подвергали их визуализации посредством UMAP. Хотя некоторые типы эпителиальных клеток имели выраженную органоспецифичность, например ацинарные (поджелудочная железа) и альвеолярные (легкое) клетки, эпителиальные клетки с аналогичными функциями обычно объединялись в совместный кластер. Например, программы экспрессии клеток плоского эпителия (легкого, желудка) объединены в совместный кластер с эпителиальными клетками роговицы и конъюнктивы (глаз), в то время как PDE1C_ACSM3-положительные клетки (желудок) объединены в совместный кластер с эпителиальными клетками кишечника (кишечник).
[00410] В эпителиальных клетках определено два нейроэндокринных скопления клеток. Более простой из них соответствовал хромаффиновым клеткам надпочечника и был маркирован специфической экспрессией HMX1 (NKX-5-3), - TF, вовлеченного в систематическую диверсификацию нейронов. Другой кластер содержал нейроэндокринные клетки из множества органов (желудка, кишечника, поджелудочной железы, легкого) и был маркирован специфической экспрессией NKX2-2, - TF, играющего ключевую роль в островках Лангерганса и энтероэндокринной дифференцировке. Выполняли дополнительный анализ последней группы, с определением пяти подгрупп: 1) бета-клетки островков Лангерганса, маркированные экспрессией инсулина; 2) альфа-/гамма-клетки островков Лангерганса, маркированные экспрессией панкреатического полипептида и глюкагона; 3) дельта-клетки островков Лангерганса, маркированные экспрессией соматостатина; 4) нейроэндокринные клетки легких (PNEC), маркированные экспрессией ASCL1, - TF, играющего ключевую роль в определении этой линии дифференцировки в легких; и 5) энтероэндокринные клетки. Энтероэндокринные клетки дополнительно содержали несколько подгрупп, включающих NEUROG-экспрессирующие клетки-предшественники эпсилон островков Лангерганса, TNF1-экспрессирующие энтерохромаффинные клетки как в желудке, так и в кишечнике, гастрин- или холецистокинин-экспрессирующие G/L/K/I-клетки. Наконец, в желудке и кишечнике наблюдались грелин-экспрессирующие энтероэндокринные клетки-предшественники, а также грелин-экспрессирующие эндокринные клетки в развивающемся легком. Поскольку разнообразные функции нейроэндокринных клеток тесно связаны с секретируемыми ими белками, определяли 1086 генов, кодирующих секретируемые белки, дифференциально экспрессирующихся среди нейроэндокринных клеток (FDR 0,05). Например, клетки PNEC демонстрировали специфическую экспрессию фактора «трилистника» 3, участвующего в защите слизистой оболочки и дифференцировке клеток легких, гастрин-высвобождающий пептид, который стимулирует высвобождение гастрина из G-клеток в желудке, и SCGB3A2, - сурфактант, ассоциированный с развитием легких.
[00411] В качестве иллюстративного примера того, как эти данные можно использовать для изучения траекторий клеток, дополнительно исследовали путь диверсификации эпителиальных клеток, ведущей к клеткам почечных канальцев. При объединении и повторном сборе в кластеры метанефрических клеток зачатка мочеточника определяли как предшественников, так и терминальные типы почечных эпителиальных клеток, причем пути дифференцировки в высокой степени соответствуют последнему исследованию почки эмбриона человека. По результатам анализа дифференциальной экспрессии генов дополнительно характеризовали TF, потенциально регулирующие их спецификацию. Например, клетки-предшественники нефронов в метанефрической траектории экспрессировали высокие уровни мезенхимального и гомеобоксного meis генов (MEOX1, MEIS1, MEIS2), тогда как подоциты специфически экспрессировали MAFB и TCF21/POD1. В качестве другого примера, HNF4A специфически экспрессируется в клетках проксимальных канальцев; мутация этого гена вызывает ренотубулярный синдром Фанкони, - заболевание, которое специфически поражает проксимальный каналец, и недавно продемонстрирована его необходимость для формирования проксимального канальца у мышей.
[00412] Сравнение атласов развития человека и мыши
[00413] Для изучения взаимосвязи развития между типами клеток затем сравнивали эти данные с последними атласами органогенеза клеток мыши (MOCA), в которых профилированы 2 миллиона клеток из всего эмбриона, охватывающие E9,5-E13,5 (окно раннего развития млекопитающего).
[00414] В качестве первого подхода сравнивали 77 основных типов человеческих клеток, определенных в настоящем документе, с траекториями развития, определенными по MOCA, посредством описанного ранее способа перекрестного сопоставления с клетками. Вкратце для выбора эквивалентных наиболее совпадающих пар типов клеток из двух наборов данных в этом способе используют регрессию неотрицательных наименьших квадратов (NNLS). Большинство типов человеческих клеток демонстрировали значительное совпадение с одной основной траекторией и субтраекторией мыши. Они в целом соответствовали ожиданиям и выступают в качестве одной формы валидации для обоих наборов аннотаций. Несколько расхождений способствовали важным исправлениям в аннотации MOCA. Многие из траекторий типов клеток человека и мыши, в которых отсутствуют значительные совпадения (комбинированный коэффициент регрессии NNLS <0,6), соответствовали тканям, исключенным из другого набора данных (например, мышиная плацента; кожа и гонады человека). Другие неоднозначности, вероятно, следуют из промежутка между исследованными периодами развития (например, типы клеток надпочечников), малой распространенности (например, биполярные клетки) и/или сложных взаимоотношений между типами клеток (например, типы клеток плода, которые происходят из множества эмбриональных траекторий).
[00415] В качестве второго подхода стремились непосредственно объединить в кластер человеческие и мышиные клетки. Вкратце брали образцы 100 000 эмбриональных клеток мыши из MOCA (случайным образом) и 65 000 клеток плода человека (до 1000 клеток каждого из 77 типов клеток) и подвергали их воздействию недавно описанной стратегии Seurat для интеграции перекрестно-видовых наборов данных scRNA-seq. Распределение клеток мыши в полученной визуализации на основе UMAP имело поразительное сходство с общим анализом MOCA. Более того, вместо распределения в соответствии с пространственным расположением органов, хотя и с некоторыми неожиданными результатами, клетки в значительной степени были распределены интуитивным образом как в отношении развития, так и во временном отношении. Например, показано, что: все фетальные эндотелиальные, гемопоэтические, печеночные, эпителиальные и мезенхимальные клетки человека картировались к соответствующим эмбриональным траекториям мыши. В то время как человеческие фетальные церебральные и мозжечковые нейроны перекрывались с траекторией эмбриональной нервной трубки мыши, производные плоского нервного гребня человека, такие как нейроны ENS, висцеральные нейроны, симпатобласты и хромаффинные клетки, группировались в кластеры отдельно от соответствующих эмбриональных траекторий мыши, возможно, из-за чрезмерных различий между видами или стадиями развития. Как и ожидалось, глия ENS человека, а также шванновские клетки, перекрывались с субтраекториями глии ПНС эмбриона мыши. Фетальные астроциты человека объединяли в кластеры с траекторией эпителия нейронов эмбриона мыши (астроциты мыши не развиваются до E18,5). Фетальные олигодендроциты человека перекрываются с редкой субтраекторией (Pdgfra+ глия) эмбриона мыши, которая в ретроспективе соответствует клеткам-предшественникам олигодендроцитов (OPC; Olig1+, Olig2+, Brinp3+) и ставит под сомнение предыдущую аннотацию другой субтраектории Oligo1+ в качестве предшественников олигодендроцитов.
[00416] Для визуализации более подробных взаимоотношений между человеческими фетальными и мышиными эмбриональными клетками применяли аналогичную стратегию интегративного анализа к клеткам человека и мыши, извлеченным из гематопоэтических, эндотелиальных и эпителиальных траекторий. Данные из этого атласа эмбриональных стволовых клеток человека легко раскладывают данные по «целому эмбриону» мыши в мелкоструктрурные функциональные или пространственные группы. Например, субпопуляции «лейкоцитарной» траектории мыши соответствуют конкретным типам клеток крови человека, таким как HSC, микроглия, макрофаги (печень и селезенка), макрофаги (другие органы) и DC. Эти подгруппы дополнительно подтверждены экспрессией связанных с ними маркеров клеток крови. Аналогично показано, что родственные субпопуляции эндотелиальных и эпителиальных клеток мыши/человека картируются друг с другом. Данный подход можно применять для получения программ экспрессии генов клеток-предшественников конкретных линий дифференцировки во временных точках развития, доступ и анатомическое разделение которых затруднены. Например, в мышиных клетках, которые ранее помечали как траекторию эпителия передней кишки, теперь можно определить вероятные факторы, влияющие на желудок по сравнению с поджелудочной железой.
[00417] Обсуждение
[00418] Успешное развитие функционального плода человека представляет собой удивительный процесс, характеризующийся процессом пролиферации и дифференцировки клеток на трех основных стадиях развития.
[00419] После короткого (две недели после оплодотворения) зародышевого периода с простым размножением клеток и имплантацией в матке стадия эмбриогенеза переходит в гаструляцию, нейруляцию и органогенез, для которых характерна интенсивная дифференцировка клеток и образование предшественников внутренних органов. К концу десятой недели гестационного возраста эмбрион приобретает свою основную форму, называемую плодом. В течение следующих двадцати недель продолжают расти и созревать различные органы с разнообразными терминальными дифференцированными типами клеток, сформированными из предшественников.
[00420] Как зародышевая стадия, так и стадия эмбриогенеза прошли интенсивное профилирование у человека или в смоделированных системах (т. е. у мышей) с использованием общих программ раннего развития. Поздняя стадия развития (стадия плода) демонстрирует различные программы и длительность развития между Homo sapiens и другими видами. Кроме того, трудности получения общего представления динамики клеток на этой стадии связаны с большей сложностью организма и ограничениями методики. Хотя недавно было опубликовано несколько исследований одиночных клеток в рамках развития плода, они в основном ограничены определенным органом или клеточной линией дифференцирования и не позволили получить общий обзор развития всего организма.
[00421] Материалы и способы
[00422] Клеточная культура млекопитающих и извлечение ядер
[00423] Все клетки млекопитающих культивировали при 37 °C в атмосфере с содержанием 5% CO2 и выдерживали в модифицированной по способу Дульбекко среде Игла (DMEM) с высоким содержанием глюкозы (Gibco, кат. № 11965), обогащенной 10% FBS и 1X пенициллином-стрептомицином (Gibco, кат. № 15140122; 100 Ед/мл пенициллина, 100 мкг/мл стрептомицина). Клетки трипсинизировали 0,25% раствором трипсин-ЭДТА (Gibco, кат. № 25200-056) и разделяли в соотношении 1 : 10 три раза в неделю.
[00424] Все клеточные линии трипсинизировали, центрифугировали при 300 x g в течение 5 мин (4 °C) и однократно промывали в 1X ледяном PBS. 5 млн клеток объединяли и лизировали с использованием 1 мл ледяного буфера для лизиса клеток (10 мМ Трис-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2 и 0,1% IGEPAL CA-630 из, модифицированного так, чтобы также включать 1% ингибитор РНазы SUPERase In и 1% BSA). Отфильтрованные ядра затем переносили в новую пробирку емкостью 15 мл (Falcon) и осаждали центрифугированием при 500 x g в течение 5 мин при 4 °C и однократно промывали 1 мл ледяного буфера для лизиса клеток. Ядра фиксировали в 4 мл ледяного раствора 4% параформальдегида (EMS) в течение 15 мин на льду. После фиксации ядра дважды промывали в 1 мл буферного раствора для отмывки ядер (буферный раствор для лизирования клеток без IGEPAAL) и ресуспендировали в 500 мкл буферного раствора для отмывки ядер. Образцы разделяли в 5 пробирок по 100 мкл в каждую пробирку и мгновенно замораживали в жидком азоте.
[00425] Подготовка тканей плода человека и извлечение ядер
[00426] Для уменьшения групповых эффектов ткани плода человека обрабатывали вместе. Каждый орган измельчали в порошок ткани молотком (на сухом льду) и перед отбором образцов перемешивали. 0,1-1 г порошков сначала инкубировали с 1 мл ледяного буфера для лизиса клеток (10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2 и 0,1% IGEPAL CA-630 из53, модифицированный так, чтобы также включать 1% SUPERase In и 1% BSA), а затем переносили в верхнюю часть клеточного сита с размером пор 40 мкм (Falcon). Ткани гомогенизировали резиновым наконечником поршня шприца (5 мл, BD) в 4 мл буферного раствора для лизирования клеток. Отфильтрованные ядра затем переносили в новую пробирку емкостью 15 мл (Falcon), и осаждали центрифугированием при 500 x g в течение 5 мин, и однократно промывали 1 мл буфера для лизирования клеток. Ядра фиксировали в 5 мл ледяного раствора 4% параформальдегида (EMS) в течение 15 мин на льду. После фиксации ядра дважды промывали в 1 мл буферного раствора для отмывки ядер (буферный раствор для лизирования клеток без IGEPAAL) и ресуспендировали в 500 мкл буферного раствора для отмывки ядер. Образцы разделяли в две пробирки по 250 мкл в каждую пробирку и мгновенно замораживали в жидком азоте. Для извлечения человеческих клеток в некоторых органах (почках, поджелудочной железе, кишечнике и желудке) и фиксации параформальдегидом.
[00427] Приготовление и секвенирование библиотек sci-RNA-seq3
[00428] Фиксированные параформальдегидом ядра обрабатывали аналогично опубликованному протоколу sci-RNA-seq3 с небольшими модификациями. Вкратце с помощью 0,2% TritonX-100 (в буфере для отмывки ядер) в течение 3 мин на льду увеличивали проницаемость мембраны размороженных ядер и подвергали краткой ультразвуковой обработке (Diagenode, 12 с в режиме малой мощности) для уменьшения слипания ядер. Ядра затем однократно промывали буфером для отмывки ядер и фильтровали через 1 мл клеточное сито Flowmi (Flowmi). Отфильтрованные ядра центрифугировали при 500 x g в течение 5 мин и ресуспендировали в буфере для отмывки ядер. Ядра из каждого образца затем распределяли в несколько отдельных лунок в четырех 96-луночных планшетах. Связи между идентификатором лунки и эмбрионом мыши регистрировали для последующей обработки данных. Для каждой лунки 80 000 ядер (16 мкл) смешивали с 8 мкл 25 мкМ фиксированного праймера олиго-dT (5′-/5Phos/CAGAGCNNNNNNNN(штрихкод 10 п. н.)TTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTT-3′ (SEQ ID NO:1), где N представляет собой любое основание; IDT) и 2 мкл 10 мМ смеси дезоксирибонуклеотидтрифосфатов (дНТФ)(Thermo), денатурировали при 55 °C в течение 5 мин и немедленно помещали на лед. Затем в каждую лунку добавляли 14 мкл реакционной смеси для синтеза первой цепи, содержащей 8 мкл буфера для первой цепи 5X Superscript IV (Invitrogen), 2 мкл 100 мМ ДТТ (Invitrogen), 2 мкл обратной транскриптазы SuperScript IV (200 Ед/мкл, Invitrogen), 2 мкл рекомбинантного ингибитора рибонуклеазы RNaseOUT (Invitrogen). Обратную транскрипцию проводили путем инкубации планшетов при градиенте температур (4 °C - 2 минуты, 10 °C - 2 минуты, 20 °C - 2 минуты, 30 °C - 2 минуты, 40 °C - 2 минуты, 50 °C - 2 минуты и 55 °C - 10 минут).
[00429] После реакции обратной транскрипции в каждую лунку добавляли 60 мкл буфера для разведения ядер (10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2 и 1% BSA). Ядра из всех лунок объединяли и центрифугировали при 500 x g в течение 10 мин. Затем ядра ресуспендировали в буфере для отмывки ядер и перераспределяли в еще четыре 96-луночных планшета, причем каждая лунка содержала 20 мкл лигазного буфера Quick (NEB), 2 мкл ДНК-лигазы Quick (NEB), 10 мкл ядер в буфере для отмывки ядер, 8 мкл штрихкодированного адаптера лигирования (100 мкМ, 5’-GCTCTG(9 п. н. или штрихкод A 10 п. н.)/дидезоксиU/ACGACGCTCTTCCGATCT(обратный комплемент штрихкода A)-3’(SEQ ID NO:2)). Реакцию лигирования проводили при 25 °C в течение 10 мин. После реакции лигирования в каждую лунку добавляли 60 мкл буфера для разведения ядер (10 мМ Tris-HCl, pH 7,4, 10 мМ NaCl, 3 мМ MgCl2 и 1% BSA). Ядра из всех лунок объединяли и центрифугировали при 600 x g в течение 10 мин.
[00430] Ядра однократно промывали буфером для отмывки ядер и однократно фильтровали 1 мл клеточного сита Flowmi (Flowmi), подсчитывали и перераспределяли в восемь 96-луночных планшетов, причем каждая лунка содержала 2500 ядер в 5 мкл буфера для отмывки ядер и 3 мкл буфера для элюирования (Qiagen). Затем в каждую лунку добавляли 1,33 мкл буфера для синтеза комплементарной цепи мРНК (NEB) и 0,66 мкл фермента для синтеза комплементарной цепи мРНК (NEB) и проводили синтез комплементарной цепи при 16 °C в течение 180 мин.
[00431] Для тагментации в каждую лунку примешивали 11 мкл буфера для тагментации ДНК (TD) Nextera (Illumina) и 1 мкл фермента 1 для тагментации ДНК (TDE1) только i7 (62,5 нМ, Illumina, разведенный в буфере Nextera TD (Illumina)), а затем инкубировали при 55 °C в течение 5 мин для выполнения тагментации. Затем реакцию останавливали посредством добавления в каждую лунку 24 мкл ДНК-связывающего буфера (Zymo) и инкубации при комнатной температуре в течение 5 мин. Затем каждую лунку очищали, используя гранулы AMPure XP 1,5х (Beckman Coulter). На стадии элюирования к каждой лунке добавляли 8 мкл воды без нуклеазной активности, 1 мкл буфера специфического для урацила вырезающего реагента (USER) 10X (NEB), 1 мкл фермента USER (NEB) и инкубировали при 37 °C в течение 15 мин. В каждую лунку добавляли еще 6,5 мкл буфера для элюирования. Гранулы AMPure XP удаляли при помощи магнитной стойки и продукт после элюирования (16 мкл) переносили в новый 96-луночный планшет.
[00432] Для ПЦР-амплификации содержимое каждой лунки (16 мкл продукта) смешивали с 2 мкл 10 мкМ индексированного праймера P5 (5′-AATGATACGGCGACCACCGAGATCTACAC(i5)ACACTCTTTCCCTACACGACGCTCTTCCGATCT-3′ (SEQ ID NO:3); IDT), 2 мкл 10 мкМ праймера P7 (5′-CAAGCAGAAGACGGCATACGAGAT(i7)GTCTCGTGGGCTCGG-3′ (SEQ ID NO:4), IDT) и 20 мкл основной смеси ПЦР 2X NEBNext High-Fidelity (NEB). Амплификацию проводили с использованием следующей программы: 72 °C в течение 5 мин, 98 °C в течение 30 с, 12-16 циклов (98 °C в течение 10 с, 66 °C в течение 30 с, 72 °C в течение 1 мин) и завершающего - 72 °C в течение 5 мин.
[00433] После ПЦР образцы объединяли и очищали с использованием 0,8 объема гранул AMPure XP. Концентрации библиотек определяли с помощью Qubit (Invitrogen) и библиотеки визуализировали посредством электрофореза на 6% полиакриламидном геле для электрофореза с буферной смесью Tris, борной кислоты и ЭДТА (TBE-PAGE). Все библиотеки секвенировали на одной платформе NovaSeq (Illumina) (чтение 1: 34 цикла, чтение 2: 52 цикла, индекс 1: 10 циклов, индекс 2: 10 циклов).
[00434] Для получения фиксированных параформальдегидом клеток их обрабатывали аналогично фиксированным ядрам с небольшими модификациями: замороженные фиксированные клетки размораживали на водяной бане при 37 °C, центрифугировали при 500 x g в течение 5 мин и инкубировали с 500 мкл PBSR (1x PBS, pH 7,4, 1% BSA, 1% SuperRnaseIn, 1% 10 мМ ДТТ), включающем 0,2% Triton X-100, в течение 3 мин на льду. Клетки осаждали и ресуспендировали в 500 мкл воды без нуклеазной активности, включающей 1% SuperRnaseIn. В клетки добавляли 3 мл 0,1 н HCl для 5 мин инкубации на льду(7). В клетки добавляли 3,5 мл Tris-HCl (pH=8,0) и 35 мкл 10% Triton X-100 для нейтрализации HCl. Клетки осаждали и промывали 1 мл PBSR. Клетки осаждали и ресуспендировали в 100 мкл PBSI (1x PBS, pH 7,4, 1% BSA, 1% SuperRnaseIn). Следующие стадии были аналогичны вышеописанному протоколу sci-RNA-seq3 (с ядрами, фиксированными параформальдегидом), с небольшими модификациями. (1) Для обратной транскрипции распределяли по 20 000 фиксированных клеток (вместо 80 000 ядер) на лунку. (2) На следующих стадиях весь буфер для отмывки ядер заменяли PBSI. (3) Весь буфер для разведения ядер заменяли PBS+1% BSA.
[00435] Обработка чтений секвенирования
[00436] Выравнивание чтений и создание матрицы для подсчета генов для RNA-seq одиночных клеток выполняли с использованием процесса подготовки, разработанного для sci-RNA-seq3 с небольшими модификациями: распознавания оснований были преобразованы в формат fastq с использованием BCLlumina bcl2fastq/версии 2.16 и демультиплексированы на основании штрихкодов i5 и i7 ПЦР с использованием пакета для демультиплексирования с максимальной правдоподобностью deML и настройками по умолчанию. Обработка последовательности ближе к 3'-концу и создание цифровой экспрессионной матрицы одиночных клеток были аналогичны sci-RNA-seq, за исключением того, что индекс обратной транскрипции (ОТ) комбинировали с индексом адаптера шпильки, и, таким образом, картированные чтения разделяли на составляющие клеточные индексы путем демультиплексирования чтений с использованием как индекса ОТ, так и индекса лигирования (расстояние редактирования (ED) < 2, включая вставки и делеции). Вкратце демультиплексированные чтения фильтровали на основании индекса ОТ и индекса лигирования (ED < 2, включая вставки и делеции) и обрезали адаптер с использованием параметров TREM_galore/версии 0.4.1 при настройках по умолчанию. Обрезанные чтения картировали с эталонным человеческим геномом (hg19) для ядер плода человека или с химерным эталонным геномом hg19 человека и mm10 мыши для смешанных ядер HEK293T и NIH/3T3 с применением STAR/версии 2.5.2b с настройками по умолчанию и аннотациями генов (GENCODE V19 для человека; GENCODE VM11 для мыши). Уникальные чтения картирования извлекали и удаляли дубликаты с применением последовательности уникального молекулярного идентификатора (UMI) (ED < 2, включая вставки и делеции), индекса обратной транскрипции (RT), индекса адаптера лигирования шпильки и конечной координаты чтения 2 (т. е. чтения с последовательностью UMI с расстоянием редактирования менее 2, индексом ОТ, индексом адаптера лигирования и сайтом тагментации считали дубликатами). Наконец, картированные чтения разделяли на составляющие клеточные индексы путем дополнительного демультиплексирования чтений с использованием индекса ОТ и шпильки лигирования (ED < 2, включая вставки и делеции). Для эксперимента со смешанными видами рассчитывали процент уникальных чтений картирования для геномов каждого вида. Клетки, у которых более 85% UMI были отнесены к одному виду, рассматривались как видоспецифические клетки, а остальные клетки были классифицированы как смешанные клетки или «коллизии». Для создания цифровых матриц экспрессии рассчитывали число специфических для цепи UMI для каждого клеточного картирования с экзонной и интронной областями каждого гена с использованием пакета HTseq56 python/версии 2.7.13. Для чтений со множеством картирований чтения относили к ближайшему гену, за исключением случаев, когда другой пересекающийся ген попадал в отрезок 100 п. о. до ближайшего конца гена, в этом случае чтение отбрасывали. Для большинства анализов включали как интронные, так и экзогенные UMI с ожидаемой цепью в матрицах экспрессии одиночных клеток для каждого гена.
[00437] После создания матрицы подсчета генов одиночных клеток, клетки с менее чем 250 UMI отфильтровывали. На основании штрихкода ОТ каждую клетку присваивали ее исходному образцу плода человека. Чтения, картирующиеся к каждому отдельному плоду, агрегировали с образованием «массового RNA-seq». Для разделения плода по полу подсчитывали чтения, картирующиеся к некодирующей РНК, специфической для особей женского пола (TSIX и XIST), или генам Y-хромосомы (за исключением генов TBL1Y, RP11-424G14.1, NLGN4Y, AC010084.1, CD24P4, PCDH11Y и TTTY14, которые обнаружены как у мужских, так и у женских особей). Плоды легко разделяли на особей женского пола (больше чтений, картирующихся к TSIX и XIST, чем к генам Y-хромосомы) и особей мужского пола (больше чтений, картирующихся к генам Y-хромосомы, чем к TSIX и XIST).
[00438] Анализ кластеризации образцов целого плода человека выполняли с помощью Monocle 3. Вкратце агрегированную матрицу экспрессии генов создавали, как описано выше для органов плода человека от каждого индивидуума. Отбирали образцы с общим содержанием UMI более 5000. Размерность данных уменьшали с помощью PCA (10 компонент), сначала на 500 наиболее высокодисперсных генах, а затем с помощью UMAP (max_components=2, n_neighbors=10, min_dist=0,5, metric = «cosine»).
[00439] Фильтрация клеток, кластеризация и определение маркерных генов
[00440] Для обнаружения потенциальных дублетных клеток сначала разделяли набор данных на подгруппы для каждого органа и индивидуума, а затем для вычисления показателя дублетов к каждой подгруппе применяли процесс scrublet/версии 0.1 с параметрами (min_count=3, min_cells=3, vscore_percentile=85, n_pc=30, expected_doublet_rate=0,06, sim_doublet_ratio=2, n_neighbors=30, scaling_method = 'log'). Клетки с показателем дублетов выше 0,2 аннотированы как обнаруженные дублеты. В полном наборе данных обнаруживали 6,4% потенциальных дублетных клеток, что соответствует общей оценочной частоте дублетов 12,6% (включая как внутрикластерные, так и межкластерные дублеты).
[00441] Для обнаружения субкластеров, полученных из дублетов, для клеток из каждого органа использовали итерационную стратегию кластеризации, как показано выше. Вкратце перед кластеризацией и уменьшением размерности исключали количество генов, картирующихся к половым хромосомам. Стадии предварительной обработки были аналогичны подходу, использованному по ссылке. Вкратце гены без подсчета отфильтровывали и каждую клетку нормализовали по общему количеству UMI на клетку. Выбирали 1000 генов с наиболее высокой дисперсией, а цифровую матрицу экспрессии генов повторно нормализовали после фильтрации генов. Данные логарифмически преобразовывали после добавления псевдоколичества и масштабировали в соответствии с единичной дисперсией и нулевым средним значением. Размерность данных уменьшали сначала с помощью PCA (30 компонент), а затем с помощью UMAP, с последующей кластеризацией Louvain, выполненной на 30 основных компонентах с параметрами по умолчанию. Для кластеризации Louvain сначала аппроксимировали первые 30 PC для вычисления графа окрестностей наблюдений с числом местных окрестностей 50 с помощью функции scanpy.api.pp.neighbors в scanpy/версии 1.0. Затем кластеризировали клетки в подгруппы с помощью алгоритма Louvain, реализованного в качестве функции scanpy.api.tl.louvain. Для визуализации UMAP подставляли матрицу PCA непосредственно в функцию scanpy.api.tl.umap с min_distance 0,1. Для определения субкластеров выбирали клетки в каждом основном типе клеток и применяли PCA, UMAP, кластеризацию Louvain аналогично анализу основного кластера. Субкластеры с обнаруженным соотношением дублетов (по данным Scrublet) более 15% аннотировали как субкластеры, полученные из дублетов.
[00442] Для визуализации данных отфильтровывали клетки, помеченные как дублеты (с помощью Scrublet), или происходящие из субкластеров, полученных из дублетов. Для каждой клетки сохраняют только кодирующие белок гены, гены длинной интергенной некодирующей РНК (lincRNA) и псевдогены. Дополнительно отфильтровывали гены, экспрессированные в менее 10 клетках, и клетки, экспрессирующие менее 100 генов. Дальнейшее уменьшение размерности и кластерный анализ проводили с помощью Monocle 3. Размерность данных уменьшали с помощью PCA (50 компонентов), сначала на 5000 наиболее высокодисперсных генах, а затем с помощью UMAP (max_components=2, n_neighbors=50, min_dist=0,1, metric = «cosine»). Скопления клеток определяли с помощью алгоритма Louvain, применяемого в Monocle 3(louvain_res=1e-04). Кластеры назначали известным типам клеток на основании маркеров, специфических для типа клеток. Обнаруживали, что вышеуказанный итерационный подход на основе кластеризации ограничен маркировкой дублетных клеток между часто встречающимися скоплениями клеток и редкими скоплениями клеток (например, менее 1% от всей популяции клеток). Для дополнительного удаления этих дублетных клеток брали скопления клеток, определенные с помощью Monocle 3, и сначала вычисляли дифференциально экспрессируемые гены по всем скоплениями клеток (в пределах органа) с помощью функции differentialGeneTest() Monocle 3. Затем выбирали набор генов, объединяющий в себе первые десять генных маркеров для каждого скопления клеток (упорядочены по q-значению и кратности разницы между экспрессией первого и второго ранжированных скоплений клеток). Клетки из каждого основного скопления клеток отбирали для уменьшения размерности с помощью PCA (10 компонент), сначала на выбранном наборе генов, наиболее специфических для скопления генных маркеров, а затем с помощью UMAP (max_component=2, n_neighbors=50, min_dist=0,1, metric = «cosine») с последующим определением кластеризации с использованием алгоритма кластеризации пиков плотности, реализованного в Monocle 3 (rho_thresh=5, delta_thresh=0,2 для большинства анализов кластеризации). Субскопления, демонстрирующие низкую экспрессию маркеров, специфических для скопления клеток-мишеней, и обогащенную экспрессию маркеров, специфических для скопления нецелевых клеток, аннотировали как полученные из дублетов субскопления и отфильтровывали их при визуализации и последующем анализе. Дифференциально экспрессируемые гены по всем типам клеток (в пределах органа) повторно вычисляли с помощью функции differentialGeneTest() Monocle 3 после удаления всех дублетов или клеток из субкластеров, полученных из дублетов.
[00443] Анализ кластеризации клеток по органам
[00444] Для анализа кластеризации 77 основных типов клеток в 15 органах отбирали по 5000 клеток из каждого типа клеток (или всех клеток для типов клеток, содержащих менее 5000 клеток в данном органе). Размерность данных сначала уменьшали с помощью PCA (50 компонент) на генном наборе, объединяющем генные маркеры, наиболее специфические для типа клеток, указанные выше (таблица S5, qval=0), а затем с помощью UMAP (max_components=2, n_neighbors=50, min_dist=0,1, metric = «cosine»). Дифференциально экспрессируемые гены во всех типах клеток определяли с помощью функции differentialGeneTest() Monocle 3. Для аннотирования специфических для типа клеток генных признаков скрещивали специфические для типа клеток гены, определенные выше, с предполагаемыми наборами генов, кодирующих секретируемый и мембранный белок, из атласа белков человека, а также с набором TF, аннотированных в данных motifAnnotations_hgnc из пакета RcisTarget/версия 1.2.1.
[00445] Для кластеризации клеток крови в 15 органах извлекали все клетки крови, включая миелоидные клетки, лимфоидные клетки, тимоциты, мегакариоциты, микроглию, антигенпредставляющие клетки, эритробласты и гемопоэтические стволовые клетки. Размерность данных сначала уменьшали с помощью PCA (40 компонент) на экспрессии набора генов, объединяющего 3000 генных маркеров, наиболее специфических для типа клеток крови (, выбирали только гены, специфически экспрессирующиеся в по меньшей мере одном типе клеток крови (q-значение < 0,05, кратность разницы экспрессии между первым и вторым ранжированными скоплениями клеток > 2), и упорядочивали по медиане qval по органам), а затем с помощью UMAP (max_components=2, n_neighbors=50, min_dist=0,1, metric = «cosine»). Скопления клеток определяли с помощью алгоритма Louvain, применяемого в Monocle 3(louvain_res=1e-04). Кластеры назначали известным типам клеток на основании маркеров, специфических для типа клеток.
[00446] Затем применяли стратегию анализа, аналогичную описанной выше, для кластеризации клеток эндотелия или эпителия в органах. В случае эндотелиальных клеток сначала извлекали клетки из клеток сосудистого эндотелия, лимфоэндотелиальных клеток и эндокардиальных клеток в органах. Размерность данных сначала уменьшали с помощью PCA (30 компонент) по набору генов, объединяющему 1000 генных маркеров, наиболее специфических для типа эндотелиальных клеток, определенных выше (выбирали только гены, специфически экспрессирующиеся в по меньшей мере одном типе эндотелиальных клеток (q-значение < 0,05, кратность разницы экспрессии между первым и вторым ранжированными скоплениями клеток > 2), и упорядочивали по медиане qval по органам), а затем с помощью UMAP с аналогичными параметрами, как и для клеток крови. Скопления клеток определяли с помощью алгоритма Louvain, реализованного в Monocle 3 (Louvain_res=1е-04), а затем аннотировали на основании тканевого происхождения эндотелиальных клеток. В случае эпителиальных клеток сначала извлекали клетки из скопления эпителиальных клеток на Фиг. S3B, с последующим уменьшением размерности с помощью PCA (50 компонент) сначала на 5000 наиболее высокодисперсных генов, а затем с помощью UMAP (max_components=2, n_neighbors=50, min_dist=0,1, metric = «cosine»).
[00447] Анализ связи «TF-ген»
[00448] Предположительно процесс регуляции генов может быть связан с крупномасштабным анализом экспрессии генов одиночных клеток. Таким образом, для прогнозирования взаимодействий «TF-ген» путем связывания их ковариации между миллионами клеток с анализом регуляторной последовательности для валидации применяли способ регуляторного вывода для одиночной клетки, аналогичный описанному в предыдущем исследовании. Рабочий процесс состоит из трех стадий. Поскольку небольшое количество таких профилей одиночных клеток усложняет задачу, сначала агрегировали количество генов из подгрупп клеток (~ 100 клеток) с высокой степенью схожести транскриптома, сгруппировав клетки (в пределах органа) в субкластеры с помощью описанной выше итерационной стратегии кластеризации, с последующей кластеризацией k-средних значений на координатах UMAP для клеток из каждого субкластера. k выбирают на основе количества клеток в каждом субкластере таким образом, чтобы среднее количество клеток на субкластер составляло 100.
[00449] Стремились определить связи между TF и их регулируемыми генами на основе ковариации экспрессии по агрегированным «псевдоклеткам» в пределах каждого органа. Выбирали клетки с обнаруженными более чем 10 000 UMI и генами (включая TF), обнаруженными в более 10% всех клеток. Полную экспрессию генов на клетку нормализовали по специфическим для клеток факторам размера библиотеки, рассчитанным на основе полной матрицы экспрессии генов с помощью estimateSizeFactors в Monocle 3, логарифмически трансформировали, центрировали, затем масштабировали по функции масштабирования в R. Для каждого обнаруженного гена строили регрессионную модель LASSO с использованием пакета glmnet/версии 2.0 для прогнозирования нормализованных уровней экспрессии каждого гена на основе нормализованной экспрессии TF, аннотированных в данных motifAnnotations_hgnc, полученных из пакета RcisTarget/версии 1.2.1, путем аппроксимации следующей модели:
[00450]
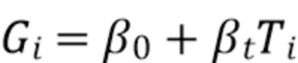 ,
,
[00451] где  представляет собой скорректированный показатель экспрессии гена i. Его вычисляют по количеству генов для каждой псевдоклетки, нормализованному по оценке специфического для клетки фактора размера (
представляет собой скорректированный показатель экспрессии гена i. Его вычисляют по количеству генов для каждой псевдоклетки, нормализованному по оценке специфического для клетки фактора размера ( ) с помощью estimateSizeFactors в Monocle 3 на полной экспрессионной матрице каждой псевдоклетки, и логарифмически преобразованному:
) с помощью estimateSizeFactors в Monocle 3 на полной экспрессионной матрице каждой псевдоклетки, и логарифмически преобразованному:
[00452]
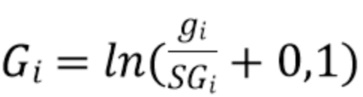
[00453] Для упрощения дальнейшего сравнения между генами стандартизируют ответ Gi перед аппроксимацией модели для каждого гена i с функцией scale() в R.
[00454] Аналогично 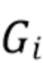 ,
, 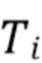 представляет собой скорректированное значение экспрессии TF для каждой псевдоклетки. Его рассчитывают по показателю экспрессии полного TF, нормализованному по оценке специфического для клетки фактора размера (
представляет собой скорректированное значение экспрессии TF для каждой псевдоклетки. Его рассчитывают по показателю экспрессии полного TF, нормализованному по оценке специфического для клетки фактора размера (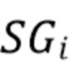 ) с помощью estimateSizeFactors в Monocle 3 на полной экспрессионной матрице каждой псевдоклетки, и логарифмически преобразованному:
) с помощью estimateSizeFactors в Monocle 3 на полной экспрессионной матрице каждой псевдоклетки, и логарифмически преобразованному:
[00455]
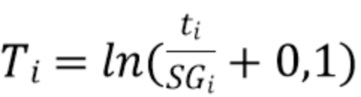
[00456] Перед аппроксимацией Ti стандартизируют с помощью функции scale() в R.
[00457] Хотя отрицательные корреляции между экспрессией TF и новой частотой синтеза гена могут отражать активность транскрипционного репрессора, считается, что более вероятное объяснение отрицательных связей, регистрируемых glmnet, заключалось во взаимоисключающих паттернах специфической для клеточного состояния экспрессии и активности TF. Таким образом, при прогнозировании исключали TF с отрицательно коррелирующей экспрессией с потенциальной частотой синтеза гена-мишени, а также связи с низким коэффициентом регрессии (< 0,03).
[00458] Целью подхода является определение TF, которые могут регулировать каждый ген, путем поиска подгруппы, которую можно использовать для прогнозирования его экспрессии в модели регрессии. Однако TF, экспрессия которого коррелирует с экспрессией гена, не подразумевает однозначно, что он непосредственно регулирует этот ген. Для определения предположительно прямых мишеней в данном наборе сначала перекрещивали связи с TF, профилированными в экспериментах ENCODE ChIP-seq. Сохраняли только наборы генов со значимым обогащением правильных сайтов связывания ChIP-seq TF (двусторонний точный тест Фишера, FDR 5%) и дополнительно сокращали для удаления непрямых генов-мишеней без поддержки данных связывания TF. Для расширения набора валидированных связей «TF-ген» дополнительно применяли пакет SCENIC, процесс для конструирования регуляторных сетей генов на основе обогащения мотивов целевого TF в отрезке в 10 т. п. н. вокруг промоторов генов. Каждый модуль совместной экспрессии, определенный с помощью регрессии LASSO, анализировали с использованием анализа цис-регуляторного мотива с использованием RcisTarget/версии 1.2.1. Сохраняли только модули со значимым обогащением мотива правильного регулятора TF и сокращали для удаления непрямых генов-мишеней без поддержки мотивов. Фильтровали связи «TF-ген» по трем пороговым значениям коэффициента корреляции (0,3, 0,4 и 0,5) и объединяли все связи, валидированные с помощью данных связывания RcisTarget36 и ChIP-seq.
[00459] Вышеописанную стратегию применяли к агрегированным псевдоклеткам в каждом органе и определяли от 1220 (тимус) до 10 059 (печень) связей «TF-ген» в органах, которые объединяли с совокупно 56 272 связями «TF-ген» между 706 TF и 12 868 генами, валидированными как ковариацией экспрессии, так и данными о связывании TF или мотивах. В качестве контрольного анализа переставляли идентификаторы клеток экспрессионной матрицы TF, и после перестановки связи не определяли. Некоторые из определенных регуляторных взаимоотношений TF и генов легко валидировать в базе данных сетей TF (TRRUST) или сетей совместной встречаемости «TF-ген» Enrichr, таких как E2F1 (наиболее обогащенный TF TRRUST из 330 связанных генов=E2F1, скорректированное р-значение=2,2e-14), HNF4A (наиболее обогащенный TF TRRUST из 745 связанных генов=HNF4A, скорректированное р-значение=0,000003) и FLI1 (наиболее обогащенная совместная встречаемость TF из 1219 связанных генов=FLI1, скорректированное р-значение=5,6e-122). 85% (48 050 из 56 272) связей «TF-ген» были органоспецифическими. Например, фосфолипид-транспортирующую АТФазу 8B1 (ATP8B1) связывали с HNF4A только в кишечнике, в соответствии с тем фактом, что она показала самые высокие корреляции с HNF4A в кишечнике (коэффициент корреляции Спирмена=0,36) по сравнению с другими органами (средний коэффициент корреляции Спирмена=0,008). 745 связей «TF-ген» обнаруживали во множестве органов (> 5). Как и ожидалось, связанные с ними гены обогащены в путях дифференцировки иммунных клеток (дифференцировка гемопоэтических стволовых клеток: скорректированное p-значение 2,5e-6; развитие легочных дендритных клеток и субпопуляций макрофагов: скорректированное р-значение 0,0001), а также основных биологических процессах, таких как ответ на стресс и клеточный цикл (повреждение ДНК ионизирующей радиацией (ИР) и клеточный ответ посредством атаксия-телеангиэктазия и Rad3-родственного белка (ATR): скорректированное р-значение 0,006, оксидативный стресс: скорректированное р-значение 0,02, контроль клеточного цикла G1-S: скорректированное р-значение 0,05). 10,5% (5935 из 56 272) связей «TF-ген» находились между двумя TF, из которых 362 пары TF продемонстрировали двунаправленные регуляторные взаимоотношения, потенциально представляющие схемы самоактивации. Например, определяли петли положительной обратной связи ключевых регуляторов, которые управляют дифференцировкой клеток скелетных мышц, включая MYOD1, MYG, TEAD4 и MYF6. Специфические для типа клеток гены, TF и их регуляторные взаимодействия можно визуализировать и исследовать на веб-сайте.
[00460] Интеграционный анализ человека и мыши
[00461] Сначала применяли слегка модифицированную стратегию для определения коррелированных типов клеток между атласом эмбриональных стволовых клеток человека и атласом клеток органогенеза мыши (MOCA). Сначала агрегировали специфические для типа клеток количества UMI, нормализованные по общему количеству, умноженному на 100 000, и логарифмически преобразованному после добавления псевдоколичества. Затем применяли регрессию неотрицательных наименьших квадратов (NNLS) для прогнозирования экспрессии генов типа клеток-мишеней ( ) в наборе данных A с экспрессией генов всех типов клеток (
) в наборе данных A с экспрессией генов всех типов клеток ( ) в наборе данных B:
) в наборе данных B:
[00462]
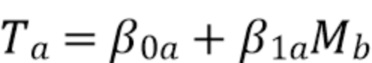 ,
,
[00463] где  и
и  представляют собой отфильтрованную экспрессию генов для типа клеток-мишеней из набора данных A и всех типов клеток из набора данных B соответственно. Для повышения точности и специфичности выбирали специфические для типа клеток гены для каждого типа клеток-мишеней путем: 1) ранжирования генов на основе кратности изменения экспрессии между типом клеток-мишеней по сравнению медианой экспрессии для всех типов клеток, а затем отбора первых 200 генов; 2) ранжирования генов на основе кратности изменения экспрессии между типом клеток-мишеней по сравнению с максимальной экспрессией для всех других типов клеток, а затем отбора первых 200 генов; 3) объединения списков генов от стадий (1) и (2).
представляют собой отфильтрованную экспрессию генов для типа клеток-мишеней из набора данных A и всех типов клеток из набора данных B соответственно. Для повышения точности и специфичности выбирали специфические для типа клеток гены для каждого типа клеток-мишеней путем: 1) ранжирования генов на основе кратности изменения экспрессии между типом клеток-мишеней по сравнению медианой экспрессии для всех типов клеток, а затем отбора первых 200 генов; 2) ранжирования генов на основе кратности изменения экспрессии между типом клеток-мишеней по сравнению с максимальной экспрессией для всех других типов клеток, а затем отбора первых 200 генов; 3) объединения списков генов от стадий (1) и (2). 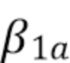 представляет собой коэффициент корреляции, рассчитанный с помощью регрессии NNLS.
представляет собой коэффициент корреляции, рассчитанный с помощью регрессии NNLS.
[00464] Аналогичным образом затем меняют порядок наборов данных A и B и прогнозируют экспрессию генов типа клеток-мишеней (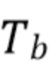 ) в наборе данных B с экспрессией генов всех типов клеток (
) в наборе данных B с экспрессией генов всех типов клеток (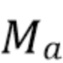 ) в наборе данных A:
) в наборе данных A:
[00465]

[00466] Таким образом, каждая клетка типа a в наборе данных A и каждая клетка типа b в наборе данных B связаны двумя коэффициентами корреляции из вышеуказанного анализа: 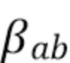 для прогнозирования клетки типа a с использованием b и
для прогнозирования клетки типа a с использованием b и 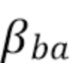 для прогнозирования клетки типа b с использованием a. Объединяют два значения с помощью:
для прогнозирования клетки типа b с использованием a. Объединяют два значения с помощью:
[00467]
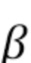 =
= 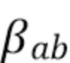 +
+ 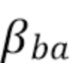 ,
,
[00468] и находят, что 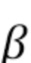 с высокой специфичностью отражает соответствие типов клеток между двумя наборами данных. Для каждого типа клеток в наборе данных A все типы клеток в наборе данных B ранжируются по
с высокой специфичностью отражает соответствие типов клеток между двумя наборами данных. Для каждого типа клеток в наборе данных A все типы клеток в наборе данных B ранжируются по 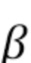 , а верхний тип верхних клеток (с
, а верхний тип верхних клеток (с 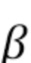 > 0,06) определяют как соответствующий тип клеток. Все типы человеческих клеток из данного исследования сравнивали с 10 основными траекториями клеток и 56 субтраекториями из атласов эмбриональных клеток мыши (MOCA).
> 0,06) определяют как соответствующий тип клеток. Все типы человеческих клеток из данного исследования сравнивали с 10 основными траекториями клеток и 56 субтраекториями из атласов эмбриональных клеток мыши (MOCA).
[00469] Затем интегрировали атлас эмбриональных клеток человека и атлас клеток органогенеза мыши (MOCA) с использованием способа интеграции Seurat версии 3 (FindAnchors и IntegrateData) с выбранной размерностью 30 из 3000 наиболее высоковариабельных генов с общими названиями генов как у человека, так и у мыши. Сначала интегрировали 65 000 фетальных клеток человека (до 1000 клеток, случайным образом отобранных из каждого из 77 типов клеток) и 100 000 эмбриональных клеток мыши, случайным образом отобранных из MOCA, с параметрами по умолчанию. Затем применяли ту же стратегию интегративного анализа для извлеченных человеческих и мышиных клеток из гемопоэтических, эндотелиальных и эпителиальных траекторий.
[00470] Пример 3
[00471] Способ профилирования доступности хроматина одиночных клеток на основе трехуровневой комбинаторного индексирования (sci-ATAC-seq)
[00472] Материалы
[00473] Реагенты и расходные материалы
[00474] 0,5 M ЭДТА (Thermo Fisher Scientific, AM9260G); лесенка 100 п. н. (New England Biolabs (NEB), N3231L); 1000X Sybr (Invitrogen (Gibco/BRL Life Tech), S7563); 10 мМ АТФ (New England Biolabs (NEB), PO756S); 10X HBSS (Gibco/BRL Life Tech, 14065-056); 10X буфер PNK (New England Biolabs (NEB), M0201L); 1 M MgCl2 (Thermo Fisher Scientific, AM9530G); 1X DPBS (Thermo Fisher Scientific, 14190-144); 5% дигитонин (Thermo Fisher Scientific, BN2006); 5 M NaCl (Thermo Fisher Scientific, AM9759); 6% TBE PAGE (Invitrogen (Gibco/BRL Life Tech), EC6265BOX); оранжевый краситель 6x (New England Biolabs (NEB), B7022S); гранулы AMPure (Beckman Coulter, A63882); BSA, класс «для молекулярной биологии» (New England Biolabs (NEB), B9000S); пробирка для ДНК LoBind объемом 1,5 мл, степень чистоты для ПЦР (Eppendorf North America, 22431021); DL-дитиотреитол, 1 M 10×0,5 мл (Sigma Aldrich, 64563-10x.5МЛ); буфер EB (Qiagen, 19086); пробирки Falcon, 15 мл (VWR Scientific, 21008-936); пробирки Falcon, 50 мл (VWR Scientific, 21008-940); Falcon®, круглодонные пробирки емкостью 5 мл с клеточным фильтром (Fisher Scientific, 352235); наконечники фильтра Green pack LTS, 200 мкл, (GP-L200F) (Rainin Instrument, 17002428); наконечники фильтра Green pack LTS, 20 мкл, (GP-L20F) (Rainin Instrument, 17002429); глицерин (Sigma Aldrich, G5516-500ML); глицин (Sigma Aldrich, 50046-250G); IGEPAL CA-630 (Sigma Aldrich, I8896-50ML); наконечники Liquidator - 10 мкл (Rainin Instrument, 17011117); наконечники Liquidator - 200 мкл (Rainin Instrument, 17010646); прозрачный 96-луночный планшет для ПЦР LoBind (Eppendorf North America, 30129512); 8-пробирочная белая пробирка Low-Profile емкостью 0,2 мл, без колпачка (Bio-rad Laboratories, TLS0851); тетрагидрат ацетата магния (Sigma Aldrich, M5661-50G); адгезивное уплотнение Microseal B (Bio-Rad Laboratories, MSB1001); модуль стерилизационного фильтра Nalgene MF 75, 0,2 мкм - 250 мл (VWR, 28199-112); модуль стерилизационного фильтра Nalgene MF 75, 0,2 мкм - 500 мл (VWR, 28198-505); основная смесь Hi-fidelity NEBNext (2x) (New England Biolabs (NEB), M0541L); набор High Output NextSeq 500 (150 циклов) (Illumina Inc., FC-404-2002); нетканая марля (Dukal, 6114); вода без нуклеазной активности (Thermo Fisher Scientific, AM9937); оптически плоские 8-колпачковые стрипы (Bio-Rad Laboratories, TCS-0803); ингибиторы протеазы (Sigma Aldrich, P8340-1 мл); RT-L250WS LTS 250 мкл с широким отверстием (Rainin Instrument, 30389249); резервуары для реагентов (Fisher Scientific, 07-200-127); спермидин (Sigma Aldrich, S2626-1G); Sybr gold (Invitrogen (Gibco/BRL Life Tech), S-11494); модуль одноразового фильтра Steriflip, пора 0,22 мкм (Fisher Scientific, SCGP00525); T4 PNK (New England Biolabs (NEB), M0201L); лигаза T7 (New England Biolabs (NEB), M0318L); лигазный буфер T7 (New England Biolabs (NEB), M0318L); Tapestation (реагент D5000) (Agilent Technologies, 5067-5589); Tapestation (screentape) (Agilent Technologies, 5067-5588); буфер TD (2x) (Illumina Inc., FC-121-1031); TDE1 (Tn5) (Illumina Inc., FC-121-1031); Tris-HCl pH 7,5 (1 M) (Thermo Fisher Scientific, 15567027); Tween-20 (Thermo Fisher Scientific, BP337-500); дистиллированная вода UltraPure (не содержащая ДНКаз, РНКаз) (Thermo Fisher Scientific, 10977023); DNA Clean and Concentrate (DCC-5) (Zymo Research, D4014).
[00475] Инструменты:
[00476] система Agilent 4200 TapeStation; гемоцитометр Bright-Line™ (Sigma); центрифуга (охлажденная до 4 °C) (Eppendorf, 5810R); магнит с боковой кромкой DynaMag™-96 (Thermo Fisher Scientific, 12027); Eppendorf Mastercycler (термоциклер); сортировщик клеток FACSAria III (BD); морозильная камера (-20 °C, -80 °C) и холодильник (4 °C); камера для электрофореза; резервуар с жидким азотом для хранения образцов; микроскоп; многоканальные пипетки (10 мкл, 200 мкл) (Rainin Instrument); платформа NextSeq 500 (Illumina); ручная система пипетирования Rainin Liquidator 96
[00477] Приготовление реагентов
[00478] Использовали рецепт ATAC-RSB. В пробирке falcon объемом 50 мл смешать 500 мкл 1 M Tris-HCl pH 7,4 (конечная 10 мМ Tris-HCl), 100 мкл 5 M NaCl (конечная концентрация 10 мМ NaCl), 300 мкл 0,5 M MgCl2 (конечная концентрация 3 мМ MgCl2) и 49,1 мл воды без нуклеазной активности. Стерилизуют посредством фильтра с помощью стерильного одноразового вакуумного фильтрующего устройства Millipore «Steriflip», мембрана из полиэфирсульфона (ПЭС); размер пор: 0,22 мкм (SCGP00525). Буфер хранят при 4 °C в течение периода до 6 месяцев.
[00479] 10% Tween-20 (хранят при 4 °C в течение периода до 6 месяцев); 10% IGEPAL CA- 630 (хранят при 4 °C в течение периода до 6 месяцев); 1% дигитонин (разбавляют 5% дигитонин до 1% водой без нуклеазной активности, хранят при 4 °C в течение периода до 6 месяцев).
[00480] Буфер для замораживания (FB). В пробирке falcon объемом 50 мл объединяют 50 мМ Tris при pH 8,0, 25% глицерин, 5 мМ Mg(OAc)2, 0,1 мМ ЭДТА и воду. Стерилизуют посредством фильтра с помощью стерильного одноразового вакуумного фильтрующего устройства Millipore «Steriflip», мембрана из полиэфирсульфона (ПЭС); размер пор: 0,22 мкм (SCGP00525). Буфер хранят при 4 °C в течение периода до 6 месяцев. В день выделения ядер смешивают 975 мкл FB, 5 мкл 5 мМ ДТТ (Sigma-Aldrich, кат. № 646563-10 X 0,5 мл) и 20 мкл 50× смесь ингибиторов протеазы (Sigma-Aldrich, кат. № P8340).
[00481] 2,5 M глицин. Готовят 2,5 M глицин, объединяют 46,92 г глицина в 250 мл воды, затем стерилизуют посредством фильтрации (система фильтрации Nalgene, мембрана из нитрата целлюлозы 0,2 мкм (VWR, 28199-112). Реагент хранят при комнатной температуре в течение периода до 6 месяцев.
[00482] 40 мМ ЭДТА. Готовят 40 мМ ЭДТА из основного раствора 0,5 M ЭДТА (Invitrogen, AM9262) с водой, а затем стерилизуют фильтрацией (VWR, 28198-505). Реагент хранят при комнатной температуре в течение периода до 6 месяцев.
[00483] Клеточная культура. Клетки GM12878 культивировали и поддерживали в среде RPMI 1640 (Thermo Fisher Scientific, кат. № 11875-093) с 15% FBS (Thermo Fisher, кат. № SH30071.03) и 1% пенициллином-стрептомицином (Thermo Fisher, кат. № 15140122). Подсчитывают и разделяют при концентрации 300 000 клеток/мл три раза в неделю. Клеточную линию CH12-LX мышей культивировали в среде RPMI 1640 с 10% FBS, 1% пенициллином-стрептомицином и 1×105 M B-ME. Клетки подсчитывали и поддерживали при плотности 1×105 клеток/мл, разделяя их три раза в неделю для поддержания концентрации клеток. Обе клеточные линии инкубировали при 37 °C в атмосфере с содержанием 5% CO2.
[00484] Выделение и фиксация ядер из клеточных линий. Для получения суспензии клеток берут ~ 10-100 миллионов клеток и осаждают клетки путем центрифугирования при 500 x g в течение 5 мин при комнатной температуре. Супернатант аспирируют и осадок ресуспендируют в 1 мл лизирующего буфера Omni-ATAC (10 мМ NaCl, 3 мМ MgCl2, 10 мМ Tris-HCl pH 7,4, 0,1% NP40, 0,1% Tween 20 и 0,01% дигитонин) и инкубируют на льду в течение 3 мин. Добавляют 5 мл 10 мМ NaCl, 3 мМ MgCl2, 10 мМ Tris-HCl pH 7,4 с 0,1% Tween 20 и осаждают ядра в течение 5 мин при 500 x g при 4 °C. Супернатант аспирируют и ресуспендируют ядра в 5 мл 1X физиологического раствора с сульфатным буфером Дульбекко (DPBS) (Thermo Fisher, кат. № 14190144). Для поперечного сшивания ядер одним впрыскиванием добавляют 140 мкл 37% формальдегида с метанолом (VWR, кат. № MK501602) при конечной концентрации 1%. Инкубируют фиксирующую смесь при комнатной температуре в течение 10 минут, переворачивая каждые 1-2 минуты. Для гашения реакции поперечного сшивания добавляют 250 мкл 2,5 M глицина и инкубируют при комнатной температуре в течение 5 минут, а затем на льду в течение 15 минут, чтобы полностью остановить поперечное сшивание. Для подсчета добавляют 20 мкл погашенной поперечносшитой смеси к 20 мкл трипанового синего. Поперечносшитые ядра центрифугируют при 500 x g в течение 5 минут при 4 °C и аспирируют супернатант. Фиксированные ядра ресуспендируют в соответствующем количестве буферного раствора для замораживания (50 мМ Tris при pH 8,0, 25% глицерин, 5 мМ Mg(OAc)2, 0,1 мМ ЭДТА, 5 мМ ДТТ (Sigma-Aldrich, кат. № 646563-10 X 0,5 мл), 1 × смесь ингибиторов протеазы (Sigma-Aldrich, кат. № P8340)) для получения 2 миллионов ядер на аликвоту 1 мл, мгновенно замораживают в жидком азоте и хранят при -80 °C.
[00485] Получение и хранение ткани
[00486] Выделяют интересующую ткань. Промывают в 1X HBSS, pH 7,4 (с Ca, с Mg), 1X HBSS с кальцием и магнием, без фенолового красного, Gibco BRL (500 мл) 14065-056. Промокают ткань насухо на полувлажной марле (влажная марля предотвращает прилипание ткани к марле). Нетканая марля Dukal № 6114. Высушенную ткань помещают на сверхпрочную фольгу (NC19180132, Fisher Scientific) или в криопробирку. Примечание. В процессе мгновенной заморозки в криопробирках может образовываться иней из водных кристаллов внутри пробирки за счет захваченного воздуха/влаги. Выполняют мгновенное замораживание с помощью жидкого азота. Хранят ткань в хранилище при -80 °C.
[00487] Измельчение в порошок и хранение. В день измельчения в порошок предварительно охлаждают предварительно маркированные пробирки и отбивают молотком на сухом льду с тканевым полотенцем между сухим льдом и металлом. Создают «подкладку», взяв сверхпрочную фольгу размером 18 дюймов x 18 дюймов (46 см х 46 см), складывают ее пополам дважды для получения прямоугольника. Складывают еще два раза, чтобы получить квадрат. Помещают замороженную ткань внутрь «подкладки» фольги, затем помещают ткань в подкладке из фольги внутрь предварительно охлажденного пластикового пакета 4 мм для предотвращения выпадания ткани на сухой лед в случае разрыва фольги. Охлаждают этот пакет с тканью между 2 пластинами сухого льда. Вручную измельчают ткань внутри пакета в порошок предварительно охлажденным молотком; совершают от 3 до 5 ударов без растирающего движения перед перерывом, чтобы не нагреть образец. Охлаждают молоток и при необходимости повторно измельчают в порошок до получения однородной ткани. Аликвоту измельченной в порошок ткани помещают в предварительно маркированные и предварительно охлажденные пробирки LoBind объемом 1,5 мл и безнуклеазные пробирки объемом 1,5 мл с защелкивающимися крышками (Eppendorf, кат. № 022431021). Аликвоты измельченных в порошок тканей можно хранить при -80 °C до дальнейшей обработки.
[00488] Выделение ядер и фиксация замороженных тканей. Перед началом готовят лизирующий буфер Omni (RSB+0,1% Tween+0,1% NP-40 и 0,01% дигитонин) и RSB с 0,1% Tween-20. В день выделения ядер добавляют лизирующий буфер непосредственно в пробирку или выливают замороженную аликвоту в посудину размером 60 мм с буфером для лизиса клеток и дополнительно измельчают лезвием. Если аликвоту не размораживали в какой-либо момент хранения, порошкообразная аликвота ткани должна легко выскальзывать из пробирки для хранения, и при этом образец не теряется. По оценке можно получать количество, составляющее ~ 20 000 клеток на мг исходного веса ткани, а эффективность может варьироваться в зависимости от ткани. Измельченную в порошок ткань ресуспендируют в 1 мл лизирующего буфера Omni (ресуспендирующий буфер (RSB) + 0,1% Tween+0,1% NP-40 и 0,01% дигитонин), затем переносят в пробирку falcon объемом 15 мл. Ядра инкубируют на льду в течение 3 минут, затем добавляют 5 мл RSB+0,1% Tween-20. Центрифугируют ядра при 500 x g в течение 5 минут при 4 °C. Супернатант аспирируют и ресуспендируют в 5 мл 1X DPBS. Для удаления комков ткани пропускают ядра в 1X DPBS через клеточное сито с размером пор 100 мкм (VWR, кат. № 10199-658).
[00489] В вытяжном шкафу выполняют поперечное сшивание ядер с помощью добавления 140 мкл 37% формальдегида (VWR, MK501602) с метанолом одним впрыскиванием до конечной концентрации 1% и быстрого перемешивания посредством переворачивания пробирки несколько раз. Инкубируют при комнатной температуре в течение ровно 10 минут, слегка переворачивая пробирку каждые 1-2 минуты. Для гашения реакции поперечного сшивания добавляют 250 мкл 2,5 M глицина (свежеприготовленного, стерилизованного с помощью фильтра), хорошо перемешивают, переворачивая пробирку несколько раз. Инкубируют в течение 5 минут при комнатной температуре, затем на льду в течение 15 минут, чтобы полностью остановить поперечное сшивание. Подсчитывают ядра с помощью гемоцитометра для определения конечного объема добавляемого буферного раствора для замораживания, цель - заморозить ~ 1-2 миллиона ядер на пробирку. Центрифугируют поперечносшитые ядра при 500 x g в течение 5 минут при 4 °C, аспирировать супернатант и ресуспендировать осадок в 1-10 мл буфера для замораживания, дополненного 1x ингибиторами протеазы и 5 мМ ДТТ. Мгновенно замораживают ядра в жидком азоте и хранят их при -80 °C.
[00490] Обработка образцов sci-ATAC-seq3 (конструирование библиотеки и контроль качества). Размораживание, увеличение проницаемости мембраны, подсчет и тагментация. Перед началом готовят лизирующий буфер Omni (RSB+0,1% Tween+0,1% NP-40 и 0,01% дигитонин) и RSB с 0,1% Tween-20. Извлекают замороженные фиксированные ядра из холодильника с температурой -80 °C и помещают на слой сухого льда. Размораживают ядра на водяной бане при 37 °C до оттаивания (~ 30 с-1 мин) и переносили в пробирку falcon объемом 15 мл. Осаждают ядра при 500 x g в течение 5 минут при 4 °C. Аспирируют супернатант, не нарушая осадок, и ресуспендируют осадок в 200 мкл лизирующего буфера Omni, а затем инкубируют на льду в течение 3 минут. Промывают лизирующий буфер 1 мл ATAC-RSB, используя 0,1% Tween -20, и осторожно переворачивают пробирку 3 раза для перемешивания. Подсчитывают ядра, взяв 20 мкл ядер и 20 мкл трипанового синего. При подсчете ядра держат на льду по возможности постоянно с этого момента. Для экспериментов с 3-уровневым индексированием при 384^3 входное число ядер составляет 4,8 миллиона по 50 000 ядер на лунку на ткань или образец, распределенный на 96 реакций. В каждой партии имеется 23 образца/тканей вместе со смесью ядер мыши и человека в качестве 24-го образца и контроля. Готовят основную смесь для реакции тагментации (таблица 1).
[00491] Таблица 1
[00492] Для каждого образца берут 225 000 ядер (на основании подсчета), центрифугируют при 500 x g в течение 5 мин при 4 °C, аспирируют супернатант и ресуспендируют осадок в 213 мкл предварительно приготовленной основной смеси для реакции тагментации. Аликвотируют 47,5 мкл ядер в смесь для тагментации с использованием наконечника с широким каналом (Rainin Instrument Co. кат. № 30389249) в 4 лунки 96-луночного планшета LoBind (Eppendorf, кат. № 30129512). Добавляют по 2,5 мкл фермента Nextera v2 (Illumina Inc., кат. № FC-121-1031) на лунку, герметизируют планшет адгезивной лентой и центрифугируют при 500 x g в течение 30 сек. Инкубируют планшет при 55 °C в течение 30 минут для тагментации ДНК. Готовят основную смесь для остановки реакции, объединяя 25 мл 40 мМ ЭДТА и 3,9 мкл 6,4 M спермидина (итог - 20 мМ ЭДТА и 1 мМ спермидина). Реакции тагментации останавливали добавлением 50 мкл смеси для остановки реакции - 40 мМ ЭДТА с 1 мМ спермидина, затем инкубировали при 37 °C в течение 15 мин.
[00493] Объединение, реакция PNK и лигирование N5. Используя наконечники с широкими каналами, тагментированные ядра объединяли (на образец) и осаждали при 500 x g в течение 5 минут при 4 °C, а затем промывали 500 мкл ATAC-RSB с 0,1% Tween-20. Ядра осаждали при 500 x g в течение 5 минут при 4 °C, аспирировали супернатант и ресуспендировали в 18 мкл ATAC-RSB с 0,1% Tween 20 на образец. Готовят основную смесь для реакции PNK (таблица 2).
[00494] Таблица 2
[00495] К каждому образцу добавляют 72 мкл основной смеси PNK. Аликвотируют по 5 мкл реакционной смеси PNK (в 16 лунок на четырех 96-луночных планшетах). Герметизируют адгезивной лентой и центрифугируют при 500 x g в течение 5 минут при 4 °C. Реакционную смесь PNK инкубировали при 37 °C в течение 30 минут. Готовят основную смесь для лигирования N5 в количестве, достаточном для 440 реакций (таблица 3).
[00496] Таблица 3
[00497] С использованием многоканальной пипетки в каждую лунку с реакцией PNK непосредственно добавляют 13,8 мкл основной смеси для лигирования. С помощью многоканального или 96-головочного дозатора (Liquidator, кат. № 17010335), добавляют 1,2 мкл 50 мкМ N5_oligo (IDT) в каждую лунку четырех 96-луночных планшетов. Запечатывают адгезивной лентой и центрифугируют при 500 x g в течение 30 секунд, затем инкубируют при 25 °C в течение 1 часа. После первого цикла лигирования для остановки реакции лигирования добавляют 20 мкл смеси ЭДТА и спермидина (20 мМ ЭДТА и 1 мМ спермидин) и инкубируют при 37 °C в течение 15 минут. С помощью наконечников с широким каналом объединяют содержимое каждой лунки в лоток и переносят в пробирку falcon объемом 50 мл. Ядра осаждают при 500 x g в течение 5 минут при 4 °C, аспирируют супернатант и ресуспендируют ядра в 1 мл ATAC-RSB с 0,1% Tween-20 для промывки остатков реакционной смеси для лигирования. Ядра осаждают при 500 x g в течение 5 минут при 4 °C и аспирируют супернатант без нарушения целостности осадка.
[00498] Лигирование N7. Готовят основную смесь для лигирования N7 в количестве, достаточном для 440 реакций (1X лигазный буфер T7, 9 мкM N7_splint (IDT), вода и ДНК-лигаза T7) и ресуспендируют ядра основной смесью для лигирования (таблица 4).
[00499] Таблица 4
[00500] Суспендированные в основной смеси ядра переносят в лоток и с помощью наконечников с широким каналом помещают аликвоты по 18,8 мкл основной смеси для лигирования в четыре 96-луночных планшета LoBind, а затем в каждую лунку четырех 96-луночных планшетов добавляют по 1,2 мкл 50 мкМ N7_oligo (IDT). Планшеты запечатывают адгезивной лентой и центрифугируют при 500 x g в течение 30 секунд, затем инкубируют при 25 °C в течение 1 часа. Останавливают лигирование путем добавления 20 мкл смеси ЭДТА и спермидина (20 мМ ЭДТА и 1 мМ спермидин) и инкубируют при 37 °C в течение 15 минут.
[00501] Объединение, подсчет и разведение. Содержимое лунок объединяют в лотке с помощью наконечников с широким каналом, а затем переносят в пробирку falcon объемом 50 мл. Ядра осаждают при 500 x g в течение 5 минут при 4 °C, аспирируют супернатант и ресуспендируют ядра в 2 мл буфера EB Qiagen (Qiagen, кат. № 19086). Фильтруют ядра с помощью пробирки FACs с фильтровальной крышкой с размером пор 40 мкм (Fisher Scientific, кат. № 352235). Для подсчета ядер берут 20 мкл ресуспендированных и отфильтрованных ядер и 20 мкл трипанового синего. Ядра разводят до концентрации 100-300 ядер на мкл и аликвотируют по 10 мкл на лунку в четырех 96-луночных планшетах LoBind.
[00502] Устранение поперечного сшивания. Для обратного поперечного сшивания ядер готовят основную смесь для обратного поперечного сшивания ядер в виде буфера EB, протеиназы k (Qiagen, кат. № 19133) и 1% додецилсульфата натрия (SDS) (по 1 мкл/0,5 мкл/0,5 мкл на лунку соответственно) и добавляют по 2 мкл в каждую лунку с ядрами. Запечатывают адгезивной лентой, центрифугируют при 500 x g в течение 30 секунд и инкубируют при 65 °C в течение 16 часов.
[00503] Контроль качества анализа ПЦР и геля. Перед началом недолго центрифугируют планшеты с устраненным поперечным сшиванием для осаждения. Готовят основную смесь для ПЦР в количестве, достаточном для 6 реакций (таблица 5).
[00504] Таблица 5
[00505] Аликвотируют 35,5 мкл основной смеси для ПЦР в белую 8-полосную пробирку без крышки (Bio-Rad Laboratories, TLS0851). Добавляют 1,25 мкл 10 мкМ праймеров P7 и P5. В смесь для ПЦР и праймеров добавляют 12 мкл ядер с устраненным поперечным сшиванием. Реакционные пробирки закрывают оптически плоскими 8-колпачковыми стрипами (Bio-Rad Laboratories, TCS-0803). Помещают в аппарат для кПЦР и отслеживают амплификацию для определения оптимального количества циклов: 72 °C - 5 мин, 98 °C - 30 с, 30 циклов при 98 °C по 10 с, 63 °C - 30 с, 72 °C 1 мин, и далее выдерживают при 10 °C. В зависимости от анализируемых лунок выбирают количество циклов таким образом, чтобы все анализируемые лунки очевидно амплифицировались, однако до насыщения интенсивности флуоресценции в любой из лунок. Берут 1 мкл продукта ПЦР для контроля качества: образцы=1 мкл+9 мкл воды без нуклеазной активности+2 мкл 6x оранжевого красителя; лесенка из 100 п. н. (1 : 10) = 1 мкл+9 мкл воды без нуклеазной активности+2 мкл 6x оранжевого красителя; пропускают 6% TBE полиакриламидный гель, 180 вольт в течение 35 мин. Окрашивают 5 мкл SYBR Gold и 50 мл 0,5X буфера TBE при комнатной температуре в течение 5 мин.
[00506] Подготовка планшета для ПЦР. Коротко центрифугируют планшет для осаждения. Откладывают на лед до получения результата анализа ПЦР. Готовят основную смесь для ПЦР (таблица 6).
[00507] Таблица 6
[00508] Обращают внимание на комбинацию колонок и рядов праймеров, используемую во время амплификации. Герметизируют адгезивной лентой, затем центрифугируют при 500 x g в течение 30 с. Запускают планшет для ПЦР с оптимальным количеством циклов на основании результата тестовой ПЦР: 72 °C - 5 мин, 98 °C - 30 с, 10-20 циклов: 98 °C - 10 с, 63 °C - 30 с, 72 °C - 1 мин, затем выдерживают при 10 °C
[00509] Очистка и контроль качества ПЦР амплификации. Очистка продуктов ПЦР с помощью Zymo Clean & Concentrator-5. Объединяют 25 мкл от каждой ПЦР-реакции (2,4 мл) в лотке, добавляют 2 объема связывающего буфера (4,8 мл), разделяют на 4 колонки C&C (600 мкл, центрифугированные 3 раза в каждой колонке), добавляют 200 мкл промывочного буфера Zymo и центрифугируют (всего 2 промывки), используют дополнительное центрифугирование для высушивания колонок в течение 1 мин после последней промывки, элюируют в 25 мкл буфера для элюирования Qiagen (позволяют буферу настояться на колонке 1 мин, затем центрифугируют 1 мин при максимальной скорости), объединяют все 4 элюата и очищают во второй раз в гранулах AMPure 1X (100 мкл), помещают на MPC (коллектор магнитных частиц) до тех пор, пока супернатант не станет прозрачным, аспирируют супернатант. Гранулы дважды промывают 200 мкл 80% этанола, высушивают гранулы в течение 30 с-1 мин до тех пор, пока цвет гранул не потускнеет, без пересушивания гранул, элюируют гранулы в 25 мкл буфера EB Qiagen, помещают в MPC и переносят супернатант в чистую пробирку для контроля качества библиотеки с использованием Tapestation, следуя спецификации производителя, с использованием анализа ScreenTape D5000. Для анализа фрагментов создают таблицу областей из 200-1000 п. н., в которой рассчитывают молярность областей. Для разведения библиотеки до 2 нМ буфером EB и 0,1% Tween-20 используют концентрацию нМ (нмоль/л). При объединении нескольких библиотек нормализуют каждую библиотеку до 2 нМ и готовят эквимолярный пул для секвенирования.
[00510] Следующее секвенирование (набор для 150 циклов). Денатурация библиотеки Разбавляют 2 н NaOH до 0,2 н NaOH (10 мкл 1 н к 90 мкл воды без нуклеазной активности), в новой пробирке Lo-Bind 1,5 переносят 10 мкл 0,1 н NaOH и добавляют 10 мкл объединенных библиотек 2 нМ, инкубируют при комнатной температуре в течение 5 минут, добавляют 980 мкл HT1 для разведения денатурированных библиотек до 20 пМ, разводят денатурированную библиотеку до концентрации загрузки 1,8 пМ (135 мкл, 20 пМ+1365 мкл HT1), разбавляют индивидуальные праймеры до 0,6 мкМ, название набора команд секвенирования NextSeq: 3LV2_sciATAC_high.
[00511] R1-50 оснований для геномной ДНК (гДНК), R2-50 оснований для гДНК.
[00512] Индекс 1-20 оснований (10 оснований для олигонуклеотида N7, 15 цикл без доступа света, штрихкод ПЦР из 10 оснований), индекс 2-20 оснований (10 оснований для олигонуклеотида N5, 15 цикл без доступа света, штрихкод ПЦР из 10 оснований).
[00513] Праймеры для секвенирования: 3L_NexteraV2_R1_seq TCGTCGGCAGCGTCAGATGTGTATAAGAGACAG (SEQ ID NO:5); L_NexteraV2_R2_seq GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG (SEQ ID NO:6); 3LV2_IDX1 CTCCGAGCCCACGAGACGACAAGTC (SEQ ID NO:7); 3LV2_IDX2 ACACATCTGACGCTGCCGACGACTGATTAC (SEQ ID NO:8).
[00514] Полное описание всех патентов, заявок на патенты и публикаций, а также материалов, доступных в электронной форме (включая, например, публикации нуклеотидных последовательностей, например, в GenBank и RefSeq, а также публикации аминокислотных последовательностей в, например, SwissProt, PIR, PRF, PDB, а также трансляции аннотированных кодирующих областей в GenBank и RefSeq), процитированные в настоящем документе, полностью включены в настоящий документ путем ссылки. Дополнительные материалы, на которые даны ссылки в публикациях (такие как дополнительные таблицы, дополнительные фигуры, дополнительные материалы и методы и/или дополнительные экспериментальные данные), также полностью включены в настоящий документ путем ссылки. В случае несоответствия между описанием настоящей заявки и описанием (-ями) каких-либо других документов, включенных в настоящий документ путем ссылки, приоритет имеет описание настоящей заявки. Указанные выше подробное описание и примеры приведены только для обеспечения лучшего понимания. Их не следует рассматривать как наложение ненужных ограничений. Описание не ограничивается исключительно приведенными и описанными деталями, и в изложение также должны быть включены изменения, очевидные специалистам в данной области, в соответствии с пунктами формулы изобретения.
[00515] Если не указано иное, все числа, выражающие количества компонентов, молекулярных масс и т. п., используемые в описании и формуле изобретения, следует понимать как модифицированные во всех случаях термином «приблизительно» Соответственно, если не указано иное, числовые параметры, указанные в описании и формуле изобретения, являются приближенными значениями, которые могут варьироваться в зависимости от желаемых свойств, которые требуется получить посредством настоящего описания. По самой меньшей мере, но не в качестве попытки ограничить применение теории эквивалентов к объему формулы изобретения, необходимо рассматривать каждый числовой параметр по меньшей мере с учетом числа представленных значащих цифр и с применением стандартных методик округления.
[00516] Хотя числовые диапазоны и параметры, устанавливающие широкий объем объекта описания, являются приблизительными, числовые значения, указанные в конкретных примерах, представлены настолько точно, насколько это возможно. Однако все числовые значения по своей природе содержат определенные ошибки, неизбежно вытекающие из стандартного отклонения, выявленного в их соответствующих тестовых измерениях.
[00517] Все заголовки предназначены для удобства читателя и не должны использоваться для ограничения смысла текста, который следует за заголовком, если не указано иное.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ILLUMINA, INC.
UNIVERSITY OF WASHINGTON
<120> ВЫСОКОПРОИЗВОДИТЕЛЬНЫЕ БИБЛИОТЕКИ ОДИНОЧНЫХ КЛЕТОК И СПОСОБЫ
ПОЛУЧЕНИЯ И ИСПОЛЬЗОВАНИЯ
<130> IP-1952-PCT-531001952WO01
<140> PCT/US2020/066013
<141> 2020-12-18
<150> 62/950,670
<151> 2019-12-19
<160> 10
<170> PatentIn, версия 3.5
<210> 1
<211> 54
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<220>
<221> modified_base
<222> (7)..(24)
<223> a, c, t, g, неизвестный или другой
<400> 1
cagagcnnnn nnnnnnnnnn nnnntttttt tttttttttt tttttttttt tttt 54
<210> 2
<211> 45
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический олигонуклеотид"
<220>
<221> источник
<223> /note="Описание объединенной молекулы ДНК/РНК:
синтетический олигонуклеотид"
<220>
<221> modified_base
<222> (7)..(16)
<223> a, c, t, g, неизвестный или другой
<220>
<221> misc_feature
<222> (7)..(16)
<223> /note="Данный участок может охватывать 9-10 нуклеотидов"
<220>
<221> modified_base
<222> (36)..(45)
<223> a, c, t, g, неизвестный или другой
<220>
<221> misc_feature
<222> (36)..(45)
<223> /note="Данный участок может охватывать 9-10 нуклеотидов"
<220>
<221> источник
<223> /note="Подробное описание замен и предпочтительных вариантов
осуществления см. в поданной спецификации"
<400> 2
gctctgnnnn nnnnnnuacg acgctcttcc gatctnnnnn nnnnn 45
<210> 3
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 3
aatgatacgg cgaccaccga gatctacac 29
<210> 4
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 4
caagcagaag acggcatacg agat 24
<210> 5
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 5
tcgtcggcag cgtcagatgt gtataagaga cag 33
<210> 6
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 6
gtctcgtggg ctcggagatg tgtataagag acag 34
<210> 7
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 7
ctccgagccc acgagacgac aagtc 25
<210> 8
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 8
acacatctga cgctgccgac gactgattac 30
<210> 9
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 9
acactctttc cctacacgac gctcttccga tct 33
<210> 10
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<221> источник
<223> /note="Описание искусственной последовательности:
синтетический праймер"
<400> 10
gtctcgtggg ctcgg 15
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЕ БИБЛИОТЕКИ ОДИНОЧНЫХ ЯДЕР И ОДИНОЧНЫХ КЛЕТОК И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2838545C2 |
| КРУПНОМАСШТАБНЫЕ МОНОКЛЕТОЧНЫЕ БИБЛИОТЕКИ ТРАНСКРИПТОМОВ И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2019 |
|
RU2773318C2 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2744175C1 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2833615C2 |
| ПОЛНОГЕНОМНЫЕ БИБЛИОТЕКИ ОТДЕЛЬНЫХ КЛЕТОК ДЛЯ БИСУЛЬФИТНОГО СЕКВЕНИРОВАНИЯ | 2018 |
|
RU2770879C2 |
| СИСТЕМА АНАЛИЗА ДЛЯ ОРТОГОНАЛЬНОГО ДОСТУПА К БИОМОЛЕКУЛАМ И ИХ МЕЧЕНИЯ В КЛЕТОЧНЫХ КОМПАРТМЕНТАХ | 2017 |
|
RU2771892C2 |
| АНАЛИЗ МНОЖЕСТВА АНАЛИТОВ С ИСПОЛЬЗОВАНИЕМ ОДНОГО АНАЛИЗА | 2019 |
|
RU2824049C2 |
| СПОСОБЫ ИНКАПСУЛИРОВАНИЯ ОДИНОЧНЫХ КЛЕТОК, ИНКАПСУЛИРОВАННЫЕ КЛЕТКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2750567C2 |
| СПОСОБЫ ИНКАПСУЛИРОВАНИЯ ОДИНОЧНЫХ КЛЕТОК, ИНКАПСУЛИРОВАННЫЕ КЛЕТКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2793717C2 |
| СПОСОБЫ И СРЕДСТВА ПОЛУЧЕНИЯ БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ | 2019 |
|
RU2815513C2 |

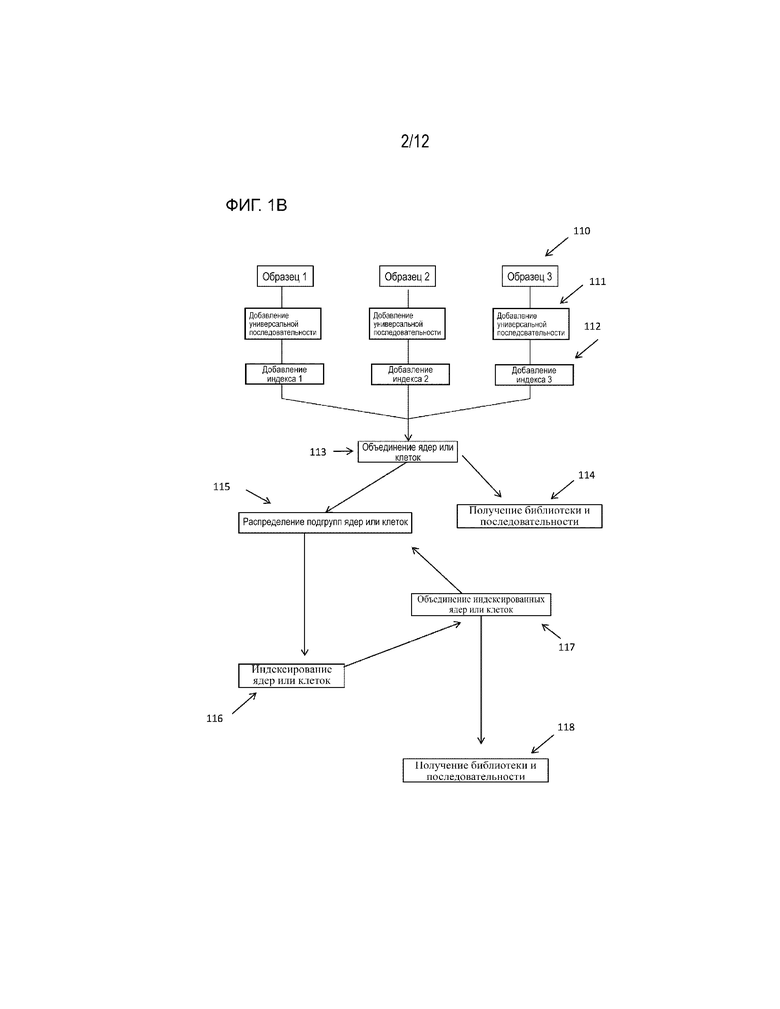
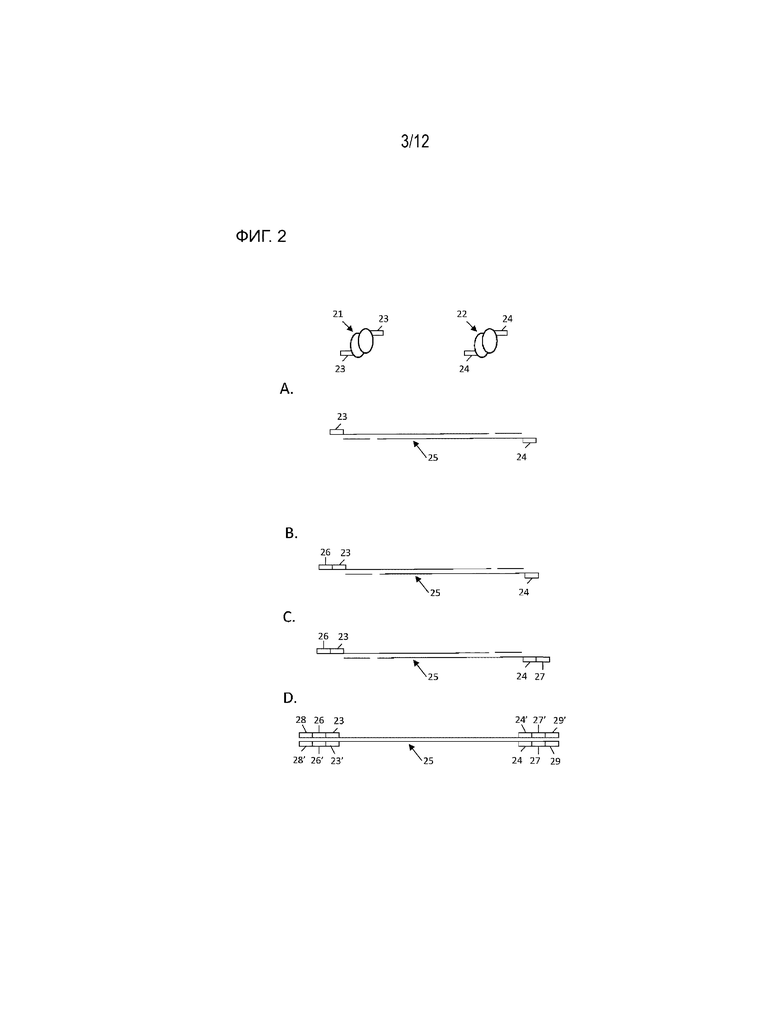
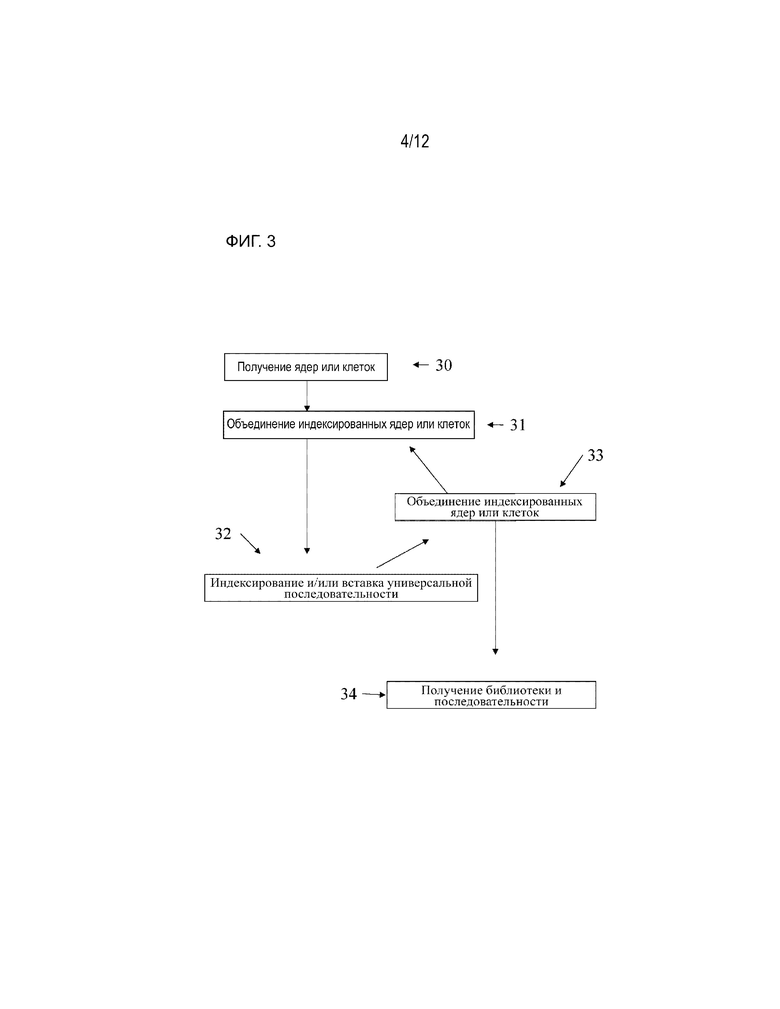

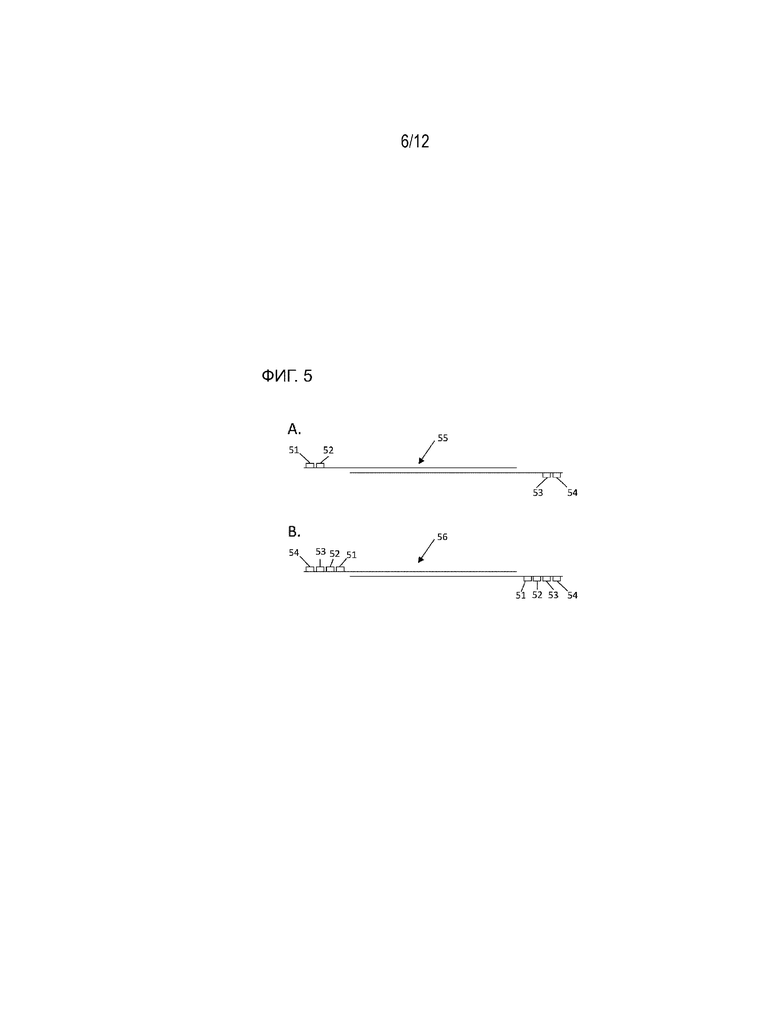
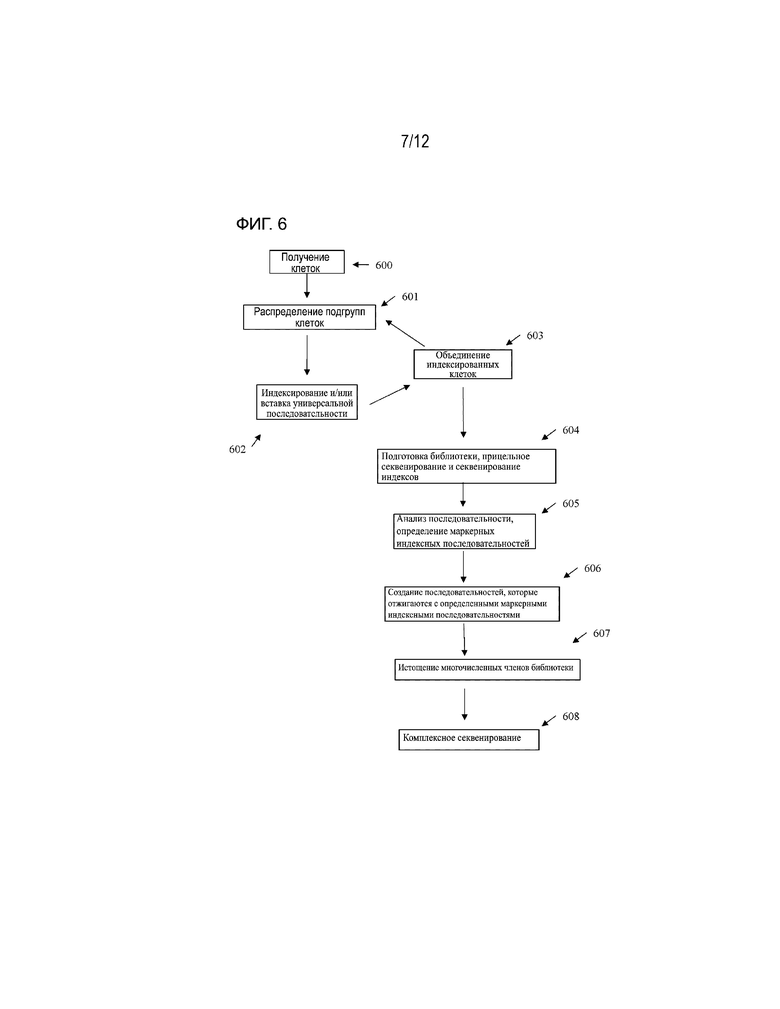
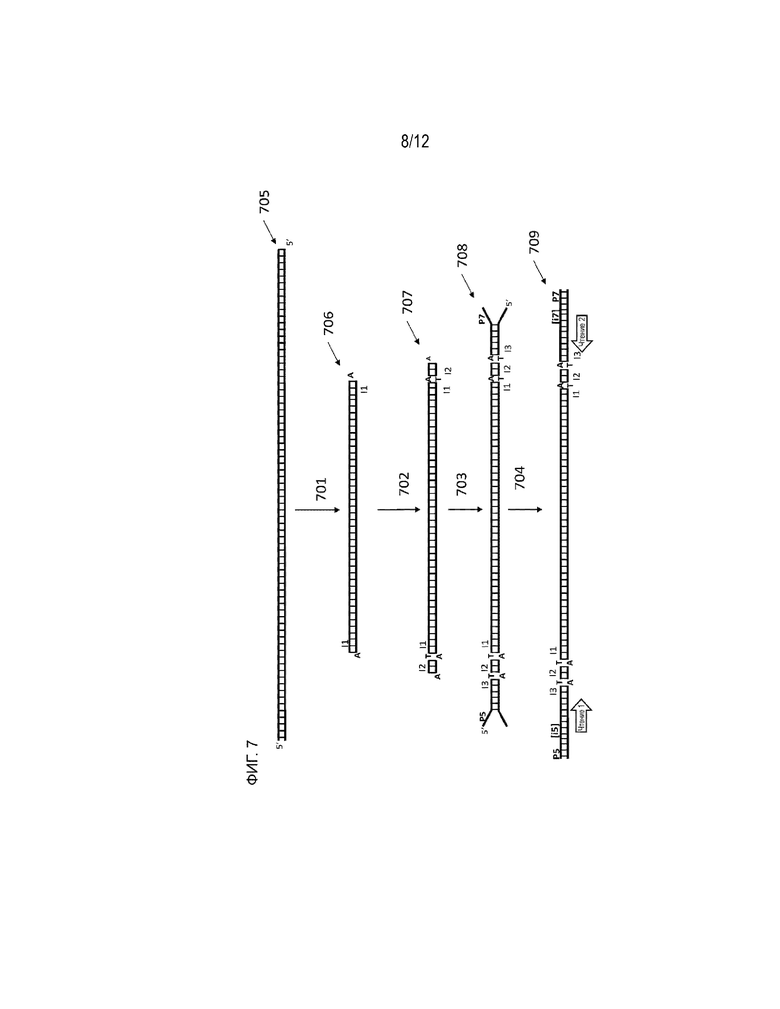

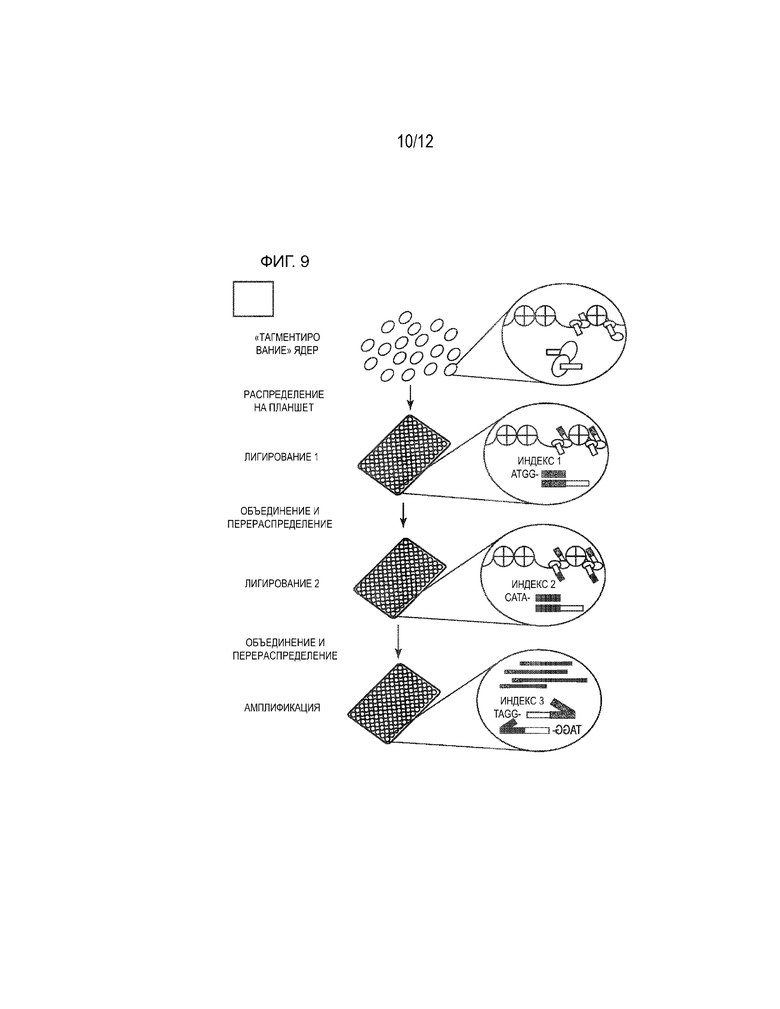
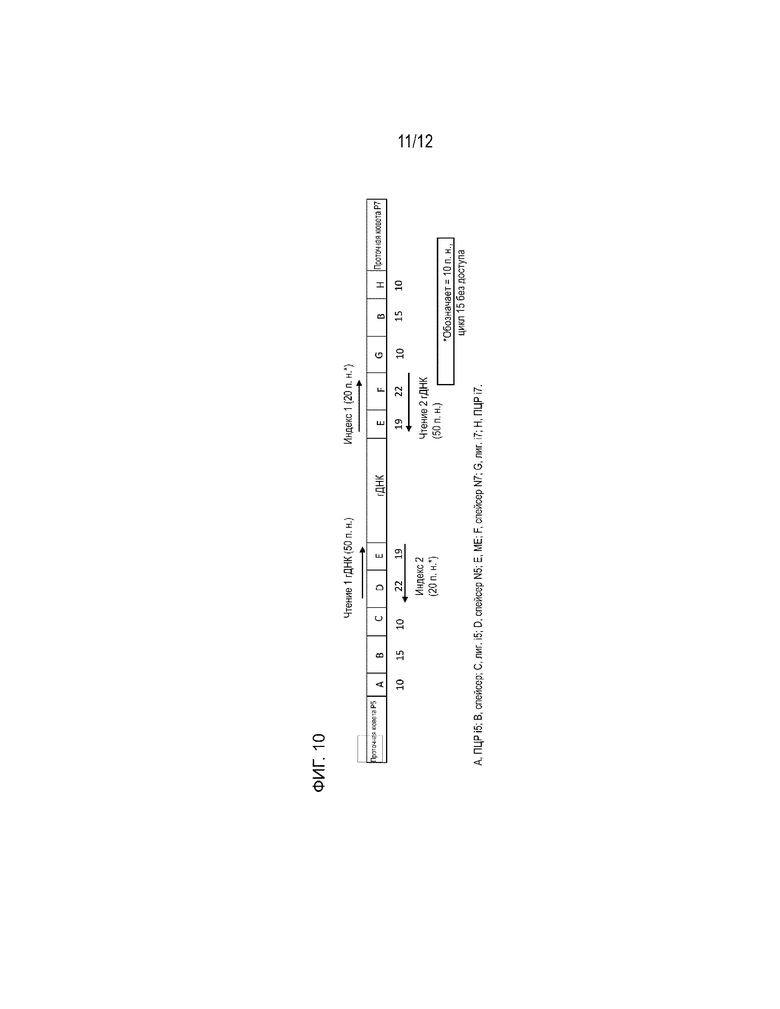
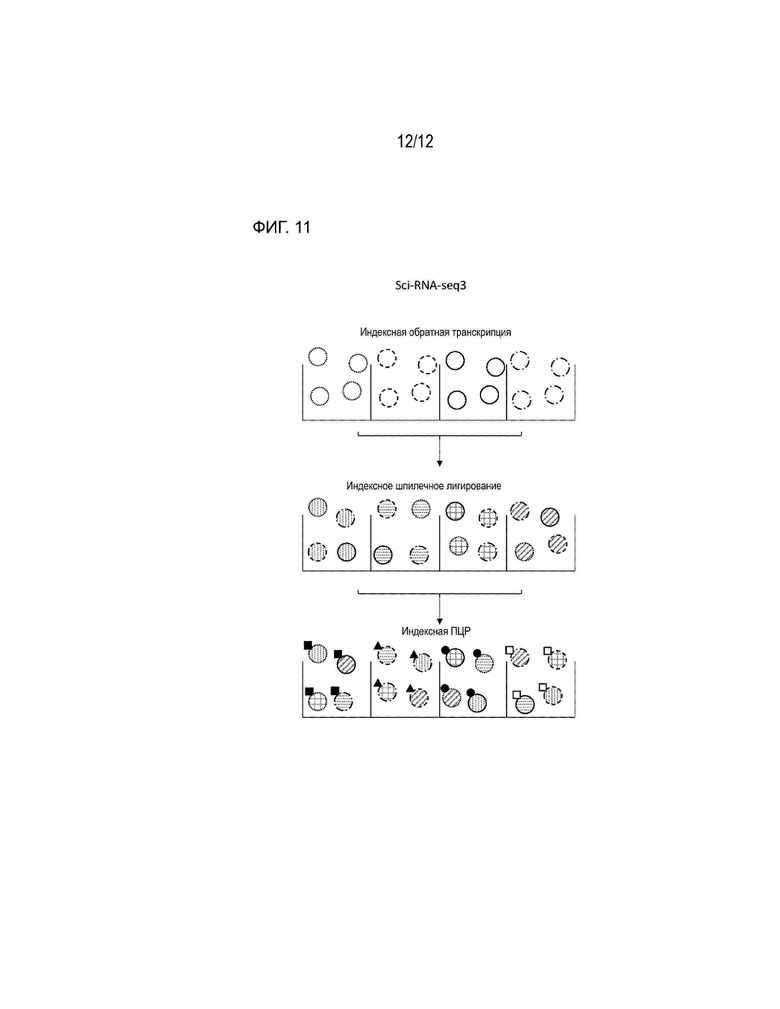
Изобретение относится к биотехнологии и молекулярной биологии, в частности к способам получения библиотеки секвенирования, включающей нуклеиновые кислоты из множества одиночных клеток. Способы по настоящему изобретению относятся к получению комбинаторных индексированных библиотек секвенирования одиночных клеток и получению из них данных о последовательности. В некоторых вариантах осуществления данные о последовательности, полученные из библиотек, являются исчерпывающими, а в других вариантах осуществления данные о последовательности, полученные из библиотек, позволяют охарактеризовать редкие события. 3 н. и 56 з.п. ф-лы, 11 ил., 6 табл., 3 пр.
1. Способ определения субпопуляции клеток или ядер, имеющей биологический признак, причем способ включает:
(a) обеспечение библиотеки секвенирования одиночных клеток,
причем библиотека секвенирования одиночных клеток содержит множество модифицированных целевых нуклеиновых кислот,
при этом модифицированные целевые нуклеиновые кислоты содержат по меньшей мере одну индексную последовательность, которая определяет членов библиотеки из одной клетки или ядра;
(b) анализ библиотеки секвенирования одиночных клеток с помощью прицельного секвенирования для определения индексных последовательностей, которые присутствуют на той же модифицированной целевой нуклеиновой кислоте, что и биологический признак,
причем индексные последовательности, связанные с биологическим признаком, представляют собой маркерную индексную последовательность,
где биологический признак включает нуклеотидную последовательность или биомолекулу;
(c) изменение библиотеки секвенирования одиночных клеток для получения подбиблиотеки,
при этом подбиблиотека содержит увеличенное представительство модифицированных целевых нуклеиновых кислот, содержащих маркерную индексную последовательность, по сравнению с другими модифицированными целевыми нуклеиновыми кислотами, присутствующими в библиотеке секвенирования одиночных клеток, которые не содержат маркерную индексную последовательность;
(d) определение нуклеотидной последовательности модифицированных целевых нуклеиновых кислот, содержащих маркерную индексную последовательность.
2. Способ по п. 1, в котором библиотека секвенирования одиночных клеток содержит нуклеиновые кислоты из множества образцов.
3. Способ по п. 2, в котором множество образцов содержит (i) образцы одной и той же ткани, полученной из разных организмов, (ii) образцы разных тканей из одного организма или (iii) образцы разных тканей из разных организмов.
4. Способ по п. 1, в котором на стадии (b) определяют более одной маркерной индексной последовательности.
5. Способ по п. 1, в котором библиотека секвенирования одиночных клеток содержит целевые нуклеиновые кислоты, представляющие весь геном клеток или ядер или подгруппу генома.
6. Способ по п. 5, в котором подгруппа генома содержит целевые нуклеиновые кислоты, представляющие транскриптом, доступный хроматин, ДНК, конформационное состояние или белки клеток или ядер.
7. Способ по любому из пп. 1-6, в котором изменение предусматривает обогащение модифицированных целевых нуклеиновых кислот, содержащих маркерную индексную последовательность.
8. Способ по п. 7, в котором обогащение включает способ на основе гибридизации.
9. Способ по п. 8, в котором способ на основе гибридизации включает гибридный захват, амплификацию или короткие палиндромные повторы, регулярно расположенные группами (CRISPR) (d)Cas9.
10. Способ по п. 9, в котором изменение предусматривает истощение модифицированных целевых нуклеиновых кислот, которые не содержат маркерную индексную последовательность.
11. Способ по п. 10, в котором истощение предусматривает способ на основе гибридизации.
12. Способ по п. 11, в котором способ на основе гибридизации включает гибридный захват, амплификацию или CRISPR (d)Cas9.
13. Способ по п. 1, в котором биологический признак включает нуклеотидную последовательность, указывающую на видовой тип.
14. Способ по п. 13, в котором видовой тип включает вид клетки.
15. Способ по п. 14, в котором биологический признак включает нуклеотиды субъединицы 16s, субъединицы 18s или нетранскрипционную область ITS.
16. Способ по п. 1, в котором биологический признак включает нуклеотидную последовательность, указывающую на класс клеток.
17. Способ по п. 16, в котором класс клеток имеет паттерн экспрессии, эпигенетический паттерн, рекомбинацию иммунных генов или их комбинацию.
18. Способ по п. 17, в котором эпигенетический паттерн содержит метку метилирования, паттерн метилирования, доступную ДНК или их комбинацию.
19. Способ по п. 1, в котором биологический признак включает нуклеотидную последовательность, указывающую на состояние или риск заболевания.
20. Способ по п. 19, в котором состояние или риск заболевания включает вариантную последовательность ДНК, вариантный паттерн экспрессии или вариантный эпигенетический паттерн, который коррелирует с заболеванием.
21. Способ по п. 20, в котором вариантная последовательность ДНК содержит по меньшей мере один однонуклеотидный полиморфизм.
22. Способ по п. 21, в котором вариантный паттерн экспрессии предусматривает экспрессию биомаркера.
23. Способ по п. 22, в котором вариантный эпигенетический паттерн включает метку метилирования, паттерн метилирования.
24. Способ по п. 1, в котором модифицированные целевые нуклеиновые кислоты содержат связный индекс по меньшей мере 2 специфических для компартмента индексных последовательностей, причем между 2 индексными последовательностями имеется не более 6 нуклеотидов.
25. Способ по п. 24, в котором связный индекс присутствует на каждом конце модифицированных целевых нуклеиновых кислот.
26. Способ по п. 24 или 25, в котором длина связного индекса составляет по меньшей мере 55 нуклеотидов.
27. Способ по любому из пп. 24-26, в котором на модифицированных целевых нуклеиновых кислотах присутствует одна копия связного индекса.
28. Способ по любому из пп. 24-26, в котором на модифицированных целевых нуклеиновых кислотах присутствуют две копии связного индекса.
29. Способ по п. 1, в котором множество модифицированных целевых нуклеиновых кислот из библиотеки секвенирования одиночных клеток представляет по меньшей мере 100 000 различных клеток или ядер.
30. Способ по п. 1, в котором обеспечение библиотеки секвенирования одиночных клеток включает:
обработку образца для получения библиотеки, причем образец представляет собой метагеномический образец, полученный из организма.
31. Способ по п. 30, в котором организм представляет собой млекопитающее.
32. Способ по п. 30 или 31, в котором метагеномический образец содержит ткань, предположительно содержащую симбиотический или патогенный микроорганизм.
33. Способ по п. 32, в котором микроорганизм является прокариотическим или эукариотическим.
34. Способ по любому из пп. 30, 31 или 33, в котором метагеномический образец содержит образец микробиома.
35. Способ по п. 1, в котором обеспечение библиотеки секвенирования одиночных клеток включает:
обработку образца для обеспечения библиотеки, причем образец получен из организма.
36. Способ по п. 35, в котором организм представляет собой млекопитающее.
37. Способ по п. 35, в котором первичный источник нуклеиновых кислот из образца содержит РНК.
38. Способ по п. 37, в котором РНК предусматривает мРНК.
39. Способ по п. 35, в котором первичный источник нуклеиновых кислот из образца содержит ДНК.
40. Способ по п. 39, в котором ДНК предусматривает геномную ДНК всей клетки.
41. Способ по п. 40, в котором геномная ДНК всей клетки содержит нуклеосомы.
42. Способ по п. 35, в котором первичный источник нуклеиновых кислот из образца содержит бесклеточную ДНК.
43. Способ по п. 35, в котором образец содержит раковые клетки.
44. Способ по п. 1, в котором обеспечение библиотеки секвенирования одиночных клеток включает получение библиотеки методом комбинаторного индексирования одиночных клеток, выбранным из секвенирования транскриптома одиночных ядер, секвенирования транскриптома одиночных клеток, секвенирования транскриптома одиночных клеток и хроматина, доступного транспозонам, полногеномного секвенирования одиночных ядер, секвенирования одиночных ядер хроматина, доступного транспозонам, секвенирования эпитопа одиночных клеток, sci-HiC и sci-MET.
45. Способ по п. 44, в котором обеспечение включает обеспечение из каждой клетки или ядра двух разных библиотек секвенирования одиночных клеток.
46. Способ по п. 45, в котором две разные библиотеки секвенирования одиночных клеток выбраны из метода комбинаторного индексирования одиночных клеток, выбранного из секвенирования транскриптома одиночных ядер, секвенирования транскриптома одиночных клеток, секвенирования транскриптома одиночных клеток и хроматина, доступного транспозонам, полногеномного секвенирования одиночных ядер, секвенирования одиночных ядер хроматина, доступного транспозонам, sci-HiC и sci-MET.
47. Способ по п. 1, дополнительно включающий выполнение процедуры секвенирования для определения нуклеотидных последовательностей для нуклеиновых кислот.
48. Способ секвенирования одиночной клетки или ядра, включающий:
(a) процессинг нуклеиновых кислот каждой клетки или ядер в образце путем добавления по меньшей мере одной индексной последовательности, которая идентифицирует членов библиотеки из одной клетки или ядра, где по меньшей мере одна индексная последовательность нуклеиновых кислот каждой клетки или ядер в образце отличается от по меньшей мере одной индексной последовательности нуклеиновых кислот других клеток или ядер в образце, с образованием таким образом индексированной библиотеки для каждой клетки или ядра;
(b) определение индексированных нуклеиновых кислот, связанных с биологическим признаком, где биологический признак включает нуклеотидную последовательность или биомолекулу, и где корреляция биологического признака и индексов, связанных с биологическим признаком, приводит к определению одной или более интересующих индексированных библиотек со стадии (a);
(c) обогащение интересующих индексированных библиотек со стадии (b) с образованием таким образом обогащенной библиотеки, где обогащение включает увеличение представления одной или нескольких представляющих интерес индексированных библиотек; и
(d) секвенирование обогащенной библиотеки со стадии (c).
49. Способ по п. 48, в котором индексированные библиотеки происходят от ДНК, РНК или белка клеток или ядер.
50. Способ по любому из пп. 48 или 49, в котором биологический признак представляет собой ДНК, РНК или белок или их комбинацию.
51. Способ по любому из пп. 48 или 49, в котором процессинг на стадии (а) включает связывание по меньшей мере двух различных индексов с нуклеиновыми кислотами клеток или ядер.
52. Способ по п. 51, в котором по меньшей мере два различных индекса представляют собой связный индекс.
53. Способ по любому из пп. 48 или 49, в котором обогащенная библиотека получена посредством позитивного обогащения.
54. Способ по п. 53, в котором позитивное обогащение предусматривает амплификацию.
55. Способ по п. 53, в котором позитивное обогащение предусматривает захватный агент.
56. Способ по п. 53, в котором позитивное обогащение предусматривает твердую подложку.
57. Способ по п. 53, в котором обогащенная библиотека получена посредством негативного обогащения.
58. Способ по любому из пп. 48 или 49, в котором определение интересующей индексированной библиотеки на стадии (c) предусматривает секвенирование индексных последовательностей.
59. Способ секвенирования одиночной клетки или ядра, включающий:
(a) обеспечение образца, причем образец содержит множество ядер или клеток;
(b) связывание первой индексной последовательности с каждым ядром или клеткой в образце;
(c) разделение образца на множество компартментов;
(d) связывание второй индексной последовательности с каждым ядром или клеткой из множества компартментов, где комбинация первой и второй индексных последовательностей идентифицирует членов библиотеки из одной клетки или ядра;
(e) объединение множества компартментов;
(f) секвенирование объединенных компартментов;
(g) определение комбинации первой и второй индексных последовательностей, связанных с биологическим признаком, где биологический признак включает нуклеотидную последовательность или биомолекулу;
(h) обогащение биологического признака из объединенных компартментов с использованием идентифицированной комбинации первой и второй индексных последовательностей со стадии (g), где обогащение включает увеличение представления одной или нескольких индексированных библиотек, включающих идентифицированную комбинацию первой и второй индексных последовательностей со стадии (g).
| VITAK S.A | |||
| et al., Sequencing thousands of single-cell genomes with combinatorial indexing, Nat Methods, 2017, v | |||
| Паровоз для отопления неспекающейся каменноугольной мелочью | 1916 |
|
SU14A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Дровопильное устройство | 1921 |
|
SU302A1 |
| JIANG L | |||
| et al., GiniClust: detecting rare cell types from single-cell gene expression data with Gini index, Genome Biol, 2016, v | |||
| Печь для сжигания твердых и жидких нечистот | 1920 |
|
SU17A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ НОВОГО ПОКОЛЕНИЯ | 2014 |
|
RU2698125C2 |
Авторы
Даты
2025-04-24—Публикация
2020-12-18—Подача