ОБЛАСТЬ ТЕХНИКИ
[1] Настоящее изобретение касается области техники «нуклеиновые кислоты», в частности молекулы олигомерной нуклеиновой кислоты, связанной с активацией гена, и ее применения.
УРОВЕНЬ ТЕХНИКИ
[2] Спинальная мышечная атрофия (СМА) - это наследственное нервно-мышечное заболевание, характеризующееся прогрессирующей слабостью и атрофией скелетных мышц и дыхательной мускулатуры, которое чаще, чем какие-либо другие наследственные заболевания, является причиной смерти у детей младше двух лет. В соответствии с клинической классификацией, СМА подразделяют на четыре типа в зависимости от возраста начала заболевания и степени его тяжести - от наиболее тяжелой СМА I типа до наиболее легкой СМА IV типа (1). СМА является одним из самых распространенных аутосомно-рецессивных заболеваний детского возраста. Частота возникновения СМА у живых новорожденных составляет 1/11 000, в то время как частота встречаемости у взрослых носителей колеблется от менее 1/67 до 1/40 (2); при этом в китайской популяции частота носительства составляет около 1/42 (3).
[3] СМА вызывается мутацией гена SMN1 (белка выживания моторных нейронов-1, Survival of Motor Neuron 1), а пациенты с СМА являются носителями различного количества копий высокогомологичного гена SMN2 (4; 5). Ген SMN1 и ген SMN2 расположены в области 5q13 хромосомы и кодируют один и тот же белок выживания моторных нейронов (SMN). Ген SMN2 отличается от гена SMN1 на 11 нуклеотидов, в то время как остальные нуклеотиды у них совпадают. Из 11 нуклеотидов только один расположен в кодирующей области SMN2: это - C (цитозин), шестой нуклеотид экзона 7, который заменен на T (C6T) и находится в области экзонного энхансера сплайсинга. Такое замещение не меняет кодирующую последовательность, но изменяет эффективность сплайсинга экзона 7 SMN2, что приводит к тому, что большинство матричных РНК-предшественников (пре-мРНК) SMN2 теряют экзон 7 при сплайсинге, а также к трансляции очень нестабильного мутантного белка SMN (SMNΔ7) и небольшого количества полноразмерного белка SMN с нормальной функцией (5; 6). Вследствие этого у пациентов с СМА функция полноразмерного белка SMN, который в небольшом количестве синтезируется с гена SMN2, оказывается недостаточной для того, чтобы компенсировать потерю белка SMN, вызванную мутацией гена SMN1. Таким образом, любой метод, способный повысить уровень экспрессии полноразмерного белка SMN, считываемого с гена SMN2, может быть полезным для лечения СМА.
[4] Эффективность стратегий модуляции сплайсинга SMN2 для увеличения количества полноразмерного белка с помощью низкомолекулярных соединений или олигонуклеотидов продемонстрирована в моделях на животных и в клинических исследованиях (7-11). Так, в 2016 году FDA одобрило антисмысловой олигонуклеотид Спинраза в качестве первого олигонуклеотидного препарата для лечения СМА у детей и взрослых. Хотя регуляция сплайсинга пре-мРНК является эффективной стратегией терапии, ее эффективность ограничена доступным количеством пре-мРНК SMN2. Если уровень экспрессии пре-мРНК SMN2 самой по себе не меняется или является низким, то количество полноразмерного белка SMN, индуцируемого терапией, также ограничено. Другая стратегия повышения уровня белка SMN состоит в повышении уровня транскрипции SMN2, с тем чтобы экспрессия полноразмерного белка SMN2 также увеличивалась. Исследования показали, что низкомолекулярные ингибиторы гистондеацетилазы (ингибиторы HDAC) могут активировать ген SMN2 и повышать уровень транскрипции SMN2 за счет подавления активности гистондеацетилазы, что приводит к выработке большего количества пре-мРНК SMN2; эффективность такой стратегии продемонстрирована в моделях СМА на животных (12; 13). Однако низкомолекулярные ингибиторы HDAC не продемонстрировали клинической эффективности у пациентов (людей) с СМА. Возможная причина состоит в том, что, подавляя активность гистондеацетилазы, ингибиторы HDAC могут активировать многие другие гены, поэтому они не проявляют специфичности и высокой активности в отношении промотора SMN2 (14; 15). Предметом настоящего изобретения является новая молекула короткой активирующей нуклеиновой кислоты, которая способна активировать транскрипцию SMN2 с высокой специфичностью, действуя на промотор гена SMN2.
РАСКРЫТИЕ ИЗОБРЕТЕНИЯ
[5] Для того чтобы решить упомянутую выше проблему, в рамках настоящего изобретения предложена молекула короткой активирующей нуклеиновой кислоты, в частности короткой активирующей РНК (каРНК), которая помогает при лечении заболеваний или состояний, вызванных дефицитом полноразмерного белка SMN, таких как СМА, путем активации/стимуляции транскрипции SMN2 и повышения уровня экспрессии полноразмерного белка SMN за счет механизма активации РНК (акт. РНК).
[6] Одним из аспектов настоящего изобретения является молекула короткой активирующей нуклеиновой кислоты (в частности молекула каРНК), активирующая/стимулирующая экспрессию гена SMN2 в клетке, причем одна цепь молекулы короткой активирующей нуклеиновой кислоты по крайней мере на 75% гомологична или комплементарна фрагменту длиной 16-35 нуклеотидов в промоторной области гена SMN2, благодаря чему она активирует или стимулирует экспрессию гена, причем промоторная область содержит последовательность из 2000 пар оснований (п. о.), расположенную выше сайта начала транскрипции. В конкретном случае, одна цепь молекулы короткой активирующей нуклеиновой кислоты по крайней мере на 75% (например, по крайней мере на примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или примерно 99%) гомологична или комплементарна фрагменту длиной 16-35 постоянных нуклеотидов в положениях от -1639 до -1481 (SEQ ID №: 476), в положениях от -1090 до -1008 (SEQ ID №: 477), в положениях от -994 до -180 (SEQ ID №: 478) или в положениях от -144 до -37 (SEQ ID №: 479) выше сайта начала транскрипции промотора гена SMN2. В более конкретном случае, одна цепь молекулы короткой активирующей нуклеиновой кислоты по крайней мере на 75% (например, по крайней мере на примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или примерно 99%) гомологична или комплементарноа любой последовательности нуклеотидов, выбранной из SEQ ID №: 315-471.
[7] Согласно настоящему изобретению, молекула короткой активирующей нуклеиновой кислоты содержит фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты, причем фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты содержат комплементарные области, способные формировать между двумя фрагментами структуру двухцепочечной нуклеиновой кислоты, которая может индуцировать экспрессию гена SMN2 в клетке по механизму акт. РНК. Фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты молекулы короткой активирующей нуклеиновой кислоты могут находиться как в двух разных цепях нуклеиновой кислоты, так и в одной цепи нуклеиновой кислоты. Если фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты находятся в двух разных цепях, то как минимум одна цепь молекулы короткой активирующей нуклеиновой кислоты имеет «липкий» 3'-конец в длиной от 0 до 6 нуклеотидов, а, предпочтительно, обе цепи имеют «липкий» 3'-конец длиной от 2 до 3 нуклеотидов, причем нуклеотидом «липкого» конца является предпочтительно dT. Если фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты находятся в одной цепи нуклеиновой кислоты, то молекула короткой активирующей нуклеиновой кислоты предпочтительно представляет собой «шпильку», образованную одной цепью нуклеиновой кислоты, причем комплементарные области фрагмента смысловой нуклеиновой кислоты и фрагмента антисмысловой нуклеиновой кислоты образуют между собой структуру двухцепочечной нуклеиновой кислоты. В упомянутой выше молекуле короткой активирующей нуклеиновой кислоты фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты имеют длину от 16 до 35 нуклеотидов (например, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 или 35 нуклеотидов).
[8] В одном варианте осуществления изобретения смысловая цепь молекулы короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, по крайней мере на 75% (например, по крайней мере на примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или примерно 99%) гомологична любой последовательности нуклеотидов, выбранной из SEQ ID №: 1-157, а антисмысловая цепь молекулы короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, по крайней мере на 75% (например, по крайней мере на примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или примерно 99%) гомологична любой последовательности нуклеотидов, выбранной из SEQ ID №: 158-314. В конкретном случае, смысловая цепь молекулы короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, содержит, представляет собой или состоит из любой последовательности нуклеотидов, выбранной из SEQ ID №: 1-157; а антисмысловая цепь молекулы короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, содержит, представляет собой или состоит из любой последовательности нуклеотидов, выбранной из SEQ ID №: 158-314.
[9] В молекуле короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, все нуклеотиды могут быть природными или модифицированными нехимическим путем, или как минимум один нуклеотид модифицирован химически, причем химическая модификация представляет собой одну или несколько модификаций из числа перечисленных ниже:
(1) модификация фосфодиэфирной связи между нуклеотидами в нуклеотидной последовательности молекулы короткой активирующей нуклеиновой кислоты;
(2) модификация 2'-OH рибозы в нуклеотидной последовательности молекулы короткой активирующей нуклеиновой кислоты; и
(3) модификация нуклеотидного основания в молекуле короткой активирующей нуклеиновой кислоты.
[10] Химическая модификация, описанная в данном документе, представляет собой модификацию, хорошо известную специалистам в данной области; при этом модификация фосфодиэфирной связи относится к модификации кислорода в фосфодиэфирной связи, в том числе фосфоротиоатной и боранофосфатной модификации. Оба типа модификации могут стабилизировать структуру каРНК и поддерживать высокую специфичность и высокую аффинность при спаривании оснований.
[11] Модификация рибозы относится к модификации 2'-OH в пентозном кольце нуклеотида, т. е. к введению определенных заместителей в гидроксильные положения рибозы, например 2'-фтор-модификация, 2'-оксиметил-модификация, 2'-оксиэтилиденметокси-модификация, 2,4'-динитрофенол-модификация, модификация для получения закрытой нуклеиновой кислоты (LNA), 2'-амино-модификация и 2'-деокси-модификация.
[12] Модификация основания относится к модификации нуклеотидного основания, например 5'-бромурацил-модификация, 5'-йодурацил-модификация, N-метилурацил-модификация и 2,6-диаминопурин-модификация.
[13] Эти модификации могут повысить биодоступность молекулы короткой активирующей нуклеиновой кислоты, увеличить аффинность связывания с целевой последовательностью, а также повысить устойчивость к гидролизу под действием нуклеаз в клетке.
[14] Кроме того, для увеличения эффекта, оказываемого короткой активирующей нуклеиновой кислотой в клетке, к концу смысловой цепи или антисмысловой цепи молекулы короткой активирующей нуклеиновой кислоты можно присоединить липофильную группу, например, холестерин, что позволит облегчить трансмембранный перенос через двухслойную липидную клеточную мембрану и оболочку ядра для ее связывания с промотором гена-мишени в клеточном ядре.
[15] Попав в клетку, молекула короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, может эффективно активировать или стимулировать экспрессию гена SMN2 в клетке, предпочтительно стимулируя экспрессию не менее чем на 10%.
[16] Одним из аспектов настоящего изобретения является нуклеиновая кислота, кодирующая молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе. В одном варианте осуществления изобретения молекула короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, представляет собой молекулу короткой активирующей РНК (каРНК). В одном варианте осуществления изобретения нуклеиновая кислота представляет собой молекулу ДНК.
[17] Одним из аспектов настоящего изобретения является клетка, содержащая молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе. В одном варианте осуществления изобретения клетка представляет собой клетку млекопитающего, предпочтительно человека. Упомянутая выше клетка может существовать in vitro, например в виде клеточной линии или клеточного штамма, либо присутствовать в организме млекопитающего, например человека.
[18] Другим аспектом настоящего изобретения является композиция, например фармацевтическая композиция, которая содержит упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, и, опционально, фармацевтически приемлемый носитель. В одном варианте осуществления изобретения фармацевтически приемлемый носитель включает водный носитель, липосому, высокомолекулярный полимер или полипептид. В одном варианте осуществления изобретения фармацевтически приемлемый носитель выбирается из водного носителя, липосомы, высокомолекулярного полимера или полипептида. В одном варианте осуществления изобретения водный носитель может представлять собой, например, воду, не содержащую РНКаз, или буфер, не содержащий РНКаз. Композиция может содержать упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, в концентрации 1-150 нМ (например, 1-100 нМ, 1-50 нМ, 1-20 нМ, 10-100 нМ, 10-50 нМ, 20-50 нМ, 20-100 нМ или 50 нМ).
[19] Другой аспект настоящего изобретения касается применения упомянутой выше молекулы короткой активирующей нуклеиновой кислоты, нуклеиновой кислоты, кодирующей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композиции, содержащей упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, для приготовления препарата, служащего для активации/стимуляции экспрессии гена SMN2 в клетке.
[20] Настоящее изобретение также касается метода активации/стимуляции экспрессии гена SMN2 в клетке, где данный метод включает введение в клетку упомянутой выше молекулы короткой активирующей нуклеиновой кислоты, нуклеиновой кислоты, кодирующей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композиции, содержащей упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе.
[21] Упомянутую выше молекулу короткой активирующей нуклеиновой кислоты, нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композицию, содержащую упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, можно ввести непосредственно в клетку или произвести в клетке по нуклеотидной последовательности, кодирующей молекулу короткой активирующей нуклеиновой кислоты, введенной в клетку. Клетка предпочтительно представляет собой клетку млекопитающего, а более предпочтительно - человека. Упомянутая выше клетка может существовать in vitro, например в виде клеточной линии или клеточного штамма, либо присутствовать в организме млекопитающего, например человека. Человеческим организмом является пациент, у которого имеется заболевание или симптом, вызванные снижением экспрессии полноразмерного белка SMN по причине мутации, делеции или недостаточной экспрессии гена SMN1 и/или вследствие недостаточной экспрессии полноразмерного белка SMN2. Молекулу короткой активирующей нуклеиновой кислоты, нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композицию, содержащую упомянутую выше молекулу короткой активирующей нуклеиновой кислоты или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, вводят в количестве, достаточном для лечения указанного заболевания или симптома. В частности, симптом, вызванный дефицитом полноразмерного белка SMN, возникшим из-за мутации или делеции гена SMN1 и/или из-за недостаточной экспрессии полноразмерного белка SMN2, включает, например, спинальную мышечную атрофию. В одном варианте осуществления изобретения заболевание, вызванное недостаточной экспрессией полноразмерного белка SMN либо мутацией или делецией гена SMN1 или недостаточной экспрессией полноразмерного белка SMN1, представляет собой спинальную мышечную атрофию. В одном варианте осуществления изобретения спинальная мышечная атрофия, описанная в данном документе, включает СМА I типа, II типа, III типа и IV типа.
[22] Другим аспектом настоящего изобретения является изолированный целевой сайт молекулы короткой активирующей нуклеиновой кислоты на гене SMN2, где целевой сайт представляет собой непрерывную нуклеотидную последовательность длиной от 16 до 35 нуклеотидов в промоторной области гена SMN2; предпочтительно целевой сайт представляет собой любую последовательность, выбранную из SEQ ID № 476-479, длиной от 16 до 35 постоянных нуклеотидов. В конкретном случае, целевой сайт содержит или выбран из любых нуклеотидных последовательностей SEQ ID №: 315-471.
[23] Другой аспект настоящего изобретения касается метода лечения заболевания, вызванного недостаточной экспрессией полноразмерного белка SMN по причине мутации или делеции гена SMN1 и/или вследствие недостаточной экспрессии полноразмерного белка SMN2, у индивидуума. Метод состоит во введении индивидууму терапевтически эффективной дозы упомянутой выше молекулы короткой активирующей нуклеиновой кислоты, нуклеиновой кислоты, кодирующей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композиции, содержащей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе. Индивидуум может быть млекопитающим, например, человеком. В одном варианте осуществления изобретения заболевание, вызванное недостаточной экспрессией полноразмерного белка SMN из-за мутации гена SMN1, может включать, например, спинальную мышечную атрофию. В одном варианте осуществления изобретения заболевание, вызванное недостаточной экспрессией полноразмерного белка SMN из-за мутации или делеции гена SMN1 и/или недостаточной экспрессии полноразмерного белка SMN2, представляет собой спинальную мышечную атрофию. В одном варианте осуществления изобретения спинальная мышечная атрофия, описанная в данном документе, включает СМА I типа, II типа, III типа и IV типа.
[24] Другой аспект настоящего изобретения касается применения молекулы короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, нуклеиновой кислоты, кодирующей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или композиции, содержащей молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, или нуклеиновую кислоту, кодирующую молекулу короткой активирующей нуклеиновой кислоты, раскрытой в данном документе, для приготовления препарата для лечения заболевания или состояния, вызванного недостаточной экспрессией полноразмерного белка SMN вследствие мутации или делеции гена SMN1 или недостаточной экспрессии полноразмерного белка SMN1 и/или недостаточной экспрессии полноразмерного белка SMN2. Индивидуум может быть млекопитающим, например человеком. В одном варианте осуществления изобретения заболевание, вызванное недостаточной экспрессией полноразмерного белка SMN вследствие мутации или делеции гена SMN1 или недостаточной экспрессии полноразмерного белка SMN1 и/или недостаточной экспрессии полноразмерного белка SMN2, может включать, например, спинальную мышечную атрофию. В одном варианте осуществления изобретения заболевание, вызванное недостаточной экспрессией полноразмерного белка SMN вследствие мутации или делеции гена SMN1 или недостаточной экспрессии полноразмерного белка SMN1 и/или недостаточной экспрессии белка SMN2, является спинальной мышечной атрофией. В одном варианте осуществления изобретения спинальная мышечная атрофия, описанная в данном документе, включает СМА I типа, II типа, III типа и IV типа.
[25] Преимущества настоящего изобретения
[26] Молекула короткой активирующей нуклеиновой кислоты, активирующая/стимулирующая экспрессию гена SMN2, которая описана в данном документе (например, молекула каРНК), может эффективно и специфично стимулировать экспрессию гена SMN2 и повышать уровень экспрессии полноразмерной мРНК SMN2, вызывая лишь незначительные токсические реакции и нежелательные эффекты. Она также может применяться для приготовления препарата для лечения расстройств, связанных с недостаточной экспрессией полноразмерного белка SMN, и заболеваний или состояний, вызванных мутацией или делецией гена SMN1 или недостаточной экспрессией полноразмерного белка SMN1 и/или недостаточной экспрессией полноразмерного белка SMN2.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
[27] РИС. 1 представляет собой схематическое изображение структуры гена SMN2 с указанием промоторной области длиной 2 kb, использованной для дизайна каРНК, и расположения праймеров для ПЦР. РИС. 1A иллюстрирует структуру гена SMN2 и промоторную область длиной 2 kb, использованную для дизайна каРНК. РИС. 1B иллюстрирует расположение праймеров для ОТ-ПЦР, используемых для амплификации мРНК SMN2. SMN F1+SMN R1 - пара праймеров для ОТ-кПЦР, используемых для высокопроизводительного скрининга каРНК; SMN F2+SMN R2 - пара праймеров для ОТ-кПЦР, используемых для валидации прототипов каРНК; SMN-экзон6-F+SMN-экзон8-R - пара праймеров для обычной ОТ-ПЦР.
[28] РИС. 2 иллюстрирует изменения уровня экспрессии мРНК SMN, опосредованные молекулами короткой активирующей нуклеиновой кислоты. 980 каРНК, направленных к промотору SMN2, были использованы для независимой трансфекции клеток почки эмбриона человека HEK293T. Через 72 ч после трансфекции определяли уровень экспрессии мРНК SMN с помощью одноэтапной ОТ-кПЦР. На рисунке показаны относительные кратные изменения уровня экспрессии мРНК SMN, вызванные каждой из 980 каРНК, которые представлены в порядке уменьшения кратности изменения.
[29] РИС. 3 иллюстрирует активные участки для каРНК к промотору SMN2. 980 каРНК, направленных к промотору SMN2, были использованы для независимой трансфекции клеток почки эмбриона человека HEK293T, а через 72 ч после трансфекции определяли уровень экспрессии мРНК SMN с помощью одноэтапной ОТ-кПЦР. На рисунке показаны кратные изменения уровня экспрессии мРНК SMN, вызванные каждой каРНК, в сравнении с контролем (холостой раствор), которые представлены в порядке приближения участка связывания каРНК в промоторе (от -1949 до -37) к сайту начала транскрипции (TSS) SMN2. На рисунке также показано расположение четырех активных участков для каРНК (прямоугольники H1-H4). Числа над прямоугольниками показывают границы активных участков (относительно TSS-сайта SMN2).
[30] РИС. 4 иллюстрирует количественную оценку способности 50 выбранных случайным образом каРНК активировать экспрессию гена SMN в клетках HEK293T. Клетки HEK293T трансфицировали указанными каРНК (n=50, конечная концентрация: 20 нМ). Через 72 ч из трансфицированных клеток выделяли РНК с помощью набора Qiagen RNeasy. После обратной транскрипции проводили кПЦР-амплификацию генов SMN с помощью системы 7500FAST для ПЦР в режиме реального времени; при этом определяли уровни мРНК генов HPRT1 и TBP, а их средние геометрические значения использовали в качестве внутренних референтных значений для нормирования нагрузки РНК. На оси Y представлены изменения уровня экспрессии мРНК SMN в каждом образце, обработанном каРНК, в сравнении с контролем (холостой раствор) после нормирования на уровни экспрессии внутренних референтных генов. dsCon2- контроль с двуспиральной РНК, а siSMN2-1 - контроль с киРНК SMN2.
[31] РИС. 5 представляет собой схематическую иллюстрацию метода ПЦР-амплификации с последующим расщеплением рестриктазой DdeI для определения экспрессии SMN. РИС. 5A иллюстрирует различия между геном SMN1 и геном SMN2. Генетическая вариация G?A в экзоне 8 гена SMN2 создает сайт рестрикции для рестриктазы Ddel. ПЦР-амплификация кДНК с использованием праймеров SMN-экзон6-F и SMN-экзон8-R позволяла получить полноразмерный продукт SMN (SMN2FL) (507 п.о.) (РИС. 5B) и/или продукт с пропуском/делецией экзона 7 (SMN2Δ7) (453 п.о.) (РИС. 5C). Чтобы различить продукты SMN2 от продуктов SMN1, продукты ПЦР расщепляли ферментом DdeI и разделяли в агарозном геле. Продукт, полученный из полноразмерного гена SMN1, не подвергался расщеплению, в то время как продукт полноразмерного гена SMN2, расщеплялся на два фрагмента (392 п.о. и 115 п.о.) (РИС. 5B), а продукт гена SMN2Δ7 расщеплялся на фрагменты длиной 338 п.о. и 115 п.о. (РИС. 5C).
[32] На РИС. 6 изображены электрофореграммы, иллюстрирующие способность 50 выбранных случайным образом каРНК повышать уровень экспрессии мРНК полноразмерного гена SMN2 в клетках HEK293T. Клетки HEK293T трансфицировали указанными каРНК (n=50, конечная концентрация: 20 нМ). В качестве контроля использовали холостой раствор, dsCon2, siSMN2-1 и вектор-опосредованную гиперэкспрессию (вектор SMN) (полосы 51, 52, 53 и 54 соответственно). Через 72 ч из трансфицированных клеток выделяли РНК с помощью набора Qiagen RNeasy. После обратной транскрипции проводили обычную амплификацию методом ОТ-ПЦР; при этом в качестве внутреннего контроля для нагрузки РНК амплифицировали ген HPRT1. Продукт амплификации гена SMN расщепляли с помощью рестриктазы DdeI, затем подвергали гель-электрофорезу и количественно оценивали интенсивность полос. Продукт амплификации гена HPRT1 анализировали методом гель-электрофореза непосредственно, то есть без расщепления. На РИС. 6A изображена гель-электрофореграмма продукта амплификации гена SMN после расщепления ферментом DdeI; на РИС. 6B изображена гель-электрофореграмма продукта амплификации гена HPRT1; на РИС. 6C приведен список названий образцов для полос на РИС. 6A и РИС. 6B. FL: продукт амплификации полноразмерного гена; SMN2Δ7: продукт с пропуском/делецией экзона 7; SMN2 partial: фрагмент после расщепления, характерный для SMN2. Черные стрелки указывают каРНК, способные увеличивать соотношение между продуктом полноразмерного гена SMN2 и продуктом гена с пропуском/делецией экзона 7.
[33] РИС. 7 иллюстрирует способность 50 выбранных случайным образом каРНК индуцировать экспрессию общей мРНК SMN2 и полноразмерного SMN2. Количественная оценка интенсивности полос на электрофореграмме на РИС. 6 позволяла определить уровни общей мРНК SMN2 (РИС. 7A) и соотношение между уровнями мРНК SMN2 и мРНК SMN2Δ7 (РИС. 7B). Для каждого образца значения нормировали на интенсивность полосы HPRT1 и представляли в виде значений в сравнении с контролем (холостой раствор).
[34] РИС. 8 показывает дозовую зависимость эффекта каРНК, то есть ее способности увеличивать экспрессию мРНК и белка, кодируемых генами SMN и SMN2FL. Две каРНК SMN2 (RAG6-281 и RAG6-550) использовали для независимой трансфекции клеток HEK293T в указанных концентрациях (1 нМ, 10 нМ, 20 нМ, 50 нМ и 100 нМ) в течение 72 ч. Из трансфицированных клеток выделяли общую РНК и подвергали ее обратной транскрипции; белок выделяли и анализировали методом вестерн-блоттинга. РИС. 8A иллюстрирует относительный уровень экспрессии общей мРНК SMN, определенный методом ОТ-кПЦР. Также амплифицировали гены TBP и HPRT1, а средние геометрические значения их уровней экспрессии использовали в качестве внутренних референтных значений. На РИС. 8B показаны уровни мРНК SMN, амплифицированных с помощью обычной ОТ-ПЦР, с последующим расщеплением ферментом DdeI и электрофорезом. Ген HPRT1 амплифицировали и использовали в качестве внутреннего контроля. Значения (после надписи SMN2 FL/Δ7), указанные под электрофореграммой, представляют собой количественные соотношения между полноразмерным SMN2 и SMN2Δ7 для каждого обработанного образца, в сравнении с соотношением для образца, обработанного холостым раствором. На РИС. 8C показана экспрессия белка SMN, выявленного методом вестерн-блоттинга; в качестве внутреннего референтного белка использовали тубулин α/β. M: контроль после трансфекции холостым раствором; C: контроль dsCon2 для дсРНК; FL: полноразмерный продукт амплификации; SMN2Δ7: продукт с пропуском/делецией экзона 7.
[35] РИС. 9 иллюстрирует результат генотипирования новорожденных мышей с экспериментальной СМА. Детеныши были получены скрещиванием мышей Smn1+/-, SMN2-/- с мышами Smn1-/-, SMN2+/+. Генотип определяли с помощью ПЦР геномной ДНК. Использовали мышей со следующими двумя генотипами: мыши с СМА типа 1 (СМА 1) - носители гомозиготной делеции гена Smn мыши и гетерозиготного нокина SMN2 человека с генотипом Smn1-/-, SMN2+/-; обычные контрольные мыши (Het) - носители гетерозиготной делеции гена Smn мыши и гетерозиготного нокина SMN2 человека с генотипом Smn1+/-, SMN2+/-. ПЦР-продукт для мышей СМА 1 обнаруживался в виде единственной полосы 160 п.о., а для мышей Het - в виде двух полос: 160 п.о. и 180 п.о.
[36] РИС. 10 иллюстрирует улучшение моторной функции мышей СМА I после терапии препаратом каРНК SMN2. Новорожденных мышей разделяли на 4 группы: контрольная группа нормальных мышей (Het), контрольная группа мышей СМА I (не получавших препарат), группа каРНК SMN2 RAG6-539 в смеси с реагентом in vivo-jetPEI (DS06-0013B, 1 мг/мл) и группа каРНК SMN2 RAG6-538 в смеси с реагентом HKP (DS06-0002B, 2 мг/мл). Новорожденные мыши получали интрацеребровентрикулярную (ИЦВ) инъекцию в 1-й день после рождения (P1). Моторную функцию у мышей оценивали с помощью теста на рефлекс переворачивания в день P7 или P8.
ПОДРОБНОЕ ОПИСАНИЕ
[37] В настоящем изобретении связанные термины определяются следующим образом.
[38] Термин «комплементарный», используемый в данном документе, относится к способности образовывать пары оснований между двумя цепями олигонуклеотида. Пары оснований в основном образуются за счет водородных связей между нуклеотидами в антипараллельных цепях олигонуклеотида. Основания комплементарных цепей олигонуклеотида могут спариваться, как описано Уотсоном - Криком (например, A с T, A с U и C с G), или любым другим способом, который позволяет сформировать двойную спираль (например, путем образования пар оснований Хугстина или обратных водородных связей Хугстина).
[39] Комплементарность включает в себя полную комплементарность и неполную комплементарность. «Полная комплементарность», или «100%-ная комплементарность», означает, что каждый нуклеотид из первой олигонуклеотидной цепи может образовывать водородную связь с нуклеотидом в соответствующем положении второй олигонуклеотидной цепи в двухцепочечной области молекулы двухцепочечного олигонуклеотида, и при этом ни одна пара оснований не будет «неправильно спаренной». «Неполная комплементарность» означает, что не все нуклеотиды двух цепей связаны между собой водородными связями. Например, в случае двух олигонуклеотидных цепей, каждая длиной 20 нуклеотидов в двухцепочечной области, если водородную связь способны образовать только две пары оснований в этой двухцепочечной области, то олигонуклеотидные цепи обладают комплементарностью в 10%. В том же примере, если водородную связь способны образовать 18 пар оснований в этой двухцепочечной области, то олигонуклеотидные цепи обладают комплементарностью в 90%. Существенная комплементарность подразумевает комплементарность не менее примерно 75%, примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или 99%.
[40] Термин «олигонуклеотид», используемый в данном документе, относится к полимерам или нуклеотидам и включает, помимо прочего, одноцепочечные или двухцепочечные молекулы ДНК, РНК или гибридной ДНК/РНК, олигонуклеотидные цепи, содержащие регулярно и нерегулярно чередующиеся дезоксирибозные и рибозные участки, а также модифицированные, природные или неприродные каркасы для таких олигонуклеотидов. Олигонуклеотидом, активирующим транскрипцию целевого гена, описанным в данном документе, является молекула короткой активирующей нуклеиновой кислоты.
[41] Термины «олигонуклеотидная цепь» и «олигонуклеотидная последовательность», используемые в данном документе, могут использоваться на равных основаниях и относятся к общему термину для коротких нуклеотидных последовательностей, имеющих менее 35 оснований (включая нуклеотиды в дезоксирибонуклеиновой кислоте (ДНК) или рибонуклеиновой кислоте (РНК)). В настоящем изобретении длина олигонуклеотидной цепи может быть любой, в пределах от 16 до 35 нуклеотидов.
[42] Термин «ген», используемый в данном документе, относится ко всем нуклеотидным последовательностям, необходимым для кодирования полипептидной цепи или транскрипции функциональной РНК. «Ген» может быть эндогенным либо частично или полностью рекомбинантным геном для клетки-хозяина (например, если в клетку-хозяина вводятся экзогенный олигонуклеотид и кодирующая промотор последовательность, или если в клетку-хозяина вводятся гетерогенный промотор, соседствующий с эндогенной кодирующей последовательностью). Например, термин «ген» включает в себя последовательность нуклеиновых кислот, состоящую из экзонов и интронов. Последовательностями, кодирующими белок, являются, например, последовательности, состоящие из экзонов в открытой рамке считывания между старт-кодоном и стоп-кодоном, а термин «ген», используемый в данном документе, может включать регуляторную последовательность гена, например последовательность промотора, энхансера и все другие последовательности, известные специалистам в данной области, служащие для контроля транскрипции, экспрессии или активности другого гена, независимо от того, содержит ли ген кодирующую последовательность или некодирующую последовательность. В одном случае, например, термин «ген» может использоваться для описания функциональной нуклеиновой кислоты, содержащей регуляторную последовательность, такую как промотор или энхансер. Экспрессия рекомбинантного гена может контролироваться гетерогенными регуляторными последовательностями одного или нескольких типов.
[43] Термин «целевой ген», используемый в данном документе, может относиться к последовательностям нуклеиновых кислот, трансгенам, вирусным или бактериальным последовательностям, хромосомам или внехромосомным генам, которые обычно присутствуют в организме и/или могут быть перенесены путем транзитной или стабильной трансфекции или инкорпорированы в клетки или клеточный хроматин. Целевой ген может быть геном, кодирующим белок, или геном, не кодирующим белок (например, ген микроРНК и геном длинной некодирующей РНК). Целевой ген, как правило, содержит последовательность промотора, а позитивная регуляция целевого гена может быть достигнута путем дизайна молекулы короткой активирующей нуклеиновой кислоты, имеющей идентичную последовательность (также называемую гомологией) с последовательностью промотора, которая характеризуется способностью стимулировать (ап-регулировать) экспрессию целевого гена. «Последовательность промотора целевого гена» относится к некодирующей последовательности целевого гена, и ссылка на последовательность промотора целевого гена во фразе «комплементарна последовательности промотора целевого гена» настоящего изобретения означает кодирующую цепь последовательности, также известную как нематричная цепь, т. е. последовательность нуклеиновых кислот, которая совпадает с кодирующей последовательностью гена. «Целевая последовательность» относится к фрагменту последовательности промотора целевого гена, которая гомологична или комплементарна смысловой олигонуклеотидной цепи или антисмысловой олигонуклеотидной цепи в молекуле короткой активирующей нуклеиновой кислоты.
[44] Термины «смысловая цепь» и «смысловая олигонуклеотидная цепь», используемые в данном документе, могут использоваться на равных основаниях, и смысловая олигонуклеотидная цепь молекулы короткой активирующей нуклеиновой кислоты относится к первой цепи нуклеиновой кислоты, имеющей последовательность, гомологичную кодирующей цепи последовательности промотора целевого гена в двуспиральной молекуле короткой активирующей нуклеиновой кислоты.
[45] Термины «антисмысловая цепь» и «антисмысловая олигонуклеотидная цепь», используемые в данном документе, могут использоваться на равных основаниях, и антисмысловая олигонуклеотидная цепь молекулы короткой активирующей нуклеиновой кислоты относится ко второй цепи нуклеиновой кислоты, которая комплементарна смысловой олигонуклеотидной цепи в двуспиральной молекуле короткой активирующей нуклеиновой кислоты.
[46] Термин «кодирующая цепь», используемый в данном документе, относится к цепи ДНК целевого гена, которую нельзя использовать для транскрипции, а нуклеотидная последовательность данной цепи совпадает с последовательностью РНК, получаемой в результате транскрипции (в РНК T из ДНК заменяется на U). Кодирующая цепь последовательности двухцепочечной ДНК промотора целевого гена, описанная в данном документе, относится к последовательности промотора той же самой цепи ДНК, что и кодирующая цепь ДНК целевого гена.
[47] Термин «матричная цепь», используемый в данном документе, относится к другой цепи, комплементарной кодирующей цепи двухцепочечной ДНК целевого гена, т. е. к цепи, которую, как матрицу, можно транскрибировать в РНК, и эта цепь комплементарна транскрибированной РНК (A с U, а G с C). В процессе транскрипции РНК-полимераза связывается с матричной цепью, двигается в направлении 3'→5' матричной цепи и за счет катализа обеспечивает синтез РНК в направлении 5'→3'. Матричная цепь последовательности двухцепочечной ДНК промотора целевого гена, описанная в данном документе, относится к последовательности промотора той же самой цепи ДНК, что и матричная цепь ДНК целевого гена.
[48] Термин «промотор», используемый в данном документе, относится к последовательности, которая пространственно связана с последовательностью нуклеиновой кислоты, кодирующей белок или РНК, и играет регуляторную роль в транскрипции последовательности нуклеиновой кислоты, кодирующей белок или РНК. Обычно промотор эукариотического гена состоит из 100-5000 пар оснований, однако этот диапазон длины не предназначен для ограничения термина «промотор», используемого в данном документе. Хотя последовательность промотора обычно расположена на 5'-конце последовательности, кодирующей белок или РНК, она также может присутствовать в последовательностях экзона и интрона.
[49] Термин «сайт начала транскрипции», используемый в данном документе, относится к нуклеотиду, обозначающему начало транскрипции на матричной цепи гена. Сайт начала транскрипции может находиться на матричной цепи в промоторной области. Ген может иметь более одного сайта начала транскрипции.
[50] Термин «идентичность» или «гомология», используемый в данном документе, означает, что одна олигонуклеотидная цепь (смысловая или антисмысловая цепь) каРНК имеет последовательность, сходную с последовательностью кодирующей цепи или матричной цепи в промоторной области целевого гена. В данном документе степень «идентичности» или «гомологии» может составлять по крайней мере примерно 75%, примерно 79%, примерно 80%, примерно 85%, примерно 90%, примерно 95% или 99%.
[51] Термин «липкий конец», используемый в данном документе, относится к неспаренным основаниям нуклеотидов на конце (5' или 3') олигонуклеотидной цепи; он формируется, когда одна цепь выступает за край другой цепи двухцепочечного олигонуклеотида. Одноцепочечный участок, выступающий за край двойной спирали на 3'-конце и/или 5'-конце, называют «липким» концом.
[52] Термины «активация гена», «активация экспрессии гена», а также «стимуляция гена» и «стимуляция экспрессии гена», используемые в данном документе, могут использоваться на равных основаниях. Они означают увеличение транскрипции, трансляции, экспрессии или активности определенной нуклеиновой кислоты, определенное путем измерения уровня транкрипции, уровня мРНК, уровня белка, ферментативной активности, статуса метилирования, состояния или конфигурации хроматина, уровня трансляции, а также активности или состояния клетки или биологической системы гена. Эти виды активности или состояния можно определять прямыми или непрямыми методами. Кроме того, термины «активация гена», «активация экспрессии гена», «стимуляция гена» и «стимуляция экспрессии гена» относятся к повышению активности, связанному с последовательностью нуклеиновой кислоты, независимо от механизма такой активации. Например, активация гена на уровне транскрипции приводит к увеличению транскрипции в РНК, а РНК транслируется в белок, повышая таким образом экспрессию белка.
[53] Термины «короткая активирующая РНК», «каРНК» и «молекула короткой активирующей нуклеиновой кислоты», используемые в данном документе, могут использоваться на равных основаниях. Они относятся к молекуле нуклеиновой кислоты, которая может стимулировать экспрессию целевого гена и состоять из первого фрагмента нуклеиновой кислоты (антисмысловая цепь, также называемая антисмысловой олигонуклеотидной цепью), содержащего нуклеотидную последовательность, идентичную некодирующей нуклеотидной последовательности целевого гена (например, промотора или энхансера), а также из второго фрагмента нуклеиновой кислоты (смысловая цепь, также называемая смысловой олигонуклеотидной цепью), содержащего нуклеотидную последовательность, комплементарную первому фрагменту нуклеиновой кислоты. При этом первый фрагмент нуклеиновой кислоты и второй фрагмент нуклеиновой кислоты формируют двойную спираль. Молекула короткой активирующей нуклеиновой кислоты также может включать синтезированную или экспрессированную посредством вектора одноцепочечную молекулу РНК, которая может формировать структуру в форме «шпильки» с помощью двух комплементарных областей внутри молекулы. При этом первая область содержит нуклеотидную последовательность, идентичную целевой последовательности промотора гена, а вторая область содержит нуклеотидную последовательность, комплементарную первой области. Длина двуспиральной области в молекуле короткой активирующей нуклеиновой кислоты обычно составляет примерно 10-50, примерно 12-48, примерно 14-46, примерно 16-44, примерно 18-42, примерно 20-40, примерно 22-38, примерно 24-36, примерно 26-34 и примерно 28-32 пар оснований, а также обычно примерно 10, примерно 15, примерно 20, примерно 25, примерно 30, примерно 35, примерно 40, примерно 45 или примерно 50 пар оснований. Кроме того, термины «каРНК», «короткая активирующая РНК» и «молекула короткой активирующей нуклеиновой кислоты» также включают другие нуклеиновые кислоты, отличные от рибонуклеиновой, в том числе, помимо прочего, модифицированные нуклеотиды и их аналоги.
[54] Термин «активный участок» относится к промоторной области гена длиной не менее 30 п.о., для которой процент функциональных молекул коротких активирующих нуклеиновых кислот увеличен, т. е. не менее 30% молекул коротких активирующих нуклеиновых кислот, созданных с целью воздействия на эту область, являются функциональными и способны индуцировать изменение экспрессии мРНК в целевом гене в 1,2 или более раз.
[55] Термин «синтез», используемый в данном документе, относится к методу получения олигонуклеотида, включая любой метод, позволяющий синтезировать РНК, например, химический синтез, транскрипция in vitro или экспрессия посредством вектора.
[56] В соответствии с настоящим изобретением, экспрессию гена SMN2 стимулируют путем активации РНК, а связанное заболевание (в частности, спинальную мышечную атрофию) лечат путем повышения уровня экспрессии полноразмерного белка SMN. Ген SMN2 в настоящем изобретении иногда также называют целевым геном.
[57] Настоящее изобретение относится к методу получения молекулы короткой активирующей нуклеиновой кислоты, который включает в себя дизайн последовательности и синтез.
[58] Молекулы короткой активирующей нуклеиновой кислоты можно химически синтезировать или получить от биотехнологической компании, специализирующейся на синтезе нуклеиновых кислот.
[59] В общих чертах, химический синтез нуклеиновых кислот включает следующие четыре этапа: (1) синтез олигомерных рибонуклеотидов; (2) удаление защитных групп; (3) очистка и выделение; (4) обессоливание и ренатурация.
[60] В качестве примера ниже приводятся конкретные этапы химического синтеза РНК, описанных в данном документе:
(1) Синтез олигомерных рибонуклеотидов
[61] Синтез 1 мкМ РНК осуществляли в автоматическом ДНК/РНК-синтезаторе (например, EXPEDITE8909 компании Applied Biosystems), причем время связывания в каждом цикле было установлено на 10-15 мин. В качестве инициатора использовали иммобилизованный на твердой фазе субстрат, 5'-O-p-диметокситрифенилметилтимидин. В первом цикле с субстратом, иммобилизованным на твердой фазе, связывалось одно основание, а в n-ном цикле (19≥n≥2) одно основание связывалось с основанием, которое связалось в цикле n-1. Этот процесс повторялся до тех пор, пока не была синтезирована вся последовательность нуклеиновой кислоты.
(2) Удаление защитных групп
[62] Иммобилизованный на твердой фазе субстрат, связанный с каРНК, помещали в пробирку для анализа, и добавляли 1 мл раствора смеси этанола и гидроксида аммония (соотношение по объему: 1:3). После этого пробирку для анализа герметично закрывали, помещали в инкубатор, и инкубировали смесь при температуре 25-70°C от 2 до 30 ч. Раствор, содержащий иммобилизованный на твердой фазе субстрат, связанный с каРНК, отфильтровывали и собирали фильтрат. Иммобилизованный на твердой фазе субстрат дважды промывали дистиллированной водой, полученной двойной перегонкой (по 1 мл за раз), и собирали фильтрат. Собранные элюенты объединяли и высушивали в вакууме в течение 1-12 ч. Затем к раствору добавляли 1 мл раствора фторида тетрабутиламмония в тетрагидрофуране (1 М), оставляли стоять при комнатной температуре на 4-12 ч, а затем добавляли 2 мл н-бутанола. Методом высокоскоростного центрифугирования собирали осадок, который представлял собой неочищенную одноцепочечную каРНК.
(3) Очистка и выделение
[63] Полученную неочищенную каРНК растворяли в 2 мл водного раствора ацетата аммония с концентрацией 1 моль/мл, и раствор отделяли с помощью жидкостной хроматографии высокого давления на колонке с обращенной фазой C18 для получения очищенной одноцепочечной каРНК.
(4) Обессоливание и ренатурация
[64] Соли удаляли гель-фильтрацией (эксклюзионной хроматографией). Олигомерную рибонуклеиновую кислоту в виде одной смысловой цепи и олигомерную рибонуклеиновую кислоту в виде одной антисмысловой цепи смешивали в 1-2 мл буфера (Tris, 10 мМ, pH=7,5-8,0; NaCl, 50 мМ) в молярном соотношении 1:1. Раствор нагревали до 95°C, а затем медленно охлаждали до комнатной температуры для получения раствора, содержащего каРНК.
[65] В этом исследовании было обнаружено, что упомянутая выше каРНК после введения в клетку способна эффективно повышать уровень экспрессии мРНК и белка, кодируемых полноразмерным геном SMN2.
[66] Настоящее изобретение далее будет проиллюстрировано конкретными примерами и рисунками. Следует понимать, что эти примеры предназначены исключительно для иллюстрации настоящего изобретения, а не для ограничения его объема. В следующих примерах использовались общие методы, которые, как правило, соответствовали стандартным методам, описанным в справочнике Sambrook, et al., Molecular Cloning: Laboratory Manual (New York: Cold Spring Harbor Laboratory Press, 1989), или методам, рекомендованным производителем.
ПРИМЕРЫ
Пример 1. Дизайн и синтез молекул коротких активирующих нуклеиновых кислот, направленных к промотору SMN2
[67] Смысловая последовательность промотора гена SMN2, ограниченная сайтом начала транскрипции (TSS) и нуклеотидом. находящимся в положении -2000 п.о. выше сайта TSS, была получена из базы данных геномов UCSC.
[68] Для выявления функциональных коротких активирующих РНК (каРНК), способных активировать экспрессию гена SMN2, была выбрана серия из 19-нуклеотидных мишеней каРНК в последовательности промотора SMN2 из 2000 п.о. (РИС. 1), начиная с положения -2000 п.о. выше сайта TSS. Продвижение в сторону сайта TSS осуществляли с шагом 1 п.о., что дало в общей сложности 1982 мишени. Затем провели фильтрацию целевых последовательностей, чтобы оставить лишь те, которые соответствовали следующим критериям: (1) содержание GC - от 35% до 65%; (2) менее 5 идентичных нуклеотидов подряд; (3) 3 или меньше динуклеотидных повторов; (4) 3 или меньше тринуклеотидных повторов. После фильтрации осталось 980 целевых последовательностей, для которых были химически синтезированы соответствующие им двухцепочечные каРНК. Каждая из смысловых цепей и антисмысловых цепей каРНК, использованных в исследовании, имела длину в 21 нуклеотид. 19 нуклеотидов в 5'-области первой цепи рибонуклеиновой кислоты (смысловая цепь) в каРНК имела последовательность, на 100% совпадающую с целевой последовательностью промотора, а 3'-конец первой цепи рибонуклеиновой кислоты имел последовательность dTdT. 19 нуклеотидов в 5'-области второй цепи рибонуклеиновой кислоты были комплементарны последовательности первой цепи рибонуклеиновой кислоты, а 3'-конец второй цепи рибонуклеиновой кислоты имел последовательность dTdT. Две цепи каРНК, упомянутые выше, смешали в молярном соотношении 1:1 и ренатурировали для получения двуспиральной каРНК.
Пример 2. Высокопроизводительный скрининг каРНК, направленных к промотору SMN2
1) Клеточная культура и трансфекция
[69] Клетки почки эмбриона человека линии HEK293T (ATCC® CRL-3216™) культивировали в среде DMEM (Gibco), содержащей 10% эмбриональной телячьей сыворотки (Gibco) и 1% пенициллина/стрептомицина (Gibco). Клетки культивировали в 5% CO2 при 37°C. Клетки HEK293T высевали в 96-луночные планшеты в количестве 5000 клеток на лунку. Каждую из каРНК использовали для независимой трансфекции клеток HEK293T в лунках; конечная концентрация каРНК составляла 10 нМ (если не указано иное), а для трансфекции использовали 0,3 мкл реагента RNAiMAX (Invitrogen, Carlsbad, CA) в соответствии с протоколом обратной трансфекции. Трансфекцию проводили в течение 72 часов. В качестве контроля использовали пустой (холостой) раствор, двуспиральный олигонуклеотид с неспецифической последовательностью (dsCon2, смысловая цепь: 5'-ACUACUGAGUGACAGUAGA[dT][dT]-3' (SEQ ID №: 472), антисмысловая цепь: 5'-UCUACUGUCACUCAGUAGU[dT][dT]-3' (SEQ ID №: 473)), короткую интерферирующую РНК к SMN2 (siMSN2-1, смысловая цепь: 5'-GGGAUGAUACAGCACUGAU[dT][dT]-3' (SEQ ID №: 474), антисмысловая цепь: 5'AUCAGUGCUGUAUCAUCCC[dT][dT]-3' (SEQ ID №: 475)); при этом контроль с пустым (холостым) раствором представлял собой трансфекцию без использования нуклеиновой кислоты.
2) Одноэтапная ОТ-кПЦР
[70] По окончании трансфекции среду выбрасывали, а каждую лунку однократно промывали 150 мкл фосфатно-солевого буфера (ФСБ). После удаления ФСБ в каждую лунку добавляли 100 мкл буфера для лизиса клеток (набор Power SYBR® Green Cells-to-Ct™, Life Technologies) и инкубировали при комнатной температуре в течение 5 мин. Из каждой лунки отбирали по 0,5 мкл полученного клеточного лизата и проводили анализ методом ОТ-кПЦР с помощью набора II для ОТ-ПЦР One Step TB Green™ PrimeScrip™ (Takara, RR086A) на приборе для ПЦР в режиме реального времени Lightcycler 480 компании Roche. Пробы для ПЦР готовили с помощью платформы Bravo Automated Liquid Handling Platform (Agilent). Каждый образец после трансфекции амплифицировали в трех параллельных лунках. Условия проведения ПЦР показаны в таблице 1.
[71] Условия реакции были следующими: реакция обратной транскрипции (этап 1): 5 мин при 42°C, 10 с при 95°C; ПЦР (этап 2): 5 с при 95°C, 20 с при 60°C, 45 циклов амплификации. HPRT1 и TBP также амплифицировали в качестве внутренних референтных генов. Праймеры для ПЦР, использованные для амплификации генов SMN, HPRT1 и TBP, показаны в таблице 4; при этом ген SMN был амплифицирован с использованием пары праймеров SMN F1/R1.
[72] Для расчета уровня экспрессии (Erel) SMN2 (целевого гена) в образце, трансфицированном каРНК, в сравнении с контролем (холостой раствор), в формуле 1 использовали значения Ct целевого гена и двух внутренних референтных генов.
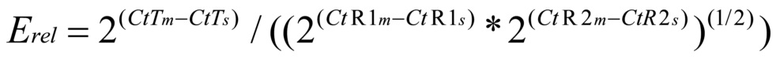
(формула 1)
где CtTm - значение Ct целевого гена для образца после после обработки холостым раствором; CtTs - значение Ct целевого гена для образца после обработки каРНК; CtR1m - значение Ct внутреннего референтного гена 1 для образца после обработки холостым раствором; CtR1s - значение Ct внутреннего референтного гена 1 для образца после обработки каРНК; CtR2m - значение Ct внутреннего референтного гена 2 для образца после обработки холостым раствором, а CtR2s - значение Ct внутреннего референтного гена 2 для образца после обработки каРНК.
3) Скрининг функциональных каРНК
[74] Для выявления каРНК, способных активировать транскрипцию SMN2, клетки HEK293T трансфицировали каждой из 980 упомянутых выше каРНК; концентрация каРНК при трансфекции составляла 10 нМ. Через 72 часа лизированные клетки анализировали с помощью одноэтапной ОТ-кПЦР, чтобы определить относительный уровень экспрессии гена SMN2 в каждом образце, обработанном каРНК (в сравнении с обработкой холостым раствором). Как видно из таблицы 2, 157 (16,02%) каРНК обладали активирующей способностью, в то время как 416 (42,45%) каРНК обладали ингибирующей способностью; 407 (41,53%) каРНК не оказывали явного эффекта на экспрессию гена SMN2. Наблюдаемый максимальный эффект активации составил 1,82 (повышение уровня экспрессии), а максимальный эффект ингибирования - 0,33 (снижение уровня экспрессии). каРНК с активирующими свойствами считаются функциональными каРНК. Их целевые последовательности, смысловые и антисмысловые последовательности, а также соответствующие относительные уровни экспрессии SMN приведены в таблице 3.
(SEQ ID №:315)
(SEQ ID №:1)
(SEQ ID №:158)
(SEQ ID №:316)
(SEQ ID №:2)
(SEQ ID №:159)
(SEQ ID №:317)
(SEQ ID №:3)
(SEQ ID №:160)
(SEQ ID №:318)
(SEQ ID №:4)
(SEQ ID №:161)
(SEQ ID №:319)
(SEQ ID №:5)
(SEQ ID №:162)
(SEQ ID №:320)
(SEQ ID №:6)
(SEQ ID №:163)
(SEQ ID №:321)
(SEQ ID №:7)
(SEQ ID №:164)
(SEQ ID №:322)
(SEQ ID №:8)
(SEQ ID №:165)
(SEQ ID №:323)
(SEQ ID №:9)
(SEQ ID №:166)
(SEQ ID №:324)
(SEQ ID №:10)
(SEQ ID №:167)
(SEQ ID №:325)
(SEQ ID №:11)
(SEQ ID №:168)
(SEQ ID №:326)
(SEQ ID №:12)
(SEQ ID №:169)
(SEQ ID №:327)
(SEQ ID №:13)
(SEQ ID №:170)
(SEQ ID №:328)
(SEQ ID №:14)
(SEQ ID №:171)
(SEQ ID №:329)
(SEQ ID №:15)
(SEQ ID №:172)
(SEQ ID №:330)
(SEQ ID №:16)
(SEQ ID №:173)
(SEQ ID №:331)
(SEQ ID №:17)
(SEQ ID №:174)
(SEQ ID №:332)
(SEQ ID №:18)
(SEQ ID №:175)
(SEQ ID №:333)
(SEQ ID №:19)
(SEQ ID №:176)
(SEQ ID №:334)
(SEQ ID №:20)
(SEQ ID №:177)
(SEQ ID №:335)
(SEQ ID №:21)
(SEQ ID №:178)
(SEQ ID №:336)
(SEQ ID №:22)
(SEQ ID №:179)
(SEQ ID №:337)
(SEQ ID №:23)
(SEQ ID №:180)
(SEQ ID №:338)
(SEQ ID №:24)
(SEQ ID №:181)
(SEQ ID №:339)
(SEQ ID №:25)
(SEQ ID №:182)
(SEQ ID №:340)
(SEQ ID №:26)
(SEQ ID №:183)
(SEQ ID №:341)
(SEQ ID №:27)
(SEQ ID №:342)
(SEQ ID №:28)
(SEQ ID №:185)
(SEQ ID №:343)
(SEQ ID №:29)
(SEQ ID №:186)
(SEQ ID №:344)
(SEQ ID №:30)
(SEQ ID №:187)
(SEQ ID №:345)
(SEQ ID №:31)
(SEQ ID №:188)
(SEQ ID №:346)
(SEQ ID №:32)
(SEQ ID №:189)
(SEQ ID №:347)
(SEQ ID №:33)
(SEQ ID №:190)
(SEQ ID №:348)
(SEQ ID №:34)
(SEQ ID №:191)
(SEQ ID №:349)
(SEQ ID №:35)
(SEQ ID №:192)
(SEQ ID №:350)
(SEQ ID №:36)
(SEQ ID №:193)
(SEQ ID №:351)
(SEQ ID №:37)
(SEQ ID №:194)
(SEQ ID №:352)
(SEQ ID №:38)
(SEQ ID №:195)
(SEQ ID №:353)
(SEQ ID №:39)
(SEQ ID №:196)
(SEQ ID №:354)
(SEQ ID №:40)
(SEQ ID №:197)
(SEQ ID №:355)
(SEQ ID №:41)
(SEQ ID №:198)
(SEQ ID №:356)
(SEQ ID №:42)
(SEQ ID №:199)
(SEQ ID №:357)
(SEQ ID №:43)
(SEQ ID №:200)
(SEQ ID №:358)
(SEQ ID №:44)
(SEQ ID №:201)
(SEQ ID №:359)
(SEQ ID №:45)
(SEQ ID №:202)
(SEQ ID №:360)
(SEQ ID №:46)
(SEQ ID №:203)
(SEQ ID №:361)
(SEQ ID №:47)
(SEQ ID №:204)
(SEQ ID №:362)
(SEQ ID №:48)
(SEQ ID №:205)
(SEQ ID №:363)
(SEQ ID №:49)
(SEQ ID №:206)
(SEQ ID №:364)
(SEQ ID №:50)
(SEQ ID №:207)
(SEQ ID №:365)
(SEQ ID №:51)
(SEQ ID №:208)
(SEQ ID №:366)
(SEQ ID №:52)
(SEQ ID №:209)
(SEQ ID №:367)
(SEQ ID №:53)
(SEQ ID №:210)
(SEQ ID №:368)
(SEQ ID №:54)
(SEQ ID №:211)
(SEQ ID №:369)
(SEQ ID №:55)
(SEQ ID №:212)
(SEQ ID №:370)
(SEQ ID №:56)
(SEQ ID №:213)
(SEQ ID №:371)
(SEQ ID №:57)
(SEQ ID №:214)
(SEQ ID №:372)
(SEQ ID №:58)
(SEQ ID №:215)
(SEQ ID №:373)
(SEQ ID №:59)
(SEQ ID №:216)
(SEQ ID №:374)
(SEQ ID №:60)
(SEQ ID №:217)
(SEQ ID №:375)
(SEQ ID №:61)
(SEQ ID №:218)
(SEQ ID №:376)
(SEQ ID №:62)
(SEQ ID №:219)
(SEQ ID №:377)
(SEQ ID №:63)
(SEQ ID №:220)
(SEQ ID №:378)
(SEQ ID №:64)
(SEQ ID №:221)
(SEQ ID №:379)
(SEQ ID №:65)
(SEQ ID №:222)
(SEQ ID №:380)
(SEQ ID №:66)
(SEQ ID №:223)
(SEQ ID №:381)
(SEQ ID №:67)
(SEQ ID №:224)
(SEQ ID №:382)
(SEQ ID №:68)
(SEQ ID №:225)
(SEQ ID №:383)
(SEQ ID №:69)
(SEQ ID №:226)
(SEQ ID №:384)
(SEQ ID №:70)
(SEQ ID №:227)
(SEQ ID №:385)
(SEQ ID №:71)
(SEQ ID №:228)
(SEQ ID №:386)
(SEQ ID №:72)
(SEQ ID №:229)
(SEQ ID №:387)
(SEQ ID №:73)
(SEQ ID №:230)
(SEQ ID №:388)
(SEQ ID №:74)
(SEQ ID №:231)
(SEQ ID №:389)
(SEQ ID №:75)
(SEQ ID №:232)
(SEQ ID №:390)
(SEQ ID №:76)
(SEQ ID №:233)
(SEQ ID №:391)
(SEQ ID №:77)
(SEQ ID №:234)
(SEQ ID №:392)
(SEQ ID №:78)
(SEQ ID №:235)
(SEQ ID №:393)
(SEQ ID №:79)
(SEQ ID №:236)
(SEQ ID №:394)
(SEQ ID №:80)
(SEQ ID №:237)
(SEQ ID №:395)
(SEQ ID №:81)
(SEQ ID №:238)
(SEQ ID №:396)
(SEQ ID №:82)
(SEQ ID №:239)
(SEQ ID №:397)
(SEQ ID №:83)
(SEQ ID №:240)
(SEQ ID №:398)
(SEQ ID №:84)
(SEQ ID №:241)
(SEQ ID №:399)
(SEQ ID №:85)
(SEQ ID №:242)
(SEQ ID №:400)
(SEQ ID №:86)
(SEQ ID №:243)
(SEQ ID №:401)
(SEQ ID №:87)
(SEQ ID №:244)
(SEQ ID №:402)
(SEQ ID №:88)
(SEQ ID №:245)
(SEQ ID №:403)
(SEQ ID №:89)
(SEQ ID №:246)
(SEQ ID №:404)
(SEQ ID №:90)
(SEQ ID №:247)
(SEQ ID №:405)
(SEQ ID №:91)
(SEQ ID №:248)
(SEQ ID №:406)
(SEQ ID №:92)
(SEQ ID №:249)
(SEQ ID №:407)
(SEQ ID №:93)
(SEQ ID №:250)
(SEQ ID №:408)
(SEQ ID №:94)
(SEQ ID №:251)
(SEQ ID №:409)
(SEQ ID №:95)
(SEQ ID №:252)
(SEQ ID №:410)
(SEQ ID №:96)
(SEQ ID №:253)
(SEQ ID №:411)
(SEQ ID №:97)
(SEQ ID №:254)
(SEQ ID №:412)
(SEQ ID №:98)
(SEQ ID №:255)
(SEQ ID №:413)
(SEQ ID №:99)
(SEQ ID №:256)
(SEQ ID №:414)
(SEQ ID №:100)
(SEQ ID №:257)
(SEQ ID №:415)
(SEQ ID №:101)
(SEQ ID №:258)
(SEQ ID №:416)
(SEQ ID №:102)
(SEQ ID №:259)
(SEQ ID №:417)
(SEQ ID №:103)
(SEQ ID №:260)
(SEQ ID №:418)
(SEQ ID №:104)
(SEQ ID №:261)
(SEQ ID №:419)
(SEQ ID №:105)
(SEQ ID №:262)
(SEQ ID №:420)
(SEQ ID №:106)
(SEQ ID №:263)
(SEQ ID №:421)
(SEQ ID №:107)
(SEQ ID №:264)
(SEQ ID №:422)
(SEQ ID №:108)
(SEQ ID №:265)
(SEQ ID №:423)
(SEQ ID №:109)
(SEQ ID №:266)
(SEQ ID №:424)
(SEQ ID №:110)
(SEQ ID №:267)
(SEQ ID №:425)
(SEQ ID №:111)
(SEQ ID №:268)
(SEQ ID №:426)
(SEQ ID №:112)
(SEQ ID №:269)
(SEQ ID №:427)
(SEQ ID №:113)
(SEQ ID №:270)
(SEQ ID №:428)
(SEQ ID №:114)
(SEQ ID №:271)
(SEQ ID №:429)
(SEQ ID №:115)
(SEQ ID №:272)
(SEQ ID №:430)
(SEQ ID №:116)
(SEQ ID №:273)
(SEQ ID №:431)
(SEQ ID №:117)
(SEQ ID №:274)
(SEQ ID №:432)
(SEQ ID №:118)
(SEQ ID №:275)
(SEQ ID №:433)
(SEQ ID №:119)
(SEQ ID №:276)
(SEQ ID №:434)
(SEQ ID №:120)
(SEQ ID №:277)
(SEQ ID №:435)
(SEQ ID №:121)
(SEQ ID №:278)
(SEQ ID №:436)
(SEQ ID №:122)
(SEQ ID №:279)
(SEQ ID №:437)
(SEQ ID №:123)
(SEQ ID №:280)
(SEQ ID №:438)
(SEQ ID №:124)
(SEQ ID №:281)
(SEQ ID №:439)
(SEQ ID №:125)
(SEQ ID №:282)
(SEQ ID №:440)
(SEQ ID №:126)
(SEQ ID №:283)
(SEQ ID №:441)
(SEQ ID №:127)
(SEQ ID №:284)
(SEQ ID №:442)
(SEQ ID №:128)
(SEQ ID №:285)
(SEQ ID №:443)
(SEQ ID №:129)
(SEQ ID №:286)
(SEQ ID №:444)
(SEQ ID №:130)
(SEQ ID №:287)
(SEQ ID №:445)
(SEQ ID №:131)
(SEQ ID №:288)
(SEQ ID №:446)
(SEQ ID №:132)
(SEQ ID №:289)
(SEQ ID №:447)
(SEQ ID №:133)
(SEQ ID №:290)
(SEQ ID №:448)
(SEQ ID №:134)
(SEQ ID №:291)
(SEQ ID №:449)
(SEQ ID №:135)
(SEQ ID №:292)
(SEQ ID №:450)
(SEQ ID №:136)
(SEQ ID №:293)
(SEQ ID №:451)
(SEQ ID №:137)
(SEQ ID №:294)
(SEQ ID №:452)
(SEQ ID №:138)
(SEQ ID №:295)
(SEQ ID №:453)
(SEQ ID №:139)
(SEQ ID №:296)
(SEQ ID №:454)
(SEQ ID №:140)
(SEQ ID №:297)
(SEQ ID №:455)
(SEQ ID №:141)
(SEQ ID №:298)
(SEQ ID №:456)
(SEQ ID №:142)
(SEQ ID №:299)
(SEQ ID №:457)
(SEQ ID №:143)
(SEQ ID №:300)
(SEQ ID №:458)
(SEQ ID №:144)
(SEQ ID №:301)
(SEQ ID №:459)
(SEQ ID №:145)
(SEQ ID №:302)
(SEQ ID №:460)
(SEQ ID №:146)
(SEQ ID №:303)
(SEQ ID №:461)
(SEQ ID №:147)
(SEQ ID №:304)
(SEQ ID №:462)
(SEQ ID №:148)
(SEQ ID №:305)
(SEQ ID №:463)
(SEQ ID №:149)
(SEQ ID №:306)
(SEQ ID №:464)
(SEQ ID №:150)
(SEQ ID №:307)
(SEQ ID №:465)
(SEQ ID №:151)
(SEQ ID №:308)
(SEQ ID №:466)
(SEQ ID №:152)
(SEQ ID №:309)
(SEQ ID №:467)
(SEQ ID №:153)
(SEQ ID №:310)
(SEQ ID №:468)
(SEQ ID №:154)
(SEQ ID №:311)
(SEQ ID №:469)
(SEQ ID №:155)
(SEQ ID №:312)
(SEQ ID №:470)
(SEQ ID №:156)
(SEQ ID №:313)
(SEQ ID №:471)
(SEQ ID №:157)
(SEQ ID №:314)
SMN-экзон8-R
SEQ ID №: 489
CCCTTCTCACAGCTCATAAAATTAC
[75] РИС. 2 иллюстрирует распределение каРНК SMN2 по активности; каРНК отсортированы в порядке уменьшения активирующей способности (от молекул с высокой активирующей способностью к молекулам с высокой ингибирующей способностью). Сортировка 980 каРНК по их расположению в промоторе SMN2 позволила показать, что распределение функциональных каРНК по промотору носило кластерный характер, т. е. они группировались лишь в определенных областях промотора: можно было выделить «активные участки», в которых число функциональных каРНК было увеличено (РИС. 3). Как видно из РИС. 3, имеется 4 активных участка, которые находятся в областях промотора от -1639 до -1481 (H1), от -1090 до -1008 (H2), от -994 до -180 (H3) и от -144 до -37 (H4) и в которых число функциональных каРНК заметно увеличено. Такой результат говорит о том, что функциональные каРНК распределялись в промоторе неслучайным образом и что в особых активных участках их число было увеличено.
[76] Последовательность активного участка H1 (от -1639 до -1481) (SEQ ID №: 476):
agtcgcactctgtcactcaggctggagtgcagtggcgtgatcttggctcactgcaacctccgcctcccgagttcaagtgattctcctggctcagcctcccaagcagctgtcattacaggcctgcaccaccacacccggctgatttttgtatttttagga (SEQ ID №: 476)
[77] Последовательность активного участка H2 (от -1090 до -1008) (SEQ ID №: 477):
aatactggaggcccggtgtggtggctcacacctgtaatcccagcactttgggaggccgaggcggtcggattacgaggtcagg (SEQ ID №: 477)
[78] Последовательность активного участка H3 (от -994 до -180) (SEQ ID №: 478):
ctggccaacatggtgaaaccccatctttactaaaaatacaaaaattagccgggtgtggtggtgggcgcctgtaatcccagctactcggggggctgaggcagaattgcttgaacctgggaggcagaggttgcagtgagctgagatcacgccactgcattccagcctgggtgacagagcaatactctgtcgcaaaaaaaaaaaagaatactggaggctgggcgaggtggctcacacctgtaatcccagcattttgggatgccagaggcgggcggaatatcttgagctcaggagttcgagaccagcctacacaatatgctccaaacgccgcctctacaaaacatacagaaactagccgggtgtggtggcgtgcccctgtggtcctagctacttgggaggttgaggcgggaggatcgcttgagctcgggaggtcgaggctgcaatgagccgagatggtgccactgcactctgacgacagagcgagactccgtctcaaaacaaacaacaaataaggttgggggatcaaatatcttctagtgtttaaggatctgccttccttcctgcccccatgtttgtctttccttgtttgtctttatatagatcaagcaggttttaaattcctagtaggagcttacatttacttttccaagggggagggggaataaatatctacacacacacacacacacacacacacacacacacactggagttcgagacgaggcctaagcaacatgccgaaaccccgtctctactaaatacaaaaaatagctgagcttggtggcgcacgcctatagtcctagctactggggaggctg (SEQ ID №: 478)
[79] Последовательность активного участка H4 (от -144 до -37) (SEQ ID №: 479):
ctgcagtgagccgagatcgcgccgctgcactccagcctgagcgacagggcgaggctctgtctcaaaacaaacaaacaaaaaaaaaaggaaaggaaatataacacagtg(SEQ ID №: 479).
Пример 3. Дополнительный скрининг и валидация функциональных каРНК, способных активировать ген SMN
[80] Для дополнительного скрининга и проверки функциональных каРНК, способных активировать ген SMN (выявленных в ходе высокопроизводительного скрининга), из 157 функциональных каРНК были случайным образом выбраны 50 каРНК для дополнительной проверки их способности активировать экспрессию генов SMN в клетках HEK293T, HS27 (линия фибробластов кожи человека) и NHDF (нормальные фибробласты дермы человека). Клетки HEK293T, HS27 и NHDF трансфицировали отдельными каРНК (n=50, конечная концентрация: 20 нМ), перечисленными в таблице 5; метод трансфекции описан в примере 2. Через 72 ч РНК из трансфицированных клеток выделяли РНК с помощью набора Qiagen RNeasy. После обратной транскрипции проводили кПЦР-амплификацию генов SMN с помощью системы 7500FAST для ПЦР в режиме реального времени; также амплифицировали гены HPRT1 и TBP, а средние геометрические значения их экспрессии использовали в качестве внутренних референтных значений. РИС. 4 иллюстрирует эффект активирующей каРНК на экспрессию гена SMN в клетках HEK293T, а таблица 5 иллюстрирует эффект активирующей каРНК на экспрессию гена SMN в клетках HS27 и NHDF. Из этих результатов видно, что такие каРНК способны активировать экспрессию генов SMN в различных клетках в различной степени, вплоть до 19-кратного увеличения уровня экспрессии.
Пример 4. Оценка экспрессии SMN2 с помощью ОТ-ПЦР и расщепления рестриктазой
[81] Поскольку ген SMN2 в высокой степени гомологичен гену SMN1, описанные выше праймеры для ОТ-кПЦР сами по себе не позволяют различить последовательности мРНК генов SMN2 и SMN1. Чтобы обнаружить именно экспрессию мРНК SMN2 после обработки каРНК, а также различить мРНК SMN2 с включением и пропуском экзона 7, кДНК из клеток, обработанных каРНК, амплифицировали с использованием пары праймеров SMN-экзон6-F и SMN-экзон8-R, а продукты такой ПЦР расщепляли рестриктазой DdeI. После гель-электрофореза продуктов расщепления оценивали уровни экспрессии полноразмерной мРНК SMN2 и мРНК SMN2 с делецией экзона 7 (SMN2Δ7) по интенсивности отдельных полос ДНК. Вкратце, клетки HEK293T высевали на 6-луночные планшеты в количестве 2-3×105 клеток на лунку и проводили обратную трансфекцию каРНК при конечной концентрации 10 нМ. По окончании трансфекции выделяли общую клеточную РНК с помощью набора RNeasy Plus Mini (Qiagen; Hilden, Германия) согласно инструкции, вложенной в набор. РНК (1 мкг) подвергали обратной транскрипции в кДНК с использованием набора PrimeScript RT, содержащего средство удаления геномной ДНК gDNA Eraser (Takara, Shlga, Япония), после чего проводили ПЦР-амплификацию с использованием праймеров SMN-экзон6-F и SMN-экзон8-R и реагента для ПЦР компании Takara (RR010A) в следующих условиях: 10 с при 98°C, 15 с при 60°C, 32 с при 72°C и 28 циклов амплификации (подробнее см. в таблице 6). После ПЦР-амплификации продукты ПЦР расщепляли ферментом DdeI для того, чтобы различить SMN1 и SMN2, после чего продукты расщепления разделяли в 2,5%-ном агарозном геле с помощью электрофореза. Интенсивность полос, соответствующих каждому продукту ПЦР или продуктам расщепления рестриктазой, анализировали с помощью системы Image Lab (BIO-RAD, система визуализации Chemistry Doctm MP). В качестве контроля для нормирования интенсивности полос исследуемых образцов использовали полосу 500 п.о. из набора маркеров для электрофореза ДНК компании Takara 100 bp DNA ladder (3407A) (в лунку геля вносили 5 мкл образца, содержащего приблизительно 150 нг ДНК). Нормированную интенсивность полос затем выражали в относительных единицах в сравнении с интенсивностью полос для образца, обработанного холостым раствором. Реакция расщепления рестриктазой и условия проведения этой реакции показаны в таблице 7. В качестве внутреннего референтного гена использовали ген HPRT1; последовательности использованных праймеров приведены в таблице 4.
[82] Ниже описана процедура конструирования вектора для гиперэкспрессии SMN2, использованного в данном примере, и трансфекции клеток этим вектором.
[83] Из клеток HEK293T выделяли общую клеточную РНК и подвергали ее обратной транскрипции в кДНК с использованием праймеров OligodT. Открытую рамку считывания (ORF) полноразмерного гена SMN2 амплифицировали с помощью праймеров для клонирования ПЦР-продуктов cSMN2-F2 (TAAGCA GGATCC ATG GCG ATG AGC AGC GGC GGC (SEQ ID №: 490)) и cSMN2-R2 (TAAGCA GAATTC TTA ATT TAA GGA ATG TGA GCA (SEQ ID №: 491)). Полученные продукты расщепляли с помощью ферментов BamHI и EcoRI. Те же ферменты использовали для расщепления плазмиды pcDNA3.1 (Invitrogen). Расщепленные плазмиды и расщепленные продукты ПЦР лигировали с помощью лигазы T4. Компетентные клетки DH5α трансформировали продуктами, полученными в результате реакции лигирования. Из клеток, оставленных на ночь для размножения, выделяли плазмиды с помощью набора Qiagen Miniprep. Полученные плазмиды (1 мкг) использовали для трансфекции клеток HEK293T с помощью реагента Lipofectamine 3000 (Invitrogen). Через 72 часа из трансфицированных клеток выделяли общую РНК и анализировали ее с помощью ОТ-ПЦР и расщепления рестриктазой.
[84] Как видно из РИС. 6 и РИС. 7A, по сравнению с пустым (холостым) контролем и контролем с двуспиральным олигонуклеотидом (dsCON2) (полосы 51-52), общий уровень экспрессии мРНК SMN2 во всех клетках, обработанных кмРНК, был увеличен; при этом максимально этот уровень повысился в 2,49 раз. Также показано, что большинство кмРНК (28, 56%) вызывали увеличение соотношения между количеством мРНК полноразмерного гена SMN2 и мРНК гена SMN2 с делецией экзона 7 (указана черными стрелками на РИС. 6 и РИС. 7B).
Пример 5. Исследование дозовой зависимости эффекта каРНК, состоящего в активации экспрессии SMN и повышении экспрессии мРНК и белка, кодируемых полноразмерным геном SMN2
[85] Чтобы установить дозовую зависимость эффекта каРНК, состоящего в активации экспрессии SMN, были выбраны 2 каРНК (RAG6-281 и RAG6-550), которые были использованы для трансфекции клеток HEK293T при концентрациях 1 нМ, 10 нМ, 20 нМ, 50 нМ и 100 нМ. Через 72 часа после трансфекции из обработанных клеток выделяли РНК и белок. РНК подвергали обратной транскрипции в кДНК, а полученную кДНК амплифицировали методом ОТ-кПЦР, после чего расщепляли рестриктазой DdeI. Белок анализировали методом вестерн-блоттинга с использованием специфического антитела к SMN для определения уровня экспрессии белка SMN. Вкратце, обработанные клетки лизировали с помощью буфера для лизиса клеток (1-кратный буфер RIPA, Cell Signaling Technology (CST), Danvers, MA, США, № 9806). В буфер для лизиса клеток добавляли ингибитор протеаз (Sigma, серия № 126M4015v). Белок количественно определяли методом с бицинхониновой кислотой (BCA) и отделяли с помощью электрофореза в полиакриламидном геле. После электрофореза белок переносили на мембрану из ПВДФ с размером пор 0,45 мкм. Блоты анализировали с использованием моноклонального мышиного антитела к SMN (CST, № 12976) или поликлонального кроличьего антитела к α/β-тубулину (CST, № 2148); в качестве второго антитела использовали связанное с пероксидазой хрена (HRP) антитело к IgG мыши (CST, № 7076) или связанное с HRP антитело к IgG кролика (CST, № 7074). Для обнаружения сигналов белка мембрану сканировали на аппарате Image Lab.
[86] Как показано на РИС. 8A, препараты RAG6-281 и RAG6-550 вызывали значительную активацию экспрессии мРНК SMN, увеличивая ее более чем в 1,5 раза, даже в случае трансфекции в концентрации 1 нМ; максимальное увеличение экспрессии SMN (в 2,38 раза и 2,16 раза соответственно) наблюдалось в том случае, когда концентрация каРНК при трансфекции составляла 50 нМ. В случае когда концентрация каРНК при трансфекции составляла 100 нМ, дополнительного увеличения экспрессии SMN не наблюдалось. Кроме того, анализ с использованием ОТ-ПЦР и расщепления рестриктазой DdeI показал, что обе каРНК (RAG6-281 и RAG6-550) стимулировали экспрессию мРНК SMN1 и SMN2, что находится в согласии с результатами ОТ-кПЦР. Количественная оценка соотношения интенсивностей полос, соответствующих продуктам полноразмерного гена SMN2 и гена SMN2Δ7, показала, что препараты RAG6-281 и RAG6-550 значительно повышали экспрессию мРНК полноразмерного гена SMN2 при всех изученных концентрациях (РИС. 8B). По сравнению с холостым контролем, каРНК RAG6-281 увеличивала отношение мРНК полноразмерного гена SMN2 к мРНК гена SMN2Δ7 в 1,9, 2,39, 2,41, 2,39 и 2,1 раза в случае трансфекции при концентрациях 1 нМ, 10 нМ, 20 нМ, 50 нМ и 100 нМ соответственно; при этом каРНК RAG6-550 увеличивала отношение мРНК полноразмерного гена SMN2 к мРНК гена SMN2Δ7 в 1,52, 1,99, 1,91, 2,3 и 1,7 раза соответственно в случае трансфекции при концентрациях, указанных выше. Величина изменений, индуцируемых препаратами RAG6-281 и RAG6-550, в случае трансфекции при концентрациях от 1 нМ до 50 нМ зависела от дозы (РИС. 8B). Изменение экспрессии белка SMN также анализировали методом вестерн-блоттинга, и полученные результаты очень хорошо соответствовали результатам анализа мРНК методом ОТ-кПЦР, из чего можно заключить, что обе каРНК (RAG6-281 и RAG6-550) значительно повышали уровень экспрессии полноразмерного белка SMN, причем эффект зависел от дозы (РИС. 8C). Однако при вестерн-блоттинге не удалось обнаружить полос, соответствующих белку SMN с делецией экзона 7 (SMN2Δ7), что, вероятно, связано с его существенной нестабильностью, о чем ранее уже сообщалось в литературе (Hua et al, PLoS Biol 2007;5(4)e73).
Пример 6. Эффективность каРНК in vivo: улучшение моторной функции у мышей с СМА I типа
1. Разведение и генотипирование мышей с СМА I типа (СМА I)
[87] Новорожденных мышей получали путем скрещивания мышей Smn1+/-, SMN2-/- (с гетерозиготным нокаутом гена Smn1 мыши) и мышей Smn1-/-, SMN2+/+ (мыши с СМА III типа и двумя копиями трансгена SMN2 человека) (предоставлены Пекинским институтом генной терапии редких заболеваний Руйси) и генотипировали путем ПЦР геномной ДНК (РИС. 9). В данном исследовании использовали детенышей со следующими генотипами: мыши с СМА I - носители гомологичной делеции гена Smn мыши и гетерозиготного трансгена SMN2 человека с генотипом Smn1-/-, SMN2+/-, гетерозиготные мыши с СМА (Het, контрольная группа нормальных мышей) - носители гетерозиготной делеции гена Smn1 мыши и гетерозиготного трансгена SMN2 человека с генотипом of Smn+/-, SMN2+/-.
2. Приготовление олигонуклеотидов в смеси с реагентами in vivo-jetPEI и HKP
[88] Подготовка лекарственной формы с помощью in-vivo jetPEI. Для приготовления маточного раствора (5 мг/мл) каРНК SMN2 RAG6-539 (DS06-0013B) растворяли в воде, не содержащей РНКаз (Invitrogen, 2063810). 5 мкл DS06-0013B и 12,5 мкл 10% раствора глюкозы (Polyplus-transfection, G181106) аккуратно перемешивали с 3,5 мкл воды, не содержащей РНКаз, для получения рабочего раствора DS06-0013B. Рабочий раствор добавляли к 4 мкл реагента in-vivo jetPEI (Polyplus-transfection, 26031A1C) и перемешивали, после чего смесь инкубировали при комнатной температуре в течение 15 мин; конечная концентрация РНК составляла 1 мг/мл.
[89] Приготовление раствора с реагентом HKP. каРНК SMN2 RAG6-538 (DS06-0002B) растворяли в воде, не содержащей РНКаз, для получения маточного раствора (4 мг/мл). Сополимер гистидина и лизина (HKP) (Suzhou Sirnaomics Biopharmaceuticals Co. Ltd., AKF271/042-79-11) растворяли в воде, не содержащей РНКаз, для получения маточного раствора (16 мг/мл). 7,5 мкл маточного раствора HKP быстро смешивали с 7,5 мкл маточного раствора DS06-0002B, после чего смесь оставляли стоять на 30 мин при комнатной температуре; конечная концентрация РНК составляла 2 мг/мл.
3. Интрацеребровентрикулярная инъекция олигонуклеотидов в смеси с реагентами in-vivo jetPEI и HKP мышам с СМА I
[90] Детенышей мышей с генотипами, указанными выше, разделили на четыре группы: контрольные мыши Het, контрольные мыши с СМА (мыши с СМА I, не получавшие никаких препаратов), DS06-0013B-J (мыши с СМА I, получавшие препарат DS06-0013B в смеси с реагентом in vivo-jetPEI, 1 мг/мл) и DS06-0002B-H (мыши с СМА I, получавшие препарат DS06-0002B в смеси с реагентом с HKP, 2 мг/мл). Новорожденным мышам проводили интрацеребровентрикулярную инъекцию (ИЦВ) в 1-й день после рождения (P1); объем введенного раствора составлял 5 мкл. Группы животных, способ введения, объем введенного раствора и время проведения инъекции показаны в таблице 8.
4. Оценка моторной функции у мышей с СМА I
[91] Моторную функцию мышей оценивали в день P7 или P8 с помощью теста на рефлекс переворачивания. Вкратце, мышей из нормального положения стоя переворачивали на спину, так что спина касалась поверхности лабораторного стола, а лапы были подняты вверх; затем их отпускали, чтобы они могли вернуться в нормальное положение стоя. Время (в секундах), необходимое мышам для возврата в нормальное положение, регистрировали и описывали как время рефлекса переворачивания или время переворота. Если мышь не могла вернуться в нормальное положение в течение 60 с, то время переворота регистрировали как > 60 с. Время переворота отражает моторную функцию мышей: чем короче время переворота, тем лучше моторная функция у мыши. В таблице 9 перечислены значения времени переворота у мышей в данном исследовании.
[92] РИС. 10 иллюстрирует улучшение моторной функции у мышей с СМА I типа после введения препаратов каРНК SMN2. Как видно из таблицы 9, время переворота со спины на лапы у нормальных мышей из контрольной группы (Het) находилось в пределах 2 с, в то время как у мышей с СМА I из контрольной группы (не получавших препарат) время переворота со спины на лапы составляло не менее 12 с; при этом две мыши из этой группы полностью утратили способность переворачиваться (время переворота > 60 с). В двух группах терапии каРНК [RAG6-538 (DS06-0002B-H) и DS06-0013B (DS06-0013B-J)] время переворота было ближе к показателям для нормальных мышей, особенно у мышей из группы DS06-0002B-H. По сравнению с контрольной группой СМА I, время переворота у мышей из группы DS06-0002B-H сократилось почти в 10 раз, а у мышей из группы DS06-0013B-J время переворота сократилось в 2,5 раза (таблица 9, РИС. 10). Этот результат демонстрирует, что моторная функция у мышей с СМА I типа после введения каРНК SMN2 значительно улучшилась, из чего можно заключить, что терапия каРНК может отсрочить начало болезни.
[93] В целом, в результате высокопроизводительного скрининга каРНК, направленных к промотору SMN2, был выявлен целый ряд каРНК, способных заметно активировать экспрессию гена SMN. Эти каРНК способны не только вызывать дозозависимую стимуляцию экспрессии гена SMN2, но также значительно увеличивать соотношение между количеством полноразмерного белка SMN2 и белка SMN2Δ7 в клетках. Кроме того, исследование эффективности in vivo доказало, что каРНК, раскрытые в данном документе, способны значительно улучшать моторную функцию у мышей с СМА I типа. Эти результаты убедительно говорят о том, что активация экспрессии SMN2 на уровне транскрипции с помощью каРНК, направленных к промотору SMN2, с целью повышения уровня экспрессии полноразмерного белка SMN, является перспективной стратегией лечения СМА.
Литература
1. Kolb SJ, Coffey CS, Yankey JW, Krosschell K, Arnold WD, et al. 2017. Natural history of infantile-onset spinal muscular atrophy. Ann Neurol 82:883-91
2. Sugarman EA, Nagan N, Zhu H, Akmaev VR, Zhou Z, et al. 2012. Pan-ethnic carrier screening and prenatal diagnosis for spinal muscular atrophy: clinical laboratory analysis of >72,400 specimens. Eur J Hum Genet 20:27-32
3. Zhang C, Lang Q, 2017. New Therapeutic medication for treating Spinal Muscular Atrophy-SPINRAZA. Journal of Clinical Phamacology 15:83-4
4. Lefebvre S, Burglen L, Reboullet S, Clermont O, Burlet P, et al. 1995. Identification and characterization of a spinal muscular atrophy-determining gene. Cell 80:155-65
5. Lorson CL, Hahnen E, Androphy EJ, Wirth B. 1999. A single nucleotide in the SMN gene regulates splicing and is responsible for spinal muscular atrophy. Proc Natl Acad Sci USA 96:6307-11
6. Monani UR, Lorson CL, Parsons DW, Prior TW, Androphy EJ, et al. 1999. A single nucleotide difference that alters splicing patterns distinguishes the SMA gene SMN1 from the copy gene SMN2. Hum Mol Genet 8:1177-83
7. Hua Y, Sahashi K, Hung G, Rigo F, Passini MA, et al. 2010. Antisense correction of SMN2 splicing in the CNS rescues necrosis in a type III SMA mouse model. Genes Dev 24:1634-44
8. Hua Y, Sahashi K, Rigo F, Hung G, Horev G, et al. 2011. Peripheral SMN restoration is essential for long-term rescue of a severe spinal muscular atrophy mouse model. Nature 478:123-6
9. Naryshkin NA, Weetall M, Dakka A, Narasimhan J, Zhao X, et al. 2014. Motor neuron disease. SMN2 splicing modifiers improve motor function and longevity in mice with spinal muscular atrophy. Science 345:688-93
10. Palacino J, Swalley SE, Song C, Cheung AK, Shu L, et al. 2015. SMN2 splice modulators enhance U1-pre-mRNA association and rescue SMA mice. Nat Chem Biol 11:511-7
11. Michelson D, Ciafaloni E, Ashwal S, Lewis E, Narayanaswami P, et al. 2018. Evidence in focus: Nusinersen use in spinal muscular atrophy: Report of the Guideline Development, Dissemination, and Implementation Subcommittee of the American Academy of Neurology. Neurology
12. Avila AM, Burnett BG, Taye AA, Gabanella F, Knight MA, et al. 2007. Trichostatin A increases SMN expression and survival in a mouse model of spinal muscular atrophy. J Clin Invest 117:659-71
13. Somers E, Riessland M, Schreml J, Wirth B, Gillingwater TH, Parson SH. 2013. Increasing SMN levels using the histone deacetylase inhibitor SAHA ameliorates defects in skeletal muscle microvasculature in a mouse model of severe spinal muscular atrophy. Neurosci Lett 544:100-4
14. Swoboda KJ, Scott CB, Crawford TO, Simard LR, Reyna SP, et al. 2010. SMA CARNI-VAL trial part I: double-blind, randomized, placebo-controlled trial of L-carnitine and valproic acid in spinal muscular atrophy. PLoS One 5:e12140
15. Kissel JT, Scott CB, Reyna SP, Crawford TO, Simard LR, et al. 2011. SMA CARNIVAL TRIAL PART II: a prospective, single-armed trial of L-carnitine and valproic acid in ambulatory children with spinal muscular atrophy. PLoS One 6:e21296.
--->
Список последовательностей
<110> Ractigen Therapeutics
<120> МОЛЕКУЛА ОЛИГОМЕРНОЙ НУКЛЕИНОВОЙ КИСЛОТЫ
И ЕЕ ПРИМЕНЕНИЕ
<130> 20191126
<160> 491
<170> PatentIn version 3.5
<210> 1
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 1
aucugugaga uguaccuuut t 21
<210> 2
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 2
cacucuguca cucaggcugt t 21
<210> 3
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 3
acucugucac ucaggcuggt t 21
<210> 4
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 4
ucugucacuc aggcuggagt t 21
<210> 5
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 5
ucaggcugga gugcaguggt t 21
<210> 6
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 6
gagugcagug gcgugaucut t 21
<210> 7
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 7
ugcaguggcg ugaucuuggt t 21
<210> 8
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 8
gcaguggcgu gaucuuggct t 21
<210> 9
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 9
ggcgugaucu uggcucacut t 21
<210> 10
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 10
gugaucuugg cucacugcat t 21
<210> 11
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 11
ugaucuuggc ucacugcaat t 21
<210> 12
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 12
aucuuggcuc acugcaacct t 21
<210> 13
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 13
cuuggcucac ugcaaccuct t 21
<210> 14
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 14
uuggcucacu gcaaccucct t 21
<210> 15
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 15
gccucccgag uucaagugat t 21
<210> 16
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 16
ccucccgagu ucaagugaut t 21
<210> 17
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 17
cucccgaguu caagugauut t 21
<210> 18
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 18
ucccgaguuc aagugauuct t 21
<210> 19
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 19
ucaagugauu cuccuggcut t 21
<210> 20
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 20
aagugauucu ccuggcucat t 21
<210> 21
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 21
agugauucuc cuggcucagt t 21
<210> 22
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 22
gugauucucc uggcucagct t 21
<210> 23
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 23
cagccuccca agcagcugut t 21
<210> 24
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 24
ccucccaagc agcugucaut t 21
<210> 25
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 25
ucccaagcag cugucauuat t 21
<210> 26
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 26
agcugucauu acaggccugt t 21
<210> 27
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 27
gcugucauua caggccugct t 21
<210> 28
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 28
cugucauuac aggccugcat t 21
<210> 29
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 29
ggagaaacag gguuucacct t 21
<210> 30
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 30
agaaacaggg uuucaccaut t 21
<210> 31
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 31
aagugcuggg auuauaggct t 21
<210> 32
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 32
uuauaggcau gagccaccgt t 21
<210> 33
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 33
auucuccccu uccuccacat t 21
<210> 34
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 34
ucuccccuuc cuccacagat t 21
<210> 35
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 35
cauuuagcaa cccuagaugt t 21
<210> 36
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 36
auuuagcaac ccuagaugct t 21
<210> 37
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 37
uuuagcaacc cuagaugcut t 21
<210> 38
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 38
uuagcaaccc uagaugcuut t 21
<210> 39
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 39
uagcaacccu agaugcuuat t 21
<210> 40
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 40
auacuggagg cccggugugt t 21
<210> 41
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 41
gugugguggc ucacaccugt t 21
<210> 42
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 42
ugguggcuca caccuguaat t 21
<210> 43
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 43
gguggcucac accuguaaut t 21
<210> 44
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 44
guggcucaca ccuguaauct t 21
<210> 45
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 45
ggcucacacc uguaauccct t 21
<210> 46
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 46
cacaccugua aucccagcat t 21
<210> 47
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 47
acaccuguaa ucccagcact t 21
<210> 48
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 48
accuguaauc ccagcacuut t 21
<210> 49
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 49
guaaucccag cacuuugggt t 21
<210> 50
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 50
uaaucccagc acuuugggat t 21
<210> 51
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 51
aaucccagca cuuugggagt t 21
<210> 52
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 52
cagcacuuug ggaggccgat t 21
<210> 53
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 53
gaggcggucg gauuacgagt t 21
<210> 54
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 54
ggcggucgga uuacgaggut t 21
<210> 55
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 55
gcggucggau uacgagguct t 21
<210> 56
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 56
cggucggauu acgaggucat t 21
<210> 57
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 57
ggucggauua cgaggucagt t 21
<210> 58
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 58
gucggauuac gaggucaggt t 21
<210> 59
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 59
ucggauuacg aggucaggat t 21
<210> 60
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 60
cggauuacga ggucaggagt t 21
<210> 61
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 61
auuacgaggu caggaguuct t 21
<210> 62
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 62
uuacgagguc aggaguucat t 21
<210> 63
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 63
uacgagguca ggaguucaat t 21
<210> 64
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 64
acgaggucag gaguucaagt t 21
<210> 65
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 65
aggucaggag uucaagacct t 21
<210> 66
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 66
aguucaagac cagccuggct t 21
<210> 67
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 67
gaaaccccau cuuuacuaat t 21
<210> 68
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 68
auuagccggg ugugguggut t 21
<210> 69
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 69
guggugggcg ccuguaauct t 21
<210> 70
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 70
ggcgccugua aucccagcut t 21
<210> 71
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 71
uaaucccagc uacucggggt t 21
<210> 72
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 72
gggcugaggc agaauugcut t 21
<210> 73
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 73
ggcagaauug cuugaaccut t 21
<210> 74
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 74
cagaauugcu ugaaccuggt t 21
<210> 75
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 75
ugaaccuggg aggcagaggt t 21
<210> 76
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 76
gaaccuggga ggcagaggut t 21
<210> 77
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 77
accugggagg cagagguugt t 21
<210> 78
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 78
ugcagugagc ugagaucact t 21
<210> 79
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 79
cugagaucac gccacugcat t 21
<210> 80
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 80
aucacgccac ugcauuccat t 21
<210> 81
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 81
gggugacaga gcaauacuct t 21
<210> 82
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 82
ugacagagca auacucugut t 21
<210> 83
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 83
agagcaauac ucugucgcat t 21
<210> 84
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 84
aaaagaauac uggaggcugt t 21
<210> 85
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 85
aaagaauacu ggaggcuggt t 21
<210> 86
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 86
auacuggagg cugggcgagt t 21
<210> 87
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 87
cgagguggcu cacaccugut t 21
<210> 88
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 88
gguggcucac accuguaaut t 21
<210> 89
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 89
ggcucacacc uguaauccct t 21
<210> 90
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 90
gcucacaccu guaaucccat t 21
<210> 91
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 91
cacaccugua aucccagcat t 21
<210> 92
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 92
uaaucccagc auuuugggat t 21
<210> 93
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 93
gggcggaaua ucuugagcut t 21
<210> 94
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 94
aauaucuuga gcucaggagt t 21
<210> 95
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 95
uucgagacca gccuacacat t 21
<210> 96
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 96
ccagccuaca caauaugcut t 21
<210> 97
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 97
acacaauaug cuccaaacgt t 21
<210> 98
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 98
cacaauaugc uccaaacgct t 21
<210> 99
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 99
acaauaugcu ccaaacgcct t 21
<210> 100
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 100
caaacgccgc cucuacaaat t 21
<210> 101
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 101
cugugguccu agcuacuugt t 21
<210> 102
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 102
gggaggaucg cuugagcuct t 21
<210> 103
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 103
gaggaucgcu ugagcucggt t 21
<210> 104
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 104
ggaggucgag gcugcaaugt t 21
<210> 105
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 105
ggucgaggcu gcaaugagct t 21
<210> 106
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 106
caaugagccg agauggugct t 21
<210> 107
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 107
aaugagccga gauggugcct t 21
<210> 108
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 108
ccgagauggu gccacugcat t 21
<210> 109
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 109
gagauggugc cacugcacut t 21
<210> 110
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 110
agauggugcc acugcacuct t 21
<210> 111
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 111
auggugccac ugcacucugt t 21
<210> 112
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 112
ccacugcacu cugacgacat t 21
<210> 113
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 113
cacugcacuc ugacgacagt t 21
<210> 114
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 114
ucugacgaca gagcgagact t 21
<210> 115
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 115
cugacgacag agcgagacut t 21
<210> 116
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 116
gagacuccgu cucaaaacat t 21
<210> 117
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 117
agacuccguc ucaaaacaat t 21
<210> 118
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 118
ucuaguguuu aaggaucugt t 21
<210> 119
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 119
uaguguuuaa ggaucugcct t 21
<210> 120
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 120
uguuuaagga ucugccuuct t 21
<210> 121
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 121
ggaucugccu uccuuccugt t 21
<210> 122
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 122
uugucuuucc uuguuuguct t 21
<210> 123
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 123
gucuuuccuu guuugucuut t 21
<210> 124
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 124
caagcagguu uuaaauucct t 21
<210> 125
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 125
gcagguuuua aauuccuagt t 21
<210> 126
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 126
acauuuacuu uuccaagggt t 21
<210> 127
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 127
acacuggagu ucgagacgat t 21
<210> 128
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 128
cuggaguucg agacgaggct t 21
<210> 129
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 129
uucgagacga ggccuaagct t 21
<210> 130
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 130
gagacgaggc cuaagcaact t 21
<210> 131
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 131
agacgaggcc uaagcaacat t 21
<210> 132
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 132
gacgaggccu aagcaacaut t 21
<210> 133
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 133
ccuaagcaac augccgaaat t 21
<210> 134
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 134
cuaagcaaca ugccgaaact t 21
<210> 135
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 135
uaagcaacau gccgaaacct t 21
<210> 136
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 136
ugguggcgca cgccuauagt t 21
<210> 137
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 137
gguggcgcac gccuauagut t 21
<210> 138
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 138
cuauaguccu agcuacuggt t 21
<210> 139
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 139
uauaguccua gcuacugggt t 21
<210> 140
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 140
ugagguggga ggaucgcuut t 21
<210> 141
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 141
cugcagugag ccgagaucgt t 21
<210> 142
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 142
ugcagugagc cgagaucgct t 21
<210> 143
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 143
ugcacuccag ccugagcgat t 21
<210> 144
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 144
cacuccagcc ugagcgacat t 21
<210> 145
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 145
acagggcgag gcucugucut t 21
<210> 146
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 146
gggcgaggcu cugucucaat t 21
<210> 147
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 147
ggcgaggcuc ugucucaaat t 21
<210> 148
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 148
gcgaggcucu gucucaaaat t 21
<210> 149
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 149
aggcucuguc ucaaaacaat t 21
<210> 150
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 150
ggcucugucu caaaacaaat t 21
<210> 151
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 151
gcucugucuc aaaacaaact t 21
<210> 152
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 152
cucugucuca aaacaaacat t 21
<210> 153
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 153
aacacaguga aaugaaaggt t 21
<210> 154
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 154
acacagugaa augaaaggat t 21
<210> 155
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 155
cagugaaaug aaaggauugt t 21
<210> 156
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 156
gugaaaugaa aggauugagt t 21
<210> 157
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 157
gaaaugaaag gauugagagt t 21
<210> 158
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 158
aaagguacau cucacagaut t 21
<210> 159
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 159
cagccugagu gacagagugt t 21
<210> 160
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 160
ccagccugag ugacagagut t 21
<210> 161
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 161
cuccagccug agugacagat t 21
<210> 162
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 162
ucaggcugga gugcaguggt t 21
<210> 163
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 163
agaucacgcc acugcacuct t 21
<210> 164
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 164
ugcaguggcg ugaucuuggt t 21
<210> 165
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 165
gccaagauca cgccacugct t 21
<210> 166
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 166
agugagccaa gaucacgcct t 21
<210> 167
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 167
ugcagugagc caagaucact t 21
<210> 168
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 168
uugcagugag ccaagaucat t 21
<210> 169
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 169
gguugcagug agccaagaut t 21
<210> 170
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 170
gagguugcag ugagccaagt t 21
<210> 171
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 171
ggagguugca gugagccaat t 21
<210> 172
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 172
ucacuugaac ucgggaggct t 21
<210> 173
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 173
aucacuugaa cucgggaggt t 21
<210> 174
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 174
aaucacuuga acucgggagt t 21
<210> 175
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 175
gaaucacuug aacucgggat t 21
<210> 176
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 176
agccaggaga aucacuugat t 21
<210> 177
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 177
ugagccagga gaaucacuut t 21
<210> 178
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 178
cugagccagg agaaucacut t 21
<210> 179
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 179
gcugagccag gagaaucact t 21
<210> 180
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 180
acagcugcuu gggaggcugt t 21
<210> 181
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 181
augacagcug cuugggaggt t 21
<210> 182
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 182
uaaugacagc ugcuugggat t 21
<210> 183
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 183
caggccugua augacagcut t 21
<210> 184
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 184
gcaggccugu aaugacagct t 21
<210> 185
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 185
ugcaggccug uaaugacagt t 21
<210> 186
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 186
ggugaaaccc uguuucucct t 21
<210> 187
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 187
auggugaaac ccuguuucut t 21
<210> 188
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 188
gccuauaauc ccagcacuut t 21
<210> 189
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 189
cgguggcuca ugccuauaat t 21
<210> 190
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 190
uguggaggaa ggggagaaut t 21
<210> 191
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 191
ucuguggagg aaggggagat t 21
<210> 192
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 192
caucuagggu ugcuaaaugt t 21
<210> 193
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 193
gcaucuaggg uugcuaaaut t 21
<210> 194
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 194
agcaucuagg guugcuaaat t 21
<210> 195
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 195
aagcaucuag gguugcuaat t 21
<210> 196
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 196
uaagcaucua ggguugcuat t 21
<210> 197
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 197
cacaccgggc cuccaguaut t 21
<210> 198
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 198
caggugugag ccaccacact t 21
<210> 199
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 199
uuacaggugu gagccaccat t 21
<210> 200
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 200
auuacaggug ugagccacct t 21
<210> 201
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 201
gauuacaggu gugagccact t 21
<210> 202
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 202
gggauuacag gugugagcct t 21
<210> 203
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 203
ugcugggauu acaggugugt t 21
<210> 204
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 204
gugcugggau uacaggugut t 21
<210> 205
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 205
aagugcuggg auuacaggut t 21
<210> 206
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 206
cccaaagugc ugggauuact t 21
<210> 207
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 207
ucccaaagug cugggauuat t 21
<210> 208
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 208
cucccaaagu gcugggauut t 21
<210> 209
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 209
ucggccuccc aaagugcugt t 21
<210> 210
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 210
cucguaaucc gaccgccuct t 21
<210> 211
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 211
accucguaau ccgaccgcct t 21
<210> 212
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 212
gaccucguaa uccgaccgct t 21
<210> 213
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 213
ugaccucgua auccgaccgt t 21
<210> 214
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 214
cugaccucgu aauccgacct t 21
<210> 215
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 215
ccugaccucg uaauccgact t 21
<210> 216
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 216
uccugaccuc guaauccgat t 21
<210> 217
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 217
cuccugaccu cguaauccgt t 21
<210> 218
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 218
gaacuccuga ccucguaaut t 21
<210> 219
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 219
ugaacuccug accucguaat t 21
<210> 220
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 220
uugaacuccu gaccucguat t 21
<210> 221
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 221
cuugaacucc ugaccucgut t 21
<210> 222
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 222
ggucuugaac uccugaccut t 21
<210> 223
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 223
gccaggcugg ucuugaacut t 21
<210> 224
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 224
uuaguaaaga ugggguuuct t 21
<210> 225
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 225
accaccacac ccggcuaaut t 21
<210> 226
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 226
gauuacaggc gcccaccact t 21
<210> 227
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 227
agcugggauu acaggcgcct t 21
<210> 228
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 228
ccccgaguag cugggauuat t 21
<210> 229
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 229
agcaauucug ccucagccct t 21
<210> 230
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 230
agguucaagc aauucugcct t 21
<210> 231
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 231
ccagguucaa gcaauucugt t 21
<210> 232
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 232
ccucugccuc ccagguucat t 21
<210> 233
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 233
accucugccu cccagguuct t 21
<210> 234
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 234
caaccucugc cucccaggut t 21
<210> 235
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 235
gugaucucag cucacugcat t 21
<210> 236
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 236
ugcaguggcg ugaucucagt t 21
<210> 237
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 237
uggaaugcag uggcgugaut t 21
<210> 238
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 238
gaguauugcu cugucaccct t 21
<210> 239
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 239
acagaguauu gcucugucat t 21
<210> 240
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 240
ugcgacagag uauugcucut t 21
<210> 241
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 241
cagccuccag uauucuuuut t 21
<210> 242
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 242
ccagccucca guauucuuut t 21
<210> 243
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 243
cucgcccagc cuccaguaut t 21
<210> 244
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 244
acagguguga gccaccucgt t 21
<210> 245
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 245
auuacaggug ugagccacct t 21
<210> 246
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 246
gggauuacag gugugagcct t 21
<210> 247
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 247
ugggauuaca ggugugagct t 21
<210> 248
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 248
ugcugggauu acaggugugt t 21
<210> 249
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 249
ucccaaaaug cugggauuat t 21
<210> 250
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 250
agcucaagau auuccgccct t 21
<210> 251
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 251
cuccugagcu caagauauut t 21
<210> 252
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 252
uguguaggcu ggucucgaat t 21
<210> 253
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 253
agcauauugu guaggcuggt t 21
<210> 254
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 254
cguuuggagc auauugugut t 21
<210> 255
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 255
gcguuuggag cauauugugt t 21
<210> 256
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 256
ggcguuugga gcauauugut t 21
<210> 257
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 257
uuuguagagg cggcguuugt t 21
<210> 258
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 258
caaguagcua ggaccacagt t 21
<210> 259
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 259
gagcucaagc gauccuccct t 21
<210> 260
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 260
ccgagcucaa gcgauccuct t 21
<210> 261
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 261
cauugcagcc ucgaccucct t 21
<210> 262
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 262
gcucauugca gccucgacct t 21
<210> 263
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 263
gcaccaucuc ggcucauugt t 21
<210> 264
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 264
ggcaccaucu cggcucauut t 21
<210> 265
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 265
ugcaguggca ccaucucggt t 21
<210> 266
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 266
agugcagugg caccaucuct t 21
<210> 267
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 267
gagugcagug gcaccaucut t 21
<210> 268
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 268
cagagugcag uggcaccaut t 21
<210> 269
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 269
ugucgucaga gugcaguggt t 21
<210> 270
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 270
cugucgucag agugcagugt t 21
<210> 271
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 271
gucucgcucu gucgucagat t 21
<210> 272
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 272
agucucgcuc ugucgucagt t 21
<210> 273
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 273
uguuuugaga cggagucuct t 21
<210> 274
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 274
uuguuuugag acggagucut t 21
<210> 275
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 275
cagauccuua aacacuagat t 21
<210> 276
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 276
ggcagauccu uaaacacuat t 21
<210> 277
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 277
gaaggcagau ccuuaaacat t 21
<210> 278
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 278
caggaaggaa ggcagaucct t 21
<210> 279
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 279
gacaaacaag gaaagacaat t 21
<210> 280
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 280
aagacaaaca aggaaagact t 21
<210> 281
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 281
ggaauuuaaa accugcuugt t 21
<210> 282
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 282
cuaggaauuu aaaaccugct t 21
<210> 283
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 283
cccuuggaaa aguaaaugut t 21
<210> 284
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 284
ucgucucgaa cuccagugut t 21
<210> 285
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 285
gccucgucuc gaacuccagt t 21
<210> 286
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 286
gcuuaggccu cgucucgaat t 21
<210> 287
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 287
guugcuuagg ccucgucuct t 21
<210> 288
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 288
uguugcuuag gccucgucut t 21
<210> 289
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 289
auguugcuua ggccucguct t 21
<210> 290
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 290
uuucggcaug uugcuuaggt t 21
<210> 291
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 291
guuucggcau guugcuuagt t 21
<210> 292
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 292
gguuucggca uguugcuuat t 21
<210> 293
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 293
cuauaggcgu gcgccaccat t 21
<210> 294
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 294
acuauaggcg ugcgccacct t 21
<210> 295
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 295
ccaguagcua ggacuauagt t 21
<210> 296
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 296
cccaguagcu aggacuauat t 21
<210> 297
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 297
aagcgauccu cccaccucat t 21
<210> 298
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 298
cgaucucggc ucacugcagt t 21
<210> 299
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 299
gcgaucucgg cucacugcat t 21
<210> 300
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 300
ucgcucaggc uggagugcat t 21
<210> 301
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 301
ugucgcucag gcuggagugt t 21
<210> 302
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 302
agacagagcc ucgcccugut t 21
<210> 303
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 303
uugagacaga gccucgccct t 21
<210> 304
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 304
uuugagacag agccucgcct t 21
<210> 305
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 305
uuuugagaca gagccucgct t 21
<210> 306
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 306
uuguuuugag acagagccut t 21
<210> 307
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 307
uuuguuuuga gacagagcct t 21
<210> 308
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 308
guuuguuuug agacagagct t 21
<210> 309
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 309
uguuuguuuu gagacagagt t 21
<210> 310
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 310
ccuuucauuu cacuguguut t 21
<210> 311
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 311
uccuuucauu ucacugugut t 21
<210> 312
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 312
caauccuuuc auuucacugt t 21
<210> 313
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 313
cucaauccuu ucauuucact t 21
<210> 314
<211> 21
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 314
cucucaaucc uuucauuuct t 21
<210> 315
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 315
atctgtgaga tgtaccttt 19
<210> 316
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 316
cactctgtca ctcaggctg 19
<210> 317
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 317
actctgtcac tcaggctgg 19
<210> 318
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 318
tctgtcactc aggctggag 19
<210> 319
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 319
tcaggctgga gtgcagtgg 19
<210> 320
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 320
gagtgcagtg gcgtgatct 19
<210> 321
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 321
tgcagtggcg tgatcttgg 19
<210> 322
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 322
gcagtggcgt gatcttggc 19
<210> 323
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 323
ggcgtgatct tggctcact 19
<210> 324
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 324
gtgatcttgg ctcactgca 19
<210> 325
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 325
tgatcttggc tcactgcaa 19
<210> 326
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 326
atcttggctc actgcaacc 19
<210> 327
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 327
cttggctcac tgcaacctc 19
<210> 328
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 328
ttggctcact gcaacctcc 19
<210> 329
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 329
gcctcccgag ttcaagtga 19
<210> 330
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 330
cctcccgagt tcaagtgat 19
<210> 331
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 331
ctcccgagtt caagtgatt 19
<210> 332
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 332
tcccgagttc aagtgattc 19
<210> 333
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 333
tcaagtgatt ctcctggct 19
<210> 334
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 334
aagtgattct cctggctca 19
<210> 335
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 335
agtgattctc ctggctcag 19
<210> 336
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 336
gtgattctcc tggctcagc 19
<210> 337
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 337
cagcctccca agcagctgt 19
<210> 338
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 338
cctcccaagc agctgtcat 19
<210> 339
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 339
tcccaagcag ctgtcatta 19
<210> 340
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 340
agctgtcatt acaggcctg 19
<210> 341
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 341
gctgtcatta caggcctgc 19
<210> 342
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 342
ctgtcattac aggcctgca 19
<210> 343
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 343
ggagaaacag ggtttcacc 19
<210> 344
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 344
agaaacaggg tttcaccat 19
<210> 345
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 345
aagtgctggg attataggc 19
<210> 346
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 346
ttataggcat gagccaccg 19
<210> 347
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 347
attctcccct tcctccaca 19
<210> 348
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 348
tctccccttc ctccacaga 19
<210> 349
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 349
catttagcaa ccctagatg 19
<210> 350
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 350
atttagcaac cctagatgc 19
<210> 351
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 351
tttagcaacc ctagatgct 19
<210> 352
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 352
ttagcaaccc tagatgctt 19
<210> 353
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 353
tagcaaccct agatgctta 19
<210> 354
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 354
atactggagg cccggtgtg 19
<210> 355
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 355
gtgtggtggc tcacacctg 19
<210> 356
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 356
tggtggctca cacctgtaa 19
<210> 357
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 357
ggtggctcac acctgtaat 19
<210> 358
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 358
gtggctcaca cctgtaatc 19
<210> 359
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 359
ggctcacacc tgtaatccc 19
<210> 360
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 360
cacacctgta atcccagca 19
<210> 361
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 361
acacctgtaa tcccagcac 19
<210> 362
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 362
acctgtaatc ccagcactt 19
<210> 363
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 363
gtaatcccag cactttggg 19
<210> 364
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 364
taatcccagc actttggga 19
<210> 365
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 365
aatcccagca ctttgggag 19
<210> 366
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 366
cagcactttg ggaggccga 19
<210> 367
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 367
gaggcggtcg gattacgag 19
<210> 368
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 368
ggcggtcgga ttacgaggt 19
<210> 369
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 369
gcggtcggat tacgaggtc 19
<210> 370
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 370
cggtcggatt acgaggtca 19
<210> 371
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 371
ggtcggatta cgaggtcag 19
<210> 372
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 372
gtcggattac gaggtcagg 19
<210> 373
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 373
tcggattacg aggtcagga 19
<210> 374
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 374
cggattacga ggtcaggag 19
<210> 375
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 375
attacgaggt caggagttc 19
<210> 376
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 376
ttacgaggtc aggagttca 19
<210> 377
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 377
tacgaggtca ggagttcaa 19
<210> 378
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 378
acgaggtcag gagttcaag 19
<210> 379
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 379
aggtcaggag ttcaagacc 19
<210> 380
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 380
agttcaagac cagcctggc 19
<210> 381
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 381
gaaaccccat ctttactaa 19
<210> 382
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 382
attagccggg tgtggtggt 19
<210> 383
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 383
gtggtgggcg cctgtaatc 19
<210> 384
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 384
ggcgcctgta atcccagct 19
<210> 385
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 385
taatcccagc tactcgggg 19
<210> 386
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 386
gggctgaggc agaattgct 19
<210> 387
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 387
ggcagaattg cttgaacct 19
<210> 388
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 388
cagaattgct tgaacctgg 19
<210> 389
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 389
tgaacctggg aggcagagg 19
<210> 390
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 390
gaacctggga ggcagaggt 19
<210> 391
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 391
acctgggagg cagaggttg 19
<210> 392
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 392
tgcagtgagc tgagatcac 19
<210> 393
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 393
ctgagatcac gccactgca 19
<210> 394
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 394
atcacgccac tgcattcca 19
<210> 395
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 395
gggtgacaga gcaatactc 19
<210> 396
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 396
tgacagagca atactctgt 19
<210> 397
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 397
agagcaatac tctgtcgca 19
<210> 398
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 398
aaaagaatac tggaggctg 19
<210> 399
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 399
aaagaatact ggaggctgg 19
<210> 400
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 400
atactggagg ctgggcgag 19
<210> 401
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 401
cgaggtggct cacacctgt 19
<210> 402
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 402
ggtggctcac acctgtaat 19
<210> 403
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 403
ggctcacacc tgtaatccc 19
<210> 404
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 404
gctcacacct gtaatccca 19
<210> 405
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 405
cacacctgta atcccagca 19
<210> 406
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 406
taatcccagc attttggga 19
<210> 407
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 407
gggcggaata tcttgagct 19
<210> 408
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 408
aatatcttga gctcaggag 19
<210> 409
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 409
ttcgagacca gcctacaca 19
<210> 410
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 410
ccagcctaca caatatgct 19
<210> 411
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 411
acacaatatg ctccaaacg 19
<210> 412
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 412
cacaatatgc tccaaacgc 19
<210> 413
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 413
acaatatgct ccaaacgcc 19
<210> 414
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 414
caaacgccgc ctctacaaa 19
<210> 415
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 415
ctgtggtcct agctacttg 19
<210> 416
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 416
gggaggatcg cttgagctc 19
<210> 417
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 417
gaggatcgct tgagctcgg 19
<210> 418
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 418
ggaggtcgag gctgcaatg 19
<210> 419
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 419
ggtcgaggct gcaatgagc 19
<210> 420
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 420
caatgagccg agatggtgc 19
<210> 421
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 421
aatgagccga gatggtgcc 19
<210> 422
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 422
ccgagatggt gccactgca 19
<210> 423
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 423
gagatggtgc cactgcact 19
<210> 424
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 424
agatggtgcc actgcactc 19
<210> 425
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 425
atggtgccac tgcactctg 19
<210> 426
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 426
ccactgcact ctgacgaca 19
<210> 427
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 427
cactgcactc tgacgacag 19
<210> 428
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 428
tctgacgaca gagcgagac 19
<210> 429
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 429
ctgacgacag agcgagact 19
<210> 430
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 430
gagactccgt ctcaaaaca 19
<210> 431
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 431
agactccgtc tcaaaacaa 19
<210> 432
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 432
tctagtgttt aaggatctg 19
<210> 433
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 433
tagtgtttaa ggatctgcc 19
<210> 434
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 434
tgtttaagga tctgccttc 19
<210> 435
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 435
ggatctgcct tccttcctg 19
<210> 436
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 436
ttgtctttcc ttgtttgtc 19
<210> 437
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 437
gtctttcctt gtttgtctt 19
<210> 438
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 438
caagcaggtt ttaaattcc 19
<210> 439
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 439
gcaggtttta aattcctag 19
<210> 440
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 440
acatttactt ttccaaggg 19
<210> 441
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 441
acactggagt tcgagacga 19
<210> 442
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 442
ctggagttcg agacgaggc 19
<210> 443
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 443
ttcgagacga ggcctaagc 19
<210> 444
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 444
gagacgaggc ctaagcaac 19
<210> 445
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 445
agacgaggcc taagcaaca 19
<210> 446
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 446
gacgaggcct aagcaacat 19
<210> 447
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 447
cctaagcaac atgccgaaa 19
<210> 448
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 448
ctaagcaaca tgccgaaac 19
<210> 449
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 449
taagcaacat gccgaaacc 19
<210> 450
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 450
tggtggcgca cgcctatag 19
<210> 451
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 451
ggtggcgcac gcctatagt 19
<210> 452
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 452
ctatagtcct agctactgg 19
<210> 453
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 453
tatagtccta gctactggg 19
<210> 454
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 454
tgaggtggga ggatcgctt 19
<210> 455
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 455
ctgcagtgag ccgagatcg 19
<210> 456
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 456
tgcagtgagc cgagatcgc 19
<210> 457
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 457
tgcactccag cctgagcga 19
<210> 458
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 458
cactccagcc tgagcgaca 19
<210> 459
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 459
acagggcgag gctctgtct 19
<210> 460
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 460
gggcgaggct ctgtctcaa 19
<210> 461
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 461
ggcgaggctc tgtctcaaa 19
<210> 462
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 462
gcgaggctct gtctcaaaa 19
<210> 463
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 463
aggctctgtc tcaaaacaa 19
<210> 464
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 464
ggctctgtct caaaacaaa 19
<210> 465
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 465
gctctgtctc aaaacaaac 19
<210> 466
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 466
ctctgtctca aaacaaaca 19
<210> 467
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 467
aacacagtga aatgaaagg 19
<210> 468
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 468
acacagtgaa atgaaagga 19
<210> 469
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 469
cagtgaaatg aaaggattg 19
<210> 470
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 470
gtgaaatgaa aggattgag 19
<210> 471
<211> 19
<212> ДНК
<213> Искусственно синтезированная
<400> 471
gaaatgaaag gattgagag 19
<210> 472
<211> 19
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 472
acuacugagu gacaguagatt 21
<210> 473
<211> 19
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 473
ucuacuguca cucaguagutt 21
<210> 474
<211> 19
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 474
gggaugauac agcacugautt 21
<210> 475
<211> 19
<212> ДНК/РНК
<213> Искусственно синтезированная
<400> 475
aucagugcug uaucauccctt 21
<210> 476
<211> 159
<212> ДНК
<213> Искусственно синтезированная
<400> 476
agtcgcactc tgtcactcag gctggagtgc agtggcgtga tcttggctca ctgcaacctc 60
cgcctcccga gttcaagtga ttctcctggc tcagcctccc aagcagctgt cattacaggc 120
ctgcaccacc acacccggct gatttttgta tttttagga 159
<210> 477
<211> 82
<212> ДНК
<213> Искусственно синтезированная
<400> 477
aatactggag gcccggtgtg gtggctcaca cctgtaatcc cagcactttg ggaggccgag 60
gcggtcggat tacgaggtca gg 82
<210> 478
<211> 815
<212> ДНК
<213> Искусственно синтезированная
<400> 478
ctggccaaca tggtgaaacc ccatctttac taaaaataca aaaattagcc gggtgtggtg 60
gtgggcgcct gtaatcccag ctactcgggg ggctgaggca gaattgcttg aacctgggag 120
gcagaggttg cagtgagctg agatcacgcc actgcattcc agcctgggtg acagagcaat 180
actctgtcgc aaaaaaaaaa aagaatactg gaggctgggc gaggtggctc acacctgtaa 240
tcccagcatt ttgggatgcc agaggcgggc ggaatatctt gagctcagga gttcgagacc 300
agcctacaca atatgctcca aacgccgcct ctacaaaaca tacagaaact agccgggtgt 360
ggtggcgtgc ccctgtggtc ctagctactt gggaggttga ggcgggagga tcgcttgagc 420
tcgggaggtc gaggctgcaa tgagccgaga tggtgccact gcactctgac gacagagcga 480
gactccgtct caaaacaaac aacaaataag gttgggggat caaatatctt ctagtgttta 540
aggatctgcc ttccttcctg cccccatgtt tgtctttcct tgtttgtctt tatatagatc 600
aagcaggttt taaattccta gtaggagctt acatttactt ttccaagggg gagggggaat 660
aaatatctac acacacacac acacacacac acacacacac acactggagt tcgagacgag 720
gcctaagcaa catgccgaaa ccccgtctct actaaataca aaaaatagct gagcttggtg 780
gcgcacgcct atagtcctag ctactgggga ggctg 815
<210> 479
<211> 108
<212> ДНК
<213> Искусственно синтезированная
<400> 479
ctgcagtgag ccgagatcgc gccgctgcac tccagcctga gcgacagggc gaggctctgt 60
ctcaaaacaa acaaacaaaa aaaaaaggaa aggaaatata acacagtg 108
<210> 480
<211> 18
<212> ДНК
<213> Искусственно синтезированная
<400> 480
cacaggccag agcgatga 18
<210> 481
<211> 23
<212> ДНК
<213> Искусственно синтезированная
<400> 481
cgaagtttca caaatgtcac cat 23
<210> 482
<211> 21
<212> ДНК
<213> Искусственно синтезированная
<400> 482
atggacagga ctgaacgtct t 21
<210> 483
<211> 20
<212> ДНК
<213> Искусственно синтезированная
<400> 483
tccagcaggt cagcaaagaa 20
<210> 484
<211> 20
<212> ДНК
<213> Искусственно синтезированная
<400> 484
ataatcccaa gcggtttgct 20
<210> 485
<211> 20
<212> ДНК
<213> Искусственно синтезированная
<400> 485
ctgccagtct ggactgttct 20
<210> 486
<211> 20
<212> ДНК
<213> Искусственно синтезированная
<400> 486
ccaccacctc ccatatgtcc 20
<210> 487
<211> 22
<212> ДНК
<213> Искусственно синтезированная
<400> 487
gctctatgcc agcatttctc ct 22
<210> 488
<211> 20
<212> ДНК
<213> Искусственно синтезированная
<400> 488
cccccaccac ctcccatatg 20
<210> 489
<211> 25
<212> ДНК
<213> Искусственно синтезированная
<400> 489
cccttctcac agctcataaa attac 25
<210> 490
<211> 33
<212> ДНК
<213> Искусственно синтезированная
<400> 490
taagcaggat ccatggcgat gagcagcggc ggc 33
<210> 491
<211> 33
<212> ДНК
<213> Искусственно синтезированная
<400> 491
taagcagaat tcttaattta aggaatgtga gca 33
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМПОЗИЦИИ И СПОСОБЫ МОДУЛЯЦИИ SMN2 СПЛАЙСИНГА У СУБЪЕКТА | 2019 |
|
RU2793459C2 |
| Генетическая конструкция, адаптированная для доставки гена SMN1 человека с помощью аденоассоциированного вируса серотипа 2 для обеспечения нейроспецифичной экспрессии | 2022 |
|
RU2801848C1 |
| Генетическая конструкция, содержащая последовательности химерных направляющих РНК для делеции гена SMN1 человека в культурах клеток человека | 2022 |
|
RU2816897C2 |
| ИСКУССТВЕННАЯ МОДИФИКАЦИЯ ГЕНОМА ДЛЯ РЕГУЛЯЦИИ ЭКСПРЕССИИ ГЕНА | 2018 |
|
RU2767201C2 |
| ИСКУССТВЕННЫЕ МОЛЕКУЛЫ НУКЛЕИНОВОЙ КИСЛОТЫ | 2014 |
|
RU2717986C2 |
| ОДНОЦЕПОЧЕЧНАЯ МОЛЕКУЛА НУКЛЕИНОВОЙ КИСЛОТЫ, ИНГИБИРУЮЩАЯ ЭКСПРЕССИЮ ГЕНА ПРОРЕНИНА ИЛИ ГЕНА РЕЦЕПТОРА ПРОРЕНИНА, И ЕЕ ПРИМЕНЕНИЕ | 2016 |
|
RU2718984C2 |
| ДНК-СОДЕРЖАЩИЕ ПОЛИНУКЛЕОТИДЫ И РУКОВОДЯЩИЕ ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ СИСТЕМ CRISPR ТИПА V И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2021 |
|
RU2832314C1 |
| Синергетическое действие SMN1 и miR-23a при лечении спинальной мышечной атрофии | 2021 |
|
RU2839898C2 |
| СТРУКТУРЫ МИРНК С ВЫСОКОЙ АКТИВНОСТЬЮ И СНИЖЕННЫМ ВОЗДЕЙСТВИЕМ ВНЕ МИШЕНИ | 2016 |
|
RU2788030C2 |
| НУКЛЕИНОВЫЕ КИСЛОТЫ ДЛЯ ИНГИБИРОВАНИЯ ЭКСПРЕССИИ LPA В КЛЕТКЕ | 2018 |
|
RU2822093C1 |
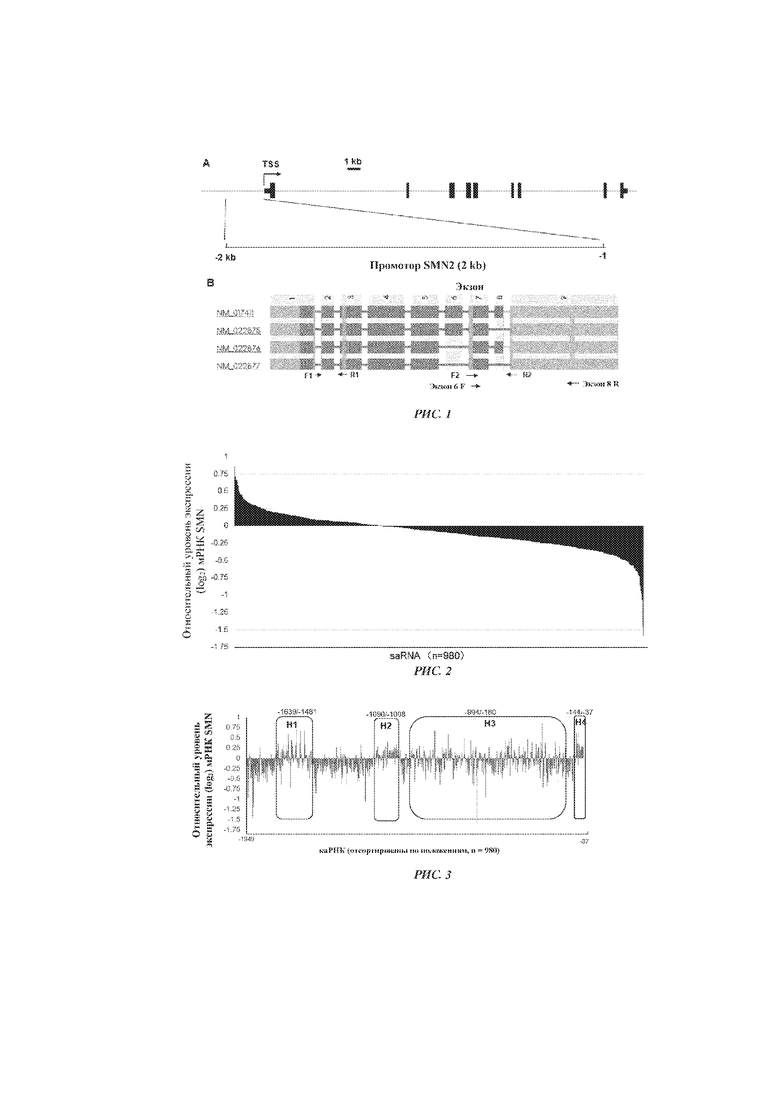
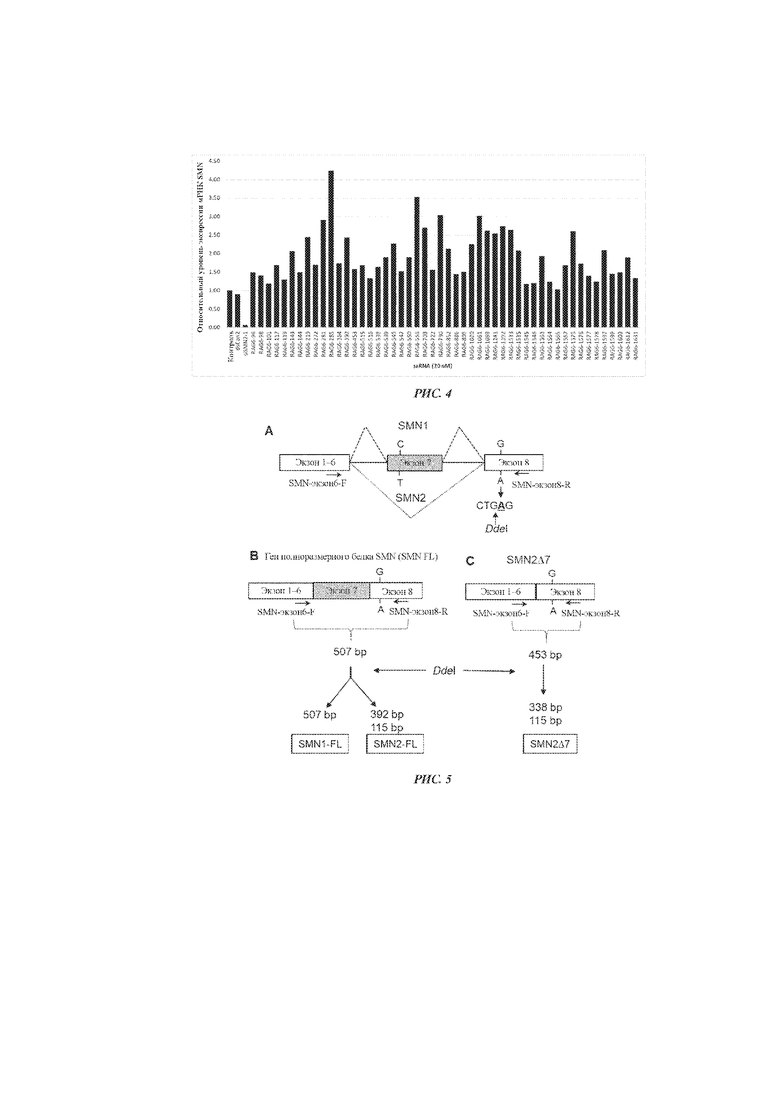
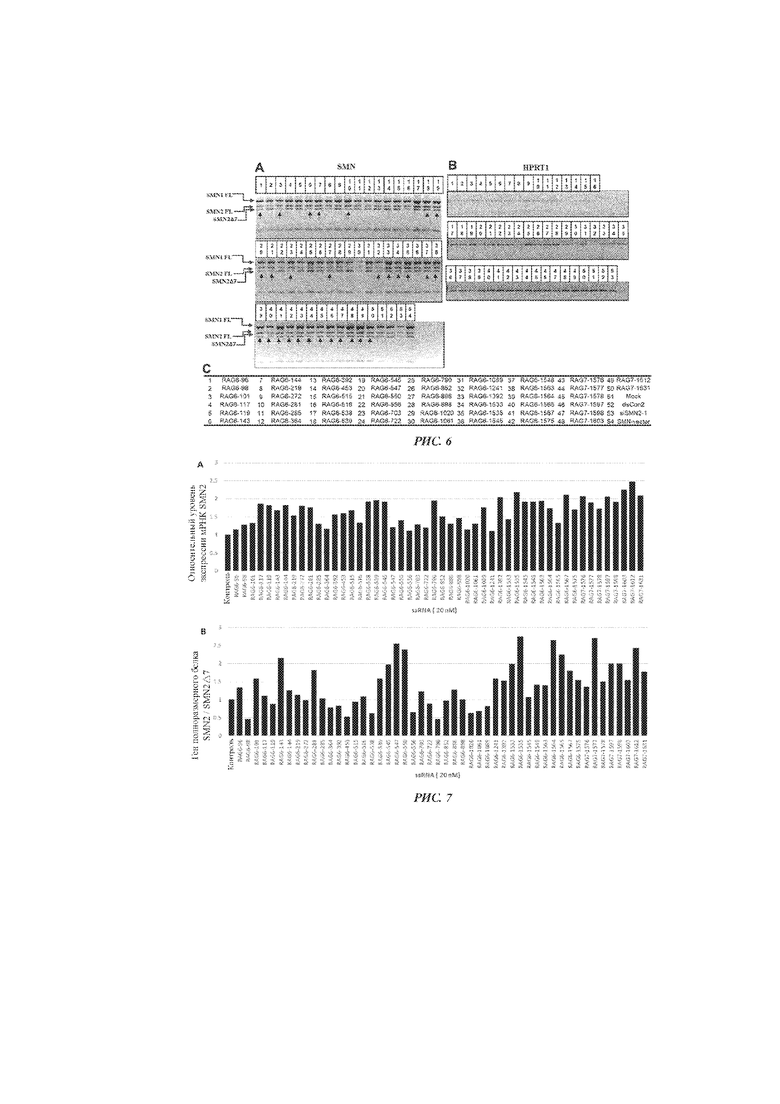
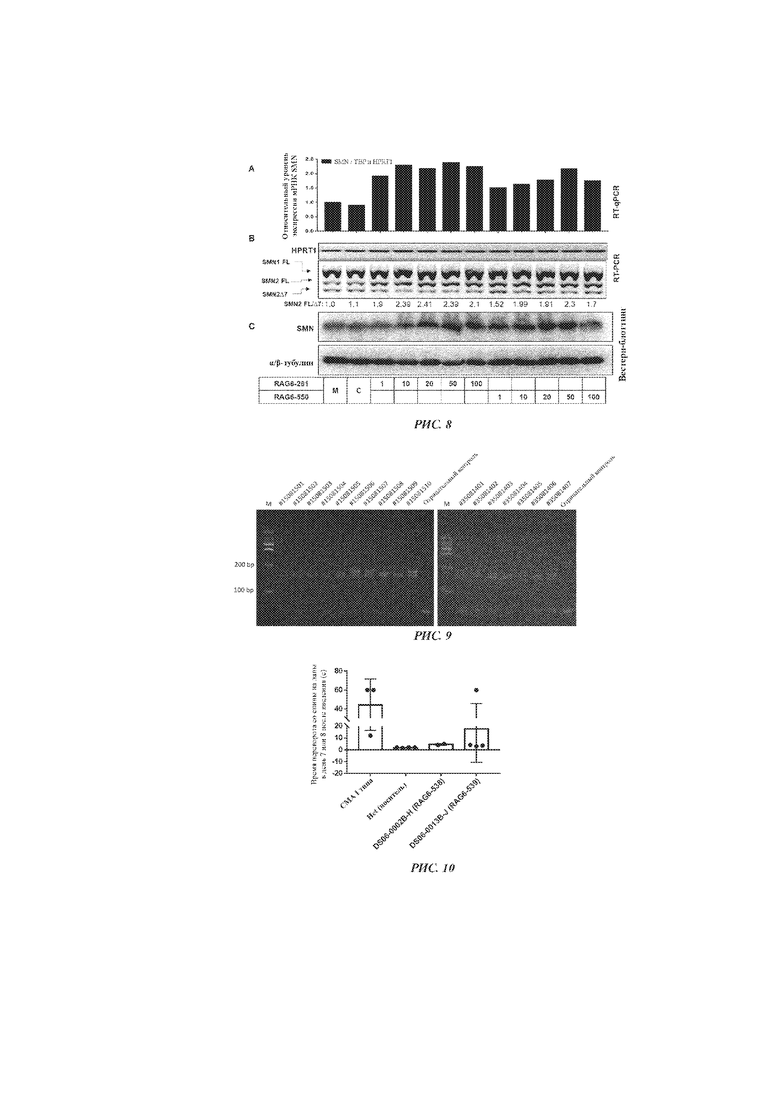
Изобретение относится к молекулярной биологии и медицине, в частности к молекуле короткой активирующей нуклеиновой кислоты для лечения СМА. Молекула короткой активирующей нуклеиновой кислоты содержит смысловую и антисмысловую цепи нуклеиновой кислоты, представляющие собой независимые олигонуклеотидные цепи длиной от 16 до 35 нуклеотидов, и одна из нуклеотидных цепей по крайней мере на 75% гомологична или комплементарна целевой последовательности нуклеотидов из промоторной области целевого гена SMN2. Изобретение также относится к фармацевтической композиции, содержащей молекулу короткой активирующей нуклеиновой кислоты, методу стимуляции экспрессии целевого гена в клетках и методам лечения заболевания, вызванного недостаточной экспрессией целевого гена. Изобретение предлагает новую молекулу короткой активирующей нуклеиновой кислоты, которая способна активировать транскрипцию SMN2 с высокой специфичностью, действуя на промотор гена SMN2. 11 н. и 32 з.п. ф-лы, 10 ил., 9 табл., 6 пр.
1. Двуцепочечная молекула нуклеиновой кислоты для активации/стимуляции экспрессии гена SMN2 в клетке, содержащая фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты, в которой фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты содержат комплементарные области, и при этом комплементарные области образуют структуру двухцепочечной нуклеиновой кислоты, которая может активировать экспрессию гена SMN2 в клетках, где фрагмент смысловой нуклеиновой кислоты или фрагмент антисмысловой нуклеиновой кислоты двуцепочечной молекулы нуклеиновой кислоты по крайней мере на 90% гомологичен или комплементарен непрерывной нуклеотидной последовательности длиной от 19 нуклеотидов в промоторной области гена SMN2 от -1639 до -1481 последовательности SEQ ID №: 476, области от -1090 до -1008 последовательности SEQ ID №: 477, области от -994 до -180 последовательности SEQ ID №: 478 или области от -144 до -37 последовательности SEQ ID №: 479 выше сайта начала транскрипции гена SMN2, где двуцепочечная молекула нуклеиновой кислоты активирует/стимулирует экспрессию гена SMN2 как минимум на 10% по сравнению с исходной экспрессией в клетке.
2. Двуцепочечная молекула нуклеиновой кислоты по п. 1, в которой фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты находятся в двух разных цепях нуклеиновой кислоты.
3. Двуцепочечная молекула нуклеиновой кислоты по п. 1, в которой фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты находятся в одной и той же цепи нуклеиновой кислоты, предпочтительно в форме «шпильки», образованной одной цепью нуклеиновой кислоты, причем комплементарные области фрагмента смысловой нуклеиновой кислоты и фрагмента антисмысловой нуклеиновой кислоты образуют структуру двухцепочечной нуклеиновой кислоты.
4. Двуцепочечная молекула нуклеиновой кислоты по п. 2, в которой как минимум одна цепь имеет липкий 3'-конец длиной от 0 до 6 нуклеотидов.
5. Двуцепочечная молекула нуклеиновой кислоты по п. 4, в которой обе цепи имеют липкий 3'-конец длиной от 2 до 3 нуклеотидов.
6. Двуцепочечная молекула нуклеиновой кислоты по любому из пп. 1-5, в которой фрагмент смысловой нуклеиновой кислоты и фрагмент антисмысловой нуклеиновой кислоты независимо имеют длину от 16 до 35 нуклеотидов.
7. Двуцепочечная молекула нуклеиновой кислоты по п. 1, в которой фрагмент смысловой нуклеиновой кислоты или фрагмент антисмысловой нуклеиновой кислоты двуцепочечной молекулы нуклеиновой кислоты по крайней мере на 75% гомологичен или комплементарен последовательности нуклеотидов, выбранной из SEQ ID №: 315-471.
8. Двуцепочечная молекула нуклеиновой кислоты по п. 7, в которой смысловой фрагмент двуцепочечной молекулы нуклеиновой кислоты по крайней мере на 75% гомологичен последовательности нуклеотидов, выбранной из SEQ ID №: 1-157, а антисмысловой фрагмент двуцепочечной молекулы нуклеиновой кислоты по крайней мере на 75% гомологичен последовательности нуклеотидов, выбранной из SEQ ID №: 158-314.
9. Двуцепочечная молекула нуклеиновой кислоты по п. 8, в которой смысловой фрагмент двуцепочечной молекулы нуклеиновой кислоты содержит последовательность нуклеотидов, выбранную из SEQ ID №: 1-157, или идентичен ей, а антисмысловой фрагмент двуцепочечной молекулы нуклеиновой кислоты содержит последовательность нуклеотидов, выбранную из SEQ ID №: 158-314, или идентичен ей.
10. Двуцепочечная молекула нуклеиновой кислоты по п. 9, где комбинацию фрагмента смысловой нуклеиновой кислоты и фрагмента антисмысловой нуклеиновой кислоты выбирают из группы, состоящей из SEQ ID NO: 1 и SEQ ID NO: 158; SEQ ID NO: 2 и SEQ ID NO: 159; SEQ ID NO: 3 и SEQ ID NO: 160; SEQ ID NO: 4 и SEQ ID NO: 161; SEQ ID NO: 5 и SEQ ID NO: 162; SEQ ID NO: 6 и SEQ ID NO: 163; SEQ ID NO: 7 и SEQ ID NO: 164; SEQ ID NO: 8 и SEQ ID NO: 165; SEQ ID NO: 9 и SEQ ID NO: 166; SEQ ID NO: 10 и SEQ ID NO: 167; SEQ ID NO: 11 и SEQ ID NO: 168; SEQ ID NO: 12 и SEQ ID NO: 169; SEQ ID NO: 13 и SEQ ID NO: 170; SEQ ID NO: 14 и SEQ ID NO: 171; SEQ ID NO: 15 и SEQ ID NO: 172; SEQ ID NO: 16 и SEQ ID NO: 173; SEQ ID NO: 17 и SEQ ID NO: 174; SEQ ID NO: 18 и SEQ ID NO: 175; SEQ ID NO: 19 и SEQ ID NO: 176; SEQ ID NO: 20 и SEQ ID NO: 177; SEQ ID NO: 21 и SEQ ID NO: 178; SEQ ID NO: 22 и SEQ ID NO: 179; SEQ ID NO: 23 и SEQ ID NO: 180; SEQ ID NO: 24 и SEQ ID NO: 181; SEQ ID NO: 25 и SEQ ID NO: 182; SEQ ID NO: 26 и SEQ ID NO: 183; SEQ ID NO: 27 и SEQ ID NO: 184; SEQ ID NO: 28 и SEQ ID NO: 185; SEQ ID NO: 29 и SEQ ID NO: 186; SEQ ID NO: 30 и SEQ ID NO: 187; SEQ ID NO: 31 и SEQ ID NO: 188; SEQ ID NO: 32 и SEQ ID NO: 189; SEQ ID NO: 33 и SEQ ID NO: 190; SEQ ID NO: 34 и SEQ ID NO: 191; SEQ ID NO: 35 и SEQ ID NO: 192; SEQ ID NO: 36 и SEQ ID NO: 193; SEQ ID NO: 37 и SEQ ID NO: 194; SEQ ID NO: 38 и SEQ ID NO: 195; SEQ ID NO: 39 и SEQ ID NO: 196; SEQ ID NO: 40 и SEQ ID NO: 197; SEQ ID NO: 41 и SEQ ID NO: 198; SEQ ID NO: 42 и SEQ ID NO: 199; SEQ ID NO: 43 и SEQ ID NO: 200; SEQ ID NO: 44 и SEQ ID NO: 201; SEQ ID NO: 45 и SEQ ID NO: 202; SEQ ID NO: 46 и SEQ ID NO: 203; SEQ ID NO: 47 и SEQ ID NO: 204; SEQ ID NO: 48 и SEQ ID NO: 205; SEQ ID NO: 49 и SEQ ID NO: 206; SEQ ID NO: 50 и SEQ ID NO: 207; SEQ ID NO: 51 и SEQ ID NO: 208; SEQ ID NO: 52 и SEQ ID NO: 209; SEQ ID NO: 53 и SEQ ID NO: 210; SEQ ID NO: 54 и SEQ ID NO: 211; SEQ ID NO: 55 и SEQ ID NO: 212; SEQ ID NO: 56 и SEQ ID NO: 213; SEQ ID NO: 57 и SEQ ID NO: 214; SEQ ID NO: 58 и SEQ ID NO: 215; SEQ ID NO: 59 и SEQ ID NO: 216; SEQ ID NO: 60 и SEQ ID NO: 217; SEQ ID NO: 61 и SEQ ID NO: 218; SEQ ID NO: 62 и SEQ ID NO: 219; SEQ ID NO: 63 и SEQ ID NO: 220; SEQ ID NO: 64 и SEQ ID NO: 221; SEQ ID NO: 65 и SEQ ID NO: 222; SEQ ID NO: 66 и SEQ ID NO: 223; SEQ ID NO: 67 и SEQ ID NO: 224; SEQ ID NO: 68 и SEQ ID NO: 225; SEQ ID NO: 69 и SEQ ID NO: 226; SEQ ID NO: 70 и SEQ ID NO: 227; SEQ ID NO: 71 и SEQ ID NO: 228; SEQ ID NO: 72 и SEQ ID NO: 229; SEQ ID NO: 73 и SEQ ID NO: 230; SEQ ID NO: 74 и SEQ ID NO: 231; SEQ ID NO: 75 и SEQ ID NO: 232; SEQ ID NO: 76 и SEQ ID NO: 233; SEQ ID NO: 77 и SEQ ID NO: 234; SEQ ID NO: 78 и SEQ ID NO: 235; SEQ ID NO: 79 и SEQ ID NO: 236; SEQ ID NO: 80 и SEQ ID NO: 237; SEQ ID NO: 81 и SEQ ID NO: 238; SEQ ID NO: 82 и SEQ ID NO: 239; SEQ ID NO: 83 и SEQ ID NO: 240; SEQ ID NO: 84 и SEQ ID NO: 241; SEQ ID NO: 85 и SEQ ID NO: 242; SEQ ID NO: 86 и SEQ ID NO: 243; SEQ ID NO: 87 и SEQ ID NO: 244; SEQ ID NO: 88 и SEQ ID NO: 245; SEQ ID NO: 89 и SEQ ID NO: 246; SEQ ID NO: 90 и SEQ ID NO: 247; SEQ ID NO: 91 и SEQ ID NO: 248; SEQ ID NO: 92 и SEQ ID NO: 249; SEQ ID NO: 93 и SEQ ID NO: 250; SEQ ID NO: 94 и SEQ ID NO: 251; SEQ ID NO: 95 и SEQ ID NO: 252; SEQ ID NO: 96 и SEQ ID NO: 253; SEQ ID NO: 97 и SEQ ID NO: 254; SEQ ID NO: 98 и SEQ ID NO: 255; SEQ ID NO: 99 и SEQ ID NO: 256; SEQ ID NO: 100 и SEQ ID NO: 257; SEQ ID NO: 101 и SEQ ID NO: 258; SEQ ID NO: 102 и SEQ ID NO: 259; SEQ ID NO: 103 и SEQ ID NO: 260; SEQ ID NO: 104 и SEQ ID NO: 261; SEQ ID NO: 105 и SEQ ID NO: 262; SEQ ID NO: 106 и SEQ ID NO: 263; SEQ ID NO: 107 и SEQ ID NO: 264; SEQ ID NO: 108 и SEQ ID NO: 265; SEQ ID NO: 109 и SEQ ID NO: 266; SEQ ID NO: 110 и SEQ ID NO: 267; SEQ ID NO: 111 и SEQ ID NO: 268; SEQ ID NO: 112 и SEQ ID NO: 269; SEQ ID NO: 113 и SEQ ID NO: 270; SEQ ID NO: 114 и SEQ ID NO: 271; SEQ ID NO: 115 и SEQ ID NO: 272; SEQ ID NO: 116 и SEQ ID NO: 273; SEQ ID NO: 117 и SEQ ID NO: 274; SEQ ID NO: 118 и SEQ ID NO: 275; SEQ ID NO: 119 и SEQ ID NO: 276; SEQ ID NO: 120 и SEQ ID NO: 277; SEQ ID NO: 121 и SEQ ID NO: 278; SEQ ID NO: 122 и SEQ ID NO: 279; SEQ ID NO: 123 и SEQ ID NO: 280; SEQ ID NO: 124 и SEQ ID NO: 281; SEQ ID NO: 125 и SEQ ID NO: 282; SEQ ID NO: 126 и SEQ ID NO: 283; SEQ ID NO: 127 и SEQ ID NO: 284; SEQ ID NO: 128 и SEQ ID NO: 285; SEQ ID NO: 129 и SEQ ID NO: 286; SEQ ID NO: 130 и SEQ ID NO: 287; SEQ ID NO: 131 и SEQ ID NO: 288; SEQ ID NO: 132 и SEQ ID NO: 289; SEQ ID NO: 133 и SEQ ID NO: 290; SEQ ID NO: 134 и SEQ ID NO: 291; SEQ ID NO: 135 и SEQ ID NO: 292; SEQ ID NO: 136 и SEQ ID NO: 293; SEQ ID NO: 137 и SEQ ID NO: 294; SEQ ID NO: 138 и SEQ ID NO: 295; SEQ ID NO: 139 и SEQ ID NO: 296; SEQ ID NO: 140 и SEQ ID NO: 297; SEQ ID NO: 141 и SEQ ID NO: 298; SEQ ID NO: 142 и SEQ ID NO: 299; SEQ ID NO: 143 и SEQ ID NO: 300; SEQ ID NO: 144 и SEQ ID NO: 301; SEQ ID NO: 145 и SEQ ID NO: 302; SEQ ID NO: 146 и SEQ ID NO: 303; SEQ ID NO: 147 и SEQ ID NO: 304; SEQ ID NO: 148 и SEQ ID NO: 305; SEQ ID NO: 149 и SEQ ID NO: 306; SEQ ID NO: 150 и SEQ ID NO: 307; SEQ ID NO: 151 и SEQ ID NO: 308; SEQ ID NO: 152 и SEQ ID NO: 309; SEQ ID NO: 153 и SEQ ID NO: 310; SEQ ID NO: 154 и SEQ ID NO: 311; SEQ ID NO: 155 и SEQ ID NO: 312; SEQ ID NO: 156 и SEQ ID NO: 313; и SEQ ID NO: 157 и SEQ ID NO: 314.
11. Двуцепочечная молекула нуклеиновой кислоты по любому из пп. 1-10, в которой как минимум один нуклеотид является химически модифицированным.
12. Двуцепочечная молекула нуклеиновой кислоты по п. 11, в которой химически модифицированный нуклеотид имеет как минимум одну из следующих модификаций:
a) модификация фосфодиэфирной связи, соединяющей нуклеотиды в нуклеотидной последовательности двуцепочечной молекулы нуклеиновой кислоты;
b) модификация 2'-OH рибозы в нуклеотидной последовательности двуцепочечной молекулы нуклеиновой кислоты; и
с) модификация основания в нуклеотидной последовательности двуцепочечной молекулы нуклеиновой кислоты.
13. Нуклеиновая кислота, кодирующая двуцепочечную молекулу нуклеиновой кислоты по любому из пп. 1-10, где нуклеиновая кислота имеет по меньшей мере 75% гомологию или комплементарность непрерывной нуклеотидной последовательности длиной от 16 до 35 в последовательности SEQ ID NO: 476, SEQ ID NO: 477, SEQ ID NO: 478 или SEQ ID NO: 479.
14. Нуклеиновая кислота по п. 13, где нуклеиновая кислота является молекулой ДНК.
15. Экспрессирующая ген SMN2 клетка, имеющая активированную/стимулированную экспрессию гена SMN2, где указанная клетка содержит терапевтически эффективное количество двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, или нуклеиновой кислоты по п. 13 или 14.
16. Композиция для активации/стимуляции экспрессии гена SMN2 в клетке, содержащая терапевтически эффективное количество двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12 или нуклеиновой кислоты по п. 13 или 14 и фармацевтически приемлемый носитель.
17. Композиция по п. 16, где композиция содержит фармацевтически приемлемый носитель.
18. Композиция по п. 16, в которой фармацевтически приемлемый носитель выбран из водного носителя, липосомы, высокомолекулярного полимера и/или полипептида.
19. Композиция по п. 16 или 17, где композиция содержит двуцепочечную молекулу нуклеиновой кислоты в концентрации 1-150 нМ.
20. Способ терапии заболевания или состояния, вызванного недостаточной экспрессией белка SMN у индивидуума, включающий введение терапевтической дозы двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19 индивидууму.
21. Способ терапии заболевания или состояния, вызванного недостаточной экспрессией полноразмерного белка SMN2 у индивидуума, который включает введение индивидууму терапевтической дозы двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19.
22. Способ по п. 20 или 21, где заболевание или состояние является наследственным нервно-мышечным заболеванием, предпочтительно спинальной мышечной атрофией.
23. Способ по п. 20 или 21, где индивидуум является млекопитающим, предпочтительно человеком.
24. Применение двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19 для приготовления препарата для лечения заболевания или состояния, вызванного недостаточной экспрессией белка SMN.
25. Применение двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19 для приготовления препарата для лечения заболевания или состояния, вызванного недостаточной экспрессией полноразмерного белка SMN2 у индивидуума.
26. Применение по п. 24 или 25, где заболевание или состояние является наследственным нервно-мышечным заболеванием, предпочтительно спинальной мышечной атрофией.
27. Применение по п. 24 или 26, где индивидуум является млекопитающим, предпочтительно человеком.
28. Применение двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19 для приготовления препарата для активации/стимуляции экспрессии гена SMN2 в клетке.
29. Применение по п. 28, где двуцепочечная молекула нуклеиновой кислоты по любому из пп. 1-12, нуклеиновая кислота по п. 13 или 14 или композиция по любому из пп. 16-19 вводится непосредственно в клетку.
30. Применение по п. 28, где двуцепочечная молекула нуклеиновой кислоты производится в клетке после введения в клетку нуклеотидной последовательности, кодирующей двуцепочечную молекулу нуклеиновой кислот.
31. Применение по любому из пп. 28-30, где клетка представляет собой клетку млекопитающего, предпочтительно клетку человека.
32. Применение по п. 31, где клетка находится в организме человека.
33. Применение по п. 32, где организм человека является пациентом, у которого имеется симптом, вызванный недостаточной экспрессией белка SMN, мутацией или делецией гена SMN1 или недостаточной экспрессией полноразмерного белка SMN1 и/или недостаточной экспрессией полноразмерного белка SMN2, причем двуцепочечная молекула нуклеиновой кислоты, нуклеиновая кислота или композиция вводится в количестве, достаточном для лечения симптома.
34. Применение по п. 33, где этот симптом, вызванный недостаточной экспрессией полноразмерного белка SMN, является наследственным нервно-мышечным заболеванием, предпочтительно спинальной мышечной атрофией.
35. Выделенный целевой сайт двуцепочечной молекулы нуклеиновой кислоты, активирующей SMN2, по п. 1, для активации/стимуляции экспрессии гена SMN2 в клетке, где выделенный целевой сайт представляет собой непрерывную последовательность длиной от 16 до 35 нуклеотидов в промоторной области гена SMN2 от -1639 до -1481 последовательности SEQ ID №: 476, области от -1090 до -1008 последовательности SEQ ID №: 477, области от -994 до -180 последовательности SEQ ID №: 478 или области от -144 до -37 последовательности SEQ ID №: 479 выше сайта начала транскрипции гена SMN2.
36. Целевой сайт двуцепочечной молекулы нуклеиновой кислоты по п. 35, где целевой сайт находится в последовательности нуклеотидов, выбранной из SEQ ID №: 315-471.
37. Способ активации/стимуляции экспрессии гена SMN2 в клетке, включающий введение в клетку двуцепочечной молекулы нуклеиновой кислоты по любому из пп. 1-12, нуклеиновой кислоты по п. 13 или 14 или композиции по любому из пп. 16-19.
38. Способ по п. 37, где двуцепочечная молекула нуклеиновой кислоты по любому из пп. 1-12, нуклеиновая кислота по п. 13 или 14 или композиция по любому из пп. 16-19 вводится непосредственно в клетку.
39. Способ по п. 37, где двуцепочечная молекула нуклеиновой кислоты производится в клетке после введения в клетку нуклеотидной последовательности, кодирующей двуцепочечную молекулу нуклеиновой кислоты.
40. Способ по любому из пп. 37-39, где клетка представляет собой клетку млекопитающего, предпочтительно клетку человека.
41. Способ по п. 40, где клетка находится в организме человека.
42. Способ по п. 41, где организм человека является пациентом, у которого имеется симптом, вызванный недостаточной экспрессией полноразмерного белка SMN, мутацией или делецией гена SMN1 или недостаточной экспрессией полноразмерного белка SMN1 и/или недостаточной экспрессией полноразмерного белка SMN2, причем двуцепочечная молекула нуклеиновой кислоты, нуклеиновая кислота или композиция вводится в количестве, достаточном для лечения симптома.
43. Способ по п. 42, где симптом, вызванный недостаточной экспрессией полноразмерного белка SMN, является наследственным нервно-мышечным заболеванием, предпочтительно спинальной мышечной атрофией.
| WO 2016170348 A2, 27.10.2016 | |||
| WO 2013173638 A1, 21.11.2013 | |||
| МАРЕТИНА М.А | |||
| Возможные генетические модификаторы спинальной мышечной атрофии, Медицинская генетика, 2017, т | |||
| Устройство для электрической сигнализации | 1918 |
|
SU16A1 |
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
Авторы
Даты
2025-04-28—Публикация
2019-12-27—Подача