Изобретение предназначено для идентификации делеции длинного плеча 6 хромосомы (6q) как молекулярно-генетического фактора неблагоприятного прогноза В-клеточных неходжкинских лимфом (В-НХЛ), в частности фолликулярной лимфомы (ФЛ), и может быть использовано в клинической медицине на этапе первичной диагностики с целью выделения групп пациентов с риском неблагоприятного течения болезни, а также в рамках фундаментальных исследований и поиска потенциальных онкогенов.
Выделение в дебюте онкологического заболевания факторов, позволяющих идентифицировать группу пациентов с неблагоприятным прогнозом, является актуальной задачей с позиций дифференциальной диагностики и возможной персонификации терапии. Определение молекулярно-генетических маркеров является оптимальным вариантом достижения подобных задач, поскольку прямо отражает биологические особенности опухоли.
Известно, что делеции 6q прогностически неблагоприятны при ряде онкологических заболеваний. В первую очередь это касается опухолей системы крови, в частности неходжкинских В-клеточных лимфом и особенно ФЛ, однако также применимо к солидным новообразованиям. Например, ранее было показано, что общая выживаемость больных фолликулярной лимфомой с делецией 6q значительно ниже, чем пациентов без данной генетической аномалии [1]. Подобные же результаты были продемонстрированы в группе больных хроническим лимфолейкозом [2], кроме того, в случае хронического лимфолейкоза делеция 6q оказалась ассоциирована с более высокими значениями лейкоцитоза и распространенной лимфаденопатией [3]. Делеция 6q является фактором неблагоприятного прогноза и риска опухолевой трансформации и рецидива заболевания при остром Т-лимфобластном лейкозе [4-6]. В области солидной онкологии отрицательное влияние делеции 6q на течение заболевания отмечено при аденокарциноме простаты [7] и неоплазиях шейки матки [8].
В области 6q расположены гены, мутации которых обладают онкогенным потенциалом. Однако на настоящий момент точно идентифицированы только некоторые их них [9]. Варианты делеций 6q, вовлекающие различные цитогенетические регионы, обладают нозологической специфичностью [10-12] и соответственно отражают уникальность патогенетических механизмов при различных типах опухолей.
Таким образом, идентификация делеций 6q и определение локализации потерь генетического материала представляется актуальной задачей как в практическом отношении диагностики прогностически неблагоприятных вариантов ФЛ, так и с позиций фундаментальных исследований и поиска ключевых онкогенов.
Данная задача может быть решена при использовании следующих вариантов лабораторного анализа:
1) цитогенетическое исследование;
2) флуоресцентная гибридизация in situ (FISH);
3) сравнительная геномная гибридизация (CGH);
4) полимеразная цепная реакция (ПЦР).
Метод 1 отличается низкой чувствительностью и недостаточной воспроизводимостью результатов, абсолютным требованием к жизнеспособности клеток материла биоптата опухолевой ткани и способности давать достаточное количество митозов в краткосрочных культурах, что исключает возможность анализа замороженных и заключенных в парафин образцов, а также накладывает ограничения на спектр анализируемых патологий, поскольку многие типы опухолей характеризуются низкой пролиферативной способностью.
Метод 2 не применим при исследовании малоклеточных образцов и образцов с низкой сохранностью генетического материала, в частности, парафиновых блоков, а также отличается технической сложностью выполнения и длительностью проведения исследования.
Метод 3 предоставляет высокоточные результаты, максимально полно характеризующие 6q, однако отличается крайне высокой стоимостью анализа одного образца, отсутствием в большинстве практических лабораторий необходимого оборудования и реактивов.
Метод 4, заключающийся в амплификации методом ПЦР искомых последовательностей ДНК, расположенных на 6q в известной локализации, характеризуется высокой чувствительностью. Он позволяет исследовать малоклеточные образцы и архивный материал (замороженные образцы опухолевых тканей и парафиновые блоки) и отличается относительной простотой выполнения и возможностью воспроизведения методики в большинстве молекулярно-генетических лабораторий.
Таким образом, данный вариант решения поставленной задачи максимально соответствует требованиям высокой чувствительности анализа, простоты технической реализации и воспроизводимости лабораторного исследования.
Возможность поиска делеций при картировании хромосом методом ПЦР реализуется при анализе полиморфных локусов микросателлитных повторов, отличающихся высоким процентом гетерозиготности, на основании различий микросателлитного профиля в материале опухолевой ткани и нормальных клетках в качестве контроля. Сравнение проводят путем сопоставления длин амплифицированных фрагментов ДНК, соответствующих разным аллелям в парных образцах при разделении и визуализации ПЦР-продукта методами горизонтального электрофореза или капиллярного электрофореза. Последний вариант является предпочтительным, поскольку обладает большей разрешающей способностью по сравнению с электрофорезом в полиакриламидном геле.
Перечни динуклеотидных микросателлитных маркеров, которые могут быть использованы для картирования хромосом, были разработаны и представлены ранее [13, 14] и послужили основой всех последующих лабораторных систем.
Наиболее близким аналогом предлагаемого изобретения можно считать системы мультиплексных реакций ABI PRISM® Linkage Mapping Set, v2.5-HD5 и v2.5-MD10 производства компании Applied Biosystems (Thermo Fisher Scientific, USA). Указанные системы подразумевают картирование всех хромосом человека, в частности региона 6q, по микросателлитным локусам, расположенным с интервалом 5 и 10 сМ, соответственно. В качестве их недостатка можно отметить тот факт, что сочетания определяемых маркеров в мультиплексных реакциях не являются сбалансированными по хромосомным регионам, в частности последовательности 6р и 6q объединены в одних и тех же пробах. Таким образом, подробный анализ 6q может быть достигнут только при использовании полной панели коммерческого набора, что с учетом стоимости избыточных реактивов и времени на проведение дополнительных лабораторных исследований представляется нерациональным для решения поставленной задачи. Кроме того, в настоящее время данный коммерческий продукт не производится и недоступен пользователям, что является основным недостатком данного аналога.
Также в работах Merup et al. [11], Inoue et al. [15] и Starostik et al. [16] представлены другие варианты аналогов систем картирования 6q. Недостатками первого исследования являются проведение ПЦР в моноплексном режиме и анализ ПЦР-продукта методом горизонтального электрофореза, что обусловливает высокий расход биологического материла в множественных реакциях и низкое разрешение результатов, соответственно. Недостаток двух других исследований заключается в недостаточном для решения поставленной задачи покрытии 6q предлагаемыми авторами панелями микросателлитных повторов. Так, в работе Inoue et al. [15] регион 6q21, на долю которого приходится большая часть делеций при онкогематологических заболеваниях [1, 9-11], представлен только 3 микросателлитными локусами. В работе Starostik et al. [16] совершенно не отражены микросателлитные повторы центромерной области 6q.
Заявляемая группа изобретений направлена на решение задачи создания простой, доступной для реализации в большинстве молекулярно-генетических лабораторий, и информативной диагностической тест-системы и способа выявления делеций 6q при ФЛ, как маркера неблагоприятного прогноза заболевания.
Поставленная задача решена разработкой тест-системы с набором праймеров и способа выявления пациентов с риском неблагоприятного течения фолликулярной лимфомы по наличию значимых делеций 6q в образце опухолевой ткани при сравнении с нормальными клетками в качестве контрольного образца путем проведения анализа изменений 20 микросателлитных локусов ДНК методом мультиплексной ПЦР с последующим фрагментным анализом ПЦР-продуктов. Выбранные микросателлиты равномерно распределены по длине 6q от центромерной до теломерной областей с результирующим интервалом 5,7 сМ в среднем (1,7 -12,5 сМ по данным [13, 14]) и характеризуются высоким уровнем гетерозиготности 82,5% в среднем (75-91% по данным [13, 14]), что также отличает предлагаемое изобретение от известных аналогов.
Результатом исследований, произведенных авторами заявляемого изобретения, является диагностическая тест-система и способ осуществления диагностики делеций 6q с определением областей их локализации при ФЛ.
В опубликованных источниках литературы авторами не обнаружено сведений, касающихся реализованного нового решения.
Использование в клинической практике заявляемого способа позволяет достичь следующих технических результатов:
1) определение прогноза заболевания на этапе первичной диагностики путем выявления прогностически неблагоприятных генетических аномалий в опухолевой ткани, а именно делеций 6q;
2) выделение значимых в патогенезе опухоли хромосомных регионов путем идентификации областей делеции микросателлитных локусов, имеющих известные хромосомные локализации, как основы направленного поиска вовлеченных онкогенов.
Сущность предлагаемого изобретения пояснена на следующих графических материалах:
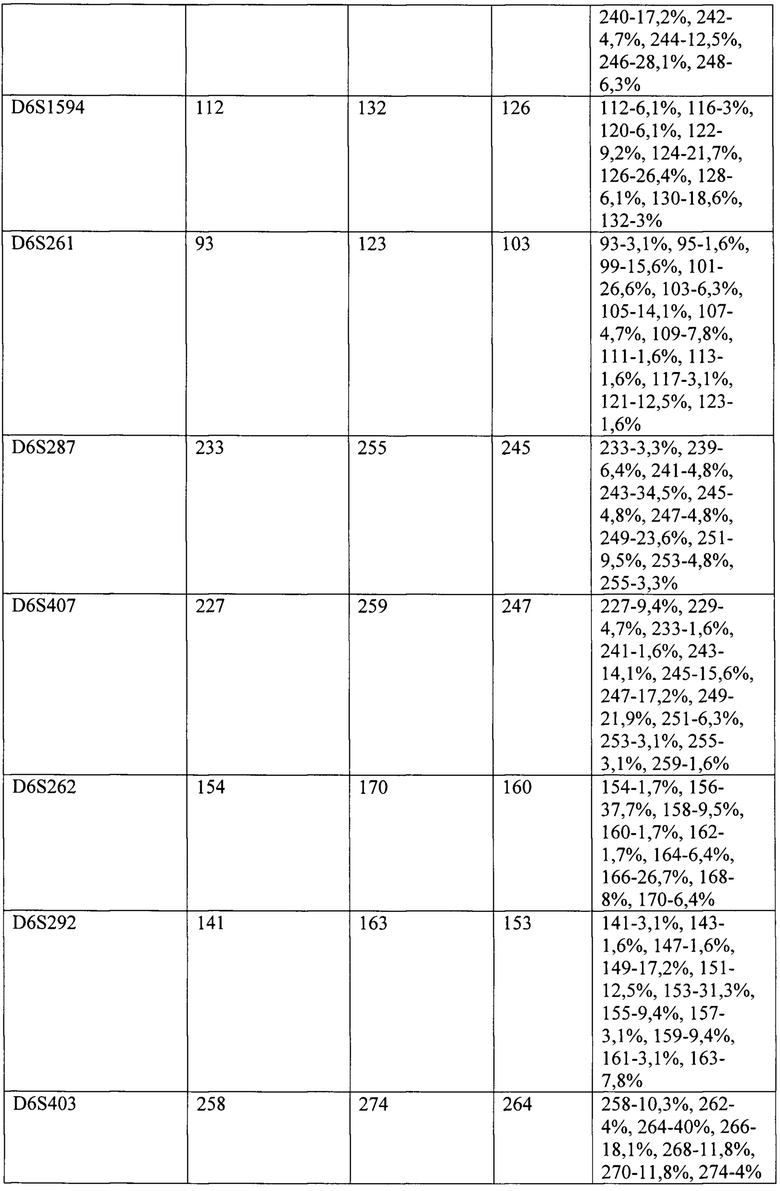
Фиг. 1 - Пул праймеров 1, относится к примеру 1 и представляет результат исследования микросателлитных локусов у здорового донора. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта.
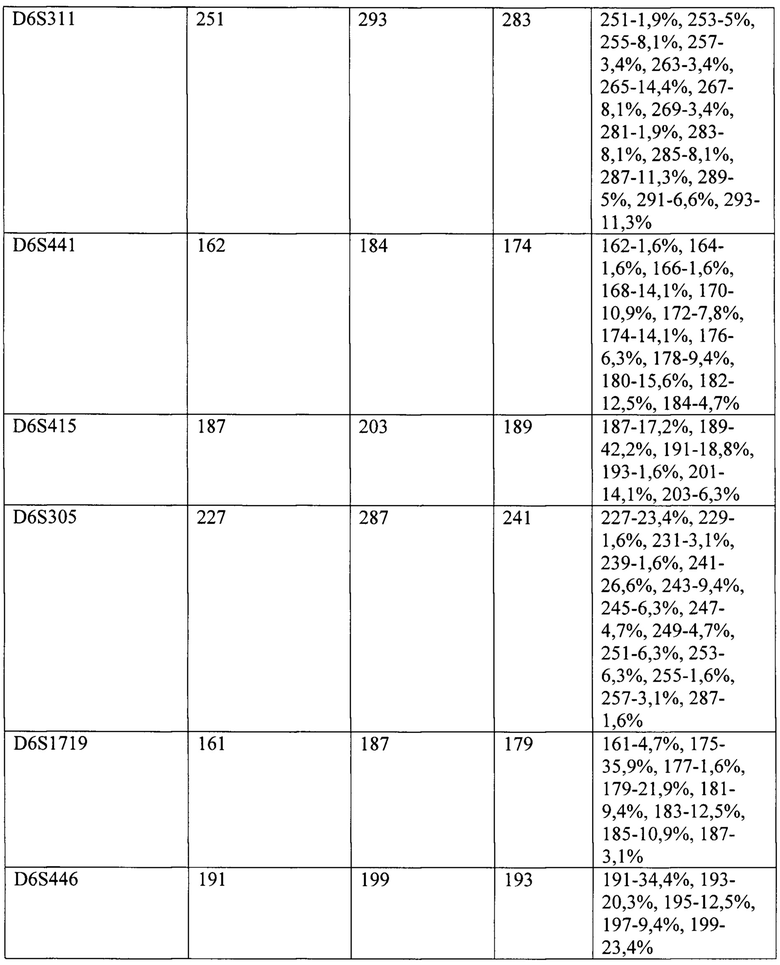
Фиг. 2 - Пул праймеров 2, относится к примеру 1 и представляет результат исследования микросателлитных локусов у здорового донора. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта.
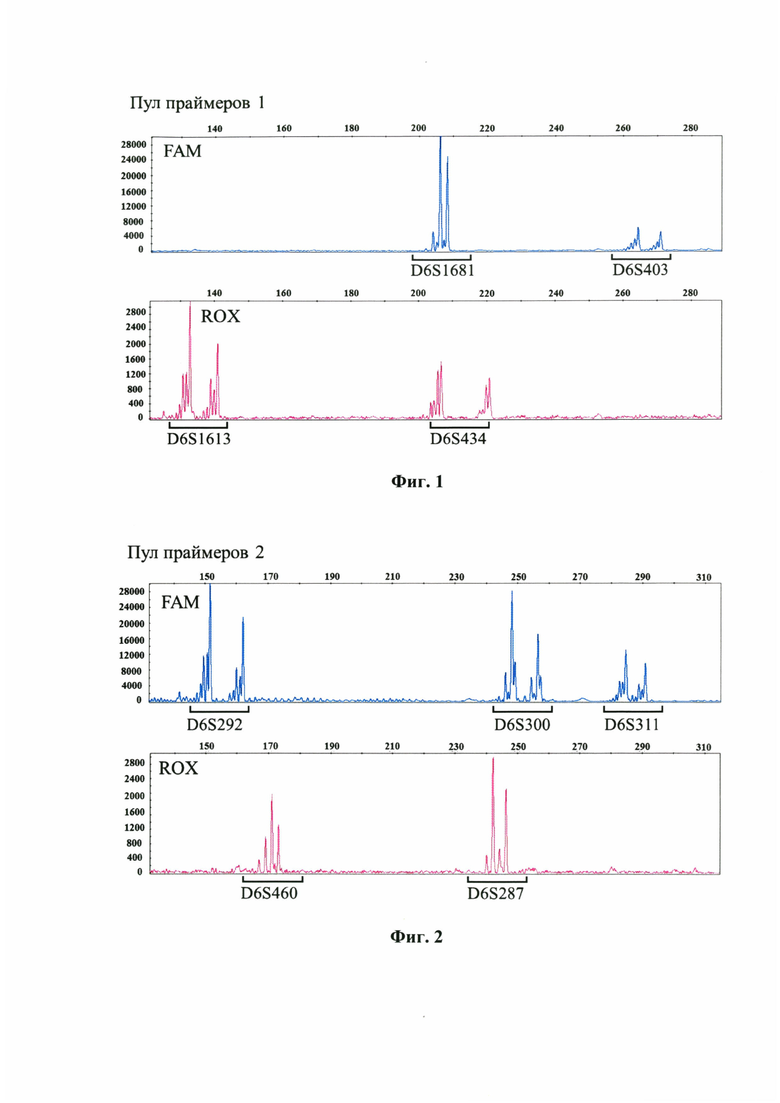
Фиг. 3 - Пул праймеров 3, относится к примеру 1 и представляет результат исследования микросателлитных локусов у здорового донора. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта.
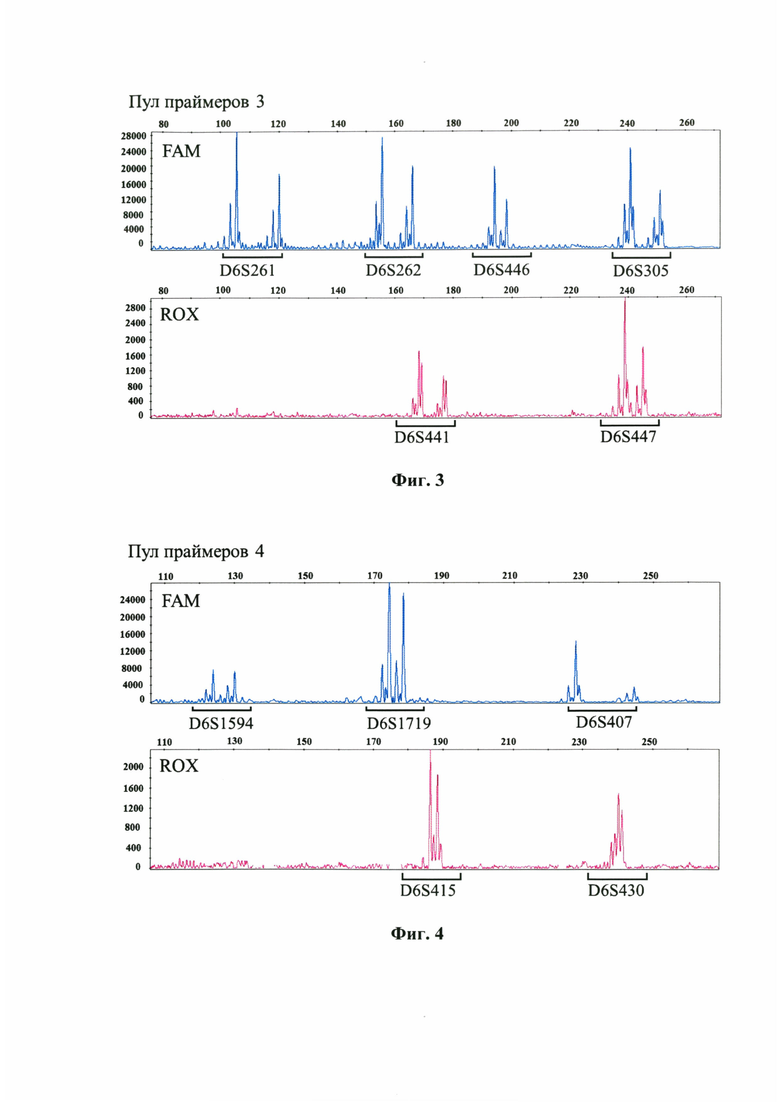
Фиг. 4 - Пул праймеров 4, относится к примеру 1 и представляет результат исследования микросателлитных локусов у здорового донора. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта.
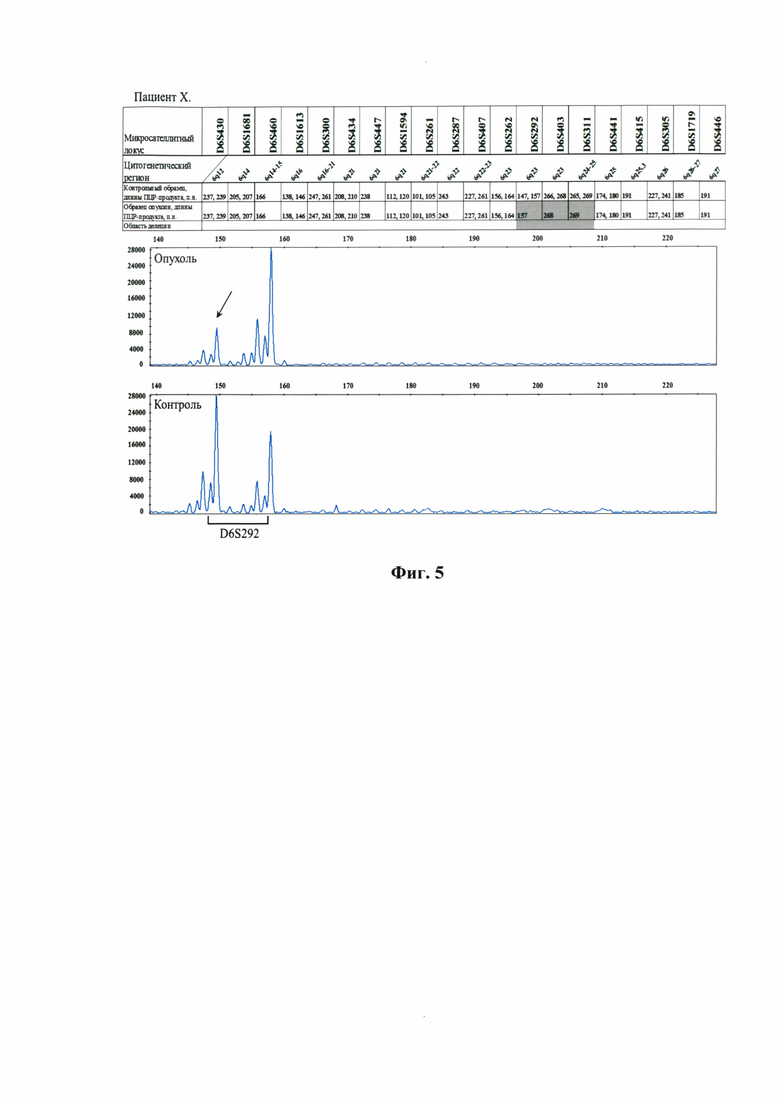
Фиг. 5 - Относится к примеру 2 и представляет результат исследования микросателлитных локусов у пациента X. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта. Стрелочками указаны локусы утраты одного из аллелей в опухолевом образце.
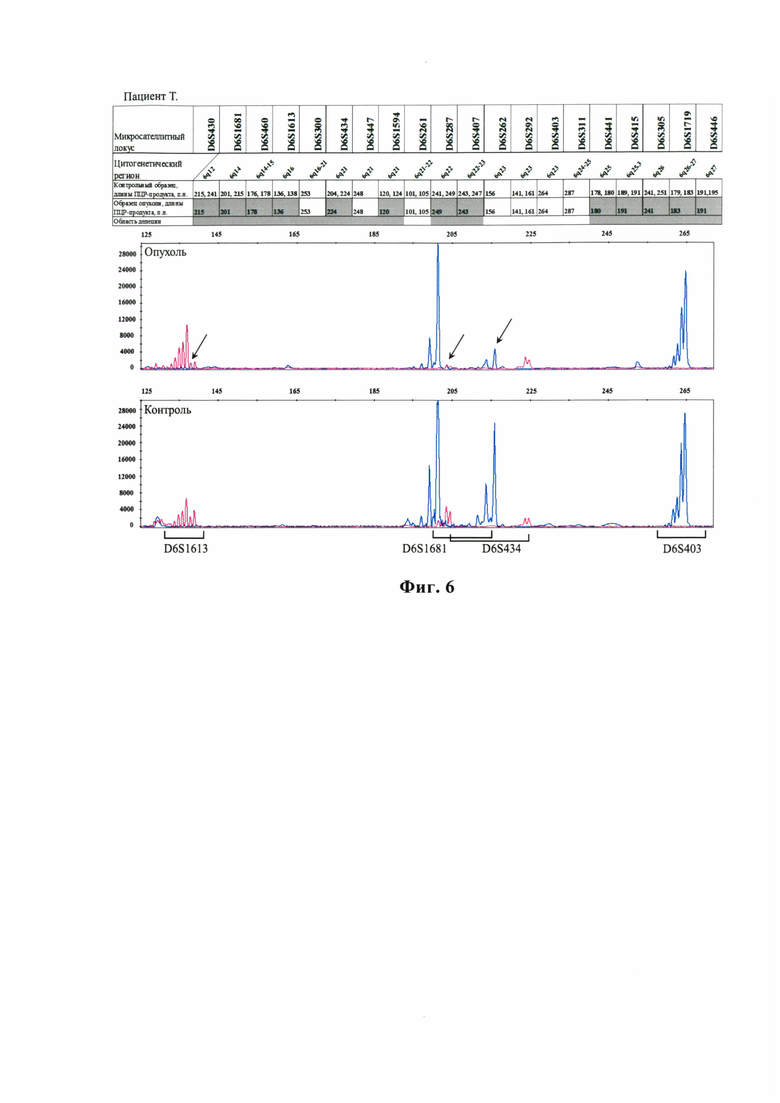
Фиг. 6 - Относится к примеру 3 и представляет результат исследования панели микросателлитных локусов у пациента Т. На графике электрофореграммы по оси X отмечена длина ПЦР-продукта, п.н. По оси Y отмечена интенсивность флуоресценции меченого ПЦР-продукта. Стрелочками указаны локусы утраты одного из аллелей в опухолевом образце.
Сущность группы изобретений пояснена следующими перечнями последовательностей:
SEQ ID NO: 1 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S1681;
SEQ ID NO: 2 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S1681;
SEQ ID NO: 3 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S434;
SEQ ID NO: 4 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S434;
SEQ ID NO: 5 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S403;
SEQ ID NO: 6 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S403;
SEQ ID NO: 7 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S1613;
SEQ ID NO: 8 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S1613;
SEQ ID NO: 9 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S292;
SEQ ID NO: 10 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S292;
SEQ ID NO: 11 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S460;
SEQ ID NO: 12 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S460;
SEQ ID NO: 13 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S287;
SEQ ID NO: 14 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S287;
SEQ ID NO: 15 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S300;
SEQ ID NO: 16 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S300;
SEQ ID NO: 17 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S311;
SEQ ID NO: 18 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S311;
SEQ ID NO: 19 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S447;
SEQ ID NO: 20 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S447;
SEQ ID NO: 21 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S262;
SEQ ID NO: 22 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S262;
SEQ ID NO: 23 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S441;
SEQ ID NO: 24 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S441;
SEQ ID NO: 25 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S305;
SEQ ID NO: 26 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S305;
SEQ ID NO: 27 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S446;
SEQ ID NO: 28 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S446;
SEQ ID NO: 29 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S261;
SEQ ID NO: 30 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S261;
SEQ ID NO: 31 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S1594;
SEQ ID NO: 32 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S1594;
SEQ ID NO: 33 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S1719;
SEQ ID NO: 34 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S1719;
SEQ ID NO: 35 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S415;
SEQ ID NO: 36 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S415;
SEQ ID NO: 37 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S430;
SEQ ID NO: 38 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S430;
SEQ ID NO: 39 последовательность нуклеотидов ДНК Homo sapiens, прямой праймер к локусу D6S407;
SEQ ID NO: 40 последовательность нуклеотидов ДНК Homo sapiens, обратный праймер к локусу D6S407.
Нуклеотидные последовательности праймеров к локусам D6S292 и D6S441 были заимствованы из работы Gyapay et al. [14], поскольку предоставляли оптимальный вариант амплификации искомой микросателлитной последовательности, удовлетворяющий требованиям разрабатываемой тест-системы. К остальным 18 микросателлитным локусам были подобраны оригинальные специфичные пары олигонуклеотидов, ранее не описанные в литературе.
Сущность группы изобретения заключается в следующем.
Разработана тест-система.
Состав тест-системы:
1) пары прямого и обратного праймеров к 20 динуклеотидным микросателлитным маркерам, объединенным в 4 мультиплексных пула соответственно 4 реакционным смесям
1 пул:
- локус D6S1681, расположенный в локусе 6q14 (прямой праймер SEQ ID NO: 1, 2,5 пмоль/мкл; обратный праймер SEQ ID NO: 2, меченный FAM, 2,5 пмоль/л);
- локус D6S434, расположенном в локусе 6q21 (прямой праймер SEQ ID NO: 3, 10 пмоль/мкл; обратный праймер SEQ ID NO: 4, меченный ROX, 10 пмоль/л);
- локус D6S403, расположенном в локусе 6q23 (прямой праймер SEQ ID NO: 5, 5 пмоль/мкл; обратный праймер SEQ ID NO: 6, меченный FAM, 5 пмоль/л);
- локус D6S1613, расположенном в локусе 6q16 (прямой праймер SEQ ID NO: 7, 5 пмоль/мкл; обратный праймер SEQ ID NO: 8, меченный ROX, 5 пмоль/л).
2 пул:
- локус D6S292, расположенном в локусе 6q23 (прямой праймер SEQ ID NO: 9, 5 пмоль/мкл; обратный праймер SEQ ID NO: 10, меченный FAM, 5 пмоль/л);
- локус D6S460, расположенном в локусе 6q14-15 (прямой праймер SEQ ID NO: 11, 10 пмоль/мкл; обратный праймер SEQ ID NO: 12, меченный ROX, 10 пмоль/л);
- локус D6S287, расположенном в локусе 6q22 (прямой праймер SEQ ID NO: 13, 10 пмоль/мкл; обратный праймер SEQ ID NO: 14, меченный ROX, 10 пмоль/л);
- локус D6S300, расположенном в локусе 6q 16-21 (прямой праймер SEQ ID NO: 15, 4 пмоль/мкл; обратный праймер SEQ ID NO: 16, меченный FAM, 4 пмоль/л);
- локус D6S311, расположенном в локусе 6q24-25 (прямой праймер SEQ ID NO: 17, 4 пмоль/мкл; обратный праймер SEQ ID NO: 18, меченный FAM, 4 пмоль/л).
3 пул:
- локус D6S447, расположенном в локусе 6q21 (прямой праймер SEQ ID NO: 19, 10 пмоль/мкл; обратный праймер SEQ ID NO: 20, меченный ROX, 10 пмоль/л);
- локус D6S262, расположенном в локусе 6q23 (прямой праймер SEQ ID NO: 21, 4 пмоль/мкл; обратный праймер SEQ ID NO: 22, меченный FAM, 4 пмоль/л);
- локус D6S441, расположенном в локусе 6q25 (прямой праймер SEQ ID NO: 23, 15 пмоль/мкл; обратный праймер SEQ ID NO: 24, меченный ROX, 15 пмоль/л);
- локус D6S305, расположенном в локусе 6q26 (прямой праймер SEQ ID NO: 25, 5 пмоль/мкл; обратный праймер SEQ ID NO: 26, меченный FAM, 5 пмоль/л);
- локус D6S446, расположенном в локусе 6q27 (прямой праймер SEQ ID NO: 27, 3,75 пмоль/мкл; обратный праймер SEQ ID NO: 28, меченный FAM, 3,75 пмоль/л);
- локус D6S261, расположенном в локусе 6q21-22 (прямой праймер SEQ ID NO: 29, 5 пмоль/мкл; обратный праймер SEQ ID NO: 30, меченный FAM, 5 пмоль/л).
4 пул:
- локус D6S1594, расположенном в локусе 6q21 (прямой праймер SEQ ID NO: 31, 3,75 пмоль/мкл; обратный праймер SEQ ID NO: 32, меченный FAM, 3,75 пмоль/л);
- локус D6S1719, расположенном в локусе 6q26-27 (прямой праймер SEQ ID NO: 33, 5 пмоль/мкл; обратный праймер SEQ ID NO: 34, меченный FAM, 5 пмоль/л);
- локус D6S415, расположенном в локусе 6q25.3 (прямой праймер SEQ ID NO: 35, 10 пмоль/мкл; обратный праймер SEQ ID NO: 36, меченный ROX, 10 пмоль/л);
- локус D6S430, расположенном в локусе 6q12 (прямой праймер SEQ ID NO: 37, 10 пмоль/мкл; обратный праймер SEQ ID NO: 38, меченный ROX, 10 пмоль/л);
- локус D6S407, расположенном в локусе 6q22-23 (прямой праймер SEQ ID NO: 39, 5 пмоль/мкл; обратный праймер SEQ ID NO: 40, меченный FAM, 5 пмоль/л).
Каждому пулу соответствует одна пробирка со смесью праймеров, которая подготавливается путем смешивания стоков синтезированных олигонуклеотидов в приведенных концентрациях, обеспечивающих наиболее благоприятные условия проведения ПЦР. Объем готовой смеси праймеров, необходимый для анализа одного образца - 1 мкл.
2) Реактивы для проведения мультиплексной ПЦР:
- 10х ПЦР-Буфер-Б (ООО "СИНТОЛ", Россия) в пробирках по 500 мкл из расчета 1 пробирка на 150 реакций;
- смесь dNTP (концентрация каждого дезоксинуклеотидтрифосфата 25 мМ) в пробирках по 500 мкл из расчета 1 пробирка на 150 реакций;
- 25 мМ раствор MgCl2 в пробирках по 500 мкл из расчета 1 пробирка на 300 реакций;
- Taq-полимераза 5 ед./мкл в пробирках по 1000 мкл из расчета 1 пробирка на 2000 реакций;
- деионизированная вода во флаконах по 50 мл из расчета 1 флакон на 3000 реакций.
Особенность заявляемого способа заключается в том, что определяют изменения совокупности 20 микросателлитных маркеров тест-системы. При реализации способа осуществляют анализ изменений микросателлитных локусов ДНК в образце опухолевой ткани при сравнении с контрольным образцом методом ПЦР на основании последующего анализа длин ПЦР-продуктов.
Амплификация исследуемых локусов производится в условиях 4 реакционных смесей соответственно 4 пулам праймеров методом мультиплексной ПЦР. Оптимальные сочетания специфичных пар праймеров к перечисленным микросателлитным локусам в 4 пулах, определенные на основании исключения вероятности взаимного отжига и образования димеров и с учетом лучшей визуализации отдельных ПЦР-продуктов, разделенных по длине и каналу флуоресценции, а также условия проведения ПЦР и соотношения концентраций реагентов являются оригинальной особенностью заявляемого изобретения.
Заявляемый способ осуществляется согласно описанной далее методике.
1. Подготовка материалов и компонентов
Исследуемый материал представлен образцом опухолевой ткани, в качестве которого допустимо использование следующих источников:
- фрагмент опухолевого биоптата (объемом не менее 0,5 мм3);
- срезы с парафиновых блоков (в количестве от 2 до 10 при толщине среза 10 мкм в зависимости от объема заключенного материала) с последующей депарафинизацией согласно стандартным методикам;
- клетки периферической крови или аспирата костного мозга при доказанном иными методами наличии лейкемизации или вовлечении в опухолевый процесс костного мозга (10 мл в пробирке с раствором ЭДТА).
Доля опухолевых клеток от общей клеточности образца должна быть не менее 10%, что определено чувствительностью молекулярного исследования.
В качестве отрицательного контроля используют внесение в состав реакционной смеси ПЦР деионизированный воды вместо матрицы ДНК.
В качестве парного нормального образца в параллельном исследовании целесообразно использовать клетки периферической крови того же пациента, а в случае лейкемизации - клетки буккального эпителия, что обеспечивает наличие контроля и достоверность анализа.
Фрагменты опухолевых биоптатов могут быть взяты в работу непосредственно после выполнения биопсии или после замораживания при температуре не выше минус 30°С без использования криоконсерванта.
2. Выделение ДНК из опухолевой ткани биоптатов и депарафинизированных срезов
Подготовку проводят по изложенной ранее методике [17]. Забор клеток аспирата костного мозга или периферической крови осуществляют в 10 мл пробирку с раствором ЭДТА. Выделение ДНК проводят по любой из стандартных методик.
ПЦР проводят в составе реакционной смеси путем смешивания следующих компонентов в 0,2 мл микропробирках:
- 3 мкл 10х буфера (ПЦР-Буфер-Б, ООО "СИНТОЛ", Россия);
- 3 мкл смеси dNTP (концентрация каждого дезоксинуклеотидтрифосфата 25 мМ);
- 1,5 мкл 25 мМ раствора MgCl2;
- не менее 20 нг геномной ДНК (в среднем в объеме водного раствора от 2 мкл до 5 мкл);
- 1 мкл одного из четырех пулов праймеров;
- 2,5 ед. Taq-полимеразы;
- деионизированная вода до общего объема реакционной смеси 25 мкл.
3. Постановка реакции
ПЦР проводят при следующих условиях:
- первичная денатурация 95°С 10 минут;
- 35 циклов денатурации при 95°С 40 секунд, отжига при 57°С 40 секунд, элонгации при 72°С 30 секунд;
- заключительная элонгация при 72°С 5 минут.
4. Учет результатов
Продукты амплификации в разведении 1:60-100 смешивают с формамидом в соотношении 1:3 и денатурируют при 95°С в течение 3 минут, после чего проводят фрагментный анализ на основе капиллярного электрофореза на генетическом анализаторе. Авторами заявляемого изобретения был использован генетический анализатор Нанофор-05 (ООО "СИНТОЛ", Россия).
5. Интерпретация результатов
Наличие делеции региона 6q определяют в случае гетерозиготного наследования на основании отсутствия пиков одного из аллелей или уменьшения интенсивности его флуоресценции по отношению к пику второго аллеля соответствующего микросателлитного локуса в опухолевой ткани при сравнении с контрольным образцом. Наличие делеции региона 6q у пациента с ФЛ относит его к группе высокого риска и позволяет предполагать у него неблагоприятное течение заболевания.
Сущность группы изобретений пояснена примерами конкретной реализации, которые не ограничивают объем прав.
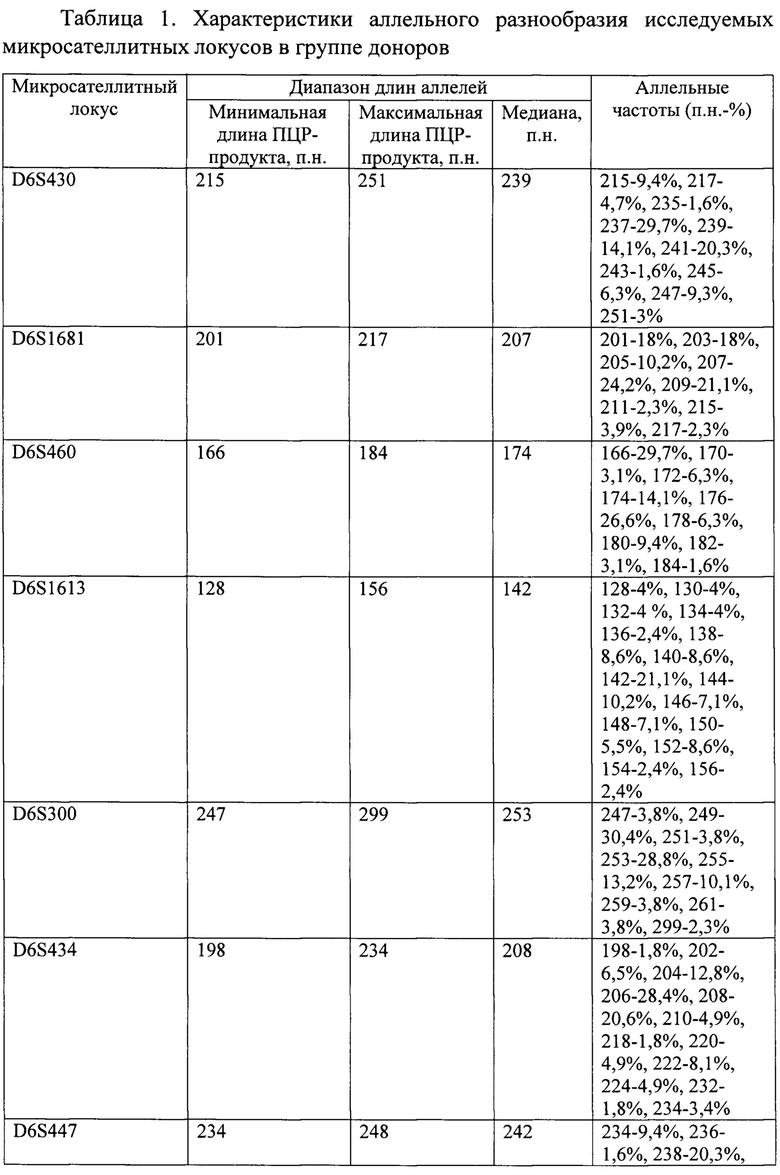
Пример 1. Технические условия заявляемого способа были разработаны и отработаны с использованием образцов ДНК клеток периферической крови 4 здоровых доноров. В дополнительной группе из 28 здоровых доноров была доказана работоспособность и воспроизводимость разработанной диагностической тест-системы.
В таблице 1 представлены данные об аллельном разнообразии исследуемых микросателлитных локусов в донорской популяции.
На Фиг. 1-4 отображены электрофореграммы, полученные при анализе ДНК одного из доноров, соответственно пулам смесей праймеров и каналам флуоресценции.
Пример 2. Пациент X. Диагноз: фолликулярная лимфома 3 морфологического типа с трансформацией в крупноклеточную лимфому, 6 курсов химиотерапии 1 линии, прогрессия заболевания после окончания лечения в течение месяца, химиотерапия 2 линии, рефрактерность, смерть.
По данным стандартного цитогенетического исследования в опухоли выявлен комплексный кариотип с множественными хромосомными нарушениями: дополнительной хромосомой X, делецией короткого плеча хромосомы 1, дериватами хромосом 3, 6 и 11 (47XY,+Xdel(1)(p36),del(3)(?q23),add(6)(q26),del(6)(q26)ort(6;6)(q26;q26),add(11)(q25)[cp1]/47,XY,+X[10], idem der(3)del(3)(?q 13) [cp 12]/47,XY,+12 [1]/46,XY[6]).
На Фиг. 5 представлены результаты исследования панели микросателлитных локусов в контрольном и опухолевом образцах пациента X., а также примеры электрофореграмм отдельных локусов. По трем микросателлитным локусам (D6S292, D6S403, D6S311) в опухолевой ткани отмечена утрата одного из аллелей, что свидетельствует о наличии делеции соответствующего хромосомного региона. В данном случае протяженность делеции распространяется на цитогенетические регионы 6q23-25.
В рассмотренном примере молекулярное и цитогенетическое исследования выявили делецию 6q как фактор неблагоприятного прогноза у пациента с ФЛ с прогрессирующим течением заболевания. Проведенный анализ микросателлитного профиля подтвердил неблагоприятный прогноз течения заболевания и представил более точные данные локализации области потери генетического материала, что имеет фундаментальное значение для изучения патогенеза опухоли.
Пример 3. Пациент Т. Фолликулярная лимфома 3А морфологического типа, 6 курсов химиотерапии 1 линии, ремиссия заболевания, ранний рецидив через год после окончания лечения, химиотерапия 2 линии, вторая ремиссия.
На Фиг. 6 представлены результаты исследования панели микросателлитных локусов в контрольном и опухолевом образцах пациента Т., а также примеры электрофореграмм отдельных локусов. По тринадцати микросателлитным локусам (D6S430, D6S1681, D6S460, D6S1613, D6S434, D6S1594, D6S287, D6S407, D6S441, D6S415, D6S305, D6S1719, D6S446) в опухолевой ткани отмечена утрата одного из аллелей, что свидетельствует о наличии делеции соответствующего хромосомного региона. В приведенном случае протяженность делеции распространяется на три независимых цитогенетического региона, разделенных областями сохранения гетерозиготности: 6q12-21, 6q22-23, 6q25-27, что свидетельствует о сложном характере генетических нарушений у данного пациента. В практическом отношении у данного пациента с делецией 6q подтвердился неблагоприятный прогноз заболевания, а именно - развитие раннего рецидива.
У данного пациента отсутствовала техническая возможность проведения цитогенетического исследования материала опухоли, доступного в качестве парафиновых блоков. Таким образом, именно предложенный метод молекулярного исследования позволил расширить информацию о генетических аберрациях опухолевого клона.
Пример 4. Пациентка П. Фолликулярная морфологического типа, 6 курсов химиотерапии 1 линии, ремиссия.
У данной пациентки при исследовании микросателлитного профиля не были выявлены аберрации микросателлитных повторов, свидетельствующие о делеции 6q, что позволяло предположить благоприятный прогноз заболевания. Действительно, несмотря на наличие экстранодальных очагов поражения, что обычно сочетается с агрессивным течением ФЛ, данная пациентка находится в длительной полной ремиссии.
Таким образом, проведенные авторами исследования позволили определить набор олигонуклеотидных праймеров, разработать диагностическую тест-систему для выявления делеций области 6q при злокачественных новообразованиях и способ выявления пациентов с риском неблагоприятного течения фолликулярной лимфомы по наличию значимых делеций длинного плеча 6 хромосомы, что реализуется путем выполнения мультиплексной ПЦР с набором оригинальных олигонуклеотидных пар праймеров 20 микросателлитных локусов с последующего фрагментным анализом ПЦР-продуктов. Результаты, получаемые при осуществлении изобретения, применимы при определении характера последующего течения опухолевого заболевания на этапе первичной диагностики и могут быть полезны в дифференциации методов терапии, а также могут быть использованы для идентификации значимых в патогенезе опухолей областей хромосомных делеций, что в дальнейшем послужит основой направленного поиска вовлеченных онкогенов.
Источники информации, принятые во внимание при составлении описания к заявке на выдачу патента РФ на изобретение «Тест-система и способ выявления делеций длинного плеча 6 хромосомы»:
1. Tilly Н, Rossi A, Stamatoullas A, Lenormand В, Bigorgne С, Kimlin А, Monconduit М, Bastard С.Prognostic value of chromosomal abnormalities in follicular lymphoma. Blood. 1994 Aug 15; 84(4): 1043-9. PMID: 8049424.
2. Jarosova M, Hruba M, Oltova A, Plevova K, Kruzova L, Kriegova E, Fillerova R, Koritakova E, Doubek M, Lysak D, Prochazka V, Mraz M, Indrak K, Papajik T. Chromosome 6q deletion correlates with poor prognosis and low relative expression of FOXO3 in chronic lymphocytic leukemia patients. Am J Hematol. 2017 Oct; 92(10):E604-E607. doi: 10.1002/ajh.24852. Epub 2017 Jul 29. PMID: 28699185.
3. Stilgenbauer S, Bullinger L, Benner A, Wildenberger K, Bentz M, Dohner K, Ho AD, Lichter P, Dohner H. Incidence and clinical significance of 6q deletions in В cell chronic lymphocytic leukemia. Leukemia. 1999 Sep; 13(9):1331-4. doi: 10.1038/sj.leu.2401499. PMID: 10482982.
4. Remke M, Pfister S, Kox C, Toedt G, Becker N, Benner A, Werft W, Breit S, Liu S, Engel F, Wittmann A, Zimmermann M, Stanulla M, Schrappe M, Ludwig WD, Bartram CR, Radlwimmer B, Muckenthaler MU, Lichter P, Kulozik AE. High-resolution genomic profiling of childhood T-ALL reveals frequent copy-number alterations affecting the TGF-beta and PI3K-AKT pathways and deletions at 6q15-16.1 as a genomic marker for unfavorable early treatment response. Blood. 2009 Jul 30; 114(5):1053-62. doi: 10.1182/blood-2008-10-186536. Epub 2009 Apr 30. PMID: 19406988.
5. Gachet S, El-Chaar T, Avran D, Genesca E, Catez F, Quentin S, Delord M,  Briot D, Meunier G, Hernandez L, Pla M, Smits WK, Buijs-Gladdines JG, Van Loocke W, Menschaert G,
Briot D, Meunier G, Hernandez L, Pla M, Smits WK, Buijs-Gladdines JG, Van Loocke W, Menschaert G,  Taghon T, Van Vlierberghe P, Meijerink JP, Baruchel A, Dombret H, Clappier E, Diaz JJ, Gazin C,
Taghon T, Van Vlierberghe P, Meijerink JP, Baruchel A, Dombret H, Clappier E, Diaz JJ, Gazin C,  Sigaux F, Soulier J. Deletion 6q Drives T-cell Leukemia Progression by Ribosome Modulation. Cancer Discov. 2018 Dec; 8(12):1614-1631. doi: 10.1158/2159-8290.CD-17-0831. Epub 2018 Sep 28. PMID: 30266814.
Sigaux F, Soulier J. Deletion 6q Drives T-cell Leukemia Progression by Ribosome Modulation. Cancer Discov. 2018 Dec; 8(12):1614-1631. doi: 10.1158/2159-8290.CD-17-0831. Epub 2018 Sep 28. PMID: 30266814.
6. Bonn BR, Rohde M, Zimmermann M, Krieger D, Oschlies I, Niggli F, Wrobel G, Attarbaschi A, Escherich G, Klapper W, Reiter A, Burkhardt B. Incidence and prognostic relevance of genetic variations in T-cell lymphoblastic lymphoma in childhood and adolescence. Blood. 2013 Apr 18; 121 (16):3153-60. doi: 10.1182/blood-2012-12-474148. Epub 2013 Feb 8. PMID: 23396305.
7. Kluth M, Jung S, Habib O, et al. Deletion lengthening at chromosomes 6q and 16q targets multiple tumor suppressor genes and is associated with an increasingly poor prognosis in prostate cancer. Oncotarget. 2017; 8(65): 108923-108935. Published 2017 Nov 11. doi:10.18632/oncotarget.22408
8. Mazurenko NN, Bliev AIu, Bidzhieva BA, Peskov DIu, Snigur NV, Savinova EB, Kiselev FL. [Loss of heterozygosity at chromosome 6 as a marker of early genetic alterations in cervical intraepithelial neoplasias and microinvasive carcinomas]. Mol Biol (Mosk). 2006 May-Jun; 40(3):436-47. Russian. PMID: 16813163.
9. Oricchio E, Katanayeva N, Donaldson MC, Sungalee S, Pasion JP, Beguelin W, Battistello E, Sanghvi VR, Jiang M, Jiang Y, Teater M, Parmigiani A, Budanov AV, Chan FC, Shah SP, Kridel R, Melnick AM, Ciriello G, Wendel HG. Genetic and epigenetic inactivation of SESTRIN1 controls mTORC1 and response to EZH2 inhibition in follicular lymphoma. Sci Transl Med. 2017 Jun 28; 9(396):eaak9969. doi: 10.1126/scitranslmed.aak9969. PMID: 28659443; PMCID: PMC5559734.
10. Offit K, Parsa NZ, Gaidano G, Filippa DA, Louie D, Pan D, Jhanwar SC, Dalla-Favera R, Chaganti RS. 6q deletions define distinct clinico-pathologic subsets of non-Hodgkin's lymphoma. Blood. 1993 Oct 1; 82(7):2157-62. PMID: 8104536.
11. Merup M, Moreno TC, Heyman M, 
 Detlofsson R, Rasool O, Liu Y,
Detlofsson R, Rasool O, Liu Y,  Juliusson G, Gahrton G, Einhorn S. 6q deletions in in acute lymphoblastic leukemia and non-Hodgkin's lymphomas. Blood. 1998 May 1; 91(9):3397-400. PMID: 9558398.
Juliusson G, Gahrton G, Einhorn S. 6q deletions in in acute lymphoblastic leukemia and non-Hodgkin's lymphomas. Blood. 1998 May 1; 91(9):3397-400. PMID: 9558398.
12. Jackson A, Carrara P, Duke V, Sinclair P, Papaioannou M, Harrison CJ, Foroni L. Deletion of 6q16-q21 in human lymphoid malignancies: a mapping and deletion analysis. Cancer Res. 2000 Jun 1; 60(11):2775-9. PMID: 10850412.
13. Broman KW, Murray JC, Sheffield VC, White RL, Weber JL. Comprehensive human genetic maps: individual and sex-specific variation in recombination. Am J Hum Genet. 1998 Sep; 63(3):861-9. doi: 10.1086/302011. PMID: 9718341; PMCID: PMC1377399.
14. Gyapay G, Morissette J, Vignal A, Dib C, Fizames C, Millasseau P, Marc S, Bernardi G, Lathrop M, Weissenbach J. The 1993-94  human genetic linkage map. Nat Genet. 1994 Jun; 7(2 Spec No):246-339. doi: 10.1038/ng0694supp-246. PMID: 7545953.
human genetic linkage map. Nat Genet. 1994 Jun; 7(2 Spec No):246-339. doi: 10.1038/ng0694supp-246. PMID: 7545953.
15. Inoue M, Marx A, Zettl A, Strobel P, Muller-Hermelink HK, Starostik P. Chromosome 6 suffers frequent and multiple aberrations in thymoma. Am J Pathol. 2002 Oct; 161(4): 1507-13. doi: 10.1016/S0002-9440(10)64426-4. PMID: 12368223; PMCID: PMC 1867301.
16. Starostik P, Greiner A, Schultz A, Zettl A, Peters K, Rosenwald A, Kolve M,  Genetic aberrations common in gastric high-grade large B-cell lymphoma. Blood. 2000 Feb 15; 95(4):1180-7. PMID: 10666188.
Genetic aberrations common in gastric high-grade large B-cell lymphoma. Blood. 2000 Feb 15; 95(4):1180-7. PMID: 10666188.
17. Sidorova JV, Biderman BV, Nikulina EE, Sudarikov AB. A simple and efficient method for DNA extraction from skin and paraffin-embedded tissues applicable to T-cell clonality assays. Exp Dermatol. 2012; 21(1):57-60. doi:10.1111/j.1600-0625.2011.01375.x
--->
Перечень последовательностей
<110> Федеральное государственное бюджетное учреждение «Национальный
медицинский исследовательский центр гематологии» Министерства
здравоохранения Российской Федерации (ФГБУ "НМИЦ гематологии" Минздрава
России)
Federal Governmental Budgetary Institution «National Research Center for
Hematology»
<120> ТЕСТ-СИСТЕМА И СПОСОБ ВЫЯВЛЕНИЯ ДЕЛЕЦИЙ ДЛИННОГО ПЛЕЧА 6 ХРОМОСОМЫ
<160> 40
<210> 1
<211> 22
<212> DNA
<213> Homo sapiens
<400> 1
ACACAGGCAT TCAAATCAGA GC
<210> 2
<211> 24
<212> DNA
<213> Homo sapiens
<400> 2
CACTCACCAC CTAAATGTTT CAGT
<210> 3
<211> 20
<212> DNA
<213> Homo sapiens
<400> 3
CAACACACAG GTAGTCCCCC
<210> 4
<211> 19
<212> DNA
<213> Homo sapiens
<400> 4
AGCTCAGGGC TTATGCCAG
<210> 5
<211> 23
<212> DNA
<213> Homo sapiens
<400> 5
TCTGAAGAAA GAGGCGTTAA CCA
<210> 6
<211> 17
<212> DNA
<213> Homo sapiens
<400> 6
CCGGCCCAAA GTCTTGT
<210> 7
<211> 23
<212> DNA
<213> Homo sapiens
<400> 7
AGGAAGACTC CACCTCATTT AAA
<210> 8
<211> 22
<212> DNA
<213> Homo sapiens
<400> 8
TCAAGATTAA GTTCCTGGCC AT
<210> 9
<211> 22
<212> DNA
<213> Homo sapiens
<400> 9
AATTCACAAG ACACAATCTC AG
<210> 10
<211> 25
<212> DNA
<213> Homo sapiens
<400> 10
AGAACTAAAG TTGCCTGTTC TTGTA
<210> 11
<211> 20
<212> DNA
<213> Homo sapiens
<400> 11
CCCAGGCTTA TAAAGGTGGC
<210> 12
<211> 20
<212> DNA
<213> Homo sapiens
<400> 12
TGGTTCTCTC ACCCTTTTGT
<210> 13
<211> 25
<212> DNA
<213> Homo sapiens
<400> 13
GCTTCTGTAA TTTTCTATAG CCTAC
<210> 14
<211> 20
<212> DNA
<213> Homo sapiens
<400> 14
GCCTTTGCCC TGAGTATTGG
<210> 15
<211> 20
<212> DNA
<213> Homo sapiens
<400> 15
TGCCTTCTTA ATGCTGCTCA
<210> 16
<211> 20
<212> DNA
<213> Homo sapiens
<400> 16
ACTTGCTTCT CTTGCCTCAA
<210> 17
<211> 20
<212> DNA
<213> Homo sapiens
<400> 17
GGCAGTGGAC ATGGAACCTT
<210> 18
<211> 20
<212> DNA
<213> Homo sapiens
<400> 18
TGGATTCAGA GCCCAGGAAG
<210> 19
<211> 20
<212> DNA
<213> Homo sapiens
<400> 19
TTCTCTCGCT CTCTCACAGC
<210> 20
<211> 20
<212> DNA
<213> Homo sapiens
<400> 20
ATTCCAGAGA GCGGGGCAAA
<210> 21
<211> 21
<212> DNA
<213> Homo sapiens
<400> 21
AGATAGCCGA ATATGCAACC A
<210> 22
<211> 23
<212> DNA
<213> Homo sapiens
<400> 22
GGACTCTCAG TTAACAAGGA GCA
<210> 23
<211> 25
<212> DNA
<213> Homo sapiens
<400> 23
AACAATATTT GGTGACTGTT AAAGG
<210> 24
<211> 25
<212> DNA
<213> Homo sapiens
<400> 24
TGGACAAATT GATTAGGAAG TAAAG
<210> 25
<211> 20
<212> DNA
<213> Homo sapiens
<400> 25
TCCTCTCCAC CAGCGTTAGA
<210> 26
<211> 20
<212> DNA
<213> Homo sapiens
<400> 26
GTTTAGATCC CAGGCCTGCA
<210> 27
<211> 20
<212> DNA
<213> Homo sapiens
<400> 27
AGGGATGTCT ACACCTACAC
<210> 28
<211> 20
<212> DNA
<213> Homo sapiens
<400> 28
GGGAGGACGT TCTGGTTTCA
<210> 29
<211> 22
<212> DNA
<213> Homo sapiens
<400> 29
TGAAACCCTG TCTCACTGCA TA
<210> 30
<211> 21
<212> DNA
<213> Homo sapiens
<400> 30
TATAGGACCA TGCCATTAGG C
<210> 31
<211> 21
<212> DNA
<213> Homo sapiens
<400> 31
GCCTGAGACT CTTCAATGCC T
<210> 32
<211> 20
<212> DNA
<213> Homo sapiens
<400> 32
AGCTTAAAGT GTAGGACGGT
<210> 33
<211> 21
<212> DNA
<213> Homo sapiens
<400> 33
GGAACTACTC CCATTCCAAC C
<210> 34
<211> 22
<212> DNA
<213> Homo sapiens
<400> 34
GAGATACAAG CAGGAGGTAG CA
<210> 35
<211> 20
<212> DNA
<213> Homo sapiens
<400> 35
TCGGCCACCA GAAGTAAAGC
<210> 36
<211> 20
<212> DNA
<213> Homo sapiens
<400> 36
GGAGGTGCAG CATCTTTCCT
<210> 37
<211> 22
<212> DNA
<213> Homo sapiens
<400> 37
CAGCATAGTT CCTTCAAGCA CA
<210> 38
<211> 21
<212> DNA
<213> Homo sapiens
<400> 38
GGCTTCCCCT TCACTTCTTC A
<210> 39
<211> 17
<212> DNA
<213> Homo sapiens
<400> 39
AAAAGTACCT CCCGCCC
<210> 40
<211> 21
<212> DNA
<213> Homo sapiens
<400> 40
GGGGACAGTT ACCTTTGGAG T
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Тест-система и способ выявления делеций гена SESN1 | 2021 |
|
RU2772504C1 |
| Набор STR-маркеров Y-хромосомы для определения этно-территориального происхождения индивида по образцу его ДНК | 2021 |
|
RU2804433C2 |
| Способ генотипирования однонуклеотидного варианта rs2072668 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835216C1 |
| Тест-система для количественной диагностики мРНК интерферонов I, II и III типов человека на основе ПЦР | 2020 |
|
RU2751791C1 |
| Способ генотипирования однонуклеотидного варианта rs3219484 (C>T) гена MUTYH человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835215C1 |
| Способ генотипирования однонуклеотидного варианта rs2304277 (G>A) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2836047C1 |
| ПАНЕЛЬ БИОМАРКЕРОВ И СПОСОБЫ ВЫЯВЛЕНИЯ МИКРОСАТЕЛЛИТНОЙ НЕСТАБИЛЬНОСТИ ПРИ РАЗНЫХ ВИДАХ РАКА | 2019 |
|
RU2795410C2 |
| Способ генотипирования однонуклеотидного варианта rs3219493 (C>G) гена MUTYH человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835214C1 |
| Способ генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2835399C1 |
| Тест-система "ESSC-tipe-1" для молекулярно-генетического типирования плоскоклеточного рака пищевода | 2020 |
|
RU2742468C1 |
Изобретение относится к набору олигонуклеотидных праймеров для специфической амплификации двадцати микросателлитных локусов длинного плеча 6 хромосомы человека (6q), который может быть использован в тест-системе для определения делеций длинного плеча 6 хромосомы (6q) методом мультиплексной полимеразной цепной реакции (ПЦР), а также в способе выявления пациентов с риском неблагоприятного течения фолликулярной лимфомы по наличию значимых делеций длинного плеча 6 хромосомы (6q) с помощью вышеупомянутой тест-системы. Наличие делеций региона 6q в случае гетерозиготного наследования на основании отсутствия пиков одного из аллелей или уменьшения интенсивности его флуоресценции по отношению к пику второго аллеля соответствующего микросателлитного локуса в опухолевой ткани при сравнении с контрольным образцом позволяет выделять группу пациентов с риском неблагоприятного течения фолликулярной лимфомы. 3 н.п. ф-лы, 6 ил., 1 табл., 4 пр.
1. Набор олигонуклеотидных пар праймеров для специфической амплификации двадцати микросателлитных локусов длинного плеча 6 хромосомы человека (6q): D6S430, расположенный в локусе 6q12, D6S1681, расположенный в локусе 6q14, D6S460, расположенный в локусе 6q14-15, D6S1613, расположенный в локусе 6q16, D6S300, расположенный в локусе 6q16-21, D6S434, расположенный в локусе 6q21, D6S447, расположенный в локусе 6q21, D6S1594, расположенный в локусе 6q21, D6S261, расположенный в локусе 6q21-22, D6S287, расположенный в локусе 6q22, D6S407, расположенный в локусе 6q22-23, D6S262, расположенный в локусе 6q23, D6S292, расположенный в локусе 6q23, D6S403, расположенный в локусе 6q23, D6S311, расположенный в локусе 6q24-25, D6S441, расположенный в локусе 6q25, D6S415, расположенный в локусе 6q25.3, D6S305, расположенный в локусе 6q26, D6S1719, расположенный в локусе 6q26-27, D6S446, расположенный в локусе 6q27, имеющих следующие нуклеотидные последовательности SEQ ID NO: 1 - SEQ ID NO: 40:
D6S430-F: 5'-CAGCATAGTTCCTTCAAGCACA-3',
D6S430-R: 5'-ROX-GGCTTCCCCTTCACTTCTTCA-3',
D6S1681 -F: 5'-ACACAGGCATTCAAATCAGAGC-3',
D6S1681 -R: 5'-FAM-CACTCACCACCTAAATGTTTCAGT-3',
D6S434-F: 5'-CAACACACAGGTAGTCCCCC-3',
D6S434-R: 5'-ROX-AGCTCAGGGCTTATGCCAG-3',
D6S261 -F: 5'-TGAAACCCTGTCTCACTGC ATA-3',
D6S261 -R: 5'-FAM-TATAGGACCATGCCATTAGGC-3',
D6S403-F: 5'-TCTGAAGAAAGAGGCGTTAACCA-3,
D6S403-R: 5'-FAM-CCGGCCCAAAGTCTTGT-3',
D6S460-F: 5'-CCCAGGCTTATAAAGGTGGC-3',
D6S460-R: 5'-ROX-TGGTTCTCTCACCCTTTTGT-3',
D6S300-F: 5'-TGCCTTCTTAATGCTGCTCA-3',
D6S300-R: 5'-F AM- ACTTGCTTCTCTTGCCTC AA-3',
D6S1594-F: 5'-GCCTGAGACTCTTCAATGCCT-3',
D6S1594-R: 5'-FAM-AGCTTAAAGTGTAGGACGGT-3',
D6S287-F: 5'-GCTTCTGTAATTTTCTATAGCCTAC-3',
D6S287-R: 5'-ROX-GCCTTTGCCCTGAGTATTGG-3',
D6S292-F: 5'-AATTCACAAGACACAATCTCAG-3',
D6S292-R: 5'-FAM-AGAACTAAAGTTGCCTGTTCTTGTA-3,
D6S311-F: 5'-GGCAGTGGACATGGAACCTT-3',
D6S311-R: 5'-FAM-TGGATTCAGAGCCCAGGAAG-3',
D6S447-F: 5'-TTCTCTCGCTCTCTCACAGC-3',
D6S447-R: 5'-ROX-ATTCCAGAGAGCGGGGCAAA-3',
D6S262-F: 5'-AGATAGCCGAATATGCAACCA-3',
D6S262-R: 5'-FAM-GGACTCTCAGTTAACAAGGAGCA-3',
D6S441-F: 5'-AAC AATATTTGGTGACTGTTAAAGG-3',
D6S441 -R: 5'-ROX-TGGACAAATTGATTAGGAAGTAAAG-3',
D6S305-F: 5'-TCCTCTCCACCAGCGTTAGA-3',
D6S305-R: 5'-FAM-GTTTAGATCCCAGGCCTGCA-3',
D6S446-F: 5'-AGGGATGTCTACACCTACAC-3',
D6S446-R: 5'-FAM-GGGAGGACGTTCTGGTTTCA-3',
D6S415-F: 5'-TCGGCCACCAGAAGTAAAGC-3',
D6S415-R: 5'-ROX-GGAGGTGCAGCATCTTTCCT-3',
D6S1719-F: 5'-GGAACTACTCCCATTCCAACC-3',
D6S1719-R: 5'-FAM-GAGATACAAGCAGGAGGTAGCA-3,
D6S407-F: 5'-AAAAGTACCTCCCGCCC-3',
D6S407-R: 5'-FAM-GGGGACAGTTACCTTTGGAGT-3',
D6S1613-F: 5'-AGGAAGACTCCACCTCATTTAAA-3',
D6S1613-R: 5'-ROX-TCAAGATTAAGTTCCTGGCCAT-3'.
2. Тест-система для определения делеций длинного плеча 6 хромосомы (6q) методом мультиплексной полимеразной цепной реакции (ПЦР), состоящая из набора олигонуклеотидных пар праймеров по п. 1, разделенного на 4 пула-смеси праймеров, исключающих вероятность взаимного отжига и образования димеров, и с учетом лучшей визуализации отдельных ПЦР-продуктов:
1 пул - D6S1681 - 2,5 пмоль/мкл, D6S434 - 10 пмоль/мкл, D6S403 - 5 пмоль/мкл, D6S1613 - 5 пмоль/мкл;
2 пул - D6S292 - 5 пмоль/мкл, D6S460 - 10 пмоль/мкл, D6S287 - 10 пмоль/мкл, D6S300 - 4 пмоль/мкл, D6S311 - 4 пмоль/мкл;
3 пул - D6S447 - 10 пмоль/мкл, D6S262 - 4 пмоль/мкл, D6S441 - 15 пмоль/мкл, D6S305 - 5 пмоль/мкл, D6S446 - 3,75 пмоль/мкл, D6S261 - 5 пмоль/мкл;
4 пул - D6S1594 - 3,75 пмоль/мкл, D6S1719 - 5 пмоль/мкл, D6S415 - 10 пмоль/мкл, D6S430 - 10 пмоль/мкл, D6S407 - 5 пмоль/мкл;
реактивов для проведения ПЦР: 10х ПЦР-Буфер-Б, смесь dNTP по 25 мМ, 25 мМ раствор MgCl2, Taq-полимераза, деионизированная вода.
3. Способ выявления пациентов с риском неблагоприятного течения фолликулярной лимфомы по наличию значимых делеций длинного плеча 6 хромосомы (6q) с помощью тест-системы по п. 2, включающий следующие этапы: сбор и подготовка парных образцов опухоли и контрольной ткани, при этом в качестве образца опухолевой ткани допустимо использование фрагментов опухолевого биоптата, срезов с парафиновых блоков с последующей депарафинизацией, клеток периферической крови или аспирата костного мозга, в качестве парного нормального образца используют клетки периферической крови того же пациента, а в случае лейкемизации - клетки буккального эпителия; выделение ДНК, проведение мультиплексной ПЦР с помощью тест-системы по п. 2 для каждого образца при следующих условиях:
первичная денатурация 95°С 10 минут,
35 циклов денатурации при 95°С 40 секунд, отжига при 57°С 40 секунд,
элонгации при 72°С 30 секунд,
заключительная элонгация при 72°С 5 минут;
продукты амплификации в разведении 1:60-100 смешивают с формамидом в соотношении 1:3 и денатурируют при 95°С в течение 3 минут, после чего проводят фрагментный анализ на основе капиллярного электрофореза на генетическом анализаторе; интерпретацию полученных данных, при этом наличие делеции региона 6q в случае гетерозиготного наследования на основании отсутствия пиков одного из аллелей или уменьшения интенсивности его флуоресценции по отношению к пику второго аллеля соответствующего микросателлитного локуса в опухолевой ткани при сравнении с контрольным образцом позволяет выделять группу пациентов с риском неблагоприятного течения фолликулярной лимфомы.
| TAKEUCH S | |||
| et al., Frequent loss of heterozygosity on the long arm of chromosome 6: identification of two distinct regions of deletion in childhood acute lymphoblastic leukemia, Cancer Res, 1998, Vol | |||
| Способ окисления боковых цепей ароматических углеводородов и их производных в кислоты и альдегиды | 1921 |
|
SU58A1 |
| Подъемное крановое устройство для железных дорог | 1925 |
|
SU2618A1 |
| GYAPAY G | |||
| et al., The 1993-94 Généthon human genetic linkage map, Nat Genet, 1994, Vol | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
Авторы
Даты
2022-04-25—Публикация
2021-07-22—Подача