Изобретение относится к микробиологии и медицине, а именно к инфекционным болезням и эпидемиологии, в частности к проблеме диагностики и генотипирования патогенных лептоспир. Диагностику и генотипирование проводят с использованием ДНК, выделенной из клинических образцов.
Лептоспироз является убиквитарным зоонозом, что связано с большим разнообразием резервуарных хозяев. Основными переносчиками считаются дикие мелкие млекопитающие (грызуны, насекомоядные) и домашние животные (КРС, МРС, собаки и т.д.). Инфекционный процесс у переносчиков чаще всего протекает в бессимптомной форме, лептоспиры колонизируют почечные канальцы и выделяются в окружающую среду с мочой, где сохраняются на протяжении нескольких месяцев [Sayanthi Y, Susanna D. Pathogenic Leptospira contamination in the environment: a systematic review. Infect Ecol Epidemiol. 2024; 14(1):2324820. Published 2024 Mar 19. doi:10.1080/20008686.2024.2324820].
Лептоспирозы считаются одним из самых распространенных зооантропонозных заболеваний, влияющих как на здоровье людей, так и животных, по всему миру. Ежегодно регистрируется порядка миллиона заболеваний, и около 60 тысяч смертельных исходов. Наибольшие показатели регулярно регистрируются в развивающихся странах с тропическим климатом [Her R, Crespin L, Etougbétché J, et al. Seroprevalence and renal carriage of pathogenic Leptospira in livestock in Cotonou, Benin. Vet Med Sci. 2024; 10(3):e1430. doi:10.1002/vms3.1430].
В последнее десятилетие в Российской Федерации наблюдается тенденция к снижению заболеваемости, однако, в значительной степени это связано с отсутствием высокоточной доступной диагностики, и с тем, что при легком течение лептоспироза инфицированные люди не обращаются в медицинские учреждения. При этом активность природных очагов лептоспироза во всех федеральных округах страны сохраняется [Транквилевский Д.В., Киселева Е.Ю., Корзун В.М. и др. Эпизоотолого-эпидемиологическая ситуация по лептоспирозам в Российской Федерации в период с 2013 по 2022 г. и прогноз на 2023 г. Проблемы особо опасных инфекций. - 2023. - №3. - С. 43-50. - DOI 10.21055/0370-1069-2023-3-43-50] и может приводить к эпидемическим вспышкам в случае природных катастроф, например, при наводнении, что в настоящее время имеет особое значение.
Диагностические ошибки при лептоспирозе часто связаны с отсутствием специфических признаков и симптомов. Легкое течение лептоспироза часто протекает под другими диагнозами, и может наблюдаться как самокупирующееся лихорадочное заболевание. Тяжелое же течение характеризуется полиорганной недостаточностью и смертностью до 40% [Alexopoulou С, Proklou A, Kokkini S, Raissaki М, Konstantinou I, Kondili E. A Fatal Case of Presumptive Diagnosis of Leptospirosis Involving the Central Nervous System. Healthcare (Basel). 2024; 12(5):568. Published 2024 Feb 29. doi:10.3390/healthcare12050568].
Специфическая лабораторная диагностика лептоспироза включает в себя как прямые, так и косвенные методы обнаружения возбудителя. К косвенным методам обнаружения лептоспир, относят иммунологические методики, направленные на поиск антител (РМА, ИФА). К прямым методам относят микроскопию в темном поле, бактериологические и молекулярные методики, направленные на поиск самого возбудителя [Verma V, Goyal М, Kala D, Gupta S, Kumar D, Kaushal A. Recent advances in the diagnosis of leptospirosis. Front Biosci (Landmark Ed). 2020; 25(9): 1655-1681. Published 2020 Mar 1. doi: 10.2741/4872]. Молекулярные методы диагностики лептоспироза, в частности ПЦР, считаются наиболее чувствительными и специфичными методами для выявления лептоспир на ранних этапах заболевания, еще до развития серологического ответа.
Развитие молекулярных методов привело к созданию таксономической классификации, основанной на генотипических характеристиках. Помимо патогенных и сапрофитных штаммов, были классифицированы виды, с умеренным уровнем патогенности, но при этом имеющие общего предка с патогенными видами [Vincent AT, Schiettekatte О, Goarant С, et al. Revisiting the taxonomy and evolution of pathogenicity of the genus Leptospira through the prism of genomics. PLoS Negl Trop Dis. 2019; 13(5):e0007270. Published 2019 May 23. doi: 10.1371/journal.pntd.0007270].
По генетической классификации, основанной на полногеномном анализе, род Leptospira включает в себя 72 вида, сгруппированных в 4 субклады: патогенные (Р1), промежуточные (Р2) и сапрофитные (S1, S2) [Са Ferreira L, de Fa Ferreira Filho L, V Cosate MR, Sakamoto T. Genetic structure and diversity of the rfb locus of pathogenic species of the genus Leptospira. Life Sci Alliance. 2024; 7(6):e202302478. Published 2024 Mar 21. doi:10.26508/lsa.202302478].
Филогенетический анализ на основе молекулярных методик, позволил выяснить что виды и серовары не имеют корреляционной связи, и один серовар может относится к разным видам. На данный момент серотипирования не достаточно, для выявления эпидемиологически значимых штаммов и генотипов [Bourhy Р, Herrmann Storck С, Theodose R, et al. Serovar diversity of pathogenic Leptospira circulating in the French West Indies. PLoS Negl Trop Dis. 2013; 7(3):e2114. doi: 10.1371/journal.pntd.0002114].
Классификация с использованием молекулярных методик, таких как ПЦР и секвенирование, являются достойной альтернативой серотипированию. Таксономическая классификация необходима для отслеживания эволюционных связей между видами. Род Leptospira характеризуется открытым пангеномом и высокой способностью к горизонтальному переносу генов, соответственно свободноживущие лептоспиры могут преобразовываться в патогенные виды и инфицировать людей и животных. Открытие новых и изучение известных патогенных и промежуточных штаммов, с генетической стороны, позволит улучшить диагностику, профилактику и контроль за лептоспирозной инфекцией [Vincent AT, Schiettekatte О, Goarant С, et al. Revisiting the taxonomy and evolution of pathogenicity of the genus Leptospira through the prism of genomics. PLoS Negl Trop Dis. 2019; 13(5):e0007270. Published 2019 May 23. doi: 10.137 l/journal.pntd.0007270].
Существует целый ряд генетических маркеров, используемых для выявления и генотипирования лептоспир. К ним относят гены 16s рРНК, lipL21, lipl32, lipL41, ligB, secY, gyrB и т.д. Отсутствие стандартного протокола, приводит к тому, что исследователи используют разные генетические мишени и сравнение результатов становится затруднительным [Di Azevedo MIN, Lilenbaum W. An overview on the molecular diagnosis of animal leptospirosis. Lett Appl Microbiol. 2021;72(5):496-508. doi:10.1111/lam.13442].
В течение долгого времени, для изучения видового разнообразия лептоспир, использовали методику секвенирования последовательности гена 16S рРНК. Однако, на данный момент, существует большое количество исследований, сообщающих о его низкой дискриминационной способности различать виды внутри клад, по причине его высокой консервативности [Baimova R, Ostankova Y, Blinova О, et al.. Molecular and genetic characterization of LEPTOSPIRA spp.collection strains from the St. Petersburg Pasteur institute based on 16S rRNA gene sequencing data. Russian Journal of Infection and Immunity. 2023; 13:6. doi: 10.15789/2220-7619-MAG-17028].
Исследование нескольких генов-мишеней позволит избежать дискриминационных ограничений, и поможет повысить актуальность разработки новых диагностических инструментов [Thibeaux R, Girault D, Bierque E, et al. Biodiversity of Environmental Leptospira: Improving Identification and Revisiting the Diagnosis. Front Microbiol. 2018; 9:816. Published 2018 May 1. doi:10.3389/fmicb.2018.00816].
Известен способ диагностики лептоспир с использованием фрагмента гена lipl32 [Latifah I, Abdul Halim A, Rahmat MS, et al. Isolation by culture and PCR identification of Lipl32 gene of pathogenic Leptospira spp.in wild rats of Kuala Lumpur. Malays J Pathol. 2017; 39(2): 161-166]. Липопротеин L32 (Upl32) - основной белок внешней мембраны лептоспир, является фактором вирулентности присущим патогенным лептоспирам. Согласно описанному методу образцы мочи, собранные у крыс, культивируют в среде, обогащенной EMJH в течение 8 недель. Контроль роста культур проверяют с помощью темнопольной микроскопии. Далее, из полученных штаммов выделяют ДНК и проводят ПНР анализ с дальнейшим секвенированием, для подтверждения патогенности лептоспир. Недостатком данного метода является трудоемкость и долгосрочность культивирования штаммов, что исключает возможность использования данной методики в качестве диагностики клинических образцов.
Известны способы генотипирования лептоспир с использованием одного гена. Vinayagamurthy Balamurugan с соавторами, проводили типирование культур патогенных лептоспир, с использованием гена rpoB кодирущего Р-субъединицу бактериальной РНК-полимеразы. [Balamurugan V, Gangadhar NL, Mohandoss N, et al. Characterization of leptospira isolates from animals and humans: phylogenetic analysis identifies the prevalence of intermediate species in India. Springerplus. 2013; 2:362. Published 2013 Jul 30. doi: 10.1186/2193-1801-2-362.]. Pascale Bourhy с соавторами, в качестве мишени для генотипирования лептоспир, использовали ген кодирующий субъединицу белка транслоказы - secY [Bourhy Р, Herrmann Storck С, Theodose R, et al. Serovar diversity of pathogenic Leptospira circulating in the French West Indies. PLoS Negl Trop Dis. 2013; 7(3):e2114. doi:10.1371/journal.pntd.0002114]. Согласно описанным методам, клинические образцы культивируют, и из изолированных штаммов (в исследовании Bourhy Р и др., так же использовали плазму человека) экстрагируют ДНК, проводят амплификацию и дальнейшее секвенирование. Недостатком описанных методик, помимо культивирования образцов, является использование одного гена для филогенетического анализа.
Наиболее близким по сущности к заявляемому изобретению и выбранным за прототип является метод, предложенный в работе Sarah Javati с соавторами [Javati S, Guernier-Cambert V, Jonduo М, Robby S, Kimopa J, Maure T, McBryde ES, Pomat W, Aplin K, Helgen KM, Abdad MY, Horwood PF. Diversity of Leptospira spp.in bats and rodents from Papua New Guinea. Transbound Emerg Dis. 2022 Nov; 69(6):4048-4054. doi: 10.1111/tbed. 14725. Epub 2022 Oct 19. PMID: 36196768; PMCID: РМС10092571]. Согласно описанному методу, образцы почек, изъятых у грызунов и летучих мышей, подвергают первичной обработке, далее экстрагируют ДНК, скрининговое исследование на наличие патогенных видов лептоспир проводят с помощью ПЦР нацеленной на фрагмент гена 16S, полученные положительные образцы амплифицируют с праймерами подобранными по фрагменту гена secY, и далее секвенируют по Сэнгеру. В данной работе не все положительные по 16S образцы удалось секвенировать по secY. Это может быть связано как с низкой специфичностью гена 16S, так и с использованием одной мишени для генотипирования. Выбор гена - мишени для видовой дифференциации является одним из самых важных этапов при проведении анализа, оптимальным можно считать подход, при котором исследуется несколько фрагментов генов.
Задачей, на решение которой направлено заявляемое изобретение, является усовершенствование и расширение арсенала способов, предназначенных для выявления и генотипирования различных видов патогенных лептоспир в целях улучшения диагностики и профилактики заболевания.
Технический результат - повышение специфичности, а соответственно и достоверности диагностики (снижение количества ложноположительных результатов), также и расширение арсенала средств, используемых для таксономической характеристики лептоспир.
Авторами предложен способ, согласно которому из клинического материала экстрагируют тотальную ДНК, далее с помощью амплификации по фрагменту гена lipl32 определяют наличие или отсутствие ДНК патогенных лептоспир. Образцы, содержащие ДНК патогенных лептоспир, амплифицируют последовательно по двум фрагментами генов rpoB и secY. Регистрируют полученные результаты посредством электрофореза в агарозном геле с последующей детекцией на трансиллюминаторе. Полученные фрагменты секвенируют по Сэнгеру.
Сущность метода заключается в том, что ДНК выделяют из мочи, крови или органов, после чего полученный образец ДНК амплифицируют по участку гена lipl32, далее положительные образцы последовательно амплифицируют по двум фрагментами генов rpoB и secY, с последующим секвенированием по Сэнгеру.
Состав амплификационной смеси представляет собой буферный раствор содержащий Трис HCl (рН 8,8), сульфат аммония, MgCl2; ЭДТА; меркаптоэтанол; БСА; dNTP; Taq ДНК-полимераза.
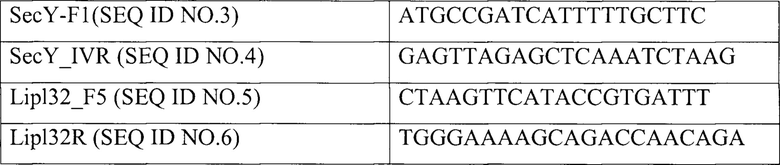
Амплификацию образца по фрагменту гена lipl32, проводят с использованием праймеров Lipl32_F5/Lipl32R, протяженность фрагмента составляет 742 нуклеотида. Для определения нуклеотидной последовательности фрагментов генов, их амплификации и последующего секвенирования используют набор олигодезоксирибонуклеотидных праймеров, комплементарных участкам выявляемых фрагментов. Для исследования по гену гроВ, используют пару праймеров Lept_1900f / Lept_2500R совместно фланкирующих фрагмент протяженностью в 600 нуклеотидов. Для исследования по гену secY, используют праймеры SecY-F1 / SecY_IVR, фланкирующих фрагмент гена длинной в 400 нуклеотидов.

Программа амплификации для lipl32 (95°, 5 мин) денатурации, 50 циклов амплификации 95° - 15 с, 60° - 20 с, 72° - 30 с. Элонгация 72° - 5 мин. Для secY состоит из (95°, 5 мин) денатурации, 40 циклов амплификации 95° -15 с, 56° - 20 с, 72° - 30 с. Элонгация 72° - 5 мин. Для rpoB (95°, 5 мин) денатурации, 35 циклов амплификации 95° - 15 с, 63° - 20 с, 72° - 40 с. Элонгация 72° - 5 мин. Продукты амплификации визуализируют в агарозном геле, окрашенным бромистым этидием, в сравнении с маркером молекулярного веса.
Результаты интерпретируют на основании наличия или отсутствия фрагмента искомого размера, в сравнении с коммерческим набором фрагментов ДНК известной длины. Результат амплификации считается положительным если при использовании праймеров lipl32, образуется фрагмент длиной 742 нуклеотида, праймеров secY фрагмент длиной 400 нуклеотидов, праймеров rpoB фрагмент длиной 600 нуклеотидов.
Сущность изобретения поясняется чертежами, где на фиг. 1 представлены фрагменты гена lipl32 размером 742 нуклеотида, полученные из ДНК, экстрагированной из различного клинического материала. На фиг. 2 представлены фрагменты гена secY, размером 400 нуклеотидов, а на фиг. 3 фрагменты гена rpoB, длиной 600 нуклеотидов.
Таким образом, заявленный способ отличается от прототипа использованием видоспецифичного гена lipl32 для выявления патогенных лептоспир и двух генов secY и rpoB для генотипирования.
Примеры конкретного использования предложенного изобретения.
Пример 1. Исследованы 38 образцов мочи, собранных у различных млекопитающих (собаки, летучие мыши). При амплификации с использованием праймеров по фрагменту гена 16S, было получено 7 положительных образцов, с использованием праймеров по фрагменту lipl32 подтвердились только 5 из 7. Дальнейшая амплификация по фрагментам генов secY и rpoB, также подтвердила наличие ДНК лептоспир в 5 образцах. Секвенирование образцов по генам secY и rpoB, позволило определить вид лептоспир как L. interrogans. Два образца, прошедших положительно по фрагменту гена 16S, при секвенировании определялись как Streptococcus spp., что говорит о ложноположительном результате, и свидетельствует о низкой специфичности данного гена. Таким образом, специфичность предложенного авторами способа выше более, чем на 25%.
Пример 2. Исследованы различные органы 190 особей, диких и синантропных мелких млекопитающих (Rattus norvegicus, Apodemus flavicollis и Myodes glareolus) с использованием праймеров по фрагменту гена 16S, было получено 12 положительных образцов, а по фрагменту гена lipl32 было выявлено 8 позитивных образцов. Дальнейшее исследование с использованием фрагментов генов secY и rpoB, и последующее секвенирование, позволило генотипировать только 8 образцов, прошедших положительно по гену lipl32, до видов L. interrogans и L. borgpetersenii. Образцы, прошедшие положительно только по фрагменту гена 16S, были идентифицированы как ложноположительные с помощью последующего секвенирования.
Пример 3. Исследованы 108 образцов сывороток крови от лихорадящих больных. С использованием праймеров по фрагменту гена lipl32 был выявлен один положительный образец. С амплификацией по фрагменту гена 16S, было выявлено два положительных образца. Дальнейшее генотипирование по генам secY и rpoB позволило идентифицировать один образец до вида L. interrogans. Образец, не подтвержденный амплификацией по фрагменту гена lipl32, так же не получилось генотипировать по генам secY и rpoB, дальнейшее секвенирование по фрагменту гена 16S, подтвердило отсутствие ДНК лептоспир в исследуемом образце, что говорит о ложноположительном результате.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="Способ детекции и генотипирования
патогенных лептоспир в клинических образцах.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2024-05-23">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>0000</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0000</ApplicationNumberText>
<FilingDate>2024-04-29</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера» Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur
Institute</ApplicantNameLatin>
<InventorName languageCode="ru">Баимова Регина
Равилевна</InventorName>
<InventorNameLatin>Baimova Regina Ravilevna</InventorNameLatin>
<InventionTitle languageCode="ru">Усовершенствование способа
детекции и генотипирования патогенных лептоспир в клинических
образцах</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctcatgggttccaacatgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcatcctcraagttgtawcctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgccgatcatttttgcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagttagagctcaaatctaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaagttcataccgtgattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgggaaaagcagaccaacaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к микробиологии и медицине, в частности к диагностике и генотипированию патогенных лептоспир. Предложен способ детекции и генотипирования патогенных лептоспир в клинических образцах. Диагностику и генотипирование проводят с использованием олигодезоксирибонуклеотидных праймеров, комплементарных участкам выявляемых фрагментов. ДНК амплифицируют по участку гена lipl32, положительные образцы последовательно амплифицируют по двум фрагментами генов rpoB и secY с последующим секвенированием по Сэнгеру. Изобретение обеспечивает повышение специфичности и достоверности диагностики, а также расширение арсенала средств, используемых для таксономической характеристики лептоспир. 3 ил., 3 пр.
Способ детекции и генотипирования патогенных лептоспир в клинических образцах, включающий экстракцию ДНК из клинического материала, проведение полимеразной цепной реакции, при этом для диагностики используют фрагмент видоспецифичного гена, а для генотипирования амплифицируют фрагменты двух генов, с использованием набора олигодезоксирибонуклеотидных праймеров, отличающийся тем, что для диагностики используют Lipl32_F5 - ctaagttcataccgtgattt, Lipl32R - tgggaaaagcagaccaacaga, для генотипирования используют Lept_1900f - cctcatgggttccaacatgca, Lept_2500R - cgcatcctcraagttgtawcctt, SecY-F1-atgccgatcatttttgcttc, SecY_IVR - gagttagagctcaaatctaag, при этом продукты амплификации визуализируют в агарозном геле, окрашенном бромистым этидием, в сравнении с маркером молекулярного веса, и визуализируют под UV, результаты интерпретируют на основании наличия или отсутствия фрагмента искомого размера, в сравнении с коммерческим набором фрагментов ДНК известной длины, результат амплификации считают положительным, если при использовании праймеров lipl32 образуется фрагмент длиной 742 нуклеотида, праймеров secY – фрагмент длиной 400 нуклеотидов, праймеров rpoB – фрагмент длиной 600 нуклеотидов, затем осуществляют прямое секвенирование по Сэнгеру полученных фрагментов и делают выводы о видовой принадлежности возбудителя.
| JAVATI S | |||
| et al | |||
| Diversity of Leptospira spp | |||
| in bats and rodents from Papua New Guinea | |||
| Transbound Emerg Dis | |||
| Способ получения продуктов конденсации фенолов с формальдегидом | 1924 |
|
SU2022A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Набор для выявления возбудителя лептоспироза в биологическом материале методом полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) | 2019 |
|
RU2744186C1 |
| BAIMOVA R.R | |||
| et al | |||
| Molecular and genetic characterization of LEPTOSPIRA spp | |||
| collection strains from the St | |||
| Устройство для электрической сигнализации | 1918 |
|
SU16A1 |
Авторы
Даты
2025-05-19—Публикация
2024-06-14—Подача