Изобретение относится к ветеринарной микробиологии, в частности к лабораторной диагностике возбудителей инфекционных болезней, а именно к средствам диагностики инфекции у животных.
Лептоспироз является одним из наиболее распространенных природно-очаговых зооантропонозных болезней в Российской Федерации (РФ). Изучение этого заболевания в России началось 80 лет тому назад, и в настоящее время лептоспироз играет важную роль в патологии человека и животных [1]. Заболевание наносит огромный экономический ущерб животноводству, что складывается уменьшением продуктивности животных, высокой летальностью, снижением воспроизводительной функции [2]. В РФ возбудителями лептоспирозов сельскохозяйственных животных и собак являются лептоспиры серогрупп Pomona, Tarassovi, Grippotyphosa, Sejroe, Icterohaemorrhagiae, CanicolA [3].
Заболевание характеризуется неспецифической клинической картиной, вариабельностью симптомов, из-за чего часто встречаются ошибки в диагностике [4]. Инфекция, вызванная лептоспирами, характеризуется весенне-летней сезонностью, энзоотичностью и периодическими вспышками [5]. Переболевшие животные становятся носителями лептоспир и выделяют возбудителя с мочой в окружающую среду, тем самым представляя опасность для здоровых животных и людей [6].
Одной из особенностей возбудителей лептоспироза является их высокое фенотипическое и генотипическое разнообразие. Геном лептоспир состоит из примерно 4768 генов, многие из которых отвечают за патогенность (синтез липополисахаридов клеточной стенки, адгезия) [7].
Диагностические мероприятия при лептоспирозе включают сбор, анализ эпизоотологических, эпидемиологических, клинических, патолого-анатомических данных с обязательным подтверждением диагноза лабораторными исследованиями [8].
Лабораторная диагностика лептоспироза включает бактериологические (выделение возбудителей из патологического материала, изучение биологических свойств), серологические (реакция микроагглютинации, иммуноферментный анализ) и молекулярно-генетические методы (ПЦР, секвенирование). С помощью реакции микроагглютинации (РМА) выявляют специфические антитела в сыворотке крови инфицированных животных. РМА отличается высокой чувствительностью и специфичностью, но не является методом ранней диагностики, так как антитела в пределах чувствительности РМА появляются не ранее 8-10 дня болезни. Иммуноферментный анализ (ИФА) также эффективен на поздних сроках инфекции [9]. Метод полимеразная цепная реакция в режиме реального времени (ПЦР РВ) является наиболее достоверным, высокочувствительным и специфичным, показывает высокую диагностическую эффективность на ранних сроках заболевания, а также на фоне проводимой антибактериальной терапии [10]. ПЦР дает возможность провести индикацию, идентификацию патогенных лептоспир, а также является инструментом экспресс-диагностики [11]. В качестве генетических маркеров используют гены, отвечающие за патогенность лептоспир, например, ген colA, кодирующий фермент микробную коллагеназу [12], lipl32 - липопротеина наружной мембраны [13]. Ген lipl32 характеризуется высокой консервативностью у патогенных лептоспир, а также детекцией данного гена можно дифференцировать патогенных лептоспир от сапрофитов, так как у свободно живущих представителей рода Leptospira ген lipl32 отсутствует [14].
Известен Способ выявления ДНК возбудителя лептоспироза (Leptospira spp.) у сельскохозяйственных животных (см. патент RU 2700478, МПК C12Q 1/68, Опубл. 17.09.2019, Бюл. №26), при котором используют специфичные возбудителям лептоспироза (Leptospira spp.) праймеры, зонды.
Из уровня техники известен Набор для выявления возбудителя лептоспироза в биологическом материале методом полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) (см. патент RU 2744186, МПК C12Q 1/68, C12Q 1/6876, Опубл. 03.03.2021, Бюл. №7). Изобретение обеспечивает эффективное выявление ДНК геномовидов лептоспир патогенных и промежуточных групп за счет высокой специфичности.
Недостатком известных решений является недостаточная чувствительность метода.
Также известна тест-система для диагностики лептоспироза животных методом гнездовой ПЦР с использованием специфичных к Leptospira interrogans внешних и внутренних праймеров, комплементарных к участку гена 16S рРНК. Детекцию продуктов реакции проводят методом горизонтального электрофореза в агарозном геле с последующим анализом посредством трансиллюминатора. Исследуемые образцы считают отрицательными, если на электрофореграмме отсутствуют четкие полосы или они не соответствуют по размеру фрагменту в контрольном образце [15]. Недостатком данного решения является недостаточная чувствительность метода, а также имеющийся риск контаминации.
Наиболее близким по техническому решению к заявляемому изобретению можно отнести способ выявления патогенного вида Leptospira interrogans в клиническом и патологическом материалах, а также в образцах воды методом ПЦР РВ (см. патент UA 101190, МПК C07K 14/20; заявл. 03.04.2015; опубл. 25.08.2015), был выбран за прототип. При данном способе в исследуемых образцах выявляются специфические фрагменты нуклеиновых кислот (ДНК) с помощью ПЦР с гибридизационно-флуоресцентной детекцией продуктов амплификации в режиме реального времени. Результат амплификации ДНК патогенных лептоспир регистрируется на канале флуоресценции FAM/Green с помощью специфических олигонуклеотидных флуоресцентно-меченных зондов и праймеров, комплементарных к участкам гена lipl32, синтезированных искусственно.
С помощью настоящего изобретения предлагается решить следующую задачу: определить наличие наиболее специфичных локусов генома возбудителя лептоспироза.
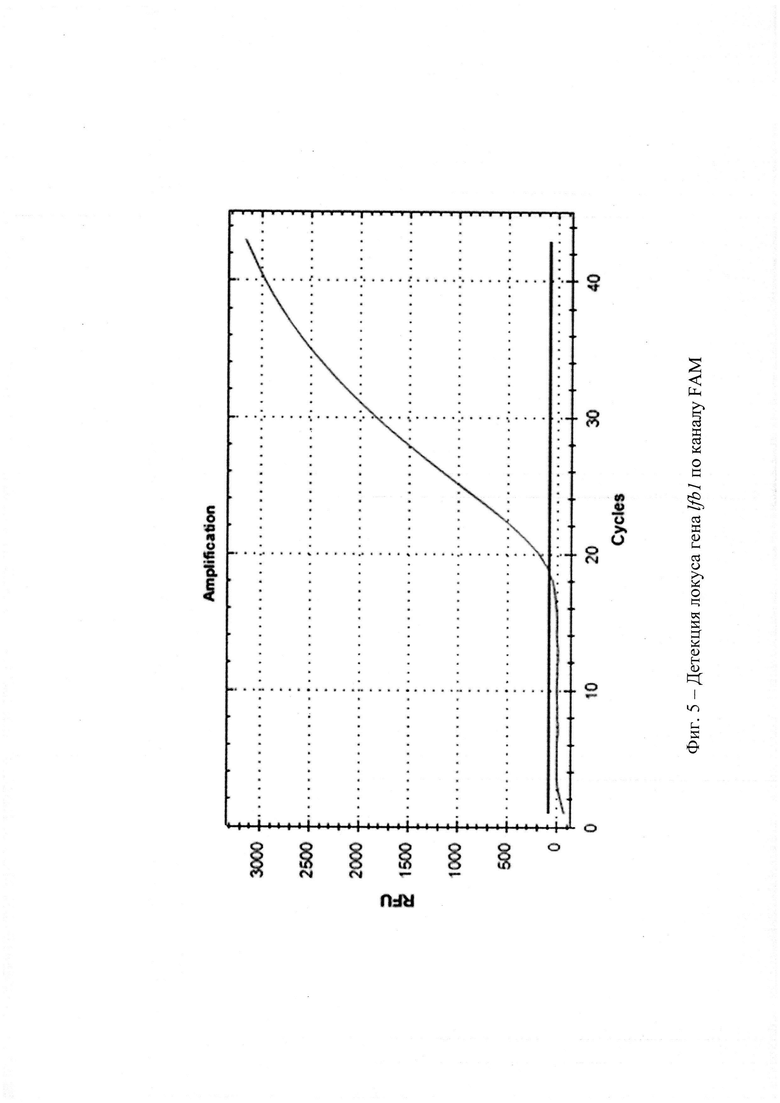
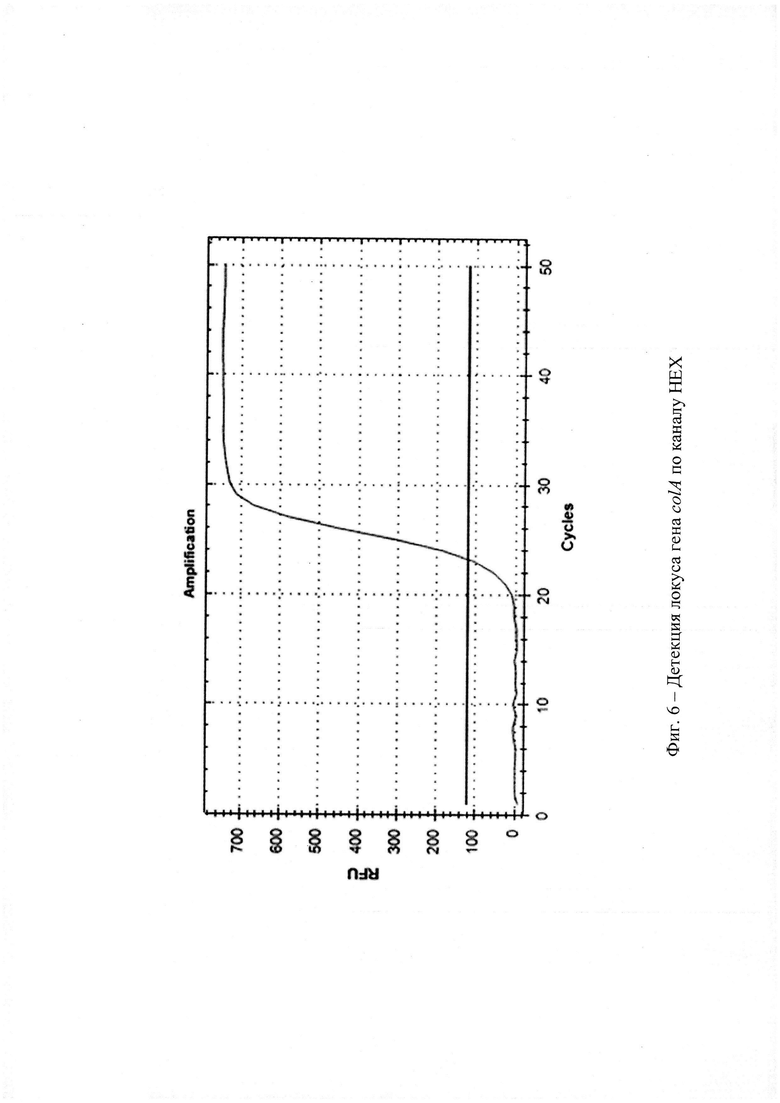
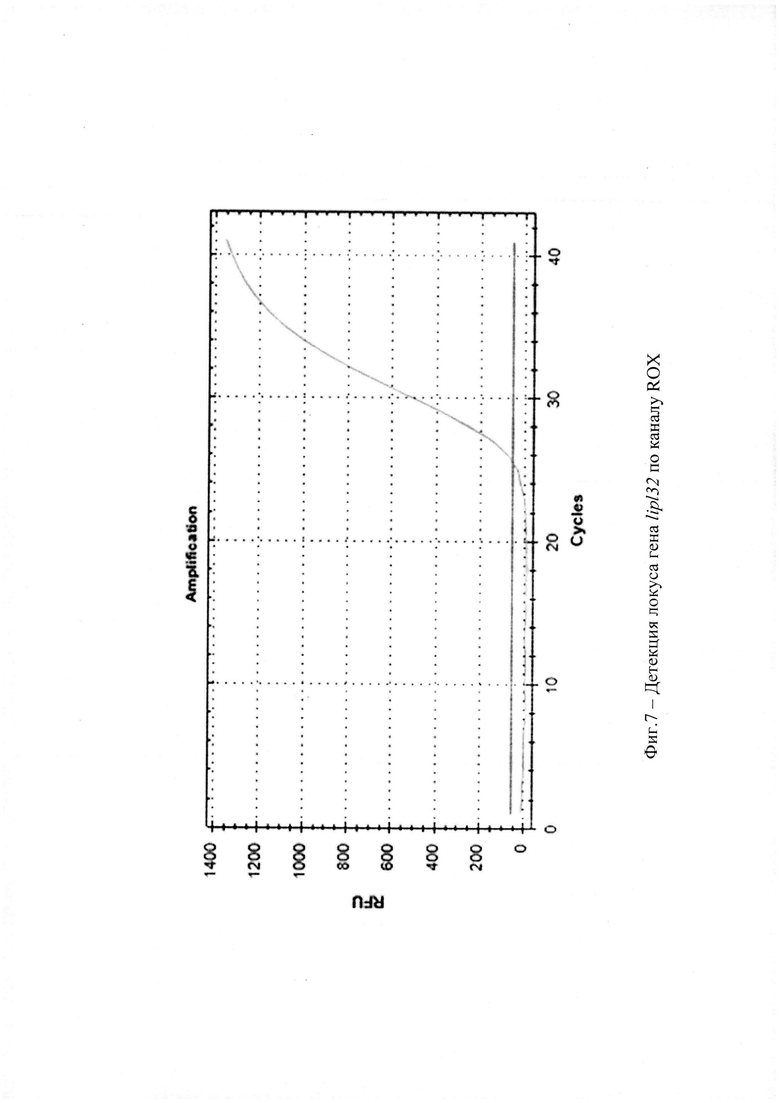
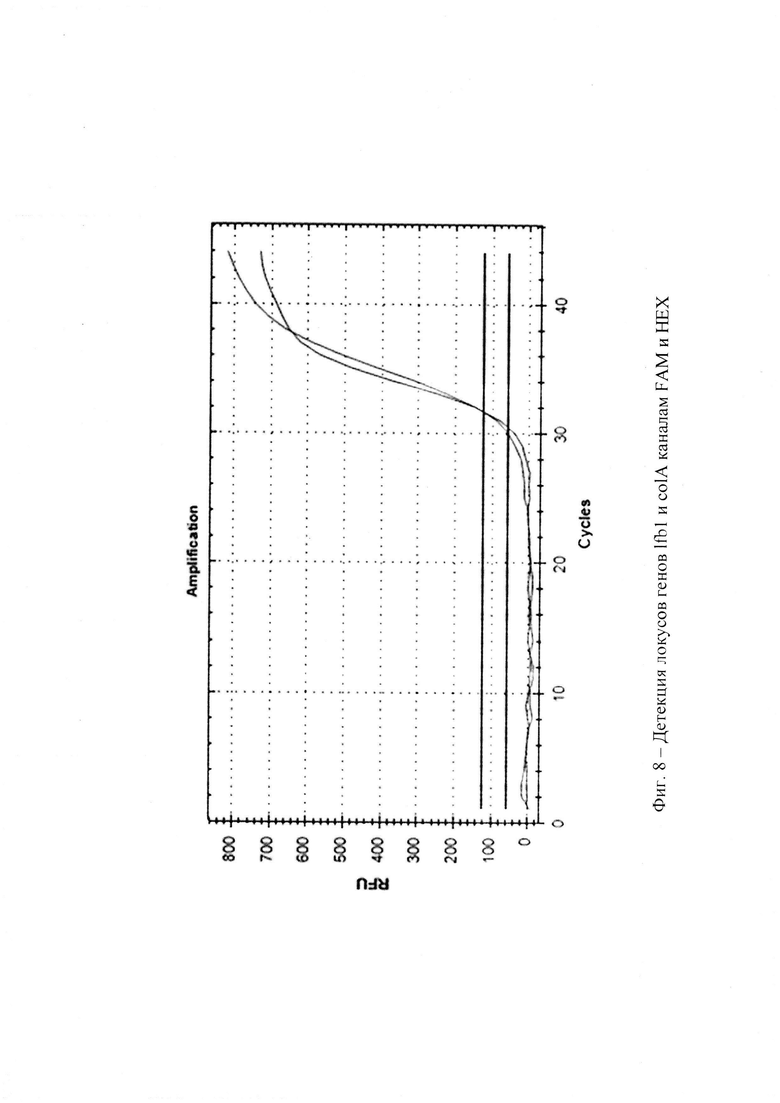
Технический результат заявляемого Способа индикации заключается в повышении специфичности и чувствительности ПЦР за счет использования способа обнаружения ДНК возбудителя лептоспироза по трем специфичным ДНК-локусам, а также в устранении таких недостатков, как выполнение электрофореза, и снижении риска контаминации. Технический результат достигается постановкой одноэтапной ПЦР в режиме реального времени по трем специфическим ДНК-локусам генома возбудителя лептоспироза. Измерение проводится по каналам накопления флуоресцентного сигнала FAM, ROX и HEX по локусам генов lfb1, lipl32, colA. Интерпретация результатов проводится на основании наличия (отсутствия) пересечения кривой флуоресценции с пороговой линией и определяется величиной порогового цикла «Ct». Если кривые накопления флуоресцентного сигнала выходят до 40 цикла (Ct<40), то ДНК возбудителей лептоспироза присутствует, и результат реакции считается положительным. Если кривые не пересекают пороговую линию или пересекают ее после 40 цикла (Ct>40), то ДНК патогенных лептоспир отсутствует и результат реакции отрицательный.
ПКО представляет собой рекомбинантную плазмиду pALT2, содержащую в своем составе три ДНК-локуса возбудителей лептоспироза - участки генов lfb1, lipl32, colA. ПКО имеет следующую нуклеотидную последовательность:

ПРИМЕЧАНИЯ.
1) Красный цвет шрифта - праймерная комбинация для амплификации гена lfb1, кодирующего фибронектин-связывающий белок;
2) Зеленый цвет шрифта - праймерная комбинация для амплификации гена lipl32, кодирующего липопротеин наружной мембраны;
3) Синий цвет шрифта - праймерная комбинация для амплификации гена colA, кодирующего ген фермента коллагеназу.
Изобретение иллюстрируется графическим материалом, где показаны: На фиг. 1 - Структура ПКО для способа индикации патогенных лептоспир методом мультиплексной ПЦР;
На фиг. 2 - Места «отжига» праймеров и зонда к гену Ifbl Leptospira interrogans serovar Lai str. 56601 chromosome I, complete sequence, GBID AEO 10300.2;
На фиг. 3 - Места «отжига» праймеров и зонда к гену Нр132 Leptospira interrogans serovar Lai str. 56601 chromosome I, complete sequence, GBID AE010300.2;
На фиг. 4 - Места «отжига» праймеров и зонда к гену colA Leptospira interrogans serovar Lai str. 56601 chromosome I, complete sequence, GBID AE010300.2;
На фиг. 5 - Детекция локуса гена lfb1 по каналу FAM;
На фиг. 6 - Детекция локуса гена colА по каналу HEX;
На фиг. 7 - Детекция локуса гена lipl32 по каналу ROX;
На фиг. 8 - Детекция локусов генов lfb1 и colA каналам FAM и HEX;
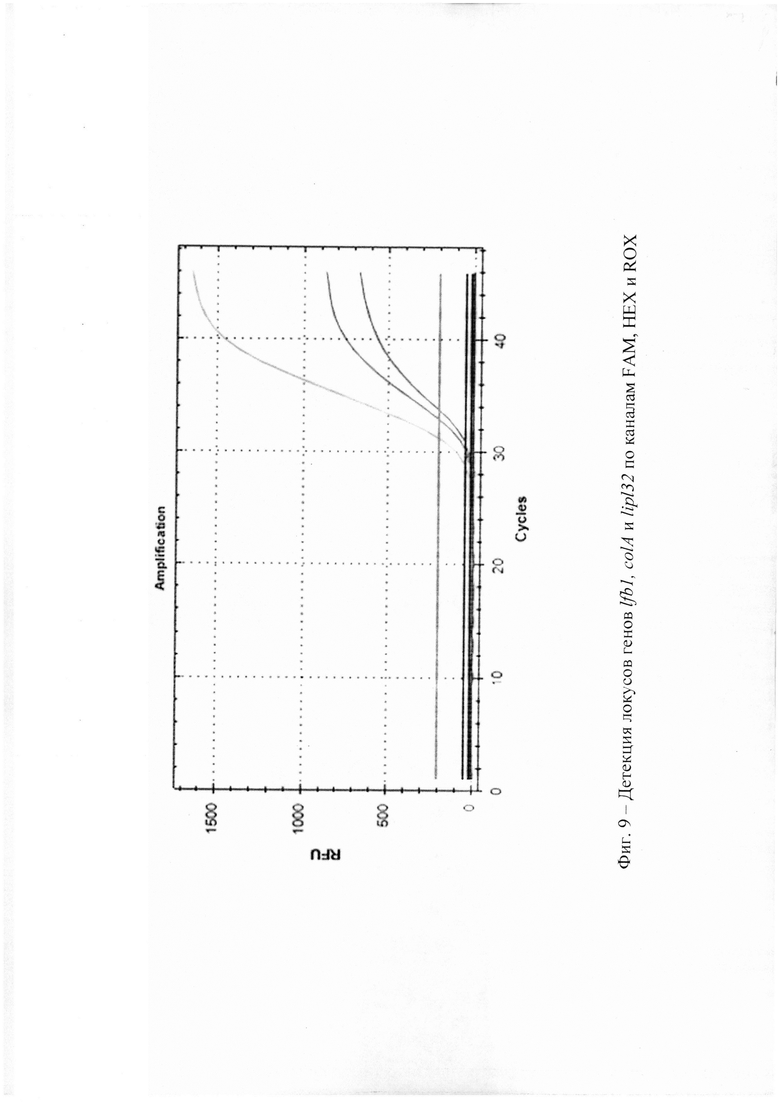
На фиг. 9 - Детекция локусов генов lfb1, colA и lipl32 по каналам FAM, HEX и ROX.
Причем на фиг. 2-4 приведены локусы генов lfb1, lipl32 и colA, по которым осуществляется обнаружение ДНК возбудителя лептоспироза, также выделены места посадки «отжига» флуоресцентно-меченных олигонуклеотидных зондов и праймеров.
Сущность изобретения поясняется графиками (фиг. 5-9), где представлены результаты детекции локусов генов lfb1, colA и lipl32 по каналам FAM, HEX, ROX соответственно. Новизна заявляемого технического решения заключается в том, что для повышения чувствительности и специфичности реакции используется обнаружение ДНК возбудителя лептоспироза по трем специфичным локусам ДНК. Также преимуществами данного технического решения является снижение риска контаминации при проведении ПЦР.
Благодаря сравнительному анализу заявляемого способа с известными способами индикации патогенных лептоспир с помощью метода мультиплексной ПЦР соответствует критерию патентоспособности изобретения «новизна» по причине отсутствия совокупности существенных признаков заявляемого изобретения среди результатов проведенного патентно-информационного поиска.
Признаки, отличающие заявляемое техническое решение от прототипа, направлены на достижение технического результата и не выявлены при изучении данной и смежной областей науки и техники и, следовательно, соответствуют критерию «изобретательский уровень».
Заявляемый способ рекомендовано использовать в ветеринарной микробиологии, так как относится к средствам выявления ДНК возбудителя лептоспироза, что соответствует критерию «промышленная применимость».
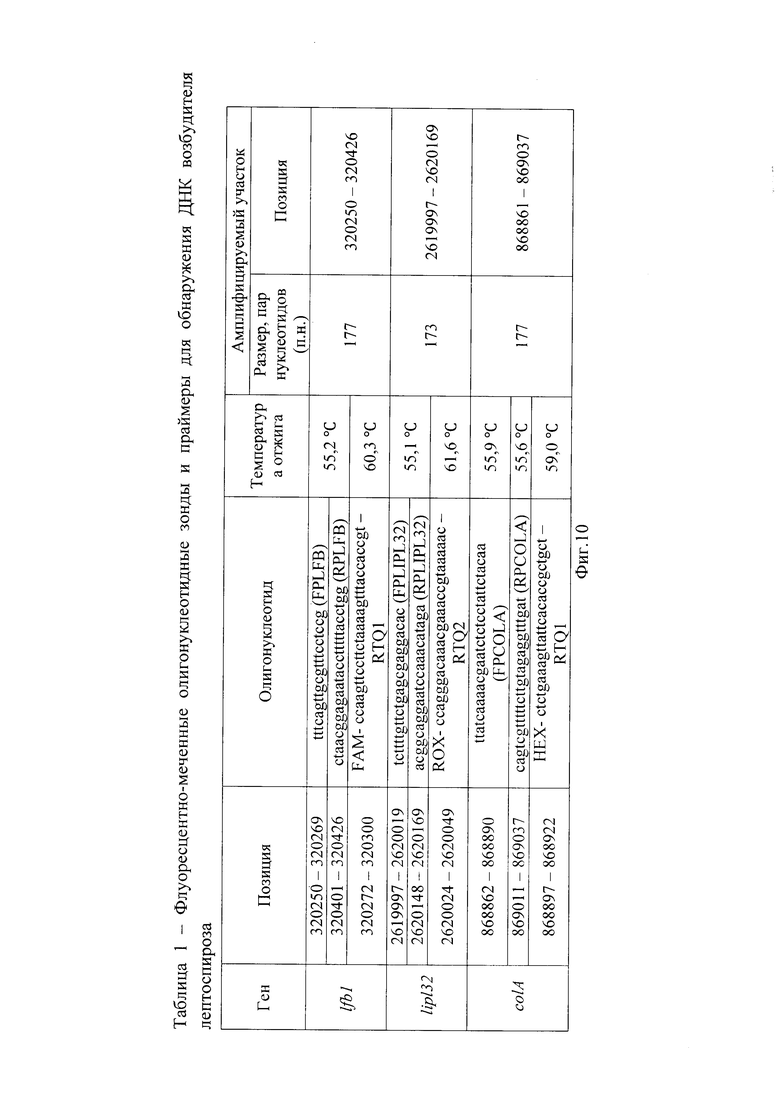
Способ выявления ДНК патогенных лептоспир из клинического материала, полученного от инфицированных животных осуществляется следующим образом. Для постановки ПЦР используются предварительно приготовленные образцы ДНК, выделенных из различных объектов окружающей среды, патологического и биологического материала (кровь, моча, вода и др.), с помощью различных методов выделения ДНК посредством различных наборов для экстракции ДНК. В качестве ПКО используют рекомбинантную плазмиду pALT2, содержащую три специфичных ДНК-локуса генома возбудителя лептоспироза. Используемые флуоресцентно-меченные олигонуклеотидные зонды и праймеры представлены в таблице 1 (Фиг. 10).
Осуществляют постановку одноэтапной ПЦР, состоящей из 40 последовательных циклов амплификации с флуоресцентной детекцией с использованием специфичных к возбудителю лептоспироза флуоресцентно-меченных олигонуклеотидных зондов, праймеров и ПКО. По каналам FAM, ROX и HEX измеряют накопление флуоресцентного сигнала, что свидетельствует о накоплении специфичных продуктов ПЦР и присутствии генома возбудителя лептоспироза в исследуемом образце. Интерпретация результатов проводится на основании наличия (отсутствия) пересечения кривой флуоресценции с пороговой линией и определяется величиной порогового цикла «Ct». Если кривые накопления флуоресцентного сигнала выходят до 40 цикла (Ct<40), то ДНК возбудителей лептоспироза присутствует, и результат реакции считается положительным. Если кривые не пересекают пороговую линию или пересекают ее после 40 цикла (Ct>40), то ДНК патогенных лептоспир отсутствует и результат реакции отрицательный. ПКО представляет собой рекомбинантную плазмиду pALT2, содержащую в своем составе три ДНК-локуса возбудителей лептоспироза - участки генов lfb1, lipl32, col А.
Флуоресцентная детекция продуктов амплификации осуществляется с использованием принципа «освобождения» флуоресцентной метки на 5' конце олигонуклеотидного зонда.
Пример конкретного осуществления способа выявления ДНК возбудителя лептоспироза. Для исследования используют предварительно приготовленные образцы ДНК, выделенных из различных объектов окружающей среды, патологического и биологического материала (кровь, моча, вода и др.), с помощью различных методов выделения ДНК посредством различных наборов для экстракции ДНК.
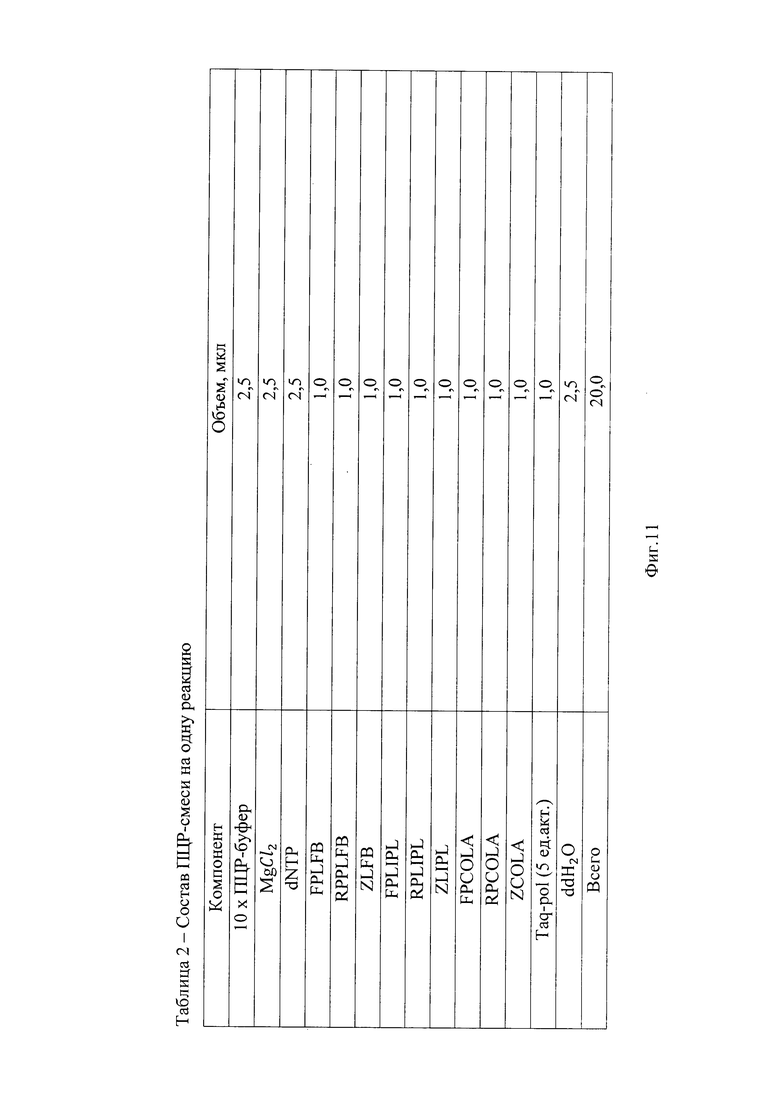
Проведение анализа. Для проведения анализа используют общие компоненты для постановки ПЦР (10 х ПЦР-буфер, магния хлорид MgCl2, синтетические дезоксирибонуклеозидтрифосфаты dNTP, полимеразу (5 ед.акт.) - Taq-pol. и диионизированную воду ddH2O), и специфические компоненты - 3 комбинации олигонуклеотидных праймеров и зондов, специфичных к локусам генов lfb1, lipl32 и colA возбудителя лептоспироза. Состав ПЦР-смеси на одну реакцию представлен в таблице 2 (фиг. 11).
Этапы анализа:
1) Проведение ПЦР с флуоресцентной детекцией в режиме реального времени;
2) Интерпретация результатов.
Подготовка образцов к проведению ПЦР Общий объем реакционной смеси - 20,0 мкл, объем ДНК-пробы - 5,0 мкл. Успешное прохождение реакции контролируют использованием ПКО. В отдельной пробирке смешивают компоненты набора из расчета на каждую реакцию ПЦР плюс еще один анализ. Перемешивают смесь на вортексе и сбрасывают капли кратковременным центрифугированием. Отбирают необходимое количество пробирок для амплификации ДНК исследуемых и контрольных образцов. Вносят по 20,0 мкл приготовленной реакционной смеси. Используя наконечники с фильтром в подготовленные пробирки вносят:
- в пробирку отрицательного контроля ПЦР - 5,0 мкл ddH2O;
- в ряд пробирок для исследуемых образцов - в каждую вносят по 5,0 мкл ДНК соответствующего образца;
- в пробирку ПКО - 5,0 мкл ПКО.
ПЦР в режиме реального времени (ПЦР РВ) с флуоресцентной детекцией проводят использованием детектирующего амплификатора.
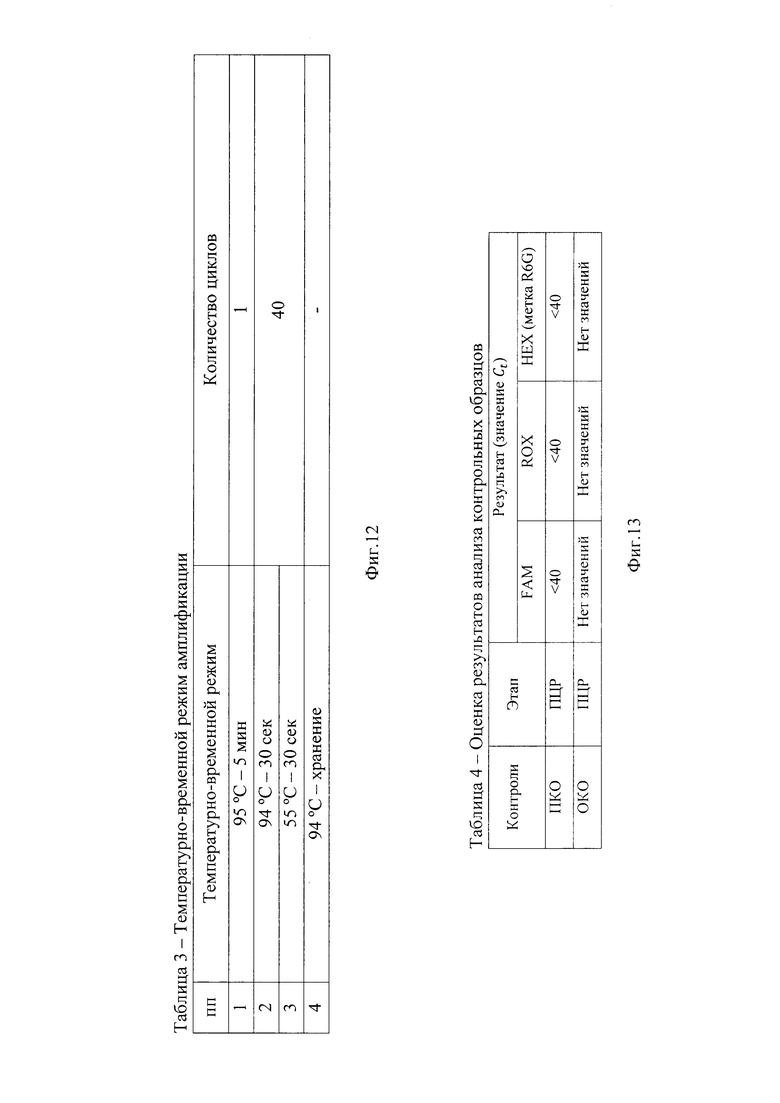
Подготовленные для проведения ПЦР пробирки помещают в его ячейки. Далее программируют прибор (таблица 3) (фиг. 12).
Интерпретацию результатов анализа проводят по полученным кривым накопления флуоресцентного сигнала, которые анализируются с помощью программного обеспечения используемого прибора для проведения ПЦР в режиме «реального времени» в соответствии с инструкцией производителя к прибору.
Учет результатов ПЦР-анализа проводится по наличию или отсутствию пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию или отсутствию значения порогового цикла «Ct» для исследуемого образца). Результат считается достоверным в случае корректного прохождения положительных и отрицательных контролей амплификации представлен в таблице 4 (фиг. 13).
В таблице 4 показано, что появление любого значения Ct результатов для отрицательного контроля свидетельствует о наличии контаминации реактивов или образцов. В этом случае результаты анализа для всех проб считаются недействительными. Требуется повторить анализ всех проб, а также предпринять меры по выявлению и ликвидации источника контаминации.
Образец считается положительным (ДНК возбудителя лептоспироза присутствует) при значении Ct меньше 40 хотя бы одному из задействованному каналу флуоресценции. Если для исследуемого образца по каналам FAM, ROX и HEX значение Ct больше или равным 40 - он считается спорным и исследуется повторно с этапа выделения ДНК. Если при повторной постановке наблюдается схожий результат, то необходимо повторно взять материал для ПЦР-анализа.
Эффективность заявляемого технического решения по сравнению с прототипом заключается в том, что для повышения чувствительности и специфичности используется обнаружение ДНК возбудителя лептоспироза по трем специфичным локусам генов lfb1, lipl32 и colA.
Список литературы
1. Ананьина, Ю.В. Лептоспирозы в России: этиологическая структура и современная этиология / Ю.В. Ананьина, Е.М. Петров // РЭТ-инфо. - 2006. - №1 (57). - С. 8-10.
2. Белоусов, В.И. Лабораторная диагностика и совершенствование профилактики лептоспироза животных в Российской Федерации / В.И. Белоусов, Г.А. Нурлыгаянова, А.А. Варенцова, С.Б. Базарбаев // Труды Всероссийского НИИ экспериментальной ветеринарии им. Я.Р. Коваленко. Т. 80 (2). - 2018. - С. 46-52.
3. Лептоспироз в Республике Татарстан. Эпидемиологические особенности / Т.А. Савицкая, В.А. Трифонов, И.В. Милова [и др.] // Проблемы особо опасных инфекций. - 2021. - №1. - С. 134-139.
4. Киселева, Е.Ю. Лабораторный скрининг лептоспирозов у людей / Е.Ю. Киселева, Н.В. Бренева // Матер. III Ежегод. Всеросс.Конгресса по инфек. Бол., Москва, 28-30 марта 2011 года / Минздрав, и соц. Разв. РФ, Фед. служ. по надзору в сфере защиты прав потреб, и благопол. человека, РАМН, Центр, науч.-иссл. инст.эпидем. Роспотребнадзора, нац. науч. общ. инфекционистов, Нац. ассоц. диетологов и нутрициологов, Федерация педиатров стран СНГ. - Москва: Без издательства, 2011. - С. 167.
5. Шабунин, С.В. Бактериальные и вирусные инфекции в патологии воспроизводительной функции коров / С.В. Шабунин, А.Г. Шахов, А.Г. Нежданов // Ветеринария. - 2012. - №10. - С. 3-8.
6. Диагностика, специфическая профилактика и лечение при бактериальных болезнях животных / М.К. Пирожков, С.В. Ленев, Е.В. Викторова [и др.] // Ветеринария. - 2011. - №1. - С. 24-27.
7. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing / Ren S.X. [et al.] // Nature. -2003. - Vol. 422(6934). - P. 888-93. doi: 10.1038/nature01597.
8. Малахов Ю.А., Панин A.H., Соболева Г.Л. Лептоспироз животных. - М.:Медгиз, 2000. - 621 с.
9. Киселева, Е.Ю. Применение полимеразной цепной реакции для лабораторной диагностики и мониторинга лептоспирозов в Сибири и на Дальнем Востоке / Е.Ю. Киселева, Н.В. Бренева, М.В. Чеснокова // Инфекционные болезни. - 2011. - Т.9, №2. - С. 64-67.
10. Попов, С.Ф. Современные подходы к диагностике и лечению больных лептоспирозом / С.Ф. Попов, Е.А. Иоанниди, О.В. Александров // Лекарственный вестник. - 2020. - Т. 14, №2 (78). - С. 31-34.
11. Методы лабораторной диагностики лептоспирозов: особенности постановки, преимущества и недостатки / Е.Ю. Киселева, Н.В. Бренева, А.К. Носков [и др.] // Бюллетень Восточно-Сибирского научного центра Сибирского отделения Российской академии медицинских наук. - 2015. - №3 (103). - С. 85-93.
12. Ваганова, А.Н. Адаптация ПЦР для диагностики лептоспироза и эпидемиологического надзора за лептоспирозной инфекцией / А. Н. Ваганова, Н.А. Стоянова, Н.К. Токаревич // Национальные приоритеты России. - 2011. - №2 (5). - С. 162-164.
13. Соболева, Г.Л. Актуальные вопросы лептоспироза людей и животных / Г.Л. Соболева, Ю.В. Ананьина, И.В. Непоклонова // Российский ветеринарный журнал. - 2017. - №8. - С. 14-18.
14. Ананьина, Ю.В. Паразитические и свободноживущие лептоспиры (Leptospiraceae): эколого-генетические особенности / Ю.В. Ананьина // Зоологический журнал. - 2010. - Т. 89, №1. - С. 48-52.
15. Викторова Е.В. Полимеразная цепная реакция при диагностике лептоспироза и изучение органотропности лептоспир у сельскохозяйственных животных: спец. 16.00.03 «ветеринарная микробиология, вирусология, эпизоотология, микология с микотоксикологией и иммунология»: автореф. дис. канд. вет. наук. Москва.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Лептоспиры.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-10-30">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024115668/10(035189)</ApplicationNumberText>
<FilingDate>2024-06-05</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение «Федеральный центр токсикологической,
радиационной и биологической безопасности»</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Scientific Institution
Federal Center for Toxicological, Radiation and Biological
Safety</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ индикации патогенных
лептоспир с помощью метода мультиплексной ПЦР</InventionTitle>
<SequenceTotalQuantity>10</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>343</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..343</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttcagttgcgtttcctccggcccaagttccttctaaaagtttaccacc
gtacttcatagattactggttctgtttctccccaatctatcttttgttctgagcgaggacacaatcccag
ggacaaacgaaaccgtaaaaacataccaggtaaaaaggtattctccgttagatattatcaaaacgaatct
ctcctattctacaattcactctgaaagttattcacaccgctgctatatctatgtttggattcctgccgtt
tccaaggtagacataagatcgttgatccttgaattgaaatcagaccacgcagagttcatcaaacctctac
aagaaaaacgactg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttcagttgcgtttcctccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaacggagaatacctttttacctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaagttccttctaaaagtttaccaccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcttttgttctgagcgaggacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acggcaggaatccaaacataga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagggacaaacgaaaccgtaaaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttatcaaaacgaatctctcctattctacaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagtcgtttttcttgtagaggtttgat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Leptospira
interrogans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctgaaagttattcacaccgctgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ детекции и генотипирования патогенных лептоспир в клинических образцах | 2024 |
|
RU2840144C1 |
| Набор для выявления возбудителя лептоспироза в биологическом материале методом полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) | 2019 |
|
RU2744186C1 |
| Способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР) | 2022 |
|
RU2798286C1 |
| Набор олигонуклеотидных праймеров и зондов для идентификации РНК вируса лихорадки Рифт-Валли методом изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2023 |
|
RU2813519C1 |
| Способ выявления ДНК возбудителя лептоспироза (Leptospira spp.) у сельскохозяйственных животных | 2018 |
|
RU2700478C1 |
| Способ индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex c помощью ПЦР-РВ | 2024 |
|
RU2837898C1 |
| Тест-система для выявления ДНК возбудителя лептоспироза (Leptospira spp.) у сельскохозяйственных животных | 2018 |
|
RU2680094C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex с помощью ПЦР-РВ | 2024 |
|
RU2839472C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |



Изобретение относится к ветеринарной микробиологии, в частности к лабораторной диагностике возбудителей инфекционных болезней, а именно к средствам диагностики инфекции у животных. Осуществляется постановка одноэтапной ПЦР в режиме реального времени по трем специфическим ДНК-локусам генома возбудителя лептоспироза. Измерение проводится по каналам накопления флуоресцентного сигнала FAM, ROX и HEX по локусам генов lfb1, lipl32, colA. Интерпретация результатов проводится на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией и определяется величиной порогового цикла «Ct». Если кривые накопления флуоресцентного сигнала выходят до 40 цикла (Ct<40), то ДНК возбудителей лептоспироза присутствует и результат реакции считается положительным. Если кривые не пересекают пороговую линию или пересекают ее после 40 цикла (Ct>40), то ДНК патогенных лептоспир отсутствует и результат реакции отрицательный. ПКО представляет собой рекомбинантную плазмиду pALT2, содержащую в своем составе три ДНК-локуса возбудителей лептоспироза - участки генов lfb1, lipl32, colA. Эффективность заявляемого технического решения заключается в том, что для повышения чувствительности и специфичности используется обнаружение ДНК возбудителя лептоспироза по трем специфичным локусам генов lfb1, lipl32 и colA. Технический результат заключается в повышении специфичности и чувствительности ПЦР за счет использования способа обнаружения ДНК возбудителя лептоспироза по трем специфичным ДНК-локусам, а также в устранении таких недостатков, как выполнение электрофореза, и снижении риска контаминации. 13 ил.
Способ индикации патогенных лептоспир с помощью метода мультиплексной ПЦР, представляющий собой одновременную амплификацию трех специфичных участков генов - lfb1, lipl32, colA - возбудителя лептоспироза с детекцией результатов в режиме реального времени по каналам накопления флуоресцентного сигнала FAM, ROX и HEX, причем праймерная комбинация для амплификации специфического участка гена lfb1 представлена нуклеотидными последовательностями: tttcagttgcgtttcctccg (FPLFB), ctaacggagaatacctttttacctgg (RPLFB), FAM-ccaagttccttctaaaagtttaccaccgt-RTQ1, а специфический фрагмент гена lipl32 амплифицируется следующими олигонуклеотидами: tcttttgttctgagcgaggacac (FPLIPL32), acggcaggaatccaaacataga (RPLIPL32), ROX-ccagggacaaacgaaaccgtaaaaac-RTQ2, где участок гена colA фланкируется следующими праймерами: ttatcaaaacgaatctctcctattctacaa (FPCOLA), cagtcgtttttcttgtagaggtttgat (RPCOLA), HEX- ctctgaaagttattcacaccgctgct-RTQ1; размер амплифицируемого фрагмента гена lfb1 составляет 177, lipl32 - 173 и colA - 111 пар нуклеотидов соответственно, интерпретация результатов проводится на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией и определяется величиной порогового цикла «Ct».
| Коконоразмоточный аппарат | 1932 |
|
SU30798A1 |
| RU 2012132517 А, 10.09.2014 | |||
| P | |||
| Khaki, F | |||
| Rahimi Zarchi, S | |||
| Moradi Bidhendi, M | |||
| Gharakhani, Application of a Multiplex PCR Assay for Molecular Identification of Pathogenic and Non-Pathogenic Leptospires based on lipL32 and 16S rRNA, Arch Razi Inst | |||
| Электромагнитный прерыватель | 1924 |
|
SU2023A1 |
Авторы
Даты
2025-03-25—Публикация
2024-06-05—Подача