Изобретение относится к сельскому хозяйству, ветеринарии, в частности к способу оценки присутствия генов токсинов, включая альфа-токсин cpa, бета-токсин cpb, эпсилон-токсин etx Clostridium perfringens и бинарный токсин cdtB, токсин А tcdA, токсин В tcdB Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота. Диагностика осуществляется с использованием молекулярно-биологического метода полимеразной цепной реакции с применением набора специфичных праймеров с последующей детекцией генов токсинов путем гель-электрофореза.
Изобретение позволяет оценить профиль генов патогенных бактерий Clostridium perfringens и Clostridioides difficile.
Известен способ диагностики патогенной микробиоты, присутствующей в ЖКТ животных, включая такие особо опасные виды микроорганизмов, как Clostridium perfringens, C. difficile, C. ramosum, C. septicum. Способ заключается в быстрой идентификации патогенной микробиоты ЖКТ животных, включая Clostridium perfringens, C. difficile, C. ramosum, C. septicum и пр. при помощи методов высокопроизводительного секвенирования и последующей биоинформатической обработки [RU 2827174, G01N 33/48].
Недостатками данного способа является его узкая специфичность и отсутствие возможности оценки присутствия токсинов патогенных микроорганизмов Clostridium perfringens и Clostridioides difficile.
Известен набор дифференцирующих и специфических олигонуклеотидов для идентификации ДНК возбудителей острых кишечных инфекций, способ идентификации острых кишечных инфекций, микрочип и диагностическая система для осуществления способа. Изобретение относится к области молекулярной биологии и может быть использовано в диагностических исследованиях, направленных на выявление возбудителей острых кишечных инфекций. Предложен набор дифференцирующих олигонуклеотидов, обеспечивающий возможность определения в биологическом образце ДНК патогенных микроорганизмов, вызывающих острые кишечные инфекции, и их видовой идентификации, где указанные микроорганизмы относятся к группе, включающей Shigella spp. и Enteroinvasive E.coli, Salmonella spp., Campylobacter jejuni, Proteus mirabilis, Klebsiella pneumoniae. Описано конструирование биочипа с иммобилизованным на нем набором зондов по изобретению, предназначенного для проведения экспресс-анализа возбудителей острых кишечных инфекций в клинических образцах и материале, полученном из объектов окружающей среды [RU 2004114259, C12N 15/11, C12Q 1/68].
Недостатками данного способа является его узкая специфичность.
Известен способ диагностики токсигенных штаммов Clostridium difficile. Изобретение относится к области медицинской микробиологии, а именно к способу диагностики токсигенных штаммов Clostridium difficile. Использование заявленного способа позволяет с высокой чувствительностью и специфичностью, простотой и доступностью диагностировать токсигенные штаммы Clostridium difficile. [RU 2548719, G01N 33/569, G01N 33/50].
Недостатками данного способа является его узкая специфичность.
Известен способ получения ботулинического токсина. Изобретение относится к биотехнологии, а именно к получению ботулинического токсина путем культивирования Clostridium botulinum, на среде, содержащей картофельный пептон, дрожжевой экстракт, глюкозу и очищенную воду в заданном соотношении. Использование заявленного способа позволяет эффективно сокращать время культивирования Clostridium botulinum в среде, не содержащей при этом продуктов животного происхождения и основных аллергенов, а также к способу получения ботулинического токсина с ее использованием. [RU 2782793, C12R1/145, C12N1/20, A61K39/08].
Недостатками данного способа является его узкая специфичность.
Наиболее близким по технической сущности к заявленному изобретению является способ детектирования и характеризации токсиногенного штамма Clostridium difficile. Изобретение относится к биотехнологии. Описан способ детектирования и характеризации токсиногенного штамма Clostridium difficile в пробе в мультиплексном ПЦР-анализе. [RU 2612789, C12Q 1/68, C12N 15/11, C12M 1/00].
Недостатками данного способа являются:
1. Выявление узкого спектра генов токсинов.
2. Способ не позволяет проводить анализ генов токсинов бактерии Clostridium perfringens.
Задача изобретения - оценка присутствия генов токсинов, включая альфа-токсин cpa, бета-токсин cpb, эпсилон-токсин etx Clostridium perfringens и бинарный токсин cdtB, токсин А tcdA, токсин В tcdB Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота.
Поставленная задача решается за счет того, что способ оценки генов токсинов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота, осуществляющийся с использованием молекулярно-биологического метода полимеразной цепной реакции с последующей детекцией генов токсинов путем гель-электрофореза с применением набора специфичных праймеров: альфа-токсин cpa, бета-токсин cpb, эпсилон-токсин etx Clostridium perfringens и бинарный токсин cdtB, токсин А tcdA, токсин В tcdB Clostridioides difficile. Используются праймеры, специфичные к участкам генов исследуемых токсинов, что позволяет проводить избирательную амплификацию искомых фрагментов ДНК при помощи полимеразной цепной реакции непосредственно из тотальной ДНК биологических образцов и детектировать фрагменты генов токсинов путем гель-электрофореза.
Новые существенные признаки
1. Широкий охват оценки присутствия генов токсинов патогенных бактерий Clostridium perfringens и Clostridioides difficile.
2. Сокращение продолжительности времени детекции за счет идентичного режима полимеразной цепной реакции для анализа присутствия генов токсинов патогенных бактерий Clostridium perfringens и Clostridioides difficile.
Предлагаемый способ является высокочувствительным, универсальным и быстрым по отношению к другим методам.
Перечисленные новые существенные признаки в совокупности с известными позволяют получить технический результат во всех случаях, на которые распространяется объем правовой охраны.
Технический результат
Технический результат заключается в быстрой идентификации присутствия генов токсинов патогенных микроорганизмов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах крупного рогатого скота, при помощи метода полимеразной цепной реакции с применением набора специфичных праймеров с последующей детекцией генов токсинов путем гель-электрофореза.
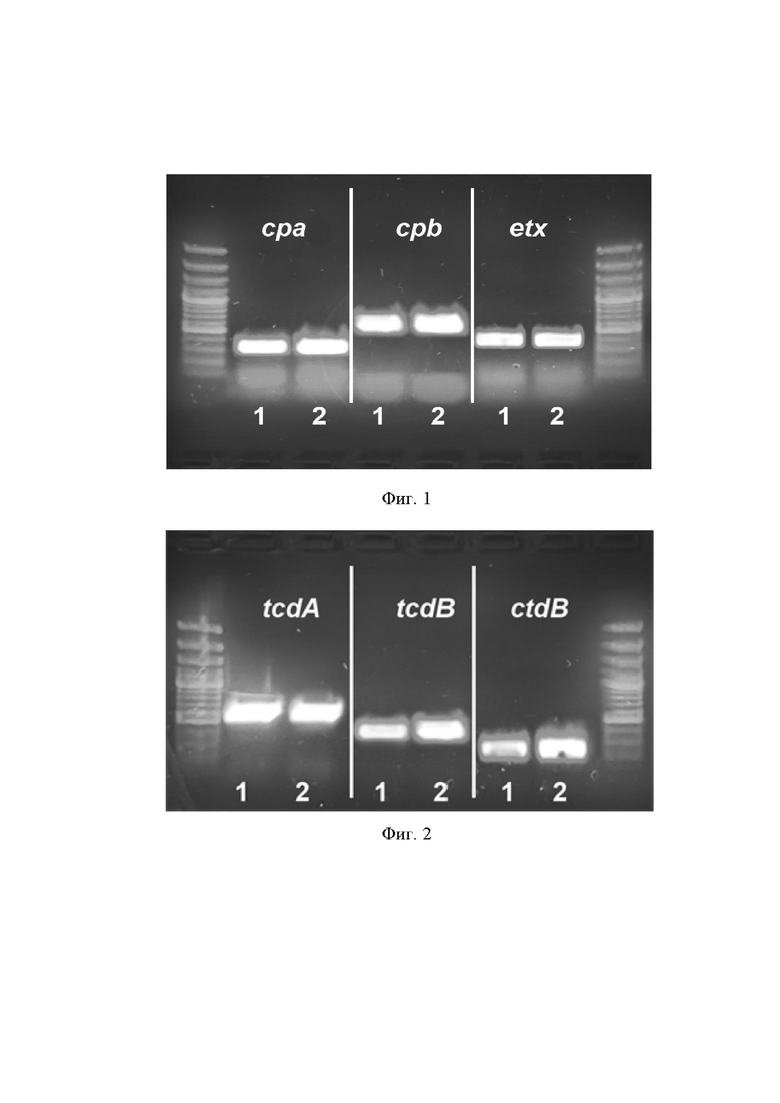
На фиг. 1 изображены результаты анализа генов токсинов в ДНК штаммов Clostridium perfringens штаммы 1 и 2.
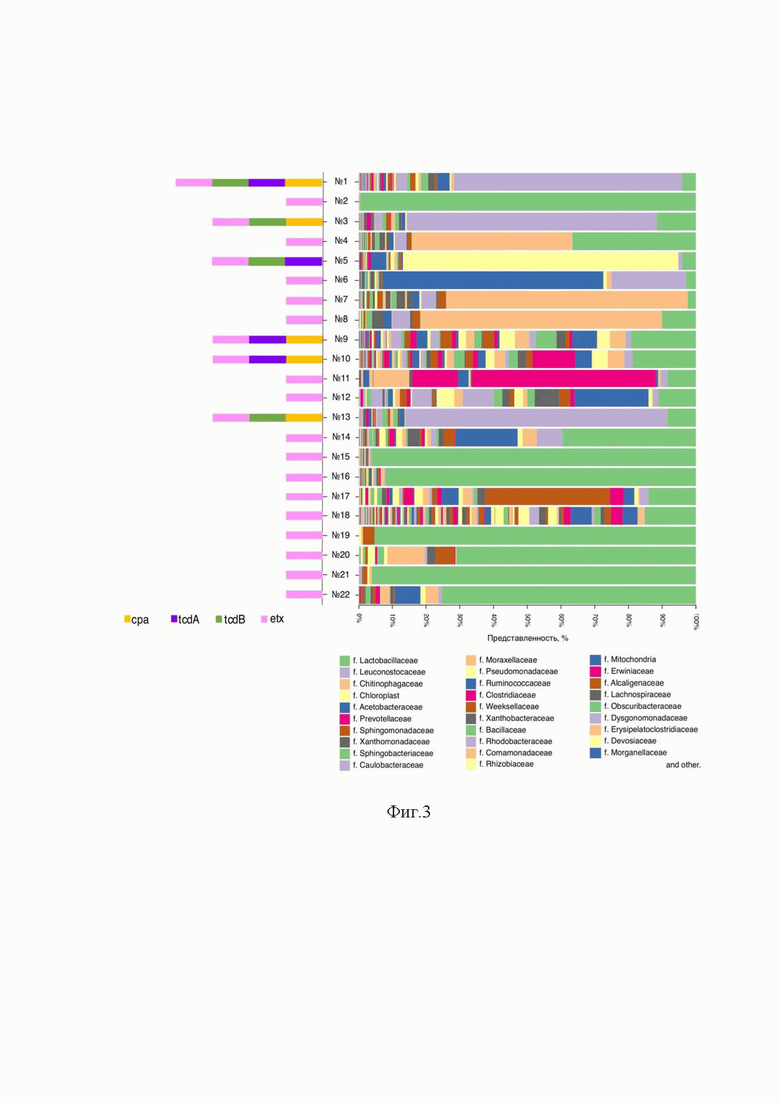
На фиг.2 изображены результаты анализа генов токсинов в ДНК штаммов Clostridioides difficile штаммы 1 и 2.
На фиг. 3 изображены гены токсинов, детектированные в кормах крупного рогатого скота и относительное количество бактерий в кормах по результатам NGS-секвенирования гена 16SрРНК, №1-22 - образцы кормов.
На фиг. 4 представлены последовательности специфических праймеров
На первом этапе выполняется выделение ДНК из образцов консервируемых объемистых кормов таких как силос, сенаж, силаж и пр. Предварительная подготовка образцов для выделения ДНК заключается в отборе их в объеме 50-100 г в стерильные пластиковые пробирки и заморозке при -20°C. Транспортировка и хранение образцов для последующего выделения ДНК осуществляются в замороженном виде.
Для выделения тотальной ДНК из образцов используется метод детергентно-опосредованного осаждения ДНК с помощью лизирующего раствора 1,2 М хлорида натрия и экстракции хлороформом.
Присутствие в образцах тотальной ДНК фрагментов генов токсинов Clostridium perfringens и Clostridioides difficile оценивается методом полимеразной цепной реакции с применением набора специфичных праймеров: альфа-токсин cpa, бета-токсин cpb, эпсилон-токсин etx Clostridium perfringens и бинарный токсин cdtB, токсин А tcdA, токсин В tcdB Clostridioides difficile (фиг.4). Режим амплификации: 3 мин при 94°С; 35 циклов - 40 с при 94°С, 60 с при 55°С, 90 с при 72°С, 5 мин при 72°С. Анализ результатов присутствия фрагментов генов токсинов выполняется посредством гель-электрофореза. Детекция результата проводится путем окрашивания бромистым этидием агарозного геля с последующим анализом в УФ трансиллюминатора. Для интерпретации результатов используются значения размеров фрагментов ДНК соответствующих токсинов, представленные на фиг.4. При обнаружении продуктов амплификации указанного размера результат присутствия генов токсинов является положительным, при отсутствии - отрицательным.
Пример 1
Для оценки работоспособности предложенного способа был проведен эксперимент по анализу присутствия генов токсинов в геноме культур штаммов Clostridium perfringens и Clostridioides difficile.
Из культур штаммов выделяли тотальную ДНК и проводили анализ генов токсинов как описано выше. Согласно полученным результатам в образцах культур штамма Clostridium perfringens (фиг.1) были выявлены гены альфа-токсина cpa, бета-токсина cpb, эпсилон-токсина etx, в образцах культур штамма Clostridioides difficile (фиг.2) были выявлены гены бинарного токсина cdtB, токсина А tcdA, токсина В tcdB.
Пример 2
Для оценки работоспособности данного способа был проведен мониторинг присутствия генов токсинов Clostridium perfringens и Clostridioides difficile в образцах консервированных объемистых кормов из предприятий Ленинградской области.
Отобранные образцы n=22 были проверены на присутствие генов альфа-токсина cpa, бета-токсина cpb и эпсилон-токсина etx Clostridium perfringens; бинарного токсина cdtB, токсина А tcdA и В tcdB Clostridioides difficile.
Параллельно проводилась сравнительная оценка микробного профиля консервированных объёмистых кормов с использованием фрагмента гена 16SрРНК с применением метода высокопроизводительного секвенирования.
Согласно полученным результатам (фиг.3) в одном образце корма №1 было найдено 4 гена токсичности клостридий, в том числе 2 гена токсичности бактерии Clostridium perfringens - альфа-токсин cpa и эпсилон-токсин etx и 2 гена токсичности Clostridioides difficile - токсин А tcdA и токсин В tcdB.
В 5 образцах корма № 3, №5, №9, №10 и №13 выявлено по три токсина.
В частности, в образцах №3 и 13 обнаружены 2 гена токсичности бактерии Clostridium perfringens - альфа-токсин cpa и эпсилон-токсин etx и ген токсичности Clostridioides difficile - токсин В tcdB.
В образцах №9 и №10 обнаружены альфа-токсин cpa бактерии Clostridium perfringens и 2 гена токсичности Clostridioides difficile - токсин А tcdA и токсин В tcdB.
В образце №5 обнаружены эпсилон-токсин etx бактерии Clostridium perfringens и 2 гена токсичности Clostridioides difficile - токсин А tcdA и токсин В tcdB.
В остальных 16 образцах обнаружен эпсилон-токсин etx бактерии Clostridium perfringens.
Итак, в 23% случаев, исследованных нами образцов, был выявлен альфа-токсин cpa Clostridium perfringens. Данный токсин является ферментом - металлофосфолипазой цинка, является основным фактором патогенности газовой гангрены. Ген cpa, кодирующий фермент - высококонсервативнй и расположен в хромосомах. Токсин является дермонекротичным и гемолитичным. Из 23% проанализированных образцов корма альфа-токсин, большая часть №1, №3 и №13 имела низкие показателями качества.
Бета-токсин, кодируемый геном cpb, относится ко второму основному токсину C. perfringens. Токсин является порообразующим цитолизином, вызывает у взрослого крупного рогатого скота некротический энтерита и энтеротоксемии у телят. Бета-токсин в исследованных образцах нами не был выявлен.
Эпсилон-токсин C. perfringens кодирует ген etx, находящийся на большой плазмиде. Токсин относится к наиболее сильнодействующим бактериальным токсинам, является дермонекротическим и летальным. У крупного рогатого скота токсин может способствовать развитию системных нарушений и энтеротоксемии. Под влиянием эпсилон-токсина C. perfringens происходит повреждение слизистого слоя кишечника. Это вызывает распространение токсина по организму, поражение сосудов, головного мозга, сердца, легких. В исследованных образцах эпсилон-токсин выявлялся в 100% случаев.
Токсины Clostridioides difficile вызывают поражение организма различных жвачных животных у взрослого скота, телят, ягнят и др., а также других животных, включая поросят, лошадей. Поражения, вызванные данным микроорганизмом млекопитающих сходны с проявлениями у человека. Известно, что штаммы бактерии Clostridioides difficile способны вырабатывать 3 вида токсинов: токсин А tcdA - энтеротоксин, токсин B tcdB - цитотоксин и бинарный токсин cdtB. При исследовании образцов кормов выявлен высокий процент контаминации кормов генами токсина А tcdA - 18% кормов и генами токсина В tcdB - 18% кормов. При этом гены cdtB бинарного токсина C. difficile в образцах нами не были обнаружены.
Согласно результатам NGS-секвенирования гена 16S рРНК бактерий (фиг.3), в 73% кормов свыше 30% микроорганизмов было представлено филой Firmicutes, включающей лактобактерии. В остальных 30% случаях в кормах доминирующими были бактерии филы Proteobacteria с процентным содержанием более 30%.
Более глубокий анализ микрофлоры кормов - на уровне семейства позволил установить, что бактерии семейства Lactobacillaceae преобладали в 32% образцов кормов, где их содержание было 35-99%. Во всех образцах наиболее представленными были бактерии рода Lactobacillus. Содержание в некоторых кормах другого семейства лактобактерий Leuconostocaceae преимущественно рода Weissella составляло 67,6% - 77,6%.
В остальных кормах преобладания лактобактерий в микробном сообществе не наблюдалось. В 16% кормов преобладали бактерии семейства Chitinophagaceae, в остальных кормах - семейства Sphingomonadaceae 37,0%, семейства Prevotellaceae 54,6%.
Согласно полученным результатам, в образцах кормов, где в микробном сообществе преобладали лактобактерии семейства Lactobacillaceae, было выявлено наименьшее количество генов токсичности Clostridium perfringens и Clostridioides difficile, а также лучшими по качеству. При отсутствии преобладания в микробном сообществе кормов лактобактерий семейства Lactobacillaceae было выявлено 3-4 гена токсичности клостридий Clostridium perfringens и Clostridioides difficile.
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ оценки генов токсинов
Clostridium perfringens и Clostridioides difficile в консервированных объемистых
кормах для крупного рогатого скота.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2025-04-17">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024135535</ApplicationNumberText>
<FilingDate/>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное бюджетное
образовательное учреждение высшего образования «Санкт-Петербургский
государственный аграрный университет» (ФГБОУ ВО СПбГАУ)</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Educational Institution of Higher
Education "Saint Petersburg State Agrarian University" (FSBEI VO
SPbSAU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ оценки генов токсинов Clostridium
perfringens и Clostridioides difficile в консервированных объемистых кормах для
крупного рогатого скота</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctaatgttactgccgttgacctctgatacatcgtgtaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcctttcttgagggaggataaatgaacctcctattttgtcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgggaacttcgatacaagcattaactcatctcccataactgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridioides difficile</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatgataaggcaacttcagtggtagttcctcctgctccatcaaatg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridioides difficile</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaaartggagtgttacaaacaggtggcatttctccattctcagcaaagta</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridioides difficile</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgacccaaagttgatgtctgattgcggatctcttgcttcagtctttatag</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ДЕТЕКТИРОВАНИЯ И ХАРАКТЕРИЗАЦИИ ТОКСИНОГЕННОГО ШТАММА CLOSTRIDIUM DIFFICILE | 2010 |
|
RU2612789C2 |
| Способ получения аминокислотных последовательностей, кодирующих наноантитела, нейтрализующие токсин А Clostridioides difficile | 2023 |
|
RU2833930C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ЗАБОЛЕВАНИЙ, ВЫЗЫВАЕМЫХ C. PERFRINGENS, У ЖИВОТНЫХ | 2018 |
|
RU2769503C2 |
| СПОСОБ АМПЛИФИКАЦИИ ДНК НА ОСНОВЕ ВНЕДРЕНИЯ В ЦЕПЬ | 2014 |
|
RU2673733C2 |
| Композиции и способы, имеющие отношение к мутантному токсину из Clostridium Difficile | 2013 |
|
RU2630671C2 |
| Способ выявления клостридий видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii | 2023 |
|
RU2826158C1 |
| Способ оценки патогенной микробиоты ЖКТ сельскохозяйственных животных, включая такие виды, как Clostridium perfringens, C. difficile, C. ramosum, C. septicum | 2023 |
|
RU2827174C1 |
| КОМПОЗИЦИИ, ОТНОСЯЩИЕСЯ К МУТАНТНОМУ ТОКСИНУ CLOSTRIDIUM DIFFICILE, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2012 |
|
RU2592686C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ИНДУКЦИИ ИММУННОГО ОТВЕТА ПРОТИВ CLOSTRIDIUM DIFFICILE | 2018 |
|
RU2781057C2 |
| РЕКОМБИНАНТНЫЕ АТТЕНУИРОВАННЫЕ МИКРООРГАНИЗМЫ CLOSTRIDIUM И ВАКЦИНА | 2007 |
|
RU2445364C2 |
Изобретение относится к сельскому хозяйству, ветеринарии, в частности к способу оценки присутствия генов токсинов. Предложен способ оценки генов токсинов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота, включая альфа-токсин cpa, бета-токсин cpb, эпсилон-токсин etx Clostridium perfringens и бинарный токсин cdtB, токсин А tcdA, токсин В tcdB Clostridioides difficile. В способе используются праймеры, специфичные к участкам генов исследуемых токсинов, что позволяет проводить избирательную амплификацию искомых фрагментов ДНК при помощи полимеразной цепной реакции непосредственно из тотальной ДНК биологических образцов и детектировать фрагменты генов токсинов путем гель-электрофореза. 4 ил., 1 пр.
Способ оценки генов токсинов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота, предусматривающий использование молекулярно-биологического метода полимеразной цепной реакции с применением набора специфичных праймеров: прямой GCTAATGTTACTGCCGTTGA и обратный CCTCTGATACATCGTGTAAG для альфа-токсина cpa, прямой TCCTTTCTTGAGGGAGGATAAA и обратный TGAACCTCCTATTTTGTATCCCA для бета- токсина cpb, прямой TGGGAACTTCGATACAAGCA и обратный TTAACTCATCTCCCATAACTGCAC для эпсилон-токсина etx Clostridium perfringens и прямой GCATGATAAGGCAACTTCAGTGGT и обратный AGTTCCTCCTGCTCCATCAAATG для бинарного токсина cdtB, прямой CCAAARTGGAGTGTTACAAACAGGTG и обратный GCATTTCTCCATTCTCAGCAAAGTA для токсина А tcdA, прямой TTGACCCAAAGTTGATGTCTGATTG и обратный CGGATCTCTTGCTTCAGTCTTTATAG для токсина В tcdB Clostridioides difficile, с последующей детекцией генов токсинов путем гель-электрофореза.
| Н.А | |||
| Безбородова и др., Разработка наборов для идентификации бактерий рода Clostridium и токсинотипов Cl | |||
| Perfringens, Siberian Journal of Life Sciences and Agriculture, Vol | |||
| Устройство для электрической сигнализации | 1918 |
|
SU16A1 |
| Оссипранди М | |||
| К | |||
| и др., Молекулярное ПЦР-типирование токсинов изолятов Clostridium perfringens и Clostridium difficile, полученных от крупного | |||
Авторы
Даты
2025-06-06—Публикация
2024-11-27—Подача