Изобретение относится к ветеринарной биотехнологии, в частности к генетической инженерии, и может быть использовано в ветеринарной практике для диагностики клостридиозов крупного рогатого скота, а также для решения научно-исследовательских задач по изучению эпизоотологии клостридиозов, оценке эффективности профилактических и лечебных мероприятий.
В последние годы в нашей стране распространение клостридиозов среди крупного рогатого скота обусловлено интенсификацией животноводства, направленной на повышение молочной продуктивности коров, которую часто осуществляют на фоне несбалансированных рационов кормления. Это приводит к нарушению обмена веществ, в частности, к развитию кетозов и ацидозов, нередко - к гибели животных. Клостридии, размножаясь на слизистой оболочке желудочно-кишечного тракта животного, вызывают некроз клеток эпителия, что способствует проникновению в кровяное русло токсинов, продуцируемых ими, а также приводит к развитию тяжелых патологических процессов в организме [Глотова Т.И., Терентьева Т.Е., Глотов А.Г. Возбудители и возрастная восприимчивость крупного рогатого скота. Сибирский вестник сельскохозяйственной науки. 2017; 47(1): 90-96]. В связи с быстротечностью инфекционного процесса, высокой токсигенной активностью возбудителей и обширностью поражения органов и тканей организма, лечение таких животных часто оказывается бесперспективным, поэтому высокопродуктивных особей приходится выбраковывать [Капустин А.В., Моторыгин А.В., Букова Н.К. Видовой состав клостридий крупного рогатого скота. Вестник ветеринарии. 2013; 1(64): 71-73].
Клостридии - анаэробные грамположительные бактерии, образующие споры, рода Clostridium, семейства Clostridiaceae, порядка Clostridials, класса Clostridia, надкласса Clostridium, типа Fusobacteria, подцарства Bacteria широко распространены в почве, воде, продуктах разложения (гниения) белковых веществ. Род Clostridium насчитывает более 100 видов, в большинстве своем являющихся представителями нормальной микрофлоры желудочно-кишечного тракта крупного рогатого скота. Однако некоторые из них патогенные и продуцируют экзотоксины, действующие местно или системно. Болезни, вызываемые токсигенными клостридиями, условно подразделяют на три основных типа: нейротоксические (ботулизм, столбняк), гистотоксические (миозит, или «черная нога», "black leg"», газовая гангрена, злокачественный отек, бациллярная гемоглобинурия, инфекционный некротический гепатит) и кишечные (заболевания, вызываемые бактерией С.perfringens типов А, В, С, D и Е, некротические энтериты, диареи, вызываемые бактерией С.difficile, абомазит и др.) [Silva R.O.S., Uzal F.A., Oliveira Jr. С.A., Lobato F.C.F. Clostridial Diseases of Animals. John Wiley & Sons, Ltd.; Hoboken N.J., USA. Gangrene Gas (Malignant Edema). 2016; 243-254].
Наибольший ущерб животноводству причиняют гистотоксические клостридии, к которым относятся С.chauvoei (эмфизематозный карбункул), С novyi (С.oedematiens) тип А, С.septicum, С.sordellii (злокачественный отек), С.haemolyticum (С.novyi тип D) (бациллярная гемоглобинурия), С.perfringens тип А (злокачественный отек, газовая гангрена, некротизирующие энтериты, метриты, мастит), С septicum, С.oedematiens (С.novyi), С.histolyticum (злокачественный отек, брадзотоподобные инфекции) [Капустин А.В., Лаишевцев А.И., Скляров О.Д., Абросимова Н.С. Разработка метода контроля иммуногенной активности ассоциированной вакцины против клостридиозов крупного рогатого скота. Russian Journal of Agricultural and Socio-Economic Sciences. 2017; 63:170-175].
Помимо описанных выше видов клостридий, от крупного рогатого скота выделяют и другие, многие из которых при благоприятных условиях также могут синтезировать экзотоксины и вызывать соответствующую патологию.
При исследовании продуктов убоя клинически здоровых животных наиболее часто идентифицируют: С.sporogenes (13%), С.cadaveris (12,5%), С. cochlearium (12%) и С.perfringens (10%), С.sordelli (8%) [Bakhtiary F., Sayevand H.R., Remely M, Hippe В., Indra A., Hosseini H., Haslberger A.G. Identification of Clostridium spp.derived from a sheep and cattle slaughterhouse by matrix-assisted laser desorption and ionization-time of flight mass spectrometry (MALDI-TOF MS) and 16S rDNA sequencing. J Food Sci Technol. 2018; 55(8):3232-3240]. Из них С.sporogenes, С.cadaveris, С.cochlearium относятся к нормальной микрофлоре кишечника крупного рогатого скота. Однако эти виды клостридий способны в некоторых случаях вызывать развитие инфекции или усиливать патогенность других клостридий, а С.perfringens, С.sporogenes и С.sordelli являются признанными патогенами крупного рогатого скота.
Следовательно, для диагностики клостридиозов крупного рогатого скота необходимо проводить видовую идентификацию выделенных бактериальных культур клостридий.
В настоящее время в России основным методом видовой идентификации выделенных бактериальных культур клостридий является определение их фенотипических и биохимических свойств [Bergey's Manual of Systematic Bacteriology. Volume 3: The Firmicutes. 2nd Ed. New York: Springer-Verlag; 2009].
К недостаткам этого способа относится длительность его выполнения (не менее 14 суток), а также сходство фенотипических и биохимических свойств некоторых видов клостридий. Следовательно, для видовой идентификации клостридий необходимо проводить дополнительные тесты, что усложняет диагностику, увеличивает сроки ее проведения, и недоступно для большинства ветеринарных лабораторий.
Известен способ идентификации культур клостридий при помощи времяпролетной масс-спектрометрии с матрично-активированной лазерной десорбцией/ионизацией (MALDI-TOF MS). Этот метод идентификации микроорганизмов доступен для некоторых диагностических лабораторий [Chean R., Kotsanas D., Francis M.J., Palombo E.A., Jadhav S.R., Awad M.M., Lyras D., Korman T.M., Jenkin G.A. Comparing the identification of Clostridium spp.by two Matrix-Assisted Laser Desorption Ionization-Time of Flight (MALDI-TOF) mass spectrometry platforms to 16S rRNA PCR sequencing as a reference standard: a detailed analysis of age of culture and sample preparation. Anaerobe. 2014; 30:85-89. doi: 10.1016/j.anaerobe.2014.09.007]. К недостаткам этого метода относится то, что он не способен идентифицировать все виды клостридий, его диагностическая эффективность зависит во многом от возраста бактериальных культур. Чувствительность метода ограничивается отсутствием видоспецифичных спектральных данных для всех видов клостридий в существующих базах данных. Поэтому его результаты должны дополняться идентификацией бактерий с помощью секвенирования гена 16S рРНК.
Близкими аналогами являются способы, основанные на выявлении фрагментов генов отдельных видов клостридий методом полимеразной цепной реакции в электрофорезном или в формате реального времени. Существуют коммерческие наборы «Hi-PCR® Clostridium sporogenes Semi-Q PCR Kit» (HiMedia Laboratories Private Limited, Индия), «AccuPower® Clostridium perfringens PCR Kit» (Bioneer, Южная Корея), «Clostridium sordellii detection kits» (Bioingentech, Чили) [Оссипранди M.K., Зербини Л., Болдырева Е. Молекулярное ПЦР-типирование токсинов изолятов Clostridium perfringens и Clostridium difficile, полученных от крупного рогатого скота. Российский ветеринарный журнал. 2013; 3: 20 - 23. Козлова А.Д., Горбачева Н.С., Климен-кова О.В., Лаишевцев А.И., Капустин А.В., Яцентюк СП. Использование молекулярно-генетических методов для типирования Clostridium perfringens. RJOAS. 2017; 3: 188-194. Ahsani M.R., Mohammadabadi M.R., Shamsaddini M.B. Clostridium perfringens isolate typing by multiplex PCR. J. Venom. Anim. Toxins incl. Trop.Dis. 2010; 16 (4): 573-578. Bhatnagar J., Deleon-Carnes M., Kellar K.L., Bandyopadhyay K., Antoniadou Z.A., Shieh W.J., Paddock CD., Zaki S.R. Rapid, simultaneous detection of Clostridium sordellii and Clostridium perfringens in archived tissues by a novel PCR-based microsphere assay: diagnostic implications for pregnancy-associated toxic shock syndrome cases. Infect Dis Obstet Gynecol. 2012; 2012: 972845]. Данные способы могут выявлять только один или два вида бактерий.
Наиболее приемлемым, с точки зрения описанных недостатков и принятым за прототип, является способ, основанный на мультиплексной полимеразной цепной реакции в реальном времени для одновременного обнаружения Clostridium beijerinckii, Clostridium sporogenes и Clostridium tyrobutyricum в пробах молока [Morandi S., Cremonesi P., Silvetti Т., Castiglioni В., Brasca M. Development of a triplex real-time PCR assay for the simultaneous detection of Clostridium beijerinckii, Clostridium sporogenes and Clostridium tyrobutyricum in milk. Anaerobe. 2015; 34: 44-49].
Способ используется в качестве быстрого альтернативного метода выявления клостридий в пробах сырого молока с пределом чувствительности от 2 КОЕ/50 мл для С.sporogenes до 300 КОЕ/50 мл для С beijerinckii.
Задачей изобретения является разработка высокоспецифичного и чувствительного способа на основе мультиплексной полимеразной цепной реакции (ПЦР) в режиме реального времени, позволяющего одновременно выявлять клостридии видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii в пробах патологического материала от больных животных, а также в смешанных и чистых бактериальных культурах.
Поставленная задача решается тем, что в способе выявления клостридий видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii, включающем предварительное обогащение на селективных питательных средах и проведение мультиплексной полимеразной цепной реакции в режиме реального времени, согласно изобретения, выявление вида Clostridium sporogenes осуществляют путем амплификации участка гена gerKA с помощью праймеров и зонда SEQ ID NO:l-SEQ ID NO:3, выявление вида Clostridium perfringens осуществляют путем амплификации участка гена pic с помощью праймеров и зонда SEQ ID NO:4-SEQ ID N0:6, выявление вида Clostridium sordellii осуществляют путем амплификации участка гена NanS с помощью праймеров и зонда SEQ ID NO:7-SEQ ID NO:9.
Изобретение иллюстрируется следующими примерами.
Пример 1. Получение синтетических олигонуклеотидных праймеров
На начальном этапе был проведен анализ нуклеотидных последовательностей геномов бактерий Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii из базы данных NCBI (http://www.ncbi.nlm.nih.gov/) и определены наиболее консервативные участки геномов. Анализ публикаций в сети интернет позволил выбрать гены, представленные у всех исследованным штаммов и изолятов соответствующего вида клостридий.
Анализ свойств олигонуклеотидных праймеров и зондов проводился с использованием программы Vector NTI 9.0.0 (InforMax).
Для идентификации генетического материала Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii методом ПЦР в реальном времени были подобраны праймеры и зонды, представленные в таблице 1.

Пример 2. Выявление бактерий Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii при помощи специфических олигонуклеотидных праймеров в пробах патологического материала и культурах бактерий.
Способ осуществляется в несколько этапов.
Этап 1. Подготовка проб биологического материала и бактериальных культур для исследования
Для исследования от павших или вынужденно убитых животных с признаками сепсиса отбирают пробы внутренних органов (мышечная ткань, брыжеечные лимфатические узлы, печень и др.) не позднее двух часов после гибели животного.
Пробы замораживают однократно и транспортируют в лабораторию в течение не более 4…12 ч в термосе со льдом, чтобы избежать повторного оттаивания и замораживания.
Пробы органов животных помещают в фарфоровые ступки, растирают и ресуспензируют в 70% этиловом спирте в соотношении 1/5-1/10, затем центрифугируют и осадок переносят в пробирку с 5 мл тиогликолевой среды, культивируют в анаэробных условиях в течение 7-10 дней.
Через 7-10 дней инкубации в анаэробных условиях при температуре 37°С пробирки просматривают, отбирают те, в которых наблюдается изменение или помутнение среды, их прогревают в течение 10 минут при 80°С и проводят посев содержимого на мясопептонный агар с добавлением 5% дефибрированной крови барана и инкубируют в течение 24-48 часов при температуре 37°С в атмосфере 5% CO2.
Через 24-48 часов инкубации просматривают чашки Петри с агаром на наличие типичных колоний (диаметром до 3 мм, округлых с ровными или изрезанными краями, слизистой консистенции, серого цвета, с наличием зоны В-гемолиза и свечением в ультрафиолете). Из этих колоний делают мазки на предметном стекле и окрашивают их по Граму для предварительной видовой идентификации. При обнаружении в мазке положительных по Граму палочек со спорами, расположенными центрально или субтерминально, колонию отбирают для исследования в ПЦР.
Для этого отдельные колонии, не смешивая, снимают с поверхности агара, переносят в отдельные стерильные стеклянные пробирки, содержащие 200 мкл стерильной дистиллированной воды, перемешивают на вортексе. Взвесь бактерий разводят стерильной дистиллированной водой до концентрации 109 КОЕ/мл и используют для выделения ДНК.
Этап 2. Выделение ДНК из бактериальной суспензии
Выделение ДНК проводят любым доступным способом, включающим лизис клеток, депротеинизацию лизата, осаждение ДНК из раствора, отмывку спиртами, высушивание и растворение ДНК в буферном растворе.
Этап 3. Постановка мультиплексной ПЦР для выявления и генотипирования бактерий Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii
Полимеразная цепная реакция. Состав реакционной смеси: ПЦР-буфер (60 mM Tris-HCl [рН 8.5], 1,5 мМ MgCl2, 25 mM KC1, 10 мМ 2-меркаптэтанола, 0,1% Тритон Х-100), 0,2 мМ dNTP, по 0,2 мкг каждого праймера и зонда, 1.25 е.а. Taq-ДНК-полимеразы, 5 мкл выделенной ДНК.
Температурный режим проведения ПЦР: 95°С - 5 мин - 1 цикл; 95°С-15 сек., 60°С - 60 сек. - 45 циклов.
Измерение флуоресценции осуществляют при температуре 60°С на всех каналах.
Этап 4. Анализ результатов полимеразной цепной реакции Анализ результатов поводят с помощью программного обеспечения прибора, используемого для проведения ПЦР с детекцией в режиме реального времени. Анализируют кривые накопления флуоресцентного сигнала по трем каналам FAM, JOE/HEX и ROX/Texas Red. Устанавливают уровень пороговой линии выше шумовых сигналов и так чтобы отсечь все кривые с не экспоненциальным подъемом и рассчитывают значение порогового цикла (cycle threshold, Ct) для каждой пробы.
Учет результатов проводят в соответствии с таблицей 2. Положительными считают образцы со значением Ct, не превышающим 35. Сомнительными считают образцы со значением Ct от 35 до 40. Результат исследования образца считают отрицательным на исследуемые бактерии, если на канале значение Ct отсутствует или превышает 40.
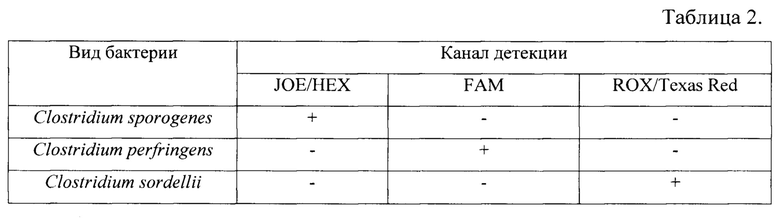
Таким образом, предлагаемый способ прост в исполнении и позволяет эффективно выявлять бактерии видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii.
Пример 3. Определение диагностической чувствительности реакции по заявленному способу
Для определения диагностической чувствительности реакции по заявленному способу суточные бактериальные культуры изолятов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii, выделенных ранее и идентифицированных с помощью «Набора для идентификации анаэробных бактерий АНАЭРОтест 23», производства Erba Mannheim (Чехия), выращивают на мясопептонном агаре с добавлением 5% дефибрированной крови барана при температуре 37°С в атмосфере 5% CO2 в течение 24 часов, переносят в бактериологическую пробирку, содержащую 1000 мкл стерильной дистиллированной воды или физиологического раствора, разводят до концентрации 1011 КОЕ/мл, титруют методом 10-кратных разведений до разведения 101 КОЕ/мл.
Результаты определения диагностической чувствительности разработанной способу представлены в таблице 3.

Таким образом, предлагаемый способ обладает высокой диагностической чувствительностью, позволяет с одинаковой эффективностью выявлять и генотипировать изоляты бактерий Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii.
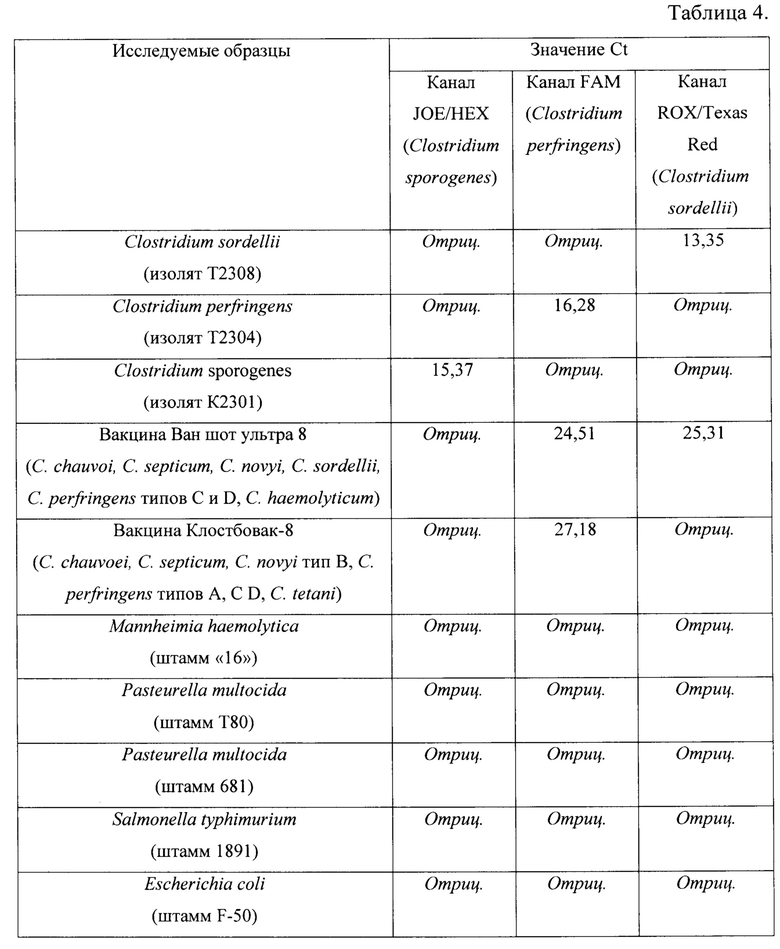
Пример 4. Определение специфичности и эффективности реакции по заявленному способу.
Для определения специфичности и эффективности реакции исследуют изоляты клостридий, идентифицированные до вида с помощью биохимических методов, вакцины против клостридиоза животных, контрольные штаммы бактерий Mannheimia haemolytica, Escherichia coli, Pasteurella multocida, выделенные от больных животных, а также пробы внутренних органов крупного рогатого скота с симптомами клостридиозов крупного рогатого скота.
Результаты опытов по определению специфичности реакции представлены в таблице 4.
Результаты опытов по определению эффективности исследования проб биологического материала, полученного от больных животных, по заявленному способу представлены в таблице 5.

Таким образом, предлагаемый способ обладает высокой специфичностью и эффективностью при выявлении и генотипировании бактерий Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii в пробах биологического материала, полученного от больных животных и культурах бактерий.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ выявления
клостридий видов Clostridium sporogenes, Clostridium perfringens и
Clostridium sordellii.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-06-01">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate>2023-06-01</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-06-01</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Сибирский федеральный научный центр
агробиотехнологий Российской академии наук (СФНЦА
РАН)</ApplicantName>
<ApplicantNameLatin>Federal'noe gosudarstvennoe byudzhetnoe
uchrezhdenie nauki Sibirskij federal'nyj nauchnyj centr
agrobiotekhnologij Rossijskoj akademii nauk (SFNCA
RAN)</ApplicantNameLatin>
<InventorName languageCode="ru">Глотов Александр
Гаврилович</InventorName>
<InventorNameLatin>Glotov Aleksandr Gavrilovich</InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления клостридий видов
Clostridium sporogenes, Clostridium perfringens и Clostridium
sordellii</InventionTitle>
<SequenceTotalQuantity>9</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sporogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagtgtggtgggtggtattat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sporogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccactgtagaaacacctact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sporogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taggtgatgcagccataagggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctagatatgaatggcaaagagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcagtaacattagcagga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
perfringens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctacattctatcttggagaggctatgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sordellii</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcagactttggcagatggtactatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sordellii</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtcccatgtttgtccattatcagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Clostridium
sordellii</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggagcaggcgatcatgcatacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ оценки генов токсинов Clostridium perfringens и Clostridioides difficile в консервированных объемистых кормах для крупного рогатого скота | 2024 |
|
RU2841390C1 |
| Способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР) | 2022 |
|
RU2798286C1 |
| Способ выявления ДНК микроорганизмов класса Mollicutes в крови и других биоматериалах методом ПЦР-РВ | 2023 |
|
RU2831277C1 |
| НАБОР ВЫСОКОСПЕЦИФИЧНЫХ ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ЗОНДОВ ДЛЯ ДЕТЕКЦИИ И ДИФФЕРЕНЦИАЦИИ ВИРУСОВ АЧС, КЧС И ВД | 2024 |
|
RU2828887C1 |
| Набор праймеров для выявления возбудителей бактериальной пневмонии человека методом мультиплексной рекомбиназной полимеразной амплификации | 2023 |
|
RU2813995C1 |
| Способ определения генотипа ротавирусов первой и второй геногрупп и реассортантов между ними методом мультиплексной ПЦР | 2024 |
|
RU2833073C1 |
| Тест-система и способ обнаружения специфических фрагментов нуклеиновых кислот 16 патогенов с использованием изотермической реакции амплификации | 2023 |
|
RU2810751C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
Изобретение относится к области биотехнологии. Описан способ выявления клостридий видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii, включающий предварительное обогащение на селективных питательных средах и проведение мультиплексной полимеразной цепной реакции в режиме реального времени. Способ отличается тем, что выявление вида Clostridium sporogenes осуществляют путем амплификации участка гена gerKA с помощью разработанных праймеров и зонда, выявление вида Clostridium perfringens осуществляют путем амплификации участка гена plc с помощью разработанных праймеров и зонда, выявление вида Clostridium sordellii осуществляют путем амплификации участка гена NanS с помощью разработанных праймеров и зонда. Технический результат заключается в разработке высокоспецифичного и чувствительного способа на основе мультиплексной полимеразной цепной реакции (ПЦР) в режиме реального времени, позволяющего одновременно выявлять клостридии видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii в пробах патологического материала от больных животных, а также в смешанных и чистых бактериальных культурах. 5 табл., 4 пр.
Способ выявления клостридий видов Clostridium sporogenes, Clostridium perfringens и Clostridium sordellii, включающий предварительное обогащение на селективных питательных средах и проведение мультиплексной полимеразной цепной реакции в режиме реального времени, отличающийся тем, что выявление вида Clostridium sporogenes осуществляют путем амплификации участка гена gerKA с помощью праймеров и зонда SEQ ID NO: 1-3, выявление вида Clostridium perfringens осуществляют путем амплификации участка гена plc с помощью праймеров и зонда SEQ ID NO: 4-6, выявление вида Clostridium sordellii осуществляют путем амплификации участка гена NanS с помощью праймеров и зонда SEQ ID NO: 7-9.
| WO 2016097491 A1, 23.06.2016 | |||
| Фундамент здания,сооружения | 1981 |
|
SU1036846A1 |
| СПОСОБ ВЫЯВЛЕНИЯ ЗАБОЛЕВАНИЙ, ВЫЗЫВАЕМЫХ C. PERFRINGENS, У ЖИВОТНЫХ | 2018 |
|
RU2769503C2 |
| Morandi S., Cremonesi P., Silvetti Т., Castiglioni В., Brasca M | |||
| Development of a triplex real-time PCR assay for the simultaneous detection of Clostridium beijerinckii, Clostridium sporogenes and Clostridium tyrobutyricum in milk | |||
| Anaerobe | |||
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
Авторы
Даты
2024-09-04—Публикация
2023-07-14—Подача