Изобретение касается способа идентификации видов неизвестного организма, предусматривающего выделение ДНК этого организма, обработку ДНК рестриктазой, гибридизацию на фильтрах фрагментов ДНК с меченым нуклеотидным зондом и детекцию гибридных последовательностей с последующим анализом ее результатов.
Известен молекулярный метод идентификации организмов (например, бактерий), заключающийся в сравнении двух геномов путем реассоциации ДНК с ДНК. Генетическое определение вида предполагает, что родство штаммов данного вида составляет 70% и более. Идентифицировать штамм путем реассоциации ДНК с ДНК удается только тогда, когда радиоактивно меченая проба ДНК и неизвестная ДНК происходят от того же самого вида. Однако практическое применение определения вида на основе родства штаммов в 70% ограничивается выбором соответствующей пробы. Это затруднение отчасти можно устранить путем выбора фенотипических признаков, которые, видимо, коррелируют с реассоциируемой группой. Однако когда они используются отдельно, определение вида на основе реассоциации ДНК с ДНК также применяется косвенно.
Бреннер в учебнике клинической микробиологии указывает, что идеальным средством для идентификации видов бактерий был бы "черный ящик", способный к разделению генов и немедленному сравнению нуклеиново-кислотных последовательностей в данном штамме с образцом-эталоном для каждого известного вида, что до какой-то степени напоминает о методе масс-спектрофотометрического анализа.
Однако Бреннер допускает, что хотя анализ с помощью рестрикционной эндонуклеазы можно осуществить для определения обычных последовательностей на изолированных генах, пока еще нет в нашем распоряжении пригодного черного ящика, в частности ящика, пригодного для применения в клинических лабораториях. Это равным образом можно отнести к любому виду организмов.
Наиболее близким к предлагаемому можно признать способ картирования транскриптов, который в принципе может быть использован для идентификации организмов, предусматривающий картирование транскриптов путем двухстадийного процесса гибридизации. ДНК получают из двух вирусов: аденовируса и вируса SV40. ДНК отдельно расщепляют рестрикционной эндонуклеазой, отделяют электрофорезом и переносят в нитроцеллюлозу. Потом применяют одну из двух разных пробных систем в зависимости от картируемого для локации транскрибированных последовательностей вирусного генома (см. фиг. 1). Обе пробы начинают с РНК, экстрагированной из клеток, зараженных гибридом аденовируса SV-40. Эти гибриды являются внутримолекулярными рекомбинантными ДНК, вводящими разные количества ДНК SV40 в ДНК аденовируса, которая подвергалась делециям различных степеней (Virology, page 465, B. N. Fields Raven Press, New York, 1985). Это значит, что гибрид РНК содержит аденовирусные последовательности в одно целое с последовательностями SV 40. Если гибрид РНК гибридизирован в аденовирусной ДНК на мембране, то участком гомологии является аденовирусная часть. Таким образом, часть SV40 остается свободной для гибридизации с меченой пробой ДНК SV40 (фиг. 2).
Если иммобилизированная ДНК представляет собой SV40, то, конечно, часть гибрида РНК, остающаяся свободной после гибридизации, представляет собой аденовирусную последовательность, детектированную с помощью меченой пробы аденовирусной ДНК (фиг. 3, A novel method to map transcripts. Evidence Homology Between an Adenovirus mPNa and Discrete Multiply Region of Viral Genome Cellulosa, v. 12, pp. 23-38, 1977).
Следует заметить, что прототип не предназначался для идентификации различных организмов. Отчасти по этой причине, известный способ недостаточно универсален и не позволяет максимально упростить и ускорить процесс идентификации.
Целью изобретения является унификация способа идентификации организма и его упрощение.
Эта цель достигается тем, что в способе при идентификации прокариотического организма в качестве зонда используют 5S, 16S или 23 S pРНК, а при идентификации эукариотического организма - 18S или 28S pРНК.
Изобретение представляет объективный способ определения организмов на основе сохранившихся последовательностей ДНК или другого генетического материала относительно известных местоположений, например сайтов рестрикционной эндонуклеазы. Обнаружение рестрикционных фрагментов, содержащих сохранившуюся последовательность, осуществляется путем гибридизации или реассоциации сегментов ДНК с нуклеиновой кислотой, содержащей информацию сохранившейся последовательности из пробы организма. Под выражением "организм", который может охарактеризовать (причем последнее выражение включает и значение "идентифицировать") с помощью предлагаемого способа, подразумевается фактически любой организм, содержащий в своем геноме ДНК или РНК. В этом отношении оказывается пригодным ссылаться на классическую таксономическую схему.
В настоящем изобретении считаются включенными все организмы, относящиеся к царствам Monera, Plantae и Animalia.
Исследуемый организм выделяют, получают известными методами его ДНК и затем полученную ДНК в определенных сайтах с помощью рестрикционных эндонуклеаз разрезают на отдельные фрагменты. Последние разделяют по размеру с помощью хроматографической системы. В качестве примера пригодной хроматографической системы можно привести гель-хроматографию. Кроме того, можно использовать и другие системы, например высоконапорную жидкостную хроматографию, электрофорез на основе зон капиллярности или другие приемы разделения. В случае использования гель-хроматографии отдельные фрагменты разделяют и гели окрашивают известными приемами, после чего их стандартизируют в соответствии с размерами фрагментов с использованием кривых стандартизации, построенных из фрагментов с известными размерами. Затем разделенные фрагменты можно перевести на бумагу из нитрованной целлюлозы с использованием метода пятна (Southern blot technique) (см. Southern, E. M. , Journal of Molecular Biology, 38: 503-517, 1975), после чего их связывают с бумагой с образованием ковалентной связи путем нагревания. Далее фрагменты, содержащие сохранившиеся последовательности, располагают по их способности к гибридизации с нуклеиново-кислотным зондом, содержащим информацию сохранившейся последовательности. Гибридизацию можно также осуществить после переваривания, но до разделения или же рестрикционное расщепление осуществляют после гибридизации с последующим разделением фрагментов.
В качестве нуклеиново-кислотного зонда используют рибосольную РНК (pРНК). Прокариотическая pРНК обычно содержит три подвида: так называемые фрагменты 5S, 16S и 23S. Эукариотическая p-РНК обычно содержит два подвида - 18S и 28S.
Следующая стадия в предлагаемом процессе представляет собой как правило, гибридизацию выделенного переваренного продукта ДНК из неизвестного организма с немеченым или (предпочтительно) радиоактивно меченым зондом РНК или ДНК. Гибридизацию осуществляют путем контактирования бумаги, содержащей ковалентно меченый переваренный продукт ДНК из неизвестного организма с гибридизационной смесью, содержащей зонд. Инкубацию осуществляют при повышенных температурах (50-70оС) в течение длительного периода времени, после чего фильтровальные бумаги для удаления несвязанной реактивности (если необходимо) промывают, высушивают на воздухе и отсчитывают для проведения детектирования. Другой весьма предпочтительный прием гибридизации, который осуществляется намного быстрее, чем описанный, представляет собой прием реассоциации с помощью феноловой эмульсии при комнатной температуре (Kohne, D. E. с сотр. , Biochemistry, 16: 5329-5341, 1977).
После гибридизации проводят избирательное детектирование соответственно гибридизированных фрагментов. Осуществляют это детектирование, пользуясь двунитевой структурой гибридизированных фрагментов и используя для этого избирательный метод (в случае немеченой пробы), авторадиографию или пригодный для измерения радиоактивного излучения прибор, необязательно связанный с ЭВМ, с помощью которого можно повысить скорость детектирования (в случае меченой пробы).
Кроме радиоактивного мечения, может использоваться хемилюминесцентная метка (биотик) или же конъюгат фермента и осадка красителя и конъюгат антитела и фермента. Эти приемы хорошо известны в данной области.
Конечным результатом указанных приемов является идентифицирующая генетическая характеристика, например хроматографический рисунок полос, имеющий определенные пики и впадины, или предпочтительно расположенные в определенных местах светлые и темные области разной интенсивности. Эти места легко можно соотнести со специфическими величинами фрагментов (в парах килооснований) с использованием для приема выделения маркера, например переваренной EcoRI ДНК бактериофага λ. Таким образом, легко можно установить и взаимное расположение полос, и абсолютную величину каждой полосы. Затем идентифицирующую генетическую характеристику неизвестного организма сравнивают с характеристиками, содержащимися в каталоге или библиотеке. Каталог или библиотека может представлять собой книгу, содержащую характеристики по крайней мере от двух до практически неограниченного числа явно отличающихся друг от друга родов и видов организмов. Например, ввиду того что известно ок. 100 патологически важных бактерий, вызывающих заболевания у человека, считается, что стандартный каталог патогенных бактерий содержит приблизительно 50-150 таких характеристик. Может быть включен также каталог типов штаммов бактерий для определения групп микроорганизмов для эпидемиологических целей. Характеристики зависят от типа и типов выбранных эндонуклеазных энзимов на конкретном организме, использованном в качестве источника меченого зонда, и от состава информационных нуклеиновых кислот сохранившейся последовательности ДНК, используемых для приготовления зонда (например, содержащих либо подтипы прокариотических pРНК 5S, 16S или 23S, либо всего лишь 16S и 23S, либо согласованные последовательности т. п. ).
Итак, каталог для каждого зонда может содержать ряд специфических относительно энзимов характеристик с указанием величины каждой приведенной полосы и относительной интенсивности. По мере понижения концентрации связи ДНК с фильтром остаются видимыми только самые интенсивные полосы и на основе величины этой полосы или этих полос можно идентифицировать вид. Любую вариацию или перестановку можно, конечно, использовать для библиотеки. Кроме того, в случае эукариотического организма библиотека может еще содержать рисунки, полученные в результате применения одного типа ДНК или любой комбинации ДНК органелл и/или ядра. Рисунок для каждого продукта переваривания ДНК зависит от состава зонда. Каталог может быть расположен так, что в случае наличия в экстрагированном образце и обнаружения с помощью зонда более одного штамма или вида можно интерпретировать получаемую характеристику. Полученную характеристику, т. е. рисунок полос, можно сравнивать визуально или с помощью одномерного цифрового прибора, управляемого на ЭВМ и запрограммированного для распознавания рисунков. Подобные приборы хорошо известны и используются обычно в супермаркетах для считывания штриховых кодов товара. В идеальном случае данные библиотеки или каталога как в виде относительных характеристик множества организмов, так и в виде абсолютных значений молекулярного веса или величины фрагментов занесены в память ЭВМ. Сравнение на основе каталога заключается в том, что неизвестную характеристику сравнивают с одной из характеристик, наличествующих в "библиотеке", с помощью одного или обоих видов хранящейся информации (относительные характеристики и/или абсолютные значения веса или величины). Кроме того, интенсивность каждой полосы при сравнении с эталоном может выявить количество гибридизированной связанной ДНК и служит для оценки степени присутствия какого-то организма, например прокариотического в эукариотическом.
Для дальнейшего доказательства природы и идентификации данного организма пользователь предлагаемого способа может переваривать неизвестный организм другой (второй) эндонуклеазой и сравнивать получаемую характеристику с содержащимися в каталоге характеристиками организма для второй выбранной эндонуклеазы. В целях точной идентификации организма этот процесс много раз можно повторять. Однако в большинстве случаев достаточен один анализ с единственной пробой.
Предлагаемый способ и его варианты можно использовать в бесчисленных применениях. Например, в выращивании растений или животных для точной их идентификации или в клинических и микробиологических лабораториях, для идентификации бактерий, паразитов или грибов, наличествующих в любой среде, в том числе в эукариотических клетках. В последнем случае предлагаемому способу отдается предпочтение перед стандартным микробиологическим анализом, поскольку он не требует микробиологических анализов, а также не нуждается в выделении и культивировании микробов. Культивирование и характеристика in vitro в случае определенных микроорганизмов, например Mycobacterium lepral (возбудителя лепры), либо невозможны, либо эти процессы в случае определенных микроорганизмов, например облигатных внутриклеточных бактерий (например, Rickettsia, Chlamydia и т. д. ), не осуществимы на стандартной среде, либо они крайне опасны (например, B, anthracis - возбудитель антракса).
Предлагаемый способ зависит от выделения нуклеиновой кислоты и с его помощью удается устранить указанные проблемы, поскольку отпадают традиционные процессы выделения и характеристики бактерий. Способ предназначен для идентификации до сих пор еще не описанных традиционным образом микроорганизмов. Кроме того, предлагаемый способ допускает различие отличных штаммов одного вида, что, например, может быть полезным для определения групп бактерий для эпидемиологических целей. Способ можно использовать в лабораториях судебной медицины для точной и однозначной идентификации тканей растений или животных в криминалистических исследованиях. Его могут использовать энтомологи для быстрой идентификации видов насекомых в процессе определения природы заражения полезных культур. Предлагаемый способ в сочетании с идентификацией нижеподвидовых таксонов (например, генов нитрогеназы в корнях растений, Hennecke, H. 291 Nature 354, 1981) можно использовать для исследования и идентификации генотипов отдельных штаммов.
Способ предпочтительно используют для идентификации микроорганизмов, где бы они ни встречались. Установление и идентификация бактериальной ДНК с помощью прокариотической пробы имеет весьма избирательный характер и осуществляется беспрепятственно даже в присутствии ДНК животного, например млекопитающего. В случае использования прокариотической пробы можно выбрать условия, сводящие в минимуму гибридизацию с митохондрической ДНК или обеспечивающие возможность вычитания митохондрических полос из рисунка. Итак, предлагаемый способ можно использовать в клинических лабораториях, местах депонирования бактерий, промышленных ферментационных лабораториях и т. п.
Особый интерес представляет возможность установить наряду с видом и штаммом заражающего микроорганизма наличие в нем любых специфических генетических последовательностей, например обнаружить на R-факторах, представляющих собой переносимые плазмиды, которые передают устойчивость к лекарствам, наличие последовательности устойчивости к антибиотикам.
В поисках возбудителей заразных болезней в конкретных особях возможно непосредственно экстрагировать нуклеиновую кислоту из особи, или сначала в целях повышения числа возбудителей культивировать особи в средах или клетках, или использовать стадию концентрирования, например центрифугирования, или провести все приемы вместе.
Настоящее изобретение создает предпосылки для разработки оборудования, включающего все необходимые для проведения процесса элементы. Подобное оборудование содержит несущее устройство, подразделенное на отсеки и предназначенное для приема в близком соседстве между собой одного или более сосудов, например трубок или ампул. Один из этих сосудов может содержать немеченый или детектируемый меченый зонд нуклеиновой кислоты. Меченый нуклеотидный зонд может быть в лиофилизованном виде или, если необходимо, в подходящем буфере. Один или более сосудов могут содержать один или более эндонуклеазных энзимов для переваривания ДНК неизвестного организма. Эти энзимы могут присутствовать в чистом виде, в виде смеси, в лиофилизованном виде или в подходящих буферах. В идеальном случае энзимы, используемые в оборудовании, представляют собой энзимы, для которых были приготовлены соответствующие каталоги. Однако пользователь, применяющий предлагаемый способ, для проведения эксперимента может приготовить и свой собственный сравнительный стандарт, т. е. если он подозревает, что неизвестный организм относится к определенному роду или виду, он может приготовить идентифицирующие признаки этого известного организма и сравнить их с признаками неизвестного организма. Итак, оборудование может также содержать все элементы, необходимые для проведения этого подпроцесса. Эти элементы могут включить один или более известных организмов, например бактерий, или выделенную ДНК известных организмов. Оборудование вдобавок может еще содержать "каталог". Это в широком смысле слова книжка, книга, брошюра, лента или диск ЭВМ, номер доступа к получению данных ЭВМ и т. п. , содержащие признаки для идентификации множества организмов определенной группы, например видов растений, млекопитающих, микробов, в частности паталогически важных бактерий, видов насекомых и т. п. Пользователю остается только приготовить признаки неизвестного организма и визуально или с помощью ЭВМ сравнить полученные признаки с признаками, приведенными в каталоге. Оборудование может также содержать в одном сосуде пробу РНК для синтеза пробы, в другом сосуде радиоактивно меченый дезоксирибонуклеозидтрифосфат, а в третьем сосуде - затравку. Таким образом, пользователь в состоянии приготовить свою собственную пробу кДНК.
Наконец, оборудование может включить добавочные элементы, необходимые для проведения предлагаемого способа, например буферы, питательные среды, энзимы, пипетки, пластинки, нуклеиновые кислоты, нуклеозидтрифосфаты, фильтровальную бумагу, гели, материалы для переноса зондов, приспособления и принадлежности для проведения детекции метки и т. п. Оно может содержать также зонды последовательностей устойчивости к антибиотикам, вирусов или другие специфические зонды.
М а т е р и а л ы и м е т о д ы
А. Бактериальные культуры.
Экстрация высокомолекулярной ДНК
Центрифугируют бактериальные бульонные культуры, клетки промывают холодным физиологическим раствором. Затем клетки взвешивают в измеряемом (в мл) объеме экстракционного буфера (0,15 М хлорида натрия, 0,1 М ЭДТА, 0,03 М трис pH 8,5), превышающем приблизительно в 10 раз вес (в г) загруженных клеток. Далее добавляют лизозим в количестве 10 мг/мл до достижения конечной концентрации 0,5 мг/мл. Потом взвесь инкубируют при 37оС в течение 30 мин. Разрыв клеток завершается путем добавления 25% SDS до достижения конечной концентрации 2,5% с повышением темпратуры до 60оС в течение 10 мин. После охлаждения в бане водопроводной воды добавляют меркаптоэтанол до достижения конечной концентрации 1% . Проназу (Pronase R) в количестве 20 мг/мл в 0,02 М трис-основания с pH - 7,4 предварительно переваривают при 37оС в течение 2 ч и затем добавляют к смеси до достижения конечной концентрации 1 мг/мл. Раствор инкубируют 18 ч при 37оС. Фенол готовят путем смешения 1 л дважды перегнанного фенола, 2,5 л дважды перегнанной воды, 270 мл насыщенного трис-основания, 12 мл меркаптоэтанола и ЭДТА до достижения конечной концентрации 10-3 М с отделением смеси при 4оС. Фенол промывают промывным буфером (10-1 М хлорида натрия, 10-3 М ЭДТА, 10 мМ трис-основания с pH 8,5). Затем добавляют одинаковый объем свежего буфера, после этого вводят меркаптоэтанол до достижения конечной концентрации 0,1% . Раствор смешивают и хранят при 4оС. Далее к раствору лизированных клеток добавляют полобъема приготовленного фенола и полобъема хлороформа. Потом встряхивают в течение приблизительно 10 мин и центрифугируют при 3400х в течение 15 мин. После этого удаляют водную фазу с помощью перевернутой стеклянной пипетки емкостью 25 мл. Процесс экстракции повторяют до образования незначительного осадка на поверхности раздела. Затем к водной фазе добавляют одну девятую ее обема 2 н. раствора ацетата натрия с значением pH 5,5. Затем вниз по боковой стенке колбы наливают двухкратный объем 95% -го этанола при -20оС, гладко запаивают пипетку Пастера и используют ее для наматывания осажденной ДНК. Высокомолекулярную ДНК растворяют в буфере (10-3 ЭДТА, 10-2 М трис-основания с pH 7,4). Концентрацию ДНК определяют за счет поглощения при 260 нм с использованием 30 мкг на единицу поглощения в качестве коэффициента пропорциональности.
Переваривание ДНК рестрикционной эндонуклеазой
Реакции с рестрикционной эндонуклеазой EcoR I проводят в 0,1 М трис-HCl с pH 7,5, 0,05 М NaCl, 0,005 М MgCl2 и 100 мкг на мл альбумина бычьей сыворотки. Реакционные смеси EcoR I содержат 5 ед. энзима на мкг ДНК и их инкубируют в течение 4 ч при 37оС. Реакции с рестрикционной нуклеазой PST I проводят в 0,006 М трис-HCl с pH 7,4, 0,05 М хлорида натрия, 0,006 М хлорида магния, 0,006 М 2-меркаптоэтанола и 100 мкг на мл альбумина бычьей сыворотки. Реакционные смеси PST I содержат 2 ед. энзима на мкг ДНК и их инкубируют 4 ч при 37оС. Обычно 10 мкг ДНК переваривают в конечном объеме 40 мкл. Затем добавляют 10-кратную концентрацию буферов. Потом в зависимости от объема ДНК добавляют дистиллированную в стерильных условиях воду. ДНК бактериофагаλ разрезают с помощью EcoR I в целях получения маркерных полос для определения величины фрагмента. Обычно 2 мкг ДНК λ переваривают с помощью 20 ед. EcoR I в конечном объеме 20 мкл.
Гель-электрофорез и перенос ДНК
К переваренным продуктам ДНК добавляют глицерин (примерно, до 20% ) и маркирущий синий краситель - тетрабромфенолсульфонафталин. В случае переваренных продуктов ДНК к каждому объему 20 мкл реакционной смеси добавляют 20 мкл буфера 1 х EcoR I. Обычно к каждому объему 40 мкл реакционной смеси добавляют 15 мкл 75% -го глицерина и 5 мкл 0,5% -го тетрабромфенолсульфонафталина.
В каждый сосуд загружают 10 мкг переваренной бактериальной ДНК или 2 мкг переваренной ДНК λ, после чего этот слой покрывают еще слоем расплавленной агарозы. Переваренные продукты подвергают электрофорезу в 0,8% -й агарозе с помощью 0,02 М ацетата натрия, 0,002 М ЭДТА, 0,018 М трис-основания и 0,028 М трис-HCl при значении pH 8,05 под напряжением 35 В до миграции красителя на 13-16 см. Затем гели погружают в бромид этидия (0,005 мг/мл) и помещают их на аппарат для облучения УФ-лучами для визуализации фрагментов λ. После этого ДНК переносят на нитроцеллюлозную фильтровальную бумагу. Потом гели обрабатывают денатурирующим раствором (1,5 М хлористого натрия, 0,5 М гидроокиси натрия) на балансирном столике в течение 20 мин. Затем денатурирующий раствор заменяют нейтрализующим раствором (3,0 М хлористого натрия, 0,5 М трис-HCl, значение pH 7,5) и через 40 мин гели проверяют с помощью бумаги для измерения значения pH. После нейтрализации гели обрабатывают буфером 6 xSSC (SSC = 0,15 М хлористого натрия, 0,015 М цитрата натрия) в течение 10 мин. Фрагменты ДНК из геля переносят на нитроцеллюлозную бумагу, продвигая через гель и нитроцеллюлозную бумагу буфер 6 xSSC с помощью стопки бумажных полотенец в течение 15 ч. Фильтры помещают в простраство между двумя листами хроматографической бумаги типа 3 ММ, обматывают алюминиевой фольгой с блестящей поверхностью наружу и высушивают в вакуумной печи при 80оС в течение 4 ч.
Синтез ДНК комплементарной относительно меченой, радиоактивным фосфором 32Р рибосомной РНК (32Р-p-РНК-к-ДНК)
Меченую 32Р ДНК, комплементарную относительно R-13 23S- и 16S-рибосомной ДНК Е. coli, синтезируют с использованием обратной транскриптазы вируса, вызывающего у птиц миелобластоз (AMV). Реакционная смесь содержит 5 мкл 0,2 М дитиотрейтола, 25 мкл 1 М трис со значением pH 8, 8,3 мкл 3 М хлористого калия, 40 мкл 0,1 М хлористого магния, 70 мкг актиномицина, 14 мкл 0,04 M dATР, 14 мкл 0,04 М dGDP, 14 мкл 0,04 М dTTP и 96,7 мкл H2O. В пластмассовую трубку загружают 137,5 мкл реакционной смеси, 15 мкл затравки из телячьего тимуса (10 мг/мл), 7 мкл H2O, 3 мкл pРНК (с использованием 40 мкг на единицу концентрации оптической плотности, т. е. 2,76 мкг/мкл), 40 мкл деоксицитидина 5'-(32P)-трифосфата (10 мКи/мл), 13 мкл AMV-полимеразы (6900 ед. /мкл). Энзиматическую реакционную массу инкубируют в течение 1,5 ч при 37оС. Затем раствор экстрагируют в хлороформе и приготовленном феноле (по 5 мл). После центрифугирования (13600 об. /мин) водную фазу непосредственно подают на колонку из Sephadex G-50 (1,5x 22 см). Для подачи на колонку используют пластмассовую пипетку емкостью 10 мл. Потом на верх колонки помещают небольшой стеклянный буртик, посредством защемляющего зажима прикрепляют резиновую трубку и добавляют дегазированный G-50, разбухший в 0,05% -ом SDS в течение ночи. Водную фазу заставляют протечь непосредственно в G-50 и элюируют 0,05% -ым SDS. В пластмассовых ампулах собирают 20 фракций по 0,5 мл каждая. Трубки, содержащие пиковые фракции, детектируют с помощью счетчика Черенкова с использованием 3Н-дискриминатора, причем время считывания на пробу составляет 0,1 мин, после этого регистрируется общее число импульсов. Потом собирают пиковые фракции. Аликвотные порции последних добавляют к доступному в торговле AquesolR и с помощью сцинтилляционного счетчика определяют 32P на мл.
Гибридизация и авторадиография
Фрагменты, содержащие последовательности гена РНК детектируют путем авторадиографии после гибридизации ДНК на фильтрах до 32P-p-РНК-к-ДНК. Фильтры замачивают в гибридизационной смеси (3x SSC, 0,1% SDS, 100 мкг/мл денатурированной, экстрагированной при воздействии ультразвука собачьей ДНК и раствор Дейнхарта - по 0,2% альбумина бычьей сыворотки, Фиколла и поливинилпирролидина) в течение часа при 68оС. Затем добавляют 32P-pРНК-кДНК при 4x106 СРМ/мл и инкубируют гибридизационную массу 48 ч при 68оС. Фильтры промывают в 3 x SSC и 0,1% SDS c интервалом по 15 мин в течение 2 ч или до того, пока промывной раствор не содержит ок. 3000 СРМ 32Р на мл. Фильтры высушивают на воздухе, завертывают их в пластмассовую обертку и авторадиографируют их примерно 1 ч при -70оС с использованием пленки типа Кодак Х-ОМАТ.
В. Эксперименты на млекопитающих.
Пробы pРНК мыши Mus musculus domesticus синтезируют из 18S и 28S и только из 28S-p-РНК. Нуклеиновую кислоту экстрагируют из печени мыши и осаждают ее. Затем наматывают и удаляют высокомолекулярную ДНК. Остающуюся нуклеиновую кислоту собирают путем центрифугирования и растворяют в буфере (50 мМ MgCl2 и 100 мМ трис, pH 7,4). Потом добавляют ДНК-зу (свободные от РНК-зы) до достижения концентрации 50 мкг/мл. Смесь инкубируют 30 мин при 37оС. Полученную в результате РНК реэкстрагируют, осаждают этанолом и растворяют в 1 мМ буфера - фосфата натрия при значении pH 6,8. Потом приготовляют градиент сахарозы концентрацией 5-20% в 0,1 М трис при значении pH 7,4 и 0,01 М ЭДТА. К нему добавляют пробу, после чего градиенты прядут 7 ч в роторе SW40 при 35К об. /мин. Фракции собирают по оптической плотности. Фракции 18S и 28S выделяют на основе сравнения с известными маркерами молекулярной массы.
Для всех экспериментов на млекопитающих используют нежесткие гибридизационные условия (54оС). Промывку осуществляют при 54оС 3 раза, каждый раз в течение 15 мин с использованием 3 x SSC с 0,05% -го SDS.
Эксперименты 1-8 описывают способы, проведенные с пробами, содержащими pРНК. В примерах 1-3 описывается моделирование с помощью ЭВМ с использованием проб, содержащих информацию гена гистона, триптофанового оперона trp D и α-фетопротеина соответственно.
Э к с п е р и м е н т 1. Определение видов бактерий на основе анализа генов рибосомной РНК с помощью рестрикционной эндонуклеазы.
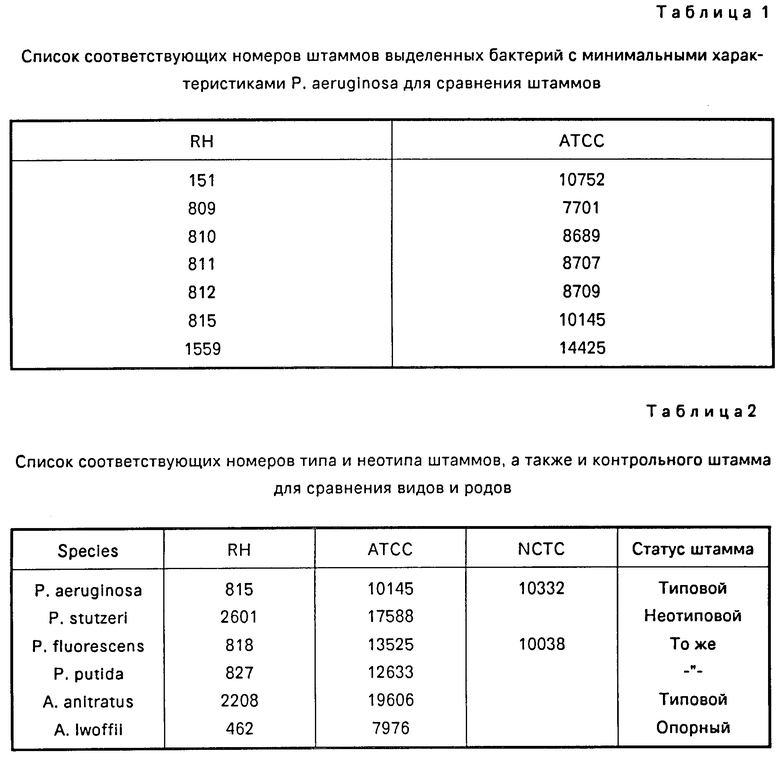
Разные применяемые в настоящем примере штаммы P. aeruginosa обладают минимальными фенотипическими характеристиками, идентифицирующими вид бактерии (см. Hugh R. H. сотр. Manual of Clinical Microbiology, 2-е изд. , 1974, с. 250-269( (табл. 1). Для сравнения видов и родов выбирают три штамма других Pseudumonas и два вида Acinetobacter (табл. 2).
Штаммы, используемые для сравнения видов Pseudomonas и Acinetobacter, приведены в табл. 2.
Виды Acinetobacter выбирают для сравнения родов, потому что они обладают некоторыми характерными признаками, которые встречаются у многих видов Pseudomonas.
Фрагменты в продуктах переваривания EcoR I имеют следующую величину (в парах килооснований): P. stutzeri 16,0; 12,0; 9,4; P. fluorescens 16,0; 10,0; 8,6; 7,8; 7,0; P. putida 24,0; 15,0; 10,0; 8,9; A. anitratus 20,0; 15,0; 12,5; 9,8; 7,8; 6,1; 5,2; 4,8; 3,8; 2,8 (величина наименьших трех фрагментов не была подсчитана);
5,3; 4,8; 4,4; 3,6; 3,2; 2,9 (величина трех наименьших фрагментов не была подсчитана).
Фрагменты в продуктах переваривания PST I имеют следующую величину (в парах килооснований): P. stutzeri 6,7; 6,1; 5,5; P. fluorescens 10,0; 9,4; 7,8; 7,0; P. putida 10,5; 9,9; 6,8; 6,3; 4,4; A. anitratus 36,0; 28,0; 20,5; 12,0; 10,0; 5,8; 3,7; 2,6; 2,4; A. lwoffi 9,9; 8,7; 7,2; 5,7; 4,0; 3,6; 3,2; 2,7.
Сравнение гибридизированных рестрикционных фрагментов из семи штаммов P. aeruginosa приводит к заключению, что этот вид можно определить посредством специфического относительно EcoR I набора фрагментов, содержащих последовательности гена в pРНК, т. е. пар килооснований (КВР) 10,1; 9,4; 7,6 и 5,9. Фрагмент 7,6 КВР EcoR I встречается в четырех из семи штаммов в этой пробе. Аналогичная ситуация наблюдается относительно некоторых фенотипических признаков штаммов названного вида. Факт, что определенные наборы фрагментов EcoR I из семи штаммов можно использовать для подразделения штаммов на две группы, говорит о том, что здесь могут иметься два вида с минимальными фенотипическими признаками P. aeruginosa. Результаты опытов, в которых ДНК переваривали с помощью PST I, приводят к заключению, что различия в штаммах, выявленные фрагментом 7.6 КВР EcoR I, представляют собой различия внутри одного вида, поскольку здесь наличествует один единственный сохранившийся набор определяющих вид фрагментов PST I, т. е. 9,4; 7,1; 6,6 и 6,4 КВР. Фрагменты PST I 9,4 и 6,6 КВР встречаются в шести из семи штаммов P. aeruginosa, фрагменты PST I 7,1 и 6,4 КВР - во всех штаммах пробы. Различия во фрагментах PST I наблюдаются у штаммов, не содержащих фрагмента EcoR I 7,6 КВР; RH 151 содержит фрагменты 10,1 и 8,2 КВР, RH 809 не содержит фрагмента 9,4 КВР, но включает фрагмент 6,0 КВР и RH 815. Типовой штамм не содержит фрагментов 6,6 КВР. Конфигурации гибридизированных фрагментов подтверждают заключение, что специфические относительно энзима сохранившиеся наборы можно использовать для определения данного вида. Штаммы какого-нибудь вида могут иметь большинство фрагментов в сохранившемся наборе. Появление различий во фрагментах у некоторых штаммов не препятствует их идентификации и может оказаться полезным в эпидемиологических исследованиях.
О появлении различий во фрагментах EcoR I 7,6 КВР у штаммов P. aeruginosa можно судить, проверяя гибридизированные фрагменты EcoR I, найденные у типовых штаммов других видов Pseudomonas. Типовые штаммы P. stutzeri, P. fluorescens и P. putida не содержат фрагмента 7,6 КВР, но они все содержат фрагменты EcoR I одинаковой величины P. aeruginosa и P. stutzeri - фрагмент 9,4 КВР, P. stutzeri и P. fluorescens - фрагмент 16 КВР и P. fluorescens и P. putida - фрагмент 10 КВР. Величины фрагментов обычно специфические для каждого из четырех видов Pseudomonas и типовые штаммы каждого вида имеют разные пределы величины фрагментов. Эти общие примечания действуют также в случае продуктов переваривания PST I.
При сравнении конфигураций фрагментов одного штамма каждого из четырех видов Pseudomonas и двух видов Acinetobacter можно установить, что виды, входящие в один род, сходные, в то время как роды отличаются друг от друга. Два вида Acinetobacter обладают более широкими пределами величины гибридизированных фрагментов, чем 4 вида Pseudomonas.
Без помощи рестрикционно-энзимных карт, доступных, например, для E. coli, Bacillus thuringiensis и B. subtilis, нельзя предсказать, где энзимы будут разрезать гены pРНК, число копий на геном и где между генами будут гетерологовые боковые участки или гетерогенность в генах. Если проба pРНК-кДНК E. coli откажет в гибридизации некоторым рестрикционным фрагментам, содержащим последовательности гена pРНК, то это свидетельствует о расстоянии на пути эволюции или о различии между исследуемым организмом и E. coli. Сохранившуюся природу pРНК можно использовать в качестве доказательства того, что это не имеет места. Это однако не играет значительной роли по сравнению с преимуществом обладания стандартной пробой, которую равным образом можно применять к любому неизвестному виду.
Э к с п е р и м е н т 2. Сравнение рестрикционного анализа с гибридизацией ДНК-ДНК в жидкой среде.
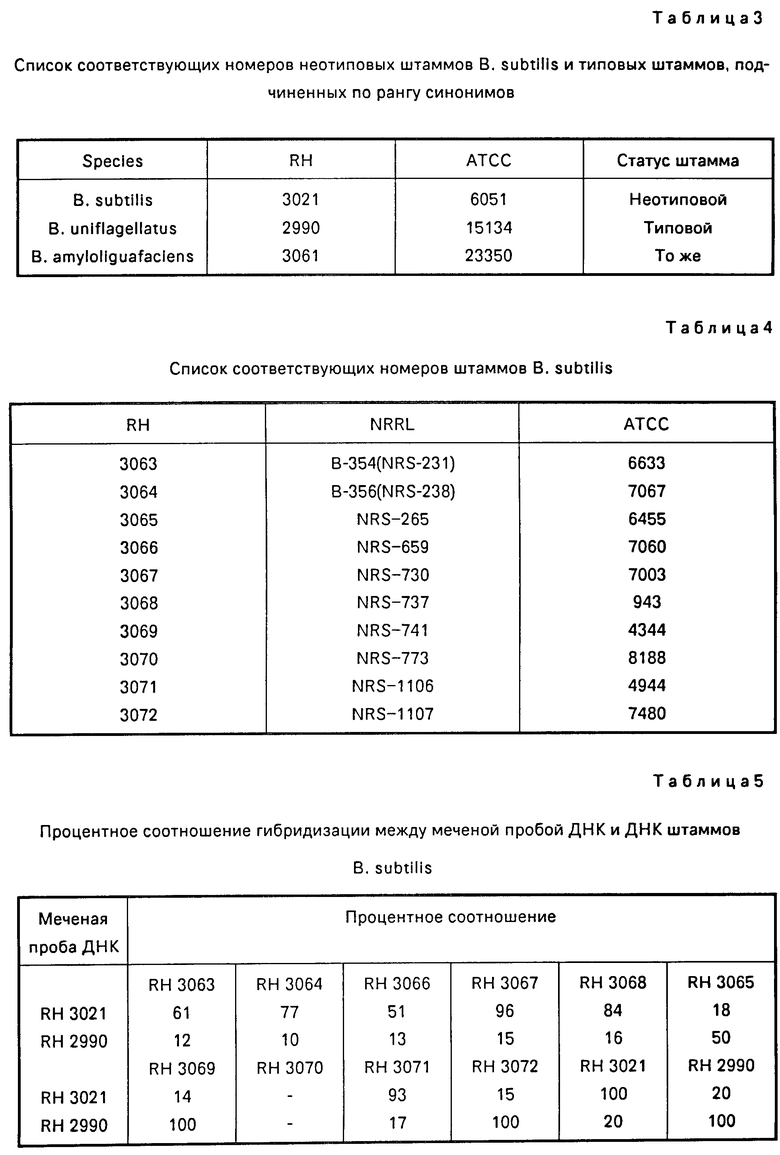
Используемые в этом эксперименте штаммы приведены в табл. 3 и 4.
Высокомолекулярную ДНК выделяют из каждого из штаммов. Данные гибридизации ДНК-ДНК в жидкой среде с использованием меченых ДНК РН 3021 и 2990 приведены в табл. 5.
Данные показывают, что речь идет о двух гибридизационных группах. Сходные данные описаны в литературе для B. subtilis (Seki c сотр. , International Journal of Systematic Bacteriology 25: 258-270, 1975). Эти две группы могут быть представлены RH 3021 и RH 2990. В случае осуществления анализа генов рибосомной РНК с помощью рестрикционной эндонуклеазы переваренные EcoR I продукты можно подразделить на две группы. Группа PH 3021 имеет два сильно гибридизированных фрагмента (2,15 и 2,1 КВР). Группа, представленная RH 2990, имеет два сильно гибридизированных фрагмента 2,6 и 2,5 КВР). Данные, полученные с ЕcoR I, можно использовать для введения штаммов B. subtilis в соответствующие группы гибридизации ДНК-ДНК. Согласно правилу 70% -й гибридизации ДНК-ДНК B. subtilis на самом деле представляет собой два вида. Однако на основе данных, полученных с PST I, возможно представить себе эти группы как две отличающиеся друг от друга популяции, восходящие к одному общему предку или процессу видообразования. Заключение, что B. subtilis представляет собой единственный вид, соответствует фенотипическим данным. Штаммы, приведенные в табл. 4, идентифицированы как B. subtilis (The Genus Bacillus. - Gordon, R. E. с сотр. - Agriculture Handbook N 427, Agricultural Research Service, U. S. Dept. of Agriculture, Washington, D. C. , s. 36-41). Рестрикционный анализ может обеспечить данные, сравнимые с данными гибридизации ДНК-ДНК, или при выборе надлежащего энзима дает возможность соответствующим образом определить вид несмотря на имеющиеся расхождения. RH 3061 был лишен сайтов PST I. Однако данные, полученные с EcoR I, показывают, что штамм представляет собой B. subtilis. Это заключение подтверждается и данными, полученными с Bgl II и Sac I.
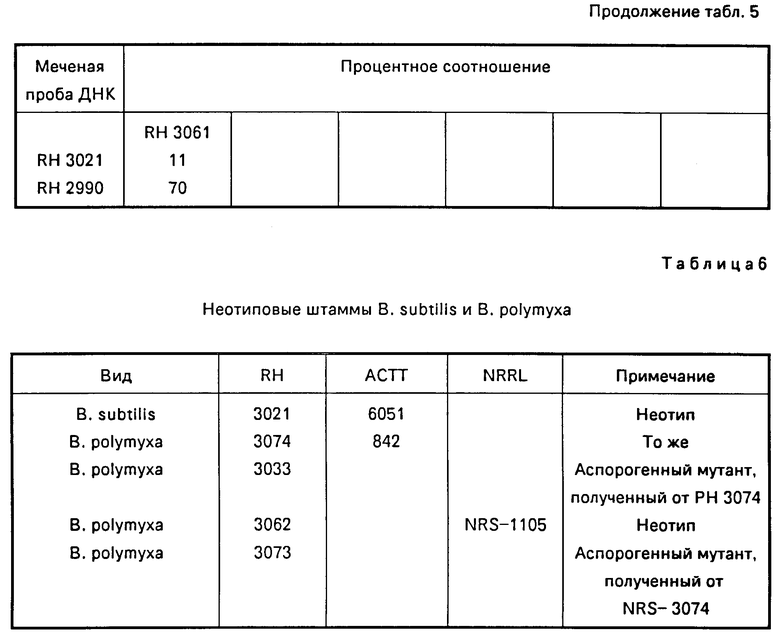
Э к с п е р и м е н т 3. Стабильность Bacillus polymyxa при рестрикционном анализе и в других экспериментах.
B. subtilis и B. polymyxa можно различить с помощью данных, полученных с EcoR I, PST I, Bgl II и Sac I. На основе значительных различий в конфигурациях полос PST I можно сделать заключение, что вид B. polymyxa отнесен не к тому роду. Хотя оба вида спорогенны, они проявляют несходство по фенотипическим признакам. Убеждающим фактом является то, что типовой штамм B. polymyxa из обеих коллекций культур ATCC и NRRL обладает теми же конфигурациями полос. Важным является тот факт, что удается идентифицировать аспорогенные мутанты. Оказывается весьма сложным, может быть даже невозможным, идентифицировать виды Bacillus, если они не способны к образованию спор.
Э к с п е р и м е н т 4. Идентификация видов бактерий в ткани мыши без их выделения.
Швейцарскую мышь Mus musculus domesticus (инбредный штамм) инокулируют внутрибрюшинно 0,5 мл мутной взвеси Streptococcus pneumoniae RH 3077 (ATCC 6303). Из погибающей мыши удаляют сердце, легкие и печень. Затем из этих тканей, S. pneumoniae RH 3077 и органов швейцарской мыши выделяют высокомолекулярную ДНК, после чего осуществляют рестрикционно-эндонуклеазный анализ генов pРНК с использованием EcoR I для переваривания ДНК. Наряду с промывкой фильтров в 3 x SSC их промывают еще в течение 2 x15 мин в 0,3 x SSC и 0,05% SDS. Авторадиографию осуществляют в течение 48 ч. Полученные данные показывают, что S. pneumoniae можно определить посредством семи гибридизированных фрагментов (17,0; 8,0; 6,0; 4,0; 3,3; 2,6 и 1,8 КВР). Проба бактериальной кДНК только мало гибридизируется с двумя фрагментами ДНК мыши (14,0 и 6,8 КВР). Гибридизированные фрагменты показывают наличие S. pneumoniae в зараженных тканях. Все семь полос обнаруживают в экстракте ДНК сердца. Они менее интенсивны, чем в экстракте ДНК печени, но все они имеются на авторадиограмме. Только полоса 6,0 КВР обнаруживается в экстракте ДНК легких. Меньшее число бактерий в легких объясняется тем, что мышь скорее поражена сепсисом, чем пневмонией. Легкие при автопсии не показывают затвердевания. В целях определения чувствительности анализа бактериальную ДНК разбавляют ДНК мыши и осуществляют электрофорез. В случае использования 0,1 мкг бактериальной ДНК в авторадиограмме обнаруживаются все 7 полос. При 10-3 мкг бактериальной ДНК становятся видимыми полосы 17,0; 8,0 и 6,0 КВР. В случае употребления числа 5˙ 10-3 мкг ДНК на 106 клеток S. pneumoniae (см. Biochim Biophys Acta, 26: 68), 10-1 мкг эквивалентно 2˙107 клеток. Итак, указанный прием пригоден для диагноза заражения на названном уровне чувствительности.
Описанный сравнительный опыт показывает, что бактериальная проба гибридизируется со специфическими относительно мыши фрагментами EcoR I (фрагменты имеют 14,0 и 6,8 КВР). Эти фрагменты соответствуют фрагментам EcoR I, обнаруженным с помощью пробы рибосомной РНК мыши 18S и 28S (фрагмент 6,8 КВР содержит последовательности 28S pРНК). Бактериальная проба не так хорошо гибридизируется с последовательностями гена рибосомной РНК млекопитающего, так что полосы менее интенсивные, система бактериальной пробы и ДНК ядра млекопитающего менее чувствительна и обнаруживается явная избирательность относительно ДНК из заражающих прокариотических организмов. В опытах, в которых бактериальная проба гибридизируется с 10 мкг переваренной бактериальной ДНК на один проход, в авторадиограммах не обнаруживается гибридизации с 10 мкг переваренной человеческой или мышиной ДНК на один проход, тогда как четко обнаружимы бактериальные полосы.
Э к с п е р и м е н т ы 5-8. Эксперименты на млекопитающих.
Нижеприведенные сравнительные эксперименты иллюстрируют, что анализ рестрикции pРНК для идентификации организмов успешно можно применить не только к бактериям, но и к сложным эукариотическим организмам.
Разные роды млекопитающих можно установить с помощью пробы 18S и 28S pРНК Mus musculus domesticus и что удается различить несколько видов рода Mus. На фиг. 13 энзимом является PST I и живые организмы и соответствующие им полосы следующие:
1. Mus musculus melossinus (мышь) 14,5; 13,5; 2,6.
2. Mus musculus domesticus (мышь) 13,5; 2,6.
3. Canis familiaris (собака) 12,0.
4. Cavia porcellus (морская свинка) 17,0; 14,0; 13,0; 8,8; 5,7; 4,7 и одна полоса ниже 3,0.
5. Cricetulus griseus (хомяк) 25,0; 4,7.
6. Homo sapiens (человек) 15,0; 5,7.
7. Felis catus (кошка) 20,0; 9,7.
8. Ratus norvegicus (крыса) 12,5.
9. Mus musculus domesticus (мышь) 13,5; 2,6.
10. Mus cervicolor cervicolor (мышь) 14,0; 2,7.
11. Mus cervicolor papeus (мышь) 13,5; 2,6.
12. Mus pahari (мышь) 13,0; 3,7.
13. Mus cookii (мышь) 13,5; 2,6.
ДНК мыши и кошки можно различить только с помощью фрагмента 28S pРНК-кДНК и что конфигурация полос гибрида зависит от состава последовательностей пробы. Энзимом является EcoR I и живые организмы и соответствующие им полосы следующие:
1. Mus musculus domesticus (мышь) 6,5 КВР.
2. Felis catus (кошка) 8,3 КВР.
Энзимом является Sac I и живые организмы и полосы следующие:
1. Erythrocebus patas (обезьяна патас) 8,5; 3,7; < 3,0.
2. Ratus norvegicus (крыса) 25,0; 9,5; 3,6; < 3,0.
3. Mus musculus domesticus (мышь) 6,8; < 3,0.
4. Felis catus (кошка) 9,5; 5,3; 4,0; < 3,0; < 3,0.
5. Homo sapiens (человек) 10,5; < 3,0.
6. Macaca mulatta (ресус) 9,8; < 3,0.
При сравнении продуктов переваривания Sac I с другими видами, относящимися к млекопитающим, становится видно, что гибридизированная конфигурация является специфической для данного энзима.
Удается различить приматы. Клеточные культуры и ткань исходного вида имеют общие полосы, и разные культуры человеческих клеток можно различить путем добавления и делеции полос.
Энзимом является EcoR I, а живые организмы и полосы следующие:
1. Erythrocebus patas (обезьяна патас) > 22,0; 11,0; 7,6; 2,6.
2. Macaca mulatta (ресус) 22,0; 11,5; 7,6.
3. Homosapiens (человек) > 22,0; 22,0; 16,0; 8,1; 6,6.
4. М 241/88 (клеточная культура лангура) 14,0; 7,2; 5,7.
5. HeLa (культура человеческих клеток) > 8,1; 6,6.
6. J96 (культура человеческих клеток) > 22,0; 22,0; 16,0; 11,0; 8,1; 6,6.
7. AО (культура человеческих клеток) 22,0; 16,0; 8,1; 6,6.
8. Х-381 (ресус) 22,0; 11,5; 7,6.
Э к с п е р и м е н т 9. Применение пробы гена гистона Н4.
С помощью ЭВМ моделируют идентификацию и характеристику двух видов животных (морского ежа и мыши) с использованием сохранившейся последовательности ДНК, полученной от гена гистона Н4.
Ниже приведена последовательность нуклеотидов гена гистона Н4 в случае морского ежа (Psammechinus miliaris), где A, T, C и G представляют собой известные нуклеотиды, а N неизвестную в данном случае позицию (788 пар оснований).
10 20 30 40 50
CAACATATTA GAGGAAGGGA GAGAGAGAGA GAGAGAGAGA GAGAGAGAGA
GTTGTATAAT CTCCTTCCCT CTCTCTCTCT CTCTCTCTCT CTCTCTCTCT
60 70 80 90 100
GGGGGGGGGG GAGGGAGAAT TGCCCAAAAC ACTGTAAATG TAGCGTTAAT
CCCCCCCCCC CTCCCTCTTA ACGGGTTTTG TGACATTTAC ATCGCAATTA
110 120 130 140 150
GAACTTTTCA TCTCATCGAC TGCGCGTGTA TAAGGATGAT TATAAGCTTT
CTTGAAAAGT AGAGTAGCTG ACGCGCACAT ATTCCTACTA ATATTCGAAA
160 170 180 190 200
TTTTCAATTT ACAGGCACTA CGTTACATTC AAATCCAATC AATCATTTGA
AAAAGTTAAA TGTCCGTGAT GCAATGTAAG TTTAGGTTAG TTAGTAAACT
210 220 230 240 250
ATCACCGTCG CAAAAGGCAG ATGTAAACTG TCAAGTTGTC AGATTGTGTG
TAGTGGCAGC GTTTTCCGTC TACATTTGAC AGTTCAACAG TCTAACACAC
260 270 280 290 300
CGCGGCCTCC AGTGAGCTAC CCACCGGGCC GTCGCGGAGG GGCGCACCTG
GCGCCGGAGG TCACTCGATG GGTGGCCCGG CAGCGCCTCC CCGCGTGGAC
310 320 330 340 350
TGCGGGAGGG GTCATCGGAG GGCGATCGAG CCTCGTCATC CAAGTCCGCA
ACGCCCTCCC CAGTAGCCTC CCGCTAGGTC GGAGCAGTAG GTTCAGGCGT
360 370 380 390 400
TACGGGTGAC AATACCCCCG CTCACCGGGA GGGTTGGTCA ATCGCTCAGC
ATGCCCACTG TTATGGGGGC GAGTGGCCCT CCCAACCAGT TAGCGAGTCG
410 420 430 440 450
GAAACGTCCA GTCGTCAGGA TCGCACTAAG ACTCTCTCTC AATCTCCATA
CTTTGCAGGT CAGCAGTCGT AGCGTGATTC TGAGAGAGAG TTAGAGGTAT
460 470 480 490 500
GAGGCGCCAA GTGGTAAAGG AGGCAAGGGG CTCGGAAAGG GAGGCGCCAA
TACAGTCCGG CACCATTTCC TCCGTTCCCC GAGCCTTTCC CTCCGCGGTT
510 520 530 540 550
GCGTCATCGC AAGGTCCTAC GAGACAACAT CCAGGGCATC ACCAAGCCTG
CGCAGTAGCG TTCCAGGATG CTCTGTTGTA GGTCCCTGAT TGGTTCGGAC
560 570 580 590 600
CAATCCGCCG ACTCNNNNNN NNNNNNNNNN NNNNNNGAAT CTCTGGTCTT
GTTAGGCGGC TGAGNNNNNN NNNNNNNNNN NNNNNNCTTA GAGACCAGAA
610 620 630 640 650
ATCTACGAGG AGACACGAGG GGTGCTGAAG GNNNNNNNNN NNNNNNNNNN
TAGATGCTCC TCTGTGCTCC CCACGACTTC CNNNNNNNNN NNNNNNNNNN
660 670 680 690 700
NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN
NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN
710 720 730 740 750
NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNGGCCGAAC ACTGTACGGC
NNNNNNNNNN NNNNNNNNNN NNNNNNNNNN NNCCGGCTTG TGACATGCCG
760 770 780
TTCGGCGGCT AAGTGAAGCA GACTTGGCTA GAATAACG
AAGCCGCCGA TTCACTTCGT CTGAACCGAT CTTATTGC
Аналогичная последовательность гена Н4 мыши приведена ниже (968 пар оснований).
10 20 30 40 50
GAATTCTCCG AGGGACTTCG GCACCATAAT TAAGAAAATC GAAAATAAAA 5. CTTAAGAGGC TCCCTGAAGC CGTGGTATTA ATTCTTTTAG CTTTTATTTT
60 70 80 90 100
AAATAAAGGC TTGAGACTGT AAGGAACCGG TAGAGGGCAG AGAAGAGAAA
TTTATTTCCG AACTCTGACA TTCCTTGGCC ATCTCCCGTC TCTTCTCTTT
110 120 130 140 150
AGAAAAACAG GAAGATGATG CAACATCCAG AGCCCGGATA ATTTAGAAAG
TCTTTTTGTC CTTCTACTAC GTTGTAGGTC TCGGGCCTAT TAAATCTTTC
10. 160 170 180 190 200
GTTCCCGCCC GCGCGCTTTC AGTTTTCAAT CTGGTCCGAT CCTCTCATAT
CAAGGGCGGG CGCGCGAAAG TCAAAAGTTA GACCAGGCTA GGAGAGTATA
210 220 230 240 250
ATTAGTGGCA CTCCACCTCC AATGCCTCAC CAGCTGGTGT TTCAGATTAC
TAATCACCGT GAGGTGGAGG TTACGGAGTG GTCGACCACA AAGTCTAATG
260 270 280 290 300
ATTAGCTATG TCTGGCAGAG GAAAGGGTGG AAAGGGTCTA GGCAAGGGTG
TAATCGATAC AGACCGTCTC CTTTCCCACC TTTCCCAGAT CCGTTCCCAC 15.
310 320 330 340 350
GCGCCAAGCG CCATCGCAAA GTCTTGCGTG ACAACATCCA GGGTATCACC
CGCGGTTCGC GGTAGCGTTT CAGAACGCAC TGTTGTAGGT CCCATAGTGG
360 370 380 390 400
AAGCCCGCCA TCCGCCGCCT GGCTCGGCGC GGTGGGGTCA AGCGCATCTC
TTCGGGCGGT AGGCGGCGGA CCGAGCCGCG CCACCCCAGT TCGCGTAGAG
410 420 430 440 450 20. CGGCCTCATC TACGAGGAGA CCCGTGGTGT GCTGAAGGTG TTCCTGGAGA
GCCGGAGTAG ATGCTCCTCT GGGCACCACA CGACTTCCAC AAGGACCTCT
460 470 480 490 500
ACGTCATCCG CGACGCAGTC ACCTACACCG AGCACGCCAA GCGCAAGACC
TGCAGTAGGC GCTGCGTCAG TGGATGTGGC TCGTCCGGTT CGCGTTCTGG
510 520 530 540 550
GTCACCGCTA TGGATGTGGT GTACCCTCTC AAGCGCCAGG GCCGCACCCT 25. CAGTGGCGAT ACCTACACCA CATGCGAGAG TTCGCGGTCC CCGCGTGGGA
560 570 580 590 600
CTACGGCTTC GGAGGCTAGA CGCCGCCGCT TCAATTCCCC CCCCCCCCCC
GATGCCGAAG CCTCCGATCT GCGGCGGCGA AGTTAAGGGG GGGGGGGGGG
610 620 630 640 650
ATCCCTAACG GCCCTTTTTA GGGCCAACCA CAGTCTCTTC AGGAGAGCTG
TAGGGATTGC CGGGAAAAAT CCCGGTTGGT GTCAGACAAG TCCTCTCGAC
660 670 680 690 700
ACACTGACTT GGGTCGTACA GGTAATAACC GCGGGTTTAG GACTCACGCT
TGTGACTGAA CCCAGCATGT CCATTATTGG CGCCCAAATC CTGAGTGCGA
710 720 730 740 750
ACTAGGTGTT CCGCTTTTAG AGCCATCCAC TTAAGTTTCT ATACCACGGC
TGATCCACAA GGCGAAAATC TCGGTAGGTG AATTCAAAGA TATGGTGCCG
760 770 780 790 800
GGATAGATAG CATCCAGCAG GGTCTGCTCA CACTGGGAAT TTTAATTCCT
CCTATCTATC GTAGGTCGTC CCAGACGAGT GTGACCCTTA AAATTAAGGA
810 820 830 840 850
ACTTAGGGTG TGAGCTGGTT GTCAGGTCAA GAGACTGGCT AAGATTTTCT
TGAATCCCAC ACTCGACCAA CAGTCCAGTT CTCTGACCGA TTCTAAAAGA
860 870 880 890 900
TTAGCTCGTT TGGAGCAGAA TTGCAATAAG GAGACCCTTT GGATGGGATG
AATCGAGCAA ACCTCGTCTT AACGTTATTC CTCTGGGAAA CCTACCCTAC
910 920 930 940 950
ACCTATGTCC ACACATCAAA TGGCTATGTG GCTGTGTCCC TGTGTTTCCA
TGGATACAGG TGTGTAGTTT ACCGATACAC CGACACAGGG ACACAAAGGT
960
ATGAGTGGCT GTGCTTGA
TACTCACCGA CACGAACT
Область гомологии для обеих указанных последовательностей указана ниже, причем звездочки означают гомологичные участки. В показанной области первые 118 пар оснований проявляют гомологию в 80,5% и их используют в этом эксперименте в качестве пробы сохранившейся последовательности ДНК: позиции оснований морского ежа 449-567 (сверху), позиции оснований мыши 257-357 (снизу):
449 TA TGTC
TGTC GGC
GGC G
G GG
GG AAAGG
AAAGG GG
GG AA
AA GGG
GGG CT
CT GG
GG AA
AA GGG
GGG GGCGCCAAGCG
GGCGCCAAGCG C 257 TA-TGTCTGGCAGAGG-AAAGGGTGG-AAAGGGTCTAGGCAA-GGGTGGCGCCAAGCGCC
C 257 TA-TGTCTGGCAGAGG-AAAGGGTGG-AAAGGGTCTAGGCAA-GGGTGGCGCCAAGCGCC
ATCGCAA GTC
GTC T
T CG
CG GACAACATCCAGGG
GACAACATCCAGGG ATCACCAAGCC
ATCACCAAGCC GC
GC ATCCGCCG
ATCCGCCG CT
CT ATCGCAAAGTCTTGCGTGACAACATCCAGGGTATCACCAAGCCCGCCATCCGCCGCCTGG
ATCGCAAAGTCTTGCGTGACAACATCCAGGGTATCACCAAGCCCGCCATCCGCCGCCTGG
NNNNNNNNNNNNNNNNNNNNNN ATCTC
ATCTC GG
GG C
C T
T ATCTACGAGGAGAC
ATCTACGAGGAGAC CG
CG GG
GG G
G
CTCGGCGCGGTGGGGTCAAGCG-CATCTCCGG-CCTCATCTACGAGGAGACCCG-TGGTG
TGCTGAAGGNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
TGCTGAAGGTGTTCCTGGAGAACGTCATCCGCGACGCAGTCACCTACACCGAGCACGCCA
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNGGCCG A
A C
C
AGCGCAAGACCGTCACCGCTATGGATGTGGTGTACGCTCTCAAGCGCCAGGGCCGCACCC CT
CT ACGGCTTCGG
ACGGCTTCGG GGCTA
GGCTA GA
GA GC
GC GAC
GAC TT
TT C
C A 780
A 780
TCT--ACGGCTTCGGAGGCTA---GACGCCGCCGCTT--CAA 584
Z = 84,503
P( 342, 289) = , 000Е+00 Е = . 000
Сайты, получаемые расщеплением рестрикционной эндонуклеазой, определяют по двум последовательностям. Список сайтов расщепления для последовательностей морского ежа и мыши приведен ниже. Номера указывают сторону 5' сайта, если название сайта не заключено в скобки, что указывает на то, что известен только сайт распознавания.
Морской еж
Последовательность Появляется в позиции
AcyI (GPCGQC) 495
AluI (AGCT) 147 267
AsuI (GGNCC) 277 514
AvaII (GGLCC) 514
CauII (CCMGG) 276 377
DdeI (CTNAG) 396 427
DpnI (GATC) 326
EcoRI* (PPATQQ) 184
EcoRII (CCLGG) 531
Fnu4HI (GCNGC) 254
FnuDII (CGCG) 125 253 285
FokI (GGATG) 148
FokI (CATCC) 324 515
HaeII (PGCGCQ) 498
HaeIII (GGCC) 256 279 459
HgaI (GCGTC) 491
HgiCI (GGQPCC) 494
HgiJII (GPGCQC) 483
HhaI (GCGC) 125 253 295 497
HindIII (AAGCTT) 145
HinfI (GANTC) 200 431 561
HpaII (CCGG) 275 376
HphI (GGTGA) 368
HphI (TCACC) 195 365 532
MboI (GATC) 324
MnlI (CCTC) 267 342
MnlI (GAGG) 4 42 54 280 299 311
372 463 484
NarI (GGCGCC) 495
NspBII (GCMGC) 256
PvuI (CGATCG) 327
ScrFI (CCNGG) 276 377 533
SfaNI (GCATC) 409 527
TaoI (TCGA) 117 327
Мышь
Последовательность Появляется в позиции
AcyI (GPCGQC) 302 571
AflII (CTTAAG) 731
AluI (AGCT) 234 256 648 815 855
AsuI (GGNCC) 184 540 611 622
AvaII (GGLCC) 184
RssHII (GCGCGC) 162
CauII (CCMGG) 135
DdeI (CTNAG) 803 840
DpnI (GATC) 190
[EcoB] (AGCANNNNNNNNTCA) 766
[EcoP1] (AGACC) 418 496 882
[EcoP1] (GGTCT) 285 771
[EcoP15] (CAGCAG) 765
EcoRI (GAATTC) 2
EcoRI* (PPATQQ) 4 790 845
EcoRII (CCLGG) 338 368 443 536
Fnu4HI (GCNGC) 366 543 574 577
FnuDII (CGCG) 162 164 380 461 682
FokI (GGATG) 526 905 910
FokI (CATCC) 111 322 346 442 537 711 748
HaeII (PGCGCQ) 305 312 537
HaeIII (GGCC) 404 542 612 624
HsaI (GACGC) 472 579
HsiAI (GLGCLC) 485
HsiCI (GGQPCC) 21 301
HsiJII (GPGCQC) 135
HnaI (GCGC) 164 166 304 311 380 395
494 536
HinfI (GANTC) 692
HpaII (CCGG) 78 135 401
HphI (TCACC) 220 339 462 495
MboI (GATC) 188
MboII (GAAGA) 105 124
MboII (TCTTC) 629
MnlI (CCTC) 202 227 236 415 559
MnlI (GAGG) 3 76 261 407 555
NarI (GGCGCC) 302
NspRII (GCMGC) 368 545 576 579
PvuII (CAGCTG) 234
RsaI (GTAC) 523 668
SacII (CCGCGG) 683
ScrFI (CCNGG) 135 340 370 445 538
SfaNI (GATGC) 127
SfaNI (GCATC) 385 751
TaQI (TCGA) 40
TthlllI (GACNNNGTC) 466
TthlllII (TGQTTG) 953
XmnI (GAANNNNTTC) 439
Последовательности морского ежа и мыши сравнивают с Hha I (GCGC) и с описанной последовательностью пробы. Последовательность морского ежа имеет сайты расщепления в позициях 295 и 497, создавая этим фрагмент 202 bp, который в денатурированном виде гибридизируется с последовательностью пробы. Сайты Hha I (GCGC) в последовательности мыши 166, 304, 311 и 380 показывают, что с помощью последовательности пробы можно обнаружить фрагменты 69 и 138.
Итак, для морского ежа получается генетическая характеристика
202 а для мыши - 69 и
138.
Э к с п е р и м е н т 10. Использование гена trpD триптофанового оперона в качестве пробы.
Тот же самый тип моделирования с помощью ЭВМ, что и в эксперименте 1, осуществляют с использованием в качестве пробы гена trpD. Это позволяет сделать заключение, что E. coli и Salmonella typhimurium можно различить с помощью рестрикционных фрагментов, содержащих сохранившуюся последовательность.
Ген trpD E. coli с фрагментами 684 bp приведены ниже.
10 20 30 40 50
GAAGCCGACG AAACCCGTAA CAAAGCTCGC GCCGTACTGC GCGCTATTGC
CTTCGGCTGC TTTGGGCATT GTTTCGAGCG CGGCATGACG CGCGATAACG
60 70 80 90 100
CACCGCGCAT CATGCACAGG AGACTTTCTG ATGGCTGACA TTCTGCTGCT
GTGGCGCGTA GTACGTGTCC TCTGAAAGAC TACCGACTGT AAGACGACGA
110 120 130 140 150
CGATAATATC GACTCTTTTA CGTACAACCT GGCAGATCAG TTGCGCAGCA
GCTATTATAG CTGAGAAAAT GCATGTTGGA CCGTCTATGC AACGCGTCGT
160 170 180 190 200
ATGGGCATAA CGTGGTGATT TACCGCAACC ATATACCGGC GCAAACCTTA
TACCCGTATT GCACCACTAA ATGGCGTTGG TATATGGCCG CGTTTGGAAT
210 220 230 240 250
ATTGAACGCT TGGCGACCAT GAGTAATCCG GTGCTGATGC TTTCTCCTGG
TAACTTGCGA ACCGCTGGTA CTCATTAGGC CACGACTACG AAAGAGGACC
260 270 280 290 300
CCCCGGTGTG CCGAGCGAAG CCGGTTGTAT GCCGGAACTC CTCACCCGCT
GGGGCCACAC GGCTCGCTTC GGCCAACATA CGGCCTTGAG GAGTGGGCGA
310 320 330 340 350
TGCGTGGCAA GCTGCCCATT ATTGGCATTT GCCTCGGACA TCAGGCGATT
ACGCACCGTT CGACGGGTAA TAACCGTAAA CGGAGCCTGT AGTCCGCTAA
360 370 380 390 400
GTCGAAGCTT ACGGGGGCTA TGTCGGTCAG GCGGGCGAAA TTCTCCACGG
CAGCTTCGAA TGCCCCCGAT ACAGCCAGTC CGCCCGCTTT AAGAGGTGCC
410 420 430 440 450
TAAAGCCTCC AGCATTGAAC ATGACGGTCA GGCGATGTTT GCCGGATTAA
ATTTCGGAGG TCGTAACTTG TACTGCCAGT CCGCTACAAA CGGCCTAATT
460 470 480 490 500
CAAACCCGCT GCCGGTGGCG CGTTATCACT CGCTGGTTGG CAGTAACATT
GTTTGGGCGA CGGCCACCGC GCAATAGTGA GCGACCAACC GTCATTGTAA
510 520 530 540 550
CCGGCCGGTT TAACCATCAA CGCCCATTTT AATGGCATGG TGATGGCAGT
GGCCGGCCAA ATTGGTAGTT GCGGGTAAAA TTACCGTACC ACTACCGTCA
560 570 580 590 600
ACGTCACGAT GCGGATCGCG TTTGTGGATT CCAGTTCCAT CCGGAATCCA
TGCAGTGCTA CGCCTAGCGC AAACACCTAA GGTCAAGGTA GGCCTTAGGT
610 620 630 640 650
TTCTCACCAC CCAGGGCGCT CGCCTGCTGG AACAAACGCT GGCCTGGGCG
AAGAGTGGTG GGTCCCGCGA GCGGACGACC TTGTTTGCGA CCGGACCCGC
660 670 680
CAGCATAAAC TAGAGCCAGC CAACACGCTG CAA
GTCGTATTTG ATCTCGGTCG GTTGTGCGAC GTT
Ниже приведен ген trpD с 683 парами оснований для S. typhimurium:
10 20 30 40 50
GAAGCCGATG AAACCCGTAA TAAAGCGCGC GCCGTATTGC GTGCTATCGC
CTTCGGCTAC TTTGGGCATT ATTTCGCGCG CGGCATAACG CACGATAGCG
60 70 80 90 100
CACCGCGCAT CATGCACAGG AGACCTTCTG ATGGCTGATA TTCTGCTGCT
GTGGCGCGTA GTACGTGTCC TCTGGAAGAC TACCGACTAT AAGACGACGA
110 120 130 140 150
CGATAACATC GACTCGTTCA CTTGGAACCT GGCAGATCAG CTACGAACCA
GCTATTGTAG CTGAGCAAGT GAACCTTGGA CCGTCTAGTC GATGCTTGGT
160 170 180 190 200
ACGGTCATAA CGTGGTGATT TACCGTAACC ATATTCCGGC GCAGACGCTT
TGCCAGTATT GCACCACTAA ATGGCATTGG TATAAGGCCG CGTCTGCGAA
210 220 230 240 250
ATCGATCGCC TGGCGACAAT GAAAAATCCT GTGCTAATGC TCTCCCCCGG
TAGCTAGCGG ACCGCTGTTA CTTTTTAGGA CACGATTACG AGAGGGGGCC
260 270 280 290 300
CCCGGGTGTT CCCAGCGAGG CAGGTTGTAT GCCGGAGCTG CTGACCCGAC
GGGCCCACAA GGGTCGCTCC GTCCAACATA CGGCCTCGAC GACTGGGCTG
310 320 330 340 350
TACGCGGCAA GTTACCGATC ATCGGCATTT GTCTGGGGCA TCAGGCGATT
ATGCGCCGTT CAATGGCTAG TAGCCGTAAA CAGACCCCGT AGTCCGCTAA
360 370 380 390 400
GTCGAAGCTT ACGGCGGTTA CGTCGGTCAG GCGGGAGAAA TCCTGCATGG
CAGCTTCGAA TGCCGCCAAT GCAGCCAGTC CGCCCTCTTT AGGACGTACC
410 420 430 440 450
CAAAGCCTCC AGCATTGAGC ATGACGGTCA GGCGATGTTC GCCGGGCTCG
GTTTCGGAGG TCGTAACTCG TACTGCCAGT CCGCTACAAG CGGCCCGAGC
460 470 480 490 500
CGAATCCGCT ACCGGTCGCG CGTTATCATT CGCTGGTCGG CAGTAATGTT
GCTTAGGCGA TGGCCAGCGC GCAATAGTAA GCGACCAGCC GTCATTACAA
510 520 530 540 550
CCTGCCGGGC TGACCATTAA CGCCCATTTC AACGGCATGG TGATGGCGGT
GGACGGCCCG ACTGGTAATT GCGGGTAAAG TTGCCCTACC ACTACCGCCA
560 570 580 590 600
ACGTCATGAT GCGGATCGCG TTTGCGGTTT TCAATTTCAT CCCGAGTCCA
TGCAGTACTA CGCCTAGCGC AAACGCCAAA AGTTAAAGTA GGGCTCAGGT
610 620 630 640 650
TCCTGACGAC ACAGGGCGCG CGTCTACTGG AGCAAACATT AGCCTGGGCG
AGGACTGCTG TGTCCCGCGC GCAGATGACC TCGTTTGTAA TCGGACCCGC
660 670 680
CTGGCGAAGC TGGAACCGAC CAACACCCTA CAG
GACCGCTTCG ACCTTGGCTG GTTGTGGGAT GTC
Затем устанавливают две гомологичные для двух последовательностей области, причем верхняя последовательность представляет собой E. coli, и нижняя - S. typhimurium.
Область 1
452 AA CCGCT
CCGCT CCGGT
CCGGT GCGCGTTATCA
GCGCGTTATCA TCGCTGGT
TCGCTGGT GGCAGTAA
GGCAGTAA TTCC
TTCC G
G C
C GG
GG 453 AA-TCCGCTACCGGTCGCGCGTTATCATTCGCTGGTCGGCAGTAATGTTCCTGCC-GGGC
453 AA-TCCGCTACCGGTCGCGCGTTATCATTCGCTGGTCGGCAGTAATGTTCCTGCC-GGGC
T A
A CCAT
CCAT AACGCCCATTTTAA
AACGCCCATTTTAA GGCATGGTGATGGC
GGCATGGTGATGGC GTACGTCA
GTACGTCA GATGCGGATCGC
GATGCGGATCGC
TGA-CCATTAACGCCCATTTCAACGGCATGGTGATGGCGGTACGTCATGATGCGGATCGC
GTTTG GG
GG TTC
TTC A
A TT
TT CATCC
CATCC GA
GA TCCAT
TCCAT CT
CT AC
AC AC
AC CAGGGCGC
CAGGGCGC CG
CG CT
CT CT GTTTGCGGTTTTC-AATTTCATCCCGAGTCCATCCTGACGACACAGGGCGCGCGTCTACT
CT GTTTGCGGTTTTC-AATTTCATCCCGAGTCCATCCTGACGACACAGGGCGCGCGTCTACT
GGA CAAAC
CAAAC T
T GCCTGGGCGC
GCCTGGGCGC GCA
GCA AA
AA CT
CT GA
GA CC
CC A
A C
C AACAC
AACAC CT
CT CA 68
CA 68
GGAGCAAACATTAGCCTGGGCGCTGGC-GAAGCTGGAACCGACC-AACACCCTACA 68
Z = 78,390
P( 236, 185) = . 000Е+ 00 Е = . 000
Область 2
1 GAAGCCGA GAAACCCGTAA
GAAACCCGTAA AA
AA GC
GC CGC
CGC CGTA
CGTA TGCG
TGCG GCTAT
GCTAT GCCACCGCGC 1 GAAGCCGATGAAACCCGTAATAAAGCGCGCGC--CGTATTGCGTGCTATCGCCACCGCGC
GCCACCGCGC 1 GAAGCCGATGAAACCCGTAATAAAGCGCGCGC--CGTATTGCGTGCTATCGCCACCGCGC
ATCATGCACAGGAGAC TT
TT CTGATGGCTGA
CTGATGGCTGA ATTCTGCTGCTCGATAA
ATTCTGCTGCTCGATAA ATCGACTC
ATCGACTC T
T
ATCATGCACAGGAGACCTT-CTGATGGCTGATATTCTGCTGCTCGATAACATCGACTCGT
T AC
AC G
G A
A A
A C
C TGGCAGATCAG
TGGCAGATCAG T
T CG
CG A
A C
C AA
AA GG
GG CATAACGTGGTGATTTA T-CACTTGGA-ACC-TGGCAGATCAGCTACG-AACCAA-CGGTCATAACGTGGTGATTTA
CATAACGTGGTGATTTA T-CACTTGGA-ACC-TGGCAGATCAGCTACG-AACCAA-CGGTCATAACGTGGTGATTTA
CCG AACCATAT
AACCATAT CCGGCGCA
CCGGCGCA AC
AC CTTA
CTTA T
T GA
GA CGC
CGC T
T G
G CGAC
CGAC ATGA
ATGA AA
AA TCC CCGTAACCATATTCCGGCGCAGACGCTTA-TCGATCGCCTGG-CGACAATGA-AAAATCC
TCC CCGTAACCATATTCCGGCGCAGACGCTTA-TCGATCGCCTGG-CGACAATGA-AAAATCC GTGCT
GTGCT ATGCT
ATGCT T
T C
C CC
CC GGCCC
GGCCC GG
GG TGT
TGT CC
CC G
G G
G A
A GC
GC GGTTGTATGCCGG TGTGCTAATGCTCTCC-CCCGGCCC-GGGTGTTCCCAGCG--AGGCAGGTTGTATGCCGG
GGTTGTATGCCGG TGTGCTAATGCTCTCC-CCCGGCCC-GGGTGTTCCCAGCG--AGGCAGGTTGTATGCCGG
* * * * ** ** * * * * * * ** *
AACTCCTCACCCG-CT-TGCGTGGCAAGCTGCCCATTATTGGCATTTGCCT-CGGACATC
AGCTGCTGACCCGACTACGCG--GCAAGTTACCGATCATCGGCATTTGTCTGGGG-CATC
AGGCGATTGTCGAAGCTTACGG GG
GG TA
TA GTCGGTCAGGCGGG
GTCGGTCAGGCGGG GAAAT
GAAAT CT
CT CA
CA GG
GG
AGGCGATTGTCGAAGCTTACGGCGG-TTACGTCGGTCAGGCGGGAGAAATCCTGCATGGC AA
AA GCCTCCAGCATTGA
GCCTCCAGCATTGA CATGACGGTCAGGCGATGTT
CATGACGGTCAGGCGATGTT GCCTT 445
GCCTT 445
AAA-GCCTCCAGCATTGAGCATGACGGTCAGGCGATGTTCGCCGG 445
Z = 80,215
P( 465, 373) = . 000Е+00 Е = . 000
Ниже приведены рестрикционные сайты двух последовательностей.
E. coli
HpaII (CCGG) 187 229 254 272 283 443 463 502
506 592
HphI (GGTGA) 177 552
HphI (TCACC) 285 597
MboI (GATC) 135 564
S. typhimurium
HpaII (CCGG) 187 248 253 283 443 463 506
HphI (GGTGA) 177 552
MboI (GATC) 135 204 317 564
MnlI (CCTC) 417
Последовательность E. coli имеет сайты MboI (GATC) в позициях 135 и 564. Имеется фрагмент 429 bp, который можно обнаружить с помощью проб и областей 1 и 2. Тот же самый энзим имеет сайты в позициях 135, 204, 317 и 564 в последовательности S. typhimurium. С помощью пробы двух гомологичных областей можно обнаружить фрагменты 69, 113 и 247 bp.
Итак, с помощью этой пробы и энзима для E. coli получается идентифицирующая генетическая характеристика
429,
а для S. tuphimurium
69,
113,
247.
Э к с п е р и м е н т 11. Применение гена α-фетопротеина в качестве пробы.
Настоящий эксперимент показывает использование области гомологии в последовательности гена α-фетопротеина человека и крысы, а также и эндонуклеазы MnlI (GAGG).
Информационная кДНК α-фетопротеина человека (1578bp) следующая:
10 20 30 40 50
AGCTTGGCAT AGCTACCATC ACCTTTACCC AGTTTGTTCC GGAAGCCACC
TCGAACCGTA TCGATGGTAG TGGAAATGGG TCAAACAAGG CCTTCGGTGG
60 70 80 90 100
GAGGAGGAAG TGAACAAAAT GACTAGCGAT GTGTTGGCTG CAATGAAGAA
CTCCTCCTTC ACTTGTTTTA CTGATCGCTA CACAACCGAC GTTACTTCTT
110 120 130 140 150
AAACTCTGGC GATGGGTGTT TAGAAAGCCA GCTATCTGTG TTTCTGGATG
TTTGAGACCG CTACCCACAA ATCTTTCGGT CGATAGACAC AAAGACCTAC
160 170 180 190 200
AAATTTGCCA TGAGACGGAA CTCTCTAACA AGTATGGACT CTCAGGCTGC
TTTAAACGGT ACTCTGCCTT GAGAGATTGT TCATACCTGA GAGTCCGACG
210 220 230 240 250
TGCAGCCAAA GTGGAGTGGA AAGACATCAG TGTCTGCTGG CACGCAAGAA
ACGTCGGTTT CACCTCACCT TTCTGTAGTC ACAGACGACC GTGCGTTCTT
260 270 280 290 300
GACTGCTCCG GCCTCTGTCC CACCCTTCCA GTTTCCAGAA CCTGCCGAGA
CTGACGAGGC CGGAGACAGG GTGGGAAGGT CAAAGGTCTT GGACGGCTCT
310 320 330 340 350
GTTGCAAAGC ACATGAAGAA AACAGGGCAG TGTTCATGAA CAGGTTCATC
CAACGTTTCG TGTACTTCTT TTGTCCCGTC ACAAGTACTT GTCCAAGTAG
360 370 380 390 400
TATGAAGTGT CAAGGAGGAA CCCCTTCATG TATGCCCCAG CCATTCTGTC
ATACTTCACA GTTCCTCCTT GGGGAAGTAC ATACGGGGTC GGTAAGACAG
410 420 430 440 450
CTTGGCTGCT CAGTACGACA AGGTCGTTCT GGCATGCTGC AAAGCTGACA
GAACCGACGA GTCATGCTGT TCCAGCAAGA CCGTACCACG TTTCGACTGT
460 470 480 490 500
ACAAGGAGGA GTGCTTCCAG ACAAAGAGAG CATCCATTGC AAAGGAATTA
TGTTCCTCCT CACGAAGGTC TGTTTCTCTC GTAGGTAACG TTTCCTTAAT
510 520 530 540 550
AGAGAAGGAA GCATGTTAAA TGAGCATGTA TGTTCAGTGA TAAGAAAATT
TCTCTTCCTT CGTACAATTT ACTCGTACAT ACAAGTCACT ATTCTTTTAA
560 570 580 590 600
TGGATCCCGA AACCTCCAGG CAACAACCAT TATTAAGCTA AGTCAAAAGT
ACCTAGGGCT TTGGAGGTCC GTTGTTGGTA ATAATTCGAT TCAGTTTTCA
610 620 630 640 650
TAACTGAAGC AAATTTTACT GAGATTCAGA AGCTGGCCCT GGATGTGGCT
ATTGACTTCG TTTAAAATGA CTCTAAGTCT TCGACCGGGA CCTACACCGA
660 670 680 690 700
CACATCCACG AGGAGTGTTG CCAAGGAAAC TCGCTGGAGT GTCTGCAGGA
GTGTAGGTGC TCCTCACAAC GGTTCCTTTG AGCGACCTCA CAGACGTCCT
710 720 730 740 750
TGGGGAAAAA GTCATGACAT ATATATGTTC TCAACAAAAT ATTCTGTCAA
ACCCCTTTTT CAGTACTGTA TATATACAAG AGTTGTTTTA TAAGACAGTT
760 770 780 790 800
GCAAAATAGC AGAGTGCTGC AAATTACCCA TGATCCAACT AGGCTTCTGC
CGTTTTATCG TCTCACGACG TTTAATGGGT ACTAGGTTGA TCCGAAGACG
810 820 830 840 850
ATAATTCACG CAGAGAATGG CGTCAAACCT GAAGGCTTAT CTCTAAATCC
TATTAAGTGC GTCTCTTACC GCAGTTTGGA CTTCCGAATA GAGATTTAGG
860 870 880 890 900
AAGCCAGTTT TTGGGAGACA GAAATTTTGC CCAATTTTCT TCAGAGGAAA
TTCGGTCAAA AACCCTCTGT CTTTAAAACG GGTTAAAAGA AGTCTCCTTT
910 920 930 940 950
AAATCATGTT CATGGCAAGC TTTCTTCATG AATACTCAAG AACTCACCCC
TTTAGTACAA GTACCGTTCG AAAGAAGTAC TTATGAGTTC TTGAGTGGGG
960 970 980 990 1000
AACCTTCCTG TCTCAGTCAT TCTAAGAATT GCTAAAACGT ACCAGGAAAT
TTGGAAGGAC AGAGTCAGTA AGATTCTTAA CGATTTTGCA TGGTCCTTTA
1010 1020 1030 1040 1050
ATTGGAGAAG TGTTCCCAGT CTGGAAATCT ACCTGGATGT CAGGACAATC
TAACCTCTTC ACAAGGGTCA GACCTTTAGA TGGACCTACA GTCCTGTTAG
1060 1070 1080 1090 1100
TGGAAGAAGA ATTGCAGAAA CACATCGAGG AGAGCCAGGC ACTGTCCAAG
ACCTTCTTCT TAACGTCTTT GTGTAGCTCC TCTCGGTCCG TGACAGGTTC
1110 1120 1130 1140 1150
CAAAGCTGCG CTCTCTACCA GACCTTAGGA GACTACAAAT TACAAAATCT
GTTTCGACGC GAGAGATGGT CTGGAATCCT CTGATGTTTA ATGTTTTAGA
1160 1170 1180 1190 1200
GTTCCTTATT GGTTACACGA GGAAAGCCCC TCAGCTGACC TCAGCAGAGC
CAAGGAATAA CCAATGTGCT CCTTTCGGGG AGTCGACTGG AGTCGTCTCG
1210 1220 1230 1240 1250
TGATCGACCT CACCGGGAAG ATGGTGAGCA TTGCCTCCAC GTGCTGCCAG
ACTAGCTGGA GTGGCCCTTC TACCACTCGT AACGGAGGTG CACGACGGTC
1260 1270 1280 1290 1300
CTCAGCGAGG AGAAATGGTC CGGCTGTGGT GAGGGAATGG CCGACATTTT
GAGTCGCTCC TCTTTACCAG GCCGACACCA CTCCCTTACC GGCTGTAAAA
1310 1320 1330 1340 1350
CATTGGACAT TTGTGTATAA GGAATGAAGC AAGCCCTGTG AACTCTGGTA
GTAACCTGTA AACACATATT CCTTACTTCG TTCGGGACAC TTGAGACCAT
1360 1370 1380 1390 1400
TCAGCCACTG CTGCAACTCT TCGTATTCCA ACAGGAGGCT ATGCATCACC
AGTCGGTGAC GACGTTGAGA AGCATAAGGT TGTCCTCCGA TACGTAGTGG
1410 1420 1430 1440 1450
AGTTTTCTGA GGGATGAAAC CTATGCCCCT CCCCCATTCT CTGAGGATAA
TCAAAAGACT CCCTACTTTG GATACGGGGA GGGGGTAAGA GACTCCTATT
1460 1470 1480 1490 1500
ATTCATCTTC CACAAGGATC GTGCCAAGCT CGGCAAAGCC CTACAGACCA
TAAGTAGAAG GTGTTCCTAG CACGGTTCGA GCCGTTTCGG GATGTCTGGT
1510 1520 1530 1540 1550
TGAAACAAGA GCTTCTCATT AACCTGGTGA AGCAAAAGCC TGAACTGACA
ACTTTGTTCT CGAAGAGTAA TTGGACCACT TCGTTTTCGG ACTTGACTGT
1560 1570
GAGGAGCAGC TGGCGGCTGT CACTGCAG
CTCCTCGTCG ACCGCCGACA GTGACGTC
Конечная 3' кДНК α-фетопротеина крыс следующая (540 bp):
10 20 30 40 50
GAGGGACTGG CCGACATTTA CATTGGACAC TTGTGTTTAA GACATGAGGC
CTCCCTGACC GGCTGTAAAT GTAACCTGTG AACACAAATT CTGTACTCCG
60 70 80 90 100
AAACCCTGTG AACTCCGGTA TCAACCACTG CTGCAGTTCC TCGTATTCCA
TTTGGGACAC TTGAGGCCAT AGTTGGTGAC GACGTCAAGG AGCATAAGGT
110 120 130 140 150
ACAGGAGGCT CTGCATCACC AGCTTTCTGA GGGACGAAAC CTACGTCCCT
TGTCCTCCGA GACGTAGTGG TCGAAAGACT CCCTGCTTTG GATGCAGGGA
160 170 180 190 200
CTACCATTCT CTGCGACAAA TTCATCTTCC ACAAGGAATC TGTGCCAAGC
GATGGTAAGA GACGCTGTTT AAGTAGAAGG TGTTCCTTAG ACACGGTTCG
210 220 230 240 250
TCAGGGCCGA GCACCACAGA CCATGAAACA AGAGCTTCTC ATTAACCTAG
AGTCCCGGCT CGTGGTGTCT GGTACTTTGT TCTCGAAGAG TAATTGGATC
260 270 280 290 300
TGAAACAAAA GCCTGAAATG ACAGAGGAGC AGCACGCGGC TGTCACTGCT
ACTTTGTTTT CGGACTTTAC TGTCTCCTCG TCGTGCGCCG ACAGTGACGA
310 320 330 340 350
GATTTCTCTG GCCTCTTGGA GAAGTGCTGC AAAGACCAGG ATCAGGAAGC
CTAAAGAGAC CGGAGAACCT CTTCACGACG TTTCTGGTCC TAGTCCTTCG
360 370 380 390 400
CTGTTTCGCA AAAGAGGTCC AAGTTGATTT CCAAACTCGT GAGGCTTTGG
GACAAAGCGT TTTCTCCAGG TTCAACTAAA GGTTTGAGCA CTCCGAAACC
410 420 430 440 450
GGGTTTAAAC ATCTCCAAGA GGAAGAAAGG ACAAAAAAAT GTGTCGACGC
CCCAAATTTG TAGAGGTTCT CCTTCTTTCC TGTTTTTTTA CACAGCTGCG
460 470 480 490 500
TTTGGTGTGA GCTTTTCGGT TTGATGGTAA CTGGTGGAGA CTTCCATGTG
AAACCACACT CGAAAAGCCA AACTACCATT GACCACCTCT GAAGGTACAC
510 520 530 540
GGATTTCTAT GCCTAAGGAA TAAAGACTTT TCAACTGTTA
CCTAAAGATA CGGATTCCTT ATTTCTGAAA AGTTGACAAT
Две последовательности имеют следующие гомологичные области (сверху - последовательность человека, а снизу - крысы):
1367 CTC GTATTCCAACAGGAGGCT
GTATTCCAACAGGAGGCT TGCATCACCAG
TGCATCACCAG TTT
TTT CTGAGGGA
CTGAGGGA GAAACCTA
GAAACCTA G
G
90 CTC---GTATTCCAACAGGAGGCTCTGCATCACCAGCTTT-CTGAGGGACGAAACCTACG CCCTC
CCCTC CC
CC ATTCTCTG
ATTCTCTG GA
GA AA
AA TTCATCTTCCACAAGGA
TTCATCTTCCACAAGGA TC
TC GTGCCAAGCTC
GTGCCAAGCTC
TCCCTCTACC-ATTCTCTG-CGACAAA-TTCATCTTCCACAAGGAATCTGTGCCAAGCTC GG
GG C
C A
A GC
GC CC
CC ACAGACCATGAAACAAGAGCTTCTCATTAACCT
ACAGACCATGAAACAAGAGCTTCTCATTAACCT GTGAA
GTGAA CAAAA
CAAAA
AGGGCCGA-GCACC-ACAGACCATGAAACAAGAGCTTCTCATTAACCTAGTGAAACAAAA
GCCTGAA TGACAGAGGAGCAGC
TGACAGAGGAGCAGC GCGGCTGTCACTGC 1576
GCGGCTGTCACTGC 1576
GCCTGAAATGACAGAGGAGCAGCACGCGGCTGTCACTGC 299
Z = 85,845
Р (219, 188) = . 000Е + 00 Е = . 000
Для человека и крысы имеются следующие рестрикционные сайты:
Человек
MboII (GAAGA) 108 261 328 1066 1069 1230
MboII (TCTTC 881 916 1361 1449
MnlI (CCTC) 273 574 1190 1200 1219 1245 1439
MnlI (GAGG) 44 47 358 449 653 887 1070 116
1250 1274 1378 1402 1436 1544
Крыса
MboII (GAAGA) 435
MboII (TCTTC) 168
MnlI (CCTC) 100 159 323
MnlI (GAGG) 39 98 122 267 357 384 412
Можно вычислить, что фрагменты, содержащие участок сохранившейся последовательности 24, 34, 104 и 108 p, описывают человеческую ДНК. Фрагменты 24, 59, 90 и 145 bp описывают ДНК крысы. Хотя обе последовательности содержат фрагмент 24 bp, значение имеет набор фрагментов (таксономическая характеристика).
Использование: молекулярная биология, а именно идентификация различных прокариотических и эукариотических организмов. Сущность изобретения: выделяют ДНК из неизвестного организма, обрабатывают рестриктазой, гибридизируют на фильтрах с меченой 5 S, 16 S или 23 S pРНК в случае прокармотного или с 18 S или 28 S pРНК в случае эукариотного организма, детектируют гибридные нуклеотидные последовательности и идентифицируют организм.
СПОСОБ ИДЕНТИФИКАЦИИ ВИДОВ НЕИЗВЕСТНОГО ОРГАНИЗМА, предусматривающий выделение ДНК неизвестного организма, обработку ДНК рестриктазой, разделение фрагментов ДНК, гибридизацию с нуклеотидным зондом, детекцию нуклеотидных последовательностей с последующей идентификацией организма, отличающийся тем, что, с целью унификации способа, в качестве зонда используют 5S, 16S или 23S рРНК при идентификации прокариотического организма или 18S или 28S рРНК при идентификации эукариотического организма.
Авторы
Даты
1994-04-30—Публикация
1984-11-20—Подача