ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Представляемое изобретение касается генов, кодирующих раффинозосинтазу, и их использования.
ОПИСАНИЕ ИЗВЕСТНОГО УРОВНЯ ТЕХНИКИ
Олигосахариды семейства раффиноз являются производными сахарозы, которые представлены общей формулой o-α-D-галактопиранозил-(1→6)n-o-α-D-глюкопиранозил-(1→2)-β-D-фруктофуранозил. Их называют "раффинозами", когда n=1, "стахиозами" при n=2, "вербаскозами" при n=3, "айюгозами" при n=4.
Было установлено, что олигосахариды семейства раффиноз обладают позитивным эффектом в отношении качества кишечной бактериофлоры, если они присутствуют в пище в соответствующем количестве. С учетом этого олигосахариды семейства раффиноз уже применяются в качестве функционального пищевого материала для добавок в некоторых видах пищевых продуктов, а также используются в специальных диетических и лечебных продуктах. С другой стороны, олигосахариды семейства раффиноз не перевариваются и не всасываются у млекопитающих, в частности у человека, но ассимилируются и разрушаются энтеробактериями с образованием газов, что приводит к метеоризму и заболеваниям, связанным с нарушением усвоения пищи. Таким образом, желательным представляется точное регулирование количества олигосахаридов семейства раффиноз в пищевых продуктах и кормах.
Олигосахариды семейства раффиноз синтезируются в соответствующей биосинтетической системе, у многих видов растений начинающейся с сахарозы. В норме эта биосинтетическая система вовлечена в реакцию последовательного прибавления галактозильных групп от галактинола с помощью связи α-(1→6) к гидроксильной группе, присоединенной к атому углерода в 6-м положении остатка D-глюкозы в молекуле сахарозы. Раффинозосинтаза - фермент, участвующий в реакции образования раффинозы за счет обеспечения способности D-галактозильной группы, производной от галактинола, образовывать связь типа α-(1→6) с гидроксильной группой, присоединенной к атому углерода в 6-м положении остатка D-глюкозы в молекуле сахарозы на первом этапе данной биосинтетической системы. Подтверждается, что этот фермент обеспечивает этап, лимитирующий скорость обозначенной системы синтеза, т.е. данный фермент весьма важен в контроле биосинтеза олигосахаридов семейства раффиноз.
Таким образом, способ контроля уровня экспрессии или активности раффинозосинтазы у растений с использованием гена раффинозосинтазы является эффективным способом контроля системы биосинтеза олигосахаридов семейства раффиноз у растений с целью увеличения или уменьшения выработки раффиноз у растений. Соответственно, выделение гена раффинозосинтазы, который может быть использован в таком способе, становится желательным.
Основным объектом данного изобретения является выделение новых генов раффинозосинтаз из растений.
Этот и другие объекты и преимущества данного изобретения станут понятны специалистам в данной области в процессе ознакомления с описанием.
С учетом вышеизложенных обстоятельств авторы интенсивно исследовали и добились успеха в выделении новых генов, кодирующих фермент раффинозосинтазу из различных видов растений. Таким образом, данное изобретение является завершенным.
Таким образом, настоящее изобретение представляет
1. Ген раффинозосинтазы, включающий нуклеотидную последовательность, гибридизующуюся с нуклеотидной последовательностью, отобранной из группы, состоящей из:
(a) нуклеотидной последовательности, кодирующей аминокислотную последовательность SEQ ID NO:1,
(b) нуклеотидной последовательности SEQ ID NO:2,
(c) нуклеотидной последовательности, кодирующей аминокислотную последовательность SEQ ID NO:3,
(d) нуклеотидной последовательности, представленной 236-2584 нуклеотидами в нуклеотидной последовательности SEQ ID NO:4,
(e) нуклеотидной последовательности, кодирующей аминокислотную последовательность SEQ ID NO:5,
(f) нуклеотидной последовательности, представленной 134-2467 нуклеотидами в нуклеотидной последовательности SEQ ID NO:6,
(g) нуклеотидной последовательности, кодирующей аминокислотную последовательность SEQ ID NO:7,
(h) нуклеотидной последовательности, представленной 1-1719 нуклеотидами в нуклеотидной последовательности SEQ ID NO 8, в жестких условиях и кодирования белка, способного связывать галактозильную группу по связи α-(1→6) с гидроксильной группой, присоединенной к атому углерода в 6-м положении остатка D-глюкозы в молекуле сахарозы с образованием раффинозы.
2. Ген раффинозосинтазы, включающий нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:1.
3. Ген раффинозосинтазы, включающий нуклеотидную последовательность SEQ ID NO:2.
4. Ген раффинозосинтазы, включающий нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:3.
5. Ген раффинозосинтазы, включающий нуклеотидную последовательность, представленной 236-2584 нуклеотидами в нуклеотидной последовательности SEQ ID NO:4.
6. Ген раффинозосинтазы, включающий нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:5.
7. Ген раффинозосинтазы, включающий нуклеотидную последовательность, представленной 134-2467 нуклеотидами в нуклеотидной последовательности SEQ ID NO:6.
8. Ген раффинозосинтазы, включающий нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:7.
9. Ген раффинозосинтазы, включающий нуклеотидную последовательность, представленную 1-1719 нуклеотидами в нуклеотидной последовательности SEQ ID NO 8.
10. Ген раффинозосинтазы, включающий нуклеотидную последовательность, представленную SEQ ID NO:6 или SEQ ID NO:8.
11. Нуклеиновая кислота, включающая частичную нуклеотидную последовательность гена раффинозосинтазы по любому из предыдущих пунктов 1-10.
12. Способ выявления нуклеиновой кислоты, содержащий ген раффинозосинтазы, который включает выявление названной нуклеиновой кислоты, с помощью гибридизации с использованием меченой нуклеиновой кислоты по п.11 в качестве зонда.
13. Способ амплификации нуклеиновой кислоты, включающей ген раффинозосинтазы, который включает амплификацию названной нуклеиновой кислоты с помощью полимеразной цепной реакции (ПЦР) с использованием нуклеиновый кислоты по п.11 в качестве праймера.
14. Способ получения гена раффинозосинтазы, который включает следующие этапы:
- выявление нуклеиновой кислоты, содержащей названный ген раффинозосинтазы, с помощью гибридизации с использованием меченой нуклеиновой кислоты по п.11 в качестве зонда и
- выделение идентифицированной нуклеиновой кислоты.
15. Способ получения гена раффинозосинтазы, включающий следующие этапы:
- амплификация нуклеиновой кислоты, содержащей названный ген раффинозосинтазы с помощью ПЦР с использованием нуклеиновой кислоты по п.11 в качестве затравки, и
- выделение амплифицированной нуклеиновой кислоты.
16. Нуклеиновая кислота, включающая ген раффинозосинтазы по любому из п.п.1-10, или нуклеиновая кислота по п.10, которая присоединена к нуклеиновой кислоте, проявляющей промоторную активность в клетке-хозяине.
17. Вектор, включающий ген раффинозосинтазы по любому из п.п.1-10.
18. Трансформант, в котором ген раффинозосинтазы по любому из п.п.1-10 введен в клетку-хозяина.
19. Трансформант, в котором нуклеиновая кислота по п.16 введена в клетку-хозяина.
20. Трансформант, в котором вектор по п.17 введен в клетку-хозяина.
21. Трансформант по любому из предыдущих п.п.18-20, в котором хозяином является микроорганизм.
22. Трансформант по любому из предыдущих п.п.18-20, в котором хозяином является растение.
23. Способ получения раффинозосинтазы, включающий следующие этапы:
- культивирование и выращивание трансформанта по любому из предыдущих п.п.18-22 с целью получения раффинозосинтазы, и
- сбор раффинозосинтазы.
24. Раффинозосинтаза, включающая аминокислотную последовательность SEQ ID NO 1.
25. Раффинозосинтаза, включающая аминокислотную последовательность SEQ ID NO 3.
26. Раффинозосинтаза, включающая аминокислотную последовательность SEQ ID NO 5.
27. Раффинозосинтаза, включающая аминокислотную последовательность SEQ ID NO 7.
Термин "нуклеиновая кислота", используемый в тексте, обозначает олигомерное соединение или высокомолекулярное соединение, которое обычно называют "ДНК" или "РНК".
Генно-инженерные методики, описываемые ниже, могут быть применены, например, в соответствии со способами, описанными в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Press, ISBN 0-87969-309-6; "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-X; "Current Protocols in Protein Science" (1995), J.Wiley & Sons Inc., ISBN 0-471-11184-8.
Гены согласно данному изобретению могут быть выделены из сои, растений, относящихся к семействам маревых, Chenopodiaceae (например, свеклы и др.), и крестоцветных, Cruciferae (такие как сарептская горчица, брюква и др.). Специфические примеры генов в настоящем изобретении включают те гены, которые имеют нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:1; нуклеотидную последовательность SEQ ID NO:2; нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:3; нуклеотидную последовательность, представленную SEQ ID NO:4 или 236-2584 нуклеотидами в нуклеотидной последовательности SEQ ID NO:4; нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:5; нуклеотидную последовательность, представленную SEQ ID NO:6 или 134-2467 нуклеотидами в нуклеотидной последовательности SEQ ID NO:6; нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO:7; нуклеотидную последовательность, представленную SEQ ID No.8 или 1-1719 нуклеотидами в нуклеотидной последовательности SEQ ID No.8.
Гены данного изобретения могут быть получены, например, следующим способом.
Соответственно, гены, данного изобретения, производные от сои, могут быть получены, например, следующим способом.
Например, ген может быть получен с помощью гибридизации с использованием нуклеиновой кислоты, имеющей нуклеотидную последовательность SEQ ID NO 2, или части нуклеотидной последовательности, используемой в качестве зонда для идентификации фрагмента нуклеиновой кислоты, который гибридизует с зондом, в ДНК, выделенным из растений сои, с последующим выделением идентифицированной нуклеиновой кислоты.
В этом способе сначала готовится нуклеиновая кислота, рассматриваемая в качестве зонда. Таковой, например, может быть нуклеиновая кислота, включающая олигонуклеотид, синтезированный химическим путем с помощью традиционных методов на основе нуклеотидной последовательности SEQ ID NO:2. В качестве специфического примера представлена нуклеиновая кислота с 800-899-й нуклеотидами в нуклеотидной последовательности SEQ ID NO:2.
Напротив, представленный в изобретении ген, производный сои, может быть получен следующим способом.
Например, ткани сои (Glycine max) замораживают в жидком азоте и измельчают физическим воздействием в ступке или аналогичном устройстве с целью получения измельченного тканевого порошка. РНК экстрагируют из этого порошка с помощью стандартного метода: для этой цели может быть использован промышленно и коммерчески доступный набор реактивов для РНК-экстракции. РНК выделяют из полученного таким методом экстракта с помощью осаждения этиловым спиртом. Фракционирование РНК с полиадениловыми "хвостами" осуществляют из выделенной таким способом РНК стандартным способом. Для этой цели может быть использована промышленно доступная олиго-дТ колонка. кДНК синтезируют на матрице полиаденилированной хвостовой РНК с помощью стандартного метода. Синтез может быть осуществлен с использованием стандартного набора реактивов для синтеза кДНК. ДНК амплифицируют с помощью ПЦР с использованием ранее выделенной кДНК в качестве матрицы и затравок, определенных и синтезированных по параметрам нуклеотидной последовательности SEQ ID NO 2. Более конкретно, затравками, например, могут быть праймеры 11 и 12, приведенные ниже в перечне 1. По окончании ПЦР с данными затравками и матрицей кДНК, полученной из сои, могут быть получены гены сои по данному изобретению (т.е. "ген раффинозосинтазы с нуклеотидной последовательностью, кодирующей аминокислотную последовательность согласно SEQ ID NO 1" и "ген раффинозосинтазы с нуклеотидной последовательностью согласно SEQ ID NO 2").
Амплифицированная ДНК может быть клонирована согласно стандартному методу, описанному, например, в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press; или "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-Х. С другой стороны, клонирование может быть проведено, например, с использованием стандартного набора реактивов для клонирования, такого как ТА набор для клонирования (Invitrogen) и стандартного плаэмидного вектора типа pBluescript II (Stratagene). Нуклеотидная последовательность ДНК-клона может быть определена способом дидезокситерминирования, например, по способу Сэйнджера (F.Sanger, S.Nicklen, A.R.Coulson, 1977, Proc. Natl. Acad. Sci. USA, 74, 5463-5467). В частности, предпочтительным является использование стандартного набора ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction, производимого фирмой Perkin-Elmer.

Гены согласно настоящему изобретению, производные от растений семейства Chenopodiaceae, таких как свекла, могут быть получены следующим способом.
Например, ткани растения семейства Chenopodiaceae, такого как обыкновенная свекла (Beta vulgaris), замораживают в жидком азоте и физически измельчают в ступке или аналогичном устройстве с получением тканевого порошка. Из этого тканевого порошка РНК экстрагируют стандартным способом. Может быть использован стандартный набор для РНК-экстракции. РНК восстанавливают из полученного РНК-экстракта с помощью осаждения этанолом. РНК с полиадениловыми "хвостами" фракционируют из восстановленной РНК с помощью стандартного способа. Стандартная олиго-дТ колонка может быть применена для такого фракционирования. кДНК синтезируют из полиаденилированной хвостовой РНК с помощью стандартного способа. Синтез может быть проведен с использованием стандартного набора для синтеза кДНК. ДНК амплифицируют с помощью ПЦР с использованием полученной кДНК в качестве матрицы и затравок, определенных и химически синтезированных по параметрам нуклеотидной последовательности SEQ ID NO 4. Более детально, что касается затравок, такие затравки 21 и 22 приведены ниже в перечне 2. По завершении ПЦР с использованием этих затравок кДНК-матрицы, выделенной из свеклы, гены свеклы согласно настоящему изобретению (т.е. "ген раффинозосинтазы с нуклеотидной последовательностью, кодирующей аминокислотную последовательность SEQ ID NO 3" и "ген раффинозосинтазы с нуклеотидной последовательностью SEQ ID NO 4") могут быть выделены. В частности ПЦР-затравки могут быть также определены и синтезированы по нуклеотидной последовательности seq ID no 4. Например, с целью амплифицирования "гена раффинозосинтазы, включающего нуклеотидную последовательность, представленную 236-2584 нуклеотидами в нуклеотидной последовательности SEQ ID NO 4" олигонуклеотиды с нуклеотидными последовательностями, представленными затравками 23 и 24 в перечне 2, лучше всего синтезировать и использовать в качестве затравок.
Амплифицированная ДНК может быть клонирована согласно стандартному методу, описанному, например, в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press или "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-Х. С другой стороны, клонирование может быть проведено, например, с использованием стандартного набора реактивов для клонирования, такого как ТА Набора для клонирования (Invitrogen) и стандартного плазмидного вектора типа pBluescript II (Stratagene). Нуклеотидная последовательность ДНК-клона может быть определена способом дидезокситерминирования, описанного F.Sanger, S.Nicklen, A.R.Coulson, 1977, Proc. Natl. Acad. Sci. USA, 74, 5463-5467. Например, предпочтительным является использование стандартного набора, такого как ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction (Perkin-Elmer).

Гены данного изобретения, выделенные из растений семейства крестоцветных, Cruciferae, таких как сарептская горчица (Brassica juncea) и брюква (Brassica napus), могут быть получены с помощью следующего способа.
Например, ткани растений семейства Cruciferae, таких как горчица или брюква, замораживают в жидком азоте и физически измельчают в ступке или аналогичном устройстве с получением тканевого порошка. Из этого тканевого порошка РНК экстрагируют стандартным способом. Может быть использован стандартный набор для РНК-экстракции. РНК восстанавливают из полученного РНК-экстракта с помощью осаждения этанолом. РНК с полиадениловыми "хвостами" фракционируют из восстановленной РНК с помощью стандартного метода. Стандартная олиго-дТ колонка может быть применена для такого фракционирования. кДНК синтезируют из полиаденилированной хвостовой РНК, полученной с помощью стандартного метода. Синтез может быть проведен с использованием стандартного набора для синтеза кДНК. ДНК амплифицируют с помощью ПЦР с использованием полученной выше кДНК в качестве матрицы и затравок, определенных и химически синтезированных по параметрам нуклеотидной последовательности SEQ ID NO 6. Например, при проведении ПЦР с использованием в качестве матрицы кДНК, полученной из горчицы (Brassica juncea), и затравок 33 и 34, приведенных ниже в перечне 3, гены растений семейства Cruciferae согласно настоящему изобретению (т.е. "ген раффинозосинтазы с нуклеотидной последовательностью, кодирующей аминокислотную последовательность SEQ ID NO 5" и "ген раффинозосинтазы с нуклеотидной последовательностью, представленной 1-2654 нуклеотидами в нуклеотидной последовательности SEQ ID NO 6") могут быть получены. В частности ПЦР-затравки могут быть определены и синтезированы на основе нуклеотидной последовательности SEQ ID NO 6.
Например, с целью амплификации ДНК, кодирующей участок открытой рамки считываний "гена раффинозосинтазы с нуклеотидной последовательностью, кодирующей белок с аминокислотной последовательностью SEQ ID NO 5" и "ген раффинозосинтазы с нуклеотидной последовательностью, представленной 134-2467 нуклеотидами SEQ ID No.6", олигонуклеотиды, с нуклеотидными последовательностями, представленными затравками 35 и 36 в перечне 3, предпочтительно синтезировать и использовать в качестве праймеров.
Амплифицированная ДНК может быть клонирована согласно стандартному методу, описанному, например, в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press или "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-Х. С другой стороны, клонирование может быть проведено, например, с использованием стандартного набора реактивов, такого как набор ТА Набор для клонирования (Invitrogen) и стандартного плазмидного вектора типа pBluescript II (Stratagene). Нуклеотидные последовательности ДНК-клона могут быть определены методом дидезокситерминирования, например, по методу Сэйнджера (F.Sanger, S.Nicklen, A.R. Coulson, 1977, Proc. Natl. Acad. Sci. USA, 74, 5463-5467). Например, предпочтительным является использование стандартного набора ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction (Perkin-Elmer).

Далее, нуклеиновую кислоту, имеющую частичную нуклеотидную последовательность гена по данному изобретению (здесь и далее определяемой как "фрагмент гена"), выделяемую описанным выше методом, метят и затем используют в качестве зонда в гибридизационном методе. Зонд может быть гибридизован, например, с ДНК, производной сои, растения семейства Chenopodiaceae или растения семейства Cruciferae с целью выявления нуклеиновой кислоты, специфически связывающейся с зондом, что позволит идентифицировать нуклеиновую кислоту, включающую ген раффинозосинтазы.
Что касается ДНК, производной сои, растения семейства Chenopodiaceae, такого как свекла, или растения семейства Cruciferae, таких как горчица или брюква, могут быть использованы, например, библиотека кДНК или геномная ДНК-библиотека этих растений. В качестве генной библиотеки могут служить стандартная генная библиотека как таковая, или библиотека, сформированная с помощью стандартного метода конструирования библиотек, например, описанного в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press или "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-X.
Что касается метода гибридизации, то могут быть применены, например, методы гибридизации на плашках или в колониях: их выбор зависит от типа вектора, используемого при конструировании библиотеки. Более детально, в случае, когда библиотека для дальнейшего применения формируется в составе фагового вектора, подходящий микроорганизм-хозяин смешивается с библиотечным фагом в инфективных условиях с целью получения трансформантов. Далее трансформант смешивают с мягкой агаровой средой, а полученную смесь распластывают на агаре. Затем смесь культивируют при 37°С до тех пор, пока не образуется плашки необходимого размера. Когда же библиотеку для дальнейшего анализа создают с помощью плазмидного вектора, плазмиду вводят в подходящий микроорганизм-хозяин с целью получения трансформантов. Полученные трансформанты разбавляют до нужной концентрации и полученный раствор помещают на агар, после чего культивируют при 37°С до тех пор, пока не образуется колонии необходимого размера. В любом варианте получения обозначенных выше библиотек, мембранный фильтр помещают на поверхность агара после указанного варианта культивирования, в результате чего фаг или трансформант перемещается на мембрану. Эту мембрану денатурируют щелочью, которую затем нейтрализуют и, например, когда используют нейлоновую мембрану, ее облучают ультрафиолетом, что проводит к фиксации ДНК фага или трансформанта на мембране. Эта мембрана становится пригодной для метода гибридизации, в котором фрагмент гена, являющийся частью нуклеотидной последовательности гена по данному изобретению и помеченный стандартным методом (здесь и далее обозначаемый как "меченый фрагмент гена"), используется в качестве зонда. Ссылку на описание этого способа можно найти в "DNA cloning, a practical approach" (1985), ed. D.M.Glover, IRL PRESS, ISBN 0-947946-18-7. Существуют различные реагенты и температурные режимы, применяемые в гибридизации. Например, в целом, предварительная гибридизация проводится путем погружения мембраны в прегибридизационный раствор [6×SSC (0,9 М NaCl, 0,09 М лимонной кислоты), 0,1-1 об.% SDS, 100 мкг/мл денатурированной ДНК сперматозоидов лосося] и инкубации при 65°С в течение 1 часа. Затем гибридизацию осуществляют путем добавления и смешивания меченого фрагмента гена и инкубирования мембраны при 42-68°С от 4 до 16 часов.
В настоящем изобретении под "жесткими условиями" подразумевается инкубация, например, при 65-68°С гибридизации.
После гибридизации мембрану вынимают и промывают раствором 2×SSC, содержащим 0,1-1 об.% SDS, еще раз промывают раствором 0,2×SSC, содержащим 0.1-1 об.% SDS и затем высушивают. Мембрану анализируют, например, авторадиографически или с помощью других способов с целью определения положения зонда на мембране и определения положения на мембране нуклеиновой кислоты, имеющей нуклеотидную последовательность, гомологичную использованному зонду. Клон, соответствующий положению нуклеиновой кислоты, идентифицированному таким способом на мембране, маркируют на исходном агаре и позитивный клон отбирают так, чтобы этот включающий нуклеиновую кислоту клон мог быть выделен. Такую процедуру идентификации повторяют с целью очистки клона, включающего нуклеиновую кислоту.
С другой стороны, стандартный набор, такой как GENE TRAPPER cDNA Positive Section System (производства GibcoBRL) может быть использован. В этом методе, во-первых, библиотеку одноцепочечных фрагментов ДНК гибридизуют с биотинилированным фрагментом гена (т.е. с зондом) с последующим добавлением связанных со стрептавидином магнитных шариков и перемешивают. Связанные со стрептавидином намагниченные шарики удаляют из смеси с помощью магнита: таким образом одноцепочечную ДНК, включающую нуклеотидную последовательность, гомологичную той, что использована в зонде, которая оказалась прикрепленной к этим шарикам опосредованно через фрагмент гена, биотин и стрептавидин, собирают и выявляют. Собранная одноцепочечная ДНК может быть преобразована в двухцепочечную форму путем проведения реакции с подходящей ДНК-полимеразой с использованием подходящего олигонуклеотида в качестве затравки.
Как было описано выше, нуклеиновая кислота, включающая ген раффинозосинтазы, может быть получена путем выявления нуклеиновой кислоты, гибридизующейся с фрагментом гена в ДНК генной библиотеки, выделенной из сои, растения семейства Chenopodiaceae или растения семейства Cruciferae, очистки клона, содержащего нуклеиновую кислоту, и выделения фаговой или плазмидной ДНК из клона. С помощью составления карты рестрикции или определения нуклеотидной последовательности нуклеиновой кислоты, полученной таким образом, с помощью традиционного метода, может быть подтверждено получение нуклеиновой кислоты, включающей ген по данному изобретению.
Например, ген по данному изобретению из растения семейства Chenopodiaceae может быть подтвержден по следующему критерию.
Аминокислота, кодируемая нуклеотидной последовательностью, определенной выше указанным способом, имеет 75%-ную или более высокую степень гомологии с аминокислотной последовательностью, представленной 103-208 аминокислотами в аминокислотной последовательности SEQ ID NO 3;
80%-ную и более высокую степень гомологии с аминокислотной последовательностью, представленной 255-271 аминокислотами в аминокислотной последовательности SEQ ID NO 3;
70%-ную и более высокую степень гомологии с аминокислотной последовательностью, представленной 289-326 аминокислотами в аминокислотной последовательности SEQ ID NO 3;
70%-ную и более высокую степень гомологии с аминокислотной последовательностью, представленной 610-696 аминокислотами в аминокислотной последовательности SEQ ID NO 3.
Ген по данному изобретению из растения семейства Cruciferae может быть подтвержден, например, по следующим критериям.
Аминокислотная последовательность, кодируемая установленной нуклеотидной последовательностью и проявляет
75%-ную или более высокую степень гомологии с аминокислотной последовательностью, представленной 111-213 аминокислотами в аминокислотной последовательности SEQ ID NO 5;
80%-ную или более высокую степень гомологии с аминокислотной последовательностью, представленной 260-275 аминокислотами в аминокислотной последовательности SEQ ID NO 5;
70%-ную или более высокую степень гомологии с аминокислотной последовательностью, представленной 293-325 аминокислотами в аминокислотной последовательности SEQ ID NO 5;
70%-ную или более высокую степень гомологии с аминокислотной последовательностью, представленной 609-695 аминокислотами в аминокислотной последовательности SEQ ID NO 5.
Термин "гомология", используемый здесь, обозначает численную пропорцию аминокислот в сегменте, которые идентичны таковым в другом сравниваемом сегменте, по отношению к общему числу аминокислот в первом сегменте путем сравнения сегментов, имеющих сходство по двум аминокислотным последовательностям. С этой точки зрения предпочтительно, чтобы сегменты, проявляющие сходство, имели большее число аминокислот. Такая гомология аминокислотных последовательностей может быть оценена с использованием стандартных программ геноанализа, таких как GENETIX (Software Kaihatu К.К.).
Далее, в соответствии с тем же подходом, который был описан выше, нуклеиновая кислота, включающая ген раффинозосинтазы, может быть выявлена путем гибридизации ДНК желательного организма с использованием фрагмента гена в качестве зонда с целью выявления нуклеиновой кислоты, с которой зонд специфически связывается (здесь и далее обозначается как способ выявления настоящего изобретения). Фрагмент гена, используемый в данном случае, может быть химически синтезирован с помощью стандартного способа на основе нуклеотидной последовательности SEQ ID NO 2, 4, 6 или 8. С другой стороны, он может быть получен с помощью ПЦР с использованием в качестве затравок олигонуклеотидов, химически синтезированных стандартным методом на основе нуклеотидных последовательностей SEQ ID NO 2, 4, 6 или 8.
Фрагмент гена может быть частью нетранслируемого участка гена раффинозосинтазы, равно как и частью открытой кодирующей рамки. Например, может быть использован олигонуклеотид, характеризующийся такой же нуклеотидной последовательностью, как часть этого 5’-расположения против хода транскрипции, такие как нуклеотиды 1-235 в нуклеотидной последовательности SEQ ID NO 4, нуклеотиды 1-133 в нуклеотидной последовательности SEQ ID NO 6 и подобные, или часть этого 3’-расположения по ходу транскрипции, такие как нуклеотиды 2588-2675 в нуклеотидной последовательности SEQ ID NO 4, нуклеотиды 2468-2676 в нуклеотидной последовательности SEQ ID NO 6 и подобные.
Когда ПЦР проводится с использованием фрагмента гена в качестве праймеров, имеется возможность амплификации нуклеиновой кислоты, включающей ген раффинозосинтазы из состава ДНК, выделенной из желательного организма (здесь и далее обозначается как амплификационный метод настоящего изобретения).
Более детально, например, олигонуклеотиды, включающие нуклеотидные последовательности фрагмента гена конструируют и химически синтезируют с помощью стандартного способа. В целом, предпочтительно, чтобы число нуклеотидов было большим с точки зрения обеспечения специфичности отжига. Также, однако, важно, чтобы число нуклеотидов не было слишком большим в связи с тем, что затравки сами по себе лабильны и могут образовывать структуры высшего порядка, что может приводить к снижению эффективности отжига, соответственно, усложняется процедура очистки после проведения синтеза. В норме, предпочтительны олигонуклеотиды, состоящие из 15-50 оснований. С этой точки зрения, основываясь на таблице кодонов, указывающих на соответствие аминокислот, кодируемых кодонами, смесь затравок может быть также синтезирована с использованием смеси множественных оснований таким образом, чтобы остаток в определенном положении в затравке менялся на другие основания в соответствии с изменчивостью кодонов, кодирующих одну и ту же аминокислоту. С другой стороны, такое основание как инозин, который может образовывать основания, способные связываться с множественными основаниями, может быть использовано вместо указанной выше смеси множественных оснований.
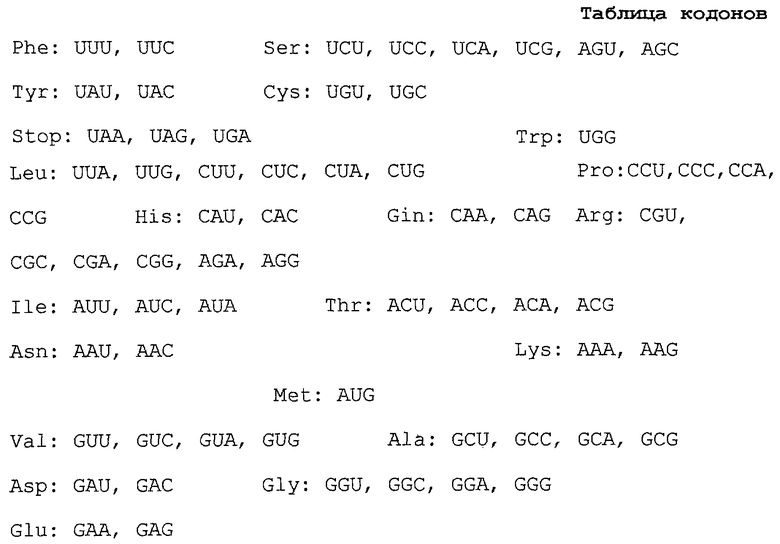
В приведенной таблице кодонов каждый кодон приведен в соответствии с нуклеотидными последовательностями в мРНК и их легких петель с 5’-конца. U представляет основание урацил в РНК, и ему соответствует основание тимин в ДНК.
Олигонуклеотид, имеющий такую же нуклеотидную последовательность, что и в кодирующей цепи двухцепочечной ДНК гена по данному изобретению, называют "смысловой затравкой", а тот, который включает нуклеотидную последовательность, комплементарную кодирующей цепи, называют "антисмысловой затравкой".
Смысловая затравка, включающая ту же нуклеотидную последовательность, что и с 5’-стороны кодирующей цепи гена по данному изобретению, и антисмысловая затравка, включающая нуклеотидную последовательность, комплементарную нуклеотидной последовательности 3’-стороны этой же кодирующей цепи, используются в сочетании с ПЦР, например с генной библиотекой, геномной ДНК или кДНК, используемыми в качестве матрицы для амплификации ДНК. С точки зрения использования генной библиотеки надо учесть, что существуют библиотеки кДНК и геномные библиотеки, выделенные из сои и растения семейства Chenopodiaceae, такого как свекла, или растений семейства Cruciferae, таких как горчица или брюква, и т.д. Генная библиотека также может быть библиотекой, сконструированной с помощью стандартного метода формирования библиотек, например, описанного в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press; "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-X, или быть стандартной библиотекой генов как таковой. Если выбор падает на геномную ДНК или кДНК, нужно учесть, что, например, такие библиотеки получены из сои, растения семейства Chenopodiceae, такого как свекла, или растений семейства Cruciferae, таких как горчица или брюква, и т.д. Например, ПЦР проводят с использованием затравок 31 и 32 в приведенном выше Перечне 3 и используемой в качестве матрицы кДНК, выделенной из горчицы, с целью амплификации ДНК, включающей нуклеотидную последовательность, представленную 749-1215 нуклеотидами нуклеотидной последовательности SEQ ID NO 6. Далее ПЦР проводят с использованием затравок и кДНК в качестве матрицы, выделенной из брюквы, с целью амплификации ДНК, включающей нуклеотидную последовательность, представленную 1-467 нуклеотидами нуклеотидной последовательности SEQ ID NO 8. Нуклеиновая кислота, амплифицированная таким образом, может быть подтверждена с использованием стандартного электрофореза. Нуклеиновая кислота может быть клонирована стандартным методом, таким как описано в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press; "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-Х. Готовят рестрикционную карту нуклеиновой кислоты или определяют ее нуклеотидную последовательность с помощью стандартного метода, таким образом, чтобы нуклеиновая кислота, включающая ген раффинозосинтазы или его часть, может быть идентифицирована. Когда нуклеиновая кислота содержит часть раффинозосинтазы, ПЦР может быть проведена на основе ее нуклеотидной последовательности с целью амплификации нуклеиновой кислоты, включающей 5’-(верхний) участок нуклеотидной последовательности или 3’-(нижний) участок нуклеотидной последовательности. Таким образом, основываясь на нуклеотидной последовательности, полученной вышеуказанным способом нуклеиновой кислоты, антисмысловую затравку определяют и синтезируют для амплификации 5’-верхней части, а смысловую затравку определяют и синтезируют для амплификации 3’-нижней части. Уже полученные нуклеотидные последовательности 5’-верхней и 3’-нижней частей могут быть определены методом RACE с использованием затравок и стандартного набора, такого как Marathon kit Clontech. Полноразмерный ген раффинозосинтазы может быть получен путем синтеза новых затравок на основе обоих концевых последовательностей в нуклеотидной последовательности, определенной таким образом, и проведением снова ПЦР.
Обозначенный выше способ определения настоящего изобретения может также быть использован для анализа генотипов у такого растения, как соя, у растения семейства Chenopodiaceae или растения семейства Cruciferae и т.д. Более детально, например, геномную ДНК, выделенную из сои, растения семейства Chenopodiaceae или растения семейства Cruciferae, приготавливают стандартным способом, например, описанным в "Cloning and Sequence (Plant Biotechnology Experiment Manual)", составленном I.Watanabe, опубликованным Noson Bunka-sha, Tokyo (1989) под редакцией M.Sugiura. Геномную ДНК расщепляют по крайней мере несколькими типами рестриктаз с последующим электрофорезом. Прошедшую электрофорезную разгонку ДНК промакивают фильтром, используя стандартный способ. Этот фильтр используют для гибридизации с зондом, приготавливаемым из ДНК, включающей фрагмент гена, с помощью стандартного способа, и в результате определяют ДНК, с которой гибридизует зонд. Выявленные молекулы ДНК сравнивают по длине по различным формам анализируемого вида растений. Различия по длине дают возможность для анализа различий по фенотипическим параметрам, ассоциированным с экспрессией олигосахаридов семейства раффиноз у различных форм. Более того, когда выявленные описанным выше способом молекулы ДНК сравнивают по длине у рекомбинантных растений и нормальных нерекомбинантных растений одних и тех же форм, рекомбинантное растение можно отличить от нерекомбинантного с помощью выявления бэндов гибридизации, больших по числу или более высоких по дозе у рекомбинантов в сравнении с нерекомбинантами. Этот способ может быть осуществлен в соответствии со способом ПДРФ (полиморфизм длин рестрикционных фрагментов), например, описанным в "Plant PCR Experiment Protocols", составленном под руководством K.Shimamoto и T.Sasaki, опубликованном Shujun-sha, Tokyo (1995), ISBN 4-87962-144-7, стр.90-94.
Далее, способ амплификации в настоящем изобретении может быть использован для анализа генов сои, растения семейства Chenopodiaceae или растения семейства Cruciferae и т.д. Более детально, например, способ амплификации в настоящем изобретении проводят с использованием геномной ДНК растения, приготовленной из сои, растения семейства Chenopodiaceae или растения семейства Cruciferae с целью амплификации ДНК. Амплифицированную ДНК смешивают с раствором формальдегида с последующей денатурацией нагреванием до 85°С в течение 5 минут и затем быстрым охлаждением на льду. Полученную пробу подвергают электрофорезу, например, в 6%-ном полиакриламидном геле, содержащем от 0% до 10% глицерина. Для такого электрофореза может быть применен промышленный электрофоретический аппарат, такой как для SSCP (одноцепочечный конформационный полиморфизм ДНК), а электрофорез может быть проведен с поддержанием геля при постоянной температуре, например при 5°С, 25°С, 37°С и т.д. Из состава электрофорезного геля ДНК выявляют, например, таким способом как окрашивание серебром стандартным реагентом. В случае наличия различия в поведении разнообразных форм при электрофорезе при выявлении ДНК выявляют мутацию в гене раффинозосинтазы и проводят анализ на поиск различий, обусловливаемых мутацией в фенотипических параметрах, связанных с экспрессией олигосахаридов семейства раффиноз. Этот способ может быть осуществлен с использованием способа идентификации SSCP, например, описанного в "Plant PCR Experiment Protocols", составленном под руководством K.Shimamoto и T.Sasaki, опубликованном Shujunsha, Tokyo (1995), ISBN 4-87962-144-7, стр.141-146.
Анализ растительного гена у сои, растения семейства Chenopodiaceae или растения семейства Cruciferae с помощью описанного выше способа выявления или способа амплификации по данному изобретению может быть использован не только для изучения различий фенотипических характеристик, связанных с экспрессией олигосахаридов семейства раффиноз, но и также, например, для отбора клонов, несущих желательные признаки с целью получения новых сортов сои, растения семейства Chenopodiaceae или растения семейства Cruciferae. Далее, он может быть также применен для идентификации клона, полученного таким образом, и включающего признаки, производные рекомбинантного растения, с целью получения сорта на основе рекомбинантного растения.
Для экспрессии гена по данному изобретению в клетках организма-хозяина предпочтительно использовать нуклеиновую кислоту, включающую фрагмент нуклеиновой кислоты, содержащий ген данного изобретения, и фрагмент нуклеиновой кислоты, обладающей промоторной активностью в клетках хозяина, присоединенный к предыдущему фрагменту нуклеиновой кислоты (здесь и далее в настоящем изобретении обозначается как экспрессионная нуклеиновая кислота).
Фрагмент нуклеиновой кислоты, обладающий промоторной активностью, в экспрессионной нуклеиновой кислоте по данному изобретению не ограничен по своей специфичности постольку, поскольку он лишь должен быть функционален в хозяине, который должен быть трансформирован. Например, существуют синтетические промоторы, функциональные в E.coli, такие как промотор лактозного оперона E.coli, промотор триптофанового оперона E.coli и tac-промотор, и т.д.; промотор гена алкогольдегидрогеназы (ADH) дрожжей, промотор позднего гена аденовируса (Ad.ML), промотор раннего гена SV40, бакуловирусный промотор и подобные. Когда хозяином является растение, промотор может включать, например, производные от Т-ДНК конститутивные промоторы, такие как промотор гена синтазы нопалина (NOS), промотор гена синтазы октопина (OCS), и т.д.; промоторы растительных вирусов, такие как промотор генов 19S и 35S вируса мозаики цветной капусты (CaMV); индуцибельные промоторы, такие как промотор гена фенилаланинаммониолиазы (PAL), промотор гена хальконсинтазы (CHS), промотор гена связанного с патогенезом белка (PR) и т.д. Более того, также может быть использован вектор pSUM-GYl (см. JP-A 06-189777/1994), который включает промотор, обеспечивающий специфическую экспрессию в соответствующих тканях растения, а именно промотор гена глицинина, являющегося запасающим белком семян, производных сои (JP-A 6-189777).
Далее, фрагмент нуклеиновой кислоты, обладающий терминаторной активностью, может быть присоединен к экспрессионной нуклеиновой кислоте по данному изобретению. В этом случае, в целом необходимым является то, чтобы экспрессионная нуклеиновая кислота по данному изобретению была сконструирована так, чтобы фрагмент с терминаторной активностью располагался ниже гена раффинозосинтазы. Выбор используемого терминатора практически не ограничен, поскольку он лишь должен быть функциональным в клетке-хозяине, подвергаемом трансформации. Например, когда хозяином является растение, такими терминаторами могут быть производные от Т-ДНК конститутивные терминаторы, такие как терминатор гена синтазы нопалина (NOS) и др.; производные растений терминаторы, такие как терминаторы вирусов GV1 или GV2 лука, и подобные.
Экспрессионная нуклеиновая кислота по данному изобретению может быть внесена в клетки-хозяева с применением стандартных генно-инженерных процедур с целью получения трансформанта. Если необходимо, экспрессионная нуклеиновая кислота по данному изобретению может быть встроена в вектор, имеющий подходящий маркер, зависящий от конкретной трансформационной методики, применяемой для внесения нуклеиновой кислоты в клетки-хозяева.
Вектор, в состав которого включают экспрессионную нуклеиновую кислоту по данному изобретению, может быть внесен в микроорганизм с использованием стандартного метода, например, описанного в "Molecular Cloning: A Laboratory Manual 2nd edition" (1989), Cold Spring Harbor Lab. Press или "Current Protocols in Molecular Biology" (1987), J.Wiley & Sons Inc., ISBN 0-471-50338-Х. Трансформированный вектором микроорганизм может быть отобран на основе селективного маркера, такого как резистентность к антибиотикам, ауксотрофия или подобное. В случае, когда ген по данному изобретению присоединен снизу к индуцибельному промотору (например, tac-промотору) в транслируемой форме в составе отобранного микроорганизма (например, в трансформированной E.coli), продукт трансляции гена по данному изобретению может быть экспрессирован в стандартной культуре при индуцибельных условиях и может быть выделен в виде белка или пептида.
Раффинозосинтазная активность продукта трансляции гена по данному изобретению, полученного описанным способом, может быть измерена, например, методом, описанным L.Lehle & W.Tanner, 1973, Eur. J. Biochem., 38, 103-110, с целью идентификации транслируемого продукта, обладающего "способностью связывать D-галактозильную группу с помощью α-(1→6) связи с гидроксильной группой, присоединенной к атому углерода в 6-м положении остатка D-глюкозы в молекуле сахарозы". Более детально, например, ген по данному изобретению клонируют в pGEX~4T3 (Pharmacia) для получения плазмиды, включающей экспрессионную нуклеиновую кислоту по данному изобретению. Полученную плазмиду вносят, например, в E.coli штамма НВ101 с целью получения трансформанта. Полученный трансформант культивируют в течение ночи и затем 1 мл культуры вносят на 100 мл культуральной среды LB. Инкубацию проводят при 37°С в течение 3 часов и затем добавляют IPTG (изопропилтио-β-D-галактозид) в конечной концентрации 1 мМ и вновь культивируют на протяжении 5 часов. Клетки выделяют из культурального бульона центрифугированием и суспензируют путем добавления 10-кратного по отношению к весу клеток количества Трис-НСl в концентрации 100 мМ (рН 7,4), 1 мМ ЭДТА, 5-мМ DTT (дитиотреитол), 1 мМ PMSF (фенилметилсульфонилфторид) и 1 мМ бензамида. Суспензию подвергают воздействию ультразвукового дизраптера (Branson) с целью разрушения клеток. Суспензию разрушенных клеток центрифугируют с целью получения раствора растворимых белков. В полученный раствор белков добавляют реактивную смесь, содержащую по конечным концентрациям 100 мМ Трис-НСl (рН 7,4), 5-мМ DTT (дитиотреитол), 0,01% BSA, 20 мкМ сахарозы, 5 мМ галактинола и 31,7 мкМ меченной [14С] сахарозы. Реактивную смесь инкубируют при 37°С с последующим добавлением полуторократного по объему количества этанола и взбалтывают. Нерастворимые частицы удаляют центрифугированием, а надосадочный слой раскапывают, например, на пластинки для HPTLC-целлюлозной тонкослойной хроматографии (пластинки HPTLC-целлюлозы фирмы Merch), затем пластинки обрабатывают смесью n-бутанола-пиридина-воды-уксусной кислоты в соотношении 60:40:30:3. Обработанные пластинки высушивают и анализируют с использованием томографического Fujix анализатора (Bio-lmage Analyzer ВАЗ-1000II manufactured by Fuji Film) с целью определения образующейся [14С]-раффинозы для измерения активности раффинозосинтазы.
Как дополнение, транслированный продукт, полученный описанным выше способом, может быть также использован в качестве антигена для получения антитела. Полученное таким образом антитело может быть использовано, например, для выявления и идентификации гена по данному изобретению в сыром белковом экстракте, приготовленном из организма, такого как растение.
Когда хозяином является растение, вектор со включенным в него геном по данному изобретению может быть внесен в растительные клетки стандартными способами, такими как способы заражения Agrobacterium (JP-B 2-58917 и JP-A 60-70080), электропорации протопластов (JP-A 60-251887 и JP-B 5-68575) или способы бомбардировки частицами (JP-A 5-508316 и JP-A 63-258525). Трансформированная внесенным вектором растительная клетка может быть отобрана с помощью селективного маркера, например, резистентности к антибиотикам, таким как канамицин или гигромицин. Из трансформированной таким образом растительной клетки растение-трансформант может быть регенерировано стандартным методом культивирования растительных клеток, например, описанным в "Plant Gene Manipulation Manual (How to Produce Transgenic Plants", подготовленном Uchimiya, 1990, Kodan-sha Sci. (ISBN 4-06-153513-7), стр.27-55. Более того, сбор семян растения-трансформанта также дает возможность получать трансформированные растения. Вдобавок, скрещивание между полученным трансформированным и нетрансформированным растениями дает возможность произвести потомство, имеющее признаки растения-трансформанта.
Что касается способов генной инженерии на сое, в целом, могут быть применены способы, описанные выше. Более детально, для трансформации растений сои могут быть применены способ "бомбардировки частицами", описанный в ЕР 301749, и методы внесения генов, например, описанные R.S.Torisky, L. Kovacs, S.Avdiushko, J.D.Newman, A.G.Hunt & G.B.Collins, 1997, "Development of a binary vector system for plant transformation based on the supervirulent Agrobacterium tumefaciens strain Chry5", Plant Cell Rep., 11, pp.102-108.
Что касается способов генной инженерии растений семейства Chenopodiaceae, указанные выше способы также могут быть применены. Более детально, могут быть применены способы внесения генов, например, описанные M.Mannerlof, S.Tuvesson, P.Steen & P.Tenning, 1997, "Transgenic sugar beet tolerant to glyphosate", Euphytica, 94, pp.83-91 и у B.K.Konwar, 1994, "Agrobacterium tumefaciens-mediated genetic transformation of sugar beet (Beta vulgaris L.)", J. Plant Biochem. & Biotechnol, 3, pp.37-41.
Что касается растений семейства Cruciferae, в целом указанные выше способы также могут быть применены. Более детально, внесение генов может быть осуществлено согласно способу, например, описанному J.Fry, A.Bamason & R.B.Horsch, 1987, "Transformation of Brassica napus with Agrobacterium tumefaciens based vector". Plant Cell Rep., 6, pp.321-325.
Например, когда внесение гена проводят с использованием способа заражения Agrobacterium, во-первых, описанную выше экспрессионную нуклеиновую кислоту по данному изобретению включают в состав бинарного вектора. Полученный вектор может быть введен, например, в Agrobacterium tumefaciens штамма LBA 4404, для конвертации которого в компетентное состояние проводят обработку хлоридом кальция. Трансформант может быть отобран соответствующим способом отбора по селективному маркеру гена вектора, например, путем культивирования штамма, включающего вектор, в культуральной среде, содержащей антибиотик, для случая, когда в качестве селективного маркера используется ген, детерминирующий резистентность к антибиотику, например к канамицину. Полученный трансформированный штамм Agrobacterium можно культивировать в жидкой культуральной среде, например в среде LB.
Соя, растение семейства Chenopodiaceae или растение семейства Cruciferae могут быть трансформированы с использованием культурального бульона с трансформированным штаммом Agrobacterium, полученным методом, описанным ниже. Например, семена сои, свеклы, брюквы или горчицы высевают в стерильных условиях, например, в среду 1/2 MS, включающую 2% сахарозы и 0,7% агара. Спустя примерно 1 неделю семядоли и черешки пророщенного растения отрезают стерильным скальпелем и пересаживают, например, в среду MS, содержащую 3% сахарозы, 4,5 мкМ ВА, 0,05 мкМ 2,4-D и 3,3 мкМ AgNO3 и культивируют в течение суток. Прокультивированные таким образом семядоли и черешки переносят в 1000-кратно разбавленный культуральный бульон Agrobacterium и оставляют на 5 минут. После этого семядоли и черешки помещают в ту же самую среду, в которой проводили предварительное культивирование, и культивируют 3-4 дня. После этого семядоли и черешки переносят, например, в среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 3,3 мкМ АgNO3 и 500 мг/л цефотаксима с последующим встряхиванием на протяжении 1 суток с целью удаления микробных клеток. Обработанные таким образом семяпочки и черешки переносят, например, в среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 3,3 мкМ АgNО3, 500 мг/л цефотаксима и 20 мг/л канамицина и культивируют в течение 3-4 недель. Затем семядоли и черешки переносят в, например, среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 100 мг/литр цефотаксима и 20 мг/литр канамицина, и культивируют. Культуру в данной среде поддерживают путем пересевов каждые 3-4 недели. Во время регенерации побегов культуру пересевают, например, в среду MS, содержащую 3% сахарозы, 0,7% агара и 20 мг/канамицина на 3-4 недели. Когда у растения образуются корешки, его переносят в смесь вермикулита и сфагнума (1:1) и аклиматизируют путем культивирования при 21-22°С и условиях освещения в соотношении 12 часов "день" и 12 часов "ночь". По мере роста растения его переносят в подходящую культивируемую почву для дальнейшего выращивания растения. Геномную ДНК экстрагируют из листьев регенерированного растения с использованием ранее описанного способа, а ПЦР проводят с использованием затравок, включающих частичные нуклеотидные последовательности экспрессионной нуклеиновой кислоты по данному изобретению с целью подтверждения встраивания гена по данному изобретению в растение.
Как уже было описано выше, с помощью внесения гена по данному изобретению в растение, например сою, растение семейства Chenopodiaceae или растение семейства Cruciferae, возможно варьировать уровень экспрессии и активности раффинозосинтазы с целью контроля количества олигосахаридов семейства раффиноз в тканях растения. Ген по данному изобретению применим в методах варьирования уровня экспрессии и активности раффинозосинтазы у сои, растения семейства Chenopodiaceae или растения семейства Cruciferae, с учетом гомологии генов, в таких методах, как метод гомологичной рекомбинации и косупрессии и подобное.
Следующие примеры иллюстрируют настоящее изобретение в деталях, но примеры не подразумевают ограничения широты настоящего изобретения.
Пример 1
Приготовление кДНК, производной сои
Примерно 2 г незрелых семян обыкновенной сои (Glycine max) сорта Williams-82 замораживают на жидком азоте и затем измельчают в ступке с добавлением 20 мл Isogen (Nippon Gene), смесь измельчают очень тщательно. Измельченный материал переносят в центрифужную пробирку, в которую добавляют 4 мл хлороформа, смесь взбалтывают в vortex миксере и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Отбирают водный слой, к которому добавляют 10 мл изопропанола, смесь взбалтывают и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Образующийся преципитат промывают 10 мл 70%-ного этанола и затем растворяют в 1 мл элюционного буфера (10мМ Трис-HCl, рН 7,5, 1 мМ ЭДТК, 0,1% SDS). Раствор оставляют на 10 минут при 60°С и затем центрифугируют при 10000 g в течение 1 минуты с целью удаления нерастворимого материала. К полученному надосадочному слою добавляют равный объем Oligotex-dt30 (Takara), смесь взбалтывают и оставляют на 5 минут при 65°С. Далее смесь помещают на лед и оставляют на 3 минуты; добавив 200 мкл 5 М NaCl и перемешав, полученную смесь оставляют на 10 минут при 37°С. Затем смесь центрифугируют при 10000 g в течение 3 минут при 4°С. Осадок отбирают и суспензируют в 1 мл буфера ТЕ, суспензию оставляют на 5 минут при 65°С. Далее суспензию помещают на лед и оставляют на 3 минуты, затем центрифугируют при 10000 g в течение 3 минут при 4°С для удаления преципитата.
К полученному надосадочному слою добавляют 100 мкл 3 М ацетата натрия и 2 мл этилового спирта с целью осаждения и отбора РНК. Собранную РНК промывают дважды 70%-ным этанолом и затем растворяют в 20 мкл стерилизованной воды, которая была использована в последствии для синтеза кДНК. Количество полученной РНК определяют путем измерения величины поглощения в участке спектра с длиной волны 260 нм.
Для синтеза кДНК используют наборы First Strand Synthesis Kit для ПЦР с ревертированием (Amersham) и cDNA Synthesis Kit (Takara), все операции проводят в соответствии с методиками, предлагаемыми инструкциями этих наборов.
Пример 2
Клонирование гена раффинозосинтазы из кДНК сои
ПЦР для амплификации фрагмента ДНК проводят с использованием кДНК, полученной от незрелых семян сои (Glycine max) сорта Williams-82 по примеру 1, как матрицы, и с затравками, определяемыми на основе аминокислотной последовательности SEQ ID NO 1, т.е. затравками, имеющими нуклеотидные последовательности, показанные ниже в перечне 4 для амплификации ДНК фрагмента. ПЦР осуществляют с использованием Gene Amp PCR Systems 2400 и DNA Thermal Cycler Model 480 (Perkin-Elmer) с использованием набора реактивов Advantage KlenTaq cDNA Kit (Clontech). Реакцию проводят путем повторения циклов в последовательности 1 минута при 94°С, 3 минуты при 50°С и 3 минуты при 72°С 40 раз с целью амплификации фрагмента ДНК. Амплифицированный фрагмент ДНК клонируют с использованием набора ТА Cloning Kit (Invitrogen) с последующим секвенированием с использованием ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer) и анализирование нуклеотидной последовательности с помощью 373S ДНК секветора ABI. На основе полученной последовательности синтезируют затравки, имеющие нуклеотидные последовательности, приведенные в перечне 5. Синтез кДНК проводят с использованием набора Marathon Kit (Clontech) с использованием мРНК, выделенной из листьев сои сорта Williams-82 по примеру 1. Полученную кДНК присоединяют к адаптеру с использованием имеющейся в наборе реактивов лигазы. Эти операции проводят, основываясь на инструкциях, прилагаемых к наборам. С использованием полученной таким образом лигированной на адаптер кДНК, ПЦР проводят с использованием затравки, приведенной в перечне 5 тем же способом, что был описан выше. Нуклеотидная последовательность терминального участка гена анализируют в соответствии с протоколом, прилагаемым к набору Marathon Kit (Clontech). В результате нуклеотидная последовательность SEQ ID NO 2 была определена.

Пример 3
Приготовление кДНК производной растения семейства Chenopodiaceae
Около 2 г листьев свеклы (Beta vulgaris) замораживают на жидком азоте и затем измельчают в ступке; после добавления 20 мл Isogen (Nippon Gene) проводят очень тщательное измельчение полученной смеси. Измельченный материал переносят в центрифужную пробирку, в которую добавляют 4 мл хлороформа, смесь взбалтывают в vortex миксере и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Водный слой отбирают, к нему добавляют 10 мл изопропанола: смесь взбалтывают и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Полученный преципитат промывают в 10 мл 70%-ного этилового спирта и затем растворяют в 180 мкл обработанной DEPC стерилизованной воды. Раствор оставляют на 5 минут при 55°С и затем добавляют в него 10 мкл 5 М NaCl. Полученный раствор очищают с использованием набора BIOMAG mRNA Purification Kit (PerSeptive Biosystems: каталожный №8-MB4003K).
К полученной мРНК добавляют 3 М ацетата натрия и этанол, РНК осаждают и собирают. Собранную РНК промывают дважды в 70%-ном этаноле и затем растворяют в 20 мкл стерилизованной воды, которая была использована для последующего синтеза кДНК. Количество полученной РНК определяют путем измерения величины поглощения в участке спектра с длиной волны 260 нм.
Для синтеза кДНК используют набор реактивов SMART PCR cDNA Synthesis Kit (Clontech), все операции проводят в соответствии с инструкцией, прилагающейся к набору.
Пример 4
Анализ нуклеотидной последовательности гена раффинозосинтазы у растения семейства Chenopodiaceae
Синтезировали синтетические ДНК-затравки, включающие нуклеотидные последовательности, приведенные в перечне 6. Метод ПЦР осуществляют на оборудовании Gene Amp PCR Systems 2400 и DNA Thermal Cycler Model 480 (Perkin-Ehner) и с набором реактивов Advantage KlenTaq cDNA Kit (Clontech). ПЦР осуществляют с использованием обозначенных выше затравок и кДНК свеклы, получение которой описано в примере 3, путем повторных циклов в режиме 1 минута при 94°С, 3 минуты при 50°С и 3 минуты при 72°С 40 раз. В результате, сочетание затравок 6-3-F и 6-8-RV и затравок 6-10-F и 6-6-RV привело к амплификации полосами длиной соответственно 0,3 и 0,6 т.п.н. Амплифицированные фрагменты ДНК клонируют с использованием реактивов ТА Cloning Kit (Invitrogen) с последующим секвенированием на ДНК-секвенсоре 373S-ABI с использованием набора реактивов ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). Основываясь на полученной нуклеотидной последовательности, готовят синтетические ДНК-затравки, включающие нуклеотидные последовательности, приведенные ниже в перечне 7, ПЦР проводят с использованием кДНК, полученной от свеклы в примере 3 тем же способом, что был описан выше. В результате на основе кДНК свеклы была получена ДНК, имеющая нуклеотидную последовательность SEQ ID NO 4.

Пример 5
Приготовление кДНК, производной растения семейства Cruciferae
Около 2 г листьев горчицы или сарептской горчицы (Brassica juncea) замораживают на жидком азоте и затем измельчают в ступке; после добавления 20 мл Isogen (Nippon Gene) проводят очень тщательное измельчение полученной смеси. Измельченный материал переносят в центрифужную пробирку, в которую добавляют 4 мл хлороформа, смесь взбалтывают в vortex миксере и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Водный слой отбирают, к нему добавляют 10 мл изопропанола, смесь взбалтывают и затем центрифугируют при 6500 g в течение 10 минут при 4°С. Полученный преципитат промывают в 10 мл 70%-ного этилового спирта и затем растворяют в 180 мкл обработанной DEPC стерилизованной воды. Раствор оставляют на 5 минут при 55°С и затем добавляют в него 10 мкл 5 М NaCl. Полученный раствор очищают с использованием набора BIOMAG mRNA Purification Kit (PerSeptive Biosystems: каталожный №8-MB4003K).
К полученному раствору мРНК добавляют 3 М ацетата натрия и этанол, РНК осаждают и собирают. Собранную РНК промывают дважды в 70%-ном этаноле и затем растворяют в 20 мкл стерилизованной воды, которая была использована для последующего синтеза кДНК. Количество полученной РНК определяют путем измерения величины поглощения в участке спектра с длиной волны 260 нм.
Для синтеза кДНК используют набор реактивов SMART PCR cDNA Synthesis Kit (Clontech), все операции проводят в соответствии с инструкцией, прилагающейся к набору.
Таким же способом, какой был описан выше, мРНК выделяют и очищают из незрелых семян брюквы (Brassica napus) сорта Westar с последующим синтезом кДНК.
Пример 6
Выделение и определение нуклеотидной последовательности гена раффинозосинтазы из растения семейства Crucuferae
Синтезируют ДНК-затравки, имеющие нуклеотидные последовательности, показанные в перечне 8. ПЦР проводят на оборудовании Gene Amp PCR Systems 2400 и DNA Thermal Cycler Model 480 (Perkin-Elmer) с использованием набора реактивов Advantage KlenTaq cDNA Kit (Clontech). ПЦР проводят с указанными выше затравками и кДНК горчицы, полученной в примере 5 с 40-кратным повторением цикла в режиме 1 минута при 94°С, 3 минуты при 50°С и 3 минуты при 72°С. Продукт полимеразной реакции анализируют с применением электрофореза в агарозном геле. В результате маркируется продукт амплификации с полосой длиной около 1,2 т.п.н. Амплифицированный фрагмент ДНК клонируют с использованием набора ТА Cloning Kit (Invitrogen) с последующим секвенированием на ДНК-секвенсоре 373S-ABI с использованием набора реактивов ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). На основе полученной нуклеотидной последовательности готовят синтетические затравки, имеющие нуклеотидные последовательности, представленные ниже в перечне 9, и проводят ПЦР на основе кДНК горчицы (Brassica juncea) и брюквы (Brassica napus) сорта Westar, полученной в примере 5 в соответствии с методами, описанными выше. В результате получают нуклеотидные последовательности, представленные 749-1215 нуклеотидами SEQ IN NO 6 и 1-467 нуклеотидами SEQ ID NO 8, определяемые соответственно по кДНК горчицы (Brassica juncea) и брюквы (Brassica napus) сорта Westar.

Пример 7
Анализ нуклеотидной последовательности полноразмерного гена раффинозосинтазы растения семейства Cruciferae.
Основываясь на нуклеотидных последовательностях, полученных в примере 6, синтезируют ДНК-затравки, имеющие нуклеотидные последовательности, приведенные ниже в перечне 10. кДНК, полученные теми же способами, что описаны в примере 5, от горчицы (Brassica juncea) и брюквы (Brassica napus) сорта Westar, лигируют на адаптеры, имеющиеся в наборе реактивов Marathon Kit (Clontech). С использованием полученных таким образом лигированных на адаптеры кДНК проводят ПЦР, используя затравки, приведенные в перечне 10.
Затравки 10-B-2RV, 10-B-3RV и 10-B-4RV используют для анализа нуклеотидов с 5'-конца цепи, а затравки 10-B-1, 10-В-8, 10-В-7 и 10-В-6 используют для анализа нуклеотидов с 3’-конца. Нуклетидные последовательности анализируют в соответствии с протоколом, прилагающимся к набору Marathon Kit (Clontech). В результате могут быть определены нуклеотидные последовательности SEQ ID NO 6 и SEQ ID NO 8, представляющие соответственно горчицу (Brassica juncea) и брюкву (Brassica napus) сорта Westar.

Пример 8
Конструирование экспрессионного вектора для растения с геном раффинозосинтазы, производным растения семейства Cruciferae
Основываясь на нуклеотидной последовательности гена раффинозосинтазы горчицы, выведенной в примере 7, готовят ДНК-затравки, имеющие нуклеотидные последовательности, приведенные в перечне 11. ПЦР проводят с использованием кДНК горчицы таким же способом, что был описан в примере 6. Амплифицированный фрагмент ДНК обрабатывают рестриктазой SacI. Затем обработанный таким образом фрагмент ДНК лигируют в состав вектора рВ1121(-), предварительно обработанный SacI, с использованием набора реактивов Ligation Kit (Takara). Плазмиду pBI121 (Clontech) обрабатывают BamHl и SacI и лигируют по линкерам, приведенным в перечне 12, для приготовления вектора pBI121(-). Полученный таким образом вектор анализируют по рестрикционной карте с помощью ПЦР, используя затравки с нуклеотидными последовательностями, приведенными в перечне 13, что позволяет подтвердить направление расположения гена раффинозосинтазы. Вектор, в котором ген раффинозосинтазы горчицы встроен в экпрессибельной ориентации, обозначают BjRS-Sac(+)-121; вектор, в котором такой же ген раффинозосинтазы встроен в обратном направлении, обозначают BjRS-Sac(-)-121.

Пример 9
Трансформация геном раффинозосинтазы, производным растения семейства Cruciferae
Векторы BjRS-Sac(+)-121 и BjRS-Sac(-)-121, приготовленные в примере 8, используют для трансформации горчицы (Brassica juncea) с использованием метода заражения Agrobacterium.
Agrobacterium tumefaciens (штамм LBA 4404, проявляющий резистентность к рифамицину и стрептомицину), предварительно конвертированный в требуемое состояние обработкой хлоридом кальция, трансфектируют независимо двумя плазмидами BjRS-Sac(+)-121 и BjRS-Sac(-)-121, полученными в примере 8. Трансформанты отбирают с использованием среды LB, содержащей 50 мкг/мл рифамицина и 25 мкг/мл канамицина с использованием признака резистентности к канамицину, детерминируемого геном резистентности к канамицину (ген неомицинфосфотрансферазы, NPTII), имеющимся в составе вносимых плазмид.
Полученные трансформанты Agrobacterium (Agrobacterium tumefaciens штамма LBA4404: резистентен к рифамицину и стрептомицину) культивируют в среде LB, содержащей 50 мкг/мл рифамицина и 25 мкг/мл канамицина, при 28°С в течение полных суток. Культуру используют для трансформации горчицы с применением описанного ниже метода.
Семена горчицы в стерильных условиях высевают в среду 1/2 MS, содержащую 2% сахарозы и 0,7% агара. Через неделю семядоли и черешки проросших растений отрезают скальпелем и переносят в среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D и 3,3 мкМ AgNO3 с последующим предварительным культивированием в течение дня. Предкультивированные семядоли и черешки переносят в 1000-кратно разбавленный культуральный бульон Agrobacterium и оставляют на 5 минут. Семядоли и черешки переносят снова в ту же среду, которую использовали для предварительного культивирования, и культивируют в течение 3-4 дней. Затем культивированные семядоли и черешки переносят в среду MS, содержащую 3% сахарозы, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 3,3 мкМ AgNO3 и 500 мг/л цефотаксима, и встряхивают на протяжении 1 дня с целью удаления микробных клеток. Семядоли и черешки, обработанные таким образом, затем переносят в среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 3,3 мкМ АgNО3, 100 мг/л цефотаксима и 20 мг/л канамицина, и культивируют на протяжении 3-4 недель. Затем семядоли и черешки переносят в среду MS, содержащую 3% сахарозы, 0,7% агара, 4,5 мкМ ВА, 0,05 мкМ 2,4-D, 100 мг/л цефотаксима и 20 мг/л канамицина, и культивируют. Дальнейшее культивирование в такой среде продолжают с использованием пересевов с интервалами в 3-4 недели. Когда начинается регенерация побегов, их переносят в среду MS, содержащую 3% сахарозы, 0,7% агара и 20 мг/л канамицина, и культивируют в течение 3-4 недель. Растения с образовавшимися корешками переносят в смесь вермикулита и сфагнума в соотношении 1:1 и культивируют при 21-22°С при соблюдении цикличности 12 часов "день" и 12 часов "ночь". По мере роста растений их пересаживают в подходящую почву.
Краткая характеристика последовательностей
SEQ ID NO 1: показана аминокислотная последовательность белка раффинозосинтазы, кодируемого раффинозосинтазным геном по данному изобретению.
SEQ ID NO 2: показана нуклеотидная последовательность гена раффинозосинтазы по данному изобретению.
SEQ ID NO 3: показана аминокислотная последовательность белка раффинозосинтазы, кодируемого раффинозосинтазным геном, выделенным у свеклы.
SEQ ID NO 4: показана нуклеотидная последовательность кДНК гена раффинозосинтазы, выделенной у свеклы.
SEQ ID NO 5: показана аминокислотная последовательность белка раффинозосинтазы, кодируемого раффинозосинтазным геном, выделенным из горчицы.
SEQ ID NO 6: показана нуклеотидная последовательность кДНК гена раффинозосинтазы, выделенного у горчицы.
SEQ ID NO 7: показана аминокислотная последовательность белка раффинозосинтазы, кодируемого раффинозосинтазным геном, выделенным у брюквы.
SEQ ID NO 8: показана нуклеотидная последовательность кДНК гена раффинозосинтазы, выделенной у брюквы.
Перечень 1
Нуклеотидные последовательности, приведенные в перечне 1, являются примерами типичных затравок, используемых для амплификации фрагмента ДНК, включающего ген раффинозосинтазы. Все эти последовательности основаны на нуклеотидной последовательности в SEQ ID NO 2. Затравка 11 является смысловой затравкой, затравка 12 - антисмысловой. В зависимости от поставленной задачи распознавательные последовательности для подходящих рестриктаз могут быть присоединены к 5’-концам этих нуклеотидных последовательностей.
Перечень 2
Нуклеотидные последовательности, приведенные в перечне 2, являются примерами типичных затравок, используемых для амплификации кДНК гена раффинозосинтазы. Затравка 21 является смысловой затравкой, соответствующей 5’-концу гена раффинозосинтазы, выделенного у свеклы. Затравка 22 является антисмысловой затравкой, соответствующей 3’-концу. В зависимости от поставленной задачи распознавательные последовательности для подходящих рестриктаз могут быть присоединены к 5’-концам этих нуклеотидных последовательностей.
Затравка 23 является смысловой затравкой, соответствующей N-концу открытой рамки считывания, а затравка 24 - антисмысловая затравка, соответствующая ее С-концу.
Перечень 3
Среди приведенных в перечне 3 нуклеотидных последовательностей затравки 31 и 32 являются типичными затравками, используемыми при амплификации ДНК, имеющей частичную нуклеотидную последовательность гена раффинозосинтазы. Затравка 31 является смысловой затравкой, используемой для амплификации ДНК, имеющей частичную нуклеотидную последовательность гена раффинозосинтазы у горчицы и брюквы, затравка 32 является антисмысловой затравкой. В зависимости от поставленной цели распознавательные последовательности для подходящих рестриктаз могут быть присоединены к 5’-концам этих нуклеотидных последовательностей.
Затравки 33 и 34 являются типичными затравками, используемыми для амплификации кДНК гена раффинозосинтазы горчицы. Обе затравки 33 и 34 основаны на нуклеотидной последовательности гена раффинозосинтазы в части его нетранслируемого участка. Затравка 33 является смысловой затравкой, соответствующей 5’-концу нетранслируемого сегмента гена раффинозосинтазы горчицы. Затравка 34 - антисмысловая затравка, соответствующая 3’-концу нетранслируемого сегмента.
Затравки 35 и 36 являются типичными затравками, используемыми для амплификации открытой рамки считывания, кодирующей аминокислотную последовательность белка раффинозосинтазы, входящей в кДНК гена раффинозосинтазы. Затравка 35 является смысловой затравкой, соответствующей 5’-концу указанной открытой рамки. Затравка 36 является антисмысловой затравкой, соответствующей 3’-концу. В зависимости от поставленной задачи распознавательные последовательности для подходящих рестриктаз могут быть присоединены к 5’-концам этих нуклеотидных последовательностей.
Перечень 4
Нуклеотидные последовательности, приведенные в перечне 4, являются затравками, используемыми в клонировании фрагмента ДНК, включающего представляемый ген раффинозосинтазы. Основание, обозначаемое символом "i", - инозин. Помещение оснований в скобки означает, что смесь этих оснований используется в синтезе. Символ "RV", проставляемый после номера затравки, означает, что эта помеченная таким образом затравка включает антисмысловую последовательность.
Перечень 5
Нуклеотидная последовательность, приведенная в перечне 5, является затравкой, используемой в анализе нуклеотидной последовательности представляемого гена раффинозосинтазы. Затравка 5-SC-2 используется в анализе 3’-концевого сегмента представляемой нуклеотидной последовательности.
Перечень 6
Нуклеотидные последовательности, приведенные в перечне 6, являются затравками, используемыми в анализе гена раффинозосинтазы свеклы. Основание, обозначаемое символом "i", - инозин. Помещение оснований в скобки означает, что смесь этих оснований используется в синтезе. Символ "RV", проставляемый после номера затравки, означает, что помеченная таким символом затравка является антисмысловой последовательностью.
Перечень 7
Нуклеотидные последовательности, приведенные в перечне 7, являются затравками, синтезируемыми по частичным нуклеотидным последовательностям гена раффинозосинтазы свеклы. Символ "RV", проставляемый после номера затравки, означает, что помеченная таким образом затравка является антисмысловой последовательностью.
Перечень 8
Нуклеотидные последовательности, приведенные в перечне 8, являются затравками, используемыми в анализе нуклеотидной последовательности кДНК гена раффинозосинтазы горчицы. Основание, обозначаемое символом "i", - инозин. Помещение оснований в скобки означает, что используется смесь этих оснований. Символ "RV", проставляемый после номера затравки, означает, что помеченная таким образом затравка является антисмысловой последовательностью.
Перечень 9
Нуклеотидные последовательности, приведенные в перечне 9, являются затравками, синтезируемыми по частичным нуклеотидным последовательностям гена раффинозосинтазы горчицы. Символ "RV", проставляемый после номера затравки, означает, что помеченная таким образом затравка является антисмысловой последовательностью.
Перечень 10
Нуклеотидные последовательности, приведенные в перечне 10, являются затравками, используемыми для анализа нуклеотидных последовательностей генов раффинозосинтазы горчицы и брюквы. Символ "RV", проставляемый после номера затравки, означает, что помеченная таким образом затравка является антисмысловой последовательностью.
Перечень 11
Нуклеотидные последовательности, приведенные в перечне 11, являются затравками, используемыми в амплификации 5’-концевого участка гена раффинозосинтазы горчицы. 11-SacI-BjN является затравкой, сайт узнавания рестриктазой SacI, в которой присоединены к нуклеотидной последовательности 8-29 нуклеотидов SEQ ID NO 6. 11-SacI-BjintRV является антисмысловой затравкой, имеющей нуклеотидную последовательность, соответствующую нуклеотидной последовательности, представленной 1164-1188 нуклеотидами SEQ ID NO 6.
Перечень 12
Нуклеотидные последовательности, приведенные в перечне 12, являются адаптерами, присоединяемыми к кДНК горчицы. Эти синтетические молекулы ДНК приобретают двухцепочечную форму тогда, когда перемешиваются, т.к. они включают комплементарные цепи. Такой адаптер имеет "липкие концы" в сайтах расщепления по рестриктазам BamHI и SacI на обоих концах и включает рестрикционные сайты по рестриктазам BamHI и SacI в двухцепочечном участке.
Перечень 13
Нуклеотидные последовательности, приведенные в перечне 13, являются затравками, используемыми для подтверждения направления встраивания гена раффинозосинтазы, выделенного у горчицы. Затравка 13-35S-3 является затравкой, смысловой по отношению к промотору 35S. Затравка 13-B-2RV является антисмысловой затравкой, имеющей нуклеотидную последовательность, представленную 593-622 нуклеотидами SEQ ID NO 6. Затравка 13-В-8 является смысловой затравкой, имеющей нуклеотидную последовательность, представленную 1110-1138 нуклеотидами SEQ ID NO 6.
Как было описано выше, в соответствии с настоящим изобретением возможным является выделить гены раффинозосинтазы, которые могут быть использованы в методах варьирования уровня экспрессии и активности раффинозосинтазы у растений.
Перечень последовательностей, не упоминающихся в тексте
SEQ ID NO 9 по SEQ ID NO 20: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы.
SEQ ID NO 21 и SEQ ID NO 22: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы; n=i (инозин), r=а или g.
SEQ ID NO 23: сконструированная олигонуклеотидная затравка для выделения гена раффинозосинтазы.
SEQ ID NO 24 по SEQ ID NO 26: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы; n=i, у=t или с, r=а или g.
SEQ ID NO 27: сконструированная олигонуклеотидная затравка для выделения гена раффиозосинтазы; n=i.
SEQ ID NO 28 no SEQ ID NO 35: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы.
SEQ ID NO 36 по SEQ ID NO 37: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы; n=i, r=а или g.
SEQ ID NO 38 по SEQ ID NO 48: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы.
SEQ ID NO 49 по SEQ ID NO 50: сконструированные олигонуклеотидные затравки для выделения гена раффинозосинтазы.
SEQ ID NO 51 по SEQ ID NO 53: сконструированные олигонуклеотидные затравки для подтверждения направленности вставленного гена раффинозосинтазы.










































Изобретение относится к биотехнологии. Раффинозосинтазу получают культивированием клетки-хозяина, трансформированной вектором, содержащим ген раффинозосинтазы. Полученную раффинозосинтазу выделяют из культуральной жидкости. Частичную последовательность гена раффинозосинтазы используют в качестве зонда или затравки. Данное изобретение позволяет варьировать уровень экспрессии и активности у растений. 9 н. и 15 з.п. ф-лы, 1 табл.
| JP 10084973, 07.04.1998 | |||
| Способ контроля бесперебойностииСКРООбРАзОВАНия СВЕчи зАжигАНия | 1979 |
|
SU849359A1 |
| РЕКОМБИНАНТНЫЙ ФРАГМЕНТ ДНК, КОДИРУЮЩИЙ СПЕЦИФИЧЕСКУЮ НИТРИЛАЗУ, ОПРЕДЕЛЯЮЩУЮ РЕЗИСТЕНТНОСТЬ РАСТЕНИЙ К ГЕРБИЦИДАМ НА ОСНОВЕ 3,5-ДИГАЛОИД-4-ОКСИБЕНЗОНИТРИЛА, И ВЕКТОР ЭКСПРЕССИИ ДЛЯ РЕКОМБИНАНТНОГО ФРАГМЕНТА ДНК (ВАРИАНТЫ) | 1989 |
|
RU2106410C1 |
| WO 9302196, 04.02.1993. | |||
Авторы
Даты
2004-12-27—Публикация
1999-04-29—Подача