ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
В данной заявке испрашивается приоритет обычной заявки США №62/088423, поданной 5 декабря 2014 г., озаглавленной «ПРИМЕНЕНИЕ ПРОИСХОДЯЩИХ ОТ ЦЕНТРАЛЬНЫХ КЛЕТОК ПАМЯТИ Т-КЛЕТОК, МОДИФИЦИРОВАННЫХ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА CS1, ДЛЯ ЛЕЧЕНИЯ МНОЖЕСТВЕННОЙ МИЕЛОМЫ», содержание которой включено в данный документ во всей ее полноте.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Иммунотерапии на основе опухолеспецифичных Т-клеток, включая терапии с использованием генетически модифицированных Т-клеток, исследовали в отношении противоопухолевого лечения. В некоторых случаях Т-клетки, используемые в таких терапиях, не остаются активными in vivo в течение достаточно длительного периода. В некоторых случаях противоопухолевая активность Т-клеток является относительно низкой. Следовательно, в данной области существует потребность в опухолеспецифичных терапиях онкологических заболеваний с более долговременным противоопухолевым функционированием.
Адоптивная T-клеточная терапия (ACT) с использованием Т-клеток, модифицированных химерным рецептором антигена (CAR), может обеспечивать безопасный и эффективный способ уменьшения частоты рецидивов разных онкологических заболеваний, так как Т-клетки с CAR могут быть генетически модифицированы для специфичного распознавания антигенно отличных популяций опухолей способом, не зависимым от МНС (главный комплекс гистосовместимости).
Множественная миелома (ММ) представляет собой B-клеточное злокачественное заболевание, характеризуемое клональным размножением плазматических клеток. На долю ММ приходится приблизительно 1 процент всех онкологических заболеваний и немного больше, чем 10 процентов гематологических злокачественных заболеваний в Соединенных Штатах. В одних Соединенных Штатах в этом году будет продиагностировано приблизительно 20000 новых случаев, и свыше 11000 людей умрут от этого заболевания. Современные терапии ММ часто индуцируют ремиссию, но почти все пациенты, в конечном счете, имеют рецидивы и умирают.
CS1 представляет собой гликопротеин поверхности клетки из семейства рецепторов сигнальных молекул активации лимфоцитов (SLAM), который сильно и селективно экспрессируется на нормальных плазматических клетках и клетках ММ, с меньшей экспрессией на клетках NK (природные киллеры) и слабой экспрессией или ее отсутствием на нормальных тканях. Элотузумаб (HuLuc63)-гуманизированное моноклональное антитело против CS1, которое вводится совместно с бортезомибом, дает у пациентов с рецидивом ММ больший или меньший PR (частичный ответ) у 48% пациентов. Высокая экспрессия CS1 на клетках ММ в сочетании с его ограничением плазматическими клетками в нормальной ткани делает CS1 целесообразной мишенью для терапии Т-клетками с CAR (Hsi et al. 2008 Clin Cancer Res 14: 2775).
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
В данном документе описываются CAR, которые содержат внеклеточный домен, трансмембранный домен и внутриклеточной домен сигнализации. Внеклеточный домен включает CS1-специфичную область scFv или ее вариант и, возможно, спейсер, содержащий, например, часть человеческого домена Fc. Внеклеточный домен обеспечивает то, что CAR, при экспрессии на поверхности Т-клетки, направляет активность Т-клетки на клетки, экспрессирующие CS1-рецептор, экспрессируемый на поверхности ММ. Трансмембранный домен включает, например, трансмембранный домен CD4, трансмембранный домен CD8, трансмембранный домен CD28 или трансмембранный домен CD3. Внутриклеточный домен сигнализации включает домен сигнализации из зета цепи комплекса человеческого CD3 (CD3ζ) и один или более чем один костимулирующий домен, например, костимулирующий домен 4-1ВВ. Включение костимулирующего домена, такого как костимулирующий домен 4-1ВВ (CD137), последовательно с CD3ζ во внутриклеточную область обеспечивает то, что Т-клетки получают костимулирующие сигналы. Т-клетки, например, специфичные для пациента аутологичные Т-клетки, могут быть генетически модифицированы для экспрессии CAR, описанных в данном документе, и данные генетически модифицированные клетки могут быть размножены и использованы в ACT. Можно использовать разные поднаборы Т-клеток, включая и альфа бета Т-клетки, и гамма дельта Т-клетки. Кроме того, CAR могут экспрессироваться в других иммунных клеках, таких как клетки NK. Когда пациента лечат иммунной клеткой, экспрессирующей CAR, описанный в данном документе, клетка может быть аутологичной Т-клеткой или аллогенной Т-клеткой. В некоторых случаях используемые клетки представляют собой популяцию клеток, которая включает и CD4+, и CD8+ центральные Т-клетки памяти (TCM), которые являются CD62L+, CCR7+, CD45RO+ и CD45RA-. Данная популяция клеток также может включать другие типы Т-клеток.
Описанный в данном документе CAR CS1 имеет некоторые полезные характеристики, например, стойкость и усиленную противоопухолевую активность после адоптивного переноса.
Т-клетки, экспрессирующие CAR, нацеленный на CS1, могут быть полезными при лечении онкологических заболеваний, таких как ММ, а также других онкологических заболеваний, при которых экспрессируется CS1. Таким образом, данное раскрытие включает способы лечения онкологического заболевания, при котором экспрессируется CS1, с использованием Т-клеток, экспрессирующих CAR, описанный в данном документе.
В данном документе описана молекула нуклеиновой кислоты, кодирующая CAR, содержащий scFv против CS1 (например,
EVQLVESGGGLVQPGGSLRLSCAASGFDFSRYWMSWVRQAPGKGLEWIGEINPDSST INYAPSLKDKFIISRDNAKNSLYLQMNSLRAEDTAVYYCARPDGNYWYFDVWGQGTLVT VSSGSTSGGGSGGGSGGGGSSDIQMTQSPSSLSASVGDRVTITCKASQDVGIAVAWY QQKPGKVPKLLIYWASTRHTGVPDRFSGSGSGTDFTLTISSLQPEDVATYYCQQYSSY PYTFGQGTKVEIK; SEQ ID NO: 1) или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен); трансмембранный домен, выбранный из: трансмембранного домена CD4 или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот (например, замен), трансмембранного домена CD8 или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот (например, замен), трансмембранного домена CD28 или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот (например, замен), и трансмембранного домена CD3ζ или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот (например, замен); костимулирующий домен (например, костимулирующий домен CD28 или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен); или костимулирующий домен 4-1ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен); или и костимулирующий домен CD28 или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен), и костимулирующий домен 4-1ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен); и домен сигнализации CD3ζ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот.
Данное раскрытие также включает молекулы нуклеиновых кислот, которые кодируют любые CAR, описанные в данном документе (например, векторы, которые включают последовательность нуклеиновой кислоты, кодирующую один из CAR), и выделенные Т-клетки, которые экспрессируют любые CAR, описанные в данном документе.
В данном документе описана молекула нуклеиновой кислоты, кодирующая химерный рецептор антигена, где химерный рецептор антигена содержит: scFV против CS1, спейсерную область, трансмембранный домен CD28 или CD4, костимулирующий домен CD28 или костимулирующий домен 4-1ВВ, возможный линкер GlyGlyGly и домен сигнализации CD3ζ.
В одном воплощении CAR CS1 состоит из или содержит аминокислотную последовательность любого из SEQ ID NO: 31, 34, 37, 40, 43 и 46 (зрелый CAR, не имеющий сигнальной последовательности), или CAR CS1 состоит из или содержит аминокислотную последовательность любого из SEQ ID NO: 30, 33, 36, 39, 42 и 45 (незрелый CAR, имеющий сигнальную последовательность GMCSFRα (рецептор α гранулоцитарно-макрофагального колониестимулирующего фактора). CAR может экспрессироваться в форме, которая включает сигнальную последовательность, например, сигнальную последовательность человеческого рецептора альфа GM-CSF (MLLLVTSLLLCELPHPAFLLIP; SEQ ID NO: 26). CAR может экспрессироваться с дополнительными последовательностями, которые являются полезными для отслеживания экспрессии, например, с последовательностью перескакивания Т2А и усеченного EGFRt (рецептор t эпидермального фактора роста). Таким образом, CAR может содержать или состоять из аминокислотной последовательности любого из SEQ ID NO: 29-46, или может содержать или состоять из аминокислотной последовательности, которая является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичной любой из SEQ ID NO: 29-46. CAR может содержать или состоять из аминокислотной последовательности любого из SEQ ID NO: 29-46 с вплоть до 1, 2, 3, 4 или 5 аминокислотными заменами (предпочтительно консервативными аминокислотными заменами).
Также раскрыта популяция человеческих Т-клеток, трансдуцированных вектором, содержащим экспрессионную кассету, кодирующую химерный рецептор антигена CS1, описанный в данном документе (например, CAR, который содержит или состоит из аминокислотной последовательности любого SEQ ID NO: 29-46, или аминокислотной последовательности, которая является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичной любой из SEQ ID NO: 29-46, или аминокислотной последовательности любого из SEQ ID NO: 29-46 с вплоть до 1, 2, 3, 4 или 5 аминокислотными заменами (предпочтительно консервативными аминокислотными заменами).
В разных воплощениях популяция человеческих Т-клеток представляет собой центральные Т-клетки памяти (TCM), например, TCM CD8+/CD4+.
Термин «модификация аминокислоты» относится к замене, вставке и/или делеции аминокислоты в последовательности белка или пептида. Термин «замена аминокислоты» или «замена» относится к замене аминокислоты в конкретном положении в последовательности родительского пептида или белка другой аминокислотой. Замену можно делать для замены аминокислоты в полученном в результате белке неконсервативным способом (т.е. посредством замены кодона от аминокислоты, принадлежащей к группе аминокислот, имеющих конкретный размер или характеристику, на аминокислоту, принадлежащую к другой группе) или консервативным способом (т.е. посредством замены кодона от аминокислоты, принадлежащей к группе аминокислот, имеющих конкретный размер или характеристику, на аминокислоту, принадлежащую к той же самой группе). Такая консервативная замена обычно приводит к меньшему изменению в структуре и функции образующегося в результате белка. Следующее представляет собой примеры разных групп аминокислот: 1) аминокислоты с неполярными группами R: аланин, валин, лейцин, изолейцин, пролин, фенилаланин, триптофан, метионин; 2) аминокислоты с незаряженными полярными группами R: глицин, серин, треонин, цистеин, тирозин, аспарагин, глутамин; 3) аминокислоты с заряженными полярными группами R (отрицательно заряженными при рН 6,0): аспарагиновая кислота, глутаминовая кислота; 4) основные аминокислоты (положительно заряженные при рН 6,0): лизин, аргинин, гистидин (при рН 6,0). Другая группа может представлять собой те аминокислоты, которые имеют фенильные группы: фенилаланин, триптофан и тирозин.
Домен scFv против CS1
Домен scFv против CS1 может представлять собой любой scFv, который связывается с CS1. В некоторых случаях домен scFv против CS1 включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной SEQ ID NO: 1. В некоторых случаях scFv против CS1 имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 1. scFv может представлять собой гуманизированный scFv.
Спейсерная область
CAR, описанный в данном документе, может включать спейсерную область, расположенную между доменом нацеливания на CS1 (т.е. scFv против CS1 или его вариант) и трансмембранным доменом. Можно использовать множество разных спейсеров. Некоторые из них включают по меньшей мере часть человеческой области Fc, например, шарнирную часть человеческой области Fc или домен СН3, или их варианты. В Таблице 1 ниже приведены разные спейсеры, которые можно использовать в CAR, описанных в данном документе.


Некоторые спейсерные области включают всю шарнирную область иммуноглобулина (например, IgG1, IgG2, IgG3, IgG4) или ее часть, т.е. последовательность, которая попадает между доменами СН1 и СН2 иммуноглобулина, например, шарнир Fc IgG4 или шарнир CD8. Некоторые спейсерные области включают домен СН3 иммуноглобулина или и домен СН3, и домен СН2. Последовательности, происходящие из иммуноглобулина, могут включать одну или более чем одну модификацию аминокислот, например, 1, 2, 3, 4 или 5 замен, например, замены, которые уменьшают связывание вне мишени.
В некоторых воплощениях спейсер представляет собой шарнир/линкер, происходящий из IgG1, IgG2, IgG3 или IgG4, который включает один или более чем один аминокислотный остаток, замененный аминокислотным остатком, отличным от остатка, который присутствует в немодифицированном шарнире. Данный один или более чем один замененный аминокислотный остаток выбран из одного или более чем одного аминокислотного остатка в положениях 220, 226, 228, 229, 230, 233, 234, 235, 234, 237, 238, 239, 243, 247, 267, 268, 280, 290, 292, 297, 298, 299, 300, 305, 309, 218, 326, 330, 331, 332, 333, 334, 336, 339 или их комбинаций, но не ограничивается ими.
В некоторых воплощениях модифицированный шарнир или шарнир/линкер происходит из IgG1, IgG2, IgG3 или IgG4, который включает одну или более чем одну из следующих замен аминокислотных остатков: C220S, C226S, S228P, C229S, P230S, Е233Р, V234A, L234V, L234F, L234A, L235A, L235E, G236A, G237A, P238S, S239D, F243L, P247I, S267E, H268Q, S280H, K290S, K290E, K290N, R292P, N297A, N297Q, S298A, S298G, S298D, S298V, Т299А, Y300L, V305I, V309L, Е318А, K326А, K326W, K326Е, L328F, A330L, A330S, A331S, P331S, I332E, Е333А, E333S, E333S, K334А, A339D, A339Q, P396L или их комбинаций, но не ограничивается ими.
В некоторых воплощениях модифицированный шарнир происходит из области шарнира/СН2/СН3 человеческого IgG4, имеющей аминокислотную последовательность SEQ ID NO: 10 или 11, или аминокислотную последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентична SEQ ID NO: 10 или 11.
В некоторых воплощениях модифицированный шарнир происходит из IgG4, который включает один или более чем один аминокислотный остаток, замененный аминокислотным остатком, отличным от остатка, присутствующего в немодифицированном шарнире. Данный один или более чем один замененный аминокислотный остаток выбран из одного или более чем одного аминокислотного остатка в положениях 220, 226, 228, 229, 230, 233, 234, 235, 234, 237, 238, 239, 243, 247, 267, 268, 280, 290, 292, 297, 298, 299, 300, 305, 309, 218, 326, 330, 331, 332, 333, 334, 336, 339 или их комбинаций, но не ограничивается ими.
В некоторых воплощениях модифицированный шарнир происходит из IgG4, который включает одну или более чем одну из следующих замен аминокислотных остатков: 220S, 226S, 228Р, 229S, 230S, 233Р, 234А, 234V, 234F, 234А, 235А, 235Е, 236А, 237А, 238S, 239D, 243L, 247I, 267Е, 268Q, 280Н, 290S, 290Е, 290N, 292Р, 297А, 297Q, 298А, 298G, 298D, 298V, 299А, 300L, 305I, 309L, 318А, 326А, 326W, 326Е, 328F, 330L, 330S, 331S, 331S, 332Е, 333А, 333S, 333S, 334А, 339D, 339Q, 396L и их комбинаций, но не ограничивается ими, где аминокислота в немодифицированном шарнире заменяется идентифицированными выше аминокислотами в указанном положении. В одном случае данная последовательность включает следующие аминокислотные замены: S228P, L235E и N297Q.
Для обсуждавшихся в данном документе положений аминокислот в иммуноглобулине нумерация осуществляется согласно индексу EU (Европейский Союз) или схеме нумерации EU (Kabat et al. 1991 Sequences of Proteins of Immunological Interest, 5th Ed., United States Public Health Service, National Institutes of Health, Bethesda, являющаяся тем самым включенной посредством ссылки). Индекс EU или индекс EU по Kabat, или схема нумерации EU относится к нумерации антитела по EU (Edelman et al. 1969 Proc Natl Acad Sci USA 63:78-85).
Шарнирная/линкерная область также может содержать шарнирную область IgG4, имеющую последовательность ESKYGPPCPSCP (SEQ ID NO: 4) или ESKYGPPCPPCP (SEQ ID NO: 3).
Шарнирная/линкерная область также может содержать последовательность ESKYGPPCPPCP (SEQ ID NO: 3), с последующей линкерной последовательностью GGGSSGGGSG (SEQ ID NO: 2), с последующей последовательностью СН3 IgG4 GQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVL DSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK (SEQ ID NO: 12). Таким образом, вся линкерная/спейсерная область может содержать последовательность:
ESKYGPPCPPCPGGGSSGGGSGGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYP SDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEA LHNHYTQKSLSLSLGK (SEQ ID NO: 11). В некоторых случаях спейсер имеет 1, 2, 3, 4 или 5 замен одиночных аминокислот (например, консервативных замен) по сравнению с SEQ ID NO: 11. В некоторых случаях шарнирная/линкерная область Fc IgG4 мутируется в двух положениях (L235E; N297Q) таким способом, который уменьшает связывание посредством рецепторов Fc (FcR).
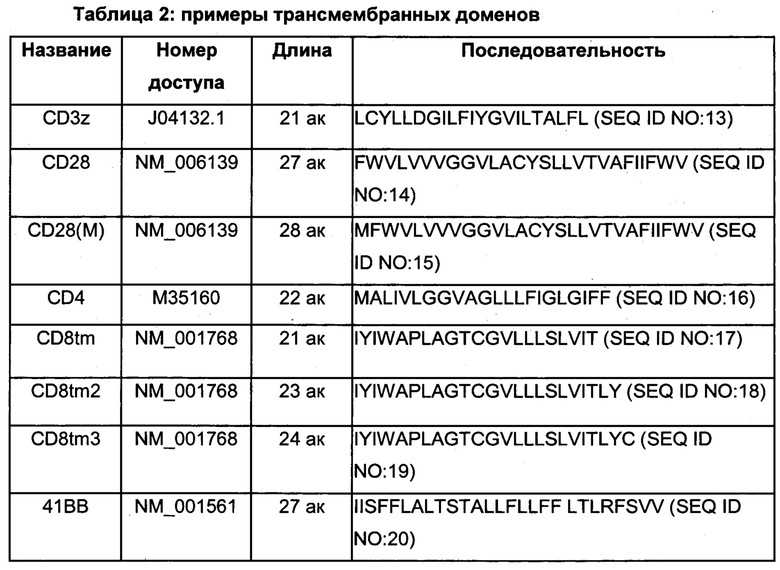
Трансмембранная область
Можно использовать целый ряд трансмембранных доменов. Таблица 2 включает примеры подходящих трансмембранных доменов. Когда присутствует спейсерная область, трансмембранный домен локализуется ближе к карбокси концу относительно спейсерной области.
Костимулирующий домен
Костимулирующий домен может представлять собой любой домен, который подходит для применения с доменом сигнализации CD3ζ. В некоторых случаях костимулирующий домен представляет собой костимулирующий домен CD28, который включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или полностью идентичной  (SEQ ID NO: 23; аминокислотная замена LL до GG дважды подчеркнута). В некоторых случаях домен косигнализации CD28 имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных и предпочтительно не в подчеркнутой последовательности GG) по сравнению с SEQ ID NO: 23. В некоторых случаях домен косигнализации представляет собой домен косигнализации 4-1ВВ, который включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL (SEQ ID NO: 24). В некоторых случаях домен косигнализации 4-1ВВ имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 24.
(SEQ ID NO: 23; аминокислотная замена LL до GG дважды подчеркнута). В некоторых случаях домен косигнализации CD28 имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных и предпочтительно не в подчеркнутой последовательности GG) по сравнению с SEQ ID NO: 23. В некоторых случаях домен косигнализации представляет собой домен косигнализации 4-1ВВ, который включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL (SEQ ID NO: 24). В некоторых случаях домен косигнализации 4-1ВВ имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 24.
Костимулирующий(щие) домен(ны) расположен(жены) между трансмембранным доменом и доменом сигнализации CD3ζ. Таблица 3 включает примеры подходящих костимулирующих доменов вместе с последовательностью домена сигнализации CD3ζ.

В разных воплощениях костимулирующий домен выбран из группы, состоящей из костимулирующего домена, показанного в Таблице 3, или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот, костимулирующего домена CD28 или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот, костимулирующего домена 4-1ВВ или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот, и костимулирующего домена ОХ40 или его варианта, имеющего 1-5 (например, 1 или 2) модификаций аминокислот. В некоторых воплощениях присутствует костимулирующий домен 4-1ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот. В некоторых воплощениях имеются два костимулирующих домена, например, костимулирующий домен CD28 или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен), и костимулирующий домен 4-1ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замен). В разных воплощениях 1-5 (например, 1 или 2) модификаций аминокислот представляют собой замены. Костимулирующий домен находится ближе к амино концу относительно домена сигнализации CD3ζ, и в некоторых случаях между костимулирующим доменом и доменом сигнализации CD3ζ располагается короткий линкер, состоящий из 2-10, например, 3 аминокислот (например, GGG).
Домен сигнализации CD3ζ
Домен сигнализации CD3ζ может представлять собой любой домен, который подходит для применения с доменом сигнализации CD3ζ. В некоторых случаях домен сигнализации CD3ζ включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или полностью идентичной RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQE GLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 21). В некоторых случаях домен сигнализации CD3ζ имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 21.
Усеченный EGFR
После домена сигнализации CD3ζ; может следовать последовательность перескакивания рибосомы (например, LEGGGEGRGSLLTCGDVEENPGPR; SEQ ID NO: 27) и усеченный EGFR, имеющий последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или полностью идентичной: LVTSLLLCELPHPAFLLIPRKVCNGIGIGEFKDSLSINATNIKHFKNCTSISGDLHILPVAFR GDSFTHTPPLDPQELDILKTVKEITGFLLIQAWPENRTDLHAFENLEIIRGRTKQHGQFSL AWSLNITSLGLRSLKEISDGDVIISGNKNLCYANTINWKKLFGTSGQKTKIISNRGENSC KATGQVCHALCSPEGCWGPEPRDCVSCRNVSRGRECVDKCNLLEGEPREFVENSECI QCHPECLPQAMNITCTGRGPDNCIQCAHYIDGPHCVKTCPAGVMGENNTLVWKYADA GHVCHLCHPNCTYGCTGPGLEGCPTNGPKIPSIATGMVGALLLLLVVALGIGLFM (SEQ ID NO: 28). В некоторых случаях усеченный EGFR имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 28.
CAR CS1
CAR CS1 может включать последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или полностью идентичной аминокислотной последовательности, показанной на Фиг. 2, Фиг. 6, Фиг. 7, Фиг. 8, Фиг. 9 или Фиг. 10 (SEQ ID NO: 29-46; либо включающие, либо исключающие сигнальную последовательность GMCSFRa, и либо включающие, либо исключающие последовательность перескакивания рибосомы Т2А и усеченный EGFRt).
Среди CAR, нацеленных на CS1, описанных в данном документе, находятся CAR, обобщенные в Таблице 4, в которой указываются спейсерная область, трансмембранный домен и костимулирующий(щие) домен(ны) для каждого CAR.
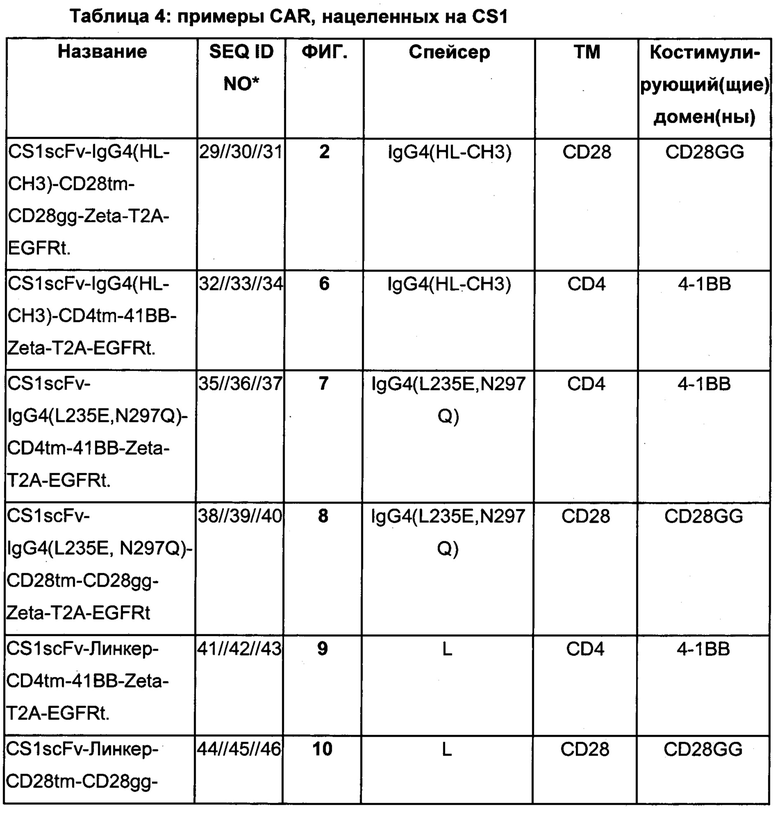

* SEQ ID NO для: полной показанной последовательности, включающей сигнальную последовательность GMCSFRa, Т2А и EGFRt // последовательности, включающей сигнальную последовательность GMCSFRa, но исключающей Т2А и EGFRt // последовательности, исключающей сигнальную последовательность GMCSFRa, Т2А и EGFRt.
ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Фиг. 1 представляет собой схематическую иллюстрацию лентивирусного вектора, экспрессирующего CAR CS1 (CS1scFv-IgG4(HL-CH3)-CD28gg-Zeta-(CO)-T2A-EGFRt_epHIV7). Конструкция CAR CS1 включает: сигнальную последовательность GMCSF, scFv против CS1, шарнирную область IgG4, линкер, домен СН3, костимулирующий домен CD28 и домен сигнализации CD3ζ. После конструкции CAR следует последовательность перескакивания рибосомы Т2А и затем последовательность, кодирующая суицидный ген EGFRt. Молекулы CAR и EGFRt экспрессируются от одного транскрипта.
На Фиг. 2 показана аминокислотная последовательность CAR CS1, которая включает сигнальный пептид, последовательность перескакивания рибосомы и EGFRt (SEQ ID NO: 29).
Фиг. 3 представляет собой пару графиков, иллюстрирующих результаты исследований, показывающих то, что TCM, перенаправленные на CAR CS1, демонстрировали цитотоксичность против клеток ММ. Цитотоксичность размноженных Т-клеток с CAR CS1 оценивали с использованием 4-часовых анализов высвобождения 51Cr после совместного культивирования с клетками-мишенями, меченными 51Cr. В качестве позитивных контролей использовали LCL, экспрессирующие OKT3, так как они занимают все TCR (рецептор Т-клетки), и CS1-негативные клетки AML (KG1a) использовали в качестве негативных контролей. CAR CS1, но не Т-клетки с имитацией модификации, не являющиеся генетически модифицированными, демонстрировали специфичную цитотоксичность против клеток ММ.
На Фиг. 4 показаны результаты исследований, демонстрирующих то, что клетки ТCM. перенаправленные на CAR CS1, демонстрировали эффекторную функцию в ответ на стимуляцию клеток ММ. Т-клетки с CAR CS1 (105) совместно культивировали 6 часов в 96-луночных планшетах для культуры ткани с 105 клеток MM.1S в качестве стимуляторов. Дегранулирование 107а и внутриклеточную продукцию IFN-гамма (интерферон-гамма) анализировали проточной цитометрией. Большинство Т-клеток с CAR, идентифицированных эрбитукс, индуцировали к дегранулированию после занятия клетками ММ, и IFN-гамма-позитивные клетки выявляли в ответ на стимуляцию антигеном.
На Фиг. 5 показаны результаты исследований, показывающих то, что клетки ТCM, перенаправленные на CAR CS1, устраняют множественную миелому in vivo. Приблизительно 2×106 клеток MM.1S, экспрессирующих люциферазу светляка, инокулировали в мышей NSG посредством инъекции в большую берцовую кость. Через 7 суток после инокуляции опухоли 1×106 Т-клеток с CAR CS1 инфундировали мышам, несущим опухоль, посредством внутривенной инъекции. Опухолевую нагрузку отслеживали визуализацией Xenogen® один раз в неделю. Мышей, которые получали клетки, не подвергавшиеся генетической модификации, использовали в качестве контроля. Т-клетки с CAR CS1 полностью устраняли опухоль ММ через 14 суток после инфузии Т-клеток, тогда как Т-клетки, не подвергавшиеся генетической модификации, не имеют влияния на ингибирование опухоли.
На Фиг. 6 показана аминокислотная последовательность CS1scFv-IgG4(HL-CH3)-CD4tm-41BB-Zeta-T2A-EGFRt (SEQ ID NO: 32).
На Фиг. 7 показана аминокислотная последовательность CS1scFv-IgG4(L235E,N297Q)-CD4tm-41BB-Zeta-T2A-EGFRt (SEQ ID NO: 35).
На Фиг. 8 показана аминокислотная последовательность CS1scFv-IgG4(L235E, N297Q)-CD28tm-CD28gg-Zeta-T2A-EGFRt (SEQ ID NO: 38).
На Фиг. 9 показана аминокислотная последовательность CS1scFv-Линкер-CD4tm-41BB-Zeta-T2A-EGFRt (SEQ ID NO: 41).
На Фиг. 10 показана аминокислотная последовательность CS1scFv-Линкер-CD28tm-CD28gg-Zeta-T2A-EGFRt (SEQ ID NO: 44).
Фиг. 11 представляет собой полную нуклеотидную последовательность CS1scFv-IgG4(HL-CH3)-CD28gg-Zeta-T2A-EGFRt_epHIV7 (SEQ ID NO: 47).
На Фиг. 12 показаны результаты исследований, показывающих то, что клетки ТCM, перенаправленные на CAR CS1, устраняют множественную миелому in vivo. 2×106 клеток MM.1S GFPffluc+ инокулировали в мышей NSG посредством инъекции в большую берцовую кость в сутки -7. 1×106 Т-клеток CAR CS1+, происходящих из центральных Т-клеток памяти (ТСM), внутривенно инфундировали мышам, несущим опухоль, в сутки 0. Мышей, не получавших Т-клетки или получавших нетрансдуцированные TCM от того же самого донора, использовали в качестве негативных контролей. Сигналы опухоли отслеживали посредством биофотонной визуализации. Показаны средние значения плюс/минус SEM (стандартная ошибка среднего) фотонов/с от многих мышей. CAR представляли собой CAR Фиг. 2 (СН2 CD28); Фиг. 6 (СН2 41ВВ); Фиг. 8 (EQ CD28); Фиг. 7 (EQ 41ВВ); Фиг. 10 (L CD28) и Фиг. 9 (L CD4 IBB).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Ниже описана структура, конструирование и характеризация нескольких CS1-специфичных химерных рецепторов антигена («CAR»). CAR представляет собой рекомбинантную биомолекулу, которая содержит внеклеточный домен распознавания, трансмембранную область и внутриклеточный домен сигнализации. Термин «антиген», следовательно, не ограничивается молекулами, которые связываются антителами, но ограничивается любой молекулой, которая может специфично связываться с любым рецептором. «Антиген», таким образом, относится к домену, распознаваемому CAR. Внеклеточный домен распознавания (также именуемый внеклеточным доменом или просто элементом распознавания, который он содержит) содержит элемент распознавания, который специфично связывается с молекулой, присутствующей на клеточной поверхности клетки-мишени. Трансмембранная область заякоривает CAR в мембране. Домен внутриклеточной сигнализации содержит домен сигнализации из зета-цепи человеческого комплекса CD3 и возможно содержит один или более чем один костимулирующий домен сигнализации. CAR может и связываться с антигеном, и трансдуцировать активацию Т-клеток, независимо от ограничения МНС. Таким образом, CAR представляют собой «универсальные» иммунорецепторы, которыми можно лечить популяцию пациентов с антиген-позитивными опухолями, независимо от их генотипа HLA (антиген лейкоцитов человека). Адоптивная иммунотерапия с использованием Т-лимфоцитов, которые экспрессируют опухолеспецифичный CAR, может быть мощной терапевтической стратегией для лечения онкологического заболевания.
В некоторых случаях CAR CS1 можно продуцировать с использованием вектора, в котором за открытой рамкой считывания CAR следует последовательность перескакивания рибосомы Т2А и усеченный EGFR (EGFRt), у которого отсутствует цитоплазматический хвост сигнализации. При данной организации коэкспрессия EGFRt дает инертный неиммуногенный маркер поверхности, который обеспечивает точное измерение генно-модифицированных клеток и обеспечивает позитивный отбор генно-модифицированных клеток, а также эффективное отслеживание терапевтических Т-клеток in vivo после адоптивного переноса. Эффективное осуществление контроля пролиферации для того, чтобы избежать цитокиновой бури и токсичности вне мишени, является важной проблемой для успеха Т-клеточной иммунотерапии. EGFRt, включенный в лентивирусный вектор с CAR CS1, может действовать в качестве суицидного гена для устранения Т-клеток CAR+ в случаях токсичности, связанной с лечением.
Описанный здесь CAR может быть получен любыми способами, известными в данной области, хотя предпочтительно он продуцируется с использованием методик генной инженерии. Нуклеиновые кислоты, кодирующие несколько областей химерного рецептора, могут быть получены и собраны в полную кодирующую последовательность стандартными методиками молекулярного клонирования, известными в данной области (скрининг геномной библиотеки, ПЦР (полимеразная цепная реакция) с перекрывающимися праймерами, лигирование с помощью праймеров, сайт-направленный мутагенез и т.д.), в зависимости от того, что является удобным. Образующуюся кодирующую область предпочтительно вставляют в экспрессионный вектор и используют для трансформации подходящей экспрессионной линии клеток-хозяев, предпочтительно, линии клеток Т-лимфоцитов и наиболее предпочтительно - линии аутологичных клеток Т-лимфоцитов.
Разные поднаборы Т-клеток, выделенных из пациента, можно трансдуцировать вектором для экспрессии CAR. Центральные Т-клетки памяти представляют собой один полезный поднабор Т-клеток. Центральные Т-клетки памяти могут быть выделены из одноядерных клеток периферической крови (РВМС) посредством отбора на клетки CD45RO+/CD62L+, используя, например, прибор CliniMACS® для иммуномагнитного отбора клеток, экспрессирующих желательные рецепторы. Клетки, обогащенные в отношении центральных Т-клеток памяти, могут быть активированы с использованием антител против CD3/CD28, трансдуцированы, например, лентивирусным вектором, который направляет экспрессию CAR CS1, а также неиммуногенного поверхностного маркера для выявления in vivo, устранения и потенциального отбора ex vivo. Активированные/генетически модифицированные центральные Т-клетки памяти с CAR CS1 могут быть размножены in vitro с использованием IL-2/IL15 и затем подвергнуты криоконсервации.
Пример 1: конструирование и структура epHIV7, используемого для экспрессии CS1-специфичного CAR
Плазмида pHIV7 представляет собой родительскую плазмиду, из которой могут быть получены клинические векторы, экспрессирующие CAR CS1. Вектор epHIV7, используемый для экспрессии CAR, был получен из вектора pHIV7 (Wang et al. 2011 Blood 118:1255). Важно то, что в данном векторе для управления экспрессией CAR используется человеческий промотор EF1. И 5', и 3'-последовательности вектора были получены из pv653RSN, который был ранее получен из провируса НХВс2. Вытесняемые последовательности ДНК полипуринового тракта (сРРТ) получали из штамма pNL4-3 ВИЧ-1 из репозитория реактивов СПИД NIH (Национальные институты здравоохранения США).
Конструирование pHIV7 проводили следующим образом. Вкратце, pv653RSN, содержащий 653 п. о. из gag-pol плюс 5' и 3' длинных концевых повторов (LTR) с расположенным между ними геном SI-3-неомицинфосфотрансферазы (Neo), субклонировали в pBluescript следующим образом: на стадии 1, последовательности от 5' LTR до элемента, отвечающего на rev (RRE), давали р5' HIV-1 51, и затем 5' LTR модифицировали посредством удаления последовательностей выше ТАТА-бокса и лигировали сначала с энхансером CMV (цитомегаловирус) и затем с репликатором SV40 (p5'HIV-2). На стадии 2 после клонирования 3' LTR в pBluescript с получением p3'HIV-1 получали делецию в 400 п.о. в 3' LTR энхансера/промотора для удаления цис-регуляторных элементов в U3 ВИЧ и образования p3'HIV-2. На стадии 3 фрагменты, выделенные из p5'HIV-3 и p3'HIV-2, лигировали с получением pHIV-3. На стадии 4 p3'HIV-2 модифицировали далее посредством удаления дополнительных расположенных выше последовательностей ВИЧ с получением p3'HIV-3, и фрагмент BamHI-Sall из 600 п.о., содержащий WPRE, добавляли к p3'HIV-3 с получением p3HIV-4. На стадии 5 размер RRE pHIV3 уменьшали посредством ПЦР и лигировали с 5'-фрагментом из pHIV-3 (не показано) и с p3'HIV-4 с получением pHIV-6. На стадии 6 фрагмент BgIII-BamHI из 190 п.о., содержащий вытесняемую последовательность ДНК сРРТ из pNL4-3 (55) ВИЧ-1, амплифицировали из pNL4-3 и помещали между последовательностями RRE и WPRE в pHIV-б с получением pHIV-7. Данную родительскую плазмиду pHIV7-GFP (GFP - зеленый флуоресцентный белок) использовали для упаковки родительского вектора с использованием четырехплазмидной системы.
Для эффективной упаковки вирусного генома в вектор требуется сигнал упаковки - пси ψ. RRE и WPRE усиливают транспорт РНК-транскрипта и экспрессию трансгена. Было продемонстрировано то, что вытесняемая последовательность в комбинации с WPRE усиливает эффективность трансдукции лентивирусного вектора в клетках млекопитающих.
Хелперные функции, требующиеся для продукции вирусного вектора, разделены на три разные плазмиды для уменьшения вероятности получения лентивируса, компетентного в отношении репликации, посредством рекомбинации: 1) pCgp кодирует белок gag/pol, требующийся для сборки вирусного вектора; 2) pCMV-Rev2 кодирует белок Rev, который действует на последовательность RRE, помогая в транспортировке вирусного генома для эффективной упаковки; и 3) pCMV-G кодирует гликопротеин вируса везикулостоматита (VSV), который требуется для инфективности вирусного вектора.
Существует минимальная гомология последовательности ДНК между геномом вектора, кодируемым pHIV7, и хелперными плазмидами. Области гомологии включают область сигнала упаковки из приблизительно 600 нуклеотидов, расположенную в последовательности gag/pol хелперной плазмиды pGgp; последовательность промотора CMV во всех трех хелперных плазмидах и последовательность RRE в хелперной плазмиде pCgp. Очень маловероятно то, что рекомбинантный вирус, компетентный в отношении репликации, мог бы быть генерирован из-за гомологии в данных областях, посколько это потребовало бы многих событий рекомбинации. Дополнительно, любые полученные в результате рекомбинанты не имели бы функциональных LTR и последовательностей tat, требующихся для репликации лентивируса.
Промотор CMV был заменен промотором EF1α-HTLV (EF1p), и новая плазмида была названа epHIV7. EF1p имеет 563 п.о., и он был введен в epHIV7 с использованием NruI и NheI после того, как промотор CMV был вырезан.
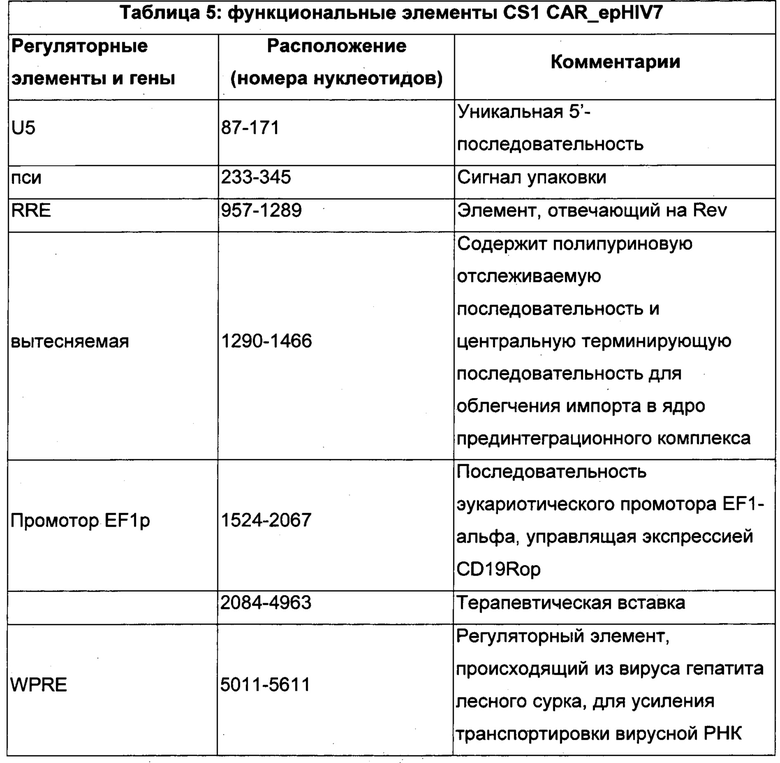
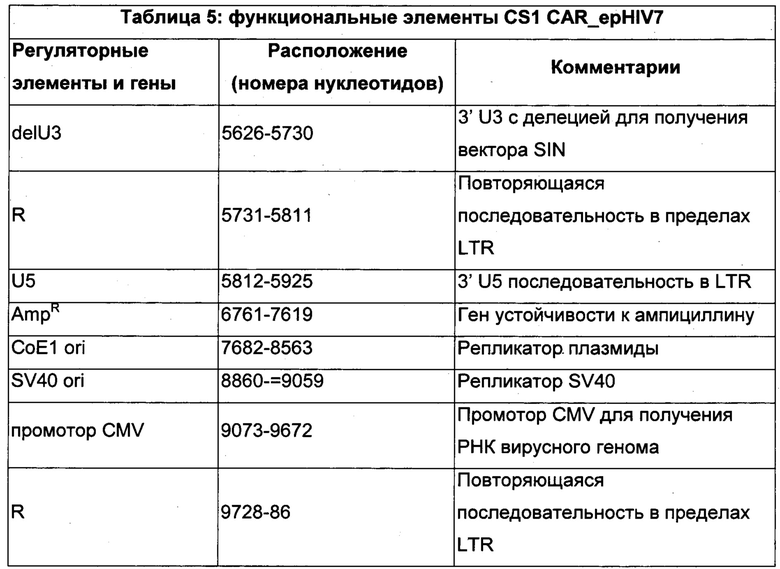
Из данной системы был удален лентивирусный геном, за исключением gag/pol и rev, которые необходимы для патогенности вируса дикого типа и требуются для продуктивной инфекции клеток-мишеней. Кроме того, конструкция вектора epHIV7 не содержит интактного промотора 3'LTR, таким образом, что образующийся в результате экспрессируемый и подвергающийся обратной транскрипции провирусный ДНК-геном в клетках-мишенях будет иметь неактивные LTR. В результате этой конструкции последовательности, происходящие от ВИЧ-1, не будут транскрибироваться от провируса, и только терапевтические последовательности будут экспрессироваться от их соответствующих промоторов. Ожидается то, что удаление промоторной активности LTR в векторе SIN значительно понизит возможность непреднамеренной активации генов хозяина. В Таблице 5 обобщены разные регуляторные элементы, присутствующие в epHIV7.
Фиг. 1 представляет собой схематическое изображение CAR CS1 (CS1scFv-IgG4(HL-CH3)-CD28gg-Zeta(CO)-T2A-EGFRt_epHIV7) - лентивирусного вектора, содержащего конструкцию CAR, состоящую scFv против CS1, шарнирной области IgG4, линкера, костимулирующего домена CD28 и домена сигнализации CD3ζ. После данной конструкции CAR следует последовательность перескакивания рибосомы Т2А и затем кодирующая последовательность суицидного гена EGFRt. Молекулы CAR и EGFRt экспрессируются от одного транскрипта. Вся нуклеотидная последовательность вектора представлена на Фиг. 11, и в Таблице 5 представлено расположение разных элементов вектора.
Пример 2: получение векторов для трансдукции Т-клеток пациента
Для каждой плазмиды (CS1 CAR_epHIV7; pCgp; pCMV-G и pCMV-Rev2) генерируется фонд вакцинных штаммов, который используется для инокуляции ферментера с получением достаточного количества плазмидной ДНК. Плазмидная ДНК тестируется на идентичность, стерильность и уровень эндотоксинов до ее применения в продуцировании лентивирусного вектора.
Вкратце, клетки размножают из рабочей клетки 293Т (WCB), которая была протестирована для подтверждения стерильности и отсутствия вирусного загрязнения. Флакон с клетками 293Т из WCB 293Т оттаивают. Клетки выращивают и размножают до существования достаточного числа клеток для засева подходящего числа 10-слойных клеточных фабрик (CF) для продукции вектора и поддержания ряда клеток. Для продукции можно использовать один ряд клеток.
Лентивирусный вектор продуцируется в подпартиях из вплоть до 10 CF. В ту же самую неделю можно продуцировать две подпартии, что приводит к продукции приблизительно 20 л лентивирусного супернатанта/неделю. Материал, полученный из всех подпартий, объединяли во время фазы последующей переработки для того, чтобы произвести одну партию продукта. Клетки 293Т высевали в CF в среду для 293Т (DMEM (среда Игла, модифицированная по Дульбекко) с 10% FBS (фетальная телячья сыворотка)). Фабрики помещали в инкубатор при 37°C и горизонтально выравнивали для того, чтобы получать равное распределение клеток на всех слоях CF. Через двое суток клетки трансфицировали четырьмя лентивирусными плазмидами, описанными выше, с использованием способа с CaPO4, который включает смесь Tris:EDTA, 2М CaCl2, 2× HBS и четырех ДНК-плазмид. В сутки 3 после трансфекции супернатант, содержащий секретированные лентивирусные векторы, отбирали, очищали и концентрировали. После удаления супернатанта из CF послепроизводственные клетки собирали из каждой CF. Клетки трипсинизировали из каждой фабрики и собирали центрифугированием. Клетки ресуспендировали в среде для замораживания и криоконсервировали. Данные клетки позднее использовали для тестирования лентивируса, компетентного в отношении репликации (RCL).
Для очистки и приготовления векторов в виде препарата неочищенный супернатант осветляли мембранной фильтрацией для удаления обломков клеток. ДНК клетки-хозяина и остаточную плазмидную ДНК деградировали эндонуклеазным расщеплением (Бензоназа®). Вирусный супернатант осветляли от клеточных обломков с использованием 0,45 мкм фильтра. Осветленный супернатант отбирали в предварительно взвешенный контейнер, в который добавляется Бензоназа® (конечная концентрация 50 U(ед)/мл). Эндонуклеазное расщепление остаточной плазмидной ДНК и геномной ДНК хозяина проводится при 37°C в течение 6 ч. Использовали исходное концентрирование супернатанта, обработанного эндонуклеазой, посредством ультрафильтрации с тангенциальным потоком (TFF) для удаления остаточных низкомолекулярных компонентов из неочищенного супернатанта при ~20-кратном концентрировании вируса. Осуществляли циркуляцию осветленного вирусного супернатанта, обработанного эндонуклеазой, через картридж на основе полых волокон с NMWCO (номинальное отсечение по молекулярной массе) 500 кДа при скорости тока, разработанной для поддержания срезывающего усилия ~4000 с-1 или менее при максимизации скорости потока. Диафильтрацию супернатанта, обработанного нуклеазой, инициировали во время процесса концентрирования для поддержания производительности картриджа. С использованием 4% лактозы в PBS (фосфатно-солевой буферный раствор) в качестве диафильтрационного буфера устанавливали скорость замены пермеата 80%. Вирусный супернатант доводили до целевого объема, представляющего собой 20-кратное концентрирование неочищенного супернатанта, и диафильтрацию продолжали в течение 4 дополнительных объемов обмена со скоростью замены пермеата 100%.
Дополнительное концентрирование вирусного продукта осуществляли с использованием методики высокоскоростного центрифугирования. Каждую подпартию лентивируса осаждали с использованием центрифуги Sorvall RC-26 plus при скорости 6000 об./мин (6088 RCF (относительная центробежная сила)) при 60°C в течение 16-20 ч. Вирусный осадок из каждой подпартии затем разводили в объеме 50 мл с использованием 4% лактозы в PBS. Растворенный в данном буфере осадок представляет собой конечную композицю для препарата вируса. Весь способ концентрирования вектора приводил приблизительно к 200-кратному уменьшению объема. После завершения получения всех подпартий материал помещали при -80°C, тогда как образцы из каждой подпартии тестировали на стерильность. После подтверждения стерильности образца подпартии быстро оттаивали при 37°C с частым встряхиванием. Вещество затем объединяли и вручную аликвотировали в боксе микробиологической безопасности класса II типа А/В3 в помещении для вирусных векторов. Использовали конфигурацию заполнения концентрированного лентивируса 1 мл в стерильных криофлаконах с внешней резьбой и О-кольцом класса 6 USP (Фармакопея США). Центр разработки прикладных технологий (CATD) систем контроля качества (QS) СОН (City of Hope) выпустил все материалы согласно Политике и стандартным технологическим регламентам для CBG (Клеточная биология и генетика) и в соответствии с современными надлежащими производственными практиками (cGMP).
Для обеспечения чистоты препарата лентивирусного вектора он тестируется на остаточные загрязнения ДНК хозяина и перенос остаточной ДНК хозяина и плазмиды. Наряду с другими анализами, идентичность вектора оценивается посредством ПЦР-ОТ (полимеразная цепная реакция с обратной транскрипцией) для обеспечения того, что присутствует правильный вектор. Для вектора, предназначенного для применения в данном исследовании, удовлетворяются все критерии выпуска.
Пример 3: приготовление клеток TCM, подходящих для применения в ACT
Т-лимфоциты получают от пациента посредством лейкафереза, и набор подходящих аллогенных или аутологичных Т-клеток, например, центральных Т-клеток памяти (TCM), генетически изменяют для экспрессии CAR, затем вводят обратно пациенту посредством любых клинически приемлемых способов для достижения противоопухолевой терапии.
ТCM, которые являются CD8+, выделяют по существу, как описано в Wang et al. (J Immunology 35:689, 2012). Вкратце, в сутки лейкафереза РВМС (одноядерные клетки периферической крови) выделяли посредством центрифугирования в градиенте плотности над Ficoll-Paque, с последующими двумя промывками в PBS/EDTA. РВМС затем один раз промывали в PBS, ресуспендировали в средах X Vivo 15, содержащих 10% фетальной телячей сыворотки (FCS), переносили в 300 см3 мешок для переноса и хранили на 3-D (трехмерный) ротаторе в течение ночи при комнатной температуре (RT). На следующие сутки вплоть до 5×109 РВМС инкубировали в 300 см3 мешке для переноса с микрошариками клинического класса качества, покрытыми антителами против CD4 (2,5 мл), против CD14 (1,25 мл) и против CD45RA (2,5 мл) (Miltenyi Biotec) в течение 30 минут при RT в X Vivo15, содержащей 10% FCS. Затем немедленно осуществляли обеднение клетками CD4+, CD14+ и CD45RA+ с использованием режима обеднения CliniMACS™ согласно инструкциям изготовителя (Miltenyi Biotec). После центрифугирования немеченую негативную фракцию клеток ресуспендировали в буфере CliniMACS™ PBS/EDTA (Miltenyi Biotec), содержащем 0,5% человеческого сывороточного альбумина (HSA), и затем метили биотинилированным mAb (моноклональное антитело) DREG56 клинического класса качества (COHNMC CBG) в соотношении 0,1 мг/106 клеток в течение 30 минут при RT. Клетки затем промывали и ресуспендировали в конечном объеме 100 мл CliniMACS™ PBS/EDTA, содержащем 0,5% HSA, и переносили в новый 300 см3 мешок для переноса. После 30 минут инкубации с 1,25 мл микрошариков против биотина (Miltenyi Biotec) фракцию РВМС CD62L+ (TCM CD8+) очищали с использованием позитивного отбора на CliniMACS™ согласно инструкциям изготовителя и ресуспендирвали в X Vivo15, содержащей 10% FCS.
ТCM, которые являются CD8+/CD4+, готовят с использованием модификации вышеописанного способа посредством модификации отбора на CD4+, CD14+ и CD45RA+ на отбор на CD14+ и CD45RA+. В данном способе используется двухэтапный способ на устройстве CliniMACS™ для исходного обеднения клетками CD14+ и CD45RA+, затем для позитивного отбора клеток CD62L+. Данная модифицированная платформа генерирует объем 50×106 TCM из одного лейкафереза.
После обогащения клетки TCM готовятся в виде препарата в полной X-Vivo15 плюс 50 IU (международная единица)/мл IL-2 и 0,5 нг/мл IL-15 и переносятся в тефлоновый мешок для культуры клеток, где они стимулируются шариками Dynal ClinEx™ Vivo CD3/CD28. Вплоть до пяти суток после стимуляции клетки трансдуцируют лентивирусным вектором, кодирующим CAR CS1, при множественности заражения (MOI) примерно 3. Культуры поддерживаются в течение вплоть до 42 суток с добавлением полной X-Vivo 15 и цитокина IL-2 и IL-15 в соответствии с потребностями для размножения клеток (поддерживая плотность клеток от 3×105 до 2×106 жизнеспособных клеток/мл и осуществляя пополнение цитокинами каждый понедельник, среду и пятницу культуры). Клетки типично размножаются при данных условиях приблизительно до 109 клеток в пределах 21 суток. В конце периода культивирования клетки отбирают, дважды промывают и готовят в виде препарата в криоконсервирующей среде клинического класса качества.
В сутки инфузии Т-клеток криоконсервированный и высвобожденный продукт будет оттаян, промыт и приготовлен в виде препарата для повторной инфузии. Криоконсервированные флаконы, содержащие высвобожденный клеточный продукт, будут удалены из хранилища с жидким азотом, оттаяны, охлаждены и промыты промывочным буфером на основе PBS/2% человеческого сывороточного альбумина (HSA). После центрифугирования супернатант будет удален, и клетки будут ресуспендированы в нормальном физиологическом растворе, не содержащем консервантов (РFNS)/разбавителе для инфузии на основе 2% HSA. Образцы будут удалены для тестирования для контроля качества.
Пример 4: аминокислотная последовательность CAR CS1 (CS1scFv-IgG4(HL-CH3)-CD28tm-CD28gg-Zeta-T2A-EGFRt)
Полная аминокислотная последовательность CS1scFv-IgG4(HL-CH3)-CD28tm-CD28gg-Zeta-T2A-EGFRt показана на Фиг. 2. Полная последовательность (SEQ ID NO: 29) включает: сигнальный пептид GMCSF (гранулоцитарно-макрофагальный колониестимулирующий фактор) из 22 аминокислот (SEQ ID NO: 26), последовательность scFv против CS1 (SEQ ID NO: 1); последовательность шарнира IgG4 (SEQ ID NO: 3; с выделенными затенением аминокислотными заменами S на Р); 10-аминокислотный линер (SEQ ID NO: 2); последовательность СН3 IgG4 (SEQ ID NO: 12); последовательность трансмембранного домена CD28 из 28 аминокислот (SEQ ID NO: 14); последовательность костимулирующего домена CD28gg (SEQ ID NO: 23; выделены аминокислотные замены от LL до GG); Gly линкер из 3 аминокислот; последовательность CD3ζ из 112 аминокислот (SEQ ID NO: 21); последовательность перескакивания рибосомы Т2А из 24 аминокислот (SEQ ID NO: 27) и последовательность EGFRt (SEQ ID NO: 28).
Пример 5: Активность CAR CS1
Цитотоксичность размноженных Т-клеток с CAR CS1, экспрессирующих CAR, показанный на Фиг. 2, оценивали с использованием 4-часовых анализов высвобождения 51Cr после сокультивирования с клетками ММ, меченными 51Cr (MM.1S). Как показано на Фиг. 3, генетически модифицированные Т-клетки с CAR CS1 демонстрируют специфичное и эффективное умерщвление клеток ММ, тогда как нетрансдуцированные Т-клетки с имитацией модификации не имели цитотоксичности в отношении клеток ММ. При сокультивировании с клетками ММ сильная эффекторная функция, опосредованная генетически модифицированными ТCM с CAR CS1, на которую указывает дегранулирование 107а и уровень IFN-гамма, показана на Фиг. 4. При адоптивном переносе мышам NSG, несущим опухоль MM, CS1-специфичные Т-клетки демонстрировали эффективную противоопухолевую активность, как показано на Фиг. 5.
В другом исследовании с дополнительным CAR CS1 (Фиг. 2 и Фиг. 6-10) 2×106 клеток MM.1S GFPffluc+ инокулировали посредством инъекции в большую берцовую кость мышам NSG в сутки -7. 1×106 Т-клеток CAR CS1+, происходящих из центральных Т-клеток памяти (TCM), внутривенно инфундировали мышам, несущим опухоль, в сутки 0. В качестве негативных контролей использовали мышей, не получавших Т-клеток, или получавших нетрансдуцированные TCM от того же самого донора. Опухолевые сигналы отслеживали биофотонной визуализацией. Показаны средние значения плюс/минус стандартная ошибка среднего фотонов/с от многих мышей. Результаты данного анализа показаны на Фиг. 12.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> City of Hope
<120> Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ НА CS1
<130> 40056-0006WO1
<140> PCT/US2015/064303
<141> 2015-12-07
<150> US 62/088,423
<151> 2014-12-05
<160> 47
<170> PatentIn version 3.5
<210> 1
<211> 244
<212> ПРТ
<213> Homo sapiens
<400> 1
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 2
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> линкер
<400> 2
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
1 5 10
<210> 3
<211> 12
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир IgG4 S228P
<400> 3
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
1 5 10
<210> 4
<211> 12
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир IgG4
<400> 4
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro
1 5 10
<210> 5
<211> 22
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир IgG4 (S228P)+ линкер
<400> 5
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser
1 5 10 15
Ser Gly Gly Gly Ser Gly
20
<210> 6
<211> 39
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир CD28
<400> 6
Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn
1 5 10 15
Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu
20 25 30
Phe Pro Gly Pro Ser Lys Pro
35
<210> 7
<211> 48
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир CD8
<400> 7
Ala Lys Pro Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
1 5 10 15
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
20 25 30
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 8
<211> 45
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир CD8
<400> 8
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 9
<211> 129
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> IgG4(HL-CH3) включает S228P в шарнире
<400> 9
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser
1 5 10 15
Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
20 25 30
Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
35 40 45
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
50 55 60
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
65 70 75 80
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys
85 90 95
Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu
100 105 110
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
115 120 125
Lys
<210> 10
<211> 229
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> IgG4(L235E,N297Q)
<400> 10
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Gln Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 11
<211> 229
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> IgG4(S228P, L235E,N297Q)
<400> 11
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe
1 5 10 15
Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Gln Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 12
<211> 107
<212> ПРТ
<213> Homo sapiens
<400> 12
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
1 5 10 15
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
20 25 30
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
35 40 45
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
50 55 60
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
65 70 75 80
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
85 90 95
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
100 105
<210> 13
<211> 21
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD3z
<400> 13
Leu Cys Tyr Leu Leu Asp Gly Ile Leu Phe Ile Tyr Gly Val Ile Leu
1 5 10 15
Thr Ala Leu Phe Leu
20
<210> 14
<211> 27
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD28
<400> 14
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 15
<211> 28
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD28M
<400> 15
Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser
1 5 10 15
Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 16
<211> 22
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD4
<400> 16
Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile
1 5 10 15
Gly Leu Gly Ile Phe Phe
20
<210> 17
<211> 21
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD8tm
<400> 17
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr
20
<210> 18
<211> 23
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD8tm2
<400> 18
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr
20
<210> 19
<211> 24
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен CD8tm3
<400> 19
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr Cys
20
<210> 20
<211> 27
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> трансмембранный домен 41BB
<400> 20
Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu
1 5 10 15
Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
20 25
<210> 21
<211> 112
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> костимулирующий домен CD3
<400> 21
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 22
<211> 41
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> костимулирующий домен CD28
<400> 22
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 23
<211> 41
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> костимулирующий домен CD28gg*
<400> 23
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 24
<211> 42
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> костимулирующий домен 41BB
<400> 24
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 25
<211> 42
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> костимулирующий домен OX40
<400> 25
Ala Leu Tyr Leu Leu Arg Arg Asp Gln Arg Leu Pro Pro Asp Ala His
1 5 10 15
Lys Pro Pro Gly Gly Gly Ser Phe Arg Thr Pro Ile Gln Glu Glu Gln
20 25 30
Ala Asp Ala His Ser Thr Leu Ala Lys Ile
35 40
<210> 26
<211> 22
<212> ПРТ
<213> Homo sapiens
<400> 26
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro
20
<210> 27
<211> 24
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> домен сигнализации CD3z
<400> 27
Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp
1 5 10 15
Val Glu Glu Asn Pro Gly Pro Arg
20
<210> 28
<211> 354
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> усеченный EGVF
<400> 28
Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu
1 5 10 15
Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys
20 25 30
Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys
35 40 45
Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly
50 55 60
Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile
65 70 75 80
Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp
85 90 95
Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile
100 105 110
Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser
115 120 125
Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp
130 135 140
Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr
145 150 155 160
Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile
165 170 175
Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys
180 185 190
His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp
195 200 205
Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys
210 215 220
Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu
225 230 235 240
Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr
245 250 255
Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile
260 265 270
Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu
275 280 285
Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His
290 295 300
Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu
305 310 315 320
Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met
325 330 335
Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu
340 345 350
Phe Met
<210> 29
<211> 960
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CAR CS1
<400> 29
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu
385 390 395 400
Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val
405 410 415
Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His
420 425 430
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
435 440 445
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
450 455 460
Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
465 470 475 480
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
485 490 495
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
500 505 510
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
515 520 525
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
530 535 540
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
545 550 555 560
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
565 570 575
Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr
580 585 590
Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val
595 600 605
Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile
610 615 620
Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser
625 630 635 640
Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser
645 650 655
Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser
660 665 670
Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys
675 680 685
Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu
690 695 700
Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly
705 710 715 720
Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn
725 730 735
Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp
740 745 750
Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn
755 760 765
Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser
770 775 780
Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala
785 790 795 800
Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val
805 810 815
Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn
820 825 830
Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile
835 840 845
Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr
850 855 860
Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly
865 870 875 880
Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn
885 890 895
Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys
900 905 910
His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys
915 920 925
Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly
930 935 940
Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met
945 950 955 960
<210> 30
<211> 579
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CAR CS1, исключающий T2A и EGFRt
<400> 30
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu
385 390 395 400
Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val
405 410 415
Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His
420 425 430
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
435 440 445
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
450 455 460
Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
465 470 475 480
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
485 490 495
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
500 505 510
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
515 520 525
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
530 535 540
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
545 550 555 560
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
565 570 575
Pro Pro Arg
<210> 31
<211> 557
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CAR CS1, исключающий сигнал GMCSFRa, T2A и EGFRt
<400> 31
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro
260 265 270
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
275 280 285
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
290 295 300
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
305 310 315 320
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
325 330 335
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
340 345 350
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
355 360 365
Leu Ser Leu Gly Lys Met Phe Trp Val Leu Val Val Val Gly Gly Val
370 375 380
Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp
385 390 395 400
Val Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met
405 410 415
Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala
420 425 430
Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys
435 440 445
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
450 455 460
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
465 470 475 480
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
485 490 495
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
500 505 510
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
515 520 525
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
530 535 540
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
545 550 555
<210> 32
<211> 955
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(HL-CH3)-CD4tm-41BB-Zeta-T2A-EGFRt
<400> 32
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val
385 390 395 400
Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe
405 410 415
Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
420 425 430
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
435 440 445
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val
450 455 460
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
465 470 475 480
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
485 490 495
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
500 505 510
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
515 520 525
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
530 535 540
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
545 550 555 560
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Leu Glu
565 570 575
Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu
580 585 590
Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val Thr Ser Leu Leu Leu
595 600 605
Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile Pro Arg Lys Val Cys
610 615 620
Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala
625 630 635 640
Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu
645 650 655
His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro
660 665 670
Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile
675 680 685
Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu
690 695 700
His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His
705 710 715 720
Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly
725 730 735
Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly
740 745 750
Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe
755 760 765
Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn
770 775 780
Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu
785 790 795 800
Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val
805 810 815
Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu
820 825 830
Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu
835 840 845
Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp
850 855 860
Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys
865 870 875 880
Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys
885 890 895
Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys Thr
900 905 910
Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro
915 920 925
Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu
930 935 940
Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met
945 950 955
<210> 33
<211> 574
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(HL-CH3)-CD4tm-41BB-Zeta
<400> 33
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val
385 390 395 400
Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe
405 410 415
Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
420 425 430
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
435 440 445
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val
450 455 460
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
465 470 475 480
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
485 490 495
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
500 505 510
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
515 520 525
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
530 535 540
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
545 550 555 560
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
565 570
<210> 34
<211> 552
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(HL-CH3)-CD4tm-41BB-Zeta, исключающий сигнал GMCSFRa
<400> 34
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro
260 265 270
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
275 280 285
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
290 295 300
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
305 310 315 320
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
325 330 335
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
340 345 350
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
355 360 365
Leu Ser Leu Gly Lys Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly
370 375 380
Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys
385 390 395 400
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
405 410 415
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
420 425 430
Gly Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala
435 440 445
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
450 455 460
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
465 470 475 480
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
485 490 495
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
500 505 510
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
515 520 525
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
530 535 540
His Met Gln Ala Leu Pro Pro Arg
545 550
<210> 35
<211> 1055
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E,N297Q)-CD4tm-41BB-Zeta-T2A-EGFRt
<400> 35
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly
500 505 510
Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
515 520 525
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
530 535 540
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly
545 550 555 560
Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys
675 680 685
Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val Thr
690 695 700
Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile Pro
705 710 715 720
Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu
725 730 735
Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile
740 745 750
Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe
755 760 765
Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr
770 775 780
Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn
785 790 795 800
Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg
805 810 815
Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile
820 825 830
Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val
835 840 845
Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp
850 855 860
Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn
865 870 875 880
Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu
885 890 895
Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser
900 905 910
Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu
915 920 925
Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln
930 935 940
Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly
945 950 955 960
Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro
965 970 975
His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr
980 985 990
Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His
995 1000 1005
Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys
1010 1015 1020
Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val
1025 1030 1035
Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu
1040 1045 1050
Phe Met
1055
<210> 36
<211> 674
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E,N297Q)-CD4tm-41BB-Zeta
<400> 36
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly
500 505 510
Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
515 520 525
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
530 535 540
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly
545 550 555 560
Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg
<210> 37
<211> 652
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E,N297Q)-CD4tm-41BB-Zeta, исключающий сигнал GMCSFRa
<400> 37
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Phe Gln Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val Leu Gly
465 470 475 480
Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys
485 490 495
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
500 505 510
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
515 520 525
Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe
530 535 540
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
545 550 555 560
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
565 570 575
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
580 585 590
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
595 600 605
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
610 615 620
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
625 630 635 640
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
645 650
<210> 38
<211> 1060
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E, N297Q)-CD28tm-CD28gg-Zeta-T2A-EGFRt
<400> 38
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
500 505 510
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
515 520 525
Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
530 535 540
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
545 550 555 560
Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly
675 680 685
Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met
690 695 700
Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala
705 710 715 720
Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu
725 730 735
Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys
740 745 750
Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe
755 760 765
Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu
770 775 780
Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln
785 790 795 800
Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu
805 810 815
Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val
820 825 830
Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile
835 840 845
Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala
850 855 860
Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr
865 870 875 880
Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln
885 890 895
Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro
900 905 910
Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val
915 920 925
Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn
930 935 940
Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn
945 950 955 960
Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His
965 970 975
Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met
980 985 990
Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val
995 1000 1005
Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro
1010 1015 1020
Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile
1025 1030 1035
Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala
1040 1045 1050
Leu Gly Ile Gly Leu Phe Met
1055 1060
<210> 39
<211> 679
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E, N297Q)-CD28tm-CD28gg-Zeta
<400> 39
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
500 505 510
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
515 520 525
Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
530 535 540
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
545 550 555 560
Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg
675
<210> 40
<211> 657
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(L235E, N297Q)-CD28tm-CD28gg-Zeta, исключающий сигнал GMCSFRa
<400> 40
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Phe Gln Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu Val Val
465 470 475 480
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
485 490 495
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp
500 505 510
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
515 520 525
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly
530 535 540
Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
545 550 555 560
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
565 570 575
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
580 585 590
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
595 600 605
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
610 615 620
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
625 630 635 640
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
645 650 655
Arg
<210> 41
<211> 836
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD4tm-41BB-Zeta-T2A-EGFRt
<400> 41
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu
275 280 285
Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys
290 295 300
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
305 310 315 320
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
325 330 335
Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly
450 455 460
Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met
465 470 475 480
Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala
485 490 495
Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu
500 505 510
Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys
515 520 525
Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe
530 535 540
Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu
545 550 555 560
Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln
565 570 575
Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu
580 585 590
Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val
595 600 605
Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile
610 615 620
Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala
625 630 635 640
Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr
645 650 655
Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln
660 665 670
Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro
675 680 685
Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val
690 695 700
Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn
705 710 715 720
Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn
725 730 735
Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His
740 745 750
Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met
755 760 765
Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val
770 775 780
Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly
785 790 795 800
Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr
805 810 815
Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile
820 825 830
Gly Leu Phe Met
835
<210> 42
<211> 455
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD4tm-41BB-Zeta
<400> 42
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu
275 280 285
Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys
290 295 300
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
305 310 315 320
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
325 330 335
Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg
450 455
<210> 43
<211> 433
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD4tm-41BB-Zeta, исключающий сигнал GMCSFRa
<400> 43
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Met Ala
245 250 255
Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu
260 265 270
Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
275 280 285
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
290 295 300
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly
305 310 315 320
Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
325 330 335
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
340 345 350
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
355 360 365
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
370 375 380
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
385 390 395 400
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
405 410 415
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
420 425 430
Arg
<210> 44
<211> 841
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD28tm-CD28gg-Zeta-T2A-EGFRt
<400> 44
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu
275 280 285
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
290 295 300
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
305 310 315 320
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
325 330 335
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly
450 455 460
Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn
465 470 475 480
Pro Gly Pro Arg Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu
485 490 495
Leu Pro His Pro Ala Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly
500 505 510
Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn
515 520 525
Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile
530 535 540
Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu
545 550 555 560
Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly
565 570 575
Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala
580 585 590
Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln
595 600 605
Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg
610 615 620
Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys
625 630 635 640
Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr
645 650 655
Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys
660 665 670
Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys
675 680 685
Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg
690 695 700
Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg
705 710 715 720
Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu
725 730 735
Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys
740 745 750
Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys
755 760 765
Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala
770 775 780
Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly
785 790 795 800
Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile
805 810 815
Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val
820 825 830
Val Ala Leu Gly Ile Gly Leu Phe Met
835 840
<210> 45
<211> 460
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD28tm-CD28gg-Zeta
<400> 45
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu
275 280 285
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
290 295 300
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
305 310 315 320
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
325 330 335
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 46
<211> 438
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CS1scFv-линкер-CD28tm-CD28gg-Zeta, исключающий сигнал GMCSFRa
<400> 46
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Met Phe
245 250 255
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
260 265 270
Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg
275 280 285
Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro
290 295 300
Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala
305 310 315 320
Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
325 330 335
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
340 345 350
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
355 360 365
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
370 375 380
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
385 390 395 400
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
405 410 415
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
420 425 430
Gln Ala Leu Pro Pro Arg
435
<210> 47
<211> 9796
<212> ДНК
<213> Искусственная последовательность
<220>
<223> CS1scFv-IgG4(HL-CH3)-CD28gg-Zeta-T2A-EGFRt_epHIV7
<400> 47
gttagaccag atctgagcct gggagctctc tggctaacta gggaacccac tgcttaagcc 60
tcaataaagc ttgccttgag tgcttcaagt agtgtgtgcc cgtctgttgt gtgactctgg 120
taactagaga tccctcagac ccttttagtc agtgtggaaa atctctagca gtggcgcccg 180
aacagggact tgaaagcgaa agggaaacca gaggagctct ctcgacgcag gactcggctt 240
gctgaagcgc gcacggcaag aggcgagggg cggcgactgg tgagtacgcc aaaaattttg 300
actagcggag gctagaagga gagagatggg tgcgagagcg tcagtattaa gcgggggaga 360
attagatcga tgggaaaaaa ttcggttaag gccaggggga aagaaaaaat ataaattaaa 420
acatatagta tgggcaagca gggagctaga acgattcgca gttaatcctg gcctgttaga 480
aacatcagaa ggctgtagac aaatactggg acagctacaa ccatcccttc agacaggatc 540
agaagaactt agatcattat ataatacagt agcaaccctc tattgtgtgc atcaaaggat 600
agagataaaa gacaccaagg aagctttaga caagatagag gaagagcaaa acaaaagtaa 660
gaaaaaagca cagcaagcag cagctgacac aggacacagc aatcaggtca gccaaaatta 720
ccctatagtg cagaacatcc aggggcaaat ggtacatcag gccatatcac ctagaacttt 780
aaatgcatgg gtaaaagtag tagaagagaa ggctttcagc ccagaagtga tacccatgtt 840
ttcagcatta tcagaaggag ccaccccaca agatttaaac accatgctaa acacagtggg 900
gggacatcaa gcagccatgc aaatgttaaa agagaccatc aatgaggaag ctgcaggcaa 960
agagaagagt ggtgcagaga gaaaaaagag cagtgggaat aggagctttg ttccttgggt 1020
tcttgggagc agcaggaagc actatgggcg cagcgtcaat gacgctgacg gtacaggcca 1080
gacaattatt gtctggtata gtgcagcagc agaacaattt gctgagggct attgaggcgc 1140
aacagcatct gttgcaactc acagtctggg gcatcaagca gctccaggca agaatcctgg 1200
ctgtggaaag atacctaaag gatcaacagc tcctggggat ttggggttgc tctggaaaac 1260
tcatttgcac cactgctgtg ccttggatct acaaatggca gtattcatcc acaattttaa 1320
aagaaaaggg gggattgggg ggtacagtgc aggggaaaga atagtagaca taatagcaac 1380
agacatacaa actaaagaat tacaaaaaca aattacaaaa attcaaaatt ttcgggttta 1440
ttacagggac agcagagatc cagtttgggg atcaattgca tgaagaatct gcttagggtt 1500
aggcgttttg cgctgcttcg cgaggatctg cgatcgctcc ggtgcccgtc agtgggcaga 1560
gcgcacatcg cccacagtcc ccgagaagtt ggggggaggg gtcggcaatt gaaccggtgc 1620
ctagagaagg tggcgcgggg taaactggga aagtgatgtc gtgtactggc tccgcctttt 1680
tcccgagggt gggggagaac cgtatataag tgcagtagtc gccgtgaacg ttctttttcg 1740
caacgggttt gccgccagaa cacagctgaa gcttcgaggg gctcgcatct ctccttcacg 1800
cgcccgccgc cctacctgag gccgccatcc acgccggttg agtcgcgttc tgccgcctcc 1860
cgcctgtggt gcctcctgaa ctgcgtccgc cgtctaggta agtttaaagc tcaggtcgag 1920
accgggcctt tgtccggcgc tcccttggag cctacctaga ctcagccggc tctccacgct 1980
ttgcctgacc ctgcttgctc aactctacgt ctttgtttcg ttttctgttc tgcgccgtta 2040
cagatccaag ctgtgaccgg cgcctacggc tagcgccgcc accatgctgc tgctcgtgac 2100
atctctgctg ctgtgcgagc tgccccaccc cgcctttctg ctgattcctg aggtgcagct 2160
ggtggaaagc ggcggaggac tggtgcagcc tggcggatct ctgagactga gctgtgccgc 2220
cagcggcttc gacttcagcc ggtactggat gagctgggtg cgccaggccc ctggcaaagg 2280
cctggaatgg atcggcgaga tcaaccccga cagcagcacc atcaactacg cccccagcct 2340
gaaggacaag ttcatcatca gccgggacaa cgccaagaac agcctgtacc tgcagatgaa 2400
ctccctgcgg gccgaggaca ccgccgtgta ctattgcgcc agacccgacg gcaactactg 2460
gtacttcgac gtgtggggcc agggcaccct cgtgacagtg tctagcggca gcacaagcgg 2520
aggcggatct ggcggaggat caggcggggg aggatccagc gatatccaga tgacccagag 2580
ccccagcagc ctgtctgcca gcgtgggcga cagagtgacc atcacatgca aggccagcca 2640
ggacgtggga atcgccgtgg cctggtatca gcagaaaccc ggcaaggtgc ccaagctgct 2700
gatctactgg gccagcacca gacacaccgg cgtgcccgat agattttccg gcagcggctc 2760
cggcaccgac ttcaccctga caatcagctc cctgcagcct gaggacgtgg ccacctacta 2820
ctgccagcag tacagcagct acccctacac cttcggacag ggcaccaagg tggaaatcaa 2880
agagtctaag tacggccctc cctgcccccc ttgtccaggc ggcggatctt ccggaggagg 2940
aagcggaggc cagcccagag aacctcaggt gtacacactg ccccctagcc aggaagagat 3000
gaccaagaat caggtgtccc tgacatgcct cgtgaagggc ttctacccct ccgatatcgc 3060
cgtggaatgg gagagcaacg gccagcctga gaacaactac aagaccaccc cccctgtgct 3120
ggacagcgac ggctcattct tcctgtacag caggctgacc gtggacaaga gccggtggca 3180
ggaaggcaac gtgttcagct gctccgtgat gcacgaggcc ctgcacaacc actacaccca 3240
gaagtccctg agcctgtccc tgggcaagat gttctgggtg ctggtggtcg tgggcggcgt 3300
gctggcctgt tatagcctgc tcgtgaccgt ggccttcatc atcttttggg tgcgcagcaa 3360
gcggagcaga ggcggccaca gcgactacat gaacatgacc cccagacggc caggccccac 3420
ccggaaacac tatcagcctt acgcccctcc cagagacttc gccgcttatc ggtccggcgg 3480
agggcgggtg aagttcagca gaagcgccga cgcccctgcc taccagcagg gccagaatca 3540
gctgtacaac gagctgaacc tgggcagaag ggaagagtac gacgtcctgg ataagcggag 3600
aggccgggac cctgagatgg gcggcaagcc tcggcggaag aacccccagg aaggcctgta 3660
taacgaactg cagaaagaca agatggccga ggcctacagc gagatcggca tgaagggcga 3720
gcggaggcgg ggcaagggcc acgacggcct gtatcagggc ctgtccaccg ccaccaagga 3780
tacctacgac gccctgcaca tgcaggccct gcccccaagg ctcgagggcg gcggagaggg 3840
cagaggaagt cttctaacat gcggtgacgt ggaggagaat cccggcccta ggatgcttct 3900
cctggtgaca agccttctgc tctgtgagtt accacaccca gcattcctcc tgatcccacg 3960
caaagtgtgt aacggaatag gtattggtga atttaaagac tcactctcca taaatgctac 4020
gaatattaaa cacttcaaaa actgcacctc catcagtggc gatctccaca tcctgccggt 4080
ggcatttagg ggtgactcct tcacacatac tcctcctctg gatccacagg aactggatat 4140
tctgaaaacc gtaaaggaaa tcacagggtt tttgctgatt caggcttggc ctgaaaacag 4200
agacttttgg catttccttt agtgtcccaa aaacgactaa gtccgaaccg gacttttgtc 4260
gacggacctc catgcctttg agaacctaga aatcatacgc ggcaggacca agcaacatgg 4320
tcagttttct cttgcagtcg tcagcctgaa cataacatcc ttgggattac gctccctcaa 4380
ggagataagt gatggagatg tgataatttc aggaaacaaa aatttgtgct atgcaaatac 4440
aataaactgg aaaaaactgt ttgggacctc cggtcagaaa accaaaatta taagcaacag 4500
aggtgaaaac agctgcaagg ccacaggcca ggtctgccat gccttgtgct cccccgaggg 4560
ctgctggggc ccggagccca gggactgcgt ctcttgccgg aatgtcagcc gaggcaggga 4620
atgcgtggac aagtgcaacc ttctggaggg tgagccaagg gagtttgtgg agaactctga 4680
gtgcatacag tgccacccag agtgcctgcc tcaggccatg aacatcacct gcacaggacg 4740
gggaccagac aactgtatcc agtgtgccca ctacattgac ggcccccact gcgtcaagac 4800
ctgcccggca ggagtcatgg gagaaaacaa caccctggtc tggaagtacg cagacgccgg 4860
ccatgtgtgc cacctgtgcc atccaaactg cacctacgga tgcactgggc caggtcttga 4920
aggctgtcca acgaatgggc ctaagatccc gtccatcgcc actgggatgg tgggggccct 4980
cctcttgctg ctggtggtgg ccctggggat cggcctcttc atgtgagcgg ccgctctaga 5040
cccgggctgc aggaattcga tatcaagctt atcgataatc aacctctgga ttacaaaatt 5100
tgtgaaagat tgactggtat tcttaactat gttgctcctt ttacgctatg tggatacgct 5160
gctttaatgc ctttgtatca tgctattgct tcccgtatgg ctttcatttt ctcctccttg 5220
tataaatcct ggttgctgtc tctttatgag gagttgtggc ccgttgtcag gcaacgtggc 5280
gtggtgtgca ctgtgtttgc tgacgcaacc cccactggtt ggggcattgc caccacctgt 5340
cagctccttt ccgggacttt cgctttcccc ctccctattg ccacggcgga actcatcgcc 5400
gcctgccttg cccgctgctg gacaggggct cggctgttgg gcactgacaa ttccgtggtg 5460
ttgtcgggga aatcatcgtc ctttccttgg ctgctcgcct gtgttgccac ctggattctg 5520
cgcgggacgt ccttctgcta cgtcccttcg gccctcaatc cagcggacct tccttcccgc 5580
ggcctgctgc cggctctgcg gcctcttccg cgtcttcgcc ttcgccctca gacgagtcgg 5640
atctcccttt gggccgcctc cccgcatcga taccgtcgac tagccgtacc tttaagacca 5700
atgacttaca aggcagctgt agatcttagc cactttttaa aagaaaaggg gggactggaa 5760
gggctaattc actcccaaag aagacaagat ctgctttttg cctgtactgg gtctctctgg 5820
ttagaccaga tctgagcctg ggagctctct ggctaactag ggaacccact gcttaagcct 5880
caataaagct tgccttgagt gcttcaagta gtgtgtgccc gtctgttgtg tgactctggt 5940
aactagagat ccctcagacc cttttagtca gtgtggaaaa tctctagcag aattcgatat 6000
caagcttatc gataccgtcg acctcgaggg ggggcccggt acccaattcg ccctatagtg 6060
agtcgtatta caattcactg gccgtcgttt tacaacgtcg tgactgggaa aaccctggcg 6120
ttacccaact taatcgcctt gcagcacatc cccctttcgc cagctggcgt aatagcgaag 6180
aggcccgcac cgatcgccct tcccaacagt tgcgcagcct gaatggcgaa tggaaattgt 6240
aagcgttaat attttgttaa aattcgcgtt aaatttttgt taaatcagct cattttttaa 6300
ccaataggcc gaaatcggca aaatccctta taaatcaaaa gaatagaccg agatagggtt 6360
gagtgttgtt ccagtttgga acaagagtcc actattaaag aacgtggact ccaacgtcaa 6420
agggcgaaaa accgtctatc agggcgatgg cccactacgt gaaccatcac cctaatcaag 6480
ttttttgggg tcgaggtgcc gtaaagcact aaatcggaac cctaaaggga gcccccgatt 6540
tagagcttga cggggaaagc cggcgaacgt ggcgagaaag gaagggaaga aagcgaaagg 6600
agcgggcgct agggcgctgg caagtgtagc ggtcacgctg cgcgtaacca ccacacccgc 6660
cgcgcttaat gcgccgctac agggcgcgtc aggtggcact tttcggggaa atgtgcgcgg 6720
aacccctatt tgtttatttt tctaaataca ttcaaatatg tatccgctca tgagacaata 6780
accctgataa atgcttcaat aatattgaaa aaggaagagt atgagtattc aacatttccg 6840
tgtcgccctt attccctttt ttgcggcatt ttgccttcct gtttttgctc acccagaaac 6900
gctggtgaaa gtaaaagatg ctgaagatca gttgggtgca cgagtgggtt acatcgaact 6960
ggatctcaac agcggtaaga tccttgagag ttttcgcccc gaagaacgtt ttccaatgat 7020
gagcactttt aaagttctgc tatgtggcgc ggtattatcc cgtattgacg ccgggcaaga 7080
gcaactcggt cgccgcatac actattctca gaatgacttg gttgagtact caccagtcac 7140
agaaaagcat cttacggatg gcatgacagt aagagaatta tgcagtgctg ccataaccat 7200
gagtgataac actgcggcca acttacttct gacaacgatc ggaggaccga aggagctaac 7260
cgcttttttg cacaacatgg gggatcatgt aactcgcctt gatcgttggg aaccggagct 7320
gaatgaagcc ataccaaacg acgagcgtga caccacgatg cctgtagcaa tggcaacaac 7380
gttgcgcaaa ctattaactg gcgaactact tactctagct tcccggcaac aattaataga 7440
ctggatggag gcggataaag ttgcaggacc acttctgcgc tcggcccttc cggctggctg 7500
gtttattgct gataaatctg gagccggtga gcgtgggtct cgcggtatca ttgcagcact 7560
ggggccagat ggtaagccct cccgtatcgt agttatctac acgacgggga gtcaggcaac 7620
tatggatgaa cgaaatagac agatcgctga gataggtgcc tcactgatta agcattggta 7680
actgtcagac caagtttact catatatact ttagattgat ttaaaacttc atttttaatt 7740
taaaaggatc taggtgaaga tcctttttga taatctcatg accaaaatcc cttaacgtga 7800
gttttcgttc cactgagcgt cagaccccgt agaaaagatc aaaggatctt cttgagatcc 7860
tttttttctg cgcgtaatct gctgcttgca aacaaaaaaa ccaccgctac cagcggtggt 7920
ttgtttgccg gatcaagagc taccaactct ttttccgaag gtaactggct tcagcagagc 7980
gcagatacca aatactgttc ttctagtgta gccgtagtta ggccaccact tcaagaactc 8040
tgtagcaccg cctacatacc tcgctctgct aatcctgtta ccagtggctg ctgccagtgg 8100
cgataagtcg tgtcttaccg ggttggactc aagacgatag ttaccggata aggcgcagcg 8160
gtcgggctga acggggggtt cgtgcacaca gcccagcttg gagcgaacga cctacaccga 8220
actgagatac ctacagcgtg agctatgaga aagcgccacg cttcccgaag ggagaaaggc 8280
ggacaggtat ccggtaagcg gcagggtcgg aacaggagag cgcacgaggg agcttccagg 8340
gggaaacgcc tggtatcttt atagtcctgt cgggtttcgc cacctctgac ttgagcgtcg 8400
atttttgtga tgctcgtcag gggggcggag cctatggaaa aacgccagca acgcggcctt 8460
tttacggttc ctggcctttt gctggccttt tgctcacatg ttctttcctg cgttatcccc 8520
tgattctgtg gataaccgta ttaccgcctt tgagtgagct gataccgctc gccgcagccg 8580
aacgaccgag cgcagcgagt cagtgagcga ggaagcggaa gagcgcccaa tacgcaaacc 8640
gcctctcccc gcgcgttggc cgattcatta atgcagctgg cacgacaggt ttcccgactg 8700
gaaagcgggc agtgagcgca acgcaattaa tgtgagttag ctcactcatt aggcacccca 8760
ggctttacac tttatgcttc cggctcgtat gttgtgtgga attgtgagcg gataacaatt 8820
tcacacagga aacagctatg accatgatta cgccaagctc gaaattaacc ctcactaaag 8880
ggaacaaaag ctggagctcc accgcggtgg cggcctcgag gtcgagatcc ggtcgaccag 8940
caaccatagt cccgccccta actccgccca tcccgcccct aactccgccc agttccgccc 9000
attctccgcc ccatggctga ctaatttttt ttatttatgc agaggccgag gccgcctcgg 9060
cctctgagct attccagaag tagtgaggag gcttttttgg aggcctaggc ttttgcaaaa 9120
agcttcgacg gtatcgattg gctcatgtcc aacattaccg ccatgttgac attgattatt 9180
gactagttat taatagtaat caattacggg gtcattagtt catagcccat atatggagtt 9240
ccgcgttaca taacttacgg taaatggccc gcctggctga ccgcccaacg acccccgccc 9300
attgacgtca ataatgacgt atgttcccat agtaacgcca atagggactt tccattgacg 9360
tcaatgggtg gagtatttac ggtaaactgc ccacttggca gtacatcaag tgtatcatat 9420
gccaagtacg ccccctattg acgtcaatga cggtaaatgg cccgcctggc attatgccca 9480
gtacatgacc ttatgggact ttcctacttg gcagtacatc tacgtattag tcatcgctat 9540
taccatggtg atgcggtttt ggcagtacat caatgggcgt ggatagcggt ttgactcacg 9600
gggatttcca agtctccacc ccattgacgt caatgggagt ttgttttggc accaaaatca 9660
acgggacttt ccaaaatgtc gtaacaactc cgccccattg acgcaaatgg gcggtaggcg 9720
tgtacggaat tcggagtggc gagccctcag atcctgcata taagcagctg ctttttgcct 9780
gtactgggtc tctctg 9796
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ НА CS1, ДЛЯ ЛЕЧЕНИЯ АМИЛОИДОЗА AL | 2018 |
|
RU2774895C2 |
| ХИМЕРНЫЕ РЕЦЕПТОРЫ АНТИГЕНА, НАЦЕЛЕННЫЕ НА HER2 | 2016 |
|
RU2753695C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА FLT3 | 2018 |
|
RU2820859C2 |
| CD20 ТЕРАПИЯ, CD22 ТЕРАПИЯ И КОМБИНИРОВАННАЯ ТЕРАПИЯ КЛЕТКАМИ, ЭКСПРЕССИРУЮЩИМИ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР (CAR) K CD19 | 2016 |
|
RU2752918C2 |
| ХИМЕРНЫЕ РЕЦЕПТОРЫ АНТИГЕНА, НАЦЕЛЕННЫЕ НА PSCA | 2016 |
|
RU2759879C2 |
| КЛЕТКИ ДЛЯ ИММУНОТЕРАПИИ, СКОНСТРУИРОВАННЫЕ ДЛЯ НАЦЕЛИВАНИЯ НА АНТИГЕН, ПРИСУТСТВУЮЩИЙ ОДНОВРЕМЕННО НА ИММУННЫХ КЛЕТКАХ И НА ПАТОЛОГИЧЕСКИХ КЛЕТКАХ | 2015 |
|
RU2714258C2 |
| СПЕЦИФИЧНЫЕ К MUC16 ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795198C2 |
| Т-КЛЕТКИ С ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ (CAR-T) ДЛЯ ЛЕЧЕНИЯ РАКА | 2019 |
|
RU2811466C2 |
| КЛЕТКА | 2014 |
|
RU2717984C2 |
| КЛЕТКА | 2014 |
|
RU2732236C2 |
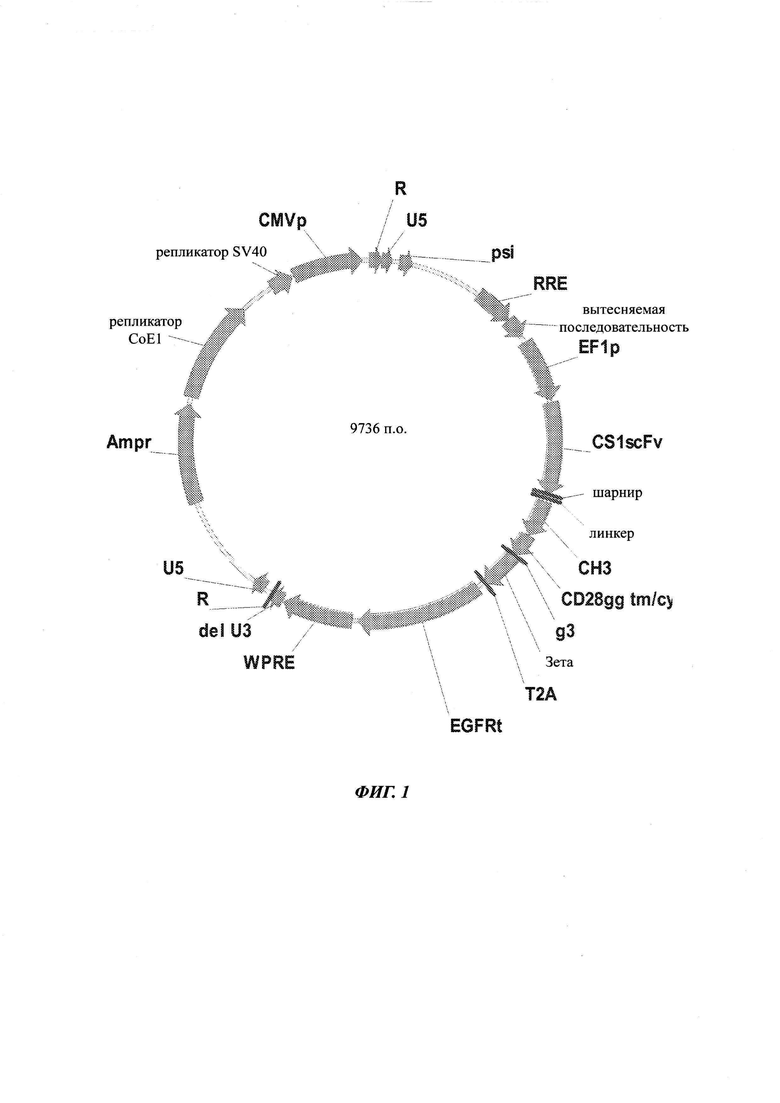

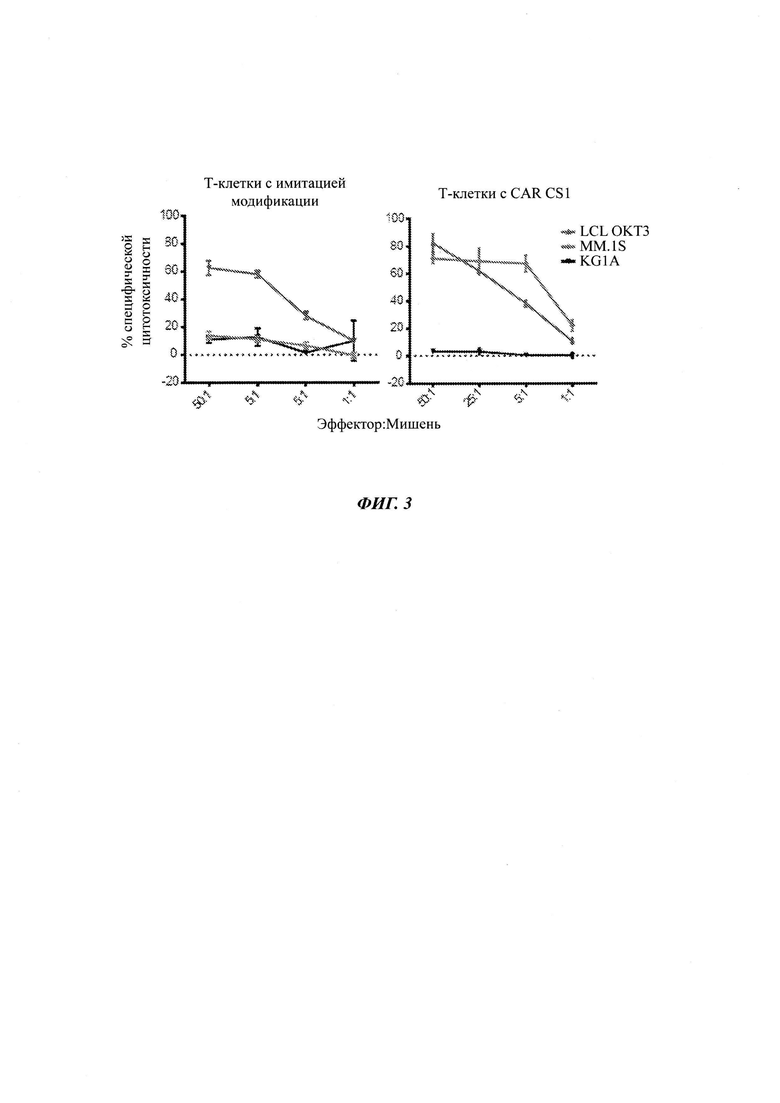
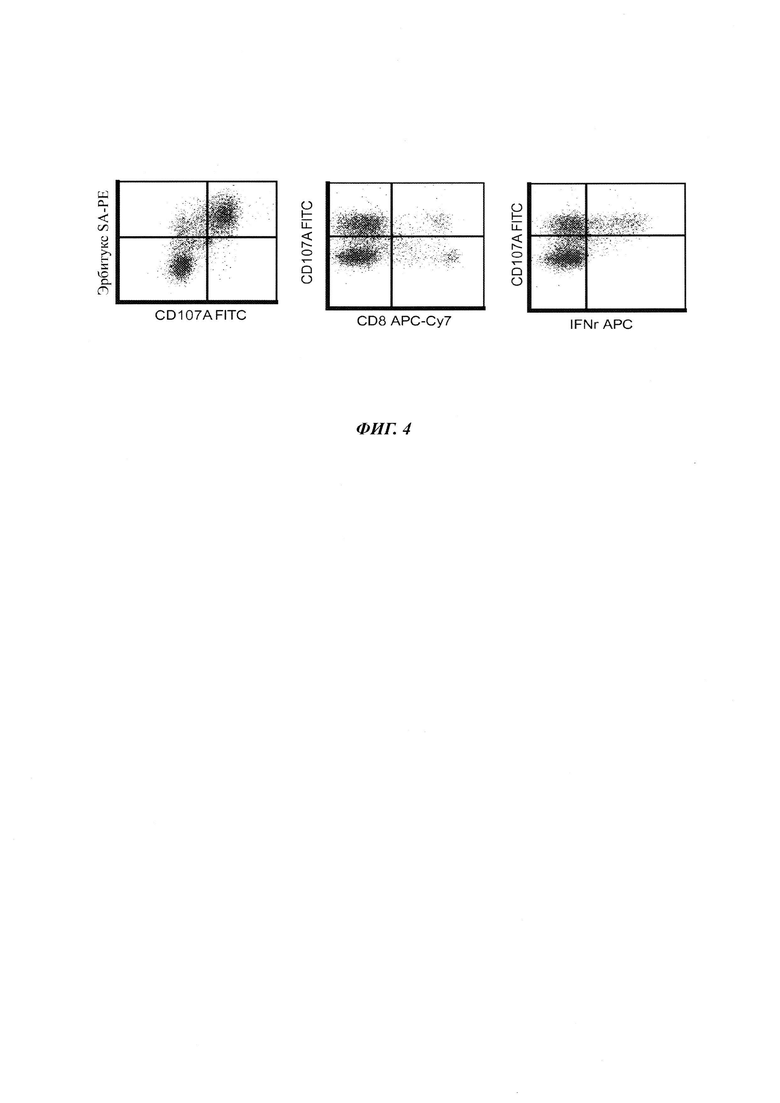







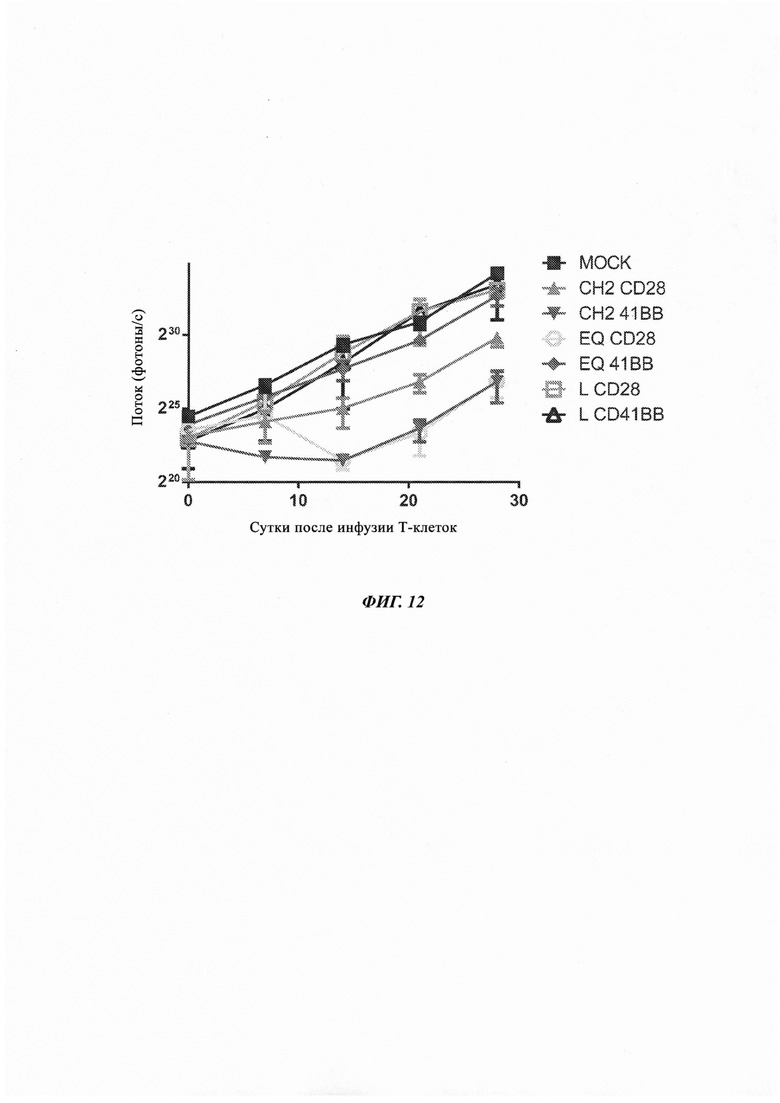
Изобретение относится к области биотехнологии, конкретно к получению химерных антигенных рецепторов, и может быть использовано в противоопухолевой терапии. Конструируют нуклеиновую кислоту, кодирующую полипептид химерного рецептора антигена, нацеленного на CS1. Вектором экспрессии трансдуцируют человеческие Т-клетки для получения популяции указанных клеток, экспрессирующих химерный рецептор антигена, нацеленный на CS1. Полученная популяция клеток эффективна в лечении онкологического заболевания, при котором экспрессируется CS1, например, такого как множественная миелома. 3 н. и 8 з.п. ф-лы, 12 ил., 5 табл., 5 пр.
.
1. Молекула нуклеиновой кислоты, кодирующая полипептид химерного рецептора антигена, где кодируемый полипептид имеет аминокислотную последовательность, представленную в любой из SEQ ID NO: 32, 33 или 34.
2. Молекула нуклеиновой кислоты по п. 1, где кодируемый полипептид имеет аминокислотную последовательность SEQ ID NO: 34.
3. Молекула нуклеиновой кислоты по п. 1, где кодируемый полипептид имеет аминокислотную последовательность SEQ ID NO: 33.
4. Молекула нуклеиновой кислоты по п. 1, где кодируемый полипептид имеет аминокислотную последовательность SEQ ID NO: 32.
5. Популяция человеческих Т-клеток, экспрессирующих химерный рецептор антигена, нацеленный на CS1, трансдуцированных вектором, кодирующим полипептид химерного рецептора антигена, где кодируемый полипептид имеет аминокислотную последовательность, выбранную из: SEQ ID NO: 33 и 34.
6. Популяция человеческих Т-клеток по п. 5, где кодируемый полипептид имеет SEQ ID NO: 34.
7. Популяция человеческих Т-клеток по п. 5, где кодируемый полипептид имеет SEQ ID NO: 33.
8. Способ лечения онкологического заболевания, при котором экспрессируется CS1, включающий введение пациенту, нуждающемуся в этом, популяции человеческих Т-клеток по любому из пп. 5-7.
9. Способ по п. 8, в котором популяция человеческих Т-клеток является аутологичной по отношению к пациенту.
10. Способ по п. 8, в котором популяция человеческих Т-клеток является аллогенной по отношению к пациенту.
11. Способ по любому из пп. 8-10, где онкологическое заболевание представляет собой множественную миелому.
| WO 2014179759 А1, 06.11.2014 | |||
| WO 2013151762 A1, 10.10.2013 | |||
| CHU J | |||
| ET AL., CS1-specific chimeric antigen receptor (CAR)-engineered natural killer cells enhance in vitro and in vivo antitumor activity against human multiple myeloma, LEUKEMIA, 2013, v.28, n.4, p.917 - 927 | |||
| ОДНОЦЕПОЧЕЧНЫЕ МУЛЬТИВАЛЕНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ЭФФЕКТОРНОЙ ФУНКЦИЕЙ | 2007 |
|
RU2487888C2 |
Авторы
Даты
2020-07-21—Публикация
2015-12-07—Подача