ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Амилоидоз легкой цепи (амилоидоз AL) отличается клональной популяцией плазматических клеток в костном мозге, которые продуцируют моноклональные легкие цепи ограничения каппа или лямбда. Амилоидгенные легкие цепи неправильно сворачиваются, образуя, посредством этого, бета-складчатые конформации, которые объединяются с образованием волокон. Данные амилоидные волокна отлагаются в тканях и органах, включающих сердце, почки и периферические нервы, где они прогрессирующе вмешиваются в структуру и функцию (Falk et al. 1997 N Engl J Med 337: 898-909). Без лечения прогноз является плохим, с медианным выживанием лишь 8 месяцев (Kyle et al. 1997 N Engl J Med 336:1202-120). Лечение амилоидоза AL, главным образом, сосредоточено на нацеливании на лежащий в его основе клон плазматических клеток для остановки продукции амилоидных волокон и обеспечения восстановления органов. Возможности для лечения включают химиотерапевтические схемы, позаимствованные от схем, используемых для лечения множественной миеломы, а также высокую дозу мелфалана, с последующей трансплантацией аутологичной стволовой клетки. Хотя пациенты с амилоидозом AL и выиграли от множества успехов, сделанных, в общем, в лечении заболеваний плазматических клеток, лечение осложняется неустойчивой природой популяции из-за дисфункции органа, связанного с амилоидом, и необходимости получать быстрый, глубокий ответ для предупреждения дальнейшего отложения волокон остаточным клоном. Несмотря на то, что возможны долговременные ремиссии с использованием как трансплантации стволовых клеток, так и новых агентов, включающих ингибиторы протеосомы и иммуномодулирующие лекарственные средства, требуются новые хорошо переносимые и эффективные лечения.
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
В данном документе описываются способы лечения амилоидоза AL с использованием химерных рецепторов антигена (CAR), нацеленных на CS1-гликопротеин клеточной поверхности, который является членом семейства рецепторов сигнальной лимфоцит-активирующей молекулы (SLAM). Ниже описываются результаты исследований, оценивающих образцы костного мозга от пациентов с заболеваниями плазматических клеток. Данные пациенты имели полные клинические оценки в отношении диагноза либо множественной миеломы (ММ), либо амилоидоза AL, включая характеристику гематологического клона, а также участие органа. Использовали анализ многоцветной проточной цитометрией для различения между злокачественными и нормальными плазматическими клетками посредством анализа нарушенных соотношений внутриклеточных цепей каппа/лямбда. Сильно отклоняющееся соотношение каппа и лямбда является надежным индикатором злокачественного клона амилоидоза AL. Клональные популяции плазматических клеток затем оценивали на экспрессию антигена созревания В-клеток (ВСМА) и экспрессию CS1. Данные исследования продемонстрировали то, что CS1 экспрессируется на клональных плазматических клетках пациентов с амилоидозом AL, и что ВСМА значимо не экспрессируется на плазматических клетках пациентов с амилоидозом AL. Это отличается от ММ, где полагают, что ВСМА является экспрессируемым повсеместно. Дополнительные исследования, описанные ниже, показывают то, что CAR, нацеленный на CS1, может эффективно устранять клетки, экспрессирующие CS1, в мышиной модели.
В данном документе описывается способ лечения амилоидоза легкой цепи, включающий введение пациенту, нуждающемуся в этом, популяции человеческих Т-клеток, трансдуцированных вектором, содержащим экспрессионную кассету, кодирующую химерный рецептор антигена, где химерный рецептор антигена содержит: scFv (одноцепочечный вариабельный фрагмент) против CS1; спейсерную область; трансмембранный домен; домен косигнализации и домен сигнализации CD3
В разных воплощениях: химерный рецептор антигена содержит: scFv против CS1; спейсерную область; трансмембранный домен CD28; домен косигнализации CD28 и домен сигнализации CD3 химерный рецептор антигена содержит: scFv против CS1; спейсерную область; трансмембранный домен CD4; домен косигнализации 4-1 ВВ и домен сигнализации CD3 химерный рецептор антигена содержит: scFv против CS1; спейсерную область, содержащую аминокислотную последовательность, выбранную из SEQ ID NO: 2-5 и 9-12; трансмембранный домен CD4; домен косигнализации 4-1 ВВ и домен сигнализации CD3 химерный рецептор антигена содержит: scFv против CS1; спейсерную область, содержащую аминокислотную последовательность, выбранную из SEQ ID NO: 2-5 и 9-12; трансмембранный домен CD28; домен косигнализации CD28 и домен сигнализации CD3 ζ; химерный рецептор антигена содержит: scFv против CS1; спейсер, содержащий аминокислотную последовательность, выбранную из SEQ ID NO: 2-5 и 9-12; трансмембранный домен CD4; домен косигнализации 4-1 ВВ и домен сигнализации CD3 ζ; химерный рецептор антигена содержит: спейсер, содержащий аминокислотную последовательность, выбранную из SEQ ID NO: 2-5 и 9-12; трансмембранный домен CD28; домен косигнализации CD28 и домен сигнализации CD3 ζ; химерный рецептор антигена содержит аминокислотную последовательность, по меньшей мере на 95% идентичную аминокислотной последовательности, выбранной из: SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45; химерный рецептор антигена содержит аминокислотную последовательность, идентичную аминокислотной последовательности, выбранной из: SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45; химерный рецептор антигена содержит аминокислотную последовательность, идентичную аминокислотной последовательности, выбранной из: SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45, причем каждая имеет не больше, чем 5 замен одиночных аминокислот; по меньшей мере 20%, 30% или 40% трансдуцированных человеческих Т-клеток представляют собой центральные Т-клетки памяти; по меньшей мере 30% трансдуцированных человеческих Т-клеток являются CD4+ и CD62L+ или CD8+ и CD62L+; популяция человеческих Т-клеток является аутологичной по отношению к пациенту; и популяция человеческих Т-клеток является аллогенной по отношению к пациенту.
ОПИСАНИЕ ЧЕРТЕЖЕЙ
На ФИГ. 1А-С показаны результаты исследований, демонстрирующих, что неопластическая плазматическая клетка при амилоидозе AL предпочтительно экспрессирует CS1. (А) Одноядерные клетки костного мозга выделяли у пациентов с диагностированным амилоидозом AL и множественной миеломой и метили антителами против CS1 и ВСМА, с последующим внутриклеточным окрашиванием κ/λ. Экспрессию CS1 и ВСМА анализировали на сортированной доминантной легкой цепи κ. (В) Представлены процентные доли CS1- и ВСМА-позитивных клеток в доминантных клонах амилоидоза AL (N равно 14). (С) Представлены процентные доли CS1- и ВСМА-позитивных клеток в доминантных клонах множественной миеломы (N равно 10).
На ФИГ. 2A-D показаны результаты исследований с использованием Т-клеток с CAR, нацеленным на CS1, показывающие то, что они являются цитотоксическими против CS1-позитивных клеток и индуцируют надежную регрессию опухоли у мышей. (А) Схемы конструкций CAR CS1, причем каждая включает антигенспецифичный scFv, шарнирную область IgG4, костимулирующий домен CD28, а также домен сигнализации CD3ζ. Шарнирная область IgG4 была сокращена делецией СН2 части. После последовательности CAR следует последовательность перескакивания рибосомы Т2А и затем кодирующая последовательность для отслеживания EGFRt/суицидного гена. (В) Очищенные центральные Т-клетки памяти (ТСМ) активировали и трансдуцировали лентивирусным вектором, кодирующим CAR CS1. Экспрессию CAR выявляли окрашиванием клеток антителом против EGFR (рецептор эпидермального фактора роста) цетуксимабом. (С) Цитотоксичность размноженных Т-клеток с CAR CS1 оценивали с использованием анализов 4-часового высвобождения 51Cr после сокультивирования с меченными 51Cr CSI-позитивными клетками-мишенями -MM.1S. LCL (лимфобластные клеточные линии), экспрессирующие OKT3, использовали в качестве позитивного контроля, и клетки миелоидного лейкоза KG1A использовали в качестве негативного контроля. Нетрансдуцированными имитирующими Т-клетками были негативные эффекторные клетки. (D) 2×106 клеток fflucGFP MM.1S, которые были генетически модифицированы для экспрессии люциферазы (ffluc) и зеленого флуоресцентного белка (GFP), инъецировали в большую берцовую кость (i.t.) мышам NOD/Scid IL2RγCnull (NSG). Через пять суток после инокуляции опухоли мышам в.в. инъецировали 1×106 Т-клеток с CAR CS1, а нетрансдуцированные имитирующие клетки инфундировали контрольным мышам. Сигналы опухоли отслеживали с использованием визуализации Xenogen один раз в неделю.
ФИГ. 3 представляет собой схематическое изображение лентивирусного вектора, экспрессирующего CAR CS1 (CS1scFv-IgG4(HL-CH3)-CD28gg-Zeta(CO)-T2A-EGFRt_epHIV7). Конструкция CAR CS1 включает следующее: сигнальная последовательность GMCSF (гранулоцитарно-макрофагальный колониестимулирующий фактор), scFv против CS1, шарнирная область IgG4, линкер, домен СН3, костимулирующий домен CD28 и домен сигнализации CD3ζ. После конструкции CAR следует последовательность перескакивания рибосомы Т2А и затем кодирующая последовательность суицидного гена EGFRt. Молекулы CAR и EGFRt экспрессируются от одного транскрипта.
На ФИГ. 4 показана аминокислотная последовательность CAR CS1, которая включает сигнальный пептид, последовательность перескакивания рибосомы и EGFRt (SEQ ID NO: 29).
На ФИГ. 5 показана аминокислотная последовательность CS1scFv-IgG4(HL-CH3)-CD4tm-41BB-Дзета-T2A-EGFRt (SEQ ID NO: 32).
На ФИГ. 6 показана аминокислотная последовательность CSIscFv-IgG4(L235E, N297Q)-CD4tm-41BB-Дзета-T2A-EGFRt (SEQ ID NO: 35).
На ФИГ. 7 показана аминокислотная последовательность CSIscFv-IgG4(L235E, N297Q)-CD28tm-CD28gg-Дзета-T2A-EGFRt (SEQ ID NO: 38).
На ФИГ. 8 показана аминокислотная последовательность CSIscFv-Линкер-CD4tm-41ВВ-Дзета-T2A-EGFRt (SEQ ID NO: 41).
На ФИГ. 9 показана аминокислотная последовательность CSIscFv-Линкер-CD28tm-CD28gg-Дзета-T2A-EGFRt (SEQ ID NO: 44).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Пример 1: экспрессия CS1 и ВСМА при амилоидозе AL и множественной миеломе
Исследовали четырнадцать пациентов с амилоидозом AL. Анализ экспрессии CS1 и ВСМА на неопластических плазматических клетках от данных пациентов выявляет то, что данные клетки предпочтительно экспрессируют CS1, но не ВСМА. Вкратце, одноядерные клетки костного мозга выделяли у пациентов с диагнозом амилоидоза AL и метили антителами против CS и ВСМА, с последующим окрашиванием на каппа/лямбда. Пример сортировки клональной популяции плазматических клеток у пациента с заболеванием, ограниченным каппа, с последующим анализом экспрессии CS1 показан на ФИГ. 1А. Все образцы амилоидоза AL экспрессировали высокие уровни CS1 (76,5 плюс/минус 4,7%), но были негативными в отношении ВСМА или демонстрировали очень низкую экспрессию ВСМА (4,9 плюс/минус 0,8%) (ФИГ. 1В).
Для сравнения анализировали образцы костного мозга от 10 пациентов с ММ на протяжении того же самого периода времени с использованием той же самой методологии (ФИГ. 1С). Клональные плазматические клетки пациентов с ММ экспрессируют CS1 аналогично экспрессии, наблюдаемой при AL; однако ВСМА является сравнительно значительно более часто экспрессируемым. Интересно то, что недостаток экспрессии ВСМА на плазматических клетках у пациентов с AL свидетельствует о том, что клональная плазматическая клетка при AL является уникальной по сравнению с миеломными клетками.
Пример 2: умерщвление клеток CAR, нацеленным на CS1
Для исследования пользы CS1 в качестве мишени для терапии амилоидоза ALT-клетками с CAR авторы изобретения анализировали второе поколение CAR CS1 (ФИГ. 2А), содержащего костимулирующий домен CD28gg, последовательность перескакивания рибосомы Т2А и усеченную последовательность рецептора EGF (EGFRt) в качестве молекулы селекции, отслеживания и отрезания, и включали в лентивирусный вектор SIN, более подробно описанный ниже. Очищенные центральные Т-клетки памяти (ТСМ) активировали и трансдуцировали лентивирусным вектором, кодирующим CAR CS1, и размножали в присутствии 50 U/мл IL-2 (интерлейкин-2) и 0,5 нг/мл IL-15 в течение 3 недель. Экспрессию CAR отслеживали окрашиванием клеток с использованием цетуксимаб-биотина и стрептавидина (SA) (ФИГ. 2В). Цитотоксичность размноженных Т-клеток с CAR против CS1 оценивали с использованием анализов 4-часового высвобождения 51Cr после совместного культивирования с меченными 51Cr клетками-мишениями CS1+ (MM.1S) (ФИГ. 2С).
Шести-десятинедельным NOD/Scid IL2RγC-Hynb мышам инъецировали в большую берцовую кость (i.t.) 2×106 fflucGFP MM.1S, которые были генетически модифицированы для экспрессии люциферазы (ffluc) и зеленого флуоресцентного белка (GFP). Через пять суток после инокуляции опухоли мышам внутривенно инъецировали (в.в.) 1×106 Т-клеток с CAR CS1, а нетрансдуцированные имитирующие клетки инфундировали контрольным мышам. Анестезированных мышей еженедельно визуализировали с использованием системы Xenogen MS серии 100 (Xenogen, Alameda, СА). Фотоны от ксенотрансплантатов опухоли ffLuc+ количественно измеряли с использованием программы Living Image (Xenogen), и сигнал биолюминисценции измеряли как общий поток фотонов, нормированный по времени воздействия и площади поверхности, и выраженный в единицах фотоны в секунду на см2 на стерадиан.
Для анализа противоопухолевой активности MM.1S инокулировали мышам NSG посредством инъекции в большую берцовую кость. Как только было подтверждено приживление опухоли, мышам, несущим опухоль, внутривенно инфундировали 1х106 Т-клеток с CAR CS1. Т-клетки с CAR CS1 демонстрируют специфичное и эффективное умерщвление CS1-позитивных клеток (MM.1S) (ФИГ. 2С). Противоопухолевые исследования в животной модели продемонстрировали то, что Т-клетки с CAR CS1 индуцировали значимую ремиссию опухоли по сравнению с мышами, обработанными имитирующими Т-клетками (ФИГ. 2D).
Эти данные поддерживают применение терапии Т-клетками с CAR, направленным на CS1, для пациентов с амилоидозом AL. AL является идеальной ситуацией для исследования терапии, опосредованной CAR. Относительно малое число злокачественных клеток, которые должны служить в качестве мишени, дает возможность успешного искоренения данного небольшого, на разрушительного клона, а также минимальный риск в отношении осложнений, связанных с синдромом высвобождения цитокинов. Кроме того, относительно безопасный профиль нацеленного на CS1 антитела элотузумаб указывает на то, что Т-клетки с CAR CS1, подобным образом, могут давать благоприятные результаты в данном отношении. Работа авторов данного изобретения представляет новое применение Т-клеток с CAR, направленным на CS1, при выявлении того, что, в отличие от доклинического опыта с ММ, ВСМА не был бы подходящей мишенью. С доклиническими данными авторов изобретения, показывающими эффективность Т-клеток с CAR, направленным на CS1, авторов данного изобретения, авторы данного изобретения планируют двигаться дальше в клиническом испытании Т-клеток с CAR CS1 в отношении AL.
Пример 3: CAR, нацеленный на CS1
CAR, нацеленный на CS1, подходящий для применения в лечении амилоидоза AL, включает CAR, который содержит внеклеточный домен, трансмембранный домен и внутриклеточный домен сигнализации. Внеклеточный домен включает CS1-специфичную область scFv или ее вариант и спейсер, содержащий, например, часть человеческого домена Fc. Внеклеточный домен позволяет CAR, при экспрессии на поверхности Т-клетки, направлять активность Т-клеток на клетки, экспрессирующие CS1. Трансмембранный домен включает, например, трансмембранный домен CD4, трансмембранный домен CD8, трансмембранный домен CD28 или трансмембранный домен CD3. Внутриклеточный домен сигнализации включает домен сигнализации из дзета цепи человеческого комплекса CD3 (CD3ζ) и один или более чем один костимулирующий домен, например, костимулирующий домен 4-1 ВВ. Включение костимулирующего домена, такого как костимулирующий домен 4-1 ВВ (CD137), последовательно с CD3ζ во внутриклеточной области обеспечивает получение Т-клеткой костимулирующих сигналов. Т-клетки, например, специфичные для пациента, аутологичные Т-клетки, можно генетически модифицировать для экспрессии CAR, описанных в данном документе, и генетически модифицированные клетки можно размножать и использовать терапевтически. Можно использовать разные поднаборы Т-клеток, включающие и альфа бета Т-клетки, и гамма дельта Т-клетки. Кроме того, CAR может экспрессироваться в других иммунных клетках, таких как NK-клетки (природные киллеры). Когда пациента лечат иммунной клеткой, экспрессирующей CAR, описанный в данном документе, данная клетка может быть аутологичной Т-клеткой или аллогенной Т-клеткой. В некоторых случаях используемыми клетками является популяция клеток, которая включает и CD4+, и CD8+ центральные Т-клетки памяти (ТСМ), которые являются CD62L+, CCR7+, CD45RO+ и CD45RA-. Данная популяция клеток также может включать другие типы Т-клеток. Несколько CAR, нацеленных на CS1, подробно описываются в WO 2016/090369.
scFv, нацеленный на CS1
CAR, нацеленный на CS1, описанный в данном документе, включает scFv, нацеленный на CS1 (например, scFv, включающий последовательность:
EVQLVESGGGLVQPGGSLRLSCAASGFDFSRYWMSWVRQAPGKGLEWIGEIN PDSSTINYAPSLKDKFIISRDNAKNSLYLQMNSLRAEDTAVYYCARPDGNYWYFDVWG QGTLVTVSSGSTSGGGSGGGSGGGGSSDIQMTQSPSSLSASVGDRVTITCKASQDVGI AVAWYQQKPGKVPKLLIYWASTRHTGVPDRFSGSGSGTDFTLTISSLQPEDVATYYCQ QYSSYPYTFGQGTKVEIK; SEQ ID NO: 1) или ее вариант, имеющий 1-5 (например, 1 или 2) модификации аминокислот (например, замены).
Полезный CAR CS1 состоит из или содержит аминокислотную последовательность любой из SEQ ID NO: 31, 34, 37, 40, 43 и 46 (зрелый CAR, не имеющий сигнальной последовательности), или CAR CS1 состоит или содержит аминокислотную последовательность любой из SEQ ID NO: 30, 33, 36, 39, 42 и 45 (незрелый CAR, имеющий сигнальную последовательность GMCSFRa). Данный CAR может экспрессироваться в форме, которая включает сигнальную последовательность, например, сигнальную последовательность человеческого рецептора GM-CSF (гранулоцитарно-макрофагальный колониестимулирующий фактор) альфа (MLLLVTSLLLCELPHPAFLLIP; SEQ ID NO: 26). Данный CAR может экспрессироваться с дополнительными последовательностями, которые являются полезными для отслеживания экспрессии, например, с последовательностью перескакивания рибосомы Т2А и усеченным EGFRt. Таким образом, CAR может содержать или состоять из аминокислотной последовательности любой из SEQ ID NO: 29-46, или может содержать или состоять из аминокислотной последовательности, которая является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичной любой из SEQ ID NO: 29-46. CAR может содержать или состоять из аминокислотной последовательности любой из SEQ ID NO: 29-46 с вплоть до 1, 2, 3, 4 или 5 аминокислотными заменами (предпочтительно консервативными аминокислотными заменами).
Спейсерная область
CAR, описанный в данном документе, может включать спейсер, расположенный между доменом нацеливания на CS1 (т.е. scFv против CS1 или его вариант) и трансмембранным доменом. Можно использовать целый ряд разных спейсеров. Некоторые из них включают по меньшей мере часть человеческой области Fc, например, шарнирную часть человеческой области Fc или домен СН3 или его варианты. В Таблице 1 ниже приводятся разные спейсеры, которые можно использовать в CAR, описанных в данном документе.
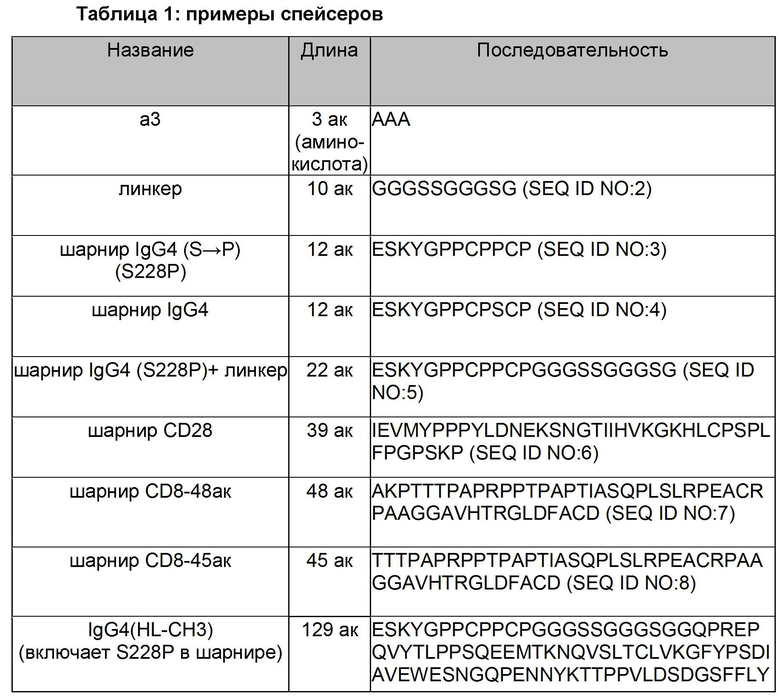

Некоторые спейсерные области включают всю или часть шарнирной области иммуноглобулина (например, IgG1, IgG2, IgG3, IgG4), т.е. последовательность, которая попадает между доменами СН1 и СН2 иммуноглобулина, например, шарнир Fc IgG4 или шарнир CD8. Некоторые спейсерные области включают домен СН3 иммуноглобулина или и домен СН3, и домен СН2. Последовательности, происходящие от иммуноглобулина, могут включать одну или более чем одну модификацию аминокислоты, например, 1, 2, 3, 4 или 5 замен, например, замен, которые уменьшают связывание вне мишени.
Шарнирная/линкерная область также может содержать шарнирную область IgG4, имеющую последовательность ESKYGPPCPSCP (SEQ ID NO: 4) или ESKYGPPCPPCP (SEQ ID NO: 3).
Шарнирная/линкерная область также может содержать последовательность ESKYGPPCPPCP (SEQ ID NO: 3), с последующей линкерной последовательностью GGGSSGGGSG (SEQ ID NO: 2), с последующей последовательностью СНЗ IgG4: GQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVL DSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK (SEQ ID NO: 12). Таким образом, вся область линкера/спейсера может содержать последовательность:
ESKYGPPCPPCPGGGSSGGGSGGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYP SDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEA LHNHYTQKSLSLSLGK (SEQ ID NO: 11). В некоторых случаях спейсер имеет 1, 2, 3, 4 или 5 одиночных аминокислотных замен (например, консервативных замен) по сравнению с SEQ ID NO: 11. В некоторых случаях шарнирная/линкерная область Fc IgG4 подвергнута мутации в двух положениях (L235E; N297Q) способом, который уменьшает связывание рецепторами Fc (FcR).
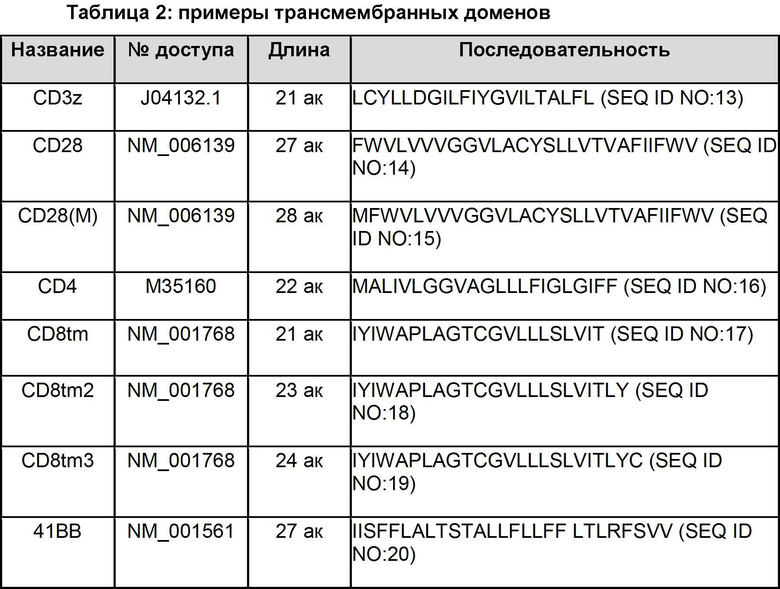
Трансмембранный домен
В CAR можно использовать целый ряд трансмембранных доменов. Таблица 2 включает примеры подходящих трансмембранных доменов. Когда присутствует спейсерная область, трансмембранный домен располагается ближе к карбокси концу относительно спейсерной области.
Костимулирующий домен
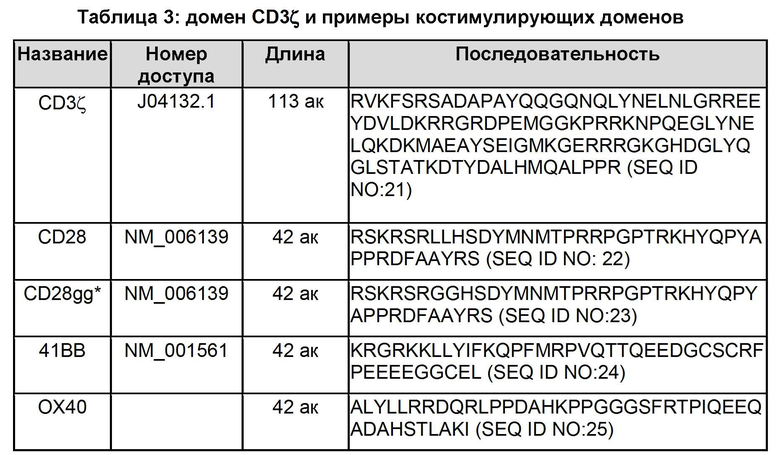
Костимулирующий домен может представлять собой любой домен, который подходит для применения с доменом сигнализации CD3ζ. В некоторых случаях костимулирующий домен представляет собой костимулирующий домен CD28, который включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной: RSKRSRGGHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS (SEQ ID NO: 23; аминокислотная замена от LL до GG подчеркнута двойной линией). В некоторых случаях домен косигнализации CD28 имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных и предпочтительно не в подчеркнутой последовательности GG) по сравнению с SEQ ID NO: 23. В некоторых случаях домен косигнализации представляет собой домен косигнализации 4-1 ВВ, который включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL (SEQ ID NO: 24). В некоторых случаях домен косигнализации 4-1 ВВ имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 24.
Костимулирующий(щие) домен(ны) располагается(ются) между трансмембранным доменом и доменом сигнализации CD3ζ. Таблица 3 включает примеры подходящих костимулирующих доменов вместе с последовательностью домена сигнализации CD3ζ.
В разных воплощениях костимулирующий домен выбран из группы, состоящей из следующих: костимулирующий домен, показанный в Таблице 3, или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот, костимулирующий домен CD28 или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот, костимулирующий домен 4-1 ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот, и костимулирующий домен или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот. В некоторых воплощениях присутствует костимулирующий домен 4-1 ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот. В некоторых воплощениях имеются два костимулирующих домена, например, костимулирующий домен CD28 или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замены), и костимулирующий домен 4-1 ВВ или его вариант, имеющий 1-5 (например, 1 или 2) модификаций аминокислот (например, замены). В разных воплощениях 1-5 (например, 1 или 2) модификаций аминокислот представляют собой замены. Костимулирующий домен является аминоконцевым относительно домена сигнализации CD3ζ, и в некоторых случаях между костимулирующим доменом и доменом сигнализации CD3ζ располагается короткий линкер, состоящий из 2-10, например, 3 аминокислот (например, GGG).
Домен сигнализации CD3ζ
Домен сигнализации CD3ζ может представлять собой любой домен, который подходит для применения с доменом сигнализации CD3ζ. В некоторых случаях домен сигнализации CD3ζ включает последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQE GLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR (SEQ ID NO: 21). В некоторых случаях домен сигнализации CD3C, имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 21.
Усеченный EGFR
После домена сигнализации CD3ζ может следовать последовательность перескакивания рибосомы (например, LEGGGEGRGSLLTCGDVEENPGPR; SEQ ID NO: 27) и усеченный EGFR, имеющий последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной LVTSLLLCELPHPAFLLIPRKVCNGIGIGEFKDSLSINATNIKHFKNCTSISGDLHILPVAFR GDSFTHTPPLDPQELDILKTVKEITGFLLIQAWPENRTDLHAFENLEIIRGRTKQHGQFSL AVVSLNITSLGLRSLKEISDGDVIISGNKNLCYANTINWKKLFGTSGQKTKIISNRGENSC KATGQVCHALCSPEGCWGPEPRDCVSCRNVSRGRECVDKCNLLEGEPREFVENSECI QCHPECLPQAMNITCTGRGPDNCIQCAHYIDGPHCVKTCPAGVMGENNTLVWKYADA GHVCHLCHPNCTYGCTGPGLEGCPTNGPKIPSIATGMVGALLLLLVVALGIGLFM (SEQ ID NO: 28). В некоторых случаях усеченный EGFR имеет 1, 2, 3, 4 или 5 аминокислотных замен (предпочтительно консервативных) по сравнению с SEQ ID NO: 28.
Пациенту, страдающему от амилоидоза AL, можно вводить популяцию человеческих Т-клеток, трансдуцированных вектором, содержащим, экспрессионную кассету, кодирующую химерный рецептор антигена CS1, описанный в данном документе (например, CAR, который содержит или состоит из аминокислотной последовательности любой из SEQ ID NO: 29-46, или аминокислотной последовательности, которая является по меньшей мере на 95%, 96%, 97%, 98% или 99% идентичной любой из SEQ ID NO: 29-46, или аминокислотной последовательности любой из SEQ ID NO: 29-46 с вплоть до 1, 2, 3, 4 или 5 аминокислотными заменами (предпочтительно консервативными аминокислотными заменами). В разных воплощениях популяция человеческих Т-клеток представляет собой центральные Т-клетки памяти (ТСМ), например, ТСМ CD8+/CD4+.
Термин «модификация аминокислоты» относится к замене, вставке и/или делеции аминокислоты в последовательности белка или пептида. Термин «замена аминокислоты» или «замена» относится к замене аминокислоты в конкретном положении в последовательности родительского пептида или белка другой аминокислотой. Замена может быть сделана для изменения аминокислоты в образующемся белке неконсервативным способом (т.е. изменением кодона от аминокислоты, принадлежащей к группе аминокислот, имеющих конкретный размер или характеристику, до аминокислоты, принадлежащей к другой группе) или консервативным способом (т.е. изменением кодона от аминокислоты, принадлежащей к группе аминокислот, имеющих конкретный размер или характеристику, до аминокислоты, принадлежащей к той же самой группе). Такая консервативная замена обычно приводит к меньшему изменению структуры и функции образующегося белка. Следующее представляет собой примеры разных групп аминокислот: 1) аминокислоты с неполярными группами R: аланин, валин, лейцин, изолейцин, пролин, фенилаланин, триптофан, метионин; 2) аминокислоты с незаряженными полярными группами R: глицин, серии, треонин, цистеин, тирозин, аспарагин, глутамин; 3) аминокислоты с заряженными полярными группами R (отрицательно заряженные при рН 6,0): аспарагиновая кислота, глутаминовая кислота; 4) основные аминокислоты (положительно заряженные при рН 6,0): лизин, аргинин, гистидин (при рН 6,0). Другой группой могут быть те аминокислоты, которые имеют фенильные группы: фенилаланин, триптофан и тирозин.
CAR CS1 может включать последовательность, которая является по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 98% идентичной или идентичной аминокислотной последовательности, показанной на ФИГ. 4-9 (SEQ ID NO: 29-46 либо включающие, либо исключающие сигнальную последовательность GMCSFRa, и либо включающие, либо исключающие последовательность перескакивания рибосомы Т2А и усеченного EGFRt).
Целый ряд CAR, нацеленных на CS1, описывается в WO 2016/090369, и данные CAR могут быть полезными для лечения амилоидоза AL.
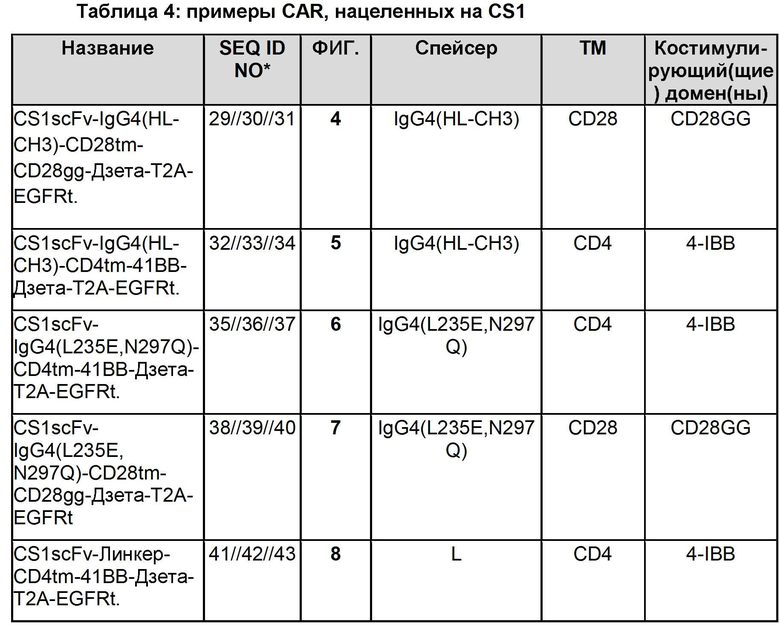
Среди CAR, нацеленных на CS1, описанных в данном документе, находятся CAR, обобщенные в Таблице 4, в которой указаны спейсерная область, трансмембранный домен и костимулирующий(щие) домен(ны) для каждого CAR.
В некоторых случаях CAR CS1 может быть получен с использованием вектора, в котором после открытой рамки считывания CAR следует последовательность перескакивания рибосомы Т2А и усеченный EGFR (EGFRt), у которого отсутствует цитоплазматический хвост сигнализации. При данной организации соэкспрессия EGFRt дает инертный, неиммуногенный поверхностный маркер, который обеспечивает точное измерение генномодифицированных клеток и позволяет осуществлять позитивную селекцию генномодифицированных клеток, а также эффективное клеточное отслеживание терапевтических Т-клеток in vivo после адоптивного переноса. Эффективный контроль пролиферации для того, чтобы избежать цитокинового шторма и токсичности вне мишени, является важным препятствием для успеха Т-клеточной иммунотерапии. EGFRt, включенный в лентивирусный вектор для CAR CS1, может действовать в качестве суицидного гена для устранения Т-клеток CAR+ в случаях токсичности, связанной с лечением.
CAR, описанный в данном документе, может быть получен любыми способами, известными в данной области, хотя предпочтительно его получают с использованием методик генной инженерии. Нуклеиновые кислоты, кодирующие несколько областей химерного рецептора, могут быть получены и собраны в полную кодирующую последовательность стандартными методиками молекулярного клонирования, известными в данной области (скрининг геномной библиотеки, перекрывающаяся ПЦР (полимеразная цепная реакция), лигирование с помощью праймера, сайт-направленный мутагенез и т.д.), в зависимости от того, что является удобным. Образующаяся кодирующая область предпочтительно вставляется в экспрессионный вектор и используется для трансформации подходящей линии клетки - хозяина экспрессии, предпочтительно линии клеток Т-лимфоцитов и наиболее предпочтительно аутологичной линии клеток Т-лимфоцитов.
Разные поднаборы Т-клеток, выделенных у пациента, можно трансдуцировать вектором для экспрессии CAR. Центральные Т-клетки памяти являются одним полезным поднабором Т-клеток. Центральная Т-клетка памяти может быть выделена из одноядерных клеток периферической крови (РВМС) посредством отбора на клетки CD45RO+/CD62L+, используя, например, прибор CliniMACS® для иммуномагнитной селекции клеток, экспрессирующих желательные рецепторы. Клетки, обогащенные центральными Т-клетками памяти, могут быть активированы антителами против CD3/CD28, трансдуцированы, например, лентивирусным вектором, который управляет экспрессией CAR CS1, а также неиммуногенным поверхностным маркером для выявления in vivo, устранения и потенциального отбора ex vivo. Активированные/генетически модифицированные центральные Т-клетки памяти, нацеленные на CS1, могут быть размножены in vitro с использованием IL-2/IL-15 (интерлейкин-2/интерлейкин-15) и затем криоконсервированы.
Пример 4: конструирование и структура epHIV7, использованного для экспрессии CS1-специфичного CAR
Плазмида pHIV7 представляет собой родительскую плазмиду, из которой могут быть получены клинические векторы, экспрессирующие CAR CS1. Вектор epHIV7, использованный для экспрессии CAR, был получен из вектора pHIV7 (Wang et al. 2011 Blood 118:1255). Важно то, что в данном векторе для управления экспрессией CAR используется человеческий промотор EF1. И 5', и 3' последовательности данного вектора были получены из pv653RSN, который был получен ранее из провируса НХВс2. Вытесняемые последовательности полиуринового тракта ДНК (сРРТ) были получены из штамма pNL4-3 HIV-1 (вирус иммунодефицита человека-1) из репозитория реактивов NIH AIDS (Национальный институт здравоохранения по СПИД).
Конструирование pHIV7 проводили следующим образом. Вкратце, pv653RSN, содержащую 653 п. о. из gag-pol плюс 5'- и 3'-длинные концевые повторы (LTR) с расположенным между ними геном SL3-неомицинфосфотрансферазы (Neo), субклонировали в pBluescript следующим образом: на стадии 1 из последовательностей от 5' LTR до элемента, отвечающего на rev (RRE), делали p5'HIV-1 51, и затем 5' LTR модифицировали посредством удаления последовательностей выше TATA бокса и лигировали сначала с энхансером CMV (цитомегаловирус) и затем с репликатором SV40 (p5'HIV-2). На стадии 2 после клонирования 3'LTR в pBluescript с получением p3'HIV-1 делали делецию 400 п.о. в 3'LTR энхансере/промоторе для удаления цис-регуляторных элементов в U3 HIV и образования p3'HIV-2. На стадии 3 фрагменты, выделенные из p5'HIV-3 и p3'HIV-2, лигировали с получением pHIV-3. На стадии 4 p3'HIV-2 дополнительно модифицировали посредством удаления дополнительных расположенных выше последовательностей HIV с получением p3'HIV-3, и фрагмент из 600 п.о. BamHI-Sall, содержащий WPRE, добавляли к p3'HIV-3 с получением p3'HIV-4. На стадии 5 уменьшали размер RRE pHIV-3 посредством ПЦР и лигировали с 5' фрагментом из pHIV-3 (не показан) и с p3'HIV-4 с получением pHIV-б. На Стадии 6 фрагмент BgIII-BamHI из 190 п. о., содержащий вытесняемую последовательность ДНК сРРТ из pNL4-3 (55) HIV-1, амплифицировали из pNL4-3 и помещали между последовательностями RRE и WPRE в pHIV6 с получением pHIV-7. Данную родительскую плазмиду - pHIV7-GFP (GFP - зеленый флуоресцентный белок) использовали для упаковки родительского вектора с использованием четырехплазмидной системы.
Упаковочный сигнал - пси (ψ) - требуется для эффективной упаковки вирусного генома в вектор. RRE и WPRE усиливают транспорт РНК-транскрипта и экспрессию трансгена. Было продемонстрировано то, что вытесняемая последовательность в комбинации с WPRE усиливает эффективность трансдукции лентивирусного вектора в клетках млекопитающих.
Хелперные функции, требующиеся для продукции вирусного вектора, разделяются на три отдельные плазмиды для уменьшения вероятности образования компетентного к репликации лентивируса посредством рекомбинации: 1) pCgp кодирует белок gag/pol, требующийся для сборки вирусного вектора; 2) pCMV-Rev2 кодирует белок Rev, который действует на последовательность RRE для помощи в транспортировке вирусного генома для эффективной упаковки; и 3) pCMV-G кодирует гликопротеин вируса везикулостоматита (VSV), который требуется для инфективности вирусного вектора.
Имеется минимальная гомология последовательности ДНК между геномом вектора, кодируемого pHIV7, и хелперными плазмидами. Области гомологии включают область упаковочного сигнала из приблизительно 600 нуклеотидов, расположенную в последовательности gag/pol хелперной плазмиды pCgp; последовательность промотора CMV во всех трех хелперных плазмидах и последовательность RRE в хелперной плазмиде pCgp. Весьма маловероятно то, что из-за гомологии в данных областях мог бы быть генерирован компетентный к репликации рекомбинантный вирус, так как это потребовало бы многочисленных событий рекомбинации. Кроме того, у любых образующихся рекомбинантов отсутствовали бы функциональные последовательности LTR и tat, требующиеся для лентивирусной репликации.
Промотор CMV был заменен промотором EF1α-HTLV (EF1p), и новая плазмида была названа epHIV7. EF1p имел 563 п.о. и был введен в epHIV7 с использованием NruI и NheI, затем был вырезан промотор CMV.
Из данной системы был удален лентивирусный геном, исключая gag/pol и rev, которые необходимы для патогенности вируса дикого типа и требуются для продуктивной инфекции клеток-мишеней. Кроме того, конструкция вектора epHIV7 не содержит интактного промотора 3'LTR, таким образом, полученная в результате экспрессируемая и обратно транскрибированная ДНК провирусного генома в клетках-мишенях будет иметь неактивные LTR. В результате данной конструкции последовательности, происходящие из HIV-1, не будут транскрибироваться из провируса, и только терапевтические последовательности будут экспрессироваться от соответствующих им промоторов. Ожидается то, что удаление промоторной активности LTR в векторе SIN значительно уменьшает возможность непреднамеренной активации генов хозяина. В Таблице 5 обобщены разные регуляторные элементы, присутствующие в epHIV7.
Фиг. 1 представляет собой схематическое изображение CAR CS1 (CS1scFv-IgG4(HL-CH3)-CD28gg-Дзета(CO)-T2A-EGFRt_epHIV7), лентивирусного вектора, содержащего данную конструкцию CAR, состоящую из scFv против CS1, шарнирной области IgG4, линкера, костимулирующего домена CD28 и домена сигнализации CD3ζ. После конструкции CAR следует последовательность перескакивания рибосомы Т2А и затем последовательность, кодирующая суицидный ген EGFRt. Молекулы CAR и EGFRt экспрессируются от одного транскрипта. В ТАБЛИЦЕ 5 представлено положение разных элементов вектора.


Пример 5: получение векторов для трансдукции Т-клеток пациента Для каждой плазмиды (CS1 CAR_epHIV7; pCgp; pCMV-G и pCMV-Rev2) получают семенной банк, который используется для инокуляции ферментера с получением достаточных количеств плазмидной ДНК. Данную плазмидную ДНК тестируют на идентичность, стерильность и присутствие эндотоксина перед ее применением в производстве лентивирусного вектора.
Вкратце, клетки размножали от рабочей клетки 293Т (WCB), которая была протестирована для подтверждения стерильности и отсутствия вирусного загрязнения. Сосуд с клетками 293Т от WCB 293Т оттаивали. Клетки выращивали и размножали до наличия достаточного числа клеток для посева в подходящее число 10-слойных клеточных фабрик (CF) для продукции вектора и поддержания линии клеток. Для продукции можно использовать одну линию клеток.
Лентивирусный вектор продуцировали в подпартиях из вплоть до 10 CF. Две подпартии можно продуцировать в ту же самую неделю, приводя к продукции приблизительно 20 л лентивирусного супернатанта/неделю. Материал, продуцированный из всех подпартий, объединяли на протяжении последующей фазы переработки для того, чтобы произвести одну партию продукта. Клетки 293Т высевали в CF в среду 293Т (DMEM (среда Игла, модифицированная по Дульбекко) с 10% FBS (фетальная телячья сыворотка)). Фабрики помещали в инкубатор при 37°С и горизонтально выравнивали для получения равномерного распределения клеток на всех слоях CF. Через двое суток клетки трансфицировали четырьмя лентивирусными плазмидами, описанными выше, с использованием способа с CaPO4, который включает смесь Tris:EDTA (этилендиаминтетрауксусная кислота), 2 М CaCl2, 2× HBS (человеческий сывороточный альбумин) и четырех ДНК плазмид. В сутки 3 после трансфекции супернатант, содержащий секретированные лентивирусные векторы, отбирали, очищали и концентрировали. После удаления супернатанта из CF из каждой CF отбирали клетки конца продуцирования. Отбирали клетки из каждой CF. Клетки из каждой фабрики трипсинизировали и отбирали центрифугированием. Клетки ресуспендировали в среде для замораживания и криоконсервировали. Данные клетки позднее использовали для тестирования на лентивирус, компетентный в отношении репликации (RCL).
Для очистки и приготовления векторов в виде препарата неочищенный супернатант осветляли мембранной фильтрацией для удаления клеточных обломков. ДНК клетки-хозяина и остаточную плазмидную ДНК деградировали посредством расщепления эндонуклеазой (Бензоназа®). Вирусный супернатант осветляли от клеточных обломков с использованием 0,45 мкм фильтра. Осветленный супернатант отбирали в предварительно взвешенный контейнер, в который добавляют Бензоназу® (конечная концентрация 50 U/мл). Эндонуклеазное расщепление остаточной плазмидной ДНК и геномной ДНК хозяина проводится при 37°С в течение 6 ч. Для удаления остаточных низкомолекулярных компонентов из неочищенного супернатанта использовали исходное концентрирование супернатанта, обработанного эндонуклеазой, посредством ультрафильтрации с тангенциальным потоком (TFF), концентрируя вирус в -20 раз. Осуществляли циркуляцию осветленного, обработанного эндонуклеазой вирусного супернатанта через картридж с полыми волокнами с NMWCO (номинальный порог отсечения по молекулярной массе) 500 кДа при скорости тока, подобранной для поддержания скорости сдвига -4000 с-1 или меньше при максимизации скорости потока. Во время процесса концентрирования запускают диафильтрацию супернатанта, обработанного нуклеазой, для поддержания эффективности картриджа. С использованием 4%-ной лактозы в PBS (фосфатно-солевой буферный раствор) в качестве диафильтрационного буфера устанавливали скорость замены пермеата 80%. Вирусный супернатант доводили до целевого объема, представляющего 20-кратное концентрирование неочищенного супернатанта, и диафильтрацию продолжали в течение 4 дополнительных смен объема со скоростью замены пермеата 100%.
Дальнейшее концентрирование вирусного продукта осуществляли с применением методики высокоскоростного центрифугирования. Каждую подпартию лентивируса осаждали с использованием центрифуги Sorvall RC-26 plus при 6000 об./мин (6088 RCF (относительная сила центрифугирования) при 6°С в течение 16-20 ч. Вирусный осадок из каждой подпартии затем растворяли в 50 мл объеме 4%-ной лактозы в PBS. Растворенный осадок в данном буфере представляет собой конечную композицию вирусного препарата. Весь процесс концентрирования вектора приводил приблизительно к 200-кратному уменьшению объема. После завершения получения всех подпартий материал затем помещали при -80°С, при тестировании образцов из каждой подпартии на стерильность. После подтверждения стерильности образца подпартии быстро оттаивали при 37°C с частым встряхиванием. Материал затем объединяли и вручную аликвотировали в боксе биологической безопасности Класса II Типа А/В3. Использовали конфигурацию заполнения 1 мл концентрированного лентивируса в стерильных криососудах класса 6 USP (Фармакопея США) с наружной резьбой и О-кольцом. Системы контроля качества (QS) Центра разработки прикладных технологий (CATD) в СОН (City of Норе) выпустили все материалы согласно политике и стандартным рабочим методикам для CBG и в соответствии с современными надлежащими производственными практиками (cGMP).
Для обеспечения чистоты препарата лентивирусного вектора его тестировали на остаточные загрязнения ДНК хозяина и перенос остаточной ДНК хозяина и плазмид. Наряду с другими тестами идентичность вектора оценивали посредством ПЦР-ОТ (полимеразная цепная реакция, сопряженная с обратной транскрипцией) для обеспечения того, что присутствует правильный вектор. Для вектора, предназначенного для применения в данном исследовании, удовлетворяются все критерии выпуска.
Пример 6: получение подходящих клеток Тем для экспрессии CAR CS1
Т-лимфоциты получают от пациента посредством лейкафереза, и подходящий поднабор аллогенных или аутологичных Т-клеток, например, центральных Т-клеток памяти (ТСм), генетически изменяется для экспрессии CAR, затем вводится пациенту посредством любых клинически приемлемых способов для достижения противораковой терапии.
Тем, которые представляют собой CD8+, выделяют по существу, как описано в Wang et al. (J Immunology 35:689, 2012). Вкратце, в сутки лейкафереза РВМС выделяли посредством центрифугирования в градиенте плотности через Ficoll-Paque, с последующими двумя промывками в PBS/EDTA. РВМС затем один раз промывали в PBS, ресуспендировали в среде X Vivo15, содержащей 10% телячьей коровьей сыворотки (FCS), переносили в 300 см3 мешок для переноса и хранили на 3-мерном вращающем устройстве при комнатной температуре (RT). На следующие сутки вплоть до 5×109 РВМС инкубировали в 300 см3 мешке для переноса с микрошариками клинического уровня качества против CD4 (2,5 мл), против CD14 (1,25 мл) и против CD45RA (2,5 мл) (Miltenyi Biotec) в течение 30 минут при RT в X Vivo15, содержащей 10% FCS. Затем немедленно осуществляли обеднение клетками CD4+, CD14+ и CD45RA+ с использованием режима обеднения CliniMACS™ согласно инструкциям изготовителя (Miltenyi Biotec). После центрифугирования немеченую негативную фракцию клеток ресуспендировали в буфере PBS/EDTA CliniMACS™ (Miltenyi Biotec), содержащем 0,5% человеческого сывороточного альбумина (HSA), и затем метили биотинилированным-DREG56 mAb (моноклональное антитело) клинического уровня качества (COHNMC CBG) в количестве 0,1 мг/106 клеток в течение 30 минут при RT. Клетки затем промывали и ресуспендировали в конечном объеме 100 мл PBS/EDTA CliniMACS™, содержащем 0,5% HSA, и переносили в новый 300 см3 мешок для переноса. После 30 минут инкубации с 1,25 мл микрошариков против биотина (Miltenyi Biotec), фракцию CD62L+ РВМС (ТСМ CD8+) очищали с использованием позитивной селекции на CliniMACS™ согласно инструкциям изготовителя и ресуспендировали в X Vivo15, содержащей 10% FCS.
Тем, которые представляют собой CD8+/CD4+, получают с использованием модификации вышеописанного способа посредством модифицирования отбора на CD4+, CD14+ и CD45RA+ на отбор на CD14+ и CD45RA+. В данном способе используется двухэтапный процесс на приборе CliniMACS™ сначала для обеднения клетками CD14+ и CD45RA+, затем для позитивной селекции на клетки CD62L+. Данная модифицированная платформа генерирует из одного лейкафереза объем 50×106 ТСм.
После обогащения клетки ТСм готовят в полной X-Vivo15 плюс 50 IU (международные единицы)/мл IL-2 и 0,5 нг/мл IL-15, и переносят в тефлоновый мешок для культуры клеток, где они стимулируются шариками Dynal ClinEx™ Vivo CD3/CD28. Вплоть до пяти суток после стимуляции клетки транедуцируются лентивирусным вектором, кодирующим CAR CS1, при множественности заражения (MOI) примерно 3. Культуры поддерживаются в течение вплоть до 42 суток с добавлением полной X-Vivo15 и цитокинов IL-2 и IL-15, как требуется для размножения клеток (поддерживая плотность клеток от 3×105 до 2×106 жизнеспособных клеток/мл и осуществляя дополнение цитокинами каждый понедельник, среду и пятницу культивирования). При данных условиях клетки типично размножаются до приблизительно 109 клеток в пределах 21 суток. В конце периода культивирования клетки отбирают, дважды промывают и готовят в виде препарата в криоконсервирующей среде клинического уровня качества.
В сутки инфузии Т-клеток криоконсервированный и выпущенный продукт будет оттаян, промыт и приготовлен в виде препарата для повторной инфузии. Криоконсервированные сосуды, содержащие выпущенный клеточный продукт, будут удалены из хранилища с жидким азотом, оттаяны, охлаждены и промыты промывочным буфером на основе PBS/2% человеческого сывороточного альбумина (HSA). После центрифугирования супернатант будет удален, и клетки будут ресуспендированы в инфузионном разбавителе на основе нормального физиологического раствора, не содержащего консервантов (PFNS)/2% HSA. Образцы будут удалены для контроля качества.
Пример 7: аминокислотная последовательность CAR CS1 (CS1scFv-IqG4(HL-CH3)-CD28tm-CD28qq-Дзета-T2A-EGFRt)
Полная аминокислотная последовательность CS1scFv-IgG4(HL-CH3)-CD28tm-CD28gg-Дзета-T2A-EGFRt показана на Фиг. 4. Полная последовательность (SEQ ID NO: 29) включает: 22-аминокислотный сигнальный пептид GMCSF (SEQ ID NO: 26), последовательность scFv против CS1 (SEQ ID NO: 1), последовательность шарнира IgG4 (SEQ ID NO: 3; с аминокислотными заменами от S до Р, выделенными затенением); 10-аминокислотный линкер (SEQ ID NO: 2), последовательность СН3 IgG4 (SEQ ID NO: 12), 28-аминокислотную последовательность трансмембранного домена CD28 (SEQ ID NO: 14); последовательность костимулирующего домена CD28gg (SEQ ID NO: 23; выделены аминокислотные замены от LL до GG); 3-аминокислотный Gly линкер; 112-аминокислотную последовательность CD3ζ (SEQ ID NO: 21); 24-аминокислотную последовательность перескакивания рибосомы Т2А (SEQ ID NO: 27) и последовательность EGFRt (SEQ ID NO: 28).
--->
<110> ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<120> Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ
НА CS1, ДЛЯ ЛЕЧЕНИЯ АМИЛОИДОЗА AL
<130> 40056-0033WO1
<140> PCT/US2018/023381
<141> 2018-03-20
<150> 62/473,980
<151> 2017-03-20
<160> 46
<170> PatentIn version 3.5
<210> 1
<211> 244
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 1
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys
<210> 2
<211> 10
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 2
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
1 5 10
<210> 3
<211> 12
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 3
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
1 5 10
<210> 4
<211> 12
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 4
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro
1 5 10
<210> 5
<211> 22
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 5
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser
1 5 10 15
Ser Gly Gly Gly Ser Gly
20
<210> 6
<211> 39
<212> ПРТ
<213> Homo sapiens
<400> 6
Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn
1 5 10 15
Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu
20 25 30
Phe Pro Gly Pro Ser Lys Pro
35
<210> 7
<211> 48
<212> ПРТ
<213> Homo sapiens
<400> 7
Ala Lys Pro Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
1 5 10 15
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
20 25 30
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 8
<211> 45
<212> ПРТ
<213> Homo sapiens
<400> 8
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 9
<211> 129
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 9
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser
1 5 10 15
Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
20 25 30
Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
35 40 45
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
50 55 60
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
65 70 75 80
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys
85 90 95
Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu
100 105 110
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
115 120 125
Lys
<210> 10
<211> 229
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 10
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Gln Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 11
<211> 229
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 11
Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe
1 5 10 15
Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Gln Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys
225
<210> 12
<211> 107
<212> ПРТ
<213> Homo sapiens
<400> 12
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
1 5 10 15
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
20 25 30
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
35 40 45
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
50 55 60
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
65 70 75 80
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
85 90 95
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
100 105
<210> 13
<211> 21
<212> ПРТ
<213> Homo sapiens
<400> 13
Leu Cys Tyr Leu Leu Asp Gly Ile Leu Phe Ile Tyr Gly Val Ile Leu
1 5 10 15
Thr Ala Leu Phe Leu
20
<210> 14
<211> 27
<212> ПРТ
<213> Homo sapiens
<400> 14
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 15
<211> 28
<212> ПРТ
<213> Homo sapiens
<400> 15
Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser
1 5 10 15
Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 16
<211> 22
<212> ПРТ
<213> Homo sapiens
<400> 16
Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile
1 5 10 15
Gly Leu Gly Ile Phe Phe
20
<210> 17
<211> 21
<212> ПРТ
<213> Homo sapiens
<400> 17
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr
20
<210> 18
<211> 23
<212> ПРТ
<213> Homo sapiens
<400> 18
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr
20
<210> 19
<211> 24
<212> ПРТ
<213> Homo sapiens
<400> 19
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr Cys
20
<210> 20
<211> 27
<212> ПРТ
<213> Homo sapiens
<400> 20
Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu
1 5 10 15
Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
20 25
<210> 21
<211> 112
<212> ПРТ
<213> Homo sapiens
<400> 21
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 22
<211> 41
<212> ПРТ
<213> Homo sapiens
<400> 22
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 23
<211> 41
<212> ПРТ
<213> Homo sapiens
<400> 23
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 24
<211> 42
<212> ПРТ
<213> Homo sapiens
<400> 24
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 25
<211> 42
<212> ПРТ
<213> Homo sapiens
<400> 25
Ala Leu Tyr Leu Leu Arg Arg Asp Gln Arg Leu Pro Pro Asp Ala His
1 5 10 15
Lys Pro Pro Gly Gly Gly Ser Phe Arg Thr Pro Ile Gln Glu Glu Gln
20 25 30
Ala Asp Ala His Ser Thr Leu Ala Lys Ile
35 40
<210> 26
<211> 22
<212> ПРТ
<213> Homo sapiens
<400> 26
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro
20
<210> 27
<211> 24
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 27
Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp
1 5 10 15
Val Glu Glu Asn Pro Gly Pro Arg
20
<210> 28
<211> 354
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 28
Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu
1 5 10 15
Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys
20 25 30
Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys
35 40 45
Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly
50 55 60
Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile
65 70 75 80
Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp
85 90 95
Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile
100 105 110
Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser
115 120 125
Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp
130 135 140
Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr
145 150 155 160
Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile
165 170 175
Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys
180 185 190
His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp
195 200 205
Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys
210 215 220
Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu
225 230 235 240
Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr
245 250 255
Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile
260 265 270
Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu
275 280 285
Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His
290 295 300
Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu
305 310 315 320
Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met
325 330 335
Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu
340 345 350
Phe Met
<210> 29
<211> 960
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 29
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu
385 390 395 400
Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val
405 410 415
Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His
420 425 430
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
435 440 445
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
450 455 460
Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
465 470 475 480
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
485 490 495
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
500 505 510
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
515 520 525
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
530 535 540
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
545 550 555 560
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
565 570 575
Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr
580 585 590
Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val
595 600 605
Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile
610 615 620
Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser
625 630 635 640
Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser
645 650 655
Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser
660 665 670
Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys
675 680 685
Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu
690 695 700
Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly
705 710 715 720
Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn
725 730 735
Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp
740 745 750
Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn
755 760 765
Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser
770 775 780
Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala
785 790 795 800
Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val
805 810 815
Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn
820 825 830
Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile
835 840 845
Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr
850 855 860
Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly
865 870 875 880
Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn
885 890 895
Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys
900 905 910
His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys
915 920 925
Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly
930 935 940
Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met
945 950 955 960
<210> 30
<211> 579
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 30
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu
385 390 395 400
Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val
405 410 415
Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His
420 425 430
Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys
435 440 445
His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
450 455 460
Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
465 470 475 480
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
485 490 495
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
500 505 510
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
515 520 525
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
530 535 540
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
545 550 555 560
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
565 570 575
Pro Pro Arg
<210> 31
<211> 557
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 31
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro
260 265 270
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
275 280 285
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
290 295 300
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
305 310 315 320
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
325 330 335
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
340 345 350
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
355 360 365
Leu Ser Leu Gly Lys Met Phe Trp Val Leu Val Val Val Gly Gly Val
370 375 380
Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp
385 390 395 400
Val Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met
405 410 415
Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala
420 425 430
Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys
435 440 445
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
450 455 460
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
465 470 475 480
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg
485 490 495
Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met
500 505 510
Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly
515 520 525
Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp
530 535 540
Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
545 550 555
<210> 32
<211> 955
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 32
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val
385 390 395 400
Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe
405 410 415
Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
420 425 430
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
435 440 445
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val
450 455 460
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
465 470 475 480
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
485 490 495
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
500 505 510
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
515 520 525
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
530 535 540
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
545 550 555 560
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Leu Glu
565 570 575
Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu
580 585 590
Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val Thr Ser Leu Leu Leu
595 600 605
Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile Pro Arg Lys Val Cys
610 615 620
Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala
625 630 635 640
Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu
645 650 655
His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro
660 665 670
Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile
675 680 685
Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu
690 695 700
His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His
705 710 715 720
Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly
725 730 735
Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly
740 745 750
Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe
755 760 765
Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn
770 775 780
Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu
785 790 795 800
Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val
805 810 815
Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu
820 825 830
Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu
835 840 845
Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp
850 855 860
Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys
865 870 875 880
Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys
885 890 895
Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys Thr
900 905 910
Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro
915 920 925
Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu
930 935 940
Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met
945 950 955
<210> 33
<211> 574
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 33
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly
275 280 285
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu
290 295 300
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
305 310 315 320
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
325 330 335
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
340 345 350
Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly
355 360 365
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
370 375 380
Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val
385 390 395 400
Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe
405 410 415
Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
420 425 430
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
435 440 445
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val
450 455 460
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
465 470 475 480
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
485 490 495
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg
500 505 510
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
515 520 525
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
530 535 540
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
545 550 555 560
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
565 570
<210> 34
<211> 552
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 34
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Gly Gln Pro Arg Glu Pro
260 265 270
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
275 280 285
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
290 295 300
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
305 310 315 320
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
325 330 335
Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
340 345 350
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
355 360 365
Leu Ser Leu Gly Lys Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly
370 375 380
Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys
385 390 395 400
Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr
405 410 415
Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu
420 425 430
Gly Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala
435 440 445
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
450 455 460
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
465 470 475 480
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
485 490 495
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
500 505 510
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
515 520 525
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
530 535 540
His Met Gln Ala Leu Pro Pro Arg
545 550
<210> 35
<211> 1055
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 35
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly
500 505 510
Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
515 520 525
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
530 535 540
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly
545 550 555 560
Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys
675 680 685
Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met Leu Leu Leu Val Thr
690 695 700
Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe Leu Leu Ile Pro
705 710 715 720
Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu
725 730 735
Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile
740 745 750
Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe
755 760 765
Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr
770 775 780
Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn
785 790 795 800
Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg
805 810 815
Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile
820 825 830
Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val
835 840 845
Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp
850 855 860
Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn
865 870 875 880
Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu
885 890 895
Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser
900 905 910
Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu
915 920 925
Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln
930 935 940
Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly
945 950 955 960
Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro
965 970 975
His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr
980 985 990
Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His
995 1000 1005
Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys
1010 1015 1020
Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val
1025 1030 1035
Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu
1040 1045 1050
Phe Met
1055
<210> 36
<211> 674
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 36
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly
500 505 510
Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
515 520 525
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
530 535 540
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly
545 550 555 560
Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln
565 570 575
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
580 585 590
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
595 600 605
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
610 615 620
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
625 630 635 640
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
645 650 655
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
660 665 670
Pro Arg
<210> 37
<211> 652
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 37
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Phe Gln Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Ala Leu Ile Val Leu Gly
465 470 475 480
Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys
485 490 495
Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg
500 505 510
Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro
515 520 525
Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe
530 535 540
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
545 550 555 560
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
565 570 575
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
580 585 590
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
595 600 605
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
610 615 620
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
625 630 635 640
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
645 650
<210> 38
<211> 1060
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 38
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
500 505 510
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
515 520 525
Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
530 535 540
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
545 550 555 560
Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly
675 680 685
Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met
690 695 700
Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala
705 710 715 720
Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu
725 730 735
Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys
740 745 750
Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe
755 760 765
Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu
770 775 780
Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln
785 790 795 800
Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu
805 810 815
Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val
820 825 830
Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile
835 840 845
Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala
850 855 860
Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr
865 870 875 880
Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln
885 890 895
Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro
900 905 910
Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val
915 920 925
Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn
930 935 940
Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn
945 950 955 960
Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His
965 970 975
Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met
980 985 990
Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val
995 1000 1005
Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro
1010 1015 1020
Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile
1025 1030 1035
Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala
1040 1045 1050
Leu Gly Ile Gly Leu Phe Met
1055 1060
<210> 39
<211> 679
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 39
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro
260 265 270
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly Gly Pro Ser Val
275 280 285
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
290 295 300
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
305 310 315 320
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
325 330 335
Thr Lys Pro Arg Glu Glu Gln Phe Gln Ser Thr Tyr Arg Val Val Ser
340 345 350
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
355 360 365
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
370 375 380
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
385 390 395 400
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
405 410 415
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
420 425 430
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
435 440 445
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
450 455 460
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
465 470 475 480
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys Met
485 490 495
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
500 505 510
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
515 520 525
Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
530 535 540
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
545 550 555 560
Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
565 570 575
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
580 585 590
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
595 600 605
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
610 615 620
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
625 630 635 640
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
645 650 655
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
660 665 670
Met Gln Ala Leu Pro Pro Arg
675
<210> 40
<211> 657
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 40
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
245 250 255
Ala Pro Glu Phe Glu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
260 265 270
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
275 280 285
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
290 295 300
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
305 310 315 320
Gln Phe Gln Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
325 330 335
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
340 345 350
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
355 360 365
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
370 375 380
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
385 390 395 400
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
405 410 415
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
420 425 430
Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
435 440 445
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
450 455 460
Lys Ser Leu Ser Leu Ser Leu Gly Lys Met Phe Trp Val Leu Val Val
465 470 475 480
Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe
485 490 495
Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp
500 505 510
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
515 520 525
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly
530 535 540
Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
545 550 555 560
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
565 570 575
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
580 585 590
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
595 600 605
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
610 615 620
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
625 630 635 640
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
645 650 655
Arg
<210> 41
<211> 836
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 41
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu
275 280 285
Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys
290 295 300
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
305 310 315 320
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
325 330 335
Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly Gly Glu Gly Arg Gly
450 455 460
Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Arg Met
465 470 475 480
Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala
485 490 495
Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu
500 505 510
Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys
515 520 525
Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe
530 535 540
Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu
545 550 555 560
Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln
565 570 575
Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu
580 585 590
Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val
595 600 605
Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile
610 615 620
Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala
625 630 635 640
Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr
645 650 655
Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln
660 665 670
Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro
675 680 685
Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val
690 695 700
Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn
705 710 715 720
Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn
725 730 735
Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His
740 745 750
Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met
755 760 765
Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val
770 775 780
Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly
785 790 795 800
Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr
805 810 815
Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile
820 825 830
Gly Leu Phe Met
835
<210> 42
<211> 455
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 42
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Ala Leu Ile Val Leu Gly Gly Val Ala Gly Leu
275 280 285
Leu Leu Phe Ile Gly Leu Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys
290 295 300
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
305 310 315 320
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
325 330 335
Gly Cys Glu Leu Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp
340 345 350
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
355 360 365
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
370 375 380
Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly
385 390 395 400
Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu
405 410 415
Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu
420 425 430
Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His
435 440 445
Met Gln Ala Leu Pro Pro Arg
450 455
<210> 43
<211> 433
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 43
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Met Ala
245 250 255
Leu Ile Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu
260 265 270
Gly Ile Phe Phe Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
275 280 285
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
290 295 300
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Gly Gly
305 310 315 320
Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
325 330 335
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
340 345 350
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
355 360 365
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
370 375 380
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
385 390 395 400
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
405 410 415
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
420 425 430
Arg
<210> 44
<211> 841
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 44
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu
275 280 285
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
290 295 300
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
305 310 315 320
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
325 330 335
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Leu Glu Gly Gly
450 455 460
Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu Glu Asn
465 470 475 480
Pro Gly Pro Arg Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu
485 490 495
Leu Pro His Pro Ala Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly
500 505 510
Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn
515 520 525
Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile
530 535 540
Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu
545 550 555 560
Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly
565 570 575
Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala
580 585 590
Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln
595 600 605
Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg
610 615 620
Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys
625 630 635 640
Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr
645 650 655
Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys
660 665 670
Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys
675 680 685
Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg
690 695 700
Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg
705 710 715 720
Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu
725 730 735
Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys
740 745 750
Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys
755 760 765
Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala
770 775 780
Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly
785 790 795 800
Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile
805 810 815
Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val
820 825 830
Val Ala Leu Gly Ile Gly Leu Phe Met
835 840
<210> 45
<211> 460
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 45
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 10 15
Ala Phe Leu Leu Ile Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly
20 25 30
Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
35 40 45
Phe Asp Phe Ser Arg Tyr Trp Met Ser Trp Val Arg Gln Ala Pro Gly
50 55 60
Lys Gly Leu Glu Trp Ile Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile
65 70 75 80
Asn Tyr Ala Pro Ser Leu Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn
85 90 95
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
100 105 110
Thr Ala Val Tyr Tyr Cys Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe
115 120 125
Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Ser Thr
130 135 140
Ser Gly Gly Gly Ser Gly Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp
145 150 155 160
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
165 170 175
Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Gly Ile Ala Val
180 185 190
Ala Trp Tyr Gln Gln Lys Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr
195 200 205
Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu
225 230 235 240
Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr
245 250 255
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Gly Gly Gly Ser Ser Gly
260 265 270
Gly Gly Ser Gly Met Phe Trp Val Leu Val Val Val Gly Gly Val Leu
275 280 285
Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
290 295 300
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
305 310 315 320
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
325 330 335
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe
340 345 350
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
355 360 365
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
370 375 380
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
385 390 395 400
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
405 410 415
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
420 425 430
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
435 440 445
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
450 455 460
<210> 46
<211> 438
<212> ПРТ
<213> Искусственная последовательность
<220>
<221> источник
<223> /примечание="Описание искусственной последовательности: синтетический
полипептид"
<400> 46
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Glu Ile Asn Pro Asp Ser Ser Thr Ile Asn Tyr Ala Pro Ser Leu
50 55 60
Lys Asp Lys Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Asp Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Ser Thr Ser Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Lys Ala Ser Gln Asp Val Gly Ile Ala Val Ala Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Val Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg His
180 185 190
Thr Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Val Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Ser Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Val Glu Ile Lys Gly Gly Gly Ser Ser Gly Gly Gly Ser Gly Met Phe
245 250 255
Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu
260 265 270
Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg
275 280 285
Gly Gly His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro
290 295 300
Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala
305 310 315 320
Tyr Arg Ser Gly Gly Gly Arg Val Lys Phe Ser Arg Ser Ala Asp Ala
325 330 335
Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu
340 345 350
Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp
355 360 365
Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu
370 375 380
Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile
385 390 395 400
Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr
405 410 415
Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met
420 425 430
Gln Ala Leu Pro Pro Arg
435
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Т-КЛЕТКИ, МОДИФИЦИРОВАННЫЕ ХИМЕРНЫМ РЕЦЕПТОРОМ АНТИГЕНА, НАЦЕЛЕННЫМ НА CS1 | 2015 |
|
RU2727451C2 |
| ХИМЕРНЫЕ РЕЦЕПТОРЫ АНТИГЕНА, НАЦЕЛЕННЫЕ НА HER2 | 2016 |
|
RU2753695C2 |
| КЛЕТКИ ДЛЯ ИММУНОТЕРАПИИ, СКОНСТРУИРОВАННЫЕ ДЛЯ НАЦЕЛИВАНИЯ НА АНТИГЕН, ПРИСУТСТВУЮЩИЙ ОДНОВРЕМЕННО НА ИММУННЫХ КЛЕТКАХ И НА ПАТОЛОГИЧЕСКИХ КЛЕТКАХ | 2015 |
|
RU2714258C2 |
| CD20 ТЕРАПИЯ, CD22 ТЕРАПИЯ И КОМБИНИРОВАННАЯ ТЕРАПИЯ КЛЕТКАМИ, ЭКСПРЕССИРУЮЩИМИ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР (CAR) K CD19 | 2016 |
|
RU2752918C2 |
| ХИМЕРНЫЕ РЕЦЕПТОРЫ АНТИГЕНА, НАЦЕЛЕННЫЕ НА PSCA | 2016 |
|
RU2759879C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА FLT3 | 2018 |
|
RU2820859C2 |
| СПЕЦИФИЧНЫЕ К MUC16 ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795198C2 |
| Т-КЛЕТКИ С ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ (CAR-T) ДЛЯ ЛЕЧЕНИЯ РАКА | 2019 |
|
RU2811466C2 |
| КЛЕТКА | 2014 |
|
RU2717984C2 |
| КЛЕТКА | 2014 |
|
RU2732236C2 |
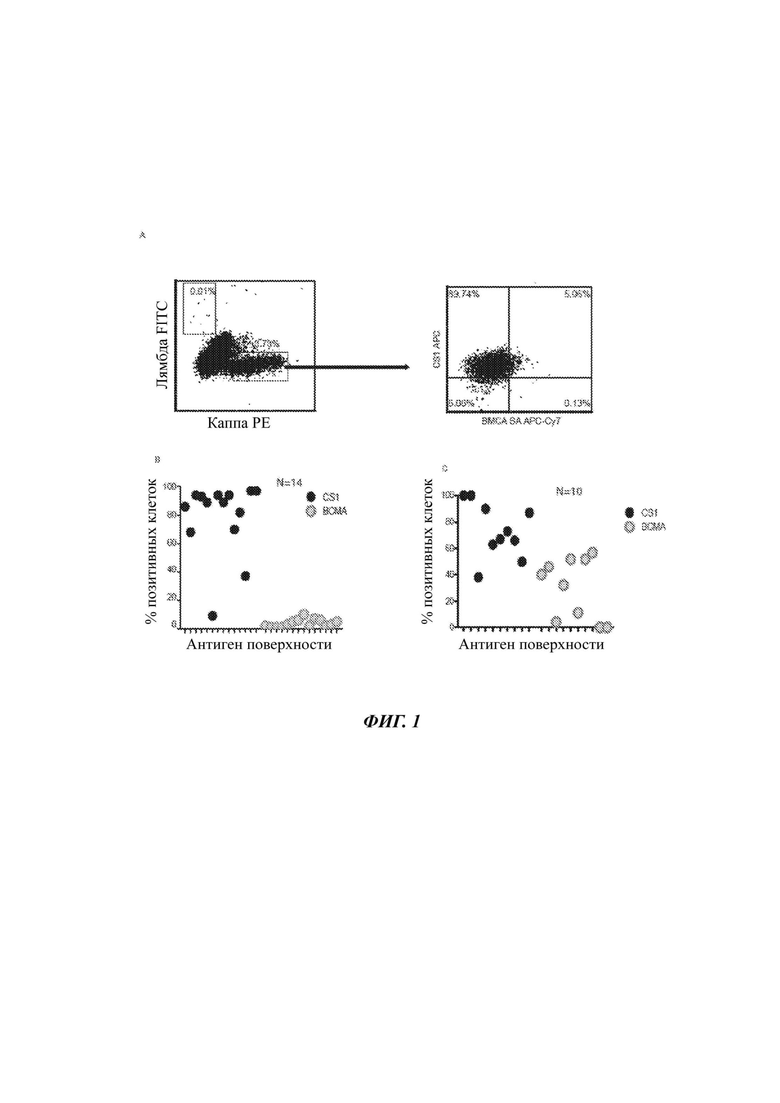
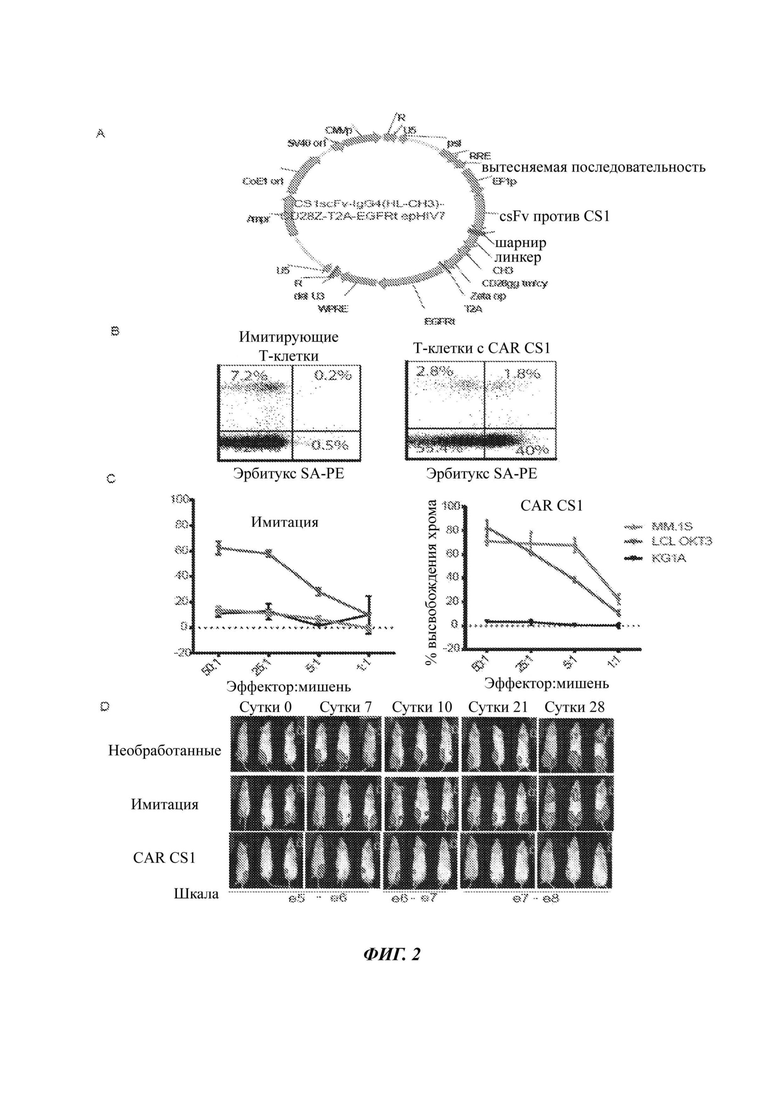







Группа изобретений относится к области генной инженерии, молекулярной биологии и медицины, в частности к способам лечения амилоидоза легкой цепи с использованием химерных антигенных рецепторов (CAR), нацеленных на CS1. Способ лечения амилоидоза легкой цепи включает введение пациенту популяции человеческих Т-клеток, трансдуцированных вектором, содержащим кассету экспрессии, кодирующую CAR, или Т-клеток, экспрессирующих CAR. Химерный антигенный рецептор содержит аминокислотную последовательность, выбранную из SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45. Изобретения позволяют повысить эффективность терапии амилоидоза легкой цепи CAR Т-клетками. 2 н. и 8 з.п. ф-лы, 9 ил., 5 табл., 7 пр.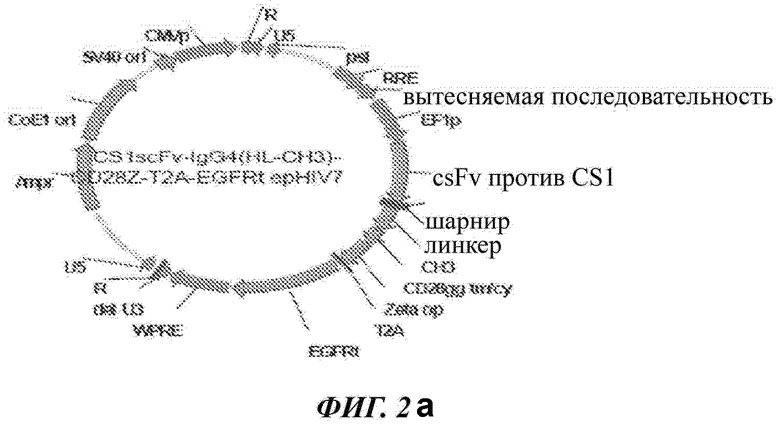
1. Способ лечения амилоидоза легкой цепи, включающий введение пациенту, нуждающемуся в этом, популяции человеческих Т-клеток, трансдуцированных вектором, содержащим кассету экспрессии, кодирующую химерный антигенный рецептор, где химерный антигенный рецептор содержит аминокислотную последовательность, выбранную из SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45.
2. Способ по п. 1, в котором по меньшей мере 20%, 30% или 40% трансдуцированных человеческих Т-клеток представляют собой центральные Т-клетки памяти.
3. Способ по п. 1, в котором по меньшей мере 30% трансдуцированных человеческих Т-клеток представляют собой CD4+ и CD62L+ или CD8+ и CD62L+.
4. Способ по п. 1, в котором популяция человеческих Т-клеток является аутологичной по отношению к данному пациенту.
5. Способ по п. 1, в котором популяция человеческих Т-клеток является аллогенной по отношению к данному пациенту.
6. Способ лечения амилоидоза легкой цепи, включающий введение пациенту, нуждающемуся в этом, популяции человеческих Т-клеток, экспрессирующих химерный антигенный рецептор, где химерный антигенный рецептор содержит аминокислотную последовательность, выбранную из SEQ ID NO: 30, 31, 33, 34, 36, 37, 39, 40, 42, 43, 44 и 45.
7. Способ по п. 6, в котором по меньшей мере 20%, 30% или 40% трансдуцированных человеческих Т-клеток представляют собой центральные Т-клетки памяти.
8. Способ по п. 6, в котором по меньшей мере 30% трансдуцированных человеческих Т-клеток представляют собой CD4+ и CD62L+ или CD8+ и CD62L+.
9. Способ по п. 6, в котором популяция человеческих Т-клеток является аутологичной по отношению к данному пациенту.
10. Способ по п. 6, в котором популяция человеческих Т-клеток является аллогенной по отношению к данному пациенту.
| AU 2013205096 B2, 03.12.2015 | |||
| Токарный резец | 1924 |
|
SU2016A1 |
| SUZUKI K | |||
| Diagnosis and treatment of multiple myeloma and AL amyloidosis with focus on improvement of renal lesion, CLINICAL AND EXPERIMENTAL NEPHROLOGY, 2012, v.16, no.5, p.659-671, DOI: 10.1007/S10157-012-0684-5 | |||
| LISENKO K | |||
| et al | |||
| Flow cytometry-based characterization of underlying | |||
Авторы
Даты
2022-06-27—Публикация
2018-03-20—Подача