Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, микробиологии и медицине и обеспечивает способ устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз чужеродной ДНК, при детекции генов blaTEM, ответственных за устойчивость к β-лактамным антибиотикам.
Уровень техники
Повсеместное распространение методов детекции, основанных на технологии амплификации нуклеиновых кислот, обусловлено их высокой чувствительностью и высокой скоростью определения по сравнению с большинством традиционных методов. Наиболее часто применяемым методом амплификации специфичных последовательностей ДНК является полимеразная цепная реакция (ПЦР). ПЦР широко используется в молекулярной биологии, вирусологии, медицине и микробиологии, в том числе для идентификации мутаций, клонирования, секвенирования и экспрессионного анализа. Лабораторная диагностика возбудителей инфекционных заболеваний является одной из главных областей, использующих высокоточные методы, основанные на ПЦР-амплификации.
За более чем 30-летнее время использования методов, основанных на ПЦР, в научных исследованиях и медицинской лабораторной диагностике были накоплены множественные факты контаминации реагентов для ПЦР, расходных материалов и реактивов, используемых для сопутствующих молекулярно-биологических исследований чужеродной бактериальной ДНК (Rand K.Н., Houck Н. Taq polymerase contains bacterial DNA of unknown origin. Mol Cell Probes. 1990, v. 4 (6), p. 445-50; Carroll N.M., Adamson P., Okhravi N. Elimination of bacterial DNA from Taq DNA polymerases by restriction endonuclease digestion. J Clin Microbiol. 1999, v. 37 (10), p. 3402-3404.; Corless C.E., Guiver M., Borrow R., et al. Contamination and sensitivity issues with a real-time universal 16S rRNA PCR. J Clin Microbiol. 2000, v. 38 (5), p. 1747-1752.; Zhang Т., Fang H.H. Applications of real-time polymerase chain reaction for quantification of microorganisms in environmental samples. Appl Microbiol Biotechnol. 2006, v. 70 (3), p. 281-289). Фермент Taq ДНК-полимераза (один из ключевых реагентов при проведении ПЦР) является наиболее уязвимым к контаминации фрагментами чужеродной ДНК, источником которых являются плазмидная и геномная ДНК бактериальных клеток, на основе которых осуществляется биоинженерное производство данного фермента (Nogami Т., Onto Т., Kawaguchi О., et al. Estimation of bacterial contamination in ultrapure water: application of the anti-DNA antibody. Anal Chem. 1998, v. 70 (24), p. 5296-5301; Kulakov L.A., McAlister M.B., Ogden K.L., et al. Analysis of bacteria contaminating ultrapure water in industrial systems. Appl Environ Microbiol. 2002, v. 68 (4), p. 1548-1555). Для селективного отбора при получении рекомбинантной ДНК-полимеразы в клетках Escherichia coli чаще всего используются векторные последовательности, содержащие гены антибиотикорезистентности (Joseph S., David W.R. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press, 3rd Edition. 2001, ISBN 0879695773). Очистка от контаминирующих последовательностей ДНК является трудоемким и дорогим процессом, при этом, добиться полной очистки на практике не представляется возможным. Даже, так называемые, свободные от ДНК коммерчески доступные реагенты высокой степени очистки («DNA-free» reagents) сопровождаются документированным интервалом копийности молекулами контаминирующей ДНК, который может быть детектирован при проведении ПЦР (Humphrey В., McLeod N., Turner С, et al. Removal of Contaminant DNA by Combined UV-EMA Treatment Allows Low Copy Number Detection of Clinically Relevant Bacteria Using Pan-Bacterial Real-Time PCR. PLoS One. 2015, v. 10 (7), p.e0132954).
Наличие в ПЦР-смеси Taq ДНК-полимеразы, содержащей фрагменты генов антибиотикорезистентности, может приводить к появлению ложноположительных результатов, драматически снижающих специфичность применяемых методов детекции и диагностики.
Известные к настоящему времени методы устранения последствий контаминации ДНК-полимераз фрагментами геномной и плазмидной чужеродной бактериальной ДНК направлены непосредственно на контаминирущюю ДНК с целью ее модификации, расщепления и последующей элиминации из коммерческого раствора фермента. Эти методы подразделяются на физические, химические, энзиматические, а также различные комбинации этих методов.
Физические методы основаны на инактивации контаминирующей ДНК путем воздействия УФ-облучения, обработки гамма-излучением или ультрафильтрации. К химическим методам относят обработку химическими реактивами, расщепляющими или инактивирующими контаминирующую ДНК. В энзиматических методах расщепление содержащейся чужеродной ДНК осуществляют ферментами.
Ниже приводятся основные недостатки этих методов:
1) Физический метод инактивации, основанный на облучении УФ-излучением (Ou C.Y., Moore J.L., Schochetman G. Use of UV irradiation to reduce false positivity in polymerase chain reaction. Biotechniques. 1991, v. 10 (4), p. 442-446), имеет следующие недостатки: малая глубина эффективного воздействия в растворе, экранирование излучения различными видами лабораторного пластика и стекла, необходимость в дополнительном оборудовании и стандартизации его характеристик, потеря активности реагентов белковой природы вследствие ионизации среды хранения;
2) Физический метод инактивации, основанный на облучении гамма-излучением (Deragon J.M., Sinnett D., Mitchell G., et al. Use of gamma irradiation to eliminate DNA contamination for PCR. Nucleic Acids Res. 1990, v. 18 (20), p. 6149.), имеет следующие недостатки: большая проникающая способность излучения требует мер по экранированию во избежание вреда здоровью, необходимость в дополнительном дорогостоящем оборудовании и стандартизации его характеристик, потеря активности реагентов белковой природы вследствие ионизации среды хранения;
3) Физический метод ультрафильтрации (Mohammadi Т., Reesink H.W., Vandenbroucke-Grauls С.М., Savelkoul Р.Н. Optimization of real-time PCR assay for rapid and sensitive detection of eubacterial 16S ribosomal DNA in platelet concentrates. J Clin Microbiol. 2003, v. 41 (10), p. 4796-4798) имеет следующие недостатки: необходимость в дополнительном оборудовании, неизбежные потери очищаемого фермента в процессе концентрации;
4) Химический метод, основанный на обработке гидрохлоридом гидроксиламина (Aslanzadeh J. Application of hydroxylamine hydrochloride for post-PCR sterilization. Mol Cell Probes. 1993, v. 7 (2), p. 145-150), имеет следующие недостатки: необходимость в деактивации гидроксиламина, токсичность вещества, невозможность использования обработанных реагентов без деактивации для последующих ПЦР;
5) Химический метод, основанный на обработке моноазидом этидия (Rueckert А., Morgan H.W. Removal of contaminating DNA from polymerase chain reaction using ethidium monoazide. J Microbiol Methods. 2007, v. 68 (3), p. 596-600) имеет следующие недостатки: необходимость в деактивации моноазида этидия, токсичность, невозможность использования обработанных реагентов без деактивации для последующих ПЦР;
6) Энзиматический метод, основанный на обработке эндонуклеазами рестрикции (Carroll N.M., Adamson P., Okhravi N. Elimination of bacterial DNA from Taq DNA polymerases by restriction endonuclease digestion. J Clin Microbiol. 1999, v. 37 (10), p. 3402-3404) имеет следующие недостатки: необходимость наличия достаточного количества сайтов рестрикции в последовательности контаминирующей ДНК и, как следствие, возможность формирования детектируемых продуктов, дающих ложноположительные результаты, необходимость в термической инактивации, риск расщепления продуктов амплификации при неполной деактивации эндонуклеазы рестрикции;
7) Энзиматический метод, основанный на обработке ДНКазой I (Rochelle Р.А., Weightman A.J., Fry J.С. DNase I treatment of Taq DNA polymerase for complete PCR decontamination. Biotechniques. 1992, v. 13 (4), p. 520) имеет следующие недостатки: необходимость в термической инактивации, риск расщепления продуктов амплификации при неполной деактивации ДНКазы I;
8) Комбинированный метод, основанный на фотосенсибилизационной обработке псораленами и последующей УФ-обработкой (Jinno Y., Yoshiura K., Niikawa N. Use of psoralen as extinguisher of contaminated DNA in PCR. Nucleic Acids Res. 1990, v. 18 (22), p. 6739) имеет следующие недостатки: необходимость в дополнительном оборудовании и стандартизации его характеристик, потеря активности реагентов белковой природы вследствие ионизации среды хранения.
Общим недостатком подобных методов и их возможных комбинации является прямое вмешательстве в реакционную смесь ПЦР, что накладывает серьезные ограничения при планировании исследования и на использование реактивов.
Одним из доступных способов избежать проведения этих дополнительных манипуляций является использование сверхчистых, свободных от чужеродной ДНК коммерчески доступных реагентов, однако, это приводит к существенному увеличению расходов, что неприемлемо для выполнения рутинных процедур лабораторной диагностики.
Предлагаемый настоящим изобретением способ направлен на препятствование или полное избежание амплификации контаминирующей ДНК без какой-либо предварительной обработки препарата фермента. Использование данного способа не оказывает прямого или косвенного влияния на эффективность проведения ПЦР, а также не требует дополнительного оборудования, специфических средств и мер защиты, помимо типичной аппаратуры и реактивов, применяемых при постановке ПЦР, и позволяет применять как в научных исследований, так и в лабораторной диагностике общедоступные, стандартные ДНК-полимеразы, не прошедшие дорогую и трудоемкую процедуру очистки.
Раскрытие сущности изобретения
Изобретением предлагается способ обнаружения чужеродной контаминирующей ДНК в коммерческих ДНК-полимеразах с целью устранения ложноположительных результатов при проведении ПЦР с использованием набора олигонуклеотидных праймеров, с последовательностями, представленными SEQ ID NO 1-4 и последующей интерпретации контаминационной картины, исследуемой аликвоты ДНК-полимеразы. Предложенный способ выгодно отличается от известных из уровня техники методов возможностью использовать в ходе научных исследований и лабораторной диагностике общедоступные ДНК-полимеразы, не прошедшие дорогую и трудоемкую процедуру очистки, не оказывает прямого или косвенного влияния на эффективность прохождения ПЦР, не требует дорогостоящего дополнительного оборудования, кроме базового, необходимого для проведения ПЦР, и не требует высококвалифицированного персонала. Данные, полученные с помощью способа, могут быть использованы как при выборе реактивов для ПЦР, в частности, ДНК-полимеразы при планировании исследования или лабораторной диагностики, касающихся генов резистентности семейства blaTEM, так и для определения мишени внутри гена blaTEM, которая не содержится в контаминирующей чужеродной ДНК полимеразы. Гены семейства blaTEM являются одними из самых распространенных генов резистентности, продукты которых расщепляют бета-лактамнымые антибиотики широкого спектра. Их своевременное выявление требуется для рационального выбора антибактериальных препаратов.
Способ основан на проведении полимеразной цепной реакции с набором специфических олигонуклеотидных праймеров с последующим разделении продуктов реакции путем электрофореза в агарозном геле, анализе электрофоретической картины разделения продуктов и интерпретации результатов.
Интерпретация результатов включает однозначную идентификацию контаминирующих фрагментов гена blaTEM, и, как следствие, позволяет выбрать пару праймеров, неспецифичных к обнаруженным фрагментам контаминирующей ДНК, но способных амплифицировать иной фрагмент гена blaTEM в изучаемом образце для устранения ложноположительных результатов.
Данное изобретение обеспечивает также оригинальный набор специфичных праймеров для осуществления идентификации участков генов резистентности семейства blaTEM. Праймеры, входящие в набор, имеют оригинальные олигонуклеотидные последовательности и позволяют специфическим образом идентифицировать участки генов резистентности семейства blaTEM.
Устранение ложноположительных результатов контаминации, основано на анализе электрофоретического разделения продуктов амплификации фрагмента гена blaTEM с оригинальным набором специфичных праймеров с последующей интерпретацией контаминационной картины конкретного образца используемой ДНК-полимеразы.
Далее изобретение будет подробно раскрыто со ссылками на фигуры и примеры, которые приводятся исключительно с целью иллюстрации и пояснения сущности заявленного изобретения, но которые не предназначены для ограничения объема притязаний.
Краткое описание таблиц и фигур
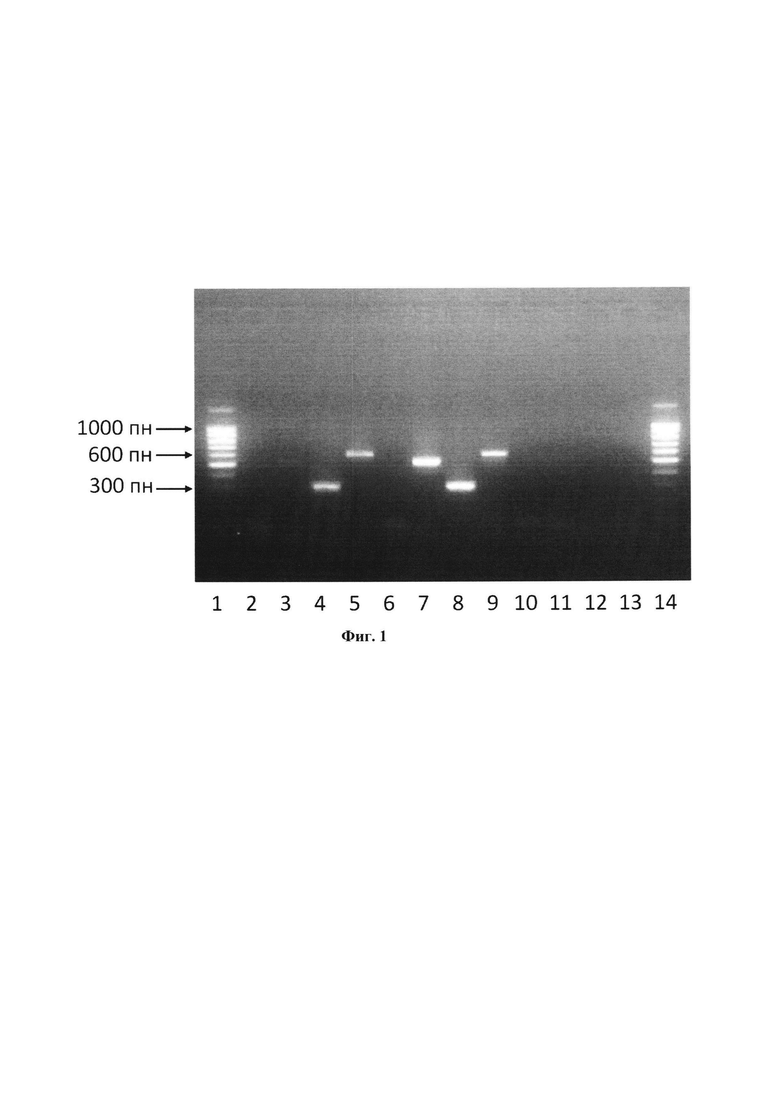
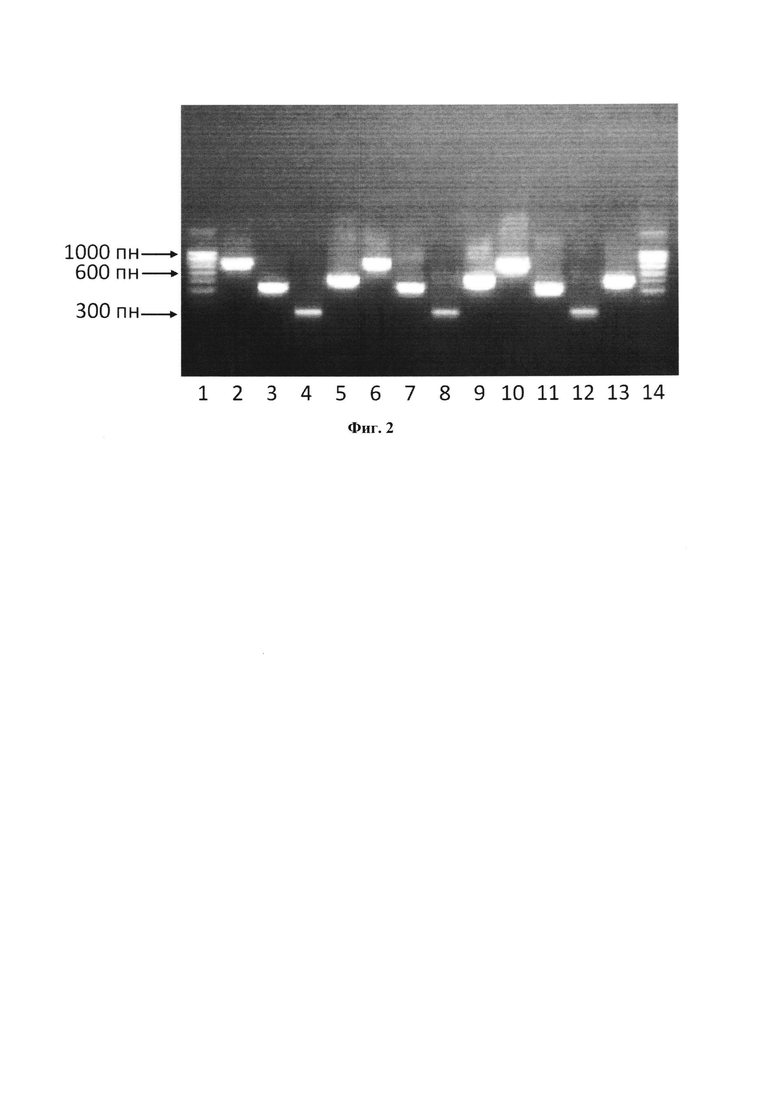
Таблица 1. Перечень олигонуклеотидных праймеров используемых в способе устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз при детекции blaTEM-генов устойчивости к β-лактамным антибиотикам. Фиг. 1. представляет картину электрофоретического разделения продуктов ПЦР, полученных при амплификации контаминирующей ДНК, содержащейся в образцах полимераз: Полимераза №1, Полимераза №2 и Полимераза №3, с соответствующими оригинальными олигонуклеотидными праймерами SEQ ID N11-4. Фиг. 2. представляет картину электрофоретического разделения продуктов ПЦР, полученных при амплификации референсного образца бактериальной ДНК содержащей ген blaTEM-1 с использованием соответствующих оригинальных нуклеотидных праймеров SEQ ID NO 1-4.
Осуществление изобретения
Целью настоящего изобретения является создание способа предотвращения ложноположительных результатов, вызванных контаминацией ДНК-полимераз чужеродной бактериальной ДНК при детекции blaTEM-генов устойчивости к β-лактамным антибиотикам.
В заявленном способе предложено использование полимеразной цепной реакции для предварительной амплификации последовательностей фрагментов blaTEM-генов устойчивости к β-лактамным антибиотикам, потенциально присутствующих в аликвоте ДНК-полимераз, с последующей интерпретацией электрофоретической картины разделения продуктов амплификации. Заявленный способ предусматривает использование оригинального набора специфических олигонуклеотидных праймеров (Таблица 1), но не ограничивается ими. В заявленном способе также могу использоваться праймеры иной нуклеотидной последовательности комплементарных участкам генов семейства blaTEM, количество используемых в способе пар праймеров также не ограничивает объем притязаний.
В качестве образца используется аликвота тестируемой ДНК-полимеразы. Метод не требует дополнительных процедур для обработки тестируемого образца и изменения его количества на единицу объема реакционной смеси ПЦР.
Подбор олигонуклеотидных праймеров выполняется на основе систематизации последовательностей, кодирующих гены семейства blaTEM. Систематизация осуществляется на основе анализа литературных источников и последовательностей генов в базах данных (GenBank). Далее проводится множественное выравнивание последовательностей генов-мишеней. Каждому выравниванию присваивается консенсусная последовательность. Выравнивание последовательностей проводится с помощью специализированного программного обеспечения, например, BioEdit Sequence Alignment Editor (версия 7.1.9) по алгоритму ClustalW, консенсусная последовательность присваивалась со значением параметра «threshold frequency)) равным 100%. Далее, используя соответствующее программное обеспечение, например, Oligo v. 6.3 (Molecular Biology Insights Inc., США) или Fast PCR (http://www.biocenter.helsinki.fi/bi/ Programs/fastpcr.htm), проводится расчет температуры отжига праймеров. При подборе праймеров руководствуются общеизвестными приемами, например, избегают выбора последовательностей, способных формировать вторичные структуры типа шпильки с высокими температурами плавления. Специфичность подобранных праймеров проверятся путем поиска по базам данных нуклеотидных последовательностей (GenBank, Silva) с использованием программного обеспечения, поддерживающего алгоритм BLAST (например, www.ncbi.nlm.nih.gov/5Zv4ST).
Продукты амплификации, полученные с помощью полимеразной цепной реакции с использованием данного набора праймеров, подвергаются разделению путем проведения электрофореза в агарозном геле. Далее проводится анализ полученных изображений электрофореграмм. По наличию и расположению специфических полос для продуктов амплификации проводится интерпретация электрофореграммы и делается однозначное заключение о наличии в анализируемом образце ДНК-полимеразы тех или иных чужеродных фрагментов blaTEM-генов, контаминирующих энзим.
На основании однозначной идентификации контаминирующих фрагментов гена blaTEM, присутствующих в качестве чужеродной ДНК в полимеразе, для последующего анализа образцов методом ПЦР выбирается мишень внутри гена blaTEM, которая отсутствует в образце энзима. Соответственно, выбирается и пара праймеров, неспецифичная к обнаруженным фрагментом контаминирующей ДНК, но способная амплифицировать иной фрагмент гена blaTEM в изучаемом образце.
Далее изобретение будет проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности заявленного изобретения, но не должны рассматриваться как ограничивающие данное изобретение.
Пример 1. Проведение полимеразной цепной реакции для исследования контаминации ДНК-полимераз фрагментами генов blaTEM.
Праймеры для проведения полимеразной цепной реакции были синтезированы на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США). Список праймеров приведен в Таблице 1.
В состав реакционной смеси для проведения ПЦР входили следующие реактивы:
- реакционный буфер, рекомендуемый производителем ДНК-полимераз;
- смесь дезоксинуклеозидтрифосфатов: дАТФ, дГТФ, дЦТФ и дУТФ, по 250 мкМ каждого (Силекс, Россия);
- смесь прямых и обратных праймеров в концентрации 5 пМ каждого;
- вода деионизированная до конечного объема в 25 мкл.
В качестве положительного контроля амплификации использовался 1 мкл раствора бактериальной ДНК, содержащей blaTEM-1 ген.
В 24,5 мкл соответствующей ПЦР-смеси вносили по 2,5 е.а образца тестируемых ДНК-полимераз - Полимераза №1 (HotStarTaa Plus DNA Polymerase, Qiagen, Chatsworth, CA, США), Полимераза №2 (HS Taq DNA polymerase, Евроген, Москва, Россия), и Полимераза №3 (Taq DNA polymerase, Fermentas, Вильнюс, Литва).
Амплификацию проводили на термоциклере С1000 Touch Thermal Cycler (Biorad, Hercules, CA, США) со следующим режимом: 95°С - 30 с, 55°С - 30 с, 72°С - 30 с; 45 циклов (в первом цикле время денатурации увеличивали до 5 мин, в последнем - время достройки до 5 мин).
10 мкл реакционной смеси, полученной после проведения ПЦР, использовали для электрофоретического разделения.
Пример 2. Проведение электрофоретического разделения продуктов амплификации контаминирующих фрагментов генов семейства blaTEM в агарозном геле.
1) готовили 1 × ТАЕ-буфер путем пятидесятикратного разведения 50 × ТАЕ буфера (2 М Трис, 1 М уксусная кислота, 50 мМ ЭДТА, рН 8,4) в объеме, достаточном для заполнения электрофорезной камеры и приготовления геля (1000 мл);
2) добавляли к 1 × ТАЕ буферу агарозу в количестве, необходимом для получения 2% раствора, и нагревали в микроволновой печи до полного расплавления агарозы;
3) охлаждали смесь до 50°С;
4) добавляли к раствору агарозы бромистый этидий до конечной концентрации 0,5 мкг/мл и осторожно перемешивали;
5) полученный раствор выливали в кювету для геля и равномерно распределяли по объему кюветы;
6) в кювету вставляли гребенку в вертикальном положении так, чтобы ее зубцы не доставали до дна примерно 1,5 мм;
7) оставляли кювету с агарозным гелем на 30 мин при комнатной температуре для застывания геля. Далее, аккуратно удаляли гребенку и помещали кювету с гелем в электрофорезную камеру, содержащую необходимое количество 1 × ТАЕ буфера.
8) по 10 мкл образов, полученных в Примере 1, смешивали с 3 мкл буфера для нанесения (10 мМ Трис-HCl, рН 7.8, 40% глицерин, 40 мМ ЭДТА, 0,01% бромфеноловый синий, 0,01% ксиленцианол) и аккуратно вносили в лунки геля;
9) проводили электрофорез при градиенте напряженности 1,5 В на 1 см геля в течение 30 мин;
10) извлекали гель из электрофорезной камеры и просматривали в УФ-свете на трансиллюминаторе. Результаты документировали путем фотографирования геля и сохранения полученных изображений.
Пример 3. Интерпретация результатов электрофоретического разделения продуктов амплификации контаминирующих фрагментов генов blaTEM.
Изображения, полученные в п. 10 Примера 2 интерпретировали следующим образом. В результате ПЦР возможна амплификация четырех типов продуктов различной длины при использовании соответствующих праймеров:
- продукт длиной 830 п.н. - фрагмент А, получаемый при использовании праймеров SEQ ID NO 1 и SEQ ID NO 4
- продукт длиной 517 п.н. - фрагмент Б, получаемый при использовании праймеров SEQ ID NO 1 и SEQ ID NO 3
- продукт длиной 290 п.н. - фрагмент В, получаемый при использовании праймеров SEQ ID NO 2 и SEQ ID NO 3
- продукт длиной 603 п.н. - фрагмент Г, получаемый при использовании праймеров SEQ ID NO 2 и SEQ ID NO 4
По наличию полосы оптической плотности, соответствующей продукту длиной 830 п.н. в дорожке на агаразном геле, делался вывод о наличии Фрагмента «А» у исследуемого образца. По наличию полосы оптической плотности, соответствующей продукту длиной 517 п.н. в дорожке на агаразном геле, делался вывод о наличии Фрагмента «Б» у исследуемого образца. По наличию полосы оптической плотности, соответствующей продукту длиной 290 п.н., в дорожке на агаразном геле, делался вывод о наличии Фрагмента «В» у исследуемого образца. По наличию полосы оптической плотности, соответствующей продукту длиной 603 п.н. в дорожке на агарозном геле, делался вывод о наличии Фрагмента «Г» у исследуемого образца. Отсутствие полосы оптической плотности свидетельствовало о том, что исследуемый образец не несет соответствующего контаминирующего фрагмента. Аликвота каждой ДНК-полимеразы была исследована в 5 повторах, относительно каждого потенциального контаминационного фрагмента.
На Фиг. 1. приведено изображение наиболее типичной картины электрофоретического разделения продуктов ПЦР в агарозном геле при анализе образцов ДНК-полимераз (Полимераза №1, Полимераза №2 и Полимераза №3) с использованием соответствующих пар праймеров. Каждому номеру соответствует следующая дорожка:
1) маркер длин ДНК 100 bp;
2) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца Полимераза №1 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «А» в образце Полимераза №1;
3) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца Полимераза №1. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «Б» в образце Полимераза №1;
4) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца Полимераза №1. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «В» в образце Полимераза №1;
5) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца Полимераза №1. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «Г» в образце Полимераза №1;
6) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца Полимераза №2 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «А» в образце Полимераза №2;
7) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца Полимераза №2. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «В» в образце Полимераза №2;
8) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца Полимераза №2. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «В» в образце Полимераза №2;
9) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца Полимераза №2. Присутствие полосы оптической плотности свидетельствует о присутствии контаминирующего Фрагмента «Г» в образце Полимераза №2;
10) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца Полимераза №3 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «А» в образце Полимераза №3;
11) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца Полимераза №3 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «В» в образце Полимераза №3;
12) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца Полимераза №3 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «В» в образце Полимераза №3;
13) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца Полимераза №3 отсутствует. Отсутствие полосы оптической плотности свидетельствует об отсутствии контаминирующего Фрагмента «Г» в образце Полимераза №3;
14) Маркер длин ДНК 100 bp.
На Фиг. 2. приведено изображение наиболее типичной картины электрофоретического разделения продуктов ПЦР в агарозном геле при анализе образцов референсной бактериальной ДНК, несущей ген blaTEM-1, с использованием ДНК-полимераз (Полимераза №1, Полимераза №2 и Полимераза №3) и соответствующих праймеров. Каждой дорожке соответствует следующий номер:
1) Маркер длин ДНК 100 bp;
2) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №1;
3) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №1;
4) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №1;
5) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №1;
6) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №2;
7) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №2;
8) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №2;
9) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №2;
10) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №3;
11) продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №3;
12) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 3 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №3;
13) продукт амплификации с праймеров SEQ ID NO 2 и SEQ ID NO 4 при анализе образца ДНК, содержащего blaTEM-1 с использованием полимеразы Полимераза №3;
14) Маркер длин ДНК 100 bp.
Пример 4. Устранение ложноположительных результатов, возникающих при использовании ДНК-полимераз, контаминированных фрагментами генов семейства blaTEM.
В ходе анализа образцов полимераз (Полимераза №1, Полимераза №2 и Полимераза №3) были выявлены следующие особенности:
1) фрагмент «А» не был найден у полимераз: Полимераза №1, Полимераза №2 и Полимераза №3;
2) фрагмент «Б» был найден у полимераз: Полимераза №1 и Полимераза №2;
3) фрагмент «В» был найден у полимераз: Полимераза №1 и Полимераза №2;
4) фрагмент «Г» был найден у полимераз: Полимераза №1 и Полимераза №2;
5) в полимеразе Полимераза №3 не было найдено фрагментов А, Б, В, Г На основании особенностей 1-5 можно сделать следующие выводы:
1) Полимераза №3 может быть использована без ограничений для исследований и лабораторной диагностики наличия генов blaTEM с использованием электрофоретического разделения в агарозном геле в качестве метода интерпретации результатов;
2) Полимеразы Полимераза №1 и Полимераза №2 имеют ограничения для исследований и лабораторной диагностики наличия генов blaTEM с использованием электрофоретического разделения в агарозном геле, в качестве метода интерпретации результатов;
3) При использовании полимераз Полимераза №1 и Полимераза №2 для исследований и лабораторной диагностики наличия генов blaTEM с использованием электрофоретического разделения в агарозном геле в качестве метода интерпретации результатов, необходимо выбирать мишень и амплифицируемый продукт так, чтобы контаминирующие последовательности не амплифицировались в ходе ПЦР и не приводили к ложноположительным результатам;
4) При использовании полимераз Полимераза №1 и Полимераза №2 для исследований и лабораторной диагностики наличия генов blaTEM с применением электрофоретического разделения в агарозном геле в качестве метода интерпретации результатов, было показано, что продукт амплификации с праймеров SEQ ID NO 1 и SEQ ID NO 4 является наиболее подходящим для детектирования генов blaTEM и не дает ложноположительных результатов при анализе наличия генов blaTEM.
Способ устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз фрагментами чужеродной ДНК, при идентификации генов blaTEM.

Способ устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз фрагментами чужерожной ДНК, при идентификации генов blaTEM
Таблица 1.
Изобретение относится к молекулярной биологии, микробиологии и медицине. Изобретение включает набор оригинальных олигонуклеотидных праймеров для осуществления способа, а также алгоритм выбора праймеров, неспецифичных к обнаруженным фрагментам контаминирующей ДНК и способных амплифицировать иной фрагмент гена blaTEM в изучаемом образце. Изобретение обеспечивает способ устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз при детекции blaTEM-генов устойчивости к β-лактамным антибиотикам. 2 н.п. ф-лы, 2 ил., 1 табл., 1 пр.
1. Способ устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз чужеродной ДНК, при детекции генов blaTEM, ответственных за устойчивость к β-лактамным антибиотикам, включающий:
1.1. амплификацию нескольких контаминирующих фрагментов генов семейства blaTEM путем проведения полимеразной цепной реакции с оригинальным набором специфических олигонуклеотидных праймеров;
1.2. разделение продуктов реакции путем электрофореза в агарозном геле;
1.3. анализ электрофоретической картины разделения продуктов амплификации;
1.4. интерпретацию результатов анализа, включающую однозначную идентификацию контаминирующих фрагментов гена blaTEM, по результатам которой для устранения ложноположительных результатов выбирается пара праймеров, неспецифичная к обнаруженным фрагментом контаминирующей ДНК, но способная амплифицировать иной фрагмент гена blaTEM в изучаемом образце.
2. Набор специфичных олигонуклеотидных праймеров для осуществления способа устранения ложноположительных результатов, вызванных контаминацией ДНК-полимераз при детекции blaTEM-генов устойчивости к β-лактамным антибиотикам, представленных последовательностями SEQ ID NO 1-4, включающий:
2.1. одну пару специфических олигонуклеотидных праймеров для амплификации контаминирующего фрагмента генов семейства blaTEM размером 830 п.н., представленных последовательностями SEQ ID NO 1 и SEQ ID NO 4;
2.2. одну пару специфических олигонуклеотидных праймеров для амплификации контаминирующего фрагмента генов семейства blaTEM размером 517 п.н., представленных последовательностями SEQ ID NO 1 и SEQ ID NO 3;
2.3. одну пару специфических олигонуклеотидных праймеров для амплификации контаминирующего фрагмента генов семейства blaTEM размером 290 п.н., представленных последовательностями SEQ ID NO 2 и SEQ ID NO 3;
2.4. одну пару специфических олигонуклеотидных праймеров для амплификации контаминирующего фрагмента генов семейства blaTEM размером 603 п.н., представленных последовательностями SEQ ID NO 2 и SEQ ID NO 4.
| WO 2012159060 A3, 22.11.2012 | |||
| СПОСОБ ОПРЕДЕЛЕНИЯ БАКТЕРИАЛЬНОЙ КОНТАМИНАЦИИ КРОВИ | 2002 |
|
RU2208645C1 |
Авторы
Даты
2020-07-29—Публикация
2019-08-08—Подача