Объект изобретения настоящей заявки относится к слитым белкам на основе белков тетраспанина, в которых внеклеточная петля заменена полностью или частично пептидными последовательностями другой структуры, а также к их продуцированию и применению в качестве транспортного носителя. Описанные слитые белки закреплены в мембране клеток и могут быть использованы для презентации слитых с ними чужеродных пептидов на поверхности клетки.
Структурный план клеток и их физиология кодируется генами. Гены состоят из ДНК и находятся либо в геноме, либо во внехромосомных элементах. Они экспрессируют первичные и вторичные генные продукты, такие как РНК и белки. Они могут быть дополнительно ферментативно модифицированы различными способами.
Продукты генов находятся в основном в цитозоле. Белки частично обнаруживаются также во внутриклеточных мембранах компартментов и во внешней клеточной мембране, а также, возможно, в клеточной стенке. В случае эукариотов белки и предшественники РНК обнаруживаются в ядре клеток. Белки сортируются во внутриклеточных мембранах в цитозоле и распределяются по клеточным компартментам, но также могут быть экспортированы из клетки секрецией. Экспортированные из эндоплазматического ретикулума (ER) белки могут быть дополнительно гликозилированы в аппарате Гольджи во время транспортировки.
Определенные виды белков, мембранозакрепленные белки, активно направляются на клеточную мембрану и закрепляются там вертикально по отношению к мембране. Горизонтально, однако, напротив, они гибко способны мигрировать внутри мембраны из-за текучести мембраны. Некоторые мембранные белки образуют с другими гетерологичными мембранными белками кластер. Таким образом, 80% человеческих белков действуют в ассоциированных белковых комплексах1. Некоторые виды взаимодействуют друг с другом в мембране и образуют там устойчивые структуры.
Множество сигнальных процессов, которые позволяют клетке взаимодействовать со своей окружающей средой, контролируется посредством мембранных белков. Также возможны мембранные переносы белков с помощью клеточной мембраны в связи с процессами наружной секреции.
Для представления эпитопов иммунная система использует мембранные белки МНС на клетках для того, чтобы идентифицировать потенциальные патогены, проникающие в клетку. Эти физиологические процессы в основном очень хорошо развиты в клетке, и для этой функции вовлеченные белки эволюционировали весьма специфическим образом, то есть они в основном весьма специфически нацелены на определенную функцию. Поэтому можно ожидать, что дополнительные искусственные аминокислотные последовательности будут мешать этим функциям.
Тетраспанины у млекопитающих относятся к суперсемейству белков из 33 представителей. Основной характеристикой тетраспанинов являются их 4 трансмембранных домена TM1-4, что нашло отражение в названии. Тетраспанины содержат дополнительно малую и большую внеклеточные белковые петли (малую и большую внеклеточные петли), известные также как SEL и LEL. LEL содержит высококонсервативный аминокислотный мотив CCG. Кроме того, в них содержится внутриклеточная белковая петля (малая внутриклеточная петля, SIL). N- и С-концы локализованы внутриклеточно34-36. С помощью латеральных белково-белковых взаимодействий (PPI), тетраспанины организуются в обогащенные тетраспанинами микродомены (ТЕМ)37,38.
Для транспортировки чужеродных белков и эпитопов гетерологичного строения целенаправленным образом, например, к поверхности клеток млекопитающих, например, клеток человека, и для их презентации требуются особенно подходящие белки-носители (носители). Они должны обладать достаточной электродвижущей силой и иметь возможность использовать мембранные потенциалы, чтобы гарантировать транспортировку белка с высокой эффективностью путем транслокации через мембрану к клеточной поверхности2-8. Ранее получили известность «дисплейные» системы, в основном, на основе мембранно-закрепленных белков, которые могут быть использованы в качестве белков-носителей для презентации чужеродных белков путем генетической модификации. Одной из наиболее известных белковых дисплейных систем является фаговый дисплей. В нем используется презентация белковых составляющих на поверхности нитевидных бактериофагов в бактериальной системе9-12. Также раньше гетерологичные белки были успешно презентированы в случае грамположительных и грамотрицательных бактерий на самой внешней мембране без использования фагового дисплея. Для направления белков к внешней мембране бактериальной клетки были использованы различные транспортные механизмы в бактериальной клетке13-20. Дальнейшие разработки дисплейных систем были сфокусированы на эукариотических системах, таких как дрожжи и клетки насекомых, для обеспечения в рекомбинантных белках фолдинга белков и модификаций, таких как, например, N-гликозилирование21-29. Первоначальные дисплейные системы также уже разрабатывались для применения в системе млекопитающих30-33.
Целью настоящего изобретения является предоставление новой возможности, исходя из текущего уровня техники, для закрепления любых искомых гетерологичных пептидов в плазматической мембране и презентации их на поверхности клетки. Неожиданно было обнаружено, что тетраспанины отлично подходят для применения в качестве носителей и обладают высокой эффективностью транспортировки чужеродных пептидов, слитых с ними, для закрепления и презентации на поверхности клеток. Они способны эффективно направлять чужеродные пептиды к плазматической мембране клеток млекопитающих через эндоплазматический ретикулум и аппарат Гольджи.
В диком типе белки семейства тетраспанинов имеют белковую петлю (большая внеклеточная петля, LEL) между трансмембранными доменами TM3 и TM4. В рамках настоящего изобретения неожиданно было обнаружено, что эта внеклеточная петля может быть полностью или частично заменена пептидными последовательностями другой структуры, что, таким образом, позволяет посредством белка-носителя тетраспанина транспортировать вставленный гетерологичный пептид к мембране и закреплять его в ней.
Изобретение основано на простоте однородной молекулярной структуры для гибкой и вариабельной модуляции последовательности в модульной системе экспрессии белка в сочетании с функцией специфического переносчика к клеточной мембране и его закрепления в ней.
Предоставляемые векторные системы позволяют гибко заменять или добавлять белковые компоненты с помощью простых стратегий клонирования (например, добавлять отдельные эпитопы, а также создавать целые библиотеки белков).
После экспрессии конструкций слияния на основе тетраспанина по изобретению (интегрированных временно или стабильно в геном) локализация слитых белков в мембране является почти количественной. Тетраспаниновые белки очень хорошо транслоцируются в мембрану. Уже известные системы такого рода (например, рецептор EGFR) и близко не приближаются к достижению этого показателя.
Экспрессия слитых белков тетраспанина по изобретению происходит относительно быстро, обычно в течение ночи, тогда как экспрессия других транспортных систем обычно происходит в течение двух дней.
Кроме того, стабильность химерных конструкций и их долговечность выше по сравнению с известными белками с семью трансмембранными доменами, поскольку накопление продуктов цитозольной деградации не обнаружено (известно, что рекомбинантные GPCR-рецепторы нестабильны).
С помощью системы по изобретению, которая имеет только четыре трансмембранных домена, можно стабильно и без каких-либо проблем транспортировать на клеточные поверхности один или несколько белков, фиксировать и иммобилизовать их там, например, в качестве стационарного партнера в экспериментах по взаимодействию с малыми молекулами, другими белками, такими как антитела, а также другими белками (например, каталитическими ферментами), ДНК, РНК и сахарами и т.д.
Вещества, взаимодействующие со слитыми белками на мембране, могут теперь гибко входить из окружающего раствора в специфические реакции с белками и гликозильными остатками на мембране.
Предпосылкой для этих реакций является функциональная конформация белков. Она полностью гарантирована в отношении слитых белков по изобретению.
Таким образом, первым объектом настоящего изобретения является слитый белок, включающий первый домен (i), второй домен (ii) и третий домен (iii), где второй домен расположен между первым и третьим доменами, где
(i) частичная последовательность тетраспанина, которая включает трансмембранный домен 1 (TM1), малую внеклеточную петлю (SEL), трансмембранный домен 2 (TM2), малую внутриклеточную петлю (SIL) и трансмембранный домен 3 (TM3) или включает последовательность, гомологичную ей, с идентичностью последовательности, по меньшей мере, 70% по всей длине,
(ii) включает пептид с заданной аминокислотной последовательностью, имеющий идентичность последовательности менее 70% по всей длине в отношении большой внеклеточной петли (LEL) тетраспанина, и
(iii) включает частичную последовательность тетраспанина, содержащую трансмембранный домен 4 (TM4) или последовательность, гомологичную ему, с идентичностью последовательности, по меньшей мере, 70% по всей длине.
В настоящем изобретении было обнаружено, что последовательности TM1-3 и TM4 отвечают за закрепление слитого белка на мембране. Следовательно, слитый белок по изобретению содержит, во-первых первый домен (i), содержащий частичную последовательность тетраспанина с трансмембранными доменами TM1-TM3 и, во-вторых, домен (iii), содержащий частичную последовательность тетраспанина с трансмембранным доменом TM4, или последовательность, гомологичную ей.
По смыслу изобретения термин «тетраспанин» описывает любые искомые белки семейства тетраспанинов, в частности, 33 тетраспанина человека. Тетраспанины могут вступать в специфические взаимодействия друг с другом в мембране. Такое поведение благоприятно для транспортировки через мембрану и закрепления в ней. Этот эффект может быть использован в слитых белках по изобретению, чтобы также эффективно переносить чужеродный пептид домена (ii) к мембране и закреплять его в ней. Предпочтительными примерами тетраспанинов по смыслу изобретения являются CD63, CD9, CD82, CD81, CD151 и CD53 (см. учетные записи для последовательностей ДНК и белков). Особенно предпочтительно, если частичные последовательности, содержащиеся в слитом белке по изобретению, происходят из тетраспанина CD63 (см. учетные записи для последовательностей ДНК и белков).
В качестве основного элемента можно выбрать частичную последовательность первого тетраспанина для домена (i) и частичную последовательность другого тетраспанина для домена (iii). Однако предпочтительно, чтобы все частичные последовательности были получены из одного и того же тетраспанина, например CD63, CD9, CD82, CD81, CD151 или CD53, предпочтительно CD63 (см. учетные записи для последовательностей ДНК и белков).
Область (i) предпочтительно содержит трансмембранный домен TM1, малую внеклеточную петлю (SEL), трансмембранный домен TM2, малую внутриклеточную петлю (SIL) и трансмембранный домен TM3 тетраспанина, где различные трансмембранные домены и петли следуют друг за другом непосредственно, без каких-либо дополнительных сегментов гетерологичной последовательности, вставленных между ними. Особенно предпочтительно, чтобы частичная последовательность соответствовала тетраспанину дикого типа без каких-либо отклонений в виде, например, замещений, вставок или делеций одной или нескольких аминокислот. Однако принципиально можно использовать модифицированные частичные последовательности тетраспанина для домена (i), если они имеют идентичность последовательности, по меньшей мере, 70% по всей длине последовательности относительно тетраспанина дикого типа. Более предпочтительно идентичность последовательности составляет, по меньшей мере, 80%, 85%, 90%, 95% или 99% по всей длине частичной последовательности тетраспинана, содержащейся в домене (i).
В особенно предпочтительном воплощении изобретения первый домен (i) состоит из вышеупомянутой частичной последовательности тетраспанина или гомологичной ему последовательности с идентичностью последовательности, по меньшей мере, 70% (более предпочтительно, по меньшей мере, 80%, 85%, 90 %, 95% или 99%) по всей длине.
Область (iii) содержит частичную последовательность тетраспанина, содержащую трансмембранный домен TM4 тетраспанина, или гомологичную ей последовательность с идентичностью последовательности, по меньшей мере, 70% по всей длине. Более предпочтительно идентичность последовательности с частичной последовательностью тетраспанина дикого типа составляет, по меньшей мере, 80%, предпочтительно, по меньшей мере, 85%, 90%, 95% или 99% по всей длине. В особенно предпочтительном воплощении домен (iii) состоит из вышеупомянутой частичной последовательности тетраспанина или последовательности, гомологичной ей.
Область (ii) содержит пептид с заданной аминокислотной последовательностью. В данном документе его также называют «чужеродным пептидом» или «чужеродным белком». Аминокислотная последовательность чужеродного пептида отличается от последовательности большой внеклеточной петли (LEL) тетраспанина дикого типа, в частности доменов (i) и (iii) тетраспанина(ов). По сравнению с LEL тетраспанина дикого типа идентичность последовательности по всей длине составляет менее 70%, предпочтительно менее 50%, более предпочтительно менее 40% или менее 30%.
Аминокислотную последовательность в домене (ii) можно в принципе выбирать произвольно. Независимо от последовательности чужеродного пептида или домена (ii), слитый белок по изобретению может быть закреплен и презентирован на поверхности клеток, в частности, клеток млекопитающих (например, HEK293T-Homo sapiens).
По смыслу настоящего изобретения «пептид» обозначает комбинацию, по меньшей мере, двух аминокислот, которые связаны между собой пептидными связями. Длина пептида в основном неограничена. Включены все олигопептиды длиной вплоть до 10 аминокислот, полипептиды с длиной более чем 10 аминокислот и макропептиды с длиной более чем 100 аминокислот. В предпочтительном воплощении размер пептида составляет менее 100 кДа, предпочтительно менее 50 кДа или менее 30 кДа, например 10-30 кДа.
По смыслу настоящего изобретения термин «аминокислота» означает предпочтительно двадцать аминокислот, обнаруженных во встречающихся в природе пептидах, полипептидах и белках, которые в каждом случае являются L-изомерами. Однако по изобретению этот термин также включает аналоги аминокислот и D-изомеры аминокислот и их аналоги.
По смыслу настоящего изобретения термины «пептид», «полипептид» и «белок» используются взаимозаменяемо. В предпочтительном воплощении пептид представляет собой белок, по меньшей мере, с 50 аминокислотами. Для этого предпочтительно, чтобы белок свернулся заданным образом, что требуется, например, для искомой функции белка.
В домене (ii) пептид с заданной аминокислотной последовательностью может включать, например, антиген или эпитоп антигена. В данном документе «эпитоп» обозначает область антигена, с которой специфически связывается антитело или Т-клеточный рецептор. Один антиген может включать один или несколько эпитопов. В одном воплощении изобретения пептид с заданной аминокислотной последовательностью включает множественные эпитопы, которые соединены вместе с помощью линкеров. Например, они могут быть множественными эпитопами антигена. Примерами эпитопов являются эпитопы злокачественных опухолей, эпитопы HIV (например, gp41 из HIV-1). Примерами этих соединений являются V5-6xHis (SEQ ID NO: 35, 36) или 2F5-4E10 (SEQ ID NO: 37, 38).
В следующем воплощении изобретения пептид с заданной аминокислотной последовательностью домена (ii) включает белок, имеющий искомую функцию, например фермент, такой как, например, протеаза.
В дополнение к пептиду с заданной аминокислотной последовательностью второй домен (ii) может содержать одну или несколько частичных последовательностей LEL тетраспанина или гомологичных им последовательностей с идентичностью последовательности, по меньшей мере, 70% по всей длине. Предпочтительно идентичность последовательности составляет, по меньшей мере, 80%, по меньшей мере, 85%, по меньшей мере, 90%, по меньшей мере, 95% или, по меньшей мере, 99%. Частичные последовательности LEL тетраспанина могут быть расположены на C- и/или N-конце пептида с заданной аминокислотной последовательностью. Предпочтительно частичные последовательности LEL в домене (ii) происходят из того же тетраспанина, что и сегменты последовательностей доменов (i) и (iii).
«Гомологичный», при использовании в данном документе, описывает сходство последовательностей между двумя полипептидными молекулами или между двумя нуклеиновыми кислотами. Если положение в обеих двух сравниваемых последовательностях занято одной и той же мономерной субъединицей основания или аминокислоты, то соответствующие молекулы в этом положении являются гомологичными. Процентная гомология между двумя последовательностями - это функция количества совпадений или гомологичных положений в двух последовательностях, разделенная на количество положений и умноженное на сто. Например, если 6 из 10 положений в двух последовательностях совпадают или гомологичны, то две последовательности гомологичны на 60%. Обычно проводится сравнение, когда две последовательности выровнены так, что получается наибольшая возможная гомология. Такое выравнивание может быть обеспечено с использованием, например, способа Niedelman et al., J. Mol. Biol. 48: 443-453, 1970, удобно реализованного в компьютерных программах.
Гомологичные последовательности имеют идентичные или сходные аминокислотные остатки, где сходные остатки являются консервативными заменами или допускаемыми точечными мутациями соответствующих аминокислотных остатков в выровненной контрольной последовательности. В этой связи «консервативная замена» остатка в эталонной последовательности представляет собой замены, которые физически или функционально схожи с соответствующими эталонными остатками, например, которые имеют одинаковый размер, форму, электрический заряд, химические свойства, включая способность к образованию ковалентных или водородных связей или т.п. Особенно предпочтительными консервативными заменами являются те, которые выполняют критерии, определенные для принятой точечной мутации в Dayhoff et al., “Atlas of Protein Sequence and Structure”, 5: Suppl. 3, chap. 22: 354-352, Nat. Biomed. Res. Foundation, Washington, D.C., 1978.
Домены слитого белка по изобретению расположены в каждом случае в порядке (i) - (ii) - (iii). В данном документе термин «слияние» относится к коллинеарному объединению доменов посредством их индивидуальных пептидных остовов.
Домены (i), (ii) и (iii) могут быть связаны необязательно напрямую или с помощью гибких линкеров. В первом воплощении домен (ii) непосредственно присоединяется к домену (i). Альтернативно или дополнительно домен (iii) непосредственно присоединяется к домену (ii).
В следующем варианте гибкие линкеры расположены между первым и вторым доменами и/или между вторым и третьим доменами. Существующие линкеры могут быть одинаковыми или разными. Типичные линкеры для применения в слитых белках по изобретению включают, например, глициновые полимеры (G)n, где n представляет собой целое число, по меньшей мере, 1, глицин-аланиновые полимеры и другие гибкие линкеры, которые известны специалисту в данной области. Предпочтительными являются линкеры без серина и аргинина; линкеры без серина и аргинина являются особенно предпочтительными. Особенно предпочтительным является линкер LQEFDIGGGG (соответствующий SEQ ID NO: 41, 42). Дополнительные линкеры описаны на фиг. 2C (см. также SEQ ID NO: 39, 40).
Область (ii) может дополнительно содержать одну или несколько последовательностей распознавания, которые могут быть расщеплены ферментом. Примерами являются сайты распознавания специфических протеаз, нуклеаз или эндогликозидаз. В ином случае сайт распознавания может содержать сайт распознавания субстрата для конкретного гидролазного фермента, например фосфатазы, гликозидазы, амидазы или эстеразы. Термин «последовательность распознавания протеазы» в данном документе кратко используется для любых последовательностей распознавания, которые могут быть расщеплены ферментативно. Они могут быть расположены на C- и/или N-конце пептида с заданной аминокислотной последовательностью. В особенно предпочтительном воплощении последовательности распознавания протеазы прямо фланкируют пептид с заданной аминокислотной последовательностью. В качестве основного элемента для этого пригодны любые искомые последовательности распознавания протеазы. Если, кроме пептида с заданной аминокислотной последовательностью, второй домен (ii) также содержит одну или несколько частичных последовательностей LEL из тетраспанина, в каждом случае предпочтительно вводить последовательности распознавания протеазы между частичной последовательностью LEL и С- или N-концом пептида с заданной аминокислотной последовательностью.
В следующем воплощении слитый белок по изобретению может дополнительно содержать четвертый домен (iv) на N-конце и/или пятый домен (v) на С-конце слитого белка. Домены (iv) и (v) могут в каждом случае быть связаны непосредственно или посредством гибких линкеров, как определено выше. Например, домен (iv) может быть связан непосредственно или посредством гибкого линкера на N-конце домена (i). Область (v) может быть связана непосредственно или посредством гибкого линкера на C-конце домена (iii). Кроме того, можно расположить последовательности распознавания протеазой, как определено выше, между доменами (iv) и (i) и/или между доменами (iii) и (v) и, в случае наличия, между линкером и одним или несколькими из числа доменов (i), (iii), (iv) и (v).
Что касается закрепления тетраспанина в плазматической мембране клетки, то С- и N-концы тетраспанина лежат на стороне цитоплазмы. То же самое относится к мембранному закреплению слитого белка по изобретению.
Четвертый и пятый домен (iv) и (v) слитого белка по изобретению могут быть выбраны, например, таким образом, чтобы они содержали маркерную группу или то, что известно как метка. В данном документе «метка» обозначает короткую пептидную последовательность, например, белковую метку для маркировки и идентификации, например, FLASH-метку или флуоресцентный репортерный белок mCherry.
Было обнаружено, что несмотря на то, что слитый белок по изобретению отличается от тетраспанина дикого типа, по меньшей мере, из-за домена (ii), тем не менее, он сворачивается правильно, аналогично тетраспанину дикого типа. Это было продемонстрировано на примере флуоресцентных белков CFP и YFP в домене (ii) (см. фиг. 3B). Эпитопы между трансмембранным доменом TM3 и TM4 с внеклеточной ориентацией могут быть обнаружены на поверхности клетки с помощью сканирующей электронной микроскопии (фиг. 4). Введение эпитопов между ТМ3 и ТМ4 тетраспанина и перенос на поверхность клетки с внеклеточной ориентацией и эпитопов на N- и С-концах продемонстрировано на примере CD63 с помощью сканирующей электронной микроскопии (фиг. 5А-5D). Успешную экспрессию флуоресцентных белков подтверждали дополнительно с помощью вестерн-блоттинга. В данном документе специфическая полоса может быть обнаружена для соответствующих слитых белков с помощью эпитопа FLAG и mCherry (фиг. 6). Аналогично показана локализация для других тетраспанинов (CD9, CD81, CD82 и CD151) на поверхности клетки (фиг. 8). Кроме того, было показано, что использование низкосериновых и низкоаргининовых линкеров повышает эффективность локализации на клеточной поверхности (фиг. 10).
Еще одним объектом настоящего изобретения является молекула нуклеиновой кислоты, которая кодирует слитый белок по изобретению. Молекула нуклеиновой кислоты по изобретению содержит сегмент кодирующей последовательности для вышеупомянутого слитого белка или включает ее. Этот сегмент последовательности описан в данном документе также как «слитый ген». Особенно предпочтительно, если молекула нуклеиновой кислоты по изобретению присутствует в выделенной форме.
«Молекула нуклеиновой кислоты» по смыслу изобретения относится к последовательности природных оснований нуклеиновых кислот, аналогов оснований, производных оснований или их комбинированных форм. Этот термин включает, в частности, ДНК и РНК, а также нуклеиновые кислоты, полученные из них, такие как, например, кДНК и мРНК. В значении изобретения нуклеиновые кислоты также включают нуклеиновые кислоты, такие как PNA (пептидная нуклеиновая кислота), LNA (блокированная нуклеиновая кислота), PSNA (фосфотиоатная нуклеиновая кислота). Эти аналоги нуклеиновых кислот могут, в принципе, иметь природные основания нуклеиновой кислоты, которые, однако, связаны друг с другом по-другому, например, в ДНК или РНК. К нуклеиновым кислотам относятся также нуклеиновые кислоты, имеющие производные нуклеиновых кислот, такие как, например, гипоксантины, 2,5-диаминопорин и/или метилцитозин, и модифицированные, конкретнее меченные нуклеиновые кислоты. Помимо производных оснований эти производные нуклеиновой кислоты могут, в принципе, также иметь природные основания нуклеиновых кислот. Метки можно вводить в основания и/или каркас. Связи могут соответствовать ДНК или РНК или аналогам нуклеиновой кислоты. Нуклеиновые кислоты могут быть двухцепочечными или одноцепочечными, линейными, разветвленными или кольцевыми.
Настоящее изобретение относится также к вектору, содержащему молекулу нуклеиновой кислоты, как описано выше. Предпочтительно молекула нуклеиновой кислоты функционально связана с последовательностью контроля экспрессии.
Термин «последовательность контроля экспрессии» по смыслу изобретения содержит как элементы, которые регулируют экспрессию на уровне транскрипции, например, энхансеры или сайленсеры, так и элементы, которые регулируют экспрессию на посттранскрипционном уровне (например, сплайсинг, ядерный экспорт или стабильность мРНК). Термин включает также несколько элементов, которые регулируют экспрессию на уровне транскрипции и посттранскрипции, а также комбинации элементов регуляции транскрипции и посттранскрипции. Способы и средства для идентификации последовательностей контроля экспрессии, которые приводят к увеличению экспрессии и к выделению, являются рутинными методами, которые, вероятно, хорошо известны специалисту в данной области техники. Последовательность контроля экспрессии может более конкретно представлять собой промотор, предпочтительно эукариотический промотор, такой как, например, промотор CMV или SV40; прокариотический промотор или фагоспецифический промотор, такой как, например, промотор SP6 или T7. Особенно предпочтительно, если промотор представляет собой промотор CMV. Вектор может дополнительно содержать сегмент последовательности для терминации и/или полиаденилирования молекулы нуклеиновой кислоты.
Вектор по изобретению дополнительно содержит предпочтительно ген селективного маркера, то есть сегмент последовательности, который кодирует селективный маркер. Примерами селективных маркеров являются антибиотикорезистентность или маркеры ауксотрофии. Предпочтительно используют амплифицируемый ген селективного маркера, такой как, например, дигидрофолатредуктаза. Необязательно, вектор по изобретению может дополнительно содержать точку начала репликации.
По смыслу настоящей заявки термин «функционально связанный» описывает связь двух или более последовательностей нуклеиновой кислоты или частичных последовательностей, которые расположены так, что они могут выполнять свою предполагаемую функцию. Например, промотор/энхансер функционально связан с кодирующей последовательностью гена, если он может контролировать или модулировать транскрипцию связанной последовательности гена в цис-положении. Как правило, хотя необязательно, функционально связанные последовательности расположены в непосредственной близости и, если две кодирующие последовательности генов связаны или в случае секреции сигнальной последовательности, в той же рамке считывания. Хотя функционально связанный промотор обычно расположен выше последовательности кодирующего гена, он не обязательно должен находиться в непосредственной близости. Энхансеры также не должны присутствовать в непосредственной близости, если они облегчают транскрипцию последовательности генов. Для этой цели они могут присутствовать как выше, так и ниже последовательности гена, возможно, также на некотором расстоянии. Сайт полиаденилирования функционально связан с последовательностью гена, если он расположен на 3'-конце последовательности гена, так что транскрипция идет по кодирующей последовательности до сигнала полиаденилирования. Связь может осуществляться в соответствии с обычными рекомбинантными способами, например, с помощью метода ПЦР, путем лигирования на подходящих сайтах рестрикции или путем сплайсинга. Если подходящие сайты рестрикции отсутствуют, синтетические олигонуклеотидные линкеры или адаптеры могут быть использованы известным образом. Предпочтительно функциональная связь не происходит посредством интронных последовательностей.
Вектор по изобретению предпочтительно подходит, по меньшей мере, для одной (хозяйской) клетки, более конкретно для репликации вектора в этой клетке и/или для экспрессии белка или пептида в этой клетке. Репликация может происходить в прокариотической и/или эукариотической клетке. Экспрессия происходит предпочтительно в эукариотической клетке. Вектор может быть плазмидой.
Векторные конструкции для эукариотической экспрессии в соответствии с предшествующим уровнем техники имеют, как правило, сильный эукариотический промотор, который, насколько это возможно, не специфичен для клеточного типа (например, промотор CMV или SV40) и часть последовательности, ответственной за терминацию транскрипции и полиаденилирование (например, терминатор SV40-полиA). Между этими двумя элементами находится CDS (кодирующая последовательность), которая должна быть экспрессирована, имеющая в начале более или менее четко определенную последовательность Козака неподалеку от стартового кодона. В случае векторной конструкции по изобретению, CDS, которая должна быть экспрессирована, представляет собой ген из суперсемейства тетраспанинов. В данном документе все тетраспаниновые гены всех видов организмов, имеющих гены этого суперсемейства, которые обеспечивают искомую способность транспорта к мембране, соответствуют изобретению. Однако слитые гены по изобретению характеризуются тем, что обладают, по меньшей мере, одной гетерологичной последовательностью ДНК, искусственно введенной внутренним, и, возможно, концевым слиянием. Слияния с концами CDS представляют собой гетерологичные последовательности ДНК, которые кодируются 5'-проксимально и/или 3'-дистально относительно гена тетраспанина для получения слитого белка или пептида. Между третьим (TM3) и четвертым (TM4) трансмембранными доменами по изобретению вводятся внутренние кодирующие гетерологичные последовательности ДНК. Трансмембранные домены тетраспанинов ответственны, среди прочего, за перенос гетерологичных кодирующих последовательностей на мембрану, транслокацию через мембрану и закрепление в ней.
Среднестатистический специалист в данной области, прошедший подготовку по молекулярной биологии, сможет создавать функциональные слитые ДНК-конструкции, возможно, с помощью соответствующей литературы39.
В частности, в данном документе следует особенно выделить более усовершенствованные векторные конструкции по изобретению. Вместо общих эукариотических промоторов они также обладают бактериальными и фагоспецифическими промоторами, а также эукариотическими клеточно-специфическими и органоспецифическими промоторами, которые позволяют осуществить экспрессию, которая зависит от клеточной дифференцировки. Такие промоторные типы могут быть использованы для применения генных конструкций по изобретению независимо от задачи. Другие варианты вектора по изобретению могут также иметь, например, промоторы фаговой РНК-полимеразы для продуцирования молекул мРНК крайне эффективным образом in vitro или, возможно, in vivo. Примерами являются фаговые промоторы, такие как SP6- или T7-специфические промоторы для получения молекул мРНК in vitro, так что мРНК слитого гена может быть получена для терапевтических целей и применений. Этот вариант приветствуется в качестве фармацевтической формы регуляторов, поскольку он должен предотвращать рекомбинацию нуклеиновых кислот в геноме.
Экспрессия производных гена тетраспанина в бактериях или дрожжах может быть применена для выделения слитых белков и использована в искусственных мембранах. Однако экспрессию, специфичную для эукариотической дифференцировки, и клеточноспецифичную экспрессию, например, в слизистой оболочке или мышечных клетках, можно также использовать для специфической экспрессии ДНК-вакцин (см., например, EP1390490 B1).
В процедурах получения разрешения для терапевтических агентов были установлены правила, которые делают такие высокоспецифичные свойства экспрессии обязательным требованием.
Кроме того, векторные системы по изобретению обладают определенным дизайном, который позволяет осуществить экспрессию множественных слитых белков в одной и той же клетке с помощью одной векторной конструкции (см. патентную заявку DE 10 2013 006 487 A9). При таком дизайне дополнительные элементы могут быть добавлены упрощенным образом.
Объектом изобретения также являются нуклеиновые кислоты и векторы, имеющие сегмент, кодирующий слитый белок, как определено выше, где, однако, вместо кодирующей последовательности для пептида с заданной аминокислотной последовательностью, присутствует только сайт множественного клонирования, который позволяет клонировать ген, кодирующий любой искомый пептид с заданной аминокислотной последовательностью, с помощью последовательностей распознавания рестрикционными эндонуклеазами. Кроме того, множественные сайты клонирования могут присутствовать в положениях 5'- и/или 3' для N- и/или С-концевых доменов (iv) и (v).
Таким образом, один из аспектов изобретения относится к молекуле нуклеиновой кислоты, содержащей первый сегмент последовательности (i), второй сегмент последовательности (ii) и третий сегмент последовательности (iii), где второй сегмент последовательности расположен между первым и третьим сегментами последовательности, где
(i) частичная последовательность тетраспанина, которая включает трансмембранный домен 1 (TM1), малую внеклеточную петлю (SEL), трансмембранный домен 2 (TM2), малую внутриклеточную петлю (SIL) и трансмембранный домен 3 (TM3), или кодирует гомологичную ей последовательность, с идентичностью последовательности, по меньшей мере, 70% по всей длине,
(ii) включает сайт множественного клонирования и
(iii) кодирует частичную последовательность тетраспанина, содержащую трансмембранный домен 4 (TM4), или последовательность, гомологичную ей, с идентичностью последовательности, по меньшей мере, 70% по всей длине.
В данном документе сегменты последовательности (i) - (iii) предпочтительно расположены от 5' до 3'. «Множественный сайт клонирования» содержит несколько сайтов рестрикции, расположенных один за другим, для рестрикционных эндонуклеаз. Сайт множественного клонирования более подходит для интеграции молекулы нуклеиновой кислоты, где интегрированная молекула нуклеиновой кислоты в рамке считывания кодирует первый сегмент (i) и третий сегмент последовательности (iii) для пептида, который должен быть презентирован. Пептид предпочтительно содержит заданную аминокислотную последовательность, имеющую идентичность последовательности менее 70% по всей длине относительно большой внеклеточной петли (LEL) тетраспанина.
Настоящее изобретение относится также к вектору, содержащему вышеупомянутую молекулу нуклеиновой кислоты, предпочтительно функционально связанную с последовательностью контроля экспрессии.
В предшествующем уровне техники многочисленные распознающие последовательности известны для самых разных рестрикционных эндонуклеаз, а также рестрикционных эндонуклеаз, имеющих значение в данном документе. Предпочтительно используют последовательности, которые состоят, по меньшей мере, из шести нуклеотидов в качестве распознающей последовательности. Список подходящих идентификационных последовательностей можно найти, например, в Sambrook et al. (1989).
Специфические элементы последовательности вектора по изобретению с сайтом множественного клонирования касаются введения гетерологичных последовательностей ДНК в соответствующие концевые и внутренние функционально приемлемые положения генов тетраспанинов (таких как, например, CD63). Таким образом, снова и снова можно вставлять новый эпитоп CDS в «уникальные», то есть индивидуальные, сайты рестрикции или различные гены и фрагменты гена аддитивно в циклической реакции в существующие векторные конструкции. Пример представлен на фиг. 2А, SEQ ID NO: 1 вектора pAE211 по изобретению. Сайты рестрикции расположены между трансмембранными доменами TM3 и TM4. Для этого положения было экспериментально показано, что оно особенно подходит для слияния с гетерологичными последовательностями ДНК, которые кодируют гетерологичный пептид, поскольку это не мешает функции мембранного транспорта. В этом сайте находится белковая петля, которая выдается над мембраной, последовательность которой может быть вариабельной, существенно при этом не мешая транспорту в плазматическую мембрану белка тетраспанина в клеточных культурах.
5'-концевое слияние с CDS для получения N-концевого слитого белка может быть проведено с помощью двух NcoI в 5'-области гена CD63, при этом в начальной области восстанавливается последовательность Козака. Это уникально.
Возможный элемент слияния CDS на 3'-конце, который приводит к синтезу С-концевого слитого белка, вставляется с помощью рестрикционной эндонуклеазы типа IIS Bsal, которая образует специфические концы для NdeI и BclI в конструкции. Эти сайты также могут быть использованы для вставки элементов генного слияния по 3'-концу.
Вышеупомянутые реализации векторных систем являются примерами, и любой другой способ или технология для модификации векторов, помимо описанного способа рестрикции-лигирования, в равной степени соответствует изобретению, например, модуляция последовательности систем с помощью подходящих систем рекомбинации, in vivo, а также in vitro.
Еще одним объектом настоящего изобретения является клетка, включающая молекулу нуклеиновой кислоты по изобретению или вектор по изобретению.
В воплощении изобретения молекула нуклеиновой кислоты или вектор, содержащий последовательность, кодирующую слитый белок и подходящие функции генетического контроля, интегрированы стабильно в геном или присутствуют внехромосомно в клетках, и гибридный белок по изобретению экспрессируется конститутивно или индуцибельно или репрессируемо.
Клетка по смыслу настоящего изобретения предпочтительно представляет собой эукариотическую клетку, предпочтительно клетку млекопитающего, такую как, в частности, клетку человека. Другими примерами подходящих клеток млекопитающих являются клеточные линии грызунов, таких как клетки мыши, крысы и хомяка или клетки обезьяны. Молекулу нуклеиновой кислоты по изобретению или вектор по изобретению можно вводить в клетку, в частности эукариотическую клетку, с помощью известных способов. Успешная трансфекция приводит к трансформированным, генетически модифицированным, рекомбинантным или трансгенным клеткам.
Трансфекция эукариотических клеток-хозяев молекулой нуклеиновой кислоты по изобретению или вектором по изобретению происходит в соответствии с обычными способами (Sambrook et al., 1989; Ausubel et al., 1994). Подходящими способами трансфекции являются, например, липосомная трансфекция, совместное осаждение фосфатом кальция, электропорация, поликатионы, (например, DEAE-декстран)-опосредованная трансфекция, слияние протопластов, микроинъекция и вирусные инфекции. Согласно изобретению клетки предпочтительно получают посредством стабильной трансфекции, где конструкции либо интегрируются в геном клетки-хозяина или в искусственную хромосому/минихромосому, либо содержатся в стабильной форме в клетке-хозяине. Для этого предпочтительным является способ трансфекции, который обеспечивает оптимальную частоту трансфекции и экспрессию гетерологичного гена в соответствующей клетке-хозяине. По определению каждая последовательность или каждый ген, который вводится в клетку-хозяина, описывается как «гетерологичная последовательность» или «гетерологичный ген» по отношению к клетке-хозяину, даже если введенная последовательность или вводимый ген идентичны эндогенной последовательности или эндогенному гену клетки-хозяина.
В воплощении по изобретению клетки могут быть клетками объединений трансгенных клеток различных типов клеток, в которых слитый ген по изобретению также экспрессируется в разных состояниях клеточной дифференцировки. Эти клетки могут быть получены ex vivo и, возможно, будут использоваться после этого терапевтически in vivo.
В следующем воплощении изобретения клетки представляют собой клетки клеточных объединений трансгенных организмов, клетки которых прошли через зародышевую линию и развились в многоклеточные системы, предпочтительно в живые организмы.
В предпочтительном воплощении клетка по изобретению дополнительно трансформируется или трансфицируется, по меньшей мере, одной дополнительной молекулой нуклеиновой кислоты или, по меньшей мере, еще одним вектором экспрессии. Другая молекула нуклеиновой кислоты или дополнительный экспрессирующий вектор предпочтительно содержит сегмент, который кодирует еще один заданный белок. Другая молекула нуклеиновой кислоты или экспрессирующий вектор может, в соответствии с одним воплощением, также быть молекулой нуклеиновой кислоты по изобретению или вектором экспрессии по изобретению, где для домена (ii) в этом случае был выбран другой пептид с другой заданной аминокислотной последовательностью. В другом воплощении дополнительная молекула нуклеиновой кислоты или дополнительный экспрессирующий вектор содержат сегмент, который кодирует фермент, например, протеазу. Если в такой клетке экспрессируется молекула нуклеиновой кислоты по изобретению и другая молекула нуклеиновой кислоты, то на поверхности мембраны будут представлены как пептид домена (ii) с заданной аминокислотной последовательностью, так и дополнительный белок. Если, например, пептид домена (ii) фланкирован последовательностями распознавания протеазы, протеаза, экспрессируемая посредством дополнительной молекулы нуклеиновой кислоты, может разрезать по этим сайтам распознавания и, таким образом, высвободить пептид.
Согласно изобретению клетки-хозяева предпочтительно разрабатывают, адаптируют и культивируют в условиях без сыворотки, возможно, в средах, свободных от белков/пептидов животных.
Другим объектом настоящего изобретения является мембранный препарат, который был получен из клетки по изобретению.
Мембранный препарат по смыслу изобретения содержит, по меньшей мере, выделенную мембрану, более предпочтительно выделенную клеточную мембрану, еще более предпочтительно выделенную клеточную плазматическую мембрану и/или выделенную клеточную мембрану других клеточных органелл. Термин «мембрана» по смыслу настоящего изобретения описывает автономную систему, содержащую, по меньшей мере, один липид и необязательно, по меньшей мере, один белок. Липиды мембраны обычно образуют липидный бислой. Мембранный препарат содержит предпочтительно мембранные частицы, более предпочтительно мембранные везикулы.
Мембранный препарат может быть получен из клетки по изобретению с помощью способов, известных специалисту в данной области, предпочтительно посредством лизиса клетки (клеточного лизата), более предпочтительно посредством лизиса клетки и последующего выделения и, возможно, очистки клеточной мембраны (выделенной и необязательно очищенной мембраны). Необязательные поверхностно-активные вещества, такие как, например, Triton X-100, могут быть использованы для солюбилизации мембран. Способ может дополнительно содержать стадию удаления клеточного дебриса. Диаметр мембранных частиц может составлять от 1 до 1000 нм, предпочтительно от 50 до 500 нм, более предпочтительно от 75 до 200 нм. По меньшей мере, 80%, по меньшей мере, 90%, по меньшей мере, 95% или, по меньшей мере, 98% мембранных частиц имеют диаметр в одном из диапазонов, описанных в данном документе.
Мембранный препарат по изобретению получают из клетки по изобретению, содержащей молекулу нуклеиновой кислоты по изобретению или вектор по изобретению. В клетке слитый белок по изобретению экспрессируется с помощью молекулы нуклеиновой кислоты или вектора. Таким образом, мембранный препарат по изобретению содержит предпочтительно, по меньшей мере, выделенную мембрану и, по меньшей мере, слитый белок по изобретению. В данном документе слитый белок по изобретению предпочтительно закрепляется на мембране, более предпочтительно на поверхности мембраны, более конкретно на внеклеточной стороне мембраны. Слитый белок по изобретению представлен на мембране, предпочтительно на поверхности мембраны, более конкретно на внеклеточной стороне мембраны. Мембранный препарат может необязательно дополнительно содержать один или несколько тетраспанинов дикого типа.
В одном воплощении мембранные системы по изобретению представляют собой наружную мембрану клеток, например, клеточных культур in vitro, в которых транзиторно экспрессируется, по меньшей мере, слитый белок по изобретению.
Настоящее изобретение относится также к синтетической мембранной системе, содержащей мембрану и слитый белок, молекулу нуклеиновой кислоты или вектор, как описано выше.
Мембрана может быть клеточной (естественной) или синтетической (искусственной) мембраной. Клеточная мембрана представляет собой, в частности, клеточную плазматическую мембрану, которая обычно располагается на поверхности (внешнем слое) клетки. Клеточная мембрана по смыслу настоящего изобретения может быть частью клетки или выделена из клетки, т.е. быть выделенной клеточной мембраной. Клеточная мембрана обычно асимметрична и имеет сторону, обращенную к цитоплазме (с плазматической или внутриклеточной стороной, с внутренней стороны) и стороной, обращенной от цитоплазмы (внеплазматическая или внеклеточная сторона, внешняя сторона).
Синтетическая мембрана может содержать синтетические и/или выделенные клеточные мембраны. Более конкретно липидная и/или белковая композиция синтетических мембран может отличаться от природных композиций клеточных мембран. Синтетическая мембрана предпочтительно не является частью клетки и присутствует более конкретно в виде выделенной синтетической мембраны. Специалист в данной области может влиять на свойства синтетической мембраны путем выбора липидов и/или белков.
В одном воплощении синтетическая мембранная система, содержащая молекулу нуклеиновой кислоты по изобретению или вектор по изобретению, дополнительно содержит систему для экспрессии молекулы нуклеиновой кислоты или вектора, посредством которой может быть экспрессирован слитый белок. Системы для экспрессии белков известны в данной области и предпочтительно не содержат клеток. Система для экспрессии предпочтительно содержит систему для трансляции молекулы нуклеиновой кислоты, в частности, РНК или мРНК; и, необязательно, систему транскрипции молекулы нуклеиновой кислоты или вектора, более конкретно для получения РНК или мРНК.
В альтернативном воплощении слитый белок может быть добавлен непосредственно к мембране для получения мембранной системы по изобретению.
В случае мембранной системы экспрессированный или добавленный слитый белок закрепляется в мембране, предпочтительно на поверхности мембраны, более конкретно на внешней стороне мембраны. Слитый белок по изобретению представлен на мембране, предпочтительно на поверхности мембраны, более конкретно на внешней стороне мембраны.
Было обнаружено, что в синтетической мембранной системе слитые белки по изобретению также могут быть направлены и транслоцированы на поверхность и закреплены там. В этом случае возможно включение искусственных неканонических аминокислот в слитые белки по изобретению in vivo или in vitro. Используя бесклеточный белковый синтез, можно получить слитые белки, которые пригодны также для заполнения синтетических мембран и презентации в закрепленном состоянии.
Еще одним аспектом настоящего изобретения является способ презентации пептида с заданной аминокислотной последовательностью на поверхности клетки, включающий стадии
(а) обеспечения клетки по изобретению, как описано выше, и
(b) культивирования клетки в условиях, при которых экспрессируется слитый белок по изобретению.
Настоящее изобретение также относится к способу закрепления пептида с заданной аминокислотной последовательностью на мембране, включающему стадии
(а) обеспечение мембраны и
(b) приведение мембраны в контакт со слитым белком, как описано выше, в условиях, при которых происходит закрепление слитого белка в мембране или приведение мембраны в контакт с нуклеиновой кислотой, как описано выше, или вектором, как описано выше, в условиях, при которых слитый белок экспрессируется и закрепляется в мембране.
Еще одним объектом изобретения является применение вышеупомянутой клетки, мембранного препарата или синтетической мембранной системы в способе скрининга партнеров пептида по взаимодействию, для изучения взаимодействий пептида с партнерами по взаимодействию (например, антителами), в способе получения пептида, в качестве реагента иммунизации в способе получения антител и/или в качестве реагента обнаружения.
Настоящее изобретение относится также к клеточному, мембранному препарату или синтетической мембранной системе, как описано выше, для применения в качестве вакцины и/или в качестве лекарственного средства.
В воплощении изобретения слитые белки удаляются из трансфицированных, временно или стабильно экспрессирующих клеток, трансгенных организмов или синтетических мембранных систем так, чтобы их можно было использовать в исследованиях и разработках, а также, возможно, поставлять как часть способа или набора для коммерциализируемого применения. Примером является производство биоаналитических-диагностических систем и терапевтических агентов.
В следующем воплощении по изобретению мембраны, содержащие слитые белки, удаляются из клеток, трансгенных или синтетических мембранных систем, чтобы их можно было использовать в исследованиях и разработках, а также, возможно, поставлять как часть способа или набора для коммерциализируемого применения. Примером является производство биоаналитических-диагностических систем и терапевтических агентов.
Экспрессируемые слитые белки по настоящему изобретению показаны на клеточной поверхности трансфицированных клеток в качестве стационарных реагентов и могут быть использованы для анализа специфических взаимодействий с другими молекулами. В данном документе интересны белково-белковые взаимодействия (например, взаимодействия моно- или поликлональных антител с паратопом-эпитопом, суперспиральные взаимодействия и многие специфические взаимодействия фармацевтического-лекарственного, в частности, молекулярно-механического значения). Молекулы слияния с тетраспанином также могут быть использованы для анализа взаимодействий с другими молекулами, таких как моно- и полисахариды, РНК, ДНК и, в частности, с низкомолекулярными молекулами, такими как природные вещества и библиотеки химических веществ.
Экспрессия множества молекул слияния с тетраспанином или вариантов видов белков в клетке столь же интересно для некоторых экспериментальных препаратов, как комбинаторное применение различных взаимодействующих белков.
В одном воплощении изобретения клетка по изобретению экспрессирует белковый домен (А), который способен очень специфично взаимодействовать с другим белковым доменом (В). В данном документе домен белка (А) соответствует пептиду с заданной аминокислотной последовательностью домена (ii) слитого белка по изобретению.
Этот внешний домен белка может затем быть связан с различными функциональными дополнительными белковыми доменами. Для всех этих модулей белковых доменов, можно гарантировать, что они, в случае их экзогенного происхождения, специфически связываются с противоположным доменом (А) экспрессирующей клетки и, таким образом, могут быть заменены белковые лиганды, которые в свою очередь могут снова связываться и, следовательно, на поверхности клетки может быть обнаружено сосуществование различных белковых модулей, опосредованных специфическим взаимодействием.
Предпочтительные примеры применения слитых белков, векторов, клеток и мембранных систем по изобретению более подробно описаны ниже.
Анализ белково-белковых взаимодействий (PPI)
Закрепленные белки, доступные на поверхностях, могут устанавливать специфические контакты с другими белками с помощью определенных структур на поверхности соответствующих белков. Эти дополнительные белки могут быть белковыми строительными блоками гомо- и гетеропротеиновых комплексов, которые могут распознавать друг друга или также специфическими белками, такими как антитела и одиночные цепи, которые могут специфически и целенаправленно связываться с определенными поверхностными структурами белка. Геномно закодированные природные белки, которые входят в специфические взаимодействия с другими белками, выполняют многие клеточные функции - от функций в ядре клетки до различных цитозольных и мембранных функций. Эти специфические и функциональные белково-белковые взаимодействия (PPI) возникли в ходе эволюции и в решающей степени отвечают целостности клеточной функции. Целевые PPI опосредуются клеточными процессами и выбором варианта белка, который имеет специфическое взаимодействие с поверхностями. Иммунная система предназначена для чувствительных реакций на изменение поверхностей в организме и специализируется на оптимизации поверхностных взаимодействий антител и цитотоксичности за очень короткие промежутки времени. При гуморальном иммунитете в короткие сроки образуются большие количества эпитоп-специфических, поликлональных антител. Известны особенные одноцепочечные антитела верблюдовых (одиночные цепи) и другие природные иммуногенные системы, которые развивают высокоспецифичные белковые взаимодействия.
В медицинских исследованиях эти процессы получения моноклональных антител были реализованы технически с помощью технологий клеточных культур, и, таким образом, стало возможным получение специфических PPI in vitro.
В разработке антител используются искусственные паратопы, созданные с помощью дисплейных технологий (рибосомный дисплей, фаговый дисплей, дрожжевой дисплей), которые могут специфически распознавать эпитопы для включения их в молекулы антител. Эффективные функции экспорта белковых единиц системы tANCHOR, описанной в данном документе, и их закрепление в мембране в высокой степени подходят для анализа PPI. Кроме того, с помощью технологии tANCHOR на поверхностях клеток могут быть презентированы белки, специфически модифицированные сахарами.
Системы детекции, с помощью которых квалифицированный специалист в данной области способен идентифицировать специфические PPI, предлагаются коммерчески как системы сортировки клеток (FACS), системы получения изображений на основе флуоресценции или биолюминесценции, а также как фотонные приборы FRET. Масс-спектроскопический анализ также является отличной возможностью в качестве аналитического инструмента.
Правильно используемая клеточная система позволяет получить качественные генерируемые данные и убедительное различение. Подобно разрешению в оптике, это является решающим параметром для всех последующих стадий процесса, которые определяют качество данных, получаемых в конце цепочки.
Система tANCHOR особенно подходит для обеспечения дисплейных функций биоаналитических диагностических приложений, а также терапевтических агентов. В этой системе целостность белковых структур может оставаться структурно интактной с сохранением, например, структурных эпитопов, а также способности связывать домены при PPI белков-клеточных регуляторов между собой.
Анализ малых молекул с помощью конструкций tANCHOR
С помощью структурно интактных белковых структур можно дополнительно провести исследования взаимодействия связывания, а также замещения, при PPI-связывании небольших молекул с белками, представленными на клетках. Интересные взаимодействия с малыми молекулами - это те, которые мешают PPI и при которых некоторые белковые агрегаты могут снова распадаться под действием идентифицированных малых молекул. PPI-анализы особенно хорошо подходят для обнаружения и разработки низкомолекулярных активных ингредиентов.
Технология tANCHOR предлагает превосходный инструмент для этого, позволяя осуществлять белковую экспрессию с одновременным закреплением белков на поверхности клеток. В сочетании с подходящими белковыми парами из другого источника экспрессии или, возможно, также путем совместной экспрессии различных молекул TANCHOR, можно получить тестовые системы для анализа взаимодействий малых молекул с белковыми комплексами1, 40-48.
Кроме того, сегменты слитых белков по изобретению, локализованных на поверхности клетки, могут быть специфически расщеплены в указанном месте посредством возможной индуцибельной коэкспрессии секретируемых протеаз с высокой специфичностью (например, TEV-протеазы) и, таким образом, переведены в раствор. Дефектные слитые белки могут постоянно заменяться в цикле ресинтеза в мембране и, посредством этого обеспечивать длительную избыточную экспрессию.
Таким образом, малые молекулы (например, из экстрактов организмов, экспрессирующих вторичные метаболиты) могут связываться с белками по изобретению и идентифицировать их как мишень.
В полученных комплексах малых молекул и презентированных сегментах белка по изобретению малые молекулы могут быть связаны либо специфически с целевыми пептидами, закрепленными на поверхности клетки, либо с уже расщепленными целевыми пептидами, присутствующими в растворе в качестве пептидов-носителей.
Полученные комплексы и, вследствие этого, малые молекулы и их связывающие партнеры затем могут быть идентифицированы с помощью масс-спектроскопии. Например, в случае непептидных малых молекул они могут быть идентифицированы с помощью масс-спектроскопии после выделения комплексов, присутствующих в растворе, и разрушения пептидов, например, посредством неспецифического ферментативного протеолиза и т.п.
Разработка и производство иммунодиагностических устройств
В соответствии с одним из воплощений изобретения интересными являются те, в которых антитела должны быть протестированы в определенном формате, например, стандартизованном 96-луночном формате, на связывание с эпитопами, которые экспонированы с помощью tANCHOR. Например, с помощью системы по изобретению, клетки высевают в микротитровальные планшеты 96-луночного формата, а слитые конструкции экспрессируются после временной трансфекции. Это также возможно со стабильными клеточными линиями, экспрессирующими слитые белки.
В качестве примера показано применение системы tANCHOR для обнаружения антител в экспериментах тИФА (фиг. 9).
Слитые белки находятся на поверхности клетки и презентируются на ней. Такая искусственная конфигурация мембраны может быть реализована непосредственно для разработки и производства аналитических и терапевтических приложений.
Таким образом, могут быть получены аналитические системы, в которых, например, подходящие сыворотки крови групп пациентов, соответствующих разрабатываемому конкретному применению, могут быть протестированы для клинических исследований. При разработке иммунодиагностических устройств и терапевтических агентов, в частности, также при разработке сопутствующей диагностики, такими системами можно качественно и количественно исследовать сыворотку пациента на предмет присутствия антител и их связывающих свойств.
Кроме того, можно фиксировать и сушить микротитровальные планшеты и другие форматы, в которых росли трансгенные клетки, с целью изготовления биоаналитических тестовых систем и диагностических устройств с ними в больших масштабах и для их производства в больших масштабах.
Кроме того, также можно использовать мембраны трансгенных организмов, которые экспрессируют продукты синтеза тетраспанина и очищать их. Кроме того, искусственные мембраны, содержащие продукты слияния по изобретению, также могут быть представлены для применений, описанных выше.
Экономия в данном случае огромна, поскольку нет сравнительно дорогого покрытия пептидами или экспрессированными и очищенными белками.
Получение чистого белка
При проведении исследований и разработок, а также при крупномасштабном производстве, получение достаточных количеств целевых белков с высокой удельной активностью посредством их экспрессии все еще является проблемой, особенно если необходимо очистить функционально свернутые белки и, возможно, модифицированную форму. Для этого выгодно использовать систему по изобретению. Данный подход может иметь большое преимущество, поскольку искомый белок-мишень локализуется вне клетки в среде, которая является намного менее сложным окружением, чем цитозоль внутри клеток. Соответственно, легче проводить обогащение и выделение. Обычно экспрессированные белки обнаруживаются внутри клетки и очищаются из цитозоля.
В одном воплощении применения по изобретению расщепление мембрано-закрепленного белка, экспрессированного с помощью tANCHOR, может быть осуществлено путем отрезания от мембраны с помощью протеиназ, специфичных к аминокислотному мотиву. С одной стороны, специфические протеазы могут подаваться в среду экзогенно или могут экспрессироваться совместно с целевым белком. Эта процедура, возможно, потребует создания специфичной к последовательности эндопротеазы, которая активируется в среде. При длительном культивировании клеток с целью выделения белка, в конечном счёте, происходит обогащение специфического белка-мишени в среде, поскольку целевой белок, высвобождаемый tANCHOR, непрерывно подается в мембрану из-за потери целостности белка.
ДНК-вакцинация в качестве терапевтического применения
В сочетании со специфическими для дифференциации и/или видоспецифическими промоторами (в том числе в ветеринарном секторе), которые экспрессируют только в очень специфических клетках-мишенях, система tANCHOR способна, благодаря эффективному мембранному транспорту, экспрессировать элементы гетерологичных слитых генов даже в интактных клетках организма. Такие элементы можно вводить в клетки экзогенно. Для инокуляции молекул ДНК или РНК слитые конструкции на основе тетраспанина можно эффективно выстреливать непосредственно в клетки с высокой скоростью с помощью так называемых «генных пушек» (gene guns или particle guns) и при попадании индуцировать в трансфецированных клетках транзиторную экспрессию слитого белка. Эти белки могут быть антигенами или субспецифическими антигенными детерминантами, эпитопами, которые ранее тестировались на предмет их пригодности. Технология tANCHOR может использоваться как для обеспечения диагностических биоанализов, пригодных для этой цели, так и для терапевтической вакцинации.
Обсуждается, что ДНК может интегрироваться в геномы клеток при попадании генных частиц, и может, возможно, вызывать повреждение геномов при интеграции, например, в ген опухолевого репрессора. Однако на практике для того, чтобы действительно вызвать высокую и значимую степень интеграции, необходимы весьма обширные гомологичные генные области, сопровождающие конструкции генов по изобретению. Такие элементы не предусмотрены в конструктах согласно настоящему изобретению. Однако из-за цепи таких ассоциаций, связанных с опасностью ДНК-вакцинации в части чувствительных регуляторов, на первое место выступают вакцины на основе мРНК. Таким образом, согласно изобретению также возможно, например, получать посредством транскрипции in vitro молекулы мРНК, которые оптимизированы для экспрессии в эукариотических клетках, стабилизировать их, фиксировать на наногранулах или упаковывать их в подходящие микро- и нанокомплексы (вирусоподобные частицы, VLP) и предоставлять мРНК в такой форме для терапевтических применений.
Из-за специфических свойств слитых конструкций на основе тетраспанинов, белки, экспрессированные таким образом, транспортируются на клеточные мембраны экспрессирующих клеток и презентируются там во внешнее пространство.
Экспонирование специфических аминокислотных последовательностей может распознаваться иммунной системой и приводить к соответствующим, целевым иммунным ответам, гуморальному или цитотоксическому.
Терапевтические применения с использованием выделенных клеток или клеточных мембран
Кроме того, возможно (в идеале из-за несовместимости групп HLA и возможных неблагоприятных иммунных реакций, связанных с ними) трансфецировать даже аутологичные клетки in vitro после экстракции ex vivo, возможно, после или во время промежуточной фазы культивирования. Трансфекция аутологичных клеток тестируемых объектов (например, людей, а также животных) ДНК-конструкциями на основе системы tANCHOR позволит использовать их при аутологичных или аутогенных вакцинациях. В связи с этим возможно использование интактных аутологичных клеток, которые были протестированы на качество, а также возможно использование выделенных клеточных мембран из таких клеток. Они могут быть получены, например, путем ультрацентрифугирования в градиенте плотности. В данном документе отношение эпитопа к общему белку можно повысить и тем самым способствовать увеличению специфичности иммунного ответа. При этом происходит снижение неспецифических сопутствующих ответов и аутоиммунных реакций.
Комбинация экспрессируемых адъювантов, коэкспрессируемых в клетке с несколькими конструкциями tANCHOR, является вариантом, который также находится в пределах диапазона возможностей для улучшения применения систем tANCHOR по изобретению.
Коэкспрессия различных белков, которые улучшают физико-химические свойства, например, растворимость мембран, может значительно улучшить для вакцинации мембраны, выделенные из клеток. Природные адъюванты также могут помочь улучшить реакцию на вакцинацию путем коэкспрессии с конструкциями слияния CD63 с антигеном/эпитопом.
Еще одним объектом изобретения является набор, содержащий
(i) молекулу нуклеиновой кислоты или вектор, описанные выше, и
(ii) клетку или мембрану.
Кроме того, в зависимости от предполагаемого применения набор по изобретению может содержать различные компоненты, такие как, например, контрольные плазмиды, контрольные белки или подобные элементы и специальные плазмиды для различных применений.
Настоящее изобретение будет описано более подробно с помощью чертежей и примеров ниже.
Фигуры и таблицы
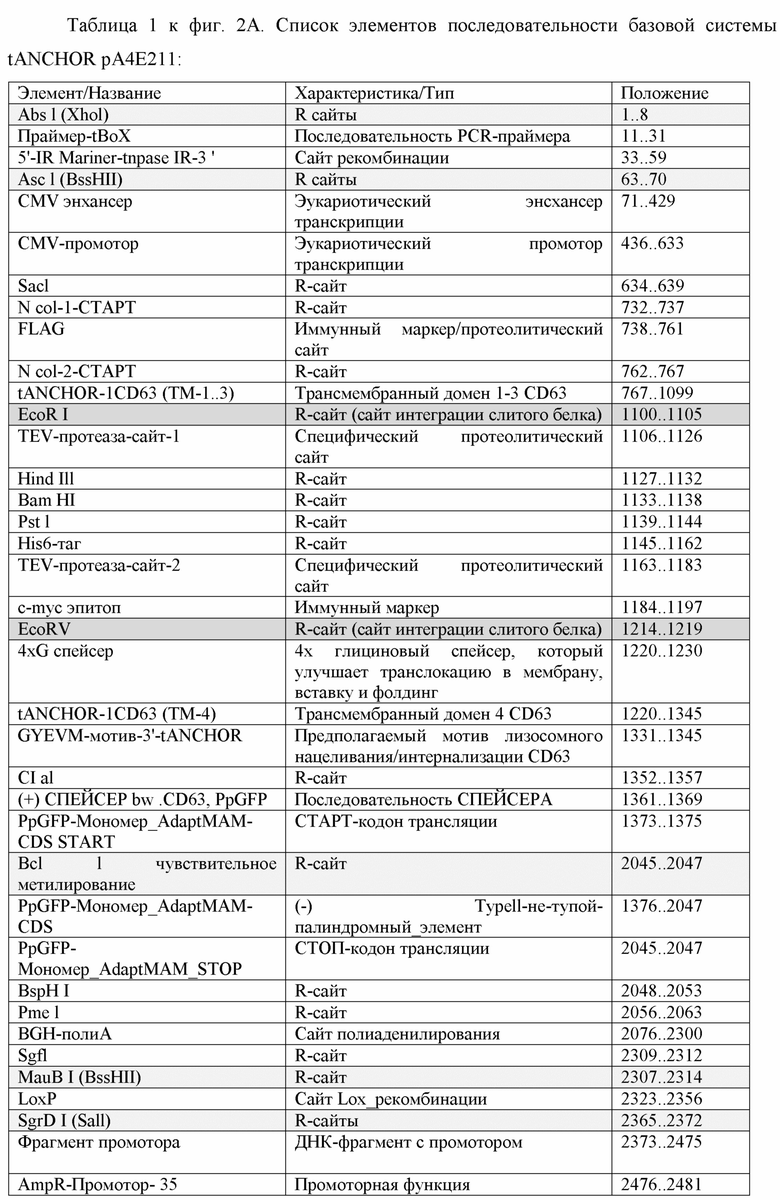
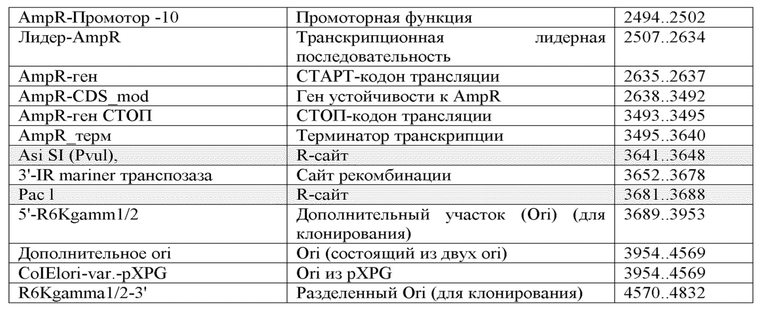
Фиг. 1. Схематическое изображение слияний по изобретению и локализация слитых белков.
(A) Положения, которые, согласно предшествующему уровню техники, могут служить различными применимыми способами разрешения проблем, связанных со слияниями и вставками. Система по изобретению, описанная также как система tANCHOR, состоит из четырех трансмембранных доменов (4TM). Последовательности ДНК, кодирующие эпитопы или белки, могут быть клонированы между TM3 и TM4. В качестве примера, метка эпитопа FLAG была имплементирована на N-конце, а, например, флуоресцентный белок mCherry был дополнительно имплементирован в качестве контроля трансфекции на С-конце CD63 в системе TANCHOR посредством слияния генов (см. также фиг. 2).
(B) С помощью системы tANCHOR, разработанной для STATUS QUO, можно четко определить эпитопы или антигены, экспрессируемые клетками человека, локализованные на плазматической мембране. Система tANCHOR способна с высокой эффективностью направлять слитые белки в плазматическую мембрану и закреплять их там.
Фиг. 2A. Схематическая структура примерной векторной системы tANCHOR.
Первая примерная векторная система tANCHOR строится на модульном мультиэкспрессирующем векторе pA4E211 (SEQ ID NO: 1) и на функциональных производных этого вектора. Дополнительно представлен обзор расположения компонентов CD63ΔLEL (фиг. 2B), а также N-концевое слияние с эпитопом FLAG, а также С-концевое слияние с флуоресцентным репортерным белком, таким как, например, mCherry или GFP из Pontellina plumata.
Фиг. 2B. Получение последовательности CD63LLEL (SEQ ID NO: 3) из кодирующей последовательности дикого типа (WT) CD63 (SEQ ID NO: 17).
Белковая последовательность CD63 дикого типа состоит из 238 аминокислот. Для получения CD63ΔLEL использовали кодирующую последовательность ДНК трансмембранных доменов 1-3 и 4 (подчеркнутая последовательность CD63 TM1-3, CD63 TM4). Линкер, который не содержит последовательности для LEL CD63 (ΔLEL), помещается между фрагментом CD63 TM1-3 и CD63 TM4 (SEQ ID NO: 42). Дополнительно в линкере добавляется мотив структуры GGGG. Последовательность гена (SEQ ID NO: 45) для линкера с аминокислотной последовательностью KLlDTVDLEK (SEQ ID NO: 46) может быть включена между геномной последовательностью CD63ΔLEL и последовательностью гена флуоресцентного белка.
Таблица 2 к фиг. 2A. Линейная организация модульно структурированных векторных систем набора TANCHOR, серия I типа: pA4E211
(0) Abs l- Asc l- | FLAG | CDXX: Эпитоп | Репортер | -MauBI-SgrDI-SELECTORS -AsiSI- P acl
(1) Abs l- Asc l- | FLAG | ----: ------ | --------- | - Mau BI- Sgr Dl-Пуромицин R- Asi SI- P acl
(2) Abs l- Asc l- | FLAG | CD63: ------- | --------- | - Mau BI- Sgr Dl-Пуромицин R - As iSI- P acl
(3) Abs l- Asc l- | FLAG | CD63: gp41 | --------- | - Mau BI- Sgr D1-Пуромицин R- Asi SI- P acl
(4) Abs l- Asc l- | FLAG | CD63: eYFP | eGFP-ген | - Mau BI- Sgr D1-Пуромицин R - As iSI- P acl
(5) Abs l- Asc l- | FLAG | CD63: bio Pep ID | eGFP-ген | - Mau BI- Sgr D1-Пуромицин R- Asi SI- P acl
(6) Abs l- Asc l- | FLAG | CD63: bio Poi ID | YFP-ген | - Mau BI- Sgr DI-Пуромицин R -AsiSI-Pacl
(7) Abs l- Asc l- | FLAG | CD63: bio Poi ID | POI-Fusionl- Mau BI- Sgr Ol-Пуромицин R- Asi SI- P acl
Векторы положительного и отрицательного контроля для системы презентации белков TANCHOR CD63 (дисплейная система). (0) Формальное представление функциональных элементов технологии tANCHOR: CDXX - (= различные виды белка тетраспанина) CDS (= кодирующая последовательность) (1) вектор tANCHOR без гена CD63, внутренняя вставка CD63 и терминальный репортер (2) вектор tANCHOR с геном CD63 без внутренней вставки CD63 и терминального репортера вектора tANCHOR с геном CD63 и, например, с эпитопом метки FLAG в качестве внутренней вставки CD63 и без терминального репортера (3) вектор tANCHOR с геном CD63 и эпитопом gp41 в качестве внутренней вставки CD63, а также терминальный репортер вектора tANCHOR, например, с геном CD63 (4) вектор tANCHOR, например, с геном CD63 и YFP-эпитопом в качестве внутренней вставки CD63, и с C-концевым репортером GFP (например, ген зеленого флуоресцентного белка). (5) вектор tANCHOR, например, с POI-bioPepID-эпитопом (эпитопами) (представляющий интерес белок/пептид), используемый для анализа в качестве внутренней вставки CD63, и с C-концевым репортером eGFP (например, ген зеленого флуоресцентного белка), а также (6) с дополнительными репортерными генами, такими как YFP (ген желтого флуоресцентного белка) и с белками-партнерами слияния соответствующего экспериментального интереса (POl-слияние). (7) с партнерами слияния POl и т.д., в зависимости от экспериментальной конструкции. bioPeplD - биопептидные библиотеки типа PeplD (EP09000893.9). Они могут использоваться в системе tANCHOR. Absl-SgrDl и Ascl-MauBI, а также AsiSI-Pacl представляют собой взаимоблокируемые эндонуклеазы рестрикции, совместимые друг с другом, с последовательностью распознавания из восьми нуклеотидов для мультиплексирования экспрессии множества генов. Любая другая реализация применений по изобретению в равной степени охватывается патентным документом.
Фиг. 2С: Примеры различных вариантов линкеров по изобретению в комбинации с CD63ΔLEL в примерном векторе pAE211_CMV-P-CD63ΔLEL. Сайты рестрикции EcoRI, EcoRV, HindIll, BamHI и Pstl можно использовать для вставки множества последовательных эпитопных блоков в соответствии со стандартным способом клонирования или способом бесшовного клонирования. (1) Seq1, только сайты рестрикции по изобретению и проверка в отношении функции (R-сайты) EcoRI и EcoRV и n * N = спейсерная последовательность (2) Seq (1) плюс His6-таг, (3) Seq (1) плюс CMyc эпитоп, (4) Seq (1) плюс His6-метка плюс C-Myc (5) сайты протеолиза TEV, включая центральные сайты клонирования, для селективного сбора экспрессированного чужеродного белка, (6) все названные транслированные и нетранслированные молекулярные функциональные элементы, (8) объединенные в конструкцию (SEQ ID NO: 39, 40). (9) Минимальный линкер для транспортировки на поверхность клетки (SEQ ID NO: 41,42).
Фиг. 3. Локализация слитых белков на поверхности клеток HEK293T с помощью cLSM.
(A) HEK293T клетки были котрансфецированы соответствующими векторами. В исследованиях локализации вектор pCMV-CD63-YFP служил контролем для экспрессии белка CD63-YFP дикого типа. Для всех белков, содержащих технологию tANCHOR (CD63ΔLEL, фиг. 2B), преобладающая локализация на поверхности клетки определялась с помощью слитого репортерного белка mCherry. Белок mCherry без сшивки с tANCHOR является цитоплазматическим, равномерно распределенным. Это указывает на отсутствие преобладания на поверхности клетки. Кроме того, на поверхности могут быть представлены дополнительные белки, такие как, например, гликопротеин gp41 из HIV-1 с трансмембранным доменом и без него (gp41ΔTM). (B) Сворачивание белка проверяли по локализации CFP и YFP на поверхности. Оба флуоресцентных белка могут быть детектированы после возбуждения диодно-аргоновым лазером при 405 нм (CFP) или 514 нм (YFP), при определенной длине волны излучения для флуорофора. (C) Экспрессированный белок mCherry, который обычно присутствует в цитозоле, с высокой эффективностью направляется в плазматическую мембрану с помощью системы tANCHOR. Таким образом, mCherry с внедренной технологией tANCHOR детектируется даже в филоподии (белая стрелка) клеток HEK293T, как в случае CD63-YFP дикого типа. Длина масштабной метки составляет 10 мкм на фиг. 3А, 3В и 1 мкм на фиг. 3С.
Фиг. 4. Схематическое изображение исследования по определению внеклеточной ориентации эпитопов
Детекцию ориентации эпитопа проводили с помощью сканирующей электронной микроскопии (SEM) на поверхности трансфецированных HEK293T. Белки связывали с помощью первичных антител и визуализировали с помощью конъюгированных вторичных антител с золотыми частицами (10 нм). В случае модельных эпитопов YFP, CFP, gp41 и gp41ΔTM связанные антитела могут быть визуализированы на поверхности с помощью иммуноглобульных частиц (фиг. 5).
Фиг. 5A-D. SEM-изображение частиц золота (10 нм) на поверхности клеток HEK293T.
Трансфецированные клетки HEK293T исследовали соответствующим образом в отношении ориентации эпитопов. Для этого соответствующие эпитопы связали со специфичными первичными антителами, а затем их детектировали с помощью вторичных антител, конъюгированных с золотом. Все эпитопы технологии tANCHOR можно было отнести к внеклеточной ориентации. Длина масштабной метки на всех фигурах составляет 300 нм.
Фиг. 6. Вестерн-блот-анализ трансфецированных клеток HEK293T с полученными векторами tANCHOR.
Трансфецированные клетки HEK293T подвергали вестерн-блот-анализу. Экспрессированные белки в отдельных клеточных лизатах выравнивали с помощью первичных антител против C-концевого репортерного белка mCherry и подвергали детекции N-концевого эпитопа FLAG. Трансфекция вектором pmCherry-N1 демонстрирует отсутствие полосы в случае инкубации с антителом против FLAG, так как этот вектор не содержит последовательности для эпитопа FLAG и служит в качестве контроля для специфической детекции экспрессированных белков.
Фиг. 7. Схематическое изображение конструкций для изучения дополнительных тетраспанинов в отношении транспорта белка, с внутренним сайтом слияния с линкером V5-6xHis.
Для исследования независимости транспорта белка от большой внеклеточной петли в суперсемействе тетраспанинов, были созданы экспрессирующие векторы, содержащие внутреннее слияние с V5-6xHis между 3-м и 4-м трансмембранным доменами, соответствующих конструкциям прототипа tANCHOR на основе CD63ΔLEL. Внутреннее слияние с V56xHis содержит линкер между EcoRI и EcoRV (SEQ ID NO: 35, 36). Слияние с FLAG интегрировано в N-концевой области; в C-концевой области флуоресцентные белки YFP слиты в случае конструктов тетраспанина дикого типа, а mCherry слит в случае конструктов ΔLEL-V5-6xHis в качестве репортерных белков.
Фиг. 8. Локализация слитых белков на поверхности клеток HEK293T.
Клетки HEK293T котрансфецировали полученными векторами (A) pCMV-CD9-YFP/pCMV-CD9ΔLEL-V5-6xHis-mCherry, (B) pCMV-CD81-YFP/pCMV-CD81ΔLEL-V5-6xHis-mCherry, (C) pCMV -CD82-YFP/pCMV-CD82ΔLEL-V5-6xHis-mCherry и (D) pCMV-CD151-YFP/pCMV-CD151ΔLEL-V5-6xHis-mCherry. Все экспрессированные белки могут детектироваться преимущественно по флуоресценции репортерного белка mCherry на поверхности клетки. Конструкция, содержащая внутреннюю сшивку с линкером V5-6xHis между 3-м и 4-м трансмембранными доменами (фиг. 7) тетраспанинов CD9, CD81, CD82 и CD151 колокализована с соответствующими экспрессированными тетраспанинами дикого типа, слитыми с флуоресцентным белком YFP. Масштабная метка на всех фигурах составляет 10 мкм.
Фиг. 9. Применение системы tANCHOR для детекции антител
(A) Результаты тИФА53 с системой tANCHOR в 96-луночном формате. Эпитоп V5 на клеточной поверхности клеток HEK293T детектировали с помощью HRP-конъюгированных антител против V5. Белковая экспрессия белков tANCHOR может быть определена зависимым от концентрации способом и в значительной степени с помощью антител против V5-HRP. (B) Схематическое изображение линкеров 2F5-4E10 между 3-м и 4-м трансмембранным доменами CD63 (SEQ ID NO: 37, 38). Линкеры вводили в вектор pCMV-CD63ΔLEL. (C). HIV-1-нейтрализующие антитела против 2F5 и против 4E10 связывались с эпитопом 2F5 и 4E10 при постоянном количестве ДНК, составляющем 0,6 мкг на лунку, при трансфекции клеток HeLa. Связанные антитела против 2F5 и против 4E10 детектируются в значительной степени, (E) экспрессированные слитые белки CD63ΔLEL-V5-6xHis, а также CD63ΔLEL-2F5-4E10, локализованы преимущественно на поверхности клеток HEK293T для контроля. Длина масштабной метки на всех фигурах составляет 10 мкм.
Фиг. 10. Повышение эффективности за счет использования линкеров с низким содержанием серина и аргинина.
(A) Используемые линкеры представлены на уровне ДНК и аминокислотной последовательности. Анализ субклеточной локализации слитых белков с двумя различными линкерами (SEQ ID NO: 41, 42 и SEQ ID NO: 43, 44) ясно показывает, что использование фланкирующих сайтов рестрикции EcoRI/EcoRV приводит к высокой эффективности транспорта белка на поверхность клеток HEK293T, и слитый белок детектируется на филоподии (белая стрелка), которая образуется на клетках (B). Длина масштабной метки на всех фигурах составляет 10 мкм.
1. Получение модельных векторов на основе CD63 для проведения экспериментов по локализации
Получение модельных векторов на основе CD63 основывалось на применении вектора pCMV-Tag2B (Stratagene). С помощью традиционных методов клонирования соответствующие последовательности ДНК вводили в этот вектор по сайтам рестрикции39. Контрольный вектор pCMV-CD63-YFP, описанный также как pCMV-CD63-YFP-FLAG49, который содержит полноразмерную последовательность CD63 (аминокислоты 1-238, GenBank регистрационный №.KF998086), использовали для введения последовательности ДНК используемого красного флуоресцентного репортерного белка mCherry с помощью праймеров PA1-01/PA1-02 и используемого в качестве шаблона вектора pmCherry-N1 (Clonteсh) с помощью сайтов рестрикции Xhol и Apal. Частичная последовательность с трансмембранными доменами TM1-3 (аминокислоты 1-110 учетный номер GenBank KF998086) вводили по сайтам рестрикции BamHI и Pstl с помощью праймеров PA1-03/PA1-04, а затем TM4 (аминокислоты 201-238, учетный номер GenBank KF998086) вводили в вектор pCMV-Tag2B по EcoRV и HindIII с помощью праймеров PA1-05/PA1-06, и используемого в качестве шаблона вектора pPR3-N-CD6349. Полученный вектор содержит последовательность гена CD63 без LEL (pCMV-CD63ΔLEL) (фиг. 2B).
Для упрощенной детекции белков вырезанный рестрикционными эндонуклеазами фрагмент ДНК mCherry вектора pCMV-CD63-mCherry лигировали в разрезанный рестрикционными эндонуклеазами вектор pCMV-CD63ΔLEL по сайтам рестрикции Xhol и Apal. Векторы pCMV-CD63ΔLEL-CFP и pCMV-CD63ΔLEL-YFP получали с помощью сайтов рестрикции EcoRI и EcoRV с помощью праймеров PA1-07/PA1-08, а также используемого в качестве шаблона вектора pSCFP3A-C1 или pSYFP2- C150. Таким же образом векторы pCMV-gp41ΔTM и pCMV-gp41 были получены с помощью праймеров PA1-09/PA1-010 и PA1-09/PA1-11 и используемого в качестве шаблона вектора pNL4-351, который содержит генную последовательность белка gp41 HIV-1.
2. Трансфекция и конфокальная лазерная сканирующая микроскопия (cLSM)
Для исследования локализации экспрессированных слитых белков, плазмиды, полученные в примере 1, трансфецировали в клетки HEK293T с помощью реагента для трансфекции. Для этого клетки HEK293T (эмбриональные клетки почки человека 293T) высевали в 8-луночные планшеты ibiTreat и культивировали в модифицированной среде Дульбекко Игла, дополненной 10% эмбриональной телячьей сывороткой, L-глутамином и пенициллином/стрептомицином. Клетки с конфлюэнтностью прибл. 50% трансфецировали полученными плазмидами с помощью Metafectene®PRO (Biontex) в соответствии с инструкциями производителя. Через 24 ч трансфецированные клетки фиксировали в 2% параформальдегиде в PBS, дважды промывали в PBS и заключали в 90% глицерина в PBS с 0,1% п-фенилендиамином и DAPI (4',6-диамидино-2-фенилиндолом) для визуализации ядра. Трансфецированные клетки исследовали с помощью обратного конфокального лазерного микроскопа LSM 780 (Carl Zeiss) в отношении субклеточной локализации (фиг. 3А-3С). Настройки длин волн возбуждения и эмиссии для флуоресцентных белков CFP, YFP и mCherry были выбраны с помощью опции Smart Setup программного обеспечения LSM 780 ZEN 2010.
3. Ориентационный анализ эпитопов на поверхности клеток HEK293T с помощью сканирующей электронной микроскопии
Экспрессированные белки детектировали с помощью сканирующей электронной микроскопии иммуно-маркированных золотом белков на плазматической мембране клеток HEK293T (фиг. 4). Для этого клетки, трансфецированные и фиксированные в 2% параформальдегиде в PBS, трижды промывали в PBS и блокировали 0,5% BSA/0,1% желатина в PBS. Белки CD63LEL-CFP и CD63ΔLEL-YFP инкубировали с кроличьими анти-GFP антителами (ab6556, Abcam), промывали, а первичные антитела детектировали с помощью козьих антикроличьих антител, конъюгированных с 10 нм частицами золота. После иммунной маркировки проводили постфиксацию глутаральдегидом (2,5% в HEPES 0,05 М). Затем следовала подготовка к сканированию. Для этого образцы постепенно высушивали с помощью этанольных стадий (30%, 50%, 70%, 90%, 96%) по 15 мин на каждой стадии, оставляли на 30 мин в абсолютном этаноле, переносили в HMDS (гексаметилдисилазан), высушивали из HMDS, и покрывали поверхность образца выпаренным углеродом. Частицы золота визуализировали с помощью сканирующего электронного микроскопа Leo 1530 Gemini с помощью детектора обратного рассеяния (Centaurus). Таким же образом детектировали белок gp41 с помощью человеческого антитела против 2F5 (National Institutes of Health, NIH), а также с использованием козьего антитела против человеческого антитела, конъюгированного с 10 нм частицами золота IgG H&L (BBlnternational) (фиг. 5A-5D).
4. Вестерн-блот-анализ экспрессированных флуоресцентных белков
Для исследования экспрессии белка, клеточные лизаты трансфецированных клеток HEK293T подвергали Вестерн-блот-анализу. Для этого клетки HEK293T с векторами трансфецировали в 6-луночном формате с помощью METAFECTENE®PRO в соответствии с инструкциями производителя. Через 48 часов среду полностью удаляли и в каждую 6-лунку помещали 100 мкл буфера для образцов Laemmli 2x и 25 Ед. эндонуклеазы из Serratia marcescens (Benzonase®). Клетки лизировали и примерно через 10 мин к образцам добавляли 5 мкл меркаптоэтанола и денатурировали при 95°С в течение 5 мин. Эти клеточные лизаты разделяли с помощью SDS-PAGE, переносили на PVDF-мембрану и детектировали с помощью мышиных антител против mCherry (ab125096, Abcam) или козьих против FLAG (NB600-344, NovusBio), а также HRP-конъюгированных антимышиных/антикозьих антител (Dako), а связанные HRP-конъюгированные антитела детектировали с помощью субстрата Pierce ECL Western Blot (Thermo Fisher Scientific). Мембрану PVDF обрабатывали с помощью стриппинг-буфера Roti®-Free Stripping Buffer 2.2 plus (Carl Roth) так, что одни и те же белки могли детектироваться с помощью дополнительного первичного антитела (фиг. 6).
5. Изучение субклеточной локализации гибридных белков на основе тетраспанина из суперсемейства тетраспанинов
Для исследования пептидной презентации на клеточной поверхности в пределах суперсемейства тетраспанинов, экспрессирующие векторы получали в соответствии с схематическим представлением (фиг. 7) с помощью генного синтеза54 (ATG: Biosynthetics), а также стандартизованных способов клонирования. Последовательности генов CD9 (SEQ ID NO: 19), CD81 (SEQ ID NO: 21), CD82 (SEQ ID NO: 23) и CD151 (SEQ ID NO: 25) использовали для замены в векторе pCMV-CD63-YFP, и последовательности генов CD9ΔLEL-V5-6xHis (SEQ ID NO: 27), CD81ΔLEL-V5-6xHis (SEQ ID NO: 29), CD82ΔLEL-V5-6xHis (SEQ ID NO: 31) и CD151ΔLEL-V5-6xHis (SEQ ID NO: ID NO: 33) были заменены в векторе pCMV-CD63-mCherry (см. 1: Получение модельных векторов на основе CD63 для проведения экспериментов по локализации) на последовательности тетраспанина CD63 с помощью сайтов рестрикции Notl/Xhol, нижеследующие экспрессирующие векторы последовательности гена дикого типа pCMV-CD9-YFP, pCMV-CD81-YFP, pCMV-CD82-YFP, pCMV-CD151-YFP, а также делеционные мутанты большой внеклеточной петли (ΔLEL) трансфецировали с внутренним линкером V5-6xHis-pCMV-CD9ΔLEL-V5-6xHis-mCherry, pCMV-CD81ΔLEL-V5-6xHis-mCherry, pCMV-CD82ΔLEL-V5-6xHis-mCherry и pCMV-CD151ΔLEL-V5-6xHismCherry, как описано в разделе 2 «Трансфекция и конфокальная лазерная сканирующая микроскопия (cLSM)», и исследовали с помощью cLSM внктриклеточную локализацию (фиг. 8).
6. Применение системы tANCHOR для детекции антител методом ИФА53
Высокоэффективная поверхностная экспрессия слитых белков с помощью системы tANCHOR подходит для детекции антител, связанных с презентированными пептидами. В качестве модели линкер V5-6xHis был выделен из конструкта pCMV-CD9ΔLEL-V5-6xHis-mCherry с помощью EcoRI и EcoRV и вставлен в вектор pCMV-CD63ΔLEL-mCherry с помощью стандартных способов клонирования, как описано в разделе 1 «Получение модельных векторов на основе CD63 для проведения экспериментов по локализации». Аналогично, эпитопы 2F5 и 4E10 (HIV-1) были вставлены в вектор pCMV-CD63ΔLEL-mCherry с помощью праймеров PA1-12/PA1-13 и используемого в качестве шаблона вектора pNL4-3 (фиг. 9B). Для детекции связанных антител на поверхности (фиг.9А) клеток НЕK293Т, клетки HEK293T и (фиг. 9C) клетки HeLa высевали в 96-луночном формате и трансфецировали плазмидами pCMV-CD63ΔLEL-V5-6xHis-mCherry (фиг. 9A) и pCMV-CD63ΔLEL-2F5-4E10-mCherry (фиг. 9C) с помощью Metafectene®PRO в соответствии с инструкциями производителя. Через 24 ч клетки фиксировали в течение 20 мин с 4% параформальдегидом (Carl Roth), дважды промывали PBS и блокировали в течение ночи раствором 3% бычьего сывороточного альбумина класса V (Carl Roth) и 2% альбумина куриного яйца (Carl Roth). После этого клетки один раз промывали PBS и инкубировали в течение 1 часа с разбавленным антителом (фиг. 9А) против V5-HRP (Invitrogen) и (фиг. 9C) человеческим антителом против 2F5/ человеческим антителом против 4E10 (NIH) в разведении 1: 3000. В случае фиг. 9C, первичные антитела связывали заячьим HRP-конъюгированным антителом против человеческих антител (DAKO) в разведении 1: 1000 и оба варианта тИФА с 3,3',5,5'-тетраметилбензидином в качестве субстрата тИФА (TMB Substrate Kit, Thermo Fisher Scientific) использовали в соответствии с инструкциями производителя, а значения OD измеряли при 450 нм с помощью спектрофотометра Multiscan ™ GO с использованием вставки для 96-луночного планшета (Thermo Fisher Scientific). Экспрессию слитых белков детектировали с помощью cLSM, как описано в разделе 1 «Получение модельных векторов на основе CD63 для проведения экспериментов по локализации».
7. Повышение эффективности транспорта белка на поверхность клеток человека за счет использования фланкирующих сайтов рестрикции EcoRI/EcoRV в системе TANCHOR
Использование фланкирующих сайтов рестрикции EcoRI/EcoRV в системе tANCHOR повышает эффективность транспорта на клеточную поверхность. Ввод дополнительных сайтов рестрикции в минимальный линкер (LQEFDIGGGG) приводит к трансляции серина и аргинина в линкере (G SS G RRS LQGGGG) между 3-м и 4-м трансмембранным доменами CD63 (фиг. 10А). Векторы с дополнительными сайтами были получены с помощью генного синтеза54 (ATG: Biosynthetics), экспрессированы в клетках HEK293T, как уже описано в разделе 2 «Трансфекция и конфокальная лазерная сканирующая микроскопия (cLSM)», и исследованы в отношении субклеточной локализации (фиг. 10B).
Список последовательностей
SEQ ID NO: 1: последовательность ДНК pA4E211 (CD63LEL)
SEQ ID NO: 2: последовательность ДНК CD63ΔLEL
SEQ ID NO: 3: белковая последовательность CD63ΔLEL
SEQ ID NO: 4: праймер PA1-01
SEQ ID NO: 5: праймер PA-02
SEQ ID NO: 6: праймер PA1-03
SEQ ID NO: 7: праймер PA1-04
SEQ ID NO: 8: праймер PA1-05
SEQ ID NO: 9: праймер PA1-06
SEQ ID NO: 10: праймер PA1-07
SEQ ID NO: 11: праймер PA1-08
SEQ ID NO: 12: праймер PA1-09
SEQ ID NO: 13: праймер PA1-10
SEQ ID NO: 14: праймер PA1-11
SEQ ID NO: 15: праймер PA1-12
SEQ ID NO: 16: праймер PA1-13
SEQ ID NO: 17: CD63, учетный номер GenBank KF998086
SEQ IN NO: 18: белковая последовательность CD63
SEQ ID NO: 19: последовательность ДНК CD9 (учетный номер GenBank NM_001769.3)
SEQ ID NO: 20: белковая последовательность CD9
SEQ ID NO: 21: последовательность ДНК CD81 (учетный номер GenBank NM_004356.3)
SEQ ID NO: 22: белковая последовательность CD81
SEQ ID NO: 23: последовательность ДНК CD82 (учетный номер GenBank NM_002231.3)
SEQ ID NO: 24: белковая последовательность CD82
SEQ ID NO: 25: последовательность ДНК CD151 (учетный номер GenBank BT007397.1)
SEQ ID NO: 26: белковая последовательность CD151
SEQ ID NO: 27: последовательность ДНК CD9ΔLEL-V5-6xHis
SEQ ID NO: 28: белковая последовательность CD9ΔLEL-V5-6xHis
SEQ ID NO: 29: последовательность ДНК CD81ΔLEL-V5-6xHis
SEQ ID NO: 30: белковая последовательность CD81ΔLEL-V5-6xHis
SEQ ID NO: 31: последовательность ДНК CD82ΔLEL-V5-6xHis
SEQ ID NO: 32: белковая последовательность CD82ΔLEL-V5-6xHis
SEQ ID NO: 33: последовательность ДНК CD151ΔLEL-V5-6xHis
SEQ ID NO: 34: белковая последовательность CD151ΔLEL-V5-6xHis
SEQ ID NO: 35: последовательность ДНК линкера V5-6xHis
SEQ ID NO: 36: белковая последовательность линкера V5-6xHis
SEQ ID NO: 37: последовательность ДНК линкера 2F5-4E10
SEQ ID NO: 38: белковая последовательность линкера 2F5-4E10
SEQ ID NO: 39: последовательность ДНК варианта линкера (8)
SEQ ID NO: 40: белковая последовательность варианта линкера (8)
SEQ ID NO: 41: последовательность ДНК линкера в векторе pCMV-CD63ΔLEL-mCherry (минимальный линкер (9))
SEQ ID NO: 42: белковая последовательность линкера в векторе pCMV-CD63ΔLEL-mCherry (минимальный линкер (9))
SEQ ID NO: 43: последовательность ДНК линкера в векторе pCMV-CD63ΔLEL-V2-mCherry
SEQ ID NO: 44: белковая последовательность линкера в векторе pCMV-CD63ΔLEL-V2-mCherry
SEQ ID NO: 45: последовательность ДНК линкера между CD63LEL и флуоресцентным белком
SEQ ID NO: 46: белковая последовательность линкера между CD63ΔLEL и флуоресцентным белком
Ниже кратко сформулированы предпочтительные аспекты изобретения:
1. Способ применения белков/пептидов с предполагаемыми искусственными аминокислотными последовательностями, которые кодируются косвенно, по меньшей мере, частично из матричных молекул, или являются полностью синтетическими, которые
а. могут состоять, по меньшей мере, частично из последовательностей представителей белкового класса тетраспанинов, таких как, например, человеческие гены CD63, CD9, CD82, CD81, CD151, CD53
b. но также, по меньшей мере, частично из гомологичных последовательностей тетраспанинов других организмов и
с. согласно a. и b., слитые продукты которых образуют молекулы, способные направлять связанные с ними природные или искусственные пептидные и белковые единицы в подходящих системах
i. к одной или нескольким поверхностям,
ii. транспортировать их по поверхностям и
iii. крепко закреплять их на поверхностях
iv. длительно презентировать их на поверхностях.
2. Химерные белковые молекулы с естественным и/или искусственным аминокислотным составом в соответствии с 1, которые косвенно служат для получения белковых единиц, которые
а. модифицированы, например, гликозилированы, на поверхностях или/и
b. могут принимать функциональную конформацию на поверхностях.
3. Молекулы в соответствии с пунктами 1 и 2, которые косвенно образуются посредством кодирующих матриц и которые возникают из слияния, по меньшей мере, частичных единиц, по меньшей мере, одной из кодирующих матриц и которые могут быть получены из комбинации различных последовательностей и возможно обогащены и выделены, а также, возможно, одновременно могут быть процессированы
а. in vivo
b. in vitro.
4. Молекулы согласно пунктам 1, 2 и 3, которые представляют собой кодирующие молекулы нуклеиновой кислоты, которые экспрессируют их in vivo и in vitro по изобретению
i. in vivo рекомбинантно, внутригеномно (система TN7) или
ii. in vivo с помощью экстрахромосомных генетических элементов (векторов) или
iii. in vivo с помощью природных или неприродных молекул нуклеиновой кислоты (РНК и т.д.) с помощью замкнутых или линейных молекул нуклеиновой кислоты или
iv in vitro в связанных системах транскрипции или трансляции
v. in vivo с включением искусственных аминокислот.
5. Поверхности для закрепления молекул в соответствии с 1-3, которые представляют собой природные клеточные мембраны природных клеток или искусственных мембранных систем.
6. Молекулы в соответствии с 1-4, генные продукты, трансфицированные клетки или части клеток которых служат для решения структурных, биоаналитических и (ветеринарно-диагностических) вопросов в отношении специфического связывания различных видов молекул (малые молекулы (библиотеки активных ингредиентов, природные вещества), РНК, белки и ДНК) и/или катализа в биотестах, а также для создания систем биоанализа (биотеста), диагностических устройств, возможно, сопутствующих диагностических устройств и/или терапии по результатам.
7. Молекулы в соответствии с 1-4, генные продукты, трансфицированные клетки или части клеток которых служат для решения вопросов в области иммуноаналитики и иммунодиагностики, для решения иммуноаналитических и диагностических вопросов и, возможно, для их обработки.
8. Молекулы в соответствии с 1-4, генные продукты, трансфицированные клетки или части клеток которых служат для разработки аналитических высокопроизводительных систем или для их применения.
9. Молекулы в соответствии с 1-4, генные продукты, трансфицированные клетки или части клеток которых прямо или косвенно служат для разработки непосредственно используемых иммунотерапевтических агентов (поликлональные или моноклональные антитела или искусственные молекулы с аналогичными возможными применениями).
10. Молекулы в соответствии с 1-3, генные продукты, трансфицированные клетки или части клеток предоставляются третьим лицам в больших количествах, полученных на основе ферментации/биопродукции для применения и использования.
Список литературы
1. Berggard,T, Linse, S. & James, P. Methods for the detection and analysis of protein-protein interactions. Proteomics 7, 2833-2842, doi:10.1002/pmic.200700131 (2007).
2. Glick, B. S. & Malhotra, V. The curious status of the Golgi apparatus. Cell 95, 883-889 (1998).
3. Call, M. E. & Wucherpfennig, K. W. The T cell receptor: critical role of the membrane environment in receptor assembly and function. Annu Rev Immunol 23, 101-125, doi:10.1146/annurev.immunoI.23.021704.115625 (2005).
4. Sachs, J. N. & Engelman, D. M. Introduction to the membrane protein reviews: the interplay of structure, dynamics, and environment in membrane protein function. Annu Rev Biochem 75, 707-712, doi:10.1146/annurev.biochem.75.110105.142336 (2006).
5. Macher, B. A. & Yen, T.-Y. Proteins at membrane surfaces-a review of approaches. Molecular BioSystems 3, 705-713, doi:10.1039/B708581H (2007).
6. Lodish, H. F. Molecular cell biology. (W.H. Freeman, 2013).
7. Nicolson, G. L. The Fluid-Mosaic Model of Membrane Structure: still relevant to understanding the structure, function and dynamics of biological membranes after more than 40 years. Biochim Biophys Acta 1838,1451-1466, doi:10.1016/j.bbamem.2013.10.019 (2014).
8. Whitelegge, J. P. Integral membrane proteins and bilayer proteomics. Anal Chem 85, 2558-2568, doi:10.1021/ac303064a (2013).
9. Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science 228, 1315-1317 (1985)
10. Crameri, R., Jaussi, R., Menz, G. & Blaser, K. Display of expression products of cDNA libraries on phage surfaces. A versatile screening system for selective isolation of genes by specific gene-product/ligand interaction. Eur J Biochem 226, 53-58 (1994).
11. Gu, H. et al. A phage display system for studying the sequence determinants of protein folding. Protein Sci 4, 1108-1117, doi:10.1002/pro.5560040609 (1995).
12. Kehoe, J. W. & Kay, B. K. Filamentous phage display in the new millennium. Chem Rev 105, 4056-4072, doi:10.1021/cr000261r (2005).
13. Chen, W. & Georgiou, G. Cell-Surface display of heterologous proteins: From high-throughput screening to environmental applications. Biotechnol Bioeng 79, 496-503, doi:10.1002/bit.10407 (2002).
14. Hoischen, C. et al. Novel bacterial membrane surface display system using cell wall-Iess L-forms of Proteus mirabilis and Escherichia coli. Appl Environ Microbiol 68, 525-531 (2002).
15. Lee, S. Y., Choi, J. H. & Xu, Z. Microbial cell-surface display. Trends Biotechnol 21, 45-52 (2003).
16. Rutherford, N. & Mourez, M. Surface display of proteins by gram-negative bacterial autotransporters. Microb Cell Fact 5, 22, doi: 10.1186/1475-2859-5-22 (2006).
17. Yang, Z., Liu, Q., Wang, Q. & Zhang, Y. Novel bacterial surface display systems based on outer membrane anchoring elements from the marine bacterium Vibrio anguillarum. Appl Environ Microbiol 74, 4359-4365, doi:10.1128/AEM.02499-07 (2008).
18. van Bloois, E., Winter, R. T., Kolmar, H. & Fraaije, M. W. Decorating microbes: surface display of proteins on Escherichia coli. Trends Biotechnol 29, 79-86, doi:10.1016/j.tibtech.2010.11.003 (2011).
19. Nicolay, T., Vanderleyden, J. & Spaepen, S. Autotransporter-based cell surface display in Gram-negative bacteria. Crit Rev Microbiol, doi:10.3109/1040841X.2013.804032 (2013).
20. Han, M.-J. & Lee, S. H. An efficient bacterial surface display system based on a novel outer membrane anchoring element from the Escherichia coli protein YiaT. (2014).
21. Boublik, Y., Di Bonito, P. & Jones, I. M. Eukaryotic virus display: engineering the major surface glycoprotein of the Autographa californica nuclear polyhedrosis virus (AcNPV) for the presentation of foreign proteins on the virus surface. Biotechnology (N Y) 13, 1079-1084 (1995).
22. Grabherr, R., Ernst, W., Doblhoff-Dier, O., Sara, M. & Katinger, H. Expression of foreign proteins on the surface of Autographa californica nuclear polyhedrosis virus. BioTechniques 22, 730-735 (1997).
23. Kim, S. Y., Sohn, J. H., Pyun, Y. R. & Choi, E. S. A cell surface display system using novel GPI-anchored proteins in Hansenula polymorpha. Yeast 19, 1153-1163, doi:10.1002/yea.911 (2002).
24. Rahman, M. M. & Gopinathan, K. P. Bombyx mori nucleopolyhedrovirus-based surface display system for recombinant proteins. J Gen Virol 84, 2023-2031 (2003).
25. Baneyx, F. & Mujacic, M. Recombinant protein folding and misfolding in Escherichia coli. Nat Biotechnol 22, 1399-1408, doi:10.1038/nbt1029 (2004).
26. Raty, J. K. et al. Enhanced gene delivery by avidin-displaying baculovirus. Mol Ther 9, 282-291, doi:10.1016/j.ymthe.2003.11.004 (2004).
27. Mao, H., Song, J., Liang, C., Yu, Z. & Chen, X. Construction of eukaryotic surface display based on the baculoviral F protein. BioTechniques 41, 266, 268, 270 passim (2006).
28. Weerapana, E. & Imperiali, B. Asparagine-linked protein glycosylation: from eukaryotic to prokaryotic systems. Glycobiology 16, 91R-101R, doi:10.1093/glycob/cwj099 (2006).
29. Wang, Q., Li, L., Chen, M., Qi, Q. & Wang, P. G. Construction of a novel system for cell surface display of heterologous proteins on Pichia pastoris. Biotechnol Lett 29, 1561-1566, doi:10.1007/s10529-007-9430-6 (2007).
30. Chesnut, J. D. et al. Selective isolation of transiently transfected cells from a mammalian cell population with vectors expressing a membrane anchored single-chain antibody. J Immunol Methods 193, 17-27 (1996).
31. Ellmark, P., Ohlin, M., Borrebaeck, C. A. & Furebring, C. A novel mammalian display system for the selection of protein-protein interactions by decoy receptor engagement. J Mol Recognit 17, 316-322, doi:10.1002/jmr.678 (2004).
32. Zhou, C., Jacobsen, F. W., Cai, L., Chen, Q. & Shen, W. D. Development of a novel mammalian cell surface antibody display platform. MAbs 2, 508-518, doi:10.4161/mabs.2.5.12970 (2010).
33. Tomimatsu, K. et al. A rapid screening and production method using a novel mammalian cell display to isolate human monoclonal antibodies. Biochem Biophys Res Commun 441, 59-64, doi: 10.1016/j.bbrc.2013.10.007 (2013).
34. Stipp, C. S., Kolesnikova, T. V. & Hemler, M. E. Functional domains in tetraspanin proteins. Trends Biochem Sci 28, 106-112 (2003).
35. Kovalenko, O. V., Metcalf, D. G., DeGrado, W. F. & Hemler, M. E. Structural organization and interactions of transmembrane domains in tetraspanin proteins. BMC Struct Biol 5, 11, doi:10.1186/1472-6807-5-11 (2005).
36. Hemler, M. E. Targeting of tetraspanin proteins--potential benefits and strategies. Nat Rev Drug Discov 7, 747-758, doi:10.1038/nrd2659 (2008).
37. Levy, S. & Shoham, T. The tetraspanin web modulates immune-signalling complexes. Nat Rev Immunol 5, 136-148, doi:10.1038/nri1548 (2005).
38. Rubinstein, E. et al. CD9, CD63, CD81, and CD82 are components of a surface tetraspan network connected to HLA-DR and VLA integrins. Eur J Immunol 26, 2657-2665, doi:10.1002/eji.1830261117 (1996).
39. Green, M. R. & Sambrook, J. Molecular cloning: a laboratory manual. 1. (Cold Spring Harbor Laboratory Press, 2012).
40. Rognan, D. Rational design of protein-protein interaction inhibitors. MedChemComm 6, 51-60, doi:10.1039/C4MD00328D (2015).
41. in, L., Wang, W. & Fang, G. Targeting protein-protein interaction by small molecules. Annu Rev Pharmacol Toxicol 54, 435-456, doi:10.1146/annurev-pharmtox-011613-140028 (2014).
42. Milroy, L. G., Grossmann, T. N., Hennig, S., Brunsveld, L. & Ottmann, C. Modulators of protein-protein interactions. Chem Rev 114, 4695-4748, doi:10.1021/cr400698c (2014).
43. Rao, V. S., Srinivas, K., Sujini, G. N. & Kumar, G. N. Protein-protein interaction detection: methods and analysis. Int J Proteomics 2014, 147648, doi:10.1155/2014/147648 (2014).
44. Arkin, M. R. & Wells, J. A. Small-molecule inhibitors of protein-protein interactions: progressing towards the dream. Nat Rev Drug Discov 3, 301-317, doi:10.1038/nrd1343 (2004).
45. Jones, S. & Thornton, J. M. Principles of protein-protein interactions. Proc Natl Acad Sci U S A 93,13-20 (1996).
46. Jones, S. & Thornton, J. M. Protein-protein interactions: a review of protein dimer structures. Prog Biophys Mol Biol 63, 31-65 (1995).
47. Sudhof, T. C. The synaptic vesicle cycle: a cascade of protein-protein interactions. Nature 375, 645-653, doi:10.1038/375645a0 (1995).
48. Mullard, A. Protein-protein interaction inhibitors get into the groove. Nat Rev Drug Discov 11, 173-175, doi:10.1038/nrd3680 (2012).
49. Ivanusic, D., Eschricht, M. & Denner, J. Investigation of membrane protein-protein interactions using correlative FRET-PLA. BioTechniques 57,188-198, doi:10.2144/000114215 (2014).
50. Kremers, G. J., Goedhart, J., van Munster, E. B. & Gadella, T. W., Jr. Cyan and yellow super fluorescent proteins with improved brightness, protein folding, and FRET Forster radius. Biochemistry 45, 6570-6580, doi:10.1021/bi0516273 (2006).
51. Adachi, A. et al. Production of acquired immunodeficiency syndrome-associated retrovirus in human and nonhuman cells transfected with an infectious molecular clone. J Virol 59, 284-291 (1986).
52. Stéphanie Charrin, François le Naour, Olivier Silvie, Pierre-Emmanuel Milhiet, Claude Boucheix, Eric Rubinstein, Lateral organization of membrane proteins: tetraspanins spin their web, Bioch. J. (2009), 420 (2) 133-154; doi: 10.1042/BJ20082422.
53. A Voller, A Bartlett, D E Bidwell, Enzyme immunoassays with special reference to ELISA techniques, J Clin Pathol., 1978 June, 31(6): 507-520.
54. Hughes RA, Miklos AE, Ellington AD, Gene synthesis: methods and applications, Methods Enzymol. 2011; 498: 277-309, doi:10.2016/B978-0-12-385120-8.00012-7
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Peter und Traudl Engelhorn-Stiftung zur Fӧrderung der
Lebenswissenschaften
<120> System zur Praesentation von Peptiden auf der Zelloberflaeche
<130> 61317P WO
<150> DE102015002851
<151> 2015-03-05
<160> 46
<170> PatentIn version 3.5
<210> 1
<211> 4832
<212> DNA
<213> artificial sequence
<220>
<223> pA4E211(CD63delLEL); küenstliche Sequenz als Shuttle Vektor
von E.
coli nach Eukaryotischen Zellen (hier: Säugetierzellen)
<400> 1
cctcgaggaa ggctactacg cctaagctac cgacaggttg gctgataagt ccccggtctc 60
aaggcgcgcc tagttattaa tagtaatcaa ttacggggtc attagttcat agcccatata 120
tggagttccg cgttacataa cttacggtaa atggcccgcc tggctgaccg cccaacgacc 180
cccgcccatt gacgtcaata atgacgtatg ttcccatagt aacgccaata gggactttcc 240
attgacgtca atgggtggag tatttacggt aaactgccca cttggcagta catcaagtgt 300
atcatctgcc aagtacgccc cctattgacg tcaatgacgg taaatggccc gcctggcatt 360
atgcccagta catgacctta tgggactttc ctacttggca gtacatctac gtattagtca 420
tcgctattac cacggtgatg cggttttggc agtacatcaa tgggcgtgga tagcggtttg 480
actcacgggg atttccaagt ctccacccca ttgacgtcaa tgggagtttg ttttggcacc 540
aaaatcaacg ggactttcca aaatgtcgta acaactccgc cccattgacg caaatgggcg 600
gtaggcgtgt acggtgggag gtctatataa gcagagctct ctggctaact agagaaccca 660
ctgcttactg gcttatcgaa attaatacga ctcactatag gaagccgcca cctgctagca 720
atttatagcc accatggatt acaaggatga cgacgataag cccatggcgg tggaaggagg 780
aatgaaatgt gtgaagttct tgctctacgt cctcctgctg gccttttgcg cctgtgcagt 840
gggactgatt gccgtgggtg tcggggcaca gcttgtcctg agtcagacca taatccaggg 900
ggctacccct ggctctctgt tgccagtggt catcatcgca gtgggtgtct tcctcttcct 960
ggtggctttt gtgggctgct gcggggcctg caaggagaac tattgtctta tgattacgtt 1020
tgccatcttt ctgtctctta tcatgttggt ggaggtggcc gcagccattg ctggctatgt 1080
gtttagagat aagctgcaag aattcgagaa tctgtacttt cagtcgaagc ttggatccct 1140
gcagcatcac catcaccatc acgaaaacct ctatttccag agcgaacaga aactgatatc 1200
cgaagaagat ctggatatcg gaggcggagg caaaaatgtg ctggtggtag ctgccgcagc 1260
ccttggaatt gcttttgtcg aggttttggg aattgtcttt gcctgctgcc tcgtgaagag 1320
tatcagaagt ggctacgagg tgatgaaact tatcgatacc gtcgatctcc atatgccagc 1380
catgaagata gaatgcagga ttacaggcac cctgaatggc gtggagttcg agctggtcgg 1440
gggtggagaa ggaacacctg agcagggccg aatgacgaac aagatgaaga gcaccaaagg 1500
agccctgaca tttagcccct atctcctctc tcatgttatg ggctacggct tttatcactt 1560
cgggacttac ccaagcggat atgaaaaccc tttccttcac gccataaaca atgggggtta 1620
caccaacacc cggattgaga aatatgagga cggtggcgtg ttgcatgtct ccttcagcta 1680
cagatatgag gctggtcgcg tgatcggaga ttttaaggtt gtgggcacag gctttccaga 1740
agactccgta atcttcaccg acaagatcat tcggtctaat gccacggtgg agcatcttca 1800
ccctatgggc gataatgtcc tcgtcgggag ctttgccaga actttttcac tgagggatgg 1860
gggatactat tcattcgtcg tagactccca catgcacttt aaatccgcaa tccacccatc 1920
aatcctgcaa aacggagggc ctatgtttgc gttcagacgt gtagaggagc tgcattctaa 1980
cactgaactc ggaatcgttg aataccagca cgctttcaaa actcccattg cattcgctgg 2040
gccctgatca tgagagttta aacataaagc ctcgactgtg ccttctagtt gccagccatc 2100
tgttgtttgc ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct 2160
ttcctaataa aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg 2220
gggtggggtg gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg 2280
ggatgcggtg ggctctatgg aaaaaacgcg cgcggatata atataacttc gtataatgta 2340
tgctatacga agttatatat aatgcgtcga cgcactcaac cctatctcgg tctattcttt 2400
tgatttataa gggattttgc cgatttcggc ctattggtta aaaaatgagc tgatttaaca 2460
aaaatttaac gcgaatttta acaaaatatt aacgcttaca atttaggtgg cacttttcgg 2520
ggaaatgtgc gcggaacccc tatttgttta tttttctaaa tacattcaaa tatgtatccg 2580
ctcatgagac aataaccctg ataaatgctt caataatatt gaaaaaggaa gagtatgagt 2640
attcaacatt tccgtgtcgc ccttattccc ttttttgcgg cattttgcct tcctgttttt 2700
gctcacccag aaacgctggt gaaagtaaaa gatgctgaag atcagttggg tgcccgagtg 2760
ggttacatcg aactggatct caacagcggt aagatccttg agagttttcg ccccgaagaa 2820
cgttttccaa tgatgagcac ttttaaagtt ctgctatgtg gcgcggtatt atcccgtatt 2880
gacgccgggc aagagcaact cggtcgccgc atacactatt ctcagaatga cttggttgag 2940
tattcaccag tcacagaaaa gcatcttacg gatggcatga cagtaagaga attatgcagt 3000
gctgccataa ccatgagtga taacactgcg gccaacttac ttctgacaac gattggagga 3060
ccgaaggagc taaccgcttt tttgcacaac atgggggatc atgtaactcg ccttgatcgt 3120
tgggaaccgg agctgaatga agccatacca aacgacgagc gtgacaccac gatgcctgta 3180
gcaatggcaa caacgttgcg taaactatta actggcgaac tacttactct agcttcccgg 3240
caacaattga tagactggat ggaggcggat aaagttgcag gaccacttct gcgctcggcc 3300
cttccggctg gctggtttat tgctgataaa tctggagccg gtgagcgtgg ttctcgcggt 3360
atcattgcag cactggggcc agatggtaag ccctcccgta tcgtagttat ctacacgacg 3420
gggagtcagg caactatgga tgaacgaaat agacagatcg ctgagatagg tgcctcactg 3480
attaagcatt ggtaactgtc agaccaagtt tactcatata tactttagat tgatttaaaa 3540
cttcattttt aatttaaaag gatctaggtg aagatccttt ttgataatct tatgaccaaa 3600
atcccttaac gtgagttttc gttccactga gcgtcagacc gcgatcgcag cagaccgggg 3660
acttatcagc caacctgtcg ttaattaacc atgtcagccg ttaagtgttc ctgtgtcact 3720
caaaattgct ttgagaggct ctaagggctt ctcagtgcgt tacatccctg gcttgttgtc 3780
cacaaccgtt aaaccttaaa ggctttaaaa gccttatata ttcttttttt tcttataaaa 3840
cttaaaacct tagaggctat ttaagttgct gatttatatt aattgaagag cattatacca 3900
aaatccctta acgtgagttt tcgttccact gagcgtcaga ccccgtagaa aagatcaaag 3960
gatcttcttg agatcctttt tttctgcgcg taatatgctg cttgcaaaca aaaaaaccac 4020
cgctaccagc ggtggtttgt ttgccggatc aagagctacc aactcttttt ccgaaggtaa 4080
ctggcttcag cagagcgcag ataccaaata ctgtccttct agtgtagccg tagttaggcc 4140
accacttcaa gaactctgta gcaccgccta catacctcgc tctgctaatc ctgttaccag 4200
tggctgctgc cagtggcgat aagtcgtgtc ttaccgggtt ggactcaaga cgatagttac 4260
cggataaggc gcagcggtcg ggctgaacgg ggggttcgtg cacacagccc agcttggagc 4320
gaacgaccta caccgaactg agatacctac agcgtgagct atgagaaagc gccacgcttc 4380
ccgaagggag aaaggcggac aggtatccgg taagcggcag ggtcggaaca ggagagcgca 4440
cgagggagct tccaggggga aacgcctggt atctttatag tcctgtcggg tttcgccacc 4500
tctgacttga gcgtcgattt ttgtgatgct cgtcaggggg gcggagccta tggaaaaacg 4560
ccagcaacgc ggccttttta cggttcctgg ccttttgctg gccttttgct catattagct 4620
cttcaaattt tattgttcaa acatgagagc ttagtacgtg aaacatgaga gcttagtacg 4680
ttagccatga gagcttagta cgttagccat gagggtttag ttcgttaaac atgagagctt 4740
agtacgttaa acatgagagc ttagtacgtg aaacatgaga gcttagtacg tactatcaac 4800
aggttgaact gctgatcttc agatcatata at 4832
<210> 2
<211> 474
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetische DNA Sequenz des humanen CD63 Gens (CD63delLEL);
Eukaryotische Zellen, hier: Säugetierzellen); kodierende Sequenz
von SEQ ID NO:1; Synthetisches CD63 - Codonen
<220>
<221> misc_feature
<222> (1)..(330)
<223> TM1-TM3
<220>
<221> misc_feature
<222> (331)..(360)
<223> Minimallinker
<220>
<221> misc_feature
<222> (361)..(474)
<223> TM4
<400> 2
atggcggtgg aaggaggaat gaaatgtgtg aagttcttgc tctacgtcct cctgctggcc 60
ttttgcgcct gtgcagtggg actgattgcc gtgggtgtcg gggcacagct tgtcctgagt 120
cagaccataa tccagggggc tacccctggc tctctgttgc cagtggtcat catcgcagtg 180
ggtgtcttcc tcttcctggt ggcttttgtg ggctgctgcg gggcctgcaa ggagaactat 240
tgtcttatga tcacgtttgc catctttctg tctcttatca tgttggtgga ggtggccgca 300
gccattgctg gctatgtgtt tagagataag ctgcaggaat tcgatatcgg aggaggagga 360
aaaaatgtgc tggtggtagc tgcagcagcc cttggaattg cttttgtcga ggttttggga 420
attgtctttg cctgctgcct cgtgaagagt atcagaagtg gctacgaggt gatg 474
<210> 3
<211> 158
<212> PRT
<213> artificial sequence
<220>
<223> CD63delLEL; Künstliche Sequenz im Shuttle Vektor - E. coli -
Baculovirus/Insekten Zellen; translatierte Sequenz von SEQ ID
NO:2
<220>
<221> MISC_FEATURE
<222> (1)..(110)
<223> TM1-TM3
<220>
<221> MISC_FEATURE
<222> (111)..(120)
<223> Minimallinker
<220>
<221> MISC_FEATURE
<222> (121)..(158)
<223> TM4
<400> 3
Met Ala Val Glu Gly Gly Met Lys Cys Val Lys Phe Leu Leu Tyr Val
1 5 10 15
Leu Leu Leu Ala Phe Cys Ala Cys Ala Val Gly Leu Ile Ala Val Gly
20 25 30
Val Gly Ala Gln Leu Val Leu Ser Gln Thr Ile Ile Gln Gly Ala Thr
35 40 45
Pro Gly Ser Leu Leu Pro Val Val Ile Ile Ala Val Gly Val Phe Leu
50 55 60
Phe Leu Val Ala Phe Val Gly Cys Cys Gly Ala Cys Lys Glu Asn Tyr
65 70 75 80
Cys Leu Met Ile Thr Phe Ala Ile Phe Leu Ser Leu Ile Met Leu Val
85 90 95
Glu Val Ala Ala Ala Ile Ala Gly Tyr Val Phe Arg Asp Lys Leu Gln
100 105 110
Glu Phe Asp Ile Gly Gly Gly Gly Lys Asn Val Leu Val Val Ala Ala
115 120 125
Ala Ala Leu Gly Ile Ala Phe Val Glu Val Leu Gly Ile Val Phe Ala
130 135 140
Cys Cys Leu Val Lys Ser Ile Arg Ser Gly Tyr Glu Val Met
145 150 155
<210> 4
<211> 30
<212> DNA
<213> artificial sequence
<220>
<223> PA1-01; konstruktiv s. Patenttext
<400> 4
tttttctcga gatggtgagc aagggcgagg 30
<210> 5
<211> 32
<212> DNA
<213> artificial sequence
<220>
<223> PA1-02; konstruktiv s. Patenttext
<400> 5
tttttgggcc catcttgtac agctcgtcca tg 32
<210> 6
<211> 33
<212> DNA
<213> artificial sequence
<220>
<223> PA1-03; konstruktiv s. Patenttext
<400> 6
ttttttggat ccatggcggt ggaaggagga atg 33
<210> 7
<211> 27
<212> DNA
<213> artificial sequence
<220>
<223> PA1-04; konstruktiv s. Patenttext
<400> 7
tttttctgca gcttatctct aaacaca 27
<210> 8
<211> 36
<212> DNA
<213> artificial sequence
<220>
<223> PA1-05; konstruktiv s. Patenttext
<400> 8
tttttgatat cggaggagga ggaaaaaatg tgctgg 36
<210> 9
<211> 33
<212> DNA
<213> artificial sequence
<220>
<223> PA1-06; konstruktiv s. Patenttext
<400> 9
ttttttaagc ttcatcacct cgtagccact tct 33
<210> 10
<211> 31
<212> DNA
<213> artificial sequence
<220>
<223> PA1-07; konstruktiv s. Patenttext
<400> 10
tttttgaatt catggtgagc aagggcgagg a 31
<210> 11
<211> 31
<212> DNA
<213> artificial sequence
<220>
<223> PA1-08; konstruktiv s. Patenttext
<400> 11
tttttgatat cctttctgag tccggacttg t 31
<210> 12
<211> 30
<212> DNA
<213> artificial sequence
<220>
<223> PA1-09; konstruktiv s. Patenttext
<400> 12
tttttgaatt cctgacggta caggccagac 30
<210> 13
<211> 31
<212> DNA
<213> artificial sequence
<220>
<223> PA1-10; konstruktiv s. Patenttext
<400> 13
tttttgatat cgttaaacca attccacaaa c 31
<210> 14
<211> 30
<212> DNA
<213> artificial sequence
<220>
<223> PA1-11; konstruktiv s. Patenttext
<400> 14
tttttgatat cctgcctaac tctattcact 30
<210> 15
<211> 30
<212> DNA
<213> artificial sequence
<220>
<223> PA1-12; konstruktiv s. Patenttext
<400> 15
tttttgaatt cattgaagaa tcgcaaaacc 30
<210> 16
<211> 31
<212> DNA
<213> artificial sequence
<220>
<223> PA1-13; konstruktiv s. Patenttext
<400> 16
tttttgatat cttttatata ccacagccaa t 31
<210> 17
<211> 714
<212> DNA
<213> homo sapiens
<220>
<221> misc_feature
<222> (1)..(3)
<223> Startkodon
<400> 17
atggcggtgg aaggaggaat gaaatgtgtg aagttcttgc tctacgtcct cctgctggcc 60
ttttgcgcct gtgcagtggg actgattgcc gtgggtgtcg gggcacagct tgtcctgagt 120
cagaccataa tccagggggc tacccctggc tctctgttgc cagtggtcat catcgcagtg 180
ggtgtcttcc tcttcctggt ggcttttgtg ggctgctgcg gggcctgcaa ggagaactat 240
tgtcttatga tcacgtttgc catctttctg tctcttatca tgttggtgga ggtggccgca 300
gccattgctg gctatgtgtt tagagataag gtgatgtcag agtttaataa caacttccgg 360
cagcagatgg agaattaccc gaaaaacaac cacactgctt cgatcctgga caggatgcag 420
gcagatttta agtgctgtgg ggctgctaac tacacagatt gggagaaaat cccttccatg 480
tcgaagaacc gagtccccga ctcctgctgc attgatgtta ctgtgggctg tgggattaat 540
ttcaacgaga aggcgatcca taaggagggc tgtgtggaga agattggggg ctggctgagg 600
aaaaatgtgc tggtggtagc tgcagcagcc cttggaattg cttttgtcga ggttttggga 660
attgtctttg cctgctgcct cgtgaagagt atcagaagtg gctacgaggt gatg 714
<210> 18
<211> 238
<212> PRT
<213> homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(238)
<223> CD63; translatierte Sequenz zu SEQ ID NO:17 (Genbank Accession
No: KF998086)
<400> 18
Met Ala Val Glu Gly Gly Met Lys Cys Val Lys Phe Leu Leu Tyr Val
1 5 10 15
Leu Leu Leu Ala Phe Cys Ala Cys Ala Val Gly Leu Ile Ala Val Gly
20 25 30
Val Gly Ala Gln Leu Val Leu Ser Gln Thr Ile Ile Gln Gly Ala Thr
35 40 45
Pro Gly Ser Leu Leu Pro Val Val Ile Ile Ala Val Gly Val Phe Leu
50 55 60
Phe Leu Val Ala Phe Val Gly Cys Cys Gly Ala Cys Lys Glu Asn Tyr
65 70 75 80
Cys Leu Met Ile Thr Phe Ala Ile Phe Leu Ser Leu Ile Met Leu Val
85 90 95
Glu Val Ala Ala Ala Ile Ala Gly Tyr Val Phe Arg Asp Lys Val Met
100 105 110
Ser Glu Phe Asn Asn Asn Phe Arg Gln Gln Met Glu Asn Tyr Pro Lys
115 120 125
Asn Asn His Thr Ala Ser Ile Leu Asp Arg Met Gln Ala Asp Phe Lys
130 135 140
Cys Cys Gly Ala Ala Asn Tyr Thr Asp Trp Glu Lys Ile Pro Ser Met
145 150 155 160
Ser Lys Asn Arg Val Pro Asp Ser Cys Cys Ile Asp Val Thr Val Gly
165 170 175
Cys Gly Ile Asn Phe Asn Glu Lys Ala Ile His Lys Glu Gly Cys Val
180 185 190
Glu Lys Ile Gly Gly Trp Leu Arg Lys Asn Val Leu Val Val Ala Ala
195 200 205
Ala Ala Leu Gly Ile Ala Phe Val Glu Val Leu Gly Ile Val Phe Ala
210 215 220
Cys Cys Leu Val Lys Ser Ile Arg Ser Gly Tyr Glu Val Met
225 230 235
<210> 19
<211> 771
<212> DNA
<213> homo sapiens
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<400> 19
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgccggtca aaggaggcac caagtgcatc aaatacctgc tgttcggatt taacttcatc 120
ttctggcttg ccgggattgc tgtccttgcc attggactat ggctccgatt cgactctcag 180
accaagagca tcttcgagca agaaactaat aataataatt ccagcttcta cacaggagtc 240
tatattctga tcggagccgg cgccctcatg atgctggtgg gcttcctggg ctgctgcggg 300
gctgtgcagg agtcccagtg catgctggga ctgttcttcg gcttcctctt ggtgatattc 360
gccattgaaa tagctgcggc catctgggga tattcccaca aggatgaggt gattaaggaa 420
gtccaggagt tttacaagga cacctacaac aagctgaaaa ccaaggatga gccccagcgg 480
gaaacgctga aagccatcca ctatgcgttg aactgctgtg gtttggctgg gggcgtggaa 540
cagtttatct cagacatctg ccccaagaag gacgtactcg aaaccttcac cgtgaagtcc 600
tgtcctgatg ccatcaaaga ggtcttcgac aataaattcc acatcatcgg cgcagtgggc 660
atcggcattg ccgtggtcat gatatttggc atgatcttca gtatgatctt gtgctgtgct 720
atccgcagga accgcgagat ggtcaagctt atcgataccg tcgacctcga g 771
<210> 20
<211> 257
<212> PRT
<213> homo sapiens
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(257)
<223> CD9; translatierte Sequenz zu SEQ ID NO:19 (Genbank Accession
No: NM_001769.3)
<400> 20
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Pro Val Lys Gly Gly Thr Lys Cys Ile Lys Tyr
20 25 30
Leu Leu Phe Gly Phe Asn Phe Ile Phe Trp Leu Ala Gly Ile Ala Val
35 40 45
Leu Ala Ile Gly Leu Trp Leu Arg Phe Asp Ser Gln Thr Lys Ser Ile
50 55 60
Phe Glu Gln Glu Thr Asn Asn Asn Asn Ser Ser Phe Tyr Thr Gly Val
65 70 75 80
Tyr Ile Leu Ile Gly Ala Gly Ala Leu Met Met Leu Val Gly Phe Leu
85 90 95
Gly Cys Cys Gly Ala Val Gln Glu Ser Gln Cys Met Leu Gly Leu Phe
100 105 110
Phe Gly Phe Leu Leu Val Ile Phe Ala Ile Glu Ile Ala Ala Ala Ile
115 120 125
Trp Gly Tyr Ser His Lys Asp Glu Val Ile Lys Glu Val Gln Glu Phe
130 135 140
Tyr Lys Asp Thr Tyr Asn Lys Leu Lys Thr Lys Asp Glu Pro Gln Arg
145 150 155 160
Glu Thr Leu Lys Ala Ile His Tyr Ala Leu Asn Cys Cys Gly Leu Ala
165 170 175
Gly Gly Val Glu Gln Phe Ile Ser Asp Ile Cys Pro Lys Lys Asp Val
180 185 190
Leu Glu Thr Phe Thr Val Lys Ser Cys Pro Asp Ala Ile Lys Glu Val
195 200 205
Phe Asp Asn Lys Phe His Ile Ile Gly Ala Val Gly Ile Gly Ile Ala
210 215 220
Val Val Met Ile Phe Gly Met Ile Phe Ser Met Ile Leu Cys Cys Ala
225 230 235 240
Ile Arg Arg Asn Arg Glu Met Val Lys Leu Ile Asp Thr Val Asp Leu
245 250 255
Glu
<210> 21
<211> 795
<212> DNA
<213> homo sapiens
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<400> 21
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggagtgg agggctgcac caagtgcatc aagtacctgc tcttcgtctt caatttcgtc 120
ttctggctgg ctggaggcgt gatcctgggt gtggccctgt ggctccgcca tgacccgcag 180
accaccaacc tcctgtatct ggagctggga gacaagcccg cgcccaacac cttctatgta 240
ggcatctaca tcctcatcgc tgtgggcgct gtcatgatgt tcgttggctt cctgggctgc 300
tacggggcca tccaggaatc ccagtgcctg ctggggacgt tcttcacctg cctggtcatc 360
ctgtttgcct gtgaggtggc cgccggcatc tggggctttg tcaacaagga ccagatcgcc 420
aaggatgtga agcagttcta tgaccaggcc ctacagcagg ccgtggtgga tgatgacgcc 480
aacaacgcga aggctgtggt gaagaccttc cacgagacgc ttgactgctg tggctccagc 540
acactgactg ctttgaccac ctcagtgctc aagaacaatt tgtgtccctc gggcagcaac 600
atcatcagca acctcttcaa ggaggactgc caccagaaga tcgacgacct cttctccggg 660
aagctgtacc tcatcggcat tgctgccatc gtggtcgctg tgatcatgat cttcgagatg 720
atcctgagca tggtgctgtg ctgtggcatc cggaacagct ccgtgtacaa gcttatcgat 780
accgtcgacc tcgag 795
<210> 22
<211> 265
<212> PRT
<213> homo sapiens
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(265)
<223> CD81; translatierte Sequenz zu SEQ ID NO: 21 (Genbank Accession
No: NM_004356.3)
<400> 22
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Val Glu Gly Cys Thr Lys Cys Ile Lys Tyr
20 25 30
Leu Leu Phe Val Phe Asn Phe Val Phe Trp Leu Ala Gly Gly Val Ile
35 40 45
Leu Gly Val Ala Leu Trp Leu Arg His Asp Pro Gln Thr Thr Asn Leu
50 55 60
Leu Tyr Leu Glu Leu Gly Asp Lys Pro Ala Pro Asn Thr Phe Tyr Val
65 70 75 80
Gly Ile Tyr Ile Leu Ile Ala Val Gly Ala Val Met Met Phe Val Gly
85 90 95
Phe Leu Gly Cys Tyr Gly Ala Ile Gln Glu Ser Gln Cys Leu Leu Gly
100 105 110
Thr Phe Phe Thr Cys Leu Val Ile Leu Phe Ala Cys Glu Val Ala Ala
115 120 125
Gly Ile Trp Gly Phe Val Asn Lys Asp Gln Ile Ala Lys Asp Val Lys
130 135 140
Gln Phe Tyr Asp Gln Ala Leu Gln Gln Ala Val Val Asp Asp Asp Ala
145 150 155 160
Asn Asn Ala Lys Ala Val Val Lys Thr Phe His Glu Thr Leu Asp Cys
165 170 175
Cys Gly Ser Ser Thr Leu Thr Ala Leu Thr Thr Ser Val Leu Lys Asn
180 185 190
Asn Leu Cys Pro Ser Gly Ser Asn Ile Ile Ser Asn Leu Phe Lys Glu
195 200 205
Asp Cys His Gln Lys Ile Asp Asp Leu Phe Ser Gly Lys Leu Tyr Leu
210 215 220
Ile Gly Ile Ala Ala Ile Val Val Ala Val Ile Met Ile Phe Glu Met
225 230 235 240
Ile Leu Ser Met Val Leu Cys Cys Gly Ile Arg Asn Ser Ser Val Tyr
245 250 255
Lys Leu Ile Asp Thr Val Asp Leu Glu
260 265
<210> 23
<211> 888
<212> DNA
<213> homo sapiens
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<400> 23
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggctcag cctgtatcaa agtcaccaaa tactttctct tcctcttcaa cttgatcttc 120
tttatcctgg gcgcagtgat cctgggcttc ggggtgtgga tcctggccga caagagcagt 180
ttcatctctg tcctgcaaac ctcctccagc tcgcttagga tgggggccta tgtcttcatc 240
ggcgtggggg cagtcactat gctcatgggc ttcctgggct gcatcggcgc cgtcaacgag 300
gtccgctgcc tgctggggct gtactttgct ttcctgctcc tgatcctcat tgcccaggtg 360
acggccgggg cactcttcta cttcaacatg ggcaagctga agcaggagat gggtggcatc 420
gtgactgagc tcattcgaga ctacaacagc agtcgcgagg acagcctgca ggatgcctgg 480
gactacgtgc aggctcaggt gaagtgctgc ggctgggtca gcttctacaa ctggacagac 540
aacgctgagc tcatgaatcg ccctgaggtc acctacccct gttcctgcga agtcaagggg 600
gaagaggaca acagcctttc tgtgaggaag ggcttctgcg aggcccccgg caacaggacc 660
cagagtggca accaccctga ggactggcct gtgtaccagg agggctgcat ggagaaggtg 720
caggcgtggc tgcaggagaa cctgggcatc atcctcggcg tgggcgtggg tgtggccatg 780
gtcgagctcc tggggatggt cctgtccatc tgcttgtgcc ggcacgtgca ttccgaagac 840
tacagcaagg tccccaagta caagcttatc gataccgtcg acctcgag 888
<210> 24
<211> 296
<212> PRT
<213> homo sapiens
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(296)
<223> CD82; translatierte Sequenz zu SEQ ID NO: 23 (Genbank Accession
No: NM_002231.3)
<400> 24
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Ser Ala Cys Ile Lys Val Thr Lys Tyr Phe
20 25 30
Leu Phe Leu Phe Asn Leu Ile Phe Phe Ile Leu Gly Ala Val Ile Leu
35 40 45
Gly Phe Gly Val Trp Ile Leu Ala Asp Lys Ser Ser Phe Ile Ser Val
50 55 60
Leu Gln Thr Ser Ser Ser Ser Leu Arg Met Gly Ala Tyr Val Phe Ile
65 70 75 80
Gly Val Gly Ala Val Thr Met Leu Met Gly Phe Leu Gly Cys Ile Gly
85 90 95
Ala Val Asn Glu Val Arg Cys Leu Leu Gly Leu Tyr Phe Ala Phe Leu
100 105 110
Leu Leu Ile Leu Ile Ala Gln Val Thr Ala Gly Ala Leu Phe Tyr Phe
115 120 125
Asn Met Gly Lys Leu Lys Gln Glu Met Gly Gly Ile Val Thr Glu Leu
130 135 140
Ile Arg Asp Tyr Asn Ser Ser Arg Glu Asp Ser Leu Gln Asp Ala Trp
145 150 155 160
Asp Tyr Val Gln Ala Gln Val Lys Cys Cys Gly Trp Val Ser Phe Tyr
165 170 175
Asn Trp Thr Asp Asn Ala Glu Leu Met Asn Arg Pro Glu Val Thr Tyr
180 185 190
Pro Cys Ser Cys Glu Val Lys Gly Glu Glu Asp Asn Ser Leu Ser Val
195 200 205
Arg Lys Gly Phe Cys Glu Ala Pro Gly Asn Arg Thr Gln Ser Gly Asn
210 215 220
His Pro Glu Asp Trp Pro Val Tyr Gln Glu Gly Cys Met Glu Lys Val
225 230 235 240
Gln Ala Trp Leu Gln Glu Asn Leu Gly Ile Ile Leu Gly Val Gly Val
245 250 255
Gly Val Ala Met Val Glu Leu Leu Gly Met Val Leu Ser Ile Cys Leu
260 265 270
Cys Arg His Val His Ser Glu Asp Tyr Ser Lys Val Pro Lys Tyr Lys
275 280 285
Leu Ile Asp Thr Val Asp Leu Glu
290 295
<210> 25
<211> 846
<212> DNA
<213> homo sapiens
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<400> 25
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggtgagt tcaacgagaa gaagacaaca tgtggcaccg tttgcctcaa gtacctgctg 120
tttacctaca attgctgctt ctggctggct ggcctggctg tcatggcagt gggcatctgg 180
acgctggccc tcaagagtga ctacatcagc ctgctggcct caggcaccta cctggccaca 240
gcctacatcc tggtggtggc gggcactgtc gtcatggtga ctggggtctt gggctgctgc 300
gccaccttca aggagcgtcg gaacctgctg cgcctgtact tcatcctgct cctcatcatc 360
tttctgctgg agatcatcgc tggtatcctc gcctacgcct actaccagca gctgaacacg 420
gagctcaagg agaacctgaa ggacaccatg accaagcgct accaccagcc gggccatgag 480
gctgtgacca gcgctgtgga ccagctgcag caggagttcc actgctgtgg cagcaacaac 540
tcacaggact ggcgagacag tgagtggatc cgctcacagg aggccggtgg ccgtgtggtc 600
ccagacagct gctgcaagac ggtggtggct ctttgtggac agcgagacca tgcctccaac 660
atctacaagg tggagggcgg ctgcatcacc aagttggaga ccttcatcca ggagcacctg 720
agggtcattg gggctgtggg gatcggcatt gcctgtgtgc aggtctttgg catgatcttc 780
acgtgctgcc tgtacaggag tctcaagctg gagcactaca agcttatcga taccgtcgac 840
ctcgag 846
<210> 26
<211> 282
<212> PRT
<213> homo sapiens
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startkodon
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (8)..(282)
<223> CD151; translatierte Sequenz zu SEQ ID No: 25 (Genbank Accession
No: BT007397.1)
<400> 26
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Glu Phe Asn Glu Lys Lys Thr Thr Cys Gly
20 25 30
Thr Val Cys Leu Lys Tyr Leu Leu Phe Thr Tyr Asn Cys Cys Phe Trp
35 40 45
Leu Ala Gly Leu Ala Val Met Ala Val Gly Ile Trp Thr Leu Ala Leu
50 55 60
Lys Ser Asp Tyr Ile Ser Leu Leu Ala Ser Gly Thr Tyr Leu Ala Thr
65 70 75 80
Ala Tyr Ile Leu Val Val Ala Gly Thr Val Val Met Val Thr Gly Val
85 90 95
Leu Gly Cys Cys Ala Thr Phe Lys Glu Arg Arg Asn Leu Leu Arg Leu
100 105 110
Tyr Phe Ile Leu Leu Leu Ile Ile Phe Leu Leu Glu Ile Ile Ala Gly
115 120 125
Ile Leu Ala Tyr Ala Tyr Tyr Gln Gln Leu Asn Thr Glu Leu Lys Glu
130 135 140
Asn Leu Lys Asp Thr Met Thr Lys Arg Tyr His Gln Pro Gly His Glu
145 150 155 160
Ala Val Thr Ser Ala Val Asp Gln Leu Gln Gln Glu Phe His Cys Cys
165 170 175
Gly Ser Asn Asn Ser Gln Asp Trp Arg Asp Ser Glu Trp Ile Arg Ser
180 185 190
Gln Glu Ala Gly Gly Arg Val Val Pro Asp Ser Cys Cys Lys Thr Val
195 200 205
Val Ala Leu Cys Gly Gln Arg Asp His Ala Ser Asn Ile Tyr Lys Val
210 215 220
Glu Gly Gly Cys Ile Thr Lys Leu Glu Thr Phe Ile Gln Glu His Leu
225 230 235 240
Arg Val Ile Gly Ala Val Gly Ile Gly Ile Ala Cys Val Gln Val Phe
245 250 255
Gly Met Ile Phe Thr Cys Cys Leu Tyr Arg Ser Leu Lys Leu Glu His
260 265 270
Tyr Lys Leu Ile Asp Thr Val Asp Leu Glu
275 280
<210> 27
<211> 648
<212> DNA
<213> artificial sequence
<220>
<223> CD9delLEL-V5-6xHis; basierend auf Genbank Accession No.:
NM_001769.3
<220>
<221> misc_feature
<222> (18)..(405)
<223> TM1-TM3
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<220>
<221> misc_feature
<222> (406)..(507)
<223> V5-6xHis-Linker
<220>
<221> misc_feature
<222> (508)..(648)
<223> TM4
<400> 27
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgccggtca aaggaggcac caagtgcatc aaatacctgc tgttcggatt taacttcatc 120
ttctggcttg ccgggattgc tgtccttgcc attggactat ggctccgatt cgactctcag 180
accaagagca tcttcgagca agaaactaat aataataatt ccagcttcta cacaggagtc 240
tatattctga tcggagccgg cgccctcatg atgctggtgg gcttcctggg ctgctgcggg 300
gctgtgcagg agtcccagtg catgctggga ctgttcttcg gcttcctctt ggtgatattc 360
gccattgaaa tagctgcggc catctgggga tattcccaca aggatgaatt cggaaagggc 420
ccgcggttcg aaggtaagcc tatccctaac cctctcctcg gtctcgattc tacgcgtacc 480
ggtcatcatc accatcacca tgatatcgga ggaggaggaa tcatcggcgc agtgggcatc 540
ggcattgccg tggtcatgat atttggcatg atcttcagta tgatcttgtg ctgtgctatc 600
cgcaggaacc gcgagatggt caagcttatc gataccgtcg acctcgag 648
<210> 28
<211> 216
<212> PRT
<213> artificial sequence
<220>
<223> CD9delLEL-V5-6xHis; translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(135)
<223> TM1-TM3
<220>
<221> MISC_FEATURE
<222> (7)..(216)
<223> CD9delLEL-V5-6xHis
<220>
<221> MISC_FEATURE
<222> (136)..(169)
<223> V5-6xHis-Linker
<220>
<221> MISC_FEATURE
<222> (170)..(216)
<223> TM4
<400> 28
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Pro Val Lys Gly Gly Thr Lys Cys Ile Lys Tyr
20 25 30
Leu Leu Phe Gly Phe Asn Phe Ile Phe Trp Leu Ala Gly Ile Ala Val
35 40 45
Leu Ala Ile Gly Leu Trp Leu Arg Phe Asp Ser Gln Thr Lys Ser Ile
50 55 60
Phe Glu Gln Glu Thr Asn Asn Asn Asn Ser Ser Phe Tyr Thr Gly Val
65 70 75 80
Tyr Ile Leu Ile Gly Ala Gly Ala Leu Met Met Leu Val Gly Phe Leu
85 90 95
Gly Cys Cys Gly Ala Val Gln Glu Ser Gln Cys Met Leu Gly Leu Phe
100 105 110
Phe Gly Phe Leu Leu Val Ile Phe Ala Ile Glu Ile Ala Ala Ala Ile
115 120 125
Trp Gly Tyr Ser His Lys Asp Glu Phe Gly Lys Gly Pro Arg Phe Glu
130 135 140
Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr Arg Thr
145 150 155 160
Gly His His His His His His Asp Ile Gly Gly Gly Gly Ile Ile Gly
165 170 175
Ala Val Gly Ile Gly Ile Ala Val Val Met Ile Phe Gly Met Ile Phe
180 185 190
Ser Met Ile Leu Cys Cys Ala Ile Arg Arg Asn Arg Glu Met Val Lys
195 200 205
Leu Ile Asp Thr Val Asp Leu Glu
210 215
<210> 29
<211> 663
<212> DNA
<213> artificial sequence
<220>
<223> CD81delLEL-V5-6xHis; basierend auf Genbank Accession No.:
NM_004356.3
<220>
<221> misc_feature
<222> (18)..(413)
<223> TM1-TM3
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<220>
<221> misc_feature
<222> (414)..(516)
<223> V5-6xHis-Linker
<220>
<221> misc_feature
<222> (517)..(663)
<223> TM4
<400> 29
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggagtgg agggctgcac caagtgcatc aagtacctgc tcttcgtctt caatttcgtc 120
ttctggctgg ctggaggcgt gatcctgggt gtggccctgt ggctccgcca tgacccgcag 180
accaccaacc tcctgtatct ggagctggga gacaagcccg cgcccaacac cttctatgta 240
ggcatctaca tcctcatcgc tgtgggcgct gtcatgatgt tcgttggctt cctgggctgc 300
tacggggcca tccaggaatc ccagtgcctg ctggggacgt tcttcacctg cctggtcatc 360
ctgtttgcct gtgaggtggc cgccggcatc tggggctttg tcaacaagga ccaggaattc 420
ggaaagggcc cgcggttcga aggtaagcct atccctaacc ctctcctcgg tctcgattct 480
acgcgtaccg gtcatcatca ccatcaccat gatatcggag gaggaggaaa gctgtacctc 540
atcggcattg ctgccatcgt ggtcgctgtg atcatgatct tcgagatgat cctgagcatg 600
gtgctgtgct gtggcatccg gaacagctcc gtgtacaagc ttatcgatac cgtcgacctc 660
gag 663
<210> 30
<211> 221
<212> PRT
<213> artificial sequence
<220>
<223> CD81delLEL-V5-6xHis; translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startkodon
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(138)
<223> TM1-TM3
<220>
<221> MISC_FEATURE
<222> (7)..(221)
<223> CD81delLEL-V5-6xHis
<220>
<221> MISC_FEATURE
<222> (139)..(172)
<223> V5-6xHis-Linker
<220>
<221> MISC_FEATURE
<222> (173)..(221)
<223> TM4
<400> 30
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Val Glu Gly Cys Thr Lys Cys Ile Lys Tyr
20 25 30
Leu Leu Phe Val Phe Asn Phe Val Phe Trp Leu Ala Gly Gly Val Ile
35 40 45
Leu Gly Val Ala Leu Trp Leu Arg His Asp Pro Gln Thr Thr Asn Leu
50 55 60
Leu Tyr Leu Glu Leu Gly Asp Lys Pro Ala Pro Asn Thr Phe Tyr Val
65 70 75 80
Gly Ile Tyr Ile Leu Ile Ala Val Gly Ala Val Met Met Phe Val Gly
85 90 95
Phe Leu Gly Cys Tyr Gly Ala Ile Gln Glu Ser Gln Cys Leu Leu Gly
100 105 110
Thr Phe Phe Thr Cys Leu Val Ile Leu Phe Ala Cys Glu Val Ala Ala
115 120 125
Gly Ile Trp Gly Phe Val Asn Lys Asp Gln Glu Phe Gly Lys Gly Pro
130 135 140
Arg Phe Glu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser
145 150 155 160
Thr Arg Thr Gly His His His His His His Asp Ile Gly Gly Gly Gly
165 170 175
Lys Leu Tyr Leu Ile Gly Ile Ala Ala Ile Val Val Ala Val Ile Met
180 185 190
Ile Phe Glu Met Ile Leu Ser Met Val Leu Cys Cys Gly Ile Arg Asn
195 200 205
Ser Ser Val Tyr Lys Leu Ile Asp Thr Val Asp Leu Glu
210 215 220
<210> 31
<211> 690
<212> DNA
<213> artificial sequence
<220>
<223> CD82delLEL-V5-6xHis; basierend auf Genbank Accession No.:
NM_002231.3
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<220>
<221> misc_feature
<222> (18)..(405)
<223> TM1-TM3
<220>
<221> misc_feature
<222> (406)..(507)
<223> V5-6xHis-Linker
<220>
<221> misc_feature
<222> (508)..(690)
<223> TM4
<400> 31
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggctcag cctgtatcaa agtcaccaaa tactttctct tcctcttcaa cttgatcttc 120
tttatcctgg gcgcagtgat cctgggcttc ggggtgtgga tcctggccga caagagcagt 180
ttcatctctg tcctgcaaac ctcctccagc tcgcttagga tgggggccta tgtcttcatc 240
ggcgtggggg cagtcactat gctcatgggc ttcctgggct gcatcggcgc cgtcaacgag 300
gtccgctgcc tgctggggct gtactttgct ttcctgctcc tgatcctcat tgcccaggtg 360
acggccgggg cactcttcta cttcaacatg ggcaagctga agcaggaatt cggaaagggc 420
ccgcggttcg aaggtaagcc tatccctaac cctctcctcg gtctcgattc tacgcgtacc 480
ggtcatcatc accatcacca tgatatcgga ggaggaggag tgcaggcgtg gctgcaggag 540
aacctgggca tcatcctcgg cgtgggcgtg ggtgtggcca tggtcgagct cctggggatg 600
gtcctgtcca tctgcttgtg ccggcacgtg cattccgaag actacagcaa ggtccccaag 660
tacaagctta tcgataccgt cgacctcgag 690
<210> 32
<211> 230
<212> PRT
<213> artificial sequence
<220>
<223> CD82delLEL-V5-6xHis; translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(135)
<223> TM1-TM3
<220>
<221> MISC_FEATURE
<222> (7)..(230)
<223> CD82delLEL-V5-6xHis
<220>
<221> MISC_FEATURE
<222> (136)..(169)
<223> V5-6xHis-Linker
<220>
<221> MISC_FEATURE
<222> (170)..(230)
<223> TM4
<400> 32
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Ser Ala Cys Ile Lys Val Thr Lys Tyr Phe
20 25 30
Leu Phe Leu Phe Asn Leu Ile Phe Phe Ile Leu Gly Ala Val Ile Leu
35 40 45
Gly Phe Gly Val Trp Ile Leu Ala Asp Lys Ser Ser Phe Ile Ser Val
50 55 60
Leu Gln Thr Ser Ser Ser Ser Leu Arg Met Gly Ala Tyr Val Phe Ile
65 70 75 80
Gly Val Gly Ala Val Thr Met Leu Met Gly Phe Leu Gly Cys Ile Gly
85 90 95
Ala Val Asn Glu Val Arg Cys Leu Leu Gly Leu Tyr Phe Ala Phe Leu
100 105 110
Leu Leu Ile Leu Ile Ala Gln Val Thr Ala Gly Ala Leu Phe Tyr Phe
115 120 125
Asn Met Gly Lys Leu Lys Gln Glu Phe Gly Lys Gly Pro Arg Phe Glu
130 135 140
Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu Asp Ser Thr Arg Thr
145 150 155 160
Gly His His His His His His Asp Ile Gly Gly Gly Gly Val Gln Ala
165 170 175
Trp Leu Gln Glu Asn Leu Gly Ile Ile Leu Gly Val Gly Val Gly Val
180 185 190
Ala Met Val Glu Leu Leu Gly Met Val Leu Ser Ile Cys Leu Cys Arg
195 200 205
His Val His Ser Glu Asp Tyr Ser Lys Val Pro Lys Tyr Lys Leu Ile
210 215 220
Asp Thr Val Asp Leu Glu
225 230
<210> 33
<211> 663
<212> DNA
<213> artificial sequence
<220>
<223> CD151delLEL-V5-6xHis; basierend auf Genbank Accession No.:
BT001769.3
<220>
<221> misc_feature
<222> (18)..(21)
<223> Startkodon
<220>
<221> misc_feature
<222> (18)..(420)
<223> TM1-TM3
<220>
<221> misc_feature
<222> (421)..(522)
<223> V5-6xHis-Linker
<220>
<221> misc_feature
<222> (523)..(663)
<223> TM4
<400> 33
gctagcgcgg ccgccaccat ggattacaag gatgacgacg ataagagccc gggcggatcc 60
atgggtgagt tcaacgagaa gaagacaaca tgtggcaccg tttgcctcaa gtacctgctg 120
tttacctaca attgctgctt ctggctggct ggcctggctg tcatggcagt gggcatctgg 180
acgctggccc tcaagagtga ctacatcagc ctgctggcct caggcaccta cctggccaca 240
gcctacatcc tggtggtggc gggcactgtc gtcatggtga ctggggtctt gggctgctgc 300
gccaccttca aggagcgtcg gaacctgctg cgcctgtact tcatcctgct cctcatcatc 360
tttctgctgg agatcatcgc tggtatcctc gcctacgcct actaccagca gctgaacacg 420
gaattcggaa agggcccgcg gttcgaaggt aagcctatcc ctaaccctct cctcggtctc 480
gattctacgc gtaccggtca tcatcaccat caccatgata tcggaggagg aggactgagg 540
gtcattgggg ctgtggggat cggcattgcc tgtgtgcagg tctttggcat gatcttcacg 600
tgctgcctgt acaggagtct caagctggag cactacaagc ttatcgatac cgtcgacctc 660
gag 663
<210> 34
<211> 221
<212> PRT
<213> artificial sequence
<220>
<223> CD151delLEL-V5-6xHis; translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> Startmethionin
<220>
<221> MISC_FEATURE
<222> (7)..(140)
<223> TM1-TM3
<220>
<221> MISC_FEATURE
<222> (7)..(221)
<223> CD151delLEL-V5-6xHis
<220>
<221> MISC_FEATURE
<222> (141)..(174)
<223> V5-6xHis-Linker
<220>
<221> MISC_FEATURE
<222> (175)..(221)
<223> TM4
<400> 34
Ala Ser Ala Ala Ala Thr Met Asp Tyr Lys Asp Asp Asp Asp Lys Ser
1 5 10 15
Pro Gly Gly Ser Met Gly Glu Phe Asn Glu Lys Lys Thr Thr Cys Gly
20 25 30
Thr Val Cys Leu Lys Tyr Leu Leu Phe Thr Tyr Asn Cys Cys Phe Trp
35 40 45
Leu Ala Gly Leu Ala Val Met Ala Val Gly Ile Trp Thr Leu Ala Leu
50 55 60
Lys Ser Asp Tyr Ile Ser Leu Leu Ala Ser Gly Thr Tyr Leu Ala Thr
65 70 75 80
Ala Tyr Ile Leu Val Val Ala Gly Thr Val Val Met Val Thr Gly Val
85 90 95
Leu Gly Cys Cys Ala Thr Phe Lys Glu Arg Arg Asn Leu Leu Arg Leu
100 105 110
Tyr Phe Ile Leu Leu Leu Ile Ile Phe Leu Leu Glu Ile Ile Ala Gly
115 120 125
Ile Leu Ala Tyr Ala Tyr Tyr Gln Gln Leu Asn Thr Glu Phe Gly Lys
130 135 140
Gly Pro Arg Phe Glu Gly Lys Pro Ile Pro Asn Pro Leu Leu Gly Leu
145 150 155 160
Asp Ser Thr Arg Thr Gly His His His His His His Asp Ile Gly Gly
165 170 175
Gly Gly Leu Arg Val Ile Gly Ala Val Gly Ile Gly Ile Ala Cys Val
180 185 190
Gln Val Phe Gly Met Ile Phe Thr Cys Cys Leu Tyr Arg Ser Leu Lys
195 200 205
Leu Glu His Tyr Lys Leu Ile Asp Thr Val Asp Leu Glu
210 215 220
<210> 35
<211> 102
<212> DNA
<213> artificial sequence
<220>
<223> V5-6xHis-Linker in Tetraspanin delLEL-V5-6xHis Konstrukten
<220>
<221> misc_feature
<222> (1)..(6)
<223> EcoRI Schnittstelle
<220>
<221> misc_feature
<222> (97)..(102)
<223> EcoRV Schnittstelle
<400> 35
gaattcggaa agggcccgcg gttcgaaggt aagcctatcc ctaaccctct cctcggtctc 60
gattctacgc gtaccggtca tcatcaccat caccatgata tc 102
<210> 36
<211> 34
<212> PRT
<213> artificial sequence
<220>
<223> V5-6xHis-Linker in Tetraspanin delLEL-V5-6xHis Konstrukten:
translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (3)..(23)
<223> V5-Epitop
<220>
<221> MISC_FEATURE
<222> (27)..(32)
<223> 6xHis-Epitop
<400> 36
Glu Phe Gly Lys Gly Pro Arg Phe Glu Gly Lys Pro Ile Pro Asn Pro
1 5 10 15
Leu Leu Gly Leu Asp Ser Thr Arg Thr Gly His His His His His His
20 25 30
Asp Ile
<210> 37
<211> 126
<212> DNA
<213> artificial sequence
<220>
<223> 2F5-4E10-Linker
<220>
<221> misc_feature
<222> (1)..(6)
<223> EcoRI Schnittstelle
<220>
<221> misc_feature
<222> (121)..(126)
<223> EcoRV Schnittstelle
<400> 37
gaattcattg aagaatcgca aaaccagcaa gaaaagaatg aacaagaatt attggaatta 60
gataaatggg caagtttgtg gaattggttt aacataacaa attggctgtg gtatataaaa 120
gatatc 126
<210> 38
<211> 42
<212> PRT
<213> artificial sequence
<220>
<223> 2F5-4E10-Linker: translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (19)..(24)
<223> 2F5-Epitop
<220>
<221> MISC_FEATURE
<222> (28)..(33)
<223> 4E10-Epitop
<400> 38
Glu Phe Ile Glu Glu Ser Gln Asn Gln Gln Glu Lys Asn Glu Gln Glu
1 5 10 15
Leu Leu Leu Glu Asp Lys Trp Ala Ser Leu Trp Asn Trp Phe Asn Ile
20 25 30
Thr Asn Trp Leu Trp Tyr Ile Lys Asp Ile
35 40
<210> 39
<211> 120
<212> DNA
<213> Artificial Sequence
<220>
<223> Linker version (8)
<220>
<221> misc_feature
<222> (1)..(6)
<223> EcoRI Schnittstelle
<220>
<221> misc_feature
<222> (7)..(26)
<223> TEV-Proteolyse-I
<220>
<221> misc_feature
<222> (27)..(33)
<223> HindIII Schnittstelle
<220>
<221> misc_feature
<222> (34)..(39)
<223> BamHI Schnittstelle
<220>
<221> misc_feature
<222> (40)..(46)
<223> PstI Schnittstelle
<220>
<221> misc_feature
<222> (46)..(63)
<223> His6-Tag
<220>
<221> misc_feature
<222> (64)..(84)
<223> TEV-Proteolyse-II
<220>
<221> misc_feature
<222> (85)..(97)
<223> c-Myc-Epitope1
<220>
<221> misc_feature
<222> (103)..(114)
<223> c-Myc-Epitope2
<220>
<221> misc_feature
<222> (115)..(120)
<223> EcoRV Schnittstelle
<400> 39
gaattcgaga atctgtactt tcagtcgaag cttggatccc tgcagcatca ccatcaccat 60
cacgaaaacc tctatttcca gagcgaacag aaactgatat ccgaagaaga tctggatatc 120
<210> 40
<211> 40
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker version (8), translatierte Sequenz
<220>
<221> MISC_FEATURE
<222> (3)..(8)
<223> TEV-Proteolysis-I
<220>
<221> MISC_FEATURE
<222> (16)..(21)
<223> His6-Tag
<220>
<221> MISC_FEATURE
<222> (22)..(28)
<223> TEV-Proteolysis-I
<220>
<221> MISC_FEATURE
<222> (29)..(33)
<223> c-Myc-Epitope1
<220>
<221> MISC_FEATURE
<222> (34)..(38)
<223> c-Myc-Epitope2
<400> 40
Glu Phe Glu Asn Leu Tyr Phe Gln Ser Lys Leu Gly Ser Leu Gln His
1 5 10 15
His His His His His Glu Asn Leu Tyr Phe Gln Ser Glu Gln Lys Leu
20 25 30
Ile Ser Glu Glu Asp Leu Asp Ile
35 40
<210> 41
<211> 27
<212> DNA
<213> artificial sequence
<220>
<223> Linker im Vektor pCMV-CD63delLEL-mCherry
(Minimallinker (9))
<220>
<221> misc_feature
<222> (1)..(6)
<223> PstI Schnittstelle
<220>
<221> misc_feature
<222> (7)..(12)
<223> EcoRI Schnittstelle
<220>
<221> misc_feature
<222> (13)..(18)
<223> EcoRV Schnittstelle
<220>
<221> misc_feature
<222> (19)..(27)
<223> 4x Glycin
<400> 41
ctgcaggaat tcgatatcgg aggagga 27
<210> 42
<211> 10
<212> PRT
<213> artificial sequence
<220>
<223> Linker im Vektor pCMV-CD63delLEL-mCherry: translatierte Sequenz
(Minimallinker (9))
<400> 42
Leu Gln Glu Phe Asp Ile Gly Gly Gly Gly
1 5 10
<210> 43
<211> 41
<212> DNA
<213> artificial sequence
<220>
<223> Linker im Vektor pCMV-CD63delLEL-V2-mCherry
<220>
<221> misc_feature
<222> (3)..(8)
<223> BamHI Schnittstelle
<220>
<221> misc_feature
<222> (18)..(23)
<223> BglIII Schnittstelle
<220>
<221> misc_feature
<222> (24)..(29)
<223> PstI Schnittstelle
<400> 43
ttggatccag cggccgcaga tctctgcagg gaggcggagg c 41
<210> 44
<211> 13
<212> PRT
<213> artificial sequence
<220>
<223> Linker im Vektor pCMV-CD63delLEL-V2-mCherry: translatierte
Sequenz (Linker)
<400> 44
Gly Ser Ser Gly Arg Arg Ser Leu Gln Gly Gly Gly Gly
1 5 10
<210> 45
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Linker zwischen CD63delLEL und Fluoreszenzprotein
<400> 45
aagcttatcg ataccgtcga cctcgagaaa 30
<210> 46
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker zwischen CD63delLEL und Fluoreszenzprotein; translatierte
Sequenz
<400> 46
Lys Leu Ile Asp Thr Val Asp Leu Glu Lys
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Конструкции для направленного редактирования генов и способы с их применением | 2020 |
|
RU2832109C2 |
| KIR3DL3 - ИНГИБИРУЮЩИЙ РЕЦЕПТОР ИММУННОЙ СИСТЕМЫ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2827900C1 |
| Т-КЛЕТКИ С ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ (CAR-T) ДЛЯ ЛЕЧЕНИЯ РАКА | 2019 |
|
RU2811466C2 |
| ИММУНОЦИТОКИНЫ НА ОСНОВЕ IL-15 И IL-15Rα ДОМЕНА SUSHI | 2012 |
|
RU2763298C2 |
| CD40L-СПЕЦИФИЧНЫЕ КАРКАСНЫЕ СТРУКТУРЫ, ПРОИСХОДЯЩИЕ ИЗ TN3, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2012 |
|
RU2826189C2 |
| МУТАНТНЫЙ БЕЛОК F RSV И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2807742C1 |
| ПОЛНОСТЬЮ ЧЕЛОВЕЧЕСКИЕ АНТИТЕЛА К МЕЗОТЕЛИНУ И ИММУННЫЕ ЭФФЕКТОРНЫЕ КЛЕТКИ, НАЦЕЛЕННЫЕ НА МЕЗОТЕЛИН | 2016 |
|
RU2748281C2 |
| АНТИТЕЛО ПРОТИВ ГЛИПИКАНА-3 И ЕГО ПРИМЕНЕНИЕ | 2016 |
|
RU2744245C2 |
| АНТИТЕЛА К PD-L1, СВЯЗЫВАЮЩИЕ PD-L1 СОБАКИ | 2015 |
|
RU2832918C2 |
| АНТИТЕЛА К PD-L1, СВЯЗЫВАЮЩИЕ PD-L1 СОБАКИ | 2015 |
|
RU2722562C2 |
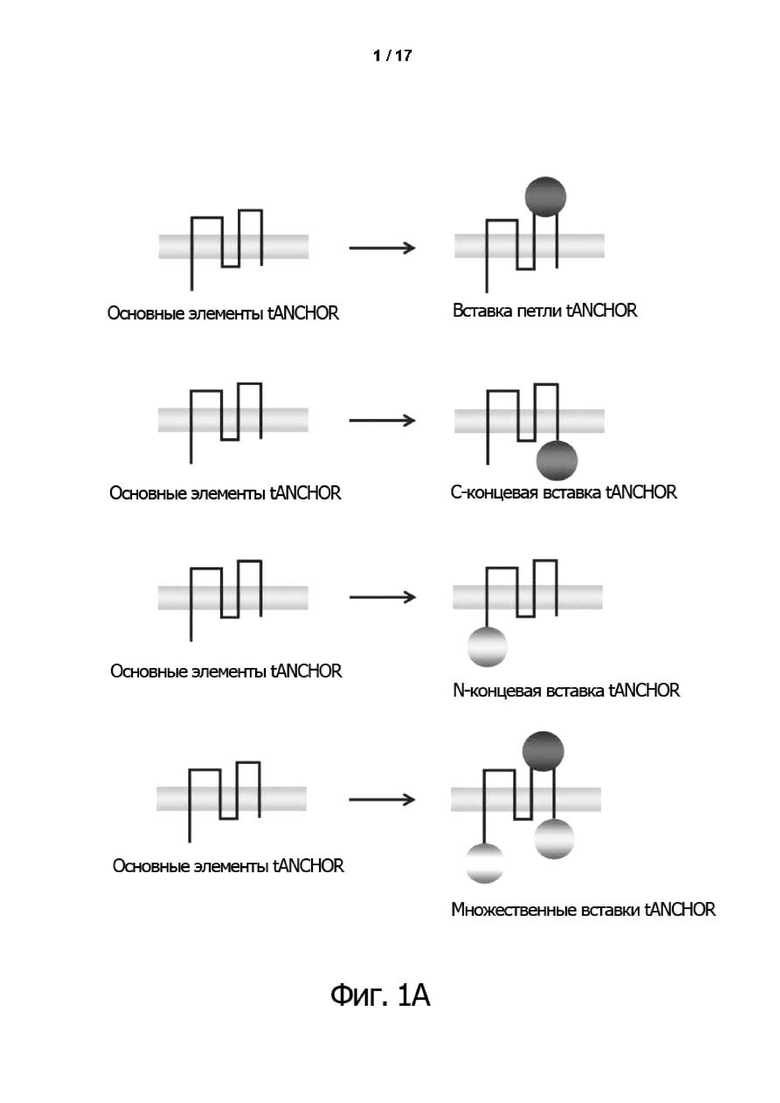
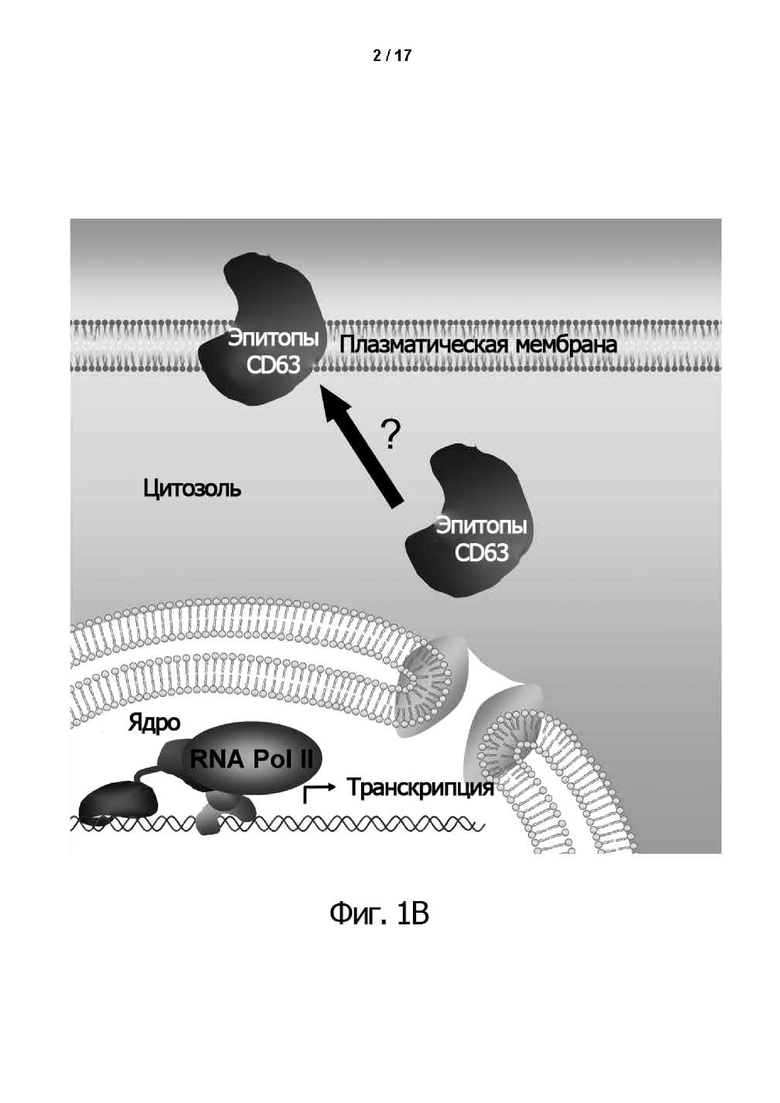


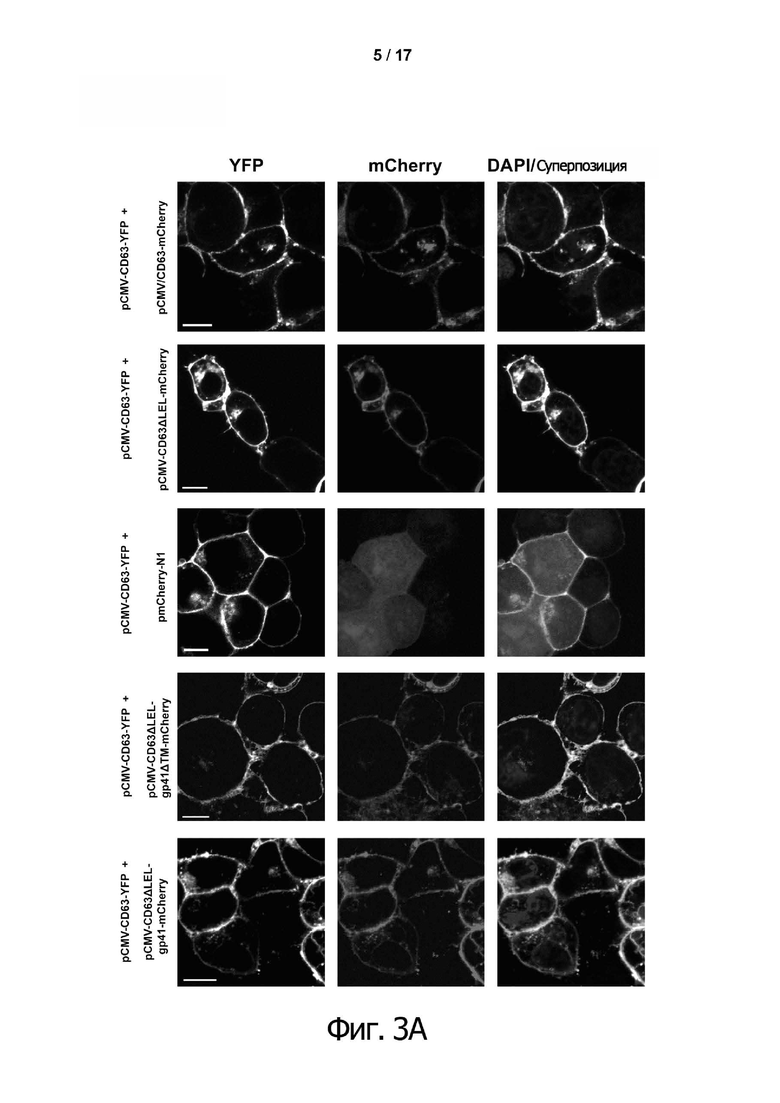
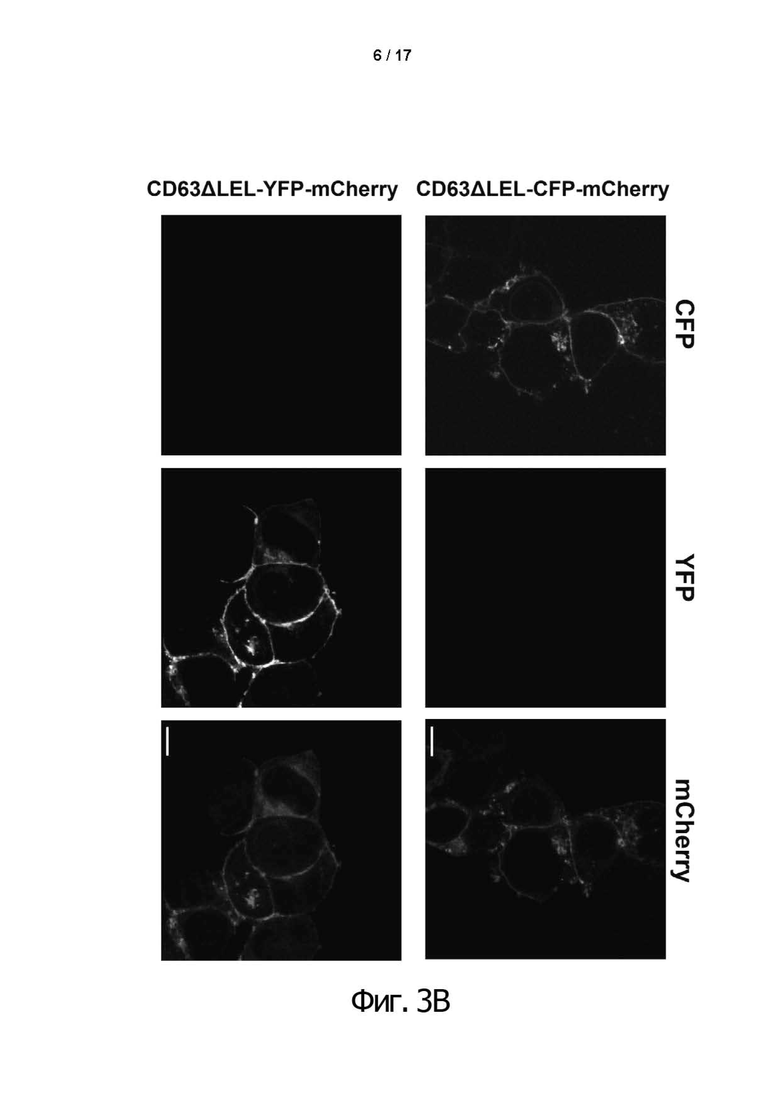
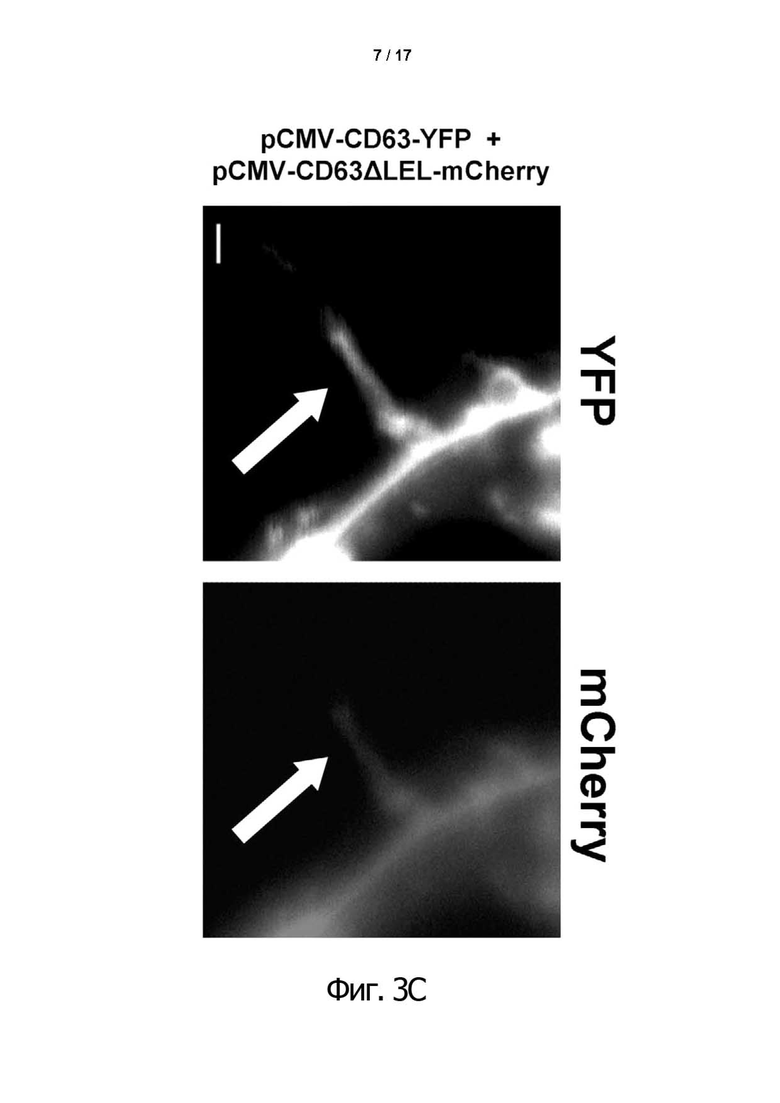
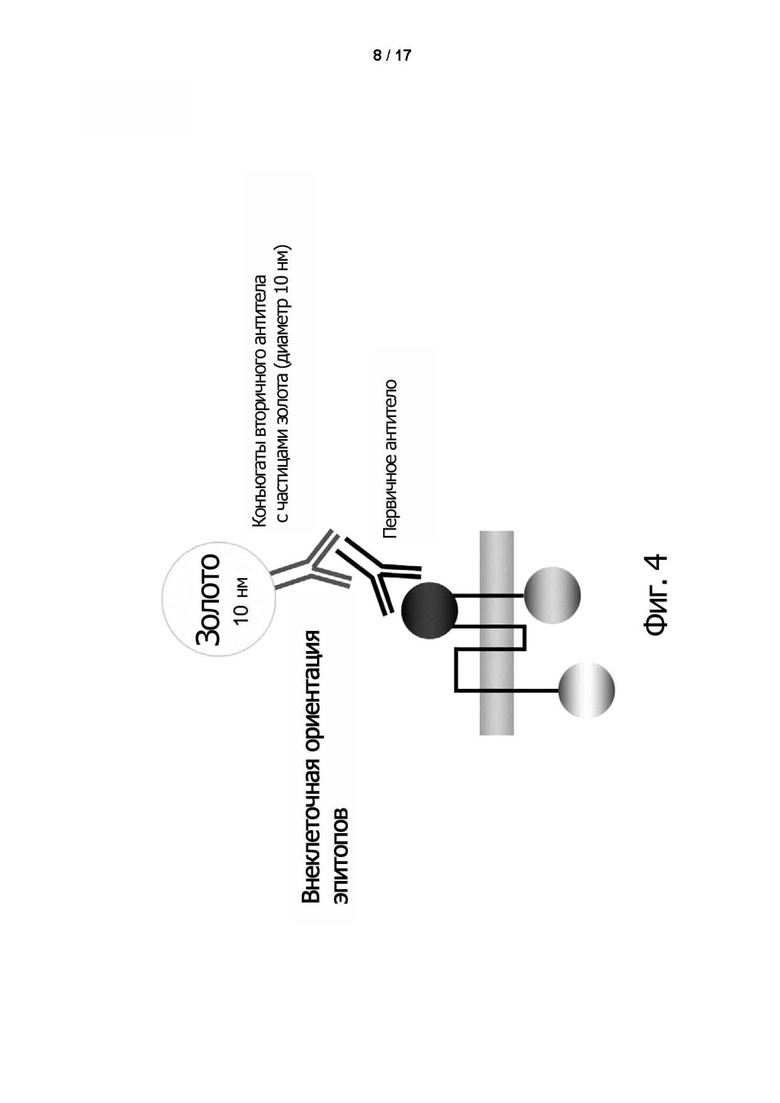
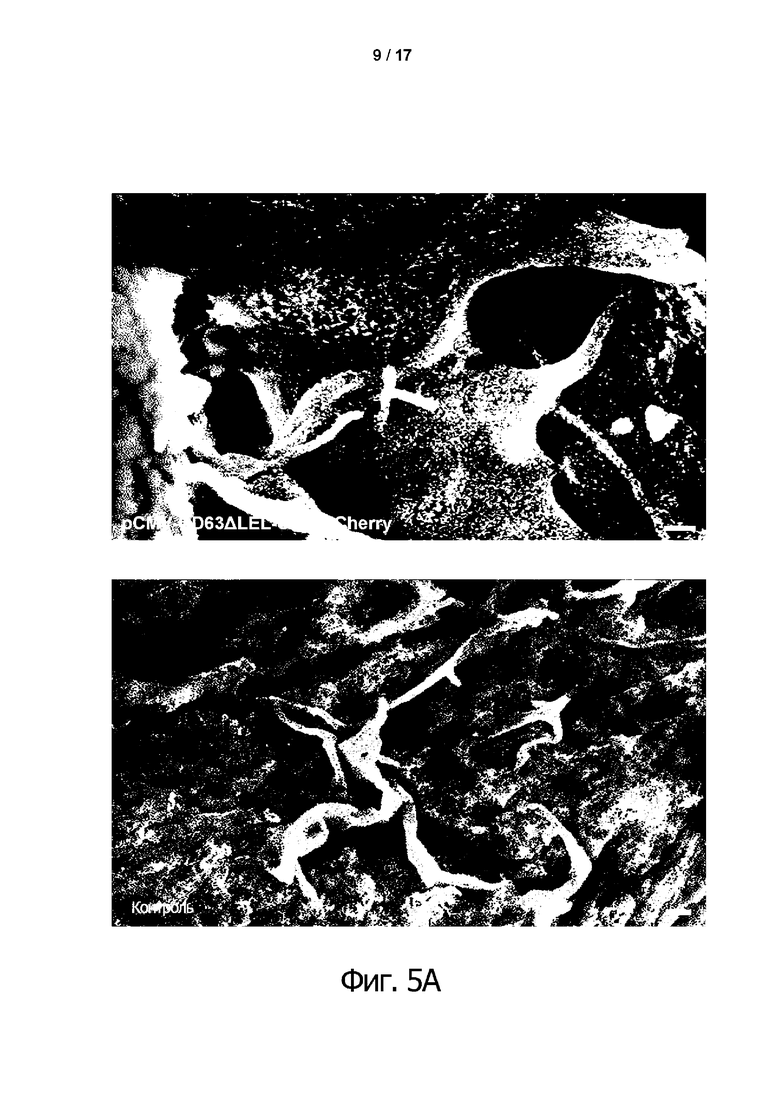
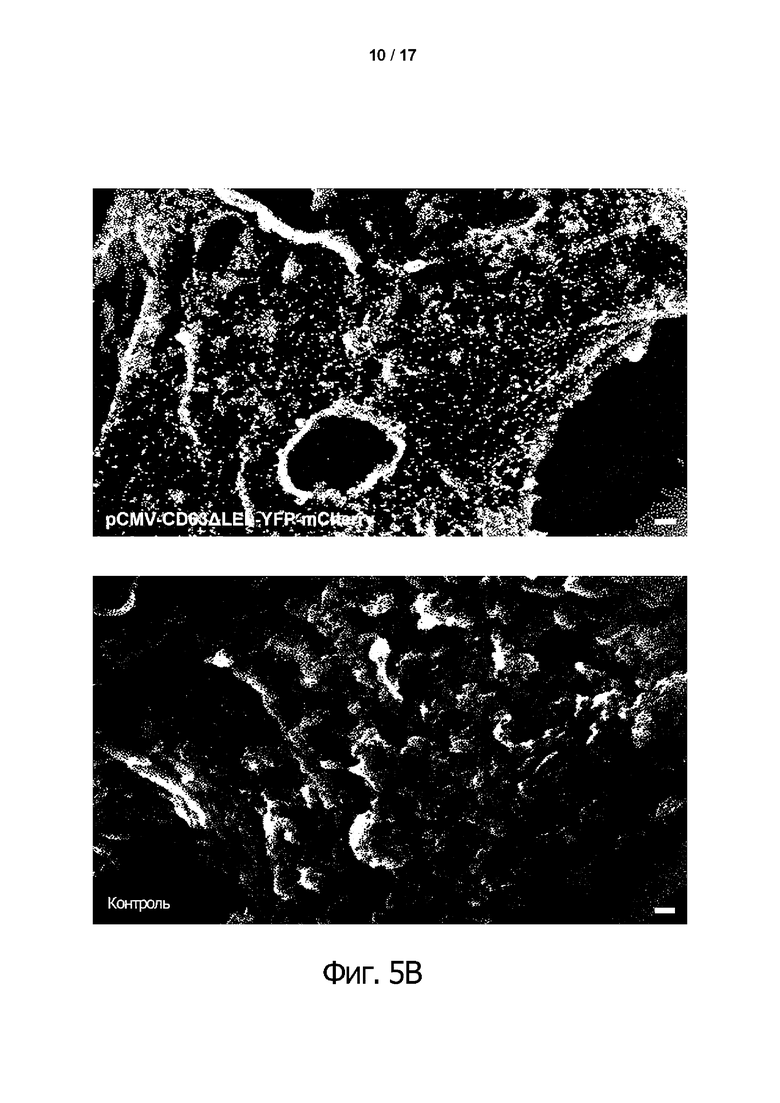

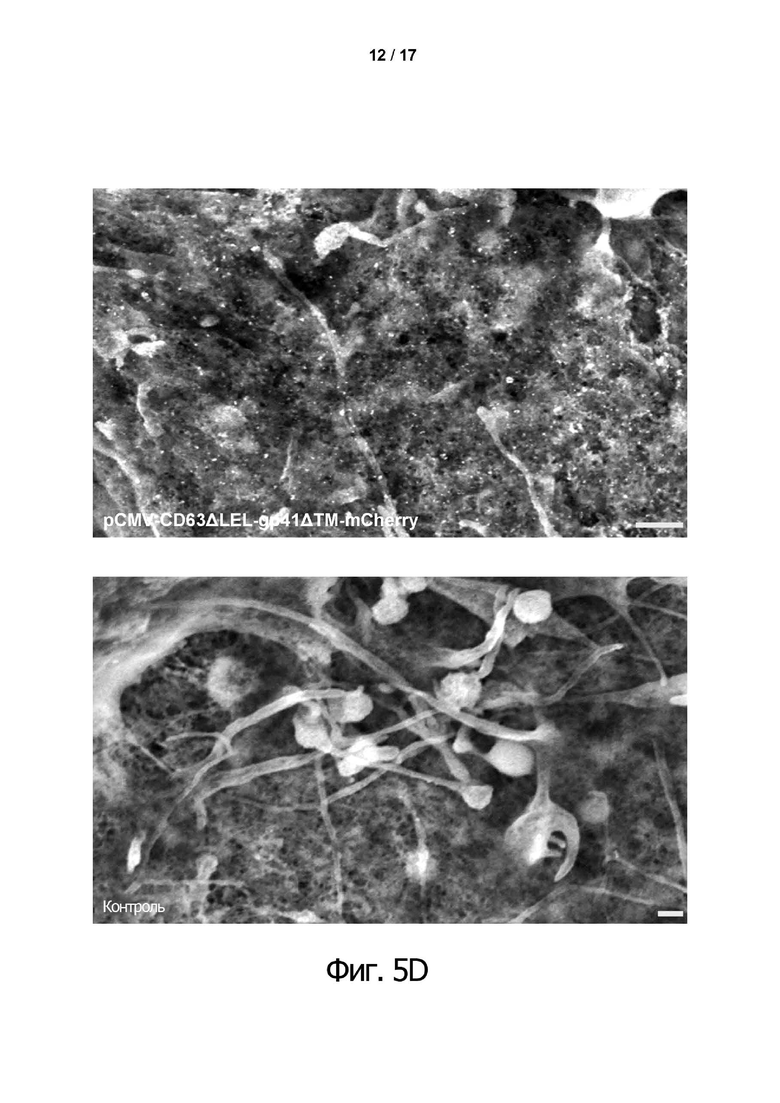


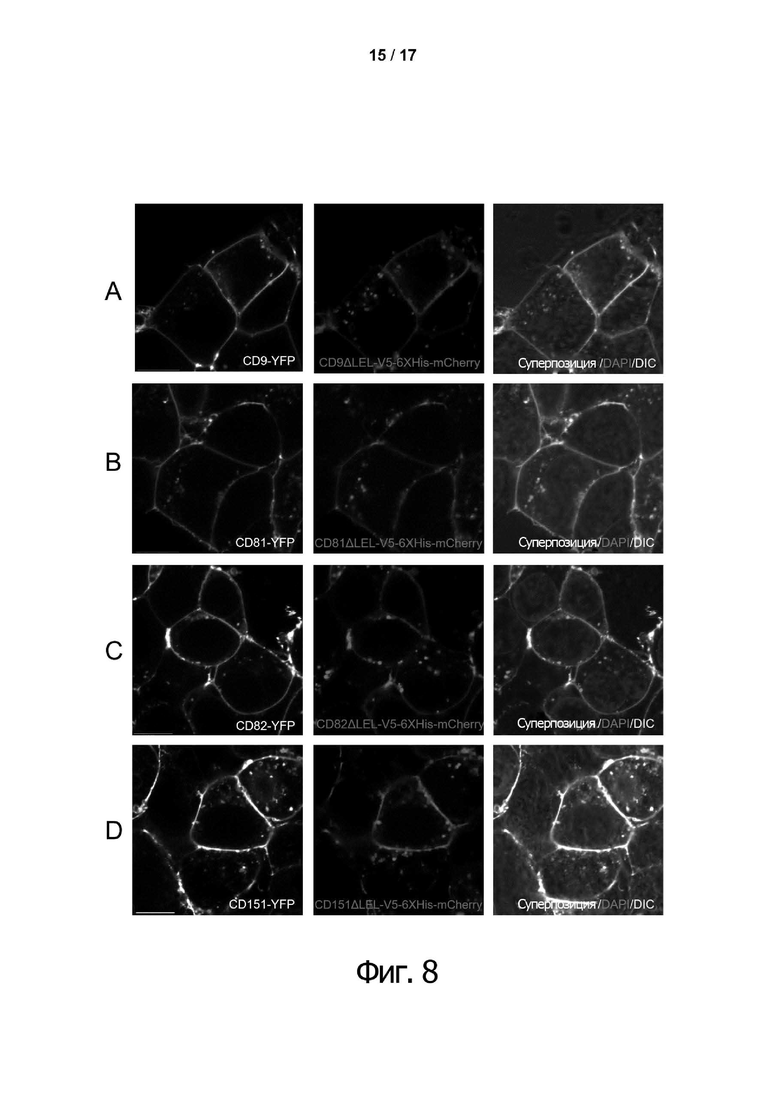
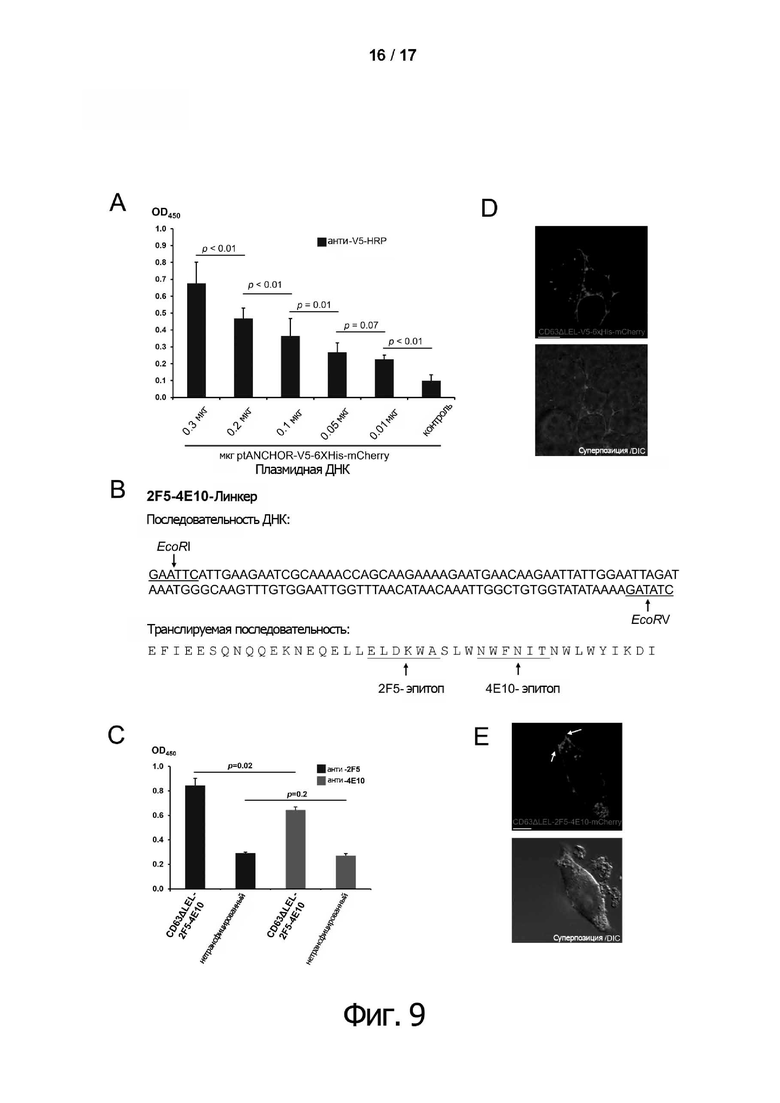
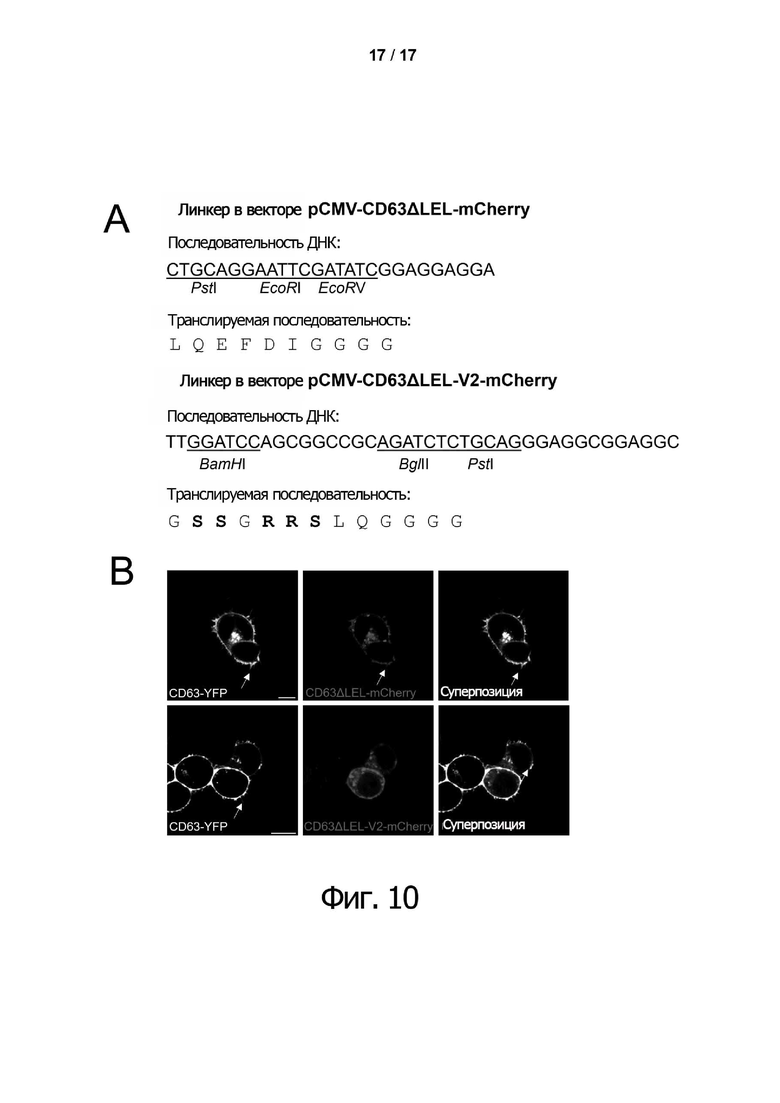
Настоящее изобретение относится к области биотехнологии, конкретно к слитым белкам на основе тетраспанина, в котором внеклеточная петля заменена полностью или частично пептидными последовательностями различного состава, и может быть использовано для их получения и применения в качестве носителя. Слитый белок состоит из первого домена (i), второго домена (ii), расположенного между первым и третьим доменами, и третьего домена (iii), гибких линкеров и, необязательно, концевого четвертого и/или пятого доменов. При этом домен (i) состоит из следующих доменов тетраспанина: трансмембранного домена 1 (TM1), малой внеклеточной петли (SEL), трансмембранного домена 2 (TM2), малой внутриклеточной петли (SIL) и трансмембранного домена 3 (TM3). Домен (ii) состоит из пептида менее 100 кДа с заданной аминокислотной последовательностью, при этом домен (ii) имеет аминокислотную последовательность, которая идентична менее чем на 70% по всей длине в отношении LEL тетраспанина. Домен (iii) состоит из трансмембранного домена 4 (TM4) тетраспанина. Изобретение позволяет презентировать пептид с заданной аминокислотной последовательностью на поверхности клетки млекопитающего. 12 н. и 7 з.п. ф-лы, 10 ил., 2 табл., 7 пр.
1. Слитый белок для презентации пептида с заданной аминокислотной последовательностью на поверхности клетки млекопитающего, состоящий из первого домена (i), второго домена (ii), расположенного между первым и третьим доменами, и третьего домена (iii), гибких линкеров, необязательно четвертого домена (iv) на N-конце указанного слитого белка и необязательно пятого домена (v) на С-конце указанного слитого белка, отличающийся тем, что
(А) домен (i) состоит из следующих доменов тетраспанина: трансмембранного домена 1 (TM1), малой внеклеточной петли (SEL), трансмембранного домена 2 (TM2), малой внутриклеточной петли (SIL) и трансмембранного домена 3 (TM3),
(B) домен (ii) состоит из указанного пептида с заданной аминокислотной последовательностью, необязательно фланкированного последовательностями сайтов распознавания одной или более протеаз, расположенных на С-конце и/или N-конце указанного пептида с заданной аминокислотной последовательностью, и необязательно одной или нескольких частичных последовательностей большой внеклеточной петли (LEL) тетраспанина, которые могут быть расположены на С- и/или N-конце этого пептида с заданной аминокислотной последовательностью, где домен (ii) имеет аминокислотную последовательность, которая идентична менее чем на 70% по всей длине в отношении LEL тетраспанина,
(С) домен (iii) состоит из трансмембранного домена 4 (TM4) тетраспанина,
где указанные гибкие линкеры представляют собой линкеры, свободные от серина и аргинина, расположенные между указанными первым и вторым доменом и/или между указанными вторым и третьим доменами, и
где размер пептида составляет менее 100 кДа, предпочтительно менее 50 кДа или более предпочтительно менее 30 кДа.
2. Слитый белок по п. 1, отличающийся тем, что второй домен (ii) также содержит одну или несколько частичных последовательностей LEL тетраспанина или гомологичных ей последовательностей с идентичностью последовательности, составляющей по меньшей мере 70% по всей длине, которые расположены в C- и/или N-концевой области пептида с заданной аминокислотной последовательностью.
3. Слитый белок по любому из предыдущих пунктов, отличающийся тем, что второй домен (ii) содержит дополнительно одну или несколько последовательностей распознавания протеазы, которые располагаются в C- и/или N-концевой области пептида с заданной аминокислотной последовательностью.
4. Слитый белок по любому из предыдущих пунктов, отличающийся тем, что пептид выбран из эпитопа, белка, антигена или фермента.
5. Слитый белок по любому из предыдущих пунктов, в котором также четвертый домен (iv) расположен на N-конце слитого белка и/или пятый домен (v) расположен на С-конце слитого белка.
6. Слитый белок по п. 5, отличающийся тем, что четвертый и/или пятый домен содержат метку, например аффинную метку или флуоресцентную метку.
7. Молекула нуклеиновой кислоты, которая кодирует слитый белок по любому из пп. 1-6 и имеет последовательность, которая кодирует аминокислотную последовательность слитого белка по любому из пп. 1-6.
8. Вектор для эукариотической экспрессии, содержащий ген из семейства тетраспанина, отличающийся тем, что указанный ген представляет собой гибридный ген, кодирующий слитый белок по любому из пп. 1-6, и этот вектор обеспечивает экспрессию слитого белка по любому из пп. 1-6 в клетке млекопитающего.
9. Клетка млекопитающего для презентирования на ее поверхности пептида с заданной аминокислотной последовательностью, содержащая молекулу нуклеиновой кислоты по п. 7, функционально связанную с последовательностью контроля экспрессии, или вектор по п. 8,
где клетка млекопитающего не является клеткой зародышевой линии человека.
10. Клетка млекопитающего по п. 9, отличающаяся тем, что является предпочтительно человеческой клеткой.
11. Способ презентации пептида с заданной аминокислотной последовательностью на поверхности клетки млекопитающего, включающий стадии:
(a) обеспечение клетки млекопитающего по любому из пп. 9, 10,
(b) культивирование клетки млекопитающего в условиях, при которых экспрессируется слитый белок по любому из пп. 1-6.
12. Применение клетки млекопитающего по любому из пп. 9, 10 в способе скрининга партнеров указанного пептида с заданной аминокислотной последовательностью по взаимодействию с этим пептидом.
13. Применение клетки млекопитающего по любому из пп. 9, 10 для изучения взаимодействий указанного пептида с заданной аминокислотной последовательностью с партнерами по взаимодействию.
14. Применение клетки млекопитающего по любому из пп. 9, 10 в способе получения указанного пептида с заданной аминокислотной последовательностью.
15. Применение клетки млекопитающего по любому из пп. 9, 10 в качестве реагента иммунизации в способе получения антител к указанному пептиду с заданной аминокислотной последовательностью.
16. Применение клетки млекопитающего по любому из пп. 9, 10 в качестве детектирующего реагента для специфического белок-белкового взаимодействия.
17. Лекарственное средство, содержащее клетку млекопитающего по любому из пп. 9, 10.
18. Лекарственное средство по п. 17, где указанное лекарственное средство представляет собой вакцину.
19. Набор для использования в способе закрепления пептида с заданной аминокислотной последовательностью, имеющего идентичность последовательности, составляющую менее чем 70% по всей длине в отношении большой внеклеточной петли (LEL) тетраспанина, в мембране, содержащий
(i) молекулу нуклеиновой кислоты по п. 7, которая функционально связана с последовательностью контроля экспрессии, или вектор по п. 8 и
(ii) клетку млекопитающего,
где клетка млекопитающего не является клеткой зародышевой линии клеток человека.
| WO 2014186649 А2, 20.11.2014 | |||
| FUMINORI RYU et al | |||
| Domain Analysis of the Tetraspanins | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Видоизменение пишущей машины для тюркско-арабского шрифта | 1923 |
|
SU25A1 |
| Кипятильник для воды | 1921 |
|
SU5A1 |
| Приспособление для обрезывания караваев теста | 1921 |
|
SU317A1 |
| ВЕКТОР, ОБЕСПЕЧИВАЮЩИЙ ЭКСПРЕССИЮ АНТИГЕНА ВИРУСА SARS НА ПОВЕРХНОСТИ КЛЕТОК, И МИКРООРГАНИЗМЫ, ТРАНСФОРМИРОВАННЫЕ ЭТИМ ВЕКТОРОМ | 2004 |
|
RU2332457C2 |
| КАМЕНСКИЙ А.А | |||
| и др | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
Авторы
Даты
2020-12-28—Публикация
2016-03-04—Подача