Область техники
Настоящее изобретение относится к области иммунотерапии или диагностики опухоли, и, в частности, к антителам, которые специфично распознают глипикан-3 (GPC3), и к их применениям.
Предшествующий уровень техники
В настоящее время адоптивная иммунотерапия на основе эффекторных иммунных клеток достигла некоторого эффекта в некоторых опухолях, и данный способ иммунотерапии может устранять дефекты лечения на основе антител, однако терапевтические эффекты в большинстве опухолей являются все еще неудовлетворительными [Grupp SA, et al. Adoptive cellular therapy. Curr Top Microbiol Immunol., 2011; 344: 149-72.]. В последние годы было обнаружено, что специфичность распознавания цитотоксических лимфоцитов (CTL) в отношении клеток-мишеней зависит от Т-клеточных рецепторов (TCR), scFv антител против антигенов, ассоциированных с опухолевыми клетками, и внутриклеточные сигнал-активирующие мотивы рецептора Т-лимфоцитов CD3ζ, или FcεRIγ сливали с химерным антигенным рецептором (CAR), и Т-лимфоцит генетически модифицировали химерными антигенными рецепторами на его поверхности посредством, например, лентивирусной инфекции. Такие CAR Т-лимфоциты способны к селективному направлению Т-лимфоцитов на опухолевые клетки и специфичному уничтожению опухоли неограничивающим образом с помощью главного комплекса гистосовместимости (МНС). CAR Т-лимфоцит представляет собой новую иммунотерапевтическую стратегию в области иммунотерапии опухолей. При конструировании CAR-модифицированных эффекторных иммунных клеток, особенно Т-клеток, гены антигенов-мишеней фактически являются критически важным выбором. Принимая во внимание сложность экспрессии генов in vivo и разные неконтролируемые факторы, отбор подходящих генов для CAR является очень важным. Кроме того, для многих опухолеспецифичных антигенов сложно найти специфичную молекулу, направленную против них и подходящую для конструирования CAR-модифицированных иммунных клеток.
Глипикан-3 (GPC3, также известный как DGSX, GTR2-2, MXR7, OCI-5, SDYS, SGB, SGBS или SGBS1) представляет собой белок поверхности клетки, принадлежащий к семейству гепарансульфатпротеогликанов. Ген GPC3 кодирует коровый белок-предшественник приблизительно 70 кДа, который расщепляется посредством фурина с образованием растворимого, доступного в крови, аминоконцевого (N-концевого) пептида приблизительно 40 кДа и мембраносвязанного карбоксиконцевого (С-концевого) пептида, содержащего приблизительно 2 гепарансульфатные (HS) сахарные цепи приблизительно 30 кДа. Белки GPC3 прикрепляются к клеточной мембране посредством гликозилфосфатидилинозитольных (GPI) якорей.
GPC3 экспрессируется на высоком уровне в фетальной печени, в то же время не экспрессируется в нормальной ткани печени у взрослых, однако его экспрессия восстанавливается при гепатоцеллюлярной карциноме и тесно связана с развитием и прогрессированием гепатоцеллюлярной карциномы. GPC3 не только на высоком уровне выявляется на ранней стадии гепатоцеллюлярной карциномы, но и степень выявления также повышается с развитием рака печени. Экспрессию GPC3 не выявляли при аденокарциноме печени, холангиокарциноме, метастазах печени ив 12 обычных солидных опухолях и 21 негепатомной клеточной линии. Корме того, GPC3 также экспрессируется в опухолях, таких как меланома, светлоклеточный рак яичников, опухоль желточного мешка и нейробластома. GPC3 считается потенциальной мишенью для иммунотерапии опухоли в виду высокоспецифичной экспрессии GPC3 в опухолях, таких как гепатоцеллюлярная карцинома и меланома.
Несмотря на то, что сообщалось о протоколах для выявления рака печени с использованием антител против GPC3 и о анализах антителозависимой (ADCC) или комплиментзависимой (CDC) цитотоксичности с использованием антител против GPC3, в настоящее время не существует доступных антител против GPC3. Только антитело GC33, описанное в CN200580000807.4, находилось на фазе I клинического исследования, в то время как клиническое применение остается неясным.
Кроме того, антитела против GPC3 можно использовать не только в качестве терапевтических антител, но также в качестве иммунотерапий, например, Т-лимфоциты для диагностики и модификации на основе химерных антигенных рецепторов (CAR). Текущее исследование показывает, что разные антитела могут обладать разной иммуногенностью, и такая иммуногенность будет приводить к терапевтическим эффектам и побочным эффектам антитела или его производного терапевтического агента. Например, CAR Т-клетки на основе мышиных антител против мезотелина могут вызывать аллергические реакции, которые оказывают влияние на их длительное выживание в организме человека. В виду вышеизложенного, в данной области существует необходимость в дальнейшей оптимизации и получении новых антител против GPC3, которые бы обладали хорошей активностью по уничтожению опухоли и прекрасными перспективами клинического применения при применении в твердых опухолях, так чтобы антитела против GPC3 могли эффективно использоваться в диагностике и лечении опухолей и приносить пользу пациентам.
Краткое описание изобретения Целью настоящего изобретения является предложение антител против глипикана-3 и их применений.
В первом аспекте настоящего изобретения предложено антитело, которое специфично распознает глипикан-3 (GPC3), и антитело имеет вариабельную область легкой цепи и вариабельную область тяжелой цепи, и
CDR1 вариабельной области легкой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 16, SEQ ID NO: 67, SEQ ID NO: 69;
CDR2 вариабельной области легкой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 18, SEQ ID NO: 68, SEQ ID NO: 70;
CDR3 вариабельной области легкой цепи имеет аминокислотную последовательность SEQ ID NO: 20;
CDR1 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 10, SEQ ID NO: 60, SEQ ID NO: 62; SEQ ID NO: 64;
CDR2 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 12, SEQ ID NO: 61, SEQ ID NO: 63; SEQ ID NO: 65; SEQ ID NO: 66; SEQ ID NO: 71;
CDR3 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 14, SEQ ID NO: 72.
В предпочтительном воплощении антитело включает:
антитело (a) (P7D4), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 12 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (б) (am4), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 60, CDR2 с последовательностью SEQ ID NO: 61 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (в) (am14), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 62, CDR2 с последовательностью SEQ ID NO: 63 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (г) (am20), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 64, CDR2 с последовательностью SEQ ID NO: 65 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (д) (am35), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 67, CDR2 с последовательностью SEQ ID NO: 68 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 66 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (е) (am42), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 69, CDR2 с последовательностью SEQ ID NO: 70 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 66 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (ж) (Т2-23), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 70 и CDR3 с последовательностью SEQ ID NO: 20, или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 71 и CDR3 с последовательностью SEQ ID NO: 72;
антитело (з), которое распознает такую же антигенную детерминанту, которую распознает любое из антител (а)-(ж).
В другом предпочтительном воплощении антитело, специфично распознающее глипикан-3 (GPC3), может представлять собой одноцепочечное антитело (scFV), моноклональное антитело, доменное антитело, Fab-фрагмент, Fd-фрагмент, Fv-фрагмент, F-(ab')2 фрагмент и их производное или другие формы антитела; предпочтительно одноцепочечное антитело.
В другом предпочтительном воплощении антитело, которое специфично распознает глипикан-3 (GPC3), является гуманизированным или полностью гуманизированным; предпочтительно, полностью гуманизированным.
В другом предпочтительном воплощении аминокислотная последовательность вариабельной области тяжелой цепи антитела (a) (P7D4) представлена в положениях 1-121 SEQ ID NO: 4; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 4;
аминокислотная последовательность вариабельной области тяжелой цепи антитела
(б) (ат4) представлена в положениях 1-121 SEQ ID NO: 25; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 25;
аминокислотная последовательность вариабельной области тяжелой цепи антитела
(в) (aml4) представлена в положениях 1-121 SEQ ID NO: 27; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 27;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (г) (am20) представлена в положениях 1-121 SEQ ID NO: 29; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 29;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (д) (ат35) представлена в положениях 1-121 SEQ ID NO: 31; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 31;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (е) (am42) представлена в положениях 1-121 SEQ ID NO: 33; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ IDNO: 33; или
аминокислотная последовательность вариабельной области тяжелой цепи антитела (ж) (Т2-23) представлена в положениях 1-121 SEQ ID NO: 35; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ IDNO: 35.
В другом аспекте настоящего изобретения предложено антитело, которое специфично распознает глипикан-3 (GPC3), которое представляет собой гуманизированное моноклональное антитело, содержащее вариабельную область тяжелой цепи и вариабельную область легкой цепи. Его вариабельная область тяжелой цепи содержит CDR1 с аминокислотной последовательностью и SEQ ID NO: 73, CDR2 с аминокислотной последовательностью SEQ ID NO: 74 и CDR3 с аминокислотной последовательностью SEQ ID NO: 75; или его вариабельная область легкой цепи содержит CDR1 с аминокислотной последовательностью SEQ ID NO: 76, CDR2 с аминокислотной последовательностью SEQ ID NO: 77 и CDR3 с аминокислотной последовательностью SEQ ID NO: 78;
В предпочтительном воплощении антитело имеет вариабельную область тяжелой цепи с аминокислотной последовательностью SEQ ID NO: 58 или вариабельную область легкой цепи с аминокислотной последовательностью SEQ ID NO: 59.
В другом аспекте изобретения предложена нуклеиновая кислота, кодирующая любое из предшествующих антител.
В другом аспекте настоящего изобретения предложен экспрессионный вектор, содержащий указанную нуклеиновую кислоту.
В другом аспекте настоящего изобретения предложена клетка-хозяин, которая содержит экспрессионный вектор или в геном которой интегрирована нуклеиновая кислота.
В другом аспекте изобретения предложено применение любого из антител, описанных выше, для получения лекарственного средства направленного действия, конъюгата антитело-лекарственное средство или полифункционального антитела, которое специфично направлено на опухолевые клетки, экспрессирующие глипикан-3; или для получения средства для диагностики опухоли, экспрессирующей глипикан-3; или для получения иммунных клеток, модифицированных химерным антигенным рецептором.
В другом аспекте настоящего изобретения предложен многофункциональный иммуноконъюгат, содержащий любое из антител, описанных выше и связанную с ним функциональную молекулу (включая ковалентно связанную, конъюгированную, присоединенную, адсорбированную); и функциональная молекула выбрана из группы, состоящей из молекулы, направленной на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, направленной на маркер поверхности иммунной клетки, или детектируемой метки.
В предпочтительном воплощении в многофункциональном иммуноконъюгате молекула, направленная на маркер поверхности опухоли, представляет собой антитело или лиганд, который связывается с маркером поверхности опухоли; или молекула, подавляющая опухоль, представляет собой противоопухолевый цитокин или противоопухолевый токсин; и, предпочтительно, цитокин включает: IL-12, IL-15, IFN (интерферон)-бета и TNF (фактор некроза опухоли)-альфа, но не ограничивается ими.
В другом предпочтительном воплощении детектируемая метка в многофункциональном иммуноконъюгате включает флуоресцентную метку и хромогенную метку.
В другом предпочтительном воплощении в многофункциональном иммуноконъюгате антитело, которое связывается с маркером поверхности опухоли, относится к антителу, которое распознает антигены, отличные от глипикана-3, где указанные отличные антигены включают: EGFR, EGFRvIII, мезотелин, HER2, EphA2, Her3, ЕрСАМ, MUC1, MUC16, СЕА, клаудин 18.2, фолатный рецептор, клаудин 6, WT1, NY-ESO-1, MAGE 3, ASGPR1 или CDH16.
В другом предпочтительном воплощении в многофункциональном иммуноконъюгате молекула, направленная на маркер поверхности иммунной клетки, представляет собой антитело, которое связывается с маркером поверхности Т-клетки, которое может образовывать бифункциональное антитело, активирующее Т-клетки, с любым из описанных выше антител (биспецифичный активатор Т-клеток, BiTE).
В другом предпочтительном воплощении в многофункциональном иммуноконъюгате антитело, которое связывается с маркером поверхности иммунной клетки, представляет собой антитело против CD3.
В другом предпочтительном воплощении антитело против CD3 представляет собой одноцепочечное антитело (scFV), моноклональное антитело, Fab-фрагмент, Fd-фрагмент, Fv-фрагмент, F(ab')2-фрагмент и их производное или антитело другой формы; предпочтительно, одноцепочечное антитело.
В другом предпочтительном воплощении антитело против CD3 является гуманизированным, химерным, полностью человеческим или мышиным.
В другом предпочтительном воплощении многофункциональный иммуноконъюгат представляет собой слитый пептид и дополнительно содержит линкерный пептид (линкер) между любым из описанных выше антител и связанной с ним функциональной молекулой.
В другом предпочтительном воплощении линкерный пептид имеет последовательность (GlyGlyGlyGlySer)n, где n представляет собой целое число от 1 до 5; более предпочтительно n равно 3.
В другом предпочтительном воплощении многофункциональный иммуноконъюгат вводят в форме полипептида или путем введения гена.
В другом аспекте изобретения предложена нуклеиновая кислота, кодирующая любой из многофункциональных иммуноконъюгатов, описанных выше.
В другом аспекте настоящего изобретения предложено применение любого из описанных выше многофункциональных иммуноконъюгатов для получения антинеопластического средства или средства для диагностики опухолей, которые экспрессируют глипикан-3; или для получения иммунных клеток, модифицированных химерным антигенным рецептором. Предпочтительно, иммунные клетки включают Т-лимфоцит, NK (естественные киллеры)-клетку или NKT лимфоцит.
В другом аспекте настоящего изобретения предложен химерный антигенный рецептор (CAR), содержащий любое из описанных выше антител, и химерный антигенный рецептор содержит: любое из описанных выше антител, трансмембранную область и внутриклеточную сигнальную область, которые последовательно связаны; и внутриклеточная сигнальная область выбрана из группы, состоящей из последовательностей внутриклеточной сигнальной области CD3ζ, FceRIγ, CD27, CD28, CD137, CD134, MyD88, CD40 или их комбинации.
В другом предпочтительном воплощении трансмембранная область в химерном антигенном рецепторе содержит трансмембранную область CD 8 или CD28.
В другом предпочтительном воплощении в химерном антигенном рецепторе химерный антигенный рецептор содержит следующие последовательно связанные антитело, трансмембранную область и внутриклеточную сигнальную область: любое из описанных выше антител, CD8 и CD3ζ; любое из выше описанных антител, CD8, CD 137 и CD3ζ; любое из выше описанных антител, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28 и CD3ζ; или любое из описанных выше антител, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28, CD137 и CD3ζ.
В другом предпочтительном воплощении в химерном антигенном рецепторе антитело представляет собой одноцепочечное антитело или доменное антитело. В другом предпочтительном воплощении химерный антигенный рецептор содержит:
SEQ ID NO: 49 или аминокислотную последовательность, представленную в положениях 22-346;
SEQ ID NO: 50 или аминокислотную последовательность, представленную в положениях 22-447;
SEQ ID NO: 51 или аминокислотную последовательность, представленную в положениях 22-491;
SEQ ID NO: 52 или аминокислотную последовательность, представленную в положениях 22-494;
SEQ ID NO: 53 или аминокислотную последовательность, представленную в положениях 22-536;
аминокислотную последовательность SEQ ID NO: 85; аминокислотную последовательность SEQ ID NO: 86; или аминокислотную последовательность SEQ ID NO: 87.
В другом аспекте изобретения предложена нуклеиновая кислота, кодирующая химерный антигенный рецептор. В другом предпочтительном воплощении нуклеиновая кислота, кодирующая химерный антигенный рецептор, содержит:
SEQ ID NO: 44 или нуклеотидную последовательность, представленную в положениях 380-1420 или 443-1420;
SEQ ID NO: 45 или нуклеотидную последовательность, представленную в положениях 380-1723 или 443-1723;
SEQ ID NO: 46 или нуклеотидную последовательность, представленную в положениях 380-1855 или 443-1855;
SEQ ID NO: 47 или нуклеотидную последовательность, представленную в положениях 380-1864 или 443-1864;
SEQ ID NO: 48 или нуклеотидную последовательность, представленную в положениях 380-1990 или 443-1990;
нуклеотидную последовательность SEQ ID NO: 85;
нуклеотидную последовательность SEQ ID NO: 87; или
нуклеотидную последовательность SEQ ID NO: 89.
В другом аспекте настоящего изобретения предложен экспрессионный вектор, содержащий нуклеиновую кислоту, кодирующую химерный антигенный рецептор. В другом предпочтительном воплощении экспрессионный вектор получают из лентивирусной плазмиды pWPT (или pWPT-eGFP).
В другом аспекте настоящего изобретения предложен вирус, содержащий указанный вектор.
Предложено применение химерного антигенного рецептора или кодирующей его нуклеиновой кислоты или экспрессионного вектора или вируса, содержащего нуклеиновую кислоту, для получения генетически модифицированных иммунных клеток, направленных на опухоль, которая экспрессирует глипикан-3.
В другом предпочтительном воплощении опухоль, экспрессирующая глипикан-3, включает рак печени, меланому, светлоклеточный рак яичников, опухоль желточного мешка и нейробластому (но не ограничивается ими).
В другом аспекте настоящего изобретения предложена генетически модифицированная иммунная клетка, которая трансдуцирована нуклеиновой кислотой, кодирующей химерный антигенный рецептор, или экспрессионным вектором или вирусом, содержащим нуклеиновую кислоту; или имеет химерный антигенный рецептор, экспрессирующийся на ее поверхности.
В другом предпочтительном воплощении иммунные клетки дополнительно несут экзогенную кодирующую последовательность для цитокинов; и предпочтительно цитокины включают IL-12, IL-15 или IL-21.
В другом предпочтительном воплощении иммунная клетка также экспрессирует другой химерный антигенный рецептор, который не содержит CD3ζ, но содержит внутриклеточный сигнальный домен CD28, внутриклеточный сигнальный домен CD137 или комбинацию обоих.
В другом предпочтительном воплощении иммунная клетка дополнительно экспрессирует хемокиновый рецептор; и предпочтительно хемокиновый рецептор включает CCR2.
В другом предпочтительном воплощении иммунная клетка дополнительно экспрессирует миРНК (малая интерферирующая РНК), которая может снижать уровень экспрессии PD-1, или белок, который блокирует PD-L1.
В другом предпочтительном воплощении иммунная клетка дополнительно экспрессирует предохранитель; и предпочтительно предохранитель включает индуцируемую каспазу-9, усеченный EGFR или RQR8.
В другом предпочтительном воплощении иммунные клетки включают Т-лимфоциты, NK-клетки или NKT-клетки.
В другом аспекте изобретения предложено применение генетически модифицированных иммунных клеток для получения лекарственного средства, ингибирующего опухоль, и опухоль представляет собой опухоль, экспрессирующую глипикан-3.
В другом аспекте изобретения предложена фармацевтическая композиция (включая лекарственное средство или диагностическое средство), содержащая:
любое из описанных выше антител или нуклеиновую кислоту, кодирующую антитело; или
любой из описанных выше иммуноконъюгатов или нуклеиновую кислоту, кодирующую конъюгат; или
любой из описанных выше химерных антигенных рецепторов или нуклеиновую кислоту, кодирующую химерный антигенный рецептор; или
любую из описанных выше генетически модифицированных иммунных клеток.
Другие аспекты изобретения будут очевидными специалистам в данной области из описания в данном документе.
Описание графических материалов
На Фиг. 1 показано связывание антител Р1В12Е и P7D4 с GPC3 человека и контрольным BSA (бычий сывороточный альбумин) в анализе ELISA (твердофазный иммуноферментный анализ) на основе одного фага. Значения для антител Р1 В12Е и P7D4 с GPC3 человека и отрицательным контролем BSA демонстрируют, что данные два отобранных антитела могут специфично связываться с GPC3 человека.
На Фиг. 2 показан электрофорез для очистки антител с GPC3 человека.
На Фиг. 3 показан анализ активности связывания антител scFv-P1B12E-Fc и scFv-P7D4-Fc с клетками HepG2, позитивными в отношении экспрессии GPC3.
На Фиг. 4 показан анализ SPR (поверхностный плазмонный резонанс) способности связывания для серии антител P7D4 с GPC3.
На Фиг. 5 показана серия антител P7D4, специфично связывающихся с рекомбинантным GPC3 человека.
На Фиг. 6 показан SDS PAGE-электрофорез (электрофорез в полиакриламидном геле в присутствии додецилсульфата натрия) экспрессированного и очищенного белка GPC3.
На Фиг. 7 показан кинетический анализ связывания антитела Y035 с GPC3 человека.
На Фиг. 8 показан кинетический анализ связывания антитела 5А5 с GPC3 человека.
На Фиг. 9 показано антитело Y035, специфично связывающееся с клетками HepG, позитивными в отношении экспрессии GPC3.
Способы реализации изобретения
После интенсивного исследования и скрининга авторы изобретения получали антитела, которые специфично распознают GPC3, включая одноцепочечные антитела и гуманизированные антитела. Антитело по настоящему изобретению можно использовать для получения разных противоопухолевых лекарственных средств направленного действия и лекарственных средств для диагностики опухолей.
Антитело против GPC3
Проводили скрининг специфичных антител с хорошими свойствами связывания с GPC3, и авторы настоящего изобретения их получали в библиотеках природных антител полного человеческого репертуара и дополнительно подвергали аминокислотным мутациям с получением антител против GPC3 со значительно повышенной аффинностью, и также авторами изобретения для них были найдены ключевые области CDR, обнаруживающие их свойства связывания.
Авторы настоящего изобретения также получали мышиное антитело против GPC3 с использованием методики на основе гибридомы, гуманизировали его и получали гуманизированное антитело против GPC3 посредством повторного сравнения с чрезвычайно превосходным свойством связывания с GPC3, и также были найдены ключевые области CDR для его эффективности связывания.
Антитела по изобретению могут представлять собой интактные молекулы иммуноглобулина или антиген-связывающие фрагменты, включая Fab-фрагменты, Fd-фрагменты, Fv-фрагменты, F(ab')2 - фрагменты, фрагменты гипервариабельных областей (CDR), одноцепочечное антитело (scFv), доменное антитело, бивалентное одноцепочечное антитело, одноцепочечное фаговое антитело, биспецифичное диатело, трехцепочечное антитело, четырехцепочечное антитело, но не ограничиваясь ими.
Антиген-связывающие свойства антитела могут быть описаны тремя специфичными областями, локализованными в вариабельных областях тяжелой и легкой цепей, называемыми гипервариабельными областями (CDR), которые разделяют вариабельные области на четыре каркасные области (FR), и аминокислотные последовательности четырех FR являются относительно консервативными, прямо не принимающими участия в реакции связывания. Данные CDR образуют петлевую структуру, в которой β-складки, образуемые FR, расположены близко друг к другу в пространстве, и антиген-связывающий сайт антитела образован CDR на тяжелой цепи и CDR на соответствующей легкой цепи. Возможно определять, какие аминокислоты составляют FR или CDR области посредством сравнения аминокислотных последовательностей одного и того же типа антитела. Области CDR представляют собой последовательности белков, представляющих иммунологический интерес, и области CDR антител по изобретению являются принципиально новыми. Антитело может содержать две, три, четыре, пять или все шесть из областей CDR, описанных в данном документе.
Другой аспект изобретения включает функциональные варианты антител, описанные в данном документе. Если вариант способен конкурировать с исходным антителом за специфичное связывание с GPC3, и его способность к распознаванию GPC3, экспрессируемому на поверхности опухолевых клеток, близка к способности к распознаванию GPC3 специфичных антител, предложенных в Примерах настоящего изобретения. Функциональные варианты могут иметь консервативные модификации последовательностей, включая нуклеотидные и аминокислотные замены, вставки и делеции. Данные модификации могут быть введены стандартными методиками, известными в данной области, такими как направленный мутагенез и случайный мутагенез на основе ПЦР, и могут включать как природные, так и неприродные нуклеотиды и аминокислоты. Предпочтительно, модификация последовательности происходит в области, находящейся вне области CDR антитела.
Иммуноконъюгат
В настоящем изобретении также предложен мультифункциональный иммуноконъюгат, содержащий антитела, описанные в данном документе, и дополнительно содержащий по меньшей мере одну функциональную молекулу другого типа. Функциональная молекула выбрана из молекулы, которая направлена на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, которая направлена на маркер поверхности иммунной клетки, или детектируемой метки, но не ограничивается ими. Антитело и функциональная молекула может образовывать конъюгат посредством ковалентного присоединения, сочетания, присоединения, поперечного сшивания или чего-то подобного.
В качестве предпочтительного способа иммуноконъюгат может содержать антитело по изобретению и по меньшей мере одну молекулу, направленную на маркер поверхности опухоли, или молекулу, подавляющую опухоль. Молекула, подавляющая опухоль, может представлять собой противоопухолевые цитокины или противоопухолевые токсины. Предпочтительно, цитокины включают IL-12, IL-15, IFN-бета, TNF-альфа, но не ограничиваются ими. Молекулы, направленные на маркеры поверхности опухоли, например, могут действовать синергетически с антителами по изобретению с более точным направлением на опухолевые клетки.
В качестве предпочтительного способа иммуноконъюгат может содержать антитело по настоящему изобретению и детектируемую метку. Такие детектируемые метки включают флуоресцентные метки, хромогенные метки, такие как ферменты, простетические группы, флуоресцентные вещества, люминесцентные вещества, биолюминесцентные вещества, радиоактивные вещества, позитрон-излучающие металлы и нерадиоактивный парамагнитный ион металла, но не ограничиваются ими. Также может быть включен более чем один маркер. Метка, используемая для мечения антитела в целях выявления и/или анализа и/или диагностики, зависит от применяемой конкретной методики и/или способа выявления/анализа/диагностики, например, иммуногистохимическое окрашивание (ткани) образцов, проточная цитометрия и тому подобное. Подходящие метки для методик и/или способов выявления/анализа/диагностики, известные в данной области, хорошо известны специалистам в данной области.
В качестве предпочтительного способа иммуноконъюгат может содержать антитело по изобретению, а также молекулу, направленную на маркер поверхности иммунной клетки. Молекула, направленная на маркеры поверхности иммунных клеток, может распознавать иммунные клетки и доставлять антитела по изобретению к иммунным клеткам, так что антитела по изобретению могут направлять иммунные клетки на опухолевые клетки и, таким образом, активировать иммуноцит для специфичного уничтожения опухоли.
Как средство химического генерирования иммуноконъюгата путем конъюгации, либо прямо, либо косвенно (например, посредством линкера), иммуноконъюгат может быть получен в виде слитого белка, содержащего антитело по изобретению и другие подходящие белки. Слитый белок может быть получен способом, известным в данной области, например, рекомбинантно получен посредством конструирования и впоследствии экспрессирования молекулы нуклеиновой кислоты, которая содержит нуклеотидную последовательность, кодирующую антитело, в рамке считывания с нуклеотидной последовательностью, кодирующей подходящую метку.
В другом аспекте изобретения предложена молекула нуклеиновой кислоты, кодирующая по меньшей мере одно антитело по изобретению, функциональный вариант или его иммуноконъюгат.Получая релевантную последовательность, способ рекомбинации можно использовать для получения релевантной последовательности в больших количествах. Это обычно осуществляют посредством клонирования ее в вектор, переноса его в клетку и затем выделения релевантной последовательности из пролиферирующих клеток-хозяев традиционными способами.
Настоящее изобретение также относится к векторам, содержащим соответствующие ДНК-последовательности, описанные выше, а также соответствующие промоторы или контрольные последовательности. Данные векторы можно использовать для трансформации соответствующей клетки-хозяина для того, чтобы сделать возможной экспрессию белка. Клетка-хозяин может представлять собой прокариотическую клетку, такую как бактериальная клетка; или клетку низших эукариот, например, дрожжевую клетку; или клетку высших эукариот, например, клетку млекопитающего.
Химерный антигенный рецептор и генетически модифицированная иммунная клетка
В настоящем изобретении предложен химерный антигенный рецептор, экспрессируемый на поверхности эффекторной иммунной клетки (иммунная клетка), где химерный антигенный рецептор содержит последовательно связанные: внеклеточную область связывания, трансмембранную область и внутриклеточную сигнальную область, и внеклеточная область связывания содержит антитело по изобретению. Посредством экспрессирования химерного антигенного рецептора на поверхности эффекторных иммунных клеток, эффекторные иммунные клетки могут оказывать специфичное цитотоксическое действие на опухолевые клетки, которые экспрессируют GPC3.
Термины «иммунные клетки» и «эффекторные иммунные клетки», в том виде, в котором они используются в данном документе, используются взаимозаменяемо и включают или NKT-клетки и тому подобное; и предпочтительно NK-клетки и Т-лимфоциты.
В качестве предпочтительного воплощения настоящего изобретения антитело, содержащееся в химерном антигенном рецепторе, представляет собой одноцепочечное антитело, которое связано с CD8 или трансмембранной областью CD28 через шарнирную область CD 8, и за трансмембранной областью сразу следует внутриклеточная сигнальная область.
Изобретение также включает нуклеиновые кислоты, кодирующие химерные антигенные рецепторы. Настоящее изобретение также относится к вариантам описанных выше полинуклеотидов, которые кодируют полипептид или фрагмент, аналог и производное полипептида, имеющее такую же аминокислотную последовательность, как и настоящее изобретение.
Трансмембранная область химерного антигенного рецептора может быть выбрана из трансмембранной области белка, такого как CD8 или CD28. Белок CD8 человека представляет собой гетеродимер, состоящий из двух цепей, αβ или γδ. В одном воплощении трансмембранная область выбрана из трансмембранной области CD8a или CD28. Кроме того, шарнир CD8a представляет собой гибкую область, так что CD8 или CD28 и трансмембранную область, а также гибкую область используют для соединения домена, распознающего мишень, scFv химерного антигенного рецептора CAR с внутриклеточной сигнальной областью.
Внутриклеточная сигнальная область может быть выбрана из группы, состоящей из внутриклеточной сигнальной области CD3ζ, FcεRIγ, CD27, CD28, CD137, CD134, MyD88, белка CD4 и их комбинаций. Молекула CD3 состоит из пяти субъединиц, в которых субъединица CD3£ (также известная как CD3 дзета, сокращенно Z) содержит 3 мотива ITAM, которые являются важными областями сигнальной трансдукции в комплексе TCR-CD3. CD3δZ представляет собой усеченную последовательность CD3ζ без мотива ITAM и обычно конструируется в настоящем изобретении в качестве отрицательного контроля. FcsRIγ главным образом распределяется на поверхности тучных клеток и базофилов, который содержит мотив ITAM, который сходен с CD3ζ по структуре, распределению и функции. Кроме того, как указано выше, CD28, CD 137 и CD 134 представляют собой костимулирующие сигнальные молекулы. Костимулирующий эффект их внутриклеточных сигнальных сегментов при связывании с соответствующими лигандами приводит к продолжительной пролиферации эффекторных иммунных клеток, главным образом Т-лимфоцитов, и повышению уровня цитокинов, таких как IL-2 и INF-γ, секретируемых эффекторными иммунными клетками, и период выживания и противоопухолевый эффект CAR эффекторных иммунных клеток in vivo увеличиваются.
Химерный антигенный рецептор по настоящему изобретению может быть последовательно связан следующим образом:
антитело по изобретению CD8 и CD3ζ,;
антитело по изобретению CD8, CD137 и CD3ζ;
антитело по изобретению, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28 и CD3ζ; или
антитела по изобретению, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28, CD137 и CD3ζ.
Их комбинации, где CD28a в релевантном химерном антигенном рецепторном белке представляет трансмембранную область молекулы CD28, и CD28b представляет внутриклеточную сигнальную область молекулы CD28. Разные химерные антигенные рецепторы, описанные выше, в совокупности называются scFv (GPC3)-CAR.
Согласно настоящему изобретению также предложен вектор, содержащий указанную выше нуклеиновую кислоту, кодирующую химерный антигенный рецепторный белок, экспрессируемый на поверхности эффекторной иммунной клетки. В конкретном воплощении вектор, используемый в настоящем изобретении, представляет собой лентивирусный плазмидный вектор pWPT-eGFP. Данная плазмида принадлежит к третьему поколению самоинактивирующей лентивирусной векторной системы. Система имеет три плазмиды: пакующая плазмида psPAX2, кодирующая белок Gag/Pol, кодирующий белок Rev; плазмида, кодирующая белки вирусной оболочки, PMD2.G, кодирующая белок VSV-G; и пустой вектор pWPT-eGFP, который может быть использован для рекомбинантного введения представляющей интерес последовательности нуклеиновой кислоты, то есть нуклеиновой кислоты, кодирующей CAR. В пустом векторе pWPT-eGFP экспрессия усиленного зеленого флуоресцентного белка (eGFP) регулируется промотором фактора элонгации-1α (EF-1α). В то время, когда в рекомбинантном экспрессионном векторе pWPT-eGFP-F2A-CAR, содержащем последовательность нуклеиновой кислоты, кодирующую CAR, коэкспрессия eGFP и CAR активируется посредством рибосомальной «перепрыгивающей» последовательности 2А (сокращенно F2A) из вируса ящура (FMDV).
Изобретение также включает вирусы, содержащие векторы, описанные выше. Вирусы по изобретению включают упакованные патогенные вирусы, а также вирусы, подлежащие упаковке, которые содержат необходимые компоненты для упаковывания в патогенные вирусы. Другие вирусы, известные в данной области, которые могут быть использованы для трансдукции экзогенных генов в эффекторные иммунные клетки, и их соответствующие плазмидные векторы также полезны в настоящем изобретении.
Настоящее изобретение дополнительно включает генетически модифицированный Т-лимфоцит, который трансдуцируют нуклеиновой кислотой по настоящему изобретению или трансдуцируют указанной выше рекомбинантной плазмидой, содержащей нуклеиновую кислоту по настоящему изобретению, или вирусной системой, содержащей плазмиду. Традиционные способы трансдукции на основе нуклеиновой кислоты в данной области, включая способы невирусной и вирусной трансдукции, можно использовать в настоящем изобретении. Способы невирусной трансдукции включают электропорацию и способы на основе транспозонов. С недавнего времени нуклеофектором, прибором для нуклеофекции, разработанным Атаха, можно прямо вводить чужеродные гены в ядро с достижением высокоэффективной трансдукции целевых генов. Кроме того, по сравнению с традиционной электропорацией, эффективность трансдукции системы транспозонов на основе системы Sleeping Beauty или транспозона PiggyBac была значительно улучшена. Сообщалось о сочетании прибора для нуклеофекции и системы транспозонов SB Sleeping Beauty [Davies JK., et al. Combining CD 19 redirection and alloanergization to generate tumor-specific human T cells for allogeneic cell therapy of B-cell malignancies. Cancer Res, 2010, 70(10): OF1-10.], и высокая эффективность трансдукции и сайт-направленная интеграция целевых генов может быть достигнута данным способом. В одном воплощении изобретения способ трансдукции Т-лимфоцита, модифицированного геном химерного антигенного рецептора, представляет собой способ трансдукции на основе вируса, такого как ретровирус или лентивирус. Способ имеет преимущества высокой эффективности трансдукции и стабильной экспрессии экзогенного гена, и может быть уменьшено время для in vitro культивирования Т-лимфоцитов до клинического уровня. Нуклеиновая кислота для трансдукции экспрессируется на поверхности трансгенных Т-лимфоцитов посредством транскрипции, трансляции. Анализ цитотоксичности in vitro, проводимый на разных культивируемых опухолевых клетках, продемонстрировал то, что эффекторные иммунные клетки по настоящему изобретению оказывают высокоспецифичное цитотоксическое действие в отношении опухолевых клеток (также известное как цитотоксичность). Вследствие этого, нуклеиновую кислоту, кодирующую химерный антигенный рецепторный белок по настоящему изобретению, плазмиду, содержащую нуклеиновую кислоту, вирус, содержащий плазмиду, и трансгенные эффекторные иммунные клетки, трансфицированные нуклеиновой кислотой, плазмидой или вирусом, описанными выше, можно эффективно использовать в иммунотерапии опухоли.
Иммунные клетки по настоящему изобретению могут также нести экзогенные кодирующие последовательности для цитокинов, включая IL-12, IL-15 или IL-21, но, не ограничиваясь ими. Данные цитокины обладают иммуномодулирующей или противоопухолевой активностью, усиливают функцию эффекторных Т-клеток и активированных NK-клеток или прямо оказывают противоопухолевое действие. Вследствие этого, специалисты в данной области поймут, что применение данных цитокинов поможет иммунным клеткам лучше функционировать.
Наряду с химерным антигенным рецептором, описанным выше, иммунные клетки по настоящему изобретению могут также экспрессировать другой химерный антигенный рецептор, который не содержит CD3ζ, а содержит внутриклеточный сигнальный домен CD28 и внутриклеточный сигнальный домен CD137 или комбинацию обоих.
Иммунные клетки по настоящему изобретению могут также экспрессировать хемокиновые рецепторы; хемокиновые рецепторы включают CCR2, но не ограничиваются им. Специалист в данной области поймет, что хемокиновый рецептор CCR2 может конкурентно связывать CCR2 в организме и является полезным для блокирования метастаза опухоли.
Иммунные клетки по настоящему изобретению могут также экспрессировать миРНК, которая может снижать уровень экспрессии PD-1, или PD-L1-блокирующий белки. Специалист в данной область поймет, что конкурентное блокирование взаимодействия между PD-L1 и его рецептором PD-1 облегчит восстановление противоопухолевых Т-клеточных ответов, ингибируя, таким образом, рост опухоли.
Иммунные клетки по настоящему изобретению могут также экспрессировать предохранитель; предпочтительно, предохранитель включает индуцируемую каспазу-9, усеченный EGFR или RQR8.
Фармацевтическая композиция
Антитела, иммуноконъюгаты, содержащие антитела, и генетически модифицированные иммунные клетки по настоящему изобретению могут быть использованы в получении фармацевтической композиции или диагностического средства. Кроме эффективного количества антитела, иммунологического конъюгата или иммунной клетки композиция может дополнительно содержать фармацевтически приемлемый носитель. Термин «фармацевтически приемлемый» означает, что когда молекулярные соединения и композиции должным образом вводят животным или людям, они не вызывают вредных, аллергических или других нежелательных реакций.
Конкретные примеры некоторых данных веществ, которые могут быть использованы в качестве фармацевтически приемлемых носителей или их компонентов, представляют собой сахара, такие как лактоза, декстроза и сахароза; крахмалы, такие как кукурузный крахмал и картофельный крахмал; целлюлоза и ее производные, такие как карбоксиметилцеллюлоза натрия, этилцеллюлоза и метилцеллюлоза; трагакантовая камедь; солод; желатин; тальк; твердые смазочные вещества, такие как стеариновая кислота и стеарат магния; сульфат кальция; растительные масла, такие как арахисовое масло, хлопковое масло, кунжутное масло, оливковое масло, кукурузное масло и какао-масло; многоатомные спирты, такие как пропиленгликоль, глицерин, сорбит, маннит и полиэтиленгликоль; альгиновую кислоту; эмульгаторы, такие как Tween®; увлажнители, такие как натрия лаурилсульфат; красители; корригенты; таблетки, стабилизаторы; антиоксиданты; консерванты; апирогенную воду; изотонические солевые растворы; и фосфатные буферы и тому подобное.
Композиция по настоящему изобретению может быть получена в разных лекарственных формах, при необходимости, и дозировка, подлежащая введению пациенту, может быть определена лечащим врачом в соответствии с факторами, такими как тип, возраст, масса тела и общее состояние заболевания пациента, способ введения и тому подобное. Например, можно использовать инъекцию или другое лечение.
Настоящее изобретение дополнительно описано ниже со ссылкой на конкретные воплощения. Следует понимать, что данные примеры предложены только для иллюстрирования настоящего изобретения и не направлены на ограничение объема настоящего изобретения. Экспериментальные процедуры в следующих примерах, где не указаны конкретные условия, обычно проводят в соответствии с условиями, описанными в общепринятых условиях, таких как условия, подобранные Sambrook et al., Molecular Cloning Experiments Guide, Third Edition, Science Press, 2002, или в соответствии с условиями, предложенными производителем.
Пример 1. Получение специфичного одноцепочечного антитела (scFv), связывающегося с GPC3 человека
1.1. Скрининг антител, специфично связывающих GPC3, на основе фагового дисплея
Используя технологию фагового дисплея, проводили скрининг антитела, специфичного к GPC3 человека (далее в данном документе называемого huGPC3), из библиотеки природных антител полного человеческого репертуара. С данной целью бактерии в глицерине (приобретенные в Shanghai Rui Jin Biotechnology Co., Ltd.) из природной библиотеки полного человеческого репертуара одноцепочечных антител на основе фагового дисплея инокулировали в 400 мл среды 2 × YT/ампициллин, так что плотность клеток достигала OD600, равной 0,1, и инкубировали при 37°С и 200 об/мин до тех пор, пока плотность клеток не достигала OD600, равной 0,5. Клетки инфицировали 1012 БОЕ (бляшкообразующая единица) хелперного фага M13KO7 (приобретеного в Invitrogen) и инкубировали при 30°С и 50 об/мин в течение 30 минут. Затем добавляли 50 мг/л канамицина, и культивирование при встряхивании проводили при 37°С и 200 об/мин в течение 30 минут, осадок отделяли посредством центрифугирования (15 минут, 1600 × g, 4°С) и ресуспендировали в 400 мл среды 2 × YT/пенициллин/канамицин и встряхивали в течение 16 часов при 37°С и 200 об/мин. Наконец, осадок отделяли посредством центрифугирования (5000 об/мин, 4°С в течение 20 минут) и отбрасывали. Супернатант фильтровали через 0,45 мкм фильтр, и добавляли 1/4 объема 20 (масс/об.) % PEG 8000, раствора 2,5 М NaCl и инкубировали на бане со льдом в течение 1 часа с осаждением осадков бактериофага. Затем осадок осаждали посредством центрифугирования (20 мин, 8000 × g, 4°С), и супернатант отбрасывали. Фаг ресуспендировали в 25 мл предварительно охлажденного PBS (фосфатно-солевой буферный раствор) (137 мМ NaCl, 2,7 мМ KCl, 8 мМ Na2HPO4, 2 мМ KH2PO4) и центрифугировали (5 минут, 20000 × g, 4°С). К супернатанту добавляли 1/4 объема 20 (масс/об.) % PEG8000, раствора 2,5 М NaCl и инкубировали на бане со льдом в течение 30 минут с повторным осаждением фаговых частиц. Осадки центрифугировали (30 мин, 20000 × g, 4°С), и осадки с фагом повторно ресуспендировали в 2 мл предварительно охлажденного PBS, держали на льду в течение 30 мин и центрифугировали (30 мин, 17000 xg, 4°С). Супернатанты смешивали с 4 (масс/об.) % BSA в PBS в соотношении 1: 1, помещали на ротационный смеситель и инкубировали в течение 30 минут при комнатной температуре перед непосредственным использованием для скрининга.
Используя описанную выше фаговую библиотеку антител, четыре круга направленного скрининга проводили на биотинилированном рекомбинантном белке GPC3 человека (приобретенном в Shanghai Rui Jin Biotechnology Co., Ltd.) по следующей схеме: фаговую библиотеку антител инкубировали с меченным биотином антигеном GPC3 при комнатной температуре в течение 2 часов и затем инкубировали с магнитными шариками, покрытыми стрептавидином, MyOne Cl (из Invitrogen), блокировали 2 (масс/об.) % BSA (бычий сывороточный альбумин, приобретенный в Shanghai Bioengineering) при комнатной температуре в течение 30 минут. Шарики затем промывали буфером PBST (содержащим 0,1% Tween-20) с удалением фагов, которые были неспецифично связаны или обладали слабой способностью к связыванию. Фаги с сильной способностью к связыванию затем элюировали с магнитных шариков раствором глицин-HCl (рН 2,2), нейтрализовали нейтрализующим раствором Tris (рН 9,1) и использовали для инфицирования Е. coli ER2738 в середине логарифмической фазы роста и для следующего круга скрининга. В четырех кругах скрининга шарики использовали в количестве 50 мкл, 20 мкл, 10 мкл и 10 мкл, и концентрации антигена GPC3, меченного биотином, составляли 200 нМ, 10 нМ, 5 нМ и 1 нМ, соответственно, и время PBST-промывания составляло 10, 10,15 и 20, соответственно.
1.2 Идентификация антител, специфично связывающих GPC3
96 клонов случайным образом отбирали в клонах, полученных из четвертого круга скрининга, и их способность к связыванию с GPC3 человека анализировали посредством ELISA (твердофазный иммуноферментный анализ) на основе одного фага. С данной целью каждую отдельную колонию инокулировали в 300 мкл среды 2 × YT/ампициллин (содержащей 2% глюкозы) в 96-луночном глубоколуночном планшете и культивировали при встряхивании при 37°С и 250 об/мин в течение 16 часов. 20 мкл культуры инокулировали в 500 мкл среды 2 × YT/ампициллин (содержащей 0,1% глюкозы) и встряхивали при 37°С и 250 об/мин в течение 1,5 часов. Для получения раствора хелперного фага брали 75 мкл M13KO7 (титр 3×1012 БОЕ/мл) и смешивали в 15 мл среды 2 × YT и добавляли в культуральный планшет в количестве 50 мкл/лунка и инкубировали при 37°С и 150 об/мин в течение 30 минут. И затем полученный раствор канамицина (брали 180 мкл 50 мг/мл канамицина и добавляли в 15 мл среды 2 × YT) добавляли в количестве 50 мкл/лунка и инкубировали при встряхивании в течение 16 часов при 37°С и 250 об/мин. Наконец, клетки осаждали посредством центрифугирования (30 мин, 5000 × g, 4°С), и супернатант переносили в новый 96-луночный глубоколуночный планшет.
Для ELISA на основе одного фага 100 нг/лунка антитегна GPC3 и белка BSA в качестве отрицательного контроля (100 мкл/лунка) использовали в 96-луночном планшете для ELISA MediSorp (приобретенном в Nunc) и осуществляли покрытие на ночь при 4°С. Каждую лунку блокировали PBST, содержащим 2 (масс./об.) % BSA. Затем лунки промывали PBST три раза, и PBST отбрасывали. Затем, каждый раствор с фагом, полученный выше, добавляли в каждую лунку планшета в количестве 100 мкл/лунка. После инкубации при 37°С в течение 2 часов планшет три раза промывали PBST. Для выявления связанного фага конъюгат антитела против М13 и пероксиддисмутазы (приобретенный в GE Healthcare) разбавляли в соотношении 1: 5000 в PBST, и отбирали 100 мкл и добавляли в каждую лунку. После инкубации при 37°С в течение 1 часа лунки три раза промывали PBST и затем три раза промывали PBS. Наконец, 50 мкл субстрата ТМВ (тетраметилбензидин) отмеряли пипеткой в лунки и проявляли в течение 10 минут при комнатной температуре с последующим добавлением 50 мкл 2М H2SO4 на лунку для остановки цветной реакции. Значения экстинкции измеряли при 450 нм с помощью иммуносорбента, связанного с ферментом (Bio-Rad).
Посредством анализа секвенирования наблюдали два разных одноцепочечных антитела Р1 В12Е (SEQ ID NO: 1 (нуклеотид), 2 (аминокислота)) и P7D4 (SEQ ID NO: 3 (нуклеотид), 4 (аминокислота)), которые демонстрировали значительно более сильный сигнал связывания с GPC3 человека (huGPC3) в анализе ELISA при отсутствии связывания с BSA (Фиг. 1).
Пример 2. Экспрессия и очистка одноцепочечного антитела против GPC3
Согласно стандартному протоколу фрагмент scFv-PlB12E амплифицировали из плазмиды (pCantab 5Е-Р1 В12Е) прошедшего скрининг клона Р1 В12Е с использованием пары праймеров V5-P1B12E-F (SEQ ID NO: 5) и V5-P1B12E-R (SEQ ID NO: 6), и фрагмент scFv-P7D4 амплифицировали из плазмиды (pCantab 5E-P7D4) прошедшего скрининг клона p7D4 (pCantab 5E-P7D4) с использованием пары праймеров V5-P7D4-F (SEQ ID NO: 7) и V5-P7D4-R (SEQ ID NO: 8), расщепляли Nhel/BamHI (приобретенной в NEB) и соединяли с векторной плазмидой pCMV-V5-Fc (Fc-фрагмент человеческого антитела IgGl сливали и экспрессировали ниже сайта множественного клонирования данного вектора, называемого ниже в данном документе V5-Fc, приобретенного в Shanghai Rui Jin Biotechnology Co., Ltd.), расщепляли NheI / BamHI с использованием ДНК лигазы Т4 (приобретенной в NEB) и трансформировали в штамм-хозяин ТОР10. Отбирали клоны, и позитивные клоны идентифицировали посредством ПЦР (полимеразная цепная реакция) и подтверждали посредством секвенирования, получая, таким образом, эукариотические экспрессионные плазмиды V5-scFv-P1B12E-Fc и V5-scFv-P7D4-Fc, соответственно.
Хорошо выросшие клетки HEK-293F, которые культивировали при 37°С, 5% СО2, 125 об/мин в течение 7 суток и центрифугировали при 4000 об/мин, соответственно трансфицировали описанными выше экспрессионными плазмидами. Осадки удаляли, и супернатант собирали и фильтровали с помощью 0,45 мкм мембраны. Процессированные образцы аффинно очищали с помощью колонки для аффинной хроматографии на основе белка А (из GE), и в конечном итоге получали очищенный слитый белок антитело-Fc scFv-P1B12E-Fc и scFv-P7D4-Fc. Результаты идентификации показаны на Фиг. 2, и их молекулярные массы составляют приблизительно 50 кДа.
Пример 3. Связывание каждой клеточной линии с одноцепочечным антителом против GPC3, анализируемое посредством проточной цитометрии
Способность каждого из антител scFv-P1B12E-Fc и scFv-P7D4-Fc к связыванию с GPC3-позитивной линией клеток гепатоцеллюлярной карциномы HepG2 (АТСС) анализировали посредством клеточного сортера с активацией флуоресценции (FACS) (BD FACSCalibur).
Конкретные способы представляют собой следующие:
1) инокулирование линии клеток гепатоцеллюлярной карциномы HepG2 в логарифмическую фазу роста в чашке 6 см при плотности клеток при инокуляции приблизительно 90% и инкубирование в течение ночи при 37°С в инкубаторе.
2) Обработка клеток 10 мМ EDTA (этилендиаминтетрауксусная кислота), сбор клеток посредством центрифугирования при 200 g × 5 мин и ресуспендирование клеток в 1% фосфатно-солевом буферном растворе (NBS PBS), содержащем телячью сыворотку в концентрации 1×106-1×107/мл, в пробирку для проточной цитометрии в количестве 100 мкл на пробирку.
3) Центрифугирование при 200 g × 5 мин и отбрасывание супернатанта.
4) Добавление антител scFv-P1B12E-Fc и scFv-P7D4-Fc, подлежащих тестированию, соответственно, при использовании PBS в качестве отрицательного контроля. Конечная концентрация антитела составляет 10 мкг/мл. 100 мкл добавляли в каждую пробирку и инкубировали на бане со льдом в течение 45 минут.
5) Добавление 2 мл 1% NBS PBS к каждой пробирке и центрифугирование при 200 g × 5 мин два раза.
6) Отбрасывание супернатанта и добавление антитела козы против человеческого антитела, флуоресцентно меченного FITC (флуоресцинизотиоцианат) (Shanghai Kangcheng Bioengineering Co., Ltd.) при разведении 1:50 при добавлении в каждую пробирку 100 мкл, инкубирование на бане со льдом в течение 45 минут.
7) Добавление 2 мл 1% NBS PBS в каждую пробирку, центрифугирование при 200 g × 5 мин два раза.
8) Отбрасывание супернатанта, ресуспендирование в 300 мкл 1% NBS PBS и выявление посредством проточной цитометрии.
9) Анализ данных с использованием программного обеспечения для анализа данных проточной цитометрии WinMDI 2.9.
Результаты показаны на Фиг. 3, и результаты проточной цитометрии демонстрировали, что антитело scFv-P7D4-Fc может специфично распознавать GPC3-экспрессирующие клетки HepG2, и антитело scFv-P1B12E-Fc не связывалось с клетками HepG2.
Пример 4. Скрининг и получение мутантов одноцепочечного антитела P7D4 с повышенной способностью к связыванию GPC3
Для усиления способности одноцепочечного антитела P7D4 к связыванию с GPC3 некоторые аминокислоты в его областях CDR1 и CDR2 тяжелой цепи или CDR1 и CDR2 легкой цепи случайным образом мутировали и конструировали соответствующие библиотеки с созревшей аффинностью H12 и L12.
4.1 Легкая и тяжелая цепи P7D4 и их CDR области
Нуклеотидная последовательность scFv P7D4 показана ниже (SEQ ID NO: 3; где положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи; положения 562-582 представляют собой CDR2 легкой цепи, положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи, положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой (Gly4Ser)3 линкер).


Аминокислотная последовательность scFv P7D4 показана ниже (SEQ ID NO: 4):

P7D4 VH CDR1:
Нуклеотидная последовательность:  (SEQ ID NO: 9);
(SEQ ID NO: 9);
аминокислотная последовательность:  (SEQ ID NO: 10).
(SEQ ID NO: 10).
P7D4 VH CDR2:
Нуклеотидная последовательность:  (SEQ IDNO: 11);
(SEQ IDNO: 11);
аминокислотная последовательность:  (SEQ ID NO: 12).
(SEQ ID NO: 12).
P7D4 VH CDR3:
Нуклеотидная последовательность:  (SEQ ID NO: 13);
(SEQ ID NO: 13);
аминокислотная последовательность: 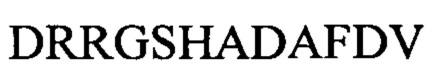 (SEQ ID NO: 14).
(SEQ ID NO: 14).
P7D4 VL CDR1:
Нуклеотидная последовательность: 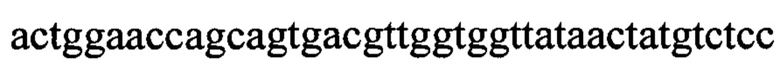 (SEQ IDNO: 15);
(SEQ IDNO: 15);
аминокислотная последовательность:  (SEQ ID NO: 16).
(SEQ ID NO: 16).
P7D4 VL CDR2:
Нуклеотидная последовательность: 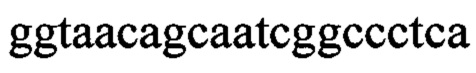 (SEQ ID NO: 17);
(SEQ ID NO: 17);
аминокислотная последовательность:  (SEQ ID NO: 18).
(SEQ ID NO: 18).
P7D4 VL CDR3:
Нуклеотидная последовательность: 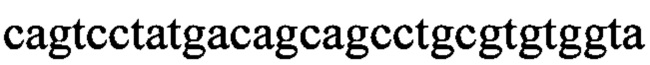 (SEQ ID NO: 19);
(SEQ ID NO: 19);
аминокислотная последовательность:  (SEQ ID NO: 20).
(SEQ ID NO: 20).
4.2 Конструирование библиотеки H12 с созревшей аффинностью
Посредством выравнивания последовательности и анализа одноцепочечного антитела P7D4 отбирали часть аминокислот в первой и второй областях CDR тяжелой цепи P7D4, и случайные мутации вводили с помощью праймеров с конструированием библиотеки тяжелых цепей с созревшей аффинностью.
Для получения фрагмента ДНК, кодирующего библиотеку мутантов P7D4, два фрагмента ДНК соответственно получали посредством ПЦР с использованием плазмиды pCantab P7D4 в качестве матрицы с последующим сплайсингом посредством обходного метода ПЦР. Конкретнее, использовали следующую процедуру: для синтеза генов ПЦР реакции проводили в объеме 50 мкл каждая с использованием плазмиды pCantab P7D4 в качестве матрицы с конечной концентрацией 0,2 мкл для каждого праймера и добавляли 5 мкл буфера 10 × KOD Plus, 4 мкл dNTP (dATP, dCTP, dGTP и dTTP, 2 мМ каждого), 2 мкл 25 мМ MgSO4 и 1 U KOD Plus (из Takara), и процедуру ПЦР начинали в термоциклере после доведения объема водой до нужного объема. Реакцию сначала нагревали до 94°С в течение 5 минут и затем инкубировали 25 циклов при 94°С в течение 30 секунд, 56°С в течение 30 секунд и 68°С в течение 30 секунд и, наконец, при 68°С в течение 10 минут. Первый фрагмент амплифицировали с использованием праймеров SI (SEQ ID NO: 21,  и 74H12F1r (SEQ ID NO: 22,
и 74H12F1r (SEQ ID NO: 22, 
 и второй фрагмент амплифицировали с использованием праймера 74H12F2f (SEQ ID NO: 23,
и второй фрагмент амплифицировали с использованием праймера 74H12F2f (SEQ ID NO: 23, 
Ожидаемые ПЦР-продукты идентифицировали посредством аналитического электрофореза в агарозном геле и очищали из образцов посредством набора Wizard SV Gel and PCR Clean-up (доступен в Promega). Данные два фрагмента добавляли в эквимолярном соотношении во второй цикл мостиковой ПЦР в качестве матрицы, и в реакционной системе еще использовалась указанная выше система KOD Plus. Реакцию сначала нагревали при 94°С в течение 5 минут и затем инкубировали 10 циклов, условия реакции каждого цикла представляли собой следующие: 94°С в течение 30 секунд, 60°С в течение 30 секунд и 68°С в течение 30 секунд и, наконец, при 68°С в течение 10 минут.Впоследствии, праймеры S1 и S6 непосредственно добавляли в реакционную систему в конечной концентрации 0,2 мкМ, и начинали программу ПЦР. Реакционную смесь сначала нагревали до 94°С в течение 5 минут и затем инкубировали 25 циклов при 94°С в течение 30 секунд, 56°С в течение 30 секунд и 68°С в течение 30 секунд и, наконец, при 68°С в течение 10 минут.Ожидаемые ПЦР-продукты разделяли посредством электрофореза в агарозном геле и очищали наборами Wizard SV Gel and PCR Clean-up в соответствии с инструкциями производителя.
В библиотеке полноразмерные фрагменты ДНК содержали сайты распознавания эндонуклеаз рестрикции sfiI и NotI на каждом конце, и обрабатывали эндонуклеазами рестрикции sfiI / NotI для расщепления рестриктазами и вставляли в фагмидный вектор pCANTAB 5Е, расщепленный теми же двумя ферментами. Продукты лигирования выделяли и обессоливали с использованием набора для электротрансформации Wizard SV Gel and PCR Clean-up.Для электротрансформации самостоятельно изготовленные компетентные клетки Е. coli ER2738 (имеющиеся в NEB) использовали с кюветой для электропорации и прибором для электропорации Gene Pulser II (из Bio-Rad). В конечном итоге подтверждали библиотеку, содержащую 8,9×109 мутантов.
4.3 Конструирование библиотеки L12 с созревшей аффинностью
Посредством выравнивания последовательности и анализа одноцепочечного антитела P7D4 отбирали часть аминокислот в первой и второй областях CDR легкой цепи P7D4, и случайные мутации вводили с помощью праймеров с конструированием библиотеки мутантов легкой цепи с созревшей аффинностью.
Для получения фрагмента ДНК, кодирующего библиотеку мутантов P7D4, два фрагмента ДНК соответственно получали посредством ПЦР с использованием в качестве матрицы плазмиды pCantab P7D4 с последующим сплайсингом посредством обходного метода ПЦР. Конкретнее, использовали следующую процедуру: для синтеза генов реакции ПЦР проводили в объеме 50 мкл каждая с использованием плазмиды pCantab P7D4 в качестве матрицы с конечной концентрацией 0,2 мкл для каждого праймера и добавляли 5 мкл буфера 10 × KOD Plus, 4 мкл dNTP (dATP, dCTP, dGTP и dTTP, 2 мМ каждого), 2 мкл 25 мМ MgSO4 1 U KOD Plus, и процедуру ПЦР начинали в термоциклере после доведения объема водой до нужного объема. Реакционную смесь сначала нагревали до 94°С в течение 5 минут и затем инкубировали 25 циклов при 94°С в течение 30 секунд, 56°С в течение 30 секунд и 68°С в течение 30 секунд и, наконец, при 68°С в течение 10 минут. Первый фрагмент амплифицировали с использованием праймеров S1 и 74L12F1r (SEQ ID NO: 83, 
 и второй фрагмент амплифицировали с использованием праймера 74L12F2f (SEQ ID NO: 84,
и второй фрагмент амплифицировали с использованием праймера 74L12F2f (SEQ ID NO: 84, 
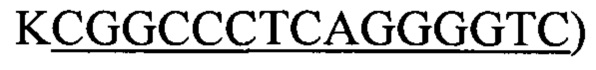 и S6.
и S6.
Ожидаемые ПЦР-продукты идентифицировали посредством аналитического электрофореза в агарозном геле и очищали из образцов посредством набора Wizard SV Gel and PCR Clean-up. Данные два фрагмента добавляли в эквимолярном соотношении во второй цикл мостиковой ПЦР в качестве матрицы, и в реакционной системе еще использовалась указанная выше система KOD Plus. В отсутствии праймеров реакцию сначала нагревали при 94°С в течение 5 минут и затем инкубировали 10 циклов, условия реакции каждого цикла представляли собой следующие: 94°С в течение 30 секунд, 60°С в течение 30 секунд и 68°С в течение 30 секунд, и, наконец, при 68°С в течение 10 минут. Далее праймеры S1 и S6 непосредственно добавляли в реакционную систему в конечной концентрации 0,2 мкМ и запускали программу ПЦР. Реакционную смесь сначала нагревали до 94°С в течение 5 минут и затем инкубировали 25 циклов при 94°С в течение 30 секунд, 56°С в течение 30 секунд и 68°С в течение 30 секунд, и, наконец, при 68°С в течение 10 минут. Ожидаемые ПЦР-продукты разделяли посредством препаративного электрофореза в агарозном геле и очищали наборами Wizard SV Gel and PCR Clean-up в соответствии с инструкциями производителя.
В библиотеке полноразмерные фрагменты ДНК на каждом конце содержали сайты распознавания эндонуклеаз рестрикции sfiI и NotI, и их расщепляли эндонуклеазами рестрикции sfiI/NotI для расщепления рестриктазами и вставляли в фагмидный вектор pCANTAB 5Е, расщепленный теми же двумя ферментами. Продукты лигирования выделяли и обессоливали с использованием набора для электротрансформации Wizard SV Gel and PCR Clean-up. Для электротрансформации самостоятельно изготовленные компетентные Е. coli ER2738 использовали с кюветой для электропорации и прибора для электропорации Gene Pulser II. В конечном итоге подтверждали библиотеку, содержащую 1,1×1010 мутантов.
Кроме того, авторы изобретения приняли метод ПЦР с ошибками для мутирования случайным образом целого фрагмента P7D4 с конструированием библиотеки Т2 с емкостью 7,9×109, в котором пара праймеров для амплификации представляет собой S1 и S6, и способ клонирования и конструирования является таким же, как способ для описанных выше H12 и L12.
Скрининг описанных выше трех библиотек с созревшей аффинностью согласуется с процедурой скрининга в Примере 1. Авторы изобретения идентифицировали шесть высокоаффинных серий P7D4 мутантных клонов am4, am14, am20, am35, am42 и Т2-23 посредством скрининга, информация о последовательностях которых показана ниже:
нуклеотидная последовательность am4 (SEQ ID NO: 24, где положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи и положения 475-516 представляют собой CDR1 легкой цепи; положения 562-582 представляют собой CDR2 легкой цепи и положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, и положения 364-408 представляют собой (Gly4Ser)3 линкер):


Аминокислотная последовательность am4 (SEQ ID NO: 25):

CDR1 тяжелой цепи:  (SEQ ID NO: 60)
(SEQ ID NO: 60)
CDR2 тяжелой цепи:  (SEQ ID NO: 61)
(SEQ ID NO: 61)
CDR3 тяжелой цепи:  (SEQ ID NO: 14)
(SEQ ID NO: 14)
CDR1 легкой цепи:  (SEQ ID NO: 16)
(SEQ ID NO: 16)
CDR2 легкой цепи:  (SEQ ID NO: 18)
(SEQ ID NO: 18)
CDR3 легкой цепи:  (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой линкерную последовательность (Gly4Ser)3.
Нуклеотидная последовательность am14 (SEQ ID NO: 26, где положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи, положения 562-582 представляют собой CDR2 легкой цепи и положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой (Gly4Ser)3 линкер):

Аминокислотная последовательность am14 (SEQ ID NO: 27):

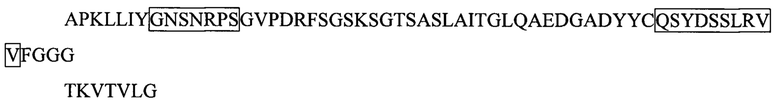
CDR1 тяжелой цепи:  (SEQ ID NO: 62)
(SEQ ID NO: 62)
CDR2 тяжелой цепи:  (SEQ ID NO: 63)
(SEQ ID NO: 63)
CDR3 тяжелой цепи:  (SEQ ID NO: 14)
(SEQ ID NO: 14)
CDR1 легкой цепи:  (SEQ ID NO: 16)
(SEQ ID NO: 16)
CDR2 легкой цепи: 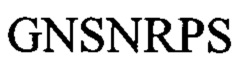 (SEQ ID NO: 18)
(SEQ ID NO: 18)
CDR3 легкой цепи:  (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой (Gly4Ser)3 линкерную последовательность.
Нуклеотидная последовательность am20 (SEQ ID NO: 28, положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи, положения 562-582 представляют собой CDR2 легкой цепи, положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи, и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой линкерную последовательность (Gly4Ser))3:


Аминокислотная последовательность am20 (SEQ ID NO: 29):

CDR1 тяжелой цепи: 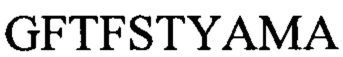 (SEQ ID NO: 64)
(SEQ ID NO: 64)
CDR2 тяжелой цепи: 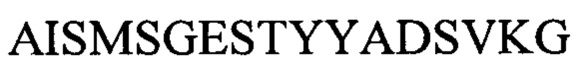 (SEQ ID NO: 65)
(SEQ ID NO: 65)
CDR3 тяжелой цепи:  (SEQ ID NO: 14)
(SEQ ID NO: 14)
CDR1 легкой цепи:  (SEQ ID NO: 16)
(SEQ ID NO: 16)
CDR2 легкой цепи:  (SEQ ID NO: 18)
(SEQ ID NO: 18)
CDR3 легкой цепи:  (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой линкерную последовательность (Gly4Ser)3.
Нуклеотидная последовательность am35 (SEQ ID NO: 30, положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи, положения 562-582 представляют собой CDR2 легкой цепи, положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи, и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой линкерную последовательность (Gly4Ser)):

Аминокислотная последовательность ат35 (SEQ ID NO: 31):

CDR1 тяжелой цепи:  (SEQ ID NO: 10)
(SEQ ID NO: 10)
CDR2 тяжелой цепи:  (SEQ ID NO: 66)
(SEQ ID NO: 66)
CDR3 тяжелой цепи:  (SEQ ID NO: 14)
(SEQ ID NO: 14)
CDR1 легкой цепи:  (SEQ ID NO: 67)
(SEQ ID NO: 67)
CDR2 легкой цепи:  (SEQ ID NO: 68)
(SEQ ID NO: 68)
CDR3 легкой цепи:  (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой линкерную последовательность (Gly4Ser)3.
Нуклеотидная последовательность am42 (SEQ ID NO: 32, положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи, положения 562-582 представляют собой CDR2 легкой цепи, положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи, и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой линкерную последовательность (Gly4Ser)3):


Аминокислотная последовательность am42 (SEQ ID NO: 33):

CDR1 тяжелой цепи:  (SEQ ID NO: 10)
(SEQ ID NO: 10)
CDR2 тяжелой цепи:  (SEQ ID NO: 66)
(SEQ ID NO: 66)
CDR3 тяжелой цепи:  (SEQ ID NO: 14)
(SEQ ID NO: 14)
CDR1 легкой цепи:  (SEQ ID NO: 69)
(SEQ ID NO: 69)
CDR2 легкой цепи:  (SEQ ID NO: 70)
(SEQ ID NO: 70)
CDR3 легкой цепи:  (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой линкерную последовательность (Gly4Ser)3.
Нуклеотидная последовательность Т2-23 (SEQ ID NO: 34, положения 76-105 представляют собой CDR1 тяжелой цепи, положения 148-198 представляют собой CDR2 тяжелой цепи, положения 295-330 представляют собой CDR3 тяжелой цепи, положения 475-516 представляют собой CDR1 легкой цепи, положения 562-582 представляют собой CDR2 легкой цепи, положения 679-708 представляют собой CDR3 легкой цепи; где положения 1-363 представляют собой нуклеотидную последовательность тяжелой цепи, и положения 409-741 представляют собой нуклеотидную последовательность легкой цепи, положения 364-408 представляют собой линкерную последовательность (Gly4Ser)3):


CDR1 тяжелой цепи:  (SEQ ID NO: 10)
(SEQ ID NO: 10)
CDR2 тяжелой цепи:  (SEQ ID NO: 71)
(SEQ ID NO: 71)
CDR3 тяжелой цепи: 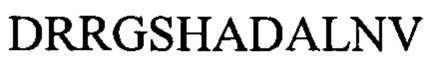 (SEQ ID NO: 72)
(SEQ ID NO: 72)
CDR1 легкой цепи:  (SEQ ID NO: 16)
(SEQ ID NO: 16)
CDR2 легкой цепи:  (SEQ ID NO: 70)
(SEQ ID NO: 70)
CDR3 легкой цепи: 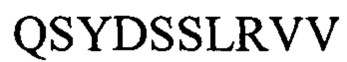 (SEQ ID NO: 20)
(SEQ ID NO: 20)
Где положения 1-121 представляют собой аминокислотную последовательность тяжелой цепи и положения 137-247 представляют собой аминокислотную последовательность легкой цепи, и положения 122-136 представляют собой линкерную последовательность (Gly4Ser)3.
4.4. Анализ SPR способности к связыванию антител серии P7D4 с GPC3
Для количественного анализа связывания антител серии P7D4 с GPC3 аффинность и кинетические параметры серии одноцепочечных антител P7D4 измеряли способом с захватом с использованием системы Biacore Т200 (из GE). Антитело против человеческого IgG (Fc) (приобретенное в GE) соединяли с поверхностью карбоксиметилдекстрана сенсорного чипа СМ5 через первичную аминогруппу посредством NHS/EDC сочетания в соответствии с инструкциями производителя. Измерения проводили в 1 × HBS-EP плюс рабочий буфер при 25°С, 30 мкл/мин, и условия регенерации представляли собой 3 М MgCl2, 10 мкл/мин в течение 30 секунд. В каждом круге цикла испытаний антитело, подлежащее тестированию, сначала захватывается на чипе. GPC3 определенной концентрации протекал по поверхности чипа. Взаимодействие между GPC3 человека и захваченным антителом вызывало изменение молекулярной концентрации на поверхности сенсорного чипа, которое измеряли в соответствии с изменениями в сигнале SPR и выражали в резонансных единицах (RU). На графике откладывали время относительно резонансных единиц (RU). На полученной сенсограмме задокументирована полная реакция, включая процессы связывания и диссоциации (Фиг. 4). Во всей кинетике одного цикла концентрации GPC3 составляли 5 нМ, 10 нМ, 20 нМ, 40 нМ и 80 нМ, соответственно. Полученные кривые оценивали с использованием пробного выпуска программы Biacore Т200 и рассчитывали значения KD аффинности. Данные по связыванию для всех ондноцепочечных антител P7D4 с GPC3 обобщены ниже в Таблице 1.

Из таблицы 1 видно, что мутантные одноцепочечные антитела серии P7D4, полученные посредством модификации, демонстрировали хорошую и значимо повышенную аффинность относительно одноцепочечного антитела P7D4.
4.5 Анализ идентификации специфичности антител серии P7D4
Для анализа специфичности связывания антитела серии P7D4 с белком GPC3 активность связывания одноцепочечного антитела серии P7D4 с членами семейства GPC человека, включая рекомбинантные человеческие GPC1 (rhGPC1), GPC2 (rhGPC2), GPC3 (rhGPC3), GPC5 (rhGPC5) и GPC6 (rhGPC6) (приобретенные в Andy Biosciences (Шанхай) Co., Ltd.), выявляли посредством ELISA, соответственно.
С данной целью, указанные выше 5 антигенов разводили 0,1 М NaHCO3 (рН 9,6) раствором для покрытия, и каждую лунку покрывали 100 нг в количестве 50 мкл/лунка в течение ночи при 4°С и блокировали 2 (масс/об.) % BSA в PBST в течение 2 часов при комнатной температуре. Затем планшет три раза промывали PBST и удаляли PBST. Далее, 100 нг каждого белка-антитела в PBST добавляли в каждую лунку планшета, и анализ каждого образца проводили в двух лунках. После инкубирования в течение 2 часов при 37°С планшеты три раза промывали PBST с последующим добавлением 100 мкл/лунка меченного HRP (пероксидазой хрена) антитела кролика против области Fc человека (приобретенного в Shanghai Rui Jin Biotechnology Co., Ltd.), разведенного в соотношении 1:20000, и инкубировали при 37°С в течение 1 часа. Для выявления лунки три раза промывали PBST с последующим трехразовым промыванием PBS и, наконец, ТМВ для проявления в течение 10 минут. Хромогенную реакцию останавливали 50 мкл 2 М H2SO4 на лунку, и значение экстинкции измеряли при 450 нм с помощью связанного с ферментом иммунодетектора (Bio-Rad).
Результаты показаны на Фиг. 5, и все одноцепочечные антитела серии P7D4 специфично связываются только с GPC3 человека, и не связываются с GPC1, GPC2, GPC5 и GPC6 человека.
Пример 5. Получение антигена GPC3 человека и гуманизированные моноклональные антитела против данного антигена
5.1 Экспрессия и очистка GPC3 человека в эукариотической экспрессионной системе
5.1.1 Конструирование и идентификация вектора GPC3
ПЦР-амплификацию проводили с использованием кДНК линии клеток гепатомы человека Huh-7 в качестве матрицы со следующими праймерами GPC3-F: 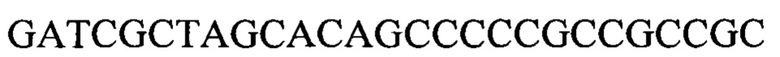 (SEQ ID NO: 54), GPC3-R:
(SEQ ID NO: 54), GPC3-R:  (SEQ ID NO: 55) с получением фрагмента GPC3 (1,6 т.п.н.) с сайтом NheI/BamHI на обоих концах. Полученный ПЦР-фрагмент дважды расщепляли эндонуклеазой NheI/BamHI (приобретенной у Fermentas), и плазмидный вектор V5H (приобретенный у Raygene) дважды расщепляли эндонуклеазой Nhel/BamHI. Вектор и вставленный фрагмент выделяли посредством электрофореза в агарозном геле и лигировали посредством лигазы ДНК Т4 (приобретенной у NEB) и трансформировали в штамм-хозяин ТОР10 (приобретенный в LIFE Co.). Проводили скрининг штаммов-хозяев на основе устойчивости к ампицилину, отбирали клоны, выделяли плазмиды и дважды расщепляли NheI/BamHI (приобретенной в Fermentas), и позитивные клоны, содержащие вставленный фрагмент, идентифицировали и проверяли посредством секвенирования с получением эукариотической экспрессионной плазмиды V5H- GPC3, содержащей правильную последовательность гена GPC3 человека.
(SEQ ID NO: 55) с получением фрагмента GPC3 (1,6 т.п.н.) с сайтом NheI/BamHI на обоих концах. Полученный ПЦР-фрагмент дважды расщепляли эндонуклеазой NheI/BamHI (приобретенной у Fermentas), и плазмидный вектор V5H (приобретенный у Raygene) дважды расщепляли эндонуклеазой Nhel/BamHI. Вектор и вставленный фрагмент выделяли посредством электрофореза в агарозном геле и лигировали посредством лигазы ДНК Т4 (приобретенной у NEB) и трансформировали в штамм-хозяин ТОР10 (приобретенный в LIFE Co.). Проводили скрининг штаммов-хозяев на основе устойчивости к ампицилину, отбирали клоны, выделяли плазмиды и дважды расщепляли NheI/BamHI (приобретенной в Fermentas), и позитивные клоны, содержащие вставленный фрагмент, идентифицировали и проверяли посредством секвенирования с получением эукариотической экспрессионной плазмиды V5H- GPC3, содержащей правильную последовательность гена GPC3 человека.
5.1.2 Экспрессия и очистка белка GPC3 человека
5.1.2.1. Липосомная трансфекция и культура плазмиды V5H-GPC3
Хорошо выросшие клетки HEK293F (HEK293F, приобретенные в LIFE Co., Ltd.) засевали в колбу для клеточной культуры при плотности 1×106 клеток/мл и инкубировали в течение ночи при 37°С и 120 об/мин для будущего использования. Плазмиду V5H-GPC3, полученную на указанной выше стадии, и липофектамин 293Fectin (приобретенный в LIFE) разводили DMEM и аккуратно перемешивали, инкубировали при комнатной температуре в течение 20 мин. Инкубированный комплекс ДНК-липосома добавляли к клеткам 293F и культивировали при 37°С и 120 об/мин в течение 120 ч. Клеточную культуру собирали и центрифугировали при 4500g в течение 15 мин. Клетки удаляли и супернатант сохраняли.
5.1.2.2. Очистка белка GPC3
Загружали 1 мл наполнителя для проведения аффинной хроматографии агарозы Ni-NTA (никель-нитрилоацетат), и колонку для проведения аффинной хроматографии Ni-NTA уравновешивали 10 объемами колонки уравновешивающего буфера (50 мМ РВ, 0,3 М NaCl, 10 мМ имидазол, рН 8,0). После центрифугирования супернатант клеточной культуры пропускали через колонку для проведения аффинной хроматографии Ni-NTA со скоростью 1 мл/мин, и фильтрат собирали и хранили при 4°С. Для промывания использовали 10 объемов колонки промывочного буфера 1 (50 мМ РВ, 0,3 М NaCl, 20 мМ имидазол, рН 8,0) и фильтрат собирали при 4°С. 4-5 объемов колонки элюирующего буфера (50 мМ РВ, 0,3 М NaCl, 250 мМ имидазол, рН 8,0) использовали для элюирования, и элюат собирали и проводили диализ в диализирующей жидкости (50 мМ РВ, рН 7,8, 0,3 М NaCl, глицерин) в течение ночи при 4°С с получением белка GPC3(H), и небольшое количество использовали в электрофорезе SDS PAGE (Фиг. 6).
5.2 Иммунизация антигеном GPC3 человека
Иммунизация рекомбинантным белком: 1 мл очищенного белка GPC3 человека GPC3(H) (1,0 мг/мл), полученного в описанном выше Примере 5.1 в качестве антигена, в достаточной степени эмульгировали и смешивали с 1 мл полного адъюванта Фрейнда (Sigma-Aldrich Co., Ltd.) для подкожной иммунизации мышей BALB/c (6-8 недельного возраста, 100 мкг человеческого белкового антигена GPC3(H) на мышь). Спустя четыре недели, человеческий антиген GPC3 эмульгировали и смешивали с неполным адъювантом Фрейнда. Мышей иммунизировали посредством внутрибрюшинной инъекции (50 мкг для каждой мыши) с последующим интервалом 2 недели и затем 50 мкг антигена внутрибрюшинно вводили для повторной иммунизации. Спустя одну неделю после четвертой повторной иммунизации, белок GPC3(H) использовали для покрытия. Титр антисыворотки мыши выявляли посредством ELISA, и повторную иммунизацию продолжали до тех пор, пока титр антисыворотки не достигал больше 105 у мышей.
Спустя три недели после последней повторной иммунизации, проводили иммунизацию 20 мкг человеческого белка GPC3(H) в селезенке для будущего использования.
5.3 Создание линий гибридомных клеток против GPC3 человека
Спустя четверо суток после повторной иммунизации в селезенке мыши, селезенки отбирали в стерильных условиях, и лимфоциты выделяли посредством фильтрации через фильтр 100 меш, сливали с линией миеломных клеток SP2/0, селективно культивировали с гипоксантином, аминоптерином и тимидином (HAT) в течение трех суток, и затем добавляли среду НТ, и данное культивирование продолжали в течение одной недели.
Антиген GPC3(H) использовали для покрытия, и проводили скрининг позитивных клонов посредством ELISA и субклонировали три раза способом предельного разведения. Клетки культивировали еще 2 месяца. Наконец, получали стабильные линии гибридомных клеток (номера клонов 5А5, 7С9 и 11D3).
5.4 Получение асцитов и очистка антитела
Мышам F1 8-10 недельного возраста инъецировали внутрибрюшинно 100 мкл пристана (приобретенного в Sigma-Aldrich), и 1 неделю спустя клоны гибридомных клеток описанного выше Примера 5.4 отбирали и инъецировали внутрибрюшинно мышам в концентрации 5×105 клеток/мышь с получением асцитов. После 7-10 суток асциты собирали и центрифугировали при 10000g в течение 10 мин, и супернатант отбирали для будущего использования. Колонку для проведения аффинной хроматографии на основе Белка G (приобретенную у GE) возвращали в комнатную температуру, и 5 объемов колонки PBS (0,01 М РВ, 0,15 М NaCl, рН 7,4) использовали для уравновешивания. Супернатант асцитов смешивали с равным объемом PBS (0,01 М РВ, 0,15 М NaCl, рН 7,4), фильтровали через 0,22 мкМ фильтр и отфильтрованный супернатант асцита загружали на колонку для проведения аффинной хроматографии на основе Белка G и промывали 5 объемами PBS. Колонку элюировали элюирующим буфером (ОД М Глицин НС1, рН 2,7), и элюирование нейтрализовали 1/10 объема нейтрализующего буфера (1M NaH2PO4, рН 9,0). Раствор диализировали против PBS (0,01 М РВ, 0,15 М NaCl, рН 7,4), во время чего два раза проводили замену PBS, и интервал между двумя сменами составлял более 5 часов. Диализированный раствор центрифугировали при 10000g в течение 10 мин и супернатант фильтровали через 0,22 мкм фильтр с получением раствора очищенного моноклонального антитела против GPC3 человека, продуцированного каждым клоном. В соответствии с анализом, 5А5 демонстрировало хорошую антиген-связывающую способность.
5.5 Гуманизация клона GPC3 5А5
5.5.1. Анализ последовательности антитела 5А5
5А5 демонстрировало хорошую антигенсвязывающую способность. Определяли последовательности вариабельных областей VL и VH антитела 5А5. Согласно схеме наименований CDR Kabat, Chothia и 1МСТопределяли последовательность шести CDR легкой и тяжелой цепи антитела.
Нуклеотидная последовательность тяжелой цепи 5А5 показана ниже (SEQ ID NO: 56, где три подчеркнутых сегмента последовательно представляют собой CDR1, CDR2, CDR3):
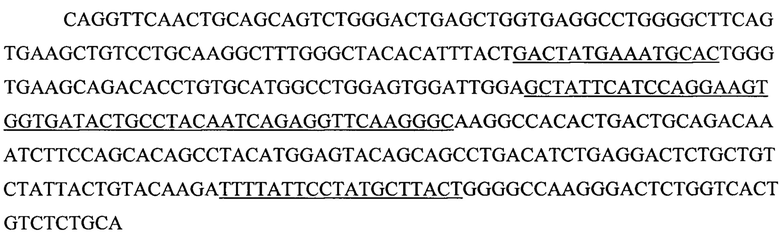
Нуклеотидная последовательность легкой цепи 5А5 показана ниже (SEQ ID NO: 57, где три подчеркнутых сегмента последовательно представляют собой CDR1, CDR2, CDR3):


Аминокислотная последовательность тяжелой цепи 5А5 (SEQ ID NO: 81, где три подчеркнутых сегмента последовательно представляют собой CDR1, CDR2, CDR3):

Аминокислотная последовательность легкой цепи 5А5 (SEQ ID NO: 82, где три подчеркнутых сегмента последовательно представляют собой CDR1, CDR2, CDR3)

5.5.2. Выбор матрицы-антитела
Существует четыре главных фактора в выборе каркасных областей матрицы-антитела: иммуногенность, антигенсвязывающие свойства, экспрессия и стабильность антитела.
(1) Определение аминокислотных остатков, поддерживающих структуру петель антитела в вариабельных областях легкой и тяжелой цепи антитела, и аминокислотных остатков областей связывания легкой и тяжелой цепи согласно WO 2008021156.
(2) Выравнивание полной длины вариабельной области легкой или тяжелой цепи 5А5 с антителами из IMGT, V BASE или NCBI.
(3) Удаление аминокислотных остатков, ассоциированных с поддержанием петли в пределах CDR областей вариабельной области легкой или тяжелой цепи 5А5 и затем выравнивание последовательности с антителами из IMGT, V BASE или NCBI.
(4) Удаление аминокислотных остатков в CDR областях в вариабельной области легкой цепи или тяжелой цепи 5А5 и затем выравнивание последовательности с антителами из IMGT, V BASE или NCBI.
(5) Сохранение только аминокислотных остатков, ассоциированных с поддержанием петли в последовательности легкой или тяжелой цепи 5А5, и затем выравнивание сходства последовательностей с антителами из IMGT, V BASE или NCBI.
(6) Сохранение только аминокислотных остатков, ассоциированных с поддержанием петли в легких цепях или каркасных областях тяжелых цепей 5А5, и затем выравнивание сходства последовательностей с антителами из IMGT, V BASE или NCBI.
(7) Выбор антитела с самым высоким сходством в качестве матрицы-антитела в соответствии с результатами выравнивания сходства последовательностей.
(8) Результаты выравнивания сходства последовательностей тяжелой цепи: на основе сходства в качестве матрицы-антитела для тяжелой цепи выбирали 5А5 VH169 * 06 (IMGT, учетные номера: L22583).
(9) Результаты выравнивания сходства последовательностей легкой цепи: на основе сходства в качестве матрицы-антитела для легкой цепи 5А5 выбирали VK2D_29 * 02 (IMGT, учетные номера: U41644).
5.5.3 Трансплантация CDR
Области CDR легкой цепи или тяжелой цепи антитела 5А5 заменяли областями CDR матрицы-антитела. В конечном итоге, идентифицировали гуманизированное антитело (Y035). Его аминокислотная последовательность показана ниже:
Тяжелая цепь гуманизированного Y035 (SEQ ID NO: 58):

аминокислотная последовательность CDR1 тяжелой цепи представляет собой: Asp Tyr Glu Met His (SEQ ID NO: 73);
аминокислотная последовательность CDR2 тяжелой цепи представляет собой: Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe Lys Gly (SEQ ID NO: 74);
аминокислотная последовательность CDR3 тяжелой цепи представляет собой: Phe Tyr Ser Tyr Ala Tyr (SEQ ID NO: 75).
Легкая цепь гуманизированного Y035 (SEQ ID NO: 59):

аминокислотная последовательность CDR1 легкой цепи представляет собой: Arg Ser Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu Gln (SEQ ID NO: 76);
аминокислотная последовательность CDR2 легкой цепи представляет собой: Lys Val Ser Asn Arg Phe Ser (SEQ ID NO: 77);
аминокислотная последовательность CDR3 легкой цепи представляет собой: Ser Gln Ser Ile Tyr Val Pro Tyr Thr (SEQ ID NO: 78).
5.5.4 Экспрессия и очистка гуманизированного антитела
(1) На основе аминокислотной последовательности гуманизированного антитела (Y035) конструировали и синтезировали нуклеотидную последовательность.
Синтезированная нуклеотидная последовательность легкой цепи (SEQ ID NO: 79, включая последовательность, кодирующую сигнальный пептид):

Синтезированная нуклеотидная последовательность тяжелой цепи (SEQ ID NO: 80, включая последовательность, кодирующую сигнальный пептид):

(2) Синтезированную нуклеотидную последовательность антитела (сигнальный пептид: положения 16-81 (легкая цепь), положения 31-84 (тяжелая цепь)) вставляли в экспрессионный вектор клетки млекопитающего, так чтобы сконструировать экспрессионный вектор для антител, содержащий тяжелую цепь и легкую цепь, соответственно, и последовательность антитела идентифицировали посредством секвенирования. В частности:
синтезированную легкую цепь и pIK-hu12L (приобретенный в Shanghai Rui Jin Biotechnology Co., Ltd.) дважды расщепляли EcoRV и BsiWI, и легкую цепь вставляли в pIK-hu12L.
Синтезированную тяжелую цепь и модифицированный pIH-hu12H (приобретенный в Shanghai Rui-Jin Biotechnology Co., Ltd., рестрикционный сайт NheI добавляли посредством синонимичной мутации на СН1) дважды расщепляли EcoRV и NheI, и тяжелую цепь вставляли в pIH-hu12H.
Компетентные клетки ТОРЮ трансформировали расщепленным и связанным вектором, и позитивные клоны идентифицировали посредством ПЦР. Корректные клоны подтверждали посредством секвенирования. Клетки 293F временно трансфицировали вектором посредством 293Fectin и экспрессировали. Супернатант культуры 293F собирали путем центрифугирования, фильтровали через 0,45 мкм фильтр и пропускали через колонку для аффинной хроматографии на основе белка А. А280 определяли посредством спектрометра для определения концентрации очищенного антитела.
5.6 Анализ SPR способности к связыванию гуманизированного антитела Y035 с GPC3 человека
Для количественного анализа связывания антитела Y035 с антигеном человеческого GPC3 параметры аффинности и кинетические параметры антитела Y035 измеряли посредством многоциклового кинетического анализа с использованием системы Biacore Т200 (от GE). Антиген GPC3 человека (приобретенный в Shanghai Rui-Jin Biotechnology Co., Ltd.) соединяли с карбоксиметилдекстрановой поверхностью сенсорного чипа СМ5 через первичную аминогруппу посредством NHS/EDC сочетания в соответствии с инструкциями производителя, и конечное количество конъюгированного лиганда составляло 305RU. Кинетические измерения проводили в 1 × HBS-EP плюс рабочий буфер при 25°С, 30 мкл/мин, и условия регенерации представляли собой следующие: 10 мМ глицин-HCl (рН 2,5), 10 мкл/мин в течение 25 секунд. В каждом круге цикла испытаний антитело Y035 определенной концентрации текло по поверхности чипа. Взаимодействие между антителом и белком-антигеном GPC3 человека, фиксированным на чипе, вызывало изменение молекулярной концентрации на поверхности сенсорного чипа, которое измеряли в соответствии с изменениями в сигнале SPR и выражали в резонансных единицах (RU). На графике откладывали время относительно резонансных единиц (RU), и после вычитания значения RU сравнительного канала без фиксированного антигена, на полученной сенсограмме задокументирована полная реакция, включающая процессы связывания и диссоциации. Кинетический анализ связывания антитела Y035 и антитела 5А5 с GPC3 человека показан на Фиг. 7-8. В разных циклах кинетического анализа концентрация антитела Y035 составляла 6,25 нМ, 12,5 нМ, 25 нМ, 50 нМ и 100 нМ, соответственно. Полученные кривые оценивали с использованием пробного выпуска программы Biacore Т200 и рассчитывали значения KD аффинности. Используя тот же самый способ, также анализировали параметры аффинности и кинетические параметры исходного мышиного моноклонального антитела 5А5 перед гуманизацией Y035. Данные по связыванию антител Y035 и 5А5 с белками GPC3 человека, соответственно, обобщены в Таблице 2, и гуманизированное антитело Y035 связывает человеческий белок GPC3 гораздо лучше, что мышиный 5А5 после гуманизации.

5.7 Анализ FACS активности антитела Y035 в отношении связывания клеток
Антитело Y035 анализировали в отношении способности к связыванию с GPC3-позитивной линией клеток гепатоцеллюлярной карциномы HepG2 (АТСС) посредством сортера клеток с активацией флуоресценции (FACS) с использованием негативных клеток 293Т (АТСС), экспрессирующих GPC3 в качестве отрицательного контроля.
Конкретный способ заключается в следующем:
1) инокуляция клеток HepG2 и 297Т в логарифмическую фазу роста в 6 см чашку соответственно при плотности клеток при инокуляции приблизительно 90%, и инкубация в течение ночи при 37°С в инкубаторе.
2) Расщепление клеток 10 мМ EDTA, сбор клеток посредством центрифугирования при 200 g × 5 мин и ресуспендирование клеток в 1% фосфатно-солевом буферном растворе (NBS PBS), содержащем телячью сыворотку при 1×106-1×107/мл, в пробирку для проточной цитометрии в количестве 100 мкл на пробирку.
3) Центрифугирование при 200 g × 5 мин и отбрасывание супернатанта.
4) Добавление антител Y035, подлежащих тестированию, в экспериментальные группы обеих линий клеток, соответственно, при использовании PBS без антитела в качестве холостой пробы. Конечная концентрация антитела составляет 20 мкг/мл. В каждую пробирку добавляли 100 мкл, и инкубировали на бане со льдом в течение 45 минут.
5) Добавление 2 мл 1% NBS PBS в каждую пробирку и центрифугирование при 200 g × 5 мин два раза.
6) Отбрасывание супернатанта и добавление антитела козы против человеческого антитела, меченного FITC (Shanghai Kangcheng Bioengineering Co., Ltd.) при разведении 1:100 с добавлением в каждую пробирку 100 мкл, инкубирование на бане со льдом в течение 45 минут.
7) Добавление 2 мл 1% NBS PBS в каждую пробирку, центрифугирование при 200 g × 5 мин два раза.
8) Отбрасывание супернатанта, ресуспендирование в 300 мкл 1% NBS PBS и выявление посредством проточной цитометрии.
Данные анализировали посредством программы для анализа данных, полученных посредством проточной цитометрии, Flowjo7.6, и результаты показаны на Фиг. 9. Результаты проточной цитометрии демонстрируют, что антитело Y035 может специфично распознавать клетки HepG2, экспрессирующие GPC3, и не связывается с GPC3-негативными клетками 293Т.
Пример 6. Конструирование экспрессионного вектора CAR на основе одноцепочечного антитела P7D4 и получение лентивируса
В качестве примера векторная система, используемая для лентивирусных плазмидных векторов, конструированных ниже, принадлежит к самоинактивирующейся лентивирусной векторной системе третьего поколения, которая имеет 4 плазмиды, а именно: пакующую плазмиду pMDLg RRE, кодирующую белок Gag/Pol (из addgene), пакующую плазмиду pRSV-REV, кодирующую белок Rev (из addgene); плазмиду, кодирующую белки вирусной оболочки, pCMV-VSV-G, кодирующую белок VSV-G (из addgene); и рекомбинантный экспрессионный вектор, кодирующий ген, представляющий интерес, CAR на основе пустого вектора pRRLSIN-cPPT.PGK-GFP.WPRE (из addgene), который может эффективно снижать риск образования реплицируемых лентивирусных частиц.
В настоящей системе автор настоящего изобретения сначала модифицировал пустой вектор pRRLSIN-cPPT.PGK-GFP.WPRE посредством традиционной методики молекулярного клонирования и заменял исходный промотор в векторе промотором фактора элонгации-1α (EF-1α) и добавлял сайт рестрикции Mlul между промотором и сигнальным пептидом CD8asp.Конкретнее, вектор pWPT-EGFP (приобретенный в Addgene) дважды расщепляли ClaI/SalI (приобретенной в NEB), и 1,1 т.п.н. ДНК-фрагмента выделяли и лигировали лигазой ДНК Т4 в ClaI/SalI расщепленный вектор pRRLSIN-cPPT.PGK-GFP.WPRE и трансформировали им штамм-хозяин ТОРЮ. Позитивные клоны отбирали посредством ПЦР в колониях и подтверждали посредством секвенирования. Получали рекомбинантную плазмиду pRRLSIN-cPPT.EF-1α-EGFP.WPRE.
Химерные антигенные рецепторы получали с использованием предварительно оптимизированного scFv P7D4. В Таблице 3 объясняется порядок соединения частей химерного антигенного рецептора, приведенного в пример в настоящем изобретении.

Примечание: CD28a представляет трансмембранную область молекулы CD28 и CD28b представляет внутриклеточную сигнальную область молекулы CD28.
1. Амплификация фрагментов нуклеиновой кислоты
(1) Амплификация последовательностей scFv
Фрагмент scFv P7D4 получали посредством ПЦР с использованием плазмиды V5-scFv-P7D4-Fc в качестве матрицы с прямым праймером P7D4fd (SEQ ID NO: 36, ctccacgccgccaggccgcaggtgcagctgcaggag, содержащая часть последовательности сигнального пептида CD8) и обратным праймером P7D4re (SEQ ID NO: 37,  содержащая часть последовательности шарнира CD8).
содержащая часть последовательности шарнира CD8).
(2) Последовательности нуклеиновой кислоты других частей химерного антигенного рецептора
Последовательности нуклеиновой кислоты других частей химерного антигенного рецепторного белка против GPC3, за исключением scFv P7D4, соответственно получали посредством ПЦР с использованием последовательностей SEQ ID NO: 26, 27, 28, 29 и 30, описанных в патентной заявке No. 201310164725.X в качестве матриц.
Конкретнее, фрагмент F1, содержащий последовательность CD8αsp, получали посредством ПЦР-амплификации с парой праймеров PWXLF (SEQ ID NO: 38, gcaggggaaagaatagtagaca) и PRRL-CD8SP-R1 (SEQ ID NO: 39, CGGCCTGGCGGCGTGGAG) с использованием плазмиды pRRLSIN-cPPT.EF-1α-EGFP.WPRE, сконструированной в данном примере в качестве матрицы.
Фрагмент F3-δZ, содержащий CD8-CD3δ дзета (δZ), получали посредством ПЦР-амплификации с парой праймеров PRRL-CD8hinge (SEQ ID NO: 40, accacgacgccagcgccg) и δZre (SEQ ID NO: 41, 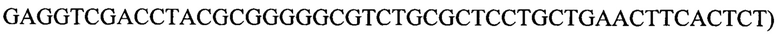 с использованием плазмиды SEQ ID NO: 26 в 201310164725.X в качестве матрицы.
с использованием плазмиды SEQ ID NO: 26 в 201310164725.X в качестве матрицы.
Фрагмент F3-Z, содержащий CD8-CD3 дзета (Z), фрагмент F3-BBZ, содержащий CD8-CD137-CD3 дзета(BBZ), фрагмент F3-28Z, содержащий CD28a-CD28b-CD3 дзета(28 г), и фрагмент F3-28BBZ, содержащий CD28a-CD28b-CD137-CD3 дзета(28 ВВг), получали посредством ПЦР-амплификации с парой праймеров PRRL-CD8hinge (SEQ ID NO: 42, accacgacgccagcgccg) и R3R (SEQ ID NO: 43, aatccagaggttgattgtcgacctagcgagggggcagggcctgc) с использованием плазмид, соответствующих SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30 в заявке №201310164725.X, соответственно.
2. Сплайсинг фрагментов нуклеиновых кислот
Фрагмент нуклеиновой кислоты F1, полученный как описано выше, и эквимолярное количество фрагмента нуклеиновой кислоты scFv P7D4 и эквимолярное количество фрагментов нуклеиновой кислоты F3-δZ или F3-Z или F3-BBZ или F3-28Z или F3-28BBZ подвергали трехсегментному сплайсингу и ПЦР, как показано на Фиг. 9, в следующих условиях: Преденатурация: 94°С в течение 4 мин; денатурация: 94°С в течение 40 с; отжиг: 60°С в течение 40 с; элонгация: 68°С в течение 140 с 5 циклов и затем общий синтез при 68°С в течение 10 мин. Добавляли ДНК-полимеразу и прямой праймер PWXLF и обратный праймер R3R (обратный праймер, соответствующий CD8-CD38 дзета, представлял собой δZ re, а другой представляет собой R3R), и затем ПЦР проводили за 30 циклов со следующими условиями амплификации: Преденатурация: 94°С в течение 4 мин; денатурация: 94°С в течение 40 с; отжиг: 60°С в течение 40 с; синтез: 68°С в течение 140 с за 30 циклов и затем общий синтез при 68°С в течение 10 мин. Фрагмент, состоящий из фрагмента F1, scFv P7D4 или мутантного scFv P7D4, и разные фрагменты F3, сконструированные в данном Примере, можно просто называть фрагментом CAR, и амплифицированные фрагменты называли следующим образом:
P7D4 scFv-δZ (SEQ ID NO: 44),
P7D4 scFv-Z (SEQ ID NO: 45),
P7D4 scFv-BBZ (SEQ ID NO: 46),
P7D4 scFv-28Z (SEQ ID NO: 47),
P7D4 scFv-28BBZ (SEQ ID NO: 48).
3. Конструирование лентивирусного плазмидного вектора
В данном примере целевой ген, состоящий из фрагмента F1, scFv P7D4 и компонентов фрагмента F3 (сокращенно CAR), дважды расщепляли рестриктазами MluI и SalI и лигировали в тот же дважды расщепленный вектор pRRLSIN-cPPT.EF-1α-EGFP.WPRE с конструированием лентивирусного вектора, экспрессирующего каждый химерный антигенный рецептор. Сконструированный вектор идентифицировали посредством расщепления MluI и SalI и корректно секвенировали, который годиться для упаковывания лентивируса. Как указано выше, релевантный ген CAR транскрибировали и транслировали в одну пептидную цепь, где химерный антигенные рецептор против GPC3 будет локализован под контролем сигнального пептида CD8α на клеточной мембране.
5 полипептидных последовательностей CAR соответственно получали посредством указанного выше конструирования, которые названы следующим образом:
P7D4-δZ (SEQ ID NO: 49);
P7D4-Z (SEQ ID NO: 50);
P7D4-BBZ (SEQ ID NO: 51);
P7D4-28Z (SEQ ID NO: 52);
P7D4-28BBZ (SEQ ID NO: 53).
4. Трансфицированные плазмидой 293T, пакующие лентивирус
Клетки НЕК-293Т (АТСС: CRL-11268), культивируемые при пассаже 6 - пассаже 10, засевали при плотности 6×106 в 10 см чашки и культивировали в течение ночи при 37°С в 5% CO2 для трансфекции. Среда представляла собой DMEM, содержащую 10% фетальную телячью сыворотку (приобретенную в Life).
Стадии трансфекции заключаются в следующем:
4.1 Получение жидкости А: растворение 5,2 мкг каждой из желательных плазмид с геном pRRLSIN-cPPT.EF-1α-CAR с 6,2 мкг пакующей плазмиды pMDLg RRE и pRSV-REV и 2,4 мкг плазмиды, кодирующей белки вирусной оболочки, pCMV-VSV-G в 800 мкл среды DMEM, не содержащей сыворотки, и тщательное перемешивание.
4.2 Получение жидкости В: растворение 60 мкг PEI (полиэтиленимин 1 мкг/мкл, приобретенный в Polysciences) в 800 мкл среды DMEM, не содержащей сыворотку, аккуратное перемешивание и инкубирование при комнатной температуре в течение 5 мин.
4.3 Образование трансфекционного комплекса: добавление жидкости А в жидкость В и аккуратное перемешивание, перемешивание с помощью вортекса или аккуратное перемешивание сразу после добавления, инкубирование при комнатной температуре в течение 20 мин.
4.4 Добавление по каплям 1,6 мл трансфекционного комплекса в клетки НЕК-293Т, и, спустя 4-5 ч, замена на DMEM с 2% FBS для трансфицированных клеток 293Т.
После 72 ч трансфекции вирус собирали посредством фильтрации с использованием 0,45 мкм фильтра (имеющегося у Millipore Corporation) и центрифугировали при 28000 об/мин с использованием ультрацентрифуги Beckman Optima L-100XP в течение 2 часов при 4°С. Супернатант отбрасывали, и полученный осадок центрифугировали при 1/10 - 1/50 стокового раствора AIM-V (приобретенного в Life) и ресуспендировали в количестве 100 мкг/пробирка в -80°С для титрования вируса или инфицирования Т-лимфоцитов.
Пример 7. Т-клетки, инфицированные рекомбинантным лентивирусом
Одноядерные клетки периферической крови человека получали из периферической крови здорового человека посредством центрифугирования в градиенте плотности (поставляемой шанхайским центром крови) и добавляли в среду для лимфоцитов AIM-V (приобретенную в Invitrogen) при плотности приблизительно 2×106/мл и добавляли. Магнитные шарики, покрытые антителами против CD3 и CD28 (Invitrogen), добавляли в соотношении клеток и магнитных частиц 1: 1, и рекомбинантный человеческий IL-2 (приобретенный в Shanghai Huaxin Biotechnology Co., Ltd.) в конечной концентрации 300 U/мл добавляли для стимуляции и культивирования в течение 48 ч. И затем Т-клетки инфицировали указанным выше рекомбинантным лентивирусом (MOI приблизительно равно 10). Инфицированные клетки пассировали раз в двое суток при плотности 5×105/мл, и рекомбинантный человеческий IL-2 в конечной концентрации 300 U/мл добавляли в культуральную среду лимфоцитов.
Инфицированные клетки CTL выявляли посредством проточной цитометрии в сутки 8 культивирования для экспрессии разных химерных антигенных рецепторов. Анализы FACS проводили с использованием человеческого рекомбинантного белка GPC3, меченного His (приобретенного в Shanghai Rui Jin Biotech Co., Ltd.); клетки сначала инкубировали с белком (50 мкг /мл) в течение 1 ч, дважды промывали D-PBS и добавляли антитело против метки His (разведение 1:50, приобретенное в Shanghai Reagent Biotechnology Co., Ltd.) и инкубировали в течение 1 ч и затем дважды промывали D-PBS и затем вторичным антителом козы против мышиного антитела, меченным FITC (Shanghai Kangcheng Biotechnology Co., Ltd.), инкубировали в течение 50 мин, промывали три раза D-PBS и тестировали посредством FACS. Неинфицированные Т-лимфоциты использовали в качестве отрицательного контроля, положительные показатели Т-клеток, инфицированных вирусом, экспрессирующих разные химерные антигенные рецепторы, показаны в Таблице 4. Результаты по положительным показателям показывают, что определенный положительный показатель CAR+T клеток можно получать с помощью лентивирусного инфицирования.

Т-клетки инфицировали вирусами, которые имели разные химерные антигенные рецепторы, упакованные, соответственно, и затем субкультивировали при плотности клеток 5×105/мл каждые два дня, считали и добавляли IL-2 (конечная концентрация 300 U/мл). На сутки 11 культивирования получали приблизительно 100-1000 кратную амплификацию, указывающую на то, что Т-клетки, экспрессирующие разные химерные антигенные рецепторы, могут быть размножены в определенном количестве in vitro, что делает возможными последующие in vitro испытания токсичности и in vivo эксперименты.
Пример 8. Испытание токсичности in vitro Т-лимфоцитов, экспрессирующих химерные антигенные рецепторы
В экспериментах по токсичности in vitro использовали следующие материалы: GPC3-позитивные клетки гепатомы (HepG2 и Huh-7) и GPC3-негативные клетки гепатомы (SK-HEP-1), как показано в Таблице 5, использовали в качестве клеток-мишеней, и эффекторные клетки представляли собой CTL, культивируемые в течение 12 суток in vitro, которые проверяли в Примере 7, и выявляли позитивную экспрессию химерного антигенного рецептора посредством FACS. Соотношения эффектор-мишень составляли 3: 1,1: 1и1:3, соответственно. Количество клеток-мишеней составляло 10000/лунка, и эффекторные клетки соответствовали разному соотношению эффектор-мишень. Каждая группа имела 5 повторных лунок, рассчитывали среднее 5 лунок, и время выявления составляло 18 ч.
Каждая экспериментальная группа и каждая контрольная группа перечислены ниже:
Каждая экспериментальная группа: каждая клетка-мишень+CTL экспрессирующие разные химерные антигенные рецепторы;
Контрольная группа 1: клетки-мишени с максимальным высвобождением LDH (лактатдегидрогеназа);
Контрольная группа 2: клетки-мишени со спонтанным высвобождением LDH;
Контрольная группа 3: эффекторные клетки со спонтанным высвобождением LDH.
Способ выявления: использовали нерадиоактивный анализ цитотоксичности CytoTox 96® (Promega), который представляет собой калориметрический анализ, который может заменять анализ высвобождения 51Cr. Анализом CytoTox 96® количественно измеряют лактатдегидрогеназу (LDH). LDH представляет собой стабильный цитозольный фермент, который высвобождается при лизисе клеток и высвобождается таким же путем, как и высвобождается радиоактивный 51 Cr. Супернатант со средой с высвобождающейся LDH можно выявить посредством 30-минутной сопряженной ферментативной реакции, в которой LDH превращает соль тетразолия (INT) в красный формазан. Количество образующегося красного продукта пропорционально числу клеток, подвергшихся лизису. Подробности можно найти в инструкциях к набору для нерадиоактивного выявления цитотоксичности CytoTox 96.
Цитотоксичность рассчитывают следующим образом:
Цитотоксичность, % = [(экспериментальная группа - контрольная группа 2 - контрольная группа 3)/(контрольная группа 1 - контрольная группа 2)]×100
Конкретнее, как показано в Таблице 5, CAR одноцепочечного антитела P7D4 по настоящему изобретению демонстрировал значимую активность уничтожения на GPC3-позитивных клетках гепатомы, и второе и третье поколения P7D4 CAR Т-клеток были чуть более сильными, чем противоопухолевая активность первого поколения. Кроме того, все CAR Т-клетки не демонстрировали цитотоксической активности на GPC3-негативных SK-HEP-1 позитивных клетках гепатомы. Данные результаты указывают на то, что P7D4 может селективно направлять на ОРС3-позитивные клетки гепатомы и эффективно их уничтожать. Кроме того, первое, второе и третье поколение CART, экспрессирующих P7D4 по настоящему изобретению, демонстрировали градиентную зависимость от соотношения эффектор-мишень, то есть чем выше соотношение эффектор-мишень, тем сильнее цитотоксический эффект.

Пример 9. Получение и сравнение CAR-T-клеток на основе Y035 и GC33
Два типа CAR-T-клеток, Y035-BBZ, Y035-28Z и Y035-28BBZ получали на основе вариабельной области легкой цепи и вариабельной области тяжелой цепи Y035, соответственно, согласно процедурам Примеров 6 и 7; и два типа CAR-T клеток, GC33-28Z и GC33-28BBZ, получали на основе вариабельной области легкой цепи и вариабельной области тяжелой цепи GC33 в качестве контроля. Части химерного антигенного рецептора соединены, как показано в Таблице 6.


Где нуклеотидная последовательность Y035-BBZ показана ниже (SEQ ID NO: 85):

Аминокислотная последовательность Y035-BBZ показана ниже (SEQ ID NO: 86):
 Нуклеотидная последовательность Y035-28Z показана ниже (SEQ ID NO: 87):
Нуклеотидная последовательность Y035-28Z показана ниже (SEQ ID NO: 87):


Аминокислотная последовательность Y035-28Z показана ниже (SEQ ID NO: 88):

Нуклеотидная последовательность Y035-28BBZ показана ниже (SEQ ID NO: 89):


Аминокислотная последовательность Y035-28BBZ показана ниже (SEQ ID NO: 90):

GPC3-позитивные клетки гепатомы Huh-7 использовали в качестве клеток-мишеней, и тест на токсичность in vitro проводили согласно способу тестирования токсичности in vitro Примера 8. Когда соотношение эффектор-мишень составляло 1:3, показатели уничтожения Y035-28Z и Y035-28BBZ на Huh-7 могут составлять выше 35%, показатель уничтожения GC33-28Z составлял только 6%, и показатель уничтожения GC33-28BZZ составлял только 23%. Когда соотношение эффектор-мишень составляло 1: 1, показатель уничтожения Y035-28Z на Huh-7 может составлять выше 60%, в то время как показатель уничтожения GC33-28Z составлял только 22%. Указанные выше результаты показывают, что активность по уничтожению клеток Y035-CAR-T клеток по настоящей заявке была значительно лучше, чем активность по уничтожению клеток GC33-CAR Т-клеток.
Пример 10. Получение биспецифичного антитела BiTE
(1) Получение нуклеотидной последовательности одноцепочечного антитела Y035 Линию гибридомных клеток, секретирующую моноклональное антитело, культивировали до логарифмической фазы роста, и считали клетки, и брали 1×107 гибридомных клеток. Из клеточного осадка выделяли тотальную РНК в соответствии с инструкцией к набору для очистки РНК TPIzol® Plus (Invitrogen, 12183-555).
а. кДНК, полученная посредством обратной транскрипции
Тотальную РНК гибридомных клеток использовали в качестве матрицы, и кДНК синтезировали посредством обратной транскрипции с использованием набора High capacity RNAto cDNA (Invitrogen, 4387406).
б. Амплификация вариабельной области антитела с помощью 5'-RACE способа
В качестве матрицы использовали кДНК и амплифицировали с помощью праймера  (SEQ ID NO: 91) и праймера
(SEQ ID NO: 91) и праймера  (SEQ ID NO: 92) с использованием набора 5'-Full RACE (TAKARA, D315). ПЦР-продукт разделяли посредством электрофореза в 1,5% агарозном геле, и полученный ПЦР-продукт представлял собой нуклеиновую кислоту одноцепочечного антитела Y035.
(SEQ ID NO: 92) с использованием набора 5'-Full RACE (TAKARA, D315). ПЦР-продукт разделяли посредством электрофореза в 1,5% агарозном геле, и полученный ПЦР-продукт представлял собой нуклеиновую кислоту одноцепочечного антитела Y035.
(2) Конструирование последовательности нуклеиновой кислоты одноцепочечного антитела CD3
ПЦР проводили с праймером (SEQ ID NO: 93, gatatcaaactgcagcagtcag) и праймером (SEQ ID NO: 94,  с использованием биспецифичного антитела против EpCAM/CD3, описанного в 201310025302.Х, в качестве матрицы со ссылкой на руководство KOD PLUS Enzyme. Продукт выделяли из геля и очищали, и полученный ПЦР-продукт представлял собой нуклеиновую кислоту одноцепочечного антитела CD3.
с использованием биспецифичного антитела против EpCAM/CD3, описанного в 201310025302.Х, в качестве матрицы со ссылкой на руководство KOD PLUS Enzyme. Продукт выделяли из геля и очищали, и полученный ПЦР-продукт представлял собой нуклеиновую кислоту одноцепочечного антитела CD3.
(3) Введение сайтов рестрикции NheI
ПЦР проводили с праймерами (ggctaactagagaacccactgc, SEQ ID NO: 95) и PH-R 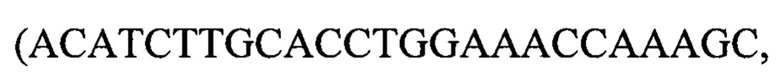 SEQ ID NO: 96) с использованием вектора PIH в качестве матрицы, и специфичная реакция может относиться к руководству KOD PLUS Enzyme. Продукт выделяли из геля, и ПЦР-продукт очищали, получая, таким образом, короткий фрагмент с сайтом рестрикции NheI.
SEQ ID NO: 96) с использованием вектора PIH в качестве матрицы, и специфичная реакция может относиться к руководству KOD PLUS Enzyme. Продукт выделяли из геля, и ПЦР-продукт очищали, получая, таким образом, короткий фрагмент с сайтом рестрикции NheI.
(4) Конструирование кодирующей последовательности нуклеиновой кислоты биспецифичного антитела Y035/CD3
ПЦР с перекрывающимися праймерами ПЦР проводили с использованием ПЦР-продуктов, полученных на стадиях (1), (2) и (3), и ПЦР проводили с праймером (SEQ ID NO: 95) и праймером (SEQ ID NO: 92). Продукт выделяли из геля, и ПЦР-продукт очищали, получая, таким образом, последовательность, кодирующую биспецифичное антитело Y035/CD3 (SEQ ID NO: 97) со следующей последовательностью:

(5) Экспрессия и очистка биспецифичных антител, направленных на Y035/CD3
Эукариотические клетки 293F временно трансфицировали плазмидами Y035 / CD3 BiTE, и трансфекцию клеток проводили со ссылкой на инструкцию по 293Fectin. Супернатант культуры очищали посредством NiSepharose™ 6 Fast Flow (приобретено в GE Healthcare Bio-Science), металлической колонки для проведения аффинной хроматографии, с получением биспецифичного антитела Y035/CD3 (SEQ ID NO: 98) со следующей последовательностью:

GPC3-позитивные клетки HepG2 и клетки SK-Hep-1 GPC3 собирали для исследования цитотоксичности in vitro Y035/CD3 BiTE. Результаты показывают, что цитотоксичность Y035/CD3 BiTE при концентрации 0,1 нг/мл на клетках HepG2 может достигать приблизительно 60%, и цитотоксичность на SK-Hep-1 GPC3 может достигать приблизительно 40%. По сравнению с GPC3/CD3 BiTE, описанным в CN103833852A, цитотоксичность Y035/CD3 BiTE значительно повышена.
Все ссылки, указанные в настоящей заявке, включены в данный документ посредством ссылки, как если бы каждая ссылка была включена по отдельности посредством ссылки. Кроме того, следует понимать, что после чтения указанных выше идей настоящего изобретения, специалисты в данной области могут вносить разные модификации или изменения в настоящее изобретение, и такие эквивалентные формы также попадают в объем прилагаемой формулы изобретения настоящей заявки.
--->
Перечень последовательностей
<110> Carsgen therapeutics limited
<120> Антитело против глипикана-3 и его применение
<130> 165497 PCWO
<150> 201510481235.1
<151> 2015-08-03
<160> 98
<170> PatentIn version 3.3
<210> 1
<211> 729
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of single chain antibody P1B12E
<400> 1
caggtgcagc tggtggaatc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc agctatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggacg aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gaagtcccca 300
tttagtggga cccccgggga gtactggggc agaggaaccc tggtcaccgt ctcgagtggt 360
ggaggcggtt caggcggagg tggttctggc ggtggcggat cggaaattgt gttgacacag 420
tctccaggca ccctgtcttt atctccaggg gaaagagcca ccctctcctg cagggccagt 480
cagagtgtta gcagcaacta cttagcctgg tatcagcaga aacctggcca ggctcccaga 540
ctcctcatct atggtgcatc ccgcagggcc actggcatcc cagacaggtt cagtggcagt 600
gggtctggga cagacttcac tctcaccatc agcagagtgg agcctgaaga ctttgcagtg 660
tatcactgtc agcagtatgg tgcctcacct aagactttcg gccaagggac caagctggag 720
atcaaacgt 729
<210> 2
<211> 243
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of single chain antibody P1B12E
<400> 2
Gln Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Ser Pro Phe Ser Gly Thr Pro Gly Glu Tyr Trp Gly Arg Gly
100 105 110
Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr
130 135 140
Leu Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser
145 150 155 160
Gln Ser Val Ser Ser Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Leu Leu Ile Tyr Gly Ala Ser Arg Arg Ala Thr Gly
180 185 190
Ile Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Thr Ile Ser Arg Val Glu Pro Glu Asp Phe Ala Val Tyr His Cys Gln
210 215 220
Gln Tyr Gly Ala Ser Pro Lys Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys Arg
<210> 3
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of single chain antibody P7D4
<400> 3
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtgg ttataactat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta tggtaacagc aatcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 4
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of single chain antibody P7D4
<400> 4
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser Asn Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 5
<211> 28
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 5
acagtgctag cacaggtgca gctggtgg 28
<210> 6
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 6
ttgtcggatc cacgtttgat ctccagc 27
<210> 7
<211> 0
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 7
<210> 8
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 8
ttgtcggatc cacctaggac ggtgacc 27
<210> 9
<211> 30
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VH CDR1
<400> 9
ggattcacct tcagtagcta tgctatgcac 30
<210> 10
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VH CDR1
<400> 10
Gly Phe Thr Phe Ser Ser Tyr Ala Met His
1 5 10
<210> 11
<211> 51
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VH CDR2
<400> 11
gctattagtg gtagtggtgg tagcacatac tacgcagact ccgtgaaggg c 51
<210> 12
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VH CDR2
<400> 12
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 13
<211> 36
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VH CDR3
<400> 13
gatcgacgag ggagccacgc tgatgctttt gatgtc 36
<210> 14
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VH CDR3
<400> 14
Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val
1 5 10
<210> 15
<211> 42
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VL CDR1
<400> 15
actggaacca gcagtgacgt tggtggttat aactatgtct cc 42
<210> 16
<211> 14
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VL CDR1
<400> 16
Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser
1 5 10
<210> 17
<211> 21
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VL CDR2
<400> 17
ggtaacagca atcggccctc a 21
<210> 18
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VL CDR2
<400> 18
Gly Asn Ser Asn Arg Pro Ser
1 5
<210> 19
<211> 30
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 VL CDR3
<400> 19
cagtcctatg acagcagcct gcgtgtggta 30
<210> 20
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4 VL CDR3
<400> 20
Gln Ser Tyr Asp Ser Ser Leu Arg Val Val
1 5 10
<210> 21
<211> 24
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 21
caacgtgaaa aaattattat tcgc 24
<210> 22
<211> 63
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<220>
<221> misc_feature
<222> (33)..(34)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (45)..(46)
<223> n is a, c, g, or t
<400> 22
ccagcccctt gcctggagcc tggcggaccc amnncatagc atamnnactg aaggtgaatc 60
cag 63
<210> 23
<211> 72
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<220>
<221> misc_feature
<222> (31)..(32)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (40)..(41)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (43)..(44)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (47)..(47)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (49)..(50)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (52)..(53)
<223> n is a, c, g, or t
<400> 23
gctccaggca aggggctgga gtgggtctca nnkattagtn nknnkgntnn knnkacatac 60
tacgcagact cc 72
<210> 24
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of am4
<400> 24
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acgtatgcta tgacgtgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcatct attagtagta gtggtgaaag tacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtgg ttataactat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta tggtaacagc aatcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 25
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am4
<400> 25
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Ser Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser Asn Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 26
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of am14
<400> 26
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acttatgcta tggcttgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagaa attagtagtt ctggtagtag gacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtgg ttataactat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta tggtaacagc aatcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 27
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am14
<400> 27
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Glu Ile Ser Ser Ser Gly Ser Arg Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser Asn Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 28
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of am20
<400> 28
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt acgtatgcta tgaattgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagcg attagtatgt ctggtgaatc tacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtgg ttataactat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta tggtaacagc aatcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 29
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am20
<400> 29
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Thr Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Met Ser Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser Asn Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 30
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of am35
<400> 30
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagct attagtagta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtca taagtttcct gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta taagaatctt ttgcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 31
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am35
<400> 31
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly His Lys Phe Pro Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Asn Leu Leu Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 32
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of am42
<400> 32
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagct attagtagta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc ttttgatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtct tatgcataat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta taagtcttcg tctcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 33
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am42
<400> 33
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Phe Asp Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Leu Met His Asn Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Lys Ser Ser Ser Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 34
<211> 741
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of T2-23
<400> 34
caggtgcagc tgcaggagtc cgggggaggc ttagttcagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtctcagct attagtagta gtggtcgtag cacatactac 180
gcagactccg tggagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaagatcga 300
cgagggagcc acgctgatgc tttaaatgtc tggggccaag gaaccctggt caccgtctcg 360
agtggtggag gcggttcagg cggaggtggt tctggcggtg gcggatcgca gtctgccctg 420
actcagcctc cctccgcgtc cgggtctcct ggacagtcag tcaccatctc ctgcactgga 480
accagcagtg acgttggtgg ttataactat gtctcctggt accaacagta cccaggcaaa 540
gcccccaaac tcctcatcta tggtaacagc aatcggccct caggggtccc tgaccgattc 600
tctggctcca agtctggcac ctcagcctcc ctggccatca ctgggctcca ggctgaggat 660
ggggctgatt attactgcca gtcctatgac agcagcctgc gtgtggtatt cggcggaggg 720
accaaggtca ccgtcctagg t 741
<210> 35
<211> 247
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of T2-23
<400> 35
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Ser Ser Gly Arg Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Glu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala Leu Asn Val Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala Leu Thr Gln Pro Pro
130 135 140
Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr Ile Ser Cys Thr Gly
145 150 155 160
Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val Ser Trp Tyr Gln Gln
165 170 175
Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Gly Asn Ser Asn Arg
180 185 190
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
195 200 205
Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala Asp Tyr
210 215 220
Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val Val Phe Gly Gly Gly
225 230 235 240
Thr Lys Val Thr Val Leu Gly
245
<210> 36
<211> 36
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 36
ctccacgccg ccaggccgca ggtgcagctg caggag 36
<210> 37
<211> 38
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 37
cggcgctggc gtcgtggtac ctaggacggt gaccttgg 38
<210> 38
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 38
gcaggggaaa gaatagtaga ca 22
<210> 39
<211> 0
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> CGGCCTGGCGGCGTGGAG
<400> 39
<210> 40
<211> 18
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 40
accacgacgc cagcgccg 18
<210> 41
<211> 48
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 41
gaggtcgacc tacgcggggg cgtctgcgct cctgctgaac ttcactct 48
<210> 42
<211> 18
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 42
accacgacgc cagcgccg 18
<210> 43
<211> 44
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 43
aatccagagg ttgattgtcg acctagcgag ggggcagggc ctgc 44
<210> 44
<211> 1442
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 scFv-¦ДZ
<400> 44
gcaggggaaa gaatagtaga cataatagca acagacatac aaactaaaga attacaaaaa 60
caaattacaa aaattcaaaa ttttatcgat ggctccggtg cccgtcagtg ggcagagcgc 120
acatcgccca cagtccccga gaagttgggg ggaggggtcg gcaattgaac cggtgcctag 180
agaaggtggc gcggggtaaa ctgggaaagt gatgtcgtgt actggctccg cctttttccc 240
gagggtgggg gagaaccgta tataagtgca gtagtcgccg tgaacgttct ttttcgcaac 300
gggtttgccg ccagaacaca ggtgtcgtga cgcggatcca ggcctaagct tacgcgtcct 360
agcgctaccg gtcgccacca tggccttacc agtgaccgcc ttgctcctgc cgctggcctt 420
gctgctccac gccgccaggc cgcaggtgca gctgcaggag tccgggggag gcttagttca 480
gcctgggagg tccctgagac tctcctgtgc agcctctgga ttcaccttca gtagctatgc 540
tatgcactgg gtccgccagg ctccaggcaa ggggctggag tgggtctcag ctattagtgg 600
tagtggtggt agcacatact acgcagactc cgtgaagggc cggttcacca tctccagaga 660
caattccaag aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt 720
atattactgt gcgaaagatc gacgagggag ccacgctgat gcttttgatg tctggggcca 780
aggaaccctg gtcaccgtct cgagtggtgg aggcggttca ggcggaggtg gttctggcgg 840
tggcggatcg cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc 900
agtcaccatc tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg 960
gtaccaacag tacccaggca aagcccccaa actcctcatc tatggtaaca gcaatcggcc 1020
ctcaggggtc cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat 1080
cactgggctc caggctgagg atggggctga ttattactgc cagtcctatg acagcagcct 1140
gcgtgtggta ttcggcggag ggaccaaggt caccgtccta ggtaccacga cgccagcgcc 1200
gcgaccacca acaccggcgc ccaccatcgc gtcgcagccc ctgtccctgc gcccagaggc 1260
gtgccggcca gcggcggggg gcgcagtgca cacgaggggg ctggacttcg cctgtgatat 1320
ctacatctgg gcgcccttgg ccgggacttg tggggtcctt ctcctgtcac tggttatcac 1380
cagagtgaag ttcagcagga gcgcagacgc ccccgcgtag gtcgacaatc aacctctgga 1440
tt 1442
<210> 45
<211> 1745
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 scFv-Z
<400> 45
gcaggggaaa gaatagtaga cataatagca acagacatac aaactaaaga attacaaaaa 60
caaattacaa aaattcaaaa ttttatcgat ggctccggtg cccgtcagtg ggcagagcgc 120
acatcgccca cagtccccga gaagttgggg ggaggggtcg gcaattgaac cggtgcctag 180
agaaggtggc gcggggtaaa ctgggaaagt gatgtcgtgt actggctccg cctttttccc 240
gagggtgggg gagaaccgta tataagtgca gtagtcgccg tgaacgttct ttttcgcaac 300
gggtttgccg ccagaacaca ggtgtcgtga cgcggatcca ggcctaagct tacgcgtcct 360
agcgctaccg gtcgccacca tggccttacc agtgaccgcc ttgctcctgc cgctggcctt 420
gctgctccac gccgccaggc cgcaggtgca gctgcaggag tccgggggag gcttagttca 480
gcctgggagg tccctgagac tctcctgtgc agcctctgga ttcaccttca gtagctatgc 540
tatgcactgg gtccgccagg ctccaggcaa ggggctggag tgggtctcag ctattagtgg 600
tagtggtggt agcacatact acgcagactc cgtgaagggc cggttcacca tctccagaga 660
caattccaag aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt 720
atattactgt gcgaaagatc gacgagggag ccacgctgat gcttttgatg tctggggcca 780
aggaaccctg gtcaccgtct cgagtggtgg aggcggttca ggcggaggtg gttctggcgg 840
tggcggatcg cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc 900
agtcaccatc tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg 960
gtaccaacag tacccaggca aagcccccaa actcctcatc tatggtaaca gcaatcggcc 1020
ctcaggggtc cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat 1080
cactgggctc caggctgagg atggggctga ttattactgc cagtcctatg acagcagcct 1140
gcgtgtggta ttcggcggag ggaccaaggt caccgtccta ggtaccacga cgccagcgcc 1200
gcgaccacca acaccggcgc ccaccatcgc gtcgcagccc ctgtccctgc gcccagaggc 1260
gtgccggcca gcggcggggg gcgcagtgca cacgaggggg ctggacttcg cctgtgatat 1320
ctacatctgg gcgcccttgg ccgggacttg tggggtcctt ctcctgtcac tggttatcac 1380
cagagtgaag ttcagcagga gcgcagacgc ccccgcgtac cagcagggcc agaaccagct 1440
ctataacgag ctcaatctag gacgaagaga ggagtacgat gttttggaca agagacgtgg 1500
ccgggaccct gagatggggg gaaagccgca gagaaggaag aaccctcagg aaggcctgta 1560
caatgaactg cagaaagata agatggcgga ggcctacagt gagattggga tgaaaggcga 1620
gcgccggagg ggcaaggggc acgatggcct ttaccagggt ctcagtacag ccaccaagga 1680
cacctacgac gcccttcaca tgcaggccct gccccctcgc taggtcgaca atcaacctct 1740
ggatt 1745
<210> 46
<211> 1877
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 scFv-BBZ
<400> 46
gcaggggaaa gaatagtaga cataatagca acagacatac aaactaaaga attacaaaaa 60
caaattacaa aaattcaaaa ttttatcgat ggctccggtg cccgtcagtg ggcagagcgc 120
acatcgccca cagtccccga gaagttgggg ggaggggtcg gcaattgaac cggtgcctag 180
agaaggtggc gcggggtaaa ctgggaaagt gatgtcgtgt actggctccg cctttttccc 240
gagggtgggg gagaaccgta tataagtgca gtagtcgccg tgaacgttct ttttcgcaac 300
gggtttgccg ccagaacaca ggtgtcgtga cgcggatcca ggcctaagct tacgcgtcct 360
agcgctaccg gtcgccacca tggccttacc agtgaccgcc ttgctcctgc cgctggcctt 420
gctgctccac gccgccaggc cgcaggtgca gctgcaggag tccgggggag gcttagttca 480
gcctgggagg tccctgagac tctcctgtgc agcctctgga ttcaccttca gtagctatgc 540
tatgcactgg gtccgccagg ctccaggcaa ggggctggag tgggtctcag ctattagtgg 600
tagtggtggt agcacatact acgcagactc cgtgaagggc cggttcacca tctccagaga 660
caattccaag aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt 720
atattactgt gcgaaagatc gacgagggag ccacgctgat gcttttgatg tctggggcca 780
aggaaccctg gtcaccgtct cgagtggtgg aggcggttca ggcggaggtg gttctggcgg 840
tggcggatcg cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc 900
agtcaccatc tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg 960
gtaccaacag tacccaggca aagcccccaa actcctcatc tatggtaaca gcaatcggcc 1020
ctcaggggtc cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat 1080
cactgggctc caggctgagg atggggctga ttattactgc cagtcctatg acagcagcct 1140
gcgtgtggta ttcggcggag ggaccaaggt caccgtccta ggtaccacga cgccagcgcc 1200
gcgaccacca acaccggcgc ccaccatcgc gtcgcagccc ctgtccctgc gcccagaggc 1260
gtgccggcca gcggcggggg gcgcagtgca cacgaggggg ctggacttcg cctgtgatat 1320
ctacatctgg gcgcccttgg ccgggacttg tggggtcctt ctcctgtcac tggttatcac 1380
cctttactgc aaacggggca gaaagaaact cctgtatata ttcaaacaac catttatgag 1440
accagtacaa actactcaag aggaagatgg ctgtagctgc cgatttccag aagaagaaga 1500
aggaggatgt gaactgagag tgaagttcag caggagcgca gacgcccccg cgtacaagca 1560
gggccagaac cagctctata acgagctcaa tctaggacga agagaggagt acgatgtttt 1620
ggacaagaga cgtggccggg accctgagat ggggggaaag ccgagaagga agaaccctca 1680
ggaaggcctg tacaatgaac tgcagaaaga taagatggcg gaggcctaca gtgagattgg 1740
gatgaaaggc gagcgccgga ggggcaaggg gcacgatggc ctttaccagg gtctcagtac 1800
agccaccaag gacacctacg acgcccttca catgcaggcc ctgccccctc gctaggtcga 1860
caatcaacct ctggatt 1877
<210> 47
<211> 1886
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 scFv-28Z
<400> 47
gcaggggaaa gaatagtaga cataatagca acagacatac aaactaaaga attacaaaaa 60
caaattacaa aaattcaaaa ttttatcgat ggctccggtg cccgtcagtg ggcagagcgc 120
acatcgccca cagtccccga gaagttgggg ggaggggtcg gcaattgaac cggtgcctag 180
agaaggtggc gcggggtaaa ctgggaaagt gatgtcgtgt actggctccg cctttttccc 240
gagggtgggg gagaaccgta tataagtgca gtagtcgccg tgaacgttct ttttcgcaac 300
gggtttgccg ccagaacaca ggtgtcgtga cgcggatcca ggcctaagct tacgcgtcct 360
agcgctaccg gtcgccacca tggccttacc agtgaccgcc ttgctcctgc cgctggcctt 420
gctgctccac gccgccaggc cgcaggtgca gctgcaggag tccgggggag gcttagttca 480
gcctgggagg tccctgagac tctcctgtgc agcctctgga ttcaccttca gtagctatgc 540
tatgcactgg gtccgccagg ctccaggcaa ggggctggag tgggtctcag ctattagtgg 600
tagtggtggt agcacatact acgcagactc cgtgaagggc cggttcacca tctccagaga 660
caattccaag aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt 720
atattactgt gcgaaagatc gacgagggag ccacgctgat gcttttgatg tctggggcca 780
aggaaccctg gtcaccgtct cgagtggtgg aggcggttca ggcggaggtg gttctggcgg 840
tggcggatcg cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc 900
agtcaccatc tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg 960
gtaccaacag tacccaggca aagcccccaa actcctcatc tatggtaaca gcaatcggcc 1020
ctcaggggtc cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat 1080
cactgggctc caggctgagg atggggctga ttattactgc cagtcctatg acagcagcct 1140
gcgtgtggta ttcggcggag ggaccaaggt caccgtccta ggtaccacga cgccagcgcc 1200
gcgaccacca acaccggcgc ccaccatcgc gtcgcagccc ctgtccctgc gcccagaggc 1260
gtgccggcca gcggcggggg gcgcagtgca cacgaggggg ctggacttcg cctgtgattt 1320
ttgggtgctg gtggtggttg gtggagtcct ggcttgctat agcttgctag taacagtggc 1380
ctttattatt ttctgggtga ggagtaagag gagcaggctc ctgcacagtg actacatgaa 1440
catgactccc cgccgccccg ggccaacccg caagcattac cagccctatg ccccaccacg 1500
cgacttcgca gcctatcgct ccagagtgaa gttcagcagg agcgcagacg cccccgcgta 1560
ccagcagggc cagaaccagc tctataacga gctcaatcta ggacgaagag aggagtacga 1620
tgttttggac aagagacgtg gccgggaccc tgagatgggg ggaaagccgc agagaaggaa 1680
gaaccctcag gaaggcctgt acaatgaact gcagaaagat aagatggcgg aggcctacag 1740
tgagattggg atgaaaggcg agcgccggag gggcaagggg cacgatggcc tttaccaggg 1800
tctcagtaca gccaccaagg acacctacga cgcccttcac atgcaggccc tgccccctcg 1860
ctaggtcgac aatcaacctc tggatt 1886
<210> 48
<211> 2012
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of P7D4 scFv-28BBZ
<400> 48
gcaggggaaa gaatagtaga cataatagca acagacatac aaactaaaga attacaaaaa 60
caaattacaa aaattcaaaa ttttatcgat ggctccggtg cccgtcagtg ggcagagcgc 120
acatcgccca cagtccccga gaagttgggg ggaggggtcg gcaattgaac cggtgcctag 180
agaaggtggc gcggggtaaa ctgggaaagt gatgtcgtgt actggctccg cctttttccc 240
gagggtgggg gagaaccgta tataagtgca gtagtcgccg tgaacgttct ttttcgcaac 300
gggtttgccg ccagaacaca ggtgtcgtga cgcggatcca ggcctaagct tacgcgtcct 360
agcgctaccg gtcgccacca tggccttacc agtgaccgcc ttgctcctgc cgctggcctt 420
gctgctccac gccgccaggc cgcaggtgca gctgcaggag tccgggggag gcttagttca 480
gcctgggagg tccctgagac tctcctgtgc agcctctgga ttcaccttca gtagctatgc 540
tatgcactgg gtccgccagg ctccaggcaa ggggctggag tgggtctcag ctattagtgg 600
tagtggtggt agcacatact acgcagactc cgtgaagggc cggttcacca tctccagaga 660
caattccaag aacacgctgt atctgcaaat gaacagcctg agagccgagg acacggccgt 720
atattactgt gcgaaagatc gacgagggag ccacgctgat gcttttgatg tctggggcca 780
aggaaccctg gtcaccgtct cgagtggtgg aggcggttca ggcggaggtg gttctggcgg 840
tggcggatcg cagtctgccc tgactcagcc tccctccgcg tccgggtctc ctggacagtc 900
agtcaccatc tcctgcactg gaaccagcag tgacgttggt ggttataact atgtctcctg 960
gtaccaacag tacccaggca aagcccccaa actcctcatc tatggtaaca gcaatcggcc 1020
ctcaggggtc cctgaccgat tctctggctc caagtctggc acctcagcct ccctggccat 1080
cactgggctc caggctgagg atggggctga ttattactgc cagtcctatg acagcagcct 1140
gcgtgtggta ttcggcggag ggaccaaggt caccgtccta ggtaccacga cgccagcgcc 1200
gcgaccacca acaccggcgc ccaccatcgc gtcgcagccc ctgtccctgc gcccagaggc 1260
gtgccggcca gcggcggggg gcgcagtgca cacgaggggg ctggacttcg cctgtgattt 1320
ttgggtgctg gtggtggttg gtggagtcct ggcttgctat agcttgctag taacagtggc 1380
ctttattatt ttctgggtga ggagtaagag gagcaggctc ctgcacagtg actacatgaa 1440
catgactccc cgccgccccg ggccaacccg caagcattac cagccctatg ccccaccacg 1500
cgacttcgca gcctatcgct ccaaacgggg cagaaagaaa ctcctgtata tattcaaaca 1560
accatttatg agaccagtac aaactactca agaggaagat ggctgtagct gccgatttcc 1620
agaagaagaa gaaggaggat gtgaactgag agtgaagttc agcaggagcg cagacgcccc 1680
cgcgtaccag cagggccaga accagctcta taacgagctc aatctaggac gaagagagga 1740
gtacgatgtt ttggacaaga gacgtggccg ggaccctgag atggggggaa agccgcagag 1800
aaggaagaac cctcaggaag gcctgtacaa tgaactgcag aaagataaga tggcggaggc 1860
ctacagtgag attgggatga aaggcgagcg ccggaggggc aaggggcacg atggccttta 1920
ccagggtctc agtacagcca ccaaggacac ctacgacgcc cttcacatgc aggccctgcc 1980
ccctcgctag gtcgacaatc aacctctgga tt 2012
<210> 49
<211> 346
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4-¦ДZ
<400> 49
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala
145 150 155 160
Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr
165 170 175
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
180 185 190
Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
195 200 205
Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu
225 230 235 240
Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val
245 250 255
Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Thr Thr Thr Pro
260 265 270
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
275 280 285
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
290 295 300
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
305 310 315 320
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
340 345
<210> 50
<211> 447
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4-Z
<400> 50
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala
145 150 155 160
Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr
165 170 175
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
180 185 190
Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
195 200 205
Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu
225 230 235 240
Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val
245 250 255
Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Thr Thr Thr Pro
260 265 270
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
275 280 285
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
290 295 300
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
305 310 315 320
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Arg Val
325 330 335
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
340 345 350
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
355 360 365
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln
370 375 380
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
385 390 395 400
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
405 410 415
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
420 425 430
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
435 440 445
<210> 51
<211> 491
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4-BBZ
<400> 51
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala
145 150 155 160
Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr
165 170 175
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
180 185 190
Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
195 200 205
Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu
225 230 235 240
Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val
245 250 255
Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Thr Thr Thr Pro
260 265 270
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
275 280 285
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
290 295 300
Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu
305 310 315 320
Ala Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr
325 330 335
Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe
340 345 350
Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg
355 360 365
Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser
370 375 380
Arg Ser Ala Asp Ala Pro Ala Tyr Lys Gln Gly Gln Asn Gln Leu Tyr
385 390 395 400
Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys
405 410 415
Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn
420 425 430
Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu
435 440 445
Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly
450 455 460
His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr
465 470 475 480
Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 52
<211> 494
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4-28Z
<400> 52
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala
145 150 155 160
Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr
165 170 175
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
180 185 190
Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
195 200 205
Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu
225 230 235 240
Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val
245 250 255
Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Thr Thr Thr Pro
260 265 270
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
275 280 285
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
290 295 300
Thr Arg Gly Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val
305 310 315 320
Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile
325 330 335
Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
340 345 350
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
355 360 365
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Val Lys
370 375 380
Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln
385 390 395 400
Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu
405 410 415
Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln Arg
420 425 430
Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys
435 440 445
Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg
450 455 460
Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys
465 470 475 480
Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
485 490
<210> 53
<211> 536
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of P7D4-28BBZ
<400> 53
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu
20 25 30
Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Thr Phe Ser Ser Tyr Ala Met His Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
65 70 75 80
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
85 90 95
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Lys Asp Arg Arg Gly Ser His Ala Asp Ala
115 120 125
Phe Asp Val Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Ser Ala
145 150 155 160
Leu Thr Gln Pro Pro Ser Ala Ser Gly Ser Pro Gly Gln Ser Val Thr
165 170 175
Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr Asn Tyr Val
180 185 190
Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
195 200 205
Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser
210 215 220
Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu
225 230 235 240
Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser Leu Arg Val
245 250 255
Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly Thr Thr Thr Pro
260 265 270
Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu
275 280 285
Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His
290 295 300
Thr Arg Gly Leu Asp Phe Ala Cys Asp Phe Trp Val Leu Val Val Val
305 310 315 320
Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile
325 330 335
Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
340 345 350
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
355 360 365
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Lys Arg Gly
370 375 380
Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val
385 390 395 400
Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu
405 410 415
Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp
420 425 430
Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn
435 440 445
Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg
450 455 460
Asp Pro Glu Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu
465 470 475 480
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
485 490 495
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
500 505 510
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
515 520 525
His Met Gln Ala Leu Pro Pro Arg
530 535
<210> 54
<211> 27
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 54
gatcgctagc acagcccccg ccgccgc 27
<210> 55
<211> 31
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 55
gtacggatcc ttcagcgggg aatgaacgtt c 31
<210> 56
<211> 345
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of 5A5 heavy chain
<400> 56
caggttcaac tgcagcagtc tgggactgag ctggtgaggc ctggggcttc agtgaagctg 60
tcctgcaagg ctttgggcta cacatttact gactatgaaa tgcactgggt gaagcagaca 120
cctgtgcatg gcctggagtg gattggagct attcatccag gaagtggtga tactgcctac 180
aatcagaggt tcaagggcaa ggccacactg actgcagaca aatcttccag cacagcctac 240
atggagtaca gcagcctgac atctgaggac tctgctgtct attactgtac aagattttat 300
tcctatgctt actggggcca agggactctg gtcactgtct ctgca 345
<210> 57
<211> 339
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of 5A5 light chain
<400> 57
gatgttgtga tgacccaaac tccactctcc ctgcctgtca gtcttggaga tcaagcctcc 60
atctcttgca gatctagtca gagccttgta cacagtaatg gaaacaccta tttacagtgg 120
tacctgcaga agccaggcca gtctccaaag ctcctgatct acaaagtttc caatcgattt 180
tctggggtcc cagacaggtt cagtggcaga ggatcaggga cagatttcac actcaagatc 240
agcagagtgg aggctgagga tctgggagtt tatttctgct ctcaaagtat atatgttccg 300
tacacgttcg gaggagggac caagctggaa ataaaacgg 339
<210> 58
<211> 115
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> Nucleotide sequence of humanized Y035 heavy chain
<400> 58
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Asp Tyr
20 25 30
Glu Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala
115
<210> 59
<211> 113
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 light chain
<400> 59
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser Gln Ser
85 90 95
Ile Tyr Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 60
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am4 heavy chain CDR1
<400> 60
Gly Phe Thr Phe Ser Thr Tyr Ala Met Thr
1 5 10
<210> 61
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am4 heavy chain CDR2
<400> 61
Ser Ile Ser Ser Ser Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 62
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am14 heavy chain CDR1
<400> 62
Gly Phe Thr Phe Ser Thr Tyr Ala Met Ala
1 5 10
<210> 63
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am14 heavy chain CDR2
<400> 63
Glu Ile Ser Ser Ser Gly Ser Arg Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 64
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am20 heavy chain CDR1
<400> 64
Gly Phe Thr Phe Ser Thr Tyr Ala Met Ala
1 5 10
<210> 65
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am20 heavy chain CDR2
<400> 65
Ala Ile Ser Met Ser Gly Glu Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 66
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am35 heavy chain CDR2
<400> 66
Ala Ile Ser Ser Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 67
<211> 14
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am35 light chain CDR1
<400> 67
Thr Gly Thr Ser Ser Asp Val Gly His Lys Phe Pro Val Ser
1 5 10
<210> 68
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am35 light chain CDR2
<400> 68
Lys Asn Leu Leu Arg Pro Ser
1 5
<210> 69
<211> 14
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am42 light chain CDR1
<400> 69
Thr Gly Thr Ser Ser Asp Val Gly Leu Met His Asn Val Ser
1 5 10
<210> 70
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of am42 light chain CDR2
<400> 70
Lys Ser Ser Ser Arg Pro Ser
1 5
<210> 71
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of T2-23 heavy chain CDR2
<400> 71
Ala Ile Ser Ser Ser Gly Arg Ser Thr Tyr Tyr Ala Asp Ser Val Glu
1 5 10 15
Gly
<210> 72
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of T2-23 heavy chain CDR3
<400> 72
Asp Arg Arg Gly Ser His Ala Asp Ala Leu Asn Val
1 5 10
<210> 73
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 heavy chain CDR1
<400> 73
Asp Tyr Glu Met His
1 5
<210> 74
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 heavy chain CDR2
<400> 74
Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe Lys
1 5 10 15
Gly
<210> 75
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 heavy chain CDR3
<400> 75
Phe Tyr Ser Tyr Ala Tyr
1 5
<210> 76
<211> 16
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 light chain CDR1
<400> 76
Arg Ser Ser Gln Ser Leu Val His Ser Asn Gly Asn Thr Tyr Leu Gln
1 5 10 15
<210> 77
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 light chain CDR2
<400> 77
Lys Val Ser Asn Arg Phe Ser
1 5
<210> 78
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of humanized Y035 light chain CDR3
<400> 78
Ser Gln Ser Ile Tyr Val Pro Tyr Thr
1 5
<210> 79
<211> 429
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of humanized antibody Y035 light chain
<400> 79
ggatcgatat ccaccatgga catgatggtg ctggcccagt tcctggcctt cctgctgctg 60
tggttcccag gcgctagatg cgacatcgtg atgacccaga cccccctgag cctgcccgtg 120
acccccggcg agcccgccag catcagctgc cggagcagcc agagcctggt gcacagcaac 180
ggcaacacct acctgcagtg gtacctgcag aagcccggcc agagccccca gctgctgatc 240
tacaaggtga gcaaccggtt cagcggcgtg cccgaccggt tcagcggcag cggcagcggc 300
accgacttca ccctgaagat cagccgggtg gaggccgagg acgtgggcgt gtactactgc 360
agccagagca tctacgtgcc ctacaccttc ggccagggca ccaagctgga gatcaaacgt 420
acggtggct 429
<210> 80
<211> 441
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of humanized antibody Y035 heavy chain
<400> 80
ggatcgatat ctgcggccta tctagccacc atgcgggtgc tgatcctgct gtggctgttt 60
accgccttcc ccggcttcct gagcgaggtg cagctggtgc agagcggcgc cgaggtgaag 120
aagcccggcg ccagcgtgaa ggtgagctgc aaggccagcg gctacacctt cagcgactac 180
gagatgcact gggtgcggca ggcccccggc cagggcctgg agtggatggg cgccatccac 240
cccggcagcg gcgacaccgc ctacaaccag cggttcaagg gccgggtgac catcaccgcc 300
gacaagagca ccagcaccgc ctacatggag ctgagcagcc tgcggagcga ggacaccgcc 360
gtgtactact gcgcccggtt ctacagctac gcctactggg gccagggcac cctggtgacc 420
gtgagcgccg ctagcaccaa a 441
<210> 81
<211> 115
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of 5A5 heavy chain
<400> 81
Gln Val Gln Leu Gln Gln Ser Gly Thr Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Leu Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Val Lys Gln Thr Pro Val His Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Tyr Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala
115
<210> 82
<211> 113
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of 5A5 light chain
<400> 82
Asp Val Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Arg Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Phe Cys Ser Gln Ser
85 90 95
Ile Tyr Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 83
<211> 68
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<220>
<221> misc_feature
<222> (42)..(43)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (46)..(46)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (48)..(49)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (52)..(52)
<223> n is a, c, g, or t
<400> 83
gtttgggggc tttgcctggg tactgttggt accaggagac mnnahnmnna hnaccaacgt 60
cactgctg 68
<210> 84
<211> 59
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<220>
<221> misc_feature
<222> (33)..(34)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (36)..(37)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (39)..(40)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (42)..(43)
<223> n is a, c, g, or t
<400> 84
acccaggcaa agcccccaaa ctcctcatct atnnknnknn knnkcggccc tcaggggtc 59
<210> 85
<211> 1398
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of Y035-BBZ
<400> 85
gaggtgcagc tggtgcagag cggcgccgag gtgaagaagc ccggcgccag cgtgaaggtg 60
agctgcaagg ccagcggcta caccttcagc gactacgaga tgcactgggt gcggcaggcc 120
cccggccagg gcctggagtg gatgggcgcc atccaccccg gcagcggcga caccgcctac 180
aaccagcggt tcaagggccg ggtgaccatc accgccgaca agagcaccag caccgcctac 240
atggagctga gcagcctgcg gagcgaggac accgccgtgt actactgcgc ccggttctac 300
agctacgcct actggggcca gggcaccctg gtgaccgtga gcgccggtgg aggcggttca 360
ggcggaggtg gttctggcgg tggcggatcg gacatcgtga tgacccagac ccccctgagc 420
ctgcccgtga cccccggcga gcccgccagc atcagctgcc ggagcagcca gagcctggtg 480
cacagcaacg gcaacaccta cctgcagtgg tacctgcaga agcccggcca gagcccccag 540
ctgctgatct acaaggtgag caaccggttc agcggcgtgc ccgaccggtt cagcggcagc 600
ggcagcggca ccgacttcac cctgaagatc agccgggtgg aggccgagga cgtgggcgtg 660
tactactgca gccagagcat ctacgtgccc tacaccttcg gccagggcac caagctggag 720
atcaaacgta ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 780
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 840
agggggctgg acttcgcctg tgatatctac atctgggcgc ccttggccgg gacttgtggg 900
gtccttctcc tgtcactggt tatcaccctt tactgcaaac ggggcagaaa gaaactcctg 960
tatatattca aacaaccatt tatgagacca gtacaaacta ctcaagagga agatggctgt 1020
agctgccgat ttccagaaga agaagaagga ggatgtgaac tgagagtgaa gttcagcagg 1080
agcgcagacg cccccgcgta caagcagggc cagaaccagc tctataacga gctcaatcta 1140
ggacgaagag aggagtacga tgttttggac aagagacgtg gccgggaccc tgagatgggg 1200
ggaaagccga gaaggaagaa ccctcaggaa ggcctgtaca atgaactgca gaaagataag 1260
atggcggagg cctacagtga gattgggatg aaaggcgagc gccggagggg caaggggcac 1320
gatggccttt accagggtct cagtacagcc accaaggaca cctacgacgc ccttcacatg 1380
caggccctgc cccctcgc 1398
<210> 86
<211> 466
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of Y035-BBZ
<400> 86
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Asp Tyr
20 25 30
Glu Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr
130 135 140
Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val
145 150 155 160
His Ser Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly
165 170 175
Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser
210 215 220
Gln Ser Ile Tyr Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
245 250 255
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
260 265 270
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
275 280 285
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
290 295 300
Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu
305 310 315 320
Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu
325 330 335
Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys
340 345 350
Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Lys
355 360 365
Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu
370 375 380
Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly
385 390 395 400
Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu
405 410 415
Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly
420 425 430
Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser
435 440 445
Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro
450 455 460
Pro Arg
465
<210> 87
<211> 1407
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of Y035-28Z
<400> 87
gaggtgcagc tggtgcagag cggcgccgag gtgaagaagc ccggcgccag cgtgaaggtg 60
agctgcaagg ccagcggcta caccttcagc gactacgaga tgcactgggt gcggcaggcc 120
cccggccagg gcctggagtg gatgggcgcc atccaccccg gcagcggcga caccgcctac 180
aaccagcggt tcaagggccg ggtgaccatc accgccgaca agagcaccag caccgcctac 240
atggagctga gcagcctgcg gagcgaggac accgccgtgt actactgcgc ccggttctac 300
agctacgcct actggggcca gggcaccctg gtgaccgtga gcgccggtgg aggcggttca 360
ggcggaggtg gttctggcgg tggcggatcg gacatcgtga tgacccagac ccccctgagc 420
ctgcccgtga cccccggcga gcccgccagc atcagctgcc ggagcagcca gagcctggtg 480
cacagcaacg gcaacaccta cctgcagtgg tacctgcaga agcccggcca gagcccccag 540
ctgctgatct acaaggtgag caaccggttc agcggcgtgc ccgaccggtt cagcggcagc 600
ggcagcggca ccgacttcac cctgaagatc agccgggtgg aggccgagga cgtgggcgtg 660
tactactgca gccagagcat ctacgtgccc tacaccttcg gccagggcac caagctggag 720
atcaaacgta ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 780
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 840
agggggctgg acttcgcctg tgatttttgg gtgctggtgg tggttggtgg agtcctggct 900
tgctatagct tgctagtaac agtggccttt attattttct gggtgaggag taagaggagc 960
aggctcctgc acagtgacta catgaacatg actccccgcc gccccgggcc aacccgcaag 1020
cattaccagc cctatgcccc accacgcgac ttcgcagcct atcgctccag agtgaagttc 1080
agcaggagcg cagacgcccc cgcgtaccag cagggccaga accagctcta taacgagctc 1140
aatctaggac gaagagagga gtacgatgtt ttggacaaga gacgtggccg ggaccctgag 1200
atggggggaa agccgcagag aaggaagaac cctcaggaag gcctgtacaa tgaactgcag 1260
aaagataaga tggcggaggc ctacagtgag attgggatga aaggcgagcg ccggaggggc 1320
aaggggcacg atggccttta ccagggtctc agtacagcca ccaaggacac ctacgacgcc 1380
cttcacatgc aggccctgcc ccctcgc 1407
<210> 88
<211> 469
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of Y035-28Z
<400> 88
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Asp Tyr
20 25 30
Glu Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr
130 135 140
Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val
145 150 155 160
His Ser Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly
165 170 175
Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser
210 215 220
Gln Ser Ile Tyr Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
245 250 255
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
260 265 270
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
275 280 285
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
290 295 300
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
305 310 315 320
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
325 330 335
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
340 345 350
Ala Tyr Arg Ser Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
355 360 365
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
370 375 380
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
385 390 395 400
Met Gly Gly Lys Pro Gln Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr
405 410 415
Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly
420 425 430
Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln
435 440 445
Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln
450 455 460
Ala Leu Pro Pro Arg
465
<210> 89
<211> 1533
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Nucleotide sequence of Y035-28BBZ
<400> 89
gaggtgcagc tggtgcagag cggcgccgag gtgaagaagc ccggcgccag cgtgaaggtg 60
agctgcaagg ccagcggcta caccttcagc gactacgaga tgcactgggt gcggcaggcc 120
cccggccagg gcctggagtg gatgggcgcc atccaccccg gcagcggcga caccgcctac 180
aaccagcggt tcaagggccg ggtgaccatc accgccgaca agagcaccag caccgcctac 240
atggagctga gcagcctgcg gagcgaggac accgccgtgt actactgcgc ccggttctac 300
agctacgcct actggggcca gggcaccctg gtgaccgtga gcgccggtgg aggcggttca 360
ggcggaggtg gttctggcgg tggcggatcg gacatcgtga tgacccagac ccccctgagc 420
ctgcccgtga cccccggcga gcccgccagc atcagctgcc ggagcagcca gagcctggtg 480
cacagcaacg gcaacaccta cctgcagtgg tacctgcaga agcccggcca gagcccccag 540
ctgctgatct acaaggtgag caaccggttc agcggcgtgc ccgaccggtt cagcggcagc 600
ggcagcggca ccgacttcac cctgaagatc agccgggtgg aggccgagga cgtgggcgtg 660
tactactgca gccagagcat ctacgtgccc tacaccttcg gccagggcac caagctggag 720
atcaaacgta ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 780
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 840
agggggctgg acttcgcctg tgatttttgg gtgctggtgg tggttggtgg agtcctggct 900
tgctatagct tgctagtaac agtggccttt attattttct gggtgaggag taagaggagc 960
aggctcctgc acagtgacta catgaacatg actccccgcc gccccgggcc aacccgcaag 1020
cattaccagc cctatgcccc accacgcgac ttcgcagcct atcgctccaa acggggcaga 1080
aagaaactcc tgtatatatt caaacaacca tttatgagac cagtacaaac tactcaagag 1140
gaagatggct gtagctgccg atttccagaa gaagaagaag gaggatgtga actgagagtg 1200
aagttcagca ggagcgcaga cgcccccgcg taccagcagg gccagaacca gctctataac 1260
gagctcaatc taggacgaag agaggagtac gatgttttgg acaagagacg tggccgggac 1320
cctgagatgg ggggaaagcc gcagagaagg aagaaccctc aggaaggcct gtacaatgaa 1380
ctgcagaaag ataagatggc ggaggcctac agtgagattg ggatgaaagg cgagcgccgg 1440
aggggcaagg ggcacgatgg cctttaccag ggtctcagta cagccaccaa ggacacctac 1500
gacgcccttc acatgcaggc cctgccccct cgc 1533
<210> 90
<211> 511
<212> PRT
<213> Artificial sequence
<400> 90
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Asp Tyr
20 25 30
Glu Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr
130 135 140
Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val
145 150 155 160
His Ser Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly
165 170 175
Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser
210 215 220
Gln Ser Ile Tyr Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys Arg Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
245 250 255
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
260 265 270
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
275 280 285
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
290 295 300
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
305 310 315 320
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
325 330 335
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
340 345 350
Ala Tyr Arg Ser Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys
355 360 365
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
370 375 380
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val
385 390 395 400
Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn
405 410 415
Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val
420 425 430
Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Gln
435 440 445
Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp
450 455 460
Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg
465 470 475 480
Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr
485 490 495
Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
500 505 510
<210> 91
<211> 45
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 91
tgctttggtt tccaggtgca agatgtgagg tgcagctggt gcaga 45
<210> 92
<211> 41
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 92
tatcggatcc accacctcca cgtttgatct ccagcttggt g 41
<210> 93
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 93
gatatcaaac tgcagcagtc ag 22
<210> 94
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 94
gagagggagt actcacccca ac 22
<210> 95
<211> 22
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 95
ggctaactag agaacccact gc 22
<210> 96
<211> 25
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> Primer
<400> 96
acatcttgca cctggaaacc aaagc 25
<210> 97
<211> 1473
<212> DNA
<213> Artificial sequence
<220>
<221> misc_feature
<223> encoding sequence of Y035/CD3 bispecific antibody
<400> 97
gaggtgcagc tggtgcagag cggcgccgag gtgaagaagc ccggcgccag cgtgaaggtg 60
agctgcaagg ccagcggcta caccttcagc gactacgaga tgcactgggt gcggcaggcc 120
cccggccagg gcctggagtg gatgggcgcc atccaccccg gcagcggcga caccgcctac 180
aaccagcggt tcaagggccg ggtgaccatc accgccgaca agagcaccag caccgcctac 240
atggagctga gcagcctgcg gagcgaggac accgccgtgt actactgcgc ccggttctac 300
agctacgcct actggggcca gggcaccctg gtgaccgtga gcgccggtgg aggcggttca 360
ggcggaggtg gttctggcgg tggcggatcg gacatcgtga tgacccagac ccccctgagc 420
ctgcccgtga cccccggcga gcccgccagc atcagctgcc ggagcagcca gagcctggtg 480
cacagcaacg gcaacaccta cctgcagtgg tacctgcaga agcccggcca gagcccccag 540
ctgctgatct acaaggtgag caaccggttc agcggcgtgc ccgaccggtt cagcggcagc 600
ggcagcggca ccgacttcac cctgaagatc agccgggtgg aggccgagga cgtgggcgtg 660
tactactgca gccagagcat ctacgtgccc tacaccttcg gccagggcac caagctggag 720
atcaaacgtg gaggtggtgg atccgatatc aaactgcagc agtcaggggc tgaactggca 780
agacctgggg cctcagtgaa gatgtcctgc aagacttctg gctacacctt tactaggtac 840
acgatgcact gggtaaaaca gaggcctgga cagggtctgg aatggattgg atacattaat 900
cctagccgtg gttatactaa ttacaatcag aagttcaagg acaaggccac attgactaca 960
gacaaatcct ccagcacagc ctacatgcaa ctgagcagcc tgacatctga ggactctgca 1020
gtctattact gtgcaagata ttatgatgat cattactgcc ttgactactg gggccaaggc 1080
accactctca cagtctcctc agtcgaaggt ggaagtggag gttctggtgg aagtggaggt 1140
tcaggtggag tcgacgacat tcagctgacc cagtctccag caatcatgtc tgcatctcca 1200
ggggagaagg tcaccatgac ctgcagagcc agttcaagtg taagttacat gaactggtac 1260
cagcagaagt caggcacctc ccccaaaaga tggatttatg acacatccaa agtggcttct 1320
ggagtccctt atcgcttcag tggcagtggg tctgggacct catactctct cacaatcagc 1380
agcatggagg ctgaagatgc tgccacttat tactgccaac agtggagtag taacccgctc 1440
acgttcggtg ctgggaccaa gctggagctg aaa 1473
<210> 98
<211> 491
<212> PRT
<213> Artificial sequence
<220>
<221> MISC_FEATURE
<223> amino acid sequence of Y035/CD3 bispecific antibody
<400> 98
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ser Asp Tyr
20 25 30
Glu Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ala Ile His Pro Gly Ser Gly Asp Thr Ala Tyr Asn Gln Arg Phe
50 55 60
Lys Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Phe Tyr Ser Tyr Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr
130 135 140
Pro Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Val
145 150 155 160
His Ser Asn Gly Asn Thr Tyr Leu Gln Trp Tyr Leu Gln Lys Pro Gly
165 170 175
Gln Ser Pro Gln Leu Leu Ile Tyr Lys Val Ser Asn Arg Phe Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu
195 200 205
Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Ser
210 215 220
Gln Ser Ile Tyr Val Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu
225 230 235 240
Ile Lys Arg Gly Gly Gly Gly Ser Asp Ile Lys Leu Gln Gln Ser Gly
245 250 255
Ala Glu Leu Ala Arg Pro Gly Ala Ser Val Lys Met Ser Cys Lys Thr
260 265 270
Ser Gly Tyr Thr Phe Thr Arg Tyr Thr Met His Trp Val Lys Gln Arg
275 280 285
Pro Gly Gln Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Ser Arg Gly
290 295 300
Tyr Thr Asn Tyr Asn Gln Lys Phe Lys Asp Lys Ala Thr Leu Thr Thr
305 310 315 320
Asp Lys Ser Ser Ser Thr Ala Tyr Met Gln Leu Ser Ser Leu Thr Ser
325 330 335
Glu Asp Ser Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Asp Asp His Tyr
340 345 350
Cys Leu Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Val
355 360 365
Glu Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Ser Gly Gly Val
370 375 380
Asp Asp Ile Gln Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro
385 390 395 400
Gly Glu Lys Val Thr Met Thr Cys Arg Ala Ser Ser Ser Val Ser Tyr
405 410 415
Met Asn Trp Tyr Gln Gln Lys Ser Gly Thr Ser Pro Lys Arg Trp Ile
420 425 430
Tyr Asp Thr Ser Lys Val Ala Ser Gly Val Pro Tyr Arg Phe Ser Gly
435 440 445
Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala
450 455 460
Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser Asn Pro Leu
465 470 475 480
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
485 490
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПОЛНОСТЬЮ ЧЕЛОВЕЧЕСКИЕ АНТИТЕЛА К МЕЗОТЕЛИНУ И ИММУННЫЕ ЭФФЕКТОРНЫЕ КЛЕТКИ, НАЦЕЛЕННЫЕ НА МЕЗОТЕЛИН | 2016 |
|
RU2748281C2 |
| ЭЛЕМЕНТЫ, СВЯЗЫВАЮЩИЕСЯ С ИЛ-1 БЕТА | 2013 |
|
RU2711118C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА, СВЯЗЫВАЮЩИЕСЯ С TFR | 2019 |
|
RU2810756C2 |
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2819627C1 |
| ИММУНОЦИТОКИНЫ НА ОСНОВЕ IL-15 И IL-15Rα ДОМЕНА SUSHI | 2012 |
|
RU2763298C2 |
| АНТИТЕЛА ПРОТИВ БЕЛКА-1 ЗАПРОГРАММИРОВАННОЙ КЛЕТОЧНОЙ СМЕРТИ (PD-1) И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2725950C1 |
| АНТИ-HLA-A2 АНТИТЕЛА И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2782276C2 |
| АНТИТЕЛА ПРОТИВ РЕЦЕПТОРА ФОЛИЕВОЙ КИСЛОТЫ 1, ИХ ИММУНОКОНЪЮГАТЫ И ИСПОЛЬЗОВАНИЕ | 2011 |
|
RU2740479C2 |
| ПРИМЕНЕНИЕ АНТИТЕЛА ПРОТИВ О-АЦЕТИЛИРОВАННОГО ГАНГЛИОЗИДА GD2 ДЛЯ УЛУЧШЕНИЯ ТЕРАПЕВТИЧЕСКОГО ПОТЕНЦИАЛА ЛЕКАРСТВЕННЫХ СРЕДСТВ | 2017 |
|
RU2771173C2 |
| Антитела против CXCR2 и их применение | 2019 |
|
RU2807067C2 |


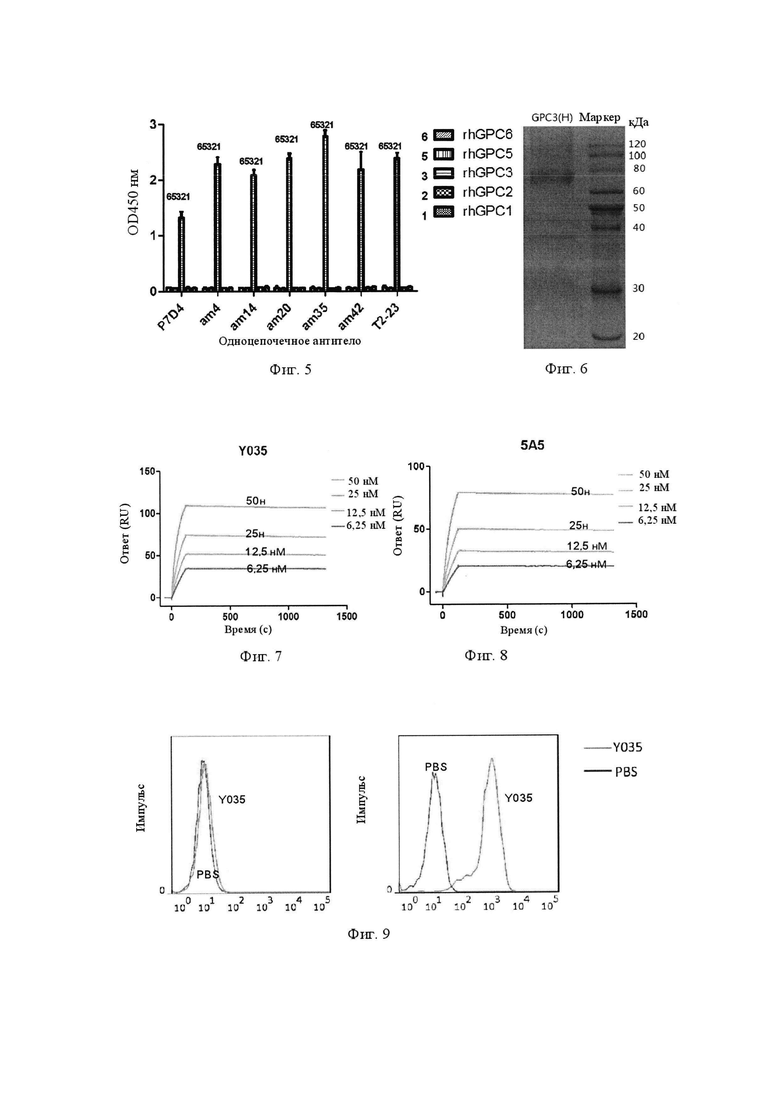
Изобретение относится к области биохимии, в частности к антителу, которое специфически связывается с глипиканом-3. Также раскрыты нуклеиновая кислота, кодирующая указанное антитело; вектор, содержащий указанную нуклеиновую кислоту; клетка-хозяин, содержащая указанный вектор; иммуноконъюгат, содержащий указанное антитело; рецептор, содержащий указанное антитело; композиция, содержащая указанное антитело. Раскрыто применение указанного антитела для получения лекарственного средства направленного действия. Изобретение позволяет эффективно лечить заболевания, ассоциированные с опухолевыми клетками, экспрессирующими глипикан-3. 15 н. и 22 з.п. ф-лы, 9 ил., 6 табл., 10 пр.
1. Антитело, специфично связывающееся с глипиканом-3, где антитело имеет вариабельную область легкой цепи и вариабельную область тяжелой цепи, и
CDR1 вариабельной области легкой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 16, SEQ ID NO: 67, SEQ ID NO: 69;
CDR2 вариабельной области легкой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 18, SEQ ID NO: 68, SEQ ID NO: 70;
CDR3 вариабельной области легкой цепи имеет аминокислотную последовательность SEQ ID NO: 20;
CDR1 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 10, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64;
CDR2 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 12, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65, SEQ ID NO: 66, SEQ ID NO: 71;
CDR3 вариабельной области тяжелой цепи имеет аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 14, SEQ ID NO: 72.
2. Антитело по п. 1, где антитело включает:
антитело (a) (P7D4), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 12 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (б) (am4), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 60, CDR2 с последовательностью SEQ ID NO: 61 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (в) (am14), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 62, CDR2 с последовательностью SEQ ID NO: 63 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (г) (am20), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 18 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 64, CDR2 с последовательностью SEQ ID NO: 65 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (д) (am35), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 67, CDR2 с последовательностью SEQ ID NO: 68 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 66 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (е) (am42), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 69, CDR2 с последовательностью SEQ ID NO: 70 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 66 и CDR3 с последовательностью SEQ ID NO: 14;
антитело (ж) (T2-23), вариабельная область легкой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 16, CDR2 с последовательностью SEQ ID NO: 70 и CDR3 с последовательностью SEQ ID NO: 20 или вариабельная область тяжелой цепи которого имеет CDR1 с последовательностью SEQ ID NO: 10, CDR2 с последовательностью SEQ ID NO: 71 и CDR3 с последовательностью SEQ ID NO: 72.
3. Антитело по п. 2, где аминокислотная последовательность вариабельной области тяжелой цепи антитела (а) (P7D4) представлена в положениях 1-121 SEQ ID NO: 4; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 4;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (б) (am4) представлена в положениях 1-121 SEQ ID NO: 25; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 25;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (в) (am14) представлена в положениях 1-121 SEQ ID NO: 27; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 27;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (г) (am20) представлена в положениях 1-121 SEQ ID NO: 29; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 29;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (д) (am35) представлена в положениях 1-121 SEQ ID NO: 31; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 31;
аминокислотная последовательность вариабельной области тяжелой цепи антитела (е) (am42) представлена в положениях 1-121 SEQ ID NO: 33; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 33; или
аминокислотная последовательность вариабельной области тяжелой цепи антитела (ж) (T2-23) представлена в положениях 1-121 SEQ ID NO: 35; или аминокислотная последовательность вариабельной области легкой цепи антитела представлена в положениях 137-247 SEQ ID NO: 35.
4. Антитело, специфично связывающееся с глипиканом-3, где антитело содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи, где его вариабельная область тяжелой цепи содержит CDR1 с аминокислотной последовательностью SEQ ID NO: 73, CDR2 с аминокислотной последовательностью SEQ ID NO: 74 и CDR3 с аминокислотной последовательностью SEQ ID NO: 75; или его вариабельная область легкой цепи содержит CDR1 с аминокислотной последовательностью SEQ ID NO: 76, CDR2 с аминокислотной последовательностью SEQ ID NO: 77 и CDR3 с аминокислотной последовательностью SEQ ID NO: 78.
5. Антитело по п. 4, где антитело представляет собой гуманизированное моноклональное антитело, содержащее вариабельную область тяжелой цепи и вариабельную область легкой цепи, где его вариабельная область тяжелой цепи содержит CDR1 с аминокислотной последовательностью SEQ ID NO: 73, CDR2 с аминокислотной последовательностью SEQ ID NO: 74 и CDR3 с аминокислотной последовательностью SEQ ID NO: 75; или его вариабельная область легкой цепи содержит CDR1 с аминокислотной последовательностью SEQ ID NO: 76, CDR2 с аминокислотной последовательностью SEQ ID NO: 77 и CDR3 с аминокислотной последовательностью SEQ ID NO: 78.
6. Антитело по п. 4, вариабельная область тяжелой цепи которого имеет аминокислотную последовательность SEQ ID NO: 58 или вариабельная область легкой цепи которого имеет аминокислотную последовательность SEQ ID NO: 59.
7. Антитело по п. 4, вариабельная область тяжелой цепи которого имеет аминокислотную последовательность SEQ ID NO: 81 или вариабельная область легкой цепи которого имеет аминокислотную последовательность SEQ ID NO: 82.
8. Нуклеиновая кислота, кодирующая антитело по любому из пп. 1-7.
9. Экспрессионный вектор, содержащий нуклеиновую кислоту по п. 8.
10. Клетка-хозяин для экспрессии антитела по любому из пп. 1-7, которая содержит экспрессионный вектор по п. 9 или в геном которой интегрирована нуклеиновая кислота по п. 8.
11. Применение антитела по любому из пп. 1-7 для получения лекарственного средства направленного действия, конъюгата антитело-лекарственное средство или полифункционального антитела, которое специфично направлено на опухолевые клетки, экспрессирующие глипикан-3.
12. Применение антитела по любому из пп. 1-7 для получения средства для диагностики опухоли, экспрессирующей глипикан-3.
13. Применение антитела любому из пп. 1-7 для получения иммунных клеток, модифицированных химерным антигенным рецептором.
14. Иммуноконъюгат для лечения опухолей, экспрессирующих глипикан-3, содержащий антитело по любому из пп. 1-7 и связанную с ним функциональную молекулу; при этом указанная функциональная молекула выбрана из группы, состоящей из молекулы, направленной на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, направленной на маркер поверхности иммунной клетки, или детектируемой метки.
15. Иммуноконъюгат для выявления опухолей, экспрессирующих глипикан-3, содержащий антитело по любому из пп. 1-7 и связанную с ним функциональную молекулу; при этом указанная функциональная молекула выбрана из группы, состоящей из молекулы, направленной на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, направленной на маркер поверхности иммунной клетки, или детектируемой метки.
16. Иммуноконъюгат для анализа опухолей, экспрессирующих глипикан-3, содержащий антитело по любому из пп. 1-7 и связанную с ним функциональную молекулу; при этом указанная функциональная молекула выбрана из группы, состоящей из молекулы, направленной на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, направленной на маркер поверхности иммунной клетки, или детектируемой метки.
17. Иммуноконъюгат для диагностики опухолей, экспрессирующих глипикан-3, содержащий антитело по любому из пп. 1-7 и связанную с ним функциональную молекулу; при этом указанная функциональная молекула выбрана из группы, состоящей из молекулы, направленной на маркер поверхности опухоли, молекулы, подавляющей опухоль, молекулы, направленной на маркер поверхности иммунной клетки, или детектируемой метки.
18. Иммуноконъюгат по любому из пп. 14-17, где молекула, направленная на маркер поверхности опухоли, представляет собой антитело или лиганд, который связывается с маркером поверхности опухоли; или молекула, подавляющая опухоль, представляет собой противоопухолевый цитокин или противоопухолевый токсин; и предпочтительно цитокин включает IL-12, IL-15, IFN (интерферон)-бета и TNF (фактор некроза опухоли)-альфа.
19. Иммуноконъюгат по любому из пп. 14-17, где детектируемая метка включает флуоресцентную метку или хромогенную метку.
20. Иммуноконъюгат по п. 18, где антитело, которое связывается с маркером поверхности опухоли, относится к антителу, которое распознает антигены, отличные от глипикана-3, причем указанные отличные антигены включают EGFR, EGFRvIII, мезотелин, HER2, EphA2, Her3, EpCAM, MUCl, MUC16, CEA, клаудин 18.2, фолатный рецептор, клаудин 6, WT1, NY-ESO-1, MAGE 3, ASGPR1 или CDH16.
21. Иммуноконъюгат по любому из пп. 14-17, где молекула, направленная на маркер поверхности иммунной клетки, представляет собой антитело, связывающееся с маркером поверхности T-клетки, которое может образовывать бифункциональное антитело, активирующее T-клетки, с антителом по любому из пп. 1-7.
22. Иммуноконъюгат по п. 21, где антитело, связывающееся с маркером поверхности иммунной клетки, представляет собой антитело против CD3.
23. Иммуноконъюгат по п. 22, где многофункциональный иммуноконъюгат представляет собой слитый пептид и дополнительно содержит линкерный пептид между антителом по любому из пп. 1-7 и связанной с ним функциональной молекулой.
24. Химерный антигенный рецептор, содержащий антитело по любому из пп. 1-7, трансмембранную область и внутриклеточную сигнальную область, которые последовательно связаны.
25. Химерный антигенный рецептор по п. 24, где внутриклеточная сигнальная область выбрана из группы, состоящей из последовательностей внутриклеточной сигнальной области CD3ζ, FcεRIγ, CD27, CD28, CD137, CD134, MyD88, CD40 или их комбинации.
26. Химерный антигенный рецептор по п. 24, где трансмембранная область содержит трансмембранную область CD8 или CD28.
27. Химерный антигенный рецептор по п. 24, где химерный антигенный рецептор содержит следующие последовательно связанные антитело, трансмембранную область и внутриклеточную сигнальную область:
антитело по любому из пп. 1-7, CD8 и CD3ζ;
антитело по любому из пп. 1-7, CD8, CD137 и CD3ζ;
антитело по любому из пп. 1-7, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28 и CD3ζ; или
антитело по любому из пп. 1-7, трансмембранная область молекулы CD28, внутриклеточная сигнальная область молекулы CD28, CD137 и CD3ζ.
28. Химерный антигенный рецептор по п. 24, где антитело представляет собой одноцепочечное антитело или доменное антитело.
29. Химерный антигенный рецептор по п. 24, где химерный антигенный рецептор содержит:
SEQ ID NO: 49 или аминокислотную последовательность, представленную в положениях 22-346 указанной последовательности;
SEQ ID NO: 50 или аминокислотную последовательность, представленную в положениях 22-447 указанной последовательности;
SEQ ID NO: 51 или аминокислотную последовательность, представленную в положениях 22-491 указанной последовательности;
SEQ ID NO: 52 или аминокислотную последовательность, представленную в положениях 22-494 указанной последовательности;
SEQ ID NO: 53 или аминокислотную последовательность, представленную в положениях 22-536 указанной последовательности;
аминокислотную последовательность SEQ ID NO: 86;
аминокислотную последовательность SEQ ID NO: 88; или
аминокислотную последовательность SEQ ID NO: 90.
30. Генетически модифицированная иммунная клетка, направленная на опухоль, экспрессирующую глипикан-3, которая имеет химерный антигенный рецептор по любому из пп. 24-29, экспрессируемый на ее поверхности.
31. Иммунная клетка по п. 30, дополнительно несущая экзогенную кодирующую последовательность для цитокинов; и предпочтительно цитокины включают IL-12, IL-15 или IL-21.
32. Иммунная клетка по п. 30, также экспрессирующая другой химерный антигенный рецептор, который не содержит CD3ζ, но содержит внутриклеточный сигнальный домен CD28, внутриклеточный сигнальный домен CD137 или их комбинацию.
33. Иммунная клетка по п. 30, дополнительно экспрессирующая хемокиновый рецептор; и предпочтительно хемокиновый рецептор включает CCR2.
34. Иммунная клетка по п. 30, дополнительно экспрессирующая миРНК, которая может снижать уровень экспрессии PD-1, или белок, который блокирует PD-L1.
35. Иммунная клетка по п. 30, дополнительно экспрессирующая предохранитель; причем предохранитель предпочтительно включает индуцируемую каспазу-9, усеченный EGFR или RQR8.
36. Иммунная клетка по п. 30, где иммунная клетка включает T-лимфоциты, NK-клетки или NKT-клетки.
37. Фармацевтическая композиция для лечения заболевания, ассоциированного с глипиканом-3, содержащая эффективное количество антитела по любому из пп. 1-7 или нуклеиновой кислоты, кодирующей указанное антитело; или
иммуноконъюгата по любому из пп. 14-23; или
генетически модифицированной иммунной клетки по любому из пп. 30-36.
| ЛЕКАРСТВЕННОЕ СРЕДСТВО ДЛЯ ЛЕЧЕНИЯ РАКА ПЕЧЕНИ | 2009 |
|
RU2523897C2 |
| АНТИТЕЛО ПРОТИВ ГЛИПИКАНА 3 | 2005 |
|
RU2427588C2 |
| CN 101633693 A, 27.01.2010 | |||
| ПНЕВМАТИЧЕСКИЙ ПОДЪЕМНИК | 2012 |
|
RU2497744C1 |
Авторы
Даты
2021-03-04—Публикация
2016-08-02—Подача