Область техники, к которой относится изобретение
Изобретение относится к области исследований в медицине, микробиологии. Данное изобретение может быть применено для выявления генов токсинов для защиты человека или животных, с целью поиска бактерий, входящих в список, определенный санитарными правилами в отношении 3-4 групп патогенности, и выяснения профиля их опасности по содержанию генов токсинов, генов антибиотикорезистентности и генно-инженерных векторов для использования в составе сети Национального интеграционного центра мониторинга биологических угроз.
Уровень техники
Из уровня техники известен способ видовой и штаммовой идентификации бифидобактерий филотипа Bifidobacterium longum, отличающийся тем, что основан на комбинации и полиморфизме генов токсин-антитоксин суперсемейства RelBE и характеризующийся тем, что для идентификации проводят амплификацию с геномной ДНК с использованием набора видо- и штаммоспецифических олигонуклеотидов, ПЦР продукты анализируют в агарозном геле, а размер полученного фрагмента определяют с помощью ДНК-маркера, видовую идентификацию проводят с использованием специфичных олигонуклеотидов, причем исследуемые штаммы относят к виду В. longum в случае наработки целевого фрагмента размером 275 п. н.; штаммовую идентификацию проводят с использованием олигонуклеотидов, исследуемый штамм признают идентичным штамму В. longum subsp.infantis АТСС 15697 в случае наработки фрагментов при использовании олигонуклеотидов ReB1N-ReB1C, ReB2N-ReB2C и ReB3.1N-ReB3.2C; исследуемый штамм признают идентичным штамму В. longum subsp.infantis АТСС 55813 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB9.1N-ReB9.1C; исследуемый штамм признают идентичным штамму В. longum subsp.infantis CCUG 52486 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C, ReB3.4N-ReB3.4C и ReB9.1N-ReB9.2C; исследуемый штамм признают идентичным штамму В. longum DJO10A в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C; исследуемый штамм признают идентичным штамму Bifidobacterium sp.(В. longum) 12_1_47BFAA в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB4N-ReB4C; исследуемый штамм признают идентичным штамму В. longum subsp.longum F8 в случае наработки фрагментов при использовании олигонуклеотидов ReB2N-ReB2C и ReB3.3N-ReBC; исследуемый штамм признают идентичным штамму В. longum NCC2705 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.2C; исследуемый штамм признают идентичным штамму В. longum subsp.longum JDM301 в случае наработки фрагментов при использовании олигонуклеотидов ReB8N-ReB8C, ReB2N-ReB2C и ReB3.1N-ReB3.1C; исследуемый штамм признают идентичным штамму В. longum subsp.longum BBMN68 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB3.4N-ReB3.4C; исследуемый штамм признают идентичным штамму В. longum subsp.infantis 157F в случае наработки фрагментов только при использовании олигонуклеотидов ReB3.1N-ReB3.1C, ReB4N-ReB4C и ReB5N-ReB5C; исследуемый штамм признают идентичным штамму В. longum subsp.longum JCM 1217 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB3.5N-ReB3.4C; исследуемый штамм признают идентичным штамму В. longum subsp.longum КАСС 91563 в случае наработки фрагментов при использовании олигонуклеотидов ReB10N-ReB10C и ReB3.1N-ReB3.4C (RU 2527069 С2, 27.08.2014).
Из уровня техники известно создание и скрининг библиотек генов http://www.bio.bsu.by/molbiol/files/lections/Razdel4.pdf. Указанный источник информации раскрывает получение библиотек ДНК: геномная библиотека - это совокупность всех нуклеотидных последовательностей геномной ДНК организма, клонированная в виде фрагментов в векторах. С последующим скринингом с помощью метода гибридизации колоний, иммуннодетекции продуктов генов. Кроме того раскрыто получение связанных библиотек - метод "прогулки по хромосоме". А также раскрыто и секвенирование - определение первичной структуры (последовательности нуклеотидов) молекулы ДНК. Данный источник информации выбран в качестве прототипа.
Недостатками ранее известных способов является их низкая производительность, сложность проведения анализа и избирательность. Например, при использовании указанных олигонуклеотидов для выявления одного штамма Bifidobacterium longum может потребоваться проведение более 10 специфических амплификаций.
Эра открытия новых патогенных микроорганизмов, не только не закончилась, а напротив, благодаря новым возможностям приобрела возможность изучать эволюционные и эпидемически значимые механизмы изменчивости, позволяющие патогенам более изощренно и с большей динамикой угрожать человечеству биологической катастрофой. Каждое новое инфекционное заболевание в 21 веке как никогда раньше может быть серьезным вызовом для современной медицины и ставит под угрозу безопасность населения отдельных территорий и суверенных государств. Изменение окружающей среды, вырубка тропических лесов, таяние льдов в Антарктике, увеличение плотности населения и повсеместное использование антибиотиков - все это не полный перечень актуальных факторов, провоцирующих появление новых патогенов и придание патогенам новых, эпидемически значимых свойств. Эпидемии гриппа, вызываемые новыми штаммами вируса; респираторные синдромы, вызываемые новыми короновирусами; вспышки инфекций, причиной которых является гемолитическая кишечная палочка; возникновение резистентных к антибиотикам супербактерий - примеры того, какие опасности могут поджидать человечество. В число первоочередных мер, которые необходимо предпринять для противодействия новым патогенам, входит разработка способов их быстрой и точной идентификации. Классические методы, широко используемые для типирования микроорганизмов в лабораториях, такие как ПЦР, ИФА и МАПДИ имеют ограничения по объему и качеству данных, которые можно получить о свойствах инфекционных агентов, содержащихся в интересуемом образце. Таким образом, остро стоит необходимость разработки методов, позволяющих осуществлять выявление и идентификацию новых патогенов методами секвенирования следующего поколения (next generation sequencing или NGS).
Осуществление изобретения
Заявленный способ включает следующие приемы:
1) на первом этапе выполняют создание ДНК-библиотек их выделенной геномной ДНК бактерий для последующего секвенирования на платформах высокопроизводительного секвенирования. Данный этап включает следующие стадии:
• ферментативной фрагментации ДНК;
• восстановления концов с целью получения двухцепочечной 5'-фосфорилированной ДНК с выступающим 3'-аденозином;
• лигирования специфических адаптеров по технологии быстрого ТА-лигирования;
• амплификации полученных ДНК-библиотек с внешних универсальных адаптерных последовательностей;
• отбор ДНК-библиотек целевой длины с применением силикатных парамагнитных частиц.
Применение различных специфических адаптеров для лигирования позволяет мультиплексировать образы для совместного секвенирования в рамках одного запуска платформы. Вариации методики отбора с использованием магнитных частиц позволяет варьировать длину получаемых ДНК-библиотек для анализа с помощью высокопроизводительного секвенирования на различных NGS и TGS платформах (в том числе Illumina, Ion Torrent, BGI, Oxford Nanopore и др.).
2) второй этап включает целевое обогащение (отбора) молекул ДНК-библиотек, несущих участки анализируемых генов токсинов с использованием метода жидкостной гибридизации ДНК-зонд. В качестве зондов используются оптимизированные по структуре синтетические одноцепочечные молекулы ДНК (олигонуклеотиды), несущие на 5' или 3' конце модификацию, позволяющую им обратимо связываться со специализированными парамагнитными силикатными частицами (например, в виде модификации биотина, алкиновой группы, азидной группы и аналогичных групп клик-химии). Олигонуклеотидные зонды в соответствующих буферных и температурных условиях образуют совместно с целевыми молекулами ДНК устойчивые комплементарные пары (дуплексы). Данные целевые дуплексы впоследствии связываются с суперпарамагнитными частицами, содержащие группы, реакционно активные относительно модификаций синтетических зондов. Например, молекулы биотина зондов образует ковалентную связь со стрептавидином на поверхности магнитных частиц, в результате чего молекулы ДНК, образовавшие дуплексы ДНК-зонд, оказываются связанными с магнитными частицами. Далее производится отмывка несвязавшихся нецелевых молекул ДНК. Целые молекулы ДНК впоследствии освобождаются с поверхности магнитных частиц и могут быть проанализированы методами NGS секвенирования.
Указанный этап включает следующие стадии:
• жидкостная гибридизация синтезированных ДНК-библиотек с зондами;
• захват гибридных дуплексов с помощью суперпарамагнитных частиц;
• отмывка несвязавшихся нецелевых молекул ДНК;
• освобождение связавшихся ДНК-библиотек с поверхности магнитных частиц;
• ПЦР амлификация обогащенных ДНК-библиотек;
• квантификация амплифицированных ДНК-библиотек для последующего NGS секвенования.
Задачей настоящего изобретения является высокоэффективное выделение целевых групп генов токсинов из сложных смесей разнородной входящей ДНК (смеси геномной ДНК различного состава и видового происхождения) с последующим целевым высокопроизводительным секвенированием на различных NGS и TGS платформах (в том числе Illumina, Ion Torrent, BGI, Oxford Nanopore и др.), для выявления фрагментов генов токсинов, присутствующих в исходной смеси.
Технический результат заключается в существенном увеличении относительной представленности целевых фрагментов генов токсинов в итоговой смеси ДНК, анализируемой методами высокопроизводительного секвенирования, по сравнению с входящей ДНК (т.н. обогащение целевыми фрагментами). Например, в результате применения метода на смеси геномной ДНК токсигенных штаммов бактерий происходит увеличение представленности целевых фрагментов с 0,001% в исходной смеси, до 50% и более в итоговой анализируемой пробе.
Раскрытие изобретения
Указанный технический результат реализуется за счет следующих приемов на примере пробоподготовки для платформы Illumina.
Способ мультиплексного выявления генов токсинов, реализуется следующим образом: на первом этапе выполняют подготовку ДНК-библиотек, при этом данный этап включает следующие приемы:
• выполняют ферментативную фрагментацию ДНК с применением неспецифических экзонуклеаз;
• выполняют восстановление концов с целью получения двухцепочечной 5'-фосфорилированной ДНК с выступающим 3'-аденозином;
• осуществляют лигирование специфических Y-образных адаптеров по технологии быстрого ТА-лигирования с применением Т4-ДНК л и газы, или аналогичных;
• осуществляют амплификации полученных ДНК-библиотек с применением праймеров, комплементарных внешним последовательностям специфических Y-образных адаптеров;
• выполняют отбор ДНК-библиотек целевой длины с применением силикатных парамагнитных частиц;
на втором этапе выполняют целевое обогащение молекул ДНК-библиотек, несущих участки анализируемых генов токсинов с использованием метода жидкостной гибридизации ДНК-зонд, при этом указанный второй этап содержит следующие приемы:
• осуществляют жидкостную гибридизацию синтезированных ДНК-библиотек с зондами, при этом смешивают ДНК-библиотеки со специфическими зондами для обогащения фрагментами целевых генов токсинов с применением коммерчески доступных буферов для гибридизации (например, HybBuf, Roche). Смесь для гибридизации инкубируют в твердотельном термостате, или амплификаторе при температуре от 35°С до 60°С в течение 10-50 часов;
• осуществляют захват гибридных дуплексов с помощью суперпарамагнитных частиц, связывающих дуплексы ДНК-зонд, при этом происходит смешивание раствора после гибридизации с суперпарамагнитными частицами;
• выполняют отмывку несвязавшихся нецелевых молекул ДНК, при этом выполняют серию промывок суперпарамагнитных частиц, несущих на себе связавшиеся дуплексы ДНК-зонд, с применением растворов для удаления несвязавшихся нецелевых молекул ДНК;
• выполняют освобождение связавшихся ДНК-библиотек с поверхности магнитных частиц, для чего отделяют обогащенные целевые фракции ДНК с суперпарамагнитных частиц, путем прогревания смеси при 95°С в течение 10-15 минут;
• затем осуществляю ПЦР амлификацию обогащенных ДНК-библиотек с внешних универсальных праймеров, комплементарных внешним последовательностям специфических Y-образных адаптеров;
• после чего выполняют квантификацию амплифицированных ДНК-библиотек для последующего NGS секвенирования флуориметрическим и/или спектрофотометрическим методами.
Подробное раскрытие осуществления изобретения
На первом этапе выполняют создание ДНК-библиотек. Для этого выделенная из исследуемого образца ДНК фрагментируется, проводится очистка фрагментированной ДНК, восстановление концов и лигирование адаптеров, амплификация с фланкирующих праймеров с последующей очисткой на магнитных частицах. В дальнейшем приготовленные библиотеки нормализуются и объединяются для последующего обогащения.
При этом данный этап включает следующие стадии:
• ферментативной фрагментации ДНК с применением неспецифических экзонуклеаз;
• восстановления концов с целью получения двухцепочечной 5'-фосфорилированной ДНК с выступающим 3'-аденозином;
• лигирования специфических адаптеров (для выбранной платформы секвенирования) по технологии быстрого ТА-лигирования с применением Т4-ДНК лигазы, или аналогичных;
• амплификации полученных ДНК-библиотек с внешних универсальных адаптерных последовательностей;
• отбор ДНК-библиотек целевой длины с применением силикатных парамагнитных частиц.
Применение различных адаптеров для лигирования позволяет мультиплексировать образы для совместного обогащения и секвенирования в рамках одного запуска платформы. На этапе после лигирования адаптеров (до ПЦР) может проводиться процедура отбора ДНК-библиотек по длине (двухсторонний size-select) с применением магнитных частиц (AMPure ХР, Beckman Coulter, или аналогичных) с получением библиотек со вставкой необходимой длины. Например, возможен отбор фракции библиотек со средней длиной вставки 450-500 п. о., что соответствует суммарной длине библиотеки 570-620 п.о. с учетом длины адаптерных последовательностей. Такая длина ДНК-библиотек оптимальна для последующего секвенирования на платформе Illumina MiSeq или HiSeq с длиной прочтения 2×250 п.о. Вариации методики отбора с использованием магнитных частиц позволяет варьировать длину получаемых ДНК-библиотек для анализа с помощью высокопроизводительного секвенирования на различных NGS и TGS платформах (в том числе Illumina, Ion Torrent, BGI, Oxford Nanopore и др.).
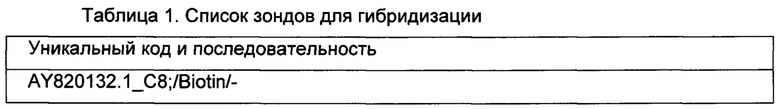
На втором этапе выполняют целевое обогащения (отбора) молекул ДНК-библиотек, несущих участки анализируемых генов токсинов с использованием метода жидкостной гибридизации «ДНК-зонд». В качестве зондов используются оптимизированные по структуре синтетические одноцепочечные молекулы ДНК (праймеры) несущие на 5' или 3' конце модификацию в виде молекулы биотина. ДНК зонды в соответствующих буферных и температурных условиях образуют совместно с целевыми молекулами ДНК устойчивые комплементарные пары (дуплексы). Данные целевые дуплексы впоследствии связываются с покрытыми стрептавидином суперпарамагнитными частицами. Молекулы биотина зондов образует ковалентную связь со стрептавидином на поверхности магнитных частиц, в результате чего молекулы ДНК, образовавшие дуплексы ДНК-зонд, оказываются связанными с магнитными частицами. Далее производится отмывка несвязавшихся нецелевых молекул ДНК. Целые молекулы ДНК впоследствии освобождаются с поверхности магнитных частиц и могут быть проанализированы методами NGS секвенирования. Панель зондов синтезирована в виде 5'-биотинилированных олигонуклеотидов (см. таблицу 1) методом твердофазного синтеза на автоматическом синтезаторе ABI 3900. Данная панель использована для отработки методики обогащения.





Указанный второй этап включает следующие стадии:
• жидкостная гибридизация синтезированных ДНК-библиотек с зондами. В рамках стадии происходит смешивание ДНК-библиотек со специфическими зондами для гибридизации в присутствии необходимых буферных условий. Смесь для гибридизации инкубируется в твердотельном термостате, или амплификаторе при выбранной температуре в течение 10-50 часов.
• захват гибридных дуплексов с помощью специфических суперпарамагнитных частиц, связывающих дуплексы ДНК-зонд. В рамках стадии происходит смешивание раствора после гибридизации со специфическими суперпарамагнитными частицами
• отмывка несвязавшихся нецелевых молекул ДНК. В рамках стадии происходит серия промывок суперпарамагнитных частиц, несущих на себе связавшиеся дуплексы ДНК-зонд, с применением растворов для удаления несвязавшихся нецелевых молекул ДНК.
• освобождение связавшихся ДНК-библиотек с поверхности магнитных частиц. В рамках стадии происходит отделение обогащенной целевой фракции ДНК с суперпарамагнитных частиц. Например, путем прогревания смеси при 95°С в течение 10-15 минут.
• ПЦР амлификация обогащенных ДНК-библиотек с внешних универсальных адаптерных последовательностей;
• квантификация амплифицированных ДНК-библиотек для последующего NGS секвенирования флуориметрическим и/или спектрофотометрическим методами.
Готовые ДНК-библиотеки подвергались процедуре гибридизации с применением разработанной панели зондов и набора для гибридизации SeqCap EZ Hybridization and Wash Kit (Roche). Образцы подвергались следующей процедуре: гибридизация ДНК-библиотек с зондами при 40°С в течение 24-48 часов, отмывку несвязавшейся ДНК, пост-гибридизацонную амплификацию с внешних фланкирующих праймеров. Набор реактивов SeqCap EZ Hybridization and Wash Kit включает буферы, блокирующие олигонуклеотиды, праймеры и полимеразу для проведения всех этапов гибридизации и ПЦР.
Промышленная применимость
Заявленный способ может быть осуществлен специалистом на практике и при осуществлении обеспечивает реализацию заявленного назначения. Возможность осуществления на практике следует из того, что для каждого признака, включенного в формулу изобретения на основании описания, известен материальный эквивалент, что позволяет сделать вывод о соответствии критерию «промышленная применимость» для изобретения и критерию «полнота раскрытия» для изобретения.
СПОСОБ МУЛЬТИПЛЕКСНОГО ВЫЯВЛЕНИЯ ГЕНОВ ТОКСИНОВ
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ





| название | год | авторы | номер документа |
|---|---|---|---|
| Технология полноэкзомного обогащения на основе различия в кинетике реассоциации фрагментов ДНК | 2023 |
|
RU2839144C1 |
| ПРЯМОЙ ЗАХВАТ, АМПЛИФИКАЦИЯ И СЕКВЕНИРОВАНИЕ ДНК-МИШЕНИ С ИСПОЛЬЗОВАНИЕМ ИММОБИЛИЗИРОВАННЫХ ПРАЙМЕРОВ | 2011 |
|
RU2565550C2 |
| Технология полноэкзомного обогащения на основе гибридизации с нормализованным транскриптомом | 2023 |
|
RU2838569C1 |
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |
| РЕАГЕНТНО-ПРОГРАММНЫЙ КОМПЛЕКС ДЛЯ ПРОВЕДЕНИЯ ТАРГЕТНОГО АНАЛИЗА | 2019 |
|
RU2744443C1 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ УЛУЧШЕНИЯ ОБОГАЩЕНИЯ БИБЛИОТЕК | 2019 |
|
RU2809771C2 |
| СПОСОБЫ И СРЕДСТВА ПОЛУЧЕНИЯ БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ | 2019 |
|
RU2815513C2 |
| Технология полноэкзомного обогащения на основе гибридизации с полноэкзомной РНК-библиотекой | 2023 |
|
RU2830886C1 |
| Способ секвенирования экзонов гена HLA-DQA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2833819C1 |
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |
Изобретение относится к области медицины и микробиологии. Предложен способ мультиплексного выявления генов токсинов. На первом этапе осуществляют синтез ДНК-библиотек. На втором этапе выполняют целевое обогащение молекул ДНК-библиотек, несущих участки анализируемых генов токсинов с использованием метода жидкостной гибридизации ДНК-зонд. Выполняют квантификацию амплифицированных ДНК-библиотек для последующего NGS секвенирования флуориметрическим методом. Изобретение обеспечивает увеличение относительной представленности целевых фрагментов генов токсинов в итоговой смеси ДНК, анализируемой методами высокопроизводительного секвенирования по сравнению с входящей ДНК. 1 табл.
Способ мультиплексного выявления генов токсинов реализуется следующим образом: на первом этапе синтез ДНК-библиотек, при этом данный этап включает следующие приемы:
- выполняют ферментативную фрагментацию ДНК с применением экзонуклеаз;
- выполняют восстановление концов с целью получения двухцепочечной 5'-фосфорилированной ДНК с выступающим 3'-аденозином;
- осуществляют лигирование Y-образных адаптеров по технологии быстрого ТА-лигирования с применением Т4-ДНК лигазы;
- осуществляют амплификацию полученных ДНК-библиотек с применением праймеров, комплементарных внешним последовательностям Y-образных адаптеров;
- выполняют отбор ДНК-библиотек целевой длины с применением силикатных парамагнитных частиц;
на втором этапе выполняют целевое обогащение молекул ДНК-библиотек, несущих участки анализируемых генов токсинов с использованием метода жидкостной гибридизации ДНК-зонд, при этом указанный второй этап включает следующие приемы:
- осуществляют жидкостную гибридизацию синтезированных ДНК-библиотек с зондами, при этом смешивают ДНК-библиотеки с зондами для гибридизации, при этом смесь для гибридизации инкубируют в амплификаторе при температуре от 35°С до 60°С в течение 10-50 часов;
- осуществляют захват гибридных дуплексов с помощью суперпарамагнитных частиц, связывающих дуплексы ДНК-зонд, при этом смешивают раствор после гибридизации с суперпарамагнитными частицами;
- выполняют отмывку несвязавшихся нецелевых молекул ДНК, при этом выполняют серию промывок суперпарамагнитных частиц, несущих на себе связавшиеся дуплексы ДНК-зонд, с применением растворов для удаления несвязавшихся нецелевых молекул ДНК;
- выполняют освобождение связавшихся ДНК-библиотек с поверхности магнитных частиц, для чего отделяют обогащенные целевые фракции ДНК с суперпарамагнитных частиц путем прогревания смеси при 95°С в течение 10-15 минут;
- затем осуществляют ПЦР амлификацию обогащенных ДНК-библиотек с применением праймеров, комплементарных внешним последовательностям Y-образных адаптеров;
- выполняют квантификацию амплифицированных ДНК-библиотек для последующего NGS секвенирования флуориметрическим методом.
| EP 3130673 A1, 15.02.2017 | |||
| US 20180030435 A1, 01.02.2018 | |||
| ОГУРЦОВ А.Н | |||
| Молекулярная биотехнология микробиологических систем: учеб | |||
| пособие | |||
| - Харьков: НТУ "ХПИ", 2012 | |||
| Приспособление в пере для письма с целью увеличения на нем запаса чернил и уменьшения скорости их высыхания | 1917 |
|
SU96A1 |
| CHARBONNEAU A.R.L | |||
| et al | |||
| Defining the ABC of gene essentiality in streptococci | |||
| BMC Genomics | |||
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| CHANDRA P.K | |||
| et al | |||
| Analyzing ligation mixtures using a | |||
Авторы
Даты
2021-04-07—Публикация
2019-12-17—Подача