Настоящее изобретение относится к области медицины, биологии, биотехнологии, клинико-лабораторной диагностики, молекулярной биологии, геномики и генетики, в частности, к способам выполнения ферментативной реакции с использованием молекулярно-генетических технологий, способам получения библиотек фрагментов нуклеиновых кислот из двуцепочечной нуклеиновой кислоты для последующего определения последовательности фрагментов ДНК методом высокопроизводительного секвенирования второго поколения, к способам процессинга молекул нуклеиновых кислот в библиотеки нуклеиновых кислот с использованием фрагментирования, гибридизации и амплификации и может быть использовано для получения библиотеки фрагментов геномной ДНК, обогащенной белок-кодирующими регионами (экзомом).
Существует множество способов и сфер применения, для которых желательно создать библиотеку молекул фрагментированной нуклеиновой кислоты (например, ДНК) из целевых молекул двуцепочечной нуклеиновой кислоты (например, двуцепочечной геномной ДНК). Часто целью является отбор (экстракция) определенных участков нуклеиновой кислоты (например - белок-кодирующих регионов генома, CoDing Sequence (CDS), Open Reading Frame (ORF)) для снижения расходов на секвенирование неинформативных для исследователя или врача участков молекулы. Так, экзом человека составляет около 1% генома, но содержит более 85% известных вариантов последовательности ДНК, связанных с фенотипом (в частности - с заболеваниями). При этом в каждой отдельной ткани человека работает 30-50% от всех генов (считывается в мРНК).
Секвенирование всего экзома (whole exome sequencing, WES) - это стратегия секвенирования ДНК, обеспечивающая избирательное определение кодирующих участков генома. Поскольку кодирующая часть генома охватывает лишь около 1% генома, этот подход представляет собой более экономически эффективную стратегию обнаружения изменений ДНК по сравнению с секвенированием всего генома.
Известно два основных способа отбора белок-кодирующих регионов геномной ДНК (обогащения экзома): амплификационный и гибридизационный [1]. Первый способ предполагает наработку белок-кодирующих регионов методом полимеразной цепной реакции (ПЦР) с праймеров, специфичных белок-кодирующим регионам. Второй способ предполагает гибридизацию фрагментов генома с заранее синтезированными ДНК или РНК-зондами, комплементарными белок-кодирующим регионам генома.
Из уровня техники известны следующие аналоги предлагаемого изобретения:
1) В научной статье Liang с соавт. (2018) Whole Exome Library Construction for Next Generation Sequencing (Создание полноэкзомной библиотеки для высокопроизводительного секвенирования) предложен протокол подготовки библиотеки полноэкзомной библиотеки (WES) с использованием набора КАРА Hyper Prep Kit с зондами Agilent Sure Select Human All Exon V5+UTR, демонстрирующий высокую эффективность преобразования ДНК в библиотеку для секвенирования на платформе Illumina HiSeq [2].
2) Патент № US 10894979 B2 Construction of next generation sequencing (NGS) libraries using competitive strand displacement (2019) «Создание библиотек для высокопроизводительного секвенирования (NGS) с использованием конкурентного смещения цепи» относится к адаптерам для секвенирования и способам создания библиотек для высокопроизводительного секвенирования ДНК (NGS) второго поколения для полногеномного секвенирования, целевого обогащения, скрининговых анализов на основе секвенирования, метагеномики или любого другого применения, требующего подготовки библиотек для NGS. Раскрытые способы состоят из двухэтапного процесса лигирования, при котором первый адаптер секвенирования лигируют с концами репарированных фрагментов ДНК посредством лигирования тупых концов, а второй адаптер секвенирования затем лигируют с первым продуктом лигирования посредством шплинтового лигирования концов [3].
Также, известно множество способов получения нормализованных (выровненных) по представленности различных транскриптов библиотек кДНК [4-6]. Как правило, данный подход используют для приготовления библиотек кДНК перед высокопроизводительным секвенированием второго поколения (NGS), либо для клонирования.
Важно отметить, что несмотря на разный состав работающих генов в каждой отдельной ткани, использование смеси мРНК из разных тканей позволяет повысить разнообразие транскриптов в библиотеке кДНК до 80-90% от всех генов человека.
Ближайшим аналогом предлагаемого изобретения является:
3) Способ экстракции примерно 50 миллионов пар оснований экзонных областей генома человека путем использования захватывающих РНК-зондов длиной около 120 оснований для захвата известных белок-кодирующих последовательностей ДНК (CDS). Полученную таким образом библиотеку фрагментов можно использовать для целевого высокопроизводительного секвенирования второго поколения [7, 8].
Настоящее изобретение относится к области медицины, биологии, биотехнологии, клинико-лабораторной диагностики, молекулярной биологии, геномики и генетики, в частности, к способам выполнения ферментативной реакции с использованием молекулярно-генетических технологий, способам получения библиотек фрагментов нуклеиновых кислот из двуцепочечной нуклеиновой кислоты для последующего определения последовательности фрагментов ДНК методом высокопроизводительного секвенирования второго поколения, к способам процессинга молекул нуклеиновых кислот в библиотеки нуклеиновых кислот с использованием фрагментирования, гибридизации и амплификации и может быть использовано для получения библиотеки фрагментов геномной ДНК, обогащенной белок-кодирующими регионами (экзомом).
Предложен способ обогащения библиотеки фрагментов генома белок-кодирующими последовательностями (экзомом) для последующего секвенирования методом высокопроизводительного секвенирования второго поколения (NGS), отличающийся тем, что для отбора белок-кодирующих регионов используют нормализованный транскриптом в форме биотинилированной библиотеки кДНК.
Предлагаемую генетическую технологию можно применять для различных видов эукариотических организмов, включая лабораторных и сельскохозяйственных животных, а также человека.
Суть изобретения.
Настоящая заявка относится к способам процессинга молекул нуклеиновых кислот в библиотеки нуклеиновых кислот с использованием фрагментирования геномной ДНК, гибридизации с библиотекой биотинилированной нормализованной кДНК и ПЦР-амплификации.
Представленный в настоящем документе способ можно применять для получения обогащенной белок-кодирующими регионами библиотеки фрагментов ДНК из целевой ДНК, представляющей собой двуцепочечную геномную ДНК из любого источника, с целью дальнейшего полноэкзомного анализа методами высокопроизводительного секвенирования второго поколения.
В соответствии с приведенным в настоящем документе описанием в одном варианте осуществления способ выполнения ферментативной реакции с использованием полноэкзомного обогащения включает:
(a) получение биотинилированной нормализованной по представленности транскриптов библиотеки кДНК (из разных тканей) по любой стандартной методике, например из [4];
(b) гибридизацию (денатурацию-реассоциацию) биотинилированной нормализованной библиотеки кДНК с фрагментированной целевой геномной ДНК, содержащей по концам адаптерные последовательности для NGS-секвенирования;
(c) отбор на колонках со стрептавидином ДНК/кДНК-гетеродуплексов;
(d) ПЦР-амплификацию обогащенной таким образом белок-кодирующими регионами библиотеки с концевых NGS-адаптеров.
Осуществление изобретения.
(а) Получение биотинилированной нормализованной по представленности транскриптов библиотеки кДНК (из разных тканей):
1. Тотальную РНК очищают любым стандартным способом из 5-7 тканей разных органов исследуемого организма, в частности для человека можно использовать следующие ткани: плацента, мозжечок, кора мозга, кровь, поперечно-полосатая мускулатура. Готовят смесь РНК в равных долях.
2. Из смеси РНК готовят библиотеку кДНК любым стандартным способом, например при помощи SMART® cDNA Library Construction Kit (Takara Bio USA).
3. Для нормализации амплифицированной кДНК, очищенную кДНК (600-1200 нг) помещают в гибридизационный буфер (50 мМ Hepes, рН 7.5, 0,4 М NaCl) в конечном объеме реакционной смеси 16 мкл.
4. Гибридизационную смесь разделяют на четыре части (S1, S1/2, S1/4 и K), в каждую пробирку добавляют каплю минерального масла, чтобы избежать испарения реакционной смеси, и после центрифугирования в течение 2 мин на максимальной скорости настольной центрифуге инкубируют реакционные смеси при 98°С в течение 2 мин в амплификаторе.
5. Понижают температуру до 68°С и продолжают инкубацию при этой температуре 5-6 ч для гибридизации денатурированной кДНК. Незадолго до окончания процедуры гибридизации готовят разведения Par_DSN до конечных концентраций 1 ед. Куница/мкл, 5 ед. Куница /мкл и 0,25 ед. Куница /мкл в 50 мМ Трис-HCl, рН 8,0.
6. По окончании гибридизации в реакционные смеси, содержащие реассоциированную кДНК, добавляют 5 мкл предварительно нагретый DSN буфер. Растворы быстро перемешивают, возвращают реакционные смеси в амплификатор и продолжают инкубацию при 68°С в течение 10 мин.
7. Добавляют в реакционные смеси по 1 мкл раствора Par_DSN: в образец SI 1 ед. Куница/мкл, в образец S1/2 0,5 ед. Куница/мкл, в образец S1/4 0,25 ед. Куница/мкл. В образец К добавляют 50 мМ Трис-HCl, рН 8,0.
8. Немедленно после добавления нуклеазы возвращают реакционные смеси в нагретый амплификатор и продолжают инкубацию при 68°С в течение 25 мин.
9. По окончании инкубации ингибируют нуклеазную активность Par_DSN добавлением в реакционные смеси 10 мкл 5 мМ ЭДТА. Содержимое пробирок перемешивают и продолжают инкубацию при 68°С в течение 5 мин.
10. Переносят пробирки с реакционными смесями на лед, добавляют в каждую реакционную смесь 20 мкл воды и используют 1 мкл аликвоты для амплификации нормализованной кДНК.
11. Амплификацию нормализованной кДНК осуществляют в объеме реакционной смеси 50 мкл с праймера, соответствующего адаптеру. Реакционные смеси подвергали предварительной денатурации при 95°С в течение 1 мин; затем проводят ПЦР в следующем режиме: 95°С - 15 с, 66°С - 20 с, 72°С - 3 мин. При этом для получения биотинидированного ПЦР-продукта в смесь добавляют γ-[N-(biotin-6-amino-hexanoyl)]-5-aminoallyl-dTTP.
12. Эффективность нормализации и накопление продуктов ПЦР оценивают с помощью электрофореза в агарозном геле с окрашиванием бромистым этидием.
(b) Гибридизация биотинилированной нормализованной библиотеки кДНК с фрагментированной целевой геномной ДНК, содержащей по концам адаптерные последовательности для NGS-секвенирования.
Фрагментация целевой ДНК на Covaris
1. На старте взять 500 нг геномной ДНК (очищенной по любой подходящей под стартовый биоматериал методике), довести объем до 130 мкл mQ/ultrapure water.
2. Поставить пробирки с ДНК в штатив прибора Covaris, опустить в воду.
3. На приборе запускать программу под названием «frag 300bp».
Отбор фрагментов нужной длины (с использованием Agencourt AMPure ХР)
1. Добавить к 130 μL реакции 78 μL бидов (0,6х от объема реакции), перемешать пипетированием;
2. Инкубировать при комнатной температуре 5 минут;
3. Сбить капли мини-центрифугой, перенести пробирки на магнитный штатив. После того как смесь станет прозрачной (на это требуется до 5 минут), нужно осторожно перенести супернатант в новые 1,5 мл пробирки, не задевая биды. Пробирки с бидами выкинуть
4. Добавить к 208 μL реакции 26 μL бидов (0,125х от объема реакции), перемешать пипетированием;
5. Инкубировать при комнатной температуре 5 минут;
6. Сбить капли мини-центрифугой, перенести пробирки на магнитный штатив;
7. Добавить 200 μL свежеприготовленного 80% EtOH, проворачивать пробирку в штативе на 180° таким образом, чтобы биды пробежали через раствор спирта несколько раз, собрать все биды на одной стенке, осторожно удалить EtOH, не задевая бидов;
8. Повторить шаг 7 один раз, чтобы в итоге получилось 2 отмывки;
9. Максимально отобрать микропипеткой 2-20 остатки раствора спирта;
10. Добавить 42 μL mQ, ресуспендировать биды на вортексе, осадить их пульс-спином, инкубировать RT 5 мин, после поместить на магнитный штатив, инкубировать RT 5 мин. Перенести супернатант в новую пробирку. В супернатанте очищенная ДНК.
Репарация концов: полировка липких концов, кинирование, А-теилинг (используются реагенты из набора FS DNA Library Prep Set User Manual)
1. Смешать следующие компоненты в стерильной PCR-пробирке:
- ERAT Enzyme mix (во время использования держать на льду) 2,9 μL
- ERAT buffer 7,1 μL
- фрагментированная ДНК 40 μL
Всего: 50 μL
2. Перемешивать пипетированием, затем сбить капли с помощью мини-центрифуги (вортекса);
3. Поместить в термоцикл ер с нагреваемой крышкой и запустить программу:
30 мин. при 37°С
15 мин. при 65°С
Хранение на 4°С
Лигирование адаптеров
(используются реагенты из набора FS DNA Library Prep Set User Manual)
1. Добавить следующие компоненты прямо к смеси после ERAT (50 μЕ):
- DNA Ligase (во время использования держать на льду) 1,6 μL
- Ligation buffer 23,4 μL
- Adaptor for MGI 5 μL
Всего: 80 μL
2. Смешать пипетированием, после сбить капли мини-центрифугой;
3. Инкубировать при 23°С 1 час в амплификаторе.
Очистка фрагментов ДНК (с использованием Agencourt AMPure ХР)
1. Добавить к 80 μL реакции 20 μL mQ до объема 100 μL.
2. Добавить к 100 μL реакции 50 μL бидов (0,5х от объема реакции), перемешать пипетированием;
3. Инкубировать при комнатной температуре 5 минут;
4. Сбить капли мини-центрифугой, перенести пробирки на магнитный штатив. После того как смесь станет прозрачной (на это требуется до 5 минут), нужно осторожно удалить супернатант, не задевая биды, которые содержат фрагменты ДНК;
5. Добавить 200 μL 80% EtOH, проворачивать пробирку в штативе на 180° таким образом, чтобы биды пробежали через раствор спирта несколько раз, собрать все биды на одной стенке, осторожно удалить EtOH, не задевая бидов;
6. Повторить шаг 4 один раз, чтобы в итоге получилось 2 отмывки;
7. Максимально отобрать микропипеткой 2-20 остатки раствора спирта. Оставить сушиться пробирки с открытой крышкой прямо на магнитном штативе на 2-5 минут, следя за тем, чтобы биды не пересушились (при пересушке они светлеют), но при этом не осталось спирта;
8. Добавить 22 μL mQ, ресуспендировать биды на вортексе, осадить их пульс-спином, инкубировать 5 мин, после поместить на магнитный штатив, инкубировать 5 мин. Перенести супернатант в новую пробирку. В супернатанте очищенная ДНК. Наработка (амплификация) библиотеки методом ПЦР
Реагенты из набора FS DNA Library Prep Set User Manual
1. Смешать следующие компоненты в стерильной PCR-пробирке:
- PCR Enzyme Mix 25 μL
- PCR Primer Mix 6 μL
- фрагменты ДНК 19 μL
Всего: 50 μL
2. Поставить в амплификатор с использованием следующей программы:

Очистка (с использованием Agencourt AMPure ХР)
1. Добавить к 50 μL реакции 50 μL бидов (1х от объема реакции), перемешать пипетированием;
2. Инкубировать при комнатной температуре 5 минут;
3. Сбить капли мини-центрифугой, перенести пробирки на магнитный штатив. После того как смесь станет прозрачной (на это требуется до 5 минут), нужно осторожно удалить супернатант, не задевая биды, которые содержат фрагменты ДНК;
4. Добавить 200 μL свежего(!) 80% EtOH, проворачивать пробирку в штативе на 180° таким образом, чтобы биды пробежали через раствор спирта несколько раз, собрать все биды на одной стенке, осторожно удалить EtOH, не задевая бидов;
5. Повторить шаг 4 один раз, чтобы в итоге получилось 2 отмывки;
6. Максимально отобрать микропипеткой 2-20 остатки раствора спирта;
7. Добавить 37 μL LowTE, ресуспендировать биды на вортексе, осадить их пульс-спином, инкубировать 5 мин, после поместить на магнитный штатив, инкубировать 5 мин. Перенести супернатант в новую пробирку. В супернатанте очищенная ДНК.
Контроль качества целевой библиотеки ДНК
1) Провести контроль качества полученных библиотек фрагментов ДНК на приборе Agilent Bioanalyzer 2100 с помощью набора High Sensitivity Kit по протоколу производителя.
Хорошим результатом является распределение длин с пиком в области 300-600 bp.
Молярность должна составлять не менее 10 nmol/l.
Гибридизация биотинилированной нормализованной библиотеки кДНК с фрагментированной целевой геномной ДНК
1. Дождаться прогрева всех компонентов гибридизации, (программа амплификации в постоянном режиме 65°С).
2. Прямо в амплификаторе открыть пробирки и перенести 5 μL нормализованной биотинилированной библиотеки кДНК к 27 мкл ранее полученной целевой библиотеки ДНК (с NGS-адаптерами), аккуратно перемешать пипетированием и закрыть крышки.
4. Добавить минеральное масло (заранее прогретое до 65°С), чтобы предотвратить выпаривание образцов (12-15 мкл). Масло добавлять по стенкам пробирок, чтобы оно аккуратно покрыло реакционную смесь. Плотно закрыть крышки.
5. Оставить гибридизацию на 16-24 часа;
Отмывка продуктов реакции обогащения
1. Приготовить Промывочный буфер Wash Buffer 3 в 1,5 mL пробирке (на 1 пул образцов), смешав:
- Hyb4 12,1 μL
- MQ Water 1200 μL
- Wash Buffer 2 300 μL
Поставить пробирку с промывочным буфером в термостат на 66°С на 45 минут.
(c) Отбор на колонках со стрептавидином ДНК/кДНК-гетеродуплексов.
1. Добавить к гибридизованной библиотеке 50 μL стрептавидиновых шариков (на 1 пул) в 1,5 mL пробирку LoBind, перемешать;
2. Инкубировать в течение 15 минут при 20°С.
3. Гранулы дважды промыть в 150 μL буфера для связывания.
4. Повторить пункт 4 дважды, чтобы в итоге получилось 3 отмывки;
5. Ресуспендировать в 40 мкл mQ.
(d) ПЦР-амплификация обогащенной белок-кодирующими регионами библиотеки с концевых NGS-адаптеров.
Постановка ПЦР-реакции
1. Смешать следующие компоненты в стерильной PCR-пробирке:
- KAPA HiFi GC Buffer (5Х) 10 μL
- KAPA dNTP Mix 1,5 μL
- MGI primer 1 5 μL
- MGI primer 2 5 μL
- KAPA HiFi HotStart DNA Polymerase 0,7 μL
- фрагменты обогащенной ДНК 20-22 μL
- mQ up to 50 μL
Всего: 50 μL
2. Поставить в амплификатор с использованием следующей программы:
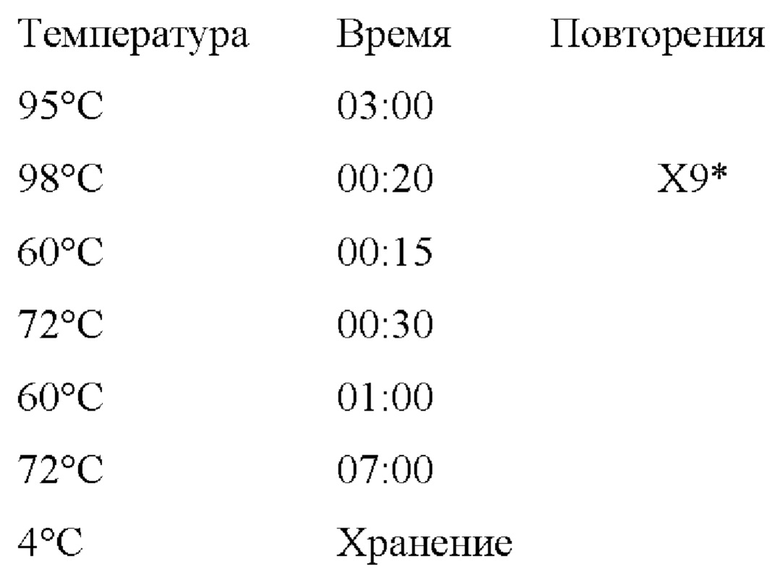
3. Отобрать 2,5 мкл ПЦР-продукта и сразу поставить ГЭФ 2% 25 мин 135 V для качественной проверки. Хорошим результатом является распределение длин с максимальным свечением в области 300-500 bp.
Очистка (с использованием MagBio)
1. Добавить к 50 μL реакции 65 μL бидов (1,3х от объема реакции), перемешать пипетированием;
2. Инкубировать при комнатной температуре 7 минут;
3. Сбить капли мини-центрифугой, перенести пробирки на магнитный штатив. После того как смесь станет прозрачной, удалить супернатант, не задевая магнитные частицы, которые содержат фрагменты ДНК;
4. Добавить 180 μL свежего (!) 80% EtOH, переставлять стрип в штативе таким образом, чтобы биды пробежали через раствор спирта несколько раз, собрать все магнитные частицы на одной стенке, осторожно удалить EtOH, не задевая бидов;
5. Повторить шаг 5 еще один раз, чтобы в итоге получилось 2 отмывки;
6. Максимально отобрать микропипеткой 2-20 остатки раствора спирта. Оставить сушиться пробирки с открытой крышкой прямо на магнитном штативе на 3-5 минут, не допуская пересушивания (при пересушивании магнитная пеллета трескается), но при этом спирт должен испариться полностью (поверхность пеллеты станет матовой);
7. Добавить 38 μL mQ, ресуспендировать биды на вортексе, осадить пульс-спином, инкубировать 7 мин. Затем поместить на магнитный штатив, инкубировать до полной агрегации бидов на стенке пробирки (до 5 мин). Перенести супернатант в новую пробирку. В супернатанте очищенная библиотека ДНК.
Ссылки на использованные источники
1. Singh RR. Target Enrichment Approaches for Next-Generation Sequencing Applications in Oncology. Diagnostics (Basel). 2022 Jun 24; 12(7): 1539. doi: 10.3390/diagnostics12071539. PMID: 35885445; PMCID: PMC9318977.
2. Liang WS, Stephenson K, Adkins J, Christofferson A, Helland A, Cuyugan L, Keats JJ. Whole Exome Library Construction for Next Generation Sequencing. Methods Mol Biol. 2018; 1706:163-174. doi: 10.1007/978-1-4939-7471-9_9. PMID: 29423798.
3. Патент № US 10894979 B2 Construction of next generation sequencing (NGS) libraries using competitive strand displacement (2019) «Создание библиотек для высокопроизводительного секвенирования (NGS)».
4. Bogdanova ЕА, Shagin DA, Lukyanov SA. Normalization of full-length enriched cDNA. MolBiosyst. 2008 Mar; 4(3):205-12. doi: 10.1039/b715110c. Epub 2008 Jan 8. PMID: 18437263.
5. Bogdanova EA, Barsova EV, Shagina IA, Scheglov A, Anisimova V, Vagner LL, Lukyanov SA, Shagin DA. Normalization of full-length-enriched cDNA. Methods Mol Biol. 2011; 729:85-98. doi: 10.1007/978-1-61779-065-2_6. PMID: 21365485.
6. Soares MB, de Fatima Bonaldo M, Hackett JD, Bhattacharya D. Expressed sequence tags: normalization and subtraction of cDNA libraries expressed sequence tags\ normalization and subtraction of cDNA libraries. Methods Mol Biol. 2009; 533:109-22. doi: 10.1007/978-1-60327-136-3_6. PMID: 19277560.
7. US 20150211047 A1 (United States) Fast hybridization for next generation sequencing target enrichment
8. Chen R, Im H, Snyder M. Whole-Exome Enrichment with the Agilent Sure Select Human All Exon Platform. Cold Spring Harb Protoc. 2015 Mar 11; 2015(7):626-33. doi: 10.1101/pdb.prot083659. PMID: 25762417; PMCID: PMC4490097.
| название | год | авторы | номер документа |
|---|---|---|---|
| Технология полноэкзомного обогащения на основе гибридизации с полноэкзомной РНК-библиотекой | 2023 |
|
RU2830886C1 |
| Технология полноэкзомного обогащения на основе различия в кинетике реассоциации фрагментов ДНК | 2023 |
|
RU2839144C1 |
| Способ создания кольцевой формы NGS библиотеки для высокопроизводительного секвенирования на платформах технологии DNBSEQ | 2020 |
|
RU2746960C1 |
| Способ создания кольцевой формы NGS библиотеки Illumina для высокопроизводительного секвенирования на платформах технологии DNBSEQ | 2021 |
|
RU2781613C1 |
| Способ определения эффективности обогащения NGS-библиотеки участками экзонов для секвенирования экзома человека | 2020 |
|
RU2743586C1 |
| Способ определения кариотипа плода беременной женщины на основании секвенирования гибридных прочтений, состоящих из коротких фрагментов внеклеточной ДНК | 2019 |
|
RU2717023C1 |
| Способ отделения клеток плацентарного трофобласта от отслоившихся клеток шейки матки беременных женщин | 2022 |
|
RU2808260C1 |
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |
| ТАГМЕНТАЦИЯ С ПРИМЕНЕНИЕМ ИММОБИЛИЗОВАННЫХ ТРАНСПОСОМ С ЛИНКЕРАМИ | 2018 |
|
RU2783536C2 |
| Метод определения уровня метилирования промоторной области гена COL1A2 для диагностики рака толстой кишки | 2015 |
|
RU2612890C1 |
Изобретение относится к биотехнологии. Описан способ обогащения библиотеки фрагментов ДНК белок-кодирующими последовательностями (экзомом) для последующего секвенирования методом высокопроизводительного секвенирования второго поколения (NGS), согласно изобретению для отбора белок-кодирующих регионов из геномной ДНК используют нормализованный транскриптом в форме биотинилированной библиотеки ДНК. Изобретение расширяет арсенал средств для получения библиотек фрагментов нуклеиновых кислот.
Способ обогащения библиотеки фрагментов ДНК белок-кодирующими последовательностями (экзомом) для последующего секвенирования методом высокопроизводительного секвенирования второго поколения (NGS), отличающийся тем, что для отбора белок-кодирующих регионов из геномной ДНК используют нормализованный транскриптом в форме биотинилированной библиотеки ДНК.
| Е.Н | |||
| Суспицын и др | |||
| Полноэкзомное секвенирование: принципы и диагностические возможности, Педиатр, 2016, том 7, Выпуск 4, страница 143, колонка 2, страница 144, колонка 1, найдено в интернет 22.07.2024 file:///C:/Users/otd1356/Downloads/polnoekzomnoe-sekvenirovanie-printsipy-i-diagnosticheskie-vozmozhnosti.pdf | |||
| David Tesin, DNA library |
Авторы
Даты
2025-04-21—Публикация
2023-12-28—Подача