Изобретение относится к биологии и медицине, а именно к генной инженерии. Данная система может быть использована в фундаментальных исследованиях клеточной биологии, а также при моделировании генетических заболеваний человека на клеточных системах.
Основным методом бесшовного внесения интересующей ДНК последовательности в клеточный геном является метод направленной гомологичной рекомбинации [Capecchi М.R. Altering the genome by homologous recombination. // Science. 1989. T. 244. №4910. C. 1288-92]. В данном методе используются донорные конструкции, содержащие выбранную для вставки ДНК последовательность между плечами гомологии - последовательностями, которые идентичны участкам, фланкирующим участок генома, в который будет проводиться вставка. Такая методика позволяет проводить очень точные модификации, но с низкой частотой: успешное встраивание последовательности в геном происходит в одной из 105-107 клеток [Capecchi М.R. Altering the genome by homologous recombination. // Science. 1989. T. 244. №4910. C. 1288-92]. Внесение двунитевого разрыва в выбранный для редактирования участок генома увеличивает эффективность вставки конструкции с плечами гомологии на несколько порядков [Bibikova М., Carroll D., Segal D.J., Trautman J.K., Smith J., Kim Y.-G., Chandrasegaran S. Stimulation of Homologous Recombination through Targeted Cleavage by Chimeric Nucleases // Mol. Cell. Biol. 2001. T. 21. №1. C. 289-297.; Plessis A., Perrin A., Haber J.E., Dujon B. Site-specific recombination determined by I-SceI, a mitochondrial group I intron-encoded endonuclease expressed in the yeast nucleus // Genetics. 1992. T. 130. №3. C. 451-460.; Rouet P., Smih F., Jasin M. Introduction of double-strand breaks into the genome of mouse cells by expression of a rare-cutting endonuclease. // Mol. Cell. Biol. 1994. T. 14. №12. C. 8096-106.; Rudin N., Sugarman E., Haber J.E. Genetic and physical analysis of double-strand break repair and recombination in Saccharomyces cerevisiae. // Genetics. 1989. T. 122. №3. C. 519-34].
В связи с этим было разработано множество специфичных ДНК-связывающих белков для внесения двунитевых разрывов в геном. Из них доминирующую роль на данный момент занимает система CRISPR-Cas9, специфичность которой задается короткой последовательность гидовой РНК (sgRNA) [Cho S.W., Kim S., Kim J.M., Kim J.-S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease // Nat. Biotechnol. 2013. T. 31. №3. C. 230-232.; Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W., Marraffini L.A., Zhang F. Multiplex Genome Engineering Using CRISPR/Cas Systems // Science. 2013. T. 339. №6121. C. 819.; Jinek M., East A., Cheng A., Lin S., Ma E., Doudna J. RNA-programmed genome editing in human cells // Elife. 2013. T. 2. С. e00471].
Репарация внесенного эндонуклеазой двунитевого разрыва ДНК может проходить несколькими путями. Среди них путь гомологичной репарации может привести к встраиванию интересующей последовательности ДНК в геном, так как в этом пути в качестве матрицы может использоваться внесенная в клетку донорная ДНК. Одним из способов повышения эффективности вставки интересующей последовательности в геном является увеличение вероятности прохождения репарации двуцепочечного разрыва по пути гомологичной репарации. Для этого можно использовать искусственные системы, привлекающие к области ДНК разрыва белки, благоприятствующие гомологичной репарации.
Среди белков, принимающих участие на ранних этапах гомологичной репарации, особый интерес вызывает CtIP белок, который связывает концевые участки ДНК и активирует резекцию 5' концов, взаимодействуя с MRN комплексом и Exol/Dna2 нуклеазами [Yeh С.D., Richardson С.D., Corn J.E. Advances in genome editing through control of DNA repair pathways // Nat. Cell Biol. 2019. T. 21. №12. C. 1468-1478].
Было показано, что использование spCas9 белка, связанного через короткий линкер с эффекторным CtIP белком, может повышать эффективность встраивания интересующей последовательности в два раза [Charpentier М., Khedher А.Н.Y., Menoret S., Brion A., Lamribet K., Dardillac Е., Boix С., Perrouault L., Tesson L., Geny S., Cian A. De, Itier J. M., Anegon I., Lopez В., Giovannangeli C., Concordet J.P. CtIP fusion to Cas9 enhances transgene integration by homology-dependent repair // Nat. Commun. 2018. T. 9. №1. C. 1-11]. Однако повышение эффективности в такой системе значительно зависит от выбранного для редактирования участка ДНК и может полностью пропадать для некоторых вариантов sgRNA [Charpentier М., Khedher А.Н.Y., Menoret S., Brion A., Lamribet K., Dardillac Е., Boix С., Perrouault L., Tesson L., Geny S., Cian A. De, Itier J. M., Anegon I., Lopez В., Giovannangeli C., Concordet J.P. CtIP fusion to Cas9 enhances transgene integration by homology-dependent repair // Nat. Commun. 2018. T. 9. №1. С. 1-11]. Разработка новой, более стабильной и эффективной системы позволяет расширить возможности применения геномного редактирования.
На момент подачи заявки из открытых источников было известно о следующих наиболее близких аналогах.
Известна система редактирования генома, использующая сшивку CtIP белка через короткий линкер с Cas9 белком (WO 2018162702, А1). Однако она показала ограниченную эффективность на клетках млекопитающих, что может объясняться конформационными ограничениями, которые накладывает прямая сшивка белков.
В качестве прототипа нами выбрана система, предполагающая привлечение эффекторных белков репарации с помощью MS2 системы адаптеров [Tran N.-T., Bashir S., Li X., Rossius J., Chu V.Т., Rajewsky K., Kiihn R. Enhancement of Precise Gene Editing by the Association of Cas9 With Homologous Recombination Factors // Front. Genet. 2019. T. 10. C. 2-5].
Использование MS2 адаптеров для привлечения белков увеличивает эффективность вставки интересующей последовательности, однако ограничивает применение этой системы клеточными линиями, в которых не используются широко применяемые MS2 адаптеры.
Проблемой, на решение которой направлено данное изобретение, является расширение арсенала средств для увеличения эффективности вставки целевых конструкций в геном человека.
Технический результат заключается в расширении арсенала средств для увеличения эффективности вставки целевых конструкций в геном человека.
Сущность изобретения заключается в следующем. Предложен рибонуклеопротеиновый комплекс для редактирования генома человека, исключая клетки зародышевой линии человека, путем вставки в него интересующей последовательности, включающий белок spCas9, связанный с sgRNA-BoxB, кодируемой последовательностью SEQ ID NO: 1, которая связана с эффекторным химерным белком, кодируемым последовательностью SEQ ID NO: 2.
Разработанный рибонуклеопротеиновый комплекс может быть доставлен в клетку с помощью трансфекции или электропорации в виде двух векторов.
В нашей работе последовательность химерного эффекторного белка (SEQ ID NO: 2) была получена за счет сшивки последовательности пептида N - пептида адаптерной системы, полученной из бактериофага λ (23 а.о.), через линкер с NLS последовательностью для локализации белка в ядре (34 а.о.), с последовательностью гена человеческого CtIP белка (897 а.о.). Для облегчения считывания успешной экспрессии химерного эффекторного белка к его концу через Р2А-Т2А разрезающиеся последовательности был добавлен репортерный красный флуоресцентный белок mRuby. Эта конструкция была клонирована в pCS2+ вектор по сайтам рестрикции BamHI/XbaI. Карта вектора, несущего последовательность химерного эффекторного белка, представлена на фиг. 1.
Конструкция, несущая sgRNA-BoxB последовательность (SEQ ID NO: 1), которая была получена внесением двух ВохВ шпилек в последовательность sgRNA, под U6 промотором, была клонирована в lentiCRISPR v2 вектор по сайтам рестрикции KpnI/NheI. Карта бицистронного вектора, несущего последовательность spCas9 белка и последовательность sgRNA-BoxB представлена на фиг. 2. Для изменения места вставки интересующей последовательности в геном, целевой участок sgRNA из первых 20 нуклеотидов может быть клонирован в вектор по сайтам рестрикции BsmBI. Последовательность целевой части sgRNA может быть получена отжигом двух олигонуклеотидов. При дизайне олигонуклеотидов для отжига следует использовать 20 нуклеотидов непосредственно перед РАМ участком в выбранном для вставки месте в геноме. При отсутствии G нуклеотида в начале целевой последовательности, этот нуклеотид нужно внести перед целевой частью sgRNA для успешной транскрипции с U6 промотора. Для клонирования по BsmBI сайтам к олигонуклеотиду прямой направленности следует добавить САСС последовательность и к олигонуклеотиду обратной направленности - АААС последовательность.
Для успешного проведения вставки в геном требуется донорный вектор, несущий интересующую последовательность. В качестве донора использовали конструкцию, несущую левое плечо гомологии к AAVS1 локусу (804 п.н.), SA сигнал акцептора сплайсинга, Т2А разрезающуюся последовательность, флуоресцентный зеленый EGFP белок (239 а.о.), сигнал полиаденилирования bGH poly(A) и правое плечо гомологии к AAVS1 локусу (804 п.н.). Эта конструкция была клонирована в pQE30 вектор по сайтам рестрикции XhoI/XbaI. Карта вектора, несущего донорную последовательность EGFP к AAVS1 локусу, представлена на фиг. 3. При изменении места вставки интересующей последовательности в геном плечи гомологии должны быть подобраны так, чтобы совпадать с последовательностями, окружающими место для вставки. При этом рекомендуется выбирать плечи гомологии так, чтобы участок генома, попадающий в область вставки между плечами гомологии, не превышал 100 п.н., в идеале не превышал 10 п.н. Плечи гомологии не должны содержать целевой последовательности sgRNA или не должны содержать РАМ участка после него. При изменении последовательности, выбранной для встраивания в геном, она должна быть помещена в донорный вектор на место EGFP последовательности.
В качестве примера использовали комплекс для проведения вставки EGFP гена в AAVS1 локус клеточной линии HEK293T. В качестве целевой последовательности был выбран TGTCCCTAGTGGCCCCACTG участок AAVS1 локуса. Для проверки эффективности комплекс был использован в сравнении с существующей MS2 системой адаптеров. Векторы, кодирующие комплексы и донорный вектор, несущий последовательность EGFP, были доставлены в клетку методом электропорации. После электропорации клетки проходили отбор пуромицином в течении трех дней. На четвертый день клетки подвергались анализу на проточном цитофлуориметре. Результаты проточной цитофлуориметрии редактируемых клеток представлены на фиг. 4 и в таблице.
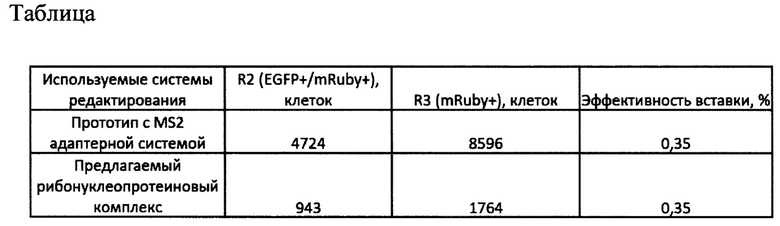
R2 областью обозначены EGFP/mRuby положительные клетки, которые прошли геномную вставку EGFP последовательности в AAVS1 локус и содержат химерный эффекторный белок. R3 областью обозначены mRuby положительные клетки, которые не прошли геномную вставку и содержат химерный эффекторный белок. Эффективность вставки рассчитывалась как отношение количества клеток в области R2 к общему количеству клеток в областях R2 и R3.
Клетки, успешно получившие компоненты предлагаемой системы, встраивают интересующую последовательность в клеточный геном, что может быть использовано для моделирования наследственных заболеваний.
Отдельно стоит отметить, что увеличенная эффективность встраивания интересующей последовательности в геном позволит с большей легкостью изучать модели полигенетических заболеваний, вызванных изменениями в нескольких генах, среди которых различные формы диабета, рака и болезней сердца.
Настоящее изобретение не предназначено для модификации генетической целостности клеток зародышевой линии человека, не предполагает использование человеческих эмбрионов.
--->
Перечень последовательностей
<110> Федеральное государственное автономное образовательное учреждение
высшего образования «Российский национальный исследовательский медицинский
университет имени Н.И. Пирогова» Министерства здравоохранения Российской
Федерации (Federalnoe gosudarstvennoe autonomnoe obrazovatelnoe uchrezhdenie
vysshego obrazovaniya «Rossijskij natsionalnyj issledovatelskij meditsinskij
universitet imeni N.I. Pirogova» Ministerstva zdravookhraneniya Rossijskoj
Federatsii)
<120> Рибонуклеопротеиновый комплекс для редактирования генома человека
путем вставки в него интересующей последовательности
<160> 2
<210> SEQ ID NO: 1
<211> 145
<212> ДНК
<213> Искусственная
<220>
<223> Последовательность, кодирующая sgRNA-BoxB
<400> 1
caccggagac gggataccgt ctctgtttta gagctaggcc gccctgaaga agggcggcct 60
agcaagttaa aataaggcta gtccgttatc aacttggccg ccctgaagaa gggcggccaa 120
gtggcaccga gtcggtgctt ttttt 145
<210> SEQ ID NO: 2
<211> 2862
<212> ДНК
<213> Искусственная
<220>
<223> Последовательность, кодирующая химерный эффекторный белок
<400> 2
atgggtaatg ctcggacccg gcgaagagag aggcgggctg agaagcaggc acagtggaag 60
gctgcaaacc gtctcctggc aagcgctgga ggaggtggaa gcggaggagg aggaagcgga 120
ggaggaggta gcggacctaa gaaaaagagg aaggtggctg ctgctaccgg tatgaacatc 180
tcgggaagca gctgtggaag ccctaactct gcagatacat ctagtgactt taaggacctt 240
tggacaaaac taaaagaatg tcatgataga gaagtacaag gtttacaagt aaaagtaacc 300
aagctaaaac aggaacgaat cttagatgca caaagactag aagaattctt caccaaaaat 360
caacagctga gggaacagca gaaagtcctt catgaaacca ttaaagtttt agaagatcgg 420
ttaagagcag gcttatgtga tcgctgtgca gtaactgaag aacatatgcg gaaaaaacag 480
caagagtttg aaaatatccg gcagcagaat cttaaactta ttacagaact tatgaatgaa 540
aggaatactc tacaggaaga aaataaaaag ctttctgaac aactccagca gaaaattgag 600
aatgatcaac agcatcaagc agctgagctt gaatgtgagg aagacgttat tccagattca 660
ccgataacag ccttctcatt ttctggcgtt aaccggctac gaagaaagga gaacccccat 720
gtccgataca tagaacaaac acatactaaa ttggagcact ctgtgtgtgc aaatgaaatg 780
agaaaagttt ccaagtcttc aactcatcca caacataatc ctaatgaaaa tgaaattcta 840
gtagctgaca cttatgacca aagtcaatct ccaatggcca aagcacatgg aacaagcagc 900
tatacccctg ataagtcatc ttttaattta gctacagttg ttgctgaaac acttggactt 960
ggtgttcaag aagaatctga aactcaaggt cccatgagcc cccttggtga tgagctctac 1020
cactgtctgg aaggaaatca caagaaacag ccttttgagg aatctacaag aaatactgaa 1080
gatagtttaa gattttcaga ttctacttca aagactcctc ctcaagaaga attacctact 1140
cgagtgtcat ctcctgtatt tggagctacc tctagtatca aaagtggttt agatttgaat 1200
acaagtttgt ccccttctct tttacagcct gggaaaaaaa aacatctgaa aacactccct 1260
tttagcaaca cttgtatatc tagattagaa aaaactagat caaaatctga agatagtgcc 1320
cttttcacac atcacagtct tgggtctgaa gtgaacaaga tcattatcca gtcatctaat 1380
aaacagatac ttataaataa aaatataagt gaatccctag gtgaacagaa taggactgag 1440
tacggtaaag attctaacac tgataaacat ttggagcccc tgaaatcatt gggaggccga 1500
acatccaaaa ggaagaaaac tgaggaagaa agtgaacatg aagtaagctg cccccaagct 1560
tcttttgata aagaaaatgc tttccctttt ccaatggata atcagttttc catgaatgga 1620
gactgtgtga tggataaacc tctggatctg tctgatcgat tttcagctat tcagcgtcaa 1680
gagaaaagcc aaggaagtga gacttctaaa aacaaattta ggcaagtgac tctttatgag 1740
gctttgaaga ccattccaaa gggcttttcc tcaagccgta aggcctcaga tggcaactgc 1800
acgttgccca aagattcccc aggggagccc tgttcacagg aatgcatcat ccttcagccc 1860
ttgaataaat gctctccaga caataaacca tcattacaaa taaaagaaga aaatgctgtc 1920
tttaaaattc ctctacgtcc acgtgaaagt ttggagactg agaatgtttt agatgacata 1980
aagagtgctg gttctcatga gccaataaaa atacaaacca ggtcagacca tggaggatgt 2040
gaacttgcat cagttcttca gttaaatcca tgtagaactg gtaaaataaa gtctctacaa 2100
aacaaccaag atgtatcctt tgaaaatatc cagtggagta tagatccggg agcagacctt 2160
tctcagtata aaatggatgt tactgtaata gatacaaagg atggcagtca gtcaaaatta 2220
ggaggagaga cagtggacat ggactgtaca ttggttagtg aaaccgttct cttaaaaatg 2280
aagaagcaag agcagaaggg agaaaaaagt tcaaatgaag aaagaaaaat gaatgatagc 2340
ttggaagata tgtttgatcg gacaacacat gaagagtatg aatcctgttt ggcagacagt 2400
ttctcccaag cagcagatga agaggaggaa ttgtctactg ccacaaagaa actacacact 2460
catggtgata aacaagacaa agtcaagcag aaagcgtttg tggagccgta ttttaaaggt 2520
gatgaaagag agactagctt gcaaaatttt cctcatattg aggtggttcg gaaaaaagag 2580
gagagaagaa aactgcttgg gcacacgtgt aaggaatgtg aaatttatta tgcagatatg 2640
ccagcagaag aaagagaaaa gaaattggct tcctgctcaa gacaccgatt ccgctacatt 2700
ccacccaaca caccagagaa tttttgggaa gttggttttc cttccactca gacttgtatg 2760
gaaagaggtt atattaagga agatcttgat ccttgtcctc gtccaaaaag acgtcagcct 2820
tacaacgcaa tattttctcc aaaaggcaag gagcagaaga ca 2862
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рибонуклеопротеиновый комплекс для редактирования генома человека | 2020 |
|
RU2749741C1 |
| Система привлечения белка к месту разрыва ДНК для увеличения эффективности редактирования генома млекопитающего | 2022 |
|
RU2808601C1 |
| Система для увеличения эффективности редактирования генома млекопитающего за счет гомологичной репарации | 2022 |
|
RU2808045C1 |
| Способ редактирования генома млекопитающих путем гомологичной репарации | 2022 |
|
RU2808600C1 |
| Набор для определения копийности вставки интересующей конструкции в AAVS1 локус генома человека | 2021 |
|
RU2786396C1 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМНОЙ ДНК ЭУКАРИОТИЧЕСКОЙ КЛЕТКИ НА ОСНОВЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩЕЙ БЕЛОК SUCAS9NLS | 2022 |
|
RU2804422C1 |
| ДОСТАВКА И ПРИМЕНЕНИЕ СИСТЕМ CRISPR-CAS, ВЕКТОРОВ И КОМПОЗИЦИЙ ДЛЯ ЦЕЛЕНАПРАВЛЕННОГО ВОЗДЕЙСТВИЯ И ТЕРАПИИ В ПЕЧЕНИ | 2014 |
|
RU2716420C2 |
| ГЕНЕТИЧЕСКИЕ КОНСТРУКЦИИ pcDEXO-GS-SSB, СОДЕРЖАЩИЕ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩИЕ ХИМЕРНЫЕ БЕЛКИ SSB И ЭКЗОНУКЛЕАЗЫ АППАРАТА ГОМОЛОГИЧНОЙ РЕКОМБИНАЦИИ БАКТЕРИОФАГОВ, ОБЪЕДИНЕННЫЕ ЧЕРЕЗ ГЛИЦИН-СЕРИНОВЫЙ ПОЛИЛИНКЕР | 2020 |
|
RU2763747C1 |
| Средство редактирования генома на основе белка LigD из бактерии Pseudomonas putida и Cas9 комплекса | 2022 |
|
RU2797049C1 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2701662C2 |


Изобретение относится к области биотехнологии, в частности к рибонуклеопротеиновому комплексу для редактирования генома человека путем вставки в него интересующей последовательности, который включает белок spCas9, связанный с sgRNA-BoxB, кодируемой последовательностью, которая связана с эффекторным химерным белком. Изобретение эффективно для создания клеточных моделей заболеваний человека, которые далее могут быть использованы для скрининга лекарственных препаратов. 4 ил.
Рибонуклеопротеиновый комплекс для редактирования генома человека, исключая клетки зародышевой линии человека, путем вставки в него интересующей последовательности, включающий белок spCas9, связанный с sgRNA-BoxB, кодируемой последовательностью SEQ ID NO: 1, которая связана с эффекторным химерным белком, кодируемым последовательностью SEQ ID NO: 2.
| NGOC-TUNG TRAN et al., Enhancement of Precise Gene Editing by the Association of Cas9 With Homologous Recombination Factors, Front Genet, 2019, Vol.10, N.365 | |||
| WO2018162702 A1, 13.09.2018 | |||
| M | |||
| CHARPENTIER et al., CtIP fusion to Cas9 enhances transgene integration by homology-dependent repair, NATURE COMMUNICATIONS, 2018, Vol.9, N.1133 | |||
| PHILIPPE |
Авторы
Даты
2021-07-06—Публикация
2020-12-11—Подача