Изобретение относится к области медицины, биотехнологии, молекулярной биологии, может быть использовано для определения и подсчета количества копий вставки интересующей конструкции в AAVS1 локус исследуемой популяции генетически модифицированных клеток. Актуальность изобретения связана с необходимостью анализа эффективности проведения вставки при проведении генетических модификаций клеток.
Настоящее изобретение не предназначено для модификации генетической целостности клеток зародышевой линии человека, а также для определения и подсчета количества копий вставки интересующей конструкции в исследуемой популяции генетически модифицированных клеток зародышевой линии человека, не предполагает использование человеческих эмбрионов.
Несмотря на быстрое развитие новых методов доставки конструкций в геном человека, наиболее распространенным методом на данный момент остается использование в качестве вектора для доставки ретровирусных конструкций. Эта методика продолжает использоваться как наиболее эффективная, однако ее использование может быть связано со значительными рисками для пациента. Так, при лечении Х-сцепленного тяжелого комбинированного иммунодефицита у пяти детей возник Т клеточный лейкоз [Howe S. J. и др. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients // J. Clin. Invest. 2008. T. 118. №9. C. 3143-3150.]. В одном из случаев лейкоз проявил резистентность к химиотерапии, что привело к гибели пациента. В другом исследовании при лечении синдрома Вискотта-Олдрича семь пациентов развили острую форму лейкоза [Braun С.J. и др. Gene therapy for Wiskott-Aldrich syndrome-long-term efficacy and genotoxicity // Sci. Transl. Med. 2014. T. 6. №227.]. При лечении Х-сцепленной хронической гранулематозной болезни два пациента развили миелодиспластический синдром [Stein S. и др. Genomic instability and myelodysplasia with monosomy 7 consequent to EVI1 activation after gene therapy for chronic granulomatous disease // Nat. Med. 2010. T. 16. №2. C. 198-204.]. Все случаи заболеваний связаны с непредсказуемостью места встраивания конструкции в геном человека при использовании ретровирусов. Это может приводить как к нарушению работы здоровых генов, так и к усилению экспрессии протоонкогенов.
Одним из возможных путей решения этой проблемы является направленное встраивание конструкции в геном человека. Для этого может использоваться быстро развивающаяся CRISPR система, которая позволяет проводить направленное встраивание конструкции при использовании подходящей донорной последовательности [Hsu P. D., Lander Е. S., Zhang F. Development and Applications of CRISPR-Cas9 for Genome Engineering // Cell. 2014. T. 157. №6. C. 1262-1278.]. Безопасное встраивание конструкции можно проводить в так называемые "тихие гавани" генома - участки, вставка в которые генетических последовательностей не нарушает нормальной работы клетки. Одним из хорошо охарактеризованных участков является интрон PPP1R12C гена на AAVS1 локусе 19 хромосомы [Sadelain М., Papapetrou Е. Р., Bushman F. D. Safe harbours for the integration of new DNA in the human genome // Nat. Rev. Cancer 2011 121. 2011. T. 12. №1. C. 51-58.]. Слабая стабильная экспрессия этого гена позволяет использовать его для встраивания конструкций без собственного промотора, что может быть использовано для экспрессии интересующей конструкции или для дальнейшего отбора отредактированных клеток с помощью флуоресцентных меток или генов устойчивости к антибиотикам.
Встраивание конструкции с использованием CRISPR системы происходит за счет репарации двунитевого разрыва ДНК выбранного участка генома. Белок CRISPR системы вносит в геномную ДНК двунитевой разрыв, специфичность которого задается короткой последовательностью гидовой РНК. При успешном встраивании интересующей конструкции в геном репарация этого разрыва проходит по пути гомологичной рекомбинации, в котором в качестве матрицы для репарации используется внесенная в клетку донорная последовательность ДНК. Значительным недостатком CRISPR системы по сравнению с использованием ретровирусных векторов остается низкая эффективность встраивания конструкции. Это объясняется особенностями работы системы, а также сложностью подбора оптимальных условий для каждого индивидуального случая, связанную со сложностью считывания успешного встраивания конструкции в геном.
На момент подачи заявки из открытых источников было известно о следующих аналогах.
В патенте US 2019203274, А1 описывается метод детекции HDR редактирования с использованием ddPCR, однако метод используется для детекции коротких изменений в геноме и не подразумевает использования для детекции встраивания полноразмерных конструкций.
В качестве прототипа нами выбран метод проверки CRISPR встраивания EGFP последовательности белка в человеческие iPS клетки с помощью системы ddPCR. В этой системе используется вставка последовательности флуоресцентного белка EGFP в локус тирозингидроксилазы для последующей визуализации дифференцированных дофаминергических нейронов [Überbacher С.и др. Application of CRISPR/Cas9 editing and digital droplet PCR in human iPSCs to generate novel knock-in reporter lines to visualize dopaminergic neurons // Stem Cell Res. 2019. T. 41.]. Для проведения ПЦР в этом методе используется две пары праймеров - одна пара для проверки наличия вставки и одна для нормировочного гена RPP30. Для проверки наличия вставки используется прямой праймер, отжигающийся выше левого плеча гомологии, использующегося в донорной конструкции, и обратный праймер, отжигающийся на Т2А последовательности донорной конструкции. В качестве зондов используются последовательность с FAM флуорофором, написанная на участок плеча гомологии и последовательность с НЕК флуорофором, написанная на последовательность RPP30 гена. Из клеток, подверженных редактированию на четвертый день выделяется геном, затем проводится пробоподготовка и постановка ddPCR, анализ результатов которого позволяет определить обогащение популяции iPS клетками с целевой вставкой.
Способ-прототип подходит для определения эффективности встраивания конструкции в ген тирозингидроксилазы, что является частным применением CRISPR редактирования. За пределами этого исследования остается определение эффективности встраивания интересующих конструкций в AAVS1 локус, что может быть использовано для широкого спектра исследований.
Проблемой, на решение которой направлено данное изобретение является создание набора, позволяющего проводить точное определение копийности вставки в AAVS1 локус интересующей конструкции в генетическом материале по отношению к копийности генома.
Технический результат, достигаемый при использовании предлагаемого набора, заключается в обеспечении высокоточного подсчета копийности вставки интересующей конструкции в генетическом материале.
Предложены оригинальные олигонуклеотидные праймеры и флуоресцентные зонды, позволяющие точно подсчитать копии вставки интересующей конструкции с нормировкой на один геном, при проведении цифровой капельной ПЦР.
Сущность изобретения заключается в следующем:
Предложен набор для определения копийности вставки интересующей конструкции в AAVS1 локусе генома человека при проведении цифровой капельной мультиплексной ПЦР, включающий прямой праймер FW-INS, имеющий последовательность SEQ ID NO: 1; обратный праймер RV-INS, имеющий последовательность SEQ ID NO: 2; флуоресцентный зонд INS-FAM, имеющий последовательность SEQ ID NO: 3, с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя 6-карбоксифлуоресцеина (6-FAM) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ1; прямой праймер FW-CCR5, имеющий последовательность SEQ ID NO: 4; обратный праймер RV-CCR5, имеющий последовательность SEQ ID NO: 5; флуоресцентный зонд CCR5-R6G, имеющий последовательность SEQ ID NO: 6, с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя Rhodamine 6G (R6G) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ2.
Таким образом, предложена система, которая позволяет быстро считывать эффективность встраивания конструкции в AAVS1 локус генома человека, что может быть использовано для эффективного подбора оптимальных условий встраивания интересующей конструкции.
Возможность точного подсчета копийности вставки интересующей конструкции в геном достигается за счет использования прямого праймера, написанного к AAVS1 участку выше левого плеча гомологии донорной конструкции и обратного праймера, написанного к Т2А последовательности:
FW-INS: 5'- CGGACCACTTTGAGCTCTAC - 3'
RV-INS: 5'- GTTAGAAGACTTCCTCTGCCC - 3'
и флуоресцентного зонда, комплементарного последовательности левого плеча гомологии донора:
INS-FAM: [6-FAM] 5' -TTCTCGGCGCTGCACCACGTGATGT-3'[BHQ1]
а также двух праймеров, фланкирующих локус участка гена CCR5:
FW-CCR5: 5'- TTGGTTTTGTGGGCAACATG - 3'
RV-CCR5: 5'- ACTTGAGTCCGTGTCACAAG - 3'
и флуоресцентного зонда, комплементарного последовательности гена CCR5:
CCR5-R6G: [R6G]5'-TGGTCCTGCCGCTGCTTGTCATGGT -3'[BHQ2]
При этом прямой праймер FW-INS для амплификации фрагмента вставки интересующей конструкции имеет последовательность SEQ ID NO: 1.
Обратный праймер RV-INS для амплификации фрагмента вставки интересующей конструкции имеет последовательность SEQ ID NO: 2.
Флуоресцентный зонд INS-FAM с флуоресцентным красителем 6-FAM и гасителем BHQ1 для детекции участка плеча гомологии интересующей конструкции имеет последовательность SEQ ID NO: 3.
Прямой праймер FW-CCR5 для амплификации фрагмента гена CCR5, имеет последовательность SEQ ID NO: 4.
Обратный праймер RV-CCR5 для амплификации фрагмента гена CCR5, имеет последовательность SEQ ID NO: 5.
Флуоресцентный зонд CCR5-R6G с флуоресцентным красителем R6G и гасителем BHQ2 детектирует участок гена CCR5 для нормировки и имеет последовательность SEQ ID NO: 6.
Для дизайна праймеров и зондов был проведен биоинформатический анализ, где в качестве исходной информации использовали последовательность генов AAVS1 и CCR5 из базы данных National Center for Biotechnology Information U.S. National Library of Medicine (Gene ID: 54776, updated on 23-Nov-2021, Gene ID: 1234, updated on 5-Dec-2021).
Далее, для реализации изобретения необходимо осуществить серию ПЦР-реакций с помощью системы (установки) для цифровой капельной ПЦР, например, Bio-Rad QX200 Droplet Digital PCR System, с использованием реагентов, предназначенных для конкретной системы (мастермикс, содержащий необходимые компоненты ПЦР, такие как Taq-полимераза, dNTP, растворы солей и т.д.). Приготовление смеси для цифровой ПЦР идет по следующему протоколу:
Состав смеси на одну лунку:
Общий объем должен быть 22 мкл, конечная концентрация праймеров 450 нМ, конечная концентрация зондов 250 нМ, раствор ДНК рекомендуется готовить так, чтобы в 4 мкл содержалось от 50 нг до 350 нг ДНК. Далее, с использованием прибора для генерации капель необходимо приготовить эмульсию капель. После завершения генерации капель планшет с каплями запаивается фольгой в течение 5 секунд при 180 градусах С. Запаянное плато помещается в любой амплификатор, поддерживающий формат 96-луночных планшетов «с юбкой» (например, С1000 Thermal Cycler, Bio-Rad). Протокол термоциклирования представлен в таблице.
После амплификации плато переносится в прибор, детектирующий флуоресценцию в каплях, по каналам FAM и R6G(HEX). Анализ результатов проводится с помощью программы, поставляемой с прибором - QuantaSoft Software Bio-Rad. В качестве типового результата программа выдает два графика флуоресценции (FAM и R6G) в формате 1D и совмещенный мультиплексный плот в формате 2D.
В качестве примера использовали метод для оценки эффективности встраивания последовательности EGFP белка в AAVS1 локус на клеточной линии Hela Kyoto. Для приготовления проб клеточная линия Hela Kyoto была трансфецирована с помощью FuGENE®HD реагента (Promega) бицистронной lentiCRISPR v2-Blast плазмидой (Addgene #83480) с гидовой РНК, направленной к AAVS1 локусу с последовательностью 5'-GTGTCCCTAGTGGCCCCACTG-3' и донорной плазмидой AAV-CAGGS-EGFP (Addgene #22212). Через пять дней после трансфекции из клеток был выделен геномный материал набором для выделения и очистки геномной ДНК ExtractDNA Blood (Евроген). Согласно представленной выше методике, была проведена оценка эффективности встраивания интересующей конструкции в геном.
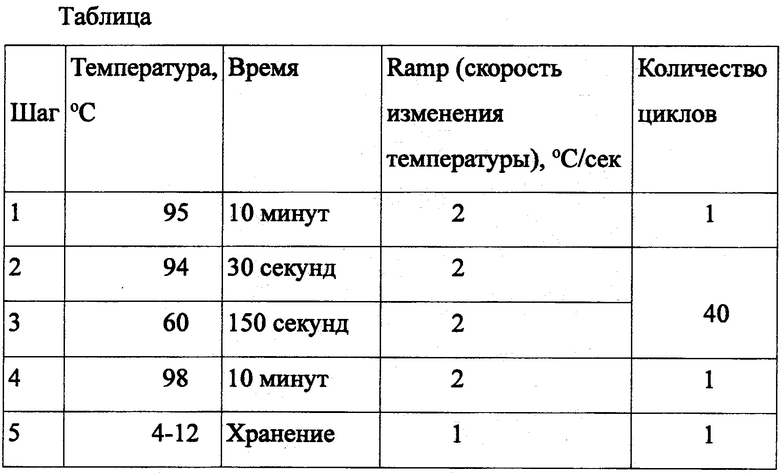
На фиг. 1 представлен пример анализа результата цифровой капельной ПЦР в виде графиков в формате 1D; верхний график - измерение флуоресценции в канале FAM; нижний график - измерение флуоресценции в канале R6G; каждая точка на графиках - проанализированная капля; по шкале ординат - интенсивность флуоресценции, по шкале абсцисс - порядковый номер проанализированной капли; нижний кластер капель (бесцветный) -капли без матрицы; верхние кластеры - положительные капли, где прошла ПЦР.
На фиг. 2 представлен пример анализа результата цифрового капельного ПЦР в виде 2D плота: по шкале ординат - интенсивность флуоресценции в канале FAM, по шкале абсцисс - интенсивность флуоресценции в канале R6G; негативный кластер капель (бесцветный) - слева внизу; кластер позитивных по FAM капель (синий) - слева вверху; кластер позитивных по R6G капель (зеленый) - справа внизу.
Таким образом, при анализе графиков в формате 1D (фиг. 1) видно два кластера капель, нижний - негативный, состоит из капель, в которые не попала матрица (ДНК) для амплификации, и верхний - позитивный, куда матрица (ДНК) попала, где успешно прошла ПЦР и появился сигнал флуоресценции. Программа автоматически разделяет оба кластера и подсчитывает количество положительных капель в каждом канале.
При анализе графиков в формате 2D (фиг. 2) кластеры капель, положительных по FAM и R6G, отчетливо различимы, и также автоматически разделяются. В случае успешного встраивания кассеты в AAVS1 локус количество капель, положительных по FAM составляет 8,8% от количества капель, положительных по R6G, что согласуется с литературными данными.
Основу способа с предлагаемым набором для измерения копийности вставки интересующей конструкции в AAVS1 локусе генома человека составляют синтезированные короткие ДНК олигонуклеотиды и приборная база для проведения цифрового капельного ПЦР. Предложенный метод считывания эффективности редактирования упрощает его оценку и может быть использован для оптимизации условий встраивания интересующей конструкции в геном.
SEQ ID NO 1: FW-INS: 5'- CGGACCACTTTGAGCTCTAC - 3'
SEQ ID NO 2: RV-INS: 5'- GTTAGAAGACTTCCTCTGCCC - 3'
SEQ ID NO 3: INS-FAM: [6-FAM] 5' -TTCTCGGCGCTGCACCACGTGATGT-3'[BHQ1]
SEQ ID NO 4: FW-CCR5: 5'- TTGGTTTTGTGGGCAACATG - 3'
SEQ ID NO 5: RV-CCR5: 5'- ACTTGAGTCCGTGTCACAAG - 3'
SEQ ID NO 6: CCR5-R6G: [R6G]5'-TGGTCCTGCCGCTGCTTGTCATGGT -3'[BHQ2]
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор для определения копийности гена АРР в геноме человека | 2021 |
|
RU2789799C1 |
| Набор для определения CCR5delta32 мутации в геноме человека | 2020 |
|
RU2748998C1 |
| Система для увеличения эффективности редактирования генома млекопитающего за счет гомологичной репарации | 2022 |
|
RU2808045C1 |
| Система привлечения белка к месту разрыва ДНК для увеличения эффективности редактирования генома млекопитающего | 2022 |
|
RU2808601C1 |
| Рибонуклеопротеиновый комплекс для редактирования генома человека путем вставки в него интересующей последовательности | 2020 |
|
RU2750939C1 |
| Рибонуклеопротеиновый комплекс для редактирования генома человека | 2020 |
|
RU2749741C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs5743708 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805861C1 |
| НАБОР РЕАГЕНТОВ И СПОСОБ ДЛЯ ВЫЯВЛЕНИЯ ДНК ВОЗБУДИТЕЛЕЙ ЧУМЫ, СИБИРСКОЙ ЯЗВЫ И ТУЛЯРЕМИИ МЕТОДОМ ПЦР С ГИБРИДИЗАЦИОННО-ФЛУОРЕСЦЕНТНЫМ УЧЕТОМ РЕЗУЛЬТАТОВ | 2013 |
|
RU2542395C1 |
| Мультиплексная ПЦР-смесь для определения серотипов 12FAB, 15BC, 22FA, 8 Streptococcus pneumoniae и способ ее применения | 2021 |
|
RU2787181C1 |
| Способ генотипирования гена TLR2 по полиморфизму rs3804100 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805860C1 |



Изобретение относится к биотехнологии, может быть использовано для определения и подсчета количества копий вставки интересующей конструкции в исследуемой популяции генетически модифицированных клеток. Предложен набор для определения копийности вставки интересующей конструкции в AAVS1 локус генома человека при проведении цифровой капельной мультиплексной полимеразной цепной реакции в режиме реального времени. Технический результат, достигаемый при использовании предлагаемого набора, заключается в обеспечении высокоточного подсчета копийности вставки интересующей конструкции в генетическом материале. 2 ил., 1 табл., 1 пр.
Набор для определения копийности вставки интересующей конструкции в AAVS1 локус генома человека, исключая клетки зародышевой линии человека, при проведении цифровой капельной мультиплексной полимеразной цепной реакции (ПЦР) в режиме реального времени, включающий прямой праймер, имеющий последовательность SEQ ID NO: 1; обратный праймер, имеющий последовательность SEQ ID NO: 2; флуоресцентный зонд INS-FAM, имеющий последовательность SEQ ID NO: 3, с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя 6-карбоксифлуоресцеина (6-FAM) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ1; прямой праймер, имеющий последовательность SEQ ID NO: 4; обратный праймер, имеющий последовательность SEQ ID NO: 5; флуоресцентный зонд CCR5-R6G, имеющий последовательность SEQ ID NO: 6, с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя Rhodamine 6G (R6G) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ2.
| Oceguera-Yanez F | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| - Т | |||
| Приспособление для записи звуковых явлений на светочувствительной поверхности | 1919 |
|
SU101A1 |
| - С | |||
| Зубчатое колесо со сменным зубчатым ободом | 1922 |
|
SU43A1 |
| Hamilton H | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Способ приготовления мыла | 1923 |
|
SU2004A1 |
| - Т | |||
| Парный автоматический сцепной прибор для железнодорожных вагонов | 0 |
|
SU78A1 |
| - No | |||
| Прибор для нагревания перетягиваемых бандажей подвижного состава | 1917 |
|
SU15A1 |
| - С | |||
Авторы
Даты
2022-12-20—Публикация
2021-12-22—Подача