Область техники, к которой относится изобретение
Одно или более воплощений изобретения относится к бактерии, конститутивно продуцирующей монофосфориллипид A (MLA), и к способу получения MLA с использованием этой бактерии.
Предшествующий уровень техники
Липополисахариды (LPS) являются одним из компонентов окружающей пептидогликан внешней мембраны у грамотрицательных бактерий. LPS представляют собой молекулы, содержащие липид А и ряд полисахаридов, конъюгированных с липидом А посредством ковалентной связи. Среди компонентов LPS липид А, также известный как эндотоксин, считают ответственным за токсичность грамотрицательных бактерий.
Липид А является очень активным стимулятором иммунной системы, активирующим клетки (например, моноциты или макрофаги) в количествах порядка пикограммов на миллилитр. Производные липида А или варианты липида А можно использовать в качестве, например, компонентов вакцин, таких как адъюванты. Монофосфориллипид A (MLA) используют в качестве адъюванта и применяют для аллерген-специфической иммунотерапии и иммунотерапии злокачественных новообразований, а также сообщали о его эффективности в профилактике и лечении деменции. Липид А, находящийся в мембране грамотрицательных бактерий, таких как Escherichia coli, образует конъюгаты с сахарами, такие как 2-кето-3-деокси-D-манно-октулозонат (Kdo). Соответственно, MLA может быть получен либо путем экстрагирования LPS из внешней мембраны бактерий, нагревания LPS в присутствии кислоты и гидролиза LPS в присутствии карбоната натрия для удаления Kdo и 1-фосфатной группы или ацильной цепи, либо способом химического синтеза. Однако в этих способах задействованы сложные процессы, и они приводят к низким выходам.
При введении в бактерии чужеродных генов с использованием методов генной инженерии бактерия может элиминировать чужеродный ген за счет естественного мутагенеза для ингибирования экспрессии чужеродного гена. Такой естественный мутагенез может вызывать снижение эффективности генно-инженерной трансформации и стабильности генетического конструирования.
Поэтому существует потребность в разработке более простого способа получения MLA и его производных по сравнению со способами из уровня техники, который не включает в себя кислотный гидролиз, уменьшает вероятность естественного мутагенеза, снижающего продукцию MLA, и приводит в результате к такой конститутивной экспрессии MLA и его производных, которая увеличивает выход.
Описание изобретения
Техническая задача изобретения
Одно или более воплощений изобретения включает бактерию, конститутивно продуцирующую монофосфориллипид A (MLA).
Одно или более воплощений изобретения включает способ получения MLA с использованием указанной бактерии, конститутивно продуцирующей ML.
Дополнительные аспекты будут отчасти представлены в следующем ниже описании, а отчасти станут очевидными на основании описания или могут быть изучены на практике представленных воплощений изобретения.
В соответствии с одним или более воплощений изобретения предложена бактерия, конститутивно продуцирующая монофосфориллипид A (MLA), которая включает в себя полипептид LpxE, и хромосома бактерии содержит мутацию полинуклеотиде, кодирующем фосфатазу ундекапренилпирофосфата (Und-PP фосфатазу), в полинуклеотиде, кодирующем фосфатазу фосфатидилглицерофосфата (PGP фосфатазу), или их комбинацию.
В соответствии с одним или более воплощений способ получения монофосфориллипида A (MLA) включает: культивирование бактерии в соответствии с любым из описанных выше воплощений изобретения с получением культуры; сбор бактерий из культуры; и выделение MLA из собранных бактерий.
Решение задачи
Данная заявка испрашивает приоритет заявки на патент Кореи 10-2017-0085406, поданной 5 июля 2017 г. в Ведомство по интеллектуальной собственности Кореи, описание которой полностью включено в настоящий документ посредством ссылки.
Используемый в настоящем документе термин «повышает экспрессию» относится к увеличению в клетке количества продукта экспрессии определенного гена, например, кодируемых этим геном мРНК или белка, которое можно обнаружить. Используемый в настоящем документе термин «родительская бактериальная клетка» относится к бактериальной клетке того же типа, но не имеющей конкретной генетической модификации. При использовании в генетической модификации клетки дикого типа родительская бактериальная клетка может представлять собой клетку «дикого типа». Например, бактерия, содержащая генетическую модификацию, повышающую экспрессию гена, может иметь уровень экспрессии продукта, превышающий этот уровень в родительской бактериальной клетке приблизительно на 5% или более, приблизительно на 10% или более, приблизительно на 15% или более, приблизительно на 20% или более, приблизительно на 30% или более, приблизительно на 40% или более, приблизительно на 50% или более, приблизительно на 60% или более, приблизительно на 70% или более, приблизительно на 80% или более, приблизительно на 90% или более, приблизительно на 95% или более или приблизительно на 100% или более. Увеличение количества продукта экспрессии в клетке может быть подтверждено любыми способами, известными в данной области техники. Уровень экспрессии продукта можно определить посредством измерения активности или количества продукта экспрессии, такого как мРНК или белок.
Генетическая модификация включает модификацию, которая вводит в клетку полинуклеотид, кодирующий полипептид; модификацию, которая приводит к заменам, добавлениям (то есть вставкам) или делециям одного или более нуклеотидов генетического материала родительской клетки, в том числе химическую модификацию (воздействие химическим веществом), приводящую в результате к изменению генетического материала родительской клетки. Генетическая модификация включает гетерологичную или гомологичную модификацию соответствующего биологического вида. Генетическая модификация включает модификацию кодирующей области полипептидов. Генетическая модификация также включает модификацию некодирующих регуляторных областей, которая изменяет экспрессию гена или функцию оперона. Некодирующие области включают 5'-некодирующую последовательность (в 5'-направлении от кодирующей последовательности) и 3'-некодирующую последовательность (в 3'-направлении от кодирующей последовательности).
Используемый в настоящем документе термин «идентичность последовательности» нуклеиновой кислоты или полипептида относится к степени идентичности нуклеотидов или аминокислотных остатков двух соответствующих последовательностей на протяжении конкретной области, измеряемой после максимально возможного выравнивания сопоставляемых последовательностей друг с другом. Идентичность последовательности представляет собой величину, которую измеряют путем сравнения двух соответствующих оптимально выровненных последовательностей конкретной сопоставимой области, в которой часть последовательности этой сопоставимой области может быть добавлена или делегирована по сравнению с референсной последовательностью. В некоторых воплощениях изобретения идентичность последовательности в процентах может быть рассчитана путем сравнения двух соответствующих оптимально выровненных последовательностей всей сопоставимой области, определения числа положений с идентичной аминокислотой или нуклеиновой кислотой в двух последовательностях с получением числа совпадающих положений, деления числа совпадающих положений на общее число (то есть диапазон размера) всех положений в пределах сопоставимого диапазона и умножения результата на 100 с получением идентичности последовательности в процентах. Идентичность последовательности в процентах можно определить, используя известные программы сравнения последовательностей, примеры которых включают BLASTN и BLASTP (NCBI), CLC Main Workbench (CLC bio.), MegAlign™ (DNASTAR Inc).
При идентификации полипептидов или полинуклеотидов различных биологических видов, которые могут иметь идентичную или сходную функцию или активность, можно использовать их подобие на основании идентичности последовательностей. Например, подобные последовательности могут иметь идентичность последовательности 50% или более, 55% или более, 60% или более, 65% или более, 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более или 100%.
Используемый в настоящем документе термин «экзогенный» и подобные термины относится к рассматриваемой молекуле (например, нуклеиновой кислоты) или рассматриваемой активности, введенной в клетку-хозяина. Нуклеиновая кислота может быть экзогенно введена в клетку-хозяина любым подходящим способом. Например, нуклеиновая кислота может быть введена в клетку-хозяина и встроена в хромосому клетки-хозяина, либо нуклеиновая кислота может быть введена в клетку-хозяина в виде нехромосомного генетического материала, такого как экспрессионный вектор (например, плазмида), который не интегрируется в хромосому клетки-хозяина. Нуклеиновую кислоту, кодирующую белок, следует вводить в форме с возможностью экспрессии (то есть так, чтобы нуклеиновая кислота могла транскрибироваться и транслироваться). Экзогенный ген может включать гомологичный ген, то есть ген, идентичный эндогенному гену, или гетерологичный ген.
В одном аспекте предложена бактерия, конститутивно продуцирующая монофосфориллипид A (MLA).
Эта бактерия может включать в себя генетическую модификацию, которая повышает экспрессию гена, кодирующего полипептид LpxE, по сравнению с родительской бактериальной клеткой. Хромосома бактерии может содержать мутацию в полинуклеотиде, кодирующем фосфатазу ундекапренилпирофосфата (Und-PP фосфатазу), мутацию в полинуклеотиде, кодирующем фосфатазу фосфатидилглицерофосфата (PGP фосфатазу), или их комбинацию.
Группировка липида А в монофосфориллипиде А состоит из двух глюкозаминов (углеводов или сахаров) с присоединенными ацильными цепями и обычно содержит одну фосфатную группу в каждом глюкозамине. Два дисахарида могут быть связаны β(1→6) связью. Ацильная цепь может быть непосредственно присоединена к гидроксильному остатку, выбранному из группы гидроксильных остатков в С-2, С-2', С-3 и С-3' положениях дисахарида глюкозамина. Ацильная цепь может иметь гидроксильный остаток, например, в ее С-3 положении, и к гидроксильному остатку, расположенному в ацильной цепи, может быть присоединена дополнительная ацильная цепь. Каждые из присоединенных ацильных цепей могут быть одинаковыми или разными. В зависимости от числа ацильных цепей липид А может представлять собой триацилированный липид А, тетраацилированный липид А, пентаацилированный липид А или гексаацилированный липид А, гептаацилированный липид А. Известно, что липид А, который эффективно активирует иммунную систему, содержит шесть ацильных цепей. Четыре ацильных цепи, присоединенных непосредственно к глюкозаминам, могут представлять собой бета-гидрокси-ацильные цепи, состоящие из 10-22 атомов углерода, а две дополнительные ацильные цепи в основном присоединены к бета-гидроксильной группе.
MLA относится к монофосфориллипиду А, в котором в С-1 или С-4' положении дисахарида глюкозамина может быть присоединена только одна фосфатная группа. MLA может представлять собой триацилированный MLA, тетраацилированный MLA, пентаацилированный MLA, гексаацилированный MLA или гептаацилированный MLA. Например, MLA может представлять собой 1-дефосфо-липид А, 1-дефосфо-тетраацилированный липид А, 1-дефосфо-пентаацилированный липид А или их комбинацию.
MLA не должен включать в себя 2-кето-3-деокси-D-манно-октулозанат (Kdo). Kdo представляет собой компонент липополисахаридов (LPS), консервативный почти во всех LPS.
MLA может присутствовать в мембране, например во внешней мембране живой бактерии.
Используемый в настоящем документе термин «бактерия» относится к прокариотической бактерии. Бактерия может представлять собой грамотрицательную бактерию. Грамотрицательные бактерии относятся к типу бактерий, которые не окрашиваются кристаллическим фиолетовым, используемым в методе окрашивания по Граму. Клеточные мембраны грамотрицательных бактерий состоят из двойной мембраны, включающей в себя внутреннюю мембрану и внешнюю мембрану, и включающей в себя тонкий пептидогликановый слой. Бактерия может быть выбрана из группы, состоящей из бактерий рода Escherichia, бактерий рода Shigella, бактерий рода Salmonella, бактерий рода Campylobacter, бактерий рода Neisseria, бактерий рода Haemophilus, бактерий рода Aeromonas, бактерий рода Francisella, бактерий рода Yersinia, бактерий рода Klebsiella, бактерий рода Bordetella, бактерий рода Legionella, бактерий рода Corynebacteria, бактерий рода Citrobacter, бактерий рода Chlamydia, бактерий рода Brucella, бактерий рода Pseudomonas, бактерий рода Helicobacter, бактерий рода Burkholderia, бактерий рода Porphyromonas, бактерий рода Rhizobium, бактерий рода Mesorhizobium и бактерий рода Vibrio. Например, бактерия может представлять собой Escherichia coli.
Бактерия в соответствии с одним или более воплощений изобретения может конститутивно продуцировать MLA. Используемый в настоящем документе термин «конститутивно» может относиться к продукции, которая осуществляется независимо от присутствия индуктора экспрессии, стимула, индуцирующего экспрессию, или антибиотиков.
Бактерия в соответствии с одним или более воплощений изобретения может включать в себя увеличенное число копий гена, кодирующего полипептид LpxE. Бактерия может включать в себя по меньшей мере один из экзогенных полинуклеотидов, кодирующих полипептид LpxE.
Полипептид LpxE относится к ЕС 3.1.3.-. LpxE относится к семейству фосфатаз фосфата липида. LpxE может содержать трехкомпонентный активный центр и шесть трансмембранных спиралей. Фосфатаза фосфата липида представляет собой гидролазу, специфично действующую на фосфомоноэфирные связи, которая может удалять фосфатную группу из липида, содержащего фосфатную группу. LpxE может представлять собой фосфатазу фосфата, специфично дефосфорилирующую 1-положение. Полипептид LpxE может представлять собой полипептид LpxE бактерии, выбранной из группы, состоящей из бактерии рода Aquifex, бактерии рода Helicobacter, бактерии рода Francisella, бактерии рода Bordetella, бактерии рода Brucella, бактерии рода Rhizobium, бактерии рода Mesorhizobium, бактерии рода Legionella, бактерии рода Agrobacterium, бактерии рода Chlorobium, бактерии рода Rhodospirillum, бактерии рода Magnetospirillum, бактерии рода Chlorobaculum, бактерии рода Pelodictyon, бактерии рода Pseudovibro, бактерии рода Phaeospirillum, бактерии рода Syntrophobacter, бактерии рода Bradyrhizobium, бактерии рода Porphyromonas, бактерии рода Ralstonia, бактерии рода Limnohabitans и бактерии рода Thermodesulfobacterium. Бактерия рода Aquifex может включать Aquifex aeolicus или Aquifex pyrophilus. Бактерия рода Aquifex представляет собой термофильную бактерию, которая может лучше всего расти при температуре в диапазоне от 85°С до 95°С. Бактерия рода Aquifex может представлять собой Aquifex aeolicus. Бактерия рода Helicobacter может представлять собой Helicobacter pylori. Например, полипептид LpxE может представлять собой полипептид LpxE, полученный из Aquifex aeolicus (AaLpxE), или полипептид LpxE, полученный из Helicobacter pylori (HpLpxE). Полипептид LpxE может представлять собой полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 12; или полипептид, обладающий идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с аминокислотной последовательностью SEQ ID NO: 12. Полипептид LpxE может представлять собой мутированный полипептид.
Полинуклеотид, кодирующий полипептид LpxE, может находиться в хромосоме бактерии. Полинуклеотид, кодирующий полипептид LpxE, может представлять собой полипептид, включающий аминокислотную последовательность, обладающую идентичностью последовательности приблизительно 90%, приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с нуклеиновокислотной последовательностью SEQ ID NO: 13. Полинуклеотид может представлять собой мутированную нуклеотидную последовательность. Например, полинуклеотид может включать в себя генную мутацию нуклеотидной последовательности, которая кодирует 17-ю аминокислоту серии (17Ser) с N-конца полипептида LpxE дикого типа, от AGC до TCG.
Полинуклеотид, кодирующий полипептид LpxE, может экспрессироваться конститутивно. Выражение «конститутивно экспрессируемый» может относиться к экспрессии гена в отсутствие индуктора экспрессии или стимула, индуцирующего экспрессию.
Бактерия в соответствии с одним или более воплощений изобретения может включать в хромосоме этой бактерии мутацию в полинуклеотиде, кодирующем фосфатазу ундекапренилпирофосфата (Und-PP фосфатазу), в полинуклеотиде, кодирующем фосфатазу фосфатидилглицерофосфата (PGP фосфатазу), или их комбинацию. Термин «бактериальная хромосома», которая содержит генетическую информацию бактерии, может представлять собой кольцевую ДНК. Бактериальная хромосома может не содержать плазмиду. Плазмида относится к кольцевой ДНК, которая физически отделена от бактериальной хромосомы и может реплицироваться независимо.
Und-PP фосфатаза представляет собой фермент, который продуцирует ундекапренилфосфат путем катализа дефосфорилирования ундекапренилпирофосфата. Ундекапренилфосфат представляет собой липидный носитель гликанового промежуточного соединения в биосинтезе углеводородного полимера, который передается в оболочку бактерии.
Полинуклеотидом, кодирующим Und-PP фосфатазу, может быть ген bacA, ген pgpB, ген ybjG или их комбинация. Ген bacA представляет собой ген, гиперэкспрессия которого придает резистентность к известному антибиотику бацитрацину. Ген pgpB представляет собой ген, кодирующий фермент, катализирующий дефосфорилирование фосфатидилглицеринфосфата (PGP) с образованием фосфатидилглицерина (PG). Этот фермент может обладать активностью Und-PP фосфатазы. Ген ybjG представляет собой ген, сверхэкспрессия которого повышает активность Und-PP фосфатазы и повышает резистентность к бацитрацину.
Полинуклеотидом, кодирующим PGP фосфатазу, может быть ген pgpB, ген pgpA, ген pgpC или их комбинация. Ген pgpA или ген pgpC представляет собой ген, кодирующий фосфатазу липида, которая дефосфорилирует PGP до PG.
Например, бактерия может включать в себя мутацию в гене bacA, мутацию в гене pgpB и мутацию в гене ybjG.
Термин «ген», означающий единицу генетической информации, может включать открытую рамку считывания (ORF), кодирующую полипептид, и регуляторную последовательность, регулирующую транскрипцию гена. Регуляторная последовательность может включать промотор, представляющий собой ДНК-домен, инициирующий транскрипцию гена, энхансер, стимулирующий транскрипцию, сайленсер, который может ингибировать транскрипцию, или их комбинацию.
Термин «мутация» относится к модификации генетического материала и может включать точечную мутацию, мутацию сдвига рамки считывания, вставку, делецию, инверсию или транслокацию. Например, мутация может представлять собой делецию, вставку, точечную мутацию, мутацию сдвига рамки считывания или их комбинацию. Точечная мутация может представлять собой миссенс-мутацию или нонсенс-мутацию. В результате мутации генетический материал может быть делетирован из генома бактерии или введен в него.
Бактерия в соответствии с одним или более воплощений изобретения может дополнительно включать в себя полинуклеотид, кодирующий полипептид, выбранный из группы, состоящей из полипептида LpxL и полипептида LpxM. Полинуклеотид может быть индуцибельным или конститутивно экспрессируемым. Индуцибельная экспрессия может относиться к экспрессии посредством индуктора экспрессии или стимула, индуцирующего экспрессию (например, термической обработки). Конститутивная экспрессия может относиться к экспрессии в отсутствие индуктора экспрессии или стимула, индуцирующего экспрессию. Полинуклеотид может экспрессироваться посредством промотора, выбранного из группы, состоящей из промотора PL, промотора PR и промотора PR'. Промотор PL, промотор PR и промотор PR' могут представлять собой промоторы, полученные из фага лямбда.
Полипептид LpxL может относиться к ЕС 2.3.1.241. Полипептид LpxL представляет собой лауроилтрансферазу биосинтеза липида А, которая катализирует перенос лаурата с белка-носителя лауроил-ацила (АСР) на Kdo2-липид IVA для синтеза Kdo2-(лауроил)-липида IVA. Полипептид LpxL может представлять собой полипептид LpxL бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium и бактерии рода Vibrio. Например, полипептид LpxL может представлять собой полипептид LpxL Escherichia coli (EcLpxL). Полипептид LpxL может представлять собой полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 1; или полипептид, обладающий идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с аминокислотной последовательностью SEQ ID NO: 1. Полипептид LpxL может кодировать нуклеиновокислотная последовательность SEQ ID NO: 2; или полинуклеотид, обладающий идентичностью последовательности приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с нуклеиновокислотной последовательностью SEQ ID NO: 2.
Полипептид LpxM может относиться к ЕС 2.3.1.243. Полипептид LpxM представляет собой миристоилтрансферазу биосинтеза липида А, которая катализирует перенос миристата с белка-носителя миристоил-ацила на Kdo2-лауроил-липид IVa для синтеза Kdo2-липида А. Полипептид LpxM может представлять собой полипептид LpxM бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium и бактерии рода Vibrio. Например, полипептид LpxM может представлять собой полипептид LpxM Escherichia coli (EcLpxM). Полипептид LpxM может представлять собой полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 5; или полипептид, обладающий идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с аминокислотной последовательностью SEQ ID NO: 5. Полипептид LpxM может кодировать нуклеиновокислотная последовательность SEQ ID NO: 6; или полинуклеотид, обладающий идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с нуклеиновокислотной последовательностью SEQ ID NO: 6.
Бактерия в соответствии с одним или более воплощений изобретения может дополнительно включать генетическую модификацию в полинуклеотиде, кодирующем полипептид LpxT, в полинуклеотиде, кодирующем полипептид PagP, в полинуклеотиде, кодирующем полипептид KdtA, или их комбинацию. Полипептид LpxT может относиться к ЕС 2.7.4.29. Полипептид LpxT может представлять собой белок внутренней мембраны LpxT. Полипептид LpxT может представлять собой полипептид, включающий в себя аминокислотную последовательность SEQ ID. NO: 18; или аминокислотную последовательность, обладающую идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с аминокислотной последовательностью SEQ ID NO: 18. Например, полипептид LpxT может представлять собой полипептид LpxT Escherichia coli (EcLpxT). Полипептид PagP может относиться к ЕС 2.1.1.251. Полипептид PagP может представлять собой пальмитоилтрансферазу липида А, требующуюся для биосинтеза гепта-ацилированных молекул липида А, содержащих пальмитат. Полипептид KdtA может относиться к ЕС 2.4.99.12 2.4.99.13 2.4.99.14 2.4.99.15. Полипептид KdtA представляет собой фермент, который связывает Kdo с липидом IVA. Полипептид KdtA может представлять собой полипептид, включающий в себя аминокислотную последовательность SEQ ID. NO: 22; или аминокислотную последовательность, обладающую идентичностью последовательности приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20%, приблизительно 10% или более с аминокислотной последовательностью SEQ ID NO: 22. Полипептид KdtA может представлять собой полипептид KdtA E.coli.
Бактерия в соответствии с одним или более воплощений изобретения может продуцировать монофосфориллипид A (MLA) без индуцирования экспрессии индуктором экспрессии, стимулом, индуцирующим экспрессию, или их комбинацией. Индуктором экспрессии может быть соединение, индуцирующее экспрессию гена. Например, индуктором экспрессии может быть изопропил-β-D-1-тиогалактопиранозид (IPTG), арабиноза, тетрациклин, триптофан или их комбинация. Стимулом, индуцирующим экспрессию, который представляет собой физический стимул, индуцирующий экспрессию гена, может быть, например, тепловая обработка или тепловой шок.
Бактерию можно культивировать без антибиотика. Антибиотик может представлять собой, например, канамицин, ампициллин, хлорамфеникол, тетрациклин, стрептомицин или их комбинацию.
В другом аспекте изобретения предложен способ получения монофосфориллипида A (MLA), включающий: культивирование бактерии в соответствии с любым из описанных выше воплощений изобретения с получением культуры; сбор бактерии из культуры; и выделение MLA из бактерии.
Термины «MLA», «конститутивно» и «бактерия», используемые в настоящем документе по отношению к способу в соответствии с одним или более воплощений изобретения, могут быть такими же, как определено выше.
Способ в соответствии с одним или более воплощений изобретения может включать культивирование бактерии в соответствии с любой из описанных выше, которые конститутивно экспрессируют MLA.
Культивирование можно осуществлять с использованием известного способа. Например, способ культивирования может представлять собой периодическую культуру, периодическую культуру с подпиткой, непрерывную культуру, ферментацию или их комбинацию.
Тип культуральной среды, температура культивирования и условия культивирования могут быть такими же, как известные в данной области техники. Температура культивирования может составлять, например, от приблизительно 10°С до приблизительно 43°С, от приблизительно 20°С до приблизительно 43°С, от приблизительно 20°С до приблизительно 40°С, от приблизительно 25°С до приблизительно 43°С, от приблизительно 25°С до приблизительно 35°С, от приблизительно 27°С до приблизительно 33°С, от приблизительно 10°С до приблизительно 15°С, от приблизительно 15°С до приблизительно 20°С, от приблизительно 20°С до приблизительно 25°С, от приблизительно 25°С до приблизительно 30°С, от приблизительно 30°С до приблизительно 33°С, от приблизительно 33°С до приблизительно 37°С, от приблизительно 37°С до приблизительно 40°С или от приблизительно 40°С до приблизительно 43°С. Бактерии можно культивировать в режиме периодической культуры, периодической культуры с подпиткой или непрерывной культуры. Культивирование можно осуществлять в стационарном состоянии или при встряхивании. Период культивирования может составлять, например, от приблизительно 1 часа до приблизительно 1 недели, от приблизительно 3 часов до приблизительно 6 дней, от приблизительно 6 часов до приблизительно 5 дней, от приблизительно 9 часов до приблизительно 4 дней, от приблизительно 12 часов до приблизительно 3 дней, от приблизительно 18 часов до приблизительно 2 дней, в течение приблизительно 1 дня или в течение ночи. Культуральная среда может включать в себя или не включать в себя антибиотик. Антибиотик может представлять собой, например, канамицин, ампициллин, хлорамфеникол, тетрациклин, стрептомицин или их комбинацию.
Культивирование бактерий можно проводить в отсутствие индуктора экспрессии, стимула, индуцирующего экспрессию, или их комбинации.
Способ в соответствии с одним или более воплощений изобретения может включать сбор бактерий из культуры. Сбор бактерий из культуры можно осуществлять с использованием известного способа. Например, сбор бактерий можно осуществлять путем центрифугирования. Собранные бактерии можно промывать буферным раствором.
Способ в соответствии с одним или более воплощений изобретения может включать выделение MLA из собранных бактерий.
MLA можно отделять от липидов бактерии. Липиды могут быть получены с использованием способа, известного в данной области техники. MLA может быть получен с использованием физического или химического способа. Физическим способом может быть многократное использование ультразвуковых импульсов или замораживание и оттаивание. Химическим способом может быть экстракция с использованием органического растворителя или осаждение. Например, органический растворитель может включать хлороформ, фенол, петролейный эфир, дихлорметан, метанол, гексан, изопропиловый спирт, этилацетат, ацетонитрил, этанол, бутанол или их комбинацию. Например, способом экстракции липидов может быть протокол экстракции липидов Блай и Дайера (см. Bligh, E.G. and Dyer, W.J., Can. J. Biochem. Physiol., 1959, vol. 37, p. 911-917). Этот способ может дополнительно включать очистку MLA от липидов. Способ может не включать стадию гидролиза для удаления группировки Kdo, поскольку полученный липид А может находиться в свободной форме, то есть не конъюгирован с группировкой Kdo.
Теперь будет приведена ссылка на детали экспериментов, примеры которых проиллюстрированы сопроводительными графическими материалами, в которых аналогичные справочные номера везде относятся к аналогичным элементам. В этом отношении воплощения настоящего изобретения могут иметь различные формы и не должны быть истолкованы как ограниченные представленными в настоящем документе описаниями. Соответственно, воплощения изобретения описаны ниже со ссылкой на графические материалы только лишь для пояснения аспектов настоящего описания. Используемые в настоящем документе термины «и/или» включают любые и все комбинации одного или более соответствующих перечисленных элементов. Выражения, такие как «по меньшей мере одно из», которым предшествует перечень элементов, модифицируют полный перечень элементов и не модифицируют отдельные элементы перечня.
Краткое описание графических материалов
Эти и/или другие аспекты станут очевидными и более понятными из последующего описания воплощений изобретения, рассмотренных в сочетании с сопроводительными графическими материалами, в которых:
На ФИГ. 1 представлено схематическое изображение, иллюстрирующее способ получения 1-дефосфо-липида А в бактерии в соответствии с одним или более воплощений изобретения;
На ФИГ. 2 представлено схематическое изображение, иллюстрирующее способ получения продукта полимеразной цепной реакции (PCR), включающего EcLpxL и EcLpxM;
На ФИГ. 3 представлено схематическое изображение, иллюстрирующее способ замещения bacA хромосомы Escherichia coli на lpxE, кодирующий фосфатазу, с использованием гомологичной рекомбинации;
На ФИГ. 4 представлено схематическое изображение, иллюстрирующее способ конструирования штамма, в котором усилена стабильность гена фосфатазы, вставленного в его хромосому;
На ФИГ. 5 представлено изображение, показывающее результаты тонкослойной хроматографии (TLC) липидов, экстрагированных из штаммов Е. coli, в которых из генома Escherichia coli удален ген bacA и заменен геном фосфатазы, где на дорожке 1 представлен KHSC0044, продуцирующий 1-дефосфо-липид А (две из 8 колоний, культивируемых в одной исходной культуре клеток), на дорожке 2 представлен KHSC0044, не продуцирующий 1-дефосфо-липид А (шесть из 8 колоний, культивируемых в одной исходной культуре клеток), на *?*-D-1-дорожке 3 представлен KHSC0031, продуцирующий 1-дефосфо-липид А (шесть из 7 колоний, культивируемых в одной исходной культуре клеток), на дорожке 4 представлен KHSC0031, не продуцирующий 1-дефосфо-липид А (одна из 7 колоний, культивируемых в одной исходной культуре клеток), на дорожке 5 представлен KHSC0045, стабильно продуцирующий 1-дефосфо-липид А (все 7 колоний, культивируемых в одной исходной культуре клеток), на дорожке 6 представлен синтетический 1-дефосфо-гекса-ацилированный липид А (InvovoGen) и на дорожке 7 представлен 1-дефосфо-липид А, выделенный и очищенный из Salmonella minnesota R595 после кислотно-основного гидролиза (InvovoGen);
На ФИГ. 6 представлено изображение, показывающее результаты TLC липидов штаммов Escherichia coli KHSC0045 и KHSC0055, где на дорожке 1 представлен синтетический 1-дефосфо-гекса-ацилированный липид A (InvovoGen), а на дорожке 2 представлен KHSC0045, культивированный с индуктором гиперэкспрессии, индуцирующим гиперэкспрессию фермента липидизации, а на дорожке 3 представлен KHSC0055, культивированный без использования индуктора гиперэкспрессии; и
На ФИГ. 7 представлено изображение, показывающее результаты TLC липидов штаммов Escherichia coli KHSC0045 и KHSC0055, где на дорожке 1 представлен KHSC0055, культивированный с антибиотиками, а на дорожке 3 представлен KHSC0055, культивированный без использования антибиотиков.
Лучший вариант осуществления изобретения
Ниже будет подробно описано одно или более воплощений настоящего описания со ссылкой на следующие примеры. Однако эти примеры приведены только в иллюстративных целях и не подразумевают ограничения объема одного или более воплощений настоящего описания.
Пример 1. Получение вектора, включающего в себя полинуклеотид, кодирующий LpxL Escherichia coli и LpxM Escherichia coli
1.1. Получение pWSK29-EcLpxLEcLpxM
Чтобы получить полинуклеотид, кодирующий полипептиды LpxL Е. coli, из генома W3110 Е. coli (GenBank, № по каталогу NC 000918.1, АТСС) амплифицировали полинуклеотид (GenBank, № по каталогу АР009048.1 (c1118159.1117239, SEQ ID NO: 2), кодирующий полипептид EcLpxL (GenBank, № по каталогу ВАА35852.1, SEQ ID NO: 1), включающий сайт связывания рибосомы (RBS), в первой полимеразной цепной реакции (PCR) с использованием пары праймеров:
прямой праймер P1 LpxL:

обратный праймер Р2 LpxL:
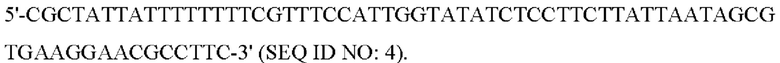
Чтобы получить полинуклеотид, кодирующий полипептиды LpxM Е. coli, из генома W3110 Е. coli амплифицировали полинуклеотид (GenBank, № по каталогу АР009048.1 (с1941907.1940936, SEQ ID NO: 6), кодирующий полипептид EcLpxM (GenBank, № по каталогу ВАА15663.1, SEQ ID NO: 5), включающий RBS, во второй PCR с использованием пары праймеров (см. ФИГ. 2):
прямой праймер Р3 LpxM:

обратный праймер Р4 LpxM:
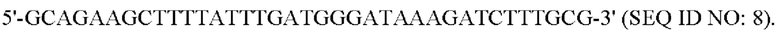
Полинуклеотид EcLpxLEcLpxM, представляющий собой слияние полинуклеотида EcLpxL и полинуклеотида EcLpxM, амплифицировали в третьей PCR с использованием прямого праймера P1 LpxL и обратного праймера Р4 LpxM и с использованием полинуклеотида EcLpxL, полученного в первой PCR, и полинуклеотида EcLpxM, полученного во второй PCR, в качестве матриц.
PCR проводили с использованием ДНК-полимеразы «горячего старта» KOD (Novagen) в термоциклере Т3000 (Biometra).
Продукты амплификации очищали с использованием набора для очистки продуктов PCR DokDo-Prep (ELPIS-BIOTECH. Inc.), и продукты очистки вводили в плазмиду pWSK29 (см. Wang, R.F., and Kushner, S.R., Gene (1991), vol. 100, p. 195-199). DH5α E. coli трансформировали этой клонированной плазмидой посредством электропорации, а затем трансформированные Е. coli отбирали на чашке со средой LB с ампициллином. Клонированная плазмида была обозначена pWSK29-EcLpxLEcLpxM (см. ФИГ. 2).
1.2. Получение pKHSC0004
Чтобы модифицировать промоторную последовательность pWSK29-EcLpxLEcLpxM, проводили сайт-направленный мутагенез промоторной последовательности PL  SEQ ID NO: 9) с использованием PCR с плазмидой pWSK29-EcLpxLEcLpxM, полученной, как описано выше в разделе 1.1, в качестве матрицы и парой праймеров:
SEQ ID NO: 9) с использованием PCR с плазмидой pWSK29-EcLpxLEcLpxM, полученной, как описано выше в разделе 1.1, в качестве матрицы и парой праймеров:
прямой праймер, амплифицирующий промотор P1:

обратный праймер, амплифицирующий промотор P1:

PCR проводили с использованием набора для сайт-направленного мутагенеза Quikchange (Agilent) в термоциклере Т3000 (Biometra).
После сайт-направленного мутагенеза продукт реакции обрабатывали ферментом рестрикции Dpn1 (ELPIS-BIOTECH. Inc.), затем трансформировали им DH5α Е. coli посредством электропорации, а затем трансформированные Е. coli отбирали на чашке со средой LB с ампициллином. Клонированная плазмида была обозначена pKHSC0004.
1.3. Получение pBAD30-HpLpxE-frt-kan-frt
1.3.1. Получение pBAD30-HpLpxE
Для делеции последовательности сайта распознавания фермента рестрикции HindIII в гене hp0021, кодирующем LpxE (HpLpxE) Helicobacter pylori, нуклеотидная последовательность, кодирующая 17-ю аминокислоту с N-конца серии (17Ser), была изменена путем мутации с AGC на TCG. Мутированный ген hp0021 синтезировали с помощью технологии Integrated DNA (mBiotech, Республика Корея).
Полинуклеотид (SEQ ID NO: 13), кодирующий аминокислотные последовательности HpLpxE (SEQ ID NO: 12), амплифицировали с использованием hp0021 в качестве матрицы и пары праймеров:
прямой праймер для амплификации мутанта HpLpxE:
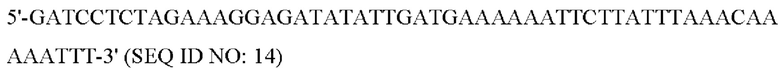
обратный праймер для амплификации мутанта LpxE Helicobacter pylori:

PCR проводили с использованием ДНК-полимеразы «горячего старта» KOD (Novagen) в термоциклере Т3000 (Biometra).
После PCR амплифицированные продукты очищали, как описано выше в разделе 1.1. Очищенный продукт клонировали в плазмиде pBAD30 (см. Guzman, L. М., Belin, D., Carson, М.J., Beckwith, J., J Bacteriol (1995). 177(14), p. 4121-4130). E.coli трансформировали этой клонированной плазмидой, а затем трансформированные Е. coli отбирали, как описано выше в разделе 1.1. Клонированная плазмида была обозначена pBAD30-HpLpxE.
1.3.2. Получение pBAD30-HpLpxE-frt-kan-frt
Полинуклеотид frt-kan-frt, имеющий последовательности сайта распознавания фермента рестрикции HindIII на обоих концах, амплифицировали с использованием плазмиды pKD4 (Kirill A. Datsenko, and Barry L. Wanner PNAS (2000), vol. 97, p. 6640-6645) в качестве матрицы и пары праймеров:
прямой праймер для амплификации frt-kan-frt:

обратный праймер для амплификации frt-kan-frt:

PCR проводили с использованием ДНК-полимеразы pfu (ELPIS-BIOTECH. Inc.) в термоциклере Т3000 (Biometra).
После PCR амплифицированный продукт очищали, как описано выше в разделе 1.1. Затем очищенный продукт клонировали в плазмиде pBAD30, как описано выше в разделе 1.3.1. Е. coli трансформировали этой клонированной плазмидой, а затем трансформированные Е. coli отбирали, как описано выше в разделе 1.1. Клонированная плазмида была обозначена pBAD30-HpLpxE-frt-kan-frt.
Пример 2. Получение штаммов Е. coli
2.1. Получение штамма Е. coli KHSC0044 (pWSK29-EcLpxLEcLpxM. ΔlpxT, ΔpagP, bacA::HpLpxE, kdtA::kan, W3110)
2.1.1 Получение E. coli, из генома которой удален ген lpxT
Был получен штамм Е. coli W3110 lpxT::kan, в котором была встроена экспрессионная кассета канамицина в ген lpxT (SEQ ID NO: 19) генома E. coli, где ген lpxT кодирует полипептид LpxT (SEQ ID NO: 18).
Затем плазмидой pCP20 (Kirill A. Datsenko, and Barry L. Wanner PNAS (2000), vol. 97, p. 6640-6645) трансформировали штамм E. coli W3110 lpxT::kan, а затем трансформированные E.coli отбирали на чашке со средой LB. Отобранные Е. coli высевали на чашку со средой LB и проводили отбор при температуре 42°С, таким образом получая штамм Е. coli ΔlpxT W3110, из которого были удалены lpxT и экспрессионная кассета канамицина.
2.1.2 Получение Е. coli, из генома которой удалены ген pagP и ген 1pxT
Фаг Р1 был получен из штамма Е. coli JW0617 (pagP::kan) (нокаут-библиотека Keio Е. coli), в котором экспрессионная кассета канамицина была встроена в ген pagP генома Е. coli. Фагом Р1 трансдуцировали штамм Е. coli ΔlpxT W3110, полученный, как описано выше в разделе 2.1.1, а затем трансдуцированные Е. coli отбирали на чашке со средой LB с канамицином, таким образом получая штамм Е. coli ΔlpxT, pagP::kan, W3110, в котором pagP::kan был встроен на место гена pagP.
Плазмидой рСР20, как описано выше в разделе 2.1.1, трансформировали штамм Е. coli ΔlpxT, pagP::kan W3110, а затем трансформированные Е. coli отбирали на чашке со средой LB с ампициллином. Отобранные Е. coli высевали на чашку со средой LB, а затем отобранные Е. coli отбирали при температуре приблизительно 42°С, таким образом получая штамм Е. coli ΔlpxT, ΔpagP W3110, из которого был удален ген pagP и экспрессионная кассета канамицина.
2.1.3 Получение Е. coli, из генома которой удален ген lpxT и ген pagP, а ген bacA генома был заменен полинуклеотидом HpLpxE-frt-kan-frt
Чтобы получить полинуклеотид bacA::HpLpxE-frt-kan-frt, способный к гомологичной рекомбинации с геном bacA, проводили PCR с плазмидой pBAD30-HpLpxE- frt-kan-frt, полученной, как описано выше в разделе 1.3.2, в качестве матрицы и пары праймеров:
прямой праймер для амплификации bacA::HpLpxE-frt-kan-frt:

обратный праймер для амплификации bacA::HpLpxE-frt-kan-frt:
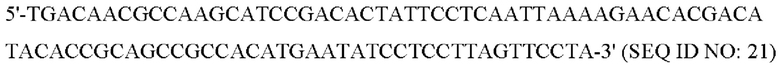
PCR проводили с использованием ДНК-полимеразы pfu (ELPIS-BIOTECH. Inc.) в термоциклере Т3000 (Biometra).
После PCR амплифицированный продукт очищали, как описано выше в разделе 1.1. Затем очищенным продуктом трансформировали DY330 Е. coli (Yu, D., et. al., PNAS. (2000). 97(11), p5978-5983) посредством электропорации, таким образом получая штамм Е. coli bacA:: Hp LpxE-frt-kan-frt DY330 посредством гомологичной рекомбинации с соседними последовательностями, расположенными справа и слева от гена bacA в геноме DY330 (см. ФИГ. 3).
Фаг Р1 был получен из штамма Е. coli DY330 bacA::HpLpxE-frt-kan-frt. Фагом P1 трансдуцировали штамм Е. coli W3110 ΔlpxT, ΔpagP, полученный, как описано выше в разделе 2.1.2, а затем трансдуцированные Е. coli отбирали на среде LB с канамицином. Отобранный штамм Е. coli был обозначен W3110 ΔlpxT, ΔpagP, bacA::HpLpxE-frt-kan-frt (см. ФИГ. 3).
2.1.4 Получение Е. coli, из генома которой удален ген lpxT и ген pagP, а ген bacA генома был заменен геном HpLpxE
Плазмидой рСР20, как описано выше в разделе 2.1.1, трансформировали штамм Е. coli W3110 ΔlpxT, ΔpagP, bacA::HpLpxE-frt-kan-frt, полученный, как описано выше в разделе 2.1.3, а затем трансформированные Е. coli отбирали на чашке со средой LB с ампициллином. Отобранные Е. coli высевали на чашку со средой LB и отбирали при температуре приблизительно 42°С, таким образом получая штамм Е. coli W3110 ΔlpxT, ΔpagP, bacA::HpLpxE, из которого был удален ген bacA и экспрессионная кассета канамицина и в который был введен ген HpLpxE (см. ФИГ. 3 и первый ряд ФИГ. 4, слева).
2.1.5 Получение штамма Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, bacA::HpLpxE, W3110
Плазмидой pWSK29-EcLpxLEcLpxM, полученной, как описано выше в разделе 1.1, трансформировали штамм Е. coli ΔlpxT, ΔpagP, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.1.4, методом электропорации. Затем трансформированные Е. coli отбирали с получением таким образом Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, bacA:: HpLpxE, W3110 (см. первый ряд ФИГ. 4, в центре).
2.1.6 Получение штамма KHSC0044 E. coli
Фаг P1 был получен из штамма Е. coli HSC1/pEcKdt, имеющего экспрессионную кассету канамицина, встроенную в ген kdtA (SEQ ID NO: 23), кодирующий полипептид KdtA (SEQ ID NO: 22) в хромосоме E. coli, и включающий в себя плазмиду pEcKdtA (Chung, Н. S., and Raetz, С.R., Biochemistry (2010), vol. 49(19), p. 4126-4137). Фагом P1 трансдуцировали штамм E.coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.1.5 (см. ФИГ. 3), а затем этот штамм Е. coli отбирали. Отобранный штамм Е. coli был обозначен Е. coli KHSC0044 (pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, bacA::HpLpxE, kdtA::kan, W3110) (см. первый ряд ФИГ. 4, справа).
2.2. Получение штамма Е. coli KHSC0031 (pWSK29-EcLpxLEcLpxM. ΔlpxT, ΔpagP, bacA::HpLpxE, kdtA::kan, W3110)
2.2.1 Получение E. coli, из генома которой удален ген lpxT, ген pagP и ген ybjG, а ген bacA генома был заменен геном HpLpxE
Фаг Р1 был получен из штамма Е. coli JW5112 (ybjG::kan) (нокаут-библиотека Keio Е. coli), в котором экспрессионная кассета канамицина была встроена в ген ybjG генома Е. coli. Фагом Р1 трансдуцировали штамм Е. coli ΔlpxT, ΔpagP, bacA:: HpLpxE, W3110, полученный, как описано выше в разделе 2.1.4, а затем этот штамм Е. coli отбирали на чашке со средой LB с канамицином, таким образом получая штамм Е. coli ΔlpxT, ΔpagP, bacA::HpLpxE, ybjG::kan, W3110, в котором кассета ybjG::kan была встроена на место гена ybjG.
Плазмидой рСР20, как описано выше в разделе 2.1.1, трансформировали штамм Е. coli ΔlpxT, ΔpagP, bacA::HpLpxE, ybjG::kan, W3110, а затем трансформированные E. coli отбирали на чашке со средой LB с ампициллином. Отобранные Е. coli высевали на чашку со средой LB с ампициллином и отбирали при температуре приблизительно 42°С, таким образом получая штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, W3110, из которого был удален ген ybjG и экспрессионная кассета канамицина (см. второй ряд ФИГ. 4, слева).
2.2.2 Получение штамма E. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, kdtA::kan, W3110
Плазмидой pWSK29-EcLpxLEcLpxM, полученной, как описано выше в разделе 1.1, трансформировали штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.1.1, методом электропорации. Трансформированные Е. coli отбирали на чашке со средой LB с ампициллином, таким образом получая штамм Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, W3110 (см. второй ряд ФИГ. 4, в центре).
2.2.3 Получение штамма Е. coli KHSC0031
Фаг Р1 был получен из Е. coli HSC1/pEcKdt, как описано выше в разделе 2.1.6. Фагом Р1 трансдуцировали штамм Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.2.2, а затем трансдуцированные Е. coli отбирали на среде LB с канамицином/ампициллином. Отобранный штамм Е. coli был обозначен KHSC0031 (pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, kdtA::kan, W3110) (см. второй ряд ФИГ. 4, справа).
2.3. Получение штамма Е. coli KHSC0045 (pWSK29-EcLpxLEcLpxM. ΔlvxT. ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, kdtA::kan, W3110)
2.3.1 Получение E.coli, из генома которой удалены гены lpxT, pagP, ybjG и pgpB, а ген bacA генома был заменен геном HpLpxE
Фаг Р1 был получен из штамма Е. coli JW1270 (pgpB::kan) (нокаут-библиотека Keio E. coli), в котором экспрессионная кассета канамицина была встроена в ген pgpB генома Е. coli. Фагом Р1 трансдуцировали штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, W3110, полученный, как описано в разделе 2.2.1, а затем трансдуцированную Е. coli отбирали на чашке со средой LB с канамицином, таким образом получая штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, pgpB::kan W3110, в котором кассета pgpB::kan была встроена на место гена pgpB.
Плазмидой рСР20, как описано выше в разделе 2.1.1, трансформировали штамм E. coli ΔlpxT, ΔpagP, ΔybjG, bacA::HpLpxE, pgpB::kan, W3110, а затем трансформированную E. coli отбирали на чашке со средой LB с ампициллином. Отобранную Е. coli высевали на чашку со средой LB, а затем отбирали при температуре приблизительно 42°С, в результате чего получили штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110, в котором были удалены pgpB и экспрессионная кассета канамицина (см. третий ряд ФИГ. 4, слева).
2.3.2 Получение штамма Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacAr::HpLyxE, kdtA::kan, W3110
Плазмидой pWSK29-EcLpxLEcLpxM, полученной, как описано выше в разделе 1.1, трансформировали штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.3.1, методом электропорации. Трансформированные Е. coli отбирали на чашке со средой LB с ампициллином, таким образом получая штамм Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110 (см. третий ряд ФИГ. 4, в центре).
2.3.3 Получение штамма KHSC0045 Е. coli
Фаг Р1 был получен из Е. coli HSC1/pEcKdt, как описано выше в разделе 2.1.6 (Chung, Н. S., and Raetz, С.R., Biochemistry (2010), vol.49(19), р.4126-4137). Фагом P1 трансдуцировали штамм Е. coli pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110, полученный, как описано в разделе 2.3.2, а затем эту Е. coli отбирали на чашке со средой LB с канамицином/ампициллином. Отобранный штамм Е. coli был обозначен KHSC0045 (pWSK29-EcLpxLEcLpxM, ΔlpxT, ΔpagP, Δ ybjG, ΔpgpB, bacA::HpLpxE, kdtA::kan, W3110) (см. третий ряд ФИГ. 4, справа). Штамм KHSC0045 был депонирован в Корейском научно-исследовательском институте биологических наук и биотехнологии, который является международным депозитарным органом, в соответствии с Будапештским договором 06 июля 2017 г. (номер доступа: КСТС 13296 ВР).
2.4. Получение штамма Е. coli KHSC0055 (pKHSC0004. ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, kdtA::kan, W3110)
2.4.1 Получение штамма E.coli pKHSC0004, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, kdtA::kan, W3110
Плазмидой pKHSC0004, полученной, как описано выше в разделе 1.2, трансформировали штамм Е. coli ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110, полученный, как описано выше в разделе 2.3.1. Трансформированную Е. coli отбирали на чашке со средой LB с ампициллином, таким образом получая штамм Е. coli pKHSC0004, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HphpxE, W3110 (см. четвертый ряд ФИГ. 4, слева).
2.4.2 Получение штамма Е. coli KHSC0055
Штамм KHSC0055 Е. coli, включающий в себя плазмиду pEcKdtA и кассету канамицина (frt-kan-frt), встроенную в ген kdtA (SEQ ID NO: 23), кодирующий полипептид KdtA (SEQ ID NO: 22) в хромосоме E. coli, был получен, как описано ниже.
Полинуклеотид kdtA::frt-kan-frt, способный к гомологичной рекомбинации с геном kdtA, амплифицировали с использованием плазмиды pKD4 (Kirill A. Datsenko, and Barry L. Wanner PNAS (2000), vol. 97, p. 6640-6645) в качестве матрицы и пары праймеров:
прямой праймер для амплификации kdtA::frt-kan-frt: 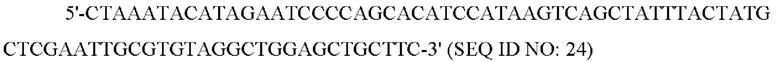
обратный праймер для амплификации kdtA::frt-kan-frt:

PCR проводили с использованием ДНК-полимеразы pfu (ELPIS-BIOTECH. Inc.) в термоциклере Т3000 (Biometra).
После PCR амплифицированный продукт очищали, как описано выше в разделе 1.1. Затем очищенным продуктом трансформировали методом электропорации штамм Е. coli DY330, включающий в себя плазмиду pEcKdtA (Chung, Н.S., and Raetz, С.R., Biochemistry (2010), vol. 49(19), p. 4126-4137), таким образом получая штамм Е. coli pEcKdtA, kdtA::frt-kan-frt, DY330 посредством гомологичной рекомбинации между соседними последовательностями, расположенными справа и слева от гена kdtA в геноме DY330 (см. ФИГ. 3).
Фаг Р1 был получен из штамма Е. coli pEcKdtA, kdtA::frt-kan-frt, DY330. Фагом P1 трансдуцировали штамм Е. coli pKHSC0004, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, W3110, полученный, как описано в разделе 2.4.1, а затем трансдуцированную Е. coli отбирали на чашке со средой LB с канамицином. Отобранный штамм Е. coli был обозначен KHSC0055 (pKHSC0004, ΔlpxT, ΔpagP, ΔybjG, ΔpgpB, bacA::HpLpxE, kdtA::frt- kan-frt, W3110) (см. четвертый ряд ФИГ. 4, справа). Штамм KHSC0055 был депонирован в Корейском научно-исследовательском институте биологических наук и биотехнологии, который является международным депозитарным органом, в соответствии с Будапештским договором 06 июля 2017 г. (номер доступа: КСТС 13297 ВР).
Пример 3. Определение липидных композиций штаммов Е. coli KHSC0044, KHSC31, KHSC0045 и KHSC0055
3.1 Культуры KHSC0044. KHSC0031 и KHSC0045
Штаммы Е. coli KHSC0044, KHSC0031 и KHSC0045 были получены, как описано выше в разделе 2.1.6, разделе 2.2.3 и разделе 2.3.3 соответственно.
Исходную культуру каждого из штаммов Е. coli высевали на чашку со средой LB, содержащей 50 мкг/мл ампициллина (EMD millipore) и 1 мМ изопропил-β-D-1-тиогалактопиранозида (IPTG) (UBP Bio), а затем культивировали. Штаммы KHSC0044 (8 колоний), KHSC0031 (7 колоний) и KHSC0045 (7 колоний) были отобраны, а затем каждый из них засевали в 3 мл жидкой среды LB, содержащей 50 мкг/мл ампициллина (EMD millipore) и 1 мМ IPTG и затем культивировали в течение ночи при приблизительно 30°С. Каждый полученный в результате культуральный раствор засевали в 200 мл свежей среды LB, содержащей 50 мкг/мл ампициллина и 1 мМ IPTG, разводили до OD600 (оптической плотности при 600 нм) от приблизительно 0,06 до приблизительно 0,1 и затем культивировали в течение ночи при приблизительно 30°С.
3.2 Культура KHSC0055
Штамм KHSC0055 Е. coli был получен, как описано выше в разделе 2.4.2.
Исходную культуру KHSC0055 засевали в 3 мл жидкой среды LB с добавлением/без добавления 50 мкг/мл ампициллина и затем инкубировали в течение ночи при приблизительно 30°С. Полученный в результате раствор культуры засевали в 200 мл свежей жидкой среды LB с добавлением/без добавления 50 мкг/мл ампициллина и затем инкубировали в течение ночи при приблизительно 30°С.
3.3 Экстракция липидов из штаммов Е. coli KHSC0044, KHSC0031, KHSC0045 и KHSC0055
Каждую из полученных культуральных сред Е. coli, как описано выше в разделе 3.1 и в разделе 3.2, центрифугировали при комнатной температуре при приблизительно 4000 g в течение приблизительно 20 минут с получением штаммов Е. coli. Каждый из полученных штаммов Е. coli промывали 30 мл PBS (забуференного фосфатом физиологического раствора) и ресуспендировали в 8 мл PBS.
К ресуспендированной Е. coli добавляли 10 мл хлороформа и 20 мл метанола и затем инкубировали при комнатной температуре в течение приблизительно 1 часа при периодическом встряхивании. Затем проинкубированную смесь центрифугировали при комнатной температуре со скоростью 2500 g в течение 30 минут и собирали супернатант. К собранному супернатанту добавляли 10 мл хлороформа и 10 мл воды, тщательно перемешивали, а затем центрифугировали при комнатной температуре при 2500 g в течение приблизительно 20 минут. После отделения органического растворителя от центрифугированной смеси слой органического растворителя дважды экстрагировали, добавляя предварительно уравновешенный слой органического растворителя к верхнему водному слою. Слой органического растворителя объединяли, а затем высушивали в роторном испарителе с получением липидов. Полученные липиды растворяли в 5 мл смеси 4:1 (об./об.) хлороформа и метанола, а затем подвергали ультразвуковой обработке на водяной бане. Обработанные ультразвуком липиды переносили в новую пробирку, и полученные липиды высушивали при комнатной температуре в атмосфере газа азота, а затем хранили при приблизительно -80°С.
3.4. Анализ липидов методом тонкослойной хроматографии (TLC)
Мембранные липиды каждого штамма Е. coli, полученные, как описано выше в разделе 3.3, анализировали, используя в качестве групп положительного контроля MPLA Synthetic (InvovoGen, код по каталогу: tlrl-mpls, синтетический липид 1-дефосфо-гекса-ацилированный липид А (дорожка 6 ФИГ. 5 и дорожка 1 ФИГ. 6) и MPLA-SM (InvovoGen, код по каталогу tlrl-mpla, 1-дефосфо-липид А, выделенный и очищенный из липополисахаридов (LPS) Salmonella minnesota R595 после кислотно-основного гидролиза) (дорожка 7 ФИГ. 5).
Для проведения тонкослойной хроматографии (TLC), получали липид, как описано выше в разделе 3.2, из 200 мл раствора каждой культуры Е. coli, и одну треть полученных суммарных липидов растворяли в 200 мкл 4:1 (об./об.) смеси хлороформа и метанола. Затем приблизительно от 5 мкл до 15 мкл этой смеси наносили на пластину 10×10 см для высокоэффективной TLC (HPTLC) (EMD Chemicals) и проявляли в смеси растворителей хлороформа, метанола, воды и гидроксида аммония (28% (об/об) аммиака) в соотношении 40:25:4:2 (об./об.). Затем проявленную пластину высушивали, визуализировали путем опрыскивания 10% (об./об.) раствором серной кислоты в этаноле с последующим нагреванием на электрической плитке при 300°С. Результаты TLC липидов представлены на ФИГ. 5, 6 и 7.
На ФИГ. 5 показано изображение результатов TLC липидов, экстрагированных из Е. coli, в которых из генома Е. coli был удален ген bacA и заменен геном фосфатазы. На ФИГ. 5, дорожка 1, представлен KHSC0044, продуцирующий 1-дефосфо-липид А (две из 8 колоний, культивируемых в одной и той же исходной культуре клеток), на дорожке 2 представлен KHSC0044, не продуцирующий 1-дефосфо-липид А (шесть из 8 колоний, культивируемых в одной и той же исходной культуре клеток), на дорожке 3 представлен KHSC0031, продуцирующий 1-дефосфо-липид А (шесть из 7 колоний, культивируемых в одной и той же исходной культуре клеток), на дорожке 4 представлен KHSC0031, не продуцирующий 1-дефосфо-липид А (одна из 7 колоний, культивируемых в одной и той же исходной культуре клеток), на дорожке 5 представлен KHSC0045, стабильно продуцирующий 1-дефосфо-липид А (все 7 колоний, культивируемых в одной и той же исходной культуре клеток), на дорожке 6 представлен синтетический 1-дефосфо-гекса-ацилированный липид A (InvovoGen, код по каталогу: tlrl-mpls) и на дорожке 7 представлен 1-дефосфо-липид А, выделенный и очищенный из Salmonella minnesota R595 после кислотно-основного гидролиза (InvovoGen, код по каталогу: tlrlmpla).
На ФИГ. 6 представлено изображение, на котором показаны результаты TLC липидов из штаммов Е. coli KHSC0045 и KHSC0055. На ФИГ. 6, дорожка 1, представлен синтетический 1-дефосфо-гекса-ацилированный липид A (InvovoGen, код по каталогу: tlrlmpls), на дорожке 2 представлен KHSC0045, культивированный с индуктором гиперэкспрессии для индуцирования гиперэкспрессии фермента липидизации, а на дорожке 3 представлен KHSC0055, культивированный без использования индуктора гиперэкспрессии.
На ФИГ. 7 представлено изображение, показывающее результаты TLC липидов штаммов Е. coli KHSC0045 и KHSC0055, где на дорожке 1 представлен штамм KHSC0055, культивированный с антибиотиками, а на дорожке 3 представлен штамм KHSC0055, культивированный без использования антибиотиков.
Со ссылкой на ФИГ. 5 было обнаружено, что даже хотя каждая из исходных культур клеток KHSC0044 и KHSC0031 была получена из одной колонии, 8 отобранных колоний KHSC0044 включали колонии, в которых был выявлен 1-дефосфо-липид А (дорожка 1, две из 8 колоний), и колонии, в которых 1-дефосфо-липид А не был выявлен (дорожка 2, шесть из 8 колоний), и 7 отобранных колоний KHSC0031 включали колонии, в которых был выявлен 1-дефосфо-липид А (дорожка 3, шесть из 7 колоний), и колонию, в которой 1-дефосфо-липид А не был выявлен (дорожка 4, одна из 7 колоний). Однако в KHSC0045 1-дефосфо-липид А был выявлен во всех 7 отобранных колониях (дорожка 5).
Эти результаты показывают, что при встраивании фосфатазы в геном Е. coli штамм Е. coli, продуцирующий 1-дефосфо-липид А, может инактивировать активность встроенной фосфатазы, таким образом легко образуя естественный мутант, неспособный продуцировать 1-дефосфо-липид А.
Таким образом, было обнаружено, что для продуцирования 1-дефосфо-липида А путем встраивания фосфатазы в геном Е. coli необходимо удалить ген фосфатазы ундекапренилпирофосфата (Und-PP фосфатазы) (bacA, pgpB или ybjG) или ген фосфатазы фосфатидилглицерофосфата (PGP фосфатазы) (pgpB, pgpA или pgpC), присутствующие в геноме Е. coli, или комбинацию этих генов (дорожка 1, дорожка 3 или дорожка 5 ФИГ. 5), чтобы стабильно продуцировать 1-дефосфо-липид А в E. coli и получить живые Е. coli с пониженной вероятностью естественной мутации.
Для Е. coli KHSC0045, чтобы эффективно продуцировать 1-дефосфо-гексаацилированный липид А, необходима гиперэкспрессия фермента липидизации во время инкубации штамма. В результате сравнения композиции мембранных липидов с использованием в качестве положительных контрольных групп синтетического 1-дефосфо-гексаацилированного липида А (дорожка 1 ФИГ. 6) и KHSC0045, инкубированного с индуцированием гиперэкспрессии фермента липидизации индуктором гиперэкспрессии, было обнаружено, что KHSC0055 Е. coli, трансформированный в pKHSC0004, представляет собой живой штамм Е. coli, эффективно продуцирующий 1-дефосфо-гексаацилированный липид А (дорожка 3 ФИГ. 6) без использования индуктора гиперэкспрессии (дорожка 3 ФИГ. 6).
Для KHSC0055 Е. coli, хотя плазмида pKHSC0004 стабильно сохранялась в KHSC0055 Е. coli, KHSC0055 Е. coli эффективно продуцировал 1-дефосфо-гекса-ацилированный липид А с антибиотиками (дорожка 1 ФИГ. 7) или без использования антибиотиков (дорожка 2 ФИГ. 7).
Как описано выше, в соответствии с одним или более воплощений изобретения, бактерия, конститутивно продуцирующая MLA, и способ получения MLA с использованием этой бактерии позволяют легко получать MLA и его производное без кислотного гидролиза, обладают пониженной вероятностью естественной мутации и позволяют получить повышенные выходы MLA и его производного за счет конститутивной экспрессии MLA и его производного.
Название депозитарного учреждения: Корейская коллекция типовых культур
Номер по каталогу: КСТС13296 ВР
Дата депозита: 6 июля 2017 г.
Название депозитарного учреждения: Корейская коллекция типовых культур
Номер по каталогу: КСТС13297 ВР
Дата депозита: 6 июля 2017 г.
Следует понимать, что описанные в настоящем документе воплощения изобретения следует рассматривать только в описательном смысле, а не в целях ограничения. Описания элементов или аспектов в пределах каждого воплощения, как правило, следует рассматривать как приемлемые для других подобных элементов или аспектов в других воплощениях изобретения.
Хотя одно или более воплощений изобретения описано со ссылкой на графические материалы, обычным специалистам в данной области техники будет понятно, что в них могут быть произведены различные изменения формы и деталей, без отклонения от сущности и объема описания, которые определены в приведенной ниже формуле изобретения.
--->
Перечень последовательностей
<110> Корейский институт научных исследований и технологий
<120> Бактерии, конститутивно продуцирующие монофосфориллипид A и
способ получения монофосфориллипида A с их
использованием
<130> PX055323PCT
<160> 25
<170> KopatentIn 2.0
<210> 1
<211> 306
<212> PRT
<213> Искусственная последовательность
<220>
<223> Полипептид LpxL Escherichia coli
<400> 1
Met Thr Asn Leu Pro Lys Phe Ser Thr Ala Leu Leu His Pro Arg Tyr
1 5 10 15
Trp Leu Thr Trp Leu Gly Ile Gly Val Leu Trp Leu Val Val Gln Leu
20 25 30
Pro Tyr Pro Val Ile Tyr Arg Leu Gly Cys Gly Leu Gly Lys Leu Ala
35 40 45
Leu Arg Phe Met Lys Arg Arg Ala Lys Ile Val His Arg Asn Leu Glu
50 55 60
Leu Cys Phe Pro Glu Met Ser Glu Gln Glu Arg Arg Lys Met Val Val
65 70 75 80
Lys Asn Phe Glu Ser Val Gly Met Gly Leu Met Glu Thr Gly Met Ala
85 90 95
Trp Phe Trp Pro Asp Arg Arg Ile Ala Arg Trp Thr Glu Val Ile Gly
100 105 110
Met Glu His Ile Arg Asp Val Gln Ala Gln Lys Arg Gly Ile Leu Leu
115 120 125
Val Gly Ile His Phe Leu Thr Leu Glu Leu Gly Ala Arg Gln Phe Gly
130 135 140
Met Gln Glu Pro Gly Ile Gly Val Tyr Arg Pro Asn Asp Asn Pro Leu
145 150 155 160
Ile Asp Trp Leu Gln Thr Trp Gly Arg Leu Arg Ser Asn Lys Ser Met
165 170 175
Leu Asp Arg Lys Asp Leu Lys Gly Met Ile Lys Ala Leu Lys Lys Gly
180 185 190
Glu Val Val Trp Tyr Ala Pro Asp His Asp Tyr Gly Pro Arg Ser Ser
195 200 205
Val Phe Val Pro Leu Phe Ala Val Glu Gln Ala Ala Thr Thr Thr Gly
210 215 220
Thr Trp Met Leu Ala Arg Met Ser Gly Ala Cys Leu Val Pro Phe Val
225 230 235 240
Pro Arg Arg Lys Pro Asp Gly Lys Gly Tyr Gln Leu Ile Met Leu Pro
245 250 255
Pro Glu Cys Ser Pro Pro Leu Asp Asp Ala Glu Thr Thr Ala Ala Trp
260 265 270
Met Asn Lys Val Val Glu Lys Cys Ile Met Met Ala Pro Glu Gln Tyr
275 280 285
Met Trp Leu His Arg Arg Phe Lys Thr Arg Pro Glu Gly Val Pro Ser
290 295 300
Arg Tyr
305
<210> 2
<211> 921
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Полинуклеотид, кодирующий полипептид LpxL Escherichia coli
<400> 2
atgacgaatc tacccaagtt ctccaccgca ctgcttcatc cgcgttattg gttaacctgg 60
ttgggtattg gcgtactttg gttagtcgtg caattgccct acccggttat ctaccgcctc 120
ggttgtggat taggaaaact ggcgttacgt tttatgaaac gacgcgcaaa aattgtgcat 180
cgcaacctgg aactgtgctt cccggaaatg agcgaacaag aacgccgtaa aatggtggtg 240
aagaatttcg aatccgttgg catgggcctg atggaaaccg gcatggcgtg gttctggccg 300
gaccgccgaa tcgcccgctg gacggaagtg atcggcatgg aacacattcg tgacgtgcag 360
gcgcaaaaac gcggcatcct gttagttggc atccattttc tgacactgga gctgggtgcg 420
cggcagtttg gtatgcagga accgggtatt ggcgtttatc gcccgaacga taatccactg 480
attgactggc tacaaacctg gggccgtttg cgctcaaata aatcgatgct cgaccgcaaa 540
gatttaaaag gcatgattaa agccctgaaa aaaggcgaag tggtctggta cgcaccggat 600
catgattacg gcccgcgctc aagcgttttc gtcccgttgt ttgccgttga gcaggctgcg 660
accacgaccg gaacctggat gctggcacgg atgtccggcg catgtctggt gcccttcgtt 720
ccacgccgta agccagatgg caaagggtat caattgatta tgctgccgcc agagtgttct 780
ccgccactgg atgatgccga aactaccgcc gcgtggatga acaaagtggt cgaaaaatgc 840
atcatgatgg caccagagca gtatatgtgg ttacaccgtc gctttaaaac acgcccggaa 900
ggcgttcctt cacgctatta a 921
<210> 3
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> прямой праймер P1 LpxL
<400> 3
cgcagtctag aaaggagata tattgatgac gaatctaccc aagttctc 48
<210> 4
<211> 67
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер P2 LpxL
<400> 4
cgctattatt ttttttcgtt tccattggta tatctccttc ttattaatag cgtgaaggaa 60
cgccttc 67
<210> 5
<211> 323
<212> PRT
<213> Искусственная последовательность
<220>
<223> полипептид LpxM Escherichia coli
<400> 5
Met Glu Thr Lys Lys Asn Asn Ser Glu Tyr Ile Pro Glu Phe Asp Lys
1 5 10 15
Ser Phe Arg His Pro Arg Tyr Trp Gly Ala Trp Leu Gly Val Ala Ala
20 25 30
Met Ala Gly Ile Ala Leu Thr Pro Pro Lys Phe Arg Asp Pro Ile Leu
35 40 45
Ala Arg Leu Gly Arg Phe Ala Gly Arg Leu Gly Lys Ser Ser Arg Arg
50 55 60
Arg Ala Leu Ile Asn Leu Ser Leu Cys Phe Pro Glu Arg Ser Glu Ala
65 70 75 80
Glu Arg Glu Ala Ile Val Asp Glu Met Phe Ala Thr Ala Pro Gln Ala
85 90 95
Met Ala Met Met Ala Glu Leu Ala Ile Arg Gly Pro Glu Lys Ile Gln
100 105 110
Pro Arg Val Asp Trp Gln Gly Leu Glu Ile Ile Glu Glu Met Arg Arg
115 120 125
Asn Asn Glu Lys Val Ile Phe Leu Val Pro His Gly Trp Ala Val Asp
130 135 140
Ile Pro Ala Met Leu Met Ala Ser Gln Gly Gln Lys Met Ala Ala Met
145 150 155 160
Phe His Asn Gln Gly Asn Pro Val Phe Asp Tyr Val Trp Asn Thr Val
165 170 175
Arg Arg Arg Phe Gly Gly Arg Leu His Ala Arg Asn Asp Gly Ile Lys
180 185 190
Pro Phe Ile Gln Ser Val Arg Gln Gly Tyr Trp Gly Tyr Tyr Leu Pro
195 200 205
Asp Gln Asp His Gly Pro Glu His Ser Glu Phe Val Asp Phe Phe Ala
210 215 220
Thr Tyr Lys Ala Thr Leu Pro Ala Ile Gly Arg Leu Met Lys Val Cys
225 230 235 240
Arg Ala Arg Val Val Pro Leu Phe Pro Ile Tyr Asp Gly Lys Thr His
245 250 255
Arg Leu Thr Ile Gln Val Arg Pro Pro Met Asp Asp Leu Leu Glu Ala
260 265 270
Asp Asp His Thr Ile Ala Arg Arg Met Asn Glu Glu Val Glu Ile Phe
275 280 285
Val Gly Pro Arg Pro Glu Gln Tyr Thr Trp Ile Leu Lys Leu Leu Lys
290 295 300
Thr Arg Lys Pro Gly Glu Ile Gln Pro Tyr Lys Arg Lys Asp Leu Tyr
305 310 315 320
Pro Ile Lys
<210> 6
<211> 972
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Полинуклеотид, кодирующий полипептид LpxM Escherichia coli
<400> 6
atggaaacga aaaaaaataa tagcgaatac attcctgagt ttgataaatc ctttcgccac 60
ccgcgctact ggggagcatg gctgggcgta gcagcgatgg cgggtatcgc tttaacgccg 120
ccaaagttcc gtgatcccat tctggcacgg ctgggacgtt ttgccggacg actgggaaaa 180
agctcacgcc gtcgtgcgtt aatcaatctg tcgctctgct ttccagaacg tagtgaagct 240
gaacgcgaag cgattgttga tgagatgttt gccaccgcgc cgcaagcgat ggcaatgatg 300
gctgagttgg caatacgcgg gccggagaaa attcagccgc gcgttgactg gcaagggctg 360
gagatcatcg aagagatgcg gcgtaataac gagaaagtta tctttctggt gccgcacggt 420
tgggccgtcg atattcctgc catgctgatg gcctcgcaag ggcagaaaat ggcagcgatg 480
ttccataatc agggcaaccc ggtttttgat tatgtctgga acacggtgcg tcgtcgcttt 540
ggcggtcgtc tgcatgcgag aaatgacggt attaaaccat tcatccagtc ggtacgtcag 600
gggtactggg gatattattt acccgatcag gatcatggcc cagagcacag cgaatttgtg 660
gatttctttg ccacctataa agcgacgttg cccgcgattg gtcgtttgat gaaagtgtgc 720
cgtgcgcgcg ttgtaccgct gtttccgatt tatgatggca agacgcatcg tctgacgatt 780
caggtgcgcc caccgatgga tgatctgtta gaggcggatg atcatacgat tgcgcggcgg 840
atgaatgaag aagtcgagat ttttgttggt ccgcgaccag aacaatacac ctggatacta 900
aaattgctga aaactcgcaa accgggcgaa atccagccgt ataagcgcaa agatctttat 960
cccatcaaat aa 972
<210> 7
<211> 67
<212> ДНК
<213> Искусственная последовательность
<220>
<223> прямой праймер P3 LpxM
<400> 7
gaaggcgttc cttcacgcta ttaataagaa ggagatatac caatggaaac gaaaaaaaat 60
aatagcg 67
<210> 8
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<223> обратный праймер P3 LpxM
<400> 8
gcagaagctt ttatttgatg ggataaagat ctttgcg 37
<210> 9
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотидная последовательность промотора PL
<400> 9
ttgacataaa taccactggc ggtgatact 29
<210> 10
<211> 77
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер, амплифицирующий промотор PL
<400> 10
ggcagtgagc gcaacgcaga attcttgaca taaataccac tggcggtgat actttcacac 60
aggaaacagc tatgacc 77
<210> 11
<211> 77
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер, амплифицирующий промотор PL
<400> 11
ggtcatagct gtttcctgtg tgaaagtatc accgccagtg gtatttatgt caagaattct 60
gcgttgcgct cactgcc 77
<210> 12
<211> 190
<212> PRT
<213> Искусственная последовательность
<220>
<223> мутант LpxE Helicobacter pylori
<400> 12
Met Lys Lys Phe Leu Phe Lys Gln Lys Phe Cys Glu Ser Leu Pro Lys
1 5 10 15
Ser Phe Ser Lys Thr Leu Leu Ala Leu Ser Leu Gly Leu Ile Leu Leu
20 25 30
Gly Ile Phe Ala Pro Phe Pro Lys Val Pro Lys Gln Pro Ser Val Pro
35 40 45
Leu Met Phe His Phe Thr Glu His Tyr Ala Arg Phe Ile Pro Thr Ile
50 55 60
Leu Ser Val Ala Ile Pro Leu Ile Gln Arg Asp Ala Val Gly Leu Phe
65 70 75 80
Gln Val Ala Asn Ala Ser Ile Ala Thr Thr Leu Leu Thr His Thr Thr
85 90 95
Lys Arg Ala Leu Asn His Val Thr Ile Asn Asp Gln Arg Leu Gly Glu
100 105 110
Arg Pro Tyr Gly Gly Asn Phe Asn Met Pro Ser Gly His Ser Ser Met
115 120 125
Val Gly Leu Ala Val Ala Phe Leu Met Arg Arg Tyr Ser Phe Lys Lys
130 135 140
Tyr Phe Trp Leu Leu Pro Leu Val Pro Leu Thr Met Leu Ala Arg Ile
145 150 155 160
Tyr Leu Asp Met His Thr Ile Gly Ala Val Leu Thr Gly Leu Gly Val
165 170 175
Gly Met Leu Cys Val Ser Leu Phe Thr Ser Pro Lys Lys Pro
180 185 190
<210> 13
<211> 573
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Полинуклеотид, кодирующий мутант LpxE Helicobacter pylori
<400> 13
atgaaaaaat tcttatttaa acaaaaattt tgtgaaagcc tgcccaaatc gttttctaaa 60
actttgttag cgctcagttt gggcttgatt ttattaggca tttttgcgcc tttccctaaa 120
gtccctaaac agcctagcgt gcctttaatg tttcatttca ccgagcatta tgcgcgcttt 180
atccctacga ttttatctgt ggcgattccc ttaatccaaa gagatgcggt agggcttttt 240
caagtcgcta acgcttctat cgctacaacc cttctcacgc acaccaccaa aagagcctta 300
aaccatgtaa caatcaacga tcagcgtttg ggcgagcgcc cttatggagg taatttcaac 360
atgccaagcg ggcattcgtc tatggtgggt ttggcggtgg cgtttttaat gcgccgctat 420
tcttttaaaa aatacttttg gctcttgccc ctagtccctt tgaccatgct cgctcgcatt 480
tatttagaca tgcacaccat tggcgcggtg ctgaccgggc ttggcgttgg aatgttgtgc 540
gtaagccttt ttacaagccc caaaaagcct taa 573
<210> 14
<211> 55
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации мутанта HpLpxE
<400> 14
gatcctctag aaaggagata tattgatgaa aaaattctta tttaaacaaa aattt 55
<210> 15
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации мутанта LpxE Helicobacter pylori
<400> 15
agctacaagc ttttaaggct ttttggggc 29
<210> 16
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации frt-kan-frt
<400> 16
gcagaagctt gtgtaggctg gagctgcttc 30
<210> 17
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации frt-kan-frt
<400> 17
gcagaagctt atgaatatcc tccttagttc ctat 34
<210> 18
<211> 237
<212> PRT
<213> Искусственная последовательность
<220>
<223> Полипептид LpxT Escherichia coli
<400> 18
Met Ile Lys Asn Leu Pro Gln Ile Val Leu Leu Asn Ile Val Gly Leu
1 5 10 15
Ala Leu Phe Leu Ser Trp Tyr Ile Pro Val Asn His Gly Phe Trp Leu
20 25 30
Pro Ile Asp Ala Asp Ile Phe Tyr Phe Phe Asn Gln Lys Leu Val Glu
35 40 45
Ser Lys Ala Phe Leu Trp Leu Val Ala Leu Thr Asn Asn Arg Ala Phe
50 55 60
Asp Gly Cys Ser Leu Leu Ala Met Gly Met Leu Met Leu Ser Phe Trp
65 70 75 80
Leu Lys Glu Asn Ala Pro Gly Arg Arg Arg Ile Val Ile Ile Gly Leu
85 90 95
Val Met Leu Leu Thr Ala Val Val Leu Asn Gln Leu Gly Gln Ala Leu
100 105 110
Ile Pro Val Lys Arg Ala Ser Pro Thr Leu Thr Phe Thr Asp Ile Asn
115 120 125
Arg Val Ser Glu Leu Leu Ser Val Pro Thr Lys Asp Ala Ser Arg Asp
130 135 140
Ser Phe Pro Gly Asp His Gly Met Met Leu Leu Ile Phe Ser Ala Phe
145 150 155 160
Met Trp Arg Tyr Phe Gly Lys Val Ala Gly Leu Ile Ala Leu Ile Ile
165 170 175
Phe Val Val Phe Ala Phe Pro Arg Val Met Ile Gly Ala His Trp Phe
180 185 190
Thr Asp Ile Ile Val Gly Ser Met Thr Val Ile Leu Ile Gly Leu Pro
195 200 205
Trp Val Leu Leu Thr Pro Leu Ser Asp Arg Leu Ile Thr Phe Phe Asp
210 215 220
Lys Ser Leu Pro Gly Lys Asn Lys His Phe Gln Asn Lys
225 230 235
<210> 19
<211> 714
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Полинуклеотид, кодирующий полипептид LpxT Escherichia coli
<400> 19
atgattaaaa atttgccgca aatagtgttg ttgaatattg tcggcctcgc gctgtttctt 60
tcctggtata tccccgttaa tcatggattc tggttgccga ttgatgcgga tattttttat 120
ttctttaatc agaaactggt cgaaagtaag gcctttttgt ggctggttgc attgaccaac 180
aatcgcgcct tcgacggttg ttcactgctg gcgatgggta tgttgatgct gagtttctgg 240
ctgaaagaaa acgcccctgg cagacgacgt atcgtgatta ttggtctggt catgctatta 300
actgcagtgg tattaaacca gctgggtcag gcattaattc ctgtaaaacg ggccagccca 360
acattgactt ttaccgatat taaccgcgtc agcgaactgc tctctgttcc cacgaaagat 420
gcctcacgag atagctttcc cggcgatcac ggcatgatgc tgcttatttt ttcggcattc 480
atgtggcgtt atttcggcaa agttgcaggc cttatcgccc ttattatttt tgtggttttt 540
gcatttccca gagtaatgat tggcgcacac tggtttactg acatcattgt cggttcgatg 600
accgtgatat tgatcggttt gccctgggtg ttgctgacgc cattaagtga tcgattaatc 660
accttttttg acaaatcact accaggaaaa aacaaacatt tccaaaacaa ataa 714
<210> 20
<211> 89
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер, амплифицирующий bacA::HpLpxE-frt-kan-frt
<400> 20
aacctggtca tacgcagtag ttcggacaag cggtacattt taataattta ggggtttatt 60
gatgaaaaaa ttcttattta aacaaaaat 89
<210> 21
<211> 89
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер, амплифицирующий bacA::HpLpxE-frt-kan-frt
<400> 21
tgacaacgcc aagcatccga cactattcct caattaaaag aacacgacat acaccgcagc 60
cgccacatga atatcctcct tagttccta 89
<210> 22
<211> 425
<212> PRT
<213> Искусственная последовательность
<220>
<223> Полипептид KdtA Escherichia coli
<400> 22
Met Leu Glu Leu Leu Tyr Thr Ala Leu Leu Tyr Leu Ile Gln Pro Leu
1 5 10 15
Ile Trp Ile Arg Leu Trp Val Arg Gly Arg Lys Ala Pro Ala Tyr Arg
20 25 30
Lys Arg Trp Gly Glu Arg Tyr Gly Phe Tyr Arg His Pro Leu Lys Pro
35 40 45
Gly Gly Ile Met Leu His Ser Val Ser Val Gly Glu Thr Leu Ala Ala
50 55 60
Ile Pro Leu Val Arg Ala Leu Arg His Arg Tyr Pro Asp Leu Pro Ile
65 70 75 80
Thr Val Thr Thr Met Thr Pro Thr Gly Ser Glu Arg Val Gln Ser Ala
85 90 95
Phe Gly Lys Asp Val Gln His Val Tyr Leu Pro Tyr Asp Leu Pro Asp
100 105 110
Ala Leu Asn Arg Phe Leu Asn Lys Val Asp Pro Lys Leu Val Leu Ile
115 120 125
Met Glu Thr Glu Leu Trp Pro Asn Leu Ile Ala Ala Leu His Lys Arg
130 135 140
Lys Ile Pro Leu Val Ile Ala Asn Ala Arg Leu Ser Ala Arg Ser Ala
145 150 155 160
Ala Gly Tyr Ala Lys Leu Gly Lys Phe Val Arg Arg Leu Leu Arg Arg
165 170 175
Ile Thr Leu Ile Ala Ala Gln Asn Glu Glu Asp Gly Ala Arg Phe Val
180 185 190
Ala Leu Gly Ala Lys Asn Asn Gln Val Thr Val Thr Gly Ser Leu Lys
195 200 205
Phe Asp Ile Ser Val Thr Pro Gln Leu Ala Ala Lys Ala Val Thr Leu
210 215 220
Arg Arg Gln Trp Ala Pro His Arg Pro Val Trp Ile Ala Thr Ser Thr
225 230 235 240
His Glu Gly Glu Glu Ser Val Val Ile Ala Ala His Gln Ala Leu Leu
245 250 255
Gln Gln Phe Pro Asn Leu Leu Leu Ile Leu Val Pro Arg His Pro Glu
260 265 270
Arg Phe Pro Asp Ala Ile Asn Leu Val Arg Gln Ala Gly Leu Ser Tyr
275 280 285
Ile Thr Arg Ser Ser Gly Glu Val Pro Ser Thr Ser Thr Gln Val Val
290 295 300
Val Gly Asp Thr Met Gly Glu Leu Met Leu Leu Tyr Gly Ile Ala Asp
305 310 315 320
Leu Ala Phe Val Gly Gly Ser Leu Val Glu Arg Gly Gly His Asn Pro
325 330 335
Leu Glu Ala Ala Ala His Ala Ile Pro Val Leu Met Gly Pro His Thr
340 345 350
Phe Asn Phe Lys Asp Ile Cys Ala Arg Leu Glu Gln Ala Ser Gly Leu
355 360 365
Ile Thr Val Thr Asp Ala Thr Thr Leu Ala Lys Glu Val Ser Ser Leu
370 375 380
Leu Thr Asp Ala Asp Tyr Arg Ser Phe Tyr Gly Arg His Ala Val Glu
385 390 395 400
Val Leu Tyr Gln Asn Gln Gly Ala Leu Gln Arg Leu Leu Gln Leu Leu
405 410 415
Glu Pro Tyr Leu Pro Pro Lys Thr His
420 425
<210> 23
<211> 1278
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Полинуклеотид, кодирующий полипептид KdtA Escherichia coli
<400> 23
atgctcgaat tgctttacac cgcccttctc taccttattc agccgctgat ctggatacgg 60
ctctgggtgc gcggacgtaa ggctccggcc tatcgaaaac gctggggtga acgttacggt 120
ttttaccgcc atccgctaaa accaggcggc attatgctgc actccgtctc cgtcggtgaa 180
actctggcgg caatcccgtt ggtgcgcgcg ctgcgtcatc gttatcctga tttaccgatt 240
accgtaacaa ccatgacgcc aaccggttcg gagcgcgtac aatcggcttt cgggaaggat 300
gttcagcacg tttatctgcc gtatgatctg cccgatgcac tcaaccgttt cctgaataaa 360
gtcgacccta aactggtgtt gattatggaa accgaactat ggcctaacct gattgcggcg 420
ctacataaac gtaaaattcc gctggtgatc gctaacgcgc gactctctgc ccgctcggcc 480
gcaggttatg ccaaactggg taaattcgtc cgtcgcttgc tgcgtcgtat tacgctgatt 540
gctgcgcaaa atgaagaaga tggtgcacgt tttgtggcgc tgggcgcaaa aaataatcag 600
gtgaccgtta ccggtagcct gaaattcgat atttctgtaa cgccgcagtt ggctgctaaa 660
gccgtgacgc tgcgccgcca gtgggcacca caccgcccgg tatggattgc caccagcact 720
cacgaaggcg aagagagtgt ggtgatcgcc gcacatcagg cattgttaca gcaattcccg 780
aatttattgc tcatcctggt accccgtcat ccggaacgct tcccggatgc gattaacctt 840
gtccgccagg ctggactaag ctatatcaca cgctcttcag gggaagtccc ctccaccagc 900
acgcaggttg tggttggcga tacgatgggc gagttgatgt tactgtatgg cattgccgat 960
ctcgcctttg ttggcggttc actggttgaa cgtggtgggc ataatccgct ggaagctgcc 1020
gcacacgcta ttccggtatt gatggggccg catactttta actttaaaga catttgcgcg 1080
cggctggagc aggcaagcgg gctgattacc gttaccgatg ccactacgct tgcaaaagag 1140
gtttcctctt tactcaccga cgccgattac cgtagtttct atggccgtca tgccgttgaa 1200
gtactgtatc aaaaccaggg cgcgctacag cgtctgcttc aactgctgga accttacctg 1260
ccaccgaaaa cgcattga 1278
<210> 24
<211> 80
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер, амплифицирующий kdtA::frt-kan-frt
<400> 24
gctaaataca tagaatcccc agcacatcca taagtcagct atttactatg ctcgaattgc 60
gtgtaggctg gagctgcttc 80
<210> 25
<211> 86
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер, амплифицирующий kdtA::frt-kan-frt
<400> 25
atcgatatga ccattggtaa tgggatcgaa agtacccgga taaatcgccc gtttttgcat 60
tgaatatcct ccttagttcc tattcc 86
<---

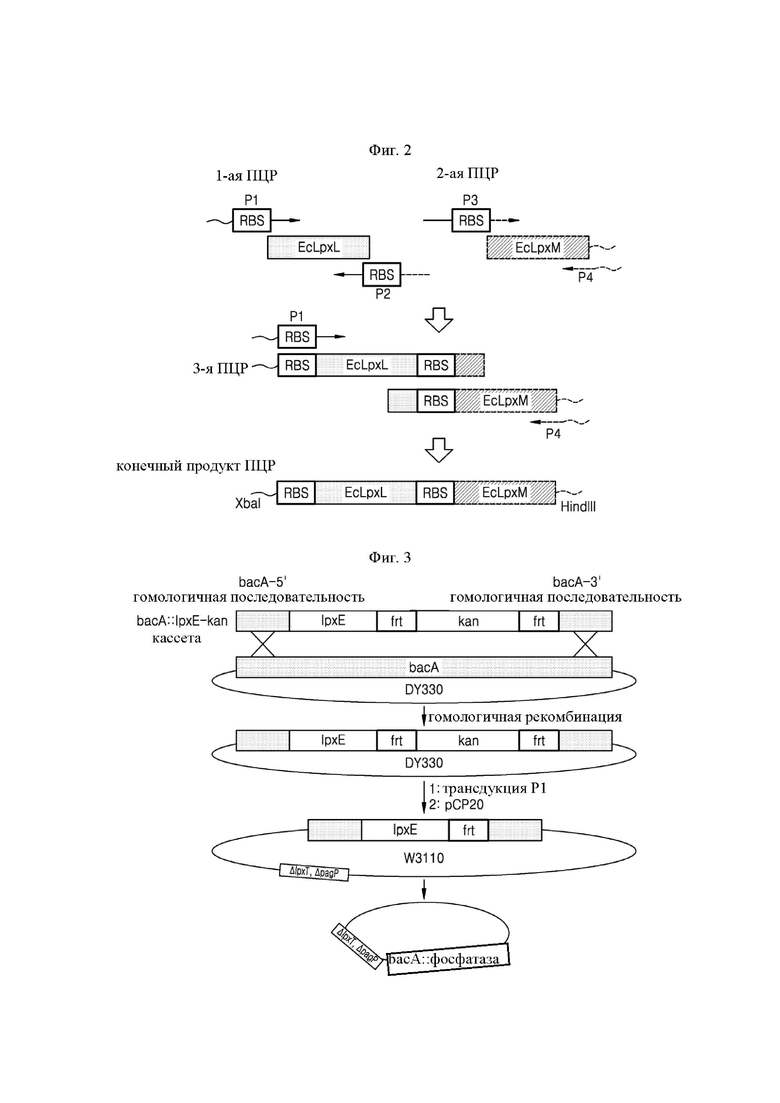
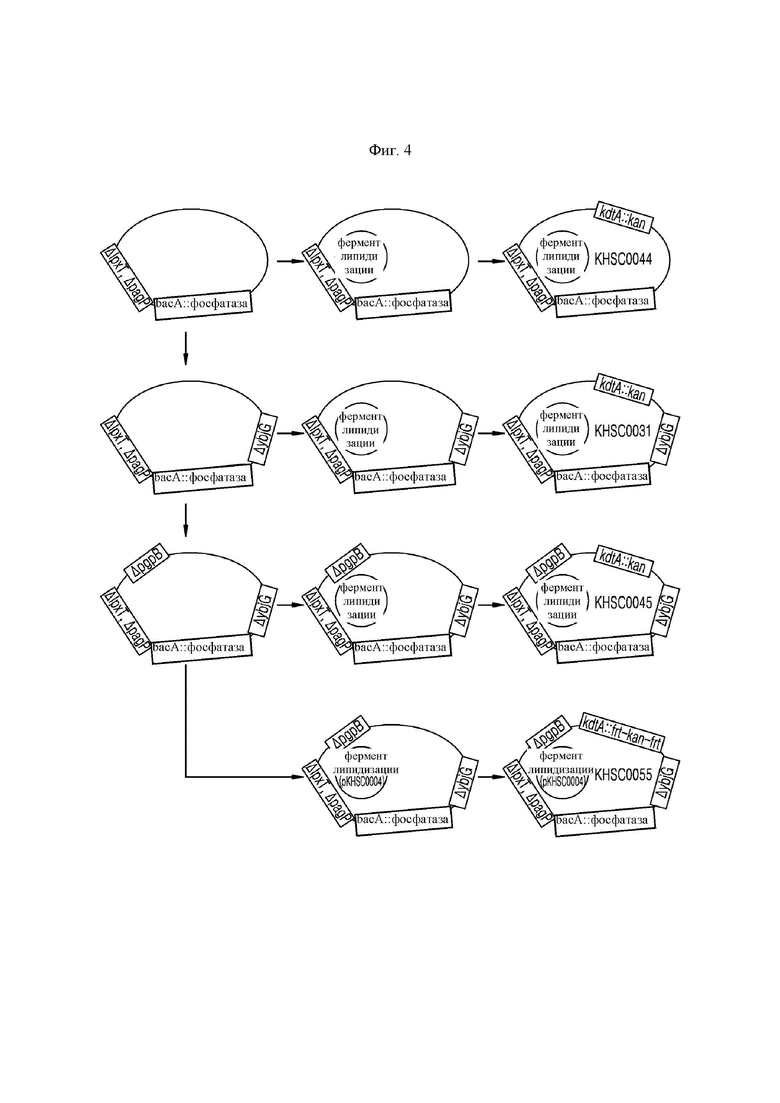

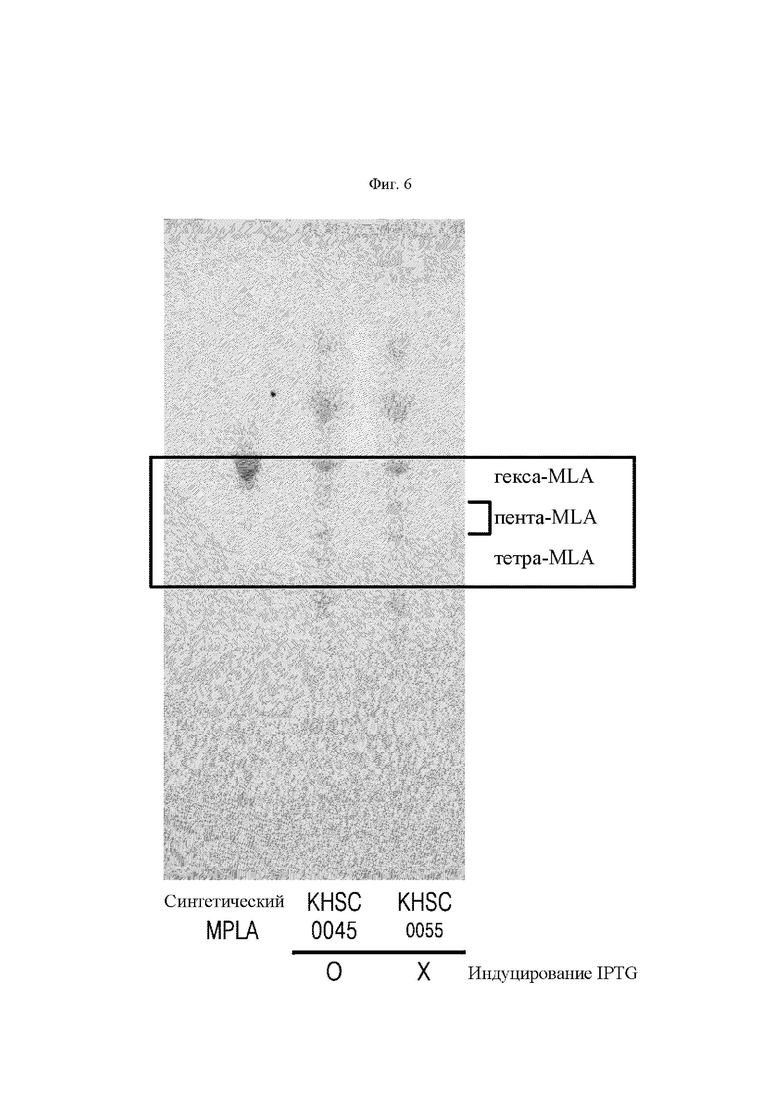

Группа изобретений относится к рекомбинантной бактерии, конститутивно продуцирующей монофосфориллипид A (MLA), не конъюгированный с 2-кето-3-деокси-D-манно-октулозонатной (Kdo) группировкой, а также способу получения MLA, не конъюгированного с Kdo группировкой, с использованием указанной бактерии. Предложена рекомбинантная бактерия, конститутивно продуцирующая MLA, не конъюгированный с Kdo группировкой, где указанная рекомбинантная бактерия имеет повышенную экспрессию гена, кодирующего полипептид липид A 1-фосфатазу (LpxE), по сравнению с родительской бактериальной клеткой. Также хромосома бактерии содержит делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего фосфатазу ундекапренилпирофосфата (Und-PP фосфатазу), делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего фосфатазу фосфатидилглицерофосфата (PGP фосфатазу), и делецию, мутацию сдвига рамки считывания или замену эндогенного полинуклеотида, кодирующего полипептид трансферазу 3-деокси-D-манно-октулозоновой кислоты (KdtA). Способ получения MLA, не конъюгированного с Kdo группировкой, включает культивирование указанной бактерии с получением культуры, сбор бактерии из культуры и выделение указанного MLA из собранных бактерий. Группа изобретений обеспечивает эффективное получение указанного MLA с использованием бактерии, конститутивно продуцирующей MLA. 2 н. и 14 з.п. ф-лы, 3 пр., 7 ил.
1. Рекомбинантная бактерия, конститутивно продуцирующая монофосфориллипид A (MLA), не конъюгированный с 2-кето-3-деокси-D-манно-октулозонатной (Kdo) группировкой, где указанная рекомбинантная бактерия имеет повышенную экспрессию гена, кодирующего полипептид липид A 1-фосфатазу (LpxE), по сравнению с родительской бактериальной клеткой, и хромосома бактерии содержит делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего фосфатазу ундекапренилпирофосфата (Und-PP фосфатазу), делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего фосфатазу фосфатидилглицерофосфата (PGP фосфатазу), и делецию, мутацию сдвига рамки считывания или замену эндогенного полинуклеотида, кодирующего полипептид трансферазу 3-деокси-D-манно-октулозоновой кислоты (KdtA).
2. Бактерия по п. 1, где MLA содержит 1-дефосфо-липид A, 1-дефосфо-тетраацилированный липид A, 1-дефосфо-пентаацилированный липид A или их комбинацию.
3. Бактерия по п. 1, где полинуклеотид, кодирующий полипептид LpxE, находится в хромосоме бактерии.
4. Бактерия по п. 1, где полинуклеотид, кодирующий Und-PP фосфатазу, представляет собой ген bacA, ген pgpB, ген ybjG или их комбинацию.
5. Бактерия по п. 1, где полинуклеотид, кодирующий PGP фосфатазу, представляет собой ген pgpB, ген pgpA, ген pgpC или их комбинацию.
6. Бактерия по п. 4, содержащая делецию, мутацию сдвига рамки считывания или замену гена bacA, делецию, мутацию сдвига рамки считывания или замену гена pgpB и делецию, мутацию сдвига рамки считывания или замену гена ybjG.
7. Бактерия по п. 1, дополнительно содержащая полинуклеотид, кодирующий полипептид, выбранный из группы, состоящей из полипептида LpxL, полипептида LpxM или их комбинации.
8. Бактерия по п. 7, где полинуклеотид является индуцибельным или конститутивно экспрессируемым.
9. Бактерия по п. 7, где полинуклеотид экспрессируется посредством промотора, выбранного из группы, состоящей из промотора PL, промотора PR и промотора PR'.
10. Бактерия по п. 1, дополнительно содержащая делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего полипептид LpxT, делецию, мутацию сдвига рамки считывания или замену полинуклеотида, кодирующего полипептид PagP, или их комбинацию.
11. Бактерия по п. 1, продуцирующая MLA без индуцирования экспрессии индуктором экспрессии, стимулом, индуцирующим экспрессию, или их комбинацией.
12. Бактерия по п. 11, где индуктор экспрессии представляет собой изопропил-β-D-1-тиогалактопиранозид (IPTG).
13. Бактерия по п. 1, продуцирующая MLA в присутствии или в отсутствие антибиотиков.
14. Способ получения монофосфориллипида A (MLA), не конъюгированного с 2-кето-3-деокси-D-манно-октулозонатной (Kdo) группировкой, включающий культивирование бактерии по любому из пп. 1-13 с получением культуры, сбор бактерии из культуры и выделение указанного MLA из собранных бактерий.
15. Способ по п. 14, где культивирование бактерии проводят в отсутствие индуктора экспрессии, стимула, индуцирующего экспрессию, или их комбинации.
16. Способ по п. 14, где культивирование бактерии проводят в присутствии или в отсутствие антибиотиков.
| CHEN J, TAO G, WANG X | |||
| Construction of an Escherichia coli mutant producing monophosphoryl lipid A | |||
| Biotechnol Lett | |||
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| EL GHACHI M., DERBISE A., BOUHSS A., MENGIN-LECREULX D | |||
| Identification of multiple genes encoding membrane proteins with undecaprenyl pyrophosphate phosphatase (UppP) activity in Escherichia coli | |||
| J Biol | |||
Авторы
Даты
2021-07-13—Публикация
2018-07-03—Подача