ОБЛАСТЬ ТЕХНИКИ ИЗОБРЕТЕНИЯ
Один или несколько вариантов осуществления относятся к бактерии, которая продуцирует монофосфориллипид A (MLA), и к способу получения MLA с применением бактерии.
УРОВЕНЬ ТЕХНИКИ ИЗОБРЕТЕНИЯ
Липополисахариды (LPS) являются одними из компонентов наружной мембраны, окружающей пептидогликан грамотрицательных бактерий. LPS представляют собой молекулы, содержащие липид А и ряд полисахаридов, конъюгированных с липидом А посредством ковалентной связи. Среди компонентов LPS, липид А, который также известен как эндотоксин, отвечает за токсичность грамотрицательных бактерий.
Липид А является очень сильным стимулятором иммунной системы, активирующим клетки (например, моноциты или макрофаги) в количествах пикограмм на миллилитр. Липид А, производные липида А или варианты липида А можно использовать, например, в качестве компонентов вакцин, таких как адъюванты. Монофосфориллипид A (MLA) используют в качестве адъювантов и используют в аллерген-специфической иммунотерапии и иммунотерапии рака, или он также является эффективным в предупреждении и лечении деменции. Кроме того, среди MLA, гексаацилированный монофосфориллипид А (гексаацилированный MLA), пентаацилированный монофосфориллипид А (пентаацилированный MLA) и 3-O-дезацил-4'-монофосфориллипид A (3D-MLA) являются эффективными в вышеупомянутом применении. Липид А является липидным компонентом, обнаруженным в мембране грамотрицательных бактерий, таких как Escherichia coli. Липид А, обнаруженный в мембране, конъюгирует с сахарами, такими как 2-кето-3-дезокси-D-маннооктулозонат (Kdo). Таким образом, для получения липида А в свободной форме он должен быть отделен от других компонентов LPS. Например, LPS можно экстрагировать из бактериальных мембран, нагретых в присутствии кислот, чтобы удалить Kdo и 1-фосфатную группу, за счет чего обеспечивается получение липида А; или MLA можно синтезировать путем химической обработки. Однако эти способы имеют недостатки, заключающиеся в том, что они предусматривают сложные стадии процессов при низком выходе.
Следовательно, существует потребность в разработке способа получения MLA и его производных, который является более простым, чем традиционные способы, без кислотного гидролиза.
ОПИСАНИЕ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ ТЕХНИЧЕСКАЯ ЗАДАЧА
Обеспечение бактерии, которая продуцирует монофосфориллипид A (MLA).
Обеспечение способа получения MLA.
РЕШЕНИЕ ЗАДАЧИ
Настоящая заявка заявляет приоритет согласно заявке на патент Кореи №10-2016-0001708, поданной 6 января 2016 года в Корейское ведомство интеллектуальной собственности, раскрытие которой включено в данный документ во всей своей полноте посредством ссылки.
Термин "повышение экспрессии", используемый в данном документе, относится к выявляемому повышению уровня продукта экспрессии определенного гена, например, mRNA или белка, кодируемого геном в клетке. Термин "родительская бактериальная клетка", используемый в данном документе, относится к бактериальной клетке того же типа, которая не имеет конкретной генетической модификации. При использовании клетки дикого типа в генетической модификации родительская бактериальная клетка может представлять собой клетку "дикого типа". Например, бактерия, содержащая генетическую модификацию, которая повышает экспрессию гена, может иметь более высокий уровень продукта экспрессии, чем таковой в родительской бактериальной клетке, на приблизительно 5% или больше, приблизительно 10% или больше, приблизительно 15% или больше, приблизительно 20% или больше, приблизительно 30% или больше, приблизительно 40% или больше, приблизительно 50% или больше, приблизительно 60% или больше, приблизительно 70% или больше, приблизительно 80% или больше, приблизительно 90% или больше, приблизительно 95% или больше или приблизительно 100% или больше. Повышение уровня продукта экспрессии в клетке можно подтвердить любыми способами, известными из уровня техники. Уровень продукта экспрессии можно определить путем измерения значений активности или количеств продукта экспрессии, такого как mRNA или белок.
Термин "снижение экспрессии", используемый в данном документе, относится к выявляемому снижению уровня продукта экспрессии определенного гена, например, mRNA или белка, кодируемого геном в клетке. Термин "родительская бактериальная клетка", используемый в данном документе, относится к бактериальной клетке того же типа, которая не имеет конкретной генетической модификации. При использовании клетки дикого типа в генетической модификации родительская бактериальная клетка может представлять собой клетку "дикого типа". Например, бактерия, содержащая генетическую модификацию, которая снижает экспрессию гена, может иметь более низкий уровень продукта экспрессии, чем таковой в родительской бактериальной клетке, на приблизительно 5% или больше, приблизительно 10% или больше, приблизительно 15% или больше, приблизительно 20% или больше, приблизительно 30% или больше, приблизительно 40% или больше, приблизительно 50% или больше, приблизительно 60% или больше, приблизительно 70% или больше, приблизительно 80% или больше, приблизительно 90% или больше, приблизительно 95% или больше или приблизительно 100% или больше. Снижение уровня продукта экспрессии в клетке можно подтвердить любыми способами, известными из уровня техники. Уровень продукта экспрессии можно определить путем измерения значений активности или количеств продукта экспрессии, такого как mRNA или белок.
Термины "нарушение", "нарушенная" и т.п., используемые в данном документе, относятся к сниженной экспрессии данного гена вследствие генетической модификации. Нарушение может быть вызвано генетической модификацией, которая полностью аннулирует экспрессию эталонного гена (далее в данном документе упоминается как "инактивация" гена.). Нарушение также предусматривает генетическую модификацию, которая вызывает экспрессию гена при сниженных уровнях без полного аннулирования экспрессии (далее в данном документе упоминается как "аттенюация" гена). Экспрессия, в этом смысле, относится к транскрипции продукта гена, а также к трансляции активного продукта гена. Таким образом, инактивация включает случай, при котором ген не является транскрибированным или транслированным, так, что продукт гена не экспрессируется, и случай, при котором, хотя ген является транскрибированным или транслированным, продукт гена не является функциональным. Подобным образом, аттенюация включает случай, при котором транскрипция и/или трансляция гена снижается, а также случай, при котором транскрипция и/или трансляция не снижается, но продукт гена характеризуется более низким уровнем активности. В данном документе термин "функциональный продукт гена" означает, что продукт гена (например, белок или фермент) обладает биохимической или физиологической функцией (например, ферментативной активностью). Нарушение гена предусматривает функциональное нарушение гена, при котором биохимическая или физиологическая функция в генетически модифицированной клетке снижена или полностью аннулирована по сравнению с родительской клеткой или клеткой дикого типа.
Генетическая модификация предусматривает модификацию, которая вводит полинуклеотид, кодирующий полипептид, в клетку; модификацию, которая замещает, добавляет (т.е. вставляет) или удаляет один или несколько нуклеотидов генетического материала родительской клетки, в том числе химическую модификацию (воздействие химического вещества), что приводит в результате к изменению генетического материала родительской клетки. Генетическая модификация предусматривает гетерологичную или гомологичную модификацию эталонных видов. Генетическая модификация предусматривает модификацию кодирующей полипептиды области. Генетическая модификация также предусматривает модификацию некодирующих регуляторных областей, которые изменяют экспрессию гена или функцию оперона. Некодирующие области включают 5'-некодирующую последовательность (5' кодирующей последовательности) и 3'-некодирующую последовательность (3' кодирующей последовательности).
Нарушение гена может быть достигнуто посредством способа генетической инженерии, таким как гомологичная рекомбинация, направленный мутагенез или направленная молекулярная эволюция. Если клетка включает множество идентичных генов или 2 или больше паралогов гена, то один или несколько генов могут быть нарушенными. Например, генетическая модификация может предусматривать трансформацию клетки вектором, включающим последовательность гена, а затем культивирование клетки, чтобы вызвать гомологичную рекомбинацию экзогенной нуклеиновой кислоты и эндогенного гена клетки, за счет чего обеспечивается нарушение эндогенного гена. Клетка, которую подвергли гомологичной рекомбинации, можно подвергать скринингу (отбирать) с использованием селективного маркера.
"Ген", используемый в данном документе, относится к фрагменту нуклеиновой кислоты, который кодирует конкретный белок, который необязательно может включать по меньшей мере одну регуляторную последовательность, такую как 5'-некодирующая последовательность и 3'-некодирующая последовательность (3' и 5' в отношении положения относительно кодирующей последовательности).
Термин "идентичность последовательностей" нуклеиновой кислоты или полипептида, используемый в данном документе, относится к степени идентичности нуклеотидов или аминокислотных остатков двух соответствующих последовательностей по конкретной области, измеренной после выравнивания последовательностей, которые должны совпадать друг с другом в максимально возможной степени. Идентичность последовательностей является значением, которое измеряют путем сравнения двух оптимально выровненных соответствующих последовательностей конкретной сравниваемой области, при этом в сравниваемой области часть последовательности может быть добавлена или удалена по сравнению с эталонной последовательностью. В некоторых вариантах осуществления процентное значение идентичности последовательностей можно рассчитать путем сравнения двух оптимально выровненных соответствующих последовательностей во всей сравниваемой области с определением числа локализаций, в которых аминокислота или нуклеиновая кислота идентичны в двух последовательностях, с получением числа совпадающих локализаций, деления числа совпадающих локализаций на общее число (то есть варьирующий размер) всех локализаций в сравниваемом диапазоне и умножения результата на 100 с получением процентного значения идентичности последовательности. Процентное значение идентичности последовательностей можно определить с использованием известных программ сравнения последовательности, примеры которых включают BLASTN и BLASTP (NCBI), CLC Main Workbench (CLC bio.), MegAlign™ (DNASTAR Inc).
При идентификации полипептидов или полинуклеотидов различных видов, которые могут обладать идентичной или подобной функцией или активностью, может быть использовано сходство в идентичности последовательностей. Например, схожие последовательности могут характеризоваться идентичностью последовательностей, составляющей 50% или больше, 55% или больше, 60% или больше, 65% или больше, 70% или больше, 75% или больше, 80% или больше, 85% или больше, 90% или больше, 95% или больше, 96% или больше, 97% или больше, 98% или больше, 99% или больше или 100%.
Термин "экзогенная" и т.п., используемый в данном документе, относится к эталонной молекуле (например, нуклеиновой кислоте) или эталонной активности, которая была введена в клетку-хозяина. Нуклеиновую кислоту можно экзогенно вводить в хозяина любым подходящим способом. Например, нуклеиновую кислоту можно вводить в клетку-хозяина и вставлять в хромосому хозяина, или нуклеиновую кислоту можно вводить в хозяина в виде нехромосомного генетического материала, такого как вектор экспрессии (например, плазмида), который не встраивается в хромосому хозяина. Нуклеиновую кислоту, кодирующую белок, следует вводить в экспрессируемой форме (т.е. так, чтобы нуклеиновая кислота могла быть транскрибированной и транслированной). Экзогенный ген может включать в себя гомологичный ген, т.е. ген, идентичный эндогенному гену, или гетерологичный ген.
В данном аспекте предусматривается бактерия, которая продуцирует монофосфориллипид A (MLA), содержащая генетическую модификацию, которая повышает экспрессию гена, кодирующего полипептид LpxE, по сравнению с родительской бактериальной клеткой. Бактерия может обладать повышенной способностью продуцировать MLA. Бактерия может быть генетически сконструированной или рекомбинантной.
Липид А состоит из глюкозаминового дисахарида с присоединенными ацильными цепями и обычно содержит одну фосфатную группу в каждом глюкозамине. Два дисахарида могут связываться Р(1→6)-связью. Ацильная цепь может быть непосредственно присоединена к гидроксильному остатку, выбранному из группы гидроксильного остатка в С-2-, С-2'-, С-3- и С-3'-положениях глюкозаминового дисахарида. Ацильная цепь может иметь гидроксильный остаток, например, в своем С-3-положении, а дополнительная ацильная цепь может быть присоединена к гидроксильному остатку, расположенному в этой ацильной цепи. Каждая присоединенная ацильная цепь может быть идентичной или отличающейся. Липид А может содержать 2, 3, 4, 5, 6 или 7 ацильных цепей. Ацильная цепь может иметь 8-30 атомов углерода. Например, ацильная цепь может иметь 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21 или больше, 25 или больше или 30 или больше атомов углерода в длину. Фрагмент липида A Escherichia coli состоит из гексаацилированного бис-1,4'-фосфорилированного глюкозаминового дисахарида, который имеет (R)-3-гидроксимиристильные остатки в С-2, С-2', С-3 и С-3'. Обе из первичных (3)-гидроксиацильных цепей на дистальном глюкозаминовом фрагменте являются эстерифицированными лауриновой и миристиновой кислотами, а первичный гидроксил в C-6-положении связан с полисахаридом посредством димерного углеводного фрагмента 3-дезокси-D-манноокт-2-улозоновой кислоты (KDO). Липид А из N. meningitidis является гексаацилированным симметричным образом, тогда как кишечные бактерии имеют асимметрично гексаацилированный липид А. Кроме того, число жирных кислот в N. meningitidis меньше по сравнению с таковым в Е. coli.
MLA относится к монофосфориллипиду А, в котором только одна фосфатная группа соединяется с С-1- или С-4'-положением глюкозаминовго дисахарида. MLA может представлять собой триацилированный MLA, тетраацилированный MLA, пентаацилированный MLA, гексаацилированный MLA или гептаацилированный MLA. Например, MLA может представлять собой 1-дефосфолипид А, 1-дефосфопентаацилированный липид А, 3-O-деацил-4'-MLA (3D-MLA) или их комбинацию. В этом случае липидом А может являться липид А из E.coli. 3D-MLA также известен как 1-дефосфо-3-О-деациллипид А.
MLA может не включать 2-кето-3-дезокси-D-маннооктулозонат (Kdo). Kdo является компонентом липополисахаридов (LPS).
MLA может присутствовать в мембране, например, в наружной мембране, живой бактерии.
Бактерия может включать повышенное число копий гена, кодирующего полипептид LpxE. Бактерия может включать по меньшей мере один экзогенный полинуклеотид, кодирующий полипептид LpxE.
Полипептид LpxE принадлежит к ЕС 3.1.3.-. LpxE принадлежит к семейству фосфатаз фосфата липида. LpxE может содержать тройной активный сайт и шесть трансмембранных спиралей. Фосфатаза фосфата липида является гидролазой, специфически действующей на связи фосфорного сложного моноэфира, которая может удалять фосфатную группу из липида, содержащего фосфатную группу. LpxE может представлять собой фосфатазу фосфата, специфически дефосфорилирующую положение 1. Полипептид LpxE может представлять собой полипептид LpxE бактерии, выбранной из группы, состоящей из бактерии рода Aquifex, бактерии рода Helicobacter, бактерии рода Francisella, бактерии рода Bordetella, бактерии рода Brucella, бактерии рода Rhizobium, бактерии рода Mesorhizobium, бактерии рода Legionella, бактерии рода Agrobacterium, бактерии рода Chlorobium, бактерии рода Rhodospirillum, бактерии рода Magnetospirillum, бактерии рода Chlorobaculum, бактерии рода Pelodictyon, бактерии рода Pseudovibro, бактерии рода Phaeospirillum, бактерии рода Syntrophobacter, бактерии рода Bradyrhizobium, бактерии рода Porphyromonas, бактерии рода Ralstonia, бактерии рода Limnohabitans и бактерии рода Thermodesulfobacterium. Бактерия рода Aquifex может включать в Aquifex aeolicus или Aquifex pyrophilus. Бактерия рода Aquifex является термофильной бактерией, которая может наилучшим образом расти при температуре в диапазоне от приблизительно 85°С до 95°С Бактерия рода Aquifex может представлять собой Aquifex aeolicus. Бактерия рода Helicobacter может представлять собой Helicobacter pylori. Например, полипептид LpxE может представлять собой полипептид LpxE, полученный из Aquifex aeolicus (AaLpxE), или полипептид LpxE, полученный из Helicobacter pylori (HpLpxE). Полипептид LpxE может представлять собой полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 9 или SEQ ID NO: 17. Полипептид LpxE может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 10 или SEQ ID NO: 16; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% идентичностью по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 10 или SEQ ID NO: 16.
Бактерия может дополнительно включать генетическую модификацию, которая повышает экспрессию гена, кодирующего полипептид LpxL, гена, кодирующего полипептид LpxM, или их комбинации по сравнению с родительской бактериальной клеткой.
Бактерия может включать повышенное число копий гена, кодирующего полипептид LpxL, гена, кодирующего полипептид LpxM, или их комбинации. Бактерия может включать экзогенный полинуклеотид, кодирующий полипептид LpxL, экзогенный полинуклеотид, кодирующий полипептид LpxL, или их комбинацию.
Полипептид LpxL может принадлежать к ЕС 2.3.1.241. Полипептид LpxL представляет собой лауроилтрансферазу биосинтеза липида А, которая катализирует перенос лаурата из лауроилацилпереносящего белка (АСР) в Kdo2-липид IVA с образованием Kdo2-(лауроил)липида IVA. Полипептид LpxL может представлять собой полипептид LpxL бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Bacteroides, бактерии рода Prevotella, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, Rhizobium, бактерии рода Mesorhizobium, бактерии рода Serratia, бактерии рода Acinetobacter, бактерии рода Shewanella, бактерии рода Xenorhabdus, бактерии рода Photobacterium, бактерии рода Lysobacter, бактерии рода Enterobacter и бактерии рода Vibrio. Например, полипептид LpxL может представлять собой полипептид LpxL из Escherichia coli (EcLpxL). Полипептид LpxL может представлять собой полипептид, который включает аминокислотную последовательность из SEQ ID NO: 1; или полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 1. Полипептид LpxL может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 2; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 2.
Полипептид LpxM может принадлежать к ЕС 2.3.1.243. Полипептид LpxM представляет собой миристоилтрансферазу биосинтеза липида А, которая катализирует перенос миристата из миристоилацилпереносящего белка в Kdo2-лауроиллипид IVA c образованием Kdo2-липида А. Полипептид LpxM может представлять собой полипептид LpxM бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Bacteroides, бактерии рода Prevotella, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium, бактерии рода Serratia, бактерии рода Acinetobacter, бактерии рода Shewanella, бактерии рода Xenorhabdus, бактерии рода Photobacterium, бактерии рода Lysobacter, бактерии рода Enterobacter и бактерии рода Vibrio. Например, полипептид LpxM может представлять собой полипептид LpxM из Escherichia coli (EcLpxM). Полипептид LpxM может представлять собой полипептид, который включает аминокислотную последовательность из SEQ ID NO: 5; или полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 5. Полипептид LpxM может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 6; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 6.
Термин "бактерия", используемый в данном документе, относится к прокариотической бактерии. Бактерией могут являться грамотрицательные бактерии. Грамотрицательные бактерии не могут удерживать краситель кристаллический фиолетовый, используемый в окрашивании по методу Грама. Клеточные мембраны грамотрицательных бактерий состоят из двойных мембран внутренней мембраны и наружной мембраны с тонким слоем пептидогликана. Бактерия может быть выбрана из группы, состоящей из бактерии рода Escherichia, бактерии рода Aquifex, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Agrobacterium, бактерии рода Chlorobium, бактерии рода Rhodospirillum, бактерии рода Magnetospirillum, бактерии рода Chlorobaculum, бактерии рода Pelodictyon, бактерии рода Pseudovibro, бактерии рода Phaeospirillum, бактерии рода Syntrophobacter, бактерии рода Bradyrhizobium, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium, бактерии рода Vibrio, бактерии рода Ralstonia, бактерии рода Limnohabitans и бактерии рода Thermodesulfobacterium. Бактерией может являться, например, Escherichia coli.
Бактерия, кроме того, может включать генетическую модификацию, которая снижает экспрессию полинуклеотида, кодирующего полипептид, вовлеченный в путь биосинтеза Kdo. Полипептид, вовлеченный в путь биосинтеза Kdo, может представлять собой полипептид, выбранный из группы, состоящей из полипептида KdtA, KdsB, KdsC, KdsA, GutQ, KpsF, KpsU и KdsD. KdtA также называют WaaA. В бактерии следующие гены могут быть нарушенными: ген, кодирующий полипептид KdtA, ген, кодирующий полипептид KdsB, ген, кодирующий полипептид KdsC, ген, кодирующий полипептид KdsA, ген, кодирующий полипептид GutQ, ген, кодирующий полипептид KpsF, ген, кодирующий полипептид KpsU, ген, кодирующий полипептид KdsD, или их комбинация. В бактерии следующие гены могут быть нарушенными: ген, кодирующий полипептид LpxT, ген, кодирующий полипептид PagP, ген, кодирующий полипептид KdtA, или их комбинация.
Полипептид LpxT может принадлежать к ЕС 2.7.4.29. Полипептид LpxT может представлять собой белок внутренней мембраны. Полипептид LpxT представляет собой фосфотрансферазу, которая катализирует перенос 1-фосфатной группы из ундекапренилпирофосфата в липид А с образованием 1-пирофосфата липида А. Полипептид LpxT может представлять собой полипептид LpxT бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium и бактерии рода Vibrio. Например, полипептид LpxT может представлять собой полипептид LpxT из Escherichia coli (EcLpxT). Полипептид LpxT может представлять собой полипептид, который включает аминокислотную последовательность из SEQ ID NO: 20; или полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 20. Полипептид LpxT может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 21; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 21.
Полипептид PagP может принадлежать к ЕС 2.3.1.251. Полипептид PagP может представлять собой пальмитоилтрансферазу липида А, которая необходима для биосинтеза видов гептаацилированного липида А, содержащих пальмитат.Полипептид PagP катализирует перенос пальмитатной цепи (16:0) из sn-1-положения глицерофосфолипида к свободной гидроксильной группе (R)-3-гидроксимиристатной цепи в положении 2 липида А. Полипептид PagP может представлять собой полипептид PagP бактерии, выбранной из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium и бактерии рода Vibrio. Например, полипептид PagP может представлять собой полипептид PagP из Escherichia coli (EcPagP). Полипептид PagP может представлять собой полипептид, который включает аминокислотную последовательность из SEQ ID NO: 26; или полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 26. Полипептид PagP может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 27; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 27.
Полипептид KdtA может принадлежать к ЕС 2.4.99.12., ЕС 2.4.99.13., ЕС 2.4.99.14. и/или ЕС 2.4.99.15. Полипептид KdtA (или WaaA) может представлять собой фермент, который катализирует перенос Kdo в липид IVA. Например, полипептид KdtA может представлять собой полипептид KdtA из Escherichia coli (EcKdtA). EcKdtA может катализировать перенос двух Kdo в липид IVA. Полипептид KdtA может представлять собой полипептид, который включает аминокислотную последовательность из SEQ ID NO: 22; или полипептид, характеризующийся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к аминокислотной последовательности из SEQ ID NO: 22. Полипептид KdtA может быть кодирован последовательностью нуклеиновой кислоты из SEQ ID NO: 23; или полинуклеотидом, характеризующимся приблизительно 99%, приблизительно 97%, приблизительно 95%, приблизительно 90%, приблизительно 80%, приблизительно 70%, приблизительно 60%, приблизительно 50%, приблизительно 40%, приблизительно 30%, приблизительно 20% или приблизительно 10% или больше идентичностью последовательности по отношению к последовательности нуклеиновой кислоты из SEQ ID NO: 23.
В другом аспекте предусматривается способ получения MLA, который предусматривает культивирование бактерии, описанной выше, с получением культуры и выделение MLA из культуры.
Способ может предусматривать культивирование бактерии, которая продуцирует монофосфориллипид A (MLA), содержащей генетическую модификацию, которая повышает экспрессию гена, кодирующего полипептид LpxE, по сравнению с родительской бактериальной клеткой, с получением культуры.
Полипептид LpxE, MLA и бактерия являются такими же, как описано в данном документе.
Культивирование можно осуществлять с использованием способа, известного из уровня техники. Тип культурального раствора, температура культивирования и условия культивирования могут являться такими, которые известны из уровня техники. Температура культивирования может составлять, например, от приблизительно 10°С до приблизительно 43°С от приблизительно 20°С до приблизительно 43°C, от приблизительно 20°С до приблизительно 40°C, от приблизительно 25°С до приблизительно 43°C, от приблизительно 25°С до приблизительно 35°С, от приблизительно 27°С до приблизительно 33°C, от приблизительно 10°С до приблизительно 15°С, от приблизительно 15°С до приблизительно 20°C, от приблизительно 20°С до приблизительно 25°C, от приблизительно 25°С до приблизительно 30°С, от приблизительно 30°С до приблизительно 33°С, от приблизительно 33°С до приблизительно 37°С, от приблизительно 37°С до приблизительно 40°С или от приблизительно 40°С до приблизительно 43°С Бактерия может быть культивирована в периодическом режиме, культуре с подпиткой или в непрерывном режиме. Культивирование можно осуществлять в стационарном условии или в условии встряхивания. Период культивирования может составлять, например, от приблизительно 1 часа до приблизительно 1 недели, от приблизительно 3 часов до приблизительно 6 дней, от приблизительно 6 часов до приблизительно 5 дней, от приблизительно 9 часов до приблизительно 4 дней, от приблизительно 12 часов до приблизительно 3 дней, от приблизительно 18 часов до приблизительно 2 дней, приблизительно 1 день или на протяжении ночи. Культуральная среда может включать антибиотики. Примеры антибиотиков могут включать канамицин, ампициллин, хлорамфеникол или их комбинацию.
Способ может предусматривать выделение MLA из культуры. Выделение может включать выделение MLA из бактериальной клетки. Выделение может включать выделение бактериальной клетки из культуры. Выделение бактериальной клетки из культуры можно осуществлять с использованием способа, известного из уровня техники. Например, бактерию можно выделять из культуры путем центрифугирования. Выделенную бактерию можно промывать буферным раствором.
Способ может предусматривать выделение MLA из бактерии.
MLA можно отделять от липида бактерии. Способом отделения липида может являться способ, который известен из уровня техники. MLA можно получать физическим или химическим способом. Физический способ может представлять собой, например, повторяющиеся ультразвуковые импульсы или повторяющееся замерзание-оттаивание. Химический способ может представлять собой экстракцию с использованием органического растворителя. Примеры органического растворителя могут включать хлороформ, фенол, петролейный эфир, дихлорметан, метанол, гексан, изопропиловый спирт, этилацетат, ацетонитрил, этанол или их комбинацию. Примеры способа экстракции липида могут представлять собой протокол экстракции липида по Bligh и Dyer (см. Bligh, E.G. and Dyer, W.J., Can. J. Biochem. Physiol., 1959, vol. 37, p. 911-917). Способ дополнительно может предусматривать очищение MLA в липиде. Способ может не предусматривать стадию гидролиза для удаления фрагмента Kdo, поскольку полученный липид А может находиться в свободной форме, т.е. неконъюгированным с фрагментом Kdo.
MLA может включать 1-дефосфолипид А, 1-дефосфотетраацилированный липид А, 1-дефосфопентаацилированный липид A, 3D-MLA или их комбинацию.
Далее приводятся подробности вариантов осуществления, примеры которых иллюстрируются в сопутствующих графических материалах, где все подобные ссылочные позиции относятся к подобным элементам. В этой связи, представленные варианты осуществления могут иметь различные формы и не должны считаться ограниченными описаниями, приведенными в данном документе. Следовательно, варианты осуществления просто описываются ниже со ссылкой на графические материалы для объяснения аспектов настоящего описания. Используемый в данном документе термин "и/или" включает любые и все комбинации одного или нескольких из ассоциированных перечисляемых объектов.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Эти и/или другие аспекты станут очевидными и более понятными из следующего описания вариантов осуществления, приведенных вместе с сопровождающими графическими материалами, из которых
фиг. 1 представляет собой схематическую диаграмму пути биосинтеза 1-дефосфо-Kdo2-липида А в бактерии;
фиг. 2А представляет собой схематическую диаграмму способа получения ПЦР-продуктов, в том числе EcLpxL и EcLpxM;
фиг. 2В представляет собой схематическую диаграмму способа получения Escherichia coli штамма KHSC003;
фиг. 3А и 3В представляют собой изображения, иллюстрирующие результаты анализа липидов с помощью тонкослойной хроматографии (TLC) (на фиг. 3А, дорожка 1: липиды, полученные кислотным гидролизом экстрагированного LPS из Escherichia coli W3110, дорожка 2: липиды, полученные кислотным гидролизом экстрагированного LPS из Escherichia coli W3110, включающей pWSK29-FnLpxE, дорожка 3: экстрагированные липиды из Escherichia coli KHSC003, включающей pBAD33.1, и дорожка 4: экстрагированные липиды из Escherichia coli KHSC003, включающей pBAD33.1-AaLpxE; и на фиг. 3В, дорожка 1: липиды, полученные кислотным гидролизом LPS, экстрагированного из Escherichia coli W3110, дорожка 2: экстрагированные липиды из Escherichia coli KHSC003, включающей pBAD33.1-HpLpxE, и дорожка 3: липиды, экстрагированные из Escherichia coli KHSC003, включающей pBAD33.1);
фиг. 4А представляет собой график, иллюстрирующий результаты анализа времяпролетной MS с лазерной десорбцией-ионизацией в присутствии матрицы (MALDI-TOF) на липиде Escherichia coli KHSC003, трансформированной с помощью pBAD33.1-AaLpxE; фиг. 4В представляет собой график, иллюстрирующий результаты анализа MALDI-TOF MS/MS на монофосфориллипиде A (m/z: 1716,36), выявляемом на фиг. 4А; и
фиг. 5 представляет собой схематическую диаграмму процесса, продуцирующего MLA в Escherichia coli KHSC003, трансформированной с помощью pBAD33.1-AaLpxE или pBAD33.1-HpLpxE.
ПРИНЦИП РАСКРЫТИЯ
Далее в данном документе настоящее раскрытие будет описываться более подробно со ссылкой на примеры. Однако эти примеры приводятся исключительно в иллюстративных целях, и настоящее раскрытие не предназначено для ограничения этими примерами.
Пример 1. Получение вектора, включающего полинуклеотид, который кодирует LpxL Escherichia coli и LpxM Escherichia coli 1.1. Получение pWSK29-EcLpxLEcLpxM
Для получения полинуклеотида, который кодирует полипептиды LpxL Escherichia coli, из генома Escherichia coli W3110 (№доступа в GenBank NC_000918.1, АТСС) полинуклеотид (№ доступа в GenBank AP009048.1 (c1118159.1117239, SEQ ID NO: 2), который кодирует полипептид EcLpxL (№ доступа в GenBank ВАА35852.1, SEQ ID NO: 1), включающий сайт связывания с рибосомой (RBS), амплифицировали с помощью первой полимеразной цепной реакции (ПЦР) с использованием пары праймеров, показанной ниже (см. фиг. 2А).
Прямой праймер P1 LpxL: SEQ ID NO: 3
Обратный праймер Р2 LpxL: SEQ ID NO: 4
Для получения полинуклеотида, который кодирует полипептиды LpxM Escherichia coli, из генома Escherichia coli W3110 полинуклеотид (№доступа в GenBank АР009048.1 (с1941907.1940936, SEQ ID NO: 6), который кодирует полипептид EcLpxM (№доступа в GenBank ВАА15663.1, SEQ ID NO: 5), включающий RBS, амплифицировали с помощью второй ПЦР с использованием пары праймеров, показанной ниже (см. фиг. 2А).
Прямой праймер Р3 LpxM: SEQ ID NO: 7
Обратный праймер Р4 LpxM: SEQ ID NO: 8
Полинуклеотид EcLpxLEcLpxM, который является слиянием полинуклеотида EcLpxL и полинуклеотида EcLpxM, амплифицировали с помощью третьей ПЦР с использованием прямого праймера P1 LpxL и обратного праймера Р4 LpxM и полинуклеотида EcLpxL, полученного из первой ПЦР, и полинуклеотида EcLpxM, полученного из второй ПЦР, в качестве матрицы.
ПЦР осуществляли с использованием KOD Hot Start DNA Polymerase (Novagen) в термоциклере T3000 (Biometra).
Амплифицированные продукты очищали с использованием набора для очистки DokDo-Prep PCR (ELPIS) и очищенные продукты вводили в сайт рестрикции XbaI и HindIII плазмиды pWSK29 (Wang, R.F., and Kushner, S.R., Gene (1991), vol. 100, p. 195-199). Клонированную плазмиду трансформировали в Escherichia coli DH5a с помощью электропорации, а затем подвергали отбору на чашке с LB-ампициллином. Клонированную плазмиду называли pWSK29-EcLpxLEcLpxM.
1.2. Получение pBAD33.1-AaLpxE
1.2.1. Получение pET21-AaLpxE
Для получения полинуклеотида, который кодирует полипептиды LpxE Aquifex aeolicus (AaLpxE), из генома Aquifex aeolicus VF5 (№доступа в GenBank NC_000918.1, АТСС) полинуклеотид (№ доступа в GenBank NC_000918.1:1199317..1199841, SEQ ID NO: 10), который кодирует полипептид AaLpxE (№доступа в GenBank NP_214169.1, SEQ ID NO: 9), амплифицировали с использованием пары праймеров, показанных ниже.
Прямой праймер AaLpxE: SEQ ID NO: 11 Обратный праймер AaLpxE: SEQ ID NO: 12
Как описывается в 1.1, осуществляли ПЦР и амплифицированные продукты очищали. Очищенные продукты вводили в сайт рестрикционного фермента NdeI и XhoI плазмиды рЕТ21а (Novagen). Клонированную плазмиду трансформировали в Е. coli DH5α и подвергали отбору, как описывается в 1.1. Клонированную плазмиду называли рЕТ21-AaLpxE.
1.2.2. Получение pBAD33.1-AaLpxE
Полинуклеотид, который кодирует AaLpxE, амплифицировали с использованием рЕТ21-AaLpxE, полученной в 1.2.1, в качестве матрицы и пары праймеров, показанных ниже.
Прямой праймер AaLpxE: SEQ ID NO: 13
Обратный праймер AaLpxE: SEQ ID NO: 14
ПЦР осуществляли с использованием KOD Hot Start DNA Polymerase (Novagen) в термоциклере T3000 (Biometra).
Как описывается в 1.1, осуществляли ПЦР и амплифицированные продукты очищали. Очищенные продукты клонировали в pBAD33.I по сайту рестрикционного фермента NdeI и HindIII (Chung, Н.S., and Raetz, С.R. Biochemistry (2010), vol. 49 (19), p. 4126-4137). Клонированную плазмиду, описанную в 1.1, трансформировали в Е. coli, а затем подвергали отбору. Клонированную плазмиду называли pBAD33.1-AaLpxE в качестве вектора экспрессии.
1.2.3. Получение pBAD33.1-HpLpxE
В hp0021, который представляет собой ген LpxE Helicobacter pylori (HpLpxE), для делеции последовательности сайта распознавания рестрикционного фермента HindIII полинуклеотидную последовательность (SEQ ID NO: 16), которая имеет кодон Ser AGC в положении, соответствующем 17 остатку Ser, и отличается от такового TCG полинуклеотидной последовательности 17 (SEQ ID NO: 15), синтезировали с помощью Integrated DNA Technologies (mBiotech, ROK). Полинуклеотид, который кодирует аминокислотные последовательности HpLpxE (SEQ ID NO: 17), амплифицировали с использованием синтезированной ДНК в качестве матрицы и пары праймеров, показанных ниже.
Прямой праймер: SEQ ID NO: 18
Обратный праймер: SEQ ID NO: 19
ПЦР осуществляли с использованием KOD Hot Start DNA Polymerase (Novagen) в термоциклере T3000 (Biometra).
Как описывается в 1.1, осуществляли ПЦР и амплифицированные продукты очищали. Очищенные продукты клонировали в pBAD33.1 по сайту рестрикционного фермента XbaI и HindIII (Chung, Н.S., and Raetz, С.R., Biochemistry (2010), vol. 49 (19), p. 4126-4137). Клонированную плазмиду, описанную в 1.1, трансформировали в Escherichia coli, а затем подвергали отбору. Клонированную плазмиду называли pBAD33.1-HpLpxE в качестве вектора экспрессии.
Пример 2. Получение штаммов Escherichia coli KHSC003 (pWSK29-EcLpxLEcLpxM, kdtA::kan, 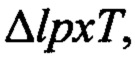 ΔpagP, W3110)
ΔpagP, W3110)
2.1. Получение Escherichia coli, в которой ген 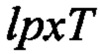 удален из генома В Escherichia coli штамма W3110
удален из генома В Escherichia coli штамма W3110 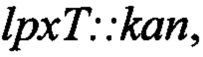 в которой кассета канамицина вставлена в ген
в которой кассета канамицина вставлена в ген 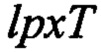 (SEQ ID NO: 21) в геноме Escherichia coli, который кодирует полипептид LpxT (SEQ ID NO: 20), трансформировали плазмиду рСР20 ((Kirill А. Datsenko, and Barry L. Wanner, PNAS (2000), vol. 97, p. 6640-6645), как описывается в 1.1., а затем подвергали отбору на твердой среде LB-ампициллин. Отобранную Escherichia coli инокулировали на твердую среду LB и подвергали отбору при температуре 42°С, за счет чего обеспечивается получение штамма Escherichia coli, из которого удаляли
(SEQ ID NO: 21) в геноме Escherichia coli, который кодирует полипептид LpxT (SEQ ID NO: 20), трансформировали плазмиду рСР20 ((Kirill А. Datsenko, and Barry L. Wanner, PNAS (2000), vol. 97, p. 6640-6645), как описывается в 1.1., а затем подвергали отбору на твердой среде LB-ампициллин. Отобранную Escherichia coli инокулировали на твердую среду LB и подвергали отбору при температуре 42°С, за счет чего обеспечивается получение штамма Escherichia coli, из которого удаляли 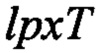 и кассету канамицина, т.е.
и кассету канамицина, т.е. 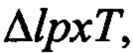 W3110 (стадия 1 на фиг. 2В).
W3110 (стадия 1 на фиг. 2В).
2.2. Получение Escherichia coli, в которой гены pagP и 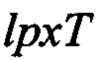 удалены из генома Из штамма Escherichia coli (JW0617 (pagP::kan)), в котором кассета канамицина вставлена в ген pagP в геноме Escherichia coli, получали вирус P1 (Current Protocols in Molecular Biology (2007), 1.16.1-1.16.24, Unit 1.16). Вирус P1 трансдуцировали в
удалены из генома Из штамма Escherichia coli (JW0617 (pagP::kan)), в котором кассета канамицина вставлена в ген pagP в геноме Escherichia coli, получали вирус P1 (Current Protocols in Molecular Biology (2007), 1.16.1-1.16.24, Unit 1.16). Вирус P1 трансдуцировали в 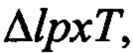 W3110, полученную в 2.1, а затем подвергали отбору на твердой среде LB-канамицин, с получением тем самым штамма Escherichia coli, в который pagP::kan вставляли вместо гена pagP, т.е.
W3110, полученную в 2.1, а затем подвергали отбору на твердой среде LB-канамицин, с получением тем самым штамма Escherichia coli, в который pagP::kan вставляли вместо гена pagP, т.е. 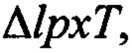 pagP::kan, W3110 (Current Protocols in Molecular Biology (2007), 1.16.1-1.16.24, Unit 1.16) (стадия 2 на фиг. 2B).
pagP::kan, W3110 (Current Protocols in Molecular Biology (2007), 1.16.1-1.16.24, Unit 1.16) (стадия 2 на фиг. 2B).
Плазмиду рСР20 (Kirill A. Datsenko, and Barry L. Wanner, PNAS (2000), vol. 97, p. 6640-6645) трансформировали в 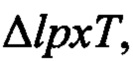 pagP::kan, W3110, а затем подвергали отбору на твердой среде LB-ампициллин. Отобранную Escherichia coli инокулировали на твердую среду LB и подвергали отбору при температуре 42°С, за счет чего обеспечивается получение штамма Escherichia coli, из которого удаляли pagP и кассету канамицина, т.е.
pagP::kan, W3110, а затем подвергали отбору на твердой среде LB-ампициллин. Отобранную Escherichia coli инокулировали на твердую среду LB и подвергали отбору при температуре 42°С, за счет чего обеспечивается получение штамма Escherichia coli, из которого удаляли pagP и кассету канамицина, т.е. 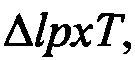 ΔpagP, W3110 (стадия 3 на фиг. 2В).
ΔpagP, W3110 (стадия 3 на фиг. 2В).
2.3. Получение Escherichia coli pWSK29-EcLpxLEcLpxM, 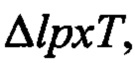 ΔpagP, штамма W3110
ΔpagP, штамма W3110
Плазмиду pWSK29-EcLpxLEcLpxM, полученную в 1.1, трансформировали в 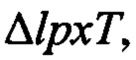 ΔpagP, W3110, полученный в 2.2, с помощью электропорации. Трансформированную Escherichia coli подвергали отбору на твердой среде LB-ампициллин, с получением тем самым Escherichia coli pWSK29-EcLpxLEcLpxM,
ΔpagP, W3110, полученный в 2.2, с помощью электропорации. Трансформированную Escherichia coli подвергали отбору на твердой среде LB-ампициллин, с получением тем самым Escherichia coli pWSK29-EcLpxLEcLpxM, 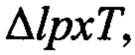 ΔpagP, штамма W3110 (стадия 4 на фиг. 2В).
ΔpagP, штамма W3110 (стадия 4 на фиг. 2В).
2.4. Получение Escherichia coli штамма KHSC003
Из Escherichia coli, включающей плазмиду pEcKdtA, в которой кассета канамицина вставлена в ген kdtA (SEQ ID NO: 23), который кодирует полипептиды KdtA (SEQ ID NO: 22), в хромосоме Escherichia coli, то есть HSCl/pEcKdt (Chung, H.S., and Raetz, C.R., Biochemistry (2010), vol. 49 (19), p. 4126-4137), получали вирус P1 (Current Protocols in Molecular Biology (2007) 1.16.1-1.16.24, Unit 1.16). Вирус P1 трансдуцировали в Escherichia coli pWSK29-EcLpxLEcLpxM, 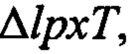 ΔpagP, штамм W3110, полученный в 2.3, а затем подвергали отбору на твердой среде LB-канамицин/ампициллин (стадия 5 на фиг. 2В). Отобранную Escherichia coli называли KHSC003 (pWSK29-EcLpxLEcLpxM,
ΔpagP, штамм W3110, полученный в 2.3, а затем подвергали отбору на твердой среде LB-канамицин/ампициллин (стадия 5 на фиг. 2В). Отобранную Escherichia coli называли KHSC003 (pWSK29-EcLpxLEcLpxM, 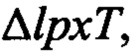 ΔpagP, kdtA::kan, W3110).
ΔpagP, kdtA::kan, W3110).
Пример 3. Тестирование липида Escherichia coli, в которую вводили AaLpxE, HpLpxE или FnLpxE
3.1. Экстрагирование липида из Escherichia coli W3110, трансформированной с помощью pWSK29-FnLpxE, путем кислотного гидролиза
Для сравнительного эксперимента получали Escherichia coli штамма W3110, в которую трансформировали pWSK29-FnLpxE, и Escherichia coli штамма W3110, в которую не трансформировали pWSK29-FnLpxE.
В частности, получали pWSK29-FnLpxE (Wang, X., Karbarz, М.J., McGrath, S.С, Cotter, R.J., and Raetz, C.R., J Biol Chem (2004), vol. 279 (47), p. 49470-49478), которая амплифицирует полинуклеотид (gb|CP000439.1|:414941-415660 Francisella novicida U112, SEQ ID NO: 25), который кодирует полипептиды FnLpxE (gi|118422929|gb|ABK89319.1, фосфатаза липида A Francisella novicida U112, SEQ ID NO: 24). Полученную pWSK29-FnLpxE трансформировали в Escherichia coli штамма W3110 с помощью электропорации, а затем трансформированную Escherichia coli подвергали отбору на твердой среде LB, содержащей 50 мкг/мл ампициллина. Отобранную колонию инокулировали в жидкую среду LB, содержащую 50 мкг/мл ампициллина, а затем культивировали на протяжении ночи при 37°С Культуральный раствор инокулировали в 200 мл свежей жидкой среды LB, содержащей 50 мкг/мл ампициллина, так, что поглощение, измеренное при длине волны 600 нм (OD600), составляло 0,06. Разбавленный культуральный раствор культивировали при 37°С до достижения OD600 1,0. Затем культуральный раствор центрифугировали при центробежной силе 4000х g при комнатной температуре в течение приблизительно 20 минут с получением тем самым культивированной Escherichia coli штамма W3110.
Нетрансформированную Escherichia coli штамма W3110 инокулировали в свежую жидкую среду LB, а затем культивировали на протяжении ночи при 37°С. Культуральный раствор разбавляли посредством 200 мл свежей жидкой среды LB так, что OD600 составляла 0,06. Разбавленный культуральный раствор культивировали при 37°С до достижения OD600 1,0. Затем культуральный раствор центрифугировали при центробежной силе 4000х g при комнатной температуре в течение приблизительно 20 минут с получением тем самым культивированной Escherichia coli штамма W3110. Каждую полученную Escherichia coli, pWSK29-FnLpxE/W3110 и W3110, промывали посредством 30 мл фосфатно-солевого буферного раствора (PBS), затем ресуспендировали с 8 мл PBS. Добавляли 10 мл хлороформа (EMD millipore) и 20 мл метанола (EMD millipore) в ресуспендированную Escherichia coli, а затем инкубировали при комнатной температуре в течение приблизительно 1 часа путем встряхивания смеси несколько раз. Затем инкубированную смесь центрифугировали при центробежной силе 2500х g в течение приблизительно 30 минут при комнатной температуре. Надосадочную жидкость центрифугированной смеси отбрасывали, а затем клеточный осадок ресуспендировали с 30 мл раствора хлороформа, метанола и PBS в соотношении 1:2:0,8 (объем/объем). Ресуспендированную смесь центрифугировали при центробежной силе 2500х g в течение приблизительно 30 минут при комнатной температуре, а надосадочную жидкость центрифугированной смеси отбрасывали. Осадок промывали еще два раза тем же способом, как описано выше, а затем надосадочную жидкость центрифугированной смеси отбрасывали. Промытый осадок повторно суспендировали с 18 мл 12,5 мМ раствора ацетата натрия (рН 4,5, Sigma-Aldrich), а затем обрабатывали ультразвуком. Обработанный ультразвуком раствор нагревали без доступа воздуха в кипящей воде при температуре 100°С в течение приблизительно 30 минут, а затем охлаждали до комнатной температуры. Добавляли 20 мл хлороформа (EMD millipore) и 20 мл метанола (EMD millipore) в охлажденный раствор, а затем полностью смешивали друг с другом с последующим центрифугированием смеси при центробежной силе 2500х g в течение приблизительно 20 минут при комнатной температуре. Слой органического растворителя отделяли от центрифугированной смеси, а затем добавляли слой предварительно кондиционированного органического растворителя к верхнему водному слою с тем, чтобы дважды экстрагировать слой органического растворителя. Слой органического растворителя объединяли, а затем высушивали в роторном испарителе с получением липида. Полученный липид растворяли в 5 мл смеси хлороформа и метанола в соотношении 4:1 (объем/объем), а затем обрабатывали ультразвуком в водяной бане. Обработанный ультразвуком липид перемещали в новую аналитическую пробирку, а затем полученный липид высушивали при комнатной температуре в атмосфере азота с последующим хранением при -80°С
3.2. Экстрагирование липида из Escherichia coli KHSC003, трансформированной с помощью pBDA33.1, pBAD33.1-AaLpxE или pBAD33.1-HpLpxE
Плазмиду, полученную в 1.2.2, pBAD33.1-AaLpxE или pBAD33.1-HpLpxE, трансформировали в Escherichia coli KHSC003, полученную в 2.4, а затем тестировали липид мембраны Escherichia coli. В качестве группы отрицательного контроля использовали Escherichia coli KHSC003, в которую трансформировали вектор pBAD33.1.
В частности, вектор pBAD33.1, pBAD33.1-AaLpxE, полученный в 1.2.2, или pBAD33.1-HpLpxE, полученный в 1.2.3, трансформировали в Escherichia coli штамма KHSC003, полученную в 2.4, с помощью электропорации. Штамм KHSC003, содержащий pBAD33.1-AaLpxE, депонировали в Корейском исследовательском институте бионаук и биотехнологии, который является международным депозитарным органом согласно Будапештскому договору от 18 ноября 2016 года (номер доступа: КСТС 13155 ВР). Штамм KHSC003, содержащий pBAD33.1-HpLpxE, депонировали в Корейском исследовательском институте бионаук и биотехнологии, который является международным депозитарным органом согласно Будапештскому договору от 18 ноября 2016 года (номер доступа: KCTC 13156 ВР).
Трансформированную Escherichia coli инокулировали в жидкую среду LB, содержащую 50 мкг/мл ампициллина (EMD millipore) и 30 мкг/мл хлорамфеникола (Sigma-Aldrich), а затем культивировали на протяжении ночи при 30°С Культуральный раствор инокулировали в 200 мл жидкой среды LB, содержащей 50 мкг/мл ампициллина, 30 мкг/мл хлорамфеникола и 1 мМ изопропил-1-тио-β-D-галактозида (IPTG, UBP Bio), и полученный таким образом культуральный раствор разбавляли так, что OD600 составляла 0,06-0,1. Культуральный раствор культивировали до достижения OD600 0,5-0,6. Культуральный раствор центрифугировали при комнатной температуре при центробежной силе 4000х g в течение приблизительно 20 минут, чтобы получить Escherichia coli. Полученную Escherichia coli промывали посредством 30 мл PBS, а затем ресуспендировали с 8 мл PBS. Добавляли 10 мл хлороформа и 20 мл метанол в ресуспендированную Escherichia coli, а затем инкубировали при комнатной температуре в течение приблизительно 1 часа со встряхиванием несколько раз. Инкубированную смесь центрифугировали при центробежной силе 2500х g в течение приблизительно 30 минут при комнатной температуре с получением тем самым в ней надосадочной жидкости. Добавляли 10 мл хлороформа и 10 мл воды в полученную надосадочную жидкость, после чего смесь полностью смешивали. Затем смесь центрифугировали при центробежной силе 2500х g в течение приблизительно 20 минут при комнатной температуре. Слой органического растворителя отделяли от центрифугированной смеси, а затем добавляли слой предварительно кондиционированного органического растворителя к верхнему водному слою с тем, чтобы дважды экстрагировать слой органического растворителя. Слой органического растворителя объединяли, а затем высушивали в роторном испарителе с получением липида. Полученный липид растворяли в 5 мл раствора хлороформа и метанола в соотношении 4:1 (объем/объем), а затем обрабатывали ультразвуком в водяной бане. Обработанный ультразвуком липид перемещали в новую аналитическую пробирку, а затем полученный липид высушивали при комнатной температуре в атмосфере азота с последующим хранением при -80°С
3.3. Анализ липида посредством тонкослойной хроматографии (TLC) Для проведения TLC, как описывается в 3.1 или 3.2, липид получали из 200 мл культурального раствора Escherichia coli и одну треть всего полученного липида растворяли в 200 мкл смеси хлороформа и метанола в соотношении 4:1 (объем/объем). Затем от 5 мкл до 15 мкл смеси наносили на пластинку HPTLC 10×10 см (EMD Chemicals) и проявляли в растворителе из хлороформа, метанола, воды и гидроксида аммония (от 30% до 20%) в соотношении 40:25:4:2 (объем/объем). Потом проявленную пластинку высушивали, визуализировали путем распыления на нее 10% (объем/объем) серной кислоты (в этаноле), а затем обугливали на горячей плите при 300°С Результаты TLC липида представлены на фиг. 3А и 3В (на фиг. 3А, дорожка 1: липиды, полученный кислотным гидролизом экстрагированного LPS из Escherichia coli W3110, дорожка 2: липиды, полученные кислотным гидролизом экстрагированного LPS из Escherichia coli W3110, содержащей pWSK29-FnLpxE, дорожка 3: экстрагированные липиды, полученные из Escherichia coli KHSC003, содержащей pBAD33.1, и дорожка 4: экстрагированные липиды, полученные из Escherichia coli KHSC003, содержащей pBAD33.1-AaLpxE; и на фиг. 3В, дорожка 1: липиды, полученные кислотным гидролизом экстрагированного LPS из Escherichia coli W3110, дорожка 2: экстрагированные липиды, полученные из Escherichia coli KHSC003, содержащей pBAD33.1-HpLpxE, и дорожка 3: экстрагированные липиды, полученные из Escherichia coli KHSC003, содержащей pBAD33.1).
Как показано на фиг. 3А и 3В, липид А выявляли из Escherichia coli W3110, которую подвергали процессу кислотного гидролиза (дорожка 1 на фиг. 3А), и 1-дефосфолипид А выявляли из Escherichia coli W3110, включающей pWSK29-FnLpxE, которую подвергали процессу кислотного гидролиза (дорожка 2 на фиг. 3А). Кроме того, выявляли липид А из Escherichia coli KHSC003 (дорожка 3 на фиг. 3А) и выявляли 1-дефосфолипид А из Escherichia coli KHSC003, включающей pBAD33.1-AaLpxE или pBAD33.1-HpLpxE (дорожка 4 на фиг. 3А или дорожка 2 на фиг. 3В). Следовательно, поскольку мембрана Escherichia coli KHSC003, трансформированной с помощью pBAD33.1-AaLpxE или pBAD33.1-HpLpxE, содержала 1-дефосфолипид А, обнаружили, что может быть получена живая Escherichia coli, содержащая 1-дефосфолипид А в мембране. Escherichia coli KHSC003, трансформированную с помощью pBAD33.1-HplLpxE, т.е. pBAD33.1-HplpxE/ KHSC003, депонировали на международном уровне 23 ноября 2016 года с номером доступа КСТС13156 ВР в Корейскую коллекцию типовых культур (КСТС), которая является международным депозитарным органом согласно Будапештскому договору.
3.4. Времяпролетная масс-спектрометрия с лазерной десорбцией-ионизацией в присутствии матрицы (MALDI-TOF) на липиде А и 1-дефосфолипиде А
Липид, полученный, как описывается в 3.2, ресуспендировали со смесью хлороформа и метанола в соотношении 4:1 (объем/объем) и образец ресуспендированного липида отправляли в Корейский институт фундаментальных наук с просьбой о масс-спектрометрии MALDI-TOF.
В частности, в качестве матрицы использовали раствор смеси раствора 10 мг/мл 2,5-дигидроксибензойной кислоты (DHB) (Sigma-Aldrich) и ацетонитрила (Sigma-Aldrich) в соотношении 1:4. 1 мкл матричного раствора распределяли по шлейфу образца и наносили на него 1 мкл образца ресуспендированного липида, с последующим высушиванием в вакууме. Масс-спектрометрию MALDI-TOF выполняли на образце липида с использованием масс-спектрометра MALDI-TOF (Shimadzu Biotech Axima Resonance). Данные масс-спектрометрии анализировали с использованием программного обеспечения mMass (http://www.mmass.org/). Результаты анализа MALDI-TOF MS показаны на фиг.4А. Результаты анализа MALDI-TOF MS-MS показаны на фиг. 4В (ось x: отношение массы к заряду (m/z), ось у: относительная интенсивность (%)).
Как показано на фиг. 4А и 4В, обнаружили, что в Escherichia coli KHSC003, трансформированной с помощью pBAD33.1-AaLpxE, выявляли монофосфориллипид А (MLA), не включающий Kdo, т.е. 1-дефосфолипид А (фактическая единица массы: 1716,25). Таким образом, с помощью экспрессии полипептидов EcLpxM, EcLpxL и AaLpxE в Escherichia coli обнаружили, что может быть получена живая Escherichia coli, содержащая 1-дефосфолипид А в мембране. Escherichia coli KHSC003, трансформированную с помощью pBAD33.1-AaLpxE, т.е. pBAD33.1-AaLpxE/ KHSC003, депонировали на международном уровне 18 ноября 2016 года с номером доступа КСТС13155 ВР в Корейскую коллекцию типовых культур (КСТС), которая является международным депозитарным органом согласно Будапештскому договору.
Как описывается выше, при применении бактерии, которая продуцирует монофосфориллипид A (MLA), в том числе полипептид LpxE, и способа получения MLA с применением бактерии согласно одному или нескольким аспектам может быть получен MLA просты способом без кислотного гидролиза и/или основного гидролиза.
Название депозитарного учреждения: Корейская коллекция типовых культур
Номер доступа: КСТС 13155 ВР
Дата депонирования: 18 ноября 2016 года
--->
Sequence Listing
<110> Korea Institute of Science and Technology
<120> Bacteria producing hexa-acylated monophosphoryl lipid A, and
method for the production of hexa-acylated monophosphoryl lipid
A using the same
<130> PX052404US
<160> 27
<170> KopatentIn 2.0
<210> 1
<211> 306
<212> PRT
<213> Artificial Sequence
<220>
<223> Escherichia coli LpxL polypeptide
<400> 1
Met Thr Asn Leu Pro Lys Phe Ser Thr Ala Leu Leu His Pro Arg Tyr
1 5 10 15
Trp Leu Thr Trp Leu Gly Ile Gly Val Leu Trp Leu Val Val Gln Leu
20 25 30
Pro Tyr Pro Val Ile Tyr Arg Leu Gly Cys Gly Leu Gly Lys Leu Ala
35 40 45
Leu Arg Phe Met Lys Arg Arg Ala Lys Ile Val His Arg Asn Leu Glu
50 55 60
Leu Cys Phe Pro Glu Met Ser Glu Gln Glu Arg Arg Lys Met Val Val
65 70 75 80
Lys Asn Phe Glu Ser Val Gly Met Gly Leu Met Glu Thr Gly Met Ala
85 90 95
Trp Phe Trp Pro Asp Arg Arg Ile Ala Arg Trp Thr Glu Val Ile Gly
100 105 110
Met Glu His Ile Arg Asp Val Gln Ala Gln Lys Arg Gly Ile Leu Leu
115 120 125
Val Gly Ile His Phe Leu Thr Leu Glu Leu Gly Ala Arg Gln Phe Gly
130 135 140
Met Gln Glu Pro Gly Ile Gly Val Tyr Arg Pro Asn Asp Asn Pro Leu
145 150 155 160
Ile Asp Trp Leu Gln Thr Trp Gly Arg Leu Arg Ser Asn Lys Ser Met
165 170 175
Leu Asp Arg Lys Asp Leu Lys Gly Met Ile Lys Ala Leu Lys Lys Gly
180 185 190
Glu Val Val Trp Tyr Ala Pro Asp His Asp Tyr Gly Pro Arg Ser Ser
195 200 205
Val Phe Val Pro Leu Phe Ala Val Glu Gln Ala Ala Thr Thr Thr Gly
210 215 220
Thr Trp Met Leu Ala Arg Met Ser Gly Ala Cys Leu Val Pro Phe Val
225 230 235 240
Pro Arg Arg Lys Pro Asp Gly Lys Gly Tyr Gln Leu Ile Met Leu Pro
245 250 255
Pro Glu Cys Ser Pro Pro Leu Asp Asp Ala Glu Thr Thr Ala Ala Trp
260 265 270
Met Asn Lys Val Val Glu Lys Cys Ile Met Met Ala Pro Glu Gln Tyr
275 280 285
Met Trp Leu His Arg Arg Phe Lys Thr Arg Pro Glu Gly Val Pro Ser
290 295 300
Arg Tyr
305
<210> 2
<211> 921
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Escherichia coli LpxL polypeptide
<400> 2
atgacgaatc tacccaagtt ctccaccgca ctgcttcatc cgcgttattg gttaacctgg 60
ttgggtattg gcgtactttg gttagtcgtg caattgccct acccggttat ctaccgcctc 120
ggttgtggat taggaaaact ggcgttacgt tttatgaaac gacgcgcaaa aattgtgcat 180
cgcaacctgg aactgtgctt cccggaaatg agcgaacaag aacgccgtaa aatggtggtg 240
aagaatttcg aatccgttgg catgggcctg atggaaaccg gcatggcgtg gttctggccg 300
gaccgccgaa tcgcccgctg gacggaagtg atcggcatgg aacacattcg tgacgtgcag 360
gcgcaaaaac gcggcatcct gttagttggc atccattttc tgacactgga gctgggtgcg 420
cggcagtttg gtatgcagga accgggtatt ggcgtttatc gcccgaacga taatccactg 480
attgactggc tacaaacctg gggccgtttg cgctcaaata aatcgatgct cgaccgcaaa 540
gatttaaaag gcatgattaa agccctgaaa aaaggcgaag tggtctggta cgcaccggat 600
catgattacg gcccgcgctc aagcgttttc gtcccgttgt ttgccgttga gcaggctgcg 660
accacgaccg gaacctggat gctggcacgg atgtccggcg catgtctggt gcccttcgtt 720
ccacgccgta agccagatgg caaagggtat caattgatta tgctgccgcc agagtgttct 780
ccgccactgg atgatgccga aactaccgcc gcgtggatga acaaagtggt cgaaaaatgc 840
atcatgatgg caccagagca gtatatgtgg ttacaccgtc gctttaaaac acgcccggaa 900
ggcgttcctt cacgctatta a 921
<210> 3
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> LpxL forward primer P1
<400> 3
cgcagtctag aaaggagata tattgatgac gaatctaccc aagttctc 48
<210> 4
<211> 67
<212> DNA
<213> Artificial Sequence
<220>
<223> LpxL reverse primer P2
<400> 4
cgctattatt ttttttcgtt tccattggta tatctccttc ttattaatag cgtgaaggaa 60
cgccttc 67
<210> 5
<211> 323
<212> PRT
<213> Artificial Sequence
<220>
<223> Escherichia coli LpxM polypeptide
<400> 5
Met Glu Thr Lys Lys Asn Asn Ser Glu Tyr Ile Pro Glu Phe Asp Lys
1 5 10 15
Ser Phe Arg His Pro Arg Tyr Trp Gly Ala Trp Leu Gly Val Ala Ala
20 25 30
Met Ala Gly Ile Ala Leu Thr Pro Pro Lys Phe Arg Asp Pro Ile Leu
35 40 45
Ala Arg Leu Gly Arg Phe Ala Gly Arg Leu Gly Lys Ser Ser Arg Arg
50 55 60
Arg Ala Leu Ile Asn Leu Ser Leu Cys Phe Pro Glu Arg Ser Glu Ala
65 70 75 80
Glu Arg Glu Ala Ile Val Asp Glu Met Phe Ala Thr Ala Pro Gln Ala
85 90 95
Met Ala Met Met Ala Glu Leu Ala Ile Arg Gly Pro Glu Lys Ile Gln
100 105 110
Pro Arg Val Asp Trp Gln Gly Leu Glu Ile Ile Glu Glu Met Arg Arg
115 120 125
Asn Asn Glu Lys Val Ile Phe Leu Val Pro His Gly Trp Ala Val Asp
130 135 140
Ile Pro Ala Met Leu Met Ala Ser Gln Gly Gln Lys Met Ala Ala Met
145 150 155 160
Phe His Asn Gln Gly Asn Pro Val Phe Asp Tyr Val Trp Asn Thr Val
165 170 175
Arg Arg Arg Phe Gly Gly Arg Leu His Ala Arg Asn Asp Gly Ile Lys
180 185 190
Pro Phe Ile Gln Ser Val Arg Gln Gly Tyr Trp Gly Tyr Tyr Leu Pro
195 200 205
Asp Gln Asp His Gly Pro Glu His Ser Glu Phe Val Asp Phe Phe Ala
210 215 220
Thr Tyr Lys Ala Thr Leu Pro Ala Ile Gly Arg Leu Met Lys Val Cys
225 230 235 240
Arg Ala Arg Val Val Pro Leu Phe Pro Ile Tyr Asp Gly Lys Thr His
245 250 255
Arg Leu Thr Ile Gln Val Arg Pro Pro Met Asp Asp Leu Leu Glu Ala
260 265 270
Asp Asp His Thr Ile Ala Arg Arg Met Asn Glu Glu Val Glu Ile Phe
275 280 285
Val Gly Pro Arg Pro Glu Gln Tyr Thr Trp Ile Leu Lys Leu Leu Lys
290 295 300
Thr Arg Lys Pro Gly Glu Ile Gln Pro Tyr Lys Arg Lys Asp Leu Tyr
305 310 315 320
Pro Ile Lys
<210> 6
<211> 972
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Escherichia coli LpxM polypeptide
<400> 6
atggaaacga aaaaaaataa tagcgaatac attcctgagt ttgataaatc ctttcgccac 60
ccgcgctact ggggagcatg gctgggcgta gcagcgatgg cgggtatcgc tttaacgccg 120
ccaaagttcc gtgatcccat tctggcacgg ctgggacgtt ttgccggacg actgggaaaa 180
agctcacgcc gtcgtgcgtt aatcaatctg tcgctctgct ttccagaacg tagtgaagct 240
gaacgcgaag cgattgttga tgagatgttt gccaccgcgc cgcaagcgat ggcaatgatg 300
gctgagttgg caatacgcgg gccggagaaa attcagccgc gcgttgactg gcaagggctg 360
gagatcatcg aagagatgcg gcgtaataac gagaaagtta tctttctggt gccgcacggt 420
tgggccgtcg atattcctgc catgctgatg gcctcgcaag ggcagaaaat ggcagcgatg 480
ttccataatc agggcaaccc ggtttttgat tatgtctgga acacggtgcg tcgtcgcttt 540
ggcggtcgtc tgcatgcgag aaatgacggt attaaaccat tcatccagtc ggtacgtcag 600
gggtactggg gatattattt acccgatcag gatcatggcc cagagcacag cgaatttgtg 660
gatttctttg ccacctataa agcgacgttg cccgcgattg gtcgtttgat gaaagtgtgc 720
cgtgcgcgcg ttgtaccgct gtttccgatt tatgatggca agacgcatcg tctgacgatt 780
caggtgcgcc caccgatgga tgatctgtta gaggcggatg atcatacgat tgcgcggcgg 840
atgaatgaag aagtcgagat ttttgttggt ccgcgaccag aacaatacac ctggatacta 900
aaattgctga aaactcgcaa accgggcgaa atccagccgt ataagcgcaa agatctttat 960
cccatcaaat aa 972
<210> 7
<211> 67
<212> DNA
<213> Artificial Sequence
<220>
<223> LpxM forward primer P3
<400> 7
gaaggcgttc cttcacgcta ttaataagaa ggagatatac caatggaaac gaaaaaaaat 60
aatagcg 67
<210> 8
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> LpxM reverse primer P3
<400> 8
gcagaagctt ttatttgatg ggataaagat ctttgcg 37
<210> 9
<211> 174
<212> PRT
<213> Artificial Sequence
<220>
<223> Aquifex aeolicus LpxE polypeptide
<400> 9
Met Val Ile Glu Asp Phe Ala Leu Asn Leu Glu Leu Phe Arg Leu Ile
1 5 10 15
Asn Asn Ala Arg His Pro Leu Leu Asp Val Phe Phe Thr His Phe Ala
20 25 30
Tyr Leu Gly Ser Gly Tyr Val Leu Phe Pro Leu Leu Ile Phe Leu Phe
35 40 45
Ile Phe Arg Lys Glu Lys Val Lys Pro Leu Ile Leu Ala Ile Ile Leu
50 55 60
Glu Thr Val Leu Val Ile Ser Leu Lys Thr Phe Phe Asn Gln Pro Arg
65 70 75 80
Pro Ala Ile Leu Leu Glu Asp Val Asn Leu Leu Phe Pro Leu His Trp
85 90 95
Arg Ser Phe Pro Ser Gly Asp Thr Ala Met Ala Phe Thr Ile Ala Thr
100 105 110
Val Leu Ser His Gly Glu Lys Leu His Ile Lys Ala Ile Leu Phe Leu
115 120 125
Tyr Ala Phe Leu Ile Gly Tyr Glu Arg Ile Tyr Ala Gly Val His Phe
130 135 140
Pro Leu Asp Val Phe Val Gly Ala Leu Ile Gly Ile Ile Cys Gly Ile
145 150 155 160
Ile Ser Leu Lys Tyr Ser Lys Gly Gly Val Tyr Glu Arg Asn
165 170
<210> 10
<211> 525
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Aquifex aeolicus LpxE polypeptide
<400> 10
atggtaatag aagactttgc tctaaacctt gaactttttc gtttgataaa caatgcgagg 60
catccacttc tggacgtttt ctttactcac tttgcttacc tcggttcggg ctatgtactg 120
tttcctttat taatctttct ttttattttc agaaaggaaa aagtaaagcc tttgatttta 180
gcgataattt tggaaacagt tttagtgatc tctctaaaaa catttttcaa ccagccgaga 240
cctgcaattt tactcgagga tgtaaactta ctttttcctt tacactggcg ttcctttccc 300
tcaggagaca ccgctatggc ttttacgata gcaactgtac tgtcacatgg tgaaaaactc 360
catataaagg caatactctt cttatacgct ttcctaatag ggtatgagag gatttacgcg 420
ggtgttcact ttcctctgga cgtttttgta ggagctctga ttggaattat ttgcggtatt 480
atttctttaa aatattccaa gggaggtgtg tatgagagaa attag 525
<210> 11
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> AaLpxE forward primer
<400> 11
gcatgccata tgatggtaat agaagacttt gctctaaac 39
<210> 12
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> AaLpxE reverse primer
<400> 12
attagcctcg agatttctct catacacacc tccctt 36
<210> 13
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> AaLpxE forward primer
<400> 13
taatacgact cactataggg 20
<210> 14
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> AaLpxE reverse primer
<400> 14
gcagaagctt ctaatttctc tcatacacac ctccc 35
<210> 15
<211> 573
<212> DNA
<213> Artificial Sequence
<220>
<223> Helicobactor pylori LpxE sequence
<400> 15
atgaaaaaat tcttatttaa acaaaaattt tgtgaaagcc tgcccaaaag cttttctaaa 60
actttgttag cgctcagttt gggcttgatt ttattaggca tttttgcgcc tttccctaaa 120
gtccctaaac agcctagcgt gcctttaatg tttcatttca ccgagcatta tgcgcgcttt 180
atccctacga ttttatctgt ggcgattccc ttaatccaaa gagatgcggt agggcttttt 240
caagtcgcta acgcttctat cgctacaacc cttctcacgc acaccaccaa aagagcctta 300
aaccatgtaa caatcaacga tcagcgtttg ggcgagcgcc cttatggagg taatttcaac 360
atgccaagcg ggcattcgtc tatggtgggt ttggcggtgg cgtttttaat gcgccgctat 420
tcttttaaaa aatacttttg gctcttgccc ctagtccctt tgaccatgct cgctcgcatt 480
tatttagaca tgcacaccat tggcgcggtg ctgaccgggc ttggcgttgg aatgttgtgc 540
gtaagccttt ttacaagccc caaaaagcct taa 573
<210> 16
<211> 573
<212> DNA
<213> Artificial Sequence
<220>
<223> Mutated Helicobactor pylori LpxE sequence (A49T, G50C, and
C51G)
<400> 16
atgaaaaaat tcttatttaa acaaaaattt tgtgaaagcc tgcccaaatc gttttctaaa 60
actttgttag cgctcagttt gggcttgatt ttattaggca tttttgcgcc tttccctaaa 120
gtccctaaac agcctagcgt gcctttaatg tttcatttca ccgagcatta tgcgcgcttt 180
atccctacga ttttatctgt ggcgattccc ttaatccaaa gagatgcggt agggcttttt 240
caagtcgcta acgcttctat cgctacaacc cttctcacgc acaccaccaa aagagcctta 300
aaccatgtaa caatcaacga tcagcgtttg ggcgagcgcc cttatggagg taatttcaac 360
atgccaagcg ggcattcgtc tatggtgggt ttggcggtgg cgtttttaat gcgccgctat 420
tcttttaaaa aatacttttg gctcttgccc ctagtccctt tgaccatgct cgctcgcatt 480
tatttagaca tgcacaccat tggcgcggtg ctgaccgggc ttggcgttgg aatgttgtgc 540
gtaagccttt ttacaagccc caaaaagcct taa 573
<210> 17
<211> 190
<212> PRT
<213> Artificial Sequence
<220>
<223> Helicobactor pylori LpxE Amino acid sequence
<400> 17
Met Lys Lys Phe Leu Phe Lys Gln Lys Phe Cys Glu Ser Leu Pro Lys
1 5 10 15
Ser Phe Ser Lys Thr Leu Leu Ala Leu Ser Leu Gly Leu Ile Leu Leu
20 25 30
Gly Ile Phe Ala Pro Phe Pro Lys Val Pro Lys Gln Pro Ser Val Pro
35 40 45
Leu Met Phe His Phe Thr Glu His Tyr Ala Arg Phe Ile Pro Thr Ile
50 55 60
Leu Ser Val Ala Ile Pro Leu Ile Gln Arg Asp Ala Val Gly Leu Phe
65 70 75 80
Gln Val Ala Asn Ala Ser Ile Ala Thr Thr Leu Leu Thr His Thr Thr
85 90 95
Lys Arg Ala Leu Asn His Val Thr Ile Asn Asp Gln Arg Leu Gly Glu
100 105 110
Arg Pro Tyr Gly Gly Asn Phe Asn Met Pro Ser Gly His Ser Ser Met
115 120 125
Val Gly Leu Ala Val Ala Phe Leu Met Arg Arg Tyr Ser Phe Lys Lys
130 135 140
Tyr Phe Trp Leu Leu Pro Leu Val Pro Leu Thr Met Leu Ala Arg Ile
145 150 155 160
Tyr Leu Asp Met His Thr Ile Gly Ala Val Leu Thr Gly Leu Gly Val
165 170 175
Gly Met Leu Cys Val Ser Leu Phe Thr Ser Pro Lys Lys Pro
180 185 190
<210> 18
<211> 55
<212> DNA
<213> Artificial Sequence
<220>
<223> HpLpxE forward primer
<400> 18
gatcctctag aaaggagata tattgatgaa aaaattctta tttaaacaaa aattt 55
<210> 19
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> HpLpxE reverse primer
<400> 19
agctacaagc ttttaaggct ttttggggc 29
<210> 20
<211> 237
<212> PRT
<213> Artificial Sequence
<220>
<223> Escherichia coli LpxT polypeptide
<400> 20
Met Ile Lys Asn Leu Pro Gln Ile Val Leu Leu Asn Ile Val Gly Leu
1 5 10 15
Ala Leu Phe Leu Ser Trp Tyr Ile Pro Val Asn His Gly Phe Trp Leu
20 25 30
Pro Ile Asp Ala Asp Ile Phe Tyr Phe Phe Asn Gln Lys Leu Val Glu
35 40 45
Ser Lys Ala Phe Leu Trp Leu Val Ala Leu Thr Asn Asn Arg Ala Phe
50 55 60
Asp Gly Cys Ser Leu Leu Ala Met Gly Met Leu Met Leu Ser Phe Trp
65 70 75 80
Leu Lys Glu Asn Ala Pro Gly Arg Arg Arg Ile Val Ile Ile Gly Leu
85 90 95
Val Met Leu Leu Thr Ala Val Val Leu Asn Gln Leu Gly Gln Ala Leu
100 105 110
Ile Pro Val Lys Arg Ala Ser Pro Thr Leu Thr Phe Thr Asp Ile Asn
115 120 125
Arg Val Ser Glu Leu Leu Ser Val Pro Thr Lys Asp Ala Ser Arg Asp
130 135 140
Ser Phe Pro Gly Asp His Gly Met Met Leu Leu Ile Phe Ser Ala Phe
145 150 155 160
Met Trp Arg Tyr Phe Gly Lys Val Ala Gly Leu Ile Ala Leu Ile Ile
165 170 175
Phe Val Val Phe Ala Phe Pro Arg Val Met Ile Gly Ala His Trp Phe
180 185 190
Thr Asp Ile Ile Val Gly Ser Met Thr Val Ile Leu Ile Gly Leu Pro
195 200 205
Trp Val Leu Leu Thr Pro Leu Ser Asp Arg Leu Ile Thr Phe Phe Asp
210 215 220
Lys Ser Leu Pro Gly Lys Asn Lys His Phe Gln Asn Lys
225 230 235
<210> 21
<211> 714
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Escherichia coli LpxT polypeptide
<400> 21
atgattaaaa atttgccgca aatagtgttg ttgaatattg tcggcctcgc gctgtttctt 60
tcctggtata tccccgttaa tcatggattc tggttgccga ttgatgcgga tattttttat 120
ttctttaatc agaaactggt cgaaagtaag gcctttttgt ggctggttgc attgaccaac 180
aatcgcgcct tcgacggttg ttcactgctg gcgatgggta tgttgatgct gagtttctgg 240
ctgaaagaaa acgcccctgg cagacgacgt atcgtgatta ttggtctggt catgctatta 300
actgcagtgg tattaaacca gctgggtcag gcattaattc ctgtaaaacg ggccagccca 360
acattgactt ttaccgatat taaccgcgtc agcgaactgc tctctgttcc cacgaaagat 420
gcctcacgag atagctttcc cggcgatcac ggcatgatgc tgcttatttt ttcggcattc 480
atgtggcgtt atttcggcaa agttgcaggc cttatcgccc ttattatttt tgtggttttt 540
gcatttccca gagtaatgat tggcgcacac tggtttactg acatcattgt cggttcgatg 600
accgtgatat tgatcggttt gccctgggtg ttgctgacgc cattaagtga tcgattaatc 660
accttttttg acaaatcact accaggaaaa aacaaacatt tccaaaacaa ataa 714
<210> 22
<211> 425
<212> PRT
<213> Artificial Sequence
<220>
<223> Escherichia coli KdtA polypeptide
<400> 22
Met Leu Glu Leu Leu Tyr Thr Ala Leu Leu Tyr Leu Ile Gln Pro Leu
1 5 10 15
Ile Trp Ile Arg Leu Trp Val Arg Gly Arg Lys Ala Pro Ala Tyr Arg
20 25 30
Lys Arg Trp Gly Glu Arg Tyr Gly Phe Tyr Arg His Pro Leu Lys Pro
35 40 45
Gly Gly Ile Met Leu His Ser Val Ser Val Gly Glu Thr Leu Ala Ala
50 55 60
Ile Pro Leu Val Arg Ala Leu Arg His Arg Tyr Pro Asp Leu Pro Ile
65 70 75 80
Thr Val Thr Thr Met Thr Pro Thr Gly Ser Glu Arg Val Gln Ser Ala
85 90 95
Phe Gly Lys Asp Val Gln His Val Tyr Leu Pro Tyr Asp Leu Pro Asp
100 105 110
Ala Leu Asn Arg Phe Leu Asn Lys Val Asp Pro Lys Leu Val Leu Ile
115 120 125
Met Glu Thr Glu Leu Trp Pro Asn Leu Ile Ala Ala Leu His Lys Arg
130 135 140
Lys Ile Pro Leu Val Ile Ala Asn Ala Arg Leu Ser Ala Arg Ser Ala
145 150 155 160
Ala Gly Tyr Ala Lys Leu Gly Lys Phe Val Arg Arg Leu Leu Arg Arg
165 170 175
Ile Thr Leu Ile Ala Ala Gln Asn Glu Glu Asp Gly Ala Arg Phe Val
180 185 190
Ala Leu Gly Ala Lys Asn Asn Gln Val Thr Val Thr Gly Ser Leu Lys
195 200 205
Phe Asp Ile Ser Val Thr Pro Gln Leu Ala Ala Lys Ala Val Thr Leu
210 215 220
Arg Arg Gln Trp Ala Pro His Arg Pro Val Trp Ile Ala Thr Ser Thr
225 230 235 240
His Glu Gly Glu Glu Ser Val Val Ile Ala Ala His Gln Ala Leu Leu
245 250 255
Gln Gln Phe Pro Asn Leu Leu Leu Ile Leu Val Pro Arg His Pro Glu
260 265 270
Arg Phe Pro Asp Ala Ile Asn Leu Val Arg Gln Ala Gly Leu Ser Tyr
275 280 285
Ile Thr Arg Ser Ser Gly Glu Val Pro Ser Thr Ser Thr Gln Val Val
290 295 300
Val Gly Asp Thr Met Gly Glu Leu Met Leu Leu Tyr Gly Ile Ala Asp
305 310 315 320
Leu Ala Phe Val Gly Gly Ser Leu Val Glu Arg Gly Gly His Asn Pro
325 330 335
Leu Glu Ala Ala Ala His Ala Ile Pro Val Leu Met Gly Pro His Thr
340 345 350
Phe Asn Phe Lys Asp Ile Cys Ala Arg Leu Glu Gln Ala Ser Gly Leu
355 360 365
Ile Thr Val Thr Asp Ala Thr Thr Leu Ala Lys Glu Val Ser Ser Leu
370 375 380
Leu Thr Asp Ala Asp Tyr Arg Ser Phe Tyr Gly Arg His Ala Val Glu
385 390 395 400
Val Leu Tyr Gln Asn Gln Gly Ala Leu Gln Arg Leu Leu Gln Leu Leu
405 410 415
Glu Pro Tyr Leu Pro Pro Lys Thr His
420 425
<210> 23
<211> 1278
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Escherichia coli KdtA polypeptide
<400> 23
atgctcgaat tgctttacac cgcccttctc taccttattc agccgctgat ctggatacgg 60
ctctgggtgc gcggacgtaa ggctccggcc tatcgaaaac gctggggtga acgttacggt 120
ttttaccgcc atccgctaaa accaggcggc attatgctgc actccgtctc cgtcggtgaa 180
actctggcgg caatcccgtt ggtgcgcgcg ctgcgtcatc gttatcctga tttaccgatt 240
accgtaacaa ccatgacgcc aaccggttcg gagcgcgtac aatcggcttt cgggaaggat 300
gttcagcacg tttatctgcc gtatgatctg cccgatgcac tcaaccgttt cctgaataaa 360
gtcgacccta aactggtgtt gattatggaa accgaactat ggcctaacct gattgcggcg 420
ctacataaac gtaaaattcc gctggtgatc gctaacgcgc gactctctgc ccgctcggcc 480
gcaggttatg ccaaactggg taaattcgtc cgtcgcttgc tgcgtcgtat tacgctgatt 540
gctgcgcaaa atgaagaaga tggtgcacgt tttgtggcgc tgggcgcaaa aaataatcag 600
gtgaccgtta ccggtagcct gaaattcgat atttctgtaa cgccgcagtt ggctgctaaa 660
gccgtgacgc tgcgccgcca gtgggcacca caccgcccgg tatggattgc caccagcact 720
cacgaaggcg aagagagtgt ggtgatcgcc gcacatcagg cattgttaca gcaattcccg 780
aatttattgc tcatcctggt accccgtcat ccggaacgct tcccggatgc gattaacctt 840
gtccgccagg ctggactaag ctatatcaca cgctcttcag gggaagtccc ctccaccagc 900
acgcaggttg tggttggcga tacgatgggc gagttgatgt tactgtatgg cattgccgat 960
ctcgcctttg ttggcggttc actggttgaa cgtggtgggc ataatccgct ggaagctgcc 1020
gcacacgcta ttccggtatt gatggggccg catactttta actttaaaga catttgcgcg 1080
cggctggagc aggcaagcgg gctgattacc gttaccgatg ccactacgct tgcaaaagag 1140
gtttcctctt tactcaccga cgccgattac cgtagtttct atggccgtca tgccgttgaa 1200
gtactgtatc aaaaccaggg cgcgctacag cgtctgcttc aactgctgga accttacctg 1260
ccaccgaaaa cgcattga 1278
<210> 24
<211> 239
<212> PRT
<213> Artificial Sequence
<220>
<223> Francisella novicida LpxE polypeptide
<400> 24
Met Leu Lys Gln Thr Leu Gln Thr Asn Phe Gln Gly Phe Lys Asp Ile
1 5 10 15
Phe Lys Lys Pro Lys Leu His Asn His Lys Leu Pro Arg Tyr Leu Gln
20 25 30
Leu Lys Tyr Thr Phe Ile Pro Leu Leu Ile Leu Val Ile Phe Ala Tyr
35 40 45
Tyr Asn Leu Asp Thr Pro Val Glu Asn Tyr Ile Lys His Ser Met Pro
50 55 60
Asn Ile Val Gly Val Ile Phe Gly Lys Ile Thr Asp Val Gly Lys Ala
65 70 75 80
Glu Tyr Ile Leu Ile Ile Cys Gly Val Ile Val Leu Ala Arg Leu Phe
85 90 95
Thr Asp Ser Gln Lys Leu Ser Ala Asn Thr Arg Ala Met Phe Asp Lys
100 105 110
Val Ser Ala Tyr Ala Gly Phe Ile Leu Ala Thr Val Ala Ile Ser Gly
115 120 125
Ile Leu Gly Gln Ile Leu Lys Met Ile Ile Gly Arg Ala Arg Pro Lys
130 135 140
Phe Phe Leu Glu Tyr Gly Ser His Tyr Phe Gln His Phe His Ala Pro
145 150 155 160
Gly Tyr Asp Phe Ala Ser Met Pro Ser Gly His Ser Ile Thr Val Gly
165 170 175
Ala Met Phe Ile Ala Phe Phe Tyr Ile Phe Pro Lys Leu Arg Tyr Phe
180 185 190
Trp Tyr Leu Leu Ile Val Val Phe Ala Gly Ser Arg Ile Met Val Gly
195 200 205
Ser His Tyr Pro Ser Asp Val Ile Phe Gly Val Ala Phe Gly Cys Tyr
210 215 220
Cys Thr Ala Tyr Ile Tyr Tyr Trp Met Arg Asn Arg Glu Ile Ile
225 230 235
<210> 25
<211> 720
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Francisella novicida LpxE polypeptide
<400> 25
atgctcaaac agacattaca aacaaacttt caaggtttta aagatatttt taaaaaacca 60
aaactacaca atcataaatt gcctagatat ctacagttga aatatacgtt tataccatta 120
ttaattttgg taatttttgc atactataac ttagataccc cagttgagaa ctatatcaag 180
cattctatgc caaatattgt tggtgtaatt tttggtaaaa taactgatgt tggtaaggcc 240
gagtatattt tgataatttg cggtgtgata gtgttagcgc gtttatttac agatagccaa 300
aaattatctg ctaatactag agctatgttt gacaaggtgt cggcatatgc gggttttatc 360
ttagcaactg tagctattag tggtattttg ggacaaatac tcaagatgat aataggtaga 420
gcgcgtccta agtttttctt ggaatatggt tcgcattatt tccaacattt tcatgcacct 480
ggatatgatt ttgcaagtat gccgtcaggg cactcaatca cagttggagc aatgtttata 540
gcattttttt atattttccc taagctaaga tatttttggt atttgctgat agtggtattt 600
gctgggagta gaattatggt tggttcacat tatcctagtg atgtaatttt tggcgttgct 660
tttggttgtt actgtacagc atatatctac tattggatga gaaatagaga gattatttag 720
720
<210> 26
<211> 186
<212> PRT
<213> Artificial Sequence
<220>
<223> Escherichia coli PagP polypeptide
<400> 26
Met Asn Val Ser Lys Tyr Val Ala Ile Phe Ser Phe Val Phe Ile Gln
1 5 10 15
Leu Ile Ser Val Gly Lys Val Phe Ala Asn Ala Asp Glu Trp Met Thr
20 25 30
Thr Phe Arg Glu Asn Ile Ala Gln Thr Trp Gln Gln Pro Glu His Tyr
35 40 45
Asp Leu Tyr Ile Pro Ala Ile Thr Trp His Ala Arg Phe Ala Tyr Asp
50 55 60
Lys Glu Lys Thr Asp Arg Tyr Asn Glu Arg Pro Trp Gly Gly Gly Phe
65 70 75 80
Gly Leu Ser Arg Trp Asp Glu Lys Gly Asn Trp His Gly Leu Tyr Ala
85 90 95
Met Ala Phe Lys Asp Ser Trp Asn Lys Trp Glu Pro Ile Ala Gly Tyr
100 105 110
Gly Trp Glu Ser Thr Trp Arg Pro Leu Ala Asp Glu Asn Phe His Leu
115 120 125
Gly Leu Gly Phe Thr Ala Gly Val Thr Ala Arg Asp Asn Trp Asn Tyr
130 135 140
Ile Pro Leu Pro Val Leu Leu Pro Leu Ala Ser Val Gly Tyr Gly Pro
145 150 155 160
Val Thr Phe Gln Met Thr Tyr Ile Pro Gly Thr Tyr Asn Asn Gly Asn
165 170 175
Val Tyr Phe Ala Trp Met Arg Phe Gln Phe
180 185
<210> 27
<211> 561
<212> DNA
<213> Artificial Sequence
<220>
<223> Polynucleotide encoding Escherichia coli pagP polypeptide
<400> 27
atgaacgtga gtaaatatgt cgctatcttt tcctttgttt ttattcagtt aatcagcgtt 60
ggtaaagttt ttgctaacgc agatgagtgg atgacaacgt ttagagaaaa tattgcacaa 120
acctggcaac agcctgaaca ttatgattta tatattcctg ccatcacctg gcatgcacgt 180
ttcgcttacg acaaagaaaa aaccgatcgc tataacgagc gaccgtgggg tggcggtttt 240
ggcctgtcgc gttgggatga aaaaggaaac tggcatggcc tgtatgccat ggcatttaag 300
gactcgtgga acaaatggga accgattgcc ggatacggat gggaaagtac ctggcgaccg 360
ctggcggatg aaaattttca tttaggtctg ggattcaccg ctggcgtaac ggcacgcgat 420
aactggaatt acatccctct cccggttcta ctgccattgg cctccgtggg ttatggccca 480
gtgacttttc agatgaccta cattccgggt acctacaaca atggcaatgt gtactttgcc 540
tggatgcgct ttcagttttg a 561
<---















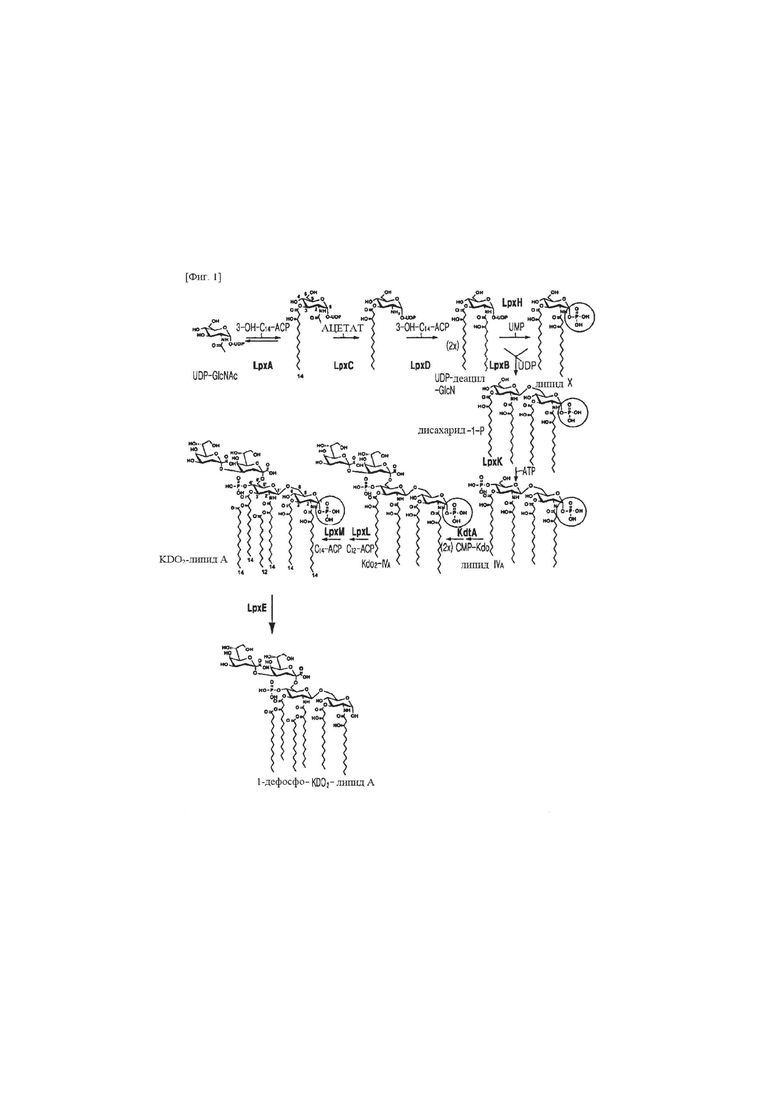
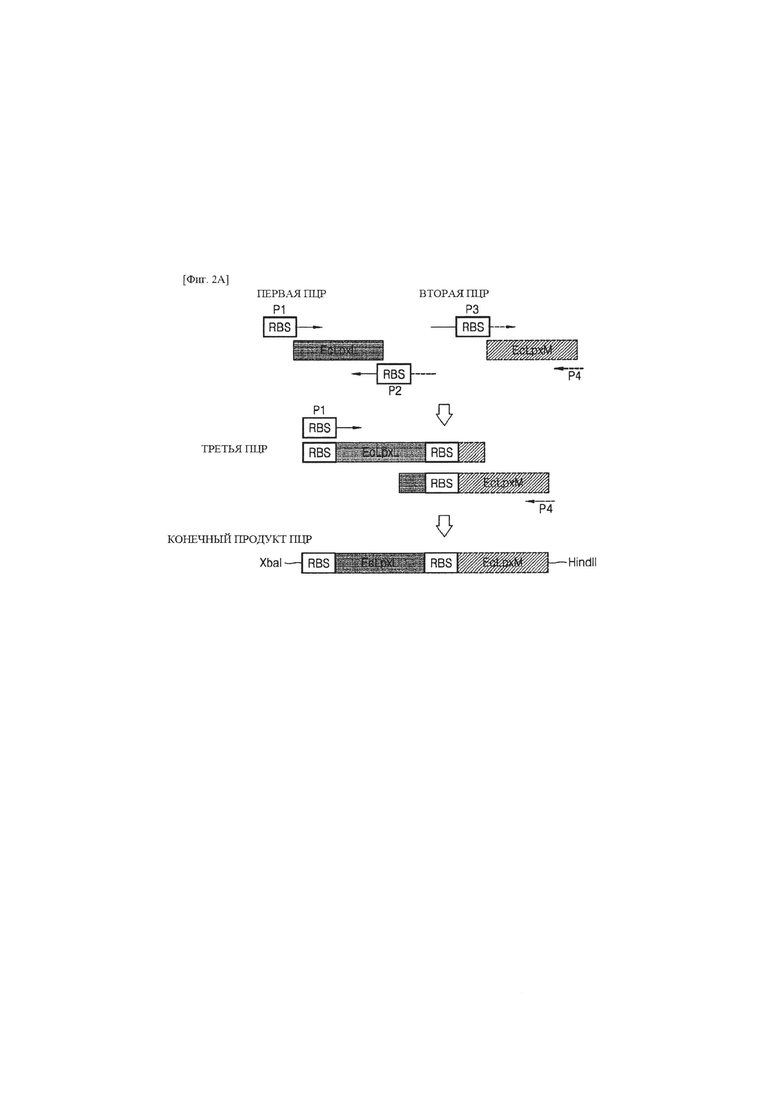
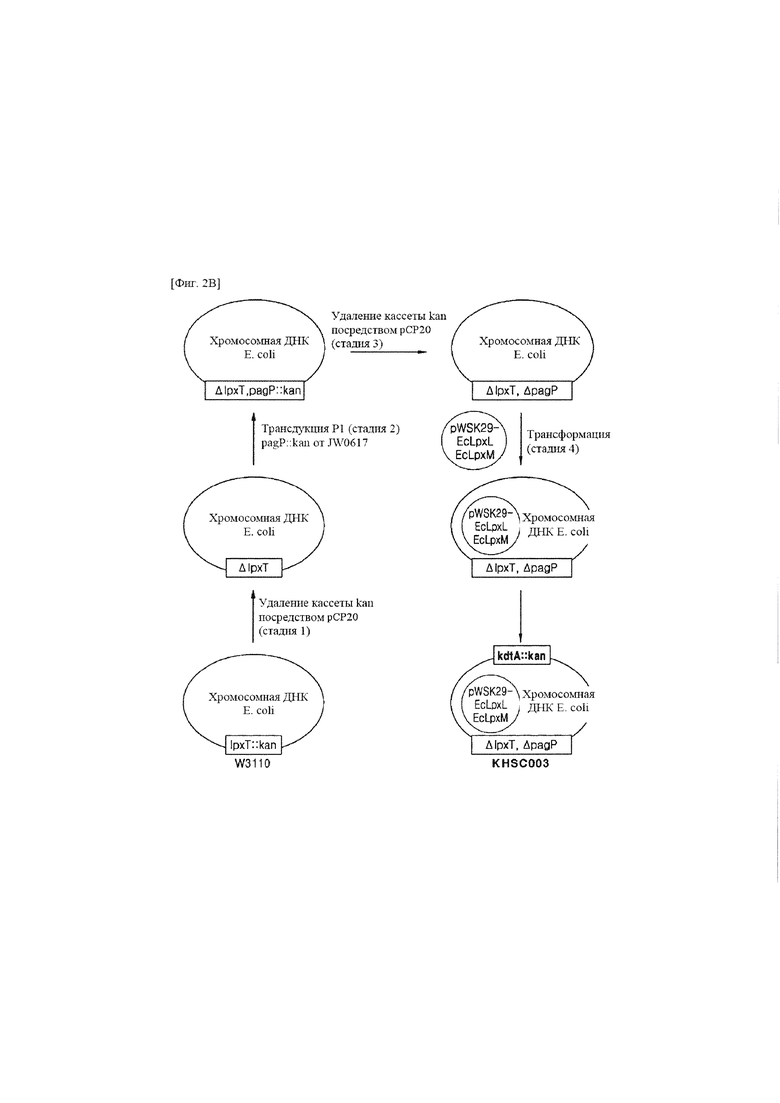
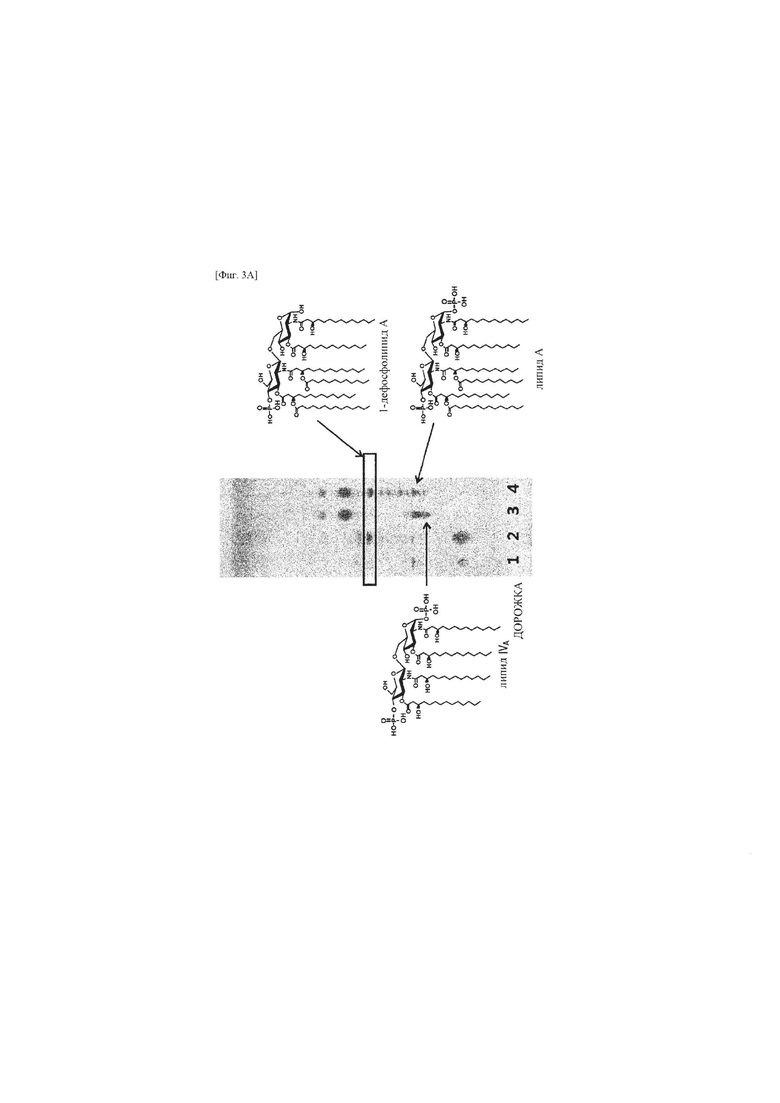
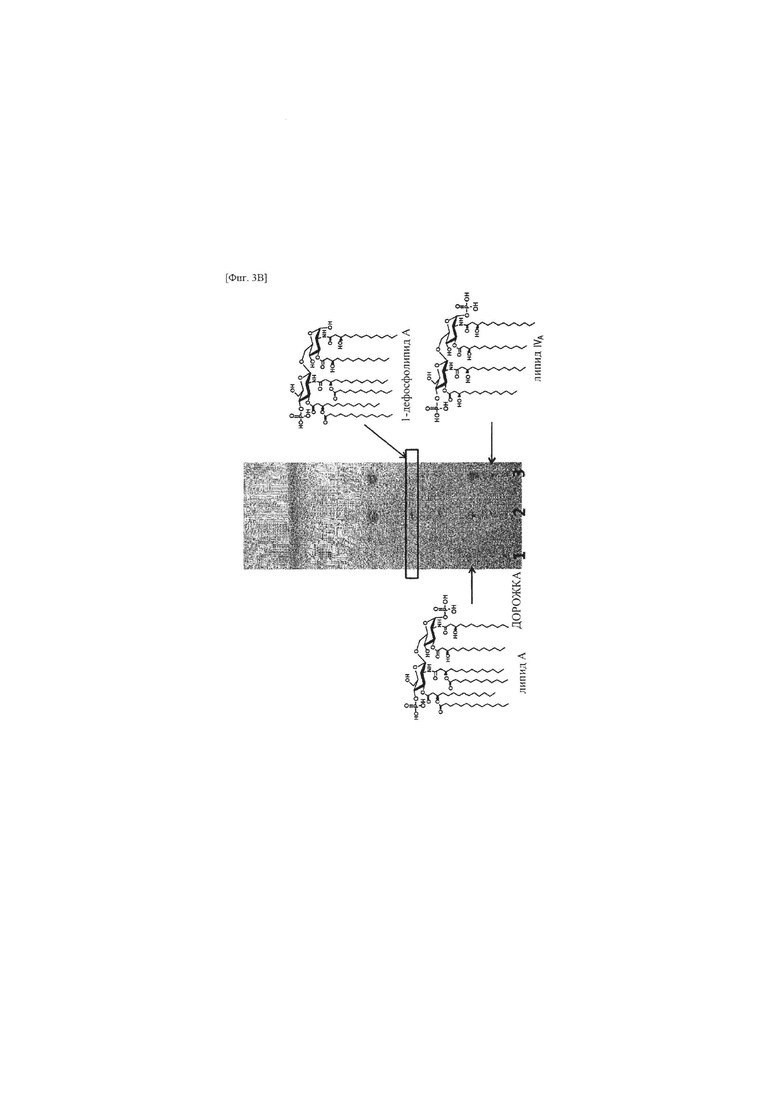
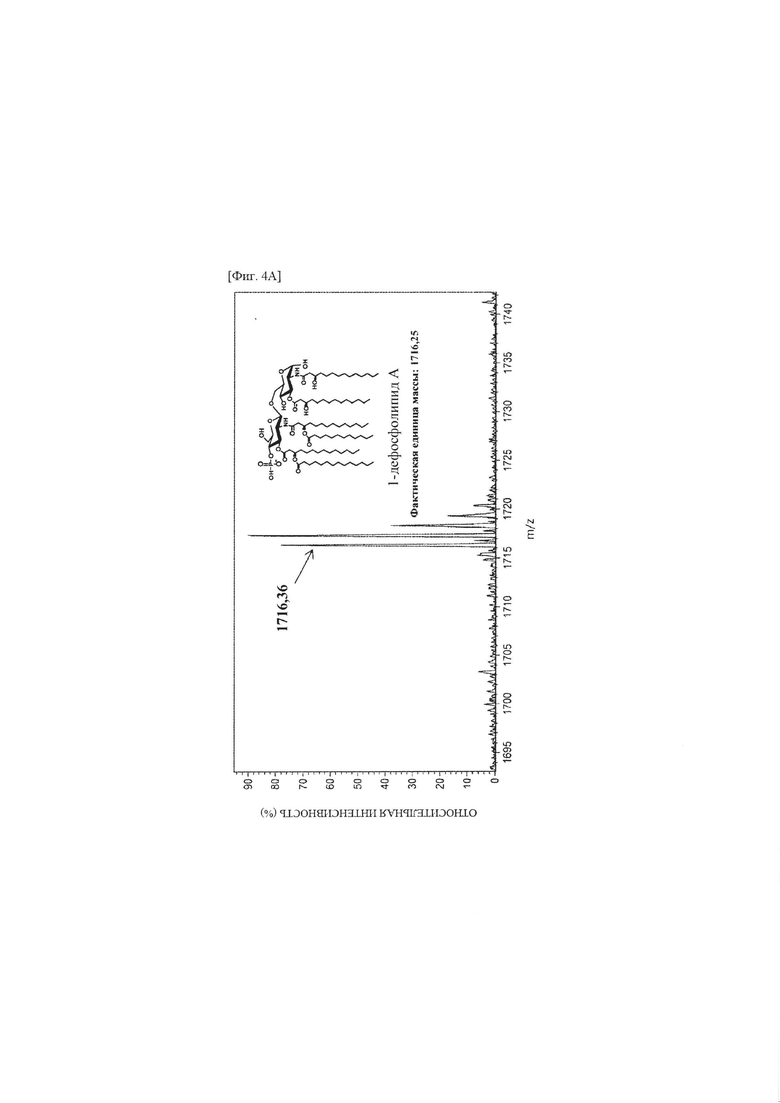
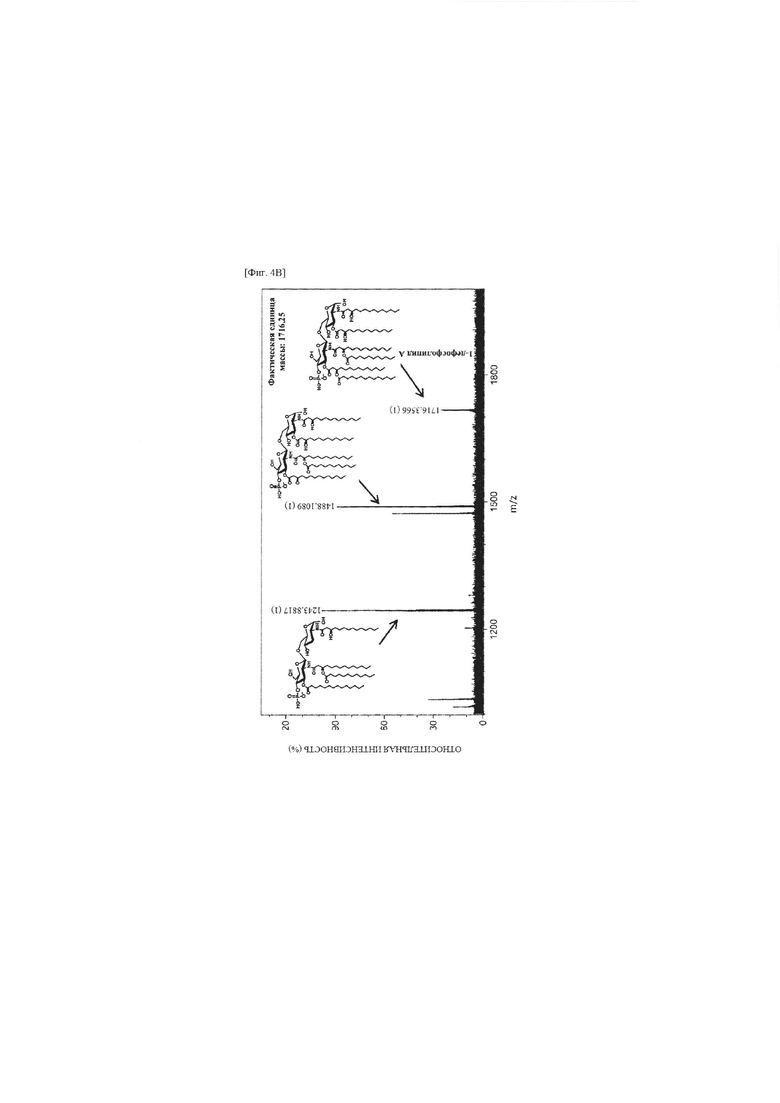
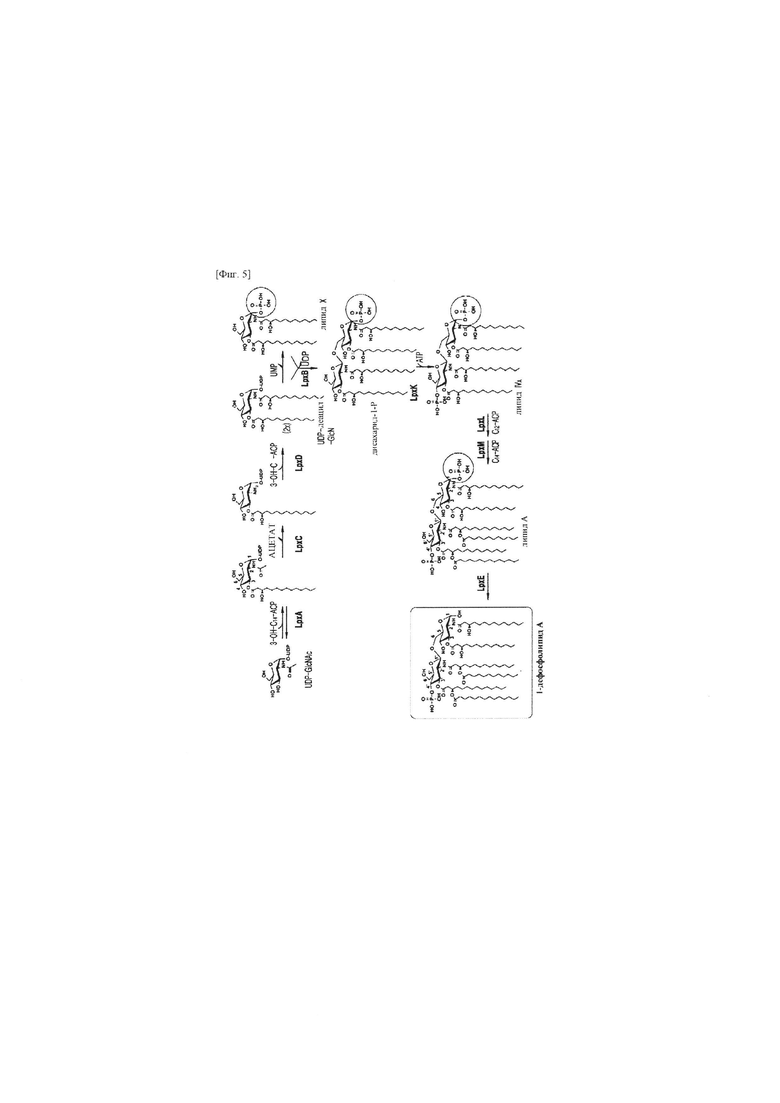
Группа изобретений относится к рекомбинантной бактерии, продуцирующей монофосфориллипид A (MLA), не конъюгированный с 2-кето-3-дезокси-D-маннооктулозонатом (Kdo), и способу получения MLA, не конъюгированного с Kdo, с использованием указанной бактерии. Предложена рекомбинантная бактерия, продуцирующая MLA, не конъюгированный с Kdo, трансформированная по меньшей мере одним экзогенным полинуклеотидом, кодирующим полипептид 1-фосфатазу липида А (LpxЕ). При этом указанная рекомбинантная бактерия модифицирована путем трансформации по меньшей мере одним из экзогенного полинуклеотида, кодирующего полипептид лауроилтрансферазу биосинтеза липида A (LpxL), экзогенного полинуклеотида, кодирующего полипептид миристоилтрансферазу биосинтеза липида A (LpxM), или их комбинацией. Указанная рекомбинантная бактерия обладает повышенной экспрессией полинуклеотида, кодирующего полипептид LpxL, полинуклеотида, кодирующего полипептид LpxM, или их комбинации по сравнению с соответствующей рекомбинантной бактерией без указанной модификации, и в которой по меньшей мере один из следующих генов удален либо экспрессия по меньшей мере одного из следующих генов снижена: ген, кодирующий полипептид KdtA, ген, кодирующий полипептид KdsB, ген, кодирующий полипептид KdsC, ген, кодирующий полипептид KdsА, ген, кодирующий полипептид GutQ, ген, кодирующий полипептид KpsF, ген, кодирующий полипептид KpsU, ген, кодирующий полипептид KdsD, или их комбинация. Предложен также способ получения MLA, не конъюгированного с Kdo, предусматривающий культивирование вышеуказанной бактерии с получением культуры и выделение MLA, не конъюгированного с Kdo, из культуры. 2 н. и 12 з.п. ф-лы, 5 ил., 3 пр.
1. Рекомбинантная бактерия, которая продуцирует монофосфориллипид A (MLA), не конъюгированный с 2-кето-3-дезокси-D-маннооктулозонатом (Kdo), где указанная рекомбинантная бактерия трансформирована по меньшей мере одним экзогенным полинуклеотидом, кодирующим полипептид 1-фосфатазу липида А (LpxЕ), где указанная рекомбинантная бактерия модифицирована путем трансформации по меньшей мере одним из экзогенного полинуклеотида, кодирующего полипептид лауроилтрансферазу биосинтеза липида A (LpxL), экзогенного полинуклеотида, кодирующего полипептид миристоилтрансферазу биосинтеза липида A (LpxM), или их комбинацией,
причем указанная рекомбинантная бактерия обладает повышенной экспрессией полинуклеотида, кодирующего полипептид LpxL, полинуклеотида, кодирующего полипептид LpxM, или их комбинации по сравнению с соответствующей рекомбинантной бактерией без указанной модификации, и
в которой по меньшей мере один из следующих генов удален либо экспрессия по меньшей мере одного из следующих генов снижена: ген, кодирующий полипептид KdtA, ген, кодирующий полипептид KdsB, ген, кодирующий полипептид KdsC, ген, кодирующий полипептид KdsА, ген, кодирующий полипептид GutQ, ген, кодирующий полипептид KpsF, ген, кодирующий полипептид KpsU, ген, кодирующий полипептид KdsD, или их комбинация.
2. Бактерия по п. 1, где MLA содержит от 2 ацильных цепей до 7 ацильных цепей.
3. Бактерия по п. 1, где MLA предусматривает 1-дефосфолипид A, 1-дефосфотетраацилированный липид A, 1-дефосфопентаацилированный липид A или их комбинацию.
4. Бактерия по п. 1, где полипептид LpxЕ принадлежит к EC 3.1.3.
5. Бактерия по п. 1, где полипептид LpxЕ представляет собой полипептид, обладающий активностью 1-фосфатазы липида А и содержащий аминокислотную последовательность, характеризующуюся приблизительно 90% или большей идентичностью последовательности по отношению к аминокислотной последовательности SEQ ID NO: 9 или SEQ ID NO: 17.
6. Бактерия по п. 1, где полипептид LpxL принадлежит к EC 2.3.1.241, а полипептид LpxM принадлежит к EC 2.3.1.243.
7. Бактерия по п. 1, где полипептид LpxL представляет собой полипептид, обладающий активностью лауроилтрансферазы биосинтеза липида A и содержащий аминокислотную последовательность, характеризующуюся приблизительно 90% или большей идентичностью последовательности по отношению к аминокислотной последовательности SEQ ID NO: 1, а полипептид LpxM представляет собой полипептид, обладающий активностью миристоилтрансферазы биосинтеза липида A и содержащий аминокислотную последовательность, характеризующуюся приблизительно 90% или большей идентичностью последовательности по отношению к аминокислотной последовательности SEQ ID NO: 5.
8. Бактерия по п. 1, где бактерия является грамотрицательной бактерией.
9. Бактерия по п. 1, где бактерия выбрана из группы, состоящей из бактерии рода Escherichia, бактерии рода Shigella, бактерии рода Salmonella, бактерии рода Campylobacter, бактерии рода Neisseria, бактерии рода Haemophilus, бактерии рода Aeromonas, бактерии рода Francisella, бактерии рода Yersinia, бактерии рода Klebsiella, бактерии рода Bordetella, бактерии рода Legionella, бактерии рода Corynebacterium, бактерии рода Citrobacter, бактерии рода Chlamydia, бактерии рода Brucella, бактерии рода Pseudomonas, бактерии рода Helicobacter, бактерии рода Burkholderia, бактерии рода Agrobacterium, бактерии рода Chlorobium, бактерии рода Rhodospirillum, бактерии рода Magnetospirillum, бактерии рода Chlorobaculum, бактерии рода Pelodictyon, бактерии рода Pseudovibro, бактерии рода Phaeospirillum, бактерии рода Syntrophobacter, бактерии рода Bradyrhizobium, бактерии рода Porphyromonas, бактерии рода Rhizobium, бактерии рода Mesorhizobium, бактерии рода Vibrio, бактерии рода Ralstonia, бактерии рода Limnohabitans и бактерии рода Thermodesulfobacterium.
10. Бактерия по п. 1, в которой по меньшей мере один из следующих генов удален либо экспрессия по меньшей мере одного из следующих генов снижена: ген, кодирующий полипептид LpxТ, ген, кодирующий полипептид PagP, ген, кодирующий полипептид KdtA, или их комбинация.
11. Способ получения MLA, не конъюгированного с 2-кето-3-дезокси-D-маннооктулозонатом (Kdo), предусматривающий культивирование бактерии по п. 1 с получением культуры и
выделение MLA, не конъюгированного с Kdo, из культуры.
12. Способ по п. 11, при этом выделение предусматривает выделение MLA, не конъюгированного с Kdo, из бактериальной клетки.
13. Способ по п. 11, где культивирование предусматривает культивирование в периодическом режиме, культуре с подпиткой или в непрерывном режиме.
14. Способ по п. 11, где MLA предусматривает 1-дефосфолипид A, 1-дефосфопентаацилированный липид A, 1-дефосфотетраацилированный липид A или их комбинацию.
| WANG B | |||
| ET AL | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| PLoS ONE, 2015, 10(12): e0144714, pp | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| US 20130230555 A1, 05.09.2013 | |||
| CN 104844665 A, 19.08.2015 | |||
| СПОСОБ ПОЛУЧЕНИЯ ЛИПОПОЛИСАХАРИДНОЙ КОМПОЗИЦИИ, СОДЕРЖАЩЕЙ ЛИПИД А, ЛИПОПОЛИСАХАРИДНАЯ КОМПОЗИЦИЯ, СОДЕРЖАЩАЯ ЛИПИД А, 3-O-ДЕЗАКТИВИРОВАННЫЙ 4`-МОНОФОСФОРИЛЛИПИД А | 2002 |
|
RU2302463C2 |
Авторы
Даты
2020-07-17—Публикация
2016-12-16—Подача