Область техники
Настоящее изобретение относится к области адаптивной клеточной иммунотерапии. Оно направлен на повышение функциональности первичных иммунных клеток против патологий, которые развивают иммунную резистентность, таких как опухоли, тем самым улучшая терапевтический потенциал этих иммунных клеток. В частности, способ по настоящему изобретению предусматривает генетическую инсерцию экзогенной кодирующей последовательности (последовательностей), кодирующих ингибиторы NK, для предотвращения отторжения аллогенных Т-клеток от атаки клеток NK пациента и содействия приживлению указанных Т-клеток, особенно если они происходят от доноров. Эти экзогенные кодирующие последовательности, в частности, встраивают в геном клетки под контроль транскрипции эндогенных генных промоторов, которые активируются при активации иммунных клеток, при микроокружении опухоли или при угрожающих жизни воспалительных состояниях, или с промоторов, которые нечувствительны к активации иммунных клеток, в частности, в локусе β2m. Настоящее изобретение также предусматривает специфичные в отношении последовательностей эндонуклеазы и ДНК-векторы, например, векторы AAV, для осуществления таких целевых инсерций в указанные определенные локусы. Способ по настоящему изобретению способствует улучшению терапевтического потенциала и безопасности сконструированных первичных иммунных клеток для их эффективного использования в клеточной терапии.
Предшествующий уровень техники
Эффективное клиническое применение первичных популяций иммунных клеток, включая гемопоэтические клеточные линии, в течение десятилетия было установлено рядом клинических испытаний против ряда патологий, в частности ВИЧ-инфекции и лейкемии (Tristen S.J. с соавт. Trends in Biotechnology. 29(11), 2011, 550-557).
Однако в большинстве таких клинических испытаний использовались иммунные клетки, в основном NK и Т-клетки, полученные от самих пациентов или от совместимых доноров, что накладывает некоторые ограничения в отношении количества доступных иммунных клеток, их пригодности и их эффективности для преодоления болезни, которая уже разработала стратегию, как обойти или уменьшить действие иммунной системы пациента.
Основным преимуществом от получения аллогенных иммунных клеток, являются универсальные иммунные клетки, которые в качестве терапевтических продуктов можно просто «взять с полки» и которые были получены путем редактирования генов (Poirot с соавт., Cancer Res. 75, 2015, 3853-3864). Эти универсальные иммунные клетки могут быть получены путем экспрессии специфической редкощепящей эндонуклеазы в иммунных клетках, происходящих от доноров, с эффектом разрушения путем двунитевого разрыва их самораспознаваемых генетических детерминант.
С момента появления на рубеже веков первых программируемых специфических в отношении последовательности реагентов, первоначально называемых мегануклеазами (Smith с соавт., Nucl. Acids Res. 34(22), 2006, е149), были разработаны различные типы специфичных в отношении последовательностей эндонуклеазных реагентов, предлагающих улучшенную специфичность, безопасность и надежность.
Нуклеазы TALE (WO2011072246), которые представляют собой продукт гибридизации TALE-связывающего домена с каталитическим доменом расщепления, были успешно применены к первичным иммунным клеткам, в частности к Т-клеткам мононуклеарных клеток периферической крови (МКПК). Такими TALE-нуклеазами, которые производят под названием TALEN®, являются те, которые в настоящее время используют для одновременной инактивации генных последовательностей в Т-клетках, происходящих от доноров, в частности, для получения аллогенных терапевтических Т-клеток, в которых нарушены гены, кодирующие рецептор Т-клеток (T-cell receptor, TCR) и CD52. Эти клетки могут быть наделены химерными антигенными рецепторами (chimeric antigen receptor, CAR) для лечения онкологических пациентов (US2013/0315884). TALE-нуклеазы являются в высокой степени специфичными реагентами, потому что они должны связывать ДНК парами в обязательной гетеродимерной форме для получения димеризации домена расщепления Fok-1.
Каждый из левого и правого гетеродимерных представителей распознает разные нуклеиновые последовательности длиной примерно от 14 до 20 п. о., вместе охватывающие последовательности-мишени со специфичностью в целом от 30 до 50 п.о.
Другие эндонуклеазные реагенты были разработаны на основе компонентов II типа прокариотических кластеризованных регулярно распределенных коротких палиндромных повторов (Clustered Regularly Interspaced Short Palindromic Repeats, CRISPR II) адаптивной иммунной системы бактерий S. pyogenes. Эта многокомпонентная система, называемая РНК-направляемой нуклеазной системой (Gasiunas с соавт.2012; Jinek с соавт.2012), включает представителей семейств эндонуклеаз Cas9 или Cpf1, связанных с молекулами направляющей РНК, которые способны направить указанную нуклеазу к некоторым специфическим последовательностям генома (Zetsche с соавт., Cell 163, 2015, 759-771). Такие программируемые РНК-направленные эндонуклеазы легко получить, потому что специфичность расщепления определяется последовательностью РНК-направляющей, которая может быть легко сконструирована и ее получение недорого. Специфичность CRISPR/Cas9, хотя и заключается в более коротких по сравнению с TAL-нуклеазами последовательностях и состоит примерно из 10 п.о., эти последовательности должны быть расположены рядом с конкретным мотивом (particular motif, РАМ) в целевой генетической последовательности. Подобные системы описаны с использованием одноцепочечного олигонуклеотида ДНК (ДНК-направляющей) в сочетании с белками Argonaute (Gao F. с соавт., 2016, doi:10.1038/nbt.3547).
Другие системы эндонуклеаз, производные от хоминг-эндонуклеаз (например, I-Onul или I-CreI), сочетаемых или не сочетаемых с TAL-нуклеазой (например, MegaTAL) или цинк-пальцевыми нуклеазами, также продемонстрировали специфичность, но пока в меньшей степени.
Параллельно, новые специфические свойства могут быть приданы иммунным клеткам посредством генетического переноса рецепторов трансгенных Т-клеток или так называемых химерных рецепторов антигенов (chimeric antigen receptor, CAR) (Jena с соавт., Blood. 116, 2010, 1035-1044). CAR являются рекомбинантными рецепторами, содержащими нацеливающий фрагмент, который связан с одним или несколькими сигнальными доменами в одной гибридизированной молекуле. В общем, связывающий фрагмент CAR состоит из антигенсвязывающего домена одноцепочечного антитела (single-chain antibody, scFv), содержащего вариабельные легкие и тяжелые фрагменты моноклонального антитела, соединенные гибким линкером. Также успешно использовали связывающие фрагменты, основанные на рецепторных или лигандных доменах. Сигнальные домены для CAR первого поколения происходят из цитоплазматической области CD3-зета или цепей Fc рецептора иммуноглобулина гамма. Было показано, что CAR первого поколения успешно перенаправляют цитотоксичность Т-клеток, однако они не способны обеспечить длительное расширение и противоопухолевую активность in vivo. Сигнальные домены из ко-стимулирующих молекул, включая CD28, ОХ-40 (CD134), ICOS и 4-1 ВВ (CD 137), были добавлены отдельно (второе поколение) или в комбинации (третье поколение) для повышения выживаемости и увеличения пролиферации CAR-модифицированных Т-клеток. CAR успешно допускают перенаправление Т-клеток против антигенов, экспрессируемых на поверхности опухолевых клеток различных злокачественных новообразований, включая лимфомы и солидные опухоли.
Недавно сконструированные Т-клетки, у которых разрушены Т-клеточные рецепторы (T-cell receptor, TCR) с использованием TALE-нуклеаз, наделенных химерным антигенным рецептором (CAR), нацеленным на злокачественный антиген CD 19, называемый продуктом «UCART19», показали терапевтический потенциал по меньшей мере у двух младенцев, у которых был не поддающийся лечению лейкоз (Nature 527, 2015, 146-147). Для получения таких клеток UCART19, TALE-нуклеазу временно экспрессировали в клетках после электропорации кэпированной мРНК для разрушения гена TCR, тогда как кассету, кодирующую рецептор химерного антигена (CAR CD19), интродуцировали случайным образом в геном с использованием ретровирусного вектора.
В этом более позднем подходе этапы инактивации генов и экспрессии рецептора химерного антигена выполняют независимо после индукции активации Т-клеток «ex-vivo».
Однако инженерия первичных иммунных клеток не обходится без каких-либо последствий для роста/физиологии таких клеток. В частности, одной из основных задач является устранение истощения/анергии клеток, которые значительно снижают их иммунную реакцию и продолжительность жизни. Это более вероятно происходит, когда клетки искусственно активируются перед введением пациенту инфузией. Это также тот случай, когда клетка наделена CAR, который излишне реакционно способен.
Чтобы избежать эти ловушки, авторы настоящего изобретения подумали о том, чтобы воспользоваться преимуществами регуляции транскрипции некоторых ключевых генов во время активации Т-клеток для экспрессии экзогенных генетических последовательностей, увеличивающих терапевтический потенциал иммунных клеток. Экзогенные генетические последовательности, которые должны экспрессироваться или совместно экспрессироваться при активации иммунных клеток, интродуцируют путем генной направленной инсерции с использованием специфических для последовательности эндонуклеазных реагентов таким образом, что их кодирующие последовательности транскрибировались под контролем эндогенных промоторов, присутствующих в указанных локусах. В другом варианте, локусы, которые не экспрессируются во время активации иммунных клеток, можно использовать в качестве «локусов безопасной гавани» для интеграции кассет экспрессии без каких-либо неблагоприятных последствий для генома.
Эти стратегии клеточной инженерии в соответствии с настоящим изобретением имеют тенденцию усиливать терапевтический потенциал первичных иммунных клеток в целом, в частности, путем увеличения их продолжительности жизни, персистенции и иммунной активности, а также путем ограничения истощения клеток. Изобретение может быть осуществлено на первичных клетках, взятых от пациентов, как часть стратегий аутологичного лечения, а также от доноров, как часть стратегий аллогенного лечения. Краткое описание изобретения
Негомологичное соединение концов (Non-homologous end-joining, NHEJ) и гомологически направленная репарация (homology-directed repair, HDR) являются двумя основными путями, используемыми для восстановления разрывов ДНК in vivo. Последний путь восстанавливает разрыв матрично-зависимым образом (HDR, естественно, использует сестринскую хроматиду в качестве матрицы для восстановления ДНК). Гомологичную рекомбинацию используют в течение десятилетий для точного редактирования геномов с целевыми модификациями ДНК с использованием экзогенно поставляемой матрицы донора. Искусственная генерация двухцепочечного разрыва (double strand break, DSB) в целевом местоположении с использованием редкощепящих эндонуклеаз значительно повышает эффективность гомологичной рекомбинации (например, US 8921332). Кроме того, совместная доставка редкощепящей эндонуклеазы вместе с матрицей донора, содержащей последовательности ДНК, гомологичные сайту разрыва, позволяет редактировать гены на основе HDR, например, осуществлять коррекцию генов или инсерцию генов. Однако такие методы не нашли широкого применения применительно к первичным иммунным клеткам, особенно CAR Т-клеткам, из-за нескольких технических ограничений: трудности трансфекции ДНК в клетки такого типа, приводящие к апоптозу, иммунные клетки имеют ограниченные продолжительность жизни и количество поколений, гомологичная рекомбинация происходит с низкой частотой.
До сих пор, специфичные в отношении последовательностей эндонуклеазы как реагенты преимущественно применяли в первичных иммунных клетках для инактивации гена (например, WO2013176915), используя NHEJ.
Адаптивный перенос CAR Т-клеток представляет многообещающую стратегию борьбы с большим количеством разных форм рака. Клинический результат такой терапии тесно связан со способностью эффекторных клеток приживаться, размножаться и специфически уничтожать опухолевые клетки у пациентов.
При оценке инфузии аллогенных CAR Т-клеток, реакций хозяин-против-трансплантата и трансплантат-против-хозяина следует избегать для предупреждения отторжения адаптивно трансфицированных клеток для минимизации повреждения ткани хозяина и выявлять значительные противоопухолевые результаты.
Настоящее изобретение предусматривает новую стратегию клеточной инженерии для решения вышеупомянутых вопросов путем успешной разработки β2m-недостаточных CAR Т-клеток, в которые экзогенная последовательность, кодирующая ингибитор NK, инсертирована сайт-направленным генным редактированием для ее экспрессии в ходе активации Т-клеток.
Важным достижением настоящего изобретения является помещение таких экзогенных последовательностей, кодирующих ингибитор NK, под контроль эндогенных промоторов, у которых способность к транскрипции не снижается в результате активации иммунных клеток.
В одном из предпочтительных вариантов осуществления настоящее изобретение основывается на проведении сайт-направленного редактирования гена в локусе β2m, в частности за счет инсерции гена (или множества генных инсерций) в клетки-мишени для того, чтобы указанная транскрипция интегрированного гена предпочтительно была под контролем эндогенного промотора указанного локуса β2m, предпочтительно для экспрессии вместо β2m. В другом варианте настоящее изобретение может основываться на осуществлении генного редактирования в первичных иммунных клетках для получения объединенной генной транскрипции под контролем эндогенного промотора, хотя поддержание экспрессии нативного гена через применение цис-регуляторных элементов (например, элементы 2А цис-активирующей гидролазы) или внутреннего участка посадки рибосомы (internal ribosome entry site, IRES) в матрице донора.
В других вариантах осуществления настоящее изобретение основывается на экспрессии химерного антигенного рецептора (chimeric antigen receptor, CAR) в локусе TCR или в отдельных локусах гена, которые активируются при активации иммунных клеток. Экзогенная последовательность (последовательности), кодирующая CAR, и последовательность (последовательности), кодирующая эндогенный ген, могут транскрибироваться совместно, например, путем разделения цис-регуляторными элементами (например, 2А цис-действующими гидролазными элементами) или внутренним участком посадки рибосомы (internal ribosome entry site, IRES), которые также интродуцированы. Например, экзогенные последовательности, кодирующие CAR, могут быть помещены под контроль транскрипции промотора эндогенных генов, которые активируются микроокружением опухоли, например, HIF1a, фактором транскрипции, а именно фактором, индуцируемым гипоксией, или арил-углеводородный рецептором (aryl hydrocarbon receptor, AhR), которые являются генными сенсорами, соответственно, индуцированными гипоксией и ксенобиотиками в тесном контакте с опухолями.
В предпочтительных вариантах осуществления настоящего изобретения метод включает стадию выработки двухнитевых разрывов в локусе, высоко транскрибируемых под действием микроокружения опухоли, путем экспрессии специфических для последовательности нуклеазных реагентов, таких как TALEN, ZFN или РНК-управляемые эндонуклеазы, в качестве одних из возможных примеров, в присутствии матрицы репарации ДНК, предпочтительно помещенной в вектор на основе AAV6. Эта матрица донора ДНК обычно включает в себя два плеча гомологии, встраивающие уникальные или множественные открытые рамки считывания (Open Reading Frames, ORF) и регуляторные генетические элементы (последовательности стоп-кодона и полиА).
Экзогенные последовательности, кодирующие ингибиторы NK, предпочтительно содержат последовательности, кодирующие не полиморфные молекулы класса I или вирусные эвазины, например, UL18 [Uniprot #F5HFB4] и UL16 [также обозначаемый ULBP1 - Uniprot #Q9BZM6], фрагменты или их гибриды.
В предпочтительном варианте осуществления настоящего изобретения указанная экзогенная последовательность кодирует полипептид, который по меньшей мере на 80% по аминокислотной последовательности идентичен HLA-G, или HLA-E, или его функциональному варианту.
Такие экзогенные последовательности могут быть интродуцированы в геном путем делеции или модификации эндогенных кодирующих последовательностей (последовательности), присутствующих в указанном локусе (нокаут за счет нокина), таким образом, что активация гена может комбинироваться с трансгенезисом.
В зависимости от нацеленного локуса и его участия в активности иммунных клеток, исходная функция нацеленного эндогенного гена может быть инактивирована или поддержана. Если целевой ген важен для активности иммунных клеток, такая процедура инсерции может привести к однократному нокину (knock-in, KI) без инактивации гена. Напротив, если целевой ген считается вовлеченным в торможение/истощение иммунных клеток, процедуру инсертирования разрабатывают для предупреждения экспрессии эндогенного гена, предпочтительно путем нокаута (knocking-out, KO) эндогенной последовательности, хотя имеется возможность экспрессии интродуцированной экзогенной кодирующей последовательности (последовательностей).
В определенных вариантах осуществления настоящее изобретение связано, при повышении регуляции с разной кинетикой, с усилением экспрессии целевого гена при активации сигнального метаболического пути CAR путем целевой интеграции (с или без разрушения нативного гена) в определенных локусах, например, PD1, PDL1, CTLA-4, TIM3, LAG3, TNFa или IFNg, но ими перечень не ограничивается.
В другом предпочтительном варианте осуществления настоящего изобретения описывают сконструированные иммунные клетки, предпочтительно первичные иммунные клетки для инфузии пациентам, содержащие экзогенные последовательности, кодирующие полипептид (полипептиды) IL-15 или IL-12, которые интегрированы в PD1, CD25 или CD69 эндогенный локус для их экспрессии под контролем эндогенных промоторов, присутствующих в этих локусах.
Иммунные клетки по настоящему изобретению могут быть [CAR]положительными, [CAR]отрицательными, [TCR]положительными или [TCR]отрицательными в зависимости от терапевтических показаний и состояния пациента. В одном из предпочтительных вариантов осуществления настоящего изобретения иммунные клетки дополнительно делают [TCR]отрицательными для аллогенной трансплантации.
Это может быть достигнуто специально путем генетического нарушения по меньшей мере одной эндогенной последовательности, кодирующей, по меньшей мере один компонент TCR, например, TRAC (локус, кодирующий TCRalpha), предпочтительно интеграцией экзогенной последовательности, кодирующей химерный антигенный рецептор (chimeric antigen receptor, CAR) или рекомбинантный TCR, или их компоненты (компонент).
В еще одном варианте осуществления настоящего изобретения иммунные клетки трансфицируют дополнительной экзогенной последовательностью дополнительно к последовательности, кодирующей ингибитор NK, чей полипептид может ассоциировать и предпочтительно интерферировать с рецептором цитокина семейства рецепторов IL-6, например, мутантным GP130. В частности, настоящее изобретение предусматривает иммунные клетки, предпочтительно Т-клетки, которые секретируют растворимый мутантный GP130, направленный на снижение синдрома выброса цитокинов (cytokine release syndrome, CRS) препятствуя, а в идеале блокируя, сигнальную трансдукцию интерлейкина-6 (IL-6). CRS является хорошо известным осложнением иммунотерапии, приводящим к аутоиммунности, которая появляется, когда трансдуцированные иммунные клетки начинают проявлять активность in-vivo. После связывания IL-6 с его рецептором IL-6R, комплекс ассоциирует с субъединицей GP130, инициируя сигнальную трансдукцию и каскад воспалительных ответов. В одном варианте осуществления настоящего изобретения димерный белок, содержащий внеклеточный домен GP130, гибридизированный с частью Fc антитела IgG1 (sgpl30Fc), экспрессируется в сконструированных иммунных клетках для связывания специфически растворимого комплекса IL-R/IL-6 с достижением частичной или полной блокировки переноса сигнала IL-6.
В другом варианте осуществления настоящего изобретения синдром выброса цитокинов (cytokine release syndrome, CRS) может быть смягчен за счет действия по другим метаболическим путям, особенно путем ингибирования синдрома активации макрофагов (macrophage activated syndrome, MAS), которые амплифицируют компонент CRS. Для достижения этой цели настоящее изобретение предусматривает интегрированные экзогенные последовательности, кодирующие антагонистов IL1 и IL18 активирующих метаболических путей, например, IL1RA и/или IL18BP. Таким образом, настоящее изобретение предусматривает методы получения терапевтических клеток, по которым экзогенные последовательности, кодирующие IL1RA и/или IL18BP, интегрированы в выбранный локус, например, один из выбранных локусов по настоящему изобретению.
Настоящее изобретение также относится к различным методам ограничения CRS при иммунотерапии, в комбинации с ингибиторами NK или без такой комбинации, в которых иммунные клетки генетически модифицированы для экспрессии растворимого полипептида, который может ассоциировать и предпочтительно интерферировать с IL1 или IL18, например, IL1RA, IL18BP или рецептором цитокина семейства рецепторов IL-6, например, sgp130Fc. В предпочтительном варианте осуществления настоящего изобретения такая последовательность, кодирующая указанный растворимый полипептид, который может ассоциировать и предпочтительно интерферировать с IL1, IL18 или рецептором цитокина из семейства рецепторов IL-6, интегрирован под контролем эндогенного промотора, предпочтительно в одном локусе, ответственном за активацию Т-клеток, например, выбранном из табл. 6, 8 или 9, особенно из локусов PD1, CD25 или CD69. Полинуклеотидные последовательности векторов, матриц доноров, содержащих экзогенные кодирующие последовательности и/или последовательности, гомологичные эндогенным локусам, последовательности, относящиеся к полученным сконструированным клеткам, а также последовательности, позволяющие обнаруживать указанных сконструированных клеток, все включены в настоящее изобретение и являются его частью.
Этап редактирования гена, включающий объединение экзогенной последовательности, кодирующей ингибитор NK, согласно настоящему уровню, может быть объединен с какой-либо другой стадией, способствующей повышению активности или безопасности сконструированных иммунных клеток. В качестве примеров, не ограничивающих рамок охвата настоящего изобретения, генетические последовательности могут быть интродуцированы для экспрессии компонентов биологических «логических элементов» («И» или «ИЛИ», или «НЕТ», или любая их комбинация) путем целевой интеграции. Подобно электронным логическим элементам, такие клеточные компоненты, экспрессируемые в разных локусах, могут обмениваться отрицательными и положительными сигналами, которые управляют, например, условиями активации иммунной клетки. Такой компонент охватывает в качестве примеров, не ограничивающих рамок охвата настоящего изобретения, положительные и отрицательные химерные антигенные рецепторы, которые можно использовать для контроля активации Т-клеток и образующейся цитотоксичности сконструированных Т-клеток, в которых они экспрессируются.
В предпочтительном варианте осуществления настоящее изобретение основано на интродукции последовательности специфичного эндонуклеазного реагента и/или матрицы донора, содержащей интересующий ген и последовательности, гомологичные целевому гену, путем трансфекции одноцепочечных ДНК (single straight DNA, ssDNA) (одним из примеров являются олигонуклеотиды), двухцепочечных ДНК (double-stranded DNA, dsDNA) (одним из примеров являются плазмидные ДНК), и более конкретно аденоассоциированный вирус (adeno-associated virus, AAV) в качестве одного из возможных примеров.
Изобретение также относится к векторам, матрицам доноров, реагентам, методам скрининга для выявления новых ингибиторов NK и к полученным в результате конструирования клеткам, относящимся к вышеуказанным методам, а также к их применению в терапии.
Краткое описание фигур и таблиц
Фиг. 1. Стратегии конструирования гемопоэтических стволовых клеток (ГСК) путем интродукции экзогенных последовательностей в определенные локусы под контролем транскрипции эндогенных промоторов, специфически активированных в определенных типах иммунных клеток. На фигуре перечислены примеры конкретных эндогенных генов, в локусы которых могут быть вставлены экзогенные кодирующие последовательности (последовательность) для экспрессии в необходимых гемопоэтических линиях согласно настоящему изобретению. Цель состоит в том, чтобы получить сконструированные ex vivo ГСК для трансплантации пациентам, чтобы они могли продуцировать иммунные клетки in vivo, которые могут экспрессировать выбранные трансгены, до тех пор, пока они дифференцируются в необходимую линию клеток.
Фиг. 2. Схематическое представление последовательностей доноров, используемых в экспериментальном разделе для вставки экзогенной кодирующей последовательности IL-15 в локусы CD25 и PD1, а также экзогенной кодирующей последовательности анти-CD22 CAR в локус TRAC. Фиг. 2А: Матрица донора (обозначенная IL-15m-CD25), предназначенная для сайт-направленной инсерции IL-15 в локус CD25 для получения совместной транскрипции CD25 и IL-15 полипептидов иммунной клеткой. Последовательности подробно описаны в примерах. Фиг. 2Б: Матрица донора (обозначенная IL-15m-PD1) предназначена для сайт-направленной инсерции IL-15 в локус PD1 для получения транскрипции IL-15 под транскрипционной активностью промотора эндогенного гена PD1. Последовательности правой и левой границ PD1 могут быть выбраны так, чтобы сохранить эндогенную кодирующую последовательность PD1 интактной или была нарушенной. В последнем случае PD1 подвергают нокауту, a IL-15 включается и транскрибируется. Фиг. 2В: Матрица донора, предназначенная для сайт-направленной инсерции химерного антигенного рецептора (например, анти-CD22 CAR) в локус TCR (например, TRAC). В общем, левую и правую границы выбирают таким образом, чтобы нарушить TCR для того, чтобы получить [TCR]отрицательный[CAR]положительный сконструированные иммунные клетки, применимые для аллогенной трансплантации пациентам.
Фиг. 3. Жидкостная цитометрия измеряет частоту целевой интеграции IL-15m в локус PD1 или CD25 с использованием, соответственно, PD1 или CD25 TALEN®, в контексте того, что анти-CD22 CAR также интегрируют в локус TRAC с использованием TRAC TALEN®. Эти результаты показывают эффективную целевую интеграцию как CAR анти-CD22 в локусе TRAC вместе, так и кодирующей последовательности IL-15 в локусах PD1 или CD25. Фиг. 3А: Ложно трансфицированные первичные Т-клетки. Фиг. 3Б: первичные Т-клетки, трансфицированные последовательностями доноров, представленных на фиг. 1 (Б и В), и специфическим TALEN® для двойной интеграции в локусах TCR и PDI. Фиг. 3В: первичные Т-клетки, трансфицированные последовательностями доноров, представленными на фиг. 1 (А и В), и специфическим TALEN® для двойной интеграции в локусы TCR и CD25.
Фиг. 4. Схематическое изображение экзогенных последовательностей, используемых в экспериментальном разделе для трансфекции первичных иммунных клеток, для получения результатов, показанных на фиг. 5 и 6.
Фиг. 5 и 6. Жидкостная цитометрия измеряет экспрессию LNGFR среди жизнеспособных Т-клеток, трансфицированных донорскими матрицами, показанными на фиг. 4, и специфическим TALEN® (TCR и CD25), при анти-CD3/CD28 неспецифической активации (Dynabeads®) и при активации CAR-зависимых опухолевых клеток (клетки опухоли Раджи). Как показано на фиг. 6, экспрессию LNGFR специфически индуцировали в [CAR анти-CD22]положительных клетках после включения CAR/опухоли.
Фиг. 7 и 8. Жидкостная цитометрия измеряет экспрессию CD25 среди жизнеспособных Т-клеток, трансфицированных матрицами доноров, показанными на фиг. 4, и специфическим TALEN® (TCR и CD25), при анти-CD3/CD28 неспецифической активации (гранулами Dynabeads®) и при активации опухолевых клеток (клетки опухоли Раджи). Как показано на фиг.8, экспрессию CD25 специфически индуцируют в [CAR анти-CD22]положительных клетках после включения CAR/опухоли.
Фиг. 9. Схематическое изображение экзогенных последовательностей, используемых в экспериментальном разделе для трансфекции первичных иммунных клеток, для получения результатов, показанных на фиг. 11 и 12.
Фиг. 10 и 11. Жидкостная цитометрия измеряет экспрессию LNGFR среди жизнеспособных Т-клеток, трансфицированных матрицами доноров, показанными на фиг. 9, и специфическим TALEN® (TCR и PD1), при анти-CD3/CD28 неспецифической активации (гранулами Dynabeads®) и при активации опухолевых клеток (клетки опухоли Раджи). Как показано на фиг. 11, экспрессию LNGFR специфически индуцируют в [CAR анти-CD22]положительных клетках после включения CAR/опухоли.
Фиг. 12. Жидкостная цитометрия измеряет экспрессию эндогенного PD1 среди жизнеспособных Т- клеток, трансфицированных матрицами доноров, показанными на фиг. 9, после анти-CD3/CD28 неспецифической активации (гранулами Dynabeads®) и активации опухолевых клеток (опухолевые клетки Раджи) с использованием и без использования TALEN® (TCR и PD1). PD1 эффективно нокаутирован обработкой TALEN (8% оставшейся экспрессии PD1 из 54%).
Фиг. 13. Диаграмма, показывающая выработку 1L-15 в [CAR]положительных (CARm) и [CAR]отрицательных сконструированных иммунных клетках в соответствии с настоящим изобретением, трансфицированных матрицей донора, описанной на фиг. 2Б, и TALEN® для вставки экзогенных кодирующих последовательностей IL-15 в локус PD1. IL15, транскрипция которого находится под контролем эндогенного промотора PD1, эффективно индуцируется после анти-CD3/CD28 неспецифической активации (гранулами Dynabeads®) и активации опухолевых клеток (клетки опухоли Раджи), и секретируется в культуральные среды.
Фиг. 14. График, показывающий количество IL-15, секретируемого в течение времени (дней) после активации иммунными клетками, сконструированными согласно настоящему изобретению. Фиг. 14А: Клетки, сконструированные путем интеграции кодирующей последовательности IL-15 в локус CD25 с использованием матриц донорных ДНК, описанных на фиг. 2А (IL-15m_CD25) и/или на фиг. 2В (CARm). Фиг. 14Б: Клетки, сконструированные путем интеграции кодирующей последовательности IL-15 в локус PD1 с использованием матриц донорных ДНК, описанных на фиг. 2Б (IL-15m_PD1) и/или 2В (CARm). Интеграции в обоих локусах показывают сходные профили секреции IL-15. Секреция IL-15 значительно увеличивается при опухолеспецифической активации CAR.
Фиг. 15. График, показывающий количество опухолевых клеток Раджи-Luc, экспрессирующих антиген CD22 (сигнал люциферазы) на протяжении времени в анализе на выживание (анализ серийного уничтожения), как описано в примере 2. Иммунные клетки (МКПК) сконструированы для интеграции кодирующих последовательностей IL-15 в локус PD1 (А) или CD25 (Б) и экспрессии анти-CD22-CAR в локусе TCR (тем самым нарушая экспрессию TCR). В этом анализе опухолевые клетки регулярно добавляются в культуральную среду, при этом они частично или полностью удаляются CAR-положительными клетками. Повторная экспрессия IL-15 в клетках PD1 или CD25 существенно помогает элиминации опухолевых клеток CAR- положительными клетками.
Фиг. 16. Схематическое представление последовательностей доноров, используемых в экспериментальном разделе для инсерции в локус PD1 экзогенных последовательностей, кодирующих IL-12 и gp130Fc. Фиг. 16А: матрицу донора (обозначенную IL-12m-PD1), предназначенную для сайт-направленной инсерции кодирующих последовательностей IL-12a и IL-12b (SEQ ID NO: 47 и 48) в локус PD1 для получения одновременной транскрипции IL-12a и IL-12b, при этом разрушая эндогенную кодирующую последовательность PD1. Последовательности правого и левого конца, гомологичные последовательностям локуса PD1, имеют длину по меньшей мере 100 п.о., предпочтительно по меньшей мере 200 п.о. и, более предпочтительно по меньшей мере 300 п.о. и представлены как SEQ ID NO: 45 и 46. Последовательности подробно описаны в табл. 5. Фиг. 16Б: матрицу донора (обозначенную gp130Fcm-PD1), предназначенную для сайт-направленной инсерции кодирующей последовательности (SEQ ID NO:51) для получения транскрипции в локусе PD1 под контролем промотора PD1, при этом разрушая эндогенную кодирующую последовательность PD1. Последовательности правого и левого конца, гомологичные последовательностям локуса PD1, имеют длину по меньшей мере 100 п.о., предпочтительно по меньшей мере 200 п.о. и, более предпочтительно по меньшей мере 300 п.о. и представлены как SEQ ID NO: 45 и 46. Последовательности подробно описаны в табл.5.
Фиг. 17. MHC-I-отрицательные Т-клетки могут быть нацелены на атаку NK-клеток. [β2m]neg Т-клетки культивируют в присутствии или в отсутствие CD2/NKp46 активированных -клеток NK при указанных соотношениях Е:Т. Данные демонстрируют более чем 50% истощение МНС I-отрицательных Т-клеток при всех протестированных соотношениях Е:Т.
Фиг.18. Диаграммы, показывающие стратегию, применяемую в соответствии с методом по настоящему изобретению, для получения сконструированных продуктов CAR Т-клеток, устойчивых к цитолитической активности как NK, так и аллогенных Т-клеток.
Фиг. 19. Схематическое изображение целевой интеграции конструкций для двойной целевой интеграции ингибиторов CAR и NK в локусы TRAC и β2m, соответственно (см. пример 3).
Фиг. 20. Общая структура тримера HLA-E, который может кодироваться экзогенной последовательностью, интегрированной в локус β2m в CAR-положительные Т-клетки по настоящему изобретению.
Фиг. 21: двойная целевая интеграция конструкций CAR и NK-ингибитора в Т-клетки с недостаточностью TRAC/B2M, полученные в соответствии с экспериментами, представленными в примере 3. Анализ с помощью жидкостной цитометрии сконструированных CAR Т-клеток, обработанных TALEN и целевыми конструкциями интеграции. Экспрессия ингибитора NK подтверждена в CAR+ Т-клетках с недостаточностью TRAC/B2M.
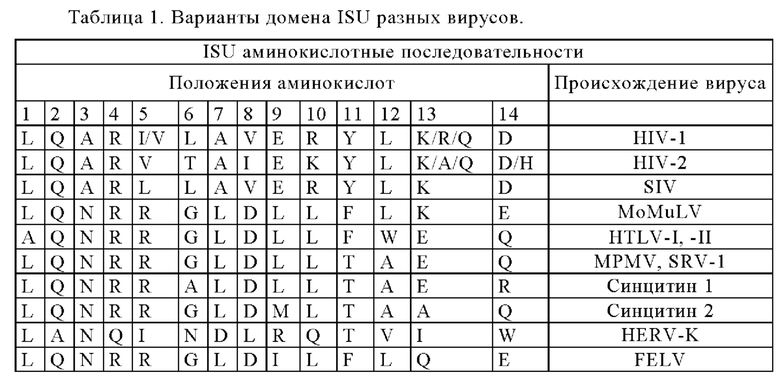
Таблица 1. Варианты домена ISU у разных вирусов.
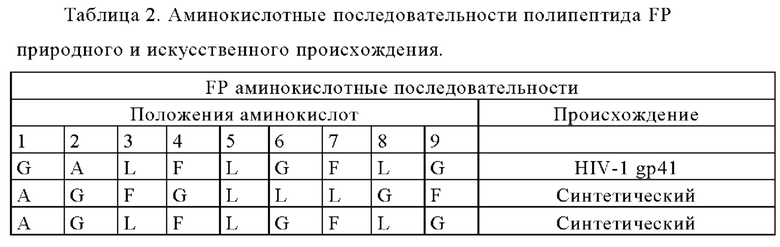
Таблица 2. Аминокислотные последовательности полипептида FP природного и искусственного происхождения.
Таблица 3. Перечень генов, участвующих в метаболических путях ингибирования иммунных клеток, которые могут быть выгодно модифицированы или инактивированы путем инсерции экзогенной кодирующей последовательности по настоящему изобретению.
Таблица 4. Последовательности, приводимые в примере 1.
Таблица 5. Последовательности, приводимые в примерах 2 и 3.
Таблица 6. Перечень генов человека, регуляция которых увеличивается при активации Т-клеток (промоторы, чувствительные к активации CAR), в которых направленная на ген инсерция осуществлена в соответствии с настоящим изобретением для улучшения терапевтического потенциала иммунных клеток.
Таблица 7. Отбор генов, которые стабильно транскрибируются в ходе активации иммунных клеток (в зависимости или независимо от активации Т-клеток).
Таблица 8. Отбор генов, регуляция которых временно повышается при активации Т-клеток.
Таблица 9. Отбор генов, регуляция которых повышается через более чем 24 ч при активации Т-клеток.
Таблица 10. Отбор генов, регуляция которых снижается при активации Т-клеток.
Таблица 11. Отбор генов, которые являются молчащими генами при активации Т-клеток (локусы как «безопасное убежище» для нацеленной генной интеграции).
Таблица 12. Перечень генных локусов, регуляция которых повышена в истощенных инфильтрующихся опухолевых лимфоцитах (по данным многих опухолей), применимых для генной интеграции экзогенных кодирующих последовательностей согласно настоящему изобретению.
Таблица 13. Перечень генных локусов, регуляция которых повышена в опухолях при гипоксии, применимых для генной интеграции экзогенных кодирующих последовательностей согласно настоящему изобретению.
Подробное описание изобретения
Если в настоящем изобретении не указано иное, то все технические и научные термины, применяемые в настоящем изобретении, имеют значения, известные специалистам в данной области генной терапии, биохимии, генетики и молекулярной биологии.
Все методы и материалы, близкие или равноценные описанным в настоящем изобретении, могут применяться в практике или для анализа по настоящему изобретению наряду с подходящими методами и материалами, описанными в настоящем изобретении. Все публикации, патентные заявки, патенты и другие источники, упоминаемые в настоящем изобретении, включены в него в виде ссылок. В случае конфликта, настоящее описание и указанные в нем понятия, преобладают. Кроме того, материалы, методы и примеры являются лишь пояснением сущности настоящего изобретения и не ограничивают рамок охвата настоящего изобретения, если не указано иное.
В практике настоящего изобретения могут быть применены традиционные методы, если не указано иное, применяемые в цитологии, для культивирования клеток, в молекулярной биологии, трансгенной биологии, микробиологии, для рекомбинации ДНК и в иммунологии, известные специалистам в данной области. Эти методы подробно описаны в литературе. См., например, кн.: Frederick М. AUSUBEL «Current Protocols in Molecular Biology», 2000, изд-во Wiley and son Inc, Библиотека Конгресса США; в кн.: Sambrook с соавт., «Molecular Cloning: A Laboratory Manual», 3-е изд., 2001, изд-во Cold Spring Harbor Laboratory Press, Cold Spring Harbor, Нью-Йорк; в кн.: «Oligonucleotide Synthesis», 1984, под ред. M.J. Gait; US 4683195; в кн.: «Nucleic Acid Hybridization», 1984, под ред. В.D. Harries и S. J. Higgins; в кн.: «Transcription And Translation», 1984, под ред. В.D. Hames и S.J. Higgins; в кн.: «Culture Of Animal Cells», 1987, изд-во R.I. Freshney, Alan R. Liss, Inc.; в кн.: «Immobilized Cells And Enzymes», 1986, изд-во IRL Press; в кн.: В. Perbal «A Practical Guide To Molecular Cloning», 1984; серийные издания «Methods In ENZYMOLOGY», под ред. J. Abelson и M. Simon, изд-во Academic Press, Inc., Нью-Йорк, тома 154, 155 (под ред. Wu с соавт.) и том 185 «Gene Expression Technology» (под ред. D. Goeddel); в кн.: «Gene Transfer Vectors For Mammalian Cells», 1987, под ред. J. H. Miller и M.P. Calos, изд-во Cold Spring Harbor Laboratory); в кн.: «Immunochemical Methods In Cell And Molecular Biology», 1987, под ред. Mayer и Walker, изд-во Academic Press, Лондон; в кн.: «Handbook Of Experimental Immunology», 1986, под ред. D.M. Weir и С.С.Blackwell, тома I-IV; в кн.: «Manipulating the Mouse Embryo», 1986, изд-во Cold Spring Harbor Laboratory Press, Cold Spring Harbor, Нью-Йорк).
Настоящее изобретение обращается к общему методу получения первичных иммунных клеток для клеточной иммунотерапии, включающему интеграцию в определенный ген экзогенной кодирующей последовательности в хромосомную ДНК указанных иммунных клеток. В некоторых вариантах осуществления настоящего изобретения эту интеграцию осуществляют таким образом, что указанную кодирующую последовательность помещают под контроль транскрипции по меньшей мере одного промотора, эндогенного по отношению к указанным клеткам, причем указанный эндогенный промотор предпочтительно не является конститутивным промотором, например, таким как транскрибирующий Т-клеточный константный рецептор альфа (TRAC - NCBI Gene ID # 28755). Конститутивный промотор по настоящему изобретению является, например, промотором, который активен независимо от активации CAR например, если Т-клетки еще не активированы.
Улучшение терапевтического потенциала иммунных клеток за счет ген-нацеленной интеграции
Методы генного редактирования с использованием реагентов, специфичных в отношении полинуклеотидной последовательности, например, редкощепящих эндонуклеаз, стали основой методов интродукции генетических модификаций в первичные клетки. Однако эти методы долго не применяли в отношении иммунных клеток для интродукции экзогенных кодирующих последовательностей под контролем транскрипции эндогенными промоторами.
Цель настоящего изобретения заключается в улучшении терапевтического потенциала иммунных клеток с помощью методик генного редактирования, особенно ген-нацеленной интеграции.
Понятие «ген-нацеленная интеграция» означает какие-либо сайт-специфические методы, позволяющие инсертировать, заменить или откорректировать геномную последовательность живой клетки. В предпочтительном варианте осуществления настоящего изобретения указанная ген-нацеленная интеграция включает гомологичную генную рекомбинацию в локусе целевого гена для получения инсерции или замещения по меньшей мере одного экзогенного нуклеотида, предпочтительно последовательности из нескольких нуклеотидов (то есть полинуклеотида) и, более предпочтительно, кодирующей последовательности.
Понятие «реагент, специфичный в отношении последовательности», означает какую-либо активную молекулу, которая обладает способностью специфически распознавать выбранную полинуклеотидную последовательность в геномном локусе, предпочтительно из 9 п.о., более предпочтительно по меньшей мере из 10 п.о., и еще более предпочтительно по меньшей мере из 12 п.о. в длину, для модификации указанного геномного локуса. В предпочтительном варианте осуществления настоящего изобретения указанный реагент, специфичный в отношении последовательности, предпочтительно является реагентом-нуклеазой, специфичным в отношении последовательности.
Понятие «иммунная клетка» означает гемопоэтическую клетку, функционально вовлеченную в инициацию и/или уничтожение врожденного и/или адаптивного иммунного ответа, например, типичную CD3 или CD4 положительную клетку. Иммунная клетка по настоящему изобретению может быть дендритной клеткой, дендритной клеткой-киллером, тучной клеткой, клеткой NK, В-клеткой или Т-клеткой, выбранной из группы, состоящей из воспалительных Т-лимфоцитов, цитотоксических Т-лимфоцитов, регуляторных Т-лимфоцитов или Т-лимфоцитов-хэлперов. Клетки могут быть получены из ряда источников (которыми перечень не ограничивается), например, мононуклеарные клетки периферической крови (МКПК), из костного мозга, ткани лимфоузлов, пуповинной крови, ткани тимуса, ткани из места инфицирования, асцита, плевральной инфузии, ткани селезенки и из опухолей, например, инфильтрованные в опухоль лимфоциты. В некоторых вариантах осуществления настоящего изобретения указанные иммунные клетки могут быть получены от здорового донора, онкологических пациентов или от пациентов с инфекционным заболеванием. В другом предпочтительном варианте осуществления настоящего изобретения указанная иммунная клетка является частью смешанной популяции иммунных клеток, презентирующих разные фенотипические свойства, например, включающие CD4, CD8 и CD56 положительные клетки.
Понятие «первичная клетка» или «первичные клетки» относится к клеткам, полученным непосредственно от живой ткани (например, материал биопсии), которую используют для роста in vitro на протяжении ограниченного времени, что означает, что они могут претерпеть ограниченное число удвоений в популяции. Первичные клетки противоположны непрерывно существующим опухолеобразующим или искусственно бессмертным линиям клеток. К примерам таких линий клеток (которыми перечень не ограничивается), относятся клетки СНО-K1, клетки HEK293, клетки Сасо2, клетки U2-OS, клетки NIH 3Т3, клетки NSO, клетки SP2, клетки CHO-S, клетки DG44, клетки K-562, клетки U-937, клетки MRC5, клетки IMR90, клетки Jurkat, клетки HepG2, клетки HeLa, клетки НТ-1080, клетки НСТ-116, клетки Hu-h7, клетки Huvec, клетки Molt 4. Первичные клетки обычно применяют в клеточной терапии, поскольку они рассматриваются в качестве более функциональных и менее опухолеобразующих.
В целом, первичные иммунные клетки получают от доноров или пациентов с помощью методов, известных в данной области, например, методов лейкафереза, описанных Schwartz J. с соавт. J Clin Apher. 28(3), 2013, 145-284.
Первичные иммунные клетки по настоящему изобретению также могут быть получены в результате дифференциации стволовых клеток, например, стволовых клеток пуповинной крови, прогениторных клеток, стволовых клеток костного мозга, гемопоэтических стволовых клеток (hematopoietic stem cell, ГСК) и индуцированных плюропотентных стволовых клеток (induced pluripotent stem cell, iPS).
Понятие «нуклеазный реагент» означает молекулу нуклеиновой кислоты, которая участвует в нуклеазной каталитической реакции в клетках-мишенях, предпочтительно эндонуклеазной реакции, причем участвует сама молекула или в качестве субъединицы комплекса, например, такого как направляющая РНК/Cas9, предпочтительно приводя к расщеплению последовательности-мишени нуклеиновой кислоты.
Нуклеазные реагенты по настоящему изобретению в общем являются «реагентами, специфичными в отношении последовательности», что означает, что они могут индуцировать расщепление ДНК в клетке в заранее определенном локусе, обозначаемым как «целевой ген». Последовательность нуклеиновой кислоты, которая распознается реагентом, специфичным в отношении последовательности, обозначается как «целевая последовательность» или «последовательность-мишень» Такую последовательность-мишень обычно выбирают как редкую или уникальную в клеточном геноме, и в более широком понятии в геноме человека, что можно определить, используя программное обеспечение и данные, доступные из базы данных генома человека, например, http://www.ensembl.org/index.html.
«Редкощепящими эндонуклеазами» называют эндонуклеазы - реагенты выбора, специфичные в отношении последовательности, причем их распознаваемые последовательности обычно варьируют от 10 до 50 последовательных пар оснований, предпочтительно от 12 до 30 п.о. и более предпочтительно от 14 до 20 п.о.
В другом предпочтительном варианте осуществления настоящего изобретения указанным эндонуклеазным реагентом является нуклеиновая кислота, кодирующая «сконструированную» или «программируемую» редкощепящую эндонуклеазу, например, хоминг-эндонуклеазу, например, описанную в WO2004067736 цинк-пальциевую нуклеазу (zing finger nuclease, ZFN), например, описанную Urnov F. с соавт. Nature 435, 2005, 646-651), TALE-нуклеазу, например, описанную Mussolino с соавт., Nucl. Acids Res. 39(21), 2011, 9283-9293), или MegaTAL нуклеазу, например, описанную Boissel с соавт., Nucleic Acids Research 42(4), 2013, 2591-2601.
В другом варианте осуществления настоящего изобретения эндонуклеазным реагентом является направляющая РНК, которую применяют вместе с РНК-направляющей эндонуклеазой, например, Cas9 или Cpf1, в соответствии с, inter alia, описанным методом Doudna J. и Chapentier Е. в статье Science 346(6213), 2014, 1077), включенной в настоящее изобретение в виде ссылки.
В предпочтительном варианте осуществления настоящего изобретения эндонуклеазный реагент временно экспрессируется в клетках, что означает, что указанный реагент не должен интегрироваться в геном или сохраняться в течение длительного периода времени, например как в случае РНК, точнее мРНК, белков или комплексов, объединяющих белки и нуклеиновые кислоты (например, рибонуклеопротеинов).
Обычно 80% эндонуклеазного реагента разрушается за 30 ч, предпочтительно за 24 ч, более предпочтительно за 20 ч после трансфекции. Эндонуклеаза в форме мРНК предпочтительно синтезируется с кэпом для повышения ее стабильности в соответствии с методами, известными в данной области, например, описанными Kore A.L. с соавт., J Am Chem Soc. 131(18), 2009, 6364-6365).
В целом стадии электропорации, которые применяют для трансфекции иммунных клеток, обычно выполняются в закрытых камерах, содержащих электроды с параллельными пластинами, вырабатывающие импульсное электрическое поле между указанными электродами с параллельными пластинами, превышающее 100 вольт/см и менее 5000 вольт/см, по существу однородное по всему объем обработки, например, описанной в патентной заявке WO/2004/083379, включенной в настоящее изобретение в виде ссылки, особенно со страницы 23, строки 25, до страницы 29, строки 11. Одна из таких камер электропорации предпочтительно имеет геометрический фактор (см-1), определяемый как отношение квадрата промежутка между электродами (см2), поделенное на объем камеры (см3), где геометрический фактор меньше или равен 0,1 см-1, где суспензия клеток и специфический в отношении последовательности реагент находятся в среде, которая регулируется таким образом, что среда имеет электропроводность в диапазоне от 0,01 до 1,0 миллиСименс. В целом, суспензия клеток подвергается воздействию одного или нескольких импульсных электрических полей. С помощью такого метода объем обработки суспензии является масштабируемым, а время обработки клеток в камере в существенной степени однородным.
Установлено, что из-за повышенной специфичности TALE-нуклеаза особенно применима для последовательности специфических нуклеазных реагентов в терапевтических целях, особенно в гетеродимерных формах - т.е. работающие в парах с «правым» мономером (также обозначаемым «5» или «прямым») и «левым» мономером (также обозначаемым «3» или «обратным»), например, согласно описанному Mussolino с соавт., Nucl. Acids Res. 42(10), 2014, 6762-6773.
Ранее было установлено, что специфический в отношении последовательности реагент предпочтительно находится в форме нуклеиновой кислоты, такой как ДНК или РНК, кодирующей субъединицу редкощепящей эндонуклеазы, но они также могут быть частью конъюгатов, включающих полинуклеотид (полинуклеотиды) и полипептид (полипептиды), которые называют «рибонуклеопротеинами». Такие конъюгаты могут быть сформированы с реагентами как Cas9 или Cpf1 (РНК-направляющие эндонуклеазы) или белками семейства Аргонавт (ДНК-направляющие эндонуклеазы), которые описаны ранее, соответственно, Zetsche В. с соавт., Cell 163(3), 2015, 759-771, и Gao F. с соавт., Nature Biotech, 2016, включающие РНК-или ДНК-направляющие, которые могут быть объединены с соответствующими нуклеазами.
Понятие «экзогенная последовательность» относится к какому-либо нуклеотиду или последовательности нуклеиновой кислоты, которые изначально не находились в определенном локусе. Такая последовательность может быть гомологичной геномной последовательности или являться ее копией, или быть чужеродной последовательностью, интродуцированной в клетку. Напротив, понятие «эндогенная последовательность» означает последовательность генома клетки, изначально присутствующую в локусе. Предпочтительно экзогенная последовательность кодирует полипептид, экспрессия которого обеспечивает терапевтическое преимущество относительно сестринских клеток, которые не содержат такой экзогенной последовательности в данном локусе. Эндогенная последовательность, которая является геном, подвергшимся редакции за счет инсерции нуклеотида или полинуклеотида в соответствии со способом настоящего изобретения, для экспрессии отличающегося полипептида в широком смысле слова называют экзогенной кодирующей последовательностью.
Способ по настоящему изобретению может быть ассоциирован с другими методами, включающими физические и генетические трансформации, например, вирусную трансдукцию или трансфекцию с использованием наночастиц, а также может сочетаться с другой инактивацией гена и/или трансгенными инсерциями.
В одном из вариантов осуществления настоящего изобретения способ по настоящему изобретению включает стадии:
- получения популяции первичных иммунных клеток,
- интродукции указанных первичных иммунных клеток в пропорции:
i) по меньшей мере, одной нуклеиновой кислоты, содержащей экзогенные нуклеотид или полинуклеотидную последовательность, интегрируют в выбранный эндогенный локус для кодирования по меньшей мере одной молекулы, улучшающей терапевтический потенциал популяции указанных клеток,
ii) по меньшей мере один специфический в отношении последовательности реагент, специфически нацеленный на указанный эндогенный локус,
причем указанные экзогенные нуклеотид или полинуклеотидную последовательность инсертируют путем целевой генной интеграции в указанный эндогенный локус таким образом, что указанные экзогенные нуклеотид или полинуклеотидную последовательность формируют экзогенную кодирующую последовательность под контролем транскрипции эндогенного промотора, присутствующего в указанном локусе.
В одном из вариантов осуществления настоящего изобретения в методе специфичный в отношении последовательности реагент является нуклеазой, и целевую интеграцию генов осуществляют посредством гомологичной рекомбинации или NHEJ в указанные иммунные клетки.
В другом варианте осуществления настоящего изобретения указанный эндогенный промотор выбран во время активации иммунных клеток и предпочтительно при повышенной регуляции. Более конкретно, настоящее изобретение относится к способу получения сконструированных первичных иммунных клеток для клеточной иммунотерапии, причем указанный способ включает:
- получение популяции первичных иммунных клеток,
- интродукцию в пропорции к указанным первичным иммунным клеткам:
i) по меньшей мере одной экзогенной нуклеиновой кислоты, содержащей экзогенную кодирующую последовательность, кодирующую по меньшей мере одну молекулу, улучшающую терапевтический потенциал указанной популяции иммунных клеток;
ii) по меньшей мере, одного специфичного для последовательности нуклеазного реагента, который специфически нацелен на ген, находящийся под контролем эндогенного промотора, активного по ходу активации иммунных клеток;
где указанная кодирующая последовательность интродуцируется в геном первичных иммунных клеток путем направленной гомологичной рекомбинации, так что указанная кодирующая последовательность находится под контролем транскрипции по меньшей мере одного эндогенного промотора указанного гена.
Под «улучшением терапевтического потенциала» подразумевают, что сконструированные иммунные клетки приобретают по меньшей мере одно полезное свойство для их применения в клеточной терапии по сравнению с их родственными не подвергнутыми конструированию иммунными клетками. Терапевтические свойства, на достижение которых направлено настоящее изобретение, могут быть оцениваемыми по каким-либо измеримым показателям, что соответствует описаниям в научной литературе.
Улучшенный терапевтический потенциал может быть точнее отражен по устойчивости иммунных клеток к лекарственному средству, увеличением их выживаемости in vitro или in vivo или более безопасным/более удобным применением во время изготовления терапевтических композиций и лечения.
В общем, указанная молекула, улучшающая терапевтический потенциал, является полипептидом, но она также может быть нуклеиновой кислотой, способной направлять или подавлять экспрессию других генов, например, таких как интерферирующие РНК или направляющие РНК. Полипептиды могут действовать прямо или косвенно, например, сигнальные трансдукторы или регуляторы транскрипции.
В одном из вариантов осуществления настоящего изобретения экзогенную последовательность интродуцируют в эндогенную хромосомальную ДНК посредством направленной гомологичной рекомбинации. Соответственно, экзогенная нуклеиновая кислота, интродуцированная в иммунную клетку, содержит по меньшей мере одну кодирующую последовательность (последовательности), наряду с последовательностями, которые могут гибридизоваться с эндогенными хромосомными последовательностями в физиологических условиях. Обычно такие гомологичные последовательности проявляют по меньшей мере 70%, предпочтительно 80% и более предпочтительно 90% идентичности с эндогенными последовательностями генов, расположенных в локусе инсерции. Эти гомологичные последовательности могут располагаться по концам кодирующей последовательности, чтобы повысить точность рекомбинации, согласно описанному, например, в US 6528313. Используя доступное программное обеспечение и онлайновые базы данных генома, можно разработать векторы, которые включают указанную кодирующую последовательность (последовательности) таким образом, что указанная последовательность (последовательности) интродуцируется в определенном локусе под контролем транскрипции по меньшей мере одного эндогенного промотора, который является промотором эндогенного гена. Затем экзогенную кодирующую последовательность (последовательности) предпочтительно инсертируют «в рамку» с указанным эндогенным геном. Последовательности, полученные в результате интеграции экзогенной полинуклеотидной последовательности (последовательностей), могут кодировать множество белков различных типов, включая гибридные белки, белки-метки или мутантные белки. Гибридные белки позволяют добавлять новые функциональные домены к белкам, экспрессируемым в клетке, например, домен димеризации, который можно использовать для включения или выключения активности указанного белка, например, переключатель каспазы-9. Белки-метки могут быть полезны для обнаружения сконструированных иммунных клеток и при наблюдениях за пациентами, которых лечат указанными клетками. Введение мутации в белки может придать устойчивость к лекарствам или агентам, истощающим иммунитет, о чем написано ниже.
Обеспечение устойчивости к лекарствам или агентам, истощающим иммунитет
В одном из вариантов осуществления настоящего изобретения экзогенная последовательность, которая интегрирована в геномный локус иммунных клеток, кодирует молекулу, которая придает устойчивость к лекарственному средству указанных иммунных клеток.
Примерами предпочтительных экзогенных последовательностей являются варианты дигидрофолатредуктазы (dihydrofolate reductase, DHFR), придающие устойчивость к аналогам фолата, таким как метотрексат, варианты инозинмонофосфатдегидрогеназы 2 (inosine monophosphate dehydrogenase 2, IMPDH2), придающие устойчивость к ингибиторам IMPDH, таким как микофеноловая кислота (mycophenolic acid, МРА) или ее пролекарство, микофенолят мофетила (mycophenolate mofetil, MMF), варианты кальциневрина или метилгуанинтрансферазы (MGMT), придающие устойчивость к ингибиторам кальциневрина, например, FK506 и/или CsA, варианты mTOR, например mTORmut, придающие устойчивость к рапамицину, и варианты Lck, например Lckmut, придающие устойчивость к иматинибу и глевеку.
В контексте настоящего изобретения понятие «лекарственное средство» означает соединение или его производное, предпочтительно стандартный химиотерапевтический агент, который обычно используют для воздействия на раковые клетки, тем самым снижая пролиферативный статус клеток или убивая их. К примерам химиотерапевтических агентов относят, но ими перечень не ограничивается, алкилирующие агенты (например, циклофосфан, ифозамид), метаболические антагонисты (например, пуриновые нуклеозидные антиметаболиты, например клофарабин, флударабин или 2'-дезоксиаденозин, метотрексат (methotrexate, МТХ), 5-фторурацил или их производные), противоопухолевые антибиотики (например, митомицин, адриамицин), противоопухолевые средства растительного происхождения (например, винкристин, виндезин, таксол), цисплатин, карбоплатин, этопозид и другие. Такие агенты могут дополнительно включать, но ими перечень не ограничивается, противораковые агенты TRIMETHOTRIXATE™ (ТМТХ), TEMOZOLOMIDE™, RALTRITREXED™, S-(4-нитробензил)-6-тиоинозин (NBMPR), 6-бензгуанидин (6-BG), бис-хлорнитрозомочевина (BCNU) и CAMPTOTHECIN™ или терапевтическое производное какого-либо из них.
В настоящем изобретении иммунная клетка становится «устойчивой или толерантной» к лекарственному средству, когда указанная клетка или популяция клеток модифицированы таким образом, что они могут пролиферировать по крайней мере in vitro, в культуральной среде, содержащей половину максимальной ингибирующей концентрации (IC50) указанного лекарственного средства (указанный IC50 определяют в отношении немодифицированной клетки (клеток) или популяции клеток).
В одном из вариантов осуществления настоящего изобретения указанная лекарственная устойчивость может быть придана иммунным клеткам посредством экспрессии по меньшей мере одной «последовательности, кодирующей лекарственную устойчивость». Указанная кодирующая последовательность лекарственной устойчивости относится к последовательности нуклеиновой кислоты, которая придает «устойчивость» агенту, например, одному из химиотерапевтических агентов, упомянутых выше. Последовательность, кодирующая лекарственную устойчивость по настоящему изобретению, может кодировать устойчивость к антиметаболитам, метотрексату, винбластину, цисплатину, алкилирующим агентам, антрациклинам, цитотоксическим антибиотикам, антииммунофилинам, их аналогам или производным и другим (Takebe N. с соавт., Mol. Ther. 3(1), 2001, 88-96; Zielske S.P. с соавт., J. Clin. Invest. 112(10), 2003, 1561-1570; Nivens M.C. с соавт., Cancer Chemother Pharmacol 53(2), 2004, 107-115; Bardenheuer W. с соавт., Leukemia 19(12), 2005, 2281-2288; Kushman M.E. с соавт., Carcinogenesis 28(1), 2007, 207-214.
Экспрессия таких экзогенных последовательностей лекарственной устойчивости в иммунных клетках согласно настоящему изобретению, более конкретно, позволяет применять указанные иммунные клетки в схемах клеточной терапии, где клеточная терапия сочетается с химиотерапией, или в случаях лечения пациентов, ранее получавших эти лекарства.
Было идентифицировано несколько последовательностей, кодирующих лекарственную устойчивость, которые потенциально могут быть применены для придания лекарственной устойчивости согласно настоящему изобретению. Одним из примеров последовательности, кодирующей лекарственную устойчивость, может быть, например, мутантная или модифицированная форма дигидрофолатредуктазы (Dihydrofolate reductase, DHFR). DHFR является ферментом, участвующим в регулировании количества тетрагидрофолата в клетке и необходимым для синтеза ДНК. Аналоги фолата, такие как метотрексат (МТХ), ингибируют DHFR и, таким образом, используются в качестве противоопухолевых агентов в клинике. Описаны различные мутантные формы DHFR, которые обладают повышенной устойчивостью к ингибированию антифолатами, используемыми в терапии. В одном из вариантов осуществления настоящего изобретения кодирующая последовательность лекарственной устойчивости в соответствии с настоящим изобретением может представлять собой последовательность нуклеиновой кислоты, кодирующую мутантную форму дикого типа DHFR человека (GenBank: ААН71996.1), которая содержит по меньшей мере одну мутацию, придающую устойчивость к лечению антифолатом, например, к метотрексату. В другом варианте осуществления настоящего изобретения мутантная форма DHFR содержит по меньшей мере одну мутантную аминокислоту в положении G15, L22, F31 или F34, предпочтительно в положениях L22 или F31 (Schweitzer с соавт., Faseb J 4 (8), 1990, 2441-2452; WO 94/24277; US 6642043). В еще одном варианте осуществления настоящего изобретения мутантная форма DHFR содержит мутантную форму, включает две мутантные аминокислоты в положении L22 и F31. Соответствие положений аминокислот, описанных в настоящем изобретении, часто выражают в положениях аминокислот в форме полипептида DHFR дикого типа. В конкретном варианте осуществления настоящего изобретения сериновый остаток в положении 15 предпочтительно заменен остатком триптофана. В другом конкретном варианте осуществления настоящего изобретения остаток лейцина в положении 22 предпочтительно заменяют аминокислотой, которая нарушает связывание мутантного DHFR с антифолатами, предпочтительно с незаряженными аминокислотными остатками, такими как фенилаланин или тирозин. В другом конкретном варианте осуществления остаток фенилаланина в положениях 31 или 34 предпочтительно заменен небольшой гидрофильной аминокислотой, такой как аланин, серин или глицин.
Другим примером последовательности, кодирующей лекарственную устойчивость, также может быть мутантная или модифицированная форма ионизин-5'-монофосфатдегидрогеназы II (IMPDH2), фермента, ограничивающего скорость синтеза de novo гуанозиновых нуклеотидов. Мутантная или модифицированная форма IMPDH2 является геном устойчивости к ингибитору IMPDH. Ингибиторами IMPDH могут быть микофеноловая кислота (mycophenolic acid, МРА) или ее пролекарство микофенолят мофетила (mycophenolate mofetil, MMF). Мутантный IMPDH2 может содержать по меньшей мере одну, предпочтительно две мутации в сайте связывания MAP дикого типа IMPDH2 человека (Genebank: NP 000875.2), приводящего к значительно повышенной устойчивости к ингибитору IMPDH. Мутации в этих вариантах предпочтительно находятся в положениях Т333 и/или S351 (Yam Р. с соавт., Mol. Ther. 14(2), 2006, 236-244; Jonnalagadda М. с соавт., PLoS One 8(6), 2013, е65519).
Другой последовательностью, кодирующей лекарственную устойчивость, является мутантная форма кальциневрина. Кальциневрин (РР2 В - NCBI: АСХ34092.1) является повсеместно экспрессируемой серин/треониновой протеинфосфатазой, которая участвует во многих биологических процессах и является центральной для активации Т-клеток. Кальциневрин представляет собой гетеродимер, состоящий из каталитической субъединицы (CnA; три изоформы) и регуляторной субъединицы (CnB; две изоформы). После включения рецептора Т-клеток кальциневрин дефосфорилирует фактор транскрипции NFAT, позволяя ему транслоцироваться в ядро и активный ключевой ген-мишень, такой как IL2. FK506 в комплексе с FKBP12 или циклоспорин A (CsA) в комплексе с СуРА блокирует доступ NFAT к активному сайту кальциневрина, предотвращая его дефосфорилирование и тем самым ингибируя активацию Т-клеток (Brewin с соавт., Blood 114(23), 2009, 4792-4803). В одном из вариантов осуществления настоящего изобретения указанная мутантная форма может содержать по меньшей мере одну мутантную аминокислоту гетеродимера кальциневрина дикого типа в положениях: V314, Y341, М347, Т351, W352, L354, K360, предпочтительно двойные мутации в положениях Т351 и L354 или V314. и Y341. В другом варианте осуществления настоящего изобретения остаток валина в положении 341 может быть заменен остатком лизина или аргинина, остаток тирозина в положении 341 может быть заменен остатком фенилаланина; метионин в положении 347 может быть заменен остатком глутаминовой кислоты, аргинина или триптофана; треонин в положении 351 может быть заменен остатком глутаминовой кислоты; остаток триптофана в положении 352 можно заменить остатком цистеина, глутаминовой кислоты или аланина, серин в положении 353 можно заменить остатком гистидина или аспарагина, лейцин в положении 354 можно заменить остатком аланина; лизин в положении 360 может быть заменен остатком аланина или фенилаланина. В другом варианте осуществления настоящего изобретения указанная мутантная форма может содержать по меньшей мере одну мутантную аминокислоту гетеродимера b кальциневрина дикого типа в положениях: V120, N123, L124 или K125, предпочтительно двойные мутации в положениях L124 и K125. В еще одном из вариантов осуществления настоящего изобретения валин в положении 120 может быть заменен остатком серина, аспарагиновой кислоты, фенилаланина или лейцина; аспарагины в положении 123 могут быть заменены триптофаном, лизином, фенилаланином, аргинином, гистидином или серином; лейцин в положении 124 может быть заменен остатком треонина; лизин в положении 125 может быть заменен аланином, глутаминовой кислотой, триптофаном, или два остатка, такие как лейцин-аргинин или изолейцин-глутаминовая кислота, могут быть добавлены после лизина в положении 125 в аминокислотной последовательности. Соответствие положений аминокислот, описанных в настоящем изобретении, часто выражают в терминах положений аминокислот в форме полипептида гетеродимера b кальциневрина дикого типа человека (NCBI: АСХ34095.1).
Еще одной последовательностью, кодирующей лекарственную устойчивость, является 0(6)-метилгуанин метилтрансфераза (MGMT -UniProtKB: Р16455), кодирующая алкилгуанинтрансферазу человека (hAGT). AGT является ДНК-репарирующим белком, который придает устойчивость к цитотоксическому действию алкилирующих агентов, таких как нитрозомочевины и темозоломид (TMZ). 6-Бензилгуанин (6-BG) является ингибитором AGT, который усиливает токсичность нитрозомочевины и вводится совместно с TMZ для усиления цитотоксического действия этого агента. Некоторые мутантные формы MGMT, которые кодируют варианты AGT, обладают высокой устойчивостью к инактивации 6-BG, но сохраняют способность репарировать повреждение ДНК (Maze R. с соавт., J. Pharmacol. Exp.Ther. 290(3), 1999 1467-1474). В одном из вариантов осуществления настоящего изобретения мутантная форма AGT может содержать мутантную аминокислоту в положении AGT дикого типа Р140. В предпочтительном варианте осуществления настоящего изобретения указанный пролин в положении 140 заменен остатком лизина.
Другой последовательностью, кодирующей лекарственную устойчивость, может быть ген белка с множественной лекарственной устойчивостью (multidrug resistance protein, MDR1). Этот ген кодирует мембранный гликопротеин, известный как Р-гликопротеин (P-GP), участвующий в транспорте метаболических побочных продуктов через клеточную мембрану. Белок P-Gp проявляет широкую специфичность по отношению к нескольким структурно не связанным химиотерапевтическим агентам. Таким образом, лекарственная устойчивость может быть придана клеткам путем экспрессии последовательности нуклеиновой кислоты, которая кодирует MDR-1 (Genebank NP_000918).
Другая последовательность, кодирующая лекарственную устойчивость, может вносить вклад в продуцирование цитотоксических антибиотиков, таких как продукты генов ble или mcrA. Эктопическая экспрессия гена ble или mcrA в иммунной клетке дает избирательное преимущество при воздействии соответствующих химиотерапевтических агентов блеомицина и митомицина С (Belcourt M.F., PNAS. 96(18), 1999, 10489-10494).
Другая последовательность, кодирующая устойчивости к лекарственному средству, может происходить от генов, кодирующих мутантные версии мишеней лекарственных средств, таких как мутантные варианты mTOR (mTOR mut), придающие устойчивость к рапамицину, такие как описанные Lorenz М.С. с соавт. The Journal of Biological Chemistry 270, 1995, 27531-27537, или некоторые мутантные варианты Lck (Lckmut), придающие устойчивость к цитостатическому препарату гливек (Lee K.С. с соавт., Leukemia, 24, 2010, 896-900).
Выше описано, что стадия генетической модификации метода может включать интродукцию в клетки экзогенной нуклеиновой кислоты, включающий по меньшей мере последовательность, кодирующую лекарственную устойчивость, и часть эндогенного гена таким образом, что гомологичная рекомбинация происходит между эндогенным геном и экзогенной нуклеиновой кислотой. В одном из вариантов осуществления настоящего изобретения указанный эндогенный ген может быть геном «устойчивости к лекарственным средствам» дикого типа, и после гомологичной рекомбинации ген дикого типа заменяется мутантной формой гена, который придает устойчивость к лекарственному средству.
Усиление устойчивости иммунных клеток in-vivo
В одном из вариантов осуществления настоящего изобретения описываемый способ подразумевает, что экзогенная последовательность, которая интегрирована в геномный локус иммунных клеток, кодирует молекулу, которая усиливает устойчивость иммунных клеток, особенно устойчивость in vivo в окружении опухоли.
Под «повышением устойчивости» подразумевают увеличение выживаемости иммунных клеток с точки зрения продолжительности жизни, особенно после того, как сконструированные иммунные клетки вводят пациенту. Например, устойчивость увеличивается, если средняя выживаемость модифицированных клеток значительно дольше по длительности, чем у немодифицированных клеток, по меньшей мере на 10%, предпочтительно на 20%, более предпочтительно на 30%, еще более предпочтительно на 50%.
Это особенно значимо, когда иммунные клетки являются аллогенными. Это может быть сделано путем создания локальной иммунной защиты путем интродукции кодирующих последовательностей, которые эктопически экспрессируют и/или секретируют иммунодепрессивные полипептиды на клеточной мембране или сквозь нее. Различные панели таких полипептидов, в частности антагонистов иммунных контрольных точек, иммуносупрессивные пептиды, производные вирусной оболочки или лиганда NKG2D, могут усиливать персистенцию и/или приживление аллогенных иммунных клеток у пациентов.
В одном из вариантов осуществления настоящего изобретения иммуносупрессивный полипептид, который кодируется указанной экзогенной кодирующей последовательностью, является лигандом цитотоксического Т-лимфоцитарного антигена 4 (CTLA-4, также обозначаемый CD152, номер в GenBank AF414120.1). Указанный лигандный полипептид предпочтительно является иммуноглобулином против CTLA-4, например, CTLA-4a Ig и CTLA-4b Ig, или их функциональными вариантами.
В другом варианте осуществления настоящего изобретения иммуносупрессивный полипептид, кодируемый указанной экзогенной последовательностью, является антагонистом PD1, например PD-L1 (также называемым CD274, запрограммированным лигандом гибели клеток 1; UniProt для последовательности полипептида человека - Q9NZQ7), который кодирует трансмембранный белок типа I из 290 аминокислот, состоящий из IgV-подобного домена, IgC-подобного домена, гидрофобного трансмембранного домена и цитоплазматического хвоста из 30 аминокислот. Такая связанная с мембраной форма лиганда PD-L1 подразумевается в настоящем изобретении в нативной форме (дикого типа) или в усеченной форме, например, за счет удаления внутриклеточного домена, или с одной или несколькими мутациями (Wang S. с соавт., J Exp Med. 2003; 197(9), 2003, 1083-1091). Следует отметить, что PD1 не рассматривается как связанная с мембраной форма лиганда PD-L1 в соответствии с настоящим изобретением. Согласно другому варианту осуществления настоящего изобретения указанный иммунодепрессивный полипептид находится в секретируемой форме. Такой рекомбинантный секретируемый PD-L1 (или растворимый PD-L1) может быть получен путем слияния внеклеточного домена PD-L1 с частью Fc иммуноглобулина (Haile S.T. с соавт., Cancer Immunol. Res. 2(7), 2014, 610-615; Song M.Y. с соавт., Gut. 64(2), 2015, 260-271). Этот рекомбинантный PD-L1 может нейтрализовать PD-1 и отменять PD-1-опосредованное Т-клеточное ингибирование. Лиганд PD-L1 может одновременно экспрессироваться с CTLA4 Ig для еще большей персистенции обоих.
В другом варианте осуществления настоящего изобретения экзогенная последовательность кодирует полипептид, содержащий иммуносупрессивный домен оболочки вируса (immusuppressive domain, ISU), который является производным, например, от HIV-1, HIV-2, SIV, MoMuLV, HTLV-I, -II, MPMV, SRV-1, Syncitin 1 или 2, HERV-K или FELV.
Приводимая ниже табл.1 представляет варианты домена ISU от разных вирусов, которые могут экспрессироваться в рамках охвата настоящего изобретения.
В другом варианте осуществления настоящего изобретения экзогенная последовательность кодирует полипептид FP, например, gp41. В приводимой ниже табл. 2 представлено несколько полипептидов FP природного и искусственного происхождения.
В другом варианте осуществления настоящего изобретения экзогенная последовательность кодирует гомолог ГКГ, не принадлежащий человеку, особенно гомолог ГКГ вируса, или химерный полипептид β2m, например, описанный Margalit А. с соавт., Int. Immunol. 15 (11), 2003, 1379-1387.
В одном из вариантов осуществления настоящего изобретения экзогенная последовательность кодирует лиганд NKG2D. Некоторые вирусы, например, цитомегаловирусы, приобрели механизмы, позволяющие избегать клеток NK, опосредовать иммунный надзор и вмешиваться в метаболический путь NKG2D, секретируя белок, способный связывать лиганды NKG2D и предотвращая их экспрессию на поверхности клетки (Welte S. А с соавт., Eur. J. Immunol., 33, 2003, 194-203). В опухолевых клетках некоторые механизмы эволюционировали, чтобы уклониться от ответа NKG2D путем секретирования NKG2D лигандов, например, ULBP2, MICB или MICA (Salih H.R. с соавт., Blood 102, 2003, 1389-1396).
В одном из вариантов осуществления настоящего изобретения экзогенная последовательность кодирует рецептор цитокина, например, рецептор IL-12. IL-12 является известным активатором активации иммунных клеток (Curtis J.H., The Journal of Immunology. 181(12), 2008, 8576-8584).
В еще одном из вариантов осуществления настоящего изобретения экзогенная последовательность кодирует антитело, которое направлено против ингибирующих пептидов или белков. Указанное антитело предпочтительно должно секретироваться в растворимой форме иммунными клетками. Нанотела от акул и верблюдов в этом отношении имеют предпочтение, поскольку они структурированы как одноцепочечные антитела (Muyldermans S., Annual Review of Biochemistry 82, 2013, 775-797). Предполагают, что они также легче гибридизируются с сигнальными полипептидами секреции и с растворимыми гидрофильными доменами.
Различные объекты, разработанные выше для усиления персистенции клеток, особенно предпочтительны, если интродуцируют экзогенную кодирующую последовательность путем разрушения эндогенного гена, кодирующего β2m или другой компонент ГКГ.
В определенных вариантах осуществления настоящего изобретения предусматривают интеграцию ингибиторов клеток NK для повышения персистенции сконструированных Т-клеток по настоящему изобретению, которые основаны на методах, подробно описанных в описании настоящего изобретения и проиллюстрированных на фиг. 17-21 и в примере 3.
В частности, настоящее изобретение дополнительно предусматривает методы получения сконструированных первичных иммунных клеток для клеточной иммунотерапии, причем указанный метод включает:
- получение популяции клеток, включающей Т-клетки, предпочтительно первичные Т-клетки,
- интродукцию в пропорции указанных Т-клеток:
i) по меньшей мере, одной нуклеиновой кислоты, содержащей экзогенную полинуклеотидную последовательность, которая должна быть интегрирована в выбранный эндогенный локус для кодирования по меньшей мере одного ингибитора клеток NK,
ii) по меньшей мере, одного специфичного для последовательности реагента, который специфически нацеливается на указанный выбранный эндогенный локус,
- причем указанную экзогенную полинуклеотидную последовательность инсертируют путем интеграции целевого гена в указанный эндогенный локус.
Под ингибитором клеток NK подразумевают полипептид, который придает аллогенным Т-клеткам защитное свойство против истощения клетками NK in vivo или при совместном культивировании с иммунными клетками. Такое истощение без ингибитора клеток NK показано, например, на фиг. 17.
Примеры ингибиторов клеток NK предусмотрены в примере 3.
Экзогенную полинуклеотидную последовательность, кодирующую ингибитор клеток NK, которая предпочтительно содержит одну из последовательностей, указанных в примере 3, предпочтительно интегрируют под контроль транскрипции эндогенного промотора, присутствующего в указанном локусе, для получения более постоянной экспрессии указанного ингибитора клеток NK.
В предпочтительном варианте осуществления настоящего изобретения указанный эндогенный промотор выбран по активности во время активации иммунных клеток, например, локусы, перечисленные в таблице 6, которые считают активно транскрибируемыми во время активации Т-клеток, по меньшей мере считают чувствительными к активации Т-клеток, наделенных химерным антигенным рецептором (chimeric antigen receptor, CAR).
В предпочтительных вариантах осуществления настоящего изобретения экзогенную последовательность, кодирующую ингибитор NK, интегрируют в эндогенный локус, регуляция которого возрастает на протяжении более 24 ч после активации Т-клеток, например, выбранный из Gzmb, Tbx21, Plek, Chekl, Slamf7, Zbtb32, Tigit, Lag3, Gzma, Weel, IL12rb2, Eeal и Dtl.
В предпочтительных вариантах осуществления настоящего изобретения экзогенную последовательность, кодирующую ингибитор NK, интегрируют в эндогенный локус, который конститутивно экспрессируется, например, в локусе TCR.
В настоящем изобретении может быть целесообразно интегрировать экзогенную последовательность, кодирующую ингибитор NK, в локус инсерции, экспрессирующей компонент ГКГ I, в частности в локус (β2m, который также является конститутивно транскрибируемым локусом.
В настоящем изобретении может быть полезно инактивировать эндогенную кодирующую последовательность β2m, в то же время иметь в этом локусе интегрированную транскрибируемую экзогенную последовательность, кодирующую ингибитор NK.
В предпочтительном варианте осуществления настоящего изобретения сконструированные Т-клетки, содержащие указанную экзогенную последовательность, кодирующую ингибитор NK, наделены химерным антигенным рецептором (CAR), как описано в тексте настоящего изобретения. Указанный химерный антигенный рецептор (CAR) может быть преимущественно интегрирован в локус TCR, тогда как экзогенная последовательность, кодирующая ингибитор NK, предпочтительно интегрирована в локус β2m, тем самым предотвращая или уменьшая как экспрессию TCR, так и/или β2m.
Реагент, специфичный в отношении последовательности, используемый в данном методе, предпочтительно является редкощепящей эндонуклеазой, описанной в настоящем изобретении выше и известной специалистам в этой области. Целевую интеграцию генов обычно осуществляют путем гомологичной рекомбинации или NHEJ в указанные иммунные клетки. Указанный специфический эндонуклеазный реагент предпочтительно выбирают из РНК-или ДНК-направляемой эндонуклеазы, например Cas9 или Cpf1, РНК-guide или ДНК-guide, TAL-эндонуклеазы, цинк-пальцевой нуклеазы, хоминг-эндонуклеазы или какой-либо их комбинации.
Еще один предпочтительный вариант осуществления настоящего изобретения проиллюстрирован в примере 3, в котором TALE-нуклеазы оптимизированы и успешно использованы для осуществления интеграции генов в локус β2m путем ограничения расщепления вне сайта в Т-клетках человека. Лучшая специфичность и эффективность были неожиданно получены с использованием TALE-нуклеазных гетеродимеров полипептидных последовательностей SEQ ID NO: 80, и/или SEQ ID NO: 81, или SEQ ID NO: 82, и/или SEQ ID NO: 83 правые и левые димеры, соответственно. Таким образом, настоящая патентная заявка конкретно относится к приведенным выше полипептидным последовательностям, кодирующим такую специфическую в отношении β2m TALEN, одну или в парах, или какую-либо эндонуклеазную последовательность, включающую повторы TAL, включающие одну из следующих последовательностей RVD:
- HD-HD-NN-NG-NN-NN-HD-HD-NG-NG-NI-NN-HD-NG-NN
- HD-HD-NI-NN-NN-HD-HD-NI-NN-NI-NI-NI-NN-NI-NN
- NG-NI-NN-HD-NG-NN-NG-NN-HD-NG-HD-NN-HD-NN-HD
- NN-NN-NI-NG-NI-NN-HD-HD-NG-HD-HD-NI-NN-NN-HD
а также к любым полинуклеотидам и векторам, кодирующим эти полипептиды.
Кроме того, задача настоящего изобретения заключается в интеграции экзогенной последовательности в геном Т-клетки с использованием эндонуклеазы, которая специфически распознает или связывает геномную последовательность β2m, содержащую одну из следующих последовательностей-мишеней SEQ ID NO. 78 и SEQ ID NO.79.
Также целью настоящего изобретения является инактивация геномной последовательности β2m в Т-клетке за счет использования эндонуклеазы, которая специфически распознает последовательность, содержащую последовательности-мишени SEQ ID NO. 78 и SEQ ID NO. 79.
Согласно настоящему изобретению, указанная экзогенная последовательность, кодирующая ингибиторы NK, предпочтительно содержит последовательности, кодирующие неполиморфные молекулы класса I, такие как HLA-G или HLA-E, или по меньшей мере фрагмент (фрагменты), содержащий тяжелую цепь этих молекул.
В предпочтительном варианте осуществления настоящего изобретения указанная экзогенная последовательность, если она интегрирована в эндогенный локус β2m, приводит к экспрессии гибридизации ее фрагмента HLA-E или HLA-G с фрагментами β2m, что обычно приводит к экспрессии димеров или тримеров HLA-E или HLA-G, например, показанных на фиг. 20 и приведенных в качестве примера в примере 3.
В соответствии с предпочтительным вариантом осуществления настоящего изобретения указанная экзогенная последовательность кодирует полипептид, который по меньшей мере на 80% идентичен аминокислотной
последовательности, выбранной из последовательностей с SEQ ID NO. 84 по SEQ ID NO. 90.
Экзогенная последовательность, кодирующая ингибиторы NK, также может содержать последовательности, кодирующие вирусные эвазины фрагментов, содержащих их эпитоп, например, из UL16 (также называемого ULBP1 - Uniprot ref.:#Q9BZM6) или UL18.
Интеграция экзогенной последовательности, кодирующей ингибитор (ингибиторы) NK, в геном Т-клетки, предпочтительно действующий на β2m, может сочетаться с инсерцией другой экзогенной последовательности, описанной в настоящем изобретении, с целью улучшения потенции и пригодности Т-клетки для адаптивной клеточной иммунотерапии.
В другом варианте осуществления настоящего изобретения экзогенная последовательность, кодирующая ингибитор (ингибиторы) NK, также может быть преимущественно интегрирована в локусы, кодирующие иммунные контрольные точки, такие как PD1 или CTLA4 (см. полный список в настоящем описании), предпочтительно с эффектом инактивации этих генов.
В настоящем изобретении описано большое количество примеров других эффективных локусов, которые могут быть полезны для придания дополнительных терапевтических свойств сконструированным Т-клеткам, например, придание устойчивости к лекарственным средствам, обычно используемым в терапии рака, например локусы DCK, HPRT или глюкокортикоидные рецепторы (Glucocorticoids receptor, GR).
Таким образом, в настоящем описании приводят сконструированные первичные иммунные клетки, которые можно получить описанным выше методом.
Такие иммунные клетки могут иметь следующие свойства:
1) Сконструированная Т-клетка содержит экзогенную последовательность, кодирующую ингибитор NK, которая интегрирована под контроль транскрипции промотором эндогенного гена.
2) Сконструированная Т-клетка по какому-либо варианту по пункту 1, в которой указанный промотор эндогенного гена выбран в одном из локусов, перечисленных в табл. 6.
3) Сконструированная Т-клетка по какому-либо из пунктов 1 или 2, в которых указанная экзогенная последовательность, кодирующая ингибитор NK, интегрирована в локус β2m.
4) Сконструированная Т-клетка по какому-либо из пунктов 1-3, в которых указанная Т-клетка наделена химерным антигенным рецептором.
5) Сконструированная Т-клетка по п. 4, которая имеет генотип [TCR]отрицательный[β2m]отрицательный[CAR]положительный.
6) Сконструированная Т-клетка по пунктам 4 или 5, в которой экзогенная последовательность (последовательности), кодирующая указанный CAR, интегрирована в локус TCR.
7) Сконструированная Т-клетка по какому-либо из пунктов 1-6, в которой указанная Т-клетка является первичной клеткой.
8) Сконструированная Т-клетка по какому-либо из пунктов 1-7 для ее использования в лечении рака или инфекции.
9) Терапевтически эффективная популяция иммунных клеток, включающая по меньшей мере 30%, предпочтительно 50%, более предпочтительно 80% сконструированных Т-клеток по какому-либо из пунктов 1-8.
Предпочтительные сконструированные Т-клетки согласно настоящему изобретению содержат полинуклеотидную последовательность, которая имеет идентичность по меньшей мере на 80%, более предпочтительно на 90%, еще более предпочтительно на 95% с одной из полинуклеотидных последовательностей SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74 или SEQ ID NO. 76 (интеграция тримерной матрицы в локус β2m, как описано в примере 3).
Примеры предпочтительных генотипов сконструированных Т-клеток по настоящему изобретению в связи с другими этапами редактирования генов, описанными в настоящем изобретении, следующие:
-[CAR]положительный[TCR]отрицательный[β2m]отрицательный[PD1]отрицательный
-[CAR]положительный[TCR]отрицательный[β2m]отрицательный[DCK]отрицательный
-[CAR]положительный[TCR]отрицательный[β2m]отрицательный[CTLA4]отрицательный
Настоящее изобретение также предусматривает терапевтические композиции, содержащие сконструированные клетки по настоящему изобретению, в частности следующие:
1) Терапевтически эффективную популяцию иммунных клеток согласно настоящему изобретению, в которой по меньшей мере 30%, предпочтительно 50%, более предпочтительно 80% клеток происходят от донора, предпочтительно от одного донора.
2) Популяцию первичных иммунных клеток в соответствии с вышеуказанным, где более 50% указанных иммунных клеток являются TCR-отрицательными Т-клетками.
3) Популяцию первичных иммунных клеток в соответствии с вышеуказанным, где более 50% указанных иммунных клеток являются CAR-положительными клетками.
4) Фармацевтическую композицию, содержащую сконструированную популяцию иммунных клеток в соответствии с вышеуказанным.
5) Метод лечения пациента, нуждающегося в этом, включающий:
- получение популяции сконструированных первичных иммунных клеток в соответствии с вышеуказанным,
- необязательно очистку или сортировку указанных сконструированных первичных иммунных клеток,
- активацию указанной популяции сконструированных первичных иммунных клеток до или после инфузии указанных клеток указанному пациенту.
6) Описанный выше метод, в котором пациента лечат от рака.
7) Описанный выше метод, в котором пациента лечат от инфекции. Настоящее изобретение дополнительно предусматривает описанный ниже метод скрининга кандидатов ингибиторов NK путем интеграции экзогенных последовательностей в Т-клетки:
1) метод идентификации соответствующей последовательности, кодирующей ингибитор NK, экспрессируемый в Т-клетке, причем указанный метод включает по меньшей мере стадии:
- обеспечения Т-клетками, в которых экспрессии как TCR, так и β2m репрессированы и/или инактивированы,
- интеграции последовательности-кандидата, кодирующей предполагаемый ингибитор NK, в эндогенный локус под контролем эндогенного промотора в указанной Т-клетке,
- культивирования полученных сконструированных Т-клеток в присутствии клеток NK,
2) метод идентификации подходящей последовательности, кодирующей ингибитор NK, экспрессируемый в Т-клетке, причем указанный метод включает по меньшей мере стадии:
- обеспечения Т-клетками, в которых экспрессия TCR репрессирована или инактивирована,
- инактивации экспрессии β2m в указанных Т-клетках путем интеграции последовательности-кандидата, кодирующей предполагаемый ингибитор NK, в локус β2m, причем экспрессия указанного предполагаемого ингибитора NK находится под контролем транскрипции эндогенного промотора указанного локуса β2m,
- культивирования полученных сконструированных Т-клеток в присутствии NK-клеток,
3) описанный выше метод, в котором дополнительно имеется стадия: придания указанным Т-клеткам химерного антигенного рецептора, 4) описанный выше метод, который дополнительно включает стадии:
- сравнения выживания указанных полученных сконструированных Т-клеток с такими же клетками, не экспрессирующими указанной последовательности-кандидата,
- необязательно выбор сконструированных клеток, которые более устойчивы к клеткам NK.
Повышение терапевтической активности иммунных клеток
В одном из вариантов осуществления настоящего изобретения в методе экзогенная последовательность, которая интегрирована в локус генома иммунных клеток, кодирует молекулу, которая повышает терапевтическую активность иммунных клеток.
Под «повышением терапевтической активности» подразумевают, что иммунные клетки или популяция иммунных клеток, сконструированных в соответствии с настоящим изобретением, становятся более агрессивными, чем не подвергшиеся конструированию клетки или популяция клеток по отношению к выбранному типу клеток-мишеней. Указанные клетки-мишени обычно принадлежат к определенному типу клеток или популяции клеток, предпочтительно характеризующихся общим поверхностным маркером (или маркерами). В настоящем описании «терапевтический потенциал» отражает терапевтическую активность, измеренную в экспериментах in vitro. В целом чувствительные линии раковых клеток, например клетки Дауди, используют для того, чтобы оценить, являются ли иммунные клетки более или менее активными по отношению к указанным клеткам, путем измерения лизиса или снижения роста клеток. Это также можно оценить путем измерения уровней дегрануляции иммунных клеток или продукции хемокинов и цитокинов. Эксперименты также могут быть выполнены на мышах с инъекцией опухолевых клеток и последующим мониторингом роста опухоли. Повышение активности считается значительным, когда число развивающихся клеток в этих экспериментах уменьшается под действием иммунных клеток более чем на 10%, более предпочтительно более чем на 20%, еще более предпочтительно более чем на 30%, и еще более предпочтительно более чем на 50%.
В одном из вариантов осуществления настоящего изобретения указанная экзогенная последовательность кодирует хемокин или цитокин, например, IL-12. Особенно целесообразно экспрессировать IL-12, поскольку в литературе этот цитокин широко упоминается как стимулирующий активацию иммунных клеток (Colombo M.P. с соавт., 13(2), 2002, 155-168).
В одном из предпочтительных вариантов осуществления настоящего изобретения экзогенная кодирующая последовательность кодирует или стимулирует секретируемые факторы, которые воздействуют на другие популяции иммунных клеток, такие как Т-регуляторные клетки, для ослабления их ингибирующего действия на указанные иммунные клетки.
В одном из вариантов осуществления настоящего изобретения указанная экзогенная последовательность, кодирующая ингибитор регуляторной активности Т-клеток, является полипептидным ингибитором фактором 3 из семейства транскрипционных ингибиторов forkhead/winged helix (FoxP3), и более предпочтительно является проникающим в клетку пептидным ингибитором FoxP3, например, ингибитором, обозначаемым Р60 (Casares N. с соавт., J Immunol 185(9), 2010, 5150-5159).
Под «ингибитором регуляторной активности Т-клеток» подразумевают молекулу или предшественника указанной молекулы, секретируемого Т-клетками и позволяющего Т-клеткам избегать понижающей регуляторной активности, оказываемой на них регуляторными Т-клетками. Обычно такой ингибитор регуляторной активности Т-клеток обладает эффектом снижения транскрипционной активности FoxP3 в указанных клетках.
В одном из вариантов осуществления настоящего изобретения указанная экзогенная последовательность кодирует секретируемый ингибитор ассоциированных с опухолью макрофагов (Tumor Associated Macrophages, ТАМ), например, агент нейтрализации CCR2/CCL2. Ассоциированные с опухолью макрофаги (ТАМ) являются критическими модуляторами микроокружения опухоли. Клинико-патологические исследования показали, что накопление ТАМ в опухолях коррелирует с плохим клиническим исходом. В соответствии с этими данными экспериментальные исследования и исследования на животных подтвердили, что ТАМ могут обеспечивать благоприятную микросреду для развития и прогрессирования опухоли (Theerawut С. с соавт., Cancers (Basel) 6(3), 2014, 1670-1690). Хемокин лиганд 2 (CCL2), также называемый моноцитарным хемоаттрактант-белком-1 (МСР1 - NCBI NP_002973.1), представляет собой небольшой цитокин, принадлежащий к семейству хемокинов СС, секретируемый макрофагами, который вызывает хемоаттракцию на моноцитах, лимфоцитах и базофилах. CCR2 (С-С хемокиновый рецептор типа 2 - NCBI NP 001116513.2) является рецептором CCL2.
Повышение специфичности и безопасности иммунных клеток
Экспрессируемые химерные антигенные рецепторы (CAR) стали современным средством для направления или улучшения специфичности первичных иммунных клеток, таких как Т-клетки и клетки NK, для лечения опухолей или инфицированных клеток. CAR, экспрессируемые этими иммунными клетками, специфически нацелены на антигенные маркеры на поверхности патологических клеток, которые дополнительно помогают указанным иммунным клеткам разрушать эти клетки in vivo (Sadelain М. с соавт., Cancer Discov. 3(4), 2013, 388-398). CAR обычно разрабатывают для включения доменов активации, которые стимулируют иммунные клетки в ответ на связывание со специфическим антигеном (так называемый положительный CAR), но они также могут содержать ингибирующий домен с противоположным эффектом (так называемый отрицательный CAR) (Fedorov, V. D., Cancer Journal 20(2), 2014, 160 165). Положительные и отрицательные CAR могут быть объединены или совместно экспрессированы для тонкой настройки иммунной специфичности клеток в зависимости от различных антигенов, присутствующих на поверхности клеток-мишеней.
Генетические последовательности, кодирующие CAR, обычно интродуцируют в геном клеток, используя ретровирусные векторы, которые обладают повышенной эффективностью трансдукции, но интегрируются в случайных местах. Согласно настоящему изобретению, компоненты химерного антигенного рецептора (CAR) можно вводить в выбранные локусы, более точно, под контроль эндогенных промоторов посредством направленной генной рекомбинации.
В одном из вариантов осуществления настоящего изобретения, если положительный CAR интродуцируют в иммунную клетку вирусным вектором, то отрицательный CAR может быть интродуцирован направленной генной инсерцией, и наоборот, причем предпочтительно активность проявляется только во время активации иммунных клеток. Соответственно, ингибирующий (т.е. отрицательный) CAR способствует повышению специфичности, предотвращая атаку иммунных клеток на данный тип клеток, который необходимо сохранить. В соответствии с этим аспектом указанный отрицательный CAR может быть апоптозным CAR, что означает, что указанный CAR содержит домен апоптоза, например, FasL (CD95 - NCBI: NP_000034.1) или его функциональный вариант, который преобразует сигнал, вызывающий гибель клетки (Eberstadt М. с соавт., Nature. 392(6679, 1998, 941-945).
Соответственно, экзогенная кодирующая последовательность, инсертированная в соответствии с настоящим изобретением, может кодировать фактор, который обладает способностью вызывать гибель клеток непосредственно, в сочетании с другим соединением или путем активации другого соединения (или соединений).
В качестве еще одного метода повышения безопасности для людей первичных иммунных клеток, экзогенная кодирующая последовательность может кодировать молекулы, которые придают чувствительность иммунным клеткам к лекарствам или другим экзогенным субстратам. Такими молекулами могут быть цитохром (цитохромы), например, из семейства Р450 (Preissner S. с соавт., Nucleic Acids Res 38, 2010, (Database issue): D237-243), например, CYP2D6-1 (NCBI - NP_000097.3), CYP2D6-2 (NCBI - NP_001020332.2), CYP2C9 (), CYP3A4 (NCBI - NP_000762.2), CYP2C19 (NCBI - NP_000760.1) или CYP1A2 (NCBI - NP_000752.2.), наделяющие гиперчувствительностью иммунные клетки к лекарству, например, к циклофосфамиду и/или изофосфамиду.
В соответствии с другим вариантом осуществления настоящего изобретения экзогенную последовательность интродуцируют в сконструированные иммунные клетки для ее экспрессии, особенно in vivo, для снижения передачи сигналов IL-6 или IL-8 с позиции контроля возможного синдрома высвобождения цитокинов (Cytokine Release Syndrome, CRS). Такая экзогенная последовательность может кодировать, например, антитела, направленные против IL-6 или IL-8 или против их рецепторов IL-6R или IL-8R (Shannon L. с соавт., Cancer J. 20(2), 2014, 119-122).
Точнее, синдром высвобождения цитокинов (CRS) можно ослабить, вмешиваясь в синдром активации макрофагов, который является одним из компонентов CRS [Lee D.W. с соавт., Blood. 124, 2014, 188-195]. Для достижения этой цели настоящее изобретение предусматривает интеграцию экзогенных последовательностей, кодирующих антагонистов активирующих метаболических путей IL1 или IL18, например, IL1RA (Uniprot # P18510) или IL18BP (Uniprot # 095998) или их активные фрагменты или варианты.
Понятие «антагонисты пути активации IL1 и IL18» означает какой-либо полипептид, способный влиять на повышенную экспрессию IL1 или IL18. Соответственно, настоящее изобретение предусматривает методы генерации терапевтических клеток, в которых экзогенные последовательности, кодирующие IL1RA или IL18BP, интегрированы в выбранные локусы, особенно PD1, CD25 или CD69, и более предпочтительно в комбинации с экспрессией CAR, необязательно интегрированного в локусы TCR и/или PD1.
Предпочтительные экзогенные последовательности кодируют антагонистов или антитела, которые одобрены фармацевтическими агентствами, такими как те, которые продаются под следующими названиями и ссылками:
- Анакинра (регистрационный номер CAS: 143090-92-0) (торговая марка Kineret) представляет собой рекомбинантную версию антагониста рецептора интерлейкина 1 (IL1-RA). Анакинра представляет собой вариант формы IL1RA, поскольку она отличается от нативного IL-1Ra человека добавлением одного остатка метионина на его амино-конце. Анакинра блокирует биологическую активность природного IL-1 [Kalliolias G.D. с соавт., Expert Opin Investig Drugs. 17(3), 2008, 349 359].
- Рилонацепт (регистрационный номер CAS: 501081-76-1), также известный как ловушка IL-1 (фирма Regeneron Pharmaceuticals, торговая марка Arcalyst), также является ингибитором интерлейкина 1. Это димерный слитый белок, состоящий из лиганд-связывающих доменов внеклеточных частей рецепторного компонента интерлейкина-1 человека (IL-1R1) и вспомогательного белка рецептора IL-1 (IL-1RAcP), связанных в линию с кристаллизуемым фрагментом (область Fc) IgG1 человека, который связывает и нейтрализует IL-1 [McDermott M.F., Drugs of Today. 45(6), 2009, 423-430].
- Канакинумаб (торговая марка Ilaris - регистрационный номер CAS: 914613-48-2) является моноклональным антителом человека, нацеленным на интерлейкин-1 бета. Он не имеет перекрестной реактивности с другими членами семейства интерлейкинов-1, включая интерлейкин-1 альфа [Rondeau J.M. с соавт., MAbs. 7(6), 2015, 1151 1160].
- Тоцилизумаб (торговая марка Actemra - регистрационный номер CAS: 375823-41-9) представляет собой гуманизированное моноклональное антитело против рецептора интерлейкина-6 (IL-6R) [Venkiteshwaran A., MAbs. 1(5), 2009, 432-438].
- Силтуксимаб (торговая марка Sylvant - регистрационный номер CAS: 541502-14-1) является химерным моноклональным антителом против IL-6 или cCLB8, представляет собой химерное полученное из белков человека и мыши моноклональное антитело, которое связывается с интерлейкином-6 [Rhee F. с соавт., Journal of Clinical Oncology 28 (23), 2010, 3701-3708].
В предпочтительном варианте осуществления настоящего изобретения указанная экзогенная последовательность может кодировать растворимый внеклеточный домен GP130, такой как домен, демонстрирующий по меньшей мере 80% идентичность с SEQ ID NO. 61.
Такой растворимый внеклеточный домен GP130 описанный, например, в публикации Rose-John S., Clinical Pharmacology & Therapeutics, 102(4), 2017, 591-598, может быть гибридизирован с фрагментами иммуноглобулинов, например, sgp130Fc (SEQ ID NO.62). Выше было отмечено, что указанная экзогенная последовательность может быть стабильно интегрирована в геном с помощью сайт-направленного мутагенеза (т.е. с использованием специфических для последовательности нуклеазных реагентов) и может находиться под транскрипционной активностью эндогенного промотора в локусе, который активен во время активации иммунных клеток, например, в одном из перечисленных в табл. 6, 8 или 9, и предпочтительно активирован при активации CAR или зависит от CAR.
В более предпочтительном варианте осуществления настоящего изобретения экзогенную последовательность интродуцируют в CAR-положительные иммунные клетки, например, анти-CD22 CAR Т-клеток экспрессирующую полинуклеотидную последовательность, например, SEQ ID NO: 31. В соответствии с некоторыми определенными вариантами осуществления настоящего изобретения указанную экзогенную последовательность, кодирующую полипептид, который может связываться и предпочтительно мешать рецептору цитокинов семейства рецепторов IL-6, такому как указанный растворимый внеклеточный домен GP130, интегрируют в локус PD1, CD25 или CD69. По настоящему изобретению, эндогенная последовательность, кодирующая локус PD1, предпочтительно нарушается указанной экзогенной последовательностью.
Таким образом, в настоящем изобретении описывают способ лечения или уменьшения синдрома высвобождения цитокинов (CRS) в клеточной иммунотерапии, по которому клетки или их терапевтические композиции вводят пациентам, причем указанные клетки генетически модифицированы для секреции полипептида (полипептидов), содержащих растворимый внеклеточный домен GP130, sGP130Fc, анти-IL-6 или анти-IL6R антитело, анти-IL-8 или анти-IL8R антитело, или какие-либо их гибриды.
Примерами предпочтительных генотипов сконструированных иммунных клеток являются:

Повышение эффективности направленной генной инсерции в первичные иммунные клетки с использованием векторов AAV
В настоящем изобретении предусматривают матрицы доноров и реагенты, специфичные в отношении последовательности, как показано на фигурах, которые полезны для эффективной инсерции кодирующей последовательности в рамку с эндогенными промоторами, в частности PD1 и CD25, а также средства и последовательности для выявления правильной инсерции указанных экзогенных последовательностей в указанных локусах.
Матрицы доноров в соответствии с настоящим изобретением обычно являются полинуклеотидными последовательностями, которые могут быть включены в различные векторы; описанные в данной области, для доставки матриц донора в ядро в то время, когда реагенты-эндонуклеазы становятся активными, чтобы получить их сайт-направленную инсерцию в геном обычно путем NHEJ или гомологичной рекомбинацией.
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии IL-15 (SEQ ID NO.59) в локусе PD1, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующую IL-15, например последовательность, идентичную SEQ ID NO: 50,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу PD1, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 45 и SEQ ID NO: 46,
- необязательно последовательность, кодирующую растворимую форму рецептора IL-15 (sIL-15R), например, последовательность, идентичную SEQ ID NO: 50,
- необязательно по меньшей мере один сайт расщепления пептида -2А, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии IL-12 (SEQ ID NO.58) в локусе PD1, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующую IL-12a, например последовательность, идентичную SEQ ID NO: 47,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу PD1, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 45 и SEQ ID NO: 46,
- необязательно последовательность, кодирующую IL-12b, например, последовательность идентичную SEQ ID NO: 48,
- необязательно по меньшей мере один сайт расщепления 2А пептида, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии растворимого GP130 (содержащего SEQ ID NO.61) в локусе PD1, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующая растворимый GP130, растворимый gp130, гибридизированный с Fc, например, идентичный SEQ ID N:62,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу PD1, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 45 и SEQ ID NO: 46,
- необязательно по меньшей мере один сайт расщепления 2А пептида, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии IL-15 (SEQ ID NO.59) в локусе CD25, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующую IL-15, например последовательность, идентичную SEQ ID NO: 50,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу CD25, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 43 и SEQ ID NO: 44,
- необязательно последовательность, кодирующую растворимую форму рецептора IL-15 (sIL-15R), например, последовательность, идентичную SEQ ID NO: 50,
- необязательно по меньшей мере один сайт расщепления 2А пептида, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии IL-12 (SEQ ID NO.58) в локусе CD25, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующую IL-12a, например последовательность, идентичную SEQ ID NO: 47,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу CD25, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 43 и SEQ ID NO: 44,
- необязательно последовательность, кодирующую IL-12b, например, последовательность идентичную SEQ ID NO: 48,
- необязательно по меньшей мере один сайт расщепления 2А пептида, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В частности, настоящее изобретение относится к специфическим полинуклеотидам доноров для экспрессии растворимого GP130 (содержащего SEQ ID NO.61) в локусе CD25, содержащем одну или несколько из следующих последовательностей:
- последовательность, кодирующую растворимый GP130, предпочтительно растворимый gp130, гибридизированный с Fc, например, идентичный SEQ ID NO:62,
- последовательности вверх и вниз по цепи (также называемые левой и правой последовательностями), гомологичные локусу CD25, содержащие предпочтительно полинуклеотидные последовательности SEQ ID NO: 43 и SEQ ID NO: 44,
- необязательно по меньшей мере один сайт расщепления 2А пептида, такой как один из SEQ ID NO: 53 (F2A), SEQ ID NO: 54 (P2A) и/или SEQ ID NO:55 (T2A).
В примерах настоящего изобретения существенно улучшили степень направленной на ген инсерции в клетки человека, используя векторы AAV, особенно векторы из семейства AAV6.
Таким образом, основным объектом настоящего изобретения является трансдукция векторов AAV в первичные иммунные клетки человека в сочетании с экспрессией специфических в отношении последовательности эндонуклеаз, применяемых в качестве реагентов, таких как эндонуклеазы TALE, более предпочтительно интродуцируемых в форме мРНК, для повышения гомологичной рекомбинации в этих клетках.
В одном из вариантов осуществления настоящего изобретения специфические в отношении последовательности эндонуклеазы, применяемые в качестве реагентов, могут быть интродуцированы в клетки путем трансфекции, более предпочтительно путем электропорации мРНК, кодирующей указанные специфические к последовательности реагенты-эндонуклеазы, такие как нуклеазы TALE.
В соответствии с таким основным объектом настоящего изобретения, в частности, предусматривают способ инсерции последовательности экзогенной нуклеиновой кислоты в последовательность эндогенного полинуклеотида в клетке, включающий по меньшей мере следующие стадии:
- трансдукции в указанную клетку вектора AAV, содержащего указанную последовательность экзогенной нуклеиновой кислоты и последовательности, гомологичные целевой эндогенной последовательности ДНК, и
- индукцию экспрессии специфического для последовательности эндонуклеазного реагента для расщепления указанной эндогенной последовательности в локусе осуществленной инсерции.
Полученная инсерция последовательности экзогенной нуклеиновой кислоты может привести к интродукции генетического материала, коррекции или замещении эндогенной последовательности, более предпочтительно «в рамке» в отношении последовательностей эндогенных генов в этом локусе.
В соответствии с другим объектом настоящего изобретения от 105 до 107, предпочтительно от 106 до 107, более предпочтительно примерно 5×106 вирусных геномов трансдуцируют в расчете на одну клетку.
Согласно другому объекту настоящего изобретения клетки могут быть обработаны ингибиторами протеосом, таких как бортезомиб, что дополнительно способствует гомологичной рекомбинации.
В качестве одного из объектов настоящего изобретения, применяемый в методе по настоящему изобретению вектор AAV может содержать экзогенную кодирующую последовательность без промотора, например, какую-либо из указанных в данном описании, для того, чтобы поместить ее под контроль эндогенного промотора в один локус, выбранный из тех, которые перечислены в настоящем описании.
В качестве одного из объектов настоящего изобретения, вектор AAV, применяемый в методе по настоящему изобретению, может содержать сайт расщепления 2А пептида, за которым следует кДНК (за исключением стартового кодона), образующая экзогенную кодирующую последовательность.
В качестве одного из объектов настоящего изобретения указанный вектор AAV содержит экзогенную последовательность, кодирующую химерный антигенный рецептор, особенно анти-CD19 CAR, анти-CD22 CAR, анти-CD123 CAR, анти-CS1 CAR, анти-CCL1 CAR, анти-HSP70 CAR, анти-GD3 CAR или анти-ROR1 CAR.
Таким образом, настоящее изобретение охватывает какие-либо векторы AAV, разработанные для осуществления описанного в настоящем изобретении способа, особенно векторы, содержащие последовательность, гомологичную локусу инсерции, расположенному в каком-либо из эндогенных генов, чувствительных к активации Т-клеток, указанных в табл. 4.
Многие другие векторы, известные в данной области, такие как плазмиды, эписомальные векторы, линейные матрицы ДНК и т.д., также могут быть применимы в соответствии с настоящим изобретением.
Ранее указывалось, что ДНК-вектор, используемый в соответствии с настоящим изобретением, предпочтительно содержит: (1) указанную экзогенную нуклеиновую кислоту, содержащую экзогенную кодирующую последовательность, которая должна быть инсертирована путем гомологичной рекомбинации, и (2) последовательность, кодирующую специфический для последовательности эндонуклеазный реагент, который способствует указанной инсерции. Согласно более предпочтительному варианту осуществления настоящего изобретения указанная экзогенная нуклеиновая кислота в соответствии с (1) не содержит какой-либо последовательности промотора, тогда как последовательность в (2) имеет свой собственный промотор. Согласно еще более предпочтительному вариант осуществления настоящего изобретения нуклеиновая кислота согласно (1) содержит внутренний сайт входа в рибосому (IRES) или «саморасщепляющиеся» 2А пептиды, такие как Т2А, Р2А, Е2А или F2A, таким образом, что эндогенный ген, в который инсертирована экзогенная кодирующая последовательность, становится мультицистронным. IRES 2A пептида может предшествовать или следовать после указанной экзогенной кодирующей последовательности.
Предпочтительными векторами по настоящему изобретению являются векторы, производные от AAV6, содержащие полинуклеотиды доноров, согласно описанному ранее в настоящем описании или проиллюстрированному в экспериментальном разделе и в фигурах. Примеры векторов по настоящему изобретению содержат или состоят из полинуклеотидов, имеющих идентичность с последовательностями SEQ ID NO: 37 (матрица для интеграции последовательности, кодирующей IL-15 в локус CD25), SEQ ID NO: 38 (матрица для интеграции последовательности, кодирующей IL-15 в локус PD1), SEQ ID NO: 39 (матрица для интеграции последовательности, кодирующей IL-12 в локус CD25), SEQ ID NO: 40 (матрица для интеграции последовательности, кодирующей IL-12 в локус PD1), SEQ ID NO: 69 (матрица для интеграции пептида HLAE VMAPRTLFL), SEQ ID NO: 71 (матрица для интеграции пептида HLAE VMAPRTLIL), SEQ ID NO: 73 (матрица для интеграции актинового пептида UL18 в локус B2m), SEQ ID NO: 75 (матрица для интеграции пептида UL18 HLACw, инсертированного в локус B2m), и SEQ ID NO: 77 (матрица для интеграции пептида UL18_βHLAG в локус β2m).
Нацеленная на гены интеграция в иммунные клетки под контролем транскрипции эндогенными промоторами
Настоящее изобретение в одном из основных вариантов его осуществления использует преимущества эндогенной транскрипционной активности иммунных клеток для экспрессии экзогенных последовательностей, которые улучшают их терапевтический потенциал.
Настоящее изобретение предусматривает несколько вариантов осуществления, основанных на профиле транскрипционной активности эндогенных промоторов и на выборе локусов промоторов, полезных для осуществления изобретения. Предпочтительными локусами являются те, у которых активность транскрипции обычно высока при активации иммунных клеток, особенно в ответ на активацию CAR (чувствительные к CAR промоторы), при условии, что клеткам придан CAR.
Соответственно, настоящее изобретение относится к способу получения аллогенных терапевтических иммунных клеток путем экспрессии первой экзогенной последовательности, кодирующей CAR в локусе TCR, тем самым нарушая экспрессию TCR, и экспрессии второй экзогенной кодирующей последовательности при транскрипционной активности эндогенного локуса, предпочтительно зависимого от следующих положений:
- активации CD3/CD28, например парамагнитными микрочастицами Dynabeads, которая полезна, например, для стимулирования размножения клеток,
- активации CAR, например, через путь CD3zeta, который полезен, например, для активации функций иммунных клеток на мишени;
- транскрипционной активности, связанной с проявлением симптома заболевания или молекулярного маркера, что полезно, например, для активации клеток in-situ в больных органах,
- дифференциации клеток, которая полезна для придания терапевтических свойств клеткам на заданном уровне дифференциации или для экспрессии белка в определенную родословную линию клеток (см. фиг.1), например, в то время, когда гемопоэтические клетки приобретают свои иммунные функции, и/или
- микроокружения опухоли (tumor microoenvironment, TME), которое полезно для перенаправления активности клеток и их амплификации в отношении специфических условий опухоли (гипоксии, низкого уровня глюкозы...) или для предотвращения истощения и/или поддержания активации,
- синдрома высвобождения цитокинов (CRS), который полезен для смягчения побочных эффектов, связанных с активностью CAR Т-клеток.
В настоящем изобретении впервые представлен разработанный список эндогенных генов (табл.6), которые, как было обнаружено, являются особенно подходящими для направленной генной рекомбинации согласно настоящему изобретению. При составлении этого списка было проанализировано несколько транскриптомных баз данных по грызунам, в частности, базы данных от Консорциума по проекту иммунологического генома, упомянутой в публикации Best J.A. с соавт., Nat. Immunol. 14 (4), 2013, 404-412, которая позволяет сравнивать уровни транскрипции различных генов при активации Т-клеток в ответ на антигены яичного альбумина. Кроме того, поскольку имеется очень мало данных, касающихся активации Т-клеток человека, были сделаны некоторые экстраполяции и анализ этих данных для сравнения с ситуацией в организме человека, изучая доступную литературу, связанную с генами человека. Выбранные локусы особенно актуальны для инсерции последовательностей, кодирующих CAR. Основываясь на первом отборе из табл.6, был сделан последующий отбор генов на основе ожидаемых профилей их экспрессии (табл. 7-10).
С другой стороны, авторы настоящего изобретения идентифицировали выбор локусов транскрипции, которые в основном неактивны, что было бы наиболее целесообразным для инсерции кассеты (кассет) экспрессии для экспрессии экзогенной кодирующей последовательности под контролем транскрипции экзогенными промоторами. Эти локусы называют «локусами безопасной гавани», так как они в основном неактивны в плане транскрипции, особенно во время активации Т-клеток. Они полезны для интеграции кодирующей последовательности за счет максимального снижения риска вмешательства в экспрессию генома иммунных клеток.
Нацеленная на ген инсерция контролем эндогенных промоторов, которые стабильно активны во время активации иммунных клеток
Выбор эндогенных генных локусов, связанных с данным вариантом осуществления настоящего изобретения, приведен в таблице 7.
Соответственно, способ по настоящему изобретению предусматривает стадию осуществления нацеленной на ген инсерции под контролем эндогенного промотора, который постоянно активен во время активации иммунных клеток, предпочтительно из эндогенного гена, выбранного из CD3G, Rn28s1, Rn18s, Rn7sk, Actg1, β2m, Rpl18a, Pabpc1, Gapdh, Rpl17, Rpl19, Rplp0, Cfl1 и Pfn1.
Понятие «стабильно активный» означает, что транскрипционная активность, наблюдаемая для этих промоторов в первичных иммунных клетках, не подвержена отрицательному регулированию при активации иммунной клетки.
В другой публикации сообщают (Acuto О., Nature Reviews Immunology 8, 2008, 699-712), что промоторы, присутствующие в локусе TCR, подвергаются различным механизмам отрицательной обратной связи после включения в TCR и, таким образом, их активность или повышенная регуляция могут не сохраняться постоянно на протяжении осуществления метода по настоящему изобретению. Настоящее изобретение в некоторой степени было разработано для того, чтобы избежать использования локуса TCR в качестве возможного сайта вставки для экзогенных кодирующих последовательностей, которые должны экспрессироваться во время активации Т-клеток. Следовательно, согласно одному из вариантов осуществления настоящего изобретения направленную инсерцию экзогенной кодирующей последовательности не осуществляют в генные локусы TCRalpha или TCRbeta.
Примерами экзогенных кодирующих последовательностей, которые могут быть полезны при интродукции в такие локусы под контроль стабильно активных эндогенных промоторов, являются те, которые кодируют или положительно регулируют выработку цитокина, рецептора хемокина, молекулы, придающей устойчивость к лекарству, совместную стимуляцию лиганда, например, 4-1BRL и OX40L, или секретируемого антитела.
Интеграция генов под эндогенными промоторами, которые зависят от активации иммунных клеток или зависят от активации CAR
Как указывалось ранее, метод по настоящему изобретению предусматривает стадию осуществления генной целевой инсерции под контролем эндогенного промотора, причем активность транскрипции предпочтительно активируется при активации иммунных клеток либо временно, либо в течение более 10 дней.
Под «активацией иммунных клеток» подразумевают выработку иммунного ответа в соответствии с механизмами, обычно описанными и общепринятыми в литературе для данного типа иммунных клеток. Например, в отношении Т-клеток активация Т-клеток обычно характеризуется одним из изменений, состоящих в экспрессии на клеточной поверхности путем продуцирования множества белков, включая CD69, CD71 и CD25 (также маркер для клеток Treg), и HLA-DR (маркер активации Т-клеток человека), высвобождение перфорина, гранзимов и гранулизина (дегрануляция) или продуцирование цитокиновых эффекторов IFN-γ, TNF и LT-альфа.
В предпочтительном варианте осуществления настоящего изобретения транскрипционная активность эндогенного гена активируется в иммунной клетке, особенно в ответ на активацию с помощью CAR. CAR может независимо экспрессироваться в иммунной клетке. Под «независимо экспрессируемым» подразумевают, что CAR может транскрибироваться в иммунной клетке из экзогенной экспрессирующей кассеты, интродуцированной, например, с использованием ретровирусного вектора, такого как лентивирусный вектор, или путем трансфекции кэпированных мРНК путем электропорации, кодирующей такой CAR. В данной области известны методы экспрессии CAR в иммунной клетке, как описано, например, (REF.).
Указанный эндогенный ген, чья транскрипционная активность повышается в результате регулирования, особенно подходит для интеграции экзогенных последовательностей, кодирующих цитокин (цитокины), такие как IL-12 и IL-15, иммуногенный пептид (пептиды) или секретируемое антитело, например, анти-IDO1, анти-IL10, анти-PD1, анти-PDL1, анти-IL6 или анти-PGE2 антитело.
В предпочтительном варианте осуществления настоящего изобретения эндогенный промотор выбран по его транскрипционной активности, которая является чувствительной и, более предпочтительно, зависит от активации CAR.
В настоящем описании показано, что CD69, CD25 и PD1 представляют собой такие локусы, которые особенно пригодны для инсерции и экспрессии экзогенных кодирующих последовательностей, которые должны экспрессироваться, когда активируются иммунные клетки, особенно в CAR-положительных иммунных клетках.
Таким образом, настоящее изобретение объединяет какие-либо методы экспрессии CAR в иммунных клетках со стадией выполнения сайт-направленной инсерции экзогенной кодирующей последовательности в локус, транскрипционная активность которого зависит от взаимодействия указанного CAR с опухолевым антигеном. В частности, способ включает стадию интродукции в CAR-положительные или рекомбинантные TCR-положительные иммунные клетки экзогенной последовательности, кодирующей IL-12 или IL-15, под контролем транскрипции одного промотора, выбранного из промоторов PD1, CD25 и CD69.
В частности, CAR-положительные клетки можно получить, следуя стадиям совместной экспрессии в иммунных клетках, предпочтительно первичных клетках и более предпочтительно в первичных Т-клетках по меньшей мере одной экзогенной последовательности, кодирующей CAR, и другой экзогенной последовательности, помещенной в зависимость от эндогенного промотора, чья транскрипционная активность зависит от указанного CAR, например, PD1, CD25 или CD71.
Выражение «зависит от указанного CAR» означает, что транскрипционную активность указанного эндогенного промотора необходимо увеличить более чем на 10%, предпочтительно более чем на 20%, более предпочтительно более чем на 50% и еще более предпочтительно более чем на 80%, поскольку результат взаимодействия CAR с его родственным антигеном в ситуации, когда, как правило, антигены превышают количество CAR, присутствующих на клеточной поверхности, и количество CAR, экспрессируемых на клеточной поверхности, составляет более 10 на клетку, предпочтительно более 100 и более предпочтительно более 1000 молекул на клетку.
Таким образом, настоящее изобретение раскрывает экспрессию последовательности CAR, предпочтительно инсертированной в локус TCR и конститутивно экспрессируемой, тогда как другая экзогенная последовательность, интегрированная в другом локусе, ко-экспрессируется в ответ или зависит от взаимодействия указанного CAR с его родственным антигеном. Указанным другим локусом является, например, CD25, PD1 или CD71 или какой-либо локус, специфически транскрибируемый при активации CAR.
Иначе говоря, настоящее изобретение обеспечивает совместную экспрессию CAR и по меньшей мере одной экзогенной кодирующей последовательности, причем экспрессия указанной экзогенной последовательности находится под контролем эндогенного промотора, транскрипционная активность которого зависит от активности CAR, причем это делается ввиду получения сконструированных иммунных клеток, обеспечивающих лучший иммунный ответ.
Ранее было описано, что это может быть выполнено путем трансфекции клеток специфичными в отношении последовательности нуклеазными реагентами, нацеленными на кодирующие области таких локусов, которые являются специфически зависимыми от CAR, наряду с матрицами доноров, содержащими последовательности, гомологичные указанным геномным областям. Нуклеазные реагенты, специфичные в отношении последовательности, помогают интегрировать матрицы доноров путем гомологичной рекомбинации или негомологичного соединение концов (Non-homologous end-joining, NHEJ).
В соответствии с предпочтительным вариантом осуществления настоящего изобретения экзогенную кодирующую последовательность интегрируют в рамку с эндогенным геном таким образом, что экспрессия указанного эндогенного гена сохраняется. Это происходит, например, в отношении CD25 и CD69 по меньшей мере в одном примере экспериментального раздела настоящего описания.
В соответствии с другим предпочтительным вариантом осуществления настоящего изобретения экзогенная кодирующая последовательность нарушает эндогенную кодирующую последовательность гена, чтобы предотвратить экспрессию одной эндогенной кодирующей последовательности, особенно когда эта экспрессия оказывает отрицательное воздействие на функции иммунных клеток, например, как в описанном в настоящем экспериментальном разделе случае с PD1.
В соответствии с еще одним более предпочтительным вариантом осуществления настоящего изобретения экзогенную кодирующую последовательность, которая нарушает последовательность эндогенного гена, помещают в рамку с эндогенным промотором таким образом, что ее экспрессия становится зависимой от эндогенного промотора, что также показано в экспериментальном разделе.
Настоящее изобретение также относится к полинуклеотидным и полипептидным последовательностям, кодирующим различные TAL-нуклеазы, приведенные в качестве примера в настоящем описании, особенно те, которые допускают сайт-направленную инсерцию в локус CD25 (SEQ ID NO: 18 и 19), а также их соответствующие мишени и последовательности RVD.
Настоящее изобретение также охватывает наборы для трансфекции иммунных клеток, содержащие полинуклеотиды, кодирующие специфичные для последовательности реагенты эндонуклеазы и последовательности доноров, разработанные для интеграции экзогенной последовательности в локус, на который нацелены указанные реагенты. Примером таких наборов является набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус PD1 (например, PD1 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-12, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус PD1 (например: PD1 TALEN®) и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-15, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус CD25 (ex: CD25 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-12, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус CD25 (например, CD25 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-15, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус PD1 (например: PD1 TALEN, ®), и вектор AAV, содержащий экзогенную последовательность, кодирующую растворимый gp130, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус CD25 (например, CD25 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую растворимый gp130, а также какие-либо наборы, включающие эндонуклеазные реагенты, нацеленные на гены, указанные в табл.6, и матрицу донора для интродукции кодирующей последовательности по настоящему изобретению.
Другими примерами таких наборов являются набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус PD1 (например, PD1 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-12, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус PD1 (например: PD1 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-15, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус CD25 (например: CD25 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-12, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус CD25 (например, CD25 TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую IL-15, набор, содержащий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус β2m (например, β2m TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую ингибитор NK, например, включающую тяжелую цепь от HLA-E или HLA-G, набор, включающий мРНК, кодирующую редкощепящую эндонуклеазу, нацеленную на локус β2m (например, β2m TALEN®), и вектор AAV, содержащий экзогенную последовательность, кодирующую растворимый gp130, и какие-либо наборы, включающие эндонуклеазные реагенты, нацеленные на гены, перечисленные в табл. 6, и матрицу донора для интродукции кодирующей последовательности, упомянутой в настоящем описании.
В настоящем изобретении также предусматривают наборы для трансфекции иммунных клеток, содержащие полинуклеотиды, кодирующие специфические в отношении последовательности эндонуклеазные реагенты, и экзогенную полинуклеотидную последовательность, предпочтительно включенную в вектор AAV, причем указанная экзогенная последовательность содержит последовательность, кодирующую антагонистов путей активации IL1 и IL18, например, IL1RA (Uniprot # Р18510), или IL18BP (Uniprot # O95998), или их активные фрагменты или варианты.
В одном из вариантов осуществления настоящего изобретения эндогенный ген выбран для слабого повышения регуляции. Экзогенная кодирующая последовательность, интродуцированная в указанный эндогенный ген, регуляция транскрипции которого слабо повышена, может быть преимущественно компонентом ингибирующего CAR или связанного с апоптозом CAR, уровень экспрессии которого обычно остается ниже, чем уровень положительного CAR. Такая комбинация экспрессии CAR, например, трансдуцированная вирусным вектором, и другая, интродуцированная в соответствии с настоящим изобретением, может значительно улучшить специфичность или безопасность CAR иммунных клеток.
У некоторых эндогенных промоторов регуляция повышается временно, иногда на протяжении менее чем 12 ч после активации иммунных клеток, например, у промоторов, выбранных из локусов эндогенных генов Spata6, Itga6, Rcbtb2, Cd1d1, St8sia4, Itgae и Fam214a (табл. 8). У других эндогенных промоторов регуляция повышается на протяжении менее чем 24 ч после активации иммунных клеток, например, у промоторов, выбранных из локусов эндогенных генов IL3, IL2, Ccl4, IL21, Gp49a, Nr4a3, Lilrb4, Cd200, Cdkn1a, Gzmc, Nr4a2, Cish, Ccr8, Lad1 и Crabp2 (табл. 9), и у других в течение более 24 ч, в более общем значении в течение более 10 дней, после активации иммунных клеток. Например, таких выбранных генов из Gzmb, Tbx21, Plek, Chek1, Slamf7, Zbtb32, Tigit, Lag3, Gzma, Weel, IL12rb2, Eea1 и Dtl (табл. 9).
В другом варианте в настоящем изобретении установлено, что эндогенный ген под контролем транскрипции промоторов, регуляция которых снижается при активации иммунных клеток, также может представлять интерес для способа по настоящему изобретению. Действительно, было высказано предположение, что экзогенные кодирующие последовательности, кодирующие анти-апоптотические факторы, такие как семейство Вс12, BclXL, NF-kB, сурвувин или анти-FAP (FAP - белок активации фибробластов, fibroblast activation protein), например, компонент CAR анти-FAP, могут быть интродуцированы в указанные локусы.
Указанный эндогенный ген под контролем транскрипции промоторов, регуляция которых понижается при активации иммунных клеток, может быть более конкретно выбран из Slc6a19, Cd55, Xkrx, Mturn, H2-Ob, Cnr2, Itgae, Raver 2, Zbtb20, Arrb1, Abca1, Tet1, Slc16a5 и Ampd3 (табл. 10).
Интеграция генов под контроль эндогенных промоторов, активированных в условиях микроокружения опухоли (МОО)
Один из объектов настоящего изобретения конкретно касается методов предупреждения истощения иммунных клеток в условиях истощения микроокружения опухоли (МОО). Истощение иммунных клеток часто происходит в ответ на истощение питательных веществ или молекулярные сигналы; фиксируемые в микроокружении опухолей, которые способствуют резистентности опухоли. Метод включает стадии конструирования клеток путем интеграции экзогенных кодирующих последовательностей под контролем эндогенных промоторов, регуляция которых активируется при истощении аргинина, цистеина, триптофана и кислорода, а также внеклеточного ацидоза (накопление лактата).
Такие экзогенные последовательности могут кодировать химерные антигенные рецепторы, интерлейкины или какой-либо полипептид, упоминаемый в тексте настоящего описания, данный в другом месте данного описания, для усиления функции или активации иммунных клеток и/или улучшения терапевтического действия.
В настоящем изобретении перечисляют ряд локусов, для которых установлено повышение регуляции в большом количестве истощенных опухолевых инфильтрующихся лимфоцитов (tumor infiltrating lymphocytes, TIL), которые перечислены в таблицах 12 и 13. В настоящем изобретении предусматривают стадию интеграции экзогенных кодирующих последовательностей в указанные предпочтительные локусы для предупреждения истощения иммунных клеток, в частности Т-клеток, в микроокружении опухоли.
Например, экзогенные последовательности, кодирующие СА6, могут быть помещены под контроль транскрипции промоторов эндогенных генов, которые активируются микроокружением опухоли, например, HIF1a, фактором транскрипции, а именно фактором, индуцируемым гипоксией, или арил-углеводородным рецептором (aryl hydrocarbon receptor, AhR). Эти гены являются сенсорами, соответственно, индуцируемыми гипоксией и ксенобиотиками, в тесном контакте с опухолями.
Таким образом, настоящее изобретение применимо для улучшения результата CAR Т-клеточной терапии путем интеграции экзогенных кодирующих последовательностей, и, в более общем плане, генетических признаков/участков под контролем эндогенных промоторов Т-клеток, на которые воздействует микроокружение опухоли (МОО).
В соответствии с настоящим изобретением повышение регуляции эндогенных генов может быть «пресечено» для повторной экспрессии соответствующих экзогенных кодирующих последовательностей для улучшения противоопухолевой активности CAR Т-клеток в микроокружении некоторых опухолей.
Нацеленная на ген инсерция и экспрессия в гемопоэтических стволовых клетках (ГСК)
Один из объектов настоящего изобретения конкретно касается инсерции трансгенов в гемопоэтические стволовые клетки (hematopoietic stem cell, ГСК).
Гемопоэтические стволовые клетки (ГСК) являются мультипотентными само обновляющимися клетками-предшественниками, от которых в результате дифференциации образуются все типы клеток крови в ходе процесса гемопоэза. К этим клеткам относятся лимфоциты, гранулоциты и макрофаги иммунной системы, а также эритроциты и тромбоциты в кровяном русле. В классическом варианте ГСК предположительно дифференцируются в две ограниченные по происхождению лимфоидные и миелоэритроидные олигопотентные клетки-предшественники. Механизмы, контролирующие самообновление ГСК и дифференциацию, предположительно подвергаются воздействию разнообразных групп цитокинов, хемокинов, рецепторов и межклеточных сигнальных молекул. В частности, дифференциация ГСК регулируется факторами роста и цитокинами, включая колониестимулирующие факторы (colony-stimulating factor, CSF) и интерлейкины (interleukin, IL), которые активируют межклеточные сигнальные пути. Известно, что факторы, отображенные ниже, влияют на мультипотенцию, распространение и приверженность к линии гемопоэтических стволовых клеток. ГСК и их дифференцированное потомство может быть идентифицировано по экспрессии специфических маркеров на поверхности клеток, например, кластера дифференциации (cluster of differentiation, CD) рецепторов белков и цитокинов в гемопоэтических стволовых клетках.
Генная терапия с применением ГСК обладает огромным потенциалом для лечения заболеваний системы кроветворения, включая иммунные заболевания. При таком подходе у пациента берут ГСК, генетически модифицируют ex-vivo, используя интегрирующие ретровирусные векторы, и затем вводят пациенту путем инфузии. До настоящего времени ретровирусные векторы были единственной системой доставки генов для ГСК генной терапии. Генная доставка в ГСК с использованием интегрирующих векторов обеспечивает эффективность для ГСК-производных зрелых гемопоэтических клеток. Однако генно-модифицированные клетки, которые вводят пациенту инфузией, представляют поликлональную популяцию, в которой разные клетки содержат векторные провирусы, интегрированные в разные места хромосомы, что может привести к большому количеству побочных мутаций, которые могут быть амплифицированы из-за некоторого преимущества по пролиферации/выживанию этих мутаций (Powers и Trobridge, J Stem Cell Res Ther 2013, S3:004. doi: 10.4172/2157-7633.S3-00).
ГСК обычно выделяют из состава периферической крови после мобилизации (пациенты получают рекомбинантный гранулоцитарный колониестимулирующий фактор человека (human granulocyte-colony stimulating factor, G-CSF). У пациента берут периферическую кровь и обогащают ГСК, используя маркер CD34+. Затем ГСК культивируют ex vivo и подвергают воздействию вирусных векторов. Время культивирования ех vivo варьирует от 1 до 4 суток. Перед инфузией генно-модифицированных ГСК пациентам могут вводить химиотерапевтические агенты или облучать для того, чтобы повысить эффективность трансплантологии. Генно-модифицированные ГСК повторно вводят пациенту внутривенной инфузией. Клетки мигрируют в костный мозг, прежде чем окончательно осесть в синусоидах и периваскулярной ткани. И наведение, и кроветворение являются неотъемлемыми составляющими приживления. Клетки, которые достигают ниши стволовых клеток благодаря самонаведению, начинают вырабатывать зрелые миелоидные и лимфоидные клетки из каждой линии крови. Гемопоэз продолжается благодаря действию долгоживущих ГСК, которые способны само обновляться в течение всей жизни зрелых клеток крови пациента, в частности, вырабатывать обычные лимфоидные клетки-предшественники, такие как Т-клетки и клетки NK, которые являются ключевыми клетками иммунной системы для элиминации инфицированных и злокачественных клеток.
Настоящее изобретение предусматривает осуществление направленной генной инсерции в ГСК для интродукции экзогенных кодирующих последовательностей под контролем эндогенных промоторов, особенно эндогенных промоторов генов, которые специфически активируются в клетках определенной гемопоэтической линии или на определенной стадии дифференциации, предпочтительно на поздней стадии дифференциации. ГСК могут быть трансдуцированы полинуклеотидным вектором (матрица донора), например, вектором AAV, во время обработки ex-vivo как описано в предыдущем разделе, тогда как специфический в отношении последовательности нуклеазный реагент экспрессируется так, чтобы способствовать инсерции кодирующих последовательностей в выбранный локус. Получаемые сконструированные ГСК затем могут быть трансплантированы пациенту, нуждающемуся в этом, для длительной выработки in-vivo сконструированных иммунных клеток, которые могут содержать указанные экзогенные кодирующие последовательности. В зависимости от активности выбранного эндогенного промотора, кодирующие последовательности могут избирательно экспрессироваться в определенных линиях или в ответ на локальное окружение иммунных клеток in-vivo, тем самым обеспечивая адаптивную иммунотерапию.
В одном из предпочтительных вариантов осуществления настоящего изобретения экзогенные кодирующие последовательности помещают под контроль промоторов генов, транскрипционная активность которых специфически индуцируется в обычных лимфоидных клетках-предшественниках, таких как CD34, CD43, Flt-3/Flk-2, IL-7 R альфа/CD127 и неприлизин/CD 10.
Более предпочтительно экзогенные кодирующие последовательности помещают под контроль промоторов генов, транскрипционная активность которых специфически индуцируется в клетках NK, например CD 161, CD229/SLAMF3, CD96, DNAM-1/CD226, Fc гамма RII/CD32, Fc гамма RII/RIII (CD32/CD16), Fc гамма RIII (CD16), IL-2 R бета, интегрин альфа 2/CD49b, KIR/CD158, NCAM-1/CD56, NKG2A/CD159a, NKG2C/CD159c, NKG2D/CD314, NKp30/NCR3, NKp44/NCR2, NKp46/NCR1, NKpSO/KLRF1, Siglec-7/CD328 и TIGIT, или индуцированные в Т-клетках, например CCR7, CD2, CD3, CD4, CD8, CD28, CD45, CD96, CD229/SLAMF3, DNAM-1/CD226, CD25/IL-2 R альфа, L-селектин/CD62L, и TIGIT.
Настоящее изобретение включает в качестве предпочтительного варианта его осуществления интродукцию экзогенной последовательности, кодирующей CAR или его компонент, в ГСК, предпочтительно под контролем транскрипции промотора гена, который не экспрессируется в ГСК, более предпочтительно гена, который экспрессируется только в кроветворных клетках, продуцируемых указанным ГСК, и еще более предпочтительно гена, который экспрессируется только в Т-клетках или клетках NK.
Вызванная CAR экспрессия ГСК для преодоления барьера тимуса
В одном из вариантов осуществления настоящего изобретения предусматривают продуцирование in vivo вышеуказанными сконструированными ГСК гемопоэтическими иммунными клетками, такими как Т-клетки или клетки NK, экспрессирующими экзогенные кодирующие последовательности, в частности CAR или его компонент.
Например, одним из основных препятствий для выработки гемопоэтических CAR-положительных клеток с помощью сконструированных ГСК является отторжение CAR-положительных клеток самой иммунной системой, особенно тимусом.
Барьер кровь-тимус регулирует обмен веществ между системой кровообращения и тимусом, создавая изолированную среду для развития незрелых Т-клеток. Этот барьер также предотвращает контакт незрелых Т-клеток с чужеродными антигенами (поскольку контакт с антигенами на этой стадии приведет к гибели Т-клеток в результате апоптоза).
Одним из решений, обеспечиваемых настоящим изобретением, является помещение последовательностей, кодирующих компоненты CAR в ГСК, под контроль транскрипции промоторов, которые незначительно транскрибируются в гемопоэтических клетках, когда они проходят через барьер тимуса. Одним из примеров гена, который обеспечивает условную экспрессию CAR в гемопоэтических клетках со сниженной или несущественной транскрипционной активностью в тимусе, является LCK (Uniprot: P06239).
В соответствии с предпочтительным вариантом осуществления настоящего изобретения экзогенную последовательность, кодирующую CAR или его компонент, вводят в ГСК под контролем транскрипции гена, который описывают как специфически экспрессируемый в Т-клетках или клетках NK, предпочтительно только в этих типах клеток.
Таким образом, настоящее изобретение предусматривает метод получения ГСК, включающих экзогенные кодирующие последовательности, которые должны экспрессироваться исключительно в выбранных гемопоэтических линиях (линии), причем указанные кодирующие последовательности предпочтительно кодируют по меньшей мере один компонент CAR или антигена для стимуляции иммунной системы.
В более широком смысле настоящее изобретение относится к способу конструирования ГСК путем нацеленной на определенный ген инсерции экзогенных кодирующих последовательностей, которые должны избирательно экспрессироваться в гемопоэтических клетках, продуцируемых указанными ГСК. В предпочтительном варианте осуществления настоящего изобретения указанные гемопоэтические клетки, продуцируемые указанными сконструированными ГСК, экспрессируют указанные экзогенные кодирующие последовательности в ответ на выбранные факторы окружающей среды или стимулы in vivo для улучшения их терапевтического потенциала.
Комбинирование инсерции (инсерции) целевой последовательности в иммунные клетки с инактивацией эндогенных геномных последовательностей
Одна из задач настоящего изобретения заключается в инактивации генов в первичных иммунных клетках в локусе путем интеграции экзогенной кодирующей последовательности в указанный локус, экспрессия которой улучшает терапевтический потенциал указанных сконструированных клеток. Примеры соответствующих экзогенных кодирующих последовательностей, которые могут быть инсертированы в соответствии с настоящим изобретением, представлены выше в связи с их положительным воздействием на терапевтический потенциал клеток. Ниже представлены эндогенные гены, которые предпочтительно являются мишенями для направленной на гены инсерции, и преимущества, связанные с их инактивацией.
В соответствии с предпочтительным вариантом осуществления настоящего изобретения инсерция кодирующей последовательности уменьшает или предотвращает экспрессию генов, участвующих в само- и не само-распознавании, чтобы уменьшить реакцию хозяина против трансплантата (host versus graft disease, GVHD) или отторжение иммунитета при введении аллогенных клеток реципиенту. Например, один из специфических в отношении последовательности реагентов, используемых в этом методе, может уменьшать или предотвращать экспрессию TCR в первичных Т-клетках, например генов, кодирующих TCR-альфа или TCR-бета.
В другом предпочтительном варианте осуществления настоящего изобретения один шаг редактирования гена заключается в уменьшении или предотвращении экспрессии белка β2m и/или другого белка, участвующего в его регуляции, например, С2ТА (Uniprot P33076), или в распознавании ГКГ, например белки HLA. Это позволяет сконструированным иммунным клеткам быть менее аллореактивными при инфузии пациентам.
Под понятием «аллогенное терапевтическое применение» подразумевают, что клетки происходят от донора с целью инфузии пациентам, имеющим другой гаплотип. Действительно, настоящее изобретение предусматривает эффективный способ получения первичных клеток, которые могут быть отредактированы в различных локусах генов, вовлеченных во взаимодействие и распознавание хозяина-трансплантата.
Другие локусы также могут быть отредактированы с целью улучшения активности, сохранения терапевтической активности сконструированных первичных клеток, о чем подробно описано в настоящем изобретении ниже.
Инактивация рецепторов контрольных точек и метаболические пути ингибирования иммунных клеток
В предпочтительном варианте осуществления настоящего изобретения инсертированная экзогенная кодирующая последовательность имеет эффект уменьшения или предотвращения экспрессии белка, участвующего в метаболических путях ингибирования иммунных клеток, в частности тех, которые в литературе называют «контрольной точкой иммунитета» (Pardoll D.M., Nature Reviews Cancer, 12, 2012, 252-264). В контексте настоящего изобретения «метаболические пути ингибирования иммунных клеток» означают какую-либо экспрессию генов в иммунных клетках, которая приводит к снижению цитотоксической активности лимфоцитов по отношению к злокачественным или инфицированным клеткам. Это может быть, например, ген, участвующий в экспрессии FOXP3, о котором известно, что он управляет активностью Tregs на Т-клетках (замедляя активность Т-клеток).
«Иммунные контрольные точки» это молекулы в иммунной системе, которые либо увеличивают сигнал (костимулирующие молекулы), либо подавляют сигнал активации иммунной клетки. Согласно настоящему изобретению иммунные контрольные точки, в частности, обозначают поверхностные белки, участвующие во взаимодействиях лиганд-рецептор между Т-клетками и антиген-презентирующими клетками (antigen-presenting cell, APC), которые регулируют ответ Т-клеток на антиген (который опосредуется комплексами молекул пептидом - главным комплексом гистосовместимости (ГКГ), которые распознаются рецептором Т-клеток (TCR)). Эти взаимодействия могут происходить при инициации ответов Т-клеток в лимфатических узлах (где основными APC являются дендритные клетки), или в периферических тканях, или в опухолях (где эффекторные реакции регулируются). Одним из значимых семейств мембраносвязанных лигандов, которые связывают как костимулирующие, так и ингибирующие рецепторы, является семейство В7. Все члены семейства В7 и их известные лиганды принадлежат к надсемейству иммуноглобулинов. Многие из рецепторов для недавно идентифицированных членов семейства В7 еще не идентифицированы. Члены семейства фактора некроза опухолей (Tumour necrosis factor, TNF), которые связываются с родственными молекулами семейства рецепторов TNF, представляют собой второе семейство регуляторных пар лиганд-рецептор. Эти рецепторы преимущественно доставляют костимулирующие сигналы, когда задействованы их родственными лигандами. Другая крупная категория сигналов, которые регулируют активацию Т-клеток, исходит от растворимых цитокинов в микроокружении. В других случаях активированные Т-клетки усиливают регуляцию лигандов, например CD40L, которые связывают родственные рецепторы на APC. A2aR, аденозиновый рецептор А2а; B7RP1, В7-родственный белок 1; BTLA, В и Т-лимфоцитарный аттенюатор; GAL9, галектин 9; HVEM, медиатор проникновения герпесвируса; ICOS, индуцибельный ко-стимулятор Т-клеток; IL, интерлейкин; KIR, иммуноглобулиноподобный рецептор клетки-киллера; LAG3, ген активации лимфоцитов 3; PD1, запрограммированный белок смерти клеток 1; PDL, лиганд PD1; TGFβ, трансформирующий фактор роста-β; TIM3, белок Т-клеточной мембраны 3.
Примеры дополнительных эндогенных генов, экспрессия которых может снижать или увеличивать активацию в сконструированных иммунных клетках по настоящему изобретению, перечислены в табл. 3.
Например, инсертированная экзогенная кодирующая последовательность (последовательности) могут снижать или предупреждать экспрессию сконструированных иммунными клетками по меньшей мере одного белка, выбранного из PD1 (Uniprot Q15116), CTLA4 (Uniprot P16410), PPP2CA (Uniprot P67775), PPP2CB (Uniprot P62714), PTPN6 (Uniprot P29350), PTPN22 (Uniprot Q9Y2R2), LAG3 (Uniprot P18627), HAVCR2 (Uniprot Q8TDQ0), BTLA (Uniprot Q7Z6A9), CD160 (Uniprot O95971), TIGIT (Uniprot Q495A1), CD96 (Uniprot P40200), CRTAM (Uniprot O95727), LAIR1 (Uniprot Q6GTX8), SIGLEC7 (Uniprot Q9Y286), SIGLEC9 (Uniprot Q9Y336), CD244 (Uniprot Q9BZW8), TNFRSF10B (Uniprot O14763), TNFRSF10A (Uniprot O00220), CASP8 (Uniprot Q14790), CASP10 (Uniprot Q92851), CASP3 (Uniprot P42574), CASP6 (Uniprot P55212), CASP7 (Uniprot P55210), FADD (Uniprot Q13158), FAS (Uniprot P25445), TGFBRII (Uniprot P37173), TGFRBRI (Uniprot Q15582), SMAD2 (Uniprot Q15796), SMAD3 (Uniprot P84022), SMAD4 (Uniprot Q13485), SMAD10 (Uniprot B7ZSB5), SKI (Uniprot P12755), SKIL (Uniprot P12757), TGIF1 (Uniprot Q15583), IL10RA (Uniprot Q13651), IL10RB (Uniprot Q08334), HMOX2 (Uniprot P30519), IL6R (Uniprot P08887), IL6ST (Uniprot P40189), EIF2AK4 (Uniprot Q9P2K8), CSK (Uniprot P41240), PAG1 (Uniprot Q9NWQ8), SIT1 (Uniprot Q9Y3P8), FOXP3 (Uniprot Q9BZS1), PRDM1 (Uniprot Q60636), BATF (Uniprot Q16520), GUCY1A2 (Uniprot P33402), GUCY1A3 (Uniprot Q02108), GUCY1B2 (Uniprot Q8BXH3) и GUCY1B3 (Uniprot Q02153). Генное редактирование, произведенное в генах, кодирующих указанные выше белки, предпочтительно сочетают с активацией TCR в CAR Т-клетках.
Предпочтение отдается инактивации PD1 и/или CTLA4 в сочетании с экспрессией неэндогенного иммунодепрессивного полипептида, такого как лиганд PD-L1 и/или Ig CTLA-4 (см. также пептиды из таблиц 1 и 2).

Ингибирование супрессивных цитокинов/метаболитов
В другом варианте осуществления настоящего изобретения инсертированная экзогенная кодирующая последовательность обладает эффектом снижения или предотвращения экспрессии генов, кодирующих или положительно регулирующих цитокины супрессии, или их метаболиты, или их рецепторы, в частности TGFbeta (Uniprot:P01137), TGFbR (Uniprot:P37173), IL10 (Uniprot:P22301), IL10R (Uniprot: Q13651 и/или Q08334), A2aR (Uniprot: P29274), GCN2 (Uniprot: P15442) и PRDM1 (Uniprot: O75626).
Предпочтение имеют сконструированные иммунные клетки, в которых последовательность, кодирующая IL-2, IL-12 или IL-15, заменяет последовательность по меньшей мере одного из указанных выше эндогенных генов.
Выработка устойчивости к химиотерапевтическим лекарственным средствам
В другом варианте осуществления настоящего изобретения инсертированная экзогенная кодирующая последовательность обладает эффектом снижения или предотвращения экспрессии гена, ответственного за чувствительность иммунных клеток к соединениям, применимым в качестве традиционных средств лечения рака или инфекций, например, препаратов -аналогов пуриновых нуклеотидов (purine nucleotide analog, PNA) или 6-меркаптопурина (6-Mercaptopurine, 6MP) и 6-тиогуанина (6 thio-guanine, 6TG), обычно используемых в химиотерапии. Уменьшение или инактивация генов, участвующих в механизме действия таких соединений (называемых «генами, повышающими чувствительность к лекарственным средствам»), улучшает устойчивость к ним иммунных клеток.
Примерами генов, сенсибилизирующих лекарственное средство, являются гены, кодирующие DCK (Uniprot P27707) в отношении активности PNA, например, клорофарабин и флударабин, HPRT (Uniprot P00492) в отношении активности пуриновых антиметаболитов, например, 6MP и 6TG, и GGH (Uniprot Q92820) в отношении активности антифолатных лекарственных средств, в частности метотрексата.
Это позволяет использовать клетки после традиционной противораковой химиотерапии или в сочетании с ней.
Устойчивость к лечению путем иммуносупрессии
В предпочтительном варианте осуществления настоящего изобретения инсертированная экзогенная кодирующая последовательность обладает эффектом уменьшения или предотвращения экспрессии рецепторов или белков, которые являются мишенями для лекарств, делая указанные клетки устойчивыми к лечению лекарственным препаратами, приводящими к истощению иммунитета.
Такой мишенью могут быть глюкокортикоидные рецепторы или антигены, чтобы сделать сконструированные иммунные клетки устойчивыми к глюкокортикоидам или к лечению, приводящему к истощению иммунитета, в котором применяют антитела, такие как алемтузумаб, который используют для истощения CD52-положительных иммунных клеток во многих методах лечения рака.
Метод по настоящему изобретению также может включать направленную инсерцию гена в эндогенный ген (гены), кодирующие или регулирующие экспрессию CD52 (Uniprot P31358) и/или GR (рецептор глюкокортикоидов, также обозначаемый NR3C1 - Uniprot P04150).
Улучшение активности и выживания CAR-положительных иммунных клеток
В другом варианте осуществления настоящего изобретения инсертированная экзогенная кодирующая последовательность может вызывать снижение или предупреждение экспрессии поверхностного антигена, такого как ВСМА, CS1 и CD38, причем такой антиген является мишенью для CAR, экспрессируемого указанными иммунными клетками.
В этом варианте осуществления настоящего изобретения может быть решена проблема антигенов, нацеленных на CAR, которые присутствуют на поверхности инфицированных или злокачественных клеток, но также в некоторой степени экспрессируются самими иммунными клетками.
В предпочтительном варианте осуществления настоящего изобретения экзогенную последовательность, кодирующую CAR или один из его компонентов, интегрируют в ген, кодирующий антиген, на который нацелен указанный CAR, чтобы избежать саморазрушения иммунных клеток.
Сконструированные иммунные клетки и популяции иммунных клеток
Настоящее изобретение также относится к разнообразию сконструированных иммунных клеток, которые можно получить согласно одному из описанных ранее методов, в отдельной форме или в качестве части популяции клеток.
В еще одном из предпочтительных вариантов осуществления настоящего изобретения сконструированные клетки являются первичными иммунными клетками, такими как клетки NK или Т-клетки, которые обычно являются частью популяций клеток, которые могут включать клетки различных типов. В общем, популяция, полученная от пациентов или доноров, выделена лейкаферезом из мононуклеарных клеток периферической крови (МКПК).
В предпочтительном варианте осуществления настоящего изобретения более 50% иммунных клеток, входящих в указанную популяцию, являются TCR-отрицательными Т-клетками. Согласно более предпочтительному варианту осуществления настоящего изобретения более 50% иммунных клеток, входящих в указанную популяцию, являются CAR-положительными Т-клетками.
Настоящее изобретение охватывает иммунные клетки, содержащие какие-либо комбинации различных экзогенных кодирующих последовательностей и инактивации генов, которые были соответственно и независимо описаны выше. Среди этих комбинаций особенно предпочтительны те, которые сочетают экспрессию CAR под контролем транскрипции эндогенного промотора, который стабильно активен во время активации иммунных клеток и предпочтительно независимо от указанной активации, и экспрессию экзогенной последовательности, кодирующей цитокин, например, IL -2, IL-12 или IL-15, под контролем транскрипции промотора, регуляция которого повышается во время активации иммунных клеток.
Другой предпочтительной комбинацией является инсерция экзогенной последовательности, кодирующей CAR или один из его компонентов, под контролем транскрипции промотора гена фактора 1, индуцируемого гипоксией (Uniprot: Q16665).
Настоящее изобретение также относится к фармацевтической композиции, содержащей сконструированную первичную иммунную клетку или популяцию иммунных клеток, как описано ранее для лечения инфекции или рака, и к методу лечения пациента, нуждающегося в этом, причем указанный метод включает:
- получение популяции сконструированных первичных иммунных клеток в соответствии с методом по настоящему изобретению, как описано ранее,
- необязательно очистку или сортировку указанных сконструированных первичных иммунных клеток,
- активацию указанной популяции сконструированных первичных иммунных клеток до или после инфузии указанных клеток указанному пациенту.
Активация и размножение Т-клеток
До или после генетической модификации, иммунные клетки по настоящему изобретению могут быть активированы или размножены, даже если они могут активировать или размножаться независимо от механизмов связывания антигена. Т-клетки, в частности, можно активировать и размножать, используя способы, описанные, например, в патентах US 6352694, 6534055, 6905680, 6692964, 5858358, 6887466,6905681, 7144575, 7067318, 7172869, 7232566, 7175843, 5883223, 6905874, 6797514, 6867041 и в патентной заявке US 20060121005. T-клетки можно размножать in vitro или in vivo. Т-клетки обычно размножаются при контакте с агентом, который стимулирует комплекс CD3 TCR и ко-стимулирующую молекулу на поверхности Т-клеток, чтобы создать сигнал активации для Т-клеток. Например, химические вещества, такие как ионофор кальция А23187, форбол-12-миристат-13-ацетат (phorbol-12-myristate 13-acetate, РМА) или митогенные лектины, такие как фитогемагглютинин (phytohemagglutinin, PHA), могут быть использованы для выработки сигнала активации Т-клеток.
В качестве примеров, не ограничивающих рамок охвата настоящего изобретения, популяции Т-клеток можно стимулировать in vitro, например, путем контакта с анти-CD3 антителом или его антигенсвязывающим фрагментом, или с анти-CD2 антителом, иммобилизованным на поверхности, или путем контакта с активатором протеинкиназы С (например, с бриостатином) в сочетании с ионофором кальция. Для ко-стимуляции вспомогательной молекулы на поверхности Т-клеток применяют лиганд, который связывает вспомогательную молекулу. Например, популяция Т-клеток может быть в контакте с анти-CD3 антителом и анти-CD28 антителом в условиях, подходящих для стимуляции пролиферации Т-клеток. Условия, подходящие для культуры Т-клеток, включают соответствующие среды (например, минимальные питательные среды, или среду RPMI 1640, или X-vivo 5 (фирма Lonza)), которые могут содержать факторы, необходимые для пролиферации и выживания, включая сыворотку (например, фетальную сыворотку теленка или сыворотку крови человека), интерлейкин-2 (IL-2), инсулин, IFN-g, 1L-4, 1L-7, GM-CSF, -10, - 2, 1L-15, TGFp и TNF, или какие-либо еще добавки, известные специалистам в данной области. Другие добавки для роста клеток включают, но не ограничиваются ими, сурфактант, плазманат и восстановители, такие как N-ацетилцистеин и 2-меркаптоэтанол. Среды могут включать RPMI 1640, A1M-V, DMEM, MEM, а-МЕМ, F-12, X-Vivo 1 и X-Vivo 20, Optimizer с добавлением аминокислот, пирувата натрия и витаминов, либо без сыворотки, либо с добавлением требуемого количеством сыворотки (или плазмы), или определенного набора гормонов, и/или цитокинов (цитокина), достаточными для роста и размножения Т-клеток. Антибиотики, например, пенициллин и стрептомицин, добавляют только в экспериментальные культуры, а не в те культуры клеток, которые предназначены для введения субъекту. Клетки-мишени поддерживают в условиях, необходимых для роста, например, при соответствующей температуре (например, 37°) и атмосфере (например, избыток 5% СО2). Т-клетки, которые неоднократно подвергают различной стимуляции, могут проявлять различные свойства.
В другом варианте осуществления настоящего изобретения указанные клетки могут быть размножены путем совместного культивирования с тканью или клетками. Указанные клетки также могут размножаться in vivo, например, в крови субъекта после введения указанных клеток субъекту.
Терапевтические композиции и применение
Метод по настоящему изобретению, описанный выше, позволяет получать сконструированные первичные иммунные клетки в течение ограниченного периода времени, составляющего приблизительно от 15 до 30 дней, предпочтительно от 15 до 20 дней и наиболее предпочтительно от 18 до 20 дней, при этом клетки сохраняют в полной мере иммунотерапевтический потенциал, особенно в отношении их цитотоксической активности.
Эти клетки образуют популяцию клеток, которые предпочтительно происходят от одного донора или пациента. Эти популяции клеток могут быть увеличены в соответствии с закрытыми культурами-реципиентами, чтобы соответствовать самым высоким требованиям производителей, и могут быть заморожены перед инфузией пациенту, тем самым обеспечивая «готовые» или «готовые к применению» терапевтические композиции.
По настоящему изобретению может быть получено существенное количество клеток в результате одного и того же лейкафереза, что является критическим для получения достаточных доз для лечения пациента. Хотя могут наблюдаться различия между популяциями клеток, происходящих от разных доноров, количество иммунных клеток, вырабатываемых лейкаферезом, обычно составляет от 108 до 1010 клеток МКПК. К МКПК относят несколько типов клеток: гранулоцитов, моноцитов и лимфоцитов, среди которых Т-клетки составляют от 30 до 60%, которые обычно составляют от 108 до 109 первичных Т-клеток от одного донора. Способ по настоящему изобретению обычно завершается получением популяции сконструированных клеток, которая обычно достигает более чем 108 Т-клеток, чаще более чем 109 Т-клеток, еще чаще более чем 1010 Т-клеток и, как правило, более чем 1011 Т-клеток.
Таким образом, настоящее изобретение, в частности, обращается к терапевтически эффективной популяции первичных иммунных клеток, в которой по меньшей мере 30%, предпочтительно 50%, более предпочтительно 80% клеток модифицировано в соответствии с каким-либо из описанных здесь методов. Указанная терапевтически эффективная популяция первичных иммунных клеток, согласно настоящему изобретению, включает иммунные клетки, в которые интегрирована по меньшей мере одна экзогенная генетическая последовательность под контролем транскрипции эндогенного промотора по меньшей мере одного из генов, перечисленных в табл. 6.
Соответственно, такие композиции или популяции клеток можно применять в качестве лекарственных средств, особенно для лечения рака, особенно для лечения лимфомы, но также для солидных опухолей, таких как меланомы, нейробластомы, глиомы или карциномы, такие как опухоли легких, молочной железы, толстой кишки, предстательной железы или яичника у пациента, нуждающегося в этом.
Точнее, настоящее изобретение относится к популяциям первичных TCR-отрицательных Т-клеток, происходящих от одного донора, где по меньшей мере 20%, предпочтительно 30%, более предпочтительно 50% клеток в указанной популяции модифицированы путем использования специфических для последовательности реагентов, по крайней мере двух, предпочтительно трех разных локусов.
В другом варианте осуществления настоящего изобретения предусматривают методы лечения пациента, нуждающегося в таком лечении, которые включают по меньшей мере одну из следующих стадий:
А) Определения специфических антигенных маркеров, присутствующих на поверхности образцов опухолей пациентов, полученных методом биопсии,
Б) Получения популяции сконструированных первичных иммунных клеток, полученных одним из ранее описанных методов по настоящему изобретению, экспрессирующих CAR, направленных против указанных специфических антигенных маркеров,
В) Введения указанной сконструированной популяции сконструированных первичных иммунных клеток указанному пациенту.
Как правило, указанные популяции клеток в основном содержат CD4- и CD8-положительные иммунные клетки, например, такие Т-клетки, которые могут устойчиво размножаться in vivo и могут сохраняться в течение продолжительного времени in vitro и in vivo.
Лечение с использованием сконструированных первичных иммунных клеток в соответствии с настоящим изобретением может быть предназначено для улучшения состояния пациента, для лечения или профилактики. Оно может быть либо частью аутологичной иммунотерапии, либо частью аллогенной иммунотерапии. Под аутологичным лечением подразумевают, что клетки, линия клеток или популяция клеток, используемые для лечения пациентов, происходят от указанного пациента или от донора, совместимого по лейкоцитарному антигену человека (Human Leucocyte Antigen, HLA). Под аллогенным лечением подразумевают, что клетки или популяция клеток, используемые для лечения пациентов, происходят не от указанного пациента, а от донора.
В другом варианте осуществления настоящего изобретения указанная выделенная клетка по настоящему изобретению или линия клеток, полученная из указанной выделенной клетки, могут применяться для лечения таких форм рака, как рак крови, предпочтительно Т-клеточного острого лимфобластного лейкоза.
Опухоли/раковые заболевания взрослых и опухоли/раковые заболевания детей также включены.
Лечение с помощью сконструированных иммунных клеток по настоящему изобретению может быть в сочетании с одной или несколькими методами лечения рака, выбранными из группы, включающей: терапию антителами, химиотерапию, цитокиновую терапию, терапию дендритными клетками, генную терапию, гормонотерапию, лазерно-лучевую терапию и радиационную терапию.
В соответствии с предпочтительным вариантом осуществления настоящего изобретения указанное лечение может назначаться пациентам, подвергающимся иммуносупрессивному лечению. Действительно, настоящее изобретение предпочтительно опирается на клетки или популяцию клеток, которые были сконструированы устойчивыми по меньшей мере к одному иммунодепрессанту из-за инактивации гена, кодирующего рецептор для такого иммунодепрессанта.
В связи с этим иммуносупрессивное лечение должно способствовать отбору и размножению Т-клеток по настоящему изобретению в организме пациента.
Введение клеток или популяции клеток в соответствии с настоящим изобретением может осуществляться любым удобным способом, включая вдыхание аэрозоля, инъекцию, прием внутрь, трансфузию, имплантацию или трансплантацию. Композиции, описанные в настоящем изобретении, могут быть введены пациенту подкожно, внутрикожно, внутрь опухоли, в узлы, интрамедуллярно, внутримышечно, внутривенно или внутрилимфатической инъекцией или внутрибрюшинно. В одном из вариантов осуществления настоящего изобретения композиции клеток по настоящему изобретению предпочтительно вводят внутривенной инъекцией.
Введение клеток или популяции клеток может заключаться во введении 104-109 клеток/кг массы тела, предпочтительно от 105 до 106 клеток/кг массы тела, включая все целые значения числа клеток в этих диапазонах. Таким образом, настоящее изобретение может обеспечить более 10, чаще более 50, еще чаще более 100 и особенно более 1000 доз, содержащих от 106 до 108 клеток с отредактированным геномом, происходящих из образца, полученного от одного донора или пациента.
Клетки или популяцию клеток можно вводить в одной или нескольких дозах. В другом варианте осуществления настоящего изобретения указанное эффективное количество клеток вводят в виде однократной дозы. В другом варианте осуществления настоящего изобретения указанное эффективное количество клеток вводят более чем в одной дозе в течение определенного времени. Время введения зависит от решения лечащего врача и зависит от клинического состояния пациента. Клетки или популяция клеток могут быть получены из какого-либо источника, такого как банк крови или донор. Поскольку индивидуальные потребности варьируют, определение оптимальных диапазонов эффективных количеств данного типа клеток. Эффективное количество означает количество, которое обеспечивает терапевтическое или профилактическое преимущество. Вводимая доза будет зависеть от возраста, состояния здоровья и веса реципиента, типа одновременно проводимого лечения, если таковое имеется, частоты лечения и характера желаемого эффекта.
В другом варианте осуществления настоящего изобретения указанное эффективное количество клеток или композиции, содержащей эти клетки, вводят парентерально. Указанное введение может быть внутривенным введением. Указанное введение может осуществляться непосредственно путем инъекции в опухоль.
В некоторых вариантах осуществления настоящего изобретения клетки вводят пациенту в сочетании (например, до, одновременно или после) с каким-либо другим значимым способом лечения, включая, но ими не ограничиваясь, противовирусную терапию такими агентами, как цидофовир и интерлейкин-2, цитарабин (также называемым ARA-C) или натализиимаб для пациентов с рассеянным склерозом (PC), или эфализтимаб для пациентов с псориазом или другие способы лечения пациентов с PML. В других вариантах осуществления настоящего изобретения Т-клетки по настоящему изобретению можно использовать в сочетании с химиотерапией, облучением, иммунодепрессантами, такими как циклоспорин, азатиоприн, метотрексат, микофенолат и FK506, антителами или другими иммуноабляционными агентами, такими как САМРАТН, анти-CD3-антитела или другими методами лечения антителами, цитоксином, флударибином, циклоспорином, FK506, рапамицином, микоплиеноловой кислотой, стероидами, FR901228, цитокинами и облучением. Эти препараты ингибируют кальций-зависимую фосфатазу кальциневрин (циклоспорин и FK506) или киназу p70S6, которая важна для передачи сигналов, индуцированных фактором роста (рапамицин). В еще одном варианте осуществления настоящего изобретения клеточные композиции по настоящему изобретению вводят пациенту в сочетании (например, до, одновременно или после) с трансплантацией костного мозга, абляционной терапией Т-клетками, с использованием какого-либо химиотерапевтического средства, например, флударабина, облучения от внешнего источника (external-beam radiation therapy, XRT), циклофосфамида или антител, таких как OKT3 или САМРАТН. В другом варианте осуществления настоящего изобретения клеточные композиции по настоящему изобретению вводят после абляционной В-клеточной терапии, например, агенты, которые реагируют с CD20, например ритуксан. Например, в одном варианте осуществления настоящего изобретения субъекты могут проходить стандартное лечение химиотерапией с высокой дозой с последующей трансплантацией стволовых клеток периферической крови. В определенных вариантах осуществления настоящего изобретения после трансплантации субъекты получают инфузию размноженных иммунных клеток по настоящему изобретению. В дополнительном варианте осуществления настоящего изобретения размноженные клетки вводят до или после операции.
Когда CAR экспрессируются в иммунных клетках или популяциях иммунных клеток в соответствии с настоящим изобретением, предпочтительными CAR являются те, которые нацелены по меньшей мере на один антиген, выбранный из CD22, CD38, CD123, CS1, HSP70, ROR1, GD3 и CLL1.
Сконструированные иммунные клетки по настоящему изобретению, наделенные CAR или модифицированным TCR, нацеленным на CD22, предпочтительно применяют для лечения лейкозов, например, острого лимфобластного лейкоза (acute lymphoblastic leukemia, ALL); клетки с CAR или модифицированным TCR, нацеленным на CD38, предпочтительно применяют для лечения лейкоза, например, острого лимфобластного лейкоза Т-клеток (Т-cell acute lymphoblastic leukemia, T-ALL) или множественной миеломы (MM);
клетки с CAR или модифицированным TCR, нацеленным на CD123, предпочтительно применяют для лечения лейкоза, например, острого миелоидного лейкоза (acute myeloid leukemia, AML) и бластного новообразования из плазмоцитоидных дендритных клеток (blastic plasmacytoid dendritic cells neoplasm, BPDCN), клеток с CAR или модифицированным TCR, нацеленным на CS1, предпочтительно применяют для лечения множественной миеломы (ММ).
Настоящее изобретение также предусматривает средства для обнаружения сконструированных клеток по настоящему изобретению, содержащих требуемые генетические инсерции, особенно методами ПЦР для обнаружения инсерций экзогенных кодирующих последовательностей в эндогенные локусы, упомянутые в настоящем описании, особенно в локусы PD1, CD25, CD69, TCR и β2m, с использованием зондов или праймеров, гибридизирующихся с какими-либо из последовательностей SEQ ID NO: 36-40.
Иммунологические исследования также могут быть выполнены для обнаружения экспрессии сконструированными клетками CAR, GP130, и для проверки отсутствия или снижения экспрессии TCR, PD1, IL-6 или IL-8 в клетках, где такие гены были удалены нокаутом или их экспрессия снижена.
Другие определения
- В настоящем изобретении аминокислотные остатки в полипептидной последовательности обозначены в соответствии с однобуквенным кодом, в котором, например, Q означает остаток Gln или глутамина, R означает остаток Arg или аргинина и D означает остаток Asp или аспарагиновой кислоты.
- Аминокислотное замещение означают замену одного аминокислотного остатка другим, например, замена остатка аргинина остатком глутамина в пептидной последовательности представляет собой «аминокислотное замещение».
- Нуклеотиды обозначаются следующим образом: однобуквенный код используют для обозначения основания нуклеозида: а обозначает аденин, t обозначает тимин, с обозначает цитозин и g обозначает гуанин. Для вырожденных нуклеотидов r обозначает g или а (пуриновые нуклеотиды), k обозначает g или t, s обозначает g или с, w обозначает а или t, m обозначает а или с, у обозначает t или с (пиримидиновые нуклеотиды), d обозначает g, а или t, v обозначает g, а или с, b обозначает g, t или с, h обозначает а, t или с, и n обозначает g, a, t или с.
- В контексте настоящего изобретения понятие «нуклеиновая кислота» или «полинуклеотид» относится к нуклеотидам и/или полинуклеотидам, таким как дезоксирибонуклеиновая кислота (ДНК) или рибонуклеиновая кислота (РНК), олигонуклеотидам, фрагментам, полученным в результате полимеразной цепной реакции (ПЦР), и фрагментам, генерируемым каким-либо действием из следующих: лигированием, расщеплением, действием эндонуклеазы и действием экзонуклеазы. Молекулы нуклеиновой кислоты могут состоять из мономеров, которые являются встречающимися в природе нуклеотидами (такие как в ДНК и РНК), или аналогами встречающихся в природе нуклеотидов (например, энантиомерных форм встречающихся в природе нуклеотидов), или их комбинаций. Модифицированные нуклеотиды могут иметь изменения во фрагментах Сахаров и/или в пиримидиновых или пуриновых основаниях. Модификации сахара включают, например, замену одной или нескольких гидроксильных групп галогенами, алкильными группами, аминами и азидогруппами, или сахара могут функционировать в виде простых или сложных эфиров. Более того, целый фрагмент сахара может быть заменен стерическими структурами или электронно-близкими структурами, такими как аза-сахара и аналоги карбоциклических сахаров. Примеры модификаций в основной части включают алкилированные пурины и пиримидины, ацилированные пурины или пиримидины или другие хорошо известные гетероциклические заместители. Мономеры нуклеиновых кислот могут быть связаны фосфодиэфирными связями или аналогами таких связей. Нуклеиновые кислоты могут быть одноцепочечными или двухцепочечными.
- Понятие «эндонуклеаза» относится к какому-либо ферменту дикого типа или варианту фермента, способному катализировать гидролиз (расщепление) связей между нуклеиновыми кислотами в молекуле ДНК или РНК, предпочтительно в молекуле ДНК. Эндонуклеазы не расщепляют молекулы ДНК или РНК независимо от их последовательности, но распознают и расщепляют молекулу ДНК или РНК в определенных полинуклеотидных последовательностях, которые также называют «целевыми последовательностями» или «сайтами-мишенями». Эндонуклеазы могут быть классифицированы как редкощепящие Эндонуклеазы, если они обычно имеют сайт распознавания полинуклеотидов длиной более 10 пар оснований (п.о.), более предпочтительно 14-55 п.о. Редкощепящие Эндонуклеазы значительно увеличивают гомологичную рекомбинацию, вызывая двухцепочечные разрывы ДНК (double-strand break, DSB) в определенном локусе, тем самым допуская терапию в виде генной репарации или инсерции генов (Pingoud A., Silva G.H., Nat. Biotechnol. 25(7), 2007, 743-744).
- Под «мишенью ДНК», «последовательностью-мишенью ДНК», «целевой последовательностью ДНК», «последовательностью-мишенью нуклеиновой кислоты», «последовательностью-мишенью» или «сайтом процессирования» подразумевают полинуклеотидную последовательность, которая может быть нацелена и процессирована редкощепящей эндонуклеазой по настоящему изобретению. Эти термины относятся к определенному местоположению ДНК, предпочтительно к геномному местоположению в клетке, но также к части генетического материала, который может существовать независимо от основной части генетического материала, такой как плазмиды, эписомы, вирусы, транспозоны или органеллы, такие как митохондрии, но этими примерами рамки охвата настоящего изобретения не ограничиваются. Примерами РНК-направляемых последовательностей-мишеней, которыми перечень не ограничивается, являются такие последовательности генома, которые могут гибридизировать направляющую РНК, которая направляет управляемую эндонуклеазой РНК в требуемый локус.
- Под понятием «мутация» подразумевают замещение, делению, инсерцию до одного, двух, трех, четырех, пяти, шести, семи, восьми, девяти, десяти, одиннадцати, двенадцати, тринадцати, четырнадцати, пятнадцати, двадцати, двадцати пяти, тридцати, сорока, пятидесяти или более нуклеотидов/аминокислот в полинуклеотидной (кДНК, ген) или полипептидной последовательности. Мутация может влиять на кодирующую последовательность гена или его регуляторную последовательность. Она также может влиять на структуру геномной последовательности или структуру/стабильность кодируемой мРНК.
- Под «вектором» подразумевают молекулу нуклеиновой кислоты, способную транспортировать другую нуклеиновую кислоту, с которой связана первая. К понятию «вектор» в настоящем изобретении относят, но ими перечень не ограничивается, вирусный вектор, плазмиду, вектор РНК или линейную или кольцевую молекулу ДНК или РНК, которая может состоять из хромосомной, не хромосомной, полусинтетической или синтетической нуклеиновой кислоты. Предпочтительными векторами являются те, которые способны к автономной репликации (эписомальный вектор) и/или экспрессии нуклеиновых кислот, с которыми они связаны (векторы экспрессии). Большое количество подходящих векторов известно специалистам в данной области, и они коммерчески доступны. К вирусным векторам относят ретровирус, аденовирус, парвовирус (например, аденоассоциированные вирусы (adenoassociated viruse, AAV), коронавирус, РНК-вирусы с отрицательной цепью, такие как ортомиксовирус (например, вирус гриппа)), рабдовирус (например, вирус бешенства и везикулярного стоматита), парамиксовирус (например, корь и Сендай), РНК-вирусы с положительной цепью, например, пикорнавирусы и альфа-вирусы, и двухцепочечные ДНК-вирусы, включая аденовирус, герпесвирус (например, вирус Herpes Simplex типа 1 и 2, Эпштейна-Барра, цитомегаловирус) и поксвирус (например, вирусы осповакцины, вирус оспы кур и вирус оспы канареек). К другим вирусам относятся, например, вирус Норфолк, тогавирус, флавивирус, реовирусы, паповавирус, гепаднавирус и вирус гепатита. Примеры ретровирусов: птичий вирус лейкоза саркомы, вирусы млекопитающих С-типа, вирусы В-типа, вирусы D-типа, вирусы группы HTLV-BLV, лентивирус, спумавирус (Coffin J.M. в кн.: «Fundamental Virology», 3-е изд., под ред. Fields B.N. с соавт., 1996, изд-во Lippincott-Raven Publishers, Филадельфия).
- В контексте настоящего изобретения понятие «локус» означает определенное физическое положение последовательности ДНК (например, гена) в геноме. Понятие «локус» может относиться к определенному физическому местоположению целевой последовательности редкощепящей эндонуклеазы в хромосоме или в последовательности генома инфекционного агента. Такой локус может содержать последовательность-мишень, которая распознается и/или расщепляется специфичной для последовательности эндонуклеазой по настоящему изобретению. Очевидно, что представляющий интерес локус настоящего изобретения может не только определять последовательность нуклеиновой кислоты, которая существует в основной части генетического материала (т.е. в хромосоме) клетки, но также и в той часть генетического материала, которая может существовать независимо от указанного основного объема генетического материала, например, в плазмиде, эписоме, вирусе, транспозоне или органелле, например, в митохондрии, но этими примерами перечень не ограничивается.
- Понятие «расщепление» относится к разрыву ковалентного каркаса полинуклеотида. Расщепление может быть инициировано различными методами, включая, но не ими ограничиваясь, ферментативный или химический гидролиз фосфодиэфирной связи. Возможны как одноцепочечное расщепление, так и двухцепочечное расщепление, и двухцепочечное расщепление может происходить в результате двух отдельных одноцепочечных событий расщепления. Гибридное расщепление двухцепочечной ДНК, РНК или ДНК/РНК может привести к образованию либо тупых концов, либо ступенчатых концов.
- Понятие «идентичность» относится к идентичности двух молекул нуклеиновой кислоты или полипептидов. Идентичность может быть определена путем сравнения положения в каждой последовательности, которое может быть выровнено для сравнения. Если в сравниваемой последовательности в определенном положении находится одно и то же основание, то молекулы в этой положении идентичны. Степень сходства или идентичности между последовательностями нуклеиновых кислот или аминокислот является функцией количества идентичных или совпадающих нуклеотидов в положениях, общих для последовательностей нуклеиновых кислот. Различные алгоритмы выравнивания и/или программы могут использоваться для вычисления идентичности между двумя последовательностями, включая FASTA или BLAST, которые доступны как часть пакета анализа последовательности GCG (Университет Висконсина, Мэдисон, Висконсин), и могут использоваться, например, с настройкой по умолчанию. Например, рассматривают полипептиды, которые по меньшей мере на 70%, 85%, 90%, 95%, 98% или 99% идентичны определенным полипептидам, описанным в настоящем изобретении, и предпочтительно проявляющим, по существу, те же функции, что и полинуклеотид, кодирующий такие полипептиды.
- В настоящем изобретении понятие «субъект» или «пациент» относится ко всем представителям царства животных, включая приматов, в том числе людей.
- Приведенное выше описание настоящего изобретения обеспечивает метод и процесс получения и применения его объектов таким образом, что какой-либо специалист в данной области может воспроизводить описанные разработку и применение, причем эта возможность предоставляется для сущности, представленной в прилагаемой формуле настоящего изобретения, составляющей часть оригинального описания.
Если в настоящем изобретении указан числовой предел или диапазон, это означает, что указаны конечные точки. Кроме того, специально включены все значения и поддиапазоны в числовом пределе или диапазоне, как если бы они были явно записаны.
После общего описания настоящего изобретения можно получить дополнительное пояснение в виде ссылок на определенные конкретные примеры его осуществления, которые представлены только с целью иллюстрации и не предназначены для ограничения области охвата настоящего изобретения.
ПРИМЕРЫ
Пример 1. AAV-управляемая гомологичная рекомбинация в первичных Т-клетках человека в различных локусах под контролем эндогенных промоторов с нокаутом эндогенного гена.
Введение
Специфичные в отношении последовательности эндонуклеазные реагенты, такие как TALEN® (фирма Cellectis, 8 rue de la Croix Jarry, 75013 PARIS), позволяют производить сайт-специфическую индукцию двухцепочечных разрывов (double-stranded break, DSB) в геноме в необходимых локусах. Восстановление DSB клеточными ферментами происходит главным образом двумя путями: негомологичным присоединением концов (non-homologous end joining, NHEJ) и репарацией, направляемой гомологией (homology directed repair, HDR). В HDR задействован гомологичный фрагмент ДНК (матричная ДНК) для восстановления DSB путем рекомбинации и, что возможно, для введения какой-либо генетической последовательности, содержащейся в матричной ДНК. Как показано в настоящем изобретении матричная ДНК может доставляться рекомбинантным аденоассоциированным вирусом (rAAV) вместе с разработанной нуклеазой, такой как TALEN®, для введения сайт-специфических DSB.
Проектирование встраиваемых матриц
1.1. Инсерция апоптоз-индуцирующего CAR в локус с повышенной регуляцией с нокаутом кодирующей последовательности эндогенного гена PD1
Местоположение сайта-мишени TALEN разработано таким образом, чтобы оно было локализовано в целевом эндогенном гене PDCD1 (запрограммированный белок смерти клеток 1, называемый PD1 - Uniprot # Q15116). Последовательность, охватывающая 1000 п.о. выше и ниже по последовательности мишеней TALEN, приведена в SEQ ID NO. 1 и SEQ ID NO. 2. Целевые последовательности TALEN (SEQ ID: SEQ ID NO. 3 и NO. 4) приведены в SEQ ID NO. 5. Интеграционная матрица состоит из последовательности (300 п.о.), гомологичной эндогенному гену выше по цепи от сайта TALEN (SEQ ID NO. 1), за которой следует регуляторный элемент 2А (SEQ ID NO. 6), за которым следует последовательность, кодирующая CAR, индуцирующий апоптоз, без стартового кодона (SEQ ID NO. 7), за которым следует STOP-кодон (TAG), за которым следует последовательность полиаденилирования (SEQ ID NO. 8), за которой следует последовательность (1000 п.о.), гомологичная эндогенному гену ниже по последовательности от сайта TALEN (SEQ ID NO. 2)). Затем инсертированную матрицу клонируют в векторе rAAV без промотора и используют для получения AAV6.
1.2 Инсерция интерлейкина в расположенный выше по цепи локус с нокаутом эндогенного гена
Локализация сайта-мишени TALEN спроектирована таким образом, чтобы этот сайт находился в целевом эндогенном гене PDCD1 (белок запрограммированной смерти клеток 1, PD1). Последовательность, охватывающая 1000 п.о. выше и ниже по цепи от мишеней TALEN, приведена в SEQ ID NO. 1 и SEQ ID NO. 2. Целевые последовательности TALEN (SEQ ID: SEQ ID NO. 3 и NO. 4) приведены в SEQ ID NO. 5. Встраиваемая матрица состоит из последовательности (300 п.о.), гомологичной эндогенному гену выше по цепи от сайта TALEN (SEQ ID NO. 1), за которой следует регуляторный элемент 2А (SEQ ID NO. 6), за которым следует последовательность, кодирующая сконструированную одноцепочечную субъединицу р35 IL-12 человека (SEQ ID NO. 9) и субъединицу р40 гибридного белка (SEQ ID NO. 10), за которой следует STOP-кодон (TAG), за которым следует последовательность полиаденилирования (SEQ ID NO. 8), за которой следует последовательность (1000 п.о.), гомологичная эндогенному гену в расположенном ниже по цепи сайте TALEN (SEQ ID NO. 2). Матрицу для инсерции затем клонируют в векторе rAAV без промотора и используют для получения AAV6.
1.3 Инсерция апоптоз-индуцирующего CAR в слабо экспрессируемом локусе без нокаута эндогенного гена - N-концевая инсерция
Локализация сайта-мишени TALEN спроектирована таким образом, чтобы сайт был расположен как можно ближе к стартовому кодону целевого эндогенного гена LCK (LCK, протоонкоген LCK, тирозинкиназа семейства Src [Homo sapiens (человек)]). Последовательность, охватывающая 1000 п.о. вверх и вниз по цепи от стартового кодона, приведена в SEQ ID NO. 11 и SEQ ID NO. 12. Матрица для интеграции разработана таким образом, что она состоит из последовательности (1000 п.о.), гомологичной эндогенному гену выше по цепи от стартового кодона, за которой следует последовательность, кодирующая апоптоз-индуцирующий CAR, содержащий стартовый кодон (SEQ ID NO. 13), затем следует 2А регуляторный элемент (SEQ ID NO. 8), затем следует последовательность (1000 п.о.), гомологичная эндогенному гену ниже по цепи от стартового кодона (SEQ ID NO. 12). Затем матрицу для инсерции клонируют в векторе rAAV без промотора и используют для получения AAV6.
1.4 Инсерция апоптоз-индуцирующего CAR в слабо экспрессируемом локусе без нокаута эндогенного гена С-концевая инсерция
Локализация сайта-мишени TALEN спроектирована таким образом, чтобы сайт был расположен как можно ближе к стоп-кодону целевого эндогенного гена LCK (LCK, протоонкоген LCK, тирозинкиназа семейства Src [Homo sapiens (человек)]).
Последовательность, охватывающая 1000 п.о. вверх и вниз по цепи от стоп-кодона, приведена в SEQ ID NO. 14 и SEQ ID NO. 15. Матрица для интеграции разработана таким образом, что она состоит из последовательности (1000 п.о.), гомологичной эндогенному гену выше по цепи от стоп-кодона, за которой следует регуляторный элемент 2А (SEQ ID NO. 8), затем следует последовательность, кодирующая апоптоз-индуцирующий CAR без стартовый кодон (SEQ ID NO. 7), затем следует STOP-кодон (TAG), за которым следует последовательность (1000 п.о.), гомологичная эндогенному гену ниже по цепи от стоп-кодона (SEQ ID NO. 15). Затем матрицу для инсерции клонируют в векторе rAAV без промотора и используют для получения AAV6.
Экспрессия специфичных в отношении последовательности нуклеазных реагентов в трансдуцированных клетках
TALEN® мРНК синтезируют с использованием набора mMessage mMachine Т7 Ultra (фирма Thermo Fisher Scientific, Гранд-Айленд, Нью-Йорк), поскольку каждый TALEN клонируют по цепи ниже промотора Т7, очищают с использованием колонок RNeasy (фирма Qiagen, Валенсия, Калифорния) и элюируют в «среду для цитопорации Т» (фирма Harvard Apparatus, Холлистон, Массачусетс). Т-клетки человека собирают и активируют из цельной периферической крови, предоставленной фирмой ALLCELLS (Аламеда, Калифорния), в среде X-Vivo-15 (фирма Lonza, Базель, Швейцария) с добавлением 20 нг/мл IL-2 человека (фирма Miltenyi Biotech, Сан-Диего, Калифорния), 5% сыворотки крови человека АВ (фирма Gemini Bio - Products, Западный Сан-Франциско, Калифорния) и гранул Dynabeads Human T- activator CD3/CD28 при соотношении гранулы:клетки = 1:1 (фирма Thermo Fisher Scientific, Гранд Айлэнд, Нью-Йорк). Гранулы удаляют через 3 дня, и 5×106 клеток подвергают электропорации с 10 мкг мРНК каждого из двух соответствующих TALEN® с использованием прибора для электропорации Cytopulse (фирма ВТХ Harvard Apparatus, Холлистон, Массачусетс), применяя два импульса 0,1 мСм при 3000 В/см, а затем четыре импульса 0,2 мСм при 325 В/см в кюветах со щелью 0,4 см в конечном объеме 200 мкл «среды Т для цитопорации» (фирма ВТХ Harvard Apparatus, Холлистон, Массачусетс). Клетки немедленно разводят в среде X-Vivo-15 с 20 нг/мл IL-2 и инкубируют при 37° в атмосфере 5% СО2. Через два часа клетки инкубируют с частицами AAV6 в количестве 3×105 вирусных геномов (vg) на клетку (37°, 16 ч). Клетки пассируют и поддерживают в среде X-Vivo-15, обогащенной 5% сыворотки АВ человека и 20 нг/мл IL-2, до анализа методом жидкостной цитометрии на экспрессию соответствующих инсертированных генных последовательностей.







Пример 2. TALEN®-опосредованная двойная целевая интеграция кодирующих матриц IL-15 и CAR в Т-клетки.
Материалы
Среду X-vivo-15 получают от фирмы Lonza (номер в каталоге: BE04-418Q), IL-2 от фирмы Miltenyi Biotech (номер в каталоге: 130-097-748), сыворотки крови человека АВ от фирмы Seralab (номер в каталоге: GEM-100-318), Т-активатор человека CD3/CD28 от фирмы Life Technology (номер в каталоге: 11132D), QBEND10-APC от фирмы R&D Systems (номер в каталоге: FAB7227A), vioblue-меченый анти-CD3, РЕ-меченый анти-LNGFR, АРС-меченый анти-CD25 и РЕ-меченый анти-PD1 от фирмы Miltenyi (номера в каталоге: 130-094-363, 130-112-790, 130-109-021 и 130-104-892, соответственно), обработанные 48-луночные планшеты (фирма CytoOne, номер в каталоге: СС7682-7548), набор для иммуноферментного анализа human IL-15 Quantikine ELISA от фирмы R&D systems (номер в каталоге: S1500), ONE-Glo от фирмы Promega (номер в каталоге: Е6110). Партии AAV6, содержащие разные матрицы, получают от фирмы Virovek, МКПК получают от фирмы Allcells (номер в каталоге: PB004F), и клетки Raji-Luciferase, полученные после трансдукции в клетки Раджи частиц лентивируса, кодирующих люциферазу светлячка, которые получают из коллекции АТСС (номер в каталоге: CCL-86).
Методы
2.1 Трансфекция-трансдукция
Двойная целевая интеграция в локусах TRAC и PD1 или CD25 выполняют следующим образом. МКПК оттаивают, промывают, ресуспендируют и культивируют в полноценной среде X-vivo-15 (X-vivo-15, 5% АВ сыворотка, 20 нг/мл IL-2). Через сутки клетки активируют гранулами Dynabeads Т-активатора человека CD3/CD28 (25 мкл гранул/1Е6 CD3-положительных клеток) и культивируют при плотности 1Е клеток/мл в течение 3 дней в полноценной среде X-vivo при 37°С при наличии 5% СО2. Затем клетки пересевают в свежую полноценную среду и не следующий день трансдуцируют/трансфицируют в соответствии со следующей процедурой. В день осуществления трансдукции-трансфекции клетки сначала освобождают от гранул путем магнитного разделения на сепараторе EasySep, дважды промывают в буфере Т для цитопорации (фирма ВТХ Harvard Apparatus, Холлистон, Массачусетс) и ресуспендируют до конечной концентрации 28Е клеток/мл в том же самом растворе. Клеточную суспензию смешивают с 5 мкг мРНК, кодирующей последовательности TRAC TALEN® (SEQ ID NO: 16 и 17), в присутствии или в отсутствие 15 мкг кодирующих последовательностей мРНК либо CD25, либо PD1 TALEN® (SEQ ID NO: 18 и 19 и SEQ ID NO: 20 и 21, соответственно), в конечном объеме 200 мкл. TALEN® представляет стандартный формат TALE-нуклеаз, полученных в результате гибридизации TALE с Fok-1. Трансфекцию проводят с использованием технологии Pulse Agile, применяя два импульса 0,1 мСм при 3000 В/см, а затем четыре импульса по 0,2 мСм при 325 В/см в кюветах со щелью 0,4 см и в конечном объеме 200 мкл буфера Т для цитопорации (фирма ВТХ Harvard Apparatus, Холлистон, Массачусетс). После электропорации клетки немедленно переносят в 12-луночный планшет, содержащий 1 мл предварительно нагретой среды X-vivo-15 без сыворотки, и инкубируют при 37°С в течение 15 мин. Затем клетки концентрируют до 8Е клеток/мл в 250 мкл той же среды в присутствии частиц AAV6 (MOI = 3Е5 vg/клетку), содержащих матрицы донора, в 48-луночных стандартно обработанных планшетах. Через 2 ч культивирования при 30°С к суспензии клеток добавляют 250 мкл среды X-vivo-15, дополненной 10% сыворотки АВ и 40 нг/мл IL-2, после чего смесь инкубируют 24 часа в тех же условиях культивирования. Через сутки клетки высевают в количестве 1Е6 клеток/мл в полноценную среду X-vivo-15 и культивируют при 37°С в присутствии 5% CO2.
2.2 Зависящая от активации экспрессия ALNGFR и секреция IL15
Сконструированные Т-клетки извлекают в процессе трансфекции-трансдукции, описанном ранее методом, и высевают в количестве 1Е6 клеток/мл отдельно или в присутствии клеток Раджи (Е:Т = 1:1) или гранул Dynabeads (12,5 мкл/1Е6 клеток) в конечном объеме 100 мкл полноценной среды X-vivo-15. Клетки культивируют в течение 48 ч, затем извлекают, метят и анализируют методом жидкостной цитометрии. На клетки наносят метку двумя независимыми наборами антител. Первые наборы антител, нацеленные на выявление ALNGFR, CAR и CD3 клеток, состояли из QBEND10-APC (в разведении 1/10), vioblue-меченного анти-CD3 (в разведении 1/25) и РЕ-меченного анти-ALNGFR (в разведении 1/25). Вторые наборы антител, нацеленные на обнаружение экспрессии эндогенных CD25 и PD1, состоят из АРС-меченного анти-CD25 (в разведении 1/25) и vioblue-меченного анти-PD1 (в разведении 1/25).
Такую же экспериментальную установку используют для изучения секреции IL-15 в среде. Смесь клеток хранят в совместной культуре в течение 2, 4, 7 и 10 дней перед отбором и анализом супернатанта с использованием набора для IL-15-специфичного метода ELISA.
2.3 Анализ серийного уничтожения клеток
Для оценки противоопухолевой активности сконструированных CAR Т-клеток проводят анализ серийного уничтожения клеток. Принцип этого анализа заключается в том, чтобы ежедневно проверять противоопухолевую активность CAR Т-клеток путем ежедневного добавления постоянного количества опухолевых клеток. Пролиферацию, уничтожение и рецидив опухолевых клеток можно контролировать с помощью люминесценции, считываемой благодаря маркеру люциферазы, стабильно интегрированному в линии опухолевых клеток.
Как правило, CAR Т-клетки смешивают с суспензией опухолевых клеток Raji-luc 2,5×105 при изменяемом соотношении Е:Т (Е:Т = 5:1 или 1:1) в общем объеме 1 мл среды X-vivo с 5% сыворотки АВ, 20 нг/мкл IL-2. Смесь инкубируют 24 часа до определения люминесценции 25 мкл суспензии клеток, используя реагент ONE-Glo. Затем смесь клеток центрифугируют, старые среду отбрасывают и заменяют 1 мл свежей полноценной среды X-vivo-15, содержащей 2,5×105 клеток Raji-Luc, после чего полученную смесь клеток инкубируют в течение 24 ч. Этот протокол повторяют 4 суток.
Эксперименты и результаты
В этом примере описывают методы улучшения терапевтического результата CAR Т-клеточной терапии путем интеграции кассеты экспрессии гетеродимера рецептора альфа (IL15/sIL15rα) IL-15/растворимого IL-15 под контролем промоторов эндогенных Т-клеток, регулирующих гены PD1 и CD25. Поскольку известно, что оба гена активируются при захвате опухоли CAR Т-клетками, они могут быть подвергнуты повторной экспрессии IL-IL15/sIL15rα только в непосредственной близости от опухоли. Этот метод направлен на снижение потенциальных побочных эффектов от системной секреции IL15/sIL15rα при сохранении способности снижать вызванную активацией гибель Т-клеток (activation induced T-cell death, AICD), способствовать выживанию Т-клеток, повышению противоопухолевой активности Т-клеток и обращению анергии Т-клеток.
Метод, разработанный для интеграции IL15/sIL15rα в локусы PD1 и CD25, заключается в создании двухцепочечного разрыва в обоих локусах с использованием TALEN в присутствии матрицы репарации ДНК, векторизованной AAV6. Эта матрица состоит из двух гомологичных плеч, встраивающих кодирующие области IL15/sIL15rα, разделенные 2А-цис-действующими элементами и регуляторными элементами (стоп-кодона и полиА последовательности). В зависимости от локуса-мишени и его участия в активности Т-клеток, целевой эндогенный ген может быть инактивирован или не инактивирован посредством специфической конструкции матрицы. Если ген CD25 рассматривают в качестве целевого локуса, инсерционную матрицу разрабатывают для включения (knock-in, KI) IL15/sIL15rα без инактивации CD25, поскольку белковый продукт этого гена считают важным для функции Т-клеток. Напротив, поскольку PD1 участвует в ингибировании Т-клеток/истощении Т-клеток, инсерционную матрицу разрабатывают для предотвращения ее экспрессии при одновременной активации экспрессии и секреции IL15/sIL15rα.
Чтобы проиллюстрировать этот подход и продемонстрировать осуществимость двойной направленной инсерции в первичные Т-клетки, разработаны три разные матрицы (рис. 2А, 2Б и 2В). Первая из них, названная CARm и представленная последовательностью SEQ ID NO: 36, разработана для инсерции анти-CD22 CAR кДНК в локус TRAC в присутствии TRAC TALEN® (SEQ ID NO: 16 и 17). Вторая, IL-15_CD25m (SEQ ID NO: 37), разработана для интеграции IL15, sIL15rα и поверхностного маркера, называемого кДНК ALNGFR, разделенных 2А цис-действующими элементами непосредственно перед стоп-кодоном эндогенной кодирующей последовательности CD25 с использованием CD25 TALEN® (SEQ ID NO: 18 и 19). Третья матрица, IL-15_PD1m (SEQ ID NO: 38), содержит ту же кассету экспрессии и предназначена для интеграции в середине открытой рамки считывания PD1 с использованием PD1 TALEN® (SEQ ID NO: 20 и 21). Три матрицы содержат дополнительный 2А цис-действующий элемент, расположенный выше по цепи экспрессионных кассет, чтобы обеспечить ко-экспрессию IL15/sIL15rα и CAR с направленным эндогенным геном.
Сначала была оценена эффективность двойной направленной инсерции в Т-клетках путем их трансдукции с помощью одного из AAV6, кодирующего матрицу IL15/sIL15rα (SEQ ID NO: 41; pCLS30519) наряду с матрицей, кодирующей CAR, и впоследствии трансфицировали соответствующий TALEN®. AAV6-ассистированная векторизация матриц в присутствии мРНК, кодирующей TRAC TALEN® (SEQ ID NO: 22 и 23) и PD1 TALEN® (SEQ ID NO: 24 и 25) или CD25 TALEN® (SEQ ID NO: 26 и 27) осуществляет экспрессию анти-CD22 CAR в сконструированных Т-клеток, количество которых доходит до 46% (фиг. 3).
Чтобы определить степень интеграции IL15m в локусы CD25 и PD1, сконструированные Т-клетки активируют либо с помощью гранул, покрытых анти-CD3/CD28, либо с помощью опухолевых клеток Раджа, экспрессирующих CD22. Через 2 суток после активации клетки извлекают и анализируют с помощью FACS с использованием экспрессии LNGFR в качестве суррогата секреции IL15/sIL15rα (фиг. 4 и 5). Наши результаты показывают, что гранулы, покрытые анти-CD3/CD28, индуцируют экспрессию ALNGFR Т-клетками, содержащими IL-15m_CD25 или IL-15m_PD1, независимо от присутствия анти-CD22 CAR (фиг 4А, Б). Опухолевые клетки, однако, индуцируют только экспрессию ALNGFR Т-клетками, обработанными как CARm, так и IL-15m. Это указывает на то, что экспрессия ALNGFR может быть специфически индуцирована посредством вовлечения опухолевых клеток с помощью CAR (фиг. 5 и 6).
Как и ожидалось, эндогенный ген CD25 продолжает экспрессироваться в активированных обработанных Т-клетках (фиг. 7 и 8), тогда как экспрессия PD1 сильно нарушена (фиг. 12).
Чтобы подтвердить, что экспрессия ALNGFR коррелирует с секрецией IL15 в среде, Т-клетки, экспрессирующие анти-CD22 CAR и ALNGFR, инкубируют в присутствии опухолевых клеток Раджи, экспрессирующих CD22 (соотношение Е:Т = 1:1), в общей сложности 10 суток. Супернатант извлекают на 2, 4, 7 и 10 день, и количественно определяют наличие IL15 с помощью анализа ELISA. Полученные в настоящем изобретении результаты показывают, что IL15 секретируется в среде только Т-клетками, которые совместно обработаны матрицами CARm и IL15m вместе с соответствующими им TALEN® (фиг. 13). Т-клетки, обработанные какой-либо из этих матриц, не могут секретировать на каком-либо существенном уровне IL15 по отношению к покоящимся Т-клеткам. Интересно, что уровень секреции IL-15 является признан временным, с максимальным пиком на 4 сутки (фиг. 14).
Чтобы оценить, может ли уровень секретируемого IL-15 (SEQ ID NO: 59) влиять на активность CAR Т-клеток, CAR Т-клетки совместно культивируют в присутствии опухолевых клеток при соотношении Е:Т = 5:1 в течение 4 суток. Их противоопухолевая активность каждый день подвергалась угрозе из-за гранулирования и ресуспендирования в культуральной среде без IL-2, содержащей свежие опухолевые клетки. Противоопухолевую активность CAR Т-клеток контролируют ежедневно, измеряя люминесценцию оставшихся опухолевых клеток Раджи, экспрессирующих люциферазу. Полученные в настоящем изобретении результаты показывают, что CAR Т-клетки, ко-экспрессирующие IL-15, обладают повышенной противоопухолевой активностью, чем те, у которых не было IL15 во всех рассматриваемых временных точках (фиг. 15).
Таким образом, в совокупности полученные результаты показывают, что разработан метод, позволяющий одновременно целенаправленно инсертировать CAR и IL15 кДНК в локусы TRAC и CD25 или PD1. Такая двойная нацеленная инсерция приводит к устойчивой экспрессии анти-CD22 CAR и к секреции IL15 в среду. Уровни секретируемого IL15 достаточны для усиления активности CAR Т-клеток.

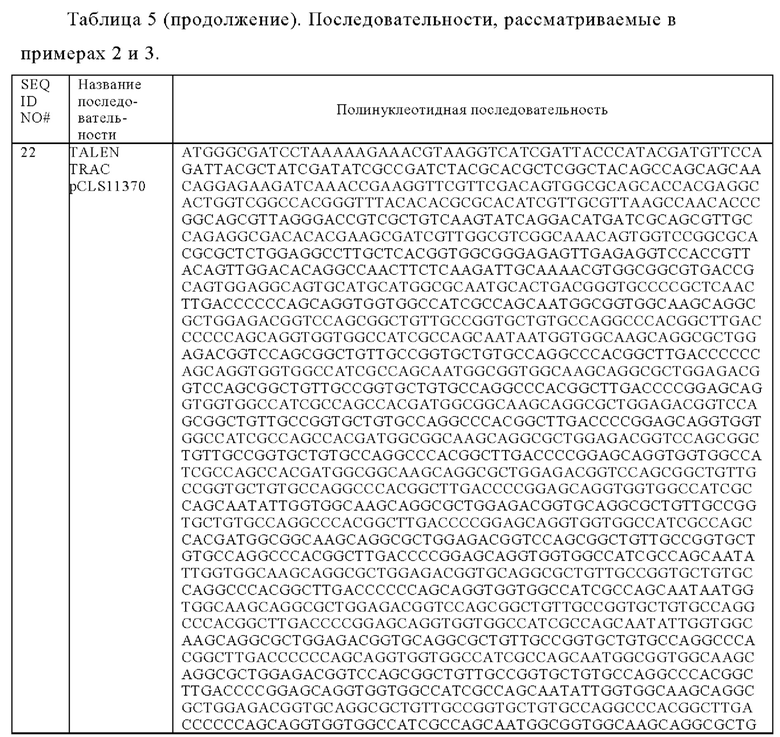














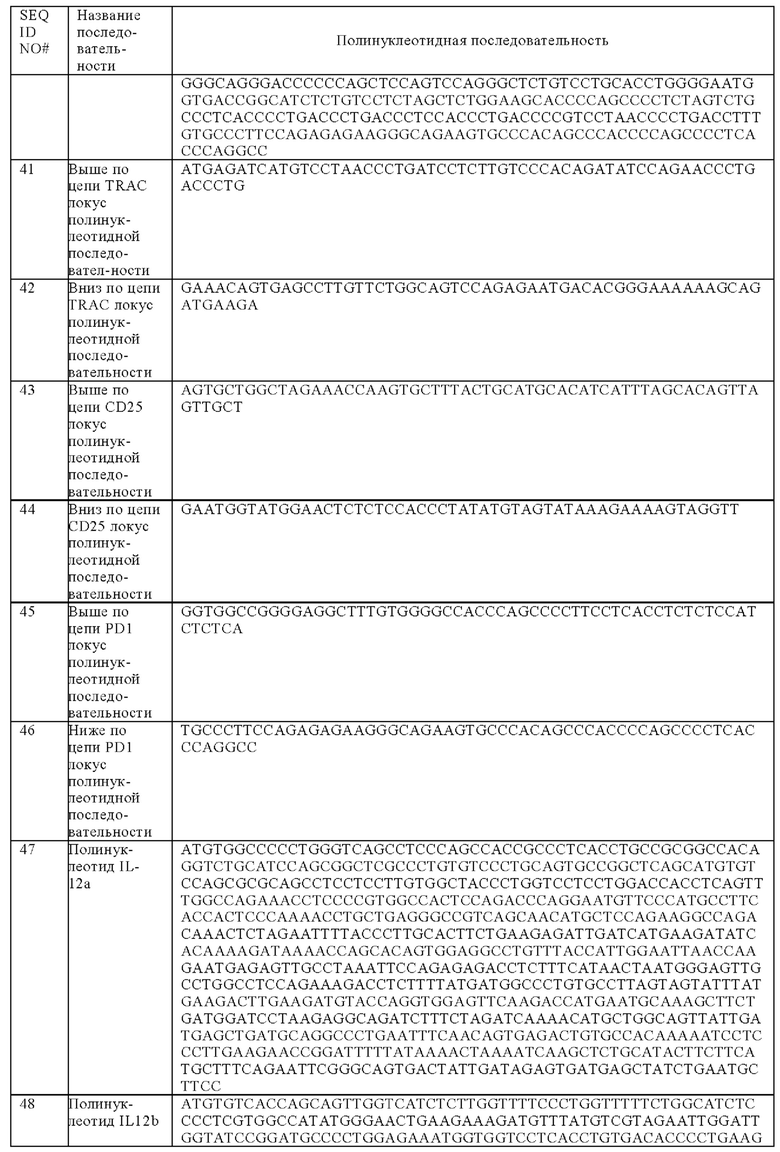
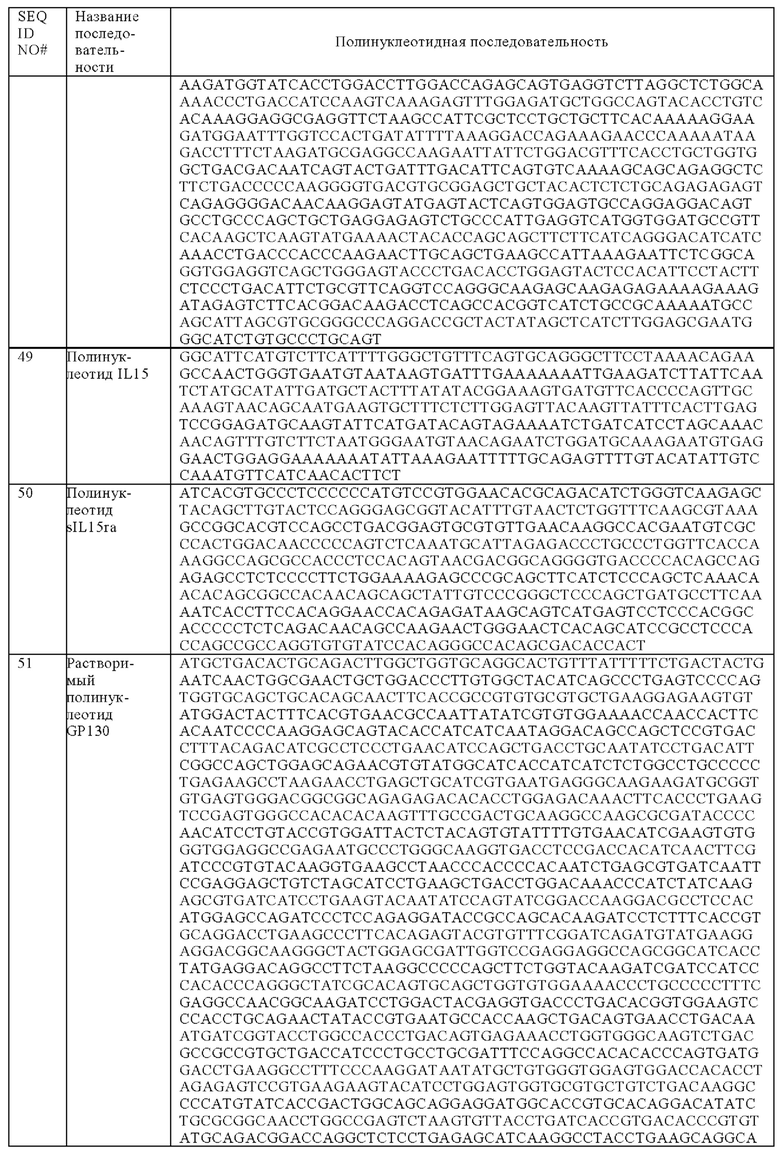
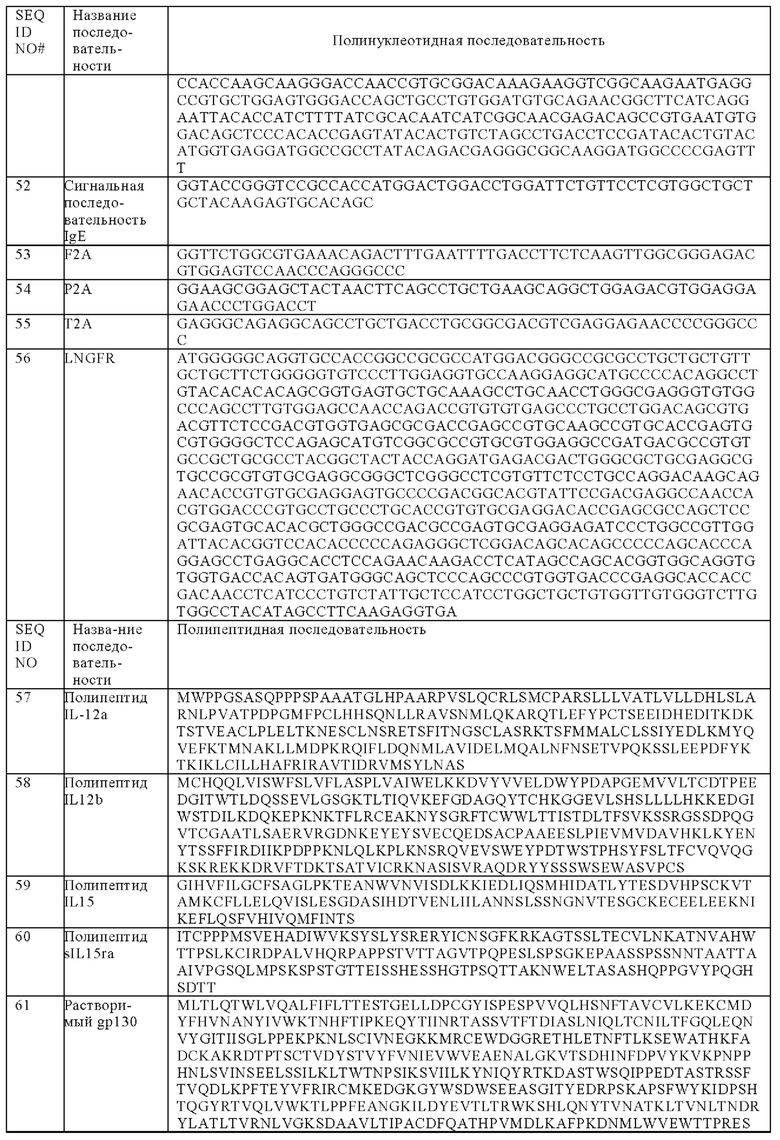





















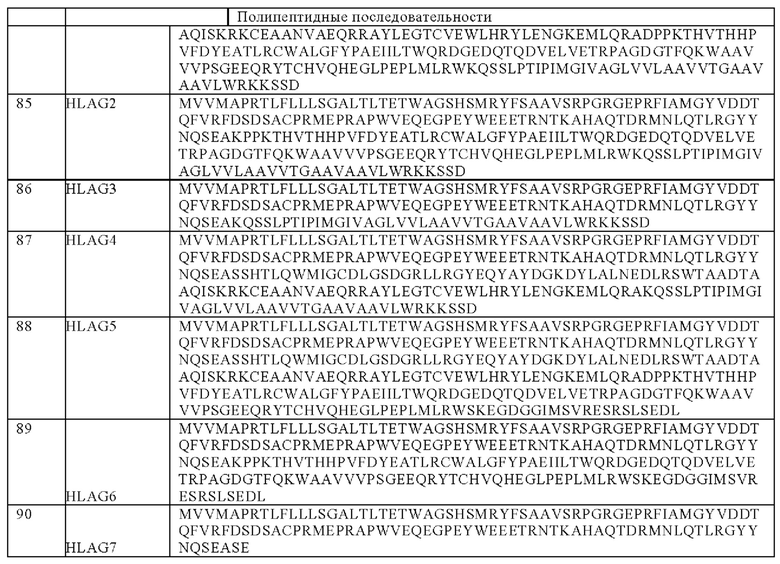
Пример 3. TALEH®-опосредованная двойная нацеленная интеграция матриц, кодирующих ингибитор NK и CAR, в локусы В2М и TRAC в первичных Т-клетках.
В этом примере описывают методы улучшения терапевтического результата CAR Т-клеточной терапии путем увеличения стойкости этих клеток in vivo. Метод состоит в одновременном TALEN®-опосредованном нокауте В2М и TCR в присутствии векторов репарации AAV6, доставляющих CAR в локус TRAC и ингибитор NK в локус В2М. Этот метод предотвращает атаку CAR Т-клеток на ткани хозяина неспецифическим и TCR-опосредованным способом (атака трансплантат-против-хозяина) и отводит опосредованное Т- и NK-клетками истощение CAR Т-клеток.
Метод, разработанный для интеграции ингибитора NK в локус B2m, заключается в создании двухцепочечного разрыва в одном из первых экзонов В2М с использованием TALEN® в присутствии матрицы репарации ДНК, направленной вектором AAV6. Эта матрица состоит из двух гомологических ветвей В2М, в которые встроена кодирующая последовательность ингибитора NK, разделенных 2A цис-действующими элементами и регуляторными элементами (последовательности стоп-кодона и полиА). Поскольку экспрессия В2М на поверхности CAR Т-клеток, вероятно, будет способствовать их истощению при переносе в аллогенную среду, инсерция репарирующей матрицы была разработана для инактивации В2М и экспрессии промотора ингибитора NK.
Чтобы проиллюстрировать этот подход и продемонстрировать осуществимость двойной направленной инсерции в первичные Т-клетки, были разработаны две разные матрицы (фиг. 19). Первая матрица, названная CARm (SEQ ID NO 31), разработана для инсерции CAR анти-CD22 кДНК в локус TRAC в присутствии TRAC TALEN® (SEQ ID NO 16 и 17). Вторая матрица, HLAEm, в двух вариантах (SEQ ID NO 69 и 71), была разработана для интеграции одноцепочечного белка, состоящего из гибридизированных пептидных фрагментов В2М, HLAE и HLAG в середине открытой рамки считывания В2М с использованием В2М TALEN® (SEQ ID NO 80 и 81 или 82 и 83 - правый и левый димеры, соответственно). Эти две матрицы содержат дополнительный 2А цис-действующий элемент, расположенный по цепи выше экспрессирующих кассет, для обеспечения совместной экспрессии одноцепочечного пептида В2М-HLAE-HLAG и CAR с нацеленным эндогенным геном. Полинуклеотидные и полипептидные последовательности перечислены в табл. 5.
Оценку эффективности проводят двойной нацеленной инсерцией в Т-клетки путем трансфекции их TRAC и В2М TALEN® и последующей их трансдукции с матрицами репарации AAV6, кодирующими анти-CD22 CAR и одноцепочечный пептид B2M-HLAE-HLAG. Такое лечение приводит к более чем 88% двойного нокаута TCR и HLA-ABC, к экспрессии примерно 68% анти-CD22 CAR среди популяции с двойным нокаутом и к примерно 68% экспрессии HLAE среди CAR-экспрессирующих Т-клеток с двойным нокаутом. В целом, этот метод позволяет вырабатывать около 40% TCR/HLA-ABC-негативных, CAR/HLAE-положительных Т-клеток (фиг. 21).
Эти сконструированные клетки могут быть проанализированы на устойчивость к NK и аллореагентным атакам Т-клеток. Тот же подход по конструированию может быть применен для получения TCR/HLA-ABC-отрицательных, CAR-положительных Т-клеток, несущих ингибиторы клеток NK, отличных от HLAE, и оценки их способности противостоять атаке клеток NK.
Такая оценка может быть выполнена на коллекции отрицательных по TCR/HLA-АВС, CAR-положительных Т-клеток, несущих различные ингибиторы клеток NK, что показано на фиг. 18. Этот подход может заключаться в трансфекции Т-клеток, например, TRAC и В2М TALEN, с последующей трансдукцией матрицей репарации AAV6, кодирующей CAR, например, анти-CD22 CAR, и библиотеку (или коллекцию) матриц репарации, кодирующих разные ингибиторы NK:
- тримерную матрицу HLAE, содержащую пептид VMAPRTLFL (SEQ ID NO.68), которая может быть инсертирована в локус B2m (SEQ ID NO.69),
- тримерную матрицу HLAE, содержащую пептид VMAPRTLIL (SEQ ID NO.70), которая может быть инсертирована в локус B2m (SEQ ID NO.71),
- тримерную матрицу UL18Тримерная матрица_Actine пептид (SEQ ID NO.72), которая может быть инсертирована в локус B2m (SEQ ID NO.73),
- тримерную матрицу UL18Тримерная матрица_HLACw пептид (SEQ ID NO.74), которая может быть инсертирована в локус B2m (SEQ ID NO.75),
- тримерную матрицу UL18Тримерная матрица_HLAG (SEQ ID NO.76), которая может быть инсертирована в локус B2m (SEQ ID NO.77),
Тримеры также могут содержать пептиды HLAG, которые можно использовать для образования этих тримеров, таких как один из следующих: HLAG1 (SEQ ID NO.84) HLAG2 (SEQ ID NO.85) HLAG3 (SEQ ID NO.86) HLAG4 (SEQ ID NO.87) HLAG5 (SEQ ID NO.88) HLAG6 (SEQ ID NO.89) HLAG7 (SEQ ID NO.90)
Тримеры HLAE или HLAG также могут содержать пептиды G (фиг. 20), выбранные из следующих пептидов, которыми перечень возможных пептидов не ограничивают:
пептид 1 VMAPRTLIL
пептид 2 VMAPRTLLL
пептид 3 VMAPRTLVL
пептид 4 AMAPRTLIL
пептид 5 VMAPRSLIL
пептид 6 VMAPRSLLL
пептид 7 VMAPRTLFL
пептид 8 VMAPRILIL
пептид 9 YLLPRRGPRL
пептид 10 ALPHAILRL
Получаемая библиотека TCR-отрицательных Т-клеток, положительных по ингибиторам CAR и NK, может культивироваться в присутствии клеток NK, а оставшиеся жизнеспособные клетки могут быть выделены и проанализированы с помощью высокопроизводительной последовательности ДНК для идентификации ингибиторов (ингибитора) NK, ответственных за устойчивость к атаке клеток NK.
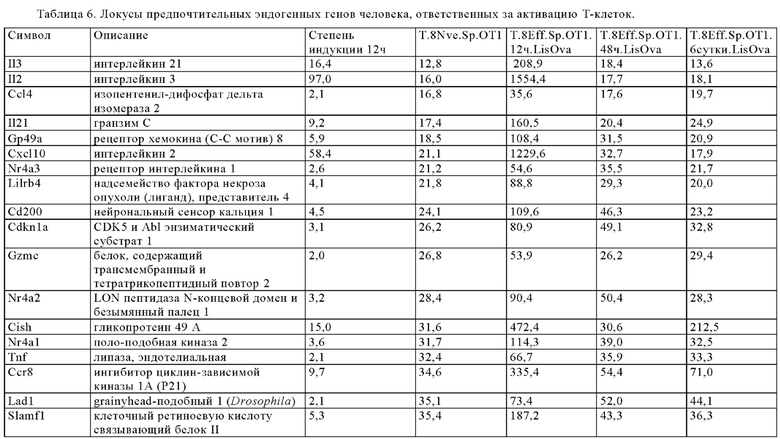
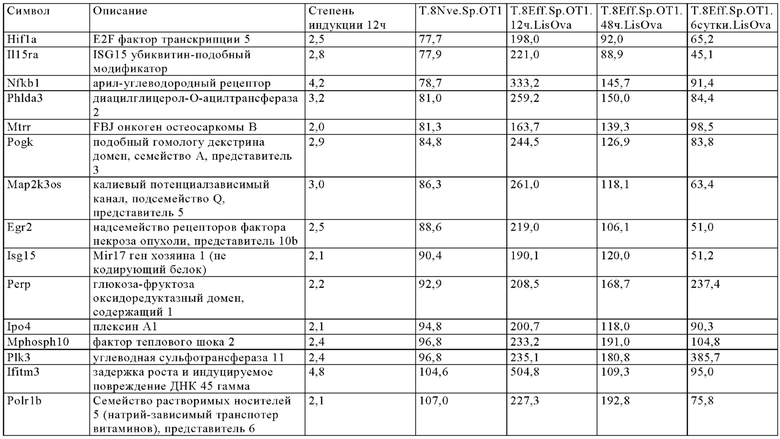
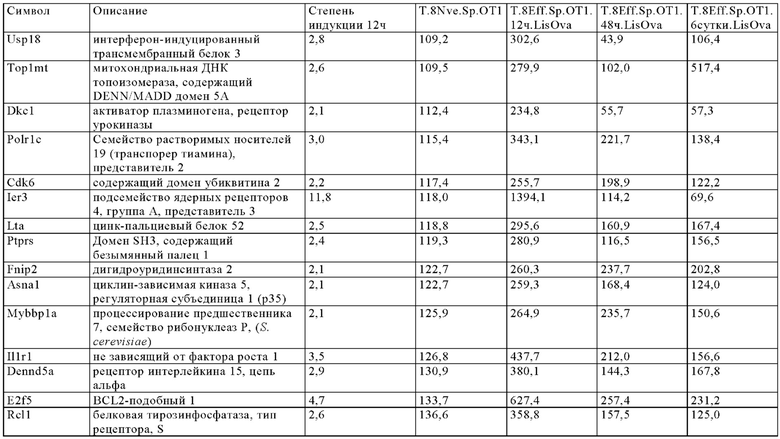
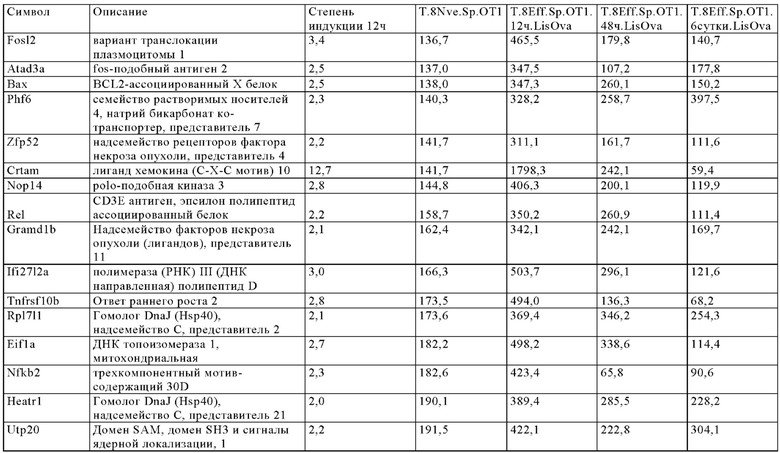
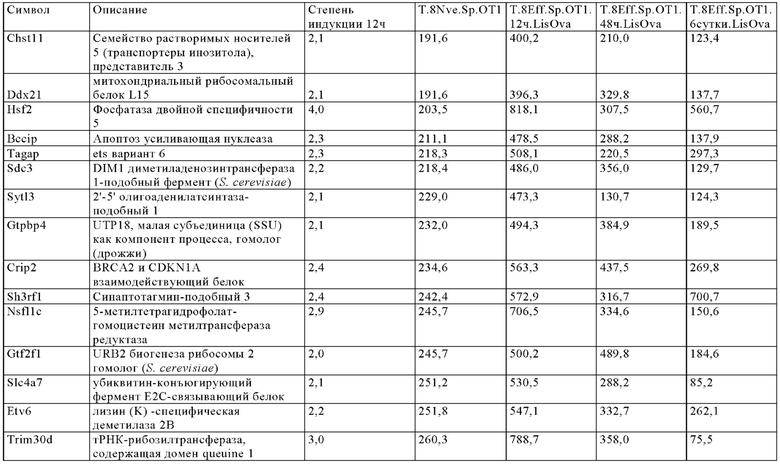
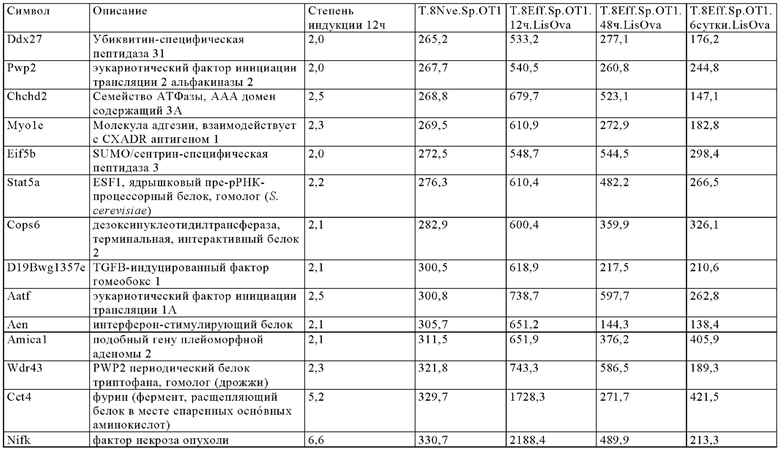
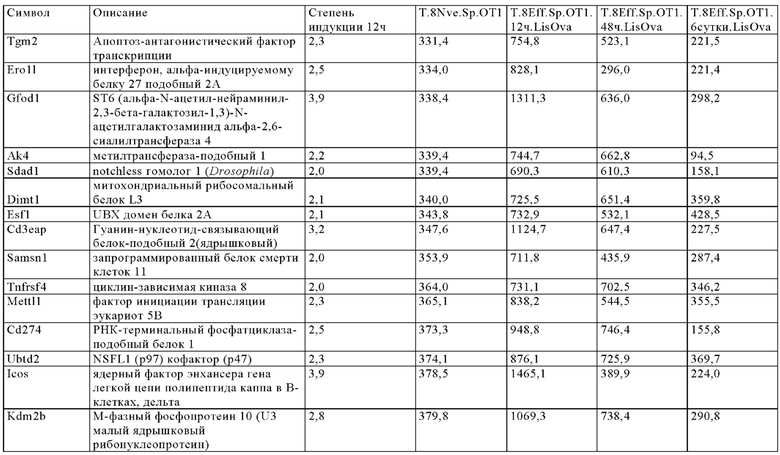
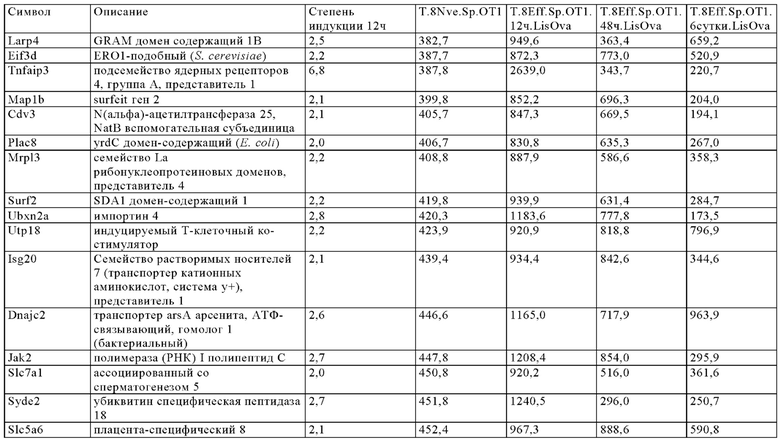
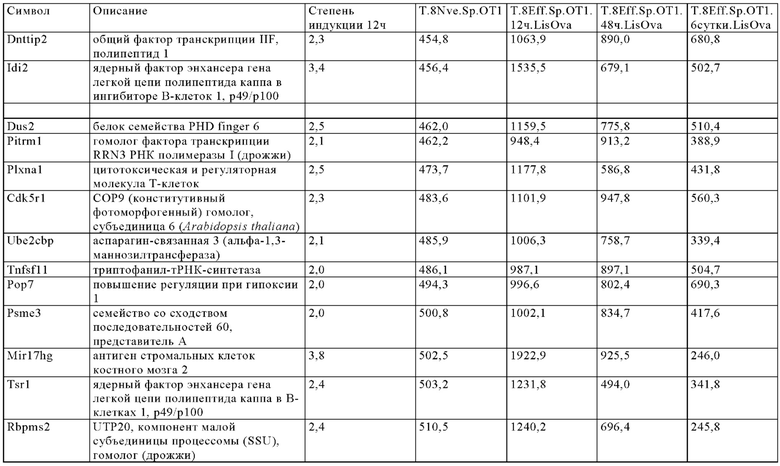
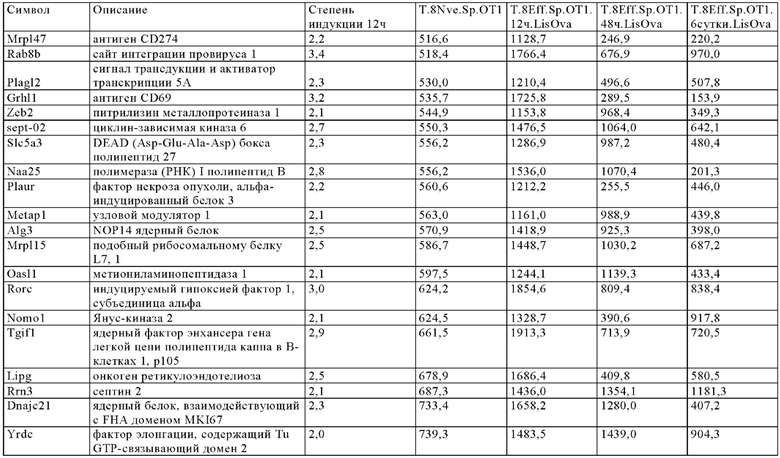
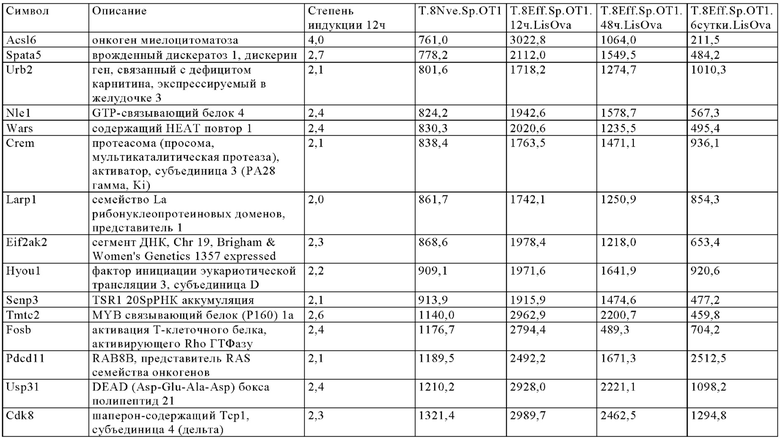

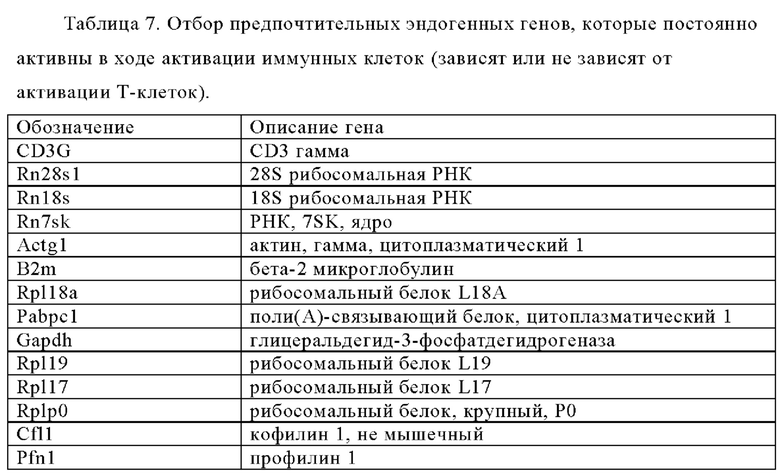
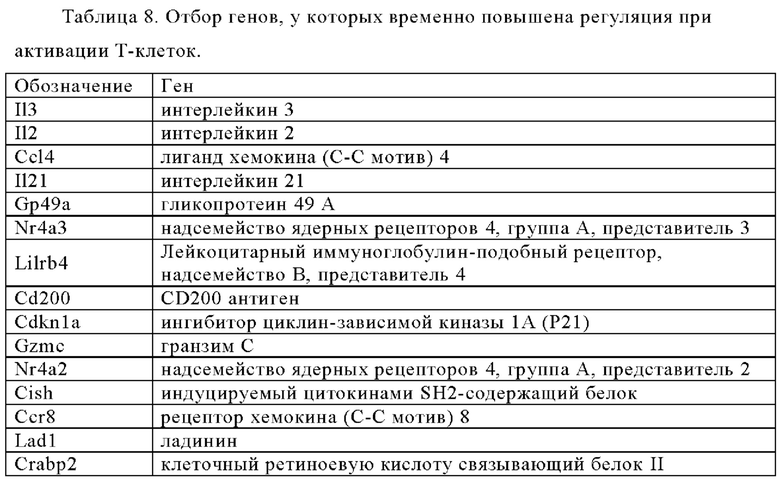

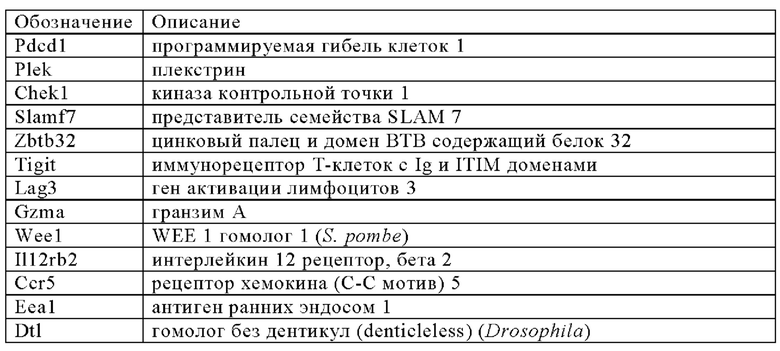
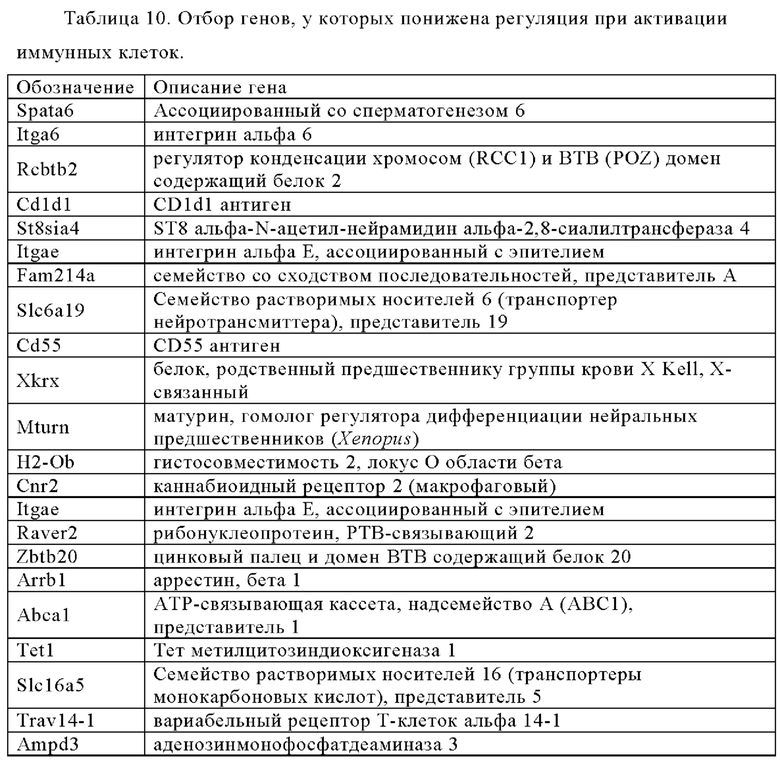
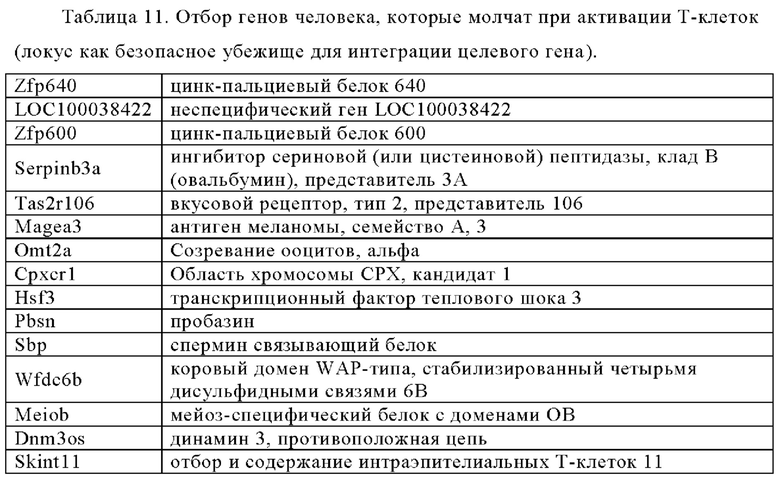
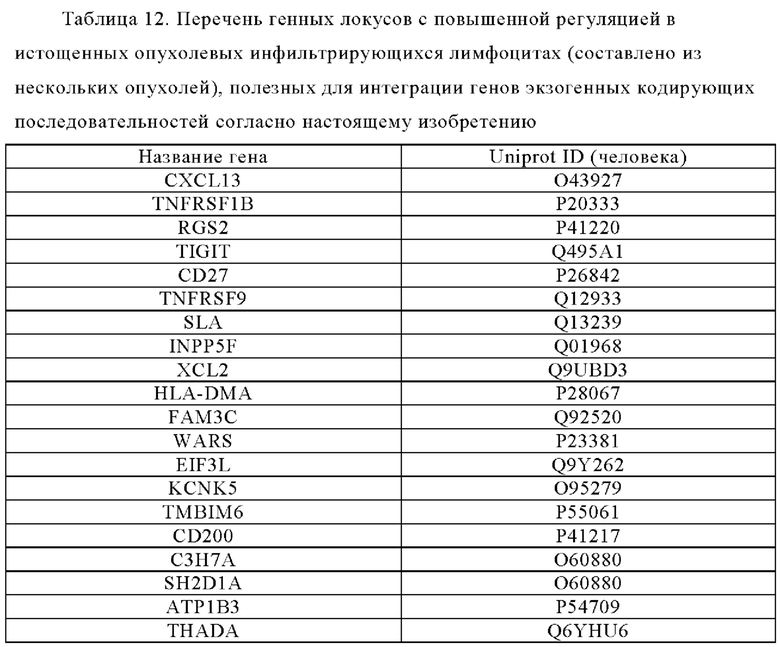
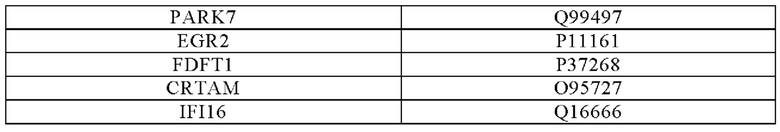
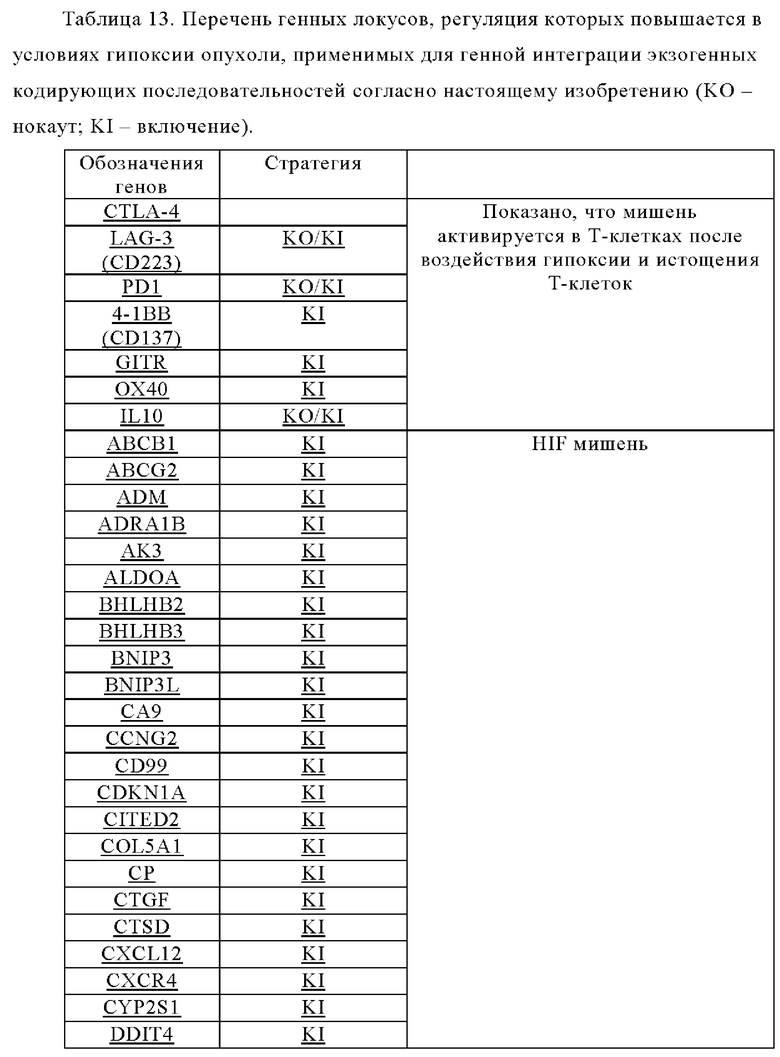
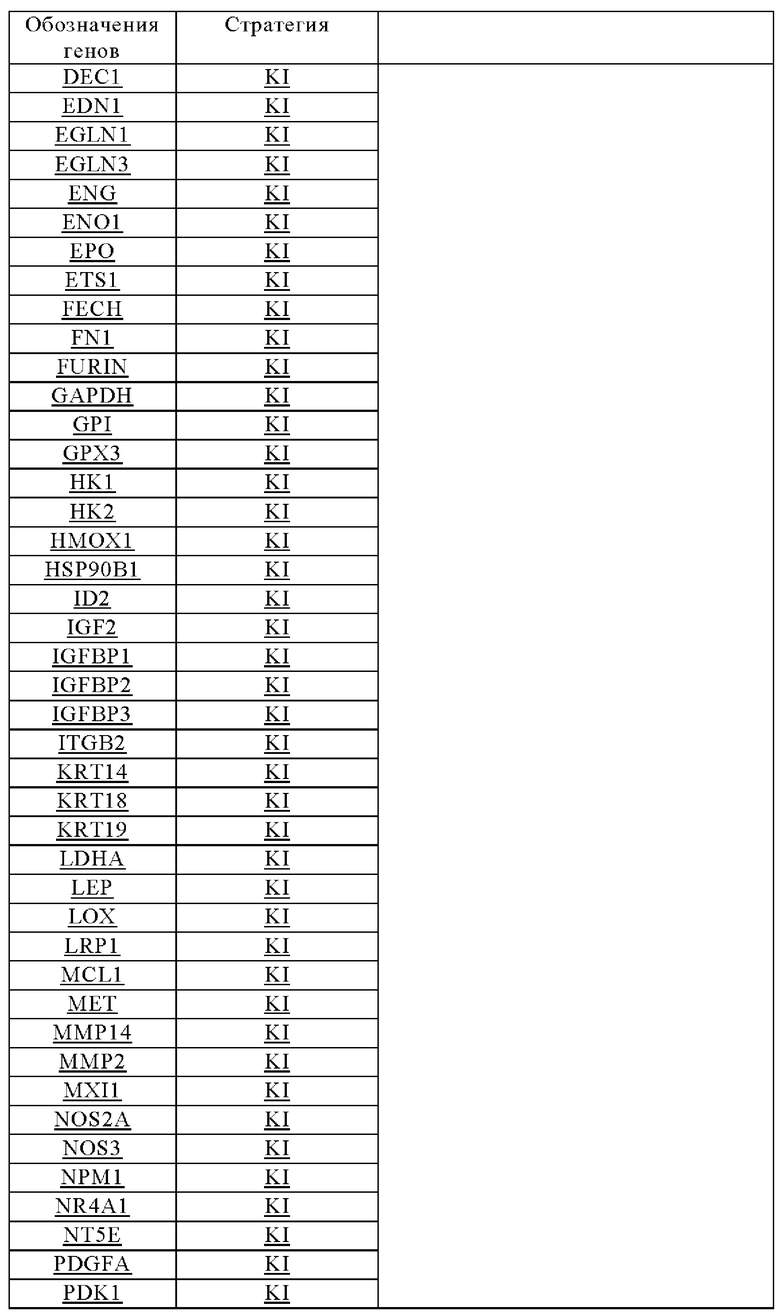
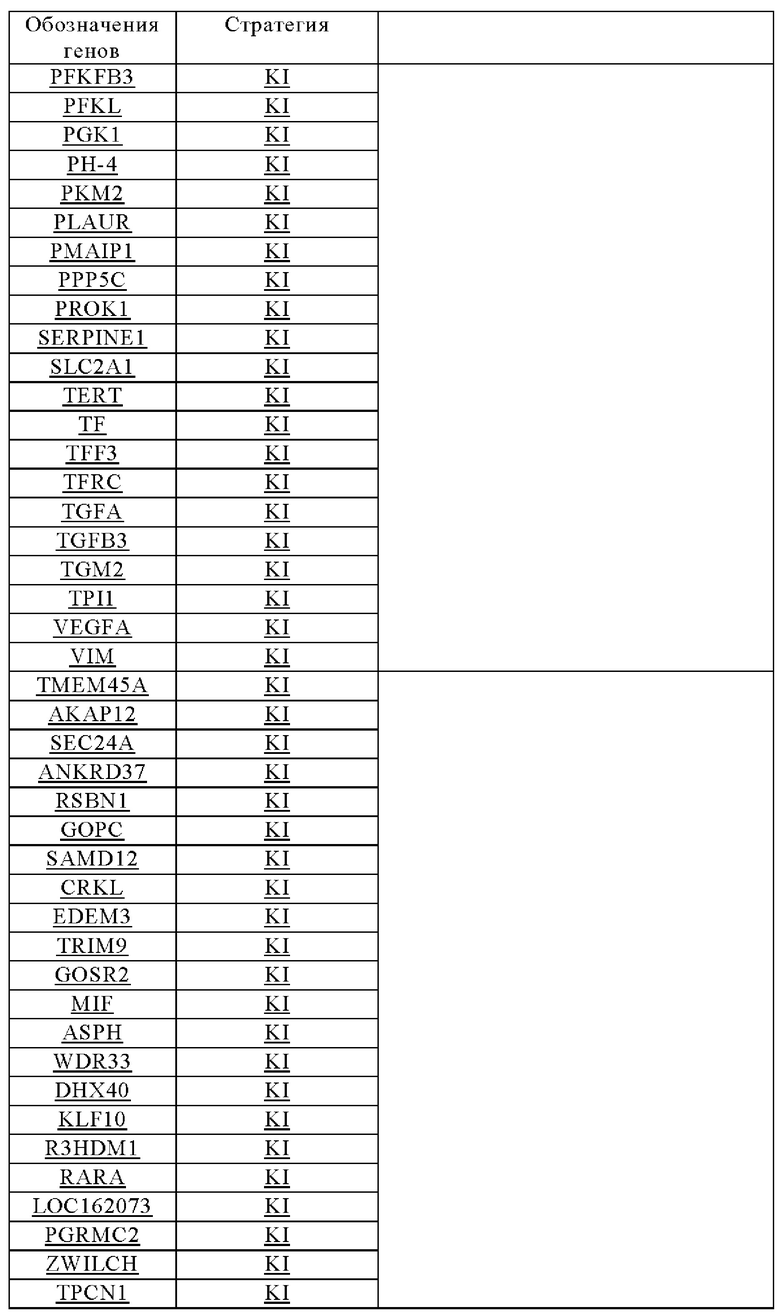
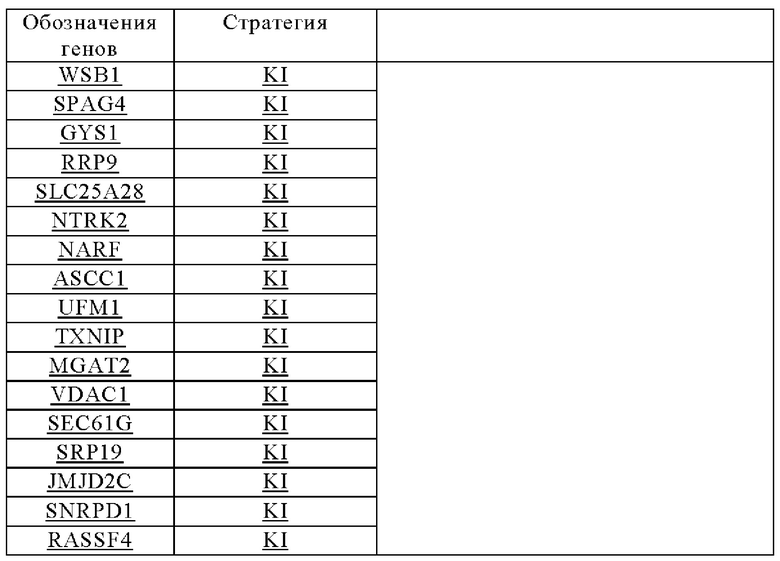
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Селлектис
<120> Целевая генная интеграция генов -ингибиторов NK для улучшенной
иммунной клеточной терапии
<130> P81604915PCT02
<150> PCT/EP2017/076798
<151> 2017-10-19
<150> PCT/EP2018/053343
<151> 2018-02-09
<160> 90
<170> PatentIn version 3.5
<210> 1
<211> 1000
<212> DNA
<213> artificial sequence
<220>
<223> PD1 left homology
<400> 1
ccaagccctg accctggcag gcatatgttt caggaggtcc ttgtcttggg agcccagggt 60
cgggggcccc gtgtctgtcc acatccgagt caatggccca tctcgtctct gaagcatctt 120
tgctgtgagc tctagtcccc actgtcttgc tggaaaatgt ggaggcccca ctgcccactg 180
cccagggcag caatgcccat accacgtggt cccagctccg agcttgtcct gaaaaggggg 240
caaagactgg accctgagcc tgccaagggg ccacactcct cccagggctg gggtctccat 300
gggcagcccc ccacccaccc agaccagtta cactcccctg tgccagagca gtgcagacag 360
gaccaggcca ggatgcccaa gggtcagggg ctggggatgg gtagccccca aacagccctt 420
tctgggggaa ctggcctcaa cggggaaggg ggtgaaggct cttagtagga aatcagggag 480
acccaagtca gagccaggtg ctgtgcagaa gctgcagcct cacgtagaag gaagaggctc 540
tgcagtggag gccagtgccc atccccgggt ggcagaggcc ccagcagaga cttctcaatg 600
acattccagc tggggtggcc cttccagagc ccttgctgcc cgagggatgt gagcaggtgg 660
ccggggaggc tttgtggggc cacccagccc cttcctcacc tctctccatc tctcagactc 720
cccagacagg ccctggaacc cccccacctt ctccccagcc ctgctcgtgg tgaccgaagg 780
ggacaacgcc accttcacct gcagcttctc caacacatcg gagagcttcg tgctaaactg 840
gtaccgcatg agccccagca accagacgga caagctggcc gccttccccg aggaccgcag 900
ccagcccggc caggactgcc gcttccgtgt cacacaactg cccaacgggc gtgacttcca 960
catgagcgtg gtcagggccc ggcgcaatga cagcggcacc 1000
<210> 2
<211> 1000
<212> DNA
<213> artificial sequence
<220>
<223> PD1 right homology
<400> 2
gcctgcgggc agagctcagg gtgacaggtg cggcctcgga ggccccgggg caggggtgag 60
ctgagccggt cctggggtgg gtgtcccctc ctgcacagga tcaggagctc cagggtcgta 120
gggcagggac cccccagctc cagtccaggg ctctgtcctg cacctgggga atggtgaccg 180
gcatctctgt cctctagctc tggaagcacc ccagcccctc tagtctgccc tcacccctga 240
ccctgaccct ccaccctgac cccgtcctaa cccctgacct ttgtgccctt ccagagagaa 300
gggcagaagt gcccacagcc caccccagcc cctcacccag gccagccggc cagttccaaa 360
ccctggtggt tggtgtcgtg ggcggcctgc tgggcagcct ggtgctgcta gtctgggtcc 420
tggccgtcat ctgctcccgg gccgcacgag gtaacgtcat cccagcccct cggcctgccc 480
tgccctaacc ctgctggcgg ccctcactcc cgcctcccct tcctccaccc ttccctcacc 540
ccaccccacc tccccccatc tccccgccag gctaagtccc tgatgaaggc ccctggacta 600
agacccccca cctaggagca cggctcaggg tcggcctggt gaccccaagt gtgtttctct 660
gcagggacaa taggagccag gcgcaccggc cagcccctgg tgagtctcac tcttttcctg 720
catgatccac tgtgccttcc ttcctgggtg ggcagaggtg gaaggacagg ctgggaccac 780
acggcctgca ggactcacat tctattatag ccaggacccc acctccccag cccccaggca 840
gcaacctcaa tccctaaagc catgatctgg ggccccagcc cacctgcggt ctccgggggt 900
gcccggccca tgtgtgtgcc tgcctgcggt ctccaggggt gcctggccca cgcgtgtgcc 960
cgcctgcggt ctctgggggt gcccggccca catatgtgcc 1000
<210> 3
<211> 2781
<212> DNA
<213> artificial sequence
<220>
<223> PD1_T3C-L2
<400> 3
atgggcgatc ctaaaaagaa acgtaaggtc atcgatatcg ccgatctacg cacgctcggc 60
tacagccagc agcaacagga gaagatcaaa ccgaaggttc gttcgacagt ggcgcagcac 120
cacgaggcac tggtcggcca cgggtttaca cacgcgcaca tcgttgcgtt aagccaacac 180
ccggcagcgt tagggaccgt cgctgtcaag tatcaggaca tgatcgcagc gttgccagag 240
gcgacacacg aagcgatcgt tggcgtcggc aaacagtggt ccggcgcacg cgctctggag 300
gccttgctca cggtggcggg agagttgaga ggtccaccgt tacagttgga cacaggccaa 360
cttctcaaga ttgcaaaacg tggcggcgtg accgcagtgg aggcagtgca tgcatggcgc 420
aatgcactga cgggtgcccc gctcaacttg acccccgagc aagtggtggc tatcgcttcc 480
aagctggggg gaaagcaggc cctggagacc gtccaggccc ttctcccagt gctttgccag 540
gctcacggac tgacccctga acaggtggtg gcaattgcct cacacgacgg gggcaagcag 600
gcactggaga ctgtccagcg gctgctgcct gtcctctgcc aggcccacgg actcactcct 660
gagcaggtcg tggccattgc cagccacgat gggggcaaac aggctctgga gaccgtgcag 720
cgcctcctcc cagtgctgtg ccaggctcat gggctgaccc cacagcaggt cgtcgccatt 780
gccagtaacg gcggggggaa gcaggccctc gaaacagtgc agaggctgct gcccgtcttg 840
tgccaagcac acggcctgac acccgagcag gtggtggcca tcgcctctca tgacggcggc 900
aagcaggccc ttgagacagt gcagagactg ttgcccgtgt tgtgtcaggc ccacgggttg 960
acaccccagc aggtggtcgc catcgccagc aatggcgggg gaaagcaggc ccttgagacc 1020
gtgcagcggt tgcttccagt gttgtgccag gcacacggac tgacccctca acaggtggtc 1080
gcaatcgcca gctacaaggg cggaaagcag gctctggaga cagtgcagcg cctcctgccc 1140
gtgctgtgtc aggctcacgg actgacacca cagcaggtgg tcgccatcgc cagtaacggg 1200
ggcggcaagc aggctttgga gaccgtccag agactcctcc ccgtcctttg ccaggcccac 1260
gggttgacac ctcagcaggt cgtcgccatt gcctccaaca acgggggcaa gcaggccctc 1320
gaaactgtgc agaggctgct gcctgtgctg tgccaggctc atgggctgac accccagcag 1380
gtggtggcca ttgcctctaa caacggcggc aaacaggcac tggagaccgt gcaaaggctg 1440
ctgcccgtcc tctgccaagc ccacgggctc actccacagc aggtcgtggc catcgcctca 1500
aacaatggcg ggaagcaggc cctggagact gtgcaaaggc tgctccctgt gctctgccag 1560
gcacacggac tgacccctca gcaggtggtg gcaatcgctt ccaacaacgg gggaaagcag 1620
gccctcgaaa ccgtgcagcg cctcctccca gtgctgtgcc aggcacatgg cctcacaccc 1680
gagcaagtgg tggctatcgc cagccacgac ggagggaagc aggctctgga gaccgtgcag 1740
aggctgctgc ctgtcctgtg ccaggcccac gggcttactc cagagcaggt cgtcgccatc 1800
gccagtcatg atggggggaa gcaggccctt gagacagtcc agcggctgct gccagtcctt 1860
tgccaggctc acggcttgac tcccgagcag gtcgtggcca ttgcctcaaa cattgggggc 1920
aaacaggccc tggagacagt gcaggccctg ctgcccgtgt tgtgtcaggc ccacggcttg 1980
acaccccagc aggtggtcgc cattgcctct aatggcggcg ggagacccgc cttggagagc 2040
attgttgccc agttatctcg ccctgatccg gcgttggccg cgttgaccaa cgaccacctc 2100
gtcgccttgg cctgcctcgg cgggcgtcct gcgctggatg cagtgaaaaa gggattgggg 2160
gatcctatca gccgttccca gctggtgaag tccgagctgg aggagaagaa atccgagttg 2220
aggcacaagc tgaagtacgt gccccacgag tacatcgagc tgatcgagat cgcccggaac 2280
agcacccagg accgtatcct ggagatgaag gtgatggagt tcttcatgaa ggtgtacggc 2340
tacaggggca agcacctggg cggctccagg aagcccgacg gcgccatcta caccgtgggc 2400
tcccccatcg actacggcgt gatcgtggac accaaggcct actccggcgg ctacaacctg 2460
cccatcggcc aggccgacga aatgcagagg tacgtggagg agaaccagac caggaacaag 2520
cacatcaacc ccaacgagtg gtggaaggtg tacccctcca gcgtgaccga gttcaagttc 2580
ctgttcgtgt ccggccactt caagggcaac tacaaggccc agctgaccag gctgaaccac 2640
atcaccaact gcaacggcgc cgtgctgtcc gtggaggagc tcctgatcgg cggcgagatg 2700
atcaaggccg gcaccctgac cctggaggag gtgaggagga agttcaacaa cggcgagatc 2760
aacttcgcgg ccgactgata a 2781
<210> 4
<211> 2778
<212> DNA
<213> artificial sequence
<220>
<223> PD1T3R
<400> 4
atgggcgatc ctaaaaagaa acgtaaggtc atcgatatcg ccgatctacg cacgctcggc 60
tacagccagc agcaacagga gaagatcaaa ccgaaggttc gttcgacagt ggcgcagcac 120
cacgaggcac tggtcggcca cgggtttaca cacgcgcaca tcgttgcgtt aagccaacac 180
ccggcagcgt tagggaccgt cgctgtcaag tatcaggaca tgatcgcagc gttgccagag 240
gcgacacacg aagcgatcgt tggcgtcggc aaacagtggt ccggcgcacg cgctctggag 300
gccttgctca cggtggcggg agagttgaga ggtccaccgt tacagttgga cacaggccaa 360
cttctcaaga ttgcaaaacg tggcggcgtg accgcagtgg aggcagtgca tgcatggcgc 420
aatgcactga cgggtgcccc gctcaacttg acccccgagc aagtcgtcgc aatcgccagc 480
catgatggag ggaagcaagc cctcgaaacc gtgcagcggt tgcttcctgt gctctgccag 540
gcccacggcc ttacccctca gcaggtggtg gccatcgcaa gtaacggagg aggaaagcaa 600
gccttggaga cagtgcagcg cctgttgccc gtgctgtgcc aggcacacgg cctcacacca 660
gagcaggtcg tggccattgc ctcccatgac ggggggaaac aggctctgga gaccgtccag 720
aggctgctgc ccgtcctctg tcaagctcac ggcctgactc cccaacaagt ggtcgccatc 780
gcctctaatg gcggcgggaa gcaggcactg gaaacagtgc agagactgct ccctgtgctt 840
tgccaagctc atgggttgac cccccaacag gtcgtcgcta ttgcctcaaa cggggggggc 900
aagcaggccc ttgagactgt gcagaggctg ttgccagtgc tgtgtcaggc tcacgggctc 960
actccacaac aggtggtcgc aattgccagc aacggcggcg gaaagcaagc tcttgaaacc 1020
gtgcaacgcc tcctgcccgt gctctgtcag gctcatggcc tgacaccaca acaagtcgtg 1080
gccatcgcca gtaataatgg cgggaaacag gctcttgaga ccgtccagag gctgctccca 1140
gtgctctgcc aggcacacgg gctgaccccc gagcaggtgg tggctatcgc cagcaatatt 1200
gggggcaagc aggccctgga aacagtccag gccctgctgc cagtgctttg ccaggctcac 1260
gggctcactc cccagcaggt cgtggcaatc gcctccaacg gcggagggaa gcaggctctg 1320
gagaccgtgc agagactgct gcccgtcttg tgccaggccc acggactcac acctgaacag 1380
gtcgtcgcca ttgcctctca cgatgggggc aaacaagccc tggagacagt gcagcggctg 1440
ttgcctgtgt tgtgccaagc ccacggcttg actcctcaac aagtggtcgc catcgcctca 1500
aatggcggcg gaaaacaagc tctggagaca gtgcagaggt tgctgcccgt cctctgccaa 1560
gcccacggcc tgactcccca acaggtcgtc gccattgcca gcaacaacgg aggaaagcag 1620
gctctcgaaa ctgtgcagcg gctgcttcct gtgctgtgtc aggctcatgg gctgaccccc 1680
gagcaagtgg tggctattgc ctctaatgga ggcaagcaag cccttgagac agtccagagg 1740
ctgttgccag tgctgtgcca ggcccacggg ctcacacccc agcaggtggt cgccatcgcc 1800
agtaacaacg ggggcaaaca ggcattggaa accgtccagc gcctgcttcc agtgctctgc 1860
caggcacacg gactgacacc cgaacaggtg gtggccattg catcccatga tgggggcaag 1920
caggccctgg agaccgtgca gagactcctg ccagtgttgt gccaagctca cggcctcacc 1980
cctcagcaag tcgtggccat cgcctcaaac ggggggggcc ggcctgcact ggagagcatt 2040
gttgcccagt tatctcgccc tgatccggcg ttggccgcgt tgaccaacga ccacctcgtc 2100
gccttggcct gcctcggcgg gcgtcctgcg ctggatgcag tgaaaaaggg attgggggat 2160
cctatcagcc gttcccagct ggtgaagtcc gagctggagg agaagaaatc cgagttgagg 2220
cacaagctga agtacgtgcc ccacgagtac atcgagctga tcgagatcgc ccggaacagc 2280
acccaggacc gtatcctgga gatgaaggtg atggagttct tcatgaaggt gtacggctac 2340
aggggcaagc acctgggcgg ctccaggaag cccgacggcg ccatctacac cgtgggctcc 2400
cccatcgact acggcgtgat cgtggacacc aaggcctact ccggcggcta caacctgccc 2460
atcggccagg ccgacgaaat gcagaggtac gtggaggaga accagaccag gaacaagcac 2520
atcaacccca acgagtggtg gaaggtgtac ccctccagcg tgaccgagtt caagttcctg 2580
ttcgtgtccg gccacttcaa gggcaactac aaggcccagc tgaccaggct gaaccacatc 2640
accaactgca acggcgccgt gctgtccgtg gaggagctcc tgatcggcgg cgagatgatc 2700
aaggccggca ccctgaccct ggaggaggtg aggaggaagt tcaacaacgg cgagatcaac 2760
ttcgcggccg actgataa 2778
<210> 5
<211> 49
<212> DNA
<213> artificial sequence
<220>
<223> PD1-T3
<400> 5
tacctctgtg gggccatctc cctggccccc aaggcgcaga tcaaagaga 49
<210> 6
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> 2A-element
<400> 6
tccggtgagg gcagaggaag tcttctaaca tgcggtgacg tggaggagaa tccgggcccc 60
<210> 7
<211> 1989
<212> DNA
<213> artificial sequence
<220>
<223> apoptosis CAR
<400> 7
gctttgcctg tcactgcctt gctgcttcca cttgctctgt tgttgcacgc cgcaagaccc 60
gaggtcaagc tccaggaaag cggaccaggg ctggtggccc ctagtcagtc attgagcgtc 120
acttgcaccg tcagcggcgt gtctctgccc gattacggcg tgagctggat cagacagccc 180
ccaaggaagg gactggagtg gctgggcgtc atctggggga gcgagactac ctactacaac 240
agcgccctga agagcaggct gaccatcatt aaggacaact ccaagtccca ggtctttctg 300
aaaatgaaca gcctgcagac tgatgacact gccatctact actgcgccaa gcattactac 360
tacgggggca gctacgctat ggactactgg gggcagggga cctctgtcac agtgtcaagt 420
ggcggaggag gcagtggcgg agggggaagt gggggcggcg gcagcgacat ccagatgacc 480
cagacaacat ccagcctctc cgcctctctg ggcgacagag tgacaatcag ctgccgggcc 540
agtcaggaca tcagcaagta tctcaattgg taccagcaga aaccagacgg gacagtgaaa 600
ttgctgatct accacacatc caggctgcac tcaggagtcc ccagcaggtt ttccggctcc 660
ggctccggga cagattacag tctgaccatt tccaacctgg agcaggagga tattgccaca 720
tacttttgcc agcaaggcaa cactctgccc tataccttcg gcggaggcac aaaactggag 780
attactcggt cggatcccga gcccaaatct cctgacaaaa ctcacacatg cccaccgtgc 840
ccagcacctc ccgtggccgg cccgtcagtg ttcctcttcc ccccaaaacc caaggacacc 900
ctcatgatcg cccggacccc tgaggtcaca tgcgtggtgg tggacgtgag ccacgaggac 960
cctgaggtca agttcaactg gtacgtggac ggcgtggagg tgcataatgc caagacaaag 1020
ccgcgggagg agcagtacaa cagcacgtac cgtgtggtca gcgtcctcac cgtcctgcac 1080
caggactggc tgaatggcaa ggagtacaag tgcaaggtgt ccaacaaagc cctcccagcc 1140
cccatcgaga aaaccatctc caaagccaaa gggcagcccc gagaaccaca ggtgtacacc 1200
ctgcccccat cccgggatga gctgaccaag aaccaggtca gcctgacctg cctggtcaaa 1260
ggcttctatc ccagcgacat cgccgtggag tgggagagca atgggcaacc ggagaacaac 1320
tacaagacca cgcctcccgt gctggactcc gacggctcct tcttcctcta cagcaagctc 1380
accgtggaca agagcaggtg gcagcagggg aacgtgttct catgctccgt gatgcatgag 1440
gccctgcaca atcactatac ccagaaatct ctgagtctga gcccaggcaa gaaggatatt 1500
ttggggtggc tttgccttct tcttttgcca attccactaa ttgtttgggt gaagagaaag 1560
gaagtacaga aaacatgcag aaagcacaga aaggaaaacc aaggttctca tgaatctcca 1620
accttaaatc ctgaaacagt ggcaataaat ttatctgatg ttgacttgag taaatatatc 1680
accactattg ctggagtcat gacactaagt caagttaaag gctttgttcg aaagaatggt 1740
gtcaatgaag ccaaaataga tgagatcaag aatgacaatg tccaagacac agcagaacag 1800
aaagttcaac tgcttcgtaa ttggcatcaa cttcatggaa agaaagaagc gtatgacaca 1860
ttgattgcag atctcaaaaa agccaatctt tgtactcttg cagagaaaat tcagactatc 1920
atcctcaagg acattactag tgactcagaa aattcaaact tcagaaatga aatccagagc 1980
ttggtcgaa 1989
<210> 8
<211> 276
<212> DNA
<213> artificial sequence
<220>
<223> BGH polyA
<400> 8
tctagagggc ccgtttaaac ccgctgatca gcctcgactg tgccttctag ttgccagcca 60
tctgttgttt gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac tcccactgtc 120
ctttcctaat aaaatgagga aattgcatcg cattgtctga gtaggtgtca ttctattctg 180
gggggtgggg tggggcagga cagcaagggg gaggattggg aagacaatag caggcatgct 240
ggggatgcgg tgggctctat gactagtggc gaattc 276
<210> 9
<211> 1000
<212> DNA
<213> artificial sequence
<220>
<223> Lck left homology
<400> 9
gggatagggg gtgcctctgt gtgtgtgtgt gagagtgtgt gtgtgtaggg tgtgtatatg 60
tatagggtgt gtgtgagtgt gtgtgtgtga gagagtgtgt gtgtggcaga atagactgcg 120
gaggtggatt tcatcttgat atgaaaggtc tggaatgcat ggtacattaa actttgagga 180
cagcgctttc caagcactct gaggagcagc cctagagaag gaggagctgc agggactccg 240
ggggcttcaa agtgagggcc ccactctgct tcaggcaaaa caggcacaca tttatcactt 300
tatctatgga gttctgcttg atttcatcag acaaaaaatt tccactgcta aaacaggcaa 360
ataaacaaaa aaaaagttat ggccaacaga gtcactggag ggttttctgc tggggagaag 420
caagcccgtg tttgaaggaa ccctgtgaga tgactgtggg ctgtgtgagg ggaacagcgg 480
gggcttgatg gtggacttcg ggagcagaag cctctttctc agcctcctca gctagacagg 540
ggaattataa taggaggtgt ggcgtgcaca cctctccagt aggggagggt ctgataagtc 600
aggtctctcc caggcttggg aaagtgtgtg tcatctctag gaggtggtcc tcccaacaca 660
gggtactggc agagggagag ggagggggca gaggcaggaa gtgggtaact agactaacaa 720
aggtgcctgt ggcggtttgc ccatcccagg tgggagggtg gggctagggc tcaggggccg 780
tgtgtgaatt tacttgtagc ctgagggctc agagggagca ccggtttgga gctgggaccc 840
cctattttag cttttctgtg gctggtgaat ggggatccca ggatctcaca atctcaggta 900
cttttggaac tttccagggc aaggccccat tatatctgat gttgggggag cagatcttgg 960
gggagcccct tcagccccct cttccattcc ctcagggacc 1000
<210> 10
<211> 219
<212> PRT
<213> artificial sequence
<220>
<223> Interleukin-12 subunit alpha
<400> 10
Met Cys Pro Ala Arg Ser Leu Leu Leu Val Ala Thr Leu Val Leu Leu
1 5 10 15
Asp His Leu Ser Leu Ala Arg Asn Leu Pro Val Ala Thr Pro Asp Pro
20 25 30
Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu Arg Ala Val
35 40 45
Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe Tyr Pro Cys
50 55 60
Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp Lys Thr Ser
65 70 75 80
Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn Glu Ser Cys
85 90 95
Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser Cys Leu Ala
100 105 110
Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser Ser Ile Tyr
115 120 125
Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met Asn Ala Lys
130 135 140
Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln Asn Met Leu
145 150 155 160
Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn Ser Glu Thr
165 170 175
Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr Lys Thr Lys
180 185 190
Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg Ala Val Thr
195 200 205
Ile Asp Arg Val Met Ser Tyr Leu Asn Ala Ser
210 215
<210> 11
<211> 328
<212> PRT
<213> artificial sequence
<220>
<223> Interleukin-12 subunit beta
<400> 11
Met Cys His Gln Gln Leu Val Ile Ser Trp Phe Ser Leu Val Phe Leu
1 5 10 15
Ala Ser Pro Leu Val Ala Ile Trp Glu Leu Lys Lys Asp Val Tyr Val
20 25 30
Val Glu Leu Asp Trp Tyr Pro Asp Ala Pro Gly Glu Met Val Val Leu
35 40 45
Thr Cys Asp Thr Pro Glu Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln
50 55 60
Ser Ser Glu Val Leu Gly Ser Gly Lys Thr Leu Thr Ile Gln Val Lys
65 70 75 80
Glu Phe Gly Asp Ala Gly Gln Tyr Thr Cys His Lys Gly Gly Glu Val
85 90 95
Leu Ser His Ser Leu Leu Leu Leu His Lys Lys Glu Asp Gly Ile Trp
100 105 110
Ser Thr Asp Ile Leu Lys Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe
115 120 125
Leu Arg Cys Glu Ala Lys Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp
130 135 140
Leu Thr Thr Ile Ser Thr Asp Leu Thr Phe Ser Val Lys Ser Ser Arg
145 150 155 160
Gly Ser Ser Asp Pro Gln Gly Val Thr Cys Gly Ala Ala Thr Leu Ser
165 170 175
Ala Glu Arg Val Arg Gly Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu
180 185 190
Cys Gln Glu Asp Ser Ala Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile
195 200 205
Glu Val Met Val Asp Ala Val His Lys Leu Lys Tyr Glu Asn Tyr Thr
210 215 220
Ser Ser Phe Phe Ile Arg Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn
225 230 235 240
Leu Gln Leu Lys Pro Leu Lys Asn Ser Arg Gln Val Glu Val Ser Trp
245 250 255
Glu Tyr Pro Asp Thr Trp Ser Thr Pro His Ser Tyr Phe Ser Leu Thr
260 265 270
Phe Cys Val Gln Val Gln Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg
275 280 285
Val Phe Thr Asp Lys Thr Ser Ala Thr Val Ile Cys Arg Lys Asn Ala
290 295 300
Ser Ile Ser Val Arg Ala Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser
305 310 315 320
Glu Trp Ala Ser Val Pro Cys Ser
325
<210> 12
<211> 1000
<212> DNA
<213> artificial sequence
<220>
<223> lck right homology
<400> 12
ggctgtggct gcagctcaca cccggaagat gactggatgg aaaacatcga tgtgtgtgag 60
aactgccatt atcccatagt cccactggat ggcaagggca cggtaagagg cgagacaggg 120
gccttggtga gggagttggg tagagaatgc aacccaggag aaagaaatga ccagcactac 180
aggcccttga aagaatagag tggccctctc ccctgaaata cagaaaggaa aagaggccca 240
gagaggggaa gggaatctcc taagatcaca cagaaagtag ttggtaaact cagggataac 300
atctaaccag gctggagagg ctgagagcag agcagggggg aagggggcca gggtctgacc 360
caatcttctg ctttctgacc ccaccctcat cccccactcc acagctgctc atccgaaatg 420
gctctgaggt gcgggaccca ctggttacct acgaaggctc caatccgccg gcttccccac 480
tgcaaggtga ccccaggcag cagggcctga aagacaaggc ctgcggatcc ctggctgttg 540
gcttccacct ctcccccacc tactttctcc ccggtcttgc cttccttgtc ccccaccctg 600
taactccagg cttcctgccg atcccagctc ggttctccct gatgcccctt gtctttacag 660
acaacctggt tatcgctctg cacagctatg agccctctca cgacggagat ctgggctttg 720
agaaggggga acagctccgc atcctggagc agtgagtccc tctccacctt gctctggcgg 780
agtccgtgag ggagcggcga tctccgcgac ccgcagccct cctgcggccc ttgaccagct 840
cggggtggcc gcccttggga caaaattcga ggctcagtat tgctgagcca gggttggggg 900
aggctggctt aaggggtgga ggggtctttg agggagggtc tcaggtcgac ggctgagcga 960
gccacactga cccacctccg tggcgcagga gcggcgagtg 1000
<210> 13
<211> 1992
<212> DNA
<213> artificial sequence
<220>
<223> apoptosis CAR
<400> 13
atggctttgc ctgtcactgc cttgctgctt ccacttgctc tgttgttgca cgccgcaaga 60
cccgaggtca agctccagga aagcggacca gggctggtgg cccctagtca gtcattgagc 120
gtcacttgca ccgtcagcgg cgtgtctctg cccgattacg gcgtgagctg gatcagacag 180
cccccaagga agggactgga gtggctgggc gtcatctggg ggagcgagac tacctactac 240
aacagcgccc tgaagagcag gctgaccatc attaaggaca actccaagtc ccaggtcttt 300
ctgaaaatga acagcctgca gactgatgac actgccatct actactgcgc caagcattac 360
tactacgggg gcagctacgc tatggactac tgggggcagg ggacctctgt cacagtgtca 420
agtggcggag gaggcagtgg cggaggggga agtgggggcg gcggcagcga catccagatg 480
acccagacaa catccagcct ctccgcctct ctgggcgaca gagtgacaat cagctgccgg 540
gccagtcagg acatcagcaa gtatctcaat tggtaccagc agaaaccaga cgggacagtg 600
aaattgctga tctaccacac atccaggctg cactcaggag tccccagcag gttttccggc 660
tccggctccg ggacagatta cagtctgacc atttccaacc tggagcagga ggatattgcc 720
acatactttt gccagcaagg caacactctg ccctatacct tcggcggagg cacaaaactg 780
gagattactc ggtcggatcc cgagcccaaa tctcctgaca aaactcacac atgcccaccg 840
tgcccagcac ctcccgtggc cggcccgtca gtgttcctct tccccccaaa acccaaggac 900
accctcatga tcgcccggac ccctgaggtc acatgcgtgg tggtggacgt gagccacgag 960
gaccctgagg tcaagttcaa ctggtacgtg gacggcgtgg aggtgcataa tgccaagaca 1020
aagccgcggg aggagcagta caacagcacg taccgtgtgg tcagcgtcct caccgtcctg 1080
caccaggact ggctgaatgg caaggagtac aagtgcaagg tgtccaacaa agccctccca 1140
gcccccatcg agaaaaccat ctccaaagcc aaagggcagc cccgagaacc acaggtgtac 1200
accctgcccc catcccggga tgagctgacc aagaaccagg tcagcctgac ctgcctggtc 1260
aaaggcttct atcccagcga catcgccgtg gagtgggaga gcaatgggca accggagaac 1320
aactacaaga ccacgcctcc cgtgctggac tccgacggct ccttcttcct ctacagcaag 1380
ctcaccgtgg acaagagcag gtggcagcag gggaacgtgt tctcatgctc cgtgatgcat 1440
gaggccctgc acaatcacta tacccagaaa tctctgagtc tgagcccagg caagaaggat 1500
attttggggt ggctttgcct tcttcttttg ccaattccac taattgtttg ggtgaagaga 1560
aaggaagtac agaaaacatg cagaaagcac agaaaggaaa accaaggttc tcatgaatct 1620
ccaaccttaa atcctgaaac agtggcaata aatttatctg atgttgactt gagtaaatat 1680
atcaccacta ttgctggagt catgacacta agtcaagtta aaggctttgt tcgaaagaat 1740
ggtgtcaatg aagccaaaat agatgagatc aagaatgaca atgtccaaga cacagcagaa 1800
cagaaagttc aactgcttcg taattggcat caacttcatg gaaagaaaga agcgtatgac 1860
acattgattg cagatctcaa aaaagccaat ctttgtactc ttgcagagaa aattcagact 1920
atcatcctca aggacattac tagtgactca gaaaattcaa acttcagaaa tgaaatccag 1980
agcttggtcg aa 1992
<210> 14
<211> 1000
<212> DNA
<213> artificial sequence
<220>
<223> Lck left homology
<400> 14
ctcataacaa ttctatgagg taggaacagt tatttactct attttccaaa taaggaaact 60
gggctcgccc aaggttccac aactaacatg tgtgtattat tgagcattta atttacacca 120
gggaagcagg ttgtggtggt gtgcacctgt tgtccagcta tttaggaggc tgaggtgaaa 180
ggatcacttg aacggaggag ttcaaatttg caatgtgcta tgattgtgcc tgtgaacagc 240
tgctgcactc cagcctgggc aacatagtga gatcccttat ctaaaacatt ttttttaagt 300
aaataatcag gtgggcacgg tggctcacgc ctgtaatcca gcactttggg aggctgaggc 360
gggcggatca cctgaggtca ggagttcaag accagcctga ccaacatgga gaaacccgtc 420
tctactaaaa atacaaaatt agcttggcgt ggtggtgcat gcctgtaatc ccagctactc 480
gagaagctga ggcaggagaa ttgtttgaac ctgggaggtg gaggttgcgg tgagccgaga 540
tcgcaccatt gcactccagc ctgggcaaca agagtgaaat tgcatctcaa aaaaaaagaa 600
aaggaaataa tctataccag gcactccaag tggtgtgact gatattcaac aagtacctct 660
agtgtgacct taccattgat gaagaccaag attcttttgg attggtgctc acactgtgcc 720
agttaaatat tccgaacatt acccttgcct gtgggcttcc agtgcctgac cttgatgtcc 780
tttcacccat caacccgtag ggatgaccaa cccggaggtg attcagaacc tggagcgagg 840
ctaccgcatg gtgcgccctg acaactgtcc agaggagctg taccaactca tgaggctgtg 900
ctggaaggag cgcccagagg accggcccac ctttgactac ctgcgcagtg tgctggagga 960
cttcttcacg gccacagagg gccagtacca gcctcagcct 1000
<210> 15
<211> 1000
<212> DNA
<213> artificial sequence
<220
<223> lck right homology
<400> 15
gaggccttga gaggccctgg ggttctcccc ctttctctcc agcctgactt ggggagatgg 60
agttcttgtg ccatagtcac atggcctatg cacatatgga ctctgcacat gaatcccacc 120
cacatgtgac acatatgcac cttgtgtctg tacacgtgtc ctgtagttgc gtggactctg 180
cacatgtctt gtacatgtgt agcctgtgca tgtatgtctt ggacactgta caaggtaccc 240
ctttctggct ctcccatttc ctgagaccac agagagaggg gagaagcctg ggattgacag 300
aagcttctgc ccacctactt ttctttcctc agatcatcca gaagttcctc aagggccagg 360
actttatcta atacctctgt gtgctcctcc ttggtgcctg gcctggcaca catcaggagt 420
tcaataaatg tctgttgatg actgttgtac atctctttgc tgtccactct ttgtgggtgg 480
gcagtggggg ttaagaaaat ggtaattagg tcaccctgag ttggggtgaa agatgggatg 540
agtggatgtc tggaggctct gcagacccct tcaaatggga cagtgctcct cacccctccc 600
caaaggattc agggtgactc ctacctggaa tcccttaggg aatgggtgcg tcaaaggacc 660
ttcctcccca ttataaaagg gcaacagcat tttttactga ttcaagggct atatttgacc 720
tcagattttg tttttttaag gctagtcaaa tgaagcggcg ggaatggagg aggaacaaat 780
aaatctgtaa ctatcctcag attttttttt ttttttgaga ctgggtctca ctttttcatc 840
caggctggag tgcagtcgca tgatcacggc tcactgtagc ctcaacctct ccagctcaaa 900
tgctcctcct gtctcagcct cccgagtacc tgggactact ttcttgaggc caggaattca 960
agaacagagt aagatcctgg tctccaaaaa aagttttaaa 1000
<210> 16
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> TALEN TRAC
<400> 16
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
260 265 270
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
290 295 300
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
325 330 335
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
355 360 365
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
565 570 575
Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 17
<211> 942
<212> PRT
<213> artificial sequence
<220>
<223> TALEN TRAC
<400> 17
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Lys Glu Thr Ala
1 5 10 15
Ala Ala Lys Phe Glu Arg Gln His Met Asp Ser Ile Asp Ile Ala Asp
20 25 30
Leu Arg Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro
35 40 45
Lys Val Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His
50 55 60
Gly Phe Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala
65 70 75 80
Leu Gly Thr Val Ala Val Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro
85 90 95
Glu Ala Thr His Glu Ala Ile Val Gly Val Gly Lys Gln Trp Ser Gly
100 105 110
Ala Arg Ala Leu Glu Ala Leu Leu Thr Val Ala Gly Glu Leu Arg Gly
115 120 125
Pro Pro Leu Gln Leu Asp Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg
130 135 140
Gly Gly Val Thr Ala Val Glu Ala Val His Ala Trp Arg Asn Ala Leu
145 150 155 160
Thr Gly Ala Pro Leu Asn Leu Thr Pro Glu Gln Val Val Ala Ile Ala
165 170 175
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
180 185 190
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
195 200 205
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
210 215 220
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
225 230 235 240
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
245 250 255
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
260 265 270
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
275 280 285
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
290 295 300
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
305 310 315 320
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
325 330 335
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
340 345 350
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
355 360 365
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
370 375 380
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
385 390 395 400
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
405 410 415
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
420 425 430
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
435 440 445
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
450 455 460
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
465 470 475 480
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
485 490 495
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
500 505 510
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
515 520 525
Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
530 535 540
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
545 550 555 560
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
565 570 575
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala
580 585 590
Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly
595 600 605
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
610 615 620
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
625 630 635 640
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
645 650 655
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
660 665 670
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
675 680 685
Gly Gly Gly Arg Pro Ala Leu Glu Ser Ile Val Ala Gln Leu Ser Arg
690 695 700
Pro Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu Val Ala Leu
705 710 715 720
Ala Cys Leu Gly Gly Arg Pro Ala Leu Asp Ala Val Lys Lys Gly Leu
725 730 735
Gly Asp Pro Ile Ser Arg Ser Gln Leu Val Lys Ser Glu Leu Glu Glu
740 745 750
Lys Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro His Glu Tyr
755 760 765
Ile Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu
770 775 780
Glu Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly
785 790 795 800
Lys His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val
805 810 815
Gly Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys Ala Tyr Ser
820 825 830
Gly Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr
835 840 845
Val Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro Asn Glu Trp
850 855 860
Trp Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe Leu Phe Val
865 870 875 880
Ser Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn
885 890 895
His Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu Glu Leu Leu
900 905 910
Ile Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu Glu Glu Val
915 920 925
Arg Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Ala Ala Asp
930 935 940
<210> 18
<211> 913
<212> PRT
<213> artificial sequence
<220>
<223> TALEN CD25
<400> 18
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
260 265 270
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
355 360 365
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
565 570 575
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ser Gly Ser
690 695 700
Gly Ser Gly Gly Asp Pro Ile Ser Arg Ser Gln Leu Val Lys Ser Glu
705 710 715 720
Leu Glu Glu Lys Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro
725 730 735
His Glu Tyr Ile Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp
740 745 750
Arg Ile Leu Glu Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly
755 760 765
Tyr Arg Gly Lys His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile
770 775 780
Tyr Thr Val Gly Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys
785 790 795 800
Ala Tyr Ser Gly Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met
805 810 815
Gln Arg Tyr Val Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro
820 825 830
Asn Glu Trp Trp Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe
835 840 845
Leu Phe Val Ser Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr
850 855 860
Arg Leu Asn His Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu
865 870 875 880
Glu Leu Leu Ile Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu
885 890 895
Glu Glu Val Arg Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Ala Ala
900 905 910
Asp
<210> 19
<211> 913
<212> PRT
<213> artificial sequence
<220>
<223> TALEN CD25
<400> 19
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
260 265 270
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
355 360 365
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
565 570 575
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ser Gly Ser
690 695 700
Gly Ser Gly Gly Asp Pro Ile Ser Arg Ser Gln Leu Val Lys Ser Glu
705 710 715 720
Leu Glu Glu Lys Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro
725 730 735
His Glu Tyr Ile Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp
740 745 750
Arg Ile Leu Glu Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly
755 760 765
Tyr Arg Gly Lys His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile
770 775 780
Tyr Thr Val Gly Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys
785 790 795 800
Ala Tyr Ser Gly Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met
805 810 815
Gln Arg Tyr Val Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro
820 825 830
Asn Glu Trp Trp Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe
835 840 845
Leu Phe Val Ser Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr
850 855 860
Arg Leu Asn His Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu
865 870 875 880
Glu Leu Leu Ile Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu
885 890 895
Glu Glu Val Arg Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Ala Ala
900 905 910
Asp
<210> 20
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> TALEN PD1
<400> 20
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Lys Leu Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
225 230 235 240
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
260 265 270
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
290 295 300
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
355 360 365
Gln Val Val Ala Ile Ala Ser Tyr Lys Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
565 570 575
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
595 600 605
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 21
<211> 941
<212> PRT
<213> artificial sequence
<220>
<223> TALEN PD1
<400> 21
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Lys Glu Thr Ala
1 5 10 15
Ala Ala Lys Phe Glu Arg Gln His Met Asp Ser Ile Asp Ile Ala Asp
20 25 30
Leu Arg Thr Leu Gly Tyr Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro
35 40 45
Lys Val Arg Ser Thr Val Ala Gln His His Glu Ala Leu Val Gly His
50 55 60
Gly Phe Thr His Ala His Ile Val Ala Leu Ser Gln His Pro Ala Ala
65 70 75 80
Leu Gly Thr Val Ala Val Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro
85 90 95
Glu Ala Thr His Glu Ala Ile Val Gly Val Gly Lys Gln Trp Ser Gly
100 105 110
Ala Arg Ala Leu Glu Ala Leu Leu Thr Val Ala Gly Glu Leu Arg Gly
115 120 125
Pro Pro Leu Gln Leu Asp Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg
130 135 140
Gly Gly Val Thr Ala Val Glu Ala Val His Ala Trp Arg Asn Ala Leu
145 150 155 160
Thr Gly Ala Pro Leu Asn Leu Thr Pro Glu Gln Val Val Ala Ile Ala
165 170 175
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
180 185 190
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
195 200 205
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
210 215 220
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
225 230 235 240
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
245 250 255
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
260 265 270
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
275 280 285
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
290 295 300
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala
305 310 315 320
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
325 330 335
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
340 345 350
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
355 360 365
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
370 375 380
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
385 390 395 400
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
405 410 415
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
420 425 430
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
435 440 445
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
450 455 460
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
465 470 475 480
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
485 490 495
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
500 505 510
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
515 520 525
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
530 535 540
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
545 550 555 560
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
565 570 575
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Gly Gly Lys Gln Ala Leu
580 585 590
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu
595 600 605
Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln
610 615 620
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His
625 630 635 640
Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly
645 650 655
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
660 665 670
Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly
675 680 685
Gly Gly Arg Pro Ala Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro
690 695 700
Asp Pro Ala Leu Ala Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala
705 710 715 720
Cys Leu Gly Gly Arg Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly
725 730 735
Asp Pro Ile Ser Arg Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys
740 745 750
Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile
755 760 765
Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu
770 775 780
Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys
785 790 795 800
His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly
805 810 815
Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly
820 825 830
Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val
835 840 845
Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp
850 855 860
Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser
865 870 875 880
Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His
885 890 895
Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile
900 905 910
Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg
915 920 925
Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Ala Ala Asp
930 935 940
<210> 22
<211> 2814
<212> DNA
<213> artificial sequence
<220>
<223> TALEN TRAC pCLS11370
<400> 22
atgggcgatc ctaaaaagaa acgtaaggtc atcgattacc catacgatgt tccagattac 60
gctatcgata tcgccgatct acgcacgctc ggctacagcc agcagcaaca ggagaagatc 120
aaaccgaagg ttcgttcgac agtggcgcag caccacgagg cactggtcgg ccacgggttt 180
acacacgcgc acatcgttgc gttaagccaa cacccggcag cgttagggac cgtcgctgtc 240
aagtatcagg acatgatcgc agcgttgcca gaggcgacac acgaagcgat cgttggcgtc 300
ggcaaacagt ggtccggcgc acgcgctctg gaggccttgc tcacggtggc gggagagttg 360
agaggtccac cgttacagtt ggacacaggc caacttctca agattgcaaa acgtggcggc 420
gtgaccgcag tggaggcagt gcatgcatgg cgcaatgcac tgacgggtgc cccgctcaac 480
ttgacccccc agcaggtggt ggccatcgcc agcaatggcg gtggcaagca ggcgctggag 540
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 600
gtggccatcg ccagcaataa tggtggcaag caggcgctgg agacggtcca gcggctgttg 660
ccggtgctgt gccaggccca cggcttgacc ccccagcagg tggtggccat cgccagcaat 720
ggcggtggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 780
cacggcttga ccccggagca ggtggtggcc atcgccagcc acgatggcgg caagcaggcg 840
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 900
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 960
ctgttgccgg tgctgtgcca ggcccacggc ttgaccccgg agcaggtggt ggccatcgcc 1020
agccacgatg gcggcaagca ggcgctggag acggtccagc ggctgttgcc ggtgctgtgc 1080
caggcccacg gcttgacccc ggagcaggtg gtggccatcg ccagcaatat tggtggcaag 1140
caggcgctgg agacggtgca ggcgctgttg ccggtgctgt gccaggccca cggcttgacc 1200
ccggagcagg tggtggccat cgccagccac gatggcggca agcaggcgct ggagacggtc 1260
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccggagca ggtggtggcc 1320
atcgccagca atattggtgg caagcaggcg ctggagacgg tgcaggcgct gttgccggtg 1380
ctgtgccagg cccacggctt gaccccccag caggtggtgg ccatcgccag caataatggt 1440
ggcaagcagg cgctggagac ggtccagcgg ctgttgccgg tgctgtgcca ggcccacggc 1500
ttgaccccgg agcaggtggt ggccatcgcc agcaatattg gtggcaagca ggcgctggag 1560
acggtgcagg cgctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 1620
gtggccatcg ccagcaatgg cggtggcaag caggcgctgg agacggtcca gcggctgttg 1680
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagcaat 1740
attggtggca agcaggcgct ggagacggtg caggcgctgt tgccggtgct gtgccaggcc 1800
cacggcttga ccccccagca ggtggtggcc atcgccagca atggcggtgg caagcaggcg 1860
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1920
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 1980
ctgttgccgg tgctgtgcca ggcccacggc ttgacccctc agcaggtggt ggccatcgcc 2040
agcaatggcg gcggcaggcc ggcgctggag agcattgttg cccagttatc tcgccctgat 2100
ccggcgttgg ccgcgttgac caacgaccac ctcgtcgcct tggcctgcct cggcgggcgt 2160
cctgcgctgg atgcagtgaa aaagggattg ggggatccta tcagccgttc ccagctggtg 2220
aagtccgagc tggaggagaa gaaatccgag ttgaggcaca agctgaagta cgtgccccac 2280
gagtacatcg agctgatcga gatcgcccgg aacagcaccc aggaccgtat cctggagatg 2340
aaggtgatgg agttcttcat gaaggtgtac ggctacaggg gcaagcacct gggcggctcc 2400
aggaagcccg acggcgccat ctacaccgtg ggctccccca tcgactacgg cgtgatcgtg 2460
gacaccaagg cctactccgg cggctacaac ctgcccatcg gccaggccga cgaaatgcag 2520
aggtacgtgg aggagaacca gaccaggaac aagcacatca accccaacga gtggtggaag 2580
gtgtacccct ccagcgtgac cgagttcaag ttcctgttcg tgtccggcca cttcaagggc 2640
aactacaagg cccagctgac caggctgaac cacatcacca actgcaacgg cgccgtgctg 2700
tccgtggagg agctcctgat cggcggcgag atgatcaagg ccggcaccct gaccctggag 2760
gaggtgagga ggaagttcaa caacggcgag atcaacttcg cggccgactg ataa 2814
<210> 23
<211> 2832
<212> DNA
<213> artificial sequence
<220>
<223> TALEN TRAC pCLS11369
<400> 23
atgggcgatc ctaaaaagaa acgtaaggtc atcgataagg agaccgccgc tgccaagttc 60
gagagacagc acatggacag catcgatatc gccgatctac gcacgctcgg ctacagccag 120
cagcaacagg agaagatcaa accgaaggtt cgttcgacag tggcgcagca ccacgaggca 180
ctggtcggcc acgggtttac acacgcgcac atcgttgcgt taagccaaca cccggcagcg 240
ttagggaccg tcgctgtcaa gtatcaggac atgatcgcag cgttgccaga ggcgacacac 300
gaagcgatcg ttggcgtcgg caaacagtgg tccggcgcac gcgctctgga ggccttgctc 360
acggtggcgg gagagttgag aggtccaccg ttacagttgg acacaggcca acttctcaag 420
attgcaaaac gtggcggcgt gaccgcagtg gaggcagtgc atgcatggcg caatgcactg 480
acgggtgccc cgctcaactt gaccccggag caggtggtgg ccatcgccag ccacgatggc 540
ggcaagcagg cgctggagac ggtccagcgg ctgttgccgg tgctgtgcca ggcccacggc 600
ttgacccccc agcaggtggt ggccatcgcc agcaatggcg gtggcaagca ggcgctggag 660
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 720
gtggccatcg ccagccacga tggcggcaag caggcgctgg agacggtcca gcggctgttg 780
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagcaat 840
attggtggca agcaggcgct ggagacggtg caggcgctgt tgccggtgct gtgccaggcc 900
cacggcttga ccccccagca ggtggtggcc atcgccagca ataatggtgg caagcaggcg 960
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1020
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 1080
ctgttgccgg tgctgtgcca ggcccacggc ttgacccccc agcaggtggt ggccatcgcc 1140
agcaatggcg gtggcaagca ggcgctggag acggtccagc ggctgttgcc ggtgctgtgc 1200
caggcccacg gcttgacccc ccagcaggtg gtggccatcg ccagcaataa tggtggcaag 1260
caggcgctgg agacggtcca gcggctgttg ccggtgctgt gccaggccca cggcttgacc 1320
ccccagcagg tggtggccat cgccagcaat aatggtggca agcaggcgct ggagacggtc 1380
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccccagca ggtggtggcc 1440
atcgccagca atggcggtgg caagcaggcg ctggagacgg tccagcggct gttgccggtg 1500
ctgtgccagg cccacggctt gaccccggag caggtggtgg ccatcgccag caatattggt 1560
ggcaagcagg cgctggagac ggtgcaggcg ctgttgccgg tgctgtgcca ggcccacggc 1620
ttgaccccgg agcaggtggt ggccatcgcc agccacgatg gcggcaagca ggcgctggag 1680
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 1740
gtggccatcg ccagcaatat tggtggcaag caggcgctgg agacggtgca ggcgctgttg 1800
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagccac 1860
gatggcggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 1920
cacggcttga ccccccagca ggtggtggcc atcgccagca ataatggtgg caagcaggcg 1980
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gacccctcag 2040
caggtggtgg ccatcgccag caatggcggc ggcaggccgg cgctggagag cattgttgcc 2100
cagttatctc gccctgatcc ggcgttggcc gcgttgacca acgaccacct cgtcgccttg 2160
gcctgcctcg gcgggcgtcc tgcgctggat gcagtgaaaa agggattggg ggatcctatc 2220
agccgttccc agctggtgaa gtccgagctg gaggagaaga aatccgagtt gaggcacaag 2280
ctgaagtacg tgccccacga gtacatcgag ctgatcgaga tcgcccggaa cagcacccag 2340
gaccgtatcc tggagatgaa ggtgatggag ttcttcatga aggtgtacgg ctacaggggc 2400
aagcacctgg gcggctccag gaagcccgac ggcgccatct acaccgtggg ctcccccatc 2460
gactacggcg tgatcgtgga caccaaggcc tactccggcg gctacaacct gcccatcggc 2520
caggccgacg aaatgcagag gtacgtggag gagaaccaga ccaggaacaa gcacatcaac 2580
cccaacgagt ggtggaaggt gtacccctcc agcgtgaccg agttcaagtt cctgttcgtg 2640
tccggccact tcaagggcaa ctacaaggcc cagctgacca ggctgaacca catcaccaac 2700
tgcaacggcg ccgtgctgtc cgtggaggag ctcctgatcg gcggcgagat gatcaaggcc 2760
ggcaccctga ccctggagga ggtgaggagg aagttcaaca acggcgagat caacttcgcg 2820
gccgactgat aa 2832
<210> 24
<211> 2745
<212> DNA
<213> artificial sequence
<220>
<223> TALEN CD25 pCLS30480
<400> 24
atgggcgatc ctaaaaagaa acgtaaggtc atcgattacc catacgatgt tccagattac 60
gctatcgata tcgccgatct acgcacgctc ggctacagcc agcagcaaca ggagaagatc 120
aaaccgaagg ttcgttcgac agtggcgcag caccacgagg cactggtcgg ccacgggttt 180
acacacgcgc acatcgttgc gttaagccaa cacccggcag cgttagggac cgtcgctgtc 240
aagtatcagg acatgatcgc agcgttgcca gaggcgacac acgaagcgat cgttggcgtc 300
ggcaaacagt ggtccggcgc acgcgctctg gaggccttgc tcacggtggc gggagagttg 360
agaggtccac cgttacagtt ggacacaggc caacttctca agattgcaaa acgtggcggc 420
gtgaccgcag tggaggcagt gcatgcatgg cgcaatgcac tgacgggtgc cccgctcaac 480
ttgacccccc agcaggtggt ggccatcgcc agcaataatg gtggcaagca ggcgctggag 540
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 600
gtggccatcg ccagcaatgg cggtggcaag caggcgctgg agacggtcca gcggctgttg 660
ccggtgctgt gccaggccca cggcttgacc ccccagcagg tggtggccat cgccagcaat 720
ggcggtggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 780
cacggcttga ccccggagca ggtggtggcc atcgccagcc acgatggcgg caagcaggcg 840
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccccag 900
caggtggtgg ccatcgccag caatggcggt ggcaagcagg cgctggagac ggtccagcgg 960
ctgttgccgg tgctgtgcca ggcccacggc ttgacccccc agcaggtggt ggccatcgcc 1020
agcaatggcg gtggcaagca ggcgctggag acggtccagc ggctgttgcc ggtgctgtgc 1080
caggcccacg gcttgacccc ccagcaggtg gtggccatcg ccagcaatgg cggtggcaag 1140
caggcgctgg agacggtcca gcggctgttg ccggtgctgt gccaggccca cggcttgacc 1200
ccccagcagg tggtggccat cgccagcaat ggcggtggca agcaggcgct ggagacggtc 1260
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccccagca ggtggtggcc 1320
atcgccagca ataatggtgg caagcaggcg ctggagacgg tccagcggct gttgccggtg 1380
ctgtgccagg cccacggctt gaccccccag caggtggtgg ccatcgccag caataatggt 1440
ggcaagcagg cgctggagac ggtccagcgg ctgttgccgg tgctgtgcca ggcccacggc 1500
ttgacccccc agcaggtggt ggccatcgcc agcaatggcg gtggcaagca ggcgctggag 1560
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ccagcaggtg 1620
gtggccatcg ccagcaatgg cggtggcaag caggcgctgg agacggtcca gcggctgttg 1680
ccggtgctgt gccaggccca cggcttgacc ccccagcagg tggtggccat cgccagcaat 1740
ggcggtggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 1800
cacggcttga ccccccagca ggtggtggcc atcgccagca atggcggtgg caagcaggcg 1860
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1920
caggtggtgg ccatcgccag ccacgatggc ggcaagcagg cgctggagac ggtccagcgg 1980
ctgttgccgg tgctgtgcca ggcccacggc ttgacccctc agcaggtggt ggccatcgcc 2040
agcaatggcg gcggcaggcc ggcgctggag agcattgttg cccagttatc tcgccctgat 2100
ccgagtggca gcggaagtgg cggggatcct atcagccgtt cccagctggt gaagtccgag 2160
ctggaggaga agaaatccga gttgaggcac aagctgaagt acgtgcccca cgagtacatc 2220
gagctgatcg agatcgcccg gaacagcacc caggaccgta tcctggagat gaaggtgatg 2280
gagttcttca tgaaggtgta cggctacagg ggcaagcacc tgggcggctc caggaagccc 2340
gacggcgcca tctacaccgt gggctccccc atcgactacg gcgtgatcgt ggacaccaag 2400
gcctactccg gcggctacaa cctgcccatc ggccaggccg acgaaatgca gaggtacgtg 2460
gaggagaacc agaccaggaa caagcacatc aaccccaacg agtggtggaa ggtgtacccc 2520
tccagcgtga ccgagttcaa gttcctgttc gtgtccggcc acttcaaggg caactacaag 2580
gcccagctga ccaggctgaa ccacatcacc aactgcaacg gcgccgtgct gtccgtggag 2640
gagctcctga tcggcggcga gatgatcaag gccggcaccc tgaccctgga ggaggtgagg 2700
aggaagttca acaacggcga gatcaacttc gcggccgact gataa 2745
<210> 25
<211> 2745
<212> DNA
<213> artificial sequence
<220>
<223> TALEN CD25 pCLS30479
<400> 25
atgggcgatc ctaaaaagaa acgtaaggtc atcgattacc catacgatgt tccagattac 60
gctatcgata tcgccgatct acgcacgctc ggctacagcc agcagcaaca ggagaagatc 120
aaaccgaagg ttcgttcgac agtggcgcag caccacgagg cactggtcgg ccacgggttt 180
acacacgcgc acatcgttgc gttaagccaa cacccggcag cgttagggac cgtcgctgtc 240
aagtatcagg acatgatcgc agcgttgcca gaggcgacac acgaagcgat cgttggcgtc 300
ggcaaacagt ggtccggcgc acgcgctctg gaggccttgc tcacggtggc gggagagttg 360
agaggtccac cgttacagtt ggacacaggc caacttctca agattgcaaa acgtggcggc 420
gtgaccgcag tggaggcagt gcatgcatgg cgcaatgcac tgacgggtgc cccgctcaac 480
ttgaccccgg agcaggtggt ggccatcgcc agcaatattg gtggcaagca ggcgctggag 540
acggtgcagg cgctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 600
gtggccatcg ccagccacga tggcggcaag caggcgctgg agacggtcca gcggctgttg 660
ccggtgctgt gccaggccca cggcttgacc ccggagcagg tggtggccat cgccagcaat 720
attggtggca agcaggcgct ggagacggtg caggcgctgt tgccggtgct gtgccaggcc 780
cacggcttga ccccccagca ggtggtggcc atcgccagca ataatggtgg caagcaggcg 840
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccccag 900
caggtggtgg ccatcgccag caataatggt ggcaagcagg cgctggagac ggtccagcgg 960
ctgttgccgg tgctgtgcca ggcccacggc ttgaccccgg agcaggtggt ggccatcgcc 1020
agcaatattg gtggcaagca ggcgctggag acggtgcagg cgctgttgcc ggtgctgtgc 1080
caggcccacg gcttgacccc ccagcaggtg gtggccatcg ccagcaataa tggtggcaag 1140
caggcgctgg agacggtcca gcggctgttg ccggtgctgt gccaggccca cggcttgacc 1200
ccccagcagg tggtggccat cgccagcaat aatggtggca agcaggcgct ggagacggtc 1260
cagcggctgt tgccggtgct gtgccaggcc cacggcttga ccccggagca ggtggtggcc 1320
atcgccagca atattggtgg caagcaggcg ctggagacgg tgcaggcgct gttgccggtg 1380
ctgtgccagg cccacggctt gaccccggag caggtggtgg ccatcgccag caatattggt 1440
ggcaagcagg cgctggagac ggtgcaggcg ctgttgccgg tgctgtgcca ggcccacggc 1500
ttgacccccc agcaggtggt ggccatcgcc agcaataatg gtggcaagca ggcgctggag 1560
acggtccagc ggctgttgcc ggtgctgtgc caggcccacg gcttgacccc ggagcaggtg 1620
gtggccatcg ccagcaatat tggtggcaag caggcgctgg agacggtgca ggcgctgttg 1680
ccggtgctgt gccaggccca cggcttgacc ccccagcagg tggtggccat cgccagcaat 1740
aatggtggca agcaggcgct ggagacggtc cagcggctgt tgccggtgct gtgccaggcc 1800
cacggcttga ccccccagca ggtggtggcc atcgccagca atggcggtgg caagcaggcg 1860
ctggagacgg tccagcggct gttgccggtg ctgtgccagg cccacggctt gaccccggag 1920
caggtggtgg ccatcgccag caatattggt ggcaagcagg cgctggagac ggtgcaggcg 1980
ctgttgccgg tgctgtgcca ggcccacggc ttgacccctc agcaggtggt ggccatcgcc 2040
agcaatggcg gcggcaggcc ggcgctggag agcattgttg cccagttatc tcgccctgat 2100
ccgagtggca gcggaagtgg cggggatcct atcagccgtt cccagctggt gaagtccgag 2160
ctggaggaga agaaatccga gttgaggcac aagctgaagt acgtgcccca cgagtacatc 2220
gagctgatcg agatcgcccg gaacagcacc caggaccgta tcctggagat gaaggtgatg 2280
gagttcttca tgaaggtgta cggctacagg ggcaagcacc tgggcggctc caggaagccc 2340
gacggcgcca tctacaccgt gggctccccc atcgactacg gcgtgatcgt ggacaccaag 2400
gcctactccg gcggctacaa cctgcccatc ggccaggccg acgaaatgca gaggtacgtg 2460
gaggagaacc agaccaggaa caagcacatc aaccccaacg agtggtggaa ggtgtacccc 2520
tccagcgtga ccgagttcaa gttcctgttc gtgtccggcc acttcaaggg caactacaag 2580
gcccagctga ccaggctgaa ccacatcacc aactgcaacg gcgccgtgct gtccgtggag 2640
gagctcctga tcggcggcga gatgatcaag gccggcaccc tgaccctgga ggaggtgagg 2700
aggaagttca acaacggcga gatcaacttc gcggccgact gataa 2745
<210> 26
<211> 2814
<212> DNA
<213> artificial sequence
<220>
<223> TALEN PD1 pCLS28959
<400> 26
atgggcgatc ctaaaaagaa acgtaaggtc atcgattacc catacgatgt tccagattac 60
gctatcgata tcgccgatct acgcacgctc ggctacagcc agcagcaaca ggagaagatc 120
aaaccgaagg ttcgttcgac agtggcgcag caccacgagg cactggtcgg ccacgggttt 180
acacacgcgc acatcgttgc gttaagccaa cacccggcag cgttagggac cgtcgctgtc 240
aagtatcagg acatgatcgc agcgttgcca gaggcgacac acgaagcgat cgttggcgtc 300
ggcaaacagt ggtccggcgc acgcgctctg gaggccttgc tcacggtggc gggagagttg 360
agaggtccac cgttacagtt ggacacaggc caacttctca agattgcaaa acgtggcggc 420
gtgaccgcag tggaggcagt gcatgcatgg cgcaatgcac tgacgggtgc cccgctcaac 480
ttgacccccg agcaagtggt ggctatcgct tccaagctgg ggggaaagca ggccctggag 540
accgtccagg cccttctccc agtgctttgc caggctcacg gactgacccc tgaacaggtg 600
gtggcaattg cctcacacga cgggggcaag caggcactgg agactgtcca gcggctgctg 660
cctgtcctct gccaggccca cggactcact cctgagcagg tcgtggccat tgccagccac 720
gatgggggca aacaggctct ggagaccgtg cagcgcctcc tcccagtgct gtgccaggct 780
catgggctga ccccacagca ggtcgtcgcc attgccagta acggcggggg gaagcaggcc 840
ctcgaaacag tgcagaggct gctgcccgtc ttgtgccaag cacacggcct gacacccgag 900
caggtggtgg ccatcgcctc tcatgacggc ggcaagcagg cccttgagac agtgcagaga 960
ctgttgcccg tgttgtgtca ggcccacggg ttgacacccc agcaggtggt cgccatcgcc 1020
agcaatggcg ggggaaagca ggcccttgag accgtgcagc ggttgcttcc agtgttgtgc 1080
caggcacacg gactgacccc tcaacaggtg gtcgcaatcg ccagctacaa gggcggaaag 1140
caggctctgg agacagtgca gcgcctcctg cccgtgctgt gtcaggctca cggactgaca 1200
ccacagcagg tggtcgccat cgccagtaac gggggcggca agcaggcttt ggagaccgtc 1260
cagagactcc tccccgtcct ttgccaggcc cacgggttga cacctcagca ggtcgtcgcc 1320
attgcctcca acaacggggg caagcaggcc ctcgaaactg tgcagaggct gctgcctgtg 1380
ctgtgccagg ctcatgggct gacaccccag caggtggtgg ccattgcctc taacaacggc 1440
ggcaaacagg cactggagac cgtgcaaagg ctgctgcccg tcctctgcca agcccacggg 1500
ctcactccac agcaggtcgt ggccatcgcc tcaaacaatg gcgggaagca ggccctggag 1560
actgtgcaaa ggctgctccc tgtgctctgc caggcacacg gactgacccc tcagcaggtg 1620
gtggcaatcg cttccaacaa cgggggaaag caggccctcg aaaccgtgca gcgcctcctc 1680
ccagtgctgt gccaggcaca tggcctcaca cccgagcaag tggtggctat cgccagccac 1740
gacggaggga agcaggctct ggagaccgtg cagaggctgc tgcctgtcct gtgccaggcc 1800
cacgggctta ctccagagca ggtcgtcgcc atcgccagtc atgatggggg gaagcaggcc 1860
cttgagacag tccagcggct gctgccagtc ctttgccagg ctcacggctt gactcccgag 1920
caggtcgtgg ccattgcctc aaacattggg ggcaaacagg ccctggagac agtgcaggcc 1980
ctgctgcccg tgttgtgtca ggcccacggc ttgacacccc agcaggtggt cgccattgcc 2040
tctaatggcg gcgggagacc cgccttggag agcattgttg cccagttatc tcgccctgat 2100
ccggcgttgg ccgcgttgac caacgaccac ctcgtcgcct tggcctgcct cggcgggcgt 2160
cctgcgctgg atgcagtgaa aaagggattg ggggatccta tcagccgttc ccagctggtg 2220
aagtccgagc tggaggagaa gaaatccgag ttgaggcaca agctgaagta cgtgccccac 2280
gagtacatcg agctgatcga gatcgcccgg aacagcaccc aggaccgtat cctggagatg 2340
aaggtgatgg agttcttcat gaaggtgtac ggctacaggg gcaagcacct gggcggctcc 2400
aggaagcccg acggcgccat ctacaccgtg ggctccccca tcgactacgg cgtgatcgtg 2460
gacaccaagg cctactccgg cggctacaac ctgcccatcg gccaggccga cgaaatgcag 2520
aggtacgtgg aggagaacca gaccaggaac aagcacatca accccaacga gtggtggaag 2580
gtgtacccct ccagcgtgac cgagttcaag ttcctgttcg tgtccggcca cttcaagggc 2640
aactacaagg cccagctgac caggctgaac cacatcacca actgcaacgg cgccgtgctg 2700
tccgtggagg agctcctgat cggcggcgag atgatcaagg ccggcaccct gaccctggag 2760
gaggtgagga ggaagttcaa caacggcgag atcaacttcg cggccgactg ataa 2814
<210> 27
<211> 2829
<212> DNA
<213> artificial sequence
<220>
<223> TALEN PD1 pCLS18792
<400> 27
atgggcgatc ctaaaaagaa acgtaaggtc atcgataagg agaccgccgc tgccaagttc 60
gagagacagc acatggacag catcgatatc gccgatctac gcacgctcgg ctacagccag 120
cagcaacagg agaagatcaa accgaaggtt cgttcgacag tggcgcagca ccacgaggca 180
ctggtcggcc acgggtttac acacgcgcac atcgttgcgt taagccaaca cccggcagcg 240
ttagggaccg tcgctgtcaa gtatcaggac atgatcgcag cgttgccaga ggcgacacac 300
gaagcgatcg ttggcgtcgg caaacagtgg tccggcgcac gcgctctgga ggccttgctc 360
acggtggcgg gagagttgag aggtccaccg ttacagttgg acacaggcca acttctcaag 420
attgcaaaac gtggcggcgt gaccgcagtg gaggcagtgc atgcatggcg caatgcactg 480
acgggtgccc cgctcaactt gacccccgag caagtcgtcg caatcgccag ccatgatgga 540
gggaagcaag ccctcgaaac cgtgcagcgg ttgcttcctg tgctctgcca ggcccacggc 600
cttacccctc agcaggtggt ggccatcgca agtaacggag gaggaaagca agccttggag 660
acagtgcagc gcctgttgcc cgtgctgtgc caggcacacg gcctcacacc agagcaggtc 720
gtggccattg cctcccatga cggggggaaa caggctctgg agaccgtcca gaggctgctg 780
cccgtcctct gtcaagctca cggcctgact ccccaacaag tggtcgccat cgcctctaat 840
ggcggcggga agcaggcact ggaaacagtg cagagactgc tccctgtgct ttgccaagct 900
catgggttga ccccccaaca ggtcgtcgct attgcctcaa acgggggggg caagcaggcc 960
cttgagactg tgcagaggct gttgccagtg ctgtgtcagg ctcacgggct cactccacaa 1020
caggtggtcg caattgccag caacggcggc ggaaagcaag ctcttgaaac cgtgcaacgc 1080
ctcctgcccg tgctctgtca ggctcatggc ctgacaccac aacaagtcgt ggccatcgcc 1140
agtaataatg gcgggaaaca ggctcttgag accgtccaga ggctgctccc agtgctctgc 1200
caggcacacg ggctgacccc cgagcaggtg gtggctatcg ccagcaatat tgggggcaag 1260
caggccctgg aaacagtcca ggccctgctg ccagtgcttt gccaggctca cgggctcact 1320
ccccagcagg tcgtggcaat cgcctccaac ggcggaggga agcaggctct ggagaccgtg 1380
cagagactgc tgcccgtctt gtgccaggcc cacggactca cacctgaaca ggtcgtcgcc 1440
attgcctctc acgatggggg caaacaagcc ctggagacag tgcagcggct gttgcctgtg 1500
ttgtgccaag cccacggctt gactcctcaa caagtggtcg ccatcgcctc aaatggcggc 1560
ggaaaacaag ctctggagac agtgcagagg ttgctgcccg tcctctgcca agcccacggc 1620
ctgactcccc aacaggtcgt cgccattgcc agcaacaacg gaggaaagca ggctctcgaa 1680
actgtgcagc ggctgcttcc tgtgctgtgt caggctcatg ggctgacccc cgagcaagtg 1740
gtggctattg cctctaatgg aggcaagcaa gcccttgaga cagtccagag gctgttgcca 1800
gtgctgtgcc aggcccacgg gctcacaccc cagcaggtgg tcgccatcgc cagtaacaac 1860
gggggcaaac aggcattgga aaccgtccag cgcctgcttc cagtgctctg ccaggcacac 1920
ggactgacac ccgaacaggt ggtggccatt gcatcccatg atgggggcaa gcaggccctg 1980
gagaccgtgc agagactcct gccagtgttg tgccaagctc acggcctcac ccctcagcaa 2040
gtcgtggcca tcgcctcaaa cggggggggc cggcctgcac tggagagcat tgttgcccag 2100
ttatctcgcc ctgatccggc gttggccgcg ttgaccaacg accacctcgt cgccttggcc 2160
tgcctcggcg ggcgtcctgc gctggatgca gtgaaaaagg gattggggga tcctatcagc 2220
cgttcccagc tggtgaagtc cgagctggag gagaagaaat ccgagttgag gcacaagctg 2280
aagtacgtgc cccacgagta catcgagctg atcgagatcg cccggaacag cacccaggac 2340
cgtatcctgg agatgaaggt gatggagttc ttcatgaagg tgtacggcta caggggcaag 2400
cacctgggcg gctccaggaa gcccgacggc gccatctaca ccgtgggctc ccccatcgac 2460
tacggcgtga tcgtggacac caaggcctac tccggcggct acaacctgcc catcggccag 2520
gccgacgaaa tgcagaggta cgtggaggag aaccagacca ggaacaagca catcaacccc 2580
aacgagtggt ggaaggtgta cccctccagc gtgaccgagt tcaagttcct gttcgtgtcc 2640
ggccacttca agggcaacta caaggcccag ctgaccaggc tgaaccacat caccaactgc 2700
aacggcgccg tgctgtccgt ggaggagctc ctgatcggcg gcgagatgat caaggccggc 2760
accctgaccc tggaggaggt gaggaggaag ttcaacaacg gcgagatcaa cttcgcggcc 2820
gactgataa 2829
<210> 28
<211> 49
<212> DNA
<213> artificial sequence
<220>
<223> TALEN target TRAC
<400> 28
ttgtcccaca gatatccaga accctgaccc tgccgtgtac cagctgaga 49
<210> 29
<211> 45
<212> DNA
<213> artificial sequence
<220>
<223> TALEN target CD25
<400> 29
tacaggagga agagtagaag aacaatctag aaaaccaaaa gaaca 45
<210> 30
<211> 49
<212> DNA
<213> artificial sequence
<220>
<223> TALEN target PD1
<400> 30
tacctctgtg gggccatctc cctggccccc aaggcgcaga tcaaagaga 49
<210> 31
<211> 2897
<212> DNA
<213> artificial sequence
<220>
<223> Matrice TRAC locus_CubiCAR CD22 pCLS30056
<400> 31
ttgctgggcc tttttcccat gcctgccttt actctgccag agttatattg ctggggtttt 60
gaagaagatc ctattaaata aaagaataag cagtattatt aagtagccct gcatttcagg 120
tttccttgag tggcaggcca ggcctggccg tgaacgttca ctgaaatcat ggcctcttgg 180
ccaagattga tagcttgtgc ctgtccctga gtcccagtcc atcacgagca gctggtttct 240
aagatgctat ttcccgtata aagcatgaga ccgtgacttg ccagccccac agagccccgc 300
ccttgtccat cactggcatc tggactccag cctgggttgg ggcaaagagg gaaatgagat 360
catgtcctaa ccctgatcct cttgtcccac agatatccag tacccctacg acgtgcccga 420
ctacgcctcc ggtgagggca gaggaagtct tctaacatgc ggtgacgtgg aggagaatcc 480
gggccccgga tccgctctgc ccgtcaccgc tctgctgctg ccactggcac tgctgctgca 540
cgctgctagg cccggagggg gaggcagctg cccctacagc aaccccagcc tgtgcagcgg 600
aggcggcggc agcggcggag ggggtagcca ggtgcagctg cagcagagcg gccctggcct 660
ggtgaagcca agccagacac tgtccctgac ctgcgccatc agcggcgatt ccgtgagctc 720
caactccgcc gcctggaatt ggatcaggca gtccccttct cggggcctgg agtggctggg 780
aaggacatac tatcggtcta agtggtacaa cgattatgcc gtgtctgtga agagcagaat 840
cacaatcaac cctgacacct ccaagaatca gttctctctg cagctgaata gcgtgacacc 900
agaggacacc gccgtgtact attgcgccag ggaggtgacc ggcgacctgg aggatgcctt 960
tgacatctgg ggccagggca caatggtgac cgtgagctcc ggaggcggcg gatctggcgg 1020
aggaggaagt gggggcggcg ggagtgatat ccagatgaca cagtccccat cctctctgag 1080
cgcctccgtg ggcgacagag tgacaatcac ctgtagggcc tcccagacca tctggtctta 1140
cctgaactgg tatcagcaga ggcccggcaa ggcccctaat ctgctgatct acgcagcaag 1200
ctccctgcag agcggagtgc catccagatt ctctggcagg ggctccggca cagacttcac 1260
cctgaccatc tctagcctgc aggccgagga cttcgccacc tactattgcc agcagtctta 1320
tagcatcccc cagacatttg gccagggcac caagctggag atcaagtcgg atcccggaag 1380
cggaggggga ggcagctgcc cctacagcaa ccccagcctg tgcagcggag gcggcggcag 1440
cgagctgccc acccagggca ccttctccaa cgtgtccacc aacgtgagcc cagccaagcc 1500
caccaccacc gcctgtcctt attccaatcc ttccctgtgt gctcccacca caacccccgc 1560
tccaaggccc cctacccccg caccaactat tgcctcccag ccactctcac tgcggcctga 1620
ggcctgtcgg cccgctgctg gaggcgcagt gcatacaagg ggcctcgatt tcgcctgcga 1680
tatttacatc tgggcacccc tcgccggcac ctgcggggtg cttctcctct ccctggtgat 1740
taccctgtat tgcagacggg gccggaagaa gctcctctac atttttaagc agcctttcat 1800
gcggccagtg cagacaaccc aagaggagga tgggtgttcc tgcagattcc ctgaggaaga 1860
ggaaggcggg tgcgagctga gagtgaagtt ctccaggagc gcagatgccc ccgcctatca 1920
acagggccag aaccagctct acaacgagct taacctcggg aggcgcgaag aatacgacgt 1980
gttggataag agaagggggc gggaccccga gatgggagga aagccccgga ggaagaaccc 2040
tcaggagggc ctgtacaacg agctgcagaa ggataagatg gccgaggcct actcagagat 2100
cgggatgaag ggggagcggc gccgcgggaa ggggcacgat gggctctacc aggggctgag 2160
cacagccaca aaggacacat acgacgcctt gcacatgcag gcccttccac cccgggaata 2220
gtctagaggg cccgtttaaa cccgctgatc agcctcgact gtgccttcta gttgccagcc 2280
atctgttgtt tgcccctccc ccgtgccttc cttgaccctg gaaggtgcca ctcccactgt 2340
cctttcctaa taaaatgagg aaattgcatc gcattgtctg agtaggtgtc attctattct 2400
ggggggtggg gtggggcagg acagcaaggg ggaggattgg gaagacaata gcaggcatgc 2460
tggggatgcg gtgggctcta tgactagtgg cgaattcccg tgtaccagct gagagactct 2520
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 2580
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 2640
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2700
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccagg taagggcagc 2760
tttggtgcct tcgcaggctg tttccttgct tcaggaatgg ccaggttctg cccagagctc 2820
tggtcaatga tgtctaaaac tcctctgatt ggtggtctcg gccttatcca ttgccaccaa 2880
aaccctcttt ttactaa 2897
<210> 32
<211> 2688
<212> DNA
<213> artificial sequence
<220>
<223> Matrice CD25 locus_IL15_2A_sIL15Ra pCLS30519
<400> 32
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 60
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 120
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 180
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 240
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 420
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 480
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 540
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 600
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 660
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 720
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 780
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 840
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 900
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 960
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1020
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1080
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1140
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1200
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1260
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1320
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1380
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1440
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1500
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1560
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1620
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1680
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1740
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1800
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1860
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1920
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 1980
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2040
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2100
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2160
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2220
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2280
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2340
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgaaa aaccaaaaga 2400
acaagaattt cttggtaaga agccgggaac agacaacaga agtcatgaag cccaagtgaa 2460
atcaaaggtg ctaaatggtc gcccaggaga catccgttgt gcttgcctgc gttttggaag 2520
ctctgaagtc acatcacagg acacggggca gtggcaacct tgtctctatg ccagctcagt 2580
cccatcagag agcgagcgct acccacttct aaatagcaat ttcgccgttg aagaggaagg 2640
gcaaaaccac tagaactctc catcttattt tcatgtatat gtgttcat 2688
<210> 33
<211> 2964
<212> DNA
<213> artificial sequence
<220>
<223> Matrice PD1 locus_IL15_2A_sIL15Ra pCLS30513
<400> 33
gactccccag acaggccctg gaaccccccc accttctccc cagccctgct cgtggtgacc 60
gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag cttcgtgcta 120
aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt ccccgaggac 180
cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa cgggcgtgac 240
ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct ctgtggggcc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 420
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 480
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 540
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 600
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 660
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 720
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 780
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 840
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 900
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 960
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1020
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1080
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1140
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1200
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1260
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1320
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1380
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1440
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1500
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1560
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1620
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1680
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1740
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1800
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1860
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1920
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 1980
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2040
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2100
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2160
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2220
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2280
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2340
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgatc tagagggccc 2400
gtttaaaccc gctgatcagc ctcgactgtg ccttctagtt gccagccatc tgttgtttgc 2460
ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct ttcctaataa 2520
aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg gggtggggtg 2580
gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg ggatgcggtg 2640
ggctctatga ctagtggcga attcggcgca gatcaaagag agcctgcggg cagagctcag 2700
ggtgacaggt gcggcctcgg aggccccggg gcaggggtga gctgagccgg tcctggggtg 2760
ggtgtcccct cctgcacagg atcaggagct ccagggtcgt agggcaggga ccccccagct 2820
ccagtccagg gctctgtcct gcacctgggg aatggtgacc ggcatctctg tcctctagct 2880
ctggaagcac cccagcccct ctagtctgcc ctcacccctg accctgaccc tccaccctga 2940
ccccgtccta acccctgacc tttg 2964
<210> 34
<211> 3363
<212> DNA
<213> artificial sequence
<220>
<223> Matrice CD25 locus_IL12a_2A_IL12b pCLS30520
<400> 34
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 60
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 120
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 180
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 240
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccatgtg gccccctggg tcagcctccc agccaccgcc ctcacctgcc 420
gcggccacag gtctgcatcc agcggctcgc cctgtgtccc tgcagtgccg gctcagcatg 480
tgtccagcgc gcagcctcct ccttgtggct accctggtcc tcctggacca cctcagtttg 540
gccagaaacc tccccgtggc cactccagac ccaggaatgt tcccatgcct tcaccactcc 600
caaaacctgc tgagggccgt cagcaacatg ctccagaagg ccagacaaac tctagaattt 660
tacccttgca cttctgaaga gattgatcat gaagatatca caaaagataa aaccagcaca 720
gtggaggcct gtttaccatt ggaattaacc aagaatgaga gttgcctaaa ttccagagag 780
acctctttca taactaatgg gagttgcctg gcctccagaa agacctcttt tatgatggcc 840
ctgtgcctta gtagtattta tgaagacttg aagatgtacc aggtggagtt caagaccatg 900
aatgcaaagc ttctgatgga tcctaagagg cagatctttc tagatcaaaa catgctggca 960
gttattgatg agctgatgca ggccctgaat ttcaacagtg agactgtgcc acaaaaatcc 1020
tcccttgaag aaccggattt ttataaaact aaaatcaagc tctgcatact tcttcatgct 1080
ttcagaattc gggcagtgac tattgataga gtgatgagct atctgaatgc ttccggaagc 1140
ggagctacta acttcagcct gctgaagcag gctggagacg tggaggagaa ccctggacct 1200
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 1260
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 1320
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 1380
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 1440
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 1500
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 1560
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 1620
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 1680
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 1740
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 1800
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 1860
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 1920
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 1980
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 2040
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 2100
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 2160
gaatgggcat ctgtgccctg cagtgagggc agaggcagcc tgctgacctg cggcgacgtc 2220
gaggagaacc ccgggcccat gggggcaggt gccaccggcc gcgccatgga cgggccgcgc 2280
ctgctgctgt tgctgcttct gggggtgtcc cttggaggtg ccaaggaggc atgccccaca 2340
ggcctgtaca cacacagcgg tgagtgctgc aaagcctgca acctgggcga gggtgtggcc 2400
cagccttgtg gagccaacca gaccgtgtgt gagccctgcc tggacagcgt gacgttctcc 2460
gacgtggtga gcgcgaccga gccgtgcaag ccgtgcaccg agtgcgtggg gctccagagc 2520
atgtcggcgc cgtgcgtgga ggccgatgac gccgtgtgcc gctgcgccta cggctactac 2580
caggatgaga cgactgggcg ctgcgaggcg tgccgcgtgt gcgaggcggg ctcgggcctc 2640
gtgttctcct gccaggacaa gcagaacacc gtgtgcgagg agtgccccga cggcacgtat 2700
tccgacgagg ccaaccacgt ggacccgtgc ctgccctgca ccgtgtgcga ggacaccgag 2760
cgccagctcc gcgagtgcac acgctgggcc gacgccgagt gcgaggagat ccctggccgt 2820
tggattacac ggtccacacc cccagagggc tcggacagca cagcccccag cacccaggag 2880
cctgaggcac ctccagaaca agacctcata gccagcacgg tggcaggtgt ggtgaccaca 2940
gtgatgggca gctcccagcc cgtggtgacc cgaggcacca ccgacaacct catccctgtc 3000
tattgctcca tcctggctgc tgtggttgtg ggtcttgtgg cctacatagc cttcaagagg 3060
tgaaaaacca aaagaacaag aatttcttgg taagaagccg ggaacagaca acagaagtca 3120
tgaagcccaa gtgaaatcaa aggtgctaaa tggtcgccca ggagacatcc gttgtgcttg 3180
cctgcgtttt ggaagctctg aagtcacatc acaggacacg gggcagtggc aaccttgtct 3240
ctatgccagc tcagtcccat cagagagcga gcgctaccca cttctaaata gcaatttcgc 3300
cgttgaagag gaagggcaaa accactagaa ctctccatct tattttcatg tatatgtgtt 3360
cat 3363
<210> 35
<211> 3639
<212> DNA
<213> artificial sequence
<220>
<223> Matrice PD1 locus_IL12a_2A_IL12b pCLS30511
<400> 35
gactccccag acaggccctg gaaccccccc accttctccc cagccctgct cgtggtgacc 60
gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag cttcgtgcta 120
aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt ccccgaggac 180
cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa cgggcgtgac 240
ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct ctgtggggcc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccatgtg gccccctggg tcagcctccc agccaccgcc ctcacctgcc 420
gcggccacag gtctgcatcc agcggctcgc cctgtgtccc tgcagtgccg gctcagcatg 480
tgtccagcgc gcagcctcct ccttgtggct accctggtcc tcctggacca cctcagtttg 540
gccagaaacc tccccgtggc cactccagac ccaggaatgt tcccatgcct tcaccactcc 600
caaaacctgc tgagggccgt cagcaacatg ctccagaagg ccagacaaac tctagaattt 660
tacccttgca cttctgaaga gattgatcat gaagatatca caaaagataa aaccagcaca 720
gtggaggcct gtttaccatt ggaattaacc aagaatgaga gttgcctaaa ttccagagag 780
acctctttca taactaatgg gagttgcctg gcctccagaa agacctcttt tatgatggcc 840
ctgtgcctta gtagtattta tgaagacttg aagatgtacc aggtggagtt caagaccatg 900
aatgcaaagc ttctgatgga tcctaagagg cagatctttc tagatcaaaa catgctggca 960
gttattgatg agctgatgca ggccctgaat ttcaacagtg agactgtgcc acaaaaatcc 1020
tcccttgaag aaccggattt ttataaaact aaaatcaagc tctgcatact tcttcatgct 1080
ttcagaattc gggcagtgac tattgataga gtgatgagct atctgaatgc ttccggaagc 1140
ggagctacta acttcagcct gctgaagcag gctggagacg tggaggagaa ccctggacct 1200
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 1260
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 1320
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 1380
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 1440
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 1500
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 1560
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 1620
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 1680
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 1740
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 1800
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 1860
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 1920
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 1980
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 2040
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 2100
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 2160
gaatgggcat ctgtgccctg cagtgagggc agaggcagcc tgctgacctg cggcgacgtc 2220
gaggagaacc ccgggcccat gggggcaggt gccaccggcc gcgccatgga cgggccgcgc 2280
ctgctgctgt tgctgcttct gggggtgtcc cttggaggtg ccaaggaggc atgccccaca 2340
ggcctgtaca cacacagcgg tgagtgctgc aaagcctgca acctgggcga gggtgtggcc 2400
cagccttgtg gagccaacca gaccgtgtgt gagccctgcc tggacagcgt gacgttctcc 2460
gacgtggtga gcgcgaccga gccgtgcaag ccgtgcaccg agtgcgtggg gctccagagc 2520
atgtcggcgc cgtgcgtgga ggccgatgac gccgtgtgcc gctgcgccta cggctactac 2580
caggatgaga cgactgggcg ctgcgaggcg tgccgcgtgt gcgaggcggg ctcgggcctc 2640
gtgttctcct gccaggacaa gcagaacacc gtgtgcgagg agtgccccga cggcacgtat 2700
tccgacgagg ccaaccacgt ggacccgtgc ctgccctgca ccgtgtgcga ggacaccgag 2760
cgccagctcc gcgagtgcac acgctgggcc gacgccgagt gcgaggagat ccctggccgt 2820
tggattacac ggtccacacc cccagagggc tcggacagca cagcccccag cacccaggag 2880
cctgaggcac ctccagaaca agacctcata gccagcacgg tggcaggtgt ggtgaccaca 2940
gtgatgggca gctcccagcc cgtggtgacc cgaggcacca ccgacaacct catccctgtc 3000
tattgctcca tcctggctgc tgtggttgtg ggtcttgtgg cctacatagc cttcaagagg 3060
tgatctagag ggcccgttta aacccgctga tcagcctcga ctgtgccttc tagttgccag 3120
ccatctgttg tttgcccctc ccccgtgcct tccttgaccc tggaaggtgc cactcccact 3180
gtcctttcct aataaaatga ggaaattgca tcgcattgtc tgagtaggtg tcattctatt 3240
ctggggggtg gggtggggca ggacagcaag ggggaggatt gggaagacaa tagcaggcat 3300
gctggggatg cggtgggctc tatgactagt ggcgaattcg gcgcagatca aagagagcct 3360
gcgggcagag ctcagggtga caggtgcggc ctcggaggcc ccggggcagg ggtgagctga 3420
gccggtcctg gggtgggtgt cccctcctgc acaggatcag gagctccagg gtcgtagggc 3480
agggaccccc cagctccagt ccagggctct gtcctgcacc tggggaatgg tgaccggcat 3540
ctctgtcctc tagctctgga agcaccccag cccctctagt ctgccctcac ccctgaccct 3600
gaccctccac cctgaccccg tcctaacccc tgacctttg 3639
<210> 36
<211> 3017
<212> DNA
<213> artificial sequence
<220>
<223> Inserted matrice TRAC locus_CubiCAR CD22 (60 nucleotides upstream and downstream)
<400> 36
atgagatcat gtcctaaccc tgatcctctt gtcccacaga tatccagaac cctgaccctg 60
ttgctgggcc tttttcccat gcctgccttt actctgccag agttatattg ctggggtttt 120
gaagaagatc ctattaaata aaagaataag cagtattatt aagtagccct gcatttcagg 180
tttccttgag tggcaggcca ggcctggccg tgaacgttca ctgaaatcat ggcctcttgg 240
ccaagattga tagcttgtgc ctgtccctga gtcccagtcc atcacgagca gctggtttct 300
aagatgctat ttcccgtata aagcatgaga ccgtgacttg ccagccccac agagccccgc 360
ccttgtccat cactggcatc tggactccag cctgggttgg ggcaaagagg gaaatgagat 420
catgtcctaa ccctgatcct cttgtcccac agatatccag tacccctacg acgtgcccga 480
ctacgcctcc ggtgagggca gaggaagtct tctaacatgc ggtgacgtgg aggagaatcc 540
gggccccgga tccgctctgc ccgtcaccgc tctgctgctg ccactggcac tgctgctgca 600
cgctgctagg cccggagggg gaggcagctg cccctacagc aaccccagcc tgtgcagcgg 660
aggcggcggc agcggcggag ggggtagcca ggtgcagctg cagcagagcg gccctggcct 720
ggtgaagcca agccagacac tgtccctgac ctgcgccatc agcggcgatt ccgtgagctc 780
caactccgcc gcctggaatt ggatcaggca gtccccttct cggggcctgg agtggctggg 840
aaggacatac tatcggtcta agtggtacaa cgattatgcc gtgtctgtga agagcagaat 900
cacaatcaac cctgacacct ccaagaatca gttctctctg cagctgaata gcgtgacacc 960
agaggacacc gccgtgtact attgcgccag ggaggtgacc ggcgacctgg aggatgcctt 1020
tgacatctgg ggccagggca caatggtgac cgtgagctcc ggaggcggcg gatctggcgg 1080
aggaggaagt gggggcggcg ggagtgatat ccagatgaca cagtccccat cctctctgag 1140
cgcctccgtg ggcgacagag tgacaatcac ctgtagggcc tcccagacca tctggtctta 1200
cctgaactgg tatcagcaga ggcccggcaa ggcccctaat ctgctgatct acgcagcaag 1260
ctccctgcag agcggagtgc catccagatt ctctggcagg ggctccggca cagacttcac 1320
cctgaccatc tctagcctgc aggccgagga cttcgccacc tactattgcc agcagtctta 1380
tagcatcccc cagacatttg gccagggcac caagctggag atcaagtcgg atcccggaag 1440
cggaggggga ggcagctgcc cctacagcaa ccccagcctg tgcagcggag gcggcggcag 1500
cgagctgccc acccagggca ccttctccaa cgtgtccacc aacgtgagcc cagccaagcc 1560
caccaccacc gcctgtcctt attccaatcc ttccctgtgt gctcccacca caacccccgc 1620
tccaaggccc cctacccccg caccaactat tgcctcccag ccactctcac tgcggcctga 1680
ggcctgtcgg cccgctgctg gaggcgcagt gcatacaagg ggcctcgatt tcgcctgcga 1740
tatttacatc tgggcacccc tcgccggcac ctgcggggtg cttctcctct ccctggtgat 1800
taccctgtat tgcagacggg gccggaagaa gctcctctac atttttaagc agcctttcat 1860
gcggccagtg cagacaaccc aagaggagga tgggtgttcc tgcagattcc ctgaggaaga 1920
ggaaggcggg tgcgagctga gagtgaagtt ctccaggagc gcagatgccc ccgcctatca 1980
acagggccag aaccagctct acaacgagct taacctcggg aggcgcgaag aatacgacgt 2040
gttggataag agaagggggc gggaccccga gatgggagga aagccccgga ggaagaaccc 2100
tcaggagggc ctgtacaacg agctgcagaa ggataagatg gccgaggcct actcagagat 2160
cgggatgaag ggggagcggc gccgcgggaa ggggcacgat gggctctacc aggggctgag 2220
cacagccaca aaggacacat acgacgcctt gcacatgcag gcccttccac cccgggaata 2280
gtctagaggg cccgtttaaa cccgctgatc agcctcgact gtgccttcta gttgccagcc 2340
atctgttgtt tgcccctccc ccgtgccttc cttgaccctg gaaggtgcca ctcccactgt 2400
cctttcctaa taaaatgagg aaattgcatc gcattgtctg agtaggtgtc attctattct 2460
ggggggtggg gtggggcagg acagcaaggg ggaggattgg gaagacaata gcaggcatgc 2520
tggggatgcg gtgggctcta tgactagtgg cgaattcccg tgtaccagct gagagactct 2580
aaatccagtg acaagtctgt ctgcctattc accgattttg attctcaaac aaatgtgtca 2640
caaagtaagg attctgatgt gtatatcaca gacaaaactg tgctagacat gaggtctatg 2700
gacttcaaga gcaacagtgc tgtggcctgg agcaacaaat ctgactttgc atgtgcaaac 2760
gccttcaaca acagcattat tccagaagac accttcttcc ccagcccagg taagggcagc 2820
tttggtgcct tcgcaggctg tttccttgct tcaggaatgg ccaggttctg cccagagctc 2880
tggtcaatga tgtctaaaac tcctctgatt ggtggtctcg gccttatcca ttgccaccaa 2940
aaccctcttt ttactaagaa acagtgagcc ttgttctggc agtccagaga atgacacggg 3000
aaaaaagcag atgaaga 3017
<210> 37
<211> 2808
<212> DNA
<213> artificial sequence
<220>
<223> Inserted matrice CD25 locus_IL15_2A_sIL15Ra (60 nucleotides upstream and downstream)
<400> 37
agtgctggct agaaaccaag tgctttactg catgcacatc atttagcaca gttagttgct 60
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 120
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 180
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 240
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 300
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 360
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 420
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 480
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 540
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 600
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 660
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 720
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 780
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 840
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 900
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 960
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 1020
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1080
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1140
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1200
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1260
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1320
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1380
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1440
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1500
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1560
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1620
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1680
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1740
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1800
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1860
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1920
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1980
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 2040
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2100
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2160
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2220
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2280
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2340
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2400
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgaaa aaccaaaaga 2460
acaagaattt cttggtaaga agccgggaac agacaacaga agtcatgaag cccaagtgaa 2520
atcaaaggtg ctaaatggtc gcccaggaga catccgttgt gcttgcctgc gttttggaag 2580
ctctgaagtc acatcacagg acacggggca gtggcaacct tgtctctatg ccagctcagt 2640
cccatcagag agcgagcgct acccacttct aaatagcaat ttcgccgttg aagaggaagg 2700
gcaaaaccac tagaactctc catcttattt tcatgtatat gtgttcatta aagcatgaat 2760
ggtatggaac tctctccacc ctatatgtag tataaagaaa agtaggtt 2808
<210> 38
<211> 3084
<212> DNA
<213> artificial sequence
<220>
<223> Inserted matrice PD1 locus_IL15_2A_sIL15Ra (60 nucleotides upstream and downstream)
<400> 38
ggtggccggg gaggctttgt ggggccaccc agccccttcc tcacctctct ccatctctca 60
gactccccag acaggccctg gaaccccccc accttctccc cagccctgct cgtggtgacc 120
gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag cttcgtgcta 180
aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt ccccgaggac 240
cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa cgggcgtgac 300
ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct ctgtggggcc 360
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 420
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 480
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 540
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 600
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 660
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 720
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 780
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 840
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 900
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 960
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 1020
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1080
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1140
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1200
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1260
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1320
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1380
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1440
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1500
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1560
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1620
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1680
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1740
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1800
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1860
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1920
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1980
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 2040
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2100
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2160
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2220
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2280
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2340
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2400
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgatc tagagggccc 2460
gtttaaaccc gctgatcagc ctcgactgtg ccttctagtt gccagccatc tgttgtttgc 2520
ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct ttcctaataa 2580
aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg gggtggggtg 2640
gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg ggatgcggtg 2700
ggctctatga ctagtggcga attcggcgca gatcaaagag agcctgcggg cagagctcag 2760
ggtgacaggt gcggcctcgg aggccccggg gcaggggtga gctgagccgg tcctggggtg 2820
ggtgtcccct cctgcacagg atcaggagct ccagggtcgt agggcaggga ccccccagct 2880
ccagtccagg gctctgtcct gcacctgggg aatggtgacc ggcatctctg tcctctagct 2940
ctggaagcac cccagcccct ctagtctgcc ctcacccctg accctgaccc tccaccctga 3000
ccccgtccta acccctgacc tttgtgccct tccagagaga agggcagaag tgcccacagc 3060
ccaccccagc ccctcaccca ggcc 3084
<210> 39
<211> 3475
<212> DNA
<213> artificial sequence
<220>
<223> Inserted matrice CD25 locus_IL12a_2A_IL12b (60 nucleotides upstream and downstream)
<400> 39
agtgctggct agaaaccaag tgctttactg catgcacatc atttagcaca gttagttgct 60
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 120
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 180
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 240
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 300
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 360
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 420
tccaacccag ggcccatgtg gccccctggg tcagcctccc agccaccgcc ctcacctgcc 480
gcggccacag gtctgcatcc agcggctcgc cctgtgtccc tgcagtgccg gctcagcatg 540
tgtccagcgc gcagcctcct ccttgtggct accctggtcc tcctggacca cctcagtttg 600
gccagaaacc tccccgtggc cactccagac ccaggaatgt tcccatgcct tcaccactcc 660
caaaacctgc tgagggccgt cagcaacatg ctccagaagg ccagacaaac tctagaattt 720
tacccttgca cttctgaaga gattgatcat gaagatatca caaaagataa aaccagcaca 780
gtggaggcct gtttaccatt ggaattaacc aagaatgaga gttgcctaaa ttccagagag 840
acctctttca taactaatgg gagttgcctg gcctccagaa agacctcttt tatgatggcc 900
ctgtgcctta gtagtattta tgaagacttg aagatgtacc aggtggagtt caagaccatg 960
aatgcaaagc ttctgatgga tcctaagagg cagatctttc tagatcaaaa catgctggca 1020
gttattgatg agctgatgca ggccctgaat ttcaacagtg agactgtgcc acaaaaatcc 1080
tcccttgaag aaccggattt ttataaaact aaaatcaagc tctgcatact tcttcatgct 1140
ttcagaattc gggcagtgac tattgataga gtgatgagct atctgaatgc ttccggaagc 1200
ggagctacta acttcagcct gctgaagcag gctggagacg tggaggagaa ccctggacct 1260
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 1320
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 1380
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 1440
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 1500
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 1560
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 1620
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 1680
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 1740
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 1800
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 1860
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 1920
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 1980
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 2040
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 2100
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 2160
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 2220
gaatgggcat ctgtgccctg cagtgagggc agaggcagcc tgctgacctg cggcgacgtc 2280
gaggagaacc ccgggcccat gggggcaggt gccaccggcc gcgccatgga cgggccgcgc 2340
ctgctgctgt tgctgcttct gggggtgtcc cttggaggtg ccaaggaggc atgccccaca 2400
ggcctgtaca cacacagcgg tgagtgctgc aaagcctgca acctgggcga gggtgtggcc 2460
cagccttgtg gagccaacca gaccgtgtgt gagccctgcc tggacagcgt gacgttctcc 2520
gacgtggtga gcgcgaccga gccgtgcaag ccgtgcaccg agtgcgtggg gctccagagc 2580
atgtcggcgc cgtgcgtgga ggccgatgac gccgtgtgcc gctgcgccta cggctactac 2640
caggatgaga cgactgggcg ctgcgaggcg tgccgcgtgt gcgaggcggg ctcgggcctc 2700
gtgttctcct gccaggacaa gcagaacacc gtgtgcgagg agtgccccga cggcacgtat 2760
tccgacgagg ccaaccacgt ggacccgtgc ctgccctgca ccgtgtgcga ggacaccgag 2820
cgccagctcc gcgagtgcac acgctgggcc gacgccgagt gcgaggagat ccctggccgt 2880
tggattacac ggtccacacc cccagagggc tcggacagca cagcccccag cacccaggag 2940
cctgaggcac ctccagaaca agacctcata gccagcacgg tggcaggtgt ggtgaccaca 3000
gtgatgggca gctcccagcc cgtggtgacc cgaggcacca ccgacaacct catccctgtc 3060
tattgctcca tcctggctgc tgtggttgtg ggtcttgtgg cctacatagc cttcaagagg 3120
tgaaaaacca aaagaacaag aatttcttgg taagaagccg ggaacagaca acagaagtca 3180
tgaagcccaa gtgaaatcaa aggtgctaaa tggtcgccca ggagacatcc gttgtgcttg 3240
cctgcgtttt ggaagctctg aagtcacatc acaggacacg gggcagtggc aaccttgtct 3300
ctatgccagc tcagtcccat cagagagcga gcgctaccca cttctaaata gcaatttcgc 3360
cgttgaagag gaagggcaaa accactagaa ctctccatct tattttcatg tatatgtgtt 3420
catgaatggt atggaactct ctccacccta tatgtagtat aaagaaaagt aggtt 3475
<210> 40
<211> 3759
<212> DNA
<213> artificial sequence
<220>
<223> Inserted matrice PD1 locus_IL12a_2A_IL12b (60 nucleotides upstream and downstream)
<400> 40
ggtggccggg gaggctttgt ggggccaccc agccccttcc tcacctctct ccatctctca 60
gactccccag acaggccctg gaaccccccc accttctccc cagccctgct cgtggtgacc 120
gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag cttcgtgcta 180
aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt ccccgaggac 240
cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa cgggcgtgac 300
ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct ctgtggggcc 360
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 420
tccaacccag ggcccatgtg gccccctggg tcagcctccc agccaccgcc ctcacctgcc 480
gcggccacag gtctgcatcc agcggctcgc cctgtgtccc tgcagtgccg gctcagcatg 540
tgtccagcgc gcagcctcct ccttgtggct accctggtcc tcctggacca cctcagtttg 600
gccagaaacc tccccgtggc cactccagac ccaggaatgt tcccatgcct tcaccactcc 660
caaaacctgc tgagggccgt cagcaacatg ctccagaagg ccagacaaac tctagaattt 720
tacccttgca cttctgaaga gattgatcat gaagatatca caaaagataa aaccagcaca 780
gtggaggcct gtttaccatt ggaattaacc aagaatgaga gttgcctaaa ttccagagag 840
acctctttca taactaatgg gagttgcctg gcctccagaa agacctcttt tatgatggcc 900
ctgtgcctta gtagtattta tgaagacttg aagatgtacc aggtggagtt caagaccatg 960
aatgcaaagc ttctgatgga tcctaagagg cagatctttc tagatcaaaa catgctggca 1020
gttattgatg agctgatgca ggccctgaat ttcaacagtg agactgtgcc acaaaaatcc 1080
tcccttgaag aaccggattt ttataaaact aaaatcaagc tctgcatact tcttcatgct 1140
ttcagaattc gggcagtgac tattgataga gtgatgagct atctgaatgc ttccggaagc 1200
ggagctacta acttcagcct gctgaagcag gctggagacg tggaggagaa ccctggacct 1260
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 1320
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 1380
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 1440
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 1500
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 1560
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 1620
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 1680
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 1740
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 1800
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 1860
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 1920
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 1980
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 2040
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 2100
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 2160
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 2220
gaatgggcat ctgtgccctg cagtgagggc agaggcagcc tgctgacctg cggcgacgtc 2280
gaggagaacc ccgggcccat gggggcaggt gccaccggcc gcgccatgga cgggccgcgc 2340
ctgctgctgt tgctgcttct gggggtgtcc cttggaggtg ccaaggaggc atgccccaca 2400
ggcctgtaca cacacagcgg tgagtgctgc aaagcctgca acctgggcga gggtgtggcc 2460
cagccttgtg gagccaacca gaccgtgtgt gagccctgcc tggacagcgt gacgttctcc 2520
gacgtggtga gcgcgaccga gccgtgcaag ccgtgcaccg agtgcgtggg gctccagagc 2580
atgtcggcgc cgtgcgtgga ggccgatgac gccgtgtgcc gctgcgccta cggctactac 2640
caggatgaga cgactgggcg ctgcgaggcg tgccgcgtgt gcgaggcggg ctcgggcctc 2700
gtgttctcct gccaggacaa gcagaacacc gtgtgcgagg agtgccccga cggcacgtat 2760
tccgacgagg ccaaccacgt ggacccgtgc ctgccctgca ccgtgtgcga ggacaccgag 2820
cgccagctcc gcgagtgcac acgctgggcc gacgccgagt gcgaggagat ccctggccgt 2880
tggattacac ggtccacacc cccagagggc tcggacagca cagcccccag cacccaggag 2940
cctgaggcac ctccagaaca agacctcata gccagcacgg tggcaggtgt ggtgaccaca 3000
gtgatgggca gctcccagcc cgtggtgacc cgaggcacca ccgacaacct catccctgtc 3060
tattgctcca tcctggctgc tgtggttgtg ggtcttgtgg cctacatagc cttcaagagg 3120
tgatctagag ggcccgttta aacccgctga tcagcctcga ctgtgccttc tagttgccag 3180
ccatctgttg tttgcccctc ccccgtgcct tccttgaccc tggaaggtgc cactcccact 3240
gtcctttcct aataaaatga ggaaattgca tcgcattgtc tgagtaggtg tcattctatt 3300
ctggggggtg gggtggggca ggacagcaag ggggaggatt gggaagacaa tagcaggcat 3360
gctggggatg cggtgggctc tatgactagt ggcgaattcg gcgcagatca aagagagcct 3420
gcgggcagag ctcagggtga caggtgcggc ctcggaggcc ccggggcagg ggtgagctga 3480
gccggtcctg gggtgggtgt cccctcctgc acaggatcag gagctccagg gtcgtagggc 3540
agggaccccc cagctccagt ccagggctct gtcctgcacc tggggaatgg tgaccggcat 3600
ctctgtcctc tagctctgga agcaccccag cccctctagt ctgccctcac ccctgaccct 3660
gaccctccac cctgaccccg tcctaacccc tgacctttgt gcccttccag agagaagggc 3720
agaagtgccc acagcccacc ccagcccctc acccaggcc 3759
<210> 41
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> upstream TRAC locus polynucleotide sequence
<400> 41
atgagatcat gtcctaaccc tgatcctctt gtcccacaga tatccagaac cctgaccctg 60
<210> 42
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> downstream TRAC locus polynucleotide sequence
<400> 42
gaaacagtga gccttgttct ggcagtccag agaatgacac gggaaaaaag cagatgaaga 60
<210> 43
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> upstream CD25 locus polynucleotide sequence
<400> 43
agtgctggct agaaaccaag tgctttactg catgcacatc atttagcaca gttagttgct 60
<210> 44
<211> 52
<212> DNA
<213> artificial sequence
<220>
<223> downstream CD25 locus polynucleotide sequence
<400> 44
gaatggtatg gaactctctc caccctatat gtagtataaa gaaaagtagg tt 52
<210> 45
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> upstream PD1 locus polynucleotide sequence
<400> 45
ggtggccggg gaggctttgt ggggccaccc agccccttcc tcacctctct ccatctctca 60
<210> 46
<211> 60
<212> DNA
<213> artificial sequence
<220>
<223> downstream PD1 locus polynucleotide sequence
<400> 46
tgcccttcca gagagaaggg cagaagtgcc cacagcccac cccagcccct cacccaggcc 60
<210> 47
<211> 759
<212> DNA
<213> artificial sequence
<220>
<223> IL-12a polynucleotide
<400> 47
atgtggcccc ctgggtcagc ctcccagcca ccgccctcac ctgccgcggc cacaggtctg 60
catccagcgg ctcgccctgt gtccctgcag tgccggctca gcatgtgtcc agcgcgcagc 120
ctcctccttg tggctaccct ggtcctcctg gaccacctca gtttggccag aaacctcccc 180
gtggccactc cagacccagg aatgttccca tgccttcacc actcccaaaa cctgctgagg 240
gccgtcagca acatgctcca gaaggccaga caaactctag aattttaccc ttgcacttct 300
gaagagattg atcatgaaga tatcacaaaa gataaaacca gcacagtgga ggcctgttta 360
ccattggaat taaccaagaa tgagagttgc ctaaattcca gagagacctc tttcataact 420
aatgggagtt gcctggcctc cagaaagacc tcttttatga tggccctgtg ccttagtagt 480
atttatgaag acttgaagat gtaccaggtg gagttcaaga ccatgaatgc aaagcttctg 540
atggatccta agaggcagat ctttctagat caaaacatgc tggcagttat tgatgagctg 600
atgcaggccc tgaatttcaa cagtgagact gtgccacaaa aatcctccct tgaagaaccg 660
gatttttata aaactaaaat caagctctgc atacttcttc atgctttcag aattcgggca 720
gtgactattg atagagtgat gagctatctg aatgcttcc 759
<210> 48
<211> 984
<212> DNA
<213> artificial sequence
<220>
<223> IL12b polynucleotide
<400> 48
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 60
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 120
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 180
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 240
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 300
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 360
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 420
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 480
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 540
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 600
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 660
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 720
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 780
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 840
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 900
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 960
gaatgggcat ctgtgccctg cagt 984
<210> 49
<211> 399
<212> DNA
<213> artificial sequence
<220>
<223> IL15 polynucleotide
<400> 49
ggcattcatg tcttcatttt gggctgtttc agtgcagggc ttcctaaaac agaagccaac 60
tgggtgaatg taataagtga tttgaaaaaa attgaagatc ttattcaatc tatgcatatt 120
gatgctactt tatatacgga aagtgatgtt caccccagtt gcaaagtaac agcaatgaag 180
tgctttctct tggagttaca agttatttca cttgagtccg gagatgcaag tattcatgat 240
acagtagaaa atctgatcat cctagcaaac aacagtttgt cttctaatgg gaatgtaaca 300
gaatctggat gcaaagaatg tgaggaactg gaggaaaaaa atattaaaga atttttgcag 360
agttttgtac atattgtcca aatgttcatc aacacttct 399
<210> 50
<211> 525
<212> DNA
<213> artificial sequence
<220>
<223> sIL15ra polynucleotide
<400> 50
atcacgtgcc ctccccccat gtccgtggaa cacgcagaca tctgggtcaa gagctacagc 60
ttgtactcca gggagcggta catttgtaac tctggtttca agcgtaaagc cggcacgtcc 120
agcctgacgg agtgcgtgtt gaacaaggcc acgaatgtcg cccactggac aacccccagt 180
ctcaaatgca ttagagaccc tgccctggtt caccaaaggc cagcgccacc ctccacagta 240
acgacggcag gggtgacccc acagccagag agcctctccc cttctggaaa agagcccgca 300
gcttcatctc ccagctcaaa caacacagcg gccacaacag cagctattgt cccgggctcc 360
cagctgatgc cttcaaaatc accttccaca ggaaccacag agataagcag tcatgagtcc 420
tcccacggca ccccctctca gacaacagcc aagaactggg aactcacagc atccgcctcc 480
caccagccgc caggtgtgta tccacagggc cacagcgaca ccact 525
<210> 51
<211> 1818
<212> DNA
<213> artificial sequence
<220>
<223> soluble GP130 polynucleotide
<400> 51
atgctgacac tgcagacttg gctggtgcag gcactgttta tttttctgac tactgaatca 60
actggcgaac tgctggaccc ttgtggctac atcagccctg agtccccagt ggtgcagctg 120
cacagcaact tcaccgccgt gtgcgtgctg aaggagaagt gtatggacta ctttcacgtg 180
aacgccaatt atatcgtgtg gaaaaccaac cacttcacaa tccccaagga gcagtacacc 240
atcatcaata ggacagccag ctccgtgacc tttacagaca tcgcctccct gaacatccag 300
ctgacctgca atatcctgac attcggccag ctggagcaga acgtgtatgg catcaccatc 360
atctctggcc tgccccctga gaagcctaag aacctgagct gcatcgtgaa tgagggcaag 420
aagatgcggt gtgagtggga cggcggcaga gagacacacc tggagacaaa cttcaccctg 480
aagtccgagt gggccacaca caagtttgcc gactgcaagg ccaagcgcga taccccaaca 540
tcctgtaccg tggattactc tacagtgtat tttgtgaaca tcgaagtgtg ggtggaggcc 600
gagaatgccc tgggcaaggt gacctccgac cacatcaact tcgatcccgt gtacaaggtg 660
aagcctaacc caccccacaa tctgagcgtg atcaattccg aggagctgtc tagcatcctg 720
aagctgacct ggacaaaccc atctatcaag agcgtgatca tcctgaagta caatatccag 780
tatcggacca aggacgcctc cacatggagc cagatccctc cagaggatac cgccagcaca 840
agatcctctt tcaccgtgca ggacctgaag cccttcacag agtacgtgtt tcggatcaga 900
tgtatgaagg aggacggcaa gggctactgg agcgattggt ccgaggaggc cagcggcatc 960
acctatgagg acaggccttc taaggccccc agcttctggt acaagatcga tccatcccac 1020
acccagggct atcgcacagt gcagctggtg tggaaaaccc tgcccccttt cgaggccaac 1080
ggcaagatcc tggactacga ggtgaccctg acacggtgga agtcccacct gcagaactat 1140
accgtgaatg ccaccaagct gacagtgaac ctgacaaatg atcggtacct ggccaccctg 1200
acagtgagaa acctggtggg caagtctgac gccgccgtgc tgaccatccc tgcctgcgat 1260
ttccaggcca cacacccagt gatggacctg aaggcctttc ccaaggataa tatgctgtgg 1320
gtggagtgga ccacacctag agagtccgtg aagaagtaca tcctggagtg gtgcgtgctg 1380
tctgacaagg ccccatgtat caccgactgg cagcaggagg atggcaccgt gcacaggaca 1440
tatctgcgcg gcaacctggc cgagtctaag tgttacctga tcaccgtgac acccgtgtat 1500
gcagacggac caggctctcc tgagagcatc aaggcctacc tgaagcaggc accaccaagc 1560
aagggaccaa ccgtgcggac aaagaaggtc ggcaagaatg aggccgtgct ggagtgggac 1620
cagctgcctg tggatgtgca gaacggcttc atcaggaatt acaccatctt ttatcgcaca 1680
atcatcggca acgagacagc cgtgaatgtg gacagctccc acaccgagta tacactgtct 1740
agcctgacct ccgatacact gtacatggtg aggatggccg cctatacaga cgagggcggc 1800
aaggatggcc ccgagttt 1818
<210> 52
<211> 72
<212> DNA
<213> artificial sequence
<220>
<223> IgE signal sequence
<400> 52
ggtaccgggt ccgccaccat ggactggacc tggattctgt tcctcgtggc tgctgctaca 60
agagtgcaca gc 72
<210> 53
<211> 75
<212> DNA
<213> artificial sequence
<220>
<223> F2A
<400> 53
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 60
tccaacccag ggccc 75
<210> 54
<211> 66
<212> DNA
<213> artificial sequence
<220>
<223> P2A
<400> 54
ggaagcggag ctactaactt cagcctgctg aagcaggctg gagacgtgga ggagaaccct 60
ggacct 66
<210> 55
<211> 54
<212> DNA
<213> artificial sequence
<220>
<223> T2A
<400> 55
gagggcagag gcagcctgct gacctgcggc gacgtcgagg agaaccccgg gccc 54
<210> 56
<211> 825
<212> DNA
<213> artificial sequence
<220>
<223> LNGFR
<400> 56
atgggggcag gtgccaccgg ccgcgccatg gacgggccgc gcctgctgct gttgctgctt 60
ctgggggtgt cccttggagg tgccaaggag gcatgcccca caggcctgta cacacacagc 120
ggtgagtgct gcaaagcctg caacctgggc gagggtgtgg cccagccttg tggagccaac 180
cagaccgtgt gtgagccctg cctggacagc gtgacgttct ccgacgtggt gagcgcgacc 240
gagccgtgca agccgtgcac cgagtgcgtg gggctccaga gcatgtcggc gccgtgcgtg 300
gaggccgatg acgccgtgtg ccgctgcgcc tacggctact accaggatga gacgactggg 360
cgctgcgagg cgtgccgcgt gtgcgaggcg ggctcgggcc tcgtgttctc ctgccaggac 420
aagcagaaca ccgtgtgcga ggagtgcccc gacggcacgt attccgacga ggccaaccac 480
gtggacccgt gcctgccctg caccgtgtgc gaggacaccg agcgccagct ccgcgagtgc 540
acacgctggg ccgacgccga gtgcgaggag atccctggcc gttggattac acggtccaca 600
cccccagagg gctcggacag cacagccccc agcacccagg agcctgaggc acctccagaa 660
caagacctca tagccagcac ggtggcaggt gtggtgacca cagtgatggg cagctcccag 720
cccgtggtga cccgaggcac caccgacaac ctcatccctg tctattgctc catcctggct 780
gctgtggttg tgggtcttgt ggcctacata gccttcaaga ggtga 825
<210> 57
<211> 253
<212> PRT
<213> artificial sequence
<220>
<223> IL-12a polypeptide
<400> 57
Met Trp Pro Pro Gly Ser Ala Ser Gln Pro Pro Pro Ser Pro Ala Ala
1 5 10 15
Ala Thr Gly Leu His Pro Ala Ala Arg Pro Val Ser Leu Gln Cys Arg
20 25 30
Leu Ser Met Cys Pro Ala Arg Ser Leu Leu Leu Val Ala Thr Leu Val
35 40 45
Leu Leu Asp His Leu Ser Leu Ala Arg Asn Leu Pro Val Ala Thr Pro
50 55 60
Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu Arg
65 70 75 80
Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe Tyr
85 90 95
Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp Lys
100 105 110
Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn Glu
115 120 125
Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser Cys
130 135 140
Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser Ser
145 150 155 160
Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met Asn
165 170 175
Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln Asn
180 185 190
Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn Ser
195 200 205
Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr Lys
210 215 220
Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg Ala
225 230 235 240
Val Thr Ile Asp Arg Val Met Ser Tyr Leu Asn Ala Ser
245 250
<210> 58
<211> 328
<212> PRT
<213> artificial sequence
<220>
<223> IL12b polypeptide
<400> 58
Met Cys His Gln Gln Leu Val Ile Ser Trp Phe Ser Leu Val Phe Leu
1 5 10 15
Ala Ser Pro Leu Val Ala Ile Trp Glu Leu Lys Lys Asp Val Tyr Val
20 25 30
Val Glu Leu Asp Trp Tyr Pro Asp Ala Pro Gly Glu Met Val Val Leu
35 40 45
Thr Cys Asp Thr Pro Glu Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln
50 55 60
Ser Ser Glu Val Leu Gly Ser Gly Lys Thr Leu Thr Ile Gln Val Lys
65 70 75 80
Glu Phe Gly Asp Ala Gly Gln Tyr Thr Cys His Lys Gly Gly Glu Val
85 90 95
Leu Ser His Ser Leu Leu Leu Leu His Lys Lys Glu Asp Gly Ile Trp
100 105 110
Ser Thr Asp Ile Leu Lys Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe
115 120 125
Leu Arg Cys Glu Ala Lys Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp
130 135 140
Leu Thr Thr Ile Ser Thr Asp Leu Thr Phe Ser Val Lys Ser Ser Arg
145 150 155 160
Gly Ser Ser Asp Pro Gln Gly Val Thr Cys Gly Ala Ala Thr Leu Ser
165 170 175
Ala Glu Arg Val Arg Gly Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu
180 185 190
Cys Gln Glu Asp Ser Ala Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile
195 200 205
Glu Val Met Val Asp Ala Val His Lys Leu Lys Tyr Glu Asn Tyr Thr
210 215 220
Ser Ser Phe Phe Ile Arg Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn
225 230 235 240
Leu Gln Leu Lys Pro Leu Lys Asn Ser Arg Gln Val Glu Val Ser Trp
245 250 255
Glu Tyr Pro Asp Thr Trp Ser Thr Pro His Ser Tyr Phe Ser Leu Thr
260 265 270
Phe Cys Val Gln Val Gln Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg
275 280 285
Val Phe Thr Asp Lys Thr Ser Ala Thr Val Ile Cys Arg Lys Asn Ala
290 295 300
Ser Ile Ser Val Arg Ala Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser
305 310 315 320
Glu Trp Ala Ser Val Pro Cys Ser
325
245 250
<210> 59
<211> 133
<212> PRT
<213> artificial sequence
<220>
<223> IL15 polypeptide
<400> 59
Gly Ile His Val Phe Ile Leu Gly Cys Phe Ser Ala Gly Leu Pro Lys
1 5 10 15
Thr Glu Ala Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu
20 25 30
Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser
35 40 45
Asp Val His Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu
50 55 60
Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp
65 70 75 80
Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn
85 90 95
Gly Asn Val Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu
100 105 110
Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met
115 120 125
Phe Ile Asn Thr Ser
130
<210> 60
<211> 175
<212> PRT
<213> artificial sequence
<220>
<223> sIL15ra polypeptide
<400> 60
Ile Thr Cys Pro Pro Pro Met Ser Val Glu His Ala Asp Ile Trp Val
1 5 10 15
Lys Ser Tyr Ser Leu Tyr Ser Arg Glu Arg Tyr Ile Cys Asn Ser Gly
20 25 30
Phe Lys Arg Lys Ala Gly Thr Ser Ser Leu Thr Glu Cys Val Leu Asn
35 40 45
Lys Ala Thr Asn Val Ala His Trp Thr Thr Pro Ser Leu Lys Cys Ile
50 55 60
Arg Asp Pro Ala Leu Val His Gln Arg Pro Ala Pro Pro Ser Thr Val
65 70 75 80
Thr Thr Ala Gly Val Thr Pro Gln Pro Glu Ser Leu Ser Pro Ser Gly
85 90 95
Lys Glu Pro Ala Ala Ser Ser Pro Ser Ser Asn Asn Thr Ala Ala Thr
100 105 110
Thr Ala Ala Ile Val Pro Gly Ser Gln Leu Met Pro Ser Lys Ser Pro
115 120 125
Ser Thr Gly Thr Thr Glu Ile Ser Ser His Glu Ser Ser His Gly Thr
130 135 140
Pro Ser Gln Thr Thr Ala Lys Asn Trp Glu Leu Thr Ala Ser Ala Ser
145 150 155 160
His Gln Pro Pro Gly Val Tyr Pro Gln Gly His Ser Asp Thr Thr
165 170 175
<210> 61
<211> 606
<212> PRT
<213> artificial sequence
<220>
<223> soluble gp130
<400> 61
Met Leu Thr Leu Gln Thr Trp Leu Val Gln Ala Leu Phe Ile Phe Leu
1 5 10 15
Thr Thr Glu Ser Thr Gly Glu Leu Leu Asp Pro Cys Gly Tyr Ile Ser
20 25 30
Pro Glu Ser Pro Val Val Gln Leu His Ser Asn Phe Thr Ala Val Cys
35 40 45
Val Leu Lys Glu Lys Cys Met Asp Tyr Phe His Val Asn Ala Asn Tyr
50 55 60
Ile Val Trp Lys Thr Asn His Phe Thr Ile Pro Lys Glu Gln Tyr Thr
65 70 75 80
Ile Ile Asn Arg Thr Ala Ser Ser Val Thr Phe Thr Asp Ile Ala Ser
85 90 95
Leu Asn Ile Gln Leu Thr Cys Asn Ile Leu Thr Phe Gly Gln Leu Glu
100 105 110
Gln Asn Val Tyr Gly Ile Thr Ile Ile Ser Gly Leu Pro Pro Glu Lys
115 120 125
Pro Lys Asn Leu Ser Cys Ile Val Asn Glu Gly Lys Lys Met Arg Cys
130 135 140
Glu Trp Asp Gly Gly Arg Glu Thr His Leu Glu Thr Asn Phe Thr Leu
145 150 155 160
Lys Ser Glu Trp Ala Thr His Lys Phe Ala Asp Cys Lys Ala Lys Arg
165 170 175
Asp Thr Pro Thr Ser Cys Thr Val Asp Tyr Ser Thr Val Tyr Phe Val
180 185 190
Asn Ile Glu Val Trp Val Glu Ala Glu Asn Ala Leu Gly Lys Val Thr
195 200 205
Ser Asp His Ile Asn Phe Asp Pro Val Tyr Lys Val Lys Pro Asn Pro
210 215 220
Pro His Asn Leu Ser Val Ile Asn Ser Glu Glu Leu Ser Ser Ile Leu
225 230 235 240
Lys Leu Thr Trp Thr Asn Pro Ser Ile Lys Ser Val Ile Ile Leu Lys
245 250 255
Tyr Asn Ile Gln Tyr Arg Thr Lys Asp Ala Ser Thr Trp Ser Gln Ile
260 265 270
Pro Pro Glu Asp Thr Ala Ser Thr Arg Ser Ser Phe Thr Val Gln Asp
275 280 285
Leu Lys Pro Phe Thr Glu Tyr Val Phe Arg Ile Arg Cys Met Lys Glu
290 295 300
Asp Gly Lys Gly Tyr Trp Ser Asp Trp Ser Glu Glu Ala Ser Gly Ile
305 310 315 320
Thr Tyr Glu Asp Arg Pro Ser Lys Ala Pro Ser Phe Trp Tyr Lys Ile
325 330 335
Asp Pro Ser His Thr Gln Gly Tyr Arg Thr Val Gln Leu Val Trp Lys
340 345 350
Thr Leu Pro Pro Phe Glu Ala Asn Gly Lys Ile Leu Asp Tyr Glu Val
355 360 365
Thr Leu Thr Arg Trp Lys Ser His Leu Gln Asn Tyr Thr Val Asn Ala
370 375 380
Thr Lys Leu Thr Val Asn Leu Thr Asn Asp Arg Tyr Leu Ala Thr Leu
385 390 395 400
Thr Val Arg Asn Leu Val Gly Lys Ser Asp Ala Ala Val Leu Thr Ile
405 410 415
Pro Ala Cys Asp Phe Gln Ala Thr His Pro Val Met Asp Leu Lys Ala
420 425 430
Phe Pro Lys Asp Asn Met Leu Trp Val Glu Trp Thr Thr Pro Arg Glu
435 440 445
Ser Val Lys Lys Tyr Ile Leu Glu Trp Cys Val Leu Ser Asp Lys Ala
450 455 460
Pro Cys Ile Thr Asp Trp Gln Gln Glu Asp Gly Thr Val His Arg Thr
465 470 475 480
Tyr Leu Arg Gly Asn Leu Ala Glu Ser Lys Cys Tyr Leu Ile Thr Val
485 490 495
Thr Pro Val Tyr Ala Asp Gly Pro Gly Ser Pro Glu Ser Ile Lys Ala
500 505 510
Tyr Leu Lys Gln Ala Pro Pro Ser Lys Gly Pro Thr Val Arg Thr Lys
515 520 525
Lys Val Gly Lys Asn Glu Ala Val Leu Glu Trp Asp Gln Leu Pro Val
530 535 540
Asp Val Gln Asn Gly Phe Ile Arg Asn Tyr Thr Ile Phe Tyr Arg Thr
545 550 555 560
Ile Ile Gly Asn Glu Thr Ala Val Asn Val Asp Ser Ser His Thr Glu
565 570 575
Tyr Thr Leu Ser Ser Leu Thr Ser Asp Thr Leu Tyr Met Val Arg Met
580 585 590
Ala Ala Tyr Thr Asp Glu Gly Gly Lys Asp Gly Pro Glu Phe
595 600 605
<210> 62
<211> 836
<212> PRT
<213> artificial sequence
<220>
<223> soluble gp130 fused to a Fc
<400> 62
Met Leu Thr Leu Gln Thr Trp Leu Val Gln Ala Leu Phe Ile Phe Leu
1 5 10 15
Thr Thr Glu Ser Thr Gly Glu Leu Leu Asp Pro Cys Gly Tyr Ile Ser
20 25 30
Pro Glu Ser Pro Val Val Gln Leu His Ser Asn Phe Thr Ala Val Cys
35 40 45
Val Leu Lys Glu Lys Cys Met Asp Tyr Phe His Val Asn Ala Asn Tyr
50 55 60
Ile Val Trp Lys Thr Asn His Phe Thr Ile Pro Lys Glu Gln Tyr Thr
65 70 75 80
Ile Ile Asn Arg Thr Ala Ser Ser Val Thr Phe Thr Asp Ile Ala Ser
85 90 95
Leu Asn Ile Gln Leu Thr Cys Asn Ile Leu Thr Phe Gly Gln Leu Glu
100 105 110
Gln Asn Val Tyr Gly Ile Thr Ile Ile Ser Gly Leu Pro Pro Glu Lys
115 120 125
Pro Lys Asn Leu Ser Cys Ile Val Asn Glu Gly Lys Lys Met Arg Cys
130 135 140
Glu Trp Asp Gly Gly Arg Glu Thr His Leu Glu Thr Asn Phe Thr Leu
145 150 155 160
Lys Ser Glu Trp Ala Thr His Lys Phe Ala Asp Cys Lys Ala Lys Arg
165 170 175
Asp Thr Pro Thr Ser Cys Thr Val Asp Tyr Ser Thr Val Tyr Phe Val
180 185 190
Asn Ile Glu Val Trp Val Glu Ala Glu Asn Ala Leu Gly Lys Val Thr
195 200 205
Ser Asp His Ile Asn Phe Asp Pro Val Tyr Lys Val Lys Pro Asn Pro
210 215 220
Pro His Asn Leu Ser Val Ile Asn Ser Glu Glu Leu Ser Ser Ile Leu
225 230 235 240
Lys Leu Thr Trp Thr Asn Pro Ser Ile Lys Ser Val Ile Ile Leu Lys
245 250 255
Tyr Asn Ile Gln Tyr Arg Thr Lys Asp Ala Ser Thr Trp Ser Gln Ile
260 265 270
Pro Pro Glu Asp Thr Ala Ser Thr Arg Ser Ser Phe Thr Val Gln Asp
275 280 285
Leu Lys Pro Phe Thr Glu Tyr Val Phe Arg Ile Arg Cys Met Lys Glu
290 295 300
Asp Gly Lys Gly Tyr Trp Ser Asp Trp Ser Glu Glu Ala Ser Gly Ile
305 310 315 320
Thr Tyr Glu Asp Arg Pro Ser Lys Ala Pro Ser Phe Trp Tyr Lys Ile
325 330 335
Asp Pro Ser His Thr Gln Gly Tyr Arg Thr Val Gln Leu Val Trp Lys
340 345 350
Thr Leu Pro Pro Phe Glu Ala Asn Gly Lys Ile Leu Asp Tyr Glu Val
355 360 365
Thr Leu Thr Arg Trp Lys Ser His Leu Gln Asn Tyr Thr Val Asn Ala
370 375 380
Thr Lys Leu Thr Val Asn Leu Thr Asn Asp Arg Tyr Leu Ala Thr Leu
385 390 395 400
Thr Val Arg Asn Leu Val Gly Lys Ser Asp Ala Ala Val Leu Thr Ile
405 410 415
Pro Ala Cys Asp Phe Gln Ala Thr His Pro Val Met Asp Leu Lys Ala
420 425 430
Phe Pro Lys Asp Asn Met Leu Trp Val Glu Trp Thr Thr Pro Arg Glu
435 440 445
Ser Val Lys Lys Tyr Ile Leu Glu Trp Cys Val Leu Ser Asp Lys Ala
450 455 460
Pro Cys Ile Thr Asp Trp Gln Gln Glu Asp Gly Thr Val His Arg Thr
465 470 475 480
Tyr Leu Arg Gly Asn Leu Ala Glu Ser Lys Cys Tyr Leu Ile Thr Val
485 490 495
Thr Pro Val Tyr Ala Asp Gly Pro Gly Ser Pro Glu Ser Ile Lys Ala
500 505 510
Tyr Leu Lys Gln Ala Pro Pro Ser Lys Gly Pro Thr Val Arg Thr Lys
515 520 525
Lys Val Gly Lys Asn Glu Ala Val Leu Glu Trp Asp Gln Leu Pro Val
530 535 540
Asp Val Gln Asn Gly Phe Ile Arg Asn Tyr Thr Ile Phe Tyr Arg Thr
545 550 555 560
Ile Ile Gly Asn Glu Thr Ala Val Asn Val Asp Ser Ser His Thr Glu
565 570 575
Tyr Thr Leu Ser Ser Leu Thr Ser Asp Thr Leu Tyr Met Val Arg Met
580 585 590
Ala Ala Tyr Thr Asp Glu Gly Gly Lys Asp Gly Pro Glu Phe Arg Ser
595 600 605
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Glu
610 615 620
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
625 630 635 640
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
645 650 655
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
660 665 670
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
675 680 685
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
690 695 700
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
705 710 715 720
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
725 730 735
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
740 745 750
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
755 760 765
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
770 775 780
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
785 790 795 800
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
805 810 815
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
820 825 830
Ser Pro Gly Lys
835
<210> 63
<211> 7711
<212> DNA
<213> artificial sequence
<220>
<223> Matrice TRAC locus_CubiCAR CD22 pCLS30056 full sequence
<400> 63
gtggcacttt tcggggaaat gtgcgcggaa cccctatttg tttatttttc taaatacatt 60
caaatatgta tccgctcatg agacaataac cctgataaat gcttcaataa tattgaaaaa 120
ggaagagtat gagtattcaa catttccgtg tcgcccttat tccctttttt gcggcatttt 180
gccttcctgt ttttgctcac ccagaaacgc tggtgaaagt aaaagatgct gaagatcagt 240
tgggtgcacg agtgggttac atcgaactgg atctcaacag cggtaagatc cttgagagtt 300
ttcgccccga agaacgtttt ccaatgatga gcacttttaa agttctgcta tgtggcgcgg 360
tattatcccg tattgacgcc gggcaagagc aactcggtcg ccgcatacac tattctcaga 420
atgacttggt tgagtactca ccagtcacag aaaagcatct tacggatggc atgacagtaa 480
gagaattatg cagtgctgcc ataaccatga gtgataacac tgcggccaac ttacttctga 540
caacgatcgg aggaccgaag gagctaaccg cttttttgca caacatgggg gatcatgtaa 600
ctcgccttga tcgttgggaa ccggagctga atgaagccat accaaacgac gagcgtgaca 660
ccacgatgcc tgtagcaatg gcaacaacgt tgcgcaaact attaactggc gaactactta 720
ctctagcttc ccggcaacaa ttaatagact ggatggaggc ggataaagtt gcaggaccac 780
ttctgcgctc ggcccttccg gctggctggt ttattgctga taaatctgga gccggtgagc 840
gtggttctcg cggtatcatt gcagcactgg ggccagatgg taagccctcc cgtatcgtag 900
ttatctacac gacggggagt caggcaacta tggatgaacg aaatagacag atcgctgaga 960
taggtgcctc actgattaag cattggtaac tgtcagacca agtttactca tatatacttt 1020
agattgattt aaaacttcat ttttaattta aaaggatcta ggtgaagatc ctttttgata 1080
atctcatgac caaaatccct taacgtgagt tttcgttcca ctgagcgtca gaccccgtag 1140
aaaagatcaa aggatcttct tgagatcctt tttttctgcg cgtaatctgc tgcttgcaaa 1200
caaaaaaacc accgctacca gcggtggttt gtttgccgga tcaagagcta ccaactcttt 1260
ttccgaaggt aactggcttc agcagagcgc agataccaaa tactgttctt ctagtgtagc 1320
cgtagttagg ccaccacttc aagaactctg tagcaccgcc tacatacctc gctctgctaa 1380
tcctgttacc agtggctgct gccagtggcg ataagtcgtg tcttaccggg ttggactcaa 1440
gacgatagtt accggataag gcgcagcggt cgggctgaac ggggggttcg tgcacacagc 1500
ccagcttgga gcgaacgacc tacaccgaac tgagatacct acagcgtgag ctatgagaaa 1560
gcgccacgct tcccgaaggg agaaaggcgg acaggtatcc ggtaagcggc agggtcggaa 1620
caggagagcg cacgagggag cttccagggg gaaacgcctg gtatctttat agtcctgtcg 1680
ggtttcgcca cctctgactt gagcgtcgat ttttgtgatg ctcgtcaggg gggcggagcc 1740
tatggaaaaa cgccagcaac gcggcctttt tacggttcct ggccttttgc tggccttttg 1800
ctcacatggt ctttcctgcg ttatcccctg attctgtgga taaccgtatt accgcctttg 1860
agtgagctga taccgctcgc cgcagccgaa cgaccgagcg cagcgagtca gtgagcgagg 1920
aagcggagag cgcccaatac gcaaaccgcc tctccccgcg cgttggccga ttcattaatg 1980
cagctggcac gacaggtttc ccgactggaa agcgggcagt gagcgcaacg caattaatgt 2040
gagttagctc actcattagg caccccaggc tttacacttt atgcttccgg ctcgtatgtt 2100
gtgtggaatt gtgagcggat aacaatttca cacaggaaac agctatgacc atgattacgc 2160
caagcgcgtc aattaaccct cactaaaggg aacaaaagct gttaattaat tgctgggcct 2220
ttttcccatg cctgccttta ctctgccaga gttatattgc tggggttttg aagaagatcc 2280
tattaaataa aagaataagc agtattatta agtagccctg catttcaggt ttccttgagt 2340
ggcaggccag gcctggccgt gaacgttcac tgaaatcatg gcctcttggc caagattgat 2400
agcttgtgcc tgtccctgag tcccagtcca tcacgagcag ctggtttcta agatgctatt 2460
tcccgtataa agcatgagac cgtgacttgc cagccccaca gagccccgcc cttgtccatc 2520
actggcatct ggactccagc ctgggttggg gcaaagaggg aaatgagatc atgtcctaac 2580
cctgatcctc ttgtcccaca gatatccagt acccctacga cgtgcccgac tacgcctccg 2640
gtgagggcag aggaagtctt ctaacatgcg gtgacgtgga ggagaatccg ggccccggat 2700
ccgctctgcc cgtcaccgct ctgctgctgc cactggcact gctgctgcac gctgctaggc 2760
ccggaggggg aggcagctgc ccctacagca accccagcct gtgcagcgga ggcggcggca 2820
gcggcggagg gggtagccag gtgcagctgc agcagagcgg ccctggcctg gtgaagccaa 2880
gccagacact gtccctgacc tgcgccatca gcggcgattc cgtgagctcc aactccgccg 2940
cctggaattg gatcaggcag tccccttctc ggggcctgga gtggctggga aggacatact 3000
atcggtctaa gtggtacaac gattatgccg tgtctgtgaa gagcagaatc acaatcaacc 3060
ctgacacctc caagaatcag ttctctctgc agctgaatag cgtgacacca gaggacaccg 3120
ccgtgtacta ttgcgccagg gaggtgaccg gcgacctgga ggatgccttt gacatctggg 3180
gccagggcac aatggtgacc gtgagctccg gaggcggcgg atctggcgga ggaggaagtg 3240
ggggcggcgg gagtgatatc cagatgacac agtccccatc ctctctgagc gcctccgtgg 3300
gcgacagagt gacaatcacc tgtagggcct cccagaccat ctggtcttac ctgaactggt 3360
atcagcagag gcccggcaag gcccctaatc tgctgatcta cgcagcaagc tccctgcaga 3420
gcggagtgcc atccagattc tctggcaggg gctccggcac agacttcacc ctgaccatct 3480
ctagcctgca ggccgaggac ttcgccacct actattgcca gcagtcttat agcatccccc 3540
agacatttgg ccagggcacc aagctggaga tcaagtcgga tcccggaagc ggagggggag 3600
gcagctgccc ctacagcaac cccagcctgt gcagcggagg cggcggcagc gagctgccca 3660
cccagggcac cttctccaac gtgtccacca acgtgagccc agccaagccc accaccaccg 3720
cctgtcctta ttccaatcct tccctgtgtg ctcccaccac aacccccgct ccaaggcccc 3780
ctacccccgc accaactatt gcctcccagc cactctcact gcggcctgag gcctgtcggc 3840
ccgctgctgg aggcgcagtg catacaaggg gcctcgattt cgcctgcgat atttacatct 3900
gggcacccct cgccggcacc tgcggggtgc ttctcctctc cctggtgatt accctgtatt 3960
gcagacgggg ccggaagaag ctcctctaca tttttaagca gcctttcatg cggccagtgc 4020
agacaaccca agaggaggat gggtgttcct gcagattccc tgaggaagag gaaggcgggt 4080
gcgagctgag agtgaagttc tccaggagcg cagatgcccc cgcctatcaa cagggccaga 4140
accagctcta caacgagctt aacctcggga ggcgcgaaga atacgacgtg ttggataaga 4200
gaagggggcg ggaccccgag atgggaggaa agccccggag gaagaaccct caggagggcc 4260
tgtacaacga gctgcagaag gataagatgg ccgaggccta ctcagagatc gggatgaagg 4320
gggagcggcg ccgcgggaag gggcacgatg ggctctacca ggggctgagc acagccacaa 4380
aggacacata cgacgccttg cacatgcagg cccttccacc ccgggaatag tctagagggc 4440
ccgtttaaac ccgctgatca gcctcgactg tgccttctag ttgccagcca tctgttgttt 4500
gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac tcccactgtc ctttcctaat 4560
aaaatgagga aattgcatcg cattgtctga gtaggtgtca ttctattctg gggggtgggg 4620
tggggcagga cagcaagggg gaggattggg aagacaatag caggcatgct ggggatgcgg 4680
tgggctctat gactagtggc gaattcccgt gtaccagctg agagactcta aatccagtga 4740
caagtctgtc tgcctattca ccgattttga ttctcaaaca aatgtgtcac aaagtaagga 4800
ttctgatgtg tatatcacag acaaaactgt gctagacatg aggtctatgg acttcaagag 4860
caacagtgct gtggcctgga gcaacaaatc tgactttgca tgtgcaaacg ccttcaacaa 4920
cagcattatt ccagaagaca ccttcttccc cagcccaggt aagggcagct ttggtgcctt 4980
cgcaggctgt ttccttgctt caggaatggc caggttctgc ccagagctct ggtcaatgat 5040
gtctaaaact cctctgattg gtggtctcgg ccttatccat tgccaccaaa accctctttt 5100
tactaagcga tcgctccggt gcccgtcagt gggcagagcg cacatcgccc acagtccccg 5160
agaagttggg gggaggggtc ggcaattgaa cgggtgccta gagaaggtgg cgcggggtaa 5220
actgggaaag tgatgtcgtg tactggctcc gcctttttcc cgagggtggg ggagaaccgt 5280
atataagtgc agtagtcgcc gtgaacgttc tttttcgcaa cgggtttgcc gccagaacac 5340
agctgaagct tcgaggggct cgcatctctc cttcacgcgc ccgccgccct acctgaggcc 5400
gccatccacg ccggttgagt cgcgttctgc cgcctcccgc ctgtggtgcc tcctgaactg 5460
cgtccgccgt ctaggtaagt ttaaagctca ggtcgagacc gggcctttgt ccggcgctcc 5520
cttggagcct acctagactc agccggctct ccacgctttg cctgaccctg cttgctcaac 5580
tctacgtctt tgtttcgttt tctgttctgc gccgttacag atccaagctg tgaccggcgc 5640
ctacctgaga tcaccggcgc caccatggct tcttaccctg gacaccagca tgcttctgcc 5700
tttgaccagg ctgccagatc caggggccac tccaacagga gaactgccct aagacccaga 5760
agacagcagg aagccactga ggtgaggcct gagcagaaga tgccaaccct gctgagggtg 5820
tacattgatg gacctcatgg catgggcaag accaccacca ctcaactgct ggtggcactg 5880
ggctccaggg atgacattgt gtatgtgcct gagccaatga cctactggag agtgctagga 5940
gcctctgaga ccattgccaa catctacacc acccagcaca ggctggacca gggagaaatc 6000
tctgctggag atgctgctgt ggtgatgacc tctgcccaga tcacaatggg aatgccctat 6060
gctgtgactg atgctgttct ggctcctcac attggaggag aggctggctc ttctcatgcc 6120
cctccacctg ccctgaccct gatctttgac agacacccca ttgcagccct gctgtgctac 6180
ccagcagcaa ggtacctcat gggctccatg accccacagg ctgtgctggc ttttgtggcc 6240
ctgatccctc caaccctccc tggcaccaac attgttctgg gagcactgcc tgaagacaga 6300
cacattgaca ggctggcaaa gaggcagaga cctggagaga gactggacct ggccatgctg 6360
gctgcaatca gaagggtgta tggactgctg gcaaacactg tgagatacct ccagtgtgga 6420
ggctcttgga gagaggactg gggacagctc tctggaacag cagtgccccc tcaaggagct 6480
gagccccagt ccaatgctgg tccaagaccc cacattgggg acaccctgtt caccctgttc 6540
agagcccctg agctgctggc tcccaatgga gacctgtaca atgtgtttgc ctgggctctg 6600
gatgttctag ccaagaggct gaggtccatg catgtgttca tcctggacta tgaccagtcc 6660
cctgctggat gcagagatgc tctgctgcaa ctaacctctg gcatggtgca gacccatgtg 6720
accacccctg gcagcatccc caccatctgt gacctagcca gaacctttgc cagggagatg 6780
ggagaggcca actaaggcgc gccactcgag cgctagctgg ccagacatga taagatacat 6840
tgatgagttt ggacaaacca caactagaat gcagtgaaaa aaatgcttta tttgtgaaat 6900
ttgtgatgct attgctttat ttgtaaccat tataagctgc aataaacaag ttaacaacaa 6960
caattgcatt cattttatgt ttcaggttca gggggaggtg tgggaggttt tttaaagcaa 7020
gtaaaacctc tacaaatgtg gtatggaagg cgcgcccaat tcgccctata gtgagtcgta 7080
ttacgtcgcg ctcactggcc gtcgttttac aacgtcgtga ctgggaaaac cctggcgtta 7140
cccaacttaa tcgccttgca gcacatcccc ctttcgccag ctggcgtaat agcgaagagg 7200
cccgcaccga aacgcccttc ccaacagttg cgcagcctga atggcgaatg ggagcgccct 7260
gtagcggcgc attaagcgcg gcgggtgtgg tggttacgcg cagcgtgacc gctacacttg 7320
ccagcgccct agcgcccgct cctttcgctt tcttcccttc ctttctcgcc acgttcgccg 7380
gctttccccg tcaagctcta aatcgggggc tccctttagg gttccgattt agtgctttac 7440
ggcacctcga ccccaaaaaa cttgattagg gtgatggttg gcctgtagtg ggccatagcc 7500
ctgatagacg gtttttcgcc ctttgacgtt ggagtccacg ttctttaata gtggactctt 7560
gttccaaact ggaacaacac tcaaccctat ctcggtctat tcttttgatt tataagggat 7620
tttgccgatt tcggcctatt ggttaaaaaa tgagctgatt taacaaaaat ttaacgcgaa 7680
ttttaacaaa atattaacgc ttacaattta g 7711
<210> 64
<211> 7502
<212> DNA
<213> artificial sequence
<220>
<223> Matrice CD25 locus_IL15_2A_sIL15Ra pCLS30519 full sequence
<400> 64
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 60
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 120
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 180
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 240
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 420
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 480
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 540
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 600
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 660
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 720
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 780
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 840
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 900
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 960
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1020
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1080
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1140
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1200
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1260
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1320
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1380
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1440
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1500
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1560
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1620
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1680
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1740
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1800
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1860
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1920
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 1980
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2040
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2100
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2160
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2220
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2280
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2340
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgaaa aaccaaaaga 2400
acaagaattt cttggtaaga agccgggaac agacaacaga agtcatgaag cccaagtgaa 2460
atcaaaggtg ctaaatggtc gcccaggaga catccgttgt gcttgcctgc gttttggaag 2520
ctctgaagtc acatcacagg acacggggca gtggcaacct tgtctctatg ccagctcagt 2580
cccatcagag agcgagcgct acccacttct aaatagcaat ttcgccgttg aagaggaagg 2640
gcaaaaccac tagaactctc catcttattt tcatgtatat gtgttcatgc gatcgctccg 2700
gtgcccgtca gtgggcagag cgcacatcgc ccacagtccc cgagaagttg gggggagggg 2760
tcggcaattg aacgggtgcc tagagaaggt ggcgcggggt aaactgggaa agtgatgtcg 2820
tgtactggct ccgccttttt cccgagggtg ggggagaacc gtatataagt gcagtagtcg 2880
ccgtgaacgt tctttttcgc aacgggtttg ccgccagaac acagctgaag cttcgagggg 2940
ctcgcatctc tccttcacgc gcccgccgcc ctacctgagg ccgccatcca cgccggttga 3000
gtcgcgttct gccgcctccc gcctgtggtg cctcctgaac tgcgtccgcc gtctaggtaa 3060
gtttaaagct caggtcgaga ccgggccttt gtccggcgct cccttggagc ctacctagac 3120
tcagccggct ctccacgctt tgcctgaccc tgcttgctca actctacgtc tttgtttcgt 3180
tttctgttct gcgccgttac agatccaagc tgtgaccggc gcctacctga gatcaccggc 3240
gccaccatgg cttcttaccc tggacaccag catgcttctg cctttgacca ggctgccaga 3300
tccaggggcc actccaacag gagaactgcc ctaagaccca gaagacagca ggaagccact 3360
gaggtgaggc ctgagcagaa gatgccaacc ctgctgaggg tgtacattga tggacctcat 3420
ggcatgggca agaccaccac cactcaactg ctggtggcac tgggctccag ggatgacatt 3480
gtgtatgtgc ctgagccaat gacctactgg agagtgctag gagcctctga gaccattgcc 3540
aacatctaca ccacccagca caggctggac cagggagaaa tctctgctgg agatgctgct 3600
gtggtgatga cctctgccca gatcacaatg ggaatgccct atgctgtgac tgatgctgtt 3660
ctggctcctc acattggagg agaggctggc tcttctcatg cccctccacc tgccctgacc 3720
ctgatctttg acagacaccc cattgcagcc ctgctgtgct acccagcagc aaggtacctc 3780
atgggctcca tgaccccaca ggctgtgctg gcttttgtgg ccctgatccc tccaaccctc 3840
cctggcacca acattgttct gggagcactg cctgaagaca gacacattga caggctggca 3900
aagaggcaga gacctggaga gagactggac ctggccatgc tggctgcaat cagaagggtg 3960
tatggactgc tggcaaacac tgtgagatac ctccagtgtg gaggctcttg gagagaggac 4020
tggggacagc tctctggaac agcagtgccc cctcaaggag ctgagcccca gtccaatgct 4080
ggtccaagac cccacattgg ggacaccctg ttcaccctgt tcagagcccc tgagctgctg 4140
gctcccaatg gagacctgta caatgtgttt gcctgggctc tggatgttct agccaagagg 4200
ctgaggtcca tgcatgtgtt catcctggac tatgaccagt cccctgctgg atgcagagat 4260
gctctgctgc aactaacctc tggcatggtg cagacccatg tgaccacccc tggcagcatc 4320
cccaccatct gtgacctagc cagaaccttt gccagggaga tgggagaggc caactaaggc 4380
gcgccactcg agcgctagct ggccagacat gataagatac attgatgagt ttggacaaac 4440
cacaactaga atgcagtgaa aaaaatgctt tatttgtgaa atttgtgatg ctattgcttt 4500
atttgtaacc attataagct gcaataaaca agttaacaac aacaattgca ttcattttat 4560
gtttcaggtt cagggggagg tgtgggaggt tttttaaagc aagtaaaacc tctacaaatg 4620
tggtatggaa ggcgcgccca attcgcccta tagtgagtcg tattacgtcg cgctcactgg 4680
ccgtcgtttt acaacgtcgt gactgggaaa accctggcgt tacccaactt aatcgccttg 4740
cagcacatcc ccctttcgcc agctggcgta atagcgaaga ggcccgcacc gaaacgccct 4800
tcccaacagt tgcgcagcct gaatggcgaa tgggagcgcc ctgtagcggc gcattaagcg 4860
cggcgggtgt ggtggttacg cgcagcgtga ccgctacact tgccagcgcc ctagcgcccg 4920
ctcctttcgc tttcttccct tcctttctcg ccacgttcgc cggctttccc cgtcaagctc 4980
taaatcgggg gctcccttta gggttccgat ttagtgcttt acggcacctc gaccccaaaa 5040
aacttgatta gggtgatggt tggcctgtag tgggccatag ccctgataga cggtttttcg 5100
ccctttgacg ttggagtcca cgttctttaa tagtggactc ttgttccaaa ctggaacaac 5160
actcaaccct atctcggtct attcttttga tttataaggg attttgccga tttcggccta 5220
ttggttaaaa aatgagctga tttaacaaaa atttaacgcg aattttaaca aaatattaac 5280
gcttacaatt taggtggcac ttttcgggga aatgtgcgcg gaacccctat ttgtttattt 5340
ttctaaatac attcaaatat gtatccgctc atgagacaat aaccctgata aatgcttcaa 5400
taatattgaa aaaggaagag tatgagtatt caacatttcc gtgtcgccct tattcccttt 5460
tttgcggcat tttgccttcc tgtttttgct cacccagaaa cgctggtgaa agtaaaagat 5520
gctgaagatc agttgggtgc acgagtgggt tacatcgaac tggatctcaa cagcggtaag 5580
atccttgaga gttttcgccc cgaagaacgt tttccaatga tgagcacttt taaagttctg 5640
ctatgtggcg cggtattatc ccgtattgac gccgggcaag agcaactcgg tcgccgcata 5700
cactattctc agaatgactt ggttgagtac tcaccagtca cagaaaagca tcttacggat 5760
ggcatgacag taagagaatt atgcagtgct gccataacca tgagtgataa cactgcggcc 5820
aacttacttc tgacaacgat cggaggaccg aaggagctaa ccgctttttt gcacaacatg 5880
ggggatcatg taactcgcct tgatcgttgg gaaccggagc tgaatgaagc cataccaaac 5940
gacgagcgtg acaccacgat gcctgtagca atggcaacaa cgttgcgcaa actattaact 6000
ggcgaactac ttactctagc ttcccggcaa caattaatag actggatgga ggcggataaa 6060
gttgcaggac cacttctgcg ctcggccctt ccggctggct ggtttattgc tgataaatct 6120
ggagccggtg agcgtggttc tcgcggtatc attgcagcac tggggccaga tggtaagccc 6180
tcccgtatcg tagttatcta cacgacgggg agtcaggcaa ctatggatga acgaaataga 6240
cagatcgctg agataggtgc ctcactgatt aagcattggt aactgtcaga ccaagtttac 6300
tcatatatac tttagattga tttaaaactt catttttaat ttaaaaggat ctaggtgaag 6360
atcctttttg ataatctcat gaccaaaatc ccttaacgtg agttttcgtt ccactgagcg 6420
tcagaccccg tagaaaagat caaaggatct tcttgagatc ctttttttct gcgcgtaatc 6480
tgctgcttgc aaacaaaaaa accaccgcta ccagcggtgg tttgtttgcc ggatcaagag 6540
ctaccaactc tttttccgaa ggtaactggc ttcagcagag cgcagatacc aaatactgtt 6600
cttctagtgt agccgtagtt aggccaccac ttcaagaact ctgtagcacc gcctacatac 6660
ctcgctctgc taatcctgtt accagtggct gctgccagtg gcgataagtc gtgtcttacc 6720
gggttggact caagacgata gttaccggat aaggcgcagc ggtcgggctg aacggggggt 6780
tcgtgcacac agcccagctt ggagcgaacg acctacaccg aactgagata cctacagcgt 6840
gagctatgag aaagcgccac gcttcccgaa gggagaaagg cggacaggta tccggtaagc 6900
ggcagggtcg gaacaggaga gcgcacgagg gagcttccag ggggaaacgc ctggtatctt 6960
tatagtcctg tcgggtttcg ccacctctga cttgagcgtc gatttttgtg atgctcgtca 7020
ggggggcgga gcctatggaa aaacgccagc aacgcggcct ttttacggtt cctggccttt 7080
tgctggcctt ttgctcacat ggtctttcct gcgttatccc ctgattctgt ggataaccgt 7140
attaccgcct ttgagtgagc tgataccgct cgccgcagcc gaacgaccga gcgcagcgag 7200
tcagtgagcg aggaagcgga gagcgcccaa tacgcaaacc gcctctcccc gcgcgttggc 7260
cgattcatta atgcagctgg cacgacaggt ttcccgactg gaaagcgggc agtgagcgca 7320
acgcaattaa tgtgagttag ctcactcatt aggcacccca ggctttacac tttatgcttc 7380
cggctcgtat gttgtgtgga attgtgagcg gataacaatt tcacacagga aacagctatg 7440
accatgatta cgccaagcgc gtcaattaac cctcactaaa gggaacaaaa gctgttaatt 7500
aa 7502
<210> 65
<211> 7778
<212> DNA
<213> artificial sequence
<220>
<223> Matrice PD1 locus_IL15_2A_sIL15Ra pCLS30513 full sequence
<400> 65
gactccccag acaggccctg gaaccccccc accttctccc cagccctgct cgtggtgacc 60
gaaggggaca acgccacctt cacctgcagc ttctccaaca catcggagag cttcgtgcta 120
aactggtacc gcatgagccc cagcaaccag acggacaagc tggccgcctt ccccgaggac 180
cgcagccagc ccggccagga ctgccgcttc cgtgtcacac aactgcccaa cgggcgtgac 240
ttccacatga gcgtggtcag ggcccggcgc aatgacagcg gcacctacct ctgtggggcc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccggtac cgggtccgcc accatggact ggacctggat tctgttcctc 420
gtggctgctg ctacaagagt gcacagcggc attcatgtct tcattttggg ctgtttcagt 480
gcagggcttc ctaaaacaga agccaactgg gtgaatgtaa taagtgattt gaaaaaaatt 540
gaagatctta ttcaatctat gcatattgat gctactttat atacggaaag tgatgttcac 600
cccagttgca aagtaacagc aatgaagtgc tttctcttgg agttacaagt tatttcactt 660
gagtccggag atgcaagtat tcatgataca gtagaaaatc tgatcatcct agcaaacaac 720
agtttgtctt ctaatgggaa tgtaacagaa tctggatgca aagaatgtga ggaactggag 780
gaaaaaaata ttaaagaatt tttgcagagt tttgtacata ttgtccaaat gttcatcaac 840
acttctggaa gcggagctac taacttcagc ctgctgaagc aggctggaga cgtggaggag 900
aaccctggac ctgggaccgg ctctgcaacc atggattgga cgtggatcct gtttctcgtg 960
gcagctgcca caagagttca cagtatcacg tgccctcccc ccatgtccgt ggaacacgca 1020
gacatctggg tcaagagcta cagcttgtac tccagggagc ggtacatttg taactctggt 1080
ttcaagcgta aagccggcac gtccagcctg acggagtgcg tgttgaacaa ggccacgaat 1140
gtcgcccact ggacaacccc cagtctcaaa tgcattagag accctgccct ggttcaccaa 1200
aggccagcgc caccctccac agtaacgacg gcaggggtga ccccacagcc agagagcctc 1260
tccccttctg gaaaagagcc cgcagcttca tctcccagct caaacaacac agcggccaca 1320
acagcagcta ttgtcccggg ctcccagctg atgccttcaa aatcaccttc cacaggaacc 1380
acagagataa gcagtcatga gtcctcccac ggcaccccct ctcagacaac agccaagaac 1440
tgggaactca cagcatccgc ctcccaccag ccgccaggtg tgtatccaca gggccacagc 1500
gacaccactg agggcagagg cagcctgctg acctgcggcg acgtcgagga gaaccccggg 1560
cccatggggg caggtgccac cggccgcgcc atggacgggc cgcgcctgct gctgttgctg 1620
cttctggggg tgtcccttgg aggtgccaag gaggcatgcc ccacaggcct gtacacacac 1680
agcggtgagt gctgcaaagc ctgcaacctg ggcgagggtg tggcccagcc ttgtggagcc 1740
aaccagaccg tgtgtgagcc ctgcctggac agcgtgacgt tctccgacgt ggtgagcgcg 1800
accgagccgt gcaagccgtg caccgagtgc gtggggctcc agagcatgtc ggcgccgtgc 1860
gtggaggccg atgacgccgt gtgccgctgc gcctacggct actaccagga tgagacgact 1920
gggcgctgcg aggcgtgccg cgtgtgcgag gcgggctcgg gcctcgtgtt ctcctgccag 1980
gacaagcaga acaccgtgtg cgaggagtgc cccgacggca cgtattccga cgaggccaac 2040
cacgtggacc cgtgcctgcc ctgcaccgtg tgcgaggaca ccgagcgcca gctccgcgag 2100
tgcacacgct gggccgacgc cgagtgcgag gagatccctg gccgttggat tacacggtcc 2160
acacccccag agggctcgga cagcacagcc cccagcaccc aggagcctga ggcacctcca 2220
gaacaagacc tcatagccag cacggtggca ggtgtggtga ccacagtgat gggcagctcc 2280
cagcccgtgg tgacccgagg caccaccgac aacctcatcc ctgtctattg ctccatcctg 2340
gctgctgtgg ttgtgggtct tgtggcctac atagccttca agaggtgatc tagagggccc 2400
gtttaaaccc gctgatcagc ctcgactgtg ccttctagtt gccagccatc tgttgtttgc 2460
ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct ttcctaataa 2520
aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg gggtggggtg 2580
gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg ggatgcggtg 2640
ggctctatga ctagtggcga attcggcgca gatcaaagag agcctgcggg cagagctcag 2700
ggtgacaggt gcggcctcgg aggccccggg gcaggggtga gctgagccgg tcctggggtg 2760
ggtgtcccct cctgcacagg atcaggagct ccagggtcgt agggcaggga ccccccagct 2820
ccagtccagg gctctgtcct gcacctgggg aatggtgacc ggcatctctg tcctctagct 2880
ctggaagcac cccagcccct ctagtctgcc ctcacccctg accctgaccc tccaccctga 2940
ccccgtccta acccctgacc tttggcgatc gctccggtgc ccgtcagtgg gcagagcgca 3000
catcgcccac agtccccgag aagttggggg gaggggtcgg caattgaacg ggtgcctaga 3060
gaaggtggcg cggggtaaac tgggaaagtg atgtcgtgta ctggctccgc ctttttcccg 3120
agggtggggg agaaccgtat ataagtgcag tagtcgccgt gaacgttctt tttcgcaacg 3180
ggtttgccgc cagaacacag ctgaagcttc gaggggctcg catctctcct tcacgcgccc 3240
gccgccctac ctgaggccgc catccacgcc ggttgagtcg cgttctgccg cctcccgcct 3300
gtggtgcctc ctgaactgcg tccgccgtct aggtaagttt aaagctcagg tcgagaccgg 3360
gcctttgtcc ggcgctccct tggagcctac ctagactcag ccggctctcc acgctttgcc 3420
tgaccctgct tgctcaactc tacgtctttg tttcgttttc tgttctgcgc cgttacagat 3480
ccaagctgtg accggcgcct acctgagatc accggcgcca ccatggcttc ttaccctgga 3540
caccagcatg cttctgcctt tgaccaggct gccagatcca ggggccactc caacaggaga 3600
actgccctaa gacccagaag acagcaggaa gccactgagg tgaggcctga gcagaagatg 3660
ccaaccctgc tgagggtgta cattgatgga cctcatggca tgggcaagac caccaccact 3720
caactgctgg tggcactggg ctccagggat gacattgtgt atgtgcctga gccaatgacc 3780
tactggagag tgctaggagc ctctgagacc attgccaaca tctacaccac ccagcacagg 3840
ctggaccagg gagaaatctc tgctggagat gctgctgtgg tgatgacctc tgcccagatc 3900
acaatgggaa tgccctatgc tgtgactgat gctgttctgg ctcctcacat tggaggagag 3960
gctggctctt ctcatgcccc tccacctgcc ctgaccctga tctttgacag acaccccatt 4020
gcagccctgc tgtgctaccc agcagcaagg tacctcatgg gctccatgac cccacaggct 4080
gtgctggctt ttgtggccct gatccctcca accctccctg gcaccaacat tgttctggga 4140
gcactgcctg aagacagaca cattgacagg ctggcaaaga ggcagagacc tggagagaga 4200
ctggacctgg ccatgctggc tgcaatcaga agggtgtatg gactgctggc aaacactgtg 4260
agatacctcc agtgtggagg ctcttggaga gaggactggg gacagctctc tggaacagca 4320
gtgccccctc aaggagctga gccccagtcc aatgctggtc caagacccca cattggggac 4380
accctgttca ccctgttcag agcccctgag ctgctggctc ccaatggaga cctgtacaat 4440
gtgtttgcct gggctctgga tgttctagcc aagaggctga ggtccatgca tgtgttcatc 4500
ctggactatg accagtcccc tgctggatgc agagatgctc tgctgcaact aacctctggc 4560
atggtgcaga cccatgtgac cacccctggc agcatcccca ccatctgtga cctagccaga 4620
acctttgcca gggagatggg agaggccaac taaggcgcgc cactcgagcg ctagctggcc 4680
agacatgata agatacattg atgagtttgg acaaaccaca actagaatgc agtgaaaaaa 4740
atgctttatt tgtgaaattt gtgatgctat tgctttattt gtaaccatta taagctgcaa 4800
taaacaagtt aacaacaaca attgcattca ttttatgttt caggttcagg gggaggtgtg 4860
ggaggttttt taaagcaagt aaaacctcta caaatgtggt atggaaggcg cgcccaattc 4920
gccctatagt gagtcgtatt acgtcgcgct cactggccgt cgttttacaa cgtcgtgact 4980
gggaaaaccc tggcgttacc caacttaatc gccttgcagc acatccccct ttcgccagct 5040
ggcgtaatag cgaagaggcc cgcaccgaaa cgcccttccc aacagttgcg cagcctgaat 5100
ggcgaatggg agcgccctgt agcggcgcat taagcgcggc gggtgtggtg gttacgcgca 5160
gcgtgaccgc tacacttgcc agcgccctag cgcccgctcc tttcgctttc ttcccttcct 5220
ttctcgccac gttcgccggc tttccccgtc aagctctaaa tcgggggctc cctttagggt 5280
tccgatttag tgctttacgg cacctcgacc ccaaaaaact tgattagggt gatggttggc 5340
ctgtagtggg ccatagccct gatagacggt ttttcgccct ttgacgttgg agtccacgtt 5400
ctttaatagt ggactcttgt tccaaactgg aacaacactc aaccctatct cggtctattc 5460
ttttgattta taagggattt tgccgatttc ggcctattgg ttaaaaaatg agctgattta 5520
acaaaaattt aacgcgaatt ttaacaaaat attaacgctt acaatttagg tggcactttt 5580
cggggaaatg tgcgcggaac ccctatttgt ttatttttct aaatacattc aaatatgtat 5640
ccgctcatga gacaataacc ctgataaatg cttcaataat attgaaaaag gaagagtatg 5700
agtattcaac atttccgtgt cgcccttatt cccttttttg cggcattttg ccttcctgtt 5760
tttgctcacc cagaaacgct ggtgaaagta aaagatgctg aagatcagtt gggtgcacga 5820
gtgggttaca tcgaactgga tctcaacagc ggtaagatcc ttgagagttt tcgccccgaa 5880
gaacgttttc caatgatgag cacttttaaa gttctgctat gtggcgcggt attatcccgt 5940
attgacgccg ggcaagagca actcggtcgc cgcatacact attctcagaa tgacttggtt 6000
gagtactcac cagtcacaga aaagcatctt acggatggca tgacagtaag agaattatgc 6060
agtgctgcca taaccatgag tgataacact gcggccaact tacttctgac aacgatcgga 6120
ggaccgaagg agctaaccgc ttttttgcac aacatggggg atcatgtaac tcgccttgat 6180
cgttgggaac cggagctgaa tgaagccata ccaaacgacg agcgtgacac cacgatgcct 6240
gtagcaatgg caacaacgtt gcgcaaacta ttaactggcg aactacttac tctagcttcc 6300
cggcaacaat taatagactg gatggaggcg gataaagttg caggaccact tctgcgctcg 6360
gcccttccgg ctggctggtt tattgctgat aaatctggag ccggtgagcg tggttctcgc 6420
ggtatcattg cagcactggg gccagatggt aagccctccc gtatcgtagt tatctacacg 6480
acggggagtc aggcaactat ggatgaacga aatagacaga tcgctgagat aggtgcctca 6540
ctgattaagc attggtaact gtcagaccaa gtttactcat atatacttta gattgattta 6600
aaacttcatt tttaatttaa aaggatctag gtgaagatcc tttttgataa tctcatgacc 6660
aaaatccctt aacgtgagtt ttcgttccac tgagcgtcag accccgtaga aaagatcaaa 6720
ggatcttctt gagatccttt ttttctgcgc gtaatctgct gcttgcaaac aaaaaaacca 6780
ccgctaccag cggtggtttg tttgccggat caagagctac caactctttt tccgaaggta 6840
actggcttca gcagagcgca gataccaaat actgttcttc tagtgtagcc gtagttaggc 6900
caccacttca agaactctgt agcaccgcct acatacctcg ctctgctaat cctgttacca 6960
gtggctgctg ccagtggcga taagtcgtgt cttaccgggt tggactcaag acgatagtta 7020
ccggataagg cgcagcggtc gggctgaacg gggggttcgt gcacacagcc cagcttggag 7080
cgaacgacct acaccgaact gagataccta cagcgtgagc tatgagaaag cgccacgctt 7140
cccgaaggga gaaaggcgga caggtatccg gtaagcggca gggtcggaac aggagagcgc 7200
acgagggagc ttccaggggg aaacgcctgg tatctttata gtcctgtcgg gtttcgccac 7260
ctctgacttg agcgtcgatt tttgtgatgc tcgtcagggg ggcggagcct atggaaaaac 7320
gccagcaacg cggccttttt acggttcctg gccttttgct ggccttttgc tcacatggtc 7380
tttcctgcgt tatcccctga ttctgtggat aaccgtatta ccgcctttga gtgagctgat 7440
accgctcgcc gcagccgaac gaccgagcgc agcgagtcag tgagcgagga agcggagagc 7500
gcccaatacg caaaccgcct ctccccgcgc gttggccgat tcattaatgc agctggcacg 7560
acaggtttcc cgactggaaa gcgggcagtg agcgcaacgc aattaatgtg agttagctca 7620
ctcattaggc accccaggct ttacacttta tgcttccggc tcgtatgttg tgtggaattg 7680
tgagcggata acaatttcac acaggaaaca gctatgacca tgattacgcc aagcgcgtca 7740
attaaccctc actaaaggga acaaaagctg ttaattaa 7778
<210> 66
<211> 8177
<212> DNA
<213> artificial sequence
<220>
<223> Matrice CD25 locus_IL12a_2A_IL12b pCLS30520 full sequence
<400> 66
gtttattatt cctgttccac agctattgtc tgccatataa aaacttaggc caggcacagt 60
ggctcacacc tgtaatccca gcactttgga aggccgaggc aggcagatca caaggtcagg 120
agttcgagac cagcctggcc aacatagcaa aaccccatct ctactaaaaa tacaaaaatt 180
agccaggcat ggtggcgtgt gcactggttt agagtgagga ccacattttt ttggtgccgt 240
gttacacata tgaccgtgac tttgttacac cactacagga ggaagagtag aagaacaatc 300
ggttctggcg tgaaacagac tttgaatttt gaccttctca agttggcggg agacgtggag 360
tccaacccag ggcccatgtg gccccctggg tcagcctccc agccaccgcc ctcacctgcc 420
gcggccacag gtctgcatcc agcggctcgc cctgtgtccc tgcagtgccg gctcagcatg 480
tgtccagcgc gcagcctcct ccttgtggct accctggtcc tcctggacca cctcagtttg 540
gccagaaacc tccccgtggc cactccagac ccaggaatgt tcccatgcct tcaccactcc 600
caaaacctgc tgagggccgt cagcaacatg ctccagaagg ccagacaaac tctagaattt 660
tacccttgca cttctgaaga gattgatcat gaagatatca caaaagataa aaccagcaca 720
gtggaggcct gtttaccatt ggaattaacc aagaatgaga gttgcctaaa ttccagagag 780
acctctttca taactaatgg gagttgcctg gcctccagaa agacctcttt tatgatggcc 840
ctgtgcctta gtagtattta tgaagacttg aagatgtacc aggtggagtt caagaccatg 900
aatgcaaagc ttctgatgga tcctaagagg cagatctttc tagatcaaaa catgctggca 960
gttattgatg agctgatgca ggccctgaat ttcaacagtg agactgtgcc acaaaaatcc 1020
tcccttgaag aaccggattt ttataaaact aaaatcaagc tctgcatact tcttcatgct 1080
ttcagaattc gggcagtgac tattgataga gtgatgagct atctgaatgc ttccggaagc 1140
ggagctacta acttcagcct gctgaagcag gctggagacg tggaggagaa ccctggacct 1200
atgtgtcacc agcagttggt catctcttgg ttttccctgg tttttctggc atctcccctc 1260
gtggccatat gggaactgaa gaaagatgtt tatgtcgtag aattggattg gtatccggat 1320
gcccctggag aaatggtggt cctcacctgt gacacccctg aagaagatgg tatcacctgg 1380
accttggacc agagcagtga ggtcttaggc tctggcaaaa ccctgaccat ccaagtcaaa 1440
gagtttggag atgctggcca gtacacctgt cacaaaggag gcgaggttct aagccattcg 1500
ctcctgctgc ttcacaaaaa ggaagatgga atttggtcca ctgatatttt aaaggaccag 1560
aaagaaccca aaaataagac ctttctaaga tgcgaggcca agaattattc tggacgtttc 1620
acctgctggt ggctgacgac aatcagtact gatttgacat tcagtgtcaa aagcagcaga 1680
ggctcttctg acccccaagg ggtgacgtgc ggagctgcta cactctctgc agagagagtc 1740
agaggggaca acaaggagta tgagtactca gtggagtgcc aggaggacag tgcctgccca 1800
gctgctgagg agagtctgcc cattgaggtc atggtggatg ccgttcacaa gctcaagtat 1860
gaaaactaca ccagcagctt cttcatcagg gacatcatca aacctgaccc acccaagaac 1920
ttgcagctga agccattaaa gaattctcgg caggtggagg tcagctggga gtaccctgac 1980
acctggagta ctccacattc ctacttctcc ctgacattct gcgttcaggt ccagggcaag 2040
agcaagagag aaaagaaaga tagagtcttc acggacaaga cctcagccac ggtcatctgc 2100
cgcaaaaatg ccagcattag cgtgcgggcc caggaccgct actatagctc atcttggagc 2160
gaatgggcat ctgtgccctg cagtgagggc agaggcagcc tgctgacctg cggcgacgtc 2220
gaggagaacc ccgggcccat gggggcaggt gccaccggcc gcgccatgga cgggccgcgc 2280
ctgctgctgt tgctgcttct gggggtgtcc cttggaggtg ccaaggaggc atgccccaca 2340
ggcctgtaca cacacagcgg tgagtgctgc aaagcctgca acctgggcga gggtgtggcc 2400
cagccttgtg gagccaacca gaccgtgtgt gagccctgcc tggacagcgt gacgttctcc 2460
gacgtggtga gcgcgaccga gccgtgcaag ccgtgcaccg agtgcgtggg gctccagagc 2520
atgtcggcgc cgtgcgtgga ggccgatgac gccgtgtgcc gctgcgccta cggctactac 2580
caggatgaga cgactgggcg ctgcgaggcg tgccgcgtgt gcgaggcggg ctcgggcctc 2640
gtgttctcct gccaggacaa gcagaacacc gtgtgcgagg agtgccccga cggcacgtat 2700
tccgacgagg ccaaccacgt ggacccgtgc ctgccctgca ccgtgtgcga ggacaccgag 2760
cgccagctcc gcgagtgcac acgctgggcc gacgccgagt gcgaggagat ccctggccgt 2820
tggattacac ggtccacacc cccagagggc tcggacagca cagcccccag cacccaggag 2880
cctgaggcac ctccagaaca agacctcata gccagcacgg tggcaggtgt ggtgaccaca 2940
gtgatgggca gctcccagcc cgtggtgacc cgaggcacca ccgacaacct catccctgtc 3000
tattgctcca tcctggctgc tgtggttgtg ggtcttgtgg cctacatagc cttcaagagg 3060
tgaaaaacca aaagaacaag aatttcttgg taagaagccg ggaacagaca acagaagtca 3120
tgaagcccaa gtgaaatcaa aggtgctaaa tggtcgccca ggagacatcc gttgtgcttg 3180
cctgcgtttt ggaagctctg aagtcacatc acaggacacg gggcagtggc aaccttgtct 3240
ctatgccagc tcagtcccat cagagagcga gcgctaccca cttctaaata gcaatttcgc 3300
cgttgaagag gaagggcaaa accactagaa ctctccatct tattttcatg tatatgtgtt 3360
catgcgatcg ctccggtgcc cgtcagtggg cagagcgcac atcgcccaca gtccccgaga 3420
agttgggggg aggggtcggc aattgaacgg gtgcctagag aaggtggcgc ggggtaaact 3480
gggaaagtga tgtcgtgtac tggctccgcc tttttcccga gggtggggga gaaccgtata 3540
taagtgcagt agtcgccgtg aacgttcttt ttcgcaacgg gtttgccgcc agaacacagc 3600
tgaagcttcg aggggctcgc atctctcctt cacgcgcccg ccgccctacc tgaggccgcc 3660
atccacgccg gttgagtcgc gttctgccgc ctcccgcctg tggtgcctcc tgaactgcgt 3720
ccgccgtcta ggtaagttta aagctcaggt cgagaccggg cctttgtccg gcgctccctt 3780
ggagcctacc tagactcagc cggctctcca cgctttgcct gaccctgctt gctcaactct 3840
acgtctttgt ttcgttttct gttctgcgcc gttacagatc caagctgtga ccggcgccta 3900
cctgagatca ccggcgccac catggcttct taccctggac accagcatgc ttctgccttt 3960
gaccaggctg ccagatccag gggccactcc aacaggagaa ctgccctaag acccagaaga 4020
cagcaggaag ccactgaggt gaggcctgag cagaagatgc caaccctgct gagggtgtac 4080
attgatggac ctcatggcat gggcaagacc accaccactc aactgctggt ggcactgggc 4140
tccagggatg acattgtgta tgtgcctgag ccaatgacct actggagagt gctaggagcc 4200
tctgagacca ttgccaacat ctacaccacc cagcacaggc tggaccaggg agaaatctct 4260
gctggagatg ctgctgtggt gatgacctct gcccagatca caatgggaat gccctatgct 4320
gtgactgatg ctgttctggc tcctcacatt ggaggagagg ctggctcttc tcatgcccct 4380
ccacctgccc tgaccctgat ctttgacaga caccccattg cagccctgct gtgctaccca 4440
gcagcaaggt acctcatggg ctccatgacc ccacaggctg tgctggcttt tgtggccctg 4500
atccctccaa ccctccctgg caccaacatt gttctgggag cactgcctga agacagacac 4560
attgacaggc tggcaaagag gcagagacct ggagagagac tggacctggc catgctggct 4620
gcaatcagaa gggtgtatgg actgctggca aacactgtga gatacctcca gtgtggaggc 4680
tcttggagag aggactgggg acagctctct ggaacagcag tgccccctca aggagctgag 4740
ccccagtcca atgctggtcc aagaccccac attggggaca ccctgttcac cctgttcaga 4800
gcccctgagc tgctggctcc caatggagac ctgtacaatg tgtttgcctg ggctctggat 4860
gttctagcca agaggctgag gtccatgcat gtgttcatcc tggactatga ccagtcccct 4920
gctggatgca gagatgctct gctgcaacta acctctggca tggtgcagac ccatgtgacc 4980
acccctggca gcatccccac catctgtgac ctagccagaa cctttgccag ggagatggga 5040
gaggccaact aaggcgcgcc actcgagcgc tagctggcca gacatgataa gatacattga 5100
tgagtttgga caaaccacaa ctagaatgca gtgaaaaaaa tgctttattt gtgaaatttg 5160
tgatgctatt gctttatttg taaccattat aagctgcaat aaacaagtta acaacaacaa 5220
ttgcattcat tttatgtttc aggttcaggg ggaggtgtgg gaggtttttt aaagcaagta 5280
aaacctctac aaatgtggta tggaaggcgc gcccaattcg ccctatagtg agtcgtatta 5340
cgtcgcgctc actggccgtc gttttacaac gtcgtgactg ggaaaaccct ggcgttaccc 5400
aacttaatcg ccttgcagca catccccctt tcgccagctg gcgtaatagc gaagaggccc 5460
gcaccgaaac gcccttccca acagttgcgc agcctgaatg gcgaatggga gcgccctgta 5520
gcggcgcatt aagcgcggcg ggtgtggtgg ttacgcgcag cgtgaccgct acacttgcca 5580
gcgccctagc gcccgctcct ttcgctttct tcccttcctt tctcgccacg ttcgccggct 5640
ttccccgtca agctctaaat cgggggctcc ctttagggtt ccgatttagt gctttacggc 5700
acctcgaccc caaaaaactt gattagggtg atggttggcc tgtagtgggc catagccctg 5760
atagacggtt tttcgccctt tgacgttgga gtccacgttc tttaatagtg gactcttgtt 5820
ccaaactgga acaacactca accctatctc ggtctattct tttgatttat aagggatttt 5880
gccgatttcg gcctattggt taaaaaatga gctgatttaa caaaaattta acgcgaattt 5940
taacaaaata ttaacgctta caatttaggt ggcacttttc ggggaaatgt gcgcggaacc 6000
cctatttgtt tatttttcta aatacattca aatatgtatc cgctcatgag acaataaccc 6060
tgataaatgc ttcaataata ttgaaaaagg aagagtatga gtattcaaca tttccgtgtc 6120
gcccttattc ccttttttgc ggcattttgc cttcctgttt ttgctcaccc agaaacgctg 6180
gtgaaagtaa aagatgctga agatcagttg ggtgcacgag tgggttacat cgaactggat 6240
ctcaacagcg gtaagatcct tgagagtttt cgccccgaag aacgttttcc aatgatgagc 6300
acttttaaag ttctgctatg tggcgcggta ttatcccgta ttgacgccgg gcaagagcaa 6360
ctcggtcgcc gcatacacta ttctcagaat gacttggttg agtactcacc agtcacagaa 6420
aagcatctta cggatggcat gacagtaaga gaattatgca gtgctgccat aaccatgagt 6480
gataacactg cggccaactt acttctgaca acgatcggag gaccgaagga gctaaccgct 6540
tttttgcaca acatggggga tcatgtaact cgccttgatc gttgggaacc ggagctgaat 6600
gaagccatac caaacgacga gcgtgacacc acgatgcctg tagcaatggc aacaacgttg 6660
cgcaaactat taactggcga actacttact ctagcttccc ggcaacaatt aatagactgg 6720
atggaggcgg ataaagttgc aggaccactt ctgcgctcgg cccttccggc tggctggttt 6780
attgctgata aatctggagc cggtgagcgt ggttctcgcg gtatcattgc agcactgggg 6840
ccagatggta agccctcccg tatcgtagtt atctacacga cggggagtca ggcaactatg 6900
gatgaacgaa atagacagat cgctgagata ggtgcctcac tgattaagca ttggtaactg 6960
tcagaccaag tttactcata tatactttag attgatttaa aacttcattt ttaatttaaa 7020
aggatctagg tgaagatcct ttttgataat ctcatgacca aaatccctta acgtgagttt 7080
tcgttccact gagcgtcaga ccccgtagaa aagatcaaag gatcttcttg agatcctttt 7140
tttctgcgcg taatctgctg cttgcaaaca aaaaaaccac cgctaccagc ggtggtttgt 7200
ttgccggatc aagagctacc aactcttttt ccgaaggtaa ctggcttcag cagagcgcag 7260
ataccaaata ctgttcttct agtgtagccg tagttaggcc accacttcaa gaactctgta 7320
gcaccgccta catacctcgc tctgctaatc ctgttaccag tggctgctgc cagtggcgat 7380
aagtcgtgtc ttaccgggtt ggactcaaga cgatagttac cggataaggc gcagcggtcg 7440
ggctgaacgg ggggttcgtg cacacagccc agcttggagc gaacgaccta caccgaactg 7500
agatacctac agcgtgagct atgagaaagc gccacgcttc ccgaagggag aaaggcggac 7560
aggtatccgg taagcggcag ggtcggaaca ggagagcgca cgagggagct tccaggggga 7620
aacgcctggt atctttatag tcctgtcggg tttcgccacc tctgacttga gcgtcgattt 7680
ttgtgatgct cgtcaggggg gcggagccta tggaaaaacg ccagcaacgc ggccttttta 7740
cggttcctgg ccttttgctg gccttttgct cacatggtct ttcctgcgtt atcccctgat 7800
tctgtggata accgtattac cgcctttgag tgagctgata ccgctcgccg cagccgaacg 7860
accgagcgca gcgagtcagt gagcgaggaa gcggagagcg cccaatacgc aaaccgcctc 7920
tccccgcgcg ttggccgatt cattaatgca gctggcacga caggtttccc gactggaaag 7980
cgggcagtga gcgcaacgca attaatgtga gttagctcac tcattaggca ccccaggctt 8040
tacactttat gcttccggct cgtatgttgt gtggaattgt gagcggataa caatttcaca 8100
caggaaacag ctatgaccat gattacgcca agcgcgtcaa ttaaccctca ctaaagggaa 8160
caaaagctgt taattaa 8177
<210> 67
<211> 6349
<212> DNA
<213> artificial sequence
<220>
<223> Matrice PD1 locus_IL12a_2A_IL12b pCLS30511 full sequence
<400> 67
tcgcgcgttt cggtgatgac ggtgaaaacc tctgacacat gcagctcccg gagacggtca 60
cagcttgtct gtaagcggat gccgggagca gacaagcccg tcagggcgcg tcagcgggtg 120
ttggcgggtg tcggggctgg cttaactatg cggcatcaga gcagattgta ctgagagtgc 180
accatatgcg gtgtgaaata ccgcacagat gcgtaaggag aaaataccgc atcaggcgcc 240
attcgccatt caggctgcgc aactgttggg aagggcgatc ggtgcgggcc tcttcgctat 300
tacgccagct ggcgaaaggg ggatgtgctg caaggcgatt aagttgggta acgccagggt 360
tttcccagtc acgacgttgt aaaacgacgg ccagtgaatt cgagctcggt acctcgcgaa 420
tgcatctaga tgactcccca gacaggccct ggaacccccc caccttctcc ccagccctgc 480
tcgtggtgac cgaaggggac aacgccacct tcacctgcag cttctccaac acatcggaga 540
gcttcgtgct aaactggtac cgcatgagcc ccagcaacca gacggacaag ctggccgcct 600
tccccgagga ccgcagccag cccggccagg actgccgctt ccgtgtcaca caactgccca 660
acgggcgtga cttccacatg agcgtggtca gggcccggcg caatgacagc ggcacctacc 720
tctgtggggc cggttctggc gtgaaacaga ctttgaattt tgaccttctc aagttggcgg 780
gagacgtgga gtccaaccca gggcccatgt ggccccctgg gtcagcctcc cagccaccgc 840
cctcacctgc cgcggccaca ggtctgcatc cagcggctcg ccctgtgtcc ctgcagtgcc 900
ggctcagcat gtgtccagcg cgcagcctcc tccttgtggc taccctggtc ctcctggacc 960
acctcagttt ggccagaaac ctccccgtgg ccactccaga cccaggaatg ttcccatgcc 1020
ttcaccactc ccaaaacctg ctgagggccg tcagcaacat gctccagaag gccagacaaa 1080
ctctagaatt ttacccttgc acttctgaag agattgatca tgaagatatc acaaaagata 1140
aaaccagcac agtggaggcc tgtttaccat tggaattaac caagaatgag agttgcctaa 1200
attccagaga gacctctttc ataactaatg ggagttgcct ggcctccaga aagacctctt 1260
ttatgatggc cctgtgcctt agtagtattt atgaagactt gaagatgtac caggtggagt 1320
tcaagaccat gaatgcaaag cttctgatgg atcctaagag gcagatcttt ctagatcaaa 1380
acatgctggc agttattgat gagctgatgc aggccctgaa tttcaacagt gagactgtgc 1440
cacaaaaatc ctcccttgaa gaaccggatt tttataaaac taaaatcaag ctctgcatac 1500
ttcttcatgc tttcagaatt cgggcagtga ctattgatag agtgatgagc tatctgaatg 1560
cttccggaag cggagctact aacttcagcc tgctgaagca ggctggagac gtggaggaga 1620
accctggacc tatgtgtcac cagcagttgg tcatctcttg gttttccctg gtttttctgg 1680
catctcccct cgtggccata tgggaactga agaaagatgt ttatgtcgta gaattggatt 1740
ggtatccgga tgcccctgga gaaatggtgg tcctcacctg tgacacccct gaagaagatg 1800
gtatcacctg gaccttggac cagagcagtg aggtcttagg ctctggcaaa accctgacca 1860
tccaagtcaa agagtttgga gatgctggcc agtacacctg tcacaaagga ggcgaggttc 1920
taagccattc gctcctgctg cttcacaaaa aggaagatgg aatttggtcc actgatattt 1980
taaaggacca gaaagaaccc aaaaataaga cctttctaag atgcgaggcc aagaattatt 2040
ctggacgttt cacctgctgg tggctgacga caatcagtac tgatttgaca ttcagtgtca 2100
aaagcagcag aggctcttct gacccccaag gggtgacgtg cggagctgct acactctctg 2160
cagagagagt cagaggggac aacaaggagt atgagtactc agtggagtgc caggaggaca 2220
gtgcctgccc agctgctgag gagagtctgc ccattgaggt catggtggat gccgttcaca 2280
agctcaagta tgaaaactac accagcagct tcttcatcag ggacatcatc aaacctgacc 2340
cacccaagaa cttgcagctg aagccattaa agaattctcg gcaggtggag gtcagctggg 2400
agtaccctga cacctggagt actccacatt cctacttctc cctgacattc tgcgttcagg 2460
tccagggcaa gagcaagaga gaaaagaaag atagagtctt cacggacaag acctcagcca 2520
cggtcatctg ccgcaaaaat gccagcatta gcgtgcgggc ccaggaccgc tactatagct 2580
catcttggag cgaatgggca tctgtgccct gcagtgaggg cagaggcagc ctgctgacct 2640
gcggcgacgt cgaggagaac cccgggccca tgggggcagg tgccaccggc cgcgccatgg 2700
acgggccgcg cctgctgctg ttgctgcttc tgggggtgtc ccttggaggt gccaaggagg 2760
catgccccac aggcctgtac acacacagcg gtgagtgctg caaagcctgc aacctgggcg 2820
agggtgtggc ccagccttgt ggagccaacc agaccgtgtg tgagccctgc ctggacagcg 2880
tgacgttctc cgacgtggtg agcgcgaccg agccgtgcaa gccgtgcacc gagtgcgtgg 2940
ggctccagag catgtcggcg ccgtgcgtgg aggccgatga cgccgtgtgc cgctgcgcct 3000
acggctacta ccaggatgag acgactgggc gctgcgaggc gtgccgcgtg tgcgaggcgg 3060
gctcgggcct cgtgttctcc tgccaggaca agcagaacac cgtgtgcgag gagtgccccg 3120
acggcacgta ttccgacgag gccaaccacg tggacccgtg cctgccctgc accgtgtgcg 3180
aggacaccga gcgccagctc cgcgagtgca cacgctgggc cgacgccgag tgcgaggaga 3240
tccctggccg ttggattaca cggtccacac ccccagaggg ctcggacagc acagccccca 3300
gcacccagga gcctgaggca cctccagaac aagacctcat agccagcacg gtggcaggtg 3360
tggtgaccac agtgatgggc agctcccagc ccgtggtgac ccgaggcacc accgacaacc 3420
tcatccctgt ctattgctcc atcctggctg ctgtggttgt gggtcttgtg gcctacatag 3480
ccttcaagag gtgatctaga gggcccgttt aaacccgctg atcagcctcg actgtgcctt 3540
ctagttgcca gccatctgtt gtttgcccct cccccgtgcc ttccttgacc ctggaaggtg 3600
ccactcccac tgtcctttcc taataaaatg aggaaattgc atcgcattgt ctgagtaggt 3660
gtcattctat tctggggggt ggggtggggc aggacagcaa gggggaggat tgggaagaca 3720
atagcaggca tgctggggat gcggtgggct ctatgactag tggcgaattc ggcgcagatc 3780
aaagagagcc tgcgggcaga gctcagggtg acaggtgcgg cctcggaggc cccggggcag 3840
gggtgagctg agccggtcct ggggtgggtg tcccctcctg cacaggatca ggagctccag 3900
ggtcgtaggg cagggacccc ccagctccag tccagggctc tgtcctgcac ctggggaatg 3960
gtgaccggca tctctgtcct ctagctctgg aagcacccca gcccctctag tctgccctca 4020
cccctgaccc tgaccctcca ccctgacccc gtcctaaccc ctgacctttg atcggatccc 4080
gggcccgtcg actgcagagg cctgcatgca agcttggcgt aatcatggtc atagctgttt 4140
cctgtgtgaa attgttatcc gctcacaatt ccacacaaca tacgagccgg aagcataaag 4200
tgtaaagcct ggggtgccta atgagtgagc taactcacat taattgcgtt gcgctcactg 4260
cccgctttcc agtcgggaaa cctgtcgtgc cagctgcatt aatgaatcgg ccaacgcgcg 4320
gggagaggcg gtttgcgtat tgggcgctct tccgcttcct cgctcactga ctcgctgcgc 4380
tcggtcgttc ggctgcggcg agcggtatca gctcactcaa aggcggtaat acggttatcc 4440
acagaatcag gggataacgc aggaaagaac atgtgagcaa aaggccagca aaaggccagg 4500
aaccgtaaaa aggccgcgtt gctggcgttt ttccataggc tccgcccccc tgacgagcat 4560
cacaaaaatc gacgctcaag tcagaggtgg cgaaacccga caggactata aagataccag 4620
gcgtttcccc ctggaagctc cctcgtgcgc tctcctgttc cgaccctgcc gcttaccgga 4680
tacctgtccg cctttctccc ttcgggaagc gtggcgcttt ctcatagctc acgctgtagg 4740
tatctcagtt cggtgtaggt cgttcgctcc aagctgggct gtgtgcacga accccccgtt 4800
cagcccgacc gctgcgcctt atccggtaac tatcgtcttg agtccaaccc ggtaagacac 4860
gacttatcgc cactggcagc agccactggt aacaggatta gcagagcgag gtatgtaggc 4920
ggtgctacag agttcttgaa gtggtggcct aactacggct acactagaag aacagtattt 4980
ggtatctgcg ctctgctgaa gccagttacc ttcggaaaaa gagttggtag ctcttgatcc 5040
ggcaaacaaa ccaccgctgg tagcggtggt ttttttgttt gcaagcagca gattacgcgc 5100
agaaaaaaag gatctcaaga agatcctttg atcttttcta cggggtctga cgctcagtgg 5160
aacgaaaact cacgttaagg gattttggtc atgagattat caaaaaggat cttcacctag 5220
atccttttaa attaaaaatg aagttttaaa tcaatctaaa gtatatatga gtaaacttgg 5280
tctgacagtt accaatgctt aatcagtgag gcacctatct cagcgatctg tctatttcgt 5340
tcatccatag ttgcctgact ccccgtcgtg tagataacta cgatacggga gggcttacca 5400
tctggcccca gtgctgcaat gataccgcga gacccacgct caccggctcc agatttatca 5460
gcaataaacc agccagccgg aagggccgag cgcagaagtg gtcctgcaac tttatccgcc 5520
tccatccagt ctattaattg ttgccgggaa gctagagtaa gtagttcgcc agttaatagt 5580
ttgcgcaacg ttgttgccat tgctacaggc atcgtggtgt cacgctcgtc gtttggtatg 5640
gcttcattca gctccggttc ccaacgatca aggcgagtta catgatcccc catgttgtgc 5700
aaaaaagcgg ttagctcctt cggtcctccg atcgttgtca gaagtaagtt ggccgcagtg 5760
ttatcactca tggttatggc agcactgcat aattctctta ctgtcatgcc atccgtaaga 5820
tgcttttctg tgactggtga gtactcaacc aagtcattct gagaatagtg tatgcggcga 5880
ccgagttgct cttgcccggc gtcaatacgg gataataccg cgccacatag cagaacttta 5940
aaagtgctca tcattggaaa acgttcttcg gggcgaaaac tctcaaggat cttaccgctg 6000
ttgagatcca gttcgatgta acccactcgt gcacccaact gatcttcagc atcttttact 6060
ttcaccagcg tttctgggtg agcaaaaaca ggaaggcaaa atgccgcaaa aaagggaata 6120
agggcgacac ggaaatgttg aatactcata ctcttccttt ttcaatatta ttgaagcatt 6180
tatcagggtt attgtctcat gagcggatac atatttgaat gtatttagaa aaataaacaa 6240
ataggggttc cgcgcacatt tccccgaaaa gtgccacctg acgtctaaga aaccattatt 6300
atcatgacat taacctataa aaataggcgt atcacgaggc cctttcgtc 6349
<210> 68
<211> 2489
<212> DNA
<213> artificial sequence
<220>
<223> HLAE trimer matrix (VMAPRTLFL peptide) inserted at the B2m locus
<400> 68
cacttagcat ctctggggcc agtctgcaaa gcgagggggc agccttaatg tgcctccagc 60
ctgaagtcct agaatgagcg cccggtgtcc caagctgggg cgcgcacccc agatcggagg 120
gcgccgatgt acagacagca aactcaccca gtctagtgca tgccttctta aacatcacga 180
gactctaaga aaaggaaact gaaaacggga aagtccctct ctctaacctg gcactgcgtc 240
gctggcttgg agacaggtga cggtccctgc gggccttgtc ctgattggct gggcacgcgt 300
ttaatataag tggaggcgtc gcgctggcgg gcattcctga agctgacagc attcgggccg 360
agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact ctctcttagc ggcctcgaag 420
ctgttatggc tccgcggact ttattcttag gtggtggcgg atccggtggt ggcggttctg 480
gtggtggcgg ctccatccag cgtacgccca aaattcaagt ctacagccga catcctgcag 540
agaacggcaa atctaatttc ctgaactgct atgtatcagg ctttcaccct agcgatatag 600
aagtggacct gctgaaaaac ggagagagga tagaaaaggt cgaacacagc gacctctcct 660
tttccaagga ctggagcttt tatcttctgt attatactga atttacaccc acggaaaaag 720
atgagtatgc gtgccgagta aaccacgtca cgctgtcaca gcccaaaata gtaaaatggg 780
atcgcgacat gggtggtggc ggttctggtg gtggcggtag tggcggcgga ggaagcggtg 840
gtggcggttc cggatctcac tccttgaagt atttccacac ttccgtgtcc cggcccggcc 900
gcggggagcc ccgcttcatc tctgtgggct acgtggacga cacccagttc gtgcgcttcg 960
acaacgacgc cgcgagtccg aggatggtgc cgcgggcgcc gtggatggag caggaggggt 1020
cagagtattg ggaccgggag acacggagcg ccagggacac cgcacagatt ttccgagtga 1080
acctgcggac gctgcgcggc tactacaatc agagcgaggc cgggtctcac accctgcagt 1140
ggatgcatgg ctgcgagctg gggcccgaca ggcgcttcct ccgcgggtat gaacagttcg 1200
cctacgacgg caaggattat ctcaccctga atgaggacct gcgctcctgg accgcggtgg 1260
acacggcggc tcagatctcc gagcaaaagt caaatgatgc ctctgaggcg gagcaccaga 1320
gagcctacct ggaagacaca tgcgtggagt ggctccacaa atacctggag aaggggaagg 1380
agacgctgct tcacctggag cccccaaaga cacacgtgac tcaccacccc atctctgacc 1440
atgaggccac cctgaggtgc tgggctctgg gcttctaccc tgcggagatc acactgacct 1500
ggcagcagga tggggagggc catacccagg acacggagct cgtggagacc aggcctgcag 1560
gggatggaac cttccagaag tgggcagctg tggtggtgcc ttctggagag gagcagagat 1620
acacgtgcca tgtgcagcat gaggggctac ccgagcccgt caccctgaga tggaagccgg 1680
cttcccagcc caccatcccc atcgtgggca tcattgctgg cctggttctc cttggatctg 1740
tggtctctgg agctgtggtt gctgctgtga tatggaggaa gaagagctca ggtggaaaag 1800
gagggagcta ctataaggct gagtggagcg acagtgccca ggggtctgag tctcacagct 1860
tgtaactgtg ccttctagtt gccagccatc tgttgtttgc ccctcccccg tgccttcctt 1920
gaccctggaa ggtgccactc ccactgtcct ttcctaataa aatgaggaaa ttgcatcgca 1980
ttgtctgagt aggtgtcatt ctattctggg gggtggggtg gggcaggaca gcaaggggga 2040
ggattgggaa gacaatagca ggcatgctgg ggatgcggtg ggctctatgt ctctttctgg 2100
cctggaggct atccagcgtg agtctctcct accctcccgc tctggtcctt cctctcccgc 2160
tctgcaccct ctgtggccct cgctgtgctc tctcgctccg tgacttccct tctccaagtt 2220
ctccttggtg gcccgccgtg gggctagtcc agggctggat ctcggggaag cggcggggtg 2280
gcctgggagt ggggaagggg gtgcgcaccc gggacgcgcg ctacttgccc ctttcggcgg 2340
ggagcagggg agacctttgg cctacggcga cgggagggtc gggacaaagt ttagggcgtc 2400
gataagcgtc agagcgccga ggttggggga gggtttctct tccgctcttt cgcggggcct 2460
ctggctcccc cagcgcagct ggagtgggg 2489
<210> 69
<211> 2289
<212> DNA
<213> artificial sequence
<220>
<223> HLAE trimer matrix (VMAPRTLFL peptide)
<400> 69
cgcgcacccc agatcggagg gcgccgatgt acagacagca aactcaccca gtctagtgca 60
tgccttctta aacatcacga gactctaaga aaaggaaact gaaaacggga aagtccctct 120
ctctaacctg gcactgcgtc gctggcttgg agacaggtga cggtccctgc gggccttgtc 180
ctgattggct gggcacgcgt ttaatataag tggaggcgtc gcgctggcgg gcattcctga 240
agctgacagc attcgggccg agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact 300
ctctcttagc ggcctcgaag ctgttatggc tccgcggact ttattcttag gtggtggcgg 360
atccggtggt ggcggttctg gtggtggcgg ctccatccag cgtacgccca aaattcaagt 420
ctacagccga catcctgcag agaacggcaa atctaatttc ctgaactgct atgtatcagg 480
ctttcaccct agcgatatag aagtggacct gctgaaaaac ggagagagga tagaaaaggt 540
cgaacacagc gacctctcct tttccaagga ctggagcttt tatcttctgt attatactga 600
atttacaccc acggaaaaag atgagtatgc gtgccgagta aaccacgtca cgctgtcaca 660
gcccaaaata gtaaaatggg atcgcgacat gggtggtggc ggttctggtg gtggcggtag 720
tggcggcgga ggaagcggtg gtggcggttc cggatctcac tccttgaagt atttccacac 780
ttccgtgtcc cggcccggcc gcggggagcc ccgcttcatc tctgtgggct acgtggacga 840
cacccagttc gtgcgcttcg acaacgacgc cgcgagtccg aggatggtgc cgcgggcgcc 900
gtggatggag caggaggggt cagagtattg ggaccgggag acacggagcg ccagggacac 960
cgcacagatt ttccgagtga acctgcggac gctgcgcggc tactacaatc agagcgaggc 1020
cgggtctcac accctgcagt ggatgcatgg ctgcgagctg gggcccgaca ggcgcttcct 1080
ccgcgggtat gaacagttcg cctacgacgg caaggattat ctcaccctga atgaggacct 1140
gcgctcctgg accgcggtgg acacggcggc tcagatctcc gagcaaaagt caaatgatgc 1200
ctctgaggcg gagcaccaga gagcctacct ggaagacaca tgcgtggagt ggctccacaa 1260
atacctggag aaggggaagg agacgctgct tcacctggag cccccaaaga cacacgtgac 1320
tcaccacccc atctctgacc atgaggccac cctgaggtgc tgggctctgg gcttctaccc 1380
tgcggagatc acactgacct ggcagcagga tggggagggc catacccagg acacggagct 1440
cgtggagacc aggcctgcag gggatggaac cttccagaag tgggcagctg tggtggtgcc 1500
ttctggagag gagcagagat acacgtgcca tgtgcagcat gaggggctac ccgagcccgt 1560
caccctgaga tggaagccgg cttcccagcc caccatcccc atcgtgggca tcattgctgg 1620
cctggttctc cttggatctg tggtctctgg agctgtggtt gctgctgtga tatggaggaa 1680
gaagagctca ggtggaaaag gagggagcta ctataaggct gagtggagcg acagtgccca 1740
ggggtctgag tctcacagct tgtaactgtg ccttctagtt gccagccatc tgttgtttgc 1800
ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct ttcctaataa 1860
aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg gggtggggtg 1920
gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg ggatgcggtg 1980
ggctctatgt ctctttctgg cctggaggct atccagcgtg agtctctcct accctcccgc 2040
tctggtcctt cctctcccgc tctgcaccct ctgtggccct cgctgtgctc tctcgctccg 2100
tgacttccct tctccaagtt ctccttggtg gcccgccgtg gggctagtcc agggctggat 2160
ctcggggaag cggcggggtg gcctgggagt ggggaagggg gtgcgcaccc gggacgcgcg 2220
ctacttgccc ctttcggcgg ggagcagggg agacctttgg cctacggcga cgggagggtc 2280
gggacaaag 2289
<210> 70
<211> 2489
<212> DNA
<213> artificial sequence
<220>
<223> HLAE trimer matrix (VMAPRTLIL peptide) inserted at the B2m locus
<400> 70
cacttagcat ctctggggcc agtctgcaaa gcgagggggc agccttaatg tgcctccagc 60
ctgaagtcct agaatgagcg cccggtgtcc caagctgggg cgcgcacccc agatcggagg 120
gcgccgatgt acagacagca aactcaccca gtctagtgca tgccttctta aacatcacga 180
gactctaaga aaaggaaact gaaaacggga aagtccctct ctctaacctg gcactgcgtc 240
gctggcttgg agacaggtga cggtccctgc gggccttgtc ctgattggct gggcacgcgt 300
ttaatataag tggaggcgtc gcgctggcgg gcattcctga agctgacagc attcgggccg 360
agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact ctctcttagc ggcctcgaag 420
ctgttatggc tccgcggact ttaattttag gtggtggcgg atccggtggt ggcggttctg 480
gtggtggcgg ctccatccag cgtacgccca aaattcaagt ctacagccga catcctgcag 540
agaacggcaa atctaatttc ctgaactgct atgtatcagg ctttcaccct agcgatatag 600
aagtggacct gctgaaaaac ggagagagga tagaaaaggt cgaacacagc gacctctcct 660
tttccaagga ctggagcttt tatcttctgt attatactga atttacaccc acggaaaaag 720
atgagtatgc gtgccgagta aaccacgtca cgctgtcaca gcccaaaata gtaaaatggg 780
atcgcgacat gggtggtggc ggttctggtg gtggcggtag tggcggcgga ggaagcggtg 840
gtggcggttc cggatctcac tccttgaagt atttccacac ttccgtgtcc cggcccggcc 900
gcggggagcc ccgcttcatc tctgtgggct acgtggacga cacccagttc gtgcgcttcg 960
acaacgacgc cgcgagtccg aggatggtgc cgcgggcgcc gtggatggag caggaggggt 1020
cagagtattg ggaccgggag acacggagcg ccagggacac cgcacagatt ttccgagtga 1080
acctgcggac gctgcgcggc tactacaatc agagcgaggc cgggtctcac accctgcagt 1140
ggatgcatgg ctgcgagctg gggcccgaca ggcgcttcct ccgcgggtat gaacagttcg 1200
cctacgacgg caaggattat ctcaccctga atgaggacct gcgctcctgg accgcggtgg 1260
acacggcggc tcagatctcc gagcaaaagt caaatgatgc ctctgaggcg gagcaccaga 1320
gagcctacct ggaagacaca tgcgtggagt ggctccacaa atacctggag aaggggaagg 1380
agacgctgct tcacctggag cccccaaaga cacacgtgac tcaccacccc atctctgacc 1440
atgaggccac cctgaggtgc tgggctctgg gcttctaccc tgcggagatc acactgacct 1500
ggcagcagga tggggagggc catacccagg acacggagct cgtggagacc aggcctgcag 1560
gggatggaac cttccagaag tgggcagctg tggtggtgcc ttctggagag gagcagagat 1620
acacgtgcca tgtgcagcat gaggggctac ccgagcccgt caccctgaga tggaagccgg 1680
cttcccagcc caccatcccc atcgtgggca tcattgctgg cctggttctc cttggatctg 1740
tggtctctgg agctgtggtt gctgctgtga tatggaggaa gaagagctca ggtggaaaag 1800
gagggagcta ctataaggct gagtggagcg acagtgccca ggggtctgag tctcacagct 1860
tgtaactgtg ccttctagtt gccagccatc tgttgtttgc ccctcccccg tgccttcctt 1920
gaccctggaa ggtgccactc ccactgtcct ttcctaataa aatgaggaaa ttgcatcgca 1980
ttgtctgagt aggtgtcatt ctattctggg gggtggggtg gggcaggaca gcaaggggga 2040
ggattgggaa gacaatagca ggcatgctgg ggatgcggtg ggctctatgt ctctttctgg 2100
cctggaggct atccagcgtg agtctctcct accctcccgc tctggtcctt cctctcccgc 2160
tctgcaccct ctgtggccct cgctgtgctc tctcgctccg tgacttccct tctccaagtt 2220
ctccttggtg gcccgccgtg gggctagtcc agggctggat ctcggggaag cggcggggtg 2280
gcctgggagt ggggaagggg gtgcgcaccc gggacgcgcg ctacttgccc ctttcggcgg 2340
ggagcagggg agacctttgg cctacggcga cgggagggtc gggacaaagt ttagggcgtc 2400
gataagcgtc agagcgccga ggttggggga gggtttctct tccgctcttt cgcggggcct 2460
ctggctcccc cagcgcagct ggagtgggg 2489
<210> 71
<211> 2289
<212> DNA
<213> artificial sequence
<220>
<223> HLAE trimer matrix (VMAPRTLIL peptide)
<400> 71
cgcgcacccc agatcggagg gcgccgatgt acagacagca aactcaccca gtctagtgca 60
tgccttctta aacatcacga gactctaaga aaaggaaact gaaaacggga aagtccctct 120
ctctaacctg gcactgcgtc gctggcttgg agacaggtga cggtccctgc gggccttgtc 180
ctgattggct gggcacgcgt ttaatataag tggaggcgtc gcgctggcgg gcattcctga 240
agctgacagc attcgggccg agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact 300
ctctcttagc ggcctcgaag ctgttatggc tccgcggact ttaattttag gtggtggcgg 360
atccggtggt ggcggttctg gtggtggcgg ctccatccag cgtacgccca aaattcaagt 420
ctacagccga catcctgcag agaacggcaa atctaatttc ctgaactgct atgtatcagg 480
ctttcaccct agcgatatag aagtggacct gctgaaaaac ggagagagga tagaaaaggt 540
cgaacacagc gacctctcct tttccaagga ctggagcttt tatcttctgt attatactga 600
atttacaccc acggaaaaag atgagtatgc gtgccgagta aaccacgtca cgctgtcaca 660
gcccaaaata gtaaaatggg atcgcgacat gggtggtggc ggttctggtg gtggcggtag 720
tggcggcgga ggaagcggtg gtggcggttc cggatctcac tccttgaagt atttccacac 780
ttccgtgtcc cggcccggcc gcggggagcc ccgcttcatc tctgtgggct acgtggacga 840
cacccagttc gtgcgcttcg acaacgacgc cgcgagtccg aggatggtgc cgcgggcgcc 900
gtggatggag caggaggggt cagagtattg ggaccgggag acacggagcg ccagggacac 960
cgcacagatt ttccgagtga acctgcggac gctgcgcggc tactacaatc agagcgaggc 1020
cgggtctcac accctgcagt ggatgcatgg ctgcgagctg gggcccgaca ggcgcttcct 1080
ccgcgggtat gaacagttcg cctacgacgg caaggattat ctcaccctga atgaggacct 1140
gcgctcctgg accgcggtgg acacggcggc tcagatctcc gagcaaaagt caaatgatgc 1200
ctctgaggcg gagcaccaga gagcctacct ggaagacaca tgcgtggagt ggctccacaa 1260
atacctggag aaggggaagg agacgctgct tcacctggag cccccaaaga cacacgtgac 1320
tcaccacccc atctctgacc atgaggccac cctgaggtgc tgggctctgg gcttctaccc 1380
tgcggagatc acactgacct ggcagcagga tggggagggc catacccagg acacggagct 1440
cgtggagacc aggcctgcag gggatggaac cttccagaag tgggcagctg tggtggtgcc 1500
ttctggagag gagcagagat acacgtgcca tgtgcagcat gaggggctac ccgagcccgt 1560
caccctgaga tggaagccgg cttcccagcc caccatcccc atcgtgggca tcattgctgg 1620
cctggttctc cttggatctg tggtctctgg agctgtggtt gctgctgtga tatggaggaa 1680
gaagagctca ggtggaaaag gagggagcta ctataaggct gagtggagcg acagtgccca 1740
ggggtctgag tctcacagct tgtaactgtg ccttctagtt gccagccatc tgttgtttgc 1800
ccctcccccg tgccttcctt gaccctggaa ggtgccactc ccactgtcct ttcctaataa 1860
aatgaggaaa ttgcatcgca ttgtctgagt aggtgtcatt ctattctggg gggtggggtg 1920
gggcaggaca gcaaggggga ggattgggaa gacaatagca ggcatgctgg ggatgcggtg 1980
ggctctatgt ctctttctgg cctggaggct atccagcgtg agtctctcct accctcccgc 2040
tctggtcctt cctctcccgc tctgcaccct ctgtggccct cgctgtgctc tctcgctccg 2100
tgacttccct tctccaagtt ctccttggtg gcccgccgtg gggctagtcc agggctggat 2160
ctcggggaag cggcggggtg gcctgggagt ggggaagggg gtgcgcaccc gggacgcgcg 2220
ctacttgccc ctttcggcgg ggagcagggg agacctttgg cctacggcga cgggagggtc 2280
gggacaaag 2289
<210> 72
<211> 2531
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _Actine peptide inserted at the B2m locus
<400> 72
cacttagcat ctctggggcc agtctgcaaa gcgagggggc agccttaatg tgcctccagc 60
ctgaagtcct agaatgagcg cccggtgtcc caagctgggg cgcgcacccc agatcggagg 120
gcgccgatgt acagacagca aactcaccca gtctagtgca tgccttctta aacatcacga 180
gactctaaga aaaggaaact gaaaacggga aagtccctct ctctaacctg gcactgcgtc 240
gctggcttgg agacaggtga cggtccctgc gggccttgtc ctgattggct gggcacgcgt 300
ttaatataag tggaggcgtc gcgctggcgg gcattcctga agctgacagc attcgggccg 360
agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact ctctcttagc ggcctcgaag 420
ctgccctgcc ccacgccatt ttgcggctcg gtggtggcgg atccggtggt ggcggttctg 480
gtggtggcgg ctccatccag cgtacgccca aaattcaagt ctacagccga catcctgcag 540
agaacggcaa atctaatttc ctgaactgct atgtatcagg ctttcaccct agcgatatag 600
aagtggacct gctgaaaaac ggagagagga tagaaaaggt cgaacacagc gacctctcct 660
tttccaagga ctggagcttt tatcttctgt attatactga atttacaccc acggaaaaag 720
atgagtatgc gtgccgagta aaccacgtca cgctgtcaca gcccaaaata gtaaaatggg 780
atcgcgacat gggtggtggc ggttctggtg gtggcggtag tggcggcgga ggaagcggtg 840
gtggcggttc cggatctatg cacgtgctga gatacggata taccggcatc ttcgacgata 900
catcccatat gactctgacc gtggtcggga tttttgacgg acagcacttc tttacatacc 960
atgtgaacag ctccgataag gcttctagtc gagcaaatgg caccatctca tggatggcca 1020
acgtgagcgc agcctacccc acatatctgg acggagaacg cgctaaaggc gatctgatct 1080
tcaatcagac cgagcagaac ctgctggagc tggaaattgc tctggggtac aggtctcaga 1140
gtgtcctgac atggactcac gaatgtaata ccacagagaa cgggagcttc gtggcaggat 1200
atgagggctt tgggtgggac ggagaaacac tgatggagct gaaggataat ctgactctgt 1260
ggaccggccc taactacgaa atcagctggc tgaagcagaa caagacttac atcgacggaa 1320
agatcaaaaa catcagcgag ggcgatacta ccatccagcg caattacctg aagggcaact 1380
gcacccagtg gagcgtgatc tactctgggt tccagacacc tgtcactcac ccagtggtca 1440
aagggggagt gcgaaaccag aatgacaacc gggccgaggc cttctgtaca tcctacggct 1500
tctttcccgg ggagatcaat attactttta tccattacgg caacaaggcc cccgacgatt 1560
ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg cacatttcac caggggtgct 1620
acgtcgctat cttctgcaat cagaactata cttgccgggt gacccatggg aactggactg 1680
tggaaatccc aatttcagtc accagccccg acgattcaag ctccggagag gtgccagatc 1740
accccaccgc aaataagaga tacaacacca tgacaatctc tagtgtgctg ctggccctgc 1800
tgctgtgcgc actgctgttc gcttttctgc attacttcac aactctgaag cagtatctgc 1860
ggaacctggc atttgcctgg cggtacagaa aagtgagatc aagctgactg tgccttctag 1920
ttgccagcca tctgttgttt gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac 1980
tcccactgtc ctttcctaat aaaatgagga aattgcatcg cattgtctga gtaggtgtca 2040
ttctattctg gggggtgggg tggggcagga cagcaagggg gaggattggg aagacaatag 2100
caggcatgct ggggatgcgg tgggctctat gtctctttct ggcctggagg ctatccagcg 2160
tgagtctctc ctaccctccc gctctggtcc ttcctctccc gctctgcacc ctctgtggcc 2220
ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag ttctccttgg tggcccgccg 2280
tggggctagt ccagggctgg atctcgggga agcggcgggg tggcctggga gtggggaagg 2340
gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc ggggagcagg ggagaccttt 2400
ggcctacggc gacgggaggg tcgggacaaa gtttagggcg tcgataagcg tcagagcgcc 2460
gaggttgggg gagggtttct cttccgctct ttcgcggggc ctctggctcc cccagcgcag 2520
ctggagtggg g 2531
<210> 73
<211> 2331
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _Actine peptide
<400> 73
cgcgcacccc agatcggagg gcgccgatgt acagacagca aactcaccca gtctagtgca 60
tgccttctta aacatcacga gactctaaga aaaggaaact gaaaacggga aagtccctct 120
ctctaacctg gcactgcgtc gctggcttgg agacaggtga cggtccctgc gggccttgtc 180
ctgattggct gggcacgcgt ttaatataag tggaggcgtc gcgctggcgg gcattcctga 240
agctgacagc attcgggccg agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact 300
ctctcttagc ggcctcgaag ctgccctgcc ccacgccatt ttgcggctcg gtggtggcgg 360
atccggtggt ggcggttctg gtggtggcgg ctccatccag cgtacgccca aaattcaagt 420
ctacagccga catcctgcag agaacggcaa atctaatttc ctgaactgct atgtatcagg 480
ctttcaccct agcgatatag aagtggacct gctgaaaaac ggagagagga tagaaaaggt 540
cgaacacagc gacctctcct tttccaagga ctggagcttt tatcttctgt attatactga 600
atttacaccc acggaaaaag atgagtatgc gtgccgagta aaccacgtca cgctgtcaca 660
gcccaaaata gtaaaatggg atcgcgacat gggtggtggc ggttctggtg gtggcggtag 720
tggcggcgga ggaagcggtg gtggcggttc cggatctatg cacgtgctga gatacggata 780
taccggcatc ttcgacgata catcccatat gactctgacc gtggtcggga tttttgacgg 840
acagcacttc tttacatacc atgtgaacag ctccgataag gcttctagtc gagcaaatgg 900
caccatctca tggatggcca acgtgagcgc agcctacccc acatatctgg acggagaacg 960
cgctaaaggc gatctgatct tcaatcagac cgagcagaac ctgctggagc tggaaattgc 1020
tctggggtac aggtctcaga gtgtcctgac atggactcac gaatgtaata ccacagagaa 1080
cgggagcttc gtggcaggat atgagggctt tgggtgggac ggagaaacac tgatggagct 1140
gaaggataat ctgactctgt ggaccggccc taactacgaa atcagctggc tgaagcagaa 1200
caagacttac atcgacggaa agatcaaaaa catcagcgag ggcgatacta ccatccagcg 1260
caattacctg aagggcaact gcacccagtg gagcgtgatc tactctgggt tccagacacc 1320
tgtcactcac ccagtggtca aagggggagt gcgaaaccag aatgacaacc gggccgaggc 1380
cttctgtaca tcctacggct tctttcccgg ggagatcaat attactttta tccattacgg 1440
caacaaggcc cccgacgatt ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg 1500
cacatttcac caggggtgct acgtcgctat cttctgcaat cagaactata cttgccgggt 1560
gacccatggg aactggactg tggaaatccc aatttcagtc accagccccg acgattcaag 1620
ctccggagag gtgccagatc accccaccgc aaataagaga tacaacacca tgacaatctc 1680
tagtgtgctg ctggccctgc tgctgtgcgc actgctgttc gcttttctgc attacttcac 1740
aactctgaag cagtatctgc ggaacctggc atttgcctgg cggtacagaa aagtgagatc 1800
aagctgactg tgccttctag ttgccagcca tctgttgttt gcccctcccc cgtgccttcc 1860
ttgaccctgg aaggtgccac tcccactgtc ctttcctaat aaaatgagga aattgcatcg 1920
cattgtctga gtaggtgtca ttctattctg gggggtgggg tggggcagga cagcaagggg 1980
gaggattggg aagacaatag caggcatgct ggggatgcgg tgggctctat gtctctttct 2040
ggcctggagg ctatccagcg tgagtctctc ctaccctccc gctctggtcc ttcctctccc 2100
gctctgcacc ctctgtggcc ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag 2160
ttctccttgg tggcccgccg tggggctagt ccagggctgg atctcgggga agcggcgggg 2220
tggcctggga gtggggaagg gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc 2280
ggggagcagg ggagaccttt ggcctacggc gacgggaggg tcgggacaaa g 2331
<210> 74
<211> 2531
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _HLACw peptide inserted at the B2m locus
<400> 74
cacttagcat ctctggggcc agtctgcaaa gcgagggggc agccttaatg tgcctccagc 60
ctgaagtcct agaatgagcg cccggtgtcc caagctgggg cgcgcacccc agatcggagg 120
gcgccgatgt acagacagca aactcaccca gtctagtgca tgccttctta aacatcacga 180
gactctaaga aaaggaaact gaaaacggga aagtccctct ctctaacctg gcactgcgtc 240
gctggcttgg agacaggtga cggtccctgc gggccttgtc ctgattggct gggcacgcgt 300
ttaatataag tggaggcgtc gcgctggcgg gcattcctga agctgacagc attcgggccg 360
agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact ctctcttagc ggcctcgaag 420
ctgttatggc tccgcggact ttaattttag gtggtggcgg atccggtggt ggcggttctg 480
gtggtggcgg ctccatccag cgtacgccca aaattcaagt ctacagccga catcctgcag 540
agaacggcaa atctaatttc ctgaactgct atgtatcagg ctttcaccct agcgatatag 600
aagtggacct gctgaaaaac ggagagagga tagaaaaggt cgaacacagc gacctctcct 660
tttccaagga ctggagcttt tatcttctgt attatactga atttacaccc acggaaaaag 720
atgagtatgc gtgccgagta aaccacgtca cgctgtcaca gcccaaaata gtaaaatggg 780
atcgcgacat gggtggtggc ggttctggtg gtggcggtag tggcggcgga ggaagcggtg 840
gtggcggttc cggatctatg cacgtgctga gatacggata taccggcatc ttcgacgata 900
catcccatat gactctgacc gtggtcggga tttttgacgg acagcacttc tttacatacc 960
atgtgaacag ctccgataag gcttctagtc gagcaaatgg caccatctca tggatggcca 1020
acgtgagcgc agcctacccc acatatctgg acggagaacg cgctaaaggc gatctgatct 1080
tcaatcagac cgagcagaac ctgctggagc tggaaattgc tctggggtac aggtctcaga 1140
gtgtcctgac atggactcac gaatgtaata ccacagagaa cgggagcttc gtggcaggat 1200
atgagggctt tgggtgggac ggagaaacac tgatggagct gaaggataat ctgactctgt 1260
ggaccggccc taactacgaa atcagctggc tgaagcagaa caagacttac atcgacggaa 1320
agatcaaaaa catcagcgag ggcgatacta ccatccagcg caattacctg aagggcaact 1380
gcacccagtg gagcgtgatc tactctgggt tccagacacc tgtcactcac ccagtggtca 1440
aagggggagt gcgaaaccag aatgacaacc gggccgaggc cttctgtaca tcctacggct 1500
tctttcccgg ggagatcaat attactttta tccattacgg caacaaggcc cccgacgatt 1560
ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg cacatttcac caggggtgct 1620
acgtcgctat cttctgcaat cagaactata cttgccgggt gacccatggg aactggactg 1680
tggaaatccc aatttcagtc accagccccg acgattcaag ctccggagag gtgccagatc 1740
accccaccgc aaataagaga tacaacacca tgacaatctc tagtgtgctg ctggccctgc 1800
tgctgtgcgc actgctgttc gcttttctgc attacttcac aactctgaag cagtatctgc 1860
ggaacctggc atttgcctgg cggtacagaa aagtgagatc aagctgactg tgccttctag 1920
ttgccagcca tctgttgttt gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac 1980
tcccactgtc ctttcctaat aaaatgagga aattgcatcg cattgtctga gtaggtgtca 2040
ttctattctg gggggtgggg tggggcagga cagcaagggg gaggattggg aagacaatag 2100
caggcatgct ggggatgcgg tgggctctat gtctctttct ggcctggagg ctatccagcg 2160
tgagtctctc ctaccctccc gctctggtcc ttcctctccc gctctgcacc ctctgtggcc 2220
ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag ttctccttgg tggcccgccg 2280
tggggctagt ccagggctgg atctcgggga agcggcgggg tggcctggga gtggggaagg 2340
gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc ggggagcagg ggagaccttt 2400
ggcctacggc gacgggaggg tcgggacaaa gtttagggcg tcgataagcg tcagagcgcc 2460
gaggttgggg gagggtttct cttccgctct ttcgcggggc ctctggctcc cccagcgcag 2520
ctggagtggg g 2531
<210> 75
<211> 2331
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _HLACw peptide
<400> 75
cgcgcacccc agatcggagg gcgccgatgt acagacagca aactcaccca gtctagtgca 60
tgccttctta aacatcacga gactctaaga aaaggaaact gaaaacggga aagtccctct 120
ctctaacctg gcactgcgtc gctggcttgg agacaggtga cggtccctgc gggccttgtc 180
ctgattggct gggcacgcgt ttaatataag tggaggcgtc gcgctggcgg gcattcctga 240
agctgacagc attcgggccg agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact 300
ctctcttagc ggcctcgaag ctgttatggc tccgcggact ttaattttag gtggtggcgg 360
atccggtggt ggcggttctg gtggtggcgg ctccatccag cgtacgccca aaattcaagt 420
ctacagccga catcctgcag agaacggcaa atctaatttc ctgaactgct atgtatcagg 480
ctttcaccct agcgatatag aagtggacct gctgaaaaac ggagagagga tagaaaaggt 540
cgaacacagc gacctctcct tttccaagga ctggagcttt tatcttctgt attatactga 600
atttacaccc acggaaaaag atgagtatgc gtgccgagta aaccacgtca cgctgtcaca 660
gcccaaaata gtaaaatggg atcgcgacat gggtggtggc ggttctggtg gtggcggtag 720
tggcggcgga ggaagcggtg gtggcggttc cggatctatg cacgtgctga gatacggata 780
taccggcatc ttcgacgata catcccatat gactctgacc gtggtcggga tttttgacgg 840
acagcacttc tttacatacc atgtgaacag ctccgataag gcttctagtc gagcaaatgg 900
caccatctca tggatggcca acgtgagcgc agcctacccc acatatctgg acggagaacg 960
cgctaaaggc gatctgatct tcaatcagac cgagcagaac ctgctggagc tggaaattgc 1020
tctggggtac aggtctcaga gtgtcctgac atggactcac gaatgtaata ccacagagaa 1080
cgggagcttc gtggcaggat atgagggctt tgggtgggac ggagaaacac tgatggagct 1140
gaaggataat ctgactctgt ggaccggccc taactacgaa atcagctggc tgaagcagaa 1200
caagacttac atcgacggaa agatcaaaaa catcagcgag ggcgatacta ccatccagcg 1260
caattacctg aagggcaact gcacccagtg gagcgtgatc tactctgggt tccagacacc 1320
tgtcactcac ccagtggtca aagggggagt gcgaaaccag aatgacaacc gggccgaggc 1380
cttctgtaca tcctacggct tctttcccgg ggagatcaat attactttta tccattacgg 1440
caacaaggcc cccgacgatt ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg 1500
cacatttcac caggggtgct acgtcgctat cttctgcaat cagaactata cttgccgggt 1560
gacccatggg aactggactg tggaaatccc aatttcagtc accagccccg acgattcaag 1620
ctccggagag gtgccagatc accccaccgc aaataagaga tacaacacca tgacaatctc 1680
tagtgtgctg ctggccctgc tgctgtgcgc actgctgttc gcttttctgc attacttcac 1740
aactctgaag cagtatctgc ggaacctggc atttgcctgg cggtacagaa aagtgagatc 1800
aagctgactg tgccttctag ttgccagcca tctgttgttt gcccctcccc cgtgccttcc 1860
ttgaccctgg aaggtgccac tcccactgtc ctttcctaat aaaatgagga aattgcatcg 1920
cattgtctga gtaggtgtca ttctattctg gggggtgggg tggggcagga cagcaagggg 1980
gaggattggg aagacaatag caggcatgct ggggatgcgg tgggctctat gtctctttct 2040
ggcctggagg ctatccagcg tgagtctctc ctaccctccc gctctggtcc ttcctctccc 2100
gctctgcacc ctctgtggcc ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag 2160
ttctccttgg tggcccgccg tggggctagt ccagggctgg atctcgggga agcggcgggg 2220
tggcctggga gtggggaagg gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc 2280
ggggagcagg ggagaccttt ggcctacggc gacgggaggg tcgggacaaa g 2331
<210> 76
<211> 2531
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _HLAG peptide inserted at the B2m locus
<400> 76
cacttagcat ctctggggcc agtctgcaaa gcgagggggc agccttaatg tgcctccagc 60
ctgaagtcct agaatgagcg cccggtgtcc caagctgggg cgcgcacccc agatcggagg 120
gcgccgatgt acagacagca aactcaccca gtctagtgca tgccttctta aacatcacga 180
gactctaaga aaaggaaact gaaaacggga aagtccctct ctctaacctg gcactgcgtc 240
gctggcttgg agacaggtga cggtccctgc gggccttgtc ctgattggct gggcacgcgt 300
ttaatataag tggaggcgtc gcgctggcgg gcattcctga agctgacagc attcgggccg 360
agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact ctctcttagc ggcctcgaag 420
ctgttatggc tccgcggact ttattcttag gtggtggcgg atccggtggt ggcggttctg 480
gtggtggcgg ctccatccag cgtacgccca aaattcaagt ctacagccga catcctgcag 540
agaacggcaa atctaatttc ctgaactgct atgtatcagg ctttcaccct agcgatatag 600
aagtggacct gctgaaaaac ggagagagga tagaaaaggt cgaacacagc gacctctcct 660
tttccaagga ctggagcttt tatcttctgt attatactga atttacaccc acggaaaaag 720
atgagtatgc gtgccgagta aaccacgtca cgctgtcaca gcccaaaata gtaaaatggg 780
atcgcgacat gggtggtggc ggttctggtg gtggcggtag tggcggcgga ggaagcggtg 840
gtggcggttc cggatctatg cacgtgctga gatacggata taccggcatc ttcgacgata 900
catcccatat gactctgacc gtggtcggga tttttgacgg acagcacttc tttacatacc 960
atgtgaacag ctccgataag gcttctagtc gagcaaatgg caccatctca tggatggcca 1020
acgtgagcgc agcctacccc acatatctgg acggagaacg cgctaaaggc gatctgatct 1080
tcaatcagac cgagcagaac ctgctggagc tggaaattgc tctggggtac aggtctcaga 1140
gtgtcctgac atggactcac gaatgtaata ccacagagaa cgggagcttc gtggcaggat 1200
atgagggctt tgggtgggac ggagaaacac tgatggagct gaaggataat ctgactctgt 1260
ggaccggccc taactacgaa atcagctggc tgaagcagaa caagacttac atcgacggaa 1320
agatcaaaaa catcagcgag ggcgatacta ccatccagcg caattacctg aagggcaact 1380
gcacccagtg gagcgtgatc tactctgggt tccagacacc tgtcactcac ccagtggtca 1440
aagggggagt gcgaaaccag aatgacaacc gggccgaggc cttctgtaca tcctacggct 1500
tctttcccgg ggagatcaat attactttta tccattacgg caacaaggcc cccgacgatt 1560
ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg cacatttcac caggggtgct 1620
acgtcgctat cttctgcaat cagaactata cttgccgggt gacccatggg aactggactg 1680
tggaaatccc aatttcagtc accagccccg acgattcaag ctccggagag gtgccagatc 1740
accccaccgc aaataagaga tacaacacca tgacaatctc tagtgtgctg ctggccctgc 1800
tgctgtgcgc actgctgttc gcttttctgc attacttcac aactctgaag cagtatctgc 1860
ggaacctggc atttgcctgg cggtacagaa aagtgagatc aagctgactg tgccttctag 1920
ttgccagcca tctgttgttt gcccctcccc cgtgccttcc ttgaccctgg aaggtgccac 1980
tcccactgtc ctttcctaat aaaatgagga aattgcatcg cattgtctga gtaggtgtca 2040
ttctattctg gggggtgggg tggggcagga cagcaagggg gaggattggg aagacaatag 2100
caggcatgct ggggatgcgg tgggctctat gtctctttct ggcctggagg ctatccagcg 2160
tgagtctctc ctaccctccc gctctggtcc ttcctctccc gctctgcacc ctctgtggcc 2220
ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag ttctccttgg tggcccgccg 2280
tggggctagt ccagggctgg atctcgggga agcggcgggg tggcctggga gtggggaagg 2340
gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc ggggagcagg ggagaccttt 2400
ggcctacggc gacgggaggg tcgggacaaa gtttagggcg tcgataagcg tcagagcgcc 2460
gaggttgggg gagggtttct cttccgctct ttcgcggggc ctctggctcc cccagcgcag 2520
ctggagtggg g 2531
<210> 77
<211> 2331
<212> DNA
<213> artificial sequence
<220>
<223> UL18Trimer matrix _HLAG peptide
<400> 77
cgcgcacccc agatcggagg gcgccgatgt acagacagca aactcaccca gtctagtgca 60
tgccttctta aacatcacga gactctaaga aaaggaaact gaaaacggga aagtccctct 120
ctctaacctg gcactgcgtc gctggcttgg agacaggtga cggtccctgc gggccttgtc 180
ctgattggct gggcacgcgt ttaatataag tggaggcgtc gcgctggcgg gcattcctga 240
agctgacagc attcgggccg agatgtctcg ctccgtggcc ttagctgtgc tcgcgctact 300
ctctcttagc ggcctcgaag ctgttatggc tccgcggact ttattcttag gtggtggcgg 360
atccggtggt ggcggttctg gtggtggcgg ctccatccag cgtacgccca aaattcaagt 420
ctacagccga catcctgcag agaacggcaa atctaatttc ctgaactgct atgtatcagg 480
ctttcaccct agcgatatag aagtggacct gctgaaaaac ggagagagga tagaaaaggt 540
cgaacacagc gacctctcct tttccaagga ctggagcttt tatcttctgt attatactga 600
atttacaccc acggaaaaag atgagtatgc gtgccgagta aaccacgtca cgctgtcaca 660
gcccaaaata gtaaaatggg atcgcgacat gggtggtggc ggttctggtg gtggcggtag 720
tggcggcgga ggaagcggtg gtggcggttc cggatctatg cacgtgctga gatacggata 780
taccggcatc ttcgacgata catcccatat gactctgacc gtggtcggga tttttgacgg 840
acagcacttc tttacatacc atgtgaacag ctccgataag gcttctagtc gagcaaatgg 900
caccatctca tggatggcca acgtgagcgc agcctacccc acatatctgg acggagaacg 960
cgctaaaggc gatctgatct tcaatcagac cgagcagaac ctgctggagc tggaaattgc 1020
tctggggtac aggtctcaga gtgtcctgac atggactcac gaatgtaata ccacagagaa 1080
cgggagcttc gtggcaggat atgagggctt tgggtgggac ggagaaacac tgatggagct 1140
gaaggataat ctgactctgt ggaccggccc taactacgaa atcagctggc tgaagcagaa 1200
caagacttac atcgacggaa agatcaaaaa catcagcgag ggcgatacta ccatccagcg 1260
caattacctg aagggcaact gcacccagtg gagcgtgatc tactctgggt tccagacacc 1320
tgtcactcac ccagtggtca aagggggagt gcgaaaccag aatgacaacc gggccgaggc 1380
cttctgtaca tcctacggct tctttcccgg ggagatcaat attactttta tccattacgg 1440
caacaaggcc cccgacgatt ctgagcctca gtgcaatccc ctgctgccta ccttcgatgg 1500
cacatttcac caggggtgct acgtcgctat cttctgcaat cagaactata cttgccgggt 1560
gacccatggg aactggactg tggaaatccc aatttcagtc accagccccg acgattcaag 1620
ctccggagag gtgccagatc accccaccgc aaataagaga tacaacacca tgacaatctc 1680
tagtgtgctg ctggccctgc tgctgtgcgc actgctgttc gcttttctgc attacttcac 1740
aactctgaag cagtatctgc ggaacctggc atttgcctgg cggtacagaa aagtgagatc 1800
aagctgactg tgccttctag ttgccagcca tctgttgttt gcccctcccc cgtgccttcc 1860
ttgaccctgg aaggtgccac tcccactgtc ctttcctaat aaaatgagga aattgcatcg 1920
cattgtctga gtaggtgtca ttctattctg gggggtgggg tggggcagga cagcaagggg 1980
gaggattggg aagacaatag caggcatgct ggggatgcgg tgggctctat gtctctttct 2040
ggcctggagg ctatccagcg tgagtctctc ctaccctccc gctctggtcc ttcctctccc 2100
gctctgcacc ctctgtggcc ctcgctgtgc tctctcgctc cgtgacttcc cttctccaag 2160
ttctccttgg tggcccgccg tggggctagt ccagggctgg atctcgggga agcggcgggg 2220
tggcctggga gtggggaagg gggtgcgcac ccgggacgcg cgctacttgc ccctttcggc 2280
ggggagcagg ggagaccttt ggcctacggc gacgggaggg tcgggacaaa g 2331
<210> 78
<211> 47
<212> DNA
<213> artificial sequence
<220>
<223> TALEN target B2m1
<400> 78
tccgtggcct tagctgtgct cgcgctactc tctctttctg gcctgga 47
<210> 79
<211> 47
<212> DNA
<213> artificial sequence
<220>
<223> TALEN target B2m2
<400> 79
ttagctgtgc tcgcgctact ctctctttct ggcctggagg ctatcca 47
<210> 80
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> pCLS31134 right TALEN B2m1
<400> 80
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
260 265 270
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
355 360 365
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
565 570 575
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
625 630 635 640
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 81
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> pCLS31135 left TALEN B2m1
<400> 81
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
260 265 270
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
325 330 335
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
355 360 365
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
500 505 510
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
565 570 575
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
625 630 635 640
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 82
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> pCLS31136 right TALEN B2m2
<400> 82
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
260 265 270
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
355 360 365
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
500 505 510
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
565 570 575
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 83
<211> 936
<212> PRT
<213> artificial sequence
<220>
<223> pCLS31137 left TALEN B2m2
<400> 83
Met Gly Asp Pro Lys Lys Lys Arg Lys Val Ile Asp Tyr Pro Tyr Asp
1 5 10 15
Val Pro Asp Tyr Ala Ile Asp Ile Ala Asp Leu Arg Thr Leu Gly Tyr
20 25 30
Ser Gln Gln Gln Gln Glu Lys Ile Lys Pro Lys Val Arg Ser Thr Val
35 40 45
Ala Gln His His Glu Ala Leu Val Gly His Gly Phe Thr His Ala His
50 55 60
Ile Val Ala Leu Ser Gln His Pro Ala Ala Leu Gly Thr Val Ala Val
65 70 75 80
Lys Tyr Gln Asp Met Ile Ala Ala Leu Pro Glu Ala Thr His Glu Ala
85 90 95
Ile Val Gly Val Gly Lys Gln Trp Ser Gly Ala Arg Ala Leu Glu Ala
100 105 110
Leu Leu Thr Val Ala Gly Glu Leu Arg Gly Pro Pro Leu Gln Leu Asp
115 120 125
Thr Gly Gln Leu Leu Lys Ile Ala Lys Arg Gly Gly Val Thr Ala Val
130 135 140
Glu Ala Val His Ala Trp Arg Asn Ala Leu Thr Gly Ala Pro Leu Asn
145 150 155 160
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
165 170 175
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
180 185 190
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
195 200 205
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
210 215 220
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser Asn
225 230 235 240
Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu Pro Val
245 250 255
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
260 265 270
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
275 280 285
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
290 295 300
Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala
305 310 315 320
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
325 330 335
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
340 345 350
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
355 360 365
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
370 375 380
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
385 390 395 400
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala
405 410 415
Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly
420 425 430
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
435 440 445
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
450 455 460
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
465 470 475 480
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
485 490 495
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
500 505 510
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
515 520 525
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
530 535 540
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
545 550 555 560
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
565 570 575
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
580 585 590
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
595 600 605
Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val
610 615 620
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
625 630 635 640
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
645 650 655
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
660 665 670
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Arg Pro Ala
675 680 685
Leu Glu Ser Ile Val Ala Gln Leu Ser Arg Pro Asp Pro Ala Leu Ala
690 695 700
Ala Leu Thr Asn Asp His Leu Val Ala Leu Ala Cys Leu Gly Gly Arg
705 710 715 720
Pro Ala Leu Asp Ala Val Lys Lys Gly Leu Gly Asp Pro Ile Ser Arg
725 730 735
Ser Gln Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg
740 745 750
His Lys Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile
755 760 765
Ala Arg Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu
770 775 780
Phe Phe Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser
785 790 795 800
Arg Lys Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr
805 810 815
Gly Val Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro
820 825 830
Ile Gly Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr
835 840 845
Arg Asn Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser
850 855 860
Ser Val Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly
865 870 875 880
Asn Tyr Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn
885 890 895
Gly Ala Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile
900 905 910
Lys Ala Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn
915 920 925
Gly Glu Ile Asn Phe Ala Ala Asp
930 935
<210> 84
<211> 338
<212> PRT
<213> artificial sequence
<220>
<223> HLAG1
<400> 84
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Ser Ser His Thr Leu Gln Trp Met Ile Gly Cys Asp Leu Gly
115 120 125
Ser Asp Gly Arg Leu Leu Arg Gly Tyr Glu Gln Tyr Ala Tyr Asp Gly
130 135 140
Lys Asp Tyr Leu Ala Leu Asn Glu Asp Leu Arg Ser Trp Thr Ala Ala
145 150 155 160
Asp Thr Ala Ala Gln Ile Ser Lys Arg Lys Cys Glu Ala Ala Asn Val
165 170 175
Ala Glu Gln Arg Arg Ala Tyr Leu Glu Gly Thr Cys Val Glu Trp Leu
180 185 190
His Arg Tyr Leu Glu Asn Gly Lys Glu Met Leu Gln Arg Ala Asp Pro
195 200 205
Pro Lys Thr His Val Thr His His Pro Val Phe Asp Tyr Glu Ala Thr
210 215 220
Leu Arg Cys Trp Ala Leu Gly Phe Tyr Pro Ala Glu Ile Ile Leu Thr
225 230 235 240
Trp Gln Arg Asp Gly Glu Asp Gln Thr Gln Asp Val Glu Leu Val Glu
245 250 255
Thr Arg Pro Ala Gly Asp Gly Thr Phe Gln Lys Trp Ala Ala Val Val
260 265 270
Val Pro Ser Gly Glu Glu Gln Arg Tyr Thr Cys His Val Gln His Glu
275 280 285
Gly Leu Pro Glu Pro Leu Met Leu Arg Trp Lys Gln Ser Ser Leu Pro
290 295 300
Thr Ile Pro Ile Met Gly Ile Val Ala Gly Leu Val Val Leu Ala Ala
305 310 315 320
Val Val Thr Gly Ala Ala Val Ala Ala Val Leu Trp Arg Lys Lys Ser
325 330 335
Ser Asp
<210> 85
<211> 246
<212> PRT
<213> artificial sequence
<220>
<223> HLAG2
<400> 85
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Lys Pro Pro Lys Thr His Val Thr His His Pro Val Phe Asp
115 120 125
Tyr Glu Ala Thr Leu Arg Cys Trp Ala Leu Gly Phe Tyr Pro Ala Glu
130 135 140
Ile Ile Leu Thr Trp Gln Arg Asp Gly Glu Asp Gln Thr Gln Asp Val
145 150 155 160
Glu Leu Val Glu Thr Arg Pro Ala Gly Asp Gly Thr Phe Gln Lys Trp
165 170 175
Ala Ala Val Val Val Pro Ser Gly Glu Glu Gln Arg Tyr Thr Cys His
180 185 190
Val Gln His Glu Gly Leu Pro Glu Pro Leu Met Leu Arg Trp Lys Gln
195 200 205
Ser Ser Leu Pro Thr Ile Pro Ile Met Gly Ile Val Ala Gly Leu Val
210 215 220
Val Leu Ala Ala Val Val Thr Gly Ala Ala Val Ala Ala Val Leu Trp
225 230 235 240
Arg Lys Lys Ser Ser Asp
245
<210> 86
<211> 154
<212> PRT
<213> artificial sequence
<220>
<223> HLAG3
<400> 86
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Lys Gln Ser Ser Leu Pro Thr Ile Pro Ile Met Gly Ile Val
115 120 125
Ala Gly Leu Val Val Leu Ala Ala Val Val Thr Gly Ala Ala Val Ala
130 135 140
Ala Val Leu Trp Arg Lys Lys Ser Ser Asp
145 150
<210> 87
<211> 246
<212> PRT
<213> artificial sequence
<220>
<223> HLAG4
<400> 87
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Ser Ser His Thr Leu Gln Trp Met Ile Gly Cys Asp Leu Gly
115 120 125
Ser Asp Gly Arg Leu Leu Arg Gly Tyr Glu Gln Tyr Ala Tyr Asp Gly
130 135 140
Lys Asp Tyr Leu Ala Leu Asn Glu Asp Leu Arg Ser Trp Thr Ala Ala
145 150 155 160
Asp Thr Ala Ala Gln Ile Ser Lys Arg Lys Cys Glu Ala Ala Asn Val
165 170 175
Ala Glu Gln Arg Arg Ala Tyr Leu Glu Gly Thr Cys Val Glu Trp Leu
180 185 190
His Arg Tyr Leu Glu Asn Gly Lys Glu Met Leu Gln Arg Ala Lys Gln
195 200 205
Ser Ser Leu Pro Thr Ile Pro Ile Met Gly Ile Val Ala Gly Leu Val
210 215 220
Val Leu Ala Ala Val Val Thr Gly Ala Ala Val Ala Ala Val Leu Trp
225 230 235 240
Arg Lys Lys Ser Ser Asp
245
<210> 88
<211> 319
<212> PRT
<213> artificial sequence
<220>
<223> HLAG5
<400> 88
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Ser Ser His Thr Leu Gln Trp Met Ile Gly Cys Asp Leu Gly
115 120 125
Ser Asp Gly Arg Leu Leu Arg Gly Tyr Glu Gln Tyr Ala Tyr Asp Gly
130 135 140
Lys Asp Tyr Leu Ala Leu Asn Glu Asp Leu Arg Ser Trp Thr Ala Ala
145 150 155 160
Asp Thr Ala Ala Gln Ile Ser Lys Arg Lys Cys Glu Ala Ala Asn Val
165 170 175
Ala Glu Gln Arg Arg Ala Tyr Leu Glu Gly Thr Cys Val Glu Trp Leu
180 185 190
His Arg Tyr Leu Glu Asn Gly Lys Glu Met Leu Gln Arg Ala Asp Pro
195 200 205
Pro Lys Thr His Val Thr His His Pro Val Phe Asp Tyr Glu Ala Thr
210 215 220
Leu Arg Cys Trp Ala Leu Gly Phe Tyr Pro Ala Glu Ile Ile Leu Thr
225 230 235 240
Trp Gln Arg Asp Gly Glu Asp Gln Thr Gln Asp Val Glu Leu Val Glu
245 250 255
Thr Arg Pro Ala Gly Asp Gly Thr Phe Gln Lys Trp Ala Ala Val Val
260 265 270
Val Pro Ser Gly Glu Glu Gln Arg Tyr Thr Cys His Val Gln His Glu
275 280 285
Gly Leu Pro Glu Pro Leu Met Leu Arg Trp Ser Lys Glu Gly Asp Gly
290 295 300
Gly Ile Met Ser Val Arg Glu Ser Arg Ser Leu Ser Glu Asp Leu
305 310 315
<210> 89
<211> 227
<212> PRT
<213> artificial sequence
<220>
<223> HLAG6
<400> 89
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Lys Pro Pro Lys Thr His Val Thr His His Pro Val Phe Asp
115 120 125
Tyr Glu Ala Thr Leu Arg Cys Trp Ala Leu Gly Phe Tyr Pro Ala Glu
130 135 140
Ile Ile Leu Thr Trp Gln Arg Asp Gly Glu Asp Gln Thr Gln Asp Val
145 150 155 160
Glu Leu Val Glu Thr Arg Pro Ala Gly Asp Gly Thr Phe Gln Lys Trp
165 170 175
Ala Ala Val Val Val Pro Ser Gly Glu Glu Gln Arg Tyr Thr Cys His
180 185 190
Val Gln His Glu Gly Leu Pro Glu Pro Leu Met Leu Arg Trp Ser Lys
195 200 205
Glu Gly Asp Gly Gly Ile Met Ser Val Arg Glu Ser Arg Ser Leu Ser
210 215 220
Glu Asp Leu
225
<210> 90
<211> 116
<212> PRT
<213> artificial sequence
<220>
<223> HLAG7
<400> 90
Met Val Val Met Ala Pro Arg Thr Leu Phe Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Thr Leu Thr Glu Thr Trp Ala Gly Ser His Ser Met Arg Tyr Phe
20 25 30
Ser Ala Ala Val Ser Arg Pro Gly Arg Gly Glu Pro Arg Phe Ile Ala
35 40 45
Met Gly Tyr Val Asp Asp Thr Gln Phe Val Arg Phe Asp Ser Asp Ser
50 55 60
Ala Cys Pro Arg Met Glu Pro Arg Ala Pro Trp Val Glu Gln Glu Gly
65 70 75 80
Pro Glu Tyr Trp Glu Glu Glu Thr Arg Asn Thr Lys Ala His Ala Gln
85 90 95
Thr Asp Arg Met Asn Leu Gln Thr Leu Arg Gly Tyr Tyr Asn Gln Ser
100 105 110
Glu Ala Ser Glu
115
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Т-КЛЕТКИ С ДВУМЯ ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ CAR | 2021 |
|
RU2839662C1 |
| Конструкции для направленного редактирования генов и способы с их применением | 2020 |
|
RU2832109C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА FLT3 | 2018 |
|
RU2820859C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛИВАЮЩИЕСЯ НА ВАРИАНТ III РЕЦЕПТОРА ЭПИДЕРМАЛЬНОГО ФАКТОРА РОСТА | 2017 |
|
RU2751662C2 |
| НЕ РЕСТРИКТИРОВАННЫЕ ПО HLA T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2812917C2 |
| CD123-СПЕЦИФИЧЕСКИЕ ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ ДЛЯ ИММУНОТЕРАПИИ РАКА | 2015 |
|
RU2727290C2 |
| СПОСОБЫ ПОЛУЧЕНИЯ ЭКСПРЕССИРУЮЩИХ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР КЛЕТОК | 2015 |
|
RU2751362C2 |
| СПЕЦИФИЧНЫЕ К MUC16 ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795198C2 |
| НЕДЕСТРУКТИВНАЯ ГЕННАЯ ТЕРАПИЯ ДЛЯ ЛЕЧЕНИЯ MMA | 2018 |
|
RU2820602C2 |
| СПОСОБЫ КОНСТРУИРОВАНИЯ АЛЛОГЕННЫХ И ВЫСОКОАКТИВНЫХ Т-КЛЕТОК ДЛЯ ИММУНОТЕРАПИИ | 2014 |
|
RU2736616C2 |
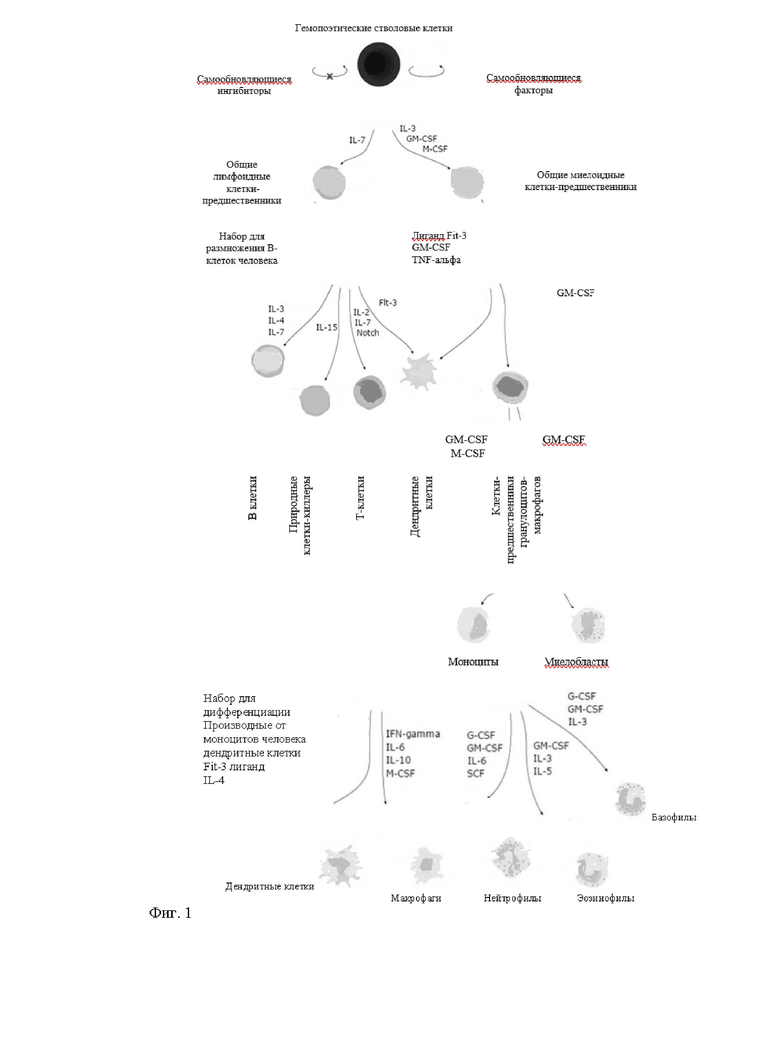
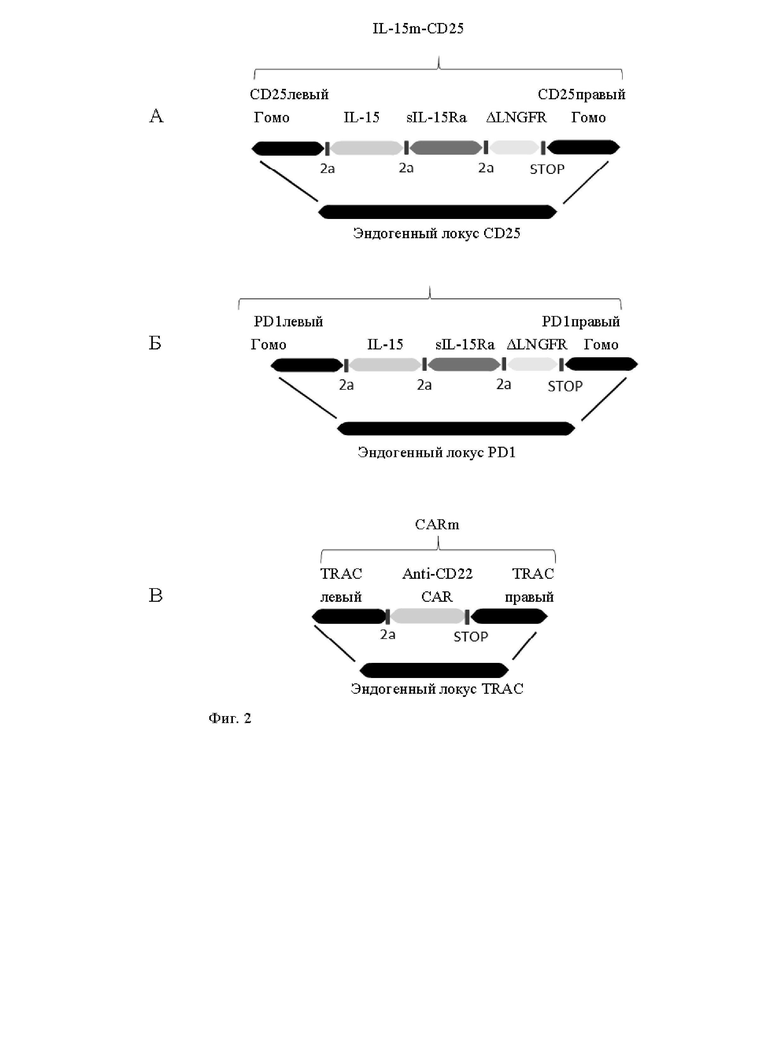
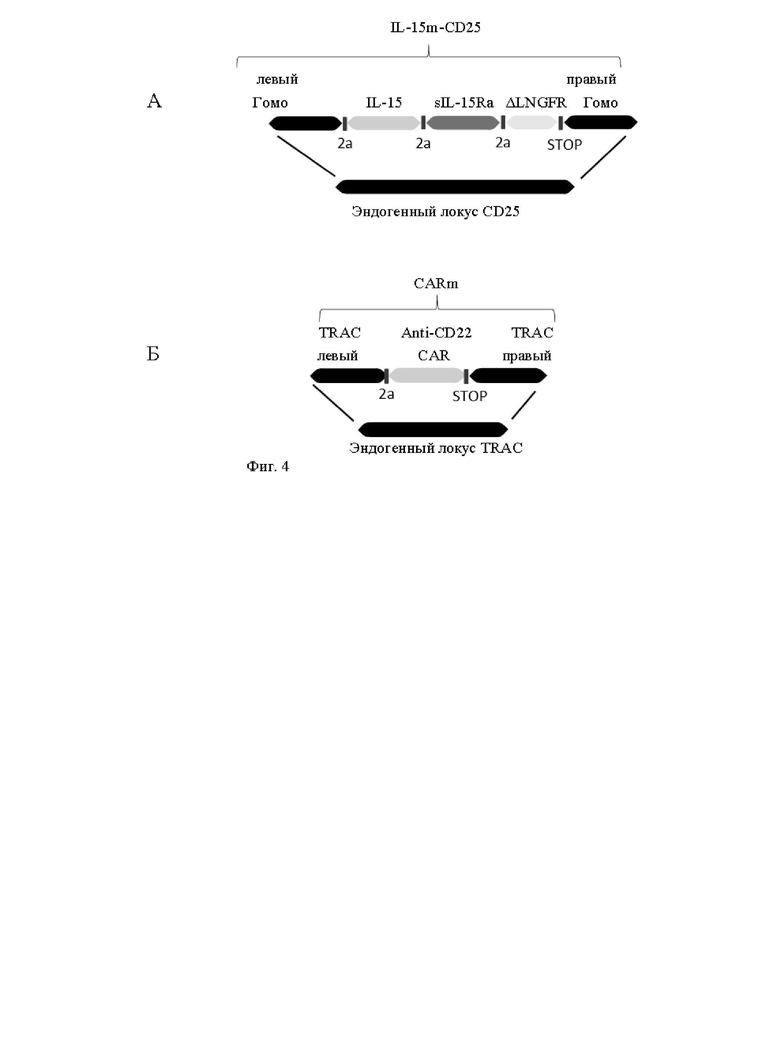
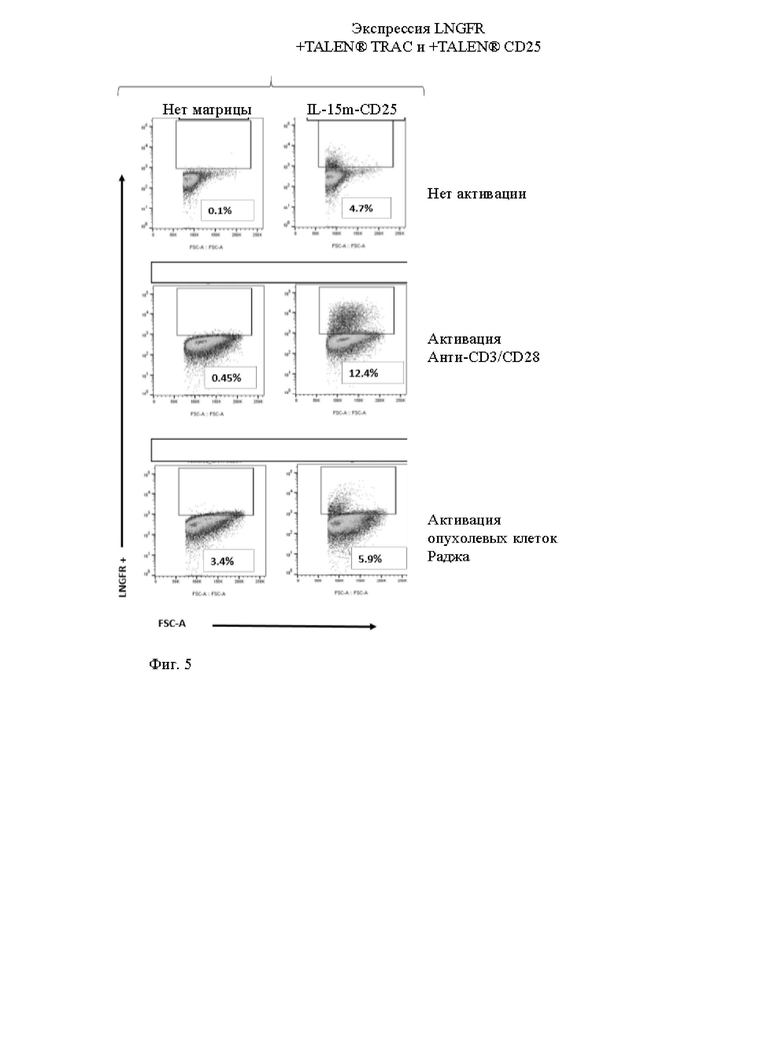
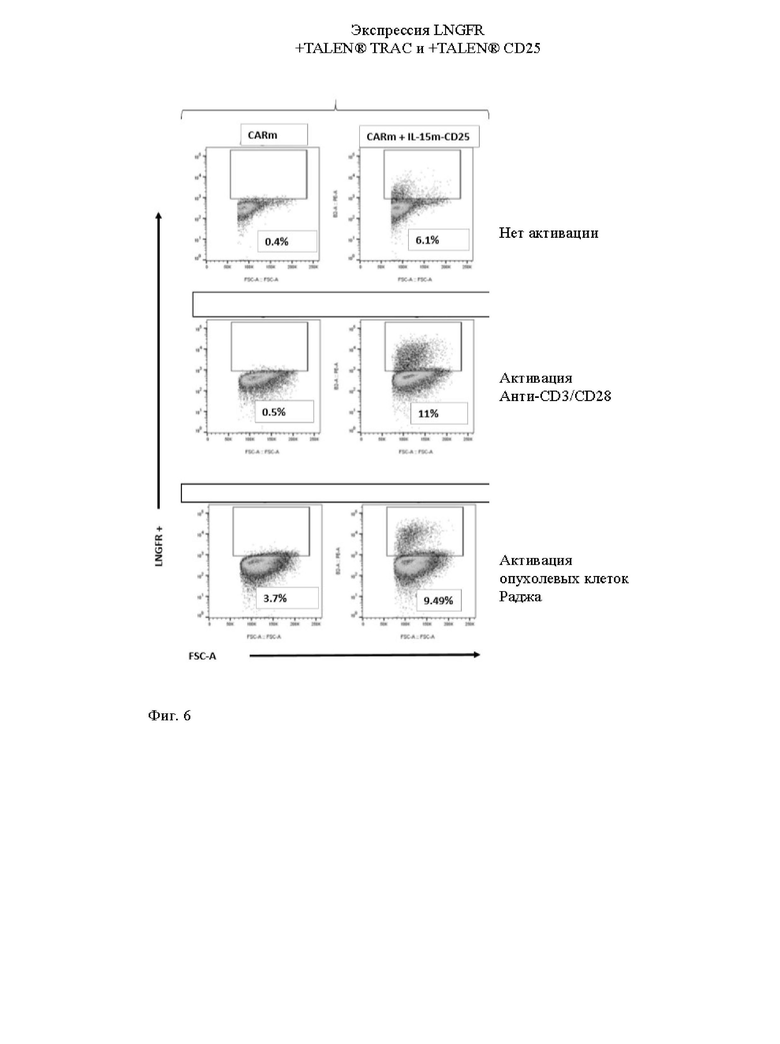
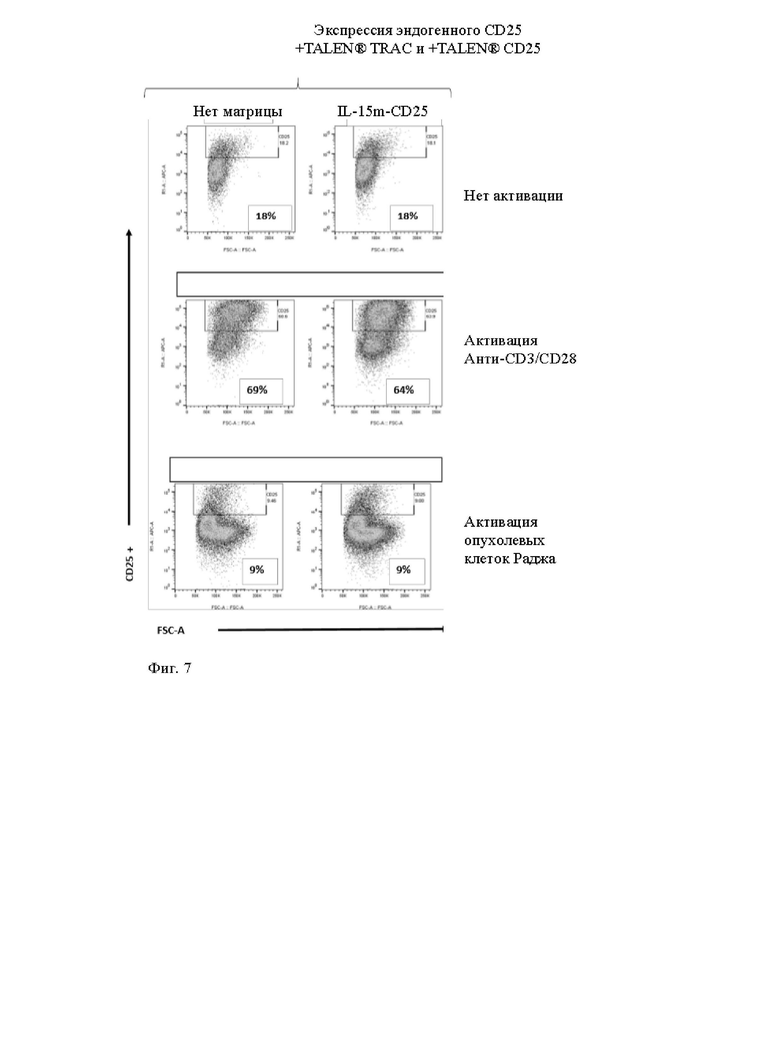
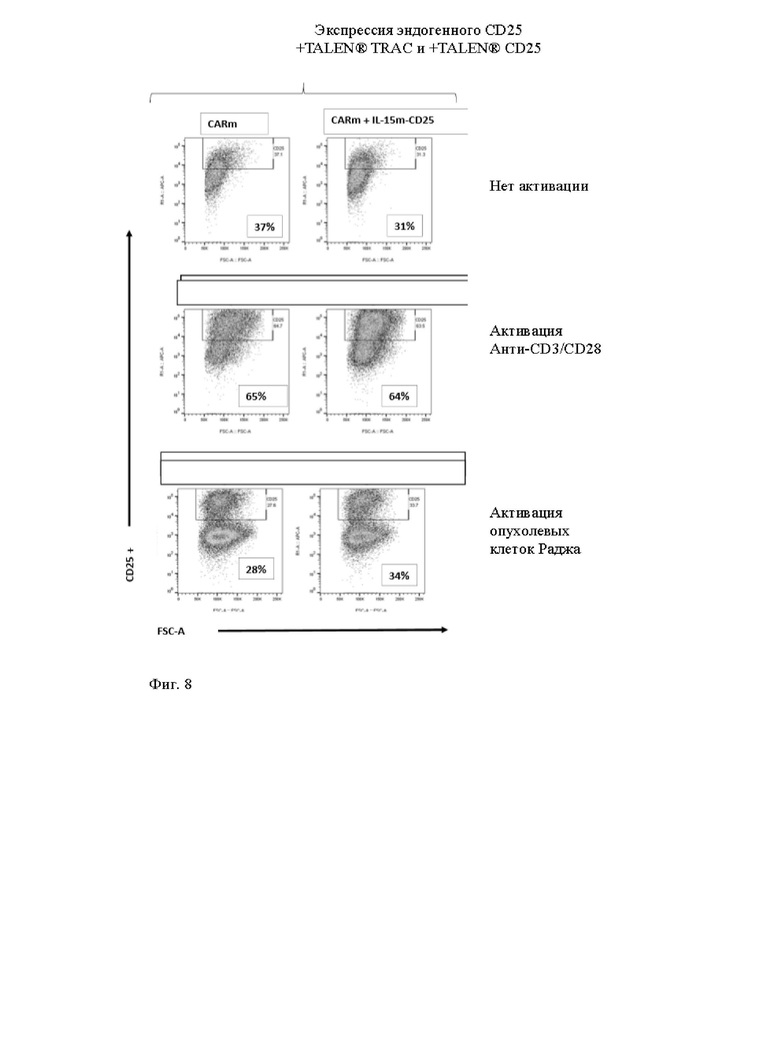
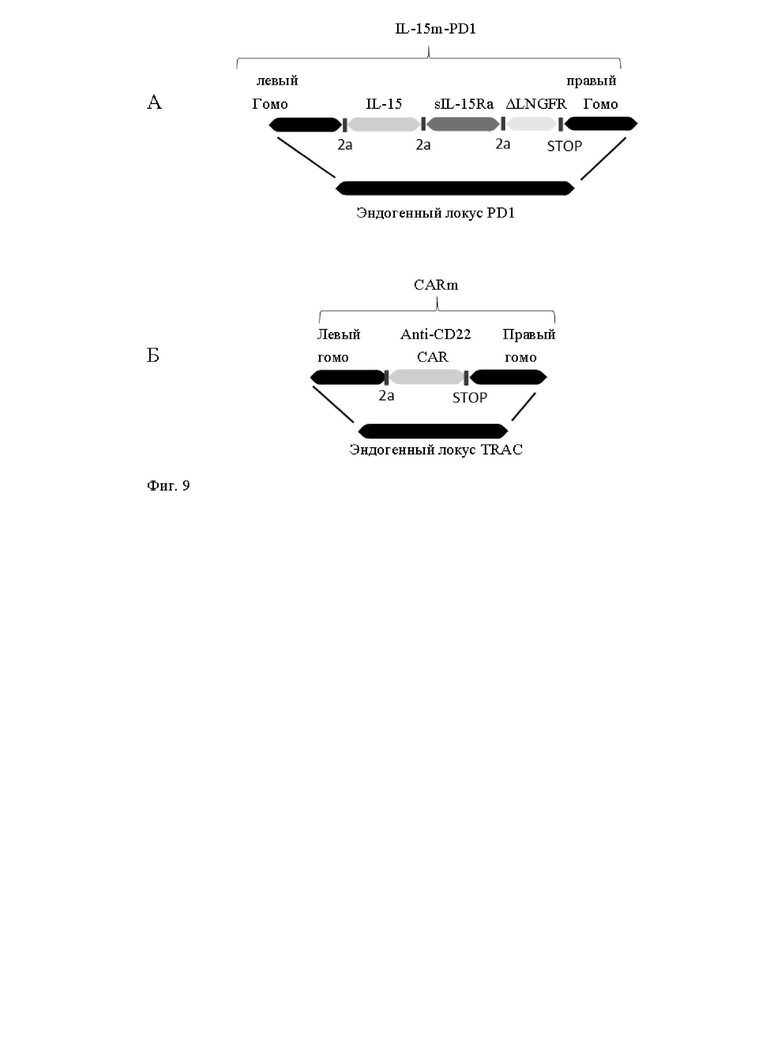
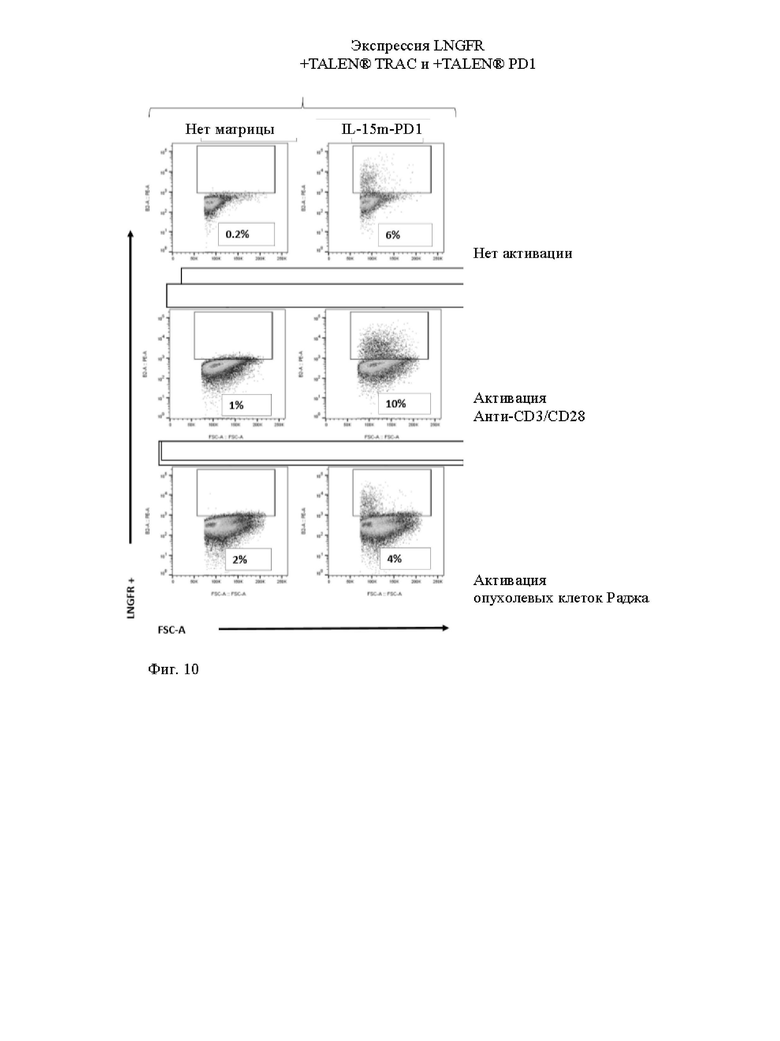
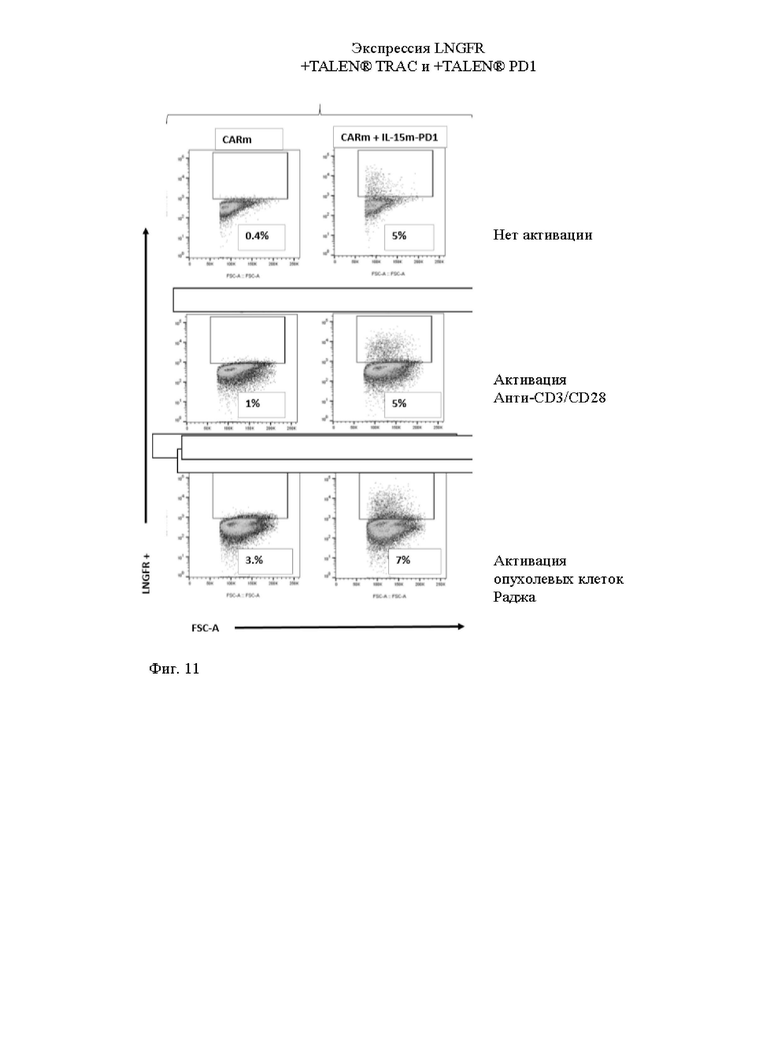
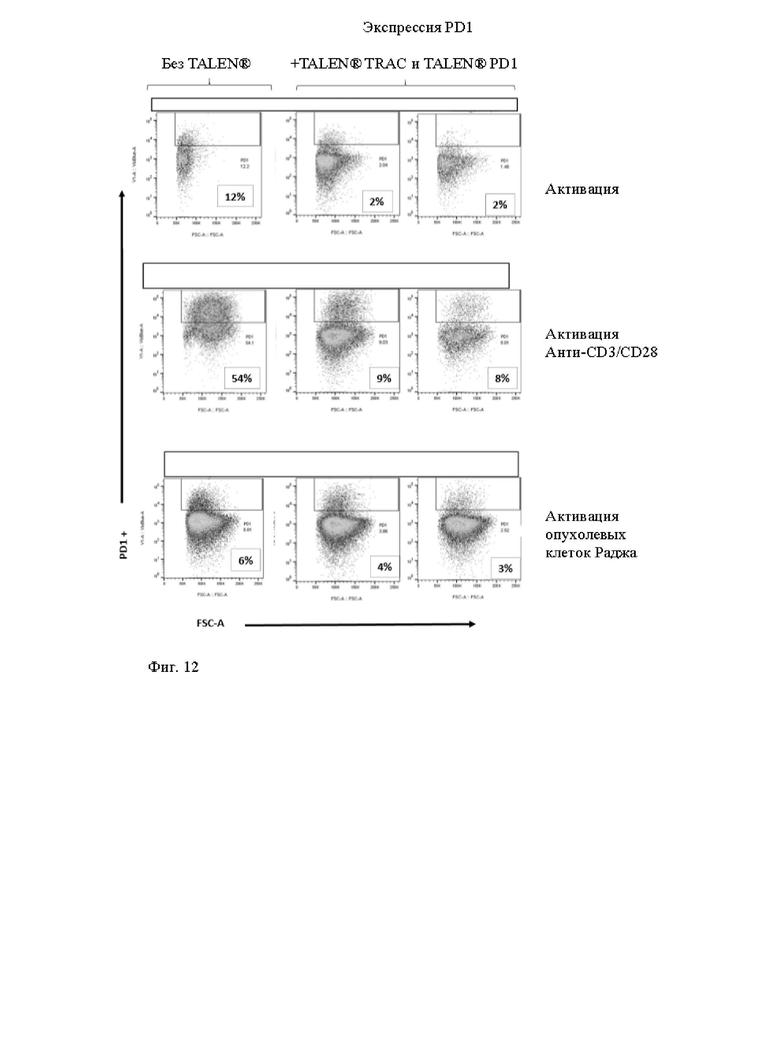
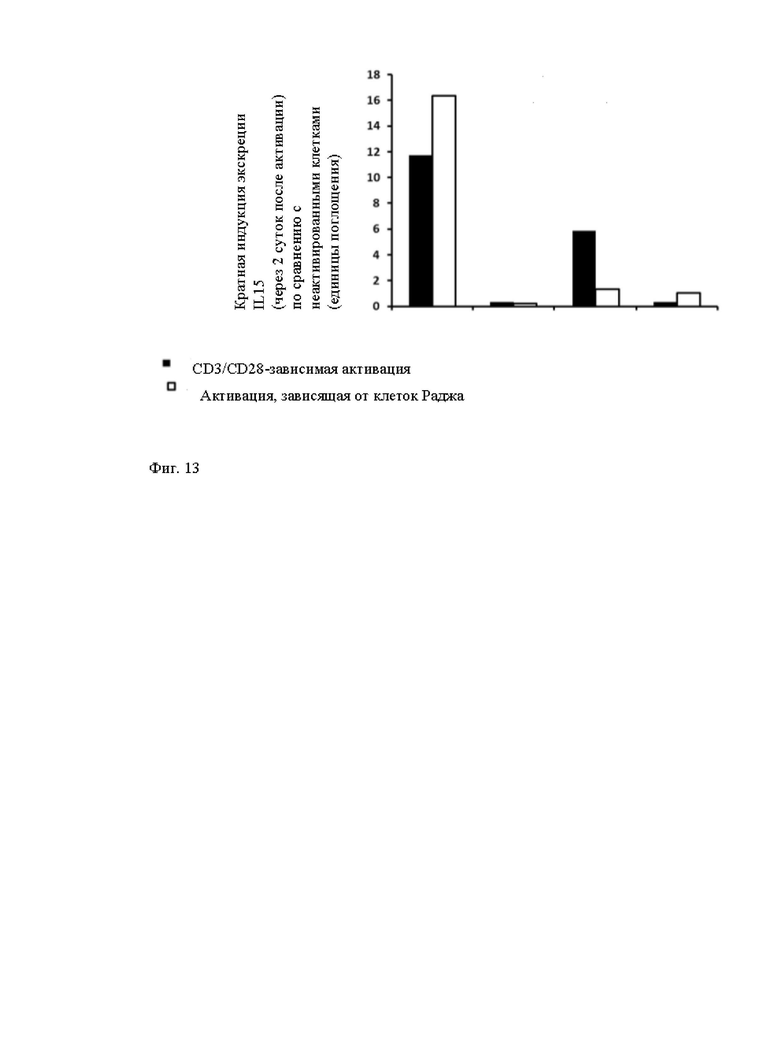
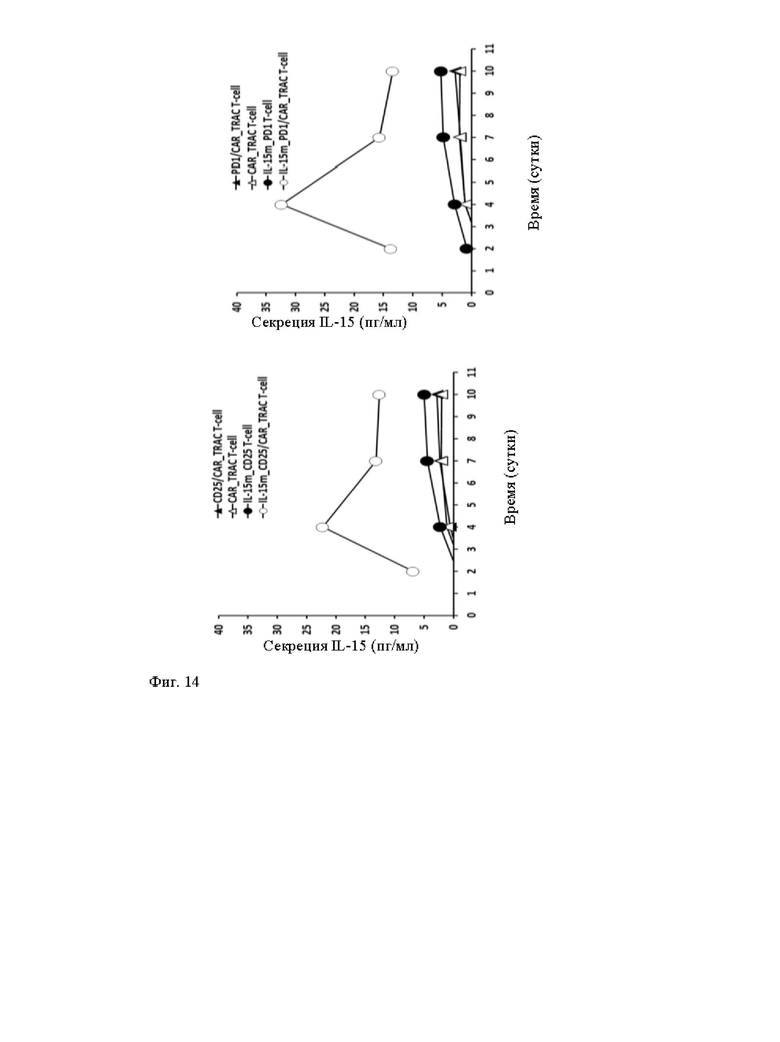
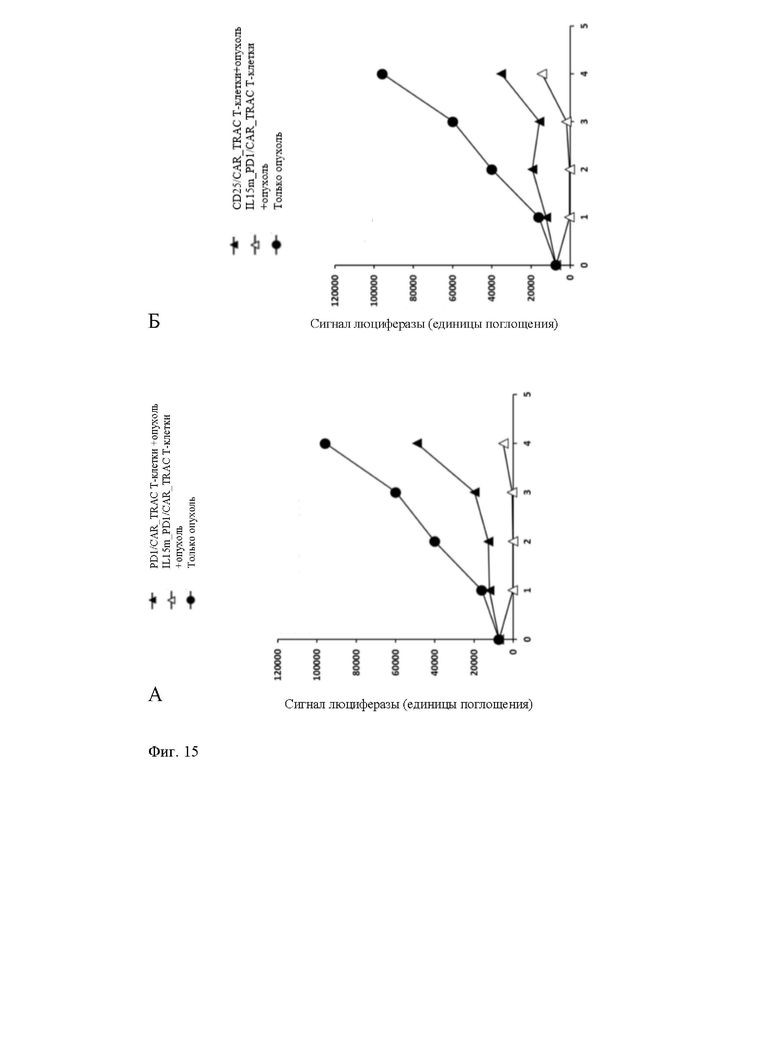
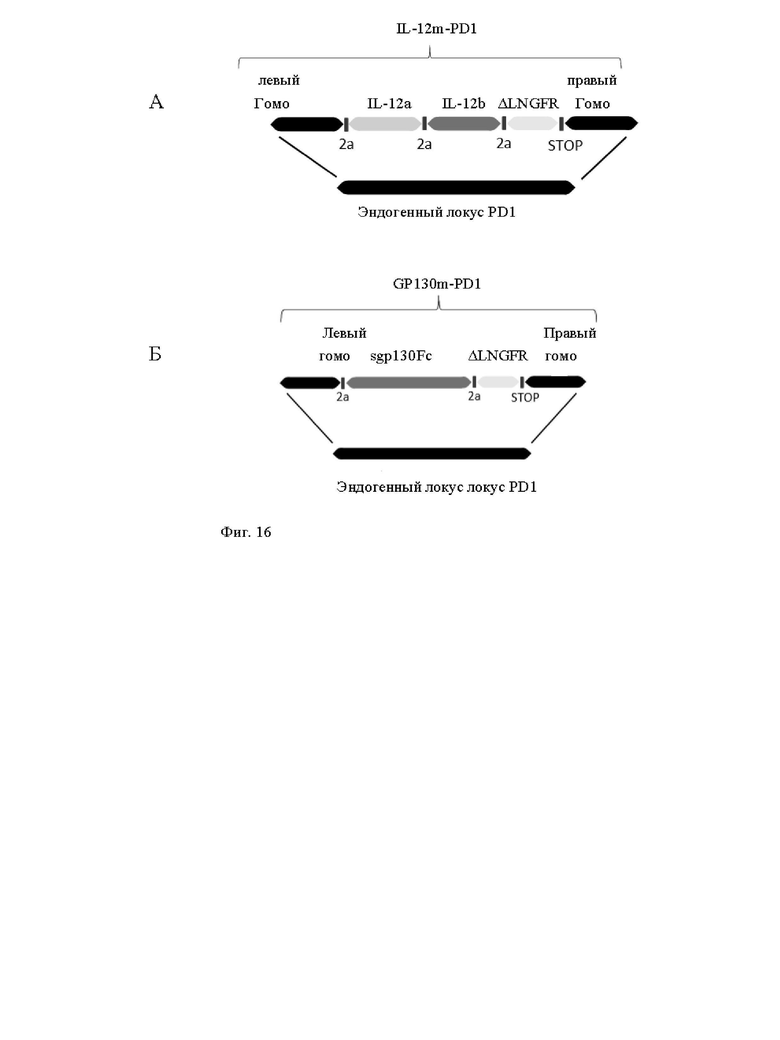
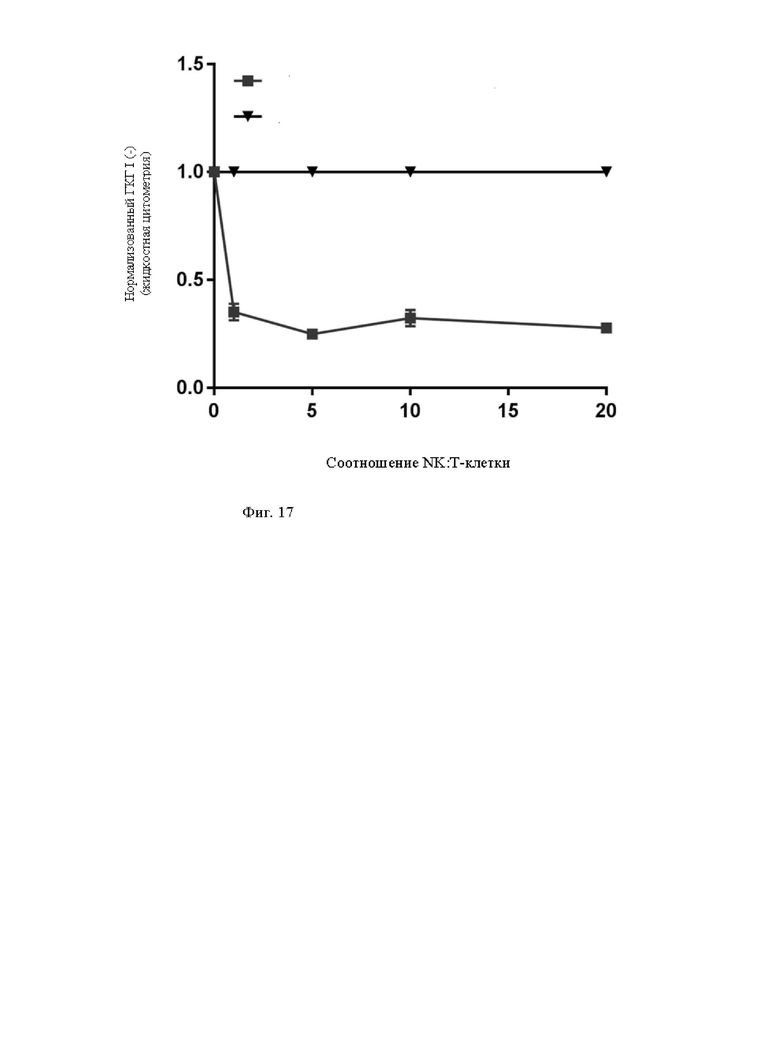
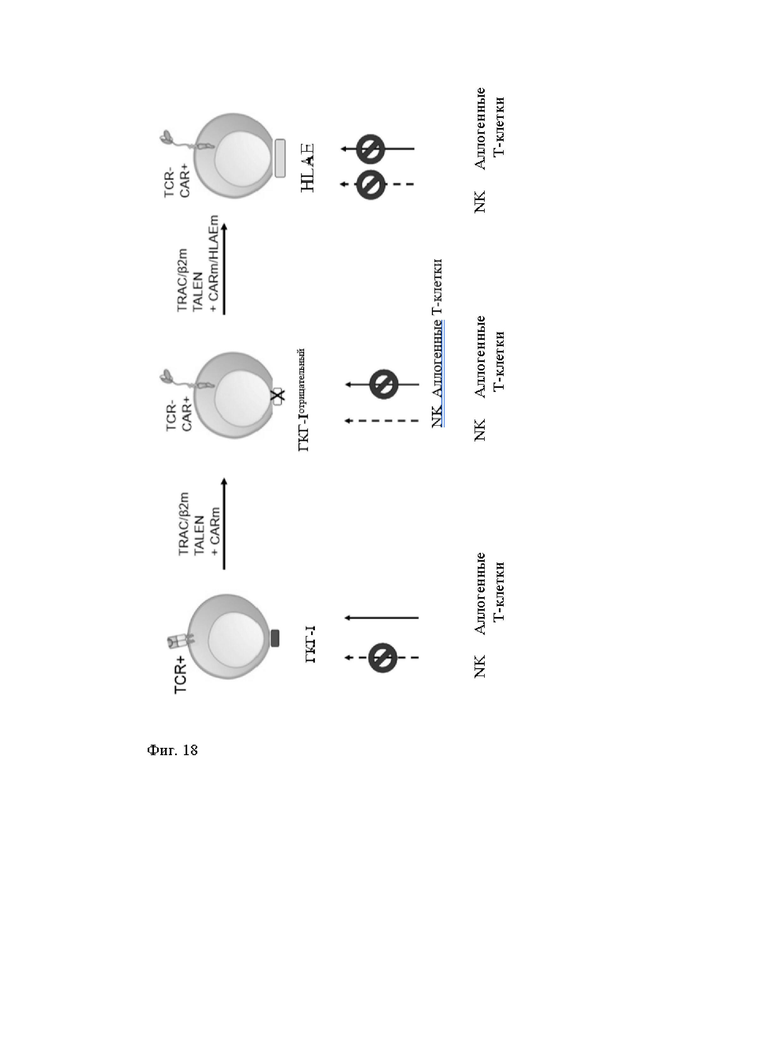
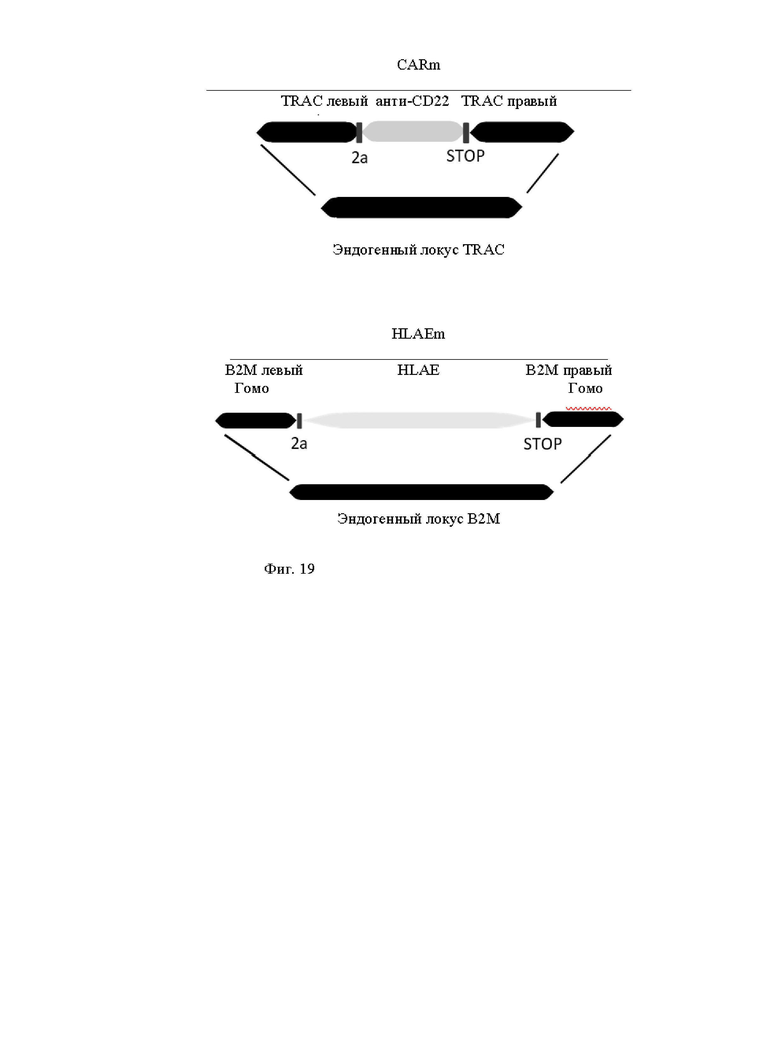


Изобретение относится к области биотехнологии, конкретно к адаптивной клеточной иммунотерапии, и может быть использовано в медицине для иммунотерапии рака. С целью получения сконструированных первичных Т-клеток для лечения рака с помощью иммунотерапии производят интродукцию в указанные первичные Т-клетки нуклеиновой кислоты, содержащей экзогенную полинуклеотидную последовательность, кодирующую HLA-G или HLA-E молекулу, которая должна быть интегрирована в эндогенный локус бета2m и специфического в отношении последовательности реагента, который специфически нацеливается на указанный выбранный эндогенный локус, а также снабжают указанные Т-клетки химерным антигенным рецептором (CAR), нацеленным на антиген, экспрессируемый на поверхности опухолевых клеток указанного рака. Изобретение позволяет получать более безопасные первичные Т-клетки с повышенным терапевтическим потенциалом. 5 н. и 13 з.п. ф-лы, 21 ил., 13 табл., 3 пр.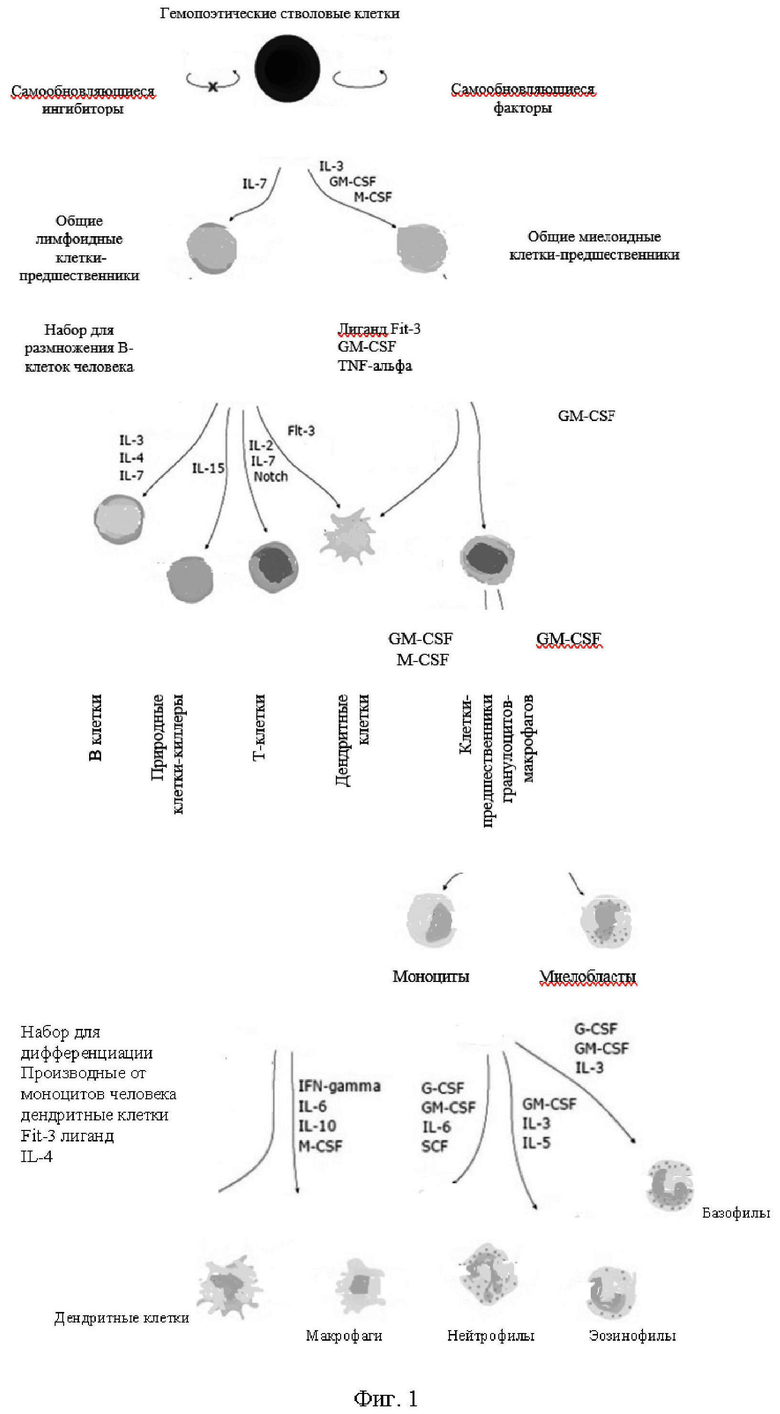
1. Способ получения сконструированных первичных Т-клеток для лечения рака с помощью иммунотерапии, включающий:
- получение популяции клеток, содержащей первичные Т-клетки,
- интродукцию в указанные первичные Т-клетки:
i) нуклеиновой кислоты, содержащей экзогенную полинуклеотидную последовательность, которая должна быть интегрирована в эндогенный локус β2m,
ii) специфического в отношении последовательности реагента, который специфически нацеливается на указанный выбранный эндогенный локус,
- снабжение указанных Т-клеток химерным антигенным рецептором (CAR), нацеленным на антиген, экспрессируемый на поверхности опухолевых клеток указанного рака,
где указанную экзогенную полинуклеотидную последовательность инсертируют путем направленной генной интеграции в указанный эндогенный локус β2m;
где указанная экзогенная полинуклеотидная последовательность включает последовательность, кодирующую по меньшей мере одну HLA-G или HLA-E молекулу, для экспрессии указанной молекулы; и
где указанный специфичный для последовательности реагент выбран из РНК- управляемой эндонуклеазы, TAL-эндонуклеазы, нуклеазы цинковых пальцев или хоминг эндонуклеазы.
2. Способ по п. 1, в котором указанную экзогенную полинуклеотидную последовательность интегрируют под контроль транскрипции эндогенного промотора, присутствующего в указанном β2m локусе.
3. Способ по п. 2, в котором указанная экзогенная последовательность включает последовательность, кодирующую HLA-G или HLA-E, и при интеграции в эндогенный локус β2m приводит к экспрессии фрагментов β2m, слитых с HLA-Е или HLA-G.
4. Способ по п. 3, в котором указанные фрагменты β2m, слитые с HLA-Е или HLA-G, приводят к экспрессии тримеров HLA-E или HLA-G.
5. Способ по любому из пп. 1-4, в котором указанная инсерция указанной экзогенной последовательности инактивирует экспрессию β2m в указанном эндогенном локусе.
6. Способ по любому из пп. 1-5, в котором экзогенная полинуклеотидная последовательность содержит полинуклеотидную последовательность SEQ ID NO: 69 или 71.
7. Способ по любому из пп. 1-6, где экзогенная последовательность, кодирующая указанный химерный антигенный рецептор (CAR), интегрирована в локус TCR.
8. Способ по п. 7, в котором указанные экзогенные последовательности, кодирующие указанный химерный антигенный рецептор (CAR), предотвращают экспрессию эндогенных последовательностей TCR.
9. Способ по любому из пп. 1-8, в котором указанные первичные Т-клетки являются первичными Т-клетками человека.
10. Способ по любому из пп. 1-9, в котором указанная популяция клеток, содержащая указанные первичные Т-клетки, была получена от донора, у которого не диагностирован рак или инфекция.
11. Сконструированная Т-клетка для применения для лечения рака с помощью иммунотерапии, которая содержит экзогенную полинуклеотидную последовательность, кодирующую HLA-Е или HLA-G молекулу, которая была интегрирована под контроль эндогенного промотора транскрипции β2m гена, и при этом указанная Т-клетка снабжена химерным антигенным рецептором (CAR), нацеленным на антиген, экспрессируемый на поверхности опухолевых клеток указанного рака.
12. Сконструированная Т-клетка по п. 11, в котором экзогенная полинуклеотидная последовательность содержит полинуклеотидную последовательность SEQ ID NO: 69 или 71.
13. Сконструированная Т-клетка по любому из пп. 11-12, в которой указанная T-клетка имеет генотип [TCR]отрицательный[β2m]отрицательный.
14. Сконструированная Т-клетка по п. 13, в которой экзогенная последовательность, кодирующая указанный CAR, интегрирована в TCR локус.
15. Сконструированная Т-клетка по любому из пп. 11-14, в которой указанная Т-клетка является первичной Т-клеткой.
16. Применение сконструированной Т-клетки по любому из пп. 11-15 для лечения рака с помощью иммунотерапии.
17. Популяция иммунных клеток для лечения рака с помощью иммунотерапии, причем указанная популяция включает по меньшей мере 30% сконструированных Т-клеток по любому из пп. 11-15.
18. Фармацевтическая композиция для лечения рака с помощью иммунотерапии, указанная популяция содержит популяцию по п. 17 в терапевтически эффективном количестве.
| WO 2016142532 A1, 15.09.2016, WO 2015136001 Al, 17.09.2015, LU P | |||
| et al., Generating hypoimmunogenic human embryonic stem cells by the disruption of beta 2-microglobulin, Stem Cell Reviews and Reports, 2013, v | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
| Н | |||
| и др., Использование мутированного варианта человеческой деоксицитидинкиназы в качестве репортерного | |||
Авторы
Даты
2023-06-01—Публикация
2018-03-09—Подача