Область техники, к которой относится изобретение
Настоящее изобретение относится к новым биспецифическим антигенсвязывающим молекулам, содержащим (а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации. Изобретение относится также к способам получения указанных молекул и способам их применения.
Предпосылки создания изобретения
Функцией нескольких представителей семейства рецепторов фактора некроза опухоли (TNFR) является поддержание Т-клеточных ответов после начальной активации Т-клеток, и поэтому они играют основную роль в организации и функционировании иммунной системы. CD27, 4-1ВВ (CD137), ОХ40 (CD134), HVEM, CD30 и GITR могут обладать костимулятрными действиями в отношении Т-клеток, это означает, что они поддерживают Т-клеточные ответы после начальной активации Т-клеток (Watts Т.Н. Annu. Rev. Immunol. 23, 2005, сс. 23-68). Воздействия указанных костимуляторных представителей семейства TNFR часто могут быть отделены функционально, по времени или в пространстве от воздействий CD28 и друг от друга. Последовательная и кратковременная регуляция сигналов активации/выживаемости Т-клеток различными костимуляторами может выполнять функцию обеспечения длительности ответа, поддерживая при этом строгий контроль выживаемости Т-клеток. В зависимости от болезненного состояния стимуляция через костимуляторных представителей семейства TNF может обострять или облегчать заболевание. Несмотря на указанные сложности, стимуляция или блокада семейства костимуляторов TNFR является перспективной для терапевтических применений при некоторых показаниях, включая рак, инфекционное заболевание, трансплантацию и аутоимунитет.
Среди нескольких костимуляторных молекул представитель семейства рецепторов фактора некроза опухоли (TNF) ОХ40 (CD134) играет основную роль в выживаемости и гомеостазе эффекторных Т-клеток и Т-клеток памяти (Croft М. и др., Immunological Reviews 229, 2009, сс. 173-191). ОХ40 (CD134) экспрессируется в нескольких типах клеток и регулирует иммунные ответы против инфекций, опухолей и аутоантигенов, его экспрессия была выявлена на поверхности Т-клеток, NKT-клеток и NK-клеток, а также нейтрофилов (Baumann R. и др., Eur. J. Immunol. 34, 2004, сс. 2268-2275), и было продемонстрировано, что она является строго индуцибельной или подвергается сильной повышающей регуляции в ответ на различные стимуляторные сигналы. Функциональная активность молекулы была выявлена в каждом экспрессирующем ОХ40 типе клеток, что позволяет предположить наличие сложной регуляции опосредуемой ОХ40 активности in vivo. В сочетании с запуском Т-клеточного рецептора контакт ОХ40 на Т-клетках с его встречающимся в естественных условиях лигандом или агонистическими антителами приводит к синергетической активации путей передачи сигналов PI3K и NFκB (Song J. и др., J. Immunology 180(11), 2008, сс. 7240-7248). В свою очередь, это приводит к усиленной пролиферации, повышенному производству цитокинового рецептора и цитокинов и улучшенной выживаемости активированных Т-клеток. В последние годы было продемонстрировано, что в дополнение к его костимуляторной активности в эффекторных CD4+- или CD8+-Т-клетках, запуск ОХ40 ингибирует развитие и иммуносупрессорную функцию регуляторных Т-клеток. Это действие, вероятно, ответственно, по меньше мере частично, за повышенную активность ОХ40 в отношении противоопухолевых или противомикробных иммунных ответов. С учетом того, что контакт с ОХ40 может приводить к увеличению популяций Т-клеток, усилению секреции цитокинов и поддержанию памяти Т-клеток, агонисты, включая антитела и растворимые формы лиганда OX40L, успешно применяли на различных доклинических моделях опухолей (Weinberg и др., J. Immunol. 164, 2000, сс. 2160-2169).
4-1ВВ (CD137), представитель суперсемейства TNF-рецепторов, впервые был идентифицирован в качестве молекулы, экспрессия которой индуцируется Т-клеточной активацией (Kwon Y.H. и Weissman S.M., Proc. Natl. Acad. Sci. USA 86, 1989, cc. 1963-1967). В последующих исследованиях продемонстрирована экспрессия 4-1ВВ в Т- и В-лимфоцитах (Snell L.M. и др., Immunol. Rev. 244, 2011, сс. 197-217 или Zhang X. и др., J. Immunol. 184, 2-10, сс. 787-795), NK-клетках (Lin W. и др., Blood 112, 2008, сс. 699-707, NKT-клетках (Kim D.H. и др., J. Immunol. 180, 2008, сс. 2062-2068), моноцитах (Kienzle G. и von Kempis J., Int. Immunol. 12, 2010, cc. 73-82 или Schwarz H. и др., Blood 85, 1995, cc. 1043-1052), нейтрофилах (Heinisch I.V. и др., Eur. J. Immunol. 30, 2000, cc. 3441-3446), тучных клетках (Nishimoto H. и др., Blood 106, 2005, cc. 4241-4248) и дендритных клетках, а также в клетках негематопоэтического происхождения, таких как эндотелиальные клетки и клетки гладких мышц (BrolI K. и др., Am. J. Clin. Pathol. 115, 2001, cc. 543-549 или Olofsson P.S. и др., Circulation 117, 2008, cc. 1292-1301). Экспрессия 4-1BB в различных типах клеток является в значительной степени индуцибельной и запускается различными стимуляторными сигналами, такими как сигналы, которые запускаются Т-клеточным рецептором (TCR) или В-клеточным рецептором, а также передачей сигналов, индуцируемой костимуляторными молекулами или рецепторами провоспалительных цитокинов (Diehl L. и др., J. Immunol. 168, 2002, сс. 3755-3762; von Kempis J. и др., Osteoarthritis Cartilage 5, 1997, сс. 394-406; Zhang X. и др., J. Immunol. 184, 2010, сс. 787-795).
Известно, что передача сигналов CD137 стимулирует секрецию IFNγ и пролиферацию NK-клеток (Buechele С. и др., Eur. J. Immunol. 42, 2012, сс. 737-748; Lin W. и др., Blood 112, 2008, сс. 699-707; Melero I. и др., Cell Immunol. 190, 1998, cc. 167-172), а также повышает активацию DC, что продемонстрировано по их увеличенной выживаемости и способности секретировать цитокины и осуществлять повышающую регуляцию костимуляторных молекул (Choi В.K. и др., J. Immunol. 182, 2009, сс. 4107-4115; Futagawa Т. и др., Int. Immunol. 14, 2002, сс. 275-286; Wilcox R. А. и др., J. Immunol. 168, 2002, сс. 4262-4267). Однако CD137 наиболее хорошо охарактеризован в качестве костимуляторной молекулы, которая модулирует индуцируемую TCR активацию как CD4+-, так и CD8+-субпопуляций Т-клеток. В сочетании с осуществляемой TCR стимуляцией агонистические 4-1ВВ-специфические антитела повышают пролиферацию Т-клеток, стимулируют секрецию лимфокинов и понижают чувствительность Т-лимфоцитов к индуцированной активации смерти клеток (Snell L.M. и др., Immunol. Rev. 244, 2011, сс. 197-217). В соответствии с указанными костимуляторными действиями антител к 4-1ВВ на Т-клетки in vitro, их введение несущим опухоли мышам приводило к эффективным противоопухолевым действиям на многих экспериментальных моделях опухолей (Melero I. и др., Nat. Med. 3, 1997, сс. 682-685; Narazaki Н. и др., Blood 115, 2010, сс. 1941-1948). В экспериментах по истощению in vivo продемонстрировано, что CD8+-Т-клетки играют наиболее важную роль в противоопухолевом воздействии 4-1ВВ-специфических антител. Однако, в зависимости от модели опухоли или комбинированной терапии, включающей антитело к 4-1ВВ, описано участие и других типов клеток, таких как DC, NK-клетки или CD4+-Т-клетки (Murillo О. и др., Eur. J. Immunol. 39, 2009, сс. 2424-2436; Stagg J. и др., Proc. Natl. Acad. Sci. USA 108, 2011, сс. 7142-7147).
Помимо их непосредственных воздействий на различные субпопуляции лимфоцитов, агонисты 4-1ВВ могут индуцировать также инфильтрацию и удерживание активированных Т-клеток в опухоли посредством опосредуемой 4-1ВВ повышающей регуляции молекулы межклеточной адгезии 1 (ICAM1) и адгезивной молекулы сосудистых клеток 1 (VCAM1) на сосудистом эндотелии опухоли (Palazon А. и др., Cancer Res. 71, 2011, сс. 801-811). Стимуляция 4-1ВВ может также обращать состояние Т-клеточной анергии, индуцированное экспозицией растворимого антигена, что может вносить вклад в нарушение иммунологической толерантности в микроокружении опухоли или при хронических инфекциях (Wilcox R.A. и др., Blood 103, 2004, сс. 177-184).
По видимому, для проявления иммуномодулирующих свойств агонистических антител к 4-1ВВ in vivo требуется присутствие Fc-области дикого типа в молекуле антитела, это свидетельствует о том, что связывание Fc-рецептора является важным событием, необходимым для фармакологической активности указанных реагентов, что описано для агонистических антител, специфических в отношении других индуцирующих апоптоз или иммуномодулирующих представителей TNFR-суперсемейства (Li F. и Ravetch J.V., Science 333, 2011, сс. 1030-1034; Teng M.W. и др., J. Immunol. 183, 2009, сс. 1911-1920). Однако системное введение 4-1ВВ-специфических агонистических антител с функционально активным Fc-доменом индуцирует также экспансию CD8+-Т-клеток, с которой ассоциирована токсичность для печени (Dubrot J. и др., Cancer Immunol. Immunother. 59, 2010, сс. 1223-1233), что снижается или в значительной степени элиминируется в отсутствии функциональных Fc-рецепторов у мышей. В клинических испытаниях на людях (Clinical Trials. gov, NCT00309023) было установлено, что Fc-компетентные агонистические антитела к 4-1ВВ (BMS-663513) при их введении один раз каждые три недели в течение 12 недель индуцировали стабилизацию болезни у пациентов с меланомой, раком яичника или почечно-клеточной карциномой. Однако это же антитело в другом испытании (NCT00612664) вызывало гепатит 4-ой степени, что привело к прекращению испытания (Simeone Е. и Ascierto P.A. J. Immunotoxicology 9, 2012, сс. 241-247). Таким образом, существует потребность в новом поколении агонистов, которые должны не только эффективно привлекать 4-1ВВ к поверхности гематопоэтических и эндотелиальных клеток, но также должны обладать способностью достигать этого посредством механизмов, отличных от связывания с Fc-рецепторами, во избежание неконтролируемых побочных действий.
Доступные результаты доклинических и клинических испытаний четко демонстрируют, что существует значительная клиническая потребность в эффективных агонистах представителей семейства костимуляторных TNFR, таких как ОХ40 и 4-1ВВ, которые обладают способностью индуцировать и повышать эффективные эндогенные иммунные ответы против рака. Однако практически никогда эффекты не ограничивались одним типом клеток или действием посредством одного механизма, и исследования, проведенные для выявления меж- и внутриклеточных механизмов передачи сигналов, выявили повышенные уровни сложности. Таким образом, существует потребность в «нацеленных (обеспечивающих таргетинг)» агонистах, которые предпочтительно действуют на один тип клеток. В антигенсвязывающих молекулах, предлагаемых в изобретении, фрагмент, который обладает способностью связываться преимущественно с опухоль специфическими или ассоциированными с опухолями мишенями, объединен с фрагментом, который обладает способностью к агонистическому связыванию с костимуляторными TNF-рецепторами. Антигенсвязывающие молекулы, предлагаемые в настоящем изобретении, могут обладать способностью запускать (стимулировать) TNF-рецепторы не только эффективно, но также и очень избирательно в требуемом сайте, снижая тем самым нежелательные побочные действия.
Краткое изложение сущности изобретения
Настоящее изобретение относится к биспецифическим антигенсвязывающим молекулам, в которых объединен по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, т.е. по меньшей мере один антигенсвязывающий сайт, мишенью которого являются костимуляторные TNF-рецепторы, по меньшей мере с одним фрагментом, обладающим способностью специфически связываться с антигеном клетки-мишени, т.е. по меньшей мере с одним антигенсвязывающим сайтом, таргетирующим антиген клетки-мишени. Эти биспецифические антигенсвязывающие молекулы обладают преимуществом, обусловленным тем, что они могут предпочтительно активировать костимуляторные TNF-рецепторы в сайте, в котором экспрессируется антиген клетки-мишени, благодаря их способности связываться с антигеном клетки-мишени. В изобретении предложены также новые антитела, обладающие способностью к специфическому связыванию с представителем семейства костимуляторных TNF-рецепторов. По сравнению с известными антителами к костимуляторным TNF-рецепторам эти антитела обладают способностями, которые дают преимущество в отношении их включения в биспецифические антигенсвязывающие молекулы в комбинации с фрагментами, которые обладают способностью специфически связываться с антигеном клетки-мишени.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
В конкретном объекте изобретения биспецифическая антигенсвязывающая молекула содержит (а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, где представитель семейства костимуляторных TNF-рецепторов выбран из группы, состоящей из ОХ40 и 4-1ВВ, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
В одном из объектов изобретения представитель семейства костимуляторных TNF-рецепторов представляет собой ОХ40. Так, в конкретном объекте изобретения фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который содержит аминокислотную последовательность SEQ ID NO: 1.
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит по меньшей мере один фрагмент, обладающий способностью специфически связываться с ОХ40, где указанный фрагмент содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 2 и SEQ ID NO: 3,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 4 и SEQ ID NO: 5, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 и SEQ ID NO: 12, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 13, SEQ ID NO: 14 и SEQ ID NO: 15,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 16, SEQ ID NO: 17 и SEQ ID NO: 18, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23 и SEQ ID NO: 24.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 и SEQ ID NO: 37, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 и SEQ ID NO: 38.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
В другом объекте изобретения представитель семейства костимуляторных TNF-рецепторов представляет собой 4-1ВВ. Так, в конкретном объекте изобретения фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который содержит аминокислотную последовательность SEQ ID NO: 39.
Кроме того, предложена биспецифическая антигенсвязывающая молекула, которая содержит по меньшей мере один фрагмент, обладающий способностью специфически связываться с 4-1ВВ, где указанный фрагмент содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 40 и SEQ ID NO: 41,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 42 и SEQ ID NO: 43, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 47 и SEQ ID NO: 48,
и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 49 и SEQ ID NO: 50,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 51 и SEQ ID NO: 52, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 56 и SEQ ID NO: 57.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 и SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 и SEQ ID NO: 67.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, в которой фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляет собой Fab-фрагмент. В одном из объектов изобретения, если биспецифическая антигенсвязывающая молекула содержит более одного фрагмента, обладающего способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, то все молекулы, обладающие способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляют собой Fab-фрагменты.
В другом объекте изобретения биспецифическая антигенсвязывающая молекула содержит (а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, где антиген клетки-мишени выбран из группы, включающей фибробласт-активирующий белок (FAP), ассоциированный с меланомой хондроитинсульфат-протеогликан (MCSP), рецептор эпидермального фактора роста (EGFR), карциноэмбриональный антиген (СЕА), CD19, CD20 и CD33, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации. Более конкретно, антиген клетки-мишени представляет собой фибробласт-активирующий белок (FAP).
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с FAP, содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 68 и SEQ ID NO: 69,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 70 и SEQ ID NO: 71, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 72 и SEQ ID NO: 73, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 74 и SEQ ID NO: 75,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 76 и SEQ ID NO: 77, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 78 и SEQ ID NO: 79.
Таким образом, следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(I) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 или SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 или SEQ ID NO: 38, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, в которой
(I) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(I) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 или SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 или SEQ ID NO: 67, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Согласно одному объекту изобретения биспецифическая антигенсвязывающая молекула представляет собой человеческое, гуманизированное или химерное антитело. В частности, Fc-домен представляет собой Fc-домен человеческого IgG, в частности, Fc-домен IgG1 или Fc-домен IgG4.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой Fc-домен содержит одну или несколько аминокислотную(ых) замену(н), которая(ые) снижает(ют) аффинность связывания антитела с Fc-рецептором и/или эффекторную функцию. В частности, Fc-домен представляет собой Fc-домен человеческого IgG1-подкласса с аминокислотными мутациями L234A, L235A и P329G (нумерация согласно EU-индексу Кэбота).
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой Fc-домен содержит модификацию, усиливающую ассоциацию первой и второй субъединиц Fc-домена. Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой первая субъединица Fc-домена содержит «выступы», а вторая субъединица Fc-домена содержит «впадины» в соответствии с технологией «knob-into-hole» («выступ-во впадину»). Более конкретно первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (нумерация согласно EU-индексу Кэбота), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация согласно EU-индексу Кэбота).
В частности, в изобретении предложена биспецифическая антигенсвязывающая молекула, содержащая
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) два фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Таким образом, предложена биспецифическая антигенсвязывающая молекула, где биспецифическая антигенсвязывающая молекула является двухвалентной как в отношении представителя семейства костимуляторных TNF-рецепторов, так и в отношении антигена клетки-мишени.
Согласно конкретному объекту изобретения биспецифическая антигенсвязывающая молекула содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен,
и
(в) два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, в которой каждый из указанных дополнительных Fab-фрагментов соединен через пептидный линкер с С-концами тяжелых цепей, указанных в подпункте (а). Более конкретно, два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кросс-Fab-фрагменты, в которых вариабельные домены VL и VH замены друг на друга и каждая из VL-CH-цепей соединена через пептидный линкер с С-концом тяжелых цепей, указанных в подпункте (а).
Согласно одному из объектов изобретения два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляют собой два Fab-фрагмента, которые обладают способностью специфически связываться с ОХ40 или 4-1ВВ, а два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кросс-Fab-фрагменты, которые обладают способностью специфически связываться с FAP.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) один фрагмент, который обладает способностью специфически связываться с антигеном клетки-мишени,
и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Таким образом, предложена биспецифическая антигенсвязывающая молекула, где биспецифическая антигенсвязывающая молекула является двухвалентной в отношении представителя семейства костимуляторных TNF-рецепторов и одновалентной в отношении антигена клетки-мишени.
Согласно конкретному объекту изобретения биспецифическая антигенсвязывающая молекула содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(б) VH- и VL-домены, обладающие способностью специфически связываться с антигеном клетки-мишени, где VH-домен соединен через пептидный линкер с С-концом одной из тяжелых цепей и где VL-домен соединен через пептидный линкер с С-концом второй тяжелой цепи.
Другим объектом изобретения является антитело, которое специфически связывается с ОХ40, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
Другим объектом изобретения является антитело, которое специфически связывается с 4-1ВВ, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
Другим объектом изобретения является выделенный полинуклеотид, который кодирует биспецифическую антигенсвязывающую молекулу, указанную выше в настоящем описании, или антитело, которое специфически связывается с ОХ40, указанное выше в настоящем описании, или антитело, которое специфически связывается с 4-1ВВ, указанное выше в настоящем описании. В изобретении предложен также вектор, в частности, экспрессионный вектор, который содержит выделенный полинуклеотид, предлагаемый в изобретении, и клетка-хозяин, которая содержит выделенный полинуклеотид или вектор, предлагаемый в изобретении. Согласно некоторым объектам изобретения клетка-хозяин представляет собой эукариротическую клетку, в частности, клетку млекопитающего.
Другим объектом изобретения является способ получения биспецифической антигенсвязывающей молекулы, указанной выше в настоящем описании, или антитела, которое специфически связывается с ОХ40, указанного выше в настоящем описании, или антитела, которое специфически связывается с 4-1ВВ, указанного выше в настоящем описании, включающий стадии, на которых (I) культивируют клетку-хозяина, предлагаемую в изобретении, в условиях, пригодных для экспрессии антигенсвязывающей молекулы, и (II) выделяют антигенсвязывающую молекулу. Изобретение относится также к биспецифической антигенсвязывающей молекуле или антителу, которое специфически связывается с ОХ40, или антителу, которое специфически связывается с 4-1ВВ, полученной/полученному с помощью способа, предлагаемого в изобретении.
В изобретении предложена также фармацевтическая композиция, содержащая биспецифическую антигенсвязывающую молекулу, указанную выше в настоящем описании, или антитело, которое специфически связывается с ОХ40, указанное выше в настоящем описании, или антитело, которое специфически связывается с 4-1ВВ, указанное выше в настоящем описании, и по меньшей мере один фармацевтически приемлемый эксципиент.
Изобретение относится также к биспецифической антигенсвязывающей молекуле, указанной выше в настоящем описании, или антителу, которое специфически связывается с ОХ40, указанному выше в настоящем описании, или антителу, которое специфически связывается с 4-1ВВ, указанному выше в настоящем описании, или фармацевтической композиции, содержащей биспецифическую антигенсвязывающую молекулу или антитело, которое специфически связывается с ОХ40, или антитело, которое специфически связывается с 4-1ВВ, для применения в качестве лекарственного средства.
Согласно одному объекту изобретения предложна биспецифическая антигенсвязывающая молекула, указанная выше в настоящем описании, или антитело, которое специфически связывается с ОХ40, указанное выше в настоящем описании, или антитело, которое специфически связывается с 4-1ВВ, указанное выше в настоящем описании, или фармацевтическая композиция, предлагаемая в изобретении, для применения для
(I) стимуляции Т-клеточного ответа,
(II) поддержания выживаемости активированных Т-клеток,
(III) лечения инфекций,
(IV) лечения рака,
(V) замедления прогрессирования рака или
(VI) пролонгирования времени жизни пациента, страдающего раком.
Конкретным вариантом осуществления изобретения является биспецифическая антигенсвязывающая молекула, указанная выше в настоящем описании, или антитело, которое специфически связывается с ОХ40, указанное выше в настоящем описании, или антитело, которое специфически связывается с 4-1ВВ, указанное выше в настоящем описании, или фармацевтическая композиция, предлагаемая в изобретении, для применения для лечения рака.
Другим конкретным вариантом осуществления изобретения является биспецифическая антигенсвязывающая молекула, указанная выше в настоящем описании, или антитело, которое специфически связывается с ОХ40, указанное выше в настоящем описании, или антитело, которое специфически связывается с 4-1ВВ, указанное выше в настоящем описании, или фармацевтическая композиция, предлагаемая в изобретении, для применения для лечения рака, в котором биспецифическую антигенсвязывающую молекулу применяют в комбинации с химиотерапевтическим средством, лучевой терапией и/или другими агентами, предназначенными для применения в противораковой иммунотерапии.
Следующим объектом изобретения является способ ингибирования роста опухолевых клеток у индивидуума, включающий введение индивидууму в эффективном количестве биспецифической антигенсвязывающей молекулы, указанной выше в настоящем описании, или антитела, которое специфически связывается с ОХ40, указанного выше в настоящем описании, или антитела, которое специфически связывается с 4-1ВВ, указанного выше в настоящем описании, или фармацевтической композиции, предлагаемой в изобретении, для ингибирования роста опухолевых клеток.
Предложено также применение биспецифической антигенсвязывающей молекулы, указанной выше в настоящем описании, или антитела, которое специфически связывается с ОХ40, указанного выше в настоящем описании, или антитела, которое специфически связывается с 4-1ВВ, указанного выше в настоящем описании, для приготовления лекарственного средства для лечения заболевания у индивидуума, который нуждается в этом, в частности, для приготовления лекарственного средства для лечения рака, а также способ лечения заболевания у индивидуума, включающий введение указанному индивидууму в терапевтически эффективном количестве композиции, которая содержит антигенсвязывающую молекулу, содержащую тримерный лиганд семейства TNF, предлагаемую в изобретении, в фармацевтически приемлемой форме. В конкретном объекте изобретения заболевание представляет собой рак. В любом из вышеуказанных объектов изобретения индивидуум представляет собой млекопитающее, прежде всего человека.
Краткое описание чертежей
На чертежах показано:
на фиг. 1А - мономерная форма Fc-сцепленного антигена TNF-рецептора, которую применяли для получения антител к TNF-рецептору. На фиг. 1Б представлена слитая молекула, содержащая антиген димерного человеческого TNF-рецептора и Fc, с С-концевой На-меткой, которую применяли для тестирования связывания антител к TNF-рецептору в присутствии лиганда для TNF (способность блокировать лиганд). Схематический план эксперимента, описанного в примере 2.4, представлен на фиг. 1В;
на фиг. 2 - данные о связывании антител к ОХ40 с активированными CD4+- и CD8+-Т-клетками. ОХ40 не экспрессировался на покоящихся человеческих РВМС (фиг. 2А и 2В). После активации человеческих РВМС обнаружена повышающая регуляция ОХ40 на CD4+- и CD8+-Т-клетках (фиг. 2Б и 2Г). Экспрессия ОХ40 на человеческих CD8+-Т-клетках ниже, чем на CD4+-T-клетках. Указанные клоны имели различную силу связывания (величины ЕС50, а также сила сигнала) с ОХ40-позитивными клетками. Представлены данные о связывании в виде медианного значения интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию на исходный уровень путем вычитания MFI «пустого» контроля. На х-оси отложена концентрация конструкций антител. Все клоны ОХ40 могли связываться с активированными экспресирующими ОХ40 человеческими CD4+-Т-клетками и в меньшей степени с активированными человеческими CD8+-Т-клетками;
на фиг. 3 - данные о связывании антител к ОХ40 с активированными мышиными CD4+- и CD8+-Т-клетками. ОХ40 не был обнаружен на покоящихся мышиных спленоцитах (фиг. 3А и 3В). После активации обнаружена повышающая регуляция ОХ40 на CD4+- и CD8+-Т-клетках (фиг. 3Б и 3Г). Мышиные спленоциты выделяли путем гемолиза с помощью АСК-буфера для лизиса механически гомогенизированных селезенок, полученных из самок мышей линии C57BL/6 возрастом 6-8 недель. Связывание антител к ОХ40 с белками клеточной поверхности определяли с использованием вторичного козьего антитела к человеческому IgG, Fc-специфического, конъюгированного с ФИТЦ, используя FACS-анализ. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию на исходный уровень путем вычитания MFI «пустого» контроля. На х-оси отложена концентрация конструкций антител. Только клон 20В7 мог связываться с активированными экспрессирующими ОХ40 мышиными CD4- и CD8 Т-клетками, но не с покоящимися Т-клетками;
на фиг. 4 - данные о связывании антител к ОХ40 с активированными CD4+- и CD8+-Т-клетками обезьян циномолгус. Указанные клоны имели различную силу связывания (величины ЕС50, а также сила сигнала) с активированными ОХ40-позитивными CD4+-Т-клетками обезьян циномолгус (фиг. 4А). Экспрессия ОХ40 на активированных CD8+-Т-клетках была ниже в этих условиях и практически никакого связывания отобранных клонов не обнаружено (фиг. 4Б). Связывание антител к ОХ40 с белками клеточной поверхности определяли с использованием вторичного козьего антитела к человеческому IgG, Fc-специфического, конъюгированного с ФИТЦ, используя FACS-анализ. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию на исходный уровень путем вычитания MFI «пустого» контроля. На х-оси отложена концентрация конструкций антител. Все клоны антител к ОХ40 могли связываться с активированными экспресирующими ОХ40 CD4+-Т-клетками обезьян циномолгус и в меньшей степени с активированными CD8+-Т-клетками обезьян циномолгус;
на фиг. 5 - данные, демонстрирующие отсутствие связывания с ОХ40-негативными опухолевыми клетками. Указанные клоны не обладали способностью связываться с ОХ40-негативными U-78 MG (фиг. 5А) и WM266-4 линиями опухолевых клеток (фиг. 5Б). Представлены данные о связывании в виде медианного значения интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию на исходный уровень путем вычитания MFI «пустого» контроля. На х-оси отложена концентрация конструкций антител. Все клоны в IgG-формате не связывались с ОХ40-негативными опухолевыми клетками. Связывание было специфическим для ОХ40 на активированных лейкоцитах;
на фиг. 6А-6Е - данные о взаимодействии между антителами к ОХ40 8Н9 (фиг. 6А), 20В7 (фиг. 6Б), 1G4 (фиг. 6В), 49В4 (фиг. 6Г), CLC-563 (фиг. 6Д) и CLC-564 (фиг. 6Е) и предварительно полученным комплексом лиганд huOX40/huOX40-Fc, измеренные с помощью поверхностного плазмонного резонанса;
на фиг. 7 - данные о воздействии антител к человеческому ОХ40, предлагаемых в изобретении, на клетки HeLa, которые экспрессируют человеческий ОХ40 и репортерный ген NF-κВ-люциферазы. Представлены данные об активации пути передачи сигнала NF-κВ в репортерной линии клеток различными анти-ОХ40-связывающими агентами в формате P329GLALA huIgG1 с (фиг. 7Б) или без (фиг. 7А) перекрестного сшивания вторичным антителом. Репортерные клетки культивировали в течение 6 ч в присутствии конструкций антител к ОХ40 в указанных концентрациях с или без перекрестносшивающего вторичного антитела в виде F(ab')2-фрагмента поликлонального козьего IgG к huIgG1, Fcγ-специфического в соотношении 1:2. Активность характеризовали, строя график количества единиц испускаемого света (URL) при измерении в течение 0,5 с в зависимости от концентрации (в нМ) тестируемой конструкции антитела к ОХ40. Испускание URL происходит в результате опосредуемого люциферазой окисления люциферина с образованием оксилюциферина. Все клоны обладали способностью индуцировать активацию NFκB, когда ось ОХ40 стимулировалась в человеческой репортерной клеточной линии ОХ40+. Таким образом, все клоны обладали агонистическим действием и осуществляли активацию в зависимости от дозы. Перекрестное сшивание вторичными Fc-фрагмент-специфическими Ат существенно повышало указанное агонистическое действие;
на фиг. 8А-8Е - данные о биологической активности антител к человеческому ОХ40 в предварительно активированных человеческих CD4-T-клетках. Костимуляция иммобилизированными на планшете анти-ОХ40-связывающими агентами (формат huIgG1 P329GLALA) усиливала клеточную пролиферацию и созревание субоптимально рестимулированных человеческих CD4-Т-клеток и индуцировала фенотип повышенной активации. Предварительно активированные ФГА-L меченные CFSE человеческие CD4-Т-клетки культивировали в течение 4 дней на планшетах, предварительно сенсибилизированных мышиными IgG, Fcγ-специфическими антителами, человеческими IgG, Fcγ-специфическими антителами (оба в концентрации 2 мкг/мл), мышиными антителами к человеческому CD3 (клон OKT3, [3 нг/мл]) и титрованными анти-ОХ40-связывающими агентами (формат huIgG1 P329GLALA). Представлены данные о количестве случаев (присутствия живых клеток) (фиг. 8А), проценте пролиферации (CFSElow)-клеток (фиг. 8Б), проценте эффекторных Т-клеток (CD127low/CD45RAlow) (фиг. 8В) и проценте CD62Llow (фиг. 8Г), ОХ40-позитивных (фиг. 8Е) или Tim-3-позитивных клеток (фиг. 8Д) в день 4. Вычитали исходные уровни, полученные для образцов, содержащих только иммобилизированное на планшете антитело к человеческому CD3. Таким образом, результаты свидетельствуют о возрастающем воздействии стимуляции ОХ40, но об отсутствии воздействия субоптимальной стимуляции антителом к CD3 per se. Все клоны обладали способностью поддерживать субоптимальную стимуляцию TCR в ОХ40-позитивных предварительно активированных CD4-T-клетках, когда ими сенсибилизировали планшет. Для клеток характерна более высокая выживаемость и более высокая пролиферация. Это могло приводить к повышенной противоопухолевой активности Т-клеток в микроокружении опухоли;
на фиг. 9 - обобщение величин ЕС50 (для всех биомаркеров) в качестве меры агонистической активности соответствующего клона (величины рассчитаны на основе кривых, представленных на фиг. 8). Эффективность возрастала слева направо. Строили график зависимости количества случаев, процента пролиферации (CFSE-low)-клеток и процента CD62L-low-, CD45RA-low- или Tim-3-позитивных клеток в день 4 от концентрации антитела к ОХ40, и величины ЕС50 рассчитывали на основе построенной с использованием котировок сигмовидной кривой доза-ответ, заложенных в программу Prism4 (программное обеспечение GraphPad, США);
на фиг. 10А-10Е - данные о биологической активности антител к человеческому ОХ40 для предварительно активированных человеческих CD4-T-клетках в растворе. Никакого воздействия на пролиферацию, созревание или статус активации субоптимально рестимулированных человеческих CD4-T-клеток не обнаружено в отсутствии иммобилизации на планшете анти-ОХ40-связывающих агентов (формат huIgG1 P329GLALA). Предварительно активированные ФГА-L меченные CFSE человеческие СВ4-Т-клетки культивировали в течение 4 дней на планшетах, предварительно сенсибилизированных мышиными IgG, Fcγ-специфическими антителами, и мышиными антителами к человеческому CD3 (клон OKT3, [3 нг/мл]). Титрованные анти-ОХ40-связывающие агенты (формат huIgG1 P329GLALA) добавляли в среды, и они присутствовали в растворе на протяжении эксперимента. Представлены данные о количестве случаев (фиг. 10А), проценте пролиферации (CFSElow)-клеток (фиг. 10Б), проценте эффекторных Т-клеток (CD127low/CD45RAlow) (фиг. 10В) и проценте CD62L-low (фиг. 10Г), ОХ40-позитивных (фиг. 10Е) или Tim-3-позитивных клеток (фиг. 10Д) в день 4. Вычитали исходные уровни для образцов, содержащих только иммобилизированное на планшете антитело к человеческому CD3. Таким образом, результаты свидетельствуют о возрастающем воздействии стимуляции ОХ40, но об отсутствии воздействия субоптимальной стимуляции антителом к CD3 per se. Не обнаружена повышенная стимуляция TCR в отсутствии сильного перекрестного сшивания (формат P329GLALA в растворе). Таким образом, перекрестное сшивание является важным для того, чтобы а ОХ40 (антитела к ОХ40) в двухвалентном формате оказывали агонистическое действие на Т-клетки. Указанное перекрестное сшивание обусловливается FAP, экспрессируемым на клеточной поверхности опухолевых клеток или клеток стромы опухоли при применении «нацеленных» (обеспечивающих таргетинг) форматов;
на фиг. 11 - данные о корреляции силы связывания и агонистической активности различных клонов антител к ОХ40. Связывание клонов антител к ОХ40 (формат huIgG1 P329GLALA) на активированных CD4-Т-клетках осуществляли согласно методу, описанному в примере 2.1.2. Уровни плато стандартизовали относительно величины, полученной при использовании клона 8Н9 (формат huIgG1 P329GLALA). Оценку биологической активности клонов антител к ОХ40 (формат huIgG1 P329GLALA) осуществляли согласно методу, описанному в примере 3.2, и уровни плато экспрессии PD-1 стандартизовали относительно уровней, полученных для клона 8Н9 (формат huIgG1 P329GLALA). Строили график зависимости стандартизованных уровней связывания относительно стандартизованной биологической активности для определения корреляции между силой связывания и агонистической активностью. Для большинства клонов обнаружена прямая корреляция (представлен график, полученный метом линейной регрессии, р-значение 0,96; угол наклона 0,91). Однако для двух клонов (49В4, 1G4) обнаружена существенно более сильная биологическая активность, чем можно было прогнозировать на основе их силы связывания. Указанная подгруппа клонов, для которой обнаружена неожиданно высокая анонисточеская способность, несмотря на низкую связывающую активность, является особенно интересной в качестве биспецифических антигенсвязывающих молекул, предлагаемых в настоящем изобретении;
на фиг. 12А - схематическое изображение приведенной в качестве примера биспецифической двухвалентной антигенсвязывающей молекулы, предлагаемой в изобретении, которая содержит два Fab-фрагмента, связывающихся с ОХ40, и два кросс-Fab-фрагмента, которые связываются с FAP (формат 2+2). На фиг. 12Б представлено схематическое изображение приведенной в качестве примера биспецифической одновалентной антигенсвязывающей молекулы (формат 1+1), предлагаемой в изобретении, которая содержит один Fab-фрагмент, связывающийся с ОХ40, и один кросс-Fab-фрагмент, который связывается с FAP. На фиг. 12В представлена схема SPR-экспериментов, демонстрирующих одновременное связывание с иммобилизированным человеческим ОХ40 или человеческим 4-1ВВ и человеческим FAP. На фиг. 12Г представлено схематическое изображение приведенной в качестве примера биспецифической антигенсвязывающей молекулы, которая является двухвалентной в отношении связывания ОХ40 и одновалентной в отношении связывания FAP. Она содержит два Fab-фрагмента, связывающихся с ОХ40, и VH- и VL-домены, связывающиеся с FAP. Жирной черной точкой обозначены модификации типа «knob-into-hole» (kih) в тяжелых цепях. На фиг. 12Д представлена схема SPR-экспериментов, демонстрирующих связывание с FAP, которые описаны в примере 5.4.1. На фиг. 12Е представлен путь измерения одновременного связывания с иммобилизованным человеческим ОХ40 и человеческим FAP (пример 5.4.1);
на фиг. 13А-13Г - SPR-диаграммы, описывающие одновременное связывание биспецифических двухвалентных конструкций 2+2 (аналит 1) с иммобилизированным человеческим ОХ40 и человеческим FAP (аналит 2). На фиг. 13Д-13З представлены данные об одновременном связывании биспецифических одновалентных конструкций 1+1 (аналит 1) с иммобилизированным человеческим ОХ40 и человеческим FAP (аналит 2). На фиг. 13К-13М представлены данные об одновременном связывании биспецифических конструкций 2+1 (аналит 1) с иммобилизированным человеческим ОХ40 и человеческим FAP (аналит 2). На фиг. 13Н представлены данные о связывании с huOX40 биспецифических конструкций 2+1, полученные с помощью клеточного FRET-анализа (фирма TagLite) (пример 5.4.2). Данные демонстрируют, что два анти-ОХ40 Fab-домена в конструкциях 2+1 связываются с huOX40 аналогично обычному антителу IgG-типа;
на фиг. 14А-14Г и 14Д-14З - данные о связывании отобранных анти-ОХ40-связывающих агентов (клон 8Н9, 1G4) в нацеленном на FAP одновалентном или двухвалентном формате с покоящимися или активированными человеческими РВМС соответственно. Характеристики связывания с ОХ40-позитивными Т-клетками (фиг. 14Б и 14Г) оказались сопоставимыми для клонов в каноническом двухвалентном huIgG-формате (незакрашенный квадрат) и нацеленном на FAP двухвалентном формате (закрашенный квадрат). Связывание одного и того же клона в нацеленном на FAP одновалентном формате (закрашенный треугольник) оказалось выражено более слабым из-за отсутствия авидности связывания. В отсутствии экспрессирующих человеческий ОХ40 клеток не удалось обнаружить никакого связывания (покоящиеся клетки, левые графики). Представлены данные о связывании в виде медианного значения интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию на исходный уровень путем вычитания MFI «пустого» контроля (см. пример 4.3.2.1). DP88 huIgG1 P329G LALA представляет собой антитело, применяемое в качестве контроля изотипа. На х-оси отложена концентрация конструкций антител. На фиг. 14А-14Г продемонстрировано, что клон 1G4 связывался с активированными экспрессирующими ОХ40 человеческими CD4-Т-клетками и в меньшей степени с активированными человеческими CD8-Т-клетками. Двухвалентная конструкция связывалась сильнее, чем одновалентная конструкция. Конструкции не связывались с ОХ40-негативными покоящимися Т-клетками. На фиг. 14Д-14З продемонстрировано, что клон 8Н9 связывался с активированными экспрессирующими ОХ40 человеческими CD4-Т-клетками и в меньшей степени с активированными человеческими CD8 Т-клетками. Двухвалентная конструкция связывалась сильнее, чем одновалентная конструкция. Конструкции не связывались с ОХ40-негативными покоящимися Т-клетками. На фиг. 14К-14Н продемонстрировано также, что двухвалентные нацеленные на FAP ОХ40-конструкции обладали характеристиками более сильного связывания с ОХ40-позитивными клетками по сравнению с соответствующим клоном в формате одновалентного антитела. На фиг. 14O-14С продемонстрировано, что различные конструкции 2+1 связывались со сходной силой с ОХ40-позитивными Т-клетками вне зависимости от второго связывающего фрагмента;
на фиг. 15А и 15Б - данные о связывании отобранных анти-ОХ40-связывающих агентов (клон 8Н9, 1G4) в нацеленном на FAP одновалентном или двухвалентном формате с FAP-позитивными опухолевыми клетками. Трансгенные модифицированные фибробласты мышиных эмбрионов линии NIH/3T3-huFAP, клон 39 или клетки WM266-4 экспрессируют высокие уровни человеческого фибробласт-активирующего белка (huFAP). Только нацеленные на FAP одно- и двухвалентные анти-ОХ40 конструкции (закрашенный квадрат и треугольник), но не этот же клон в формате человеческого IgG1 P329GLALA (незакрашенный квадрат) связывались с клетками NIH/3T3-huFAP, клон 39 (фиг. 15А) и клетками WM266-4 (фиг. 15Б) соответственно. Представлены данные о связывании в виде медианного значения интенсивности флуоресценции (MFI) меченного с помощью флуоресцеинизотиоцианата (ФИТЦ) F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела. MFI измеряли с помощью проточной цитометрии. На х-оси отложена концентрация конструкций антител. Двухвалентная FAP-конструкция связывалась сильнее, чем одновалентная конструкция. Данные о связывании с экспрессирующими человеческий FAP опухолевыми клетками представлены также на фиг. 15В, 15Г, 15Д и 15Е (более подробно см. в примере 4.5.4.2);
на фиг. 16А-16Ж - данные об активации NFκB отобранными связывающими агентами (8Н9, 1G4) в одновалентном или двухвалентном нацеленном на FAP формате в присутствии перекрестной гипер-сшивки или без нее. Представлены данные об активации пути передачи сигнала NF-κВ в репортерных клетках отобранными связывающими агентами 1G4 (фиг. 16А-16Г) и 8Н9 (фиг. 16Д-16Ж) в одновалентном (закрашенный треугольник) или двухвалентном (закрашенный квадрат) нацеленном на FAP формате или в виде «ненацеленных» антител huIgG P329GLALA (незакрашенный квадрат). Перекрестную гиперсшивку обеспечивали либо с помощью анти-hu IgG, Fcγ-специфических вторичных антител (соотношение первичных и вторичных антител 1:2), либо посредством экспрессирующих FAP опухолевых клеток NIH/3T3-huFAP, клон 39 и WM266-4 (соотношение опухолевых FAP+-клеток и репортерных клеток 2:1). Опосредуемую NF-κВ люциферазную активность характеризовали путем блоттинга единиц испускаемого света (URL) при измерении в течение 0,5 с в зависимости от концентрации (в нМ) тестируемой конструкции антитела к ОХ40. Испускание URL происходит в результате опосредуемого люциферазой окисления люциферина с образованием оксилюциферина. Осуществляли коррекцию значений на исходный уровень путем вычитания URL «пустого» контроля. На фиг. 16А продемонстрировано, что все конструкции, содержащие клон 1G4, обладали способностью индуцировать активацию NFkB в репортерных ОХ40+-клетках HeLa. Перекрестное сшивание вторичным анти-IgG, Fcγ-специфическим антителом сильно повышало активацию NFkB. Однако добавление FAP-позитивных клеток (NIH или WM266-4) приводило к повышению только агонистического потенциала нацеленных на FAP молекул, но не молекул формата P329GLALA IgG. Двухвалентные конструкции обладали выраженно более высокой активностью, чем одновалентные конструкции. На фиг. 16Д-16Ж продемонстрировано, что все конструкции, содержащие клон 8Н9, обладали способностью индуцировать активацию NFkB в репортерных ОХ40+-клетках HeLa. Перекрестное сшивание вторичным анти-IgG, Fcγ-специфическим антителом сильно повышала активацию NFkB. Однако добавление FAP-позитивных клеток (NIH) приводило к повышению только агонистического потенциала нацеленных на FAP молекул, но не молекул формата P329GLALA IgG. Двухвалентные конструкции обладали выраженно более высокой активностью, чем одновалентные конструкции. На фиг. 16З, 16К и 16Л продемонстрировано, что конструкции с одновалентным связыванием с ОХ40 (1+1) оказались менее эффективными, чем конструкции с двухвалентным связыванием с ОХ40 (конструкции 2+1 и 2+2). Дополнительные данные представлены на фиг. 16М, 16Н и 16O и в примере 5.1.
На фиг. 17А и 17Б и фиг. 18А-18Г - данные о поддержании субоптимальной рестимуляции TCR предварительно активированных CD4-Т-клеток иммобилизированными на планшете нацеленными на FAP одно- и двухвалентными конструкциями анти-ОХ40 (1G4 и 8Н9). Костимуляция иммобилизированными на планшете анти-ОХ40-связывающими агентами (формат huIgG1 P329GLALA) усиливала клеточную пролиферацию и созревание субоптимально рестимулированных человеческих CD4-Т-клеток и индуцировала фенотип повышенной активации. Предварительно активированные ФГА-L меченные CFSE человеческие CD4-Т-клетки культивировали в течение 4 дней на планшетах, предварительно сенсибилизированных мышиными IgG, Fcγ-специфическими антителами, человеческими IgG, Fcγ-специфическими антителами (оба в концентрации 2 мкг/мл), мышиными антителами к человеческому CD3 (клон OKT3, [3 нг/мл]) и титрованными анти-ОХ40-связывающими агентами (формат huIgG1 P329GLALA). Представлены данные о количестве случаев, проценте пролиферации (CFSElow)-клеток, проценте эффекторных Т-клеток (CD127low/CD45RAlow) и проценте CD62Llow, ОХ40-позитивных или Tim-3-позитивных клеток в день 4. Вычитали исходные уровни для образцов, содержащих только иммобилизированное на планшете антитело к человеческому CD3. Таким образом, результаты свидетельствуют о возрастающем воздействии стимуляции ОХ40, но об отсутствии воздействии субоптимальной стимуляции антителом к CD3 per se. На фиг. 17 продемонстрировано, что все конструкции, содержащие клон 8Н9, обладали способностью поддерживать субоптимальную стимуляцию TCR предварительно активированных ОХ40+ CD4-Т-клеток при их применении для сенсибилизации планшета. Клетки имели более активированный (Tim3+ FSC+) фенотип. Двухвалентные конструкции обладали несколько более высокой активностью, чем одновалентные конструкции. На фиг. 18 продемонстрировано, что все конструкции, содержащие клон 1G4, обладали способностью поддерживать субоптимальную стимуляцию TCR предварительно активированных ОХ40+ CD4-Т-клеток при применении для сенсибилизации планшета. Клетки характеризовались более высоким уровнем пролиферации и активированным (Tim3+ Ох40+) фенотипом. Двухвалентные конструкции обладали несколько более высокой активностью, чем одновалентные конструкции. Обе двухвалентные конструкции обладали сопоставимой активностью при применении для сенсибилизации планшета;
на фиг. 19 - величины EC50, рассчитанные на основе поддержания субоптимальной стимуляции TCR иммобилизированными на планшете нацеленными на FAP одно- и двухвалентными конструкциями анти-ОХ40 (клон 1G4). Строили график зависимости процента пролиферирующих (CFSElow) клеток и процента CD127Llow, Tim-3-позитивных и ОХ40-позитивных клеток в день 4 от концентрации антитела к ОХ40, и рассчитывали величины ЕС50 в качестве меры агонистической активности на основе построенной с помощью котировок сигмовидной кривой доза-ответ в программе Prism4 (программное обеспечение GraphPad, США). Все конструкции, содержащие клон 1G4, обладали способностью поддерживать субоптимальную стимуляцию TCR предварительно активированных ОХ40+ CD4- Т-клеток при применении для сенсибилизации планшета. Однако двухвалентные конструкции (2+2) превышали по активности одновалентные (1+1) конструкции;
на фиг. 20А-20З - данные об опосредуемой ОХ40 костимуляции субоптимально стимулированных TCR покоящихся человеческих РВМС и роли перекрестной гипер-сшивки с помощью находящегося на клеточной поверхности FAP. Только конструкции, содержащие FAP-связывающий фрагмент, обладали способностью поддерживать субоптимальную стимуляцию TCR предварительно активированных ОХ40+ CD4-Т-клеток, когда перекрестное сшивание обеспечивалось FAP-позитивными клетками (NIH). Представлены данные либо о количестве случаев (фиг. 20Б и 20Г), проценте клеток с низким уровнем пролиферации (CFSE-high) (фиг. 20А и 20В), либо MFI для CD62L (фиг. 20Е и 20З), CD127 (фиг. 20Д) или содержащих гранзим В живых CD4+-клеток (фиг. 20Ж) и СВ8+-Т-клеток. Вычитали фоновые значения, полученные для образцов, содержащих только антитело к человеческому CD3 (клон V9, huIgG1), покоящиеся человеческие РВМС и клетки NIH/3T3-huFAP, клон 39. Таким образом, продемонстрировано усиливающее действие костимуляции ОХ40, но отсутствие воздействия субоптимальной стимуляции антителом к CD3 per se. У клеток обнаружен более высокий уровень выживаемости, пролиферции и фенотип, характерный для более сильной активации (CD62L и CD127low). Нацеленные двухвалентные конструкции обладали лишь несколько более высокой активностью по сравнению с одновалентными конструкциями. Клон 8Н9 в «ненацеленной» конструкции huIgG1P329GLALA не обладал способностью поддерживать субоптимальную стимуляцию TCR в отсутствии дополнительного перекрестного сшивания. В микроокружении FAP-позитивных опухолей это могло приводить к повышенной противоопухолевой активности Т-клеток, избегая при этом системной активации ОХ40;
на фиг. 21А-21В - данные об активации покоящейся человеческой CD4-клетки с использованием иммобилизированных на поверхности нацеленных на FAP одно- и двухвалентных анти-ОХ40 (1G4) конструкций. Костимуляция «ненацеленной» конструкцией анти-ОХ40 (1G4) huIgG1 не обладала способностью поддерживать субоптимально стимулированные TCR CD4- и CD8-Т-клеток (данные не представлены). Перекрестная гипер-сшивка нацеленных на FAP одно- и двухвалентных анти-ОХ40 конструкций благодаря присутствию NIH/3T3-huFAP, клон 39 сильно увеличивала пролиферацию, выживаемость (данные не представлены) и индуцировала повышенный активированный фенотипа человеческих CD4-клеток. Представлены данные о величинах MFI, характеризующих экспрессию CD25 на CD4-Т-клетках, и о проценте CD25+-CD4-Т-клеток. Вычитали фоновые значения, полученные для образцов, содержащих только антитело к человеческому CD3 (клон V9, huIgG1), покоящиеся человеческие РВМС и NIH/3T3-huFAP, клон 39. Агонистическое действие соединений оценивали количественно по площади под кривой с использованием программного обеспечения со встроенной функцией GraphPad, и данные представлены для трех различных конструкций анти-ОХ40 (1G4). Нацеленные двухвалентные (2+2) конструкции обладали более высокой активностью, чем одновалентные (1+1) конструкции;
на фиг. 21Г-21З и 21К-21О соответственно - данные, полученные во втором эксперименте. Одновалентная анти-ОХ40 конструкция (1+1; закрашенный треугольник) в меньшей степени обладала способностью поддерживать стимуляцию TCR, чем двухвалентные анти-ОХ40 нацеленные конструкции (наполовину закрашенный кружок, закрашенный квадрат). Двухвалентная FAP-связывающая конструкция 2+2 обладала способностью уже в более низких концентрациях поддерживать субоптимальную стимуляцию TCR по сравнению с одновалентными FAP-связывающими конструкциями 2+1. В формате 2+1 обладающий высокой аффинностью связывания с FAP клон 4В9 выраженно превышал по активности низкоаффинный клон 28Н1 (фиг. 21К-21O). Это позволяет предположить, что величины ЕС50, характеризующие биологическую активность, обусловливались связыванием с FAP (2+2>2+1(4В9)>2+1(28Н1). На фиг. 21Р представлены данные об агонистической активности каждой конструкции, полученные путем количественной оценки анализируемых маркеров по площади под кривой, и представлены на графике относительно друг друга. Обнаруженная биологическая активность оказалась наиболее высокой для конструкции FAP (28Н1) (2+2), за ней в порядке уменьшения следовала конструкция FAP (4В9) (2+1), а затем конструкция FAP (28Н1) (2+1);
на фиг. 22А и 22Б - данные, демонстрирующие, что антитела к ОХ40, содержащие Fc-области человеческих IgG1, могут индуцировать лизис ОХ40-позитивных клеток. Меченные PkH26 клетки HeLa_hOx40_NFkB_Luc 1 и свежевыделенные NK-клетки совместно культивировали при соотношении Е:Т 3:1 в течение 24 ч в присутствии антител к ОХ40 (человеческий IgG1 и человеческий IgG1 P329GLALA). Содержание LDH анализировали через 4 ч, используя набор для детекции цитотоксичности-LDH (фирма Roche, каталожный №11644793001). Через 24 ч клетки окрашивали Dapi и анализировали с помощью проточной цитометрии. Процент Dapi-позитивных мертвых клеток использовали для расчета специфического лизиса. Анти-ОХ40-связывающие агенты в формате человеческого IgG связывались с Fc-рецепторами на NK-клетках и индуцировали ADCC ОХ40-позитивных клеток-мишеней. Использование вместо указанного выше формата hu IgG1 P329GLALA предупреждало ADCC ОХ40-позитивных клеток (например, непосредственно перед этим активированных Т-клеток);
на фиг. 23А-23Г - данные о связывании с покоящимися и активированными Т-клетками четырех клонов, специфических в отношении человеческого 4-1ВВ, представленных в виде формата huIgG1 P329G LALA (закрашенный ромб: клон 25G7, закрашенный квадрат: клон 12В3, закрашенная звездочка: клон 11D5, треугольник вершиной кверху: клон 9В11), и одного специфического в отношении мышиного 4-1ВВ клона 20G2, представленного в виде формата huIgG1 P329G LALA (треугольник вершиной вниз). В качестве отрицательного контроля применяли не специфическое в отношении 4-1ВВ антитело huIgG1 P329G LALA, клон DP47 (незакрашенный серый кружок). На верхних панелях представлены данные о связывании с покоящимися CD4+-Т-клетками (фиг. 23А) и активированными CD4+-Т-клетками (фиг. 23Б), а на нижних панелях представлены данные о связывании с покоящимися CD8+-Т-клетками (фиг. 23В) и активированными CD8+-Т-клетками (фиг. 23Г). Связывание характеризовали с помощью графика зависимости медианной интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ или меченного с помощью РЕ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела, от концентрации (в нМ) тестируемых первичных анти-4-1ВВ-связывающих антител huIgG1 P329G LALA. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию значений на исходный уровень путем вычитания MFI «пустого» (без первичного антитела);
на фиг. 24А-24Г - данные о связывании с экспрессирующими 4-1ВВ мышиными Т-клеткам. Представлены данные о связывании с покоящимися и активированными мышиными Т-клетками четырех клонов связывающегося с человеческим 4-1ВВ антитела huIgG1 P329G LALA (закрашенный ромб: клон 25G7, закрашенный квадрат: клон 12В3, закрашенная звездочка: клон 11D5, треугольник вершиной кверху: клон 9В11) и одного связывающегося с мышиным 4-1ВВ антитела huIgG1 P329G LALA клона 20G2 (треугольник вершиной вниз). В качестве отрицательного контроля применяли неспецифическое для 4-1ВВ антитело DP47 huIgG1 P329G LALA (незакрашенный серый кружок). На верхних панелях представлены данные о связывании с покоящимися мышиными CD4+-Т-клетками (фиг. 24А) и активированными CD4+-Т-клетками (фиг. 24Б), а на нижних панелях представлены данные о связывании с покоящимися мышиными CD8+-T-клетками (фиг. 24В) и активированными CD8+-Т-клетками (фиг. 24Г). Связывание характеризовали с помощью графика зависимости медианной интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела, от концентрации (в нМ) тестируемых первичных анти-4-1ВВ-связывающих антител huIgG1 P329G LALA. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию значений на исходный уровень путем вычитания MFI «пустого» (без первичного антитела);
на фиг. 25А-25Г - данные о связывании мышиных IgG с экспрессирующими 4-1ВВ мышиными Т-клетками. Представлены данные о связывании с покоящимися и активированными мышиными Т-клетками связывающегося с мышиным 4-1ВВ антитела клона 20G2, представленного в виде форматов мышиный IgG1 DAPG и мышиный IgG1 дикого типа (wt). В качестве отрицательного контроля применяли поступающий в продажу несвязывающийся с 4-1ВВ мышиный IgG1 wt (контроль изотипа) (незакрашенный серый кружок, фирма BioLegend, каталожный №400153). На верхних панелях представлены данные о связывании с покоящимися CD4+-Т-клетками (фиг. 25А) и активированными CD4+-Т-клетками (фиг. 25Б), а на нижних панелях представлены данные о связывании с покоящимися CD8+-Т-клетками (фиг. 25В) и активированными CD8+-Т-клетками (фиг. 25Г). Связывание характеризовали с помощью графика зависимости медианной интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к мышиному IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела, от концентрации в нМ тестируемых первичных анти-4-1ВВ-связывающих антител moIgG. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию значений на исходный уровень путем вычитания MFI «пустого» (без первичного антитела);
на фиг. 26А и 26Б - данные о связывании с экспрессирующими 4-1ВВ Т-клетками обезьян циномолгус. Представлены данные о связывании с активированными Т-клетками обезьян циномолгус четырех клонов связывающегося с человеческим 4-1ВВ антитела huIgG1 P329G LALA (закрашенный ром: клон 25G7, закрашенный квадрат: клон 12В3, закрашенная звездочка: клон 11D5, треугольник вершиной кверху: клон 9В11). В качестве отрицательного контроля применяли несвязывающееся с 4-1ВВ антитело DP47 huIgGl P329G LALA (незакрашенный серый кружок). Представлены данные о связывании с активированными CD4+-Т-клетками (фиг. 26А) и с активированными CD8+-Т-клетками (фиг. 26Б) соответственно. Связывание характеризовали с помощью графика зависимости медианной интенсивности флуоресценции (MFI) меченного с помощью ФИТЦ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического, которое применяли в качестве вторичного идентифицирующего антитела, от концентрации (в нМ) тестируемых первичных анти-4-1ВВ-связывающих антител huIgG1 P329G LALA. MFI измеряли с помощью проточной цитометрии и осуществляли коррекцию значений на исходный уровень путем вычитания MFI «пустого» (без первичного антитела);
на фиг. 27А-27Д - данные о способности связываться с лигандом антител к 4-1ВВ, предлагаемых в изобретении, определенной с помощью поверхностного плазмонного резонанса. Представлены данные о взаимодействии между человеческими антителами к 4-1ВВ IgG-типа 25G7, 11D5, 9В11 и 12В3 и предварительно полученным комплексом лиганд hu4-1BB/hu4-1BB, а также о взаимодействии мышиного анти-4-1ВВ клона 20G2 и предварительно полученного комплекса лиганд mu4-1BB/mu4-1BB;
на фиг. 28А-28 В и 28Г-28Е - результаты экспериментов по конкурентному связыванию. На фиг. 28А-28В - данные о взаимодействие между анти-4-1ВВ IgG клонами 12В3, 11D5 и 25G7 и предварительно полученным комплексом клона 9В11 и hu4-1BB. На фиг. 28Г-28Е - данные о взаимодействии анти-4-1ВВ IgG клонами 12В3, 9В11 и 25G7 и предварительно полученным комплексом клона 11D5 и hu4-1BB. Продемонстрировано, что анти-4-1ВВ клоны 12В3, 11D5 и 9В11 характеризуются отличным от 25G7 пространственным эпитопом, поскольку два антитела могли связываться одновременно с человеческим 4-1ВВ;
на фиг. 29А-29Г - данные о связывании гибридных вариантов 4-1ВВ Fc(kih) с антителами к 4-1ВВ, т.е. связывании вариантов hu4-1BBD1/mu4-1BBD2-Fc(kih) и mu4-1BBD1/hu4-1BBD2-Fc(kih) с антителами к 4-1ВВ. Подчеркнут 4-1ВВ-домен, распознаваемый антителом;
на фиг. 30А-30Г - данные о связывании антител к человеческому 4-1ВВ 11D5, 12В3, 25G7 и 9В11 с доменом 1 человеческого 4-1ВВ. Антитела к человеческому 4-1ВВ 11D5, 12В3 и 9В11 связывались с конструкциями, содержащими домен 1 человеческого 4-1ВВ;
на фиг. 31А-31Г - данные о функциональных свойствах различных клонов антител к человеческому 4-1ВВ in vitro. Предварительно активированные человеческие CD8+-Т-клетки активировали с помощью взятых в различных концентрациях иммобилизированных на поверхности специфических в отношении человеческого 4-1ВВ антител huIgG1 P329G LALA в отсутствии антитела к человеческому CD3 (фиг. 31А и 31В) или в присутствии в субоптимальной концентрации иммобилизированного на поверхности антитела к человеческому CD3 (фиг. 31Б и 31Г). Представлены данные о частоте встречаемости IFNγ+ (А и Б) и TNFα+ (В и Г) CD8+-Т-клеток во всей популяции CD8+-Т-клеток в зависимости от концентрации иммобилизированного на поверхности 4-1ВВ-связывающего huIgG1 P329G LALA (в пМ). В присутствии CD3-стимуляции костимуляция 4-1ВВ могла повышать секрецию IFNγ+ (фиг. 31Б) и TNFα+ (фиг. 31Г) в зависимости от концентрации. В отсутствии CD3-стимуляции активация 4-1ВВ не оказывала воздействия на секрецию IFNγ+ (фиг. 31А) и TNFα+ (фиг. 31В);
на фиг. 32А и 32Б - данные о функциональных свойствах антитела к мышиному 4-1ВВ клона 20G2 in vitro. Мышиные спленоциты инкубировали в присутствии 0,5 мкг/мл IgG хомяка к мышиному IgG1-CD3 (клон 145-2С11) и взятых в различных концентрациях антител к мышиному 4-1ВВ (закрашенный черный ромб: мышиный IgG, незакрашенный черный ромб: мышиный IgG DAPG) или соответствующих контролей изотипов (закрашенный серый кружок: мышиный IgG1, незакрашенный серый кружок: мышиный IgG1 DAPG) в растворе. Концентрация отложена на х-оси в нМ. Только, если антитело к мышиному 4-1ВВ в виде клона 20G2 мышиного IgG1 (черные ромбы) было перекрестно сшито через экспрессирующие FcR клетки, активация гранзима В (фиг. 32А) и эомезодермина (фиг. 32Б) могли возрастать в зависимости от концентрации;
на фиг. 33 - данные о функциональных свойствах антитела к мышиному 4-1ВВ клона 20G2 in vivo. Представлены результаты, полученные при использовании трех мышей на группу. После обработки мышиным IgG1 к мышиному 4-1ВВ клона 20G2 (серые прямоугольники) происходило накапливание общего количества CD8+-Т-клеток в печени (а). Кроме того, обнаружена повышающая регуляция маркера пролиферации Ki67 в понятиях частоты встречаемости (б) и общего количества (в) на CD8+-Т-клетках. Обработка индуцировала также петлю положительной обратной связи путем повышающей регуляции 4-1ВВ (CD137) в понятиях частоты встречаемости (г) и общего количества (д) на CD8+-Т-клетках. Наиболее сильное действие обнаружено в день 1 после третьей инъекции. Если мышей обрабатывали мышиным IgG1 DAPG к мышиному 4-1ВВ клона 20G2 (черные прямоугольники), то перекрестное сшивание антитела предупреждалось и не происходило активации 4-1ВВ. Следовательно, CD8+-Т-клетки в печени оказались сходными по количеству и фенотипу с клетками обработанных ЗФР мышей (белые прямоугольники);
на фиг. 34А - схематическое изображение приведенной в качестве примера биспецифической двухвалентной антигенсвязывающей молекулы, предлагаемой в изобретении, которая содержит два Fab-фрагмента, которые связываются с 4-1ВВ, и два кросс-Fab-фрагмента, которые связываются с FAP (формат 2+2). На фиг. 34Б представлена схема SPR-экспериментов, демонстрирующих одновременное связывание с иммобилизированным человеческим 4-1ВВ и человеческим FAP. На фиг. 34В представлено схематическое изображение приведенной в качестве примера биспецифической одновалентной антигенсвязывающей молекулы, предлагаемой в изобретении, которая содержит один Fab-фрагмент, который связывается с 4-1ВВ, и один кросс-Fab-фрагмент, который связывается с FAP (формат 1+1);
на фиг. 35А-35Г - данные об одновременном связывании биспецифических двухвалентных анти-4-1ВВ/анти-РАР конструкций. Биспецифические конструкции применяли в качестве аналита 1 к иммобилизированному человеческому 4-1ВВ и человеческий FAP применяли в качестве аналита 2. Все биспецифические конструкции могли связываться одновременно с человеческим 4-1ВВ и человеческим FAP;
на фиг. 36А и 36Б - изображение приведенных в качестве примеров биспецифических антигенсвязывающих молекул, которые представляют собой двухвалентную анти-4-1ВВ и одновалентную анти-FAP huIgG1 P329GLALA конструкции, обозначенные также как формат 2+1. Биспецифические антигенсвязывающие молекулы содержат два Fab-фрагмента, которые связываются с 4-1ВВ, и VH- и VL-домены, связывающиеся с FAP;
на фиг. 37А-37В - данные об одновременном связывании биспецифических анти-4-1ВВ- и анти-FAP-конструкций в формате 2+1. На фиг. 37А представлена пиктограмма схемы анализа; на фиг. 37Б и 37В - данные об обнаруженном одновременном связывании биспецифических антигенсвязывающих молекул в формате 2+1 (аналит 1) с иммобилизированными человеческим 4-1ВВ и человеческим FAP;
на фиг. 38А-38Е - данные о связывании с покоящимися CD4+-(верхние панели) и CD8+-Т-клетками (нижние панели) специфических в отношении человеческого 4-1ВВ клона 11D5 (фиг. 38А и В), 12В3 (фиг. 38Б и Г) и 25G7 (фиг. 38Д и Е). Данные о связывании представлены в виде среднего геометрического интенсивности флуоресценции вторичного идентифицирующего конъюгированного с РЕ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического в зависимости от концентрации первичного 4-1ВВ-связывающего антитела. На всех графиках в качестве отрицательного контроля представлены данные для DP47-«ненацеленного» huIgG1 P329G LALA (незакрашенный черный кружочек, пунктирная линия). Ни для одной из конструкций не выявлено специфическое связывание с покоящимися человеческими CD4+-T -клетками (фиг. 38 А, Б и Д) или покоящимися CD8+-Т-клетками (фиг. 38 В, Г и Е);
на фиг. 39А-39Е - данные о связывании с активированными CD4+- (верхние панели) и CD8+-Т-клетками (нижние панели) специфического в отношении человеческого 4-1ВВ клона 11D5 (фиг. 39А и В), 12В3 (фиг. 39Б и Г) и 25G7 (фиг. 39Д и Е). Данные о связывании представлены в виде среднего геометрического интенсивности флуоресценции вторичного идентифицирующего конъюгированного с РЕ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического в зависимости от концентрации первичного 4-1ВВ-связывающего антитела. На всех графиках в качестве отрицательного контроля представлены данные для DP47-«ненацеленного» huIgG1 P329G LALA (незакрашенный черный кружочек, пунктирная линия). Все конструкции связывались главным образом с активированными человеческими CD8+-Т-клетками (фиг. 39 В, Г и Е), для которых характерен более высокий уровень экспрессии 4-1ВВ, чем для активированных человеческих CD4+-T-клеток (фиг. 40 А. Б и Д);
на фиг. 40 - обобщение данных о связывании с активированными человеческими CD8+-Т-клетками различных клонов и различных форматов, полученные по площади под кривой (AUC) для кривых связывания. Различные форматы и применяемые анти-РАР-связывающие клоны указаны в виде пиктограмм под графиком, 4-1ВВ-связывающие клоны обозначены раскраской колонок: контрольная молекула DP47 обозначена белым цветом, 25G7-содержащие молекулы черным цветом, если «ненацеленный» DP47 обозначен черным цветом с белыми полосками, то клон 11D5 обозначен серым цветом, клон 12В3 обозначен бело/черной шахматной клеткой;
на фиг. 41А-41Е - данные о связывании с экспрессирующей человеческий FAP линии клеток меланомы WM-266-4 (фиг. 42 А, Б и Д) и клеток NIH/3T3-huFAP, клон 19 (фиг. 42 В, Г и Д). Данные о связывании представлены в виде среднего геометрического интенсивности флуоресценции вторичного идентифицирующего конъюгированного с РЕ F(ab')2-фрагмента козьего IgG к человеческому IgG, Fcγ-специфического в зависимости от концентрации первичного 4-1ВВ-связывающего антитела. Кривые связывания, полученные с использованием конструкций, содержащих 4-1ВВ-связывающий клон 11D5, представлены на фиг 41А и 41В, клон 12В3 - на фиг. 41Б и 41Г, а клон 25G7 - на фиг. 41Д и 41Е. На всех графиках в качестве отрицательного контроля представлены данные для DP47-«ненацеленного» huIgG1 P329G LALA (незакрашенный черный кружочек, пунктирная линия). Только нацеленные на FAP форматы связывались с экспрессирующими FAP клетками и не связывались с их родительскими антителами к 4-1ВВ huIgG1P329G LALA. Таким образом, продемонстрировано, что вне зависимости от формата все нацеленные на FAP молекулы обладали FAP-специфическим таргетирующим свойством. В зависимости от формата FAP-связывающего клона и таргетирующего фрагмента некоторые молекулы обладали лучшим FAP-таргетирующим свойством по сравнению с другими;
на фиг. 42 - обобщение данных о связывании с клетками NIH/3T3-huFAP. Представлены площади под кривой (AUC) для кривых связывания. Применяемые антитела указаны в виде пиктограмм под графиком, 4-1ВВ-связывающие клоны обозначены цветом колонок: контрольная молекула DP47 обозначена белым цветом, 25G7-содержащие молекулы черным цветом, если ненацеленный DP47 обозначен черным цветом с белыми полосками, то клон 11D5 обозначен серым цветом, а клон 12В3 бело/черной шахматной клеткой. На графике продемонстрировано, что только нацеленные на FAP молекулы, но ни их 4-1ВВ-связывающий родительский huIgG1 P329G LALA, ни нацеленные на DP47 4-1ВВ (25G7)-связывающие молекулы не могли связываться с экспрессирующими FAP клетками;
на фиг. 43А-43И - данные об опосредуемой NF-κВ люциферазной активности в экспрессирующей 4-1ВВ репортерной клеточной линии HeLa-hu4-1BB-NFkB-luc. Люциферазная активность отложена на у-оси в виде единиц испускаемого света (URL) относительно добавленной концентрации агонистических связывающих человеческий 4-1ВВ молекул после инкубации в течение 6 ч. На фиг. 43А, Г и Ж представлены результаты, полученные без добавления экспрессирующих FAP опухолевых клеток. На фиг. 43Б, Д и З представлены результаты, полученные при добавлении к репортерным клеткам экспрессирующей FAP клеточной линии человеческой меланомы WM-266-4, а на фиг. 43В, Е и И - при добавлении трансфектированных человеческим FAP клеток NIH/3T3 в соотношении 5:1 и инкубации в течение 6 ч. Кривые активации, полученные с использованием конструкций, содержащих 4-1ВВ связывающий клон 11D5, представлены на фиг. 43А, 43Б и 43В, клон 12В3-на фиг. 43Г, 43Д и 43Е, а клон 25G7 - на фиг. 43Ж, 43З и 43И. Только нацеленные на FAP форматы индуцировали люциферазную активность в присутствии экспрессирующих FAP опухолевых клеток. Уровни активации зависели от клона, формата и экспрессирующей FAP линии опухолевых клеток;
на фиг. 44А и 44Б - обобщение данных об опосредуемой NF-κB люциферазной активности в экспрессирующей 4-1ВВ репортерной клеточной линии HeLa-hu4-1BB-NFkB-luc в присутствии клеток NIH/3T3-huFAP. Данные представлены в виде площади под кривой (AUC) для кривых активации в присутствии клеток NIH/3T3-huFAP. Применяемые форматы антител и клоны антител к FAP указаны в виде пиктограмм под графиком, различные клоны агонистических антител к 4-1ВВ обозначены цветом колонок: контрольная молекула DP47 обозначена белым цветом, 25G7-содержащие молекулы - черным цветом, клон 11D5 - серым цветом, а клон 12В3-бело/черной шахматной клеткой. На графике продемонстрировано, что только нацеленные на FAP молекулы могли индуцировать сильную активацию, превышающую фоновый уровень. Уровни активации зависели от клона, FAP-таргетирования и формата.
Подробное описание изобретения
Определения
Если не указано иное, то технические и научные понятия, применяемые в настоящем описании, имеют значения, которые обычно используют в области техники, к которой относится изобретение. Для целей интерпретации настоящей заявки следует применять приведенные ниже определения, и если это возможно, то понятия, применяемые в единственном числе, включают также их применение во множественном числе и наоборот.
В контексте настоящего описания понятие «антигенсвязывающая молекула» в наиболее широком смысле относится к молекуле, которая специфически связывается с антигенной детерминантой. Примерами антигенсвязывающих молекул являются антитела, фрагменты антител и антигенсвязывающие белки-каркасы («скаффолд-белки»).
В контексте настоящего описания понятие «фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени» относится к полипептидной молекуле, которая специфически связывается с антигенной детерминантой. В одном из объектов изобретения антигенсвязывающий фрагмент обладает способностью активировать передачу сигнала через антиген его клетки-мишени. В конкретном объекте изобретения антигенсвязывающий фрагмент обладает способностью направлять субстанцию, с которой он связан (например, тримерный лиганд семейства TNF) к сайту-мишени, например, к определенному типу опухолевых клеток или стромы опухоли, несущих антигенную детерминанту. Фрагменты, обладающие способностью специфически связываться с антигеном клетки-мишени, включают антитела и их фрагменты, более подробно представленные в настоящем описания. Кроме того, фрагменты, обладающие способностью специфически связываться с антигеном клетки-мишени, включают антигенсвязывающие белки-каркасы, более подробно представленные в настоящем описании, например, связывающие домены, основой который являются созданные повторы белков или созданные повторы доменов (см., например, WO 2002/020565).
Касательно антитела или его фрагмента понятие «фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени» относится к части молекулы, которая содержит область, специфически связывающуюся и комплементарную части антигена или всему антигену. Фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, может представлять собой, например, один или несколько вариабельных доменов антитела (которые называют также вариабельными областями антитела). В частности, фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, содержит вариабельную область легкой цепи антитела (VL) и вариабельную область тяжелой цепи антитела (VH). В конкретном объекте изобретения «фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени» представляет собой Fab-фрагмент или кросс-Fab-фрагмент.
Понятие «фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов» относится к полипептидной молекуле, которая специфически связывается с представителем семейства костимуляторных TNF-рецепторов. В одном из объектов изобретения антигенсвязывающий фрагмент обладает способностью активировать передачу сигналов через представителя семейства костимуляторных TNF-рецепторов.
Фрагменты, обладающие способностью специфически связываться с антигеном клетки-мишени, включают антитела и их фрагменты, дополнительно указанные в настоящем описании. Кроме того, фрагменты, обладающие способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, включают каркасные антигенсвязывающие белки, дополнительно указанные в настоящем описании, например, связывающие домены, сконструированные основанные на повторах белки или сконструированные основанные на повторах домены (см., например, WO 2002/020565). В частности, фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, содержит вариабельную область легкой цепи антитела (VL) и вариабельную область тяжелой цепи антитела (VH). В конкретном объекте изобретения «фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов», представляет собой Fab-фрагмент или кросс-Fab-фрагмент.
В контексте настоящего описания понятие «антитело» используется в его наиболее широком смысле, и оно относится к различным структурам антител, включая (но не ограничиваясь только ими) моноклональные антитела, поликлональные антитела, моноспецифические и мультиспецифические антитела (например, биспецифические антитела) и фрагменты антител, при условии, что они обладают требуемой антигенсвязывающей активностью.
Понятие «моноклональное антитело» в контексте настоящего описания относится к антителу, полученному из популяции практически гомогенных антител, т.е. индивидуальные антитела, входящие в популяцию, идентичны и/или связываются с одним и тем же эпитопом, за исключением возможных вариантов антител, например, содержащих мутации, встречающиеся в естественных условиях или возникающие в процессе производства препарата моноклонального антитела, указанные варианты, как правило, присутствуют в минорных количествах. В отличие от препаратов поликлональных антител, которые, как правило, включают различные антитела к различным детерминантам (эпитопам), каждое моноклональное антитело из препарата моноклонального антитела направлено против одной детерминанты на антигене.
В контексте настоящего описания понятие «моноспецифическое» антитело означает антитело, которое имеет один или несколько сайтов связывания, каждый из которых связывается с одним и тем же эпитопом одного и того же антигена. Понятие «биспецифическая» означает, что антигенсвязывающая молекула обладает способностью специфически связываться по меньшей мере с двумя различными антигенными детерминантами. Как правило, биспецифическая антигенсвязывающая молекула содержит два антигенсвязывающих сайта, каждый из которых является специфическим в отношении различных антигенных детерминант. В некоторых вариантах осуществления изобретения биспецифическая антигенсвязывающая молекула обладает способностью одновременно связываться с двумя антигенными детерминантами, прежде всего с двумя антигенными детерминантами, экспрессируемыми на двух различных клетках.
В контексте настоящего описания понятие «валентность» означает наличие определенного количества сайтов связывания, специфических в отношении одной конкретной антигенной детерминанты в антигенсвязывающей молекуле, которая является специфической в отношении одной конкретной антигенной детерминанты. Таким образом, понятия «двухвалентная», «четырехвалентная» и «шестивалентная» означает присутствие двух сайтов связывания, четырех сайтов связывания и шести сайтов связывания, специфических в отношении определенной антигенной детерминанты соответственно в антигенсвязывающей молекуле. В конкретных объектах изобретения биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, могут являться одновалентными в отношении определенной антигенной детерминанты, это означает, что они имеют только один сайт связывания с указанной антигенной детерминантой, или они могут являться двухвалентными в отношении определенной антигенной детерминанты, это означает, что они имеют два сайта связывания с указанной антигенной детерминантой.
Понятия «полноразмерное антитело», «интактное антитело» и «цельное антитело» в контексте настоящего описания используют взаимозаменяемо для обозначения антитела, имеющего структуру, практически сходную с нативной структурой антитела. Понятие «нативные антитела» относится к встречающимся в естественных условиях молекулам иммуноглобулинов с различными структурами. Например, нативные антитела IgG-класса представляют собой гетеротетрамерные гликопротеины с молекулярной массой примерно 150000 Да, состоящие из двух идентичных легких цепей и двух идентичных тяжелых цепей, связанных дисульфидными мостиками. Начиная с N- конца к С-концу, каждая тяжелая цепь имеет вариабельную область (VH), которую называют также вариабельным тяжелым доменом или вариабельным доменом тяжелой цепи, за которой следует три константных домена (CH1, СН2 и СН3), которые называют также константной областью тяжелой цепи. Аналогично этому, начиная с N-конца к С-концу, каждая легкая цепь имеет вариабельную область (VL), которую называют также вариабельным легким доменом или вариабельным доменом легкой цепи, за которой следует константный домен легкой цепи (CL), который называют также константной областью легкой цепи. Тяжелая цепь антитела может относиться к одному из пяти типов, которые обозначают как α (IgA), δ (IgD), ε (IgE), γ (IgG) или μ (IgM), некоторые из которых можно дополнительно подразделять на подтипы, например, γ1 (IgG1), γ2 (IgG2), γ3 (IgG3), γ4 (IgG4), α1 (IgA1) и α2 (IgA2). Легкая цепь антитела может принадлежать к одному из двух типов, называемых каппа (κ) и лямбда (λ), в зависимости от аминокислотной последовательности ее константного домена.
Понятие «фрагмент антитела» относится к молекуле, отличной от интактного антитела, которая содержит часть интактного антитела, обладающую способностью связываться с антигеном, с которым связывается интактное антитело. Примерами фрагментов антител являются (но не ограничиваясь только ими) Fv, Fab, Fab', Fab'-SH, F(ab')2, димерные антитела (диабоди), тримерные антитела (триабоди), тетрамерные антитела (тетрабоди), кросс-Fab-фрагменты; линейные антитела: одноцепочечные молекулы антител (например, scFv); и однодоменные антитела. Обзор некоторых фрагментов антител см., например, у Hudson и др., Nat Med 9, 2003, сс.129-134. Обзор scFv-фрагментов см., например, у  в: The Pharmacology of Monoclonal Antibodies, т. 113, под ред. Rosenburg и Moore, изд-во Springer-Verlag, New York, 1994, cc. 269-315; см. также WO 93/16185; и US №№5571894 и 5587458. Обсуждение Fab- и F(ab')2-фрагментов, содержащих остатки эпитопа, связывающегося с рецептором спасения, и обладающих удлиненным временем полужизни in vivo, см. в US №5869046. Димерные антитела представляют собой фрагменты антител с двумя антигенсвязывающими сайтами, которые могут быть двухвалентными или биспецифическими (см., например, ЕР 404097; WO 1993/01161; Hudson и др., Nat Med 9, 2003, сс. 129-134; и Hollinger и др., Proc Natl Acad Sci USA 90, 1993, сс. 6444-6448. Тримерные и тетрамерные антитела описаны также у Hudson и др., Nat Med 9, 2003, сс. 129-134. Однодоменные антитела представляют собой фрагменты антител, содержащие весь вариабельный домен тяжелой цепи или его часть или весь вариабельный домен легкой цепи антитела или его часть. В некоторых вариантах осуществления изобретения однодоменное антитело представляет собой человеческое однодоменное антитело (фирма Domantis, Inc., Валтам, шт. Массачусетс; см., например, US №6248516 В1). Фрагменты антител можно создавать с помощью различных методик, включая (но не ограничиваясь только ими) протеолитическое расщепление интактного антитела, а также получать с использованием рекомбинантных клеток-хозяев (например, Е. coli или фага), как указано в настоящем описании.
в: The Pharmacology of Monoclonal Antibodies, т. 113, под ред. Rosenburg и Moore, изд-во Springer-Verlag, New York, 1994, cc. 269-315; см. также WO 93/16185; и US №№5571894 и 5587458. Обсуждение Fab- и F(ab')2-фрагментов, содержащих остатки эпитопа, связывающегося с рецептором спасения, и обладающих удлиненным временем полужизни in vivo, см. в US №5869046. Димерные антитела представляют собой фрагменты антител с двумя антигенсвязывающими сайтами, которые могут быть двухвалентными или биспецифическими (см., например, ЕР 404097; WO 1993/01161; Hudson и др., Nat Med 9, 2003, сс. 129-134; и Hollinger и др., Proc Natl Acad Sci USA 90, 1993, сс. 6444-6448. Тримерные и тетрамерные антитела описаны также у Hudson и др., Nat Med 9, 2003, сс. 129-134. Однодоменные антитела представляют собой фрагменты антител, содержащие весь вариабельный домен тяжелой цепи или его часть или весь вариабельный домен легкой цепи антитела или его часть. В некоторых вариантах осуществления изобретения однодоменное антитело представляет собой человеческое однодоменное антитело (фирма Domantis, Inc., Валтам, шт. Массачусетс; см., например, US №6248516 В1). Фрагменты антител можно создавать с помощью различных методик, включая (но не ограничиваясь только ими) протеолитическое расщепление интактного антитела, а также получать с использованием рекомбинантных клеток-хозяев (например, Е. coli или фага), как указано в настоящем описании.
Расщепление интактных антител папаином приводит к образованию двух идентичных антигенсвязывающих фрагментов, которые называют «Fab»-фрагментами, каждый из которых содержит вариабельные домены тяжелой и легкой цепей, а также константный домен легкой цепи и первый константный домен (СН1) тяжелой цепи. В контексте настоящего описания понятие «Fab-фрагмент» относится к фрагменту антитела, содержащему фрагмент легкой цепи, который содержит VL-домен и константный домен легкой цепи (CL), и VH-домен и первый константный домен (СН1) тяжелой цепи. Fab'-фрагменты отличаются от Fab-фрагментов добавлением нескольких остатков на карбоксильный конец СН1-домена тяжелой цепи, включая один или несколько остатков цистеина из шарнирной области антитела. Fab'-SH обозначает Fab'-фрагменты, в который остаток(тки) цистеина константных доменов несет(ут) свободную тиольную группу. Обработка пепсином позволяет получать F(ab')2-фрагменты, которые имеют два антигенсвязывающих сайта (два Fab-фрагмента) и часть Fc-области. Согласно настоящему изобретению понятие «Fab-фрагмент» включает также «кросс-Fab-фрагменты» или «кроссовер-Fab-фрагменты», описанные ниже.
Понятие «кросс-Fab-фрагмент» или «xFab-фрагмент» или «кроссовер-Fab-фрагмент» относится к Fab-фрагменту, в котором либо вариабельные области, либо константные области тяжелой и легкой цепи обменены. Возможны две различные композиции цепи кроссовер-молекулы Fab, и они могут входить в биспецифические антитела, предлагаемые в изобретении: с одной стороны, имеет место обмен вариабельными областями тяжелой и легкой цепи Fab, т.е. кроссовер-молекула Fab содержит пептидную цепь, состоящую из вариабельной области легкой цепи (VL) и константной области тяжелой цепи (СН1), и пептидную цепь, состоящую из вариабельной области тяжелой цепи (VH) и константной области легкой цепи (CL). Указанную кроссовер-молекулу Fab обозначают также как CrossFab(VLVH). С другой стороны, когда друг на друга обменены константные области тяжелой и легкой цепи Fab, то кроссовер-молекула Fab содержит пептидную цепь, состоящую из вариабельной области тяжелой цепи (VH) и константной области легкой цепи (CL), и пептидную цепь, состоящую из вариабельной области легкой цепи (VL) и константной области тяжелой цепи (СН1). Указанную кроссовер-молекулу Fab обозначают также как
CrossFab(CLCH1).
«Одноцепочечный Fab-фрагмент» или «scFab» представляет собой полипептид, состоящий из вариабельного домена тяжелой цепи (VH) антитела, константного домена 1 (СН1) антитела, вариабельного домена легкой цепи (VL) антитела, константного домена легкой цепи (CL) антитела и линкера, в котором указанные домены антитела и указанный линкер имеют один из следующих порядков расположения в направлении от N-конца к С-концу: a) VH-CH1-линкер-VL-CL, б) VL-CL-линкер-VH-СН1, в) VH-CL-линкер-VL-СН1 или г) VL-СН1-линкер-VH-CL; и в котором указанный линкер представляет собой полипептид, состоящий по меньшей мере из 30 аминокислот, предпочтительно примерно от 32 до 50 аминокислот. Указанные одноцепочечные Fab-фрагменты стабилизируют с помощью встречающейся в естественных условиях дисульфидной связи между CL-доменом и СН1-доменом. Кроме того, указанные одноцепочечные молекулы Fab можно дополнительно стабилизировать посредством создания межцепочечных дисульфидных связей путем встраивания остатков цистеина (например, в положение 44 в вариабельной тяжелой цепи и в положение 100 в вариабельной легкой цепи согласно нумерации Кэбота).
«Одноцепочечный кроссовер-Fab-фрагмент» или «х-scFab» представляет собой полипептид, состоящий из вариабельного домена тяжелой цепи (VH) антитела, константного домена 1 (СН1) антитела, вариабельного домена легкой цепи (VL) антитела, константного домена легкой цепи (CL) антитела и линкера, в котором указанные домены антитела и указанный линкер имеют один из следующих порядков расположения в направлении от N-конца к С-концу: a) VH-CL-линкер-VL-CH1 и б) VL-CH1-линкер-VH-CL; где VH и VL вместе образуют антигенсвязывающий сайт, который специфически связывается с антигеном, и в котором указанный линкер представляет собой полипептид, состоящий по меньшей мере из 30 аминокислот. Кроме того, указанные молекулы x-scFab можно дополнительно стабилизировать посредством создания межцепочечных дисульфидных связей путем встраивания остатков цистеина (например, в положение 44 в вариабельной тяжелой цепи и в положение 100 в вариабельной легкой цепи согласно нумерации Кэбота).
«Одноцепочечный вариабельный фрагмент (scFv)» представляет собой слитый белок вариабельных областей тяжелой (VH) и легкой (VL) цепей антитела, соединенных коротким линкерным пептидом, который содержит от 10 до примерно 25 аминокислот. Линкер, как правило, представляет собой богатый глицином линкер для придания гибкости, а также богатый серином или треонином для обеспечения растворимости, и он может соединять либо N-конец VH с С-концом VL, либо наоборот. Указанный белок сохраняет специфичность исходного антитела, несмотря на удаление константных областей и интродукцию линкера. Антитела в виде scFv описаны, например, у Houston J.S., Methods in Enzymol. 203, 1991, сс. 46-96). Кроме того, фрагменты антител содержат одноцепочечные полипептиды, которые имеют характеристики VH-домена, а именно, обладают способностью к сборке с VL-доменом, или характеристики VL, а именно, обладают способностью к сборке с VH-доменом с образованием антигенсвязывающего сайта, и тем самым обладают антигенсвязывающим свойством полноразмерных антител.
«Антигенсвязывающие белки-каркасы (скаффолд-белки)») известны в данной области, например, фибронектин и созданные содержащие анкириновые повторы белки (DARPin), применяли в качестве альтернативных каркасов для антигенсвязывающих доменов (см. Gebauer и Skerra, Engineered protein scaffolds as next-generation antibody Therapeutics. Curr Opin Chem Biol 13, 2009, cc. 245-255 и Stumpp и др., Darpins: A new generation of protein Therapeutics. Drug Discov Today 13, 2008, cc. 695-701). В одном из объектов изобретения антигенсвязывающий белок-каркас выбран из группы, включающей CTLA-4 (эвибоди (Evibody)), липокаины (антикалин (Anticalin)), полученную из белка А молекулу, такую как Z-домен белка A (Affibody, SpA), А-домен (авимер/максибоди (Avimer/Maxibody)), сывороточный трансферрин (транс-боди), сконструированный белок, содержащий анкириновые повторы (DARPin), вариабельный домен легкой цепи или тяжелой цепи антитела (однодоменное антитело, sdAb), вариабельный домен тяжелой цепи антитела (нанободи, aVH), VNAR-фрагменты, фибронектин (аднектин (AdNectin)), лектиновый домен С-типа (тетранектин), вариабельный домен нового антигенного рецептора бета-лактамазы (VNAR-фрагменты), человеческий гамма-кристаллин или убиквитин (молекулы-аффилины (Affilin), домен Кунитц-типа человеческих ингибиторов протеаз, микрободи, такие как белки из семейства knottin (кноттины, ингибиторы цистеинового узла), пептидные аптамеры и фибронектин (аднектин). CTLA-4 (ассоциированный с цитотоксическими Т-лимфоцитами антиген 4) относится к CD28-семейству рецепторов, которые главным образом экспрессируются на CD4+-Т-клетках. Его внеклеточный домен имеет укладку, напоминающую укладку вариабельного домена Ig. Петли, соответствующие CDR антител, можно заменять на гетерологичную последовательность для придания различных связывающих свойств. Молекулы CTLA-4, сконструированные таким образом, что они обладают различными связывающими специфичностями, известны также как эвибоди (см., например, US №7166697 В1). Эвибоди имеют примерно такой же размер, что и выделенная вариабельная область антитела (например, доменного антитела. Дополнительные сведения см. в Journal of Immunological Methods 248 (1-2), 2001, cc. 31-45. Липокаины относятся к семейству внеклеточных белков, которые транспортируют малые гидрофобные молекулы, такие как стероиды, билины, ретиноиды и липиды. Они имеют жесткую вторичную структуру в виде бета-складки с рядом петель на открытом конце конической структуры, их можно создавать для связывания с различными антигенами-мишенями. Антикалины состоят из 160-180 аминокислот и их получают из липокаинов. Дополнительные сведения см. в Biochim Biophys Acta 1482, 2000, сс. 337-350, в US №7250297 В1 и US №2007/0224633. Аффибоди представляет собой каркас, полученный из белка A Staphylococcus aureus, который можно создавать для связывания с антигеном. Домен состоит из трехспирального пучка, содержащего примерно 58 аминокислот. Библиотеки были созданы путем рандомизации поверхностных остатков. Дополнительные сведения см. в Protein Eng. Des. SeI. 17, 2004, cc. 455-462 и ЕР 1641818 A1. Авимеры представляют собой мультидоменные белки, полученные из А-домена семейства белков-каркасов. Нативные домены, состоящие примерно из 35 аминокислот, адоптируют определенную связанную дисульфидами структуру. Разнообразие создают путем «перестановки» встречающихся в естественных условиях вариаций, характерных для семейства А-доменов. Дополнительные сведения см. в Nature Biotechnology 23(12), 2005, сс.1556-1561 и Expert Opinion on Investigational Drugs 16(6), июнь 2007, cc. 909-917. Трансферрин представляет собой мономерный сывороточный транспортный гликопротеин. Трансферрины можно создавать для связывания с различными антигенами-мишенями путем встраивания пептидных последовательностей в приемлемую поверхностную петлю. Примеры сконструированных трансферинновых каркасов включают транс-боди. Дополнительные сведения см. в J. Biol. Chem 274, 1999, сс. 24066-24073. Сконструированные белки, содержащие анкириновые повторы (DARPin), получают из анкирина, который относится к семейству белков, опосредующих присоединение интегральных мембранных белков к цитоскелету. Один анкириновый повтор представляет собой состоящий из 33 остатков мотив, включающий две альфа-спирали и бета-изгиб. Их можно создавать для связывания с различными антигенами-мишенями путем рандомизации остатков в первой альфа-спирали и бета-изгибе каждого повтора. Их связывающую поверхность раздела можно увеличивать путем увеличения количества модулей (метод созревания аффинности). Дополнительные сведения см. в J. MoI. Biol. 332, 2003, сс. 489-503, PNAS 100(4), 2003, сс. 1700-1705 и J. MoI. Biol. 369, 2007, сс. 1015-1028, а также в US №2004/0132028 А1. Однодоменное антитело представляет собой фрагмент антитела, состоящий из единичного мономерного вариабельного домена антитела. Впервые единичный домен был получен из вариабельного домена тяжелой цепи антитела верблюдов (нанабоди или VhH-фрагменты). Кроме того, понятие однодоменное антитело включает автономный вариабельный домен человеческой тяжелой цепи (aVH) или VNAR-фрагменты, полученные из акул. Фибронектин представляет собой каркас, который можно создавать для связывания антигена. Аднектины состоят из каркаса из встречающейся в естественных условиях аминокислотной последовательности 10-го домена из 15 повторяющихся единиц человеческого фибронектина типа III (FN3). Можно создавать три петли на одном конце «бета-сэндвича», что позволяет аднектину специфически распознавать представляющую интерес терапевтическую мишень. Дополнительные сведения см. в Protein Eng. Des. SeI. 18, 2005, сс. 435-444, US №2008/0139791, WO 2005/056764 и US №6818418 B1. Пептидные аптамеры представляют собой комбинаторные распознающие молекулы, которые состоят из константного белка-каркаса, как правило, тиоредоксина (TrxA), который содержит ограниченную вариабельную пептидную петлю, встроенную в активный сайт. Дополнительные сведения см. в Expert Opin. Biol. THER. 5, 2005, сс. 783-797. Микрободи получают из встречающихся в естественных условиях микробелков, состоящих из 25-50 аминокислот, которые содержат 3-4 цистеиновых мостика - примеры микробелков включают KalataBI и конотоксин и кноттины. Микробелки имеют петлю, которую можно подвергать инженерии таким образом, чтобы она включала вплоть до 25 аминокислот, без воздействия на общую укладку микробелка. Дополнительные сведения о создании кноттин-доменов см. в WO 2008/098796.
Понятие «антигенсвязывающая молекула, которая связывается с таким же эпитопом», что и референс-молекула, относится к антигенсвязывающей молекуле, которая блокирует связывание референс-молекулы с ее антигеном на 50% или более при оценке в условиях конкуренции, и, наоборот, референс-молекула блокирует связывание антигенсвязывающей молекулы с ее антигеном на 50% или более при оценке в условиях конкуренции.
Понятие «антигенсвязывающий домен» или «антигенсвязывающий сайт (центр)» относится к части антигенсвязывающей молекулы, которая содержит область, специфически связывающуюся и являющуюся комплементарной части антигена или полному антигену. Если антиген является крупным, то антигенсвязывающая молекула может связываться только с конкретной частью антигена, которую называют эпитопом. Антигенсвязывающий домен может представлять собой, например, один или несколько вариабельных доменов антитела (которые называют также вариабельными областями).
Предпочтительно антигенсвязывающий домен содержит вариабельную область легкой цепи (VL) антитела и вариабельную область тяжелой цепи (VH) антитела.
В контексте настоящего описания понятие «антигенная детерминанта» является синонимом понятий «антиген» и «эпитоп» и относится к сайту (например, участку, состоящему из смежных аминокислот, или конформационной конфигурации, состоящей из различных областей несмежных аминокислот) на полипептидной макромолекуле, с которой связывается антигенсвязывающий фрагмент с образованием комплекса антигенсвязывающий фрагмент-антиген. Пригодные антигенные детерминанты могут присутствовать, например, на поверхности опухолевых клеток, на поверхности инфицированных вирусом клеток, на поверхности других больных клеток, на поверхности иммунных клеток, клеток, находящихся в свободном состоянии в сыворотке крови и/или во внеклеточном матриксе (ЕСМ). Белки, которые можно применять в качестве антигенов в настоящем изобретении, если не указано иное, могут представлять собой любые нативные формы белков из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди) и грызуны (например, мыши и крысы). В конкретном варианте осуществления изобретения антиген представляет собой человеческий белок. В контексте настоящего описания при ссылке на конкретный белок подразумевается «полноразмерный», непроцессированный белок, а также любая форма белка, полученная в результате процессинга в клетке. Под понятие подпадают также встречающиеся в естественных условиях варианты белка, например, сплайсинговые варианты или аллельные варианты.
Понятие «специфически связывается» означает, что связывание является избирательным в отношении антигена и его можно отличать от нежелательных или неспецифических взаимодействий. Способность антигенсвязывающей молекулы связываться со специфическим антигеном можно определять с помощью твердофазного иммуноферментного анализа (ELISA) или других методик, известных специалисту в данной области, например, с помощью методики на основе поверхностного плазмонного резонанса (осуществляя анализ с помощью устройства BIAcore) (Liljeblad и др., Glyco J 17, 2000, сс. 323-329), и традиционных анализов связывания (Heeley, Endocr Res 28, 2002, сс. 217-229). В одном из вариантов осуществления изобретения уровень связывания антигенсвязывающей молекулы с неродственным белком составляет менее чем примерно 10% от уровня связывания антигенсвязывающей молекулы с антигеном, при измерении, например, с помощью SPR. В некоторых вариантах осуществления изобретения молекула, которая связывается с антигеном, характеризуется величиной константы диссоциации (KD), составляющей ≤1 мкМ, ≤100 нМ, ≤10 нМ, ≤1 нМ, ≤0,1 нМ, ≤0,01 нМ или ≤0,001 нМ (например, 10-8 М или менее, например, от 10-8 М до 10-13 М, например, от 10-9 М до 10-13 М).
Понятие «аффинность» или «аффинность связывания» относится к суммарной силе всех нековалентных взаимодействий между индивидуальным сайтом связывания молекулы (например, антитела) и ее партнера по связыванию (например, антигена). Если не указано иное, то в контексте настоящего описания понятие «аффинность связывания» относится к присущей компонентам связывающейся пары (например, антителу и антигену) аффинности связывания, отражающей взаимодействие по типу 1:1. Аффинность молекулы X к ее партнеру Y можно, как правило, характеризовать с помощью константы диссоциации (KD), которая представляет собой отношение констант скорости реакции диссоциации и ассоциации (koff и kon соответственно). Таким образом, эквивалентные аффинности могут соответствовать различным константам скорости, если соотношение констант скорости остается таким же. Аффинность можно оценивать общепринятыми методами, известными в данной области, включая представленные в настоящем описании методы. Конкретным методом измерения аффинности является метод поверхностного плазмонного резонанса (SPR).
Антитело «с созревшей аффинностью» относится к антителу с одним или несколькими изменения в одном или нескольких гиперварибельных участках (HVR) по сравнению с родительским антителом, которое не несет указанных изменений, указанные изменения приводят к улучшению аффинности антитела к антигену.
В контексте настоящего описания понятие «антиген клетки-мишени» относится к антигенной детерминанте, присутствующей на поверхности клетки-мишени, например, клетке в опухоли, такой как раковая клетка или клетка стромы опухоли. В некоторых вариантах осуществления изобретения антиген клетки-мишени представляет собой антиген на поверхности опухолевой клетки.
В одном из вариантов осуществления изобретения антиген клетки-мишени выбирают из группы, включающей фибробласт-активирующий белок (FAP), карциноэмбриональный антиген (СЕА), ассоциированный с меланомой хондроитинсульфат-протеогликан (MCSP), рецептор эпидермального фактора роста (EGFR), CD19, CD20 и CD33. В частности, антиген клетки-мишени представляет собой фибробласт-активирующий белок (FAP).
Понятие «фибробласт-активирующий белок (FAP)», известный также как пролилэндопептидаза FAP или сепраза (КФ 3.4.21), если не указано иное, относится к любой нативной форме FAP из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус (яванский макак-крабоед) и грызуны (например, мыши и крысы). Понятие относится к полноразмерному непроцессированному FAP, а также к любой форме FAP, полученной в результате процессинга в клетке. Под понятие подпадают также встречающиеся в естественных условиях варианты FAP, например, сплайсинговые варианты или аллельные варианты. В одном из вариантов осуществления изобретения антигенсвязывающая молекула, предлагаемая в изобретении, обладает способностью специфически связываться с FAP человека, мышей и/или обезьян циномолгус. Аминокислотная последовательность человеческого FAP представлена в UniProt (www.uniprot.org), код доступа № Q12884 (версия 149, SEQ ID NO: 84) или в NCBI (www.ncbi.nlm.nih.gov/) RefSeq NP_004451.2. Внеклеточный домен (ECD) человеческого FAP простирается от аминокислотного положения 26 до 760. Аминокислотная и нуклеотидная последовательности меченного His человеческого FAP-ECD представлены в SEQ ID NO: 85 и 86 соответственно. Аминокислотная последовательность мышиного FAP представлена в UniProt, код доступа №Р97321 (версия 126, SEQ ID NO: 87) или NCBI RefSeq NP 032012.1. Внеклеточный домен (ECD) мышиного FAP простирается от аминокислотного положения 26 до 761. В SEQ ID NO: 88 и 89 представлены аминокислотная и нуклеотидная последовательности меченного His мышиного FAP-ECD соответственно. В SEQ ID NO: 90 и 91 представлены аминокислотная и нуклеотидная последовательности соответственно меченного FAP-ECD обезьян циномолгус. Предпочтительно анти-РАР-связывающая молекула, предлагаемая в изобретении, связывается с внеклеточным доменом FAP.
Понятие «карциноэмбриональный антиген (СЕА)», известный также как родственный молекуле клеточной адгезии 5 карциноэмбриональный антиген (СЕАСАМ5), если не указано иное, относится к любой нативной форме СЕА из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого СЕА представлена в UniProt, код доступа № Р06731 (версия 151, SEQ ID NO: 92). Понятие «ассоциированный с меланомой хондроитинсульфат-протеогликан (MCSP), известный также как хонроитинсульфат-протеогликан 4 (CSPG4), если не указано иное, относится к любой нативной форме MCSP из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого MCSP представлена в UniProt, код доступа № Q6UVK1 (версия 103, SEQ ID NO: 93). Понятие «рецептор эпидермального фактора роста (EGFR)» известный также как протоонкоген с-ErbB-1 или рецепторная тирозинпротеинкиназа erbB-1, относится, если не указано иное, к любой нативной форме EGFR из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого EGFR представлена в UniProt, код доступа № Р00533 (версия 211, SEQ ID NO: 94). Понятие «CD19» относится к антигену CD19 лимфоцитов, известному также как поверхностный антиген В4 В-лимфоцитов или антиген Т-клеточной поверхности Leu-12, и относится, если не указано иное, к любой нативной форме CD19 из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого CD19 представлена в UniProt, код доступа № P15391 (версия 160, SEQ ID NO: 95). Понятие «CD20» относится к антигену В-лимфоцитов CD20, известному также как трансмембранный четырехдоменный представитель 1 подсемейства A (MS4A1), поверхностный антиген В1 В-лимфоцитов или антиген поверхности лейкоцитов Leu-16, и относится, если не указано иное, к любой нативной форме CD20 из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого CD20 представлена в UniProt, код доступа № Р11836 (версия 149, SEQ ID NO: 96). Понятие «CD33» относится к поверхностному антигену миелоидных клеток CD33, известному также как SIGLEC3 или gp67, и относится, если не указано иное, к любой нативной форме CD33 из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы). Аминокислотная последовательность человеческого CD33 представлена в UniProt, код доступа №. Р20138 (версия 157, SEQ ID NO: 97).
Понятие «вариабельная область» или «вариабельный домен» относится к домену тяжелой или легкой цепи антитела, который участвует в связывании антигенсвязывающей молекулы с антигеном. Вариабельные домены тяжелой цепи и легкой цепи (VH и VL соответственно) нативного антитела, как правило, имеют сходные структуры, при этом каждый домен содержит четыре консервативных каркасных участка (FR) и три гипервариабельных участка (HVR) (см., например, Kindt и др., Kuby Immunology, 6-ое изд., изд-во W.H. Freeman and Co., 2007, с. 91). Одного VH- или VL-домена может быть достаточно для обеспечения специфичности связывания антигена.
Понятие «гипервариабельный участок» или «HVR» в контексте настоящего описания относится к каждому из участков вариабельного домена антитела, последовательности которых являются гипервариабельными, и/или которые образуют структуры в виде петель («гипервариабельные петли»). Как правило, нативные четырехцепочечные антитела содержат шесть HVR; три в VH (H1, Н2, Н3) и три в VL (L1, L2, L3). HVR, как правило, содержат аминокислотные остатки из гипервариабельных петель и/или из «определяющих комплементарность участков» (CDR), последние отличаются наиболее выраженной вариабельностью последовательности и/или участвуют в распознавании антигенов. Приведенные в качестве примера гипервариабельные петли включают аминокислотные остатки 26-32 (L1), 50-52 (L2), 91-96 (L3), 26-32 (H1), 53-55 (Н2), и 96-101 (Н3) (Chothia и Lesk, J. Mol. Biol. 196, 1987, сс. 901-917). Приведенные в качестве примера CDR (CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 и CDR-H3) включают аминокислотные остатки 24-34 (L1), 50-56 (L2), 89-97 (L3), 31-35 В (H1), 50-65 (Н2) и 95-102 (Н3) (Kabat Е.А. и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991). Гипервариабельные участки (HVR) часто обозначают как «определяющие комплементарность участки» (CDR), и эти понятия в контексте настоящего описания используют взаимозаменяемо, и они относятся к участкам вариабельной области, которые образуют антигенсвязывающие области. Эта конкретная область описана у Kabat и др., U.S. Dept. of Health and Human Services, ((Sequences of Proteins of Immunological Interest)), 1983 и у Chothia и др., J. Mol. Biol. 196, 1987, cc. 901-917, причем эти определения относятся к перекрывающимся аминокислотным остаткам или поднаборам аминокислотных остатков при их сравнении друг с другом. Однако в контексте настоящего описания подразумевается возможность применения любого определения CDR антитела или его вариантов. Соответствующие аминокислотные остатки, из которых состоят CDR, как они определены в каждой из процитированных выше ссылок, представлены в сравнении ниже в таблице А. Точные номера остатков, которые образуют конкретный CDR, должны варьироваться в зависимости от последовательности и размера CDR. Специалисты в данной области на основе данных об аминокислотной последовательности вариабельной области антитела легко могут определять, какие остатки входят в конкретный CDR.
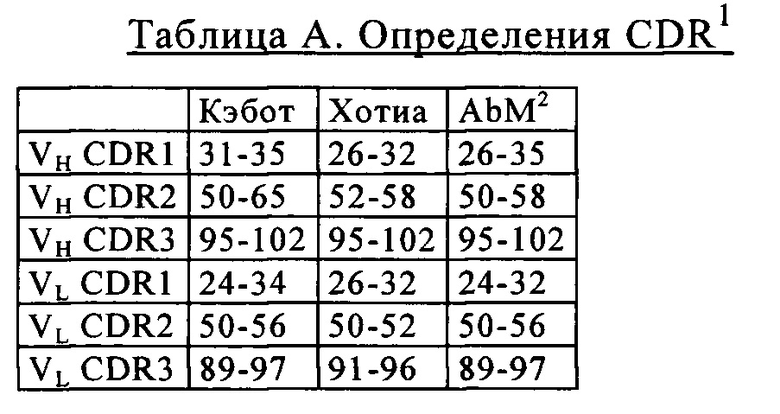
1 Нумерация всех входящих в CDR остатков в таблице А дана в соответствии с номенклатурой, предложенной Кэботом с соавторами (см. ниже).
2 Обозначение «AbM» с прописной буквой «Ь», использованное в таблице А, относится к CDR, как они определены программой для моделирования антител «AbM» компании Oxford Molecular Group.
Кэбот с соавторами предложили также систему нумерации (номенклатуру) последовательностей вариабельных областей, которую можно применять для любого антитела. Обычный специалист в данной области может однозначно применять эту систему «нумерации по Кэботу» к любой последовательности вариабельной области, не имея никаких экспериментальных данных, кроме сведений о самой последовательности. В контексте настоящего описания понятие «нумерация по Кэботу» относится к системе нумерации, описанной у Kabat и др., «Sequence of Proteins of Immunological Interest)), изд-во U.S. Dept. of Health and Human Services, 1983. Если не указано иное, то ссылки на нумерацию положений конкретных аминокислотных остатков в вариабельной области антитела даны в соответствии с системой нумерации по Кэботу.
За исключением CDR1 в VH, CDR, как правило, содержат аминокислотные остатки, которые образуют гипервариабельные петли. CDR содержат также «определяющие специфичность остатки» или «SDR», которые представляют собой остатки, контактирующие с антигеном. SDR, содержащиеся в областях CDR, обозначают как укороченные -CDR или a-CDR. Приведенные в качестве примера a-CDR (a-CDR-L1, a-CDR-L2, a-CDR-L3, a-CDR-H1, a-CDR-H2 и a-CDR-H3) содержат аминокислотные остатки 31-34 (L1), 50-55 (L2), 89-96 (L3), 31-35B (H1), 50-58 (H2) и 95-102 (Н3) (см. Almagro и Fransson, Front. Biosci. 13, 2008, cc. 1619-1633). Если не указано иное, остатки HVR и другие остатки в вариабельном домене (например, остатки FR) в настоящем описании нумеруют согласно Kabat и др., выше.
«Каркасные участки» или «FR» представляют собой участки вариабельных доменов, отличные от остатков гипервариабельных участков (HVR). FR вариабельного домена, как правило, представлены четырьмя FR-доменами: FR1, FR2, FR3 и FR4. Таким образом, последовательности HVR и FR, как правило, расположены в VH (или VL) в следующем порядке: FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4.
Понятие «химерное» антитело относится к антителу, в котором часть тяжелой и/или легкой цепи происходит из конкретного источника или конкретных видов, а остальная часть тяжелой и/или легкой цепи происходит из другого источника или других видов.
Понятие «класс» антитела относится к типу константного домена или константной области, входящего/входящей в тяжелую цепь. Существует пять основных классов антител: IgA, IgD, IgE, IgG и IgM, а некоторые из них можно дополнительно подразделять на подклассы (изотипы), например, IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2. Константные домены тяжелых цепей, соответствующие различным классам иммуноглобулинов, обозначают как α, δ, ε, γ и μ соответственно.
Понятие «гуманизированное» антитело относится к химерному антителу, которое содержит аминокислотные остатки из нечеловеческих HVR и аминокислотные остатки из человеческих FR. В некоторых вариантах осуществления изобретения гуманизированное антитело может содержать практически все из по меньшей мере одного и, как правило, двух вариабельных доменов, в которых все или практически все HVR (например, CDR) соответствуют участкам нечеловеческого антитела, а все или практически все FR соответствуют участкам человеческого антитела. Гуманизированное антитело необязательно может содержать по меньшей мере часть константной области антитела, происходящей из человеческого антитела. Понятие «гуманизированная форма» антитела, например, нечеловеческого антитела, относится к антителу, которое подвергали гуманизации. Другие формы «гуманизированных антител», подпадающие под объем настоящего изобретения, представляют собой антитела, в которых константная область дополнительно модифицирована или изменена по сравнению с исходным антителом для создания свойств, которые требуются согласно изобретению, прежде всего касательно связывания C1q и/или связывания с Fc-рецептора (FcR).
«Человеческое антитело» представляет собой антитело, которое имеет аминокислотную последовательность, соответствующую последовательности антитела, продуцируемого человеком или человеческой клеткой, или полученную из источника, отличного от человека, с использованием спектра человеческих антител или других кодирующих человеческое антитело последовательностей. Из указанного определения человеческого антитела специально исключено гуманизированное антитело, содержащее нечеловеческие антигенсвязывающие остатки.
В контексте настоящего описания понятие «Fc-домен» или «Fc-область» относится к С-концевой области тяжелой цепи антитела, которая содержит по меньшей мере часть константной области. Понятие относится к нативной последовательности Fc-областей и вариантам Fc-областей. Fc-область IgG содержит СН2-домен IgG и СН3-домен IgG. «СН2-домен» Fc-области человеческого IgG, как правило, простирается от аминокислотного остатка, находящегося примерно в положении 231, до аминокислотного остатка, находящегося примерно в положении 340. В одном из вариантов осуществления изобретения к СН2-домену присоединена углеводная цепь. В контексте настоящего описания СН2-домен может иметь нативную последовательность СН2-домена или последовательность варианта СН2-домена. «СН3-домен» содержит сегмент С-концевых остатков до СН2-домена в Fc-области (т.е. от аминокислотного остатка, находящегося примерно в положении 341, до аминокислотного остатка, находящегося примерно в положении 447 IgG). В контексте настоящего описания СН3-домен может иметь нативную последовательность СН3-домена или последовательность варианта СН3-домена (например, СН3-домен с интродуцированной «выпуклостью» («выступ») в одной его цепи и с соответствующей интродуцированной «полостью» («впадина») в другой его цепи; см. US №5821333, специально включенный в настоящее описание в качестве ссылки). Указанные варианты СН3-доменов можно применять для усиления гетеродимеризации двух неидентичных тяжелых цепей антитела, согласно описанному в настоящем описании методу. В одном из вариантов осуществления изобретения Fc-область тяжелой цепи человеческого IgG простирается от Cys226 или от Pro230 до карбоксильного конца тяжелой цепи. Однако С-концевой лизин (Lys447) Fc-области может либо присутствовать, либо отсутствовать. Если специально не указано иное, то нумерация аминокислотных остатков в Fc-области или константном участке соответствует системе EU-нумерации, которую называют также EU-индексом, описанной у Kabat и др., Sequences of Proteins of Immunological Interest, 5-oe изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991.
Технология «knob-into-hole» описана, например, в US №5731168; US №7695936; у Ridgway и др., Prot Eng 9, 1996, cc. 617-621 и Carter, J Immunol Meth 248, 2001, cc. 7-15. В целом, метод включает интродукцию выпуклости («выступ») на поверхности раздела первого полипептида и соответствующей полости («впадина») в поверхности раздела второго полипептида, в результате выпуклость может помещаться в полость, усиливая тем самым образование гетеродимера и препятствуя образованию гомодимера. Выпуклости конструируют путем замены аминокислот с небольшими боковыми цепями на поверхности раздела первого полипептида на аминокислоты с более крупными боковыми цепями (например, на тирозин или триптофан). Компенсирующие полости идентичного или сходного с выпуклостями размера конструируют в поверхности раздела второго полипептида путем замены аминокислот с крупными боковыми цепями на аминокислоты с менее крупными боковыми цепями (например, аланин или треонин). Выпуклость и полость можно создавать, изменяя нуклеиновую кислоту, которая кодирует полипептиды, например, с помощью сайтспецфического мутагенеза или с использованием пептидного синтеза. В конкретном варианте осуществления изобретения модификация, приводящая к образованию «выступа», включает аминокислотную замену T366W в одной из двух субъединиц Fc-домена, и модификация, приводящая к образованию «впадины», включает аминокислотные замены T366S, L368A и Y407V в другой одной из двух субъединиц Fc-домена. В другом конкретном варианте осуществления изобретения субъединица Fc-домена, которая содержит модификацию, приводящую к образованию «выступа», дополнительно содержит аминокислотную замену S354C, а субъединица Fc-домена, которая содержит модификацию, приводящую к образованию «впадины», дополнительно содержит аминокислотную замену Y349C. Интродукция этих двух остатков цистеина приводит к образованию дисульфидного мостика между двумя субъединицами Fc-домена, дополнительно стабилизирующего димер (Carter, J Immunol Methods 248, 2001, cc. 7-15).
Подразумевается, что «область, эквивалентная Fc-области иммуноглобулина» включает встречающиеся в естественных условиях аллельные варианты Fc-области иммуноглобулина, а также варианты, имеющие изменения, приводящие к заменам, добавлениям или делециям, которые не снижают существенно способность иммуноглобулина опосредовать эффекторные функции (такие как антитело-обусловленная клеточнозависимая цитотоксичность). Например, одну или несколько аминокислот можно изымать путем делеции из N-конца или С-конца Fc-области иммуноглобулина без существенного снижения биологической функции. Указанные варианты можно выбирать согласно общим правилам, известным в данной области, так, чтобы оказывать минимальное воздействие на активность (см., например Bowie J.U. и др., Science 247, 1990, сс. 1306-1310).
Понятие «эффекторные функции» относится к видам биологической активности, присущим Fc-области антитела, которые варьируются в зависимости от изотипа антитела. Примерами эффекторных функций антитела являются: способность связываться с C1q и комплементзависимая цитотоксичность (CDC), способность связываться с Fc-рецептором, антитело-обусловленная клеточнозависимая цитотоксичность (ADCC), антитело-обусловленный клеточнозависимый фагоцитоз (ADCP), секреция цитокинов, опосредованное иммунным комплексом поглощение антигена антигенпрезентирующими клетками, понижающая регуляция рецепторов клеточной поверхности (например, В-клеточного рецептора) и активация В-клеток.
Зависящие от связывания Fc-рецептора эффекторные функции могут опосредоваться взаимодействием Fc-области антитела с Fc-рецепторами (FcR), которые представляют собой специализированные рецепторы клеточной поверхности гематопоэтических клеток. Fc-рецепторы принадлежат к суперсемейству иммуноглобулинов и, как установлено, опосредуют как удаление «покрытых» антителом патогенов путем фагоцитоза иммунных комплексов, так и лизис эритроцитов и различных других клеточных мишеней (например, опухолевых клеток), «покрытых» соответствующим антителом посредством антитело-обусловленной клеточнозависимой цитотоксичности (ADCC) (см., например, Van de Winkel J.G. и Anderson C.L., J. Leukoc. Biol. 49, 1991, cc. 511-524). FcR классифицируют на основе их специфичности в отношении изотипов иммуноглобулинов: Fc-рецепторы для антител IgG-тип обозначают как FcγR. Связывание с Fc-рецепторами описано, например, у Ravetch J.V. и Kinet J.P., Annu. Rev. Immunol. 9, 1991, cc. 457-492; Capel P.J. и др., Immunomethods 4, 1994, cc. 25-34; de Haas M. и др., J. Lab. Clin. Med. 126, 1995, cc. 330-341; и Gessner J.E. и др., Ann. Hematol. 76, 1998, cc. 231-248.
Перекрестное сшивание рецепторов для Fc-области антител IgG-типа (FcγR) запускает широкое разнообразие эффекторных функций, включая фагоцитоз, антитело-обусловленную клеточнозависимую цитотоксичность и высвобождение медиаторов воспаления, а также клиренс иммунного комплекса и регуляцию производства антител. У человека охарактеризовано три следующих класса FcγR:
- FcγRI (CD64) связывается с мономерным IgG с высокой аффинностью и экспрессируется на макрофагах, моноцитах, нейтрофилах и эозинофилах. Модификация в Fc-области IgG по меньшей мере одного из аминокислотных остатков E233-G236, Р238, D265, N297, А327 и Р329 (нумерация согласно EU-индексу Кэбота) снижает связывание с FcγRI. Остатки в IgG2 в положениях 233-236, замененные на остатки, присутствующие в IgG1 и IgG4, снижают связывание с FcγRI в 103 раз и элиминируют ответ человеческих моноцитов на сенсибилизированные антителом эритроциты (Armour K.L. и др., Eur. J. Immunol. 29, 1999, cc. 2613-2624).
- FcγRII (CD32) связывается с входящим в комплекс IgG с аффинностью от средней до низкой и характеризуется широким спектром экспрессии. Этот рецептор можно подразделять на два подтипа, FcγRIIA и FcγRIIB. FcγRIIA обнаружен на многих клетках, участвующих в цитолизе (например, макрофаги, макроциты, нейтрофилы) и, вероятно, может активировать процесс цитолиза. FcγRIIB, вероятно, играет роль в процессах ингибирования и обнаружен на В-клетках, макрофагах и тучных клетках и эозинофилах. На В-клетках его функцией, вероятно, является подавление дополнительного производства иммуноглобулинов и переключение изотипа, например, на IgE-класс. На макрофагах действие FcγRIIB заключается в ингибировании фагоцитоза, опосредуемого FcγRIIA. На эозинофилах и тучных клетках В-форма может способствовать подавлению активации этих клеток через IgE-связывание с его отдельным рецептором. Пониженное связывание FcγRIIA обнаружено, например, у антител, содержащих Fc-область IgG с мутациями по меньшей мере одного из аминокислотных остатков E233-G236, Р238, D265, N297, А327, Р329, D270, Q295, А327, R292 и K414 (нумерация согласно EU-индексу Кэбота).
- FcγRIII (CD16) связывается с IgG с аффинностью от средней до низкой и существует в виде двух типов. FcγRIIIA обнаружен на NK-клетках, макрофагах, эозинофилах и некоторых моноцитах и на Т-клетках и опосредует ADCC. Для FcγRIIIB характерен высокий уровень экспрессии на нейтрофилах. Пониженное связывание с FcγRIIIA обнаружено, например, у антител, которые содержат Fc-область IgG с мутацией по меньшей мере одного из аминокислотных остатков E233-G236, Р238, D265, N297, А327, Р329, D270, Q295, А327, S239, Е269, Е293, Y296, V303, А327, K338 и D376 (нумерация согласно EU-индексу Кэбота).
Картирование сайтов связывания на человеческом IgG1 для Fc-рецепторов, указанные выше сайты мутации и методы измерения связывания с FcγRI и FcγRIIA описаны у Shields R.L. и др., J. Biol. Chem. 276, 2001, сс. 6591-6604.
Понятие «ADCC» или «антитело-обусловленная клеточнозависимая цитотоксичность» относится к функции, опосредуемой связыванием с Fc-рецептором и относится к лизису клеток-мишеней антителом, указанным в настоящем описании, в присутствии эффекторных клеток. Способность антитела индуцировать начальные стадии, опосредующие ADCC, изучают путем измерения их связывания с экспрессирующими Fcγ-рецепторы клетками, такими как клетки, рекомбинантно экспрессирующие FcγRI и/или FcγRIIA, или NK-клетки (экспрессирующие в основном FcγRIIIA). В частности, измеряют связывание с FcγR на NK-клетках.
«Активирующий Fc-рецептор» представляет собой Fc-рецептор, который после взаимодействия с Fc-областью антитела осуществляет процесс передачи сигналов, которые стимулируют осуществление несущей рецептор клеткой эффекторных функций. Активирующие Fc-рецепторы включают FcγRIIIa (CD16a), FcγRI (CD64), FcγRIIa (CD32) и FcαRI (CD89). Конкретный активирующий Fc-рецептор представляет собой человеческий FcγRIIIa (см. UniProt, код доступа № Р08637, версия 141).
«Суперсемейство рецепторов фактора некроза опухоли» или «суперсемейство TNF-рецепторов» в настоящее время состоит из 27 рецепторов. Оно представляет собой группу цитокиновых рецепторов, характеризующуюся способностью связывать факторы некроза опухоли (TNF) через внеклеточный богатый цистеином домен (CRD). Указанные псевдо повторы отличаются наличием внутри цепочечных дисульфидных связей, образованных высоко консервативными остатками цистеина в цепях рецепторов. За исключением фактора роста нервов (NGF) все TNF являются гомологами архетипа TNF-альфа. В другой активной форме большинство TNF-рецепторов образуют тримерные комплексы в плазматической мембране. Таким образом, большинство TNF-рецепторов содержат трансмембранные домены (TMD). Некоторые из этих рецепторов содержат также внутриклеточные домены смерти (DD), которые осуществляют рекрутмент взаимодействующих с каспазой белков после связывания с лигандом, инициируя внешний путь активации каспазы. Другие рецепторы суперсемейства TNF, у которых отсутствуют домены смерти, связывают ассоциированные с TNF-рецептором факторы и активируют внутриклеточные пути передачи сигналов, которые могут приводить к пролиферации и дифференцировке. Эти рецепторы могут инициировать также апоптоз, но они осуществляют это через опосредованные механизмы. В дополнение к регуляции апоптоза некоторые рецепторы суперсемейства TNF участвуют в регуляции функций иммунных клеток, таких как гомеостаз и активация В-клеток, активация естественных клеток-киллеров и костимуляция Т-клеток. Некоторые другие регулируют специфические в отношении типа клеток ответы, такие как развитие фолликул волоса и развитие остеокластов. Представители суперсемейства TNF-рецепторов включают: рецептор фактора некроза опухоли 1 (1А) (TNFRSF1A, CD120a), рецептор фактора некроза опухоли 2 (1B) (TNFRSF1B, CD120b), рецептор лимфотоксина бета (LTBR, CD18), ОХ40 (TNFRSF4, CD134), CD40 (Вр50), Fas-рецептор (Аро-1, CD95, FAS), рецептор-ловушку 3 (TR6, М68, TNFRSF6B), CD27 (S152, Тр55), CD30 (Ki-1, TNFRSF8), 4-1ВВ (CD137, TNFRSF9), DR4 (TRAILR1, Аро-2, CD261, TNFRSF10A), DR5 (TRAILR2, CD262, TNFRSF10B), рецептор-ловушку 1 (TRAILR3, CD263, TNFRSF10C), рецептор-ловушку 2 (TRAILR4, CD264, TNFRSF10D), RANK (CD265, TNFRSF11A), остеопротегерин (OCIF, TR1, TNFRSF11B), TWEAK-рецептор (Fn14, CD266, TNFRSF12A), TACI (CD267, TNFRSF13B), BAFF-рецептор (CD268, TNFRSF13C), медиатор проникновения вируса герпеса (HVEM, TR2, CD270, TNFRSF14), рецептор фактора роста нервов (p75NTR, CD271, NGFR), антиген В-клеточного созревания (CD269, TNFRSF17), индуцируемый глюкокортикоидом родственный TNFR (GITR, AITR, CD357, TNFRSF18), TROY (TNFRSF19), DR6 (CD358, TNFRSF21), DR3 (Аро-3, TRAMP, WS-1, TNFRSF25) и рецептор эктодисплазина А2 (XEDAR, EDA2R).
Некоторые представители семейства рецепторов фактора некроза опухоли (TNFR) функционируют после начальной Т-клеточной активации, поддерживая ответы Т-клеток. Понятие «представитель семейства костимуляторных TNF-рецепторов» или «семейство костимуляторного TNF-рецептора» относится к подгруппе представителей семейства TNF-рецепторов, которые обладают способностью костимулировать пролиферацию и производство цитокинов Т-клетками. Понятие относится к любому нативному представителю семейства TNF-рецепторов из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди), приматы кроме человека (например, обезьяны циномолгус) и грызуны (например, мыши и крысы), если не указано иное. В конкретных вариантах осуществления изобретения представитель семейства костимуляторных TNF-рецепторов выбирают из группы, состоящей из ОХ40 (CD134), 4-1ВВ (CD137), CD27, HVEM (CD270), CD30 и GITR, которые все могут оказывать костимуляторные действия на Т-клетки. Более конкретно представитель семейства костимуляторных TNF-рецепторов выбирают из группы, состоящей из ОХ40 и 4-1ВВ.
Дополнительную информацию, в частности, о последовательностях представителей семейства TNF-рецепторов, можно почерпнуть из публично доступных баз данных, таких как Uniprot (www.uniprot.org). Например, человеческие костимуляторные TNF-рецепторы имеют следующие аминокислотные последовательности: человеческий ОХ40 (UniProt, код доступа № Р43489, SEQ ID NO: 98), человеческий 4-1ВВ (UniProt, код доступа № Q07011, SEQ ID NO: 99), человеческий CD27 (UniProt, код доступа № Р26842, SEQ ID NO: 100), человеческий HVEM (UniProt, код доступа № Q92956, SEQ ID NO: 101), человеческий CD30 (UniProt, код доступа № Р28908, SEQ ID NO: 102) и человеческий GITR (UniProt, код доступа № Q9Y5U5, SEQ ID NO: 103).
В контексте настоящего описания понятие «ОХ40» относится к любому нативному ОХ40 из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди) и грызуны (например, мыши и крысы), если не указано иное. Понятие подразумевает «полноразмерный», непроцессированный ОХ40, а также любую форму ОХ40, полученную в результате процессинга в клетке. Под понятие подпадают также встречающиеся в естественных условиях варианты ОХ40, например, сплайсинговые варианты или аллельные варианты. Аминокислотная последовательность приведенного в качестве примера человеческого ОХ40 представлена в SEQ ID NO: 98 (Uniprot Р43489, версия 112), а аминокислотная последовательность приведенного в качестве примера мышиного ОХ40 представлена в SEQ ID NO: 104 (Uniprot Р47741, версия 101).
Понятия «антитело анти-ОХ40», «анти-ОХ40», «антитело к ОХ40» и «антитело, которое специфически связывается с ОХ40» относится к антителу, которое обладает способностью связываться с ОХ40 с аффинностью, достаточной для применения антитела к качестве диагностического и/или терапевтического агента, мишенью которого является ОХ40. В одном из вариантов осуществления изобретения уровень связывания антитела к ОХ40 с неродственным белком, не представляющим собой ОХ40, составляет менее чем примерно 10% от уровня связывания антитела к ОХ40 при измерении, например, с помощью радиоиммуноанализа (РИА) или проточной цитометрии (FACS). В некоторых вариантах осуществления изобретения антитело, которое связывается с ОХ40, характеризуется величиной константы диссоциации (KD), составляющей ≤1 мкМ, ≤100 нМ, ≤10 нМ, ≤1 нМ, ≤0,1 нМ, ≤0,01 нМ или ≤0,001 нМ (например, 10-6 М или менее, например, от 10-8 М до 10-13 М, например, от 10-8 М до 10-10 М).
В контексте настоящего описания понятие «4-1ВВ» относится к любому нативному 4-1ВВ из любого применяемого в качестве источника позвоночного животного, включая млекопитающих, таких как приматы (например, люди) и грызуны (например, мыши и крысы), если не указано иное. Понятие подразумевает «полноразмерный», непроцессированный 4-1ВВ, а также любую форму 4-1ВВ, полученную в результате процессинга в клетке. Под понятие подпадают также встречающиеся в естественных условиях варианты 4-1ВВ, например, сплайсинговые варианты или аллельные варианты. Аминокислотная последовательность приведенного в качестве примера человеческого 4-1ВВ представлена в SEQ ID NO: 99 (Uniprot Р43489, код доступа № Q07011), аминокислотная последовательность приведенного в качестве примера мышиного 4-1ВВ представлена в SEQ ID NO: 105 (Uniprot, код доступа № P20334), а аминокислотная последовательность приведенного в качестве примера 4-1ВВ обезьян циномолгус (из Масаса mulatta) представлена в SEQ ID NO: 106 (Uniprot, код доступа № F6W5G6).
Понятия «антитело анти-4-1ВВ», «анти-4-1ВВ», «антитело к 4-1ВВ» и «антитело, которое специфически связывается с 4-1ВВ» относится к антителу, которое обладает способностью связываться с 4-1ВВ с аффинностью, достаточной для применения антитела к качестве диагностического и/или терапевтического агента, мишенью которого является 4-1ВВ. В одном из вариантов осуществления изобретения уровень связывания антитела к 4-1ВВ с неродственным белком, не представляющим собой 4-1ВВ, составляет менее чем примерно 10% от уровня связывания антитела к 4-1ВВ при измерении, например, с помощью радиоиммуноанализа (РИА) или проточной цитометрии (FACS). В некоторых вариантах осуществления изобретения антитело, которое связывается с 4-1 ВВ, характеризуется величиной константы диссоциации (KD), составляющей ≤1 мкМ, ≤100 нМ, ≤10 нМ, ≤1 нМ, ≤0,1 нМ, ≤0,01 нМ или ≤0,001 нМ (например, 10-6 М или менее, например, от 10-8 М до 10-13 М, например, от 10-8 М до 10-10 М).
Понятие «пептидный линкер» относится к пептиду, содержащему одну или несколько аминокислот, как правило, примерно 2-20 аминокислот. Пептидные линкеры известны в данной области и указаны в настоящем описании Приемлемыми неиммуногенными линкерными пептидами являются, например, пептидные линкеры (G4S)n, (SG4)n или G4(SG4)n, в которых «n», как правило, обозначает число от 1 до 10, как правило, от 1 до 4, в частности 2, т.е. пептиды, выбранные из группы, состоящей из GGGGS (SEQ ID NO: 107) GGGGSGGGGS (SEQ ID NO: 108), SGGGGSGGGG (SEQ ID NO: 109) и GGGGSGGGGSGGGG (SEQ ID NO: 110), но также включают последовательности GSPGSSSSGS (SEQ ID NO: 111), (G4S)3 (SEQ ID NO: 112), (G4S)4 (SEQ ID NO: 113), GSGSGSGS (SEQ ID NO: 114), GSGSGNGS (SEQ ID NO: 115), GGSGSGSG (SEQ ID NO: 116), GGSGSG (SEQ ID NO: 117), GGSG (SEQ ID NO: 118), GGSGNGSG (SEQ ID NO: 119), GGNGSGSG (SEQ ID NO: 120) и GGNGSG (SEQ ID NO: 121). Наибольший интерес представляют пептидные линкеры (G4S) (SEQ ID NO: 107), (G4S)2 или GGGGSGGGGS (SEQ ID NO: 108) и GSPGSSSSGS (SEQ ID NO: 111).
Понятие «аминокислота» в контексте настоящего описания обозначает группу встречающихся в естественных условиях карбокси-α-аминокислот, таких как аланин (трехбуквенный код: ala, однобуквенный код: А), аргинин (arg, R), аспарагин (asn, N), аспарагиновая кислота (asp, D), цистеин (cys, С), глутамин (gln, Q), глутаминовая кислота (glu, Е), глицин (gly, G), гистидин (his, Н), изолейцин (ile, I), лейцин (leu, L), лизин (lys, K), метионин (met, М), фенилаланин (phe, F), пролин (pro, Р), серии (ser, S), треонин (thr, Т), триптофан (trp, W), тирозин (tyr, Y) и валин (val, V).
Понятие «слитый» или «соединенный» подразумевает, что компоненты (например, тяжелая цепь антитела и Fab-фрагмент) сцеплены пептидными связями либо непосредственно, либо через один или несколько пептидных линкеров.
«Процент (%) идентичности аминокислотной последовательности» относительно полипептидной (белковой) референс-последовательности определяют как процент аминокислотных остатков в последовательности-кандидате, которые идентичны аминокислотным остаткам в полипептидной референс-последовательности, после выравнивания последовательностей и интродукции при необходимости брешей для достижения максимального процента идентичности последовательностей, и при этом какие-либо консервативные замены не учитываются при оценке идентичности последовательностей. Сравнительный анализ для определения процента идентичности аминокислотных последовательностей можно осуществлять различными путями, которые находятся в компетенции специалиста в данной области, например, с использованием публично доступных компьютерных программ, таких как программа BLAST, BLAST-2, ALIGN SAWI или Megalign (DNASTAR). Специалисты в данной области могут определять соответствующие параметры для выравнивания последовательностей, включая любые алгоритмы, необходимые для достижения максимального выравнивания по всей длине сравниваемых последовательностей. Однако для целей настоящего изобретения величину % идентичности аминокислотных последовательностей получают с использованием предназначенной для сравнения последовательностей компьютерной программы ALIGN-2. Предназначенная для сравнения последовательностей компьютерная программа ALIGN-2 разработана фирмой Genentech, Inc., и исходный код помещен на хранение вместе с документацией для пользователя в U.S. Copyright Office, Washington D.C., 20559, где он зарегистрирован под регистрационным номером U.S. Copyright Registration, № TXU510087. Программа ALIGN-2 представляет собой публично доступную программу фирмы Genentech, Inc., Южный Сан-Франциско, шт. Калифорния, или ее можно компилировать из исходного кода. Программу ALIGN-2 можно компилировать для применения в операционной системе UNIX, включая цифровую версию UNIX V4.0D. В программе ALIGN-2 все параметры для сравнения последовательностей являются заданными и не должны изменяться. В ситуациях, когда ALIGN-2 применяют для сравнения аминокислотных последовательностей, % идентичности аминокислотных последовательностей данной аминокислотной последовательности А относительно или по сравнению с данной аминокислотной последовательностью Б (которую другими словами можно обозначать как данная аминокислотная последовательность А, которая имеет или отличается определенным % идентичности аминокислотной последовательности относительно или по сравнению с данной аминокислотной последовательностью Б), рассчитывают следующим образом:
100 × частное X/Y,
где X обозначает количество аминокислотных остатков, оцененных программой сравнительного анализа последовательностей ALIGN-2 как идентичные совпадения при сравнительном анализе последовательностей А и Б с помощью указанной программы, и где Y обозначает общее количество аминокислотных остатков в Б. Должно быть очевидно, что, когда длина аминокислотной последовательности А не равна длине аминокислотной последовательности Б, то % идентичности аминокислотной последовательности А относительно аминокислотной последовательности Б не должен быть равен % идентичности аминокислотной последовательности Б относительно аминокислотной последовательности А. Если специально не указано иное, то в контексте настоящего описания все величины % идентичности аминокислотных последовательностей получают согласно процедуре, описанной в последнем из предшествующих параграфов, с помощью компьютерной программы ALIGN-2.
Некоторыми вариантами осуществления изобретения являются «варианты аминокислотных последовательностей» содержащих тримерный лиганд TNF антигенсвязывающих молекул, представленных в настоящем описании. Например, может оказаться желательным повышать аффинность связывания и/или другие биологические свойства содержащих тримерный лиганд TNF антигенсвязывающих молекул. Варианты аминокислотных последовательностей содержащих тримерный лиганд TNF антигенсвязывающих молекул можно получать путем интродукции соответствующих модификаций в нуклеотидную последовательность, кодирующую молекулы, или путем пептидного синтеза. Указанные модификации включают, например, делеции и/или инсерции, и/или замены остатков в аминокислотных последовательностях антитела. Для получения конечной конструкции можно использовать любую комбинацию делеций, инсерций и замен, при условии, что конечная конструкция обладает требуемыми характеристиками, например, способностью связываться с антигеном. Представляющие интерес сайты для замещающего мутагенеза включают HVR и каркасные участки (FR). Консервативные замены представлены в таблице Б под заголовком «предпочтительные замены» и дополнительно описаны ниже со ссылкой на классы (1)-(6) боковых цепей аминокислот. Аминокислотные замены можно интродуцировать в представляющую интерес молекулу и продукты подвергать скринингу в отношении требуемой активности, например, сохранения/повышения способности к связыванию антигена, пониженной иммуногенности или улучшенной ADCC или CDC.
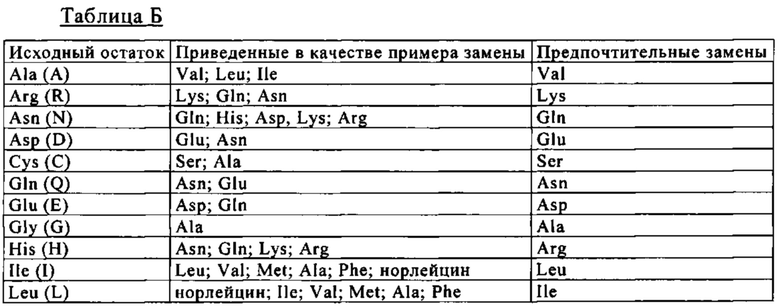

Аминокислоты можно группировать на основе общих свойств боковых цепей следующим образом:
(1) гидрофобные: норлейцин, Met, Ala, Val, Leu, Ile;
(2) нейтральные гидрофильные: Cys, Ser, Thr, Asn, Gln;
(3) кислотные: Asp, Glu;
(4)  : His, Lys, Arg;
: His, Lys, Arg;
(5) остатки, которые влияют на ориентацию цепи: Gly, Pro;
(6) ароматические: Trp, Tyr, Phe.
Под неконсервативными заменами подразумевают замену представителя одного из указанных классов на представителя из другого класса.
Понятие «варианты аминокислотной последовательности» включает полученные в результате замены варианты, в которых имеют место аминокислотные замены одного или нескольких остатков в гипервариабельном участке родительской антигенсвязывающей молекулы (например, гуманизированного или человеческого антитела). Как правило, образовавшийся(еся) вариант(ы), выбранный(ые) для дальнейшего изучения, должен(ны) иметь модификации (например, улучшения) определенных биологических свойств (например, повышенная аффинность, пониженная иммуногенность) относительно родительской антигенсвязывающей молекулы и/или должен(ны) практически сохранять определенные биологические свойства родительской антигенсвязывающей молекулы. Примером полученного в результате замены варианта является антитело с созревшей аффинностью, которое можно легко получать, например, с помощью методов созревания аффинности на основе фагового дисплея, которые указаны в настоящем описании. В целом, метод состоит в следующем: один или несколько остатков HVR подвергают мутации и вариант антигенсвязывающих молекул экспонируют на фаге и подвергают скринингу в отношении конкретной биологической активности (например, аффинности связывания). В некоторых вариантах осуществления изобретения замены, инсерции или делеции могут затрагивать один или несколько HVR, если указанные изменения не снижают существенно способность антигенсвязывающей молекулы связываться с антигеном. Например, в HVR могут быть сделаны консервативные изменения (например, консервативные замены, указанные в настоящем описании), которые не снижают существенно аффинность связывания. Ценным методом идентификации остатков или областей антитела, которые можно подвергать мутагенезу, является так называемый «аланин-сканирующий мутагенез», описанный у Cunningham B.C. и Wells J.A., Science 244, 1989, сс. 1081-1085. При осуществлении этого метода остаток или группу остатков-мишеней (например, заряженные остатки, такие как Arg, Asp, His, Lys и Glu) идентифицируют и заменяют на нейтральную или отрицательно заряженную аминокислоту (например, аланин или полиаланин) для решения вопроса о том, будет ли это влиять на взаимодействие антитела с антигеном. Дополнительные замены можно интродуцировать в те положения аминокислот, для которых продемонстрирована функциональная чувствительность к начальным заменам. В альтернативном или дополнительном варианте изучают кристаллическую структуру комплекса антиген-антигенсвязывающая молекула для идентификации точек контакта между антителом и антигеном. Указанные контактирующие остатки и соседние остатки можно рассматривать в качестве мишеней или исключать из рассмотрения в качестве кандидатов для замены. Варианты можно подвергать скринингу для решения вопроса о том, обладают ли они требуемыми свойствами.
Аминокислотные инсерции включают амино- и/или карбоксиконцевые слияния, варьирующие по длине от одного остатка до полипептидов, содержащих сто или большее количество остатков, а также инсерции одного или нескольких аминокислотных остатков внутрь последовательности. Примерами результата осуществления концевых инсерции являются биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, с концевыми метионильным остатком. Другие инсерционные варианты молекулы включают слияние N- или С-концов с полипептидом, который удлиняет время полужизни в сыворотке биспецифических антигенсвязывающих молекул.
В некоторых вариантах осуществления изобретения биспецифические антигенсвязывающие молекулы, представленные в настоящем описании, изменяют с целью повышения или понижения степени гликозилирования антитела. Варианты гликозилирования молекул легко можно получать путем такого изменения аминокислотной последовательности, которое позволяет создавать или удалять один или несколько сайтов гликозилирования. Если содержащая тримерный лиганд TNF антигенсвязывающая молекула содержит Fc-область, то можно изменять присоединенный к ней углевод. Нативные антитела, которые продуцируются клетками млекопитающих, как правило, содержат разветвленный биантенный олигосахарид, который, как правило, присоединен с помощью N-связи к Asn297 СН2-домена Fc-области (см., например, Wright А. и Morrison S.L., TIBTECH 15, 1997, сс. 26-32). Олигосахарид может включать различные углеводы, например, маннозу, N-ацетилглюкозамин (GlcNAc), галактозу и сиаловую кислоту, а также фукозу, присоединенную к GlcNAc в «стебле» биантенной олигосахаридной структуры. В некоторых вариантах осуществления изобретения модификации олигосахарида в содержащей тримерный лиганд семейства TNF антигенсвязывающей молекуле, предлагаемой в изобретении, можно осуществлять для создания вариантов с определенными улучшенными свойствами. Одним из объектов изобретения являются содержащие тримерный лиганд семейства TNF антигенсвязывающие молекулы, имеющие углеводную структуру, в которой снижено содержание фукозы, присоединенной (прямо или косвенно) к Fc-области. Указанные фукозилированные варианты могут обладать улучшенной ADCC-функцией (см., например, US №2003/0157108 (Presta L.) или US №2004/0093621 (фирма Kyowa Hakko Kogyo Co., Ltd). Другим объектом изобретения являются варианты биспецифических антигенсвязывающих молекул, предлагаемых в изобретении, содержащие двурассекающие олигосахариды, например, в которых биантенный олигосахарид, присоединенный к Fc-области, двурассекается с помощью GlcNAc. Указанные варианты могут обладать пониженным уровнем фукозилирования и/или улучшенной ADCC-функцией, см., например, WO 2003/011878 (Jean-Mairet и др.); US №6602684 (Umana и др.) и US 2005/0123546 (Umana и др.). Предложены также варианты по меньшей мере с одним остатком галактозы в олигосахариде, присоединенном к Fc-области. Такие варианты антител могут обладать повышенной CDC-функцией, и они описаны, например, WO 1997/30087 (Patel и др.); WO 1998/58964 (Raju S.) и WO 1999/22764 (Raju S.).
В некоторых вариантах осуществления изобретения может оказаться желательным создавать «сконструированные с помощью цистеина варианты» биспецифических антигенсвязывающих молекул, предлагаемых в изобретении, например, «тиоМАт», в которых один или несколько остатков в молекуле заменены на остатки цистеина. В конкретных вариантах осуществления изобретения замененные остатки находятся в доступных сайтах молекулы. Путем замены этих остатков на цистеин реакционноспособные тиольные группы помещаются в доступные сайты антитела, их можно использовать для конъюгации антитела с другими фрагментами, такими как фрагменты, представляющие собой лекарственное средство, или фрагменты, представляющие собой линкер, связанный с лекарственным средством, с созданием иммуноконъюгата, указанного ниже в настоящем описании. В некоторых вариантах осуществления изобретения любой(ые) один или несколько следующих остатков может(гут) быть заменен(ы) на цистеин: V205 (нумерация по Кэботу) легкой цепи; А118 (EU-нумерация) тяжелой цепи и S400 (EU-нумерация) Fc-области тяжелой цепи. Сконструированные с помощью цистеина антигенсвязывающие молекулы можно получать согласно методу, например, описанному в US №7521541.
Понятие «полинуклеотид» относится к выделенной молекуле нуклеиновой кислоты или конструкции, например, матричной РНК (мРНК), РНК вирусного происхождения или плазмидной ДНК (пДНК). Полинуклеотид может содержать обычную фосфодиэфирную связь или нетрадиционную связь (например, амидную связь, такую, которая присутствует в пептидных нуклеиновых кислотах (ПНК)). Понятие «молекула нуклеиновой кислоты» относится к любому одному или нескольким сегментам нуклеиновой кислоты, например, фрагментам ДНК или РНК, присутствующим в полинуклеотиде.
Под «выделенной» молекулой нуклеиновой кислоты или полинуклеотидом подразумевается молекула нуклеиновой кислоты, т.е. ДНК или РНК, которая отделена от ее нативного окружения. Например, рекомбинантный полинуклеотид, кодирующий входящий в вектор полипептид, рассматривается как выделенный для целей настоящего изобретения. Другими примерами выделенного полинуклеотида являются рекомбинантные полинуклеотиды, присутствующие в гетерологичных клетках-хозяевах, или очищенные (частично или полностью) полинуклеотиды, находящиеся в растворе. Выделенный полинуклеотид включает молекулу полинуклеотида, входящую в клетки, которые в норме содержат молекулу полинуклеотида, но молекула полинуклеотида присутствует вне хромосомы или имеет локализацию в хромосоме, отличную от ее локализации в хромосоме в естественных условиях. Выделенные молекулы РНК включают полученные in vivo или in vitro РНК-транскрипты, предлагаемые в настоящем изобретении, а также формы с позитивной и негативной цепью и двухцепочечные формы. Выделенные полинуклеотиды или нуклеиновые кислоты, предлагаемые в настоящем изобретении, включают также указанные молекулы, полученные с помощью синтеза. Кроме того, полинуклеотид или нуклеиновая кислота может представлять собой или может включать регуляторный элемент, такой как промотор, сайт связывания рибосом или терминатор транскрипции.
Под нуклеиновой кислотой или полинуклеотидом, имеющей/имеющим нуклеотидную последовательность, которая, например, на 95% «идентична» нуклеотидной референс-последовательности, предлагаемой в настоящем изобретении, подразумевается, что нуклеотидная последовательность полинуклеотида идентична референс-последовательности за исключением того, что полинуклеотидная последовательность может включать вплоть до 5 точечных мутаций на каждые 100 нуклеотидов нуклеотидной референс-последовательности. Другими словами, для получения полинуклеотида, имеющего нуклеотидную последовательность, которая идентична по меньшей мере на 95% нуклеотидной референс-последовательности, вплоть до 5% нуклеотидов в референс-последовательности можно изымать путем делеции или заменять на другой нуклеотид, или вплоть до 5% нуклеотидов от общего количества нуклеотидов в референс-последовательности можно встраивать в референс-последовательность. Эти изменения референс-последовательности могут иметь место в положениях на 5'- или 3'-конце нуклеотидной референс-последовательности или в ином положении между этими концевыми положениями, и их встраивают либо индивидуально между остатками в референс-последовательности, либо их встраивают в референс-последовательность в виде одной или нескольких смежных групп. На практике решение вопроса о том, идентична ли конкретная полинуклеотидная последовательность по меньшей мере на 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% нуклеотидной последовательности, предлагаемой в настоящем изобретении, можно решать, как правило, с использованием известных компьютерных программ, например, указанных выше для полипептидов (например, ALIGN-2).
Понятие «кассета экспрессии» относится к полинуклеотиду, полученному с помощью рекомбинации или синтеза, который содержит серии специфических нуклеотидных элементов, которые обеспечивают транскрипцию конкретной нуклеиновой кислоты в клетке-мишени. Рекомбинантную кассету экспрессии можно встраивать в плазмиду, хромосому, митохондриальную ДНК, пластидную ДНК, вирус или фрагмент нуклеиновой кислоты. Как правило, рекомбинантная кассета экспрессии, представляющая собой часть экспрессионного вектора, включает среди прочих последовательностей подлежащую транскрипции нуклеотидную последовательность и промотор. В некоторых вариантах осуществления изобретения кассета экспрессии, предлагаемая в изобретении, содержит полинуклеотидные последовательности, которые кодируют биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, или их фрагменты.
Понятие «вектор» или «экспрессионный вектор» является синонимом понятия «экспрессионная конструкция» и относится к молекуле ДНК, которую применяют для интродукции и обеспечения экспрессии конкретного гена, с которой он функционально связан в клетке-мишени. Понятие включает вектор, представляющий собой самореплицирующуюся структуру нуклеиновой кислоты, а также вектор, встроенный в геном клетки-хозяина, в которую он интродуцирован. Экспрессионный вектор, предлагаемый в настоящем изобретении, содержит кассету экспрессии. Экспрессионные векторы позволяют осуществлять транскрипцию стабильной мРНК в больших количествах. Когда экспрессионный вектор находится внутри клетки-мишени, то молекула рибонуклеиновой кислоты или белок, который кодируется геном, продуцируется в результате клеточного механизма транскрипции и/или трансляции. В одном из вариантов осуществления изобретения экспрессионный вектор, предлагаемый в изобретении, содержит кассету экспрессии, которая включает полинуклеотидные последовательности, которые кодируют биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, или их фрагменты.
В контексте настоящего описания понятия «клетка-хозяин», «линия клеток-хозяев» и «культура клеток-хозяев» используются взаимозаменяемо, и они относятся к клеткам, в которые интродуцирована экзогенная нуклеиновая кислота, включая потомство указанных клеток. К клеткам-хозяевам относятся «трансформанты» и «трансформированные клетки», которые включают первичные трансформированные клетки, а также потомство, выведенное из них, независимо от количества пересевов. Потомство может не быть строго идентичным родительской клетке по составу нуклеиновых кислот, а может нести мутации. Под данное понятие подпадает мутантное потомство, которое обладает такой же функцией или биологической активностью, что и отобранная путем скрининга или селекции исходная трансформированная клетка. Клетка-хозяин представляет собой любой тип клеточной системы, которую можно применять для создания биспецифических антигенсвязывающих молекул, предлагаемых в настоящем изобретении. Клетки-хозяева включают культивируемые клетки, например, культивируемые клетки млекопитающих, такие как (но не ограничиваясь только ими) СНО-клетки, BHK-клетки, NS0-клетки, SP2/0-клетки, клетки миеломы линии YO, клетки мышиной миеломы линии Р3Х63, PER-клетки, PER.С6-клетки или клетки гибридомы, клетки дрожжей, клетки насекомых и клетки растений, но также клетки, находящиеся в трансгенном животном, трансгенном растении или в культивируемой растительной или животной ткани.
Понятие «эффективное количество» агента относится к количеству, необходимому для обеспечения физиологического изменения в клетке или ткани, в которую его вводят.
Понятие «терапевтически эффективное количество» агента, например, фармацевтической композиции, относится к количеству, эффективному при применении в дозах и в течение периодов времени, необходимых для достижения требуемого терапевтического или профилактического результата. Терапевтически эффективное количество агента, например, элиминирует, снижает, замедляет, минимизирует или предупреждает нежелательные явления заболевания.
«Индивидуум» или «субъект» представляет собой млекопитающее. Млекопитающие представляют собой (но не ограничиваясь только ими) одомашненных животных (например, коровы, овцы, кошки, собаки и лошади), приматов (например, люди и приматы кроме человека, такие как мартышки), кроликов и грызунов (например, мыши и крысы). Предпочтительно индивидуум или субъект представляет собой человека.
Понятие «фармацевтическая композиция» относится к препарату, который находится в такой форме, что он обеспечивает биологическую активность входящего в его состав действующего вещества, которое должно обладать эффективностью, и который не содержит дополнительных компонентов, которые обладают неприемлемой токсичностью для индивидуума, которому следует вводить композицию.
«Фармацевтически приемлемый носитель» относится к ингредиенту в фармацевтической композиции, отличному от действующего вещества, который является нетоксичным для индивидуума. Фармацевтически приемлемые носители включают (но не ограничиваясь только ими) буфер, эксципиент, стабилизатор или консервант.
Понятие «листовка-вкладыш в упаковку» в контексте настоящего описания относится к инструкциям, которые обычно помещают в поступающие в продажу упаковки терапевтических продуктов, которые содержат информацию о показаниях, применении, дозе, пути введения, комбинированной терапии, противопоказаниях и/или мерах предосторожности при применении указанных терапевтических продуктов.
В контексте настоящего описания понятие «лечение» (и его грамматические варианты, такие как «лечить» или «процесс лечения») относится к клиническому вмешательству с целью изменения естественного течения болезни у индивидуума, подлежащего лечению, и его можно осуществлять либо для профилактики или в процессе развития клинической патологии. Требуемыми действиями лечения являются (но не ограничиваясь только ими) предупреждение возникновения или рецидива болезни, облегчение симптомов, уменьшение любых прямых или косвенных патологических последствий болезни, предупреждение метастазов, снижение скорости развития болезни, облегчение или временное ослабление болезненного состояния и ремиссия или улучшение прогноза. В некоторых вариантах осуществления изобретения молекулы, предлагаемые в изобретении, применяют для задержки развития болезни или замедления прогрессирования болезни.
В контексте настоящего описания понятие «рак» относится к пролиферативным заболеваниям, таким как лимфомы, лимфолейкозы, рак легкого, немелкоклеточный рак легкого (NSCL), альвеолярно-клеточный рак легкого, рак кости, рак поджелудочной железы, рак кожи, рак головы или шеи, кожная или внутриглазная меланома, рак матки, рак яичника, ректальный рак, рак анальной области, рак желудка, гастральный рак, рак ободочной кишки, рак молочной железы, рак матки, карцинома фаллопиевых труб, карцинома эндометрия, карцинома шейки матки, карцинома влагалища, карцинома вульвы, болезнь Ходжкина, рак пищевода, рак тонкого кишечника, рак эндокринной системы, рак щитовидной железы, рак паращитовидной железы, рак надпочечников, саркома мягких тканей, рак мочеиспускательного канала, рак пениса, рак предстательной железы, рак мочевого пузыря, рак почки или мочеточника, почечно-клеточная карцинома, карцинома почечной лоханки, мезотелиома, печеночно-клеточный рак, билиарный рак, неоплазмы центральной нервной системы (ЦНС), опухоли позвоночника, глиома ствола головного мозга, мультиформная глиобластома, астроцитомы, шванномы, эпендимомы, медуллобластомы, менингиомы, плоскоклеточные карциномы, аденома гипофиза и саркома Юинга, включая рефрактерные варианты любого из указанных выше видов рака и комбинации одного или нескольких видов рака.
Биспецифические антитела, предлагаемые в изобретении
В изобретении предложены новые биспецифические антигенсвязывающие молекулы с наиболее предпочтительными свойствами, такими как технологичность, стабильность, аффинность связывания, биологическая активность, эффективность целенаправленного действия (таргетинг) и пониженная токсичность.
Примеры биспецифических антигенсвязывающих молекул
Одним объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
В конкретном объекте изобретения эти биспецифические антигенсвязывающие молекулы отличаются способностью агонистически связываться с представителем семейства костимуляторных TNF-рецепторов. В частности, представителя семейства костимуляторных TNF-рецепторов выбирают из ОХ40 и 4-1ВВ.
Биспецифические антигенсвязывающие молекулы, которые связываются с ОХ40
Согласно одному из объектов изобретения представитель семейства костимуляторных TNF-рецепторов представляет собой ОХ40. В частности, в изобретении предложены биспецифические антигенсвязывающие молекулы, в которых фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который имеет аминокислотную последовательность SEQ ID NO: 1.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая по меньшей мере один фрагмент, обладающий способностью специфически связываться с ОХ40, в которой указанный фрагмент содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 2 и SEQ ID NO: 3,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 4 и SEQ ID NO: 5, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 и SEQ ID NO: 12, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 13, SEQ ID NO: 14 и SEQ ID NO: 15,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 16, SEQ ID NO: 17 и SEQ ID NO: 18, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23 и SEQ ID NO: 24.
В частности, предложена биспецифическая антигенсвязывающая молекула, содержащая по меньшей мере один фрагмент, обладающий способностью специфически связываться с ОХ40, в которой указанный фрагмент содержит
(а) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 2, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 4, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 6, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 13, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 19,
(б) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 2, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 4, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 7, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 13, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 20,
(в) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 2, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 4, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 8, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 13, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 21,
(г) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 2, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 4, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 9, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 13, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 22,
(д) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 3, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 5, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 10, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 14, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 17, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 23,
(е) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 3, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 5, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 11, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 14, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 17, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 23, или
(ж) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 3, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 5, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 12, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 15, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 18, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 24.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая по меньшей мере один фрагмент, обладающий способностью специфически связываться с ОХ40, в которой указанный фрагмент содержит VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 2, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 4, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 7, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 13, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 20.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 и SEQ ID NO: 37, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 и SEQ ID NO: 38.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит
(I) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 28.
Биспецифические антигенсвязывающие молекулы, которые связываются с 4-1ВВ
Согласно одному из объектов изобретения представитель семейства костимуляторных TNF-рецепторов представляет собой 4-1ВВ. В частности, в изобретении предложены биспецифические антигенсвязывающие молекулы, в которых фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который имеет аминокислотную последовательность SEQ ID NO: 39.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая по меньшей мере один фрагмент, обладающий способностью специфически связываться с 4-1ВВ, в которой указанный фрагмент содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 40 и SEQ ID NO: 41,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 42 и SEQ ID NO: 43, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 47 и SEQ ID NO: 48,
и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 49 и SEQ ID NO: 50,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 51 и SEQ ID NO: 52, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 56 и SEQ ID NO: 57.
В частности, предложена биспецифическая антигенсвязывающая молекула, содержащая по меньшей мере один фрагмент, обладающий способностью специфически связываться с 4-1ВВ, в которой указанный фрагмент содержит
(а) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 40, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 42, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 44, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 49, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 53,
(б) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 41, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 43, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 45, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 50, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 52, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 54,
(в) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 40, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 42, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 46, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 49, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 55,
(г) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 40, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 42, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 47, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 49, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 56, или
(д) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 40, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 42, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 48, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 49, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 57.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 и SEQ ID NO: 66, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 и SEQ ID NO: 67.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с 1ВВ, содержит
(I) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность SEQ ID NO: 67.
Биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, дополнительно отличаются тем, что содержат по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени. Таким образом, биспецифические антигенсвязывающие молекулы обладают преимуществом по сравнению с каноническими антителами, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, состоящим в том, что они могут избирательно индуцировать ответ костимуляторных Т-клеток на клетках-мишенях, которые, как правило, представляют собой раковые клетки. В одном из объектов изобретению антиген клетки-мишени выбирают из группы, состоящей из фибробласт-активирующего белка (FAP), ассоциированного с меланомой хондроитинсульфат-протеогликана (MCSP), рецептора эпидермального фактора роста (EGFR), карциноэмбрионального антигена (СЕА), CD19, CD20 и CD33.
Биспецифические антигенсвязывающие молекулы, у которых антиген клетки-мишени представляет собой FAP
В конкретном объекте изобретения антиген клетки-мишени представляет собой фибробласт-активирующий белок (FAP). FAP-связывающие фрагмент описаны в WO 2012/02006, которая полностью включена в настоящее описание в качестве ссылки. Представляющие особый интерес FAP-связывающие фрагменты описаны ниже.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с FAP, содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 68 и SEQ ID NO: 69,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 70 и SEQ ID NO: 71, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 72 и SEQ ID NO: 73, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 74 и SEQ ID NO: 75,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 76 и SEQ ID NO: 77, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 78 и SEQ ID NO: 79.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с FAP, содержит
(а) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 68, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 70, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 72, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 74, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 76, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 78, или
(б) VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 69, CDR-H2, который содержит аминокислотную последовательность SEQ ID NO: 71, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 73, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 75, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 77, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 79.
В конкретном объекте изобретения фрагмент, обладающий способностью специфически связываться с FAP, содержит VH-домен, содержащий CDR-H1, который содержит аминокислотную последовательность SEQ ID NO: 69, CDR-Н2, который содержит аминокислотную последовательность SEQ ID NO: 71, CDR-H3, который содержит аминокислотную последовательность SEQ ID NO: 73, и VL-домен, содержащий CDR-L1, который содержит аминокислотную последовательность SEQ ID NO: 75, CDR-L2, который содержит аминокислотную последовательность SEQ ID NO: 77, и CDR-L3, который содержит аминокислотную последовательность SEQ ID NO: 79.
В частности, предложена биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с FAP, содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 81, или
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 83.
Биспецифические антигенсвязывающие молекулы, которые связываются с ОХ40 и FAP
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(I) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 или SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 или SEQ ID NO: 38, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(а) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(б) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(в) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(г) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(д) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 30, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(е) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 30, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(ж) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 32, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(з) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 32, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(и) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 34, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(к) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 34, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(л) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 36, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(м) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 36, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(н) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81, или
(o) фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81, или в которой фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83.
Биспецифические антигенсвязывающие молекулы, которые связываются с 4-1ВВ и FAP
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(I) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 или SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 или SEQ ID NO: 67, и
(II) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая идентична по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой
(а) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 59, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(б) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 59, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(в) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 61, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(г) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 61, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(д) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 63, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(е) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 63, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(ж) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 65, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81,
(з) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 65, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83,
(и) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 67, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 80, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 81, или
(к) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 67, а фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 83.
Биспецифические одновалентные антигенсвязывающие молекулы (формат 1+1)
Одним из объектов изобретения являются биспецифические антигенсвязывающие молекулы, которые содержат (а) один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, (б) один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с ОХ40,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с ОХ40,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с FAP, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 303, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 182,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(б) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 231, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 186,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(в) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 233, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 190,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(г) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 235, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 194,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(д) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 237, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 198,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(е) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 239, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 202,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с 4-1ВВ,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Следующим объектом изобретения является биспецифическая антигенсвязывающая молекула, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с 4-1ВВ,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с FAP, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 295, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 261,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(б) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 297, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 265,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(в) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 299, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 269,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(г) первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 301, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 273,
вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 229, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217.
Биспецифические двухвалентные антигенсвязывающие молекулы (формат 2+2)
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) два фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Согласно одному из объектов изобретения биспецифическая антигенсвязывающая молекула является двухвалентной как в отношении представителя семейства костимуляторных TNF-рецепторов, так и в отношении антигена клетки-мишени.
Согласно одному из объектов изобретения биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(б) два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, где каждый из указанных двух дополнительных Fab-фрагментов соединен через пептидный линкер с С-концами тяжелых цепей, указанных в подпункте (а).
В конкретном объекте изобретения пептидный линкер представляет собой (G4S)4.
В другом объекте изобретения два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кроссовер-Fab-фрагменты, в которых вариабельные домены VL и VH заменены друг на друга и каждая из VL-CH-цепей соединена через пептидный линкер с С-концом тяжелых цепей, указанных в подпункте (а).
В частности, изобретение относится к биспецифическим антигенсвязывающим молекулам, в которых два Fab-фрагмента, обладающих способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляют собой два Fab-фрагмента, которые обладают способностью специфически связываться с ОХ40 или 4-1ВВ, и два дополнительных Fab-фрагмента, обладающих способностью специфически связываться с антигеном клетки-мишени, представляют собой кроссовер-Fab-фрагменты, обладающие способностью специфически связываться с FAP.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) два фрагмента, которые обладают способностью специфически связываться с ОХ40, (б) два фрагмента, которые обладают способностью специфически связываться с FAP, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным вариантом осуществления изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 216, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 182, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(б) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 219, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 186, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(в) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 221, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 190, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(г) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 223, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 194, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(д) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 225, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 198, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(е) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 227, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 202, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) два фрагмента, которые обладают способностью специфически связываться с 4-1ВВ, (б) два фрагмента, которые обладают способностью специфически связываться с FAP, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным вариантом осуществления изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 287, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 261, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(б) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 289, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 265, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(в) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 291, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 269, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217, или
(г) две тяжелые цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 293, первую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 273, и вторую легкую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 217.
Биспецифические антигенсвязывающие молекулы, которые являются двухвалентными в отношении связывания с представителем семейства костимуляторных TNF-рецепторов и одновалентными в отношении связывания с антигеном клетки-мишени (формат 2+1)
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) один фрагмент, который обладает способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Таким образом, предложена биспецифическая антигенсвязывающая молекула, где биспецифическая антигенсвязывающая молекула является двухвалентной в отношении связывания с представителем семейства костимуляторных TNF-рецепторов и одновалентной в отношении связывания с антигеном клетки-мишени.
В конкретном объекте изобретения биспецифическая антигенсвязывающая молекула содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(б) VH- и VL-домены, которые обладают способностью специфически связываться с антигеном клетки-мишени, где VH-домен соединен через пептидный линкер с С-концом одной из тяжелых цепей и где VL-домен соединен через пептидный линкер с С-концом второй тяжелой цепи.
В другом конкретном объекте изобретения биспецифическая антигенсвязывающая молекула содержит (а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и (б) VH- и VL-домены, которые обладают способностью специфически связываться с антигеном клетки-мишени, где VH-домен соединен через пептидный линкер с С-концом тяжелой цепи Fc с «выступом» и VL-домен соединении через пептидный линкер с С-концом тяжелой цепи Fc с «впадиной».
В другом конкретном объекте изобретения биспецифическая антигенсвязывающая молекула содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(б) VH- и VL-домены, которые обладают способностью специфически связываться с антигеном клетки-мишени, где VH-домен соединен через пептидный линкер с С-концом тяжелой цепи Fc с «впадиной» и VL-домен соединен через пептидный линкер с С-концом тяжелой цепи Fc с «выступом». В частности, в изобретении предложены биспецифические антигенсвязывающие молекулы, в которых два Fab-фрагмента, обладающие способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляют собой два Fab-фрагмента, которые обладают способностью специфически связываться с ОХ40 или 4-1ВВ, и VH- и VL-домены, обладающие способностью специфически связываться с антигеном клетки-мишени, обладают способностью связываться с FAP.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) два Fab-фрагмента, которые обладают способностью специфически связываться с ОХ40, (б) VH- и VL-домены, которые обладают способностью специфически связываться с FAP, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 186, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 306, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 307, или
(б) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 186, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 310, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 311.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая (а) два Fab-фрагмента, которые обладают способностью специфически связываться с 4-1ВВ, (б) VH- и VL-домены, которые обладают способностью специфически связываться с FAP, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая
(а) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 261, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 318, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 319, или
(б) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 265, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 322, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 323, или
(в) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 269, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 326, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 327, или
(г) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 273, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 330, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 331, или
(д) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 261, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 334, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 335, или
(е) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 265, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 338, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 339, или
(ж) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 269, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 342, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 343, или
(з) две легкие цепи, каждая из которых содержит аминокислотную последовательность SEQ ID NO: 273, первую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 346, и вторую тяжелую цепь, которая содержит аминокислотную последовательность SEQ ID NO: 347.
Модификации Fc-домена, приводящие к снижению связывания с Fc-рецептором и/или эффекторной функции
Биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, содержат также Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
В некоторых объектах изобретения одну или несколько аминокислотных модификаций можно интродуцировать в Fc-область антитела, представленного в настоящем описании, создавая тем самым вариант Fc-области. Вариант Fc-области может сдержать последовательность человеческой Fc-области (например, Fc-области человеческого IgG1, IgG2, IgG3 или IgG4), содержащую аминокислотную модификацию (например, замену) в одном или нескольких аминокислотных положениях.
Fc-домен придает биспецифическим антителам, предлагаемым в изобретении, предпочтительные фармакокинетические свойства, включая продолжительное время полужизни в сыворотке, что обеспечивает хорошее накопление в ткани-мишени и предпочтительное соотношение распределения в ткани-крови. Однако в то же время он может приводить к нежелательной направленности биспецифических антител, предлагаемых в изобретении, к клеткам, которые экспрессируют Fc-рецепторы, а не к предпочтительным несущим антиген клеткам. Таким образом, согласно конкретным вариантам осуществления изобретения Fc-домен биспецифических антител, предлагаемых в изобретении, обладает пониженной аффинностью связывания с Fc-рецептором и/или пониженной эффекторной функцией по сравнению с нативным Fc-доменом IgG, в частности Fc-доменом IgG1 или Fc-доменом IgG4. Более предпочтительно Fc-домен представляет собой Fc-домен IgG1.
В одном из объектов изобретения Fc-домен (или биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержащая указанный Fc-домен) характеризуется аффинностью связывания, составляющей менее чем 50%, предпочтительно менее чем 20%, более предпочтительно менее чем 10% и наиболее предпочтительно менее чем 5% от аффинности связывания с Fc-рецептором нативного Fc-домена IgG1 (или биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, содержащей указанный нативный Fc-домен IgG1) и/или эффекторной функцией, составляющей менее чем 50%, предпочтительно менее чем 20%, более предпочтительно менее чем 10% и наиболее предпочтительно менее чем 5% от эффекторной функции нативного Fc-домена IgG1 (или биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, содержащей указанный нативный Fc-домен IgG1). В одном из объектов изобретения Fc-домен (или биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержащая указанный Fc-домен) практически не связывается с Fc-рецептором и/или не индуцирует эффекторную функцию. В конкретном объекте изобретения Fc-рецептор представляет собой Fcγ-рецептор. В одном из объектов изобретения Fc-рецептор представляет собой человеческий Fc-рецептор. В одном из объектов изобретения Fc-рецептор представляет собой активирующий Fc-рецептор. В конкретном объекте изобретения Fc-рецептор представляет собой активирующий человеческий Fcγ-рецептор, более конкретно человеческий FcγRIIIa, FcγRI или FcγRIIa, наиболее предпочтительно человеческий FcγRIIIa. В одном из объектов изобретения Fc-рецептор представляет собой ингибирующий Fc-рецептор. В конкретном объекте изобретения Fc-рецептор представляет собой ингибирующий человеческий Fcγ-рецептор, более конкретно человеческий FcγRIIB. В конкретном объекте изобретения эффекторная функция представляет собой одну или несколько функций, выбранных из группы, включающей CDC, ADCC, ADCP и секрецию цитокинов. В одном из объектов изобретения эффекторная функция представляет собой ADCC. В одном из объектов изобретения Fc-домен характеризуется практически такой же аффинностью связывания с неонатальным Fc-рецептором (FcRn), что и нативный Fc-домен IgG1. Практически такое же связывание с FcRn достигается, когда Fc-домен (или биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержащая указанный Fc-домен), характеризуется аффинностью связывания, составляющей более чем примерно 70%, предпочтительно более чем примерно 80%, более предпочтительно более чем примерно 90%, от аффинности связывания нативного Fc-домена IgG1 (или биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, содержащей нативный Fc домен IgG1) с FcRn.
Согласно конкретному объекту изобретения Fc-домен конструируют так, чтобы он обладал пониженной аффинностью связывания с Fc-рецептором и/или пониженной эффекторной функцией по сравнению с не подвергнутым инженерии Fc-доменом. В конкретном объекте изобретения Fc-домен биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, содержит одну или несколько аминокислотных мутаций, которые снижают аффинность связывания Fc-домена с Fc-рецептором и/или эффекторную функцию. Как правило, в каждой из двух субъединиц Fc-домена присутствует(ют) одна или несколько одинаковых аминокислотных мутаций. В одном из объектов изобретения аминокислотная мутация снижает аффинность связывания Fc-домена с Fc-рецептором. В другом объекте изобретения аминокислотная мутация снижает аффинность связывания Fc-домена с Fc-рецептором по меньшей мере в 2 раза, по меньшей мере в 5 раз или по меньшей мере в 10 раз. В одном из объектов изобретения биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержащая сконструированный Fc-домен, характеризуется аффинностью связывания, составляющей менее чем 20%, в частности, менее чем 10%, более предпочтительно менее чем 5% от аффинности связывания с Fc-рецептором, характерной для биспецифических антител, предлагаемых в изобретении, содержащих не подвергнутый инженерии Fc-домен. В конкретном объекте изобретения Fc-рецептор представляет собой Fcγ-рецептор. В других объектах изобретения Fc-рецептор представляет собой человеческий Fc-рецептор. В одном из объектов изобретения Fc-рецептор представляет собой ингибирующий Fc-рецептор. В конкретном объекте изобретения Fc-рецептор представляет собой ингибирующий человеческий Fc-рецептор, более конкретно человеческий FcγRIIB. В некоторых объектах изобретения Fc-рецептор представляет собой активирующий Fc-рецептор. В конкретном объекте изобретения Fc-рецептор представляет собой активирующий человеческий Fc-рецептор, более конкретно человеческий FcγRIIIa, FcγRI или FcγRIIa, наиболее предпочтительно человеческий FcγRIIIa. Предпочтительно уменьшается связывание с каждым из этих рецепторов. Согласно некоторым объектам изобретения снижается также аффинность связывания с компонентом системы комплемента, в частности, аффинность связывания с C1q. Согласно одному из объектов изобретения не снижается аффинность связывания с неонатальным Fc-рецептором (FcRn). Практически такое же связывание с FcRn, т.е. сохранение аффинности связывания Fc-домена с указанным рецептором, достигается, когда Fc-домен (или биспецифическая антигенсвязывающая молекула, предлагаемая в изобретении, содержащая указанный Fc-домен) характеризуется аффинностью связывания с FcRn, составляющей более чем примерно 70% от аффинности связывания с FcRn не подвергнутой инженерии формы Fc-домена (или биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, содержащей указанную не подвергнутую инженерии форму Fc-домена). Fc-домен или биспецифическая антигенсвязывающая молекула, предлагаемый(ая) в изобретении, содержащий(ая) указанный Fc-домен, может характеризоваться аффинностью, составляющей более чем примерно 80% и даже более чем примерно 90% от указанной выше аффинности. В некоторых вариантах осуществления изобретения Fc-домен биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, создают так, чтобы он обладал пониженной эффекторной функцией по сравнению с не подвергнутым инженерии Fc-доменом Пониженная эффекторная функция может представлять собой (но не ограничиваясь только ими) пониженную(ые) одну или несколько из следующих функций: пониженная комплементзависимая цитотоксичность (CDC), пониженная антитело-обусловленная клеточнозависимая цитотоксичность (ADCC), пониженный антитело-обусловленный клеточнозависимый фагоцитоз (ADCP), пониженная секреция цитокинов, пониженное опосредованное иммунным комплексом поглощение антигена антигенпрезентирующими клетками, пониженное связывание с NK-клетками, пониженное связывание с макрофагами, пониженное связывание с моноцитами, пониженное связывание с полиморфноядерными клетками, пониженная непосредственная передача сигнала, индуцирующего апоптоз, пониженное созревание дендритных клеток или пониженное Т-клеточное примирование.
Антитела с пониженной эффекторной функцией включают также Fc-домены с заменой одного или нескольких остатков 238, 265, 269, 270, 297, 327 и 329 (US №637056). К указанным мутантам Fc относятся мутанты Fc с заменами в двух или большем количестве из аминокислотных положений 265, 269, 270, 297 и 327, включая так называемый мутант Fc-домена «DANA» с заменой остатков 265 и 297 на аланин (US №7332581). Описаны некоторые варианты антител с повышенной или сниженной способностью связываться с FcR (например, в US №6737056; WO 2004/056312 и у Shields R.L. и др., J. Biol. Chem. 276, 2001, сс. 6591-6604).
В одном из объектов изобретения Fc-домен содержит аминокислотную замену в положении Е233, L234, L235, N297, Р331 и Р329. В некоторых объектах Fc-домен содержит аминокислотные замены L234A и L235A («LALA»). В одном из указанных вариантов осуществления изобретения Fc-домен представляет собой Fc-домен IgG1, в частности Fc-домен человеческого IgG1. В одном из объектов изобретения Fc-домен содержит аминокислотную замену в положении Р329. В более конкретном объекте изобретения аминокислотная замена представляет собой Р329А или P329G, в частности P329G. В одном из вариантов осуществления изобретения Fc-домен содержит аминокислотную замену в положении Р329 и дополнительно аминокислотную замену, выбранную из группы, которая состоит из Е233Р, L234A, L235A, L235E, N297A, N297D или P331S. В более конкретных вариантах осуществления изобретения Fc-домен содержит аминокислотные мутации L234A, L235A и P329G («P329G LALA»). Комбинация аминокислотных замен «P329G LALA» практически полностью элиминирует связывание с Fcγ-рецептором Fc-домена человеческого IgG1, и она описана в опубликованной заявке на патент WO 2012/130831 А1. В указанном документе описаны методы получения указанных мутантных Fc-доменов и методы определения их свойства, таких как связывание с Fc-рецептором или эффекторные функции. Указанное антитело представляет собой IgG1 с мутациями L234A и L235A или мутациями L234A, L235A и P329G (согласно EU-нумерации Кэбота, описанной у Kabat и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во Public Health Service, National Institutes of Health, Bethesda, MD, 1991).
В одном из объектов изобретения Fc-домен представляет собой Fc-домен IgG4. В более конкретном варианте осуществления изобретения Fc-домен представляет собой Fc-домен IgG4, который содержит аминокислотные замены в положении S228 (нумерация по Кэботу), конкретно аминокислотную замену S228P. В более конкретном варианте осуществления изобретения Fc-домен представляет собой Fc-домен IgG4, который содержит аминокислотные замены в положениях L235E и S228P, и P329G. Указанная аминокислотная замена снижает in vivo обмен в Fab-плече антител в виде IgG4 (см. Stubenrauch и др., Drug Metabolism and Disposition 38, 2010, cc. 84-91).
Антитела с удлиненным временем полужизни и повышенной способностью к связыванию с неонатальным Fc-рецептором (FcRn), который ответствен за перенос материнских IgG эмбриону (Guyer R.L. и др., J. Immunol. 117, 1976, сс. 587-593 и Kim J.K. и др., J. Immunol. 24, 1994, сс. 2429-2434), описаны в US 2005/0014934. Эти антитела содержат Fc-область с одной или несколькими заменами, которые повышают связывание Fc-области с FcRn. Указанные варианты Fc-области включают варианты с заменами одного или нескольких следующих остатков Fc-области: 238, 256, 265, 272, 286, 303, 305, 307, 311, 312, 317, 340, 356, 360, 362, 376, 378, 380, 382, 413, 424 или 434, например, замену остатка 434 в Fc-области (US №7371826). Дополнительные примеры, касающиеся других вариантов Fc-области, описаны также у Duncan A.R. и Winter G., Nature 322, 1988, сс. 738-740; в US №5648260; US №5624821 и WO 94/29351.
Связывание с Fc-рецепторами можно легко определять, например, с помощью ELISA или поверхностного плазмонного резонанса (SPR), используя стандартный инструментарий, такой как устройство BIAcore (фирма GE Healthcare), и Fc-рецепторы, например, которые можно получать с помощью рекомбинантной экспрессии. В настоящем описании представлен приемлемый такой анализ связывания. Альтернативно этому, аффинность связывания Fc-доменов или клеток с активирующими биспецифическими антигенсвязывающими молекулами, которые содержат Fc-домен для Fc-рецепторов, можно оценивать, используя клеточные линии, для которых известно, что они экспрессируют конкретные Fc-рецепторы, такие как человеческие NK-клетки, экспрессирующие FcγIII-рецептор. Эффекторную функцию Fc-домена или биспецифических антител, предлагаемых в изобретении, которые содержат Fc-домен, можно оценивать методами, известными в данной области. Приемлемый анализ для оценки ADCC представлен в настоящем описании. Другие примеры анализов in vitro, предназначенных для оценки ADCC-активности представляющей интерес молекулы, описаны в US №5500362; у Hellstrom и др., Proc Natl Acad Sci USA 83, 1986, сс. 7059-7063 и Hellstrom и др., Proc Natl Acad Sci USA 82, 1985, cc. 1499-1502; US №5821337; у Bruggemann и др., J Exp Med 166, 1987, cc. 1351-1361. Альтернативно этому, можно применять методы, основанные на нерадиоактивном анализе (см., например, ACTI™ - нерадиоактивный анализ цитотоксичности с помощью проточной цитометрии (фирма CellTechnology, Inc. Маунтин-Вью, шт. Калифорния); и CytoTox 96® - нерадиоактивный анализ цитотоксичности (фирма Promega, Мэдисон, шт. Висконсин)). Приемлемыми эффекторными клетками для таких анализов являются мононуклеарные клетки периферической крови (РВМС) и естественные клетки-киллеры (NK). В альтернативном или дополнительном варианте ADCC-активность представляющей интерес молекулы можно оценивать in vivo, например, с использованием созданных на животных моделей, описанных у Clynes и др., Proc Natl Acad Sci USA 95, 1998, cc. 652-656.
В следующем разделе описаны предпочтительные аспекты биспецифических антигенсвязывающих молекул, предлагаемых в изобретении, которые содержат модификации Fc-домена, снижающие связывание Fc-рецептора и/или эффекторную функцию. Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, содержащая (а) по меньшей мере один фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, в которой Fc-домен содержит одну или несколько аминокислотную(ых) замену(н), которая(ые) снижает(ют) аффинность связывания антитела с Fc-рецептором, в частности, Fcγ-рецептором. Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, содержащая (а) по меньшей мере один фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, в которой Fc-домен содержит одну или несколько аминокислотную(ых) замену(н), которая(ые) снижает(ют) эффекторную функцию. В конкретном объекте изобретения Fc-домен представляет собой Fc-домен человеческого IgG1-подкласса с аминокислотными мутациями L234A, L235A и P329G (нумерация согласно EU-индексу Кэбота).
Модификации Fc-домена, способствующие гетеродимеризации
Биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, содержат различные антигенсвязывающие сайты, слитые с одной или другой из двух субъединиц Fc-домена, при этом две субъединицы Fc-домена, как правило, содержатся в двух неидентичных полипептидных цепях. Рекомбинантная совместная экспрессия этих полипептидов и последующая димеризация приводит к нескольким возможным комбинациям двух полипептидов. Для повышения выхода и чистоты биспецифических антител, предлагаемых в изобретении, при их получении методами рекомбинации целесообразно интродуцировать в Fc-домен биспецифических антигенсвязывающих молекул, предлагаемых в изобретении, модификацию, которая способствует ассоциации требуемых полипептидов.
Таким образом, конкретными объектами изобретения является биспецифическая антигенсвязывающая молекула, содержащая (а) по меньшей мере один фрагмент, который обладает способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, который обладает способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, в которой Fc-домен содержит модификацию, которая способствует ассоциации первой и второй субъединиц Fc-домена. Сайт наиболее сильного белок-белкового взаимодействия двух субъединиц Fc-домена человеческого IgG находится в СН3-домене Fc-домена. Таким образом, в одном из объектов изобретения указанная модификация находится в СН3-домене Fc-домена.
В конкретном объекте изобретения указанная модификация представляет собой так называемую модификацию типа «knob-in-hole» (типа «выступ-во впадину»), которая включает модификацию, приводящую к образованию «выступа» на одной из двух субъединиц Fc-домена и модификацию, приводящую к образованию «впадины» в другой одной из двух субъединиц Fc-домена. Таким образом, изобретение относится к биспецифической антигенсвязывающей молекуле, содержащей (а) по меньшей мере один фрагмент, который обладает способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) по меньшей мере один фрагмент, который обладает способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, в котором первая субъединица Fc-домена содержит «выступы», а вторая субъединица Fc-домена содержит «впадины» в соответствии с методом «knobs into holes». В конкретном объекте изобретения первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (EU-нумерация), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация согласно EU-индексу Кэбота).
Технология «knob-into-hole» описана, например, в US №5731168; US №7695936; у Ridgway и др., Prot Eng 9, 1996, сс. 617-621 и Carter, J Immunol Meth 248, 2001, сс. 7-15. В целом, метод включает интродукцию выпуклости («выступ») на поверхности раздела первого полипептида и соответствующей полости («впадина») в поверхности раздела второго полипептида, в результате выпуклость может помещаться в полость, способствуя тем самым образованию гетеродимера и препятствуя образованию гомодимера. Выпуклости конструируют путем замены аминокислот с небольшими боковыми цепями на поверхности раздела первого полипептида на аминокислоты с более крупными боковыми цепями (например, на тирозин или триптофан). Компенсирующие полости идентичного или сходного с выпуклостями размера создают в поверхности раздела второго полипептида путем замены аминокислот с крупными боковыми цепями на аминокислоты с менее крупными боковыми цепями (например, аланин или треонин).
Согласно одному из объектов изобретения в СН3-домене первой субъединицы Fc-домена биспецифических антигенсвязывающих молекул, предлагаемых в изобретении, аминокислотный остаток заменен аминокислотным остатком, который имеет больший объем боковой цепи, что приводит к образованию выпуклости в СН3-домене первой субъединицы, которая может помещаться в полость в СН3-домене второй субъединицы, и в СН3-домене второй субъединицы Fc-домена аминокислотный остаток заменен аминокислотным остатком, который имеет меньший объем боковой цепи, что приводит к образованию полости в СН3-домене второй субъединицы, в которую может помещаться выпуклость в СН3-домене первой субъединицы. Выпуклость и полость можно создавать путем изменения нуклеиновых кислот, кодирующих полипептиды, например, с помощью сайтнаправленного мутагенеза или путем пептидного синтеза. В конкретном объекте изобретения в СН3-домене первой субъединицы Fc-домена остаток треонина в положении 366 заменен остатком триптофана (T366W), и в СН3-домене второй субъединицы Fc-домена остаток тирозина в положении 407 заменен остатком валина (Y407V). В одном из объектов изобретения во второй субъединице Fc-домена дополнительно остаток треонина в положении 366 заменен остатком серина (T366S) и остаток лейцина в положении 368 заменен остатком аланина (L368A).
В следующем объекте изобретения в первой субъединице Fc-домена дополнительно остаток серина в положении 354 заменен остатком цистеина (S354C) и во второй субъединице Fc-домена дополнительно остаток тирозина в положении 349 заменен остатком цистеина (Y349C). Интродукция этих двух остатков цистеина приводит к образованию дисульфидного мостика между двумя субъединицами Fc-домена, дополнительно стабилизирующего димер (Carter, J Immunol Methods 248, 2001, сс. 7-15). В конкретном объекте изобретения первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (EU-нумерация), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация согласно EU-индексу Кэбота).
В альтернативном объекте изобретения модификация, способствующая ассоциации первой и второй субъединиц Fc-домена, представляет собой модификацию, которая опосредует определяемые электростатическими силами воздействия, например, описанные в публикации РСТ WO 2009/089004. В целом, указанный метод включает замену одного или нескольких аминокислотных остатков на поверхности раздела двух субъединиц Fc-домена на заряженные аминокислотные остатки, в результате чего образование гомодимера становится электростатически невыгодным, а гетеродимеризация становится электростатически выгодной.
С-конец тяжелой цепи биспецифического антитела, представленного в настоящем описании, может представлять собой полный С-конец, заканчивающийся аминокислотными остатками PGK. С-конец тяжелой цепи может представлять собой укороченный С-конец, в котором один или два С-концевой(ых) аминокислотный(ых) остаток(ка) удален(ы). В одном из предпочтительных объектов изобретения С-конец тяжелой цепи представляет собой укороченный С-конец, заканчивающийся PG. В одном из объектов всех указанных в настоящем описании объектов изобретения биспецифическое антитело, содержащее тяжелую цепь, которая включает С-концевой СН3-домен, указанный в настоящем описании, содержит С-концевой глицин-лизиновый дипептид (G446 и K447, нумерация согласно EU-индексу Кэбота). В одном из вариантов осуществления всех объектов изобретения, указанных в настоящем описании, биспецифическое антитело, содержащее тяжелую цепь, которая включает С-концевой СН3-домен, указанный в настоящем описании, содержит С-концевой остаток глицина (G446, нумерация согласно EU-индексу Кэбота).
Модификации в Fab-доменах
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит (а) первый Fab-фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, где в одном из Fab-фрагментов либо вариабельные домены VH и VL, либо константные домены СН1 и CL обменены. Биспецифические антитела получают согласно Crossmab-технологии.
Мультиспецифические антитела с заменой/обменом доменов в одном связывающем плече (CrossMabVH-VL или CrossMabCH-CL) подробно описаны в WO 2009/080252 и у Schaefer W. и др., Proc. Natl. Acad. Sci. USA, 108, 2011, сс. 11187-11191. При их применении выражение снижается образование побочных продуктов, связанное с ошибочным спариванием легкой цепи, связывающейся с первым антигеном, с «неправильной» тяжелой цепью, связывающейся с вторым антигеном (по сравнению с подходами, в которых не использован указанный обмен доменами).
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит (а) первый Fab-фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, где в одном из Fab-фрагментов константные домены CL и СН1 заменены друг на друга, в результате чего СН1-домен является частью легкой цепи, а CL-домен является частью тяжелой цепи. Более конкретно, во втором Fab-фрагменте, который обладает способностью специфически связываться с антигеном клетки-мишени, константные домены CL и СН1 заменены друг на друга, в результате чего СН1-домен является частью легкой цепи, а CL-домен является частью тяжелой цепи.
Одним из объектов изобретения является биспецифическая антигенсвязывающая молекула, которая содержит (а) первый Fab-фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, в котором константные домены CL и СН1 заменены друг на друга, в результате чего СН1-домен является частью легкой цепи, а CL-домен является частью тяжелой цепи. Указанную молекулу называют одновалентной биспецифической антигенсвязывающей молекулой.
Другим объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит (а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и (б) два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, где каждый из указанных дополнительных Fab-фрагментов соединен через пептидный линкер с С-концами тяжелых цепей, указанных в подпункте (а). В конкретном объекте изобретения дополнительные Fab-фрагменты представляют собой Fab-фрагменты, в которых вариабельные домены VL и VH заменены друг на друга, в результате чего VH-домен является частью легкой цепи, а VL-домен является частью тяжелой цепи.
Таким образом, конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, которая содержит (а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и (б) два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, где указанные два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кроссовер-Fab-фрагменты, в которых вариабельные домены VL и VH заменены друг на друга и каждая из VL-CH-цепей соединена через пептидный линкер с С-концом тяжелых цепей, указанных в подпункте (а).
В другом объекте изобретения для дальнейшего улучшения правильного спаривания биспецифическая антигенсвязывающая молекула, которая содержит (а) первый Fab-фрагмент, обладающий способностью связываться с представителем семейства костимуляторных TNF-рецепторов, (б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, может включать различные замены заряженных аминокислот (так называемые «заряженные остатки»). Эти модификации интродуцируют в скрещенные или нескрещенные СН1- и CL-домены. Конкретным объектом изобретения является биспецифическая антигенсвязывающая молекула, в которой в одном из CL-доменов аминокислота в положении 123 (EU-нумерация) заменена на аргинин (R) и аминокислота в положении 124 (EU-нумерация) заменена на лизин (K) и в которой в одном из СН1-доменов аминокислоты в положении 147 (EU-нумерация) и положении 213 (EU-нумерация) заменены на глутаминовую кислоту (Е).
Более конкретно, изобретение относится к биспецифической связывающей молекуле, содержащей Fab, в котором в CL-домене, примыкающем к представителю семейства лигандов TNF, аминокислота в положении 123 (EU-нумерация) заменена на аргинин (R) и аминокислота в положении 124 (EU-нумерация) заменена на лизин (K), и в которой в СН1-домене, примыкающем к представителю семейства лигандов TNF, аминокислоты в положении 147 (EU-нумерация) и положении 213 (EU-нумерация) заменены на глутаминовую кислоту (Е).
Примеры антител, предлагаемых в изобретении
Одним из объектов изобретения являются новые антитела и фрагменты антител, которые специфически связываются с OX40. Эти антитела обладают существенными преимуществами по сравнению с известными антителами к OX40, что делает их наиболее пригодными для включения в биспецифические антигенсвязывающие молекулы, которые содержат другой антигенсвязывающий фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени.
В частности, предложено антитело, которое специфически связывается с OX40, где указанное антитело содержит
(а) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 2, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 4, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 6, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 13, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 19,
(б) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 2, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 4, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 7, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 13, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 20,
(в) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 2, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 4, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 8, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 13, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 21,
(г) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 2, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 4, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 9, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 13, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 16, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 22,
(д) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 3, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 5, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 10, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 14, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 17, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 23,
(е) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 3, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 5, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 11, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 14, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 17, и CDR-:3, содержащий аминокислотную последовательность SEQ ID NO: 23,
или
(ж) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 3, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 5, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 12, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 15, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 18, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 24,
Одним из объектов изобретения является антитело, которое специфически связывается с OX40, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
Следующим объектом изобретения является антитело, которое конкурирует за связывание с антителом, которое специфически связывается с OX40, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
Одним из объектов изобретения является антитело, которое конкурирует за связывание с антителом, которое специфически связывается с OX40, где указанное антитело содержит вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28. В частности предложено антитело, которое специфически связывается с OX40, где антитело содержит вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28.
Другим объектом изобретения является антитело, которое специфически связывается с OX40 и обладает перекрестной реактивностью с человеческим и мышиным OX40, где указанное антитело содержит вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32.
Другим объектом изобретения являются новые антитела и фрагменты антител, которые специфически связываются с 4-1ВВ. Эти антитела обладают существенными преимуществами по сравнению с известными антителами к 4-1ВВ, что делает их наиболее пригодными для включения в биспецифические антигенсвязывающие молекулы, которые содержат другой антигенсвязывающий фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени.
В частности, предложено антитело, которое специфически связывается с 4-1ВВ, где указанное антитело содержит
(а) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 40, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 42, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 44, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 49, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 53,
(б) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 41, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 43, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 45, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 50, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 52, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 54,
(в) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 40, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 42, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 46, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 49, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 55,
(г) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 40, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 42, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 47, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 49, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 56, или
(д) VH-домен, который содержит CDR-H1, содержащий аминокислотную последовательность SEQ ID NO: 40, CDR-H2, содержащий аминокислотную последовательность SEQ ID NO: 42, CDR-H3, содержащий аминокислотную последовательность SEQ ID NO: 48, и VL-домен, который содержит CDR-L1, содержащий аминокислотную последовательность SEQ ID NO: 49, CDR-L2, содержащий аминокислотную последовательность SEQ ID NO: 51, и CDR-L3, содержащий аминокислотную последовательность SEQ ID NO: 57.
Одним из объектов изобретения является антитело, которое специфически связывается с 4-1ВВ, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
Следующим объектом изобретения является антитело, которое конкурирует за связывание с антителом, которое специфически связывается с 4-1ВВ, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
Полинуклеотиды
Изобретение относится также к выделенным полинуклеотидам, которые кодируют биспецифическую антигенсвязывающую молекулу, указанную в настоящем описании, или ее фрагмент.
Выделенные полинуклеотиды, кодирующие биспецифические антитела, предлагаемые в изобретении, можно экспрессировать в виде индивидуального полинуклеотида, который кодирует полную антигенсвязывающую молекулу, или в виде нескольких (например, двух или большего количества) совместно экспрессируемых полинуклеотидов. Полипептиды, кодируемые совместно экспрессируемыми полинуклеотидами, можно соединять, например, через дисульфидные связи или другими путями с получением функциональной антигенсвязывающей молекулы. Например, часть иммуноглобулина, представляющая собой легкую цепь, может кодироваться полинуклеотидом, отличным от полинуклеотида, кодирующего часть иммуноглобулина, представляющую собой тяжелую цепь. При совместной экспрессии полипептиды тяжелой цепи должны связываться с полипептидами легкой цепи с образованием иммуноглобулина.
В некоторых объектах изобретения выделенный полинуклеотид кодирует полипептид, который содержится в биспецифической молекуле, предлагаемой в изобретении, которая указана в настоящем описании.
Одним из объектов настоящего изобретения является выделенный полинуклеотид, который кодирует биспецифическую антигенсвязывающую молекулу, содержащую (а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с OX40, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Другим объектом изобретения является выделенный полинуклеотид, который кодирует биспецифическую антигенсвязывающую молекулу, содержащую (а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с 4-1ВВ, (б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и (в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
Следующим объектом изобретения является выделенный полинуклеотид, содержащий последовательность, которая кодирует антитело или фрагмент антитела, которое/который специфически связывается с OX40.
Другим объектом изобретения является выделенный полинуклеотид, содержащий последовательность, которая кодирует антитело или фрагмент антитела, которое/который специфически связывается с 4-1ВВ.
В некоторых вариантах осуществления изобретения полинуклеотид или нуклеиновая кислота представляет собой ДНК. В других вариантах осуществления изобретения полинуклеотид, предлагаемый в настоящем изобретении, представляет собой РНК, например, в форме матричной РНК (мРНК). РНК, предлагаемая в настоящем изобретении, может быть одноцепочечной или двухцепочечной.
Методы рекомбинации
Биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, можно получать, например, путем твердофазного пептидного синтеза (например, твердофазный синтез Меррифилда) или методом рекомбинации. Для рекомбинантного получения один или несколько полинуклеотидов, кодирующих биспецифическую антигенсвязывающую молекулу или ее полипептидные фрагменты, например, описанные выше, выделяют и встраивают в один или несколько векторов для дополнительного клонирования и/или экспрессии в клетке-хозяине. Указанный полинуклеотид можно легко выделять и секвенировать с помощью общепринятых процедур. Одним из объектов изобретения является вектор, предпочтительно экспрессионный вектор, содержащий один или несколько полинуклеотидов, предлагаемых в изобретении. Методы, хорошо известные специалистам в данной области, можно применять для конструирования экспрессионных векторов, содержащих кодирующую последовательность биспецифической антигенсвязывающей молекулы (фрагмента) наряду с приемлемыми контролирующими транскрипцию/трансляцию сигналами. Эти методы включают технологии рекомбинантной ДНК in vitro, методы синтеза и рекомбинации/генетической рекомбинации in vivo (см., например, методы, описанные у Maniatis и др., Maniatis и др., Molecular Cloning A Laboratory Manual, изд-во Cold Spring Harbor Laboratory, N.Y., 1989; и Ausubel и др., Current Protocols in Molecular Biology, изд-во Greene Publishing Associates and Wiley Interscience, N.Y., 1989). Экспрессионный вектор может представлять собой часть плазмиды, вируса или может представлять собой фрагмент нуклеиновой кислоты. Экспрессионный вектор включает кассету экспрессии, в которой полинуклеотид, кодирующий биспецифическую антигенсвязывающую молекулу или ее полипептидные фрагменты (т.е. кодирующую область), клонируют с обеспечением функциональной связи с промотором и/или другими элементами, контролирующими транскрипцию или трансляцию. В контексте настоящего описания «кодирующая область» представляет собой часть нуклеиновой кислоты, которая состоит из кодонов, транслируемых в аминокислоты. Хотя «стоп-кодон» (TAG, TGA или ТАА) не транслируется в аминокислоту, он, в случае его присутствия, может рассматриваться как часть кодирующей области, однако любые фланкирующие последовательности, например, промоторы, сайты связывания рибосом, терминаторы транскрипции, интроны, 5'- и 3'-нетранслируемые области и т.п., не являются частью кодирующей области. Две или большее количество кодирующих областей может присутствовать в индивидуальной полинуклеотидной конструкции, например, в индивидуальном векторе, или в отдельных полинуклеотидных конструкциях, например, в отдельных (различных) векторах. Кроме того, любой вектор может содержать одну кодирующую область или может содержать две или большее количество кодирующих областей, например, вектор, предлагаемый в настоящем изобретении, может кодировать один или несколько полипептидов, которые пост- или котранляционно разделяются на конечные белки посредством протеолитического расщепления. Кроме того, вектор, полинуклеотид или нуклеиновая кислота, предлагаемый/предлагаемая в изобретении, может кодировать гетерологичные кодирующие области, либо слитые, либо не слитые с полинуклеотидом, который кодирует биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении, или ее полипептидные фрагменты или варианты, или производные. Гетерологичные кодирующие области включают (но не ограничиваясь только ими) специализированные элементы или мотивы, такие как секреторный сигнальный пептид или гетерологичный функциональный домен. Функциональная связь имеет место, когда кодирующая область генного продукта, например, полипептида, ассоциирована с одной или несколькими регуляторными последовательностями таким образом, чтобы экспрессия генного продукта находилась под воздействием или контролем регуляторной(ых) последовательности(ей). Два ДНК-фрагмента (такие как кодирующая область полипептида и ассоциированный с ней промотор) являются «функционально связанными», если индукция промоторной функции приводит к транскрипции мРНК, кодирующей требуемый генный продукт, и если природа связи между двумя ДНК-фрагментами не оказывает воздействия на способность регулирующих экспрессию последовательностей направлять экспрессию генного продукта, или не оказывает воздействия на способность ДНК-матрицы к транскрипции. Таким образом, промоторная область должна быть функционально связана с нуклеиновой кислотой, кодирующей полипептид, если промотор обладает способностью осуществлять транскрипцию нуклеиновой кислоты. Промотор может представлять собой специфический для клетки промотор, который обеспечивает значительную транскрипцию ДНК только в предварительно отобранных клетках. Другие контролирующие транскрипцию элементы, помимо промотора, например, энхансеры, операторы, репрессоры и сигналы терминации транскрипции, можно функционально связывать с полинуклеотидом для обеспечения специфической для клетки транскрипции.
Приемлемые промоторы и другие контролирующие транскрипцию области представлены в настоящем описании. Специалистам в данной области известно широкое разнообразие контролирующих транскрипцию областей. Они включают (но не ограничиваясь только ими) контролирующие транскрипцию области, которые функционируют в клетках позвоночных животных, такие как (но не ограничиваясь только ими) сегменты промоторов и энхансеров из цитомегаловирусов (например, немедленно-ранний промотор в сочетании с интроном-А), обезьяньего вируса 40 (например, ранний промотор) и ретровирусов (таких как вирус саркомы Рауса). Другие контролирующие транскрипцию области включают области, выведенные из генов позвоночных животных, таких как ген актина, белка теплового шока, бычьего гормона роста и кроличьего β-глобина, а также другие последовательности, которые могут контролировать экспрессию генов в эукариотических клетках. Дополнительные приемлемые контролирующие транскрипцию области включают тканеспецифические промоторы и энхансеры, а также индуцибельные промоторы (например, промоторы, индуцируемые тетрациклинами). Аналогично этому, обычным специалистам в данной области известно широкое разнообразие контролирующих трансляцию элементов. Они включают (но не ограничиваясь только ими) сайты связывания рибосом, кодоны инициации трансляции и терминирующие кодоны и элементы, выведенные из вирусных систем (в частности внутренний сайт связывания (посадки) рибосом или IRES, который обозначают также как CITE-последовательность). Кассета экспрессии может включать также другие характерные структуры, такие как сайт инициации репликации и/или интегрированные в хромосому элементы, такие как длинные концевые повторы (LTR) ретровирусов, или инвертированные концевые повторы (ITR) аденоассоциированного вируса (AAV).
Кодирующие области полинуклеотида и нуклеиновой кислоты, предлагаемые в настоящем изобретении, могут быть ассоциированы с дополнительными кодирующими областями, которые кодируют секреторные или сигнальные пептиды, которые направляют секрецию полипептида, кодируемого полинуклеотидом, предлагаемым в настоящем изобретении. Например, если требуется секреция биспецифической антигенсвязывающей молекулы или ее полипептидных фрагментов, то ДНК, кодирующую сигнальную последовательность, можно помещать против хода транскрипции относительно нуклеиновой кислоты, кодирующей биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении, или ее полипептидные фрагменты. Согласно гипотезе, касающейся сигналов, белки, секретируемые клетками млекопитающих, имеют сигнальный пептид или секреторную лидерную последовательность, который/которая отщепляется от зрелого белка после инициации экспорта растущей белковой цепи через шероховатый эндоплазматический ретикулум. Обычным специалистам в данной области должно быть очевидно, что полипептиды, секретируемые клетками позвоночных животных, как правило, имеют сигнальный пептид, слитый с N-концом полипептида, который отщепляется от транслируемого полипептида с образованием секретируемой или «зрелой» формы полипептида. В некоторых вариантах осуществления изобретения используют нативный сигнальный пептид, например, сигнальный пептид тяжелой цепи или легкой цепи иммуноглобулина или функциональное производное указанной последовательности, которое сохраняет способность обеспечивать секрецию полипептида, функционально связанного с ним. Альтернативно этому, можно применять гетерологичный сигнальный пептид млекопитающих или его функциональное производное. Например, лидерную последовательность дикого типа можно заменять на лидерную последовательность человеческого тканевого активатора плазминогена (ТРА) или мышиной β-глюкуронидазы.
ДНК, кодирующую короткую белковую последовательность, которую можно применять для облегчения дальнейшей очистки (например, гистидиновую метку), или предназначенную для мечения слитого белка, можно включать внутрь или на концы полинуклеотида, кодирующего биспецифическую антигенсвязывающую молекулу или ее полипептидные фрагменты.
Другим объектом изобретения является клетка-хозяин, содержащая один или несколько полинуклеотидов, предлагаемых в изобретении. Некоторыми объектами изобретения является клетка-хозяин, содержащая один или несколько векторов, предлагаемых в изобретении. Полинуклеотиды и векторы могут обладать любыми особенностями, индивидуально или в сочетании, указанными в настоящем описании касательно полинуклеотидов и векторов соответственно. В одном из объектов изобретения клетка-хозяин содержит (например, трансформирована или трансфектирована) вектором, содержащим полинуклеотид, который кодирует биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении (или ее часть). В контексте настоящего описания понятие «клетка-хозяин» относится к любому типу клеточной системы, которую можно конструировать для получения слитых белков, предлагаемых в изобретении, или их фрагментов. Клетки-хозяева, пригодные для репликации и для поддержания экспрессии антигенсвязывающих молекул, хорошо известны в данной области. Такие клетки можно трансфектировать или трансдуцировать соответствующим образом конкретным экспрессионным вектором и можно выращивать большее количество содержащих вектор клеток с целью внесения в ферментеры для крупномасштабных процессов получения антигенсвязывающей молекулы в достаточных для клинических применений количествах. Приемлемыми клетками-хозяевами являются прокариотические микроорганизмы, такие как Е.coli, или различные эукариотические клетки, такие как клетки яичника китайского хомячка (СНО), клетки насекомых или т.п. Например, полипептиды можно получать в бактериях, в частности, когда отсутствует потребность в гликозилировании. После экспрессии полипептид можно выделять из пасты бактериальных клеток в растворимую фракцию и можно дополнительно очищать. Помимо прокариот, в качестве хозяев для клонирования или экспрессии векторов, которые кодируют полипептид, можно использовать эукариотические микроорганизмы, такие как нитчатые грибы или дрожжи, включая штаммы грибов и дрожжей, пути гликозилирования которых были «гуманизированы», что позволяет получать полипептид с частично или полностью человеческой схемой гликозилирования (см. Gerngross, Nat. Biotech. 22, 2004, сс. 1409-1414 и Li и др., Nat. Biotech. 24, 2006, сс. 210-215).
Клетки-хозяева, которые можно использовать для экспрессии (гликозилированных) полипептидов, получают также из многоклеточных организмов (беспозвоночных и позвоночных животных). Примерами клеток беспозвоночных являются клетки насекомых, а также можно применять клетки растений. Были выявлены многочисленные бакуловирусные штаммы и соответствующие пригодные для них в качестве хозяев клетки насекомых, прежде всего для трансфекции клеток Spodoptera frugiperda. В качестве хозяев можно применять также культуры растительных клеток (см., например, US №№5959177, 6040498, 6420548, 7125978 и 6417429 (описание технологии PLANTIBODIES™ для получения антител в трансгенных растениях). В качестве хозяев можно применять также клетки позвоночных животных. Например, можно использовать клеточные линии млекопитающих, которые адаптированы к росту в суспензии. Другими примерами приемлемых линий клеток-хозяев млекопитающих являются линия клеток почки обезьяны CV1, трансформированная с помощью SV40 (COS-7); линия клеток почки эмбриона человека (293 или клетки линии 293, субклонированные с целью выращивания в суспензионной культуре, Graham и др., J. Gen. Virol., 36, 1977, с. 59); клетки почки детеныша хомяка (ВНК); клетки Сертоли мыши (ТМ4-клетки, описанные, например, у Mather, Biol. Reprod., 23, 1980, сс. 243-251); клетки почки обезьяны (CV1); клетки почки африканской зеленой мартышки (VERO-76,); клетки карциномы шейки матки человека (HELA); клетки почки собаки (MDCK); клетки печени бычьей крысы (BRL 3А); клетки легкого человека (W138); клетки печени человека (Нер G2); клетки опухоли молочной железы мыши (ММТ 060562); клетки TRI, описанные, например, у Mather и др.. Annals N.Y. Acad. Sci., 383, 1982, сс. 44-68); клетки MRC 5 и клетки FS4. Другими ценными линиями клеток-хозяев млекопитающих являются клетки яичника китайского хомячка (СНО), включая dhfr--CHO-клетки (Urlaub и др., Proc. Natl. Acad. Sci. USA, 77, 1980, с. 4216); и клеточные линии миеломы, такие как Y0, NS0 и Sp2/0. Обзор конкретных линий клеток-хозяев млекопитающих, которые можно применять для производства белка, см., например, у Yazaki и Wu, в: Methods in Molecular Biology под ред. В.K.С. Lo, изд-во Humana Press, Totowa, NJ, т. 248, 2003, сс. 255-268. Клетки-хозяева включают культивируемые клетки, например, культивируемые клетки млекопитающих, клетки дрожжей, клетки насекомых, клетки бактерий и клетки растений (но не ограничиваясь только ими), а также клетки, находящиеся в организме трансгенного животного, трансгенного растения или культивируемой растительной или животной ткани. В одном из вариантов осуществления изобретения клетка-хозяин представляет собой эукариотическую клетку, предпочтительно клетку млекопитающего, такую как клетка яичника китайского хомячка (СНО), клетка почки человеческого эмбриона (HEK) или лимфоидная клетка (например, клетка Y0, NS0, Sp20). В данной области известны стандартные технологии для экспрессии чужеродных генов в этих системах. Клетки, экспрессирующие полипептид, содержащий либо тяжелую, либо легкую цепь иммуноглобулина, можно конструировать таким образом, чтобы в них происходила экспрессия других цепей иммуноглобулина, например, таким образом, чтобы экспрессируемый продукт представлял собой иммуноглобулин, который имеет как тяжелую, так и легкую цепь.
Одним из объектов изобретения является способ получения биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, или ее полипептидных фрагментов, где способ включает культивирование клетки-хозяина, содержащей полинуклеотиды, которые кодируют биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении, или ее полипептидные фрагменты, представленные в настоящем описании, в условиях, пригодных для экспрессии биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, или ее полипептидных фрагментов и выделение биспецифической антигенсвязывающей молекулы, предлагаемой в изобретении, или ее полипептидных фрагментов из клетки-хозяина (или культуральной среды клетки-хозяина).
Биспецифические молекулы, предлагаемые в изобретении, полученные с помощью представленных в настоящем описании методов, можно очищать с использованием известных в данной области методик, таких как жидкостная хроматография высокого разрешения, ионообменная хроматография, гель-электрофорез, аффинная хроматография, гель-фильтрация и т.п. Фактические условия, применяемые для очистки конкретного белка, зависят, в частности, от таких факторов, как чистый заряд, гидрофобность, гидрофильность и т.д., и они должны быть очевидны специалисту в данной области. Для очистки с помощью аффинной хроматографии можно использовать антитело, лиганд, рецептор или антиген, с которым связывается биспецифическая антигенсвязывающая молекула. Например, для очистки с помощью аффинной хроматографии слитых белков, предлагаемых в изобретении, можно использовать матрикс с белком А или белком G. Например, последовательное применение аффинной хроматографии на белке А или G и гель-фильтрации можно применять для выделения антигенсвязывающей молекулы практически согласно методу, описанному в разделе «Примеры». Чистоту биспецифической антигенсвязывающей молекулы или ее фрагментов можно определять с помощью любого из широкого разнообразия хорошо известных аналитических методов, включая гель-электрофорез, жидкостную хроматографию высокого давления и т.п. Например, установлено, что биспецифические антигенсвязывающие молекулы, экспрессия которых описана в разделе «Примеры», являются интактными и правильно собранными, что продемонстрировано с помощью ДСН-ПААГ в восстанавливающих и невосстанавливающих условиях.
Анализы
Антигенсвязывающие молекулы, представленные в настоящем описании, можно идентифицировать, подвергать скринингу или характеризовать их физические/химические свойства и/или виды биологической активности с помощью различных анализов, известных в данной области.
1. Анализы аффинности
Аффинность биспецифических антигенсвязывающих молекул, антител и фрагментов антител, представленных в настоящем описании, к соответствующему TNF-рецептору можно определять с помощью методов, изложенных в разделе «Примеры», с использованием поверхностного плазменного резонанса (SPR), применяя стандартную инструментальную базу, например, устройство BIAcore (фирма GE Healthcare), и рецепторы или белки-мишени, которые можно получать с помощью рекомбинантной экспрессии. Аффинность биспецифической антигенсвязывающей молекулы к антигену клетки-мишени можно определять также с помощью поверхностного плазменного резонанса (SPR), применяя стандартную инструментальную базу, например, устройство BIAcore (фирма GE Healthcare), и рецепторы или белки-мишени, которые можно получать с помощью рекомбинантной экспрессии. Конкретный приведенный с целью иллюстрации и примера вариант измерения аффинности связывания представлен в примере 2. Согласно одному из объектов изобретения величину KD измеряли методом поверхностного плазменного резонанса с помощью устройства BIACORE® Т100 (фирма GE Healthcare) при 25°С.
2. Анализы связывания и другие анализы
Связывание биспецифической антигенсвязывающей молекулы, представленной в настоящем описании, с клетками, экспрессирующими соответствующий рецептор, можно оценивать с использованием клеточных линий, которые экспрессируют конкретный рецептор или антиген-мишень, например, с помощью проточной цитометрии (FACS). В одном из объектов изобретения в анализе связывания используют мононуклеарные клетки периферической крови (РВМС), которые экспрессируют TNF-рецептор. Эти клетки применяют непосредственно после выделения (наивные РМВС) или после стимуляции (активированные РМВС). Согласно другому объекту изобретения для демонстрации связывания биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, с клетками, экспрессирующими соответствующий TNF-рецептор, используют активированные мышиные спленоциты (экспрессирующие молекулу TNF-рецептора). Согласно другому объекту изобретения для демонстрации связывания биспецифической антигенсвязывающей молекулы или антитела с клетками, экспрессирующими соответствующий TNF-рецептор обезьян циномолгус, используют РВМС, выделенные из гепаринизированной крови здоровых Масаса fascicularis.
Согласно следующему объекту изобретения для демонстрации связывания антигенсвязывающих молекул с антигеном клетки-мишени используют линии раковых клеток, экспрессирующих антиген-мишень, например, FAP.
Согласно другому объекту изобретения для идентификации антигенсвязывающей молекулы, которая конкурирует со специфическим антителом или антигенсвязывающей молекулой за связывание с мишенью или TNF-рецептором соответственно, можно применять анализы в условиях конкуренции. В некоторых вариантах осуществления изобретения указанная конкурирующая антигенсвязывающая молекула связывается с тем же эпитопом (например, линейным или конформационным эпитопом), с которым связывается специфическое антитело к мишени или специфическое антитело к TNF-рецептору. Подробные приведенные в качестве примеров методы картирования эпитопа, с которым связывается антитело, представлены у Morris, «Epitope Mapping Protocols», в: Methods in Molecular Biology, изд-во Humana Press, Totowa, NJ, T. 66, 1996.
3. Анализы активности
Одним из объектов изобретения являются анализы, предназначенные для идентификации биспецифических антигенсвязывающих молекул, которые связываются со специфическим антигеном клетки-мишени и со специфическим TNF-рецептором, которые обладают биологической активностью. Биологическая активность может включать, например, агонистическую активность в отношении передачи сигнала через TNF-рецептор на клетках, которые экспрессируют антиген клетки-мишени. Предложены также антигенсвязывающие молекулы, содержащие тримерный лиганд семейства TNF, которые идентифицированы с помощью анализов как обладающие биологической активностью in vitro.
В некоторых объектах изобретения биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении, тестируют в отношении указанной биологической активности. Кроме того, анализы для детекции клеточного лизиса (например, путем измерения высвобождения LDH), кинетики индукции апоптоза (например, путем измерения активности каспазы 3/7) или апоптоза (например, с использованием TUNEL-анализа) хорошо известны в данной области. Кроме того, биологическую активность указанных комплексов можно оценивать по их воздействию на выживаемость, пролиферацию и секрецию лимфокинов различными субпопуляциями лимфоцитов, такими как NK-клетки, NKT-клетки или γδT-клетки, или оценивать по их способности модулировать фенотип и функцию антигенпрезентирующих клеток, таких как дендритные клетки, моноциты/макрофаги или В-клетки.
Фармацевтические композиции, препаративные формы и пути введения
Следующим объектом изобретения являются фармацевтические композиции, содержащие любую/любое из биспецифических антигенсвязывающих молекул или антител, представленных в настоящем описании, например, предназначенные для применения в любом из указанных ниже терапевтических способов. В одном из вариантов осуществления изобретения фармацевтическая композиция содержит любую из биспецифических антигенсвязывающих молекул, представленных в настоящем описании, и по меньшей мере один фармацевтически приемлемый носитель. В другом варианте осуществления изобретения фармацевтическая композиция содержит любую из биспецифических антигенсвязывающих молекул, представленных в настоящем описании, и по меньшей мере одно дополнительное терапевтическое средство, например, указанное ниже.
Фармацевтические композиции, предлагаемые в настоящем изобретении, содержат в терапевтически эффективном количестве одну или несколько биспецифических антигенсвязывающих молекул, которая(ые) растворена(ы) или диспергирована(ы) в фармацевтически приемлемом носителе. Понятия «фармацевтически или фармакологически приемлемый» относится к молекулярным субстанциям и композициям, которые, в целом, нетоксичны для реципиентов в применяемых дозах и концентрациях, т.е. не вызывают вредные, аллергические или другие нежелательные реакции при введении при необходимости животному, такому, например, как человек. Приготовление фармацевтической композиции, которая содержит по меньшей мере одну биспецифическую антигенсвязывающую молекулу и необязательно дополнительное действующее вещество, должно быть очевидно специалистам в данной области в свете настоящего описания, например, из справочника Remington's Pharmaceutical Sciences, 18-oe изд., изд-во Mack Printing Company, 1990, включенного в настоящее описание в качестве ссылки. В частности, композиции представляют собой лиофилизированные препаративные формы или водные растворы. В контексте настоящего описания «фармацевтически приемлемый носитель» включает любые и все растворители, буферы, дисперсионные среды, покрытия, поверхностно-активные вещества, антиоксиданты, консерванты (например, антибактериальные агенты, противогрибные агенты), агенты для придания изотоничности, соли, стабилизаторы и их комбинации, которые должны быть известны обычному специалисту в данной области.
Парентеральные композиции включают композиции, созданные для введения путем инъекции, например, подкожной, внутрикожной, внутрь повреждения, внутривенной, внутриартериальной, внутримышечной, подоболочечной или внутрибрюшинной инъекции. Для инъекции биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, можно включать в препаративные формы в виде водных растворов, предпочтительно в физиологически совместимых буферах, таких как раствор Хэнкса, раствор Рингера или физиологический соляной буфер. Раствор может содержать предназначенные для получения препаративной формы агенты, такие как суспендирующие, стабилизирующие и/или диспергирующие агенты. Альтернативно этому, биспецифические антигенсвязывающие молекулы или антитела могут находиться в порошкообразной форме, предназначенной для восстановления перед применением приемлемым наполнителем, например, стерильной не содержащей пирогенов водой. Стерильные инъецируемые растворы приготавливают путем включения антигенсвязывающих молекул, предлагаемых в изобретении, в требуемом количестве в соответствующий растворитель при необходимости в сочетании с различными другими ингредиентами, перечисленными ниже. Стерильность можно легко обеспечивать, например, путем фильтрации через стерильные фильтрующие мембраны. Как правило, дисперсии получают путем включения различных стерилизованных действующих веществ в стерильный наполнитель, который содержит основную дисперсионную среду и/или другие ингредиенты. В случае стерильных порошков для получения стерильных инъецируемых растворов, суспензий или эмульсий предпочтительными методами получения являются вакуумная сушка или сушка вымораживанием, которые позволяют получать порошок действующего вещества в сочетании с любым дополнительным требуемым ингредиентом из предварительно стерилизованной фильтрацией жидкой среды. При необходимости жидкая среда перед осуществлением инъекции может быть соответствующим образом забуферена и жидкому разбавителю сначала придана изотоничность с помощью достаточного количества соляного раствора или глюкозы. Композиция должны быть стабильной в условиях приготовления и хранения и защищена от загрязняющего действия микроорганизмов, таких как бактерии и грибы. Принято поддерживать загрязнение эндотоксинами на минимальном безопасном уровне, например, менее 0,5 нг/мг белка. Пригодные фармацевтически приемлемые носители включают (но не ограничиваясь только ими): буферы, такие как фосфатный, цитратный и буферы на основе других органических кислот; антиоксиданты, включая аскорбиновую кислоту и метионин; консерванты (такие как октадецилдиметилбензиламмонийхлорид; гексаметонийхлорид; бензалконийхлорид; бензетонийхлорид; фенол, бутиловый или бензиловый спирт; алкилпарабены, такие как метил- или пропилпарабен; катехол, резорцинол; циклогексанол; 3-пентанол и мета-крезол); низкомолекулярные (содержащие менее примерно 10 остатков) полипептиды; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включая глюкозу, маннозу или декстрины; хелатирующие агенты, такие как ЭДТА; сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; комплексы с металлами (например, комплексы Zn-белок); и/или неионогенные поверхностно-активные вещества, такие как полиэтиленгликоль (ПЭГ). Водные суспензии для инъекций могут содержать соединения, которые повышают вязкость суспензии, такие как натриевая соль карбоксиметилцеллюлозы, сорбит, декстран или т.п. Необязательно суспензия может содержать также стабилизаторы или агенты, которые повышают растворимость соединений, что позволяет получать высококонцентрированные растворы. Кроме того, суспензии действующих веществ можно получать в виде соответствующих масляных предназначенных для инъекции суспензий. Приемлемые липофильные растворители или наполнители включают жирные нелетучие масла, такие как кунжутное масло, или синтетические эфиры жирных кислот, такие как этилолеаты или триглицериды, или липосомы.
Действующие вещества можно заключать в микрокапсулы, например, полученные с помощью методов коацервации или межфазной полимеризации, например в гидроксипропилметилцеллюлозные или желатиновые микрокапсулы и поли(метилметакрилатные) микрокапсулы соответственно, в коллоидные системы введения лекарственного средства (например, в липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы) или в макроэмульсии. Такие методы описаны в Remington's Pharmaceutical Sciences (18-ое изд., изд-во Mack Printing Company, 1990). Приемлемыми примерами препаратов с замедленным высвобождением являются полупроницаемые матрицы из твердых гидрофобных полимеров, включающие полипептид, такие матрицы представляют собой изделия определенной формы, например, пленки или микрокапсулы. В конкретном варианте осуществления изобретения для достижения пролонгированной абсорбции инъецируемой композиции можно применять в композиции агенты, замедляющие абсорбцию, такие, например, как моностеарат алюминия, желатин или их комбинации.
В контексте настоящего описания фармацевтически приемлемые носители включают также диспергирующие агенты интерстициальных лекарственных средств, такие как растворимые активные в нейтральной среде гликопротеины гиалуронидаз (sHASEGP), например, человеческие растворимые гликопротеины гиалуронидазы РН-20, такие как rHuPH20 (HYLENEX®, фирма Baxter International, Inc.). Некоторые примеры sHASEGP и методы их применения, в том числе rHuPH20, описаны в публикациях патентов США 2005/0260186 и 2006/0104968. Согласно одному из объектов изобретения sHASEGP объединяют с одним или несколькими дополнительными гликозаминогликаназами, такими как хондроитиназы.
Примеры лиофилизированных композиций антител описаны в US №6267958. Водные композиции антител включают композиции, описанные в №US 6171586 и WO 2006/044908, последние композиции включают гистидин-ацетатный буфер.
Помимо описанных выше композиций, антигенсвязывающие молекулы можно приготавливать также в виде препарата в форме депо. Указанные препаративные формы длительного действия можно применять путем имплантации (например, подкожной или внутримышечной) или внутримышечной инъекции. Так, например, слитые белки можно включать в препаративные формы в сочетании с приемлемыми полимерными или гидрофобными материалами (например, в виде эмульсии в приемлемом масле) или ионообменными смолами, или в виде умеренно растворимых производных, например, умеренно растворимой соли.
Фармацевтические композиции, содержащие биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, можно приготавливать с помощью общепринятых процессов смешения, растворения, эмульгирования, капсулирования, захвата или лиофилизации. Фармацевтические композиции можно включать в препаративные формы с помощью общепринятого метода с использованием одного или нескольких физиологически приемлемых носителей, разбавителей, эксципиентов или вспомогательных веществ, которые облегчают обработку белков, с получением препаратов, которые можно применять в фармацевтических целях. Соответствующая форма зависит от выбранного пути введения.
Биспецифические антигенсвязывающие молекулы можно включать в композиции в виде свободной кислоты или свободного основания, в нейтральной форме или в форме соли. Фармацевтически приемлемые соли представляют собой соли, которые практически сохраняют биологическую активность свободной кислоты или свободного основания. Они включают кислотно-аддитивные соли, например, соли, образованные со свободными аминогруппами белковой композиции, или образованные с неорганическими кислотами, такими, например, как соляная или фосфорная кислота, или такими органическими кислотами, как уксусная, щавелевая, винная или миндальная кислота. Соли, образованные со свободной карбоксильной группой, можно получать также из неорганических оснований, таких, например, как гидроксиды натрия, калия, аммония, кальция или железа; или таких органических оснований как изопропиламин, триметиламин, гистидин или прокаин. Фармацевтические соли имеют тенденцию к более высокой растворимости в водных и других протонных растворителях по сравнению с соответствующими формами в виде свободных оснований.
Композиция, представленная в настоящем описании, может содержать также более одного действующего вещества, если это необходимо для конкретного подлежащего лечению показания, предпочтительно вещества с дополнительными видами активности, которые не оказывают отрицательного воздействия друг на друга. Такие действующие вещества должны присутствовать в комбинации в количествах, эффективных для решения поставленной задачи.
Композиции, предназначенные для применения in vivo, как правило, должны быть стерильными. Это легко можно осуществлять путем фильтрации через стерильные фильтрующие мембраны.
Способы и композиции для терапевтического применения
Любую из биспецифических антигенсвязывающих молекул, представленных в настоящем описании, можно применять в терапевтических способах.
Для применения в терапевтических способах биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, можно включать в состав препаративных форм, дозировать и вводить в соответствии с надлежащей клинической практикой. Рассматриваемые в этом контексте факторы включают конкретное нарушение, подлежащее лечению, конкретное млекопитающее, подлежащее лечению, клиническое состояние индивидуального пациента, причину заболевания, область введения агента, метод введения, схему введения и другие факторы, известные практикующим медикам.
Одним из объектов изобретения являются биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, предназначенные для применения в качестве лекарственного средства.
В следующих объектах изобретения биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, предназначены для применения (I) для стимуляции или повышения Т-клеточного ответа, (II) для поддержания выживаемости активированных Т-клеток, (III) для применения лечения инфекций, (IV) для применения для лечения рака, (V) для применения для замедления прогрессирования рака или (VI) для применения для пролонгирования времени жизни пациента, страдающего раком. Одним из объектов изобретения являются антигенсвязывающие молекулы или антитела, содержащие тримерный лиганд семейства TNF, предлагаемые в изобретении, предназначенные для применения при лечении заболевания у индивидуума, в частности для применения для лечения рака.
В некоторых объектах изобретения биспецифические антигенсвязывающие молекулы или антитела, предлагаемые в изобретении, предназначены для применения в способе лечения. Одним из объектов изобретения являются биспецифические антигенсвязывающие молекулы или антитела, представленные в настоящем описании, для применения для лечения заболевания у индивидуума, который нуждается в этом. Некоторыми объектами изобретения являются биспецифические антигенсвязывающие молекулы или антитела для применения в способе лечения индивидуума, который имеет заболевание, включающем введение индивидууму в терапевтически эффективном количестве биспецифической антигенсвязывающей молекулы или антитела. В некоторых объектах изобретения заболевание представляет собой рак. Субъект, пациент или «индивидуум», нуждающийся в лечении, представляет собой млекопитающее, более конкретно человека.
Одним из объектов изобретения является способ (I) стимуляции или повышения Т-клеточного ответа, (II) поддержания выживаемости активированных Т-клеток, (III) лечения инфекций, (IV) лечения рака, (V) замедления прогрессирования рака или (VI) пролонгирования времени жизни пациента, страдающего раком, где способ включает введение в терапевтически эффективном количестве биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, индивидууму, который нуждается в этом. Следующим объектом изобретения является применение биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, для получения или производства лекарственного средства, предназначенного для лечения заболевания у индивидуума, который нуждается в этом. В одном из объектов изобретения лекарственное средство предназначено для применения в способе лечения заболевания, включающем введение индивидууму, имеющему заболевание, лекарственного средства в терапевтически эффективном количестве. В некоторых объектах изобретения заболевание, подлежащее лечению, представляет собой пролиферативное нарушение, в частности рак. Примеры видов рака включают (но не ограничиваясь только ими) рак мочевого пузыря, рак головного мозга, рак головы и шеи, рак поджелудочной железы, рак легкого, рак молочной железы, рак яичника, рак матки, рак шейки матки, рак эндометрия, рак пищевода, рак ободочной кишки, колоректальный рак, ректальный рак, рак желудка, рак предстательной железы, рак крови, рак кожи, плоскоклеточную карциному, рак кости и рак почки. Другие примеры рака включают карциному, лимфому (например, лимфому Ходжкина и неходжкинскую лимфому), бластому, саркому и лейкоз. Другие связанные с пролиферацией клеток нарушения, которые можно лечить с помощью биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, включают (но не ограничиваясь только ими) неоплазмы, локализованные в: брюшном отделе, кости, молочной железе, пищеварительной системе, печени, поджелудочной железе, брюшной полости, эндокринных железах (надпочечники, паращитовидная, гипофиз, яички, яичник, тимус, щитовидная железа), глазах, голове и шеи, нервной системе (центральной и периферической), лимфатической системе, тазовой области, коже, мягкой ткани, селезенке, грудной области и мочеполовой системе. Также они включают предраковые состояния или повреждения и метастазы рака. В некоторых вариантах осуществления изобретения рак выбирают из группы, состоящей из почечно-клеточного рака, рака кожи, рака легкого, колоректального рака, рака молочной железы, рака головного мозга, рака головы и шеи. Специалисту в данной области должно быть очевидно, что во многих случаях биспецифическая антигенсвязывающая молекула или антитело, предлагаемая/предлагаемое в изобретении, не может обеспечивать исцеление, а может только оказывать благоприятное воздействие. В некоторых объектах изобретения физиологические изменения, характеризующиеся некоторым благоприятным воздействием, рассматриваются также как терапевтически ценные. Таким образом, в некоторых объектах изобретения количество биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, которое обеспечивает физиологическое изменение, рассматривается как «эффективное количество» или «терапевтически эффективное количество».
Для предупреждения или лечения заболевания соответствующая доза биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении (при применении индивидуально или в комбинации с одним или несколькими другими дополнительными терапевтическими агентами) должна зависеть от типа заболевания, подлежащего лечению, пути введения, веса тела пациента, специфичности молекулы, серьезности и течения заболевания, от того, вводят ли биспецифическую антигенсвязывающую молекулу или антитело в превентивных или терапевтических целях, предшествующих или осуществляемых одновременно терапевтических вмешательств, истории болезни пациента и ответа на слитый белок, и от предписания лечащего врача. Практикующий специалист, ответственный за введение, в любом случае, должен определять концентрацию действующего(их) вещества(в) в композиции и соответствующую(ие) дозу(ы) для индивидуального пациента. Различные схемы введения доз включают (но не ограничиваясь только ими) однократное введение или несколько введений в различные моменты времени, болюсное введение и пульсирующую инфузию.
Биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, можно вводить пациенту в виде одной обработки или серий обработок. В зависимости от типа и серьезности заболевания возможная начальная доза антигенсвязывающей молекулы, содержащей тримерный лиганд семейства TNF, для введения пациенту, например, с использованием одного или нескольких индивидуальных введений или с помощью непрерывной инфузии, может составлять примерно от 1 мкг/кг до 15 мг/кг (например, 0,1-10 мг/кг). Типичная суточная доза может составлять от примерно 1 мкг/кг до 100 мг/кг или более в зависимости от отмеченных выше факторов. Для повторных введений в течение нескольких дней или более продолжительного периода в зависимости от состояния лечение, как правило, должно продолжаться до достижения требуемого подавления имеющихся симптомов заболевания. В качестве одного примера, доза биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, может составлять от примерно 0,005 до примерно 10 мг/кг. В другом примере (но не ограничиваясь только указанным) доза на одно введение может составлять от примерно 1, примерно 5, примерно 10, примерно 50, примерно 100, примерно 200, примерно 350, примерно 500 мкг/кг веса тела, примерно 1, примерно 5, примерно 10, примерно 50, примерно 100, примерно 200, примерно 350, примерно 500 до примерно 1000 мг/кг веса тела или более, и находиться в любом указанном диапазоне. В качестве примеров (но не ограничиваясь только ими) указанного диапазона значений, можно вводить от примерно 5 до примерно 100 мг/кг веса тела, от примерно 5 мкг/кг веса тела до примерно 500 мг/кг веса тела и т.д. с учетом указанных выше уровней доз. Так, пациенту можно вводить одну или несколько доз, составляющих примерно 0,5, 2,0, 5,0 или 10 мг/кг (или любую их комбинацию). Указанные дозы можно вводить прерывисто, например, каждую неделю или каждые три недели (например, таким образом, чтобы пациент получал от примерно двух до примерно двадцати или, например, примерно шесть доз слитого белка). Можно вводить начальную более высокую ударную дозу, после которой применять одну или несколько более низких доз. Однако можно использовать другие схемы введения доз. Успех такой терапии легко оценивать с помощью общепринятых методик и анализов.
Биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, как правило, следует применять в количестве, эффективном для достижения поставленной цели. При применении для лечения или предупреждения болезненного состояния биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, или их фармацевтические композиции, вводят или применяют в терапевтически эффективном количестве. Определение терапевтически эффективного количества находится в компетенции специалистов в данной области, прежде всего в свете представленного подробного описания изобретения.
Для системного введения терапевтически эффективную дозу можно сначала определять с помощью анализов in vitro, например, анализов с использованием клеточных культур. Затем дозу можно включать в форму для изучения на животных моделях для достижения концентрации в кровотоке, находящейся в диапазоне, включающем значение IC50, определенное на клеточной культуре. Указанную информацию можно использовать для более точного определения доз, которые можно применять на людях.
Начальные дозы можно оценивать также, исходя из данных, полученных in vivo, например, на животных моделях, используя методики, хорошо известные в данной области. Обычный специалист в данной области легко может оптимизировать применение на людях на основе данных, полученных на животных.
Уровень доз и интервал можно регулировать индивидуально для получения уровней в плазме биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, которые являются достаточными для поддержания терапевтического действия. Обычные дозы, предназначенные для введения пациенту путем инъекции, составляют от примерно 0,1 до 50 мг/кг/день, как правило, от примерно 0,5 до 1 мг/кг/день. Для достижения терапевтически эффективных уровней в плазме можно вводить несколько доз каждый день. Уровни в плазме можно оценивать, например, с помощью ЖХВР.
В случаях местного применения или избирательного поглощения эффективная местная концентрация биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, может не соответствовать концентрации в плазме. Специалист в данной области может оптимизировать терапевтически эффективные местные дозы без чрезмерных экспериментов.
Применение в терапевтически эффективной дозе биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, которые представлены в настоящем описании, должно, как правило, обеспечивать терапевтическую пользу, не вызывая существенной токсичности. Токсичность и терапевтическую эффективность слитого белка можно определять с помощью стандартных фармацевтических процедур на культурах клеток или экспериментальных животных. Анализы на клеточных культурах или опыты на животных можно применять для определения значений LD50 (доза, смертельная для 50% популяции) и ED50 (доза, терапевтически эффективная для 50% популяции). Соотношение доз, характеризующих токсические и терапевтические действия, обозначают как терапевтический индекс, который можно выражать в виде соотношения LD50/ED50. Биспецифические антигенсвязывающие молекулы, имеющие высокие терапевтические индексы, являются предпочтительными. В одном из вариантов осуществления изобретения биспецифическая антигенсвязывающая молекула или антитело, предлагаемая/предлагаемое в изобретении, характеризуется высоким терапевтическим индексом. Данные, полученные в анализах с использованием клеточных культур и в опытах на животных, можно использовать для определения диапазона доз, которые можно применять на людях. Доза лежит предпочтительно в диапазоне концентраций в кровотоке, которые включают ED50, обладающих невысокой токсичностью или не обладающих токсичностью. Доза может варьироваться в указанном диапазоне в зависимости от различных факторов, например, от применяемой лекарственной формы, применяемого пути введения, состояния индивидуума и т.п. Точную препаративную форму, путь введения и дозу может выбирать индивидуально врач в зависимости от состояния пациента (см., например, Fingl и др. в: The Pharmacological Basis of Therapeutics, гл. 1, 1975, с. 1, публикация полностью включена в настоящее описание в качестве ссылки).
Лечащему врачу пациентов, которым вводят слитые белки, предлагаемые в изобретении, должно быть очевидно, как и когда заканчивать, прерывать или регулировать введение из-за токсичности, дисфункции органов и т.п. И, наоборот, лечащему врачу должно быть известно, как регулировать лечение в сторону применения более высоких доз, если клинический ответ является неадекватным (предотвращая токсичность). Величина вводимой дозы при лечении представляющего интерес нарушения должна варьироваться в зависимости от серьезности состояния, подлежащего лечению, пути введения и т.п. Серьезность состояния можно, например, оценивать среди прочего с помощью стандартных прогностических методов оценки. Кроме того, доза и предполагаемая частота введения дозы должны также варьироваться в зависимости от возраста, веса тела и ответа индивидуального пациента.
Другие средства и варианты лечения
Биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, можно вводить в комбинации с одним или несколькими другими агентами для осуществления терапии. Например, биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, можно вводить совместно по меньшей мере с одним дополнительным терапевтическим средством. Понятие «терапевтическое средство» включает любое средство, которое вводят для лечения симптома или заболевания у индивидуума, который нуждается в таком лечении. Указанное дополнительное терапевтическое средство может представлять собой любое действующее вещество, которое можно применять при конкретном показании, подлежащем лечению, предпочтительно с дополнительными видами активности, которые не оказывают отрицательное действие друг на друга. В некоторых вариантах осуществления изобретения дополнительное терапевтическое средство представляет собой другой противораковый агент, например, агент, разрушающий микротрубочки, антиметаболит, ингибитор топоизомеразы, ДНК-интеркалятор, алкилирующий агент, средство гормональной терапии, ингибитор киназ, антагонист рецептора, активатор апоптоза опухолевых клеток или антиангиогенный агент. В некоторых объектах изобретения дополнительное терапевтическое средство представляет собой иммуномодулятор, цитостатический агент, ингибитор клеточной адгезии, цитотоксический или цитостатический агент, активатор апоптоза клеток или агент, который повышает чувствительность клеток к индукторам апоптоза.
Указанные другие средства могут присутствовать в комбинации в количествах, эффективных для указанных целей. Эффективное количество указанных других средств зависит от количества применяемого слитого белка, типа нарушения или лечения, и других указанных выше факторов. Биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении, как правило, применяют в таких же дозах и с использованием указанных в настоящем описании путей введения, или в дозах, составляющих примерно от 1 до 99% от указанных в настоящем описании доз, или в любой дозе и с использованием любого пути введения, которые согласно эмпирическим/клиническим данным рассматриваются как приемлемые.
Отмеченные выше комбинированные терапии предусматривают совместное введение (когда два или большее количество терапевтических средств включают в одну и ту же или в отдельные композиции) и раздельное введение, в этом случае введение биспецифической антигенсвязывающей молекулы или антитела, предлагаемой/предлагаемого в изобретении, можно осуществлять до, одновременно и/или после введения дополнительного терапевтического средства и/или адъюванта.
Изделия
Другим объектом изобретения является изделие, которое содержит продукты, применяемые для лечения, предупреждения и/или диагностирования указанных выше нарушений. Изделие представляет собой контейнер и этикетку или листовку-вкладыш в упаковку, которые размещены на контейнере или прилагаются к нему. Приемлемыми контейнерами являются, например банки, пузырьки, шприцы, пакеты для внутривенного (IV) раствора и т.д. Контейнеры можно изготавливать из различных материалов, таких как стекло или пластмасса. Контейнер содержит композицию, которая сама по себе или в сочетании с другой композицией является эффективной для лечения, предупреждения и/или диагностирования состояния, и может иметь стерильный порт доступа (например, контейнер может представлять собой пакет для внутривенного раствора или пузырек, снабженный пробкой, которую можно прокалывать с помощью иглы для подкожных инъекций). По меньшей мере одно действующее вещество в композиции представляет собой биспецифическую антигенсвязывающую молекулу или антитело, предлагаемую/предлагаемое в изобретении.
На этикетке или листовке-вкладыше в упаковку указано, что композицию применяют для лечения выбранного состояния. Кроме того, изделие может включать (а) первый контейнер с находящейся в нем композицией, где композиция содержит биспецифическую антигенсвязывающую молекулу, предлагаемую в изобретении; и (б) второй контейнер с находящейся в нем композицией, где композиция содержит дополнительное цитотоксическое или иное терапевтическое средство. Согласно этому варианту осуществления изобретения изделие может содержать листовку-вкладыш в упаковку, которая содержит информацию о том, что композиции можно использовать для лечения конкретного состояния.
В альтернативном или дополнительном варианте изделие может дополнительно включать второй (или третий) контейнер с фармацевтически приемлемым буфером, таким как бактериостатическая вода для инъекций (БСВИ), забуференный фосфатом физиологический раствор, раствор Рингера и раствор декстрозы. Кроме того, оно может включать другие материалы, необходимые с коммерческой точки зрения и с точки зрения потребителя, в частности, другие буферы, разбавители, фильтры, иглы и шприцы.
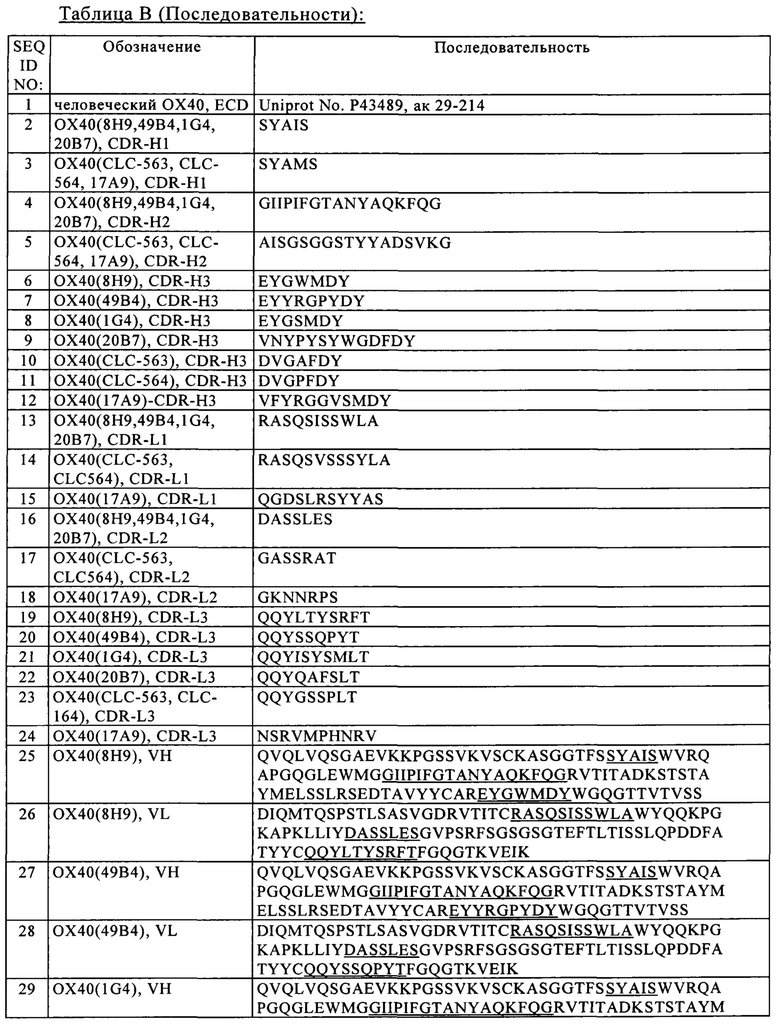






Все нуклеотидные последовательности представлены без соответствующих последовательностей стоп-кодона.
Ниже перечислены специфические варианты осуществления изобретения:
1. Биспецифическая антигенсвязывающая молекула, которая содержит
(а) по меньшей мере один фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
2. Биспецифическая антигенсвязывающая молекула по п. 1, где представитель семейства костимуляторных TNF-рецепторов выбран из группы, состоящей из OX40 и 4-1ВВ.
3. Биспецифическая антигенсвязывающая молекула по п. 1 или п. 2, где представитель семейства костимуляторных TNF-рецепторов представляет собой OX40.
4. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-3, в которой фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который содержит аминокислотную последовательность SEQ ID NO: 1.
5. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-4, которая содержит по меньшей мере один фрагмент, обладающий способностью специфически связываться с OX40, где указанный фрагмент содержит VH-домен, содержащий
(I) CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 2 и SEQ ID NO: 3,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 4 и SEQ ID NO: 5, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 и SEQ ID NO: 12, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 13, SEQ ID NO: 14 и SEQ ID NO: 15,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 16, SEQ ID NO: 17 и SEQ ID NO: 18, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23 и SEQ ID NO: 24.
6. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-5, в которой фрагмент, обладающий способностью связываться с OX40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 и SEQ ID NO: 37, и вариабельную область легкой цепи VL, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит из SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 и SEQ ID NO: 38.
7. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-5, в которой фрагмент, обладающий способностью специфически связываться с OX40, содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26.
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
8. Биспецифическая антигенсвязывающая молекула по п. 1 или п. 2, где представитель семейства костимуляторных TNF-рецепторов представляет собой 4-1ВВ.
9. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1, 2 или 8, в которой фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, связывается с полипептидом, который содержит аминокислотную последовательность SEQ ID NO: 39.
10. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1, 2, 8 или 9, которая содержит по меньшей мере один фрагмент, обладающий способностью специфически связываться с 4-1ВВ, где указанный фрагмент содержит VH-домен, содержащий
(I) a CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 40 и SEQ ID NO: 41,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 42 и SEQ ID NO: 43, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 44, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 47 и SEQ ID NO: 48,
и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 49 и SEQ ID NO: 50,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 51 и SEQ ID NO: 52, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 53, SEQ ID NO: 54, SEQ ID NO: 55, SEQ ID NO: 56 и SEQ ID NO: 57.
11. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1, 2, 8, 9 или 10, в которой фрагмент, обладающий способностью связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 и SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности, выбранной из группы, которая состоит SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 и SEQ ID NO: 67.
12. Биспецифическая антигенсвязывающая молекула по п.п. 1, 2, и 8-11, в которой фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
13. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-12, где антиген клетки-мишени выбран из группы, включающей фибробласт-активирующий белок (FAP), ассоциированный с меланомой хондроитинсульфат-протеогликан (MCSP), рецептор эпидермального фактора роста (EGFR), карциноэмбриональный антиген (СЕА), CD19, CD20 и CD33.
14. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-13, где антиген клетки-мишени представляет собой фибробласт-активирующий белок (FAP).
15. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-13, в которой фрагмент, обладающий способностью специфически связываться с FAP, содержит VH-домен, содержащий
(I) a CDR-H1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 68 и SEQ ID NO: 69,
(II) CDR-H2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 70 и SEQ ID NO: 71, и
(III) CDR-H3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 72 и SEQ ID NO: 73, и VL-домен, содержащий
(IV) CDR-L1, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 74 и SEQ ID NO: 75,
(V) CDR-L2, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 76 и SEQ ID NO: 77, и
(VI) CDR-L3, который содержит аминокислотную последовательность, выбранную из группы, которая состоит из SEQ ID NO: 78 и SEQ ID NO: 79.
16. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-7, в которой
(I) фрагмент, обладающий способностью специфически связываться с OX40, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 25, SEQ ID NO: 27, SEQ ID NO: 29, SEQ ID NO: 31, SEQ ID NO: 33, SEQ ID NO: 35 или SEQ ID NO: 37, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 26, SEQ ID NO: 28, SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 36 или SEQ ID NO: 38, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
17. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1, 2 или 8-12, в которой
(I) фрагмент, обладающий способностью специфически связываться с 4-1ВВ, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 58, SEQ ID NO: 60, SEQ ID NO: 62, SEQ ID NO: 64 или SEQ ID NO: 66, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 59, SEQ ID NO: 61, SEQ ID NO: 63, SEQ ID NO: 65 или SEQ ID NO: 67, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 80 или SEQ ID NO: 82, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере примерно на 95%, 96%, 97%, 98%, 99% или 100% идентична аминокислотной последовательности SEQ ID NO: 81 или SEQ ID NO: 83.
18. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-17, где указанная молекула содержит
(а) первый Fab-фрагмент, обладающий способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) второй Fab-фрагмент, обладающий способностью специфически связываться с антигеном клетки-мишени, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
19. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-18, в которой Fc-домен представляет собой Fc-домен человеческого IgG, в частности, Fc-домен IgG1 или Fc-домен IgG4.
20. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-19, в которой Fc-домен содержит одну или несколько аминокислотную(ых) замену(н), которая(ые) снижает(ют) аффинность связывания антитела с Fc-рецептором и/или эффекторную функцию.
21. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-20, в которой Fc-домен представляет собой Fc-домен человеческого IgG1-подкласса с аминокислотными мутациями L234A, L235A и P329G (нумерация согласно EU-индексу Кэбота).
22. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-21, в которой Fc-домен содержит модификацию, способствующую ассоциации первой и второй субъединиц Fc-домена.
23. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-22, в которой первая субъединица Fc-домена содержит «выступы», а вторая субъединица Fc-домена содержит «впадины» в соответствии с технологией «knob-into-hole».
24. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-23, в которой первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (нумерация согласно EU-индексу Кэбота), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация согласно EU-индексу Кэбота).
25. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-24, содержащая
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) два фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени,
и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
26. Биспецифическая антигенсвязывающая молекула по п. 25, где биспецифическая антигенсвязывающая молекула является двухвалентной как в отношении представителя семейства костимуляторных TNF-рецепторов, так и в отношении антигена клетки-мишени.
27. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-24, которая содержит
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(в) два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, в которой каждый из указанных дополнительных Fab-фрагментов соединен через пептидный линкер с С-концами тяжелых цепей, указанных в подпункте (а).
28. Биспецифическая антигенсвязывающая молекула по п. 27, в которой два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кроссовер-Fab-фрагменты, в которых вариабельные домены VL и VH замены друг на друга и каждая из VL-CH-цепей соединена через пептидный линкер с С-концами тяжелых цепей, указанных в подпункте (а).
29. Биспецифическая антигенсвязывающая молекула по п. 27 или п. 28, в которой два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, представляют собой два Fab-фрагмента, которые обладают способностью специфически связываться с OX40 или 4-1ВВ, а два дополнительных Fab-фрагмента, которые обладают способностью специфически связываться с антигеном клетки-мишени, представляют собой кроссовер-Fab-фрагменты, которые обладают способностью специфически связываться с FAP.
30. Биспецифическая антигенсвязывающая молекула по одному из предыдущих пунктов, содержащая
(а) два фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов,
(б) один фрагмент, который обладает способностью специфически связываться с антигеном клетки-мишени,
и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации.
31. Биспецифическая антигенсвязывающая молекула по п. 30, где биспецифическая антигенсвязывающая молекула является двухвалентной в отношении представителя семейства костимуляторных TNF-рецепторов и одновалентной в отношении антигена клетки-мишени.
32. Биспецифическая антигенсвязывающая молекула по одному из предыдущих пунктов, содержащая
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с представителем семейства костимуляторных TNF-рецепторов, и Fc-домен, и
(б) VH- и VL-домены, обладающие способностью специфически связываться с антигеном клетки-мишени, где VH-домен соединен через пептидный линкер с С-концом одной из тяжелых цепей и где VL-домен соединен через пептидный линкер с С-концом второй тяжелой цепи.
33. Полинуклеотид, кодирующий биспецифическую антигенсвязывающую молекулу по одному из п.п. 1-32.
34. Антитело, которое специфически связывается с OX40, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34,
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36, или
(VII) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 37, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 38.
35. Антитело, которое специфически связывается с 4-1ВВ, где указанное антитело содержит
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 58, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 59,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 60, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 61,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 62, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 63,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 65, или
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 66, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 67.
36. Полинуклеотид, кодирующий антитело по п. 34 или п. 35.
37. Фармацевтическая композиция, содержащая биспецифическую антигенсвязывающую молекулу по одному из п.п. 1-32 или антитело по п. 34 или п. 35 и по меньшей мере один фармацевтически приемлемый эксципиент.
38. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-32, или антитело по п. 34 или п. 35, или фармацевтическая композиция по п. 37, предназначенные для применения в качестве лекарственного средства.
39. Биспецифическая антигенсвязывающая молекула по одному из п.п. 1-32, или антитело по п. 34 или п. 35, или фармацевтическая композиция по п. 37, предназначенные для применения для
(I) стимуляции Т-клеточного ответа,
(II) поддержания выживаемости активированных Т-клеток,
(III) лечения инфекций,
(IV) лечения рака,
(V) замедления прогрессирования рака или
(VI) пролонгирования времени жизни пациента, страдающего раком.
40. Биспецифическая антигенсвязывающая молекула по одному п.п. 1-32 или антитело по п. 34 или п. 35, или фармацевтическая композиция по п. 37, предназначенные для применения для лечения рака.
41. Биспецифическая антигенсвязывающая молекула по одному п.п. 1-32 или антитело по п. 34 или п. 35, или фармацевтическая композиция по п. 37, предназначенные для применения для лечения рака, в котором биспецифическую антигенсвязывающую молекулу вводят в комбинации с химиотерапевтическим средством, лучевой терапией и/или другими агентами, предназначенными для применения в противораковой иммунотерапии.
42. Способ ингибирования роста опухолевых клеток у индивидуума, включающий введение индивидууму в эффективном количестве биспецифической антигенсвязывающей молекулы по одному из п.п. 1-32 или антитела по п. 34 или п. 35, или фармацевтической композиции по п. 37 для ингибирования роста опухолевых клеток.
Примеры
Ниже представлены примеры способов и композиций, предлагаемых в изобретении. Как должно быть очевидно, можно воплощать на практике различные другие варианты осуществления изобретения в целом с учетом представленного выше описания изобретения.
Методы рекомбинантной ДНК
Для манипуляций с ДНК использовали стандартные методы, описанные у Sambrook J. и др., Molecular cloning: A laboratory manual; изд-во Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, 1989. Реагенты для молекулярной биологии применяли согласно инструкциям производителей. Общую информацию, касающуюся нуклеотидных последовательностей легких и тяжелых цепей человеческих иммуноглобулинов, см. у: Kabat E.A. и др., Sequences of Proteins of Immunological Interest, 5-ое изд., изд-во NIH, публикация N 91-3242, 1991.
Секвенирование ДНК
Последовательности ДНК определяли путем секвенирования двух цепей.
Синтез генов
Требуемые сегменты генов либо создавали с помощью ПЦР с использованием соответствующих матриц, либо синтезировали на фирме Geneart AG (Регенсбург, Германия) из синтетических олигонуклеотидов и ПЦР-продуктов посредством автоматического синтеза генов. В тех случаях, когда точная генная последовательность не была доступна, создавали олигонуклеотидные праймеры на основе последовательностей ближайших гомологов и гены выделяли с помощью ОТ-ПЦР из РНК, полученной из соответствующей ткани. Сегменты генов, фланкированные единичными сайтами, распознаваемыми рестрикционными эндонуклеазами, клонировали в стандартных клонирующих/секвенирующих векторах. Плазмидную ДНК очищали из трансформированных бактерий и определяли концентрацию с помощью УФ-спектроскопии. Последовательность ДНК субклонированных фрагментов генов подтверждали ДНК-секвенированием. Создавали сегменты генов с требуемыми сайтами рестрикции, позволяющими субклонировать их в соответствующих экспрессионных векторах. Все конструкции создавали с 5'-концевой последовательностью ДНК, кодирующей лидерный пептид, который направляет секрецию белков в эукариотических клетках.
Очистка белков
Белки очищали из профильтрованных супернатантов клеточных культур согласно стандартному протоколу. В целом, метод состоял в следующем: антитела вносили в колонку с белок А-Сефарозой (фирма GE Healthcare, Швеция) и промывали с помощью ЗФР. Элюцию белков осуществляли при значении рН 2,8, после чего немедленно проводили нейтрализацию образца. Агрегированные белки отделяли от мономерных антител с помощью гель-фильтрации (Супердекс 200, фирма GE Healthcare) в ЗФР или в буфере, содержащем 20 мМ гистидин, 150 мМ NaCl, рН 6,0. Фракции мономерных антител объединяли, концентрировали (при необходимости), используя, например, центрифужный концентратор MILLIPORE Amicon Ultra (30 MWCO), замораживали и хранили при температуре -20°С или -80°С. Часть образцов использовали для последующего аналитического анализа белков и аналитической характеризации, например, с помощью ДСН-ПААГ, гель-фильтрации (SEC) или масс-спектрометрии.
ДСН-ПААГ
Гелевую систему NuPAGE® Pre-Cast (фирма Invitrogen) применяли согласно инструкции производителя. В частности, применяли 10% или 4-12% гели NuPAGE® Novex® Bis-TRIS Pre-Cast (рН 6,4) и подвижный буфер NuPAGE® MES (восстановленные гели с антиоксидантной добавкой для подвижного буфера NuPAGE®) или MOPS (невосстановленные гели).
Аналитическая гель-фильтрация
Гель-фильтрацию (SEC) для определения агрегированного и олигомерного состояния антител осуществляли с помощью ЖХВР-хроматографии. В целом, метод состоял в следующем: очищенные на белке А антитела вносили в колонку Tosoh TSKgel G3000SW в 300 мМ NaCl, 50 мМ KH2PO4/K2HPO4, рН 7,5, в ЖХВР-системе Agilent 1100 или в колонку Супердекс 200 (фирма GE Healthcare) в 2 × ЗФР в ЖХВР-системе Dionex. Элюированные белки оценивали количественно с помощью УФ-абсорбции и интегрирования площадей пиков. В качестве стандарта служил BioRad Gel Filtration Standard 151-1901.
Масс-спектрометрия
В этом разделе описана характеризация мультиспецифических антител с VH/VL-обменом или CH/CL-обменом (CrossMab) в отношении их правильной сборки. Ожидаемые первичные структуры анализировали с помощью масс-спектрометрии с использованием ионизация электроспреем (ESI-MC) дегликозилированных интактных CrossMab и дегликозилированных расщепленных плазмином или в альтернативном варианте дегликозилированных/ограничено расщепленных LysC CrossMab.
CrossMab дегликозилировали с помощью N-гликозидазы F в фосфатном или Трис-буфере при 37°С в течение вплоть до 17 ч с использованием белка в концентрации 1 мг/мл. Расщепление плазмином или ограниченное расщепление LysC (фирма Roche) осуществляли с использованием 100 мкг дегликозилированных VH/VL-CrossMab в Трис-буфере, рН 8 при комнатной температуре в течение 120 ч и при 37°С в течение 40 мин соответственно. Перед осуществлением масс-спектрометрии образцы обессоливали посредством ЖХВР на колонке Сефадекс G25 (фирма GE Healthcare). Общую массу определяли с помощью ESI-MC с использованием МС-системы maXis 4G UHR-QTOF (фирма Bruker Daltonik), снабженной источником TriVersa NanoMate (фирма Advion).
Пример 1
Создание антител к OX40 и применяемых в качестве «инструментов» связывающих агентов
1.1 Получение, очистка и характеризация антигенов и скрининг «инструментов» для создания новых ОХ40-связывающих агентов с помощью фагового дисплея
Последовательности ДНК, кодирующие эктодомены OX40 человека, мышей или обезьян циномолгус (таблица 1), субклонировали в рамке считывания с СН2- и СН3-доменами тяжелой цепи человеческого IgG1 на цепи с «выступом» (Merchant и др., 1998). Сайт расщепления протеазой AcTEV интродуцировали между экто доменом антигена и Fc человеческого IgG1. Avi-метку для направленного биотинилирования интродуцировали на С-конец конструкции антиген-Fc с «выступом». Комбинация цепи антиген-Fc с «выступом», которая содержит мутации S354C/T366W, с Fc-цепью с «впадиной», которая содержит мутации Y349C/T366S/L368A/Y407V, позволяет создавать гетеродимер, который включает единичную копию содержащей эктодомен OX40 цепи, получая тем самым мономерную форму сцепленного с Fc антигена (фиг. 1А). В таблице 1 представлены аминокислотные последовательности различных эктодоменов OX40. В таблице 2 представлены последовательности кДНК и аминокислотные последовательности мономерных содержащих конструкцию слитых молекул антиген-Fc(kih), представленных на фиг. 1.



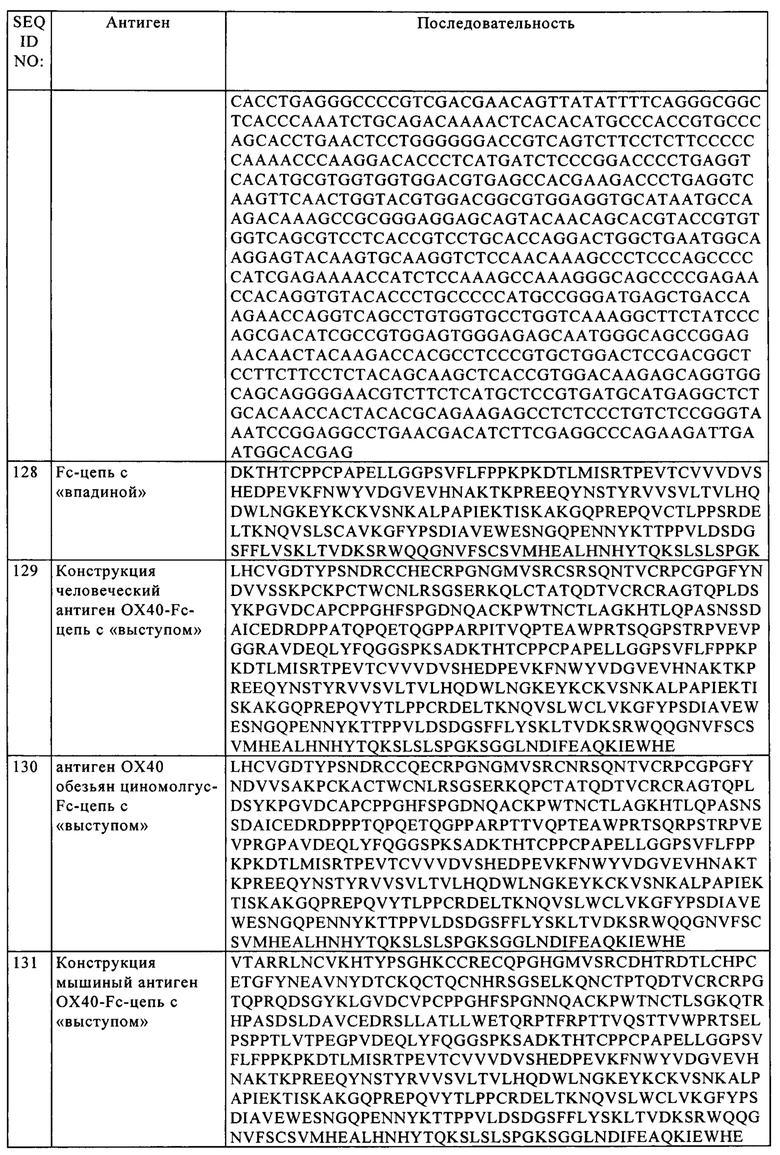
Все последовательности, кодирующие слияния OX40-Fc, клонировали в плазмидном векторе, в котором экспрессия вставки контролируется промотором MPSV и который содержит синтетическую сигнальную полиА-последовательность, локализованную на 3'-конце CDS. Кроме того, вектор содержал последовательность OriP EBV для поддержки эписомальной формы плазмиды.
Для получения биотинилированных мономерных слитых молекул антиген/Fc растущие в суспензии клетки HEK293 EBNA на экспоненциальной фазе роста совместно трансфектировали тремя векторами, кодирующими два компонента слитого белка (цепи с «выступом» и «впадиной», а также BirA, фермент, необходимый для реакции биотинилирования. Соответствующие векторы применяли в соотношении 2:1:0,05 («BCD антигена-AcTEV-Fc с «выступом» : «Fc с «впадиной» : «BirA»).
Для получения белка в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD CHO. Экспрессионные векторы ресуспендировали в 20 мл CD CHO, содержащей 200 мкг векторной ДНК. После добавления 540 мкл полиэтиленимина (ПЭИ) раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Секретированные белки очищали из супернатантов клеточных культур с помощью аффинной хроматографии с использованием белка А с последующей гель-фильтрацией. Для осуществления аффинной хроматографии супернатант вносили в колонку HiTrap ProteinA HP (CV=5 мл, фирма GE Healthcare), уравновешенную 40 мл буфера, содержащего 20 мМ фосфат натрия, 20 мМ цитрат натрия (рН 7,5). Несвязанный белок удаляли путем промывки буфером, содержащим 20 мМ фосфат натрия, 20 мМ цитрат натрия и 0,5М хлорид натрия (рН 7,5), используя его в объеме, кратном по меньшей 10 объемам колонки. Связанный белок элюировали, используя линейный градиент хлорида натрия (от 0 до 500 мМ), созданный с помощью раствора, содержащего 20 мМ цитрат натрия, 0,01 об. % Твин-20, рН 3,0, который применяли в объеме, кратном 20 объемам колонки. Затем колонку промывали с помощью раствора, содержащего 20 мМ цитрат натрия, 500 мМ хлорид натрия и 0,01 об. % Твин-20, рН 3,0, который применяли в объеме, кратном 10 объемам колонки.
Значение рН собранных фракций регулировали, добавляя 1/40 (об./об.) 2М Трис, рН 8,0. Белок концентрировали и фильтровали перед внесением в колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 2 мМ MOPS, 150 мМ хлорид натрия, 0,02% (мас./об.) азида натрия, рН 7,4.
1.2 Отбор ОХ40-специфических антител 8Н9, 20В7, 49В4, 1G4, CLC-563, CLC-564 и 17А9 из родовых библиотек Fab и библиотек клонов с общей легкой цепью
Антитела к OX40 отбирали из трех различных родовых фаговых дисплейных библиотек: DP88-4 (клоны 20В7, 8H91G4 и 49В4), библиотека клонов с общей легкой цепью Vk3_20/VH3_23 (клоны CLC-563 и CLC-564) и лямбда-DP47 (клон 17А9).
Библиотеку DP88-4 конструировали на основе генов человеческой зародышевой линии, используя V-домен со спаренными Vk1_5 (легкая каппа-цепь) и VH1_69 (тяжелая цепь), содержащим рандомизированный участок последовательности в CDR3 легкой цепи (L3, 3 различные длины) и CDR3 тяжелой цепи (Н3, 3 различные длины). Создание библиотек осуществляли путем сборки 3 амплифицированных с помощью ПЦР фрагментов, применяя ПЦР для «сращивания (генома) перекрывающимися расширениями» (SOE). Фрагмент 1 содержал 5'-конец гена антитела, включая рандомизированный L3, фрагмент 2 представлял собой центральный константный фрагмент, простирающийся от L3 до Н3, а фрагмент 3 содержал рандомизированный Н3 и 3'-область гена антитела. Следующие комбинации праймеров применяли для создания указанных фрагментов библиотек для библиотеки DP88-4: фрагмент 1 (прямой праймер LMB3 в комбинации с обратными праймерами Vk1_5_L3r_S или Vk1_5_L3r_SY или Vk1_5_L3r_SPY), фрагмент 2 (прямой праймер RJH31 в комбинации с обратными праймер RJH32) и фрагмент 3 (прямые праймеры DP88-v4-4 или DP88-v4-6, или DP88-v4-8 в комбинации с обратным праймером fdseqlong) соответственно. Параметры ПЦР для получения фрагментов библиотек были следующими: 5 мин начальная денатурация при 94°С, 25 циклов по 1 мин при 94°С, 1 мин при 58°С, 1 мин при 72°С и финальное удлинение в течение 10 мин при 72°С. Для ПЦР-сборки при использовании эквимолярных соотношений очищенных на геле единичных фрагментов в качестве матрицы применяли следующие параметры: 3 мин начальная денатурация при 94°С и 5 циклов по 30 с при 94°С, 1 мин при 58°С, 2 мин при 72°С. На этой стадии добавляли внешние праймеры (LMB3 и fdseqlong) и осуществляли 20 дополнительных циклов перед финальным удлинением в течение 10 мин при 72°С. После сборки в достаточном количестве полноразмерных рандомизированных конструкций Fab их расщепляли с помощью NcoI/NheI и встраивали путем лигирования в обработанный аналогичным образом акцепторный фагмидный вектор. Очищенные полученные лигированием конструкции применяли для ~60 трансформаций электрокомпетентных клеток Е.coli TG1. Фагмидные частицы экспонирующие библиотеку Fab, выделяли («спасали») и очищали с помощью ПЭГ/NaCl для использования для селекции. Эти стадии конструирования библиотек повторяли три раза с получением конечной библиотеки размером 4,4×109. Проценты функциональных клонов, определенные на основе детекции С-концевой метки методом дот-блоттинга, составляли 92,6% для легкой цепи и 93,7% для тяжелой цепи соответственно.
Библиотеку с общей легкой цепью Vk3_20/VH3_23 конструировали на основе генов человеческой зародышевой линии, используя V-домен со спаренными Vk3_20 (легкая каппа-цепь) и VH3_23 (тяжелая цепь), содержащим общую константную нерандомизированную легкую цепь Vk3_20 и рандомизированный участок последовательности в CDR3 тяжелой цепи (Н3, 3 различные длины). Создание библиотек осуществляли путем сборки 2 амплифицированных с помощью ПЦР фрагментов, применяя ПЦР для «сращивания (генома) перекрывающимися расширениями» (SOE). Фрагмент 1 представлял собой константный фрагмент, простирающийся от L3 до Н3, а фрагмент 2 содержал рандомизированный Н3 и 3'-область гена антитела. Следующие комбинации праймеров применяли для создания указанных фрагментов библиотеки для библиотеки с общей легкой цепью Vk3_20/VH3_23: фрагмент 1 (прямой праймер MS64 в комбинации с обратным праймером DP47CDR3_ba (mod.)) и фрагмент 2 (прямые праймеры DP47-v4-4, DP47-v4-6, DP47-v4-8 в комбинации с обратным праймером fdseqlong) соответственно. Параметры ПЦР для получения фрагментов библиотек были следующими: 5 мин начальная денатурация при 94°С, 25 циклов по 1 мин при 94°С, 1 мин при 58°С, 1 мин при 72°С и финальное удлинение в течение 10 мин при 72°С. Для ПЦР-сборки при использовании эквимолярных соотношений очищенных на геле единичных фрагментов в качестве матрицы применяли следующие параметры: 3 мин начальная денатурация при 94°С и 5 циклов по 30 с при 94°С, 1 мин при 58°С, 2 мин при 72°С. На этой стадии добавляли внешние праймеры (MS64 и fdseqlong) и осуществляли 18 дополнительных циклов перед финальным удлинением в течение 10 мин при 72°С. После сборки в достаточном количестве полноразмерных рандомизированных конструкций VH их расщепляли с помощью MunI/NotI и встраивали путем лигирования в обработанный аналогичным образом акцепторный фагмидный вектор. Очищенные полученные лигированием конструкции применяли для ~60 трансформаций электрокомпетентных клеток Е.coli TG1. Фагмидные частицы экспонирующие библиотеку Fab, «спасали» и очищали с помощью ПЭГ/NaCl для использования для селекции. Получали конечную библиотеку размером 3,75×109. Проценты функциональных клонов, определенные на основе детекции С-концевой метки методом дот-блоттинга, составляли 98,9% для легкой цепи и 89,5% для тяжелой цепи соответственно.
Библиотеку лямбда-DP47 конструировали на основе генов человеческой зародышевой линии, используя V-домен со спаренными: V13_19 (легкая лямбда-цепь) и VH3_23 (тяжелая цепь). Библиотеку рандомизировали в CDR3 легкой цепи (L3) и CDR3 тяжелой цепи (Н3) и собирали из 3 фрагментов, применяя ПЦР для «сращивания (генома) перекрывающимися расширениями» (SOE). Фрагмент 1 содержал 5'-конец гена антитела, включая рандомизированный L3, фрагмент 2 представлял собой центральный константный фрагмент, простирающийся от конца L3 до начала H3, а фрагмент 3 содержал рандомизированный H3 и 3'-область Fab-фрагмента. Следующие комбинации праймеров применяли для создания указанных фрагментов библиотеки для библиотеки: фрагмент 1 (LMB3-V1_3_19_L3r_V/V1_3_19_L3r_HV/V1_3_19_L3r_HLV), фрагмент 2 (RJH80 - DP47CDR3_ba (mod)) и фрагмент 3 (DP47-v4-4/DP47-v4-6/DP47-v4-8 - fdseqlong). Параметры ПЦР для получения фрагментов библиотек были следующими: 5 мин начальная денатурация при 94°С, 25 циклов по 60 с при 94°С, 60 с при 55°С, 60 с при 72°С и финальное удлинение в течение 10 мин при 72°С. Для ПЦР-сборки при использовании в качестве матрицы 3 фрагментов в эквимолярных соотношениях применяли следующие параметры: 3 мин начальная денатурация при 94°С и 5 циклов по 60 с при 94°С, 60 с при 55°С, 120 с при 72°С. На этой стадии добавляли внешние праймеры и осуществляли 20 дополнительных циклов перед финальным удлинением в течение 10 мин при 72°С. После сборки в достаточном количестве полноразмерных рандомизированных Fab-фрагментов их расщепляли с помощью NcoI/NheI параллельно с аналогичной обработкой акцепторного фагмидного вектора. 15 мкг вставки в виде библиотеки Fab встраивали путем лигирования в 13,3 мкг фагмидного вектора. Очищенные полученные лигированием конструкции применяли для 60 трансформаций с получением 1,5×109 трансформантов. Фагмидные частицы, экспонирующие библиотеку Fab, «спасали» и очищали с помощью ПЭГ/NaCl для использования для селекции.
Человеческий OX40 (CD134) в качестве антигена для селекции методом фагового дисплея кратковременно экспрессировали в виде N-концевого слияния с мономерным Fc в клетках HEK EBNA и осуществляли in vivo сайтспецифическое биотинилирование посредством совместной экспрессии биотин-лигазы BirA в распознаваемой последовательности с avi-меткой, локализованной на С-конце Fc-области, несущей цепь рецептора (Fc-цепь с «выступом»).
Раунды селекции (биопэннинг) осуществляли в растворе согласно следующей схеме:
1. преклиринг ~ 1012 фагмидных частиц на планшетах maxisorp, сенсибилизированных 10 мкг/мл нерелевантного человеческого IgG для истощения библиотек антител, распознающих Fc-область антигена,
2. инкубация несвязывающихся фагмидных частиц с 100 нМ биотинилированным человеческим OX40 в течение 0,5 ч в присутствии 100 нМ нерелевантной небиотинилированной конструкции Fc «выступ»-во-«впадину» для дальнейшего истощения связывающих Fc агентов в общем объеме 1 мл,
3. захват биотинилированного huOX40 и присоединение специфически связывающего фага путем переноса в 4 лунки предварительно сенсибилизированного нейтравидином титрационного микропланшета в течение 10 мин (в раундах 1 и 3),
4. промывка соответствующих лунок с использованием 5 × ЗФР/Твин20 и 5 × ЗФР,
5. элюция фаговых частиц путем добавления 250 мкл 100 мМ ТЭА (триэтиламин) на лунку в течение 10 мин и нейтрализация путем добавления 500 мкл 1М Трис/HCl, рН 7,4 к объединенным элюатам из 4 лунок,
6. пост-клиринг нейтрализованных элюатов путем инкубации на предварительно сенсибилизированном нейтравидином титрационном микропланшете с 100 нМ захваченной биотином конструкции Fc «выступ»-во-«впадину» для окончательного удаления связывающих Fc агентов,
7. повторное заражение на log-фазе клеток Е.coli TG1 супернатантом элюированных фаговых частиц, заражение хелперным фагом VCSM13, инкубация при встряхивании при 30°С в течение ночи и последующее осаждение с помощью ПЭГ/NaCl фагмидных частиц для применения в следующем раунде селекции.
Селекции осуществляли с использованием 3 или 4 раундов, применяя постоянную концентрацию антигена 100 нМ. Для повышения вероятности того, что связывающие агенты будут обладать перекрестной реактивность не только с OX40 обезьян циномолгус, но и с мышиным OX40, в некоторых раундах селекции вместо человеческого OX40 в качестве мишени применяли мышиный. В раундах 2 и 4 для того, чтобы избегать обогащения связывающими нейтравидин агентами, осуществляли захват комплексов антиген : фаг, добавляя 5,4×107 покрытых стрептавидином магнитных гранул. Специфические связывающие агенты идентифицировали с помощью ELISA следующим образом: 100 мкл 25 нМ биотинилированного человеческого OX40 и 10 мкг/мл человеческого IgG применяли для сенсибилизации нейтравидиновых планшетов и maxisorp-планшетов соответственно. Добавляли содержащие Fab бактериальные супернатанты и связывание Fab оценивали посредством их Flag-меток, использую вторичное антитело к Flag/HRP. Клоны, дающие сигналы на человеческий OX40 и являющиеся отрицательными в отношении человеческого IgG, включали в шорт-лист для дополнительных анализов и тестировали также аналогичным образом в отношении OX40 обезьян циномолгус и мышей. Их экспрессировали в бактериях в объеме культуры 0,5 л, очищали на основе аффинности и дополнительно характеризовали с помощью SPR-анализа, используя биосенсор ProteOn XPR36 фирмы BioRad.
В таблице 3 представлена последовательность родовой фаговой дисплейной библиотеки антител (DP88-4), в таблице 4 представлены последовательности кДНК и аминокислотные последовательности библиотечной матрицы зародышевой линии DP88-4, а в таблице 5 представлены праймерные последовательности, применяемые для создания матрицы зародышевой линии DP88-4.





В таблице 6 представлена последовательность родовой фаговой дисплейной библиотеки антител с общей легкой цепью (Vk3_20/VH3_23), в таблице 7 представлены последовательности кДНК и аминокислотные последовательности матрицы библиотеки зародышевой линии с общей легкой цепью (Vk3_20/VH3_23), а в таблице 8 представлены праймерные последовательности, применяемые для создания библиотеки с общей легкой цепью (Vk3_20/VH3_23).





1: G/D=20%, E/V/S=10%, A/P/R/L/T/Y=5%; 2: G/Y/S=15%, A/D/T/R/P/L/V/N/W/F/I/E=4,6%; 3: G/A/Y=20%, P/W/S/D/T=8%; 4: F=46%, L/M=15%, G/I/Y=8%; 5: K=70%, R=30%.
В таблице 9 представлена последовательность родовой фаговой дисплейной библиотеки лямбда-DP47 (V13_19/VH3_23), применяемой в качестве матрицы для ПЦР, в таблице 10 представлены последовательности кДНК и аминокислотные последовательности матрицы библиотеки зародышевой линии лямбда-DP47 (V13_19/VH3_23), а в таблице 11 представлены праймерные последовательности, применяемые для создания библиотеки лямбда-DP47 (V13_19/VH3_23).





Дополнительные праймеры, применяемые для конструирования библиотеки лямбда-DP47, т.е. DP47CDR3_ba (mod.), DP47-v4-4, DP47-v4-6, DP47-v4-8 и fdseqlong, идентичны праймерам, которые применяли для конструирования библиотеки общей легкой цепи (Vk3_20/VH3_23), и они уже перечислены в таблице 8.
С помощью описанной выше процедуры клоны 8Н9, 20В7, 49В4, 1G4, CLC-563, CLC-564 и 17А9 идентифицированы в качестве специфических связывающих человеческий OX40 агентов. Последовательности кДНК их вариабельных областей представлены ниже в таблице 12, соответствующие аминокислотные последовательности представлены в таблице В.


1.3 Получение, очистка и характеризация антител к OX40 в формате IgG1 P329G LALA
Последовательности ДНК вариабельных областей тяжелых и легких цепей выбранных анти-ОХ40-связывающих агентов субклонировали в рамке считывания либо с константной тяжелой цепью, либо с константной легкой цепью человеческого IgG1. Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей с «выступом» и «впадиной» для элиминации связывания с рецепторами Fc-гамма согласно методу, описанному в международной заявке на патент WO 2012/130831 А1.
Последовательности кДНК и аминокислотные последовательности анти-OX40 клонов, представлены в таблице 13. Все последовательности, кодирующие слияние анти-ОХ40-Fc клонировали в плазмидном векторе, в котором экспрессия вставки контролируется промотором MPSV и который содержит синтетическую сигнальную полиА-последовательность, локализованную на 3'-конце CDS. Кроме того, вектор содержал последовательность OriP EBV для поддержки эписомальной формы плазмиды.






Антитела к OX40 получали путем котрансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих, используя полиэтиленимин. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1 («вектор тяжелой цепи» : «вектор легкой цепи»),
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн. клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD CHO. Экспрессионные векторы (200 мкг общей ДНК) смешивали с 20 мл среды CD CHO. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку молекул антител из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слияний антиген-Fc.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, pH 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу антител анализировали с помощью капиллярного электрофореза в присутствии додецилсульфата натрия (КЭ-ДСН) в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, pH 6,7, при 25°С.
В таблице 14 обобщены данные о выходе и конечном содержании антител к OX40 в формате P329G LALA IgG1.
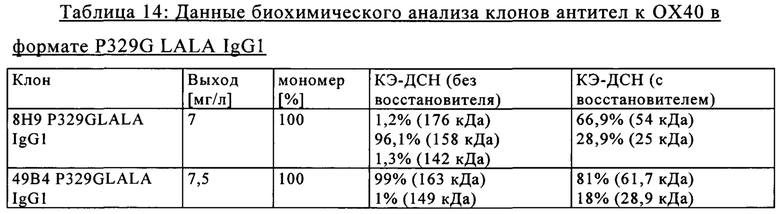
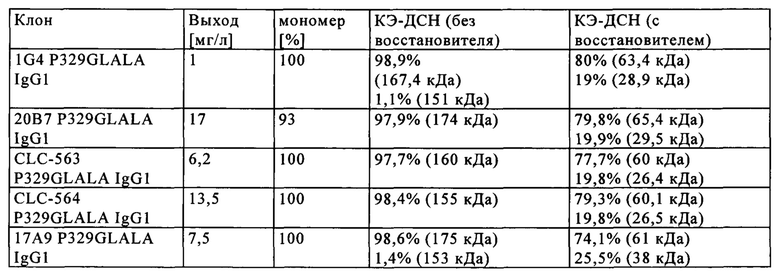
Пример 2
Характеризация антител к OX40
2.1 Связывание с человеческим OX40
2.1.1 Поверхностный плазменный резонанс (авидность + аффинность)
Связывание полученных с помощью фагов ОХ40-специфических антител с конструкцией рекомбинантный OX40-Fc(kih) оценивали с помощью поверхностного плазменного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore T200 при 25°C с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0.15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
В этом же эксперименте определяли видовую избирательность и авидность взаимодействия между полученными с помощью фагового дисплея клонами антител к OX40 8Н9, 49В4, 1G4, 20В7, CLC-563, CLC-564 и 17А9 (все в формате человеческого IgG1 P329GLALA) и рекомбинантным OX40 (человека, обезьян циномолгус (супо) и мышей). Конструкции, содержащие биотинилированный OX40 человека, обезьян циномолгус и мышей в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 600 резонансных единиц (RU).
Полученные с помощью фагового дисплея антитела к OX40 в формате человеческого IgG1 P329GLALA пропускали через проточные ячейки, используя диапазон концентраций от 2 до 500 нМ (3-кратное разбавление), со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации комплекса осуществляли в течение 210 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 16).
В этом же эксперименте определяли аффинность взаимодействия полученных с помощью фагового дисплея антител 8Н9, 49В4, 1G4, 20В7, CLC-563 и CLC-564 (формат человеческого IgG1 P329GLALA) с рекомбинантным OX40. Для этой цели эктодомен человеческого или мышиного OX40 также субклонировали в рамке считывания с avi-меткой (GLNDIFEAQKIEWHE) и гексагистидиновой меткой (последовательности см. в таблице 15) или получали путем расщепления протеазой AcTEV и удаления Fc хроматографическим методом.
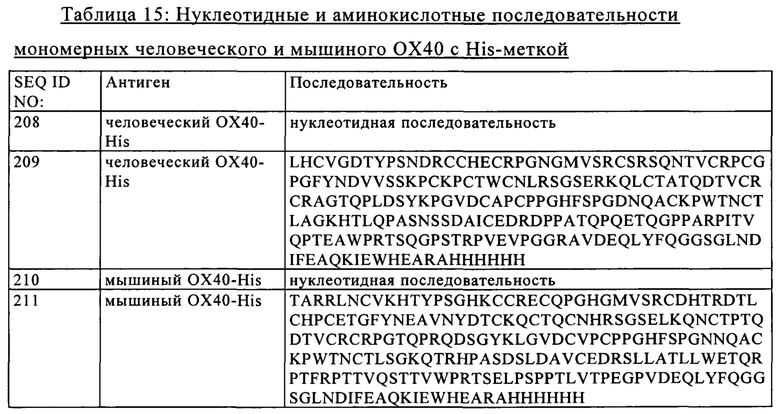
Очистку белков осуществляли согласно методу, описанному выше для слитого с Fc белка. Секретированные белки очищали из супернатантов клеточных культур с помощью хелатирующей хроматографии с последующей гель-фильтрацией.
Первую хроматографическую стадию осуществляли с использованием картриджа NiNTA Superflow (5 мл, фирма Qiagen) уравновешенного в буфере, содержащем 20 мМ фосфат натрия, 500 нМ хлорид натрия, рН 7,4. Элюцию осуществляли с использованием градиента от 5% до 45% буфера для элюции (20 мМ фосфат натрия, 500 нМ хлорид натрия, 500 мМ имидазол, рН 7,4), применяя объем, кратный 12 объемам колонки.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 75 (фирма GE Healthcare), уравновешенную раствором, содержащим 2 мМ MOPS, 150 мМ хлорид натрия, 0,02% (мас./об.) азида натрия, рН 7,4.
Определение аффинности осуществляли с использованием двух стадий.
Стадия 1) Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 9000 RU. Полученные с помощью фагового дисплея антитела к OX40 захватывали в течение 60 с в концентрациях от 25 до 50 нМ. Конструкцию рекомбинантный человеческий OX40-Fc(kih) пропускали через проточные ячейки в диапазоне концентраций от 4 до 1000 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-ЕР, а не антитела.
Стадия 2) Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 8000 RU. Полученные с помощью фагового дисплея антитела к OX40 захватывали в течение 60 с в концентрации 20 нМ. Конструкцию рекомбинантный человеческий OX40-avi-His пропускали через проточные ячейки в диапазоне концентраций от 2,3 до 600 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-ЕР, а не антитела.
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.
Клоны 49В4, 1G4 и CLC-564 связывались с конструкцией человеческий OX40-Fc(kih) с более низкой аффинностью, чем клоны 8Н9, 20В7 и CLC-563.
Константы аффинности, характеризующие взаимодействие между анти-OX40 P329GLALA IgG1 и конструкцией человеческий OX40-Fc(kih), определяли, осуществляя подгонку к модели связывания 1:1 Ленгмюра
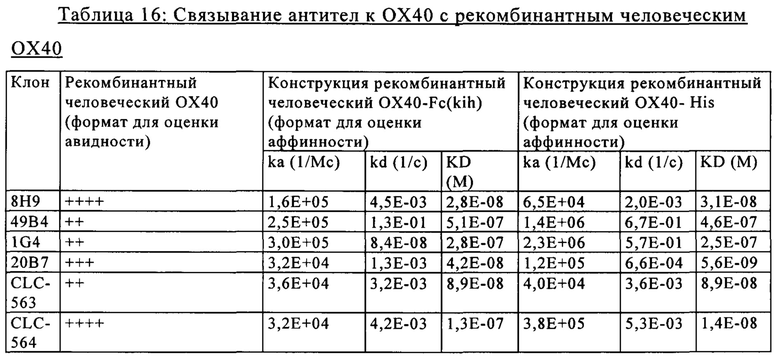
2.1.2 Связывание с экспрессирующими человеческий OX40 клетками: наивные и активированные мононуклеарные лейкоциты периферической крови человека (РВМС)
Лейкоцитарные пленки получали из центра донорской крови Цюриха. Для выделения свежих мононуклеарных клеток периферической крови (РВМС) лейкоцитарную пленку разводили таким же объемом D-ЗФР (фирма Gibco на фирме Life Technologies, каталожный №14190326). В 50-миллилитровые полипропиленовые центрифужные пробирки (фирма ТРР, каталожный №91050) вносили 15 мл Histopaque 1077 (фирма SIGMA Life Science, каталожный №10771, полисахароза и диатризоат натрия, плотность доведена до 1,077 г/мл) и наслаивали раствор разведенной лейкоцитарной пленки на Histopaque 1077. Пробирки центрифугировали при комнатной температуре в течение 30 мин при 400 × g, при небольшом ускорении и без перерыва. Затем РВМС собирали из интерфазы, промывали трижды D-ЗФР и ресуспендировали в среде для Т-клеток, состоящей из среды RPMI 1640 (фирма Gibco на фирме Life Technology, каталожный №42401-042), дополненной 10% фетальной бычьей сыворотки (FBS, фирма Gibco на фирме Life Technology, каталожный №16000-044, партия 941273, обработана гамма-лучами, свободная от микоплазм и инактивированная тепловой обработкой при 56°С в течение 35 мин), 1 об. % GlutaMAX-I (фирма GIBCO на фирме Life Technologies, каталожный №35050038), 1 мМ пируватом натрия (фирма SIGMA, каталожный №S8636), 1 об. % содержащей заменимые аминокислоты среды MEM (фирма SIGMA, каталожный № М7145) и 50 мкМ β-меркаптоэтанолом (фирма SIGMA, M3148).
РВМС использовали непосредственно после выделения (связывание с наивными человеческими РВМС) или стимулировали для индукции сильной экспрессии человеческого ОХ-40 на клеточной поверхности Т-клеток (связывание с активированными человеческими РВМС). Для этого наивные РВМС культивирования в течение 5 дней в среде для Т-клеток, дополненной 200 ед./мл пролейкина и 2 мкг/мл ФГА-L, в 6-луночном планшете для культуры ткани и затем в течение 1 дня в предварительно сенсибилизированных 6-луночных планшетах (2 мкг/мл антитела к человеческому CD3 (клон OKT3) и 2 мкг/мл антитела к человеческому CD28 (клон CD28.2)) в среде для Т-клеток, содержащей 200 ед./мл пролейкина, при 37°С и 5% СО2.
Для детекции OX40 смешивали наивные человеческие РВМС и активированные человеческие РВМС. Для того, чтобы отличать наивные клетки от активированных человеческих РВМС, покоящиеся клетки метили перед анализом связывания с помощью красителя для оценки пролиферации клеток eFluor670 (фирма eBioscience, каталожный №65-0840-85).
Для осуществления мечения клетки собирали, промывали предварительно нагретым (37°С) D-ЗФР и плотность клеток доводили до 1×107 клеток/мл в D-ЗФР. Краситель для оценки пролиферации клеток eFluor670 (фирма eBioscience, каталожный №65-0840-85) добавляли в суспензию наивных человеческих РВМС в конечной концентрации 2,5 мМ и при конечной плотности клеток 0,5×107 клеток/мл в D-ЗФР. Затем клетки инкубировали в течение 10 мин при комнатной температуре в темноте. Для прекращения реакции мечения добавляли 2 мл FBS и клетки промывали трижды средой для Т-клеток. Затем смесь 1:1 1×105 наивных клеток, меченных eFluor670 человеческих РВМС и немеченых активированных человеческих РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, Cellstar, каталожный №650185).
Планшеты центрифугировали в течение 4 мин при 400 × g и 4°С и супернатант отбрасывали. Клетки промывали однократно, используя 200 мкл холодного (4°С) FACS-буфера (D-ЗФР, дополненный 2% FBS, 5 мМ ЭДТА, рН 8 (фирма Amresco, каталожный № Е177) и 7,5 мМ азидом натрия (фирма Sigma-Aldrich, S2002)). Клетки инкубировали в 50 мкл/лунку холодного (4°С) FACS-буфера, содержащего титрованные конструкции антитела к OX40, в течение 120 мин при 4°С. Планшеты промывали четыре раза, используя 200 мкл/лунку холодного (4°С) FACS-буфера для удаления несвязанной конструкции.
Клетки окрашивали в течение 30 мин при 4°С в темноте, используя 25 мкл/лунку холодного (4°С) FACS-буфера, содержащего флуоресцентно меченое антитело к человеческому CD4 (клон RPA-T4, мышиный IgG1 k, фирма BioLegend, каталожный №300532), антитело к человеческому CD8 (клон RPa-Т8, мышиный IgG1k, фирма BioLegend, каталожный №3010441), антитело к человеческому CD45 (клон HI30, мышиный IgG1k, фирма BioLegend, каталожный №304028) и конъюгированный с флуоресцеинизотиоцианатом (ФИТЦ) F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098).
Планшеты промывали дважды, используя 200 мкл/лунку FACS-буфера (4°С), окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598) и получали данные в этот же день с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 2А-2Г, ни одна из конструкций антител, специфических в отношении OX40, не связывалась с покоящимися человеческими CD4+-T-клетками или CD8+-Т-клетками, которые не экспрессировали OX40. В противоположность этому, все конструкции связывались с активированными CD8+- или CD4+-Т-клетками, которые могли экспрессировать OX40. Связывание с CD4+-Т-клетками оказалось существенно более сильным, чем с CD8+-Т-клетками. Активированные человеческие CD8+-Т-клетки могли экспрессировать только часть от уровней OX40, обнаруженных на активированных CD4+-Т-клетках. Различие зависело от донора, а также от времени. Проанализированные клоны антител к OX40 варьировались по силе связывания. Величины ЕС50 представлены в таблице 17. Для дополнительного изучения двухвалентных и одновалентных нацеленных на FAP конструкций были выбраны клоны с высокой (8Н9) и низкой (49В4/ 1G4) способностью к связыванию.

2.2 Связывание с мышиным OX40
2.2.1 Поверхностный плазменный резонанс (авидность + аффинность)
Связывание полученных с помощью фагов ОХ40-специфического антитела 20В7 с конструкцией рекомбинантный OX40-Fc(kih) оценивали с помощью поверхностного плазменного резонанса (SPR), описанного выше для связывания с человеческим OX40 (см. пример 2.1.1).
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 18).
Для определения аффинности из-за неспецифического взаимодействия слитого с Fc белка с проточной референс-ячейкой применяли конструкцию мышиный OX40 His (см. пример 2.1.2) или OX40-Fc(kih), расщепленную протеазой AcTEV. Антитело к человеческому Fc-домену (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 8000 RU. Полученные с помощью фагового дисплея антитела к OX40 захватывали в течение 60 с в концентрации 25 нМ. Рекомбинантный мышиный OX40 (расщепленный AcTEV согласно инструкции поставщика) пропускали через проточные ячейки в диапазоне концентраций от 4,1 до 1000 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-EP, а не антитела.
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра. Установлено, что клон 20В7 связывался с мышиным OX40 (таблица 18).
Константы аффинности взаимодействия между молекулами анти-ОХ40 P329GLALA IgG1 и мышиным OX40 определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.

2.2.2 Связывание с экспрессирующими мышиный OX40 клетками: наивные и активированные мышиные спленоциты (отобранные клоны)
Селезенки мышей собирали в 3 мл ЗФР и получали суспензию единичных клеток с использованием мягких пробирок MACS (фирма Miltenyi Biotec, каталожный №130-096-334) и мягкого разрушителя MACS Octo (фирма Miltenyi Biotec). Затем спленоциты фильтровали через 30-микрометровые фильтры для предварительного разделения (фирма Miltenyi Biotec, каталожный №130-041-407) и центрифугировали в течение 7 мин при 350 × g и 4°С. Супернатант удаляли аспирацией и клетки ресуспендировали в среде RPMI 1640, дополненной 10 об. % FBS, 1 об. % GlutaMAX-I, 1 мМ пируватом натрия, 1 об. % содержащей заменимые аминокислоты среды MEM, 50 мкМ β-меркаптоэтанолом и 10% пенициллина-стрептомицина (фирма SIGMA, каталожный № Р4333). Культивировали 106 клеток/мл в течение 3 дней в 6-луночном планшете для культуры ткани, сенсибилизированном 10 мкг/мл IgG армянского хомяка к мышиному CD3ε (клон 145-2С11, BioLegend, каталожный №100331) и 2 мкг/мл IgG сирийского хомяка к мышиному CD28 (клон 37.51, фирма BioLegend, каталожный №102102). Активированные или свежие спленоциты мышей собирали, промывали в D-ЗФР, подсчитывали количество клеток и 0,1×106 клеток переносили в каждую лунку 96-луночного планшета с U-образным дном, не обработанного тканевой культурой. Клетки промывали ЗФР и окрашивали в 50 мкл FACS-буфера, содержащего в различных концентрациях антитела к OX40 в формате человеческого IgG1 P329GLALA (только отобранные связывающие агенты). Клетки инкубировали в течение 120 мин при 4°С. Затем клетки промывали дважды FACS-буфером и окрашивали, используя 25 мкл/лунку FACS-буфера, содержащего антитело к мышиному CD8b в виде крысиного IgG2bκ-APC-Cy7 (фирма BioLegend, каталожный №100714, клон 53-6.7), антитело к мышиному CD4 в виде крысиного IgG2bκ-PE-Cy7 (фирма BioLegend, каталожный №100422, клон GK1.5) и конъюгированный с ФИТЦ F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), в течение 30 мин при 4°С. Планшеты промывали дважды, используя 200 мкл/лунку FACS-буфера (4°С), клетки окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и получали данные в этот же день с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 3А-3Г, только клон 20В7 и хорошо охарактеризованное мышиное специфическое эталонное антитело OX86 связывались с активированными мышиными CD4+- и CD8+-Т-клетками. Не обнаружено никакого связывания с покоящимися мышиными спленоцитами.
2.3 Связывание с OX40 обезьян циномолгус
Для оценки реактивности отобранных анти-ОХ40-связывающих агентов с клетками обезьян циномолгус выделяли РВМС здоровых Масаса fascicularis из гепаринизированной крови с помощью центрифугирования в градиенте плотности согласно методу, описанному для человеческих клеток, с незначительными модификациями. РВМС обезьян циномолгус выделяли путем центрифугирования в градиенте плотности из гепаринизированной свежей крови, используя среду Lymphoprep (90 об. %, фирма Axon Lab, каталожный №1114545), разведенную в D-ЗФР. Центрифугирование осуществляли при 520×g без перерыва при комнатной температуре в течение 30 мин. Сопредельное центрифугирование при 150×g при комнатной температуре в течение 15 мин осуществляли для уменьшения количества тромбоцитов с последующими несколькими стадиями центрифугирования при 400×g при комнатной температуре в течение 10 мин для промывки РВМС стерильным D-ЗФР. РВМС стимулировали для достижения сильной экспрессии OX40 на клеточной поверхности Т-клеток (связывание с активированными РВМС обезьян циномолгус). Для этого наивные РВМС культивировали в течение 72 ч на предварительно сенсибилизированных 12-луночных планшетах для культуры ткани [10 мкг/мл обладающего перекрестной реактивностью в отношении обезьян циномолгус антитела к человеческому CD3 (клон SP34)] и 2 мкг/мл обладающего перекрестной реактивностью в отношении обезьян циномолгус антитела к человеческому CD28 (клон CD28.2)] в среде для Т-клеток, дополненной 200 ед./мл пролейкина, при 37°С и 5% CO2.
Затем добавляли 0,5×105 активированных РВМС обезьян циномолгус в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185). Клетки промывали однократно, используя 200 мкл холодного (4°С) FACS-буфера и инкубировали в 50 мкл/лунку холодного (4°С) FACS-буфера, содержащего титрованные конструкции антител к OX40, в течение 120 мин при 4°С. Затем планшеты промывали четыре раза, используя 200 мкл/лунку холодного (4°С) FACS-буфера. Клетки ресуспендировали в 25 мкл/лунку холодного (4°С) FACS-буфера, содержащего флуоресцентно меченое обладающее перекрестной реактивностью в отношении обезьян циномолгус антитело к человеческому CD4 (клон OKT-4, мышиный IgG1k, фирма BD, каталожный №. 317428), антитело к человеческому CD8 (клон HIT8a, мышиный IgG1k, фирма BD, каталожный №555369) и конъюгированный с ФИТЦ F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), и инкубировали в течение 30 мин при 4°С в темноте. Планшеты промывали дважды, используя 200 мкл/лунку FACS-буфера (4°С), клетки окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и получали данные в этот же день с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 4А и 4Б, большинство конструкций связывались с активированными CD4+-Т-клетками обезьян циномолгус. Связывание с CD4+-Т-клетками было существенно более сильным, чем с CD8+-Т-клетками. Кинетические характеристики и сила стимуляции зависели от уровней экспрессии OX40, и они были оптимизированными для CD4+-Т-клеток обезьян циномолгус, но не для CD8+-Т-клеток обезьян циномолгус, так, что лишь незначительная экспрессия OX40 индуцировалась на CD8+-Т-клетках. Проанализированные клоны антител к OX40 варьировались по силе связывания. Величины ЕС50 представлены в таблице 19. Из-за нетипичной подгонки кривой не удалось рассчитать величины ЕС50 для клонов 8Н9, 49В4, 21Н4.

2.3.1 Поверхностный плазменный резонанс (авидность + аффинность)
Связывание полученных с помощью фагов ОХ40-специфических антител (все в формате человеческого IgG1 P329GLALA) с конструкцией рекомбинантный OX40 обезьян циномолгус-Fc(kih) оценивали с помощью поверхностного плазменного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore T200 при 25°C с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0.15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
Конструкции, содержащие биотинилированный OX40 обезьян циномолгус в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 800 резонансных единиц (RU).
Полученные с помощью фагового дисплея антитела к OX40 в формате человеческого IgG1 P329GLALA пропускали через проточные ячейки, используя диапазон концентраций от 2 до 500 нМ (3-кратное разведение), со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации комплекса осуществляли в течение 210 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 20).
В этом же эксперименте определяли аффинность взаимодействия полученных с помощью фагового дисплея антител (в формате человеческого IgG1 P329GLALA) с конструкцией рекомбинантный OX40 обезьян циномолгус-Fc(kih). Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 9000 RU. Полученные с помощью фагового дисплея антитела к OX40 захватывали в течение 60 с в концентрациях от 25 до 50 нМ. Конструкцию рекомбинантный OX40 обезьян циномолгус-Fc(kih) пропускали через проточные ячейки в диапазоне концентраций от 4 до 1000 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-ЕР, а не антитела.
Кинетические константы определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра (таблица 20).
Клоны 49В4, 1G4 и CLC-564 связывались с конструкцией OX40 обезьян циномолгус-Fc(kih) с более низкой аффинностью, чем клоны 8Н9, 20В7 и CLC-563.
Константы аффинности, характеризующие взаимодействие между анти-OX40 P329GLALA IgG1 и конструкцией OX40 обезьян циномолгус-Fc(kih) определяли с помощью программного обеспечения Biacore T200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.
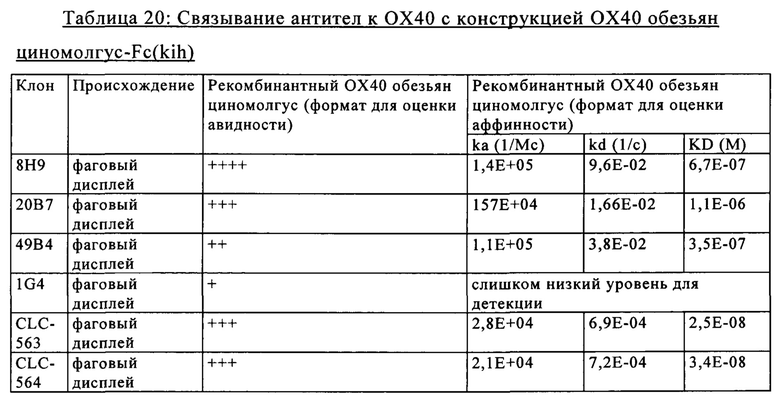
2.3.2 Связывание с экспрессирующими OX40 обезьян циномолгус клетками: активированные мононуклеарные лейкоциты периферической крови (РВМС) обезьян циномолгус
Связывание с ОХ40-негативными опухолевыми клетками
Отсутствие связывания с ОХ40-негативными опухолевыми клетками тестировали с использованием клеток WM266-4 (АТСС CRL-1676) и опухолевых клеток U-87 MG (АТСС НТВ-14). Для получения возможности различать оба типа опухолевых клеток WM266-4-клетки предварительно метили с помощью набора PKH-26 Red Fluorescence Cell linker (фирма Sigma, каталожный № PKH26GL). Клетки собирали и промывали трижды средой RPMI 1640. Дебрис окрашивали в течение 5 мин при комнатной температуре в темноте при конечной клеточной плотности 1×107 клеток в свежеприготовленном растворе красителя PKH26-Red (конечная концентрация [1 нМ] в указанном разбавителе В). Избыток FBS добавляли для прекращения реакции мечения и клетки промывали 4 раза средой RPMI 1640, дополненной 10 об. % FBS, 1 об. % GlutaMAX-I, для удаления избытка красителя.
Смесь, содержащую 5×104 меченных PKH26 клеток WM266-4 и немеченых клеток U-87 MG, добавляли в D-ЗФР в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток. Планшеты центрифугировали в течение 4 мин при 400 × g, 4°C и супернатант отбрасывали. Клетки промывали однократно 200 мкл D-ЗФР и дебрис ресуспендировали в помощью кратковременного и осторожного перемешивания. Все образцы ресуспендировали, используя 50 мкл/лунку холодного (4°C) FACS-буфера, содержащего титрованные концентрации конструкций антител к OX40 в формате человеческого IgG1 P329GLALA в течение 120 мин при 4°С. Планшеты промывали 4 раза, используя 200 мкл/на лунку FACS-буфера (4°С). Клетки ресуспендировали, используя 25 мкл/на лунку холодного (4°С) FACS-буфера, содержащего конъюгированный с ФИТЦ F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), и инкубировали в течение 30 мин при 4°С в темноте. Планшеты промывали дважды, используя 200 мкл/лунку FACS-буфера (4°С), клетки окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и получали данные в этот же день с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 5А и 5Б, ни одна из конструкций антител, специфических в отношении OX40, не связывалась с ОХ40-негативными человеческими опухолевыми клетками WM266-4 и U-78 MG.
2.4 Способность блокировать лиганд
Для определения способности молекул ОХ40-специфических человеческих антител IgG1 P329GLALA оказывать влияние на взаимодействия ОХ40/лиганд OX40 применяли человеческий лиганд OX40 (фирма R&D systems). Из-за низкой аффинности взаимодействия между OX40 и лигандом OX40 приготавливали слитую молекулу, содержащую димерный человеческий OX40-Fc с С-концевой На-меткой (фиг. 1Б). Нуклеотидная и аминокислотная последовательности указанной слитой молекулы, содержащей димерный человеческий OX40-Fc, представлены в таблице 21. Получение и очистку осуществляли согласно методу, описанному для мономерной формы OX40-Fc(kih) в примере 1.1.

Человеческий лиганд OX40 (фирма R&D systems) непосредственно сшивали с двумя проточным ячейками СМ5-чипа с уровнем, соответствующим примерно 2500 RU, при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Конструкцию рекомбинантный человеческий OX40-Fc пропускали через вторую проточную ячейку в концентрации 200 нМ со скоростью потока 30 мкл/мин в течение 90 с. Диссоциацию исключали и полученное с помощью фагов антитело к OX40 в формате человеческого IgG1P329LALA пропускали через обе проточные ячейки в концентрации 500 нМ со скоростью потока 30 мкл/мин в течение 90 с. Мониторинг диссоциации осуществляли в течение 60 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антитела пропускали по поверхности с иммобилизированным человеческим лигандом OX40, но инъецировали HBS-EP вместо конструкции рекомбинантный человеческий OX40-Fc. На фиг. 1 В представлена схеме эксперимента.
Полученный с помощью фагов клон 20В7 связывался с комплексом человеческого OX40 с его лигандом OX40 (таблица 22, фиг. 6Б). Таким образом, это антитело не могло конкурировать с лигандом за связывание с человеческим OX40, и поэтому его обозначали как «неблокирующее лиганд». В противоположность этому, клоны 8Н9, 1G4, 49В4, CLC-563 и CLC-564 не связывались с человеческим OX40 в комплексе с его лигандом, и поэтому их обозначали как «блокирующие лиганд».
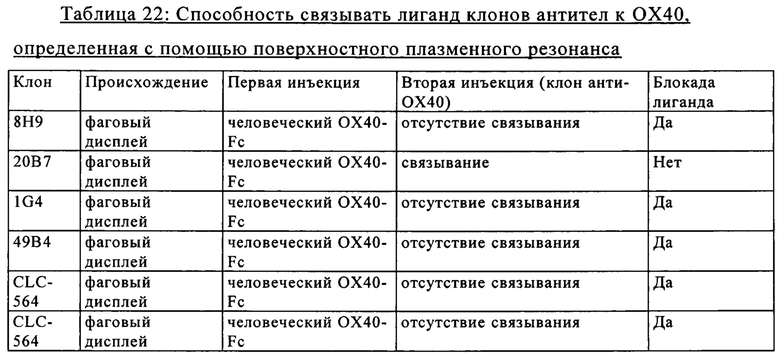
Пример 3
Функциональные свойства клонов антител, связывающихся с человеческим OX40
3.1 Клетки HeLa, экспрессирующие человеческий OX40 и репортерный ген NF-κВ-люциферазы
Агонистическое связывание OX40 с его лигандом индуцирует передачу сигналов в прямом направлении посредством активации ядерного фактора каппа В (NFκB) (A.D. Weinberg и др., J. Leukoc. Biol. 75(6), 2004, сс. 962-972). Рекомбинантную репортерную линию клеток HeLa_hOx40_NFκB_Luc1 создавали для экспрессии человеческого OX40 на их поверхности. Она несла также репортерную плазмиду, содержащую ген люциферазы под контролем чувствительного к NFκB энхансерного сегмента. Стимуляция OX40 индуцирует зависящую от дозы активацию NFκB, который транслоцируется в ядро, в котором он связывается с чувствительным к NFκB энхансером репортерной плазмиды, повышая экспрессию белка люциферазы. Люцифераза катализирует окисление люциферина, что приводит к образованию оксилюциферина, испускающего свет. Это можно оценивать количественно с помощью люминометра. Таким образом, в одном эксперименте анализировали способность различных анти-ОХ40-связывающих агентов в формате P329GLALA huIgG1 индуцировать активацию NFκB в репортерных клетках HeLa_hOx40_NFκB_Luc1.
Прикрепленные клетки HeLa_hOx40_NFκB_Luc1 собирали, используя буфер для диссоциации клеток (фирма Invitrogen, каталожный №13151-014), в течение 10 мин при 37°С. Клетки однократно промывали D-ЗФР и плотность клеток доводили до 2×105 в среде для анализа, содержащей MEM (фирма Invitrogen, каталожный №22561-021), 10 об. % инактивированной тепловой обработкой FBS, 1 мМ пируват натрия и 1 об. % заменимых аминокислот. Клетки высевали с плотностью 0,3×105 клеток на лунку в стерильный белый плоскодонный 96-луночный планшет для культуры ткани с крышкой (фирма Greiner bio-one, каталожный №655083) и выдерживали в течение ночи при 37°С в инкубаторе (Hera Cell 150) в атмосфере с 5% СО2.
На следующий день HeLa_hOx40_NFκB_Luc1 стимулировали в течение 6 ч, добавляя среду для анализа, содержащую в титрованных количествах различные анти-ОХ40-связывающие агенты в формате P329GLALA huIgG1. Для тестирования воздействия перекрестной гипер-сшивки на антитела к OX40 добавляли 50 мкл/лунку среды, содержащей в качестве вторичного антитела F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-специфический (фирма Jackson ImmunoResearch, каталожный №109-006-098) в соотношении 1:2 (2-кратное превышение количества вторичного антитела относительно первичного единичного анти-ОХ40 P329GLALA huIgG1). После инкубации супернатант аспирировали и планшеты промывали два раза D-ЗФР. Количественное определение испускания света осуществляли с использованием системы для анализа люциферазы 100 и репортерного лизирующего буфера (оба фирмы Promega, каталожный № Е4550 и каталожный № Е3971) согласно инструкциям производителя. В целом, метод состоял в следующем: клетки лизировали в течение 10 мин при -20°С путем добавления 30 мкл на лунку 1-кратного лизирующего буфера. Клетки подвергали оттаиванию в течение 20 мин при 37°С, после чего добавляли 90 мкл на лунку реагента для анализа люциферазы. Испускание света немедленно оценивали количественно с помощью ридера для микропланшетов SpectraMax M5/M5e (фирма Molecular Devices, США), для чего осуществляли интегрирование в течение 500 мс без использования какого-либо фильтра для учета всех длин волн. Уровень испускаемого света (в относительных единицах испускаемого света, URL) корректировали с учетом исходной люминесценции клеток HeLa_hOx40_NFκB_Luc1 и строили график зависимости от логарифма концентрации первичного антитела, используя программу Prism4 (фирма GraphPad Software, США). Кривые аппроксимировали на основе заложенного в программу сигмоидального дозового ответа.
Как продемонстрировано на фиг. 7А и 7Б, ограниченная зависящая от дозы NFkB-активация индуцировалась уже при добавлении антител к OX40 P329GLALA huIgG1 (левый график) к репортерной линии клеток. Перекрестная гипер-сшивка антител к OX40 с помощью античеловеческих IgG-специфических вторичных антител приводила к сильному повышению индукции опосредуемой NFκB активации люциферазы в зависимости от концентрации (правый график). Величины ЕС50, характеризующие активацию, обобщены в таблице 23.

3.2 Опосредуемая OX40 костимуляция субоптимально стимулированных TCR предварительно активированных человеческих CD4-Т-клеток
Лигирование OX40 обеспечивает синергетический костимуляторный сигнал, повышающий деление и выживаемость Т-клеток после субоптимальной стимуляции Т-клеточного рецептора (TCR) (M. Croft и др., Immunol. Rev., 229(1), 2009, cc. 173-191). Кроме того, повышается производство некоторых цитокинов и поверхностная экспрессия маркеров Т-клеточной активации (I. Gramaglia и др., J. Immunol., 161(12), 1998, сс. 6510-6517; S.М. Jensen и др., Seminars in Oncology, 37(5), 2010, сс. 524-532).
Для тестирования агонистических свойств различных анти-ОХ40-связывающих агентов предварительно активированные ОХ40-позитивные CD4-Т-клетки стимулировали в течение 72 ч применяемыми в субоптимальной концентрации иммобилизированными на планшете антителами к CD3 в присутствии антител к ОХ40 либо в растворе, либо в иммобилизированной форме на поверхности планшета. Воздействия на выживаемость и пролиферацию Т-клеток анализировали, осуществляя мониторинг общего количества клеток и разведения CFSE в живых клетках с помощью проточной цитометрии. Дополнительно клетки окрашивали совместно флуоресцентно мечеными антителами против маркеров активации и дифференцировки Т-клеток, например, CD127, CD45RA, Tim-3, CD62L и самого ОХ40.
Человеческие РВМС выделяли путем центрифугирования в градиенте плотности фикола и стимулировали в течение 3 дней с использованием ФГА-L [2 мкг/мл] и пролейкина [200 ед./мл] согласно методу, описанному в примере 2.1.2. Затем клетки метили CFSE при клеточной плотности 1×106 клеток/мл с использованием CFDA-SE (сложный N-сукцинимидиловый эфир 5(6)-карбоксифлуоресцеиндиацетата, фирма Sigma-Aldrich, каталожный №2188) в конечной концентрации [50 нМ] в течение 10 мин при 37°C. Затем клетки промывали дважды избытком D-ЗФР, содержащим FBS (10 об. %). Меченые клетки выдерживали в средах для Т-клеток при 37°С в течение 30 мин. Затем неконвертированный CFDA-SE удаляли путем двух дополнительных стадий промывки с помощью D-ЗФР. Выделение CD4-Т-клеток из предварительно активированных меченных CFSE человеческих РВМС осуществляли с использованием набора для выделения CD4-Т-клеток на основе негативной селекции MACS (MACS negative CD4 T-cell) (фирма Miltenyi Biotec.) согласно инструкциям производителя.
Morris с соавторами продемонстрировали, что агонистическая костимуляция с помощью канонических антител к ОХ40 основана на поверхностной иммобилизации (N.P. Morris и др., Mol. Immunol., 44(12), 2007, сс. 3112-3121). Так, козьи антимышиные антитела, Fcγ-специфические (фирма Jackson Immuno Research, каталожный №111-500-5008) наносили на поверхность 96-луночного планшета для культуры клеток с U-образным дном (фирма Greiner Bio One) в концентрация 2 мкг/мл в ЗФР в течение ночи при 4°С в присутствии (антитело к ОХ40, иммобилизированное на поверхности) или в отсутствии (антитело к ОХ40 в растворе) козьего античеловеческого, Fcγ-специфического антитела (фирма Jackson Immuno Research, каталожный №109-006-098). Затем поверхность планшета блокировали D-ЗФР, содержащим БСА (1% (об./мас.). Все Т-клетки подвергали стадиям инкубации при 37°С в течение 90 мин в ЗФР, содержащем БСА (1% (об./мас). Между стадиями инкубации планшеты промывали D-ЗФР.
Мышиное антитело к человеческому CD3 (клон ОКТЗ, фирма eBioscience, каталожный №16-0037-85, фиксированная концентрация [3 нг/мл]) захватывали на последующей стадии инкубации посредством нанесенных на поверхность антимышиных, Fcγ-специфических антител. В одном эксперименте затем на планшете иммобилизировали титрованные человеческие антитела к ОХ40 (человеческий IgG1 P329G LALA) с помощью дополнительной стадии инкубации в D-ЗФР. Во втором эксперименте антитела к ОХ40 добавляли в процессе анализа активации непосредственно в среду к планшетам, которые не сенсибилизировали предварительно антителами к человеческому IgG, Fc- специфическими.
Меченные CFSE предварительно активированные CD4+-Т-клетки добавляли в предварительно сенсибилизированные планшеты при клеточной плотности 0,6×105 клеток на лунку в 200 мкл среды для Т-клеток и культивировали в течение 96 ч. Клетки окрашивали комбинацией меченных флуорохромом мышиных антител к человеческому ОХ40 (клон BerACT35, фирма Bioledgend, каталожный №35008), TIM-3 (клон F38-2E2, фирма Biolegend, каталожный №345008), CD127 (клон A019D5, фирма Biolegend, каталожный №351234), CD62L (клон DREG 56, фирма Biolegend, каталожный №304834) и CD45RA (клон HI100, фирма BD Biosciences, каталожный №555489) в течение 20 мин при 4°С в темноте. Планшеты промывали дважды, используя 200 мкл/на лунку (4°С) FACS-буфера, клетки окончательно ресуспендировали в 80 мкл/на лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и получали данные в этот же день с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
DAPI-негативные живые клетки анализировали в отношении снижения медианной CFSE-флуоресценции в качестве маркера пролиферации. Осуществляли мониторинг процента ОХ40-позитивных, CD62Llow и TIM-3-позитивных Т-клеток в качестве маркера Т-клеточной активации. Экспрессию CD45RA и CD127 анализировали для определения изменений в статусе созревания Т-клеток, a CD45RAlow CD127low-клетки относили к категории эффекторных Т-клеток.
Костимуляция с помощью иммобилизированных на планшете антител сильно в зависимости от дозы повышала субоптимальную стимуляцию предварительно активированных человеческих CD4-Т-клеток при использовании иммобилизированного на планшете антитела к человеческому CD3 (фиг. 8А-8Е). Пролиферация Т-клеток была более сильной, она характеризовалась более зрелым фенотипом с более высоким процентом эффекторных Т-клеток и более высокими процентами CD62Llow, Tim-3-позитивных и ОХ40-позитивных активированных клеток. Связывание некоторых клонов (8Н9, 20В7) с клеточным ОХ40 не поддавалось оценке с использованием поступающих в продажу средств для детекции антител. Для них оказалось невозможным рассчитать величину EC50 и поэтому все величины EC50, характеризующие индукцию ОХ40, были исключены при расчете всех величин EC50. Изменения, составляющие половину от максимального всех других параметров Т-клеточной активации, достигались в диапазоне концентраций от 3 до 700 пМ, данные обобщены на фиг. 9 и в таблице 24. Никакого усиления субоптимальной стимуляции TCR не обнаружено, когда антитела к ОХ40 добавляли в раствор в отсутствии иммобилизации на поверхности (фиг. 10А-10Е). Эти результаты вновь демонстрируют сильную зависимость оси ОХ40-активации от перекрестной гипер-сшивки ОХ40-рецептора.
Корреляция между силой связывания и агонистической активностью (биологическая активность) антител к ОХ40 (формат IgG1 P329GLALA) продемонстрирована на фиг. 11. Для большинства клонов обнаружена прямая корреляция, однако для двух клонов (49В4, 1G4) неожиданно обнаружена более сильная биологическая активность по сравнению с предсказанной на основе их силы связывания.

Пример 4
Создание биспецифических конструкций, мишенями которых являются ОХ40 и фибробласт-активирующий белок (ТАР)
4.1 Создание биспецифических двухвалентных антигенсвязывающих молекул, мишенями которых являются ОХ40 и фибробласт-активирующий белок (FAP) (формат 2+2)
Получали биспецифические агонистические антитела к ОХ40, обладающие двухвалентным связыванием с ОХ40 и FAP. Технологию Crossmab, описанную в международной заявке на патент WO 2010/145792 А1, применяли для снижения образования неправильно спаренных легких цепей.
Разработка и получение связывающих FAP агентов описаны в WO 2012/020006 А2, которая включена в настоящее описание в качестве ссылки.
В этом примере «скрещенный» модуль Fab (VHCL) связывающего FAP агента 28Н1 сливали на С-конце с тяжелой цепью антитела к ОХ40 huIgG1, используя последовательность-коннектор (G4S)4. Указанное содержащее тяжелую цепь слияние совместно экспрессировали с легкой цепью антитела к ОХ40 и соответствующей «скрещенной» легкой цепью антитела к FAP (VLCH1). Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей для элиминации связывания с Fc гамма рецепторами согласно методу, описанному в опубликованной международной заявке на патент WO 2012/130831 А1. Образовавшаяся биспецифическая двухвалентная конструкция представлена на фиг. 12А.
В таблице 25 представлены соответственно нуклеотидные и аминокислотные последовательности зрелых биспецифических двухвалентных анти-ОХ40/анти-FAP человеческих антител в формате IgG1 P329GLALA.







Все гены кратковременно экспрессировали под контролем химерного промотора MPSV, состоящего из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр).
Биспецифические анти-ОХ40, анти-БАР-конструкции получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1 («вектор тяжелой цепи» : «вектор легкой цепи1» : «вектор легкой цепи2»).
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку биспецифических конструкций из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слияний антиген-Fc и антител.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, рН 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нМ с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу биспецифических молекул анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, который содержал 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С.

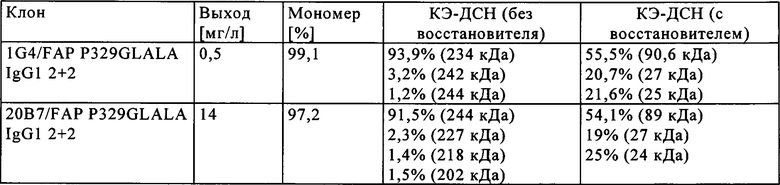
4.2 Создание биспецифических одновалентных антигенсвязывающих молекул, мишенями которых являются ОХ40 и фибробласт-активирующий белок (FAP) (Формат 1+1)
Получали биспецифические агонистические антитела к ОХ40 с одновалентным связыванием с ОХ40 и FAP. Технологию Crossmab, описанную в международной заявке на патент WO 2010/145792 А1, применяли для снижения образования неправильно спаренных легких цепей.
В этом примере «скрещенный» модуль Fab (VHCL) связывающего FAP агента 28Н1 сливали с содержащей «впадину» тяжелой цепью huIgG1. Fab против анти-ОХ40 сливали с содержащей «выступ» тяжелой цепью. Комбинация нацеленной конструкции анти-FAP-Fc, содержащей «впадину», с конструкцией анти-ОХ40-Fc, содержащей «выступ», позволяла получать гетеродимер, который включал FAP-связывающий Fab и ОХ40-связывающий Fab (фиг. 12Б).
Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей для элиминации связывания с рецепторами Fc гамма согласно методу, описанному в опубликованной международной заявке на патент WO 2012/130831 А1.
Полученная биспецифическая одновалентная конструкция представлена на фиг. 12А, а нуклеотидные и аминокислотные последовательности представлены в таблице 27.





Все гены кратковременно экспрессировали под контролем химерного промотора MPSV, состоящего из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр).
Биспецифические анти-ОХ40, анти-РАР-конструкции получали путем котрансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1:1 («вектор тяжелой цепи с «впадиной» : «вектор тяжелой цепи с «выступом» : «вектор легкой цепи1» : «вектор легкой цепи2»).
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°C в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку биспецифических конструкций из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слияний антиген-Fc и антител.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, рН 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу биспецифических молекул анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, который содержал 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С.
В таблице 28 обобщены результаты биохимического анализа биспецифических одновалентных анти-Ох40/анти-FAP антигенсвязывающих молекула в формате IgG1 P329G LALA kih.
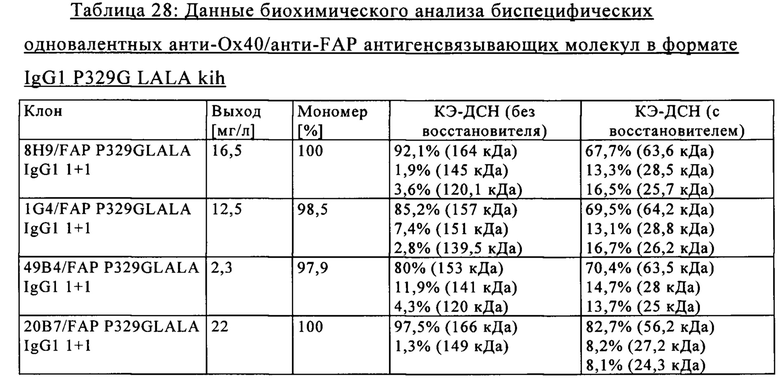
4.3 Характеризация биспецифических конструкций, мишенями которых являются ОХ40 и FAP
4.3.1 Поверхностный плазмонный резонанс (одновременное связывание)
Способность связываться одновременно с конструкцией человеческий OX40-Fc(kih) и с человеческим FAP оценивали с помощью поверхностного плазмонного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°C с использованием HBS-EP в качестве подвижного буфера (0,01M HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
Конструкции, содержащие биотинилированный человеческий ОХ40 в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 1000 резонансных единиц (RU).
Биспецифические конструкции, мишенями которых являются ОХ40 и FAP, пропускали через проточные ячейки, используя концентрацию порядка 250 нМ, со скоростью потока 30 мкл/мин и диссоциацию устанавливали на 0 с. Человеческий FAP инъецировали в проточные ячейки в качестве второго аналита со скоростью потока 30 мкл/мин в течение 90 с в концентрации 250 нМ (фиг. 12В). Мониторинг диссоциации комплекса осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Как продемонстрировано на графиках, представленных на фиг 13А-13З, все биспецифические конструкции обладали способностью связываться одновременно с человеческим ОХ40 и человеческим FAP.
4.3.2 Связывание на клетках
4.3.2.1 Связывание на наивных в сравнении с активированными человеческими РВМС нацеленных на FAP антител к ОХ40
Человеческие РВМС выделяли путем центрифугирования в градиенте плотности фикола согласно методу, описанному в примере 2.1.2. РВМС использовали непосредственно после выделения (связывание с покоящимися человеческими РВМС) или их стимулировали для достижения высокого уровня экспрессии человеческого ОХ40 на клеточной поверхности Т-клеток (связывание с активированными человеческими РВМС). Для этого наивные РВМС культивировали в течение 4-7 дней в среде для Т-клеток, дополненной 200 ед./мл пролейкина и 2 мкг/мл ФГА-L, в 6-луночном планшете для культуры ткани и затем 1 день в предварительно сенсибилизированных 6-луночных планшетах для культуры ткани [10 мкг/мл антитела к человеческому CD3 (клон ОКТЗ) и 2 мкг/мл антитела к человеческому CD28 (клон CD28.2)] в среде для Т-клеток, дополненной 200 ед./мл пролейкина, при 37°С и 5% СО2.
Для детекции ОХ40 наивные человеческие РВМС и активированные человеческие РВМС смешивали. Для того, чтобы отличать наивные клетки от активированных человеческих РВМС, покоящиеся клетки метили перед анализом связывания с помощью красителя для оценки пролиферации клеток eFluor670 (фирма eBioscience, каталожный №65-0840-85). Затем смесь 1:1 1×105 наивных клеток, меченных eFluor670 человеческих РВМС, и немеченых активированных человеческих РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185) и осуществляли анализ связывания согласно методу, описанному в примере 2.1.2. Затем смесь 1:1 1×105 наивных клеток, меченных eFluor670 человеческих РВМС, и немеченых активированных человеческих РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185) и осуществляли анализ связывания согласно методу, описанному в разделе 2.1.2.
Первичные антитела представляли собой титрованные конструкции антител к ОХ40, инкубированные в течение 120 мин при 4°С. Раствор вторичного антитела представлял собой смесь флуоресцентно меченого антитела к человеческому CD4 (клон RPA-T4, мышиный IgG1k, фирма BioLegend, каталожный №300532, антитело к человеческому CD8 (клон RPa-T8, мышиный IgG1, фирма BioLegend, каталожный №3010441) и конъюгированный с флуоресцеинизотиоцианатом (ФИТЦ) F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), инкубированную в течение 30 мин при 4°С в темноте. Содержимое планшетов окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598) и получали данные в этот же день с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 14А-14С, ни одна из антигенсвязывающих молекул, специфических в отношении ОХ40, не связывалась с покоящимися человеческими CD4+-Т-клетками или CD8+-Т-клетками. В противоположность этому, все антигенсвязывающие молекулы связывались с активированными CD8+- или CD4+-Т-клетками. Связывание с CD4+-Т-клетками оказалось существенно более сильным, чем с CD8+-Т-клетками, аналогично тому, что уже было описано в примере 2.1.2. Как продемонстрировано на фиг. 14А-14С, двухвалентная нацеленная на FAP конструкция ОХ40 обладала сходными характеристиками связывания с ОХ40-позитивными и негативными клетками с соответствующими клонами в формате канонических антитела IgG-типа, в то время как одновалентные антитела обладали существенно более низкой способностью связываться с ОХ40-позитивными клетками из-за снижения авидности.
4.3.2.2 Связывание с экспрессирующими человеческий FAP опухолевыми клетками
Связывание с находящимся на клеточной поверхности FAP тестировали с использованием клеток, экспрессирующих человеческий фибробласт-активирующий белок (huFAP) линии NIH/3T3-huFAP, клон 39 или клеток WM266-4 cells (АТСС CRL-1676). Линию NIH/3T3-huFAP, клон 39 создавали путем трансфекции линии клеток фибробластов мышиных эмбрионов NIH/3T3 (АТСС CRL-1658) экспрессионным вектором pETR4921 для экспрессии huFAP при селекции пуромицином в концентрации 1,5 мкг/мл. В некоторых анализах клетки WM266-4 предварительно метили с помощью набора PKH-26 Red Fluorescence Cell linker (фирма Sigma, каталожный № PKH26GL) согласно методу, описанному в примере 2.3.2, для получения возможности отличать эти опухолевые клетки от других присутствующих клеток (например, человеческих РВМС).
Затем 0,5×105 клеток NIH/3T3-huFAP, клон 39 или WM266-4, добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (Greiner bio-one, cellstar, каталожный №650185) и осуществляли анализ связывания аналогично методу, описанному в примере 2.3.2. Планшеты центрифугировали в течение 4 мин при 400 × g при 4°С и супернатанты отбрасывали. Клетки промывали однократно 200 мкл D-ЗФР и дебрисы ресуспендировали путем кратковременного и осторожного встряхивания. Все образцы ресуспендировали в 50 мкл/на лунку холодного (4°С) FACS-буфера, содержащего биспецифические антигенсвязывающие молекулы (первичное антитело) в указанном диапазоне концентраций (титрованных) и инкубировали в течение 120 мин при 4°С. Затем клетки промывали 4 раза, используя 200 мкл (4°С) FACS-буфера и ресуспендировали путем кратковременного встряхивания. Затем клетки окрашивали, используя 25 мкл/на лунку холодного (4°С) раствора конъюгированного с флуоресцеинизотиоцианатом (ФИТЦ) F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098) в качестве вторичного антитела, и инкубировали в течение 30 мин при 4°С в темноте. Содержимое планшетов окончательно ресуспендировали в 80 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598) и получали данные в этот же день с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 15А и 15Б, нацеленные на FAP одно- и двухвалентные анти-ОХ40 антигенсвязывающие молекулы, в отличие от таких же клонов в формате huIgG1 P329GLALA, эффективно связывались с экспрессирующими человеческий FAP клетками-мишенями. При этом только нацеленные на FAP одно- и двухвалентные анти-ОХ40-антигенсвязывающие молекулы обладали непосредственными таргетирующими опухоль свойствами. У двухвалентной конструкции (закрашенный квадрат) обнаружено более сильное связывание с FAP, чем у одновалентных конструкций, что объясняется повышением авидности в двухвалентном формате по сравнению с одновалентным форматом. Это является более выраженным для отличающихся высоким уровнем экспрессии FAP клеток NIH/3T3-huFAP, клон 39 (фиг. 15А) по сравнению с имеющими более низкий уровень экспрессии FAP клетками WM266-4 (фиг. 15Б). Более низкая плотность FAP на поверхности клеток WM266-4 может не обеспечивать тесную близость с молекулами FAP для того, чтобы всегда осуществлять двухвалентное связывание анти-ОХ40-конструкций. Величины ЕС50, характеризующие связывание с активированными человеческими CD4-Т-клетками и FAP-позитивными опухолевыми клетками, обобщены в таблице 29.
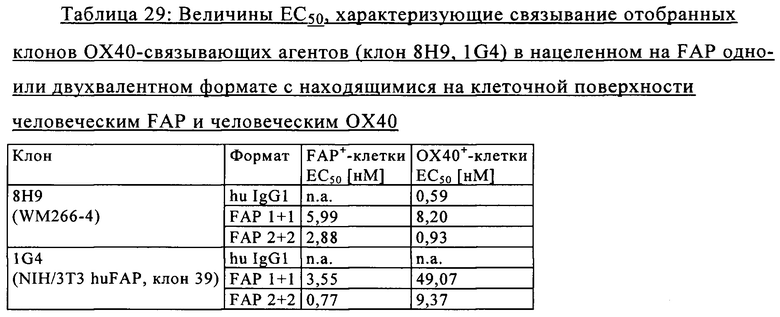
4.4 Создание биспецифических антигенсвязывающих молекул, мишенями которых являются ОХ40 и фибробласт-активирующий белок (FAP), которые являются двухвалентными в отношении ОХ40 и одновалентными в отношении FAP (формат 2+1)
Биспецифические агонистические антитела к ОХ40 с двухвалентным связыванием в отношении ОХ40 и одновалентным связыванием в отношении FAP, получали, используя технологию «выступ»-во-«впадину», которая позволяла осуществлять сборку двух различных тяжелых цепей.
В этом примере первая тяжелая цепь (НС 1) содержала один модуль Fab (VHCH1) анти-ОХ40-связывающего агента 49В4, за которым располагалась Fc-цепь с «выступом», слитая с помощью (C4S)-линкера с VH-доменом анти-FAP-связывающего агента 28Н1 или 4В9. Вторая тяжелая цепь (НС 2) конструкции содержала один модуль Fab (VHCH1) анти-ОХ40-связывающего агента 49В4, за которым располагалась Fc-цепь с «впадиной», слитая с помощью (C4S)-линкера с VL-доменом анти-FAP-связывающего агента 28Н1 или 4В9.
Конструирование и получение связывающих FAP агентов описано в WO 2012/020006 А2, которая включена в настоящее описание в качестве ссылки.
Мутации Pro329Gly, Leu234Ala и Leu235Ala интродуцировали в константные области тяжелых цепей для элиминации связывания с рецепторами Fc гамма согласно методу, описанному в опубликованной международной заявке на патент WO 2012/130831 А1. Слитые белки тяжелых цепей совместно экспрессировали с легкой цепью анти-ОХ40-связывающего агента 49В4 (CLVL). Полученные биспецифические конструкции двухвалентные в отношении связывания с ОХ40 представлены на фиг. 12Г, а описание их нуклеотидных и аминокислотных последовательностей представлено в таблице 30.
Кроме того, получали «ненацеленную» конструкцию 2+1, в которой VH- и VL-домены анти-FAP-связывающего агента заменяли на контрольную зародышевую линию, обозначенную как DP47, которая не связывалась с антигеном.





Все гены кратковременно экспрессировали под контролем химерного промотора MPSV, состоящего из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр).
Биспецифические анти-ОХ40/анти-FAP (2+1)-конструкции получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина (ПЭИ; фирма Polysciences Inc.). Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:2 («вектор НС1» : «вектор НС2» : «вектор LC»).
Для получения 200 мл в 500-миллилитровые встряхиваемые колбы засевали 250 млн клеток НЕК293 EBNA за 24 ч до трансфекции в среду Excell (фирма Sigma) с добавками. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ (1 мг/мл) (фирма Polysciences Inc.) раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2 и встряхивали при 165 об/мин. После инкубации добавляли 160 мл среды Excell с добавками (1 мМ вальпроевая кислота, 5 г/л PepSoy (соевый пептид), 6 мМ L-глутамин) и клетки культивировали в течение 24 ч. Через 24 ч после трансфекции к клеткам добавляли содержащую аминокислоты и глюкозу добавку 12% в конечном объеме (24 мл) и 3 г/л глюкозы (1,2 мл из маточного раствора с концентрацией 500 г/л). После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 45 мин при 2000-3000 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку биспецифических конструкций из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием MabSelectSure. Белок концентрировали и фильтровали перед внесением в колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, 0,01% Твин-20, рН 6,0.
Для осуществления аффинной хроматографии супернатант вносили в колонку ProtA MabSelect Sure (CV=5 мл, фирма GE Healthcare), уравновешенную 30 мл раствора, содержащего 20 мМ цитрат натрия, 20 мМ фосфат натрия, рН 7.5. Несвязанный белок удаляли путем промывки буфером, содержащим 20 мМ фосфат натрия, 20 мМ цитрат натрия (рН 7,5), используя его в объеме, кратном 6-10 объемам колонки. Связанный белок элюировали, используя линейный рН-градиент, 20 CV (от 0 до 100%) буфера, содержащего 20 мМ цитрат натрия, 100 мМ хлорид натрия, 100 мМ глицин, 0,01 об. % Твин-20, рН 3,0. Затем колонку промывали, применяя в объеме, кратном 10 объемам колонки, раствор, содержащий 20 мМ цитрат натрия, 100 мМ хлорид натрия, 100 мМ глицин, 0,01 об. % Твин-20, рН 3,0, с последующей стадией повторного уравновешивания.
Значение рН собранных фракций регулировали, добавляя 1/10 (об./об.) 0,5М фосфата натрия, рН 8,0. Белок концентрировали и фильтровали перед внесением в колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ хлорид натрия, 0,01 об. % Твин20, рН 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нМ с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу биспецифических конструкций анализировали с помощью КЭ-ДСН в присутствии и в отсутствии восстановителя (фирма Invitrogen), используя устройство LabChipGXII (фирма Caliper). Содержание агрегатов биспецифических конструкций анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ фосфат калия, 125 мМ хлорид натрия, 200 мМ моногидрохлорид аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С (таблица 31).
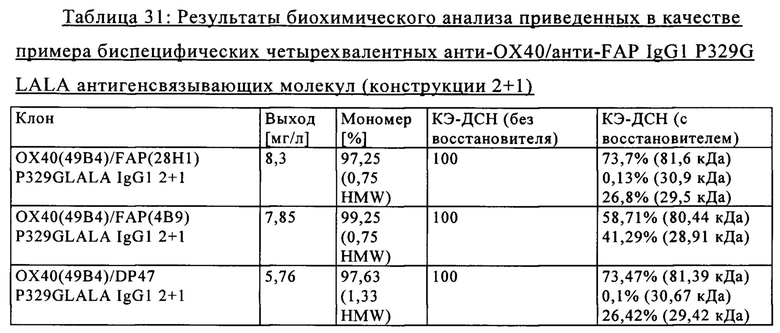
4.5 Характеризация биспецифических конструкций, мишенями которых являются ОХ40 и FAP. в формате 2+1
4.5.1 Связывание с FAP (поверхностный плазмонный резонанс)
Способность биспецифических конструкций связываться с FAP человека, мышей и обезьян циномолгус оценивали с помощью поверхностного плазмонного резонанса (SPR). Все SPR-эксперименты осуществляли с помощью устройства Biacore Т200 (фирма Biacore) при 25°С, используя HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore).
Меченный с помощью His димерный FAP человека, мышей или обезьян циномолгус захватывали на СМ5-чипе (фирма GE Healthcare) с иммобилизированным антителом к His (фирма Qiagen, каталожный №34660) путем инъекции 500 нМ huFAP в течение 60 с со скоростью потока 10 мкл/мин, 10 нМ мышиного FAP в течение 20 с со скоростью потока 20 мкл/мин и 10 нМ cynoFAP в течение 20 с со скоростью потока 20 мкл/мин. Применяемые уровни иммобилизации антитела к His соответствовали вплоть до 18000 резонансных единиц (RU). Схема анализа представлена на фиг. 12Д.
После стадии захвата биспецифические конструкции, а также контрольные молекулы немедленно пропускали по поверхности чипа в концентрациях в диапазоне от 0,78 до 100 нМ со скоростью потока 30 мкл/мин в течение 280 с и с фазой диссоциации 180 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный FAP. Аффинность определяли, осуществляя подгонку кривой к модели связывания 1:1 Ленгмюра. Для двухвалентного связывания использования такую же подгонку к модели 1:1, что позволяло получать кажущуюся величину KD.

Примечание: все величины KD зависели от конкретных условий эксперимента.
4.5.2 Связывание с человеческим ОХ40 - конкурентное связывание с двухвалентным связыванием ОХ40
Для подтверждения способности обоих двух анти-ОХ40 Fab-доменов связываться с huOX40 аналогично связыванию IgG, применяли клеточный FRET (флуоресцентный резонансный перенос энергии)-анализ (фирма TagLite). Для этого 10000 Hek293 EBNA клеток/на лунку, трансфектированных слиянием huOX40-SNAP и меченных с помощью применяемого в качестве донора в FRET-анализе тербия (фирма Cisbio), смешивали с 0,2 нМ (49В4) IgG, меченным применяемым в FRET-анализе акцептором d2 (фирма Cisbio). Кроме того, добавляли, используя разведение концентраций от 0,004 до 750 нМ, либо (49В4) IgG, либо биспецифическую конструкцию 49В4/28Н1 (2+1), и инкубировали в течение 2-4 ч при КТ. Флуоресцентный сигнал измеряли при 620 нМ для донора флуоресценции (тербий) и при 665 нМ для красителя-акцептора флуоресценции (М100 Pro, фирма Tecan). Рассчитывали соотношение 665/620×1000 и вычитали значение для контроля (только клетки) (фиг. 13Н). Для определения ЕС50 результаты анализировали с помощью Graph Pad Prism5. Установленные величины ЕС50 представлены в таблице 33. Для всех конструкций в формате 2+1 обнаружены величины ЕС50, сходные с величинами для двухвалентного IgG, в указанных экспериментальных условиях.

4.5.3 Одновременное связывание с ОХ40 и FAP
Способность связываться одновременно с конструкцией человеческий OX40-Fc (kih) и с человеческим FAP оценивали с помощью поверхностного плазмонного резонанса (SPR). Все SPR-эксперименты осуществляли с помощью устройства Biacore Т200 (фирма Biacore) при 25°С, используя HBS-EP в качестве подвижного буфер (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore).
Конструкцию биотинилированный человеческий ОХ40 в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровни иммобилизации, соответствующие вплоть до 1000 резонансных единиц (RU).
Биспецифические антитела, мишенями которых являются ОХ40 и FAP, пропускали через проточные ячейки в концентрации 250 нМ со скоростью потока 30 мкл/мин в течение 90 с и режим диссоциации устанавливали на 0 с. Человеческий FAP инъецировали в проточные ячейки в качестве второго аналита со скоростью потока 30 мкл/мин в течение 90 с в концентрации 250 нМ (фиг. 12Е). Мониторинг диссоциации комплекса осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Все биспецифические конструкции обладали способностью связываться одновременно с человеческим ОХ40 и человеческим FAP (фиг. 13К-13М).
4.5.4 Связывание на клетках
4.5.4.1 Связывание с наивными в сравнении с активированными человеческими РВМС биспецифических антител, мишенями которых являются ОХ40 и FAP
Человеческие РВМС выделяли путем центрифугирования в градиенте плотности фикола согласно методу, описанному в примере 2.1.2. РВМС применяли непосредственно после выделения (связывание на покоящихся человеческих РВМС) или их стимулировали для обеспечения высокого уровня экспрессии человеческого ОХ40 на клеточной поверхности Т-клеток (связывание на активированных человеческих РВМС). Для этого наивные РВМС культивировали в течение 4 дней в среде для Т-клеток, дополненной 200 ед./мл пролейкина и 2 мкг/мл ФГА-L, в 6-луночном планшете для культуры ткани и затем 1 день в предварительно сенсибилизированных 6-луночных планшетах для культуры ткани [4 мкг/мл антитела к человеческому CD3 (клон ОКТЗ) и 2 мкг/мл антитела к человеческому CD28 (клон CD28.2)] в среде для Т-клеток, дополненной 200 ед./мл пролейкина, при 37°С и 5% СО2.
Для детекции ОХ40 наивные человеческие РВМС и активированные человеческие РВМС смешивали. Для того, чтобы отличать наивные клетки от активированных человеческих РВМС, покоящиеся клетки метили перед анализом связывания с помощью красителя для оценки пролиферации клеток eFluor670 (фирма eBioscience, каталожный №65-0840-85). Затем смесь 1:1 1×105 наивных клеток, меченных eFluor670 человеческих РВМС, и немеченых активированных человеческих РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185) и осуществляли анализ связывания согласно методу, описанному в примере 2.1.2. Затем смесь 1:1 1×105 наивных клеток, меченных eFluor670 человеческих РВМС, и немеченых активированных человеческих РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185) и осуществляли анализ связывания согласно методу, описанному в разделе 2.1.2
Первичные антитела представляли собой титрованные конструкции антител к ОХ40, инкубированные в течение 120 мин при 4°С. Раствор вторичного антитела представлял собой смесь флуоресцентно меченого антитела к человеческому CD4 (клон RPA-T4, мышиный IgG1k, фирма BioLegend, каталожный №300532), антитела к человеческому CD8 (клон RPa-T8, мышиный IgG1k, фирма BioLegend, каталожный №3010441) и конъюгированного с флуоресцеинизотиоцианатом (ФИТЦ) F(ab')2-фрагментом козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), инкубированного в течение 30 мин при 4°С в темноте. Содержимое планшетов окончательно ресуспендировали в 90 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598) и получали данные в этот же день с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 14К и14С, ни одна из антигенсвязывающих молекул, специфических в отношении ОХ40, не связывалась с покоящимися человеческими CD4+-Т-клетками или CD8+-T-клетками. В противоположность этому, все антигенсвязывающие молекулы связывались с активированными CD8+- или CD4+-Т-клетками. Связывание с CD4+-Т-клетками оказалось существенно более сильным, чем с CD8+-T-клетками, аналогично тому, что уже было описано в примере 4.3.2.1. Как продемонстрировано на фиг. 14Л и 14Н, двухвалентные нацеленные на FAP конструкции антитела к ОХ40 обладали более сильными характеристиками связывания с ОХ40-позитивными клетками, чем соответствующие клоны в формате одновалентного антитела, благодаря повышению авидности. Все сконструированные форматы 2+1 связывались со сходной силой с ОХ40-позитивными клетками вне зависимости от связывающего фрагмента второй специфичности (фиг. 14П и 14С).
4.5.4.2 Связывание с экспрессирующими человеческий FAP опухолевыми клетками
Связывание с находящемся на клеточной поверхности FAP тестировали с использованием экспрессирующих человечески фибробласт-активированный белок (huFAP) клеток WM266-4 (АТСС CRL-1676). Отсутствие связывания с ОХ40-негативными FAP-негативными опухолевыми клетками тестировали с использованием клеток А549 NucLight™ Red (фирма Essenbioscience, каталожный №4491), экспрессирующих флуоресцентный белок NucLight Red, окрашивание которым ограничено ядрами, что позволяло отличать их от немеченых позитивных по человеческому FAP клеток WM266-4. Родительские клетки А549 (АТСС CCL-185) трансдуцировали лентивирусом Essen CellPlayer NucLight Red (фирма Essenbioscience, каталожный №4476; EF1α, пуромицин) при MOI 3 (ТЕ/клетку) в присутствии 8 мкг/мл полибрена, следуя стандартному протоколу Essen. Это позволяло достигать эффективности трансдукции ≥70%.
Смесь из 5×104 немеченых клеток WM266-4 и немеченых клеток А549 NucLight™ Red в FACS-буфере добавляли в каждую лунку круглодонных 96- луночных планшетов для суспензионных клеток (фирма Greiner bio-one, Cellstar, каталожный №650185) и анализ связывания осуществляли согласно методу, описанному в примере 4.3.2.2. Планшеты центрифугировали в течение 4 мин при 400 × g при 4°С и супернатанты отбрасывали. Клетки промывали однократно 200 мкл D-ЗФР и дебрисы ресуспендировали путем кратковременного и осторожного встряхивания. Все образцы ресуспендировали в 50 мкл/на лунку холодного (4°С) FACS-буфера, содержащего биспецифические антигенсвязывающие молекулы (первичное антитело) в указанном диапазоне концентраций (титрованных) и инкубировали в течение 120 мин при 4°С. Затем клетки промывали 4 раза, используя 200 мкл (4°С) FACS-буфера, и ресуспендировали путем кратковременного встряхивания. Затем клетки окрашивали, используя 25 мкл/на лунку холодного (4°С) раствора конъюгированного с флуоресцеинизотиоцианатом (ФИТЦ) F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098) в качестве вторичного антитела и инкубировали в течение 30 мин при 4°С в темноте. Содержимое планшетов окончательно ресуспендировали в 90 мкл/лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и получали данные в этот же день с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 15В и 15Д, нацеленные на FAP одно- и двухвалентные анти-ОХ40 антигенсвязывающие молекулы эффективно связывались с экспрессирующими человеческий FAP клетками-мишенями. При этом, только у нацеленных на FAP одно- и двухвалентных анти-ОХ40 антигенсвязывающих молекул обнаружены непосредственные таргетирующие опухоль свойства. Для обладающего высокой аффинностью связывания с FAP клона 4В9 в одновалентной конструкции продемонстрировано более сильное связывание с человеческим FAP, чем для соответствующего связывающего FAP клона 28Н1 в таком же одновалентном формате (фиг. 15Д). Никакого связывания с FAP не обнаружено у 2+1-конструкций, у которых отсутствовал FAP-связывающий домен (незакрашенный кружок, фиг. 15Е). Величины ЕС50, характеризующие связывание с активированными человеческими CD4+-T-клетками и FAP-позитивными опухолевыми клетками, обобщены в таблице 34.
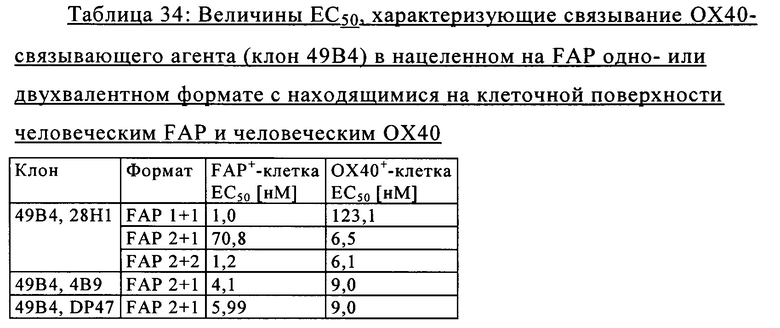
Пример 5
Функциональные свойства биспецифических молекул антител, связывающих человеческий ОХ40
5.1 Клетки HeLa, экспрессирующие человеческий ОХ40 и репортерный ген NF-κВ-люциферазы
Как продемонстрировано в примере 3.1, ограниченная зависящая от дозы NFkB-активация индуцировалась при добавлении антител к ОХ40 P329GLALA huIgG1 к репортерной линии клеток HeLa_hOX40_NFkB_Luc1. Перекрестная гипер-сшивка антител к ОХ40 с помощью античеловеческих IgG-специфических вторичных антител сильно повышала индукцию опосредуемой NFκB активации люциферазы в зависимости от концентрации. Таким образом, при создании изобретения тестировали активирующую способность NFκB отобранных анти-ОХ40-связывающих агентов (8Н9, 1G4, 49В9) в одновалентном и двухвалентном нацеленном на FAP формате индивидуально и с перекрестной гипер-сшивкой конструкций с помощью либо вторичного антитела, либо FAP+-линии опухолевых клеток.
Как описано в примере 3.1, прикрепленные клетки HeLa_hOx40_ NFκB_Luc1 культивировали в течение ночи при клеточной плотности 0,3×105 клеток на лунку и стимулировали в течение 5-6 ч средой для анализа, содержащей титрованные анти-ОХ40-связывающие агенты (клон 8Н9 и 1G4) в нацеленном на FAP одновалентном (FAP 1+1) и двухвалентном (FAP 2+2) формате и в виде конструкций P329GLALA hu IgG1. Для тестирования воздействия перекрестной гипер-сшивки с помощью вторичных антител добавляли 25 мкл/лунку среды, содержащей в качестве вторичного антитела F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-специфический (фирма Jackson ImmunoResearch, каталожный №109006098) в соотношении 1:2 (первичное антитело к вторичному антителу). Для оценки воздействия перекрестной гипер-сшивки с помощью связывания с находящемся на клеточной поверхности FAP 25 мкл/на лунку среды, содержащей опухолевые FAP+-клетки (WM266-4 и/или NIH/3T3-huFAP, клон 39), совместно культивировали в соотношении 2:1 (двукратное превышение опухолевых FAP+-клеток относительно репортерных клеток на лунку). Количество активированного NFκB оценивали путем измерения испускания света с использованием системы для анализа люциферазы 1000 и репортерного лизирующего буфера (оба фирмы Promega, каталожный № Е4550 и каталожный № Е3971) согласно методу, описанному в примере 3.1.
Как продемонстрировано на фиг. 16А-16Ж, присутствие всех конструкций анти-ОХ40 индуцировало ограниченную активацию NFkB. Перекрестная гипер-сшивка через вторичное huIgG Fcγ-специфическое антитело повышало активацию NFkB при использовании обоих связывающих агентов в формате huIgG1 P329GLALA, а также в нацеленном на FAP одно/двухвалентном формате. При этом одновалентное связывание с ОХ40 оказалось менее эффективным, чем двухвалентное связывание с ОХ40, что демонстрирует необходимость в олигомеризации ОХ40-рецептора для полной активации оси передачи сигналов ОХ40. Экспрессирующая FAP опухолевая клетка сильно повышала индукцию опосредуемой NFκB активации люциферазы в зависимости от концентрации, когда применяли нацеленные на FAP молекулы (закрашенный квадрат и треугольник). Такое действие не обнаружено, когда эти же клоны применяли в «ненацеленном» формате huIgG1 P329GLALA, поскольку конструкция не могла подвергаться дополнительной перекрестной гипер-сшивке с помощью опухолевых FAP+-клеток. И в этом случае двухвалентная молекула превышала по активности одновалентную молекулу. Высокий уровень экспрессии FAP гарантировал более высокое перекрестное сшивание и в результате более высокое агонистическое действие нацеленной на FAP конструкции (по сравнению с обозначенной закрашенным ромбом линией с высоким уровнем экспрессии FAP NIH/3T3-huFAP, клон 39 и FAP-позитивными клетками WM266-4). Для двухвалентной нацеленной на FAP конструкции обнаружен в присутствии опухолевых FAP+-клеток пик активности при концентрации ~ от 0,1 до 1 нМ. Дальнейшей повышение концентрации соединения фактически снижало его способность индуцировать NFκB. Наиболее вероятно, двухвалентные конструкции, связывающиеся только с одной мишенью (FAP или ОХ40), присутствовали в более высоких концентрациях, превосходя конструкции, связывающиеся одновременно с FAP и ОХ40. В свою очередь, указанное уменьшение перекрестного сшивания снижало агонистическую передачу сигналов ОХ40.
В другом эксперименте прикрепленные клетки HeLa_hOX40_NFκB_Luc1 культивировали в течение ночи при клеточной плотности 0,2×105 клеток на лунку и стимулировали в течение 5 ч средой для анализа, содержащей титрованные анти-ОХ40-связывающие агенты (клон 49В9) в нацеленном на FAP одновалентном (FAP 1+1) и двухвалентном (FAP 2+1 и FAP 2+2) формате и в виде конструкций P329GLALA hu IgG1. Таргетинг FAP в формате 2+1 обеспечивался двумя различными связывающими FAP клонами, 28Н1 с низкой аффинностью к FAP и 4В9 с высокой аффинностью к FAP. Для оценки воздействия перекрестной гипер-сшивки с помощью вторичных антител добавляли 25 мкл/лунку среды, содержащей в качестве вторичного антитела F(ab')2-фрагмент козьего IgG против человеческого IgG, Fcγ-специфический (фирма Jackson ImmunoResearch, каталожный №109006098) в соотношении 1:2 (первичное антитело к вторичному антителу). Для оценки воздействия перекрестной гипер-сшивки с помощью связывания с находящимся на клеточной поверхности FAP 25 мкл/на лунку среды, содержащей опухолевые FAP+-клетки (NIH/3T3-huFAP, клон 19), совместно культивировали в соотношении 2:1 (четырехкратное превышение количества опухолевых FAP+-клеток относительно количества репортерных клеток на лунку). Количество активированного NFκB оценивали путем измерения испускания света с использованием системы для анализа люциферазы 1000 и репортерного лизирующего буфера (оба фирмы Promega, каталожный № Е4550 и каталожный № Е3971) согласно методу, описанному в примере 3.1.
Как продемонстрировано на фиг. 16З-16О, в этом эксперименте также продемонстрировано, что присутствие всех анти-ОХ40 конструкций индуцировало ограниченную активацию NFκB. Перекрестная гипер-сшивка через вторичное huIgG Fcγ-специфическое антитело повышало указанную активацию NFκB при использовании всех связывающих агентов в нацеленном на FAP одно/двухвалентном формате. При этом одновалентное связывание с ОХ40 оказалось менее эффективным, чем двухвалентное связывание с ОХ40, что демонстрирует необходимость в олигомеризации ОХ40-рецептора для полной активации оси передачи сигналов ОХ40 (фиг. 16К, сравнение величин ЕС50). Экспрессирующие FAP опухолевые клетки сильно повышали индукцию опосредуемой NFκB активации люциферазы в зависимости от концентрации, когда применяли нацеленные на FAP молекулы (закрашенный квадрат, треугольник, наполовину закрашенные кружки). Такое действие не обнаружено, когда 2+1-формат FAP-связывающего фрагмента заменяли на несвязывающий модуль DP47 (незакрашенный кружок), поскольку конструкция не могла подвергаться дополнительной перекрестной гипер-сшивке с помощью опухолевых FAP+-клеток. И в этом случае двухвалентная молекула превышала по активности одновалентную молекулу (при сравнении величин ЕС50). Для одновалентной нацеленной на FAP и двухвалентной нацеленной на ОХ40 конструкции (2+1) обнаружена в присутствии опухолевых FAP+-клеток наиболее высокая активность на уровне плато. В противоположность одновалентной ОХ40-связывающей 1+1-конструкции двухвалентная связывающая ОХ40 конструкция в формате 2+1 обладала повышенной агонистической активностью в результате олигомеризации ОХ40 (воздействие на величину ЕС50). Однако, вероятно, что из-за одновалентного связывания 2+1-конструкций с FAP могло перекрестно сшиваться удвоенное количество указанных молекул по сравнению с двухвалентным FAP-связывающим форматом 2+2, что можно объяснить более высокой активацией ОХ40 на уровне плато (фиг. 16Л, более высокий уровень плато).
5.2 Опосредуемая ОХ40 костимуляция субоптимально индуцированных TCR предварительно активированных человеческих СР4-Т-клеток
Отобранные связывающие агенты (клоны 8Н9 и 1G4) в нацеленном на FAP одновалентном или двухвалентном формате тестировали в отношении их способности совместно активировать Т-клетки при их иммобилизации на поверхности.
Как описано в примере 3.2, предварительно активированные меченные CFSE ОХ40-позитивные CD4-Т-клетки стимулировали в течение 72 ч взятыми в субоптимальной концентрации иммобилизированными на планшете антителами к CD3 в присутствии титрованных антител к ОХ40, иммобилизированных на поверхности планшета. Воздействия на выживаемость и пролиферацию Т-клеток анализировали, осуществляя мониторинг общего количества клеток и разведения CFSE в живых клетках с помощью проточной цитометрии. Дополнительно клетки окрашивали совместно флуоресцентно мечеными антителами против маркеров активации и дифференцировки Т-клеток, например CD127, CD45RA, Tim-3, CD62L и самого ОХ40.
Костимуляция с помощью иммобилизированных на планшете биспецифических анти-ОХ40 антигенсвязывающих молекул сильно повышала в зависимости от дозы субоптимальную стимуляцию предварительно активированных человеческих CD4-Т-клеток при использовании иммобилизированного на планшете антитела к человеческому CD3 (фиг. 17А и 17Б). Пролиферация Т-клеток была более сильной, характеризуясь более зрелым фенотипом с более высоким процентом CD62Llow, Tim-3-позитивных и ОХ40- позитивных активированных клеток (повышение гранулярности (SSC) обнаружено только для клона 8Н9). При этом одновалентное связывание (символ в виде треугольника) с ОХ40 оказалось менее эффективным, чем двухвалентное связывание (символ в виде квадрата) с ОХ40, что свидетельствует о необходимости олигомеризации ОХ40-рецептора для полной активации оси передачи сигналов ОХ40. Изменения, составляющие половину от максимального всех проанализированных параметров Т-клеточной активации, достигались в диапазоне концентраций от 3 до 2300 пМ, и они обобщены для клона 1G4 на фиг.
18А-18Г и в таблице 35.

5.3 Опосредуемая ОХ40 костимуляция субоптимально индуцированных TCR покоящихся человеческих РВМС и перекрестная гипер-сшивка с помощью FAP, присутствующего на клеточной поверхности
Как было продемонстрировано в примере 5.1, добавление опухолевых FAP+-клеток может приводить к сильному повышению активности NFkB, индуцируемой нацеленными на FAP одно- и двухвалентными конструкциями анти-ОХ40, в человеческих ОХ40-позитивных репортерных линиях клеток посредством выраженной олигомеризации рецепторов ОХ40. Кроме того, при создании изобретения тестировали нацеленные на FAP одновалентные (1+1) и двухвалентные (2+1, 2+2) конструкции анти-ОХ40 в присутствии клеток NIH/3T3-huFAP, клон 39, в отношении их способности поддерживать субоптимальную стимуляцию TCR покоящихся человеческих РВМС-клеток.
Препараты человеческих РВМС содержали (1) покоящиеся ОХ40- негативные CD4+- и CD8+-Т-клетки и (2) антигенпрезентирующие клетки, несущие различные молекулы Fcγ-рецептора на своей поверхности, например, В-клетки и моноциты. Антитело к человеческому CD3 человеческого IgG1-изотипа могло связываться посредством своей Fc-области с присутствующими молекулами Fcγ-рецептора и опосредовать пролонгированную активацию TCR на покоящихся ОХ40-негативных CD4+- и CD8+-Т-клетках. Затем эти клетки в течение нескольких часов начинали экспрессировать ОХ40. Функциональные соединения-агонисты ОХ40 могут передавать сигнал через рецептор ОХ40, присутствующий на активированных CD8+- и CD4+-Т-клетках, и поддерживать опосредуемую TCR стимуляцию.
Покоящиеся меченные CFSE человеческие РВМС стимулировали в течение 4-5 дней взятым в субоптимальной концентрации антителом к CD3 в присутствии подвергнутых облучению клеток FAP+-NIH/3T3-huFAP, клон 39 и титрованных анти-ОХ40 конструкций. Анализировали воздействия на выживание и пролиферацию Т-клеток, осуществляя с помощью проточной цитометрии мониторинг общего количества клеток и концентрации CFSE в живых клетках. Дополнительно клетки совместно окрашивали флуоресцентно мечеными антителами к маркеру Т-клеточной активации и созревания CD25, гранзиму В, CD62L и CD127. Во втором эксперименте клетки совместно окрашивали флуоресцентно мечеными антителами к маркеру Т-клеточной активации и созревания CD25 и Tim-3.
Собирали клетки мышиных эмбриональных фибробластов NIH/3T3-huFAP, клон 39 (см. пример 4.3.2.2) с использованием буфера для диссоциации клеток (фирма Invitrogen, каталожный №13151-014) в течение 10 мин при 37°С. Клетки однократно промывали D-ЗФР. Клетки NIH/3T3-huFAP, клон 39 культивировали при плотности 0,2×105 клеток/лунку в среде для Т-клеток в стерильном круглодонном 96-луночном планшете для прикрепленной культуры ткани (фирма ТРР, каталожный №92097) в течение ночи в инкубаторе (Hera Cell 150) при 37°С и в атмосфере, содержащей 5% СО2. На следующий день клетки облучали в рентгеновском облучателе дозой 4500 рад для предупреждения последующего избыточного разрастания человеческих РВМС под влиянием линии опухолевых клеток.
Человеческие РВМС выделяли путем центрифугирования в градиенте плотности фиколла и метили с помощью CFSE согласно методу, описанному в примере 2.1.2. Клетки добавляли при плотности 0,75×105 клеток на лунку (первый эксперимент) или 0,5×105 клеток на лунку (второй эксперимент). Добавляли антитело к человеческому CD3 (клон V9, человеческий IgG1) в конечной концентрации 10 нМ и нацеленные на FAP одно- и двухвалентные анти-ОХ40 антигенсвязывающие молекулы в указанных концентрациях. Клетки активировали в течение 4-5 дней при 37°С и в атмосфере, содержащей 5% СО2, в инкубаторе (Hera Cell 150). Затем окрашивали поверхность клеток конъюгированными с флуоресцентным красителем антителами к человеческому CD4 (клон RPA-T4, фирма BioLegend, каталожный №300532), CD8 (клон RPa-Т8, фирма BioLegend, каталожный №3010441), CD25 (клон М-А251, фирма BioLegend, каталожный №356112), CD127 (клон A019D5, фирма Biolegend, каталожный №351234) и CD62L (клон DREG 56, фирма Biolegend, каталожный №304834) или Tim-3 (клон F38-E2E, фирма Biolegend, каталожный №345012) в течение 30 мин при 4°С. Для повышения проницаемости клеточной мембраны клеточный дебрис промывали дважды FACS-буфером, затем ресуспендировали в 50 мкл/на лунку свежеприготовленного буфера FoxP3 Fix/Perm (фирма eBioscience, каталожный №00-5123 и 00-5223) в течение 45 мин при комнатной температуре в темноте. После трехкратной промывки Perm-Wash-буфером (фирма eBioscience, каталожный №00-8333-56) осуществляли внутриклеточное окрашивание клеток, используя 25 мкл/на лунку Perm-Wash-буфера, содержащего антитело к человеческому гранзиму В (клон GB-11, фирма BD Bioscience, каталожный №561142), в течение 1 ч при комнатной температуре в темноте. Клетки ресуспендировали в 85 мкл/лунку FACS-буфера и данные получали с использованием проточного цитометра Fortessa с 5 лазерами (фирма BD Bioscience, программное обеспечение DIVA).
Как продемонстрировано на фиг. 20А-203, костимуляция «ненацеленной» конструкцией анти-ОХ40 (8Н9) huIgG1 P329GLALA (незакрашенный квадрат) не поддерживала субоптимально стимулированные TCR CD4- и CD8-Т-клетки. Перекрестная гипер-сшивка нацеленных на FAP одно- (закрашенный треугольник) и двухвалентных (закрашенный квадрат) анти-ОХ40 конструкций благодаря присутствию клеток NIH/3T3-huFAP, клон 39 сильно повышала пролиферацию, выживаемость и индуцировала повышенный активированный фенотип человеческих CD4- и CD8-Т-клеток. В случае высоко аффинного клона 8Н9 (фиг. 20А-203) одновалентная и двухвалентная биспецифическая антигенсвязывающая молекула обладала сопоставимой способностью сохранять субоптимальную стимуляцию TCR. У обеих конструкций и в этом случае обнаружен пик активности при ~0,1-1 нМ и пониженный ответ при более высокой концентрации. Аналогично данным, полученным при применении NFκB-репортерной клеточной линии (фиг. 16А-16Н), это может являться следствием конкуренции за связывание с мишенью между конструкциями, которые связывались только с FAP или ОХ40, и теми, которые связывались одновременно с обеими мишенями и таким образом, имели необходимое перекрестное сшивание. Этот эффект является менее выраженным, когда тестировали обладающий низкой аффинностью клон 1G4 в нацеленных на FAP одновалентных конструкциях в сравнении с двухвалентными анти-ОХ40 конструкциями (фиг. 21А-21В). При этом одновалентное антитело четко уступало по активности двухвалентной конструкции. Это удалось наиболее четко продемонстрировать, когда агонистическую способность каждой конструкции количественно оценивали по площади под кривой и строили график одной относительно другой (фиг. 21А-21В).
Результаты, полученные при осуществлении второго эксперимента, представлены на фиг. 21Г-21З и 21К-21О соответственно. Как продемонстрировано на фиг. 21Г-213, костимуляция ненацеленным форматом анти-ОХ40 (49В4) 2+1 DP47 (незакрашенный кружок) не поддерживала субоптимально стимулированные TCR CD4- и CD8-Т-клетки. Перекрестная гипер-сшивка нацеленных на FAP одно- (закрашенный треугольник) и двухвалентных (закрашенный квадрат, наполовину закрашенный кружок) анти-ОХ40 конструкций благодаря присутствию клеток NIH/3T3-huFAP, клон 39 сильно повышала выживаемость и индуцировала повышенный активированный фенотип человеческих CD4- и CD8-Т-клеток. Одновалентная анти-ОХ40 конструкция (1+1; закрашенный треугольник) обладала меньшей способностью сохранять TCR-стимуляцию, чем двухвалентные анти-ОХ40 нацеленные конструкции (наполовину закрашенный кружок, закрашенный квадрат). Двухвалентная в отношении связывания FAP 2+2-конструкция в более низких концентрациях уже обладала способностью сохранять субоптимальную TCR-стимуляцию по сравнению с одновалентными касательно связывания с FAP 2+1-конструкциями. В формате 2+1 обладающий высокой аффинностью связывания FAP клон 4В9 явно превышал по силе связывания обладающий низкой аффинностью клон 28Н1 (фиг. 21К-21О). Это позволяет предположить, что величины ЕС50, характеризующие биологическую активность, были обусловлены связыванием с FAP (2+2>2+1 (4В9)>2+1 (28Н1)). Это удалось наиболее четко продемонстрировать, когда агонистическую способность каждой конструкции количественно оценивали для анализируемого маркера по площади под кривой и сравнивали друг с другом соответствующие графики (фиг. 21Р).
5.4 Предупреждение ADCC путем применения формата человеческого IgG1 P329GLALA
Антитела человеческого IgG1-класса могут индуцировать антитело- обусловленную клеточнозависимую цитотоксичность (ADCC) позитивных по антигену клеток-мишеней благодаря связыванию с Fcγ-рецепторами (FcγR) на ADCC-компетентных клетках, например NK-клетках. Таким образом, компетентное в отношении ADCC антитело к ОХ40 может опосредовать лизис недавно активированных ОХ40+-Т-клеток и уменьшать пул реактивных в отношении опухоли Т-клеток. Интродукция мутации IgG1P329GLALA в Fc-область антитела предупреждает связывание с FcγR (публикация международной заявки на патент WO 2012/130831 А1) и тем самым ADCC в присутствии NK-клеток. Однако связывание с FcN-рецептором не изменяется, что гарантирует IgG-подобную фармакокинетику антитела.
Отобранные клоны конвертировали в канонический формат человеческого IgG1 для оценки их способности индуцировать ADCC. Для оценки компетентности в отношении ADCC меченные с помощью PKh26 ОХ40-позитивные опухолевые клетки (репортерная клеточная линия HeLa_hOX40_NFkB_Luc1) и свежевыделенные NK-клетки совместно культивировали при соотношении Е к Т 3:1 в присутствии ряда серийных разведений антител к ОХ40 (формат человеческого IgG1 или человеческого IgG1-P329GLALA). Количественную оценку опосредуемой NK-клетками ADCC осуществляли на основе высвобождения лактатдегидрогеназы (LDH) и количества DAPI-позитивных клеток.
В целом, метод состоял в следующем: клетки HeLa_hOX40_NFkB_Luc1 (пример 3.1) метили с помощью набора PKH-26 Red Fluorescence Cell linker (фирма Sigma, каталожный № PKH26GL) согласно методу, описанному в примере 2.3.2. Меченные PKH-26 клетки HeLa_hOx40_NFkB_luc1 высевали с плотностью 0,5×105 клеток на лунку в среду AIM V (фирма Gibco, каталожный №12055-09) стерильного 96-луночного круглодонного планшета для адгезионной культуры ткани (фирма ТРР, каталожный №92097) в течение ночи при 37°С и 5% CO2 в инкубаторе (Hera Cell 150). На следующий день человеческие РВМС выделяли путем центрифугирования в градиенте плотности фикола согласно методу, описанному в примере 2.1.2. Выделение NK-клеток осуществляли с используя набор для выделения MACS-негативных человеческих NK-клеток (фирма Miltenyi Biotec, каталожный №130-092-657) согласно инструкциям производителя. NK-клетки добавляли с плотностью 1,5×105 клеток на лунку в среду AIM V до достижения соотношения Е к Т, составляющего 3:1. Антитела к ОХ40 (формат человеческого IgG1 или человеческого IgG1 P329GLALA) добавляли в указанных концентрациях и планшеты инкубировали в течение ночи при 37°С и 5% СО2 в инкубаторе (Hera Cell 150). Через 4 ч отбирали пробы супернатанта по 100 мкл для осуществления анализа LDH и среду заменяли свежей средой AIM-V. LDH-активность количественно оценивали, используя набор для детекции цитотоксичности - LDH (фирма Roche, каталожный №11644793001) согласно инструкциям производителя, применяя ридер для микропланшетов SpectraMax М5/М5е (фирма Molecular Devices) (фильтр: 490-650 нм, время интегрирования 1 мс).
Через 24 ч клетки открепляли с помощью трипсина. Клетки окрашивали мечеными антителами к человеческому CD56 (клон NCAM16.2, мышиный IgG1 к, фирма BioLegend, каталожный №562751), человеческому CD25 (клон МА251, мышиный IgG1 к, фирма BioLegend, каталожный №356116) и человеческому CD69 (клон FN50, мышиный IgG1 к, фирма BioLegend, каталожный №356116), инкубировали в течение 20 мин при 4°С в темноте. Содержимое планшетов окончательно ресуспендировали в 80 мкл/на лунку FACS-буфера, содержащего 0,2 мкг/мл DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), данные получали с использованием устройства LSR-Fortessa с 5 лазерами (фирма BD Bioscience, программное обеспечение DIVA).
Все антитела в формате ADCC-компетентного человеческого IgG1 обладали способностью индуцировать лизис ОХ40-позитивных клеток в степени, аналогичной степени, характерной для ADCC-компетентного референс-антитела (GA201) против EGFR. Клоны в формате IgGP329GLALA не могли опосредовать ADCC (см. фиг. 22А и 22Б).
Интродукция мутации IgG1 P329GLALA в Fc-область указанных в настоящем описании нацеленных форматов предупреждала связывание с FcγR (публикация международной заявки на патент WO 2012/130831 А1), и тем самым ADCC в присутствии NK-клеток. Однако связывание с FcN-рецептором не изменялось, что обеспечивало IgG-подобную фармакокинетику антитела. В противоположность уже известным антителам к ОХ40, перекрестная гипер-сшивка, необходимая для оптимальной агонистической передачи сигналов ОХ40, присутствовала в биспецифических антителах, предлагаемых в изобретении, благодаря связыванию с FAP-позитивными опухолевыми клетками или фибробластами. Наличие FAP-позитивности либо на ассоциированных с опухолью фибробластах, либо на самих опухолевых клетках, описано при многих опухолевых состояниях. Таким образом, биспецифический формат обладает потенциалом сильного опосредуемого ОХ40 агонистического действия в микроокружении опухоли в отсутствии системной активации, которая может предупреждать связанную с иммунитетом токсичность. В отличие от канонических антител к ОХ40, биспецифические антигенсвязывающие молекулы, предлагаемые в изобретении, не индуцировали ADCC активированных незадолго до этого ОХ40-позитивных эффекторных Т-клеток. Таким образом, биспецифические антитела потенциально могут реактивировать ранее существовавший, но подавленный адаптивный иммунный ответ против опухолевых клеток и их можно эффективно применять для лечения страдающих раком пациентов.
Пример 6
Создание антител к 4-1ВВ и применяемых в качестве «инструментов» связывающих агентов
6.1 Получение, очистка и характеризация антигенов и скрининг «инструментов» для создания новых 4-1ВВ-связывающих агентов с помощью фагового дисплея
Последовательности ДНК, кодирующие эктодомены 4-1ВВ человека, мышей или обезьян циномолгус (таблица 36), субклонировали в рамке считывания с СН2- и СН3-доменами тяжелой цепи человеческого IgG1 на цепи с «выступом» (Merchant и др., 1998). Сайт расщепления протеазой AcTEV интродуцировали между эктодоменом антигена и Fc человеческого IgG1. Avi-метку для направленного биотинилирования интродуцировали на С-конец конструкции антиген-Fc с «выступом». Комбинация конструкции антиген-Fc- цепь с «выступом», которая содержит мутации S354C/T366W, с Fc-цепью с «впадиной», которая содержит мутации Y349C/T366S/L368A/Y407V, позволяет создавать гетеродимер, который включает единичную копию содержащей эктодомен 4-1ВВ цепи, получая тем самым мономерную форму сцепленного с Fc антигена (фиг. 1А). В таблице 37 представлены последовательности кДНК и аминокислотные последовательности слитых конструкций антиген-Fc.



Все последовательности, кодирующие слитые молекулы 4-1BB-Fc, клонировали в плазмидном векторе, в котором экспрессия вставки контролируется промотором MPSV и который содержит синтетическую сигнальную полиА-последовательность, локализованную на 3'-конце CDS. Кроме того, вектор содержал последовательность OriP EBV для поддержки эписомальной формы плазмиды.
Для получения биотинилированных мономерных слитых молекул антиген/Fc растущие в суспензии клетки HEK293 EBNA на экспоненциальной фазе роста совместно трансфектировали тремя векторами, кодирующими два компонента слитого белка (цепи с «выступом» и «впадиной», а также BirA, фермент, необходимый для реакции биотинилирования. Соответствующие векторы применяли в соотношении 2:1:0,05 («ECD антигена-AcTEV-Fc с «выступом»: «Fc с «впадиной»: «BirA»).
Для получения белка в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210×g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы ресуспендировали в 20 мл CD СНО, содержащей 200 мкг векторной ДНК. После добавления 540 мкл полиэтиленимина (ПЭИ) раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Среду для производства дополняли 5 мкМ кифунензином. Через 1 день после трансфекции в культуру добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210×g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Секретированные белки очищали из супернатантов клеточных культур с помощью аффинной хроматографии с использованием белка А с последующей гель-фильтрацией. Для осуществления аффинной хроматографии супернатант вносили в колонку HiTrap ProteinA HP (CV=5 мл, фирма GE Healthcare), уравновешенную 40 мл буфера, содержащего 20 мМ фосфат натрия, 20 мМ цитрат натрия, рН 7,5. Несвязанный белок удаляли путем промывки буфером, содержащим 20 мМ фосфат натрия, 20 мМ цитрат натрия и 0,5М хлорид натрия (рН 7,5), используя его в объеме, кратном по меньшей 10 объемам колонки. Связанный белок элюировали, используя линейный градиент хлорида натрия (от 0 до 500 мМ), созданный с помощью раствора, содержащего 20 мМ цитрат натрия, 0,01 об. % Твин-20, рН 3,0, который применяли в объеме, кратном 20 объемам колонки. Затем колонку промывали с помощью раствора, содержащего 20 мМ цитрат натрия, 500 мМ хлорид натрия и 0,01 об. % Твин-20, рН 3,0, который применяли в объеме, кратном 10 объемам колонки. Значение рН собранных фракций регулировали, добавляя 1/40 (об./об.) 2М Трис, рН 8,0. Белок концентрировали и фильтровали перед внесением в колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 2 мМ MOPS, 150 мМ хлорид натрия, 0,02% (мас./об.) азида натрия, рН 7,4.
6.2 Отбор 4-1ВВ-специфических антител 12В3, 25G7, 11D5, 9В11 и 20G2 из родовых библиотек F(ab)
Антитела 11D5, 9В11 и 12В3, обладающие специфичностью в отношении 4-1ВВ человека и обезьян циномолгус, отбирали из родовой фаговой дисплейной библиотеки антител (DP88-4) в Fab-формате. Из этой библиотеки отбирали также дополнительное антитело, клон 20G2, обладающее реактивностью в отношении мышиного 4-1ВВ. Эту библиотеку конструировали на основе генов человеческой зародышевой линии, используя V-домен со спаренными Vk1_5 (легкая каппа-цепь) и VH1_69 (тяжелая цепь), содержащий рандомизированный участок последовательности в CDR3 легкой цепи (L3, 3 различные длины) и CDR3 тяжелой цепи (Н3, 3 различные длины). Создание библиотек осуществляли путем сборки 3 амплифицированных с помощью ПЦР фрагментов, применяя ПЦР для «сращивания (генома) перекрывающимися расширениями» (SOE). Фрагмент 1 содержал 5'-конец гена антитела, включая рандомизированный L3, фрагмент 2 представлял собой центральный константный фрагмент, простирающийся от L3 до Н3, а фрагмент 3 содержал рандомизированный Н3 и 3'-область гена антитела. Для создания указанных фрагментов библиотек для библиотеки DP88-4 применяли следующие комбинации праймеров: фрагмент 1 (прямой праймер LMB3 в комбинации с обратными праймерами Vk1_5_L3r_S или Vk1_5_L3r_SY или Vk1_5_L3r_SPY), фрагмент 2 (прямой праймер RJH31 в комбинации с обратными праймер RJH32) и фрагмент 3 (прямые праймеры DP88-v4-4 или DP88-v4-6, или DP88-v4-8 в комбинации с обратным праймером fdseqlong) соответственно. Параметры ПЦР для получения фрагментов библиотек были следующими: 5 мин начальная денатурация при 94°С, 25 циклов по 1 мин при 94°С, 1 мин при 58°С, 1 мин при 72°С и финальное удлинение в течение 10 мин при 72°С. Для ПЦР-сборки при использовании эквимолярных соотношений очищенных на геле единичных фрагментов в качестве матрицы применяли следующие параметры: 3 мин начальная денатурация при 94°С и 5 циклов по 30 с при 94°С, 1 мин при 58°С, 2 мин при 72°С. На этой стадии добавляли внешние праймеры (LMB3 и fdseqlong) и осуществляли 20 дополнительных циклов перед финальным удлинением в течение 10 мин при 72°С. После сборки в достаточном количестве полноразмерных рандомизированных конструкций Fab их расщепляли с помощью NcoI/NheI и встраивали путем лигирования в обработанный аналогичным образом акцепторный фагмидный вектор. Очищенные полученные лигированием конструкции применяли для ~60 трансформаций электрокомпетентных клеток Е. coli TG1. Фагмидные частицы, экспонирующие библиотеку Fab, «спасали» и очищали с помощью ПЭГ/NaCl для использования для селекции. Эти стадии конструирования библиотек повторяли три раза с получением конечной библиотеки размером 4,4×109. Процентные содержания функциональных клонов, определенные на основе детекции С-концевой метки методом дот-блоттинга, составляли 92,6% для легкой цепи и 93,7% для тяжелой цепи соответственно.
Антитело 25G7, обладающее специфичностью в отношении 4-1ВВ человека и обезьян циномолгус, отбирали из родовой фаговой дисплейной библиотеки антител (λ-DP47) в Fab-формате. Эту библиотеку конструировали на основе генов человеческой зародышевой линии, используя V-домен со спаренными: V13_19 (легкая лямбда-цепь) и VH3_23 (тяжелая цепь). Библиотеку рандомизировали в CDR3 легкой цепи (L3) и CDR3 тяжелой цепи (Н3) и собирали из 3 фрагментов, применяя ПЦР для «сращивания (генома) перекрывающимися расширениями» (SOE). Фрагмент 1 содержал 5'-конец гена антитела, включая рандомизированный L3, фрагмент 2 представлял собой центральный константный фрагмент, простирающийся от конца L3 до начала Н3, а фрагмент 3 содержал рандомизированный Н3 и 3'-область Fab-фрагмента. Следующие комбинации праймеров применяли для создания указанных фрагментов библиотеки для библиотеки: фрагмент 1 (LMB3-Vl_3_19_L3r_V/Vl_3_19_L3r_HV/Vl_3_19_L3r_HLV), фрагмент 2 (RJH80-DP47CDR3_ba (mod)) и фрагмент 3 (DP47-v4-4/DP47-v4-6/DP47-v4-8-fdseqlong). Параметры ПЦР для получения фрагментов библиотек были следующими: 5 мин начальная денатурация при 94°С, 25 циклов по 1 мин при 94°С, 1 мин при 58°С, 1 мин при 72°С. Для ПЦР-сборки при использовании эквимолярных соотношений очищенных на геле единичных фрагментов в качестве матрицы применяли следующие параметры: 3 мин начальная денатурация при 94°С и 5 циклов по 30 с при 94°С, 1 мин при 58°С, 2 мин при 72°С. На этой стадии добавляли внешние праймеры и осуществляли 20 дополнительных циклов перед финальным удлинением в течение 10 мин при 72°С. После сборки в достаточном количестве полноразмерных рандомизированных Fab-конструкций их расщепляли с помощью NcoI/NheI параллельно с аналогичной обработкой акцепторного фагмидного вектора. Очищенные полученные лигированием конструкции применяли для ~60 трансформаций электрокомпетентных клеток Е. coli TG1. Фагмидные частицы, экспонирующие библиотеку Fab, «спасали» и очищали с помощью ПЭГ/NaCl для использования для селекции. Получали конечную библиотеку размером 9,5×109. Проценты функциональных клонов, определенные на основе детекции С-концевой метки методом дот-блоттинга, составляли 81,1% для легкой цепи и 83,2% для тяжелой цепи соответственно.
В таблице 38 представлена последовательность родовой фаговой дисплейной библиотеки антител (DP88-4), в таблице 39 представлены последовательности кДНК и аминокислотные последовательности матрицы для библиотеки зародышевой линии DP88-4, а в таблице 40 представлены праймерные последовательности, применяемые для создания матрицы зародышевой линии DP88-4.





В таблице 41 представлена последовательность родовой фаговой дисплейной библиотеки лямбда-DP47 (Vl3_19/VH3_23), применяемой в качестве матрицы для ПЦР. В таблице 42 представлены последовательности кДНК и аминокислотные последовательности матрицы библиотеки зародышевой линии лямбда-DP47 (Vl3_19/VH3_23), а в таблице 43 представлены праймерные последовательности, применяемые для создания библиотеки лямбда-DP47 (Vl3_19/VH3_23).





1: G/D=20%, E/V/S=10%, A/P/R/L/T/Y=5%; 2: G/Y/S=15%, A/D/T/R/P/L/V/N/W/F/I/E=4,6%; 3: G/A/Y=20%, P/W/S/D/T=8%; 4: F=46%, L/M=15%, G/I/Y=8%; 5: K=70%, R=30%.
4-1BB (CD137) человека, мышей и обезьян циномолгус в качестве антигена для селекции методом фагового дисплея и скринингов на основе ELISA и SPR кратковременно экспрессировали в виде N-концевого слияния с мономерным Fc в клетках HEK EBNA и осуществляли in vivo сайтспецифическое биотинилирование посредством совместной экспрессии биотин-лигазы BirA в распознаваемой последовательности с avi-меткой, локализованной на С-конце Fc-области, несущей рецепторную цепь (Fc-цепь с «выступом»).
Раунды селекции (биопэннинг) осуществляли в растворе согласно следующей процедуре. Первая стадия - преклиринг ~1012 фагмидных частиц на планшетах maxisorp, сенсибилизированных 10 мкг/мл нерелевантного человеческого IgG для истощения библиотек антител, распознающих Fc-область антигена; вторая стадия - инкубация несвязывающихся фагмидных частиц с 100 нМ биотинилированным человеческим или мышиным 4-1ВВ в течение 0,5 ч в присутствии 100 нМ нерелевантной небиотинилированной конструкции Fc «выступ»-во-«впадину» для дальнейшего истощения связывающих Fc агентов в общем объеме 1 мл; третья стадия - захват биотинилированного hu4-1BB и присоединение специфически связывающего фага путем переноса в 4 лунки предварительно сенсибилизированного нейтравидином титрационного микропланшета в течение 10 мин (в раундах 1 и 3); четвертая стадия - промывка соответствующих лунок с использованием 5×ЗФР/Твин20 и 5×ЗФР; пятая стадия - элюция фаговых частиц путем добавления 250 мкл 100 мМ ТЭА (триэтиламин) на лунку в течение 10 мин и нейтрализация путем добавления 500 мкл 1М Трис/HCl, рН 7,4 к объединенным элюатам из 4 лунок; шестая стадия - пост-клиринг нейтрализованных элюатов путем инкубации на предварительно сенсибилизированном нейтравидином титрационном микропланшете с 100 нМ захваченной биотином конструкции Fc «выступ»-во-«впадину» для окончательного удаления связывающих Fc агентов; седьмая стадия - повторное заражение на log-фазе клеток Е. coli TG1 супернатантом элюированных фаговых частиц, заражение хелперным фагом VCSM13, инкубация при встряхивании при 30°С в течение ночи и последующее осаждение с помощью ПЭГ/NaCl фагмидных частиц для применения в следующем раунде селекции. Селекции осуществляли с использованием 3 или 4 раундов, применяя постоянную концентрацию антигена 100 нМ. В раундах 2 и 4 для того, чтобы избегать обогащения связывающими нейтравидин агентами, захват комплексов антиген: фаг осуществляли, добавляя 5,4×107 покрытых стрептавидином магнитных гранул. Специфические связывающие агенты идентифицировали с помощью ELISA следующим образом: 100 мкл 25 нМ биотинилированного человеческого или мышиного 4-1ВВ и 10 мкг/мл человеческого IgG применяли для сенсибилизации нейтравидиновых планшетов и maxisorp-планшетов соответственно. Добавляли содержащие Fab бактериальные супернатанты и связывание Fab оценивали посредством их Flag-меток, использую вторичное антитело к Flag/HRP. Клоны, дающие сигналы на человеческий или мышиный 4-1ВВ и являющиеся отрицательными в отношении человеческого IgG, включали в шорт-лист для дополнительных анализов и тестировали также аналогичным образом в отношении двух остальных видов 4-1ВВ. Их экспрессировали в бактериях в объеме культуры 0,5 л, очищали на основе аффинности и дополнительно характеризовали с помощью SPR-анализа, используя биосенсор фирмы BioRad ProteOn XPR36.
С использованием описанной выше процедуры клоны 12В3, 25G7, 11D5 и 9В11 идентифицированы в качестве специфических для человеческого 4-1ВВ связывающих агентов. С использованием описанной выше процедуры клон 20G идентифицирован в качестве специфического для мышиного 4-1ВВ связывающего агента. Последовательности кДНК их вариабельных областей представлены ниже в таблице 44, соответствующие аминокислотные последовательности можно найти в таблице В.


6.3 Получение, очистка и характеризация антител к 4-1ВВ IgG1 P329G LALA
Последовательности ДНК вариабельных областей тяжелых и легких цепей отобранных анти-4-1ВВ-связывающих агентов субклонировали в рамке считывания либо се константной тяжелой цепью, либо с константной легкой цепью человеческого IgG1. Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей с «выступом» и «впадиной» для элиминации связывания с рецепторами Fc-гамма согласно методу, описанному в публикации международной заявки на патент WO 2012/130831 А1.
Нуклеотидные и аминокислотные последовательности анти-4-1ВВ клонов представлены в таблице 45. Все кодирующие слияние анти-4-1BB-Fc последовательности клонировали в плазмидном векторе, в котором экспрессия вставки контролируется промотором MPSV и который содержит синтетическую сигнальную полиА-последовательность, локализованную на 3'-конце CDS. Кроме того, вектор содержал последовательность OriP EBV для поддержки эписомальной формы плазмиды.







Антитела к 4-1ВВ получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих, используя полиэтиленимин. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1 («вектор тяжелой цепи»: «вектор легкой цепи»).
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы (200 мкг общей ДНК) смешивали с 20 мл среды CD СНО. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% CO2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210×g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку молекул антител из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слияний антиген-Fc.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, рН 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу антител анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С.
В таблице 46 обобщены данные о выходе и конечном содержании антител к 4-1ВВ в формате P329G LALA IgG1.

Пример 7
Характеризация антител к 4-1ВВ
7.1 Связывание с человеческим 4-1ВВ
7.1.1 Поверхностный плазмонный резонанс (авидность + аффинность)
Связывание полученных с помощью фагов 4-1ВВ-специфических антител с конструкцией рекомбинантный 4-1BB-Fc(kih) оценивали с помощью поверхностного плазмонного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
В этом же эксперименте определяли видовую избирательность и авидность взаимодействия между полученными с помощью фагового дисплея клонами антител к 4-1ВВ 12В3, 25G7, 11D5, 9В11 и 20G2 (все в формате человеческого IgG1 P329GLALA) и рекомбинантным 4-1ВВ (человека, cyno и мышей). Конструкции, содержащие биотинилированный 4-1ВВ человека, обезьян циномолгус и мышей в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 100 резонансных единиц (RU). Полученные с помощью фагового дисплея антитела к 4-1ВВ в формате человеческого IgG1 P329GLALA пропускали через проточные ячейки, используя диапазон концентраций от 4 до 450 нМ (3-кратное разведение), со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации комплекса осуществляли в течение 220 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Кинетические константы определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 47).
В этом же эксперименте определяли аффинность взаимодействия полученных с помощью фагового дисплея антител (человеческий IgG1 P329GLALA) с рекомбинантным 4-1ВВ (человека, супо и мышей). Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 7500 RU. Полученные с помощью фагового дисплея антитела к 4-1ВВ захватывали в течение 60 с в концентрациях порядка 25 нМ. Конструкцию рекомбинантный человеческий 4-1BB-Fc(kih) пропускали через проточные ячейки в диапазоне концентраций от 4,1 до 1000 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-EP, а не антитела. Кинетические константы определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.
Клоны 25G7 и 9В11 связывались с конструкцией человеческий 4-1ВВ-Fc(kih) с более низкой аффинностью, чем клоны 12В3 и 11D5. Клон 20G2 не связывался с человеческим 4-1ВВ. Константы аффинности, характеризующие взаимодействие между анти-4-1ВВ P329GLALA IgG1 и конструкцией человеческий 4-1BB-Fc(kih), определяли, осуществляя подгонку к модели связывания 1:1 Ленгмюра.
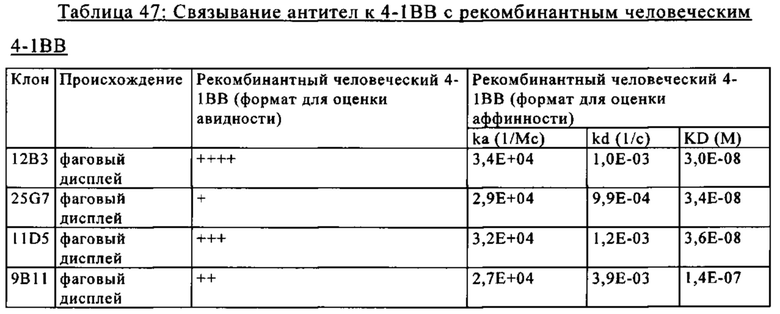
7.1.2 Связывание с экспрессирующими человеческий 4-1ВВ клетками: покоящиеся и активированные мононуклеарные лейкоциты периферической крови (РВМС) человека
Экспрессия человеческого 4-1ВВ отсутствует на покоящихся и наивных человеческих Т-клетках (Kienzle G. и von Kempis J., Int. Immunol. 12(1), 2000, cc. 73-82, Wen Т. и др., J. Immunol. 168, 2002, cc. 4897-4906). После активации иммобилизированным агонистическим антителом к человеческому CD3 происходит повышающая регуляция 4-1ВВ на CD4+- и CD8+-Т-клетках. Экспрессия 4-1ВВ описана также на активированных человеческих NK-клетках (Baessler Т. и др., Blood 115(15), 2010, сс. 3058-3069), активированных человеческих NKT-клетках (Cole S.L. и др., J. Immunol. 192(8), 2014, сс. 3898-3907), активированных человеческих В-клетках (Zhang и др., J. Immunol. 184(2), 2010, сс. 787-795), активированных человеческих эозинофилах (Heinisch и др., 2001), конститутивно на человеческих нейтрофилах (Heinisch I.V., J Allergy Clin Immunol. 108(1), 2000, сс.21-28), активированных человеческих моноцитах (Langstein J. и др., J Immunol. 160(5), 1998, сс. 2488-2494, Kwajah М. и Schwarz Н., Eur J Immunol. 40(7), 2010, сс. 1938-1949), конститутивно на человеческих регуляторных Т-клетках (Bacher Р. и др., Mucosal Immunol. 7(4), 2014, сс. 916-928), человеческих фолликулярных дендритных клетках (Pauly S. и др., J Leukoc Biol. 72(1), 2002, сс.35-42), активированных человеческих дендритных клетках (Zhang L. и др., Cell Mol Immunol. 1(1), 2004, сс. 71-76) и на кровеносных сосудах злокачественных человеческих опухолей (Broil K. и др., Am J Clin Pathol. 115(4), 2001, сс. 543-549).
Для оценки связывания полученных при создании изобретения клонов анти-4-1ВВ с встречающимися в естественных условиях клетками, экспрессирующими человеческий 4-1ВВ, применяли свежевыделенные покоящиеся мононуклеарные клетки периферической крови (РВМС) или предварительно активированные ФГА-L/пролейкином и реактивированные CD3/CD28 РВМС. РВМС из лейкоцитарных пленок, полученных из центра донорской крови Цюриха, выделяли путем центрифугирования в градиенте плотности фикола, используя Histopaque 1077 (фирма SIGMA Life Science, каталожный №10771, полисахароза и диатризоат натрия, плотность доведена до 1,077 г/мл)), и ресуспендировали в среде для Т-клеток, состоящей из среды RPMI 1640, фирма Gibco на фирме Life Technology, каталожный №42401-042), дополненной 10% фетальной бычьей сыворотки (FBS, фирма Gibco на фирме Life Technology, каталожный №16000-044, партия 941273, обработана гамма-лучами, свободная от микоплазм и инактивированная тепловой обработкой при 56°С в течение 35 мин), 1 об. % GlutaMAX-I (фирма GIBCO на фирме Life Technologies, каталожный №35050038), 1 мМ пируватом натрия (фирма SIGMA, каталожный № S8636), 1 об. % содержащей заменимые аминокислоты среды MEM (фирма SIGMA, каталожный № M7145) и 50 мкМ β-меркаптоэтанолом (фирма SIGMA, М3148). РВМС использовали непосредственно после выделения (покоящиеся клетки) или стимулировали для индукции экспрессии 4-1ВВ на клеточной поверхности Т-клеток путем культивирования в течение 3-5 дней в среде для Т-клеток, дополненной 200 ед./мл пролейкина (фирма Novartis Pharma Schweiz AG, CHCLB-P-476-700-10340) и 2 мкг/мл ФГА-L (фирма SIGMA, каталожный №L2769), в 6-луночном планшете для культуры ткани и затем в течение 2 дней в 6-луночном планшете для культуры ткани, сенсибилизированном 10 мкг/мл антитела к человеческому CD3 (клон OKT3, фирма BioLegend, каталожный №317315) и 2 мкг/мл антитела к человеческому CD28 (клон CD28.2, фирма BioLegend, каталожный №302928), в среде для Т-клеток при 37°С и 5% CO2.
Для определения связывания с человеческим 4-1ВВ, экспрессируемом человеческими РВМС, 0,1-0,2×106 свежевыделенных или активированных РВМС добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185). Планшеты центрифугировали в течение 4 мин при 400×g при 4°С и супернатант отбрасывали. Клетки промывали, используя 200 мкл/на лунку D-ЗФР, и затем инкубировали в течение 30 мин при 4°С с 100 мкл/мл D-ЗФР, содержащего разведенный в соотношении 1:5000 краситель для оценки жизнеспособности клеток (Fixable Viability Dye) eFluor 450 (фирма eBioscience, каталожный №64-0863-18) или Fixable Viability Dye eFluor 660 (фирма eBioscience, каталожный №65-0864-18). Затем клетки промывали однократно, используя 200 мкл/на лунку холодного FACS-буфера (D-ЗФР, дополненный 2 об. % FBS, 5 мМ ЭДТА, рН 8 (фирма Amresco, каталожный № Е177) и 7,5 мМ азидом натрия (фирма Sigma-Aldrich, S2002)). Затем добавляли 50 мкл/на лунку холодного (4°С) FACS-буфера, содержащего титрованные связывающие агенты к человеческому 4-1ВВ и клетки инкубировали в течение 120 мин при 4°С. Клетки промывали четыре раза, используя 200 мкл/лунку холодного (4°С) FACS-буфера для удаления несвязанных молекул. После этого клетки дополнительно инкубировали с 50 мкл/на лунку холодного (4°С) FACS-буфера, содержащего 2,5 мкг/мл конъюгированного с РЕ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-116-098) или 30 мкг/мл конъюгированного с ФИТЦ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098, антитело к человеческому CD45 AF488 (клон HI30, фирма BioLegend, каталожный №304019), 0,67 мкг/мл конъюгированного с АРС/Су7 moIgG1κ к человеческому CD3, (клон UCH1, фирма BioLegend, каталожный №300426) или 0,125 мкг/мл конъюгированного с РЕ мышиного IgG1κ к человеческому CD3 (клон SK7, фирма BioLegend, каталожный №344806) или 0,67 мкг/мл конъюгированного с PerCP/Су5.5 мышиного IgG1κ к человеческому CD3 (клон UCHT1, фирма BioLegend, каталожный №300430), 0,125 мкг/мл конъюгированного с BV421 moIgG1κ к человеческому CD4 (клон RPA-T4, фирма BioLegend, каталожный №300532), или 0,23 мкг/мл конъюгированного с BV421 мышиного IgG2bκ к человеческому CD4 (клон OKT4, фирма BioLegend, каталожный №317434), или 0,08 мкг/мл конъюгированного с РЕ/Су7 мышиного IgG1κ к человеческому CD4 (клон SK3, фирма BioLegend каталожный №344612), 0,17 мкг/мл конъюгированного с АРС/Су7 антитела к человеческому CD8 (мышиный IgG1κ, клон RPA-T8, фирма BioLegend, каталожный №301016) или 0,125 мкг/мл конъюгированного с РЕ/Су7 антитела к человеческому CD8a (moIgG1κ, клон RPA-T8, фирма BioLegend, каталожный №301012), или 0,33 мкг/мл антитела к человеческому CD8 BV510 (moIgG1κ, клон SK1, фирма BioLegend, каталожный №344732) и 0,25 мкг/мл конъюгированного с АРС антитела к человеческому CD56 (мышиный IgG1κ, клон HCD56, фирма BioLegend, каталожный №318310), или 1 мкл конъюгированного с AF488 антитела к человеческому CD56 (moIgG1κ, клон В159, фирма BD Pharmingen, каталожный №557699) и 0,67 мкг/мл антитела к человеческому CD19-PE/Cy7 (moIgG1κ, клон HIB19, фирма BioLegend, каталожный №302216), и инкубировали в течение 30 мин при 4°С.
Клетки промывали дважды, используя 200 мкл FACS-буфера/на лунку, и фиксировали путем ресуспендирования в 50 мкл/на лунку D-ЗФР, содержащего 1% формальдегида (фирма Sigma, НТ501320-9.5L). Получали данные для клеток в этот же или на следующий день с использованием устройства с 3 лазерами Canto II (фирма BD Bioscience, программное обеспечение DIVA) или с 5 лазерами Fortessa (фирма BD Bioscience, программное обеспечение DIVA), или снабженного 3-лазерами анализатора MACSQuant 10 (фирма Miltenyi Biotech). Дискриминационные окна устанавливали на CD8+- и CD4+-Т-клетки и определяли медианное значение интенсивности флуоресценции (MFI) или геометрическое среднее значение интенсивности флуоресценции вторичного идентифицирующего антитела, которое применяли для анализа связывания первичных антител. С использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.) данные стандартизовали относительно фонового уровня путем вычитания значений для «пустого» контроля (без добавления первичного антитела) и рассчитывали величины ЕС50 на основе подгонки кривой нелинейной регрессии (надежный подбор).
На человеческих Т-клетках в покоящемся состоянии отсутствовала экспрессия 4-1ВВ, но имела место повышающая регуляция 4-1ВВ после активации. Для человеческих CD8+-Т-клеток характерна более сильная повышающая регуляция, чем для CD4+-Т-клеток. Созданные антитела, специфические в отношении человеческого 4-1ВВ, могли связываться с человеческим 4-1ВВ, экспрессируемым активированными человеческими Т-клетками, что продемонстрировано на фиг. 23А-23Г. Представленные клоны антител к человеческому 4-1ВВ можно классифицировать как обладающие сильным связыванием (клоны 12В3 и 11D5) и слабым связыванием (клоны 25G7 и 9В11). Различия обнаружены не только по величине ЕС50, но также и по MFI. Величины ЕС50, характеризующие связывание с активированными CD8-T-клетками, представлены в таблице 48. Антимышиный 4-1ВВ-специфический клон 20G2 не связывался с человеческим 4-1ВВ и, следовательно, не обладал перекрестной реактивностью с человеческим антигеном.

7.2 Связывание с мышиным 4-1ВВ
7.2.1 Поверхностный плазмонный резонанс (авидность + аффинность)
Связывание полученного с помощью фагов 4-1ВВ-специфического антитела 20G2 с конструкцией рекомбинантный 4-1BB-Fc(kih) оценивали с помощью поверхностного плазмонного резонанса (SPR), описанного выше для конструкции человеческий 4-1BB-Fc(kih) (см. пример 7.1.1). Кинетические константы определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 44).
Для определения аффинности из-за неспецифического взаимодействия слитого с Fc белка с проточной референс-ячейкой применяли конструкцию мышиный 4-1BB-Fc(kih), расщепленную протеазой AcTEV, и Fc-область удаляли хроматографическим методом. Антитело к человеческому Fc-домену (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 7500 RU. Полученные с помощью фагового дисплея антитела к 4-1ВВ захватывали в течение 60 с в концентрации 25 нМ. Рекомбинантный мышиный 4-1ВВ, расщепленный AcTEV, пропускали через проточные ячейки в диапазоне концентраций от 4,1 до 1000 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антигены пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-ЕР, а не антитела.
Кинетические константы определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра. Установлено, что клон 20G2 связывался с мышиным 4-1ВВ (таблица 49).
Константы аффинности взаимодействия между молекулами анти-4-1ВВ P329GLALA IgG1 и мышиным 4-1ВВ определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore AB, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.

7.2.2 Связывание с экспрессирующими мышиный 4-1ВВ клетками: покоящиеся и активированные мышиные спленоциты (отобранные клоны)
Аналогично человеческим Т-клеткам, свежевыделенные покоящиеся мышиные Т-клетки не экспрессируют 4-1ВВ, но экспрессию можно индуцировать путем активации TCR с помощью подвергнутых импульсной обработке пептидом АРС (Cannons J. и др., J. Immunol. 167(3), 2001, сс. 1313-1324) или иммобилизированным антителом к мышиному CD3 или комбинацией антитела к мышиному CD3 и антитела к мышиному CD28 (Pollok K. и др., European J. Immunol. 25(2), 1995, сс. 488-494). Установлено, что после активации с помощью иммобилизированного на поверхности антитела к CD3 экспрессия 4-1ВВ была более высокой на CD8+-Т-клетках (Shuford W. и др., J. of Experimental Med. 186(1), 1997, сс. 47-55), но это могло зависеть от протокола активации. Кроме того, экспрессия 4-1ВВ описана также на активированных мышиных NK-клетках (Melero и др., Cell Immunol. 190(2), 1998, сс. 167-172), активированных мышиных NKT-клетках (Vinay D. и др. J Immunol. 173(6), 2004, сс. 4218-4229), активированных мышиных В-клетках (Vinay, Kwon, Cell Mol Immunol. 8(4), 2011, сс. 281-284), конститутивно на мышиных нейтрофилах (Lee S. и др., Infect Immun. 73(8), 2005, сс. 5144-5151) и мышиных регуляторных Т-клетках (Gavin и др., Nat Immunol. 3(1), 2002, сс. 33-41), мышиных IgE-стимулированных тучных клетках (Nishimoto и др., Blood 106(13), 2005, сс. 4241-4248), мышиных клетках миелоидной линии дифференцировки (Lee и др., Nat Immunol. 9(8), 2008, сс. 917-926), мышиных фолликулярных дендритных клетках (Middendorp и др., Blood 114(11), 2009, сс. 2280-2289) и активированных мышиных дендритных клетках (Wilcox, Chapoval и др., J Immunol. 168(9), 2002, сс. 4262-4267).
Связывание специфических антител к 4-1ВВ тестировали непосредственно после выделения спленоцитов из здоровых самок мышей линии C57BL/6, а также после активации in vitro в течение 72 ч с помощью иммобилизированных на поверхности агонистического антитела к мышиному CD3 и антитела к мышиному CD28. Самок мышей линии C57BL/6 (возрастом 7-9 недель) покупали у фирмы Charles River, Франция. После доставки животных выдерживали в течение 1 недели для акклиматизации к новым условиям окружающей среды и для обследования. Мышей содержали в условиях отсутствия специфических патогенов с суточным циклом 12 ч света/12 ч темноты, и они имели свободный доступ к корму и воде согласно соответствующим руководствам (GV-Solas; Felasa; TierschG). Постоянный мониторинг состояния здоровья осуществляли на регулярной основе. Мышей умерщвляли путем смещения шейных позвонков. Селезенки иссекали и хранили на льду в среде RPMI 1640, дополненной 10 об. % инактивированной тепловой обработкой FBS и 1 об. % GlutaMAX-I. Для получения раствора единичных клеток селезенки гомогенизировали, пропуская через 70-микрометровое клеточное сито (фирма BD Falcon; Германия) и подвергали гемолизу в течение 10 мин при 37°С в лизирующем буфере АСК (0,15М NH4Cl, 10 мМ KHCO3, 0,1 мМ ЭДТК в ddH2O, рН 7,2). После двух стадий промывки стерильным D-ЗФР спленоциты восстанавливали в среде для Т-клеток. Либо применяли свежевыделенные клетки (покоящиеся), либо 106 спленоцитов/мл дополнительно стимулировали в течение 72 ч в среде для Т-клеток, состоящей из среды RPMI 1640, дополненной 10 об. % FBS, 1 об. % GlutaMAX-I, 1 мМ пируватом натрия, 1 об. % содержащей заменимые аминокислоты среды MEM и 50 мкМ β-меркаптоэтанолом, в 6-луночных планшетах для культуры клеток, сенсибилизированных 1 мкг/мл антитела к мышиному CD3 (крысиный IgG2b, клон 17А2, фирма BioLegend, каталожный №100223) и 2 мкг/мл антитела к мышиному CD28 (антитело сирийского хомяка, клон 37.51, фирма BioLegend, каталожный №102112).
Для определения связывания с мышиным 4-1ВВ свежевыделенные или активированные мышиные спленоциты ресуспендировали в D-ЗФР и 0,1×106 спленоцитов/на лунку переносили в круглодонный 96-луночный планшет для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185). Клетки центрифугировали в течение 4 мин при 400×g при 4°С и супернатант удаляли. После ресуспендирования в 100 мкл/лунку D-ЗФР, содержащего разведенный в соотношении 1:5000 краситель для оценки жизнеспособности клеток (Fixable Viability Dye) eFluor 450 (фирма eBioscience, каталожный №65-0863-18) или Fixable Viability Dye eFluor 660 (фирма eBioscience, каталожный №65-0864-18), клетки инкубировали в течение 30 мин при 4°С. Клетки промывали FACS-буфером и добавляли 50 мкл/на лунку FACS-буфера, содержащего титрованные концентрации антител к человеческому 4-1ВВ huIgG1 P329G LALA, клоны 12В3, 25G7, 11D5, 9В11, и специфический в отношении мышиного 4-1ВВ клон 20G2 в виде huIgG1 P329G LALA или мышиного IgG1, или мышиного IgG1 DAPG. После инкубации в течение 1 ч при 4°С клетки промывали 4 раза для удаления избытка антител. Если оценивали связывание антимышиного 20G2 в формате мышиного IgG1 и мышиного IgG1 DAPG, то клетки инкубировали в 50 мкл FACS-буфера/на лунку, содержащего 30 мкг/мл конъюгированного с ФИТЦ F(ab')2-фрагмента козьего IgG против мышиного IgG, Fcγ-фрагмент-специфического AffiniPure в течение 30 мин при 4°С и промывали дважды FACS-буфером. Если оценивали связывание анти-4-1ВВ связывающих агентов, содержащих Fc-фрагмент человеческого IgG1 P329G LALA, то это стадию пропускали. После этого клетки инкубировали в 50 мкл FACS-буфера/на лунку, содержащего 0,67 мкг/мл конъюгированного с РЕ антитела к мышиному CD3 (крысиный IgG2bκ, клон 17А2, фирма BD Pharmingen, каталожный №555275) или конъюгированного с АРС-Су7 антитела к мышиному CD3 (крысиный IgG2aκ, клон 53-6.7, фирма BioLegend, каталожный №100708), 0,67 мкг/мл конъюгированного с РЕ/Су7 антитела к мышиному CD4 (крысиный IgG2bκ, клон GK1.5, фирма BioLegend, каталожный №100422), 0,67 мкг/мл конъюгированного с АРС/Су7 антитела к мышиному CD8 (крысиный IgG2aκ, клон 53-6.7, фирма BioLegend, каталожный №1007141) или конъюгированного с РЕ антитела к мышиному CD8 (крысиный IgG2aκ, клон 53-6.7, фирма BioLegend, каталожный №100708), 2 мкг/мл конъюгированного с АРС антитела к мышиному NK1.1 (мышиный IgG2a, κ, клон PK136, фирма BioLegend, каталожный №108710) или конъюгированного с PerCP/Су5.5 антитела к мышиному NK1.1 (мышиный IgG2a, κ, клон PK136, фирма BioLegend, каталожный №108728) и 10 мкг/мл антитела к мышиным CD16/CD32 (мышиный Fc-блок, крысиный IgG2b κ, клон 2.4G2, фирма BD Bioscience, каталожный №553142). Если оценивали связывание анти-4-1ВВ связывающих агентов, содержащих Fc-фрагмент человеческого IgG1 P329G LALA, то добавляли также 30 мкг/мл конъюгированного с ФИТЦ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109096098). Клетки инкубировали в течение 30 мин при 4°С, промывали дважды, используя 200 мкл/на лунку FACS-буфера, и ресуспендировали для фиксации с помощью 50 мкл/на лунку D-ЗФР, содержащего 1 об. % формальдегида. На следующий день клетки ресуспендировали в 200 мкл/на лунку FACS-буфера и получали данные с использованием устройства с 2 лазерами CantoII (фирма BD Bioscience, программное обеспечение DIVA) или с 5 лазерами Fortessa (фирма BD Bioscience, программное обеспечение DIVA). Дискриминационные окна устанавливали на CD8+- и CD4+-Т-клетки и определяли медианное значение интенсивности флуоресценции (MFI) вторичного идентифицирующего антитела, которое применяли для анализа связывания первичных антител. С использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.) данные стандартизовали относительно фонового уровня путем вычитания значений для «пустого» контроля (без добавления первичного антитела) и рассчитывали величины ЕС50 на основе подгонки кривой нелинейной регрессии (надежный подбор).
Как продемонстрировано на фиг. 24Б и 24Г, только связывающийся с мышиным 4-1ВВ клон 20G2 связывался с активированными мышиными CD8+- и CD4+-Т-клетками, в то время, как связывающиеся с человеческим 4-1ВВ клоны 9В11, 11D5, 12В3 и 25G7 не связывались с мышиным 4-1ВВ, и, следовательно, не обладали перекрестной реактивностью с мышиным антигеном. Только клон 20G2 из протестированных клонов можно применять в качестве мышиного аналога. Как и ожидалось, ни один из анти-4-1ВВ-связывающих клонов не связывался со свежевыделенными покоящимися мышиными Т-клетками (фиг. 24А и 24В). Аналогично активированным человеческим CD4+-Т-клеткам, также и активированные мышиные CD4+-Т-клетки экспрессировали более низкие уровни 4-1ВВ по сравнению с активированными мышиными CD8+-Т-клетками. Однако различие было не столь значительным, как в случае человеческих Т-клеток, что может быть связано также с другим протоколом активации. Величины ЕС50 представлены в таблице 50.

Как продемонстрировано на фиг. 25Б и 25Г, связывающийся с мышиным 4-1ВВ клон 20G2 связывался с активированными мышиными CD8+- и CD4+-T-клетками аналогично связыванию moIgG1 дикого типа (wt) или антитела в формате moIgG1 DAPG. Связывание оказалось сходным со связыванием, продемонстрированным на фиг. 24Б и 24Г; следовательно, изменение формата не влияло на особенности связывания. При создании изобретения клон 20G2 конвертировали в moIgG для предупреждения запуска образования антител к лекарственным средствам (ADA) у иммунокомпетентных мышей. Мутация DAPG эквивалента мутации P329G LALA в конструкциях человеческого IgG1, например, она препятствует перекрестному сшиванию через иммунные FcR+-клетки. Величины ЕС50 представлены в таблице 51.

7.3 Связывание с 4-1ВВ обезьян циномолгус
7.3.1 Поверхностный плазмонный резонанс (авидность + аффинность)
Связывание полученных с помощью фагов 4-1ВВ-специфических антител 12В3, 25G7, 11D5 и 9В11 с конструкцией рекомбинантный 4-1ВВ обезьян циномолгус-Fc(kih) оценивали с помощью поверхностного плазменного резонанса (SPR) согласно методу, описанному для выше для конструкции человеческий 4-1BB-Fc(kih) (см. пример 7.1.1). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия). Кинетические константы определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра, и применяли для качественной оценки авидности (таблица 52).
В этом же эксперименте определяли аффинность взаимодействия полученных с помощью фагового дисплея антител (человеческий IgG1 P329GLALA) с конструкцией рекомбинантный 4-1ВВ обезьян циномолгус-Fc(kih). Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 9000 RU. Полученные с помощью фагового дисплея антитела к 4-1ВВ захватывали в течение 60 с в концентрациях от 25 до 100 нМ. Эксперимент осуществляли согласно методу, описанному для конструкции человеческий 4-1BB-Fc(kih) (пример 7.1.1).
Клоны 12В3, 25G7, 11D5 и 9В11 связывались с конструкций 4-1ВВ обезьяны циномолгус-Fc(kih) со сходными аффинностями (таблица 52), а клоны 12В3 и 11D5 связывались с более высокой авидностью с клетками, экспрессирующими 4-1ВВ обезьян циномолгус. Константы аффинности взаимодействия между антителами к 4-1ВВ в формате P329GLALA IgG1 и конструкцией 4-1ВВ обезьян циномолгус-Fc(kih) определяли с помощью программного обеспечения Biacore Т200 Evaluation (vAA, фирма Biacore АВ, Уппсала/Швеция), осуществляя подгонку путем численного интегрирования уравнений для скорости к модели связывания 1:1 Ленгмюра.


7.3.2 Связывание на экспрессирующих 4-1ВВ обезьян циномолгус клетками: активированные мононуклеарные лейкоциты периферической крови (РВМС) обезьян циномолгус
Для оценки перекрестной реактивности клонов антител, связывающихся с человеческим 4-1ВВ, с клетками обезьян циномолгус РВМС здоровых Cynomolgus fascicularis выделяли из гепаринизированной крови центрифугированием в градиенте плотности, согласно методу, описанному для человеческих РВМС (7.1.2) с небольшими изменениями. Выделенные РВМС культивировали в течение 72 ч при клеточной плотности 1,5×106 клеток/мл в среде для Т-клеток, содержащей среду RPMI 1640, дополненную 10% FBS, 1 об. % GlutaMAX-I, 1 мМ пируватом натрия, 1 об. % содержащей заменимые аминокислоты среды MEM и 50 мкМ β-меркаптоэтанолом, в 60-луночных планшетах для культуры клеток (фирма Greiner Bio-One, Германия), сенсибилизированных обладающим перекрестной реактивностью для обезьян циномолгус антителом к CD3 в концентрации 10 мкг/мл (mo IgG3 λ, антитело к человеческому CD3, клон SP34, фирма BD Pharmingen, каталожный №556610) и обладающим перекрестной реактивностью для обезьян циномолгус антителом к CD28 в концентрации 2 мкг/мл (moIgG1κ, антитело к человеческому CD28, клон CD28.2, фирма BioLegend, каталожный №140786). Через 72 ч после стимуляции клетки собирали и высевали в круглодонный 96-луночный планшет для суспензионных клеток (фирма Greiner bio-one, cellstar, каталожный №650185) в концентрации 0,1×106 клеток/на лунку. Клетки инкубировали в 50 мкл/на лунку FACS-буфера, содержащего в различных концентрациях первичные античеловеческие 4-1ВВ-специфические антитела в формате huIgG P32G LALA, в течение 2 ч при 4°С. Затем клетки промывали 4 раза, используя 200 мкл/на лунку FACS-буфера и инкубировали в течение еще 30 мин при 4°С с 50 мкл/на лунку FACS-буфера, содержащего 2 мкл конъюгированного с РЕ обладающего перекрестной реактивностью для обезьян циномолгус антитела к CD4 (moIgG2aκ, антитело к человеческому CD4, клон М-Т477, фирма BD Pharmingen, каталожный №.556616), 1 мкг/мл конъюгированного с PerCP/Су5.5 обладающего перекрестной реактивностью для обезьян циномолгус антитела к CD8 (moIgG1κ, антитело к человеческому CD8, клон RPA-T8, фирма BioLegend, каталожный №301032) и 30 мкг/мл конъюгированного с ФИТЦ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109096098). Клетки промывали дважды FACS-буфером и ресуспендировали в 100 мкл/на лунку FACS-буфера, дополненного 0,2 мкг/мл DAPI, для дискриминации мертвых и живых клеток. Клетки немедленно анализировали с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA). Дискриминационные окна устанавливали на CD8+- и CD4+-Т-клетки и медианное значение интенсивности флуоресценции (MFI) для вторичного идентифицирующего антитела применяли для анализа связывания первичных антител. С использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.) данные стандартизовали относительно фонового уровня путем вычитания значений для «пустого» контроля (без добавления первичного антитела) и рассчитывали величины ЕС50 на основе подгонки кривой нелинейной регрессии (надежный подбор).
Как продемонстрировано на фиг. 26А и 26Б, по меньшей мере три клона антител к человеческому 4-1ВВ, а именно, 12В3, 11D5 и 25G7, обладали перекрестной реактивностью к 4-1ВВ обезьян циномолгус, который экспрессировался на активированных CD4+- и CD8+-Т-клетках. Важно отметить, что кривые связывания выглядели подобно кривым для человеческого 4-1ВВ, например, для клонов 12В3 и 11D5 обнаружены наиболее высокие значения MFI и наиболее низкие значения ЕС50 для всех протестированных конструкций, а 25G7 был самым слабым связывающим агентом, что характеризовалось наиболее низким значением MFI и наиболее высоким значением ЕС50 (таблица 53). Клон 9В11 связывался только при наиболее высокой концентрации 100 нМ. Это отличается от связывания с человеческим 4-1ВВ, при анализе которого установлено, что клон 9В11 превосходил клон 25G7. Другие различия уровней экспрессии 4-1ВВ на активированных CD4+- и CD8+-Т-клетках обезьян циномолгус были сходными с различиями для активированных человеческих Т-клеток, например, CD4+-Т-клетки экспрессировали существенно более низкие уровни 4-1ВВ, чем CD8+-Т-клетки.

7.4 Способность блокировать лиганд
Для определения способности молекул 4-1ВВ-специфических человеческих антител IgG1 P329GLALA влиять на взаимодействия 4-1ВВ/лиганд 4-1ВВ применяли человеческий лиганд 4-1ВВ (фирма R&D systems). Аналогично этому, мышиный лиганд 4-1ВВ (фирма R&D systems) применяли для оценки способности блокировать лиганд антимышиного 4-1ВВ-специфического антитела 20G2 IgG1 P329GLALA.
Человеческий или мышиный лиганд 4-1ВВ непосредственно сшивали с двумя проточным ячейками СМ5-чипа с уровнем, соответствующим примерно 1500 RU, при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Конструкцию рекомбинантный человеческий или мышиный 4-1BB-Fc(kih) пропускали через вторую проточную ячейку в концентрации 500 нМ со скоростью потока 30 мкл/мин в течение 90 с. Диссоциацию исключали и полученное с помощью фагов антитело к 4-1ВВ в формате человеческий IgG1P329LALA пропускали через обе проточные ячейки в концентрации 200 нМ со скоростью потока 30 мкл/мин в течение 90 с. Мониторинг диссоциации осуществляли в течение 60 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антитела пропускали по поверхности с иммобилизированным человеческим или мышиным лигандом 4-1ВВ, но инъецировали HBS-EP вместо конструкции рекомбинантный человеческий 4-1BB-Fc(kih).
Полученный с помощью фагов клон 25G7 связывался с комплексом человеческого 4-1ВВ с его лигандом 4-1ВВ (таблица 54, фиг. 27А). Таким образом, это антитело не могло конкурировать с лигандом за связывание с человеческим 4-1ВВ, и поэтому его обозначали как «неблокирующее лиганд». В противоположность этому, клоны 12В3, 11D5 и 9В11 не связывались с человеческим 4-1ВВ в комплексе с его лигандом, и поэтому их обозначали как «блокирующие лиганд». Мышиный аналог 20G2 не связывался с мышиным 4-1ВВ, ассоциированным с его лигандом, и поэтому также был обозначен как «блокирующий лиганд».

7.5 Характеризация эпитопа
Эпитоп, распознаваемый полученным с помощью фага антителом к 4-1ВВ, характеризовали с помощью поверхностного плазмонного резонанса. Во-первых, способность антител конкурировать за связывание с человеческим 4-1ВВ оценивали с помощью поверхностного плазмонного резонанса. Во-вторых, оценивали связывание антител к 4-1ВВ с двумя различными доменами человеческого и мышиного 4-1ВВ с помощью SPR, указанного выше в настоящем описании. Для этой цели создавали гибридные слитые белки 4-1ВВ-Fc(kih). Они содержали эктодомены 4-1ВВ либо человеческого, либо мышиного домена. Гибридные варианты 4-1ВВ сливали с цепью с «выступом» Fc(kih), аналогично слиянию антигены-Fc, которое применяли для селекции с помощью фагового дисплея и которое описано выше. Целью этих экспериментов было определение «группы эпитопа». Антитела, которые конкурируют за сходный или перекрывающийся эпитоп не могут связываться одновременно с 4-1ВВ и принадлежат к «группе эпитопа». Антитела к 4-1ВВ, которые могут связываться одновременно с 4-1ВВ, не имеют общего эпитопа или его части и поэтому их относят к другой группе эпитопа.
7.5.1 Конкурентное связывание (SPR)
Для изучения конкурентного связывания с человеческим или мышиным рецептором антител к человеческому 4-1ВВ в формате человеческого IgG1 P329GLALA (фиг. 28А-28Е) полученные с помощью фагов анти-4-1ВВ клоны 9В11 и 11D5 непосредственно сшивали с СМ5-чипом с уровнем иммобилизации, который соответствовал 1400 и 2200 RU, при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Конструкцию рекомбинантный человеческий 4-1BB-Fc(kih) инъецировали в концентрации 200 нМ со скоростью потока 30 мкл/мин в течение 180 с. Диссоциацию исключали и второе антитело к 4-1ВВ в формате человеческого IgG1 P329GLALA пропускали в концентрации 100 нМ со скоростью потока 30 мкл/мин в течение 90 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке.
SPR-эксперименты осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия). Эксперимент по оценке конкурентного связывания продемонстрировал, что полученные с помощью фагов анти-4-1ВВ клоны 12В3, 11D5 и 9В11 распознают пространственный эпитоп, отличный от эпитопа 25G7, поскольку два антитела могли связываться одновременно с конструкцией человеческий 4-1BB-Fc(kih) (таблица 55).

0 обозначает отсутствие связывания; 1 обозначает наличие связывания; X обозначает, что связывание не определяли, поскольку для второй инъекции использовали такое же антитело, что и антитело, иммобилизированное на чипе.
7.5.2 Связывание с вариантами рецептора (SPR)
Эпитопы, распознаваемые полученными с помощью фагов клонами антител к 4-1ВВ 12В3, 25G7, 11D5, 9В11 и 20G2, характеризовали с использованием гибридных вариантов слитых молекул 4-1BB-Fc(kih), которые содержали комбинацию человеческого и мышиного доменов или только один единичный домен (таблица 56). Гибридный 4-1ВВ сливали с Fc(kih) согласно методу, описанному в пример 6.1 для антигенов 4-1BB-Fc(kih). В таблице 52 представлены последовательности доменов 4-1ВВ, которые использовали для получения гибридных вариантов 4-1ВВ.
Конструкция 1 состоит из N-концевой области (обозначена как домен 1) мышиного 4-1ВВ, слитой с С-концевой областью (обозначена как домен 2) человеческого 4-1ВВ (таблица 56). Конструкция 2 состоит из N-концевой области (обозначена как домен 1) человеческого 4-1ВВ, слитой с С-концевой областью (обозначена как домен 2) мышиного 4-1ВВ (таблица 57). Такие гибридные молекулы создавали, поскольку экспрессия домена 2 не приводила к получению функциональной молекулы. N-концевую область (домен 1) человеческого 4-1ВВ экспрессировали также в виде слитой с Fc(kih) молекулы (конструкция 3). Экспрессию и очистку гибридных вариантов 4-1BB-Fc(kih) осуществляли согласно методу, описанному в примере 6.1.


SPR-эксперименты осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01 М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия). Антитело к человеческому Fab (фирма Biacore, Фрейбург/Германия) непосредственно сшивали с СМ5-чипом при рН 5,0, используя набор для стандартного аминного сочетания (фирма Biacore, Фрейбург/Германия). Уровень иммобилизации соответствовал примерно 8000 RU. Полученные с помощью фагового дисплея антитела к 4-1ВВ захватывали в течение 60 с в концентрации 100 нМ. Варианты конструкций рекомбинантный гибридный человеческий/мышиный 4-1BB-Fc(kih) пропускали через проточные ячейки в концентрации 200 нМ со скоростью потока 30 мкл/мин в течение 120 с. Мониторинг диссоциации осуществляли в течение 120 с (фиг. 29А-29Г).
Человеческий 4-1BB-D1 (конструкция 3) сшивали непосредственно с СМ5-чипом с уровнем иммобилизации, соответствующем примерно 1000 RU. Антитела к 4-1ВВ пропускали в концентрации 200 нМ со скоростью 30 мкл/мин в течение 180 с и мониторинг диссоциации осуществляли в течение 60 с (фиг. 30А-30Г). Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке. В ней антитела пропускали по поверхности с иммобилизированным антителом к человеческому Fab, но инъецировали HBS-EP вместо антител.
Полученные с помощью фагов клоны 12В3, 11D5 и 9В11 связывались с обеими содержащими человеческий домен 1 молекулами 4-1BB-Fc(kih). Полученные с помощью фагов клоны 25G7 связывались с содержащей человеческий домен 2 гибридной молекулой 4-1BB-Fc(kih). Полученные с помощью фагов клоны 20G2 связывались с содержащей мышиный домен 1 гибридной молекулой 4-1BB-Fc(kih). Обобщение результатов представлено в таблице 58.

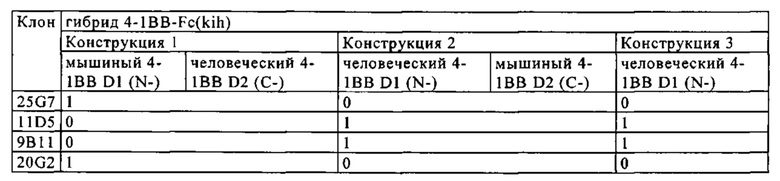
Пример 8
Функциональные свойства связывающих 4-1ВВ клонов антител
8.1 Функциональные свойства связывающих человеческий 4-1ВВ клонов антител
4-1ВВ служит в качестве костимуляторного рецептора и повышает экспансию, производство цитокинов и функциональные свойства Т-клеток в зависимости от TCR. Активация TCR с помощью иммобилизированного на поверхности антитела к CD3 или импульсная обработка пептидом антигенпрезентирующих клеток индуцируют повышающую регуляцию 4-1ВВ на Т-клетках (Pollok и др., J. Immunol. 150(3), 1993, сс. 771-781) и поддерживает после контакта иммунный ответ путем усиления экспансии и высвобождения цитокинов (Hurtado и др., J Immunology 155(7), 1995, сс. 3360-3367). Для оценки усиливающей иммунный ответ способности созданных антител к человеческому 4-1ВВ круглодонные 96-луночные планшеты для суспензий (фирма Greiner bio-one, cellstar, каталожный №650185) сенсибилизировали в течение ночи D-ЗФР, содержащим 2 мкг/мл F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson Immunoresearch, каталожный №109-006-008) и 2 мкг/мл козьего IgG против мышиного IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson Immunoresearch, каталожный №115-005-008). Планшеты промывали D-ЗФР для удаления избыточных молекул и блокировали в течение 90 мин при 37°С D-ЗФР, содержащим 1% (мас./об.) БСА (фирма SIGMA-Aldrich, каталожный №А3059-100G). Супернатант удаляли и планшеты инкубировали с D-ЗФР, дополненным 1% (мас./об.) БСА и 10 нг/мл антитела к человеческому CD3 (фирма BioLegend, каталожный №317315, клон OKT3), или без указанного антитела в течение 90 мин при 37°С. Планшеты промывали D-ЗФР и инкубировали с D-ЗФР, дополненным 1% (мас./об.) БСА и применяемыми в различных концентрациях титрованными антителами к человеческому 4-1ВВ IgG1 Р329 G LALA. Планшеты промывали D-ЗФР и супернатант удаляли аспирацией.
Человеческие РВМС выделяли согласно описанному выше методу (7.1.2) и активировали в среде RPMI 1640, содержащей 10 об. % FBS, 1 об. % GlutaMAX-I, 2 мкг/мл ФГА-L и 200 ед./мл пролейкина, в течение 5 дней. Клетки дополнительно культивировали в RPMI 1640, содержащей 10 об. % FBS, 1 об. % GlutaMAX-I и 200 ед./мл пролейкина, в течение еще 21 дня при плотности 1-2×106 клеток/мл. После длительного культивирования РВМС собирали, промывали и выделяли CD8-Т-клетки согласно протоколу производителя, используя набор для выделения человеческих CD8+-Т-клеток (фирма Miltenyi Biotec, каталожный №130-096-495). Ргеактивированные и отсортированные CD8+-Т-клетки высевали с плотностью 7×104 клеток/на лунку в 200 мкл/на лунку среды для Т-клеток, состоящей из среды RPMI 1640, дополненной 10% FBS, 1 об. % GlutaMAX-I, 1 мМ пируватом натрия, 1 об. % содержащей заменимые аминокислоты среды MEM и 50 мкМ β-меркаптоэтанолом. Клетки инкубировали в течение 72 ч, последние 4 ч в присутствии стоп-реагента Гольджи (Golgi stop) (ингибитор белкового транспорта, содержащий монезин, фирма BD Bioscience, каталожный №554724). Клетки промывали D-ЗФР и инкубировали в течение 30 мин при 4°С в 100 мкл/на лунку D-ЗФР, содержащего разведенный в 1:5000 раз зеленый фиксирующий краситель для определения жизнеспособности (LIVE/DEAD Fixable Green Dead Cell Stain) (фирма Molecular Probes, Life Technologies, каталожный № L-23101). Затем клетки промывали и инкубировали в течение 30 мин при 4°С в 50 мкл/на лунку FACS-буфера, содержащего 0,5 мкг/мл конъюгированного с PerCP/Су5.5 антитела к человеческому CD8 (мышиный IgG1 κ, клон RPA-T8, фирма BioLegend, каталожный №301032), 0,5 мкг/мл конъюгированного с РЕ/Су7 антитела к человеческому CD25 (мышиный IgG1 κ, клон ВС96, фирма BioLegend, каталожный №302612) и 1 мкг/мл конъюгированного с АРС/Су7 антитела к человеческому PD-1 (мышиный IgG1 κ, клон ЕН12.2Н7, фирма BioLegend, каталожный №329922). Клетки промывали FACS-буфером и ресуспендировали в 50 мкл/на лунку свежеприготовленного раствора для фиксации/пермеабилизации (фирма eBioscience, каталожный №00-5523-00). После инкубации в течение 30 мин при 4°С клетки промывали свежеприготовленным буфером для пермеабилизации (фирма eBioscience, каталожный №-00-5523-00) и инкубировали в течение 1 ч при 4°С в 50 мкл/на лунку Perm-буфера, содержащего 2 мкг/мл меченного с помощью АРС антитела к человеческому IFNγ (mo IgG1 κ, клон В27, фирма BD Pharmingen, каталожный №554702) и 2 мкг/мл конъюгированного с РЕ антитела к человеческому TNFα (mo IgG1 κ, клон MAb11, фирма BD Pharmingen, каталожный №554513). Клетки промывали и фиксировали с помощью D-ЗФР, содержащего 1% формальдегида. Клетки анализировали на следующий день с использованием устройства с 2 лазерами Canto II (фирма BD, программное обеспечение DIVA). Дискриминационные окна устанавливали на CD8+-Т-клетки и определяли частоту встречаемости секретирующих TNFα и IFNγ CD8+-Т-клеток. С использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.) данные наносили на графики и кривые рассчитывали на основе подгонки кривой по методу нелинейной регрессии (надежный подбор).
Как описано выше, в отсутствии TCR- или CD3-стимуляции контакт с 4-1ВВ не влиял на функцию CD8+-Т-клеток (Pollok 1995), в то время как при субоптимальной CD3-активации костимуляции 4-1ВВ возрастала секреция цитокинов (фиг. 31А или 31В). Субоптимальная активация с помощью антитела к CD3 индуцировала секрецию IFNγ в 30% CD8+-Т-клеток во всей популяции CD8+-Т-клеток. Добавление костимуляции 4-1ВВ повышало популяцию IFNγ+ CD8-T-клеток вплоть до 55% в зависимости от концентрации (фиг. 31Б). В таблице 59 представлены соответствующие величины ЕС50. Секреция TNF(могла повышаться с 23% до 39% во всей популяции CD8+-Т-клеток (фиг. 31Г). Аналогично их способности к связыванию клоны 12В3 и 11D5 повышали частоту экспрессии INFγ в большей степени, чем клоны 25G7 и 9В11 и величины ЕС50. Таким образом, разработанные при создании изобретения клоны были функциональными и могли повышать TCR-опосредуемую активацию и функцию Т-клеток. Если 4-1ВВ-специфические антитела не были иммобилизированы на поверхности, они не могли повышать активацию CD8+-Т-клеток (данные не представлены).

8.2 Функциональные свойства связывающего мышиный 4-1ВВ клона 20G2 in vitro
Для предупреждения перекрестного связывания агонистических антител к 4-1ВВ через Fc-рецепторы интродуцировали мутацию в Fc-область антител. Аналогично мутации P329G LALA в человеческих антителах IgG1-типа, мутацию DAPG интродуцировали в Fc-область мышиных молекул IgG1. Для решения вопроса о том, предупреждает ли это перекрестное сшивание через FcR и следовательно способность к активации, мышиные спленоциты активировали с помощью антитела к мышиному CD3 в субоптимальной концентрации и применяемого в различных концентрациях клона 20G2 в качестве мышиного IgG1 и мышиного IgG1 DAPG.
Селезенки выделяли из здоровых самок мышей C57BL/6 (возраст 7-9 недель, фирма Charles River, Франция) и гомогенизировали в 3 мл MACS-буфера с использованием С-пробирок (Miltenyi Biotec, каталожный №130-096-334) и мягкого диссоциатора MACS Octo (фирма Miltenyi Biotec, каталожный №130-095-937). После центрифугирования (400×g, 8 мин, КТ) клеточный дебрис ресуспендировали в 7 мл буфера для лизиса ACK и гемолиз осуществляли в течение 10 мин при 37°С. Гемолиз прекращали, добавляя Т-клеточную среду, состоящую из среды RPMI 1640, дополненной 10% FBS, 1 об. % GlutaMAX-I, 1 мМ пируватом натрия, 1 об. % содержащей заменимые аминокислоты среды MEM и 50 мкМ β-меркаптоэтанолом. Спленоциты промывали D-ЗФР и фильтровали через 70-микрометровое клеточное сито (фирма Corning, каталожный №-431751). Спленоциты ресуспендировали до плотности 2×107 клеток/мл в 37°С в D-ЗФР и добавляли краситель для оценки клеточной пролиферации eFluor 670 (фирма eBioscience, каталожный №-65-0840-90) до конечной концентрации 2,5 мМ. Клетки инкубировали в течение 10 мин при 37°С, процесс мечения прекращали, добавляя FBS, и клетки промывали. Добавляли 200 мкл/на лунку среды для Т-клеток, содержащей 0,5 мкг/мл антимышиного CD3-специфического IgG хомяка (клон 145-2С11, фирма BD Bioscience, каталожный №553057), 0,01-50 нМ титрованный антимышиный 4-1ВВ-специфический клон 20G2 мышиного IgG1 или мышиного IgG1 DAPG, или применяемого в качестве контроля изотипа МОРС-21 мышиного IgG1 (фирма BD Bioscience, каталожный №554121) или применяемого в качестве контроля изотипа DP47 moIgG1 DAPG, и 1×105 спленоцитов переносили в лунки 96-луночного круглодонного планшета для культуры ткани (фирма ТРР, каталожный №2097). Планшеты инкубировали в течение 48 ч. Клетки промывали, ресуспендировали в 25 мкл/на лунку FACS-буфера, содержащего 1 мкг/мл конъюгированного с BV711 крысиного IgG2a против мышиного CD8 (клон 53-6.7, фирма BioLegend, каталожный №100748) и 2 мкг/мл конъюгированного с BV421 крысиного IgG2a против мышиного CD4 (клон RM4-5, фирма BioLegend, каталожный №100544), и инкубировали в течение 20 мин при 4°С. Клетки промывали и ресуспендировали в 100 мкл/на лунку свежеприготовленном растворе для фиксации/пермеабилизации (фирма eBioscience, каталожный №00-5523-00). После инкубации в течение 30 мин при 4°С клетки промывали D-ЗФР и ресуспендировали в 100 мкл/на лунку свежеприготовленного буфера для пермеабилизации (фирма eBioscience, каталожный №00-5523-00), содержащего 10 мкг/мл конъюгированного с Fluor 488 крысиного IgG2a против мышиного Eomes (эомезодермин) (клон DAN11MAG, фирма eBioscience, каталожный №53-4875-82) и 2 мкг/мл конъюгированного с РЕ крысиного IgG2a против мышиного гранзима В (клон NGZB, фирма eBioscience, каталожный №12-8898-82). Клетки инкубировали в течение 1 ч при КТ в темноте, промывали буфером для пермеабилизации, ресуспендировали в FACS-буфере и получали данные с помощью устройства с 5 лазерами Fortessa (фирма BD Bioscience, программное обеспечение DIVA). Дискриминационные окна устанавливали на CD8+-T-клетки и определяли частоту встречаемости экспрессирующих Eomes и гранзим В CD8+-Т-клеток. С использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.) данные наносили на графики и кривые рассчитывали на основе подгонки кривой нелинейной регрессии (надежный подбор).
Оптимальная активация CD8+-Т-клеток индуцировали экспрессию эомезодермина (Eomes), фактора транскрипции Т-бокса, регулирующего цитолитические эффекторные механизмы CD8+-Т-клеток (Glimcher и др., 2004), и цитолитических ферментов типа гранзима В. Как продемонстрировано на фиг. 32А и 32Б, применяемая концентрация 0,5 мкг/мл антитела к мышиному CD3 была субоптимальной и частота экспрессирующих гранзим В CD8+-Т-клеток не превышала 30%, а частота встречаемости экспрессирующих эомезодермин (eomes) CD8+-Т-клеток не превышала 18%. Только при добавлении в достаточном количестве клона 20G2 мышиного IgG1 против мышиного 4-1ВВ увеличивалось количество CD8+-Т-клеток, которые начинали экспрессировать гранзим В (фиг. 32А) и эомезодермин (фиг.32Б). Это доказывает, что клон 20G2 является агонистическим связывающим агентом и обладает способностью активировать Т-клетки путем привлечения мышиного 4-1ВВ. Если перекрестное сшивание клона 20G2 нарушали с помощью мутации DAPG, то никакого повышения активации не обнаружено, это свидетельствует о том, что и в случае мышиных клеток перекрестное сшивание антитела к 4-1ВВ необходимо для индукции передачи сигналов 4-1ВВ в прямом направлении. Таким образом, мутация DAPG препятствует достаточному перекрестному сшиванию и презентации антитела через Fc-связывающий рецептор. Неспецифические в отношении мышиного 4-1ВВ контроли изотипов не оказывали воздействия на Т-клеточную активацию.
8.3 Функциональные свойства связывающего мышиный 4-1ВВ клона 20G2 in vivo
Обработка мышей агонистическим крысиным IgG2a к мышиному 4-1ВВ приводит к накоплению активированных Т-клеток в печени у не имеющих опухоли мышей (Dubrot и др., Cancer Immunol Immunother. 59(8), 2010, cc. 1223-1233; Niu и др., J Immunol. 178(7), 2007, cc. 4194-4213) и у несущих опухоли мышей (Wang и др. J Immunol. 185(12), 2010, cc. 7654-7662; Kocak и др., Cancer Res. 66(14), 2006, cc. 7276-7284) и индуцирует воспаление печени. Аналогично этому, фаза II клинических испытаний с использованием антитела к 4-1ВВ урелумаба (huIgG4), которую проводили на пациентах с меланомой IV стадии, была прекращена из-за необычно высокой частоты встречаемости гепатита четвертой стадии (Ascierto Р. и др., Semin Oncol. 37(5), 2010, сс. 508-516). Для решения вопроса о том, может ли мышиный IgG1 против мышиного 4-1ВВ индуцировать накопление Т-клеток в печени, что описано для агонистических антител к мышиному 4-1ВВ в виде крысиных IgG2a, а также для решения вопроса о том, может ли мутация DAPG препятствовать этому, наивных здоровых самок мышей линии C57BL/6 (возраст 7-9 недель, фирма Charles River, Франция) обрабатывали IgG1 к мышиному 4-1ВВ и клоном IgG DAPG 20G2. Мышей выдерживали и размещали согласно представленному выше в настоящем описании протоколу. 150 мкг антитела инъецировали внутривенно в хвостовую вену еженедельно в течение 3 недель. Мышей умерщвляли путем смещения шейных позвонков через 1 день после второй инъекции и через 1 день и 8 дней после третьей инъекции. Селезенки и печень иссекали и хранили на льду в среде RPMI 1640, дополненной 10 об. % инактивированной тепловой обработкой FBS и 1 об. % GlutaMAX-I. Спленоциты выделяли согласно описанному выше методу (раздел 8.2). Лейкоциты из печени выделяли следующим образом: каждую печень переносили в С-пробирку (фирма Miltenyi Biotec, каталожный №130-096-334), содержащую 5 мл RPMI 1640, дополненной 0,3 мг/мл диспазы II (фирма Roche Applied Science, каталожный №04942078001), 1 мкг/мл ДНКазы I (фирма Roche Applied Science, каталожный №11284932001) и 2 мг/мл коллагеназы D (фирма Roche Applied Science, каталожный №11088882001), ткань разрушали с помощью мягкого диссоциатора MACS Octo, используя программу «m_liver_01» (фирма Miltenyi Biotec, каталожный №130-095-937) и печени инкубировали в течение 30 мин при 37°С. Расщепленные печени разрушали второй раз с помощью диссоциатора MACS Octo, используя программу «m_liver_02». После центрифугирования (400×g, 8 мин, КТ) клеточный дебрис ресуспендировали в 7 мл буфера для лизиса АСК и гемолиз осуществляли в течение 10 мин при 37°С. Гемолиз прекращали, добавляя среду RPMI 1640, дополненную 10% FBS, 1 об. % GlutaMAX-I. 106 клеток/на лунку спленоцитов или лейкоцитов из печени переносили в круглодонный 96-луночный планшет для суспензии клеток (фирма Greiner bio-one, cellstar, каталожный №650185) и однократно промывали D-ЗФР.
Клетки ресуспендировали в 100 мкл/на лунку раствора, содержащего разведенный в 1:5000 раз зеленый фиксирующий краситель для определения жизнеспособности (LIVE/DEAD Fixable Green Dead Cell Stain) (фирма Live Technologies, каталожный № L-23101), и инкубировали в течение 30 мин при 4°С. Затем клетки промывали и ресуспендировали в 50 мкл/на лунку в FACS-буфере, содержащем 6 мкг/мл конъюгированного с РЕ IgG сирийского хомяка против мышиного CD137 (клон 17В5, фирма BioLegend, каталожный №106106) или применяемого в качестве контроля изотипа антитела сирийского хомяка (клон SHG-1, фирма BioLegend, каталожный №402008), 2 мкг/мл конъюгированного с PerCP/Cy5.5 IgG армянского хомяка против мышиного TCR-((клон Н57-597, фирма BioLegend, каталожный №109228), 5 мкг/мл конъюгированного с РЕ/Су7 крысиного IgG1 против мышиного CD25 (клон РС61, фирма BD Bioscience, каталожный №552880) или применяемого в качестве контроля изотипа крысиного IgG1 λ (клон А110-1, фирма BD Bioscience, каталожный №552869), 2 мкг/мл меченного с помощью Alexa Fluor 700 крысиного IgG2a (против мышиного CD8a (клон 53-6.7, фирма BD Bioscience, каталожный №557959), 0,25 мкг/мл конъюгированного с BV421 крысиного IgG2b (против мышиного CD4 (клон GK1.5, фирма BioLegend, каталожный №100438), 4 мкг/мл конъюгированного с BV605 IgG армянского хомяка против мышиного CD11c (клон N418, фирма BioLegend, каталожный №117333) или применяемого в качестве контроля изотипа антитела армянского хомяка (клон HTK888, фирма BioLegend, каталожный №400943), и инкубировали в течение 30 мин при 4°С. Клетки промывали и ресуспендировали в 100 мкл/на лунку свежеприготовленного раствора для фиксации / пермеабилизации (фирма eBioscience, каталожный №00-5523-00). После инкубации в течение 30 мин при 4°С клетки промывали с помощью свежеприготовленного буфера для пермеабилизации (фирма eBioscience, каталожный №00-5523-00) и ресуспендировали в 50 мкл/на лунку буфера для пермеабилизации, содержащего 0,2 мкг/мл конъюгированного с eF660 крысиного IgG2a (против мышиного Ki67 (клон SolA15, фирма eBioscience, каталожный №50-5698-82). Клетки инкубировали в течение 30 мин при 4°С, промывали дважды буфером для пермеабилизации и фиксировали, используя 50 мкл/на лунку D-ЗФР, содержащего 1% формальдегида. На следующий день получали данные для клеток с помощью устройства с 5 лазерами Fortessa (фирма BD Bioscience, программное обеспечение DIVA). Данные анализировали с помощью FlowJo (FlowJo, фирма LLC), дискриминационные окна устанавливали на CD8+- или CD4+-Т-клетки и определяли частоту встречаемости CD137+-, CD25+-, CD11c+- и Ki67+-Т-клеток. Данные наносили на графики с использованием программы Graph Pad Prism (фирма Graph Pad Software Inc.).
Как продемонстрировано на фиг.33, только обработка мышей клоном 20G2 мышиного IgG1 против мышиного 4-1ВВ индуцировала накопление, пролиферацию и повышение экспрессии 4-1ВВ (CD137) на CD8+-Т-клетках в печени, в то время как предупреждение перекрестного сшивания посредством FcR-связывания с помощью мутации DAPG не приводило к активации CD8+-T-клеток. Предупреждение FcR-перекрестного сшивания с помощью мутации DAPG препятствовало активации CD8+-Т-клеток в печени (фиг. 33), хотя обе молекулы имели сходные особенности связывания с мышиным CD137, экспрессируемым на Т-клетках (фиг. 25Б и 25Г), и поэтому обладали одинаковыми потенциальными способностями к активации.
Пример 9
Получение, очистка и характеризация биспецифических двухвалентных антител, мишенями которых являются 4-1ВВ и ассоциированный с опухолью антиген (ТАА)
9.1 Создание биспецифических двухвалентных антител, мишенями которых являются 4-1ВВ и фибробласт-активирующий белок (FAP) (формат 2+2)
Получали биспецифические агонистические антитела к 4-1ВВ, обладающие двухвалентным связыванием с 4-1ВВ и FAP. Технологию Crossmab, описанную в международной заявке на патент WO 2010/145792 А1, применяли для снижения образования неправильно спаренных легких цепей.
Создание и получение связывающих FAP агентов описано в WO 2012/020006 А2, которая включена в настоящее описание в качестве ссылки.
В этом примере «скрещенный» модуль Fab (VHCL) связывающего FAP агента 28Н1 сливали на С-конце с содержащей «впадину» тяжелой цепью антитела к 4-1ВВ hu IgG1 с помощью коннекторной последовательности (G4S)4. Это слияние тяжелой цепи совместно экспрессировали с легкой цепью антитела к 4-1ВВ и соответствующей «скрещенной» легкой цепью антитела к FAP (VLCH1). Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей для элиминации связывания с рецепторами Fc гамма согласно методу, описанному в опубликованной международной заявке на патент WO 2012/130831 А1. Указанная полученная биспецифическая двухвалентная конструкция является аналогом одной из указанных на фиг. 34А.
В таблице 60 представлены соответственно нуклеотидные и аминокислотные последовательности зрелых биспецифических двухвалентных анти-4-1ВВ/анти-FAP человеческих антител IgG1 P329GLALA.





Все гены кратковременно экспрессировали под контролем химерного промотора MPSV, состоящего из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр).
Биспецифические анти-4-1ВВ/ анти-FAP-конструкции получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1 («вектор тяжелой цепи : «вектор легкой цепи 1» : «вектор легкой цепи 2»).
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% CO2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку биспецифических конструкций из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слияний антиген-Fc и антител.
Белок концентрировали и фильтровали перед загрузкой на колонку HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенную раствором, содержащим 20 мМ гистидин, 140 мМ NaCl, рН 6,0.
Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу биспецифических конструкций анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С (таблица 61).
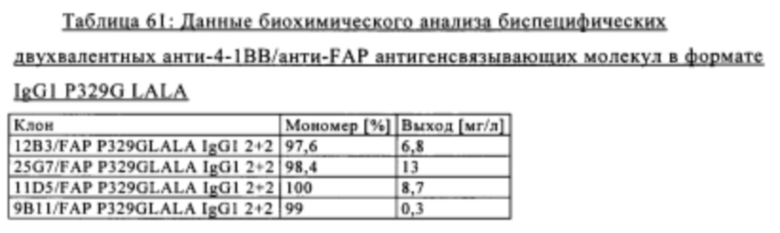
9.2 Связывание биспецифических двухвалентных антител, мишенями которых являются 4-1ВВ и FAP
9.2.1 Поверхностный плазмонный резонанс (одновременное связывание)
Способность связываться одновременно с конструкцией человеческий 4-1BB-Fc(kih) и человеческим FAP оценивали с помощью поверхностного плазмонного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01 М HEPES, рН 7,4, 0,15 М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
Конструкцию, содержащую биотинилированный человеческий 4-1ВВ в слиянии с Fc(kih), непосредственно сшивали с различными проточными ячейками стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 1000 резонансных единиц (RU). Биспецифические антитела, мишенями которых являются 4-1ВВ и FAP, пропускали через проточные ячейки, используя концентрацию порядка 250 нМ, со скоростью потока 30 мкл/мин и диссоциацию устанавливали на 0 с. Человеческий FAP инъецировали в проточные ячейки в качестве второго аналита со скоростью потока 30 мкл/мин в течение 90 с в концентрации 250 нМ (фиг. 34Б). Мониторинг диссоциации комплекса осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок. Все биспецифические конструкции обладали способностью связываться одновременно с человеческим 4-1ВВ и человеческим FAP (фиг. 35А-35Г).
9.2.2 Связывание на клетках
9.2.2.1 Связывание на экспрессирующих человеческий 4-1ВВ клетках: покоящиеся и активированные мононуклеарные лейкоциты периферической крови (РВМС) человека
Человеческие РВМС выделяли, активировали для индукции 4-1ВВ и применяли для оценки способности к связыванию антител с человеческим 4-1ВВ согласно методу, описанному выше в примере 7.1.2.
Для демонстрации того, что связанный с Fc FAP-таргетинг не влияет на связывание 4-1ВВ-специфических клонов антител или на детекцию посредством конъюгированного с РЕ F(ab')-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического при создании изобретения оценивали способность к связыванию нацеленных на FAP или «ненацеленных» DP47-конструкций в формате 2+2 по сравнению с родительскими форматами huIgG P329G LALA с покоящимися человеческими CD4+- и CD8+-Т-клетки (фиг. 38А-38Е) и активированными человеческими CD4+- и CD8+-Т-клетками (фиг. 39А-39Е). Аналогично результатам, представленным на фиг. 23, и в этом случае нацеленные на FAP или нацеленные на DP47 специфические в отношении человеческого 4-1ВВ молекулы не связывались с покоящимися Т-клетками (фиг. 38А-38Е), а связывались в основном с активированными CD8+-Т-клетками (фиг. 39Г, Д и Е). Превращение в нацеленные на FAP или «ненацеленные» DP47-форматы 2+2 не изменяло значительно особенности связывания с экспрессирующими 4-1ВВ Т-клетками. Различия можно объяснить тем, что для детекции применяли Fc-специфическое поликлональное вторичное антитело (например, Fc-слияние может маскировать эпитопы и снижать детекцию). Величины ЕС50 варьировались от донора к донору в зависимости от количества экспрессируемого 4-1ВВ после активации. Это объясняет различия величин ЕС50, представленных в таблице 62, по сравнению с величинами для родительских антител, которые приведены в таблице 48. Площади под кривой связывания с активированными CD8+-Т-клетками представлены на фиг. 40.


9.2.2.2 Связывание с экспрессирующими человеческий FAP опухолевыми клетками
Для анализов связывания с экспрессируемым на клеточной поверхности фибробласт-активирующим белком (FAP) применяли клеточную линию NIH/3T3-huFAP, клон 19 или клеточную линию человеческой меланомы WM-266-4 (АТСС CRL-1676). Линию NIH/3T3-huFAP, клон 19 создавали путем трансфекции линии клеток фибробластов мышиных эмбрионов NIH/3T3 (АТСС CRL-1658) экспрессионной плазмидой pETR4921, кодирующей человеческий FAP под контролем промотора CMV. Клетки поддерживали в присутствии 1,5 мкг/мл пуромицина (фирма InvivoGen, каталожный № ant-pr-5). 2×105 экспрессирующих FAP опухолевых клеток добавляли в каждую лунку круглодонных 96-луночных планшетов для суспензионных клеток (Greiner bio-one, cellstar, каталожный №650185). Клетки промывали однократно 200 мкл D-ЗФР, ресуспендировали в 100 мкл/на лунку холодного (4°С) D-ЗФР-буфера, содержащего разведенный в 1:5000 раз зеленый фиксирующий краситель для определения жизнеспособности Fixable Viability Dye eFluor 450 (фирма eBioscience, каталожный №65086318) или Fixable Viability Dye eFluor 660 (фирма eBioscience, каталожный №65-0864-18), и инкубировали в течение 30 мин при 4°С. Клетки промывали однократно 200 мкл холодного (4°С) D-ЗФР-буфера, ресуспендировали в 50 мкл на лунку холодного (4°С) FACS-буфера, содержащего в различных титрованных концентрациях 4-1ВВ-специфические нацеленные на FAP и «ненацеленные» антитела, с последующей инкубацией в течение 1 ч при 4°С. После четырехкратной промывки с использованием 200 мкл/на лунку клетки окрашивали с использованием 50 мкл/на лунку холодного (4°С) FACS-буфера, содержащего 2,5 мкг/мл конъюгированного с РЕ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-116-098) или 30 мкг/мл конъюгированного с ФИТЦ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического AffiniPure (фирма Jackson ImmunoResearch, каталожный №109-096-098), в течение 30 мин при 4°С. Клетки промывали дважды, используя 200 мкл холодного (4°С) FACS-буфера, и клетки ресуспендировали в 50 мкл/на лунку D-ЗФР, содержащего 1% формальдегида для фиксации. В этот же или на следующий день клетки ресуспендировали в 100 мкл FACS-буфера и получали данные с помощью устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA) или анализатора MACSQuant 10 (фирма Miltenyi Biotec).
Как продемонстрировано на фиг. 41А-41Е, все протестированные нацеленные на FAP молекулы в формате 2+2, в отличие от нацеленных на DP47 или родительских форматов huIgG1 P293G LALA, включая 4-1ВВ-связывающие клоны 25G7, 11D5 и 12В3, связывались эффективно с экспрессирующими человеческий FAP клетками. Таким образом, изученные FAP (28Н1)-нацеленные конструкции 2+2 могли специфически связываться с экспрессирующими FAP клетками. На фиг. 42 обобщены данные о площадях под кривой (AUC) для кривых, характеризующих связывание с NIH/3T3-huFAP. Изученные форматы и FAP-связывающие клоны представлены ниже на графике в виде пиктограмм. Из них видно, что все изученные нацеленные на FAP форматы 2+2 характеризовались сходными AUC.
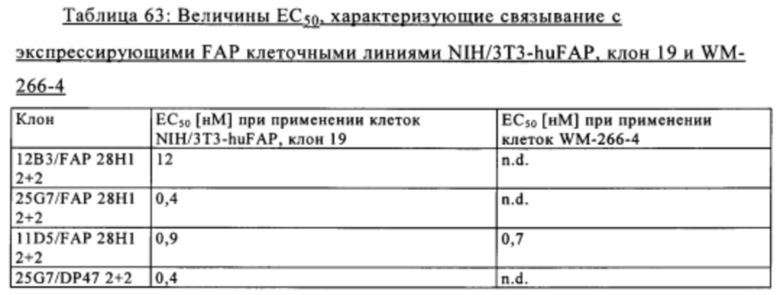
9.2.3 Активация NFκB
9.2.3.1 Создание клеток HeLa, экспрессирующих человеческий 4-1ВВ и NF-κВ-люциферазу
Клеточную линию карциномы шейки матки HeLa (АТСС CCL-2) трансдуцировали плазмидой, основой которой является экспрессионный вектор pETR10829, содержащий последовательность человеческого 4-1ВВ (Uniprot, код доступа Q07011) под контролем промотора CMV, и ген, обусловливающий устойчивость к пуромицину. Клетки культивировали в среде DMEM, дополненной 10 об. % FBS, 1 об. % GlutaMAX-I и 3 мкг/мл пуромицина.
Трансдуцированные 4-1ВВ клетки HeLa тестировали в отношении экспрессии 4-1ВВ с помощью проточной цитометрии: 0,2×106 живых клеток ресуспендировали в 100 мкл FACS-буфера, содержащего 0,1 мкг конъюгированного с PerCP/Су5.5 мышиного IgG1κ, клон 4В4-1 к человеческому 4-1ВВ (BioLegend, каталожный №309814) или контроль изотипа (конъюгированное с PerCP/Су5.5 применяемое в качестве контроля изотипа мышиное антитело IgG1κ, клон МОРС-21, фирма BioLegend, каталожный №400150), и инкубировали в течение 30 мин при 4°С. Клетки промывали дважды FACS-буфером, ресуспендировали в 300 мкл FACS-буфера, содержащего 0,06 мкг DAPI (фирма Santa Cruz Biotec, каталожный № Sc-3598), и данные получали с использованием устройства с 5 лазерами LSR-Fortessa (фирма BD Bioscience, программное обеспечение DIVA). Осуществляли серийные разведения для создания единичных клонов с использованием следующего метода: трансдуцированные человеческим 4-1ВВ клетки HeLa ресуспендировали в среде до достижения плотности 10, 5 и 2,5 клеток/мл и 200 мкл клеточных суспензий переносили в обработанные круглодонные 96-луночные планшеты для культуры ткани (по 6 планшетов для каждой концентрации клеток, фирма ТРР, каталожный №92697). Единичные клоны собирали, размножали и тестировали в отношении экспрессии 4-1ВВ согласно описанному выше методу. Клон с наиболее высоким уровнем экспрессии 4-1ВВ (клон 5) отбирали для последующей трансфекции экспрессионным вектором для NF-κВ-люциферазы 5495р Tranlucent HygB. Вектор придавал трансфектированным клеткам как устойчивость к гигромицину В, так и способность экспрессировать люциферазу под контролем NF-kB-реагирующего элемента (фирма Panomics, каталожный № LR0051). Для трансфекции клон 5 клеток HeLa с человеческим 4-1ВВ культивировали до 70% конфлюэнтности. Вносили 50 мкг (40 мкл) линеаризованного (с помощью рестриктаз AseI и SalI) экспрессионного вектора 5495р Tranlucent HygB в стерильную кювету модульной системы для электропорации Gene Pulser/MicroPulser с 0,4-сантиметровой щелью (фирма Biorad, каталожный №65-2081). Добавляли 2,5×106 клеток клона 5 HeLa с человеческим 4-1 ВВ в 400 мкл среды DMEM без добавок и осторожно перемешивали с раствором плазмиды. Трансфекцию клеток осуществляли с помощью общей системы Gene Pulser Xcell (фирма Biorad, каталожный №165-2660), применяя следующие установки: экспоненциальный импульс, емкость 500 мкФ, напряжение 160 В, сопротивлениие ∞. Немедленно после импульсной обработки трансфектированные клетки переносили в 75 см2 - колбу для культуры ткани (фирма ТРР, каталожный №90075) с 15 мл теплой (37°С) среды DMEM, дополненной 10 об. % FBS и 1 об. % GlutaMAX-I. На следующий день добавляли культуральную среду, содержащую 3 мкг/мл пуромицина и 200 мкг/мл гигромицина В (фирма Roche, каталожный №10843555001). Выжившие клетки размножали и осуществляли их серийное разведение согласно описанному выше методу с получением единичных клонов.
Клоны тестировали в отношении экспрессии 4-1ВВ согласно описанному выше методу и в отношении NF-κВ-люциферазной активности следующим образом: клоны собирали в селекционной среде и подсчитывали с помощью устройства Cell Counter Vi-cell xr 2.03 (фирма Beckman Coulter, каталожный №731050). Плотность клеток доводили до 0,33×106 клеток/мл и 150 мкл указанной клеточной суспензии переносили в каждую лунку стерильного белого плоскодонного 96-луночного планшета для культуры ткани с крышкой (фирма Greiner bio-one, каталожный №655083). Клетки инкубировали при 37°С и 5% СО2 в течение ночи в инкубаторе для клеток (Hera Cell). На следующий день добавляли 50 мкл среды, содержащей в различных концентрациях рекомбинантный человеческий фактор некроза опухоли альфа (rhTNF-α, фирма PeproTech, каталожный №300-01А), в каждую лунку 96-луночного планшета, получая конечную концентрацию rhTNF-α 100, 50, 25, 12,5, 6,25 и 0 нг/лунку. Клетки инкубировали в течение 6 ч при 37°С и 5% СО2 и затем промывали три раза, применяя 200 мкл/лунку D-ЗФР. Добавляли репортерный буфер для лизиса (фирма Promega, каталожный №Е3971) в каждую лунку (40 мкл) и планшеты выдерживали в течение ночи при -20°С. На следующий день замороженные клетки и идентифицирующий буфер (система для анализа люциферазы 1000, фирма Promega, каталожный № Е4550) подвергали оттаиванию, доводя до комнатной температуры. Добавляли 100 мкл идентифицирующего буфера в каждую лунку и планшет анализировали максимально быстро с использованием ридера для микропланшетов SpectraMax М5/М5е и программного обеспечения SoftMax Pro (фирма Molecular Devices). Люциферазную активность оценивали по количеству измеренных единиц испускаемого света (URL) в течение 500 мс/лунку, превышающему уровень в контроле (без добавления rhTNF-α). Для дальнейшего исследования был отобран клон 26 NF-κB-luc-4-1BB-HeLa, характеризующийся наиболее высокой люциферазной активностью и значительным уровнем экспрессии 4-1ВВ.
9.2.3.2 Активация NF-κВ в репортерных клетках Hela, экспрессирующих человеческий 4-1ВВ, при совместном культивировании с экспрессирующими FAP опухолевыми клетками
Клетки HeLa-hu4-1BB-NF-κB-luc, клон 26 собирали и ресуспендировали в среде DMEM, дополненной 10 об. % FBS и 1 об. % GlutaMAX-I, до концентрации 0,2×106 клеток/мл. 100 мкл (2×104 клеток) указанной клеточной суспензии переносили в каждую лунку стерильного белого плоскодонного 96-луночного планшета для культуры ткани с крышкой (фирма Greiner bio-one, каталожный №655083) и планшет инкубировали при 37°С и 5% СО2 в течение ночи. На следующий день добавляли 50 мкл среды, содержащей в титрованных концентрациях нацеленные на FAP конструкции к человеческому 4-1ВВ или их родительские антитела huIgG1 P329G LALA. Экспрессирующие FAP клетки NIH/3T3-huFAP, клон 19 или WM-266-4 ресуспендировали в среде DMEM, дополненной 10 об. % FBS и 1 об. % GlutaMAX-I, до концентрации 2×106 клеток/мл.
Суспензию экспрессирующих FAP опухолевых клеток (50 мкл) или только среду в качестве отрицательного контроля (без опухолевых FAP+-клеток) добавляли в каждую лунку и планшеты инкубировали в течение 6 ч при 37°С и 5% СО2 в инкубаторе для клеток. Клетки промывали дважды, используя 200 мкл/лунку D-ЗФР. Добавляли в каждую лунку 40 мкл свежеприготовленного репортерного буфера для лизиса (фирма Promega, каталожный № Е3971) и планшет выдерживали в течение ночи при -20°С. На следующий день планшеты с замороженными клетками и идентифицирующий буфер (система для анализа люциферазы 1000, фирма Promega, каталожный № Е4550) подвергали оттаиванию, доводя до комнатной температуры. Добавляли 100 мкл идентифицирующего буфера в каждую лунку и максимально быстро измеряли люциферазную активность с помощью ридера для микропланшетов SpectraMax М5/М5е и программного обеспечения SoftMax Pro (фирма Molecular Devices).
Нацеленные на FAP (28Н1) конструкции 2+2 (клон 25G7, 11D5 или 12В3) инициировали активацию пути передачи сигналов NFκB в репортерной клеточной линии в присутствии клеток NIH/3T3-huFAP (фиг. 43В, Е и И) в зависимости от концентрации. В противоположность этому, «ненацеленная» контрольная молекула (4-1ВВ (25G7) × DP47 2+2) или родительские антитела P329G LALA не инициировали такое же действие при применении в любых протестированных концентрациях. Указанная активность изученных нацеленных на FAP антител к человеческому 4-1ВВ в формате 2+2 строго зависела от экспрессии FAP на клеточной поверхности добавленных перекрестносшивающих клеток, поскольку никакой активации NF-kB не удалось обнаружить в отсутствии экспрессирующих FAP опухолевых клеток (фиг. 43А, Г и Ж) или в присутствии линии опухолевых клеток, экспрессирующей низкие уровни FAP (WM-266-4) (фиг. 43Б, Д и З). Таким образом, активация зависела от уровня экспрессии FAP, а не от силы аффинности анти-4-1ВВ-связывающего агента, поскольку степень активации при сравнении конструкций 25G7 × FAP 2+2, 11D5 × FAP 2+2 и 12В3 × FAP 2+2 (фиг. 44А и 44Б и таблица 64) не коррелировала с их аффинностью связывания с 4-1ВВ (фиг. 42).

Пример 10
Получение, очистка и характеризация биспецифических одновалентных антител, мишенями которых являются 4-1ВВ и ассоциированный с опухолью антиген (ТАА)
10.1 Создание биспецифических антител, мишенями которых являются 4-1ВВ и фибробласт-активируюший белок (FAP), в одновалентном формате (формат 1+1)
Получали биспецифические агонистические антитела к 4-1ВВ, обладающие одновалентным связыванием с 4-1ВВ и FAP. Для снижения образования неправильно спаренных легких цепей применяли технологию Crossmab, описанную в международной заявке на патент WO 2010/145792 А1.
Создание и получение связывающих FAP агентов описано в WO 2012/020006 А2, которая включена в настоящее описание в качестве ссылки.
Биспецифическая конструкция характеризовалась одновалентным связыванием с 4-1ВВ и с FAP (фиг. 34В). Она содержала «скрещенный» модуль Fab (VHCL) связывающего FAP агента 28Н1, слитый с содержащей «выступ» тяжелой цепью антитела к 4-1ВВ huIgG1 (содержащего мутации S354C/T366W). Тяжелую цепь Fc с «впадиной» (содержащую мутации Y349C/T366S/L368A/Y407V) сливали с Fab-фрагментом антитела к 4-1ВВ. Комбинация нацеленной анти-FAP-Fc содержащей «выступ» конструкции с анти-4-1BB-Fc содержащей «впадину» конструкцией позволяла получать гетеродимер, который включал специфически связывающийся с FAP Fab и специфически связывающийся с 4-1ВВ Fab.
Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей для элиминации связывания с рецепторами Fc гамма согласно методу, описанному в опубликованной международной заявке на патент WO 2012/130831 А1.
Биспецифические одновалентные анти-4-1ВВ и анти-FAP huIgG1 P329GLALA конструкции получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих, используя полиэтиленимин. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1:1 («вектор тяжелой цепи с «выступом) : «вектор легкой цепи1» : «вектор тяжелой цепи с «впадиной» : «вектор легкой цепи2).
В результате получали биспецифические одновалентные конструкции и очищали их согласно методу, описанному для биспецифических двухвалентных анти-4-1ВВ и анти-FAP huIgG1 P329GLALA (см. пример 9.1). Нуклеотидные и аминокислотные последовательности представлены в таблице 65.



Все гены кратковременно экспрессировали под контролем химерного промотора MPSV, состоящего из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр).
Биспецифические анти-4-1ВВ/анти-FAP-конструкции получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1:1 («вектор тяжелой цепи с «выступом»: «вектор тяжелой цепи с «впадиной» : «вектор легкой цепи1» : «вектор легкой цепи2»).
Для получения в 500-миллилитровые встряхиваемые колбы засевали 400 млн клеток HEK293 EBNA за 24 ч до трансфекции. Для трансфекции клетки центрифугировали в течение 5 мин при 210 × g и супернатант заменяли предварительно нагретой средой CD СНО. Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2. После инкубации добавляли 160 мл среды F17 и клетки культивировали в течение 24 ч. Через 1 день после трансфекции добавляли 1 мМ вальпроевую кислоту и 7% Feed. После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 15 мин при 210 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку биспецифических конструкций из супернатантов клеточной культуры осуществляли с помощью аффинной хроматографии с использованием белка А согласно методу, описанному выше для очистки слитых молекул антиген-Fc или антител. Концентрацию белка определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу биспецифических конструкций анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen, США) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов в образцах анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ K2HPO4, 125 мМ NaCl, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С.
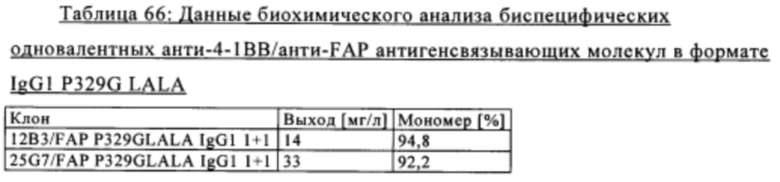
10.2 Получение, очистка и характеризация антигенов FAP в качестве «инструмента» для скрининга
Для того чтобы оценивать связывание с FAP, последовательности ДНК, кодирующие эктодомены FAP человека, мышей или обезьян циномолгус, слитые с С-концевой His-меткой, клонировали в экспрессионном векторе, который содержит химерный промотор MPSV, состоящий из корового промотора MPSV, объединенного с энхансерным фрагментом промотора CMV. Экспрессионный вектор содержал также область oriP для эписомальной формы репликации в клетках-хозяевах, содержащих EBNA (ядерный антиген вируса Эпштейна-Барр). Аминокислотная и нуклеотидная последовательности меченного His ECD человеческого FAP представлены в SEQ ID NO: 85 и 86 соответственно. В SEQ ID NO: 88 и 89 представлены аминокислотная и нуклеотидная последовательности соответственно меченного His ECD мышиного FAP. В SEQ ID NO: 90 и 91 представлены аминокислотная и нуклеотидная последовательности соответственно меченного His ECD FAP обезьян циномолгус.
FAP-антигены получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих с использованием полиэтиленимина (ПЭИ; фирма Polysciences Inc.).
Для получения 200 мл в 500-миллилитровые встряхиваемые колбы засевали 300 млн клеток HEK293 EBNA за 24 ч до трансфекции в среду 100% F17 + 6 мМ глутамин. Для трансфекции 400 млн клеток центрифугировали в течение 5 мин при 210×g и супернатант заменяли 20 мл предварительно нагретой среды CD СНО (фирма Gibco). Экспрессионные векторы смешивали в 20 мл среды CD СНО до достижения конечного количества ДНК 200 мкг. После добавления 540 мкл ПЭИ (1 мг/мл) (фирма Polysciences Inc.) раствор интенсивно перемешивали в течение 15 с и инкубировали в течение 10 мин при комнатной температуре. Затем клетки смешивали с раствором ДНК/ПЭИ, переносили в 500-миллилитровую встряхиваемую колбу и инкубировали в течение 3 ч при 37°С в инкубаторе в атмосфере с 5% СО2 и встряхивали при 165 об/мин. После инкубации добавляли 160 мл среды F171 и добавки (1 мМ вальпроевая кислота, 5 г/л PepSoy (соевый пептид). 6 мМ L-глутамин) и клетки культивировали в течение 24 ч. Через 24 ч после трансфекции к клеткам добавляли содержащую аминокислоты и глюкозу добавку в количестве, составлявшем 12% от конечного объема (24 мл). После культивирования в течение 7 дней супернатант собирали центрифугированием в течение 45 мин при 2000-3000 × g. Раствор стерилизовали фильтрацией (0,22-микрометровый фильтр), дополняли азидом натрия до конечной концентрации 0,01% (мас./об.) и выдерживали при 4°С.
Очистку антигенов из супернатантов клеточных культур осуществляли с помощью двух стадий очистки с использованием первой стадии аффинной хроматографии с применением либо 5-миллилитровой колонки IMAC (фирма Roche), либо 5-миллилитровой колонки NiNTA (фирма Qiagen) с последующим применением гель фильтрации с использованием колонки HiLoad Супердекс 200 (фирма GE Healthcare), уравновешенной буфером, содержащим 20 мМ гистидин, 140 мМ NaCl, рН 6,0 или 2 мМ MOPS, 150 мМ NaCl, 0,02% NaN3, рН 7,3, соответственно.
При применении аффинной хроматографии на колонке IMAC (фирма Roche) уравновешивание осуществляли буфером, содержащим 25 мМ Трис-HCl, 500 мМ хлорид натрия, 20 мМ имидазол, рН 8,0, используя 8 CV. Вносили супернатант и несвязанный белок отмывали 10 CV буфера, содержащего 25 мМ Трис-HCl, 500 мМ хлорид натрия, 20 мМ имидазол, рН 8,0. Связанный белок элюировали с помощью линейного градиента 20 CV (от 0 до 100%) буфера, содержащего 25 мМ Трис-HCl, 500 мМ хлорид натрия, 20 мМ имидазол, рН 8,0, с последующей стадий при 100%, используя 8 CV буфера.
При применении аффинной хроматографии на колонке NiNTA (фирма Qiagen) уравновешивание осуществляли буфером, содержащим 50 мМ фосфат натрия, 300 мМ хлорид натрия, рН 8,0, используя 8 CV. Вносили супернатант и несвязанный белок удаляли путем промывки буфером, содержащим 50 мМ фосфат натрия, 300 мМ хлорид натрия, рН 8,0 используя его в объеме, кратном 10 объемам колонки. Связанный белок элюировали с помощью линейного градиента 20 CV (от 0 до 100%) буфера, содержащего 50 мМ фосфат натрия, 300 мМ хлорид натрия, 500 мМ имидазол, рН 7,4, с последующей стадией, на которой использовали 5CV буфера, содержащего 50 мМ фосфат натрия, 300 мМ хлорид натрия, 500 мМ имидазол, рН 7,4. Затем колонку вновь уравновешивали, используя 8 CV буфера, содержащего 50 мМ фосфат натрия, 300 мМ хлорид натрия, рН 8,0.
К собранным фракциям затем добавляли 1/10 (об./об.) 0,5М ЭДТК, рН 8,0. Белок концентрировали с помощью колонок Vivaspin (номинальная отсекаемая масса 30 кДа, фирма Sartorius) и фильтровали перед внесением в колонку HiLoad Супердекс 200 (фирма GE Healthcare) уравновешенную буфером, содержащим 2 мМ MOPS, 150 мМ NaCl, 0,02% NaN3, pH 7,3 или 20 мМ гистидин, 140 мМ NaCl, рН 6,0.
Концентрацию очищенных антигенов определяли на основе измерения оптической плотности (ОП) при 280 нм с использованием коэффициента молярной экстинкции, рассчитанного на основе аминокислотной последовательности. Чистоту и молекулярную массу антигенов анализировали с помощью КЭ-ДСН в присутствии восстановителя (фирма Invitrogen) или без него с помощью устройства LabChipGXII (фирма Caliper). Содержание агрегатов антигенов анализировали, используя колонку для аналитической гель-фильтрации TSKgel G3000 SW XL (фирма Tosoh), уравновешенную подвижным буфером, содержащим 25 мМ фосфат натрия, 125 мМ хлорид натрия, 200 мМ моногидрохлорид L-аргинина, 0,02% (мас./об.) NaN3, рН 6,7, при 25°С.
10.3 Связывание биспецифических одновалентных антител, мишенями которых являются 4-1ВВ и FAP
10.3.1 Поверхностный плазмонный резонанс (одновременное связывание)
Способность связываться одновременно с конструкцией человеческий 4-1BB-Fc(kih) и человеческим FAP оценивали с помощью поверхностного плазмонного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01М HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
Конструкцию, содержащую биотинилированный человеческий 4-1ВВ в слиянии с Fc(kih), непосредственно сшивали с проточной ячейкой стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 1000 резонансных единиц (RU).
Биспецифические антитела, мишенями которых являются 4-1ВВ и FAP, пропускали через проточные ячейки, используя концентрацию порядка 250 нМ, со скоростью потока 30 мкл/мин и диссоциацию устанавливали на 0 с. Человеческий FAP инъецировали в проточные ячейки в качестве второго аналита со скоростью потока 30 мкл/мин в течение 90 с в концентрации 250 нМ. Мониторинг диссоциации комплекса осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок. Все биспецифические конструкции обладали способностью связываться одновременно с человеческим 4-1ВВ и человеческим FAP.
10.3.2 Связывание на клетках
10.3.2.1 Связывание на экспрессирующих человеческий 4-1ВВ клетках: покоящиеся и активированные мононуклеарные лейкоциты периферической крови (РВМС) человека
Человеческие РВМС выделяли, активировали для индукции 4-1ВВ и применяли для оценки способности к связыванию антител с человеческим 4-1ВВ согласно методу, описанному выше в примере 7.1.2.
Для сравнения одновалентно связывающих 4-1ВВ нацеленных на 4-1BB/FAP конструкций 1+1 и двухвалентно связывающих 4-1ВВ родительских анти-4-1ВВ-специфических клонов huIgG1 P329G LALA конструкции инкубировали с покоящимися или активированными экспрессирующими человеческий 4-1ВВ РВМС и осуществляли детекцию с помощью конъюгированного с РЕ F(ab')2-фрагмента козьего IgG против человеческого IgG, Fcγ-фрагмент-специфического согласно описанному ранее методу. Как продемонстрировано на фиг. 39Г и 39Е, конверсия в одновалентные нацеленные на 4-1ВВ/FAP форматы 1+1 повышала величины ЕС50, характеризующие связывание с 4-1ВВ (таблица 67), и снижали значение MFI (фиг. 39Б/Д и 39В/Е) по сравнению с их родительскими конструкциями huIgG1 P329G LALA. Указанное увеличение EC50 и снижение MFI приводило к получению в большей или меньшей степени делению пополам площади под кривой (AUC) кривых связывания, как продемонстрировано на фиг. 40.

10.3.2.2 Связывание с экспрессирующими человеческий FAP клетками
Связывание с экспрессирующей FAP опухолевой клеткой уже описано выше в разделе 9.2.2.2.
Как продемонстрировано на фиг. 41, нацеленные на FAP молекулы в формате 1+1 в отличие от нацеленных на DP47 молекул в формате 1+1 или клонов родительских антител huIgG1 P293G LALA 25G7 и 12В3, связывались эффективно с экспрессирующими человеческий FAP клетками (фиг. 41Б/Г и 41Д/Е). Таким образом, конструкции в формате 1+1 также могли специфически связываться с экспрессирующими FAP клетками, хотя в результате одновалентного таргетинга FAP с несколько более высокими величинами ЕС50 по сравнению с нацеленными на FAP форматами 2+2 (таблица 68 и фиг. 41Б/Г и 41Д/Е). Однако это оказывало лишь минимальное действие на AUC кривых связывания (фиг. 42).
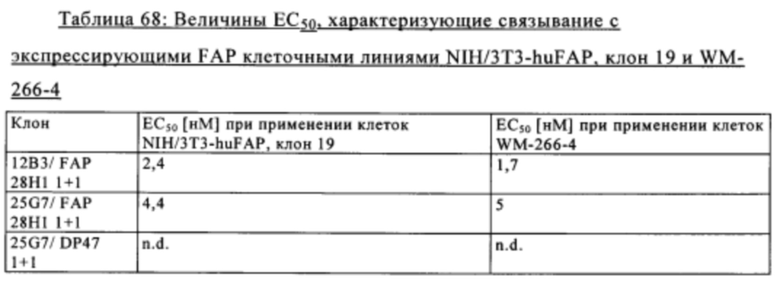
10.3.3. Активация NFκB
Протокол анализа функциональной способности с использованием экспрессирующей человеческий 4-1ВВ репортерной клеточной линии HeLa-hu4-1BB-NFκB-luc уже описан подробно в примере 9.2.3.2.
Нацеленные на FAP (28Н1) конструкции клона 25G7 или 12В3 в формате 1+1 инициировали активацию передачи сигнала NFkB в репортерной клеточной линии в присутствии клеток NIH/3T3-huFAP (фиг. 43Е и И). В противоположность этому, «ненацеленная» контрольная молекула (4-1ВВ (25G7) × DP47 1+1) или родительские антитела P329G LALA не инициировали такое же действие при применении в любых тестируемых концентрациях. Указанная активность нацеленных на FAP антител к человеческому 4-1ВВ в формате 1+1 строго зависела от высокого уровня экспрессии FAP на клеточной поверхности добавленных FAP+-клеток, поскольку никакой активации NF-kB не удалось обнаружить в отсутствии экспрессирующих FAP опухолевых клеток (фиг. 43Г и Ж) или в присутствии линии опухолевых клеток, экспрессирующей низкие уровни FAP (WM-266-4) (фиг. 43Д и З). При сравнении установлено различное поведение клонов 12В3 и 25G7 нацеленных на FAP молекул в формате 1+1 и нацеленных на FAP молекул в формате 2+2. Клон 12В3 нацеленной на FAP конструкции 1+1 имел более высокую величину ЕС50, а также плато на кривой активации, расположенное на более высоком уровне, чем у клона 12В3 нацеленной на FAP конструкция 2+2 (фиг. 43Е) и, таким образом, обе конструкции имели сходные AUC (фиг. 44А), в то время как клон 25G7 нацеленной на FAP конструкции 1+1 имел более высокую величину ЕС50 и плато на кривой активации, расположенное на таком же уровне, что и у клона 25G7 нацеленной на FAP конструкции 2+2 (фиг. 44И), и в результате меньшую AUC (фиг. 44А), например, клон 25G7 нацеленной на FAP молекулы 1+1 явно обладал меньшей эффективностью, чем клон 25G7 нацеленной на FAP молекулы 2+2. Причинами этого могли быть различия между сильным (например, 12В3) и слабым (например, 25G7) 4-1ВВ-связывающим агентом, которые могли становиться более выраженными при одновалентном 4-1ВВ-связывании.

Пример 11
Получение, очистка и характеризация биспецифических антител с двухвалентным связыванием с 4-1ВВ и одновалентным связыванием с ассоциированным с опухолью антигеном (ТАА)
11.1 Создание биспецифических антител с двухвалентным связыванием с 4-1ВВ и одновалентным связыванием с ассоциированным с опухолью антигеном (ТАА) (формат 2+1)
Получали биспецифические агонистические антитела к 4-1ВВ, обладающие двухвалентным связыванием с 4-1ВВ и одновалентным связыванием с FAP (обозначены также как антитела в формате 2+1), согласно процессу, представленному на фиг. 36.
В этом примере первая тяжелая цепь НС1 конструкции содержала следующие компоненты: VHCH1 анти-4-1ВВ-связывающего агента, за которым находилась Fc-область с «выступом», с С-концом которой слит VL- или VH aHTH-FAP-связывающего агента. Вторая тяжелая цепь НС2 содержала VHCH1 анти-4-1ВВ, за которым находилась Fc-область с «впадиной», с С-концом которой слит VH- или VL соответственно анти-FAP-связывающего агента (клон 4В9). Конструирование и получение связывающего FAP агента 4В9 описано в WO 2012/020006 А2, которая включена в настоящее описание в качестве ссылки. Связывающие 4-1ВВ агенты (12В3, 9В11, 11D5 и 25G7) создавали согласно методу, описанному в примере 6. Комбинация цепей нацеленной конструкции анти-FAP-Fc-цепь с «выступом» и конструкции анти-4-1ВВ-Fc-цепь с «впадиной» позволяла получать гетеродимер, который включал связывающий FAP фрагмент и два связывающих 4-1ВВ Fab (фиг. 36А и 36Б).
Интродуцировали мутации Pro329Gly, Leu234Ala и Leu235Ala в константную область тяжелых цепей с «выступом» и «впадиной» для элиминации связывания с рецепторами Fc-гамма согласно методу, описанному в публикации международной заявки на патент WO 2012/130831 А1.
Биспецифические антитела в формате 2+1 анти-4-1BB/анти-FAP huIgG1 P329GLALA получали путем совместной трансфекции клеток HEK293-EBNA экспрессионными векторами для клеток млекопитающих, используя полиэтиленимин. Клетки трансфектировали соответствующими экспрессионными векторами в соотношении 1:1:1 («вектор тяжелой цепи с «выступом» : «вектор легкой цепи» : «вектор тяжелой цепи с «впадиной»). Конструкции получали и очищали согласно методу, описанному для биспецифических двухвалентных антител анти-4-1ВВ и анти-FAP huIgG1 P329GLALA (см. пример 9.1).
Последовательности пар оснований и аминокислотные последовательности анти-4-1ВВ, анти-FAP-конструкций в формате 2+1 с VH a-FAP, слитой с цепью с «выступом», и VL, слитой с цепью с «впадиной», представлены соответственно в таблице 70.










Последовательности пар оснований и аминокислотные последовательности анти-4-1ВВ, анти-FAP-конструкций в формате 2+1 с VL a-FAP, слитой с цепью с «выступом», и VH, слитой с цепью с «впадиной», представлены соответственно в таблице 71.










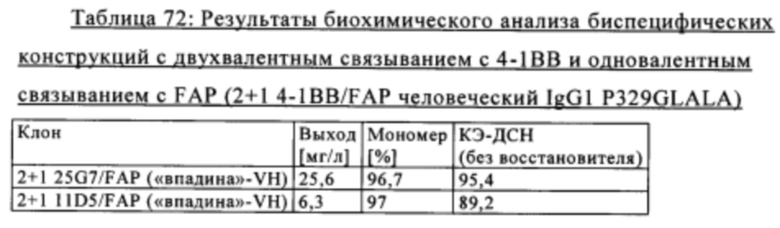
11.2. Связывание биспецифических одновалентных антител, мишенями которых являются 4-1ВВ и FAP
11.2.1. Поверхностный плазмонный резонанс (одновременное связывание) Способность связываться одновременно с конструкцией человеческий 4-1BB-Fc(kih) и человеческим FAP оценивали с помощью поверхностного плазмонного резонанса (SPR). Все эксперименты на основе SPR осуществляли с помощью устройства Biacore Т200 при 25°С с использованием HBS-EP в качестве подвижного буфера (0,01M HEPES, рН 7,4, 0,15М NaCl, 3 мМ ЭДТА, 0,005% сурфактанта Р20, фирма Biacore, Фрейбург/Германия).
Конструкцию, содержащую биотинилированный человеческий 4-1ВВ в слиянии с Fc(kih), непосредственно сшивали с проточной ячейкой стрептавидинового (SA) сенсорного чипа. Использовали уровень иммобилизации, соответствующий вплоть до 400 резонансных единиц (RU). Биспецифические антитела, мишенями которых являются 4-1ВВ и FAP, пропускали через проточные ячейки, используя концентрацию порядка 200 нМ, со скоростью потока 30 мкл/мин и диссоциацию устанавливали на 0 с. Человеческий FAP инъецировали в проточные ячейки в качестве второго аналита со скоростью потока 30 мкл/мин в течение 90 с в концентрации 500 нМ. Мониторинг диссоциации комплекса осуществляли в течение 120 с. Различия всех показателей преломления корректировали путем вычитания ответа, полученного в проточной референс-ячейке, в которой отсутствовал иммобилизированный белок.
Как продемонстрировано на фиг. 37Б и 37В, все биспецифические конструкции обладали способностью связываться одновременно с человеческим 4-1ВВ и человеческим FAP.
11.2.2. Активация NFκB
Создание NFκB-репортерной клеточной линии HeLa-huCD137-NFκB-luc, клон 26, а также протокол анализа активации уже описаны подробно в примере 9.2.3.2.
Изученные нацеленные на FAP (4В9) конструкции в формате 2+1, содержащие клон 11D5 (фиг. 44А-В) или клон 25G7 (фиг. Ж-И), инициировали активацию передачи сигнала NFkB в репортерной клеточной линии в присутствии экспрессирующих FAP опухолевых клеток. Указанная активность строго зависела от уровня экспрессии FAP на клеточной поверхности экспрессирующих FAP опухолевых клеток, поскольку никакой активации NF-kB не удалось обнаружить в отсутствии экспрессирующих FAP опухолевых клеток (фиг. 43Г и Ж). В отличие от других протестированных форматов (например, 2+2, 1+1) нацеленные на FAP (4В9) конструкции в формате 2+1 могли индуцировать также активацию в присутствии отличающихся более низким уровнем экспрессии FAP клеток WM-266-4 (фиг. 44Д и З). В присутствии клеток NIH/3T3-huFAP, клон 19 связывающие агенты в формате 2+1 также превышали по активности нацеленные на FAP (28Н1) конструкции 2+2 или 1+1 (фиг. 43Е и И, фиг. 44Б). Это можно объяснить более сильной способностью агента к связыванию FAP (4В9>28Н1) и различным соотношением между FAP-связывающим участками и 4-1ВВ-связывающими участками (1:2 относительно 1:1).
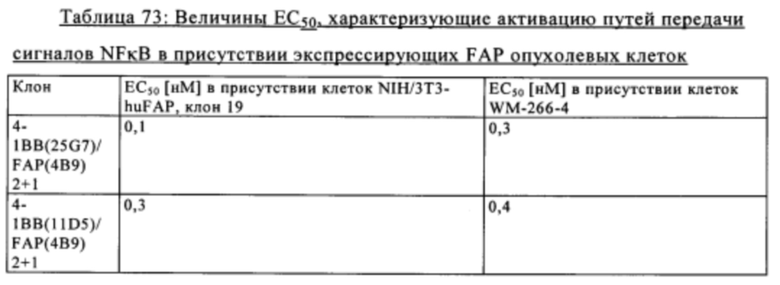
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Ф.ХОФФМАНН-ЛЯ РОШ АГ
<120> Биспецифические антитела, специфические в отношении
костимуляторного TNF-рецептора
<130> P33116-WO
<150> EP15188095.2
<151> 2015-10-02
<150> EP16170363.2
<151> 2016-05-19
<160> 347
<170> PatentIn version 3.5
<210> 1
<211> 186
<212> PRT
<213> Homo sapiens
<400> 1
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys His
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Ser Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ser Lys Pro Cys Lys Pro Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Leu Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Ala Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Ile Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Gly Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Gly Gly Arg Ala
180 185
<210> 2
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9,49B4,1G4, 20B7) CDR-H1
<400> 2
Ser Tyr Ala Ile Ser
1 5
<210> 3
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563, CLC-564, 17A9) CDR-H1
<400> 3
Ser Tyr Ala Met Ser
1 5
<210> 4
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9,49B4,1G4, 20B7) CDR-H2
<400> 4
Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 5
<211> 17
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563, CLC-564, 17A9) CDR-H2
<400> 5
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 6
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9) CDR-H3
<400> 6
Glu Tyr Gly Trp Met Asp Tyr
1 5
<210> 7
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(49B4) CDR-H3
<400> 7
Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr
1 5
<210> 8
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(1G4) CDR-H3
<400> 8
Glu Tyr Gly Ser Met Asp Tyr
1 5
<210> 9
<211> 13
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(20B7) CDR-H3
<400> 9
Val Asn Tyr Pro Tyr Ser Tyr Trp Gly Asp Phe Asp Tyr
1 5 10
<210> 10
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563) CDR-H3
<400> 10
Asp Val Gly Ala Phe Asp Tyr
1 5
<210> 11
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-564) CDR-H3
<400> 11
Asp Val Gly Pro Phe Asp Tyr
1 5
<210> 12
<211> 11
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9)-CDR-H3
<400> 12
Val Phe Tyr Arg Gly Gly Val Ser Met Asp Tyr
1 5 10
<210> 13
<211> 11
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9,49B4,1G4, 20B7) CDR-L1
<400> 13
Arg Ala Ser Gln Ser Ile Ser Ser Trp Leu Ala
1 5 10
<210> 14
<211> 12
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563, CLC564) CDR-L1
<400> 14
Arg Ala Ser Gln Ser Val Ser Ser Ser Tyr Leu Ala
1 5 10
<210> 15
<211> 11
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9) CDR-L1
<400> 15
Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala Ser
1 5 10
<210> 16
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9,49B4,1G4, 20B7) CDR-L2
<400> 16
Asp Ala Ser Ser Leu Glu Ser
1 5
<210> 17
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563, CLC564) CDR-L2
<400> 17
Gly Ala Ser Ser Arg Ala Thr
1 5
<210> 18
<211> 7
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9) CDR-L2
<400> 18
Gly Lys Asn Asn Arg Pro Ser
1 5
<210> 19
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9) CDR-L3
<400> 19
Gln Gln Tyr Leu Thr Tyr Ser Arg Phe Thr
1 5 10
<210> 20
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(49B4) CDR-L3
<400> 20
Gln Gln Tyr Ser Ser Gln Pro Tyr Thr
1 5
<210> 21
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(1G4) CDR-L3
<400> 21
Gln Gln Tyr Ile Ser Tyr Ser Met Leu Thr
1 5 10
<210> 22
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(20B7) CDR-L3
<400> 22
Gln Gln Tyr Gln Ala Phe Ser Leu Thr
1 5
<210> 23
<211> 9
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563, CLC-164) CDR-L3
<400> 23
Gln Gln Tyr Gly Ser Ser Pro Leu Thr
1 5
<210> 24
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9) CDR-L3
<400> 24
Asn Ser Arg Val Met Pro His Asn Arg Val
1 5 10
<210> 25
<211> 116
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9) VH
<400> 25
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Trp Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 26
<211> 108
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(8H9) VL
<400> 26
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Thr Tyr Ser Arg
85 90 95
Phe Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 27
<211> 118
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(49B4) VH
<400> 27
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser
115
<210> 28
<211> 107
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(49B4) VL
<400> 28
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Gln Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 29
<211> 116
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(1G4) VH
<400> 29
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Ser Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 30
<211> 108
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(1G4) VL
<400> 30
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ile Ser Tyr Ser Met
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 31
<211> 122
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(20B7) VH
<400> 31
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Asn Tyr Pro Tyr Ser Tyr Trp Gly Asp Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 32
<211> 107
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(20B7) VL
<400> 32
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Gln Ala Phe Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 33
<211> 116
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563) VH
<400> 33
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Asp Val Gly Ala Phe Asp Tyr Trp Gly Gln Gly Ala Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 34
<211> 108
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-563) VL
<400> 34
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 35
<211> 116
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-564) VH
<400> 35
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Phe Asp Val Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 36
<211> 108
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(CLC-564) VL
<400> 36
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 37
<211> 120
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9) VH
<400> 37
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Phe Tyr Arg Gly Gly Val Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 38
<211> 106
<212> PRT
<213> Artificial sequence
<220>
<223> OX40(17A9) VL
<400> 38
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Val Met Pro His Asn Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val
100 105
<210> 39
<211> 163
<212> PRT
<213> Homo sapiens
<400> 39
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Asp
85 90 95
Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr
115 120 125
Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro
130 135 140
Gly Ala Ser Ser Val Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His
145 150 155 160
Ser Pro Gln
<210> 40
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3,11D5, 9B11, 20G2) CDR-H1
<400> 40
Ser Tyr Ala Ile Ser
1 5
<210> 41
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-H1
<400> 41
Ser Tyr Ala Met Ser
1 5
<210> 42
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3,11D5, 9B11, 20G2) CDR-H2
<400> 42
Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 43
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-H2
<400> 43
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 44
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3) CDR-H3
<400> 44
Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr
1 5 10
<210> 45
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-H3
<400> 45
Asp Asp Pro Trp Pro Pro Phe Asp Tyr
1 5
<210> 46
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(11D5) CDR-H3
<400> 46
Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr
1 5 10
<210> 47
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(9B11) CDR-H3
<400> 47
Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr
1 5 10
<210> 48
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(20G2) CDR-H3
<400> 48
Ser Tyr Tyr Trp Glu Ser Tyr Pro Phe Asp Tyr
1 5 10
<210> 49
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3,11D5, 9B11, 20G2) CDR-L1
<400> 49
Arg Ala Ser Gln Ser Ile Ser Ser Trp Leu Ala
1 5 10
<210> 50
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-L1
<400> 50
Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala Ser
1 5 10
<210> 51
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3,11D5, 9B11, 20G2) CDR-L2
<400> 51
Asp Ala Ser Ser Leu Glu Ser
1 5
<210> 52
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-L2
<400> 52
Gly Lys Asn Asn Arg Pro Ser
1 5
<210> 53
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3) CDR-L3
<400> 53
Gln Gln Tyr His Ser Tyr Pro Gln Thr
1 5
<210> 54
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) CDR-L3
<400> 54
Asn Ser Leu Asp Arg Arg Gly Met Trp Val
1 5 10
<210> 55
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(11D5) CDR-L3
<400> 55
Gln Gln Leu Asn Ser Tyr Pro Gln Thr
1 5
<210> 56
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(9B11) CDR-L3
<400> 56
Gln Gln Val Asn Ser Tyr Pro Gln Thr
1 5
<210> 57
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(20G2) CDR-L3
<400> 57
Gln Gln Gln His Ser Tyr Tyr Thr
1 5
<210> 58
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3) VH
<400> 58
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 59
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(12B3) VL
<400> 59
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr His Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 60
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) VH
<400> 60
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 61
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(25G7) VL
<400> 61
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Leu Asp Arg Arg Gly Met Trp
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val
100 105
<210> 62
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(11D5) VH
<400> 62
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 63
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(11D5) VL
<400> 63
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu Asn Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 64
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(9B11) VH
<400> 64
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 65
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(9B11) VL
<400> 65
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Asn Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 66
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(20G2) VH
<400> 66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Tyr Tyr Trp Glu Ser Tyr Pro Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 67
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> 4-1BB(20G2) VL
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gln His Ser Tyr Tyr Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 68
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-H1
<400> 68
Ser His Ala Met Ser
1 5
<210> 69
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-H1
<400> 69
Ser Tyr Ala Met Ser
1 5
<210> 70
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-H2
<400> 70
Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val Lys Gly
1 5 10 15
<210> 71
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-H2
<400> 71
Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 72
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-H3
<400> 72
Gly Trp Leu Gly Asn Phe Asp Tyr
1 5
<210> 73
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-H3
<400> 73
Gly Trp Phe Gly Gly Phe Asn Tyr
1 5
<210> 74
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-L1
<400> 74
Arg Ala Ser Gln Ser Val Ser Arg Ser Tyr Leu Ala
1 5 10
<210> 75
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-L1
<400> 75
Arg Ala Ser Gln Ser Val Thr Ser Ser Tyr Leu Ala
1 5 10
<210> 76
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-L2
<400> 76
Gly Ala Ser Thr Arg Ala Thr
1 5
<210> 77
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-L2
<400> 77
Val Gly Ser Arg Arg Ala Thr
1 5
<210> 78
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) CDR-L3
<400> 78
Gln Gln Gly Gln Val Ile Pro Pro Thr
1 5
<210> 79
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) CDR-L3
<400> 79
Gln Gln Gly Ile Met Leu Pro Pro Thr
1 5
<210> 80
<211> 116
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) VH
<400> 80
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser
115
<210> 81
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(28H1) VL
<400> 81
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Ile Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Gln Val Ile Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 82
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) VH
<400> 82
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 83
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> FAP(4B9) VL
<400> 83
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile Met Leu Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 84
<211> 760
<212> PRT
<213> Homo sapiens
<400> 84
Met Lys Thr Trp Val Lys Ile Val Phe Gly Val Ala Thr Ser Ala Val
1 5 10 15
Leu Ala Leu Leu Val Met Cys Ile Val Leu Arg Pro Ser Arg Val His
20 25 30
Asn Ser Glu Glu Asn Thr Met Arg Ala Leu Thr Leu Lys Asp Ile Leu
35 40 45
Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe Pro Asn Trp Ile Ser Gly
50 55 60
Gln Glu Tyr Leu His Gln Ser Ala Asp Asn Asn Ile Val Leu Tyr Asn
65 70 75 80
Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu Ser Asn Arg Thr Met Lys
85 90 95
Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser Pro Asp Arg Gln Phe Val
100 105 110
Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp Arg Tyr Ser Tyr Thr Ala
115 120 125
Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly Glu Phe Val Arg Gly Asn
130 135 140
Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys Trp Ser Pro Val Gly Ser
145 150 155 160
Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile Tyr Leu Lys Gln Arg Pro
165 170 175
Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn Gly Arg Glu Asn Lys Ile
180 185 190
Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu Glu Glu Met Leu Ala Thr
195 200 205
Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly Lys Phe Leu Ala Tyr Ala
210 215 220
Glu Phe Asn Asp Thr Asp Ile Pro Val Ile Ala Tyr Ser Tyr Tyr Gly
225 230 235 240
Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile Pro Tyr Pro Lys Ala Gly
245 250 255
Ala Lys Asn Pro Val Val Arg Ile Phe Ile Ile Asp Thr Thr Tyr Pro
260 265 270
Ala Tyr Val Gly Pro Gln Glu Val Pro Val Pro Ala Met Ile Ala Ser
275 280 285
Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp Val Thr Asp Glu Arg Val
290 295 300
Cys Leu Gln Trp Leu Lys Arg Val Gln Asn Val Ser Val Leu Ser Ile
305 310 315 320
Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp Asp Cys Pro Lys Thr Gln
325 330 335
Glu His Ile Glu Glu Ser Arg Thr Gly Trp Ala Gly Gly Phe Phe Val
340 345 350
Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile Ser Tyr Tyr Lys Ile Phe
355 360 365
Ser Asp Lys Asp Gly Tyr Lys His Ile His Tyr Ile Lys Asp Thr Val
370 375 380
Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys Trp Glu Ala Ile Asn Ile
385 390 395 400
Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr Ser Ser Asn Glu Phe Glu
405 410 415
Glu Tyr Pro Gly Arg Arg Asn Ile Tyr Arg Ile Ser Ile Gly Ser Tyr
420 425 430
Pro Pro Ser Lys Lys Cys Val Thr Cys His Leu Arg Lys Glu Arg Cys
435 440 445
Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr Ala Lys Tyr Tyr Ala Leu
450 455 460
Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser Thr Leu His Asp Gly Arg
465 470 475 480
Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu Asn Lys Glu Leu Glu Asn
485 490 495
Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu Glu Ile Lys Lys Leu Glu
500 505 510
Val Asp Glu Ile Thr Leu Trp Tyr Lys Met Ile Leu Pro Pro Gln Phe
515 520 525
Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile Gln Val Tyr Gly Gly Pro
530 535 540
Cys Ser Gln Ser Val Arg Ser Val Phe Ala Val Asn Trp Ile Ser Tyr
545 550 555 560
Leu Ala Ser Lys Glu Gly Met Val Ile Ala Leu Val Asp Gly Arg Gly
565 570 575
Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr Ala Val Tyr Arg Lys Leu
580 585 590
Gly Val Tyr Glu Val Glu Asp Gln Ile Thr Ala Val Arg Lys Phe Ile
595 600 605
Glu Met Gly Phe Ile Asp Glu Lys Arg Ile Ala Ile Trp Gly Trp Ser
610 615 620
Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu Ala Ser Gly Thr Gly Leu
625 630 635 640
Phe Lys Cys Gly Ile Ala Val Ala Pro Val Ser Ser Trp Glu Tyr Tyr
645 650 655
Ala Ser Val Tyr Thr Glu Arg Phe Met Gly Leu Pro Thr Lys Asp Asp
660 665 670
Asn Leu Glu His Tyr Lys Asn Ser Thr Val Met Ala Arg Ala Glu Tyr
675 680 685
Phe Arg Asn Val Asp Tyr Leu Leu Ile His Gly Thr Ala Asp Asp Asn
690 695 700
Val His Phe Gln Asn Ser Ala Gln Ile Ala Lys Ala Leu Val Asn Ala
705 710 715 720
Gln Val Asp Phe Gln Ala Met Trp Tyr Ser Asp Gln Asn His Gly Leu
725 730 735
Ser Gly Leu Ser Thr Asn His Leu Tyr Thr His Met Thr His Phe Leu
740 745 750
Lys Gln Cys Phe Ser Leu Ser Asp
755 760
<210> 85
<211> 748
<212> PRT
<213> Artificial Sequence
<220>
<223> hu FAP ectodomain+poly-lys-tag+his6-tag
<400> 85
Arg Pro Ser Arg Val His Asn Ser Glu Glu Asn Thr Met Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe
20 25 30
Pro Asn Trp Ile Ser Gly Gln Glu Tyr Leu His Gln Ser Ala Asp Asn
35 40 45
Asn Ile Val Leu Tyr Asn Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu
50 55 60
Ser Asn Arg Thr Met Lys Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly
100 105 110
Glu Phe Val Arg Gly Asn Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn
145 150 155 160
Gly Arg Glu Asn Lys Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly
180 185 190
Lys Phe Leu Ala Tyr Ala Glu Phe Asn Asp Thr Asp Ile Pro Val Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Val Val Arg Ile Phe Ile
225 230 235 240
Ile Asp Thr Thr Tyr Pro Ala Tyr Val Gly Pro Gln Glu Val Pro Val
245 250 255
Pro Ala Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Thr Asp Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp
290 295 300
Asp Cys Pro Lys Thr Gln Glu His Ile Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Asn Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Glu Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Ser Tyr Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu
465 470 475 480
Glu Ile Lys Lys Leu Glu Val Asp Glu Ile Thr Leu Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Arg Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Ser Tyr Leu Ala Ser Lys Glu Gly Met Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Ile Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Lys Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Val Tyr Thr Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Leu Ser Gly Leu Ser Thr Asn His Leu Tyr Thr
705 710 715 720
His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly Lys
725 730 735
Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 86
<211> 2244
<212> DNA
<213> Artificial Sequence
<220>
<223> hu FAP ectodomain+poly-lys-tag+his6-tag
<400> 86
cgcccttcaa gagttcataa ctctgaagaa aatacaatga gagcactcac actgaaggat 60
attttaaatg gaacattttc ttataaaaca ttttttccaa actggatttc aggacaagaa 120
tatcttcatc aatctgcaga taacaatata gtactttata atattgaaac aggacaatca 180
tataccattt tgagtaatag aaccatgaaa agtgtgaatg cttcaaatta cggcttatca 240
cctgatcggc aatttgtata tctagaaagt gattattcaa agctttggag atactcttac 300
acagcaacat attacatcta tgaccttagc aatggagaat ttgtaagagg aaatgagctt 360
cctcgtccaa ttcagtattt atgctggtcg cctgttggga gtaaattagc atatgtctat 420
caaaacaata tctatttgaa acaaagacca ggagatccac cttttcaaat aacatttaat 480
ggaagagaaa ataaaatatt taatggaatc ccagactggg tttatgaaga ggaaatgctt 540
gctacaaaat atgctctctg gtggtctcct aatggaaaat ttttggcata tgcggaattt 600
aatgatacgg atataccagt tattgcctat tcctattatg gcgatgaaca atatcctaga 660
acaataaata ttccataccc aaaggctgga gctaagaatc ccgttgttcg gatatttatt 720
atcgatacca cttaccctgc gtatgtaggt ccccaggaag tgcctgttcc agcaatgata 780
gcctcaagtg attattattt cagttggctc acgtgggtta ctgatgaacg agtatgtttg 840
cagtggctaa aaagagtcca gaatgtttcg gtcctgtcta tatgtgactt cagggaagac 900
tggcagacat gggattgtcc aaagacccag gagcatatag aagaaagcag aactggatgg 960
gctggtggat tctttgtttc aacaccagtt ttcagctatg atgccatttc gtactacaaa 1020
atatttagtg acaaggatgg ctacaaacat attcactata tcaaagacac tgtggaaaat 1080
gctattcaaa ttacaagtgg caagtgggag gccataaata tattcagagt aacacaggat 1140
tcactgtttt attctagcaa tgaatttgaa gaataccctg gaagaagaaa catctacaga 1200
attagcattg gaagctatcc tccaagcaag aagtgtgtta cttgccatct aaggaaagaa 1260
aggtgccaat attacacagc aagtttcagc gactacgcca agtactatgc acttgtctgc 1320
tacggcccag gcatccccat ttccaccctt catgatggac gcactgatca agaaattaaa 1380
atcctggaag aaaacaagga attggaaaat gctttgaaaa atatccagct gcctaaagag 1440
gaaattaaga aacttgaagt agatgaaatt actttatggt acaagatgat tcttcctcct 1500
caatttgaca gatcaaagaa gtatcccttg ctaattcaag tgtatggtgg tccctgcagt 1560
cagagtgtaa ggtctgtatt tgctgttaat tggatatctt atcttgcaag taaggaaggg 1620
atggtcattg ccttggtgga tggtcgagga acagctttcc aaggtgacaa actcctctat 1680
gcagtgtatc gaaagctggg tgtttatgaa gttgaagacc agattacagc tgtcagaaaa 1740
ttcatagaaa tgggtttcat tgatgaaaaa agaatagcca tatggggctg gtcctatgga 1800
ggatacgttt catcactggc ccttgcatct ggaactggtc ttttcaaatg tggtatagca 1860
gtggctccag tctccagctg ggaatattac gcgtctgtct acacagagag attcatgggt 1920
ctcccaacaa aggatgataa tcttgagcac tataagaatt caactgtgat ggcaagagca 1980
gaatatttca gaaatgtaga ctatcttctc atccacggaa cagcagatga taatgtgcac 2040
tttcaaaact cagcacagat tgctaaagct ctggttaatg cacaagtgga tttccaggca 2100
atgtggtact ctgaccagaa ccacggctta tccggcctgt ccacgaacca cttatacacc 2160
cacatgaccc acttcctaaa gcagtgtttc tctttgtcag acggcaaaaa gaaaaagaaa 2220
aagggccacc accatcacca tcac 2244
<210> 87
<211> 761
<212> PRT
<213> Mus musculus
<400> 87
Met Lys Thr Trp Leu Lys Thr Val Phe Gly Val Thr Thr Leu Ala Ala
1 5 10 15
Leu Ala Leu Val Val Ile Cys Ile Val Leu Arg Pro Ser Arg Val Tyr
20 25 30
Lys Pro Glu Gly Asn Thr Lys Arg Ala Leu Thr Leu Lys Asp Ile Leu
35 40 45
Asn Gly Thr Phe Ser Tyr Lys Thr Tyr Phe Pro Asn Trp Ile Ser Glu
50 55 60
Gln Glu Tyr Leu His Gln Ser Glu Asp Asp Asn Ile Val Phe Tyr Asn
65 70 75 80
Ile Glu Thr Arg Glu Ser Tyr Ile Ile Leu Ser Asn Ser Thr Met Lys
85 90 95
Ser Val Asn Ala Thr Asp Tyr Gly Leu Ser Pro Asp Arg Gln Phe Val
100 105 110
Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp Arg Tyr Ser Tyr Thr Ala
115 120 125
Thr Tyr Tyr Ile Tyr Asp Leu Gln Asn Gly Glu Phe Val Arg Gly Tyr
130 135 140
Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys Trp Ser Pro Val Gly Ser
145 150 155 160
Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile Tyr Leu Lys Gln Arg Pro
165 170 175
Gly Asp Pro Pro Phe Gln Ile Thr Tyr Thr Gly Arg Glu Asn Arg Ile
180 185 190
Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu Glu Glu Met Leu Ala Thr
195 200 205
Lys Tyr Ala Leu Trp Trp Ser Pro Asp Gly Lys Phe Leu Ala Tyr Val
210 215 220
Glu Phe Asn Asp Ser Asp Ile Pro Ile Ile Ala Tyr Ser Tyr Tyr Gly
225 230 235 240
Asp Gly Gln Tyr Pro Arg Thr Ile Asn Ile Pro Tyr Pro Lys Ala Gly
245 250 255
Ala Lys Asn Pro Val Val Arg Val Phe Ile Val Asp Thr Thr Tyr Pro
260 265 270
His His Val Gly Pro Met Glu Val Pro Val Pro Glu Met Ile Ala Ser
275 280 285
Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp Val Ser Ser Glu Arg Val
290 295 300
Cys Leu Gln Trp Leu Lys Arg Val Gln Asn Val Ser Val Leu Ser Ile
305 310 315 320
Cys Asp Phe Arg Glu Asp Trp His Ala Trp Glu Cys Pro Lys Asn Gln
325 330 335
Glu His Val Glu Glu Ser Arg Thr Gly Trp Ala Gly Gly Phe Phe Val
340 345 350
Ser Thr Pro Ala Phe Ser Gln Asp Ala Thr Ser Tyr Tyr Lys Ile Phe
355 360 365
Ser Asp Lys Asp Gly Tyr Lys His Ile His Tyr Ile Lys Asp Thr Val
370 375 380
Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys Trp Glu Ala Ile Tyr Ile
385 390 395 400
Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr Ser Ser Asn Glu Phe Glu
405 410 415
Gly Tyr Pro Gly Arg Arg Asn Ile Tyr Arg Ile Ser Ile Gly Asn Ser
420 425 430
Pro Pro Ser Lys Lys Cys Val Thr Cys His Leu Arg Lys Glu Arg Cys
435 440 445
Gln Tyr Tyr Thr Ala Ser Phe Ser Tyr Lys Ala Lys Tyr Tyr Ala Leu
450 455 460
Val Cys Tyr Gly Pro Gly Leu Pro Ile Ser Thr Leu His Asp Gly Arg
465 470 475 480
Thr Asp Gln Glu Ile Gln Val Leu Glu Glu Asn Lys Glu Leu Glu Asn
485 490 495
Ser Leu Arg Asn Ile Gln Leu Pro Lys Val Glu Ile Lys Lys Leu Lys
500 505 510
Asp Gly Gly Leu Thr Phe Trp Tyr Lys Met Ile Leu Pro Pro Gln Phe
515 520 525
Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile Gln Val Tyr Gly Gly Pro
530 535 540
Cys Ser Gln Ser Val Lys Ser Val Phe Ala Val Asn Trp Ile Thr Tyr
545 550 555 560
Leu Ala Ser Lys Glu Gly Ile Val Ile Ala Leu Val Asp Gly Arg Gly
565 570 575
Thr Ala Phe Gln Gly Asp Lys Phe Leu His Ala Val Tyr Arg Lys Leu
580 585 590
Gly Val Tyr Glu Val Glu Asp Gln Leu Thr Ala Val Arg Lys Phe Ile
595 600 605
Glu Met Gly Phe Ile Asp Glu Glu Arg Ile Ala Ile Trp Gly Trp Ser
610 615 620
Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu Ala Ser Gly Thr Gly Leu
625 630 635 640
Phe Lys Cys Gly Ile Ala Val Ala Pro Val Ser Ser Trp Glu Tyr Tyr
645 650 655
Ala Ser Ile Tyr Ser Glu Arg Phe Met Gly Leu Pro Thr Lys Asp Asp
660 665 670
Asn Leu Glu His Tyr Lys Asn Ser Thr Val Met Ala Arg Ala Glu Tyr
675 680 685
Phe Arg Asn Val Asp Tyr Leu Leu Ile His Gly Thr Ala Asp Asp Asn
690 695 700
Val His Phe Gln Asn Ser Ala Gln Ile Ala Lys Ala Leu Val Asn Ala
705 710 715 720
Gln Val Asp Phe Gln Ala Met Trp Tyr Ser Asp Gln Asn His Gly Ile
725 730 735
Ser Ser Gly Arg Ser Gln Asn His Leu Tyr Thr His Met Thr His Phe
740 745 750
Leu Lys Gln Cys Phe Ser Leu Ser Asp
755 760
<210> 88
<211> 749
<212> PRT
<213> Artificial Sequence
<220>
<223> Murine FAP ectodomain+poly-lys-tag+his6-tag
<400> 88
Arg Pro Ser Arg Val Tyr Lys Pro Glu Gly Asn Thr Lys Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Tyr Phe
20 25 30
Pro Asn Trp Ile Ser Glu Gln Glu Tyr Leu His Gln Ser Glu Asp Asp
35 40 45
Asn Ile Val Phe Tyr Asn Ile Glu Thr Arg Glu Ser Tyr Ile Ile Leu
50 55 60
Ser Asn Ser Thr Met Lys Ser Val Asn Ala Thr Asp Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Gln Asn Gly
100 105 110
Glu Phe Val Arg Gly Tyr Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Tyr Thr
145 150 155 160
Gly Arg Glu Asn Arg Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asp Gly
180 185 190
Lys Phe Leu Ala Tyr Val Glu Phe Asn Asp Ser Asp Ile Pro Ile Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Gly Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Val Val Arg Val Phe Ile
225 230 235 240
Val Asp Thr Thr Tyr Pro His His Val Gly Pro Met Glu Val Pro Val
245 250 255
Pro Glu Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Ser Ser Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp His Ala Trp
290 295 300
Glu Cys Pro Lys Asn Gln Glu His Val Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Ala Phe Ser Gln Asp Ala Thr
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Tyr Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Gly Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Asn Ser Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Tyr Lys
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Leu Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Gln Val Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ser Leu Arg Asn Ile Gln Leu Pro Lys Val
465 470 475 480
Glu Ile Lys Lys Leu Lys Asp Gly Gly Leu Thr Phe Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Lys Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Thr Tyr Leu Ala Ser Lys Glu Gly Ile Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Phe Leu His
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Leu Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Glu Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Ile Tyr Ser Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Ile Leu Ser Gly Arg Ser Gln Asn His Leu Tyr
705 710 715 720
Thr His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly
725 730 735
Lys Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 89
<211> 2247
<212> DNA
<213> Artificial Sequence
<220>
<223> Murine FAP ectodomain+poly-lys-tag+his6-tag
<400> 89
cgtccctcaa gagtttacaa acctgaagga aacacaaaga gagctcttac cttgaaggat 60
attttaaatg gaacattctc atataaaaca tattttccca actggatttc agaacaagaa 120
tatcttcatc aatctgagga tgataacata gtattttata atattgaaac aagagaatca 180
tatatcattt tgagtaatag caccatgaaa agtgtgaatg ctacagatta tggtttgtca 240
cctgatcggc aatttgtgta tctagaaagt gattattcaa agctctggcg atattcatac 300
acagcgacat actacatcta cgaccttcag aatggggaat ttgtaagagg atacgagctc 360
cctcgtccaa ttcagtatct atgctggtcg cctgttggga gtaaattagc atatgtatat 420
caaaacaata tttatttgaa acaaagacca ggagatccac cttttcaaat aacttatact 480
ggaagagaaa atagaatatt taatggaata ccagactggg tttatgaaga ggaaatgctt 540
gccacaaaat atgctctttg gtggtctcca gatggaaaat ttttggcata tgtagaattt 600
aatgattcag atataccaat tattgcctat tcttattatg gtgatggaca gtatcctaga 660
actataaata ttccatatcc aaaggctggg gctaagaatc cggttgttcg tgtttttatt 720
gttgacacca cctaccctca ccacgtgggc ccaatggaag tgccagttcc agaaatgata 780
gcctcaagtg actattattt cagctggctc acatgggtgt ccagtgaacg agtatgcttg 840
cagtggctaa aaagagtgca gaatgtctca gtcctgtcta tatgtgattt cagggaagac 900
tggcatgcat gggaatgtcc aaagaaccag gagcatgtag aagaaagcag aacaggatgg 960
gctggtggat tctttgtttc gacaccagct tttagccagg atgccacttc ttactacaaa 1020
atatttagcg acaaggatgg ttacaaacat attcactaca tcaaagacac tgtggaaaat 1080
gctattcaaa ttacaagtgg caagtgggag gccatatata tattccgcgt aacacaggat 1140
tcactgtttt attctagcaa tgaatttgaa ggttaccctg gaagaagaaa catctacaga 1200
attagcattg gaaactctcc tccgagcaag aagtgtgtta cttgccatct aaggaaagaa 1260
aggtgccaat attacacagc aagtttcagc tacaaagcca agtactatgc actcgtctgc 1320
tatggccctg gcctccccat ttccaccctc catgatggcc gcacagacca agaaatacaa 1380
gtattagaag aaaacaaaga actggaaaat tctctgagaa atatccagct gcctaaagtg 1440
gagattaaga agctcaaaga cgggggactg actttctggt acaagatgat tctgcctcct 1500
cagtttgaca gatcaaagaa gtaccctttg ctaattcaag tgtatggtgg tccttgtagc 1560
cagagtgtta agtctgtgtt tgctgttaat tggataactt atctcgcaag taaggagggg 1620
atagtcattg ccctggtaga tggtcggggc actgctttcc aaggtgacaa attcctgcat 1680
gccgtgtatc gaaaactggg tgtatatgaa gttgaggacc agctcacagc tgtcagaaaa 1740
ttcatagaaa tgggtttcat tgatgaagaa agaatagcca tatggggctg gtcctacgga 1800
ggttatgttt catccctggc ccttgcatct ggaactggtc ttttcaaatg tggcatagca 1860
gtggctccag tctccagctg ggaatattac gcatctatct actcagagag attcatgggc 1920
ctcccaacaa aggacgacaa tctcgaacac tataaaaatt caactgtgat ggcaagagca 1980
gaatatttca gaaatgtaga ctatcttctc atccacggaa cagcagatga taatgtgcac 2040
tttcagaact cagcacagat tgctaaagct ttggttaatg cacaagtgga tttccaggcg 2100
atgtggtact ctgaccagaa ccatggtata ttatctgggc gctcccagaa tcatttatat 2160
acccacatga cgcacttcct caagcaatgc ttttctttat cagacggcaa aaagaaaaag 2220
aaaaagggcc accaccatca ccatcac 2247
<210> 90
<211> 748
<212> PRT
<213> Artificial Sequence
<220>
<223> Cynomolgus FAP ectodomain+poly-lys-tag+his6-tag
<400> 90
Arg Pro Pro Arg Val His Asn Ser Glu Glu Asn Thr Met Arg Ala Leu
1 5 10 15
Thr Leu Lys Asp Ile Leu Asn Gly Thr Phe Ser Tyr Lys Thr Phe Phe
20 25 30
Pro Asn Trp Ile Ser Gly Gln Glu Tyr Leu His Gln Ser Ala Asp Asn
35 40 45
Asn Ile Val Leu Tyr Asn Ile Glu Thr Gly Gln Ser Tyr Thr Ile Leu
50 55 60
Ser Asn Arg Thr Met Lys Ser Val Asn Ala Ser Asn Tyr Gly Leu Ser
65 70 75 80
Pro Asp Arg Gln Phe Val Tyr Leu Glu Ser Asp Tyr Ser Lys Leu Trp
85 90 95
Arg Tyr Ser Tyr Thr Ala Thr Tyr Tyr Ile Tyr Asp Leu Ser Asn Gly
100 105 110
Glu Phe Val Arg Gly Asn Glu Leu Pro Arg Pro Ile Gln Tyr Leu Cys
115 120 125
Trp Ser Pro Val Gly Ser Lys Leu Ala Tyr Val Tyr Gln Asn Asn Ile
130 135 140
Tyr Leu Lys Gln Arg Pro Gly Asp Pro Pro Phe Gln Ile Thr Phe Asn
145 150 155 160
Gly Arg Glu Asn Lys Ile Phe Asn Gly Ile Pro Asp Trp Val Tyr Glu
165 170 175
Glu Glu Met Leu Ala Thr Lys Tyr Ala Leu Trp Trp Ser Pro Asn Gly
180 185 190
Lys Phe Leu Ala Tyr Ala Glu Phe Asn Asp Thr Asp Ile Pro Val Ile
195 200 205
Ala Tyr Ser Tyr Tyr Gly Asp Glu Gln Tyr Pro Arg Thr Ile Asn Ile
210 215 220
Pro Tyr Pro Lys Ala Gly Ala Lys Asn Pro Phe Val Arg Ile Phe Ile
225 230 235 240
Ile Asp Thr Thr Tyr Pro Ala Tyr Val Gly Pro Gln Glu Val Pro Val
245 250 255
Pro Ala Met Ile Ala Ser Ser Asp Tyr Tyr Phe Ser Trp Leu Thr Trp
260 265 270
Val Thr Asp Glu Arg Val Cys Leu Gln Trp Leu Lys Arg Val Gln Asn
275 280 285
Val Ser Val Leu Ser Ile Cys Asp Phe Arg Glu Asp Trp Gln Thr Trp
290 295 300
Asp Cys Pro Lys Thr Gln Glu His Ile Glu Glu Ser Arg Thr Gly Trp
305 310 315 320
Ala Gly Gly Phe Phe Val Ser Thr Pro Val Phe Ser Tyr Asp Ala Ile
325 330 335
Ser Tyr Tyr Lys Ile Phe Ser Asp Lys Asp Gly Tyr Lys His Ile His
340 345 350
Tyr Ile Lys Asp Thr Val Glu Asn Ala Ile Gln Ile Thr Ser Gly Lys
355 360 365
Trp Glu Ala Ile Asn Ile Phe Arg Val Thr Gln Asp Ser Leu Phe Tyr
370 375 380
Ser Ser Asn Glu Phe Glu Asp Tyr Pro Gly Arg Arg Asn Ile Tyr Arg
385 390 395 400
Ile Ser Ile Gly Ser Tyr Pro Pro Ser Lys Lys Cys Val Thr Cys His
405 410 415
Leu Arg Lys Glu Arg Cys Gln Tyr Tyr Thr Ala Ser Phe Ser Asp Tyr
420 425 430
Ala Lys Tyr Tyr Ala Leu Val Cys Tyr Gly Pro Gly Ile Pro Ile Ser
435 440 445
Thr Leu His Asp Gly Arg Thr Asp Gln Glu Ile Lys Ile Leu Glu Glu
450 455 460
Asn Lys Glu Leu Glu Asn Ala Leu Lys Asn Ile Gln Leu Pro Lys Glu
465 470 475 480
Glu Ile Lys Lys Leu Glu Val Asp Glu Ile Thr Leu Trp Tyr Lys Met
485 490 495
Ile Leu Pro Pro Gln Phe Asp Arg Ser Lys Lys Tyr Pro Leu Leu Ile
500 505 510
Gln Val Tyr Gly Gly Pro Cys Ser Gln Ser Val Arg Ser Val Phe Ala
515 520 525
Val Asn Trp Ile Ser Tyr Leu Ala Ser Lys Glu Gly Met Val Ile Ala
530 535 540
Leu Val Asp Gly Arg Gly Thr Ala Phe Gln Gly Asp Lys Leu Leu Tyr
545 550 555 560
Ala Val Tyr Arg Lys Leu Gly Val Tyr Glu Val Glu Asp Gln Ile Thr
565 570 575
Ala Val Arg Lys Phe Ile Glu Met Gly Phe Ile Asp Glu Lys Arg Ile
580 585 590
Ala Ile Trp Gly Trp Ser Tyr Gly Gly Tyr Val Ser Ser Leu Ala Leu
595 600 605
Ala Ser Gly Thr Gly Leu Phe Lys Cys Gly Ile Ala Val Ala Pro Val
610 615 620
Ser Ser Trp Glu Tyr Tyr Ala Ser Val Tyr Thr Glu Arg Phe Met Gly
625 630 635 640
Leu Pro Thr Lys Asp Asp Asn Leu Glu His Tyr Lys Asn Ser Thr Val
645 650 655
Met Ala Arg Ala Glu Tyr Phe Arg Asn Val Asp Tyr Leu Leu Ile His
660 665 670
Gly Thr Ala Asp Asp Asn Val His Phe Gln Asn Ser Ala Gln Ile Ala
675 680 685
Lys Ala Leu Val Asn Ala Gln Val Asp Phe Gln Ala Met Trp Tyr Ser
690 695 700
Asp Gln Asn His Gly Leu Ser Gly Leu Ser Thr Asn His Leu Tyr Thr
705 710 715 720
His Met Thr His Phe Leu Lys Gln Cys Phe Ser Leu Ser Asp Gly Lys
725 730 735
Lys Lys Lys Lys Lys Gly His His His His His His
740 745
<210> 91
<211> 2244
<212> DNA
<213> Artificial Sequence
<220>
<223> Cynomolgus FAP ectodomain+poly-lys-tag+his6-tag
<400> 91
cgccctccaa gagttcataa ctctgaagaa aatacaatga gagcactcac actgaaggat 60
attttaaatg ggacattttc ttataaaaca ttttttccaa actggatttc aggacaagaa 120
tatcttcatc aatctgcaga taacaatata gtactttata atattgaaac aggacaatca 180
tataccattt tgagtaacag aaccatgaaa agtgtgaatg cttcaaatta tggcttatca 240
cctgatcggc aatttgtata tctagaaagt gattattcaa agctttggag atactcttac 300
acagcaacat attacatcta tgaccttagc aatggagaat ttgtaagagg aaatgagctt 360
cctcgtccaa ttcagtattt atgctggtcg cctgttggga gtaaattagc atatgtctat 420
caaaacaata tctatttgaa acaaagacca ggagatccac cttttcaaat aacatttaat 480
ggaagagaaa ataaaatatt taatggaatc ccagactggg tttatgaaga ggaaatgctt 540
gctacaaaat atgctctctg gtggtctcct aatggaaaat ttttggcata tgcggaattt 600
aatgatacag atataccagt tattgcctat tcctattatg gcgatgaaca atatcccaga 660
acaataaata ttccataccc aaaggccgga gctaagaatc cttttgttcg gatatttatt 720
atcgatacca cttaccctgc gtatgtaggt ccccaggaag tgcctgttcc agcaatgata 780
gcctcaagtg attattattt cagttggctc acgtgggtta ctgatgaacg agtatgtttg 840
cagtggctaa aaagagtcca gaatgtttcg gtcttgtcta tatgtgattt cagggaagac 900
tggcagacat gggattgtcc aaagacccag gagcatatag aagaaagcag aactggatgg 960
gctggtggat tctttgtttc aacaccagtt ttcagctatg atgccatttc atactacaaa 1020
atatttagtg acaaggatgg ctacaaacat attcactata tcaaagacac tgtggaaaat 1080
gctattcaaa ttacaagtgg caagtgggag gccataaata tattcagagt aacacaggat 1140
tcactgtttt attctagcaa tgaatttgaa gattaccctg gaagaagaaa catctacaga 1200
attagcattg gaagctatcc tccaagcaag aagtgtgtta cttgccatct aaggaaagaa 1260
aggtgccaat attacacagc aagtttcagc gactacgcca agtactatgc acttgtctgc 1320
tatggcccag gcatccccat ttccaccctt catgacggac gcactgatca agaaattaaa 1380
atcctggaag aaaacaagga attggaaaat gctttgaaaa atatccagct gcctaaagag 1440
gaaattaaga aacttgaagt agatgaaatt actttatggt acaagatgat tcttcctcct 1500
caatttgaca gatcaaagaa gtatcccttg ctaattcaag tgtatggtgg tccctgcagt 1560
cagagtgtaa ggtctgtatt tgctgttaat tggatatctt atcttgcaag taaggaaggg 1620
atggtcattg ccttggtgga tggtcgggga acagctttcc aaggtgacaa actcctgtat 1680
gcagtgtatc gaaagctggg tgtttatgaa gttgaagacc agattacagc tgtcagaaaa 1740
ttcatagaaa tgggtttcat tgatgaaaaa agaatagcca tatggggctg gtcctatgga 1800
ggatatgttt catcactggc ccttgcatct ggaactggtc ttttcaaatg tgggatagca 1860
gtggctccag tctccagctg ggaatattac gcgtctgtct acacagagag attcatgggt 1920
ctcccaacaa aggatgataa tcttgagcac tataagaatt caactgtgat ggcaagagca 1980
gaatatttca gaaatgtaga ctatcttctc atccacggaa cagcagatga taatgtgcac 2040
tttcaaaact cagcacagat tgctaaagct ctggttaatg cacaagtgga tttccaggca 2100
atgtggtact ctgaccagaa ccacggctta tccggcctgt ccacgaacca cttatacacc 2160
cacatgaccc acttcctaaa gcagtgtttc tctttgtcag acggcaaaaa gaaaaagaaa 2220
aagggccacc accatcacca tcac 2244
<210> 92
<211> 702
<212> PRT
<213> Homo sapiens
<400> 92
Met Glu Ser Pro Ser Ala Pro Pro His Arg Trp Cys Ile Pro Trp Gln
1 5 10 15
Arg Leu Leu Leu Thr Ala Ser Leu Leu Thr Phe Trp Asn Pro Pro Thr
20 25 30
Thr Ala Lys Leu Thr Ile Glu Ser Thr Pro Phe Asn Val Ala Glu Gly
35 40 45
Lys Glu Val Leu Leu Leu Val His Asn Leu Pro Gln His Leu Phe Gly
50 55 60
Tyr Ser Trp Tyr Lys Gly Glu Arg Val Asp Gly Asn Arg Gln Ile Ile
65 70 75 80
Gly Tyr Val Ile Gly Thr Gln Gln Ala Thr Pro Gly Pro Ala Tyr Ser
85 90 95
Gly Arg Glu Ile Ile Tyr Pro Asn Ala Ser Leu Leu Ile Gln Asn Ile
100 105 110
Ile Gln Asn Asp Thr Gly Phe Tyr Thr Leu His Val Ile Lys Ser Asp
115 120 125
Leu Val Asn Glu Glu Ala Thr Gly Gln Phe Arg Val Tyr Pro Glu Leu
130 135 140
Pro Lys Pro Ser Ile Ser Ser Asn Asn Ser Lys Pro Val Glu Asp Lys
145 150 155 160
Asp Ala Val Ala Phe Thr Cys Glu Pro Glu Thr Gln Asp Ala Thr Tyr
165 170 175
Leu Trp Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg Leu Gln
180 185 190
Leu Ser Asn Gly Asn Arg Thr Leu Thr Leu Phe Asn Val Thr Arg Asn
195 200 205
Asp Thr Ala Ser Tyr Lys Cys Glu Thr Gln Asn Pro Val Ser Ala Arg
210 215 220
Arg Ser Asp Ser Val Ile Leu Asn Val Leu Tyr Gly Pro Asp Ala Pro
225 230 235 240
Thr Ile Ser Pro Leu Asn Thr Ser Tyr Arg Ser Gly Glu Asn Leu Asn
245 250 255
Leu Ser Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser Trp Phe
260 265 270
Val Asn Gly Thr Phe Gln Gln Ser Thr Gln Glu Leu Phe Ile Pro Asn
275 280 285
Ile Thr Val Asn Asn Ser Gly Ser Tyr Thr Cys Gln Ala His Asn Ser
290 295 300
Asp Thr Gly Leu Asn Arg Thr Thr Val Thr Thr Ile Thr Val Tyr Ala
305 310 315 320
Glu Pro Pro Lys Pro Phe Ile Thr Ser Asn Asn Ser Asn Pro Val Glu
325 330 335
Asp Glu Asp Ala Val Ala Leu Thr Cys Glu Pro Glu Ile Gln Asn Thr
340 345 350
Thr Tyr Leu Trp Trp Val Asn Asn Gln Ser Leu Pro Val Ser Pro Arg
355 360 365
Leu Gln Leu Ser Asn Asp Asn Arg Thr Leu Thr Leu Leu Ser Val Thr
370 375 380
Arg Asn Asp Val Gly Pro Tyr Glu Cys Gly Ile Gln Asn Lys Leu Ser
385 390 395 400
Val Asp His Ser Asp Pro Val Ile Leu Asn Val Leu Tyr Gly Pro Asp
405 410 415
Asp Pro Thr Ile Ser Pro Ser Tyr Thr Tyr Tyr Arg Pro Gly Val Asn
420 425 430
Leu Ser Leu Ser Cys His Ala Ala Ser Asn Pro Pro Ala Gln Tyr Ser
435 440 445
Trp Leu Ile Asp Gly Asn Ile Gln Gln His Thr Gln Glu Leu Phe Ile
450 455 460
Ser Asn Ile Thr Glu Lys Asn Ser Gly Leu Tyr Thr Cys Gln Ala Asn
465 470 475 480
Asn Ser Ala Ser Gly His Ser Arg Thr Thr Val Lys Thr Ile Thr Val
485 490 495
Ser Ala Glu Leu Pro Lys Pro Ser Ile Ser Ser Asn Asn Ser Lys Pro
500 505 510
Val Glu Asp Lys Asp Ala Val Ala Phe Thr Cys Glu Pro Glu Ala Gln
515 520 525
Asn Thr Thr Tyr Leu Trp Trp Val Asn Gly Gln Ser Leu Pro Val Ser
530 535 540
Pro Arg Leu Gln Leu Ser Asn Gly Asn Arg Thr Leu Thr Leu Phe Asn
545 550 555 560
Val Thr Arg Asn Asp Ala Arg Ala Tyr Val Cys Gly Ile Gln Asn Ser
565 570 575
Val Ser Ala Asn Arg Ser Asp Pro Val Thr Leu Asp Val Leu Tyr Gly
580 585 590
Pro Asp Thr Pro Ile Ile Ser Pro Pro Asp Ser Ser Tyr Leu Ser Gly
595 600 605
Ala Asn Leu Asn Leu Ser Cys His Ser Ala Ser Asn Pro Ser Pro Gln
610 615 620
Tyr Ser Trp Arg Ile Asn Gly Ile Pro Gln Gln His Thr Gln Val Leu
625 630 635 640
Phe Ile Ala Lys Ile Thr Pro Asn Asn Asn Gly Thr Tyr Ala Cys Phe
645 650 655
Val Ser Asn Leu Ala Thr Gly Arg Asn Asn Ser Ile Val Lys Ser Ile
660 665 670
Thr Val Ser Ala Ser Gly Thr Ser Pro Gly Leu Ser Ala Gly Ala Thr
675 680 685
Val Gly Ile Met Ile Gly Val Leu Val Gly Val Ala Leu Ile
690 695 700
<210> 93
<211> 2322
<212> PRT
<213> Homo sapiens
<400> 93
Met Gln Ser Gly Pro Arg Pro Pro Leu Pro Ala Pro Gly Leu Ala Leu
1 5 10 15
Ala Leu Thr Leu Thr Met Leu Ala Arg Leu Ala Ser Ala Ala Ser Phe
20 25 30
Phe Gly Glu Asn His Leu Glu Val Pro Val Ala Thr Ala Leu Thr Asp
35 40 45
Ile Asp Leu Gln Leu Gln Phe Ser Thr Ser Gln Pro Glu Ala Leu Leu
50 55 60
Leu Leu Ala Ala Gly Pro Ala Asp His Leu Leu Leu Gln Leu Tyr Ser
65 70 75 80
Gly Arg Leu Gln Val Arg Leu Val Leu Gly Gln Glu Glu Leu Arg Leu
85 90 95
Gln Thr Pro Ala Glu Thr Leu Leu Ser Asp Ser Ile Pro His Thr Val
100 105 110
Val Leu Thr Val Val Glu Gly Trp Ala Thr Leu Ser Val Asp Gly Phe
115 120 125
Leu Asn Ala Ser Ser Ala Val Pro Gly Ala Pro Leu Glu Val Pro Tyr
130 135 140
Gly Leu Phe Val Gly Gly Thr Gly Thr Leu Gly Leu Pro Tyr Leu Arg
145 150 155 160
Gly Thr Ser Arg Pro Leu Arg Gly Cys Leu His Ala Ala Thr Leu Asn
165 170 175
Gly Arg Ser Leu Leu Arg Pro Leu Thr Pro Asp Val His Glu Gly Cys
180 185 190
Ala Glu Glu Phe Ser Ala Ser Asp Asp Val Ala Leu Gly Phe Ser Gly
195 200 205
Pro His Ser Leu Ala Ala Phe Pro Ala Trp Gly Thr Gln Asp Glu Gly
210 215 220
Thr Leu Glu Phe Thr Leu Thr Thr Gln Ser Arg Gln Ala Pro Leu Ala
225 230 235 240
Phe Gln Ala Gly Gly Arg Arg Gly Asp Phe Ile Tyr Val Asp Ile Phe
245 250 255
Glu Gly His Leu Arg Ala Val Val Glu Lys Gly Gln Gly Thr Val Leu
260 265 270
Leu His Asn Ser Val Pro Val Ala Asp Gly Gln Pro His Glu Val Ser
275 280 285
Val His Ile Asn Ala His Arg Leu Glu Ile Ser Val Asp Gln Tyr Pro
290 295 300
Thr His Thr Ser Asn Arg Gly Val Leu Ser Tyr Leu Glu Pro Arg Gly
305 310 315 320
Ser Leu Leu Leu Gly Gly Leu Asp Ala Glu Ala Ser Arg His Leu Gln
325 330 335
Glu His Arg Leu Gly Leu Thr Pro Glu Ala Thr Asn Ala Ser Leu Leu
340 345 350
Gly Cys Met Glu Asp Leu Ser Val Asn Gly Gln Arg Arg Gly Leu Arg
355 360 365
Glu Ala Leu Leu Thr Arg Asn Met Ala Ala Gly Cys Arg Leu Glu Glu
370 375 380
Glu Glu Tyr Glu Asp Asp Ala Tyr Gly His Tyr Glu Ala Phe Ser Thr
385 390 395 400
Leu Ala Pro Glu Ala Trp Pro Ala Met Glu Leu Pro Glu Pro Cys Val
405 410 415
Pro Glu Pro Gly Leu Pro Pro Val Phe Ala Asn Phe Thr Gln Leu Leu
420 425 430
Thr Ile Ser Pro Leu Val Val Ala Glu Gly Gly Thr Ala Trp Leu Glu
435 440 445
Trp Arg His Val Gln Pro Thr Leu Asp Leu Met Glu Ala Glu Leu Arg
450 455 460
Lys Ser Gln Val Leu Phe Ser Val Thr Arg Gly Ala Arg His Gly Glu
465 470 475 480
Leu Glu Leu Asp Ile Pro Gly Ala Gln Ala Arg Lys Met Phe Thr Leu
485 490 495
Leu Asp Val Val Asn Arg Lys Ala Arg Phe Ile His Asp Gly Ser Glu
500 505 510
Asp Thr Ser Asp Gln Leu Val Leu Glu Val Ser Val Thr Ala Arg Val
515 520 525
Pro Met Pro Ser Cys Leu Arg Arg Gly Gln Thr Tyr Leu Leu Pro Ile
530 535 540
Gln Val Asn Pro Val Asn Asp Pro Pro His Ile Ile Phe Pro His Gly
545 550 555 560
Ser Leu Met Val Ile Leu Glu His Thr Gln Lys Pro Leu Gly Pro Glu
565 570 575
Val Phe Gln Ala Tyr Asp Pro Asp Ser Ala Cys Glu Gly Leu Thr Phe
580 585 590
Gln Val Leu Gly Thr Ser Ser Gly Leu Pro Val Glu Arg Arg Asp Gln
595 600 605
Pro Gly Glu Pro Ala Thr Glu Phe Ser Cys Arg Glu Leu Glu Ala Gly
610 615 620
Ser Leu Val Tyr Val His Arg Gly Gly Pro Ala Gln Asp Leu Thr Phe
625 630 635 640
Arg Val Ser Asp Gly Leu Gln Ala Ser Pro Pro Ala Thr Leu Lys Val
645 650 655
Val Ala Ile Arg Pro Ala Ile Gln Ile His Arg Ser Thr Gly Leu Arg
660 665 670
Leu Ala Gln Gly Ser Ala Met Pro Ile Leu Pro Ala Asn Leu Ser Val
675 680 685
Glu Thr Asn Ala Val Gly Gln Asp Val Ser Val Leu Phe Arg Val Thr
690 695 700
Gly Ala Leu Gln Phe Gly Glu Leu Gln Lys Gln Gly Ala Gly Gly Val
705 710 715 720
Glu Gly Ala Glu Trp Trp Ala Thr Gln Ala Phe His Gln Arg Asp Val
725 730 735
Glu Gln Gly Arg Val Arg Tyr Leu Ser Thr Asp Pro Gln His His Ala
740 745 750
Tyr Asp Thr Val Glu Asn Leu Ala Leu Glu Val Gln Val Gly Gln Glu
755 760 765
Ile Leu Ser Asn Leu Ser Phe Pro Val Thr Ile Gln Arg Ala Thr Val
770 775 780
Trp Met Leu Arg Leu Glu Pro Leu His Thr Gln Asn Thr Gln Gln Glu
785 790 795 800
Thr Leu Thr Thr Ala His Leu Glu Ala Thr Leu Glu Glu Ala Gly Pro
805 810 815
Ser Pro Pro Thr Phe His Tyr Glu Val Val Gln Ala Pro Arg Lys Gly
820 825 830
Asn Leu Gln Leu Gln Gly Thr Arg Leu Ser Asp Gly Gln Gly Phe Thr
835 840 845
Gln Asp Asp Ile Gln Ala Gly Arg Val Thr Tyr Gly Ala Thr Ala Arg
850 855 860
Ala Ser Glu Ala Val Glu Asp Thr Phe Arg Phe Arg Val Thr Ala Pro
865 870 875 880
Pro Tyr Phe Ser Pro Leu Tyr Thr Phe Pro Ile His Ile Gly Gly Asp
885 890 895
Pro Asp Ala Pro Val Leu Thr Asn Val Leu Leu Val Val Pro Glu Gly
900 905 910
Gly Glu Gly Val Leu Ser Ala Asp His Leu Phe Val Lys Ser Leu Asn
915 920 925
Ser Ala Ser Tyr Leu Tyr Glu Val Met Glu Arg Pro Arg His Gly Arg
930 935 940
Leu Ala Trp Arg Gly Thr Gln Asp Lys Thr Thr Met Val Thr Ser Phe
945 950 955 960
Thr Asn Glu Asp Leu Leu Arg Gly Arg Leu Val Tyr Gln His Asp Asp
965 970 975
Ser Glu Thr Thr Glu Asp Asp Ile Pro Phe Val Ala Thr Arg Gln Gly
980 985 990
Glu Ser Ser Gly Asp Met Ala Trp Glu Glu Val Arg Gly Val Phe Arg
995 1000 1005
Val Ala Ile Gln Pro Val Asn Asp His Ala Pro Val Gln Thr Ile
1010 1015 1020
Ser Arg Ile Phe His Val Ala Arg Gly Gly Arg Arg Leu Leu Thr
1025 1030 1035
Thr Asp Asp Val Ala Phe Ser Asp Ala Asp Ser Gly Phe Ala Asp
1040 1045 1050
Ala Gln Leu Val Leu Thr Arg Lys Asp Leu Leu Phe Gly Ser Ile
1055 1060 1065
Val Ala Val Asp Glu Pro Thr Arg Pro Ile Tyr Arg Phe Thr Gln
1070 1075 1080
Glu Asp Leu Arg Lys Arg Arg Val Leu Phe Val His Ser Gly Ala
1085 1090 1095
Asp Arg Gly Trp Ile Gln Leu Gln Val Ser Asp Gly Gln His Gln
1100 1105 1110
Ala Thr Ala Leu Leu Glu Val Gln Ala Ser Glu Pro Tyr Leu Arg
1115 1120 1125
Val Ala Asn Gly Ser Ser Leu Val Val Pro Gln Gly Gly Gln Gly
1130 1135 1140
Thr Ile Asp Thr Ala Val Leu His Leu Asp Thr Asn Leu Asp Ile
1145 1150 1155
Arg Ser Gly Asp Glu Val His Tyr His Val Thr Ala Gly Pro Arg
1160 1165 1170
Trp Gly Gln Leu Val Arg Ala Gly Gln Pro Ala Thr Ala Phe Ser
1175 1180 1185
Gln Gln Asp Leu Leu Asp Gly Ala Val Leu Tyr Ser His Asn Gly
1190 1195 1200
Ser Leu Ser Pro Arg Asp Thr Met Ala Phe Ser Val Glu Ala Gly
1205 1210 1215
Pro Val His Thr Asp Ala Thr Leu Gln Val Thr Ile Ala Leu Glu
1220 1225 1230
Gly Pro Leu Ala Pro Leu Lys Leu Val Arg His Lys Lys Ile Tyr
1235 1240 1245
Val Phe Gln Gly Glu Ala Ala Glu Ile Arg Arg Asp Gln Leu Glu
1250 1255 1260
Ala Ala Gln Glu Ala Val Pro Pro Ala Asp Ile Val Phe Ser Val
1265 1270 1275
Lys Ser Pro Pro Ser Ala Gly Tyr Leu Val Met Val Ser Arg Gly
1280 1285 1290
Ala Leu Ala Asp Glu Pro Pro Ser Leu Asp Pro Val Gln Ser Phe
1295 1300 1305
Ser Gln Glu Ala Val Asp Thr Gly Arg Val Leu Tyr Leu His Ser
1310 1315 1320
Arg Pro Glu Ala Trp Ser Asp Ala Phe Ser Leu Asp Val Ala Ser
1325 1330 1335
Gly Leu Gly Ala Pro Leu Glu Gly Val Leu Val Glu Leu Glu Val
1340 1345 1350
Leu Pro Ala Ala Ile Pro Leu Glu Ala Gln Asn Phe Ser Val Pro
1355 1360 1365
Glu Gly Gly Ser Leu Thr Leu Ala Pro Pro Leu Leu Arg Val Ser
1370 1375 1380
Gly Pro Tyr Phe Pro Thr Leu Leu Gly Leu Ser Leu Gln Val Leu
1385 1390 1395
Glu Pro Pro Gln His Gly Ala Leu Gln Lys Glu Asp Gly Pro Gln
1400 1405 1410
Ala Arg Thr Leu Ser Ala Phe Ser Trp Arg Met Val Glu Glu Gln
1415 1420 1425
Leu Ile Arg Tyr Val His Asp Gly Ser Glu Thr Leu Thr Asp Ser
1430 1435 1440
Phe Val Leu Met Ala Asn Ala Ser Glu Met Asp Arg Gln Ser His
1445 1450 1455
Pro Val Ala Phe Thr Val Thr Val Leu Pro Val Asn Asp Gln Pro
1460 1465 1470
Pro Ile Leu Thr Thr Asn Thr Gly Leu Gln Met Trp Glu Gly Ala
1475 1480 1485
Thr Ala Pro Ile Pro Ala Glu Ala Leu Arg Ser Thr Asp Gly Asp
1490 1495 1500
Ser Gly Ser Glu Asp Leu Val Tyr Thr Ile Glu Gln Pro Ser Asn
1505 1510 1515
Gly Arg Val Val Leu Arg Gly Ala Pro Gly Thr Glu Val Arg Ser
1520 1525 1530
Phe Thr Gln Ala Gln Leu Asp Gly Gly Leu Val Leu Phe Ser His
1535 1540 1545
Arg Gly Thr Leu Asp Gly Gly Phe Arg Phe Arg Leu Ser Asp Gly
1550 1555 1560
Glu His Thr Ser Pro Gly His Phe Phe Arg Val Thr Ala Gln Lys
1565 1570 1575
Gln Val Leu Leu Ser Leu Lys Gly Ser Gln Thr Leu Thr Val Cys
1580 1585 1590
Pro Gly Ser Val Gln Pro Leu Ser Ser Gln Thr Leu Arg Ala Ser
1595 1600 1605
Ser Ser Ala Gly Thr Asp Pro Gln Leu Leu Leu Tyr Arg Val Val
1610 1615 1620
Arg Gly Pro Gln Leu Gly Arg Leu Phe His Ala Gln Gln Asp Ser
1625 1630 1635
Thr Gly Glu Ala Leu Val Asn Phe Thr Gln Ala Glu Val Tyr Ala
1640 1645 1650
Gly Asn Ile Leu Tyr Glu His Glu Met Pro Pro Glu Pro Phe Trp
1655 1660 1665
Glu Ala His Asp Thr Leu Glu Leu Gln Leu Ser Ser Pro Pro Ala
1670 1675 1680
Arg Asp Val Ala Ala Thr Leu Ala Val Ala Val Ser Phe Glu Ala
1685 1690 1695
Ala Cys Pro Gln Arg Pro Ser His Leu Trp Lys Asn Lys Gly Leu
1700 1705 1710
Trp Val Pro Glu Gly Gln Arg Ala Arg Ile Thr Val Ala Ala Leu
1715 1720 1725
Asp Ala Ser Asn Leu Leu Ala Ser Val Pro Ser Pro Gln Arg Ser
1730 1735 1740
Glu His Asp Val Leu Phe Gln Val Thr Gln Phe Pro Ser Arg Gly
1745 1750 1755
Gln Leu Leu Val Ser Glu Glu Pro Leu His Ala Gly Gln Pro His
1760 1765 1770
Phe Leu Gln Ser Gln Leu Ala Ala Gly Gln Leu Val Tyr Ala His
1775 1780 1785
Gly Gly Gly Gly Thr Gln Gln Asp Gly Phe His Phe Arg Ala His
1790 1795 1800
Leu Gln Gly Pro Ala Gly Ala Ser Val Ala Gly Pro Gln Thr Ser
1805 1810 1815
Glu Ala Phe Ala Ile Thr Val Arg Asp Val Asn Glu Arg Pro Pro
1820 1825 1830
Gln Pro Gln Ala Ser Val Pro Leu Arg Leu Thr Arg Gly Ser Arg
1835 1840 1845
Ala Pro Ile Ser Arg Ala Gln Leu Ser Val Val Asp Pro Asp Ser
1850 1855 1860
Ala Pro Gly Glu Ile Glu Tyr Glu Val Gln Arg Ala Pro His Asn
1865 1870 1875
Gly Phe Leu Ser Leu Val Gly Gly Gly Leu Gly Pro Val Thr Arg
1880 1885 1890
Phe Thr Gln Ala Asp Val Asp Ser Gly Arg Leu Ala Phe Val Ala
1895 1900 1905
Asn Gly Ser Ser Val Ala Gly Ile Phe Gln Leu Ser Met Ser Asp
1910 1915 1920
Gly Ala Ser Pro Pro Leu Pro Met Ser Leu Ala Val Asp Ile Leu
1925 1930 1935
Pro Ser Ala Ile Glu Val Gln Leu Arg Ala Pro Leu Glu Val Pro
1940 1945 1950
Gln Ala Leu Gly Arg Ser Ser Leu Ser Gln Gln Gln Leu Arg Val
1955 1960 1965
Val Ser Asp Arg Glu Glu Pro Glu Ala Ala Tyr Arg Leu Ile Gln
1970 1975 1980
Gly Pro Gln Tyr Gly His Leu Leu Val Gly Gly Arg Pro Thr Ser
1985 1990 1995
Ala Phe Ser Gln Phe Gln Ile Asp Gln Gly Glu Val Val Phe Ala
2000 2005 2010
Phe Thr Asn Phe Ser Ser Ser His Asp His Phe Arg Val Leu Ala
2015 2020 2025
Leu Ala Arg Gly Val Asn Ala Ser Ala Val Val Asn Val Thr Val
2030 2035 2040
Arg Ala Leu Leu His Val Trp Ala Gly Gly Pro Trp Pro Gln Gly
2045 2050 2055
Ala Thr Leu Arg Leu Asp Pro Thr Val Leu Asp Ala Gly Glu Leu
2060 2065 2070
Ala Asn Arg Thr Gly Ser Val Pro Arg Phe Arg Leu Leu Glu Gly
2075 2080 2085
Pro Arg His Gly Arg Val Val Arg Val Pro Arg Ala Arg Thr Glu
2090 2095 2100
Pro Gly Gly Ser Gln Leu Val Glu Gln Phe Thr Gln Gln Asp Leu
2105 2110 2115
Glu Asp Gly Arg Leu Gly Leu Glu Val Gly Arg Pro Glu Gly Arg
2120 2125 2130
Ala Pro Gly Pro Ala Gly Asp Ser Leu Thr Leu Glu Leu Trp Ala
2135 2140 2145
Gln Gly Val Pro Pro Ala Val Ala Ser Leu Asp Phe Ala Thr Glu
2150 2155 2160
Pro Tyr Asn Ala Ala Arg Pro Tyr Ser Val Ala Leu Leu Ser Val
2165 2170 2175
Pro Glu Ala Ala Arg Thr Glu Ala Gly Lys Pro Glu Ser Ser Thr
2180 2185 2190
Pro Thr Gly Glu Pro Gly Pro Met Ala Ser Ser Pro Glu Pro Ala
2195 2200 2205
Val Ala Lys Gly Gly Phe Leu Ser Phe Leu Glu Ala Asn Met Phe
2210 2215 2220
Ser Val Ile Ile Pro Met Cys Leu Val Leu Leu Leu Leu Ala Leu
2225 2230 2235
Ile Leu Pro Leu Leu Phe Tyr Leu Arg Lys Arg Asn Lys Thr Gly
2240 2245 2250
Lys His Asp Val Gln Val Leu Thr Ala Lys Pro Arg Asn Gly Leu
2255 2260 2265
Ala Gly Asp Thr Glu Thr Phe Arg Lys Val Glu Pro Gly Gln Ala
2270 2275 2280
Ile Pro Leu Thr Ala Val Pro Gly Gln Gly Pro Pro Pro Gly Gly
2285 2290 2295
Gln Pro Asp Pro Glu Leu Leu Gln Phe Cys Arg Thr Pro Asn Pro
2300 2305 2310
Ala Leu Lys Asn Gly Gln Tyr Trp Val
2315 2320
<210> 94
<211> 1210
<212> PRT
<213> Homo sapiens
<400> 94
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Val Cys Gln
20 25 30
Gly Thr Ser Asn Lys Leu Thr Gln Leu Gly Thr Phe Glu Asp His Phe
35 40 45
Leu Ser Leu Gln Arg Met Phe Asn Asn Cys Glu Val Val Leu Gly Asn
50 55 60
Leu Glu Ile Thr Tyr Val Gln Arg Asn Tyr Asp Leu Ser Phe Leu Lys
65 70 75 80
Thr Ile Gln Glu Val Ala Gly Tyr Val Leu Ile Ala Leu Asn Thr Val
85 90 95
Glu Arg Ile Pro Leu Glu Asn Leu Gln Ile Ile Arg Gly Asn Met Tyr
100 105 110
Tyr Glu Asn Ser Tyr Ala Leu Ala Val Leu Ser Asn Tyr Asp Ala Asn
115 120 125
Lys Thr Gly Leu Lys Glu Leu Pro Met Arg Asn Leu Gln Glu Ile Leu
130 135 140
His Gly Ala Val Arg Phe Ser Asn Asn Pro Ala Leu Cys Asn Val Glu
145 150 155 160
Ser Ile Gln Trp Arg Asp Ile Val Ser Ser Asp Phe Leu Ser Asn Met
165 170 175
Ser Met Asp Phe Gln Asn His Leu Gly Ser Cys Gln Lys Cys Asp Pro
180 185 190
Ser Cys Pro Asn Gly Ser Cys Trp Gly Ala Gly Glu Glu Asn Cys Gln
195 200 205
Lys Leu Thr Lys Ile Ile Cys Ala Gln Gln Cys Ser Gly Arg Cys Arg
210 215 220
Gly Lys Ser Pro Ser Asp Cys Cys His Asn Gln Cys Ala Ala Gly Cys
225 230 235 240
Thr Gly Pro Arg Glu Ser Asp Cys Leu Val Cys Arg Lys Phe Arg Asp
245 250 255
Glu Ala Thr Cys Lys Asp Thr Cys Pro Pro Leu Met Leu Tyr Asn Pro
260 265 270
Thr Thr Tyr Gln Met Asp Val Asn Pro Glu Gly Lys Tyr Ser Phe Gly
275 280 285
Ala Thr Cys Val Lys Lys Cys Pro Arg Asn Tyr Val Val Thr Asp His
290 295 300
Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser Tyr Glu Met Glu Glu
305 310 315 320
Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly Pro Cys Arg Lys Val
325 330 335
Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn
340 345 350
Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp
355 360 365
Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr
370 375 380
Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu
385 390 395 400
Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp
405 410 415
Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln
420 425 430
His Gly Gln Phe Ser Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu
435 440 445
Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser
450 455 460
Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu
465 470 475 480
Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu
485 490 495
Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro
500 505 510
Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn
515 520 525
Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly
530 535 540
Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro
545 550 555 560
Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro
565 570 575
Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp Gly Pro His Cys Val
580 585 590
Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp
595 600 605
Lys Tyr Ala Asp Ala Gly His Val Cys His Leu Cys His Pro Asn Cys
610 615 620
Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly
625 630 635 640
Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu
645 650 655
Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe Met Arg Arg Arg His
660 665 670
Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu Gln Glu Arg Glu Leu
675 680 685
Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro Asn Gln Ala Leu Leu
690 695 700
Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile Lys Val Leu Gly Ser
705 710 715 720
Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp Ile Pro Glu Gly Glu
725 730 735
Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu Arg Glu Ala Thr Ser
740 745 750
Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala Tyr Val Met Ala Ser
755 760 765
Val Asp Asn Pro His Val Cys Arg Leu Leu Gly Ile Cys Leu Thr Ser
770 775 780
Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe Gly Cys Leu Leu Asp
785 790 795 800
Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser Gln Tyr Leu Leu Asn
805 810 815
Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr Leu Glu Asp Arg Arg
820 825 830
Leu Val His Arg Asp Leu Ala Ala Arg Asn Val Leu Val Lys Thr Pro
835 840 845
Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala Lys Leu Leu Gly Ala
850 855 860
Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys Val Pro Ile Lys Trp
865 870 875 880
Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr Thr His Gln Ser Asp
885 890 895
Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu Met Thr Phe Gly Ser
900 905 910
Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile Ser Ser Ile Leu Glu
915 920 925
Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys Thr Ile Asp Val Tyr
930 935 940
Met Ile Met Val Lys Cys Trp Met Ile Asp Ala Asp Ser Arg Pro Lys
945 950 955 960
Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met Ala Arg Asp Pro Gln
965 970 975
Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met His Leu Pro Ser Pro
980 985 990
Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp Glu Glu Asp Met Asp
995 1000 1005
Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro Gln Gln Gly Phe
1010 1015 1020
Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu Ser Ser Leu
1025 1030 1035
Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp Arg Asn
1040 1045 1050
Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln Arg
1055 1060 1065
Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
1070 1075 1080
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro
1085 1090 1095
Lys Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln
1100 1105 1110
Pro Leu Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro
1115 1120 1125
His Ser Thr Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln
1130 1135 1140
Pro Thr Cys Val Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala
1145 1150 1155
Gln Lys Gly Ser His Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln
1160 1165 1170
Gln Asp Phe Phe Pro Lys Glu Ala Lys Pro Asn Gly Ile Phe Lys
1175 1180 1185
Gly Ser Thr Ala Glu Asn Ala Glu Tyr Leu Arg Val Ala Pro Gln
1190 1195 1200
Ser Ser Glu Phe Ile Gly Ala
1205 1210
<210> 95
<211> 556
<212> PRT
<213> Homo sapiens
<400> 95
Met Pro Pro Pro Arg Leu Leu Phe Phe Leu Leu Phe Leu Thr Pro Met
1 5 10 15
Glu Val Arg Pro Glu Glu Pro Leu Val Val Lys Val Glu Glu Gly Asp
20 25 30
Asn Ala Val Leu Gln Cys Leu Lys Gly Thr Ser Asp Gly Pro Thr Gln
35 40 45
Gln Leu Thr Trp Ser Arg Glu Ser Pro Leu Lys Pro Phe Leu Lys Leu
50 55 60
Ser Leu Gly Leu Pro Gly Leu Gly Ile His Met Arg Pro Leu Ala Ile
65 70 75 80
Trp Leu Phe Ile Phe Asn Val Ser Gln Gln Met Gly Gly Phe Tyr Leu
85 90 95
Cys Gln Pro Gly Pro Pro Ser Glu Lys Ala Trp Gln Pro Gly Trp Thr
100 105 110
Val Asn Val Glu Gly Ser Gly Glu Leu Phe Arg Trp Asn Val Ser Asp
115 120 125
Leu Gly Gly Leu Gly Cys Gly Leu Lys Asn Arg Ser Ser Glu Gly Pro
130 135 140
Ser Ser Pro Ser Gly Lys Leu Met Ser Pro Lys Leu Tyr Val Trp Ala
145 150 155 160
Lys Asp Arg Pro Glu Ile Trp Glu Gly Glu Pro Pro Cys Leu Pro Pro
165 170 175
Arg Asp Ser Leu Asn Gln Ser Leu Ser Gln Asp Leu Thr Met Ala Pro
180 185 190
Gly Ser Thr Leu Trp Leu Ser Cys Gly Val Pro Pro Asp Ser Val Ser
195 200 205
Arg Gly Pro Leu Ser Trp Thr His Val His Pro Lys Gly Pro Lys Ser
210 215 220
Leu Leu Ser Leu Glu Leu Lys Asp Asp Arg Pro Ala Arg Asp Met Trp
225 230 235 240
Val Met Glu Thr Gly Leu Leu Leu Pro Arg Ala Thr Ala Gln Asp Ala
245 250 255
Gly Lys Tyr Tyr Cys His Arg Gly Asn Leu Thr Met Ser Phe His Leu
260 265 270
Glu Ile Thr Ala Arg Pro Val Leu Trp His Trp Leu Leu Arg Thr Gly
275 280 285
Gly Trp Lys Val Ser Ala Val Thr Leu Ala Tyr Leu Ile Phe Cys Leu
290 295 300
Cys Ser Leu Val Gly Ile Leu His Leu Gln Arg Ala Leu Val Leu Arg
305 310 315 320
Arg Lys Arg Lys Arg Met Thr Asp Pro Thr Arg Arg Phe Phe Lys Val
325 330 335
Thr Pro Pro Pro Gly Ser Gly Pro Gln Asn Gln Tyr Gly Asn Val Leu
340 345 350
Ser Leu Pro Thr Pro Thr Ser Gly Leu Gly Arg Ala Gln Arg Trp Ala
355 360 365
Ala Gly Leu Gly Gly Thr Ala Pro Ser Tyr Gly Asn Pro Ser Ser Asp
370 375 380
Val Gln Ala Asp Gly Ala Leu Gly Ser Arg Ser Pro Pro Gly Val Gly
385 390 395 400
Pro Glu Glu Glu Glu Gly Glu Gly Tyr Glu Glu Pro Asp Ser Glu Glu
405 410 415
Asp Ser Glu Phe Tyr Glu Asn Asp Ser Asn Leu Gly Gln Asp Gln Leu
420 425 430
Ser Gln Asp Gly Ser Gly Tyr Glu Asn Pro Glu Asp Glu Pro Leu Gly
435 440 445
Pro Glu Asp Glu Asp Ser Phe Ser Asn Ala Glu Ser Tyr Glu Asn Glu
450 455 460
Asp Glu Glu Leu Thr Gln Pro Val Ala Arg Thr Met Asp Phe Leu Ser
465 470 475 480
Pro His Gly Ser Ala Trp Asp Pro Ser Arg Glu Ala Thr Ser Leu Gly
485 490 495
Ser Gln Ser Tyr Glu Asp Met Arg Gly Ile Leu Tyr Ala Ala Pro Gln
500 505 510
Leu Arg Ser Ile Arg Gly Gln Pro Gly Pro Asn His Glu Glu Asp Ala
515 520 525
Asp Ser Tyr Glu Asn Met Asp Asn Pro Asp Gly Pro Asp Pro Ala Trp
530 535 540
Gly Gly Gly Gly Arg Met Gly Thr Trp Ser Thr Arg
545 550 555
<210> 96
<211> 297
<212> PRT
<213> Homo sapiens
<400> 96
Met Thr Thr Pro Arg Asn Ser Val Asn Gly Thr Phe Pro Ala Glu Pro
1 5 10 15
Met Lys Gly Pro Ile Ala Met Gln Ser Gly Pro Lys Pro Leu Phe Arg
20 25 30
Arg Met Ser Ser Leu Val Gly Pro Thr Gln Ser Phe Phe Met Arg Glu
35 40 45
Ser Lys Thr Leu Gly Ala Val Gln Ile Met Asn Gly Leu Phe His Ile
50 55 60
Ala Leu Gly Gly Leu Leu Met Ile Pro Ala Gly Ile Tyr Ala Pro Ile
65 70 75 80
Cys Val Thr Val Trp Tyr Pro Leu Trp Gly Gly Ile Met Tyr Ile Ile
85 90 95
Ser Gly Ser Leu Leu Ala Ala Thr Glu Lys Asn Ser Arg Lys Cys Leu
100 105 110
Val Lys Gly Lys Met Ile Met Asn Ser Leu Ser Leu Phe Ala Ala Ile
115 120 125
Ser Gly Met Ile Leu Ser Ile Met Asp Ile Leu Asn Ile Lys Ile Ser
130 135 140
His Phe Leu Lys Met Glu Ser Leu Asn Phe Ile Arg Ala His Thr Pro
145 150 155 160
Tyr Ile Asn Ile Tyr Asn Cys Glu Pro Ala Asn Pro Ser Glu Lys Asn
165 170 175
Ser Pro Ser Thr Gln Tyr Cys Tyr Ser Ile Gln Ser Leu Phe Leu Gly
180 185 190
Ile Leu Ser Val Met Leu Ile Phe Ala Phe Phe Gln Glu Leu Val Ile
195 200 205
Ala Gly Ile Val Glu Asn Glu Trp Lys Arg Thr Cys Ser Arg Pro Lys
210 215 220
Ser Asn Ile Val Leu Leu Ser Ala Glu Glu Lys Lys Glu Gln Thr Ile
225 230 235 240
Glu Ile Lys Glu Glu Val Val Gly Leu Thr Glu Thr Ser Ser Gln Pro
245 250 255
Lys Asn Glu Glu Asp Ile Glu Ile Ile Pro Ile Gln Glu Glu Glu Glu
260 265 270
Glu Glu Thr Glu Thr Asn Phe Pro Glu Pro Pro Gln Asp Gln Glu Ser
275 280 285
Ser Pro Ile Glu Asn Asp Ser Ser Pro
290 295
<210> 97
<211> 364
<212> PRT
<213> Homo sapiens
<400> 97
Met Pro Leu Leu Leu Leu Leu Pro Leu Leu Trp Ala Gly Ala Leu Ala
1 5 10 15
Met Asp Pro Asn Phe Trp Leu Gln Val Gln Glu Ser Val Thr Val Gln
20 25 30
Glu Gly Leu Cys Val Leu Val Pro Cys Thr Phe Phe His Pro Ile Pro
35 40 45
Tyr Tyr Asp Lys Asn Ser Pro Val His Gly Tyr Trp Phe Arg Glu Gly
50 55 60
Ala Ile Ile Ser Arg Asp Ser Pro Val Ala Thr Asn Lys Leu Asp Gln
65 70 75 80
Glu Val Gln Glu Glu Thr Gln Gly Arg Phe Arg Leu Leu Gly Asp Pro
85 90 95
Ser Arg Asn Asn Cys Ser Leu Ser Ile Val Asp Ala Arg Arg Arg Asp
100 105 110
Asn Gly Ser Tyr Phe Phe Arg Met Glu Arg Gly Ser Thr Lys Tyr Ser
115 120 125
Tyr Lys Ser Pro Gln Leu Ser Val His Val Thr Asp Leu Thr His Arg
130 135 140
Pro Lys Ile Leu Ile Pro Gly Thr Leu Glu Pro Gly His Ser Lys Asn
145 150 155 160
Leu Thr Cys Ser Val Ser Trp Ala Cys Glu Gln Gly Thr Pro Pro Ile
165 170 175
Phe Ser Trp Leu Ser Ala Ala Pro Thr Ser Leu Gly Pro Arg Thr Thr
180 185 190
His Ser Ser Val Leu Ile Ile Thr Pro Arg Pro Gln Asp His Gly Thr
195 200 205
Asn Leu Thr Cys Gln Val Lys Phe Ala Gly Ala Gly Val Thr Thr Glu
210 215 220
Arg Thr Ile Gln Leu Asn Val Thr Tyr Val Pro Gln Asn Pro Thr Thr
225 230 235 240
Gly Ile Phe Pro Gly Asp Gly Ser Gly Lys Gln Glu Thr Arg Ala Gly
245 250 255
Val Val His Gly Ala Ile Gly Gly Ala Gly Val Thr Ala Leu Leu Ala
260 265 270
Leu Cys Leu Cys Leu Ile Phe Phe Ile Val Lys Thr His Arg Arg Lys
275 280 285
Ala Ala Arg Thr Ala Val Gly Arg Asn Asp Thr His Pro Thr Thr Gly
290 295 300
Ser Ala Ser Pro Lys His Gln Lys Lys Ser Lys Leu His Gly Pro Thr
305 310 315 320
Glu Thr Ser Ser Cys Ser Gly Ala Ala Pro Thr Val Glu Met Asp Glu
325 330 335
Glu Leu His Tyr Ala Ser Leu Asn Phe His Gly Met Asn Pro Ser Lys
340 345 350
Asp Thr Ser Thr Glu Tyr Ser Glu Val Arg Thr Gln
355 360
<210> 98
<211> 277
<212> PRT
<213> Homo sapiens
<400> 98
Met Cys Val Gly Ala Arg Arg Leu Gly Arg Gly Pro Cys Ala Ala Leu
1 5 10 15
Leu Leu Leu Gly Leu Gly Leu Ser Thr Val Thr Gly Leu His Cys Val
20 25 30
Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys His Glu Cys Arg Pro
35 40 45
Gly Asn Gly Met Val Ser Arg Cys Ser Arg Ser Gln Asn Thr Val Cys
50 55 60
Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val Ser Ser Lys Pro
65 70 75 80
Cys Lys Pro Cys Thr Trp Cys Asn Leu Arg Ser Gly Ser Glu Arg Lys
85 90 95
Gln Leu Cys Thr Ala Thr Gln Asp Thr Val Cys Arg Cys Arg Ala Gly
100 105 110
Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp Cys Ala Pro Cys
115 120 125
Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala Cys Lys Pro Trp
130 135 140
Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln Pro Ala Ser Asn
145 150 155 160
Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro Ala Thr Gln Pro
165 170 175
Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Ile Thr Val Gln Pro Thr
180 185 190
Glu Ala Trp Pro Arg Thr Ser Gln Gly Pro Ser Thr Arg Pro Val Glu
195 200 205
Val Pro Gly Gly Arg Ala Val Ala Ala Ile Leu Gly Leu Gly Leu Val
210 215 220
Leu Gly Leu Leu Gly Pro Leu Ala Ile Leu Leu Ala Leu Tyr Leu Leu
225 230 235 240
Arg Arg Asp Gln Arg Leu Pro Pro Asp Ala His Lys Pro Pro Gly Gly
245 250 255
Gly Ser Phe Arg Thr Pro Ile Gln Glu Glu Gln Ala Asp Ala His Ser
260 265 270
Thr Leu Ala Lys Ile
275
<210> 99
<211> 255
<212> PRT
<213> Homo sapiens
<400> 99
Met Gly Asn Ser Cys Tyr Asn Ile Val Ala Thr Leu Leu Leu Val Leu
1 5 10 15
Asn Phe Glu Arg Thr Arg Ser Leu Gln Asp Pro Cys Ser Asn Cys Pro
20 25 30
Ala Gly Thr Phe Cys Asp Asn Asn Arg Asn Gln Ile Cys Ser Pro Cys
35 40 45
Pro Pro Asn Ser Phe Ser Ser Ala Gly Gly Gln Arg Thr Cys Asp Ile
50 55 60
Cys Arg Gln Cys Lys Gly Val Phe Arg Thr Arg Lys Glu Cys Ser Ser
65 70 75 80
Thr Ser Asn Ala Glu Cys Asp Cys Thr Pro Gly Phe His Cys Leu Gly
85 90 95
Ala Gly Cys Ser Met Cys Glu Gln Asp Cys Lys Gln Gly Gln Glu Leu
100 105 110
Thr Lys Lys Gly Cys Lys Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln
115 120 125
Lys Arg Gly Ile Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys
130 135 140
Ser Val Leu Val Asn Gly Thr Lys Glu Arg Asp Val Val Cys Gly Pro
145 150 155 160
Ser Pro Ala Asp Leu Ser Pro Gly Ala Ser Ser Val Thr Pro Pro Ala
165 170 175
Pro Ala Arg Glu Pro Gly His Ser Pro Gln Ile Ile Ser Phe Phe Leu
180 185 190
Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu
195 200 205
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
210 215 220
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
225 230 235 240
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
245 250 255
<210> 100
<211> 260
<212> PRT
<213> Homo sapiens
<400> 100
Met Ala Arg Pro His Pro Trp Trp Leu Cys Val Leu Gly Thr Leu Val
1 5 10 15
Gly Leu Ser Ala Thr Pro Ala Pro Lys Ser Cys Pro Glu Arg His Tyr
20 25 30
Trp Ala Gln Gly Lys Leu Cys Cys Gln Met Cys Glu Pro Gly Thr Phe
35 40 45
Leu Val Lys Asp Cys Asp Gln His Arg Lys Ala Ala Gln Cys Asp Pro
50 55 60
Cys Ile Pro Gly Val Ser Phe Ser Pro Asp His His Thr Arg Pro His
65 70 75 80
Cys Glu Ser Cys Arg His Cys Asn Ser Gly Leu Leu Val Arg Asn Cys
85 90 95
Thr Ile Thr Ala Asn Ala Glu Cys Ala Cys Arg Asn Gly Trp Gln Cys
100 105 110
Arg Asp Lys Glu Cys Thr Glu Cys Asp Pro Leu Pro Asn Pro Ser Leu
115 120 125
Thr Ala Arg Ser Ser Gln Ala Leu Ser Pro His Pro Gln Pro Thr His
130 135 140
Leu Pro Tyr Val Ser Glu Met Leu Glu Ala Arg Thr Ala Gly His Met
145 150 155 160
Gln Thr Leu Ala Asp Phe Arg Gln Leu Pro Ala Arg Thr Leu Ser Thr
165 170 175
His Trp Pro Pro Gln Arg Ser Leu Cys Ser Ser Asp Phe Ile Arg Ile
180 185 190
Leu Val Ile Phe Ser Gly Met Phe Leu Val Phe Thr Leu Ala Gly Ala
195 200 205
Leu Phe Leu His Gln Arg Arg Lys Tyr Arg Ser Asn Lys Gly Glu Ser
210 215 220
Pro Val Glu Pro Ala Glu Pro Cys His Tyr Ser Cys Pro Arg Glu Glu
225 230 235 240
Glu Gly Ser Thr Ile Pro Ile Gln Glu Asp Tyr Arg Lys Pro Glu Pro
245 250 255
Ala Cys Ser Pro
260
<210> 101
<211> 283
<212> PRT
<213> Homo sapiens
<400> 101
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Lys Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Cys Ser Pro Gly His Phe Cys Ile
115 120 125
Val Gln Asp Gly Asp His Cys Ala Ala Cys Arg Ala Tyr Ala Thr Ser
130 135 140
Ser Pro Gly Gln Arg Val Gln Lys Gly Gly Thr Glu Ser Gln Asp Thr
145 150 155 160
Leu Cys Gln Asn Cys Pro Pro Gly Thr Phe Ser Pro Asn Gly Thr Leu
165 170 175
Glu Glu Cys Gln His Gln Thr Lys Cys Ser Trp Leu Val Thr Lys Ala
180 185 190
Gly Ala Gly Thr Ser Ser Ser His Trp Val Trp Trp Phe Leu Ser Gly
195 200 205
Ser Leu Val Ile Val Ile Val Cys Ser Thr Val Gly Leu Ile Ile Cys
210 215 220
Val Lys Arg Arg Lys Pro Arg Gly Asp Val Val Lys Val Ile Val Ser
225 230 235 240
Val Gln Arg Lys Arg Gln Glu Ala Glu Gly Glu Ala Thr Val Ile Glu
245 250 255
Ala Leu Gln Ala Pro Pro Asp Val Thr Thr Val Ala Val Glu Glu Thr
260 265 270
Ile Pro Ser Phe Thr Gly Arg Ser Pro Asn His
275 280
<210> 102
<211> 595
<212> PRT
<213> Homo sapiens
<400> 102
Met Arg Val Leu Leu Ala Ala Leu Gly Leu Leu Phe Leu Gly Ala Leu
1 5 10 15
Arg Ala Phe Pro Gln Asp Arg Pro Phe Glu Asp Thr Cys His Gly Asn
20 25 30
Pro Ser His Tyr Tyr Asp Lys Ala Val Arg Arg Cys Cys Tyr Arg Cys
35 40 45
Pro Met Gly Leu Phe Pro Thr Gln Gln Cys Pro Gln Arg Pro Thr Asp
50 55 60
Cys Arg Lys Gln Cys Glu Pro Asp Tyr Tyr Leu Asp Glu Ala Asp Arg
65 70 75 80
Cys Thr Ala Cys Val Thr Cys Ser Arg Asp Asp Leu Val Glu Lys Thr
85 90 95
Pro Cys Ala Trp Asn Ser Ser Arg Val Cys Glu Cys Arg Pro Gly Met
100 105 110
Phe Cys Ser Thr Ser Ala Val Asn Ser Cys Ala Arg Cys Phe Phe His
115 120 125
Ser Val Cys Pro Ala Gly Met Ile Val Lys Phe Pro Gly Thr Ala Gln
130 135 140
Lys Asn Thr Val Cys Glu Pro Ala Ser Pro Gly Val Ser Pro Ala Cys
145 150 155 160
Ala Ser Pro Glu Asn Cys Lys Glu Pro Ser Ser Gly Thr Ile Pro Gln
165 170 175
Ala Lys Pro Thr Pro Val Ser Pro Ala Thr Ser Ser Ala Ser Thr Met
180 185 190
Pro Val Arg Gly Gly Thr Arg Leu Ala Gln Glu Ala Ala Ser Lys Leu
195 200 205
Thr Arg Ala Pro Asp Ser Pro Ser Ser Val Gly Arg Pro Ser Ser Asp
210 215 220
Pro Gly Leu Ser Pro Thr Gln Pro Cys Pro Glu Gly Ser Gly Asp Cys
225 230 235 240
Arg Lys Gln Cys Glu Pro Asp Tyr Tyr Leu Asp Glu Ala Gly Arg Cys
245 250 255
Thr Ala Cys Val Ser Cys Ser Arg Asp Asp Leu Val Glu Lys Thr Pro
260 265 270
Cys Ala Trp Asn Ser Ser Arg Thr Cys Glu Cys Arg Pro Gly Met Ile
275 280 285
Cys Ala Thr Ser Ala Thr Asn Ser Cys Ala Arg Cys Val Pro Tyr Pro
290 295 300
Ile Cys Ala Ala Glu Thr Val Thr Lys Pro Gln Asp Met Ala Glu Lys
305 310 315 320
Asp Thr Thr Phe Glu Ala Pro Pro Leu Gly Thr Gln Pro Asp Cys Asn
325 330 335
Pro Thr Pro Glu Asn Gly Glu Ala Pro Ala Ser Thr Ser Pro Thr Gln
340 345 350
Ser Leu Leu Val Asp Ser Gln Ala Ser Lys Thr Leu Pro Ile Pro Thr
355 360 365
Ser Ala Pro Val Ala Leu Ser Ser Thr Gly Lys Pro Val Leu Asp Ala
370 375 380
Gly Pro Val Leu Phe Trp Val Ile Leu Val Leu Val Val Val Val Gly
385 390 395 400
Ser Ser Ala Phe Leu Leu Cys His Arg Arg Ala Cys Arg Lys Arg Ile
405 410 415
Arg Gln Lys Leu His Leu Cys Tyr Pro Val Gln Thr Ser Gln Pro Lys
420 425 430
Leu Glu Leu Val Asp Ser Arg Pro Arg Arg Ser Ser Thr Gln Leu Arg
435 440 445
Ser Gly Ala Ser Val Thr Glu Pro Val Ala Glu Glu Arg Gly Leu Met
450 455 460
Ser Gln Pro Leu Met Glu Thr Cys His Ser Val Gly Ala Ala Tyr Leu
465 470 475 480
Glu Ser Leu Pro Leu Gln Asp Ala Ser Pro Ala Gly Gly Pro Ser Ser
485 490 495
Pro Arg Asp Leu Pro Glu Pro Arg Val Ser Thr Glu His Thr Asn Asn
500 505 510
Lys Ile Glu Lys Ile Tyr Ile Met Lys Ala Asp Thr Val Ile Val Gly
515 520 525
Thr Val Lys Ala Glu Leu Pro Glu Gly Arg Gly Leu Ala Gly Pro Ala
530 535 540
Glu Pro Glu Leu Glu Glu Glu Leu Glu Ala Asp His Thr Pro His Tyr
545 550 555 560
Pro Glu Gln Glu Thr Glu Pro Pro Leu Gly Ser Cys Ser Asp Val Met
565 570 575
Leu Ser Val Glu Glu Glu Gly Lys Glu Asp Pro Leu Pro Thr Ala Ala
580 585 590
Ser Gly Lys
595
<210> 103
<211> 241
<212> PRT
<213> Homo sapiens
<400> 103
Met Ala Gln His Gly Ala Met Gly Ala Phe Arg Ala Leu Cys Gly Leu
1 5 10 15
Ala Leu Leu Cys Ala Leu Ser Leu Gly Gln Arg Pro Thr Gly Gly Pro
20 25 30
Gly Cys Gly Pro Gly Arg Leu Leu Leu Gly Thr Gly Thr Asp Ala Arg
35 40 45
Cys Cys Arg Val His Thr Thr Arg Cys Cys Arg Asp Tyr Pro Gly Glu
50 55 60
Glu Cys Cys Ser Glu Trp Asp Cys Met Cys Val Gln Pro Glu Phe His
65 70 75 80
Cys Gly Asp Pro Cys Cys Thr Thr Cys Arg His His Pro Cys Pro Pro
85 90 95
Gly Gln Gly Val Gln Ser Gln Gly Lys Phe Ser Phe Gly Phe Gln Cys
100 105 110
Ile Asp Cys Ala Ser Gly Thr Phe Ser Gly Gly His Glu Gly His Cys
115 120 125
Lys Pro Trp Thr Asp Cys Thr Gln Phe Gly Phe Leu Thr Val Phe Pro
130 135 140
Gly Asn Lys Thr His Asn Ala Val Cys Val Pro Gly Ser Pro Pro Ala
145 150 155 160
Glu Pro Leu Gly Trp Leu Thr Val Val Leu Leu Ala Val Ala Ala Cys
165 170 175
Val Leu Leu Leu Thr Ser Ala Gln Leu Gly Leu His Ile Trp Gln Leu
180 185 190
Arg Ser Gln Cys Met Trp Pro Arg Glu Thr Gln Leu Leu Leu Glu Val
195 200 205
Pro Pro Ser Thr Glu Asp Ala Arg Ser Cys Gln Phe Pro Glu Glu Glu
210 215 220
Arg Gly Glu Arg Ser Ala Glu Glu Lys Gly Arg Leu Gly Asp Leu Trp
225 230 235 240
Val
<210> 104
<211> 272
<212> PRT
<213> Mus musculus
<400> 104
Met Tyr Val Trp Val Gln Gln Pro Thr Ala Leu Leu Leu Leu Ala Leu
1 5 10 15
Thr Leu Gly Val Thr Ala Arg Arg Leu Asn Cys Val Lys His Thr Tyr
20 25 30
Pro Ser Gly His Lys Cys Cys Arg Glu Cys Gln Pro Gly His Gly Met
35 40 45
Val Ser Arg Cys Asp His Thr Arg Asp Thr Leu Cys His Pro Cys Glu
50 55 60
Thr Gly Phe Tyr Asn Glu Ala Val Asn Tyr Asp Thr Cys Lys Gln Cys
65 70 75 80
Thr Gln Cys Asn His Arg Ser Gly Ser Glu Leu Lys Gln Asn Cys Thr
85 90 95
Pro Thr Gln Asp Thr Val Cys Arg Cys Arg Pro Gly Thr Gln Pro Arg
100 105 110
Gln Asp Ser Gly Tyr Lys Leu Gly Val Asp Cys Val Pro Cys Pro Pro
115 120 125
Gly His Phe Ser Pro Gly Asn Asn Gln Ala Cys Lys Pro Trp Thr Asn
130 135 140
Cys Thr Leu Ser Gly Lys Gln Thr Arg His Pro Ala Ser Asp Ser Leu
145 150 155 160
Asp Ala Val Cys Glu Asp Arg Ser Leu Leu Ala Thr Leu Leu Trp Glu
165 170 175
Thr Gln Arg Pro Thr Phe Arg Pro Thr Thr Val Gln Ser Thr Thr Val
180 185 190
Trp Pro Arg Thr Ser Glu Leu Pro Ser Pro Pro Thr Leu Val Thr Pro
195 200 205
Glu Gly Pro Ala Phe Ala Val Leu Leu Gly Leu Gly Leu Gly Leu Leu
210 215 220
Ala Pro Leu Thr Val Leu Leu Ala Leu Tyr Leu Leu Arg Lys Ala Trp
225 230 235 240
Arg Leu Pro Asn Thr Pro Lys Pro Cys Trp Gly Asn Ser Phe Arg Thr
245 250 255
Pro Ile Gln Glu Glu His Thr Asp Ala His Phe Thr Leu Ala Lys Ile
260 265 270
<210> 105
<211> 256
<212> PRT
<213> Mus musculus
<400> 105
Met Gly Asn Asn Cys Tyr Asn Val Val Val Ile Val Leu Leu Leu Val
1 5 10 15
Gly Cys Glu Lys Val Gly Ala Val Gln Asn Ser Cys Asp Asn Cys Gln
20 25 30
Pro Gly Thr Phe Cys Arg Lys Tyr Asn Pro Val Cys Lys Ser Cys Pro
35 40 45
Pro Ser Thr Phe Ser Ser Ile Gly Gly Gln Pro Asn Cys Asn Ile Cys
50 55 60
Arg Val Cys Ala Gly Tyr Phe Arg Phe Lys Lys Phe Cys Ser Ser Thr
65 70 75 80
His Asn Ala Glu Cys Glu Cys Ile Glu Gly Phe His Cys Leu Gly Pro
85 90 95
Gln Cys Thr Arg Cys Glu Lys Asp Cys Arg Pro Gly Gln Glu Leu Thr
100 105 110
Lys Gln Gly Cys Lys Thr Cys Ser Leu Gly Thr Phe Asn Asp Gln Asn
115 120 125
Gly Thr Gly Val Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Arg
130 135 140
Ser Val Leu Lys Thr Gly Thr Thr Glu Lys Asp Val Val Cys Gly Pro
145 150 155 160
Pro Val Val Ser Phe Ser Pro Ser Thr Thr Ile Ser Val Thr Pro Glu
165 170 175
Gly Gly Pro Gly Gly His Ser Leu Gln Val Leu Thr Leu Phe Leu Ala
180 185 190
Leu Thr Ser Ala Leu Leu Leu Ala Leu Ile Phe Ile Thr Leu Leu Phe
195 200 205
Ser Val Leu Lys Trp Ile Arg Lys Lys Phe Pro His Ile Phe Lys Gln
210 215 220
Pro Phe Lys Lys Thr Thr Gly Ala Ala Gln Glu Glu Asp Ala Cys Ser
225 230 235 240
Cys Arg Cys Pro Gln Glu Glu Glu Gly Gly Gly Gly Gly Tyr Glu Leu
245 250 255
<210> 106
<211> 254
<212> PRT
<213> cynomolgus
<400> 106
Met Gly Asn Ser Cys Tyr Asn Ile Val Ala Thr Leu Leu Leu Val Leu
1 5 10 15
Asn Phe Glu Arg Thr Arg Ser Leu Gln Asp Leu Cys Ser Asn Cys Pro
20 25 30
Ala Gly Thr Phe Cys Asp Asn Asn Arg Ser Gln Ile Cys Ser Pro Cys
35 40 45
Pro Pro Asn Ser Phe Ser Ser Ala Gly Gly Gln Arg Thr Cys Asp Ile
50 55 60
Cys Arg Gln Cys Lys Gly Val Phe Lys Thr Arg Lys Glu Cys Ser Ser
65 70 75 80
Thr Ser Asn Ala Glu Cys Asp Cys Ile Ser Gly Tyr His Cys Leu Gly
85 90 95
Ala Glu Cys Ser Met Cys Glu Gln Asp Cys Lys Gln Gly Gln Glu Leu
100 105 110
Thr Lys Lys Gly Cys Lys Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln
115 120 125
Lys Arg Gly Ile Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys
130 135 140
Ser Val Leu Val Asn Gly Thr Lys Glu Arg Asp Val Val Cys Gly Pro
145 150 155 160
Ser Pro Ala Asp Leu Ser Pro Gly Ala Ser Ser Ala Thr Pro Pro Ala
165 170 175
Pro Ala Arg Glu Pro Gly His Ser Pro Gln Ile Ile Phe Phe Leu Ala
180 185 190
Leu Thr Ser Thr Val Val Leu Phe Leu Leu Phe Phe Leu Val Leu Arg
195 200 205
Phe Ser Val Val Lys Arg Ser Arg Lys Lys Leu Leu Tyr Ile Phe Lys
210 215 220
Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys
225 230 235 240
Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
245 250
<210> 107
<211> 5
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 107
Gly Gly Gly Gly Ser
1 5
<210> 108
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 108
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 109
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 109
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10
<210> 110
<211> 14
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10
<210> 111
<211> 10
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 111
Gly Ser Pro Gly Ser Ser Ser Ser Gly Ser
1 5 10
<210> 112
<211> 15
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 112
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 113
<211> 20
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 113
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 114
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 114
Gly Ser Gly Ser Gly Ser Gly Ser
1 5
<210> 115
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 115
Gly Ser Gly Ser Gly Asn Gly Ser
1 5
<210> 116
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 116
Gly Gly Ser Gly Ser Gly Ser Gly
1 5
<210> 117
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 117
Gly Gly Ser Gly Ser Gly
1 5
<210> 118
<211> 4
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 118
Gly Gly Ser Gly
1
<210> 119
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 119
Gly Gly Ser Gly Asn Gly Ser Gly
1 5
<210> 120
<211> 8
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 120
Gly Gly Asn Gly Ser Gly Ser Gly
1 5
<210> 121
<211> 6
<212> PRT
<213> Artificial sequence
<220>
<223> Peptide linker
<400> 121
Gly Gly Asn Gly Ser Gly
1 5
<210> 122
<211> 186
<212> PRT
<213> cynomolgus
<400> 122
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys Gln
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Asn Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ala Lys Pro Cys Lys Ala Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Pro Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Pro Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Thr Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Arg Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Arg Gly Pro Ala
180 185
<210> 123
<211> 192
<212> PRT
<213> Mus musculus
<400> 123
Val Thr Ala Arg Arg Leu Asn Cys Val Lys His Thr Tyr Pro Ser Gly
1 5 10 15
His Lys Cys Cys Arg Glu Cys Gln Pro Gly His Gly Met Val Ser Arg
20 25 30
Cys Asp His Thr Arg Asp Thr Leu Cys His Pro Cys Glu Thr Gly Phe
35 40 45
Tyr Asn Glu Ala Val Asn Tyr Asp Thr Cys Lys Gln Cys Thr Gln Cys
50 55 60
Asn His Arg Ser Gly Ser Glu Leu Lys Gln Asn Cys Thr Pro Thr Gln
65 70 75 80
Asp Thr Val Cys Arg Cys Arg Pro Gly Thr Gln Pro Arg Gln Asp Ser
85 90 95
Gly Tyr Lys Leu Gly Val Asp Cys Val Pro Cys Pro Pro Gly His Phe
100 105 110
Ser Pro Gly Asn Asn Gln Ala Cys Lys Pro Trp Thr Asn Cys Thr Leu
115 120 125
Ser Gly Lys Gln Thr Arg His Pro Ala Ser Asp Ser Leu Asp Ala Val
130 135 140
Cys Glu Asp Arg Ser Leu Leu Ala Thr Leu Leu Trp Glu Thr Gln Arg
145 150 155 160
Pro Thr Phe Arg Pro Thr Thr Val Gln Ser Thr Thr Val Trp Pro Arg
165 170 175
Thr Ser Glu Leu Pro Ser Pro Pro Thr Leu Val Thr Pro Glu Gly Pro
180 185 190
<210> 124
<211> 681
<212> DNA
<213> Artificial Sequence
<220>
<223> Nucleotide sequence Fc hole chain
<400> 124
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 60
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 120
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 180
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 240
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 300
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 360
gggcagcccc gagaaccaca ggtgtgcacc ctgcccccat cccgggatga gctgaccaag 420
aaccaggtca gcctctcgtg cgcagtcaaa ggcttctatc ccagcgacat cgccgtggag 480
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 540
gacggctcct tcttcctcgt gagcaagctc accgtggaca agagcaggtg gcagcagggg 600
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 660
ctctccctgt ctccgggtaa a 681
<210> 125
<211> 1335
<212> DNA
<213> Artificial Sequence
<220>
<223> Nucleotide sequence human OX40 antigen Fc knob chain
<400> 125
ctgcactgcg tgggcgacac ctaccccagc aacgaccggt gctgccacga gtgcagaccc 60
ggcaacggca tggtgtcccg gtgcagccgg tcccagaaca ccgtgtgcag accttgcggc 120
cctggcttct acaacgacgt ggtgtccagc aagccctgca agccttgtac ctggtgcaac 180
ctgcggagcg gcagcgagcg gaagcagctg tgtaccgcca cccaggatac cgtgtgccgg 240
tgtagagccg gcacccagcc cctggacagc tacaaacccg gcgtggactg cgccccttgc 300
cctcctggcc acttcagccc tggcgacaac caggcctgca agccttggac caactgcacc 360
ctggccggca agcacaccct gcagcccgcc agcaatagca gcgacgccat ctgcgaggac 420
cgggatcctc ctgccaccca gcctcaggaa acccagggcc ctcccgccag acccatcacc 480
gtgcagccta cagaggcctg gcccagaacc agccaggggc ctagcaccag acccgtggaa 540
gtgcctggcg gcagagccgt cgacgaacag ttatattttc agggcggctc acccaaatct 600
gcagacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 660
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 720
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 780
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 840
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 900
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 960
aaagggcagc cccgagaacc acaggtgtac accctgcccc catgccggga tgagctgacc 1020
aagaaccagg tcagcctgtg gtgcctggtc aaaggcttct atcccagcga catcgccgtg 1080
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1140
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1200
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1260
agcctctccc tgtctccggg taaatccgga ggcctgaacg acatcttcga ggcccagaag 1320
attgaatggc acgag 1335
<210> 126
<211> 1335
<212> DNA
<213> Artificial Sequence
<220>
<223> Nucleotide sequence cynomolgus OX40 antigen Fc knob chain
<400> 126
ctccactgtg tcggggacac ctaccccagc aacgaccggt gctgtcagga gtgcaggcca 60
ggcaacggga tggtgagccg ctgcaaccgc tcccagaaca cggtgtgccg tccgtgcggg 120
cccggcttct acaacgacgt ggtcagcgcc aagccctgca aggcctgcac atggtgcaac 180
ctcagaagtg ggagtgagcg gaaacagccg tgcacggcca cacaggacac agtctgccgc 240
tgccgggcgg gcacccagcc cctggacagc tacaagcctg gagttgactg tgccccctgc 300
cctccagggc acttctcccc gggcgacaac caggcctgca agccctggac caactgcacc 360
ttggccggga agcacaccct gcagccagcc agcaatagct cggacgccat ctgtgaggac 420
agggaccccc cacccacaca gccccaggag acccagggcc ccccggccag gcccaccact 480
gtccagccca ctgaagcctg gcccagaacc tcacagagac cctccacccg gcccgtggag 540
gtccccaggg gccctgcggt cgacgaacag ttatattttc agggcggctc acccaaatct 600
gcagacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 660
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 720
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 780
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 840
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 900
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 960
aaagggcagc cccgagaacc acaggtgtac accctgcccc catgccggga tgagctgacc 1020
aagaaccagg tcagcctgtg gtgcctggtc aaaggcttct atcccagcga catcgccgtg 1080
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1140
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1200
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1260
agcctctccc tgtctccggg taaatccgga ggcctgaacg acatcttcga ggcccagaag 1320
attgaatggc acgag 1335
<210> 127
<211> 1353
<212> DNA
<213> Artificial Sequence
<220>
<223> Nucleotide sequence murine OX40 antigen Fc knob chain
<400> 127
gtgaccgcca gacggctgaa ctgcgtgaag cacacctacc ccagcggcca caagtgctgc 60
agagagtgcc agcccggcca cggcatggtg tccagatgcg accacacacg ggacaccctg 120
tgccaccctt gcgagacagg cttctacaac gaggccgtga actacgatac ctgcaagcag 180
tgcacccagt gcaaccacag aagcggcagc gagctgaagc agaactgcac ccccacccag 240
gataccgtgt gcagatgcag acccggcacc cagcccagac aggacagcgg ctacaagctg 300
ggcgtggact gcgtgccctg ccctcctggc cacttcagcc ccggcaacaa ccaggcctgc 360
aagccctgga ccaactgcac cctgagcggc aagcagacca gacaccccgc cagcgacagc 420
ctggatgccg tgtgcgagga cagaagcctg ctggccaccc tgctgtggga gacacagcgg 480
cccaccttca gacccaccac cgtgcagagc accaccgtgt ggcccagaac cagcgagctg 540
cccagtcctc ctaccctcgt gacacctgag ggccccgtcg acgaacagtt atattttcag 600
ggcggctcac ccaaatctgc agacaaaact cacacatgcc caccgtgccc agcacctgaa 660
ctcctggggg gaccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 720
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 780
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 840
gagcagtaca acagcacgta ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg 900
ctgaatggca aggagtacaa gtgcaaggtc tccaacaaag ccctcccagc ccccatcgag 960
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtacac cctgccccca 1020
tgccgggatg agctgaccaa gaaccaggtc agcctgtggt gcctggtcaa aggcttctat 1080
cccagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 1140
acgcctcccg tgctggactc cgacggctcc ttcttcctct acagcaagct caccgtggac 1200
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 1260
aaccactaca cgcagaagag cctctccctg tctccgggta aatccggagg cctgaacgac 1320
atcttcgagg cccagaagat tgaatggcac gag 1353
<210> 128
<211> 227
<212> PRT
<213> Artificial Sequence
<220>
<223> Fc hole chain
<400> 128
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Cys Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro Gly Lys
225
<210> 129
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> human OX40 antigen Fc knob chain
<400> 129
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys His
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Ser Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ser Lys Pro Cys Lys Pro Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Leu Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Ala Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Ile Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Gly Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Gly Gly Arg Ala Val Asp Glu Gln Leu Tyr
180 185 190
Phe Gln Gly Gly Ser Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro
195 200 205
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
210 215 220
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
225 230 235 240
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
245 250 255
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
260 265 270
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
275 280 285
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
290 295 300
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
305 310 315 320
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg
325 330 335
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly
340 345 350
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
355 360 365
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
370 375 380
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
385 390 395 400
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
405 410 415
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ser Gly Gly Leu
420 425 430
Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu
435 440 445
<210> 130
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> cynomolgus OX40 antigen
Fc knob chain
<400> 130
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys Gln
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Asn Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ala Lys Pro Cys Lys Ala Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Pro Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Pro Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Thr Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Arg Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Arg Gly Pro Ala Val Asp Glu Gln Leu Tyr
180 185 190
Phe Gln Gly Gly Ser Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro
195 200 205
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
210 215 220
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
225 230 235 240
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
245 250 255
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
260 265 270
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
275 280 285
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
290 295 300
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
305 310 315 320
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg
325 330 335
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly
340 345 350
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
355 360 365
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
370 375 380
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
385 390 395 400
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
405 410 415
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ser Gly Gly Leu
420 425 430
Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu
435 440 445
<210> 131
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<223> murine OX40 antigen Fc knob chain
<400> 131
Val Thr Ala Arg Arg Leu Asn Cys Val Lys His Thr Tyr Pro Ser Gly
1 5 10 15
His Lys Cys Cys Arg Glu Cys Gln Pro Gly His Gly Met Val Ser Arg
20 25 30
Cys Asp His Thr Arg Asp Thr Leu Cys His Pro Cys Glu Thr Gly Phe
35 40 45
Tyr Asn Glu Ala Val Asn Tyr Asp Thr Cys Lys Gln Cys Thr Gln Cys
50 55 60
Asn His Arg Ser Gly Ser Glu Leu Lys Gln Asn Cys Thr Pro Thr Gln
65 70 75 80
Asp Thr Val Cys Arg Cys Arg Pro Gly Thr Gln Pro Arg Gln Asp Ser
85 90 95
Gly Tyr Lys Leu Gly Val Asp Cys Val Pro Cys Pro Pro Gly His Phe
100 105 110
Ser Pro Gly Asn Asn Gln Ala Cys Lys Pro Trp Thr Asn Cys Thr Leu
115 120 125
Ser Gly Lys Gln Thr Arg His Pro Ala Ser Asp Ser Leu Asp Ala Val
130 135 140
Cys Glu Asp Arg Ser Leu Leu Ala Thr Leu Leu Trp Glu Thr Gln Arg
145 150 155 160
Pro Thr Phe Arg Pro Thr Thr Val Gln Ser Thr Thr Val Trp Pro Arg
165 170 175
Thr Ser Glu Leu Pro Ser Pro Pro Thr Leu Val Thr Pro Glu Gly Pro
180 185 190
Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro Lys Ser Ala Asp
195 200 205
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
210 215 220
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
225 230 235 240
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
245 250 255
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
260 265 270
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
275 280 285
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
290 295 300
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
305 310 315 320
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
325 330 335
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
340 345 350
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
355 360 365
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
370 375 380
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
385 390 395 400
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
405 410 415
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
420 425 430
Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu
435 440 445
Trp His Glu
450
<210> 132
<211> 1613
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of library DP88-4
<400> 132
tgaaatacct attgcctacg gcagccgctg gattgttatt actcgcggcc cagccggcca 60
tggccgacat ccagatgacc cagtctcctt ccaccctgtc tgcatctgta ggagaccgtg 120
tcaccatcac ttgccgtgcc agtcagagta ttagtagctg gttggcctgg tatcagcaga 180
aaccagggaa agcccctaag ctcctgatct atgatgcctc cagtttggaa agtggggtcc 240
catcacgttt cagcggcagt ggatccggga cagaattcac tctcaccatc agcagcttgc 300
agcctgatga ttttgcaact tattactgcc aacagtataa tagttattct acgtttggcc 360
agggcaccaa agtcgagatc aagcgtacgg tggctgcacc atctgtcttc atcttcccgc 420
catctgatga gcagttgaaa tctggaactg cctctgttgt gtgcctgctg aataacttct 480
atcccagaga ggccaaagta cagtggaagg tggataacgc cctccaatcg ggtaactccc 540
aggagagtgt cacagagcag gacagcaagg acagcaccta cagcctcagc agcaccctga 600
cgctgagcaa agcagactac gagaaacaca aagtctacgc ctgcgaagtc acccatcagg 660
gcctgagctc gcccgtcaca aagagcttca acaggggaga gtgtggagcc gcagaacaaa 720
aactcatctc agaagaggat ctgaatggag ccgcagacta caaggacgac gacgacaagg 780
gtgccgcata ataaggcgcg ccaattctat ttcaaggaga cagtcatatg aaatacctgc 840
tgccgaccgc tgctgctggt ctgctgctcc tcgctgccca gccggcgatg gcccaggtgc 900
aattggtgca gtctggggct gaggtgaaga agcctgggtc ctcggtgaag gtctcctgca 960
aggcctccgg aggcacattc agcagctacg ctataagctg ggtgcgacag gcccctggac 1020
aagggctcga gtggatggga gggatcatcc ctatctttgg tacagcaaac tacgcacaga 1080
agttccaggg cagggtcacc attactgcag acaaatccac gagcacagcc tacatggagc 1140
tgagcagcct gagatctgag gacaccgccg tgtattactg tgcgagacta tccccaggcg 1200
gttactatgt tatggatgcc tggggccaag ggaccaccgt gaccgtctcc tcagctagca 1260
ccaaaggccc atcggtcttc cccctggcac cctcctccaa gagcacctct gggggcacag 1320
cggccctggg ctgcctggtc aaggactact tccccgaacc ggtgacggtg tcgtggaact 1380
caggcgccct gaccagcggc gtgcacacct tcccggctgt cctacagtcc tcaggactct 1440
actccctcag cagcgtggtg accgtgccct ccagcagctt gggcacccag acctacatct 1500
gcaacgtgaa tcacaagccc agcaacacca aagtggacaa gaaagttgag cccaaatctt 1560
gtgacgcggc cgcaagcact agtgcccatc accatcacca tcacgccgcg gca 1613
<210> 133
<211> 723
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of Fab light chain Vk1_5
<400> 133
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctacgtt tggccagggc 300
accaaagtcg agatcaagcg tacggtggct gcaccatctg tcttcatctt cccgccatct 360
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 420
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 480
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 540
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 600
agctcgcccg tcacaaagag cttcaacagg ggagagtgtg gagccgcaga acaaaaactc 660
atctcagaag aggatctgaa tggagccgca gactacaagg acgacgacga caagggtgcc 720
gca 723
<210> 134
<211> 241
<212> PRT
<213> Artificial Sequence
<220>
<223> Fab light chain Vk1_5
<400> 134
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Asn Ser Tyr Ser Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys Gly Ala Ala Glu Gln Lys Leu Ile Ser Glu Glu
210 215 220
Asp Leu Asn Gly Ala Ala Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala
225 230 235 240
Ala
<210> 135
<211> 720
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of Fab heavy chain VH1_69
<400> 135
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagactatcc 300
ccaggcggtt actatgttat ggatgcctgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca aaggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaaag tggacaagaa agttgagccc 660
aaatcttgtg acgcggccgc aagcactagt gcccatcacc atcaccatca cgccgcggca 720
<210> 136
<211> 240
<212> PRT
<213> Artificial Sequence
<220>
<223> Fab heavy chain VH1_69
<400> 136
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Leu Ser Pro Gly Gly Tyr Tyr Val Met Asp Ala Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Ala Ala Ala Ser Thr Ser Ala His His His His His His Ala Ala Ala
225 230 235 240
<210> 137
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> LMB3 Primer
<400> 137
caggaaacag ctatgaccat gattac 26
<210> 138
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<223> Vk1_5_L3r_S Primer
<400> 138
ctcgactttg gtgccctggc caaacgtsba atacgaatta tactgttggc agtaataagt 60
tgcaaaatca t 71
<210> 139
<211> 74
<212> DNA
<213> Artificial Sequence
<220>
<223> Vk1_5_L3r_SY Primer
<400> 139
ctcgactttg gtgccctggc caaacgtmhr sgratacgaa ttatactgtt ggcagtaata 60
agttgcaaaa tcat 74
<210> 140
<211> 77
<212> DNA
<213> Artificial Sequence
<220>
<223> Vk1_5_L3r_SPY
<400> 140
ctcgactttg gtgccctggc caaacgtmhh msssgratac gaattatact gttggcagta 60
ataagttgca aaatcat 77
<210> 141
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> RJH31 Primer
<400> 141
acgtttggcc agggcaccaa agtcgag 27
<210> 142
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> RJH32 Primer
<400> 142
tctcgcacag taatacacgg cggtgtcc 28
<210> 143
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP88-v4-4
<400> 143
ggacaccgcc gtgtattact gtgcgagaga ctactggggc caagggacca ccgtgaccgt 60
ctcc 64
<210> 144
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP88-v4-6
<400> 144
ggacaccgcc gtgtattact gtgcgagaga ctactggggc caagggacca ccgtgaccgt 60
ctcc 64
<210> 145
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP88-v4-8
<400> 145
ggacaccgcc gtgtattact gtgcgagaga ctactggggc caagggacca ccgtgaccgt 60
ctcc 64
<210> 146
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> fdseqlong Primer
<400> 146
gacgttagta aatgaatttt ctgtatgagg 30
<210> 147
<211> 1596
<212> DNA
<213> Artificial Sequence
<220>
<223> (Vk3_20/VH3_23) template
<400> 147
atgaaatacc tattgcctac ggcagccgct ggattgttat tactcgcggc ccagccggcc 60
atggccgaaa tcgtgttaac gcagtctcca ggcaccctgt ctttgtctcc aggggaaaga 120
gccaccctct cttgcagggc cagtcagagt gttagcagca gctacttagc ctggtaccag 180
cagaaacctg gccaggctcc caggctcctc atctatggag catccagcag ggccactggc 240
atcccagaca ggttcagtgg cagtggatcc gggacagact tcactctcac catcagcaga 300
ctggagcctg aagattttgc agtgtattac tgtcagcagt atggtagctc accgctgacg 360
ttcggccagg ggaccaaagt ggaaatcaaa cgtacggtgg ctgcaccatc tgtcttcatc 420
ttcccgccat ctgatgagca gttgaaatct ggaactgcct ctgttgtgtg cctgctgaat 480
aacttctatc ccagagaggc caaagtacag tggaaggtgg ataacgccct ccaatcgggt 540
aactcccagg agagtgtcac agagcaggac agcaaggaca gcacctacag cctcagcagc 600
accctgacgc tgagcaaagc agactacgag aaacacaaag tctacgcctg cgaagtcacc 660
catcagggcc tgagctcgcc cgtcacaaag agcttcaaca ggggagagtg tggagccgca 720
catcaccatc accatcacgg agccgcagac tacaaggacg acgacgacaa gggtgccgca 780
taataaggcg cgccaattct atttcaagga gacagtcata tgaaatacct gctgccgacc 840
gctgctgctg gtctgctgct cctcgctgcc cagccggcga tggccgaggt gcaattgctg 900
gagtctgggg gaggcttggt acagcctggg gggtccctga gactctcctg tgcagcctcc 960
ggattcacct ttagcagtta tgccatgagc tgggtccgcc aggctccagg gaaggggctg 1020
gagtgggtct cagctattag tggtagtggt ggtagcacat actacgcaga ctccgtgaag 1080
ggccggttca ccatctccag agacaattcc aagaacacgc tgtatctgca gatgaacagc 1140
ctgagagccg aggacacggc cgtatattac tgtgcgaaac cgtttccgta ttttgactac 1200
tggggccaag gaaccctggt caccgtctcg agtgctagca ccaaaggccc atcggtcttc 1260
cccctggcac cctcctccaa gagcacctct gggggcacag cggccctggg ctgcctggtc 1320
aaggactact tccccgaacc ggtgacggtg tcgtggaact caggcgccct gaccagcggc 1380
gtgcacacct tcccggctgt cctacagtcc tcaggactct actccctcag cagcgtggtg 1440
accgtgccct ccagcagctt gggcacccag acctacatct gcaacgtgaa tcacaagccc 1500
agcaacacca aagtggacaa gaaagttgag cccaaatctt gtgacgcggc cgcagaacaa 1560
aaactcatct cagaagagga tctgaatgcc gcggca 1596
<210> 148
<211> 714
<212> DNA
<213> Artificial Sequence
<220>
<223> Fab light chain Vk3_20
<400> 148
gaaatcgtgt taacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcttgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggagcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg atccgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcaccgct gacgttcggc 300
caggggacca aagtggaaat caaacgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgtggagc cgcacatcac 660
catcaccatc acggagccgc agactacaag gacgacgacg acaagggtgc cgca 714
<210> 149
<211> 238
<212> PRT
<213> Artificial Sequence
<220>
<223> Fab light chain Vk3_20
<400> 149
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys Gly Ala Ala His His His His His His
210 215 220
Gly Ala Ala Asp Tyr Lys Asp Asp Asp Asp Lys Gly Ala Ala
225 230 235
<210> 150
<211> 711
<212> DNA
<213> Artificial Sequence
<220>
<223> Fab heavy chain VH3_23
<400> 150
gaggtgcaat tgctggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gaaaccgttt 300
ccgtattttg actactgggg ccaaggaacc ctggtcaccg tctcgagtgc tagcaccaaa 360
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 420
ctgggctgcc tggtcaagga ctacttcccc gaaccggtga cggtgtcgtg gaactcaggc 480
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 540
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 600
gtgaatcaca agcccagcaa caccaaagtg gacaagaaag ttgagcccaa atcttgtgac 660
gcggccgcag aacaaaaact catctcagaa gaggatctga atgccgcggc a 711
<210> 151
<211> 237
<212> PRT
<213> Artificial Sequence
<220>
<223> Fab heavy chain VH3_23 (DP47)
<400> 151
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Phe Pro Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Ala Ala Ala Glu
210 215 220
Gln Lys Leu Ile Ser Glu Glu Asp Leu Asn Ala Ala Ala
225 230 235
<210> 152
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> MS64 Primer
<400> 152
acgttcggcc aggggaccaa agtgg 25
<210> 153
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> DP47CDR3_ba Primer
<400> 153
cgcacagtaa tatacggccg tgtcc 25
<210> 154
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP47-v4-4
<400> 154
cgaggacacg gccgtatatt actgtgcgga ctactggggc caaggaaccc tggtcaccgt 60
ctcg 64
<210> 155
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP47-v4-6
<400> 155
cgaggacacg gccgtatatt actgtgcgga ctactggggc caaggaaccc tggtcaccgt 60
ctcg 64
<210> 156
<211> 64
<212> DNA
<213> Artificial Sequence
<220>
<223> DP47-v4-8
<400> 156
cgaggacacg gccgtatatt actgtgcgga ctactggggc caaggaaccc tggtcaccgt 60
ctcg 64
<210> 157
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> fdseqlong
<400> 157
gacgttagta aatgaatttt ctgtatgagg 30
<210> 158
<211> 1605
<212> DNA
<213> Artificial Sequence
<220>
<223> Vl3_19/VH3_23 library template
<400> 158
atgaaatacc tattgcctac ggcagccgct ggattgttat tactcgcggc ccagccggcc 60
atggcctcgt ctgagctgac tcaggaccct gctgtgtctg tggccttggg acagacagtc 120
aggatcacat gccaaggaga cagcctcaga agttattatg caagctggta ccagcagaag 180
ccaggacagg cccctgtact tgtcatctat ggtaaaaaca accggccctc agggatccca 240
gaccgattct ctggctccag ctcaggaaac acagcttcct tgaccatcac tggggctcag 300
gcggaagatg aggctgacta ttactgtaac tcccgtgata gtagcggtaa tcatgtggta 360
ttcggcggag ggaccaagct gaccgtccta ggacaaccca aggctgcccc cagcgtgacc 420
ctgttccccc ccagcagcga ggaattgcag gccaacaagg ccaccctggt ctgcctgatc 480
agcgacttct acccaggcgc cgtgaccgtg gcctggaagg ccgacagcag ccccgtgaag 540
gccggcgtgg agaccaccac ccccagcaag cagagcaaca acaagtacgc cgccagcagc 600
tacctgagcc tgacccccga gcagtggaag agccacaggt cctacagctg ccaggtgacc 660
cacgagggca gcaccgtgga gaaaaccgtg gcccccaccg agtgcagcgg agccgcagaa 720
caaaaactca tctcagaaga ggatctgaat ggagccgcag actacaagga cgacgacgac 780
aagggtgccg cataataagg cgcgccaatt ctatttcaag gagacagtca tatgaaatac 840
ctgctgccga ccgctgctgc tggtctgctg ctcctcgctg cccagccggc gatggccgag 900
gtgcaattgc tggagtctgg gggaggcttg gtacagcctg gggggtccct gagactctcc 960
tgtgcagcct ccggattcac ctttagcagt tatgccatga gctgggtccg ccaggctcca 1020
gggaaggggc tggagtgggt ctcagctatt agtggtagtg gtggtagcac atactacgca 1080
gactccgtga agggccggtt caccatctcc agagacaatt ccaagaacac gctgtatctg 1140
cagatgaaca gcctgagagc cgaggacacg gccgtatatt actgtgcgaa accgtttccg 1200
tattttgact actggggcca aggaaccctg gtcaccgtct cgagtgctag caccaaaggc 1260
ccatcggtct tccccctggc accctcctcc aagagcacct ctgggggcac agcggccctg 1320
ggctgcctgg tcaaggacta cttccccgaa ccggtgacgg tgtcgtggaa ctcaggcgcc 1380
ctgaccagcg gcgtgcacac cttcccggct gtcctacagt cctcaggact ctactccctc 1440
agcagcgtgg tgaccgtgcc ctccagcagc ttgggcaccc agacctacat ctgcaacgtg 1500
aatcacaagc ccagcaacac caaagtggac aagaaagttg agcccaaatc ttgtgacgcg 1560
gccgcaagca ctagtgccca tcaccatcac catcacgccg cggca 1605
<210> 159
<211> 726
<212> DNA
<213> Artificial Sequence
<220>
<223> Fab light chain Vl3_19
<400> 159
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactcccgt gatagtagcg gtaatcatgt ggtattcggc 300
ggagggacca agctgaccgt cctaggacaa cccaaggctg cccccagcgt gaccctgttc 360
ccccccagca gcgaggaatt gcaggccaac aaggccaccc tggtctgcct gatcagcgac 420
ttctacccag gcgccgtgac cgtggcctgg aaggccgaca gcagccccgt gaaggccggc 480
gtggagacca ccacccccag caagcagagc aacaacaagt acgccgccag cagctacctg 540
agcctgaccc ccgagcagtg gaagagccac aggtcctaca gctgccaggt gacccacgag 600
ggcagcaccg tggagaaaac cgtggccccc accgagtgca gcggagccgc agaacaaaaa 660
ctcatctcag aagaggatct gaatggagcc gcagactaca aggacgacga cgacaagggt 720
gccgca 726
<210> 160
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> Fab light chain Vl3_19
<400> 160
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp Ser Ser Gly Asn His
85 90 95
Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys
100 105 110
Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln
115 120 125
Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly
130 135 140
Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly
145 150 155 160
Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala
165 170 175
Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser
180 185 190
Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val
195 200 205
Ala Pro Thr Glu Cys Ser Gly Ala Ala Glu Gln Lys Leu Ile Ser Glu
210 215 220
Glu Asp Leu Asn Gly Ala Ala Asp Tyr Lys Asp Asp Asp Asp Lys Gly
225 230 235 240
Ala Ala
<210> 161
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> LMB3 lambda-DP47 library
<400> 161
caggaaacag ctatgaccat gattac 26
<210> 162
<211> 85
<212> DNA
<213> Artificial Sequence
<220>
<223> Vl_3_19_L3r_V lambda-DP47 library
<400> 162
ggacggtcag cttggtccct ccgccgaata cvhvattacc gctactatca cgggagttac 60
agtaatagtc agcctcatct tccgc 85
<210> 163
<211> 88
<212> DNA
<213> Artificial Sequence
<220>
<223> Vl_3_19_L3r_HV lambda-DP47
<400> 163
ggacggtcag cttggtccct ccgccgaata ccmmatgatt accgctacta tcacgggagt 60
tacagtaata gtcagcctca tcttccgc 88
<210> 164
<211> 91
<212> DNA
<213> Artificial Sequence
<220>
<223> Vl_3_19_L3r_HLV lambda-DP47
<400> 164
ggacggtcag cttggtccct ccgccgaata crhmvwgatg attaccgcta ctatcacggg 60
agttacagta atagtcagcc tcatcttccg c 91
<210> 165
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> RJH80 lambda-DP47
<400> 165
ttcggcggag ggaccaagct gaccgtcc 28
<210> 166
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (8H9 ) VL
<400> 166
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactcccgt gttatgcctc ataatcgcgt attcggcgga 300
gggaccaagc tgaccgtc 318
<210> 167
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (8H9) VH
<400> 167
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgttttc 300
taccgtggtg gtgtttctat ggactactgg ggccaaggaa ccctggtcac cgtctcgagt 360
<210> 168
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (49B4) VL
<400> 168
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatagttcgc agccgtatac gtttggccag 300
ggcaccaaag tcgagatcaa g 321
<210> 169
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (49B4) VH
<400> 169
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctca 354
<210> 170
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (1G4) VL
<400> 170
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatatttcgt attccatgtt gacgtttggc 300
cagggcacca aagtcgagat caag 324
<210> 171
<211> 348
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (1G4) VH
<400> 171
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttctatgg actactgggg ccaagggacc accgtgaccg tctcctca 348
<210> 172
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (20B7) VL
<400> 172
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatcaggctt tttcgcttac gtttggccag 300
ggcaccaaag tcgagatcaa g 321
<210> 173
<211> 366
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (20B7) VH
<400> 173
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagttaac 300
tacccgtact cttactgggg tgacttcgac tactggggcc aagggaccac cgtgaccgtc 360
tcctca 366
<210> 174
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-563) VL
<400> 174
gagatcgtgc tgacccagag ccccggcaca ctctccctgt ctcctgggga aagggccacc 60
ctttcatgca gagccagcca gtccgtctct agtagctacc tggcatggta tcagcagaag 120
ccaggacaag ccccccgcct cctgatttac ggcgcttcct ctcgggcaac tggtatccct 180
gacaggttct cagggagcgg aagcggaaca gattttacct tgactatttc tagactggag 240
ccagaggact tcgccgtgta ttactgtcag cagtacggta gtagccccct cacctttggc 300
caggggacaa aagtcgaaat caag 324
<210> 175
<211> 348
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-563) VH
<400> 175
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcttgacgtt 300
ggtgctttcg actactgggg ccaaggagcc ctggtcaccg tctcgagt 348
<210> 176
<211> 324
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-564) VL
<400> 176
gagatcgtgc tgacccagag ccccggcaca ctctccctgt ctcctgggga aagggccacc 60
ctttcatgca gagccagcca gtccgtctct agtagctacc tggcatggta tcagcagaag 120
ccaggacaag ccccccgcct cctgatttac ggcgcttcct ctcgggcaac tggtatccct 180
gacaggttct cagggagcgg aagcggaaca gattttacct tgactatttc tagactggag 240
ccagaggact tcgccgtgta ttactgtcag cagtacggta gtagccccct cacctttggc 300
caggggacaa aagtcgaaat caag 324
<210> 177
<211> 348
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-564) VH
<400> 177
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gttcgacgtt 300
ggtccgttcg actactgggg ccaaggaacc ctggtcaccg tctcgagt 348
<210> 178
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (17A9) VL
<400> 178
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactcccgt gttatgcctc ataatcgcgt attcggcgga 300
gggaccaagc tgaccgtc 318
<210> 179
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (17A9) VH
<400> 179
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgttttc 300
taccgtggtg gtgtttctat ggactactgg ggccaaggaa ccctggtcac cgtctcgagt 360
<210> 180
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 8H9 P329GLALA IgG1 light chain
<400> 180
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatttgacgt attcgcggtt tacgtttggc 300
cagggcacca aagtcgagat caagcgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 645
<210> 181
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 8H9 P329GLALA IgG1 heavy chain
<400> 181
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttggatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 420
ctgggctgcc tggtcaagga ctacttcccc gaaccggtga cggtgtcgtg gaactcaggc 480
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 540
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 600
gtgaatcaca agcccagcaa caccaaggtg gacaagaaag ttgagcccaa atcttgtgac 660
aaaactcaca catgcccacc gtgcccagca cctgaagctg cagggggacc gtcagtcttc 720
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 780
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatccc gggatgagct gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 182
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<223> 8H9 P329GLALA IgG1 light chain
<400> 182
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Thr Tyr Ser Arg
85 90 95
Phe Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 183
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> 8H9 P329GLALA IgG1 Heavy chain
<400> 183
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Trp Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 184
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 49B4 P329GLALA IgG1 light chain
<400> 184
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatagttcgc agccgtatac gtttggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 185
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 49B4 P329GLALA IgG1 heavy chain
<400> 185
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg taaa 1344
<210> 186
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> 49B4 P329GLALA IgG1 light chain
<400> 186
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Ser Gln Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 187
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> 49B4 heavy chain
<400> 187
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 188
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 1G4 P329GLALA IgG1 light chain
<400> 188
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatatttcgt attccatgtt gacgtttggc 300
cagggcacca aagtcgagat caagcgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 645
<210> 189
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 1G4 P329GLALA IgG1 heavy chain
<400> 189
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttctatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 420
ctgggctgcc tggtcaagga ctacttcccc gaaccggtga cggtgtcgtg gaactcaggc 480
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 540
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 600
gtgaatcaca agcccagcaa caccaaggtg gacaagaaag ttgagcccaa atcttgtgac 660
aaaactcaca catgcccacc gtgcccagca cctgaagctg cagggggacc gtcagtcttc 720
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 780
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatccc gggatgagct gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 190
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<223> 1G4 P329GLALA IgG1 light chain
<400> 190
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ile Ser Tyr Ser Met
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 191
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> 1G4 P329GLALA IgG1 heavy chain
<400> 191
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Ser Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 192
<211> 642
<212> DNA
<213> Artificial sequence
<220>
<223> DNA 20B7 P329GLALA IgG1 light chain
<400> 192
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatcaggctt tttcgcttac gtttggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 193
<211> 1356
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 20B7 P329GLALA IgG1 heavy chain
<400> 193
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagttaac 300
tacccgtact cttactgggg tgacttcgac tactggggcc aagggaccac cgtgaccgtc 360
tcctcagcta gcaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 420
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga accggtgacg 480
gtgtcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 540
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 600
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagaaagtt 660
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaagctgca 720
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 780
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 840
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 900
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 960
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcg gcgcccccat cgagaaaacc 1020
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1080
gatgagctga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1140
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1200
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 1260
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1320
tacacgcaga agagcctctc cctgtctccg ggtaaa 1356
<210> 194
<211> 214
<212> PRT
<213> Artificial
<220>
<223> 20B7 P329GLALA IgG1 Light chain
<400> 194
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Gln Ala Phe Ser Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 195
<211> 452
<212> PRT
<213> Artificial
<220>
<223> 20B7 P329GLALA IgG1 heavy chain
<400> 195
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Asn Tyr Pro Tyr Ser Tyr Trp Gly Asp Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 196
<211> 645
<212> DNA
<213> Artificial
<220>
<223> DNA CLC-563 P329GLALA IgG1 light chain
<400> 196
gagatcgtgc tgacccagag ccccggcaca ctctccctgt ctcctgggga aagggccacc 60
ctttcatgca gagccagcca gtccgtctct agtagctacc tggcatggta tcagcagaag 120
ccaggacaag ccccccgcct cctgatttac ggcgcttcct ctcgggcaac tggtatccct 180
gacaggttct cagggagcgg aagcggaaca gattttacct tgactatttc tagactggag 240
ccagaggact tcgccgtgta ttactgtcag cagtacggta gtagccccct cacctttggc 300
caggggacaa aagtcgaaat caagcgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 645
<210> 197
<211> 1338
<212> DNA
<213> Artificial
<220>
<223> DNA CLC-563 P329GLALA IgG1 heavy chain
<400> 197
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcttgacgtt 300
ggtgctttcg actactgggg ccaaggagcc ctggtcaccg tctcgagtgc tagcaccaag 360
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 420
ctgggctgcc tggtcaagga ctacttcccc gaaccggtga cggtgtcgtg gaactcaggc 480
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 540
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 600
gtgaatcaca agcccagcaa caccaaggtg gacaagaaag ttgagcccaa atcttgtgac 660
aaaactcaca catgcccacc gtgcccagca cctgaagctg cagggggacc gtcagtcttc 720
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 780
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatccc gggatgagct gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 198
<211> 215
<212> PRT
<213> Artificial
<220>
<223> CLC-563 P329GLALA IgG1 light chain
<400> 198
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 199
<211> 446
<212> PRT
<213> Artificial
<220>
<223> CLC-563 P329GLALA IgG1 heavy chain
<400> 199
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Asp Val Gly Ala Phe Asp Tyr Trp Gly Gln Gly Ala Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 200
<211> 645
<212> DNA
<213> Artificial
<220>
<223> DNA CLC-564 P329GLALA IgG1 light chain
<400> 200
gagatcgtgc tgacccagag ccccggcaca ctctccctgt ctcctgggga aagggccacc 60
ctttcatgca gagccagcca gtccgtctct agtagctacc tggcatggta tcagcagaag 120
ccaggacaag ccccccgcct cctgatttac ggcgcttcct ctcgggcaac tggtatccct 180
gacaggttct cagggagcgg aagcggaaca gattttacct tgactatttc tagactggag 240
ccagaggact tcgccgtgta ttactgtcag cagtacggta gtagccccct cacctttggc 300
caggggacaa aagtcgaaat caagcgtacg gtggctgcac catctgtctt catcttcccg 360
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 420
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 480
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 540
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 600
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 645
<210> 201
<211> 1338
<212> DNA
<213> Artificial
<220>
<223> DNA CLC-564 P329GLALA IgG1 heavy chain
<400> 201
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gttcgacgtt 300
ggtccgttcg actactgggg ccaaggaacc ctggtcaccg tctcgagtgc tagcaccaag 360
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 420
ctgggctgcc tggtcaagga ctacttcccc gaaccggtga cggtgtcgtg gaactcaggc 480
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 540
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 600
gtgaatcaca agcccagcaa caccaaggtg gacaagaaag ttgagcccaa atcttgtgac 660
aaaactcaca catgcccacc gtgcccagca cctgaagctg cagggggacc gtcagtcttc 720
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 780
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 840
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatccc gggatgagct gaccaagaac 1080
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 202
<211> 215
<212> PRT
<213> Artificial
<220>
<223> CLC-564 P329GLALA IgG1 light chain
<400> 202
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 203
<211> 446
<212> PRT
<213> Artificial
<220>
<223> CLC-564 P329GLALA IgG1 heavy chain
<400> 203
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Phe Asp Val Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 204
<211> 639
<212> DNA
<213> Artificial
<220>
<223> DNA 17A9 P329GLALA IgG1 light chain
<400> 204
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactcccgt gttatgcctc ataatcgcgt attcggcgga 300
gggaccaagc tgaccgtcct aggtcaaccc aaggctgccc ccagcgtgac cctgttcccc 360
cccagcagcg aggaactgca ggccaacaag gccaccctgg tctgcctgat cagcgacttc 420
tacccaggcg ccgtgaccgt ggcctggaag gccgacagca gccccgtgaa ggccggcgtg 480
gagaccacca cccccagcaa gcagagcaac aacaagtacg ccgccagcag ctacctgagc 540
ctgacccccg agcagtggaa gagccacagg tcctacagct gccaggtgac ccacgagggc 600
agcaccgtgg agaaaaccgt ggcccccacc gagtgcagc 639
<210> 205
<211> 1350
<212> DNA
<213> Artificial
<220>
<223> DNA 17A9 P329GLALA IgG1 heavy chain
<400> 205
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgttttc 300
taccgtggtg gtgtttctat ggactactgg ggccaaggaa ccctggtcac cgtctcgagt 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 206
<211> 213
<212> PRT
<213> Artificial
<220>
<223> 17A9 P329GLALA IgG1 light chain
<400> 206
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Val Met Pro His Asn Arg
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Cys Ser
210
<210> 207
<211> 450
<212> PRT
<213> Artificial
<220>
<223> 17A9 P329GLALA IgG1 heavy chain
<400> 207
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Phe Tyr Arg Gly Gly Val Ser Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 208
<211> 663
<212> DNA
<213> Artificial Sequence
<220>
<223> human OX40 His nucleotide sequence
<400> 208
ctgcactgcg tgggcgacac ctaccccagc aacgaccggt gctgccacga gtgcagaccc 60
ggcaacggca tggtgtcccg gtgcagccgg tcccagaaca ccgtgtgcag accttgcggc 120
cctggcttct acaacgacgt ggtgtccagc aagccctgca agccttgtac ctggtgcaac 180
ctgcggagcg gcagcgagcg gaagcagctg tgtaccgcca cccaggatac cgtgtgccgg 240
tgtagagccg gcacccagcc cctggacagc tacaaacccg gcgtggactg cgccccttgc 300
cctcctggcc acttcagccc tggcgacaac caggcctgca agccttggac caactgcacc 360
ctggccggca agcacaccct gcagcccgcc agcaatagca gcgacgccat ctgcgaggac 420
cgggatcctc ctgccaccca gcctcaggaa acccagggcc ctcccgccag acccatcacc 480
gtgcagccta cagaggcctg gcccagaacc agccaggggc ctagcaccag acccgtggaa 540
gtgcctggcg gcagagccgt cgacgaacag ttatattttc agggcggctc aggcctgaac 600
gacatcttcg aggcccagaa gatcgagtgg cacgaggctc gagctcacca ccatcaccat 660
cac 663
<210> 209
<211> 221
<212> PRT
<213> Artificial Sequence
<220>
<223> human OX40 His
<400> 209
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys His
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Ser Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ser Lys Pro Cys Lys Pro Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Leu Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Ala Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Ile Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Gly Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Gly Gly Arg Ala Val Asp Glu Gln Leu Tyr
180 185 190
Phe Gln Gly Gly Ser Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile
195 200 205
Glu Trp His Glu Ala Arg Ala His His His His His His
210 215 220
<210> 210
<211> 681
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA murine OX40 His
<400> 210
gtgaccgcca gacggctgaa ctgcgtgaag cacacctacc ccagcggcca caagtgctgc 60
agagagtgcc agcccggcca cggcatggtg tccagatgcg accacacacg ggacaccctg 120
tgccaccctt gcgagacagg cttctacaac gaggccgtga actacgatac ctgcaagcag 180
tgcacccagt gcaaccacag aagcggcagc gagctgaagc agaactgcac ccccacccag 240
gataccgtgt gcagatgcag acccggcacc cagcccagac aggacagcgg ctacaagctg 300
ggcgtggact gcgtgccctg ccctcctggc cacttcagcc ccggcaacaa ccaggcctgc 360
aagccctgga ccaactgcac cctgagcggc aagcagacca gacaccccgc cagcgacagc 420
ctggatgccg tgtgcgagga cagaagcctg ctggccaccc tgctgtggga gacacagcgg 480
cccaccttca gacccaccac cgtgcagagc accaccgtgt ggcccagaac cagcgagctg 540
cccagtcctc ctaccctcgt gacacctgag ggccccgtcg acgaacagtt atattttcag 600
ggcggctcag gcctgaacga catcttcgag gcccagaaga tcgagtggca cgaggctcga 660
gctcaccacc atcaccatca c 681
<210> 211
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> murine OX40 His
<400> 211
Thr Ala Arg Arg Leu Asn Cys Val Lys His Thr Tyr Pro Ser Gly His
1 5 10 15
Lys Cys Cys Arg Glu Cys Gln Pro Gly His Gly Met Val Ser Arg Cys
20 25 30
Asp His Thr Arg Asp Thr Leu Cys His Pro Cys Glu Thr Gly Phe Tyr
35 40 45
Asn Glu Ala Val Asn Tyr Asp Thr Cys Lys Gln Cys Thr Gln Cys Asn
50 55 60
His Arg Ser Gly Ser Glu Leu Lys Gln Asn Cys Thr Pro Thr Gln Asp
65 70 75 80
Thr Val Cys Arg Cys Arg Pro Gly Thr Gln Pro Arg Gln Asp Ser Gly
85 90 95
Tyr Lys Leu Gly Val Asp Cys Val Pro Cys Pro Pro Gly His Phe Ser
100 105 110
Pro Gly Asn Asn Gln Ala Cys Lys Pro Trp Thr Asn Cys Thr Leu Ser
115 120 125
Gly Lys Gln Thr Arg His Pro Ala Ser Asp Ser Leu Asp Ala Val Cys
130 135 140
Glu Asp Arg Ser Leu Leu Ala Thr Leu Leu Trp Glu Thr Gln Arg Pro
145 150 155 160
Thr Phe Arg Pro Thr Thr Val Gln Ser Thr Thr Val Trp Pro Arg Thr
165 170 175
Ser Glu Leu Pro Ser Pro Pro Thr Leu Val Thr Pro Glu Gly Pro Val
180 185 190
Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Gly Leu Asn Asp Ile Phe
195 200 205
Glu Ala Gln Lys Ile Glu Trp His Glu Ala Arg Ala His His His His
210 215 220
His His
225
<210> 212
<211> 1317
<212> DNA
<213> Artificial Sequence
<220>
<223> Nucleotide sequence dimeric human OX40 antigen Fc
<400> 212
ctgcactgcg tgggcgacac ctaccccagc aacgaccggt gctgccacga gtgcagaccc 60
ggcaacggca tggtgtcccg gtgcagccgg tcccagaaca ccgtgtgcag accttgcggc 120
cctggcttct acaacgacgt ggtgtccagc aagccctgca agccttgtac ctggtgcaac 180
ctgcggagcg gcagcgagcg gaagcagctg tgtaccgcca cccaggatac cgtgtgccgg 240
tgtagagccg gcacccagcc cctggacagc tacaaacccg gcgtggactg cgccccttgc 300
cctcctggcc acttcagccc tggcgacaac caggcctgca agccttggac caactgcacc 360
ctggccggca agcacaccct gcagcccgcc agcaatagca gcgacgccat ctgcgaggac 420
cgggatcctc ctgccaccca gcctcaggaa acccagggcc ctcccgccag acccatcacc 480
gtgcagccta cagaggcctg gcccagaacc agccaggggc ctagcaccag acccgtggaa 540
gtgcctggcg gcagagccgt cgacgaacag ttatattttc agggcggctc acccaaatct 600
gcagacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 660
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 720
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 780
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 840
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 900
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 960
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga tgagctgacc 1020
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1080
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1140
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1200
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1260
agcctctccc tgtctccggg taaatccggc tacccatacg atgttccaga ttacgct 1317
<210> 213
<211> 439
<212> PRT
<213> Artificial Sequence
<220>
<223> dimeric human OX40 antigen Fc
<400> 213
Leu His Cys Val Gly Asp Thr Tyr Pro Ser Asn Asp Arg Cys Cys His
1 5 10 15
Glu Cys Arg Pro Gly Asn Gly Met Val Ser Arg Cys Ser Arg Ser Gln
20 25 30
Asn Thr Val Cys Arg Pro Cys Gly Pro Gly Phe Tyr Asn Asp Val Val
35 40 45
Ser Ser Lys Pro Cys Lys Pro Cys Thr Trp Cys Asn Leu Arg Ser Gly
50 55 60
Ser Glu Arg Lys Gln Leu Cys Thr Ala Thr Gln Asp Thr Val Cys Arg
65 70 75 80
Cys Arg Ala Gly Thr Gln Pro Leu Asp Ser Tyr Lys Pro Gly Val Asp
85 90 95
Cys Ala Pro Cys Pro Pro Gly His Phe Ser Pro Gly Asp Asn Gln Ala
100 105 110
Cys Lys Pro Trp Thr Asn Cys Thr Leu Ala Gly Lys His Thr Leu Gln
115 120 125
Pro Ala Ser Asn Ser Ser Asp Ala Ile Cys Glu Asp Arg Asp Pro Pro
130 135 140
Ala Thr Gln Pro Gln Glu Thr Gln Gly Pro Pro Ala Arg Pro Ile Thr
145 150 155 160
Val Gln Pro Thr Glu Ala Trp Pro Arg Thr Ser Gln Gly Pro Ser Thr
165 170 175
Arg Pro Val Glu Val Pro Gly Gly Arg Ala Val Asp Glu Gln Leu Tyr
180 185 190
Phe Gln Gly Gly Ser Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro
195 200 205
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
210 215 220
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
225 230 235 240
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
245 250 255
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
260 265 270
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
275 280 285
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
290 295 300
Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
305 310 315 320
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
325 330 335
Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
340 345 350
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
355 360 365
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
370 375 380
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
385 390 395 400
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
405 410 415
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys Ser Gly Tyr Pro
420 425 430
Tyr Asp Val Pro Asp Tyr Ala
435
<210> 214
<211> 2064
<212> DNA
<213> Artificial Sequence
<220>
<223> (8B9) VHCH1-Heavy chain-(28H1) VHCL
<400> 214
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttggatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggcccatccg tgttccctct ggccccttcc agcaagtcta cctctggcgg cacagccgct 420
ctgggctgcc tcgtgaagga ctacttcccc gagcctgtga cagtgtcctg gaactctggc 480
gccctgacat ccggcgtgca cacctttcca gctgtgctgc agtcctccgg cctgtactcc 540
ctgtcctccg tcgtgacagt gccctccagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agccctccaa caccaaggtg gacaagaagg tggaacccaa gtcctgcgac 660
aagacccaca cctgtccccc ttgtcctgcc cctgaagctg ctggcggccc tagcgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatctccc ggacccccga agtgacctgc 780
gtggtggtgg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagcct agagaggaac agtacaactc cacctaccgg 900
gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga gtacaagtgc 960
aaggtgtcca acaaggccct gggagccccc atcgaaaaga ccatctccaa ggccaagggc 1020
cagcctcgcg agcctcaggt gtacaccctg ccccctagca gagatgagct gaccaagaac 1080
caggtgtccc tgacctgtct cgtgaaaggc ttctacccct ccgatatcgc cgtggaatgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc cccctgtgct ggactccgac 1200
ggctcattct tcctgtactc taagctgaca gtggacaagt cccggtggca gcagggcaac 1260
gtgttctcct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagtccctg 1320
tccctgtctc ccgggggagg cggaggatct ggcggaggcg gatccggtgg tggcggatct 1380
gggggcggtg gatctgaggt gcagctgctg gaatctgggg gaggactggt gcagccaggc 1440
ggatctctga ggctgtcctg cgctgcttcc ggctttacct tctccagcca cgccatgagt 1500
tgggtgcgcc aggcacccgg aaaaggactg gaatgggtgt cagccatctg ggcctccggc 1560
gagcagtact acgccgatag cgtgaagggc cggttcacca tctctcggga taacagcaag 1620
aatactctgt acctgcagat gaactccctg cgcgctgaag ataccgctgt gtattactgc 1680
gccaagggct ggctgggcaa cttcgattac tggggccagg gaaccctcgt gactgtctcg 1740
agcgcttctg tggccgctcc ctccgtgttc atcttcccac cttccgacga gcagctgaag 1800
tccggcactg cctctgtcgt gtgcctgctg aacaacttct accctcggga agccaaggtg 1860
cagtggaaag tggataacgc cctgcagtcc ggcaactccc aggaatccgt gaccgagcag 1920
gactccaagg acagcaccta ctccctgagc agcaccctga ccctgtccaa ggccgactac 1980
gagaagcaca aggtgtacgc ctgtgaagtg acccaccagg gcctgtccag ccccgtgacc 2040
aagtccttca accggggcga gtgc 2064
<210> 215
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA VLCH1-Light chain 2 (28H1)
<400> 215
gagatcgtgc tgacccagtc tcccggcacc ctgagcctga gccctggcga gagagccacc 60
ctgagctgca gagccagcca gagcgtgagc cggagctacc tggcctggta tcagcagaag 120
cccggccagg cccccagact gctgatcatc ggcgccagca cccgggccac cggcatcccc 180
gatagattca gcggcagcgg ctccggcacc gacttcaccc tgaccatcag ccggctggaa 240
cccgaggact tcgccgtgta ctactgccag cagggccagg tgatcccccc caccttcggc 300
cagggcacca aggtggaaat caagagctcc gctagcacca agggcccctc cgtgtttcct 360
ctggccccca gcagcaagag cacctctggc ggaacagccg ccctgggctg cctggtgaaa 420
gactacttcc ccgagcccgt gaccgtgtcc tggaactctg gcgccctgac cagcggcgtg 480
cacacctttc cagccgtgct gcagagcagc ggcctgtact ccctgagcag cgtggtgaca 540
gtgccctcca gcagcctggg cacccagacc tacatctgca acgtgaacca caagcccagc 600
aacaccaaag tggacaagaa ggtggaaccc aagagctgcg ac 642
<210> 216
<211> 688
<212> PRT
<213> Artificial Sequence
<220>
<223> (8B9) VHCH1-Heavy chain-(28H1) VHCL
<400> 216
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Trp Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
450 455 460
Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
465 470 475 480
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser
485 490 495
His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
500 505 510
Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
530 535 540
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
545 550 555 560
Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu
565 570 575
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
580 585 590
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
595 600 605
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
610 615 620
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
625 630 635 640
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
645 650 655
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
660 665 670
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
675 680 685
<210> 217
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> VLCH1-Light chain 2 (28H1)
<400> 217
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Arg Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Ile Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Gln Val Ile Pro
85 90 95
Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Ser Ser Ala Ser
100 105 110
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
115 120 125
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
130 135 140
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
145 150 155 160
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
165 170 175
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
180 185 190
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
195 200 205
Glu Pro Lys Ser Cys Asp
210
<210> 218
<211> 2070
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (49B4) VHCH1-Heavy chain-(28H1) VHCL
<400> 218
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catccgtgtt ccctctggcc ccttccagca agtctacctc tggcggcaca 420
gccgctctgg gctgcctcgt gaaggactac ttccccgagc ctgtgacagt gtcctggaac 480
tctggcgccc tgacatccgg cgtgcacacc tttccagctg tgctgcagtc ctccggcctg 540
tactccctgt cctccgtcgt gacagtgccc tccagctctc tgggcaccca gacctacatc 600
tgcaacgtga accacaagcc ctccaacacc aaggtggaca agaaggtgga acccaagtcc 660
tgcgacaaga cccacacctg tcccccttgt cctgcccctg aagctgctgg cggccctagc 720
gtgttcctgt tccccccaaa gcccaaggac accctgatga tctcccggac ccccgaagtg 780
acctgcgtgg tggtggatgt gtcccacgag gaccctgaag tgaagttcaa ttggtacgtg 840
gacggcgtgg aagtgcacaa tgccaagacc aagcctagag aggaacagta caactccacc 900
taccgggtgg tgtccgtgct gaccgtgctg caccaggatt ggctgaacgg caaagagtac 960
aagtgcaagg tgtccaacaa ggccctggga gcccccatcg aaaagaccat ctccaaggcc 1020
aagggccagc ctcgcgagcc tcaggtgtac accctgcccc ctagcagaga tgagctgacc 1080
aagaaccagg tgtccctgac ctgtctcgtg aaaggcttct acccctccga tatcgccgtg 1140
gaatgggaga gcaacggcca gcccgagaac aactacaaga ccaccccccc tgtgctggac 1200
tccgacggct cattcttcct gtactctaag ctgacagtgg acaagtcccg gtggcagcag 1260
ggcaacgtgt tctcctgctc cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgtccc tgtctcccgg gggaggcgga ggatctggcg gaggcggatc cggtggtggc 1380
ggatctgggg gcggtggatc tgaggtgcag ctgctggaat ctgggggagg actggtgcag 1440
ccaggcggat ctctgaggct gtcctgcgct gcttccggct ttaccttctc cagccacgcc 1500
atgagttggg tgcgccaggc acccggaaaa ggactggaat gggtgtcagc catctgggcc 1560
tccggcgagc agtactacgc cgatagcgtg aagggccggt tcaccatctc tcgggataac 1620
agcaagaata ctctgtacct gcagatgaac tccctgcgcg ctgaagatac cgctgtgtat 1680
tactgcgcca agggctggct gggcaacttc gattactggg gccagggaac cctcgtgact 1740
gtctcgagcg cttctgtggc cgctccctcc gtgttcatct tcccaccttc cgacgagcag 1800
ctgaagtccg gcactgcctc tgtcgtgtgc ctgctgaaca acttctaccc tcgggaagcc 1860
aaggtgcagt ggaaagtgga taacgccctg cagtccggca actcccagga atccgtgacc 1920
gagcaggact ccaaggacag cacctactcc ctgagcagca ccctgaccct gtccaaggcc 1980
gactacgaga agcacaaggt gtacgcctgt gaagtgaccc accagggcct gtccagcccc 2040
gtgaccaagt ccttcaaccg gggcgagtgc 2070
<210> 219
<211> 690
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1-Heavy chain-(28H1) VHCL
<400> 219
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp
515 520 525
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
530 535 540
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
545 550 555 560
Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly
565 570 575
Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe
580 585 590
Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val
595 600 605
Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp
610 615 620
Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr
625 630 635 640
Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr
645 650 655
Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val
660 665 670
Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly
675 680 685
Glu Cys
690
<210> 220
<211> 2064
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (1G4) VHCH1-Heavy chain-(28H1) VHCL
<400> 220
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttctatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggcccatccg tgttccctct ggccccttcc agcaagtcta cctctggcgg cacagccgct 420
ctgggctgcc tcgtgaagga ctacttcccc gagcctgtga cagtgtcctg gaactctggc 480
gccctgacat ccggcgtgca cacctttcca gctgtgctgc agtcctccgg cctgtactcc 540
ctgtcctccg tcgtgacagt gccctccagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agccctccaa caccaaggtg gacaagaagg tggaacccaa gtcctgcgac 660
aagacccaca cctgtccccc ttgtcctgcc cctgaagctg ctggcggccc tagcgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatctccc ggacccccga agtgacctgc 780
gtggtggtgg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagcct agagaggaac agtacaactc cacctaccgg 900
gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga gtacaagtgc 960
aaggtgtcca acaaggccct gggagccccc atcgaaaaga ccatctccaa ggccaagggc 1020
cagcctcgcg agcctcaggt gtacaccctg ccccctagca gagatgagct gaccaagaac 1080
caggtgtccc tgacctgtct cgtgaaaggc ttctacccct ccgatatcgc cgtggaatgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc cccctgtgct ggactccgac 1200
ggctcattct tcctgtactc taagctgaca gtggacaagt cccggtggca gcagggcaac 1260
gtgttctcct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagtccctg 1320
tccctgtctc ccgggggagg cggaggatct ggcggaggcg gatccggtgg tggcggatct 1380
gggggcggtg gatctgaggt gcagctgctg gaatctgggg gaggactggt gcagccaggc 1440
ggatctctga ggctgtcctg cgctgcttcc ggctttacct tctccagcca cgccatgagt 1500
tgggtgcgcc aggcacccgg aaaaggactg gaatgggtgt cagccatctg ggcctccggc 1560
gagcagtact acgccgatag cgtgaagggc cggttcacca tctctcggga taacagcaag 1620
aatactctgt acctgcagat gaactccctg cgcgctgaag ataccgctgt gtattactgc 1680
gccaagggct ggctgggcaa cttcgattac tggggccagg gaaccctcgt gactgtctcg 1740
agcgcttctg tggccgctcc ctccgtgttc atcttcccac cttccgacga gcagctgaag 1800
tccggcactg cctctgtcgt gtgcctgctg aacaacttct accctcggga agccaaggtg 1860
cagtggaaag tggataacgc cctgcagtcc ggcaactccc aggaatccgt gaccgagcag 1920
gactccaagg acagcaccta ctccctgagc agcaccctga ccctgtccaa ggccgactac 1980
gagaagcaca aggtgtacgc ctgtgaagtg acccaccagg gcctgtccag ccccgtgacc 2040
aagtccttca accggggcga gtgc 2064
<210> 221
<211> 688
<212> PRT
<213> Artificial Sequence
<220>
<223> (1G4) VHCH1-Heavy chain-(28H1) VHCL
<400> 221
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Ser Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
450 455 460
Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
465 470 475 480
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser
485 490 495
His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
500 505 510
Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
530 535 540
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
545 550 555 560
Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu
565 570 575
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
580 585 590
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
595 600 605
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
610 615 620
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
625 630 635 640
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
645 650 655
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
660 665 670
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
675 680 685
<210> 222
<211> 2082
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (20B7) VHCH1-Heavy chain-(28H1) VHCL
<400> 222
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagttaac 300
tacccgtact cttactgggg tgacttcgac tactggggcc aagggaccac cgtgaccgtc 360
tcctcagcta gcaccaaggg cccatccgtg ttccctctgg ccccttccag caagtctacc 420
tctggcggca cagccgctct gggctgcctc gtgaaggact acttccccga gcctgtgaca 480
gtgtcctgga actctggcgc cctgacatcc ggcgtgcaca cctttccagc tgtgctgcag 540
tcctccggcc tgtactccct gtcctccgtc gtgacagtgc cctccagctc tctgggcacc 600
cagacctaca tctgcaacgt gaaccacaag ccctccaaca ccaaggtgga caagaaggtg 660
gaacccaagt cctgcgacaa gacccacacc tgtccccctt gtcctgcccc tgaagctgct 720
ggcggcccta gcgtgttcct gttcccccca aagcccaagg acaccctgat gatctcccgg 780
acccccgaag tgacctgcgt ggtggtggat gtgtcccacg aggaccctga agtgaagttc 840
aattggtacg tggacggcgt ggaagtgcac aatgccaaga ccaagcctag agaggaacag 900
tacaactcca cctaccgggt ggtgtccgtg ctgaccgtgc tgcaccagga ttggctgaac 960
ggcaaagagt acaagtgcaa ggtgtccaac aaggccctgg gagcccccat cgaaaagacc 1020
atctccaagg ccaagggcca gcctcgcgag cctcaggtgt acaccctgcc ccctagcaga 1080
gatgagctga ccaagaacca ggtgtccctg acctgtctcg tgaaaggctt ctacccctcc 1140
gatatcgccg tggaatggga gagcaacggc cagcccgaga acaactacaa gaccaccccc 1200
cctgtgctgg actccgacgg ctcattcttc ctgtactcta agctgacagt ggacaagtcc 1260
cggtggcagc agggcaacgt gttctcctgc tccgtgatgc acgaggccct gcacaaccac 1320
tacacccaga agtccctgtc cctgtctccc gggggaggcg gaggatctgg cggaggcgga 1380
tccggtggtg gcggatctgg gggcggtgga tctgaggtgc agctgctgga atctggggga 1440
ggactggtgc agccaggcgg atctctgagg ctgtcctgcg ctgcttccgg ctttaccttc 1500
tccagccacg ccatgagttg ggtgcgccag gcacccggaa aaggactgga atgggtgtca 1560
gccatctggg cctccggcga gcagtactac gccgatagcg tgaagggccg gttcaccatc 1620
tctcgggata acagcaagaa tactctgtac ctgcagatga actccctgcg cgctgaagat 1680
accgctgtgt attactgcgc caagggctgg ctgggcaact tcgattactg gggccaggga 1740
accctcgtga ctgtctcgag cgcttctgtg gccgctccct ccgtgttcat cttcccacct 1800
tccgacgagc agctgaagtc cggcactgcc tctgtcgtgt gcctgctgaa caacttctac 1860
cctcgggaag ccaaggtgca gtggaaagtg gataacgccc tgcagtccgg caactcccag 1920
gaatccgtga ccgagcagga ctccaaggac agcacctact ccctgagcag caccctgacc 1980
ctgtccaagg ccgactacga gaagcacaag gtgtacgcct gtgaagtgac ccaccagggc 2040
ctgtccagcc ccgtgaccaa gtccttcaac cggggcgagt gc 2082
<210> 223
<211> 694
<212> PRT
<213> Artificial Sequence
<220>
<223> (20B7) VHCH1-Heavy chain-(28H1) VHCL
<400> 223
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Asn Tyr Pro Tyr Ser Tyr Trp Gly Asp Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
450 455 460
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
465 470 475 480
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
485 490 495
Gly Phe Thr Phe Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro
500 505 510
Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln
515 520 525
Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
530 535 540
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
545 550 555 560
Thr Ala Val Tyr Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr
565 570 575
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala
580 585 590
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
595 600 605
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
610 615 620
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
625 630 635 640
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
645 650 655
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
660 665 670
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
675 680 685
Phe Asn Arg Gly Glu Cys
690
<210> 224
<211> 2064
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-563) VHCH1-Heavy chain-(28H1) VHCL
<400> 224
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcttgacgtt 300
ggtgctttcg actactgggg ccaaggagcc ctggtcaccg tctcgagtgc tagcaccaag 360
ggcccatccg tgttccctct ggccccttcc agcaagtcta cctctggcgg cacagccgct 420
ctgggctgcc tcgtgaagga ctacttcccc gagcctgtga cagtgtcctg gaactctggc 480
gccctgacat ccggcgtgca cacctttcca gctgtgctgc agtcctccgg cctgtactcc 540
ctgtcctccg tcgtgacagt gccctccagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agccctccaa caccaaggtg gacaagaagg tggaacccaa gtcctgcgac 660
aagacccaca cctgtccccc ttgtcctgcc cctgaagctg ctggcggccc tagcgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatctccc ggacccccga agtgacctgc 780
gtggtggtgg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagcct agagaggaac agtacaactc cacctaccgg 900
gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga gtacaagtgc 960
aaggtgtcca acaaggccct gggagccccc atcgaaaaga ccatctccaa ggccaagggc 1020
cagcctcgcg agcctcaggt gtacaccctg ccccctagca gagatgagct gaccaagaac 1080
caggtgtccc tgacctgtct cgtgaaaggc ttctacccct ccgatatcgc cgtggaatgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc cccctgtgct ggactccgac 1200
ggctcattct tcctgtactc taagctgaca gtggacaagt cccggtggca gcagggcaac 1260
gtgttctcct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagtccctg 1320
tccctgtctc ccgggggagg cggaggatct ggcggaggcg gatccggtgg tggcggatct 1380
gggggcggtg gatctgaggt gcagctgctg gaatctgggg gaggactggt gcagccaggc 1440
ggatctctga ggctgtcctg cgctgcttcc ggctttacct tctccagcca cgccatgagt 1500
tgggtgcgcc aggcacccgg aaaaggactg gaatgggtgt cagccatctg ggcctccggc 1560
gagcagtact acgccgatag cgtgaagggc cggttcacca tctctcggga taacagcaag 1620
aatactctgt acctgcagat gaactccctg cgcgctgaag ataccgctgt gtattactgc 1680
gccaagggct ggctgggcaa cttcgattac tggggccagg gaaccctcgt gactgtctcg 1740
agcgcttctg tggccgctcc ctccgtgttc atcttcccac cttccgacga gcagctgaag 1800
tccggcactg cctctgtcgt gtgcctgctg aacaacttct accctcggga agccaaggtg 1860
cagtggaaag tggataacgc cctgcagtcc ggcaactccc aggaatccgt gaccgagcag 1920
gactccaagg acagcaccta ctccctgagc agcaccctga ccctgtccaa ggccgactac 1980
gagaagcaca aggtgtacgc ctgtgaagtg acccaccagg gcctgtccag ccccgtgacc 2040
aagtccttca accggggcga gtgc 2064
<210> 225
<211> 688
<212> PRT
<213> Artificial Sequence
<220>
<223> (CLC-563) VHCH1-Heavy chain-(28H1) VHCL
<400> 225
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Asp Val Gly Ala Phe Asp Tyr Trp Gly Gln Gly Ala Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
450 455 460
Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
465 470 475 480
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser
485 490 495
His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
500 505 510
Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
530 535 540
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
545 550 555 560
Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu
565 570 575
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
580 585 590
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
595 600 605
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
610 615 620
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
625 630 635 640
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
645 650 655
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
660 665 670
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
675 680 685
<210> 226
<211> 2064
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-564) VHCH1-Heavy chain-(28H1) VHCL
<400> 226
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gttcgacgtt 300
ggtccgttcg actactgggg ccaaggaacc ctggtcaccg tctcgagtgc tagcaccaag 360
ggcccatccg tgttccctct ggccccttcc agcaagtcta cctctggcgg cacagccgct 420
ctgggctgcc tcgtgaagga ctacttcccc gagcctgtga cagtgtcctg gaactctggc 480
gccctgacat ccggcgtgca cacctttcca gctgtgctgc agtcctccgg cctgtactcc 540
ctgtcctccg tcgtgacagt gccctccagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agccctccaa caccaaggtg gacaagaagg tggaacccaa gtcctgcgac 660
aagacccaca cctgtccccc ttgtcctgcc cctgaagctg ctggcggccc tagcgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatctccc ggacccccga agtgacctgc 780
gtggtggtgg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagcct agagaggaac agtacaactc cacctaccgg 900
gtggtgtccg tgctgaccgt gctgcaccag gattggctga acggcaaaga gtacaagtgc 960
aaggtgtcca acaaggccct gggagccccc atcgaaaaga ccatctccaa ggccaagggc 1020
cagcctcgcg agcctcaggt gtacaccctg ccccctagca gagatgagct gaccaagaac 1080
caggtgtccc tgacctgtct cgtgaaaggc ttctacccct ccgatatcgc cgtggaatgg 1140
gagagcaacg gccagcccga gaacaactac aagaccaccc cccctgtgct ggactccgac 1200
ggctcattct tcctgtactc taagctgaca gtggacaagt cccggtggca gcagggcaac 1260
gtgttctcct gctccgtgat gcacgaggcc ctgcacaacc actacaccca gaagtccctg 1320
tccctgtctc ccgggggagg cggaggatct ggcggaggcg gatccggtgg tggcggatct 1380
gggggcggtg gatctgaggt gcagctgctg gaatctgggg gaggactggt gcagccaggc 1440
ggatctctga ggctgtcctg cgctgcttcc ggctttacct tctccagcca cgccatgagt 1500
tgggtgcgcc aggcacccgg aaaaggactg gaatgggtgt cagccatctg ggcctccggc 1560
gagcagtact acgccgatag cgtgaagggc cggttcacca tctctcggga taacagcaag 1620
aatactctgt acctgcagat gaactccctg cgcgctgaag ataccgctgt gtattactgc 1680
gccaagggct ggctgggcaa cttcgattac tggggccagg gaaccctcgt gactgtctcg 1740
agcgcttctg tggccgctcc ctccgtgttc atcttcccac cttccgacga gcagctgaag 1800
tccggcactg cctctgtcgt gtgcctgctg aacaacttct accctcggga agccaaggtg 1860
cagtggaaag tggataacgc cctgcagtcc ggcaactccc aggaatccgt gaccgagcag 1920
gactccaagg acagcaccta ctccctgagc agcaccctga ccctgtccaa ggccgactac 1980
gagaagcaca aggtgtacgc ctgtgaagtg acccaccagg gcctgtccag ccccgtgacc 2040
aagtccttca accggggcga gtgc 2064
<210> 227
<211> 688
<212> PRT
<213> Artificial Sequence
<220>
<223> (CLC-564) VHCH1-Heavy chain-(28H1) VHCL
<400> 227
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Phe Asp Val Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
450 455 460
Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
465 470 475 480
Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser
485 490 495
His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
500 505 510
Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val
515 520 525
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
530 535 540
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
545 550 555 560
Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu
565 570 575
Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe
580 585 590
Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys
595 600 605
Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val
610 615 620
Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln
625 630 635 640
Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser
645 650 655
Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His
660 665 670
Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
675 680 685
<210> 228
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (28H1) VHCL-heavy chain hole
<400> 228
gaagtgcagc tgctggaatc cggcggaggc ctggtgcagc ctggcggatc tctgagactg 60
tcctgcgccg cctccggctt caccttctcc tcccacgcca tgtcctgggt ccgacaggct 120
cctggcaaag gcctggaatg ggtgtccgcc atctgggcct ccggcgagca gtactacgcc 180
gactctgtga agggccggtt caccatctcc cgggacaact ccaagaacac cctgtacctg 240
cagatgaact ccctgcgggc cgaggacacc gccgtgtact actgtgccaa gggctggctg 300
ggcaacttcg actactgggg acagggcacc ctggtcaccg tgtccagcgc tagcgtggcc 360
gctcccagcg tgttcatctt cccacccagc gacgagcagc tgaagtccgg cacagccagc 420
gtggtgtgcc tgctgaacaa cttctacccc cgcgaggcca aggtgcagtg gaaggtggac 480
aacgccctgc agagcggcaa cagccaggaa tccgtgaccg agcaggacag caaggactcc 540
acctacagcc tgagcagcac cctgaccctg agcaaggccg actacgagaa gcacaaggtg 600
tacgcctgcg aagtgaccca ccagggcctg tccagccccg tgaccaagag cttcaaccgg 660
ggcgagtgcg acaagaccca cacctgtccc ccttgccctg cccctgaagc tgctggtggc 720
ccttccgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatcag ccggaccccc 780
gaagtgacct gcgtggtggt cgatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaatgcc aagaccaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtgcaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctctcgtgc gcagtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctcgtg agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 229
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> (28H1) VHCL-heavy chain hole
<400> 229
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe Ile Phe Pro
115 120 125
Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu
130 135 140
Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp
145 150 155 160
Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp
165 170 175
Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys
180 185 190
Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln
195 200 205
Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 230
<211> 1343
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (49B4) VHCH1-heavy chain knob
<400> 230
aggtgcaatt ggtgcagtct ggggctgagg tgaagaagcc tgggtcctcg gtgaaggtct 60
cctgcaaggc ctccggaggc acattcagca gctacgctat aagctgggtg cgacaggccc 120
ctggacaagg gctcgagtgg atgggaggga tcatccctat ctttggtaca gcaaactacg 180
cacagaagtt ccagggcagg gtcaccatta ctgcagacaa atccacgagc acagcctaca 240
tggagctgag cagcctgaga tctgaggaca ccgccgtgta ttactgtgcg agagaatact 300
accgtggtcc gtacgactac tggggccaag ggaccaccgt gaccgtctcc tcagctagca 360
ccaagggccc tagcgtgttc cctctggccc ctagcagcaa gagcacaagt ggaggaacag 420
ccgccctggg ctgcctggtc aaggactact tccccgagcc cgtgaccgtg tcctggaatt 480
ctggcgccct gacaagcggc gtgcacacat ttccagccgt gctgcagagc agcggcctgt 540
actctctgag cagcgtcgtg accgtgccct ctagctctct gggcacccag acctacatct 600
gcaacgtgaa ccacaagccc agcaacacca aagtggacaa gaaggtggaa cccaagagct 660
gcgacaagac ccacacctgt cccccttgcc ctgcccctga agctgctggt ggcccttccg 720
tgttcctgtt ccccccaaag cccaaggaca ccctgatgat cagccggacc cccgaagtga 780
cctgcgtggt ggtcgatgtg tcccacgagg accctgaagt gaagttcaat tggtacgtgg 840
acggcgtgga agtgcacaat gccaagacca agccgcggga ggagcagtac aacagcacgt 900
accgtgtggt cagcgtcctc accgtcctgc accaggactg gctgaatggc aaggagtaca 960
agtgcaaggt ctccaacaaa gccctcggcg cccccatcga gaaaaccatc tccaaagcca 1020
aagggcagcc ccgagaacca caggtgtaca ccctgccccc atgccgggat gagctgacca 1080
agaaccaggt cagcctgtgg tgcctggtca aaggcttcta tcccagcgac atcgccgtgg 1140
agtgggagag caatgggcag ccggagaaca actacaagac cacgcctccc gtgctggact 1200
ccgacggctc cttcttcctc tacagcaagc tcaccgtgga caagagcagg tggcagcagg 1260
ggaacgtctt ctcatgctcc gtgatgcatg aggctctgca caaccactac acgcagaaga 1320
gcctctccct gtctccgggt aaa 1343
<210> 231
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1-heavy chain knob
<400> 231
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 232
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (1G4) VHCH1-heavy chain knob
<400> 232
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttctatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggccctagcg tgttccctct ggcccctagc agcaagagca caagtggagg aacagccgcc 420
ctgggctgcc tggtcaagga ctacttcccc gagcccgtga ccgtgtcctg gaattctggc 480
gccctgacaa gcggcgtgca cacatttcca gccgtgctgc agagcagcgg cctgtactct 540
ctgagcagcg tcgtgaccgt gccctctagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agcccagcaa caccaaagtg gacaagaagg tggaacccaa gagctgcgac 660
aagacccaca cctgtccccc ttgccctgcc cctgaagctg ctggtggccc ttccgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatcagcc ggacccccga agtgacctgc 780
gtggtggtcg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatgcc gggatgagct gaccaagaac 1080
caggtcagcc tgtggtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 233
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> (1G4) VHCH1-heavy chain knob
<400> 233
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Ser Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 234
<211> 1356
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (20B7) VHCH1-heavy chain knob
<400> 234
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagttaac 300
tacccgtact cttactgggg tgacttcgac tactggggcc aagggaccac cgtgaccgtc 360
tcctcagcta gcaccaaggg ccctagcgtg ttccctctgg cccctagcag caagagcaca 420
agtggaggaa cagccgccct gggctgcctg gtcaaggact acttccccga gcccgtgacc 480
gtgtcctgga attctggcgc cctgacaagc ggcgtgcaca catttccagc cgtgctgcag 540
agcagcggcc tgtactctct gagcagcgtc gtgaccgtgc cctctagctc tctgggcacc 600
cagacctaca tctgcaacgt gaaccacaag cccagcaaca ccaaagtgga caagaaggtg 660
gaacccaaga gctgcgacaa gacccacacc tgtccccctt gccctgcccc tgaagctgct 720
ggtggccctt ccgtgttcct gttcccccca aagcccaagg acaccctgat gatcagccgg 780
acccccgaag tgacctgcgt ggtggtcgat gtgtcccacg aggaccctga agtgaagttc 840
aattggtacg tggacggcgt ggaagtgcac aatgccaaga ccaagccgcg ggaggagcag 900
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 960
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcg gcgcccccat cgagaaaacc 1020
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatgccgg 1080
gatgagctga ccaagaacca ggtcagcctg tggtgcctgg tcaaaggctt ctatcccagc 1140
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1200
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 1260
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1320
tacacgcaga agagcctctc cctgtctccg ggtaaa 1356
<210> 235
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<223> (20B7) VHCH1-heavy chain knob
<400> 235
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Asn Tyr Pro Tyr Ser Tyr Trp Gly Asp Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val
355 360 365
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 236
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-563) VHCH1-heavy chain knob
<400> 236
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcttgacgtt 300
ggtgctttcg actactgggg ccaaggagcc ctggtcaccg tctcgagtgc tagcaccaag 360
ggccctagcg tgttccctct ggcccctagc agcaagagca caagtggagg aacagccgcc 420
ctgggctgcc tggtcaagga ctacttcccc gagcccgtga ccgtgtcctg gaattctggc 480
gccctgacaa gcggcgtgca cacatttcca gccgtgctgc agagcagcgg cctgtactct 540
ctgagcagcg tcgtgaccgt gccctctagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agcccagcaa caccaaagtg gacaagaagg tggaacccaa gagctgcgac 660
aagacccaca cctgtccccc ttgccctgcc cctgaagctg ctggtggccc ttccgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatcagcc ggacccccga agtgacctgc 780
gtggtggtcg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatgcc gggatgagct gaccaagaac 1080
caggtcagcc tgtggtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 237
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> (CLC-563) VHCH1-heavy chain knob
<400> 237
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Asp Val Gly Ala Phe Asp Tyr Trp Gly Gln Gly Ala Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 238
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (CLC-564) VHCH1-heavy chain knob
<400> 238
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gttcgacgtt 300
ggtccgttcg actactgggg ccaaggaacc ctggtcaccg tctcgagtgc tagcaccaag 360
ggccctagcg tgttccctct ggcccctagc agcaagagca caagtggagg aacagccgcc 420
ctgggctgcc tggtcaagga ctacttcccc gagcccgtga ccgtgtcctg gaattctggc 480
gccctgacaa gcggcgtgca cacatttcca gccgtgctgc agagcagcgg cctgtactct 540
ctgagcagcg tcgtgaccgt gccctctagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agcccagcaa caccaaagtg gacaagaagg tggaacccaa gagctgcgac 660
aagacccaca cctgtccccc ttgccctgcc cctgaagctg ctggtggccc ttccgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatcagcc ggacccccga agtgacctgc 780
gtggtggtcg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatgcc gggatgagct gaccaagaac 1080
caggtcagcc tgtggtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 239
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> (CLC-564) VHCH1-heavy chain knob
<400> 239
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Phe Asp Val Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 240
<211> 163
<212> PRT
<213> cynomolgus
<400> 240
Leu Gln Asp Leu Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Ser Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Lys Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Ile Ser Gly Tyr His Cys Leu Gly Ala Glu Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Asp
85 90 95
Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr
115 120 125
Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro
130 135 140
Gly Ala Ser Ser Ala Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His
145 150 155 160
Ser Pro Gln
<210> 241
<211> 164
<212> PRT
<213> Mus musculus
<400> 241
Val Gln Asn Ser Cys Asp Asn Cys Gln Pro Gly Thr Phe Cys Arg Lys
1 5 10 15
Tyr Asn Pro Val Cys Lys Ser Cys Pro Pro Ser Thr Phe Ser Ser Ile
20 25 30
Gly Gly Gln Pro Asn Cys Asn Ile Cys Arg Val Cys Ala Gly Tyr Phe
35 40 45
Arg Phe Lys Lys Phe Cys Ser Ser Thr His Asn Ala Glu Cys Glu Cys
50 55 60
Ile Glu Gly Phe His Cys Leu Gly Pro Gln Cys Thr Arg Cys Glu Lys
65 70 75 80
Asp Cys Arg Pro Gly Gln Glu Leu Thr Lys Gln Gly Cys Lys Thr Cys
85 90 95
Ser Leu Gly Thr Phe Asn Asp Gln Asn Gly Thr Gly Val Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Arg Ser Val Leu Lys Thr Gly Thr
115 120 125
Thr Glu Lys Asp Val Val Cys Gly Pro Pro Val Val Ser Phe Ser Pro
130 135 140
Ser Thr Thr Ile Ser Val Thr Pro Glu Gly Gly Pro Gly Gly His Ser
145 150 155 160
Leu Gln Val Leu
<210> 242
<211> 1266
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA human 4-1BB antigen Fc knob chain
<400> 242
ctgcaggacc cctgcagcaa ctgccctgcc ggcaccttct gcgacaacaa ccggaaccag 60
atctgcagcc cctgcccccc caacagcttc agctctgccg gcggacagcg gacctgcgac 120
atctgcagac agtgcaaggg cgtgttcaga acccggaaag agtgcagcag caccagcaac 180
gccgagtgcg actgcacccc cggcttccat tgtctgggag ccggctgcag catgtgcgag 240
caggactgca agcagggcca ggaactgacc aagaagggct gcaaggactg ctgcttcggc 300
accttcaacg accagaagcg gggcatctgc cggccctgga ccaactgtag cctggacggc 360
aagagcgtgc tggtcaacgg caccaaagaa cgggacgtcg tgtgcggccc cagccctgct 420
gatctgtctc ctggggccag cagcgtgacc cctcctgccc ctgccagaga gcctggccac 480
tctcctcagg tcgacgaaca gttatatttt cagggcggct cacccaaatc tgcagacaaa 540
actcacacat gcccaccgtg cccagcacct gaactcctgg ggggaccgtc agtcttcctc 600
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 660
gtggtggacg tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 720
gaggtgcata atgccaagac aaagccgcgg gaggagcagt acaacagcac gtaccgtgtg 780
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 840
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaagggcag 900
ccccgagaac cacaggtgta caccctgccc ccatgccggg atgagctgac caagaaccag 960
gtcagcctgt ggtgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 1020
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1080
tccttcttcc tctacagcaa gctcaccgtg gacaagagca ggtggcagca ggggaacgtc 1140
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacgcagaa gagcctctcc 1200
ctgtctccgg gtaaatccgg aggcctgaac gacatcttcg aggcccagaa gattgaatgg 1260
cacgag 1266
<210> 243
<211> 1266
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA cynomolgus 4-1BB antigen Fc knob chain
<400> 243
ttgcaggatc tgtgtagtaa ctgcccagct ggtacattct gtgataataa caggagtcag 60
atttgcagtc cctgtcctcc aaatagtttc tccagcgcag gtggacaaag gacctgtgac 120
atatgcaggc agtgtaaagg tgttttcaag accaggaagg agtgttcctc caccagcaat 180
gcagagtgtg actgcatttc agggtatcac tgcctggggg cagagtgcag catgtgtgaa 240
caggattgta aacaaggtca agaattgaca aaaaaaggtt gtaaagactg ttgctttggg 300
acatttaatg accagaaacg tggcatctgt cgcccctgga caaactgttc tttggatgga 360
aagtctgtgc ttgtgaatgg gacgaaggag agggacgtgg tctgcggacc atctccagcc 420
gacctctctc caggagcatc ctctgcgacc ccgcctgccc ctgcgagaga gccaggacac 480
tctccgcagg tcgacgaaca gttatatttt cagggcggct cacccaaatc tgcagacaaa 540
actcacacat gcccaccgtg cccagcacct gaactcctgg ggggaccgtc agtcttcctc 600
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacatgcgtg 660
gtggtggacg tgagccacga agaccctgag gtcaagttca actggtacgt ggacggcgtg 720
gaggtgcata atgccaagac aaagccgcgg gaggagcagt acaacagcac gtaccgtgtg 780
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaatg gcaaggagta caagtgcaag 840
gtctccaaca aagccctccc agcccccatc gagaaaacca tctccaaagc caaagggcag 900
ccccgagaac cacaggtgta caccctgccc ccatgccggg atgagctgac caagaaccag 960
gtcagcctgt ggtgcctggt caaaggcttc tatcccagcg acatcgccgt ggagtgggag 1020
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1080
tccttcttcc tctacagcaa gctcaccgtg gacaagagca ggtggcagca ggggaacgtc 1140
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacgcagaa gagcctctcc 1200
ctgtctccgg gtaaatccgg aggcctgaac gacatcttcg aggcccagaa gattgaatgg 1260
cacgag 1266
<210> 244
<211> 1269
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA murine 4-1BB antigen Fc knob chain
<400> 244
gtgcagaaca gctgcgacaa ctgccagccc ggcaccttct gccggaagta caaccccgtg 60
tgcaagagct gcccccccag caccttcagc agcatcggcg gccagcccaa ctgcaacatc 120
tgcagagtgt gcgccggcta cttccggttc aagaagttct gcagcagcac ccacaacgcc 180
gagtgcgagt gcatcgaggg cttccactgc ctgggccccc agtgcaccag atgcgagaag 240
gactgcagac ccggccagga actgaccaag cagggctgta agacctgcag cctgggcacc 300
ttcaacgacc agaacgggac cggcgtgtgc cggccttgga ccaattgcag cctggacggg 360
agaagcgtgc tgaaaaccgg caccaccgag aaggacgtcg tgtgcggccc tcccgtggtg 420
tccttcagcc ctagcaccac catcagcgtg acccctgaag gcggccctgg cggacactct 480
ctgcaggtcc tggtcgacga acagttatat tttcagggcg gctcacccaa atctgcagac 540
aaaactcaca catgcccacc gtgcccagca cctgaactcc tggggggacc gtcagtcttc 600
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 660
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 720
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 780
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 840
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggg 900
cagccccgag aaccacaggt gtacaccctg cccccatgcc gggatgagct gaccaagaac 960
caggtcagcc tgtggtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1020
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1080
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1140
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1200
tccctgtctc cgggtaaatc cggaggcctg aacgacatct tcgaggccca gaagattgaa 1260
tggcacgag 1269
<210> 245
<211> 422
<212> PRT
<213> Artificial Sequence
<220>
<223> human 4-1BB antigen Fc knob chain
<400> 245
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Asp
85 90 95
Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr
115 120 125
Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro
130 135 140
Gly Ala Ser Ser Val Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His
145 150 155 160
Ser Pro Gln Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro Lys
165 170 175
Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
180 185 190
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
195 200 205
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
210 215 220
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
225 230 235 240
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
245 250 255
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
260 265 270
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
275 280 285
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
290 295 300
Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln
305 310 315 320
Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
325 330 335
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
340 345 350
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
355 360 365
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
370 375 380
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
385 390 395 400
Leu Ser Pro Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln
405 410 415
Lys Ile Glu Trp His Glu
420
<210> 246
<211> 422
<212> PRT
<213> Artificial Sequence
<220>
<223> cynomolgus 4-1BB antigen
Fc knob chain
<400> 246
Leu Gln Asp Leu Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Ser Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Lys Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Ile Ser Gly Tyr His Cys Leu Gly Ala Glu Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Asp
85 90 95
Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr
115 120 125
Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro
130 135 140
Gly Ala Ser Ser Ala Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His
145 150 155 160
Ser Pro Gln Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro Lys
165 170 175
Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
180 185 190
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
195 200 205
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
210 215 220
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
225 230 235 240
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
245 250 255
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
260 265 270
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
275 280 285
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
290 295 300
Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln
305 310 315 320
Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
325 330 335
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
340 345 350
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
355 360 365
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
370 375 380
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
385 390 395 400
Leu Ser Pro Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln
405 410 415
Lys Ile Glu Trp His Glu
420
<210> 247
<211> 423
<212> PRT
<213> Artificial Sequence
<220>
<223> murine 4-1BB antigen Fc knob chain
<400> 247
Val Gln Asn Ser Cys Asp Asn Cys Gln Pro Gly Thr Phe Cys Arg Lys
1 5 10 15
Tyr Asn Pro Val Cys Lys Ser Cys Pro Pro Ser Thr Phe Ser Ser Ile
20 25 30
Gly Gly Gln Pro Asn Cys Asn Ile Cys Arg Val Cys Ala Gly Tyr Phe
35 40 45
Arg Phe Lys Lys Phe Cys Ser Ser Thr His Asn Ala Glu Cys Glu Cys
50 55 60
Ile Glu Gly Phe His Cys Leu Gly Pro Gln Cys Thr Arg Cys Glu Lys
65 70 75 80
Asp Cys Arg Pro Gly Gln Glu Leu Thr Lys Gln Gly Cys Lys Thr Cys
85 90 95
Ser Leu Gly Thr Phe Asn Asp Gln Asn Gly Thr Gly Val Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Arg Ser Val Leu Lys Thr Gly Thr
115 120 125
Thr Glu Lys Asp Val Val Cys Gly Pro Pro Val Val Ser Phe Ser Pro
130 135 140
Ser Thr Thr Ile Ser Val Thr Pro Glu Gly Gly Pro Gly Gly His Ser
145 150 155 160
Leu Gln Val Leu Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro
165 170 175
Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
180 185 190
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
195 200 205
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
210 215 220
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
225 230 235 240
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
245 250 255
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
260 265 270
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
275 280 285
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
290 295 300
Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn
305 310 315 320
Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
325 330 335
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
340 345 350
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
355 360 365
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
370 375 380
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
385 390 395 400
Ser Leu Ser Pro Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala
405 410 415
Gln Lys Ile Glu Trp His Glu
420
<210> 248
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Primer MS63
<400> 248
tttcgcacag taatatacgg ccgtgtcc 28
<210> 249
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(12B3) VL
<400> 249
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatcattcgt atccgcagac gtttggccag 300
ggcaccaaag tcgagatcaa g 321
<210> 250
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(12B3) VH
<400> 250
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
<210> 251
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(25G7) VL
<400> 251
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactccctt gataggcgcg gtatgtgggt attcggcgga 300
gggaccaagc tgaccgtc 318
<210> 252
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(25G7) VH
<400> 252
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagt 354
<210> 253
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(11D5) VL
<400> 253
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag cttaattcgt atcctcagac gtttggccag 300
ggcaccaaag tcgagatcaa g 321
<210> 254
<211> 357
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(11D5) VH
<400> 254
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctca 357
<210> 255
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(9B11) VL
<400> 255
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag gttaattctt atccgcagac gtttggccag 300
ggcaccaaag tcgagatcaa g 321
<210> 256
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(9B11) VH
<400> 256
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
<210> 257
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(20G2) VL
<400> 257
gacatccaga tgacccagtc tccatccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag cagcactcgt attatacgtt tggccagggc 300
accaaagtcg agatcaag 318
<210> 258
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 4-1BB(20G2) VH
<400> 258
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttac 300
tactgggaat cttacccgtt cgactactgg ggccaaggga ccaccgtgac cgtctccagc 360
<210> 259
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 12B3 P329GLALA IgG1 light chain
<400> 259
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag tatcattcgt atccgcagac gtttggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 260
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 12B3 P329GLALA IgG1 heavy chain
<400> 260
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 261
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> 12B3 P329GLALA IgG1 light chain
<400> 261
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr His Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 262
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> 12B3 P329GLALA IgG1 heavy chain
<400> 262
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 263
<211> 639
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 25G7 P329GLALA IgG1 light chain
<400> 263
tcgtctgagc tgactcagga ccctgctgtg tctgtggcct tgggacagac agtcaggatc 60
acatgccaag gagacagcct cagaagttat tatgcaagct ggtaccagca gaagccagga 120
caggcccctg tacttgtcat ctatggtaaa aacaaccggc cctcagggat cccagaccga 180
ttctctggct ccagctcagg aaacacagct tccttgacca tcactggggc tcaggcggaa 240
gatgaggctg actattactg taactccctt gataggcgcg gtatgtgggt attcggcgga 300
gggaccaagc tgaccgtcct aggtcaaccc aaggctgccc ccagcgtgac cctgttcccc 360
cccagcagcg aggaactgca ggccaacaag gccaccctgg tctgcctgat cagcgacttc 420
tacccaggcg ccgtgaccgt ggcctggaag gccgacagca gccccgtgaa ggccggcgtg 480
gagaccacca cccccagcaa gcagagcaac aacaagtacg ccgccagcag ctacctgagc 540
ctgacccccg agcagtggaa gagccacagg tcctacagct gccaggtgac ccacgagggc 600
agcaccgtgg agaaaaccgt ggcccccacc gagtgcagc 639
<210> 264
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 25G7 P329GLALA IgG1 heavy chain
<400> 264
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagtgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg taaa 1344
<210> 265
<211> 213
<212> PRT
<213> Artificial Sequence
<220>
<223> 25G7 P329GLALA IgG1 light chain
<400> 265
Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Leu Asp Arg Arg Gly Met Trp
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln Pro Lys Ala
100 105 110
Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu Leu Gln Ala
115 120 125
Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr Pro Gly Ala
130 135 140
Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys Ala Gly Val
145 150 155 160
Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr Ala Ala Ser
165 170 175
Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His Arg Ser Tyr
180 185 190
Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys Thr Val Ala
195 200 205
Pro Thr Glu Cys Ser
210
<210> 266
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> 25G7 P329GLALA IgG1 heavy chain
<400> 266
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 267
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 11D5 P329GLALA IgG1 light chain
<400> 267
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag cttaattcgt atcctcagac gtttggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 268
<211> 1347
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 11D5 P329GLALA IgG1 heavy chain
<400> 268
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctcagct 360
agcaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagaaagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagctgc agggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatcccg ggatgagctg 1080
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtaaa 1347
<210> 269
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> 11D5 P329GLALA IgG1 light chain
<400> 269
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Leu Asn Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 270
<211> 449
<212> PRT
<213> Artificial Sequence
<220>
<223> 11D5 P329GLALA IgG1 heavy chain
<400> 270
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 271
<211> 642
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 9B11 P329GLALA IgG1 light chain
<400> 271
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag gttaattctt atccgcagac gtttggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 642
<210> 272
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 9B11 P329GLALA IgG1 heavy chain
<400> 272
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 273
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> 9B11 P329GLALA IgG1 light chain
<400> 273
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Val Asn Ser Tyr Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 274
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> 9B11 P329GLALA IgG1 heavy chain
<400> 274
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 275
<211> 639
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 20G2 P329GLALA IgG1 light chain
<400> 275
gacatccaga tgacccagtc tccatccacc ctgtctgcat ctgtaggaga ccgtgtcacc 60
atcacttgcc gtgccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgat gcctccagtt tggaaagtgg ggtcccatca 180
cgtttcagcg gcagtggatc cgggacagaa ttcactctca ccatcagcag cttgcagcct 240
gatgattttg caacttatta ctgccaacag cagcactcgt attatacgtt tggccagggc 300
accaaagtcg agatcaagcg tacggtggct gcaccatctg tcttcatctt cccgccatct 360
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 420
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 480
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 540
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 600
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 639
<210> 276
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA 20G2 P329GLALA IgG1 heavy chain
<400> 276
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttac 300
tactgggaat cttacccgtt cgactactgg ggccaaggga ccaccgtgac cgtctccagc 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 277
<211> 213
<212> PRT
<213> Artificial Sequence
<220>
<223> 20G2 P329GLALA IgG1 light chain
<400> 277
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Ser Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Gln His Ser Tyr Tyr Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala Pro
100 105 110
Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr
115 120 125
Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys
130 135 140
Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu
145 150 155 160
Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser
165 170 175
Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala
180 185 190
Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe
195 200 205
Asn Arg Gly Glu Cys
210
<210> 278
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> 20G2 P329GLALA IgG1 heavy chain
<400> 278
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Tyr Tyr Trp Glu Ser Tyr Pro Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 279
<211> 421
<212> PRT
<213> Artificial Sequence
<220>
<223> mu4-1BB D1 / hu4-1BB D2 Fc knob
<400> 279
Val Gln Asn Ser Cys Asp Asn Cys Gln Pro Gly Thr Phe Cys Arg Lys
1 5 10 15
Tyr Asn Pro Val Cys Lys Ser Cys Pro Pro Ser Thr Phe Ser Ser Ile
20 25 30
Gly Gly Gln Pro Asn Cys Asn Ile Cys Arg Val Cys Ala Gly Tyr Phe
35 40 45
Arg Phe Lys Lys Phe Cys Ser Ser Thr His Asn Ala Glu Cys Glu Cys
50 55 60
Ile Glu Gly Phe His Cys Leu Gly Pro Gln Cys Thr Arg Cys Glu Lys
65 70 75 80
Asp Cys Arg Pro Gly Gln Glu Leu Thr Lys Gln Gly Cys Lys Asp Cys
85 90 95
Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro Trp
100 105 110
Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr Lys
115 120 125
Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro Gly
130 135 140
Ala Ser Ser Val Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His Ser
145 150 155 160
Pro Gln Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro Lys Ser
165 170 175
Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
180 185 190
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
195 200 205
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
210 215 220
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
225 230 235 240
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
245 250 255
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
260 265 270
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
275 280 285
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
290 295 300
Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val
305 310 315 320
Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
325 330 335
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
340 345 350
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
355 360 365
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
370 375 380
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
385 390 395 400
Ser Pro Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys
405 410 415
Ile Glu Trp His Glu
420
<210> 280
<211> 424
<212> PRT
<213> Artificial Sequence
<220>
<223> hu4-1BB D1 / mu4-1BB D2 Fc knob
<400> 280
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Thr
85 90 95
Cys Ser Leu Gly Thr Phe Asn Asp Gln Asn Gly Thr Gly Val Cys Arg
100 105 110
Pro Trp Thr Asn Cys Ser Leu Asp Gly Arg Ser Val Leu Lys Thr Gly
115 120 125
Thr Thr Glu Lys Asp Val Val Cys Gly Pro Pro Val Val Ser Phe Ser
130 135 140
Pro Ser Thr Thr Ile Ser Val Thr Pro Glu Gly Gly Pro Gly Gly His
145 150 155 160
Ser Leu Gln Val Leu Val Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser
165 170 175
Pro Lys Ser Ala Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
180 185 190
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
195 200 205
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
210 215 220
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
225 230 235 240
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
245 250 255
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
260 265 270
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
275 280 285
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
290 295 300
Glu Pro Gln Val Tyr Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys
305 310 315 320
Asn Gln Val Ser Leu Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
325 330 335
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
340 345 350
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
355 360 365
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
370 375 380
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
385 390 395 400
Leu Ser Leu Ser Pro Gly Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu
405 410 415
Ala Gln Lys Ile Glu Trp His Glu
420
<210> 281
<211> 354
<212> PRT
<213> Artificial Sequence
<220>
<223> hu 4-1BB D1 Fc knob
<400> 281
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Val
85 90 95
Asp Glu Gln Leu Tyr Phe Gln Gly Gly Ser Pro Lys Ser Ala Asp Lys
100 105 110
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
115 120 125
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
130 135 140
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
145 150 155 160
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
165 170 175
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
180 185 190
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
195 200 205
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
210 215 220
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
225 230 235 240
Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp
245 250 255
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
260 265 270
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
275 280 285
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
290 295 300
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
305 310 315 320
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
325 330 335
Lys Ser Gly Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp
340 345 350
His Glu
<210> 282
<211> 94
<212> PRT
<213> Artificial Sequence
<220>
<223> Murine 4-1BB domain D1 (N-terminus)
<400> 282
Val Gln Asn Ser Cys Asp Asn Cys Gln Pro Gly Thr Phe Cys Arg Lys
1 5 10 15
Tyr Asn Pro Val Cys Lys Ser Cys Pro Pro Ser Thr Phe Ser Ser Ile
20 25 30
Gly Gly Gln Pro Asn Cys Asn Ile Cys Arg Val Cys Ala Gly Tyr Phe
35 40 45
Arg Phe Lys Lys Phe Cys Ser Ser Thr His Asn Ala Glu Cys Glu Cys
50 55 60
Ile Glu Gly Phe His Cys Leu Gly Pro Gln Cys Thr Arg Cys Glu Lys
65 70 75 80
Asp Cys Arg Pro Gly Gln Glu Leu Thr Lys Gln Gly Cys Lys
85 90
<210> 283
<211> 68
<212> PRT
<213> Artificial Sequence
<220>
<223> Human 4-1BB domain D2 (C-terminus)
<400> 283
Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg
1 5 10 15
Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly
20 25 30
Thr Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser
35 40 45
Pro Gly Ala Ser Ser Val Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly
50 55 60
His Ser Pro Gln
65
<210> 284
<211> 95
<212> PRT
<213> Artificial Sequence
<220>
<223> Human 4-1BB domain D1 (N-terminus)
<400> 284
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys
85 90 95
<210> 285
<211> 70
<212> PRT
<213> Artificial Sequence
<220>
<223> Murine 4-1BB domain D2 (C-terminus)
<400> 285
Thr Cys Ser Leu Gly Thr Phe Asn Asp Gln Asn Gly Thr Gly Val Cys
1 5 10 15
Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Arg Ser Val Leu Lys Thr
20 25 30
Gly Thr Thr Glu Lys Asp Val Val Cys Gly Pro Pro Val Val Ser Phe
35 40 45
Ser Pro Ser Thr Thr Ile Ser Val Thr Pro Glu Gly Gly Pro Gly Gly
50 55 60
His Ser Leu Gln Val Leu
65 70
<210> 286
<211> 2076
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1-Heavy chain-(28H1) VHCL
<400> 286
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc cgtgttccct ctggcccctt ccagcaagtc tacctctggc 420
ggcacagccg ctctgggctg cctcgtgaag gactacttcc ccgagcctgt gacagtgtcc 480
tggaactctg gcgccctgac atccggcgtg cacacctttc cagctgtgct gcagtcctcc 540
ggcctgtact ccctgtcctc cgtcgtgaca gtgccctcca gctctctggg cacccagacc 600
tacatctgca acgtgaacca caagccctcc aacaccaagg tggacaagaa ggtggaaccc 660
aagtcctgcg acaagaccca cacctgtccc ccttgtcctg cccctgaagc tgctggcggc 720
cctagcgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatctc ccggaccccc 780
gaagtgacct gcgtggtggt ggatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaatgcc aagaccaagc ctagagagga acagtacaac 900
tccacctacc gggtggtgtc cgtgctgacc gtgctgcacc aggattggct gaacggcaaa 960
gagtacaagt gcaaggtgtc caacaaggcc ctgggagccc ccatcgaaaa gaccatctcc 1020
aaggccaagg gccagcctcg cgagcctcag gtgtacaccc tgccccctag cagagatgag 1080
ctgaccaaga accaggtgtc cctgacctgt ctcgtgaaag gcttctaccc ctccgatatc 1140
gccgtggaat gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggactccg acggctcatt cttcctgtac tctaagctga cagtggacaa gtcccggtgg 1260
cagcagggca acgtgttctc ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgtccctgtc tcccggggga ggcggaggat ctggcggagg cggatccggt 1380
ggtggcggat ctgggggcgg tggatctgag gtgcagctgc tggaatctgg gggaggactg 1440
gtgcagccag gcggatctct gaggctgtcc tgcgctgctt ccggctttac cttctccagc 1500
cacgccatga gttgggtgcg ccaggcaccc ggaaaaggac tggaatgggt gtcagccatc 1560
tgggcctccg gcgagcagta ctacgccgat agcgtgaagg gccggttcac catctctcgg 1620
gataacagca agaatactct gtacctgcag atgaactccc tgcgcgctga agataccgct 1680
gtgtattact gcgccaaggg ctggctgggc aacttcgatt actggggcca gggaaccctc 1740
gtgactgtct cgagcgcttc tgtggccgct ccctccgtgt tcatcttccc accttccgac 1800
gagcagctga agtccggcac tgcctctgtc gtgtgcctgc tgaacaactt ctaccctcgg 1860
gaagccaagg tgcagtggaa agtggataac gccctgcagt ccggcaactc ccaggaatcc 1920
gtgaccgagc aggactccaa ggacagcacc tactccctga gcagcaccct gaccctgtcc 1980
aaggccgact acgagaagca caaggtgtac gcctgtgaag tgacccacca gggcctgtcc 2040
agccccgtga ccaagtcctt caaccggggc gagtgc 2076
<210> 287
<211> 692
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1-Heavy chain-(28H1) VHCL
<400> 287
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr
515 520 525
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
530 535 540
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
545 550 555 560
Val Tyr Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly
565 570 575
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
580 585 590
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
595 600 605
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
610 615 620
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
625 630 635 640
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
645 650 655
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
660 665 670
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
675 680 685
Arg Gly Glu Cys
690
<210> 288
<211> 2070
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1-Heavy chain-(28H1) VHCL
<400> 288
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagtgctagc 360
accaagggcc catccgtgtt ccctctggcc ccttccagca agtctacctc tggcggcaca 420
gccgctctgg gctgcctcgt gaaggactac ttccccgagc ctgtgacagt gtcctggaac 480
tctggcgccc tgacatccgg cgtgcacacc tttccagctg tgctgcagtc ctccggcctg 540
tactccctgt cctccgtcgt gacagtgccc tccagctctc tgggcaccca gacctacatc 600
tgcaacgtga accacaagcc ctccaacacc aaggtggaca agaaggtgga acccaagtcc 660
tgcgacaaga cccacacctg tcccccttgt cctgcccctg aagctgctgg cggccctagc 720
gtgttcctgt tccccccaaa gcccaaggac accctgatga tctcccggac ccccgaagtg 780
acctgcgtgg tggtggatgt gtcccacgag gaccctgaag tgaagttcaa ttggtacgtg 840
gacggcgtgg aagtgcacaa tgccaagacc aagcctagag aggaacagta caactccacc 900
taccgggtgg tgtccgtgct gaccgtgctg caccaggatt ggctgaacgg caaagagtac 960
aagtgcaagg tgtccaacaa ggccctggga gcccccatcg aaaagaccat ctccaaggcc 1020
aagggccagc ctcgcgagcc tcaggtgtac accctgcccc ctagcagaga tgagctgacc 1080
aagaaccagg tgtccctgac ctgtctcgtg aaaggcttct acccctccga tatcgccgtg 1140
gaatgggaga gcaacggcca gcccgagaac aactacaaga ccaccccccc tgtgctggac 1200
tccgacggct cattcttcct gtactctaag ctgacagtgg acaagtcccg gtggcagcag 1260
ggcaacgtgt tctcctgctc cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgtccc tgtctcccgg gggaggcgga ggatctggcg gaggcggatc cggtggtggc 1380
ggatctgggg gcggtggatc tgaggtgcag ctgctggaat ctgggggagg actggtgcag 1440
ccaggcggat ctctgaggct gtcctgcgct gcttccggct ttaccttctc cagccacgcc 1500
atgagttggg tgcgccaggc acccggaaaa ggactggaat gggtgtcagc catctgggcc 1560
tccggcgagc agtactacgc cgatagcgtg aagggccggt tcaccatctc tcgggataac 1620
agcaagaata ctctgtacct gcagatgaac tccctgcgcg ctgaagatac cgctgtgtat 1680
tactgcgcca agggctggct gggcaacttc gattactggg gccagggaac cctcgtgact 1740
gtctcgagcg cttctgtggc cgctccctcc gtgttcatct tcccaccttc cgacgagcag 1800
ctgaagtccg gcactgcctc tgtcgtgtgc ctgctgaaca acttctaccc tcgggaagcc 1860
aaggtgcagt ggaaagtgga taacgccctg cagtccggca actcccagga atccgtgacc 1920
gagcaggact ccaaggacag cacctactcc ctgagcagca ccctgaccct gtccaaggcc 1980
gactacgaga agcacaaggt gtacgcctgt gaagtgaccc accagggcct gtccagcccc 2040
gtgaccaagt ccttcaaccg gggcgagtgc 2070
<210> 289
<211> 690
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1-Heavy chain-(28H1) VHCL
<400> 289
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp
515 520 525
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
530 535 540
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
545 550 555 560
Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly
565 570 575
Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val Phe
580 585 590
Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser Val
595 600 605
Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp
610 615 620
Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val Thr
625 630 635 640
Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr
645 650 655
Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu Val
660 665 670
Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg Gly
675 680 685
Glu Cys
690
<210> 290
<211> 2073
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1-Heavy chain-(28H1) VHCL
<400> 290
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctcagct 360
agcaccaagg gcccatccgt gttccctctg gccccttcca gcaagtctac ctctggcggc 420
acagccgctc tgggctgcct cgtgaaggac tacttccccg agcctgtgac agtgtcctgg 480
aactctggcg ccctgacatc cggcgtgcac acctttccag ctgtgctgca gtcctccggc 540
ctgtactccc tgtcctccgt cgtgacagtg ccctccagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gccctccaac accaaggtgg acaagaaggt ggaacccaag 660
tcctgcgaca agacccacac ctgtccccct tgtcctgccc ctgaagctgc tggcggccct 720
agcgtgttcc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 780
gtgacctgcg tggtggtgga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caatgccaag accaagccta gagaggaaca gtacaactcc 900
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 960
tacaagtgca aggtgtccaa caaggccctg ggagccccca tcgaaaagac catctccaag 1020
gccaagggcc agcctcgcga gcctcaggtg tacaccctgc cccctagcag agatgagctg 1080
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1140
gtggaatggg agagcaacgg ccagcccgag aacaactaca agaccacccc ccctgtgctg 1200
gactccgacg gctcattctt cctgtactct aagctgacag tggacaagtc ccggtggcag 1260
cagggcaacg tgttctcctg ctccgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctgt ccctgtctcc cgggggaggc ggaggatctg gcggaggcgg atccggtggt 1380
ggcggatctg ggggcggtgg atctgaggtg cagctgctgg aatctggggg aggactggtg 1440
cagccaggcg gatctctgag gctgtcctgc gctgcttccg gctttacctt ctccagccac 1500
gccatgagtt gggtgcgcca ggcacccgga aaaggactgg aatgggtgtc agccatctgg 1560
gcctccggcg agcagtacta cgccgatagc gtgaagggcc ggttcaccat ctctcgggat 1620
aacagcaaga atactctgta cctgcagatg aactccctgc gcgctgaaga taccgctgtg 1680
tattactgcg ccaagggctg gctgggcaac ttcgattact ggggccaggg aaccctcgtg 1740
actgtctcga gcgcttctgt ggccgctccc tccgtgttca tcttcccacc ttccgacgag 1800
cagctgaagt ccggcactgc ctctgtcgtg tgcctgctga acaacttcta ccctcgggaa 1860
gccaaggtgc agtggaaagt ggataacgcc ctgcagtccg gcaactccca ggaatccgtg 1920
accgagcagg actccaagga cagcacctac tccctgagca gcaccctgac cctgtccaag 1980
gccgactacg agaagcacaa ggtgtacgcc tgtgaagtga cccaccaggg cctgtccagc 2040
cccgtgacca agtccttcaa ccggggcgag tgc 2073
<210> 291
<211> 691
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1-Heavy chain-(28H1) VHCL
<400> 291
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
450 455 460
Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
465 470 475 480
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
485 490 495
Phe Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
500 505 510
Leu Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala
515 520 525
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
530 535 540
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
545 550 555 560
Tyr Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln
565 570 575
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser Val
580 585 590
Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala Ser
595 600 605
Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val Gln
610 615 620
Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser Val
625 630 635 640
Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr Leu
645 650 655
Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys Glu
660 665 670
Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn Arg
675 680 685
Gly Glu Cys
690
<210> 292
<211> 2076
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1-Heavy chain-(28H1) VHCL
<400> 292
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc cgtgttccct ctggcccctt ccagcaagtc tacctctggc 420
ggcacagccg ctctgggctg cctcgtgaag gactacttcc ccgagcctgt gacagtgtcc 480
tggaactctg gcgccctgac atccggcgtg cacacctttc cagctgtgct gcagtcctcc 540
ggcctgtact ccctgtcctc cgtcgtgaca gtgccctcca gctctctggg cacccagacc 600
tacatctgca acgtgaacca caagccctcc aacaccaagg tggacaagaa ggtggaaccc 660
aagtcctgcg acaagaccca cacctgtccc ccttgtcctg cccctgaagc tgctggcggc 720
cctagcgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatctc ccggaccccc 780
gaagtgacct gcgtggtggt ggatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaatgcc aagaccaagc ctagagagga acagtacaac 900
tccacctacc gggtggtgtc cgtgctgacc gtgctgcacc aggattggct gaacggcaaa 960
gagtacaagt gcaaggtgtc caacaaggcc ctgggagccc ccatcgaaaa gaccatctcc 1020
aaggccaagg gccagcctcg cgagcctcag gtgtacaccc tgccccctag cagagatgag 1080
ctgaccaaga accaggtgtc cctgacctgt ctcgtgaaag gcttctaccc ctccgatatc 1140
gccgtggaat gggagagcaa cggccagccc gagaacaact acaagaccac cccccctgtg 1200
ctggactccg acggctcatt cttcctgtac tctaagctga cagtggacaa gtcccggtgg 1260
cagcagggca acgtgttctc ctgctccgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgtccctgtc tcccggggga ggcggaggat ctggcggagg cggatccggt 1380
ggtggcggat ctgggggcgg tggatctgag gtgcagctgc tggaatctgg gggaggactg 1440
gtgcagccag gcggatctct gaggctgtcc tgcgctgctt ccggctttac cttctccagc 1500
cacgccatga gttgggtgcg ccaggcaccc ggaaaaggac tggaatgggt gtcagccatc 1560
tgggcctccg gcgagcagta ctacgccgat agcgtgaagg gccggttcac catctctcgg 1620
gataacagca agaatactct gtacctgcag atgaactccc tgcgcgctga agataccgct 1680
gtgtattact gcgccaaggg ctggctgggc aacttcgatt actggggcca gggaaccctc 1740
gtgactgtct cgagcgcttc tgtggccgct ccctccgtgt tcatcttccc accttccgac 1800
gagcagctga agtccggcac tgcctctgtc gtgtgcctgc tgaacaactt ctaccctcgg 1860
gaagccaagg tgcagtggaa agtggataac gccctgcagt ccggcaactc ccaggaatcc 1920
gtgaccgagc aggactccaa ggacagcacc tactccctga gcagcaccct gaccctgtcc 1980
aaggccgact acgagaagca caaggtgtac gcctgtgaag tgacccacca gggcctgtcc 2040
agccccgtga ccaagtcctt caaccggggc gagtgc 2076
<210> 293
<211> 692
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1-Heavy chain-(28H1) VHCL
<400> 293
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr
515 520 525
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
530 535 540
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
545 550 555 560
Val Tyr Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly
565 570 575
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Val Ala Ala Pro Ser
580 585 590
Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly Thr Ala
595 600 605
Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala Lys Val
610 615 620
Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln Glu Ser
625 630 635 640
Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser Ser Thr
645 650 655
Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr Ala Cys
660 665 670
Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser Phe Asn
675 680 685
Arg Gly Glu Cys
690
<210> 294
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1-heavy chain knob
<400> 294
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggccctag cgtgttccct ctggccccta gcagcaagag cacaagtgga 420
ggaacagccg ccctgggctg cctggtcaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaattctg gcgccctgac aagcggcgtg cacacatttc cagccgtgct gcagagcagc 540
ggcctgtact ctctgagcag cgtcgtgacc gtgccctcta gctctctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaaag tggacaagaa ggtggaaccc 660
aagagctgcg acaagaccca cacctgtccc ccttgccctg cccctgaagc tgctggtggc 720
ccttccgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatcag ccggaccccc 780
gaagtgacct gcgtggtggt cgatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaatgcc aagaccaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatg ccgggatgag 1080
ctgaccaaga accaggtcag cctgtggtgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 295
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1-heavy chain knob
<400> 295
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 296
<211> 1344
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1-heavy chain knob
<400> 296
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagtgctagc 360
accaagggcc ctagcgtgtt ccctctggcc cctagcagca agagcacaag tggaggaaca 420
gccgccctgg gctgcctggt caaggactac ttccccgagc ccgtgaccgt gtcctggaat 480
tctggcgccc tgacaagcgg cgtgcacaca tttccagccg tgctgcagag cagcggcctg 540
tactctctga gcagcgtcgt gaccgtgccc tctagctctc tgggcaccca gacctacatc 600
tgcaacgtga accacaagcc cagcaacacc aaagtggaca agaaggtgga acccaagagc 660
tgcgacaaga cccacacctg tcccccttgc cctgcccctg aagctgctgg tggcccttcc 720
gtgttcctgt tccccccaaa gcccaaggac accctgatga tcagccggac ccccgaagtg 780
acctgcgtgg tggtcgatgt gtcccacgag gaccctgaag tgaagttcaa ttggtacgtg 840
gacggcgtgg aagtgcacaa tgccaagacc aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc catgccggga tgagctgacc 1080
aagaaccagg tcagcctgtg gtgcctggtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct ctacagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg taaa 1344
<210> 297
<211> 448
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1-heavy chain knob
<400> 297
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 298
<211> 1347
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1-heavy chain knob
<400> 298
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctcagct 360
agcaccaagg gccctagcgt gttccctctg gcccctagca gcaagagcac aagtggagga 420
acagccgccc tgggctgcct ggtcaaggac tacttccccg agcccgtgac cgtgtcctgg 480
aattctggcg ccctgacaag cggcgtgcac acatttccag ccgtgctgca gagcagcggc 540
ctgtactctc tgagcagcgt cgtgaccgtg ccctctagct ctctgggcac ccagacctac 600
atctgcaacg tgaaccacaa gcccagcaac accaaagtgg acaagaaggt ggaacccaag 660
agctgcgaca agacccacac ctgtccccct tgccctgccc ctgaagctgc tggtggccct 720
tccgtgttcc tgttcccccc aaagcccaag gacaccctga tgatcagccg gacccccgaa 780
gtgacctgcg tggtggtcga tgtgtcccac gaggaccctg aagtgaagtt caattggtac 840
gtggacggcg tggaagtgca caatgccaag accaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatgccg ggatgagctg 1080
accaagaacc aggtcagcct gtggtgcctg gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctctacagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtaaa 1347
<210> 299
<211> 449
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1-heavy chain knob
<400> 299
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Lys
<210> 300
<211> 1350
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1-heavy chain knob
<400> 300
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggccctag cgtgttccct ctggccccta gcagcaagag cacaagtgga 420
ggaacagccg ccctgggctg cctggtcaag gactacttcc ccgagcccgt gaccgtgtcc 480
tggaattctg gcgccctgac aagcggcgtg cacacatttc cagccgtgct gcagagcagc 540
ggcctgtact ctctgagcag cgtcgtgacc gtgccctcta gctctctggg cacccagacc 600
tacatctgca acgtgaacca caagcccagc aacaccaaag tggacaagaa ggtggaaccc 660
aagagctgcg acaagaccca cacctgtccc ccttgccctg cccctgaagc tgctggtggc 720
ccttccgtgt tcctgttccc cccaaagccc aaggacaccc tgatgatcag ccggaccccc 780
gaagtgacct gcgtggtggt cgatgtgtcc cacgaggacc ctgaagtgaa gttcaattgg 840
tacgtggacg gcgtggaagt gcacaatgcc aagaccaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatg ccgggatgag 1080
ctgaccaaga accaggtcag cctgtggtgc ctggtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctctac agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtaaa 1350
<210> 301
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1-heavy chain knob
<400> 301
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 302
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (8H9) VHCH1-heavy chain knob
<400> 302
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
ggttggatgg actactgggg ccaagggacc accgtgaccg tctcctcagc tagcaccaag 360
ggccctagcg tgttccctct ggcccctagc agcaagagca caagtggagg aacagccgcc 420
ctgggctgcc tggtcaagga ctacttcccc gagcccgtga ccgtgtcctg gaattctggc 480
gccctgacaa gcggcgtgca cacatttcca gccgtgctgc agagcagcgg cctgtactct 540
ctgagcagcg tcgtgaccgt gccctctagc tctctgggca cccagaccta catctgcaac 600
gtgaaccaca agcccagcaa caccaaagtg gacaagaagg tggaacccaa gagctgcgac 660
aagacccaca cctgtccccc ttgccctgcc cctgaagctg ctggtggccc ttccgtgttc 720
ctgttccccc caaagcccaa ggacaccctg atgatcagcc ggacccccga agtgacctgc 780
gtggtggtcg atgtgtccca cgaggaccct gaagtgaagt tcaattggta cgtggacggc 840
gtggaagtgc acaatgccaa gaccaagccg cgggaggagc agtacaacag cacgtaccgt 900
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 960
aaggtctcca acaaagccct cggcgccccc atcgagaaaa ccatctccaa agccaaaggg 1020
cagccccgag aaccacaggt gtacaccctg cccccatgcc gggatgagct gaccaagaac 1080
caggtcagcc tgtggtgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1140
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1200
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1260
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1320
tccctgtctc cgggtaaa 1338
<210> 303
<211> 446
<212> PRT
<213> Artificial Sequence
<220>
<223> (8H9) VHCH1-heavy chain knob
<400> 303
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Gly Trp Met Asp Tyr Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
115 120 125
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
130 135 140
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
145 150 155 160
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
165 170 175
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
180 185 190
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
195 200 205
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr
210 215 220
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe
225 230 235 240
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
245 250 255
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
260 265 270
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
275 280 285
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
290 295 300
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
305 310 315 320
Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr Ile Ser
325 330 335
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
340 345 350
Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys Leu Val
355 360 365
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
370 375 380
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
385 390 395 400
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
405 410 415
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
420 425 430
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 304
<211> 1752
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of (49B4) VHCH1 Fc knob VH (4B9)
<400> 304
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc cctgcagaga tgagctgacc 1080
aagaaccagg tgtccctgtg gtgtctggtc aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgacggca gcttcttcct gtactccaaa ctgaccgtgg acaagagccg gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg cggaggcggc ggaagcggag gaggaggatc tgggggcgga 1380
ggttccggag gcggaggatc cgaggtgcag ctgctcgaaa gcggcggagg actggtgcag 1440
cctggcggca gcctgagact gtcttgcgcc gccagcggct tcaccttcag cagctacgcc 1500
atgagctggg tccgccaggc ccctggcaag ggactggaat gggtgtccgc catcatcggc 1560
tctggcgcca gcacctacta cgccgacagc gtgaagggcc ggttcaccat cagccgggac 1620
aacagcaaga acaccctgta cctgcagatg aacagcctgc gggccgagga caccgccgtg 1680
tactactgcg ccaagggatg gttcggcggc ttcaactact ggggacaggg caccctggtc 1740
accgtgtcca gc 1752
<210> 305
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of (49B4) VHCH1 Fc hole VL (4B9)
<400> 305
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtgc accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctctc gtgcgcagtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct cgtgagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg tggaggcggc ggaagcggag gaggaggatc cggcggcgga 1380
ggttccggag gcggtggatc tgagatcgtg ctgacccagt ctcccggcac cctgtctctg 1440
agccctggcg agagagccac cctgtcctgc agagcctccc agtccgtgac ctcctcctac 1500
ctcgcctggt atcagcagaa gcccggccag gcccctcggc tgctgatcaa cgtgggcagt 1560
cggagagcca ccggcatccc tgaccggttc tccggctctg gctccggcac cgacttcacc 1620
ctgaccatct cccggctgga acccgaggac ttcgccgtgt actactgcca gcagggcatc 1680
atgctgcccc ccacctttgg ccagggcacc aaggtggaaa tcaag 1725
<210> 306
<211> 584
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc knob VH (4B9)
<400> 306
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala
515 520 525
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
530 535 540
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
545 550 555 560
Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln
565 570 575
Gly Thr Leu Val Thr Val Ser Ser
580
<210> 307
<211> 575
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc hole VL (4B9)
<400> 307
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
465 470 475 480
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
485 490 495
Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
500 505 510
Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp
515 520 525
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
530 535 540
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile
545 550 555 560
Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 308
<211> 1749
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of (49B4) VHCH1 Fc knob VH (28H1)
<400> 308
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc cctgcagaga tgagctgacc 1080
aagaaccagg tgtccctgtg gtgtctggtc aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgacggca gcttcttcct gtactccaaa ctgaccgtgg acaagagccg gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg cggaggcggc ggaagcggag gaggaggatc cggaggaggg 1380
ggaagtggcg gcggaggatc tgaggtgcag ctgctggaat ccggcggagg cctggtgcag 1440
cctggcggat ctctgagact gtcctgcgcc gcctccggct tcaccttctc ctcccacgcc 1500
atgtcctggg tccgacaggc tcctggcaaa ggcctggaat gggtgtccgc catctgggcc 1560
tccggcgagc agtactacgc cgactctgtg aagggccggt tcaccatctc ccgggacaac 1620
tccaagaaca ccctgtacct gcagatgaac tccctgcggg ccgaggacac cgccgtgtac 1680
tactgtgcca agggctggct gggcaacttc gactactggg gccagggcac cctggtcacc 1740
gtgtccagc 1749
<210> 309
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> nuclelotide sequence of (49B4) VHCH1 Fc hole VL (28H1)
<400> 309
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtgc accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctctc gtgcgcagtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct cgtgagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg tggaggcggc ggaagcggag gaggaggatc cggtggtggc 1380
ggatctgggg gcggtggatc tgagatcgtg ctgacccagt ctcccggcac cctgagcctg 1440
agccctggcg agagagccac cctgagctgc agagccagcc agagcgtgag ccggagctac 1500
ctggcctggt atcagcagaa gcccggccag gcccccagac tgctgatcat cggcgccagc 1560
acccgggcca ccggcatccc cgatagattc agcggcagcg gctccggcac cgacttcacc 1620
ctgaccatca gccggctgga acccgaggac ttcgccgtgt actactgcca gcagggccag 1680
gtgatccccc ccaccttcgg ccagggcacc aaggtggaaa tcaag 1725
<210> 310
<211> 583
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc knob VH (28H1)
<400> 310
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser His Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Trp Ala Ser Gly Glu Gln Tyr Tyr Ala Asp
515 520 525
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr
530 535 540
Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr
545 550 555 560
Tyr Cys Ala Lys Gly Trp Leu Gly Asn Phe Asp Tyr Trp Gly Gln Gly
565 570 575
Thr Leu Val Thr Val Ser Ser
580
<210> 311
<211> 575
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc hole VL (28H1)
<400> 311
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
465 470 475 480
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
485 490 495
Ser Arg Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
500 505 510
Arg Leu Leu Ile Ile Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Asp
515 520 525
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
530 535 540
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Gln
545 550 555 560
Val Ile Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 312
<211> 1746
<212> DNA
<213> Artificial Sequence
<220>
<223> nucleotide sequence of (49B4) VHCH1 Fc knob VH (DP47)
<400> 312
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc cctgcagaga tgagctgacc 1080
aagaaccagg tgtccctgtg gtgtctggtc aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgacggca gcttcttcct gtactccaaa ctgaccgtgg acaagagccg gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg cggaggcggc ggaagcggag gaggaggatc cggaggaggg 1380
ggaagtggcg gcggaggatc tgaggtgcaa ttgttggagt ctgggggagg cttggtacag 1440
cctggggggt ccctgagact ctcctgtgca gccagcggat tcacctttag cagttatgcc 1500
atgagctggg tccgccaggc tccagggaag gggctggagt gggtctcagc tattagtggt 1560
agtggtggta gcacatacta cgcagactcc gtgaagggcc ggttcaccat ctccagagac 1620
aattccaaga acacgctgta tctgcagatg aacagcctga gagccgagga cacggccgta 1680
tattactgtg cgaaaggcag cggatttgac tactggggcc aaggaaccct ggtcaccgtc 1740
tcgagc 1746
<210> 313
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> nucelotide sequence of (49B4) VHCH1 Fc hole VL (DP47)
<400> 313
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagagaatac 300
taccgtggtc cgtacgacta ctggggccaa gggaccaccg tgaccgtctc ctcagctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtgc accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctctc gtgcgcagtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct cgtgagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg tggaggcggc ggaagcggag gaggaggatc cggaggcggc 1380
ggaagcggag ggggaggctc tgaaattgtg ctgacccaga gccccggcac cctgtcactg 1440
tctccaggcg aaagagccac cctgagctgc agagccagcc agagcgtgtc cagctcttac 1500
ctggcctggt atcagcagaa gcccggacag gcccccagac tgctgatcta cggcgcctct 1560
tctagagcca ccggcatccc cgatagattc agcggcagcg gctccggcac cgacttcacc 1620
ctgacaatca gcagactgga acccgaggac tttgccgtgt attactgcca gcagtacggc 1680
agcagccccc tgacctttgg ccagggcacc aaggtggaaa tcaaa 1725
<210> 314
<211> 582
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc knob VH (DP47)
<400> 314
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala
515 520 525
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
530 535 540
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
545 550 555 560
Tyr Tyr Cys Ala Lys Gly Ser Gly Phe Asp Tyr Trp Gly Gln Gly Thr
565 570 575
Leu Val Thr Val Ser Ser
580
<210> 315
<211> 575
<212> PRT
<213> Artificial Sequence
<220>
<223> (49B4) VHCH1 Fc hole VL (DP47)
<400> 315
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Tyr Tyr Arg Gly Pro Tyr Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
465 470 475 480
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
485 490 495
Ser Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
500 505 510
Arg Leu Leu Ile Tyr Gly Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp
515 520 525
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
530 535 540
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly
545 550 555 560
Ser Ser Pro Leu Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 316
<211> 1758
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1 Fc knob VH (4B9) (HC1)
<400> 316
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctggagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcgagt 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccctg cagagatgag 1080
ctgaccaaga accaggtgtc cctgtggtgt ctggtcaagg gcttctaccc cagcgatatc 1140
gccgtggagt gggagagcaa cggccagcct gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac tccaaactga ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgagcctgag ccccggcgga ggcggcggaa gcggaggagg aggatctggg 1380
ggcggaggtt ccggaggcgg tggatctgag gtgcagctgc tcgaaagcgg cggaggactg 1440
gtgcagcctg gcggcagcct gagactgtct tgcgccgcca gcggcttcac cttcagcagc 1500
tacgccatga gctgggtccg ccaggcccct ggcaagggac tggaatgggt gtccgccatc 1560
atcggctctg gcgccagcac ctactacgcc gacagcgtga agggccggtt caccatcagc 1620
cgggacaaca gcaagaacac cctgtacctg cagatgaaca gcctgcgggc cgaggacacc 1680
gccgtgtact actgcgccaa gggatggttc ggcggcttca actactgggg acagggcacc 1740
ctggtcaccg tgtccagc 1758
<210> 317
<211> 1731
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1 Fc hole VL (4B9)
<400> 317
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctggagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcgagt 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtgcaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctctcgtgc gcagtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctcgtg agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtgga ggcggcggaa gcggaggagg aggatccggc 1380
ggcggaggtt ccggaggcgg aggatccgag atcgtgctga cccagtctcc cggcaccctg 1440
tctctgagcc ctggcgagag agccaccctg tcctgcagag cctcccagtc cgtgacctcc 1500
tcctacctcg cctggtatca gcagaagccc ggccaggccc ctcggctgct gatcaacgtg 1560
ggcagtcgga gagccaccgg catccctgac cggttctccg gctctggctc cggcaccgac 1620
ttcaccctga ccatctcccg gctggaaccc gaggacttcg ccgtgtacta ctgccagcag 1680
ggcatcatgc tgccccccac ctttggccag ggcaccaagg tggaaatcaa g 1731
<210> 318
<211> 586
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1 Fc knob VH (4B9)
<400> 318
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 319
<211> 577
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1 Fc hole VL (4B9)
<400> 319
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 320
<211> 1752
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1 Fc knob VH (4B9)
<400> 320
gaggtgcagc tgctggaatc tggcggcgga ctggtgcagc ctggcggatc tctgagactg 60
agctgtgccg ccagcggctt caccttcagc agctacgcca tgagctgggt gcgccaggcc 120
cctggaaaag gcctggaatg ggtgtccgcc atctctggca gcggcggcag cacctactac 180
gccgattctg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgcg ggccgaggac accgccgtgt actattgcgc cagggacgac 300
ccctggcccc cctttgatta ttggggacag ggcaccctcg tgaccgtgtc cagcgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc cctgcagaga tgagctgacc 1080
aagaaccagg tgtccctgtg gtgtctggtc aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgacggca gcttcttcct gtactccaaa ctgaccgtgg acaagagccg gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg cggaggcggc ggaagcggag gaggaggatc tgggggcgga 1380
ggttccggag gcggtggatc tgaggtgcag ctgctcgaaa gcggcggagg actggtgcag 1440
cctggcggca gcctgagact gtcttgcgcc gccagcggct tcaccttcag cagctacgcc 1500
atgagctggg tccgccaggc ccctggcaag ggactggaat gggtgtccgc catcatcggc 1560
tctggcgcca gcacctacta cgccgacagc gtgaagggcc ggttcaccat cagccgggac 1620
aacagcaaga acaccctgta cctgcagatg aacagcctgc gggccgagga caccgccgtg 1680
tactactgcg ccaagggatg gttcggcggc ttcaactact ggggacaggg caccctggtc 1740
accgtgtcca gc 1752
<210> 321
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1 Fc hole VL (4B9)
<400> 321
gaggtgcagc tgctggaatc tggcggcgga ctggtgcagc ctggcggatc tctgagactg 60
agctgtgccg ccagcggctt caccttcagc agctacgcca tgagctgggt gcgccaggcc 120
cctggaaaag gcctggaatg ggtgtccgcc atctctggca gcggcggcag cacctactac 180
gccgattctg tgaagggccg gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga acagcctgcg ggccgaggac accgccgtgt actattgcgc cagggacgac 300
ccctggcccc cctttgatta ttggggacag ggcaccctcg tgaccgtgtc cagcgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtgc accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctctc gtgcgcagtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct cgtgagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg tggaggcggc ggaagcggag gaggaggatc cggcggcgga 1380
ggttccggag gcggaggatc cgagatcgtg ctgacccagt ctcccggcac cctgtctctg 1440
agccctggcg agagagccac cctgtcctgc agagcctccc agtccgtgac ctcctcctac 1500
ctcgcctggt atcagcagaa gcccggccag gcccctcggc tgctgatcaa cgtgggcagt 1560
cggagagcca ccggcatccc tgaccggttc tccggctctg gctccggcac cgacttcacc 1620
ctgaccatct cccggctgga acccgaggac ttcgccgtgt actactgcca gcagggcatc 1680
atgctgcccc ccacctttgg ccagggcacc aaggtggaaa tcaag 1725
<210> 322
<211> 584
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1 Fc knob VH
<400> 322
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala
515 520 525
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
530 535 540
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
545 550 555 560
Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln
565 570 575
Gly Thr Leu Val Thr Val Ser Ser
580
<210> 323
<211> 575
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1 Fc hole VL (4B9)
<400> 323
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
465 470 475 480
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
485 490 495
Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
500 505 510
Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp
515 520 525
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
530 535 540
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile
545 550 555 560
Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 324
<211> 1755
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1 Fc knob VH (4B9)
<400> 324
caggtgcagc tggtgcagtc tggcgccgaa gtgaagaaac ccggcagcag cgtgaaggtg 60
tcctgcaagg cttccggcgg caccttcagc agctacgcca tttcttgggt gcgccaggcc 120
cctggacagg gcctggaatg gatgggcggc atcatcccca tcttcggcac cgccaactac 180
gcccagaaat tccagggcag agtgaccatc accgccgaca agagcaccag caccgcctac 240
atggaactga gcagcctgcg gagcgaggac accgccgtgt actactgtgc cagaagcacc 300
ctgatctacg gctacttcga ctactggggc cagggcacca ccgtgaccgt gtctagcgct 360
agcaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagaaagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagctgc agggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccctgcag agatgagctg 1080
accaagaacc aggtgtccct gtggtgtctg gtcaagggct tctaccccag cgatatcgcc 1140
gtggagtggg agagcaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gacagcgacg gcagcttctt cctgtactcc aaactgaccg tggacaagag ccggtggcag 1260
cagggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctga gcctgagccc cggcggaggc ggcggaagcg gaggaggagg atctgggggc 1380
ggaggttccg gaggcggtgg atctgaggtg cagctgctcg aaagcggcgg aggactggtg 1440
cagcctggcg gcagcctgag actgtcttgc gccgccagcg gcttcacctt cagcagctac 1500
gccatgagct gggtccgcca ggcccctggc aagggactgg aatgggtgtc cgccatcatc 1560
ggctctggcg ccagcaccta ctacgccgac agcgtgaagg gccggttcac catcagccgg 1620
gacaacagca agaacaccct gtacctgcag atgaacagcc tgcgggccga ggacaccgcc 1680
gtgtactact gcgccaaggg atggttcggc ggcttcaact actggggaca gggcaccctg 1740
gtcaccgtgt ccagc 1755
<210> 325
<211> 1728
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1 Fc hole VL (4B9)
<400> 325
caggtgcagc tggtgcagtc tggcgccgaa gtgaagaaac ccggcagcag cgtgaaggtg 60
tcctgcaagg cttccggcgg caccttcagc agctacgcca tttcttgggt gcgccaggcc 120
cctggacagg gcctggaatg gatgggcggc atcatcccca tcttcggcac cgccaactac 180
gcccagaaat tccagggcag agtgaccatc accgccgaca agagcaccag caccgcctac 240
atggaactga gcagcctgcg gagcgaggac accgccgtgt actactgtgc cagaagcacc 300
ctgatctacg gctacttcga ctactggggc cagggcacca ccgtgaccgt gtctagcgct 360
agcaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagaaagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagctgc agggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tgcaccctgc ccccatcccg ggatgagctg 1080
accaagaacc aggtcagcct ctcgtgcgca gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctcgtgagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtggaggc ggcggaagcg gaggaggagg atccggcggc 1380
ggaggttccg gaggcggagg atccgagatc gtgctgaccc agtctcccgg caccctgtct 1440
ctgagccctg gcgagagagc caccctgtcc tgcagagcct cccagtccgt gacctcctcc 1500
tacctcgcct ggtatcagca gaagcccggc caggcccctc ggctgctgat caacgtgggc 1560
agtcggagag ccaccggcat ccctgaccgg ttctccggct ctggctccgg caccgacttc 1620
accctgacca tctcccggct ggaacccgag gacttcgccg tgtactactg ccagcagggc 1680
atcatgctgc cccccacctt tggccagggc accaaggtgg aaatcaag 1728
<210> 326
<211> 585
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1 Fc knob VH (4B9)
<400> 326
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
450 455 460
Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
465 470 475 480
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
485 490 495
Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
500 505 510
Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr
515 520 525
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
530 535 540
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
545 550 555 560
Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly
565 570 575
Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 327
<211> 576
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1 Fc hole VL (4B9)
<400> 327
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser
355 360 365
Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
450 455 460
Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser
465 470 475 480
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
485 490 495
Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala
500 505 510
Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro
515 520 525
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
530 535 540
Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly
545 550 555 560
Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 328
<211> 1758
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1 Fc knob VH (4B9)
<400> 328
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccctg cagagatgag 1080
ctgaccaaga accaggtgtc cctgtggtgt ctggtcaagg gcttctaccc cagcgatatc 1140
gccgtggagt gggagagcaa cggccagcct gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac tccaaactga ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgagcctgag ccccggcgga ggcggcggaa gcggaggagg aggatctggg 1380
ggcggaggtt ccggaggcgg tggatctgag gtgcagctgc tcgaaagcgg cggaggactg 1440
gtgcagcctg gcggcagcct gagactgtct tgcgccgcca gcggcttcac cttcagcagc 1500
tacgccatga gctgggtccg ccaggcccct ggcaagggac tggaatgggt gtccgccatc 1560
atcggctctg gcgccagcac ctactacgcc gacagcgtga agggccggtt caccatcagc 1620
cgggacaaca gcaagaacac cctgtacctg cagatgaaca gcctgcgggc cgaggacacc 1680
gccgtgtact actgcgccaa gggatggttc ggcggcttca actactgggg acagggcacc 1740
ctggtcaccg tgtccagc 1758
<210> 329
<211> 1731
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1 Fc hole VL (4B9)
<400> 329
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtgcaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctctcgtgc gcagtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctcgtg agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtgga ggcggcggaa gcggaggagg aggatccggc 1380
ggcggaggtt ccggaggcgg aggatccgag atcgtgctga cccagtctcc cggcaccctg 1440
tctctgagcc ctggcgagag agccaccctg tcctgcagag cctcccagtc cgtgacctcc 1500
tcctacctcg cctggtatca gcagaagccc ggccaggccc ctcggctgct gatcaacgtg 1560
ggcagtcgga gagccaccgg catccctgac cggttctccg gctctggctc cggcaccgac 1620
ttcaccctga ccatctcccg gctggaaccc gaggacttcg ccgtgtacta ctgccagcag 1680
ggcatcatgc tgccccccac ctttggccag ggcaccaagg tggaaatcaa g 1731
<210> 330
<211> 586
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1 Fc knob VH (4B9)
<400> 330
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 331
<211> 577
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1 Fc hole VL (4B9)
<400> 331
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 332
<211> 1731
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1 Fc knob VL (4B9)
<400> 332
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctggagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcgagt 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccctg cagagatgag 1080
ctgaccaaga accaggtgtc cctgtggtgt ctggtcaagg gcttctaccc cagcgatatc 1140
gccgtggagt gggagagcaa cggccagcct gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac tccaaactga ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgagcctgag ccccggcgga ggcggcggaa gcggaggagg aggatctggg 1380
ggcggaggtt ccggaggcgg tggatctgag atcgtgctga cccagtctcc cggcaccctg 1440
tctctgagcc ctggcgagag agccaccctg tcctgcagag cctcccagtc cgtgacctcc 1500
tcctacctcg cctggtatca gcagaagccc ggccaggccc ctcggctgct gatcaacgtg 1560
ggcagtcgga gagccaccgg catccctgac cggttctccg gctctggctc cggcaccgac 1620
ttcaccctga ccatctcccg gctggaaccc gaggacttcg ccgtgtacta ctgccagcag 1680
ggcatcatgc tgccccccac ctttggccag ggcaccaagg tggaaatcaa g 1731
<210> 333
<211> 1758
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (12B3) VHCH1 Fc hole VH (4B9)
<400> 333
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctggagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctgaa 300
ttccgtttct acgctgactt cgactactgg ggccaaggga ccaccgtgac cgtctcgagt 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtgcaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctctcgtgc gcagtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctcgtg agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtgga ggcggcggaa gcggaggagg aggatccggc 1380
ggcggaggtt ccggaggcgg aggatccgag gtgcagctgc tcgaaagcgg cggaggactg 1440
gtgcagcctg gcggcagcct gagactgtct tgcgccgcca gcggcttcac cttcagcagc 1500
tacgccatga gctgggtccg ccaggcccct ggcaagggac tggaatgggt gtccgccatc 1560
atcggctctg gcgccagcac ctactacgcc gacagcgtga agggccggtt caccatcagc 1620
cgggacaaca gcaagaacac cctgtacctg cagatgaaca gcctgcgggc cgaggacacc 1680
gccgtgtact actgcgccaa gggatggttc ggcggcttca actactgggg acagggcacc 1740
ctggtcaccg tgtccagc 1758
<210> 334
<211> 577
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1 Fc knob VL (4B9)
<400> 334
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 335
<211> 586
<212> PRT
<213> Artificial Sequence
<220>
<223> (12B3) VHCH1 Fc hole VH (4B9)
<400> 335
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Phe Arg Phe Tyr Ala Asp Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 336
<211> 1725
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1 Fc knob VL (4B9)
<400> 336
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagtgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtac accctgcccc cctgcagaga tgagctgacc 1080
aagaaccagg tgtccctgtg gtgtctggtc aagggcttct accccagcga tatcgccgtg 1140
gagtgggaga gcaacggcca gcctgagaac aactacaaga ccaccccccc tgtgctggac 1200
agcgacggca gcttcttcct gtactccaaa ctgaccgtgg acaagagccg gtggcagcag 1260
ggcaacgtgt tcagctgcag cgtgatgcac gaggccctgc acaaccacta cacccagaag 1320
tccctgagcc tgagccccgg cggaggcggc ggaagcggag gaggaggatc tgggggcgga 1380
ggttccggag gcggtggatc tgagatcgtg ctgacccagt ctcccggcac cctgtctctg 1440
agccctggcg agagagccac cctgtcctgc agagcctccc agtccgtgac ctcctcctac 1500
ctcgcctggt atcagcagaa gcccggccag gcccctcggc tgctgatcaa cgtgggcagt 1560
cggagagcca ccggcatccc tgaccggttc tccggctctg gctccggcac cgacttcacc 1620
ctgaccatct cccggctgga acccgaggac ttcgccgtgt actactgcca gcagggcatc 1680
atgctgcccc ccacctttgg ccagggcacc aaggtggaaa tcaag 1725
<210> 337
<211> 1752
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (25G7) VHCH1 Fc hole VH (4B9)
<400> 337
gaggtgcaat tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctccggatt cacctttagc agttatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcagatga acagcctgag agccgaggac acggccgtat attactgtgc gcgtgacgac 300
ccgtggccgc cgttcgacta ctggggccaa ggaaccctgg tcaccgtctc gagtgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aagctgcagg gggaccgtca 720
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 780
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 840
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 900
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 960
aagtgcaagg tctccaacaa agccctcggc gcccccatcg agaaaaccat ctccaaagcc 1020
aaagggcagc cccgagaacc acaggtgtgc accctgcccc catcccggga tgagctgacc 1080
aagaaccagg tcagcctctc gtgcgcagtc aaaggcttct atcccagcga catcgccgtg 1140
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1200
tccgacggct ccttcttcct cgtgagcaag ctcaccgtgg acaagagcag gtggcagcag 1260
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1320
agcctctccc tgtctccggg tggaggcggc ggaagcggag gaggaggatc cggcggcgga 1380
ggttccggag gcggaggatc cgaggtgcag ctgctcgaaa gcggcggagg actggtgcag 1440
cctggcggca gcctgagact gtcttgcgcc gccagcggct tcaccttcag cagctacgcc 1500
atgagctggg tccgccaggc ccctggcaag ggactggaat gggtgtccgc catcatcggc 1560
tctggcgcca gcacctacta cgccgacagc gtgaagggcc ggttcaccat cagccgggac 1620
aacagcaaga acaccctgta cctgcagatg aacagcctgc gggccgagga caccgccgtg 1680
tactactgcg ccaagggatg gttcggcggc ttcaactact ggggacaggg caccctggtc 1740
accgtgtcca gc 1752
<210> 338
<211> 575
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1 Fc knob VL (4B9)
<400> 338
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu
465 470 475 480
Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val
485 490 495
Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
500 505 510
Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro Asp
515 520 525
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
530 535 540
Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly Ile
545 550 555 560
Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 339
<211> 584
<212> PRT
<213> Artificial Sequence
<220>
<223> (25G7) VHCH1 Fc hole VH (4B9)
<400> 339
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Pro Trp Pro Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser Cys
355 360 365
Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Gly
435 440 445
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
450 455 460
Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln
465 470 475 480
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
485 490 495
Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
500 505 510
Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr Ala
515 520 525
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn
530 535 540
Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
545 550 555 560
Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly Gln
565 570 575
Gly Thr Leu Val Thr Val Ser Ser
580
<210> 340
<211> 1728
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1 Fc knob VL (4B9)
<400> 340
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctcagct 360
agcaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagaaagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagctgc agggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccctgcag agatgagctg 1080
accaagaacc aggtgtccct gtggtgtctg gtcaagggct tctaccccag cgatatcgcc 1140
gtggagtggg agagcaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1200
gacagcgacg gcagcttctt cctgtactcc aaactgaccg tggacaagag ccggtggcag 1260
cagggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 1320
aagtccctga gcctgagccc cggcggaggc ggcggaagcg gaggaggagg atctgggggc 1380
ggaggttccg gaggcggtgg atctgagatc gtgctgaccc agtctcccgg caccctgtct 1440
ctgagccctg gcgagagagc caccctgtcc tgcagagcct cccagtccgt gacctcctcc 1500
tacctcgcct ggtatcagca gaagcccggc caggcccctc ggctgctgat caacgtgggc 1560
agtcggagag ccaccggcat ccctgaccgg ttctccggct ctggctccgg caccgacttc 1620
accctgacca tctcccggct ggaacccgag gacttcgccg tgtactactg ccagcagggc 1680
atcatgctgc cccccacctt tggccagggc accaaggtgg aaatcaag 1728
<210> 341
<211> 1755
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (11D5) VHCH1 Fc hole VH (4B9)
<400> 341
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatctact 300
ctgatctacg gttacttcga ctactggggc caagggacca ccgtgaccgt ctcctcagct 360
agcaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 420
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 480
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 540
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 600
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagaaagt tgagcccaaa 660
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaagctgc agggggaccg 720
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 780
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 840
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 900
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 960
tacaagtgca aggtctccaa caaagccctc ggcgccccca tcgagaaaac catctccaaa 1020
gccaaagggc agccccgaga accacaggtg tgcaccctgc ccccatcccg ggatgagctg 1080
accaagaacc aggtcagcct ctcgtgcgca gtcaaaggct tctatcccag cgacatcgcc 1140
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1200
gactccgacg gctccttctt cctcgtgagc aagctcaccg tggacaagag caggtggcag 1260
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1320
aagagcctct ccctgtctcc gggtggaggc ggcggaagcg gaggaggagg atccggcggc 1380
ggaggttccg gaggcggagg atccgaggtg cagctgctcg aaagcggcgg aggactggtg 1440
cagcctggcg gcagcctgag actgtcttgc gccgccagcg gcttcacctt cagcagctac 1500
gccatgagct gggtccgcca ggcccctggc aagggactgg aatgggtgtc cgccatcatc 1560
ggctctggcg ccagcaccta ctacgccgac agcgtgaagg gccggttcac catcagccgg 1620
gacaacagca agaacaccct gtacctgcag atgaacagcc tgcgggccga ggacaccgcc 1680
gtgtactact gcgccaaggg atggttcggc ggcttcaact actggggaca gggcaccctg 1740
gtcaccgtgt ccagc 1755
<210> 342
<211> 576
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1 Fc knob VL (4B9)
<400> 342
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Trp
355 360 365
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
450 455 460
Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser
465 470 475 480
Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser
485 490 495
Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala
500 505 510
Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile Pro
515 520 525
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
530 535 540
Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Gly
545 550 555 560
Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
565 570 575
<210> 343
<211> 585
<212> PRT
<213> Artificial Sequence
<220>
<223> (11D5) VHCH1 Fc hole VH (4B9)
<400> 343
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Thr Leu Ile Tyr Gly Tyr Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro
225 230 235 240
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu Lys
325 330 335
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys Thr
340 345 350
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Ser
355 360 365
Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp Lys
405 410 415
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
450 455 460
Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
465 470 475 480
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
485 490 495
Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly
500 505 510
Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr Tyr
515 520 525
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
530 535 540
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
545 550 555 560
Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp Gly
565 570 575
Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<210> 344
<211> 1731
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1 Fc knob VL (4B9)
<400> 344
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccctg cagagatgag 1080
ctgaccaaga accaggtgtc cctgtggtgt ctggtcaagg gcttctaccc cagcgatatc 1140
gccgtggagt gggagagcaa cggccagcct gagaacaact acaagaccac cccccctgtg 1200
ctggacagcg acggcagctt cttcctgtac tccaaactga ccgtggacaa gagccggtgg 1260
cagcagggca acgtgttcag ctgcagcgtg atgcacgagg ccctgcacaa ccactacacc 1320
cagaagtccc tgagcctgag ccccggcgga ggcggcggaa gcggaggagg aggatctggg 1380
ggcggaggtt ccggaggcgg tggatctgag atcgtgctga cccagtctcc cggcaccctg 1440
tctctgagcc ctggcgagag agccaccctg tcctgcagag cctcccagtc cgtgacctcc 1500
tcctacctcg cctggtatca gcagaagccc ggccaggccc ctcggctgct gatcaacgtg 1560
ggcagtcgga gagccaccgg catccctgac cggttctccg gctctggctc cggcaccgac 1620
ttcaccctga ccatctcccg gctggaaccc gaggacttcg ccgtgtacta ctgccagcag 1680
ggcatcatgc tgccccccac ctttggccag ggcaccaagg tggaaatcaa g 1731
<210> 345
<211> 1758
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA (9B11) VHCH1 Fc hole VH (4B9)
<400> 345
caggtgcaat tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cctccggagg cacattcagc agctacgcta taagctgggt gcgacaggcc 120
cctggacaag ggctcgagtg gatgggaggg atcatcccta tctttggtac agcaaactac 180
gcacagaagt tccagggcag ggtcaccatt actgcagaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac accgccgtgt attactgtgc gagatcttct 300
ggtgcttacc cgggttactt cgactactgg ggccaaggga ccaccgtgac cgtctcctca 360
gctagcacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 420
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaaccggt gacggtgtcg 480
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 540
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 600
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagaa agttgagccc 660
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaagc tgcaggggga 720
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 780
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 840
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 900
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 960
gagtacaagt gcaaggtctc caacaaagcc ctcggcgccc ccatcgagaa aaccatctcc 1020
aaagccaaag ggcagccccg agaaccacag gtgtgcaccc tgcccccatc ccgggatgag 1080
ctgaccaaga accaggtcag cctctcgtgc gcagtcaaag gcttctatcc cagcgacatc 1140
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1200
ctggactccg acggctcctt cttcctcgtg agcaagctca ccgtggacaa gagcaggtgg 1260
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1320
cagaagagcc tctccctgtc tccgggtgga ggcggcggaa gcggaggagg aggatccggc 1380
ggcggaggtt ccggaggcgg aggatccgag gtgcagctgc tcgaaagcgg cggaggactg 1440
gtgcagcctg gcggcagcct gagactgtct tgcgccgcca gcggcttcac cttcagcagc 1500
tacgccatga gctgggtccg ccaggcccct ggcaagggac tggaatgggt gtccgccatc 1560
atcggctctg gcgccagcac ctactacgcc gacagcgtga agggccggtt caccatcagc 1620
cgggacaaca gcaagaacac cctgtacctg cagatgaaca gcctgcgggc cgaggacacc 1680
gccgtgtact actgcgccaa gggatggttc ggcggcttca actactgggg acagggcacc 1740
ctggtcaccg tgtccagc 1758
<210> 346
<211> 577
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1 Fc knob VL (4B9)
<400> 346
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Cys Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Trp Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu
465 470 475 480
Ser Leu Ser Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln
485 490 495
Ser Val Thr Ser Ser Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
500 505 510
Ala Pro Arg Leu Leu Ile Asn Val Gly Ser Arg Arg Ala Thr Gly Ile
515 520 525
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
530 535 540
Ile Ser Arg Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln
545 550 555 560
Gly Ile Met Leu Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile
565 570 575
Lys
<210> 347
<211> 586
<212> PRT
<213> Artificial Sequence
<220>
<223> (9B11) VHCH1 Fc hole VH (4B9)
<400> 347
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Gly Ala Tyr Pro Gly Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Gly Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Cys
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Ser Cys Ala Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Val Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
450 455 460
Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
465 470 475 480
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
485 490 495
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
500 505 510
Gly Leu Glu Trp Val Ser Ala Ile Ile Gly Ser Gly Ala Ser Thr Tyr
515 520 525
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
530 535 540
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
545 550 555 560
Ala Val Tyr Tyr Cys Ala Lys Gly Trp Phe Gly Gly Phe Asn Tyr Trp
565 570 575
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
580 585
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО АНТИ-EGFR/АНТИ-4-1ВВ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2831836C2 |
| HER-2-НАПРАВЛЕННЫЕ АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ, СОДЕРЖАЩИЕ 4-1BBL | 2019 |
|
RU2815451C2 |
| МОЛЕКУЛЫ, СВЯЗЫВАЮЩИЕСЯ С 4-1ВВ | 2011 |
|
RU2551963C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| БИСПЕЦИФИЧЕСКИЕ 2+1 КОНТОРСТЕЛА | 2018 |
|
RU2797305C2 |
| Т-КЛЕТКИ С ДВУМЯ ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ CAR | 2021 |
|
RU2839662C1 |
| ВАРИАНТЫ СЛИТОГО БЕЛКА SIRP-альфа-4-1BBL И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2815515C2 |
| УСЕЧЕННЫЕ ХИМЕРНЫЕ РЕЦЕПТОРЫ NKG2D И ПУТИ ИХ ПРИМЕНЕНИЯ В ИММУНОТЕРАПИИ С ИСПОЛЬЗОВАНИЕМ ЕСТЕСТВЕННЫХ КЛЕТОК-КИЛЛЕРОВ | 2018 |
|
RU2830556C2 |
| Т-КЛЕТКИ С КОСТИМУЛИРУЮЩИМ ХИМЕРНЫМ АНТИГЕННЫМ РЕЦЕПТОРОМ, НАЦЕЛЕННЫЕ НА IL13Rα2 | 2015 |
|
RU2749922C2 |
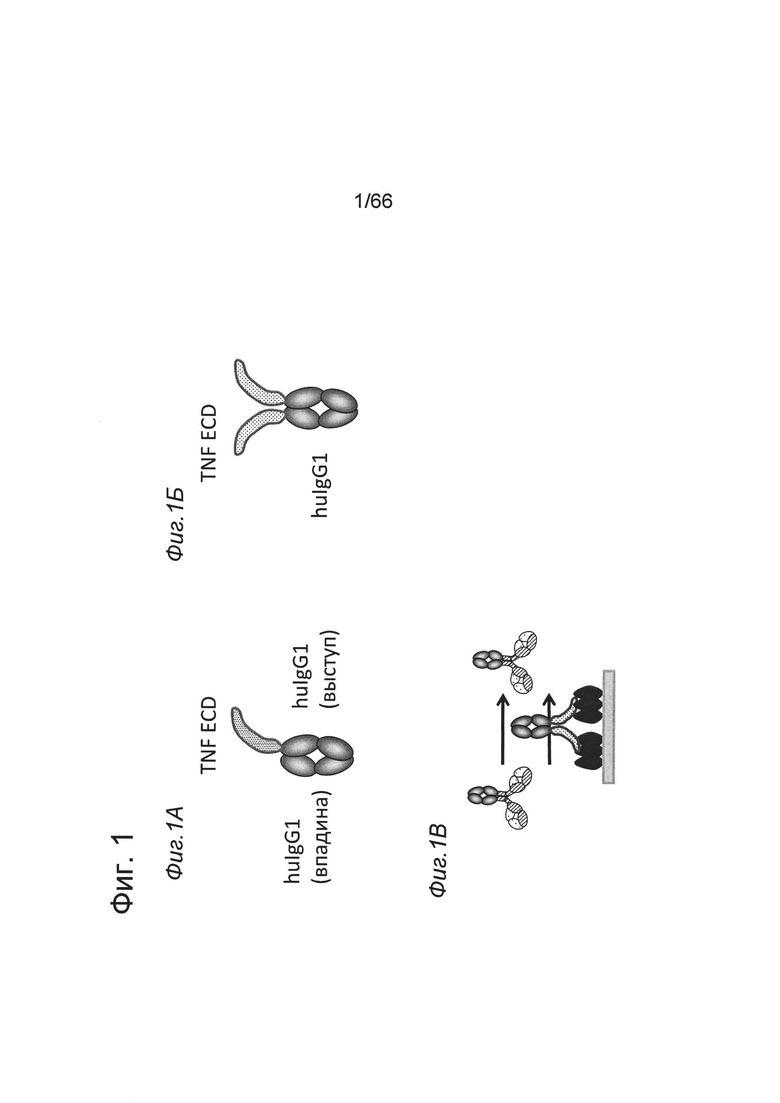

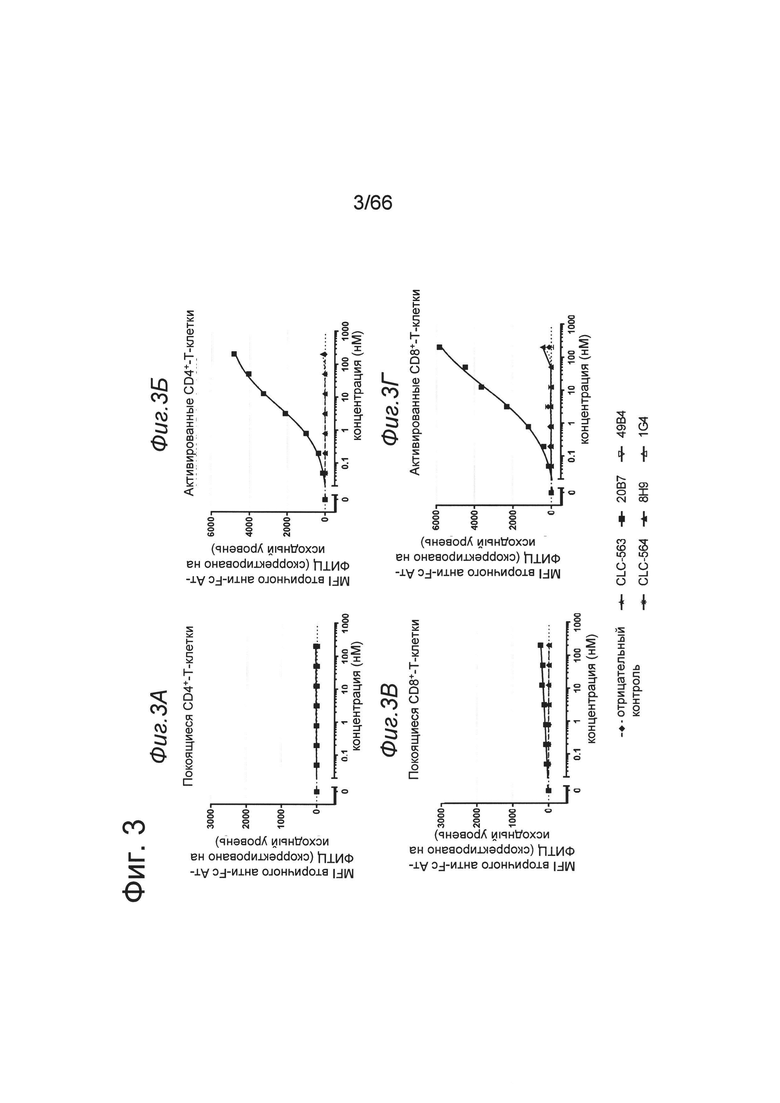
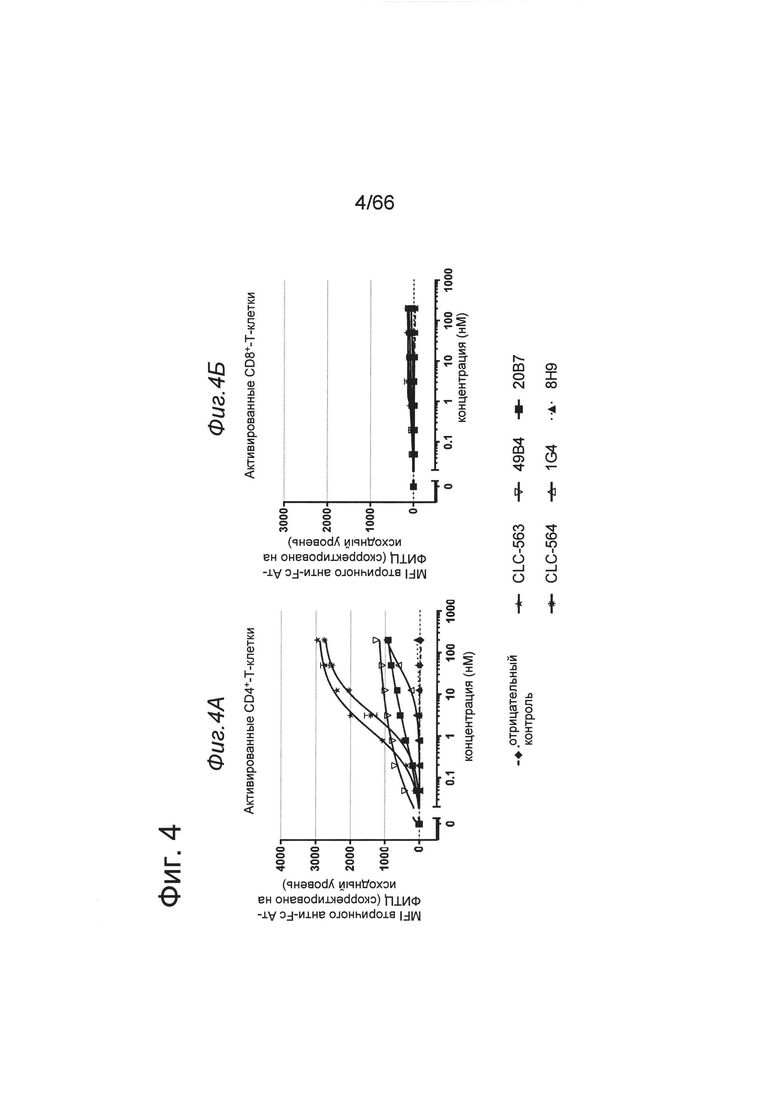
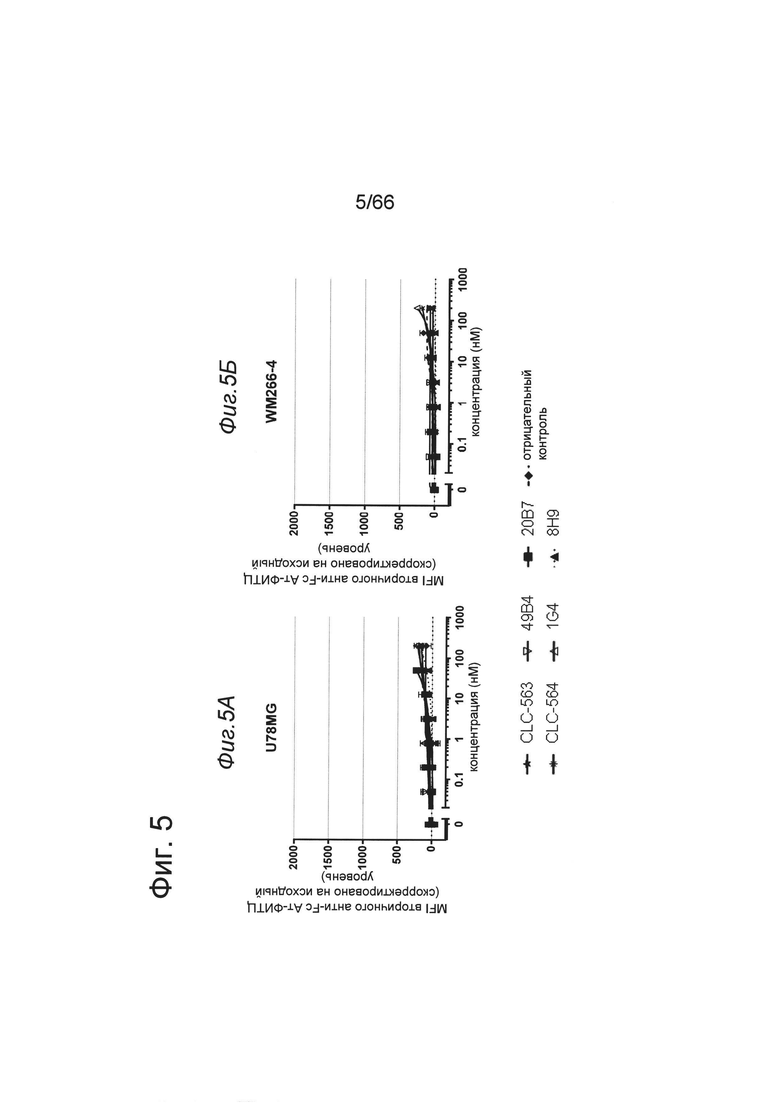
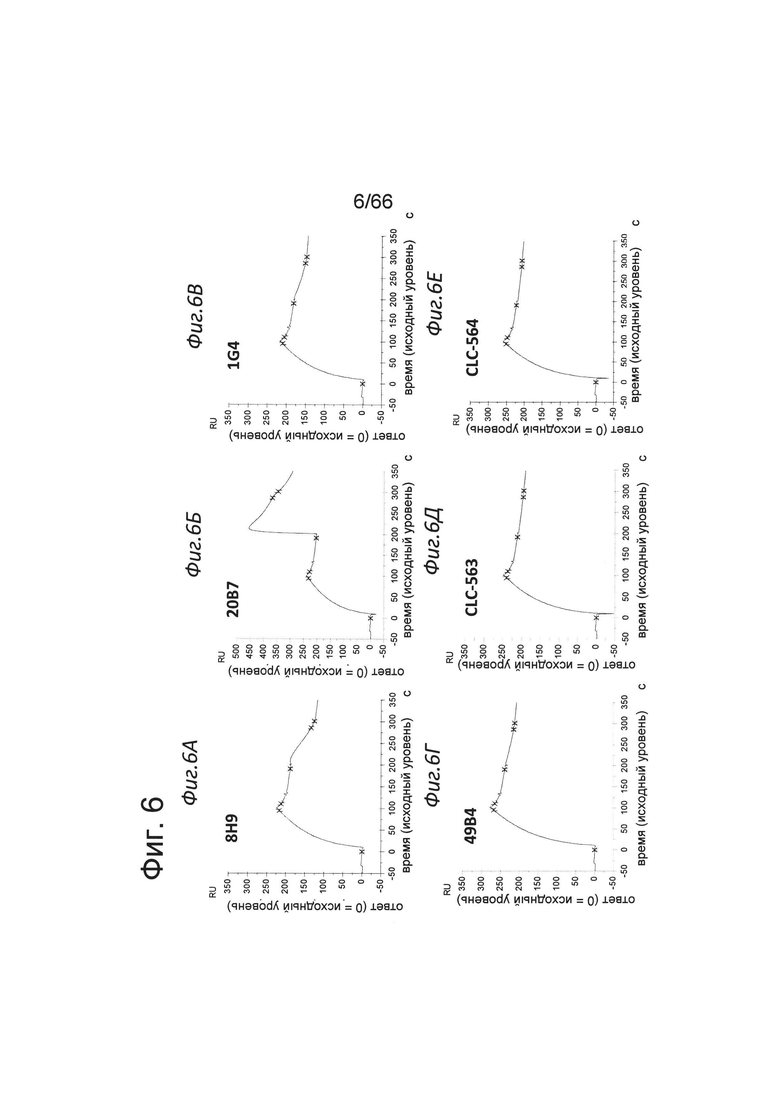
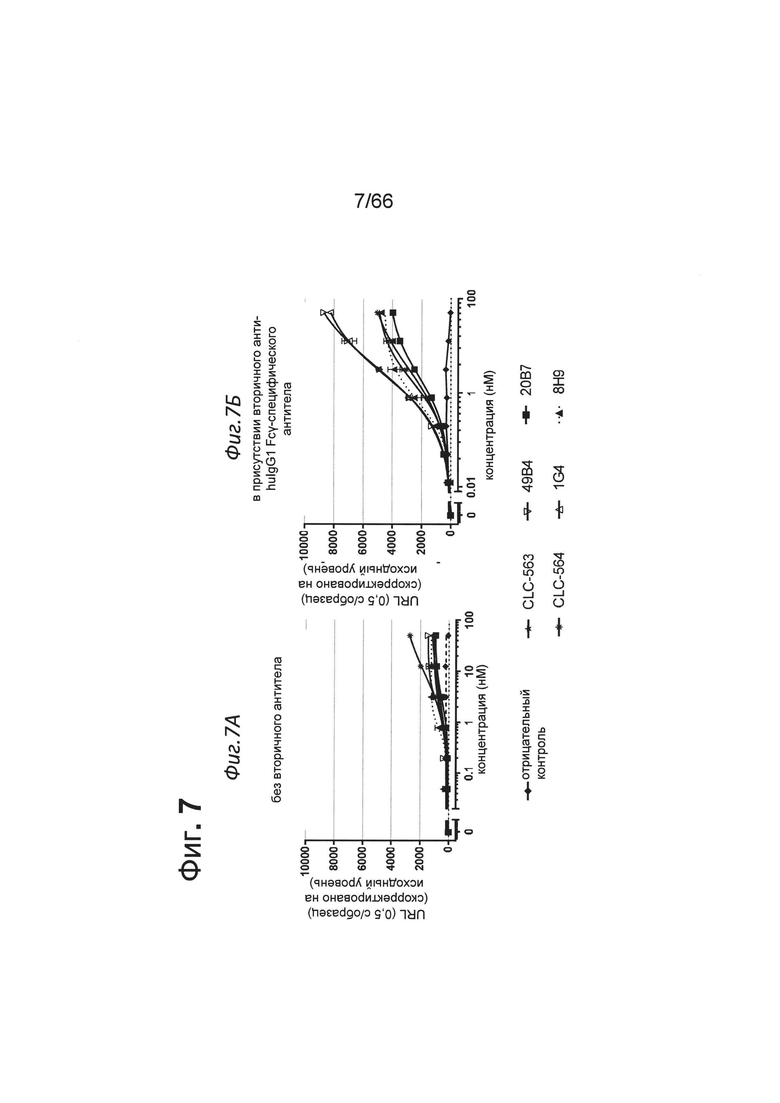
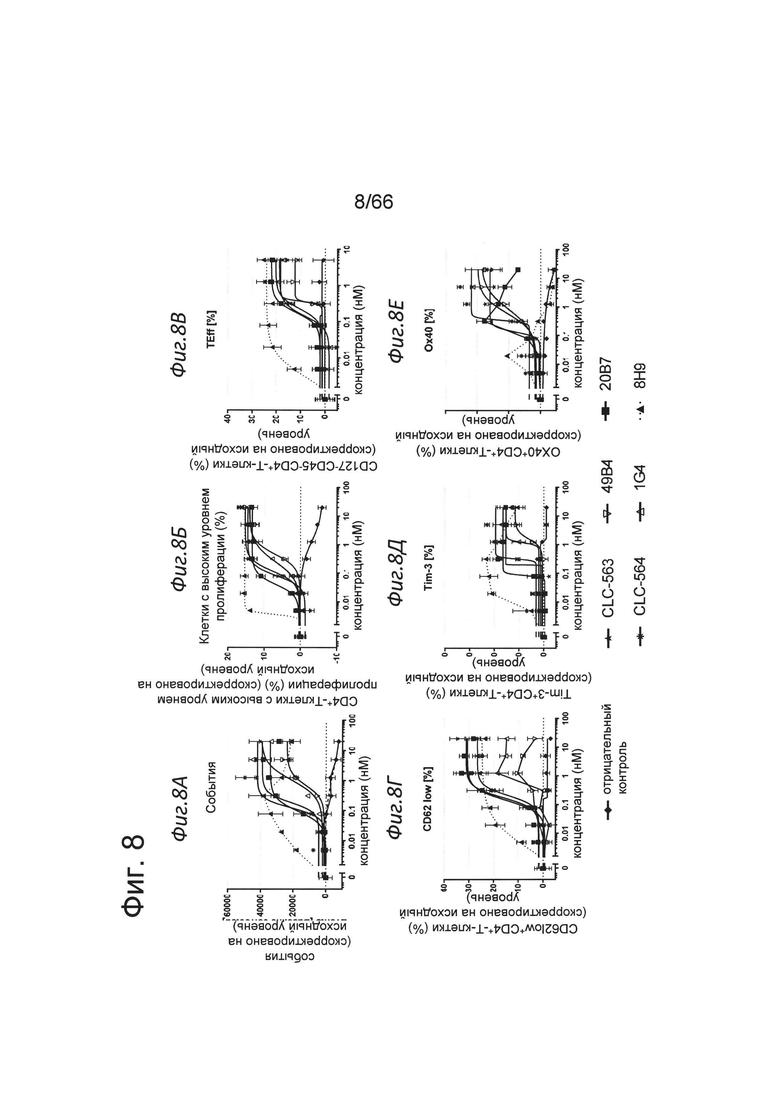
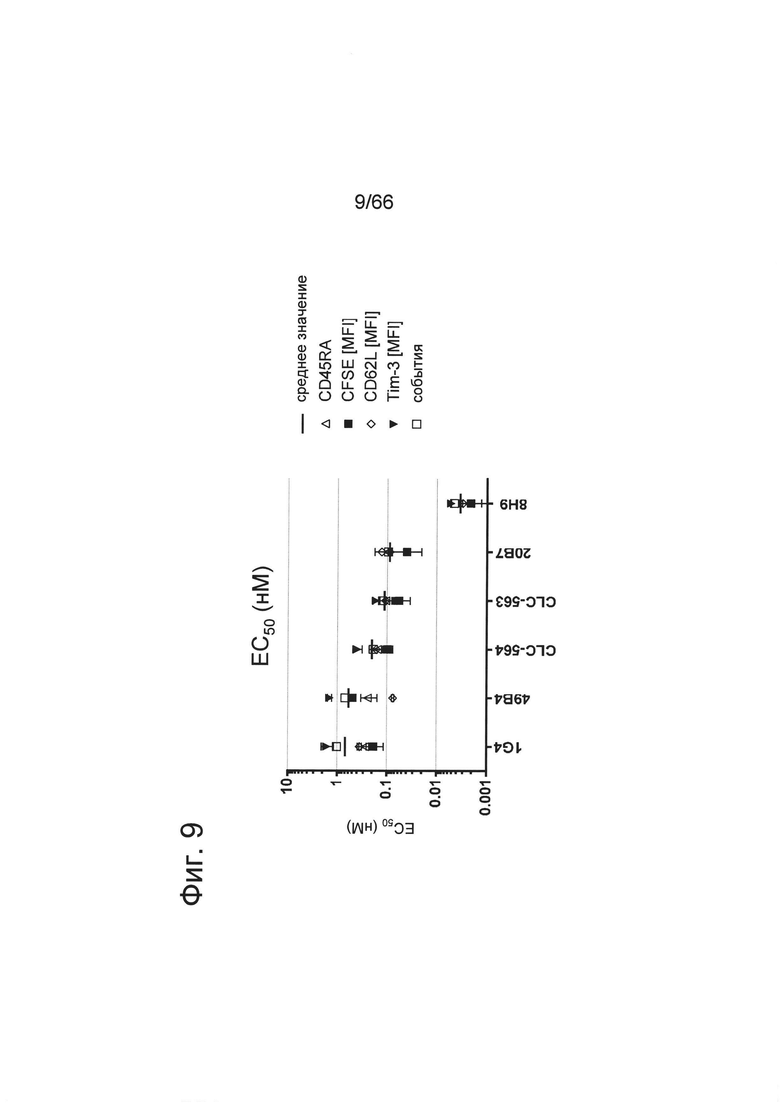
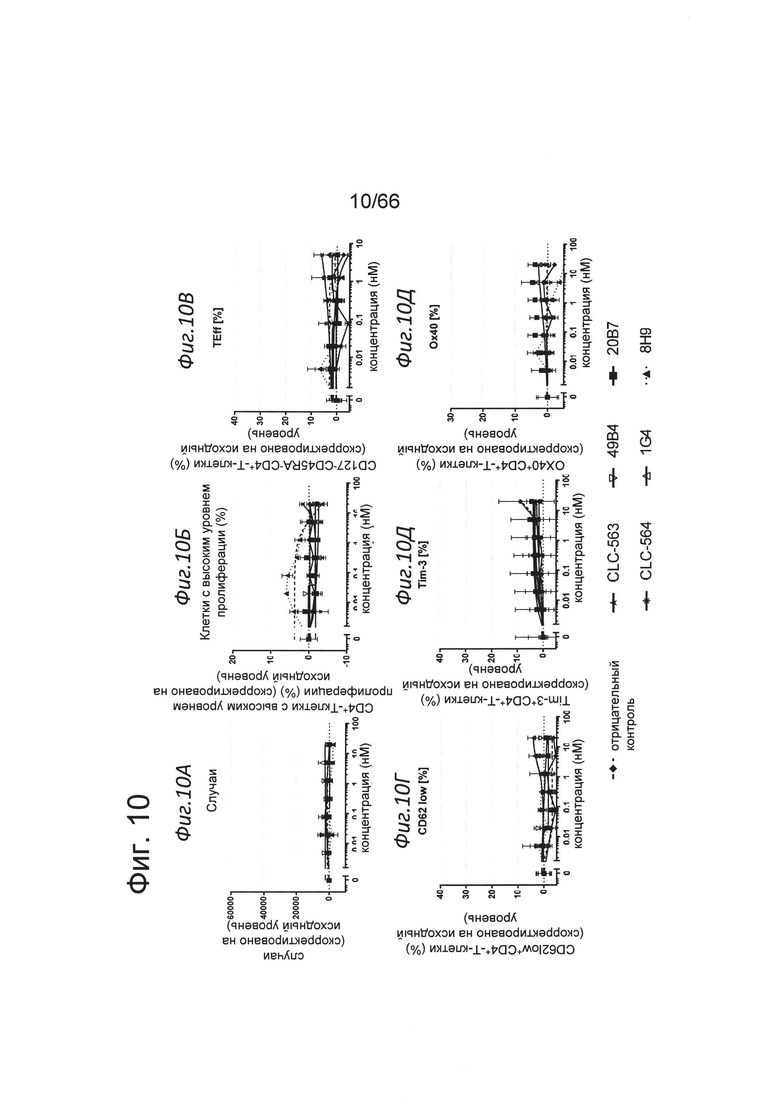
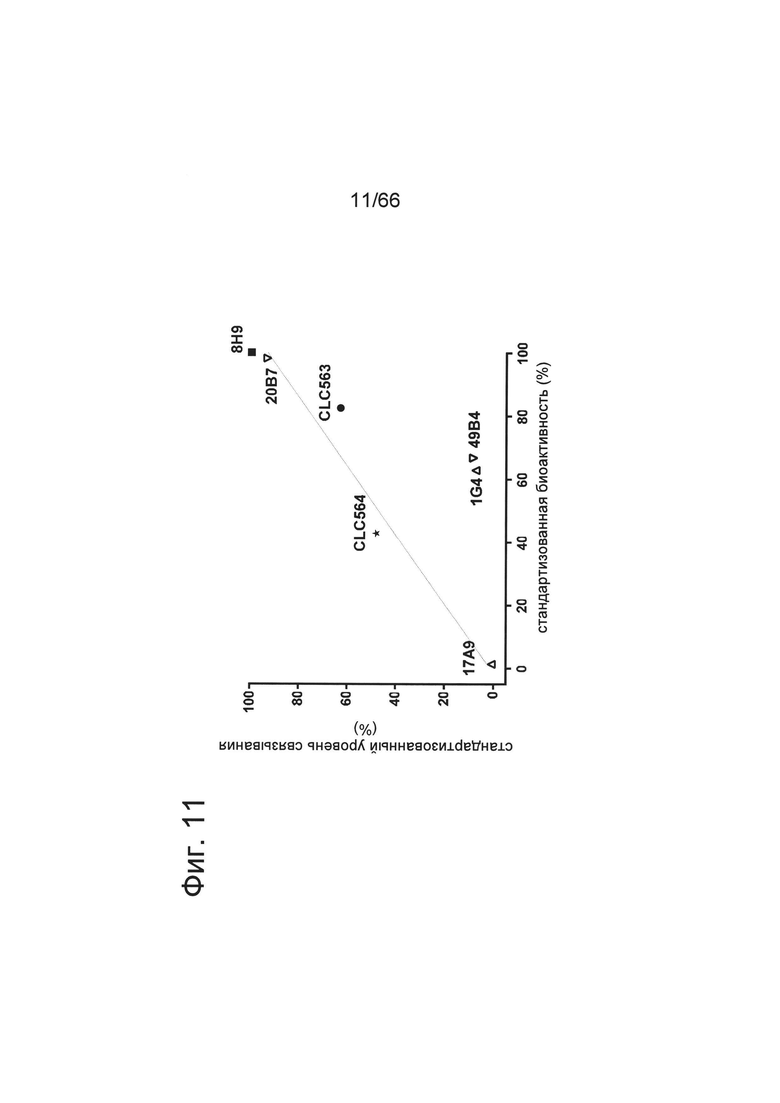
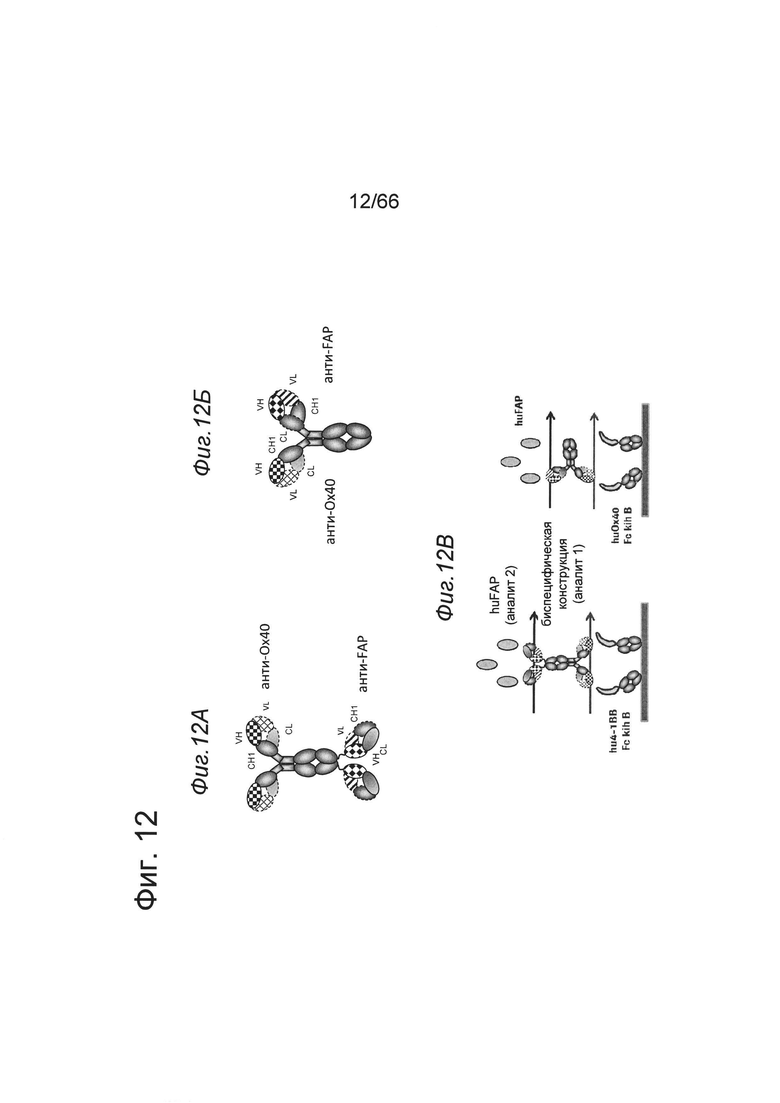
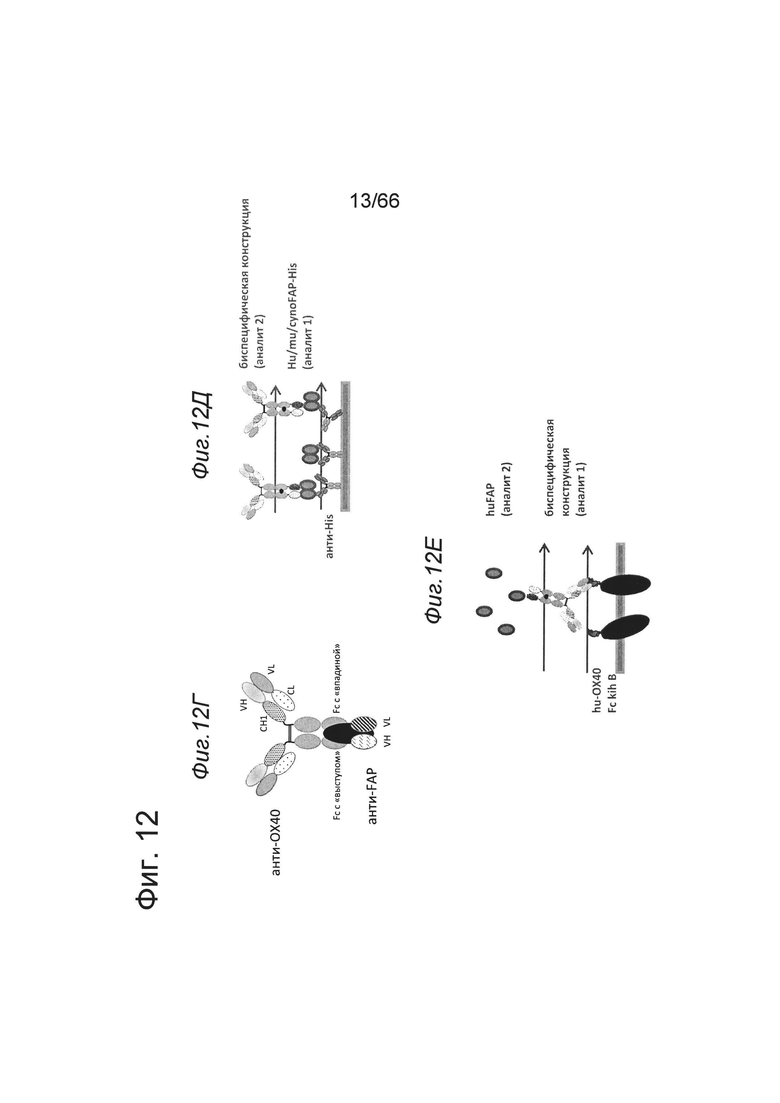
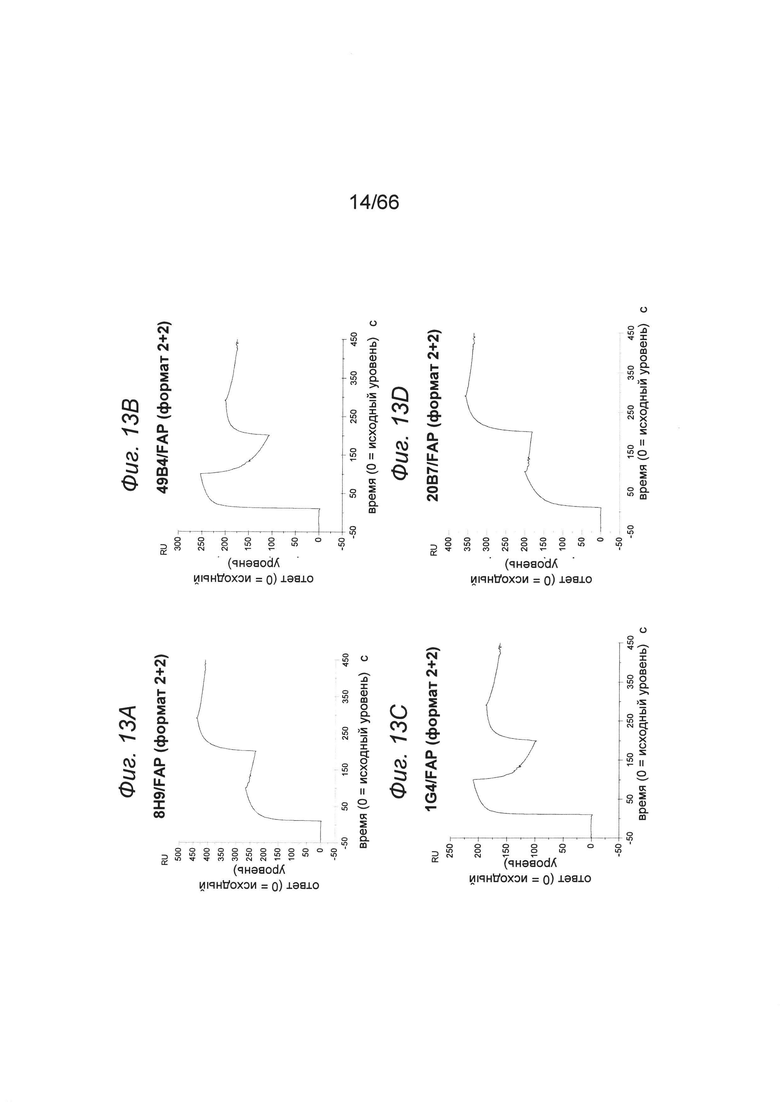
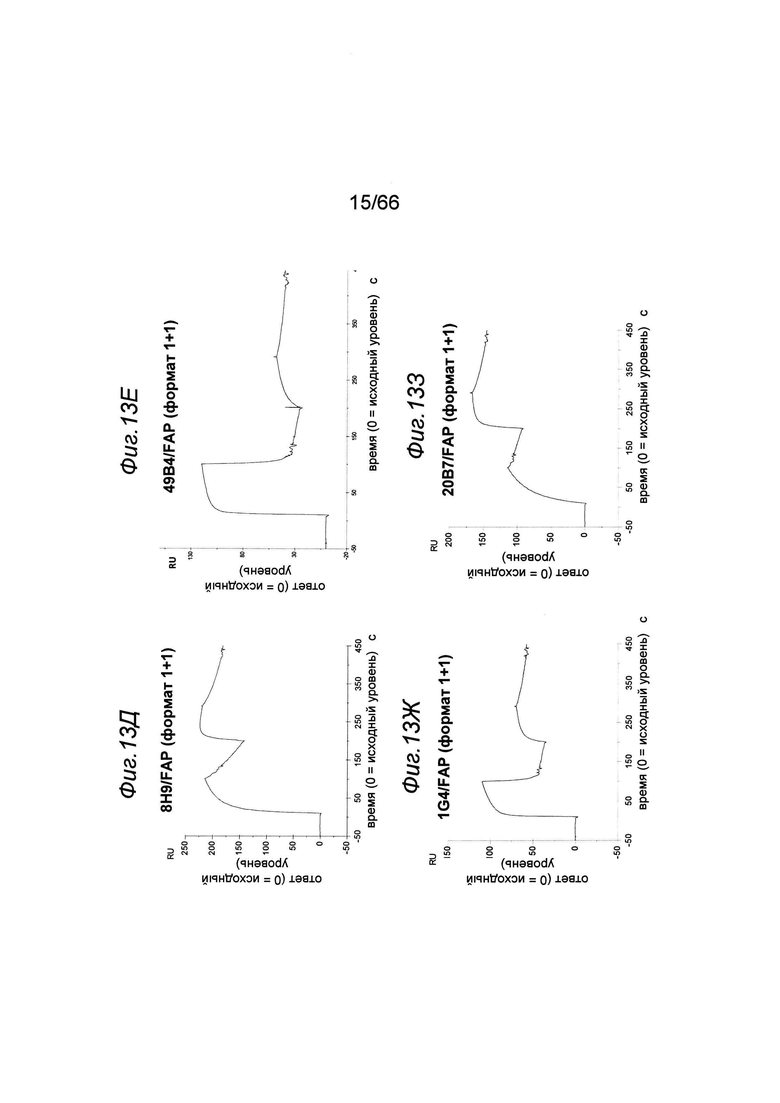
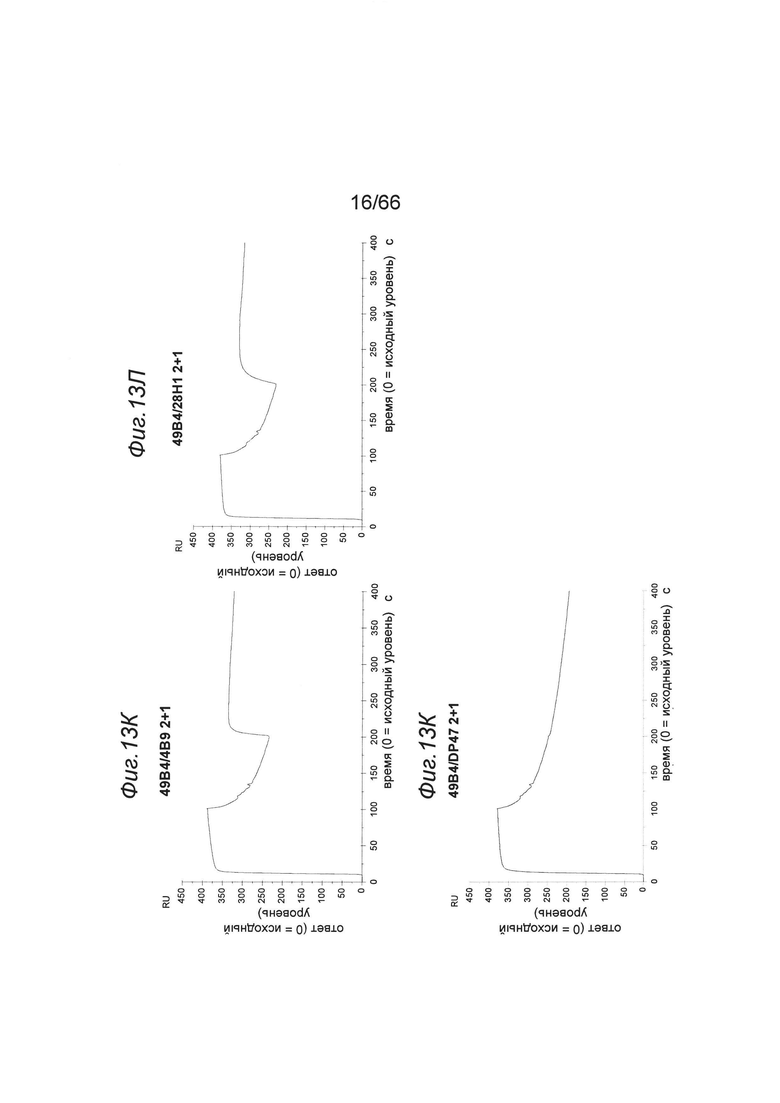
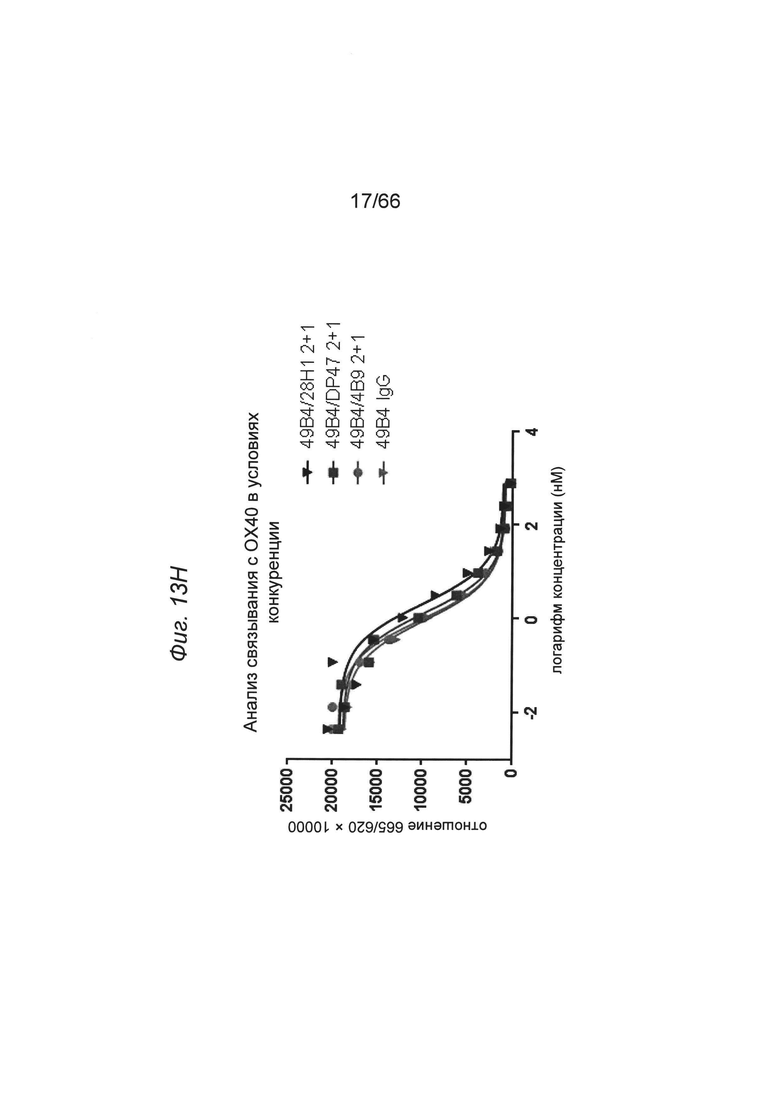

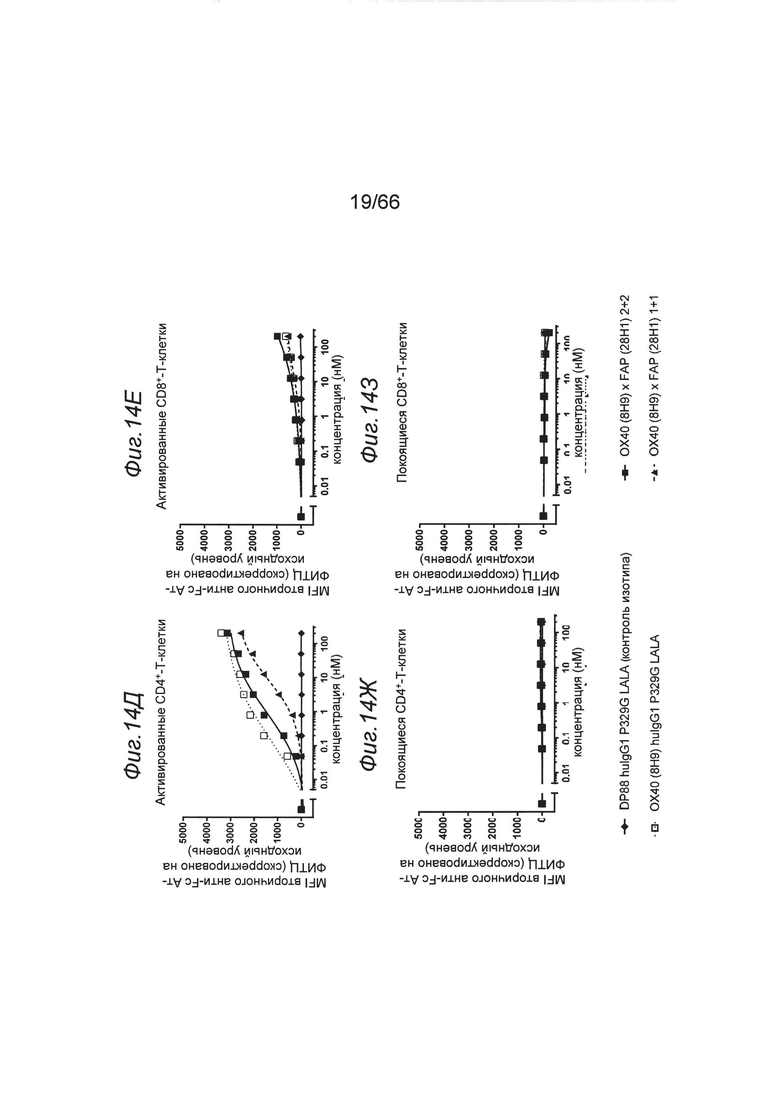



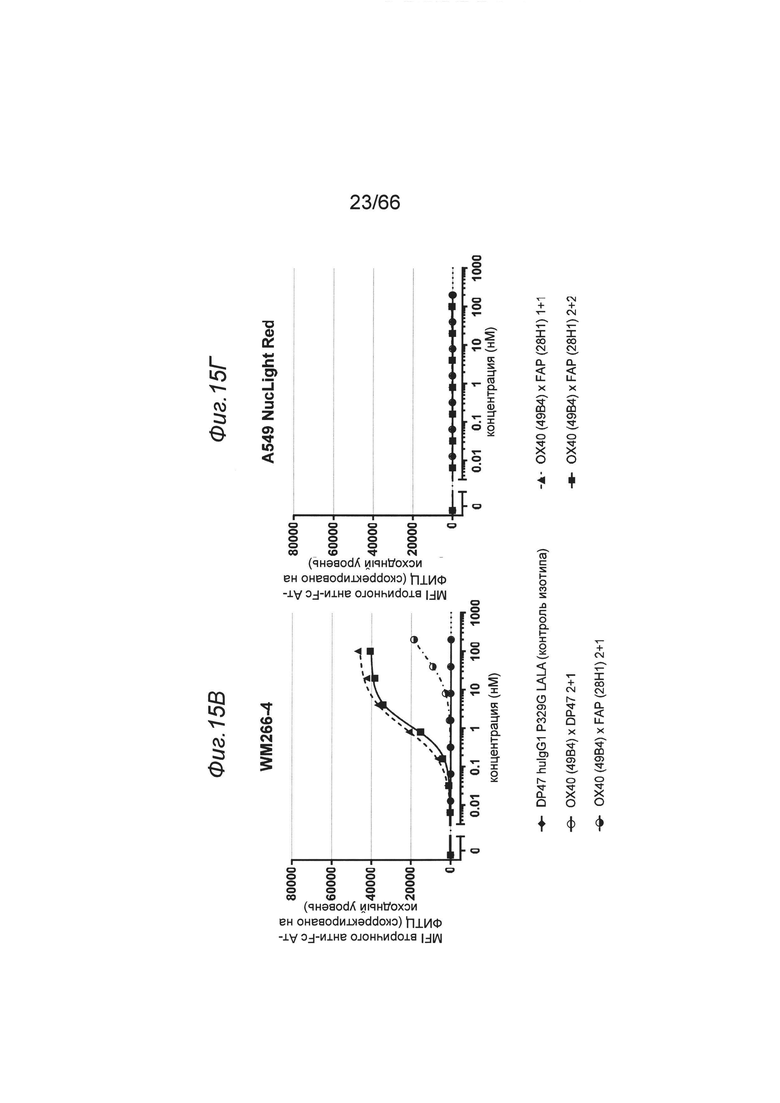
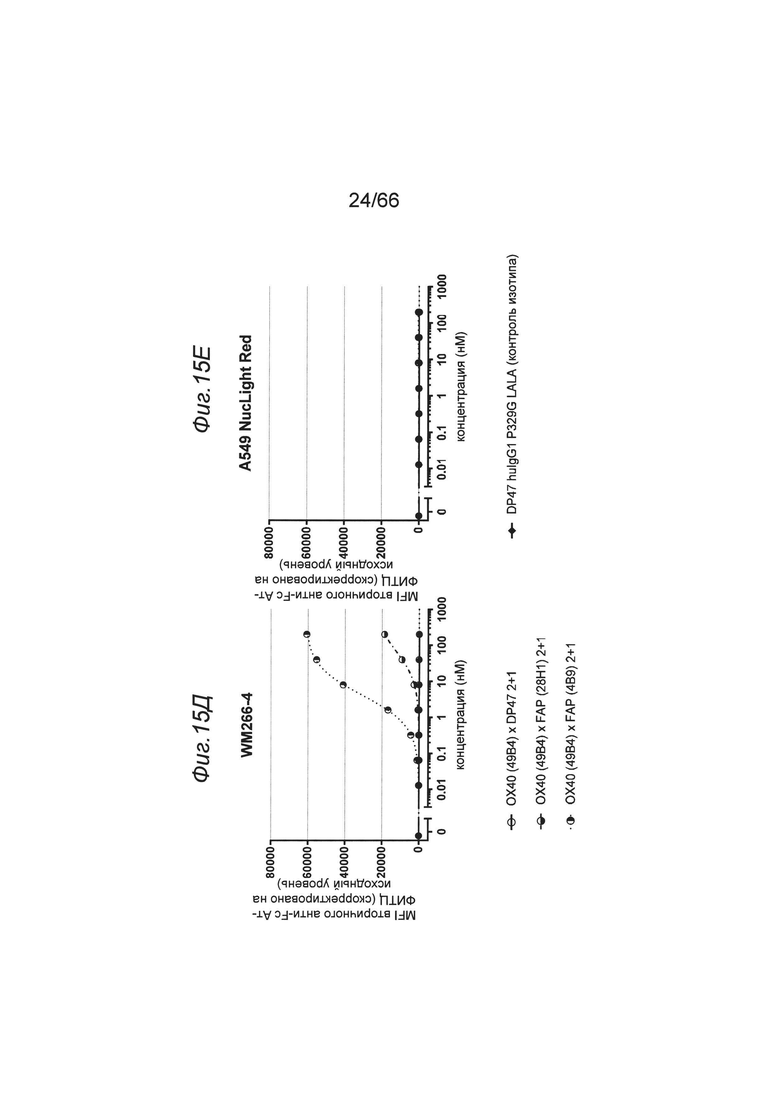
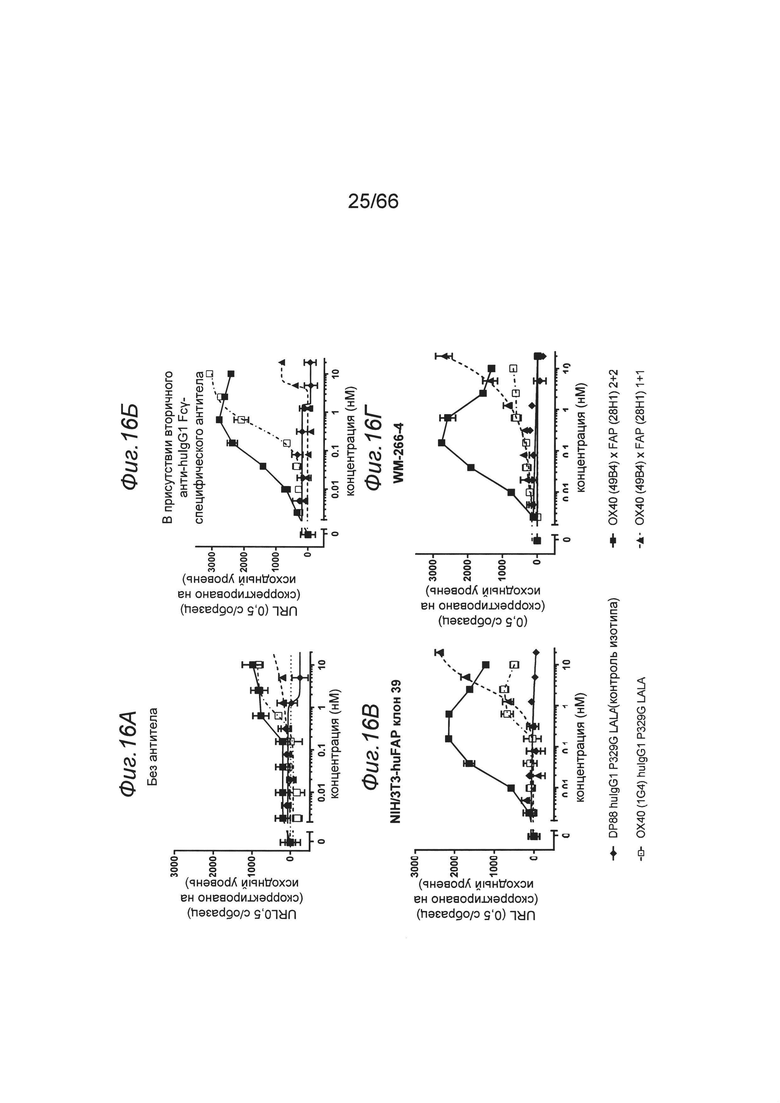
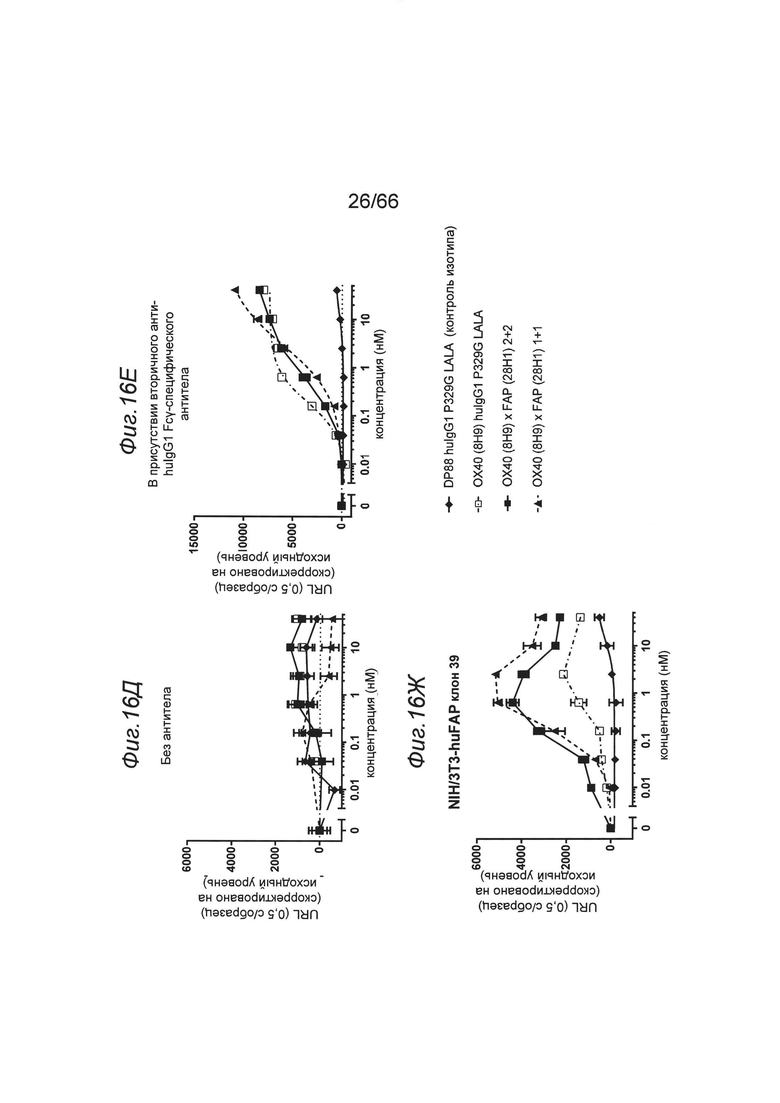
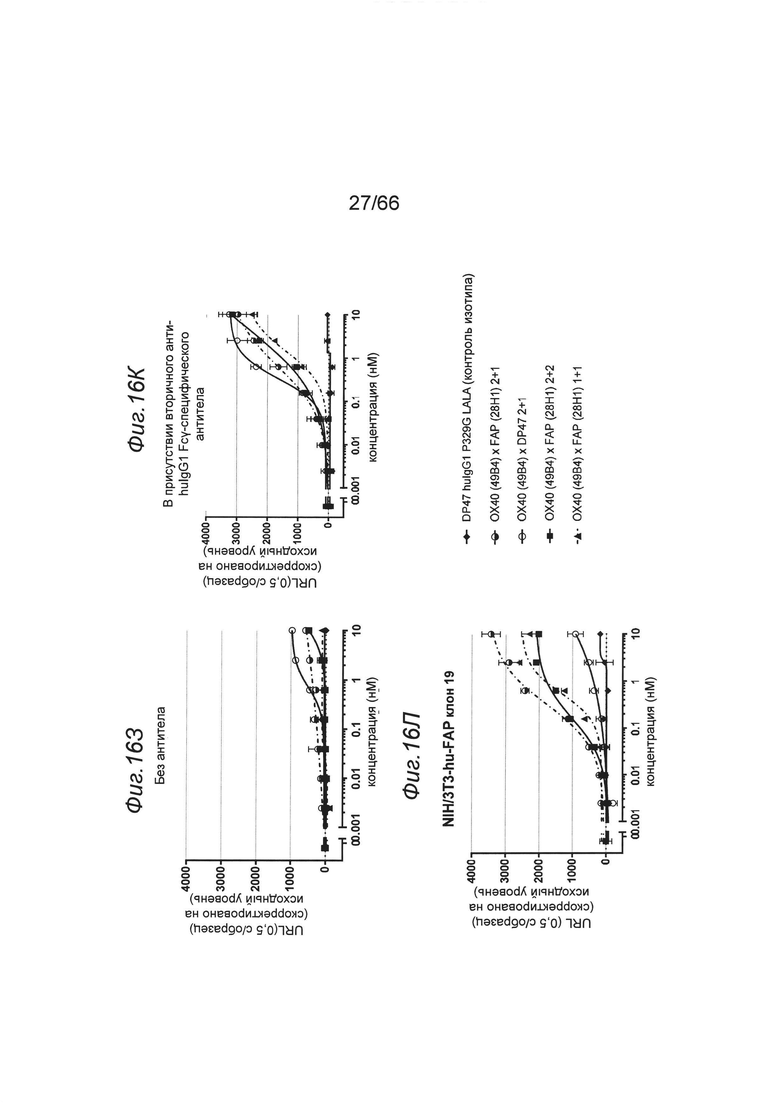
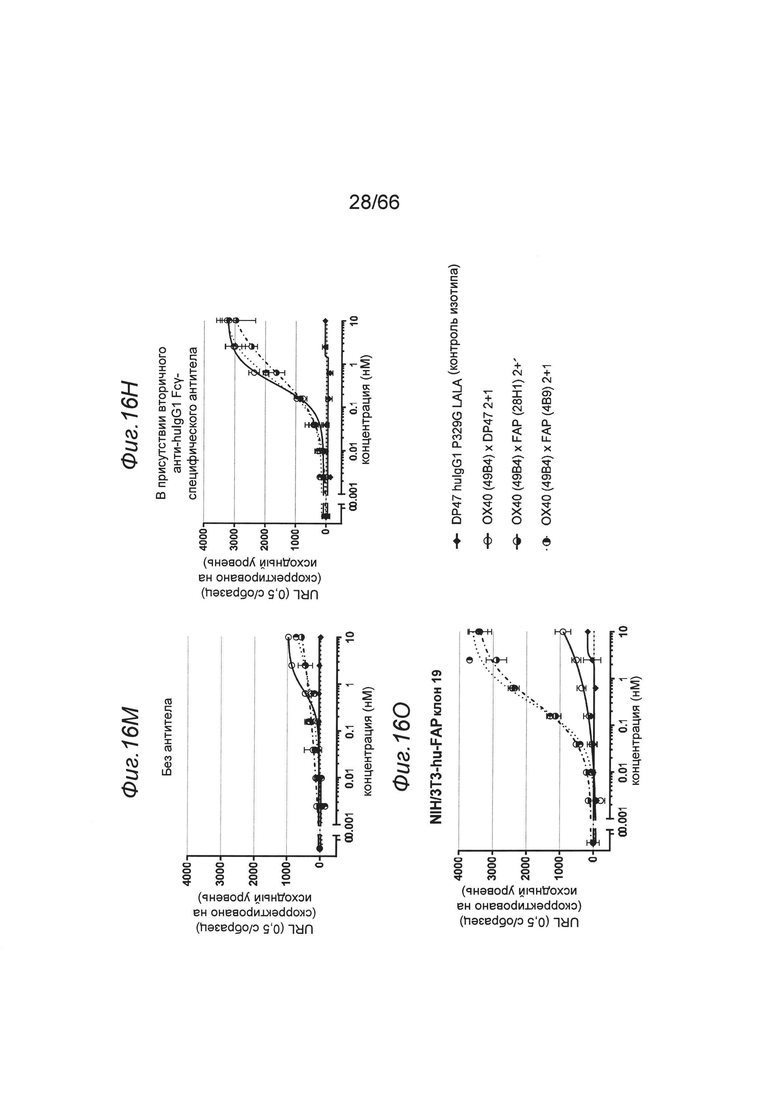

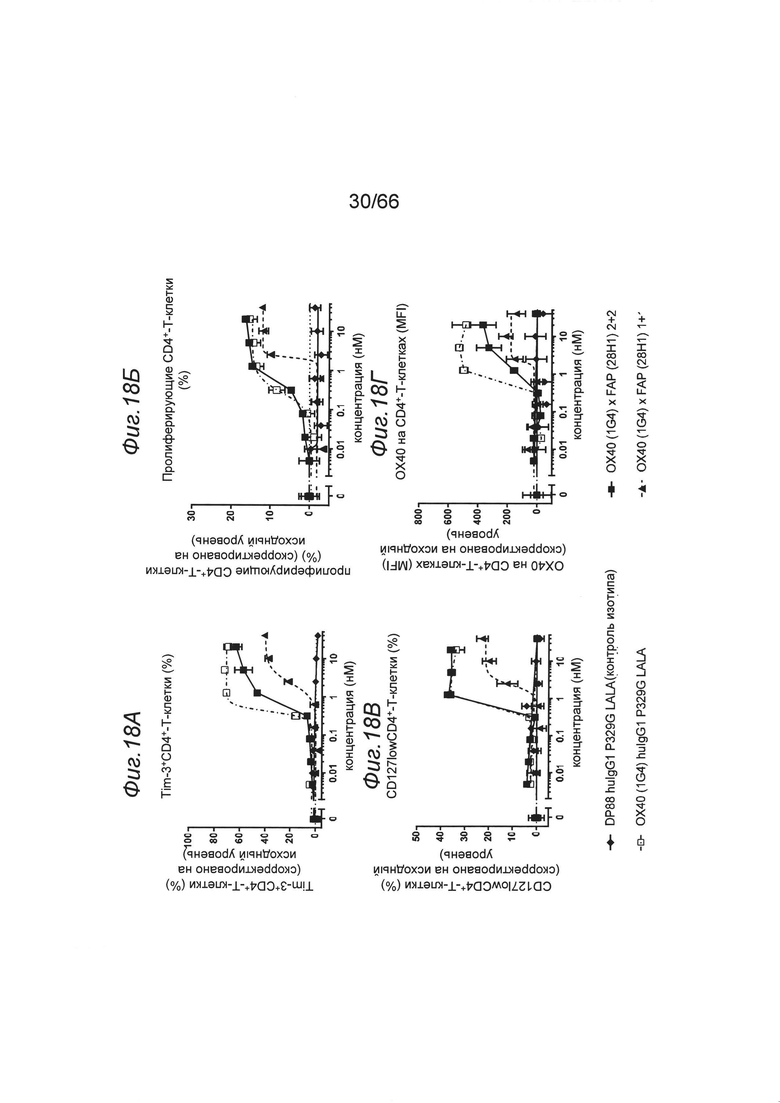

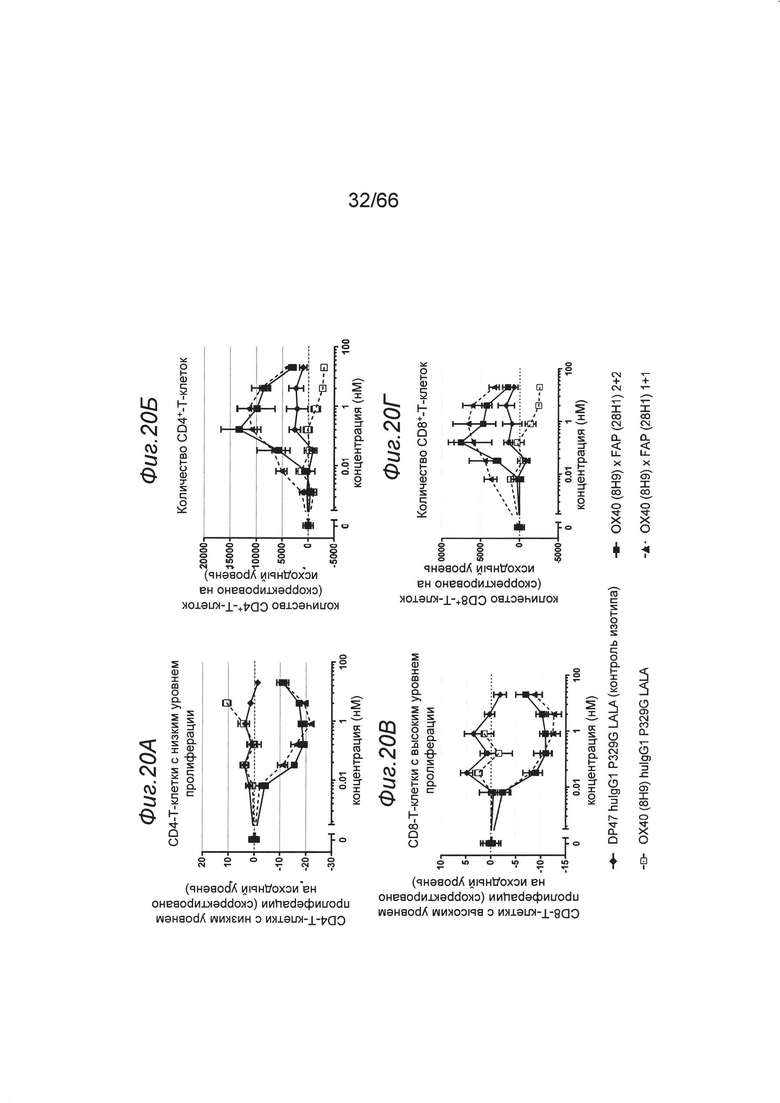

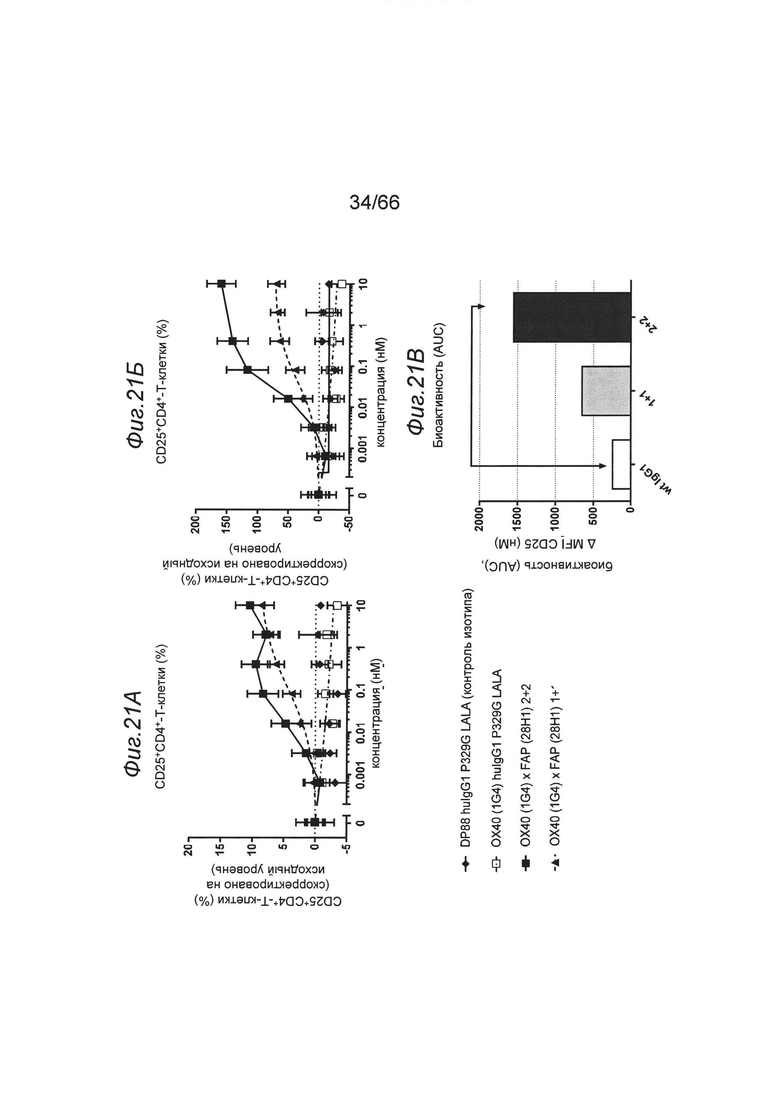
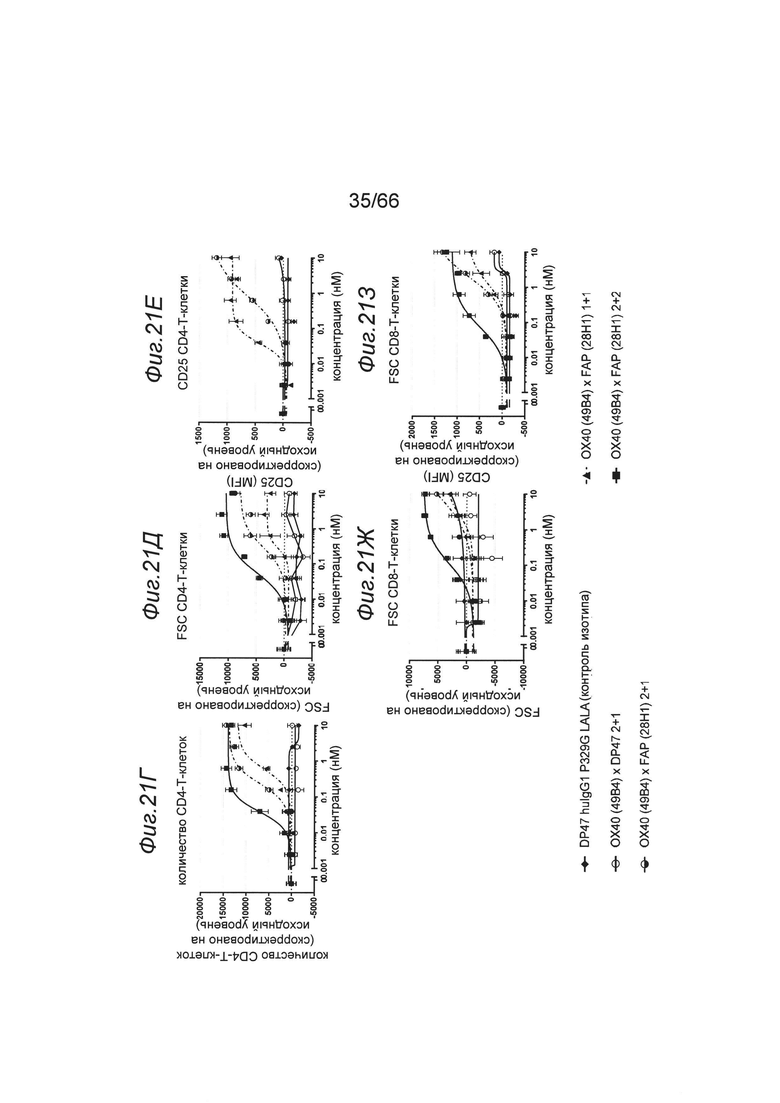
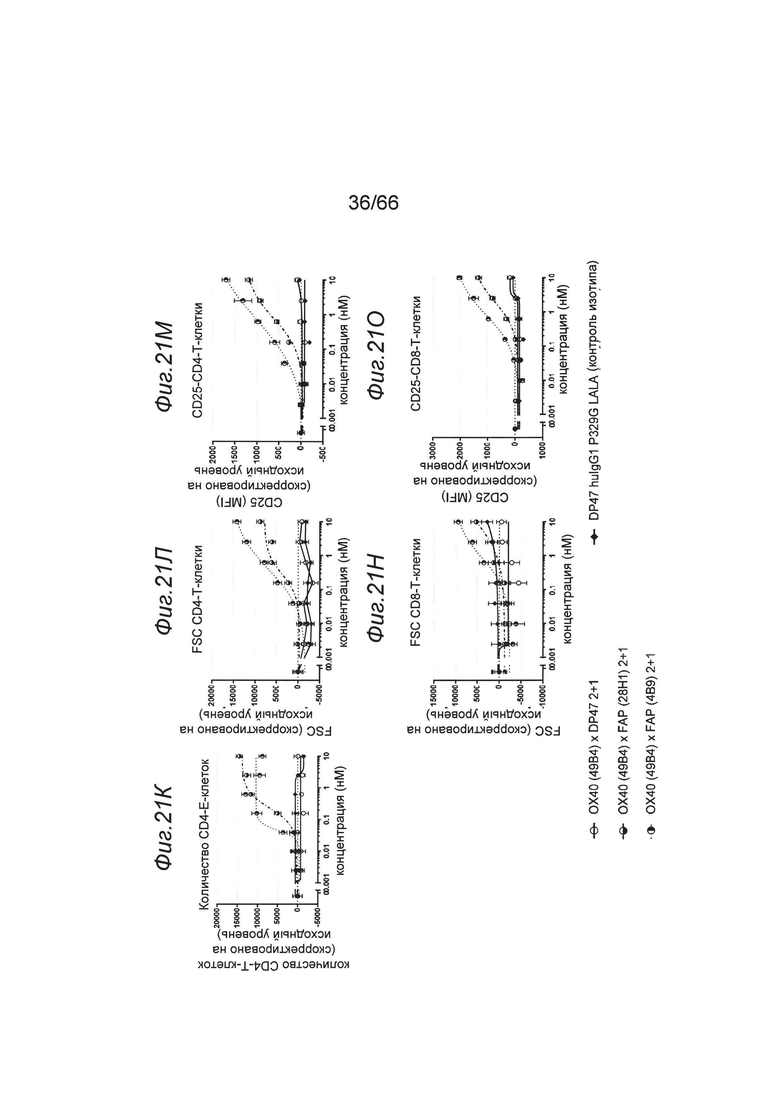
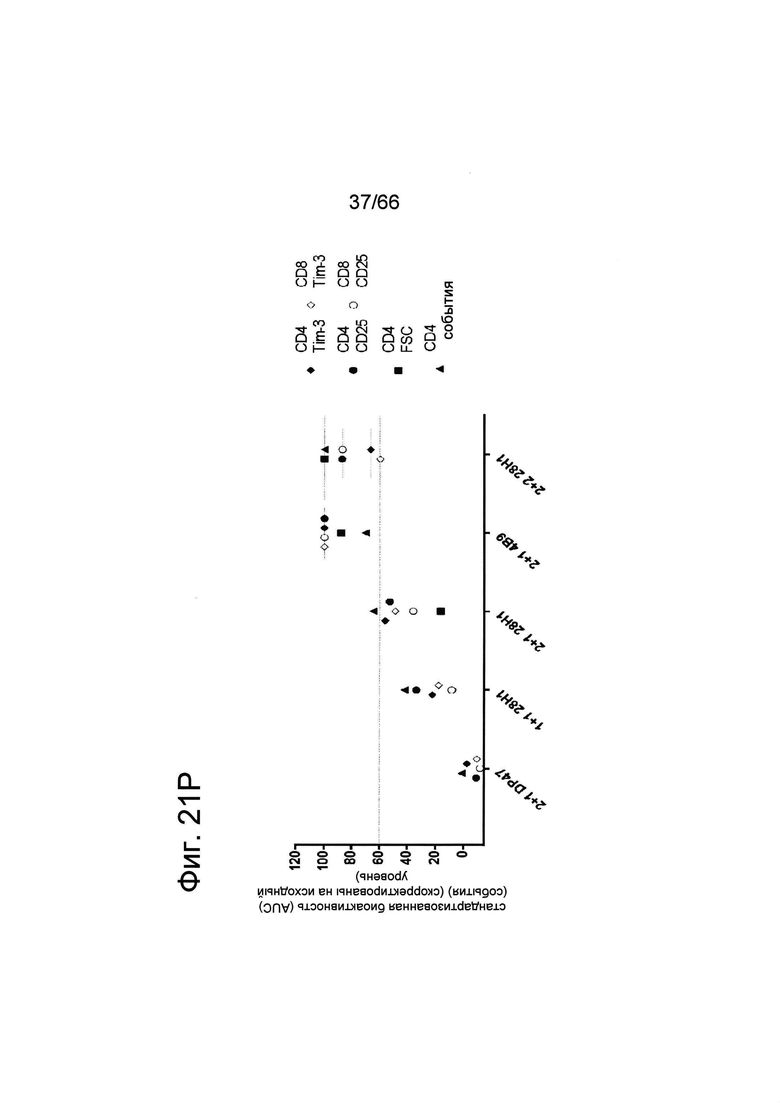
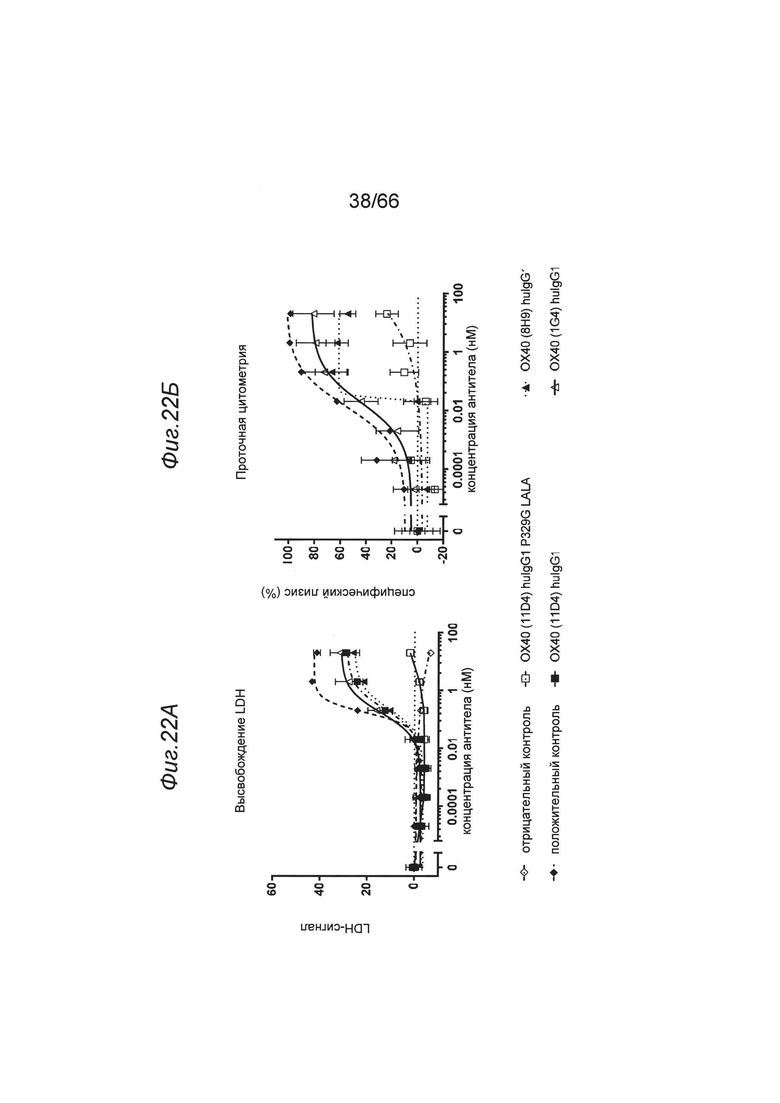

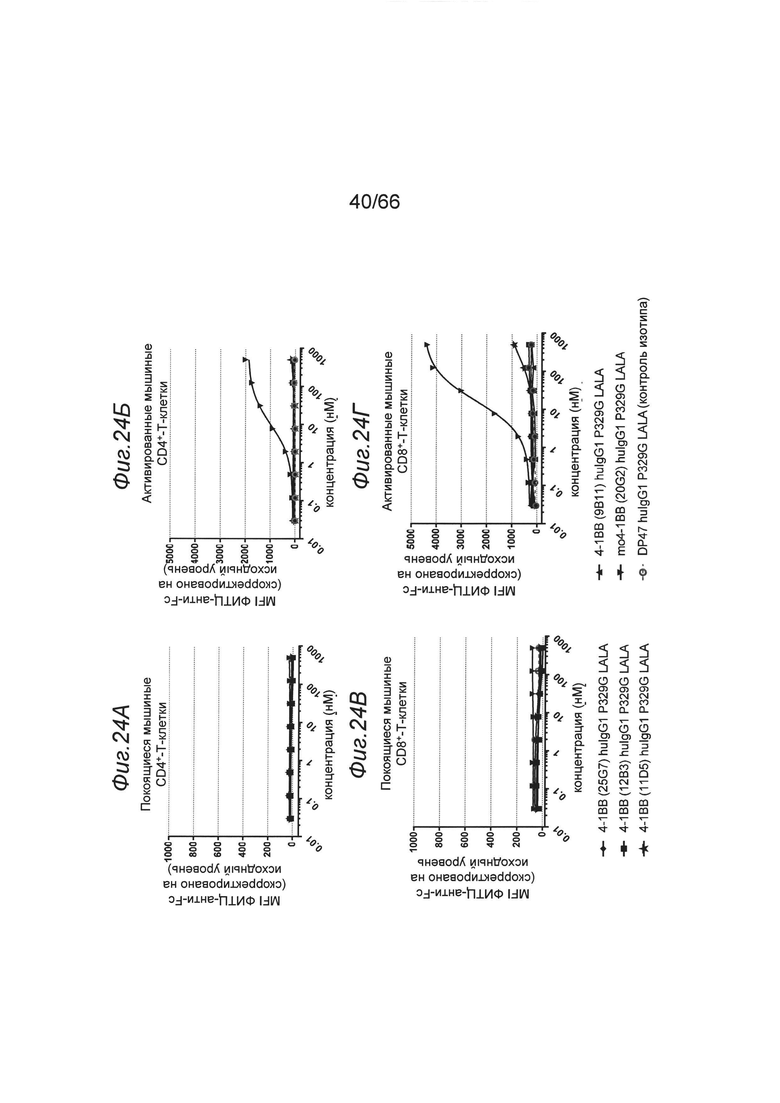
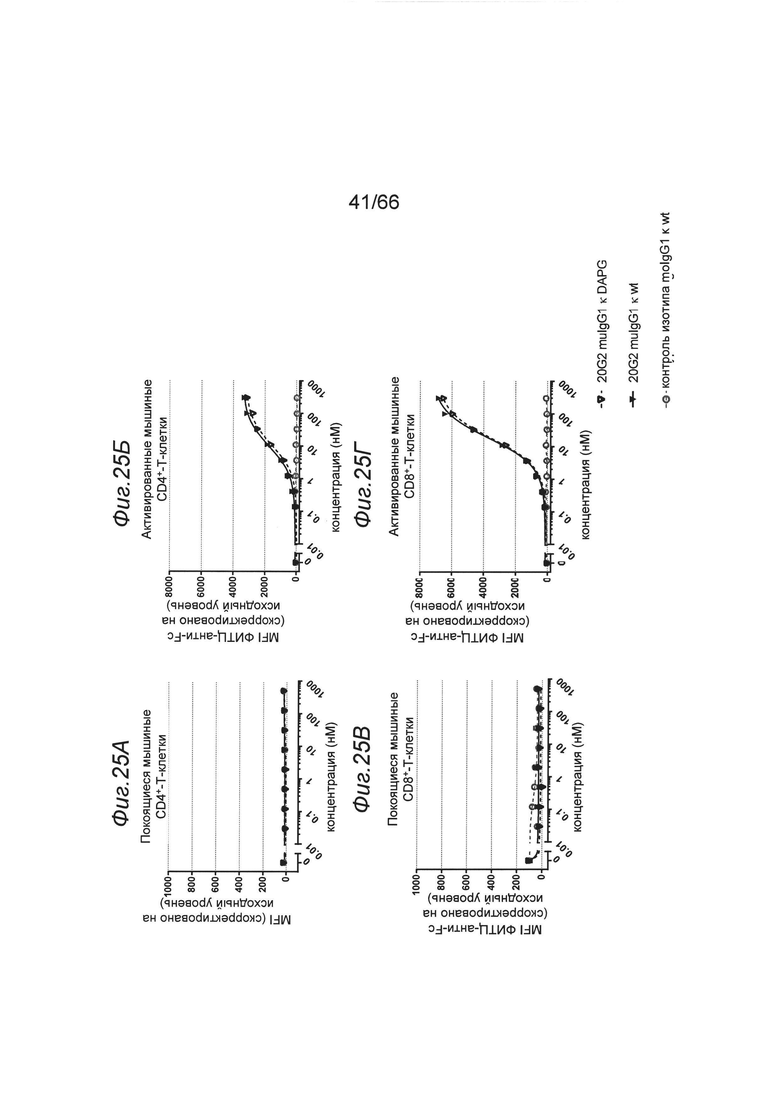
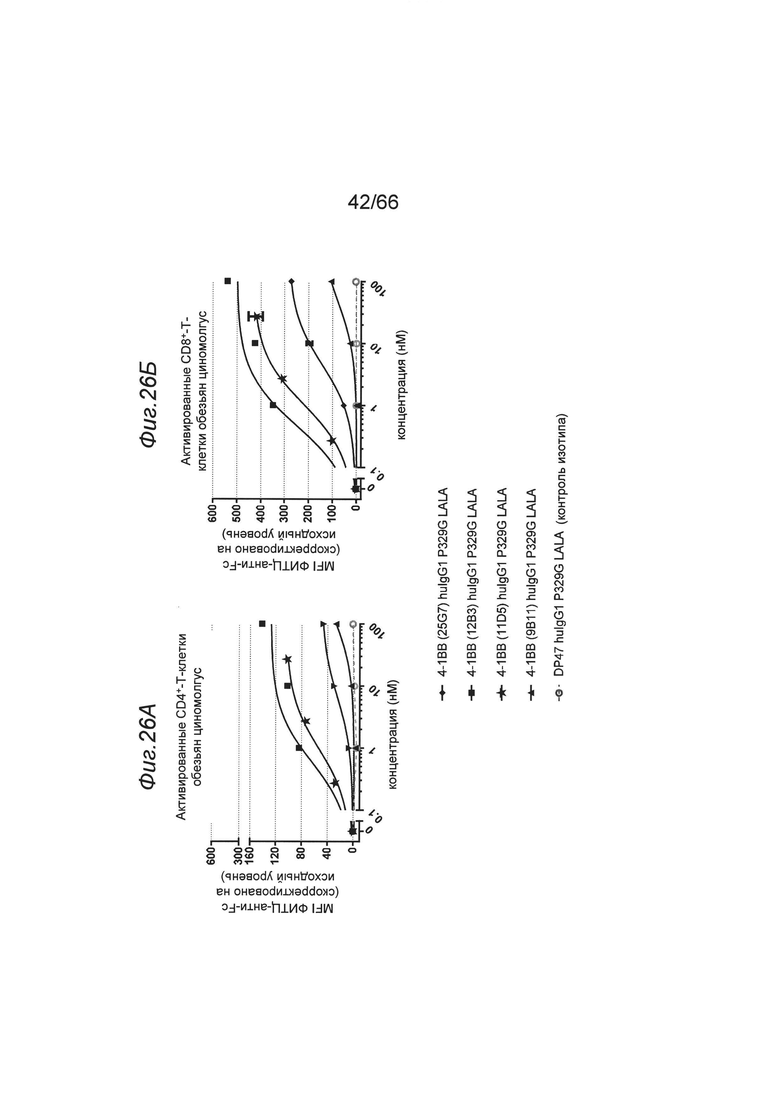
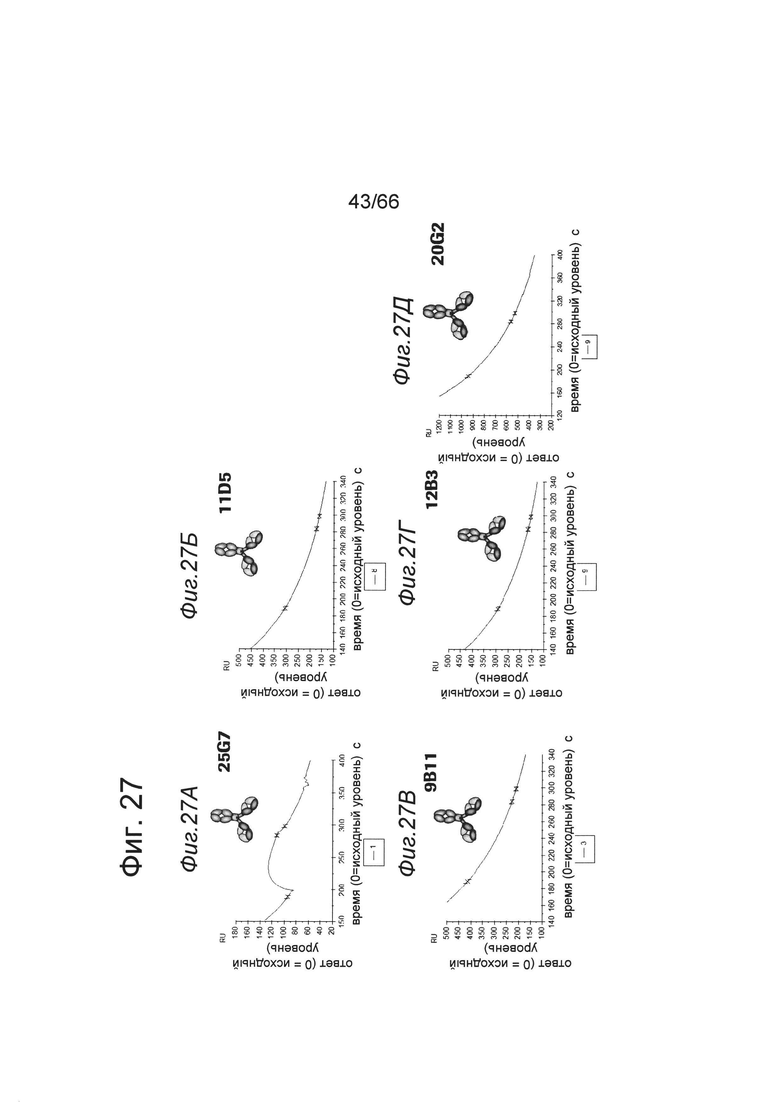

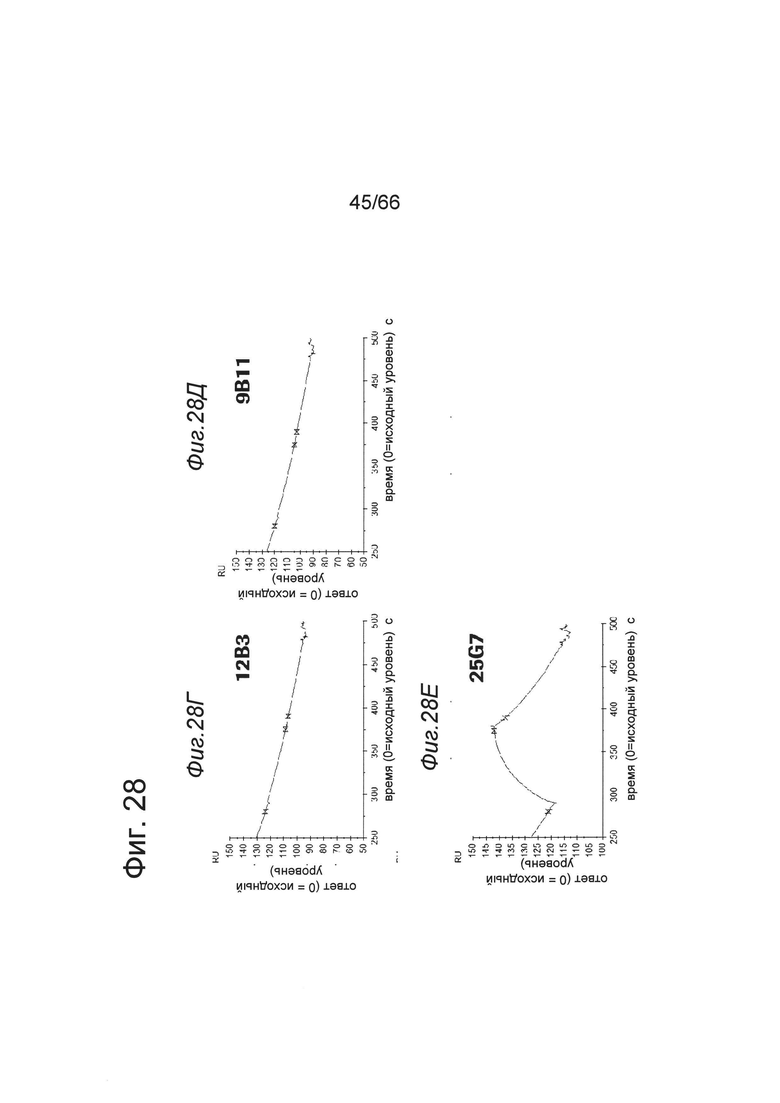


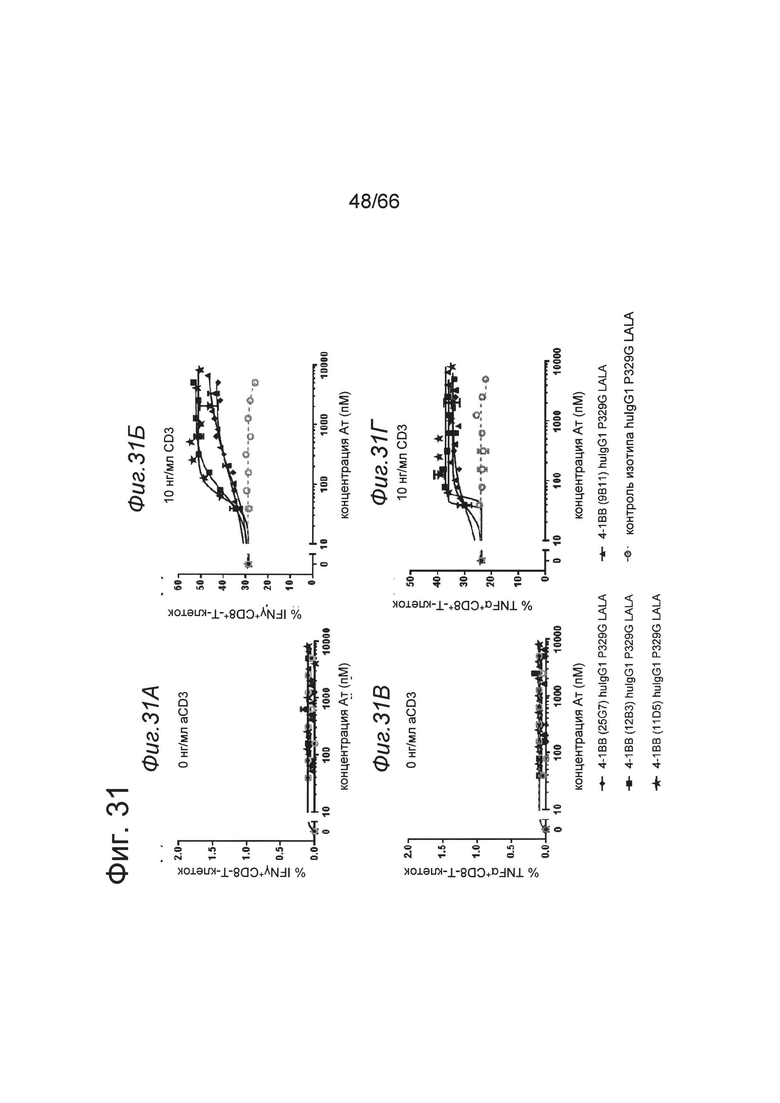
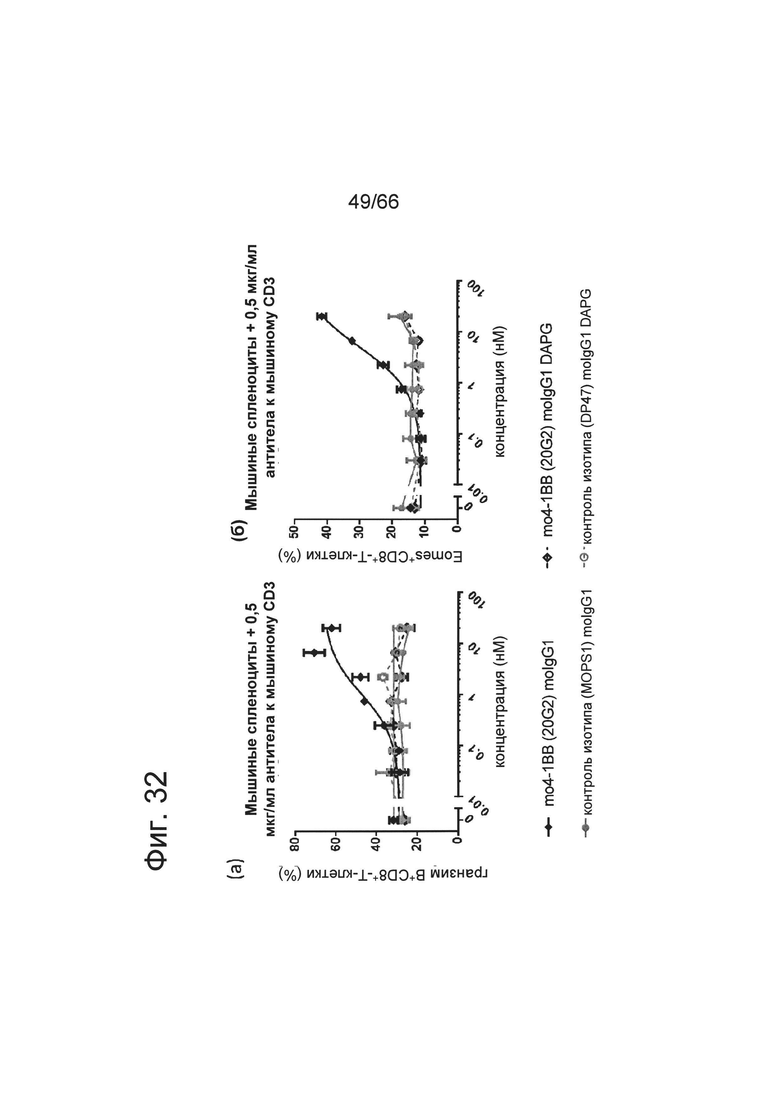
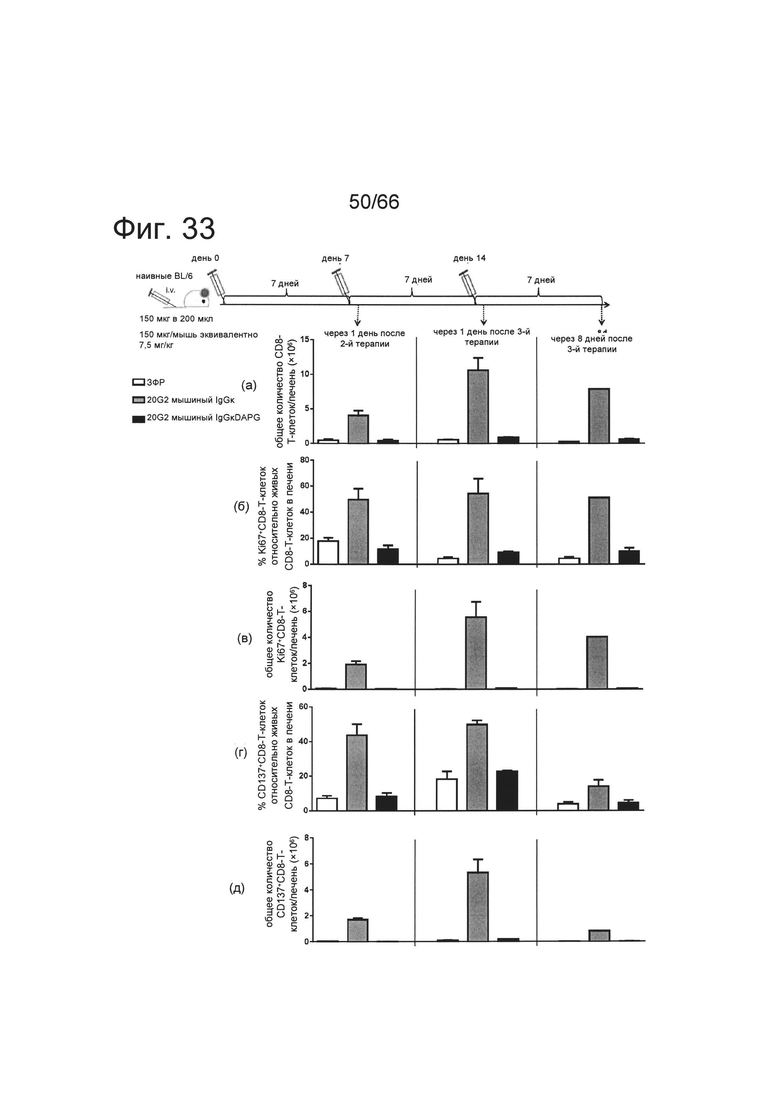
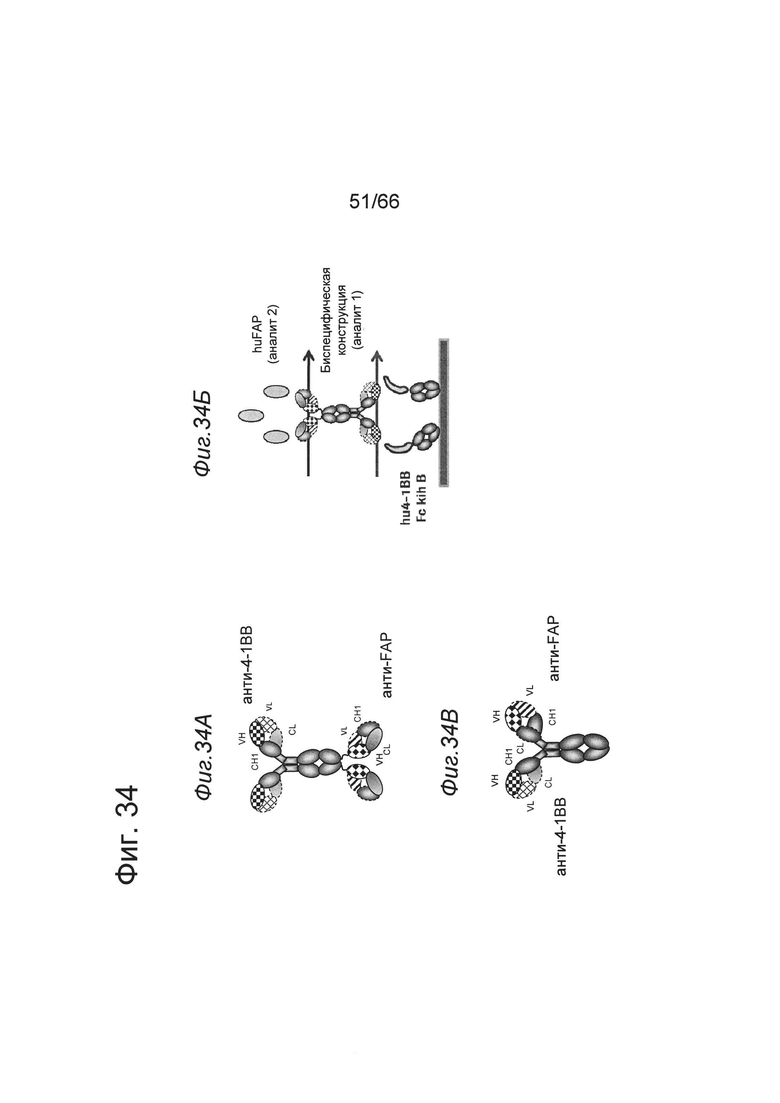
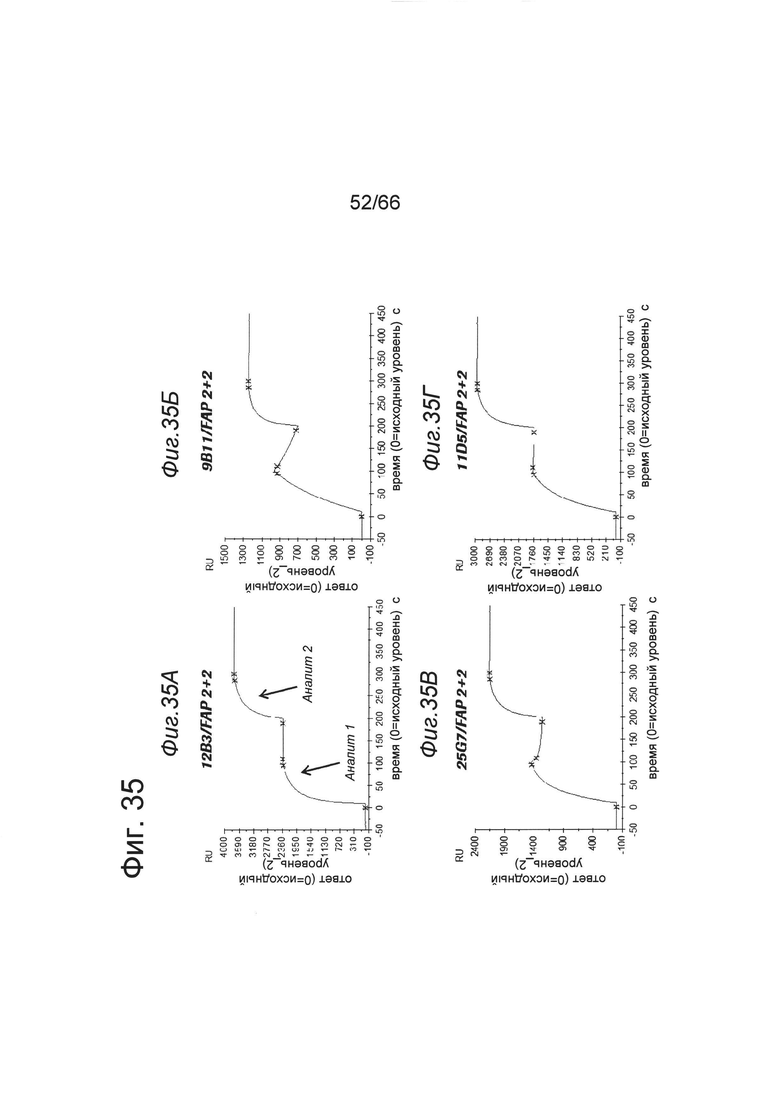
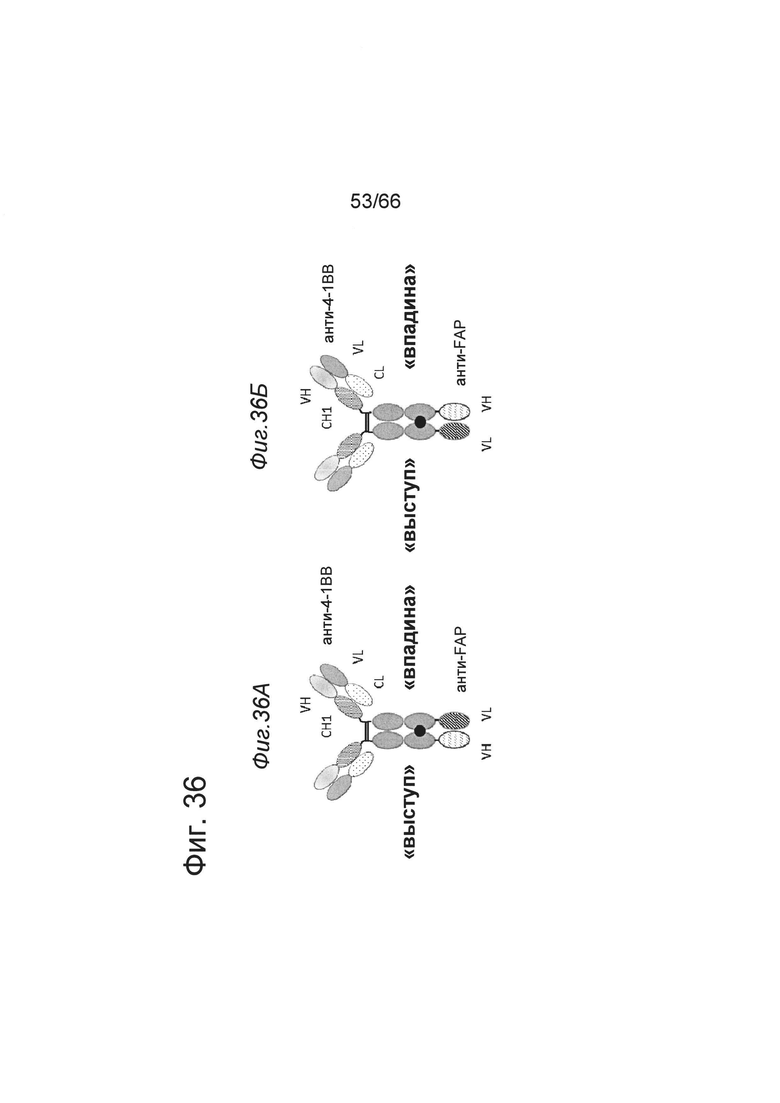
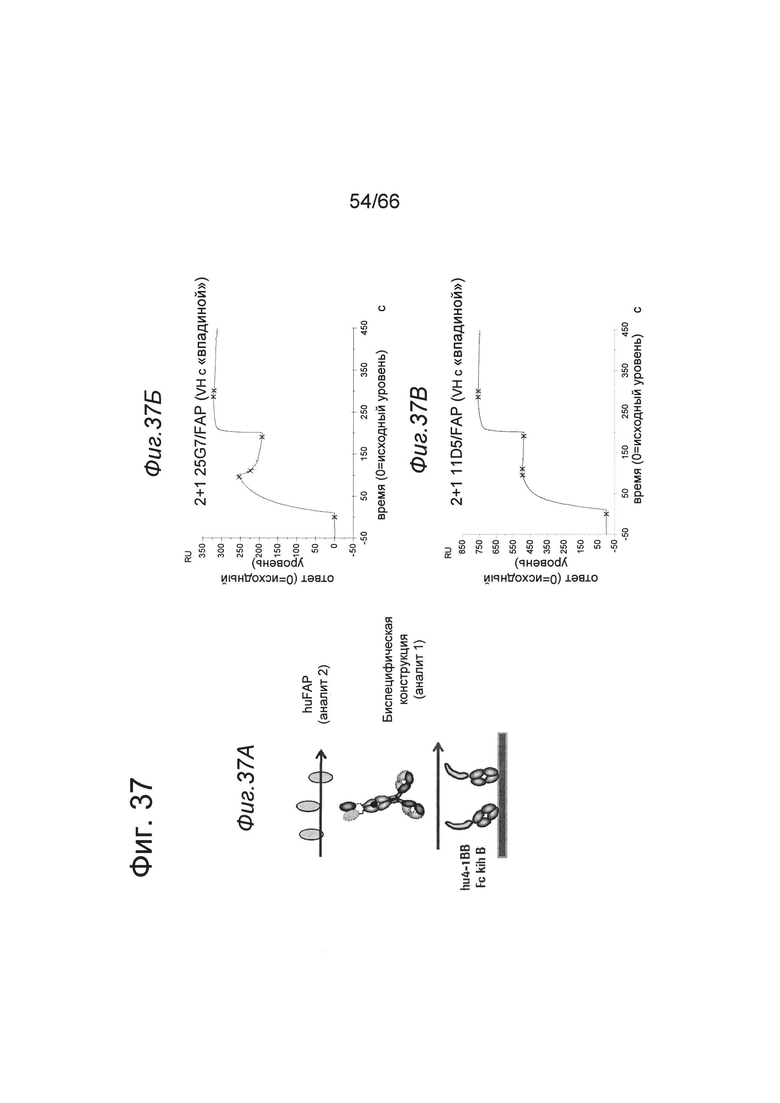


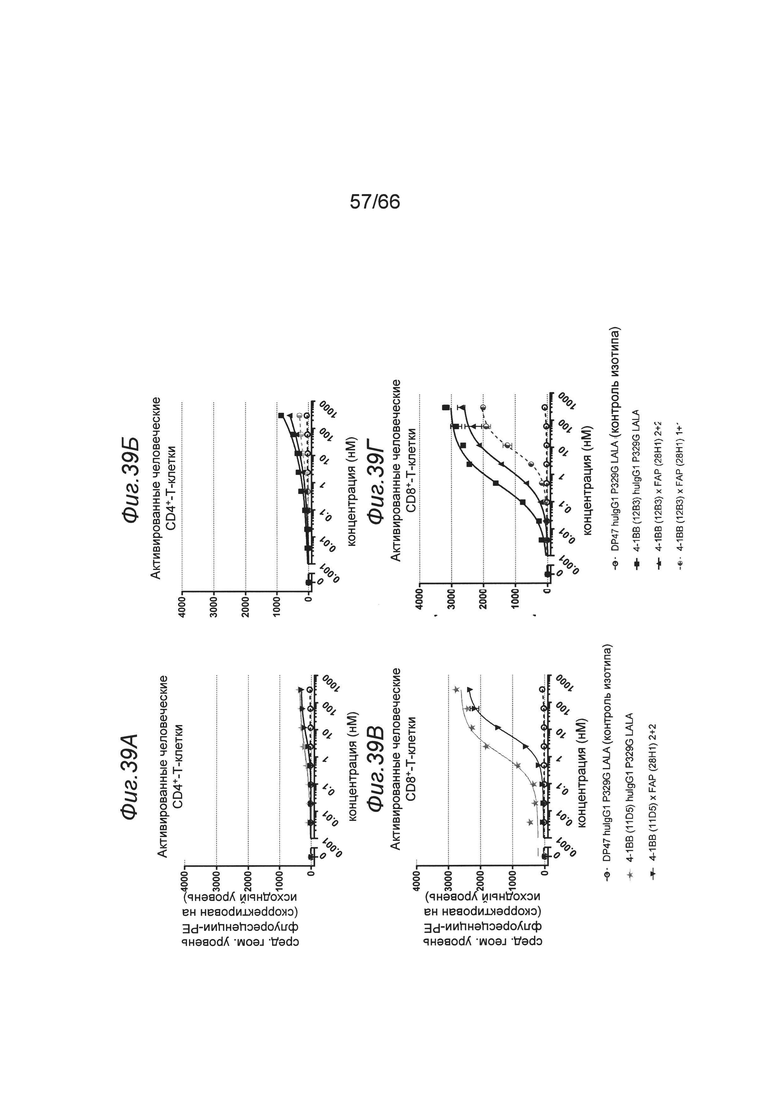

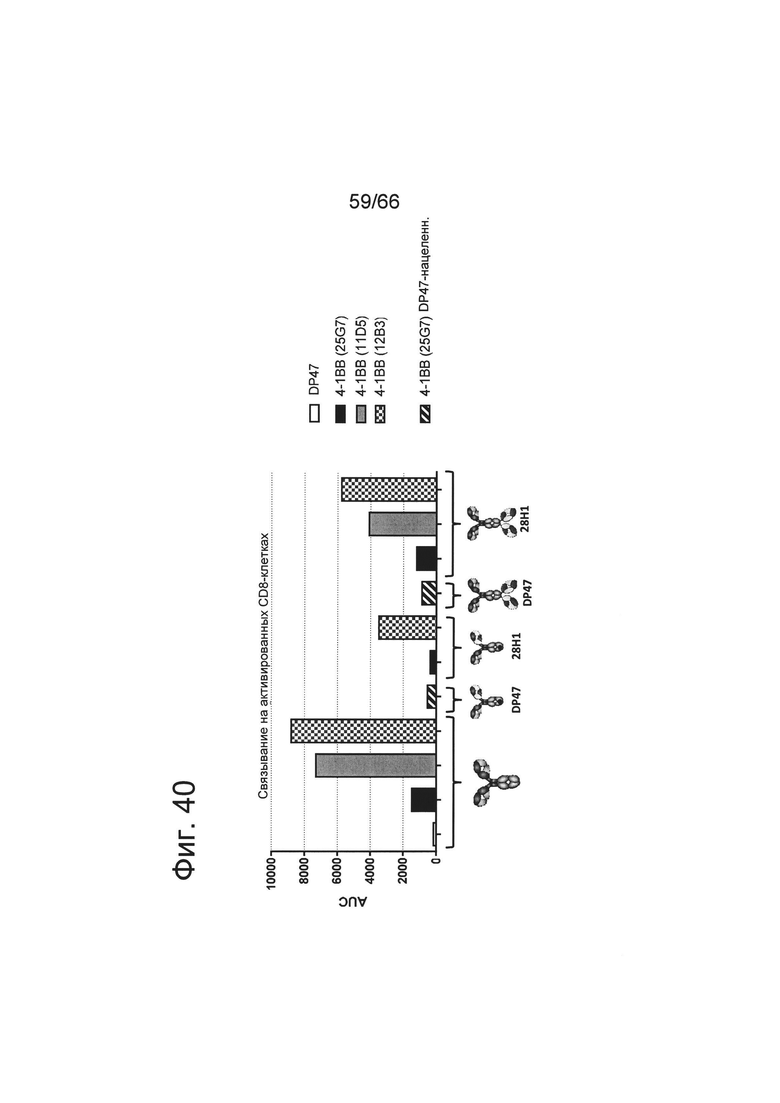
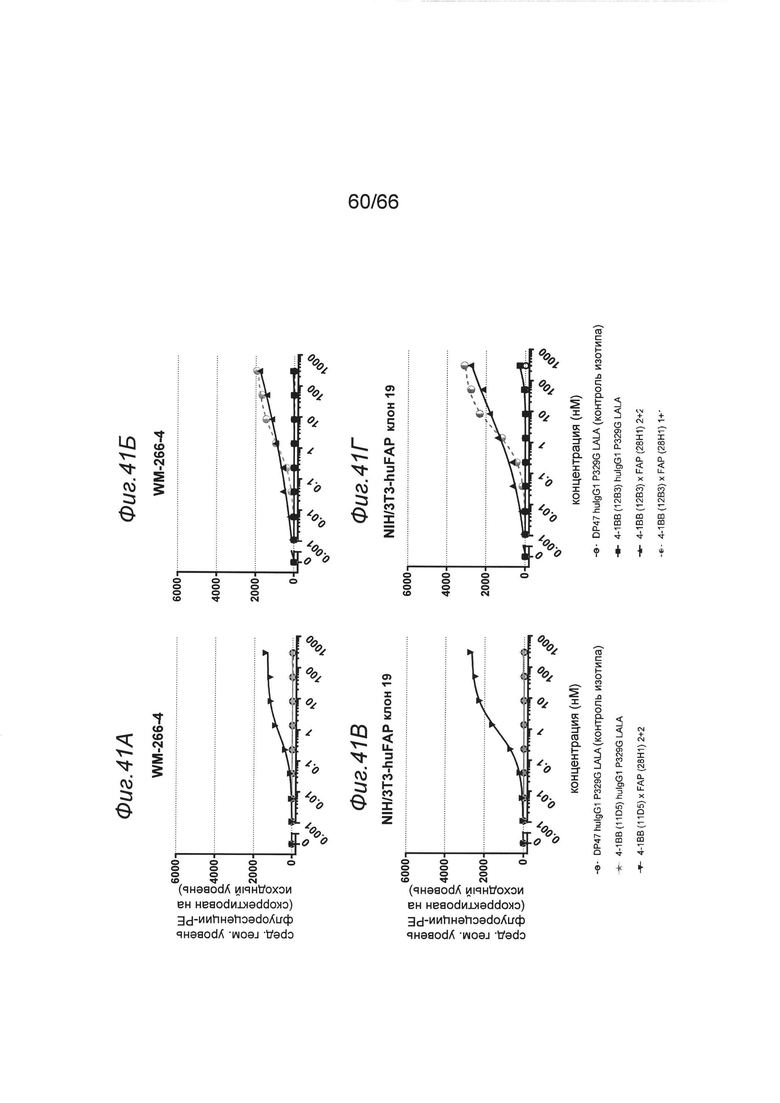

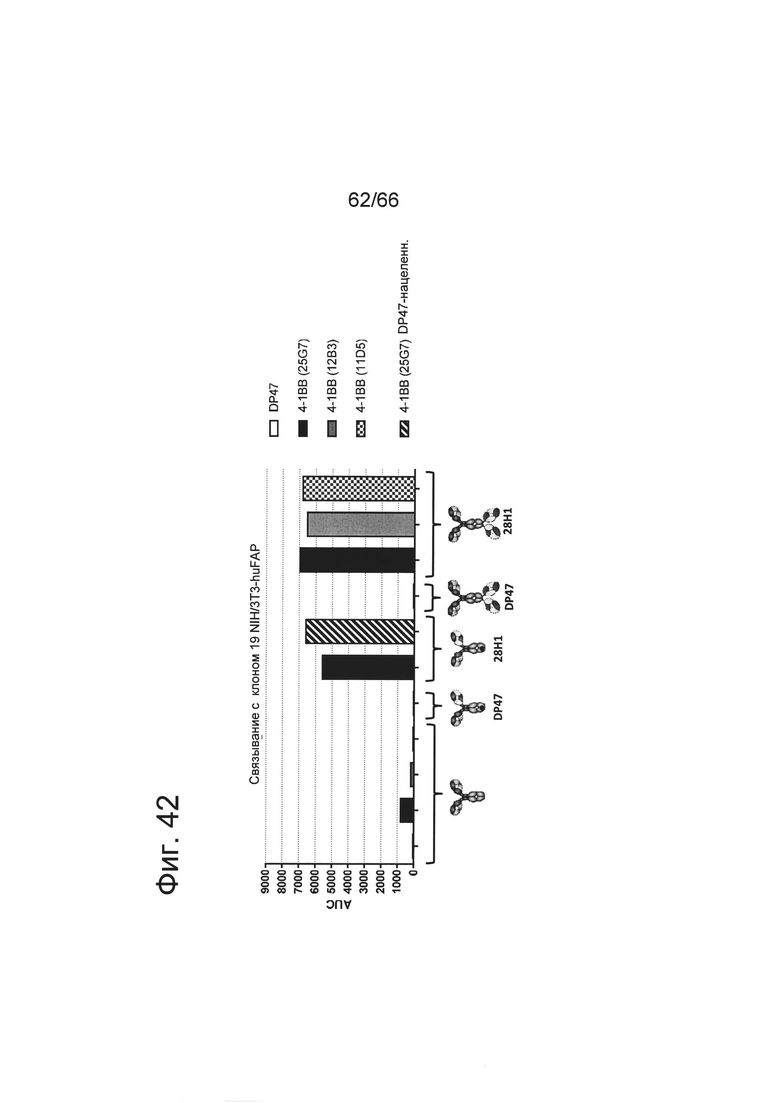
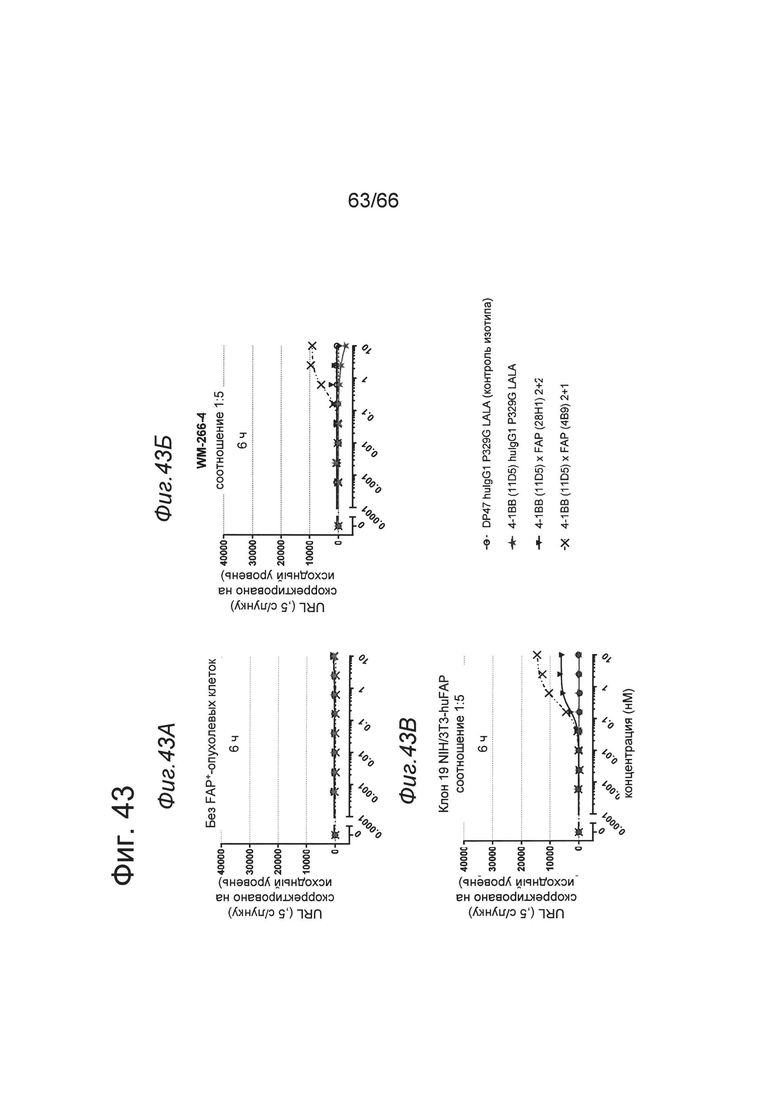


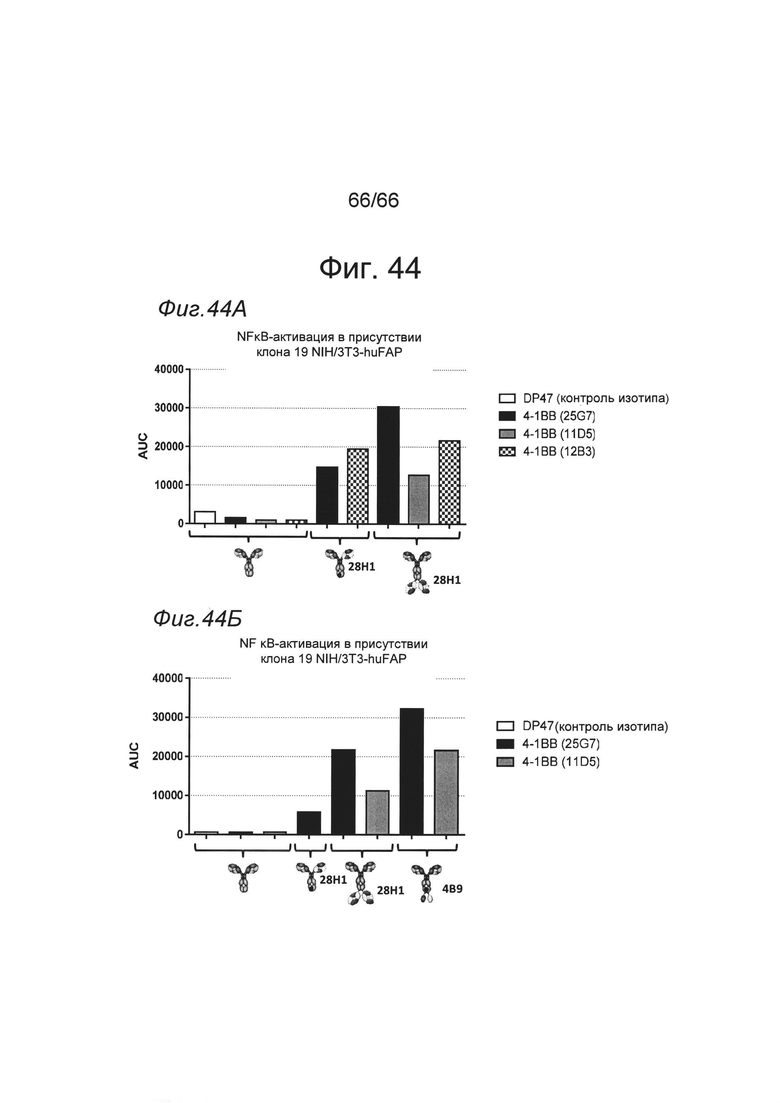
Изобретение относится к области биотехнологии, в частности к биспецифической антигенсвязывающей молекуле, связывающейся с ОХ40 и FAP, которая содержит по меньшей мере один Fab-фрагмент, обладающий способностью специфически связываться с ОХ40, и по меньшей мере один фрагмент, обладающий способностью специфически связываться с фибробласт-активирующим белком (FAP), а также к содержащей ее композиции. Также раскрыт полинуклеотид, кодирующий вышеуказанную молекулу. Изобретение эффективно для стимулирования Т-клеточного ответа. 4 н. и 7 з.п. ф-лы, 44 ил., 73 табл., 11 пр.
1. Биспецифическая антигенсвязывающая молекула, связывающаяся с ОХ40 и FAP, которая содержит
(а) по меньшей мере один Fab-фрагмент, обладающий способностью специфически связываться с ОХ40, где указанный Fab-фрагмент включает
(I) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 26,
(II) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28,
(III) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 29, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 30,
(IV) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 31, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 32,
(V) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 33, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 34, или
(VI) вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 35, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 36,
(б) по меньшей мере один фрагмент, обладающий способностью специфически связываться с фибробласт-активирующим белком (FAP), где указанный фрагмент, обладающий способностью специфически связываться с FAP, включает вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO:83, и
(в) Fc-домен, состоящий из первой и второй субъединиц, которые обладают способностью к стабильной ассоциации, где Fc-домен относится к подклассу человеческого IgGl с аминокислотными заменами L234A, L235A и P329G (нумерация согласно EU-индексу Кэбота).
2. Биспецифическая антигенсвязывающая молекула по п. 1, в которой
(I) Fab-фрагмент, обладающий способностью специфически связываться с ОХ40, содержит вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 27, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 28, и
(II) фрагмент, обладающий способностью специфически связываться с FAP, содержит вариабельную область тяжелой цепи VH, которая содержит аминокислотную последовательность SEQ ID NO: 82, и вариабельную область легкой цепи VL, которая содержит аминокислотную последовательность SEQ ID NO: 83.
3. Биспецифическая антигенсвязывающая молекула по п. 1 или 2, в которой Fc-домен содержит модификацию, усиливающую ассоциацию первой и второй субъединиц Fc-домена.
4. Биспецифическая антигенсвязывающая молекула по любому из пп. 1-3, в которой первая субъединица Fc-домена содержит «выступы», а вторая субъединица Fc-домена содержит «впадины» в соответствии с технологией «knob-into-hole».
5. Биспецифическая антигенсвязывающая молекула по любому из пп. 1-4, в которой первая субъединица Fc-домена содержит аминокислотные замены S354C и T366W (нумерация согласно EU-индексу Кэбота), а вторая субъединица Fc-домена содержит аминокислотные замены Y349C, T366S и Y407V (нумерация согласно EU-индексу Кэбота).
6. Биспецифическая антигенсвязывающая молекула по п. 1, где биспецифическая антигенсвязывающая молекула является двухвалентной как в отношении ОХ40, так и FAP.
7. Биспецифическая антигенсвязывающая молекула по п. 1, где биспецифическая антигенсвязывающая молекула является двухвалентной в отношении ОХ40 и одновалентной в отношении FAP.
8. Биспецифическая антигенсвязывающая молекула по любому из пп. 1-5 или 7, содержащая
(а) две легкие цепи и две тяжелые цепи антитела, содержащего два Fab-фрагмента, которые обладают способностью специфически связываться с FAP, и Fc-домен, и
(б) VH- и VL-домен, обладающие способностью специфически связываться с FAP, где VH-домен соединен через пептидный линкер с С-концом одной из тяжелых цепей и где VL-домен соединен через пептидный линкер с С-концом второй тяжелой цепи.
9. Полинуклеотид, кодирующий биспецифическую антигенсвязывающую молекулу по любому из пп. 1-8.
10. Фармацевтическая композиция для применения в стимулировании Т-клеточного ответа, содержащая терапевтически эффективное количество биспецифической антигенсвязывающей молекулы по любому из пп. 1-8 и по меньшей мере один фармацевтически приемлемый эксципиент.
11. Применение биспецифической антигенсвязывающей молекулы по любому из пп. 1-8 для стимуляции Т-клеточного ответа.
| WO 2013092001 A1, 27.06.2013 | |||
| US 2014370019 A1, 18.12.2014 | |||
| US 2013243772 A1, 19.09.2013 | |||
| WO 2010145792 A1, 23.12.2010 | |||
| WO 2012020006 A2, 16.02.2012 | |||
| WO 2012130831 A1, 04.10.2012 | |||
| RU 2014109551 A, 27.09.2015. |
Авторы
Даты
2021-12-06—Публикация
2016-09-29—Подача