Область изобретения
Настоящее изобретение относится к вакцинным конструктам и их применениям против инфекций стафилококка. Более конкретно, настоящее изобретение относится к вакцинным конструктам, сочетающим антигены, и их применению против инфекций стафилококка, таких как внутримаммарные инфекции крупного рогатого скота (IMI).
Ссылка на перечень последовательностей
В соответствии с 37 C.F.R. 1.821 (c), перечень последовательностей представлен в настоящей заявке в виде ASCII-совместимого текстового файла с именем Перечень последовательностей USP62411120_ST25, созданного 5 октября 2017 года и имеющего размер 278 килобайт. Содержимое вышеупомянутого файла полностью включено в настоящее описание посредством ссылки.
Уровень техники
Мастит крупного рогатого скота является наиболее частым и дорогостоящим заболеванием для производителей молочных продуктов, а Staphylococcus aureus считается переносимой бактерией, которая наиболее часто ответственна за развитие заболевания (Sears et al., 2003). Стафилококковая IMI, которая может привести к маститу, трудно поддается лечению, и частые рецидивы встречаются (Sandholm et al., 1990).
Разработка вакцин для профилактики и борьбы с IMI S. aureus была тщательно исследована, хотя ни одна формула не продемонстрировала защитной эффективности до настоящего времени. Вероятно, это связано с неадекватными вакцинными мишенями (Middleton, 2008; Middleton 2009), большим разнообразием штаммов, способных провоцировать мастит (Buzzola, 2007; Kerro-Dego, 2006; Middleton, 2008) или неспособностью вызвать соответствующий иммунный ответ (Bharathan, 2011; Ference, 2000; Fowler, 2014; Proctor, 2012). Становится все более очевидным, что иммунитет, основанный исключительно на антителах, индуцированных вакциной, может быть важен, но, тем не менее, недостаточен для индукции защиты от S. aureus (Middleton 2008; Middleton 2009). Похоже, что клеточный иммунитет (CMI) на основе ответов типа Th1 и Th17 может быть необходим для завершения защиты (Fowler, 2014; Lin, 2009; Proctor, 2012; Spellberg, 2012).
Восприимчивость бактерий к антибиотикам in vitro является плохим показателем терапевтической эффективности у хронически инфицированных коров (Owens et al., 1997). Хотя инфекции, которые следуют за лечением мастита, могут быть вызваны вновь приобретенными штаммами, они часто являются результатом персистенции исходного инфекционного организма (Sandholm et al., 1990; Myllys et al., 1997). Таким образом, существующие методы лечения часто не в состоянии устранить инфекцию, и было бы крайне желательно найти новые подходы для профилактики или лечения стафилококковой IMI.
Недостаток эффективности и защитной способности вакцины был отмечен для коммерчески доступных вакцин против S. aureus (Middleton, 2008). Таким образом, было бы крайне желательно использовать высокоэффективные антигены S. aureus, которые, как известно, экспрессируются во время IMI, в качестве компонентов вакцины для защиты от IMI и мастита.
Настоящее изобретение предназначено для удовлетворения этих и других потребностей.
Настоящее описание ссылается на ряд документов, содержание которых включено в настоящий документ посредством ссылки во всей их полноте.
Сущность изобретения
Первым объектом настоящего изобретения являются слитые полипептиды, проявляющие повышенную иммуногенность, и их применение в качестве вакцины против стафилококковой IMI.
Характерной чертой стафилококка, такого как IMI S. aureus, является способность S. aureus сохраняться в клетках хозяина. В частности, варианты малых колоний S. aureus (SCV) обычно не вызывают инвазивных инфекций и могут быть интернализованы в клетках-хозяевах. Таким образом, следующим объектом настоящего изобретения являются живые аттенуированные штаммы S. aureus для целей вакцины на основе фенотипических аспектов SCV, чтобы обеспечить иммунный ответ против таких штаммов и повысить защитную эффективность вакцины.
Настоящее изобретение обеспечивает следующие объекты:
Пункт 1: Слитный конструкт формулы (I): X-A-линкер-B-Z (I), где: (1) A и B являются идентичными или различными и независимо друг от друга представляют собой: (a) полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131), SACOL0264 полипептид (SEQ ID NO: 185), SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92), SACOL0718 полипептид (SEQ ID NO: 186), SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120), SACOL1353 полипептид (SEQ ID NO: 187), SACOL1416 полипептид (SEQ ID NO: 188), SACOL1611 полипептид (SEQ ID NO: 189), SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164), SACOL1912 полипептид (SEQ ID NO: 43), SACOL1944 полипептид (SEQ ID NO: 190), SACOL2144 полипептид (SEQ ID NO: 191), SACOL2365 полипептид (SEQ ID NO: 192), SACOL2385 полипептид (SEQ ID NO: 50) или SACOL2599 полипептид (SEQ ID NO: 193), на основе номенклатуры гена из генома Staphylococcus aureus COL (SACOL), изложенного в NCBI эталонной последовательности NC_002951.2; (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (c); (2) линкер представляет собой аминокислотную последовательность из по меньшей мере одной аминокислоты или отсутствует; (3) X отсутствует или представляет собой аминокислотную последовательность из по меньшей мере одной аминокислоты; и (4) Z отсутствует или представляет собой аминокислотную последовательность из по меньшей мере одной аминокислоты.
Пункт 2: Конструкт по пункту 1, где (1) (a) представляет собой полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131), SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92), SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120), или SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164).
Пункт 3: Конструкт по пункту 2, где по меньшей мере один из A и B представляет собой (a) полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131); (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (d); и другой один из A и B представляет собой (a') полипептид, содержащий SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164); (b') полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a'); (c') полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a') или (b'); (d') полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из (a') - (c'); или (e') полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 12 последовательных аминокислот согласно любому из (a') - (d').
Пункт 4: Конструкт по пункту 2, где по меньшей мере один из A и B представляет собой (a) полипептид, содержащий SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92); (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (d); и другой один из A и B представляет собой (a') полипептид, содержащий SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120); (b') полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a'); (c') полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a') или (b'); (d') полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из (a') - (c'); или (e') полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 12 последовательных аминокислот согласно любому из (a') - (d').
Пункт 5: Конструкт по пункту 2, где A и B являются идентичными или различными и представляют собой (a) полипептид, содержащий SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120); (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (d).
Пункт 6: Конструкт по любому из п.п. 1-2 и 4, где указанный иммуногенный фрагмент (d) содержит одну или более из следующих аминокислотных последовательностей: KDTINGKSNKSRNW (SEQ ID NO: 34); и KDGGKYTLESHKELQ (SEQ ID NO: 1).
Пункт 7: Конструкт по пункту 6, где указанный иммуногенный фрагмент (d) содержит одну или более из следующих аминокислотных последовательностей: STQNSSSVQDKQLQKVEEVPNNSEKALVKKLYDRYSKDTINGKSNKSRNWVYSERPLNENQVRI HLEGTYTVAGRVYTPKRNITLNKEVVTLKELDHIIRFAHISYGLYMGEHLPKGNIVINTKDGGKY TLESHKELQKDRENVKINTADIKNVTFKLVKSVNDIEQV (SEQ ID NO: 30); DKQLQKVEEVPNNSEKALVKKLYDRYSKDTINGKSNKSRNWVYSERPLNENQVRIHLEGTYTVA GRVYTPKRNITLNKEVVTLKELDHIIRFAHISYGLYMGEHLPKGNIVINTK (SEQ ID NO :32); и DKQLQKVEEVPNNSEKALVKKLYDRYSKDTINGKSNKSRNWVYSERPLNENQVRIHLEGTYTVA GRVYTPKRNITLNKEVVTLKELDHIIRFAHISYGLYMGEHLPKGNIVINTKDGGKYTLESHKELQK DRENVKINTADIKNVTFKLVKSVNDIEQV (SEQ ID NO: 33).
Пункт 8: Конструкт по любому из п.п. 1-2 и 4-5, где указанный иммуногенный фрагмент (d) содержит одну или более из следующих аминокислотных последовательностей: QFGFDLKHKKDALA (SEQ ID NO: 21); TIKDQQKANQLAS (SEQ ID NO: 22); KDINKIYFMTDVDL (SEQ ID NO: 23); и DVDLGGPTFVLND (SEQ ID NO: 24).
Пункт 9: Конструкт по пункту 8, где указанный иммуногенный фрагмент (d) содержит одну или более из следующих аминокислотных последовательностей: RASLSSEIKYTAPHDVTIKDQQKANQLASELNNQKIPHFYNYKEVIHTKLYKDNLFDVKAKEPYN VTITSDKYIPNTDLKRGQADLFVAEGSIKDLVKHKKHGKAIIGTKKHHVNIKLRKDINKIYFMTDV DLGGPTFVLNDKDYQEIRKYTKAKHIVSQFGFDLKHKKDALALEKAKNKVDKSIETRSEAISSISS LTG (SEQ ID NO: 12);
ASLSSEIKYTAPHDVTIKDQQKANQLASELNNQKIPHFYNYKEVIHTKLYKDNLFDVKAK EPYNVTITSDKYIPNTDLKRGQADLFVAEGSIKDLVKHKKHGKAIIGTKKHHVNIKLRKDINKIYF MTDVDLGGPTFVLNDKDYQEIRKYTKAKHIVSQFGFDLKHKKDALALEKAKNKVDKSIETRSEA ISSISSLTG (SEQ ID NO: 13);
ASLSSEIKYTAPHDVTIKDQQKANQLASELNNQKIPHFYNYKEVIHTKLYKDNLFDVKAK EPYNVTITSDKYIPNTDLKRGQADLFVAEGSIKDLVKHKKHGKAIIGTKKHHVNIKLRKDINKIYF MTDVDLGGPTFVLNDKDYQE (SEQ ID NO: 14);
KDINKIYFMTDVDLGGPTFVLNDKDYQEIRKYTKAKHIVSQFGFDLKHKKDALA (SEQ ID NO: 15); KDINKIYFMTDVDLGGPTFVLNDKD (SEQ ID NO: 16);
KDINKIYFMTDVDLGGPTFVLNDKDY (SEQ ID NO: 17);
KDINKIYFMTDVDLGGPTFVLND (SEQ ID NO: 19);
SQFGFDLKHKKDALA (SEQ ID NO: 18); и KHIVSQFGFDLKHKKDALA (SEQ ID NO: 20).
Пункт 10: Конструкт по любому из п.п. 1-3, где указанный иммуногенный фрагмент (c') содержит одну или более из следующих аминокислотных последовательностей: PYNGVVSFKDATGF (SEQ ID NO: 165); AHPNGDKGNGGIYK (SEQ ID NO: 167); SISDYPGDEDISVM (SEQ ID NO: 169); RGPKGFNFNENVQA (SEQ ID NO: 172); QFESTGTIKRIKDN (SEQ ID NO: 175); и GNSGSPVLNSNNEV (SEQ ID NO: 178).
Пункт 11: Конструкт по пункту 10, где указанный иммуногенный фрагмент (d) содержит следующую аминокислотную последовательность: TQVKDTNIFPYNGVVSFKDATGFVIGKNTIITNKHVSKDYKVGDRITAHPNGDKGNGGIYKIKSIS DYPGDEDISVMNIEEQAVERGPKGFNFNENVQAFNFAKDAKVDDKIKVIGYPLPAQNSFKQFEST GTIKRIKDNILNFDAYIEPGNSGSPVLNSNNEVIGVVYGGIGKIGSEYNGAVYFTPQIKDFIQKHIEQ (SEQ ID NO: 39).
Пункт 12: Конструкт по любому из п.п. 1 - 11, где указанный линкер содержит по меньшей мере четыре идентичные или различные аминокислоты, выбранные из группы, состоящей из глицина, серина, аланина, аспартата, глутамата и лизина.
Пункт 13: Конструкт по любому из п.п. 1 - 12, где указанный линкер содержит (GGGGS)n (SEQ ID NO: 67), (ERKYK)n (SEQ ID NO: 61); или (EAAAK)n (SEQ ID NO: 63), где n=1 - 5.
Пункт 14: Конструкт по любому из п.п. 1 - 13, где указанный X содержит полигистидин из от 6 до 10 аминокислот.
Пункт 15: Конструкт по любому из п.п. 1 - 13, где указанный X отсутствует.
Пункт 16: Конструкт по любому из п.п. 1 - 15, где указанный Z отсутствует.
Пункт 17: Выделенная молекула нуклеиновой кислоты, кодирующая конструкт, как определено в любом из п.п. 1 - 16.
Пункт 18: Вектор, содержащий выделенную нуклеиновую кислоту, определенную в пункте 17.
Пункт 19: Клетка-хозяин, содержащая вектор, определенный в пункте 18.
Пункт 20: Клетка по пункту 19, которая представляет собой живую аттенуированную форму Staphylococcus aureus.
Пункт 21: Клетка по пункту 20, где живая аттенуированная форма Staphylococcus aureus имеет стабилизированный фенотип варианта мелких колоний (SCV).
Пункт 22: Клетка по пункту 21, где живая аттенуированная форма of Staphylococcus aureus, имеющая стабилизированный фенотип SCV, представляет собой ΔhemBΔ720 S. aureus.
Пункт 23: Композиция, содержащая: (A) по меньшей мере один из конструктов, как определено в любом из п.п. 1 - 16; по меньшей мере одну из молекул нуклеиновой кислоты, определенных в пункте 17; по меньшей мере один из векторов, определенных в пункте 18; или по меньшей мере одну из клеток, как определено в любом из п.п. 19 - 22; и (B) (i) полипептид, как определено в любом из п.п. 1 - 11; (ii) живой аттенуированный Staphylococcus aureus; (iii) фармацевтически приемлемый эксципиент; (iv) адъювант; или (v) комбинацию по меньшей мере двух из (i) - (iv).
Пункт 24: Композиция по пункту 23, где живая аттенуированная форма Staphylococcus aureus экспрессирует: (a) полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131), SACOL0264 полипептид (SEQ ID NO: 185), SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92), SACOL0718 полипептид (SEQ ID NO: 186), SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120), SACOL1353 полипептид (SEQ ID NO: 187), SACOL1416 полипептид (SEQ ID NO: 188), SACOL1611 (SEQ ID NO: 189), SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164), SACOL1912 полипептид (SEQ ID NO: 43), SACOL1944 (SEQ ID NO: 190), SACOL2144 полипептид (SEQ ID NO: 191), SACOL2365 полипептид (SEQ ID NO: 192), SACOL2385 полипептид (SEQ ID NO: 50) или SACOL2599 полипептид (SEQ ID NO: 193) на основе номенклатуры гена из генома Staphylococcus aureus COL (SACOL), изложенного в NCBI эталонной последовательности NC_002951.2; (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (c).
Пункт 25: Композиция по пункту 23 или 24, где живая аттенуированная форма Staphylococcus aureus имеет стабилизированный фенотип варианта мелких колоний (SCV).
Пункт 26: Композиция по любому из п.п. 23 - 25, где адъювант содержит алюм, масло (например, эмульгированное масло, минеральное масло), сапонин (например, Quil-A™), циклический дигуанозин-5'-монофосфат (c-di-GMP), полифосфазин, индолицидин, патоген-ассоциированный молекулярный паттерн (PAMPS) или комбинация по меньшей мере двух из них.
Пункт 27: Способ профилактики и/или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего, причем указанный способ включает введение указанному млекопитающему эффективного количества конструкта, как определено в любом из п.п. 1 - 16; молекулы нуклеиновой кислоты, определенной в пункте 17; вектора, определенного в пункте 18; клетки, как определено в любом из п.п. 19 - 22; или композиции, как определено в любом из п.п. 23 - 26.
Пункт 28: Способ по пункту 27, где указанная стафилококковая IMI вызвана одним или более штаммами Staphylococcus aureus.
Пункт 29: Способ по пункту 27 или 28, где указанный млекопитающим является корова.
Пункт 30: Применение эффективного количества (i) конструкта, как определено в любом из п.п. 1 - 16; (ii), молекулы нуклеиновой кислоты, определенной в пункте 17; (iii) вектора, определенного в пункте 18; клетки, как определено в любом из п.п. 19 - 22; (iv) композиции, как определено в любом из п.п. 23 - 26; или (v) комбинации по меньшей мере двух из (i) - (iv), для профилактики и/или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего.
Пункт 31: Применение по пункту 30, где указанная стафилококковая IMI вызвана одним или более штаммами Staphylococcus aureus.
Пункт 32: Применение по пункту 30 или 31, где указанный млекопитающим является корова.
Пункт 33: Конструкт, как определено в любом из п.п. 1 - 16; молекула нуклеиновой кислоты, определенная в пункте 17; вектор, определенный в пункте 18; клетка, как определено в любом из п.п. 19 - 22; композиция, как определено в любом из п.п. 23 - 26, или комбинация по меньшей мере двух из них, для применения для профилактики и/или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего.
Пункт 34: Конструкт, молекула нуклеиновой кислоты, вектор, клетка или композиция по пункту 33, где указанная стафилококковая IMI вызвана одним или более штаммами Staphylococcus aureus.
Пункт 35: Конструкт, молекула нуклеиновой кислоты, вектор, клетка или композиция по пункту 33 или 34, где указанный млекопитающим является корова.
Пункт 36: Набор для профилактики и/или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего, содержащий: (A) (i) по меньшей мере один из конструктов, как определено в любом из п.п. 1 - 16; (ii) по меньшей мере одну из молекул нуклеиновой кислоты, определенных в пункте 17; (iii) по меньшей мере один из векторов, определенных в пункте 18; (iv) по меньшей мере одну из клеток, как определено в любом из п.п. 19 - 22; или (v) комбинацию по меньшей мере двух из (i) - (iv), и (B) (i) полипептид, как определено в любом из п.п. 1 - 11; (ii) живой аттенуированный Staphylococcus aureus; (iii) фармацевтически приемлемый эксципиент; (iv) адъювант; (v) инструкции по применению набора для профилактики и/или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего; или (vi) комбинацию по меньшей мере двух из (i) - (v).
Краткое описание чертежей
На приложенных чертежах:
Фиг. 1A-D показывают общие сывороточные титры IgG (Фиг. 1A), IgG1 (Фиг. 1B) и IgG2 (Фиг. 1C) для вакцинированных (9) и плацебо (10) коров для каждого антигена вакцины, а именно SACOL0029, SACOL0442, SACOL0720, SACOL1867, SACOL1912 и SACOL2385, через четыре недели после второй иммунизации (непосредственно перед экспериментальной инфекцией). На Фиг.1D соотношение IgG2/IgG1 представлено для вакцинированных коров. На Фиг. 1А, В и С, незакрашенные кружки (○) представляют данные для вакцинированных коров, черные квадраты (■) представляют данные для коров плацебо. Каждый символ представляет титр для одной коровы. Горизонтальные линии представляют медианы: пунктирные линии представляют медианы для вакцинированных коров, в то время как непрерывные линии представляют медианы для коров плацебо. На Фиг. 1A, B и C, титры для вакцинированных коров выше, чем титры для коров плацебо (P <0,0001). На Фиг. 1D символы представляют соотношение IgG2/IgG1 для каждой коровы. Горизонтальные линии представляют медианы. Различные буквы показывают статистические различия. ***, P <0,001.
Фиг. 2 показывает антиген-зависимую пролиферацию клеток CD4 + крови вакцинированных коров (9) и плацебо (10) через четыре недели после второй иммунизации каждым антигеном. Каждый символ представляет процент клеток CD4 +, которые пролиферировали для каждой коровы после недели инкубации с положительным контролем (ConA) или каждым антигеном, а именно SACOL0029, SACOL0442, SACOL0720, SACOL1867, SACOL1912 и SACOL2385. Незакрашенные кружки (○) представляют данные для вакцинированных коров, черные квадраты (■) представляют данные для коров плацебо. Горизонтальные линии представляют медианы: пунктирные линии представляют медианы для вакцинированных коров, в то время как непрерывные линии представляют медианы для коров плацебо. Статистический анализ: смешанная процедура SAS. Символ * показывает статистические различия между вакцинированными и плацебо-группами для антигенов SACOL0029, SACOL0442, SACOL0720 и SACOL1912 (*, P <0,05). Кроме того, пролиферация клеток CD8 + была подобной для вакцинированных и плацебо-коров для всех антигенов, за исключением антигена SACOL0720, для которого у вакцинированных коров наблюдалась более высокая пролиферация клеток CD8 + (данные не показаны).
Фиг. 3 показывает экспериментальную интрамаммарную инфекцию S. aureus у молочных коров. Через четыре недели и 4 дня после второй иммунизации 63 колониеобразующих единиц (КОЕ) S. aureus были инфузированы в 3 из 4 доли вымени вакцинированных (9) и плацебо коров (10) при вечернем доении (день 1, стрелка на Фиг. 3). Пробы асептического молока отбирали во время утреннего доения, и количество соматических клеток (SCC) определяли с помощью Valacta (Ste-Anne-de-Bellevue, QC). Незаполненные кружки (○) и пунктирная линия представляют данные для вакцинированных коров, в то время как черные квадраты (■) и непрерывная линия представляют данные для коров плацебо. Каждый кружок представляет среднее значение SCC для всех зараженных долей вымени вакцинированных коров (27), в то время как каждый квадрат представляет среднее значение SCC для всех зараженных долей вымени коров плацебо (30 долей вымени). Было обнаружено, что в течение периода заражения количество соматических клеток в молоке было значительно ниже у вакцинированных коров, чем у плацебо (***; P <0,001).
Фиг. 4A-C. Фиг. 4A показывает корреляцию между КОЕ и SCC для каждой коровы, и на Фиг.4В показана корреляция между титром сывороточного IgG1 против SACOL0442 и SCC для каждой коровы. Каждый символ представляет данные для одной коровы. На Фиг. 4А он представляет среднее значение SCC и КОЕ для 3 зараженных кварталов от начала до конца инфекции. На Фиг.4В он представляет среднее значение SCC для 3 инфицированных долей вымени от начала до конца инфекции и титр сывороточного IgG1 против SACOL0442 через четыре недели после второй иммунизации и непосредственно перед экспериментальной инфекцией. Незакрашенные кружки представляют данные для вакцинированных коров, а черные квадраты представляют данные для коров плацебо. Существует сильная корреляция между SCC/мл и КОЕ/мл и отрицательная корреляция между SCC и титром IgG1 против SACOL0442. На Фиг. 4C, корреляция между SCC или КОЕ относительно титра IgG2 молока против SACOL0029 показана для каждой коровы через десять дней после экспериментальной инфекции (через 6 недель после второй иммунизации). Каждый символ представляет данные для одной коровы через десять дней после экспериментальной инфекции. Пробы асептического молока отбирали во время утреннего доения, а жизнеспособные количества S. aureus определяли с помощью 10-кратных разведений и высевания на чашки с триптическим соевым агаром (TSA), тогда как SCC определяли с помощью Valacta (Ste-Anne-de-Bellevue, QC). Данные SCC и CFU для каждой коровы являются средними данными для 3 зараженных долей вымени через десять дней после экспериментальной инфекции. Образцы молока для определения титров IgG2 молока представляют собой смесь эквивалентного объема молока из 4 долей вымени каждой коровы через 10 дней после экспериментальной инфекции (через 6 недель после второй иммунизации). Черные квадраты (■) представляют данные для коров плацебо, незакрашенные кружки (○) представляют данные для вакцинированных коров.
Фиг. 5 показывает титры общего IgG в сыворотке для вакцинированных коров для каждого антигена вакцины, содержащей слитые антигены SACOL0029 и SACOL1867 (показаны как SACOL0029-1867) и антигены SACOL0442 и SACOL0720. В ELISA целевыми антигенами были SACOL0029-1867, SACOL0029, SACOL1867, SACOL0442 и SACOL0720. Каждый открытый кружок представляет титр через четыре недели после второй иммунизации для каждой из 11 коров, тогда как каждый черный ромб представляет преиммунный титр. Горизонтальные линии представляют медианы: сплошная линия для преиммунных сывороток, пунктирная линия для образцов, взятых через четыре недели после иммунизации. Титры вакцинированных коров выше, чем титры преиммунных сывороток (**, P <0,01; ***, P <0,001 для других протестированных антигенов).
Фиг. 6 показывает общие титры IgG в сыворотке против антигена SACOL1867 мышей, иммунизированных слитым белком (SACOL0029-1867; слияние), комбинацией отдельных белков (SACOL0029 + SACOL1867; комбинация), только белком SACOL0029 (0029) или только белком SACOL1867 (1867), в эквивалентных молярных количествах. Незакрашенные кружки (○) представляют данные для преиммунных титров, черные квадраты (■) представляют данные для иммунных титров. Что касается преиммунных титров, преиммунные сыворотки смешивали поровну между 5 мышами в каждой группе иммунизации, чтобы получить титр преиммунного пула, представленный одним открытым кружком на группу. Для иммунных титров каждый квадратный символ представляет титр для одной мыши. Горизонтальные линии представляют медианы: черные линии представляют медианы для иммунных сывороток, а пунктирные линии (и открытый круг) представляют медианы для пула преиммунных сывороток. Титры для вакцинированных мышей в группах слияния, комбинации и 1867 выше, чем титры для преиммунных мышей (P <0,001), и было обнаружено, что титры мышей, которые получали только моновалентный антиген SACOL0029, значительно не отличались от титров преиммунного пула против SACOL1867. Показана статистическая значимость между иммунными титрами группы слияния по сравнению с комбинацией и двумя группами моновалентных вакцин (***: P <0,001).
Фиг. 7. Общий IgG в сыворотке (измеренный по OD 450 нм в анализе ELISA), направленный против последовательности эпитопа B-клеток KDGGKYTLESHKELQ (SEQ ID NO: 1), содержащейся в фрагменте аминокислотной последовательности SACOL0442 (GEHLPKGNIVINTKDGGKYTLESHKELQKDRENVKINTAD, SEQ ID NO: 2) и полученные от мышей, иммунизированных слиянием пептидов, кодируемых из SACOL0442 и SACOL0720 KDGGKYTLESHKELQEAAAKEAAAKKDINKIYFMTDVDLGGPTFVLND (SEQ ID NO: 3) (группа 1), или пептидом KDGGKYTLESHKELQ (SEQ ID NO: 1), кодируемым из SACOL0442 (группа 2). Каждая группа состояла из 4 животных (n = 4), которым два раза вводили эквимолярные количества последовательности KDGGKYTLESHKELQ (SEQ ID NO: 1) (что соответствует 100 мкг KDGGKYTLESHKELQEAAAKEAAAKKDINKIYFMTDVDLGGPTFVLND (SEQ ID NO: 3) для группы 1 и 31.25 мкг KDGGKYTLESHKELQ (SEQ ID NO: 1) для группы 2) с интервалом в 2 недели, и сыворотки готовили из крови, собранной через неделю после последней инъекции. Анализ ELISA проводили с образцами сыворотки, разведенными в 100 000 раз, и фрагмент из SACOL0442 (GEHLPKGNIVINTKDGGKYTLESHKELQKDRENVKINTAD (SEQ ID NO: 2)) применяли в качестве антигена-мишени, содержащей пептидный эпитоп KDGGKYTLESHKELQ (SEQ ID NO: 1). Отдельные данные представлены в виде кружков на графике, а медианы - в виде столбцов. Различие между группами было обнаружено статистически значимым (P <0,0286, тест Кускала-Уоллиса, GraphPad Prism™ 7.00).
Фиг. 8A-B. Делеция hemB у штаммов ATCC29213 и Δ720 Staphylococcus aureus. (Фиг. 8A) Ген hemB в штамме ATCC29213 дикого типа (WT) и его изогенный мутант Δ720 были удалены путем гомологичной рекомбинации и замены кассетой ermA для создания мутантных штаммов ΔhemB и Δ720ΔhemB соответственно. Толстые линии и цифры обозначают области, амплифицированные ПЦР, обозначенные на B для родительских штаммов (1) и штаммов с делецией hemB (2). (Фиг. 8B) Продукты ПЦР штамма WT и его изогенного мутанта ΔhemB (аналогичные результаты были получены со штаммами Δ720 и Δ720ΔhemB).
Фиг. 9A-C. показывают влияние мутаций S. aureus ΔhemB, Δ720 и ΔhemBΔ720 на инфекционность клеток MAC-T. Клетки MAC-T инфицировали каждым из четырех штаммов в течение 3 часов, затем инкубировали с лизостафином дополнительно 30 минут (t = 3 часа), 12 часов или 24 часа и лизировали для измерения жизнеспособных внутриклеточных бактерий (КОЕ). (Фиг. 9A) Относительное восстановление исходного инокулята, обнаруженного в клетках в момент времени t 3ч для Δ720, и (Фиг. 9B) для мутантов ΔhemBΔ720. Результаты нормированы в соответствии с результатами, полученными для ATCC 29213 (WT) или ΔhemB, соответственно, и выражены как среднее значение с SD (**, P ≤ 0,01; ***, P ≤ 0,001; непарный t-критерий). (Фиг. 9C) Средние значения и SD внутриклеточных КОЕ для WT и мутантов через 12 ч (слева) и 24 ч (справа). Был использован двусторонний тест ANOVA и критерий множественных сравнений Тьюки (*: P ≤ 0,05; ***: P ≤ 0,001). Все значения указывают на среднее значение трех независимых экспериментов, каждый из которых выполнен в трех экземплярах.
Фиг. 10. показывает персистенцию S. aureus ATCC 29213 (WT) и изогенных мутантов в клетках MAC-T с течением времени. Клетки MAC-T инфицировали каждым из четырех штаммов в течение 3 часов, затем инкубировали с лизостафином дополнительно 30 минут, 12 часов или 24 часа и лизировали для измерения внутриклеточных бактерий (КОЕ). Внутриклеточные бактериальные КОЕ выражены в процентах от исходного инокулята после преобразования в логарифмические значения с основанием 10 (Log10 КОЕ/мл). Значения указывают на среднее значение трех независимых трехкратных экспериментов со стандартными отклонениями.
Фиг. 11. показывает жизнеспособность клеток MAC-T, инфицированных S. aureus ATCC 29213 (WT) и изогенными мутантами. Клетки MAC-T инфицировали каждым из четырех штаммов в течение 3 часов, затем инкубировали с лизостафином в течение 12 часов (A) или 24 часов (B). Затем анализ жизнеспособности МТТ проводили с помощью метода, описанного в Kubica et al., 2008. Результаты представлены в виде процента жизнеспособности по отношению к неинфицированным клеткам и выражены как среднее значение с SD для трех независимых экспериментов, проведенных в трех экземплярах. Статистическая значимость с символом (Φ) сравнивается с WT (двусторонний анализ ANOVA и критерий множественных сравнений Тьюки: * или Φ: P ≤ 0,05; **: P≤ 0,01; ***: P ≤ 0,001; ΦΦΦΦ: P ≤ 0,0001).
Фиг. 12. Показаны мышиные IMI с родительским (WT) и ΔhemBΔ720 (ΔΔ) штаммами. Мышей заражали, как описано ранее, и собирали железы в указанный час (ч) или день (D) после заражения. Каждый столбец представляет среднее значение количества КОЕ бактерий для группы желез, а диапазоны указаны столбцами. Для штамма WT на D7 использовалось минимум шесть желез на группу (2 железы: выжила только одна мышь). Смертность мышей в определенные моменты времени обозначена стрелками. Звездочкой обозначена очистка ΔhemBΔ720 от желез (ниже предела обнаружения 10 КОЕ/железа).
Фиг. 13. Двойной мутант (Δ720ΔhemB) стимулирует приток нейтрофилов в молочные железы до аналогичных уровней по сравнению с WT в первые 24 часа после заражения. Мышей заражали, как описано в материалах и способах, и контрольной группе (PBS) мышей вводили стерильную инъекцию PBS. Железы собирали в указанное время, гомогенизировали и кинетически анализировали на активность МРО, как описано в материалах и методах. Каждая точка представляет Единицы MPO для одной железы, которые показаны как необработанные значения, скорректированные на грамм железы. Средние значения представлены жирными линиями.
Фиг. 14. Визуальное воспаление крупных молочных желез R4 и L4 через 24 ч после IMI мыши с S. aureus ATCC 29213 (WT) и двойным мутантом Δ720ΔhemB (ΔΔ). Мыши были инфицированы, как описано в материалах и методах, и мыши контрольной группы (PBS) получали стерильную инъекцию PBS. На снимках показаны железы, которые были собраны через 24 часа. На каждой панели показаны железы R4 (слева) и L4 (справа).
Фиг. 15A. После очистки двойного мутанта Δ720ΔhemB инфильтрация нейтрофилов возвращается к нормальным уровням. Мышей заражали, как описано в материалах и способах, и контрольной группе (PBS) мышей вводили стерильную инъекцию PBS. Железы собирали в указанное время, гомогенизировали и кинетически анализировали на активность МРО, как описано в материалах и методах. Столбцы представляют собой средние значения единиц MPO группы из 6 желез (4 для контроля PBS), скорректированные на грамм железы, а столбцы ошибок иллюстрируют стандартное отклонение. Статистическая значимость между группами 4 и 12 дня после заражения показана символом (Φ). Использовались односторонние ANOVA и множественные сравнительные тесты Тьюки (ΦΦ: P ≤ 0,01; NS: нет существенных различий между группами).
Фиг. 15B-C. Иммунизация мышей живым аттенуированным двойным мутантом (Δ720ΔhemB) стимулирует сильный гуморальный ответ против изолятов бычьего мастита S. aureus широко распространенных spa-типов. Мышей иммунизировали, как описано ранее: сыворотки собирали до первичной иммунизации (Preimmune) и через десять дней после бустерной иммунизации (Immune). Фиг. 15В. Титры IgG повышаются с увеличением дозы живого аттенуированного штамма Δ720GΔhemB: каждая точка представляет общий титр IgG одной мыши против экстракта цельных клеток Δ720GΔhemB. Медианы представлены жирными линиями для титров Immune и пунктирными линиями для титров Preimmune. Титры сравнивали с соответствующими им преиммунными титрами (двухфакторный ANOVA и критерий множественных сравнений Тьюки: ****: P ≤ 0,0001). Фиг. 15C. Иммунизация живым аттенуированным мутантом Δ720GΔhemB придает титры IgG против компонентов, которые являются общими для штаммов мастита часто встречающихся spa-типов. Каждая точка представляет общий титр IgG одной мыши против цельноклеточного экстракта указанного штамма. Медианы представлены жирными линиями для титров Immune и пунктирными линиями для титров Preimmune. Все иммунные титры сравнивали с их соответствующим преиммунным титром (P ≤ 0,0001) и между клиническими штаммами (двухфакторный ANOVA и тест множественных сравнений Sidak: NS: нет существенной разницы).
Фиг. 16 показывает общие сывороточные титры IgG против слитого белка SACOL0029-1867 мышей, иммунизированных белковой смесью (состоящей из 5 мкг антигенов SACOL0029, SACOL0442, SACOL0720 и слияния SACOL0029-1867), 105 КОЕ аттенуированного живого штамма Δ720ΔhemB отдельно или в комбинации с белковой смесью и штаммом Δ720ΔhemB. Незакрашенные кружки (○) представляют данные для преиммунных титров, черные квадраты (■) представляют данные для иммунных титров. Каждый символ представляет титр для одной мыши. Горизонтальные линии представляют медианы: черные линии представляют медианы для иммунных сывороток, а пунктирные линии представляют медианы для преиммунных сывороток. Титры для вакцинированных мышей в группе смешивания белков и в группе комбинирования выше, чем титры для преиммунных мышей (P <0,001). Статистическая значимость между иммунными титрами комбинации по сравнению с двумя другими вакцинированными группами мышей показана (**: P <0,01).
Фиг. 17 показывает общие сывороточные титры IgG против SACOL0029 мышей, иммунизированных белковой смесью (состоящей из 5 мкг антигенов SACOL0029, SACOL0442, SACOL0720 и слияния SACOL0029-1867), 105 КОЕ аттенуированного живого штамма Δ720ΔhemB отдельно или комбинации с белковой смесью и штаммом Δ720ΔhemB. Незакрашенные кружки (○) представляют данные для преиммунных титров, черные квадраты (■) представляют данные для иммунных титров. Каждый символ представляет общий титр IgG для одной мыши. Горизонтальные линии представляют медианы: черные линии представляют медианы для иммунных сывороток, а пунктирные линии представляют медианы для преиммунных сывороток. Статистическая значимость между иммунными и преиммунными титрами трех групп мышей показана (**: P <0,01).
Фиг. 18 показывает общие сывороточные титры IgG против стафилококкового поверхностного белка ClfA для мышей, иммунизированных белковой смесью SACOL0029, SACOL0442, SACOL0720 и SACOL0029-1867, 105 КОЕ аттенуированного живого штамма Δ720ΔhemB отдельно или комбинации с белковой смесью и штаммом Δ2020ΔhemB. Незакрашенные кружки (○) представляют данные для преиммунных титров, черные квадраты (■) представляют данные для иммунных титров. Каждый символ представляет титр для одной мыши. Горизонтальные линии представляют медианы: черные линии представляют медианы для иммунных сывороток, а пунктирные линии представляют медианы для преиммунных сывороток. Статистическая значимость между преиммунными титрами и иммунными титрами показана (*: P <0,05).
Фиг. 19 ниже показывает сывороточное соотношение титров IgG2a/IgG1 против слитого полипептида SACOL0029-1867 для мышей, иммунизированных белковой смесью или комбинацией белковой смеси и аттенуированного живого штамма Δ720ΔhemB.
Фиг. 20 показывает сывороточные соотношения против антигена SACOL0029. Открытые квадраты (□) представляют данные для преиммунных титров, черные квадраты (■) представляют данные для иммунных титров. Каждый символ представляет соотношение титров для одной мыши. Горизонтальные линии представляют медианы. Показана статистическая значимость между соотношениями группы смеси белков и группы комбинации (*: P <0,05; **: P <0,01).
Фиг. 21A-J. I. SACOL0029 полинуклеотиды (последовательность полной длины SEQ ID NO: 4) и полипептиды (последовательности полной длины, фрагмент(ы) и вариант(ы) SEQ ID NO: 5 - 9). Выделенные эпитопы показаны затененными и/или жирными; II. SACOL0720 полинуклеотиды (последовательность полной длины SEQ ID NO: 10) и полипептиды (последовательности полной длины, фрагмент(ы) и вариант(ы) SEQ ID NO: 11 - 27). Выделенные эпитопы показаны затененными; III. SACOL0442 полинуклеотиды (последовательность полной длины SEQ ID NO: 28) и полипептиды (последовательности полной длины, фрагмент(ы) и вариант(ы) SEQ ID NO: 29 - 36 и 1). Выделенные эпитопы показаны затененными; IV. SACOL1867 полинуклеотиды (последовательность полной длины SEQ ID NO: 37) и полипептиды (последовательности полной длины, фрагмент(ы) и вариант(ы) SEQ ID NO: 38 - 41). Выделенные эпитопы показаны затененными. Предсказанный трансмембранный (http://www.enzim.hu/hmmtop/html/submit.html) домен показан жирным; V. SACOL1912 полинуклеотид (последовательность полной длины SEQ ID NO: 42) и полипептиды (последовательности полной длины и вариант(ы) SEQ ID NO: 43 - 44). Выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 45-48); VI. SACOL2385 полинуклеотид (SEQ ID NO: 49 полной длины) и полипептиды (последовательности полной длины и вариант(ы) SEQ ID NO: 50 - 51). Выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 52-53); VII. Слияния: (i) SACOL0029-1867 полинуклеотидные последовательности слияния (SEQ ID NO: 54 и 56) и полипептидные последовательности (SEQ ID NO: 55, 57-58). В полинуклеотидных и полипептидных последовательностях дважды подчеркнутая последовательность, если таковая имеется, представляет собой последовательность полигистидина, выделенная курсивом последовательность представляет собой последовательность фрагмента SACOL0029, одиночно подчеркнутая последовательность представляет собой последовательность линкера, а выделенными жирным последовательностями является последовательность фрагмента SACOL1867; (ii) SACOL0720-720 полипептидная последовательность слияния (SEQ ID NO: 27). В полипептидной последовательности дважды подчеркнутой, если таковая имеется, является последовательность полигистидина, выделенными курсивом последовательностями являются последовательности фрагментов SACOL0720, и однократно подчеркнутой последовательностью является последовательность линкера; (iii) полипептидная последовательность слияния SACOL0442-720 (SEQ ID NO: 3). В полинуклеотидной и полипептидной последовательностях дважды подчеркнутой последовательностью, если таковая имеется, является последовательность полигистидина, выделенной курсивом последовательностью является последовательность фрагмента SACOL0442 однократно подчеркнутой последовательностью является последовательность линкера, а жирная последовательность представляет собой последовательность фрагмента SACOL0720; и VIII. Последовательности линкеров (SEQ ID NO: 59-70).
Фиг. 22A-D I. Выравнивание множества полинуклеотидных последовательностей (SEQ ID NO: 71-72, 28, 73 - 81) для полной длины SACOL0442 и ортологов; II. Выравнивание множества полипептидных последовательностей (SEQ ID NO: 29 и 82 - 92) для полной длины SACOL0442, ортологов и полученных из них консенсусных последовательностей представлены. В этих последовательностях «*» означает, что остатки в этом столбце идентичны во всех последовательностях выравнивания, «:» означает, что наблюдались консервативные замены, а «.» означает, что наблюдались полуконсервативные замены. Консенсусные последовательности, полученные из этих выравниваний, также представлены, где X представляет собой любую аминокислоту. В полипептидных последовательностях выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 1, 34, 93-97).
Фиг. 23A-K I. Выравнивание множества полинуклеотидных последовательностей (SEQ ID NO: 98 - 104, 10, и 105 - 108) для полной длины SACOL0720 и ортологов; II. Выравнивание множества полипептидных последовательностей (SEQ ID NO: 11 и 109 - 120) для полной длины SACOL0720, ортологов и полученных из них консенсусных последовательностей представлены. В этих последовательностях «*» означает, что остатки в этом столбце идентичны во всех последовательностях выравнивания, «:» означает, что наблюдались консервативные замены, а «.» означает, что наблюдались полуконсервативные замены. Консенсусные последовательности, полученные из этих выравниваний, также представлены, где X представляет собой любую аминокислоту. В полипептидных последовательностях выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 22, 19 и 21).
Фиг. 24. Выравнивание множества полипептидных последовательностей (SEQ ID NO: 5 и 121 - 131) для полной длины SACOL0029, ортологов и полученных из них консенсусных последовательностей представлены. В этих последовательностях «*» означает, что остатки в этом столбце идентичны во всех последовательностях выравнивания, «:» означает, что наблюдались консервативные замены, а «.» означает, что наблюдались полуконсервативные замены. Консенсусные последовательности, полученные из этих выравниваний, также представлены, где X представляет собой любую аминокислоту. В полипептидных последовательностях выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 132-139). Выделенный жирным эпитоп идентифицирован посредством BCPred™. Выявлены затененным эпитопы идентифицированы посредством AAp прогнозирований.
Фиг. 25A-D. I - Выравнивание множества полинуклеотидных последовательностей (SEQ ID NO: 140 - 151) для полной длины SACOL1867 и ортологов; II - Выравнивание множества полипептидных последовательностей (SEQ ID NO: 152 - 164) для полной длины SACOL1867, ортологов и полученных из них консенсусных последовательностей представлены. В этих последовательностях «*» означает, что остатки в этом столбце идентичны во всех последовательностях выравнивания, «:» означает, что наблюдались консервативные замены, а «.» означает, что наблюдались полуконсервативные замены. Консенсусные последовательности, полученные из этих выравниваний, также представлены, где X представляет собой любую аминокислоту. В полипептидных последовательностях выделенные эпитопы показаны затененными (смотрите, например, SEQ ID NO: 165-180) и конец сигнального пептидного домена и/или трансмембранного домена отмечен линией (отделяет сигнальный пептидный и/или трансмембранный домен от секретируемой формы).
Фиг. 26. I - полинуклеотидная последовательность (SEQ ID NO: 181) для полной длины SACOL1715 (hemB); и II - аминокислотная последовательность (SEQ ID NO: 182) для полной длины SACOL1715 (hemB).
Фиг. 27. I - полинуклеотидная последовательность (SEQ ID NO: 183) для полной длины ClfA (NWMN_0756, newman); и II - аминокислотная последовательность (SEQ ID NO: 184) для полной длины ClfA.
Описание иллюстративных вариантов выполнения настоящего изобретения
Настоящее изобретение показало, что слияние двух антигенов создает неожиданный синергизм при иммунном ответе.
Кроме того, настоящее изобретение также стабилизировало фенотип SCV стафилококка через hemB (полная делеция, таким образом, ухудшая возможность возврата к инвазивному фенотипу (Tuchscherr, 2011)), что позволяет использовать его в качестве системы доставки вакцины. HemB кодирует белок/мономер HemB, который объединяется для создания фермента порфобилиногенсинтазы или аминолевулинатдегидратазы [EC 4.2.1.24]. Кроме того, дальнейшая аттенуация была вызвана инактивацией антигена согласно настоящему изобретению, а именно гена SACOL0720, который, как было ранее показано, играет важную роль для S. aureus в устойчивости к катионным пептидам (Falord, 2012; Kawada-Matsuo, 2011; Meehl, 2007) и in vivo в ходе IMI (Allard, 2013). Следовательно, эти аттенуированные конструкты экспрессирующие штамм двойного мутанта, согласно настоящему изобретению, можно использовать для иммунизации и защиты от IMI.
Общие определения
Заголовки и другие идентификаторы, например (а), (b), (i), (ii) и т. д., представлены просто для удобства чтения описания и формулы изобретения. Использование заголовков или других идентификаторов в описании или формуле изобретения не обязательно требует выполнения шагов или элементов в алфавитном или числовом порядке или в порядке их представления.
В настоящем описании ряд терминов широко используется. Чтобы обеспечить четкое и непротиворечивое понимание описания и формулы изобретения, включая объем, который должен быть дан таким терминам, предоставляются следующие определения.
Использование формы единственного числа при использовании в сочетании с термином «содержащий» в формуле изобретения и/или описании может означать «один», но это также согласуется со значением «один или более», «по меньшей мере один» и «один или более чем один».
Во всей этой заявке термин «около» используется для указания того, что значение включает стандартное отклонение ошибки для устройства или метода, используемого для определения значения. В общем, термин «около» предназначен для обозначения возможного отклонения до 10%. Следовательно, вариация 1, 2, 3, 4, 5, 6, 7, 8, 9 и 10% значения включена в термин «около». Если не указано иное, использование термина «около» перед диапазоном применяется к обоим концам диапазона.
Как используется в данном описании и пункте(-ах) формулы изобретения, слова «содержащий» (и любая форма слова «содержащий», такая как «содержат» и «содержит»), «имеющий» (и любая форма слова «имеющий», такая как «иметь» и «имеет»), «включая» (и любая форма слова «включая», такая как «включает» и «включать») или «включающий» (и любая форма слова «включающий», такая как «содержит» и «содержать») являются включающими или открытыми и не исключают дополнительные, не перечисленные элементы или стадии способа.
Как применяется в настоящей заявке, термин «состоит из» или «состоящий из» означает включение только элементов, стадий или ингредиентов, конкретно перечисленных в конкретном заявленном варианте выполнения настоящего изобретения или формулы изобретения.
Полипептиды, нуклеиновые кислоты и системы доставки
Как применяется в настоящей заявке, термин «вакцина» относится к любому соединению/агенту («компоненту вакцины») или их комбинациям, способным индуцировать/вызывать иммунный ответ у хозяина и позволяющим лечить и/или предотвращать инфекцию и/или заболевание. Следовательно, неограничивающие примеры такого агента включают белки, полипептиды, фрагменты белков/полипептидов, иммуногены, антигены, пептидные эпитопы, эпитопы, смеси белков, пептидов или эпитопов, а также нуклеиновые кислоты, гены или части генов (кодирующие полипептид или представляющий интерес белок или его фрагмент), добавляемый отдельно или в непрерывной последовательности, как например в вакцинах на основе нуклеиновых кислот, и тому подобное.
Объектом настоящего изобретения является слитный конструкт формулы I:
X-A-линкер-B-Z (формула (I),
Где A и B являются идентичными или различными и каждая независимо представляет собой антигенный полипептид (т.e. нативный, фрагмент или его вариант) согласно настоящему изобретению.
В конкретном варианте выполнения настоящего изобретения, A и/или B представляет собой (a) полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131), SACOL0264 полипептид (SEQ ID NO: 185), SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92), SACOL0718 полипептид (SEQ ID NO: 186), SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-J (SEQ ID NO: 11 и 109 - 120), SACOL1353 полипептид (SEQ ID NO: 187), SACOL1416 полипептид (SEQ ID NO: 188), SACOL1611 (SEQ ID NO: 189), SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164), SACOL1912 полипептид, как изложено на Фиг. 21G-V (SEQ ID NO: 43), SACOL1944 (SEQ ID NO: 190), SACOL2144 полипептид (SEQ ID NO: 191), SACOL2365 полипептид (SEQ ID NO: 192), SACOL2385 полипептид, как изложено в VI на Фиг. 21H (SEQ ID NO: 50) или SACOL2599 полипептид (SEQ ID NO: 193). В конкретном варианте выполнения настоящего изобретения, вышеуказанный полипептид (а) является секретируемым или внеклеточным фрагментом полипептида, определенного выше. Трансмембранные домены можно прогнозировать, используя, например, программное обеспечение TMpred™ (ExPASy) http://www.ch.embnet.org/software/TMPRED_form.html, http://www.psort.org/psortb/index.html http://www.enzim.hu/hmmtop/html/submit.html и/или SignlP 4.1 (http://www.cbs.dtu.dk/services/SignalP). TMpred™ и SignalIP 4.1 предсказали внеклеточный домен для: SACOL0720: AA 310-508; SACOL0442 AA 36-203. Enzim предсказал трансмембранный домен SACOL1867 (1-40), чтобы внеклеточный домен был: AA 41-239, в то время как http://www.psort.org/psortb/index.html предсказал, что SACOL1867 был внеклеточным белком. Поскольку вышеупомянутые трансмембранные и/или сигнальные пептидные домены являются предполагаемыми, настоящее изобретение охватывает случаи, когда представленные здесь антигены (например, SACOL1867) имеют или не имеют сигнальный пептид и/или трансмембранный домен и охватывают соответствующие внеклеточные фрагменты. В варианте выполнения настоящего изобретения вышеупомянутый полипептид представляет собой полипептид, обычно секретируемый или экспрессируемый на поверхности бактерий (например, Staphylococcus aureus).
Номера доступа Genbank для S. aureus генов, перечисленные в настоящей заявке, и их кодируемые антигенные полипептиды, охватываемые настоящим изобретения, приведены в Таблице I ниже:
Таблица I: Номера доступа Genbank для IMI-связаных S. aureus генов и кодируемых полипептидов, описанных в настоящей заявке
(SEQ ID NO: 5)
WP_000570071
(SEQ ID NO: 185)
(SEQ ID NO: 29)
WP_000985996
(SEQ ID NO: 186)
(SEQ ID NO: 11)
WP_000603968
(SEQ ID NO: 187)
WP_000548932
(SEQ ID NO: 188)
WP_001095260
(SEQ ID NO: 189)
(SEQ ID NO: 38)
(SEQ ID NO: 43)
WP_000149064
(SEQ ID NO: 190)
WP_000908177
(SEQ ID NO: 191)
WP_000827000
(SEQ ID NO: 192)
(SEQ ID NO: 50)
AAW38600
(SEQ ID NO: 193)
Согласия, полученные на основе выравнивания некоторых перечисленных выше полипептидов, представлены на Фиг. 21-25. В конкретном варианте выполнения этих согласований каждый Х в консенсусных последовательностях (например, согласования на Фиг. 21-25) определяется как любая аминокислота или отсутствует, когда эта позиция отсутствует в одном или более ортологах, представленных в выравнивание. В конкретном варианте выполнения я этих согласований каждый Х в консенсусных последовательностях определяется как любая аминокислота, которая представляет собой консервативную или полуконсервативную замену любой аминокислоты в соответствующем положении в ортологах, представленных в выравнивании, или отсутствует, когда эта позиция отсутствует в одном или более ортологах, представленных в выравнивании. На Фиг. 21-25, консервативные замены обозначены символом «:», а полуконсервативные замены обозначены символом “.”. В другом варианте выполнения настоящего изобретения каждый X относится к любой аминокислоте, принадлежащей к тому же классу, что и любой из аминокислотных остатков в соответствующем положении в ортологах, представленных в выравнивании, или отсутствует, когда это положение отсутствует в одном или более из ортологов, представленных в выравнивании. В другом варианте выполнения настоящего изобретения каждый X относится к любой аминокислоте в соответствующем положении ортологов, представленных в выравнивании, или отсутствует, когда это положение отсутствует в одном или более ортологах, представленных в выравнивании. В частности, A и/или B представляет собой полипептид, удовлетворяющий любому из этих утверждений или его фрагмент.
Консервативная аминокислотная мутация может включать добавление, делецию или замену аминокислоты; консервативная аминокислотная замена определяется в настоящей заявке как замена аминокислотного остатка другим аминокислотным остатком со сходными химическими свойствами (например, размером, зарядом или полярностью). Такая консервативная аминокислотная замена может представлять собой замену основной, нейтральной, гидрофобной или кислотной аминокислоты на другую из той же группы (смотрите, например, Таблицу II ниже). Под термином «основная аминокислота» подразумеваются гидрофильные аминокислоты, имеющие значение pK боковой цепи более 7, которые обычно заряжены положительно при физиологическом pH. Основные аминокислоты включают гистидин (His или H), аргинин (Arg или R) и лизин (Lys или K). Под термином «нейтральная аминокислота» (также «полярная аминокислота») подразумеваются гидрофильные аминокислоты, имеющие боковую цепь, которая не заряжена при физиологическом рН, но которая имеет по меньшей мере одну связь, в которой пара электронов, совместно используемых двумя атомами, находится ближе к одному из атомов. Полярные аминокислоты включают серина (Ser или S), треонин (Thr или T), цистеин (Cys или C), тирозин (Tyr или Y), аспарагин (Asn или N) и глютамин (GIn или Q). Термин «гидрофобная аминокислота» (также «неполярная аминокислота»), как означает, включает аминокислоты, проявляющие гидрофобность больше нуля, согласно нормированной консенсусной шкале гидрофобности Эйзенберга (1984). Гидрофобные аминокислоты включают пролин (Pro или P), изолейцин (He или I), фенилаланин (Phe или F), валин (VaI или V), лейцин (Leu или L), триптофан (Trp или W), метионин (Met или M), аланин (Ala или A) и глицин (Gyy или G). «Кислотная аминокислота» относится к гидрофильным аминокислотам, имеющим значение pK боковой цепи менее 7, которые обычно заряжены отрицательно при физиологическом pH. Кислотные аминокислоты включают глутамат (GIu или E) и аспартат (Asp или D).
Полуконсервативная аминокислота заменяет один остаток другим, который имеет сходную стерическую конформацию, но не обладает общими химическими свойствами. Примеры полуконсервативных замен включают замену цистеина на аланина или лейцин; замену серина на аспарагин; замену валина на треонин; или замену пролина на аланина.
Таблица II ниже указывает, какая аминокислота относится к каждому классу аминокислот.
Сходство и идентичность аминокислотных или нуклеотидных последовательностей можно определить путем сравнения каждого положения в выровненных последовательностях. Оптимальное выравнивание последовательностей для сравнения сходства и/или идентичности может проводиться с использованием различных алгоритмов, например, с использованием программы/программного обеспечения для выравнивания множества последовательностей, хорошо известных в данной области, таких как ClustalW™, SAGA™, UGENE™ или T-coffee™. Примеры выравнивания множества последовательностей описаны в приведенных ниже примерах и изображены на Фиг.21А-25.
Оперон гена
В другом варианте выполнения настоящего изобретения, A и/или B представляет собой (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a), как определено выше. Например, SACOL0718 представляет собой ген из того же оперона как SACOL0720.
Фрагмент
В другом варианте выполнения настоящего изобретения, A и/или B представляет собой (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b), как определено выше.
Иммуногенный фрагмент белка/полипептида определяется как часть белка/полипептида, который способен индуцировать/вызывать иммунный ответ у хозяина. В соответствии с настоящим изобретением, иммуногенный фрагмент способен вызывать тот же иммунный ответ таким же образом, хотя и не обязательно в том же количестве, что белок/полипептид. Иммуногенный фрагмент белка/полипептида предпочтительно содержит один или более эпитопов указанного белка/полипептида. Эпитоп белка/полипептида определяется как фрагмент указанного белка/полипептида длиной около 4 или 5 аминокислот, способный вызывать специфическое антитело и/или иммунную клетку (например, Т-клетку или В-клетку), несущую рецептор, способный специфически связываться с указанным эпитопом. Существуют два разных типа эпитопов: линейные эпитопы и конформационные эпитопы. Линейный эпитоп включает отрезок последовательных аминокислот. Конформационный эпитоп обычно образован несколькими участками последовательных аминокислот, которые сложены в определенном положении и вместе образуют эпитоп в правильно свернутом белке. Иммуногенный фрагмент, как применяется в настоящей заявке, относится либо к одному, либо к обоим из указанных типов эпитопов. В варианте выполнения настоящего изобретения, когда иммуногенные фрагменты используются по отдельности (то есть не слиты с более крупным полипептидным конструктом (например, слияние с другим антигенным фрагментом)), иммуногенный фрагмент белка/полипептида содержит по меньшей мере 16 аминокислотных остатков. В другом вариант выполнения настоящего изобретения, иммуногенный фрагмент содержит по меньшей мере 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, или 160 последовательных аминокислот нативного белка/полипептида. В конкретном варианте выполнения настоящего изобретения, фрагмент имеет по меньшей мере 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46 ,47, 48, 49, 50, или 50 или более последовательных аминокислот нативного белка/полипептида. В варианте выполнения настоящего изобретения, где по меньшей мере один иммуногенный фрагмент образует часть большего полипептидного конструкта (например, слияние с другим антигенным полипептидом, его фрагментом или вариантом), иммуногенный фрагмент содержит по меньшей мере 13 последовательных аминокислотных остатков полипептида. Без ограничения, фрагменты, охватываемые настоящим изобретением, содержат иммуногенные фрагменты из по меньшей мере 13 последовательных аминокислот SACOL029, как показано на Фиг. 21 (и соответствующие фрагменты в SACOL029 ортологах (например, показанных на Фиг. 24); SACOL0442, как показано на Фиг. 21 (и соответствующие фрагменты в SACOL0442 ортологах (например, как изложено на Фиг. 22); SACOL0720, как показано на Фиг. 21, (и соответствующие фрагменты в SACOL0720 ортологах (например, как изложено на Фиг. 23); и SACOL1867, как показано на Фиг. 21 (и соответствующие фрагменты в SACOL1867 ортологах (например, как изложено на Фиг. 25). В другом варианте выполнения настоящего изобретения, фрагменты, охватываемые настоящим изобретением, включают иммуногенные фрагменты, содержащие по меньшей мере один эпитоп антигенных белков/полипептидов согласно настоящему изобретению (полипептид (a), определенный выше). В другом варианте выполнения настоящего изобретения, фрагменты, охватываемые настоящим изобретением, включают иммуногенные фрагменты, содержащие по меньшей мере один эпитоп, который изображен (заштрихован) в любом из антигенных белков/полипептидов, изображенных на любой из Фиг. 21-25. Без ограничения к этому, эпитопы в последовательности могут быть предсказаны с помощью таких программ, как BCPred™, AAP™, FBCPred™ и ABCPred™.
В варианте выполнения настоящего изобретения, вышеупомянутый иммуногенный фрагмент содержит последовательность, которая является консервативной (то есть идентичной) у по меньшей мере двух различных штаммов Staphylococcus aureus. В дополнительном варианте выполнения настоящего изобретения вышеупомянутый иммуногенный фрагмент содержит последовательность, которая является консервативной (то есть идентичной) у по меньшей мере 3, 4, 5, 6, 7, 8, 9 или 10 различных штаммов Staphylococcus aureus. В соответствии с настоящим изобретением вышеупомянутыми штаммами Staphylococcus aureus являются COL, RF122, NCTC 8325, JH1, JH9, Newman, Mu3, Mu50, USA300-FPR3757, N315, MW2 или MSSA476. В варианте выполнения настоящего изобретения вышеупомянутые штаммы Staphylococcus aureus связаны с бычьим маститом (например, RF122).
Варианты
В другом варианте выполнения настоящего изобретения, вышеупомянутый полипептид, или полипептид, по существу идентичный указанному полипептиду, экспрессируется в по меньшей мере двух различных штаммах Staphylococcus aureus. По существу идентичные, как применяется в настоящей заявке, относится к полипептидам, имеющим по меньшей мере 60% идентичности, в вариантах выполнения настоящего изобретения на по меньшей мере 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92% 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности в их аминокислотных последовательностях. В других вариантах выполнения настоящего изобретения полипептиды имеют по меньшей мере 60%, 65%, 70%, 71%, 72%, 73%, 74%, 75%, 80%, 81%, 82%, 83%, 84 %, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности в их аминокислотных последовательностях с другими полипептидами, с которыми они сравниваются.
В другом варианте выполнения настоящего изобретения, A и/или B представляет собой (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (с), определенных выше. В других вариантах выполнения настоящего изобретения аминокислота на по меньшей мере 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентична (a) (например, по всей ее длине). В других вариантах выполнения настоящего изобретения аминокислота на по меньшей мере 60%, 65%, 70%,71%, 72%, 73%, 74%, 75%, 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентична (a). Например, антигены-ортологи, представленные на Фиг. 21-25 не являются идентичными, но представляют определенную идентичность с антигенами или фрагментами, с которыми они сравниваются. Согласования, представленные на этих чертежах, воплощают такие процентные идентичности.
В другом варианте выполнения настоящего изобретения, A и/или B представляет собой (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a)-(d). Иммуногенный вариант белка/полипептида определяется как часть белка/полипептида, который способен индуцировать/вызывать иммунный ответ у хозяина. Как будет понятно специалисту в данной области, агенты (белки/полипептиды, их фрагменты), имеющие не встречающиеся в природе модификации (например, иммуногенные варианты) и которые способны индуцировать иммунный ответ, специфичный для немодифицированного агента (например, способны к индукции продуцирования антител, способных распознавать немодифицированный агент), также входят в объем термина «вакцинный компонент». Например, вакцинные компонентов согласно настоящему изобретению могут быть модифицированы для повышения их активности, стабильности и/или биодоступности, и/или для снижения их токсичности. Консервативные аминокислотные замены могут быть сделаны, такие как, например, замена аминокислоты, содержащей кислотную боковую цепь, другой аминокислотой, содержащей кислотную боковую цепь, замена объемной аминокислоты другой объемной аминокислотой, замена аминокислоты, содержащей основную боковую цепь другой аминокислотой, содержащей основную боковую цепь, и тому подобное. Специалист в данной области вполне способен генерировать варианты белка/полипептида. Это делается, например, с помощью скрининга библиотеки пептидов или с помощью программ замены пептидов. Иммуногенный вариант в соответствии с настоящим изобретением обладает по существу такими же иммуногенными свойствами указанного белка таким же образом, но необязательно в таком же количестве. Иммуногенный вариант белка/полипептида согласно настоящему изобретению может, например, включать слитый белок и/или химерный белок. Например, биологическая функция идентифицированного здесь белка, который, как предсказывают, является экзотоксином, энтеротоксином или суперантигеном (например, SACOL0442), может потенциально влиять на иммунную систему млекопитающих и выработку антител, и/или демонстрировать некоторую токсичность у хозяина. Хотя такого вмешательства не наблюдалось, когда полипептид SACOL0442 использовался в сочетании с, например, SACOL0720 во время иммунизации, может быть полезно модифицировать белок или полипептид, используемый для вакцинации, так, чтобы биологическая активность экзотоксина снижалась. Для этой цели можно инактивировать экзотоксин химическими веществами (например, формальдегидом). Также возможно использовать методы молекулярной биологии для делеции или мутации предполагаемой области (областей), вовлеченных в активность экзотоксинов, без потери иммуногенности (Chang et al., 2008). Другим примером является конъюгация или смесь компонентов на основе аминокислот с нуклеиновыми кислотами (например, гены или части генов, добавленные отдельно или в непрерывной последовательности) углеводов, таких как углеводы, содержащиеся в микробных полисахаридных капсулах или биопленках. Другие примеры вариантов включают антигены, описанные здесь, или их фрагменты, содержащие на своем N- или С-конце или встроенные в их последовательность антигена, олигопептид, пригодный для очистки (например, аффинная очистка) или полезный в качестве спейсера или линкера. Примеры олигопептидов, полезных для аффинной очистки, включают метки полигистидина (например, 6-10 остатков гистидина, включая или нет метки RGS (например, HHHHHH, RGSHHHHHH или RGSHHHHHGS). За меткой his может также следовать аминокислотная последовательность, подходящая для облегчения удаления полигистидиновой метки с использованием эндопептидаз. Сегменты «X» и/или «Z», как указано в формуле (I), также могут содержать такой олигопептид, пригодный для очистки, и/или последовательность, подходящую для облегчения удаления такого олигопептида, полезного для очистки.
В конкретных вариантах выполнения настоящего изобретения, иммуногенный фрагмент содержит по меньшей мере один эпитоп полипептида (a). Без ограничения таким образом, в определенных вариантах выполнения настоящего изобретения, иммуногенный фрагмент содержит по меньшей мере один эпитоп полипептида (a), как показано (затемнением) в последовательностях, представленных на Фиг. 21-25, В других конкретных вариантах выполнения настоящего изобретения, варианты являются такими, как раскрыто на Фиг. 21-25.
Линкер
Вставка линкеров между доменами слитого белка может увеличить биологическую активность, увеличивая расстояние между доменами, уменьшая потенциальные силы отталкивания между различными сегментами (например, антигенными фрагментами) конструкта, что приводит к улучшенному и/или восстановленному сворачиванию белка. Могут быть использованы различные последовательности полипептидных линкеров, которые, как известно, обладают различными свойствами, такие как гибкие, жесткие или расщепляемые линкеры. Настоящее изобретение охватывает использование любых таких линкеров, включая любой из перечисленных в Chen et. al., например, Adv Drug Deliv Rev. (2013), 65 (10): 1357-69. Приведенные здесь примеры предоставляют иллюстрации конкретных линкеров, которые использовались (т.е. GGGGSGGGGSGGGGS (SEQ ID NO: 60), ERKYK (SEQ ID NO: 61) или и EAAAKEAAAK (SEQ ID NO: 62)), то есть гибкие линкерные структуры, обогащенные небольшими гидрофильные аминокислотами, которые поддерживают расстояние между двумя связанными доменами и улучшают их складывание.
В другом конкретном варианте выполнения настоящего изобретения Fc содержит домен CH2, домен CH3 и шарнирную область. В другом конкретном варианте выполнения настоящего изобретения Fc представляет собой константный домен иммуноглобулина, выбранный из группы, состоящей из IgG-1, IgG-2, IgG-3, IgG-3 и IgG-4. В другом конкретном варианте выполнения настоящего изобретения Fc представляет собой константный домен иммуноглобулина IgG-1
Линкеры могут быть включены между смежными антигенами слияния (например, 1 линкер в слиянии, содержащем два антигена, 2 линкера в слиянии, содержащем три антигена, три линкера в слиянии, содержащем четыре антигена и т.д.). В слияниях, где используются большие белковые домены, линкер может быть больше и может содержать фрагмент кристаллизуемой области (Fc).
В конкретном варианте выполнения настоящего изобретения, линкер представляет собой аминокислотную последовательность из по меньшей мере одной аминокислоты или отсутствует. В конкретном варианте выполнения настоящего изобретения, линкер содержит по меньшей мере три (по меньшей мере 4, 5, 6,7, 8, 9 или 10) аминокислот, выбранных из группы, состоящей из глицина, серина, аланина, аспартата, глутамата и лизина. В конкретном варианте выполнения настоящего изобретения, линкер представляет собой (EAAAK)n (SEQ ID NO: 63); (GGGGS)n (SEQ ID NO: 67); или (XPXPXP)n (SEQ ID NO: 69), где x представляет собой любую аминокислоту; где n равно любому из 1 - 5, более конкретно 1, 2, 3, 4 или 5; EAAAKEAAAK (SEQ ID NO: 62); EAAAKEAAAKEAAAK (SEQ ID NO: 64); GGGGS (SEQ ID NO: 67); GGGGSGGGGS (SEQ ID NO: 68); GGGGSGGGGSGGGGS (SEQ ID NO: 60); XPXPXP (SEQ ID NO: 69), где x представляет собой любую аминокислоту; XPXPXPXPXPXP (SEQ ID NO: 70), где x представляет собой любую аминокислоту; ERKYK (SEQ ID NO: 61); ERKYKERKYK (SEQ ID NO: 65); ERKYKERKYKERKYK (SEQ ID NO: 66). В более конкретном варианте выполнения настоящего изобретения, линкер представляет собой GGGGSGGGGSGGGGS (SEQ ID NO: 60), ERKYK (SEQ ID NO: 61), или EAAAKEAAAK (SEQ ID NO: 62).
N и C концы конструкта
X и Z каждый независимо отсутствует или представляет собой аминокислотную последовательность из по меньшей мере одной аминокислоты. Не ограничиваясь этим, они могут представлять собой один или более аминокислот, полученных в результате стратегии клонирования, которые используются для облегчения очистки конструкта (например, полигистидин), и пригодны для облегчения удаления метки очистки с использованием эндопептидаз. В конкретных вариантах выполнения настоящего изобретения, где слитный конструкт включает три или более полипептида антигена, любой из X и/или Z может также включать последовательность другого антигена (антиген C, антиген D и т.д.) и, необязательно, последовательность по меньшей мере одного другого линкера. Такие варианты выполнения настоящего изобретения, в которых X и/или Z содержат один или более других антигенов и необязательно линкер(ы), могут быть более подробно проиллюстрированы как, например, формула (II) или (III) следующим образом: X'-C-линкер1-A-линкер2-B-Z' (II), когда слияние содержит по меньшей мере 3 антигена; или X'-C-линкер1-A-линкер2-B-линкер3-D-Z' (III), когда слияние содержит по меньшей мере 4 антигена. В обеих формулах (II) и (III) X', Z', линкер1, линкер2, и, в случае линкер3, являются идентичными или различными и независимо друг от друга определены как X, Z м линкер в формуле (I), как определено в настоящей заявке.
Следовательно, в конкретных вариантах выполнения настоящего изобретения, слитный конструкт содержит 2, 3, 4 или более полипептидов антигена (и в случае других линкеров). В более конкретном варианте выполнения настоящего изобретения, и без ограничения таким образом, слитный конструкт может представлять собой SACOL0029_SACOL0442; SACOL0029_SACOL0720; SACOL0029_SACOL1867; SACOL0029_SACOL0720_SACOL1867; SACOL0029_SACOL1867_SACOL0442; SACOL0029_SACOL0720_SACOL0442; SACOL0442_SACOL0029_SACOL0720; SACOL0442_SACOL0029_SACOL1867; SACOL0442_SACOL1867_SACOL0720; SACOL0720_SACOL0442_SACOL1867; или SACOL0029_SACOL1867_SACOL0720_SACOL0442, или любой из вышеуказанных конструктов, где полипептиды антигена находятся в любом другом порядке.
Комбинация
Конструкты согласно настоящему изобретению могут использоваться в качестве единственного иммуногенного компонента композиции (например, вакцины) согласно настоящему изобретению или в сочетании с одним или более дополнительными слитными конструктами, иммуногенными полипептидами, их фрагментами или вариантами и/или живыми аттенуированными бактериями (например, S. aureus) (экспрессирующими или нет слитные конструкты и/или полипептид(ы), их фрагмент(ы) или вариант(ы)).
Один или более слитных конструктов могут быть любыми иммуногенными слитными конструктами, включая дополнительный слитный конструкт, как определено выше (смотрите, например, Пример 14).
Один или более иммуногенных полипептидов, их фрагментов или вариантов для использования в композициях согласно настоящему изобретению могут представлять собой любой полипептид(ы), фрагмент(ы) или вариант(ы), который (которые) вносят вклад в иммуногенность композиций согласно настоящему критерию, как определено в настоящей заявке. Без ограничения к этому, такой полипептид(ы), фрагмент(ы) или вариант(ы) включает (a) полипептид, содержащий SACOL0029 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 24 (SEQ ID NO: 5 и 121 - 131), SACOL0264 полипептид (SEQ ID NO: 185), SACOL0442 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 22D (SEQ ID NO: 29 и 82 - 92), SACOL0718 полипептид (SEQ ID NO: 186), SACOL0720 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 23I-K (SEQ ID NO: 11 и 109 - 120), SACOL1353 полипептид (SEQ ID NO: 187), SACOL1416 полипептид (SEQ ID NO: 188), SACOL1611 (SEQ ID NO: 189), SACOL1867 полипептид, как изложено в любой из последовательностей, показанных на Фиг. 25D (SEQ ID NO: 152 - 164), SACOL1912 полипептид (SEQ ID NO: 43), SACOL1944 полипептид (SEQ ID NO: 190), SACOL2144 полипептид (SEQ ID NO: 191), SACOL2365 полипептид (SEQ ID NO: 192), SACOL2385 полипептид (SEQ ID NO: 50) или SACOL2599 полипептид (SEQ ID NO: 193); (b) полипептид, кодируемый геном из того же оперона, что и ген, кодирующий полипептид согласно (a); (c) полипептид, содержащий иммуногенный фрагмент из по меньшей мере 13 последовательных аминокислот согласно (a) или (b); (d) полипептид, содержащий аминокислотную последовательность в общем на по меньшей мере 60% идентичную последовательности полипептида согласно любому из п.п. (a) - (c); или (e) полипептид, содержащий иммуногенный вариант, содержащий по меньшей мере 13 последовательных аминокислот согласно любому из (a) - (c), как определено выше. Без ограничения этим, любой такой полипептид(ы), фрагмент(ы) или вариант(ы) охватывает те, которые включены в композицию (например, вакцины №1 - №8), примеры которых приведены в Примерах 1 - 14 и 21-26.
Живые аттенуированные бактерии
Живые аттенуированные бактерии (например, S. aureus) для использования в композициях согласно настоящему изобретению могут быть независимыми от конструктов слияния и/или полипептида(ов), их фрагмента(ов) или варианта(ов) согласно настоящему изобретению или быть сосудом для (то есть могут экспрессировать) таких слитных конструктов и/или полипептида(ов), их фрагмента(ов) или варианта(ов) согласно настоящему изобретению.
Без ограничения к этому, как проиллюстрировано в настоящей заявке, полезные живые аттенуированные бактерии согласно настоящему изобретению включают бактерии Staphylococcus (например, aureus), имеющие по меньшей мере один ген, способствующий вирулентности (например, Δ720) или способствующий физической форме в хозяине (например, метаболический ген), мутированный или удаленный. Без ограничения к этому, таким геном может быть любой из генов, идентифицированных в Novick 2003, Novick 2008, или Maresso and Schneewind 2008.
В другом варианте выполнения настоящего изобретения, живые аттенуированные бактерии могут быть дополнительно аттенуированы благодаря наличию стабилизированного фенотипа SCV. Как применяются в настоящей заявке термин «фенотип SCV» относится к бактериям, имеющим дисфункциональный окислительный метаболизм, вызывающий медленный рост, изменение экспрессии факторов вирулентности и способность к интернализации в клетки-хозяева. Как применяется в настоящей заявке термин «стабилизированный фенотип SCV» используется для обозначения штамма SCV, сохраняющего фенотип SCV, то есть неспособный, чтобы продуцировать инвазивные ревертанты (то есть возврат к нормальному фенотипу роста). Такой стабилизированный SCV S. aureus может быть получен путем мутации или удаления любого из генов (например, ΔhemB), перечисленных в таблице III ниже. Не ограничиваясь этим, настоящее изобретение охватывает использование стабилизированного SCV S. aureus, приведенного в качестве примеров в примерах 15-25. Мутация, которая имеет место в настоящей заявке, включает замену, делецию и/или вставку одного или более нуклеотидов, которые предотвращают экспрессию полипептида, кодируемого геном согласно настоящему изобретению, или предотвращают экспрессию функционального полипептида. В предпочтительном варианте выполнения настоящего изобретения, мутация предотвращает экспрессию полипептида. В другом конкретном варианте выполнения настоящего изобретения две мутации в одном и том же аттенуированном живом или инактивированном штамме S. aureus представляют собой делецию или вставку. Ожидается, что мутантный штамм S. aureus, имеющий мутацию в любом положении одного из генов согласно настоящему изобретению, которая предотвращает экспрессию полипептида, может быть использован в качестве аттенуированной живой вакцины в соответствии с настоящим изобретением. Живые аттенуированные вакцины, то есть вакцины, содержащие бактерию согласно изобретению, в живой аттенуированной форме, имеют преимущество перед инактивированными вакцинами в том, что они наилучшим образом имитируют естественный способ заражения. Кроме того, их репликативные способности позволяют делать вакцинацию с низким содержанием бактерий; их количество будет автоматически увеличиваться, пока не достигнет уровня запуска иммунной системы. С этого момента иммунная система запускается и, наконец, уничтожает бактерии. Небольшой недостаток использования живых аттенуированных бактерий, однако, может заключаться в том, что по сути сохраняется определенный уровень вирулентности. Это не должно быть реальным недостатком, пока уровень вирулентности приемлем, то есть пока вакцина по меньшей мере уменьшает симптомы бактериальной инфекции (например, IMI). Конечно, чем ниже остаточная вирулентность живой аттенуированной вакцины, тем меньше влияние вакцинации на увеличение веса во время/после вакцинации.
Таблица III: Номера доступа Genbank для генов S. aureus, связанных с SCV фенотипом (Kahl, 2014)
WP_000667126.1
GI:446589780
EC:4.2.1.24
WP_000526687.1
GI:446448832
EC:2.2.1.9
WP_000667126.1
GI:446589780
EC:2.1.1.45
GI:57285609
GI:57285005
GI:57284701
EC:2.7.6.5
GI:57285285
GI:57284807
EC:4.99.1.1
GI:57285910
Нуклеиновые кислоты
Нуклеиновая кислота согласно настоящему изобретению предпочтительно содержит нуклеотидную последовательность, которая кодирует один или более вышеуказанных белков/полипептидов (или их фрагментов), функционально связанных с регуляторными элементами, необходимыми для экспрессии генов, такими как промотор, инициирующий кодон, стоп-кодон, энхансеры и сигнал полиаденилирования. Регуляторные элементы предпочтительно выбирают так, чтобы они работали у видов, которым они должны вводиться. В конкретных вариантах выполнения настоящего изобретения нуклеиновая кислота является такой, как изображено на Фиг. 21-25.
В контексте настоящего изобретения нуклеиновую кислоту согласно настоящему изобретению млекопитающему вводят in vivo, так что один или более представляющих интерес белков/полипептиды (или их фрагмент) экспрессируются/экспрессируются у млекопитающего (например, вакцина на основе нуклеиновой кислоты, ДНК или РНК-вакцина).
Системы доставки
Нуклеиновая кислота вакцины согласно настоящему изобретению может представлять собой «голую» ДНК или может быть функционально включена в вектор. Нуклеиновую кислоту можно доставлять в клетки in vivo с применением таких методов, как прямая инъекция ДНК, опосредуемый рецептором захват ДНК, вирус-опосредуемая трансфекция или невирусная трансфекция и липидная трансфекция, при этом все они могут включать применение векторов с терапевтическими генами. Прямую инъекцию применяют для введения депротеинизированной ДНК в клетки in vivo (см., напр., Acsadi et al. (1991) Nature 332:815-818; Wolff et al. (1990) Science 247:1465-1468). Для инъекции ДНК в клетки in vivo можно применять устройство для доставки (напр., "генная пушка"). Для этого можно применить коммерчески доступный аппарат (напр., от BioRad). Депротеинизированную ДНК можно вводить в клетки путем комплексообразования ДНК с катионом, таким как полилизин, который связывается с лигандом рецептора поверхности клетки (см., напр., Wu, G. and Wu, С. Н. (1988) J. Biol. Chem. 263:14621; Wilson el al. (1992) J. Biol. Chem. 267:963-967; и Патент США № 5,166,320). Связывание комплекса ДНК-лиганд с рецептором может облегчать захват ДНК по муханизму рецептор-опосредуемого эндоцитоза. Чтобы избежать разрушения этого комплекса межклеточными лизосомами можно применять комплекс ДНК-лиганд, связанный с аденовирусными капсидами, которые расщепляют эндосомы, что приводит к высвобождению материала в цитоплазму (см., например, Curiel el al. (1991) Proc. Natl. Acad. Sci. USA 88:8850; Cristiano et al. (1993) Proc. Natl. Acad. Sci. USA 90:2122-2126).
Полезные векторы доставки включают биоразлагаемые микрокапсулы, иммуностимулирующие комплексы (ISCOM) или липосомы и генно-инженерные аттенуированные живые векторы, такие как клетки, вирусы или бактерии.
Липосомные векторы представляют собой униламеллярные или мультиламеллярные везикулы, имеющие образованную липофильным материалом мембранную часть и внутреннюю водную часть. В настоящем изобретении водная часть используется для того, чтобы в ней содержался полинуклеотидный материал, который должен быть доставлен к клеткам-мишеням. Обычно предпочтительно, чтобы образующие липосому материалы имели катионную группу, такую как группа четвертичного аммония, и одну или более липофильных групп, таких как насыщенные или ненасыщенные алкильные группы, имеющие приблизительно от 6 и приблизительно до 30 атомов углерода. Группа подходящих материалов описана в Европейской патентной публикации No. 0187702 и дополнительно обсуждается в патенте США No. 6228844, выданном на имя Wolff et al., описание соответствующей части которого включено здесь посредством ссылки. Многие другие подходящие образующие липосому катионные липидные соединения описаны в литературе. См., например, L. Stamatatos, et al., Biochemistry 27: 3917-3925 (1988); и H. Eibl, et al., Biophysical Chemistry 10: 261-271 (1979). Альтернативно можно использовать микросферы, такие как биодеградируемые полиактид-когликолидные микросферы. Конструкция нуклеиновой кислоты инкапсулируется в липосомы или микросферы или иным способом образует с ними комплекс для доставки нуклеиновой кислоты к ткани, как это практикуется в данной области.
Предпочтительные вирусные векторы включают бактериофаги, вирус герпеса, аденовирус, вирус полиомиелита, вирус Vaccinia, дефектные ретровирусы, аденоассоциированный вирус (AAV) и Avipox. Способы трансформации вирусного вектора экзогенной ДНК-конструкцией также хорошо описаны в данной области. Смотри Sambrook и Russell, выше.
Как указано выше, нуклеиновая кислота (например, ДНК или РНК) может быть включена в хозяин, такого как клетка-хозяин, in vitro или ex vivo (например, в иммунную клетку, такую как дендритная клетка) или, как указано выше, в аттенуированный микробный хозяин (например, аттенуированный S. aureus, SCV и т.д., смотрите, например, примеры 25-26) путем трансфекции или трансформации, и трансфицированная или трансформированная клетка или микроорганизм, который экспрессирует представляющие интерес полипептид (например, слияние множества антигенов или их фрагментов и/или отдельные антигены или их фрагменты) могут быть введены субъекту. После введения клетка будет экспрессировать интересующий белок или полипептид (или его вариант или фрагмент) у субъекта, что, в свою очередь, приведет к индукции иммунного ответа, направленного против белка, полипептида или его фрагмента.
Использование аттенуированных живых бактерий для иммунизации и/или для доставки специфических конструктов или смеси антигенов согласно настоящему изобретению представляет интересный подход для улучшения иммунных ответов (Griffiths and Khader, 2014). Живые аттенуированные организмы, которые имитируют естественную инфекцию, мощно стимулируют иммунную систему, вызывая широкие и устойчивые иммунные ответы, которые продуцируют как сывороточные антитела, так и антитела слизистой оболочки, а также эффекторные клетки и Т-клетки памяти, которые действуют синергетически для защиты от болезней (Detmer and Glenting, 2006; Kollaritsch et al., 2000; Pasetti et al., 2011). Примеры подходящих аттенуированных живых бактериальных векторов включают S. aureus, Salmonella typhimurium, Salmonella typhi, Shigella, Bacillus, Lactobacillus, Bacille Calmette-Guerin (BCG), Escherichia coli, Vibrio cholerae, Campylobacter или любой другой подходящий в качестве бактериального вектора в искусстве. Способы трансформации живых бактериальных векторов экзогенной ДНК-конструкцией хорошо описаны в данной области. Смотрите, например, Joseph Sambrook and David W. Russell, Molecular Cloning, A Laboratory Manual, 3rd Ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. (2001). Настоящее изобретение охватывает применение композиции, содержащей аттенуированную живую бактерию (например, ΔhemBΔ720 S. aureus, экспрессирующую конструкт согласно настоящему изобретению в качестве единственного иммуногенного компонента или в сочетании с другими аттенуированными живыми бактериями, каждая из которых экспрессирует другой полипептид, фрагмент или вариант согласно настоящему изобретению (например, SACOL0442, SACOL0720 или их фрагменты или варианты).
Композиции
Описанные здесь полипептиды, нуклеиновые кислоты и системы доставки (например, клетки-хозяева, содержащие указанные нуклеиновые кислоты или векторы) могут быть составлены в композиции. Как применяется в настоящей заявке, термин «фармацевтически приемлемый» относится к компонентам вакцины (например, эксципиенты, носители, адъюванты) и композициям, которые являются физиологически переносимыми и обычно не вызывают аллергическую или подобную неблагоприятную реакцию, такую как расстройство желудка, головокружение и тому подобное, при введении субъекту. Предпочтительно, используемый в настоящей заявке термин «фармацевтически приемлемый» означает одобренный регулирующим органом федерального правительства или правительства штата или внесенный в список фармакопеи США или другой общепризнанной фармакопеи для применения у животных и людей. Термин «эксципиент» относится к разбавителю, носителю или среде, с помощью которых могут вводиться компоненты вакцины согласно настоящему изобретению. В качестве носителей можно использовать стерильную воду или водные солевые растворы, а также водные растворы декстрозы и глицерина, особенно для инъекционных растворов.
В варианте выполнения настоящего изобретения, агент согласно настоящему значению вводят в сочетании с адъювантом или иммуностимулятором. Подходящий адъювант или иммуностимулятор, который может улучшить эффективность компонентов для повышения иммунного ответа, включает, но не ограничивается ими, масла (например, минеральное масло, эмульгированное масло, такое как MONTANIDE™ или EMULSIGEN™-D), соли металлов (например, алюм, алюминия гидроксид или фосфат алюминия), катионные пептиды (Bowdish et al., 2005; Hancock, et al., 2000), такие как индолицидин, катионный пептид, продуцируемый иммунными клетками коровы (Falla et al., 1996), природные и искусственные микробные компоненты (например, бактериальные липосахариды, адъюванты Фрейнда, мурамилдипептид (MDP), циклический дигуанозин-5'-монофосфат (c-di-GMP), патоген-ассоциированный молекулярный паттерн (PAMPS), такие как поверхностные полисахариды, липополисахариды, гликаны, пептидогликан или микробная ДНК (например, CpG), растительные компоненты, такие как, например, сапонины (например, Quil-A™) и/или одно или более веществ, обладающих эффектом носителя (например, бентонит, частицы латекса, липосомы, ISCOM™, сополимеры ДНК и полифосфазина (PCPP)). Также возможна иммунизация синтетическими наночастицами (например, полученными из биоразлагаемого синтетического полимера, такого как поли(D,L-молочная-гликолевая кислота)), содержащими антигены плюс лиганды, которые передают сигналы через TLR для стимуляции провоспалительных цитокинов (Kasturi et al., 2011).
Вакцинные компоненты согласно настоящему изобретению могут быть введены в фармацевтической композиции. Фармацевтические композиции можно вводить в стандартной лекарственной форме. Может быть использован любой подходящий путь введения, например, парентеральное, подкожное, внутримышечное, внутримаммарное, внутричерепное, внутриглазное, офтальмологическое, внутрижелудочковое, внутрикапсулярное, внутрисуставное, внутриспинальное, внутрицистернальное, внутрибрюшинное, интраназальное, аэрозольное или пероральное. Примеры конкретных путей введения включают парентеральный, например, внутривенный, внутрикожный, подкожный, интрамаммарный; оральный (например, ингаляционный); трансдермальный (актуальный); трансмукозальное и ректальное введение.
Обычная фармацевтическая практика может применяться для обеспечения подходящих составов или композиций для введения таких компонентов вакцины с или без адъювантами субъектам. Способы, хорошо известные в данной области для изготовления фармацевтических композиций и составов, можно найти, например, в Remington: The Science and Practice of Pharmacy, (20th ed.) ed. A. R. Gennaro A R., 2000, Lippincott: Philadelphia. Составы для парентерального введения могут, например, содержать наполнители, стерильную воду или физиологический раствор, полиалкиленгликоли, такие как полиэтиленгликоль, миглиол, масла растительного происхождения или гидрогенизированные нафталины. Биосовместимый, биоразлагаемый лактидный полимер, лактид/гликолидный сополимер или полиоксиэтилен-полиоксипропиленовые сополимеры могут быть использованы для контроля высвобождения соединений. Другие потенциально полезные системы парентеральной доставки для соединений согласно настоящему изобретению включают частицы сополимера этиленвинилацетата, осмотические насосы, имплантируемые инфузионные системы и липосомы. Составы для ингаляций или внутримаммарных инъекций могут содержать наполнители, например, лактозу, или могут быть водными растворами, содержащими, например, полиоксиэтилен-9-лауриловый эфир, миглиол, гликохолат и дезоксихолат, или могут быть масляными растворами (например, парафиновым маслом) для введения в форме капель для носа или в виде геля.
Терапевтические составы могут быть в форме жидких растворов или суспензии; для перорального введения составы могут быть в форме таблеток или капсул; и для интраназальных составов в форме порошков, капель для носа или аэрозолей. Растворы или суспензии, используемые для парентерального, внутрикожного, внутримаммарного или подкожного применения, могут включать следующие компоненты: стерильный разбавитель, такой как вода для инъекций, физиологический раствор, жирные масла (например, парафиновое масло), полиэтиленгликоли, глицерин, пропиленгликоль, миглиол или другие синтетические растворители; антибактериальные агенты, такие как бензиловый спирт или метилпарабены; антиоксиданты, такие как аскорбиновая кислота или бисульфит натрия; хелатообразующие агенты, такие как этилендиаминтетрауксусная кислота; восстанавливающие агенты, такие как дитиотреитол, буферы, такие как ацетаты, цитраты или фосфаты, и агенты для регулирования тонуса, такие как хлорид натрия или декстроза. Значение рН можно регулировать с помощью кислот или оснований, таких как соляная кислота или гидроксид натрия. Парентеральный препарат может быть заключен в ампулы, одноразовые шприцы или флаконы с множественными дозами, сделанные из стекла или пластика.
Фармацевтические композиции, подходящие для инъекционного применения, включают стерильные водные растворы (когда растворимы в воде) или дисперсии и стерильные порошки для немедленного приготовления стерильных инъекционных растворов или дисперсий. Для внутривенного или внутримаммарного введения подходящие носители включают физиологический солевой раствор, бактериостатическую воду, Cremophor™ ELTM (BASF, Parsippany, N.J.) или фосфатно-буферный солевой раствор (PBS).
Пероральные композиции обычно включают инертный разбавитель или пищевой носитель. Они могут быть заключены в желатиновые капсулы или спрессованы в таблетки или корм. Для перорального введения вакцины активные компоненты могут быть включены с наполнителями и использоваться в форме таблеток, пастилок, капсул или в корме. Фармацевтически совместимые связующие агенты и/или вспомогательные вещества могут быть включены в состав композиции. Таблетки, пилюли, капсулы, пастилки и тому подобное могут содержать любой из следующих ингредиентов или соединения аналогичной природы: связующее, такое как микрокристаллическая целлюлоза, трагакантовая камедь или желатин; эксципиент, такой как крахмал или лактоза, дезинтегрирующий агент, такой как альгиновая кислота, Primogel™ или кукурузный крахмал; смазывающее вещество, такое как стеарат магния или стероты; глидант, такой как коллоидный диоксид кремния; подсластитель, такой как сахароза или сахарин; или ароматизатор, такой как мята перечная, метилсалицилат или апельсиновый ароматизатор.
Для введения путем ингаляции компоненты вакцины доставляются в виде аэрозольного спрея из находящегося под давлением контейнера или дозатора, который содержит подходящий пропеллент, например газ, такой как диоксид углерода, или распылитель.
Системное введение также может осуществляться трансмукозальным или трансдермальным способом. Для трансмукозального или трансдермального введения в составе используются пенетранты, соответствующие барьеру для проникновения. Такие пенетранты обычно известны в данной области техники и включают, например, для трансмукозального введения, детергенты, соли желчных кислот и производные фузидиевой кислоты. Трансмукозальное введение может быть достигнуто путем использования назальных спреев или суппозиториев. Для трансдермального введения активные соединения формуют в мази, бальзамы, гели или кремы, как в общем известно в данной области техники.
Липосомные суспензии (включая липосомы, нацеленные на конкретные типы клеток) также можно использовать в качестве фармацевтически приемлемых носителей.
Фармацевтические композиции могут также содержать консерванты, солюбилизирующие агенты, стабилизирующие агенты, смачивающие агенты, эмульгаторы, подсластители, красители, отдушки, соли для изменения осмотического давления, буферы, покрывающие агенты или антиоксиданты. Они также могут содержать другие терапевтически ценные агенты.
Внутривенное, внутримышечное, подкожное, внутримаммарное или пероральное введение является предпочтительной формой применения. Дозировки, в которых компоненты согласно настоящему изобретению вводятся в эффективных количествах, зависят от природы конкретного активного ингредиента, хозяина и требований субъекта и способа введения.
Микробиологические мишени
Полипептиды, нуклеиновые кислоты и системы доставки согласно настоящему изобретению могут быть использованы в качестве противомикробных средств против стафилококковых инфекций, включая те, которые вызывают внутримаммарную инфекцию (IMI). В предпочтительном варианте выполнения настоящего изобретения стафилококковые инфекции вызваны Staphylococcus aureus.
Способы иммунизации полипептидами, нуклеиновыми кислотами, векторами, клетками, композициями и системами доставки
Охватываемые способы, применения, фармацевтические композиции и наборы согласно настоящему изобретению предназначены для пассивной и активной иммунизациеи.
Пассивная иммунизация представляет собой инъекцию антител или антисыворотки, ранее созданных против патогена (или антигенов, описанных в настоящем документе), для защиты или лечения животного-реципиента от инфекции или будущей инфекции. Защита ослабевает в течение нескольких недель, в течение которых активная иммунизация полипептидами, нуклеиновыми кислотами или системами доставки (например, как описано выше) будет иметь время для создания стойкого защитного ответа. Сыворотка для пассивной иммунизации может быть получена путем иммунизации животных-доноров с использованием полипептидов, нуклеиновых кислот или систем доставки, как описано здесь. Эта сыворотка, которая содержит антитела против антигенов, может использоваться немедленно или храниться в соответствующих условиях. Она может использоваться для борьбы с острыми инфекциями (например, IMI) или в качестве профилактического средства (Tuchscherr et al., 2008). Использование антител или сывороток при пассивной иммунизации может сочетаться с другими агентами, такими как антибиотик, для увеличения скорости излечения инфекции, которая развивается в настоящее время, или для повышения защиты от неминуемой инфекции.
Активная иммунизация представляет собой введение полипептидов, нуклеиновых кислот или систем доставки, как описано здесь, субъекту.
Компоненты, идентифицированные в соответствии с указаниями согласно настоящему изобретению, имеют профилактическое и/или терапевтическое значение, так как они могут быть использованы для повышения иммунного ответа для предотвращения и/или борьбы с заболеваниями или состояниями и, в частности, заболеваниями или состояниями, связанными с микробными инфекциями.
Термины «предотвращать/предотвращение/профилактика» или «лечить/лечение/обработка», используемые в настоящей заявке, относятся к вызову желаемого биологического ответа, то есть профилактического и терапевтического эффекта, соответственно, у субъекта. В соответствии с настоящим изобретением терапевтический эффект включает одно или более из уменьшения/сокращения тяжести, интенсивности и/или продолжительности микробной инфекции (например, стафилококковой инфекции) или любого ее симптома после введения полипептида, нуклеиновой кислоты или системы доставки (агента/композиции согласно настоящему изобретению) согласно настоящему изобретению по сравнению с его тяжестью, интенсивностью и/или продолжительностью у субъекта до лечения или по сравнению с таковыми у не подвергавшегося лечению контрольного пациента, страдающего инфекцией или любым другим симптом ее. В соответствии с изобретением профилактический эффект может включать задержку начала микробной инфекции (например, стафилококковой инфекции) или любого ее симптома у бессимптомного субъекта с риском возникновения микробной инфекции (например, стафилококковой инфекции) или любого ее симптома в будущем времени; или уменьшение/сокращение тяжести, интенсивности и/или продолжительности микробной инфекции (например, стафилококковой инфекции) или любого ее симптома, происходящее после введения агента/композиции согласно настоящему изобретению, по сравнению со сроками их появления или их тяжестью, интенсивностью и/или продолжительностью у необработанного контрольного субъекта (то есть бессимптомного субъекта с риском возникновения микробной (например, бактериальной) инфекции (например, стафилококковой инфекции) или любого ее симптома); и/или уменьшение/сокращение прогрессирования любой ранее существовавшей микробной инфекции (например, стафилококковая инфекция) или любого ее симптома у субъекта после введения агента/композиции согласно настоящему изобретению по сравнению с прогрессированием микробной инфекции (например, стафилококковой инфекции) или любого ее симптома у необработанного контрольного субъекта, имеющего такую ранее существовавшую микробную инфекцию (например, стафилококковую инфекцию), или любой ее симптом. Как применяется в настоящей заявке, при терапевтическом лечении агент/композиция согласно настоящему изобретению вводятся после начала микробной инфекции (например, стафилококковой инфекции) или любого ее симптома. Как применяется в настоящей заявке, при профилактическом лечении агент/композицию согласно настоящему изобретению вводят до начала микробной инфекции (например, стафилококковой инфекции) или любого ее симптома или после их появления, но до прогрессирования.
Как применяется в настоящей заявке, «уменьшение» или «сокращение» микробной инфекции (например, стафилококковой инфекции) или любого ее симптома относится к уменьшению симптома по меньшей мере на 10% по сравнению с контрольным субъектом (субъект, не обработанный агентом/композицией согласно настоящему изобретению), в варианте выполнения настоящего изобретения на по меньшей мере 20% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 30% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 40% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 50% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 60% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 70% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 80% ниже, в другом варианте выполнения настоящего изобретения на по меньшей мере 90% ниже, в другом варианте выполнения настоящего изобретения на 100% (полное ингибирование).
Как применяется в настоящей заявке, термин «симптом» применительно к стафилококковой инфекции относится к любому симптому стафилококковой инфекции, таким как боль, воспаление, лихорадка, рвота, диарея, усталость, мышечные боли, анорексия, обезвоживание, низкое кровяное давление, целлюлит, импетиго, фурункулы и синдром ошпаренной кожи. Более конкретно, применительно к стафилококковой IMI, симптом стафилококковой IMI относится, например, к визуальным нарушениям молока (например, водянистому внешнему виду, хлопьям, сгусткам, неприятным запахом из-за присутствия крови), покраснению вымени, отечности вымя, болезненности вымени, повышенной ректальной температуре (>39,0°C), анорексии, снижению моторики рубца и утомляемости. Увеличение количества соматических клеток молока (SCC) является еще одним симптомом стафилококковой IMI. Молочные соматические клетки включают лейкоциты, такие как лейкоциты или нейтрофилы, а также эпителиальные клетки. Общепринято, что SCC>200000/мл может представлять собой симптом стафилококковой IMI или свидетельствует о стафилококковой IMI.
Доза
Токсичность или эффективность компонентов вакцины для вызывания иммунного ответа можно определить стандартными процедурами на клеточных культурах или экспериментальных животных. Соотношение доз между токсическим и иммуностимулирующим эффектами может быть измерено. Компоненты, которые показывают большие соотношения, являются предпочтительными. Хотя могут использоваться компоненты, которые проявляют токсические побочные эффекты, следует позаботиться о разработке системы доставки, чтобы минимизировать потенциальное повреждение клеток и, таким образом, уменьшить побочные эффекты.
Данные, полученные из анализов клеточных культур и лабораторных исследований на животных, могут быть использованы при составлении диапазона дозировок для применения у крупных животных и людей. Дозировка таких компонентов предпочтительно находится в диапазоне вводимых концентраций, которые включают эффективность при малой токсичности или ее отсутствии. Дозировка может варьироваться в этом диапазоне в зависимости от используемой лекарственной формы и используемого пути введения.
Любое подходящее количество фармацевтической композиции может быть введено субъекту. Дозировки будут зависеть от многих факторов. Как правило, количество активного ингредиента, содержащегося в одной дозе, представляет собой количество, которое эффективно предотвращает или лечит IMI, не вызывая значительной токсичности. Специалист в данной области поймет, что определенные факторы могут влиять на дозировку, необходимую для эффективного повышения иммунного ответа у субъекта. Кроме того, для терапевтически эффективного количества антигенов (например, слитный конструкт) согласно настоящему результату может потребоваться ряд доз. В общем, количество около 0,01 мг - 500 мг антигенов, включая слитный конструкт на дозу, принимается во внимание. В конкретном варианте выполнения настоящего изобретения учитывается количество около 0,1 мг - 1 мг антигенов, включая слитный конструкт на дозу. Обычно одна, две или три дозы вакцины могут способствовать оптимальному развитию иммунитета. Время между двумя дозами может составлять три или четыре недели, но может быть предпочтительным разделить первичную дозу (первую дозу) и бустерную дозу (вторую дозу) на пять, шесть, семь, восемь, девять или десять недель до стимуляции иммунной системы с помощью бустер-инъекции. Последующая бустер-инъекция (повторный выстрел) также может быть оптимальным для обеспечения устойчивого иммунитета. Этот вторичный иммунный ответ может происходить, например, каждые полгода (6 месяцев), ежегодно, каждые два года, каждые три или каждые пять лет.
«Образец» или «биологический образец» относится к любому твердому или жидкому образцу, выделенному из живого существа. В конкретном варианте выполнения настоящего изобретения оно относится к любому твердому (например, образцу ткани) или жидкому образцу, выделенному из млекопитающего, например молоку, материалу для биопсии (например, образцу твердой ткани), крови (например, плазме, сыворотке или цельной крови), слюне, синовиальной жидкости, моче, амниотической жидкости и спинномозговой жидкости. Такой образец может быть, например, свежим, фиксированным (например, фиксированным формалином, спиртом или ацетоном), погруженным в парафин или замороженным до анализа уровня экспрессии инфекционного агента.
Пациенты
Как применяется в настоящей заявке термин «субъект» или «пациент» относится к животному, предпочтительно млекопитающему, такому как, но не ограничиваясь этим, человек, корова (например, телка, многоплодная, первородящая, теленок), коза, овца, агница, осел, лошадь, свинья, курица, кошка, собака и т.д., которые являются объектом лечения, наблюдения или эксперимента. В конкретном варианте выполнения настоящего изобретения, это корова (например, с риском возникновения стафилококковой (например, IMI) инфекции).
Как применяется в настоящей заявке термины «субъект с риском развития стафилококковой инфекции (например, стафилококковой инфекции (например, IMI) или любого ее симптома в будущем» относится к млекопитающему (например, корова (например, телка, многоплодная, первородящая, теленок), коза, овца), которое используется для производства молока или мяса.
В варианте выполнения настоящего изобретения, вышеупомянутом млекопитающим является корова.
Способ обнаружения
Примеры методов измерения количества/уровня выбранных белков/полипептидов включают, но не ограничиваются ими: вестерн-блот, иммуноблот, энзим-связанный иммуносорбентный анализ (ELISA), радиоиммуноанализ (RIA), иммунопреципитацию, поверхностный плазмонный резонанс, хемилюминесценцию, флуоресцентная поляризация, фосфоресценция, иммуногистохимический анализ, масс-спектрометрия на основе матричной лазерной десорбции/времени ионизации (MALDI-TOF), микроцитометрия, микроматричный анализ, микроскопия, проточная цитометрия и анализы, основанные на свойстве белка, включая, но не ограничиваясь этим, связывание ДНК, связывание лигандом, взаимодействие с другими белковыми партнерами или ферментативная активность.
В варианте выполнения настоящего изобретения, количество полипептида/белка в способах согласно настоящему изобретению определяется с использованием антител, которые направлены специфически против полипептида/белка. Термин «антитело», как применяется в настоящей заявке, охватывает моноклональные антитела, поликлональные антитела, мультиспецифичные антитела (например, биспецифичные антитела) и фрагменты антител, при условии, что они проявляют желаемую биологическую активность или специфичность. «Фрагменты антител» содержат часть полноразмерного антитела, обычно его антигенсвязывающую или вариабельную область. Взаимодействия между антителами и целевым полипептидом выявляются радиометрическими, колориметрическими или флуорометрическими методами. Обнаружение комплексов антиген-антитело может быть достигнуто путем добавления вторичного антитела, которое связано с детектируемой меткой, такой как, например, фермент, флуорофор или хромофор.
Способы получения антител хорошо известны в данной области техники. Поликлональные антитела могут быть получены путем иммунизации подходящего субъекта (например, кролика, козы, мыши или другого млекопитающего) интересующим полипептидом/белком или его фрагментом в качестве иммуногена. «Фрагмент», «часть» или «сегмент» полипептида/белка представляет собой полоску аминокислотных остатков из по меньшей мере около 5, 7, 10, 14, 15, 20, 21 или более аминокислот полипептида, отмеченных выше. Титр антител у иммунизированного субъекта можно контролировать с течением времени стандартными методами, такими как иммуносорбентный анализ (ELISA) с использованием иммобилизованного экзосомального маркерного полипептида или его фрагмента. В подходящее время после иммунизации, например, когда титры антител являются наивысшими, продуцирующие антитела клетки можно получить от животного, обычно мыши, и их можно использовать для получения моноклональных антител стандартными методами, такими как методика гибридомы, первоначально описанная Kohler and Milstein (1975) Nature 256: 495-497, the human B cell hybridoma technique (Kozbor et al. (1983) Immunol. Today 4: 72), методика EBV-гибридомы (Cole et al. (1985) in Monoclonal Antibodies and Cancer Therapy, ed. Reisfeld and Sell (Alan R. Liss, Inc., New York, NY), pp. 77-96) или методика триомы. Методика получения гибридом хорошо известна (смотрите, в общем, Coligan et al., eds. (1994) Current Protocols in Immunology, John Wiley & Sons, Inc., New York, NY).
Альтернативно, для получения гибридом, секретирующих моноклональное антитело, моноклональное антитело может быть модифицировано и изолировано путем скрининга рекомбинантной комбинаторной библиотеки иммуноглобулинов (например, библиотеки антител фагового дисплея) с полипептидом или его фрагментом, чтобы, таким образом, выделить члены иммуноглобулиновой библиотеки, которые связывают полипептид. Наборы для генерации и скрининга библиотек фагового дисплея доступны коммерчески (например, у Pharmacia Recombinant Phage Antibody System, каталожный номер 27-9400-01; и Stratagene SurfZAP™ Phage Display Kit, каталожный номер 240612).
Кроме того, антитела, направленные против одного или более полипептидов/белков, описанных в настоящей заявке, могут быть получены из коммерческих источников.
Использование иммобилизованных антител, специфичных к полипептидам/белкам, также предусмотрено настоящим изобретением и хорошо известно специалисту в данной области. Антитела могут быть иммобилизованы на многообразии твердых носителей, таких как магнитные частицы или частицы хроматографической матрицы, поверхность места анализа (например, лунки для микротитрования), кусочки материала твердого субстрата (такого как пластик, нейлон, бумага) и подобное. Пластину для анализа можно приготовить, путем нанесения антитела или множества антител в массиве в виде покрытия на твердую подложку. Затем эту полоску можно окунуть в тестовый образец и затем быстро обработать с помощью промывок и стадий обнаружения для создания измеримого сигнала, такого как цветное пятно.
Анализ множества (2 или более) полипептидов/белков может проводиться отдельно или одновременно с одним тестируемым образцом. Несколько полипептидов/белков могут быть объединены в один тест для эффективной обработки множества образцов.
Анализ полипептидов/белков также может проводиться в различных физических форматах. Например, использование микротитровальных планшетов или автоматизация могут быть использованы для облегчения обработки большого количества тестируемых образцов. В качестве альтернативы могут быть разработаны форматы единичных образцов для своевременного лечения и диагностики. Особенно полезные физические форматы содержат поверхности, имеющие множество дискретных, адресуемых местоположений для обнаружения множества различных аналитов. Такие форматы включают белковые микрочипы или «белковые чипы» (смотрите, например, Ng and Ilag, J. Cell Mol. Med. 6: 329-340, 2002) и капиллярные устройства.
В варианте выполнения настоящего изобретения, вышеуказанный уровень экспрессии определяется путем измерения уровня экспрессии мРНК, транскрибированной с указанного одного или более генов.
Методы определения уровней нуклеиновой кислоты (мРНК) известны в данной области и включают, например, полимеразную цепную реакцию (ПЦР), ПЦР с обратной транскриптазой (ОТ-ПЦР), SAGE, количественную ПЦР (q-ПЦР), Саузерн-блот, Нозерн-блот, анализ последовательностей, микроматричный анализ, обнаружение репортерного гена или другие платформы гибридизации ДНК/РНК. Для экспрессии РНК предпочтительные способы включают, но не ограничиваются ими: экстракцию клеточной мРНК и нозерн-блоттинг с использованием меченых зондов, которые гибридизуются с транскриптами, кодирующими всю или часть одной или более нуклеиновых кислот, кодирующих белки/полипептиды по настоящему изобретению; амплификацию мРНК, экспрессируемой из одной или более нуклеиновых кислот, кодирующих белки/полипептиды данного изобретения, с использованием специфических праймеров, полимеразной цепной реакции (ПЦР), количественной ПЦР (q-ПЦР) и полимеразной цепной реакции с обратной транскриптазой (ОТ-ПЦР)) с последующим количественным определением продукта любым из множества способов; выделение тотальной РНК из биологического образца, который затем метят и используют для зондирования кДНК или олигонуклеотидов, кодирующих все или часть нуклеиновых кислот, кодирующих белки/полипептиды данного изобретения, расположенные на любой из множества поверхностей.
Наборы
Настоящее изобретение также охватывает наборы, содержащие компоненты настоящего изобретения. Например, набор может содержать один или более компонентов. Компоненты могут быть упакованы в подходящий контейнер и устройство для введения. Набор может дополнительно содержать инструкции по использованию набора.
Настоящее изобретение также обеспечивает набор или упаковку, содержащую реагенты, полезные для введения одного или более конструктов, полипептидов, нуклеиновой кислоты, вектора, хозяина, композиций согласно настоящему изобретению или комбинации по меньшей мере двух из них субъекту, нуждающемуся в этом, для лечения и/или профилактики стафилококковой IMI. Такой набор может дополнительно содержать, например, инструкции по профилактике и/или лечению стафилококковой IMI, контейнеры, реагенты, полезные для выполнения способов. Набор может дополнительно включать, когда это необходимо, агенты для уменьшения фоновых помех в тесте, агенты для увеличения сигнала, программное обеспечение и алгоритмы для объединения и интерполяции значений маркеров для получения прогнозируемого клинического исхода, представляющего интерес, аппарат для проведения теста, калибровочные кривые и диаграммы, кривые и диаграммы стандартизации и тому подобное.
Способ(ы) осуществления изобретения
Настоящее изобретение проиллюстрировано более подробно следующими неограничивающими примерами.
Пример 1: Материалы и способы для вакцины, включающей SACOL0029, SACOL0442, SACOL0720, SACOL1867, SACOL1912 и SACOL2385 (Вакцина №1)
Получение антигенов. Шесть антигенов, которые являются высокоэкспрессированными в ходе S. aureus интрамаммарной инфекции крупного рогатого скота отобрали для включения в первую бычью вакцину (Вакцина №1). Этими антигенами являются: SACOL0029 (GenBank номер доступа: YP_184940.1) (SEQ ID NO: 5), SACOL0442 (YP_185332.1) (SEQ ID NO: 29), SACOL0720 (YP_185601.1) (SEQ ID NO: 11), SACOL1867 (GenBank номер доступа: YP_186695.1) (SEQ ID NO: 38), SACOL1912 (GenBank номер доступа: YP_186737.1) (SEQ ID NO: 43), и SACOL2385 (GenBank номер доступа: YP_187189.1) (SEQ ID NO: 50). His-нацеленные рекомбинантные белки SACOL0029, SACOL1867, SACOL1912, и SACOL2385 были разработаны и произведены компанией GenScript, Inc. (Piscataway, NJ). His-нацеленные рекомбинантные белки SACOL0442 и SACOL0720 были разработаны и произведены с применением QIA экспрессионной методики (pQE30 плазмида) от Qiagen Inc. (Mississauga, ON, Canada), согласно рекомендациям производителя. Смотрите Фиг. 21I-VI для his-нацеленных последовательностей антигенов. Примеры 2-5 и Фиг. 1-4 относятся к этой вакцине №1.
Иммунизация молочных коров. Девятнадцать здоровых многократно рожавших голштинских коров в середине лактации содержались в биобезопасном сарае уровня II в Канадском научно-исследовательском центре сельского хозяйства и агропродовольственной отрасли молочной промышленности и свиней (Sherbrooke, QC). Коровы были случайным образом разделены на 2 группы: одна группа (10 коров) получала физиологический раствор (группа плацебо); другая группа (9 коров) получала вакцину № 1 (вакцинированная группа). Вакцина состояла из 300 мг каждого из шести антигенов (SACOL0029, SACOL0720, SACOL1867, SACOL1912, и SACOL2385), объединенных с EmulsigenTM-D (MVP Technologies, Omaha, NE), CpG ODN 2007 (TCGTCGTTGTCGTTTTGTCGTT (SEQ ID NO: 194), патоген-связанного молекулярного паттерна(PAMP), VIDO, Saskatoon, SW) и катионного пептида индолицидина (ILPWKWPWWPWRR (SEQ ID NO: 195), применяемая для индукции иммунного ответа у коров, (Chemprep Inc., Miami, FL). Две иммунизации были выполнены с интервалом в 10 недель, подкожно в области шеи. Побочных эффектов не наблюдалось. Кровь из хвостовой вены и пробы молока брали перед первой иммунизацией (преиммунная сыворотка), а затем каждые две недели для выявления общего IgG, IgG1 и IgG2. Большие объемы крови из яремной вены (150 мл отбирали до первой иммунизации и через 14 недель после первой иммунизации (то есть через 4 недели после второй иммунизации) для выделения мононуклеарных клеток периферической крови (РВМС) и анализа клеточных иммунных ответов.
Обнаружение общего IgG, IgG1 и IgG2 посредством ELISA. Обнаружение общего IgG, IgG1 и IgG2 против каждого из антигенов в сыворотке и молоке проводили как описано ранее с некоторыми модификациями (Ster et al., Vet. Immunol. Immunopathol. (2010), 136: 311-318). Nunc MaxiSorp™ 96-луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрыли тестовым антигеном (5 мкг/мл разбавленного в карбонатном/бикарбонатном буфере, Sigma Aldrich, Oakville, ON) и инкубировали всю ночь при 37°C. Планшеты затем насытили PBS, содержащим 0.5% желатина (BD, Franklin Lakes, NJ) в течение 1 часа при 37°C. Сто микролитров двухкратных серийных разбавлений сыворотки в PBS, содержащем 0.5% желатина и 0.1% Tween™ 20 загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты инкубировали три раза PBS, содержащим 0.1% Tween™ 20. Сто микролитров вторичных антител, конъюгированных с пероксидазой хрена (HRP) добавили на планшет. Применяемыми вторичными антителами были козлиные анти-бычьи IgG (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), a овечьи анти-бычьи IgG1 (AbD Serotec, Raleigh, NC) или овечьи анти-бычьи IgG2 (AbD Serotec), разбавленные 1/50,000 1/20,000 и 1/20,000 соответственно в PBS, содержащем 0.5% желатина и 0.1% Tween™ 20. Через 1 час инкубации при 37°C с последующими тремя промывками, пероксидазную активность обнаружили с помощью 3,3′,5,5′- тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD) согласно рекомендациям производителя.
Обнаружение общего IgG, IgG1 и IgG2 в молоке проводили с применением той же методики с несколькими модификациями. Образцы молока разбавили в PBS, содержащем 0.5% желатина. Овечьи анти-бычьи IgG2 разбавили 1/10,000 в PBS, содержащем 0.5% желатина и 0.1% TweenTM 20.
Развитие клеточного иммунного ответа. PBMC выделили из крови яремной вены и пометили карбоксифлуоресцеин диацетатом, сукцинимидиловым простым эфиром (CFDA-SE; Molecular Probes Inc., Eugene, OR) как описано ранее (Loiselle et al., J. Dairy. Sci. (2009), 92:1900-1912). В конце процедуры внесения метки CFDA-SE, PBMC суспендировали в среде RPMI, содержащей 5% FBS и 1X антибиотика/ антимикотика (A5955, Sigma Chemical Aldrich). PBMC (5×106 клеток на лунку) стимулировали митогеном конканавалином A (ConA; положительный контроль; Sigma Aldrich) при конечной концентрации 1 мкг/мл, или каждым антигеном (5 мкг на лунку) и инкубировали в течение 7 дней при 37°C. В качестве отрицательного контроля, PBMC инкубировали без какого-либо митогена. Стимуляции проводили дважды (Ster et al., 2010).
Пролиферацию CD4+ и CD8+ клеток оценивали после инкубации различными митогенами. Клетки центрифугировали при 300 × g в течение 5 мин, суспендировали в PBS, содержащем 0.5% BSA. Мышиные анти-бычьи CD8, связанные с Alexa Fluor™ 647 (разбавленные 1/20, AbD serotec) и мышиные анти-бычьи CD4, связанные с rPE (разбавленные 1/20, AbD serotec) затем добавили. После 20 мин инкубации на льду клетки трижды промывали PBS, содержащим 0,5% BSA. Затем клетки суспендировали в PBS с 0,5% формальдегидом. Процентное содержание пролиферативных популяций определяли с помощью проточной цитометрии на проточном цитометре BD FACS Canto II с использованием программного обеспечения BD FACS Diva.
Экспериментальные S. aureus при IMI у молочных коров. Перед их использованием при экспериментальной IMI была определена взаимосвязь между поглощением бактериальных культур (A600 нм) и КОЕ. В день заражения объем ночной культуры S. aureus в бульоне Мюллера-Хинтона (MHB; BD) переносили в 200 мл свежей MHB с получением A600 нм, равного 0,1 и затем выращивали при 35°C до достижения A600 нм значения, соответствующего 108 КОЕ /мл в экспоненциальной фазе роста. Штамм, который будет использоваться при этой экспериментальной инфекции (CLJ08-3), ранее был охарактеризован в лаборатории соавтора (Allard et al., Vet. Microbiol. (2013) 162: 761-770). Для внутримаммарных инфузий бактерии обычно разводили в стерильном PBS (Sigma Aldrich) для получения приблизительно 50 КОЕ в 3 мл. В этом эксперименте инокулят высевали на TSA и обнаружили, что он содержит 63 КОЕ в 3 мл.
Определение количества соматических клеток (SCC) и бактериальный анализ асептических образцов молока из вымя проводили до экспериментальной IMI, чтобы гарантировать, что все коровы не имели IMI. Экспериментальная инфузия вымя бактериями была выполнена в трех (произвольно выбранных) из четырех долей вымя каждой коровы после вечернего доения в соответствии с процедурой, описанной ранее (Petitclerc et al., J. Dairy. Sci. (2007), 90: 2778-2787) с небольшими изменениями. Вкратце, перед инокуляцией соски чистили марлей, пропитанной 70% этанолом. Соскам давали возможность высохнуть на воздухе перед внутримаммарной инфузией 3 мл бактериальной суспензии (содержащей 63 КОЕ) в три из четырех долей вымя. Сразу же после вливания все доли вымя тщательно массажировали, а соски погружали в дезинфицирующее средство для сосков на основе йодофора. Одноразовые перчатки носили на протяжении всей процедуры и дезинфицировали перед тем, как перейти к следующему животному. Все доли вымя, зараженные S. aureus, заразились, и у всех коров наблюдались клинические признаки (воспаление и/или плохое появление молока) мастита через некоторое время в течение первых нескольких дней после инфузии S. aureus.
Оценка количества жизнеспособных микроорганизмов S. aureus после экспериментальных инфекций. Пробы асептического молока отбирали до утреннего доения три раза в неделю в течение 3 первых недель после экспериментальной инфекции, а затем два раза в неделю в течение 2 оставшихся недель. После того, как первое молоко было выброшено и соски были продезинфицированы 70% этанолом, 10-мл пробу молока в асептических условиях собирали в 50-мл стерильный флакон для каждой отдельной доли вымя. Образцы молока серийно разводили и 100 мкл каждого разведения высевали на чашки с триптическим соевым агаром (Becton Dickinson) и чашки с агаром с солью маннита (Becton Dickinson) для определения КОЕ и идентификации S. aureus. Затем чашки инкубировали в течение 24 часов при 35°С до подсчета колоний. Разведения, которые показывали от 30 до 300 колоний, использовали для расчета концентрации бактерий. Каждое разведение высевали в двух экземплярах.
Оценка количества соматических клеток. С той же частотой, что и для образцов асептического молока, молоко собирали с использованием отдельных доильных аппаратов при утреннем доении и взвешивали для определения производства молока в доле вымя. Неасептический 50-мл образец также отбирали из каждой четверти доильных установок для определения SCC в коммерческой лаборатории (Valacta Inc., Ste-Anne-de-Bellevue, QC, Canada). Доильное оборудование тщательно промывали и дезинфицировали с помощью йодсодержащего бактерицидного моющего средства (K.O. Dyne®, GEA Farm Technologies, Westmoreland, NY) между их использованием на каждой корове. Все остальные материалы, соприкасающиеся с молоком, были продезинфицированы 70% этанолом.
Статистический анализ. Статистический анализ данных экспериментальной инфекции выполняли, используя процедуру MIXED SAS (SAS Institute Inc., Cary, NC) в качестве повторных измерений. Для анализа SCC и КОЕ данные были преобразованы в log10 до анализа. Статистический анализ титров антител и корреляции между КОЕ и SCC проводили с использованием GraphPad Prism™ v6.05.
Этические заявления. Все эксперименты на животных были одобрены местным институциональным комитетом по уходу за животными и проводились в соответствии с руководящими принципами Канадского совета по уходу за животными.
Пример 2: Титры общего IgG1 в сыворотке после вакцинации - вакцина №1
Рекомбинантные His-нацеленные антигены для SACOL0029 (GenBank номер доступа: YP_184940.1) (SEQ ID NO: 5), SACOL0442 (SEQ ID NO: 29), SACOL0720 (SEQ ID NO: 11), SACOL1867 (GenBank номер доступа: YP_186695.1) (SEQ ID NO: 38), SACOL1912 (GenBank номер доступа: YP_186737.1) (SEQ ID NO: 43), и SACOL2385 (GenBank номер доступа: YP_187189.1) (SEQ ID NO: 50), получали и вводили здоровым коровам, как описано в Примере 1 (Получение антигенов и Иммунизация молочных коров). Девять коров получали вакцину, и 10 коров получали физиологический раствор (плацебо). Титры общего IgG, IgG1 и IgG2 в сыворотке обнаруживали, как описано в Примере 1 (Обнаружение общего IgG, IgG1 и IgG2 посредством ELISA).
Как и ожидалось, и, как показано на фиг.1B-C, иммунизация индуцировала повышенную продукцию антиген-специфических сывороточных IgG1 и IgG2 для вакцинированной группы по сравнению с группой плацебо. Интересно, что соотношение IgG2 / IgG1 было 1 для SACOL0442 (см. Фиг. 1D), что свидетельствует о сбалансированном иммунном ответе Th1 / Th2 на этот антиген. Для антигенов SACOL0029, SACOL0720, SACOL1912 и SACOL2385 соотношение IgG2 / IgG1 значительно ниже, чем соотношение для SACOL0442, что указывает на то, что эти антигены индуцируют в основном ответ антител IgG1 через путь Th2.
Пример 3: Антиген-зависимая пролиферация клеток CD4+ и CD8+ в крови после вакцинации - вакцина №1
Антиген-зависимую пролиферацию клеток CD4 + и CD8 + в крови вакцинированных коров (9) и плацебо (10) оценивали, как описано в Примере 1 (Развитие клеточного иммунного ответа) через четыре недели после второй иммунизации (непосредственно перед экспериментальной инфекцией) для каждого антигена. Результаты для клеток CD4 + показаны на Фиг. 2, где каждый символ представляет процент клеток CD4 +, которые пролиферировали для каждой коровы после недели инкубации с положительным контролем (ConA) или каждым антигеном. Незакрашенные кружки (○) представляют данные для вакцинированных коров, черные квадраты (■) представляют данные для коров плацебо. Горизонтальные линии представляют медианы: пунктирные линии представляют медианы для вакцинированных коров, в то время как непрерывные линии представляют медианы для коров плацебо.
Символ * показывает статистические различия между группами вакцинированных коров и коров плацебо для антигенов SACOL0029, SACOL0442, SACOL0720 и SACOL1912 (*, P<0.05).
Кроме того, пролиферация CD8 + клеток была сходной для вакцинированных и плацебо-коров для всех антигенов, за исключением антигена SACOL0720 с более высокой пролиферацией клеток CD8 +, наблюдаемой для вакцинированных коров (данные не показаны). Индукция клеток CD8 + также, по-видимому, важна для исключения инфекции (Riollet et al., 2001; Burton and Erskine, 2003). Вакцина способна стимулировать как клеточный (CD8 +), так и гуморальный (CD4 +) иммунный ответ. Вакцина №1 с различными антигенами приводит к сбалансированному иммунному ответу.
Пример 4: Защитный эффект вакцины, как оценено посредством следующей оценки количества соматических клеток (SCC) - вакцина №1
Экспериментальную инфекцию S. aureus IMI у молочных коров проводили и оценивали, как описано в Примере 1 (Экспериментальная IMI S. aureus у молочных коров, Оценка количества жизнеспособных микроорганизмов S. aureus после экспериментальных инфекций, Оценка количества соматических клеток и Статистический анализ). Через четыре недели и 4 дня после второй иммунизации 63 КОЕ S. aureus были введены в 3 из 4 долей вымя вакцинированных (9) и плацебо коров (10) при вечернем доении (день 1, стрелка на Фиг. 3) , Образцы асептического молока отбирали во время утреннего доения и определяли SCC по Valacta (Ste-Anne-de-Bellevue, QC). Результаты показаны на Фиг. 3, на котором незакрашенные кружки (○) и пунктирная линия представляют данные для вакцинированных коров, в то время как черные квадраты (■) и непрерывная линия представляют данные для коров плацебо. Каждый кружок представляет среднее значение SCC для всех зараженных долей вымя вакцинированных коров (27), в то время как каждый квадрат представляет среднее значение SCC для всех зараженных долей вымя коров плацебо (30 долей).
В течение периода заражения было установлено, что SCC в молоке значительно ниже для вакцинированных коров, чем для плацебо (***; P <0,001), что указывает на меньшее воспаление и лучший контроль над инфекцией у вакцинированных коров.
Пример 5: Корреляция между SCC или количеством жизнеспособных микроорганизмов S. aureus (CFU) относительно титров IgG в сыворотке или молоке против конкретных антигенов - вакцина №1
Как показано на Фиг. 4A-B, SCC положительно коррелировали с КОЕ S. aureus периода заражения (Фиг. 4A, r = 0,82, P <0,0001) и отрицательно коррелировали с сывороточным титром IgG1 против SACOL0442, измеренным до инфекции (Фиг. 4B, r = -0,49, P <0,05). Таким образом, вакцинация снизила этот критерий воспаления, вызванного заражением. Аналогичный анализ был проведен с образцами, собранными в день 10. Те же корреляции наблюдались, как и ранее, но в этот конкретный момент времени SCC и КОЕ S. aureus также коррелировали с титром IgG2 в молоке против SACOL0029 (Фиг. 4C, r = -0,48. , P <0,05 и r = -0,58, P <0,05 соответственно). Эти результаты показывают, что более чем один антиген участвует в иммунном ответе против инфекции.
Пример 6: Материалы и способы для вакцины, включающей SACOL0442, SACOL0720, и слияние между SACOL1867 и SACOL0029 - вакцина №2
Получение антигенов. Четыре антигена, которые являются высоко экспрессированными в ходе S. aureus интрамаммарной инфекции крупного рогатого скота, отбирали для включения в вакцину №2. Антигены представляют собой полипептиды, кодируемые: SACOL0029 (GenBank номер доступа: YP_184940.1) (SEQ ID NO: 5), SACOL1867 (GenBank номер доступа: YP_186695.1) (SEQ ID NO: 38), SACOL0442 (SEQ ID NO: 29), и SACOL0720 (SEQ ID NO: 11). SACOL0029 и SACOL1867 антигены включили в форму слияния. His-нацеленные рекомбинантные белки SACOL0720 и SACOL0029-1867 были разработаны и произведены компанией GenScript, Inc. (Piscataway, NJ). His-нацеленные рекомбинантные белки SACOL0442 были разработаны и произведены с применением QIA экспрессионной методики (pQE30 плазмида) от Qiagen Inc. (Mississauga, ON, Canada), согласно рекомендациям производителя (смотрите Фиг. 21D-E и I, и пункты II, III и VII для SACOL0720, SACOL0442, и SACOL0029-1867 his-нацеленных последовательностей). Поверхностный белок ClfA (SEQ ID NO: 184) также дополнительно получали с использованием экспрессионного вектора QIA путем клонирования гена clfA из S. aureus ATCC 25904. Последний рекомбинантный белок не был частью вакцинной композиции, но был использован в ELISA анализах для определения титров IgG сыворотки против других белков S. aureus, таких как ClfA.
Вакцина состояла из 300 мкг каждого из 3 антигенов (SACOL0442 и SACOL0720 как определено в примере 1 и слияния SACOL0029-1867) и с Emulsigen™-D (MVP Technologies, Omaha, NE), CpG ODN 2007 (т.e. TCGTCGTTGTCGTTTTGTCGTT (SEQ ID NO: 194) (IDT, Coralville, IA)), и индолицидином (ILPWKWPWWPWRR (SEQ ID NO: 195, GenScript, Piscataway, NJ, Chemprep Inc., Miami, FL) (ванцина№2).
Иммунизация молочных коров. Одиннадцать здоровых многородящих голштинских коров в середине лактации содержались в биобезопасном сарае уровня II в Канадском научно-исследовательском центре сельского хозяйства и агропромышленного комплекса «Молочные продукты и свиньи» (Sherbrooke, QC). Коровы получили вакцину № 2 (вакцинированная группа). Две иммунизации были выполнены с интервалом в 10 недель, подкожно в области шеи. Побочных эффектов не наблюдалось. Кровь из хвостовой вены и пробы молока брали до первой иммунизации (преиммунная сыворотка), а затем каждую неделю для обнаружения общего IgG.
Обнаружение общего IgG посредством ELISA. Обнаружение общего IgG против каждого из антигенов в сыворотке проводили как описано ранее с некоторыми модификациями (Ster et al., Vet. Immunol. Immunopathol. (2010), 136: 311-318). Nunc MaxiSorp™ 96-ти луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрыли тестовым антигеном (5 мкг/мл разбавленного в карбонатном/бикарбонатном буфере, Sigma Aldrich, Oakville, ON) и инкубировали всю ночь при 37°C. Планшеты затем насытили PBS, содержащим 0.5% желатина (BD, Franklin Lakes, NJ) в течение 1 часа при 37°C. Сто микролитров двухкратных серийных разбавлений сыворотки в PBS, содержащем 0.5% желатина и 0.1% Tween™ 20 загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты инкубировали три раза PBS, содержащим 0.1% Tween™ 20. Сто микролитров вторичных антител, конъюгированных с пероксидазой хрена (HRP)добавили на планшет. Применяемыми вторичными антителами были козлиные анти-бычьи IgG (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), разбавленные 1/1000,000 в PBS, содержащем 0.5% желатина и 0.1% Tween™ 20. Через 1 час инкубации при 37°C с последующими тремя промывками, пероксидазную активность обнаружили с помощью 3,3′,5,5′-тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD) согласно рекомендациям производителя.
Пример 7: Слияние антигенов индуцирует высокие титры антител - вакцина №2
Фиг. 5 показывает титры общего IgG в сыворотке для вакцинированных коров для каждого антигена вакцины (включая слитые антигены SACOL0029 и SACOL1867 (помеченные SACOL0029-1867 на Фиг. 5). Каждый открытый кружок представляет титр через четыре недели после второй иммунизации для каждой коровы (непосредственно перед экспериментальным заражением), в то время как каждый черный ромб представляет преиммунный титр. Горизонтальные линии представляют медианы: сплошная линия для преиммунных сывороток, пунктирная линия для образцов, взятых через четыре недели после иммунизации. Титры для вакцинированных коров выше, чем титры преиммунных сывороток (**, P <0,01; ***, P <0,001 для других протестированных антигенов).
Фиг. 5, таким образом, показывает, что вакцина, состоящая из трех отдельных антигенов, включая слитый пептид, вызывает сильный иммунный ответ у коров. Кроме того, Фиг. 5 неожиданно показывает, что слитые антигены SACOL0029 и SACOL1867 (слияние SACOL0029-SACOL1867) повышают титры антител, которые были выше, чем титры, вызываемые каждым из антигенов отдельно (сравните Фиг. 1A и Фиг. 5), и что такие слитые антигены обеспечивают дополнительное преимущество вакцине.
Более конкретно, при индивидуальном введении в вакцине титры иммунных коров против SACOL0029 и SACOL1867 достигали 3200 и 51200 соответственно (Фиг. 1A), тогда как когда эти антигены вводили в виде слияния, титры иммунных коров достигали 12800 и 409600, соответственно (Фиг. 5), показывая, что слияние создает неожиданный синергизм в иммунном ответе.
Пример 8: Материалы и методы для вакцины, содержащей SACOL0029, SACOL1867, и слияние между SACOL1867 и SACOL0029 - вакцина №3
Получение антигенов. Три антигена, происходящие из двух генов, которые высоко экспрессированы в ходе S. aureus интрамаммарной инфекции крупного рогатого скота, отбирали для включения в вакцину. Этими антигенами были: SACOL0029 (GenBank номер доступа: YP_184940.1) (SEQ ID NO: 5), SACOL1867 (GenBank номер доступа: YP_186695.1) (SEQ ID NO: 38) и слияние между SACOL1867 и SACOL0029 (GenBank номер доступа: YP_184940.1) (SEQ ID NO: 5). His-нацеленные рекомбинантные белки SACOL0029, SACOL1867 и SACOL0029-1867 были разработаны и произведены компанией GenScript, Inc. (Piscataway, NJ) (смотрите Фиг. 21A, F и I, пункты I, IV и VII для SACOL0029, SACOL1867, и SACOL0029-1867 his-нацеленных последовательностей).
Иммунизация мыши. Иммуногенные свойства рекомбинантных белков S. aureus, кодируемых генами SACOL0029, SACOL1867 и слиянием SACOL0029 и SACOL1867, оценивали на мышах. Четыре группы мышей, получивших точное эквимолярное количество белков, либо в одновалентной форме (SACOL0029 или SACOL1867), либо в мультивалентной форме (слитый SACOL0029-1867 или SACOL0029 вместе с SACOL1867 в комбинации), сравнивали.
Вкратце, теоретическая молекулярная масса каждой аминокислотной последовательности, соответствующая всему слиянию или части слияния SACOL0029 или SACOL1867, была рассчитана с использованием портала биоинформационных ресурсов ExPASy ™ (http://web.expasy.org/cgi-bin/compute_pi/pi_tool).
Пять микрограммов слияния вводили одной группе мышей. Было определено, что соответствующее молярное количество 5 мкг слияния SACOL0029-1867 составляет 168,55 пмоль относительно его теоретической молекулярной массы. Количество 1,15 мкг и 3,69 мкг SACOL0029 и SACOL1867, соответственно, вводили двум другим группам мышей, чтобы обеспечить 168,55 пмоль каждого антигена соответственно. Последняя группа мышей получала комбинацию двух отдельных антигенов (168,55 пмоль каждого).
Для приготовления иммунизационных доз SACOL0029, SACOL1867 и гибридные полипептиды SACOL0029-1867 индивидуально смешивали и суспендировали в PBS для получения конечного эквимолярного количества каждой антигенной дозы в объеме 100 мкл. Двадцать самок мышей CD-1 были случайным образом разделены на 4 группы: группа А (5 мышей) получала 5 мкг слитого белка SACOL0029-1867 (Fusion); группа B (5 мышей) получала 1,15 мкг SACOL0029 и 3,69 мкг SACOL1867 (комбинация); группа C (5 мышей) получала 1,15 мкг SACOL0029 (0029), а группа D получала 3,69 мкг SACOL1867 (1867). Мышей CD-1 иммунизировали двумя подкожными инъекциями в шею с интервалом в две недели. Никаких побочных эффектов не наблюдалось в течение всего периода экспериментальной иммунизации. Пробы крови брали непосредственно перед первой первичной инъекцией (преиммунные сыворотки) и через десять дней после бустерной иммунизации (иммунные сыворотки). Аликвоты крови оставляли для свертывания при комнатной температуре в течение часа, центрифугировали при 10000 g в течение 10 минут при 4°С. Сыворотки собирали и хранили при -20°С до последующего анализа.
Обнаружение общего IgG посредством ELISA. Обнаружение общего сывороточного IgG против SACOL0029 и SACOL1867 рекомбинантных белков проводили, как описано ранее с некоторыми модификациями (Ster et al., Vet. Immunol. Immunopathol. (2010), 136: 311-318). Nunc MaxiSorp™ 96-ти луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрыли с 75 мкл каждого тестируемого антигена (6.67 мкг/мл разбавленного в карбонатном/бикарбонатном буфере, Sigma Aldrich, Oakville, ON) и инкубировали всю ночь при комнатной температуре. Планшеты затем насытили PBS, содержащего 5% сухого обезжиренного молока, в течение 1 часа при 37°C. Сто микролитров четырехкратных серийных разбавлений в PBS, содержащем 3% молока и 0.025% Tween™ 20, загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты инкубировали три раза PBS, содержащим 0.05% Tween™ 20. Сто микролитров вторичных антител, конъюгированных с пероксидазой хрена (HRP) затем добавили на планшет. Вторичным применяемым антителом был коммерчески доступный козлиный анти-мышиный IgG (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), разбавленный 1/5000 в PBS, содержащем 3% молока и 0.025% Tween™ 20. Через 1 час инкубации при 37°C с последующими тремя промывками, пероксидазную активность обнаружили с помощью добавления ста микролитров 3,3′,5,5′-тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD, согласно рекомендациям производителя.
Статистический анализ. Статистический анализ титров антител и корреляции проводили с применением GraphPad Prism™ v6.05.
Пример 9: Слияние антигенов индуцирует значительно более высокие титры антител по сравнению с одновалентными антигенами или комбинацией антигенов - вакцина №3
Фиг. 6 показывает, что антиген (SACOL1867), включенный в слитый полипептид (то есть слитый белок SACOL0029-1867), может вызывать сильный и специфический иммунный ответ антител против этого специфического антигена (SACOL1867) и, что более важно, этот ответ может быть значительно выше, чем полученный с одновалентной формой этого антигена (SACOL1867, вводимый отдельно) или с мультивалентной комбинацией отдельных полипептидов, которые являются частью слияния (комбинация SACOL1867 плюс SACOL0029). Таким образом, в дополнение к преимуществу создания иммунного ответа против множества полипептидных мишеней такие слитые антигены также обеспечивают дополнительное преимущество значительного улучшения титров антител против этих мишеней.
Пример 10: Материалы и способы для вакцин, включающих SACOL0720 фрагмент(ы) и/или SACOL442 фрагмент(ы) - вакцины №4-6
Получение антигенов. Пептиды и аминокислотные фрагменты длиной от 15 до 50 аминокислот, полученные из последовательностей SACOL0442 и / или SACOL0720, были отобраны на основании присутствия В-клеточных эпитопов. Также были сконструированы слияния пептидных эпитопов, в которых аминокислотный линкер (например, EAAAKEAAAK (SEQ ID NO: 62), или ERKYK (SEQ ID NO: 61), или KDYERKYKKHIVS (SEQ ID NO: 196)) присоединялся к различным эпитопам. Пептиды и аминокислотные фрагменты были синтезированы компанией Biomatik, Inc. (Cambridge, ON). После получения лиофилизированные пептиды и аминокислотные фрагменты суспендировали в стерильной воде при концентрации 5 мг/мл и хранили при -80°C до дня использования.
Иммунизация мыши. Пептиды и аминокислотные фрагменты использовали в качестве антигенов для иммунизации мышей. Для приготовления доз для иммунизации каждый пептид и аминокислотный фрагмент или их комбинацию смешивали и суспендировали в PBS, содержащем 20% адъюванта эмульсии типа масло-в-воде EMULSIGEN-D, для получения конечной дозы 100 мкг полипептида на дозу, если не указано иное. Самок мышей CD-1 случайным образом разделили на разные группы из 3-4 животных. Мышей иммунизировали двумя подкожными инъекциями в шею с интервалом в две недели. В течение всего экспериментального периода побочных эффектов не наблюдалось. Пробы крови брали непосредственно перед первой первичной инъекцией (преиммунные сыворотки) и через десять дней после бустерной иммунизации (иммунные сыворотки). Аликвоты крови оставляли для свертывания при комнатной температуре в течение часа, а затем центрифугировали при 10000 g в течение 10 минут при 4°С. Сыворотки собирали и хранили при -20°С до последующего анализа.
Обнаружение общего IgG посредством ELISA. Обнаружение общего сывороточного IgG, против специфических аминокислотных последовательностей, обнаруживаемых в антигенах, применяемых для иммунизации мышей, проводили как описано ранее с некоторыми модификациями (Ster et al., Vet. Immunol. Immunopathol. (2010), 136: 311-318). Nunc MaxiSorp™ 96-ти луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрывали 100 мкг каждой из целевых аминокислотных последовательностей, разбавленных при конечной концентрации 5 мкг/мл в карбонатном/бикарбонатном буфере (Sigma Aldrich, Oakville, ON) и инкубировали всю ночь при комнатной температуре. Планшеты затем насыщаля PBS, содержащим 5% сухого обезжиренного молока в течение 1 часа при 37°C. Сто микролитров четырехкратных или двукратных разбавлении сыворотки в PBS, содержащем 1% молока и 0.025% Tween™ 20 загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты затем промыли трижды PBS, содержащим 0.05% Tween™ 20. Сто микролитров вторичных антител, конъюгированных с пероксидазой хрена (HRP), затем добавили на планшет. Применяемыми вторичными антителами были козлиные анти-мышиные IgG (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), разбавленные 1/5000 в PBS, содержащем 1% молока и 0.025% Tween™ 20. Через 1 час инкубации при 37°C с последующими тремя промывками с PBS Tween™ 20 и конечной промывкой с PBS, пероксидазную активность обнаружили с помощью 3,3′,5,5′-тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD) согласно рекомендациям производителя.
Статистический анализ. Статистический анализ титров антител и оптических плотностей проводили с использованием GraphPad Prism™ v6.05.
Пример 11: Иммунный ответ против слияния пептидов, который включает эпитопы, кодируемые последовательностями SACOL0442 и SACOL0720 - вакцина №4
Слияние пептидных эпитопов, кодируемых SACOL0442 и SACOL0720, применяли для вакцинации мышей (n = 4). Последовательностью слияния пептидов была KDGGKYTLESHKELQEAAAKEAAAKKDINKIYFMTDVDLGGPTFVLND (SEQ ID NO: 3) (вакцина №4), где линкер выделен курсивом, и различные эпитопы выделены жирным шрифтом. Эпитопами были KDGGKYTLESHKELQ (SEQ ID NO: 1), кодируемый SACOL0442, KDINKIYFMTDVDL (SEQ ID NO: 23), кодируемый SACOL0720, и DVDLGGPTFVLND (SEQ ID NO: 24), также кодируемый SACOL0720. Антитела IgG из сывороток, собранных у животных, были способны связывать аминокислотные фрагменты, содержащие В-клеточные эпитопы, из либо SACOL0442 (т.е. KDGGKYTLESHKELQ (SEQ ID NO: 1)), и / или SACOL0720 (т.е. QFGFDLKHKKDALA (SEQ ID NO: 21); KDINKIYFMTDVDL (SEQ ID NO: 23), DVDLGGPTFVLND (SEQ ID NO: 24)) в анализах ELISA с титрами антител 1/6400 или выше. Слияние пептидов, используемых для иммунизации, и аминокислотных фрагментов или полипептидов, используемых в качестве мишеней антител в анализах ELISA, показано в таблице III ниже. В этой таблице эпитопы выделены жирным шрифтом, а последовательность линкера выделена курсивом.
Таблица III - Полипептидная вакцина и мишени ответа антител
Все мишени антител, показанные выше, были связаны в анализе ELISA посредством IgG из мышей, вакцинированных слитым антигеном выше.
Это демонстрирует, что слияние пептидных эпитопов, кодируемых как SACOL0442, так и SACOL0720, может быть использовано для иммунизации и вызова иммунного ответа у млекопитающего. Полученный иммунный ответ включает продукцию антител, которые распознают SACOL0442 или SACOL0720, аминокислотные фрагменты или варианты, кодированные SACOL0442 или SACOL0720.
Пример 12: Слияние мультиплетных эпитопов, применяемое в качестве антигена при иммунизации, значительно усиливает иммунный ответ против одного эпитопа - вакцина №4
Слияние пептидных эпитопов, кодируемых SACOL0442 и SACOL0720, применяли для вакцинации мышей (n = 4). Последовательность слияния пептидов представляла собой KDGGKYTLESHKELQEAAAKEAAAKKDINKIYFMTDVDLGGPTFVLND (SEQ ID NO: 3), где линкер выделен курсивом, а различные эпитопы выделены жирным шрифтом. Эпитопами были KDGGKYTLESHKELQ (SEQ ID NO: 1), кодированные SACOL0442, KDINKIYFMTDVDL (SEQ ID NO: 23), кодированные SACOL0720, и DVDLGGPTFVLND (SEQ ID NO: 24), также кодированные SACOL0720. Другую группу мышей (n = 4) иммунизировали одним пептидным эпитопом KDGGKYTLESHKELQ (SEQ ID NO: 1), кодируемым SACOL0442.
Сыворотки собирали у животных и тестировали на наличие антител IgG, направленных на аминокислотный фрагмент, кодированный SACOL0442 (GEHLPKGNIVINTKDGGKYTLESHKELQKDRENVKINTAD) (SEQ ID NO: 2), который содержит пептидный эпитоп KDGGKYTLESHKELQ (SEQ ID NO: 1).
Как показано на Фиг. 7, иммунизация слиянием трех пептидных эпитопов (один из которых кодируется SACOL0442, а два - SACOL0720) значительно увеличивала выработку антител против аминокислотного фрагмента, кодированного SACOL0442 (GEHLPKGNIVINTKDGGKYTLESHKELQKDRENVKINTAD) (SEQ ID NO: 2), который содержит пептидный эпитоп KDGGKYTLESHKELQ (SEQ ID NO: 1), по сравнению с уровнем антител, полученным при применении только пептидного эпитопа KDGGKYTLESHKELQ (SEQ ID NO: 1) в качестве антигена для иммунизации.
Пример 13: Иммунный ответ против полипептидного фрагмента из 50 аминокислот, кодируемого вариантом SACOL0720 - вакцина №5
Группу мышей (n = 3) вакцинировали пептидным фрагментом из 50-аминокислот (KDINKIYFMTDVDLGGPTFVLNDKDYERKYKKHIVSQFGFDLKHKKDALA (SEQ ID NO: 27)) (вакцина №5), содержащим B-клеточные эпитопы (выделено жирным шрифтом) кодируемое последовательностью SACOL0720, более специфично эпитопы KDINKIYFMTDVDL (SEQ ID NO: 23), DVDLGGPTFVLND (SEQ ID NO: 24) и QFGFDLKHKKDALA (SEQ ID NO: 21). Общая последовательность KDINKIYFMTDVDLGGPTFVLNDKDYERKYKKHIVSQFGFDLKHKKDALA (SEQ ID NO: 27) отличается от нативной последовательности SACOL0720 четырьмя аминокислотами в области связывания эпитопов DVDLGGPTFVLND (SEQ ID NO: 24) и QFGFDLKHKKDALA (SEQ ID NO: 21). Таким образом, вакцину № 5 можно рассматривать как вариантный фрагмент SACOL0720 или слияние эпитопов из SACOL0720, которые разделены линкером ERKYK (SEQ ID NO: 61).
Сыворотки тестировали на присутствие антител IgG, направленных на фрагмент нативного белка, кодируемого SACOL0720 (SEQ ID NO: 25) (то есть полигистидиновая версия, показанная на Фиг. 21D, пункт II). Обе мыши, вакцинированные пептидным фрагментом, соответствующим вариантной последовательности аминокислот (или слияния SACOL0720-720), продуцировали антитела, которые распознавали эпитопы в исходной последовательности аминокислот в анализе ELISA с титрами 1/6400 или выше.
Это демонстрирует, что аминокислотный фрагмент, который содержит эпитопы, кодированные последовательностью SACOL0720, может вызывать иммунный ответ у млекопитающего. Это также дополнительно демонстрирует, что вариант нативной последовательности обладает способностью стимулировать иммунную систему против исходной последовательности фрагмента, содержащей B-клеточные эпитопы.
Пример 14: Иммунный ответ против комбинации слияний (пептидное слияние 0442-0720 и полипептидное слияние 0029-1867) - вакцина №6
Слияние пептидных эпитопов, кодируемых SACOL0442 и SACOL0720 (смотрите последовательность на Фиг. 21I, пункт VII - слияния), объединяли с полипептидным слиянием, содержащим последовательности SACOL0029 и SACOL1867 (смотрите последовательность на Фиг. 21I, пункт VII слияния) и использовали, чтобы вакцинировать мышей (вакцина №6).
Для приготовления доз иммунизации пептидное слияние 0442-0720 и полипептидное слияние 1867-0029 смешивали и суспендировали в PBS, содержащем 20% адъюванта эмульсии типа масло-в-воде EMULSIGEN®-D, чтобы получить конечную дозу 100 мкг и 5 мкг на дозу пептидного слияния (0442-0720) и полипептидного слияния (0029-1867), соответственно. Самок мышей CD-1 (n = 3) иммунизировали тремя подкожными инъекциями в шею. Первые две инъекции были сделаны с интервалом в одну неделю, а третья инъекция через 3 недели после второй. В течение всего экспериментального периода побочных эффектов не наблюдалось. Образцы крови брали непосредственно перед первой первичной инъекцией (преиммунные сыворотки) и через четырнадцать дней после последней иммунизации (иммунные сыворотки). Аликвоты крови оставляли для свертывания при комнатной температуре в течение часа, а затем центрифугировали при 10000 g в течение 10 минут при 4°С. Сыворотки собирали и хранили при -20°С до последующего анализа.
Антитела IgG из сывороток, собранных у животных, были способны связывать аминокислотные фрагменты, содержащие эпитопы из SACOL0442 или SACOL0720, или полипептиды SACOL0029 или SACOL1867 в анализах ELISA, с титрами антител 1/6400 или выше. Слияние пептидов и полипептидов, применяемое для иммунизации, и полипептиды или аминокислотные фрагменты, используемые в качестве мишеней антител в анализах ELISA, показаны в Таблице IV ниже.
Таблица IV - Смешанная полипептидная слитая вакцина и мишени ответа антител
Эпитопы выделены жирным шрифтом, а последовательность линкера выделена курсивом
SACOL1867 (SEQ ID NO: 40) (смотрите his-нацеленную последовательность на Фиг. 21F, Пункт IV);
Это демонстрирует, что комбинация слияний (например, пептидный слияние 0442-0720 в смеси с полипептидным слиянием 0029-1867) может быть использована для иммунизации и выявления иммунного ответа у млекопитающего. Полученный иммунный ответ включает продуцирование антител, которые распознают аминокислотные последовательности, кодируемые либо SACOL0442, либо SACOL0720, либо SACOL0029, либо SACOL1867.
Пример 15: Материалы и способы для аттенуированного живого мутанта
Бактериальные штаммы и условия роста. Штаммы, использованные в примерах 15-25, перечислены в таблице V. S. aureus ATCC 29213 и его изогенный мутант 720 были описаны ранее (Allard et al., 2013). Если не указано иное, штаммы S. aureus выращивали в триптическом соевом бульоне (TSB) и агаре (TSA) (BD, ON, Канада), а Escherichia coli DH5α выращивали в среде LB и LBA (BD). При необходимости на агаровые планшеты добавляли ампициллин (100 мкг/мл) (Sigma, Oakville, Ontario, Canada), хлорамфеникол (20 мкг/мл) (ICN Biomedicals, Irvine, CA) и эритромицин (10 мкг/мл) (Sigma). Для иммунологических тестов были отобраны четыре различных изолята бычьего мастита, соответствующие некоторым из преобладающих типов S.aureus spa, обнаруженных в канадских молочных стадах и в других странах мира (Veh et al., 2015; Mitra et al., 2013). Штамм SHY97-3906 (spa t529) был выделен из случая клинического мастита крупного рогатого скота, имевшего место в период лактации, а CLJ08-3 (spa t359) был первоначально выделен у коровы с персистирующим маститом при осушении (Allard et al. , 2013). Штаммы Sa3151 (spa t13401) и Sa3181 (spa t267) были получены из Канадской сети исследований качества говядины и мастита (CBMMQRN), коллекции патогенных культур мастита (Университет Монреаля, Faculté de Médecine Vétérinaire, St-Hyacinthe, QC, Canada) и были выделены из случаев субклинических внутримаммарных инфекций.
Таблица V - Штаммы и плазмиды, применяемые в примерах 15-25
Условия клеточной культуры. Установленная линия эпителиальных клеток молочной железы крупного рогатого скота (BMEC), MAC-T (Huynh et al., 1991), была использована в качестве модели инфекции на клеточной культуре. Клетки MAC-T обычно культивировали и содержали в модифицированной Дульбекко среде Игла (DMEM), содержащей 10% инактивированной нагреванием фетальной бычьей сыворотки (FBS), с добавлением 5 мкг/мл инсулина (Roche Diagnostics Inc., Laval, Canada) и 1 мкг/мл гидрокортизона (Sigma), и инкубировали при 37°C в увлажненном инкубаторе с 5% CO2. Реагенты для клеточных культур были приобретены у Wisent (St-Bruno, QC, Canada).
Манипуляции с ДНК. Следовали рекомендациям производителей наборов для выделения геномной ДНК (Sigma), выделения плазмидной ДНК (Qiagen, ON, Canada), выделения фрагментов ДНК из агарозных гелей (Qiagen) и очистки продуктов ПЦР и расщепленных фрагментов ДНК (Qiagen). Дополнительную обработку в течение 1 часа лизостафином (Sigma) при концентрации 200 мкг/мл использовали для достижения эффективного лизиса клеток S. aureus при выделении геномной и плазмидной ДНК. Праймеры (IDT® Integrated DNA Technologies; Coraville, Iowa, USA) были разработаны для добавления сайтов рестрикции в 3'-5' направлении и в 5'-3' направлении от амплифицированных продуктов. ПЦР проводили с использованием ДНК-полимеразы Taq (NEB, Pickering, ON, Canada) для обычной ПЦР или высококачественной ДНК-полимеразы Q5 (NEB) для клонирования, и время и температуры цикла оптимизировали для каждой пары праймеров. Плазмидные конструкции были получены с использованием E. coli DH5a (Invitrogen, Burlington, ON, Canada), рестрикционных ферментов (NEB) и ДНК-лигазы T4 (NEB). Плазмидные конструкты были подтверждены с помощью паттернов рестрикционного расщепления и секвенирования ДНК перед электропорацией в S. aureus RN4220 (Kreiswirth et al., 1983) и в конечных штаммах-хозяевах. Плазмиды, использованные в примерах 15-25, перечислены в таблице V выше.
Образование аттенуированного штамма S. aureus Δ720 и ΔhemB. Был сконструирован изогенный мутант hemB штамма ATCC 29213, в котором ген hemB был удален и заменен вставкой кассеты emrA путем гомологичной рекомбинации. Мутант S. aureus ATCC 29213 для гена SACOL0720 (Δ720) был создан с использованием системы нокаут гена TargeTron ™ (с TargeTron ™ Vector pNL9164 (Sigma-Aldrich Canada Ltd.) (Chen et al., 2007) для разрушения бактериальных генов посредством вставки интронов группы II (размер фрагмента около 2 т.о., как описано ранее (Allard et al., 2013) между нуклеотидами 803 и 804 в S. aureus ATCC29213. Следовали протоколам и рекомендациям производителя.
Образование ΔhemBΔ720. Для достижения второй мутации в гене hemB с целью получения фенотипа SCV на генетическом фоне мутанта Δ720 была использована другая стратегия: чувствительный к температуре pBT2-hemB: emrA (pBT-E: hemB) был использован в стратегии, описанной ранее (Mitchell et al., 2008), с некоторыми модификациями. Вкратце, плазмида pBT-E была сконструирована вставкой кассеты ermA между сайтами XbaI и SalI чувствительного к температуре шаттл-вектора pBT2 (Brückner, 1997). Фланкирующие участки фрагментов ДНК гена hemB (SACOL1715) были амплифицированы из S. aureus ATCC 29213 и были клонированы с обеих сторон кассеты ermA в плазмиду pBT-E. Затем плазмиду переносили для размножения в S. aureus RN4220 (res-). После бактериального лизиса лизостафином (200 мкг/мл в течение 1 часа при комнатной температуре) выделяли плазмидную ДНК и использовали для трансформации ATCC 29213 и Δ720 путем электропорации. Для интеграции плазмиды и генерации мутанта бактерии сначала выращивали в течение ночи при 30°C с добавлением 10 мкг/мл эритромицина и добавкой гемина 1 мкг/мл (Sigma-Aldrich, ON, Canada). Бактерии затем разбавляли 1: 1000 и выращивали в течение ночи при 42°С с 2,5 мкг/мл эритромицина и 1 мкг/мл гемина. Этот шаг был повторен дважды. Наконец, бактерии разводили 1: 1000 и выращивали в течение ночи при 42°С без антибиотиков. Мутанты с инактивированным геном hemB были отобраны как устойчивые к эритромицину и чувствительные к хлорамфениколу, вместе с фенотипом SCV, который может быть дополнен (то есть возвращение к нормальному фенотипу роста) добавкой гемина 5 мкг/мл на чашки с агаром. Удаление hemB в штаммах АТСС 29213 (т.е. ΔhemB) и Δ720 (то есть ΔhemBΔ720) было подтверждено с помощью ПЦР (см. Фиг. 8A и B).
Добавка гемина в бульонную культуру. Чтобы оценить способность гемина восстанавливать оптимальную кинетику роста S. aureus ΔhemB и двойного мутанта Δ720ΔhemB, ночные бактериальные культуры разводили до A600нм приблизительно 0,1 в пробирках для культивирования, содержащих свежий BHI, дополненный гемином (Sigma), добавляемый при различных концентрациях. A600нм культур контролировали в разные моменты времени в течение периода инкубации при 35°C (225 об. в инфекция эпителиальных клеток молочной железы крупного рогатого скота (BMEC). BMEC MAC-T были использованы для характеристики внутриклеточной инфекционности и устойчивости ATCC 29213 (WT) и его изогенных мутантов. За сорок восемь часов до заражения на обработанный 24-ти луночный планшет (Corning) высевали 1x105/мл клеток MAC-T с получением 30% слияния. Монослои выращивали до слияния под 10% CO2 при 37°С. За шесть часов до заражения монослои промывали DMEM и инкубировали с инвазивной средой (IM) (ростовая среда без антибиотиков, содержащая 1% инактивированной нагреванием FBS). Ночные бактериальные культуры разводили 1:20 в свежем TSB и выращивали до середины логарифмической фазы роста, затем промывали PBS и разводили в IM до множественности инфекции, равной 10. Инвазию достигали путем инкубации монослоев с бактериями в течение 3 часов. Монослои затем промывали DMEM и инкубировали с IM, содержащим 20 мг/мл лизостафина, для уничтожения внеклеточных бактерий. Использование лизостафина для уничтожения внеклеточных нормальных клеток и SCV S. aureus ранее было подтверждено в анализах клеточной инвазии (Moisan et al., 2006 и Tuchscherr et al, 2011). Обработку проводили в течение 30 минут для определения КОЕ через 3 часа после заражения или в течение дополнительных 12 или 24 часов. Затем, после интенсивной промывки фосфатно-солевым буфером Дульбекко (DPBS), монослои отделяли трипсинизацией и лизировали с помощью 0,05% Triton X-100 и добавляли PBS для получения конечной концентрации 1X. Лизат серийно разводили и высевали на TSA для определения КОЕ.
Жизнеспособность и анализ метаболической активности BMEC. Чтобы определить цитотоксическое повреждение, вызванное S. aureus ATCC 29213 (WT) и его изогенными мутантами на клетках MAC-T, проводили анализ метаболической активности клеток MTT, который измеряет восстановление 3-[4,5-диметилтиазол-2-ил] - 2,5-дифенилтетразолийбромид (МТТ) в нерастворимый продукт формазан в жизнеспособных клетках. Анализ проводился по методу Kubica et al. (Kubica et al., 2008) с некоторыми модификациями. Вкратце, заражение клеток S. aureus было достигнуто, как описано в анализе на устойчивость, но вместо лизиса через 12 или 24 часа клетки инкубировали с 100 мкл реагента МТТ (5 мг / мл) (Sigma) в DPBS в течение 2 часов при 37°С. После этого добавляли раствор кислого растворителя из 16% SDS и 40% PMF, pH 4,7, чтобы лизировать клетки и растворить кристаллы формазана в течение ночи. Образцы считывали с использованием устройства для считывания микропланшетов Epoch (Biotek Instruments Inc.) на длине волны 570 нм. Все анализы были выполнены в трех экземплярах, и контрольные лунки с неинфицированными клетками (контроль высокой жизнеспособности) или лизированными WT-инфицированными клетками (бактериальный фоновый контроль; обработанные 0,05% тритоном Х-100 в течение 10 минут перед добавлением МТТ) были включены в каждый планшет. Уровень метаболической активности рассчитывали с использованием следующей формулы: (оптическая плотность образца - фоновый контроль) / высокий контроль) ×100.
Вирулентность на модели мышиного мастита. Модель заражения мышиным маститом основана на ранее описанной (Brouillette, 2005; Brouillette, 2004). Все эксперименты, проведенные на мышах, были одобрены этическим комитетом по экспериментам на животных Факультета наук Университета Шербрука и проводились в соответствии с руководящими принципами Канадского совета по уходу за животными. Вкратце, спустя один час после удаления 12-14-дневного потомства, кормящих мышей CD-1 (Charles River Laboratories) анестезировали кетамином и ксилазином при концентрации 87 и 13 мг/кг массы тела соответственно, и молочные железы инокулировали под биноклем. Молочные протоки обнажали небольшим разрезом на ближних концах сосков и 100 мл бактериальной суспензии, содержащей 102 КОЕ в эндотоксин-свободном фомфатно-буферизированном солевом растворе (PBS, Sigma) вводили через канал соска с применением 32-миллилитровой тупой иглы. Для каждого животного были инокулированы две железы (четвертая справа [R4] и четвертая слева [L4] от головы до хвоста). Молочные железы асептически собирали в указанное время, взвешивали и визуально оценивали на предмет воспаления. Бактериальную нагрузку оценивали после механической гомогенизации ткани в PBS, серийных разведений и посева на агаре для определения КОЕ. Во втором эксперименте гомогенизированные железы были сохранены для экстракции белка для ферментативных анализов активности миелопероксидазы (MPO).
Экстракция белка молочной железы. Выделение общего белка из молочных желез проводили оптимизированным способом, описанным ранее (Pulli et al., 2013), с некоторыми модификациями. Ткани молочных желез гомогенизировали в буфере, содержащем конечную концентрацию фосфата калия 50 мМ, рН 6,0 и гексадецилтриметиламмония бромид (CTAB) 50 мМ (Sigma). Затем образцы обрабатывали ультразвуком, размораживали в жидком азоте и центрифугировали при 2000 g в течение 15 минут при 4°C. Наконец, жировой слой удаляли аспирацией, и супернатанты сохраняли для конечного центрифугирования в течение 15 минут при 15000g, чтобы удалить каждый клеточный дебрис. Супернатанты распределяли в аликвотах и хранили при -80°С до использования для ферментативных анализов или определения концентрации белка при измерении с помощью набора для анализа белка по методу бицинхониновой кислоты (BCA) (Thermo-Scientific).
Анализ активности MPO. Рекрутмент нейтрофилов в тканях молочной железы измеряли количественным определением активности фермента МРО по методу о-дианизидин- H2O2, модифицированному для микропланшетов (Bradley, RD. and Rothstein, GPPC., 1982). В 96-луночном микропланшете 10 мкл супернатантов экстракции ткани инкубировали с раствором гидрохлорида о-дианизидина (0,167 мг/мл) (Sigma) и 0,0005% H2O2 (Sigma) в 50 мМ фосфатном буфере CTAB 50 мМ, pH 6,0. Активность MPO измеряли кинетически с интервалами 15 с в течение 5 мин в устройстве для считывания микропланшетов Epoch при 460 нм. Единицей MPO считали количество фермента, которое разлагает 1 мкмоль H2O2/мин при 25°C, предполагая коэффициент поглощения 11,3 мМ−1 см−1 при 460 нм для о-дианизидина (Zhang et al., 2004). Результаты выражали в единицах МРО на г железы.
Иммунизации мышей живым аттенуированным мутантом Δ720ΔhemB. Иммуногенные свойства аттенуированного штамма Δ720ΔhemB, вводимого в виде живой вакцины, оценивали на мышах. В предварительных исследованиях мыши хорошо переносили внутримышечные и подкожные (SC) инъекции аттенуированного штамма. Дозы 106, 107 м 108 КОЕ и маршрут SC были выбраны для последующих экспериментов. Для приготовления бактериального инокулята колонии S. aureus Δ720ΔhemB, ранее выращенные на чашках BHIA, дважды промывали в ледяном PBS и суспендировали в PBS, содержащем 15% глицерина, затем аликвотировали и хранили при -80°C до последующего использования. Количество жизнеспособных бактерий в препарате инокулята было подтверждено серийным разведением с нанесением на BHIA. Мыши CD-1 были случайным образом разделены на 3 группы: группа 1 (n = 3) получала дозу of 106 КОЕ; группа 2 (n=3), 107 КОЕ, и группа 3 (n=3), 108 КОЕ. Мышей иммунизировали двумя подкожными инъекциями бактерий в PBS (100 мкл) в шею с интервалом в две недели. Образцы крови отбирали непосредственно перед первичной инъекцией (преиммунные сыворотки) и через десять дней после бустерной иммунизации (иммунные сыворотки). Аликвоты крови оставляли для свертывания при комнатной температуре в течение часа и затем центрифугировали при 10000g в течение 10 минут при 4°C. Сыворотки собирали и хранили при -20°С до последующего анализа.
Получение клеточных экстрактов S. aureus. Приготовление цельноклеточных экстрактов S. aureus было выполнено, как описано ранее с некоторыми модификациями (Asli et al., 2016). Вкратце, бактериальные культуры в течение ночи разводили 1/1000 в свежем бульоне BHI и затем инкубировали при 35°C (225 об/мин) до достижения A600 нм ~0,8. Бактериальные клетки центрифугировали, и пеллеты дважды промывали в ледяном PBS и суспендировали с добавлением 5 мл PBS на мл пеллеты. Бактериальные суспензии сначала обрабатывали лизостафином (Sigma) (100 мкг/мл пеллеты) в течение 1 часа при 37°C, а затем 3 мкг коктейля протеазных ингибиторов (Sigma), 8 мкг РНКазы A (Sigma) и 8 мкг ДНКазы (Qiagen) на мл пеллеты добавляли к суспензии. Через 30 минут при комнатной температуре клетки были механически разрушены за 3-4 прохода в разрушителе SLM Aminco ™ French, а затем центрифугированы при 12000×g и 4°C в течение 10 минут для удаления неразбитых клеток. Супернатант собирали, и общую концентрацию белка определяли, как описано ранее, с помощью набора для анализа белка BCA.
Обнаружение мышиного общего IgG посредством ELISA. Обнаружение общего сывороточного IgG против штамма вакцинации Δ720ΔhemB и каждого из изолятов IMI крупного рогатого скота проводили для демонстрации и измерения системного гуморального ответа, генерируемого иммунизацией мышей. Для целевых антигенов Nunc MaxiSorp ™ 96-луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрывали 100 мкл каждого из цельноклеточных экстрактов S. aureus (10 мкг/мл, разбавленных в карбонатном/бикарбонатном буфере, Sigma), и инкубировали всю ночь при комнатной температуре. Затем планшеты насыщали PBS, содержащим 5% сухого обезжиренного молока, в течение 1 часа при 37°C, после чего выполняли вторую стадию блокирования с добавлением 5% сыворотки свиньи, чтобы предотвратить неспецифические взаимодействия белка A aureus S. Сто микролитров двухкратных серийных разбавлений сыворотки в буфере разбавления (PBS с 2% молока, 5% сыворотка свиньи и 0.025% Tween™ 20) загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты затем промыли три раза с помощью PBS, содержащего 0.05% Tween™ 20, и загрузили 100 мкл козлиного анти-мышиного Ig, конъюгированного с пероксидазой хрена (HRP) (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), разбавленного 1/5000 в буфере разбавления. Через 1 час инкубации при 37°C с последующими тремя промывками, пероксидазную активность обнаружили с помощью 3,3′,5,5′-тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD) согласно рекомендациям производителя.
Статистический анализ. Статистический анализ проводили с помощью программного обеспечения GraphPad Prism ™ (v.6.02). Внутриклеточные бактериальные КОЕ и бактериальные КОЕ/г железы (IMI у мышей) были преобразованы в значения логарифма с основанием 10 перед использованием для статистического анализа. Статистические тесты, используемые для анализа каждого эксперимента и значимости, указаны в подписях к Фигурам.
Пример 16: Конструкт штамма S. aureus ATCC 29213 Δ720, ΔhemB и Δ720ΔhemB
Живые аттенуированные организмы, которые имитируют естественную инфекцию, мощно стимулируют иммунную систему, вызывая широкие и устойчивые иммунные ответы, которые продуцируют как сывороточные, так и слизистые антитела, а также эффекторные Т-клетки и Т-клетки памяти, которые действуют синергетически для защиты от болезней (Detmer, 2006; Kollaritsch, 2000; Pasetti, 2011).
Мутация в гене SACOL0720, как показано, изменяет вирулентность S. aureus в экспериментальных IMI инфекциях у коровы (Allard et al., 2013).
Другие живые аттенуированные штаммы были приготовлены для целей вакцинации на основе фенотипических аспектов SCV S. aureus. SCV обычно не вызывают инвазивных инфекций (то есть дополнительного ослабления) и могут быть интернализованы в клетках-хозяевах и, следовательно, будут стимулировать клеточный иммунный ответ в дополнение к гуморальному иммунному ответу.
Стабильный SCV S. aureus был впервые создан путем делеции гена hemB (ΔhemB) (смотрите пример 15, Получение живых аттенуированных штаммов S. aureus Δ720 и ΔhemB). Дальнейшее ослабление этого SCV было затем достигнуто путем инактивации гена SACOL0720 (Δ720) (смотрите пример 15, образование ΔhemBΔ720).
После заражения MAC-T эпителиальными клетками молочной железы крупного рогатого скота двойной мутант (Δ720ΔhemB) демонстрировал значительно более низкую интернализацию и разрушение клеток по сравнению с тем, что наблюдали с ΔhemB и Δ720 соответственно.
Пример 17: Штамм S. aureus ΔhemBΔ720 аттенуирован в MAC-T клетках
Инфекционность штаммов АТСС 29213 (WT), Δ720, ΔhemB и ΔhemBΔ720 затем сравнивали в анализ внутриклеточной персистенции с использованием клеток MAC-T. Сравнивая три мутантных штамма с их изогенным родителем WT, наблюдали различные эффекты мутаций в генах hemB и SACOL0720. Короткая 3-часовая инкубация бактерий с клеточными монослоями с последующим добавлением лизостафина для устранения внеклеточных бактерий продемонстрировала высокий уровень интернализации в клетки MAC-T для штаммов WT и ΔhemB, на основе восстановления жизнеспособных внутриклеточных бактерий (КОЕ) (ФИГ. 9А и В). Однако один мутант Δ720 показал значительно меньшую (P ≤0,01) интернализацию по сравнению с его родительским штаммом WT (фиг. 9A). Снижение интернализации, наблюдаемое у Δ720, было еще более выраженным при сравнении двойного мутанта ΔhemBΔ720 с ΔhemB, с 10-кратным уменьшением восстановления инокулята в этом 3-часовом анализе интернализации (P ≤ 0,001, Фиг. 9B). Это первоначальное восстановление интернализованной бактериальной загрузки было все еще очевидным через 12 и 24 ч после инвазии (PI) для штамма с двойным мутантом ΔhemBΔ720 (фиг. 9C), что иллюстрируется снижением КОЕ/мл на 1-log10 в обеих временных точках по сравнению с наблюдаемым для ΔhemB (P ≤ 0,001). Разница в исходных внутриклеточных бактериальных нагрузках между штаммом с одиночным мутантном Δ720 и штаммами WT (Фиг. 9А) постепенно исчезала при увеличении времени инкубации (Фиг. 9C), так как оба штамма плохо сохранялись в клетках MAC-T (Фиг. 10). Напротив, внутриклеточные КОЕ, выделенные для одного штамма ΔhemB, были значительно выше по сравнению с таковыми для трех других штаммов через 24 ч PI (Фиг. 9C, P ≤ 0.001 против всех). В целом и как и ожидалось для фенотипа SCV, штамм ΔhemB показал более высокую внутриклеточную персистенцию, чем любой другой штамм со временем (Фиг. 10). Эти результаты предполагают, что мутация Δ720 в основном уменьшает процесс интернализации в клетки MAC-T. Результаты также показывают, что мутант ΔhemBΔ720 по-прежнему способен к интернализации и персистенции в BMEC, но в гораздо меньшей степени, чем та, которая наблюдается с одним мутантом ΔhemB.
SCV ΔhemBΔ720 и ΔhemB вызывают низкое разрушение BMEC. Как сообщалось выше, штаммы ΔhemB и ΔhemBΔ720 SCV демонстрировали большую персистенцию во времени в клетках MAC-T, о чем свидетельствует их устойчивая жизнеспособность при 12 и 24 ч PI по сравнению со штаммами WT и Δ720 (ФИГ. 9C и 10). Процент инокулята, извлеченного из клеток, оставался практически одинаковым через 0-24 часа после добавления лизостафина, как для двойных, так и для одиночных мутантов hemB, с небольшим увеличением через 12 часов, что указывает на внутриклеточный рост (Фиг. 10). Оба штамма начали медленно снижаться после этого времени заражения. Однако очевидное снижение внутриклеточных КОЕ для штаммов WT и Δ720 сопровождалось визуальным наблюдением увеличения повреждения клеточных монослоев с течением времени по сравнению с тем, которое наблюдалось у штаммов фенотипа SCV.
Жизнеспособность клеток MAC-T также оценивали после заражения каждым из четырех исследованных штаммов. Жизнеспособность клеток MAC-T оценивали по методу МТТ (Kubica et al., 2008). Результаты показывают, что оба штамма SCV (ΔhemB и ΔhemBΔ720) вызывали значительно меньшее уничтожение MAC-T в этом анализе в отличие от штаммов WT и Δ720. По сравнению с ΔhemB штамм WT уменьшил почти вдвое жизнеспособность клеток через 12 ч (фиг. 11A: WT: 25,4%; ΔhemB: 48,4%). Эта разница все еще была очевидна через 24 часа (фиг. 11B: 16,3% против 34,5% соответственно), даже если бактериальная нагрузка была в 10 раз выше для мутанта ΔhemB (фиг. 9C). Клетки MAC-T были более повреждены ΔhemB, чем двойным мутантом Δ720ΔhemB, но разница была значимой только через 24 часа (P ≤0.01). При прямом сравнении со штаммом WT двойной мутант Δ720ΔhemB поддерживал жизнеспособность эпителиальных клеток в 2,3 раза больше за 12 ч (фиг. 11A) и в 2,7 раза больше за 24 ч (фиг. 11B) (12 и 24 ч: P ≤ 0,0001). Следовательно, более высокая внутриклеточная персистенция обоих штаммов SCV по сравнению со штаммами WT и Δ720 во времени (Фиг. 10), вероятно, объясняется более низкой токсичностью SCV для клеток MAC-T (Фиг. 11). Взятые вместе, результаты анализов инфекции BMEC предоставили доказательства аддитивного эффекта обеих мутаций ΔhemB и Δ720 для ослабления штамма WT.
Пример 18: Штамм S. aureus ΔhemBΔ720 аттенуирован в мышиной модели IMI
Чтобы засвидетельствовать аттенуирование ΔhemBΔ720 в модели инфекции in vivo, вирулентность двойного мутанта оценивали и сравнивали со штаммом WT в мышиной модели IMI (Brouillette and Malouin, 2005). Для обоих штаммов экспоненциальная фаза инфекции имела место в основном в течение первых 12 ч после заражения, тогда как максимальная бактериальная нагрузка была достигнута через 24 ч для двойного мутанта и 48 ч (день 2 [D2]) для штамма WT (Фиг. 12). Через 24 часа у двойного мутанта наблюдалось снижение среднего 1,9 log10 КОЕ/г железы по сравнению с WT (P ≤0,05). Кроме того, через 24 часа бактериальная нагрузка мутанта показала постоянное снижение до достижения полного бактериального клиренса на 12-й день (отмечена звездочкой на Фиг. 12). Напротив, родительский штамм WT вызывал тяжелые инвазивные инфекции по сравнению с мутантом, убивая 3 из 9 оставшихся мышей на 2-й день и 2 из 3-х мышей на 7-й день (Фиг. 12; стрелки), прежде чем железы могли быть собраны для этих групп. Мыши, выжившие после инфекции WT, сохраняли высокое количество жизнеспособных микроорганизмов (9 log10 КОЕ/г железы) на 7-й день, около 5 log10 разницы в бактериальной нагрузке по сравнению с двойным мутантом. Эти результаты ясно демонстрируют заметно сниженную способность штамма ΔhemBΔ720 размножаться и выживать в молочной железе. Следовательно, двойной мутант ΔhemBΔ720 сильно ослабляется в модели мышиной интрамаммарной инфекции (IMI) и эффективно очищается из молочных желез.
Аттенуированный штамм ΔhemBΔ720 представляется идеальным для целей вакцинации и для внутриклеточной доставки антигенов. Действительно, низкая и временная интернализация ΔhemBΔ720 должна помочь стимуляции клеточного иммунитета, компонента иммунного ответа, который важен для защиты от S. aureus (Fowler and Proctor, 2014).
Пример 19: Воспалительная реакция на Δ720ΔhemB и WT штаммы после IMI
Для контроля воспалительного ответа (иммунного ответа) мышей на инфекции, вызванные WT и мутантными штаммами, нейтрофильную инфильтрацию в железах оценивали по ферментативной активности MPO общих белковых экстрактов гомогенатов желез. Активность МРО в биологических образцах ранее была тесно связана с абсолютным числом нейтрофилов (Xia, 1997) и, следовательно, является адекватным маркером. В течение первых часов после заражения рекрутирование нейтрофилов проводилось по аналогичным профилям для желез, пораженных двойным мутантом и WT (Фиг. 13), с экспоненциальной интенсификацией очевидной инфильтрации нейтрофилов от 12 ч до 24 ч после заражения, совпадающей с ростом бактерий, хотя и с некоторой задержкой. Ранее было показано, что абсолютные количества полиморфноядерных клеток в связи с бактериальной нагрузкой в молочных железах не всегда достигают пика в одно и то же время (Brouillette, 2005). В 6, 12 и 24 ч между железами, инфицированными мутантными штаммами и штаммами WT, значительных различий в активности MPO не наблюдалось (Фиг. 13). Эта эквивалентность в видимой инфильтрации нейтрофилов, однако, не коррелирует с визуальным наблюдением воспаления через 24 часа, после чего инфекция WT вызывает обширное покраснение инфицированных желез по сравнению с двойным мутантом (фотографии на фиг. 14). Напротив, инфицированные мутантом железы не были визуально изменены на макроскопическом уровне по сравнению с контролями PBS. Несоответствие между визуальной оценкой результатов воспаления и проникновения нейтрофилов может быть связано с различиями в бактериальных нагрузках (ФИГ. 9A-C) и цитотоксической активностью штамма WT (Фиг. 11) и может быть связано с высокоинвазивной способностью и способность к распространению штамма через экспрессию токсинов и ферментов. Следовательно, эти результаты указывают на то, что рекрутирование нейтрофилов в железах, инфицированных мутантным штаммом, было эквивалентно тому, что наблюдалось у штамма WT, и что этого было достаточно, чтобы позволить последующее снижение и очистку мутантных бактериальных нагрузок.
Наконец, для подтверждения безопасности штамма и оценки того, что этот воспалительный ответ не является следствием недопустимого реактогенного штамма, активность MPO контролировали в инфицированных Δ720ΔhemB железах через 4 и 12 дней после заражения. Затем уровень активности сравнивали с уровнями, полученными у мышей, которым инъецировали PBS. Как показано на Фиг. 15, явное присутствие нейтрофилов в железах, инфицированных мутантами, все еще оставалось высоким через 4 дня после заражения, при этом активность MPO составляла от 8 до 21 Ед./г железы. Кроме того, инволюция железы, процесс, с помощью которого лактирующая железа возвращается в морфологически близкое к предбеременному состоянию, обычно связана с нейтрофильным рекрутингом, который позволяет фагоцитоз апоптотических клеток во время ремоделирования ткани (Stein, 2007). Через несколько дней после заражения в этой модели мышиные железы уже находятся в нормальном состоянии модификации, о чем свидетельствует их быстрое сокращение. Тем не менее, уровни MPO в инфицированных мутантом железах значительно снизились в период с 4 по 12 день (P ≤0,01). Уровни MPO затем рассматривали, как возвращающиеся к нормальному уровню на 12-й день, не показывая значительных отличий от уровня, достигнутого у мышей, которым инъецировали PBS. Воспалительный ответ желез, инфицированных Δ720ΔhemB, возвращается к нормальному уровню с бактериальным клиренсом (Фиг. 15).
Пример 20: Иммунизация Δ720GΔhemB создает сильный гуморальный ответ на несколько изолятов S. aureus интрамаммарной инфекции крупного рогатого скота.
Чтобы подтвердить, что иммунизация живым Δ720ΔhemB действительно может генерировать сильный иммунный ответ, подходящий для его использования в качестве предполагаемой живой вакцины против интрамаммарных инфекций S. aureus, мышей иммунизировали различными дозами живой вакцины и анализировали общие IgG сыворотки на связывание с цельноклеточными экстрактами различных изолятов S. aureus крупного рогатого скота. Во-первых, дозы 106, 107 и 108 КОЕ, вводимые подкожно в шею, не вызывали побочных эффектов, таких как изменение поведения мышей или признаки воспаления или некроза в месте иммунизации в течение всего периода иммунизации. Кроме того, иммунизация с использованием возрастающих количеств живого двойного мутанта ATCC 29213 Δ720ΔhemB дала увеличение титров системных антител IgG против цельноклеточного экстракта его собственных антигенов (фиг. 15B). Титры иммунных сывороток были значительно выше, чем титры преиммунных сывороток, что свидетельствует о специфичности продукции антител против антигенов S. aureus, присутствующих в живой вакцине. Что наиболее важно, иммунизация с использованием увеличивающихся количеств Δ720ΔhemB также привела к последовательному повышению титров антител против различных штаммов S. aureus, выделенных из бычьего мастита, включая штаммы из основных spa типов, найденных в Канаде и других странах мира (Фиг. 15C). Эти результаты ясно показывают, что (i) иммунизация двойным мутантом может вызывать иммунный ответ, и что (ii) фоновый штамм (ATCC 29213) имеет достаточно общих признаков со штаммами бычьего мастита, так что ответ антител также сильно распознает штаммы основных spa типов.
Иммунизация мышей с использованием подкожных инъекций живого Δ720ΔhemB вызвала сильный гуморальный ответ, судя по высоким титрам общего IgG, измеренным в отношении цельноклеточного бактериального экстракта. Кроме того, вакцинный штамм Δ720ΔhemB имел достаточно общих признаков со штаммами бычьего мастита, так что ответ антител также четко распознавал штаммы из множества распространенных spa типов, связанных с маститом.
Хотя это продемонстрировало, что фоновый двойной мутант (ATCC 29213) имеет много общих признаков со штаммами бычьего мастита, такой двойной мутант может быть создан на любом желаемом генетическом фоне, если это желательно, особенно в любом штамме, который был выделен из бычьего мастита, таком как, но не ограничиваясь этим, штамм S. aureus RF122.
Эти результаты показывают, что штамм SCV, имеющий некоторые остаточные внутриклеточные способности, может позволить рекруитинг иммунных клеток без установления тяжелой инфекции. Такой штамм SCV может действовать как живая аттенуированная вакцина, которая адекватно стимулирует иммунный ответ на борьбу с патогенами с внутриклеточными способностями.
Пример 21: Материалы и способы - SACOL0442, SACOL0720, SACOL0029 и слияние между SACOL1867 и SACOL0029 + аттенуированная живая вакцина (вакцина №7)
Получение антигенов. Получение антигенов проводили, как описано в Примере 6, в части получение антигенов, за исключением дополнительного присутствия антигена SACOL0029. His-нацеленные рекомбинантные белки SACOL0029 были разработаны и произведены компанией GenScript, Inc. (Piscataway, NJ) (см. ФИГ. 21A, пункт I, his-нацеленная последовательность).
Образование живого атеннуированного S. aureus штамма Δ720ΔhemB. Живого аттенуированного штамма S. aureus проводили, как описано в Примере 15 (Образование ΔhemBΔ720).
Иммунизация мышей. Иммунногенные свойства рекмбинантных белков S. aureus, кодируемых SACOL0442, SACOL0720, SACOL0029 генами и слиянием SACOL0029 и SACOL1867 генов, в комбинации, или нет, с живым аттенуированным бактериальным штамом S. aureus Δ720ΔhemB оценивали на мышах. Мыши хорошо переносили дозы 103, 105, 106, 107 и 108 КОЕ путем подкожных инъекций в шею и внутримышечных инъекций в бедро. Доза 105 и подкожный путь были выбраны для следующих экспериментов.
Для приготовления бактериального инокулята колонии S. aureus Δ720ΔhemB, ранее выращенные на чашках BHIA, дважды промывали в ледяном PBS и ресуспендировали в PBS, содержащем 15% глицерина, затем аликвотировали и хранили при -80°C до последующего использования. Чтобы получить конечную дозу иммунизации мышей, соответствующую 105 КОЕ аттенуированных бактерий, концентрацию замороженного инокулята в бактериях оценивали путем серийного разведения на BHIA и затем разбавляли до конечной концентрации 105 КОЕ/мл в PBS в день иммунизации.
Для приготовления доз белка SACOL0029, SACOL0442, SACOL0720 и слитый полипептид SACOL0029-1867 смешивали и суспендировали в PBS для получения конечной дозы 5 мкг каждая. Самок мышей CD-1 случайным образом разделили на 3 группы: группа 1 (5 мышей) получала смешанную дозу белка (белковая смесь); группа 2 (5 мышей) получала дозу аттенуированных бактерий (Δ720ΔhemB); группа 3 (6 мышей) получала комбинацию белковой смеси и аттенуированных бактерий (комбинация). Самок мышей CD-1 иммунизировали двумя подкожными инъекциями в шею с интервалом в две недели. Дозы белков и бактериальных штаммов разводили в PBS как указано ранее и вводили в конечном объеме 100 мкл для каждой группы мышей. Никаких побочных эффектов не наблюдалось в течение всего периода экспериментальной иммунизации. Пробы крови брали непосредственно перед первой первичной инъекцией (преиммунные сыворотки) и через десять дней после бустерной иммунизации (иммунные сыворотки). Аликвоты крови оставляли для свертывания при комнатной температуре в течение часа, центрифугировали при 10000 g в течение 10 минут при 4°С. Сыворотки собирали и хранили при -20°С до последующего анализа.
Обнаружение общего IgG, IgG1 и IgG2 посредством ELISA. обнаружение общего сывороточного IgG, IgG1 и IgG2 против каждого из антигенов, ранее применяемых для иммунизации, проводили, как описано ранее с некоторыми модификациями (Ster et al., Vet. Immunol. Immunopathol. (2010), 136: 311-318). Кроме того, обнаружение IgG против стафилококкового поверхностного белка ClfA проводили, чтобы продемонстрировать дополнительные преимущества применения живого штамма для усиленного и сбалансированного иммунного ответа против S. aureus. Nunc MaxiSorp™ 96-ти луночный планшет (Thermo Fisher Scientific Inc., Rochester, NY) покрыли 75 мкл каждого тестируемого антигена (6.67 мкг/мл разбавленного в карбонатном/бикарбонатном буфере, Sigma Aldrich, Oakville, ON) и инкубировали всю ночь при комнатной температуре. Планшеты затем насытили PBS, содержащим 5% сухого обезжиренного молока в течение 1 часа при 37°C. Что микролитров четырехкратных серийных разбавлений сыворотки в PBS, содержащем 3% молока, и 0.025% Tween™ 20 загрузили на планшеты и инкубировали в течение 1 часа при 37°C. Планшеты инкубировали три раза PBS, содержащим 0.05% Tween™ 20. Сто микролитров вторичных антител, конъюгированных с пероксидазой хрена (HRP) затем добавили на планшет. Применяемыми вторичными антителами были козлиные анти-мышиные IgG, IgG2a и IgG1 (Jackson ImmunoResearch Laboratories Inc., West Grove, PA), разбавленные 1/5000 соответственно в PBS, содержащем 3% молока и 0.025% Tween™ 20. Через 1 час инкубации при 37°C с последующими тремя промывками, пероксидазную активность обнаружили с помощью 3,3′,5,5′-тетраметидбензидина (TMB) реагента (KPL Inc., Gaithersburg, MD) согласно рекомендациям производителя.
Статистический анализ. Статистический анализ титров антител и корреляции проводили с использованием GraphPad Prism™ v6.05.
Пример 22: Слияние антигенов и комбинация с живым аттенуированным штаммом S. aureus вызывает высокие титры антител у мышей - вакцина №7
Антигены и живой аттенуированный штамм S. aureus Δ720ΔhemB получают, как описано в примере 21. Мышей иммунизируют, и IgG обнаруживают, как описано в примере 21.
Результаты на Фиг. 16 показывают, что иммунизация слиянием SACOL0029-1867 (при совместном введении либо с другими антигенами, либо с живым аттенуированным штаммом) вызывает высокий и специфический к антителам ответ у мышей.
Пример 23: Живой аттенуированный штамм S. aureus неожиданно улучшает иммунный ответ антител против некоторых специфических антигенов - вакцина №7
Антигены и живой аттенуированный штамм S. aureus Δ720ΔhemB получают, как описано в примере 21. Мышей иммунизируют, и IgG обнаруживают, как описано в примере 21.
Результаты на Фиг. 17 показывают, что иммунизация аттенуированным живым штаммом Δ720ΔhemB значительно увеличивает выработку специфических антител IgG против антигена SACOL0029 по сравнению с иммунизацией антителами IgG мышей, иммунизированных только белковой смесью.
Пример 24: Живой аттенуированный штамм S. aureus индуцировал значительные титры антител против дополнительных поверхностных белков S. aureus - вакцина №7
Антигены и живой аттенуированный штамм S. aureus Δ720ΔhemB получают, как описано в примере 21. Мышей иммунизируют, и IgG обнаруживают, как описано в примере 21.
Результаты на Фиг. 18 показывают, что иммунизация аттенуированным живым штаммом Δ720ΔhemB (отдельно или при совместном введении с полипептидными антигенами) значительно увеличивает продукцию специфических антител против стафилококкового поверхностного белка ClfA по сравнению с той, которая достигается с помощью только белковой смеси, состоящей из SACOL0029, SACOL0442, SACOL0720 и SACOL0029-1867.
Пример 25: Живой аттенуированный штамм S. aureus значительно сбалансировал Th1/Th2 иммунный ответ - вакцина №7
Антигены и живой аттенуированный штамм S. aureus Δ720ΔhemB получают, как описано в Примере 21.
Изотипы сывороточных IgG2a и IgG1 против слитого белка SACOL0029-1867 были обнаружены в сыворотках вакцинированных мышей, как было указано ранее, и было определено соотношение титров IgG2a и IgG1 каждой мыши. Изотип IgG2a связан с иммунным ответом Th1 у мышей, тогда как IgG1 является маркером ответа Th2. Как описано в Примере 5, индукция продукции IgG2 у коров и степень титров IgG2 в молоке значительно коррелируют с защитой коров от заражения S. aureus, судя по уровням соответствующих соматических клеток (SCC) или количеству жизнеспособных бактерий (КОЕ) в молоке коров (Фиг. 4C).
Результаты, показанные в ФИГ. 19 и 20, показывают, что аттенуированный живой штамм Δ720ΔhemB, включенный в комбинированную иммунизационную вакцину (Δ720ΔhemB S. aureus, вводимую с SACOL0029, SACOL0442, SACOL0720 и SACOL0029-1867), индуцирует значительно более высокое соотношение антител IgG2a/IgG1 против слияния SACOL0029-1867 и белков SACOL0029, чем то, которое наблюдается при иммунизации белковой смесью (SACOL0029, SACOL0442, SACOL0720 и SACOL0029-1867), что приводит к значительно более сбалансированному ответу Th1/Th2.
Приведенные выше примеры 21-25 показывают, что даже если сильный иммунный ответ был получен путем иммунизации различными антигенами (включая, например, слияние SACOL0029-1867) в композиции белковой смеси, иммунизация мышей комбинацией этих антигенов с живым аттенуированным штамм значительно улучшала иммунные ответы против S. aureus, индуцируя более высокие титры антител против некоторых специфических антигенов (например, SACOL0029), продуцируя антитела против других стафилококковых белков (например, ClfA) и достигая более сбалансированного соотношения IgG2a/IgG1, хорошего маркера более сильного ответа типа Th1, против антигенов, вводимых совместно с живым штаммом.
Пример 26: Экспрессия рекомбинантных белков в штамме S. aureus ΔhemBΔ720 - вакцины №8, 9, 10 и т.д.
Гены SACOL0442, SACOL0720, SACOL0029, и/или SACOL1867, а также слияние (например, размер 50 AA или более) генов (или их фрагментов) SACOL0029 и SACOL1867 (SACOL0029-SACOL1867), слияния фрагментов (например, эпитов) SACOL720 и/или SACOL0442 (слияние 720-720) (слияние 442-720) или любое другое слияние генов или их фрагментов, например, SACOL0029-SACOL0442, SACOL0029-SACOL0720, SACOL0029-SACOL0720-SACOL0442, SACOL0029-SACOL0720-SACOL1867, SACOL0029-SACOL1867-SACOL0442 SACOL0442-SACOL0029-SACOL0720, SACOL0442-SACOL0029-SACOL0720, SACOL0442-SACOL1867-SACOL0720, SACOL0720-SACOL0442-SACOL1867, SACOL04029-SACOL1867-SACOL0720-SACOL0442, клонировали в плазмиду pCN36 (Charpentier et al., 2004) при конститутивном промоторе (PblaZ из плазмиды pCN40) (Charpentier et al., 2004) и экспрессировали в штамме S. aureus ΔhemBΔ720. Предполагается, что определенные белковые антигены, обеспечиваемые в настоящей заявке, являются экзотоксином, энтеротоксином или суперантигеном (например, SACOL0442) или белками, полезными для защиты от иммунной защиты хозяина (например, SACOL0720), и могут потенциально влиять на иммунную систему млекопитающих и выработку антител, и/или показывать некоторую токсичность у хозяина. Хотя такое вмешательство не наблюдалось в отношении вакцинной композиции и составов, описанных в настоящей заявке, может быть полезно модифицировать белок или полипептид, экспрессируемый в штамме S. aureus ΔhemBΔ720, чтобы клонированные гены не дополняли его вирулентность. Для этой цели можно использовать методы молекулярной биологии для удаления или мутации предполагаемых областей, участвующих в такой белковой активности, без потери иммуногенности (Chang et al., 2008). Именно такой подход заявители использовали для приготовления антигенов вакцинных композиций согласно настоящему изобретению.
Экспрессия отдельных рекомбинантных белковых продуктов каждым из штаммов S. aureus ΔhemBΔ720, несущих один из сконструированных экспрессионных векторов, подтверждена с помощью анализов LC-MS/MS. Вкратце, штаммы, выращенные в BHI с 15 мкг/мл тетрациклина до средней логарифмической фазы, были центрифугированы, и пеллеты были инактивированы этанолом. Образцы хранили при -20°С до процедур лизиса клеток и расщепления трипсином. Образцы инкубировали с лизостафином и трипсином при 37°С с последующим разрушением клеток путем механической гомогенизации с использованием стеклянных шариков и гранулометра. Затем лизаты центрифугировали в течение 25 минут при 13 000 об. в минуту при 4°С, чтобы удалить клеточный дебрис, перед тем как перейти к расщеплению белка трипсином, восстановление и алкилирование проводили стандартными процедурами перед инъекцией образца для обнаружения белка с использованием MRM метода LC-MS/MS.
Альтернативно, экспрессия рекомбинантного белка также подтверждается вестерн-блоттингом бактериальных лизатов.
Пример 27: Иммунизация мышей аттенуированными штаммами, экспрессирующими антигены - вакцины №8 и т.д.
Самок мышей CD-1 вакцинируют двумя подкожными инъекциями с интервалом в две недели. Каждый из штаммов S. aureus ΔhemBΔ720, несущий или не нет один из сконструированных векторов экспрессии, разбавляют физиологическим раствором и вводят в конечном объеме 100 мкл на дозу. Группа 1 получает только двойной мутантный штамм; группа 2 получает штамм с двойным мутантом, экспрессирующий слияние SACOL0029-1867; группа 3 получает двойной мутантный штамм, экспрессирующий слияние SACOL0029-0442; группа 4 получает двойной мутантный штамм, экспрессирующий слияние SACOL0029-0720; группа 5 получает смесь двойных мутантных штаммов, один из которых экспрессирует слитый SACOL0029-1867, а другой экспрессирует слитый SACOL0029-0442; группа 6 получает смесь двойных мутантных штаммов, один из которых экспрессирует слитый SACOL0029-1867, а другой экспрессирует слитый SACOL0029-0720; и группа 7 получает смесь двойных мутантных штаммов. Образцы крови берут непосредственно перед первой инъекцией и через двенадцать дней после второй инъекции. Образцам дают сгуститься при комнатной температуре в течение часа, затем центрифугируют при 2000 g в течение 10 минут при 4°С. Супернатанты (сыворотки) собирают и хранят при -20°С до последующего анализа. Мышей умерщвляют на 27 день, и кровь собирают путем пункции сердца. Иммунные сыворотки извлекаются, делятся на аликвоты и хранятся как для преиммунных сывороток.
Иммунный ответ на вакцинацию оценивают с помощью иммуноферментного анализа (ELISA) на наличие поликлональных антител IgG в сыворотке, направленных на целые клетки S. aureus (штамм Вуда) или специфические рекомбинантные белки. Анти-мышиный IgG-HRP (HRP: пероксидаза хрена) используют в качестве вторичного антитела для определения колориметрической продукции 3,3',5,5'-тетраметидбензидина (TMB), окисления субстрата под действием пероксидазы с использованием спектрофотометра.
Ссылочные источники
Allard M, Ster C., Jacob CL, Scholl D, Diarra MS, Lacasse P, and Malouin F, 2013. The expression of a putative exotoxin and an ABC transporter during bovine intramammary infection contributes to the virulence of Staphylococcus aureus. Vet. Microbiol. 162:761-70.
Allard, M., C. Ster, L. St-James, P. Lacasse, M.S. Diarra, C. L. Jacob, and F. Malouin. 2008. Transcriptional Analysis of In Vivo-Expressed Genes in Staphylococcus aureus During Bovine Mastitis. American Society for Microbiology General Meeting. Boston, USA. June 1-5, 2008 (Poster)
Allard, M., H. Moisan, E. Brouillette, A.L. Gervais, M. Jacques, P. Lacasse, M.S. Diarra, and F. Malouin. 2006. Transcriptional modulation of some Staphylococcus aureus iron-regulated genes during growth in vitro and in a tissue cage model in vivo. Microbes Infect. 7:1679-1690.
Asli A, Brouillette E, Krause KM, Nichols WW, Malouin F. Distinctive binding of avibactam to penicillin-binding proteins of gram-negative and gram-positive bacteria. Antimicrob Agents Chemother. 2016;60: 752-756.
Atalla, H., C. Gyles, and B. Mallard. 2010. Persistence of a Staphylococcus aureus small colony variants (S. aureus SCV) within bovine mammary epithelial cells. Vet. Microbiol. Elsevier B.V. 143:319-28.
Atalla, H., C. Gyles, and B. Mallard. 2011. Staphylococcus aureus small colony variants (SCVs) and their role in disease. Anim. Health Res. Rev. 12:33-45.
Atalla, H., C. Gyles, B. Wilkie, K. Leslie, and B. Mallard. 2009. Somatic cell scores and clinical signs following experimental intramammary infection of dairy cows with a Staphylococcus aureus small colony variant (S. aureus SCV) in comparison to other bovine strains. Vet. Microbiol. 137:326-34.
Atalla, H., C. Gyles, C.L. Jacob, H. Moisan, F. Malouin, and B. Mallard. 2008. Characterization of a Staphylococcus aureus small colony variant (SCV) associated with persistent bovine mastitis. Foodborne Pathog 5:785-799.
Barbagelata, M. S., L. Alvarez, M. Gordiola, L. Tuchscherr, C. von Eiff, K. Becker, D. Sordelli, and F. Buzzola. 2011. Auxotrophic mutant of Staphylococcus aureus interferes with nasal colonization by the wild type. Microbes Infect. 13:1081-90.
Barkema, H.W., Y.H. Schukken, and R.N. Zadoks. 2006. Invited Review: The role of cow, pathogen, and treatment regimen in the therapeutic success of bovine Staphylococcus aureus mastitis. J Dairy Sci. 89:1877-1895.
Barrio, M. B., P. Rainard, F.B.Gilbert, B. Poutrel. 2003. Assessment of the opsonic activity of purified bovine sIgA following intramammary immunization of cows with Staphylococcus aureus. J. Dairy Sci. 86 :2884-2894.
Bharathan, M., and I. K. Mullarky. 2011. Targeting mucosal immunity in the battle to develop a mastitis vaccine. J. Mammary Gland Biol. Neoplasia 16:409-19.
Bowdish, D.M.E., Davidson, D.J., Scott, M.G. and Hancock, R.E.W., 2005. Immunomodulatory Activities of Small Host Defense Peptides. Antimicrobial Agents and Chemotherapy, 49(5): 1727-1732.
Bradley RD Rothstein, G, P. P. C. 1982. Cellular and extracellular myeloperoxidase in pyogenic inflammation. Blood 60:618-622.
Bradley, A. 2002. Bovine mastitis: an evolving disease. Vet J. 164:116-128.
Brouillette, E., A. Martinez, B. J. Boyll, N. E. Allen, and F. Malouin. 2004. Persistence of a Staphylococcus aureus small-colony variant under antibiotic pressure in vivo. FEMS Immunol. Med. Microbiol. 41:35-41.
Brouillette, E., and F. Malouin. 2005. The pathogenesis and control of Staphylococcus aureus-induced mastitis: study models in the mouse. Microbes Infect. 7:560-8.
Brouillette, E., G. Grondin, B. G. Talbot, and F. Malouin. 2005. Inflammatory cell infiltration as an indicator of Staphylococcus aureus infection and therapeutic efficacy in experimental mouse mastitis. Vet. Immunol. Immunopathol. 104:163-9.
Brouillette, E., M. Hyodo, Y. Hayakawa, D. K. Karaolis, and F. Malouin. 2005. 3',5'-cyclic diguanylic acid reduces the virulence of biofilm-forming Staphylococcus aureus strains in a mouse model of mastitis infection. Antimicrob. Agents Chemother. 49:3109-3113.
Brückner, R. 1997. Gene replacement in Staphylococcus carnosus and Staphylococcus xylosus. FEMS Microbiol. Lett. 151:1-8.
Burlak, C., C.H. Hammer, M.A. Robinson, A.R. Whitney, M.J. McGavin, B.N. Kreiswirth, and F.R. Deleo. 2007. Global analysis of community-associated methicillin-resistant Staphylococcus aureus exoproteins reveals molecules produced in vitro and during infection. Cell Microbiol. 9:1172-1190.
Buzzola, F. R., L. P. Alvarez, L. P. N. Tuchscherr, M. S. Barbagelata, S. M. Lattar, L. Calvinho, and D. O. Sordelli. 2007. Differential abilities of capsulated and noncapsulated Staphylococcus aureus isolates from diverse agr groups to invade mammary epithelial cells. Infect. Immun. 75:886-91.
Buzzola, F. R., M. S. Barbagelata, R. L. Caccuri, and D. O. Sordelli. 2006. Attenuation and persistence of and ability to induce protective immunity to a Staphylococcus aureus aroA mutant in mice. Infect. Immun. 74:3498-506.
Chang, B.S., J.S. Moon, H.M. Kang, Y.I. Kim, H.K. Lee, J.D. Kim, B.S. Lee, H.C. Koo, Y.H. Park. 2008. Protective effects of recombinant staphylococcal enterotoxin type C mutant vaccine against experimental bovine infection by a strain of Staphylococcus aureus isolated from subclinical mastitis in dairy cattle. Vaccine. 26:2081-2091.
Charpentier E, Anton AI, Barry P, Alfonso B, Fang Y, Novick RP. 2004. Novel cassette-based shuttle vector system for gram-positive bacteria. Appl Environemental Microbiol 70:6076-6085.
Chen J., H Liu., J. Yang, K. Chou. 2007. Prediction of linear B-cell epitopes using amino acid pair antigenicity scale. Amino Acids 33: 423-428.
Chen et. al, Adv Drug Deliv Rev. (2013), 65(10):1357-69.
Chen Y., L. Caruso, B. McClane, D. Fisher, P. Gupta. 2007. Disruption of a toxin by introduction of a foreign gene into the chromosome of Clostridium perfringens using targetron induced mutagenesis. Plasmid. 58:182-189.
Cui, L., J. Lian, and H. Neoh. 2005. DNA microarray-based identification of genes associated with glycopeptide resistance in Staphylococcus aureus. Antimicrob. agents … 49:3404-3413.
De Groot, A.S., J. McMurry, and L. Moise. 2008. Prediction of immunogenicity: in silico paradigms, ex vivo and in vivo correlates. Curr Opinion in Pharmacol. 8:620-626.
Dehal PS, Joachimiak MP, Price MN, Bates JT, Baumohl JK, Chivian D, Friedland GD, Huang KH, Keller K, Novichkov PS, Dubchak IL, Alm EJ, Arkin AP. MicrobesOnline: an integrated portal for comparative and functional genomics. Nucleic Acids Res. 2010 Jan; 38(Database issue): D396-400. Epub 2009 Nov 11.
Detmer, A., and J. Glenting. 2006. Live bacterial vaccines-a review and identification of potential hazards. Microb. Cell Fact. 12:1-12.
Diarra, M. S., D. Petitclerc, and P. Lacasse. 2002. Response of Staphylococcus aureus isolates from bovine mastitis to exogenous iron sources. J. Dairy Sci. 85:2141-2148.
EL-Manzalawy Y, Dobbs D, Honavar V. 2008a. Predicting linear B-cell epitopes using string kernels. J Mol Recognit 21: 243-255.
EL-Manzalawy Y, Dobbs D, Honavar V. 2008b. Predicting flexible length linear B-cell epitopes. 7th International Conference on Computational Systems Bioinformatics, Stanford, CA. pp. 121-131
Eng, N.F., S. Garlapati, V. Gerdts, A. Potter, L.A. Babiuk, and G.K. Mutwiri. 2010. The Potential of Polyphosphazenes for Delivery of Vaccine Antigens and Immunotherapeutic Agents. Curr Drug Deliv. 7(1):13-30.
Falla, T.J., D. Karunaratne, N., Hancock, R.E.W. 1996. Mode of Action of the Antimicrobial Peptide Indolicidin. The Journal of Biological Chemistry, 271(32), Issue of August 9, pp. 19298 -19303.
Falord, M., G. Karimova, A. Hiron, and T. Msadek. 2012. GraXSR proteins interact with the VraFG ABC transporter to form a five-component system required for cationic antimicrobial peptide sensing and resistance in Staphylococcus aureus. Antimicrob. Agents Chemother. 56:1047-58.
Falord, M., U. Mäder, A. Hiron, M. Débarbouillé, and T. Msadek. 2011. Investigation of the Staphylococcus aureus GraSR Regulon Reveals Novel Links to Virulence, Stress Response and Cell Wall Signal Transduction Pathways. PLoS One 6:e21323.
Ferens, W. a, and G. a Bohach. 2000. Persistence of Staphylococcus aureus on mucosal membranes: superantigens and internalization by host cells. J. Lab. Clin. Med. 135:225-30.
Foster, T. J. 2009. Colonization and infection of the human host by staphylococci: adhesion, survival and immune evasion. Vet. Dermatol. 20:456-70.
Fowler, V., and R. Proctor. 2014. Where does a Staphylococcus aureus vaccine stand? Clin. Microbiol. Infect. 20 Suppl 5:66-75.
García, V., M. Gómez, M. Iglesias, N. Sanjuan, M. Gherardi, M. C. Cerquetti, and D. Sordelli. 1996. Intramammary immunization with live-attenuated Staphylococcus aureus: microbiological and immunological studies in a mouse mastitis model. FEMS Immunol. Med. Microbiol. 14:45-51.
Gardy, J. L., M. R. Laird, F. Chen, S. Rey, C. J. Walsh, M. Ester and F. S. L. Brinkman. 2005. PSORTb v.2.0: Expanded prediction of bacterial protein subcellular localization and insights gained from comparative proteome analysis. Bioinformatics 21(5):617-623; doi:10.1093/bioinformatics/bti057
Garzoni, C., P. Francois, A. Huyghe, S. Couzinet, C. Tapparel, Y. Charbonnier, A. Renzoni, S. Lucchini, D.P. Lew, P. Vaudaux, W,L. Kelley, and J. Schrenzel. 2007. A global view of Staphylococcus aureus whole genome expression upon internalization in human epithelial cells. BMC Genomics. 8:171.
Gasteiger E., Hoogland C., Gattiker A., Duvaud S., Wilkins M.R., Appel R.D., Bairoch A. Protein Identification and Analysis Tools on the ExPASy Server; (In) John M. Walker (ed): The Proteomics Protocols Handbook, Humana Press (2005), pp. 571-607.
Gaupp, R., N. Ledala, and G. a Somerville. 2012. Staphylococcal response to oxidative stress. Front. Cell. Infect. Microbiol. 2:33.
Gläser, R., K. Becker, C. von Eiff, U. Meyer-Hoffert, and J. Harder. 2014. Decreased Susceptibility of Staphylococcus aureus Small-Colony Variants Toward Human Antimicrobial Peptides. J. Invest. Dermatol. 134:2347-50.
Goerke, C., S. Campana, M.G. Bayer, G. Döring, K. Botzenhart, and C. Wolz. 2000. Direct quantitative transcript analysis of the agr regulon of Staphylococcus aureus during human infection in comparison to the expression profile in vitro. Infect Immun. 68:1304-1311.
Griffiths, K. L., and S. a Khader. 2014. Novel vaccine approaches for protection against intracellular pathogens. Curr. Opin. Immunol. Elsevier Ltd 28:58-63.
Guidry, A.J., L.M. Berning, C.N. Hambleton.1993. Opsonization of Staphylococcus aureus by bovine immunoglobulin isotypes. J. of Dairy Sci. 76:1285-1289.
Hancock, R.E.W., and Diamond, G., 2000. The role of cationic antimicrobial peptides in innate host defences. Trends In Microbiology. 8(9):402.
Haveri, M., A. Roslöf, L. Rantala, and S. Pyörälä. 2007. Virulence genes of bovine Staphylococcus aureus from persistent and nonpersistent intramammary infections with different clinical characteristics. J Appl Microbiol. 103:993-1000.
Hogarth, C.J., J.L. Fitzpatrick, A.M. Nolan, F.J. Young, A. Pitt, and P.D. Eckersall. 2004. Differential protein composition of bovine whey: a comparison of whey from healthy animals and from those with clinical mastitis. Proteomics. 4:2094-2100.
Horsburgh, M. J., J. L. Aish, I. J. White, J. K. Lithgow, S. J. Foster, and L. Shaw. 2002. σ B Modulates Virulence Determinant Expression and Stress Resistance: Characterization of a Functional rsbU Strain Derived from Staphylococcus aureus 8325-4. J. Bacteriol. 184:5457-5467.
Jayarao, B.M., D.R. Henning. 2001. Prevalence of foodborne pathogens in bulk tank milk. J Dairy Sci. 84:2157-2162.
Kafala, B., and a Sasarman. 1997. Isolation of the Staphylococcus aureus hemCDBL gene cluster coding for early steps in heme biosynthesis. Gene 199:231-9.
Kahl B.C., 2014. Small colony variants (SCVs) of Staphylococcus aureus - A bacterial survival strategy. Infection, Genetics and Evolution 21: 515-522.
Karaolis, D.K., T.K. Means, D. Yang, M. Takahashi, T. Yoshimura, E. Muraille, D. Philpott, J.T. Schroeder, M. Hyodo, Y. Hayakawa, B.G. Talbot, E. Brouillette, and F. Malouin. 2007. Bacterial c-di-GMP is an immunostimulatory molecule. J Immunol. 178:2171-2181.
Kasturi, S.P. et al. 2011. Programming the magnitude and persistence of antibody responses with innate immunity. Nature 470:543-547.
Kawada-Matsuo, M., and Y. Yoshida. 2011. Role of two-component systems in the resistance of Staphylococcus aureus to antibacterial agents. Virulence 2:427-430.
Kawada-Matsuo, M., Y. Yoshida, T. Zendo, J. Nagao, Y. Oogai, Y. Nakamura, K. Sonomoto, N. Nakamura, and H. Komatsuzawa. 2013. Three distinct two-component systems are involved in resistance to the class I bacteriocins, Nukacin ISK-1 and nisin A, in Staphylococcus aureus. PLoS One 8: e69455.
Kerro-Dego, O., T. Prysliak, A. a Potter, and J. Perez-Casal. 2006. DNA-protein immunization against the GapB and GapC proteins of a mastitis isolate of Staphylococcus aureus. Vet. Immunol. Immunopathol. 113:125-38.
Kollaritsch, H., S. J. Cryz, a B. Lang, C. Herzog, J. U. Que, and G. Wiedermann. 2000. Local and systemic immune responses to combined vibrio cholerae CVD103-HgR and salmonella typhi ty21a live oral vaccines after primary immunization and reimmunization. Vaccine 18:3031-9.
Koo, S. P., A. S. Bayer, H. G. Sahl, R. A. Proctor, M. R. Yeaman, S. Koo, A. S. Bayer, H. Sahl, and R. A. Proctor. 1996. Staphylocidal action of thrombin-induced platelet microbicidal protein is not solely dependent on transmembrane potential. These include: Staphylocidal Action of Thrombin-Induced Platelet Microbicidal Protein Is Not Solely Dependent on Transmembrane Pot 64.
Kraus, D., S. Herbert, S. a Kristian, A. Khosravi, V. Nizet, F. Götz, and A. Peschel. 2008. The GraRS regulatory system controls Staphylococcus aureus susceptibility to antimicrobial host defenses. BMC Microbiol. 8:85.
Kreiswirth, B. N., S. Löfdahl, M. J. Betley, M. O'Reilly, P. M. Schlievert, M. S. Bergdoll, and R. P. Novick. 1983. The toxic shock syndrome exotoxin structural gene is not detectably transmitted by a prophage. Nature 305:709-12.
Kubica, M., K. Guzik, J. Koziel, M. Zarebski, W. Richter, B. Gajkowska, A. Golda, A. Maciag-Gudowska, K. Brix, L. Shaw, T. Foster, and J. Potempa. 2008. A potential new pathway for Staphylococcus aureus dissemination: the silent survival of S. aureus phagocytosed by human monocyte-derived macrophages. PLoS One 3:e1409.
Kuroda, M., K. Kuwahara-Arai, and K. Hiramatsu. 2000. Identification of the up- and down-regulated genes in vancomycin-resistant Staphylococcus aureus strains Mu3 and Mu50 by cDNA differential hybridization method. Biochem. Biophys. Res. Commun. 269:485-90.
Lammers, A., E. Kruijt, K. C. van de, P. J. Nuijten, and H. E. Smith. 2000. Identification of Staphylococcus aureus genes expressed during growth in milk: a useful model for selection of genes important in bovine mastitis? Microbiology. 146:981-987.
Larkin M.A., Blackshields G., Brown N.P., Chenna R., McGettigan P.A., McWilliam H.*, Valentin F.*, Wallace I.M., Wilm A., Lopez R., Thompson J.D., Gibson T.J. and Higgins D.G. 2007. ClustalW and ClustalX version 2. Bioinformatics 2007 23(21): 2947-2948.
Leitner, G., O. Krifucks, M. D. Kiran, and N. Balaban. 2011. Vaccine development for the prevention of staphylococcal mastitis in dairy cows. Vet. Immunol. Immunopathol. Elsevier B.V. 142:25-35.
Li, M., D. J. Cha, Y. Lai, A. E. Villaruz, D. E. Sturdevant, and M. Otto. 2007. The antimicrobial peptide-sensing system aps of Staphylococcus aureus. Mol. Microbiol. 66:1136-47.
Lin, L., A. S. Ibrahim, X. Xu, J. M. Farber, V. Avanesian, B. Baquir, Y. Fu, S. W. French, J. E. Edwards, and B. Spellberg. 2009. Th1-Th17 cells mediate protective adaptive immunity against Staphylococcus aureus and Candida albicans infection in mice. PLoS Pathog. 5:e1000703.
Linghua, Z., T. Xingshan, Z. Fengzhen. 2006. The efficacy of CpG oligodinucleotides, in combination with conventional adjuvants, as immunological adjuvants to swine streptococcic septicemia vaccine in piglets in vivo. Int Immunopharmacol. 6:1267-76.
Löffler, B., L. Tuchscherr, S. Niemann, and G. Peters. 2013. Staphylococcus aureus persistence in non-professional phagocytes. Int. J. Med. Microbiol. Elsevier GmbH.
Loiselle, M.C., C. Ster, B.G. Talbot, X. Zhao, G.F. Wagner, Y.R. Boisclair, and P. Lacasse. 2009. Impact of postpartum milking frequency on the immune system and the blood metabolite concentration of dairy cows. J Dairy Sci. 92:1900-1912.
Lowe, A.M., D.T. Beattie, and R.L. Deresiewicz. 1998. Identification of novel staphylococcal virulence genes by in vivo expression technology. Mol Microbiol. 27:967-976.
Maresso, A.W., and O. Schneewind. 2006. Iron acquisition and transport in Staphylococcus aureus. Biometals. 19:193-203.
Maresso AW, Schneewind O. Sortase as a target of anti-infective therapy. Pharmacol Rev. 2008 Mar;60(1):128-41.
Mayer, S.J., A.E. Waterman, P.M. Keen, N. Craven, and F.J. Bourne. 1988. Oxygen concentration in milk of healthy and mastitic cows and implications of low oxygen tension for the killing of Staphylococcus aureus by bovine neutrophils. J Dairy Res 55:513-519.
Meehl, M., S. Herbert, F. Götz, and A. Cheung. 2007. Interaction of the GraRS two-component system with the VraFG ABC transporter to support vancomycin-intermediate resistance in Staphylococcus aureus. Antimicrob. Agents Chemother. 51:2679-89.
Melchior, M.B., M.H. vanOsch, R.M. Graat, E. van Duijkeren, D.J. Mevius, N. Nielen, W. Gaastra, J. Fink-Gremmels. 2009. Biofilm formation and genotyping of Staphylococcus aureus bovine mastitis isolates: evidence for lack of penicillin-resistance in Agr-type II strains. Vet. Microbiol. 137:83-89.
Merino, N., A. Toledo-Arana, M. Vergara-Irigaray, J. Valle, C. Solano, E. Calvo, J. A. Lopez, T. J. Foster, J. R. Penadés, and I. Lasa. 2009. Protein A-mediated multicellular behavior in Staphylococcus aureus. J. Bacteriol. 191:832-43.
Middleton, J. R., C. D. Luby, and D. S. Adams. 2009. Efficacy of vaccination against staphylococcal mastitis: a review and new data. Vet. Microbiol. 134:192-8.
Middleton, J.R. 2008. Staphylococcus aureus antigens and challenges in vaccine development. Expert Rev Vaccines. 7(6):805-815.
Mitchell, G., A. Fugère, K. Pépin Gaudreau, E. Brouillette, E. H. Frost, A. M. Cantin, and F. Malouin. 2013. SigB is a dominant regulator of virulence in Staphylococcus aureus small-colony variants. PLoS One 8:e65018.
Mitchell, G., C.A. Lamontagne, ,E. Brouillette, G. Grondin , B.G. Talbot, M. Grandbois, F. Malouin. 2008. Staphylococcus aureus SigB activity promotes a strong fibronectin-bacterium interaction which may sustain host tissue colonization by small-colony variants isolated from cystic fibrosis patients. Mol Microbiol 70:1540-1555.
Mitchell, G., C.-A. Lamontagne, E. Brouillette, G. Grondin, B. G. Talbot, M. Grandbois, and F. Malouin. 2008. Staphylococcus aureus SigB activity promotes a strong fibronectin-bacterium interaction which may sustain host tissue colonization by small-colony variants isolated from cystic fibrosis patients. Mol. Microbiol. 70:1540-55.
Mitchell, G., G. Grondin, G. Bilodeau, A. M. Cantin, and F. Malouin. 2011. Infection of Polarized Airway Epithelial Cells by Normal and Small-Colony Variant Strains of Staphylococcus aureus Is Increased in Cells with Abnormal Cystic Fibrosis Transmembrane Conductance Regulator Function and Is Influenced by NF-{kappa}B. Infect. Immun. 79:3541-51.
Mitra SD, Velu D, Bhuvana M, Krithiga N, Banerjee A, Shome R, et al. Staphylococcus aureus spa type t267, clonal ancestor of bovine subclinical mastitis in India. J Appl Microbiol. 2013;114: 1604-1615.
Moisan, H., E. Brouillette, C.L. Jacob, P. Langlois-Bégin, S. Michaud, and F. Malouin. 2006. Transcription of virulence factors in Staphylococcus aureus small-colony variants isolated from cystic fibrosis patients is influenced by SigB. J Bacteriol. 188:64-76.
Myllys, V., J. Ridell, J. Bjorkroth, I. Biese, and S. Pyorala. 1997. Persistence in bovine mastitis of Staphylococcus aureus clones as assessed by random amplified polymorphic DNA analysis, ribotyping and biotyping. Vet Microbiol. 57:245-251.
National Mastitis Council. 1996. Current Concept of Bovine Mastitis. 4 ed. National Mastitis Council, Madison, WI.
Nickerson, S. C., W. E. Owens, L. K. Fox, C. C. Scheifinger, T. R. Shryock, and T. E. Spike. 1999. Comparison of tilmicosin and cephapirin as therapeutics for Staphylococcus aureus mastitis at dry-off. J Dairy Sci. 82:696-703.
Novick RP.Autoinduction and signal transduction in the regulation of staphylococcal virulence. Mol Microbiol. 2003 Jun;48(6):1429-49.
Novick RP, Geisinger E. Quorum sensing in staphylococci. Annu Rev Genet. 2008;42: 541-64.
Overton, I. M., S. Graham, K. a Gould, J. Hinds, C. H. Botting, S. Shirran, G. J. Barton, and P. J. Coote. 2011. Global network analysis of drug tolerance, mode of action and virulence in methicillin-resistant S. aureus. BMC Syst. Biol. BioMed Central Ltd 5:68.
Owens, W.E., C.H. Ray, J.L. Watts, and R.J. Yancey. 1997. Comparison of success of antibiotic therapy during lactation and results of antimicrobial susceptibility tests for bovine mastitis. J Dairy Sci. 80:313-317.
Park, Y.K., H.C. Koo, S.H. Kim, S.Y. Hwang, W.K. Jung, J. Kim, S. Shin, R. Kim, and Y. Park. 2007. The analysis of milk components and pathogenic bacteria isolated from bovine raw milk in Korea. J Dairy Sci. 90:5405-5414.
Pasetti, M. F., J. K. Simon, M. B. Sztein, and M. M. Levine. 2011. Immunology of gut mucosal vaccines. Immunol. Rev. 239:125-48.
Peles, F., M. Wagner, L. Varga, I. Hein, P. Rieck, K. Gutser, P. Keresztúri, G. Kardos, I. Turcsányi, B. Béri, and A. Szabó. 2007. Characterization of Staphylococcus aureus strains isolated from bovine milk in Hungary. Int J Food Microbiol. 118:186-93.
Pellegrino, M., J. Giraudo, C. Raspanti, L. Odierno, and C. Bogni. 2010. Efficacy of immunization against bovine mastitis using a Staphylococcus aureus avirulent mutant vaccine. Vaccine 28:4523-8.
Pellegrino, M., J. Giraudo, C. Raspanti, R. Nagel, L. Odierno, V. Primo, and C. Bogni. 2008. Experimental trial in heifers vaccinated with Staphylococcus aureus avirulent mutant against bovine mastitis. Vet. Microbiol. 127:186-90.
Peterson, J. D., Umayam, L. A., Dickinson, T., Hickey, E. K., White, O. 2001. The Comprehensive Microbial Resource. Nucleic Acids Res. 29(1): 123-5.
Petitclerc,D., K. Lauzon, A. Cochu, C. Ster, M.S. Diarra, and P. Lacasse. 2007. Efficacy of a lactoferrin-penicillin combination to treat {beta}-lactam-resistant Staphylococcus aureus mastitis. J Dairy Sci. 90:2778-2787.
Pragman, A.A., and P.M. Schlievert. 2004. Virulence regulation in Staphylococcus aureus: the need for in vivo analysis of virulence factor regulation. FEMS Immunol Med Microbiol. 42:147-154.
Proctor, R. a, C. von Eiff, B. C. Kahl, K. Becker, P. McNamara, M. Herrmann, and G. Peters. 2006. Small colony variants: a pathogenic form of bacteria that facilitates persistent and recurrent infections. Nat. Rev. Microbiol. 4:295-305.
Proctor, R. a. 2012. Challenges for a universal Staphylococcus aureus vaccine. Clin. Infect. Dis. 54:1179-86.
Proctor, R. a., A. Kriegeskorte, B. C. Kahl, K. Becker, B. Löffler, and G. Peters. 2014. Staphylococcus aureus Small Colony Variants (SCVs): a road map for the metabolic pathways involved in persistent infections. Front. Cell. Infect. Microbiol. 4:1-8.
Pulli, B., M. Ali, R. Forghani, S. Schob, K. L. C. Hsieh, G. Wojtkiewicz, J. J. Linnoila, and J. W. Chen. 2013. Measuring myeloperoxidase activity in biological samples. PLoS One 8: e67976.
Reyher, K. K., S. Dufour, H. W. Barkema, L. Des Côteaux, T. J. Devries, I. R. Dohoo, G. P. Keefe, J.-P. Roy, and D. T. Scholl. 2011. The National Cohort of Dairy Farms--a data collection platform for mastitis research in Canada. J. Dairy Sci. Elsevier 94:1616-26.
Sadowska, B., A. Bonar, C. von Eiff, R. A. Proctor, M. Chmiela, W. Rudnicka, and B. Róźalska. 2002. Characteristics of Staphylococcus aureus, isolated from airways of cystic fibrosis patients, and their small colony variants. FEMS Immunol. Med. Microbiol. 32:191-7.
Saha, S and Raghava G.P.S., (2006) Prediction of Continuous B-cell Epitopes in an Antigen Using Recurrent Neural Network. Proteins, 65(1),40-48.
Saha.S and Raghava G.P.S. BcePred: Prediction of Continuous B-Cell Epitopes in Antigenic Sequences Using Physico-chemical Properties. In G.Nicosia, V.Cutello, P.J. Bentley and J.Timis (Eds.) ICARIS 2004, LNCS 3239, 197-204, Springer, 2004.
Sandholm, M., L. Kaartinen, and S. Pyorala. 1990. Bovine mastitis - why does antibiotic therapy not always work? An overview. J Vet Phamacol Therap. 13:248-260.
Schaffer, A.C., and J.C. Lee. 2009. Staphylococcal vaccines and immunotherapies. Infect Dis Clin North Am. 23:153-171.
Sears, P.M. and McCarthy, K.K. 2003. Management and treatment of staphylococcal mastitis. Vet Clin North Am Food Anim Pract 19:171-185.
Sendi, P., and R. a Proctor. 2009. Staphylococcus aureus as an intracellular pathogen: the role of small colony variants. Trends Microbiol. 17:54-8.
Senn, M. M., M. Bischoff, C. Von Eiff, B. Berger-bächi, and B. Berger-ba. 2005. σ B Activity in a Staphylococcus aureus hemB Mutant. J. Bacteriol. 187:7397-7406.
Sibbald, M.J., A.K. Ziebandt, S. Engelmann, M. Hecker, A. de Jong, H.J. Harmsen, G.C. Raangs, I. Stokroos, J.P. Arends, J.Y. Dubois, and J.M. van Dijl. 2006. Mapping the pathways to staphylococcal pathogenesis by comparative secretomics. Microbiol Mol Biol Rev. 70:755-788.
Silanikove, N., F. Shapiro, and G. Leitner. 2007. Posttranslational ruling of xanthine oxidase activity in bovine milk by its substrates. Biochem Biophys Res Commun. 363:561-565.
Somerville, G.A., and R.A. Proctor. 2009. At the crossroads of bacterial metabolism and virulence factor synthesis in Staphylococci. Microbiol Mol Biol Rev. 73:233-248.
Spellberg, B., and R. Daum. 2012. Development of a vaccine against Staphylococcus aureus. Semin. Immunopathol. 34:335-48.
Sprickler A.R. and J.A. Roth. Adjuvants in veterinary vaccines: mode of action and adverse effects. 2003. 17:273-281.
Srinivasan, V., A.A. Sawant, B.E. Gillespie, S.J. Headrick, L. Ceasaris, and S.P. Oliver. 2006. Prevalence of enterotoxin and toxic shock syndrome toxin genes in Staphylococcus aureus isolated from milk of cows with mastitis. Foodborne Pathog Dis. 3:274-83.
Srivastava S, Singh V, Kumar V, Verma PC, Srivastava R, Basu V, Gupta V, Rawat AK. Identification of regulatory elements in 16S rRNA gene of Acinetobacter species isolated from water sample. Bioinformation. 2008; 3(4):173-6. Epub 2008 Dec 6.
Ster, C., M. Allard, S. Boulanger, M. Lamontagne Boulet, J. Mulhbacher, D. a Lafontaine, E. Marsault, P. Lacasse, and F. Malouin. 2013. Experimental treatment of Staphylococcus aureus bovine intramammary infection using a guanine riboswitch ligand analog. J. Dairy Sci. Elsevier 96:1000-8.
Sutra, L., and B. Poutrel. 1994. Virulence factors involved in the pathogenesis of bovine intramammary infections due to Staphylococcus aureus. J. Med. Microbiol. 40:79-89.
Taverna, F., A. Negri, R. Piccinini, A. Zecconi, S. Nonnis, S. Ronchi, and G. Tedeschi. 2007. Characterization of cell wall associated proteins of a Staphylococcus aureus isolated from bovine mastitis case by a proteomic approach. Vet Microbiol. 119:240-247
Tollersrud, T., A.H. Kampen, and K. Kenny. 2006. Staphylococcus aureus enterotoxin D is secreted in milk and stimulates specific antibody responses in cows in the course of experimental intramammary infection. Infect Immun. 74:3507-3512.
Tuchscherr, L., E. Medina, M. Hussain, W. Völker, V. Heitmann, S. Niemann, D. Holzinger, J. Roth, R. a Proctor, K. Becker, G. Peters, and B. Löffler. 2011. Staphylococcus aureus phenotype switching: an effective bacterial strategy to escape host immune response and establish a chronic infection. EMBO Mol. Med. 3:129-41.
Tuchscherr, L., V. Heitmann, M. Hussain, D. Viemann, J. Roth, C. von Eiff, G. Peters, K. Becker, and B. Löffler. 2010. Staphylococcus aureus small-colony variants are adapted phenotypes for intracellular persistence. J. Infect. Dis. 202:1031-40.
Tuchscherr, L.P., F.R. Buzzola, L.P. Alvarez, J.C. Lee, and D.O. Sordelli. 2008. Antibodies to capsular polysaccharide and clumping factor A prevent mastitis and the emergence of unencapsulated and small-colony variants of Staphylococcus aureus in mice. Infect Immun. 76:5738-5744.
Tusnády, G.E. and Simon, I. 2001. The HMMTOP transmembrane topology prediction server" Bioinformatics 17, 849-850.
Veh KA, Klein RC, Ster C, Keefe G, Lacasse P, Scholl D, et al. Genotypic and phenotypic characterization of Staphylococcus aureus causing persistent and nonpersistent subclinical bovine intramammary infections during lactation or the dry period. J Dairy Sci. 2015;98: 155-168
Von Eiff, C., P. McNamara, K. Becker, X. Lei, M. Ziman, B. R. Bochner, G. Peters, and R. A. Proctor. 2006. Phenotype Microarray Profiling of Staphylococcus aureus menD and hemB Mutants with the Small-Colony-Variant Phenotype Phenotype Microarray Profiling of Staphylococcus aureus menD and hemB Mutants with the Small-Colony-Variant Phenotype †. J. Bacteriol. 188:687-693.
Voyich, J.M., K.R. Braughton, D.E. Sturdevant, A.R. Whitney, B. Saïd-Salim, S.F. Porcella, R.D. Long, D.W. Dorward, D.J. Gardner, B.N. Kreiswirth, J.M. Musser, and F.R. DeLeo. 2005. Insights into mechanisms used by Staphylococcus aureus to avoid destruction by human neutrophils. J Immunol. 175:3907-3919.
Watson, D. L. 1984. Evaluation of attenuated, live staphylococcal mastitis vaccine in lactating heifers. J. Dairy Sci. 67:2608-13.
Whist, A. C., O. Osterås, and L. Sølverød. 2009. Association between isolation of Staphylococcus aureus one week after calving and milk yield, somatic cell count, clinical mastitis, and culling through the remaining lactation. J. Dairy Res. 76:24-35.
WO/2003/091279
WO/2004/043405
WO/2005/007683
WO/2006/059846
WO/2008/152447
Xia, Y., and J. L. Zweier. 1997. Measurement of Myeloperoxidase in Leukocyte-Containing Tissues. Anal. Biochem. 245:93-96.
Yang, S.-J., A. S. Bayer, N. N. Mishra, M. Meehl, N. Ledala, M. R. Yeaman, Y. Q. Xiong, and A. L. Cheung. 2012. The Staphylococcus aureus Two-Component Regulatory System, GraRS, Senses and Confers Resistance to Selected Cationic Antimicrobial Peptides. Infect. Immun. 80:74-81.
Zecconi, A., R. Piccinini, and L. K. Fox. 2003. Epidemiologic study of intramammary infections with Staphylococcus aureus during a control program in nine commercial dairy herds. J. Am. Vet. Med. Assoc. 223:684-8.
Zhang, C., J. Yang, and L. K. Jennings. 2004. Non - Leukocyte-Derived Reactive Oxygen Species. Diabetes 53:2950-2959.
Ziebandt, A.K., H. Kusch, M. Degner, S. Jaglitz, M.J. Sibbald, J.P. Arends, M.A. Chlebowicz, D. Albrecht, R. Pantucek, J. Doškar, W. Ziebuhr, B.M. Bröker, M. Hecker, J.M. van Dijl, and S. Engelmann. 2010. Proteomics uncovers extreme heterogeneity in the Staphylococcus aureus exoproteome due to genomic plasticity and variant gene regulation. Proteomics 285(47)36794-36803.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> SOCPRA SCIENCES ET GENIE S.E.C.
MALOUIN, FRANCOIS
STER, CELINE
COTE-GRAVEL, JULIE
BROUILLETTE, ERIC
<120> ВАКЦИННЫЕ КОНСТРУКТЫ И ИХ ПРИМЕНЕНИЯ ПРОТИВ ИНФЕКЦИЙ СТАФИЛОКОККА
<130> 193 481 m2/m14/mfe
<140>
<141>
<150> 62/411,120
<151> 2016-10-21
<160> 196
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> PRT
<213> Staphylococcus aureus
<400> 1
Lys Asp Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln
1 5 10 15
<210> 2
<211> 40
<212> PRT
<213> Staphylococcus aureus
<400> 2
Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp Gly
1 5 10 15
Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg Glu
20 25 30
Asn Val Lys Ile Asn Thr Ala Asp
35 40
<210> 3
<211> 48
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 3
Lys Asp Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Glu
1 5 10 15
Ala Ala Ala Lys Glu Ala Ala Ala Lys Lys Asp Ile Asn Lys Ile Tyr
20 25 30
Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu Asn Asp
35 40 45
<210> 4
<211> 168
<212> ДНК
<213> Staphylococcus aureus
<400> 4
atgctagaat ctagagagca attatcagtc gaagaatacg aaacattctt taacagattt 60
gataatcaag aatttgattt cgaacgtgaa ttgacacaag atccatattc aaaagtatac 120
ttatacagta tagaagacca tatcagaaca tataagatag agaaataa 168
<210> 5
<211> 55
<212> PRT
<213> Staphylococcus aureus
<400> 5
Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe
1 5 10 15
Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr
20 25 30
Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile
35 40 45
Arg Thr Tyr Lys Ile Glu Lys
50 55
<210> 6
<211> 165
<212> ДНК
<213> Staphylococcus aureus
<400> 6
ctagaatcta gagagcaatt atcagtcgaa gaatacgaaa cattctttaa cagatttgat 60
aatcaagaat ttgatttcga acgtgaattg acacaagatc catattcaaa agtatactta 120
tacagtatag aagaccatat cagaacatat aagatagaga aataa 165
<210> 7
<211> 54
<212> PRT
<213> Staphylococcus aureus
<400> 7
Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe Phe
1 5 10 15
Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr Gln
20 25 30
Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile Arg
35 40 45
Thr Tyr Lys Ile Glu Lys
50
<210> 8
<211> 61
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 8
Met His His His His His His Leu Glu Ser Arg Glu Gln Leu Ser Val
1 5 10 15
Glu Glu Tyr Glu Thr Phe Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp
20 25 30
Phe Glu Arg Glu Leu Thr Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr
35 40 45
Ser Ile Glu Asp His Ile Arg Thr Tyr Lys Ile Glu Lys
50 55 60
<210> 9
<211> 66
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 9
Met Arg Gly Ser His His His His His His Gly Ser Leu Glu Ser Arg
1 5 10 15
Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe Phe Asn Arg Phe Asp
20 25 30
Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr Gln Asp Pro Tyr Ser
35 40 45
Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile Arg Thr Tyr Lys Ile
50 55 60
Glu Lys
65
<210> 10
<211> 1889
<212> ДНК
<213> Staphylococcus aureus
<400> 10
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cgttaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gctcataaac taaacatgac agagtcatat ccaattataa aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc gaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac atctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt acacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac tagtgataaa 1140
tacatcccta atactgattt gaaacgtggg caagctgatt tatttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaggcaa ttataggaac gaaaaaacat 1260
catgttaata ttaagttacg taaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaggcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagcgaaaaa taaagttgat aaatctattg aaacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac ataaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatttt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa cattagacat tccatataa 1889
<210> 11
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 11
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Thr Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Ala Lys Asn Lys Val Asp Lys Ser Ile Glu Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 12
<211> 200
<212> PRT
<213> Staphylococcus aureus
<400> 12
Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala Pro His Asp Val
1 5 10 15
Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala Ser Glu Leu Asn
20 25 30
Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu Val Ile His Thr
35 40 45
Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala Lys Glu Pro Tyr
50 55 60
Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn Thr Asp Leu Lys
65 70 75 80
Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser Ile Lys Asp Leu
85 90 95
Val Lys His Lys Lys His Gly Lys Ala Ile Ile Gly Thr Lys Lys His
100 105 110
His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys Ile Tyr Phe Met
115 120 125
Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu Asn Asp Lys Asp
130 135 140
Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His Ile Val Ser Gln
145 150 155 160
Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu Ala Leu Glu Lys
165 170 175
Ala Lys Asn Lys Val Asp Lys Ser Ile Glu Thr Arg Ser Glu Ala Ile
180 185 190
Ser Ser Ile Ser Ser Leu Thr Gly
195 200
<210> 13
<211> 199
<212> PRT
<213> Staphylococcus aureus
<400> 13
Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala Pro His Asp Val Thr
1 5 10 15
Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala Ser Glu Leu Asn Asn
20 25 30
Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu Val Ile His Thr Lys
35 40 45
Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala Lys Glu Pro Tyr Asn
50 55 60
Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn Thr Asp Leu Lys Arg
65 70 75 80
Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser Ile Lys Asp Leu Val
85 90 95
Lys His Lys Lys His Gly Lys Ala Ile Ile Gly Thr Lys Lys His His
100 105 110
Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr
115 120 125
Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr
130 135 140
Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His Ile Val Ser Gln Phe
145 150 155 160
Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu Ala Leu Glu Lys Ala
165 170 175
Lys Asn Lys Val Asp Lys Ser Ile Glu Thr Arg Ser Glu Ala Ile Ser
180 185 190
Ser Ile Ser Ser Leu Thr Gly
195
<210> 14
<211> 146
<212> PRT
<213> Staphylococcus aureus
<400> 14
Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala Pro His Asp Val Thr
1 5 10 15
Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala Ser Glu Leu Asn Asn
20 25 30
Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu Val Ile His Thr Lys
35 40 45
Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala Lys Glu Pro Tyr Asn
50 55 60
Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn Thr Asp Leu Lys Arg
65 70 75 80
Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser Ile Lys Asp Leu Val
85 90 95
Lys His Lys Lys His Gly Lys Ala Ile Ile Gly Thr Lys Lys His His
100 105 110
Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr
115 120 125
Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr
130 135 140
Gln Glu
145
<210> 15
<211> 54
<212> PRT
<213> Staphylococcus aureus
<400> 15
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly
1 5 10 15
Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr
20 25 30
Thr Lys Ala Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His
35 40 45
Lys Lys Asp Ala Leu Ala
50
<210> 16
<211> 25
<212> PRT
<213> Staphylococcus aureus
<400> 16
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly
1 5 10 15
Pro Thr Phe Val Leu Asn Asp Lys Asp
20 25
<210> 17
<211> 26
<212> PRT
<213> Staphylococcus aureus
<400> 17
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly
1 5 10 15
Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr
20 25
<210> 18
<211> 15
<212> PRT
<213> Staphylococcus aureus
<400> 18
Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu Ala
1 5 10 15
<210> 19
<211> 23
<212> PRT
<213> Staphylococcus aureus
<400> 19
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly
1 5 10 15
Pro Thr Phe Val Leu Asn Asp
20
<210> 20
<211> 19
<212> PRT
<213> Staphylococcus aureus
<400> 20
Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp
1 5 10 15
Ala Leu Ala
<210> 21
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 21
Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu Ala
1 5 10
<210> 22
<211> 13
<212> PRT
<213> Staphylococcus aureus
<400> 22
Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala Ser
1 5 10
<210> 23
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 23
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu
1 5 10
<210> 24
<211> 13
<212> PRT
<213> Staphylococcus aureus
<400> 24
Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu Asn Asp
1 5 10
<210> 25
<211> 153
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 25
Met His His His His His His Ala Ser Leu Ser Ser Glu Ile Lys Tyr
1 5 10 15
Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln
20 25 30
Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr
35 40 45
Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val
50 55 60
Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile
65 70 75 80
Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu
85 90 95
Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile
100 105 110
Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile
115 120 125
Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe
130 135 140
Val Leu Asn Asp Lys Asp Tyr Gln Glu
145 150
<210> 26
<211> 158
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 26
Met Arg Gly Ser His His His His His His Gly Ser Ala Ser Leu Ser
1 5 10 15
Ser Glu Ile Lys Tyr Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln
20 25 30
Gln Lys Ala Asn Gln Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro
35 40 45
His Phe Tyr Asn Tyr Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp
50 55 60
Asn Leu Phe Asp Val Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr
65 70 75 80
Ser Asp Lys Tyr Ile Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp
85 90 95
Leu Phe Val Ala Glu Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys
100 105 110
His Gly Lys Ala Ile Ile Gly Thr Lys Lys His His Val Asn Ile Lys
115 120 125
Leu Arg Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu
130 135 140
Gly Gly Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu
145 150 155
<210> 27
<211> 50
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 27
Lys Asp Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly
1 5 10 15
Pro Thr Phe Val Leu Asn Asp Lys Asp Tyr Glu Arg Lys Tyr Lys Lys
20 25 30
His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala
35 40 45
Leu Ala
50
<210> 28
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 28
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctggcaga gtgtatacac ctaagaggaa tattactctt 360
aataaagaag ttgtcacttt aaaagaattg gatcatatca taagatttgc tcatatttcc 420
tatggcttgt atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggtggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 29
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 29
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 30
<211> 168
<212> PRT
<213> Staphylococcus aureus
<400> 30
Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu Gln Lys Val
1 5 10 15
Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys Lys Leu Tyr
20 25 30
Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn Lys Ser Arg
35 40 45
Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln Val Arg Ile
50 55 60
His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr Thr Pro Lys
65 70 75 80
Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys Glu Leu Asp
85 90 95
His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr Met Gly Glu
100 105 110
His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp Gly Gly Lys
115 120 125
Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg Glu Asn Val
130 135 140
Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys Leu Val Lys
145 150 155 160
Ser Val Asn Asp Ile Glu Gln Val
165
<210> 31
<211> 116
<212> PRT
<213> Staphylococcus aureus
<400> 31
Gln Asp Lys Gln Leu Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu
1 5 10 15
Lys Ala Leu Val Lys Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile
20 25 30
Asn Gly Lys Ser Asn Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro
35 40 45
Leu Asn Glu Asn Gln Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val
50 55 60
Ala Gly Arg Val Tyr Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu
65 70 75 80
Val Val Thr Leu Lys Glu Leu Asp His Ile Ile Arg Phe Ala His Ile
85 90 95
Ser Tyr Gly Leu Tyr Met Gly Glu His Leu Pro Lys Gly Asn Ile Val
100 105 110
Ile Asn Thr Lys
115
<210> 32
<211> 115
<212> PRT
<213> Staphylococcus aureus
<400> 32
Asp Lys Gln Leu Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys
1 5 10 15
Ala Leu Val Lys Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn
20 25 30
Gly Lys Ser Asn Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu
35 40 45
Asn Glu Asn Gln Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala
50 55 60
Gly Arg Val Tyr Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val
65 70 75 80
Val Thr Leu Lys Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser
85 90 95
Tyr Gly Leu Tyr Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile
100 105 110
Asn Thr Lys
115
<210> 33
<211> 159
<212> PRT
<213> Staphylococcus aureus
<400> 33
Asp Lys Gln Leu Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys
1 5 10 15
Ala Leu Val Lys Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn
20 25 30
Gly Lys Ser Asn Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu
35 40 45
Asn Glu Asn Gln Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala
50 55 60
Gly Arg Val Tyr Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val
65 70 75 80
Val Thr Leu Lys Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser
85 90 95
Tyr Gly Leu Tyr Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile
100 105 110
Asn Thr Lys Asp Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu
115 120 125
Gln Lys Asp Arg Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn
130 135 140
Val Thr Phe Lys Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
145 150 155
<210> 34
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 34
Lys Asp Thr Ile Asn Gly Lys Ser Asn Lys Ser Arg Asn Trp
1 5 10
<210> 35
<211> 166
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 35
Met His His His His His His Asp Lys Gln Leu Gln Lys Val Glu Glu
1 5 10 15
Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys Lys Leu Tyr Asp Arg
20 25 30
Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn Lys Ser Arg Asn Trp
35 40 45
Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln Val Arg Ile His Leu
50 55 60
Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr Thr Pro Lys Arg Asn
65 70 75 80
Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys Glu Leu Asp His Ile
85 90 95
Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr Met Gly Glu His Leu
100 105 110
Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp Gly Gly Lys Tyr Thr
115 120 125
Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg Glu Asn Val Lys Ile
130 135 140
Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys Leu Val Lys Ser Val
145 150 155 160
Asn Asp Ile Glu Gln Val
165
<210> 36
<211> 171
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 36
Met Arg Gly Ser His His His His His His Gly Ser Asp Lys Gln Leu
1 5 10 15
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
20 25 30
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
35 40 45
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
50 55 60
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr
65 70 75 80
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
85 90 95
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
100 105 110
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
115 120 125
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
130 135 140
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
145 150 155 160
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
165 170
<210> 37
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 37
atgaataaaa atatagtcat taaaagcatg gcagcattag ccattctaac ctcagtaact 60
ggaataaatg ctgcagtcgt tgaagagaca caacaaatag caaatgcaga gaagaatgtt 120
acgcaagtta aagatacaaa tatttttcca tataatggcg tcgtttcatt taaagatgcg 180
acaggttttg taattggaaa aaatacaatt atcaccaata aacatgtatc aaaagattat 240
aaagttggcg atagaattac tgcccatcca aacggtgaca aaggaaatgg tggtatatat 300
aaaattaaaa gcatttctga ttatccgggt gatgaagaca tctctgtcat gaatattgaa 360
gaacaagcag tcgaacgtgg accaaaaggc tttaatttta atgaaaatgt ccaagcattc 420
aattttgcga aagatgctaa agttgatgac aaaattaaag ttattggtta cccattacct 480
gctcaaaata gttttaaaca gtttgaatct acaggaacta taaaaagaat caaagacaat 540
attttaaatt ttgatgcata cattgaaccc gggaattcag gatcaccagt tctaaattct 600
aacaatgagg tcataggtgt ggtgtatggc ggtattggaa aaattggttc tgaatataat 660
ggtgccgtat actttacgcc tcaaatcaaa gattttattc aaaagcacat tgaacaataa 720
<210> 38
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 38
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 39
<211> 199
<212> PRT
<213> Staphylococcus aureus
<400> 39
Thr Gln Val Lys Asp Thr Asn Ile Phe Pro Tyr Asn Gly Val Val Ser
1 5 10 15
Phe Lys Asp Ala Thr Gly Phe Val Ile Gly Lys Asn Thr Ile Ile Thr
20 25 30
Asn Lys His Val Ser Lys Asp Tyr Lys Val Gly Asp Arg Ile Thr Ala
35 40 45
His Pro Asn Gly Asp Lys Gly Asn Gly Gly Ile Tyr Lys Ile Lys Ser
50 55 60
Ile Ser Asp Tyr Pro Gly Asp Glu Asp Ile Ser Val Met Asn Ile Glu
65 70 75 80
Glu Gln Ala Val Glu Arg Gly Pro Lys Gly Phe Asn Phe Asn Glu Asn
85 90 95
Val Gln Ala Phe Asn Phe Ala Lys Asp Ala Lys Val Asp Asp Lys Ile
100 105 110
Lys Val Ile Gly Tyr Pro Leu Pro Ala Gln Asn Ser Phe Lys Gln Phe
115 120 125
Glu Ser Thr Gly Thr Ile Lys Arg Ile Lys Asp Asn Ile Leu Asn Phe
130 135 140
Asp Ala Tyr Ile Glu Pro Gly Asn Ser Gly Ser Pro Val Leu Asn Ser
145 150 155 160
Asn Asn Glu Val Ile Gly Val Val Tyr Gly Gly Ile Gly Lys Ile Gly
165 170 175
Ser Glu Tyr Asn Gly Ala Val Tyr Phe Thr Pro Gln Ile Lys Asp Phe
180 185 190
Ile Gln Lys His Ile Glu Gln
195
<210> 40
<211> 206
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 40
Met His His His His His His Thr Gln Val Lys Asp Thr Asn Ile Phe
1 5 10 15
Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val Ile
20 25 30
Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr Lys
35 40 45
Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn Gly
50 55 60
Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu Asp
65 70 75 80
Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro Lys
85 90 95
Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys Asp
100 105 110
Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro Ala
115 120 125
Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg Ile
130 135 140
Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn Ser
145 150 155 160
Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val Tyr
165 170 175
Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr Phe
180 185 190
Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
195 200 205
<210> 41
<211> 211
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 41
Met Arg Gly Ser His His His His His His Gly Ser Thr Gln Val Lys
1 5 10 15
Asp Thr Asn Ile Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala
20 25 30
Thr Gly Phe Val Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val
35 40 45
Ser Lys Asp Tyr Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly
50 55 60
Asp Lys Gly Asn Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr
65 70 75 80
Pro Gly Asp Glu Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val
85 90 95
Glu Arg Gly Pro Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe
100 105 110
Asn Phe Ala Lys Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly
115 120 125
Tyr Pro Leu Pro Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly
130 135 140
Thr Ile Lys Arg Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile
145 150 155 160
Glu Pro Gly Asn Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val
165 170 175
Ile Gly Val Val Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn
180 185 190
Gly Ala Val Tyr Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His
195 200 205
Ile Glu Gln
210
<210> 42
<211> 600
<212> ДНК
<213> Staphylococcus aureus
<400> 42
atggcaatga actttaaagt ctttgacaat agtcaacttg tagcagaata tgctgctgat 60
attattagaa agcaatttaa caataatcct actacaattg caggttttca tttagataca 120
gatcaagcgc cagttctaga tgaattaaag aaaaatgttg aaaaacatgc tgttgatttt 180
agccaaataa atattttaga ttatgacgat aaaaaatcat atttcgaagc gttaggtgta 240
ccagcaggtc aagtttatcc aattgcttat gaaaaagatg caatcgaatt aatcgctgat 300
aagattaaaa ctaaagaaaa taaagggaaa ttaacattac aagttgtttc tatcgatgag 360
caaggtaagt taaatgttag tattcgtcaa ggactaatgg aagcaagaga aattttctta 420
gtagtgacag gtgctaataa acgagatgta gttgaaaaat tatatcaaga aaatggtaaa 480
acaagcttcg aaccagccga tttaaaagca catagaatgg taaatgttat tcttgataaa 540
gaagcggctg caggtttacc tgaagatgtt aaagcttact ttacgtcacg ctttgcttaa 600
<210> 43
<211> 199
<212> PRT
<213> Staphylococcus aureus
<400> 43
Met Ala Met Asn Phe Lys Val Phe Asp Asn Ser Gln Leu Val Ala Glu
1 5 10 15
Tyr Ala Ala Asp Ile Ile Arg Lys Gln Phe Asn Asn Asn Pro Thr Thr
20 25 30
Ile Ala Gly Phe His Leu Asp Thr Asp Gln Ala Pro Val Leu Asp Glu
35 40 45
Leu Lys Lys Asn Val Glu Lys His Ala Val Asp Phe Ser Gln Ile Asn
50 55 60
Ile Leu Asp Tyr Asp Asp Lys Lys Ser Tyr Phe Glu Ala Leu Gly Val
65 70 75 80
Pro Ala Gly Gln Val Tyr Pro Ile Ala Tyr Glu Lys Asp Ala Ile Glu
85 90 95
Leu Ile Ala Asp Lys Ile Lys Thr Lys Glu Asn Lys Gly Lys Leu Thr
100 105 110
Leu Gln Val Val Ser Ile Asp Glu Gln Gly Lys Leu Asn Val Ser Ile
115 120 125
Arg Gln Gly Leu Met Glu Ala Arg Glu Ile Phe Leu Val Val Thr Gly
130 135 140
Ala Asn Lys Arg Asp Val Val Glu Lys Leu Tyr Gln Glu Asn Gly Lys
145 150 155 160
Thr Ser Phe Glu Pro Ala Asp Leu Lys Ala His Arg Met Val Asn Val
165 170 175
Ile Leu Asp Lys Glu Ala Ala Ala Gly Leu Pro Glu Asp Val Lys Ala
180 185 190
Tyr Phe Thr Ser Arg Phe Ala
195
<210> 44
<211> 206
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 44
Met His His His His His His Met Ala Met Asn Phe Lys Val Phe Asp
1 5 10 15
Asn Ser Gln Leu Val Ala Glu Tyr Ala Ala Asp Ile Ile Arg Lys Gln
20 25 30
Phe Asn Asn Asn Pro Thr Thr Ile Ala Gly Phe His Leu Asp Thr Asp
35 40 45
Gln Ala Pro Val Leu Asp Glu Leu Lys Lys Asn Val Glu Lys His Ala
50 55 60
Val Asp Phe Ser Gln Ile Asn Ile Leu Asp Tyr Asp Asp Lys Lys Ser
65 70 75 80
Tyr Phe Glu Ala Leu Gly Val Pro Ala Gly Gln Val Tyr Pro Ile Ala
85 90 95
Tyr Glu Lys Asp Ala Ile Glu Leu Ile Ala Asp Lys Ile Lys Thr Lys
100 105 110
Glu Asn Lys Gly Lys Leu Thr Leu Gln Val Val Ser Ile Asp Glu Gln
115 120 125
Gly Lys Leu Asn Val Ser Ile Arg Gln Gly Leu Met Glu Ala Arg Glu
130 135 140
Ile Phe Leu Val Val Thr Gly Ala Asn Lys Arg Asp Val Val Glu Lys
145 150 155 160
Leu Tyr Gln Glu Asn Gly Lys Thr Ser Phe Glu Pro Ala Asp Leu Lys
165 170 175
Ala His Arg Met Val Asn Val Ile Leu Asp Lys Glu Ala Ala Ala Gly
180 185 190
Leu Pro Glu Asp Val Lys Ala Tyr Phe Thr Ser Arg Phe Ala
195 200 205
<210> 45
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 45
Asp Ile Ile Arg Lys Gln Phe Asn Asn Asn Pro Thr Thr Ile
1 5 10
<210> 46
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 46
Tyr Phe Glu Ala Leu Gly Val Pro Ala Gly Gln Val Tyr Pro
1 5 10
<210> 47
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 47
Ala Asp Lys Ile Lys Thr Lys Glu Asn Lys Gly Lys Leu Thr
1 5 10
<210> 48
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 48
Gln Glu Asn Gly Lys Thr Ser Phe Glu Pro Ala Asp Leu Lys
1 5 10
<210> 49
<211> 429
<212> ДНК
<213> Staphylococcus aureus
<400> 49
atgatattga acttcaatca attcgagaat caaaactttt ttaacggtaa tccaagtgat 60
acatttaaag atttaggtaa acaagtattt aattactttt caacaccttc atttgtaacg 120
aatatatatg aaacagacga attatattac ttagaagctg aactagcagg tgtaaataaa 180
gaagatatta gtatcgattt caataataat acgctcacta ttcaagctac tagaagcgca 240
aaatacaaat ctgaacaact cattttagat gagcgtaact tcgaatcatt aatgcgtcaa 300
tttgattttg aagctgttga taagcaacat attactgcta gttttgaaaa tgggttatta 360
accattacct tgcctaaaat caaaccaagc aatgaaacta cttcatcaac atctattcca 420
atttcatag 429
<210> 50
<211> 93
<212> PRT
<213> Staphylococcus aureus
<400> 50
Met Ile Leu Asn Phe Asn Gln Phe Glu Asn Gln Asn Phe Phe Asn Gly
1 5 10 15
Asn Pro Ser Asp Thr Phe Lys Asp Leu Gly Lys Gln Val Phe Asn Tyr
20 25 30
Phe Ser Thr Pro Ser Phe Val Thr Asn Ile Tyr Glu Thr Asp Glu Leu
35 40 45
Tyr Tyr Leu Glu Ala Glu Leu Ala Gly Val Asn Lys Glu Asp Ile Ser
50 55 60
Ile Asp Phe Asn Asn Asn Thr Leu Thr Ile Gln Ala Thr Arg Ser Ala
65 70 75 80
Lys Tyr Lys Ser Glu Gln Leu Ile Leu Asp Glu Arg Asn
85 90
<210> 51
<211> 100
<212> PRT
<213> Staphylococcus aureus
<400> 51
Met His His His His His His Met Ile Leu Asn Phe Asn Gln Phe Glu
1 5 10 15
Asn Gln Asn Phe Phe Asn Gly Asn Pro Ser Asp Thr Phe Lys Asp Leu
20 25 30
Gly Lys Gln Val Phe Asn Tyr Phe Ser Thr Pro Ser Phe Val Thr Asn
35 40 45
Ile Tyr Glu Thr Asp Glu Leu Tyr Tyr Leu Glu Ala Glu Leu Ala Gly
50 55 60
Val Asn Lys Glu Asp Ile Ser Ile Asp Phe Asn Asn Asn Thr Leu Thr
65 70 75 80
Ile Gln Ala Thr Arg Ser Ala Lys Tyr Lys Ser Glu Gln Leu Ile Leu
85 90 95
Asp Glu Arg Asn
100
<210> 52
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 52
Asn Gly Asn Pro Ser Asp Thr Phe Lys Asp Leu Gly Lys Gln
1 5 10
<210> 53
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 53
Glu Asp Ile Ser Ile Asp Phe Asn Asn Asn Thr Leu Thr Ile
1 5 10
<210> 54
<211> 807
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 54
ctagaatcta gagagcaatt atcagtcgaa gaatacgaaa cattctttaa cagatttgat 60
aatcaagaat ttgatttcga acgtgaattg acacaagatc catattcaaa agtatactta 120
tacagtatag aagaccatat cagaacatat aagatagaga aaggaggtgg cggttcagga 180
ggtggaggat ctggaggcgg tggatcaacg caagttaaag atacaaatat ttttccatat 240
aatggcgtcg tttcatttaa agatgcgaca ggttttgtaa ttggaaaaaa tacaattatc 300
accaataaac atgtatcaaa agattataaa gttggcgata gaattactgc ccatccaaac 360
ggtgacaaag gaaatggtgg tatatataaa attaaaagca tttctgatta tccgggtgat 420
gaagacatct ctgtcatgaa tattgaagaa caagcagtcg aacgtggacc aaaaggcttt 480
aattttaatg aaaatgtcca agcattcaat tttgcgaaag atgctaaagt tgatgacaaa 540
attaaagtta ttggttaccc attacctgct caaaatagtt ttaaacagtt tgaatctaca 600
ggaactataa aaagaatcaa agacaatatt ttaaattttg atgcatacat tgaacccggg 660
aattcaggat caccagttct aaattctaac aatgaggtca taggtgtggt gtatggcggt 720
attggaaaaa ttggttctga atataatggt gccgtatact ttacgcctca aatcaaagat 780
tttattcaaa agcacattga acaataa 807
<210> 55
<211> 268
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 55
Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe Phe
1 5 10 15
Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr Gln
20 25 30
Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile Arg
35 40 45
Thr Tyr Lys Ile Glu Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
50 55 60
Gly Gly Gly Gly Ser Thr Gln Val Lys Asp Thr Asn Ile Phe Pro Tyr
65 70 75 80
Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val Ile Gly Lys
85 90 95
Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr Lys Val Gly
100 105 110
Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn Gly Gly Ile
115 120 125
Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu Asp Ile Ser
130 135 140
Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro Lys Gly Phe
145 150 155 160
Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys Asp Ala Lys
165 170 175
Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro Ala Gln Asn
180 185 190
Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg Ile Lys Asp
195 200 205
Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn Ser Gly Ser
210 215 220
Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val Tyr Gly Gly
225 230 235 240
Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr Phe Thr Pro
245 250 255
Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
260 265
<210> 56
<211> 825
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 56
atgcaccacc accaccacca cctggaatcc cgtgaacaac tgtccgtcga agaatacgaa 60
accttcttta accgctttga taaccaagaa tttgatttcg aacgtgaact gacccaggat 120
ccgtattcta aagtgtatct gtacagtatc gaagatcata ttcgcacgta caaaatcgaa 180
aaaggcggtg gcggttctgg cggtggcggt agtggcggtg gcggtagcac ccaggtgaaa 240
gatacgaata tctttccgta taacggcgtg gtttctttta aagatgcgac cggcttcgtt 300
atcggtaaaa acaccatcat cacgaacaaa catgtgagca aagattacaa agttggcgat 360
cgtattaccg cccacccgaa tggcgataag ggtaacggcg gtatctacaa aatcaaaagc 420
atctctgatt acccgggtga tgaagatatc agcgtgatga atattgaaga acaggcagtt 480
gaacgcggcc cgaaaggttt taacttcaat gaaaacgttc aggcgtttaa tttcgcgaaa 540
gatgccaaag tggatgataa aatcaaagtt attggctatc cgctgccggc ccagaacagc 600
tttaaacagt tcgaatctac cggtacgatc aaacgtatca aagataacat cctgaacttc 660
gatgcatata ttgaaccggg caatagtggt agcccggtgc tgaacagtaa caatgaagtt 720
attggtgtgg tttatggcgg tatcggcaaa attggtagcg aatacaacgg tgctgtgtat 780
tttacgccgc agatcaaaga cttcatccag aaacatatcg aacaa 825
<210> 57
<211> 275
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 57
Met His His His His His His Leu Glu Ser Arg Glu Gln Leu Ser Val
1 5 10 15
Glu Glu Tyr Glu Thr Phe Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp
20 25 30
Phe Glu Arg Glu Leu Thr Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr
35 40 45
Ser Ile Glu Asp His Ile Arg Thr Tyr Lys Ile Glu Lys Gly Gly Gly
50 55 60
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Gln Val Lys
65 70 75 80
Asp Thr Asn Ile Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala
85 90 95
Thr Gly Phe Val Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val
100 105 110
Ser Lys Asp Tyr Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly
115 120 125
Asp Lys Gly Asn Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr
130 135 140
Pro Gly Asp Glu Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val
145 150 155 160
Glu Arg Gly Pro Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe
165 170 175
Asn Phe Ala Lys Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly
180 185 190
Tyr Pro Leu Pro Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly
195 200 205
Thr Ile Lys Arg Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile
210 215 220
Glu Pro Gly Asn Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val
225 230 235 240
Ile Gly Val Val Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn
245 250 255
Gly Ala Val Tyr Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His
260 265 270
Ile Glu Gln
275
<210> 58
<211> 280
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 58
Met Arg Gly Ser His His His His His His Gly Ser Leu Glu Ser Arg
1 5 10 15
Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe Phe Asn Arg Phe Asp
20 25 30
Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr Gln Asp Pro Tyr Ser
35 40 45
Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile Arg Thr Tyr Lys Ile
50 55 60
Glu Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
65 70 75 80
Ser Thr Gln Val Lys Asp Thr Asn Ile Phe Pro Tyr Asn Gly Val Val
85 90 95
Ser Phe Lys Asp Ala Thr Gly Phe Val Ile Gly Lys Asn Thr Ile Ile
100 105 110
Thr Asn Lys His Val Ser Lys Asp Tyr Lys Val Gly Asp Arg Ile Thr
115 120 125
Ala His Pro Asn Gly Asp Lys Gly Asn Gly Gly Ile Tyr Lys Ile Lys
130 135 140
Ser Ile Ser Asp Tyr Pro Gly Asp Glu Asp Ile Ser Val Met Asn Ile
145 150 155 160
Glu Glu Gln Ala Val Glu Arg Gly Pro Lys Gly Phe Asn Phe Asn Glu
165 170 175
Asn Val Gln Ala Phe Asn Phe Ala Lys Asp Ala Lys Val Asp Asp Lys
180 185 190
Ile Lys Val Ile Gly Tyr Pro Leu Pro Ala Gln Asn Ser Phe Lys Gln
195 200 205
Phe Glu Ser Thr Gly Thr Ile Lys Arg Ile Lys Asp Asn Ile Leu Asn
210 215 220
Phe Asp Ala Tyr Ile Glu Pro Gly Asn Ser Gly Ser Pro Val Leu Asn
225 230 235 240
Ser Asn Asn Glu Val Ile Gly Val Val Tyr Gly Gly Ile Gly Lys Ile
245 250 255
Gly Ser Glu Tyr Asn Gly Ala Val Tyr Phe Thr Pro Gln Ile Lys Asp
260 265 270
Phe Ile Gln Lys His Ile Glu Gln
275 280
<210> 59
<211> 45
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 59
ggaggtggcg gttcaggagg tggaggatct ggaggcggtg gatca 45
<210> 60
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 60
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 61
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 61
Glu Arg Lys Tyr Lys
1 5
<210> 62
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 62
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
1 5 10
<210> 63
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 63
Glu Ala Ala Ala Lys
1 5
<210> 64
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 64
Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
1 5 10 15
<210> 65
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 65
Glu Arg Lys Tyr Lys Glu Arg Lys Tyr Lys
1 5 10
<210> 66
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 66
Glu Arg Lys Tyr Lys Glu Arg Lys Tyr Lys Glu Arg Lys Tyr Lys
1 5 10 15
<210> 67
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 67
Gly Gly Gly Gly Ser
1 5
<210> 68
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 68
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 69
<211> 6
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(1)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (3)..(3)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (5)..(5)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 69
Xaa Pro Xaa Pro Xaa Pro
1 5
<210> 70
<211> 12
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(1)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (3)..(3)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (5)..(5)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (7)..(7)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (9)..(9)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (11)..(11)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 70
Xaa Pro Xaa Pro Xaa Pro Xaa Pro Xaa Pro Xaa Pro
1 5 10
<210> 71
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 71
atgttcaaaa aaaatgactc gaaaaattca attctattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
ccaagtgtac aagataaaca attccaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agccaaaata caataaacgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtataaat 300
ttagaaggaa catacagagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 72
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 72
atgttcaaaa aaaatgactc gaaaaattca attctattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
ccaagtgtac aagataaaca attccaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agccaaaata caataaacgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtataaat 300
ttagaaggaa catacagagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 73
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 73
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctggcaga gtgtatacac ctaagaggaa tattactctt 360
aataaagaag ttgtcacttt aaaagaattg gatcatatca taagatttgc tcatatttcc 420
tatggcttgt atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggtggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 74
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 74
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctggcaga gtgtatacac ctaagaggaa tattactctt 360
aataaagaag ttgtcacttt aaaagaattg gatcatatca taagatttgc tcatatttcc 420
tatggcttgt atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggtggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 75
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 75
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctggcaga gtgtatacac ctaagaggaa tattactctt 360
aataaagaag ttgtcacttt aaaagaattg gatcatatca taagatttgc tcatatttcc 420
tatggcttgt atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggtggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 76
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 76
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 77
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 77
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 78
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 78
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 79
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 79
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 80
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 80
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attacaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaacttta cgatagatac agcaaggata caataaatgg aaaatctaat 240
aaatctagga attgggttta ttcagagaga cctttaaatg aaaaccaagt tcgtatacat 300
ttagaaggaa catacacagt tgctgataga gtatatacac ctaagagaaa tattactctt 360
aataaagaag ttgtcacttt aaaggaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaaagat 480
ggcggtaaat atacattaga gtcgcataaa gagctacaaa aagataggga aaatgtaaaa 540
attaatacag ccgatataaa aaatgtaact ttcaaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 81
<211> 612
<212> ДНК
<213> Staphylococcus aureus
<400> 81
atgttcaaaa aatatgactc aaaaaattca atcgtattaa aatctattct atcgctaggt 60
atcatctatg ggggaacatt tggaatatat ccaaaagcag acgcgtcaac acaaaattcc 120
tcaagtgtac aagataaaca attccaaaaa gttgaagaag taccaaataa ttcagaaaaa 180
gctttggtta aaaaactgta cgatagatac agccaaaata caataaacgg aaaatctaat 240
aaagctagga attgggttta ttcagagaga cctttaaatg aaaatcaagt tcgcatacat 300
ttagaaggta catacagagt tgctgataga gtgtatacac ctaagaggaa cattactctt 360
aataaagaag ttgtcacttt aaaagaattg gatcatatca taagatttgc tcatatttct 420
tatggcttat atatgggaga acatttgcct aaaggtaaca tcgtcataaa tacaaagaat 480
ggcggtaaat atacattaga gtcgcacaaa gagttacaaa agaataggga aaatgtagaa 540
attaatactg atgatataaa aaatgtaact ttcgaacttg tgaaaagtgt taatgacatt 600
gaacaagttt ga 612
<210> 82
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 82
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 83
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 83
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 84
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 84
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Gly Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 85
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 85
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 86
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 86
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 87
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 87
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 88
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 88
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 89
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 89
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Leu
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Lys Asp Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Thr Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 90
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 90
Met Phe Lys Lys Asn Asp Ser Lys Asn Ser Ile Leu Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Pro Ser Val Gln Asp Lys Gln Phe
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Gln Asn Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ser Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile Asn Leu Glu Gly Thr Tyr Arg Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asp
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asp Arg
165 170 175
Glu Asn Val Lys Ile Asn Thr Ala Asp Ile Lys Asn Val Thr Phe Lys
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 91
<211> 203
<212> PRT
<213> Staphylococcus aureus
<400> 91
Met Phe Lys Lys Tyr Asp Ser Lys Asn Ser Ile Val Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Ser Ser Val Gln Asp Lys Gln Phe
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Gln Asn Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Ala Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile His Leu Glu Gly Thr Tyr Arg Val Ala Asp Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Asn
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Asn Arg
165 170 175
Glu Asn Val Glu Ile Asn Thr Asp Asp Ile Lys Asn Val Thr Phe Glu
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Glu Gln Val
195 200
<210> 92
<211> 203
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (5)..(5)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (12)..(12)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (41)..(41)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (48)..(48)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (72)..(73)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (82)..(82)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (100)..(100)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (106)..(106)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (109)..(109)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (160)..(160)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (175)..(175)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (180)..(180)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (184)..(184)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (192)..(192)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (201)..(203)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 92
Met Phe Lys Lys Xaa Asp Ser Lys Asn Ser Ile Xaa Leu Lys Ser Ile
1 5 10 15
Leu Ser Leu Gly Ile Ile Tyr Gly Gly Thr Phe Gly Ile Tyr Pro Lys
20 25 30
Ala Asp Ala Ser Thr Gln Asn Ser Xaa Ser Val Gln Asp Lys Gln Xaa
35 40 45
Gln Lys Val Glu Glu Val Pro Asn Asn Ser Glu Lys Ala Leu Val Lys
50 55 60
Lys Leu Tyr Asp Arg Tyr Ser Xaa Xaa Thr Ile Asn Gly Lys Ser Asn
65 70 75 80
Lys Xaa Arg Asn Trp Val Tyr Ser Glu Arg Pro Leu Asn Glu Asn Gln
85 90 95
Val Arg Ile Xaa Leu Glu Gly Thr Tyr Xaa Val Ala Xaa Arg Val Tyr
100 105 110
Thr Pro Lys Arg Asn Ile Thr Leu Asn Lys Glu Val Val Thr Leu Lys
115 120 125
Glu Leu Asp His Ile Ile Arg Phe Ala His Ile Ser Tyr Gly Leu Tyr
130 135 140
Met Gly Glu His Leu Pro Lys Gly Asn Ile Val Ile Asn Thr Lys Xaa
145 150 155 160
Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln Lys Xaa Arg
165 170 175
Glu Asn Val Xaa Ile Asn Thr Xaa Asp Ile Lys Asn Val Thr Phe Xaa
180 185 190
Leu Val Lys Ser Val Asn Asp Ile Xaa Xaa Xaa
195 200
<210> 93
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 93
Gln Asn Thr Ile Asn Gly Lys Ser Asn Lys Ser Arg Asn Trp
1 5 10
<210> 94
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 94
Gln Asn Thr Ile Asn Gly Lys Ser Asn Lys Ala Arg Asn Trp
1 5 10
<210> 95
<211> 15
<212> PRT
<213> Staphylococcus aureus
<400> 95
Lys Asn Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln
1 5 10 15
<210> 96
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(2)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (11)..(11)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 96
Xaa Xaa Thr Ile Asn Gly Lys Ser Asn Lys Xaa Arg Asn Trp
1 5 10
<210> 97
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (2)..(2)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 97
Lys Xaa Gly Gly Lys Tyr Thr Leu Glu Ser His Lys Glu Leu Gln
1 5 10 15
<210> 98
<211> 1755
<212> ДНК
<213> Staphylococcus aureus
<400> 98
atgacagagt catatccaat tattaaggaa ggctcacaag tcggaagcta ctttctattt 60
ttcatcataa ttgcattttt gttatatgcc aatgtgttat ttattaaacg acgaagttat 120
gagcttgcat tatatcaaac attaggttta tctaaattca acattattta tatactaatg 180
ctcgaacaat tactaatatt tataattacg gcaatattag gtattattat tggtattttt 240
ggttcaaaac tgttattaat gattgtcttt acattattag gaattaaaga aaaggttcca 300
attattttta gtttgagggc ggtatttgaa acattaatgt taatcggtgt cgcttatttt 360
ttaacctctg ctcaaaattt tatattagtg ttcaaacaat ctatttcaca gatgtcaaag 420
aataaccagg ttaaagaaac aaatcataat aaaattacat ttgaagaggt tgttttaggc 480
atcttaggta tagtattgat tatcacagga tactatctat ctttgaacat tgttcaatat 540
tatgattcta tcggtatact tatgtttatt ttattgtcaa ctgtgattgg ggcatactta 600
ttttttaaaa gctctgtttc tctagttttt aaaatggtga agaagtttag aaaaggtgtt 660
ataagtgtaa atgatgtcat gttctcatca tctattatgt atcgtattaa gaaaaatgct 720
ttttcactta cggtcatggc aatcatttca gcgattactg tttcagttct ttgctttgct 780
gctataagta gagcgtcctt atcaagtgaa ataaaatata ctgcaccaca cgacgttaca 840
attaaagacc aacaaaaagc taatcaatta gcaagtgaat taaacaatca aaaaattcct 900
catttttata attataaaga agtaattcat acgaaattgt ataaagataa tttatttgat 960
gtaaaagcga aagaaccata caatgtaaca attactagtg ataaatatat ccctaatact 1020
gatttgaaac gtggacaagc tgatttgttt gtagcggaag gttctatcaa agatttagtg 1080
aaacataaga agcatggtaa ggcaattata ggaacgaaaa aacatcatgt taatattaag 1140
ttacggaaag atattaataa aatctatttt atgacagatg ttgatttagg tggaccaacg 1200
tttgtcttaa atgacaaaga ctatcaagaa ataagaaagt atacaaaagc aaagcatatc 1260
gtctctcaat ttggattcga tttgaaacat aaaaaagatg ctttagcatt agaaaaagtg 1320
aaaaataaag ttgataaatc tattaaaaca agaagtgaag cgataagctc aatatcaagt 1380
ttaaccggaa tattattatt tgtaacatca tttttaggta ttacattctt gattgctgta 1440
tgttgcatta tatacattaa gcaaatagat gaaaccgaag atgagttaga gaattatagt 1500
atattgagaa agcttggatt tacacaaaaa gatatggcaa ggggactaaa gtttaaaatt 1560
atgtttaatt ttgggttacc tttagttatt gcactatcac atgcatattt tacatcatta 1620
gcatatatga aattaatggg tacaacgaat caaataccgg ttttcatagt aatgggatta 1680
tacatttgta tgtatgctgt ttttgcagtg acggcttata atcattccaa gcgaacaatt 1740
agacattcca tataa 1755
<210> 99
<211> 1755
<212> ДНК
<213> Staphylococcus aureus
<400> 99
atgacagagt catatccaat tattaaggaa ggctcacaag tcggaagcta ctttctattt 60
ttcatcataa ttgcattttt gttatatgcc aatgtgttat ttattaaacg acgaagttat 120
gagcttgcat tatatcaaac attaggttta tctaaattca acattattta tatactaatg 180
ctcgaacaat tactaatatt tataattacg gcaatattag gtattattat tggtattttt 240
ggttcaaaac tgttattaat gattgtcttt acattattag gaattaaaga aaaggttcca 300
attattttta gtttgagggc ggtatttgaa acattaatgt taatcggtgt cgcttatttt 360
ttaacctctg ctcaaaattt tatattagtg ttcaaacaat ctatttcaca gatgtcaaag 420
aataaccagg ttaaagaaac aaatcataat aaaattacat ttgaagaggt tgttttaggc 480
atcttaggta tagtattgat tatcacagga tactatctat ctttgaacat tgttcaatat 540
tatgattcta tcggtatact tatgtttatt ttattgtcaa ctgtgattgg ggcatactta 600
ttttttaaaa gctctgtttc tctagttttt aaaatggtga agaagtttag aaaaggtgtt 660
ataagtgtaa atgatgtcat gttctcatca tctattatgt atcgtattaa gaaaaatgct 720
ttttcactta cggtcatggc aatcatttca gcgattactg tttcagttct ttgctttgct 780
gctataagta gagcgtcctt atcaagtgaa ataaaatata ctgcaccaca cgacgttaca 840
attaaagacc aacaaaaagc taatcaatta gcaagtgaat taaacaatca aaaaattcct 900
catttttata attataaaga agtaattcat acgaaattgt ataaagataa tttatttgat 960
gtaaaagcga aagaaccata caatgtaaca attactagtg ataaatatat ccctaatact 1020
gatttgaaac gtggacaagc tgatttgttt gtagcggaag gttctatcaa agatttagtg 1080
aaacataaga agcatggtaa ggcaattata ggaacgaaaa aacatcatgt taatattaag 1140
ttacggaaag atattaataa aatctatttt atgacagatg ttgatttagg tggaccaacg 1200
tttgtcttaa atgacaaaga ctatcaagaa ataagaaagt atacaaaagc aaagcatatc 1260
gtctctcaat ttggattcga tttgaaacat aaaaaagatg ctttagcatt agaaaaagtg 1320
aaaaataaag ttgataaatc tattaaaaca agaagtgaag cgataagctc aatatcaagt 1380
ttaaccggaa tattattatt tgtaacatca tttttaggta ttacattctt gattgctgta 1440
tgttgcatta tatacattaa gcaaatagat gaaaccgaag atgagttaga gaattatagt 1500
atattgagaa agcttggatt tacacaaaaa gatatggcaa ggggactaaa gtttaaaatt 1560
atgtttaatt ttgggttacc tttagttatt gtactatcac atgcatattt tacatcatta 1620
gcatatatga aattaatggg tacaacgaat caaataccgg ttttcatagt aatgggatta 1680
tacatttgta tgtatgctgt ttttgcagtg acggcttata atcattccaa gcgaacaatt 1740
agacattcca tataa 1755
<210> 100
<211> 1755
<212> ДНК
<213> Staphylococcus aureus
<400> 100
atgacagagt catatccaat tattaaggaa ggctcacaag tcggaagcta ctttctattt 60
ttcatcataa ttgcattttt gttatatgcc aatgtgttat ttattaaacg acgaagttat 120
gagcttgcat tatatcaaac attaggttta tctaaattca acattattta tatactaatg 180
ctcgaacaat tactaatatt tataattacg gcaatattag gtattattat tggtattttt 240
ggttcaaaac tgttattaat gattgtcttt acattattag gaattaaaga aaaggttcca 300
attattttta gtttgagggc ggtatttgaa acattaatgt taatcggtgt cgcttatttt 360
ttaacctctg ctcaaaattt tatattagtg ttcaaacaat ctatttcaca gatgtcaaag 420
aataaccagg ttaaagaaac aaatcataat aaaattacat ttgaagaggt tgttttaggc 480
atcttaggta tagtattgat taccacagga tactatctat ctttgaacat tgttcaatat 540
tatgattcta tcggtatact tatgtttatt ttattgtcaa ctgtgattgg ggcatactta 600
ttttttaaaa gctctgtttc tctagttttt aaaatggtga agaagtttag aaaaggtgtt 660
ataagtgtaa atgatgtcat gttctcatca tctattatgt atcgtattaa gaaaaatgct 720
ttttcactta cggtcatggc aatcatttca gcgattactg tttcagttct ttgctttgct 780
gctataagta gagcgtcctt atcaagtgaa ataaaatata ctgcaccaca cgacgttaca 840
attaaagacc aacaaaaagc taatcaatta gcaagtgaat taaacaatca aaaaattcct 900
catttttata attataaaga agtaattcat acgaaattgt ataaagataa tttatttgat 960
gtaaaagcga aagaaccata caatgtaaca attactagtg ataaatatat ccctaatact 1020
gatttgaaac gtggacaagc tgatttgttt gtagcggaag gttctatcaa agatttagtg 1080
aaacataaga agcatggtaa ggcaattata ggaacgaaaa aacatcatgt taatattaag 1140
ttacggaaag atattaataa aatctatttt atgacagatg ttgatttagg tggaccaacg 1200
tttgtcttaa atgacaaaga ctatcaagaa ataagaaagt atacaaaagc aaagcatatc 1260
gtctctcaat ttggattcga tttgaaacat aaaaaagatg ctttagcatt agaaaaagtg 1320
aaaaataaag ttgataaatc tattaaaaca agaagtgaag cgataagctc aatatcaagt 1380
ttaaccggaa tattattatt tgtaacatca tttttaggta ttacattctt gattgctgta 1440
tgttgcatta tatacattaa gcaaatagat gaaaccgaag atgagttaga gaattatagt 1500
atattgagaa agcttggatt tacacaaaaa gatatggcaa ggggactaaa gtttaaaatt 1560
atgtttaatt ttgggttacc tttagttatt gcactatcac atgcatattt tacatcatta 1620
gcatatatga aattaatggg tacaacgaat caaataccgg ttttcatagt aatgggatta 1680
tacatttgta tgtatgctgt ttttgcagtg acggcttata atcattccaa gcgaacaatt 1740
agacattcca tataa 1755
<210> 101
<211> 1840
<212> ДНК
<213> Staphylococcus aureus
<400> 101
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca gctttgtagc 60
attaaaatac gcgcataaac taaacatgac agagtcatat ccaattatta aggaaggctc 120
acaagtcgga agctactttc tatttttcat cataattgca tttttgttat atgccaatgt 180
gttatttatt aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa 240
attcaacatt atttatatac taatgctcga acaattacta atatttataa ttacggcaat 300
attaggtatt attattggta tttttggttc aaaactgtta ttaatgattg tctttacatt 360
attaggaatt aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt 420
aatgttaatc ggtgtcgctt attttttaac ctctgctcaa aattttatat tagtgttcaa 480
acaatctatt tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat 540
tacatttgaa gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta 600
tctatctttg aacattgttc aatattatga ttctatcggt atacttatgt ttattttatt 660
gtcaactgtg attggggcat acttattttt taaaagctct gtttctctag tttttaaaat 720
ggtgaagaag tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat 780
tatgtatcgt attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat 840
tactgtttca gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa 900
atatactgca ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag 960
tgaattaaac aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa 1020
attgtataaa gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac 1080
tagtgataaa tatatcccta atactgattt gaaacgtgga caagctgatt tgtttgtagc 1140
ggaaggttct atcaaagatt tagtgaaaca taagaagcat ggtaaggcaa ttataggaac 1200
gaaaaaacat catgttaata ttaagttacg gaaagatatt aataaaatct attttatgac 1260
agatgttgat ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag 1320
aaagtataca aaagcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa 1380
agatgcttta gcattagaaa aagtgaaaaa taaagttgat aaatctatta aaacaagaag 1440
tgaagcgata agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt 1500
aggtattaca ttcttgattg ctgtatgttg cattatatac attaagcaaa tagatgaaac 1560
cgaagatgag ttagagaatt atagtatatt gagaaagctt ggatttacac aaaaagatat 1620
ggcaagggga ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact 1680
atcacatgca tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat 1740
accggttttc atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc 1800
ttataatcat tccaagcgaa caattagaca ttccatataa 1840
<210> 102
<211> 1890
<212> ДНК
<213> Staphylococcus aureus
<400> 102
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cattaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gcgcataaac taaacatgac agagtcatat ccaattatta aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc aaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac ctctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt atacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac tagtgataaa 1140
tatatcccta atactgattt gaaacgtgga caagctgatt tgtttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaggcaa ttataggaac gaaaaaacat 1260
catgttaata ttaagttacg gaaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaagcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagtgaaaaa taaagttgat aaatctatta aaacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac attaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatatt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa caattagaca ttccatataa 1890
<210> 103
<211> 1890
<212> ДНК
<213> Staphylococcus aureus
<400> 103
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cattaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gcgcataaac taaacatgac agagtcatat ccaattatta aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc aaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac ctctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt atacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac tagtgataaa 1140
tatatcccta atactgattt gaaacgtgga caagctgatt tgtttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaagcag ttataggaac gaaaaaacat 1260
catgttaata ttaagttgcg gaaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaagcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagtgaaaaa taaagttgat aaatctatta aaacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac ataaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatttt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa caattagaca ttccatataa 1890
<210> 104
<211> 1890
<212> ДНК
<213> Staphylococcus aureus
<400> 104
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cattaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gcgcataaac taaacatgac agagtcatat ccaattatta aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc aaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac ctctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt atacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac tagtgataaa 1140
tatatcccta atactgattt gaaacgtgga caagctgatt tgtttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaagcag ttataggaac gaaaaaacat 1260
catgttaata ttaagttgcg gaaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaagcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagtgaaaaa taaagttgat aaatctatta aaacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac ataaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatttt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa caattagaca ttccatataa 1890
<210> 105
<211> 1890
<212> ДНК
<213> Staphylococcus aureus
<400> 105
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cgttaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gctcataaac taaacatgac agagtcatat ccaattataa aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc gaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac atctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt acacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa agcgaaagaa ccatacaatg taacaattac tagtgataaa 1140
tacatcccta atactgattt gaaacgtggg caagctgatt tatttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaggcaa ttataggaac gaaaaaacat 1260
catgttaata ttaagttacg taaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaggcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagcgaaaaa taaagttgat aaatctattg aaacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac ataaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatttt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa caattagaca ttccatataa 1890
<210> 106
<211> 1800
<212> ДНК
<213> Staphylococcus aureus
<400> 106
ttgtatttta gctttgtagc attaaaatac gctcataaac taaacatgac agagtcatat 60
ccaattataa aggaaggctc acaagtcgga agctactttc tatttttcat cataattgca 120
tttttgttat atgccaatgt gttatttatt aaacgacgaa gttatgagct tgcattatat 180
caaacattag gtttatctaa attcaacatt atttatatac taatgctcga acaattacta 240
atatttataa ttacggcaat attaggtatt attattggta tttttggttc gaaactgtta 300
ttaatgattg tctttacatt attaggaatt aaagaaaagg ttccaattat ttttagtttg 360
agggcggtat ttgaaacatt aatgttaatc ggtgtcgctt attttttaac atctgctcaa 420
aattttatat tagtgttcaa acaatctatt tcacagatgt caaagaataa ccaggttaaa 480
gaaacaaatc ataataaaat tacatttgaa gaggttgttt taggcatctt aggtatagta 540
ttgattacca caggatacta tctatctttg aacattgttc aatattatga ttctatcggt 600
acacttatgt ttattttatt gtcaactgtg attggggcat acttattttt taaaagctct 660
gtttctctag tttttaaaat ggtgaagaag tttagaaaag gtgttataag tgtaaatgat 720
gtcatgttct catcatctat tatgtatcgt attaagaaaa atgctttttc acttacggtc 780
atggcaatca tttcagcgat tactgtttca gttctttgct ttgctgctat aagtagagcg 840
tccttatcaa gtgaaataaa atatactgca ccacacgacg ttacaattaa agaccaacaa 900
aaagctaatc aattagcaag tgaattaaac aatcaaaaaa ttcctcattt ttataattat 960
aaagaagtaa ttcatacgaa attgtataaa gataatttat ttgatgtaaa agcgaaagaa 1020
ccatacaatg taacaattac tagtgataaa tacatcccta atactgattt gaaacgtggg 1080
caagctgatt tatttgtagc ggaaggttct atcaaagatt tagtgaaaca taagaagcat 1140
ggtaaggcaa ttataggaac gaaaaaacat catgttaata ttaagttacg taaagatatt 1200
aataaaatct attttatgac agatgttgat ttaggtggac caacgtttgt cttaaatgac 1260
aaagactatc aagaaataag aaagtataca aaggcaaagc atatcgtctc tcaatttgga 1320
ttcgatttga aacataaaaa agatgcttta gcattagaaa aagcgaaaaa taaagttgat 1380
aaatctattg aaacaagaag tgaagcgata agctcaatat caagtttaac cggaatatta 1440
ttatttgtaa catcattttt aggtattaca ttcttgattg ctgtatgttg cattatatac 1500
ataaagcaaa tagatgaaac cgaagatgag ttagagaatt atagtatttt gagaaagctt 1560
ggatttacac aaaaagatat ggcaagggga ctaaagttta aaattatgtt taattttggg 1620
ttacctttag ttattgcact atcacatgca tattttacat cattagcata tatgaaatta 1680
atgggtacaa cgaatcaaat accggttttc atagtaatgg gattatacat ttgtatgtat 1740
gctgtttttg cagtgacggc ttataatcat tccaagcgaa caattagaca ttccatataa 1800
<210> 107
<211> 1755
<212> ДНК
<213> Staphylococcus aureus
<400> 107
atgacagagt catatccaat tataaaggaa ggctcacaag tcggaagcta ctttctattt 60
ttcatcataa ttgcattttt gttatatgcc aatgtgttat ttattaaacg acgaagttat 120
gagcttgcat tatatcaaac attaggttta tctaaattca acattattta tatactaatg 180
ctcgaacaat tactaatatt tataattacg gcaatattag gtattattat tggtattttt 240
ggttcgaaac tgttattaat gattgtcttt acattattag gaattaaaga aaaggttcca 300
attattttta gtttgagggc ggtatttgaa acattaatgt taatcggtgt cgcttatttt 360
ttaacatctg ctcaaaattt tatattagtg ttcaaacaat ctatttcaca gatgtcaaag 420
aataaccagg ttaaagaaac aaatcataat aaaattacat ttgaagaggt tgttttaggc 480
atcttaggta tagtattgat taccacagga tactatctat ctttgaacat tgttcaatat 540
tatgattcta tcggtacact tatgtttatt ttattgtcaa ctgtgattgg ggcatactta 600
ttttttaaaa gctctgtttc tctagttttt aaaatggtga agaagtttag aaaaggtgtt 660
ataagtgtaa atgatgtcat gttctcatca tctattatgt atcgtattaa gaaaaatgct 720
ttttcactta cggtcatggc aatcatttca gcgattactg tttcagttct ttgctttgct 780
gctataagta gagcgtcctt atcaagtgaa ataaaatata ctgcaccaca cgacgttaca 840
attaaagacc aacaaaaagc taatcaatta gcaagtgaat taaacaatca aaaaattcct 900
catttttata attataaaga agtaattcat acgaaattgt ataaagataa tttatttgat 960
gtaaaagcga aagaaccata caatgtaaca attactagtg ataaatacat ccctaatact 1020
gatttgaaac gtgggcaagc tgatttattt gtagcggaag gttctatcaa agatttagtg 1080
aaacataaga agcatggtaa ggcaattata ggaacgaaaa aacatcatgt taatattaag 1140
ttacgtaaag atattaataa aatctatttt atgacagatg ttgatttagg tggaccaacg 1200
tttgtcttaa atgacaaaga ctatcaagaa ataagaaagt atacaaaggc aaagcatatc 1260
gtctctcaat ttggattcga tttgaaacat aaaaaagatg ctttagcatt agaaaaagcg 1320
aaaaataaag ttgataaatc tattgaaaca agaagtgaag cgataagctc aatatcaagt 1380
ttaaccggaa tattattatt tgtaacatca tttttaggta ttacattctt gattgctgta 1440
tgttgcatta tatacataaa gcaaatagat gaaaccgaag atgagttaga gaattatagt 1500
attttgagaa agcttggatt tacacaaaaa gatatggcaa ggggactaaa gtttaaaatt 1560
atgtttaatt ttgggttacc tttagttatt gcactatcac atgcatattt tacatcatta 1620
gcatatatga aattaatggg tacaacgaat caaataccgg ttttcatagt aatgggatta 1680
tacatttgta tgtatgctgt ttttgcagtg acggcttata atcattccaa gcgaacaatt 1740
agacattcca tataa 1755
<210> 108
<211> 1890
<212> ДНК
<213> Staphylococcus aureus
<400> 108
atgaccttta acgagataat atttaaaaat ttccgtcaaa atttatcaca ttatgccatc 60
tatctttttt cattaattac gagtgtagta ttgtatttta gctttgtagc attaaaatac 120
gctcataaac taaacatgac agagtcatat ccaattataa aggaaggctc acaagtcgga 180
agctactttc tatttttcat cataattgca tttttgttat atgccaatgt gttatttatt 240
aaacgacgaa gttatgagct tgcattatat caaacattag gtttatctaa attcaacatt 300
atttatatac taatgctcga acaattacta atatttataa ttacggcaat attaggtatt 360
attattggta tttttggttc gaaactgtta ttaatgattg tctttacatt attaggaatt 420
aaagaaaagg ttccaattat ttttagtttg agggcggtat ttgaaacatt aatgttaatc 480
ggtgtcgctt attttttaac atctgctcaa aattttatat tagtgttcaa acaatctatt 540
tcacagatgt caaagaataa ccaggttaaa gaaacaaatc ataataaaat tacatttgaa 600
gaggttgttt taggcatctt aggtatagta ttgattacca caggatacta tctatctttg 660
aacattgttc aatattatga ttctatcggt acacttatgt ttattttatt gtcaactgtg 720
attggggcat acttattttt taaaagctct gtttctctag tttttaaaat ggtgaagaag 780
tttagaaaag gtgttataag tgtaaatgat gtcatgttct catcatctat tatgtatcgt 840
attaagaaaa atgctttttc acttacggtc atggcaatca tttcagcgat tactgtttca 900
gttctttgct ttgctgctat aagtagagcg tccttatcaa gtgaaataaa atatactgca 960
ccacacgacg ttacaattaa agaccaacaa aaagctaatc aattagcaag tgaattaaac 1020
aatcaaaaaa ttcctcattt ttataattat aaagaagtaa ttcatacgaa attgtataaa 1080
gataatttat ttgatgtaaa atcgaaacaa ccatacaatg taacaattac tagtgataaa 1140
tacatcccta gtactgattt gaaacgtggg caagctgatt tgtttgtagc ggaaggttct 1200
atcaaagatt tagtgaaaca taagaagcat ggtaaagcag ttataggaac gaaaaaacat 1260
catgttaata ttaagttacg taaagatatt aataaaatct attttatgac agatgttgat 1320
ttaggtggac caacgtttgt cttaaatgac aaagactatc aagaaataag aaagtataca 1380
aaggcaaagc atatcgtctc tcaatttgga ttcgatttga aacataaaaa agatgcttta 1440
gcattagaaa aagcgaaaaa taaagttgat aaatctattg agacaagaag tgaagcgata 1500
agctcaatat caagtttaac cggaatatta ttatttgtaa catcattttt aggtattaca 1560
ttcttgattg ctgtatgttg cattatatac attaagcaaa tagatgaaac cgaagatgag 1620
ttagagaatt atagtatatt gagaaagctt ggatttacac aaaaagatat ggcaagggga 1680
ctaaagttta aaattatgtt taattttggg ttacctttag ttattgcact atcacatgca 1740
tattttacat cattagcata tatgaaatta atgggtacaa cgaatcaaat accggttttc 1800
atagtaatgg gattatacat ttgtatgtat gctgtttttg cagtgacggc ttataatcat 1860
tccaagcgaa caattagaca ttccatataa 1890
<210> 109
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 109
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Thr Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Ala Lys Asn Lys Val Asp Lys Ser Ile Glu Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 110
<211> 599
<212> PRT
<213> Staphylococcus aureus
<400> 110
Met Tyr Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met
1 5 10 15
Thr Glu Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr
20 25 30
Phe Leu Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu
35 40 45
Phe Ile Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly
50 55 60
Leu Ser Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu
65 70 75 80
Ile Phe Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly
85 90 95
Ser Lys Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu
100 105 110
Lys Val Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met
115 120 125
Leu Ile Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu
130 135 140
Val Phe Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys
145 150 155 160
Glu Thr Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile
165 170 175
Leu Gly Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile
180 185 190
Val Gln Tyr Tyr Asp Ser Ile Gly Thr Leu Met Phe Ile Leu Leu Ser
195 200 205
Thr Val Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val
210 215 220
Phe Lys Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp
225 230 235 240
Val Met Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe
245 250 255
Ser Leu Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu
260 265 270
Cys Phe Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr
275 280 285
Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln
290 295 300
Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr
305 310 315 320
Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val
325 330 335
Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile
340 345 350
Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu
355 360 365
Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile
370 375 380
Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile
385 390 395 400
Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe
405 410 415
Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala
420 425 430
Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp
435 440 445
Ala Leu Ala Leu Glu Lys Ala Lys Asn Lys Val Asp Lys Ser Ile Glu
450 455 460
Thr Arg Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu
465 470 475 480
Leu Phe Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys
485 490 495
Cys Ile Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu
500 505 510
Asn Tyr Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala
515 520 525
Arg Gly Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val
530 535 540
Ile Ala Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu
545 550 555 560
Met Gly Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr
565 570 575
Ile Cys Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys
580 585 590
Arg Thr Ile Arg His Ser Ile
595
<210> 111
<211> 584
<212> PRT
<213> Staphylococcus aureus
<400> 111
Met Thr Glu Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser
1 5 10 15
Tyr Phe Leu Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val
20 25 30
Leu Phe Ile Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu
35 40 45
Gly Leu Ser Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu
50 55 60
Leu Ile Phe Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe
65 70 75 80
Gly Ser Lys Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys
85 90 95
Glu Lys Val Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu
100 105 110
Met Leu Ile Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile
115 120 125
Leu Val Phe Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val
130 135 140
Lys Glu Thr Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly
145 150 155 160
Ile Leu Gly Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn
165 170 175
Ile Val Gln Tyr Tyr Asp Ser Ile Gly Thr Leu Met Phe Ile Leu Leu
180 185 190
Ser Thr Val Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu
195 200 205
Val Phe Lys Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn
210 215 220
Asp Val Met Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala
225 230 235 240
Phe Ser Leu Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val
245 250 255
Leu Cys Phe Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys
260 265 270
Tyr Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn
275 280 285
Gln Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn
290 295 300
Tyr Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp
305 310 315 320
Val Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr
325 330 335
Ile Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala
340 345 350
Glu Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala
355 360 365
Ile Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp
370 375 380
Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr
385 390 395 400
Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys
405 410 415
Ala Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys
420 425 430
Asp Ala Leu Ala Leu Glu Lys Ala Lys Asn Lys Val Asp Lys Ser Ile
435 440 445
Glu Thr Arg Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile
450 455 460
Leu Leu Phe Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val
465 470 475 480
Cys Cys Ile Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu
485 490 495
Glu Asn Tyr Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met
500 505 510
Ala Arg Gly Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu
515 520 525
Val Ile Ala Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys
530 535 540
Leu Met Gly Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu
545 550 555 560
Tyr Ile Cys Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser
565 570 575
Lys Arg Thr Ile Arg His Ser Ile
580
<210> 112
<211> 584
<212> PRT
<213> Staphylococcus aureus
<400> 112
Met Thr Glu Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser
1 5 10 15
Tyr Phe Leu Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val
20 25 30
Leu Phe Ile Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu
35 40 45
Gly Leu Ser Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu
50 55 60
Leu Ile Phe Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe
65 70 75 80
Gly Ser Lys Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys
85 90 95
Glu Lys Val Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu
100 105 110
Met Leu Ile Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile
115 120 125
Leu Val Phe Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val
130 135 140
Lys Glu Thr Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly
145 150 155 160
Ile Leu Gly Ile Val Leu Ile Ile Thr Gly Tyr Tyr Leu Ser Leu Asn
165 170 175
Ile Val Gln Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu
180 185 190
Ser Thr Val Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu
195 200 205
Val Phe Lys Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn
210 215 220
Asp Val Met Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala
225 230 235 240
Phe Ser Leu Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val
245 250 255
Leu Cys Phe Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys
260 265 270
Tyr Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn
275 280 285
Gln Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn
290 295 300
Tyr Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp
305 310 315 320
Val Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr
325 330 335
Ile Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala
340 345 350
Glu Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala
355 360 365
Ile Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp
370 375 380
Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr
385 390 395 400
Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys
405 410 415
Ala Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys
420 425 430
Asp Ala Leu Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile
435 440 445
Lys Thr Arg Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile
450 455 460
Leu Leu Phe Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val
465 470 475 480
Cys Cys Ile Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu
485 490 495
Glu Asn Tyr Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met
500 505 510
Ala Arg Gly Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu
515 520 525
Val Ile Ala Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys
530 535 540
Leu Met Gly Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu
545 550 555 560
Tyr Ile Cys Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser
565 570 575
Lys Arg Thr Ile Arg His Ser Ile
580
<210> 113
<211> 584
<212> PRT
<213> Staphylococcus aureus
<400> 113
Met Thr Glu Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser
1 5 10 15
Tyr Phe Leu Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val
20 25 30
Leu Phe Ile Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu
35 40 45
Gly Leu Ser Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu
50 55 60
Leu Ile Phe Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe
65 70 75 80
Gly Ser Lys Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys
85 90 95
Glu Lys Val Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu
100 105 110
Met Leu Ile Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile
115 120 125
Leu Val Phe Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val
130 135 140
Lys Glu Thr Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly
145 150 155 160
Ile Leu Gly Ile Val Leu Ile Ile Thr Gly Tyr Tyr Leu Ser Leu Asn
165 170 175
Ile Val Gln Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu
180 185 190
Ser Thr Val Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu
195 200 205
Val Phe Lys Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn
210 215 220
Asp Val Met Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala
225 230 235 240
Phe Ser Leu Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val
245 250 255
Leu Cys Phe Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys
260 265 270
Tyr Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn
275 280 285
Gln Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn
290 295 300
Tyr Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp
305 310 315 320
Val Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr
325 330 335
Ile Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala
340 345 350
Glu Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala
355 360 365
Ile Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp
370 375 380
Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr
385 390 395 400
Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys
405 410 415
Ala Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys
420 425 430
Asp Ala Leu Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile
435 440 445
Lys Thr Arg Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile
450 455 460
Leu Leu Phe Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val
465 470 475 480
Cys Cys Ile Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu
485 490 495
Glu Asn Tyr Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met
500 505 510
Ala Arg Gly Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu
515 520 525
Val Ile Val Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys
530 535 540
Leu Met Gly Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu
545 550 555 560
Tyr Ile Cys Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser
565 570 575
Lys Arg Thr Ile Arg His Ser Ile
580
<210> 114
<211> 584
<212> PRT
<213> Staphylococcus aureus
<400> 114
Met Thr Glu Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser
1 5 10 15
Tyr Phe Leu Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val
20 25 30
Leu Phe Ile Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu
35 40 45
Gly Leu Ser Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu
50 55 60
Leu Ile Phe Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe
65 70 75 80
Gly Ser Lys Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys
85 90 95
Glu Lys Val Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu
100 105 110
Met Leu Ile Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile
115 120 125
Leu Val Phe Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val
130 135 140
Lys Glu Thr Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly
145 150 155 160
Ile Leu Gly Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn
165 170 175
Ile Val Gln Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu
180 185 190
Ser Thr Val Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu
195 200 205
Val Phe Lys Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn
210 215 220
Asp Val Met Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala
225 230 235 240
Phe Ser Leu Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val
245 250 255
Leu Cys Phe Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys
260 265 270
Tyr Thr Ala Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn
275 280 285
Gln Leu Ala Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn
290 295 300
Tyr Lys Glu Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp
305 310 315 320
Val Lys Ala Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr
325 330 335
Ile Pro Asn Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala
340 345 350
Glu Gly Ser Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala
355 360 365
Ile Ile Gly Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp
370 375 380
Ile Asn Lys Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr
385 390 395 400
Phe Val Leu Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys
405 410 415
Ala Lys His Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys
420 425 430
Asp Ala Leu Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile
435 440 445
Lys Thr Arg Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile
450 455 460
Leu Leu Phe Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val
465 470 475 480
Cys Cys Ile Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu
485 490 495
Glu Asn Tyr Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met
500 505 510
Ala Arg Gly Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu
515 520 525
Val Ile Ala Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys
530 535 540
Leu Met Gly Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu
545 550 555 560
Tyr Ile Cys Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser
565 570 575
Lys Arg Thr Ile Arg His Ser Ile
580
<210> 115
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 115
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile Lys Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 116
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 116
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Ile Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile Lys Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 117
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 117
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Val Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile Lys Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 118
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 118
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Ile Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ala
355 360 365
Lys Glu Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Asn
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Val Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Val Lys Asn Lys Val Asp Lys Ser Ile Lys Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 119
<211> 629
<212> PRT
<213> Staphylococcus aureus
<400> 119
Met Thr Phe Asn Glu Ile Ile Phe Lys Asn Phe Arg Gln Asn Leu Ser
1 5 10 15
His Tyr Ala Ile Tyr Leu Phe Ser Leu Ile Thr Ser Val Val Leu Tyr
20 25 30
Phe Ser Phe Val Ala Leu Lys Tyr Ala His Lys Leu Asn Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Thr Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Thr Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Ser
355 360 365
Lys Gln Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Ser
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Val Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Ala Lys Asn Lys Val Asp Lys Ser Ile Glu Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Ala
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 120
<211> 629
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(45)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (213)..(213)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (231)..(231)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (368)..(368)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (370)..(370)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (384)..(384)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (414)..(414)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (486)..(486)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (494)..(494)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (576)..(576)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 120
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
20 25 30
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Met Thr Glu
35 40 45
Ser Tyr Pro Ile Ile Lys Glu Gly Ser Gln Val Gly Ser Tyr Phe Leu
50 55 60
Phe Phe Ile Ile Ile Ala Phe Leu Leu Tyr Ala Asn Val Leu Phe Ile
65 70 75 80
Lys Arg Arg Ser Tyr Glu Leu Ala Leu Tyr Gln Thr Leu Gly Leu Ser
85 90 95
Lys Phe Asn Ile Ile Tyr Ile Leu Met Leu Glu Gln Leu Leu Ile Phe
100 105 110
Ile Ile Thr Ala Ile Leu Gly Ile Ile Ile Gly Ile Phe Gly Ser Lys
115 120 125
Leu Leu Leu Met Ile Val Phe Thr Leu Leu Gly Ile Lys Glu Lys Val
130 135 140
Pro Ile Ile Phe Ser Leu Arg Ala Val Phe Glu Thr Leu Met Leu Ile
145 150 155 160
Gly Val Ala Tyr Phe Leu Thr Ser Ala Gln Asn Phe Ile Leu Val Phe
165 170 175
Lys Gln Ser Ile Ser Gln Met Ser Lys Asn Asn Gln Val Lys Glu Thr
180 185 190
Asn His Asn Lys Ile Thr Phe Glu Glu Val Val Leu Gly Ile Leu Gly
195 200 205
Ile Val Leu Ile Xaa Thr Gly Tyr Tyr Leu Ser Leu Asn Ile Val Gln
210 215 220
Tyr Tyr Asp Ser Ile Gly Xaa Leu Met Phe Ile Leu Leu Ser Thr Val
225 230 235 240
Ile Gly Ala Tyr Leu Phe Phe Lys Ser Ser Val Ser Leu Val Phe Lys
245 250 255
Met Val Lys Lys Phe Arg Lys Gly Val Ile Ser Val Asn Asp Val Met
260 265 270
Phe Ser Ser Ser Ile Met Tyr Arg Ile Lys Lys Asn Ala Phe Ser Leu
275 280 285
Thr Val Met Ala Ile Ile Ser Ala Ile Thr Val Ser Val Leu Cys Phe
290 295 300
Ala Ala Ile Ser Arg Ala Ser Leu Ser Ser Glu Ile Lys Tyr Thr Ala
305 310 315 320
Pro His Asp Val Thr Ile Lys Asp Gln Gln Lys Ala Asn Gln Leu Ala
325 330 335
Ser Glu Leu Asn Asn Gln Lys Ile Pro His Phe Tyr Asn Tyr Lys Glu
340 345 350
Val Ile His Thr Lys Leu Tyr Lys Asp Asn Leu Phe Asp Val Lys Xaa
355 360 365
Lys Xaa Pro Tyr Asn Val Thr Ile Thr Ser Asp Lys Tyr Ile Pro Xaa
370 375 380
Thr Asp Leu Lys Arg Gly Gln Ala Asp Leu Phe Val Ala Glu Gly Ser
385 390 395 400
Ile Lys Asp Leu Val Lys His Lys Lys His Gly Lys Ala Xaa Ile Gly
405 410 415
Thr Lys Lys His His Val Asn Ile Lys Leu Arg Lys Asp Ile Asn Lys
420 425 430
Ile Tyr Phe Met Thr Asp Val Asp Leu Gly Gly Pro Thr Phe Val Leu
435 440 445
Asn Asp Lys Asp Tyr Gln Glu Ile Arg Lys Tyr Thr Lys Ala Lys His
450 455 460
Ile Val Ser Gln Phe Gly Phe Asp Leu Lys His Lys Lys Asp Ala Leu
465 470 475 480
Ala Leu Glu Lys Ala Xaa Asn Lys Val Asp Lys Ser Ile Xaa Thr Arg
485 490 495
Ser Glu Ala Ile Ser Ser Ile Ser Ser Leu Thr Gly Ile Leu Leu Phe
500 505 510
Val Thr Ser Phe Leu Gly Ile Thr Phe Leu Ile Ala Val Cys Cys Ile
515 520 525
Ile Tyr Ile Lys Gln Ile Asp Glu Thr Glu Asp Glu Leu Glu Asn Tyr
530 535 540
Ser Ile Leu Arg Lys Leu Gly Phe Thr Gln Lys Asp Met Ala Arg Gly
545 550 555 560
Leu Lys Phe Lys Ile Met Phe Asn Phe Gly Leu Pro Leu Val Ile Xaa
565 570 575
Leu Ser His Ala Tyr Phe Thr Ser Leu Ala Tyr Met Lys Leu Met Gly
580 585 590
Thr Thr Asn Gln Ile Pro Val Phe Ile Val Met Gly Leu Tyr Ile Cys
595 600 605
Met Tyr Ala Val Phe Ala Val Thr Ala Tyr Asn His Ser Lys Arg Thr
610 615 620
Ile Arg His Ser Ile
625
<210> 121
<211> 55
<212> PRT
<213> Staphylococcus aureus
<400> 121
Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe
1 5 10 15
Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr
20 25 30
Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile
35 40 45
Arg Thr Tyr Lys Ile Glu Lys
50 55
<210> 122
<211> 55
<212> PRT
<213> Staphylococcus aureus
<400> 122
Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe
1 5 10 15
Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr
20 25 30
Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His Ile
35 40 45
Arg Thr Tyr Lys Ile Glu Lys
50 55
<210> 123
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 123
Cys Leu Phe Ser Tyr Gly Ser Gly Ala Val Gly Glu Ile Phe Ser Gly
1 5 10 15
Ser Ile Val Lys Gly Tyr Asp Lys Ala Leu Asp Lys Glu Lys His Leu
20 25 30
Asn Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr
35 40 45
Phe Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu
50 55 60
Thr Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His
65 70 75 80
Ile Arg Thr Tyr Lys Ile Glu Lys
85
<210> 124
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 124
Cys Leu Phe Ser Tyr Gly Ser Gly Ala Val Gly Glu Ile Phe Ser Gly
1 5 10 15
Ser Ile Val Lys Gly Tyr Asp Lys Ala Leu Asp Lys Glu Lys His Leu
20 25 30
Asn Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr
35 40 45
Phe Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu
50 55 60
Thr Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His
65 70 75 80
Ile Arg Thr Tyr Lys Ile Glu Lys
85
<210> 125
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 125
Cys Leu Phe Ser Tyr Gly Ser Gly Ala Val Gly Glu Ile Phe Ser Gly
1 5 10 15
Ser Ile Val Lys Gly Tyr Asp Lys Ala Leu Asp Lys Glu Lys His Leu
20 25 30
Asn Met Leu Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr
35 40 45
Phe Phe Asn Arg Phe Asp Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu
50 55 60
Thr Gln Asp Pro Tyr Ser Lys Val Tyr Leu Tyr Ser Ile Glu Asp His
65 70 75 80
Ile Arg Thr Tyr Lys Ile Glu Lys
85
<210> 126
<211> 78
<212> PRT
<213> Staphylococcus aureus
<400> 126
Gly Glu Phe Tyr Ser Ala Thr Leu Val Glu Gly Tyr Lys Asp His Leu
1 5 10 15
Asp Gln Ala Ala His Lys Ala Leu Leu Asn Asn Arg Thr Glu Val Ser
20 25 30
Val Asp Ala Tyr Glu Thr Phe Phe Lys Arg Phe Asp Asp Val Asp Phe
35 40 45
Asp Glu Gln Gln Asp Ala Val His Glu Asp Arg Arg Ile Phe Tyr Leu
50 55 60
Ser Asn Ile Glu Asn Asn Val Arg Glu Tyr His Arg Pro Glu
65 70 75
<210> 127
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 127
Gly Leu Phe Ser Tyr Gly Ser Gly Ser Val Gly Glu Phe Tyr Ser Ala
1 5 10 15
Thr Leu Val Glu Gly Tyr Lys Asp His Leu Asp Gln Ala Ala His Lys
20 25 30
Ala Leu Leu Asn Asn Arg Thr Glu Val Ser Val Asp Ala Tyr Glu Thr
35 40 45
Phe Phe Lys Arg Phe Asp Asp Val Asp Phe Asp Glu Gln Gln Asp Ala
50 55 60
Val His Glu Asp Arg Arg Ile Phe Tyr Leu Ser Asn Ile Glu Asn Asn
65 70 75 80
Val Arg Glu Tyr His Arg Pro Glu
85
<210> 128
<211> 78
<212> PRT
<213> Staphylococcus aureus
<400> 128
Gly Glu Phe Tyr Ser Ala Thr Leu Val Glu Gly Tyr Lys Asp His Leu
1 5 10 15
Asp Gln Ala Ala His Lys Ala Leu Leu Asn Asn Arg Thr Glu Val Ser
20 25 30
Val Asp Ala Tyr Glu Thr Phe Phe Lys Arg Phe Asp Asp Val Asp Phe
35 40 45
Asp Glu Glu Gln Asp Ala Val His Glu Asp Arg His Ile Phe Tyr Leu
50 55 60
Ser Asn Ile Glu Asn Asn Val Arg Glu Tyr His Arg Pro Glu
65 70 75
<210> 129
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 129
Gly Leu Phe Ser Tyr Gly Ser Gly Ser Val Gly Glu Phe Tyr Ser Ala
1 5 10 15
Thr Leu Val Glu Gly Tyr Lys Asp His Leu Asp Gln Ala Ala His Lys
20 25 30
Ala Leu Leu Asn Asn Arg Thr Glu Val Ser Val Asp Ala Tyr Glu Thr
35 40 45
Phe Phe Lys Arg Phe Asp Asp Val Glu Phe Asp Glu Glu Gln Asp Ala
50 55 60
Val His Glu Asp Arg His Ile Phe Tyr Leu Ser Asn Ile Glu Asn Asn
65 70 75 80
Val Arg Glu Tyr His Arg Pro Glu
85
<210> 130
<211> 88
<212> PRT
<213> Staphylococcus aureus
<400> 130
Gly Leu Phe Ser Tyr Gly Ser Gly Ser Val Gly Glu Phe Tyr Ser Ala
1 5 10 15
Thr Leu Val Glu Gly Tyr Lys Asp His Leu Asp Gln Ala Ala His Lys
20 25 30
Ala Leu Leu Asn Asn Arg Thr Glu Val Ser Val Asp Ala Tyr Glu Thr
35 40 45
Phe Phe Lys Arg Phe Asp Asp Val Glu Phe Asp Glu Glu Gln Asp Ala
50 55 60
Val His Glu Asp Arg His Ile Phe Tyr Leu Ser Asn Ile Glu Asn Asn
65 70 75 80
Val Arg Glu Tyr His Arg Pro Glu
85
<210> 131
<211> 88
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(34)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (36)..(37)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (39)..(41)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (44)..(45)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (51)..(51)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (55)..(57)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (60)..(72)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (75)..(76)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (79)..(81)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (83)..(83)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (85)..(88)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 131
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
20 25 30
Xaa Xaa Leu Xaa Xaa Arg Xaa Xaa Xaa Ser Val Xaa Xaa Tyr Glu Thr
35 40 45
Phe Phe Xaa Arg Phe Asp Xaa Xaa Xaa Phe Asp Xaa Xaa Xaa Xaa Xaa
50 55 60
Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Tyr Leu Xaa Xaa Ile Glu Xaa Xaa
65 70 75 80
Xaa Arg Xaa Tyr Xaa Xaa Xaa Xaa
85
<210> 132
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 132
Glu Ser Arg Glu Gln Leu Ser Val Glu Glu Tyr Glu Thr Phe
1 5 10
<210> 133
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 133
Asn Asn Arg Thr Glu Val Ser Val Asp Ala Tyr Glu Thr Phe
1 5 10
<210> 134
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 134
Asn Gln Glu Phe Asp Phe Glu Arg Glu Leu Thr Gln Asp Pro
1 5 10
<210> 135
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 135
Asp Val Asp Phe Asp Glu Gln Gln Asp Ala Val His Glu Asp
1 5 10
<210> 136
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 136
Asp Val Asp Phe Asp Glu Glu Gln Asp Ala Val His Glu Asp
1 5 10
<210> 137
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 137
Asp Val Glu Phe Asp Glu Glu Gln Asp Ala Val His Glu Asp
1 5 10
<210> 138
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(2)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (4)..(6)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (9)..(10)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 138
Xaa Xaa Arg Xaa Xaa Xaa Ser Val Xaa Xaa Tyr Glu Thr Phe
1 5 10
<210> 139
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(3)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (6)..(14)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 139
Xaa Xaa Xaa Phe Asp Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 140
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 140
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
taatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtc tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 141
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 141
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
taatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtc tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttgctgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 142
<211> 740
<212> ДНК
<213> Staphylococcus aureus
<400> 142
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcga ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
aaaagcacat tgaacaataa 740
<210> 143
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 143
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcga ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 144
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 144
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
taatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcga ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 145
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 145
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcga ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaata tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 146
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 146
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcctta aatgaaacga cgccattata tggaaaatta tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 147
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 147
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcctta aatgaaacga cgccattata tggaaaatta tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 148
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 148
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcctta aatgaaacga cgccattata tggaaaatta tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 149
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 149
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcctta aatgaaacga cgccattata tggaaaatta tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 150
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 150
ttattgttca atgtgctttt gaataaaatc tttgatttga ggcgtaaagt atacggcacc 60
attatattca gaaccaattt ttccaatacc gccatacacc acacctatga cctcattgtt 120
agaatttaga actggtgatc ctgaattccc gggttcaatg tatgcatcaa aatttaaaat 180
attgtctttg attcttttta tagttcctgt agattcaaac tgtttaaaac tattttgagc 240
aggtaatggg taaccaataa ctttaatttt gtcatcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattaaa gccttttggt ccacgttcaa ctgcttgttc 360
ttcaatattc atgacagaga tgtcttcatc acccggataa tcagaaatgc ttttaatttt 420
atatatacca ccatttcctt tgtcaccgtt tggatgggca gtaattctat cgccaacttt 480
ataatctttt gatacatgtt tattggtgat aattgtattt tttccaatta caaaacctgt 540
cgcatcctta aatgaaacga cgccattata tggaaaatta tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcttca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 151
<211> 720
<212> ДНК
<213> Staphylococcus aureus
<400> 151
ttattgttca atgtgctttt gaataaattc tttgatttga ggcgtaaaat aaactgcacc 60
attgtattct gatccgattt ttccaatacc tccatacaca actcctacga cttcattatt 120
taaatttaaa actggggatc ctgaatttcc aggctcgata tatgcatcaa aatttaaatt 180
attatcttta atacttttta cagttccagt tgattcaaat tgtttgaatg tattttgagc 240
tggtaaaggg tatccaataa ctttaatttt gtcgtcaact ttagcatctt tcgcaaaatt 300
gaatgcttgg acattttcat taaaattata accatttgct ccacgttcaa cagcattttc 360
ttcaacgttc attactgata tatcctcatt acctggataa tcagaaatat ttttaatttt 420
ataaattcca ccgttgcctt tgtcaccatt tgggtgggca gtaattctat cgccaacttt 480
atagtccttt gatacatgtt tattggtgat aattgtattt ttttcaattg caaaacctgt 540
cgcatcttta aatgaaacga cgccattata tggaaaaaca tttgtatctt taacttgcgt 600
aacattcttc tctgcatttg ctatttgttg tgtctcatca acgactgcag catttattcc 660
agttactgag gttagaatgg ctaatgctgc catgctttta atgactatat ttttattcat 720
<210> 152
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 152
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asp Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Leu Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 153
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 153
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Ala Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asp Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Leu Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 154
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 154
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 155
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 155
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 156
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 156
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 157
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 157
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Ile
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 158
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 158
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Asn
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 159
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 159
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Asn
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 160
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 160
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Asn
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 161
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 161
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Asn
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 162
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 162
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Glu Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Asn
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Val
50 55 60
Ile Gly Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Ser Ile Ser Asp Tyr Pro Gly Asp Glu
100 105 110
Asp Ile Ser Val Met Asn Ile Glu Glu Gln Ala Val Glu Arg Gly Pro
115 120 125
Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Ser Phe Lys Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg
165 170 175
Ile Lys Asp Asn Ile Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val Ile Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Asp Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 163
<211> 239
<212> PRT
<213> Staphylococcus aureus
<400> 163
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Val Thr Gly Ile Asn Ala Ala Val Val Asp Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Val
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Ala
50 55 60
Ile Glu Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Asn Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Asn Ile Ser Asp Tyr Pro Gly Asn Glu
100 105 110
Asp Ile Ser Val Met Asn Val Glu Glu Asn Ala Val Glu Arg Gly Ala
115 120 125
Asn Gly Tyr Asn Phe Asn Glu Asn Val Gln Ala Phe Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Thr Phe Lys Gln Phe Glu Ser Thr Gly Thr Val Lys Ser
165 170 175
Ile Lys Asp Asn Asn Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Leu Asn Asn Glu Val Val Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Glu Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 164
<211> 239
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (19)..(19)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (28)..(28)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (48)..(48)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (64)..(64)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (66)..(66)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (91)..(91)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (104)..(104)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (111)..(111)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (119)..(119)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (122)..(122)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (128)..(129)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (131)..(131)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (140)..(140)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (164)..(164)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (174)..(174)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (176)..(176)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (181)..(181)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (200)..(200)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (205)..(205)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (231)..(231)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 164
Met Asn Lys Asn Ile Val Ile Lys Ser Met Ala Ala Leu Ala Ile Leu
1 5 10 15
Thr Ser Xaa Thr Gly Ile Asn Ala Ala Val Val Xaa Glu Thr Gln Gln
20 25 30
Ile Ala Asn Ala Glu Lys Asn Val Thr Gln Val Lys Asp Thr Asn Xaa
35 40 45
Phe Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe Xaa
50 55 60
Ile Xaa Lys Asn Thr Ile Ile Thr Asn Lys His Val Ser Lys Asp Tyr
65 70 75 80
Lys Val Gly Asp Arg Ile Thr Ala His Pro Xaa Gly Asp Lys Gly Asn
85 90 95
Gly Gly Ile Tyr Lys Ile Lys Xaa Ile Ser Asp Tyr Pro Gly Xaa Glu
100 105 110
Asp Ile Ser Val Met Asn Xaa Glu Glu Xaa Ala Val Glu Arg Gly Xaa
115 120 125
Xaa Gly Xaa Asn Phe Asn Glu Asn Val Gln Ala Xaa Asn Phe Ala Lys
130 135 140
Asp Ala Lys Val Asp Asp Lys Ile Lys Val Ile Gly Tyr Pro Leu Pro
145 150 155 160
Ala Gln Asn Xaa Phe Lys Gln Phe Glu Ser Thr Gly Thr Xaa Lys Xaa
165 170 175
Ile Lys Asp Asn Xaa Leu Asn Phe Asp Ala Tyr Ile Glu Pro Gly Asn
180 185 190
Ser Gly Ser Pro Val Leu Asn Xaa Asn Asn Glu Val Xaa Gly Val Val
195 200 205
Tyr Gly Gly Ile Gly Lys Ile Gly Ser Glu Tyr Asn Gly Ala Val Tyr
210 215 220
Phe Thr Pro Gln Ile Lys Xaa Phe Ile Gln Lys His Ile Glu Gln
225 230 235
<210> 165
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 165
Pro Tyr Asn Gly Val Val Ser Phe Lys Asp Ala Thr Gly Phe
1 5 10
<210> 166
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 166
Ala His Pro Asp Gly Asp Lys Gly Asn Gly Gly Ile Tyr Lys
1 5 10
<210> 167
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 167
Ala His Pro Asn Gly Asp Lys Gly Asn Gly Gly Ile Tyr Lys
1 5 10
<210> 168
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (4)..(4)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 168
Ala His Pro Xaa Gly Asp Lys Gly Asn Gly Gly Ile Tyr Lys
1 5 10
<210> 169
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 169
Ser Ile Ser Asp Tyr Pro Gly Asp Glu Asp Ile Ser Val Met
1 5 10
<210> 170
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 170
Asn Ile Ser Asp Tyr Pro Gly Asn Glu Asp Ile Ser Val Met
1 5 10
<210> 171
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (1)..(1)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (8)..(8)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 171
Xaa Ile Ser Asp Tyr Pro Gly Xaa Glu Asp Ile Ser Val Met
1 5 10
<210> 172
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 172
Arg Gly Pro Lys Gly Phe Asn Phe Asn Glu Asn Val Gln Ala
1 5 10
<210> 173
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 173
Arg Gly Ala Asn Gly Tyr Asn Phe Asn Glu Asn Val Gln Ala
1 5 10
<210> 174
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (3)..(4)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (6)..(6)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 174
Arg Gly Xaa Xaa Gly Xaa Asn Phe Asn Glu Asn Val Gln Ala
1 5 10
<210> 175
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 175
Gln Phe Glu Ser Thr Gly Thr Ile Lys Arg Ile Lys Asp Asn
1 5 10
<210> 176
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 176
Gln Phe Glu Ser Thr Gly Thr Val Lys Ser Ile Lys Asp Asn
1 5 10
<210> 177
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (8)..(8)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<220>
<221> misc_признак
<222> (10)..(10)
<223> Xaa может представлять собой любую аминокислоту
природного происхождения
<400> 177
Gln Phe Glu Ser Thr Gly Thr Xaa Lys Xaa Ile Lys Asp Asn
1 5 10
<210> 178
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 178
Gly Asn Ser Gly Ser Pro Val Leu Asn Ser Asn Asn Glu Val
1 5 10
<210> 179
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 179
Gly Asn Ser Gly Ser Pro Val Leu Asn Leu Asn Asn Glu Val
1 5 10
<210> 180
<211> 14
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<220>
<221> misc_признак
<222> (10)..(10)
<223> Xaa может представлять собой любую аминокислоту природного
происхождения
<400> 180
Gly Asn Ser Gly Ser Pro Val Leu Asn Xaa Asn Asn Glu Val
1 5 10
<210> 181
<211> 975
<212> ДНК
<213> Staphylococcus aureus
<400> 181
atgaaatttg atagacatag aagattgaga tcatcagcga caatgagaga tatggttaga 60
gagaatcatg taagaaaaga agatttaata tatccaattt ttgtagttga aaaagacgat 120
gtgaaaaaag aaattaagtc attgccaggt gtataccaaa tcagtttgaa tttacttgaa 180
agtgaattaa aagaagctta tgacttaggc atacgtgcca ttatgttttt cggtgttcca 240
aactcaaaag atgatatagg tactggtgca tacattcacg atggtgttat tcaacaggca 300
acacgtattg ctaaaaaaat gtatgatgac ttattaattg ttgcagacac ttgtttatgt 360
gaatatactg atcatggtca ttgtggcgtg attgatgacc atacacatga cgttgacaat 420
gataaatcat tgccactact tgttaaaaca gcaatttctc aagtggaagc tggtgctgat 480
attattgcgc caagtaatat gatggatggt tttgttgctg aaattcgtcg tggattagat 540
gaagccggct attacaatat tcctataatg agttatggtg tcaagtatgc atcaagtttc 600
tttggacctt ttagagatgc agcagattca gcgccatcat ttggggatag aaaaacgtat 660
cagatggacc ctgctaaccg tttggaagca cttcgtgaat tagaaagtga tcttaaagaa 720
gggtgcgaca tgatgattgt taaacctgct ctaagttatt tagatatagt tcgagatgtt 780
aaaaatcata cgaatgttcc agttgttgca tataatgtga gtggagaata tagtatgact 840
aaagcagcgg cacaaaatgg ttggatagat gaagaacgtg tcgttatgga acaaatggtt 900
tcaatgaaac gtgcaggtgc tgatatgatt attacgtatt ttgcaaagga catttgtcgc 960
tatttagata aataa 975
<210> 182
<211> 324
<212> PRT
<213> Staphylococcus aureus
<400> 182
Met Lys Phe Asp Arg His Arg Arg Leu Arg Ser Ser Ala Thr Met Arg
1 5 10 15
Asp Met Val Arg Glu Asn His Val Arg Lys Glu Asp Leu Ile Tyr Pro
20 25 30
Ile Phe Val Val Glu Lys Asp Asp Val Lys Lys Glu Ile Lys Ser Leu
35 40 45
Pro Gly Val Tyr Gln Ile Ser Leu Asn Leu Leu Glu Ser Glu Leu Lys
50 55 60
Glu Ala Tyr Asp Leu Gly Ile Arg Ala Ile Met Phe Phe Gly Val Pro
65 70 75 80
Asn Ser Lys Asp Asp Ile Gly Thr Gly Ala Tyr Ile His Asp Gly Val
85 90 95
Ile Gln Gln Ala Thr Arg Ile Ala Lys Lys Met Tyr Asp Asp Leu Leu
100 105 110
Ile Val Ala Asp Thr Cys Leu Cys Glu Tyr Thr Asp His Gly His Cys
115 120 125
Gly Val Ile Asp Asp His Thr His Asp Val Asp Asn Asp Lys Ser Leu
130 135 140
Pro Leu Leu Val Lys Thr Ala Ile Ser Gln Val Glu Ala Gly Ala Asp
145 150 155 160
Ile Ile Ala Pro Ser Asn Met Met Asp Gly Phe Val Ala Glu Ile Arg
165 170 175
Arg Gly Leu Asp Glu Ala Gly Tyr Tyr Asn Ile Pro Ile Met Ser Tyr
180 185 190
Gly Val Lys Tyr Ala Ser Ser Phe Phe Gly Pro Phe Arg Asp Ala Ala
195 200 205
Asp Ser Ala Pro Ser Phe Gly Asp Arg Lys Thr Tyr Gln Met Asp Pro
210 215 220
Ala Asn Arg Leu Glu Ala Leu Arg Glu Leu Glu Ser Asp Leu Lys Glu
225 230 235 240
Gly Cys Asp Met Met Ile Val Lys Pro Ala Leu Ser Tyr Leu Asp Ile
245 250 255
Val Arg Asp Val Lys Asn His Thr Asn Val Pro Val Val Ala Tyr Asn
260 265 270
Val Ser Gly Glu Tyr Ser Met Thr Lys Ala Ala Ala Gln Asn Gly Trp
275 280 285
Ile Asp Glu Glu Arg Val Val Met Glu Gln Met Val Ser Met Lys Arg
290 295 300
Ala Gly Ala Asp Met Ile Ile Thr Tyr Phe Ala Lys Asp Ile Cys Arg
305 310 315 320
Tyr Leu Asp Lys
<210> 183
<211> 2802
<212> ДНК
<213> Staphylococcus aureus
<400> 183
atgaatatga agaaaaaaga aaaacacgca attcggaaaa aatcgattgg cgtggcttca 60
gtgcttgtag gtacgttaat cggttttgga ctactcagca gtaaagaagc agatgcaagt 120
gaaaatagtg ttacgcaatc tgatagcgca agtaacgaaa gcaaaagtaa tgattcaagt 180
agcgttagtg ctgcacctaa aacagacgac acaaacgtga gtgatactaa aacatcgtca 240
aacactaata atggcgaaac gagtgtggcg caaaatccag cacaacagga aacgacacaa 300
tcatcatcaa caaatgcaac tacggaagaa acgccggtaa ctggtgaagc tactactacg 360
acaacgaatc aagctaatac accggcaaca actcaatcaa gcaatacaaa tgcggaggaa 420
ttagtgaatc aaacaagtaa tgaaacgact tttaatgata ctaatacagt atcatctgta 480
aattcacctc aaaattctac aaatgcggaa aatgtttcaa caacgcaaga tacttcaact 540
gaagcaacac cttcaaacaa tgaatcagct ccacagagta cagatgcaag taataaagat 600
gtagttaatc aagcggttaa tacaagtgcg cctagaatga gagcatttag tttagcggca 660
gtagctgcag atgcaccggc agctggcaca gatattacga atcagttgac gaatgtgaca 720
gttggtattg actctggtac gactgtgtat ccgcaccaag caggttatgt caaactgaat 780
tatggttttt cagtgcctaa ttctgctgtt aaaggtgaca cattcaaaat aactgtacct 840
aaagaattaa acttaaatgg tgtaacttca actgctaaag tgccaccaat tatggctgga 900
gatcaagtat tggcaaatgg tgtaatcgat agtgatggta atgttattta tacatttaca 960
gactatgtaa atactaaaga tgatgtaaaa gcaactttga ccatgcccgc ttatattgac 1020
cctgaaaatg ttaaaaagac aggtaatgtg acattggcta ctggcatagg tagtacaaca 1080
gcaaacaaaa cagtattagt agattatgaa aaatatggta agttttataa cttatctatt 1140
aaaggtacaa ttgaccaaat cgataaaaca aataatacgt atcgtcagac aatttatgtc 1200
aatccaagtg gagataacgt tattgcgccg gttttaacag gtaatttaaa accaaatacg 1260
gatagtaatg cattaataga tcagcaaaat acaagtatta aagtatataa agtagataat 1320
gcagctgatt tatctgaaag ttactttgtg aatccagaaa actttgagga tgtcactaat 1380
agtgtgaata ttacattccc aaatccaaat caatataaag tagagtttaa tacgcctgat 1440
gatcaaatta caacaccgta tatagtagtt gttaatggtc atattgatcc gaatagcaaa 1500
ggtgatttag ctttacgttc aactttatat gggtataact cgaatataat ttggcgctct 1560
atgtcatggg acaacgaagt agcatttaat aacggatcag gttctggtga cggtatcgat 1620
aaaccagttg ttcctgaaca acctgatgag cctggtgaaa ttgaaccaat tccagaggat 1680
tcagattctg acccaggttc agattctggc agcgattcta attcagatag cggttcagat 1740
tcgggtagtg attctacatc agatagtggt tcagattcag cgagtgattc agattcagca 1800
agtgattcag actcagcgag tgattcagat tcagcaagcg attccgactc agcgagcgat 1860
tccgactcag acaatgactc ggattcagat agcgattctg actcagacag tgactcagat 1920
tccgacagtg actcagattc agatagcgat tctgactcag acagtgactc agattcagat 1980
agcgattcag attcagatag cgattcagat tccgacagtg attccgactc agacagcgat 2040
tctgactccg acagtgattc cgactcagac agcgattcag attccgacag tgattccgac 2100
tcagatagcg attccgactc agatagcgac tcagattcag acagcgattc agattcagac 2160
agcgattcag attcagatag cgattcagat tccgacagtg actcagattc cgacagtgac 2220
tcggattcag atagcgattc agattccgac agtgactcag attccgacag tgactcagac 2280
tcagacagtg attcggattc agcgagtgat tcggattcag atagtgattc cgactccgac 2340
agtgactcgg attcagatag cgactcagac tcggatagcg actcggattc agatagcgat 2400
tcggactcag atagcgattc agaatcagac agcgattcag aatcagacag cgattcagat 2460
tcagacagcg actcagacag tgactcagat tcagatagtg actcggattc agcgagtgat 2520
tcagactcag gtagtgactc cgattcatca agtgattccg actcagaaag tgattcaaat 2580
agcgattccg agtcaggttc taacaataat gtagttccgc ctaattcacc taaaaatggt 2640
actaatgctt ctaataaaaa tgaggctaaa gatagtaaag aaccattacc agatacaggt 2700
tctgaagatg aagcaaatac gtcactaatt tggggattat tagcatcaat aggttcatta 2760
ctacttttca gaagaaaaaa agaaaataaa gataagaaat aa 2802
<210> 184
<211> 933
<212> PRT
<213> Staphylococcus aureus
<400> 184
Met Asn Met Lys Lys Lys Glu Lys His Ala Ile Arg Lys Lys Ser Ile
1 5 10 15
Gly Val Ala Ser Val Leu Val Gly Thr Leu Ile Gly Phe Gly Leu Leu
20 25 30
Ser Ser Lys Glu Ala Asp Ala Ser Glu Asn Ser Val Thr Gln Ser Asp
35 40 45
Ser Ala Ser Asn Glu Ser Lys Ser Asn Asp Ser Ser Ser Val Ser Ala
50 55 60
Ala Pro Lys Thr Asp Asp Thr Asn Val Ser Asp Thr Lys Thr Ser Ser
65 70 75 80
Asn Thr Asn Asn Gly Glu Thr Ser Val Ala Gln Asn Pro Ala Gln Gln
85 90 95
Glu Thr Thr Gln Ser Ser Ser Thr Asn Ala Thr Thr Glu Glu Thr Pro
100 105 110
Val Thr Gly Glu Ala Thr Thr Thr Thr Thr Asn Gln Ala Asn Thr Pro
115 120 125
Ala Thr Thr Gln Ser Ser Asn Thr Asn Ala Glu Glu Leu Val Asn Gln
130 135 140
Thr Ser Asn Glu Thr Thr Phe Asn Asp Thr Asn Thr Val Ser Ser Val
145 150 155 160
Asn Ser Pro Gln Asn Ser Thr Asn Ala Glu Asn Val Ser Thr Thr Gln
165 170 175
Asp Thr Ser Thr Glu Ala Thr Pro Ser Asn Asn Glu Ser Ala Pro Gln
180 185 190
Ser Thr Asp Ala Ser Asn Lys Asp Val Val Asn Gln Ala Val Asn Thr
195 200 205
Ser Ala Pro Arg Met Arg Ala Phe Ser Leu Ala Ala Val Ala Ala Asp
210 215 220
Ala Pro Ala Ala Gly Thr Asp Ile Thr Asn Gln Leu Thr Asn Val Thr
225 230 235 240
Val Gly Ile Asp Ser Gly Thr Thr Val Tyr Pro His Gln Ala Gly Tyr
245 250 255
Val Lys Leu Asn Tyr Gly Phe Ser Val Pro Asn Ser Ala Val Lys Gly
260 265 270
Asp Thr Phe Lys Ile Thr Val Pro Lys Glu Leu Asn Leu Asn Gly Val
275 280 285
Thr Ser Thr Ala Lys Val Pro Pro Ile Met Ala Gly Asp Gln Val Leu
290 295 300
Ala Asn Gly Val Ile Asp Ser Asp Gly Asn Val Ile Tyr Thr Phe Thr
305 310 315 320
Asp Tyr Val Asn Thr Lys Asp Asp Val Lys Ala Thr Leu Thr Met Pro
325 330 335
Ala Tyr Ile Asp Pro Glu Asn Val Lys Lys Thr Gly Asn Val Thr Leu
340 345 350
Ala Thr Gly Ile Gly Ser Thr Thr Ala Asn Lys Thr Val Leu Val Asp
355 360 365
Tyr Glu Lys Tyr Gly Lys Phe Tyr Asn Leu Ser Ile Lys Gly Thr Ile
370 375 380
Asp Gln Ile Asp Lys Thr Asn Asn Thr Tyr Arg Gln Thr Ile Tyr Val
385 390 395 400
Asn Pro Ser Gly Asp Asn Val Ile Ala Pro Val Leu Thr Gly Asn Leu
405 410 415
Lys Pro Asn Thr Asp Ser Asn Ala Leu Ile Asp Gln Gln Asn Thr Ser
420 425 430
Ile Lys Val Tyr Lys Val Asp Asn Ala Ala Asp Leu Ser Glu Ser Tyr
435 440 445
Phe Val Asn Pro Glu Asn Phe Glu Asp Val Thr Asn Ser Val Asn Ile
450 455 460
Thr Phe Pro Asn Pro Asn Gln Tyr Lys Val Glu Phe Asn Thr Pro Asp
465 470 475 480
Asp Gln Ile Thr Thr Pro Tyr Ile Val Val Val Asn Gly His Ile Asp
485 490 495
Pro Asn Ser Lys Gly Asp Leu Ala Leu Arg Ser Thr Leu Tyr Gly Tyr
500 505 510
Asn Ser Asn Ile Ile Trp Arg Ser Met Ser Trp Asp Asn Glu Val Ala
515 520 525
Phe Asn Asn Gly Ser Gly Ser Gly Asp Gly Ile Asp Lys Pro Val Val
530 535 540
Pro Glu Gln Pro Asp Glu Pro Gly Glu Ile Glu Pro Ile Pro Glu Asp
545 550 555 560
Ser Asp Ser Asp Pro Gly Ser Asp Ser Gly Ser Asp Ser Asn Ser Asp
565 570 575
Ser Gly Ser Asp Ser Gly Ser Asp Ser Thr Ser Asp Ser Gly Ser Asp
580 585 590
Ser Ala Ser Asp Ser Asp Ser Ala Ser Asp Ser Asp Ser Ala Ser Asp
595 600 605
Ser Asp Ser Ala Ser Asp Ser Asp Ser Ala Ser Asp Ser Asp Ser Asp
610 615 620
Asn Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
625 630 635 640
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
645 650 655
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
660 665 670
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
675 680 685
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
690 695 700
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
705 710 715 720
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
725 730 735
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
740 745 750
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Ala
755 760 765
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
770 775 780
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
785 790 795 800
Ser Asp Ser Asp Ser Asp Ser Glu Ser Asp Ser Asp Ser Glu Ser Asp
805 810 815
Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp Ser Asp
820 825 830
Ser Asp Ser Asp Ser Ala Ser Asp Ser Asp Ser Gly Ser Asp Ser Asp
835 840 845
Ser Ser Ser Asp Ser Asp Ser Glu Ser Asp Ser Asn Ser Asp Ser Glu
850 855 860
Ser Gly Ser Asn Asn Asn Val Val Pro Pro Asn Ser Pro Lys Asn Gly
865 870 875 880
Thr Asn Ala Ser Asn Lys Asn Glu Ala Lys Asp Ser Lys Glu Pro Leu
885 890 895
Pro Asp Thr Gly Ser Glu Asp Glu Ala Asn Thr Ser Leu Ile Trp Gly
900 905 910
Leu Leu Ala Ser Ile Gly Ser Leu Leu Leu Phe Arg Arg Lys Lys Glu
915 920 925
Asn Lys Asp Lys Lys
930
<210> 185
<211> 219
<212> PRT
<213> Staphylococcus aureus
<400> 185
Met Ile Glu Ile Asn Asn Leu Ser Lys Arg Tyr Arg Asn Lys Gln Ile
1 5 10 15
Phe Asn His Leu Thr Met Ser Phe Asp Ser Asn Arg Leu Thr Val Leu
20 25 30
Leu Gly Asp Asn Gly Ala Gly Lys Ser Thr Leu Leu Arg Met Ile Ala
35 40 45
Gly Ile Glu Lys Ala Asn Asp Gly Thr Ile Asn Tyr Phe Gly Glu Lys
50 55 60
Trp Asn Gln Arg Gln Ile Gln Asn His Ile Gly Tyr Val Pro Gln Asp
65 70 75 80
Ile Ala Leu Phe Glu His Met Thr Val Ala Glu Asn Ile Lys Phe Phe
85 90 95
Lys Ser Leu Cys Lys Asn Pro Ile Asn Asp Thr Thr Ile Asn Glu Tyr
100 105 110
Leu Gln Gln Leu Asn Phe Asp Asp Thr Ser Ala Lys Val Ser Thr Leu
115 120 125
Ser Gly Gly Asn Lys Arg Lys Ile Asn Ile Leu Val Gly Leu Leu Gly
130 135 140
Gln Pro Arg Ile Leu Ile Leu Asp Glu Pro Thr Val Gly Ile Asp Leu
145 150 155 160
Lys Ser Arg His Asp Ile His Gln Leu Leu Asn Ile Met Lys Ser Lys
165 170 175
Cys Leu Ile Ile Leu Thr Thr His His Leu Asp Glu Val Glu Ala Leu
180 185 190
Ala Asp Asp Ile Lys Leu Ile Gly Gln Asp Pro Phe Tyr Gln His Val
195 200 205
Leu Glu Asp Lys Gln Trp Thr Tyr Thr Tyr Tyr
210 215
<210> 186
<211> 253
<212> PRT
<213> Staphylococcus aureus
<400> 186
Met Ala Ile Leu Glu Val Lys Gln Leu Thr Lys Ile Tyr Gly Thr Lys
1 5 10 15
Lys Met Ala Gln Glu Val Leu Arg Asp Ile Asn Met Ser Ile Glu Glu
20 25 30
Gly Glu Phe Ile Ala Ile Met Gly Pro Ser Gly Ser Gly Lys Thr Thr
35 40 45
Leu Leu Asn Val Leu Ser Ser Ile Asp Tyr Ile Ser Gln Gly Ser Ile
50 55 60
Thr Leu Lys Gly Lys Lys Leu Glu Lys Leu Ser Asn Lys Glu Leu Ser
65 70 75 80
Asp Ile Arg Lys His Asp Ile Gly Phe Ile Phe Gln Glu Tyr Asn Leu
85 90 95
Leu His Thr Leu Thr Val Lys Glu Asn Ile Met Leu Pro Leu Thr Val
100 105 110
Gln Lys Leu Asp Lys Glu His Met Leu Asn Arg Tyr Glu Lys Val Ala
115 120 125
Glu Ala Leu Asn Ile Leu Asp Ile Ser Asp Lys Tyr Pro Ser Glu Leu
130 135 140
Ser Gly Gly Gln Arg Gln Arg Thr Ser Ala Ala Arg Ala Phe Ile Thr
145 150 155 160
Leu Pro Ser Ile Ile Phe Ala Asp Glu Pro Thr Gly Ala Leu Asp Ser
165 170 175
Lys Ser Thr Gln Asp Leu Leu Lys Arg Leu Thr Arg Met Asn Glu Ala
180 185 190
Phe Lys Ser Thr Ile Ile Met Val Thr His Asp Pro Val Ala Ala Ser
195 200 205
Tyr Ala Asn Arg Val Val Met Leu Lys Asp Gly Gln Ile Phe Thr Glu
210 215 220
Leu Tyr Gln Gly Asp Asp Asp Lys His Thr Phe Phe Lys Glu Ile Ile
225 230 235 240
Arg Val Gln Ser Val Leu Gly Gly Val Asn Tyr Asp Leu
245 250
<210> 187
<211> 243
<212> PRT
<213> Staphylococcus aureus
<400> 187
Met Ile Leu Ser Tyr Leu Lys Ile Glu Phe Lys Val Ile Met Arg Lys
1 5 10 15
Lys Thr Thr Leu Ile Leu Ser Ile Leu Phe Pro Val Ile Phe Tyr Ile
20 25 30
Leu Phe Thr Ser Ile Leu Glu Leu Pro Glu Asp Val Lys Pro Lys Phe
35 40 45
Tyr Lys Glu Tyr Met Tyr Ser Met Thr Val Tyr Ser Leu Leu Ser Phe
50 55 60
Ser Leu Leu Thr Phe Pro Leu Asp Ile Ile Asn Glu Lys Gln Asn Glu
65 70 75 80
Trp Arg Gln Arg Leu Met Val Thr Pro Phe Thr Phe Thr Ser Tyr Tyr
85 90 95
Ile Ser Lys Val Val Lys Thr Met Leu Gln Phe Ala Ile Ala Ile Leu
100 105 110
Val Ile Phe Met Val Gly His Phe Tyr Lys Gly Val Ala Met Ser Ala
115 120 125
Val Gln Trp Leu Glu Ser Gly Ile Phe Leu Trp Leu Gly Ala Ser Leu
130 135 140
Leu Ile Thr Phe Gly Ile Leu Phe Ser Leu Leu Asn Asp Ile Gln Lys
145 150 155 160
Thr Ser Ala Leu Ala Asn Ile Val Thr Ile Gly Leu Ala Val Leu Gly
165 170 175
Gly Leu Trp Phe Pro Ile Asn Thr Phe Pro Asn Trp Leu Gln His Val
180 185 190
Ala His Val Leu Pro Ser Tyr His Leu Arg Lys Leu Gly Val Asp Ile
195 200 205
Ala Ser Asn His His Ile Asn Leu Ile Ser Phe Ala Ile Ile Leu Leu
210 215 220
Tyr Ala Leu Gly Ser Ile Ile Ala Val Tyr Cys Ile Ser His Phe Lys
225 230 235 240
Arg Ala Glu
<210> 188
<211> 276
<212> PRT
<213> Staphylococcus aureus
<400> 188
Met His Lys Ile Phe Ser Lys Asn Asn Leu Ile Phe Phe Val Phe Val
1 5 10 15
Ala Phe Ile Phe Val Val Ile Val Leu Gln Phe Phe Val Ser Ser Glu
20 25 30
Asn Ala Thr Lys Val Asn Leu Ser Gln Thr Phe Glu Pro Ile Ser Trp
35 40 45
Leu His Leu Leu Gly Thr Asp Asp Tyr Gly Arg Asp Leu Phe Thr Arg
50 55 60
Ile Ile Ile Gly Ala Arg Ser Thr Leu Phe Val Thr Val Leu Thr Leu
65 70 75 80
Ile Ala Ile Val Val Ile Gly Val Thr Leu Gly Leu Phe Ala Gly Tyr
85 90 95
Lys Lys Gly Trp Ile Glu Arg Leu Val Leu Arg Phe Ile Asp Val Gly
100 105 110
Leu Ser Ile Pro Glu Phe Ile Ile Met Ile Ala Leu Ala Ser Phe Phe
115 120 125
Gln Pro Ser Leu Trp Asn Leu Val Ile Ser Ile Thr Leu Ile Lys Trp
130 135 140
Met Asn Tyr Thr Arg Leu Thr Arg Ser Ile Val Asn Ser Glu Met Asn
145 150 155 160
Lys Pro Tyr Ile Lys Met Ala Gln Leu Phe His Val Pro Thr Arg Thr
165 170 175
Ile Leu Ile Arg His Leu Thr Pro Lys Ile Ile Pro Ala Ile Ile Val
180 185 190
Leu Met Val Val Asp Phe Gly Lys Ile Ile Leu Tyr Ile Ser Ser Leu
195 200 205
Ser Phe Ile Gly Leu Gly Ala Gln Pro Pro Thr Pro Glu Trp Gly Ala
210 215 220
Met Leu Gln Gln Gly Arg Asp Phe Ile Ser Ser His Pro Ile Met Leu
225 230 235 240
Ile Ala Pro Ala Ser Val Ile Ala Ile Thr Ile Leu Ile Phe Asn Leu
245 250 255
Thr Gly Asp Ala Leu Arg Asp Arg Leu Leu Lys Gln Arg Gly Glu Tyr
260 265 270
Asp Glu Ser His
275
<210> 189
<211> 136
<212> PRT
<213> Staphylococcus aureus
<400> 189
Met Asn Thr Asn Asp Ala Ile Lys Ile Leu Lys Glu Asn Gly Leu Lys
1 5 10 15
Tyr Thr Asp Lys Arg Lys Asp Met Leu Asp Ile Phe Val Glu Glu Asp
20 25 30
Lys Tyr Ile Asn Ala Lys Tyr Ile Gln Gln Val Met Asp Glu Asn Tyr
35 40 45
Pro Gly Ile Ser Phe Asp Thr Ile Tyr Arg Asn Leu His Leu Phe Lys
50 55 60
Asp Leu Gly Ile Ile Glu Asn Thr Glu Leu Asp Gly Glu Met Lys Phe
65 70 75 80
Arg Ile Ala Cys Thr Asn His His His His His Phe Ile Cys Glu Lys
85 90 95
Cys Gly Asp Thr Lys Val Ile Asp Tyr Cys Pro Ile Asp Gln Ile Lys
100 105 110
Leu Ser Leu Pro Gly Val Asn Ile His Lys His Lys Leu Glu Val Tyr
115 120 125
Gly Val Cys Glu Ser Cys Gln Asp
130 135
<210> 190
<211> 233
<212> PRT
<213> Staphylococcus aureus
<400> 190
Met Thr His Lys Tyr Ile Ser Thr Gln Met Leu Ile Ile Phe Thr Ala
1 5 10 15
Leu Met Ile Ile Ala Asn Phe Tyr Tyr Ile Phe Phe Glu Lys Ile Gly
20 25 30
Phe Leu Leu Val Leu Leu Leu Gly Cys Val Leu Val Tyr Val Gly Tyr
35 40 45
Leu Tyr Phe His Lys Ile Arg Gly Leu Leu Ala Phe Trp Ile Gly Ala
50 55 60
Leu Leu Ile Ala Phe Thr Leu Leu Ser Asn Lys Tyr Thr Ile Ile Ile
65 70 75 80
Leu Phe Val Phe Leu Leu Leu Leu Ile Val Arg Tyr Leu Ile His Lys
85 90 95
Phe Lys Pro Lys Lys Val Val Ala Thr Asp Glu Val Met Thr Ser Pro
100 105 110
Ser Phe Ile Lys Gln Lys Trp Phe Gly Glu Gln Arg Thr Pro Val Tyr
115 120 125
Val Tyr Lys Trp Glu Asp Val Gln Ile Gln His Gly Ile Gly Asp Leu
130 135 140
His Ile Asp Leu Thr Lys Ala Ala Asn Ile Lys Glu Asn Asn Thr Ile
145 150 155 160
Val Val Arg His Ile Leu Gly Lys Val Gln Val Ile Leu Pro Val Asn
165 170 175
Tyr Asn Ile Asn Leu His Val Ala Ala Phe Tyr Gly Ser Thr Tyr Val
180 185 190
Asn Glu Lys Ser Tyr Lys Val Glu Asn Asn Asn Ile His Ile Glu Glu
195 200 205
Met Met Lys Pro Asp Asn Tyr Thr Val Asn Ile Tyr Val Ser Thr Phe
210 215 220
Ile Gly Asp Val Glu Val Ile Tyr Arg
225 230
<210> 191
<211> 260
<212> PRT
<213> Staphylococcus aureus
<400> 191
Met Leu Ile Gln Leu Asp Gln Ile Gly Arg Met Lys Gln Gly Lys Thr
1 5 10 15
Ile Leu Lys Lys Ile Ser Trp Gln Ile Ala Lys Gly Asp Lys Trp Ile
20 25 30
Leu Tyr Gly Leu Asn Gly Ala Gly Lys Thr Thr Leu Leu Asn Ile Leu
35 40 45
Asn Ala Tyr Glu Pro Ala Thr Ser Gly Thr Val Asn Leu Phe Gly Lys
50 55 60
Met Pro Gly Lys Val Gly Tyr Ser Ala Glu Thr Val Arg Gln His Ile
65 70 75 80
Gly Phe Val Ser His Ser Leu Leu Glu Lys Phe Gln Glu Gly Glu Arg
85 90 95
Val Ile Asp Val Val Ile Ser Gly Ala Phe Lys Ser Ile Gly Val Tyr
100 105 110
Gln Asp Ile Asp Asp Glu Ile Arg Asn Glu Ala His Gln Leu Leu Lys
115 120 125
Leu Val Gly Met Ser Ala Lys Ala Gln Gln Tyr Ile Gly Tyr Leu Ser
130 135 140
Thr Gly Glu Lys Gln Arg Val Met Ile Ala Arg Ala Leu Met Gly Gln
145 150 155 160
Pro Gln Val Leu Ile Leu Asp Glu Pro Ala Ala Gly Leu Asp Phe Ile
165 170 175
Ala Arg Glu Ser Leu Leu Ser Ile Leu Asp Ser Leu Ser Asp Ser Tyr
180 185 190
Pro Thr Leu Ala Met Ile Tyr Val Thr His Phe Ile Glu Glu Ile Thr
195 200 205
Ala Asn Phe Ser Lys Ile Leu Leu Leu Lys Asp Gly Gln Ser Ile Gln
210 215 220
Gln Gly Ala Val Glu Asp Ile Leu Thr Ser Glu Asn Met Ser Arg Phe
225 230 235 240
Phe Gln Lys Asn Val Ala Val Gln Arg Trp Asn Asn Arg Phe Ser Met
245 250 255
Ala Met Leu Glu
260
<210> 192
<211> 209
<212> PRT
<213> Staphylococcus aureus
<400> 192
Met Lys Arg Leu Val Thr Gly Leu Leu Ala Leu Ser Leu Phe Leu Ala
1 5 10 15
Ala Cys Gly Gln Asp Ser Asp Gln Gln Lys Asp Gly Asn Lys Glu Lys
20 25 30
Asp Asp Lys Ala Lys Thr Glu Gln Gln Asp Lys Lys Thr Asn Asp Ser
35 40 45
Ser Lys Asp Lys Lys Asp Asn Lys Asp Asp Ser Lys Asp Val Asn Lys
50 55 60
Asp Asn Lys Asp Asn Ser Ala Asn Asp Asn Gln Gln Gln Ser Asn Ser
65 70 75 80
Asn Ala Thr Asn Asn Asp Gln Asn Gln Thr Asn Asn Asn Gln Ser Ser
85 90 95
Asn Asn Gln Ala Asn Asn Asn Gln Lys Ser Ser Tyr Val Ala Pro Tyr
100 105 110
Tyr Gly Gln Asn Ala Ala Pro Val Ala Arg Gln Ile Tyr Pro Phe Asn
115 120 125
Gly Asn Lys Asn Gln Ala Leu Gln Gln Leu Pro Asn Phe Gln Thr Ala
130 135 140
Leu Asn Ala Ala Asn Asn Glu Ala Asn Lys Phe Gly Ser Asn Asn Lys
145 150 155 160
Val Tyr Asn Asp Tyr Ser Ile Glu Glu His Asn Gly Asn Tyr Lys Tyr
165 170 175
Val Phe Ser Phe Lys Asp Pro Asn Ala Asn Gly Lys Tyr Ser Ile Val
180 185 190
Thr Val Asp Tyr Thr Gly Gln Ala Met Val Thr Asp Pro Asn Tyr Gln
195 200 205
Gln
<210> 193
<211> 439
<212> PRT
<213> Staphylococcus aureus
<400> 193
Met Arg Thr Gly His Tyr Thr Pro Ile Pro Asn Glu Pro His Tyr Leu
1 5 10 15
Val Ile Ser His Ala Asp Lys Leu Thr Ala Thr Glu Lys Ala Lys Leu
20 25 30
Arg Leu Leu Ile Ile Lys Gln Lys Leu Asp Ile Ser Leu Ala Glu Ser
35 40 45
Val Val Ser Ser Pro Ile Ala Ser Glu His Val Ile Glu Gln Leu Thr
50 55 60
Leu Phe Gln His Glu Arg Arg His Leu Arg Pro Lys Ile Ser Ala Thr
65 70 75 80
Phe Leu Ala Trp Leu Leu Ile Phe Leu Met Phe Ala Leu Pro Ile Gly
85 90 95
Ile Ala Tyr Gln Phe Ser Asp Trp Phe Gln Asn Gln Tyr Val Ser Ala
100 105 110
Trp Ile Glu Tyr Leu Thr Gln Thr Thr Leu Leu Asn His Asp Ile Leu
115 120 125
Gln His Ile Leu Phe Gly Asp Tyr Gly Val Leu Ser Leu Gly Thr Tyr
130 135 140
Ser Leu Val Trp Ala Leu Pro Val Val Ile Leu Ile Ser Leu Ser Thr
145 150 155 160
Ala Ile Ile Asp Gln Thr Gly Leu Lys Ser Trp Met Ile Trp Ala Ile
165 170 175
Glu Pro Ser Met Leu Trp Ile Gly Leu Gln Gly Asn Asp Ile Val Pro
180 185 190
Leu Leu Glu Gly Phe Gly Cys Asn Ala Ala Ala Ile Ser Gln Ala Ala
195 200 205
His Gln Cys His Thr Cys Thr Lys Thr Gln Cys Met Ser Leu Ile Ser
210 215 220
Phe Gly Ser Ser Cys Ser Tyr Gln Ile Gly Ala Thr Leu Ser Ile Phe
225 230 235 240
Ser Val Ala Gly Lys Ser Trp Leu Phe Met Pro Tyr Leu Ile Leu Val
245 250 255
Leu Leu Gly Gly Ile Leu His Asn Arg Ile Trp Leu Lys Lys Asn Asp
260 265 270
Gln Gln Leu Ser Val Pro Leu Pro Tyr Asp Arg Gln Leu His Met Pro
275 280 285
Asn Ile Arg Gln Met Leu Leu Gln Met Trp Gln Asn Ile Gln Met Phe
290 295 300
Ile Val Gln Ala Leu Pro Ile Phe Ile Thr Ile Cys Leu Ile Val Ser
305 310 315 320
Ile Leu Ser Leu Thr Pro Ile Leu Asn Val Leu Ser Gln Ile Phe Thr
325 330 335
Pro Ile Leu Ser Leu Leu Gly Ile Ser Ser Glu Leu Ser Pro Gly Ile
340 345 350
Leu Phe Ser Met Ile Arg Lys Asp Gly Met Leu Leu Phe Asn Leu His
355 360 365
Gln Gly Ala Leu Leu Gln Gly Met Thr Ala Thr Gln Leu Leu Leu Leu
370 375 380
Val Phe Phe Ser Ser Thr Phe Thr Ala Cys Ser Val Thr Met Thr Met
385 390 395 400
Leu Leu Lys His Leu Gly Gly Gln Ser Ala Leu Lys Leu Ile Gly Lys
405 410 415
Gln Met Val Thr Ser Leu Ser Leu Val Ile Gly Val Gly Ile Ile Val
420 425 430
Lys Ile Val Met Leu Ile Ile
435
<210> 194
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 194
tcgtcgttgt cgttttgtcg tt 22
<210> 195
<211> 13
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 195
Ile Leu Pro Trp Lys Trp Pro Trp Trp Pro Trp Arg Arg
1 5 10
<210> 196
<211> 13
<212> PRT
<213> Искусственная последовательность
<220>
<223> Синтетическая последовательность
<400> 196
Lys Asp Tyr Glu Arg Lys Tyr Lys Lys His Ile Val Ser
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Мутантные иммуноглобулин-связывающие полипептиды | 2015 |
|
RU2712882C2 |
| РЕКОМБИНАНТНЫЕ МОДИФИЦИРОВАННЫЕ ФАКТОРЫ РОСТА ФИБРОБЛАСТОВ И ИХ ТЕРАПЕВТИЧЕСКОЕ ПРИМЕНЕНИЕ | 2018 |
|
RU2811435C2 |
| CD131-СВЯЗЫВАЮЩИЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2773927C2 |
| МОНОКЛОНАЛЬНЫЕ АГЕНТЫ, СВЯЗЫВАЮЩИЕ сМЕТ, ИХ КОНЪЮГАТЫ С ЛЕКАРСТВЕННЫМИ СРЕДСТВАМИ И ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2744914C2 |
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2819627C1 |
| IL-17A-связывающие полипептиды | 2016 |
|
RU2749712C2 |
| АНТИТЕЛА, НАПРАВЛЕННЫЕ ПРОТИВ РЕЦЕПТОРА ИНТЕРЛЕЙКИНА 36 (IL-36R) | 2016 |
|
RU2745898C2 |
| РЕКРУТИРУЮЩИЕ Т-КЛЕТКИ ПОЛИПЕПТИДЫ, СПОСОБНЫЕ СВЯЗЫВАТЬ CD123 И TCR АЛЬФА/БЕТА | 2017 |
|
RU2775063C2 |
| CD123-СПЕЦИФИЧЕСКИЕ ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ ДЛЯ ИММУНОТЕРАПИИ РАКА | 2015 |
|
RU2727290C2 |
| МОЛЕКУЛЫ, СВЯЗЫВАЮЩИЕ ADAM9, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2783619C2 |
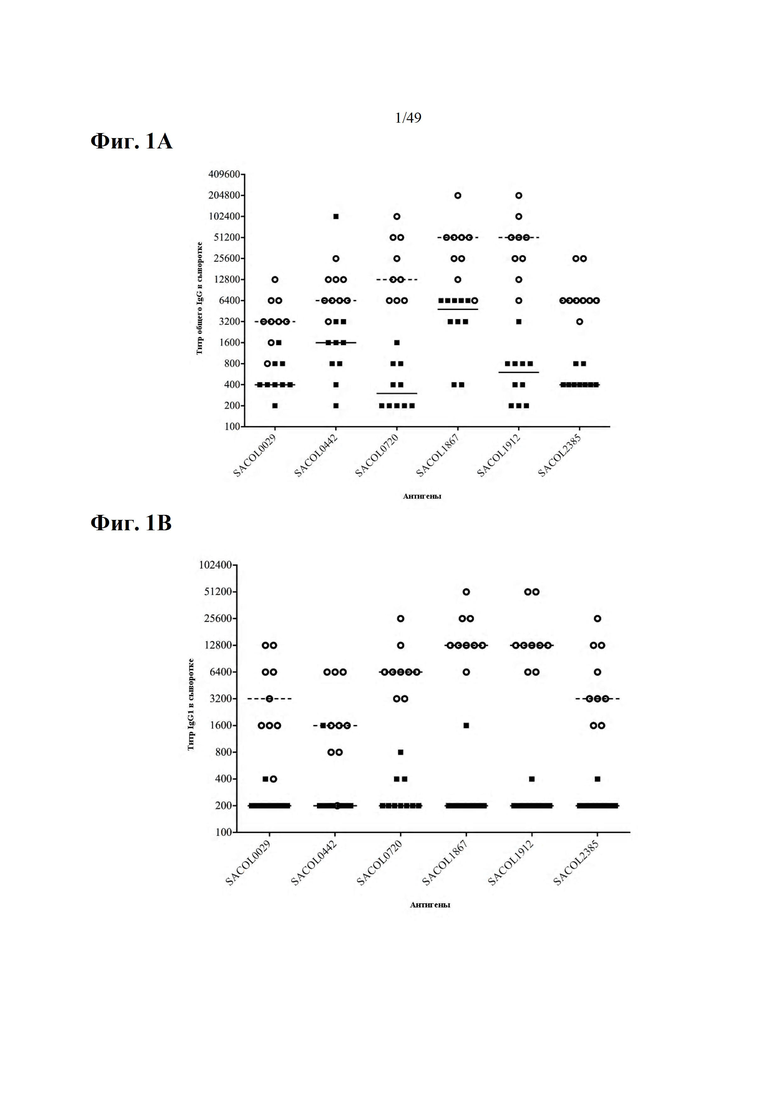
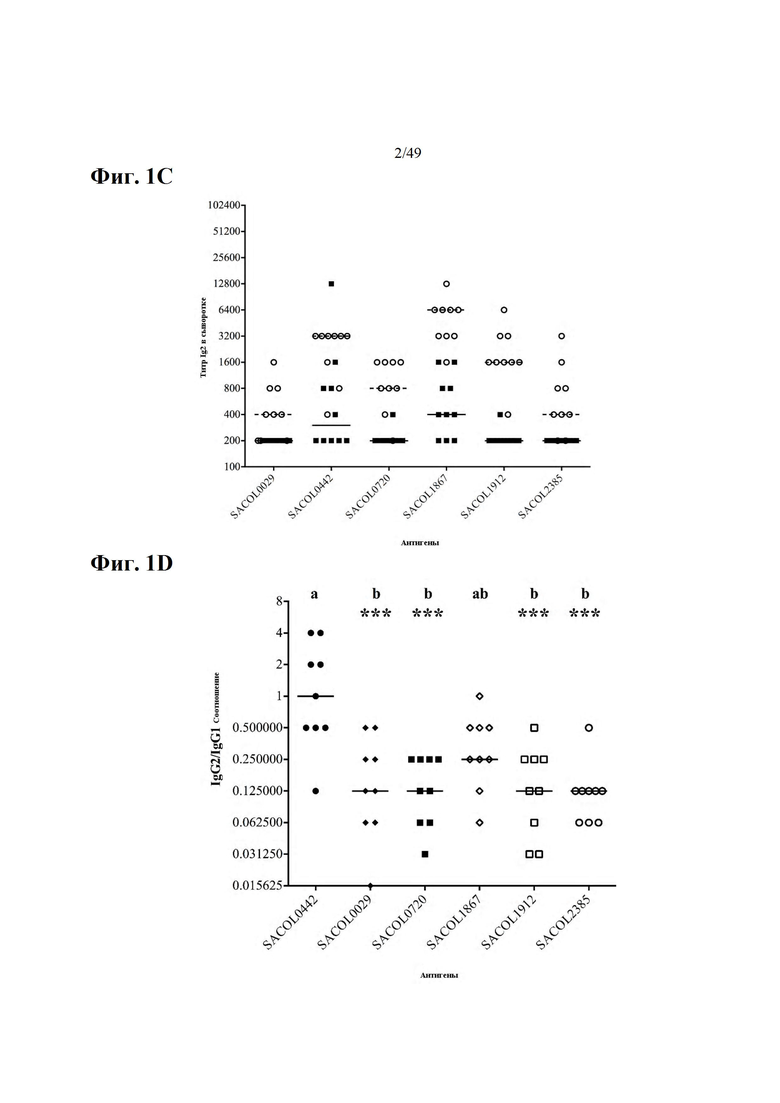
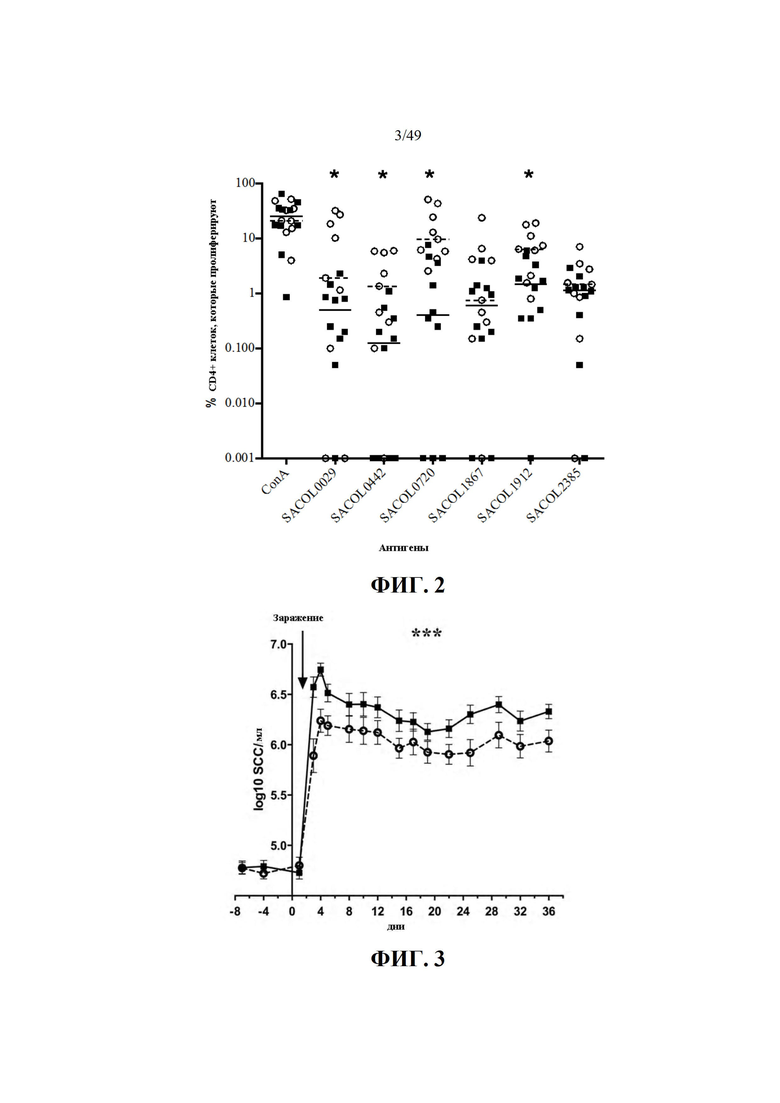
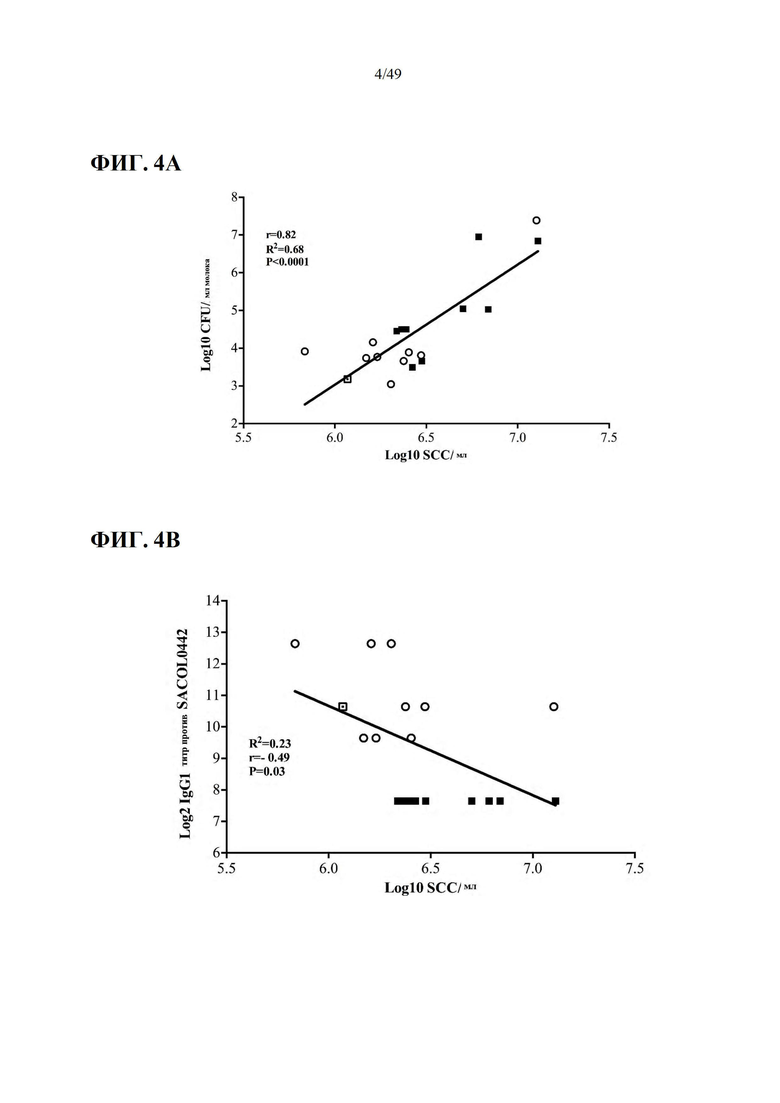
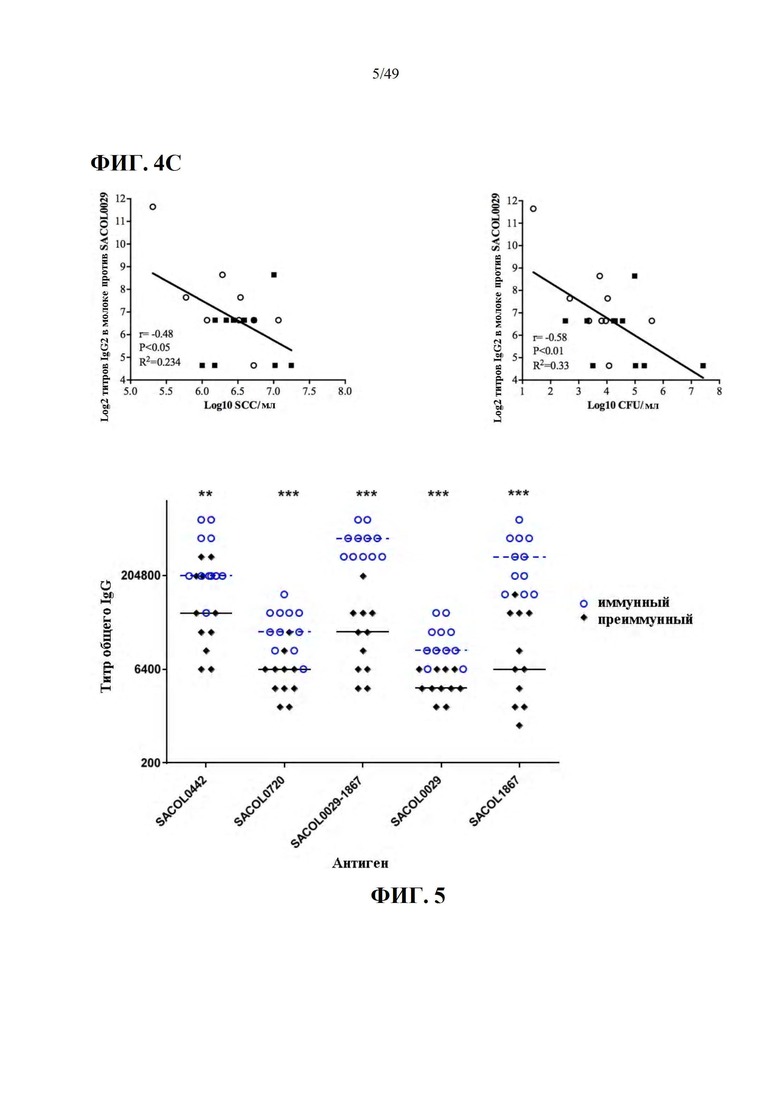
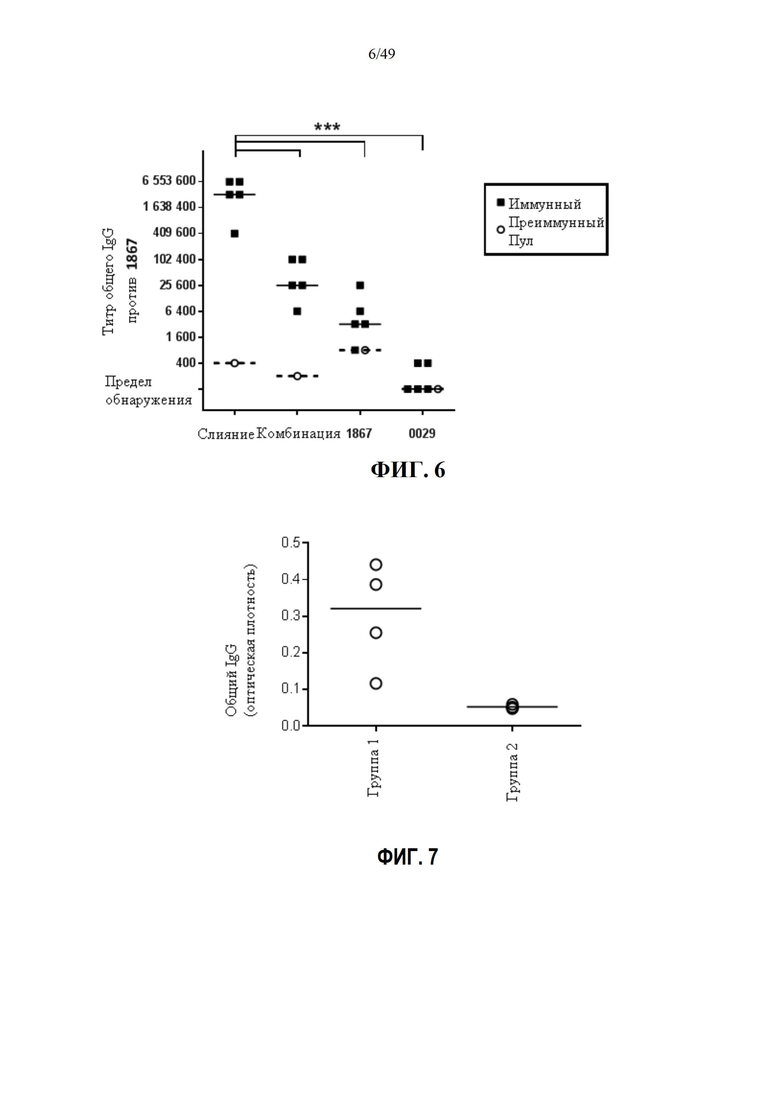
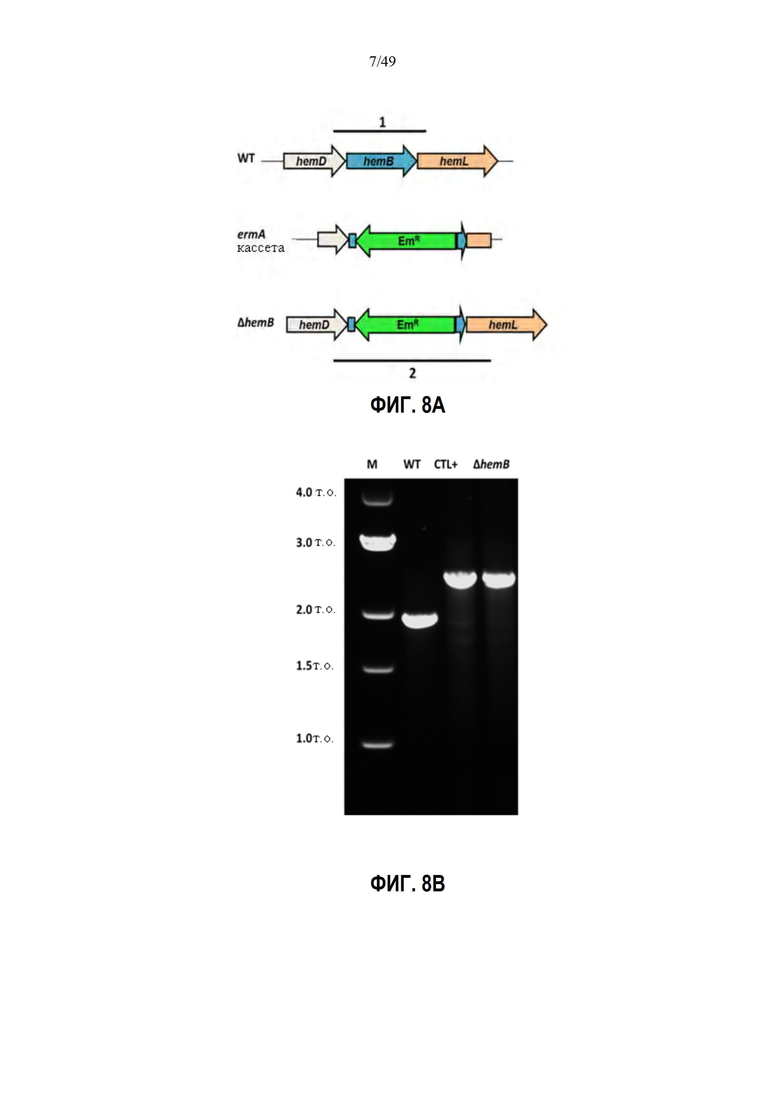
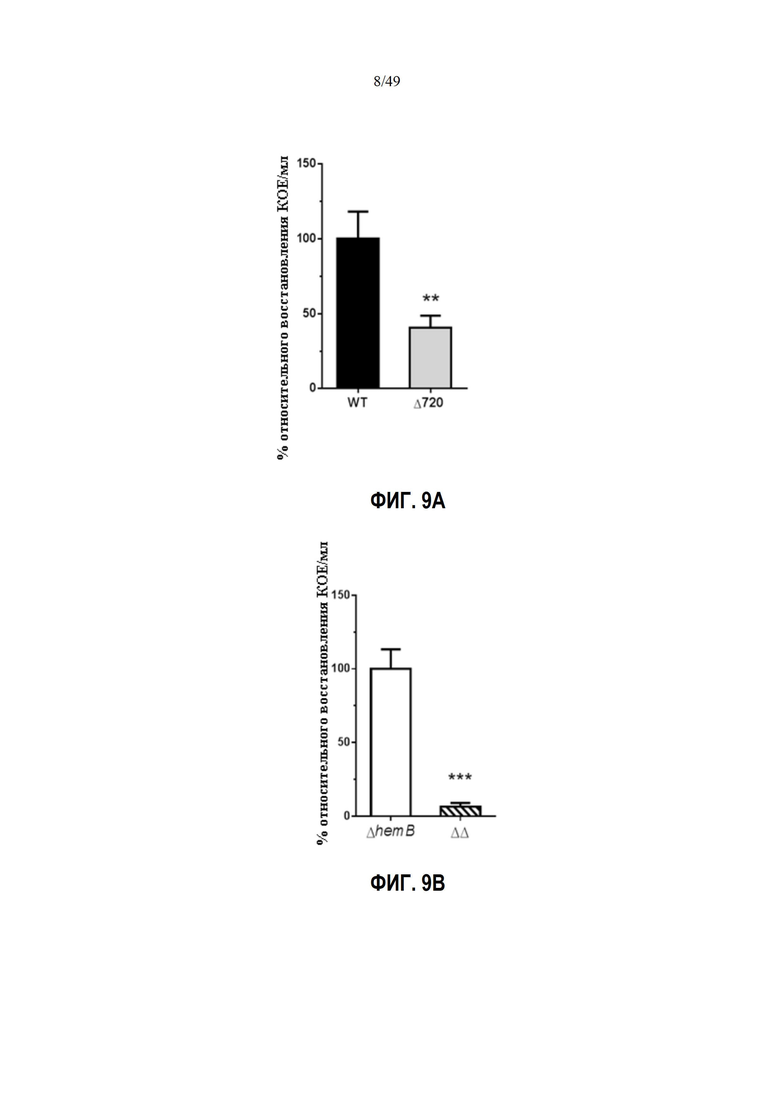
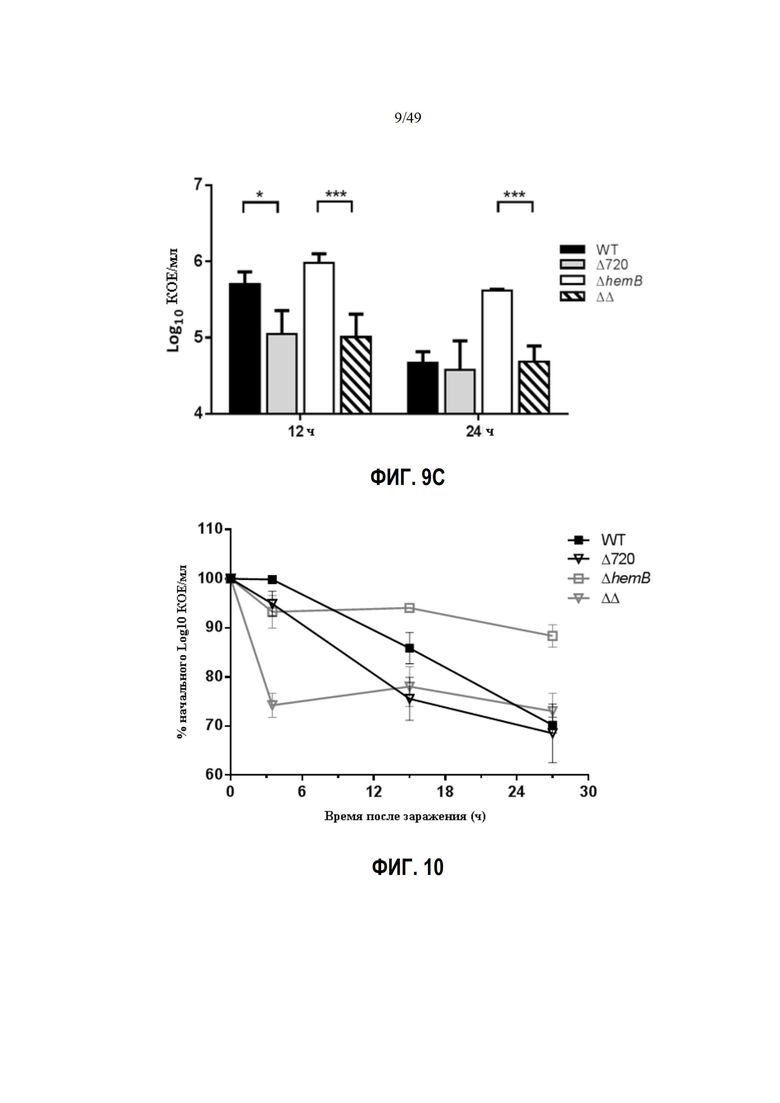
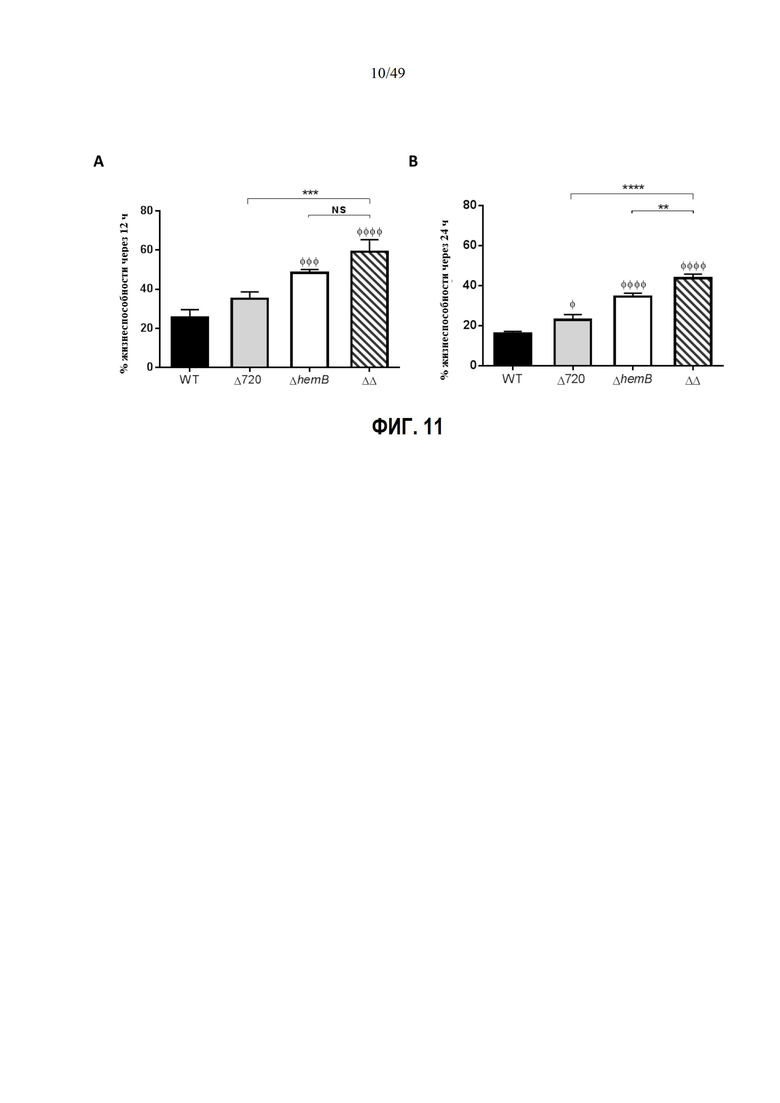
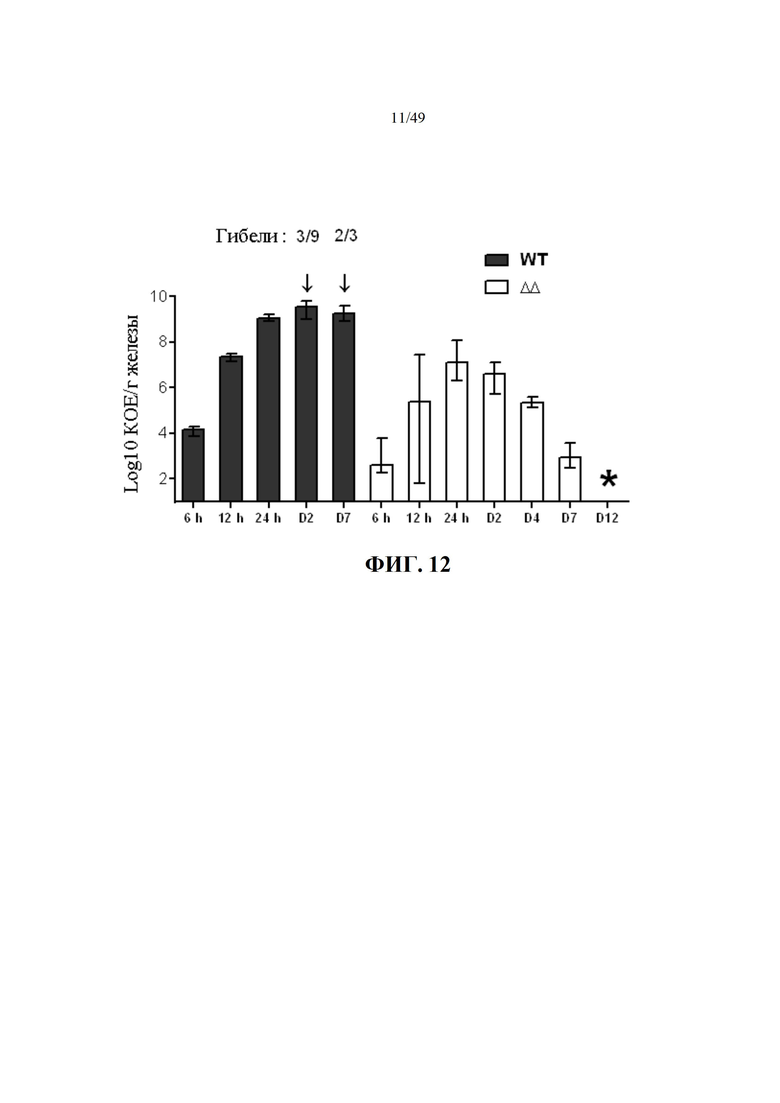
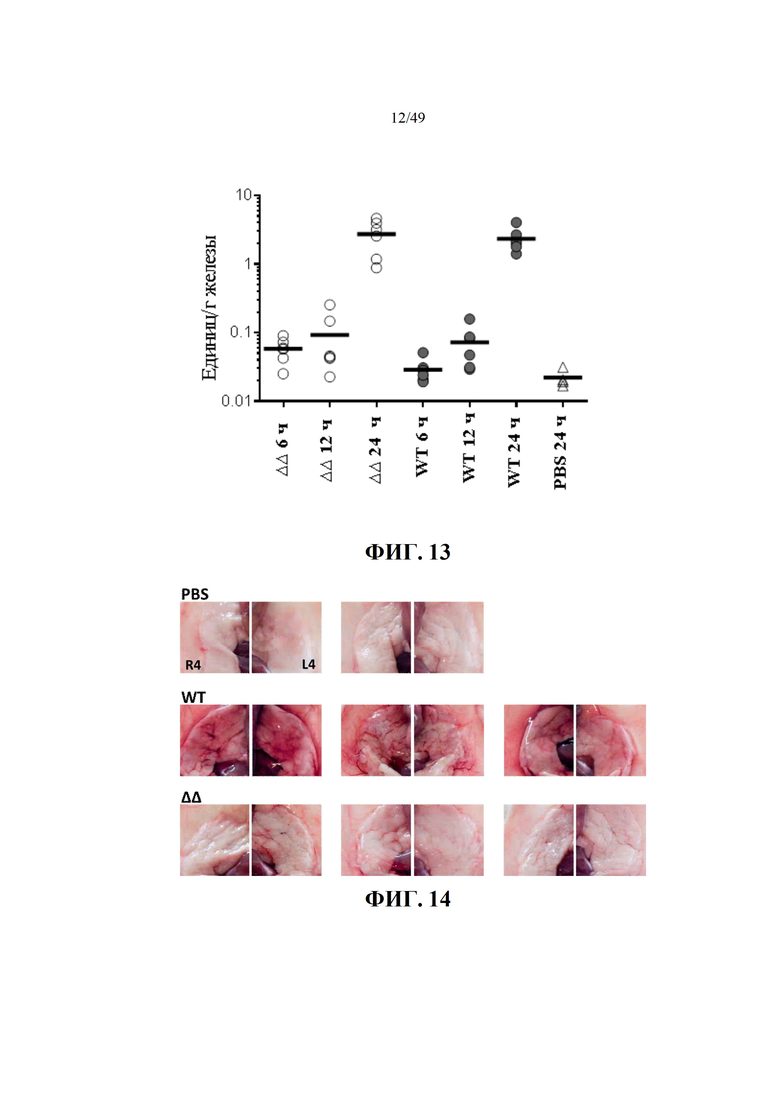
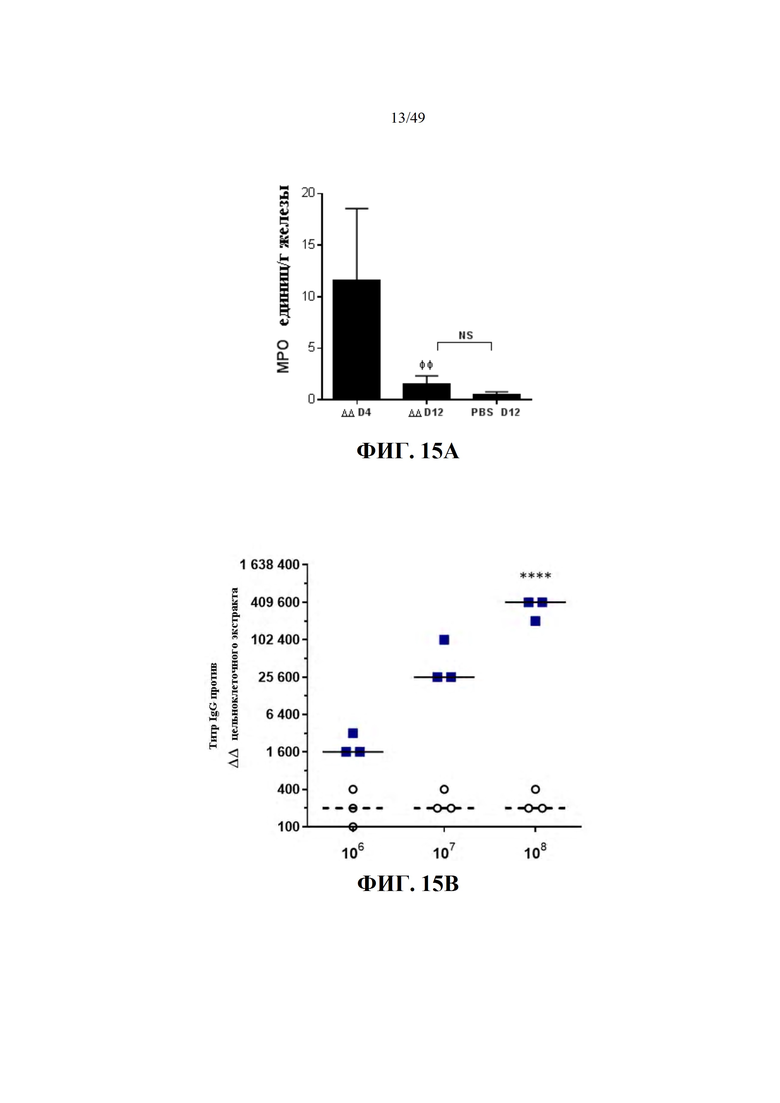
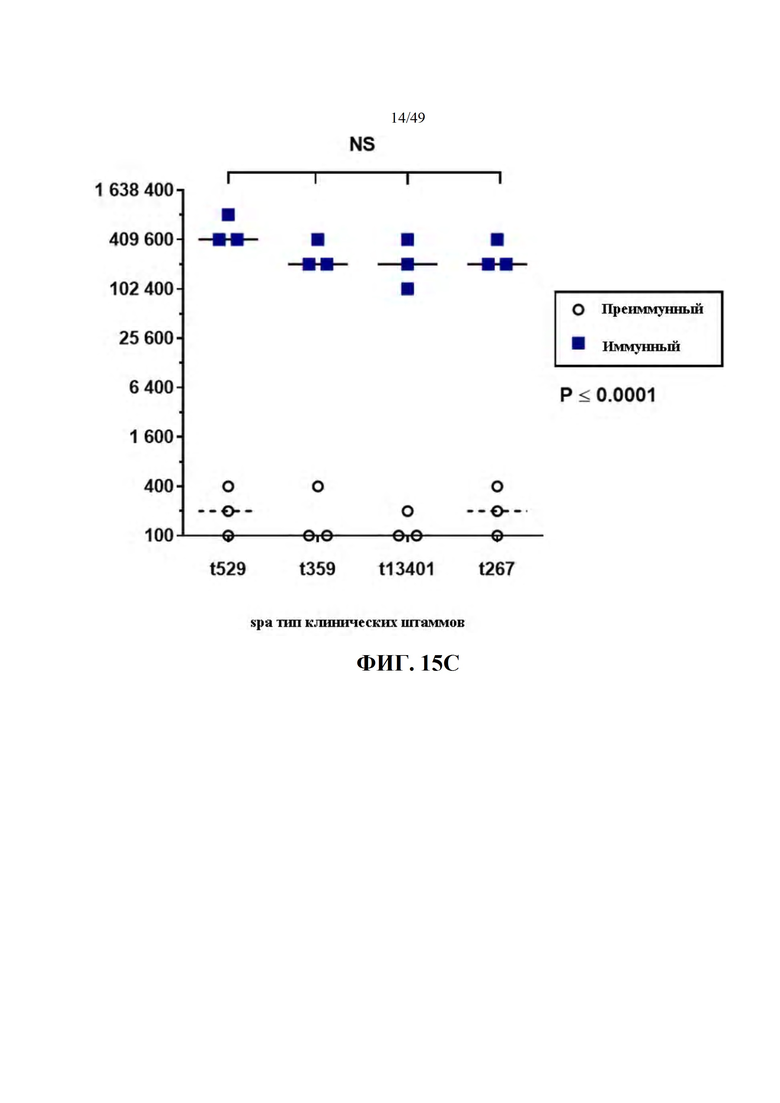
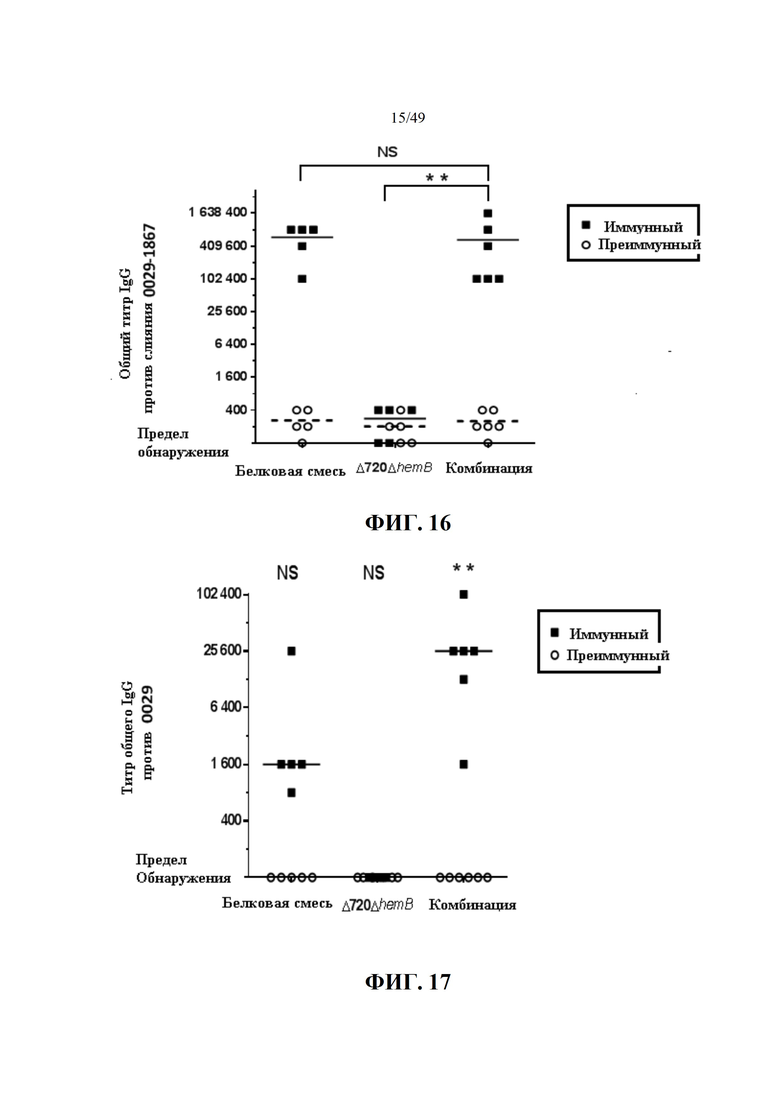
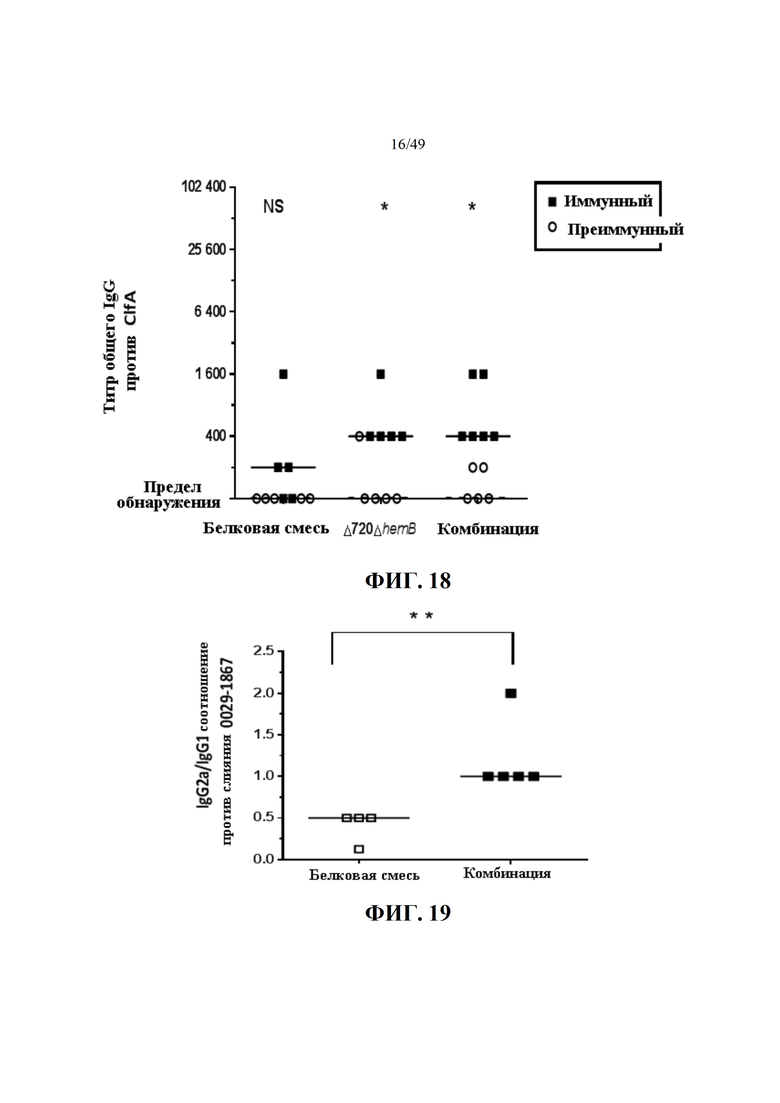
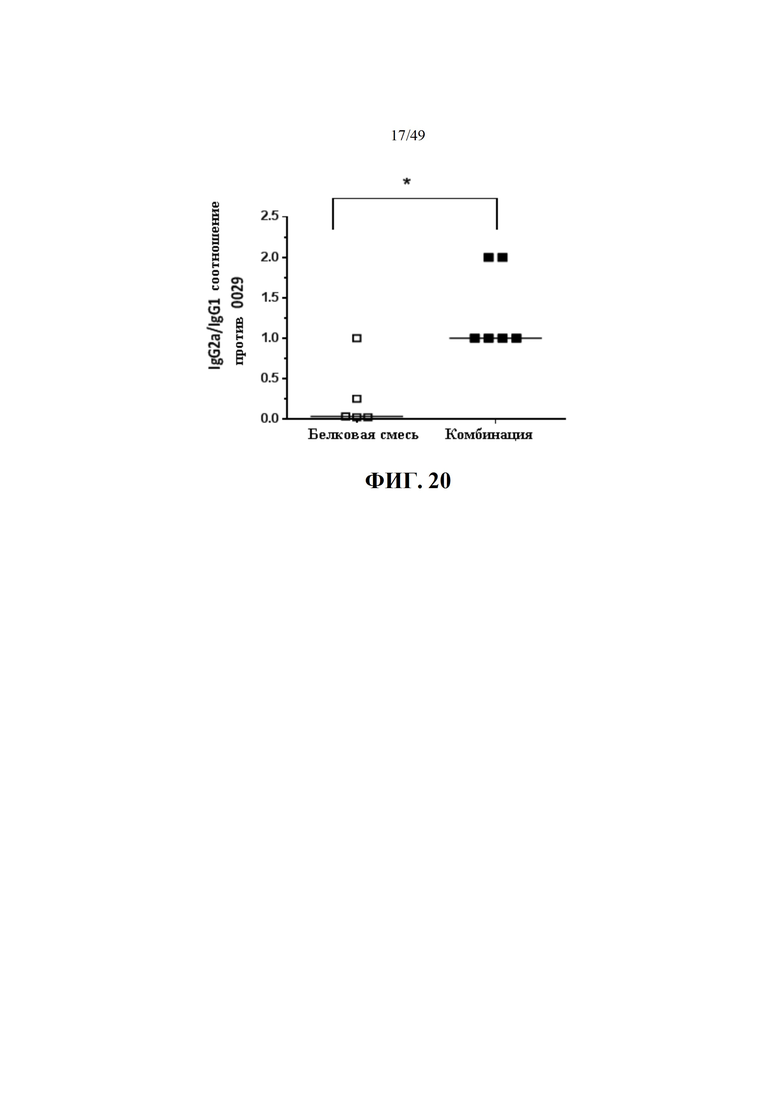
































Изобретение относится к области биотехнологии, конкретно к получению вакцинных конструктов против стафилококковой интрамаммарной инфекции (IMI). Полученные конструкты, содержащие аминокислотную последовательность SEQ ID NO: 3 или SEQ ID NO: 55, могут быть использованы для эффективной профилактики или лечения стафилококковой интрамаммарной инфекции (IMI), вызванной Staphylococcus aureus, у млекопитающего. 7 н. и 7 з.п. ф-лы, 27 ил., 6 табл., 27 пр.
1. Вакцинный конструкт против стафилококковой интрамаммарной инфекции (IMI), содержащий аминокислотную последовательность SEQ ID NO: 3 или SEQ ID NO: 55.
2. Выделенная молекула нуклеиновой кислоты, кодирующая конструкт, определенный в п. 1.
3. Вектор экспрессии, содержащий выделенную молекулу нуклеиновой кислоты, определенную в п. 2.
4. Клетка-хозяин, содержащая вектор, определенный в п. 3.
5. Клетка по п. 4, которая представляет собой живую аттенуированную форму Staphylococcus aureus.
6. Клетка по п. 5, где живая аттенуированная форма Staphylococcus aureus имеет стабилизированный фенотип варианта мелких колоний (SCV).
7. Клетка по п. 5, где живая аттенуированная форма Staphylococcus aureus, имеющая стабилизированный фенотип SCV, представляет собой ΔhemBΔ720 S. aureus.
8. Композиция для профилактики или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего, содержащая:
(a) конструкт по п. 1;
(b) молекулу нуклеиновой кислоты по п. 2;
(с) клетку-хозяин по любому из пп. 4 - 6; или
(d) комбинацию по меньшей мере двух из (а) - (с),
в эффективном количестве.
9. Композиция по п. 8, дополнительно содержащая живую аттенуированную форму Staphylococcus aureus, где живая аттенуированная форма Staphylococcus aureus имеет стабилизированный фенотип варианта мелких колоний (SCV).
10. Композиция по п. 8 или 9, дополнительно содержащая адъювант, где адъювант содержит алюм, масло, сапонин, циклический дигуанозин-5’-монофосфат (c-di-GMP), полифосфазин, индолицидин, патоген-ассоциированные молекулярные паттерны (PAMPS), липосому или комбинацию по меньшей мере двух из них.
11. Применение конструкта по п. 1 для профилактики или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего.
12. Применение по п. 11, где указанная стафилококковая IMI вызвана одним или более штаммами Staphylococcus aureus.
13. Применение по п. 11 или 12, где указанным млекопитающим является корова.
14. Набор для профилактики или лечения стафилококковой интрамаммарной инфекции (IMI) у млекопитающего, включающий композицию по любому из пп.8 - 10.
| RU 2013154779 A, 20.06.2015 | |||
| ИММУНОГЕННАЯ КОМПОЗИЦИЯ ДЛЯ ПРИМЕНЕНИЯ В ВАКЦИНАЦИИ ПРОТИВ СТАФИЛОКОККОВ | 2009 |
|
RU2521501C2 |
| SPENCER A | |||
| J | |||
| et al., Fusion of the Mycobacterium tuberculosis antigen 85A to an oligomerization domain enhances its immunogenicity in both mice and non-human primates, PloS one, 2012, V | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| CHEN X | |||
| et al., Fusion protein linkers: property, design and functionality, Advanced | |||
Авторы
Даты
2022-03-15—Публикация
2017-10-20—Подача