Область техники
Изобретение относится к биотехнологии, а именно, к новым ферментам - Cas нуклеазам систем CRISPR-Cas, применяемым для разрезания ДНК и редактирования геномов различных организмов. Данная технология может применяться в будущем для генной терапии наследственных заболеваний человека, а также для редактирования геномов других организмов.
Работа выполнена при финансовой поддержке Министерства Науки и Высшего образования Российской Федерации в рамках Соглашения №075-15-2019-1661 от 31.10.2019.
Уровень техники
Изменение последовательности ДНК - одна из актуальных задач биотехнологии на сегодняшний день. Редактирование и изменение геномов эукариотических и прокариотических организмов, а также манипуляции с ДНК in vitro, требуют направленного внесения двунитевых разрывов в последовательности ДНК.
Для решения этой задачи в настоящее время используют следующие методики: искусственные нуклеазные системы, содержащей домены типа «цинковые пальцы» (ZFN), эффекторные нуклеазы, подобные активатору транскрипции (TALEN-системы) и бактериальные CRISPR-Cas системы. Первые два метода требуют трудозатратой оптимизации аминокислотной последовательности нуклеазы для узнавания конкретной последовательности ДНК. В отличие от них в случае CRISPR-Cas систем структурами, узнающими ДНК мишень, являются не белки, а короткие направляющие РНК. Разрезание конкретной ДНК мишени не требует синтеза нуклеазы или ее гена de novo, а обеспечивается за счет использования направляющих РНК, комплементарных целевой последовательности. Это делает CRISPR-Cas системы удобными и эффективными инструментами разрезания различных ДНК-последовательностей. Методика позволяет осуществлять единовременное разрезание ДНК в нескольких участках при использовании направляющих РНК разной последовательностей. Такой подход используется в том числе для одновременного изменения нескольких генов в эукариотических организмах.
По своей природе CRISPR-Cas системы являются иммунными системами прокариот, способными высоко специфично вносить разрывы в генетический материал вирусов (Mojica F. J. M., 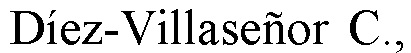 García-Martínez J. & Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). Аббревиатура CRISPR-Cas расшифровывается как «Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR associated genes» (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575), что переводе с английского обозначает «короткие палиндромные повторы, регулярно расположенные группами, и ассоциированные с ними гены». Все CRISPR-Cas системы состоят из CRISPR кассет и генов, кодирующих различные Cas белки (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). CRISPR кассеты состоят из последовательностей спейсеров, каждый из которых имеет уникальную нуклеотидную последовательность, и повторяющихся палиндромных повторов (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). В результате транскрипции CRISPR кассет и их последующего процессинга образуются направляющие крРНК, которые вместе с Cas белками формируют эффекторный комплекс
García-Martínez J. & Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). Аббревиатура CRISPR-Cas расшифровывается как «Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR associated genes» (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575), что переводе с английского обозначает «короткие палиндромные повторы, регулярно расположенные группами, и ассоциированные с ними гены». Все CRISPR-Cas системы состоят из CRISPR кассет и генов, кодирующих различные Cas белки (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). CRISPR кассеты состоят из последовательностей спейсеров, каждый из которых имеет уникальную нуклеотидную последовательность, и повторяющихся палиндромных повторов (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). В результате транскрипции CRISPR кассет и их последующего процессинга образуются направляющие крРНК, которые вместе с Cas белками формируют эффекторный комплекс
(Brouns S. J., Jore M. M., Lundgren M., Westra E. R., Slijkhuis R. J., Snijders A. P., Dickman M. J., Makarova K. S., Koonin E. V. & van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes // Science. - 2008. - Т. 321. - №. 5891. - С. 960-964). За счет комплементарного спаривания крРНК с целевым участком ДНК, именуемым протоспейсером, Cas-нуклеаза узнает ДНК-мишень и высоко специфично вносит в нее разрыв.
В зависимости от типа эффекторов все CRISPR-Cas системы разделяют на два класса. И далее эти классы подразделяются на шесть различных типов (от I до VI). CRISPR-Cas системы класса 2 представлены одиночным белком-эффектором.
CRISPR системы с эффекторным белком Cas12a (ранее обозначаемым как cpf1) являются представителями класса 2 и типа V. Система CRISPR-Cas12a типа отличается простотой состава и механизма работы. В отличие от систем CRISPR-Cas9 для ее функционирования необходимо формирование эффекторного комплекса, помимо самой нуклеазы включающего только одну направляющую РНК - крРНК (crRNA). Трейсерная РНК (tracrRNA, трРНК), комплементарно спаривающаяся с участком крРНК, при этом отсутствует. Эффекторный белок Cas12a является РНК-зависимой ДНК эндонуклеазой, вносящей разрывы в комплементарные нити целевой ДНК, таким образом образуя двунитевой разрыв ДНК. Продукты расщепления нуклеазой Cas12a имеют 5'-липкие концы, что также отличается от продуктов расщепления нуклеазой Cas9, образующей «тупые» концы (Zetsche B., Gootenberg J. S., Abudayyeh O. O., Slaymaker I. M., Makarova K. S., Essletzbichler P., Volz S. E., Joung J., van der Oost J., Regev A., Koonin E. V. & Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system // Cell. - 2015. - Т. 163. - № 3. - С. 759-771). Образующиеся 5'-липкие концы делают данную нуклеазу более удобным инструментом геномного редактирования, так как они позволяют осуществлять вставку фрагментов ДНК, обладающих комплементарными липкими участками, посредством лигации.
Эффекторному рибонуклеиновому комплексу, состоящему из Cas12a и крРНК, для распознавания и последующего гидролиза ДНК помимо комплементарного соответствия спейсера крРНК и протоспейсера необходимо присутствие PAM (от англ. «PAM» - Protospacer Adjusted Motif) на ДНК мишени (Shah S. A., Erdmann S., Mojica F. J. M. & Garrett R. A. Protospacer recognition motifs // RNA Biology. - 2013. - Т. 10. - № 5. - С. 891-899). PAM представляет собой строго определенную последовательность из нескольких нуклеотидов, расположенных в случае CRISPR-Cas12a систем вплотную к 5'-концу протоспейсера на нетаргетной цепи. При отсутствии PAM гидролиза связей в ДНК с образованием двунитевого разрыва не происходит. Необходимость присутствия PAM последовательности на мишени повышает специфичность узнавания, но в то же время накладывает ограничение в выборе целевых участков ДНК, в которые необходимо внести разрыв. Таким образом, наличие нужной PAM последовательности, фланирующей ДНК-мишень с 5'-конца, является характеристикой, ограничивающей применение CRISPR-Cas12a систем на любых участках ДНК.
На сегодняшний день описано порядка 40 различных нуклеаз V-A типа. Что значительно уступает количеству описанных представителей упомянутых ранее Cas9 нуклеаз. При этом активность в клетках человека показана только для трети охарактеризованных Cas12a нуклеаз: ArCas12a, AsCas12a, BsCas12a, CeCas12a, FnCas12a, HkCas12a, LbCas12a, MbCas12a, Mb2Cas12a, Mb3Cas12a, PiCas12a, PrCas12a, PxCas12a, TsCas12a (Teng F. et al. Enhanced mammalian genome editing by new Cas12a orthologs with optimized crRNA scaffolds // Genome Biology. - 2019. - Т. 20. - № 15; Chen P. et al. A Cas12a ortholog with stringent PAM recognition followed by low off-target editing rates for genome editing // Genome Biology. - 2020. - Т. 21. - № 78; Zetsche B. et al. A Survey of Genome Editing Activity for 16 Cas12a Orthologs // The Keio Journal of Medicine. - 2020. - Т. 69. - № 3. - С. 59-65).
Использование новых разнообразных CRISPR-Cas белков необходимо для обеспечения возможности изменения любого участка ДНК, как in vitro, так и в геноме живых организмов. Несмотря на известность ряда способов разрезания ДНК и изменения последовательности геномной ДНК, на сегодняшний день сохраняется потребность в новых эффективных инструментах для модификации ДНК в различных организмах и в строго определенных местах последовательности ДНК.
Сущность изобретения
Задачей настоящего изобретения является создание новых инструментов для изменения последовательности геномной ДНК одноклеточных или многоклеточных организмов на основе системы CRISPR-Cas12a. Поиск новых ферментов Cas12a позволит расширить арсенал имеющихся средств для образования двунитевого разрыва в необходимых, строго определенных местах в молекулах ДНК разных организмов. Для решения этой задачи авторами была охарактеризована ранее биоинформатически предсказанная система из бактерии Sedimentisphaera cyanobacteriorum L21-RPul-D3.
CRISPR нуклеаза V типа ScCas12a, которая может быть применена для внесения направленных изменений в геном как этого, так и других организмов. Существенным признаком, отличающим настоящее изобретение, является небольшой процент сходства ScCas12a с ранее описанными нуклеазами данного типа по аминокислотной последовательности.
Указанная задача решается путем применения белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного непосредственно после нуклеотидной последовательности 5'-(T/C)TTN-3' в указанной молекуле ДНК.
Указанная задача также решается путем создания способа изменения последовательности геномной ДНК одноклеточного или многоклеточного организма, включающего введение в по меньшей мере одну клетку этого организма эффективного количества: а) либо белка, содержащего аминокислотную последовательность SEQ ID NO: 1, либо нуклеиновой кислоты, кодирующей белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и б) либо крРНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, непосредственно примыкающей к нуклеотидной последовательности 5'-(T/C)TTN-3', и взаимодействующей с указанным белком после образования дуплекса, либо последовательности ДНК, кодирующей указанную крРНК; при этом взаимодействие указанного белка с крРНК и нуклеотидной последовательностью 5'-(T/C)TTN-3' приводит к образованию двунитевого разрыва в последовательности геномной ДНК, непосредственно примыкающей к последовательности 5'-(T/C)TTN-3'. В некоторых вариантах изобретения данный способ характеризуется тем, что дополнительно включающий введение экзогенной последовательности ДНК одновременно с крРНК.
Изобретение может быть использовано как для разрезания целевой ДНК in vitro, так и для модификации генома какого-либо живого организма. Модификация генома может проводиться прямым способом - разрезанием генома в соответствующем сайте, а также вставкой экзогенной последовательности ДНК за счет гомологичной репарации.
В качестве экзогенной последовательности ДНК может быть использован любой участок двунитевой или однонитевой ДНК из генома организма, отличного от организма, используемого при введении (или смесь таких участков между собой и с другими фрагментами ДНК), при этом этот участок (или смесь участков) предназначен для интеграции в место двуцепочечного разрыва в целевой ДНК, образованного под действием нуклеазы ScCas12a. В некоторых вариантах изобретения в качестве экзогенной последовательности ДНК может быть использован участок двуцепочечной ДНК из генома организма, используемого при введении белка ScCas12a, но при этом измененный мутациями (заменой нуклеотидов), а также вставками или делециями одного, или нескольких нуклеотидов.
Техническим результатом настоящего изобретения является повышение универсальности доступных CRISPR систем, позволяющее использовать нуклеазы для разрезания геномной или плазмидной ДНК в большем количестве специфических сайтов и специфических условий.
Краткое описание рисунков
Фиг. 1. Схема устройства локуса CRISPR ScCas12a системы.
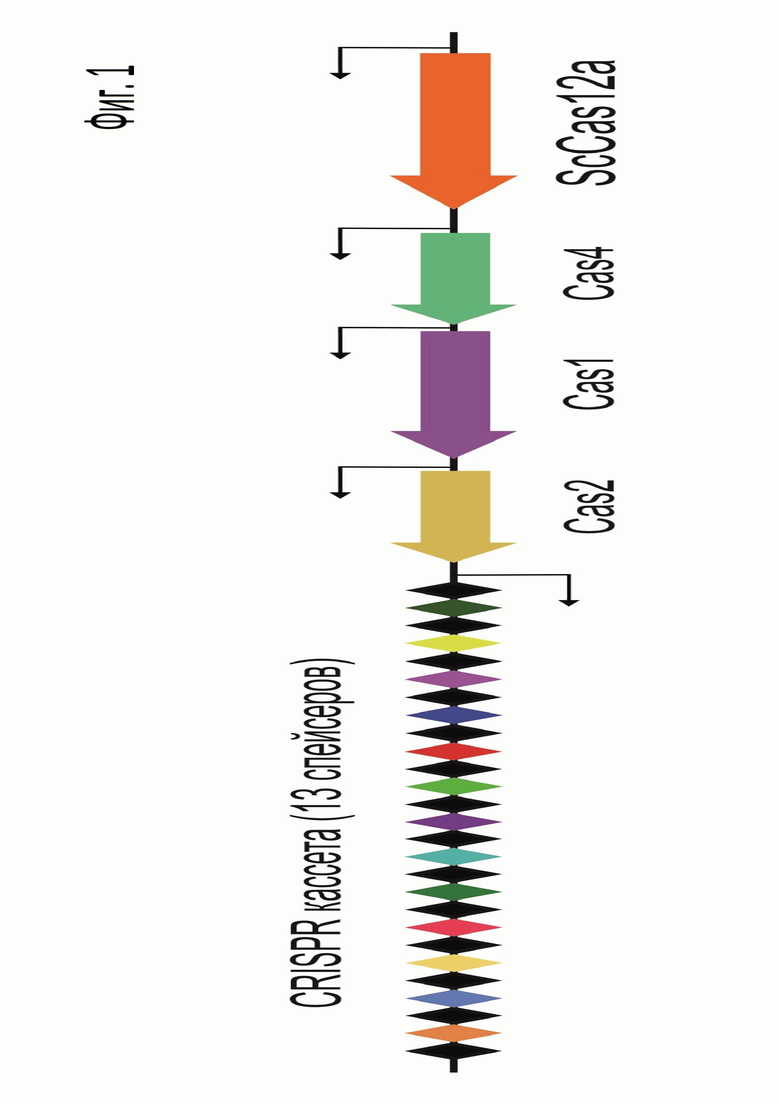
Фиг. 2. Результаты поиска протоспейсеров геномов бактериофагов, комплементарных последовательностям спейсеров CRISPR кассеты ScCas12a системы с использованием программы CRISPRTarget. Красными рамками отмечены предполагаемые участки с последовательностями PAM.
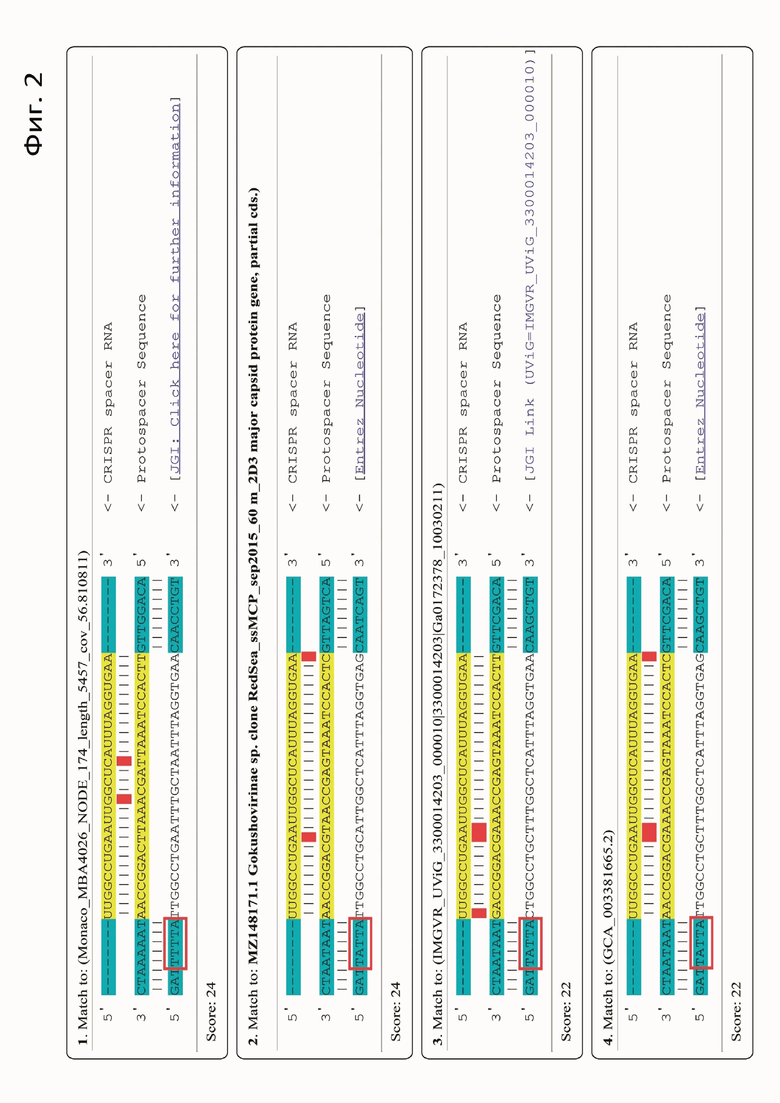
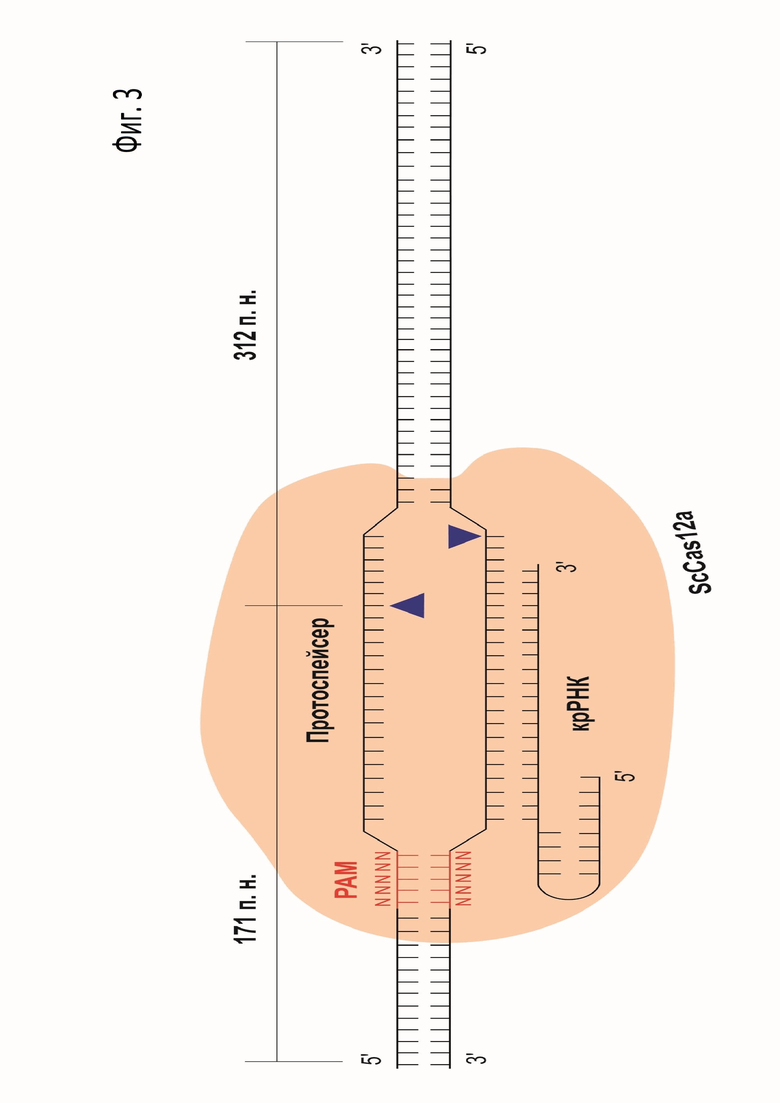
Фиг. 3. PAM скрининг in vitro. Схема эксперимента.
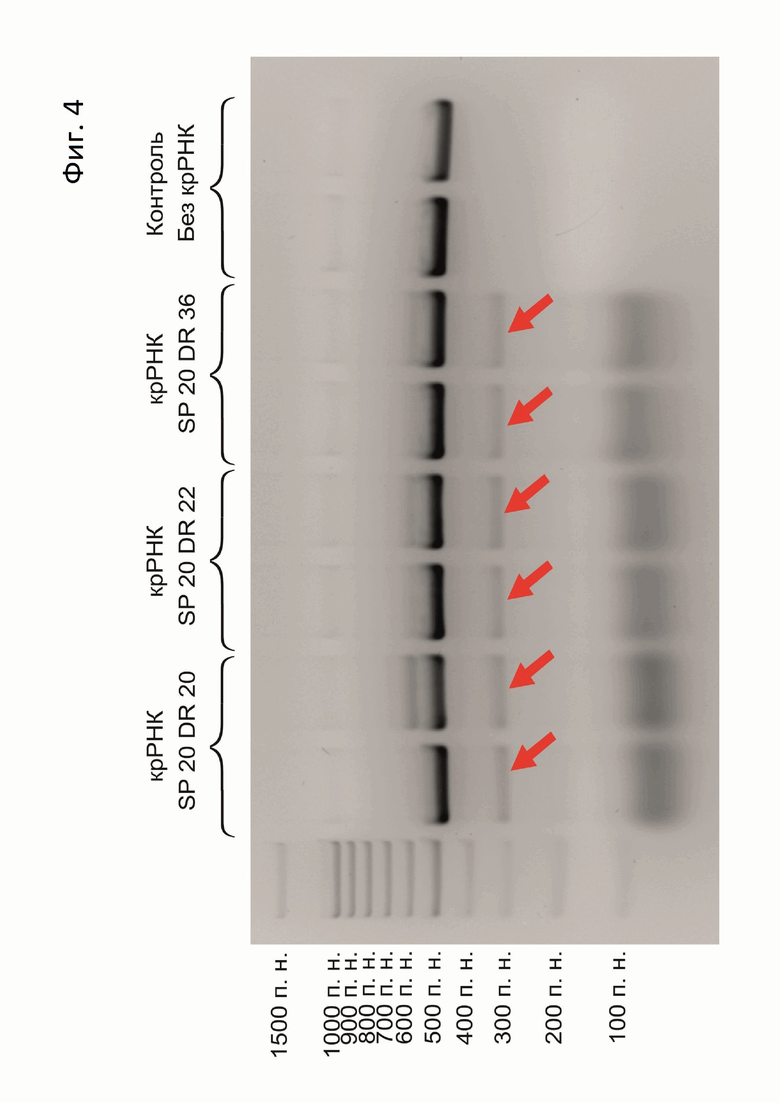
Фиг. 4. Разрезание нуклеазой ScCas12a фрагментов 5N библиотеки при использовании крРНК с разными длинами прямого повтора (DR) и длиной спейсерной части 20 нуклеотидов (SP 20). Красными стрелками отмечено положение разрезанного фрагмента.
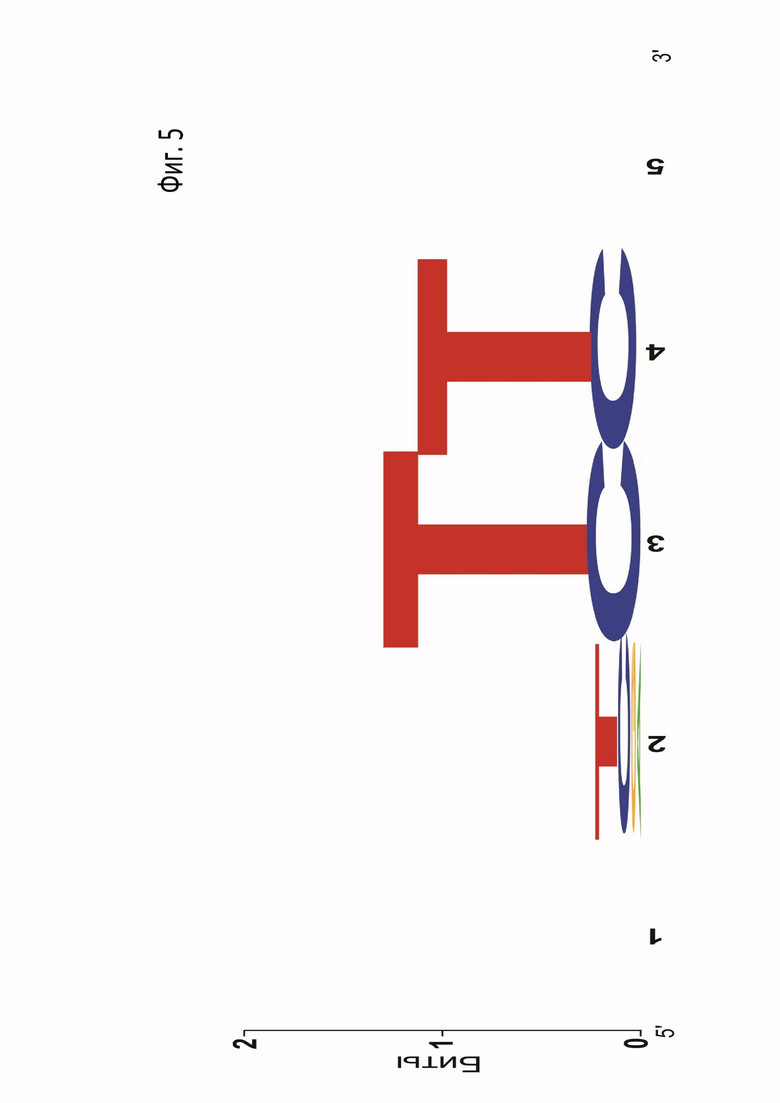
Фиг. 5. PAM Logo нуклеазы ScCas12a, полученное в результате in vitro скрининга 5N PAM библиотек.
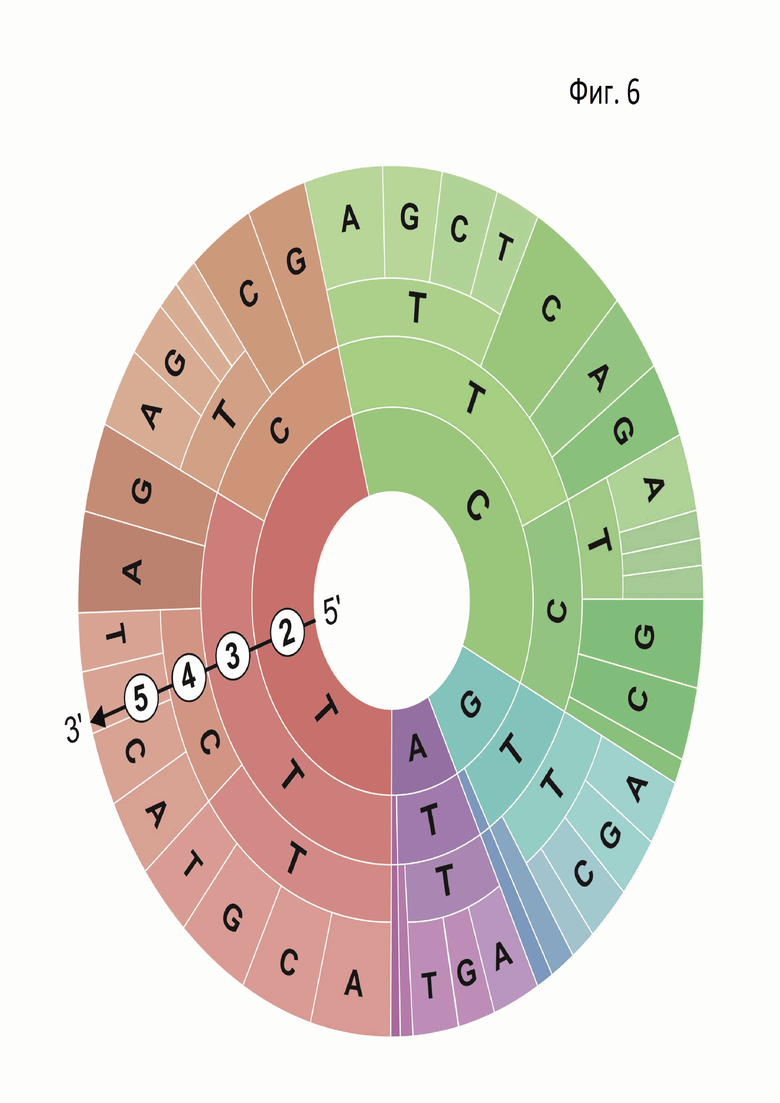
Фиг. 6. Круговая диаграмма PAM нуклеазы ScCas12a («PAM колесо») для 2, 3, 4 и 5 позиций PAM.
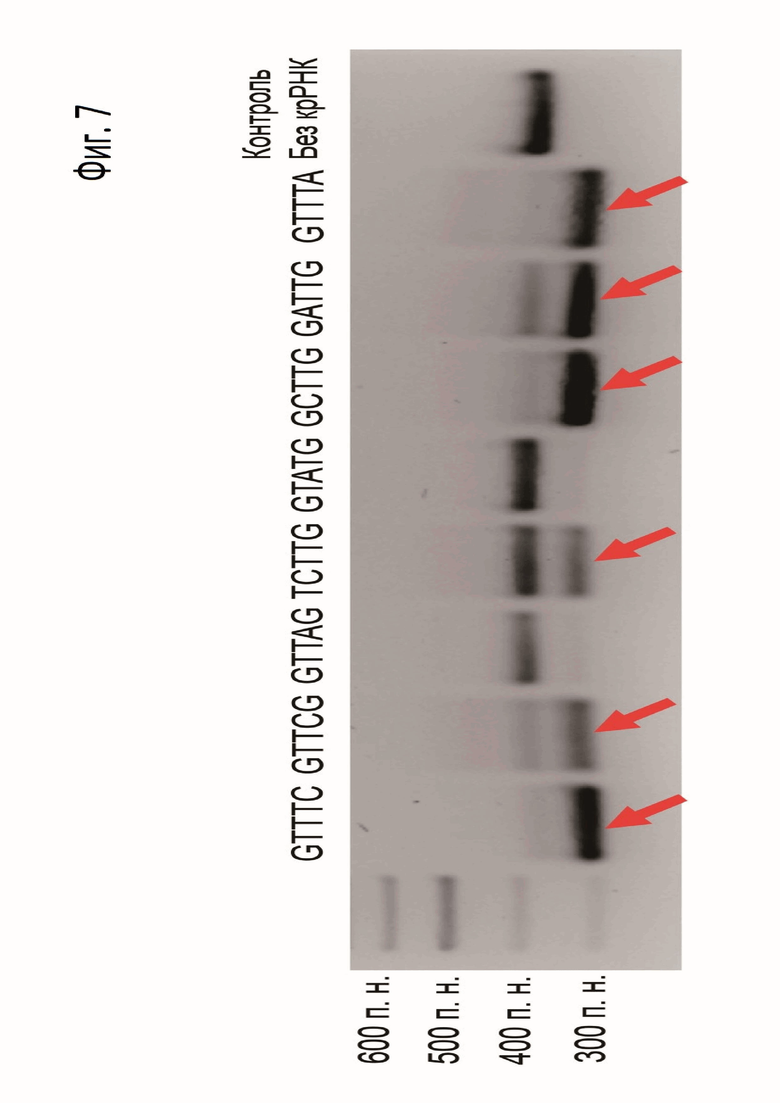
Фиг. 7. Разрезание нуклеазой ScCas12a ДНК-мишеней с конкретными PAM последовательностями (1-5 PAM позиции). Красными стрелками отмечено положение разрезанного фрагмента ДНК.
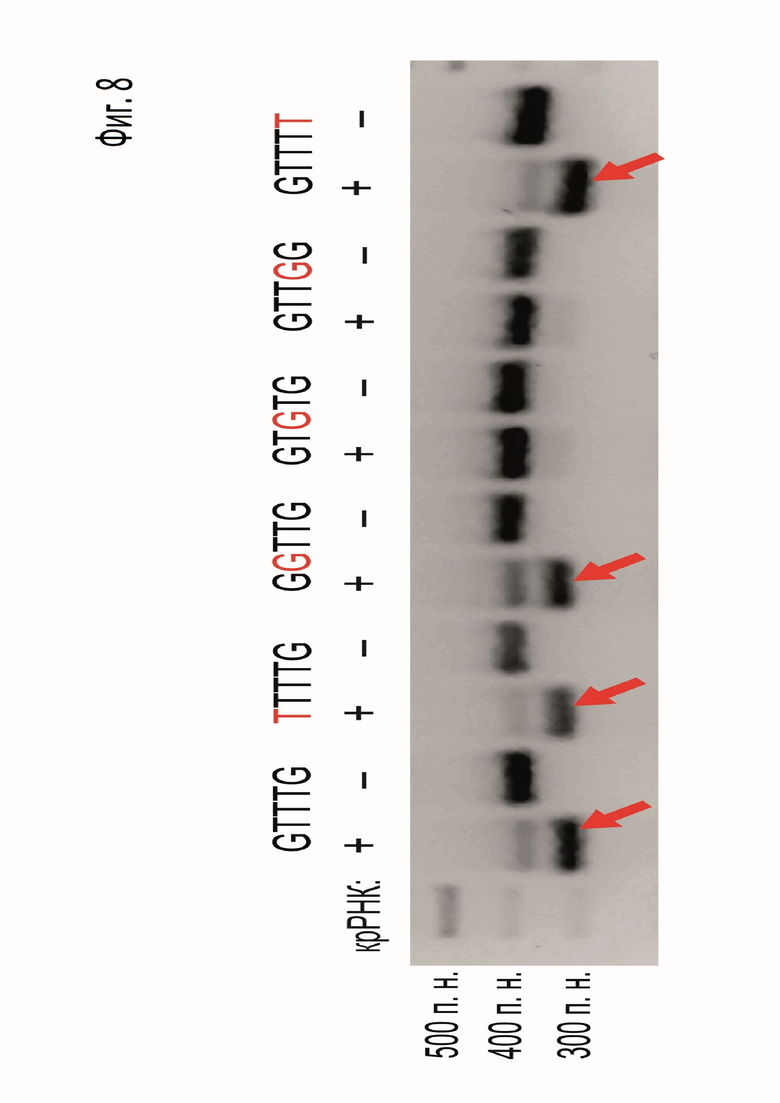
Фиг. 8. Проверка значимости нуклеотидных позиций в PAM последовательности ScCas12a.
Красными стрелками отмечено положение разрезанного фрагмента ДНК.
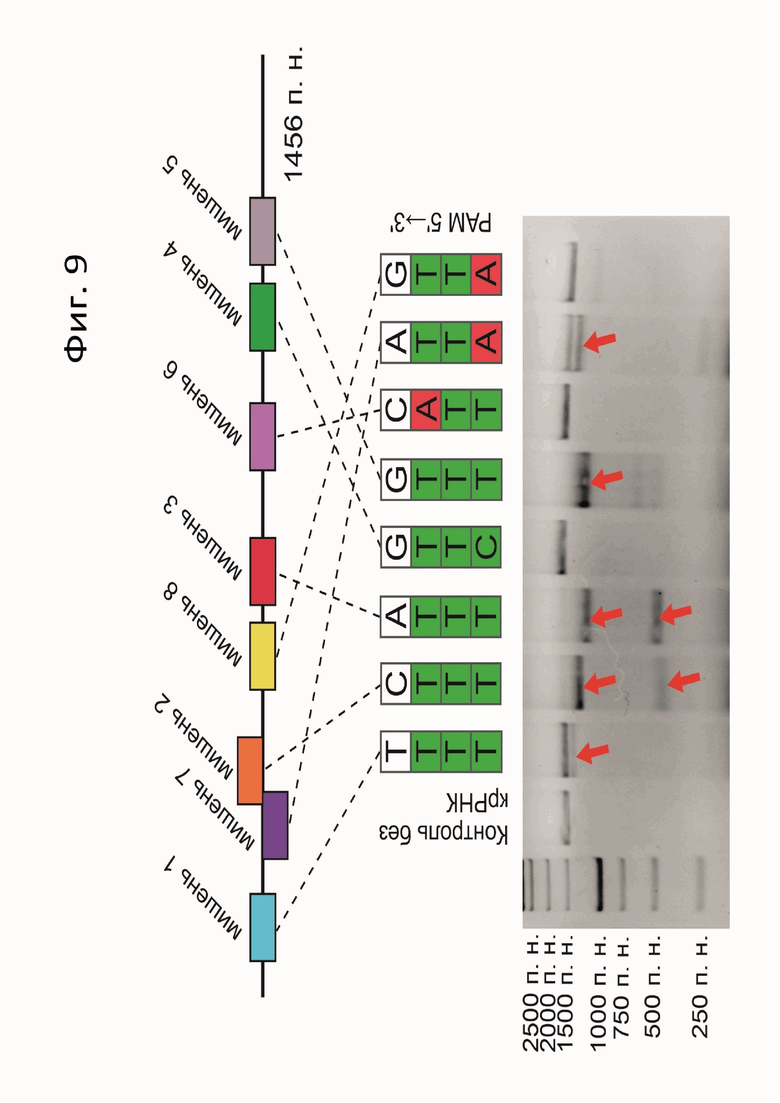
Фиг. 9. Разрезание различных сайтов ДНК с помощью белка ScCas12a. Красными стрелками отмечено положение разрезанного фрагмента ДНК.
Фиг. 10. Выравнивание аминокислотных последовательностей белков ScCas12a и TsCas12a из Thiomicrospira sp. XS5. Желтым цветом выделен домен, отвечающий за взаимодействие с PAM последовательностью.
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Используемый здесь термин «процент гомологии двух последовательностей» эквивалентен термину «процент идентичности двух последовательностей». Идентичность последовательностей определяется на основании референсной последовательности. Алгоритмы для анализа последовательности известны в данной области, такие как BLAST, описанный в Altschul et al. (Basic local alignment search tool // Journal of Molecular Biology. - 1990. - Т. 215. - С. 403-410). Для целей настоящего изобретения для определения уровня идентичности и сходства между нуклеотидными последовательностями и аминокислотными последовательностями может быть использовано сравнение нуклеотидных и аминокислотных последовательностей, производимое с помощью пакета программ BLAST, предоставляемого National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov/blast) с использованием содержащего разрывы выравнивания со стандартными параметрами. Процент идентичности двух последовательностей определяется числом положений идентичных аминокислот в этих двух последовательностях с учетом числа пробелов и длины каждого пробела, которые необходимо ввести для оптимального сопоставления двух последовательностей путем выравнивания. Процент идентичности равен числу идентичных аминокислот в данных положениях с учетом выравнивания последовательностей, разделенному на общее число положений и умноженному на 100.
Термин «специфически гибридизуется» относится к ассоциации между двумя одноцепочечными молекулами нуклеиновых кислот или в достаточной степени комплементарными последовательностями, что разрешает такую гибридизацию в предопределенных условиях, обычно использующихся в данной области.
Фраза «двунитевой разрыв, расположенный после нуклеотидной последовательности РАМ» означает, что двунитевой разрыв в целевой последовательности ДНК будет произведен на расстоянии от 5 до 25 нуклеотидов после нуклеотидной последовательности РАМ.
Под экзогенной последовательностью ДНК, вводимой одновременно с крРНК, следует понимать последовательность ДНК, подготовленную специально для специфической модификации двуцепочечной целевой ДНК в месте разрыва, определяемого специфичностью крРНК. Подобной модификацией может быть, например, вставка или делеция определенных нуклеотидов в месте разрыва целевой ДНК. Экзогенной ДНК может служить как участок ДНК из другого организма, так и участок ДНК из того же организма, что и целевая ДНК.
Под белком, содержащим определенную аминокислотную последовательность следует понимать белок, имеющий аминокислотную последовательность, составленную из указанной аминокислотной последовательности и, возможно, других последовательностей, соединенных пептидными связями с указанной аминокислотной последовательностью. Примером других последовательностей может служить последовательность сигнала ядерной локализации (NLS), или другие последовательности, обеспечивающие повышенную функциональность для указанной аминокислотной последовательности.
Под эффективным количеством вводимых в клетку белка и РНК следует понимать такое количество белка и РНК, которое при попадании в указанную клетку будет способно образовать функциональный комплекс, то есть комплекс, который будет специфически связываться с целевой ДНК и производить в ней двунитевой разрыв в месте, определяемом крРНК и РАМ последовательностью на ДНК. Эффективность этого процесса может быть оценена при помощи анализа целевой ДНК, выделенной из указанной клетки с помощью стандартных методов, известных специалистам.
Доставка белка и крРНК в клетку может быть осуществлена различными способами, известными специалистам. Например, белок может быть доставлен в виде ДНК-плазмиды, которая кодирует ген этого белка, как мРНК для трансляции этого белка в цитоплазме клетки, или как рибонуклеопротеидный комплекс, включающий этот белок и крРНК.
Нуклеиновая кислота, кодирующая компоненты системы, может быть введена в клетку, непосредственно или опосредованно: за счет трансфекции или трансформации клеток известными специалистам способами, за счет использования рекомбинантного вируса, за счет манипуляций с клеткой, таких как микроинъекция ДНК и т. п.
Молекула нуклеиновой кислоты, кодирующая белок, который необходимо ввести в клетку, может быть интегрирована в хромосому или может представлять собой внехромосомно реплицирующуюся ДНК. В некоторых вариантах для обеспечения эффективной экспрессии гена белка с вводимой в клетку ДНК необходимо изменить последовательность этой ДНК в соответствии с типом клетки в целях оптимизации кодонов при экспрессии, обусловленное неравномерностью частот встречаемости синонимичных кодонов в кодирующих областях генома различных организмов. Оптимизация кодонов необходима для увеличения экспрессии в клетках животных, растений, грибов или микроорганизмов.
Для функционирования белка, имеющего последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1, в эукариотической клетке необходимо, чтобы этот белок оказался в ядре этой клетки. Поэтому, в некоторых вариантах изобретения, для образования двунитевых разрывов в целевой ДНК используют белок, имеющий последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1, и который дополнительно модифицирован с одного или с обоих концов добавлением одного или нескольких сигналов ядерной локализации. Например, может быть использован сигнал ядерной локализации из вируса SV40. Для эффективной доставки в ядро сигнал ядерной локализации может быть отделен от основной последовательности белка спейсерной последовательностью, например, описанной в Shen B. et al. (Generation of gene-modified mice via Cas9/RNA-mediated gene targeting // Cell Research. - 2013. - Т. 23. - №5. - С. 720-723). Также, в других вариантах осуществления, может быть использован другой сигнал ядерной локализации, или альтернативный метод доставки указанного белка в ядро клетки.
Настоящее изобретение охватывает применение белка из организма Sedimentisphaera cyanobacteriorum, гомологичного ранее охарактеризованным белкам Cas12a, для внесения двуцепочечных разрывов в молекулы ДНК в строго определенных положениях. Использование CRISPR нуклеаз для внесения направленных изменений в геном имеет ряд преимуществ. Во-первых, специфичность действия системы определяется последовательностью крРНК, что позволяет использовать один тип нуклеазы для всех локусов-мишеней. Во-вторых, методика позволяет доставить в клетку сразу несколько РНК, комплементарных разным генам-мишеням, что позволяет осуществлять единовременное изменение сразу нескольких генов.
ScCas12a - Cas нуклеаза, найденная в бактериях Sedimentisphaera cyanobacteriorum L21-RPul-D3, выделенных из гиперсоленого озера. Оптимальная температура роста данных бактерий соответствует 35°C, что может свидетельствовать о вероятном оптимуме для работы данной нуклеазы на температурах, приближенных к температуре тела человека. Sedimentisphaera cyanobacteriorum CRISPR-Cas12a система (далее CRISPR ScCas12a) относится к V-A типу CRISPR Cas систем и состоит из CRISPR кассеты, несущей 14 прямых повторов (direct repeats, DR) последовательностью
5'-GTCTAAAGGCCTTATAAAATTTCTACTGTCGTAGAT-3', разделенных последовательностями уникальных спейсеров. К CRISPR кассете прилегают гены белков адаптационного модуля Cas2, Cas1, Cas4 и эффекторного Cas12a белка, ScCas12a (Фиг. 1).
Обнаруженные в CRISPR кассете спейсеры были проанализировали при помощи программы CRISPRTarget (Biswas A., Gagnon J. N., Brouns S. J. J., Fineran P. C. & Brown C. M. CRISPRTarget // RNA Biology. - 2013. - Т. 10. - №5. - С. 817-827). Анализ участков PAM регионов в последовательностях бактериофагов, соответствующих проанализованным спейсерам, указывает на вероятно T-богатый PAM нуклеазы ScCas12a (Фиг. 2).
Знание характерной архитектуры РНК-Cas белкового комплекса систем типа V-5 позволила предсказать направление транскрипции CRISPR кассеты: пре-крРНК транскрибируется в том же направлении, что и Cas гены (Фиг. 1).
Таким образом, анализ последовательности локуса ScCas12a позволил предсказать последовательности крРНК:
5'-GUCUAAAGGCCUUAUAAAAUUUCUACUGUCGUAGAUxxxxxxxxxxxxxxxxxxxx-3'
Жирным шрифтом обозначена последовательность прямого повтора DR длиной 36 п. н. Знаками «x» обозначены нуклеотиды вариабельной спейсерной части крРНК.
Для проверки активности ScCas12a нуклеазы и определения требуемого ScCas12a PAM мотива были проведены эксперименты по воссозданию реакции разрезания ДНК in vitro. Для определения PAM последовательности белка ScCas12a использовали in vitro разрезание двунитевых PAM библиотек. Для этого необходимо было получить все компоненты эффекторного комплекса ScCas12a: крРНК и нуклеазу в рекомбинантной форме. Определенная ранее последовательность крРНК была синтезирована in vitro. Синтез осуществляли с помощью набора NEB HiScribe T7 RNA synthesis. Двунитевые ДНК библиотеки представляли собой фрагменты размером 483 п. н., содержащие последовательность протоспейсера, фланкированную рандомизированными пятью нуклеотидами (5'-NNNNN-3') c 5' конца:

Для разрезания этой мишени использовали следующие крРНК:
Длина спейсера 20 (SP 20), длина прямого повтора 20 (DR 20)
5'-AAAUUUCUACUGUCGUAGAUgugcucaaugaaaggagaua-3' (SEQ ID NO: 3)
Длина спейсера 20 (SP 20), длина прямого повтора 22 (DR 22)
5'-UAAAAUUUCUACUGUCGUAGAUgugcucaaugaaaggagaua-3' (SEQ ID NO: 4)
Длина спейсера 20 (SP 20), длина прямого повтора 36 (DR 36)
5'-GUCUAAAGGCCUUAUAAAAUUUCUACUGUCGUAGAUgugcucaaugaaaggagaua-3' (SEQ ID NO: 5)
Жирным шрифтом выделена последовательность крРНК, комплементарная протоспейсеру (целевой ДНК последовательности).
Для получения рекомбинантного белка ScCas12a его ген был клонирован в плазмиду pET21a-6xHis. В качестве кодирующей ген ДНК, использовалась ДНК, амплифицированная с геномной ДНК Sedimentisphaera cyanobacteriorum L21-RPul-D3, заказанной из коллекции DSMZ (Leibniz Institute DSMZ-German Collection of Microorganisms and Cell Cultures GmbH). Клетки E. coli Rosetta были трансформированы полученной плазмидой pET21a-6xHis-ScCas12a.
500 мкл ночной культуры разводили в 500 мл среды LB, и растили клетки при температуре 37°C до достижения оптической плотности 0.6 отн. ед. Синтез целевого белка индуцировали добавлением ИПТГ до концентрации 1 мМ, после чего клетки инкубировали при температуре 16°C в течение 16 часов. Затем проводили центрифугирование клеток на скорости 5000 g в течение 30 минут, полученные осадки клеток замораживали при температуре -20°C.
Осадки размораживали на льду в течение 30 минут, ресуспензировали в 15 мл лизисного буфера (Tris-HCl 40 мМ pH 8, 500 мМ NaCl, β-меркаптоэтанол 1 мМ, имидазол 10 мМ) с добавлением 15 мг лизоцима и снова инкубировали на льду в течение 30 минут. Затем клетки разрушали воздействием ультразвука в течение 30 минут и центрифугировали в течение 60 минут на скорости 16000 g. Полученный супернатант пропускали через фильтр 0.2 мкм и наносили на колонку HisTrap HP 1 mL (GE Healthcare) на скорости 1 мл/мин.
Хроматографию проводили при помощи FPLC хроматографа AKTA (GE Healthcare) на скорости 0.5 мл/мин. Колонку с нанесенным белком промывали 20 мл лизисного буфера с добавлением 10 мМ имидазола, после чего белок смывали лизисным буфером с добавлением 300 мМ имидазола.
Затем, фракцию белка, полученную в ходе афинной хроматографии, пропускали через гель-фильтрационную колонку Superdex 200 10/300 GL, уравновешенную следующим буфером: Tris-HCl 40 мМ pH 8, 500 мМ NaCl, 1 мМ DTT. При помощи концентратора Аmicon (с фильтром на 100 кДа) фракции, соответствующие мономерной форме белка ScCas12a, сконцентрировали до 2 мг/мл, после чего очищенный белок хранили при температуре -80°C в буфере, содержащем 10% глицерин.
In vitro реакцию порезки линейных PAM библиотек проводили в объеме 20 мкл в следующих условиях. Реакционная смесь состояла из: 1X CutSmart буфера (NEB), 5 мМ DTT, 100 нМ PAM-библиотеки, 4 мкМ крРНК, 400 нМ белка ScCas12a. В качестве контроля аналогичным образом были приготовлены пробы, не содержащие крРНК. Пробы инкубировали при температуре 37°C. Реакцию останавливали добавлением 1 мкл протеиназы К (NEB) с последующим инкубированием в течение 10 мин при 37°С и анализировали методом гель-электрофореза в 2% агарозном геле. В случае правильного узнавания и специфического разрезания ДНК белком ScCas12a должны формироваться два фрагмента ДНК длиной порядка 171 и 312 пар оснований (см. Фиг. 3).
Результаты опыта показали, что ScCas12a обладает нуклеазной активностью и разрезает часть фрагментов PAM библиотеки (Фиг. 4). При этом все три варианта крРНК с разными длинами DR оказались одинаково эффективны. Для дальнейшей работы использовался вариант с длиной прямого повтора 20 п. н.
Реакцию разрезания библиотеки повторяли в подобранных условиях. Продукты реакции наносили на 2% агарозный гель и подвергали электрофорезу. Непорезанные фрагменты ДНК длиной 483 п.н. экстрагировали из геля и подготавливали для высокоэффективного секвенирования с помощью набора NEB NextUltra II. Образцы секвенировали на платформе Illumina и далее проводили анализ последовательностей биоформатическими методами: определяли разницу в представленности нуклеотидов в отдельных позициях PAM (NNNNN) в сравнении с контрольным образцом. Для анализа результатов были построены PAM лого (Фиг. 5) и PAM колесо (Фиг. 6).
Анализ данных указывают на значимость 2, 3 и 4 позиций PAM при использовании 5N рандомизированной библиотеки. Таким образом, в результате in vitro анализа удалось установить предположительную PAM последовательность для ScCas12a: 5'-N(T/C)TTN-3'. Эта последовательность является предположительной в силу неточности результатов, получаемых скрининговыми подходами к определению PAM.
Для подтверждения наличия нуклеазной активности у ScCas12a проводили реакции разрезания ДНК фрагментов, содержащих ДНК-мишень 5'-gtgctcaatgaaaggagata-3', фланкированную PAM последовательностями, соответствующих предполагаемому консенсусу PAM (5'-GTTTC-3', 5'-GTTCG-3', 5'-GTTAG-3', 5'-TCTTG-3', 5'-GTATG-3', 5'-GCTTG-3', 5'-GATTG-3', 5'-GTTTA-3') (Фиг. 7). ScCas12a разрезал все мишени, фланкированные выбранными PAM. Менее эффективно была разрезана мишень, фланкированная последовательностью 5'-TCTTG-3'. Что может указывать на большее предпочтение к тимину во второй позиции в сравнении с цитозином.
Далее для уточнения PAM последовательности была произведена проверка значимости отдельных нуклеотидных позиций.
Для этого эксперимента была выбрана последовательность PAM 5'-GTTTG-3'.
Реакции разрезания (Фиг. 8) проводили in vitro с использованием ДНК фрагментов, содержащих ДНК-мишень 5'-gtgctcaatgaaaggagata-3', фланкированную PAM последовательностью (например, 5'-GTTTG-3'):

Все реакции разрезания ДНК проводили в следующих условиях:
1x CutSmart буфер
5 мМ DTT
400 нM ScCas12a
40 нM ДНК
4 мкM крРНК
Время инкубации - 10 минут, температура проведения реакции 37°C.
Замена нуклеотидов в каждом положении PAM (пурин на пиримидин и наоборот) показала, что значимыми являются только 2, 3, 4 позиции, подтвердив результаты PAM скрининга (Фиг. 8).
В результате проведенных исследований удалось сделать следующий вывод: PAM, распознаваемый нуклеазой ScCas12a, соответствует следующей формуле: 5'-(T/C)TTN-3'.
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Тестирование активности белка ScCas12a в разрезании различных ДНК мишеней.
Для того, чтобы проверить способность ScCas12a узнавать различные последовательности ДНК, фланкированные 5'-(T/C)TTN-3' последовательностью, были проведены эксперименты по in vitro разрезанию ДНК-мишеней из последовательности гена grin2b человека (см. Таблицу 1).

В реакции разрезания в качестве мишени использовался фрагмент гена grin2b (ПЦР продукт), несущий сайты узнавания (Таблица 1), предположительно распознаваемые ScCas12a в соответствии с PAM консенсусом 5'-(T/C)TTN-3'. Для узнавания этих последовательностей были синтезированы крРНК, направляющие ScCas12a на данные сайты.
Реакции разрезания проводились в подобранных для ScCas12a условиях, результат представлен на Фиг. 9. Из Фиг. 9 видно, что фермент ScCas12a порезал четыре из пяти мишеней (мишени 1, 2, 3 и 5) с подходящим PAM. Мишень 4 имела PAM последовательность CTTG, которая согласно предсказаниям на основании результатов «depletion analysis» должна эффективно распознаваться белком. Однако в in vitro эксперименте узнавание данного фрагмента не произошло. Вероятно, это связано с тем, что белок имеет некие дополнительные предпочтения к последовательности ДНК мишени, возможно связанные со вторичной структурой ДНК.
Две из трех мишеней (мишени 6 и 8), выбранных в качестве отрицательных контролей не были порезаны нуклеазой ScCas12a. Мишень 7, фланкированная последовательностью ATTA, не должна распознаваться белком согласно биоинформатическим предсказаниям. Однако по результатам in vitro эксперимента видно слабое разрезание данного фрагмента (Фиг. 9). Это не протеворечит данным PAM скриннинга, свидетельствующем о большей толерантности нуклеазы ScCas12a к заменам в четвертой позиции (считая от начала протоспейсера), чем в третьей и во второй.
В результате, можно сделать вывод о том, что использование последовательностей PAM, соответствующих консенсусу 5'-(T/C)TTN-3' с большей вероятностью приводит к эффективному разрезанию выбранной мишени.
Проведенные исследовательские испытания показали наличие нуклеазной активности у ScCas12a, а также позволили определить его PAM последовательность и последовательности крРНК.
ScCas12a рибонуклеопротеиновый комплекс специфически вносит разрывы в мишени, ограниченные PAM 5'-(T/C)TTN-3' с 5' конца, фланкирующих протоспейсер.
Пример 2. Белки, относящиеся к одному с ScCas12a типу V-A.
На сегодняшний день в Sedimentisphaera cyanobacteriorum не охарактеризовано ни одного фермента системы CRISPR-ScCas12a.
Из описанных ранее представителей типа V-A наиболее близка к ScCas12a нуклеаза TsCas12a из Thiomicrospira sp. XS5. При этом они идентичны всего лишь на 41.38% (Фиг. 10, степень идентичности была рассчитана по программе BLASTp, default parameters). Значительная часть отличий нуклеаз приходится на домен, взаимодействующий с PAM последовательностью (степень идентичности обоих доменов 37.78%).
Таким образом, белок ScCas12a существенно отличается по аминокислотной последовательности от других Cas12a белков, изученных на сегодняшний день.
Специалисту в области генетической инженерии очевидно, что полученный и охарактеризованный в данном Описании вариант последовательности белка ScCas12a может быть изменен без изменения функции самого белка (например, направленным мутагенезом аминокислотных остатков, напрямую не влияющих на функциональную активность. В частности, специалисту известно, что могут быть изменены неконсервативные аминокислотные остатки, не затрагивающие остатки, определяющие функциональность белка (определяющие его функцию или структуру). Примерами таких изменений могут служить замены неконсервативных аминокислотных остатков на гомологичные. В некоторых вариантах осуществления изобретения возможно использование белка, содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного непосредственно после нуклеотидной последовательности 5'-(T/C)TTN-3' в указанной молекуле ДНК. Гомологичные белки могут быть получены путем мутагенеза (например, сайт-направленного или ПЦР-опосредуемого мутагенеза) соответствующих молекул нуклеиновых кислот с последующим тестированием кодируемого модифицированного белка Cas12a на сохранение его функций в соответствии с описанными здесь функциональными анализами.
Пример 3. Описанная в настоящем изобретении система ScCas12a в комплексе с крРНК может быть использована для изменения последовательности геномной ДНК многоклеточного организма, в том числе эукариотического. Для введения системы ScCas12a в комплексе с крРНК в клетки этого организма (во все клетки или в часть клеток) могут быть применены различные подходы, известные специалистам. Например, методы доставки CRISPR систем в клетки организмов раскрыты в источниках (Liu C., Zhang L., Liu H. & Cheng K. Delivery strategies of the CRISPR-Cas9 gene-editing system for therapeutic applications // Journal of Controlled Release. - 2017. - Т. 28. - № 266. - С. 17-26; Lino C. A., Harper J. C., Carney J. P. & Timlin J. A. Delivering CRISPR: a review of the challenges and approaches // Drug Delivery. - 2018. - Т. 25. - №1. - С. 1234-1257; и в источниках, раскрытых внутри этих источников.)
Для эффективной экспрессии нуклеазы ScCas12a в эукариотических клетках будет желательно провести оптимизацию кодонов для аминокислотной последовательности белка ScCas12a методами, известными специалистам (например, IDT codon optimization tool).
Для эффективной работы нуклеазы ScCas12a в эукариотических клетках необходимо обеспечить импорт этого белка внутрь ядра эукариотической клетки. Для этого можно использовать сигнал ядерной локализации из Т-антигена вируса SV40 (Lanford R. E., Kanda P. & Kennedy R. C. Induction of nuclear transport with a synthetic peptide homologous to the SV40 T antigen transport signal // Cell. - 1986. - Т. 46. - №4. - С. 575-582), соединенный с последовательностью ScCas12a с помощью спейсерной последовательности, описанной в Shen B et al. «Generation of gene-modified mice via Cas9/RNA-mediated gene targeting», Cell Research. - 2013. - Т. 23. - №5. - С. 720-723 или без нее. Таким образом, полная аминокислотная последовательность нуклеазы, транспортируемой внутрь ядра эукариотической клетки, будет представлять собой следующую последовательность: MAPKKKRKVGIHGVPAA-ScCas12a-KRPAATKKAGQAKKKK (далее ScCas12a NLS). Для доставки белка с приведенной выше аминокислотной последовательностью, могут быть использованы по меньшей мере два подхода.
Доставка в виде гена осуществляется путем создания плазмиды, несущей ген ScCas12a NLS под регуляцией промотора (например, CMV промотора) и последовательности, кодирующей крРНК под регуляцией U6 промотора. В качестве ДНК-мишеней используются ДНК последовательности, фланкированные 5'-(T/C)TTN-3', например, последовательности гена grin2b человека:
5'-GTGCTCAATGAAAGGAGATA-3'
Плазмидную ДНК очищают и трансфицируют в клетки человека HEK293 c помощью реагента Lipofectamine 2000 (Thermo Fisher Scientific). Клетки инкубируют в течение 72 часов, после чего из них выделяется геномная ДНК с помощью колонок для очистки геномной ДНК (Thermo Fisher Scientific). Целевой ДНК сайт анализируется с помощью секвенирования на платформе Illumina с целью определения числа вставок-делеций в ДНК, происходящих в целевом сайте по причине направленного двунитевого разрыва и последующей его репарации.
Для амплификации целевых фрагментов используют праймеры, фланкирующие предположительное место внесения разрыва.
После амплификации пробы готовятся по протоколу реагента Ultra II DNA Library Prep Kit for Illumina (NEB) для подготовки образцов к высокопроизводительному секвенированию. Затем проводится секвенирование на платформе Illumina 300cycles, прямое прочтение. Результаты секвенирования анализируются биоинформатическими методами. В качестве детекции разрезания принимается вставка или делеция нескольких нуклеотидов в целевой последовательности ДНК.
Доставка в виде рибонуклеинового комплекса осуществляется путем инкубации рекомбинантной формы ScCas12a NLS c крРНК в CutSmart буфере (NEB). Рекомбинантный белок получают из бактериальных клеток-продуцентов, очищая его с помощью аффинной хроматографии (NiNTA, Qiagen) разделением по размеру (Superdex 200).
Белок смешивают с РНК в соотношении 1:2 (ScCas12a NLS : крРНК), инкубируют в течение 10 минут на комнатной температуре, затем смесь трансфицируют в клетки.
Далее проводится анализ экстрагированной из них ДНК на предмет вставок-делеций в целевом ДНК сайте (как описано выше).
ScCas12a - новая Cas нуклеаза, сильно отличающаяся по аминокислотной последовательности от описанных ранее Cas12a нуклеаз и имеющая простой в использовании PAM. Белок ScCas12a разрезает с высокой эффективностью ДНК-мишени, в том числе и при 37°С, и может стать основой нового инструмента геномного редактирования.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные случаи приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть, понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
--->
<110> Федеральное государственное бюджетное учреждение науки Институт
биологии гена Российской академии наук (Institute of Gene Biology
Russian Academy of Sciences)
<120> Средство разрезания ДНК на основе ScCas12a белка из бактерии
Sedimentisphaera cyanobacteriorum
<160> 6
<210> 1
<211> 1310
<212> PRT
<213> Sedimentisphaera cyanobacteriorum
<400> 1
MKEFTNQYSL TKTLRFELRP VGETAEKIED FKSGGLKQTV EKDRERTEAY KQLKEVIDSY 60
HRDFIEQAFA RQQTLSEEDF KQTYQLYKEA QKEKDGETLT KQYEHLRKKI AAMFSKATKE 120
WAVMGENNEL IGKNKESKLY QWLEKNYRAG RIEKEEFDHN AGLIEYFEKF STYFVGFDKN 180
RANMYSKEAK ATAISFRTIN ENMVKHFDNC QRLEKIKSKY PDLAEELKDF EEFFKPSYFI 240
NCMNQSGIDY YNISAIGGKD EKDQKANMKI NLFTQKNHLK GSDKPPFFAK LYKQILSDRE 300
KSVVIDEFEK DSELTEALKN VFSKDGLINE EFFTKLKSAL ENFMLPEYQG QLYIRNAFLT 360
KISANIWGSG SWGIIKDAVT QAAENNFTRK SDKEKYAKKD FYSIAELQQA IDEYIPTLEN 420
GVQNASLIEY FRKMNYKPRG SEEDAGLIEE INNNLRQAGI VLNQAELGSG KQREENIEKI 480
KNLLDSVLNL ERFLKPLYLE KEKMRPKAAN LNKDFCESFD PLYEKLKTFF KLYNKVRNYA 540
TKKPYSKDKF KINFDTATLL YGWSLDKETA NLSVIFRKRE KFYLGIINRY NSQIFNYKIA 600
GSESEKGLER KRSLQQKVLA EEGEDYFEKM VYHLLLGASK TIPKCSTQLK EVKAHFQKSS 660
EDYIIQSKSF AKSLTLTKEI FDLNNLRYNT ETGEISSELS DTYPKKFQKG YLTQTGDVSG 720
YKTALHKWID FCKEFLRCYR NTEIFTFHFK DTKEYESLDE FLKEVDSSGY EISFDKIKAS 780
YINEKVNAGE LYLFEIYNKD FSEYSKGKPN LHTIYWKSLF ETQNLLDKTA KLNGKAEIFF 840
RPRSIKHNDK IIHRAGETLK NKNPLNEKPS SRFDYDITKD RRFTKDKFFL HCPITLNFKQ 900
DKPVRFNEQV NLYLKDNPDV NIIGIDRGER HLLYYTLINQ NGEILQQGSL NRIGEEESRP 960
TDYHRLLDER EKQRQQARET WKAVEGIKDL KAGYLSRVVH KLAGLMVQNN AIVVLEDLNK 1020
GFKRGRFAVE KQVYQNFEKA LIQKLNYLVF KEVNSKDAPG HYLKAYQLTA PFISFEKLGT 1080
QSGFLFYVRA WNTSKIDPAT GFTDQIKPKY KNQKQAKDFM SSFDSVRYNR KENYFEFEAD 1140
FEKLAQKPKG RTRWTICSYG QERYSYSPKE RKFVKHNVTQ NLAELFNSEG ISFDSGQCFK 1200
DEILKVEDAS FFKSIIFNLR LLLKLRHTCK NAEIERDFII SPVKGNNSSF FDSRIAEQEN 1260
ITSIPQNADA NGAYNIALKG LMNLHNISKD GKAKLIKDED WIEFVQKRKF 1310
<210> 2
<211> 483
<212> ДНК
<213> artificial sequence
<220>
<223> ДНК библиотека, содержащая последовательность протоспейсера,
фланкированную рандомизированными пятью нуклеотидами (5'-NNNNN-3')
c 5' конца
<400> 2
ttgtctctgc ctgtagctgc caatgactat agcaatagca ccttttattg ccttgttcaa 60
ggatttctga ggcttttgaa agtttcattt tctctcattc tgcagagcaa ataccagaga 120
taagagagta ggctggtaga tggagttggn nnnngtgctc aatgaaagga gataaggtcc 180
ttgaattgca gtatctagcc tcttctaaga caggttacgt gatgtagatc ctattttaac 240
atgctctttc tttgtgtttg cagggagtcg acgagttgaa gatgaagccc agagcggagt 300
gctgttctcc caagttctgg ttggtgttgg ccgtcctggc cgtgtcaggc agcagagctc 360
gttctcagaa gagccccccc agcattggca ttgctgtcat cctcgtgggc acttccgacg 420
aggtggccat caaggatgcc cacgagaaag atgatttcca ccatctctcc gtggtacccc 480
ggg 483
<210> 3
<211> 40
<212> РНК
<213> artificial sequence
<220>
<223> крРНК: Длина спейсера 20 (SP 20), длина прямого повтора 20 (DR 20)
<400> 3
aaatttctac tgtcgtagat gtgctcaatg aaaggagata 40
<210> 4
<211> 42
<212> РНК
<213> artificial sequence
<220>
<223> крРНК: Длина спейсера 20 (SP 20), длина прямого повтора 22 (DR 22)
<400> 4
taaaatttct actgtcgtag atgtgctcaa tgaaaggaga ta 42
<210> 5
<211> 56
<212> РНК
<213> artificial sequence
<220>
<223> крРНК: Длина спейсера 20 (SP 20), длина прямого повтора 36 (DR 36)
<400> 5
gtctaaaggc cttataaaat ttctactgtc gtagatgtgc tcaatgaaag gagata 56
<210> 6
<211> 374
<212> ДНК
<213> artificial sequence
<220>
<223> ДНК, содержащая ДНК-мишень 5'-gtgctcaatgaaaggagata-3', фланкированную
PAM последовательностью (например, 5'-GTTTG-3')
<400> 6
aataccagag ataagagagt aggctggtag atggagttgg gtttcgtgct caatgaaagg 60
agataaggtc cttgaattgc agtatctagc ctcttctaag acaggttacg tgatgtagat 120
cctattttaa catgctcttt ctttgtgttt gcagggagtc gacgagttga agatgaagcc 180
cagagcggag tgctgttctc ccaagttctg gttggtgttg gccgtcctgg ccgtgtcagg 240
cagcagagct cgttctcaga agagcccccc cagcattggc attgctgtca tcctcgtggg 300
cacttccgac gaggtggcca tcaaggatgc ccacgagaaa gatgatttcc accatctctc 360
cgtggtaccc cggg 374
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Средство обнаружения нуклеиновых кислот на основе ScCas12a белка из бактерии Sedimentisphaera cyanobacteriorum | 2023 |
|
RU2820345C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Streptococcus uberis NCTC3858 | 2022 |
|
RU2788197C1 |
| Средство разрезания ДНК на основе Cas9 белка из биотехнологически значимой бактерии Clostridium cellulolyticum | 2018 |
|
RU2712497C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| ПРИМЕНЕНИЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA ДЛЯ МОДИФИКАЦИИ ГЕНОМНОЙ ДНК В КЛЕТКАХ | 2019 |
|
RU2724470C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМНОЙ ДНК ЭУКАРИОТИЧЕСКОЙ КЛЕТКИ НА ОСНОВЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩЕЙ БЕЛОК SUCAS9NLS | 2022 |
|
RU2804422C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Capnocytophaga ochracea | 2021 |
|
RU2778156C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |

Группа изобретений относится к биотехнологии. Настоящее изобретение описывает применение бактериальной нуклеазы системы CRISPR-Cas12a из бактерии Sedimentisphaera cyanobacteriorum L21-RPul-D3 для образования строго специфичных двунитевых разрывов в молекуле ДНК. Данная нуклеаза может быть использована в качестве инструмента для внесения изменений в строго определенных местах в последовательности геномной ДНК одноклеточных или многоклеточных организмов. Таким образом, достигается повышение универсальности доступных CRISPR систем, что позволит использовать нуклеазы Cas12a из различных организмов для разрезания геномной или плазмидной ДНК в большем количестве специфических сайтов и при различных условиях. 2 н. и 3 з.п. ф-лы, 1 табл., 10 ил., 3 пр.
1. Применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1 или содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного после нуклеотидной последовательности 5'-(T/C)TTN-3' в указанной молекуле ДНК.
2. Применение по п. 1, характеризующееся тем, что образование двунитевого разрыва в молекуле ДНК происходит при температуре 37°C.
3. Применение белка по п. 1, где белок содержит аминокислотную последовательность SEQ ID NO: 1.
4. Способ изменения последовательности геномной ДНК одноклеточного или многоклеточного организма, включающий введение в по меньшей мере одну клетку этого организма эффективного количества: а) либо белка, содержащего аминокислотную последовательность SEQ ID NO: 1, либо нуклеиновой кислоты, кодирующей белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и б) либо крРНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, непосредственно примыкающей к нуклеотидной последовательности 5'-(T/C)TTN-3' и взаимодействующей с указанным белком после образования дуплекса, либо последовательности ДНК, кодирующей указанную крРНК; при этом взаимодействие указанного белка с крРНК и нуклеотидной последовательностью 5'-(T/C)TTN-3' приводит к образованию двунитевого разрыва в последовательности геномной ДНК, примыкающей к последовательности 5'-(T/C)TTN-3'.
5. Способ по п. 4, дополнительно включающий введение экзогенной последовательности ДНК одновременно с крРНК.
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| WO 2014093661 A9, 19.06.2014 | |||
| DELTCHEVA E | |||
| et al | |||
| "CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III.", Nature 471.7340 (2011): р.602-607 | |||
| ASTRID WENINGER et al | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
Авторы
Даты
2023-03-07—Публикация
2022-06-23—Подача