Область техники
Изобретение относится к биотехнологии, а именно, к новым ферментам - Cas нуклеазам систем CRISPR-Cas, применяемым для разрезания ДНК и редактирования геномов различных организмов. Данная технология может применяться в будущем для генной терапии наследственных заболеваний человека, а также для редактирования геномов других организмов.
Уровень техники
Изменение последовательности ДНК - одна из актуальных задач биотехнологии на сегодняшний день. Редактирование и изменение геномов эукариотических и прокариотических организмов, а также манипуляции с ДНК in vitro, требуют направленного внесения двунитевых разрывов в последовательности ДНК.
Для решения этой задачи в настоящее время используют следующие методики: искусственные нуклеазные системы, содержащей домены типа «цинковые пальцы» (ZFN), эффекторные нуклеазы, подобные активатору транскрипции (TALEN-системы) и бактериальные CRISPR-Cas системы. Первые два метода требуют трудозатратой оптимизации аминокислотной последовательности нуклеазы для узнавания конкретной последовательности ДНК. В отличие от них в случае CRISPR-Cas систем структурами, узнающими ДНК мишень, являются не белки, а короткие направляющие РНК. Разрезание конкретной ДНК мишени не требует синтеза нуклеазы или ее гена de novo, а обеспечивается за счет использования направляющих РНК, комплементарных целевой последовательности. Это делает CRISPR-Cas системы удобными и эффективными инструментами разрезания различных ДНК-последовательностей. Методика позволяет осуществлять единовременное разрезание ДНК в нескольких участках при использовании направляющих РНК разной последовательностей. Такой подход используется в том числе для одновременного изменения нескольких генов в эукариотических организмах.
По своей природе CRISPR-Cas системы являются иммунными системами прокариот, способными высоко специфично вносить разрывы в генетический материал вирусов (Mojica F. J. M., 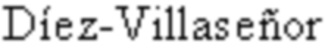 C.,
C., 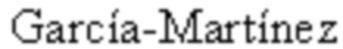 J. & Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). Аббревиатура CRISPR-Cas расшифровывается как «Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR associated genes» (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575), что переводе с английского обозначает «короткие палиндромные повторы, регулярно расположенные группами, и ассоциированные с ними гены». Все CRISPR-Cas системы состоят из CRISPR кассет и генов, кодирующих различные Cas белки (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). CRISPR кассеты состоят из последовательностей спейсеров, каждый из которых имеет уникальную нуклеотидную последовательность, и повторяющихся палиндромных повторов (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). В результате транскрипции CRISPR кассет и их последующего процессинга образуются направляющие крРНК, которые вместе с Cas белками формируют эффекторный комплекс
J. & Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). Аббревиатура CRISPR-Cas расшифровывается как «Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR associated genes» (Jansen R., Embden J. D., Gaastra W. & Schouls L. M. Identification of genes that are associated with DNA repeats in prokaryotes // Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575), что переводе с английского обозначает «короткие палиндромные повторы, регулярно расположенные группами, и ассоциированные с ними гены». Все CRISPR-Cas системы состоят из CRISPR кассет и генов, кодирующих различные Cas белки (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). CRISPR кассеты состоят из последовательностей спейсеров, каждый из которых имеет уникальную нуклеотидную последовательность, и повторяющихся палиндромных повторов (Jansen R. et al., Molecular microbiology. - 2002. - Т. 43. - №. 6. - С. 1565-1575). В результате транскрипции CRISPR кассет и их последующего процессинга образуются направляющие крРНК, которые вместе с Cas белками формируют эффекторный комплекс
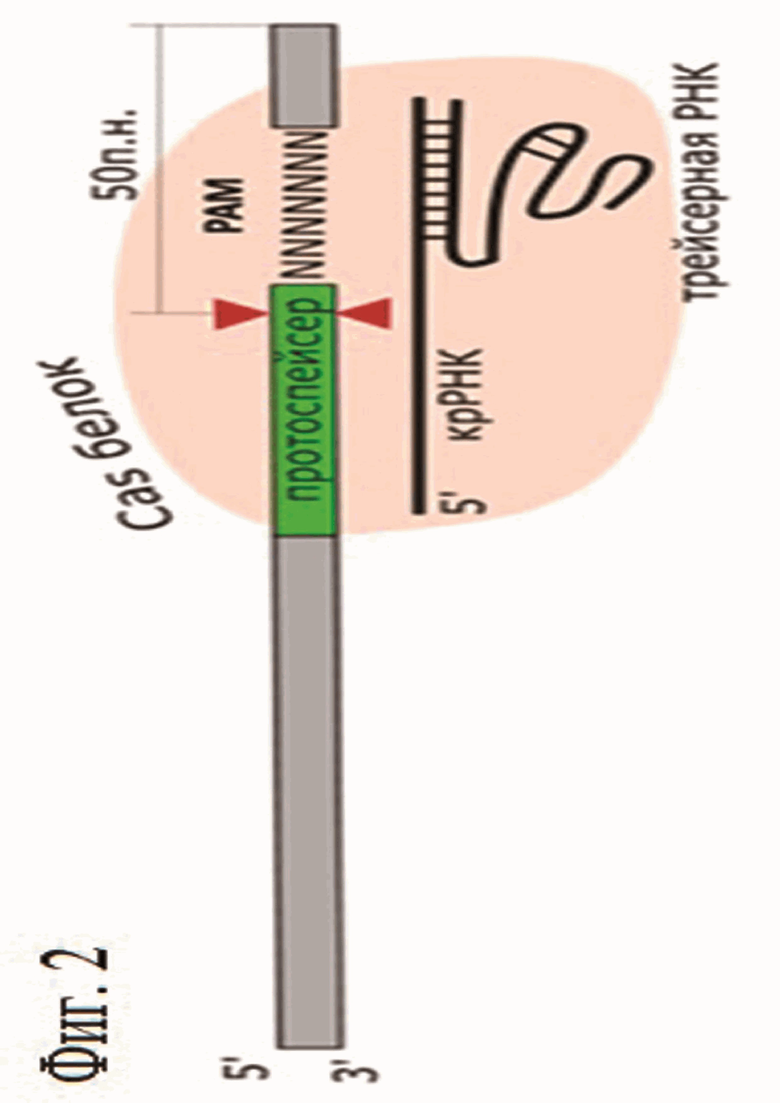
(Brouns S. J., Jore M. M., Lundgren M., Westra E. R., Slijkhuis R. J., Snijders A. P., Dickman M. J., Makarova K. S., Koonin E. V. & van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes // Science. - 2008. - Т. 321. - №. 5891. - С. 960-964). За счет комплементарного спаривания крРНК с целевым участком ДНК, именуемым протоспейсером, Cas-нуклеаза узнает ДНК-мишень и высоко специфично вносит в нее разрыв.
CRISPR-Cas системы, представленными одиночным белком-эффектором, разделяют на шесть различных типов (от I до VI) в зависимости от Cas белков, входящих в состав систем. В 2013 году впервые было предложено использовать систему CRISPR-Cas9, относящуюся к типу II, для редактирования геномной ДНК клеток человека (Cong L., Ran F. A., Cox D., Lin S., Barretto R., Habib N., Hsu P. D., Wu X., Jiang W., Marraffini L. A. & Zhang F. Multiplex genome engineering using CRISPR/Cas systems // Science. - 2013. - Т. 339. - № 6121. - С. 819-823). Система CRISPR-Cas9 II типа отличается простотой состава и механизма работы: для ее функционирования необходимо формирование эффекторного комплекса, состоящего лишь из одного белка Cas9 и двух коротких РНК: крРНК (crRNA) и трейсерной РНК (tracrRNA, трРНК). Трейсерная РНК комплементарно спаривается с участком крРНК, происходящим из CRISPR повтора, образуя вторичную структуру, необходимую для связывания направляющих РНК с Cas эффектором. Определение последовательности направляющих РНК является важным шагом в характеризации неизученных ранее Cas-ортологов. Эффекторный белок Cas9 является РНК-зависимой ДНК эндонуклеазой с двумя нуклеазными доменами (HNH и RuvC), вносящими разрывы в комплементарные нити целевой ДНК, таким образом образуя двунитевой разрыв ДНК (Deltcheva E., Chylinski K., Sharma C. M., Gonzales K., Chao Y., Pirzada Z. A., Eckert M. R., Vogel J. & Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III // Nature. - 2011. - Т. 471. - №. 7340. - С. 602).
Технология CRISPR-Cas9 является одной из самых современных и быстроразвивающихся методик внесения разрывов в ДНК различных организмов, начиная от бактериальных штаммов и заканчивая клетками человека, а также in vitro (Song M. The CRISPR/Cas9 system: Their delivery, in vivo and ex vivo applications and clinical development by startups // Biotechnology Progress. - 2017. - Т. 33. - № 4. - С. 1035-1045).
Эффекторному рибонуклеиновому комплексу, состоящему из Cas9 и дуплекса крРНК и трейсерной РНК, для распознавания и последующего гидролиза ДНК помимо комплементарного соответствия спейсера крРНК и протоспейсера необходимо присутствие PAM (от англ. «PAM» - protospacer adjusted motif) на ДНК мишени (Mojica F. J. M. et al., Journal of molecular evolution. - 2005. - Т. 60. - №. 2. - С. 174-182). PAM представляет собой строго определенную последовательность из нескольких нуклеотидов, расположенных в системах типа II вплотную либо в нескольких нуклеотидах от 3'-конца протоспейсера на нетаргетной цепи. При отсутствии PAM гидролиза связей в ДНК с образованием двунитевого разрыва не происходит. Необходимость присутствия PAM последовательности на мишени повышает специфичность узнавания, но в то же время накладывает ограничение в выборе целевых участков ДНК, в которые необходимо внести разрыв. Таким образом, наличие нужной PAM последовательности, фланирующей ДНК-мишень с 3'-конца, является характеристикой, ограничивающей применение CRISPR-Cas систем на любых участках ДНК.
На сегодняшний день известно несколько CRISPR-Cas нуклеаз, способных направлено и специфично вносить двунитевые разрывы в ДНК. Например, NmeCas9 из Neisseria meningitidis strain 8013 (Esvelt K. M., Mali P., Braff J. L., Moosburner M., Yaung S. J. & Church G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing // Nature Methods. - 2013. - Т. 10. - С. 1116-1121), Nme2Cas9 из Neisseria meningitidis strain De11444 (Edraki A., Mir A., Ibraheim R., Gainetdinov I., Yoon Y., Song C.-Q., Cao Y., Gallant J., Xue W., Rivera-Perez J. A. & Sontheimer E. J. A compact, high-accuracy Cas9 with a dinucleotide PAM for in vivo genome editing // Molecular Cell. - 2019. - Т. 73. - С. 714-726), CjCas9 из Campylobacter jejuni (Kim E., Koo T., Park S. W., Kim D., Kim K., Cho H. Y., Song D. W., Lee K. J., Jung M. H., Kim S., Kim J. H., Kim J. H. & Kim J. S. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni // Nature Communications. - 2017. - Т. 8. - С. 14500), CdCas9 из Corynebacterium diphtheriae (Hirano S., Abudayyeh O. O., Gootenberg J. S., Horii T., Ishitani R., Hatada I., Zhang F., Nishimasu H. & Nureki O. Structural basis for the promiscuous PAM recognition by Corynebacterium diphtheriae Cas9 // Nature Communications. - 2019. - Т. 10. - С. 1968), GeoCas9 из Geobacillus stearothermophilus (Harrington L. B., Paez-Espino D., Staahl B. T., Chen J. S., Ma E., Kyrpides N. C. & Doudna J. A. A thermostable Cas9 with increased lifetime in human plasma // Nature Communications. - 2017. - Т. 8. - С. 1424.), SaCas9 из Staphylococcus aureus (Ran F. A., Cong L., Yan W. X., Scott D. A., Gootenberg J. S., Kriz A. J., Zetsche B., Shalem O., Wu X., Makarova K. S., Koonin E. V., Sharp P. A. & Zhang F. In vivo genome editing using Staphylococcus aureus Cas9 // Nature. - 2015. - Т. 520. - С. 186-191), SauriCas9 из Staphylococcus auricularis (Hu Z., Wang S., Zhang C., Gao N., Li M., Wang D., Wang D., Liu D., Liu H., Ong S.-G., Wang H., Wang Y. A compact Cas9 ortholog from Staphylococcus Auricularis (SauriCas9) expands the DNA targeting scope // PLOS Biology. - 2020. - Т. 18. - e3000686).
Различные CRISPR-Cas белки используют для своей работы разные, оригинальные PAM последовательности. Однако большинство из описанных характеризуются длинной и сложной PAM последовательностью.
Использование CRISPR-Cas белков с новыми разнообразными PAM последовательностями необходимо для обеспечения возможности изменения любого участка ДНК, как in vitro, так и в геноме живых организмов. Изменение эукариотических геномов также требует использования нуклеаз малого размера для обеспечения доставки CRISPR-Cas систем в клетки посредством AAV вирусов.
Несмотря на известность ряда способов разрезания ДНК и изменения последовательности геномной ДНК, на сегодняшний день сохраняется потребность в новых эффективных инструментах для модификации ДНК в различных организмах и в строго определенных местах последовательности ДНК.
Сущность изобретения
Задачей настоящего изобретения является создание новых инструментов для изменения последовательности геномной ДНК одноклеточных или многоклеточных организмов на основе систем CRISPR-Cas9. Существующие в настоящее время системы имеют ограниченное применение из-за специфичной последовательности РАМ, которая должна присутствовать на 3'-конце участка ДНК, подвергающегося модификации. Поиск новых ферментов Cas9 с другими РАМ последовательностями позволит расширить арсенал имеющихся средств для образования двунитевого разрыва в необходимых, строго определенных местах в молекулах ДНК разных организмов. Для решения этой задачи авторами была охарактеризована ранее биоинформатически предсказанная система из бактерии Capnocytophaga ochracea.
CRISPR нуклеаза II типа CoCas9, которая может быть применена для внесения направленных изменений в геном как этого, так и других организмов. Существенным признаком, отличающим настоящее изобретение, является отличающаяся от других известных последовательность PAM.
Указанная задача решается путем применения белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного непосредственно перед нуклеотидной последовательностью 5'-N(A/G)(A/G)(A/T)C-3' в указанной молекуле ДНК. В некоторых вариантах изобретения данное применение характеризуется тем, что образование двунитевого разрыва в молекуле ДНК происходит при температуре от 35°C до 45°C.
Указанная задача также решается путем создания способа изменения последовательности геномной ДНК одноклеточного или многоклеточного организма, включающего введение в по меньшей мере одну клетку этого организма эффективного количества: а) либо белка, содержащего аминокислотную последовательность SEQ ID NO: 1, либо нуклеиновой кислоты, кодирующей белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и б) либо направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, непосредственно примыкающей к нуклеотидной последовательности 5'-N(A/G)(A/G)(A/T)C-3', и взаимодействующей с указанным белком после образования дуплекса, либо последовательности ДНК, кодирующей указанную направляющую РНК; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью 5'-N(A/G)(A/G)(A/T)C-3' приводит к образованию двунитевого разрыва в последовательности геномной ДНК, непосредственно примыкающей к последовательности 5'-N(A/G)(A/G)(A/T)C-3'. В некоторых вариантах изобретения данный способ характеризуется тем, что дополнительно включающий введение экзогенной последовательности ДНК одновременно с направляющей РНК.
В качестве направляющей РНК может быть использована смесь из крРНК (crRNA) и трейсерной РНК (tracrRNA), способных образовать комплекс с участком целевой ДНК и белком CoCas9. В предпочтительных вариантах изобретения в качестве направляющей РНК может быть использована гибридная РНК, сконструированная на основе крРНК и трейсерной РНК. Методы конструирования гибридной направляющей РНК известны специалистам (Hsu P. D., Scott D. A., Weinstein J. A., Ran F. A., Konermann S., Agarwala V., Li Y., Fine E. J., Wu X., Shalem O., Cradick T. J., Marraffini L. A., Bao G. & Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases // Nature Biotechnology. - 2013. - Т. 31. - №9. - С. 827-832). Один из вариантов конструирования гибридной РНК раскрыт в Примерах ниже.
Изобретение может быть использовано как для разрезания целевой ДНК in vitro, так и для модификации генома какого-либо живого организма. Модификация генома может проводиться прямым способом - разрезанием генома в соответствующем сайте, а также вставкой экзогенной последовательности ДНК за счет гомологичной репарации.
В качестве экзогенной последовательности ДНК может быть использован любой участок двунитевой или однонитевой ДНК из генома организма, отличного от организма, используемого при введении (или смесь таких участков между собой и с другими фрагментами ДНК), при этом этот участок (или смесь участков) предназначен для интеграции в место двуцепочечного разрыва в целевой ДНК, образованного под действием нуклеазы CoCas9. В некоторых вариантах изобретения в качестве экзогенной последовательности ДНК может быть использован участок двуцепочечной ДНК из генома организма, используемого при введении белка CoCas9, но при этом измененный мутациями (заменой нуклеотидов), а также вставками или делециями одного или нескольких нуклеотидов.
Техническим результатом настоящего изобретения является повышение универсальности доступных систем CRISPR-Cas9, позволяющее использовать нуклеазу Cas9 для разрезания геномной или плазмидной ДНК в большем количестве специфических сайтов и специфических условий.
Подробное раскрытие изобретения
В описании данного изобретения термины «включает» и «включающий» интерпретируются как означающие «включает, помимо всего прочего». Указанные термины не предназначены для того, чтобы их истолковывали как «состоит только из». Если не определено отдельно, технические и научные термины в данной заявке имеют стандартные значения, общепринятые в научной и технической литературе.
Используемый здесь термин «процент гомологии двух последовательностей» эквивалентен термину «процент идентичности двух последовательностей». Идентичность последовательностей определяется на основании референсной последовательности. Алгоритмы для анализа последовательности известны в данной области, такие как BLAST, описанный в Altschul et al. (Basic local alignment search tool // Journal of Molecular Biology. - 1990. - Т. 215. - С. 403-410). Для целей настоящего изобретения для определения уровня идентичности и сходства между нуклеотидными последовательностями и аминокислотными последовательностями может быть использовано сравнение нуклеотидных и аминокислотных последовательностей, производимое с помощью пакета программ BLAST, предоставляемого National Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov/blast) с использованием содержащего разрывы выравнивания со стандартными параметрами. Процент идентичности двух последовательностей определяется числом положений идентичных аминокислот в этих двух последовательностях с учетом числа пробелов и длины каждого пробела, которые необходимо ввести для оптимального сопоставления двух последовательностей путем выравнивания. Процент идентичности равен числу идентичных аминокислот в данных положениях с учетом выравнивания последовательностей, разделенному на общее число положений и умноженному на 100.
Термин «специфически гибридизуется» относится к ассоциации между двумя одноцепочечными молекулами нуклеиновых кислот или в достаточной степени комплементарными последовательностями, что разрешает такую гибридизацию в предопределенных условиях, обычно использующихся в данной области.
Фраза «двунитевой разрыв, расположенный непосредственно перед нуклеотидной последовательностью РАМ» означает, что двунитевой разрыв в целевой последовательности ДНК будет произведен на расстоянии от 0 до 25 нуклеотидов перед нуклеотидной последовательностью РАМ.
Под экзогенной последовательностью ДНК, вводимой одновременно с направляющей РНК, следует понимать последовательность ДНК, подготовленную специально для специфической модификации двуцепочечной целевой ДНК в месте разрыва, определяемого специфичностью направляющей РНК. Подобной модификацией может быть, например, вставка или делеция определенных нуклеотидов в месте разрыва целевой ДНК. Экзогенной ДНК может служить как участок ДНК из другого организма, так и участок ДНК из того же организма, что и целевая ДНК.
Под белком, содержащим определенную аминокислотную последовательность следует понимать белок, имеющий аминокислотную последовательность, составленную из указанной аминокислотной последовательности и, возможно, других последовательностей, соединенных пептидными связями с указанной аминокислотной последовательностью. Примером других последовательностей может служить последовательность сигнала ядерной локализации (NLS), или другие последовательности, обеспечивающие повышенную функциональность для указанной аминокислотной последовательности.
Под экзогенной последовательностью ДНК, вводимой одновременно с направляющей РНК, следует понимать последовательность ДНК, подготовленную специально для специфической модификации двуцепочечной целевой ДНК в месте разрыва, определяемого специфичностью направляющей РНК. Подобной модификацией может быть, например, вставка или делеция определенных нуклеотидов в месте разрыва целевой ДНК. Экзогенной ДНК может служить как участок ДНК из другого организма, так и участок ДНК из того же организма, что и целевая ДНК.
Под эффективным количеством вводимых в клетку белка и РНК следует понимать такое количество белка и РНК, которое при попадании в указанную клетку будет способно образовать функциональный комплекс, то есть комплекс, который будет специфически связываться с целевой ДНК и производить в ней двунитевой разрыв в месте, определяемом направляющей РНК и РАМ последовательностью на ДНК. Эффективность этого процесса может быть оценена при помощи анализа целевой ДНК, выделенной из указанной клетки с помощью стандартных методов, известных специалистам.
Доставка белка и РНК в клетку может быть осуществлена различными способами. Например, белок может быть доставлен в виде ДНК-плазмиды, которая кодирует ген этого белка, как мРНК для трансляции этого белка в цитоплазме клетки, или как рибонуклеопротеидный комплекс, включающий этот белок и направляющую РНК. Доставка может быть осуществлена различными методами, известными специалистам.
Нуклеиновая кислота, кодирующая компоненты системы, может быть введена в клетку, непосредственно или опосредованно: за счет трансфекции или трансформации клеток известными специалистам способами, за счет использования рекомбинантного вируса, за счет манипуляций с клеткой, таких как микроинъекция ДНК и т. п.
Доставка рибонуклеинового комплекса, состоящего из нуклеазы и направляющих РНК и экзогенной ДНК (при необходимости) может осуществляться путем трансфекции комплексов в клетку или за счет механического введения комплекса внутрь клетки, например, микроинъекции.
Молекула нуклеиновой кислоты, кодирующая белок, который необходимо ввести в клетку, может быть интегрирована в хромосому или может представлять собой внехромосомно реплицирующуюся ДНК. В некоторых вариантах для обеспечения эффективной экспрессии гена белка с вводимой в клетку ДНК необходимо изменить последовательность этой ДНК в соответствии с типом клетки в целях оптимизации кодонов при экспрессии, обусловленное неравномерностью частот встречаемости синонимичных кодонов в кодирующих областях генома различных организмов. Оптимизация кодонов необходима для увеличения экспрессии в клетках животных, растений, грибов или микроорганизмов.
Для функционирования белка, имеющего последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1, в эукариотической клетке необходимо, чтобы этот белок оказался в ядре этой клетки. Поэтому, в некоторых вариантах изобретения, для образования двунитевых разрывов в целевой ДНК используют белок, имеющий последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1, и который дополнительно модифицирован с одного или с обоих концов добавлением одного или нескольких сигналов ядерной локализации. Например, может быть использован сигнал ядерной локализации из вируса SV40. Для эффективной доставки в ядро сигнал ядерной локализации может быть отделен от основной последовательности белка спейсерной последовательностью, например, описанной в Shen B, et al. (Generation of gene-modified mice via Cas9/RNA-mediated gene targeting // Cell Research. - 2013. - Т. 23. - №5. - С. 720-723). Также, в других вариантах осуществления, может быть использован другой сигнал ядерной локализации, или альтернативный метод доставки указанного белка в ядро клетки.
Настоящее изобретение охватывает применение белка из организма Capnocytophaga ochracea, гомологичного ранее охарактеризованным белкам Cas9, для внесения двуцепочечных разрывов в молекулы ДНК в строго определенных положениях. Использование CRISPR нуклеаз для внесения направленных изменений в геном имеет ряд преимуществ. Во-первых, специфичность действия системы определяется последовательностью крРНК, что позволяет использовать один тип нуклеазы для всех локусов-мишеней. Во-вторых, методика позволяет доставить в клетку сразу несколько направляющих РНК, комплементарных разным генам-мишеням, что позволяет осуществлять единовременное изменение сразу нескольких генов.
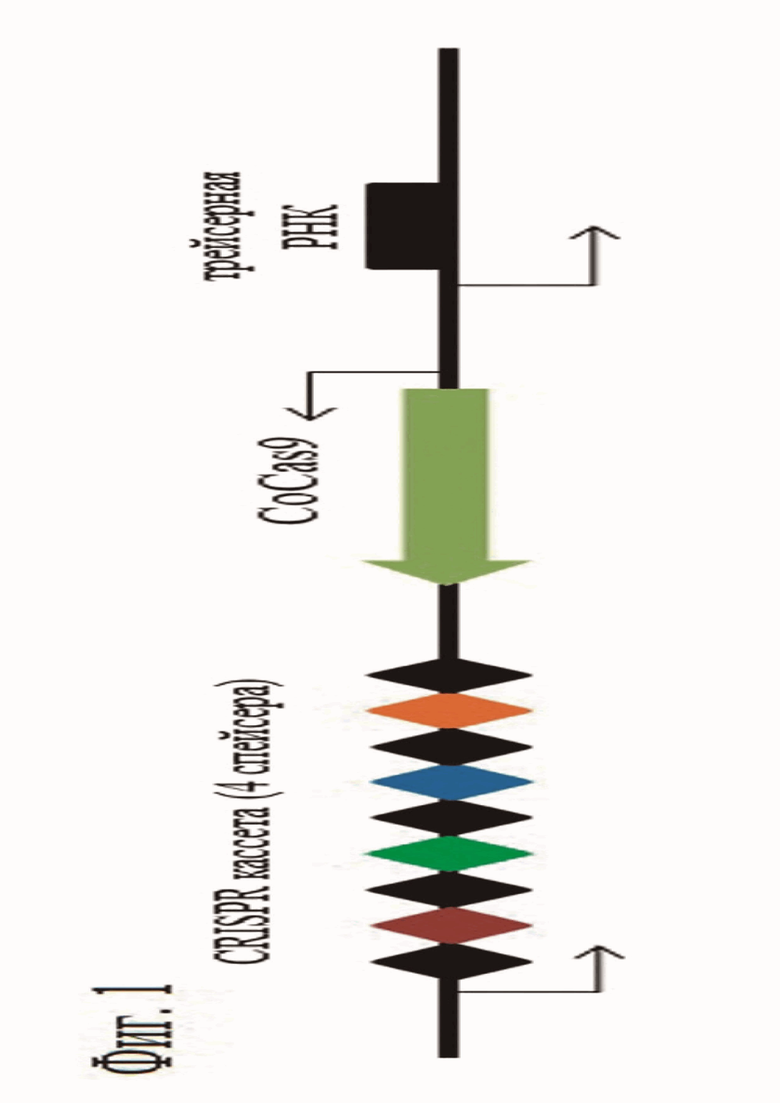
CoCas9 - Cas нуклеаза, найденная в бактериях Capnocytophaga ochracea DSM 7271, являющихся оппортунистическими патогенами человека, найденными в ротовой полости. Capnocytophaga ochracea CRISPR-Cas9 система (далее CRISPR CoCas9) относится к II C типу CRISPR Cas систем и состоит из CRISPR кассеты, несущей пять прямых повторов (direct repeats, DR) последовательностью 5'-GTTGTGAATTGCTTTCAAATTTTGTAGTTTTGCGATTGATAACAAC-3' разделенных последовательностями уникальных спейсеров. Ни один из спейсеров системы не совпадает по последовательности с известными на сегодня бактериофагами или плазмидами, что не позволяет определить требуемый CoCas9 PAM биоинформатическим анализом. К CRISPR кассете прилегает ген эффекторного Cas9 белка CoCas9. Рядом с Cas геном была обнаружена последовательность, частично комплементарная прямым повторам, складывающаяся в характерную вторичную структуру, - предполагаемая трейсерная РНК (tracrRNA, трРНК) (Фиг. 1)
Знание характерной архитектуры РНК-Cas белкового комплекса систем II-C типа позволила предсказать направление транскрипции CRISPR кассеты: пре-крРНК транскрибируется в противоположном от Cas генов направлении (Фиг. 1)
Таким образом, анализ последовательности локуса CoCas9 позволил предсказать последовательности трейсерной и направляющих РНК (Таблица 1).
Для проверки активности CoCas9 нуклеазы и определения требуемого CoCas9 PAM мотива, были проведены эксперименты по воссозданию реакции разрезания ДНК in vitro. Для определения PAM последовательности белка CoCas9 использовали in vitro разрезание двунитевых PAM библиотек. Для этого необходимо было получить все компоненты эффекторного комплекса CoCas9: направляющие РНК и нуклеазу в рекомбинантной форме. Определение последовательности направляющих РНК позволило синтезировать in vitro молекулы крРНК и трРНК. Синтез осуществляли с помощью набора NEB HiScribe T7 RNA synthesis. Двунитевые ДНК библиотеки представляли собой фрагменты размером 374 пар нуклеотидов (п. н.), содержащие последовательность протоспейсера, фланкированную рандомизированными семью нуклеотидами (5'-NNNNNNN-3') c 3' конца: 5'-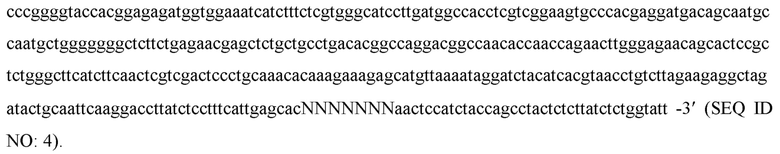
Для разрезания этой мишени использовали направляющие РНК следующей последовательности: в качестве трРНК последовательность SEQ ID NO: 2; в качестве крРНК:
5'-uaucuccuuucauugagcacGUUGUGAAUUGCUUUCAAAUUUUGUAGUUUUGCGAUUGAUAACAA-3' (SEQ ID NO: 5).
Жирным шрифтом выделена последовательность крРНК, комплементарная протоспейсеру (целевой ДНК последовательности).
Для получения рекомбинантного белка CoCas9 его ген был клонирован в плазмиду pET21a. В качестве кодирующей ген ДНК, использовалась ДНК, амплифицированная с геномной ДНК Capnocytophaga ochracea DSM 7271, заказанной из коллекции DSMZ (Leibniz Institute DSMZ-German Collection of Microorganisms and Cell Cultures GmbH). Клетки E. coli Rosetta были трансформированы полученной плазмидой pET21a-6xHis-CoCas9. 500 мкл ночной культуры разводили в 500 мл среды LB, и растили клетки при температуре 37°C до достижения оптической плотности 0.6 отн. ед. Синтез целевого белка индуцировали добавлением ИПТГ до концентрации 1 мМ, после чего клетки инкубировали при температуре 16°C в течение 16 часов. Затем проводили центрифугирование клеток на скорости 5000 g в течение 30 минут, полученные осадки клеток замораживали при температуре -20°C. Осадки размораживали на льду в течение 30 минут, ресуспензировали в 15 мл лизисного буфера (Tris-HCl 50мМ pH 8, 500 мМ NaCl, β-меркаптоэтанол 1мМ, имидазол 10 мМ) с добавлением 15 мг лизоцима и снова инкубировали на льду в течение 30 минут. Затем клетки разрушали воздействием ультразвука в течение 30 минут и центрифугировали в течение 40 минут на скорости 16000 g. Полученный супернатант пропускали через фильтр 0.2 мкм и наносили на колонку HisTrap HP 1 mL (GE Healthcare) на скорости 1 мл/мин.
Хроматографию проводили при помощи FPLC хроматографа AKTA (GE Healthcare) на скорости 1 мл/мин. Колонку с нанесенным белком промывали 20 мл лизисного буфера с добавлением 10 мМ имидазола, после чего белок смывали лизисным буфером с добавлением 300 мМ имидазола.
Затем, фракцию белка, полученную в ходе афинной хроматографии, пропускали через гель-фильтрационную колонку Superdex 200 10/300 GL (24 мл), уравновешенную следующим буфером: Tris-HCl 50 мМ pH 8, 500 мМ NaCl, 1 мМ DTT. При помощи концентратора Amicon (с фильтром на 30 кДа) фракции, соответствующие мономерной форме белка CoCas9, сконцентрировали до 1.6 мг/мл, после чего очищенный белок хранили при температуре -80°C в буфере, содержащем 10% глицерин.
In vitro реакцию порезки линейных PAM библиотек проводили в объеме 20 мкл в следующих условиях. Реакционная смесь состояла из: 1X CutSmart буфера (NEB), 5 мМ DTT, 100 нМ PAM-библиотеки, 2 мкМ трРНК/крРНК, 400 нМ белка CoCas9. В качестве контроля аналогичным образом были приготовлены пробы, не содержащие РНК. Пробы инкубировали при различных температурах и анализировали методом гель-электрофореза в 2% агарозном геле. В случае правильного узнавания и специфического разрезания ДНК белком CoCas9 должны формироваться два фрагмента ДНК длиной порядка 326 и 48 пар оснований (см. Фиг. 2).
Результаты опыта показали, что CoCas9 обладает нуклеазной активностью и разрезает часть фрагментов PAM библиотеки (Фиг. 3).
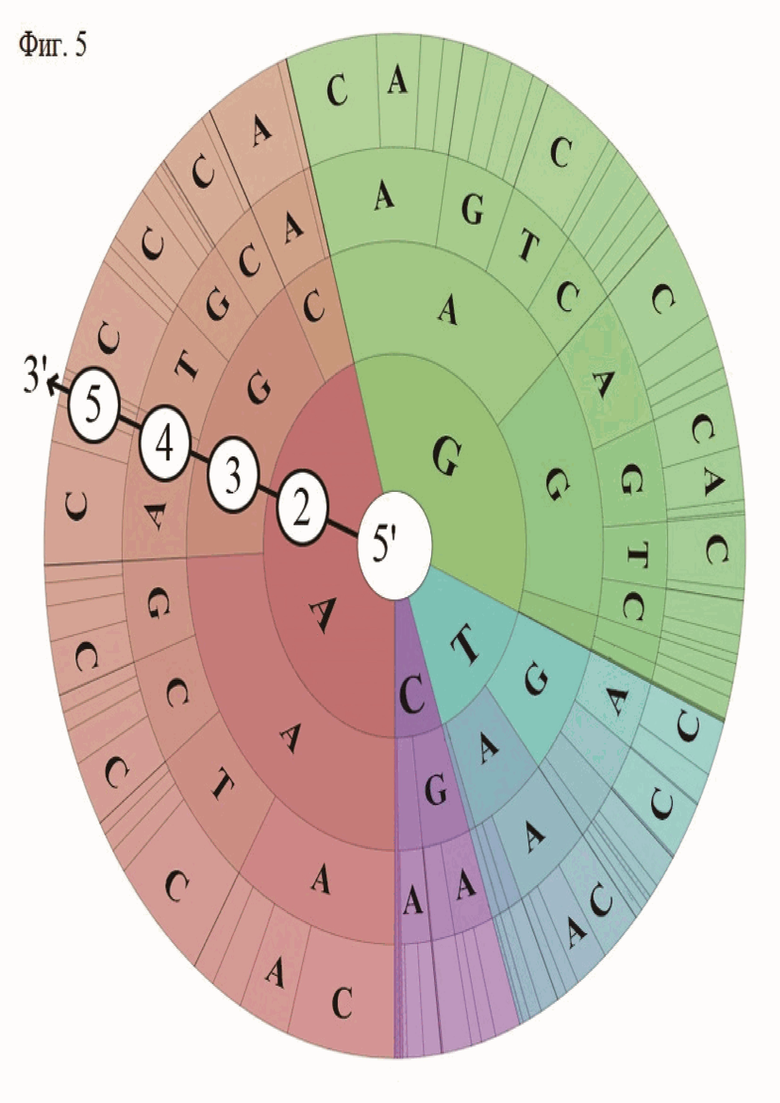
Реакцию разрезания библиотеки повторяли в подобранных условиях. Продукты реакции наносили на 2% агарозный гель и подвергали электрофорезу. Непорезанные фрагменты ДНК длиной 374 п.н. экстрагировали из геля и подготавливали для высокоэффективного секвенирования с помощью набора NEB NextUltra II. Образцы секвенировали на платформе Illumina и далее проводили анализ последовательностей биоформатическими методами: определяли разницу в представленности нуклеотидов в отдельных позициях PAM (NNNNNNN) в сравнении с контрольным образцом. Для анализа результатов были построены PAM лого (Фиг. 4) и PAM колесо (Фиг. 5).
Анализ данных указывают на значимость 2, 3, 4 и 5 позиций PAM. Таким образом, в результате in vitro анализа удалось установить предположительную PAM последовательность для CoCas9: 5'-N(A/G)A(A/T)(C/A)-3'. Эта последовательность является предположительной в силу неточности результатов, получаемых скрининговыми подходами к определению PAM.
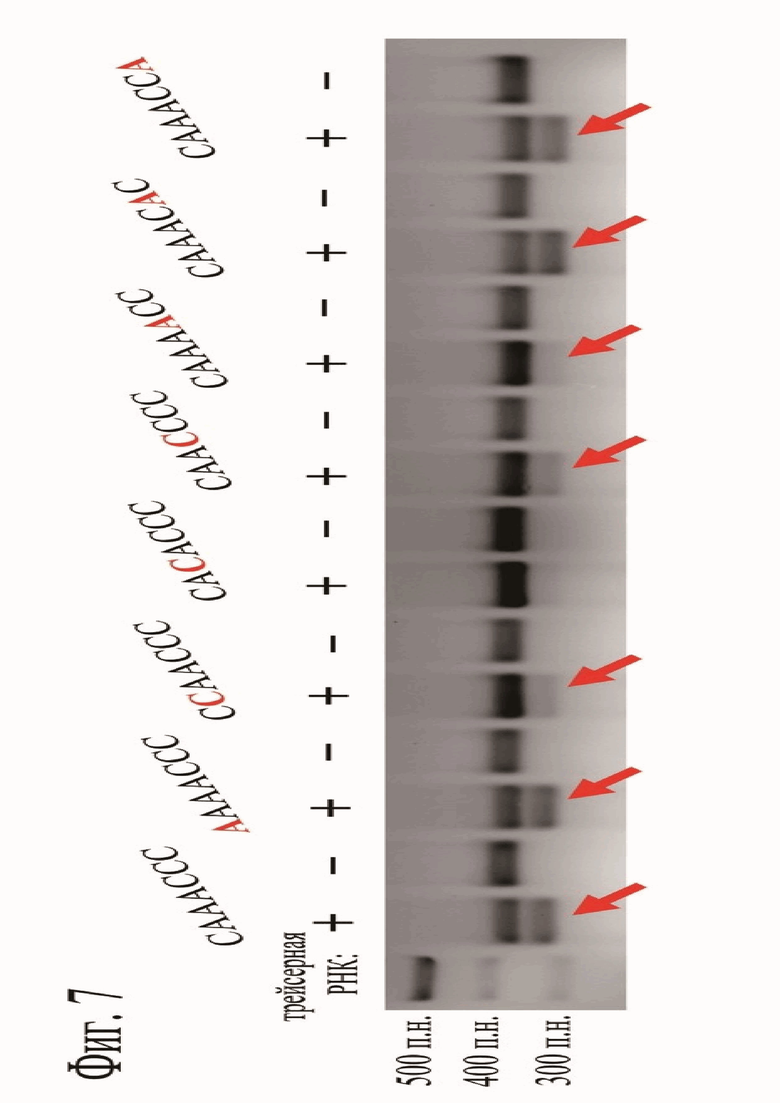
Для подтверждения наличия нуклеазной активности у CoCas9 проводили реакции разрезания ДНК фрагментов, содержащих ДНК-мишень 5'-atctcctttcattgagcac-3', фланкированную PAM последовательностями, выбранными по результатам PAM скрининга (5'-AACAACG-3', 5'-CAAACCC-3', 5'-CAAACAA-3', 5'-CAAACTA-3', 5'-CAAACAC-3', 5'-AAATCCA-3', 5'-AAAACCC-3', 5'-CAAACCG-3', 5'-AAAACTC-3', 5'-CAAACAG-3', 5'-AAAAACG-3', 5'-CAAAACC-3', последовательности PAM расположены в порядке уменьшения эффективности узнавания согласно результатам анализа).
CoCas9 разрезал большинство мишеней, фланкированных выбранными PAM, менее эффективно разрезав 5'-AAAAACG-3', 5'-CAAAACC-3', показавшие меньшую долю разрезания в PAM скрининге (Фиг. 6).
Далее для уточнения PAM последовательности была произведена проверка значимости отдельных нуклеотидных позиций.
Для этого эксперимента была выбрана последовательность PAM 5'-CAAACCC-3', на которой CoCas9 продемонстрировал высокую нуклеазную активность (Фиг. 6).
Реакции разрезания (Фиг. 7 и Фиг. 8) проводили in vitro с использованием ДНК фрагментов, содержащих ДНК-мишень 5'-atctcctttcattgagcac-3', фланкированную PAM последовательностью 5'-CAAACCC-3' (или ее производных): 5'-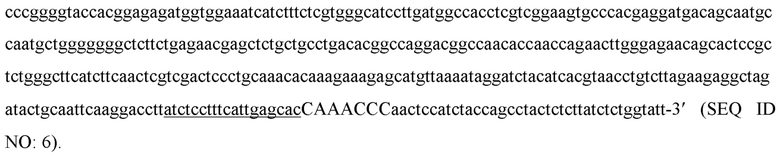
Все реакции разрезания ДНК проводили в следующих условиях:
1x CutSmart буфер
400 нМ CoCas9
40 нМ ДНК
2 мкМ крРНК
2 мкМ трРНК
Время инкубации - 30 минут, температура проведения реакции 37°C.
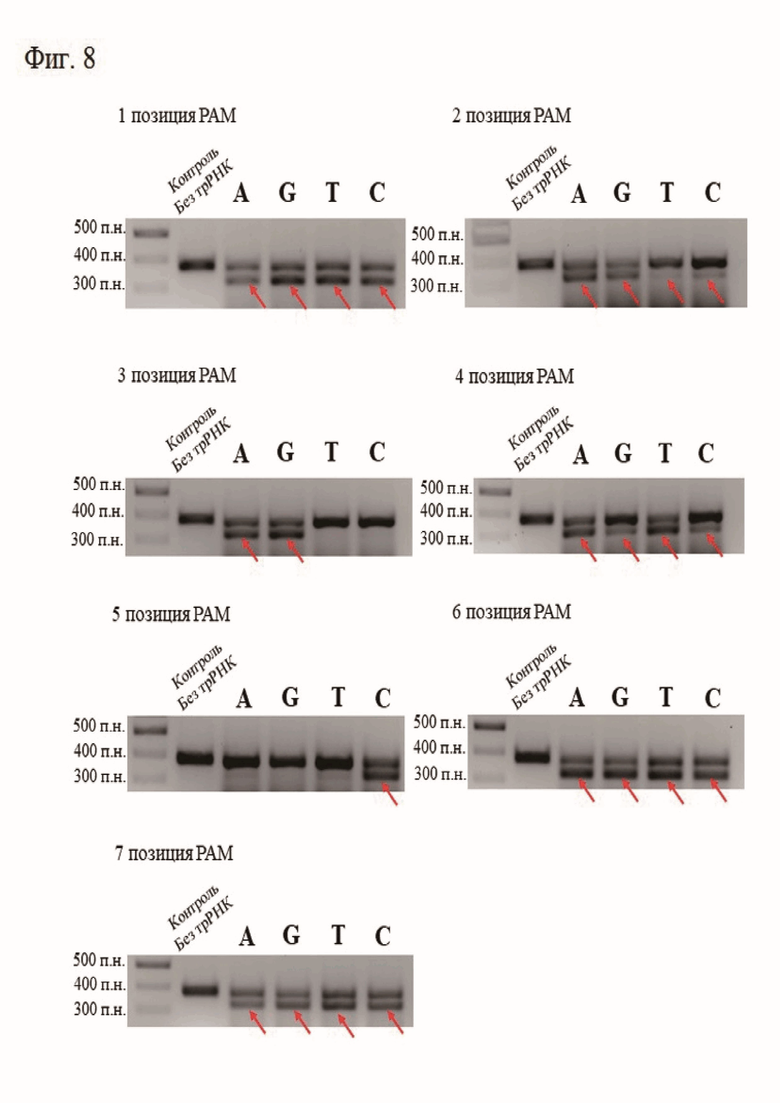
Замена нуклеотидов в каждом положении PAM (пурин на пиримидин и наоборот) показала, что значимыми являются только 2, 3, 4 5 позиции, подтвердив результаты PAM скрининга (Фиг. 7).
Далее каждый нуклеотид PAM был заменен на все возможные варианты нуклеотидов (Фиг. 8). Замена незначимых 1, 6 и 7 позиции PAM на все четыре возможные варианта нуклеотидов не повлияла на эффективность работы белка (Фиг. 8).
В позициях 2 и 3 CoCas9 требует наличия аденина или гуанина, в позиции 4 требует аденин или тимин. А при замене цитозина в пятой позиции белок практически переставал работать.
Эти данные согласуются с результатами PAM скрининга (Фиг. 5)
В результате проведенных исследований удалось сделать следующий вывод: PAM, распознаваемый нуклеазой CoCas9, соответствует следующей формуле 5'-N(A/G)(A/G)(A/T)C-3' (5'-NRRWC-3').
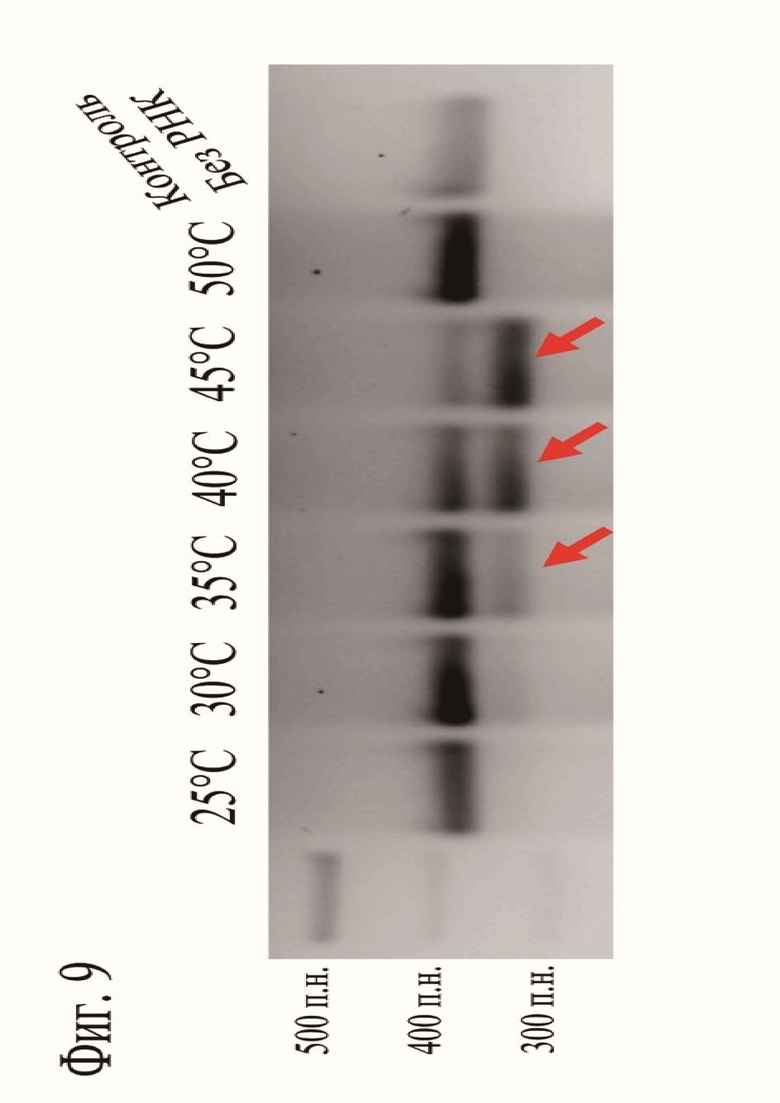
Дополнительно был исследован температурный оптимум нуклеазной активности белка CoCas9 (Фиг. 9). В результате было показано, что белок активен в диапазоне температур 35-45°С.
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Пример 1. Использование гибридной направляющей РНК для разрезания ДНК мишени.
sgRNA - форма направляющих РНК, которая представляет собой слитые воедино трРНК (трейсерная РНК) и крРНК. Для подбора оптимальной sgRNA были сконструированы два варианта этой последовательности, отличающиеся длиной трРНК - крРНК дуплекса. РНК синтезировали in vitro и проводили с ними эксперименты по разрезанию ДНК -мишени.
В качестве гибридных РНК были использованы следующие РНК последовательности:
1 - sgRNA1 28DR: UAUCUCCUUUCAUUGAGCACGUUGUGAAUUGCUUUCAAAUUUUGUAGUGAAAGUCGCACAAUUUGAAAGCAAUUCACAAUAAGGAUUAUUCCGUUGUGAAAACAUUUAAAGGAGCCCUAUCAUUAUAUUAGUGAUAGGGUUCUUUUUU (SEQ ID NO: 7);
2 - sgRNA2 35DR:
UAUCUCCUUUCAUUGAGCACGUUGUGAAUUGCUUUCAAAUUUUGUAGUUUUGCGAGAAAGUCGCACAAUUUGAAAGCAAUUCACAAUAAGGAUUAUUCCGUUGUGAAAACAUUUAAAGGAGCCCUAUCAUUAUAUUAGUGAUAGGGUUCUUUUUU (SEQ ID NO: 8).
Жирным шрифтом обозначена 20-нуклеотидная последовательность, обеспечивающая спаривание с ДНК -мишенью (вариабельная часть sgRNA). Кроме того, в эксперименте делали контрольную пробу без РНК, а также положительный контроль - разрезание мишени с помощью крРНК+трРНК.
В качестве ДНК мишени использовалась последовательность, содержащая сайт узнавания 5'-tatctcctttcattgagcac-3' с соответствующим консенсусу PAM CAAACCC: 5'-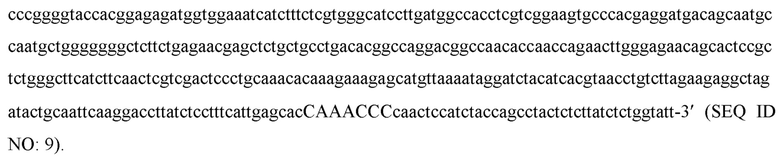
Жирным шрифтом обозначен сайт узнавания, заглавными буквами PAM.
Реакцию проводили в следующих условиях: концентрация ДНК последовательности, содержащей PAM (CAAACCC) - 40 нМ, концентрация белка - 400 нМ, концентрация РНК - 2 мкМ; время инкубирования - 30 минут, температура инкубирования - 37°С.
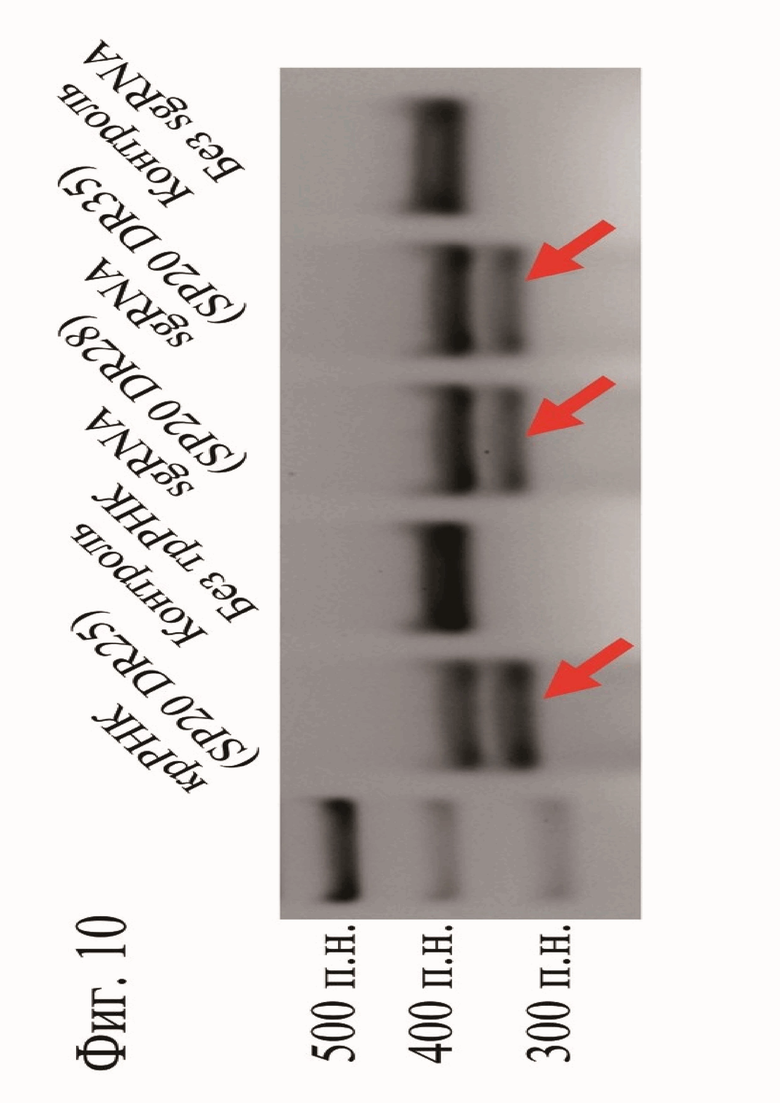
Подобранные sgRNA1 и sgRNA2 оказались так же эффективны, как и нативные последовательности трРНК и крРНК (Фиг. 10).
Эти варианты гибридной РНК могут быть использованы для разрезания любой другой целевой ДНК при изменении последовательности, непосредственно спаривающейся с ДНК -мишенью.
Пример 2. Белки Cas9 из близкородственных организмов, относящихся к Capnocytophaga ochracea.
На сегодняшний день в Capnocytophaga ochracea не охарактеризовано ни одного фермента системы CRISPR-Cas9. Сравнимый по размерам белок Cca1 из Capnocytophaga canis (также относится к белкам Cas9) идентичен CoCas9 на 66.34% (Фиг. 11, степень идентичности была рассчитана по программе BLASTp, default parameters). При этом значительная часть отличий нуклеаз приходится на домен, взаимодействующий с PAM последовательностью (степень идентичности обоих доменов 67%). Различия этих доменов обуславливают то, что нуклеазы взаимодействуют с разными PAM (PAM Cca1 5'-BRTTTTT-3').
Таким образом, белок CoCas9 существенно отличается по аминокислотной последовательности от других Cas9 белков, изученных на сегодняшний день.
Специалисту в области генетической инженерии очевидно, что полученный и охарактеризованный в данном Описании вариант последовательности белка CoCas9 может быть изменен без изменения функции самого белка (например, направленным мутагенезом аминокислотных остатков, напрямую не влияющих на функциональную активность (Sambrook et al., Molecular Cloning: A Laboratory Manual, (1989), CSH Press, pp. 15.3-15.108)). В частности, специалисту известно, что могут быть изменены неконсервативные аминокислотные остатки, не затрагивающие остатки, определяющие функциональность белка (определяющие его функцию или структуру). Примерами таких изменений могут служить замены неконсервативных аминокислотных остатков на гомологичные. В некоторых вариантах осуществления изобретения возможно использование белка, содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного непосредственно перед нуклеотидной последовательностью 5'-N(A/G)(A/G)(A/T)C-3' в указанной молекуле ДНК. Гомологичные белки могут быть получены путем мутагенеза (например, сайт-направленного или ПЦР-опосредуемого мутагенеза) соответствующих молекул нуклеиновых кислот с последующим тестированием кодируемого модифицированного белка Cas9 на сохранение его функций в соответствии с описанными здесь функциональными анализами.
Пример 3. Описанная в настоящем изобретении система CoCas9 в комплексе с направляющими РНК может быть использована для изменения последовательности геномной ДНК многоклеточного организма, в том числе эукариотического. Для введения система CoCas9 в комплексе с направляющими РНК в клетки этого организма (во все клетки или в часть клеток) могут быть применены различные подходы, известные специалистам. Например, методы доставки CRISPR-Cas9 систем в клетки организмов раскрыты в источниках (Liu C et al., Delivery strategies of the CRISPR-Cas9 gene-editing system for therapeutic applications. J Control Release. 2017 Nov 28;266:17-26; Lino CA et al., Delivering CRISPR: a review of the challenges and approaches. Drug Deliv. 2018 Nov;25(1):1234-1257), и в источниках, раскрытых внутри этих источников.
Для эффективной экспрессии нуклеазы CoCas9 в эукариотических клетках будет желательно провести оптимизацию кодонов для аминокислотной последовательности белка CoCas9 методами, известными специалистам (например, IDT codon optimization tool).
Для эффективной работы нуклеазы CoCas9 в эукариотических клетках необходимо обеспечить импорт этого белка внутрь ядра эукариотической клетки. Для этого можно использовать сигнал ядерной локализации из Т-антигена вируса SV40 (Lanford et al., Cell, 1986, 46: 575-582), соединенный с последовательностью CoCas9 с помощью спейсерной последовательности, описанной в Shen B, et al. "Generation of gene-modified mice via Cas9/RNA-mediated gene targeting", Cell Res. 2013 May;23(5):720-3 или без нее. Таким образом, полная аминокислотная последовательность нуклеазы, транспортируемой внутрь ядра эукариотической клетки, будет представлять собой следующую последовательность: MAPKKKRKVGIHGVPAA-CoCas9-KRPAATKKAGQAKKKK (далее CoCas9 NLS). Для доставки белка с приведенной выше аминокислотной последовательностью, могут быть использованы по меньшей мере два подхода.
Доставка в виде гена осуществляется путем создания плазмиды, несущей ген CoCas9 NLS под регуляцией промотора (например, CMV промотора) и последовательности, кодирующей направляющие РНК под регуляцией U6 промотора. В качестве ДНК- мишеней используются ДНК последовательности фланкированные 5'-N(A/G)(A/G)(A/T)C-3', например, последовательности гена grin2b человека:
5'-CAGCTGAAGTAATGTTAGAG-3'
Таким образом, кассета для экспрессии sgРНК выглядит следующим образом:
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccg CAGCTGAAGTAATGTTAGAGGTTGTGAATTGCTTTCAAATTTTGTAGTGAAAGTCGCACAATTTGAAAGCAATTCACAATAAGGATTATTCCGTTGTGAAAACATTTAAAGGAGCCCTATCATTATATTAGTGATAGGGTTCTTTTTT (SEQ ID NO: 10).
Жирным шрифтом выделена последовательность U6 промотора, далее идет последовательность, необходимая для узнавания целевой ДНК (заглавными буквами), а далее идет последовательность, образующая структуру sgRNA (заглавные буквы жирным шрифтом).
Плазмидную ДНК очищают и трансфицируют в клетки человека HEK293 c помощью реагента Lipofectamine 2000 (Thermo Fisher Scientific). Клетки инкубируют в течение 72 часов, после чего из них выделяется геномная ДНК с помощью колонок для очистки геномной ДНК (Thermo Fisher Scientific). Целевой ДНК сайт анализируется с помощью секвенирования на платформе Illumina с целью определения числа вставок-делеций в ДНК, происходящих в целевом сайте по причине направленного двунитевого разрыва и последующей его репарации.
Для амплификации целевых фрагментов используют праймеры, фланкирующие предположительное место внесения разрыва.
После амплификации пробы готовятся по протоколу реагента Ultra II DNA Library Prep Kit for Illumina (NEB) для подготовки образцов к высокопроизводительному секвенированию. Затем проводится секвенирование на платформе Illumina 300cycles, прямое прочтение. Результаты секвенирования анализируются биоинформатическими методами. В качестве детекции разрезания принимается вставка или делеция нескольких нуклеотидов в целевой последовательности ДНК.
Доставка в виде рибонуклеинового комплекса осуществляется путем инкубации рекомбинантной формы CoCas9 NLS c направляющими РНК в CutSmart буфере (NEB). Рекомбинантный белок получают из бактериальных клеток-продуцентов, очищая его с помощью аффинной хроматографии (NiNTA, Qiagen) разделением по размеру (Superdex 200).
Белок смешивают с РНК в соотношении 1:2 (CoCas9 NLS : sgRNA), инкубируют в течение 10 минут на комнатной температуре, затем смесь трансфицируют в клетки.
Далее проводится анализ экстрагированной из них ДНК на предмет вставок-делеций в целевом ДНК сайте (как описано выше).
Охарактеризованная в настоящем изобретении нуклеаза CoCas9 из бактерии Capnocytophaga ochracea имеет ряд преимуществ относительно ранее охарактеризованных Cas9 белков.
CoCas9 обладает коротким отличным от других известных Cas нуклеаз PAM мотивом, необходимым для функционирования системы.
Известные на сегодняшний день большинство Cas нуклеаз, способных вносить двунитевые разрывы в ДНК, имеют сложные многобуквенные PAM последовательности, ограничивающие выбор последовательностей, пригодных для разрезания. Среди изученных Cas нуклеаз, распознающих короткие PAM, только CoCas9 может распознавать последовательности, фланкированные NRRWC мотивом.
CoCas9 - новая Cas нуклеаза, имеющая простой в использовании PAM, отличающийся от известных на сегодняшний день PAM последовательностей других нуклеаз. Белок CoCas9 разрезает с высокой эффективностью различные ДНК-мишени, в том числе и при 37°С, и может стать основой нового инструмента геномного редактирования.
Несмотря на то, что изобретение описано со ссылкой на раскрываемые варианты воплощения, для специалистов в данной области должно быть очевидно, что конкретные подробно описанные случаи приведены лишь в целях иллюстрирования настоящего изобретения, и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения. Должно быть, понятно, что возможно осуществление различных модификаций без отступления от сути настоящего изобретения.
--->
<110> Федеральное государственное бюджетное учреждение науки Институт
биологии гена Российской академии наук (Institute of Gene Biology Russian
Academy of Sciences)
<120> Средство разрезания ДНК на основе Cas9 белка из бактерии
Capnocytophaga ochracea
<160> 10
<210> 1
<211> 1426
<212> PRT
<213> Capnocytophaga ochracea
<400> 1
Met Lys Asn Ile Leu Gly Leu Asp Leu Gly Thr Thr Ser Ile Gly Phe 16
5 10 15
Ala His Ile Val Glu Asp Glu Asn Lys Glu Lys Ser Glu Ile Lys Glu 32
20 25 30
Leu Gly Val Arg Ile Val Ser Leu Thr Thr Asp Glu Gln Ser Asp Phe 48
35 40 45
Glu Lys Gly Lys Ser Ile Thr Thr Asn Ala Asn Arg Thr Leu Lys His 64
50 55 60
Gly Ala Arg Leu Asn Leu Asp Arg Tyr Gln Gln Arg Arg Lys Tyr Leu 80
65 70 75 80
Ile Asp Leu Leu Gln Lys Ala Asn Leu Ile Thr Pro Ser Ser Ile Leu 96
85 90 95
Ala Glu Asn Gly Lys Asn Thr Thr His Ser Thr Trp Gln Leu Arg Ala 112
100 105 110
Lys Ala Val Thr Glu Arg Ile Glu Lys Glu Glu Phe Ala Arg Ile Leu 128
115 120 125
Leu Ala Ile Asn Lys Lys Arg Gly Tyr Lys Ser Ser Arg Lys Ala Lys 144
130 135 140
Thr Glu Asp Glu Gly Gln Ala Ile Asp Gly Met Ala Ile Ala Lys Arg 160
145 150 155 160
Leu Tyr Asp Glu Asn Leu Thr Pro Gly Gln Leu Ser Leu Gln Leu Leu 176
165 170 175
Gln Gln Asn Lys Lys Leu Leu Pro Asp Phe Tyr Arg Ser Asp Leu Gln 192
180 185 190
Lys Glu Phe Asp Leu Val Trp Asn Phe Gln Lys Gln Phe Tyr Pro Asp 208
195 200 205
Ile Leu Thr Asp Ile Phe Tyr Lys Glu Leu Gln Gly Lys Gly Lys Asp 224
210 215 220
Ala Thr Ser Lys Ala Phe Ser Lys Arg Tyr His Phe Asp Thr Thr Glu 240
225 230 235 240
Asn Lys Gly Ser Lys Glu Ser Val Arg Leu Gln Ala Tyr Gln Trp Arg 256
245 250 255
Ala Glu Ala Ile Ser Lys Gln Leu Ser Lys Glu Glu Val Ala Tyr Val 272
260 265 270
Leu Thr Glu Ile Asn Asn Asn Leu Asn Asn Ala Ser Gly Tyr Leu Gly 288
275 280 285
Ala Ile Ser Asp Arg Ser Lys Glu Leu Tyr Phe Asn Arg Gln Thr Val 304
290 295 300
Gly Gln Tyr Leu Tyr Ala Lys Leu Gln Glu Asn Arg His Asn Ser Leu 320
305 310 315 320
Lys Asn Lys Val Phe Tyr Arg Gln Asp Tyr Leu Asp Glu Phe Glu Arg 336
325 330 335
Ile Trp Glu Thr Gln Ala Ser Phe His Lys Glu Leu Thr Asp Glu Leu 352
340 345 350
Lys Lys Gln Ile Arg Asp Val Val Ile Phe Tyr Gln Arg Lys Pro Lys 368
355 360 365
Ser Gln Lys Gly Leu Ile Ser Phe Cys Glu Phe Glu Ser Lys Glu Ile 384
370 375 380
Glu Ile Glu Lys Asp Gly Lys Thr Ile Thr Lys Asn Ile Gly Ala Arg 400
385 390 395 400
Val Val Pro Lys Ser Ser Pro Leu Phe Gln Glu Phe Lys Ile Trp Gln 416
405 410 415
Ile Leu Asn Asn Val Ile Cys Lys Arg Lys Gly Ile Arg Lys Lys Lys 432
420 425 430
Ile Ser Ala Lys Thr Thr Gln Leu Asp Leu Leu Asn Glu Ser Ser Gln 448
435 440 445
Thr Ile Phe Ser Leu Asp Met Glu Cys Lys Gln Leu Leu Phe Asp Glu 464
450 455 460
Leu Asn Leu Lys Gly Asp Leu Lys Ser Asp Lys Val Leu Lys Leu Leu 480
465 470 475 480
Gly Tyr Ser Pro Gln Glu Trp Glu Ile Asn Tyr Asn Gln Leu Glu Gly 496
485 490 495
Asn Arg Thr Gln Lys Ala Leu Tyr Glu Ala Tyr Leu Lys Ile Val Glu 512
500 505 510
Met Glu Ala His Asp Val Lys Asp Ile Leu Gln Ile Lys Ser Ala Lys 528
515 520 525
Asp Asp Trp Ser Leu Asp Glu Ser Pro Leu Ser Ala Ser Glu Ile Arg 544
530 535 540
Glu Lys Val Lys Ala Ile Phe Gln Thr Leu Gly Ile Cys Thr Lys Ile 560
545 550 555 560
Leu Tyr Phe Asp Pro Leu Leu Pro Val Lys Glu Phe Glu Glu Gln Asp 576
565 570 575
Ser Tyr Gln Leu Trp His Leu Leu Tyr Ser Tyr Glu Ser Asp Asp Ser 592
580 585 590
Thr Ser Gly Asn Glu Thr Leu Tyr Arg Ile Leu Glu Lys Lys Tyr Ala 608
595 600 605
Phe Lys Arg Glu His Ala Arg Ile Leu Ala Asn Val Ala Leu Gln Asp 624
610 615 620
Asp Tyr Gly Ser Leu Ser Thr Lys Ala Ile Arg Lys Ile Tyr Pro Asn 640
625 630 635 640
Ile Lys Glu Asn Gln Tyr Ser Thr Ala Cys Glu Lys Ala Gly Tyr Lys 656
645 650 655
His Ser Lys Leu Ser Leu Thr Thr Glu Glu Leu Glu Ala Arg Glu Leu 672
660 665 670
Lys Asn Ile Ile Pro Leu Leu Lys Lys Asn Ala Leu Arg Asn Pro Val 688
675 680 685
Val Glu Lys Ile Leu Asn Gln Met Ile Asn Val Val Asn Ala Leu Ile 704
690 695 700
Glu Lys Asn Ser Glu Arg Asp Ala Glu Gly Lys Ile Thr Lys Tyr Phe 720
705 710 715 720
His Phe Asp Glu Ile Arg Ile Glu Leu Ala Arg Glu Leu Lys Lys Asn 736
725 730 735
Ala Gln Lys Arg Tyr Glu Met Thr Gln Asn Ile Asn Lys Ala Lys Leu 752
740 745 750
Glu His Gln Lys Ile Ser Glu Ile Leu Gln Lys Glu Phe Gly Ile Lys 768
755 760 765
Asn Pro Thr Lys Ser Asp Ile Ile Arg Tyr Arg Leu Tyr Gln Glu Leu 784
770 775 780
Glu His Asn Gly Tyr Lys Glu Leu Tyr Thr Asn Ala Pro Ile Ala Arg 800
785 790 795 800
Asp Met Leu Phe Ser Lys Asn Ile Glu Ile Glu His Ile Val Pro Lys 816
805 810 815
Ala Arg Val Phe Asp Asp Ser Phe Ser Asn Lys Thr Leu Thr Phe His 832
820 825 830
Arg Ile Asn Ser Asp Lys Gly Glu Tyr Thr Ala Phe Asp Tyr Ile Thr 848
835 840 845
Ser Leu Asn Ser Glu Glu Glu Leu Asn Gln Tyr Leu Thr Arg Val Glu 864
850 855 860
Asn Ala Tyr Lys Thr Lys Ser Ile Ser Pro Thr Lys Tyr Lys Asn Leu 880
865 870 875 880
Leu Lys Lys Ala Ser Glu Ile Gly Asp Asp Phe Ile Asn Arg Asp Leu 896
885 890 895
Arg Asp Thr Gln Tyr Ile Ala Lys Lys Ala Lys Glu Ile Leu Phe Gln 912
900 905 910
Val Thr Lys Asn Val Leu Ser Thr Ser Gly Ser Ile Thr Asp Arg Leu 928
915 920 925
Arg Glu Asp Trp Gly Leu Val Asp Val Met Lys Glu Leu Asn Met Pro 944
930 935 940
Lys Tyr Gln Ser Leu Gly Leu Thr Glu Val Glu Glu Arg Lys Asp Gly 960
945 950 955 960
Asn Lys Val Thr Val Ile Lys Asn Trp Thr Lys Arg Asn Asp His Arg 976
965 970 975
His His Ala Met Asp Ala Leu Thr Val Ala Phe Thr Lys Pro Ser Tyr 992
980 985 990
Ile Gln Tyr Leu Asn His Leu Asn Ala Arg Lys Asp Glu Asn Asn Lys 1008
995 1000 1005
Asn Tyr Ser Val Ile Leu Ala Ile Glu Glu Lys Glu Thr Ile Lys Val 1024
1010 1015 1020
Pro Thr Asn Asn Gly Lys Asn Lys Arg Val Phe Ile Glu Pro Ile Pro 1040
1025 1030 1035 1040
Asn Phe Arg Gln Val Ala Lys Lys His Leu Glu Glu Ile Phe Ile Ser 1056
1045 1050 1055
His Lys Ala Lys Asn Lys Val Val Thr Lys Asn Thr Asn Lys Pro Ala 1072
1060 1065 1070
Gly Thr Asp Lys Gln Gln Ile Thr Leu Thr Pro Arg Gly Gln Leu His 1088
1075 1080 1085
Lys Glu Thr Ile Tyr Gly Lys Tyr Gln Tyr Tyr Ile Asn Lys Glu Glu 1104
1090 1095 1100
Lys Ile Gly Val Lys Phe Asp Glu Arg Thr Ile Ala Lys Val Ser Asn 1120
1105 1110 1115 1120
Pro Val Tyr Arg Glu Ala Leu Leu Lys Arg Leu Gln Ala Asn Asp Asn 1136
1125 1130 1135
Asp Pro Lys Lys Ala Phe Ala Gly Lys Asn Ala Leu Ser Lys Asn Pro 1152
1140 1145 1150
Ile Tyr Leu Asp Glu Ser Lys Thr Lys Thr Leu Pro Glu Lys Val Asn 1168
1155 1160 1165
Leu Thr Tyr Leu Glu Glu Asp Phe Ser Ile Arg Lys Asp Ile Ser Pro 1184
1170 1175 1180
Asp Asn Phe Lys Asp Leu Lys Ser Ile Glu Lys Val Ile Asp Gln Gly 1200
1185 1190 1195 1200
Val Lys Arg Ile Leu Ile Lys Arg Leu Gln Ala Tyr Asp Asn Asp Pro 1216
1205 1210 1215
Lys Lys Ala Phe Val Asp Leu Glu Lys Asn Pro Ile Trp Leu Asn Lys 1232
1220 1225 1230
Glu Lys Gly Ile Ala Ile Lys Arg Val Thr Ile Ser Gly Val Asn Asn 1248
1235 1240 1245
Ala Gln Pro Leu His Ile Gly Lys Asp His Leu Gly Lys Thr Thr Leu 1264
1250 1255 1260
Asn Lys Glu Gly Lys Glu Ile Pro Val Asp Tyr Val Ser Thr Gly Asn 1280
1265 1270 1275 1280
Asn His His Val Ala Ile Tyr Arg Asp Lys Glu Gly Asn Leu Gln Glu 1296
1285 1290 1295
Gln Ile Val Ser Phe Phe Asp Ala Val Val Arg Ala Gln Gln Gly Ile 1312
1300 1305 1310
Pro Ile Ile Asp Lys Thr Tyr Lys Gln Ala Glu Gly Trp Gln Phe Leu 1328
1315 1320 1325
Phe Thr Met Lys Gln Asn Glu Met Phe Val Phe Pro Asn Ala Thr Thr 1344
1330 1335 1340
Gly Phe Asn Pro Ala Glu Ile Asp Leu Leu Asp Pro Lys Asn Lys Lys 1360
1345 1350 1355 1360
Leu Ile Ser Pro Asn Leu Phe Arg Val Gln Lys Ile Ala Thr Lys Asp 1376
1365 1370 1375
Tyr Phe Phe Arg His His Leu Glu Thr Asn Val Glu Thr Asp Asn Ile 1392
1380 1385 1390
Leu Lys Asn Val Thr Trp Lys Arg Glu Gly Leu Ser Gly Leu Lys Asp 1408
1395 1400 1405
Ile Val Lys Val Arg Ile Asn His Leu Gly Asp Ile Val Ser Ile Gly 1424
1410 1415 1420
Glu Tyr 1426
1425
<210> 2
<211> 96
<212> РНК
<213> artificial sequence
<220>
<223> CoCas9 трРНК
<400> 2
gucgcacaau uugaaagcaa uucacaauaa ggauuauucc guugugaaaa cauuuaaagg 60
agcccuauca uuauauuagu gauaggguuc uuuuuu 96
<210> 3
<211> 66
<212> РНК
<213> artificial sequence
<220>
<223> CoCas9 крРНК
<400> 3
nnnnnnnnnn nnnnnnnnnn guugugaauu gcuuucaaau uuuguaguuu ugcgauugau 60
aacaac 66
<210> 4
<211> 374
<212> ДНК
<213> artificial sequence
<220>
<223> ДНК-библиотека
<400> 4
cccggggtac cacggagaga tggtggaaat catctttctc gtgggcatcc ttgatggcca 60
cctcgtcgga agtgcccacg aggatgacag caatgccaat gctggggggg ctcttctgag 120
aacgagctct gctgcctgac acggccagga cggccaacac caaccagaac ttgggagaac 180
agcactccgc tctgggcttc atcttcaact cgtcgactcc ctgcaaacac aaagaaagag 240
catgttaaaa taggatctac atcacgtaac ctgtcttaga agaggctaga tactgcaatt 300
caaggacctt atctcctttc attgagcacN NNNNNNaact ccatctacca gcctactctc 360
ttatctctgg tatt 374
<210> 5
<211> 65
<212> РНК
<213> artificial sequence
<220>
<223> крРНК
<400> 5
uaucuccuuu cauugagcac guugugaauu gcuuucaaau uuuguaguuu ugcgauugau 60
aacaa 65
<210> 6
<211> 374
<212> ДНК
<213> artificial sequence
<220>
<223> ДНК-библиотека, содержащая ДНК-мишень 5'-atctcctttcattgagcac-3',
фланкированную PAM последовательностью 5’-CAAACCC-3’
<400> 6
cccggggtac cacggagaga tggtggaaat catctttctc gtgggcatcc ttgatggcca 60
cctcgtcgga agtgcccacg aggatgacag caatgccaat gctggggggg ctcttctgag 120
aacgagctct gctgcctgac acggccagga cggccaacac caaccagaac ttgggagaac 180
agcactccgc tctgggcttc atcttcaact cgtcgactcc ctgcaaacac aaagaaagag 240
catgttaaaa taggatctac atcacgtaac ctgtcttaga agaggctaga tactgcaatt 300
caaggacctt atctcctttc attgagcacC AAACCCaact ccatctacca gcctactctc 360
ttatctctgg tatt 374
<210> 7
<211> 148
<212> РНК
<213> artificial sequence
<220>
<223> sgRNA1 28DR
<400> 7
uaucuccuuu cauugagcac guugugaauu gcuuucaaau uuuguaguga aagucgcaca 60
auuugaaagc aauucacaau aaggauuauu ccguugugaa aacauuuaaa ggagcccuau 120
cauuauauua gugauagggu ucuuuuuu 148
<210> 8
<211> 155
<212> РНК
<213> artificial sequence
<220>
<223> sgRNA2 35DR
<400> 8
uaucuccuuu cauugagcac guugugaauu gcuuucaaau uuuguaguuu ugcgagaaag 60
ucgcacaauu ugaaagcaau ucacaauaag gauuauuccg uugugaaaac auuuaaagga 120
gcccuaucau uauauuagug auaggguucu uuuuu 155
<210> 9
<211> 375
<212> ДНК
<213> artificial sequence
<220>
<223> ДНК-библиотека, содержащая сайт узнавания 5'-tatctcctttcattgagcac-3'
с соответствующим консенсусу PAM CAAACCC
<400> 9
cccggggtac cacggagaga tggtggaaat catctttctc gtgggcatcc ttgatggcca 60
cctcgtcgga agtgcccacg aggatgacag caatgccaat gctggggggg ctcttctgag 120
aacgagctct gctgcctgac acggccagga cggccaacac caaccagaac ttgggagaac 180
agcactccgc tctgggcttc atcttcaact cgtcgactcc ctgcaaacac aaagaaagag 240
catgttaaaa taggatctac atcacgtaac ctgtcttaga agaggctaga tactgcaatt 300
caaggacctt atctcctttc attgagcacC AAACCCcaac tccatctacc agcctactct 360
cttatctctg gtatt 375
<210> 10
<211> 398
<212> ДНК
<213> artificial sequence
<220>
<223> кассета для экспрессии sgРНК
<400> 10
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg cagctgaagt aatgttagag gttgtgaatt gctttcaaat tttgtagtga 300
aagtcgcaca atttgaaagc aattcacaat aaggattatt ccgttgtgaa aacatttaaa 360
ggagccctat cattatatta gtgatagggt tctttttt 398
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| ПРИМЕНЕНИЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA ДЛЯ МОДИФИКАЦИИ ГЕНОМНОЙ ДНК В КЛЕТКАХ | 2019 |
|
RU2724470C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| СПОСОБ СОЗДАНИЯ НОВЫХ МУТАЦИЙ В ОРГАНИЗМАХ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2833881C1 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9 II ТИПА И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2794774C1 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |




Группа изобретений относится к биотехнологии и описывает новую бактериальную нуклеазу системы CRISPR-Cas9 из бактерии Capnocytophaga ochracea, а также ее применение для образования строго специфичных двунитевых разрывов в молекуле ДНК. Данная нуклеаза обладает необычными свойствами и может быть использована в качестве инструмента для внесения изменений в строго определенных местах в последовательности геномной ДНК одноклеточных или многоклеточных организмов. Изобретение позволяет повысить универсальность доступных систем CRISPR-Cas9, что позволит использовать нуклеазы Cas9 из различных организмов для разрезания геномной или плазмидной ДНК в большем количестве специфических сайтов и при различных условиях. 2 н. и 3 з.п. ф-лы, 11 ил., 3 пр.
1. Применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1 или содержащего аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности SEQ ID NO: 1 и имеет отличия по сравнению с SEQ ID NO: 1 только в неконсервативных аминокислотных остатках, для образования двунитевого разрыва в молекуле ДНК, расположенного непосредственно перед нуклеотидной последовательностью 5'-N(A/G)(A/G)(A/T)C-3' в указанной молекуле ДНК.
2. Применение по п. 1, характеризующееся тем, что образование двунитевого разрыва в молекуле ДНК происходит при температуре от 35°C до 45°C.
3. Применение белка по п. 1, где белок содержит аминокислотную последовательность SEQ ID NO: 1.
4. Способ изменения последовательности геномной ДНК одноклеточного или многоклеточного организма, включающий введение в по меньшей мере одну клетку этого организма эффективного количества: а) либо белка, содержащего аминокислотную последовательность SEQ ID NO: 1, либо нуклеиновой кислоты, кодирующей белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и б) либо направляющей РНК, содержащей последовательность, образующую дуплекс с нуклеотидной последовательностью участка геномной ДНК организма, непосредственно примыкающей к нуклеотидной последовательности 5'-N(A/G)(A/G)(A/T)C-3', и взаимодействующей с указанным белком после образования дуплекса, либо последовательности ДНК, кодирующей указанную направляющую РНК; при этом взаимодействие указанного белка с направляющей РНК и нуклеотидной последовательностью 5'-N(A/G)(A/G)(A/T)C-3' приводит к образованию двунитевого разрыва в последовательности геномной ДНК, непосредственно примыкающей к последовательности 5'-N(A/G)(A/G)(A/T)C-3'.
5. Способ по п. 4, дополнительно включающий введение экзогенной последовательности ДНК одновременно с направляющей РНК.
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ DEFLUVIIMONAS SP. | 2018 |
|
RU2712492C1 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ МОДИФИКАЦИИ ЗАДАННОЙ ПОСЛЕДОВАТЕЛЬНОСТИ НУКЛЕИНОВОЙ КИСЛОТЫ-МИШЕНИ | 2012 |
|
RU2663354C2 |
| WO 2014093661 A9, 19.06.2014 | |||
| База данных: NCBI Reference Sequence: WP_015781852.1, 20.05.2013. | |||
Авторы
Даты
2022-08-15—Публикация
2021-10-19—Подача