ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к лечению и диагностике рака легких и его метастазов. Более конкретно, изобретение относится к источникам антигена, обеспечивающим, по меньшей мере, иммуногенную часть белка, ассоциированного со злокачественной клеткой, который предпочтительно представляет собой белок блестящей оболочки (ZP), особенно белок ZP3. Такие источники антигенов можно использовать в вакцинах и фармацевтических композициях для терапевтического и профилактического лечения первичного и/или рецидивирующего рака легких и его метастазов.
ПРЕДШЕСТВУЮЩИЙ ИЗОБРЕТЕНИЮ УРОВЕНЬ ТЕХНИКИ
Рак легких относится к злокачественным опухолям, происходящим из ткани легкого, которые могут возникать в любой части легкого, хотя подавляющим большинством (приблизительно 90-95%) раков легких являются карциномы - злокачественные новообразования, которые возникают из эпителиальных клеток. Существует два основных типа карциномы легкого, классифицируемые гистопатологическим анализом на немелкоклеточную (80,4%) и мелкоклеточную (16,8%) карциному легкого. Злокачественные опухоли также могут возникать из плевры (называются мезотелиомами) или редко из поддерживающих тканей в пределах легких, например, кровеносных сосудов. Основными причинами любой злокачественной опухоли являются не генетические факторы, такие как воздействие канцерогенов (например, табачный дым, асбест, мышьяк, твердые частицы), ионизирующая радиация и вирусная инфекция. Эпидемиологические исследования показали, что генетические факторы могут способствовать риску развития рака легких.
Рак легких является одной из самых распространенных злокачественных опухолей. В 2007 году на долю рака легких приходилось примерно 15% всех случаев диагноза злокачественной опухоли и 28% всех смертей от злокачественной опухоли. Это четвертая наиболее диагностируемая злокачественная опухоль у мужчин и женщин, но это причина номер один ежегодной смерти от злокачественной опухоли у мужчин и женщин. Большинство случаев рака легких диагностируется на поздней стадии, что свидетельствует о неблагоприятном прогнозе. Рак легких обладает высокой летальностью, при этом 5-летний показатель выживаемости пациентов в США составляет всего 14%. Лечение рака легких зависит от специфического типа клеток злокачественной опухоли, метастазов и состояния пациента. Общие методы лечения включают (комбинации) хирургическое вмешательство, химиотерапию (например, цисплатин, карбоплатин, гемцитабин, паклитаксел, доцетаксел, этопозид, винрорелбин, топотекан, ирринотекан), лучевую терапию, иммунотерапию (например, атезолизумаб, ниволумаб, пембролизумаб) и «нацеленную терапию» (например, гефитиниб, эрлотиниб, бевацизумаб). Ряд «нацеленных агентов» находится на ранних стадиях клинической разработки, например ингибиторы циклооксигеназы-2, промотор апоптоза экзисулинд, ингибиторы протеасомы, бексаротин и ингибитор рецептора эпидермального фактора роста цетуксимаб.
Мелкоклеточную карциному легкого сначала лечат облучением и химиотерапией с цисплатином или этопозидом, иногда в комбинации с карбоплатином, гемцитабином, паклитакселом, винорелбином, топотеканом или иринотеканом. Хирургическое лечение не оказывает заметного влияния на выживаемость при мелкоклеточной карциноме легкого. При запущенной стадии мелкоклеточного рака легких можно комбинировать целекоксиб с этопозидом для улучшения исходов.
Немелкоклеточная карцинома легких (НМКРЛ) представляет собой любой тип эпителиального рака легких, кроме мелкоклеточной карциномы легкого (МКРЛ). На НМКРЛ C приходится около 85% всех случаев рака легких. Как класс, НМКРЛ относительно нечувствительны к химиотерапии, по сравнению с МКРЛ. Когда это возможно, их, в основном, лечат хирургической резекцией с лечебными намерениями, хотя химиотерапия все чаще используется как до операции (неоадъювантная химиотерапия), так и послеоперационно (адъювантная химиотерапия). В настоящее время первичное лечение НМКРЛ включает хирургическое вмешательство и химиотерапию цисплатином или карбоплатином, часто в комбинации с гемцитабином, паклитакселом, доцетакселом, этопозидом или винорелбином. Операция, как правило, является единственным вариантом при НМКРЛ, ограниченным одним легким, вплоть до стадии IIIA. Если операция больше не возможна, дают первичную химиотерапию.
В то время как существующие методы лечения приносят некоторую пользу при лечении рака легких, перспективы все еще, в основном, плохие, и все еще абсолютно необходимо найти способы лечения, которые являются более эффективными и менее токсичными. Выявление новых терапевтических мишеней для лечения рака легких является решающим шагом для реализации этой задачи.
Достижения в области молекулярной медицины повысили интерес к опухолеспецифичным антигенам, которые могут служить мишенями для различных иммунотерапевтических стратегий или стратегий с низкомолекулярными соединениями.
Чтобы служить подходящей мишенью для иммунотерапевтических стратегий против злокачественной опухоли, такие антигены должны быть высоко экспрессированы в тканях злокачественной опухоли и, в идеале, не в нормальных тканях. Однако может быть приемлемой экспрессия в тканях, которые не являются необходимыми для жизни.
Концепция лечения опухолей, экспрессирующих ZP, путем активной иммунизации была ранее описана в данной области. Например, Rahman et al. (A novel treatment strategy for ovarian cancer based on immunization with zona pellucida protein (ZP) 3. FASEB J. 2012 Jan; 26 (1): 324-33) доказывает принцип лечения злокачественных опухолей яичников у трансгенных мышей путем выявления гуморальной и клеточной вакцинации против белков ZP3 и клеток, экспрессирующих ZP3, соответственно. Кроме того, активная иммунизация белками ZP является распространенной практикой у крупных диких животных как способ стерилизации, который демонстрирует ее осуществимость и безопасность (см. например, Gupta and Minhas, Frontiers In Bioscience, 2017, Scholar, 9, 357-374, http://www.bioscience.org/2017/v9c/af/492/2.htm; Mask et al., 2015, Theriogenology; 84 (2): 261-267). Многочисленные исследования указывают на то, что снижение фертильности было связано с титрами антител (Shresta et al., 2015, Vaccine 33, 133-140; Harris et al., 1999, Protein Expr Purif; 16 (2): 298-307; Martinez and Harris, 2000, J Reprod Fertil; 120 (1): 19-32). Дополнительные дегенеративные патологии яичников были описаны для иммунизации рекомбинантными ZP3 и ZP4, в частности (например, Govind et al., 2002, Vaccine 21, 78-88; Paterson et al., 1998, Am J Reprod Immunol 40, 198-209; и Srivastava et al., 2002, Reproduction 123, 847-857). В исследовании аутоиммунного заболевания яичников исследователи продемонстрировали, что у мышей можно индуцировать оофорит (повреждение ткани яичника), вызывая ZP3-специфический CD4+ T-клеточный ответ путем вакцинации полипептидом ZP3 из 15 аминокислот, содержащим эпитоп, связывающий MHC класса II. (Rhim et al., 1992, J. Clin. Invest. 89, 28-35). Однако нет сообщений об экспрессии белка ZP при раке легких.
Целью настоящего изобретения является создание новых иммунотерапевтических стратегий для лечения и/или рецидивирующих форм рака легких и его метастазов.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Авторы настоящего изобретения неожиданно обнаружили, что белки блестящей оболочки (ZP), особенно белок Zona pellucida 3 (ZP3), экспрессируются в злокачественных клетках легкого и представляют собой подходящую мишень для иммунотерапевтических стратегий при лечении рака легких и его метастазов, а также при его диагностике.
ZP3 в норме выявляют у женщин в так называемой «zona pellucida», которая образует внеклеточный матрикс, окружающий ооцит. Эта zona pellucida (блестящая оболочка) вызывает акросомную реакцию сперматозоидов, определяет видоспецифичность оплодотворения и предотвращает полиспермию у млекопитающих. Блестящая оболочка содержит четыре главных гликопротеина, ZP1, ZP2, ZP3 и ZP4. Для удобства мы будем ссылаться в данном документе на белки блестящей оболочки или белки ZP, под которыми подразумевается как гликозилированные, так и негликозилированные формы белков блестящей оболочки, если специально не указано иное.
Настоящее изобретение основано на открытии, что злокачественные клетки легкого могут демонстрировать экспрессию белка ZP, значительную до такой степени, что могут применяться иммунотерапевтические стратегии, например, пассивная иммунизация, активная иммунизация и нацеленная терапия с использованием иммуноконъюгатов, для получения, по меньшей мере, одного из i) снижения роста или размера первичных опухолей, ii) снижения роста или размера метастазов, происходящих из них, iii) ликвидации опухолей, и iv) повышения выживаемости.
Настоящая стратегия в равной степени подходит для предотвращения метастазирования рака легких, а также для предотвращения рецидива рака легких у ранее леченных индивидуумов. Таким образом, понятно, что термины «злокачественная клетка легкого» и «злокачественная клетка легкого, экспрессирующая ZP» также включают такие клетки, полученные из злокачественных клеток легкого, поскольку они могут встречаться в других частях организма, иных чем легкие, например, в результате метастазирования.
Пример 4 в данном документе описывает доклиническое исследование гуманизированных HLA-A2 трансгенных мышей с hZP3-экспрессирующими опухолями. Вакцинация этих трансгенных мышей полипептидом hZP3 (а/к 1-383) и hZP3 HLA-A2-рестриктированными T-клеточными эпитопами индуцировала CD8+ T-клетки. Кроме того, не наблюдали увеличение размера опухоли, тогда как размер опухоли увеличился у контрольных мышей, получавших PBS.
Экспрессия белка ZP в злокачественных клетках легкого никогда не была установлена ранее. Таким образом, на известном уровне техники нет никаких указаний на то, что злокачественные клетки легкого могут на самом деле быть объектом иммунотерапии на основе ZP. Настоящее изобретение, таким образом, впервые предлагает способы лечения рака легких у индивидуума, нацеленные на белок ZP как опухолеспецифический антиген. Как будет понятно специалистам в данной области на основании настоящего описания, такие методы лечения включают пассивную иммунизацию, активную иммунизацию, а также нацеленную терапию с использованием иммуноконъюгатов.
Эти и другие аспекты настоящего изобретения будут более подробно описаны далее.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Первый аспект настоящего изобретения относится к способу терапевтического и/или профилактического лечения или диагностики первичного и/или рецидивирующего рака легких и его метастазов у нуждающегося в этом индивидуума, где способ включает введение указанному индивидууму, по меньшей мере, одного из следующих:
источник иммуногенного полипептида, способный вызывать клеточный или гуморальный иммунный ответ против человеческого белка блестящей оболочки (hZP), hZP3 или hZP3 (23-350);
Т-клетка, содержащая Т-клеточный рецептор, который связывается с комплексом МНС-пептид, где пептид является пептидом из hZP, а также из hZP3 или hZP3 (23-350);
антитело или его фрагмент, который специфически связывается с hZP, либо hZP3 или hZP3 (23-350); и
генетическая конструкция, содержащая последовательность нуклеиновых кислот, кодирующую указанный полипептид или антитело, где генетическая конструкция настроена для доставки и экспрессии в человеке.
Как применяют в настоящем документе термин «рак легких» относится к первичному и/или рецидивирующему раку легких, а также к его метастазам, которые могут быть локализованы в любом месте в организме. Предпочтительно, настоящий способ представляет собой способ лечения злокачественных опухолей, происходящих из эпителиальных клеток, такие злокачественные опухоли в совокупности также обозначаются как карциномы. Более конкретно, изобретение относится к лечению НМКРЛ, который включает аденокарциномы, плоскоклеточные карциномы и крупные клеточные карциномы, а также их метастазы.
Как правило, для целей настоящего изобретения термин «рак легких» относится к злокачественным заболеваниям или состояниям и может использоваться взаимозаменяемо с некоторыми другими терминами, такими как «(злокачественная) опухоль легкого» и «(злокачественное) неопластическое заболевание». Как правило, для целей настоящего изобретения термин «рак легких» не охватывает доброкачественное новообразование и/или доброкачественные опухоли. Однако настоящее изобретение в самом широком смысле может также охватывать лечение индивидуумов, страдающих такими доброкачественными состояниями, с целью предотвращения прогрессирования или развития злокачественных новообразований (рака легких). Стадия рака легких может быть определена с использованием классификации Американского объединенного комитета по злокачественной опухоли TNM (Tumor, Node, Metastases – опухоль, узел, метастазы) следующим образом:
Стадия I: опухоль обнаружена только в одном легком и не распространилась ни на один лимфоузел;
Стадия II: опухоль распространилась на лимфоузлы, которые содержатся в окружении легкого;
Стадия IIIa: опухоль распространилась на лимфоузлы за пределами легкого, в области трахеи, включая грудную стенку и диафрагму на той же стороне, с которой началась злокачественная опухоль;
Стадия IIIb: опухоль распространилась на лимфоузлы на противоположном легком или в шее;
Стадия IV: опухоль распространилась на другие части легких или отдаленно на всем протяжении тела.
Вышеупомянутая система определения стадии TNM не часто используется для пациентов с МКРЛ, потому что в большинстве случаев подозревают или определяют метастатическое заболевание во время диагноза. Выживание у этих пациентов, как правило, не зависит от незначительных различий в степени поражения опухолью. Вместо этого большинство экспертов используют простую двухстадийную систему, созданную Исследовательской группой по раку легких при совете ветеранов. Эта система определяет МКРЛ как «ограниченную» или «распространенную» стадию. Настоящий способ обеспечивает значимую частоту ответов на всех стадиях, и, таким образом, в широком смысле он касается лечения рака легких на любой из вышеуказанных стадий.
Способ по изобретению может представлять собой первичное лечение или применяться в качестве дополнительной терапии до, во время или после лечения пациентов с использованием любого другого способа лечения, включая, например:
операцию;
лучевую терапию;
химиотерапию, например, с использованием лекарственных средств на основе платины, таких как цисплатин и карбоплатин; аналогов нуклеозидов, таких как гемцитабин, таксанов, таких как паклитаксел и доцетаксел; ингибиторов топоизомеразы I, таких как топотекан и иринотекан; ингибиторов топоизомеразы II, таких как этопозид; и антимитотических лекарственных средств, таких как винорелбин;
«нацеленную терапию», например, с применением ингибиторов EGFR, таких как гефитиниб; ингибиторов тирозинкиназы, таких как эрлотиниб; ингибиторов VEGF-A, таких как бевацизумаб; ингибиторов циклооксигеназы-2; ингибиторов фосфодиэстеразы циклического гуанозинмонофосфата, таких как эксисулинд; ингибиторов протеасомы; агонистов RXR, таких как бексаротин; и ингибиторов EGFR, таких как цетуксимаб;
иммуномодулирующую терапию, например, как описано ниже; и
их сочетания.
Способ по изобретению предпочтительно комбинируют с иммуномодулирующей терапией. Особенно предпочтительно, что когда способ включает в себя введение источника иммуногенного полипептида, способного вызывать клеточный или гуморальный иммунный ответ против hZP, такой способ комбинируют с иммуномодулирующей терапией. Иммуномодулирующую терапию, которую можно комбинировать со способом по изобретению, можно выбирать из одной или нескольких из следующих:
применение ингибитора контрольной точки, такого как, например, антитело против PD1, PDL1, CTLA4, TIM-3 и/или LAG-3;
применение направленной доставки антител к выбранным членам семейства рецепторов TNF, таких как, например, антитело против CD40, 4-1 BB, CD137, OX-40/CD134 и/или CD27;
применение иммуносупрессивного цитокина, такого как, например, IL-10, TGF-β и/или IL-6;
применение цитокина γC, такого как, например, IL-7, IL-15, и IL-21 и/или IL-2, так как эти цитокины обладают способностью наращивать коммитированные Т-клетки;
применение агониста TLR, такого как, например, описанный Kaczanowska et al. (J Leukoc Biol. 2013 Jun; 93 (6): 847–863) и/или лиганда TLR, как описано в настоящем документе ниже;
применение агониста инвариантных Т-клеток-естественных киллеров (iNKT), такого как, например, синтетический агонист iNKT, описанный Cerundolo et al. (Curr Opin Immunol. 2010; 22 (3): 417-24).
Изобретение относится к способам, которые подходящим образом используют для лечения первичного рака легких, рецидивирующего рака легких и его метастазов, что считают в настоящем документе «терапевтическим лечением» или «радикальным лечением», а также для предотвращения метастазов и/или рецидивов рака легких необязательно после или в сочетании с другими способами лечения, такими как описанные ранее, что считают в настоящем документе «профилактическим лечением».
Для способов по изобретению индивидуумом, подлежащим лечению, является человек, и он может быть мужчиной или женщиной. Когда индивидуумом, подлежащим лечению, является (пременопаузальная) женщина, клиническую эффективность способа можно контролировать путем мониторинга аутоиммунитета против ткани яичника. Например, уровни гормонов в крови можно затем использовать в качестве биомаркера для клинической эффективности и замены клинической конечной точки (PFS, OS) для сокращения сроков вывода данных.
Настоящее изобретение основано на обнаружении экспрессии белков ZP злокачественной клеткой, что представляет различные варианты для иммунотерапевтических стратегий. Однако, поскольку разные опухоли могут иметь разные или измененные паттерны экспрессии гена, как будет понятно специалисту, могут также возникать определенные виды рака легких, не экспрессирующие ZP белки в какой-либо значительной степени. Таким образом, как правило, изобретение относится к лечению рака легких или его метастазов, экспрессирующих белки ZP, предпочтительно ZP3.
Наименование гликопротеиновых компонентов ZP было довольно противоречивым на протяжении многих лет и использовало несколько критериев, в том числе очевидную молекулярную массу, длину последовательности белка и сравнение идентичности, что привело к запутанной номенклатуре. Harris et al. [(1994) DNA seq. 96: 829-834] предложили единую систему номенклатуры, в которой гены ZP гены были названы в порядке длины их последовательности кодированного белка от самой длинной до самой короткой. Поскольку по этим критериям мышиные гены ZP расположились в порядке ZP2, ZP1, а затем ZP3, была введена новая система, где ZP2 стал ZPA, ZP1 стал ZPB, а ZP3 стал ZPC. Совсем недавно Hughes et al [(1999) BBA-Gene Structure and Expression 1447: 303-306], среди прочего, сообщили, что истинный человеческий ортолог известного гена мыши ZP1 не является ZPB, но существует отдельный ген человека ZP1. В настоящее время общепринято, что существует четыре различных (человеческих) семейства гликопротеинов ZP - ZP1, ZP2, ZP3 и ZPB [ср. Lefievre et al (2004) Hum. Reprod. 19:1580-1586]. Гликопротеин ZPB согласно этой номенклатуре теперь также обозначается как ZP4. Эту номенклатуру используют, например, в базах данных Uniprot/SWISSprot, ensEMBL, BLAST (NCBI), SOURCE, SMART, STRING, PSORT2, CDART, UniGene и SOSUI, все они задействованы в Bioinformatic Harvester (http://harvester.embl.de).
В соответствии с этим, термины ZP1, ZP2, ZP3 и ZP4 используют в настоящем документедля обозначения четырех семейств гликопротеинов ZP, где ZP2, ZP3 и ZP4 соответствуют ZPA, ZPC и ZPB, соответственно, согласно номенклатуре, предложенной Harris et al. Более конкретно, термины hZP1, hZP2, hZP3 и hZP4 применяют в настоящем документе по отношению к белкам, имеющим полипептидные остовы, перечисленные в SEQ ID NO. 1, SEQ ID NO. 2, SEQ ID NO. 3 и SEQ ID NO. 4, соответственно, и их аллельные варианты.
В соответствии с изобретением, белок ZP, как правило, представляет собой белок ZP3.
Аллельные варианты ZP, которые могут встречаться в природе, также охватываются соответствующими терминами ZP и hZP. Аллельные варианты включают в себя, в частности, варианты, являющиеся результатом однонуклеотидных полиморфизмов (SNP). SNP могут попадать в кодирующие последовательности генов, некодирующие области генов или в межгенные области. SNP в пределах кодирующей последовательности не обязательно изменят аминокислотную последовательность белка, который производится. SNP, в котором обе формы приводят к одной и той же полипептидной последовательности, называется синонимическим (иногда называется молчащей мутацией) - если создается другая полипептидная последовательность, они не синонимичны. Чтобы вариант считался SNP, он должен встречаться, по меньшей мере, у 1% в популяции. В контексте настоящего изобретения «аллельные варианты» также могут включать полипептидную последовательность вариантов, возникающую в результате (несинонимичных) мутаций, т.е. варианты полипептида, возникающие в результате точечных мутаций, вставок, делции и т. д., встречающихся менее чем у 1% в популяции.
Таким образом, в соответствии с настоящим изобретением термины hZP1, hZP2, hZP3 и hZP4 включают в себя белки ZP, которые отличаются от SEQ ID NO. 1, SEQ ID NO. 2, SEQ ID NO. 3 и SEQ ID NO. 4, соответственно, по минорным модификациям последовательности. Такие модификации в неограничивающих примеров включают в себя изменения в одной или нескольких аминокислотах, включая делеции (например, усеченную версию пептида), вставки и/или замены.
Под «аллельным вариантом» в настоящем документе понимают вариант, который имеет, по меньшей мере, 90%, предпочтительно, по меньшей мере, 95%, более предпочтительно, по меньшей мере, 97%, еще более предпочтительно, по меньшей мере, 98%, еще более предпочтительно, по меньшей мере, 99%, еще более предпочтительно, по меньшей мере, 99,5% и наиболее предпочтительно, по меньшей мере, 99,9% идентичности аминокислотных последовательностей с любой из SEQ ID NO. 1, SEQ ID NO. 2, SEQ ID NO. 3 и SEQ ID NO. 4.
Каждый из белков ZP содержит сигнальный пептид для направления белка по секреторному пути, зональный домен и трансмембранный домен рядом с каррбоксильным концом, за которым следует короткий цитоплазматический хвост. В вариантах осуществления изобретения, которые включают введение антитела или его фрагмента, который специфически связывается с hZP, антитела или его фрагменты направлены против внеклеточного домена ZP. В вариантах осуществления изобретения, которые касаются активной иммунизации, в качестве источника иммуногенного полипептида, способного вызывать клеточный иммунный ответ против hZP, в принципе можно использовать любую часть hZP, которая содержит T-клеточный эпитоп, рестрицированный по MHC класса I и/или класса II.
Аминокислотная последовательность образующегося белка hZP3 содержит N-концевую последовательность сигнального пептида (положения аминокислот 1-22 в SEQ ID NO: 3), консервативный "внеклеточный домен ZP" (положения аминокислот 23-350 в SEQ ID NO: 3), и про-пептид (положения аминокислот 351-424 в SEQ ID NO: 3), состоящий из консенсусного фуринового участка расщепления (CFCS; положения аминокислот 351-352 в SEQ ID NO: 3), блокирующего полимеризацию внешнего гидрофобного участка (EHP) и C-концевого трансмембранного домена (положения аминокислот 353-424 в SEQ ID NO: 3). Сигнальный пептид hZP3 отщепляется во время трансляции, а расщепление по CFCS отделяет внеклеточный домен зрелого белка hZP3 (состоящий из положений 23-350 аминокислот в SEQ ID NO: 3) от EHP, что позволяет ему включаться в образующиеся филаменты ZP. Таким образом, аминокислотный остов внеклеточного домена зрелого hZP3 имеет аминокислотную последовательность, состоящую из положений аминокислот 23-350 в SEQ ID NO: 3 hZP3. Этот фрагмент внеклеточного домена обозначен в настоящем документе как hZP3 (23-350) (SEQ ID NO: 5).
В предпочтительном варианте осуществления настоящее изобретение относится к способу лечения рака легких и его метастазов у индивидуума путем активной иммунизации. Способ активной иммунизации включает введение источника иммуногенного полипептида, способного вызывать клеточный иммунный ответ против hZP3 или против клеток, экспрессирующих (любую часть) hZP3, и/или гуморальный иммунный ответ против hZP3 или hZP3 (23-350).
Источником иммуногенного полипептида может быть белковый источник, нуклеиновая кислота или их сочетание. Белковый источник может, например, представлять собой композицию, включающую один или несколько пептидов, полипептидов или белков, которые действуют в качестве иммуногена. Альтернативно источником иммуногенных полипептидов могут быть молекула нуклеиновых кислот, кодирующая одну или несколько иммуногенных пептидов, полипептидов или белков, которая при введении индивидууму, которого лечат, экспрессирует иммуногенные пептиды, полипептиды или белки. Молекула нуклеиновой кислоты может представлять собой ДНК, кДНК, РНК, мРНК, их вариант, их фрагмент или их сочетание, как, например, описано в WO2014/165291 и WO2013/087083. Источником иммуногенного полипептида дополнительно может быть клетка, предпочтительно живая клетка, которая экспрессирует иммуногенный полипептид или презентирует эпитоп иммуногенного полипептида. Предпочтительно, клетка является микробной, более предпочтительно, бактериальной, такой как, например, живая аттенуированная Listeria monocytogenes, как, например, описанная в WO 2015/164121. Альтернативно, клеточный источник иммуногенного полипептида по изобретению представляет собой аутологичную или аллогенную дендритную клетку, которая презентирует, по меньшей мере, один эпитоп иммуногенного полипептида в HLA-молекуле на своей поверхности.
В предпочтительном варианте осуществления иммуногенный полипептид содержит непрерывную аминокислотную последовательность, выбранную из аминокислотной последовательности белка ZP, где непрерывная аминокислотная последовательность предпочтительно содержит, по меньшей мере, один из Т-клеточных эпитопов, рестрицированный по MHC класса I и класса II. Более предпочтительно, иммуногенный полипептид содержит непрерывную аминокислотную последовательность, выбранную из аминокислотной последовательности белка hZP3 (т.е. SEQ ID NO: 3), где непрерывная аминокислотная последовательность предпочтительно содержит, по меньшей мере, один из Т-клеточных эпитопов, рестрицированный по MHC класса I и класса II, например, выбранный из таблиц 2, 3 и 5 соответственно. Более предпочтительно, непрерывная аминокислотная последовательность содержит, по меньшей мере, один из Т-клеточных эпитопов, рестрицированных по МНС класса I и МНС класса II, с низким процентильным рангом (см. Moutaftsi et al., Nat Biotechnol. 2006 Jul; 24(7): 817-9; и Kotturi et al., J Virol. 2007 May; 81 (10): 4928-40). Т-клеточный эпитоп, рестрицированный по МНС класса I, с низким процентильным рангом представляет собой эпитоп с процентильным рангом, который не превышает 1,00, 0,80, 0,40, 0,30, 0,20, 0,15 , 0,10 или 0,05 (см. например, таблицы 2 и 5). Т-клеточный эпитоп, рестрицированный по MHC класса II, с низким процентильным рангом представляет собой эпитоп с процентильным рангом, который не превышает 2,50, 2,40, 2,05, 2,00, 1,80, 1,60 , 1,40, 1,20, 1,10, 1,00, 0,90, 0,70, 0,60, 0,50, 0,40, 0,20. 0,15, 0,10, 0,05 или 0,02. Предпочтительно, непрерывная аминокислотная последовательность выбрана из аминокислотной последовательности из группы аминокислотных последовательностей из протеолитических фрагментов hZP3, состоящих из последовательности N-концевого сигнального пептида (положения 1-22 в SEQ ID NO: 3), последовательности зрелого внеклеточного домена (положения 23-350 в SEQ ID NO: 3, т.е. SEQ ID NO: 5), и последовательности пропептида (положения аминокислот 351-424 в SEQ ID NO: 3). Примеры таких непрерывных аминокислотных последовательностей показаны на фигуре 4, а комбинации таких непрерывных аминокислотных последовательностей показаны в таблице 4.
Непрерывная аминокислотная последовательность из белка hZP (3), содержащаяся в иммуногенном полипептиде, содержит иммунологически активный (последовательность) фрагмент белка hZP (3). Термин «его иммунологически активные фрагменты», как правило, в данной области понимают как относящийся к фрагменту антигена белка hZP (3), содержащему по меньшей мере эпитоп, что означает, что иммуногенный полипептид, по меньшей мере, включает 4, 5, 6, 7. или 8 непрерывных аминокислот из последовательности антигена белка hZP (3). По настоящему изобретению фрагмент содержит, по меньшей мере, пептид, связывающий MHC класса I или MHC класса II, презентируемый такой молекулой MHC иммунной системе. «Иммунологически активный фрагмент» по настоящему изобретению включает в себя 7, 8, 9, 10, 11, 12, 13, 14, 15, 16 или 17 непрерывных аминокислот из последовательности антигена белка ZP или его гомолога или аналога. В то время как большинство MHC-связывающих пептидов имеют длину 9 аминокислот, более длинные пептиды могут быть приспособлены за счет выпуклости их центральной части (Guo et al., 1992, Nature; 360 (6402): 364–366; Speir et al. ., 2001, Immunity; 14 (1): 81–92), что приводит к связыванию пептидов длиной от 8 до 15 аминокислот (Schumacher et al., 1991, Nature; 350 (6320): 703–706). Примеры пептидов в последовательности hZP3, связывающих МНС класса I, приведены в таблицах 2 и 5. Пептиды, связывающиеся с белками класса II, не ограничены по размеру (Nelson et al., 1999, Rev Immunogenet; 1 (1): 47–59; Yassai et al., 2002, J Immunol; 168 (3): 1281–1285) и могут варьироваться от 11 до 30 аминокислот в длину (Rammensee 1995, Immunogenetics; 41 (4): 178–228), возможны даже целые белки. Однако мотив связывания имеет длину около 9 аминокислот. MHC класса II может вместить гораздо более длинные пептиды, чем MHC класса I, потому что концы связующей канавки MHC II открыты, таким образом, эпитоп (связывающийся в канавке) может быть окружен дополнительными участками аминокислот на любом конце. Примеры пептидов в последовательности hZP3, связывающих МНС класса II, приведены в таблице 3. Еще более предпочтительный фрагмент включает эпитоп как для цитотоксического T-лимфоцита (ЦТЛ), так и для хелперного T-лимфоцита (ХТЛ). Однако наиболее предпочтительным фрагментом является пептид, который требует процессирования антигенпредставляющей клеткой, т.е. фрагмент имеет длину, по меньшей мере, приблизительно 18 аминокислот, которые необязательно являются непрерывной последовательностью из антигена белка hZP (3).
Длина непрерывной аминокислотной последовательности из белка hZP (3), входящей в состав иммуногенного полипептида, или длина самого иммуногенного полипептида, таким образом, предпочтительно составляет, по меньшей мере, 18, 19, 20, 21, 22, 25, 27, 30, 33 или 35 аминокислот и предпочтительно не более, чем 100, 80, 60, 50, 45, 40, 35, 33 или 30 аминокислот. Предпочтительно длина непрерывной аминокислотной последовательности из белка hZP (3), входящей в состав иммуногенного полипептида, или длина самого иммуногенного полипептида, составляет 19-50 или 19-45, более предпочтительно 25-40 аминокислот, даже более предпочтительно 25-35 и наиболее предпочтительно 25-30 аминокислот. С точки зрения технологичности оптимальным является иммуногенный полипептид с длиной около 25 аминокислот, при этом он достаточно длинный, чтобы содержать несколько эпитопов и обеспечивать презентирование с помощью антигенпредставляющих клеток. Подходящие примеры таких иммуногенных полипептидов, включающих непрерывную аминокислотную последовательность из белка hZP3, каждый из которых содержит один или несколько пептидов, связывающих МНС класса I и/или МНС класса II, представлены на фигуре 4.
Термины «их гомологи», как применяют в настоящем документе, относятся к полипептидам, которые отличаются от природного полипептида незначительными модификациями, но которые поддерживают структуру основного полипептида и боковой цепи природной формы. Такие изменения в качестве неограничивающих примеров включают изменения в одной или нескольких боковых цепях аминокислот; изменения в одной или нескольких аминокислотах, в том числе делеции (например, усеченная версия пептида), вставки и/или замены; изменения в стереохимии одного или нескольких атомов; и/или незначительные дериватизации, включая в качестве неограничивающих примеров метилирование, гликозилирование, фосфорилирование, ацетилирование, миристоилирование, пренилирование, пальмитацию, амидирование и/или добавление гликозилфосфатидилинозитола. Как применяют в настоящем документе, гомолог или аналог имеет либо усиленную, либо по существу аналогичную функциональность по сравнению с природным полипептидом. Как правило, при оптимальном выравнивании, таком как при помощи программ GAP или BESTFIT с использованием параметров по умолчанию, природный полипептид и его гомолог имеют, по меньшей мере, определенный процент идентичности последовательностей. GAP использует алгоритм глобального выравнивания Needleman и Wunsch для выравнивания двух последовательностей по всей их длине, максимизируя количество совпадений и минимизируя количество пропусков. В основном, параметры GAP по умолчанию применяются с штрафом за создание пропуска= 8 и штрафом за расширение пропуска = 2. Для белковоценочной матрицей по умолчанию является Blosum62 (Henikoff & Henikoff, 1992, PNAS 89, 915-919). Выравнивание последовательностей и баллы для процента идентичности последовательностей можно определить с помощью компьютерных программ, таких как пакет GCG Wisconsin, версия 10.3, доступный от Accelrys Inc., 9685 Scranton Road, San Diego, CA 92121-3752, USA. Альтернативно процент сходства или идентичности можно определить путем поиска по базам данных, таким как FASTA, BLAST и т. д.
Под гомологом в настоящем документе понимают иммуногенный полипептид, имеющий, по меньшей мере,, 70%, предпочтительно, по меньшей мере, 80%, более предпочтительно, по меньшей мере, 90%, еще более предпочтительно, по меньшей мере, 95%, еще более предпочтительно, по меньшей мере, 98% и наиболее предпочтительно, по меньшей мере, 99% идентичности аминокислотных последовательностей с природными полипептидами ZP, упомянутыми выше, и все еще способный вызывать, по меньшей мере, получаемый при этом иммунный ответ. Гомолог или аналог в настоящем документе могут содержать замены, вставки, делеции, дополнительные N- или C-концевые аминокислоты и/или дополнительные химические группы, такие как углеводы, для повышения стабильности, растворимости и иммуногенности.
Изобретение явным образом включает применение иммуногенного полипептида, содержащего белки, которые являются гомологом белка дикого типа hZP (3), но отличаются от него в результате опухолеспецифических мутаций (которые могут быть специфичными для пациента или общими), которые приводят к измененным аминокислотным последовательностям, т.е. так называемым неоантигенам.
В соответствии с настоящим изобретением, иммуногенный полипептид, который вводят человеку в соответствии с настоящим способом, может представлять собой белок или гликопротеин или содержать белок или гликопротеин, быть продуктом расщепления белка или гликопротеина и/или их фрагментами, которые могут находиться в очищенной форме или могут содержаться внутри неочищенной композиции, предпочтительно биологического происхождения, такой как лизаты, соникаты или фиксаты прокариотических или эукариотических клеточных линий. Более того, иммуногенный полипептид представляет собой или содержит химически синтезированные (поли) пептиды или (поли) пептиды, которые были получены ферментативно in vitro, которые могут быть в очищенной форме или могут содержаться в неочищенной композиции.
Как применяют в настоящем документе термин «эпитоп» относится к части антигена, как правило, определяемой коротким пептидом, который способен вызывать клеточный или гуморальный иммунный ответ при презентировании в физиологически соответствующем контексте in vivo. «T-клеточный эпитоп» относится к короткому пептиду или его части, который связывается с молекулами MHC и распознается определенными T-клетками при презентировании в определенном фрагменте MHC. Т-клеточный эпитоп способен индуцировать клеточный опосредованный иммунный ответ путем прямого или непрямого презентирования в гетеродимерной мембране молекул МНС. Предпочтительно, в иммуногенном полипептиде, по меньшей мере, один эпитоп, рестрицированный по МНС класса II, и эпитоп, рестриктицированный по MHC класса I, присутствуют в пределах непрерывной последовательности из аминокислотной последовательности белка hZP (3), в силу чего предпочтительно, что эпитопы, рестрицированные по МНС класса II, и, по меньшей мере, один эпитоп, рестрицированный по MHC класса I, выбраны из таблиц 3, 2 и 5, соответственно. С целью ясности, пептид по изобретению предпочтительно включает, по меньшей мере, один эпитоп, презентируемый МНС класса I, а также, по меньшей мере, один эпитоп, презентируемый МНС класса II. Каждый из этих эпитопов может презентироваться и будет связываться с соответствующими специфическими молекулами MHC, присутствующими на клетках, после того, как был процессирован, как описано в настоящем документе. Таким образом, каждый рестрицированный по МНС эпитоп можно также назвать МНС-связывающим и/или МНС-презентирующимся эпитопом. Предпочтительно, специфический протеасомный участок расщепления, создающий C-конец такого эпитопа, присутствует точно после аминокислотной последовательности эпитопа для того, чтобы быть освобожденным от иммуногенного полипептида и представленным на молекуле MHC класса I. Требования к длине намного менее строгие для эпитопов МНС класса II, поэтому потребность в точной ферментативной генерации пептида, связывающего класс II, менее безоговорочна. В кратком изложении, MHC молекулы преимущественно связывают определенные аминокислотные остатки, известные как «якорные» остатки (K. Falk et al., Nature 351: 290-96 (1991)). Эта характеристика позволяет распознавать мотивы связывания МНС класса I и II в любой известной последовательности пептида (см. например, таблицы 2, 5 и 3).
В настоящем контексте термин «эпитоп, рестрицированный по MHC» является синонимом T-клеточного эпитопа. Как применяют в настоящем документе, термин «эпитоп, рестрицированный по МНС класса I» относится к пептиду, распознаваемому цитотоксическими Т-лимфоцитами (также называемыми CD8+-клетками, TCD8 или ЦТЛ), в ассоциации с МНС класса I. Как применяют в данном документе, термин «эпитоп, рестрицированный по МНС класса II», относится к пептиду, распознаваемому хелперными Т-клетками (также называемыми CD4+-клетки, TCD4 или ХТЛ) в ассоциации с МНС класса II. «B-клеточный эпитоп» представляет собой часть антигена, которая способна связываться с антигенсвязывающим участком иммуноглобулина и, таким образом, способна стимулировать гуморальный ответ без презентации при помощи молекулы МНС. Как объяснено в настоящем документе ранее, полипептид, подходящий для настоящего изобретения, или нуклеиновая кислота, кодирующая указанный полипептид, включает в себя, по меньшей мере, один Т-клеточный эпитоп. Однако применение полипептидов, которые также содержат В-клеточный эпитоп, не исключено из настоящего изобретения. Такие иммуногенные полипептиды также могут включать множественные Т-клеточные и, необязательно, B-клеточный эпитоп. Когда в пептиде присутствует несколько эпитопов, они могут быть ориентированы тандемно или во вложенной или перекрывающейся конфигурации, где, по меньшей мере, один аминокислотный остаток может быть использован двумя или более эпитопами.
Иммуногенный полипептид по изобретению предпочтительно включает один или несколько эпитопов, рестрицированных по МНС класса I. Как, в основном, известно специалисту, антиген, содержащий один эпитоп, рестрицированный по MHC, будет полезен только для лечения (небольшой) группы пациентов, которые экспрессируют аллель MHC, способный связывать этот конкретный пептид. Подсчитано, что у людей, вакцины, содержащие эпитопы ЦТЛ, рестрицированные по HLA-A1, -A2, -A3, -A24 и -B7, могут охватывать примерно 80% индивидуумов большинства этнических групп. Таким образом, если настоящий способ применяют для лечения человека, особенно предпочтительно, чтобы способ включал введение композиции, содержащей один или несколько различных полипептидов, включающих один, более предпочтительно два, наиболее предпочтительно три МНС класса I-связывающих эпитопа из нативного ZP, предпочтительно hZP3, выбранные из эпитопов, рестрицированных по HLA-A1, HLA-A2, HLA-A3, HLA-A24 и HLA-B7; или их гомологи.
Согласно другому варианту осуществления иммуногенный полипептид по изобретению предпочтительно включает один или несколько эпитопов, рестрицированных по МНС класса II. Наиболее часто встречающиеся продукты аллелей MHC класса II у людей включают HLA-DR1, -DR3, -DR4 и -DR7. Таким образом, предпочтительно, чтобы способ включал введение композиции, содержащей один или несколько различных полипептидов, включающих один, более предпочтительно два, наиболее предпочтительно три МНС класса II-связывающих эпитопа из нативного ZP, предпочтительно hZP3, выбранные из эпитопов, рестрицированных по HLA-DR1, HLA-DR3, HLA-DR4 и HLA-DR7; или их гомологи.
В другом варианте осуществления способ по изобретению включает введение композиции, содержащей один или несколько полипептидов, включающих один или несколько эпитопов, рестрицированных по МНС класса I, и один или несколько эпитопов, рестрицированных по МНС класса II, как описано выше и/или в таблицах выше 2, 5 и 3 соответственно; или их гомологи. Даже более предпочтительно, указанная композиция содержит эффективное количество одного или нескольких различных полипептидов, которые вместе включают, по существу, все эпитопы, связывающие MHC класса I и II, содержащиеся в одном из нативных гликопротеинов ZP, предпочтительно hZP3; или гомологи указаного одного или нескольких полипептидов.
В предпочтительном варианте осуществления настоящий способ включает введение композиции, содержащей один или несколько иммуногенных пептидов. Предпочтительно, указанные один или несколько различных полипептидов вместе составляют, по меньшей мере, 50%, более предпочтительно, по меньшей мере, 70%, еще более предпочтительно, по меньшей мере, 80%, более предпочтительно, по меньшей мере, 90% и наиболее предпочтительно, по меньшей мере, 95% из эпитопов, рестрицированных по MHC класса I и MHC класса II, содержащихся в нативном ZP, предпочтительно hZP3 или hZP3 (23-350) или гомологи указанных одного или нескольких полипептидов.
В предпочтительном варианте осуществления настоящий способ включает введение композиции, содержащей один или несколько иммуногенных пептидов, выбранных из пептидов, представленных на фигуре 4, например, иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности из одного или нескольких из SEQ ID NO: 66-75. Таким образом, композиция может содержать один из иммуногенных пептидов, содержащий или состоящий из аминокислотной последовательности, выбранной из SEQ ID NO: 66-75. Предпочтительно, однако, композиция содержит комбинацию, по меньшей мере, двух иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO. 66-75. В частности, композиция содержит комбинацию иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из комбинаций последовательностей, перечисленных в таблице 4, или комбинацию всех 10 из SEQ ID NO. 66-75.
В дополнительном предпочтительном варианте осуществления настоящий способ включает введение композиции, содержащей один или несколько иммуногенных пептидов, содержащих или состоящих из эпитопа, выбранного из эпитопов, представленных в таблице 5, т.е. SEQ ID NO. 76-85. Композиция, таким образом, может включать один из иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO. 76-85. Предпочтительно, однако, композиция содержит комбинацию, по меньшей мере, двух иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO. 76-85. В частности, композиция содержит комбинацию иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из комбинаций последовательностей, перечисленных в таблице 6, или комбинацию всех 10 из SEQ ID NO. 76-85. Особенно предпочтительная композиция содержит один или несколько или все из иммуногенных пептидов, содержащих или состоящих из аминокислотных последовательностей из SEQ ID NO. 79, 80, 81, 83 и 84.
В предпочтительном варианте осуществления настоящий способ включает введение источника иммуногенного полипептида, при этом полипептид содержит, по меньшей мере, 50%, более предпочтительно, по меньшей мере, 70%, еще более предпочтительно, по меньшей мере, 80%, еще более предпочтительно, по меньшей мере, 90% и наиболее предпочтительно, по меньшей мере, 95% полного аминокислотного остова нативного гликопротеина ZP, предпочтительно hZP3 или hZP3(23-350); или гомолог указанного полипептида.
В особенно предпочтительном варианте осуществления настоящий способ включает введение композиции, содержащей источник иммуногенного полипептида, при этом полипептид содержит 90, 95, 97, 98, 99 или 100% полного аминокислотного остова внеклеточного домена нативного ZP, а именно hZP3 или hZP3 (23-350) или гомолог указанного полипептида. Такие иммуногенные полипептиды, как определено в настоящем документе ранее, могут быть гликозилированы. Не желая быть связанными теорией, мы выдвигаем гипотезу, что за счет гликозилирования этих полипептидов их иммуногенность, по меньшей мере, в той мере, в какой они вызывают гуморальный ответ (В-клеточный ответ), увеличивается. Таким образом, иммуногенный полипептид, как определено в настоящем документе ранее, является гликозилированным, с содержанием углеводов в пределах 10-80, 15-70 или 20-60% масс., на основании общей массы гликопротеина или гликозилированного полипептида. Предпочтительно, указанный гликозилированный иммуногенный полипептид включает в себя паттерн гликозилирования, сходный с таковым соответствующего природного участка ZP гликопротеина человека.
В другом особенно предпочтительном варианте осуществления, способ включает введение источника иммуногенного полипептида, который представляет собой композицию, содержащую эффективное количество множества различных перекрывающихся полипептидных фрагментов нативного гликопротеина ZP, hZP3 или hZP3 (23-350), при этом длина различных перекрывающихся полипептидных фрагментов составляет от 18 до 60 аминокислот, и вместе они включают, по меньшей мере, 50%, более предпочтительно, по меньшей мере, 70%, еще более предпочтительно, по меньшей мере, 80%, еще более предпочтительно, по меньшей мере, 90%, и наиболее предпочтительно, по меньшей мере, 95% от полного аминокислотного остова указанного нативного ZP или внеклеточного домена указанного нативного ZP, hZP3 или hZP3 (23-350) или гомологов указанных полипептидов. Более предпочтительно, различные перекрывающиеся полипептидные фрагменты имеют длину 25-40 аминокислот, даже более предпочтительно 25-35 и наиболее предпочтительно 25-30 аминокислот. Как правило, аминокислотная последовательность перекрывается между различными последовательными полипептидными фрагментами размером 18-60 аминокислот, по меньшей мере, по семи аминокислотам, предпочтительно, по меньшей мере, по восьми аминокислотам, более предпочтительно, по меньшей мере, по девяти аминокислотам и наиболее предпочтительно, по меньшей мере, по десяти аминокислотам.
Описаны мотивы связывания МНС для наиболее распространенных аллелей МНС класса I и II. Эти мотивы детализируют аминокислотные остатки, которые служат якорями связывания MHC для конкретных аллелей MHC класса I и II. Для предсказания и количественной оценки аффинности связывания пептид/MHC используют сложные компьютерные алгоритмы, которые учитывают якоря связывания MHC, а также аминокислотную последовательность пептида. Таким образом, из входных данных известной аминокислотной последовательности белков блестящей оболочки эти алгоритмы перечисляют все потенциальные Т-клеточные эпитопы, каждый со своим соответствующим прогнозирующим показателем связывания (см. например, таблицы 2, 5 и 3). Общеизвестные инструменты биоинформатики для этих целей включают, например, HLA_BIND (Parker et al., 1994, J. Immunol. 152: 163), SYFPEITHI (Rammensee et al., 1995, Immunogenetics 41, 178-228; Rammensee et al., Landes Bioscience 1997, международный дистрибьютор: Springer Verlag GmbH&Co. KG, Гейдельберг, Германия; http://www.syfpeithi.de/), NetMHC (Buus et al., 2003, Tissue Antigens., 62: 378-84; Nielsen et al., 2003, Белок Sci., 12: 1007-17; Nielsen et al., 2004, Bioinformatics, 20 (9): 1388-97; http://www.cbs.dtu.dk/services/NetMHC/), TEPITOPE 2000 (Sturniolo et al., 1999, Nature Biotechnology 17, 555-562; http://www.vaccinome.com/pages/597444/), и (постоянно обновляемые) ресурсы анализа IEDB - инструменты прогнозирования Т-клеточных эпитопов: http://tools.iedb.org/main/tcell/.
Альтернативно, специалисты в данной области смогут определить ХТЛ- и ЦТЛ-связывающие эпитопы экспериментально, используя стандартные эксперименты (Current Protocols in Immunology, Wiley Interscience 2004). В предпочтительном варианте осуществления способ включает введение композиции, содержащей эффективное количество множества полипептидных фрагментов длиной от 18 до 100 аминокислот из нативного гликопротеина ZP, hZP3 или hZP3 (23-350), где каждый полипептидный фрагмент содержит один или несколько из указанных предсказанных потенциальных эпитопов, рестрицированных по MHC класса I или MHC класса II. Предпочтительно, аминокислотная последовательность указанных предсказанных потенциальных эпитопов, рестрицированных по MHC класса I или MHC класса II, в различных полипептидных фрагментах не перекрывается. Предпочтительно, множество различных полипептидных фрагментов в совокупности составляют, по меньшей мере, 50, 70, 80, 90 или 95% потенциальных эпитопов MHC класса I или MHC класса II, прогнозируемых одним или несколькими из указанных выше инструментов биоинформатики.
В некоторых случаях было замечено, что один и тот же пептид может связываться с несколькими аллельными продуктами MHC I или II (см. например, таблицу 2, 5 и 3). В одном из вариантов осуществления применение таких «беспорядочных» MHC-связывающих пептидов в настоящем способе является особенно предпочтительным.
Настоящий способ иммунизации предпочтительно включает в себя введение источника иммуногенно активных полипептидных фрагментов, указанные полипептидные фрагменты выбраны из белковых фрагментов блестящей оболочки и/или их гомологов, как определено в настоящем документе ранее, указанные полипептидные фрагменты содержат эпитопы ЦТЛ и/или ХТЛ, рестрицированные по ряду молекул HLA, и эти фрагменты имеют длину от 18 до 45 аминокислот. Было обнаружено, что пептиды длиной от 18 до 45 аминокислот обеспечивают превосходные иммуногенные свойства, как описано в WO 02/070006. Пептиды предпочтительно могут быть химически синтезированы и необязательно могут (частично) перекрываться, и/или могут быть лигированы с другими молекулами, пептидами или белками. Также может быть выгодно добавлять к амино- или карбокси-концу пептида химические группы или дополнительные (модифицированные или D-) аминокислоты, чтобы увеличить устойчивость и/или уменьшить биоразлагаемость пептида. Для улучшения иммуногенности/иммуностимулирующих свойств можно присоединять составные группы, например, путем липидирования, удлинения и/или конъюгации (см. ниже). Например, пептид может быть удлинен путем добавления заряженных или полярных аминокислот, чтобы повысить его растворимость и/или повысить его стабильность in vivo.
Для целей иммунизации указанные выше иммуногенные полипептиды по изобретению также могут быть слиты с белками, в качестве неограничивающих примеров, с такими как столбнячный токсин/анатоксин, дифтерийный токсин/анатоксин или другие молекулы-носители. Полипептиды по изобретению также могут быть предпочтительно слиты с белками теплового шока, такими как рекомбинантный эндогенный (мышиный) gp96 (GRP94) в качестве носителя для иммунодоминантных пептидов, как описано в (ссылки: Rapp UK и Kaufmann SH, Int Immunol. 2004 Apr; 16 (4: 597-605; Zugel U, Infect Immun. 2001; 69 (6): 4164-7) или слитые белки с Hsp70 (Triebel et al .; WO99/54464). Иммуногенные полипептиды по изобретению также могут быть конъюгированы с молекулами, обладающими адъювантной активностью, как указано ниже, в частности, с лигандами/агонистами TLR, перечисленными ниже.
Индивидуальные аминокислотные остатки настоящих иммуногенных (поли)пептидов по изобретению могут быть включены в пептид посредством пептидной связи или миметика пептидной связи. Миметик пептидной связи по изобретению включает модификации пептидного остова, хорошо известные специалистам в данной области. Такие модификации включают модификации амидного азота, α-углерода, амидного карбонила, полную замену амидной связи, удлинения, делеции или перекрестные сшивки в остове. См., в основном, Spatola, Chemistry and Biochemistry of Amino Acids, Peptides and Proteins, Vol. VII (Weinstein ed., 1983). Известно несколько модификаций пептидного остова, включая ψ [CH2S], ψ [CH2NH], ψ [CSNH2], ψ [NHCO], ψ [COCH2] и ψ [(E) или (Z) CH=CH]. Используемая выше номенклатура следует той, которая была предложена у Spatola выше. В этом контексте ψ указывает на отсутствие амидной связи. Структура, которая заменяет амидную группу, указана в скобках.
Также в полипептиды могут быть включены миметики аминокислот. «Миметик аминокислоты» представляет собой составную группу, отличную от природной аминокислоты, которая конформационно и функционально служит заменой аминокислоты в полипептиде по настоящему изобретению. Такая составная группа служит заменой аминокислотного остатка, если она не мешает способности пептида вызывать иммунный ответ против Т-клеточных эпитопов нативного белка ZP. Миметики аминокислот могут включать небелковые аминокислоты, такие как β-, γ-, δ-аминокислоты, β-, γ-, δ-иминокислоты (такие как пиперидин-4-карбоновая кислота), а также многие производные L-α-аминокислот. Ряд подходящих миметиков аминокислот известен специалистам в данной области, они включают циклогексилаланин, 3-циклогексилпропионовую кислоту, L-адамантил аланин, адамантилуксусную кислоту и т.п. Пептидные миметики, подходящие для пептидов по настоящему изобретению, обсуждаются у Morgan и Gainor, (1989) Ann. Repts. Med. Chem 24: 243-252.
Согласно предпочтительному варианту осуществления, настоящий способ включает введение композиции, содержащей один или несколько данных иммуногенных полипептидов, как определенно в настоящем документе, и, по меньшей мере, один эксципиент. Эксципиенты хорошо известны в данной области фармации и, их, например, можно найти в учебниках, таких как Remington's Pharmaceutical Sciences, Mack Publishing, 1995.
В другом предпочтительном варианте осуществления источником иммуногенного полипептида по изобретению, который можно вводить, является молекула нуклеиновой кислоты, кодирующая иммуногенный полипептид. Источник или композиция, содержащая молекулу нуклеиновой кислоты, кодирующую иммуногенный полипептид, может содержать одну или несколько различных молекул нуклеиновой кислоты, кодирующей любой из иммуногенных полипептидов, полипептидных фрагментов и/или пептидов, как определено в настоящем документе выше. Кроме того, молекула нуклеиновой кислоты может кодировать большую часть нативного белка ZP. Молекула нуклеиновой кислоты может, например, кодировать полипептид, содержащий, по меньшей мере, 50, 70, 80, 90, 95 или 100% полного аминокислотного остова ZP, белка hZP3, более предпочтительно hZP3 (23-350) или гомолога указанного полипептида. Предпочтительно молекула нуклеиновой кислоты кодирует непрерывный участок, по меньшей мере, из 50, 70, 80, 90, 95 или 99% полного аминокислотного остова. Также возможно, что молекула нуклеиновой кислоты кодирует более одного (содержащего Т-клеточный эпитоп) иммунологически активного фрагмента непрерывных аминокислотных последовательностей из hZP (3), как определенно в настоящем документе выше, в результате чего в кодируемых аминокислотных последовательностях различные иммунологически активные фрагменты разделены спейсером или линкером в виде бус на нитке.
Молекула нуклеиновой кислоты, кодирующая иммуногенный полипептид по изобретению, может быть молекулой ДНК, предпочтительно генетической конструкцией, где нуклеотидная последовательность, кодирующая иммуногенный полипептид (кДНК), функционально связана с соответствующей регуляторной последовательностью экспрессии, обеспечивающей функциональную экспрессию иммуногенного полипептида в клетках-мишенях человеческого индивидуума, например, в том числе, по меньшей мере, с сильным (например, вирусным) промотором. Генетические конструкции для применения в качестве ДНК-вакцин, в том числе, например, плазмиды или вирусные векторы, среди прочего, описаны в WO2014/165291, WO2016/123285 и WO2017/136758. Альтернативно, молекула нуклеиновой кислоты, кодирующая иммуногенный полипептид по изобретению, может быть молекулой РНК, например, мРНК, оцРНК, дцРНК или их сочетаниями. Молекула РНК может, например, быть сформулирована в частицу, содержащую молекулу. Подходящими вариантами осуществления вакцин на основе РНК являются, например, описанные в WO2013/087083.
Пути введения вакцин на основе нуклеиновой кислоты в качестве неограничивающих примеров включают внутримышечный, интраназальный, интраперитонеальный, интрадермальный, подкожный, внутривенный, внутриартериальный, внутриглазной и пероральный, а также местный, трансдермальный, путем ингаляции или суппозитория или в слизистую ткань, таким как лаваж в вагинальную, ректальную, уретральную, буккальную и сублингвальную ткань. Предпочтительные пути введения включают внутримышечную, интраперитонеальную, интрадермальную и подкожную инъекцию. Генетические конструкции могут быть введены способами, включая в качестве неограничивающих примеров, способы и устройства для электропорации, обычные шприцы, безыгольные инъекционные устройства или «бомбардировку микрочастицами из ионных пушек".
Источником иммуногенного полипептида по изобретению может дополнительно быть живая клетка, которая экспрессирует и/или презентирует иммуногенный полипептид. Клетка может представлять собой аутологичную или аллогенную иммунную клетку, например, дендритную клетку, полученную от индивидуума, которого преполагается лечить, или клетка может представлять собой микробную клетку, более предпочтительно бактериальную, например, живую аттенуированную Listeria monocytogenes. Экспрессирующийся иммуногенный полипептид предпочтительно представляет собой иммуногенный полипептид, определенный в документе выше. Иммуногенный полипептид можно экспрессировать в виде части слитого белка, где предпочтительно иммуногенный полипептид слит с белком, который является эндогенным для организма, например, N-концевым фрагментом L. monocytogenes LLO или белок ActA. Подходящие варианты осуществления вакцина на основе листерии, например, описаны в WO 2015/164121. Бактерию, экспрессирующую иммуногенный полипептид по изобретению, можно вводить перорально или парентерально, предпочтительно внутривенно.
В другом варианте осуществления изобретения, источником иммуногенного полипептида по изобретению является аутологичная или аллогенная дендритная клетка (ДК), которая презентирует, по меньшей мере, один рестрицированный по МНС эпитоп иммуногенного полипептида в HLA молекуле на своей поверхности. Такие дендритные клетки, например, можно получать ex vivo путем контакта и/или загрузки ДК из крови пациента, например ДК, выделенных из мононуклеарных клеток у пациента/индивидуума, с композицией, содержащей иммуногенные полипептиды по изобретению. Иммуногенный полипептид, контактирующий с мононуклеарными клетками или ДК, рпедставляет собой иммуногенный полипептид, как определено в настоящем документе выше. Можно использовать фармацевтический препарат для облегчения сбора ДК, такой как ProgenipoietinTM (Monsanto, St. Louis, Mo.) или GM-CSF/IL-4. После примирования ДК с пептидами и промывкой для удаления несвязанных пептидов ДК повторно вливают пациенту. В этом варианте осуществления предлагается композиция, содержащая пептид-примированные ДК, которые презентируют сенсибилизирующие пептидные эпитопы в HLA-молекулах на своей поверхности. Альтернативно, вместо использования аутологичных клеток, полученных от индивидуума, можно использовать аллогенные ДК, которые получены из человеческой линии предшественников дендритных клеток и предназначены для доставки антигенов, ассоциированных с опухолью, такие как, например, описанные в WO2009/019320, WO2014/090795 и WO2014/006058. Способы индуцирования иммунного ответа с использованием пептид-примированных ex vivo ДК хорошо известны специалисту.
Настоящий способ иммунизации может дополнительно включать в себя введение, предпочтительно совместное введение, по меньшей мере, одного адъюванта. Адъюванты могут включать любой адъювант, известный в данной области вакцинации, и их можно выбирать с использованием учебников, таких как Current Protocols in Immunology, Wiley Interscience, 2004.
В настоящем документе подразумевается, что адъюванты включают любое вещество или соединение, которое при использовании в комбинации с антигеном для иммунизации человека или животного стимулирует иммунную систему, тем самым провоцируя, усиливая или облегчая иммунный ответ на антиген, а не генерируя специфический иммунный ответ на сам адъювант. Предпочтительные адъюванты усиливают иммунный ответ против данного антигена, по меньшей мере, в 1,5, 2, 2,5, 5, 10 или 20 раз по сравнению с иммунным ответом, генерируемым против антигена в тех же условиях, но в отсутствие адъюванта. Тесты для определения статистического среднего усиления иммунного ответа против данного антигена, продуцируемого адъювантом, в группе животных или людей по сравнению с контрольной группой доступны в данной области. Адъювант предпочтительно способен усиливать иммунный ответ, по меньшей мере, против двух разных антигенов. Адъювант по изобретению, как правило, будет являться чужеродным для человека соединением, исключая, тем самым, иммуностимулирующие соединения, которые являются эндогенными для людей, такие как, например, интерлейкины, интерфероны и другие гормоны.
Ряд адъювантов хорошо известен специалисту в данной области. Подходящие адъюванты включают, например, гранулоцитарно-макрофагальный колониестимулирующий фактор (GM-CSF), неполный адъювант Фрейнда (IFA), MontanideTM ISA-51, MontanideTM ISA 720 (адъюванты, производимые Seppic, Франция), альфа-галактозилцерамид, квасцы, фосфат алюминия, гидроксид алюминия, N-ацетил-мурамил-L-треонил-D-изоглутамин (thr-MDP), N-ацетил-нор-мурамил-L-аланил-D-изоглутамин (CGP 11637, обозначаемый как нор-MDP), N-ацетилмурамил-L-аланил-D-изоглутаминил-L-аланин-2- (1'-2'-дипальмитоил-sn-глицеро-3-гидрокси-фосфорилокси)-этиламин (CGP 19835A, обозначаемый как MTP-PE), DDA (2 диметилдиоктадециламмоний бромид), полиI:C, поли-А-поли-U, RIBI™, GERBU™, Pam3™, Карбопол™, Specol™, Titermax™, столбнячный токсин, дифтерийный токсоид, белки наружной мембраны менингококков, дифтерийный белок CRM197. Предпочтительные адъюванты содержат лиганд, который распознается толл-подобным рецептором (TLR), присутствующим на антигенпрезентирующих клетках. В данной области известны различные лиганды, распознаваемые TLR, и они включают, например, липопептиды (см., например, WO 04/110486), липополисахариды, пептидогликаны, лиоптейхоевые кислоты, липоарабиноманнаны, липопротеины (из микоплазм или спирохет), двухцепочечную РНК (поли I:C), поли ICLC (HiltonolTM, производства Oncovir, Inc., США), неметилированную ДНК, флагеллин, CpG-содержащие олигонуклеотиды, Pam3CysSK4 и имидазохинолины, а также производные этих лигандов, имеющие химические модификации. Использование поли I:C является особенно предпочтительным.
Для терапевтического применения настоящие иммуногенные полипептиды, последовательности нуклеиновой кислоты, кодирующие их, клетки, экспрессирующие или презентирующие их, или настоящие композиции, включающие эти полипептиды, последовательности нуклеиновой кислоты или клетки, вводят пациенту, страдающему от рака легких и, возможно, от метастазов, или пациенту, который получил другие способы лечения рака легких, описанные в настоящем документе ранее, в количестве, достаточном для индукции первичного аутоиммунного ответа, направленного против нативных гликопротеинов ZP и клеток тканей, экспрессирующих белки ZP. Количество, достаточное для достижения этой цели, определяется как «терапевтически» или «профилактически эффективная» доза. Такие эффективные дозы будут зависеть от ряда факторов, включая заболевание и общее состояние здоровья пациента. Таким образом, режимы дозирования могут определяться и корректироваться квалифицированным медицинским персоналом для обеспечения оптимального терапевтического или профилактического эффекта.
В настоящем способе один или несколько иммуногенных полипептидов, как правило, вводят в дозировке приблизительно 1, 2, 5, 10, 20, 50, 100, 200, 500 или 1000 мкг на иммуногенный полипептид или молекулу нуклеиновой кислоты или более, по меньшей мере, один раз. Предпочтительно введение дозы повторяют один, два, три или более раз с интервалом в 2, 3 или 4 недели.
Согласно одному предпочтительному варианту осуществления, типичные режимы дозирования включают введение дозы 1-1000 мкг на пептид на иммунизацию, более предпочтительно 10-500 мкг на пептид на иммунизацию, еще более предпочтительно 5-150 мкг на пептид на иммунизацию, по меньшей мере, один раз. Предпочтительно введение дозы повторяют один, два, три или более раз с интервалом в 2, 3 или 4 недели. Согласно предпочтительному варианту осуществления вводят 5-150 мкг на пептид на иммунизацию, и повторяют в течение 2-3 недель от одного до нескольких раз на курс лечения.
Настоящий способ предпочтительно включает введение настоящих иммуногенных полипептидов и включающих их композиций парентеральным или пероральным путем, предпочтительно парентеральным путем. Предпочтительные пути введения в качестве неограничивающих примеров включают внутриопухолевое, внутримышечное, интраназальное, интраперитонеальное, интрадермальное, подкожное, внутривенное, внутриартериальное, внутриглазное и пероральное введение, а также местное введение, трансдермальное введение, путем ингаляции или суппозитория или введение в слизистую оболочку, например, через лаваж в ректальую, уретральную, буккальную и сублингвальнаю ткань. Предпочтительные пути введения включают внутримышечную, интраперитонеальную, интрадермальную и подкожную инъекцию. В другом варианте осуществления введение производят в анатомический участок, который стекает в лимфатический коллектор. В другом варианте осуществления введение производят в несколько лимфатических коллекторов.
Особенно предпочтительным является сочетание интрадермального и подкожного введения лекарственного средства по изобретению. ДК в эпидермисе четко отличается от ДК в дерме и в подкожной клетчатке. Внутрикожная (интрадермальная) иммунизация вызовет процессинг антигена и активацию эпидермальных ДК (лангерин-положительных клеток Лангерганса), которые через свою дендритную сеть находятся в тесном контакте с кератиноцитами. Это также оптимально активирует воспалительные пути во взаимодействиях между клеткой Лангерганса и кератиноцитами с последующим перемещением загруженной антигеном и активированной клетки Лангерганса к лимфоузлам, дренирующим кожу. Подкожное введение активирует другие подгруппы ДК, которые также будут загружены антигеном и путешествуют независимо от лимфоузлов, дренирующих кожу. Вероятно, использование лекарственных средств, которые можно вводить как интрадермально, так и подкожно, может привести к синергетической стимуляции Т-клеток в этих дренирующих узлах различными подгруппами ДК.
Лекарственное средство по изобретению имеет еще одно преимущество, заключающееся в том, что при интрадермальном введении малых количеств лекарственного средства, предпочтительно пептида, как определено ранее в данном документе, иммуногенный эффект все же может быть достигнут. Количество каждого используемого пептида находится в диапазоне от 0,3 до 1000 мкг, более предпочтительно от 1 до 500 мкг, даже более предпочтительно от 5 до 150 мкг. Другой аспект изобретения относится к фармацевтическому препарату, содержащему в качестве активного ингредиента данный источник полипептида, как определено в данном документе ранее. Более конкретно фармацевтический препарат содержит в качестве активного ингредиента один или несколько указанных выше иммуногенных полипептидов, выбранных из группы белков ZP, их гомологов и фрагментов указанных белков ZP и их гомологов, или, альтернативно, генотерапевтический вектор, как определенно в настоящем документе выше.
Согласно первому варианту осуществления предлагается фармацевтический препарат, содержащий один или несколько иммуногенных полипептидов по изобретению. Концентрация указанного полипептида в фармацевтической композиции может широко варьироваться, т.е. от менее чем приблизительно 0,1% по массе, как правило, по меньшей мере, приблизительно 1% по массе, до целых 20% по массе или более.
Композиция предпочтительно, по меньшей мере, содержит фармацевтически приемлемый носитель в дополнение к активному ингредиенту. Фармацевтический носитель может быть любым совместимым, нетоксичным веществом, подходящим для доставки иммуногенных полипептидов или генотерапевтических векторов пациенту. Для полипептидов в качестве носителя можно использовать стерильную воду, спирт, жиры, воски и инертные твердые вещества. Также в фармацевтические композиции могут быть включены фармацевтически приемлемые адъюванты, буферные средства, диспергирующие средства и т.п.
В соответствии с особенно предпочтительным вариантом осуществления, настоящая фармацевтическая композиция содержит адъювант, как определено более подробно в настоящем документе ранее. Адъюванты для включения в настоящую композицию предпочтительно выбраны из группы лигандов, которые распознаются толл-подобным рецептором (TLR), присутствующим на антигенпрезентирующих клетках, и включают липопептиды (см. например, WO 04/110486), липополисахариды, пептидогликаны, лиоптейхоевые кислоты, липоарабиноманнаны, липопротеины (из микоплазм или спирохет), двухцепочечную РНК (поли I:C), неметилированную ДНК, флагеллин, CpG-содержащую ДНК, Pam3cysSK4, имидазохинолины, а также производные этих лигандов, имеющие химические модификации. Специалист сможет определить точное количество любого из этих адъювантов, которые будут включены в настоящие фармацевтические препараты, чтобы сделать их достаточно иммуногенными. Согласно другому предпочтительному варианту осуществления настоящий фармацевтический препарат может содержать один или несколько дополнительных ингредиентов, которые применяют для усиления иммунитета ЦТЛ, как объяснено в настоящем документе ранее.
Состав лекарственных средств по изобретению, например, композиция, содержащая источник иммуногенного полипептида, пути введения и применение фармацевтически приемлемых эксципиентов известны и общеприняты в данной области и, например, описаны в «Remington: The Science and Practice of Pharmacy» (Ed. Allen, L. V. 22nd edition, 2012, www.pharmpress.com).
Иммуногенные полипептиды, нуклеиновые кислоты, кодирующие их или клетки, экспрессирующие их для использования в настоящем изобретении, можно получать с использованием рекомбинантных способов, таких как описанные в Ausubel et al., «Current Protocols in Molecular Biology», Greene Publishing и Wiley Interscience, New York (1987) и в Sambrook and Russell (2001) «Molecular Cloning: A Laboratory Manual» (3rd edition), Cold Spring Harbor Laboratory, Cold Spring Harbor Laboratory Press, New York; сожержание которых включено в настоящий документ в качестве ссылки в полном объеме. Также см., Kunkel (1985), Proc. Natl. Акад. Sci. 82: 488 (описание сайт-направленного мутагенеза) и Roberts et al. (1987) Nature 328: 731 734 или Wells, J.A., et al. (1985) Gene 34: 315 (описывающие кассетный мутагенез). Однако, более предпочтительно, иммуногенные полипептиды по изобретению или нуклеиновые кислоты, кодирующие их, получают путем химического синтеза. Химический синтез пептидов или нуклеиновых кислот является обычной практикой, и специалисту известы различные подходящие способы. Химический синтез пептидов или нуклеиновых кислот также решает проблемы, связанные с рекомбинантной выработкой, которую сложнее стандартизировать и которая требует обширной очистки и мер контроля качества.
В еще одном аспекте изобретение относится к T-клетке, содержащей Т-клеточный рецептор, которая связывает комплекс MHC-пептид, где пептид предпочтительно является пептидом, содержащим или включающим эпитопы, рестрицированные по MHC класса I и MHC класса II и содержащиеся в нативном ZP, предпочтительно hZP3 или hZP3 (23-350), или гомологом указанного одного или нескольких полипептидов. Такую Т-клетку можно получать, например, способом, включающим контактирование Т-клетки с антигенпрезентирующей клеткой, экспрессирующей полинуклеотид, кодирующий иммуногенный полипептид по изобретению и/или контактирование Т-клетки с антигенпрезентирующей клеткой, нагруженной иммуногенным полипептидом по изобретению; и, необязательно, культивирование указанной Т-клетки. Антигенпрезентирующая клетка (АПК) предпочтительно является дендритной клеткой (ДК). Т-клетка предпочтительно является CD8+ цитотоксической Т-клеткой или CD4+ хелперной Т-клеткой. Введение полинуклеотида, кодирующего иммуногенный полипептид, в АПК или ДК можно проводить любым способом, известным специалисту в данной области, предпочтительно полинуклеотид по изобретению вводят в АПК или ДК с использованием трансфекции. Предпочтительно полинуклеотид, кодирующий иммуногенный полипептид, снабжен надлежащими контрольными последовательностями или должен находиться в надлежащем экспрессирующем векторе. Контактирование Т-клеток с иммуногенным полипептидом по изобретению можно проводить любым способом, известным специалисту в данной области. Предпочтительно, иммуногенный полипептид или эпитоп, содержащийся в иммуногенном полипептиде, представлен CD8+ цитотоксической T-клетке или CD4+ хелперной Т-клетке молекулами MHC класса I или MHC класса II на поверхности АПК, или ДК. Специалист в данной области знает, как загрузить АПК или ДК пептидом. Культивирование указанной T-клетки можно проводить любым способом, известным специалисту в данной области. Поддержание Т-клеток в условиях, необходимых для для сохранения клеток живыми, в настоящем документе также следует рассматривать как культивирование. Предпочтительно, Т-клетка в соответствии с этим аспектом изобретения связывается с иммуногенным полипептидом по изобретению, определенным в первом аспекте изобретения. Способы получения и активации Т-клеток, специфических к опухолевым антигенам, ex vivo более подробно описаны, например, в WO2017/173321. В этом аспекте изобретение также относится к композиции, включающей (активированную) Т-клетку по изобретению, а также к способам по изобретению для терапевтического и/или профилактического лечения рака легких и/или метастазов, включающим введение индивидууму терапевтического количества (активированной) опухолеспецифической Т-клетки, описываемой в настоящем документе, или получение способом, описываемым в настоящем документе (см., например, пример 4). В вариантах осуществления введение включает введение приблизительно от 106 до 1012, приблизительно от 108 до 1011 или приблизительно от 109 до 1010 (активированных) опухолеспецифических Т-клеток. При этом Т-клетку или композицию предпочтительно вводят посредством внутривенного, интраперитонеального, внутриопухолевого, интрадермального или подкожного введения. В другом варианте осуществления Т-клетку или композицию вводят в анатомический участок, который стекает в лимфатический коллектор. В другом варианте осуществления введение производят в несколько лимфатических коллекторов.
Дополнительный аспект изобретения относится к способу лечения или диагностики рака легких и его метастазов у индивидуума путем введения антитела или его фрагмента, которые специфически связываются с эпитопом человеческого белка блестящей оболочки (hZP), предпочтительно антитело или его фрагмент специфически связываются с эпитопом hZP3 или hZP3 (23-350).
Антитело, «которое связывает» интересующий антиген, например, ассоциированный с опухолью антиген белка hZP или его эпитоп, представляет собой антитело, связывающее антиген с достаточной аффинностью, такой, что антитело пригодно в качестве терапевтического средства при нацеливании на клетку или ткань, экспрессирующую антиген, и существенно не реагирует перекрестно с другими белками. В таких вариантах осуществления степень связывания антитело с «нецелевым» белком будет составлять менее чем приблизительно 10% от связывания антитела с его конкретным белком-мишенью, что определяется с помощью анализа активируемой флуоресценцией сортировки клеток (FACS) или радиоиммунологического анализа (RIA). Что касается связывания антитела с молекулой-мишенью, термин "специфическое связывание" или "специфически связывается с" или "является специфичным для" конкретного полипептида или эпитопа на конкретном полипептиде, означает связывание, которое заметно отличается от неспецифического взаимодействия. Специфическое связывание можно измерять, например, путем определения связывания молекулы по сравнению со связыванием контрольной молекулы, которая, как правило, представляет собой необходимое вещество аналогичной структуры, не обладающее активностью связывания. Например, специфическое связывание можно определить путем конкуренции с контрольной молекулой, которая похожа на мишень, например, избытком немеченой мишени. В этом случае, специфическое связывание определяют, если связывание меченой мишени с зондом конкурентно ингибируется избытком немеченой мишени. Как применяют в настоящем документе термин «специфическое связывание» или «специфически связывается с» или «специфичен» для конкретного полипептида или эпитопа на конкретной полипептидной цели может быть продемонстрирован, например, молекулой, имеющей для мишени Kd (которую можно определить, как описано ниже), по меньшей мере, приблизительно 10-4 M, альтернативно, по меньшей мере, приблизительно 10-5 M, альтернативно, по меньшей мере, приблизительно 10-6 M, альтернативно, по меньшей мере, приблизительно 10-7 M, альтернативно, по меньшей мере, приблизительно 10-8 M, альтернативно, по меньшей мере, приблизительно 10-9 M, альтернативно, по меньшей мере, приблизительно 10-10 M, альтернативно, по меньшей мере, приблизительно 10-11 M, альтернативно, по меньшей мере, приблизительно 10-12 M, или больше. В одном из вариантов осуществления термин «специфическое связывание» относится к связыванию, при котором молекула связывается с определенным полипептидом или эпитопом на определенном полипептиде по существу без связывания с любым другим полипептидом или полипептидным эпитопом.
«Kd» или «значение Kd» можно измерить с помощью анализов поверхностного плазмонного резонанса с использованием BIAcore™-2000 или BIAcore™-3000 (BIAcore, Inc., Piscataway, NJ) при 25°C с чипами CM5 с иммобилизованными антигенами при ~ 10-50 единиц ответа (РЕ). В кратком изложении, биосенсорные чипы с карбоксиметилированным декстраном (CM5, BIAcore Inc.) активируют с помощью N-этил-N’-(3-диметиламинопропил)-карбодиимид гидрохлорида (EДК) и N-гидроксисукцинимида (NHS) по инструкциям поставщика. Антиген разбавляют 10 мМ ацетата натрия, pH 4,8, до 5 мкг/мл (± 0,2 мкМ) перед инъекцией при скорости потока 5 мкл/минуту для достижения приблизительно 10 единиц ответа (РЕ) связанного белка. После инъекции антигена вводят 1М этаноламин, чтобы блокировать непрореагировавшие группы. Для измерения кинетики двукратные серийные разведения антитела или Fab (от 0,78 до 500 нМ) вводят в PBS с 0,05% Твин 20 (PBST) при 25°C при скорости потока приблизительно 25 мкл/мин. Скорости ассоциации (kon) и скорости диссоциации (koff) рассчитывают с использованием простой однозначной модели связывания Ленгмюра (BIAcore Evaluation Software, версия 3.2) путем одновременного подбора сенсограммы ассоциации и диссоциации. Равновесную константу диссоциации (Kd) рассчитывают как отношение koff/kon. См., например, Chen, Y. et al., (1999) J. Mol. Biol. 293: 865-881. Если скорость прямой реакции превышает 106 M-1с-1 по анализу поверхностного плазмонного резонанса выше, то скорость прямой реакции можно определить с помощью способа гашения флуоресценции, который измеряет увеличение или уменьшение интенсивности флуоресценции (возбуждение = 295 нм; излучение = 340 нм, полоса пропускания 16 нм) при 25°C с 20 нМ антитела к антигену (Fab-форма) в PBS, pH 7,2, в присутствии увеличивающихся концентраций антигена, измеренных в спектрометре, таком как спектрофотометр с остановленным потоком (Aviv Instruments) или спектрофотометр SLM-Aminco серии 8000 (ThermoSpectronic) с красной кюветой с мешалкой. «Скорость прямой реакции» или «скорость ассоциации» или "kon" по настоящему изобретению также можно определить с помощью той же методики поверхностного плазмонного резонанса, описанной выше, с использованием BIAcore™-2000 или BIAcore™-3000 (BIAcore , Inc., Piscataway, NJ), как описано выше.
Как применяют в настоящем документе, термин «антитело» рассматривается в широком смысле и относится к любому типу молекулы иммуноглобулина, включая поликлональные антитела, моноклональные антитела, в том числе мыши, человека, адаптированные к человеку, гуманизированные и химерные моноклональные антитела, фрагменты антител, биспецифические или полиспецифические антитела, димерные, тетрамерные или мультимерные антитела и одноцепочечные антитела.
Один из вариантов осуществления изобретения относится к способу лечения рака легких и его метастазов у индивидуума, включающему введение указанному индивидууму композиции, содержащей антитело к ZP, анти-hZP3 или антитело к hZP3 (23-350), где антитело вызывает гибель злокачественных клеток легких путем антителозависимой клеточной цитотоксичности (ADCC), антитело-зависимого клеточного фагоцитоза (ADCP), обусловленной комплементом цитотоксичности (CDC) или апоптоза. Как, в основном, понимают специалисты в данной области, эти эффекторные функции антитела могут быть опосредованы Fc-частью антитела, например, путем связывания эффекторного домена (-ов) Fc с Fc-рецептором на иммунной клетке с фагоцитирующей или литической активностью или путем связывания эффекторного домена (-ов) Fc с компонентами системы комплемента. Как правило, эффект (-ы), опосредуемый Fc-связывающими клетками или компонентами комплемента, в конечном итоге приводит к ингибированию и/или истощению клеток-мишеней, т.е. ZP-экспрессирующих клеток. Изотипы человеческого IgG - IgG1, IgG2, IgG3 и IgG4 проявляют дифференциальную способность к эффекторным функциям. ADCC может быть опосредована IgG1 и IgG3, ADCP может быть опосредована IgG1, IgG2, IgG3 и IgG4, а CDC может быть опосредована IgG1 и IgG3. В способах, описываемых в настоящем документе, антитело к hZP3 или антитело к hZP3 (23-350) предпочтительно является антителом IgG1, IgG2, IgG3 или IgG4.
В способах, описываемых в настоящем документе, антитело к hZP3 или антитело к hZP3 (23-350) индуцирует in vitro и/или in vivo гибель злокачественных клеток легких, которые экспрессируют белок ZP3, посредством антителозависимой клеточной цитотоксичности (ADCC). В способах, описываемых в настоящем документе, антитело к hZP3 или антитело к hZP3 (23-350) индуцирует in vitro и/или in гибель злокачественных клеток легких, которые экспрессируют белок ZP, путем комплементзависимой цитотоксичности (CDC). В способах, описываемых в настоящем документе, антитело к hZP3 или антитело к hZP3 (23-350) индуцирует in vitro и/или in гибель злокачественных клеток легких, которые экспрессируют белок ZP3, путем антителозависимого клеточного фагоцитоза (ADCP).
В способах, описываемых в настоящем документе, антитело к hZP3 или антитело к hZP3 (23-350) индуцирует in vitro и/или in гибель злокачественных клеток легких, которые экспрессируют белок ZP3, путем апоптоза.
В способах, описываемых в настоящем документе, антитело к ZP может связывать человеческий ZP с диапазоном аффинностей (KD). В одном из вариантов осуществления изобретения антитело к ZP связывается с ZP с высокой аффинностью, например, с KD, равной или менее чем приблизительно 10-7 М, такой как, но не ограничиваясь, 1-9,9 (или любым диапазоном или значением в его пределах, таким как 1, 2, 3, 4, 5, 6, 7, 8, или 9)×10−8, 10−9, 10−10, 10−11, 10−12, 10−13, 10−14, 10−15 или любой диапазон или значение в нем, как определено поверхностным плазмонным резонансом или способом Kinexa (анализа кинетического исключения), как практикуют специалисты в данной области. В одном из примеров аффинность равна или менее чем 1×10−9 М. В другом из примеров аффинность равна или менее чем 1×10−9 М.
Предпочтительными антителами для использования в настоящем изобретении являются моноклональные антитела. Подходящие (моноклональные) антитела можно получать, проверять и производить способами, которые хорошо известны в данной области и, например, описаны в учебниках, таких как «Antibodies: A Laboratory Manual, Второй edition, Edited by E.A. Greenfield, 2014, Cold Spring Harbor Laboratory Press. Более конкретно, Ranking et al. (1998, Development 125, 2415-2424) описывают генерацию моноклонального антитела H3.1 против C-концевого пептида внеклеточного домена hZP3, содержащего аминокислоты 335-350 из SEQ ID NO: 3. Более предпочтительно, антитела для использования в настоящем изобретении являются гуманизированными, или даже более предпочтительно человеческими моноклональными антителами, которые могут быть получены с помощью методов, хорошо известных в данной области, таких как например, описанные соответственно, в Olimpieri et al. (Bioinformatics. 2015; 31 (3): 434–435) и Sheehan and Marasco (Microbiol Spectr. 2015; 3 (1): AID-0028-2014) и в ссылках, цитируемых в них.
Любые антитела, которые связываются с внеклеточным доменом белка ZP, таким как внеклеточный домен hZP3, т.е. hZP3 (23-350), у которого внеклеточный белок имеет аминокислотную последовательность SEQ ID NO: 5, можно использовать для настоящего изобретения. Примеры подходящих антител, которые можно использовать для настоящего изобретения, включают, например, антитела, которые способны перекрестно блокировать связывание одного или нескольких референсных антител, которые, как известно, связываются с hZP3 (23-350). Одним из таких референсных антител, которое, как известно, специфически связывается с hZP3 (23-350), является антитело H3.1, описанное Ranking et al. (1998, выше), гибридома для которого может быть получена от ATCC под номером доступа: ATCC CRL-2569.
Таким образом, предпочтительное антитело для использования в настоящем изобретении обладает способностью перекрестно блокировать связывание, по меньшей мере, одного антитела, которое, как известно, специфически связывается с ZP, предпочтительно с hZP3, более предпочтительно с hZP3 (23-350) или гомологом указанного полипептида. Более предпочтительно, антитело обладает способностью перекрестно блокировать связывание антитела H3.1 с номером доступа ATCC CRL-2569. Способность антитела к ZP перекрестно блокировать связывание референсного антитела в настоящем документе определяют как способность уменьшать связывание референсного антитела с подходящей молекулой-мишенью, включающей аминокислотные последовательности ZP, hZP3 или hZP3 (23-350), по меньшей мере, на 10, 20, 50, 75, 90, 95, 99, 99,9 или 99,99%, когда молекула-мишень сначала связывается с антителом к ZP, или наоборот (т.е. связывание антитела к ZP уменьшается, когда молекула-мишень сначала связывается с референсным антителом). Способность к перекрестной блокировке в принципе можно определять с использованием иммунологического анализа любого типа, предпочтительно, конкурентного иммунологического анализа, включая, например, ELISA, прямой или непрямой радиоиммунологический анализ (RIA), прямой или непрямой твердофазный ферментный иммунологический анализ (EIA), конкурентный сэндвич-анализ (см. Stahli et al., Methods in Enzymology 9:242-253 (1983)); твердофазный прямой биотин-авидиновый EIA (см. Kirkland et al 20, J. Immunol. 137: 3614-3619 (1986)); твердофазный прямой анализ с меткой, твердофазный прямой сэндвич-анализ с меткой (см. "Antibodies, A Laboratory Manual," Second edition, 2014; выше); твердофазный прямой RIA с меткой с использованием I125 (см. Morel et al., Molec. Immunol. 25 (1): 7-15 (1988)); твердофазный прямой биотин-авидиновый EIA (Cheung et al., Virology 176: 546-552 (1990)); и прямой RIA с меткой (Moldenhauer et al., Scand. J. Immunol. 32: 77-82 (1990)). Как правило, такой анализ включает использование очищенной молекулы-мишени, связанной с твердой поверхностью, немеченого исследуемого антитела и меченного контрольного антитела. Конкурентное ингибирование измеряют путем определения количества метки, связанной с твердой поверхностью в присутствии исследуемого антигенсвязывающего белка. Как правило, исследуемое антитело присутствует в избытке.
В способах по изобретению, описываемых в настоящем документе, антитело к ZP может быть предоставлено в подходящих фармацевтических композициях, включающих антитело к ZP и фармацевтически приемлемый носитель. Подходяще носители и составы, включающие в себя другие человеческие белки, например, сывороточный альбумин человека, описаны, например, в The Science and Practice of Pharmacy, 21st Edition, Troy, D. B. ed., Lipincott Williams and Wilkins, Philadelphia, Pa. 2006, Part 5, Pharmaceutical Manufacturing pp 691-1092, см. особенно pp. 958-989, см. особенно с. 958-989.
Способ введения антитела к ZP в способах по изобретению, описываемых в настоящем документе, может быть любым подходящим способом, таким как парентеральное введение, например, интрадермальное, внутримышечное, интраперитонеальное, внутривенное или подкожное, легочное, трансмукозальное (пероральное, интраназальное, интравагинальное или ректальное) или другие способы введения, понятные специалистами в данной области, поскольку они хорошо известны в данной области. Антитело к ZP в способах по изобретению, описываемых в настоящем документе, можно вводить пациенту любым подходящим путем, например, путем внутривенной (в/в) инфузии или болюсной инъекции, внутримышечно или подкожно, или интраперитонеально.
В способах по изобретению антитело к ZP вводят в терапевтически эффективном количестве. Термин «терапевтическое эффективное количество» относится к количеству, эффективному в дозировках и для периодов времени, необходимых для достижения желаемого терапевтического результата. Терапевтически эффективное количество может варьироваться в зависимости от факторов, таких как состояние болезни, возраст, пол, масса индивидуума и способность терапевтического средства или комбинации терапевтических средств вызывать желаемый ответ у индивидуума. Примерные показатели эффективного терапевтического средства или сочетания терапевтических средств включают, например, улучшение самочувствия пациента, уменьшение массы опухоли, остановку или замедление роста опухоли и/или отсутствие метастазирования злокачественных клеток в другие места в организме.
Доза, назначаемая индивидууму, страдающему от рака легких, достаточна для облегчения или, по меньшей мере, частичного купирования заболевания, подвергаемого лечению («терапевтическое количество»), и иногда может составлять от 0,005 мг до приблизительно 100 мг/кг, например, приблизительно от 0,05 мг до приблизительно 30 мг/кг или приблизительно от 5 мг до приблизительно 25 мг/кг, или приблизительно 4 мг/кг, приблизительно 8 мг/кг, приблизительно 16 мг/кг или приблизительно 24 мг/кг, или приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 мг/кг, но может быть даже выше, например, приблизительно 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 40 , 50, 60, 70, 80, 90 или 100 мг/кг.
Настоящее изобретение также относится к способу лечения рака легких и его метастазов у индивидуума с помощью нацеленной терапии, как правило, путем введения композиции, содержащей антитело или его фрагмент, способный к иммуноспецифическому связыванию с эпитопом человеческого белка блестящей оболочки (hZP), предпочтительно антитело или его фрагмент способны к иммуноспецифическому связыванию с эпитопом человеческого белка блестящей оболочки 3 (hZP3), где антитело или фрагмент является частью иммуноконъюгата, такого как иммунотоксин, конъюгат антитело-лекарственное средство или антитело, переносящее радиоактивный изотоп.
Термин «иммуноконъюгат» относится к конъюгатам, в которых антитело или его фрагмент химически связаны с другой молекулой. Когда молекула, связанная с антителом или его фрагментом, является токсином, иммуноконъюгат известен как иммунотоксин. Когда молекула, связанная с антителом или его фрагментом, является (цитотоксическим) лекарственным средством, иммуноконъюгат часто обозначают как «конъюгат антитело-лекарственное средство». Применение иммуноконъюгатов для местной доставки цитотоксических или цитостатических агентов, т.е. лекарственных средств для уничтожения или ингибирования опухолевых клеток при лечении злокачественной опухоли может позволить направленную доставку лекарственного средства к опухолям, и внутриклеточн накопление в них, при этом системное введение этих неконъюгированных лекарственных средств может привести к недопустимому уровню токсичности для нормальных клетокв то время как опухолевые клетки избегали гибели. Тем самым достигается максимальная эффективность с минимальной токсичностью. Усилия по разработке и усовершенствованию конъюгатов атител были сосредоточены на селективности моноклональных антител (mAb), а также на свойствах свзяывания и высвобождения лекарственного средства.
В некоторых вариантах осуществления радиоактивный изотоп может быть связан с антителом, полученный иммуноконъюгат часто обозначают как иммунорадиоактивный изотоп.
Как будет понятно специалистам в данной области, антитело к части ZP может представлять собой антитело к ZP, имеющее какие-либо или все характеристики, описанные в данном документе ранее, в отношении варианта осуществления пассивной иммунизации. Иммуноконъюгаты могут также содержать антитело или его фрагмент, который просто способен к иммуноспецифическому связыванию с эпитопом с эпитопом человеческого белка блестящей оболочки (hZP), предпочтительно антитело или его фрагмент способно к иммуноспецифическому связыванию с эпитопом с эпитопом человеческого белка блестящей оболочки 3 (hZP3), без индуцирования каких-либо эффекторных механизмов.
Настоящее изобретение дополнительно относится к иммуноконъюгатам, содержащим антитело или его фрагмент, конъюгированные с радиоактивным изотопом или радионуклидом или с группой (способной содержать), содержащей такой радионуклид. Опытный врач поймет, что существует множество радионуклидов, которые могут быть связаны с опухолеспецифическими антителами с помощью хорошо известных способов и доставлены к нужному участку для специфического повреждения опухолевых клеток и тканей. Например, пригодные для использования реагенты включают 125I, 123I. 111In, 177Lu, 90Y, 213Bi, 225Ac и 99mTc. Среди радионуклидов, особенно подходящим для радиоиммунотерапии может быть иттрий-90 (90Y), поскольку иттрий-90 (90Y) обеспечивает преимущества по сравнению с йодом-131 (131I), т.к. он обеспечивает более высокую энергию бета (2,3 МэВ по сравнению с 0,61 МэВ) в отношении опухоли и имеет длину пути от 5 до 10 мм, что приводит к улучшенной способности уничтожать как целевые, так и соседние клетки, что является преимуществом, в частности, при объемной или плохо васкуляризованной опухоли.
В дополнительном варианте осуществления изобретение относится к способу диагностики и/или прогнозирования рака легких и/или его метастазов и/или его рецидива у человеческого индивидуума. Способ предпочтительно применяют для диагностики и/или прогнозирования немелкоклеточного рака легких и/или его метастазов и/или его рецидива. Способ диагностики и/или прогнозирования предпочтительно включает введение антитела или его фрагмента, который специфически связывается с эпитопом hZP, а также с hZP3 или hZP3 (23-350), как описано выше. Антитело или его фрагмент предпочтительно конъюгировано с визуализирующим средством или детектируемым средством, предпочтительно средство является визуализирующим средством in vivo. Неограничивающие примеры таких средств включают ферменты, радиоактивные метки, гаптены, флуоресцентные метки, фосфоресцентные молекулы, хемилюминесцентные молекулы, хромофоры, люминесцентные молекулы, фотоаффинные молекулы, цветные частицы или лиганды, такие как биотин, парамагнитные ионы, радиоактивные изотопы, фторхромы, ЯМР-обнаруживаемые вещества и рентгеновские визуализирующие средства (см. например, US20170240637 A1).
Фраза «визуализация in vivo», как применяют в настоящем документе, относится к способам детекции присутсвия детектируемого меченого антитела по изобретению у живого млекопитающего. Оптически детектируемые белки, такие как флуоресцентные антитела и антитела, связанные с люциферазой, могут быть обнаружены с помощью визуализации in vivo. Изображения in vivo можно использовать для получения 2-D и 3-D изображений млекопитающего. Меченые радиоактивным изотопом антитела, например, можно вводить индивидууму и получать изображения индивидуума с помощью гамма-камеры. Для получения изображений in vivo можно использовать камеры на приборах с зарядовой связью, CMOS или 3D-томографы. Способы визуализации in vivo с использованием компьютерной томографии, магнитно-резонансной томографии, ультрасонографии, позитронно-эмиссионной томографии, однофотонной эмиссионной компьютерной томографии (SPECT) и т.п. хорошо известны в данной области. Информация от многих способов визуализации in vivo, как описано выше, может предоставить данные о злокачественных клетках в индивидууме.
В диагностических и/или прогностических способах детектируемое меченое антитело по изобретению можно использовать, чтобы определить, есть ли у индивидуума рак легких, и/или его метастазы, и/или его рецидив, который более или менее поддается лечению, описываемому в настоящем документе, а также, чтобы следить за ходом лечения и/или ответом на терапию у индивидуума. Также его можно использовать для оценки курса других (комбинированных) методов лечения. Таким образом, диагностические/прогностические способы могут информировать врача о выборе терапии и схемы лечения.
В этом документе и в его формуле изобретения глагол «содержать» и его спряжения применяют в неограничивающем смысле для обозначения того, что элементы, следующие за словом, включены, но элементы, конкретно не упомянутые, не исключены. Кроме того, ссылка на элемент неопределенным артиклем «a» или «an» не исключает возможности того, что присутствует более одного элемента, если контекст явно не требует наличия одного и только одного из элементов. Неопределенный артикль a» или «an», таким образом, как правило, означает «по меньшей мере, один».
Изобретение далее иллюстрируется следующими примерами, которые не предназначены для ограничения объема изобретения каким-либо образом.
ОПИСАНИЕ РИСУНКОВ
Фигура 1: Экспрессия ZP3 в клетках плоскоклеточной карциномы рака легких от трех разных пациентов. Каждая горизонтальная полоса представляет результаты одного пациента. Слева направо, соответственно, гистопатология с окраской гематоксилином-эозином (HEX5); гибридизация in situ экспрессии мРНК ZP3 (объем РНК X40); и иммуногистохимия экспрессии белка ZP3 (ИГХ X20). Избыточная экспрессия ZP3 обнаруживается исключительно в клетках плоскоклеточной карциномы клетках (SCC; стрелки), и экспрессия ZP3 не наблюдается в стромальных клетках (S) или здоровых клетках.
Фигура 2: Экспрессия ZP3 в клетках аденокарциномы рака легких от трех разных пациентов. Каждая горизонтальная полоса представляет результаты одного пациента. Слева направо, соответственно, гистопатология с окраской гематоксилином-эозином (HEX5); гибридизация in situ экспрессии мРНК ZP3 (объем РНК X40); и иммуногистохимия экспрессии белка ZP3 (ИГХ X20). Избыточная экспрессия ZP3 обнаруживается исключительно в клетках аденокарциномы (ACC; стрелки), и экспрессия ZP3 не наблюдается в стромальных клетках (S) или здоровых клетках.
Фигура 3: Экспрессия ZP3 в яичнике (верхний ряд) и в селезенке (нижний ряд), в качестве, соответственно, положительного и отрицательного контролей. Слева направо, соответственно, гистопатология с окраской гематоксилином-эозином (HEX5); гибридизация in situ экспрессии мРНК ZP3 (объем РНК X40); и иммуногистохимия экспрессии белка ZP3 (ИГХ X20). Обилие мРНК ZP3 обнаружили в ооцитах, а белок ZP3 обнаружили в ZP, окружающей ооциты человека, а также в цитоплазме ооцитов. В селезенке экспрессия мРНК ZP3 или белок не обнаружены.
Фигура 4: Выбранные последовательности пептида hZP3. Коробки и овалы содержат предсказанные вещества, связывающие МНС класса I и II, как указано в таблицах 2 и 5, и 3 соответственно. Звездочками обозначены потенциальные N-связанные участки гликозилирования, а в скобках над последовательностями обозначены области с кластером O-связанных гликанов.
Фигура 5: График введения hZP3-положительных B16-HLA-A2 опухолевых клеток меланомы и hZP3-пептидной смеси (включающей пептиды 4, 5, 6, 8 и 9 в таблице 5) или белка rhZP31-383 трансгенным мышам HLA-A2. На Сутки 0 (D0) была сделана подкожно (s.c.) инъекция опухоли 106 клеток/мышь. После этого вводили первую инъекцию с 7 до 10 суток (D7-10) и бустерную инъекцию на сутки 14 до суток 17 (D14-17). Размер опухоли наблюдали после лечения. В сутки с 30 до 35 мышей умерщвляли, чтобы определить hZP3-специфические CD8+ ответы (ELISpot).
Фигура 6: Размер опухоли (в мм3) как функция суток после заражения опухолью у контрольных мышей, получающих PBS, и у мышей после 2 подкожных иммунизаций либо белком rhZP31-383, либо комбинацией 5 пептидов hZP3 (включая пептиды 4, 5, 6, 8 и 9 в таблице 5).
фигура 7: Ответы CD8+ T-клеток, определенные по ELISpot с интерферон-γ (IFNg), измеренные в отношении пептидов (все 5 или 4, 5, 6, 8 или 9 пептидов индивидуально) после иммунизации смесью из 5 пептидов hZP3 (пептид) или после иммунизации белком ZP3 rhZP31-383 (белок).
ПРИМЕРЫ
Пример 1
Экспрессию ZP3 в ткани рака легких человека определяли с помощью иммуногистохимии (ИГХ). Опухолевые микрочипы (TMA) из тканей рака легких, происходящих из разных пациентов, получали из института патологии в Нидерландах.
Иммуногистохимические определения проводили с человеческими моноклональными антителами H3.1 к ZP3 в двух разных концентрациях, 1,0 мкг мл и 0,1 мкг/мл.
Срезы человеческого яичника, содержащие ооциты, использовали в качестве тканевых положительных контролей. Кроме того, образец ткани селезенки использовали как отрицательный контроль.
Следующий протокол ИГХ использовали для всех образцов:
Сутки 1
1. Депарафинизация и гидратация:
a) Ксилен – 3X3 мин,
b) Абсолютный EtOH – 2X3 мин
c) 96% EtOH – 2X3 мин
d) 70% EtOH – 2X3 мин
е) dH20–1– 1X10 мин
2. Извлечение антигена:
а) Буфер с 10 мМ лимонной кислотой с 0,05% Твин 20 (рН 6,0) в извлекателе в течение 2,5 часов
б) охладить
2. Промыть срезы в Трис-забуференном физиологическом растворе (TBS) с 0,05% Tween 20 (TBST) 3 раза в течение 5 минут каждый.
3. Инкубировать срезы в 3% H2О2 (разбавленном в TBS) в течение 15 минут, чтобы погасить активность эндогенной пероксидазы при комнатной температуре (RT), в темноте.
4. Промыть срезы в TBST (TBS с 0,05% Tween 20) 2 раза в течение 5 минут каждый.
5. Инкубировать срезы в 1 мг/мл раствора борогидрида натрия (разведенного в TBS) в течение 15 минут, чтобы погасить свободные группы альдегидов (RT, в темноте, более важно для ИФ-окрашивания, совместная локализация ZP3).
6. Промыть срезы в TBST (TBS с 0,05% Tween 20) 3 раза в течение 5 минут каждый.
7. Инкубировать срезы в блокирующем растворе - 3% BSA в TBST (1 час; RT; увлажненная камера).
8. Инкубировать срезы с первичным антителом против hZP3 (1,0, 0,125 или 0,1 мкг/мл), разведенным в блокирующем растворе (3% BSA в TBST) в течение ночи в увлажненной камере в холодной комнате.
Сутки 2
1. Промыть срезы в TBST 3 раза в течение 5 минут каждый.
2. Инкубировать срезы с анти-мышиным полимером DAKO 30 минут (EnVision # 4001).
3. Промыть срезы в TBST 3 раза в течение 5 минут каждый.
4. Инкубировать срезы с системой хромогена (DAKO, 1 капля на 1 мл буфера # K3468) - 10 мин, остановить реакцию, окунув слайды в контейнер с водопроводной водой.
5. Инкубировать срезы в водопроводной воде в течение 2 минут.
6. Промыть слайды с ddH2O перед контрастным окрашиванием
7. Контрастное окрашивание срезов в гематоксилине - 20-30 секунд.
8. Инкубировать срезы в водопроводной воде, чтобы удалить избыток гематоксилина.
9. Дегидратация срезов:
a) 70% EtOH - 2X3 мин
b) 96% EtOH - 2X3 мин
с) Абсолютный EtOH - 2X3 мин
d) Ксилол - 2X3 мин
10. Крепление с помощью Pertex.
В положительных контролях, антитела обнаруживают белки в ZP, окружающей ооцит человека. Белки ZP3 также присутствуют в цитоплазме ооцитов. Не обнаружено положительного окрашивания в срезах образцов тканей рака легких, если первичное антитело отсутствует. Не наблюдали положительного окрашивания с моноклональным антителом к ZP в ткани селезенки и печени.
В образцах рака легких (TMA) присутствие ZP3 подтверждается областями положительного окрашивания ткани на ZP3, причем интенсивности варьируют среди образцов, полученных от разных пациентов (см. результаты TMA плоскоклеточных злокачественных опухолей в таблице 1). Такие опухоли, окрашенные положительно в отношении экспрессии ZP3, можно лечить путем иммунизации антигенами ZP3 в соответствии с настоящим изобретением.
(1,0 мкг/мл)
(0,1 мкг/мл)
Пример 2
Экспрессию ZP3 в тканях рака легких человека определяли с помощью ИГХ (плоскоклеточная злокачественная опухоль, n=10 и аденокарцинома легкого, n=10) и гибридизации объема РНК in situ (плоскоклеточная злокачественная опухоль, n=4 и аденокарцинома легкого, n=4). Образцы ткани от разных пациентов были получены от института патологии в Польше.
Иммуногистохимические определения проводили с моноклональными антителами человека к ZP3 в двух разных концентрациях. Следовали тому же протоколу ИГХ, что и описанный в примере 1.
Срезы яичника человека использовали в качестве тканевого положительного контроля. Кроме того, образец ткани селезенки использовали в качестве отрицательного контроля.
Гибридизацию in situ образцов яичника, фиксированых в формалине с погружением в парафин (FFPE), проводили с помощью RNAscope FFPE 2.0 HD Detection Kit Brown [Advanced Cell Diagnostics (ACD), Hayward, California, USA, кат. № 310033] по протоколу производителя.
Депарафинизация срезов FFPE
1. В вытяжном шкафу заполнить две емкости для очищающих средств ~ 200 мл свежего ксилола и две - 200 мл свежего 100% этанола.
2. Поместите предметные стекла в первую емкость для очищающих средств с ксилолом и инкубируйте в течение 5 минут при комнатной температуре. Перемешайте стекла, периодически двигая подставку со стеклами вверх и вниз в емкости с чистящим средством.
3. Извлеките подставку со стеклами из первой емкости для очищающих средств с ксилолом и сразу же поместите во вторую емкость для очищающих средств с ксилолом в вытяжной шкаф и повторите инкубацию.
4. Извлеките подставку со стеклами из второй емкости для очищающих средств с ксилолом и немедленно поместите в емкость для окрашивания, содержащую 100% этанол. Инкубируйте стекла в 100% этаноле в течение 1 минуты при комнатной температуре с перемешиванием.
5. Повторите шаг 4 со свежим 100% этанолом.
6. Извлеките стекла из подставки и положите их на впитывающую бумагу срезом вверх. Сушите на воздухе в течение 5 минут при комнатной температуре.
Уравновесьте оборудование
1. Включите печь HybEZ™ и установите температуру на 40°C.
2. Поместите увлажняющую бумагу в лоток для контроля влажности и полностью смочите дистиллированной водой.
3. Вставьте закрытый лоток в печь и закройте дверцу печи. Грейте лоток в течение 30 минут при 40°C перед использованием.
Подготовьте 1X предварительную обработку 2
Приготовьте 700 мл свежей 1X предварительной обработки 2, добавив 630 мл дистиллированной воды в 1 флакон (70 мл) 10X раствора предварительной обработки 2 в химическом стакане на 1 л. Хорошо перемешать. Поместите стакан, содержащий 1X предварительную обработку 2 на горячую плитуу. Накройте стакан фольгой и включите горячую плиту на высокий уровень на 10–15 минут. Как только 1X предварительная обработка 2 достигнет кипения, поверните ручку нагревательной плиты до 100–104°C, чтобы поддерживать равномерное кипение.
Предварительная обработка 1 и 2
1. Нанесите предварительную обработку, чтобы покрыть весь срез и инкубировать в течение 10 минут при комнатной температуре.
2. Удалите раствор предварительной обработки 1 с одного предметного стекла за один раз, прижав предметное стекло к впитывающей бумаге, и/или щелкнув. Немедленно вставьте предметное стекло в подставку для предметных стекол, погруженную в емкость для окрашивания, заполненную дистиллированной водой.
3. Промойте стекла дважды в дистиллированной воде.
4. Нанесите предварительную обработку 2. Убедитесь, что раствор 1X предварительной обработки 2 находится при слабом кипении. С помощью пары щипцов очень медленно погрузите подставку, содержащую стекла, в кипящий раствор 1X предварительной обработки 2. Накройте стакан фольгой и кипятите стекла в течение 15 минут. После того, как время предварительной обработки закончилось, используйте щипцы, чтобы немедленно перенести подставку с горячими стеклами из 1X предварительной обработки 2 в емкость для окрашивания, содержащую дистиллированную воду. Не позволяйте стеклам остыть в предварительной обработке 2.
5. Промойте стекла дважды в дистиллированной воде.
6. Промойте стекла в свежем 100% этаноле, перемещая подставку вверх и вниз 3–5 раз. Высушите стекла на воздухе. Создайте гидрофобный барьер, используя ручку для гидрофобного барьера.
7. Поместите высушенные стекла на подставку для стекол и добавьте предварительную обработку 3, чтобы полностью покрыть каждый срез. Извлеките лоток для контроля влажности HybEZ™ из печи HybEZ™ и поместите подставку для стекол HybEZ™ в лоток. Закройте крышку и вставьте лоток обратно в печь.
8. Инкубируйте стекла при температуре 40°С в течение 30 минут.
9. Выймите лоток контроля влажности HybEZ™ тз духовки.
10. Промойте стекла дважды дистиллированной водой.
11. Добавьте соответствующий зонд, чтобы полностью покрыть каждый срез.
Зонд к человеческому ZP3 (каталожный номер 442631), зонд положительного контроля для транскриптов низкой численности (Mm-Polr-2a, # 312471) и зонд отрицательного контроля (DapB, ACD-310043).
Поместите подставку для стекол HybEZ™ в лоток для контроля влажности HybEZ™, закройте крышкой и поставьте в печь на 2 часа при 40°C.
12. Извлеките лоток контроля HybEZ™ из печи и удалите подставку для стекол HybEZ™.
13. Промойте стекла в 1X промывочном буфере в течение 2 минут при комнатной температуре. Перемешайте стекла, перемещая подставку для стекол вверх и вниз в емкости.
14. Повторите шаг 13 со свежим 1X промывочным буфером.
15. Инкубируйте стекла с Amp 1 с полностью покрытым каждым срезом в течение 30 минут при 40°C.
16. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
17. Инкубируйте стекла с Amp 2 с полностью покрытым каждым срезом в течение 15 минут при 40°C.
18. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
19. Инкубируйте стекла с Amp 3 с полностью покрытым каждым срезом в течение 30 минут при 40°C.
20. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
21. Инкубируйте стекла с Amp 4 с полностью покрытым каждым срезом в течение 15 минут при 40°C.
22. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
23. Инкубируйте стекла с Amp 5 с полностью покрытым каждым срезом в течение 30 минут при комнатной температуре.
24. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
25. Инкубируйте стекла с Amp 6 с полностью покрытым каждым срезом в течение 15 минут при комнатной температуре.
26. Промойте стекла дважды в 1X промывочном буфере в течение 2 минут при комнатной температуре с периодическим перемешиванием.
27. Смешайте равные объемы коричневого-A и коричневого-B (субстрат DAB) в пробирке соответствующего размера, дозируя одинаковое количество капель (2 капли каждого реагента, всего 4) для каждого раствора. Пипеттируйте DAB на каждый срез ткани. Убедитесь, что срезы покрыты, и инкубируйте в течение 10 минут при комнатной температуре.
28. Промойте предметные стекла в дистиллированной воде, перемещая подставку для стекол вверх и вниз 3–5 раз. Замените свежей дистиллированной водой.
29. Контрастное окрашивание стекол в окрашивающем растворе 50% гематоксилина в течение 2 минут при комнатной температуре.
30. Промойте предметные стекла в дистиллированной воде, перемещая подставку для стекол вверх и вниз 3–5 раз. Повторяйте со свежей дистиллированной водой до тех пор, пока стекла не станут прозрачными, а участки останутся фиолетовыми.
31. Замените дистиллированную воду в емкости для окрашивания окрашивающей посуде на 0,02% аммиачную воду. Переместите подставку вверх и вниз 2–3 раза. Срез должен стать синим. Замените аммиачную воду на дистиллированную воду. Промойте стекла 3–5 раз.
32. Дегидратируйте стекла (70% этанол в течение 2 минут, 100%-70% этанол в течение 2 минут, ксилол в течение 5 минут).
33. Закрепите образцы с помощью Pertex.
В положительных контролях антитела обнаруживают белки в ZP, окружающей ооцит человека, а также в цитоплазме ооцита (фигура 3, верхний ряд ИГХ X20). Не было выявлено положительного окрашивания в срезах образцах тканей рака легких, если отсутствует первичное антитело (не показано). Не наблюдали положительного окрашивания с моноклональным антителом к ZP в ткани селезенки (фигура 3, нижний ряд ИГХ X20).
В образцах рака легких, в подтипах плоскоклеточной злокачественной опухоли (фигура 1) и аденокарциномы (фигура 2) присутствие ZP3 подтверждается областями положительного окрашивания ткани на ZP3, как по ИХС, так и по объему РНК, причем интенсивности варьируются среди полученных образцов от разных пациентов Опухоли, такие как эти, которые окрашиваются положительно в отношении экспрессии ZP3, можно лечить путем иммунизации антигенами ZP3 в соответствии с настоящим изобретением.
Пример 3
Последовательность hZP3 анализировали на эпитопы цитотоксических Т-клеток (ЦТЛ), рестрицированные по HLA класса I, и вспомогательные эпитопы, рестрицированные по HLA класса II, с использованием алгоритмов, которые предсказывают связывание пептидов с HLA классов I и II.
Предсказания по связыванию MHC класса I доступны с использованием сервера NetCTLpan от Центра анализа биологических последовательностей (CBS) Технического университета Дании (http://www.cbs.dtu.dk/services/NetCTLpan/) (Xu et al., 2012, Clin Dev Immunol; 12: 831010).
Прогнозы связывания MHC класса II были сделаны с использованием инструмента Consensus на ресурсе для анализа IEDB (http://tools.immuneepitope.org/main/html/tcell_tools.html) (Stranzl et al., 2010, Immunogenetics; 62: 357-368; Wang et al., 2008, PLoS Comput Biol. 4 (4): e1000048). Этот инструмент использует различные способы прогнозирования эпитопов MHC класса II, в том числе консенсусный подход, который сочетает в себе NN-выравнивание, SMM-выравнивание и способы комбинаторных библиотек. Низкий процентильный ранг равен хорошим связывающим агентам.
Предложенные аллели принадлежали к супертипам HLA класса I и II (Wang et al., 2010, BMC Bioinformatics. 11: 568; Sidney et al., 2008, BMC Immunol .; 9: 1. Doi: 10,1186 / 1471-2172-9-1). Молекулы MHC чрезвычайно полиморфны, но, несмотря на этот полиморфизм, молекулы HLA класса I могут быть сгруппированы в группы, обозначенные как супертипы, представляющие наборы молекул, которые имеют в значительной степени перекрывающуюся специфичность связывания пептидов (Wang et al., 2010 BMC Bioinformatics. 11: 568). Таким же образом для HLA класса II определяют семь различных супертипов (основной DR, DR4, DRB3, основной DQ, DQ7, основной DP и DP2) (Greenbaum et al., 2011, Immunogenetics .; 63 (6): 325- 35).
Последовательность пептида hZP3 была представлена без сигнального пептида и трансмембранного домена, то есть с 23 по 387 аминокислоту. Результаты анализа содержатся в таблицах 2 и 5 (лиганды MHC класса I) и в таблице 3 (лиганды MHC класса II).
Лиганды MHC классов I и II, представленные в таблице 2 и 3 соответственно, служат руководством для разработки иммуногенных пептидов по изобретению, например, композиции, содержащей один или несколько полипептидов, где указаные один или несколько полипептидов включают один или несколько эпитопов, рестрицированных по MHC класса I и выбранных из таблицы 2, и/или эпитопов, рестрицированных по MHC класса II и выбранных из таблицы 3.
Серии пептидов hZP3, охватывающих последовательности hZP3, для применения в клинических условиях представлены на фигуре 4. Прогнозируемые последовательности, связывающие МНС класса I и II из таблиц 2 и 3, а также возможные участки гликозилирования были учтены для определения пептидов, каждый из который содержит ряд предсказанных связывающих последовательностей для МНС класса I и II с низким процентильным рангом для индукции хорошего иммунного ответа. Эти пептидные фрагменты hZP3 включают hZP330-78 (SEQ ID NO: 66), hZP376-101 (SEQ ID NO: 67), hZP399-141 (SEQ ID NO: 68), hZP3138-187 (SEQ ID NO: 69), hZP3185-225 (SEQ ID NO: 70), hZP3224-267 (SEQ ID NO: 71), hZP3257-290 (SEQ ID NO: 72), hZP3298-317 (SEQ ID NO: 73), hZP3331-357 (SEQ ID NO: 74) и hZP3358-383 (SEQ ID NO: 75). Комбинации этих пептидных фрагментов для составления композиции, содержащей комбинации, по меньшей мере, двух пептидных фрагментов, перечислены в таблице 4.
Таблица 2: Предсказанные лиганды MHC класса I, содержащиеся в белке hZP3.
¶Лиганд содержит N-связанный участок гликозилирования (положение 125, 147 и 272, соответственно).
Таблица 3: Предсказанные лиганды MHC класса II, содержащиеся в белке hZP3
§Процентильный ранг приведен в скобках после соответствующего аллеля HLA; рассматриваются только лиганды с процентильными рангами > 2,5.
‡N-связанный участок гликозилирования в предшествующем положении 125.
¶Лиганд содержит N-связанный участок гликозилирования (положение 272).
Пример 4
С программным обеспечением для прогнозирования in silico были идентифицированы потенциальные Т-клеточные эпитопы, рестрицированные по HLA-A2, из белка rhZP3. Программное обеспечение представляло собой программное обеспечение для прогнозирования in silico NETMHCPAN 3.0.
Таблица 5: Т-клеточные эпитопы из белка rhZP3, рестрицированные по HLA-A2.
Было проведено доклиническое исследование эффективности in vivo оценки иммунного ответа и противоопухолевого ответа после иммунизации полипептидом rhZP3 (1-383 с his-меткой) и пятью различными эпитопами ZP3. Эпитопы hZP3 выбирали на основе ответов CD8+ T-клеток, наблюдаемых в ходе ранее выполненных иммуногенных экспериментов с полипептидом rhZP3 (а/к 1-383) и с совокупностью пептидов, содержащих эпитопы hZP3, как указано (см. таблицу 5), в сочетании с различными адъювантами (данные не показаны). Пептиды синтезировали в Pepscan (Нидерланды) с чистотой >96%.
В этом эксперименте в терапевтических условиях трансгенных мыейи HLA-A2 с опухолью, экспрессирующей ZP3, обрабатывали белком rhZP3, совокупностью пептидов hZP3 или PBS в качестве контроля.
Для этого исследования использовали модель трансгенных мышей HLA-A2. Чтобы создать модель опухоли, клеточную линию меланомы трансгенных мышей B16-HLA-A2 трансфицировали rhZP3 (см. пример в Tran et al., Clin. Cancer Res, 2016; 22 (16): 4133-44).
Три группы самок трансгенных мышей HLA-A2 (n=6) с опухолями, экспрессирующими ZP3, обрабатывали:
1. PBS;
2. Совокупность пяти пептидов hZP3 (5 доминантных пептидов, ранее идентифицированных у HLA-A2/DR1 мышей, включая эпитопы, идентифицированные как 4, 5, 6, 8 и 9 в таблице 5; 0,5 нмоль на пептид) плюс GM-CSF/CpG в качестве адъюванта (20 мкг GM-CSF и 50 мкг CpG); и,
3. белок rhZP3 (100 мкг, подкожно) плюс GM-CSF/CpG в качестве адъюванта (20 мкг GM-CSF и 50 мкг CpG).
На сутки 0 (D0) была сделана инъекция опухоли 106 клеток/мышь (подкожно). После этого примирующую инъекцию вводили в интервале с 7 до 10 суток (D7-10) и стимулирующую инъекцию на сутки от 14 до 17 (D14-17). Размер опухоли контролировали после лечения. На сутки с 30 до 35 мышей умерщвляли, чтобы определить ZP3-специфический ответ TCD8+ (ELISpot). См. график на фигуре 5.
После введения PBS размер опухоли увеличился, тогда как через две подкожных иммунизации либо белком rhZP3, либо комбинацией из пяти пептидов hZP3 роста опухоли не наблюдали (см. фигуру 6).
Кроме того, как показано на фигуре 7, измерили сильный CD8+ Т-клеточный ответ (IFNg) против пептидов после иммунизации пятью пептидами hZP3. Также после иммунизации белком ZP3 наблюдали ответ TCD8 (см. фигуру 7).
Таблица 4. Комбинации аминокислотных последовательности из фигуры 4, которые должны быть включены в иммуногенные пептиды для составления композиции из синтетических длинных пептидов в соответствии с изобретением. Различные комбинации разделены точкой с запятой.
Комбинация из 2 пептидов
SEQ ID NO. 66, SEQ ID NO. 67; SEQ ID NO. 66, SEQ ID NO. 68; SEQ ID NO. 66, SEQ ID NO. 69; SEQ ID NO. 66, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68; SEQ ID NO. 67, SEQ ID NO. 69; SEQ ID NO. 67, SEQ ID NO. 70; SEQ ID NO. 67, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69; SEQ ID NO. 68, SEQ ID NO. 70; SEQ ID NO. 68, SEQ ID NO. 71; SEQ ID NO. 68, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 3 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 4 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 5 пептидов
SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 6 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 7 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 8 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75;
Комбинация из 9 пептидов
SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 67, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 66, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75; SEQ ID NO. 67, SEQ ID NO. 68, SEQ ID NO. 69, SEQ ID NO. 70, SEQ ID NO. 71, SEQ ID NO. 72, SEQ ID NO. 73, SEQ ID NO. 74, SEQ ID NO. 75.
Таблица 6. Комбинации аминокислотных последовательностей из Таблицы 5, которые должны быть включены в иммуногенные пептиды для составления композиции синтетических длинных пептидов в соответствии с изобретением. Различные комбинации разделены точкой с запятой.
Комбинация из 2 пептидов
SEQ ID NO. 76, SEQ ID NO. 77; SEQ ID NO. 76, SEQ ID NO. 78; SEQ ID NO. 76, SEQ ID NO. 79; SEQ ID NO. 76, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78; SEQ ID NO. 77, SEQ ID NO. 79; SEQ ID NO. 77, SEQ ID NO. 80; SEQ ID NO. 77, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79; SEQ ID NO. 78, SEQ ID NO. 80; SEQ ID NO. 78, SEQ ID NO. 81; SEQ ID NO. 78, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 3 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 4 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 5 пептидов
SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 6 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 7 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 8 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85;
Комбинация из 9 пептидов
SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 77, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 76, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85; SEQ ID NO. 77, SEQ ID NO. 78, SEQ ID NO. 79, SEQ ID NO. 80, SEQ ID NO. 81, SEQ ID NO. 82, SEQ ID NO. 83, SEQ ID NO. 84, SEQ ID NO. 85.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Pantarhei Bioscience B.V.
<120> Иммунотерапевтические способы лечения и/или профилактики рака легких
<130> P6066722PCT
<150> EP17199441.1
<151> 2017-10-31
<160> 85
<170> PatentIn version 3.5
<210> 1
<211> 638
<212> Белок
<213> Homo sapiens
<400> 1
Met Ala Gly Gly Ser Ala Thr Thr Trp Gly Tyr Pro Val Ala Leu Leu
1 5 10 15
Leu Leu Val Ala Thr Leu Gly Leu Gly Arg Trp Leu Gln Pro Asp Pro
20 25 30
Gly Leu Pro Gly Leu Arg His Ser Tyr Asp Cys Gly Ile Lys Gly Met
35 40 45
Gln Leu Leu Val Phe Pro Arg Pro Gly Gln Thr Leu Arg Phe Lys Val
50 55 60
Val Asp Glu Phe Gly Asn Arg Phe Asp Val Asn Asn Cys Ser Ile Cys
65 70 75 80
Tyr His Trp Val Thr Ser Arg Pro Gln Glu Pro Ala Val Phe Ser Ala
85 90 95
Asp Tyr Arg Gly Cys His Val Leu Glu Lys Asp Gly Arg Phe His Leu
100 105 110
Arg Val Phe Met Glu Ala Val Leu Pro Asn Gly Arg Val Asp Val Ala
115 120 125
Gln Asp Ala Thr Leu Ile Cys Pro Lys Pro Asp Pro Ser Arg Thr Leu
130 135 140
Asp Ser Gln Leu Ala Pro Pro Ala Met Phe Ser Val Ser Thr Pro Gln
145 150 155 160
Thr Leu Ser Phe Leu Pro Thr Ser Gly His Thr Ser Gln Gly Ser Gly
165 170 175
His Ala Phe Pro Ser Pro Leu Asp Pro Gly His Ser Ser Val His Pro
180 185 190
Thr Pro Ala Leu Pro Ser Pro Gly Pro Gly Pro Thr Leu Ala Thr Leu
195 200 205
Ala Gln Pro His Trp Gly Thr Leu Glu His Trp Asp Val Asn Lys Arg
210 215 220
Asp Tyr Ile Gly Thr His Leu Ser Gln Glu Gln Cys Gln Val Ala Ser
225 230 235 240
Gly His Leu Pro Cys Ile Val Arg Arg Thr Ser Lys Glu Ala Cys Gln
245 250 255
Gln Ala Gly Cys Cys Tyr Asp Asn Thr Arg Glu Val Pro Cys Tyr Tyr
260 265 270
Gly Asn Thr Ala Thr Val Gln Cys Phe Arg Asp Gly Tyr Phe Val Leu
275 280 285
Val Val Ser Gln Glu Met Ala Leu Thr His Arg Ile Thr Leu Ala Asn
290 295 300
Ile His Leu Ala Tyr Ala Pro Thr Ser Cys Ser Pro Thr Gln His Thr
305 310 315 320
Glu Ala Phe Val Val Phe Tyr Phe Pro Leu Thr His Cys Gly Thr Thr
325 330 335
Met Gln Val Ala Gly Asp Gln Leu Ile Tyr Glu Asn Trp Leu Val Ser
340 345 350
Gly Ile His Ile Gln Lys Gly Pro Gln Gly Ser Ile Thr Arg Asp Ser
355 360 365
Thr Phe Gln Leu His Val Arg Cys Val Phe Asn Ala Ser Asp Phe Leu
370 375 380
Pro Ile Gln Ala Ser Ile Phe Pro Pro Pro Ser Pro Ala Pro Met Thr
385 390 395 400
Gln Pro Gly Pro Leu Arg Leu Glu Leu Arg Ile Ala Lys Asp Glu Thr
405 410 415
Phe Ser Ser Tyr Tyr Gly Glu Asp Asp Tyr Pro Ile Val Arg Leu Leu
420 425 430
Arg Glu Pro Val His Val Glu Val Arg Leu Leu Gln Arg Thr Asp Pro
435 440 445
Asn Leu Val Leu Leu Leu His Gln Cys Trp Gly Ala Pro Ser Ala Asn
450 455 460
Pro Phe Gln Gln Pro Gln Trp Pro Ile Leu Ser Asp Gly Cys Pro Phe
465 470 475 480
Lys Gly Asp Ser Tyr Arg Thr Gln Met Val Ala Leu Asp Gly Ala Thr
485 490 495
Pro Phe Gln Ser His Tyr Gln Arg Phe Thr Val Ala Thr Phe Ala Leu
500 505 510
Leu Asp Ser Gly Ser Gln Arg Ala Leu Arg Gly Leu Val Tyr Leu Phe
515 520 525
Cys Ser Thr Ser Ala Cys His Thr Ser Gly Leu Glu Thr Cys Ser Thr
530 535 540
Ala Cys Ser Thr Gly Thr Thr Arg Gln Arg Arg Ser Ser Gly His Arg
545 550 555 560
Asn Asp Thr Ala Arg Pro Gln Asp Ile Val Ser Ser Pro Gly Pro Val
565 570 575
Gly Phe Glu Asp Ser Tyr Gly Gln Glu Pro Thr Leu Gly Pro Thr Asp
580 585 590
Ser Asn Gly Asn Ser Ser Leu Arg Pro Leu Leu Trp Ala Val Leu Leu
595 600 605
Leu Pro Ala Val Ala Leu Val Leu Gly Phe Gly Val Phe Val Gly Leu
610 615 620
Ser Gln Thr Trp Ala Gln Lys Leu Trp Glu Ser Asn Arg Gln
625 630 635
<210> 2
<211> 745
<212> Белок
<213> Homo sapiens
<400> 2
Met Ala Cys Arg Gln Arg Gly Gly Ser Trp Ser Pro Ser Gly Trp Phe
1 5 10 15
Asn Ala Gly Trp Ser Thr Tyr Arg Ser Ile Ser Leu Phe Phe Ala Leu
20 25 30
Val Thr Ser Gly Asn Ser Ile Asp Val Ser Gln Leu Val Asn Pro Ala
35 40 45
Phe Pro Gly Thr Val Thr Cys Asp Glu Arg Glu Ile Thr Val Glu Phe
50 55 60
Pro Ser Ser Pro Gly Thr Lys Lys Trp His Ala Ser Val Val Asp Pro
65 70 75 80
Leu Gly Leu Asp Met Pro Asn Cys Thr Tyr Ile Leu Asp Pro Glu Lys
85 90 95
Leu Thr Leu Arg Ala Thr Tyr Asp Asn Cys Thr Arg Arg Val His Gly
100 105 110
Gly His Gln Met Thr Ile Arg Val Met Asn Asn Ser Ala Ala Leu Arg
115 120 125
His Gly Ala Val Met Tyr Gln Phe Phe Cys Pro Ala Met Gln Val Glu
130 135 140
Glu Thr Gln Gly Leu Ser Ala Ser Thr Ile Cys Gln Lys Asp Phe Met
145 150 155 160
Ser Phe Ser Leu Pro Arg Val Phe Ser Gly Leu Ala Asp Asp Ser Lys
165 170 175
Gly Thr Lys Val Gln Met Gly Trp Ser Ile Glu Val Gly Asp Gly Ala
180 185 190
Arg Ala Lys Thr Leu Thr Leu Pro Glu Ala Met Lys Glu Gly Phe Ser
195 200 205
Leu Leu Ile Asp Asn His Arg Met Thr Phe His Val Pro Phe Asn Ala
210 215 220
Thr Gly Val Thr His Tyr Val Gln Gly Asn Ser His Leu Tyr Met Val
225 230 235 240
Ser Leu Lys Leu Thr Phe Ile Ser Pro Gly Gln Lys Val Ile Phe Ser
245 250 255
Ser Gln Ala Ile Cys Ala Pro Asp Pro Val Thr Cys Asn Ala Thr His
260 265 270
Met Thr Leu Thr Ile Pro Glu Phe Pro Gly Lys Leu Lys Ser Val Ser
275 280 285
Phe Glu Asn Gln Asn Ile Asp Val Ser Gln Leu His Asp Asn Gly Ile
290 295 300
Asp Leu Glu Ala Thr Asn Gly Met Lys Leu His Phe Ser Lys Thr Leu
305 310 315 320
Leu Lys Thr Lys Leu Ser Glu Lys Cys Leu Leu His Gln Phe Tyr Leu
325 330 335
Ala Ser Leu Lys Leu Thr Phe Leu Leu Arg Pro Glu Thr Val Ser Met
340 345 350
Val Ile Tyr Pro Glu Cys Leu Cys Glu Ser Pro Val Ser Ile Val Thr
355 360 365
Gly Glu Leu Cys Thr Gln Asp Gly Phe Met Asp Val Glu Val Tyr Ser
370 375 380
Tyr Gln Thr Gln Pro Ala Leu Asp Leu Gly Thr Leu Arg Val Gly Asn
385 390 395 400
Ser Ser Cys Gln Pro Val Phe Glu Ala Gln Ser Gln Gly Leu Val Arg
405 410 415
Phe His Ile Pro Leu Asn Gly Cys Gly Thr Arg Tyr Lys Phe Glu Asp
420 425 430
Asp Lys Val Val Tyr Glu Asn Glu Ile His Ala Leu Trp Thr Asp Phe
435 440 445
Pro Pro Ser Lys Ile Ser Arg Asp Ser Glu Phe Arg Met Thr Val Lys
450 455 460
Cys Ser Tyr Ser Arg Asn Asp Met Leu Leu Asn Ile Asn Val Glu Ser
465 470 475 480
Leu Thr Pro Pro Val Ala Ser Val Lys Leu Gly Pro Phe Thr Leu Ile
485 490 495
Leu Gln Ser Tyr Pro Asp Asn Ser Tyr Gln Gln Pro Tyr Gly Glu Asn
500 505 510
Glu Tyr Pro Leu Val Arg Phe Leu Arg Gln Pro Ile Tyr Met Glu Val
515 520 525
Arg Val Leu Asn Arg Asp Asp Pro Asn Ile Lys Leu Val Leu Asp Asp
530 535 540
Cys Trp Ala Thr Ser Thr Met Asp Pro Asp Ser Phe Pro Gln Trp Asn
545 550 555 560
Val Val Val Asp Gly Cys Ala Tyr Asp Leu Asp Asn Tyr Gln Thr Thr
565 570 575
Phe His Pro Val Gly Ser Ser Val Thr His Pro Asp His Tyr Gln Arg
580 585 590
Phe Asp Met Lys Ala Phe Ala Phe Val Ser Glu Ala His Val Leu Ser
595 600 605
Ser Leu Val Tyr Phe His Cys Ser Ala Leu Ile Cys Asn Arg Leu Ser
610 615 620
Pro Asp Ser Pro Leu Cys Ser Val Thr Cys Pro Val Ser Ser Arg His
625 630 635 640
Arg Arg Ala Thr Gly Ala Thr Glu Ala Glu Lys Met Thr Val Ser Leu
645 650 655
Pro Gly Pro Ile Leu Leu Leu Ser Asp Asp Ser Ser Phe Arg Gly Val
660 665 670
Gly Ser Ser Asp Leu Lys Ala Ser Gly Ser Ser Gly Glu Lys Ser Arg
675 680 685
Ser Glu Thr Gly Glu Glu Val Gly Ser Arg Gly Ala Met Asp Thr Lys
690 695 700
Gly His Lys Thr Ala Gly Asp Val Gly Ser Lys Ala Val Ala Ala Val
705 710 715 720
Ala Ala Phe Ala Gly Val Val Ala Thr Leu Gly Phe Ile Tyr Tyr Leu
725 730 735
Tyr Glu Lys Arg Thr Val Ser Asn His
740 745
<210> 3
<211> 424
<212> Белок
<213> Homo sapiens
<400> 3
Met Glu Leu Ser Tyr Arg Leu Phe Ile Cys Leu Leu Leu Trp Gly Ser
1 5 10 15
Thr Glu Leu Cys Tyr Pro Gln Pro Leu Trp Leu Leu Gln Gly Gly Ala
20 25 30
Ser His Pro Glu Thr Ser Val Gln Pro Val Leu Val Glu Cys Gln Glu
35 40 45
Ala Thr Leu Met Val Met Val Ser Lys Asp Leu Phe Gly Thr Gly Lys
50 55 60
Leu Ile Arg Ala Ala Asp Leu Thr Leu Gly Pro Glu Ala Cys Glu Pro
65 70 75 80
Leu Val Ser Met Asp Thr Glu Asp Val Val Arg Phe Glu Val Gly Leu
85 90 95
His Glu Cys Gly Asn Ser Met Gln Val Thr Asp Asp Ala Leu Val Tyr
100 105 110
Ser Thr Phe Leu Leu His Asp Pro Arg Pro Val Gly Asn Leu Ser Ile
115 120 125
Val Arg Thr Asn Arg Ala Glu Ile Pro Ile Glu Cys Arg Tyr Pro Arg
130 135 140
Gln Gly Asn Val Ser Ser Gln Ala Ile Leu Pro Thr Trp Leu Pro Phe
145 150 155 160
Arg Thr Thr Val Phe Ser Glu Glu Lys Leu Thr Phe Ser Leu Arg Leu
165 170 175
Met Glu Glu Asn Trp Asn Ala Glu Lys Arg Ser Pro Thr Phe His Leu
180 185 190
Gly Asp Ala Ala His Leu Gln Ala Glu Ile His Thr Gly Ser His Val
195 200 205
Pro Leu Arg Leu Phe Val Asp His Cys Val Ala Thr Pro Thr Pro Asp
210 215 220
Gln Asn Ala Ser Pro Tyr His Thr Ile Val Asp Phe His Gly Cys Leu
225 230 235 240
Val Asp Gly Leu Thr Asp Ala Ser Ser Ala Phe Lys Val Pro Arg Pro
245 250 255
Gly Pro Asp Thr Leu Gln Phe Thr Val Asp Val Phe His Phe Ala Asn
260 265 270
Asp Ser Arg Asn Met Ile Tyr Ile Thr Cys His Leu Lys Val Thr Leu
275 280 285
Ala Glu Gln Asp Pro Asp Glu Leu Asn Lys Ala Cys Ser Phe Ser Lys
290 295 300
Pro Ser Asn Ser Trp Phe Pro Val Glu Gly Ser Ala Asp Ile Cys Gln
305 310 315 320
Cys Cys Asn Lys Gly Asp Cys Gly Thr Pro Ser His Ser Arg Arg Gln
325 330 335
Pro His Val Met Ser Gln Trp Ser Arg Ser Ala Ser Arg Asn Arg Arg
340 345 350
His Val Thr Glu Glu Ala Asp Val Thr Val Gly Pro Leu Ile Phe Leu
355 360 365
Asp Arg Arg Gly Asp His Glu Val Glu Gln Trp Ala Leu Pro Ser Asp
370 375 380
Thr Ser Val Val Leu Leu Gly Val Gly Leu Ala Val Val Val Ser Leu
385 390 395 400
Thr Leu Thr Ala Val Ile Leu Val Leu Thr Arg Arg Cys Arg Thr Ala
405 410 415
Ser His Pro Val Ser Ala Ser Glu
420
<210> 4
<211> 540
<212> Белок
<213> Homo sapiens
<400> 4
Met Trp Leu Leu Arg Cys Val Leu Leu Cys Val Ser Leu Ser Leu Ala
1 5 10 15
Val Ser Gly Gln His Lys Pro Glu Ala Pro Asp Tyr Ser Ser Val Leu
20 25 30
His Cys Gly Pro Trp Ser Phe Gln Phe Ala Val Asn Leu Asn Gln Glu
35 40 45
Ala Thr Ser Pro Pro Val Leu Ile Ala Trp Asp Asn Gln Gly Leu Leu
50 55 60
His Glu Leu Gln Asn Asp Ser Asp Cys Gly Thr Trp Ile Arg Lys Gly
65 70 75 80
Pro Gly Ser Ser Val Val Leu Glu Ala Thr Tyr Ser Ser Cys Tyr Val
85 90 95
Thr Glu Trp Asp Ser His Tyr Ile Met Pro Val Gly Val Glu Gly Ala
100 105 110
Gly Ala Ala Glu His Lys Val Val Thr Glu Arg Lys Leu Leu Lys Cys
115 120 125
Pro Met Asp Leu Leu Ala Arg Asp Ala Pro Asp Thr Asp Trp Cys Asp
130 135 140
Ser Ile Pro Ala Arg Asp Arg Leu Pro Cys Ala Pro Ser Pro Ile Ser
145 150 155 160
Arg Gly Asp Cys Glu Gly Leu Gly Cys Cys Tyr Ser Ser Glu Glu Val
165 170 175
Asn Ser Cys Tyr Tyr Gly Asn Thr Val Thr Leu His Cys Thr Arg Glu
180 185 190
Gly His Phe Ser Ile Ala Val Ser Arg Asn Val Thr Ser Pro Pro Leu
195 200 205
Leu Leu Asp Ser Val Arg Leu Ala Leu Arg Asn Asp Ser Ala Cys Asn
210 215 220
Pro Val Met Ala Thr Gln Ala Phe Val Leu Phe Gln Phe Pro Phe Thr
225 230 235 240
Ser Cys Gly Thr Thr Arg Gln Ile Thr Gly Asp Arg Ala Val Tyr Glu
245 250 255
Asn Glu Leu Val Ala Thr Arg Asp Val Lys Asn Gly Ser Arg Gly Ser
260 265 270
Val Thr Arg Asp Ser Ile Phe Arg Leu His Val Ser Cys Ser Tyr Ser
275 280 285
Val Ser Ser Asn Ser Leu Pro Ile Asn Val Gln Val Phe Thr Leu Pro
290 295 300
Pro Pro Phe Pro Glu Thr Gln Pro Gly Pro Leu Thr Leu Glu Leu Gln
305 310 315 320
Ile Ala Lys Asp Lys Asn Tyr Gly Ser Tyr Tyr Gly Val Gly Asp Tyr
325 330 335
Pro Val Val Lys Leu Leu Arg Asp Pro Ile Tyr Val Glu Val Ser Ile
340 345 350
Leu His Arg Thr Asp Pro Tyr Leu Gly Leu Leu Leu Gln Gln Cys Trp
355 360 365
Ala Thr Pro Ser Thr Asp Pro Leu Ser Gln Pro Gln Trp Pro Ile Leu
370 375 380
Val Lys Gly Cys Pro Tyr Ile Gly Asp Asn Tyr Gln Thr Gln Leu Ile
385 390 395 400
Pro Val Gln Lys Ala Leu Asp Leu Pro Phe Pro Ser His His Gln Arg
405 410 415
Phe Ser Ile Phe Thr Phe Ser Phe Val Asn Pro Thr Val Glu Lys Gln
420 425 430
Ala Leu Arg Gly Pro Val His Leu His Cys Ser Val Ser Val Cys Gln
435 440 445
Pro Ala Glu Thr Pro Ser Cys Val Val Thr Cys Pro Asp Leu Ser Arg
450 455 460
Arg Arg Asn Phe Asp Asn Ser Ser Gln Asn Thr Thr Ala Ser Val Ser
465 470 475 480
Ser Lys Gly Pro Met Ile Leu Leu Gln Ala Thr Lys Asp Pro Pro Glu
485 490 495
Lys Leu Arg Val Pro Val Asp Ser Lys Val Leu Trp Val Ala Gly Leu
500 505 510
Ser Gly Thr Leu Ile Leu Gly Ala Leu Leu Val Ser Tyr Leu Ala Val
515 520 525
Lys Lys Gln Lys Ser Cys Pro Asp Gln Met Cys Gln
530 535 540
<210> 5
<211> 328
<212> Белок
<213> Homo sapiens
<400> 5
Gln Pro Leu Trp Leu Leu Gln Gly Gly Ala Ser His Pro Glu Thr Ser
1 5 10 15
Val Gln Pro Val Leu Val Glu Cys Gln Glu Ala Thr Leu Met Val Met
20 25 30
Val Ser Lys Asp Leu Phe Gly Thr Gly Lys Leu Ile Arg Ala Ala Asp
35 40 45
Leu Thr Leu Gly Pro Glu Ala Cys Glu Pro Leu Val Ser Met Asp Thr
50 55 60
Glu Asp Val Val Arg Phe Glu Val Gly Leu His Glu Cys Gly Asn Ser
65 70 75 80
Met Gln Val Thr Asp Asp Ala Leu Val Tyr Ser Thr Phe Leu Leu His
85 90 95
Asp Pro Arg Pro Val Gly Asn Leu Ser Ile Val Arg Thr Asn Arg Ala
100 105 110
Glu Ile Pro Ile Glu Cys Arg Tyr Pro Arg Gln Gly Asn Val Ser Ser
115 120 125
Gln Ala Ile Leu Pro Thr Trp Leu Pro Phe Arg Thr Thr Val Phe Ser
130 135 140
Glu Glu Lys Leu Thr Phe Ser Leu Arg Leu Met Glu Glu Asn Trp Asn
145 150 155 160
Ala Glu Lys Arg Ser Pro Thr Phe His Leu Gly Asp Ala Ala His Leu
165 170 175
Gln Ala Glu Ile His Thr Gly Ser His Val Pro Leu Arg Leu Phe Val
180 185 190
Asp His Cys Val Ala Thr Pro Thr Pro Asp Gln Asn Ala Ser Pro Tyr
195 200 205
His Thr Ile Val Asp Phe His Gly Cys Leu Val Asp Gly Leu Thr Asp
210 215 220
Ala Ser Ser Ala Phe Lys Val Pro Arg Pro Gly Pro Asp Thr Leu Gln
225 230 235 240
Phe Thr Val Asp Val Phe His Phe Ala Asn Asp Ser Arg Asn Met Ile
245 250 255
Tyr Ile Thr Cys His Leu Lys Val Thr Leu Ala Glu Gln Asp Pro Asp
260 265 270
Glu Leu Asn Lys Ala Cys Ser Phe Ser Lys Pro Ser Asn Ser Trp Phe
275 280 285
Pro Val Glu Gly Ser Ala Asp Ile Cys Gln Cys Cys Asn Lys Gly Asp
290 295 300
Cys Gly Thr Pro Ser His Ser Arg Arg Gln Pro His Val Met Ser Gln
305 310 315 320
Trp Ser Arg Ser Ala Ser Arg Asn
325
<210> 6
<211> 9
<212> Белок
<213> Homo sapiens
<400> 6
His Pro Glu Thr Ser Val Gln Pro Val
1 5
<210> 7
<211> 9
<212> Белок
<213> Homo sapiens
<400> 7
Pro Glu Thr Ser Val Gln Pro Val Leu
1 5
<210> 8
<211> 9
<212> Белок
<213> Homo sapiens
<400> 8
Gln Glu Ala Thr Leu Met Val Met Val
1 5
<210> 9
<211> 9
<212> Белок
<213> Homo sapiens
<400> 9
Ala Thr Leu Met Val Met Val Ser Lys
1 5
<210> 10
<211> 9
<212> Белок
<213> Homo sapiens
<400> 10
Leu Ile Arg Ala Ala Asp Leu Thr Leu
1 5
<210> 11
<211> 9
<212> Белок
<213> Homo sapiens
<400> 11
Thr Glu Asp Val Val Arg Phe Glu Val
1 5
<210> 12
<211> 9
<212> Белок
<213> Homo sapiens
<400> 12
Gln Val Thr Asp Asp Ala Leu Val Tyr
1 5
<210> 13
<211> 9
<212> Белок
<213> Homo sapiens
<400> 13
Ala Leu Val Tyr Ser Thr Phe Leu Leu
1 5
<210> 14
<211> 9
<212> Белок
<213> Homo sapiens
<400> 14
Phe Leu Leu His Asp Pro Arg Pro Val
1 5
<210> 15
<211> 9
<212> Белок
<213> Homo sapiens
<400> 15
Arg Pro Val Gly Asn Leu Ser Ile Val
1 5
<210> 16
<211> 9
<212> Белок
<213> Homo sapiens
<400> 16
Tyr Pro Arg Gln Gly Asn Val Ser Ser
1 5
<210> 17
<211> 9
<212> Белок
<213> Homo sapiens
<400> 17
Ser Ser Gln Ala Ile Leu Pro Thr Trp
1 5
<210> 18
<211> 9
<212> Белок
<213> Homo sapiens
<400> 18
Ala Ile Leu Pro Thr Trp Leu Pro Phe
1 5
<210> 19
<211> 9
<212> Белок
<213> Homo sapiens
<400> 19
Trp Leu Pro Phe Arg Thr Thr Val Phe
1 5
<210> 20
<211> 9
<212> Белок
<213> Homo sapiens
<400> 20
Val Phe Ser Glu Glu Lys Leu Thr Phe
1 5
<210> 21
<211> 9
<212> Белок
<213> Homo sapiens
<400> 21
Ser Glu Glu Lys Leu Thr Phe Ser Leu
1 5
<210> 22
<211> 9
<212> Белок
<213> Homo sapiens
<400> 22
Arg Leu Met Glu Glu Asn Trp Asn Ala
1 5
<210> 23
<211> 9
<212> Белок
<213> Homo sapiens
<400> 23
Phe His Leu Gly Asp Ala Ala His Leu
1 5
<210> 24
<211> 9
<212> Белок
<213> Homo sapiens
<400> 24
Ile His Thr Gly Ser His Val Pro Leu
1 5
<210> 25
<211> 9
<212> Белок
<213> Homo sapiens
<400> 25
Ser Pro Tyr His Thr Ile Val Asp Phe
1 5
<210> 26
<211> 9
<212> Белок
<213> Homo sapiens
<400> 26
Gly Leu Thr Asp Ala Ser Ser Ala Phe
1 5
<210> 27
<211> 9
<212> Белок
<213> Homo sapiens
<400> 27
Leu Thr Asp Ala Ser Ser Ala Phe Lys
1 5
<210> 28
<211> 9
<212> Белок
<213> Homo sapiens
<400> 28
Val Pro Arg Pro Gly Pro Asp Thr Leu
1 5
<210> 29
<211> 9
<212> Белок
<213> Homo sapiens
<400> 29
Arg Pro Gly Pro Asp Thr Leu Gln Phe
1 5
<210> 30
<211> 9
<212> Белок
<213> Homo sapiens
<400> 30
Gln Phe Thr Val Asp Val Phe His Phe
1 5
<210> 31
<211> 9
<212> Белок
<213> Homo sapiens
<400> 31
Phe Thr Val Asp Val Phe His Phe Ala
1 5
<210> 32
<211> 9
<212> Белок
<213> Homo sapiens
<400> 32
Ala Asn Asp Ser Arg Asn Met Ile Tyr
1 5
<210> 33
<211> 9
<212> Белок
<213> Homo sapiens
<400> 33
Met Ile Tyr Ile Thr Cys His Leu Lys
1 5
<210> 34
<211> 9
<212> Белок
<213> Homo sapiens
<400> 34
Ile Tyr Ile Thr Cys His Leu Lys Val
1 5
<210> 35
<211> 9
<212> Белок
<213> Homo sapiens
<400> 35
Phe Ser Lys Pro Ser Asn Ser Trp Phe
1 5
<210> 36
<211> 9
<212> Белок
<213> Homo sapiens
<400> 36
Lys Pro Ser Asn Ser Trp Phe Pro Val
1 5
<210> 37
<211> 9
<212> Белок
<213> Homo sapiens
<400> 37
Arg Gln Pro His Val Met Ser Gln Trp
1 5
<210> 38
<211> 9
<212> Белок
<213> Homo sapiens
<400> 38
Arg Gly Asp His Glu Val Glu Gln Trp
1 5
<210> 39
<211> 9
<212> Белок
<213> Homo sapiens
<400> 39
Glu Ala Thr Leu Met Val Met Val Ser
1 5
<210> 40
<211> 9
<212> Белок
<213> Homo sapiens
<400> 40
Leu Met Val Met Val Ser Lys Asp Leu
1 5
<210> 41
<211> 9
<212> Белок
<213> Homo sapiens
<400> 41
Met Val Met Val Ser Lys Asp Leu Phe
1 5
<210> 42
<211> 9
<212> Белок
<213> Homo sapiens
<400> 42
Leu Ile Arg Ala Ala Asp Leu Thr Leu
1 5
<210> 43
<211> 9
<212> Белок
<213> Homo sapiens
<400> 43
Leu Val Met Ser Asp Thr Glu Asp Val
1 5
<210> 44
<211> 9
<212> Белок
<213> Homo sapiens
<400> 44
Val Ser Met Asp Thr Glu Asp Val Val
1 5
<210> 45
<211> 9
<212> Белок
<213> Homo sapiens
<400> 45
Val Val Arg Phe Glu Val Gly Leu His
1 5
<210> 46
<211> 9
<212> Белок
<213> Homo sapiens
<400> 46
Gln Val Thr Asp Asp Ala Leu Val Tyr
1 5
<210> 47
<211> 9
<212> Белок
<213> Homo sapiens
<400> 47
Ala Leu Val Tyr Ser Thr Phe Leu Leu
1 5
<210> 48
<211> 9
<212> Белок
<213> Homo sapiens
<400> 48
Leu Val Tyr Ser Thr Phe Leu Leu His
1 5
<210> 49
<211> 9
<212> Белок
<213> Homo sapiens
<400> 49
Phe Leu Leu Asp Pro His Arg Pro Val
1 5
<210> 50
<211> 9
<212> Белок
<213> Homo sapiens
<400> 50
Leu Leu His Asp Pro Arg Pro Val Gly
1 5
<210> 51
<211> 9
<212> Белок
<213> Homo sapiens
<400> 51
Leu Ser Ile Val Arg Thr Asn Arg Ala
1 5
<210> 52
<211> 9
<212> Белок
<213> Homo sapiens
<400> 52
Ile Val Arg Thr Asn Arg Ala Glu Ile
1 5
<210> 53
<211> 9
<212> Белок
<213> Homo sapiens
<400> 53
Pro Phe Arg Thr Thr Val Phe Ser Glu
1 5
<210> 54
<211> 9
<212> Белок
<213> Homo sapiens
<400> 54
Phe Arg Thr Thr Val Phe Ser Glu Glu
1 5
<210> 55
<211> 9
<212> Белок
<213> Homo sapiens
<400> 55
Thr Phe Ser Leu Arg Leu Met Glu Glu
1 5
<210> 56
<211> 9
<212> Белок
<213> Homo sapiens
<400> 56
Phe His Leu Gly Asp Ala Ala His Leu
1 5
<210> 57
<211> 9
<212> Белок
<213> Homo sapiens
<400> 57
Leu Arg Leu Phe Val Asp His Cys Val
1 5
<210> 58
<211> 9
<212> Белок
<213> Homo sapiens
<400> 58
Leu Phe Val Asp His Cys Val Ala Thr
1 5
<210> 59
<211> 9
<212> Белок
<213> Homo sapiens
<400> 59
Gln Phe Thr Val Asp Val Phe His Phe
1 5
<210> 60
<211> 9
<212> Белок
<213> Homo sapiens
<400> 60
Phe Ala Asn Asp Ser Asn Arg Met Ile
1 5
<210> 61
<211> 9
<212> Белок
<213> Homo sapiens
<400> 61
Ile Tyr Ile Thr Cys His Leu Lys Val
1 5
<210> 62
<211> 9
<212> Белок
<213> Homo sapiens
<400> 62
Val Met Ser Gln Trp Ser Arg Ser Ala
1 5
<210> 63
<211> 9
<212> Белок
<213> Homo sapiens
<400> 63
Trp Ser Arg Ser Ala Ser Arg Asn Arg
1 5
<210> 64
<211> 9
<212> Белок
<213> Homo sapiens
<400> 64
Leu Ile Phe Leu Asp Arg Arg Gly Asp
1 5
<210> 65
<211> 9
<212> Белок
<213> Homo sapiens
<400> 65
Ile Phe Leu Asp Arg Arg Gly Asp His
1 5
<210> 66
<211> 49
<212> Белок
<213> Homo sapiens
<400> 66
Gly Gly Ala Ser His Pro Glu Thr Ser Val Gln Pro Val Leu Val Glu
1 5 10 15
Cys Gln Glu Ala Thr Leu Met Val Met Val Ser Lys Asp Leu Phe Gly
20 25 30
Thr Gly Lys Leu Ile Arg Ala Ala Asp Leu Thr Leu Gly Pro Glu Ala
35 40 45
Cys
<210> 67
<211> 26
<212> Белок
<213> Homo sapiens
<400> 67
Glu Ala Cys Glu Pro Leu Val Ser Met Asp Thr Glu Asp Val Val Arg
1 5 10 15
Phe Glu Val Gly Leu His Glu Cys Gly Asn
20 25
<210> 68
<211> 43
<212> Белок
<213> Homo sapiens
<400> 68
Cys Gly Asn Ser Met Gln Val Thr Asp Asp Ala Leu Val Tyr Ser Thr
1 5 10 15
Phe Leu Leu His Asp Pro Arg Pro Val Gly Asn Leu Ser Ile Val Arg
20 25 30
Thr Asn Arg Ala Glu Ile Pro Ile Glu Cys Arg
35 40
<210> 69
<211> 50
<212> Белок
<213> Homo sapiens
<400> 69
Ile Glu Cys Arg Tyr Pro Arg Gln Gly Asn Val Ser Ser Gln Ala Ile
1 5 10 15
Leu Pro Thr Trp Leu Pro Phe Arg Thr Thr Val Phe Ser Glu Glu Lys
20 25 30
Leu Thr Phe Ser Leu Arg Leu Met Glu Glu Asn Trp Asn Ala Glu Lys
35 40 45
Arg Ser
50
<210> 70
<211> 41
<212> Белок
<213> Homo sapiens
<400> 70
Lys Arg Ser Pro Thr Phe His Leu Gly Asp Ala Ala His Leu Gln Ala
1 5 10 15
Glu Ile His Thr Gly Ser His Val Pro Leu Arg Leu Phe Val Asp His
20 25 30
Cys Val Ala Thr Pro Thr Pro Asp Gln
35 40
<210> 71
<211> 44
<212> Белок
<213> Homo sapiens
<400> 71
Asp Gln Asn Ala Ser Pro Tyr His Thr Ile Val Asp Phe His Gly Cys
1 5 10 15
Leu Val Asp Gly Leu Thr Asp Ala Ser Ser Ala Phe Lys Val Pro Arg
20 25 30
Pro Gly Pro Asp Thr Leu Gln Phe Thr Val Asp Val
35 40
<210> 72
<211> 34
<212> Белок
<213> Homo sapiens
<400> 72
Gly Pro Asp Thr Leu Gln Phe Thr Val Asp Val Phe His Phe Ala Asn
1 5 10 15
Asp Ser Arg Asn Met Ile Tyr Ile Thr Cys His Leu Lys Val Thr Leu
20 25 30
Ala Glu
<210> 73
<211> 20
<212> Белок
<213> Homo sapiens
<400> 73
Lys Ala Cys Ser Phe Ser Lys Pro Ser Asn Ser Trp Phe Pro Val Glu
1 5 10 15
Gly Pro Ala Asp
20
<210> 74
<211> 27
<212> Белок
<213> Homo sapiens
<400> 74
Ser His Ser Arg Arg Gln Pro His Val Met Ser Gln Trp Ser Arg Ser
1 5 10 15
Ala Ser Arg Asn Arg Arg His Val Thr Glu Glu
20 25
<210> 75
<211> 26
<212> Белок
<213> Homo sapiens
<400> 75
Ala Asp Val Thr Val Gly Pro Leu Ile Phe Leu Asp Arg Arg Gly Asp
1 5 10 15
His Glu Val Glu Gln Trp Ala Leu Pro Ser
20 25
<210> 76
<211> 9
<212> Белок
<213> Homo sapiens
<400> 76
Phe Leu Leu His Asp Pro Arg Pro Val
1 5
<210> 77
<211> 9
<212> Белок
<213> Homo sapiens
<400> 77
Arg Leu Met Glu Glu Asn Trp Asn Ala
1 5
<210> 78
<211> 9
<212> Белок
<213> Homo sapiens
<400> 78
Ala Leu Val Tyr Ser Thr Phe Leu Leu
1 5
<210> 79
<211> 9
<212> Белок
<213> Homo sapiens
<400> 79
Phe Thr Val Asp Val Phe His Phe Ala
1 5
<210> 80
<211> 10
<212> Белок
<213> Homo sapiens
<400> 80
Leu Arg Leu Met Glu Glu Asn Trp Asn Ala
1 5 10
<210> 81
<211> 10
<212> Белок
<213> Homo sapiens
<400> 81
Phe Leu Asp Arg Arg Gly Asp His Glu Val
1 5 10
<210> 82
<211> 9
<212> Белок
<213> Homo sapiens
<400> 82
Cys Leu Val Asp Gly Leu Thr Asp Ala
1 5
<210> 83
<211> 9
<212> Белок
<213> Homo sapiens
<400> 83
Asn Met Ile Tyr Ile Thr Cys His Leu
1 5
<210> 84
<211> 10
<212> Белок
<213> Homo sapiens
<400> 84
Thr Phe Leu Leu His Asp Pro Arg Pro Val
1 5 10
<210> 85
<211> 10
<212> Белок
<213> Homo sapiens
<400> 85
Lys Leu Ile Arg Ala Ala Asp Leu Thr Leu
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ДРОЖЖИ, ПРОДУЦИРУЮЩИЕ ЭКТОИН | 2018 |
|
RU2745157C1 |
| АНТИ-ROR1/АНТИ-CD3 БИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2020 |
|
RU2831608C2 |
| СВЯЗЫВАЮЩИЕСЯ С РЕЦЕПТОРАМИ АГОНИСТЫ TNF | 2016 |
|
RU2821343C2 |
| АНТИТЕЛА К ПОВЕРХНОСТНЫМ ДЕТЕРМИНАНТАМ S. AUREUS | 2013 |
|
RU2808018C2 |
| АНТИТЕЛА К ПОВЕРХНОСТНЫМ ДЕТЕРМИНАНТАМ S. AUREUS | 2013 |
|
RU2698131C2 |
| Выделенное антитело к CD45RC человека, нуклеиновая кислота, вектор экспрессии, клетка, фармацевтическая композиция, их применение, in vitro способ обнаружения hCD45RC | 2019 |
|
RU2826421C2 |
| АНТИТЕЛО К EPHA4 | 2016 |
|
RU2719158C2 |
| ИНСЕКТИЦИДНЫЕ БЕЛКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2740312C2 |
| АНТИТЕЛА ПРОТИВ OX40 И ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2783314C2 |
| НОВЫЕ СЛИТЫЕ БЕЛКИ, СПЕЦИФИЧЕСКИЕ В ОТНОШЕНИИ CD137 И GPC3 | 2020 |
|
RU2814653C2 |
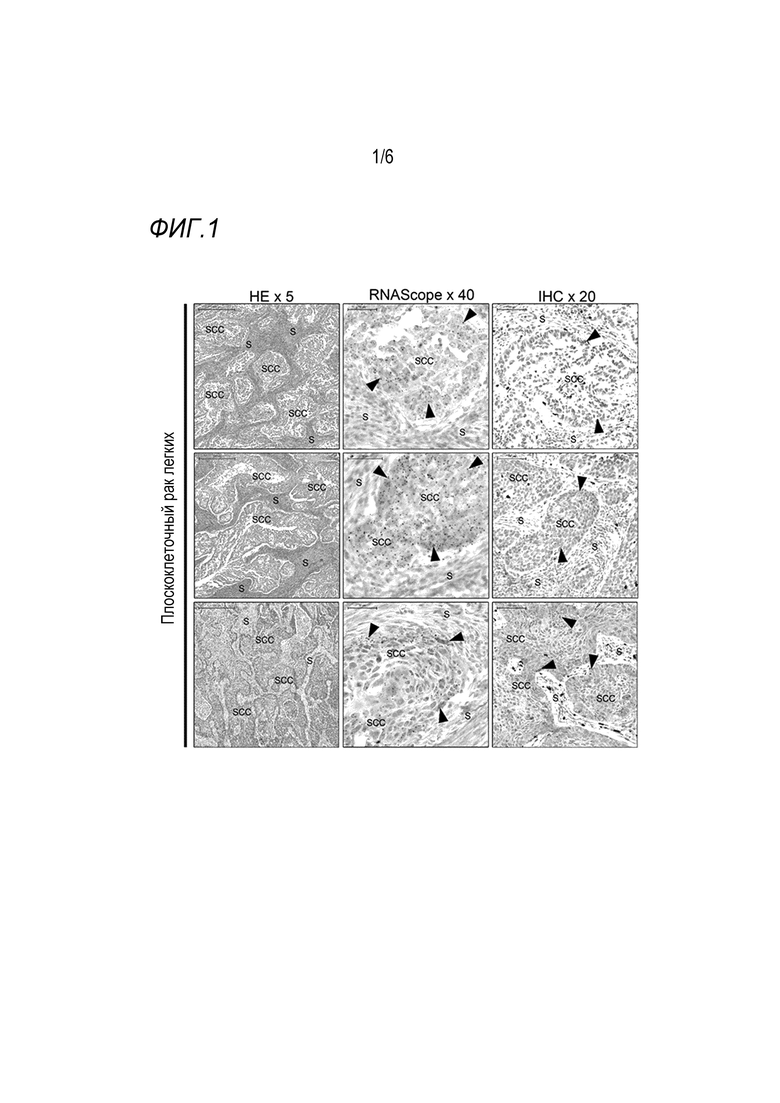
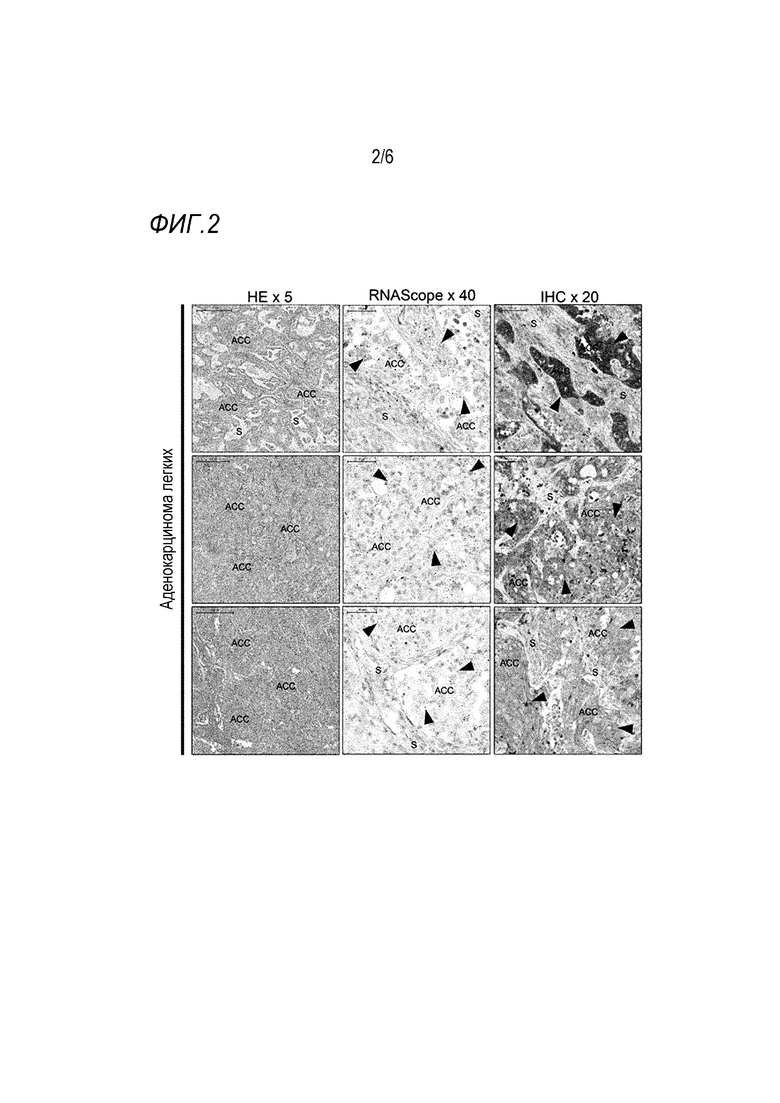
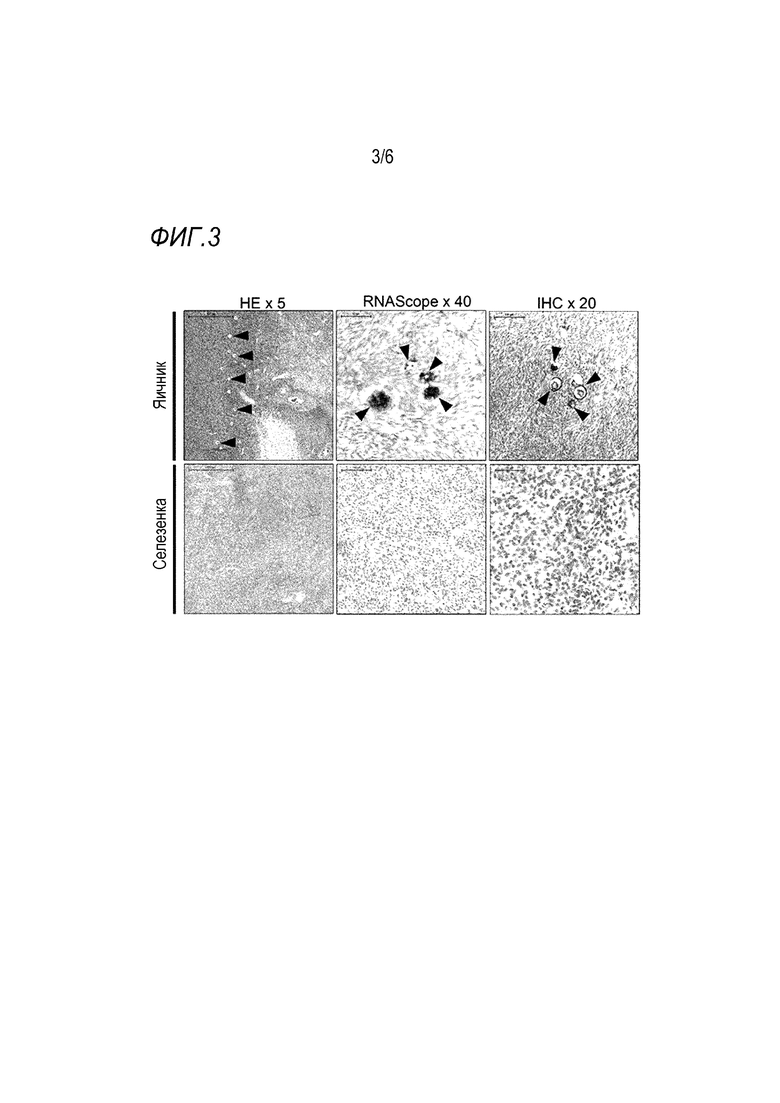

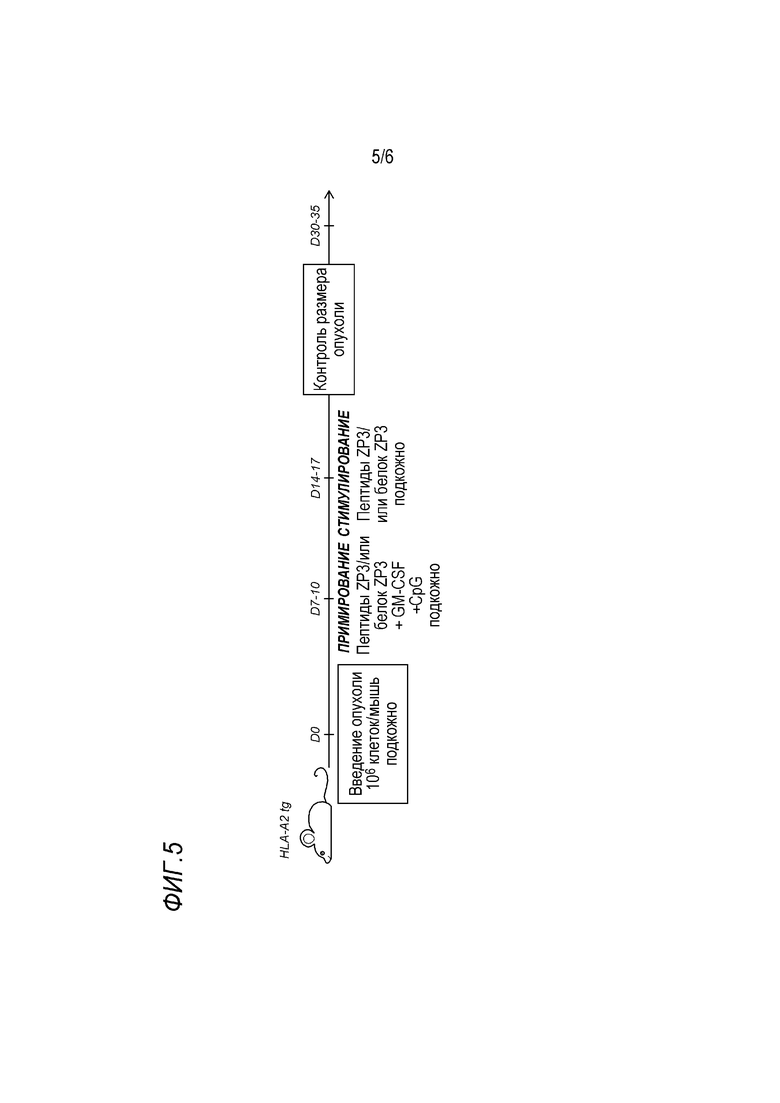
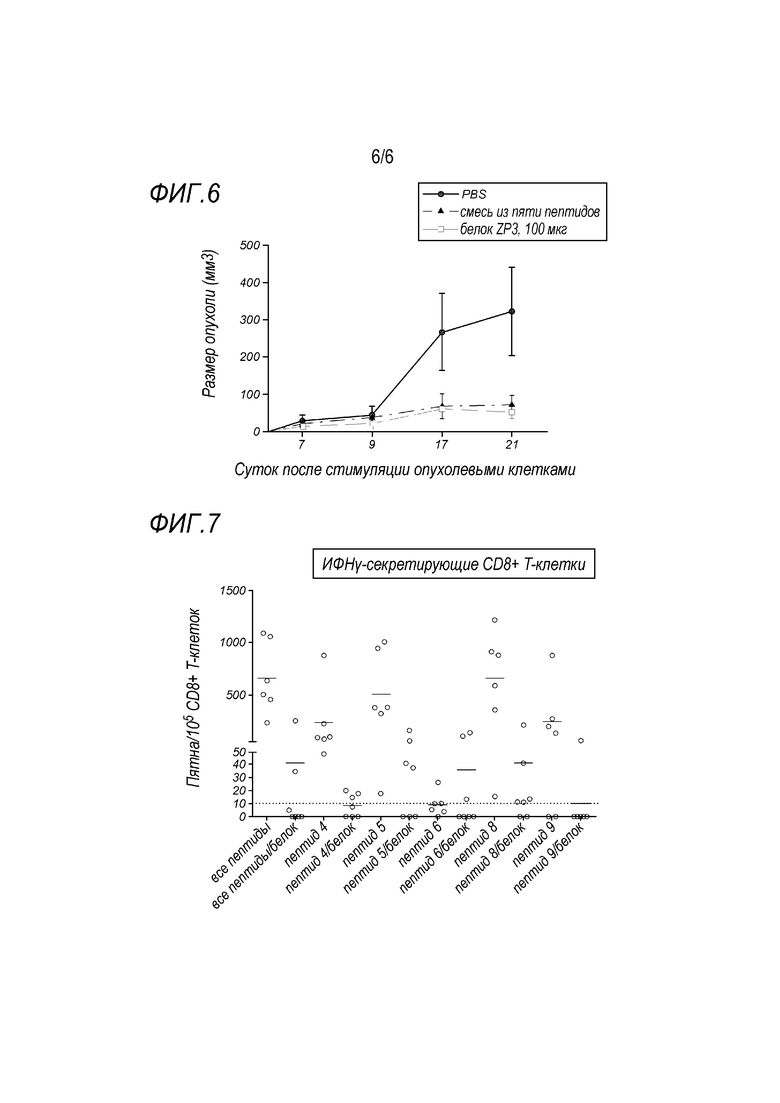
Изобретение относится к лечению и диагностике рака легких и его метастазов. Предложена фармацевтическая композиция для применения в способе терапевтического или профилактического лечения или для диагностики рака легких, экспрессирующего белок ZP3, и/или его метастазов у человека. Композиция содержит источник иммуногенного полипептида, включающий по меньшей мере один из эпитопов, рестрицированных по MHC класса I и класса II, из аминокислотной последовательности человеческого белка блестящей оболочки 3 (hZP3), и по меньшей мере один эксципиент. Источник иммуногенного полипептида включает по меньше мере один иммуногенный пептид, содержащий или состоящий из аминокислотной последовательности SEQ ID NO: 83, молекулу нуклеиновой кислоты, кодирующую указанный иммуногенный полипептид и/или клетку, экспрессирующую указанный иммуногенный полипептид. Изобретение обеспечивает диагностику и лечение первичного и/или рецидивирующего рака легких, экспрессирующего белок ZP3, и его метастазов. 11 з.п. ф-лы, 7 ил., 6 табл., 4 пр.
1. Фармацевтическая композиция для применения в способе терапевтического или профилактического лечения, или для диагностики рака легких, экспрессирующего белок ZP3, и/или его метастазов у человека, где композиция содержит:
источник иммуногенного полипептида, включающий по меньшей мере один из эпитопов, рестрицированных по MHC класса I и класса II, из аминокислотной последовательности человеческого белка блестящей оболочки 3 (hZP3), и по меньшей мере один эксципиент, где источник иммуногенного полипептида представляет собой композицию, включающую:
a) белковую композицию, содержащую по меньше мере один иммуногенный пептид, содержащий или состоящий из аминокислотной последовательности SEQ ID NO: 83, и по меньшей мере один эксципиент,
b) молекулу нуклеиновой кислоты, кодирующую аминокислотную последовательность указанного иммуногенного полипептида; и/или
c) клетку, экспрессирующую указанный иммуногенный полипептид.
2. Фармацевтическая композиция для применения по п. 1, где источником иммуногенного полипептида является композиция, содержащая
a) белковую композицию, дополнительно содержащую все из иммуногенных пептидов, содержащих или состоящих из аминокислотных последовательностей SEQ ID NO: 79, 80, 81 и 84;
b) одну или более молекул нуклеиновой кислоты, кодирующих аминокислотную последовательность указанных иммуногенных полипептидов; и/или
c) клетку, экспрессирующую указанные иммуногенные полипептиды.
3. Фармацевтическая композиция для применения по п. 1 или 2, где
a) белковая композиция дополнительно содержит один или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 66-75; и/или один или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 76-78, 82 и 85, и где один или более иммуногенных пептидов необязательно дополнительно содержат эпитоп, рестрицированный по MHC класса II, из аминокислотной последовательности белка hZP3;
b) одна или более молекул нуклеиновой кислоты дополнительно кодируют аминокислотную последовательность одного или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 66-75, и/или одного или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 76-78, 82 и 85, и где один или более иммуногенных пептидов необязательно дополнительно содержат эпитоп, рестрицированный по MHC класса II, из аминокислотной последовательности белка hZP3; и/или
c) клетка дополнительно экспрессирует один или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 66-75, и/или один или более иммуногенных пептидов, содержащих или состоящих из аминокислотной последовательности, выбранной из SEQ ID NO: 76-78, 82 и 85, и где один или более иммуногенных пептидов необязательно дополнительно содержат эпитоп, рестрицированный по MHC класса II, из аминокислотной последовательности белка hZP3.
4. Фармацевтическая композиция для применения по любому из пп. 1-3, где способ лечения представляет собой способ профилактики метастазов и/или рецидива рака легких, где, предпочтительно, рак легких представляет собой немелкоклеточный рак легких.
5. Фармацевтическая композиция для применения по любому из пп. 1-4, где способ лечения сочетается с иммуномодулирующей терапией, хирургической операцией, лучевой терапией, химиотерапией, нацеленной терапией или их сочетанием.
6. Фармацевтическая композиция для применения по п. 5, где иммуномодулирующая терапия включает применение по меньшей мере одного из ингибитора контрольной точки, антитела, нацеленного на член семейства рецепторов TNF, иммуносупрессивного цитокина, цитокина γC, агониста TLR и агониста клеток iNKT.
7. Фармацевтическая композиция для применения по любому из пп. 1-6, где:
a) белковая композиция содержит по меньшей мере один иммуногенный полипептид, как указано в любом из пп. 1-3, включающий непрерывную аминокислотную последовательность по меньшей мере из 18 аминокислот, выбранных из аминокислотной последовательности hZP3, и где непрерывная аминокислотная последовательность содержит по меньшей мере один из Т-клеточных эпитопов, рестрицированных по MHC класса I и MHC класса II;
b) молекула нуклеиновой кислоты представляет собой:
i) молекулу ДНК, которая является экспрессирующей конструкцией для экспрессии указанного иммуногенного полипептида в клетке человека; или
ii) молекулу РНК, которую можно транслировать в указанный иммуногенный полипептид в клетке человека;
c) клетка представляет собой микробную клетку, предпочтительно клетку Listeria.
8. Фармацевтическая композиция для применения по п. 7, где белковая композиция содержит более одного различных иммуногенных полипептидов, каждый из которых содержит непрерывную аминокислотную последовательность по меньшей мере из 18 аминокислот, выбранных из аминокислотной последовательности hZP3, и имеет длину в диапазоне 18-100 аминокислот, предпочтительно, длину в диапазоне 18-60 аминокислот.
9. Фармацевтическая композиция для применения по любому из предшествующих пунктов, где hZP3 содержит последовательность SEQ ID NO: 5.
10. Фармацевтическая композиция для применения по любому из предшествующих пунктов, где источник одного или нескольких иммуногенных пептидов в совокупности содержит по меньшей мере одно из:
a) по меньшей мере 50, 70, 80, 90 или 95% полной аминокислотной последовательности hZP3 или аминокислотной последовательности аминокислот 23 - 350 в SEQ ID NO: 3; и
b) по меньшей мере 50, 70, 80, 90 или 95% потенциальных эпитопов MHC I и/или MHC II, предсказанных биоинформатическим инструментом с компьютерным алгоритмом.
11. Фармацевтическая композиция для применения по любому из предшествующих пунктов, где способ дополнительно включает введение, предпочтительно совместное введение, по меньшей мере одного адъюванта.
12. Фармацевтическая композиция для применения по любому из предшествующих пунктов, где способ лечения включает введение эффективного количества композиции, где композицию вводят по меньшей мере одним из внутриопухолевого, внутримышечного, интраперитонеального, интрадермального, подкожного, трансдермального путей введения, и введения в анатомический участок, который дренируется по меньшей мере в один лимфатический коллектор.
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| ИММУНОТЕРАПЕВТИЧЕСКИЙ СПОСОБ ЛЕЧЕНИЯ РАКА ПРОСТАТЫ | 2011 |
|
RU2586774C2 |
| WO 2016080830 A2, 26.05.2016 | |||
| US 5672488 A, 30.09.1997 | |||
| RAHMAN N | |||
| A | |||
| et al | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
Авторы
Даты
2023-04-11—Публикация
2018-10-31—Подача