Область техники
Настоящее изобретение относится к области техники генной инженерии и, в частности, относится к способу получения сайт-специфичной мутации в организме в отсутствие матрицы искусственной ДНК и его применению.
Уровень техники
Технология генной инженерии для модификации генома организмов широко используется в промышленном и сельскохозяйственном производстве, например, генетически модифицированные микроорганизмы, обычно используемые в фармацевтической и химической областях, и генетически модифицированные культуры с резистентностью к насекомым и гербицидам в сельском хозяйстве. После открытия сайт-специфичных нуклеаз путем введения таргетной фрагментации в геном организма-реципиента и вызова спонтанной репарации стало возможным достичь сайт-специфичного редактирования генома и более точной модификации генома.
Инструменты редактирования генов в основном включают три типа сиквенс-специфичных нуклеаз (SSN): цинк-пальцевую нуклеазу (ZFN), нуклеазу на основе эффектора, подобную активатору транскрипции (TALEN) и ассоциированную с кластеризованными регулярными промежуточными короткими палиндромными повторами (CRISPR) систему Cas (систему CRISPR/Cas). Сиквенс-специфичными нуклеазами являются программируемые нуклеазы, которые могут создавать двухцепочечные разрывы ДНК (DSB) в специфичных сайтах генома. Двухцепочечные разрывы ДНК активируют эндогенный путь репарации ДНК для репарации повреждений ДНК в клетке, но процесс репарации легко приводит к изменениям в последовательности ДНК в сайтах-мишенях, тем самым обеспечивая введение мутаций в представляющие интерес сайты. Эта технология позволяет биологам точно таргетировать ген-мишень и редактировать его. Среди них, как ZFN, так и TALEN должны создать специфические модули белка распознавания для последовательности-мишени, что приводит к низкой пропускной способности и сложным операциям. Однако, белок Cas является универсальным в системе CRISPR/Cas, в которой направляющая РНК (gРНК) может быть образована специфичной CRISPR-РНК (crРНК), созданной только для сайта-мишени или в сочетании с трансактивирующей РНК (tracrРНК), или достаточно только одной направляющей РНК (sgРНК), crРНК и tracrРНК вместе или только sgРНК могут быть собраны с белком Cas с образованием рибонуклеопротеинового комплекса (RNP), последовательность-мишень идентифицируется на основе мотива, примыкающего к протоспейсеру (PAM) в геноме, тем самым реализуя сайт-специфичное редактирование. Таким образом, он стал основным инструментом редактирования генов из-за его простой работы, широкого диапазона применения и высокой производительности.
Сиквенс-специфичная нуклеаза может продуцировать двухцепочечные разрывы ДНК в специфичных сайтах в геноме. Эти двухцепочечные разрывы ДНК могут быть репарированы в различные типы репарации, которыми в основном являются вставки или делеции оснований. Например, двумя наиболее распространенными типами событий редактирования CRISPR/Cas9 являются вставка основания в месте разрыва или делеция основания в месте разрыва (Shen et al. 2018. Predictable and precise template-free CRISPR editing of pathogenic variants. Nature. DOI: 10.1038/s41586-018-0686-x). Вставка или делеция оснований в кодирующей области вызывает мутации со сдвигом рамки считывания, что приводит к потере функции гена. Следовательно, основной целью вышеупомянутых инструментов редактирования генов по-прежнему является нокаут генов.
Все время считалось, что использование только сиквенс-специфичных нуклеаз не может привести к мутациям типа замены оснований. С этой целью, известный уровень техники предлагает три решения: 1) добавление фрагмента экзогенной ДНК в качестве матрицы репарации для инициации пути репарации гомологичной рекомбинации; 2) слияние дезаминазы с Cas9 для последовательной разработки одноосновных инструментов редактирования для C - T и A - G; 3) слияние обратной транскриптазы с Cas9 с использованием pegРНК для управления синтезом и заменой малой цепи ДНК. Однако, эффективность редактирования этих трех растворов значительно ниже эффективности нокаута гена, и одновременное введение фрагмента экзогенной ДНК и обратной транскриптазы вполне может вызвать опасения по поводу биологической безопасности. Эффект вне мишени одноосновного редактирования также ограничивает его потенциальное применение в клеточной терапии. Особенно для долгосрочных проектов по селекции растений, то, как повысить эффективность замены оснований на сайте-мишени при одновременном снижении опасений регулирующих органов по поводу биобезопасности, является проблемой, которую необходимо решить при применении технологии редактирования генов.
Таким образом, в области клеточной терапии и биологической селекции существует острая техническая потребность в сайт-специфичной замене оснований с использованием только таргетного нокаута сиквенс-специфичной нуклеазы без введения фрагмента чужеродной ДНК, особенно с помощью не трансгенной системы временного редактирования для эффективного завершения редактирования сайт-специфичной замены основания.
Сущность изобретения
Изобретение представляет способ создания сайт-специфичной мутации в организме только путем создания двухцепочечных разрывов в геноме и без предоставления матрицы искусственной ДНК, и применение этого способа.
Технические решения, принятые настоящим изобретением, следующие:
Способ создания новой мутации в организме, который включает следующие стадии: последовательное создание двух или нескольких разрывов ДНК в специфичном сайте генома организма и их спонтанную репарацию, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, созданной из предыдущей репарации разрыва ДНК.
В конкретном варианте осуществления, «разрыв ДНК» достигается путем доставки нуклеазы с таргетным свойством в клетку организма для контакта со специфичным сайтом геномной ДНК.
В конкретном варианте осуществления, «нуклеазой с таргетным свойством» является система ZFN, TALEN или CRISPR/Cas.
В конкретном варианте осуществления, «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» относится к тому, что на основе новой последовательности, созданной в результате предыдущего события репарации разрыва ДНК, вызванного редактированием ZFN или TALEN, создается новый белок ZFN или TALEN для того, чтобы снова разрезать сайт.
В другом конкретном варианте осуществления, «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» относится к тому, что на основе новой последовательности, созданной в результате предыдущего события репарации разрыва ДНК, вызванного системой CRISPR/Cas, создается новая РНК-мишень для того, чтобы снова разрезать сайт. Например, второе разрезание проводят в этом сайте снова путем конструирования новой РНК-мишени на основе новой последовательности, сгенерированной в результате события репарации первого разрыва при редактировании Cas9. Аналогичным образом, в этом сайте проводят третье разрезание путем конструирования новой РНК-мишени на основе новой последовательности, полученной в результате второго события репарации разрыва и так далее, как показано на фигуре 1.
В конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются путем последовательной доставки разных таргетных нуклеаз в клетки-реципиенты разных поколений, где мутантная клетка, которая завершила предыдущее редактирование, используется в качестве реципиента для доставки таргетной нуклеазы для последующего редактирования, тем самым выполняя второе редактирование для создания сайт-специфичной мутации. Этот способ предпочтительно использовать для систем редактирования ZFN и TALEN.
В другом конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются путем доставки разных таргетных нуклеаз для разных мишеней в одну и ту же клетку-реципиент. Этот способ предпочтительно использовать для системы редактирования CRISPR/Cas.
В конкретном варианте осуществления, «два или более несколько ДНК» создаются, когда комплексы RNP образуются одной и той же CRISPR/Cas нуклеазой, соответственно, где разные gРНК или sgРНК последовательно разрезают соответствующие последовательности-мишени.
В другом конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, образованные каждой из двух или нескольких нуклеаз CRISPR/Cas, которые распознают разные последовательности PAM с соответствующими gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени. Например, последовательностью PAM, распознаваемой Cas9 Streptococcus pyogenes, является «NGG» или «NAG» (Jinek et al., "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity", Science 2012, 337:816-821), последовательностью PAM, распознаваемой Cas9 Staphylococcus aureus, является «NNGRRT» или «NNGRR(N)», последовательностью PAM, распознаваемой Cas9 Neisseria meningitidis, является NNNNGATT, и последовательностью PAM, распознаваемой Cas9 Streptococcus thermophilus, является NNAGAAW. Таким образом, редактируемое окно молекулы ДНК становится больше.
В конкретном варианте осуществления, таргетной нуклеазой является любая нуклеаза CRISPR/Cas, способная осуществлять редактирование генома.
В конкретном варианте осуществления, таргетная нуклеаза имеет форму ДНК.
В другом конкретном варианте осуществления, таргетная нуклеаза имеет форму mРНК или белка вместо ДНК. Белковая форма является предпочтительной.
В конкретном варианте осуществления, способ доставки таргетных нуклеаз в клетку выбран, но не ограничен ими, из: 1) способа ПЭГ-опосредованной трансфекции клеток; 2) способа липосомно-опосредованной трансфекции клеток; 3) способа электропорационной трансформации; 4) микроинъекции; 5) бомбардировки генной пушкой; 6) способа Agrobacterium-опосредованной трансформации.
В этом способе, новую мишень создают на основе новой последовательности, созданной в результате предыдущей репарации разрыва ДНК, и, таким образом, мутации могут последовательно образовываться много раз в специфичном сайте генома, тем самым экспоненциально обогащая типы событий репарации после разрывов ДНК и создавая новые типы мутаций с заменой основания, делецией и вставкой, которые нельзя получить путем редактирования одного гена, так что этот способ подходит для использования в качестве инструмента для создания новых мутаций. Этот способ можно кратко описать как способ запрограммированного последовательного отрезания/редактирования или последовательного отрезания/редактирования.
В конкретном варианте осуществления, новую мишень создают на основе новой специфической последовательности, которая, по прогнозам, будет получена в результате предыдущего восстановления разрыва в специфичном сайте генома организма, затем выполняют последовательное редактирование, и, таким образом, окончательная возможная мутация в этом сайте может быть создана заранее, чтобы добиться ожидаемого редактирования.
В другом конкретном варианте осуществления, новую мишень создают на основе новой последовательности, которая, по прогнозам, будет создаваться в результате предыдущего восстановления разрыва в специфичном сайте генома организма, затем выполняют последовательное редактирование, и, в дополнение к ожидаемому событию редактирования, различные в конечном итоге на сайте могут быть созданы различные мутации, так что способ можно использовать в качестве инструмента для создания различных мутаций.
В другом аспекте настоящее изобретение дополнительно обеспечивает способ создания новой мутации в организме, который включает следующую стадию: последовательное создание двух или более разрывов ДНК в специфическом участке гена на уровне генома или хромосомы организма., тем самым достигается точная замена основания, делеция или вставка.
В конкретном варианте осуществления, «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» относится к тому, что новая РНК-мишень создана на основе новой последовательности, созданной из предыдущего события репарации разрыва, и отрезание выполняют снова на том же сайте.
В конкретном варианте осуществления, «разрыв ДНК» достигается с помощью нуклеазы с таргетным свойством.
Настоящее изобретение дополнительно представляет новую мутацию, полученную вышеупомянутым способом.
Настоящее изобретение дополнительно относится к белку или его биологически активному фрагменту, который имеет вышеупомянутую новую мутацию.
Настоящее изобретение дополнительно относится к нуклеиновой кислоте, которая содержит последовательность нуклеиновой кислоты или комплементарную ей последовательность, которая кодирует белок или его биологически активный фрагмент.
Настоящее изобретение дополнительно представляет нуклеиновую кислоту, которая содержит:
(а) нуклеотидную последовательность, кодирующую РНК-мишень, где
РНК-мишень содержит, по меньшей мере, две РНК-мишени, в которых первая РНК-мишень таргетирует ДНК, вызывая разрыв в ДНК, и последняя РНК-мишень таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и снова создает разрыв.
В конкретном варианте осуществления, нуклеиновая кислота дополнительно содержит (b) нуклеотидную последовательность, кодирующую Cas полипептид.
В конкретном варианте осуществления, РНК-мишенью является sgРНК или gРНК.
В конкретном варианте осуществления, Cas полипептид и РНК-мишень присутствуют в клетке in vitro или клетке ex vivo.
Настоящее изобретение дополнительно представляет рекомбинантный вектор экспрессии, который содержит вышеупомянутую нуклеиновую кислоту и функционально связанный с ней промотор.
Настоящее изобретение дополнительно представляет кассету экспрессии, которая содержит вышеупомянутую нуклеиновую кислоту.
Настоящее изобретение дополнительно представляет клетку-хозяина, которая содержит вышеупомянутую кассету экспрессии.
Настоящее изобретение дополнительно представляет организм, регенерируемый с использованием вышеупомянутой клетки-хозяина.
Настоящее изобретение дополнительно представляет способ лизиса ДНК-мишени, который включает контакт ДНК-мишени с комплексом, где комплекс содержит:
(а) Cas полипептид; и
(b) по меньшей мере, две РНК-мишени, где первая РНК-мишень таргетирует ДНК, чтобы вызвать разрыв в ДНК, и последняя РНК-мишень таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и снова создает разрыв.
В конкретном варианте осуществления, РНК-мишенью является sgРНК или gРНК.
В конкретном варианте осуществления, ДНК-мишень присутствует в бактериальной клетке, эукариотической клетке, растительной клетке или животной клетке.
В конкретном варианте осуществления, ДНК-мишенью является хромосомная ДНК.
В конкретном варианте осуществления, Cas полипептид и РНК-мишень присутствуют в клетке in vitro или клетке ex vivo.
В конкретном варианте осуществления, контакт включает введение в клетку следующего: (а) Cas полипептида или полинуклеотида, кодирующего Cas полипептид, и (b) РНК-мишени или полинуклеотида ДНК, кодирующего РНК-мишень.
Настоящее изобретение дополнительно представляет композицию, которая содержит:
(а) Cas полипептид или полинуклеотид, кодирующий Cas полипептид; и
(b) по меньшей мере, две РНК-мишени или полинуклеотиды ДНК, кодирующие РНК-мишени, где первая РНК таргетирует ДНК, чтобы вызвать разрыв в ДНК, и последняя РНК-мишень таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и создает новый разрыв.
В конкретном варианте осуществления, РНК-мишенью является sgРНК или gРНК.
В конкретном варианте осуществления, Cas полипептид и РНК-мишень присутствуют в клетке in vitro или клетке ex vivo.
Изобретение дополнительно относится к применению композиции в производстве лекарственного средства для лечения заболевания.
Заболевание, которое можно лечить с помощью композиции по настоящему изобретению, включает, но не ограничено ими, заболевание, вызванное мутацией одного гена, такое как генетическая тирозинемия 1 типа, фенилкетонурия, прогерия, серповидно-клеточная анемия и т. д. Самопроизвольная репарация клеток индуцируется путем доставки в клетку Cas белка и композиции crРНК или sgРНК, которая, как ожидается, репарирует сайт патогенной мутации с продуцированием нормального функционального белка, и, таким образом, достигается терапевтический эффект.
Настоящее изобретение дополнительно представляет набор, который включает:
(а) Cas полипептид или нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую Cas полипептид; и
(b) по меньшей мере, две РНК-мишени или нуклеиновые кислоты, содержащие нуклеотидные последовательности, кодирующие РНК-мишени, где первая РНК-мишень таргетирует ДНК, чтобы вызвать разрыв в ДНК, и последняя РНК-мишень таргетирует последовательность, созданную в результате предыдущего события репарации разрыва и снова создает разрыв;
где (а) и (b) находятся в одном и том же контейнере или отдельных контейнерах.
В конкретном варианте осуществления, РНК-мишенью является sgРНК или gРНК.
В конкретном варианте осуществления, РНК-мишени в (b) находятся в одном контейнере или отдельных контейнерах.
Настоящее изобретение дополнительно представляет способ скрининга событий редактирования, независимых от экзогенных трансгенных маркеров, включающий следующие стадии:
1) два или несколько разрывов ДНК последовательно создают в специфичном сайте первого гена-мишени клетки-реципиента и спонтанно репарируют, соответственно, где более поздний разрыв ДНК создают на основе новой последовательности, полученной в результате репарации предыдущего разрыва ДНК;
2) определенные события редактирования создают после того, как конкретный сайт первого гена-мишени последовательно отрезают и репарируют, что может придать мутантной клетке резистентность к определенному давлению отбора для продуцирования фенотипического селектируемого признака, соответствующее давление отбора применяют для селекции признака, и выделяют клетку, ткань, орган или целый организм, который содержит такие события редактирования;
3) необязательно, в дополнение к первому гену-мишени, таргетную нуклеазу для, по меньшей мере, одного второго гена-мишени используют для одновременного редактирования другого сайта-мишени, и событие редактирования второго гена-мишени синхронно обогащают и подвергают скринингу посредством скрининга селектируемого признака, созданного мутациями первого гена-мишени, и выделяют клетки, ткани, органа или целого организма, которые одновременно содержат события редактирования первого гена-мишени и, по меньшей мере, одного второго гена-мишени.
В конкретном варианте осуществления, «первым геном-мишенью» является локус гена, кодирующий, по меньшей мере, один фенотипический селектируемый признак, где, по меньшей мере, одним фенотипическим селектируемым признаком является признак резистентности/толерантности или признак преимущества роста.
В конкретном варианте осуществления, «специфический сайт первого гена-мишени» относится к сайту, в котором возникает определенный тип мутации после последовательных разрезов и репараций, который способен придать клетке-реципиенту резистентность к определенному давлению отбора для получения, по меньшей мере, одного фенотипически селектируемого признака резистентности/толерантности или признака преимущества роста.
В конкретном варианте осуществления, «мутация определенного типа» включает замену одного основания, замену множества оснований или вставку или делецию неопределенного числа оснований.
В конкретном варианте осуществления, «определенным давлением отбора» может быть давление окружающей среды или давление, возникающее в результате добавления соединения; например, давлением окружающей среды является высокая температура, низкая температура или гипоксия и подобные; давлением, возникающим в результате добавления соединения, может быть давление, возникающее в результате концентрации иона соли, антибиотика, цитотоксина, гербицида и т. д.
В конкретном варианте осуществления, «разрыв ДНК» достигается путем доставки нуклеазы с таргетным свойством в клетку организма для контакта со специфичным сайтом геномной ДНК.
В конкретном варианте осуществления, «нуклеазой с таргетным свойством» является любая нуклеаза CRISPR/Cas, способная осуществлять редактирование генома.
В конкретном варианте осуществления, признак «два или несколько разрывов ДНК последовательно создаются в определенной последовательности в специфичном сайте» относится к тому, что основано на новой последовательности, образованной предыдущим событием репарации разрыва ДНК, созданным системой CRISPR/Cas, новая РНК-мишень создана для повторного разрезания сайта.
В конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, образованные одной и той же CRISPR/Cas нуклеазой, соответственно, с разными gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
В другом конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, соответственно, образованные каждой из двух или нескольких нуклеаз CRISPR/Cas, которые распознают разные последовательности PAM с соответствующими gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени. Таким образом, редактируемое окно молекулы ДНК становится больше.
В конкретном варианте осуществления, «второй ген-мишень» относится к другому гену, кодирование которого отличается от первого гена-мишени.
В конкретном варианте осуществления, «таргетная нуклеаза, по меньшей мере, для одного второго гена-мишени» и нуклеаза CRISPR/Cas, используемые для создания разрыва ДНК в специфичном сайте первого гена-мишени, являются одинаковыми.
В другом конкретном варианте осуществления, «таргетная нуклеаза, по меньшей мере, для одного второго гена-мишени» и нуклеаза CRISPR/Cas, используемые для создания разрыва ДНК в специфичном сайте первого гена-мишени, являются разными. Таким образом, на втором гене-мишени имеется больше сайтов селектируемого редактирования.
В конкретном варианте осуществления, таргетная нуклеаза имеет форму ДНК.
В другом конкретном варианте осуществления, таргетная нуклеаза имеет форму mРНК или белка вместо ДНК. Белковая форма является предпочтительной.
В конкретном варианте осуществления, способ доставки таргетной нуклеазы в клетку выбран, но не ограничен ими, из: 1) способа ПЭГ-опосредованной трансфекции клеток; 2) способа липосомно-опосредованный трансфекции клеток; 3) способа электропорационной трансформации; 4) микроинъекции; 5) бомбардировки генной пушкой; или 6) способом Agrobacterium-опосредованной трансформации.
Настоящее изобретение также представляет способ не трансгенного временного редактирования генома организма, включающий следующие стадии:
1) создания и синтеза комбинации, по меньшей мере, двух фрагментов crРНК или комбинации, по меньшей мере, двух фрагментов sgРНК для специфичного сайта первого гена-мишени клетки-реципиента, где комбинация crРНК в комбинации с tracrРНК или только комбинация sgРНК способны управлять соответствующим Cas белком для последовательного создания двух или нескольких разрывов ДНК в конкретном сайте в первом гене-мишени клетки-реципиента и их спонтанной репарации, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, создаваемой из предыдущей репарации разрыва ДНК;
2) смешивания соответствующего количества белка CRISPR/Cas или его соответствующей mРНК с комбинацией фрагментов crРНК и фрагмента tracrРНК или только с комбинацией фрагментов sgРНК, как указано выше, созданной и синтезированной заранее, которая способна направлять сайт-специфическое редактирование первого гена-мишени для создания эндогенных селективных маркеров, необязательно, последующего добавления, по меньшей мере, одного из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней, и проведения инкубации in vitro с образованием комплекса RNP;
3) доставки вышеуказанного комплекса RNP в клетку-реципиент и контакта со специфичным сайтом геномной ДНК для осуществления редактирования гена;
4) в соответствии с фенотипическим селектируемым признаком, создаваемым сайт-специфическим редактированием первого гена-мишени комплексом RNP, применения соответствующего давления отбора, чтобы провести селекцию признака и выделения клетки, ткани, органа или всего организма, который содержит событие редактирования, и, необязательно, выделения клетки, ткани, органа или полного организма, которые одновременно содержат события редактирования первого гена-мишени и, по меньшей мере, одного из второго, третьего или нескольких генов-мишеней.
В конкретном варианте осуществления «первым геном-мишенью» является локус гена, кодирующий, по меньшей мере, один фенотипический селектируемый признак, где, по меньшей мере, одним фенотипическим селектируемым признаком является признак резистентности/толерантности или признака преимущества роста.
В конкретном варианте осуществления, «специфичный сайт первого гена-мишени» относится к сайту, в котором создается определенный тип мутации после последовательных разрезов и репараций, которые способны придать клетке-реципиенту резистентность к определенному давлению отбора для получения, по меньшей мере, одного фенотипически селектируемого признака резистентности/толерантности или признака преимущества роста.
В конкретном варианте осуществления, «мутация определенного типа» включает замену одного основания, замену множества оснований или вставку или делецию неопределенного числа оснований.
В конкретном варианте осуществления, «определенным давлением отбора» может быть давление окружающей среды или давление, возникающее в результате добавления соединения; например, давлением окружающей среды является высокая температура, низкая температура или гипоксия и подобные; давлением, возникающим в результате добавления соединения, может быть давление, возникающее в результате концентрации иона соли, антибиотика, цитотоксина или гербицида и подобных.
В конкретном варианте осуществления, белком CRISPR/Cas является любая нуклеаза CRISPR/Cas, способная осуществлять редактирование генома.
В конкретном варианте осуществления, признак «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» означает, что на основе новой последовательности, образованной предыдущим событием репарации разрыва ДНК, сгенерированным системой CRISPR/Cas, создается новая РНК-мишень для повторного разрезания сайта.
В конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, образованные одной и той же нуклеазой CRISPR/Cas, соответственно, с различными gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
В другом конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, соответственно, образованные каждой из двух или нескольких нуклеаз CRISPR/Cas, которые распознают разные последовательности PAM с соответствующими gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени. Таким образом, редактируемое окно молекулы ДНК становится больше.
В конкретном варианте осуществления, «второй, третий или несколько генов-мишеней» относятся к другим генам, кодирование которых отличается от первого гена-мишени.
В конкретном варианте осуществления, «по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней» имеет тот же самый Cas белок, что и crРНК или sgРНК, таргетирующие первый ген-мишень.
В другом конкретном варианте осуществления, «по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней», и crРНК или sgРНК, таргетирующих первый ген-мишень, используют Cas белки, которые распознают разные последовательности PAM. Таким образом, на втором гене-мишени имеется больше селектируемых сайтов редактирования.
В конкретном варианте осуществления, способ доставки RNP комплекса в клетки выбран, но не ограничен ими, из: 1) способа ПЭГ-опосредованной трансфекции клеток; 2) способа липосомно-опосредованной трансфекции клеток; 3) способа электропорационной трансформации; 4) микроинъекции; 5) бомбардировки генной пушкой; и так далее.
Настоящее изобретение также представляет способ не трансгенного временного редактирования генома растения, включающий следующие стадии:
1) создания и синтеза комбинации, по меньшей мере, двух фрагментов crРНК или комбинации, по меньшей мере, двух фрагментов sgРНК для специфичного сайта первого гена-мишени растительной клетки-реципиента, где комбинация crРНК в комбинации с tracrРНК или только комбинация sgРНК способны управлять соответствующим Cas белком для последовательного создания двух или нескольких разрывов ДНК в конкретном сайте в первом гене-мишени клетки-реципиента и их спонтанной репарации, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, создаваемой из предыдущей репарации разрыва ДНК;
2) смешивания соответствующего количества белка CRISPR/Cas или его соответствующей mРНК с комбинацией фрагментов crРНК и фрагмента tracrРНК или только с комбинацией фрагментов sgРНК, как указано выше, созданной и синтезированной заранее, которая способна направлять сайт-специфическое редактирование первого гена-мишени для создания эндогенных селективных маркеров, необязательно, последующего добавления, по меньшей мере, одного из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней, и проведения инкубации in vitro с образованием комплекса RNP;
3) доставки вышеуказанного комплекса RNP в растительную клетку или ткань-реципиент и контакта со специфичным сайтом геномной ДНК для осуществления редактирования гена;
4) в соответствии с фенотипическим селектируемым признаком, создаваемым сайт-специфическим редактированием первого гена-мишени комплексом RNP, применения соответствующего давления отбора, чтобы провести селекцию признака и выделения клетки, ткани, органа или всего растения, которое содержит событие редактирования, и, необязательно, выделения клетки, ткани, органа или полного растения, которые одновременно содержат события редактирования первого гена-мишени и, по меньшей мере, одного из второго, третьего или нескольких генов-мишеней.
В конкретном варианте осуществления «первым геном-мишенью» является локус гена, кодирующий, по меньшей мере, один фенотипический селектируемый признак, где, по меньшей мере, одним фенотипическим селектируемым признаком является признак резистентности/толерантности или признак преимущества роста.
В конкретном варианте осуществления, «специфичный сайт первого гена-мишени» относится к сайту, в котором мутация определенного типа создается после последовательного разрезания и репарации в сайте, что может придать клетке-реципиенту резистентность к определенному давлению отбора для получения, по меньшей мере, одного фенотипически селектируемого признака резистентности/толерантности или признака преимущества роста.
В конкретном варианте осуществления «мутация определенного типа» включает замену одного основания, замену множества оснований или вставку или делецию неопределенного числа оснований.
В конкретном варианте осуществления «определенным давлением отбора» может быть давление окружающей среды или давление, возникающее в результате добавления соединения; например, давлением окружающей среды предпочтительно является высокая температура, низкая температура или гипоксия и т.п.; давлением, возникающим в результате добавления соединения может быть давление, вызванное концентрацией ионов соли, антибиотиком, цитотоксином, гербицидом и т.д.
В конкретном варианте осуществления, «реципиентной растительной клеткой или тканью» является любая клетка или ткань, которые могут служить реципиентом для временной экспрессии и могут быть регенерированы в полноценное растение через культуру ткани. В частности, клеткой является протопластная клетка или суспензионная клетка; тканью предпочтительно является каллюс, незрелый зародыш, зрелый зародыш, лист, верхушка побега, молодой колос, гипокотиль и т.д.
В конкретном варианте осуществления, белком CRISPR/Cas является любая нуклеаза CRISPR/Cas, способная осуществлять редактирование генома.
В конкретном варианте осуществления функция «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» означает, что на основе новой последовательности, образованной предыдущим событием репарации разрыва ДНК, созданным системой CRISPR/Cas, создается новая РНК-мишень для повторного разрезания сайта.
В конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда комплексы RNP, образованные одной и той же CRISPR/Cas нуклеазой, соответственно, с различными gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
В другом конкретном варианте осуществления, «два или несколько разрывов ДНК» создаются, когда, комплексы RNP, соответственно, образованные каждой из двух или нескольких CRISPR/Cas нуклеаз, которые распознают разные последовательности PAM, с соответствующими gРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени. Таким образом, редактируемое окно молекулы ДНК становится больше.
В конкретном варианте осуществления, «второй, третий или несколько генов-мишеней» относятся к другим генам, кодирование которых отличается от первого гена-мишени.
В конкретном варианте осуществления «по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней» имеет общий белок Cas с crРНК или sgРНК, таргетирующими первый ген-мишень.
В другом конкретном варианте осуществления «по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней», и crРНК или sgРНК, таргетирующие первый ген-мишень, используют белки Cas, которые распознают разные последовательности PAM. Таким образом, на втором гене-мишени имеется больше селектируемых сайтов редактирования.
В конкретном варианте осуществления, способ доставки комплекса RNP в растительные клетки выбран, но не ограничен ими, из: 1) способа ПЭГ-опосредованной трансформации протопластов; 2) микроинъекции; 3) бомбардировки генной пушкой; 4) способа, опосредованного волокнами карбида кремния; 5) способа вакуумной инфильтрации или любого другого способа кратковременного введения. Бомбардировка генной пушкой является предпочтительной.
В конкретном варианте осуществления, «первым геном-мишенью» является, по меньшей мере, один эндогенный ген, который кодирует, по меньшей мере, один фенотипический селектируемый признак, выбранный из резистентности/толерантности к гербицидам, где резистентность/толерантность к гербицидам выбрана из группы, состоящей из резистентности/толерантности к ингибитору EPSPS (включая глифосат); резистентности/толерантности к ингибитору синтеза глутамина (включая глюфосинат); резистентности/толерантности к ингибитору ALS или AHAS (включая имидазолин или сульфонилмочевину); резистентности/толерантности к ингибитору АССазы (включая арилоксифеноксипропионовую кислоту (FOP)); резистентности/толерантности к ингибитору биосинтеза каротиноидов, включая ингибиторы биосинтеза каротиноидов на стадии фитоендесатуразы (PDS), ингибиторы 4-гидроксифенилпируватдиоксигеназы (HPPD) или другие таргетные ингибиторы биосинтеза каротиноидов; резистентности/толерантности к ингибитору целлюлозы; резистентности/толерантности к ингибитору синтеза жиров; резистентности/толерантности к ингибитору длинноцепочечных жирных кислот; резистентности/толерантности к ингибитору сборки микротрубочек; резистентности/толерантности к электрон-шунтирующему агенту фотосистемы I; резистентности/толерантности к ингибитору фотосистемы II (включая карбаматы, триазины и триазоны); резистентности/толерантности к ингибитору PPO; и резистентности/толерантности к синтетическому гормону роста (включая дикамба, 2,4-D (т.е. 2,4-дихлорфеноксиуксусную кислоту)). Где первый ген-мишень выбран из PsbA, ALS, EPSPS, ACCase, PPO, HPPD, PDS, GS, DOXPS, TIR1, AFB5, и некоторые типы мутаций, созданных после последовательных разрезаний и репараций в специфичных сайтах этих гербицидных генов-мишеней, могут придать клеткам растения-реципиента резистентность/толерантность к соответствующим гербицидам.
В конкретном варианте осуществления, «первым геном-мишенью» является ALS, и «специфичный сайт гена» относится к сайту A122, P197, R198, D204, A205, D376, R377, W574, S653 или G654 в аминокислотной последовательности белка Arabidopsis AtALS (например, показанной в SEQ ID NO:1) и аминокислотным сайтам в белке ALS другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам при использовании аминокислотной последовательности AtALS в качестве эталонного стандарта. crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности белка AtALS, выбранный из группы, состоящей из A122, P197, R198, D204, A205, D376, R377, W574, S653, G654 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке ALS другого растения, который соответствует вышеупомянутым аминокислотным сайтам, и любую их комбинацию, с использованием аминокислотной последовательности AtALS в качестве эталонного стандарта. Сайт ALS W574 является предпочтительным. Давлением отбора предпочтительно является обработка пироксуламом или никосульфуроном.
В конкретном варианте осуществления, «первым геном-мишенью» является АССаза, и «специфичный сайт гена» относится к сайтам I1781, E1874, N1878, W1999, W2027, I2041, D2078, C2088 или G2096 аминокислотной последовательности белка Alopecurus myosuroides AmACCase (например, показанной в SEQ ID NO: 3, и последовательности гена, показанной в SEQ ID NO: 4), и аминокислотные сайты в белке АССазы другого однодольного растения, которые соответствуют вышеупомянутым аминокислотным сайтам с использованием аминокислотной последовательности AmACCase в качестве эталонного стандарта. crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности AmACCase, выбранный из группы, состоящей из I1781, E1874, N1878, W1999, W2027, I2041, D2078, C2088, G2096 или любую их комбинацию, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке АССазы другого однодольного растения, которая соответствует указанному выше аминокислотному сайту, и любую их комбинацию, с использованием аминокислотной последовательности AmACCase в качестве эталонного стандарта. Сайт W2027 ACCазы является предпочтительным. Давлением отбора предпочтительно является обработка хизалофоп-п-этилом.
В конкретном варианте осуществления, «первым геном-мишенью» является HPPD, и «специфичный сайт гена» относится к сайту H141, L276, P277, N338, G342, R346, D370, P386, K418 или G419 в аминокислотной последовательности белка Oryza sativa OsHPPD (показанной в SEQ ID NO: 5, и последовательности генома, показанной в SEQ ID NO: 6), и аминокислотные сайты в белке HPPD другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам при использовании аминокислотной последовательности OsHPPD в качестве эталонного стандарта. crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsHPPD, выбранный из группы, состоящей из H141, L276, P277, N338, G342, R346, D370, P386, K418, G419 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке HPPD другого растения, который соответствует указанному выше аминокислотному сайту, и любую их комбинацию, с использованием аминокислотной последовательности OsHPPD в качестве эталонного стандарта. Давлением отбора предпочтительно является обработка бискарфентразоном.
В конкретном варианте осуществления, «первым геном-мишенью» является РРО, и «специфичный сайт гена» относится к сайту S128, V217, S223, V364, K373, L423, Y425 или W470 в аминокислотной последовательности белка Oryza sativa OsPPO1 (показанной в SEQ ID NO: 7, и последовательности генома, показанной в SEQ ID NO: 8), и аминокислотным сайтам в белке РРО другого растения, которые соответствуют вышеупомянутым аминокислотным сайтом, при использовании аминокислотной последовательности OsPPO1 в качестве эталонного стандарта. crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsPPO1, выбранный из группы, состоящей из S128, V217, S223, V364, K373, L423, Y425, W470, или любой их комбинации, и последовательность-мишень, содержащую последовательность вышеупомянутых аминокислотных сайтов, соответствующих белку РРО другого растения, и любую их комбинацию при использовании аминокислотной последовательности OsPPO1 в качестве эталонного стандарта. Давлением отбора предпочтительно является лечение сафлуфенацилом.
В конкретном варианте осуществления, «первым геном-мишенью» является TIR1, и «специфичный сайт гена» относится к сайт F93, F357, C413 или S448 в аминокислотной последовательности белка Oryza sativa OsTIR1 (показанной в SEQ ID NO: 9, и последовательности генома, показанной в SEQ ID NO: 10), и аминокислотным сайтам в белке TIR1 другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам при использовании аминокислотной последовательности OsTIR1 в качестве эталонного стандарта. crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsTIR1, выбранный из группы, состоящей из F93, F357, C413, S448 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке TIR1 другого растения, который соответствует указанному выше аминокислотному сайту, и любую их комбинацию, при использовании аминокислотной последовательности OsTIR1 в качестве эталонного стандарта. Давлением отбора предпочтительно является обработка 2,4-D.
Настоящее изобретение дополнительно представляет не трансгенную систему временного редактирования, использующую вышеупомянутый способ.
Настоящее изобретение дополнительно представляет использование вышеупомянутой не трансгенной системы временного редактирования в качестве маркера селекции.
Настоящее изобретение дополнительно представляет использование вышеупомянутой не трансгенной системы временного редактирования для лечения заболевания.
Настоящее изобретение дополнительно представляет использование вышеупомянутой не трансгенной системы временного редактирования в биологической селекции.
Настоящее изобретение дополнительно представляет генетически модифицированное растение, полученное вышеупомянутым способом, геном которого содержит событие редактирования первого гена-мишени, и генетически модифицированное растение получено не трансгенным способом.
Настоящее изобретение дополнительно представляет генетически модифицированное растение, полученное вышеупомянутым способом, геном которого содержит событие редактирования первого гена-мишени и дополнительно содержит, по меньшей мере, одно событие редактирования второго гена-мишени, и генетически модифицированное растение получено в не трансгенным способом.
Настоящее изобретение дополнительно представляет генетически модифицированное растение, полученное вышеупомянутым способом, геном которого содержит, по меньшей мере, одно второе событие редактирования гена-мишени, и генетически модифицированное растение получено не трансгенным способом, где первое событие редактирования гена-мишени было удалено путем генетического разделения.
Настоящее изобретение дополнительно представляет геном генетически модифицированного растения, полученного вышеупомянутым способом, где геном содержит: 1) событие редактирования первого гена-мишени; 2) событие редактирования первого гена-мишени и событие редактирования, по меньшей мере, одного второго гена-мишени; или 3), по меньшей мере, одно событие редактирования второго гена-мишени, где событие редактирования первого гена-мишени было удалено путем генетического разделения; где генетически модифицированное растение получено не трансгенным способом.
Другой аспект настоящего изобретения представляет новую мутацию гена растения, полученную вышеупомянутым способом.
Настоящее изобретение также представляет новую мутацию, созданную в растении, которая включает один или комбинацию двух или нескольких из следующих типов:
замена аспарагиновой кислоты в сайте, соответствующем Arabidopsis ALS376, на любую другую аминокислоту, замена триптофана в сайте, соответствующем Arabidopsis ALS574, на любую другую аминокислоту, замена серина в сайте, соответствующем Arabidopsis ALS653, на любую другую аминокислоту, или замена серина в сайте, соответствующем Arabidopsis ALS654, на любую другую аминокислоту; или замена триптофана в сайте, соответствующем Alopecurus myosuroides ACCase2027, на любую другую аминокислоту.
В конкретном варианте осуществления, в котором аспарагиновая кислота на сайте, соответствующем Arabidopsis ALS376, заменена глутаминовой кислотой (D376E), триптофан на сайте, соответствующем Arabidopsis ALS574, заменена лейцином или метионином (W574L или W574M), серин на сайте, соответствующем Arabidopsis ALS653, заменен аспарагином или аргинином (S653N или S653R), или глицин на сайте, соответствующем Arabidopsis ALS654, заменен аспарагиновой кислотой (G654D), где аминокислотные сайты указаны с использованием соответствующих аминокислотных сайтов в Arabidopsis thalianan в качестве эталона; или триптофан на сайте, соответствующем Alopecurus myosuroides АССаза2027, заменен лейцином или цистеином (W2027L или W2027C), где аминокислотный сайт указан с использованием соответствующего аминокислотного сайта в Alopecurus myosuroides в качестве эталона.
В другом конкретном варианте осуществления, типом мутации является S653R/G654D, где аминокислотные сайты указаны с использованием соответствующих аминокислотных сайтов в Arabidopsis thalianan в качестве эталона.
В конкретном варианте осуществления, аспарагиновая кислота на сайте 350 Oryza sativa ALS заменена любой другой аминокислотой, триптофан на сайте 548 Oryza sativa ALS заменен любой другой аминокислотой, или триптофан на сайте 561 Solanum tuberosum L. ALS 2 заменен любой другой аминокислотой; или триптофан на сайте 2038 Oryza sativa ACCase2 заменен любой другой аминокислотой.
В другом конкретном варианте осуществления, аспарагиновая кислота на сайте 350 Oryza sativa ALS заменена глутаминовой кислотой (D350E), триптофан на сайте 548 Oryza sativa ALS заменена лейцином или метионином (W548L или W548M), или триптофан на сайте 561 Solanum tuberosum L. ALS 2 заменен лейцином или метионином (W561L или W561M); или триптофан на сайте 2038 Oryza sativa АССаза2 заменен лейцином или цистеином (W2038L или W2038C), где аминокислотная последовательность белка Oryza sativa ALS показана в SEQ ID NO: 11, аминокислотная последовательность белка Solanum tuberosum L. StALS2 показана в SEQ ID NO: 19, и аминокислотная последовательность белка Oryza sativa ACCase2 показана в SEQ ID NO: 13.
Настоящее изобретение дополнительно представляет белок или его биологически активный фрагмент, который имеет вышеупомянутую новую мутацию.
Настоящее изобретение также представляет нуклеиновую кислоту, которая содержит последовательность нуклеиновой кислоты или комплементарную ей последовательность, которая кодирует белок или его биологически активный фрагмент.
Настоящее изобретение дополнительно представляет рекомбинантный вектор экспрессии, который содержит нуклеиновую кислоту и функционально связанный с ней промотор.
Настоящее изобретение дополнительно представляет кассету экспрессии, содержащую нуклеиновую кислоту.
Настоящее изобретение дополнительно представляет растительную клетку, содержащую кассету экспрессии.
Настоящее изобретение дополнительно представляет растение, регенерированное с использованием растительной клетки.
Другой аспект настоящего изобретения представляет способ получения растения с улучшенной резистентностью или толерантностью к гербицидам, который включает регенерацию растительной клетки в растение.
В другом аспекте настоящего изобретения, представлен способ борьбы с сорняками на участке выращивания растений, где растение включает указанное выше растение или растение, полученное указанным выше способом, где способ включает внесение на участок выращивания одного или нескольких гербицидов в эффективном количестве для борьбы с сорняками.
Другой аспект настоящего изобретения также представляет применение новой мутации, белка или его биологически активного фрагмента, нуклеиновой кислоты, рекомбинантного вектора экспрессии или кассеты экспрессии для повышения резистентности или толерантности растительной клетки, растительной ткани, части растения или растения к гербицидам.
Настоящее изобретение имеет следующие превосходные технические эффекты:
На основе последовательностей, созданных из нового события репарации, созданного последовательным редактированием, могут быть разработаны новые мишени, которые могут последовательно формировать мутации много раз на специфичном сайте генома, тем самым экспоненциально обогащая типы событий репарации после разрывов ДНК, и реализуя новые типы мутаций замен оснований, делеции и вставки, которые не могут быть получены путем редактирования одного гена. То есть, запрограммированная последовательная схема разрезания/редактирования, принятая в настоящем изобретении, которая использует последовательность, созданную из предыдущей репарации редактирования гена в качестве более поздней мишени редактирования гена, может наделить CRISPR/Cas новыми функциями редактирования одного основания и сайт-точной делеции и вставки через простой нокаут.
Изобретение может реализовать скрининг событий редактирования генов в отсутствие экзогенных маркеров, дополнительно реализовать не трансгенное редактирование генов и проводить эффективный скрининг событий редактирования, и может значительно уменьшить проблемы биологической безопасности способа в клеточной терапии и биологической селекции.
В частности, способ не трансгенного временного редактирования растений, представленный настоящим изобретением, вовлекает только Cas белок и искусственно синтезированные малые фрагменты gРНК или sgРНК, без участия экзогенной ДНК во всем процессе, и продуцирует эндогенные маркеры селекции резистентности путем редактирования первого гена-мишени через последовательное разрезание/редактирование, так что событие редактирования может быть эффективно подвергнуто скринингу, фактически без участия операций генетической модификации, и, таким образом, способ эквивалентен химическому мутагенезу или радиационно-индуцированной селекции, а также не требует непрерывного множественного создания сепарации и обнаружения экзогенных трансгенных компонентов, тем самым сокращая цикл селекции, обеспечивая биологическую безопасность, экономя затраты на надзор и регистрацию и открывая большие перспективы применения в точной селекции растений.
Подробное описание изобретения
В настоящем изобретении, если не указано иное, используемые здесь научные и технические термины имеют значения, обычно понятные специалистам в данной области техники. Кроме того, термины, связанные с химией белков и нуклеиновых кислот, молекулярной биологией, культурой клеток и тканей, микробиологией, иммунологией и лабораторными процедурами, используемые здесь, являются терминами и рутинными процедурами, широко используемыми в соответствующих областях техники. В то же время, чтобы лучше понять настоящее изобретение, ниже приведены определения и пояснения связанных терминов.
Используемый здесь термин «геном» относится ко всем дополнениям генетического материала (генам и не кодирующим последовательностям), присутствующим в каждой клетке или вирусе или органелле организма, и/или полному геному, унаследованному от родителя, как единице (гаплоидной).
Термин «редактирование генов» относится к стратегиям и методам таргетной специфической модификации любой генетической информации или генома живых организмов. Следовательно, этот термин включает редактирование областей, кодирующих ген, но также включает редактирование областей, отличных от областей, кодирующих ген, в геноме. Это также включает редактирование или модификацию другой генетической информации ядер (если они есть) и клеток.
Термин «CRISPR/Cas нуклеаза» может быть нуклеазой на основе CRISPR или последовательностью нуклеиновой кислоты, кодирующей ее, включая, но не ограничиваясь ими: 1) Cas9, включая SpCas9, ScCas9, SaCas9, xCas9, VRER-Cas9, EQR-Cas9, SpG-Cas9, SpRY-Cas9, SpCas9-NG, NG-Cas9, NGA-Cas9 (VQR) и т.д.; 2) Cas12, включая LbCpf1, FnCpf1, AsCpf1, MAD7 и т. д., или любой вариант или производное вышеупомянутой нуклеазы на основе CRISPR; предпочтительно, когда, по меньшей мере, одна нуклеаза на основе CRISPR содержит мутацию по сравнению с соответствующей последовательностью дикого типа, так что полученная нуклеаза на основе CRISPR распознает другую последовательность PAM. Используемый здесь термин «нуклеаза на основе CRISPR» является любой нуклеазой, которая была идентифицирована в существующей в природе системе CRISPR, которая впоследствии была выделена из своей природной среды и, предпочтительно, была модифицирована или объединена в представляющую интерес рекомбинантную конструкцию, подходящую в качестве инструмента для таргетного конструирования генома. Поскольку исходная нуклеаза на основе CRISPR дикого типа обеспечивает распознавание ДНК, т.е. свойства связывания, можно использовать любую нуклеазу на основе CRISPR и, необязательно, перепрограммировать или иным образом мутировать так, чтобы она подходила для различных вариантов осуществления изобретения.
Термин «CRISPR» относится к методу сиквенс-специфичных генетических манипуляций, который основан на кластеризованных регулярных промежуточных коротких палиндромных повторах, что отличается от РНК-интерференции, которая регулирует экспрессию генов на уровне транскрипции.
«Cas9 нуклеаза» и «Cas9» используются в настоящем документе взаимозаменяемо и относятся к РНК-управляемой нуклеазе, содержащей белок Cas9 или его фрагмент (например, белок, содержащий активный домен расщепления ДНК Cas9 и/или gРНК связывающий домен Cas9). Cas9 является компонентом системы редактирования генома CRISPR/Cas (кластеризованные регулярные промежуточные короткие палиндромные повторы и ассоциированные системы). Он может таргетировать и разрезать последовательности-мишени ДНК под руководством направляющей РНК с образованием двухцепочечных разрывов ДНК (DSB).
«Cas белок» или «Cas полипептид» относится к полипептиду, кодируемому геном Cas (CRISPR-ассоциированным). Cas белок включает Cas эндонуклеазу. Cas белок может быть бактериальным или простейшим белком. Например, белки CRISPR Cas типов с I - III в настоящем документе обычно происходят от прокариотов; белки Cas типа I и типа III могут быть получены из бактерий или видов простейших, и белок Cas типа II (т.е. Cas9) может быть получен из видов бактерий. «Cas белки» включают Cas9 белок, Cpf1 белок, C2c1 белок, C2c2 белок, C2c3 белок, Cas3, Cas3-HD, Cas5, Cas7, Cas8, Cas10, Cas12a, Cas12b или их комбинацию или комплекс.
«Вариант Cas9» или «вариант Cas9 эндонуклеазы» относится к варианту исходной Cas9 эндонуклеазы, где, будучи связанным с crРНК и tracРНК или с sgРНК, вариант Cas9 эндонуклеазы сохраняет способность распознавать, связываться со всей или частью последовательности ДНК-мишени и, необязательно, развертывать всю или часть последовательности ДНК-мишени, никовать всю или часть последовательности ДНК-мишени или разрезать всю или часть последовательности ДНК-мишени. Варианты Cas9 эндонуклеазы включают варианты Cas9 эндонуклеазы, описанные в настоящем документе, где варианты Cas9 эндонуклеазы отличаются от исходной Cas9 эндонуклеазы следующим образом: варианты Cas9 эндонуклеазы (в комплексе с gРНК с образованием полинуклеотид-направленного эндонуклеазного комплекса, способного модифицировать сайт-мишень) обладают, по меньшей мере, одним улучшенным свойством, таким как, но не ограничиваясь ими, повышенная эффективность трансформации, повышенная эффективность редактирования ДНК, пониженное разрезание вне мишени или любая их комбинация по сравнению с исходной Cas9 эндонуклеазой (в комплексе с той же gРНК с образованием полинуклеотидом-направленного эндонуклеазного комплекса, способного модифицировать один и тот же сайт-мишень).
Варианты Cas9 эндонуклеазы, описанные в настоящем документе, включают варианты, которые могут связываться и никовать сайты-мишени двухцепочечной ДНК, будучи ассоциированной с crРНК и tracrРНК или с sgРНК, в то время как исходная Cas эндонуклеаза может связываться с сайтом-мишенью и приводить к двухцепочечному разрыву (расщеплению), будучи ассоциированной с crРНК и tracrРНК или с sgРНК.
«Направляющая РНК» и «gРНК» используются в настоящем документе взаимозаменяемо и относятся к последовательности направляющей РНК, используемой для таргетирования конкретного гена для коррекции с использованием технологии CRISPR, которая обычно состоит из молекул crРНК и tracrРНК, частично комплементарных для образования комплекса, где crРНК содержит последовательность, которая имеет достаточную комплементарность с последовательностью-мишенью, чтобы гибридизоваться с последовательностью-мишенью и направлять комплекс CRISPR (Cas9+crРНК+tracrРНК) на специфическое связывание с последовательностью-мишенью. Однако в данной области техники известно, что можно сконструировать одну направляющую РНК (sgРНК), которая обладает свойствами как crРНК, так и tracrРНК.
Термины «одиночная направляющая РНК» и «sgРНК» используются в данном документе взаимозаменяемо и относятся к синтетическому слиянию двух молекул РНК, которое включает слияние crРНК (CRISPR РНК) вариабельного таргетирующего домена (связанного с последовательностью спаривания tracr, гибридизированной с tracrРНК) и tracrРНК (трансактивирующей CRISPR РНК). sgРНК может содержать crРНК или фрагменты crРНК и tracrРНК или фрагменты tracrРНК системы CRISPR/Cas типа II, которые могут образовывать комплекс с Cas эндонуклеазой типа II, где комплекс направляющей РНК/Cas эндонуклеазы может направлять Cas эндонуклеазу к сайту-мишени ДНК таким образом, чтобы Cas эндонуклеаза могла распознавать, необязательно связываться с сайтом-мишенью ДНК и необязательно никовать сайт-мишень ДНК или разрезать (вводить одноцепочечный или двухцепочечный разрыв) сайт-мишень ДНК.
В некоторых вариантах осуществления направляющие РНК и Cas9 могут быть доставлены в клетку в виде комплекса рибонуклеопротеина (RNP). RNP состоит из очищенного Cas9 белка в комплексе с gРНК, и в данной области техники хорошо известно, что RNP можно эффективно доставлять во многие типы клеток, включая, но не ограничиваясь ими, стволовые клетки и иммунные клетки (Addgene, Cambridge, MA, Mirus Bio LLC, Madison, WI).
Примыкающий к протоспейсеру мотив (PAM) в настоящем документе относится к короткой нуклеотидной последовательности, примыкающей к (таргетной) последовательности-мишени (преспейсеру), распознаваемой эндонуклеазной системой gРНК/Cas. Если последовательность ДНК-мишени не примыкает к соответствующей последовательности PAM, Cas эндонуклеаза не сможет успешно распознать последовательность ДНК-мишени. Последовательность и длина PAM в настоящем документе могут различаться в зависимости от используемого Cas белка или комплекса Cas белков. Последовательность PAM может быть любой длины, но обычно имеет длину 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 нуклеотидов.
Используемый в настоящем документе термин «организм» или «живой организм» включает животных, растения, грибы, бактерии и подобные.
Используемый в настоящем документе термин «клетка-хозяин» включает клетки растений, клетки животных, клетки грибов, клетки бактерий и подобные.
В настоящем изобретении, «животное» включает, но не ограничено ими, позвоночных, таких как человек, млекопитающие, отличные от человека, птицы, рыбы, рептилии, земноводные и т.д., а также беспозвоночных, таких как насекомые.
В настоящем изобретении, под «растением» следует понимать любой дифференцированный многоклеточный организм, способный осуществлять фотосинтез, в частности однодольные или двудольные растения, например, (1) пищевые культуры: Oryza spp., такие как Oryza sativa, Oryza latifolia, Oryza sativa, Oryza glaberrima; Triticum spp., такие как Triticum aestivum, T. Turgidumssp. durum; Hordeum spp., такие как Hordeum vulgare, Hordeum arizonicum; злаковые Secale; Avena spp., такие как Avena sativa, Avena fatua, Avena byzantine, Avena fatua var.sativa, Avena hybrida; Echinochloa spp., такие как Pennisetum glaucum, Sorghum, Sorghum bicolor, Sorghum vulgare, Triticale, Zea mays или Maize, Millet, Rice, Foxtail millet, Proso millet, Sorghum bicolor, Panicum, Fagopyrum spp., Panicum miliaceum, Setaria italica, Zizania palustris, Eragrostis tef, Panicum miliaceum, Eleusine coracana; (2) бобовые культуры: Glycine spp., такие как Glycine max, Soja hispida, Soja max, Vicia spp., Vigna spp., Pisum spp., конские бобы, Lupinus spp., Vicia, Tamarindus indica, Lens culinaris, Lathyrus spp., Lablab, кормовые бобы, маш, красная фасоль, нут; (3) масличные культуры: Arachis hypogaea, Arachis spp., Sesamum spp., Helianthus spp., такие как Helianthus annuus, Elaeis, такие как Eiaeis guineensis, Elaeis oleifera, соя, Brassicanapus, Brassica oleracea, Sesamum orientale, Brassica juncea, Oilseed rape, Camellia oleifera, масличная пальма, олива, клещевина, Brassica napus L., рапс; (4) волокнистые культуры: Agave sisalana, Gossypium spp. такие как Gossypium, Gossypium barbadense, Gossypium hirsutum, Hibiscus cannabinus, Agave sisalana, Musa textilis Nee, Linum usitatissimum, Corchorus capsularis L, Boehmeria nivea (L.), Cannabis sativa, Cannabis sativa; (5) плодовые культуры: Ziziphus spp., Cucumis spp., Passiflora edulis, Vitis spp., Vaccinium spp., Pyrus communis, Prunus spp., Psidium spp., Punica granatum, Malus spp., Citrullus lanatus, Citrus spp., Ficus carica, виды Fortunella, виды Fragaria, виды Crataegus, виды Diospyros, Eugenia unifora, Eriobotrya japonica, Dimocarpus longan, Carica papaya, Cocos spp., Averrhoa carambola, Actinidia spp., Prunus amygdalus, Musa spp. (musa acuminate), Persea spp. (Persea Americana), Psidium guajava, Mammea Americana, Mangifera indica, Canarium album (Oleaeuropaea), Caricapapaya, Cocos nucifera, Malpighia emarginata, Manilkara zapota, Ananas comosus, Annona spp., Citrus reticulate (Citrus spp.), Artocarpus spp., Litchi chinensis, Ribes spp., Rubus spp., груша, персик, абрикос, слива, красная восковница, лимон, кумкват, дуриан, апельсин, клубника, черника, дыня хами, мускусная дыня, финиковая пальма, ореховое дерево, вишня; (6) корневищные культуры: Manihot spp., Ipomoea batatas, Colocasia esculenta, горчица клубневая, Allium cepa (лук), eleocharis tuberose (водяной орех), Cyperus rotundus, Rhizoma dioscoreae; (7) овощные культуры: Spinacia spp., Phaseolus spp., Lactuca sativa, Momordica spp, Petroselinum crispum, Capsicum spp., Solanum spp. (например, Solanum tuberosum, Solanum integrifolium, Solanum lycopersicum), Lycopersicon spp. (например, Lycopersicon esculentum, Lycopersicon lycopersicum, Lycopersicon pyriforme), Macrotyloma spp., Kale, Luffa acutangula, чечевица, бамия, лук, картофель, артишоки, спаржа, брокколи, Brussels sprouts, капуста, морковь, цветная капуста, сельдерей, листовая капуста, кабачки, Benincasa hispida, Asparagus officinalis, Apiumgraveolens, Amaranthus spp., Allium spp., Abelmoschus spp., Cichorium endivia, Cucurbita spp., Coriandrum sativum, B.carinata, Rapbanus sativus, Brassica spp. (например, Brassica napus, Brassica rapa ssp., рапс, масличный рапс, турнепс, турнепс, листовая горчица, капуста, черная горчица, рапс (рапс), Brussels sprout, Solanaceae (баклажан), Capsicum annuum (сладкий перец), огурец, люффа, китайская капуста, рапс, капуста, калебас, китайский зеленый лук, лотос, корень лотоса, салат; (8) цветочные культуры: Tropaeolum minus, Tropaeolum majus, Canna indica, Opuntia spp., Tagetes spp., Cymbidium (орхидея), Crinum asiaticum L., Clivia, Hippeastrum rutilum, Rosa rugosa, Rosa Chinensis, Jasminum sambac, Tulipa gesneriana L., Cerasus sp., Pharbitis nil (L.) Choisy, Calendula officinalis L., Nelumbo sp., Bellis perennis L., Dianthus caryophyllus, Petunia hybrida, Tulipa gesneriana L., Lilium brownie, Prunus mume, Narcissus tazetta L., Jasminum nudiflorum Lindl., Primula malacoides, Daphne odora, Camellia japonica, Michelia alba, Magnolia liliiflora, Viburnum macrocephalum, Clivia miniata, Malus spectabilis, Paeonia suffruticosa, Paeonia lactiflora, Syzygium aromaticum, Rhododendron simsii, Rhododendron hybridum, Michelia figo (Lour.) Spreng., Cercis chinensis, Kerria japonica, Weigela florida, Fructus forsythiae, Jasminum mesnyi, Parochetus communis, Cyclamen persicum Mill., Phalaenophsis hybrid, Dendrobium nobile, Hyacinthus orientalis, Iris tectorum Maxim, Zantedeschia aethiopica, Calendula officinalis, Hippeastrum rutilum, Begonia semperflorenshybr, Fuchsia hybrida, Begonia maculataRaddi, Geranium, Epipremnum aureum; (9) лекарственные культуры: Carthamus tinctorius, Mentha spp., Rheum rhabarbarum, Crocus sativus, Lycium chinense, Polygonatum odoratum, Polygonatum Kingianum, Anemarrhena asphodeloides Bunge, Radix ophiopogonis, Fritillaria cirrhosa, Curcuma aromatica, Amomum villosum Lour., Polygonum multiflorum, Rheum officinale, Glycyrrhiza uralensis Fisch, Astragalus membranaceus, Panax ginseng, Panax notoginseng, Acanthopanax gracilistylus, Angelica sinensis, Ligusticum wallichii, Bupleurum sinenses DC., Datura stramonium Linn., Datura metel L., Mentha haplocalyx, Leonurus sibiricus L., Agastache rugosus, Scutellaria baicalensis, Prunella vulgaris L., Pyrethrum carneum, Ginkgo biloba L., Cinchona ledgeriana, Hevea brasiliensis (дикая), Medicago sativa Linn, Piper Nigrum L., Radix Isatidis, Atractylodes macrocephala Koidz; (10) сырьевые культуры: Hevea brasiliensis, Ricinus communis, Vernicia fordii, Morus alba L., Hops Humulus lupulus, Betula, Alnus cremastogyne Burk., Rhus verniciflua stokes; (11) пастбищные культуры: Agropyron spp., Trifolium spp., Miscanthus sinensis, Pennisetum sp., Phalaris arundinacea, Panicum virgatum, спороболусы, Indiangrass, Big bluestem grass, Phleum pratense, turf, Cyperaceae (Kobresia pygmaea, Carex pediformis, Carex humilis), Medicago sativa Linn, Phleum pratense L., Medicago sativa, Melilotus suavcolen, Astragalus sinicus, Crotalaria juncea, Sesbania cannabina, Azolla imbircata, Eichhornia crassipes, Amorpha fruticosa, Lupinus micranthus, Trifolium, Astragalus adsurgens pall, Pistia stratiotes linn, Alternanthera philoxeroides, Lolium; (12) сахарные культуры: Saccharum spp., Beta vulgaris; (13) культуры для производства напитков: Camellia sinensis, Camellia Sinensis, чай, кофе (Coffea spp.), Theobroma cacao, Humulus lupulus Linn.; (14) газонные растения: Ammophila arenararia, Poa spp. (Poa pratensis (мятлик)), Agrostis spp. (Agrostis matsumurae, Agrostis palustris), Lolium spp. (Lolium), Festuca spp. (Festuca ovina L.), Zoysia spp. (Zoysiajaponica), Cynodon spp. (Cynodon dactylon/bermudagrass), Stenotaphrum secundatum (Stenotaphrum secundatum), Paspalum spp., Eremochloa ophiuroides (эремохлоя змеехвостная), Axonopus spp. (мутовчатка), Bouteloua dactyloides (бизонова трава), Bouteloua var. spp. (Bouteloua gracilis), Digitaria sanguinalis, Cyperusrotundus, Kyllingabrevifolia, Cyperusamuricus, Erigeron canadensis, Hydrocotylesibthorpioides, Kummerowiastriata, Euphorbia humifusa, Viola arvensis, Carex rigescens, Carex heterostachya, дерн; (15) древесные культуры: Pinus spp., Salix spp., Acer spp., Hibiscus spp., Eucalyptus spp., Ginkgo biloba, Bambusa sp., Populus spp., Prosopis spp., Quercus spp., Phoenix spp., Fagus spp., Ceiba pentandra, Cinnamomum spp., Corchorus spp., Phragmites australis, Physalis spp., Desmodium spp., Populus, Hedera helix, Populus tomentosa Carr, Viburnum odoratissinum, Ginkgo biloba L., Quercus, Ailanthus altissima, Schima superba, Ilex pur-purea, Platanus acerifolia, ligustrum lucidum, Buxus megistophylla Levl., Dahurian larch, Acacia mearnsii, Pinus massoniana, Pinus khasys, Pinus yunnanensis, Pinus finlaysoniana, Pinus tabuliformis, Pinus koraiensis, Juglans nigra, Citrus limon, Platanus acerifolia, Syzygium jambos, Davidia involucrate, Bombax malabarica L., Ceiba pentandra (L.), Bauhinia blakeana, Albizia saman, Albizzia julibrissin, Erythrina corallodendron, Erythrina indica, Magnolia gradiflora, Cycas revolute, Lagerstroemia indica, хвойные, макрофанерофиты, Frutex; (16) орехоплодные культуры: Bertholletia excelsea, Castanea spp., Corylus spp., Carya spp., Juglans spp., Pistacia vera, Anacardium occidentale, Macadamia (Macadamia integrifolia), Carya illinoensis Koch, Macadamia, Pistachio, Badam, другие растения, которые дают орехи; (17) другие: arabidopsis thaliana, Brachiaria eruciformis, Cenchrus echinatus, Setaria faberi, eleusine indica, Cadaba farinose, водоросли, Carex elata, декоративные растения, Carissa macrocarpa, Cynara spp., Daucus carota, Dioscorea spp., Erianthus sp., Festuca arundinacea, Hemerocallis fulva, Lotus spp., Luzula sylvatica, Medicago sativa, Melilotus spp., Morus nigra, Nicotiana spp., Olea spp., Ornithopus spp., Pastinaca sativa, Sambucus spp., Sinapis sp., Syzygium spp., Tripsacum dactyloides, Triticosecale rimpaui, Viola odorata и подобные.
В конкретном варианте осуществления, растение выбирают из риса, кукурузы, пшеницы, сои, подсолнечника, сорго, рапса, люцерны, хлопка, ячменя, проса, сахарного тростника, томатов, табака, маниоки, картофеля, сладкого картофеля, пекинской капусты, белокочанной капусты, огурца, китайской розы, Scindapsus aureus, арбуза, дыни, клубники, черники, винограда, яблока, цитрусовых, персика, груши, банана и т. д.
Используемый в настоящем документе термин «растение» включает целое растение и любое потомство, клетку, ткань или часть растения. Термин «часть растения» включает любую часть растения, включая, например, но не ограничиваясь ими: семена (включая зрелые семена, незрелые зародыши без семенной оболочки и незрелые семена); черенки растений; растительную клетку; культуру растительных клеток; орган растения (например, пыльцу, зародыш, цветок, плод, почку, лист, корень, стебель и родственный эксплантат). Тканью растения или органом растения могут быть семена, каллюсная ткань или любая другая популяция растительных клеток, организованная в структурную или функциональную единицу. Растительная клетка или культура ткани может регенерировать растение, имеющее физиологические и морфологические характеристики растения, из которого получены клетка или ткань, и может регенерировать растение, имеющее по существу тот же генотип, что и растение. Напротив, некоторые растительные клетки не могут регенерировать растения. Регенерируемые клетки в растительных клетках или культурах тканей могут представлять собой зародыши, протопласты, меристематические клетки, каллюс, пыльцу, листья, пыльники, корни, кончики корней, шелк, цветки, зерна, колосья, початки, шелуху или стебли.
Части растения включают собираемые части и части, которые можно использовать для размножения растений-потомков. Части растения, которые можно использовать для размножения, включают, например, но не ограничены ими: семена; плоды; черенки; рассаду; клубни; и подвои. Собираемыми частями растений могут быть любые полезные части растений, включая, например, но не ограничиваясь ими: цветы; пыльца; рассада; клубни; листья; стебли; плоды; семена; и корни.
Клетки растений являются структурными и физиологическими единицами растений. В настоящем документе, растительные клетки включают протопласты и протопласты с частичными клеточными стенками. Растительные клетки могут быть в форме выделенных одиночных клеток или клеточных агрегатов (например, рыхлого каллюса и культивируемых клеток) и могут быть частью тканевых единиц более высокого порядка (например, растительных тканей, органов растений и растений). Следовательно, растительные клетки могут быть протопластами, клетками, продуцирующими гаметы, или клетками или набором клеток, способных регенерировать целое растение. Следовательно, в вариантах осуществления настоящего изобретения семя, содержащее множество растительных клеток и способное регенерировать в целое растение, рассматривается как «часть растения».
Используемый в настоящем документе термин «протопласт» относится к растительной клетке, у которой полностью или частично удалена клеточная стенка и открыта жировая двухслойная мембрана. Как правило, протопластом является выделенная растительная клетка без клеточной стенки, которая может регенерировать клеточную культуру или целое растение.
«Потомство» растения включает любые последующие поколения растения.
Термин «бактерии» означает всех прокариотов, включая все организмы царства Procaryotae. Термин «бактерия» включает все микроорганизмы, считающиеся бактериями, включая Mycoplasma, Chlamydia, Actinomyces, Streptomyce и Rickettsia. В это определение включены все формы бактерий, включая кокки, бациллы, спириллы, сферопласты, протопласты и т. д. Этот термин также включает прокариоты, которые являются грамотрицательными или грамположительными. «Грамотрицательный» и «грамположительный» означают картину окрашивания с использованием методов окрашивания по Граму, хорошо известных в данной области техники (см., например, Finegold and Martin, Diagnostic Microbiology, 6th Ed., CV Mosby St. Louis, pp. 13-15 [1982]). «Грамположительными бактериями» являются бактерии, которые могут сохранять исходный краситель, используемый для окрашивания по Граму, в результате чего окрашенные клетки под микроскопом имеют цвет от темно-синего до пурпурного. «Грамотрицательные бактерии» не сохраняют первоначальный краситель, используемый для окрашивания по Граму, но могут быть окрашены контр-окрашиванием. Поэтому грамотрицательные бактерии окрашивают в красный цвет после реакции окрашивания по Граму.
Используемый в настоящем документе термин «грибы» относится к эукариотическим организмам, таким как плесень и дрожжи, включая диморфные грибы.
Термины «толерантность к гербицидам» и «резистентность к гербицидам» могут использоваться взаимозаменяемо, и оба относятся к толерантности к гербицидам и резистентности к гербицидам. «Улучшение толерантности к гербицидам» и «улучшение резистентности к гербицидам» означают, что толерантность или резистентность к гербицидам улучшаются по сравнению с растением, содержащим ген дикого типа.
Термин «дикий тип» относится к молекуле нуклеиновой кислоты или белку, которые можно найти в природе.
В настоящем изобретении термин «участок культивирования» включает участок, где выращивают растение по настоящему изобретению, такой как почва, а также включает, например, семена растений, рассаду растений и выращенные растения. Термин «эффективное для борьбы с сорняками количество» относится к количеству гербицида, достаточному для воздействия на рост или развитие сорняка-мишени, например, для предотвращения или ингибирования роста или развития сорняка-мишени, или для уничтожения сорняка. Предпочтительно, эффективное для борьбы с сорняками количество не оказывает существенного влияния на рост и/или развитие семян растений, рассады растений или растений по настоящему изобретению. Специалисты в данной области техники могут определить такое эффективное количество для борьбы с сорняками с помощью обычных экспериментов.
Термин «ДНК-мишень», используемый в настоящем документе, относится к полинуклеотиду ДНК, содержащему «сайт-мишень» или «последовательность-мишень».
Термин «лизис» означает расщепление ковалентного остова молекулы ДНК. Лизис можно инициировать различными способами, включая, но не ограничиваясь ими, ферментативный или химический гидролиз фосфодиэфирных связей. Возможен и одноцепочечный, и двухцепочечный лизис, и двухцепочечный лизис может происходить из-за двух отдельных событий одноцепочечного лизиса. Лизис ДНК может привести к тупым или смещенным концам. В некоторых вариантах осуществления, для таргетного лизиса двухцепочечной ДНК используют комплекс, содержащий РНК, таргетирующую ДНК, и сайт-специфической полипептид модификации используют для таргетного лизиса двухцепочечной ДНК.
Термин «ген» включает фрагмент нуклеиновой кислоты, экспрессирующий функциональную молекулу (такую, как, но не ограниченную ими, специфичный белок), включая регуляторные последовательности до (5' не кодирующие последовательности) и после (3' не кодирующие последовательности) кодирующей последовательности.
Последовательностью ДНК, которая «кодирует» конкретную РНК, является последовательность нуклеиновой кислоты ДНК, которая может быть транскрибирована в РНК. Полинуклеотиды ДНК могут кодировать РНК (mРНК), которая может быть транслирована в белок, или полинуклеотиды ДНК могут кодировать РНК, которая не может быть транслирована в белок (например, tРНК, rРНК или ДНК-таргетную РНК; которые также известна как «не кодирующая» РНК или «ncРНК»).
Термины «полипептид», «пептид» и «белок» используются в настоящем изобретении взаимозаменяемо и относятся к полимеру аминокислотных остатков. Эти термины применяют к аминокислотным полимерам, в которых один или несколько аминокислотных остатков являются искусственными химическими аналогами соответствующих и встречающихся в природе аминокислот, а также к полимерам аминокислот природного происхождения. Термины «полипептид», «пептид», «аминокислотная последовательность» и «белок» могут также включать их формы модификации, включая, но не ограничиваясь ими, гликозилирование, липидную связь, сульфатирование, γ-карбоксилирование остатка глутаминовой кислоты, гидроксилирование и ADP-рибозилирование.
Термин «биологически активный фрагмент» относится к фрагменту, который имеет один или несколько аминокислотных остатков, удаленных с N- и/или C-конца белка, при этом сохраняющему свою функциональную активность.
Для терминов, связанных с аминокислотной заменой, используемых в описании, первая буква представляет встречающуюся в природе аминокислоту в определенном положении в определенной последовательности, следующее число представляет положение в соответствующей последовательности, и вторая буква представляет другую аминокислоту для замены встречающейся в природе аминокислоты. Например, W574L означает, что триптофан в положении 574 заменен лейцином. Для двойных или множественных мутаций каждая мутация отделяется знаком «/».
Термины «полинуклеотид» и «нуклеиновая кислота» используются взаимозаменяемо и включают ДНК, РНК или их гибриды, которые могут быть двухцепочечными или одноцепочечными.
Термины «нуклеотидная последовательность» и «последовательность нуклеиновой кислоты» относятся к последовательности оснований в ДНК или РНК.
Используемые в настоящем изобретении термины «кассета экспрессии», «вектор экспрессии» и «конструкция экспрессии» относятся к вектору, такому как рекомбинантный вектор, подходящему для экспрессии представляющей интерес нуклеотидной последовательности в растении. Термин «экспрессия» относится к продуцированию функционального продукта. Например, экспрессия нуклеотидной последовательности может относиться к транскрипции нуклеотидной последовательности (такой как транскрипция для получения mРНК или функциональной РНК) и/или трансляции РНК в предшественник или зрелый белок.
«Конструкцией экспрессии» по настоящему изобретению может быть фрагмент линейной нуклеиновой кислоты, кольцевая плазмида, вирусный вектор или, в некоторых вариантах осуществления, может быть РНК (такая как mРНК), которая может быть транслирована.
«Конструкция экспрессии» по настоящему изобретению может содержать представляющие интерес регуляторные последовательности и нуклеотидные последовательности из разных источников, или представляющие интерес регуляторные последовательности и нуклеотидные последовательности из одного и того же источника, но расположенные иначе, чем обычно встречающиеся в природе.
Термины «рекомбинантный вектор экспрессии» или «конструкция ДНК» используются в настоящем документе взаимозаменяемо и относятся к молекуле ДНК, содержащей вектор и, по меньшей мере, одну вставку. Рекомбинантные векторы экспрессии обычно получают с целью экспрессии и/или размножения вставки или для конструирования других рекомбинантных нуклеотидных последовательностей. Вставка может быть функционально или не функционально связана с промоторной последовательностью, и может быть функционально или не функционально связана с регуляторной последовательностью ДНК.
Термины «регуляторная последовательность» и «регуляторный элемент» могут использоваться взаимозаменяемо и относятся к нуклеотидной последовательности, расположенной выше (5' не кодирующая последовательность), в середине или ниже (3' не кодирующая последовательность) кодирующей последовательности, и влияет на транскрипцию, процессинг РНК, стабильность или трансляцию родственной кодирующей последовательности. Регуляторные элементы экспрессии растений относятся к нуклеотидным последовательностям, которые могут контролировать транскрипцию, процессинг РНК или стабильность или трансляцию представляющей интерес нуклеотидной последовательности в растениях.
Регуляторные последовательности могут включать, но не ограничены ими, промоторы, лидерные последовательности трансляции, интроны и последовательности распознавания поли А.
Термин «промотор» относится к фрагменту нуклеиновой кислоты, способному контролировать транскрипцию другого фрагмента нуклеиновой кислоты. В некоторых вариантах осуществления настоящего изобретения, промотором является промотор, способный контролировать транскрипцию гена в растительных клетках, независимо от того, происходит ли он из растительных клеток. Промотор может быть конститутивным промотором, или тканеспецифическим промотором, или промотором, регулируемым развитием, или индуцируемым промотором.
Термин «конститутивный промотор» относится к промотору, который обычно вызывает экспрессию гена в большинстве типов клеток в большинстве случаев. «Тканеспецифический промотор» и «тканепредпочтительный промотор» используются взаимозаменяемо и относятся к промотору, который в основном, но не обязательно исключительно, экспрессируется в ткани или органе, а также экспрессируется в конкретной клетке или типе клеток. «Промотор, регулируемый развитием» относится к промотору, активность которого определяется событием развития. «Индуцируемый промотор» отвечает на эндогенный или экзогенный стимул (окружающую среду, гормон, химический сигнал и т.д.) для селективной экспрессии функционально связанной последовательности ДНК.
Используемый в настоящем документе термин «функционально связанный» относится к соединению регуляторного элемента (например, но не ограничиваясь ими, последовательности промотора, последовательности терминации транскрипции и т.д.) с последовательностью нуклеиновой кислоты (например, кодирующей последовательностью или открытой рамкой считывания), так что транскрипция нуклеотидной последовательности контролируется и регулируется регуляторным элементом транскрипции. Методы функционального связывания области регуляторного элемента с молекулой нуклеиновой кислоты известны в данной области техники.
«Введение» молекулы нуклеиновой кислоты (такой как плазмида, линейный фрагмент нуклеиновой кислоты, РНК и т.д.) или белка в растение относится к трансформации клетки растения нуклеиновой кислотой или белком таким образом, чтобы нуклеиновая кислота или белок могли функционировать в растительной клетке. Термин «трансформация», используемый в настоящем изобретении, включает стабильную трансформацию и кратковременную трансформацию.
Термин «стабильная трансформация» относится к тому, что введение экзогенной нуклеотидной последовательности в геном растения приводит к стабильному наследованию экзогенного гена. После стабильной трансформации последовательность экзогенной нуклеиновой кислоты стабильно интегрируется в геном растения и любых его последующих поколений.
Термин «временная трансформация» относится к тому, что введение молекулы нуклеиновой кислоты или белка в растительную клетку для выполнения функции не приводит к стабильному наследованию чужеродного гена. При временной трансформации последовательность экзогенной нуклеиновой кислоты не интегрируется в геном растения.
Если не указано иное, все используемые в настоящем документе технические и научные термины имеют те же значения, которые обычно понимаются специалистами в области техники, к которой относится настоящее изобретение. Хотя любые способы и материалы, подобные или эквивалентные описанным в настоящем документе, также могут быть использованы при практическом применении или тестировании настоящего изобретения, теперь описаны предпочтительные способы и материалы.
Все публикации и патенты, цитируемые в этом описании, включены в настоящее описание посредством ссылки, как если бы каждая отдельная публикация или патент были точно и индивидуально указаны для включения посредством ссылки, и включены в настоящее описание посредством ссылки для раскрытия и описания методов и/или материалов, связанных с цитированными публикациями. Ссылка на любую публикацию, которая была опубликована до даты подачи заявки, не должна интерпретироваться как признание того, что настоящее изобретение не может предшествовать публикациям существующего изобретения. Кроме того, указанная дата публикации может отличаться от фактической даты публикации, что может потребовать независимой проверки.
Если специально не указано или не подразумевается, используемые в настоящем документе термины «а», «а/an» и «the» означают «по меньшей мере, один». Все патенты, патентные заявки и публикации, упомянутые или цитируемые в данном документе, включены в настоящий документ посредством ссылки во всей их полноте с той же степенью цитирования, как если бы они цитировались по отдельности.
Краткое описание чертежей
На фигуре 1 показана схематическая диаграмма способа получения новых мутаций в организмах по настоящему изобретению. На фигуре в качестве примера взят только Cas9 с NGG в качестве PAM; таким же образом можно использовать и другие варианты Cas9 с другим PAM (например, NG).
На фигуре 2 показана схема дизайна gРНК в сайтах W574 и S653 гена ALS Arabidopsis thaliana.
На фигуре 3 показано Т2 поколение Arabidopsis thaliana резистентных к гербициду штаммов, трансформированных двумя различными запрограммированными векторами последовательного разрезания/редактирования. pQY743 и pQY745 являются номерами векторов. Резистентные штаммы были способны нормально укореняться, но Col-0 дикого типа и не резистентные устойчивые штаммы не способны.
На фигуре 4 представлена диаграмма пиков секвенирования гена ALS штамма T2 поколения Arabidopsis thaliana, резистентного к имазапику, в котором T, обозначенный стрелкой, мутирован из G, что приводит к мутации W574L.
На фигуре 5 показан дизайн запрограммированной схемы последовательного разрезания/редактирования в сайте W574 гена ALS Arabidopsis thaliana. Последовательность Т-ДНК экспрессирует четыре гена, sgРНК1, sgРНК2, Cas9 и HygR. Среди них, sgРНК1 и Cas9 образуют комплекс, который разрезает кодон W574 ALS в геноме и, как ожидается, образует -G генотип посредством клеточной спонтанной репарации. Эта новая последовательность может быть распознана и разрезана комплексом, образованным sgРНК2 и Cas9, и формирует +T генотип посредством спонтанной клеточной репарации, приводящей к W574L.
На фигуре 6 показаны резистентная рассада Arabidopsis thaliana, после скрининга имазапиком и результаты секвенирования сайта ALS W574.
На фигуре 7 показан дизайн запрограммированной схемы последовательного разрезания/редактирования в сайте S653 гена ALS Arabidopsis thaliana. Последовательность Т-ДНК экспрессирует четыре гена, sgРНК1, sgРНК2, Cas9 и HygR. Среди них sgРНК1 и Cas9 образуют комплекс, разрезающий кодон S653 ALS в геноме. После спонтанной клеточной репарации формируется -G генотип. Эта последовательность может быть распознана и разрезана комплексом, образованным sgРНК2 и Cas9. После спонтанной клеточной репарации формируется +A генотип с получением S653N.
На фигуре 8 показана резистентная рассада Arabidopsis thaliana после скрининга имазапиком и результаты секвенирования сайта ALS S653. На левой панели показаны результаты скрининга резистентной рассады и доля резистентной рассады, и на правой панели показана диаграмма пиков секвенирования и типа мутации сайта S653.
На фигуре 9 показан дизайн запрограммированной схемы последовательного разрезания/редактирования в сайте W574 гена ALS Arabidopsis thaliana. Последовательность Т-ДНК экспрессирует четыре гена, sgРНК1, sgРНК2, Cas9 и HygR. Среди них sgРНК1 и Cas9 образуют комплекс, разрезающий кодон W574 ALS в геноме. После клеточной спонтанной репарации формируется +А генотип. Эта последовательность может быть распознана и разрезана комплексом, образованным sgРНК2 и Cas9. После спонтанной клеточной репарации формируется -G генотип с получением W574M. Для двух разрезов используют разные сайты PAM.
На фигуре 10 показан дизайн запрограммированной схемы последовательного разрезания/редактирования в сайте W2038 гена ACCase2 Oryza sativa. Этот сайт соответствует сайту W2027 гена ACCase2 Alopecurus myosuroides. Последовательность Т-ДНК экспрессирует четыре гена, sgРНК1, sgРНК2, Cas9 и HygR. Среди них sgРНК1 и Cas9 образуют комплекс, разрезающий кодон W2038 АССазы в геноме. Генотип A-G формируется после спонтанной клеточной репарации. Эта последовательность может быть распознана и разрезана комплексом, образованным sgРНК2 и Cas9. После клеточного спонтанного восстановления формируется +T генотип с получением W2038L.
На фигуре 11 показан резистентный каллюс Oryza sativa, подвергнутый совместному скринингу с гигромицином (50 мкг/л) и квизалофопом-р (50 мкг/л), и результаты секвенирования сайта W2038.
На фигуре 12 показана диаграмма электрофореза в полиакриламидном геле белков SpCas9 и NGA-Cas9, которые получены с помощью прокариотической экспрессии и очищены. Полоса, указанная стрелкой, является полосой белка Cas9.
На фигуре 13 показана in vitro активность разрезания очищенного белка Cas9 на фрагментах ДНК, содержащих сайт-мишень OsALS W548 и сайт-мишень OsACCase2 W2038, которые были обнаружены электрофорезом в агарозном геле, и можно увидеть, что только когда белок Cas9 и фрагмент sgРНК были добавлены одновременно, фрагменты ДНК можно было разрезать до ожидаемого размера.
На фигуре 14 показана диаграмма пиков секвенирования сайта-мишени OsALS W548 RNP-трансформированных протопластов Oryza sativa, и этот сайт соответствует сайту ALS W574 Arabidopsis thaliana. В месте, указанном стрелкой, в дополнение к исходному сигнальному пику G основания, имеется сигнальный пик T основания, полученный путем мутации, которая привела к мутации W548L.
На фигуре 15 показан резистентный каллюс Oryza sativa, отредактированный RNP на сайте OsACCase2 W2038 и подвергнутый скринингу с 50 мкг/л квизалофопа-p. Стрелка указывает на резистентный каллюс.
На фигуре 16 представлена диаграмма пиков секвенирования сайта-мишени OsACCase2 W2038 рассады Oryza sativa, дифференцированных из резистентного каллюса. Т, указанный стрелкой, мутирован из G, что приводит к мутации W2038L.
На фигуре 17 показан тест на резистентность рассады поколения T1, отредактированных OsACCase2 W2038L, к Галоксифопу-p. Слева направо: рассада № 1 показывает результаты контрольной обработки водой дикого типа Huaidao № 5 (сорт риса), и рассада № 2-4 показывает результаты дикого типа Huaidao No.5 и двух трансформированных RNP генной пушкой W2038L T1 поколения отредактированных штаммов QY367-7-12 и QY367-7-18, все которые обработаны 5 г/му активного ингредиента Галоксифоп-p (му, единица площади, 1 му=1/15 гектара). Очевидно, что у отредактированного штамма развилась резистентность к Галоксифопу-p.
На фигуре 18 показан тест на резистентность рассады поколения T1 обработанных OsACCase2 W2038L к квизалохоп-p. Слева направо: рассада № 1 показывает результаты контрольной обработки водой дикого типа Huaidao № 5, и рассада № 2-4 показывает результаты дикого типа Huaidao No.5 и двух трансформированных RNP генной пушкой W2038L T1 поколения отредактированных штаммов QY367-5-10 и QY367-5-21, все которые обработаны 5 г/му активного ингредиента квизалохопа-p. Очевидно, что у отредактированного штамма развилась резистентность к квизалохопу-p.
На фигуре 19 показан резистентный каллюс Oryza sativa, отредактированный RNP одновременно на сайте OsACCase2 W2038 и гене OsBADH2 и подвергнутый скринингу с использованием 50 мкг/л квизалофоп-p. Стрелка указывает на резистентный каллюс.
На фигуре 20 представлена диаграмма пиков секвенирования сайта-мишени OsACCase2 W2038 поколения T0 отредактированной на двойном сайте рассады. Т, указанный стрелкой, мутирован из G, что дает мутацию W2038L.
На фигуре 21 показана диаграмма пиков секвенирования сайта-мишени OsBADH2 поколения T0 отредактированной на двойном сайте рассады. +А гомозиготная мутация имеет место в точке, указанной стрелкой.
На фигуре 22 показан резистентный каллюс Oryza sativa, отредактированный RNP одновременно на сайте OsALS W548 и гене OsSWEET14, и подвергнутый скринингу с 5 мг/л пироксулама. Стрелка указывает на резистентный каллюс.
На фигуре 23 показана диаграмма пиков секвенирования сайта-мишени OsALS W548 поколения T0 отредактированной на двойном сайте рассады. Сигнальные пики обоих оснований G и T присутствуют в месте, указанном стрелкой, что дает мутацию W548L.
На фигуре 24 показана диаграмма пиков секвенирования сайта-мишени OsSWEET14 поколения T0 отредактированной на двойном сайте рассады. -С гомозиготная мутация имеет место в точке, указанном стрелкой.
На фигуре 25 показан тест резистентности рассады поколения T1 отредактированной на сайте OsALS W548 и двойном сайте OsSWEET14 рассады, к никосульфурону. Слева направо: рассада № 1 показывает результаты контрольной обработки водой Huaidao № 5 дикого типа, и рассада № 2-3 показывает результаты Huaidao No.5 дикого типа и трансформированного RNP генной пушкой поколения T1 W548L отредактированного штамма QY360-7-11, оба которых обработаны 4 г/му активного ингредиента никосульфурона. Видно, что у отредактированного штамма развилась резистентность к никосульфурону.
На фигуре 26 показан тест резистентности рассады поколения T1 отредактированной на сайте OsALS W548 и двойном сайте OsSWEET14 рассады к флукарбазону-Na. Слева направо: рассада № 1 показывает результаты контрольной обработки водой Huaidao № 5 дикого типа, и рассада № 2-3 показывает результаты Huaidao No.5 дикого типа и трансформированного RNP генной пушкой поколения T1 W548L отредактированного штамма QY360-7-9, оба которых обработаны 2 г/му активного ингредиента флукарбазона-Na. Видно, что у отредактированного штамма развилась резистентность к флукарбазону-Na.
На фигуре 27 показан тест резистентности рассады поколения T1 отредактированной на сайте OsALS W548 и двойном сайте OsSWEET14 рассады, к имазапику. Слева направо: рассада № 1 показывает результаты контрольной обработки водой Huaidao № 5 дикого типа, и рассада № 2-3 показывает результаты Huaidao No.5 дикого типа и трансформированного RNP генной пушкой поколения T1 W548L отредактированного штамма QY360-7-11, оба которых обработаны 7 г/му активного ингредиента имазапика. Видно, что у отредактированного штамма развилась резистентность к имазапику.
На фигуре 28 показан тест резистентности рассады поколения T1 отредактированной на сайте OsALS W548 и двойном сайте OsSWEET14 рассады, к пироксуламу. Слева направо: рассада № 1 показывает результаты контрольной обработки водой Huaidao № 5 дикого типа, и рассада № 2-4 показывает результаты Huaidao No.5 дикого типа и трансформированного RNP генной пушкой поколения T1 W548L отредактированных штаммов QY360-7-2 и QY360-7-11, все которые обработаны 2 г/му активного ингредиента пироксулама. Видно, что у отредактированного штамма развилась резистентность к пироксуламу.
На фиг. 29 показана схема запрограммированного последовательного разрезания/редактирования гена HBB в клетках 293T. А. Показан дизайн сайтов гена HBB для запрограммированного последовательного разрезания/редактирования в клетках 293T; B. Эффективность преобразования запрограммированного последовательного разрезания/редактирования гена HBB в клетках 293T после 48 часов трансформации каждого вектора редактирования; C. Соотношение типов редактирования генов, полученных из запрограммированного последовательного разрезания/редактирования гена HBB и разрезания/редактирования одного сайта гена HBB в клетках 293T; WT: дикий тип, indel: генотип для делеции или вставки, C->T SNP: генотип с заменой основания C на T при разрезании.
Описание списка последовательностей
Основные последовательности, задействованные в настоящем изобретении, суммированы следующим образом, и родственные последовательности представлены в списке последовательностей.
Варианты осуществления изобретения
Настоящее изобретение будет дополнительно объяснено в связи со следующими примерами. Следующие примеры проиллюстрированы посредством примеров, но объем правовой охраны настоящего изобретения не должен ограничиваться этими примерами. Экспериментальные способы в следующих примерах, если не указано иное, представляют собой способы, описанные в широко используемых руководствах по молекулярной биологии, технологии культуры тканей и агрономии. Например, конкретные стадии можно найти в: "Molecular Cloning: A Laboratory Manual (3rd edition)" (Sambrook, J., Russell, David W., 2001, Cold Spring Harbor), "Plant Propagation by Tissue Culture" (Edwin F. George , Michael A. Hall, Geert-Jan De Klerk, 2008, Springer). Материалы, реагенты, инструменты и т.д., используемые в следующих примерах, могут быть получены из коммерческих источников, если не указано иное.
Пример 1: Создание предсказуемых замен оснований, введенных запрограммированным последовательным разрезанием/редактированием для сайтов W574 и S653 гена ALS Arabidopsis thaliana
А. Экспериментальные материалы
1. Материал Arabidopsis thaliana
Arabidopsis thaliana дикого типа Col-0 является модельным сортом двудольного растения, его оригинальные семена предоставлены Department of Weeds, College of Plant Protection, China Agricultural University, и их размножение и сохранение проводят в нашей лаборатории в соответствии со стандартными способами в этой области техники.
2. Векторы
Векторные плазмиды pCBC-dT1T2 (Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ 2014. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biol. Nov 29;14(1):327. Более подробно см. в https://www.addgene.org/50590/), pHEE401E (см. Wang ZP, Xing HL, Dong L, Zhang HY, Han CY, Wang XC, Chen QJ Genome Biol. 2015 Jul 21; 16:144. doi: 10.1186/s13059-015-0715-0. Конкретную информацию см. в https://www.addgene.org/71287/) и pHEE401E-NG (мутацию, описанную в Nishimasu et al. 2018 Engineered CRISPR-Cas9 nuclease with expanded Targeting space. Science 361(6408):1259-1262. doi: 10.1126/science.aas9129, введенную в pHEE401E для создания вектора pHEE401E-NG, способного распознавать NG PAM), приобретают на веб-сайте Addgene или создают в нашей лаборатории в соответствии с обычными способами молекулярной биологии и хранят в нашей лаборатории.
3. Основное оборудование
Капельная пушка, водяная баня, прибор для ПЦР (Bio-rad T100), прибор для электрофореза (WIX-EP600), аппарат для визуализации на геле, электрическая взрывная сушилка, центрифуга (Eppendorf 5424R), высокопроизводительный lyser (не нашла адекватный перевод) тканей, шейкер, электронные весы, рН метр, и т.д.
4. Основные реагенты
ДНК полимераза с высокой точностью (приобретается у Tsingke Bio), набор для восстановления из агарозного геля и набор для выделения плазмид (приобретен у Sparkjade), BsaI и T4 ДНК лигазы (приобретены у NEB), Trans5α-компетентные клетки и EHA105-компетентные клетки (приобретены у TransGen Biotech, Beijing, China), GV3101 Agrobacterium-компетентные клетки (приобретены у Shanghai AngyuBio), Tris, EDTA, канамицин, цефалоспорин, гигромицин, агароза, порошковые дрожжи, триптон, NaCl (приобретен у Sangon Biotech), порошок MS, сахароза, Silwet-77, гигромицин (приобретен у Solarbio), краситель нуклеиновой кислоты (Dured), абсолютный этанол (приобретен у Sinopharm) и т. д.
5. Приготовление основных растворов
1) Дезинфицирующее средство для семян: 4 мл 10% SDS, 20 мл NaClO, добавляют воду до объема 200 мл.
2) Буфер для экстракции SDS: 40 мл 1M Tris⋅HCl (pH 8,0), 50 мл 0,1M EDTA, 10 мл 5M NaCl, 10 мл 10% SDS, добавляют воду до объема 200 мл.
3) Инфекционный раствор: 1,5 г сахарозы, 9 мкл Silwet-77, с добавлением 30 мл сверхчистой воды.
4) Твердая среда LB: 5 г порошковых дрожжей, 10 г триптона, 10 г NaCl, 15 г агара, добавляют воду до 1 л, стерилизуют при 121°C в течение 15 минут, выливают на планшет для последующего использования.
5) 50× исходный раствор TAE: 242 г Tris, 37,2 г Na2EDTA⋅2H2O, добавляют 800 мл ультрачистой воды, хорошо перемешивают до растворения, добавляют 57,1 мл уксусной кислоты, хорошо перемешивают, наконец разбавляют до 1 л деионизированной водой и хранят при комнатной температуре.
6) Твердая среда MS: взвешивают 4,42 г порошка MS и 10 г сахарозы, добавляют 800 мл сверхчистой воды, доводят pH до 5,8, добавляют воду до объема 1 л, добавляют 10 г фитагеля, стерилизуют при 121°C в течение 15 минут и выливают на планшет для последующего использования.
B. Способы эксперимента
1. Дизайн и конструирование вектора двойной мишени CRISPR/Cas9
1.1 Дизайн мишени
Последовательность гена ALS Arabidopsis thaliana представлена в SEQ ID NO: 2. Последовательность-мишень gРНК1 (5'-GCATGGTTATGCAATGGGA-3') из 19 оснований конструируют с использованием AGA рядом с сайтом ALS574 Arabidopsis thaliana в качестве PAM, по прогнозу, одно основание G будет удалено между первыми 3-4 сайтами PAM после редактирования, затем на основе последовательности, созданной при удалении, конструируют вторую последовательность-мишень gРНК2 (5'-GGCATGGTTATGCAATGGA-3'), и по прогнозу одно основание T будет вставлено посредством второго редактирования, тем самым осуществляя превращение TGG-TTG, как показано на фигуре 2.
Аналогично, последовательность-мишень gРНК3 (5'-TGCCGATGATCCCGAGTGG-3') из 19 оснований конструируют с использованием TGG рядом с сайтом ALS653 Arabidopsis thaliana в качестве PAM, по прогнозу одно основание G будет удалено между первыми 3-4 положениями PAM после редактирования, затем конструируют вторую последовательность-мишень gРНК4 (5'-TTGCCGATGATCCCGATGG-3') на основе последовательности, созданной в результате делеции, и по прогнозу одно основание А будет вставлено после второго редактирования, тем самым осуществляя превращение AGT-AAT, как показано на фигуре 2.
1.2 Векторная конструкция
Используют способ, описанный Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ 2014. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biol. Nov 29;14(1): 327. В частности, плазмиду dT1T2 используют в качестве матрицы для амплификации ALS574 и 653 сайтов фрагмента с двумя мишенями, соответственно, для конструирования кассеты экспрессии sgРНК. Векторные скелеты pHEE401E и pHEE401E-NG переваривают BsaI, полосы вырезают из геля и восстанавливают, и фрагмент-мишень непосредственно используют для реакции лигирования после перевара. Т4 ДНК лигазу используют для лигирования скелета вектора и фрагмента-мишени, продукты лигирования превращают в Trans5α-компетентные клетки, разные моноклоны отбирают для секвенирования. После подтверждения с помощью секвенирования, набор Sparkjade High Purity Plasmid Mini Extraction Kit используют для экстракции плазмид с получением рекомбинантных плазмид, которые были соответственно называют как pQY743 и pQY745.
2. Дизайн праймеров для обнаружения мишеней
Праймеры для обнаружения мишени берут сайты-мишени ALS574 и ALS653 в качестве центров, где праймер для восходящего обнаружения находится примерно в 100 п.н. от сайта-мишени ALS574, и праймер для нисходящего обнаружения находится примерно в 280 п.н. от сайта-мишени ALS653. Последовательности праймеров следующие: 574/653checking-F: 5'ATTGACGGAGATGGAAGCTT3' и 574/653checking-R: 5'CCAAACTGAGCCAGTCACAA3'.
3. Получение системы генетической трансформации Arabidopsis thaliana
3.1 Трансформация Agrobacterium
Сконструированные рекомбинантные плазмиды трансформируют в Agrobacterium GV3101-компетентные клетки для получения рекомбинантных клеток Agrobacterium.
3.2 Приготовление инфекционного раствора Agrobacterium
1) Активированные Agrobacterium собирают и инокулируют в 30 мл жидкой среды YEP (содержащей 25 мг/л Rif и 50 мг/л Kan) и культивируют при встряхивании при 200 об/мин и 28°С в течение ночи до значения OD600, равного примерно 1,0-1,5.
2) После центрифугирования при 6000 об/мин в течение 10 минут собирают Agrobacterium и отбрасывают супернатант.
3) Agrobacterium ресуспендируют в инфекционном растворе (нет необходимости регулировать pH) до OD600=0,8 для последующего использования.
3.3 Трансформация Arabidopsis thaliana
1) Перед трансформацией растения обращают внимание на то, хорошо ли росло растение, было ли пышно соцветие, не было ли реакции на стресс. Первую трансформацию проводят, когда высота растения составляет около 20 см. Полив осуществляют должным образом, когда почва высыхает. Выросшие стручки срезают ножницами за день до трансформации.
2) Соцветие трансформируемого растения погружают в вышеуказанный раствор на период от 30 секунд до 1 минуты при осторожном перемешивании, при этом на инфильтрированном растении должен быть слой жидкой мембраны.
3) После завершения трансформации растение культивируют в темноте в течение 24 часов, и затем вынимают и помещают в нормальную световую среду для роста.
4) Через неделю таким же образом можно провести вторую трансформацию.
3.4 Сбор семян
После созревания семян их собирают. После сбора семена повторно сушат в печи при 37°С в течение примерно одной недели.
4. Выбор трансгенного растения
Семена обрабатывают дезинфицирующим средством в течение 5 минут, 5 раз промывают деионизированной водой и равномерно распределяют на селекционной среде MS (содержащей 30 мкг/мл гигромицина, 100 мкг/мл цефалоспорина), среду помещают в световой инкубатор (температура 22°, 16 часов света, 8 часов темноты, интенсивность света 100-150 мкмоль/м2/с, влажность 75%), и через неделю положительную рассаду отбирают и пересаживают в почву.
5. Обнаружение мутантных растений T1
5.1 Экстракция геномной ДНК
1) Листья Arabidopsis thaliana срезают и помещают в 2 мл центрифужную пробирку, добавляют стальные шарики, и листья измельчают с помощью высокопроизводительного lyser ткани.
2) После завершения измельчения добавляют 400 мкл буфера для экстракции SDS, перемешивают перевертыванием и инкубируют на водяной бане при 65° в течение 15 минут и перемешивают перевертыванием каждые 5 минут.
3) Центрифугирование проводят при 13000 об/мин в течение 5 минут.
4) 300 мкл супернатанта отбирают пипеткой и переносят в новую 1,5 мл центрифужную пробирку, в центрифужную пробирку добавляют равный объем изопропанола, предварительно охлажденного до -20°С, и центрифужную пробирку выдерживают при -20°С в течение 1 часа или в течение ночи.
5) Центрифугирование проводят при 13000 об/мин в течение 10 минут, и супернатант отбрасывают.
6) В центрифужную пробирку добавляют 500 мкл 70% этанола для промывки лепешки, промывочный раствор удаляют после центрифугирования (будьте осторожны, чтобы не выбросить лепешку), сушат при комнатной температуре, добавляют 30 мкл сверхчистой воды для растворения ДНК, и ДНК хранят при -20°С.
5.2 ПЦР амплификация
Извлеченный геном растения T1 используют в качестве матрицы, определяющие праймеры используют для амплификации фрагмента-мишени, 5 мкл амплифицированного продукта отбирают пипеткой и определяют с помощью электрофореза в 1% агарозном геле, и визуализируют с помощью аппарата для визуализации на геле. Оставшийся продукт доставляют в компанию, занимающуюся секвенированием, для непосредственного проведения секвенирования.
6. Обнаружение мутантного растения Т2
После сбора семян штамма T1 с одного растения, выбирают семена разных штаммов двух векторов и высевают на среду для селекции с имазапиком (среда MS+0,24 мкг/мл имазапика) для проведения селекции, и положительную рассаду пересаживают в почву через неделю и подвергают молекулярному обнаружению, где способ такой же, как на стадии 5.
Используемые праймеры и их последовательности :
С. Результаты эксперимента
1. Определение генотипа растения T1
Семена T1 выбирают с помощью резистентной к гигромицину среды MS, всего 32 положительных сеянца получают для вектора pQY743 и всего 18 положительных сеянцев получают для вектора pQY745. Для каждого вектора отбирают 10 сеянцев и экстрагируют геномную ДНК из листьев для обнаружения сайта-мишени. Было обнаружено, что в поколении T1 на сайте ALS574 редактирование не происходит, и на сайте ALS653 происходят события редактирования, которые соответствуют ожиданиям проекта. Результаты обнаружения представлены в таблице 1:
2. Результаты селекции семян поколения Т2
После сбора семян поколения Т2 одного растения, их высевают на резистентную к имазапику среду для проведения селекции. Было видно, что Col-0 дикого типа не может расти на резистентной среде, в то время как положительные растения мутантных штаммов могут нормально расти на резистентной среде, как показано на фигуре 3.
Для каждого вектора отбирают 10 сеянцев и выделяют их геномную ДНК из листьев для молекулярного обнаружения. Было обнаружено, что для вектора pQY743 имеется 6 штаммов, которые имеют гомозиготную мутацию в соответствии с ожиданиями, т. е. мутацию от TGG к TTG, как показано на фигуре 4. Кроме того, имеется 1 штамм с гетерозиготной мутацией, и имеются 3 штамма с химерной мутацией. Результаты обнаружения представлены в таблице 2.
Вышеприведенные результаты показали, что с помощью технического решения запрограммированного последовательного разрезания/редактирования по настоящему изобретению можно достичь дизайна и реализации ожидаемых мутаций в сайте-мишени, а мутации замены оснований можно реализовать путем конструирования последовательных комбинаций sgРНК только с Cas9 белком.
Пример 2. Получение множественных типов мутаций путем запрограммированного последовательного разрезания/редактирования в сайтах W574 и S653 ALS гена Arabidopsis thaliana
Технологические стадии дизайна вектора, конструирования и трансформации и селекции Arabidopsis thaliana проводят согласно примеру 1. Для сайта AtALS W574 дизайн вектора был таким же, как и в примере 1. Схематическая диаграмма вектора показана на фигуре 5, и ожидается, что мутация W574L должна была быть реализована сначала -G, затем +T в специфичном сайте. Никаких событий редактирования не было обнаружено в поколении T1 вектора, трансформированного Arabidopsis thaliana. Поколение T2 трансгенного Arabidopsis thaliana выбирают с 0,24 мг/л имазапика, и получают большое количество резистентных к гербицидам растений, как показано на левой панели фигуры 6, проводят молекулярное обнаружение этих резистентных растений, результаты секвенирования продукта ПЦР показывают, что не только ожидаемая мутация W574L появляется в сайте W574, но также появляется другая резистентная мутация W574M, как показано на правой панели фигуры 6.
Для сайта AtALS S653, дизайн вектора такой же, как в примере 1. Диаграмма вектора показана на фигуре 7, и ожидается, что мутация S653N должна быть реализована сначала -G, затем +A в специфичном сайте. Ожидаемое событие редактирования S653N обнаружено в поколении T1 трансформированного вектором Arabidopsis thaliana. Поколение T2 трансгенного Arabidopsis thaliana непрерывно выбирают с 0,24 мг/л имазапика, и получают большое количество резистентных к гербицидам растений, как показано на левой панели фигуры 8, для этих резистентных растений проводят молекулярное обнаружение, результаты секвенирования продукта ПЦР показывают, что в сайте S653 произошла не только ожидаемая мутация S653N, но также произошли две другие резистентные мутации S653R и S653R/G654D, как показано на правой панели фигуры 8.
Вышеприведенные результаты показывают, что с помощью технического решения запрограммированного последовательного разрезания/редактирования по настоящему изобретению, в дополнение к ожидаемым мутациям, предназначенным для сайта-мишени, можно также создавать несколько типов функциональных мутаций путем конструирования последовательных комбинаций sgРНК только с соответствующим Cas9 белком, так что он является подходящим инструментом для создания новых функциональных мутаций.
Пример 3: Создание мутации резистентности W574M с использованием различных PAM для выполнения запрограммированного последовательного разрезания/редактирования рядом с сайтом W574 гена ALS Arabidopsis thaliana
Технологические стадии проектирования вектора, конструирования и трансформации и селекции Arabidopsis thaliana проводят в соответствии с примером 1, за исключением того, что последовательность 5'CTTGGCATGGTTATGCAATGgg3' рядом с сайтом AtALS W574, GG, расположенный ближе к сайту W574, используют первым в качестве PAM для конструирования sgРНК1: 5'CTTGGCATGGTTATGCAATG3', в котором сайт W574 был подчеркнут, и NG PAM выделен курсивом. По прогнозу, новая последовательность 5'CTTGGCATGGTTATGCAAATGGGAag3' должна образоваться с помощью +A после разрезания и репарации. AG используют в качестве нового сайта PAM для разработки sgРНК2: 5'CTTGGCATGGTTATGCAAATGGGA3', и генотип -G образуется после спонтанной репарации клеток, с получением W574M. То есть, разные сайты PAM используют для двух разрезов в этой схеме. Диаграмма вектора показана на фигуре 9. Вектор используют для трансформации Arabidopsis thaliana, определяют генотип трансгенного штамма поколения T1 подвергают, из чего определяют ожидаемое событие редактирования W574M, и растение демонстрирует резистентность к обработке имазапиком.
Результаты показали, что при использовании технического решения настоящего изобретения и использовании различных PAM для выполнения запрограммированного последовательного разрезания/редактирования, редактирование может быть выполнено в более широком диапазоне последовательностей с получением аминокислотной замены.
Пример 4. Разработка прогнозируемых замен оснований для сайтов D350 и W548 ALS гена Oryza sativa
Мутацию, описанную в Nishimasu et al. 2018 Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science 361(6408):1259-1262. doi: 10.1126/science.aas9129, вводят в pHUE411 (набор инструментов CRISPR/Cas9 для мультиплексного редактирования генома растений. Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ. BMC Plant Biol. 2014 Nov 29;14(1):327. 10.1186/s12870-014-0327-y, более подробно см. в https://www.addgene.org/71287/) для конструирования вектора pHUE411-NG, способного распознавать NG PAM.
Последовательность гена ALS Oryza sativa представлена в SEQ ID NO:12. Используя 5'GGCGTGCGGTTTGATGATCG3' (подчеркнутая часть является сайтом OSALS-D350, соответствующим Arabidopsis thaliana ALS-D376) и новую последовательность 5'GGCGTGCGGTTTGATGACG3', которая, по прогнозам должна быть получена в результате редактирования в качестве мишеней, конструируют вектор с двойной мишенью согласно способу, описанному Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ. BMC Plant Biol. 2014, и ожидают получить превращение GAT-GAA, приводящее к мутации OsALS D350E.
Используя 5'GGTATGGTTGTGCAATGGGA3' (подчеркнутая часть является сайтом OsALS-W548, соответствующим Arabidopsis thaliana ALS-W574) и новую последовательность 5'GGTATGGTTGTGCAATGGA 3', которая, по прогнозам должна быть получена в результате редактирования в качестве мишеней, конструируют вектор с двойной мишенью, и ожидают получить превращение TGG-TTG, приводящее к мутации OsALS W548L.
Затем два вектора переносят в Oryza sativa для получения трансгенных растений, и идентификация показывает, что получены растения с ожидаемыми заменами D350E и W548L. Результаты биотестирования резистентности к гербицидам в полевых условиях показали, что мутанты D350E и W548L приобрели резистентность к гербицидам-ингибиторам ALS.
Пример 5. Разработка прогнозируемых замен оснований и выбор множественных типов мутаций для W2038 сайта ACCase2 гена Oryza sativa
Последовательность гена ACCase2 Oryza sativa представлена в SEQ ID NO:14, в которой сайт OsACCase2 W2038 соответствует сайту ACCase W2027 Alopecurus myosuroides. AGG, близкий к этому сайту, используют в качестве PAM для конструирования sgРНК1: 5'TTCATCCTCGCTAAC-TGAG3', и по прогнозу новая последовательность будет образовываться посредством -G после разрезания и репарации. AGG непрерывно используют в качестве PAM для конструирования sgРНК2: 5'CTTC-ATCCTCGCTAACTGAG3', и +T генотип формируется после разрезания и репарации снова, что дает мутацию W2038L. sgРНК1 и sgРНК2 конструируют на векторе pHUE411 для получением вектора редактирования, и диаграмма вектора показана на фигуре 10.
Вектор редактирования используют для трансформации каллюса Huaidao № 5 (сорт риса), и после 3 недель совместной селекции с 50 мкг/л гигромицина и 50 мкг/л квизалопа-p получают большое количество резистентных каллюсов, как показано на левой панели фигуры 11. Резистентный каллюс берут для идентификации генотипа, и обнаруживают, что произошла не только ожидаемая мутация W2038L, но также и мутация W2038C, как показано на правой панели фигуры 11.
Приведенные выше результаты запрограммированного последовательного разрезания/редактирования гена Oryza sativa показали, что техническое решение настоящего изобретения применимо как к однодольным, так и к двудольным растениям.
Пример 6. Экспрессия и очистка белков SpCas9 и NGA-Cas9.
1. Экспериментальные приборы и реагенты
2. Экспериментальный способ
2.1 Конструирование вектора экспрессии pET15b-Cas9
Последовательности ДНК белков SpCas9 и NGA-Cas9, полученные после оптимизации растительных кодонов, показаны в SEQ ID NO: 15 и SEQ ID NO: 16 соответственно, и последовательности синтезируют с помощью GenScript в качестве матричных ДНК.
После раздельной амплификации последовательностей NG-Cas9 и NGA-Cas9 два фрагмента лигируют в вектор экспрессии pET15b способом инфузии, трансформируют в DH5a и секвенируют после проверки.
2.2 Экспрессия и очистка белков
Сконструированные векторы экспрессии трансформируют в Escherichia coli Rosetta (DE3), их экспрессию индуцируют с помощью IPTG, и бактерии собирают, лизируют и очищают на колонке Ni-NTA. Конкретный способ описан ниже:
а) Рекомбинантные векторы экспрессии трансформируют в штамм Rosetta (DE3), одну колонию собирают в 10 мл среды LB, резистентной к CmR+Amp (pET15b) или CmR+Kana (pET28a), культивируют при 200 об/мин при 37° в течение ночи, переносят в 2 л встряхиваемую колбу, содержащую 1 л среды LB, культивируют при 37°C и 200 об/мин до достижения OD600 d 0,6-0,8, охлаждают до 18°C, экспрессию индуцируют 0,5 мМ IPTG в течение ночи, и бактерии собирают центрифугированием при 4000g.
b) Собранные бактерии ресуспендируют в Ni-буфере А: 50 мМ HEPES, pH 7,4, 500 мМ NaCl, 20 мМ имидазола, 5 мМ β-меркаптоэтанола, с добавлением PMSF с конечной концентрацией 1 мМ и 250 мкл коктейля ингибиторов, и затем хорошо перемешивают.
с) после разрушения ресуспендированных клеток ультразвуковым дезинтегратором, их центрифугируют при 40000g в течение 30 минут при 4°C, супернатант собирают и пропускают через колонку Ni-NTA.
d) Очистка на колонке Ni-NTA: надосадочную жидкость лизата объединяют со смолой в течение 20 минут, элюируют буфером А, содержащим 50 мМ имидазола, для удаления примесей и, наконец, элюируют буфером для элюирования, содержащим 400 мМ имидазола.
е) SDS-PAGE систему гель-электрофореза используют для определения эффекта очистки белка.
f) Диализ проводят, заменяя буфер на 50 мМ HEPES pH 7,5, 150 мМ KCl, 1 мМ ДТТ, 3% глицерин.
h) Конечный образец подвергают SDS-PAGE гель-электрофорезу для определения эффекта очистки белка. После концентрации ультрафильтрацией белок замораживают при -80°С для последующего использования.
2.3 Результаты экспрессии и очистки слитых белков Cas9
Результаты очистки белков SpCas9 и NGA-Cas9 показаны на фигуре 12. Стрелки указывают на полосы белка Cas9, указывая на то, что была достигнута более высокая чистота.
Пример 7. In vitro активность расщепления белков SpCas9 и NGA Cas9, обнаруженная отдельно в отношении сайтов-мишеней OsACCase2 W2038 и OsALS W548
1. Последовательности ДНК генов OsALS и OsACCase2 вводят в онлайн-инструмент CRISPOR (http://crispor.tefor.net/), следующие sgРНК разрабатывают для W548 OsALS (548 аминокислота OsALS, аминокислотная последовательность белка ALS Oryza sativa представлена в SEQ ID NO: 11), соответствующей аминокислотному сайту 574 белка ALS Arabidopsis thaliana и W2038 OsACCase2 (2038 аминокислота OsACCase2, аминокислотная последовательность белка ACCase2 Oryza sativa показана в SEQ ID NO: 13), соответствующей аминокислотному сайту 2027 Alopecurus myosuroides, и эти sgРНК синтезируют с помощью GenScript:
>sgRNA1-548-G:
5’GGGUAUGGUUGUGCAAUGGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’
>sgRNA1-2038-G:
5’GUUCAUCCUCGCUAACUGGAGguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’
2. Специфические праймеры обнаружения, OsALS265AA-F: 5’ggtcttgcgtctggttggc3’ и OsALS-end-R: 5’ccatgccaagcacatcaaacaag3’, используют для амплификации фрагмента, содержащего сайт-мишень OsALS W548, и продукт ПЦР имеет длину 1200 п.н.
Специфические праймеры обнаружения, OsACC1750AA-F: 5'gcgaagaagactatgctcgtattgg3' и OsACC2196AA-R: 5'cttaatcacacctttcgcagcc3', используют для амплификации фрагмента, содержащего сайт-мишень OsACCase W2038, и продукт ПЦР имеет длину 1500 п.н.
Система ПЦР показана в следующей таблице:
3. Проводят реакцию ПЦР, и условия реакции показаны в следующей таблице:
4. Продукты ПЦР обнаруживают электрофорезом в агарозном геле и подвергают секвенированию для дальнейшей проверки. После правильной проверки гель разрезают для извлечения фрагментов ДНК. Восстановленные фрагменты ДНК растворяют, добавляя 30 мкл сверхчистой воды, не содержащей РНКазы, и измеряют их концентрацию.
5. Для определения активности белка Cas9 используют следующую систему обнаружения, и после того, как система приготовлена, ее инкубируют при 37°C в течение 1 часа.
6. После завершения реакции, систему обрабатывают при 65°C в течение 10 минут, добавляли 4 мкл 6× буфера для загрузки ДНК и запускают на 2% агарозном геле для определения размера полосы.
Результаты представлены на фигуре 13, и видно, что очищенные белки Cas9 могут разрезать двухцепочечную ДНК на сконструированных сайтах-мишенях только в присутствии sgРНК.
Пример 8: Получение мутации W548L в сайте OsALS548 путем трансформации протопласта Oryza sativa двумя различными таргетными RNP комплексами
1. Получение протопластов Oryza sativa и PEG-опосредованную трансформацию выполняют на основе частично модифицированного метода, который был опубликован (Bart et al., 2006), и конкретные стадии получения включают следующие:
(1) Сначала готовят рассаду Oryza sativa для протопластов. Сорт риса - Nipponbare. Сначала семена риса очищают от шелухи, и очищенные семена промывают 75% этанолом в течение 1 минуты, обрабатывают 5% (об./об.) гипохлоритом натрия в течение 20 минут, затем промывают стерильной водой более 5 раз, помещают на сверхчистый стол и высушивают обдуванием, и затем помещают в колбу для тканевых культур, содержащую 1/2 среды MS, по 20 семян в каждой колбе. Протопласты получают путем инкубации семян при 26°С и 12-часовом освещении в течение примерно 10 дней.
(2) Выбирают листовое влагалище рассады, разрезают острым лезвием бритвы Gillette на кусочки размером примерно 1 мм и помещают в 0,6М маннит и культуральную среду MES (состав: 0,6М маннит, 0,4М MES, рН 5,7) для последующего использования. После разрезания всех материалов их переносят в 20 мл раствора для ферментолиза (состав: 1,5% целлюлаза R10/RS (YaKult Honsha), 0,5% Мецерозим R10 (YaKult Honsha), 0,5М маннит, 20 мМ KCl, 20 мМ MES, pH5,7, 10 мМ CaCl2, 0,1% BSA, 5 мМ β-меркаптоэтанол), заворачивают в алюминиевую фольгу и помещают в шейкер при 28°. Ферментолиз проводят при 50 об/мин в темноте в течение примерно 4 часов, и последние 2 минуты скорость повышают до 100 об/мин;
(3) После ферментолиза добавляют равный объем раствора W5 (состав: 154 мМ NaCl, 125 мМ CaCl2, 5 мМ KCl, 15 мМ MES), встряхивают горизонтально в течение 10 секунд для высвобождения протопласта. Клетки, полученные в результате ферментолиза, фильтруют через сито 300 меш и центрифугируют при 150 g в течение 5 минут для сбора протопласта;
(4) Клетки дважды промывают раствором W5 и собирают протопласт центрифугированием при 150 g в течение 5 минут;
(5) Протопласт ресуспендируют с соответствующим количеством раствора MMG (состав: 3,05 г/л MgCl2, 1 г/л MES, 91,2 г/л маннита), и концентрация протопласта составляет около 2×106 клеток/мл.
2. Приготовление комплекса RNP:
В соответствии с последовательностью сайта-мишени OsALS548 GGGTATGGTTGTGCAATGGGAgga (сайт OsALS W548 подчеркнут, в соответствии с Arabidopsis thaliana ALS W574, и сайт PAM, распознаваемый Cas9 белком, показан курсивом в нижнем регистре), очищенный белок NGA-Cas9 выбирают для получения комплекса RNP. GGA используют в качестве PAM для конструирования >CrRNA1-548-G: 5'-GGGUAUGGUUGUGCAAUGGGAguuuuagagcuaugcu-3', было спрогнозировано, что одно основание G будет удалено между первыми 3-4 положениями PAM после редактирования, и затем последовательность, полученную в результате вышеуказанной делеции, используют для конструирования второй >CrRNA2-548+T: 5'-UGGGUAUGGUUGUGCAAUGGAguuuuagagcuaugcu-3', было спрогнозировано, что одно основание T будет вставлено после второго редактирования с получением превращения TGG-TTG.
>CrRNA1-548-G и >CrRNA1-548+T синтезированы компанией GenScript Biotechnology Company, и также синтезируют sgРНК:
>sgRNA1-548-G:
5’GGGUAUGGUUGUGCAAUGGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’
>sgRNA2-548+T:
5’UGGGUAUGGUUGUGCAAUGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’.
Синтезированные crРНК и GenCRISPR tracrРНК (GenScript SC1933) смешивают эквимолярно, добавляют буфер для отжига crРНК и tracrРНК (GenScript SC1957-B) и отжигают для получения gРНК в соответствии с инструкциями. Последовательностью tracrРНК является 5'-agcauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugcuuu-3'.
Реакционную систему RNP получают в соответствии со следующей таблицей, и после получения реакционной системы ее инкубируют при 25°С в течение 10 минут.
3. Трансформация протопластов
(1) 200 мкл ресуспендированного протопласта MMG, полученного выше, берут и добавляют комплекс RNP (20 мкг Cas9 белка, 20 мкг sgРНК), созданный после инкубации, и осторожно встряхивают для хорошего перемешивания.
(2) Равный объем 40% (масса/объем) раствора PEG (состав: 40% (масса/объем) PEG, 0,5М маннита, 100 мМ CaCl2), осторожно встряхивают для хорошего перемешивания и выдерживают при 28°С в темноте в течение 15 минут;
(3) После индукции и трансформации, медленно добавляют 1,5 мл раствора W5, осторожно взбалтывают для тщательного перемешивания клеток, и клетки собирают центрифугированием при 150 g в течение 3 минут. Эту стадию повторяют один раз;
(4) Клетки ресуспендируют добавлением 1,5 мл раствора W5, помещают в инкубатор при 28°C и культивируют в темноте в течение 12-16 часов. Клетки следует инкубировать от 48 до 60 часов, если они использовались для экстракции геномной ДНК протопластов.
4. Обнаружение события редактирования генома-мишени
(1) ДНК протопласта экстрагируют с использованием частично модифицированного способа CTAB, и этот способ конкретно описан следующим образом: протопласт центрифугируют, и супернатант отбрасывают, добавляют 500 мкл раствора для экстракции ДНК, тщательно встряхивают и перемешивают, и инкубируют на водяной бане при 65°C в течение 1 часа; образец охлаждают после инкубации на водяной бане, добавляют равный объем хлороформа, несколько раз переворачивают для тщательного перемешивания, и центрифугируют при 10000 об/мин в течение 10 минут; 400 мкл супернатанта берут и переносят в новую 1,5 мл центрифужную пробирку, добавляют 1 мл 70% (об./об.) этанола и выдерживают для осаждения при -20°С в течение 20 минут; ДНК осаждают центрифугированием при 12000 об/мин в течение 15 минут, осадок сушат на воздухе, затем растворяют, добавляя 50 мкл сверхчистой воды, и хранят при -20°С для дальнейшего использования.
(2) Ген-специфические праймеры используют для амплификации фрагмента, содержащего сайт-мишень W548, и для сайта-мишени конструируют следующие праймеры для выявления:
OsALS500AA-F: 5’GGCTAACCCAGGTGTCACAG3’,
OsALS-3’UTR-R: 5’CCATGCCAAGCACATCAAACAAG3’
Реакционная система ПЦР представлена в следующей таблице:
(3) Проводят реакцию ПЦР была установлена, и общие условия реакции показаны в следующей таблице:
(4) Обнаружение проводят электрофорезом в агарозном геле, и фрагменты ПЦР выделяют и секвенируют.
5. Результаты эксперимента:
Либо с помощью gРНК, полученной с синтетическими crРНК и tracrРНК путем отжига, либо непосредственно с использованием синтетической sgРНК, может быть получен активный комплекс RNP с очищенным NGA-Cas9 белком. Путем секвенирования сайта-мишени OsALS548, может быть обнаружена мутация из TGG в TTG, что демонстрирует то, что сайт-специфическая мутация сайта-мишени в клетке может быть достигнута путем запрограммированного последовательного разрезания/редактирования, созданного из комплекса RNP в комбинации с crРНК или sgРНК в последовательном порядке. Как показано на фигуре 14, в точке, указанной стрелкой на диаграмме пиков секвенирования протопласта OsALS548-мишени, в дополнение к исходному пику сигнала основания G, также имеется пик сигнала основания T, созданный в результате мутации, которая приводит к мутации W548L на сайте в OsALS548.
Пример 9. Получение мутации W2038L в сайте OsACCase2 W2038 путем бомбардировки каллюса Oryza sativa двумя различными таргетными комплексами RNP.
1. Получение комплексов RNP:
В соответствии с последовательностью сайта-мишени OsACCase2 W2038 GTTCATCCTCGCTAACTGGAGagg, в которой подчеркнут сайт OsACCase2 W2038, соответствующий Alopecurus myosuroides ACCase W2027, и сайт PAM, распознаваемый Cas9 белком, выделен курсивом строчными буквами, очищенный SpCas9 белок был выбран для получения RNP комплексов. GGA используют в качестве PAM для конструирования >CrRNA1-2038-G: 5'-GUUCAUCCUCGCUAACUGGAGguuuuagagcuaugcu-3', было спрогнозировано, что одно основание G будет удалено между первыми 3-4 положениями PAM после редактирования, затем последовательность, образовавшуюся при делеции, используют для конструирования второй CrRNA2-2038+T: 5'-UGUUCAUCCUCGCUAACUGAGguuuuagagcuaugcu-3', было спрогнозировано, что после второго редактирования будет вставлено одно основание Т, таким образом, получают превращение TGG-TTG.
>CrRNA1-2038-G и >CrRNA2-2038+T синтезированы компанией GenScript Biotechnology Company, и также синтезируют sgРНК:
>sgRNA1-2038-G:
5’GUUCAUCCUCGCUAACUGGAGguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc 3’
>sgRNA2-2038+T:
5’UGUUCAUCCUCGCUAACUGAGguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc 3’.
Синтезированные crРНК и GenCRISPR tracrРНК (GenScript SC1933) смешивают эквимолярно, добавляют буфер для отжига crРНК и tracrРНК (GenScript SC1957-B) и подвергают отжигу для получения gРНК в соответствии с инструкциями.
Комплексы RNP получают путем инкубации в той же реакционной системе, что и в примере 8. Берут количество 10 бомбардировок из генной пушки для трансформации, например: 20 мкг Cas9 белка, 20 мкг gРНК или sgРНК, 10 мкл 10× Cas9 реакционного буфера, общий объем доводят до 100 мкл ультрачистой водой, не содержащей РНКазы, инкубируют при 25°C в течение 10 минут и осторожно перемешивают.
2. Индукция каллюса Oryza sativa
Зрелые и разбухшие семена Oryza sativa отбирают, очищают от шелухи и дезинфицируют в соответствии со следующими стадиями:
(1) Семена риса (Oryza sativa) очищают от шелухи, сорт риса Huaidao No.5 куплен на рынке семян.
(2) Семена промывают стерильной водой неограниченное количество раз до тех пор, пока промытая вода не становится прозрачной.
(3) После стерилизации 70% спиртом в течение 1 минуты, семена помещают в 10% гипохлорит натрия на горизонтальном шейкере и инкубируют в течение 25 минут при встряхивании.
(4) После дезинфекции гипохлоритом натрия, семена 5 раз промывают стерильной водой. Семена высевают на среду для индукции каллюса (состав: порошок MS (4,42 г/л) + 2,4-D (2 мг/л) + сахароза (30 г/л) + фитагель (4 г/л)) и культивируют в темноте при 28°С для индукции каллюса.
3. RNP бомбардировка генной пушкой:
(1) Гипертоническая культивация
Каллюс с хорошей эмбриогенностью переносят на гипертоническую среду (состав: MS порошок (4,42 г/л) + 2,4-D (2 мг/л) + сахароза (30 г/л) + D-маннит (0,4 М)) + фитагель (4 г/л)), стерильную операцию проводят на ультрачистом лабораторном столе, и культивирование проводят в темноте при 25° в течение 4-6 часов.
(2) Получение суспензии золотого порошка: с помощью импортированной 1,5 мл пробирки EP взвешивают 30 мг золотого порошка (диаметр 0,6 мкм); добавляют 1 мл 70% этанола, тщательно встряхивают и выдерживают на льду в течение 10 минут. После центрифугирования в течение 1 минуты, супернатант удаляют; добавляют 1 мл стерильной воды, тщательно встряхивают, центрифугируют в течение 1 минуты, супернатант удаляют и повторяют описанную выше операцию 3 раза. Добавляют 500 мкл стерилизованного глицерина (50%), тщательно встряхивают для приготовления суспензии золотого порошка с концентрацией 60 мкг/мкл, которую хранят при -20°С.
(3) Берут 50 мкл суспензии золотого порошка (60 мкг/мл), добавляют 100 мкл полученного комплекса RNP и осторожно перемешивают.
(4) Берут 15 мкл смеси RNP/золотой порошок и помещают в центр расщепляемой мембраны настольной генетической пушки PDS-1000 (Bio-Rad), сушат обдувом, и затем подвергают бомбардировке в соответствии с инструкциями для инструмента.
Параметры бомбардировки: степень вакуума 26-28, расстояние 6 см, давление воздуха 1100 ф./кв.д. или 1350 ф./кв.д.
(5) После окончания бомбардировки, каллюс культивируют в гипертонической среде при 25°С в течение ночи (16 часов) в темноте.
4. Выбор, дифференциация и укоренение:
(1) После культивирования подвергнутого бомбардировке каллюса в течение ночи, его переносят в индукционную среду и культивируют для восстановления при 28°C в течение одной недели.
(2) После одной недели восстановления, каллюс переносят в среду для селекции (состав: 4,1 г/л N6 порошок+0,3 г/л гидролизованный казеин+2,8 г/л пролин+2 мг/л 2,4-D+3% сахароза+50 мкг/л квизалофоп-р+500 мг/л Cef (цефалоспорин) + 0,1 г/л инозит+0,35% фитогель, рН 5,8) для проведения селекции в течение 3-4 недель; для предполагаемого редактирования OsACCase2 W2038, для селекции используют 50 мкг/л квизалофопа-p, как показано на фигуре 15.
(3) Отобранный резистентный каллюс с хорошим статусом роста переносят на среду для дифференциации (состав: MS порошок (4,42 г/л) + KT (1 мг/л) + сахароза (30 г/л) + фитагель (4,5 г/л).) + 50 мкг/л квизалофоп-p, pH 5,8) и культивируют при 28° на свету для индукции дифференциации в течение 2-4 недель.
(4) Дифференцированную рассаду переносят на среду для укоренения (состав: 1/2 MS порошок (2,3 г/л) + сахароза (30 г/л) + фитагель (4,5 г/л)) для укоренения, рассаду закаляют после завершения укоренения, и затем переносят в горшки, наполненные почвой, и помещают в теплицу для выращивания.
5. Обнаружение событий редактирования мишени в резистентных каллюсах и рассаде культур тканей T0:
Всего при селекции получают 11 резистентных каллюсов, ДНК которых была экстрагирована способом СТАВ. Праймеры для выявления для сайта-мишени конструируют следующим образом: OsACC2038test-F: 5'CTGTAGGCATTTGAAACTGCAGTG3', OsACC2038test-R: 5'GCAATCCTGGAGTTCCT-CTGACC3', и ПЦР фрагмент, содержащий сайт OsACCase2 W2038, амплифицируют, выделяют и секвенируют. Обнаружение секвенирования показало, что 10 из них имели мутацию от TGG к TTG на сайте OsACCase2 W2038, где 3 образца являются гомозиготными мутантами.
Для рассады культуры ткани поколения Т0, полученной путем дифференциации резистентного каллюса, экстрагируют их ДНК для обнаружения последовательности сайта-мишени редактирования, и также обнаруживают гомозиготную мутацию из TGG в TTG на сайте OsACCase2 W2038, как показано на фигуре 16.
6. Тест на резистентность рассады поколения Т1 к гербицидам, ингибиторам АССазы.
После размножения штамма T0, содержащего мутацию W2038L, мутантную рассаду поколения T1 тестируют на резистентность к гербицидам с квизалофопом-p и галоксифопом-p в рабочих концентрациях. Можно увидеть, что мутантный штамм OsACCase2 W2038L обладает значительной резистентностью к этим двум гербицидам, ингибиторам АССазы, как показано на фигурах 17-18.
Таким образом, либо используя gРНК, полученную с синтетическими crРНК и tracrРНК путем отжига, либо непосредственно используя синтетическую sgРНК, активный комплекс RNP может быть образован с очищенным SpCas9 белком; каллюсы, подвергнутые бомбардировке генной пушкой, могут быть отобраны с использованием квизалофопа-p на стадии культуры ткани; гомозиготная мутация от TGG до TTG может быть обнаружена путем секвенирования сайта-мишени OsACCase2 W2038 рассады тканевой культуры поколения T0; мутация может быть унаследована в поколении T1 и показала резистентность к гербицидам, ингибиторам АССазы, что также демонстрирует то, что запрограммированное последовательное разрезание/редактирование, созданное из комплекса RNP в сочетании с последовательным таргетированием crРНК или sgРНК, может привести к сайт-специфической мутации сайта-мишени в клетке и может управлять продуцированием клеточно-эндогенных маркеров селекции для селекции тканевых культур, тем самым создавая резистентные к гербицидам культуры.
Пример 10: Различные таргетные комплексы RNP одновременно редактируют сайт OsACCase2 W2038 и ген OsBADH2 каллюса Oryza sativa
Комплекс RNP получают способом по примеру 8, процедуры бомбардировки генной пушкой и культуры ткани такие же, как в примере 9. Кроме crРНК или sgРНК, таргетирующих сайт OsACCase2 W2038, одновременно добавляют crРНК или sgРНК, таргетирующие ген OsBADH2, и их инкубируют с SpCas9 белком с образованием таргетного комплекса RNP для второго гена-мишени OsBADH2. Проводят бомбардировку генной пушкой, рассаду культуры тканей поколения T0 получают после выделения культуры, скрининга, дифференциации и укоренения, и поколение T1 получают путем размножения.
Последовательность генома Oryza sativa OsBADH2 представлена в SEQ ID NO: 17. Согласно онлайн-инструменту CRISPOR (http://crispor.tefor.net/) выбирают последовательность сайта-мишени CCAAGTACCTCCGCGCAATCGcgg, в котором сайт PAM, распознаваемый Cas9 белком, показан курсивом строчными буквами, и очищенный SpCas9 белок выбирают для получения RNP комплекса. CGG используют в качестве PAM для конструирования >CrRNA1-OsBADH2: 5’-CCAAGUACCUCCGCGCAAUCGguuuuagagcuaugcu-3’, было спрогнозировано, что резистентная мутация OsACCase2 W2038L и событие нокаутной мутации OsBADH2 могут быть одновременно обнаружены в резистентном каллюсе, полученном селекцией квизалофопа-p.
>CrRNA1-OsBADH2 был синтезирован GenScript Biotechnology Company, также синтезируют sgРНК:
>OsBADH2-sgRNA:5’CCAAGUACCUCCGCGCAAUCGguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’.
Резистентный каллюс отбирают в соответствии со стадиями трансформации в примере 9, как показано на фигуре 19, и резистентный каллюс отбирают и подвергают дифференциации и появлению проростков, и последовательности сайтов-мишеней OsACCase2 и OsBADH2 каллюса и рассады культуры тканей поколения Т0 секвенируют. Праймеры для выявления мишени OsBADH2 следующие:
OsBADH2-check F: 5’CATCGGTACCCTCCTCTTC3’
OsBADH2-check R: 5’ATCGATCGATTTGGGGCTCA3’
В результате, всего путем селекции получают 13 резистентных каллюсов, событие мутации OsACCase2 W2038L обнаруживают в 11 из них, и обнаружение последовательности-мишени OsBADH2 для этих 11 образцов каллюсов показывает, что 8 из них одновременно содержат событие редактирования в сайте-мишени OsBADH2. В рассаде культуры ткани поколения T0, полученной дифференциацией, обнаружено наличие мутации OsACCase2 W2038L и гомозиготной мутации OsBADH2 +A, как показано на фигурах 20-21.
В общем, после того, как каллюс риса был подвергнут бомбардировки генной пушкой с последовательным таргетированием crРНК- или sgРНК-таргетным RNP комплексом для проведения сайт-специфического редактирования OsACCase2 W2038 и одновременным добавлением таргетного RNP-комплекса, таргетирующего второй ген-мишень OsBADH2, селекция каллюса может быть проведена на стадии культуры ткани с квизалофопом-р; мутация OsACCase2 W2038 и таргетный нокаут OsBADH2 одновременно возникают в 61% резистентного каллюса, и штаммы, содержащие гомозиготную мутацию OsBADH2 обнаружены в рассаде культуры тканей поколения Т0. Это показывает, что последовательное разрезание/редактирование в сочетании с трансформацией RNP для сайт-специфического редактирования генов резистентности может создавать маркеры эндогенной селекции, и добавление соответствующего давления отбора может одновременно отбирать события редактирования второго гена-мишени, тем самым достигая сайт-специфического редактирования генома не трансгенными средствами.
Пример 11: Различные таргетные комплексы RNP одновременно редактируют сайт OsALS548 и ген OsSWEET14 каллюса Oryza sativa
Таргетные комплексы RNP для сайта OsALS548 получают способом по примеру 8, где последовательности crРНК и sgРНК такие же, как в примере 8, соответственно:
>CrRNA1-548-G: 5’-GGGUAUGGUUGUGCAAUGGGAguuuuagagcuaugcu-3’,
>CrRNA2-548+T: 5’-UGGGUAUGGUUGUGCAAUGGAguuuuagagcuaugcu-3’,
>sgRNA1-548-G:
5’GGGUAUGGUUGUGCAAUGGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’,
>sgRNA2-548+T:
5’UGGGUAUGGUUGUGCAAUGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’.
Согласно примеру 8, gРНК или sgРНК инкубируют с NGA-Cas9 для получения комплексов RNP, таргетирующих сайт OsALS548.
В дополнение к таргетному комплексу RNP для сайта OsALS548, одновременно добавляют crРНК или sgРНК для гена OsSWEET14, и формируют таргетный комплекс RNP для второго гена-мишени OsSWEET14 путем инкубации с SpCas9 белком. Рассаду тканевой культуры поколения T0 получают путем бомбардировки генной пушкой, выделения культуры, селекции, дифференциации, укоренения, и поколение T1 получают путем размножения. Давление отбора составляет 5 мг/л пироксулама.
Последовательность генома Oryza sativa OsSWEET14 представлена в SEQ ID NO: 18. Согласно онлайн-инструменту CRISPOR (http://crispor.tefor.net/) выбирают последовательность сайта-мишени GAGCTTAGCACCTGGTTGGAGggg, в которой сайты PAM, распознаваемые SpCas9 белком, показаны курсивом строчными буквами, и очищенный SpCas9 белок выбирают для получения комплекса RNP. GGG используют в качестве PAM для конструирования:
>CrRNA1-OsSWEET14::
5'-GAGCUUAGCACCUGGUUGGAGguuuuagagcuaugcu-3', и было спрогнозировано, что резистентная мутация OsALS W548L и нокаутная мутация OsSWEET14 могут быть одновременно обнаружены в резистентном каллюсе, полученном селекцией пироксулама.
>CrRNA1-Os SWEET14 был синтезирован компанией GenScript Biotechnology Company, и также синтезируют sgРНК:
>Os SWEET14-sgRNA:
5’GAGCUUAGCACCUGGUUGGAGguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’.
Резистентный каллюс выбирают в соответствии с процедурами бомбардировки генной пушкой и культуры ткани, как описано в примере 9, как показано на фигуре 22, и отбирают резистентный каллюс для проведения дифференциации и появления проростков, где состав среды для селекции следующий : 4,1 г/л порошка N6+0,3 г/л гидролизата казеина+2,8 г/л пролина+2 мг/л 2,4-D+3% сахарозы+5 мг/л пироксулама+500 мг/л Cef (цефалоспорина) + 0,1 г/л инозита+0,35% фитагеля, рН 5,8.
Последовательность с в сайте OsALS548 и сайте-мишени OsSWEET14 каллюса и проростках культуры ткани поколения T0 секвенируют. Праймеры для выявления сайта-мишени OsSWEET14 следующие:
OsSWEET14-check F: 5’ ATGGGTGCTGATGATTATCTTGTAT3’
OsSWEET14-check R: 5’ TGAAGAGACATGCCAGCCATTG3’
В результате, селекция дает 9 резистентных каллюсов, событие мутации OsALS W548L обнаруживают в 8 из них, и обнаружение последовательности-мишени OsSWEET14 в этих 8 образцах каллюсов показало, что 5 из них также содержат событие редактирования в сайте-мишени OsSWEET14. В рассаде культуры тканей поколения T0, полученных дифференциацией, можно обнаружить как мутацию OsALS W548L, так и гомозиготную мутацию OsSWEET14-C, как показано на фигурах 23-24.
После размножения штаммов T0, в которых была обнаружена мутация OsALS W548L, мутантные штаммы рассады поколения T1 тестируют на резистентность к гербицидам с пироксуламом, имазапиком, никосульфуроном и флукарбазоном-Na в рабочих концентрациях. Можно видеть, что мутантный штамм OsALS W548L проявляет значительную резистентность ко всем этим 4 гербицидам-ингибиторам ALS, как показано на фигурах. 25-28.
Таким образом, либо используя gРНК, полученную с синтетическими crРНК и tracrРНК путем отжига, либо непосредственно используя синтетическую sgРНК, активный комплекс RNP может быть образован с очищенным NGA Cas9 белком, сайт-специфическая мутация сайта-мишени в клетках может быть достигнута с помощью запрограммированного последовательного разрезания/редактирования, создаваемого из комплекса RNP в комбинации с последовательным таргетированием crРНК или sgРНК, селекция каллюса, бомбардированного генной пушкой, может быть выполнена с пироксуламом на стадии культуры ткани, мутация от TGG до TTG обнаруживается путем секвенирования сайта-мишени OsALS548 рассады культуры тканей поколения T0, и эта мутация могла быть унаследована поколением T1 и показала резистентность к гербицидам, ингибиторам ALS.
После бомбардировки каллюса риса генной пушкой с одновременным добавлением таргетного комплекса RNP, таргетирующего второй ген-мишень OsSWEET14, и с использованием SpCas9 белка, который распознает NGG PAM, проводят селекцию каллюса пироксуламом на стадии культуры ткани. Мутация OsALS W548L и таргетный нокаут OsSWEET14 происходят одновременно в 55% резистентного каллюса, появление гомозиготной мутации OsSWEET14 может быть обнаружено в рассаде культуры ткани поколения T0, что дополнительно указывает на то, что маркеры эндогенной селекции могут быть созданы запрограммированным последовательным разрезанием/редактированием в комбинации с сайт-специфическим редактированием резистентных генов, созданных при трансформации PNP, и события редактирования второго гена-мишени могут быть выбраны одновременно с одновременным использованием Cas9 белков, которые распознают разные сайты PAM, и добавлением соответствующего давления отбора, тем самым достигая сайт-специфического редактирования генома не трансгенными средствами.
Пример 12: Создание мутации W561L в сайте StALS561 при бомбардировке эксплантатов картофеля (Solanum tuberosum L.) двумя различными таргетными комплексами RNP
Аминокислотная последовательность белка StALS2 Solanum tuberosum L. показана в SEQ ID NO: 19, и последовательность гена StALS2 Solanum tuberosum L. показана в SEQ ID NO: 20. Способы получения комплексов RNP и бомбардировки генной пушкой описаны в примерах 8-9, и sgРНК для исходной последовательности и отредактированной последовательности, сконструированной для сайта StALS2W561 Solanum tuberosum L. StALS2, соответствующего сайту ALS574 Arabidopsis thaliana следующая:
>StALS561- G:
5’GGGAAUGGUGGUUCAGUGGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’
>StALS561 +T:
5’UGGGAAUGGUGGUUCAGUGGAguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugc3’,
И комплексы RNP получают в соответствии со способом, описанным в Примере 8.
Реципиентным сортом картофеля является Atlantic или Favorita, и его листья, стебли и пазушные почки используют, соответственно, в качестве эксплантатов. Способы бомбардировки генной пушкой и селекции и дифференциации следующие:
(1) Листья: весь лист разрезают и расправляют на гипертонической среде М6 (состав: 4,42 г/л порошок MS+1 мл/л витамины В5 (Phytotechlab, G219) + 30 г/л сахароза+8 г/л агар+2мг/л 2,4-D+0,8 мг/л зеатинрибозид+0,2 М маннит+250 мг/л Cef), для каждой бомбардировки используют около 5-6 листьев. После предварительной инкубации в течение 24 часов проводят бомбардировку золотым порошком. После бомбардировки их непрерывно культивируют в гипертонической среде М6 в темноте в течение 2 дней, затем переносят в среду М6 (4,42 г/л порошок MS+1 мл/л витамины B5 (Phytotechlab, G219) + 30 г/л сахароза+8 г/л агар+2мг/л 2,4-D+0,8 мг/л зеатинрибозид+250 мг/л Cef) и непрерывно культивируют в течение 1 недели, затем через 1 неделю переносят в среду M6 с давлением отбора, селекцию резистентного каллюса проводят с давлением отбора 20 мкг/л хлорсульфурона (состав: 4,42 г/л порошок MS+1 мл/л витамины B5 (Phytotechlab, G219) + 30 г/л сахароза+8 г/л агар+2 мг/л 2,4-D+0,8 мг/л зеатинрибозид+250 мг/л Cef+20 мкг/л хлорсульфурон), 4 недели спустя их переносят в среду для индукции глазков R4, имеющую давление отбора (состав: 4,42 г/л порошок MS+1 мл/л витамины B5 (Phytotechlab, G219) + 30г/л сахароза+8 г/л агар+2 мг/л GA3+0,8 мг/л зеатинрибозид+250 мг/л Cef+20 мкг/л хлорсульфурон) до появления всходов.
2) Стебли: берут стебли картофеля (без пазушных почек), разрезают их вдоль надрезом вверх и помещают на среду CIMI (4,42 г/л порошок MS+20 г/л сахароза+8 г/л агар+0,5 мг/л зеатиннуклеозид+2 мг/л 2,4-D+250 мг/л Cef+20 мкг/л хлорсульфурон), с последующей бомбардировкой генной пушкой, культивируют в темноте в течение 1 дня после бомбардировки, стебли переносят в свежую среду CIMI и непрерывно культивируют в течение 1 недели, затем стебли переносят в среду SIMI, имеющую давление отбора (состав: 4,42 г/л порошок MS+20 г/л сахароза+8 г/л агар+1 мг/л зеатинрибозид+0,1 мг/л GA3+250 мг/л Cef+20 мкг/л хлорсульфурон) для селекции резистентных почек до появления всходов.
(3) Пазушные почки: пазушные почки берут из стеблей картофеля, разрезают продольно и переносят в среду CIMI разрезом вверх, помещают в среду CIMI с последующей бомбардировкой генной пушкой, культивируют в темноте в течение 1 дня после бомбардировки, их переносят в свежую среду CIMI и непрерывно культивируют в течение 1 недели, и затем переносят в среду SIMI, содержащую давление отбора, для проведения селекции резистентных почек до появления всходов.
Праймеры для выявления сайта StALS2 W561:
StALS561-Check F: 5’GTGGATTAGGAGCAATGGGATTT3’
StALS561-Check R: 5’ TTATTTTAGATAATACAATGCCTCG3’
После обнаружения, событие редактирования W561L происходит в сайте StALS2 W561 подвергнутой скринингу на резистентность рассады культур ткани картофеля поколения T0, что демонстрирует, что способ не трансгенного временного редактирования генов, предложенный в настоящем изобретении, подходит для сельскохозяйственных культур, таких как картофель, от которых трудно отделить и удалить экзогенный трансгенный элемент путем самоопыления или гибридизации.
Пример 13: Успешное использование запрограммированной схемы последовательного разрезания/редактирования для замены оснований в клетках 293T человека
Отбирают ген HBB (бета-субъединица гемоглобина) в эмбриональной клетке почки человека 293T (его последовательность ДНК показана в SEQ ID NO: 21, его последовательность CDS показана в SEQ ID NO: 22, и его аминокислотная последовательность показана в SEQ ID NO: 23), сайт-мишень для последовательного разрезания/редактирования sgРНК конструируют в области первого экзона, в котором первой мишенью является catggtgcaCctgactcctgAGG. sgРНК, которая распознает эту мишень, названа sgHBB, и по прогнозам, делеция одного основания C может быть создана на sgРНК разрезе этого сайта. Второй мишенью является ccatggtgcatctgactctgAGG, которая распознает последовательность с делецией одного основания C в результате разрезания/редактирования первой мишени, и sgРНК этой мишени названа sgHBB-c. Конструируют sgРНК без сайта-мишени в клетках 293T и называют sgNOTAR, ее используют в качестве комплементарной плазмиды для трансфекции в эксперименте.
В соответствии с описанной выше конструкцией, синтезируют комплементарные одноцепочечные фрагменты ДНК, соответственно. После отжига их лигируют в плазмиду рх458 (addgene: 48138), переваренную ферментом BbsI, и трансформируют в компетентную E. coli DH5a. После подтверждения полученных одиночных колоний E. coli секвенированием, плазмиду экстрагируют и очищают с помощью набора для экстракции плазмиды без эндотоксина (Tiangen Bio).
Энергично растущие клетки 293T переваривают и выделяют с 0,05% трипсином (Gibico), разбавляют средой DMEM (10% эмбриональной телячьей сыворотки; двойная резистентность к пенициллину+стрептомицину) и инокулируют в 24-луночные культуральные планшеты и помещают в инкубаторе с диоксидом углерода на ночь. На следующий день, их смешивают отдельно с, согласно последовательному разрезанию/редактированию: плазмиды sgHBB и sgHBB-c по 0,5 мкг; разрезание/редактирование одной мишени: плазмиды sgHBB и sgNOTAR по 0,5 мкг каждая; без контроля мишени: плазмида pEGFP-c1 1 мкг. Трансформацию проводят липофектамином 3000 (Invitrogen). Делают 3 повтора для каждой группы.
Через 48 часов после трансформации получают изображения с помощью флуоресцентного микроскопа для регистрации эффективности трансформации, и общую ДНК из каждой лунки экстрагируют с помощью набора для экстрагирования нуклеиновых кислот (Omega).
Сконструированы следующие праймеры секвенирования Hi-tom:
Hi-HBB-F: gaggtgagtacggtgtgcGCTTACATTTGCTTCTGACACAACT;
Hi-HBB-R: gagttggatgctggatggTCTATTGGTCTCCTTAAACCTGTCTTG.
Эти праймеры используют для проведения ПЦР для каждого образца ДНК. Высокопроизводительное секвенирование продуктов ПЦР проводят с использованием способа Hi-tom (Sci China Life Sci. 2019 Jan;62(1):1-7. doi: 10.1007/s11427-018-9402-9).
Статистические данные результатов секвенирования представлены в таблице 6 и таблице 7. Эти данные указывают на то, что способ последовательного разрезания/редактирования на сайте HBB дал результат редактирования от C к T (что приводит к мутации P6S) в соотношении примерно 1,67%, в то время как способ разрезания/редактирования одной мишени не может привести к такой замене оснований.
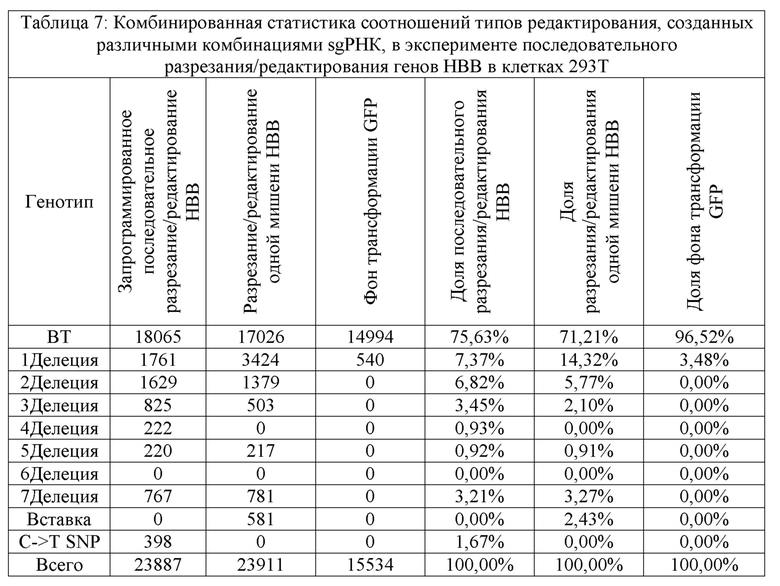
Примечание: WT представляет дикий тип; Делеция представляет генотип делеции; Вставка представляет генотип вставки; C->T SNP представляет генотип с заменой основания с C на T на разрезе; Всего представляет общий итог.
Кроме того, серповидно-клеточная анемия и β-талассемия являются наследственными анемиями, вызванными мутациями в гене HBB, кодирующем β-субъединицу гемоглобина взрослых. Пациентам с этими заболеваниями может потребоваться переливание крови или другие способы лечения на протяжении всей жизни. Результаты вышеуказанного эксперимента показали что посредством технического решения запрограммированного последовательного разрезания/редактирования, представленного в настоящем изобретении, можно сконструировать комбинацию crРНК или sgРНК для сайта мутации гена HBB, чтобы вызвать ожидаемую репарацию в сайте мутации, чтобы клетка продуцировала активный гемоглобин и восстановила функцию, тем самым достигнув терапевтического эффекта. То есть, композиция по настоящему изобретению может быть использована для лечения заболеваний.
После множества одновременных испытаний, поскольку этот новый способ полностью основан на существующих функциях Cas9, способ по настоящему изобретению будет полностью применим для достижения новых функций замены оснований, делеции и вставки специфических фрагментов в другие организмы (растения, животные, грибы или бактерии и т.д.), где Cas9 может хорошо работать.
Все публикации и заявки на патенты, упомянутые в описании, включены в настоящий документ посредством ссылки, как если бы каждая публикация или заявка на патент были индивидуально и специально включены в настоящий документ посредством ссылки.
Хотя вышеупомянутое изобретение было описано более подробно посредством примеров и вариантов осуществления для ясного понимания, очевидно, что определенные изменения и модификации могут быть осуществлены в пределах объема прилагаемой формулы изобретения, и все такие изменения и модификации входят в объем настоящего изобретения.
--->
Список последовательностей
<110> Qingdao Kingagroot Chemical Compound Co., Ltd.
<120> Способ создания новых мутаций в организмах и его применение
<130> IEC200334
<150> 201911081617X
<151> 2019-11-07
<150> 2020108218772
<151> 2020-08-15
<150> 2020109741512
<151> 2020-09-16
<160> 23
<170> PatentIn version 3.5
<210> 1
<211> 670
<212> БЕЛОК
<213> Аминокислотная последовательность белка ALS Arabidopsis thaliana
(аминокислотная последовательность)
<400> 1
Met Ala Ala Ala Thr Thr Thr Thr Thr Thr Ser Ser Ser Ile Ser Phe
1 5 10 15
Ser Thr Lys Pro Ser Pro Ser Ser Ser Lys Ser Pro Leu Pro Ile Ser
20 25 30
Arg Phe Ser Leu Pro Phe Ser Leu Asn Pro Asn Lys Ser Ser Ser Ser
35 40 45
Ser Arg Arg Arg Gly Ile Lys Ser Ser Ser Pro Ser Ser Ile Ser Ala
50 55 60
Val Leu Asn Thr Thr Thr Asn Val Thr Thr Thr Pro Ser Pro Thr Lys
65 70 75 80
Pro Thr Lys Pro Glu Thr Phe Ile Ser Arg Phe Ala Pro Asp Gln Pro
85 90 95
Arg Lys Gly Ala Asp Ile Leu Val Glu Ala Leu Glu Arg Gln Gly Val
100 105 110
Glu Thr Val Phe Ala Tyr Pro Gly Gly Ala Ser Met Glu Ile His Gln
115 120 125
Ala Leu Thr Arg Ser Ser Ser Ile Arg Asn Val Leu Pro Arg His Glu
130 135 140
Gln Gly Gly Val Phe Ala Ala Glu Gly Tyr Ala Arg Ser Ser Gly Lys
145 150 155 160
Pro Gly Ile Cys Ile Ala Thr Ser Gly Pro Gly Ala Thr Asn Leu Val
165 170 175
Ser Gly Leu Ala Asp Ala Leu Leu Asp Ser Val Pro Leu Val Ala Ile
180 185 190
Thr Gly Gln Val Pro Arg Arg Met Ile Gly Thr Asp Ala Phe Gln Glu
195 200 205
Thr Pro Ile Val Glu Val Thr Arg Ser Ile Thr Lys His Asn Tyr Leu
210 215 220
Val Met Asp Val Glu Asp Ile Pro Arg Ile Ile Glu Glu Ala Phe Phe
225 230 235 240
Leu Ala Thr Ser Gly Arg Pro Gly Pro Val Leu Val Asp Val Pro Lys
245 250 255
Asp Ile Gln Gln Gln Leu Ala Ile Pro Asn Trp Glu Gln Ala Met Arg
260 265 270
Leu Pro Gly Tyr Met Ser Arg Met Pro Lys Pro Pro Glu Asp Ser His
275 280 285
Leu Glu Gln Ile Val Arg Leu Ile Ser Glu Ser Lys Lys Pro Val Leu
290 295 300
Tyr Val Gly Gly Gly Cys Leu Asn Ser Ser Asp Glu Leu Gly Arg Phe
305 310 315 320
Val Glu Leu Thr Gly Ile Pro Val Ala Ser Thr Leu Met Gly Leu Gly
325 330 335
Ser Tyr Pro Cys Asp Asp Glu Leu Ser Leu His Met Leu Gly Met His
340 345 350
Gly Thr Val Tyr Ala Asn Tyr Ala Val Glu His Ser Asp Leu Leu Leu
355 360 365
Ala Phe Gly Val Arg Phe Asp Asp Arg Val Thr Gly Lys Leu Glu Ala
370 375 380
Phe Ala Ser Arg Ala Lys Ile Val His Ile Asp Ile Asp Ser Ala Glu
385 390 395 400
Ile Gly Lys Asn Lys Thr Pro His Val Ser Val Cys Gly Asp Val Lys
405 410 415
Leu Ala Leu Gln Gly Met Asn Lys Val Leu Glu Asn Arg Ala Glu Glu
420 425 430
Leu Lys Leu Asp Phe Gly Val Trp Arg Asn Glu Leu Asn Val Gln Lys
435 440 445
Gln Lys Phe Pro Leu Ser Phe Lys Thr Phe Gly Glu Ala Ile Pro Pro
450 455 460
Gln Tyr Ala Ile Lys Val Leu Asp Glu Leu Thr Asp Gly Lys Ala Ile
465 470 475 480
Ile Ser Thr Gly Val Gly Gln His Gln Met Trp Ala Ala Gln Phe Tyr
485 490 495
Asn Tyr Lys Lys Pro Arg Gln Trp Leu Ser Ser Gly Gly Leu Gly Ala
500 505 510
Met Gly Phe Gly Leu Pro Ala Ala Ile Gly Ala Ser Val Ala Asn Pro
515 520 525
Asp Ala Ile Val Val Asp Ile Asp Gly Asp Gly Ser Phe Ile Met Asn
530 535 540
Val Gln Glu Leu Ala Thr Ile Arg Val Glu Asn Leu Pro Val Lys Val
545 550 555 560
Leu Leu Leu Asn Asn Gln His Leu Gly Met Val Met Gln Trp Glu Asp
565 570 575
Arg Phe Tyr Lys Ala Asn Arg Ala His Thr Phe Leu Gly Asp Pro Ala
580 585 590
Gln Glu Asp Glu Ile Phe Pro Asn Met Leu Leu Phe Ala Ala Ala Cys
595 600 605
Gly Ile Pro Ala Ala Arg Val Thr Lys Lys Ala Asp Leu Arg Glu Ala
610 615 620
Ile Gln Thr Met Leu Asp Thr Pro Gly Pro Tyr Leu Leu Asp Val Ile
625 630 635 640
Cys Pro His Gln Glu His Val Leu Pro Met Ile Pro Ser Gly Gly Thr
645 650 655
Phe Asn Asp Val Ile Thr Glu Gly Asp Gly Arg Ile Lys Tyr
660 665 670
<210> 2
<211> 2013
<212> ДНК
<213> ДНК последовательность гена ALS Arabidopsis thaliana (ДНК
последовательность)
<400> 2
atggcggcgg caacaacaac aacaacaaca tcttcttcga tctccttctc caccaaacca 60
tctccttcct cctccaaatc accattacca atctccagat tctccctccc attctcccta 120
aaccccaaca aatcatcctc ctcctcccgc cgccgcggta tcaaatccag ctctccctcc 180
tccatctccg ccgtgctcaa cacaaccacc aatgtcacaa ccactccctc tccaaccaaa 240
cctaccaaac ccgaaacatt catctcccga ttcgctccag atcaaccccg caaaggcgct 300
gatatcctcg tcgaagcttt agaacgtcaa ggcgtagaaa ccgtattcgc ttaccctgga 360
ggtgcatcaa tggagattca ccaagcctta acccgctctt cctcaatccg taacgtcctt 420
cctcgtcacg aacaaggagg tgtattcgca gcagaaggat acgctcgatc ctcaggtaaa 480
ccaggtatct gtatagccac ttcaggtccc ggagctacaa atctcgttag cggattagcc 540
gatgcgttgt tagatagtgt tcctcttgta gcaatcacag gacaagtccc tcgtcgtatg 600
attggtacag atgcgtttca agagactccg attgttgagg taacgcgttc gattacgaag 660
cataactatc ttgtgatgga tgttgaagat atccctagga ttattgagga agctttcttt 720
ttagctactt ctggtagacc tggacctgtt ttggttgatg ttcctaaaga tattcaacaa 780
cagcttgcga ttcctaattg ggaacaggct atgagattac ctggttatat gtctaggatg 840
cctaaacctc cggaagattc tcatttggag cagattgtta ggttgatttc tgagtctaag 900
aagcctgtgt tgtatgttgg tggtggttgt ttgaattcta gcgatgaatt gggtaggttt 960
gttgagctta cggggatccc tgttgcgagt acgttgatgg ggctgggatc ttatccttgt 1020
gatgatgagt tgtcgttaca tatgcttgga atgcatggga ctgtgtatgc aaattacgct 1080
gtggagcata gtgatttgtt gttggcgttt ggggtaaggt ttgatgatcg tgtcacgggt 1140
aagcttgagg cttttgctag tagggctaag attgttcata ttgatattga ctcggctgag 1200
attgggaaga ataagactcc tcatgtgtct gtgtgtggtg atgttaagct ggctttgcaa 1260
gggatgaata aggttcttga gaaccgagcg gaggagctta agcttgattt tggagtttgg 1320
aggaatgagt tgaacgtaca gaaacagaag tttccgttga gctttaagac gtttggggaa 1380
gctattcctc cacagtatgc gattaaggtc cttgatgagt tgactgatgg aaaagccata 1440
ataagtactg gtgtcgggca acatcaaatg tgggcggcgc agttctacaa ttacaagaaa 1500
ccaaggcagt ggctatcatc aggaggcctt ggagctatgg gatttggact tcctgctgcg 1560
attggagcgt ctgttgctaa ccctgatgcg atagttgtgg atattgacgg agatggaagc 1620
tttataatga atgtgcaaga gctagccact attcgtgtag agaatcttcc agtgaaggta 1680
cttttattaa acaaccagca tcttggcatg gttatgcaat gggaagatcg gttctacaaa 1740
gctaaccgag ctcacacatt tctcggggat ccggctcagg aggacgagat attcccgaac 1800
atgttgctgt ttgcagcagc ttgcgggatt ccagcggcga gggtgacaaa gaaagcagat 1860
ctccgagaag ctattcagac aatgctggat acaccaggac cttacctgtt ggatgtgatt 1920
tgtccgcacc aagaacatgt gttgccgatg atcccgagtg gtggcacttt caacgatgtc 1980
ataacggaag gagatggccg gattaaatac tga 2013
<210> 3
<211> 2320
<212> БЕЛОК
<213> Аминокислотная последовательность белка ACCase Alopecurus
myosuroides (аминокислотная последовательность)
<400> 3
Met Gly Ser Thr His Leu Pro Ile Val Gly Phe Asn Ala Ser Thr Thr
1 5 10 15
Pro Ser Leu Ser Thr Leu Arg Gln Ile Asn Ser Ala Ala Ala Ala Phe
20 25 30
Gln Ser Ser Ser Pro Ser Arg Ser Ser Lys Lys Lys Ser Arg Arg Val
35 40 45
Lys Ser Ile Arg Asp Asp Gly Asp Gly Ser Val Pro Asp Pro Ala Gly
50 55 60
His Gly Gln Ser Ile Arg Gln Gly Leu Ala Gly Ile Ile Asp Leu Pro
65 70 75 80
Lys Glu Gly Ala Ser Ala Pro Asp Val Asp Ile Ser His Gly Ser Glu
85 90 95
Asp His Lys Ala Ser Tyr Gln Met Asn Gly Ile Leu Asn Glu Ser His
100 105 110
Asn Gly Arg His Ala Ser Leu Ser Lys Val Tyr Glu Phe Cys Thr Glu
115 120 125
Leu Gly Gly Lys Thr Pro Ile His Ser Val Leu Val Ala Asn Asn Gly
130 135 140
Met Ala Ala Ala Lys Phe Met Arg Ser Val Arg Thr Trp Ala Asn Asp
145 150 155 160
Thr Phe Gly Ser Glu Lys Ala Ile Gln Leu Ile Ala Met Ala Thr Pro
165 170 175
Glu Asp Met Arg Ile Asn Ala Glu His Ile Arg Ile Ala Asp Gln Phe
180 185 190
Val Glu Val Pro Gly Gly Thr Asn Asn Asn Asn Tyr Ala Asn Val Gln
195 200 205
Leu Ile Val Glu Ile Ala Glu Arg Thr Gly Val Ser Ala Val Trp Pro
210 215 220
Gly Trp Gly His Ala Ser Glu Asn Pro Glu Leu Pro Asp Ala Leu Thr
225 230 235 240
Ala Lys Gly Ile Val Phe Leu Gly Pro Pro Ala Ser Ser Met Asn Ala
245 250 255
Leu Gly Asp Lys Val Gly Ser Ala Leu Ile Ala Gln Ala Ala Gly Val
260 265 270
Pro Thr Leu Ala Trp Ser Gly Ser His Val Glu Ile Pro Leu Glu Leu
275 280 285
Cys Leu Asp Ser Ile Pro Glu Glu Met Tyr Arg Lys Ala Cys Val Thr
290 295 300
Thr Ala Asp Glu Ala Val Ala Ser Cys Gln Met Ile Gly Tyr Pro Ala
305 310 315 320
Met Ile Lys Ala Ser Trp Gly Gly Gly Gly Lys Gly Ile Arg Lys Val
325 330 335
Asn Asn Asp Asp Glu Val Lys Ala Leu Phe Lys Gln Val Gln Gly Glu
340 345 350
Val Pro Gly Ser Pro Ile Phe Ile Met Arg Leu Ala Ser Gln Ser Arg
355 360 365
His Leu Glu Val Gln Leu Leu Cys Asp Glu Tyr Gly Asn Val Ala Ala
370 375 380
Leu His Ser Arg Asp Cys Ser Val Gln Arg Arg His Gln Lys Ile Ile
385 390 395 400
Glu Glu Gly Pro Val Thr Val Ala Pro Arg Glu Thr Val Lys Glu Leu
405 410 415
Glu Gln Ala Ala Arg Arg Leu Ala Lys Ala Val Gly Tyr Val Gly Ala
420 425 430
Ala Thr Val Glu Tyr Leu Tyr Ser Met Glu Thr Gly Glu Tyr Tyr Phe
435 440 445
Leu Glu Leu Asn Pro Arg Leu Gln Val Glu His Pro Val Thr Glu Ser
450 455 460
Ile Ala Glu Val Asn Leu Pro Ala Ala Gln Val Ala Val Gly Met Gly
465 470 475 480
Ile Pro Leu Trp Gln Ile Pro Glu Ile Arg Arg Phe Tyr Gly Met Asp
485 490 495
Asn Gly Gly Gly Tyr Asp Ile Trp Arg Lys Thr Ala Ala Leu Ala Thr
500 505 510
Pro Phe Asn Phe Asp Glu Val Asp Ser Gln Trp Pro Lys Gly His Cys
515 520 525
Val Ala Val Arg Ile Thr Ser Glu Asn Pro Asp Asp Gly Phe Lys Pro
530 535 540
Thr Gly Gly Lys Val Lys Glu Ile Ser Phe Lys Ser Lys Pro Asn Val
545 550 555 560
Trp Gly Tyr Phe Ser Val Lys Ser Gly Gly Gly Ile His Glu Phe Ala
565 570 575
Asp Ser Gln Phe Gly His Val Phe Ala Tyr Gly Glu Thr Arg Ser Ala
580 585 590
Ala Ile Thr Ser Met Ser Leu Ala Leu Lys Glu Ile Gln Ile Arg Gly
595 600 605
Glu Ile His Thr Asn Val Asp Tyr Thr Val Asp Leu Leu Asn Ala Pro
610 615 620
Asp Phe Arg Glu Asn Thr Ile His Thr Gly Trp Leu Asp Thr Arg Ile
625 630 635 640
Ala Met Arg Val Gln Ala Glu Arg Pro Pro Trp Tyr Ile Ser Val Val
645 650 655
Gly Gly Ala Leu Tyr Lys Thr Ile Thr Thr Asn Ala Glu Thr Val Ser
660 665 670
Glu Tyr Val Ser Tyr Leu Ile Lys Gly Gln Ile Pro Pro Lys His Ile
675 680 685
Ser Leu Val His Ser Thr Ile Ser Leu Asn Ile Glu Glu Ser Lys Tyr
690 695 700
Thr Ile Glu Ile Val Arg Ser Gly Gln Gly Ser Tyr Arg Leu Arg Leu
705 710 715 720
Asn Gly Ser Leu Ile Glu Ala Asn Val Gln Thr Leu Cys Asp Gly Gly
725 730 735
Leu Leu Met Gln Leu Asp Gly Asn Ser His Val Ile Tyr Ala Glu Glu
740 745 750
Glu Ala Gly Gly Thr Arg Leu Leu Ile Asp Gly Lys Thr Cys Leu Leu
755 760 765
Gln Asn Asp His Asp Pro Ser Arg Leu Leu Ala Glu Thr Pro Cys Lys
770 775 780
Leu Leu Arg Phe Leu Ile Ala Asp Gly Ala His Val Asp Ala Asp Val
785 790 795 800
Pro Tyr Ala Glu Val Glu Val Met Lys Met Cys Met Pro Leu Leu Ser
805 810 815
Pro Ala Ala Gly Val Ile Asn Val Leu Leu Ser Glu Gly Gln Ala Met
820 825 830
Gln Ala Gly Asp Leu Ile Ala Arg Leu Asp Leu Asp Asp Pro Ser Ala
835 840 845
Val Lys Arg Ala Glu Pro Phe Glu Gly Ser Phe Pro Glu Met Ser Leu
850 855 860
Pro Ile Ala Ala Ser Gly Gln Val His Lys Arg Cys Ala Ala Ser Leu
865 870 875 880
Asn Ala Ala Arg Met Val Leu Ala Gly Tyr Asp His Ala Ala Asn Lys
885 890 895
Val Val Gln Asp Leu Val Trp Cys Leu Asp Thr Pro Ala Leu Pro Phe
900 905 910
Leu Gln Trp Glu Glu Leu Met Ser Val Leu Ala Thr Arg Leu Pro Arg
915 920 925
Arg Leu Lys Ser Glu Leu Glu Gly Lys Tyr Asn Glu Tyr Lys Leu Asn
930 935 940
Val Asp His Val Lys Ile Lys Asp Phe Pro Thr Glu Met Leu Arg Glu
945 950 955 960
Thr Ile Glu Glu Asn Leu Ala Cys Val Ser Glu Lys Glu Met Val Thr
965 970 975
Ile Glu Arg Leu Val Asp Pro Leu Met Ser Leu Leu Lys Ser Tyr Glu
980 985 990
Gly Gly Arg Glu Ser His Ala His Phe Ile Val Lys Ser Leu Phe Glu
995 1000 1005
Glu Tyr Leu Ser Val Glu Glu Leu Phe Ser Asp Gly Ile Gln Ser Asp
1010 1015 1020
Val Ile Glu Arg Leu Arg Leu Gln Tyr Ser Lys Asp Leu Gln Lys Val
1025 1030 1035 1040
Val Asp Ile Val Leu Ser His Gln Gly Val Arg Asn Lys Thr Lys Leu
1045 1050 1055
Ile Leu Ala Leu Met Glu Lys Leu Val Tyr Pro Asn Pro Ala Ala Tyr
1060 1065 1070
Arg Asp Gln Leu Ile Arg Phe Ser Ser Leu Asn His Lys Arg Tyr Tyr
1075 1080 1085
Lys Leu Ala Leu Lys Ala Ser Glu Leu Leu Glu Gln Thr Lys Leu Ser
1090 1095 1100
Glu Leu Arg Thr Ser Ile Ala Arg Asn Leu Ser Ala Leu Asp Met Phe
1105 1110 1115 1120
Thr Glu Glu Lys Ala Asp Phe Ser Leu Gln Asp Arg Lys Leu Ala Ile
1125 1130 1135
Asn Glu Ser Met Gly Asp Leu Val Thr Ala Pro Leu Pro Val Glu Asp
1140 1145 1150
Ala Leu Val Ser Leu Phe Asp Cys Thr Asp Gln Thr Leu Gln Gln Arg
1155 1160 1165
Val Ile Gln Thr Tyr Ile Ser Arg Leu Tyr Gln Pro Gln Leu Val Lys
1170 1175 1180
Asp Ser Ile Gln Leu Lys Tyr Gln Asp Ser Gly Val Ile Ala Leu Trp
1185 1190 1195 1200
Glu Phe Thr Glu Gly Asn His Glu Lys Arg Leu Gly Ala Met Val Ile
1205 1210 1215
Leu Lys Ser Leu Glu Ser Val Ser Thr Ala Ile Gly Ala Ala Leu Lys
1220 1225 1230
Asp Ala Ser His Tyr Ala Ser Ser Ala Gly Asn Thr Val His Ile Ala
1235 1240 1245
Leu Leu Asp Ala Asp Thr Gln Leu Asn Thr Thr Glu Asp Ser Gly Asp
1250 1255 1260
Asn Asp Gln Ala Gln Asp Lys Met Asp Lys Leu Ser Phe Val Leu Lys
1265 1270 1275 1280
Gln Asp Val Val Met Ala Asp Leu Arg Ala Ala Asp Val Lys Val Val
1285 1290 1295
Ser Cys Ile Val Gln Arg Asp Gly Ala Ile Met Pro Met Arg Arg Thr
1300 1305 1310
Phe Leu Leu Ser Glu Glu Lys Leu Cys Tyr Glu Glu Glu Pro Ile Leu
1315 1320 1325
Arg His Val Glu Pro Pro Leu Ser Ala Leu Leu Glu Leu Asp Lys Leu
1330 1335 1340
Lys Val Lys Gly Tyr Asn Glu Met Lys Tyr Thr Pro Ser Arg Asp Arg
1345 1350 1355 1360
Gln Trp His Ile Tyr Thr Leu Arg Asn Thr Glu Asn Pro Lys Met Leu
1365 1370 1375
His Arg Val Phe Phe Arg Thr Leu Val Arg Gln Pro Ser Ala Gly Asn
1380 1385 1390
Arg Phe Thr Ser Asp His Ile Thr Asp Val Glu Val Gly His Ala Glu
1395 1400 1405
Glu Pro Leu Ser Phe Thr Ser Ser Ser Ile Leu Lys Ser Leu Lys Ile
1410 1415 1420
Ala Lys Glu Glu Leu Glu Leu His Ala Ile Arg Thr Gly His Ser His
1425 1430 1435 1440
Met Tyr Leu Cys Ile Leu Lys Glu Gln Lys Leu Leu Asp Leu Val Pro
1445 1450 1455
Val Ser Gly Asn Thr Val Val Asp Val Gly Gln Asp Glu Ala Thr Ala
1460 1465 1470
Cys Ser Leu Leu Lys Glu Met Ala Leu Lys Ile His Glu Leu Val Gly
1475 1480 1485
Ala Arg Met His His Leu Ser Val Cys Gln Trp Glu Val Lys Leu Lys
1490 1495 1500
Leu Val Ser Asp Gly Pro Ala Ser Gly Ser Trp Arg Val Val Thr Thr
1505 1510 1515 1520
Asn Val Thr Gly His Thr Cys Thr Val Asp Ile Tyr Arg Glu Val Glu
1525 1530 1535
Asp Thr Glu Ser Gln Lys Leu Val Tyr His Ser Thr Ala Leu Ser Ser
1540 1545 1550
Gly Pro Leu His Gly Val Ala Leu Asn Thr Ser Tyr Gln Pro Leu Ser
1555 1560 1565
Val Ile Asp Leu Lys Arg Cys Ser Ala Arg Asn Asn Lys Thr Thr Tyr
1570 1575 1580
Cys Tyr Asp Phe Pro Leu Thr Phe Glu Ala Ala Val Gln Lys Ser Trp
1585 1590 1595 1600
Ser Asn Ile Ser Ser Glu Asn Asn Gln Cys Tyr Val Lys Ala Thr Glu
1605 1610 1615
Leu Val Phe Ala Glu Lys Asn Gly Ser Trp Gly Thr Pro Ile Ile Pro
1620 1625 1630
Met Gln Arg Ala Ala Gly Leu Asn Asp Ile Gly Met Val Ala Trp Ile
1635 1640 1645
Leu Asp Met Ser Thr Pro Glu Phe Pro Ser Gly Arg Gln Ile Ile Val
1650 1655 1660
Ile Ala Asn Asp Ile Thr Phe Arg Ala Gly Ser Phe Gly Pro Arg Glu
1665 1670 1675 1680
Asp Ala Phe Phe Glu Ala Val Thr Asn Leu Ala Cys Glu Lys Lys Leu
1685 1690 1695
Pro Leu Ile Tyr Leu Ala Ala Asn Ser Gly Ala Arg Ile Gly Ile Ala
1700 1705 1710
Asp Glu Val Lys Ser Cys Phe Arg Val Gly Trp Thr Asp Asp Ser Ser
1715 1720 1725
Pro Glu Arg Gly Phe Arg Tyr Ile Tyr Met Thr Asp Glu Asp His Asp
1730 1735 1740
Arg Ile Gly Ser Ser Val Ile Ala His Lys Met Gln Leu Asp Ser Gly
1745 1750 1755 1760
Glu Ile Arg Trp Val Ile Asp Ser Val Val Gly Lys Glu Asp Gly Leu
1765 1770 1775
Gly Val Glu Asn Ile His Gly Ser Ala Ala Ile Ala Ser Ala Tyr Ser
1780 1785 1790
Arg Ala Tyr Glu Glu Thr Phe Thr Leu Thr Phe Val Thr Gly Arg Thr
1795 1800 1805
Val Gly Ile Gly Ala Tyr Leu Ala Arg Leu Gly Ile Arg Cys Ile Gln
1810 1815 1820
Arg Ile Asp Gln Pro Ile Ile Leu Thr Gly Phe Ser Ala Leu Asn Lys
1825 1830 1835 1840
Leu Leu Gly Arg Glu Val Tyr Ser Ser His Met Gln Leu Gly Gly Pro
1845 1850 1855
Lys Ile Met Ala Thr Asn Gly Val Val His Leu Thr Val Pro Asp Asp
1860 1865 1870
Leu Glu Gly Val Ser Asn Ile Leu Arg Trp Leu Ser Tyr Val Pro Ala
1875 1880 1885
Asn Ile Gly Gly Pro Leu Pro Ile Thr Lys Ser Leu Asp Pro Ile Asp
1890 1895 1900
Arg Pro Val Ala Tyr Ile Pro Glu Asn Thr Cys Asp Pro Arg Ala Ala
1905 1910 1915 1920
Ile Ser Gly Ile Asp Asp Ser Gln Gly Lys Trp Leu Gly Gly Met Phe
1925 1930 1935
Asp Lys Asp Ser Phe Val Glu Thr Phe Glu Gly Trp Ala Lys Thr Val
1940 1945 1950
Val Thr Gly Arg Ala Lys Leu Gly Gly Ile Pro Val Gly Val Ile Ala
1955 1960 1965
Val Glu Thr Gln Thr Met Met Gln Leu Val Pro Ala Asp Pro Gly Gln
1970 1975 1980
Pro Asp Ser His Glu Arg Ser Val Pro Arg Ala Gly Gln Val Trp Phe
1985 1990 1995 2000
Pro Asp Ser Ala Thr Lys Thr Ala Gln Ala Met Leu Asp Phe Asn Arg
2005 2010 2015
Glu Gly Leu Pro Leu Phe Ile Leu Ala Asn Trp Arg Gly Phe Ser Gly
2020 2025 2030
Gly Gln Arg Asp Leu Phe Glu Gly Ile Leu Gln Ala Gly Ser Thr Ile
2035 2040 2045
Val Glu Asn Leu Arg Thr Tyr Asn Gln Pro Ala Phe Val Tyr Ile Pro
2050 2055 2060
Lys Ala Ala Glu Leu Arg Gly Gly Ala Trp Val Val Ile Asp Ser Lys
2065 2070 2075 2080
Ile Asn Pro Asp Arg Ile Glu Cys Tyr Ala Glu Arg Thr Ala Lys Gly
2085 2090 2095
Asn Val Leu Glu Pro Gln Gly Leu Ile Glu Ile Lys Phe Arg Ser Glu
2100 2105 2110
Glu Leu Lys Glu Cys Met Gly Arg Leu Asp Pro Glu Leu Ile Asp Leu
2115 2120 2125
Lys Ala Arg Leu Gln Gly Ala Asn Gly Ser Leu Ser Asp Gly Glu Ser
2130 2135 2140
Leu Gln Lys Ser Ile Glu Ala Arg Lys Lys Gln Leu Leu Pro Leu Tyr
2145 2150 2155 2160
Thr Gln Ile Ala Val Arg Phe Ala Glu Leu His Asp Thr Ser Leu Arg
2165 2170 2175
Met Ala Ala Lys Gly Val Ile Arg Lys Val Val Asp Trp Glu Asp Ser
2180 2185 2190
Arg Ser Phe Phe Tyr Lys Arg Leu Arg Arg Arg Leu Ser Glu Asp Val
2195 2200 2205
Leu Ala Lys Glu Ile Arg Gly Val Ile Gly Glu Lys Phe Pro His Lys
2210 2215 2220
Ser Ala Ile Glu Leu Ile Lys Lys Trp Tyr Leu Ala Ser Glu Ala Ala
2225 2230 2235 2240
Ala Ala Gly Ser Thr Asp Trp Asp Asp Asp Asp Ala Phe Val Ala Trp
2245 2250 2255
Arg Glu Asn Pro Glu Asn Tyr Lys Glu Tyr Ile Lys Glu Leu Arg Ala
2260 2265 2270
Gln Arg Val Ser Arg Leu Leu Ser Asp Val Ala Gly Ser Ser Ser Asp
2275 2280 2285
Leu Gln Ala Leu Pro Gln Gly Leu Ser Met Leu Leu Asp Lys Met Asp
2290 2295 2300
Pro Ser Lys Arg Ala Gln Phe Ile Glu Glu Val Met Lys Val Leu Lys
2305 2310 2315 2320
<210> 4
<211> 6963
<212> ДНК
<213> ДНК последовательность гена ACCase Alopecurus myosuroides
(ДНК последовательность)
<400> 4
atgggatcca cacatctgcc cattgtcggg tttaatgcat ccacaacacc atcgctatcc 60
actcttcgcc agataaactc agctgctgct gcattccaat cttcgtcccc ttcaaggtca 120
tccaagaaga aaagccgacg tgttaagtca ataagggatg atggcgatgg aagcgtgcca 180
gaccctgcag gccatggcca gtctattcgc caaggtctcg ctggcatcat cgacctccca 240
aaggagggcg catcagctcc agatgtggac atttcacatg ggtctgaaga ccacaaggcc 300
tcctaccaaa tgaatgggat actgaatgaa tcacataacg ggaggcacgc ctctctgtct 360
aaagtttatg aattttgcac ggaattgggt ggaaaaacac caattcacag tgtattagtc 420
gccaacaatg gaatggcagc agctaagttc atgcggagtg tccggacatg ggctaatgat 480
acatttgggt cagagaaggc gattcagttg atagctatgg caactccgga agacatgaga 540
ataaatgcag agcacattag aattgctgat cagtttgttg aagtacctgg tggaacaaac 600
aataacaact atgcaaatgt ccaactcata gtggagatag cagagagaac tggtgtctcc 660
gccgtttggc ctggttgggg ccatgcatct gagaatcctg aacttccaga tgcactaact 720
gcaaaaggaa ttgtttttct tgggccacca gcatcatcaa tgaacgcact aggcgacaag 780
gttggttcag ctctcattgc tcaagcagca ggggttccca ctcttgcttg gagtggatca 840
catgtggaaa ttccattaga actttgtttg gactcgatac ctgaggagat gtataggaaa 900
gcctgtgtta caaccgctga tgaagcagtt gcaagttgtc agatgattgg ttaccctgcc 960
atgatcaagg catcctgggg tggtggtggt aaagggatta gaaaggttaa taatgatgac 1020
gaggtgaaag cactgtttaa gcaagtacag ggtgaagttc ctggctcccc gatatttatc 1080
atgagacttg catctcagag tcgtcatctt gaagtccagc tgctttgtga tgaatatggc 1140
aatgtagcag cacttcacag tcgtgattgc agtgtgcaac gacgacacca aaagattatc 1200
gaggaaggac cagttactgt tgctcctcgt gaaacagtga aagagctaga gcaagcagca 1260
aggaggcttg ctaaggccgt gggttacgtc ggtgctgcta ctgttgaata tctctacagc 1320
atggagactg gtgaatacta ttttctggag cttaatccac ggttgcaggt tgagcaccca 1380
gtcaccgagt cgatagctga agtaaatttg cctgcagccc aagttgcagt tgggatgggt 1440
ataccccttt ggcagattcc agagatcaga cgtttctacg gaatggacaa tggaggaggc 1500
tatgatattt ggaggaaaac agcagctctc gctactccat tcaactttga tgaagtagat 1560
tctcaatggc cgaagggtca ttgtgtggca gttaggataa ccagtgagaa tccagatgat 1620
ggattcaagc ctactggtgg aaaagtaaag gagataagtt ttaaaagtaa gccaaatgtc 1680
tggggatatt tctcagttaa gtctggtgga ggcattcatg aatttgcgga ttctcagttt 1740
ggacacgttt ttgcctatgg agagactaga tcagcagcaa taaccagcat gtctcttgca 1800
ctaaaagaga ttcaaattcg tggagaaatt catacaaacg ttgattacac ggttgatctc 1860
ttgaatgccc cagacttcag agaaaacacg atccataccg gttggctgga taccagaata 1920
gctatgcgtg ttcaagctga gaggcctccc tggtatattt cagtggttgg aggagctcta 1980
tataaaacaa taaccaccaa tgcggagacc gtttctgaat atgttagcta tctcatcaag 2040
ggtcagattc caccaaagca catatccctt gtccattcaa ctatttcttt gaatatagag 2100
gaaagcaaat atacaattga gattgtgagg agtggacagg gtagctacag attgagactg 2160
aatggatcac ttattgaagc caatgtacaa acattatgtg atggaggcct tttaatgcag 2220
ctggatggaa atagccatgt tatttatgct gaagaagaag cgggtggtac acggcttctt 2280
attgatggaa aaacatgctt gctacagaat gaccatgatc cgtcaaggtt attagctgag 2340
acaccctgca aacttcttcg tttcttgatt gccgatggtg ctcatgttga tgctgatgta 2400
ccatacgcgg aagttgaggt tatgaagatg tgcatgcccc tcttgtcgcc tgctgctggt 2460
gtcattaatg ttttgttgtc tgagggccag gcgatgcagg ctggtgatct tatagcgaga 2520
cttgatctcg atgacccttc tgctgtgaag agagccgagc catttgaagg atcttttcca 2580
gaaatgagcc ttcctattgc tgcttctggc caagttcaca aaagatgtgc tgcaagtttg 2640
aacgctgctc gaatggtcct tgcaggatat gaccatgcgg ccaacaaagt tgtgcaagat 2700
ttggtatggt gccttgatac acctgctctt cctttcctac aatgggaaga gcttatgtct 2760
gttttagcaa ctagacttcc aagacgtctt aagagcgagt tggagggcaa atacaatgaa 2820
tacaagttaa atgttgacca tgtgaagatc aaggatttcc ctaccgagat gcttagagag 2880
acaatcgagg aaaatcttgc atgtgtttcc gagaaggaaa tggtgacaat tgagaggctt 2940
gttgaccctc tgatgagcct gctgaagtca tacgagggtg ggagagaaag ccatgcccac 3000
tttattgtca agtccctttt tgaggagtat ctctcggttg aggaactatt cagtgatggc 3060
attcagtctg acgtgattga acgcctgcgc ctacaatata gtaaagacct ccagaaggtt 3120
gtagacattg ttttgtctca ccagggtgtg agaaacaaaa caaagctgat actcgcgctc 3180
atggagaaac tggtctatcc aaaccctgct gcctacagag atcagttgat tcgcttttct 3240
tccctcaacc ataaaagata ttataagttg gctcttaaag ctagtgaact tcttgaacaa 3300
accaagctca gcgaactccg cacaagcatt gcaaggaacc tttcagcgct ggatatgttc 3360
accgaggaaa aggcagattt ctccttgcaa gacagaaaat tggccattaa tgagagcatg 3420
ggagatttag tcactgcccc actgccagtt gaagatgcac ttgtttcttt gtttgattgt 3480
actgatcaaa ctcttcagca gagagtgatt cagacataca tatctcgatt ataccagcct 3540
caacttgtga aggatagcat ccagctgaaa tatcaggatt ctggtgttat tgctttatgg 3600
gaattcactg aaggaaatca tgagaagaga ttgggtgcta tggttatcct gaagtcacta 3660
gaatctgtgt caacagccat tggagctgct ctaaaggatg catcacatta tgcaagctct 3720
gcgggcaaca cggtgcatat tgctttgttg gatgctgata cccaactgaa tacaactgaa 3780
gatagtggtg ataatgacca agctcaagac aagatggata aactttcttt tgtactgaaa 3840
caagatgttg tcatggctga tctacgtgct gctgatgtca aggttgttag ttgcattgtt 3900
caaagagatg gagcaatcat gcctatgcgc cgtaccttcc tcttgtcaga ggaaaaactt 3960
tgttacgagg aagagccgat tcttcggcat gtggagcctc cactttctgc acttcttgag 4020
ttggataaat tgaaagtgaa aggatacaat gagatgaagt atacaccgtc acgtgatcgt 4080
cagtggcata tatacacact tagaaatact gaaaatccaa aaatgctgca cagggtattt 4140
ttccgaacac ttgtcagaca acccagtgca ggcaacaggt ttacatcaga ccatatcact 4200
gatgttgaag taggacacgc agaggaacct ctttcattta cttcaagcag catattaaaa 4260
tcgttgaaga ttgctaaaga agaattggag cttcacgcga tcaggactgg ccattctcat 4320
atgtacttgt gcatattgaa agagcaaaag cttcttgacc ttgttcctgt ttcagggaac 4380
actgttgtgg atgttggtca agatgaagct actgcatgct ctcttttgaa agaaatggct 4440
ttaaagatac atgaacttgt tggtgcaaga atgcatcatc tttctgtatg ccagtgggaa 4500
gtgaaactta agttggtgag cgatgggcct gccagtggta gctggagagt tgtaacaacc 4560
aatgttactg gtcacacctg cactgtggat atctaccggg aggtcgaaga tacagaatca 4620
cagaaactag tataccactc caccgcattg tcatctggtc ctttgcatgg tgttgcactg 4680
aatacttcgt atcagccttt gagtgttatt gatttaaaac gttgctctgc caggaacaac 4740
aaaactacat actgctatga ttttccattg acatttgaag ctgcagtgca gaagtcgtgg 4800
tctaacattt ccagtgaaaa caaccaatgt tatgttaaag cgacagagct tgtgtttgct 4860
gaaaagaatg ggtcgtgggg cactcctata attcctatgc agcgtgctgc tgggctgaat 4920
gacattggta tggtagcctg gatcttggac atgtccactc ctgaatttcc cagcggcaga 4980
cagatcattg ttatcgcaaa tgatattaca tttagagctg gatcatttgg cccaagggaa 5040
gatgcatttt tcgaagctgt aaccaacctg gcttgtgaga agaagcttcc acttatctac 5100
ttggctgcaa actctggtgc tcggattggc attgctgatg aagtaaaatc ttgcttccgt 5160
gttggatgga ctgatgatag cagccctgaa cgtggattta ggtacattta tatgactgac 5220
gaagaccatg atcgtattgg ctcttcagtt atagcacaca agatgcagct agatagtggc 5280
gagatcaggt gggttattga ttctgttgtg ggaaaagagg atggactagg tgtggagaac 5340
atacatggaa gtgctgctat tgccagtgcc tattctaggg cgtacgagga gacatttaca 5400
cttacattcg ttactggacg aactgttgga atcggagcct atcttgctcg acttggcata 5460
cggtgcatac agcgtattga ccagcccatt attttgaccg ggttttctgc cctgaacaag 5520
cttcttgggc gggaggtgta cagctcccac atgcagttgg gtggtcccaa aatcatggcg 5580
acgaatggtg ttgtccatct gactgttcca gatgaccttg aaggtgtttc taatatattg 5640
aggtggctca gctatgttcc tgcaaacatt ggtggacctc ttcctattac aaaatctttg 5700
gacccaatag acagacccgt tgcatacatc cctgagaata catgtgatcc tcgtgcagcc 5760
atcagtggca ttgatgacag ccaagggaaa tggttgggtg gcatgtttga caaagacagt 5820
tttgtggaga catttgaagg atgggcgaag acagtagtta ctggcagagc aaaacttgga 5880
gggattcctg ttggtgttat agctgtggag acacagacca tgatgcagct cgtccccgct 5940
gatccaggcc agcctgattc ccacgagcgg tctgttcctc gtgctgggca agtttggttt 6000
ccagattctg ctaccaagac agcgcaggcg atgttggact tcaaccgtga aggattacct 6060
ctgttcatac ttgctaactg gagaggcttc tctggagggc aaagagatct ttttgaagga 6120
attctgcagg ctgggtcaac aattgttgag aaccttagga catacaatca gcctgccttt 6180
gtatatatcc ccaaggctgc agagctacgt ggaggagcct gggtcgtgat tgatagcaag 6240
ataaacccag atcgcatcga gtgctatgct gagaggactg caaagggtaa tgttctcgaa 6300
cctcaagggt tgattgagat caagttcagg tcagaggaac tcaaagaatg catgggtagg 6360
cttgatccag aattgataga tctgaaagca agactccagg gagcaaatgg aagcctatct 6420
gatggagaat cccttcagaa gagcatagaa gctcggaaga aacagttgct gcctctgtac 6480
acccaaatcg cggtacgttt tgcggaattg cacgacactt cccttagaat ggctgctaaa 6540
ggtgtgatca ggaaagttgt agactgggaa gactctcggt ctttcttcta caagagatta 6600
cggaggaggc tatccgagga cgttctggca aaggagatta gaggtgtaat tggtgagaag 6660
tttcctcaca aatcagcgat cgagctgatc aagaaatggt acttggcttc tgaggcagct 6720
gcagcaggaa gcaccgactg ggatgacgac gatgcttttg tcgcctggag ggagaaccct 6780
gaaaactata aggagtatat caaagagctt agggctcaaa gggtatctcg gttgctctca 6840
gatgttgcag gctccagttc ggatttacaa gccttgccgc agggtctttc catgctacta 6900
gataagatgg atccctctaa gagagcacag tttatcgagg aggtcatgaa ggtcctgaaa 6960
tga 6963
<210> 5
<211> 446
<212> БЕЛОК
<213> Аминокислотная последовательность белка HPPD Oryza sativa L.
(аминокислотная последовательность)
<400> 5
Met Pro Pro Thr Pro Thr Pro Thr Ala Thr Thr Gly Ala Val Ser Ala
1 5 10 15
Ala Ala Ala Ala Gly Glu Asn Ala Gly Phe Arg Leu Val Gly His Arg
20 25 30
Arg Phe Val Arg Ala Asn Pro Arg Ser Asp Arg Phe Gln Ala Leu Ala
35 40 45
Phe His His Val Glu Leu Trp Cys Ala Asp Ala Ala Ser Ala Ala Gly
50 55 60
Arg Phe Ala Phe Ala Leu Gly Ala Pro Leu Ala Ala Arg Ser Asp Leu
65 70 75 80
Ser Thr Gly Asn Ser Ala His Ala Ser Leu Leu Leu Arg Ser Ala Ser
85 90 95
Val Ala Phe Leu Phe Thr Ala Pro Tyr Gly Gly Asp His Gly Val Gly
100 105 110
Ala Asp Ala Ala Thr Thr Ala Ser Ile Pro Ser Phe Ser Pro Gly Ala
115 120 125
Ala Arg Arg Phe Ala Ala Asp His Gly Leu Ala Val His Ala Val Ala
130 135 140
Leu Arg Val Ala Asp Ala Ala Asp Ala Phe Arg Ala Ser Val Ala Ala
145 150 155 160
Gly Ala Arg Pro Ala Phe Gln Pro Ala Asp Leu Gly Gly Gly Phe Gly
165 170 175
Leu Ala Glu Val Glu Leu Tyr Gly Asp Val Val Leu Arg Phe Val Ser
180 185 190
His Pro Asp Gly Ala Asp Ala Pro Phe Leu Pro Gly Phe Glu Gly Val
195 200 205
Ser Asn Pro Gly Ala Val Asp Tyr Gly Leu Arg Arg Phe Asp His Val
210 215 220
Val Gly Asn Val Pro Glu Leu Ala Pro Val Ala Ala Tyr Ile Ser Gly
225 230 235 240
Phe Thr Gly Phe His Glu Phe Ala Glu Phe Thr Ala Glu Asp Val Gly
245 250 255
Thr Ala Glu Ser Gly Leu Asn Ser Val Val Leu Ala Asn Asn Ala Glu
260 265 270
Thr Val Leu Leu Pro Leu Asn Glu Pro Val His Gly Thr Lys Arg Arg
275 280 285
Ser Gln Ile Gln Thr Tyr Leu Asp His His Gly Gly Pro Gly Val Gln
290 295 300
His Ile Ala Leu Ala Ser Asp Asp Val Leu Gly Thr Leu Arg Glu Met
305 310 315 320
Arg Ala Arg Ser Ala Met Gly Gly Phe Glu Phe Leu Ala Pro Pro Pro
325 330 335
Pro Asn Tyr Tyr Asp Gly Val Arg Arg Arg Ala Gly Asp Val Leu Ser
340 345 350
Glu Glu Gln Ile Asn Glu Cys Gln Glu Leu Gly Val Leu Val Asp Arg
355 360 365
Asp Asp Gln Gly Val Leu Leu Gln Ile Phe Thr Lys Pro Val Gly Asp
370 375 380
Arg Pro Thr Phe Phe Leu Glu Met Ile Gln Arg Ile Gly Cys Met Glu
385 390 395 400
Lys Asp Glu Ser Gly Gln Glu Tyr Gln Lys Gly Gly Cys Gly Gly Phe
405 410 415
Gly Lys Gly Asn Phe Ser Glu Leu Phe Lys Ser Ile Glu Glu Tyr Glu
420 425 430
Lys Ser Leu Glu Ala Lys Gln Ala Pro Thr Val Gln Gly Ser
435 440 445
<210> 6
<211> 2099
<212> ДНК
<213> ДНК последовательность генома HPPD Oryza sativa L. (ДНК
последовательность)
<400> 6
atgcctccca ctcccacccc caccgccacc accggcgccg tctcggccgc tgcggcggcg 60
ggggagaacg cggggttccg cctcgtcggg caccgccgct tcgtccgcgc caacccgcgg 120
agcgaccggt tccaggcgct cgcgttccac cacgtcgagc tctggtgcgc cgacgccgcg 180
tccgccgcgg gccggttcgc cttcgccctg ggcgcgccgc tcgccgccag gtccgacctc 240
tccacgggga actccgcgca cgcctccctc ctcctccgct ccgcctccgt cgcgttcctc 300
ttcaccgccc cctacggcgg cgaccacggc gtcggcgcgg acgcggccac caccgcctcc 360
atcccttcct tctccccagg cgccgcgcgg aggttcgccg cggaccacgg cctcgcggtg 420
cacgccgtgg cgctgcgcgt cgccgacgcg gccgacgcct tccgcgccag cgtcgcggcc 480
ggtgcgcgcc cggcgttcca gcccgccgac ctcggcggtg gcttcggcct cgcggaggtg 540
gagctctacg gcgacgtcgt gctccgcttc gtcagccacc cggacggcgc cgacgcgccc 600
ttcctcccgg gtttcgaggg cgtcagcaac ccgggcgccg tggactacgg cctccgccgg 660
ttcgaccacg tcgtcggcaa cgtgccggag ctcgctccgg tagccgcgta catctccggg 720
ttcaccgggt tccacgagtt cgccgagttc accgccgagg acgtgggcac cgccgagagc 780
ggcctcaact cggtggtgct cgccaacaac gcggagaccg tgctgctgcc gctcaacgag 840
ccggtgcacg gcaccaagcg gcggagccag atacagacgt acctggacca ccacggcggc 900
ccgggggtgc agcacatcgc gctggccagc gacgacgtgc tcgggacgct gagggagatg 960
cgggcgcgct ccgccatggg cggcttcgag ttcttggcgc cgccgccgcc caactactac 1020
gacggcgtgc ggcggcgcgc cggggacgtg ctctcggagg agcagatcaa cgagtgccag 1080
gagctcgggg tgctcgtgga cagggatgac cagggggtgt tgctccagat cttcaccaag 1140
ccagtaggag acaggtaaaa tcctcacctc tttcatgatg aaaatggctt atgaattcag 1200
atttgcagtt atttgttggc acatagcatc gattaggcgc agaaaggtgt caagcattat 1260
gaaattaatc cagaatgctt gaataataca gtataatata tgatagtgag ctctgtgata 1320
ctccatggat actctttatg tgtctccatg aatccatgat gcgcctttct gaagattgtg 1380
acactagaaa gggaataaag ctgaatgtgc ataggaaaaa aatgaaaagc caatgtgtgt 1440
ctgtttatgc cttcttgcaa gcatatccca gttccttttt gccggcatgt tgtaatgcag 1500
atagccagcc acatatagct acttaattag tgagtactcc ctctcacaat gtaagtcatt 1560
ctagtatttt ccacattcat attgatgcta atctatctag attcattagc atcaatatga 1620
atatgggaaa tactagaatg acttacattg tgaaacggag gaagtattac ttactacatc 1680
taaggtccat ggattccttt ttttacaaaa gaaagaaaga atcttatggc aactccatca 1740
gcataaacca gcaatgctgc tgggaacaac ttaaacttta ggttcaggag gttgtaattg 1800
tctttaagct taatagtctg attcagtcag tattctaatt tctgctgcat ctttgctatt 1860
gttatttcct ctctgtgact ccaaatctaa ctggatcagc tatttcactc aggccaacct 1920
ttttcttgga gatgatacaa aggattgggt gcatggagaa ggatgagagt gggcaggagt 1980
accagaaggg cggctgcggc gggtttggga agggcaactt ctcggagctg ttcaagtcca 2040
ttgaggagta tgagaaatcc cttgaagcca agcaagcccc tacagttcaa ggatcctag 2099
<210> 7
<211> 536
<212> БЕЛОК
<213> Аминокислотная последовательность белка PPO1 Oryza sativa L.
(аминокислотная последовательность)
<400> 7
Met Ala Ala Ala Ala Ala Ala Met Ala Thr Ala Thr Ser Ala Thr Ala
1 5 10 15
Ala Pro Pro Leu Arg Ile Arg Asp Ala Ala Arg Arg Thr Arg Arg Arg
20 25 30
Gly His Val Arg Cys Ala Val Ala Ser Gly Ala Ala Glu Ala Pro Ala
35 40 45
Ala Pro Gly Ala Arg Val Ser Ala Asp Cys Val Val Val Gly Gly Gly
50 55 60
Ile Ser Gly Leu Cys Thr Ala Gln Ala Leu Ala Thr Lys His Gly Val
65 70 75 80
Gly Asp Val Leu Val Thr Glu Ala Arg Ala Arg Pro Gly Gly Asn Ile
85 90 95
Thr Thr Ala Glu Arg Ala Gly Glu Gly Tyr Leu Trp Glu Glu Gly Pro
100 105 110
Asn Ser Phe Gln Pro Ser Asp Pro Val Leu Thr Met Ala Val Asp Ser
115 120 125
Gly Leu Lys Asp Asp Leu Val Phe Gly Asp Pro Asn Ala Pro Arg Phe
130 135 140
Val Leu Trp Glu Gly Lys Leu Arg Pro Val Pro Ser Lys Pro Gly Asp
145 150 155 160
Leu Pro Phe Phe Asp Leu Met Ser Ile Pro Gly Lys Leu Arg Ala Gly
165 170 175
Leu Gly Ala Leu Gly Val Arg Ala Pro Pro Pro Gly Arg Glu Glu Ser
180 185 190
Val Glu Asp Phe Val Arg Arg Asn Leu Gly Ala Glu Val Phe Glu Arg
195 200 205
Leu Ile Glu Pro Phe Cys Ser Gly Val Tyr Ala Gly Asp Pro Ser Lys
210 215 220
Leu Ser Met Lys Ala Ala Phe Gly Lys Val Trp Arg Leu Glu Asp Thr
225 230 235 240
Gly Gly Ser Ile Ile Gly Gly Thr Ile Lys Thr Ile Gln Glu Arg Gly
245 250 255
Lys Asn Pro Lys Pro Pro Arg Asp Pro Arg Leu Pro Thr Pro Lys Gly
260 265 270
Gln Thr Val Ala Ser Phe Arg Lys Gly Leu Thr Met Leu Pro Asp Ala
275 280 285
Ile Thr Ser Arg Leu Gly Ser Lys Val Lys Leu Ser Trp Lys Leu Thr
290 295 300
Ser Ile Thr Lys Ser Asp Asn Lys Gly Tyr Ala Leu Val Tyr Glu Thr
305 310 315 320
Pro Glu Gly Val Val Ser Val Gln Ala Lys Thr Val Val Met Thr Ile
325 330 335
Pro Ser Tyr Val Ala Ser Asp Ile Leu Arg Pro Leu Ser Ser Asp Ala
340 345 350
Ala Asp Ala Leu Ser Ile Phe Tyr Tyr Pro Pro Val Ala Ala Val Thr
355 360 365
Val Ser Tyr Pro Lys Glu Ala Ile Arg Lys Glu Cys Leu Ile Asp Gly
370 375 380
Glu Leu Gln Gly Phe Gly Gln Leu His Pro Arg Ser Gln Gly Val Glu
385 390 395 400
Thr Leu Gly Thr Ile Tyr Ser Ser Ser Leu Phe Pro Asn Arg Ala Pro
405 410 415
Ala Gly Arg Val Leu Leu Leu Asn Tyr Ile Gly Gly Ser Thr Asn Thr
420 425 430
Gly Ile Val Ser Lys Thr Glu Ser Glu Leu Val Glu Ala Val Asp Arg
435 440 445
Asp Leu Arg Lys Met Leu Ile Asn Pro Lys Ala Val Asp Pro Leu Val
450 455 460
Leu Gly Val Arg Val Trp Pro Gln Ala Ile Pro Gln Phe Leu Ile Gly
465 470 475 480
His Leu Asp His Leu Glu Ala Ala Lys Ser Ala Leu Gly Lys Gly Gly
485 490 495
Tyr Asp Gly Leu Phe Leu Gly Gly Asn Tyr Val Ala Gly Val Ala Leu
500 505 510
Gly Arg Cys Val Glu Gly Ala Tyr Glu Ser Ala Ser Gln Ile Ser Asp
515 520 525
Tyr Leu Thr Lys Tyr Ala Tyr Lys
530 535
<210> 8
<211> 3246
<212> ДНК
<213> ДНК последовательность генома PPO1 Oryza sativa L. (ДНК
последовательность)
<400> 8
atggccgccg ccgccgcagc catggccacc gccacctccg ccacggcagc gccgccgctc 60
cgcattcgcg acgccgcgag gaggacccgc cgacgcggcc acgttcgctg cgccgtcgcc 120
agcggcgcgg ccgaggcgcc cgcggcgccc ggggcgcggg tgtcggcgga ctgcgtcgtg 180
gtgggcggcg gcatcagcgg gctctgcacc gcgcaggcgc tggccacaaa gcacggcgtc 240
ggcgacgtgc tcgtcacgga ggcccgcgcc cgccccggcg gcaacatcac caccgccgag 300
cgcgccggcg agggctacct ctgggaggag gggcccaaca gcttccagcc ttccgacccc 360
gtcctcacca tggccgtacg tcttcttgct ccccttctct tctgattctc tcgcggcgga 420
gacgacatga atgggaatgg tggtgcatgg attggggcgc gcaggtggac agcgggctca 480
aggacgatct cgtgttcggg gaccccaacg cgccgcggtt cgtgctgtgg gaggggaagc 540
taaggccggt gccgtccaag cccggcgacc tgccgttctt cgacctcatg agcatccccg 600
gcaagctcag ggccggcctt ggcgcgctcg gcgttcgagc gccacctcca gtttgtgtgc 660
tctccccgct gtgcattctt gattcacttg tgaaattcga ttgtgctgag cgtttccggc 720
gaaggtttca ggggcgtgag gagtcggtgg aggacttcgt gcggcgcaac ctcggcgcgg 780
aggtctttga gcgcctcatt gagcctttct gctcaggtgt ttattgtagt gtgcaattgc 840
tgttttgttt ttgatgattc agataagaat acggtgattt cggtgcttag gtgtgtatgc 900
tggtgatcct tcaaagctca gtatgaaggc tgcatttggg aaggtgtgga ggctggagga 960
tactggaggt agcattattg gtggaaccat caaaacaatc caggagaggg ggaaaaaccc 1020
caaaccgccg agggatccgt gagtgagaaa ttgccttctt tgttggatta attgtccatt 1080
gtgttacact gatatgcctt caccattttt agccgccttc caacgccaaa ggggcagaca 1140
gttgcatctt tcaggaaggg tctgactatg ctcccggatg ctattacatc taggtttgtt 1200
atcattgtct ttgtaattta cctagttctt caactatgga tattaggtgc tgtagattgt 1260
tcagatagat gcacattgta caacaatcta ggtagattga ttgctatggc ttgttgaatt 1320
aactgtttca cttgcatcat tgcctcagca catatgaagc atatggatag attcttcaat 1380
catttatccc tcaataacac aattttgaca ccagtgcttc tccttttttt catcctgctt 1440
catctggcta tccacaatat aattaagcat acaaaagagg cactctttga tggacaattc 1500
atagtgttgt gggttaatat tcatttgcat tctttgaggg acaattcgtt acaccctaac 1560
atgaactagt aatgattggg gtgcttaacc atttgttctg catttcctcc attttcaggt 1620
tgggtagcaa agtcaaactt tcatggaagt tgacaagcat tacaaagtca gacaacaaag 1680
gatatgcatt agtgtatgaa acaccagaag gggtggtctc ggtgcaagct aaaactgttg 1740
tcatgaccat cccatcatat gttgctagtg atatcttgcg gccactttca gtaagttata 1800
tatatttaat taactttctg ttcccaaaat acactgcagc acttcattgc ttcctgaggt 1860
cctcgattca tttttcggta gacaggaagt agtattcatt tgcacttttt aagggattaa 1920
ttcaacatat ccactggaaa tatacatatc ctacacatcc tgtcaacata cttgctaaac 1980
agcattttgt ttgagttgac tggcatctca gcagccaatt actatcttta ggggacaagc 2040
cacattctta ataaatcctg tcggaaatca ctttttgatt tttatagatt agtgttgtca 2100
tagaatttgg cttggtgatc tacttggtaa ggttaactga ttcacaagtc ggacaatttc 2160
ttcaccaatc tagcagtatg tttaagtgtg ttggtactta attctaaatg tcctgcgcat 2220
ggtaacatat catatgcaaa aattcctcag taaccgaaat ttatactgta agttttaact 2280
gtctttacac tgttaatttt agacatactt cttccttgct tgttcattga acttgtttcc 2340
cccttccaca gagtgatgca gcagatgctc tgtcaatatt ctattatcca ccagttgctg 2400
ctgtaactgt ttcatatcca aaagaagcaa ttagaaaaga atgcttaatt gacggagagc 2460
tccagggttt cggccagctg catccgcgta gtcagggagt tgagacttta ggtacttata 2520
ggaattcaac cttattattc ttctaacata taaatgaact aatctttctt gtctagtttg 2580
catttattgt ggattaagtt tggttatatt gttcttacaa gtttgtggta ttattttgta 2640
taggaacaat atatagctca tcactctttc caaatcgtgc tccagctgga agggtgttac 2700
ttctgaacta cataggaggt tctacaaata cagggattgt ttccaaggta tcgctgtcaa 2760
gttgtttatt ttgcgactat atgattacag tatcctgttt ttcaactcca gctgctgtta 2820
gactgtcata ataaatctgc tactacatgt ttgcacacta tttgactgca tttaaaaact 2880
cagatagcct atatttttag ttgcctgcta ctgggtgtat ttctaatgat cccatcatgt 2940
ttgcagactg aaagtgagct ggtagaagca gttgaccgtg acctcaggaa gatgctgata 3000
aatcctaaag cagtggaccc tttggtcctt ggcgtccggg tatggccaca agccatacca 3060
cagttcctca ttggccatct tgatcatctt gaggctgcaa aatctgccct gggcaaaggt 3120
ggttatgatg gattgttcct cggagggaac tatgttgcag gagttgccct gggccgatgc 3180
gttgaaggtg catatgagag tgcctcacaa atatctgact acttgaccaa gtacgcctac 3240
aagtga 3246
<210> 9
<211> 587
<212> БЕЛОК
<213> Аминокислотная последовательность белка TIR1 Oryza sativa L.
(аминокислотная последовательность)
<400> 9
Met Gly Arg Gly Gly Ser Arg Ala Ala Cys Ala Ala Ala Ala Pro Pro
1 5 10 15
Trp His Ser Leu Pro Asp Glu Val Trp Glu His Ala Phe Ser Phe Leu
20 25 30
Pro Ala Ala Ala Asp Arg Gly Ala Ala Ala Gly Ala Cys Ser Ser Trp
35 40 45
Leu Arg Ala Glu Arg Arg Ser Arg Arg Arg Leu Ala Val Ala Asn Cys
50 55 60
Tyr Ala Ala Ala Pro Arg Asp Ala Val Glu Arg Phe Pro Ser Val Arg
65 70 75 80
Ala Ala Glu Val Lys Gly Lys Pro His Phe Ala Asp Phe Gly Leu Val
85 90 95
Pro Pro Ala Trp Gly Ala Ala Ala Ala Pro Trp Ile Ala Ala Ala Ala
100 105 110
Asp Gly Trp Pro Leu Leu Glu Glu Leu Ser Phe Lys Arg Met Val Val
115 120 125
Thr Asp Glu Cys Leu Glu Met Ile Ala Ala Ser Phe Arg Asn Phe Gln
130 135 140
Val Leu Arg Leu Val Ser Cys Asp Gly Phe Ser Thr Ala Gly Leu Ala
145 150 155 160
Ala Ile Ala Ala Gly Cys Arg His Leu Arg Glu Leu Asp Leu Gln Glu
165 170 175
Asn Glu Ile Glu Asp Cys Ser Ile His Trp Leu Ser Leu Phe Pro Glu
180 185 190
Ser Phe Thr Ser Leu Val Thr Leu Asn Phe Ser Cys Leu Glu Gly Glu
195 200 205
Val Asn Ile Thr Val Leu Glu Arg Leu Val Thr Arg Cys His Asn Leu
210 215 220
Lys Thr Leu Lys Leu Asn Asn Ala Ile Pro Leu Asp Lys Leu Ala Ser
225 230 235 240
Leu Leu His Lys Ala Pro Gln Leu Val Glu Leu Gly Thr Gly Lys Phe
245 250 255
Ser Ala Asp Tyr His Ser Asp Leu Phe Ala Lys Leu Glu Ala Ala Phe
260 265 270
Gly Gly Cys Lys Ser Leu Arg Arg Leu Ser Gly Ala Trp Asp Ala Val
275 280 285
Pro Asp Tyr Leu Pro Ala Phe Tyr Cys Val Cys Glu Gly Leu Thr Ser
290 295 300
Leu Asn Leu Ser Tyr Ala Thr Val Arg Gly Pro Glu Leu Ile Lys Phe
305 310 315 320
Ile Ser Arg Cys Arg Asn Leu Gln Gln Leu Trp Val Met Asp Leu Ile
325 330 335
Glu Asp His Gly Leu Ala Val Val Ala Ser Ser Cys Asn Lys Leu Gln
340 345 350
Glu Leu Arg Val Phe Pro Ser Asp Pro Phe Gly Ala Gly Phe Leu Thr
355 360 365
Glu Arg Gly Leu Val Asp Val Ser Ala Ser Cys Pro Met Leu Glu Ser
370 375 380
Val Leu Tyr Phe Cys Arg Arg Met Thr Asn Glu Ala Leu Ile Thr Ile
385 390 395 400
Ala Lys Asn Arg Pro Asn Phe Thr Cys Phe Arg Leu Cys Ile Leu Glu
405 410 415
Pro His Thr Pro Asp Tyr Ile Thr Arg Glu Pro Leu Asp Ala Gly Phe
420 425 430
Ser Ala Ile Val Glu Ser Cys Arg Gly Leu Arg Arg Leu Ser Ile Ser
435 440 445
Gly Leu Leu Thr Asp Leu Val Phe Lys Ser Ile Gly Ala His Ala Asp
450 455 460
Arg Leu Glu Met Leu Ser Ile Ala Phe Ala Gly Asn Ser Asp Leu Gly
465 470 475 480
Leu His Tyr Ile Leu Ser Gly Cys Lys Ser Leu Lys Lys Leu Glu Ile
485 490 495
Arg Asp Cys Pro Phe Gly Asp Lys Pro Leu Leu Ala Asn Ala Ala Lys
500 505 510
Leu Glu Thr Met Arg Ser Leu Trp Met Ser Ser Cys Leu Leu Thr Leu
515 520 525
Gly Ala Cys Arg Gln Leu Ala Arg Lys Met Pro Arg Leu Ser Val Glu
530 535 540
Ile Met Asn Asp Pro Gly Arg Ser Cys Pro Leu Asp Ser Leu Pro Asp
545 550 555 560
Glu Thr Pro Val Glu Lys Leu Tyr Val Tyr Arg Thr Ile Ala Gly Pro
565 570 575
Arg Ser Asp Thr Pro Ala Cys Val Gln Ile Val
580 585
<210> 10
<211> 5791
<212> ДНК
<213> ДНК последовательность генома TIR1 Oryza sativa L. (ДНК
последовательность)
<400> 10
atggggcgcg gcggctcgcg cgcggcgtgc gccgccgcgg cgccgccgtg gcactcgctc 60
ccggacgagg tctgggagca cgccttctcc ttcctccccg ccgccgcgga caggggcgcc 120
gcggcggggg cgtgcagctc gtggctccgc gccgagcgcc ggtcgcgccg ccgcctcgcc 180
gtcgccaact gctacgccgc cgcgccgcgg gacgccgtcg agcggttccc gtccgtgcgc 240
gccgccgagg tcaagggcaa gccccacttc gccgacttcg gcctcgtccc ccccgcctgg 300
ggcgccgccg cggcgccgtg gatcgccgcc gccgccgacg ggtggccgct gctcgaggag 360
ctcagcttca agcgcatggt cgtcaccgac gagtgcctcg agatgatcgc cgcgtccttc 420
aggaacttcc aggtgctccg cctcgtctcc tgcgacggct tcagcaccgc gggcctcgcc 480
gccattgctg ccggttgcag gtgaggtgga gagtgttgtt tgggtcaaat ctttttagct 540
aaataagcat gggatctttg ggttgttctt ggatttgaga ggaatctttc acggcccaag 600
ctttgattcg tcttcttttt agctaaaaca tgacagattt cttggtgcca tttctttctt 660
cacagtgttc tttagtgagt tcatggtcta gctgaagtga tttgttcttc catgattcct 720
atattaactt tgttttgaac ttcattgagt agtatgtgga agtagatgtg gctttctgct 780
aagatttgat ctattcttgg tgtttaattt gcaaagggac agtttttttt tctgtttact 840
tgtgtatgat ctggatttac ttttggtgtt tcctaagttg aagaccaaga agtcttcttg 900
tgacagtatt ttagatgctt ataacgtgtt aatgtcaaag gaatgattcc catgaaaatt 960
ctgctgtgat ttgtacattt tattaggagt taagtaacag agtgaattga gtgatgctca 1020
cgtcttcttt aaatcttagc tatgatttgt tcctaggaag ctttgacttg tatttgagtg 1080
cctggtttgc gaagatgtga ttctcttgca cggatctgtg tgattgacag aagaaatatt 1140
tctacctgtt taagcaatga tggatccatc tctttgtttc tttttgtgct ttttacccaa 1200
aagaatggga tatttcaagt ttacaaacaa ctggtgatgc gcctttataa ttttgggctt 1260
ttttaaaaga atgtattctt cccatcatga gttactagat ctgagttgaa ctgttttcct 1320
ttatattttc ctgatattta agtgaatttg ttgagttcct atcctgattt gaacttccag 1380
attctaaaga accaggtctt ttaagcatgt ttttgtcatg tagattccca gtttatcccc 1440
tccccctcct tttgtgctca agaatctact accttttatc ttgcttttgg tgagggaaag 1500
tgatttttgc tccaatattt gtgatgtcct catgtaatgg acagttaaaa caaacagatt 1560
tgttcttgcg agtatcttct ggactccccc cagaaaagaa ttaggaatgt gtgggattcc 1620
tatcattggg gaatttatat cctaaaccag atagaaggat ctaatagtgg gagtttcgat 1680
cctgattcat ccattagtag tagtttaata tgccaaagca gtatcatttg catcattttg 1740
aggaactcga aacagttgaa ctgtgaagtt taactactgt acacttcaaa caggcagatt 1800
agaataggtt gtgagataac taaatttggt tcggtgatcg tataaggcca tattcttgga 1860
tattaggacg aaatgagcct gagcaccaca tgtgtacttg ctgatggttg agaacttagc 1920
atagatacta gtttttatgc tagactagag ggctactggt acattcactt gtttgataaa 1980
tccagatgta tttaagtttt gattcaaaat attttgactc tagattggag actacatttt 2040
agagcattca aaatttacaa tggaagcagg attttagcaa aactattttc atacatttac 2100
tctgttattg agccagaatc attttttagt tcttgctact tttttgttaa cactgttatg 2160
catgtaaact cagcgttaca tttatttcaa ttacaaggat ctacacttgt agctctgggt 2220
gcagatggta gttaacattg atgaatctta gctatatcga aatactatct tctaagtaca 2280
aacttttctt gtgtgattct tatccttgtt gttactgttg ctacttatat ccattgattg 2340
agcatgcttt gaattacctt aagaatttct ctctccagtc aaatttattt gactttgaga 2400
gaactgtgtt tatttctccc aacgtcaaat gttttgaatt ttgaatggac agagtatgta 2460
gtagtgggca gcacattggt gccctagctg caaataaaac cttttttttt tgttaaaaca 2520
atgattatga ggatgtcggt ttacactgtc cactcagaaa tttgacttga ttatggatag 2580
ggtacacctt gttgttttag atggttcctt cttaacaaga tgctagattt tgatgatttg 2640
attctttaca aggagtagtc taaaatactg gaagtgtcaa cttaaaaagt tcaaacttgc 2700
cctagccata cgtgatacat tagacaactg atgtgctgtg ttatctgcaa ctgacaatta 2760
ttgttgttgc tatctccttt ttttgcttta gagtaggcag gtattttgtg tcaactaaaa 2820
gtgtgtttca cgtgttgcct ttcttaatac cttttgaata atagggtata agaaaatttt 2880
catgaatggt ttgctttcca attgttgatg ttgctttcct tcaccttatt ttttaaacca 2940
ccatgattaa tctatttatc taggtaatat tgctcatgct gattcatgat acgtggtaaa 3000
aaaataggtc ttttcaactt tcagaggctg ctggactcca tttatttagt tgattttgat 3060
ccctacaccg attttatcca ttggtttata ttcactgcta acatatgata ctcgctatca 3120
tttacttcac tgttgatcat caatgtttta attttgacca ttattaactt ttatatgatt 3180
aatttagtat taatgaaagt tgtgcggttg tatttatggc atgacacaca tttctaacat 3240
ggcatacttt taagcataca taactttttt tgtaaaaagg acaagcgatc aaaggtaaaa 3300
aggtgaatag catacagtga gaagtatttg agaccaaagg gagtataatc attatctact 3360
aaacaaatac aataccattc attataagct gagttcgcat agtgtaattg aaagtgggga 3420
atgctagtat agctcacaca tgaaatcaag tccctgtgaa atgatagtaa accctacgtg 3480
ttcattgttt tagtcttatg atatgttcca gttatgtttg ccatggttgt tgaatgcagt 3540
agtcaaagtt tgcttctgtt tgtttgagtg gattctcatt taccttgcct tttccaccat 3600
atactcatat gggctttgtt tcacaagtga taacattggt taatggctat atgctatcct 3660
tacagtttca ttcttctttt aagatatgag tcaccatgca catttcttca acttctgatt 3720
ttgtctaata ttgcagacac ctaagagaac ttgacctgca agagaacgag attgaggatt 3780
gttctattca ttggctcagc ctcttcccgg aatcgttcac ttctctagta actctaaact 3840
tttcatgctt agagggggag gtcaatatca ctgtacttga acggttagtg accagatgtc 3900
acaacctgaa gactcttaag ctcaacaatg ctatccccct tgacaagctt gctagcctcc 3960
ttcataaggc tcctcagcta gttgaactcg gaactggcaa attctctgct gattaccatt 4020
ccgatctgtt tgcaaagctg gaggcggcgt ttggaggttg taaaagcttg agaaggcttt 4080
ctggggcttg ggatgctgtt ccagattatc tgccagcatt ctattgtgta tgtgaaggcc 4140
tcacatcact taatctgagt tatgctactg tgcgaggtcc tgagctcatc aaattcatta 4200
gtagatgcag aaatttgcaa caattatggg tatgcaactc ttgtcaattc ttttttacta 4260
ctatgtattc atcccattcg aatctgttaa cttttgaaaa actgttgcat atctctttgc 4320
aatacaagca cttgattccc tacaagtgga ttttgcaaat tttttctttt cttctgtgac 4380
ctccagtaaa aaagttcctg atgttttctt acttatcatg gtcactagta aaagtccgac 4440
tacatgtatt cttttttcac aatgtagttt tatcaattat attggtatag ggctgcaata 4500
gcattttatg atgaaactga ctgcactttt cccatgacaa caagaatttg tggcttggaa 4560
taatctattg agtgttaaat catgcatatg tttttcatgt ttgtcaatac agttttgtag 4620
gatgcatgtt acattcaatg gcatgctgtt gggtgcctcc aatgaataga ttgaaattac 4680
cactcttatc ttttggggaa gtaggatatt tatatggaaa aaacatagat gacaatggta 4740
tcctatcctt attagaatca ttgtgaaaag actgccagat gaatccattt gctctagggc 4800
agcattcttt gcctttcttg gcaaactgag ttattgtctg aaccctcata gtttccatcc 4860
tttaactgtt aactgctaga tgagttgctt gctctacggc agatatttta aactgaatta 4920
tgatctgaac cccaatggct attaacctgg tcatgttagc tctttatgca atcttttttc 4980
tctatatttt tcttctaaaa gttaagctgc aatttttcag gtgatggacc tcattgagga 5040
tcatggttta gctgttgtgg catcatcttg caataaactt caagagttgc gggtcttccc 5100
ttctgaccct tttggtgcag gattcttgac tgaaagaggt cttgttgatg tctctgcaag 5160
ttgtccaatg ttggagtcag tgctctactt ctgcagacgg atgacaaatg aggcacttat 5220
taccattgca aagaaccgtc ccaacttcac ttgcttccgc ctatgcatcc ttgagccaca 5280
cactccagac tacatcacac gggagcctct tgatgcaggt ttcagcgcca ttgtggagtc 5340
atgcaggggc cttaggcgtc tctctatctc aggccttctc acagatcttg tgtttaaatc 5400
cattggggca catgctgatc gtcttgagat gctttcaatc gccttcgctg ggaacagcga 5460
cttgggcctg cattacatcc tctcaggctg caagagcctg aagaaactgg agatcaggga 5520
ctgcccattt ggtgataagc cattgctggc gaacgcagca aagctggaga caatgcgatc 5580
cctttggatg tcgtcgtgct tgttgaccct gggcgcatgc cgacagcttg cacgcaagat 5640
gccccgcctt agtgtggaga tcatgaacga tcctggaagg tcatgcccct tggattcgct 5700
tccggatgaa acacctgttg agaaactgta cgtctaccgg acgatcgcag gtccaaggtc 5760
cgacacacca gcctgcgtcc agattgtgta g 5791
<210> 11
<211> 644
<212> БЕЛОК
<213> Аминокислотная последовательность белка ALS Oryza sativa L.
(аминокислотная последовательность)
<400> 11
Met Ala Thr Thr Ala Ala Ala Ala Ala Ala Thr Leu Ser Ala Ala Ala
1 5 10 15
Thr Ala Lys Thr Gly Arg Lys Asn His Gln Arg His His Val Leu Pro
20 25 30
Ala Arg Gly Arg Val Gly Ala Ala Ala Val Arg Cys Ser Ala Val Ser
35 40 45
Pro Val Thr Pro Pro Ser Pro Ala Pro Pro Ala Thr Pro Leu Arg Pro
50 55 60
Trp Gly Pro Ala Glu Pro Arg Lys Gly Ala Asp Ile Leu Val Glu Ala
65 70 75 80
Leu Glu Arg Cys Gly Val Ser Asp Val Phe Ala Tyr Pro Gly Gly Ala
85 90 95
Ser Met Glu Ile His Gln Ala Leu Thr Arg Ser Pro Val Ile Thr Asn
100 105 110
His Leu Phe Arg His Glu Gln Gly Glu Ala Phe Ala Ala Ser Gly Tyr
115 120 125
Ala Arg Ala Ser Gly Arg Val Gly Val Cys Val Ala Thr Ser Gly Pro
130 135 140
Gly Ala Thr Asn Leu Val Ser Ala Leu Ala Asp Ala Leu Leu Asp Ser
145 150 155 160
Val Pro Met Val Ala Ile Thr Gly Gln Val Pro Arg Arg Met Ile Gly
165 170 175
Thr Asp Ala Phe Gln Glu Thr Pro Ile Val Glu Val Thr Arg Ser Ile
180 185 190
Thr Lys His Asn Tyr Leu Val Leu Asp Val Glu Asp Ile Pro Arg Val
195 200 205
Ile Gln Glu Ala Phe Phe Leu Ala Ser Ser Gly Arg Pro Gly Pro Val
210 215 220
Leu Val Asp Ile Pro Lys Asp Ile Gln Gln Gln Met Ala Val Pro Val
225 230 235 240
Trp Asp Thr Ser Met Asn Leu Pro Gly Tyr Ile Ala Arg Leu Pro Lys
245 250 255
Pro Pro Ala Thr Glu Leu Leu Glu Gln Val Leu Arg Leu Val Gly Glu
260 265 270
Ser Arg Arg Pro Ile Leu Tyr Val Gly Gly Gly Cys Ser Ala Ser Gly
275 280 285
Asp Glu Leu Arg Arg Phe Val Glu Leu Thr Gly Ile Pro Val Thr Thr
290 295 300
Thr Leu Met Gly Leu Gly Asn Phe Pro Ser Asp Asp Pro Leu Ser Leu
305 310 315 320
Arg Met Leu Gly Met His Gly Thr Val Tyr Ala Asn Tyr Ala Val Asp
325 330 335
Lys Ala Asp Leu Leu Leu Ala Phe Gly Val Arg Phe Asp Asp Arg Val
340 345 350
Thr Gly Lys Ile Glu Ala Phe Ala Ser Arg Ala Lys Ile Val His Ile
355 360 365
Asp Ile Asp Pro Ala Glu Ile Gly Lys Asn Lys Gln Pro His Val Ser
370 375 380
Ile Cys Ala Asp Val Lys Leu Ala Leu Gln Gly Leu Asn Ala Leu Leu
385 390 395 400
Asp Gln Ser Thr Thr Lys Thr Ser Ser Asp Phe Ser Ala Trp His Asn
405 410 415
Glu Leu Asp Gln Gln Lys Arg Glu Phe Pro Leu Gly Tyr Lys Thr Phe
420 425 430
Gly Glu Glu Ile Pro Pro Gln Tyr Ala Ile Gln Val Leu Asp Glu Leu
435 440 445
Thr Lys Gly Glu Ala Ile Ile Ala Thr Gly Val Gly Gln His Gln Met
450 455 460
Trp Ala Ala Gln Tyr Tyr Thr Tyr Lys Arg Pro Arg Gln Trp Leu Ser
465 470 475 480
Ser Ala Gly Leu Gly Ala Met Gly Phe Gly Leu Pro Ala Ala Ala Gly
485 490 495
Ala Ser Val Ala Asn Pro Gly Val Thr Val Val Asp Ile Asp Gly Asp
500 505 510
Gly Ser Phe Leu Met Asn Ile Gln Glu Leu Ala Leu Ile Arg Ile Glu
515 520 525
Asn Leu Pro Val Lys Val Met Val Leu Asn Asn Gln His Leu Gly Met
530 535 540
Val Val Gln Trp Glu Asp Arg Phe Tyr Lys Ala Asn Arg Ala His Thr
545 550 555 560
Tyr Leu Gly Asn Pro Glu Cys Glu Ser Glu Ile Tyr Pro Asp Phe Val
565 570 575
Thr Ile Ala Lys Gly Phe Asn Ile Pro Ala Val Arg Val Thr Lys Lys
580 585 590
Ser Glu Val Arg Ala Ala Ile Lys Lys Met Leu Glu Thr Pro Gly Pro
595 600 605
Tyr Leu Leu Asp Ile Ile Val Pro His Gln Glu His Val Leu Pro Met
610 615 620
Ile Pro Asn Gly Gly Ala Phe Lys Asp Met Ile Leu Asp Gly Asp Gly
625 630 635 640
Arg Thr Met Tyr
<210> 12
<211> 1935
<212> ДНК
<213> ДНК последовательность гена ALS Oryza sativa L. (ДНК
последовательность)
<400> 12
atggctacga ccgccgcggc cgcggccgcc accttgtccg ccgccgcgac ggccaagacc 60
ggccgtaaga accaccagcg acaccacgtc cttcccgctc gaggccgggt gggggcggcg 120
gcggtcaggt gctcggcggt gtccccggtc accccgccgt ccccggcgcc gccggccacg 180
ccgctccggc cgtgggggcc ggccgagccc cgcaagggcg cggacatcct cgtggaggcg 240
ctggagcggt gcggcgtcag cgacgtgttc gcctacccgg gcggcgcgtc catggagatc 300
caccaggcgc tgacgcgctc cccggtcatc accaaccacc tcttccgcca cgagcagggc 360
gaggcgttcg cggcgtccgg gtacgcgcgc gcgtccggcc gcgtcggggt ctgcgtcgcc 420
acctccggcc ccggggcaac caacctcgtg tccgcgctcg ccgacgcgct gctcgactcc 480
gtcccgatgg tcgccatcac gggccaggtc ccccgccgca tgatcggcac cgacgccttc 540
caggagacgc ccatagtcga ggtcacccgc tccatcacca agcacaatta ccttgtcctt 600
gatgtggagg acatcccccg cgtcatacag gaagccttct tcctcgcgtc ctcgggccgt 660
cctggcccgg tgctggtcga catccccaag gacatccagc agcagatggc tgtgccagtc 720
tgggacacct cgatgaatct accggggtac attgcacgcc tgcccaagcc acccgcgaca 780
gaattgcttg agcaggtctt gcgtctggtt ggcgagtcac ggcgcccgat tctctatgtc 840
ggtggtggct gctctgcatc tggtgatgaa ttgcgccggt ttgttgagct gaccggcatc 900
ccagttacaa ccactctgat gggcctcggc aatttcccca gtgatgatcc gttgtccctg 960
cgcatgcttg ggatgcatgg cacggtgtac gcaaattatg cggtggataa ggctgacctg 1020
ttgcttgcat ttggcgtgcg gtttgatgat cgtgtgacag ggaaaattga ggcttttgca 1080
agcagggcca agattgtgca cattgacatt gatccagcgg agattggaaa gaacaagcaa 1140
ccacatgtgt caatttgcgc agatgttaag cttgctttac agggcttgaa tgctctgcta 1200
gaccagagca caacaaagac aagttctgat tttagtgcat ggcacaatga gttggaccag 1260
cagaagaggg agtttcctct ggggtacaag acttttggtg aagagatccc accgcaatat 1320
gctattcagg tgctggatga gctgacgaaa ggggaggcaa tcatcgctac tggtgttgga 1380
cagcaccaga tgtgggcggc acaatattac acctacaagc ggccacggca gtggctgtct 1440
tcggctggtc tgggcgcaat gggatttggg ctgcctgctg cagctggtgc ttctgtggct 1500
aacccaggtg tcacagttgt tgatattgat ggggatggta gcttcctcat gaacattcag 1560
gagttggcat tgatccgcat tgagaacctc ccggtgaagg tgatggtgtt gaacaaccaa 1620
catttgggta tggttgtgca atgggaggat aggttttaca aggcaaatag ggcgcataca 1680
tacttgggca acccagaatg tgagagcgag atatatccag attttgtgac tattgctaaa 1740
gggttcaata ttcctgcagt ccgtgtaaca aagaagagtg aagtccgtgc cgccatcaag 1800
aagatgctcg agaccccagg gccatacttg ttggatatca tcgtcccaca ccaggagcat 1860
gtgctgccta tgatcccaaa tgggggcgca ttcaaggaca tgatcctgga tggtgatggc 1920
aggactatgt attaa 1935
<210> 13
<211> 2327
<212> БЕЛОК
<213> Аминокислотная последовательность белка ACCase2 Oryza sativa L.
(аминокислотная последовательность)
<400> 13
Met Thr Ser Thr His Val Ala Thr Leu Gly Val Gly Ala Gln Ala Pro
1 5 10 15
Pro Arg His Gln Lys Lys Ser Ala Gly Thr Ala Phe Val Ser Ser Gly
20 25 30
Ser Ser Arg Pro Ser Tyr Arg Lys Asn Gly Gln Arg Thr Arg Ser Leu
35 40 45
Arg Glu Glu Ser Asn Gly Gly Val Ser Asp Ser Lys Lys Leu Asn His
50 55 60
Ser Ile Arg Gln Gly Leu Ala Gly Ile Ile Asp Leu Pro Asn Asp Ala
65 70 75 80
Ala Ser Glu Val Asp Ile Ser His Gly Ser Glu Asp Pro Arg Gly Pro
85 90 95
Thr Val Pro Gly Ser Tyr Gln Met Asn Gly Ile Ile Asn Glu Thr His
100 105 110
Asn Gly Arg His Ala Ser Val Ser Lys Val Val Glu Phe Cys Thr Ala
115 120 125
Leu Gly Gly Lys Thr Pro Ile His Ser Val Leu Val Ala Asn Asn Gly
130 135 140
Met Ala Ala Ala Lys Phe Met Arg Ser Val Arg Thr Trp Ala Asn Asp
145 150 155 160
Thr Phe Gly Ser Glu Lys Ala Ile Gln Leu Ile Ala Met Ala Thr Pro
165 170 175
Glu Asp Leu Arg Ile Asn Ala Glu His Ile Arg Ile Ala Asp Gln Phe
180 185 190
Val Glu Val Pro Gly Gly Thr Asn Asn Asn Asn Tyr Ala Asn Val Gln
195 200 205
Leu Ile Val Glu Ile Ala Glu Arg Thr Gly Val Ser Ala Val Trp Pro
210 215 220
Gly Trp Gly His Ala Ser Glu Asn Pro Glu Leu Pro Asp Ala Leu Thr
225 230 235 240
Ala Lys Gly Ile Val Phe Leu Gly Pro Pro Ala Ser Ser Met His Ala
245 250 255
Leu Gly Asp Lys Val Gly Ser Ala Leu Ile Ala Gln Ala Ala Gly Val
260 265 270
Pro Thr Leu Ala Trp Ser Gly Ser His Val Glu Val Pro Leu Glu Cys
275 280 285
Cys Leu Asp Ser Ile Pro Asp Glu Met Tyr Arg Lys Ala Cys Val Thr
290 295 300
Thr Thr Glu Glu Ala Val Ala Ser Cys Gln Val Val Gly Tyr Pro Ala
305 310 315 320
Met Ile Lys Ala Ser Trp Gly Gly Gly Gly Lys Gly Ile Arg Lys Val
325 330 335
His Asn Asp Asp Glu Val Arg Thr Leu Phe Lys Gln Val Gln Gly Glu
340 345 350
Val Pro Gly Ser Pro Ile Phe Ile Met Arg Leu Ala Ala Gln Ser Arg
355 360 365
His Leu Glu Val Gln Leu Leu Cys Asp Gln Tyr Gly Asn Val Ala Ala
370 375 380
Leu His Ser Arg Asp Cys Ser Val Gln Arg Arg His Gln Lys Ile Ile
385 390 395 400
Glu Glu Gly Pro Val Thr Val Ala Pro Arg Glu Thr Val Lys Glu Leu
405 410 415
Glu Gln Ala Ala Arg Arg Leu Ala Lys Ala Val Gly Tyr Val Gly Ala
420 425 430
Ala Thr Val Glu Tyr Leu Tyr Ser Met Glu Thr Gly Glu Tyr Tyr Phe
435 440 445
Leu Glu Leu Asn Pro Arg Leu Gln Val Glu His Pro Val Thr Glu Trp
450 455 460
Ile Ala Glu Val Asn Leu Pro Ala Ala Gln Val Ala Val Gly Met Gly
465 470 475 480
Ile Pro Leu Trp Gln Ile Pro Glu Ile Arg Arg Phe Tyr Gly Met Asn
485 490 495
His Gly Gly Gly Tyr Asp Leu Trp Arg Lys Thr Ala Ala Leu Ala Thr
500 505 510
Pro Phe Asn Phe Asp Glu Val Asp Ser Lys Trp Pro Lys Gly His Cys
515 520 525
Val Ala Val Arg Ile Thr Ser Glu Asp Pro Asp Asp Gly Phe Lys Pro
530 535 540
Thr Gly Gly Lys Val Lys Glu Ile Ser Phe Lys Ser Lys Pro Asn Val
545 550 555 560
Trp Ala Tyr Phe Ser Val Lys Ser Gly Gly Gly Ile His Glu Phe Ala
565 570 575
Asp Ser Gln Phe Gly His Val Phe Ala Tyr Gly Thr Thr Arg Ser Ala
580 585 590
Ala Ile Thr Thr Met Ala Leu Ala Leu Lys Glu Val Gln Ile Arg Gly
595 600 605
Glu Ile His Ser Asn Val Asp Tyr Thr Val Asp Leu Leu Asn Ala Ser
610 615 620
Asp Phe Arg Glu Asn Lys Ile His Thr Gly Trp Leu Asp Thr Arg Ile
625 630 635 640
Ala Met Arg Val Gln Ala Glu Arg Pro Pro Trp Tyr Ile Ser Val Val
645 650 655
Gly Gly Ala Leu Tyr Lys Thr Val Thr Ala Asn Thr Ala Thr Val Ser
660 665 670
Asp Tyr Val Gly Tyr Leu Thr Lys Gly Gln Ile Pro Pro Lys His Ile
675 680 685
Ser Leu Val Tyr Thr Thr Val Ala Leu Asn Ile Asp Gly Lys Lys Tyr
690 695 700
Thr Ile Asp Thr Val Arg Ser Gly His Gly Ser Tyr Arg Leu Arg Met
705 710 715 720
Asn Gly Ser Thr Val Asp Ala Asn Val Gln Ile Leu Cys Asp Gly Gly
725 730 735
Leu Leu Met Gln Leu Asp Gly Asn Ser His Val Ile Tyr Ala Glu Glu
740 745 750
Glu Ala Ser Gly Thr Arg Leu Leu Ile Asp Gly Lys Thr Cys Met Leu
755 760 765
Gln Asn Asp His Asp Pro Ser Lys Leu Leu Ala Glu Thr Pro Cys Lys
770 775 780
Leu Leu Arg Phe Leu Val Ala Asp Gly Ala His Val Asp Ala Asp Val
785 790 795 800
Pro Tyr Ala Glu Val Glu Val Met Lys Met Cys Met Pro Leu Leu Ser
805 810 815
Pro Ala Ser Gly Val Ile His Val Val Met Ser Glu Gly Gln Ala Met
820 825 830
Gln Ala Gly Asp Leu Ile Ala Arg Leu Asp Leu Asp Asp Pro Ser Ala
835 840 845
Val Lys Arg Ala Glu Pro Phe Glu Asp Thr Phe Pro Gln Met Gly Leu
850 855 860
Pro Ile Ala Ala Ser Gly Gln Val His Lys Leu Cys Ala Ala Ser Leu
865 870 875 880
Asn Ala Cys Arg Met Ile Leu Ala Gly Tyr Glu His Asp Ile Asp Lys
885 890 895
Val Val Pro Glu Leu Val Tyr Cys Leu Asp Thr Pro Glu Leu Pro Phe
900 905 910
Leu Gln Trp Glu Glu Leu Met Ser Val Leu Ala Thr Arg Leu Pro Arg
915 920 925
Asn Leu Lys Ser Glu Leu Glu Gly Lys Tyr Glu Glu Tyr Lys Val Lys
930 935 940
Phe Asp Ser Gly Ile Ile Asn Asp Phe Pro Ala Asn Met Leu Arg Val
945 950 955 960
Ile Ile Glu Glu Asn Leu Ala Cys Gly Ser Glu Lys Glu Lys Ala Thr
965 970 975
Asn Glu Arg Leu Val Glu Pro Leu Met Ser Leu Leu Lys Ser Tyr Glu
980 985 990
Gly Gly Arg Glu Ser His Ala His Phe Val Val Lys Ser Leu Phe Glu
995 1000 1005
Glu Tyr Leu Tyr Val Glu Glu Leu Phe Ser Asp Gly Ile Gln Ser Asp
1010 1015 1020
Val Ile Glu Arg Leu Arg Leu Gln His Ser Lys Asp Leu Gln Lys Val
1025 1030 1035 1040
Val Asp Ile Val Leu Ser His Gln Ser Val Arg Asn Lys Thr Lys Leu
1045 1050 1055
Ile Leu Lys Leu Met Glu Ser Leu Val Tyr Pro Asn Pro Ala Ala Tyr
1060 1065 1070
Arg Asp Gln Leu Ile Arg Phe Ser Ser Leu Asn His Lys Ala Tyr Tyr
1075 1080 1085
Lys Leu Ala Leu Lys Ala Ser Glu Leu Leu Glu Gln Thr Lys Leu Ser
1090 1095 1100
Glu Leu Arg Ala Arg Ile Ala Arg Ser Leu Ser Glu Leu Glu Met Phe
1105 1110 1115 1120
Thr Glu Glu Ser Lys Gly Leu Ser Met His Lys Arg Glu Ile Ala Ile
1125 1130 1135
Lys Glu Ser Met Glu Asp Leu Val Thr Ala Pro Leu Pro Val Glu Asp
1140 1145 1150
Ala Leu Ile Ser Leu Phe Asp Cys Ser Asp Thr Thr Val Gln Gln Arg
1155 1160 1165
Val Ile Glu Thr Tyr Ile Ala Arg Leu Tyr Gln Pro His Leu Val Lys
1170 1175 1180
Asp Ser Ile Lys Met Lys Trp Ile Glu Ser Gly Val Ile Ala Leu Trp
1185 1190 1195 1200
Glu Phe Pro Glu Gly His Phe Asp Ala Arg Asn Gly Gly Ala Val Leu
1205 1210 1215
Gly Asp Lys Arg Trp Gly Ala Met Val Ile Val Lys Ser Leu Glu Ser
1220 1225 1230
Leu Ser Met Ala Ile Arg Phe Ala Leu Lys Glu Thr Ser His Tyr Thr
1235 1240 1245
Ser Ser Glu Gly Asn Met Met His Ile Ala Leu Leu Gly Ala Asp Asn
1250 1255 1260
Lys Met His Ile Ile Gln Glu Ser Gly Asp Asp Ala Asp Arg Ile Ala
1265 1270 1275 1280
Lys Leu Pro Leu Ile Leu Lys Asp Asn Val Thr Asp Leu His Ala Ser
1285 1290 1295
Gly Val Lys Thr Ile Ser Phe Ile Val Gln Arg Asp Glu Ala Arg Met
1300 1305 1310
Thr Met Arg Arg Thr Phe Leu Trp Ser Asp Glu Lys Leu Ser Tyr Glu
1315 1320 1325
Glu Glu Pro Ile Leu Arg His Val Glu Pro Pro Leu Ser Ala Leu Leu
1330 1335 1340
Glu Leu Asp Lys Leu Lys Val Lys Gly Tyr Asn Glu Met Lys Tyr Thr
1345 1350 1355 1360
Pro Ser Arg Asp Arg Gln Trp His Ile Tyr Thr Leu Arg Asn Thr Glu
1365 1370 1375
Asn Pro Lys Met Leu His Arg Val Phe Phe Arg Thr Leu Val Arg Gln
1380 1385 1390
Pro Ser Val Ser Asn Lys Phe Ser Ser Gly Gln Ile Gly Asp Met Glu
1395 1400 1405
Val Gly Ser Ala Glu Glu Pro Leu Ser Phe Thr Ser Thr Ser Ile Leu
1410 1415 1420
Arg Ser Leu Met Thr Ala Ile Glu Glu Leu Glu Leu His Ala Ile Arg
1425 1430 1435 1440
Thr Gly His Ser His Met Tyr Leu His Val Leu Lys Glu Gln Lys Leu
1445 1450 1455
Leu Asp Leu Val Pro Val Ser Gly Asn Thr Val Leu Asp Val Gly Gln
1460 1465 1470
Asp Glu Ala Thr Ala Tyr Ser Leu Leu Lys Glu Met Ala Met Lys Ile
1475 1480 1485
His Glu Leu Val Gly Ala Arg Met His His Leu Ser Val Cys Gln Trp
1490 1495 1500
Glu Val Lys Leu Lys Leu Asp Cys Asp Gly Pro Ala Ser Gly Thr Trp
1505 1510 1515 1520
Arg Ile Val Thr Thr Asn Val Thr Ser His Thr Cys Thr Val Asp Ile
1525 1530 1535
Tyr Arg Glu Met Glu Asp Lys Glu Ser Arg Lys Leu Val Tyr His Pro
1540 1545 1550
Ala Thr Pro Ala Ala Gly Pro Leu His Gly Val Ala Leu Asn Asn Pro
1555 1560 1565
Tyr Gln Pro Leu Ser Val Ile Asp Leu Lys Arg Cys Ser Ala Arg Asn
1570 1575 1580
Asn Arg Thr Thr Tyr Cys Tyr Asp Phe Pro Leu Ala Phe Glu Thr Ala
1585 1590 1595 1600
Val Arg Lys Ser Trp Ser Ser Ser Thr Ser Gly Ala Ser Lys Gly Val
1605 1610 1615
Glu Asn Ala Gln Cys Tyr Val Lys Ala Thr Glu Leu Val Phe Ala Asp
1620 1625 1630
Lys His Gly Ser Trp Gly Thr Pro Leu Val Gln Met Asp Arg Pro Ala
1635 1640 1645
Gly Leu Asn Asp Ile Gly Met Val Ala Trp Thr Leu Lys Met Ser Thr
1650 1655 1660
Pro Glu Phe Pro Ser Gly Arg Glu Ile Ile Val Val Ala Asn Asp Ile
1665 1670 1675 1680
Thr Phe Arg Ala Gly Ser Phe Gly Pro Arg Glu Asp Ala Phe Phe Glu
1685 1690 1695
Ala Val Thr Asn Leu Ala Cys Glu Lys Lys Leu Pro Leu Ile Tyr Leu
1700 1705 1710
Ala Ala Asn Ser Gly Ala Arg Ile Gly Ile Ala Asp Glu Val Lys Ser
1715 1720 1725
Cys Phe Arg Val Gly Trp Ser Asp Asp Gly Ser Pro Glu Arg Gly Phe
1730 1735 1740
Gln Tyr Ile Tyr Leu Ser Glu Glu Asp Tyr Ala Arg Ile Gly Thr Ser
1745 1750 1755 1760
Val Ile Ala His Lys Met Gln Leu Asp Ser Gly Glu Ile Arg Trp Val
1765 1770 1775
Ile Asp Ser Val Val Gly Lys Glu Asp Gly Leu Gly Val Glu Asn Ile
1780 1785 1790
His Gly Ser Ala Ala Ile Ala Ser Ala Tyr Ser Arg Ala Tyr Lys Glu
1795 1800 1805
Thr Phe Thr Leu Thr Phe Val Thr Gly Arg Thr Val Gly Ile Gly Ala
1810 1815 1820
Tyr Leu Ala Arg Leu Gly Ile Arg Cys Ile Gln Arg Leu Asp Gln Pro
1825 1830 1835 1840
Ile Ile Leu Thr Gly Tyr Ser Ala Leu Asn Lys Leu Leu Gly Arg Glu
1845 1850 1855
Val Tyr Ser Ser His Met Gln Leu Gly Gly Pro Lys Ile Met Ala Thr
1860 1865 1870
Asn Gly Val Val His Leu Thr Val Ser Asp Asp Leu Glu Gly Val Ser
1875 1880 1885
Asn Ile Leu Arg Trp Leu Ser Tyr Val Pro Ala Tyr Ile Gly Gly Pro
1890 1895 1900
Leu Pro Val Thr Thr Pro Leu Asp Pro Pro Asp Arg Pro Val Ala Tyr
1905 1910 1915 1920
Ile Pro Glu Asn Ser Cys Asp Pro Arg Ala Ala Ile Arg Gly Val Asp
1925 1930 1935
Asp Ser Gln Gly Lys Trp Leu Gly Gly Met Phe Asp Lys Asp Ser Phe
1940 1945 1950
Val Glu Thr Phe Glu Gly Trp Ala Lys Thr Val Val Thr Gly Arg Ala
1955 1960 1965
Lys Leu Gly Gly Ile Pro Val Gly Val Ile Ala Val Glu Thr Gln Thr
1970 1975 1980
Met Met Gln Thr Ile Pro Ala Asp Pro Gly Gln Leu Asp Ser Arg Glu
1985 1990 1995 2000
Gln Ser Val Pro Arg Ala Gly Gln Val Trp Phe Pro Asp Ser Ala Thr
2005 2010 2015
Lys Thr Ala Gln Ala Leu Leu Asp Phe Asn Arg Glu Gly Leu Pro Leu
2020 2025 2030
Phe Ile Leu Ala Asn Trp Arg Gly Phe Ser Gly Gly Gln Arg Asp Leu
2035 2040 2045
Phe Glu Gly Ile Leu Gln Ala Gly Ser Thr Ile Val Glu Asn Leu Arg
2050 2055 2060
Thr Tyr Asn Gln Pro Ala Phe Val Tyr Ile Pro Met Ala Ala Glu Leu
2065 2070 2075 2080
Arg Gly Gly Ala Trp Val Val Val Asp Ser Lys Ile Asn Pro Asp Arg
2085 2090 2095
Ile Glu Cys Tyr Ala Glu Arg Thr Ala Lys Gly Asn Val Leu Glu Pro
2100 2105 2110
Gln Gly Leu Ile Glu Ile Lys Phe Arg Ser Glu Glu Leu Gln Asp Cys
2115 2120 2125
Met Ser Arg Leu Asp Pro Thr Leu Ile Asp Leu Lys Ala Lys Leu Glu
2130 2135 2140
Val Ala Asn Lys Asn Gly Ser Ala Asp Thr Lys Ser Leu Gln Glu Asn
2145 2150 2155 2160
Ile Glu Ala Arg Thr Lys Gln Leu Met Pro Leu Tyr Thr Gln Ile Ala
2165 2170 2175
Ile Arg Phe Ala Glu Leu His Asp Thr Ser Leu Arg Met Ala Ala Lys
2180 2185 2190
Gly Val Ile Lys Lys Val Val Asp Trp Glu Glu Ser Arg Ser Phe Phe
2195 2200 2205
Tyr Lys Arg Leu Arg Arg Arg Ile Ser Glu Asp Val Leu Ala Lys Glu
2210 2215 2220
Ile Arg Ala Val Ala Gly Glu Gln Phe Ser His Gln Pro Ala Ile Glu
2225 2230 2235 2240
Leu Ile Lys Lys Trp Tyr Ser Ala Ser His Ala Ala Glu Trp Asp Asp
2245 2250 2255
Asp Asp Ala Phe Val Ala Trp Met Asp Asn Pro Glu Asn Tyr Lys Asp
2260 2265 2270
Tyr Ile Gln Tyr Leu Lys Ala Gln Arg Val Ser Gln Ser Leu Ser Ser
2275 2280 2285
Leu Ser Asp Ser Ser Ser Asp Leu Gln Ala Leu Pro Gln Gly Leu Ser
2290 2295 2300
Met Leu Leu Asp Lys Met Asp Pro Ser Arg Arg Ala Gln Leu Val Glu
2305 2310 2315 2320
Glu Ile Arg Lys Val Leu Gly
2325
<210> 14
<211> 11927
<212> ДНК
<213> ДНК последовательность генома ACCase2 Oryza sativa L. (ДНК
последовательность)
<400> 14
atgacatcca cacatgtggc gacattggga gttggtgccc aggcacctcc tcgtcaccag 60
aaaaagtcag ctggcactgc atttgtatca tctgggtcat caagaccctc ataccgaaag 120
aatggtcagc gtactcggtc acttagggaa gaaagcaatg gaggagtgtc tgattccaaa 180
aagcttaacc actctattcg ccaaggtgac cactagctac tttacatatg ctataatttg 240
tgccaaacat aaacatgcaa tggctgctat tatttaaacg ttaatgttga aatagctgct 300
ataggataca gcaaaaatat ataattgact gggcaagatg caacaattgt ttttcactaa 360
agttagttat cttttgctgt aaaagacaac tgttttttac ataaaatggt attaataacc 420
ttgtaatatt caatgcaaca tgttctcaag taaaaaaaaa cattgcctgg ttgtataagc 480
aaatgtgtcg ttgtagacat cttattaaac ctttttgtga tatctattac cgtagggaac 540
aggggagctg tttaaatctg ttatcataga gtaatatgag aaaagtggat tgtgcgactt 600
tggcatgtat acctgctcaa tttcaaatat atgtctatgt gcaggtcttg ctggcatcat 660
tgacctccca aatgacgcag cttcagaagt tgatatttca cagtaaggac tttatatttt 720
ataataatta ttatataatt ttctgacatg ttttgagaac ctcaaaacat gtgattgcac 780
cttccttttt tatgtctggt tcagaaactg ataagttttg acagtgttta ggatggatct 840
ttgatgcgca cagtgctttc taatgttttc atttttgaaa gtaatgtttt aggaagaaat 900
atctgattaa atttatactt tatctttaca aaagtcaaat gcgttctgta tcaattgcgg 960
tttgtaatat ggcaagaaca tgctttcaga atttgttcat acaatgcttt ctttctatta 1020
ttatgtagaa caaataccta atactttgtt caccttttat agtggacacc tctcacagct 1080
ttttcagtaa gtgatgcaat tttgtacatt tgtaagatgt gttccagaaa ccttttctcc 1140
tgcaattcta atgtacccac tcaaactggt atcaccaaag atctccatct gattgaaaaa 1200
aagctgcgtg aagtatgctt atttatgcta accatacatg atttatactg ttttatagta 1260
caatgcttat ttatgctaac catacataat tttattctgt tttctagtac attatttgtg 1320
cccctgacca taaatgatcc tttcttttac agtggttccg aagatcccag ggggcctacg 1380
gtcccaggtt cctaccaaat gaatgggatt atcaatgaaa cacataatgg gaggcatgct 1440
tcagtctcca aggttgttga gttttgtacg gcacttggtg gcaaaacacc aattcacagt 1500
gtattagtgg ccaacaatgg aatggcagca gctaagttca tgcggagtgt ccgaacatgg 1560
gctaatgata cttttggatc agagaaggca attcagctga tagctatggc aactccggag 1620
gatctgagga taaatgcaga gcacatcaga attgccgatc aatttgtaga ggtacctggt 1680
ggaacaaaca acaacaacta tgcaaatgtc caactcatag tggaggttag ttcagctcat 1740
ccctcaacac aacattttcg tttctattta agttagggaa aaatctctac gaccctccaa 1800
tttctgaaca tccaattttc accatcaact gcaatcacag atagcagaga gaacaggtgt 1860
ttctgctgtt tggcctggtt ggggtcatgc atctgagaat cctgaacttc cagatgcgct 1920
gactgcaaaa ggaattgttt ttcttgggcc accagcatca tcaatgcatg cattaggaga 1980
caaggttggc tcagctctca ttgctcaagc agctggagtt ccaacacttg cttggagtgg 2040
atcacatgtg agccttgtct tctctttttt agcttatcat cttatctttt cggtgatgca 2100
ttatcccaat gacactaaac cataggtgga agttcctctg gagtgttgct tggactcaat 2160
acctgatgag atgtatagaa aagcttgtgt tactaccaca gaggaagcag ttgcaagttg 2220
tcaggtggtt ggttatcctg ccatgattaa ggcatcttgg ggtggtggtg gtaaaggaat 2280
aaggaaggtt tgttcttctt gtagttatca agagattgtt tggattgcaa gtgtttagtg 2340
cccatagtta actctggtct ttctaacatg agtaactcaa ctttcttgca ggttcataat 2400
gatgatgagg ttaggacatt atttaagcaa gttcaaggcg aagtacctgg ttccccaata 2460
tttatcatga ggctagctgc tcaggtgggg ccttttatgg aagttacacc ttttccctta 2520
atgttgagtt attccggagt tattatggtt atgttctgta tgtttgatct gtaaattatt 2580
gaaattcacc tccattggtt ctccagatta gcagacctac aattctacat atggtttata 2640
ctttataaat actaggattt agggatcttc atatagttta tacatggtat ttagatttca 2700
tttgtaaccc tattgaagac atcctgattg ttgtcttatg tagagtcgac atcttgaagt 2760
tcagttgctt tgtgatcaat atggcaacgt agcagcactt cacagtcgag attgcagtgt 2820
acaacggcga caccaaaagg tctgctgtct cagttaaatc acccctctga atgatctact 2880
tcttgcctgc tgcgttggtc agaggaataa tggttgtatt ctactgaaca gataatcgag 2940
gaaggaccag ttactgttgc tcctcgtgag actgtgaaag agcttgagca ggcagcacgg 3000
aggcttgcta aagctgtggg ttatgttggt gctgctactg ttgaatacct ttacagcatg 3060
gaaactggtg aatattattt tctggaactt aatccacggc tacaggtcgg ctcctttgac 3120
attcttcagg aattaatttc tgttgaccac atgatttaca ttgtcaaatg gtctcacagg 3180
ttgagcatcc tgtcactgag tggatagctg aagtaaattt gcctgcggct caagttgctg 3240
ttggaatggg tatacccctt tggcagattc caggtaatgc ttcttcattt agttcctgct 3300
ctttgttaat tgaatgagct cttatacaga ccatgagaca cattctactg ttaattcata 3360
gtatcccctg acttgttagt gttagagata cagagatgta tcacaaattc attgtatctc 3420
ctcaaggact gtaaaaatcc tataattaaa tttctgaaaa tttgttcttt taagcagaaa 3480
aaaaatctct aaattatctc cctgtataca gagatcaggc gcttctacgg aatgaaccat 3540
ggaggaggct atgacctttg gaggaaaaca gcagctctag cgactccatt taactttgat 3600
gaagtagatt ctaaatggcc aaaaggccac tgcgtagctg ttagaataac tagcgaggat 3660
ccagatgatg ggtttaagcc tactggtgga aaagtaaagg tgcggtttcc tgatgttagg 3720
tgtatgaatt gaacacattg ctatattgca gctagtgaaa tgactggatc atggttctct 3780
tattttcagg agataagttt caagagtaaa ccaaatgttt gggcctattt ctcagtaaag 3840
gtagtcctca atattgttgc actgccacat tatttgagtt gtcctaacaa ttgtgctgca 3900
attgttagtt ttcaactatt tgttgttctg tttggttgac tggtaccctc tctttgcagt 3960
ctggtggagg catccatgaa ttcgctgatt ctcagttcgg tatgtaaagt taaaagagta 4020
atattgtctt tgctatttat gtttgtcctc acttttaaaa gatattgcct tccattacag 4080
gacatgtttt tgcgtatgga actactagat cggcagcaat aactaccatg gctcttgcac 4140
taaaagaggt tcaaattcgt ggagaaattc attcaaacgt agactacaca gttgacctat 4200
taaatgtaag gactaaatat ctgcttattg aaccttgctt tttggttccc taatgccatt 4260
ttagtctggc tactgaagaa cttatccatc atgccatttc tgttatctta aattcaggcc 4320
tcagatttta gagaaaataa gattcatact ggttggctgg ataccaggat agccatgcgt 4380
gttcaagctg agaggcctcc atggtatatt tcagtcgttg gaggggcttt atatgtaaga 4440
caaactatgc cactcattag catttatgtg aagcaaatgc ggaaaacatg atcaatatgt 4500
cgtcttattt aaatttattt atttttgtgc tgcagaaaac agtaactgcc aacacggcca 4560
ctgtttctga ttatgttggt tatcttacca agggccagat tccaccaaag gtactattct 4620
gttttttcag gatatgaatg ctgtttgaat gtgaaaacca ttgaccataa atccttgttt 4680
gcagcatata tcccttgtct atacgactgt tgctttgaat atagatggga aaaaatatac 4740
agtaagtgtg acattcttaa tggggaaact taatttgttg taaataatca atatcatatt 4800
gactcgtgta tgctgcatca tagatcgata ctgtgaggag tggacatggt agctacagat 4860
tgcgaatgaa tggatcaacg gttgacgcaa atgtacaaat attatgtgat ggtgggcttt 4920
taatgcaggt aatatcttct tcctagttaa agaagatata tcttgttcaa agaattctga 4980
ttattgatct tttaatgttt tcagctggat ggaaacagcc atgtaattta tgctgaagaa 5040
gaggccagtg gtacacgact tcttattgat ggaaagacat gcatgttaca ggtaatgata 5100
gccttgttct ttttagttct agtcacggtg tttgcttgct atttgttgta tctatttaat 5160
gcattcacta attactatat tagtttgcat catcaagtta aaatggaact tctttcttgc 5220
agaatgacca tgacccatca aagttattag ctgagacacc atgcaaactt cttcgtttct 5280
tggttgctga tggtgctcat gttgatgctg atgtaccata tgcggaagtt gaggttatga 5340
agatgtgcat gcccctctta tcacccgctt ctggtgtcat acatgttgta atgtctgagg 5400
gccaagcaat gcaggtacat tcctacattc cattcattgt gctgtgctga catgaacatt 5460
tcaagtaaat acctgtaact tgtttattat tctaggctgg tgatcttata gctaggctgg 5520
atcttgatga cccttctgct gttaagagag ctgagccgtt cgaagatact tttccacaaa 5580
tgggtctccc tattgctgct tctggccaag ttcacaaatt atgtgctgca agtctgaatg 5640
cttgtcgaat gatccttgcg gggtatgagc atgatattga caaggtaaac atcatgtcct 5700
cttgtttttt cttttgttta tcatgcattc ttatgttcat catgtcctct ggcaaatcta 5760
gattccgctg tcgtttcaca cagatttttc tcattctcat aatggtgcca aacataaata 5820
tgctgctata ttcatcaatg ttttcactcg atttctaatt ttgcttttga gttttaaact 5880
ttagtacaat ccatatctaa tctcctttgg caacagtgaa tccattatat atatttttat 5940
taaactgctt tctttttcag gttgtgccag agttggtata ctgcctagac actccggagc 6000
ttcctttcct gcagtgggag gagcttatgt ctgttttagc aactagactt ccaagaaatc 6060
ttaaaagtga ggtatattat ggttgacaag atagctagtc tcatgctcta aggacttgta 6120
catttcgcca cataggttaa ttttccatat caagttctaa tgtacgatat aaaagtagta 6180
ctggcctaaa acagtattgg tggttgacta tctttgttgt gtaagatcaa gtatttcttt 6240
ttcatgctta gtttgtcaat acttcacatt tatcactgac ttgtcgagct aaatgagatt 6300
ttatttgatt tctgtgctcc attatttttg tatatatata tatatattta actatgacta 6360
tatgttatgc ctcaaacgtt tcaaactctt tcagttggag ggcaaatatg aggaatacaa 6420
agtaaaattt gactctggga taatcaatga tttccctgcc aatatgctac gagtgataat 6480
tgaggtcagt tattcaattt gttgtgataa tcactgcctt aactgttcgt tcttttaaca 6540
agcggtttta taggaaaatc ttgcatgtgg ttctgagaag gagaaggcta caaatgagag 6600
gcttgttgag cctcttatga gcctactgaa gtcatatgag ggtgggagag aaagtcatgc 6660
tcactttgtt gtcaagtccc tttttgagga gtatctctat gttgaagaat tgttcagtga 6720
tggaattcag gttaacttac ctattcgcat taaacaaatc atcagttgtt ttatgataaa 6780
gtcaaaatgt ttatatttcc cattcttctg tggatcaaat atatcacgga catgatatag 6840
tttccttagg ctatataatg gttcttcatc aaataatatt gcaggaaaca gtatagcaaa 6900
ctatttgtat atactcgaga tggaaattgt tagaaacatc attgactaaa tctgtccttt 6960
gttacgctgt ttttgtagtc tgatgtgatt gagcgtctgc gccttcaaca tagtaaagac 7020
ctacagaagg tcgtagacat tgtgttgtcc caccaggtaa atttcttcat ggtctgatga 7080
cttcactgcg aatggttact gaactgtctt cttgttctga caatgtgact tttctttgta 7140
gagtgttaga aataaaacta agctgatact aaaactcatg gagagtctgg tctatccaaa 7200
tcctgctgcc tacagggatc aattgattcg cttttcttcc cttaatcaca aagcgtatta 7260
caaggtgacc aggataaaca taaataaacg tgaatttttc aatgaccttt tcttctgaca 7320
tctgaatctg atgaatttct tgcatattaa tacagttggc acttaaagct agtgaacttc 7380
ttgaacaaac aaaacttagt gagctccgtg caagaatagc aaggagcctt tcagagctgg 7440
agatgtttac tgaggaaagc aagggtctct ccatgcataa gcgagaaatt gccattaagg 7500
agagcatgga agatttagtc actgctccac tgccagttga agatgcgctc atttctttat 7560
ttgattgtag tgatacaact gttcaacaga gagtgattga gacttatata gctcgattat 7620
accaggtatg agaagaaaga ccttttgaaa ttatttatat taacatatcc tagtaaaaca 7680
gcatgctcat catttcttaa aaaaagttta cagcacctga tgtttggtta ctgaccgcat 7740
cattaaaata aagttacttg ttgtggagag atgtattttg gaacttgtgg cacatgcagt 7800
aacatgctac tgctcgatat gtttgctaac ttgacaacaa tatttttcag cctcatcttg 7860
taaaggacag tatcaaaatg aaatggatag aatcgggtgt tattgcttta tgggaatttc 7920
ctgaagggca ttttgatgca agaaatggag gagcggttct tggtgacaaa agatggggtg 7980
ccatggtcat tgtcaagtct cttgaatcac tttcaatggc cattagattt gcactaaagg 8040
agacatcaca ctacactagc tctgagggca atatgatgca tattgctttg ttgggtgctg 8100
ataataagat gcatataatt caagaaaggt atgttcatat gctatgttgg tgctgaaata 8160
gttatatatg tagttagctg gtggagttct ggtaattaac ctatcccatt gttcagtggt 8220
gatgatgctg acagaatagc caaacttccc ttgatactaa aggataatgt aaccgatctg 8280
catgcctctg gtgtgaaaac aataagtttc attgttcaaa gagatgaagc acggatgaca 8340
atgcgtcgta ccttcctttg gtctgatgaa aagctttctt atgaggaaga gccaattctc 8400
cggcatgtgg aacctcctct ttctgcactt cttgagttgg tacgtgatat catcaaaatg 8460
ataatgtttt ggtatggcat tgattatctt ctatgctctt tgtatttatt cagcctattg 8520
tggatacagg acaagttgaa agtgaaagga tacaatgaaa tgaagtatac cccatcacgg 8580
gatcgtcaat ggcatatcta cacacttaga aatactgaaa accccaaaat gttgcaccgg 8640
gtatttttcc gaacccttgt caggcaaccc agtgtatcca acaagttttc ttcgggccag 8700
attggtgaca tggaagttgg gagtgctgaa gaacctctgt catttacatc aaccagcata 8760
ttaagatctt tgatgactgc tatagaggaa ttggagcttc acgcaattag aactggccat 8820
tcacacatgt atttgcatgt attgaaagaa caaaagcttc ttgatcttgt tccagtttca 8880
gggtaagtgc gcatatttct ttttgggaac atatgcttgc ttatgaggtt ggtcttctca 8940
atgatcttct tatcttactc aggaatacag ttttggatgt tggtcaagat gaagctactg 9000
catattcact tttaaaagaa atggctatga agatacatga acttgttggt gcaagaatgc 9060
accatctttc tgtatgccaa tgggaagtga aacttaagtt ggactgcgat ggtcctgcca 9120
gtggtacctg gaggattgta acaaccaatg ttactagtca cacttgcact gtggatgtaa 9180
gtttaatcct ctagcatttt gttttctttg gaaaagcatg tgattttaag ccggctggtc 9240
ctcataccca gacctagtga tctttatata gtgtagacat ttttctaact gcttttaatt 9300
gttttagatc taccgtgaga tggaagataa agaatcacgg aagttagtat accatcccgc 9360
cactccggcg gctggtcctc tgcatggtgt ggcactgaat aatccatatc agcctttgag 9420
tgtcattgat ctcaaacgct gttctgctag gaataataga actacatact gctatgattt 9480
tccactggtg agttgactgc tcccttatat tcaatgcatt accatagcaa attcatattc 9540
gttcatgttg tcaaaataag ccgatgaaaa ttcaaaactg taggcatttg aaactgcagt 9600
gaggaagtca tggtcctcta gtacctctgg tgcttctaaa ggtgttgaaa atgcccaatg 9660
ttatgttaaa gctacagagt tggtatttgc ggacaaacat gggtcatggg gcactccttt 9720
agttcaaatg gaccggcctg ctgggctcaa tgacattggt atggtagctt ggaccttgaa 9780
gatgtccact cctgaatttc ctagtggtag ggagattatt gttgttgcaa atgatattac 9840
gttcagagct ggatcatttg gcccaaggga agatgcattt tttgaagctg ttaccaacct 9900
agcctgtgag aagaaacttc ctcttattta tttggcagca aattctggtg ctcgaattgg 9960
catagcagat gaagtgaaat cttgcttccg tgttgggtgg tctgatgatg gcagccctga 10020
acgtgggttt cagtacattt atctaagcga agaagactat gctcgtattg gcacttctgt 10080
catagcacat aagatgcagc tagacagtgg tgaaattagg tgggttattg attctgttgt 10140
gggcaaggaa gatggacttg gtgtggagaa tatacatgga agtgctgcta ttgccagtgc 10200
ttattctagg gcatataagg agacatttac acttacattt gtgactggaa gaactgttgg 10260
aataggagct tatcttgctc gacttggcat ccggtgcata cagcgtcttg accagcctat 10320
tattcttaca ggctattctg cactgaacaa gcttcttggg cgggaagtgt acagctccca 10380
catgcagttg ggtggtccca aaatcatggc aactaatggt gttgtccatc ttactgtttc 10440
agatgacctt gaaggcgttt ctaatatatt gaggtggctc agttatgttc ctgcctacat 10500
tggtggacca cttccagtaa caacaccgtt ggacccaccg gacagacctg ttgcatacat 10560
tcctgagaac tcgtgtgatc ctcgagcggc tatccgtggt gttgatgaca gccaagggaa 10620
atggttaggt ggtatgtttg ataaagacag ctttgtggaa acatttgaag gttgggctaa 10680
gacagtggtt actggcagag caaagcttgg tggaattcca gtgggtgtga tagctgtgga 10740
gactcagacc atgatgcaaa ctatccctgc tgaccctggt cagcttgatt cccgtgagca 10800
atctgttcct cgtgctggac aagtgtggtt tccagattct gcaaccaaga ctgcgcaggc 10860
attgctggac ttcaaccgtg aaggattacc tctgttcatc ctcgctaact ggagaggctt 10920
ctctggtgga caaagagatc tttttgaagg aattcttcag gctggctcga ctattgttga 10980
gaaccttagg acatacaatc agcctgcctt tgtctacatt cccatggctg cagagctacg 11040
aggaggggct tgggttgtgg ttgatagcaa gataaaccca gaccgcattg agtgctatgc 11100
tgagaggact gcaaaaggca atgttctgga accgcaaggg ttaattgaga tcaagttcag 11160
gtcagaggaa ctccaggatt gcatgagtcg gcttgaccca acattaattg atctgaaagc 11220
aaaactcgaa gtagcaaata aaaatggaag tgctgacaca aaatcgcttc aagaaaatat 11280
agaagctcga acaaaacagt tgatgcctct atatactcag attgcgatac ggtttgctga 11340
attgcatgat acatccctca gaatggctgc gaaaggtgtg attaagaaag ttgtggactg 11400
ggaagaatca cgatctttct tctataagag attacggagg aggatctctg aggatgttct 11460
tgcaaaagaa attagagctg tagcaggtga gcagttttcc caccaaccag caatcgagct 11520
gatcaagaaa tggtattcag cttcacatgc agctgaatgg gatgatgacg atgcttttgt 11580
tgcttggatg gataaccctg aaaactacaa ggattatatt caatatctta aggctcaaag 11640
agtatcccaa tccctctcaa gtctttcaga ttccagctca gatttgcaag ccctgccaca 11700
gggtctttcc atgttactag ataaggtaat tagcttactg atgcttatat aaattctttt 11760
tcattacata tggctggaga actatctaat caaataatga ttataattcc aatcgttctt 11820
tttatgccat tatgatcttc tgaaatttcc ttctttggac acttattcag atggatccct 11880
ctagaagagc tcaacttgtt gaagaaatca ggaaggtcct tggttga 11927
<210> 15
<211> 4203
<212> ДНК
<213> ДНК последовательность SpCas9 (ДНК последовательность)
<400> 15
atggctccga agaagaagag gaaggttggc atccacgggg tgccagctgc tgacaagaag 60
tactcgatcg gcctcgatat tgggactaac tctgttggct gggccgtgat caccgacgag 120
tacaaggtgc cctcaaagaa gttcaaggtc ctgggcaaca ccgatcggca ttccatcaag 180
aagaatctca ttggcgctct cctgttcgac agcggcgaga cggctgaggc tacgcggctc 240
aagcgcaccg cccgcaggcg gtacacgcgc aggaagaatc gcatctgcta cctgcaggag 300
attttctcca acgagatggc gaaggttgac gattctttct tccacaggct ggaggagtca 360
ttcctcgtgg aggaggataa gaagcacgag cggcatccaa tcttcggcaa cattgtcgac 420
gaggttgcct accacgagaa gtaccctacg atctaccatc tgcggaagaa gctcgtggac 480
tccacagata aggcggacct ccgcctgatc tacctcgctc tggcccacat gattaagttc 540
aggggccatt tcctgatcga gggggatctc aacccggaca atagcgatgt tgacaagctg 600
ttcatccagc tcgtgcagac gtacaaccag ctcttcgagg agaaccccat taatgcgtca 660
ggcgtcgacg cgaaggctat cctgtccgct aggctctcga agtctcggcg cctcgagaac 720
ctgatcgccc agctgccggg cgagaagaag aacggcctgt tcgggaatct cattgcgctc 780
agcctggggc tcacgcccaa cttcaagtcg aatttcgatc tcgctgagga cgccaagctg 840
cagctctcca aggacacata cgacgatgac ctggataacc tcctggccca gatcggcgat 900
cagtacgcgg acctgttcct cgctgccaag aatctgtcgg acgccatcct cctgtctgat 960
attctcaggg tgaacaccga gattacgaag gctccgctct cagcctccat gatcaagcgc 1020
tacgacgagc accatcagga tctgaccctc ctgaaggcgc tggtcaggca gcagctcccc 1080
gagaagtaca aggagatctt cttcgatcag tcgaagaacg gctacgctgg gtacattgac 1140
ggcggggcct ctcaggagga gttctacaag ttcatcaagc cgattctgga gaagatggac 1200
ggcacggagg agctgctggt gaagctcaat cgcgaggacc tcctgaggaa gcagcggaca 1260
ttcgataacg gcagcatccc acaccagatt catctcgggg agctgcacgc tatcctgagg 1320
aggcaggagg acttctaccc tttcctcaag gataaccgcg agaagatcga gaagattctg 1380
actttcagga tcccgtacta cgtcggccca ctcgctaggg gcaactcccg cttcgcttgg 1440
atgacccgca agtcagagga gacgatcacg ccgtggaact tcgaggaggt ggtcgacaag 1500
ggcgctagcg ctcagtcgtt catcgagagg atgacgaatt tcgacaagaa cctgccaaat 1560
gagaaggtgc tccctaagca ctcgctcctg tacgagtact tcacagtcta caacgagctg 1620
actaaggtga agtatgtgac cgagggcatg aggaagccgg ctttcctgtc tggggagcag 1680
aagaaggcca tcgtggacct cctgttcaag accaaccgga aggtcacggt taagcagctc 1740
aaggaggact acttcaagaa gattgagtgc ttcgattcgg tcgagatctc tggcgttgag 1800
gaccgcttca acgcctccct ggggacctac cacgatctcc tgaagatcat taaggataag 1860
gacttcctgg acaacgagga gaatgaggat atcctcgagg acattgtgct gacactcact 1920
ctgttcgagg accgggagat gatcgaggag cgcctgaaga cttacgccca tctcttcgat 1980
gacaaggtca tgaagcagct caagaggagg aggtacaccg gctgggggag gctgagcagg 2040
aagctcatca acggcattcg ggacaagcag tccgggaaga cgatcctcga cttcctgaag 2100
agcgatggct tcgcgaaccg caatttcatg cagctgattc acgatgacag cctcacattc 2160
aaggaggata tccagaaggc tcaggtgagc ggccaggggg actcgctgca cgagcatatc 2220
gcgaacctcg ctggctcgcc agctatcaag aaggggattc tgcagaccgt gaaggttgtg 2280
gacgagctgg tgaaggtcat gggcaggcac aagcctgaga acatcgtcat tgagatggcc 2340
cgggagaatc agaccacgca gaagggccag aagaactcac gcgagaggat gaagaggatc 2400
gaggagggca ttaaggagct ggggtcccag atcctcaagg agcacccggt ggagaacacg 2460
cagctgcaga atgagaagct ctacctgtac tacctccaga atggccgcga tatgtatgtg 2520
gaccaggagc tggatattaa caggctcagc gattacgacg tcgatcatat cgttccacag 2580
tcattcctga aggatgactc cattgacaac aaggtcctca ccaggtcgga caagaaccgg 2640
ggcaagtctg ataatgttcc ttcagaggag gtcgttaaga agatgaagaa ctactggcgc 2700
cagctcctga atgccaagct gatcacgcag cggaagttcg ataacctcac aaaggctgag 2760
aggggcgggc tctctgagct ggacaaggcg ggcttcatca agaggcagct ggtcgagaca 2820
cggcagatca ctaagcacgt tgcgcagatt ctcgactcac ggatgaacac taagtacgat 2880
gagaatgaca agctgatccg cgaggtgaag gtcatcaccc tgaagtcaaa gctcgtctcc 2940
gacttcagga aggatttcca gttctacaag gttcgggaga tcaacaatta ccaccatgcc 3000
catgacgcgt acctgaacgc ggtggtcggc acagctctga tcaagaagta cccaaagctc 3060
gagagcgagt tcgtgtacgg ggactacaag gtttacgatg tgaggaagat gatcgccaag 3120
tcggagcagg agattggcaa ggctaccgcc aagtacttct tctactctaa cattatgaat 3180
ttcttcaaga cagagatcac tctggccaat ggcgagatcc ggaagcgccc cctcatcgag 3240
acgaacggcg agacggggga gatcgtgtgg gacaagggca gggatttcgc gaccgtcagg 3300
aaggttctct ccatgccaca agtgaatatc gtcaagaaga cagaggtcca gactggcggg 3360
ttctctaagg agtcaattct gcctaagcgg aacagcgaca agctcatcgc ccgcaagaag 3420
gactgggatc cgaagaagta cggcgggttc gacagcccca ctgtggccta ctcggtcctg 3480
gttgtggcga aggttgagaa gggcaagtcc aagaagctca agagcgtgaa ggagctgctg 3540
gggatcacga ttatggagcg ctccagcttc gagaagaacc cgatcgattt cctggaggcg 3600
aagggctaca aggaggtgaa gaaggacctg atcattaagc tccccaagta ctcactcttc 3660
gagctggaga acggcaggaa gcggatgctg gcttccgctg gcgagctgca gaaggggaac 3720
gagctggctc tgccgtccaa gtatgtgaac ttcctctacc tggcctccca ctacgagaag 3780
ctcaagggca gccccgagga caacgagcag aagcagctgt tcgtcgagca gcacaagcat 3840
tacctcgacg agatcattga gcagatttcc gagttctcca agcgcgtgat cctggccgac 3900
gcgaatctgg ataaggtcct ctccgcgtac aacaagcacc gcgacaagcc aatcagggag 3960
caggctgaga atatcattca tctcttcacc ctgacgaacc tcggcgcccc tgctgctttc 4020
aagtacttcg acacaactat cgatcgcaag aggtacacaa gcactaagga ggtcctggac 4080
gcgaccctca tccaccagtc gattaccggc ctctacgaga cgcgcatcga cctgtctcag 4140
ctcgggggcg acaagcggcc agcggcgacg aagaaggcgg ggcaggcgaa gaagaagaag 4200
tga 4203
<210> 16
<211> 4203
<212> ДНК
<213> ДНК последовательность NGA-Cas9 (ДНК последовательность)
<400> 16
atggctccga agaagaagag gaaggttggc atccacgggg tgccagctgc tgacaagaag 60
tactcgatcg gcctcgatat tgggactaac tctgttggct gggccgtgat caccgacgag 120
tacaaggtgc cctcaaagaa gttcaaggtc ctgggcaaca ccgatcggca ttccatcaag 180
aagaatctca ttggcgctct cctgttcgac agcggcgaga cggctgaggc tacgcggctc 240
aagcgcaccg cccgcaggcg gtacacgcgc aggaagaatc gcatctgcta cctgcaggag 300
attttctcca acgagatggc gaaggttgac gattctttct tccacaggct ggaggagtca 360
ttcctcgtgg aggaggataa gaagcacgag cggcatccaa tcttcggcaa cattgtcgac 420
gaggttgcct accacgagaa gtaccctacg atctaccatc tgcggaagaa gctcgtggac 480
tccacagata aggcggacct ccgcctgatc tacctcgctc tggcccacat gattaagttc 540
aggggccatt tcctgatcga gggggatctc aacccggaca atagcgatgt tgacaagctg 600
ttcatccagc tcgtgcagac gtacaaccag ctcttcgagg agaaccccat taatgcgtca 660
ggcgtcgacg cgaaggctat cctgtccgct aggctctcga agtctcggcg cctcgagaac 720
ctgatcgccc agctgccggg cgagaagaag aacggcctgt tcgggaatct cattgcgctc 780
agcctggggc tcacgcccaa cttcaagtcg aatttcgatc tcgctgagga cgccaagctg 840
cagctctcca aggacacata cgacgatgac ctggataacc tcctggccca gatcggcgat 900
cagtacgcgg acctgttcct cgctgccaag aatctgtcgg acgccatcct cctgtctgat 960
attctcaggg tgaacaccga gattacgaag gctccgctct cagcctccat gatcaagcgc 1020
tacgacgagc accatcagga tctgaccctc ctgaaggcgc tggtcaggca gcagctcccc 1080
gagaagtaca aggagatctt cttcgatcag tcgaagaacg gctacgctgg gtacattgac 1140
ggcggggcct ctcaggagga gttctacaag ttcatcaagc cgattctgga gaagatggac 1200
ggcacggagg agctgctggt gaagctcaat cgcgaggacc tcctgaggaa gcagcggaca 1260
ttcgataacg gcagcatccc acaccagatt catctcgggg agctgcacgc tatcctgagg 1320
aggcaggagg acttctaccc tttcctcaag gataaccgcg agaagatcga gaagattctg 1380
actttcagga tcccgtacta cgtcggccca ctcgctaggg gcaactcccg cttcgcttgg 1440
atgacccgca agtcagagga gacgatcacg ccgtggaact tcgaggaggt ggtcgacaag 1500
ggcgctagcg ctcagtcgtt catcgagagg atgacgaatt tcgacaagaa cctgccaaat 1560
gagaaggtgc tccctaagca ctcgctcctg tacgagtact tcacagtcta caacgagctg 1620
actaaggtga agtatgtgac cgagggcatg aggaagccgg ctttcctgtc tggggagcag 1680
aagaaggcca tcgtggacct cctgttcaag accaaccgga aggtcacggt taagcagctc 1740
aaggaggact acttcaagaa gattgagtgc ttcgattcgg tcgagatctc tggcgttgag 1800
gaccgcttca acgcctccct ggggacctac cacgatctcc tgaagatcat taaggataag 1860
gacttcctgg acaacgagga gaatgaggat atcctcgagg acattgtgct gacactcact 1920
ctgttcgagg accgggagat gatcgaggag cgcctgaaga cttacgccca tctcttcgat 1980
gacaaggtca tgaagcagct caagaggagg aggtacaccg gctgggggag gctgagcagg 2040
aagctcatca acggcattcg ggacaagcag tccgggaaga cgatcctcga cttcctgaag 2100
agcgatggct tcgcgaaccg caatttcatg cagctgattc acgatgacag cctcacattc 2160
aaggaggata tccagaaggc tcaggtgagc ggccaggggg actcgctgca cgagcatatc 2220
gcgaacctcg ctggctcgcc agctatcaag aaggggattc tgcagaccgt gaaggttgtg 2280
gacgagctgg tgaaggtcat gggcaggcac aagcctgaga acatcgtcat tgagatggcc 2340
cgggagaatc agaccacgca gaagggccag aagaactcac gcgagaggat gaagaggatc 2400
gaggagggca ttaaggagct ggggtcccag atcctcaagg agcacccggt ggagaacacg 2460
cagctgcaga atgagaagct ctacctgtac tacctccaga atggccgcga tatgtatgtg 2520
gaccaggagc tggatattaa caggctcagc gattacgacg tcgatcatat cgttccacag 2580
tcattcctga aggatgactc cattgacaac aaggtcctca ccaggtcgga caagaaccgg 2640
ggcaagtctg ataatgttcc ttcagaggag gtcgttaaga agatgaagaa ctactggcgc 2700
cagctcctga atgccaagct gatcacgcag cggaagttcg ataacctcac aaaggctgag 2760
aggggcgggc tctctgagct ggacaaggcg ggcttcatca agaggcagct ggtcgagaca 2820
cggcagatca ctaagcacgt tgcgcagatt ctcgactcac ggatgaacac taagtacgat 2880
gagaatgaca agctgatccg cgaggtgaag gtcatcaccc tgaagtcaaa gctcgtctcc 2940
gacttcagga aggatttcca gttctacaag gttcgggaga tcaacaatta ccaccatgcc 3000
catgacgcgt acctgaacgc ggtggtcggc acagctctga tcaagaagta cccaaagctc 3060
gagagcgagt tcgtgtacgg ggactacaag gtttacgatg tgaggaagat gatcgccaag 3120
tcggagcagg agattggcaa ggctaccgcc aagtacttct tctactctaa cattatgaat 3180
ttcttcaaga cagagatcac tctggccaat ggcgagatcc ggaagcgccc cctcatcgag 3240
acgaacggcg agacggggga gatcgtgtgg gacaagggca gggatttcgc gaccgtcagg 3300
aaggttctct ccatgccaca agtgaatatc gtcaagaaga cagaggtcca gactggcggg 3360
ttctctaagg agtcaattct gcctaagcgg aacagcgaca agctcatcgc ccgcaagaag 3420
gactgggatc cgaagaagta cggcgggttc gtcagcccca ctgtggccta ctcggtcctg 3480
gttgtggcga aggttgagaa gggcaagtcc aagaagctca agagcgtgaa ggagctgctg 3540
gggatcacga ttatggagcg ctccagcttc gagaagaacc cgatcgattt cctggaggcg 3600
aagggctaca aggaggtgaa gaaggacctg atcattaagc tccccaagta ctcactcttc 3660
gagctggaga acggcaggaa gcggatgctg gcttccgctg gcgagctgca gaaggggaac 3720
gagctggctc tgccgtccaa gtatgtgaac ttcctctacc tggcctccca ctacgagaag 3780
ctcaagggca gccccgagga caacgagcag aagcagctgt tcgtcgagca gcacaagcat 3840
tacctcgacg agatcattga gcagatttcc gagttctcca agcgcgtgat cctggccgac 3900
gcgaatctgg ataaggtcct ctccgcgtac aacaagcacc gcgacaagcc aatcagggag 3960
caggctgaga atatcattca tctcttcacc ctgacgaacc tcggcgcccc tgctgctttc 4020
aagtacttcg acacaactat cgatcgcaag cagtaccgga gcactaagga ggtcctggac 4080
gcgaccctca tccaccagtc gattaccggc ctctacgaga cgcgcatcga cctgtctcag 4140
ctcgggggcg acaagcggcc agcggcgacg aagaaggcgg ggcaggcgaa gaagaagaag 4200
tga 4203
<210> 17
<211> 5860
<212> ДНК
<213> ДНК последовательность генома OsBADH2 Oryza sativa L. (ДНК
последовательность)
<400> 17
atggccacgg cgatcccgca gcggcagctc ttcgtcgccg gcgagtggcg cgcccccgcg 60
ctcggccgcc gcctccccgt cgtcaacccc gccaccgagt cccccatcgg taccctcctc 120
ttcaccctct ccaccctctg cttctgcctc tgattagcct ttttgttgtt gttgttgttg 180
ctgctgtttt ttgcgtgtcg gtgcgcaggc gagatcccgg cgggcacggc ggaggacgtg 240
gacgcggcgg tggcggcggc gcgggaggcg ctgaagagga accggggccg cgactgggcg 300
cgcgcgccgg gcgccgtccg ggccaagtac ctccgcgcaa tcgcggccaa ggtagggtgg 360
tgactacccc cacccccccc ccccccccca acgcgacccg cgtgcgtgtg ttccgtacag 420
ggggaggagc tccgcgtggc tctccagtag gtttttgagc cccaaatcga tcgatatgct 480
ctagttttaa gtttgctgct taaattcctc aagggtttag tttgcaacca aatccttatt 540
ttagcttcgg tataagcccc ccatatgatg tgcgtgcgtc ggcatcggaa gtgcgtatcc 600
tctgttctgg actaggaatt ggccataggt tgatcgacag ttcgagtatt ctgcttctgt 660
ttggaataag ttggaagcat ggctgattgt gtatctggat gctgtttttg tggtgattcg 720
tttcaagctc ttgttaattg atgggttcaa gcggagaggg tgcgcaacaa caagtgtata 780
tggctcacgg ccatgggtgt gcacatttga ttggtgcgca acaacaagtg tatattgttt 840
gtgtgcttcg ttagttggca ggtcctagtc actaaatcac tattggattg gtactagtta 900
cttttgtgcc ttgacgatgg gactggatta ctagcctttt ggttgccttt gtggtattcc 960
gttgttatgg gcctgttgat ggatggatcc ctttaatttc tagtgccaaa tgcatgctag 1020
atttctcaca gtttttctct tcaggttata tttctcgtat ttccttttcc taaaggattg 1080
ctttttcatg tattttctgg catatatagg ttattattat tattattctc cagaacaaga 1140
ttacccatat tatggatcac tagtgtacac ttttttggat gaaaaaccta cttactgaaa 1200
gtaaaacagt gaccagtgca cactttactt gaactgtcaa accatcaatt ttctagcaaa 1260
gcaggggatg ctagccttcc agtctaaatg acagtaaact actatacttt tgtccgtagg 1320
tttggaaata tgctaatttc tatcataaaa attttcatgg catatgcgag cattttatga 1380
tcaccttttc cctttttctt cagataatcg agaggaaatc tgagctggct agactagaga 1440
cgcttgattg tgggaagcct cttgatgaag cagcatggga catggtatgt ggccagttat 1500
ccactgtatg aatatgtagt tgcctacaca gcaatctttc ctgaacatga atcctgatgt 1560
atgatattcc atttgtcagg acgatgttgc tggatgcttt gagtactttg cagatcttgc 1620
agaatccttg gacaaaaggc aaaatgcacc tgtctctctt ccaatggaaa actttaaatg 1680
ctatcttcgg aaagagccta tcggtgtagt tgggttgatc acaccttggt atttcacatt 1740
tttctctcat cctgcgctta tatttattta tgacccaagc atggtactaa atagtactag 1800
taacatgcat atactgaatg agtttacaac tttacatgat ttttttgaac tatgaaagtt 1860
gaagacattt gagattttat tcctcttctc ttgtgcaaac atattattgt ctcacaaatt 1920
gtacctagca gctactctct ccgtttcata ttataagtcg tttgactttt ttcctagtca 1980
aaatgtgtta agtttgacca agtttataga aaaatttagc aacatctaaa atatcaaagt 2040
catgttttag tgttttttca ggctctcatg taagcaattt tgatgtgccc tctcctttct 2100
tcttaatata atgatacaca gctcttgtgt attcaaagga aaatatatat atatataatg 2160
atacacacct ctcctccgtg ttaatgcagc tcatttgttc tgtcccggtt caaatatcta 2220
tttttctcat atgttgtcag catgattcac ttaatttagt atatagaaga tgccattatt 2280
tatgtctgga atcttactgc agaagggaaa acaattgata acggaattga ttgcattcta 2340
atttgttgtt tctttgttat gttcttatcg acaattacaa atttgattct gagaatcatg 2400
ttcgggatgt gtatttctac tgcaggaact atcctctcct gatggcaaca tggaaggtag 2460
ctcctgccct ggctgctggc tgtacagctg tactaaaacc atctgaattg gcttccgtgt 2520
aagtttaaca tgttaacttg ttaatgtcat acccatgcta gttgcaatga catttgattt 2580
taaaatgttg tggcatgtcc atgctgcaag caatgtaatt tgaaatctct ctctatcatt 2640
aattaccagg acttgtttgg agcttgctga tgtgtgtaaa gaggttggtc ttccttcagg 2700
tgtgctaaac atagtgactg gattaggttc tgaagccggt gctcctttgt catcacaccc 2760
tggtgtagac aaggtacagc tattcctcct gtaatcatgt ataccccatc aatggaaatg 2820
atattcctct caatacatgg tttatgtttt ctgttaggtt gcatttactg ggagttatga 2880
aactggtaaa aagattatgg cttcagctgc tcctatggtt aaggtttgtt tccaaatttc 2940
tgtggatatt ttttgttctc tttctactaa ctctctatta tcaattctca atgttgtcct 3000
tttcttttaa ctcctttact ttttagaatt gtgatcaaga cactttgagc atcattctag 3060
tagccagttc tatcctgttt cttacctttt tatggttcgt cttttcttga cagcctgttt 3120
cactggaact tggtggaaaa agtcctatag tggtgtttga tgatgttgat gttgaaaaag 3180
gtacatgcca cttgctatga ttaactaatt ctgaagtgcg ggactttgta aagcacttaa 3240
ctgagctgga tgctagaccc ccaaaagccc tttttggtgt cttgggcttg ttgcagaaat 3300
actggtccca gacgagcagg atgcaagaaa attaactact tttgccactg attagtattt 3360
cttagaagtt acacctcaag gattagcaat actttcttaa aatgtgctat tgattaaaaa 3420
gatgtcctgt attattttga gcagatcttg tactggttga tcggcttgca tgaaaatatt 3480
gttgaggatt ataatgccat gccaactgag taaagaaaag agttgtaaaa tatgttatgc 3540
aacatgaata tatatgtgat ttcatttttc ctttttcttt tcgtggcaag gaaggcagtt 3600
aggaaggact gatgtgaaaa gcacaagtac tattcttagt tctggaaaac tgtgttcttt 3660
attttcctaa ctacaattca ccttgattag tcagtaactt gatattggca attctagctg 3720
attatgaatt ctgtttatat ttcactaatt ttgaatcttt aattacattt tatggttgaa 3780
atttaacgtt ttgtctggtt atggactctg tttgtattca ctcaatttgg atcttccatt 3840
agatttcatt gttggtcctt cttcttgtac agctgttgag tggactctct ttggttgctt 3900
ttggaccaat ggccagattt gcagtgcaac atcgcgtctt attcttcatg taagcattga 3960
atatatccgt caatcataat ctattgttgt acttgatttt ttttctgatc aactcctgag 4020
ttcagattat tatatgatgc cattactatt gcacagagcg aataaaattg tatttatgca 4080
cagcatgtat tttgagtaat atatgcattg cctattattt aatatataga ttgtagcact 4140
taattttgtg tccatgtctc tatgatgttt attactttat tattgccggc atgaagcaac 4200
tttgaactct atgttgatct tgaactaaaa ttgaaattaa ttggcttatt gctattaatg 4260
atatagcttt cagcttcttg ctcctgacca tgaaagtttt gcagaaaaaa atcgctaaag 4320
aatttcaaga aaggatggtt gcatgggcca aaaatattaa ggtgtcagat ccacttgaag 4380
agggttgcag gcttgggccc gttgttagtg aaggacaggt accacatgta aactttttct 4440
aaattcaaaa aagaaatgcc actgatcaat ggtaggtcct tccaagcctt attgctggat 4500
tgttgcactg ttttgtcaat tttgtgtaat atagttctga atgaattagt cggtgtatgc 4560
tcttgctagt tgctagtatg tggtacaggg tcttcctact ttgagcaaat tcgtgttaaa 4620
atgcattgat gaaaaggcca cctttccgta ggtttatctt gtcataattt aaaccccaat 4680
aaaattttaa ttttttgttt tgaccccatg gcactttaat gaaatcactt agccatgagc 4740
ttttgtatat attttcaaag caccagaatg tttagatggt ttgttggaaa tcttacacat 4800
cctattgcct tgtgtcagta tgagaagatt aagcaatttg tatctaccgc caaaagccaa 4860
ggtgctacca ttctgactgg tggggttaga cccaaggtaa taatctacta cacggttgta 4920
tatataggta cccacatatc attatgaagt agaaataatc ttgtatgttt ttgtcagcat 4980
ctggagaaag gtttctatat tgaacccaca atcattactg atgtcgatac atcaatgcaa 5040
atttggaggg aagaagtttt tggtccagtg ctctgtgtga aagaatttag cactgaagaa 5100
gaagccattg aattggccaa cgatactcag tgagtttttt ttttaataca gttcattgtc 5160
ctgttcaatc ttgcagcata tgtatatact ctgtggcata tgaacttatt ctgctactac 5220
tacttttgat agttatggtc tggctggtgc tgtgctttcc ggtgaccgcg agcgatgcca 5280
gagattaact gaggtatatc caagtgaagg gggttggcat tgtttgattc atatgacatg 5340
gttgcatcaa gctgatattc aagaatctca tttattactt gcattctatg catctccagt 5400
tcttccctgg actccggtca atgttaatat agtttgtttg ctagtagtat gctactccaa 5460
ttaagttgct cttcacttcc acatcatctg atccatgact ttatatttga cccctttttt 5520
ttgcaaaaga aagggaaata cttaacgaaa atttcctact gcaggagatc gatgccggaa 5580
ttatctgggt gaactgctcg caaccctgct tctgccaagc tccatggggc gggaacaagc 5640
gcagcggctt tggacgcgag ctcggagaag ggtgggtagc acacaacaat ctcactttaa 5700
aacaccattt cgatcgtctg atgatctcga cctgacatca tgcctttggt attttcattc 5760
acttttcagg ggcattgaca actacctaag cgtcaagcaa gtgacggagt acgcctccga 5820
tgagccgtgg ggatggtaca aatccccttc caagctgtaa 5860
<210> 18
<211> 2192
<212> ДНК
<213> ДНК последовательность генома OsSWEET14 Oryza sativa L. (ДНК
последовательность)
<400> 18
atggctggca tgtctcttca gcatccctgg gccttcgcct ttggtctcct aggtgtgttg 60
cctttgatct gatccaagga attctcttga gaattaatct tgcatggtta tttacttttg 120
ttgttattat tctctacatt tttaatcatg tacttttcca tgttccactt ttgttgccaa 180
agctactata tttttcctac caattcatcc aaaactacta tattatagca aggcagctag 240
tggatcgact ttgcactttt ggatgcaatt gtgagtggtt tacattagag gggccatgca 300
tagccaaata aattgtttgg caaaatatga tttcccttgt agttagcaca atattgtgat 360
ctttcattcc tttactcttt ttgttccacc accctcatta ttgcaaagcc ccctctttta 420
gaaccaaact aaagataaga aacatttgat tcatcattag aggagattag atatacatac 480
atactacaca tgcattgctg tgataagcct agccaggcac agaaagagcc aagacaaaag 540
gagctcaaaa gaaaaggggc tccaatatct tgcccggcct gatcagtgta acatgacgtc 600
acccacacaa ttattaaaag gatagatgca tatgtgttcg ttacgtatct ccttgtgtca 660
cgttgctgtg catggttcac tcgcttaatt ataacgtccg tcgcatttta tgcatgcact 720
tcgtttttag ttaatcaaat taaacaagat cctaacatat gatttctatt ttattctctt 780
tgcaggcaac atcatctcct tcatgaccta cctggcccca ctgtaagtcc cacacatccg 840
gatattttct tttctttagc gaaattttac attgctccag aaagtacctt ttacactaaa 900
atttcttata caccaccata aaattaatgt ggtactattg gtaccttatc aaaaatgata 960
aaattacctt tttcttcaga taaaaacact ccccgtctct gtataactaa gatgtagaca 1020
accaatacac atgtctagag ctaatcctat caattaacat tgctagtaaa tacaaggaaa 1080
aatataaaga attaaccaat tgcttggatg tcttgcaggc cgacgttcta caggatctac 1140
aagagcaagt cgacgcaggg gttccagtcg gtaccctacg tggtggcgct gttcagcgcg 1200
atgctgtgga tctactacgc gctgctcaag tccgacgagt gcctcctcat caccatcaac 1260
tccgctggct gcgtcatcga gaccatctac atcgccgtct acctcgtcta cgcccccaag 1320
aaggccaaga tgttcaccgc caagctcctc ctcctcgtca acgtcggcgt cttcggcctc 1380
atcctcctcc tcaccctcct cctctccgcc ggcgaccgcc gcatcgtggt tcttggttgg 1440
gtctgcgttg gcttctccgt cagcgtcttc gtcgcccccc ttagcatcat cgtaagaaac 1500
cctagaattg cctgtaaaca ataataactt tcggtctctg caccaactaa ggatgcacat 1560
atttacgcac cgcttttgct atctctgcag aggctggtgg tgcgcaccaa gagcgtggag 1620
ttcatgccgt tctcgctctc cttctccctc accatcagcg ccgtcgtctg gttcctctac 1680
ggcctcctca tcaaggacaa atatgtcgct gtgagtagct ccgattcgac ccgttcttct 1740
tcctaaattt tccgctgctc gatttaattt ctatttctaa ttgtggaaga ttgtttaatt 1800
atagtgatta tggctagctg ataaccgatt ttaatttctg atggtgatcc aaaatggaac 1860
agcttcccaa cgtgctgggc ttctccttcg gcgtcatcca gatggggctg tacgccatgt 1920
acaggaactc gacgcccaag gccgtgctga ccaaggaggt cgaggcggcg acggccaccg 1980
gcgacgacga ccactccgcc gccggcgtca aggagcacgt cgtcaacatc gccaagctct 2040
ctgccgccgt cgacgtcgtc aagacccgcg aggtgcaccc cgtcgacgtc gagtccccgc 2100
cggcagaggc gccgcctgag gaggacgaca aggccgccgc cgccaccgcc gccgccgtcg 2160
ccggcgccgg cgagaagaag gtagctgcat ga 2192
<210> 19
<211> 657
<212> БЕЛОК
<213> Аминокислотная последовательность белка StALS2 Solanum tuberosum
L. (аминокислотная последовательность)
<400> 19
Met Ala Ala Ala Ser Pro Ser Pro Cys Phe Ser Lys Asn Leu Pro Pro
1 5 10 15
Ser Ser Ser Lys Ser Ser Ile Leu Leu Pro Lys Ser Thr Phe Thr Phe
20 25 30
His Asn His Pro Lys Asn Thr Ser Pro Leu His Leu Thr His Thr Gln
35 40 45
His His Ser Arg Phe Thr Val Ser Asn Val Ile Leu Ser Thr Thr Thr
50 55 60
His Asn Asp Val Ser Glu Pro Glu Ile Phe Val Ser Arg Phe Ala Pro
65 70 75 80
Asp Glu Pro Arg Lys Gly Cys Asp Val Leu Val Glu Ala Leu Glu Arg
85 90 95
Glu Gly Val Lys Asp Val Phe Ala Tyr Pro Gly Gly Ala Ser Met Glu
100 105 110
Ile His Gln Ala Leu Thr Arg Ser Asn Ile Ile Arg Asn Val Leu Pro
115 120 125
Arg His Glu Gln Gly Gly Val Phe Ala Ala Glu Gly Tyr Ala Arg Ala
130 135 140
Thr Gly Phe Pro Gly Val Cys Ile Ala Thr Ser Gly Pro Gly Ala Thr
145 150 155 160
Asn Leu Val Ser Gly Leu Ala Asp Ala Leu Leu Asp Ser Ile Pro Ile
165 170 175
Val Ala Ile Thr Gly Gln Val Pro Arg Arg Met Ile Gly Thr Asp Ala
180 185 190
Phe Gln Glu Thr Pro Ile Val Glu Val Thr Arg Ser Ile Thr Lys His
195 200 205
Asn Tyr Leu Val Met Asp Val Glu Asp Ile Pro Arg Val Val Arg Glu
210 215 220
Ala Phe Phe Leu Ala Lys Ser Gly Arg Pro Gly Pro Val Leu Ile Asp
225 230 235 240
Val Pro Lys Asp Ile Gln Gln Gln Leu Val Ile Pro Asn Trp Asp Gln
245 250 255
Pro Met Arg Leu Pro Gly Tyr Ile Ser Arg Leu Pro Lys Leu Pro Asn
260 265 270
Glu Met Leu Leu Glu Gln Ile Val Arg Leu Ile Ser Glu Ser Lys Lys
275 280 285
Pro Val Leu Tyr Val Gly Gly Gly Cys Thr Gln Ser Ser Glu Glu Leu
290 295 300
Arg Arg Phe Val Glu Leu Thr Gly Ile Pro Val Ala Ser Thr Leu Met
305 310 315 320
Gly Leu Gly Thr Phe Pro Cys Gly Asp Glu Leu Ser Leu Gln Met Leu
325 330 335
Gly Met His Gly Thr Val Tyr Ala Asn Tyr Ala Val Asp Ser Ser Asp
340 345 350
Leu Leu Leu Ala Phe Gly Val Arg Phe Asp Asp Arg Val Thr Gly Lys
355 360 365
Leu Glu Ala Phe Ala Ser Arg Ala Lys Ile Val His Ile Asp Ile Asp
370 375 380
Ser Ala Glu Ile Gly Lys Asn Lys Gln Pro His Val Ser Ile Cys Ala
385 390 395 400
Asp Ile Lys Leu Ala Leu Gln Gly Leu Asn Ser Ile Leu Glu Gly Lys
405 410 415
Glu Gly Lys Leu Lys Leu Asp Phe Ser Ala Trp Arg Gln Glu Leu Thr
420 425 430
Glu Gln Lys Val Lys Tyr Pro Leu Asn Tyr Lys Thr Phe Gly Glu Ala
435 440 445
Ile Pro Pro Gln Tyr Ala Ile Gln Val Leu Asp Glu Leu Thr Asn Gly
450 455 460
Asn Ala Ile Ile Ser Thr Gly Val Gly Gln His Gln Met Trp Ala Ala
465 470 475 480
Gln Tyr Tyr Lys Tyr Lys Lys Pro Arg Gln Trp Leu Thr Ser Gly Gly
485 490 495
Leu Gly Ala Met Gly Phe Gly Leu Pro Ala Ala Ile Gly Ala Ala Val
500 505 510
Gly Arg Pro Gly Glu Ile Val Val Asp Ile Asp Gly Asp Gly Ser Phe
515 520 525
Ile Met Asn Val Gln Glu Leu Ala Thr Ile Lys Val Glu Asn Leu Pro
530 535 540
Val Lys Ile Met Leu Leu Asn Asn Gln His Leu Gly Met Val Val Gln
545 550 555 560
Trp Glu Asp Arg Phe Tyr Lys Ala Asn Arg Ala His Thr Tyr Leu Gly
565 570 575
Asn Pro Ala Asn Glu Glu Glu Ile Phe Pro Asn Met Leu Lys Phe Ala
580 585 590
Glu Ala Cys Gly Val Pro Ala Ala Arg Val Ser His Arg Asp Asp Leu
595 600 605
Arg Ala Ala Ile Gln Lys Met Leu Asp Thr Pro Gly Pro Tyr Leu Leu
610 615 620
Asp Val Ile Val Pro His Gln Glu His Val Leu Pro Met Ile Pro Ser
625 630 635 640
Gly Gly Ala Phe Lys Asp Val Ile Thr Glu Gly Asp Gly Arg Arg Ser
645 650 655
Tyr
<210> 20
<211> 1974
<212> ДНК
<213> ДНК последовательность гена StALS2 Solanum tuberosum L. (ДНК
последовательность)
<400> 20
atggcggctg catctccatc tccttgtttt tccaaaaacc tacctccatc ttcatcaaaa 60
tcttccatcc ttcttcccaa atctaccttt actttccaca atcaccccaa aaatacctca 120
ccccttcacc ttacccacac ccaacatcat agccgtttca ctgtctcaaa tgtcatccta 180
tcaaccacga cccataacga cgtttctgaa cccgaaatct tcgtttcacg tttcgcccct 240
gacgaaccca gaaagggttg tgatgttctt gtggaggcac ttgaaaggga aggggttaag 300
gatgtatttg catacccagg aggtgcttcc atggagattc atcaggcttt gacacgttcc 360
aatattattc gtaatgtgct gccacgtcat gaacagggtg gtgtgtttgc tgcagagggt 420
tacgcacggg ccactgggtt ccctggtgtt tgcattgcta catctggtcc gggagctacg 480
aatcttgtta gcggtcttgc ggatgctttg ttggatagta ttccgattgt tgctattacg 540
ggtcaagtgc cgaggaggat gattggtact gatgcgtttc aggaaactcc tattgttgag 600
gtaacgagat ccattacgaa gcataattat cttgttatgg atgtagagga tattcctagg 660
gttgttcgtg aagcgttttt tctagcgaaa tcgggacggc ctggaccggt tctgattgat 720
gttcctaagg atattcagca acaattggtg atacctaatt gggatcagcc aatgaggttg 780
cctggttaca tctctaggtt gcctaaattg cctaatgaga tgcttttgga acaaattgtt 840
aggctgattt cggagtcgaa gaagcctgtt ttgtatgtgg gtggtgggtg tacacaatcg 900
agtgaggagc tgagacgatt tgtggagctt acgggtattc ctgtggcgag tactttgatg 960
ggtcttggaa cttttccatg tggggatgag ctttctcttc aaatgttggg tatgcatggg 1020
actgtgtatg ctaattatgc ggtggatagt agtgatttgt tgcttgcatt tggggtgagg 1080
tttgatgatc gagttactgg taaattggaa gcttttgcta gccgagcgaa aattgtccac 1140
attgatattg attcggctga gattggaaag aacaagcaac ctcatgtttc catttgtgca 1200
gatatcaagt tggcattaca gggtttgaat tccatattgg agggtaaaga aggtaagctg 1260
aagttggact tttctgcttg gaggcaggag ttaacggagc agaaggtgaa gtacccattg 1320
aattataaga cttttggtga agccatccct ccacaatatg ctattcaggt tcttgatgag 1380
ttaactaacg gaaatgccat tattagtact ggtgtggggc aacaccaaat gtgggctgcc 1440
caatactata agtacaaaaa gccacgccaa tggttgacat ctggtggatt aggagcaatg 1500
ggatttggtt tgcctgctgc tataggtgcg gctgttggaa gaccgggtga gattgtggtt 1560
gacattgatg gtgacgggag ttttatcatg aatgtgcagg agttagcaac aattaaggtg 1620
gagaatctcc cagttaagat tatgttactg aataatcaac acttgggaat ggtggttcag 1680
tgggaggatc gattctataa ggctaacaga gcacacactt acttgggtaa tcctgctaat 1740
gaggaagaaa tcttccctaa tatgctgaaa tttgcagagg cttgtggcgt acctgctgca 1800
agagtgtcac acagggatga tcttagagct gccattcaaa agatgttaga cactcctggg 1860
ccatacttgt tggatgtgat tgtacctcat caggagcacg ttctacctat gattcccagt 1920
ggtggcgctt tcaaagatgt gattacggag ggtgatggga gacgttccta ttga 1974
<210> 21
<211> 1424
<212> ДНК
<213> ДНК последовательность гена HBB (содержащая интрон)(ДНК
последовательность)
<400> 21
atggtgcacc tgactcctga ggagaagtct gccgttactg ccctgtgggg caaggtgaac 60
gtggatgaag ttggtggtga ggccctgggc aggttggtat caaggttaca agacaggttt 120
aaggagacca atagaaactg ggcatgtgga gacagagaag actcttgggt ttctgatagg 180
cactgactct ctctgcctat tggtctattt tcccaccctt aggctgctgg tggtctaccc 240
ttggacccag aggttctttg agtcctttgg ggatctgtcc actcctgatg ctgttatggg 300
caaccctaag gtgaaggctc atggcaagaa agtgctcggt gcctttagtg atggcctggc 360
tcacctggac aacctcaagg gcacctttgc cacactgagt gagctgcact gtgacaagct 420
gcacgtggat cctgagaact tcagggtgag tctatgggac gcttgatgtt ttctttcccc 480
ttcttttcta tggttaagtt catgtcatag gaaggggata agtaacaggg tacagtttag 540
aatgggaaac agacgaatga ttgcatcagt gtggaagtct caggatcgtt ttagtttctt 600
ttatttgctg ttcataacaa ttgttttctt ttgtttaatt cttgctttct ttttttttct 660
tctccgcaat ttttactatt atacttaatg ccttaacatt gtgtataaca aaaggaaata 720
tctctgagat acattaagta acttaaaaaa aaactttaca cagtctgcct agtacattac 780
tatttggaat atatgtgtgc ttatttgcat attcataatc tccctacttt attttctttt 840
atttttaatt gatacataat cattatacat atttatgggt taaagtgtaa tgttttaata 900
tgtgtacaca tattgaccaa atcagggtaa ttttgcattt gtaattttaa aaaatgcttt 960
cttcttttaa tatacttttt tgtttatctt atttctaata ctttccctaa tctctttctt 1020
tcagggcaat aatgatacaa tgtatcatgc ctctttgcac cattctaaag aataacagtg 1080
ataatttctg ggttaaggca atagcaatat ctctgcatat aaatatttct gcatataaat 1140
tgtaactgat gtaagaggtt tcatattgct aatagcagct acaatccagc taccattctg 1200
cttttatttt atggttggga taaggctgga ttattctgag tccaagctag gcccttttgc 1260
taatcatgtt catacctctt atcttcctcc cacagctcct gggcaacgtg ctggtctgtg 1320
tgctggccca tcactttggc aaagaattca ccccaccagt gcaggctgcc tatcagaaag 1380
tggtggctgg tgtggctaat gccctggccc acaagtatca ctaa 1424
<210> 22
<211> 444
<212> ДНК
<213> CDS последовательность гена HBB (ДНК последовательность)
<400> 22
atggtgcacc tgactcctga ggagaagtct gccgttactg ccctgtgggg caaggtgaac 60
gtggatgaag ttggtggtga ggccctgggc aggctgctgg tggtctaccc ttggacccag 120
aggttctttg agtcctttgg ggatctgtcc actcctgatg ctgttatggg caaccctaag 180
gtgaaggctc atggcaagaa agtgctcggt gcctttagtg atggcctggc tcacctggac 240
aacctcaagg gcacctttgc cacactgagt gagctgcact gtgacaagct gcacgtggat 300
cctgagaact tcaggctcct gggcaacgtg ctggtctgtg tgctggccca tcactttggc 360
aaagaattca ccccaccagt gcaggctgcc tatcagaaag tggtggctgg tgtggctaat 420
gccctggccc acaagtatca ctaa 444
<210> 23
<211> 147
<212> БЕЛОК
<213> Аминокислотная последовательность гена HBB (аминокислотная
последовательность)
<400> 23
Met Val His Leu Thr Pro Glu Glu Lys Ser Ala Val Thr Ala Leu Trp
1 5 10 15
Gly Lys Val Asn Val Asp Glu Val Gly Gly Glu Ala Leu Gly Arg Leu
20 25 30
Leu Val Val Tyr Pro Trp Thr Gln Arg Phe Phe Glu Ser Phe Gly Asp
35 40 45
Leu Ser Thr Pro Asp Ala Val Met Gly Asn Pro Lys Val Lys Ala His
50 55 60
Gly Lys Lys Val Leu Gly Ala Phe Ser Asp Gly Leu Ala His Leu Asp
65 70 75 80
Asn Leu Lys Gly Thr Phe Ala Thr Leu Ser Glu Leu His Cys Asp Lys
85 90 95
Leu His Val Asp Pro Glu Asn Phe Arg Leu Leu Gly Asn Val Leu Val
100 105 110
Cys Val Leu Ala His His Phe Gly Lys Glu Phe Thr Pro Pro Val Gln
115 120 125
Ala Ala Tyr Gln Lys Val Val Ala Gly Val Ala Asn Ala Leu Ala His
130 135 140
Lys Tyr His
145
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9 II ТИПА И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2794774C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| МУТАНТНАЯ П-ГИДРОКСИФЕНИЛПИРУВАТДИОКСИГЕНАЗА, НУКЛЕИНОВАЯ КИСЛОТА, КОДИРУЮЩАЯ ЕЕ, И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2781830C2 |
| ШТАММ ESCHERICHIA COLI BL21(DE3)PLYSS/PET15B-HISCPF1 - ПРОДУЦЕНТ РНК-НАПРАВЛЯЕМОЙ ЭНДОНУКЛЕАЗЫ CRISPR/CPF1 | 2021 |
|
RU2774120C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Capnocytophaga ochracea | 2021 |
|
RU2778156C1 |
| ЭКСПРЕССИРУЮЩИЕ CAS МЫШИНЫЕ ЭМБРИОНАЛЬНЫЕ СТВОЛОВЫЕ КЛЕТКИ И МЫШИ И ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2782358C2 |
| ВЫСОКОАФФИННЫЙ Т-КЛЕТОЧНЫЙ РЕЦЕПТОР ДЛЯ РАСПОЗНАВАНИЯ АНТИГЕНА AFP | 2019 |
|
RU2836365C2 |
| АНТАГОНИСТЫ | 2019 |
|
RU2839429C2 |
| ВАРИАНТЫ, КОМПОЗИЦИИ И СПОСОБЫ ПРИМЕНЕНИЯ ЭНДОНУКЛЕАЗЫ CBLB | 2018 |
|
RU2779097C2 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |


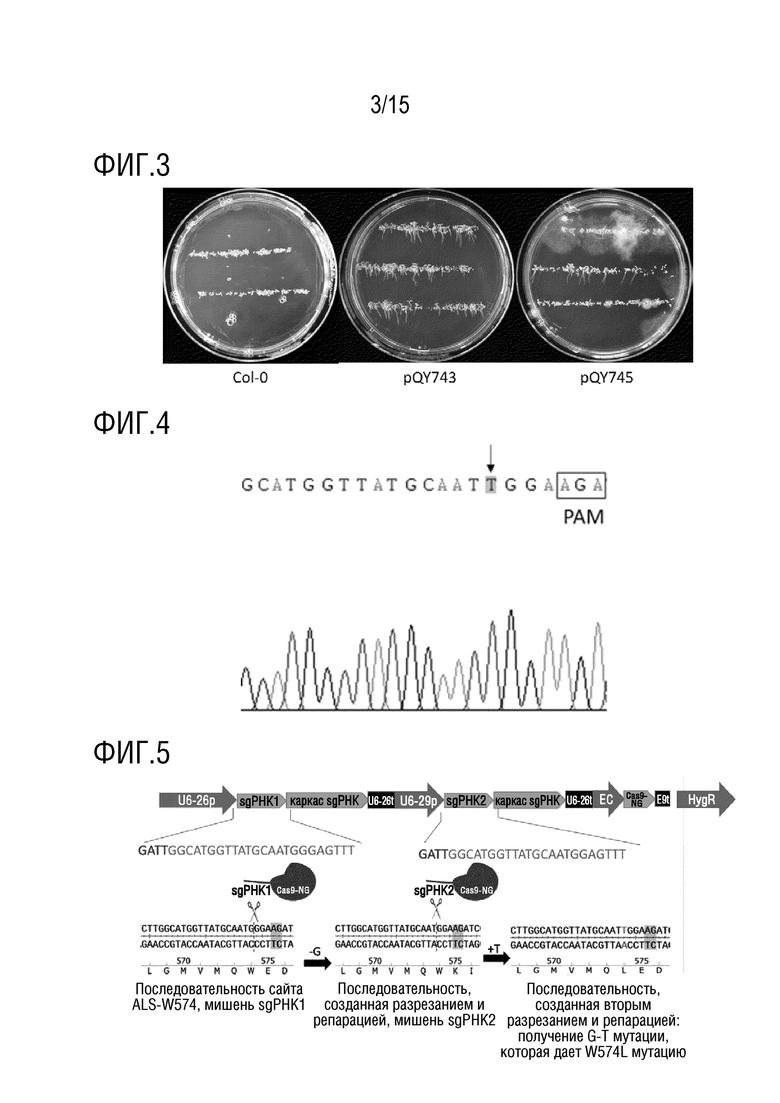
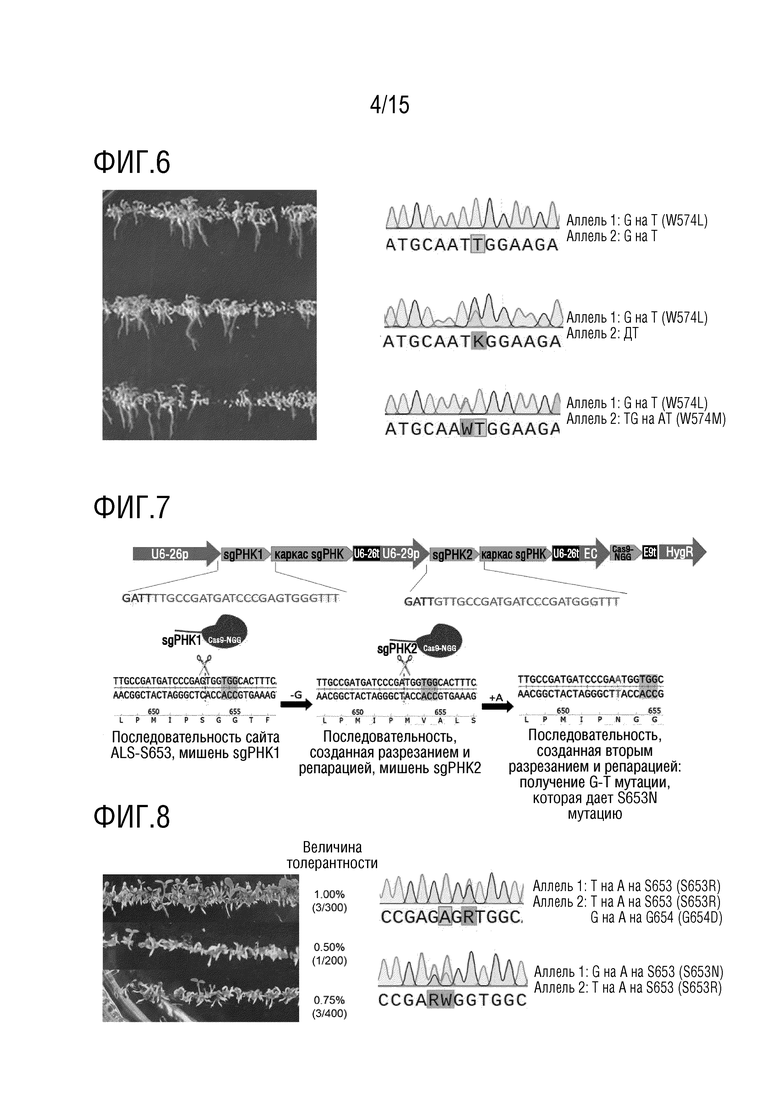
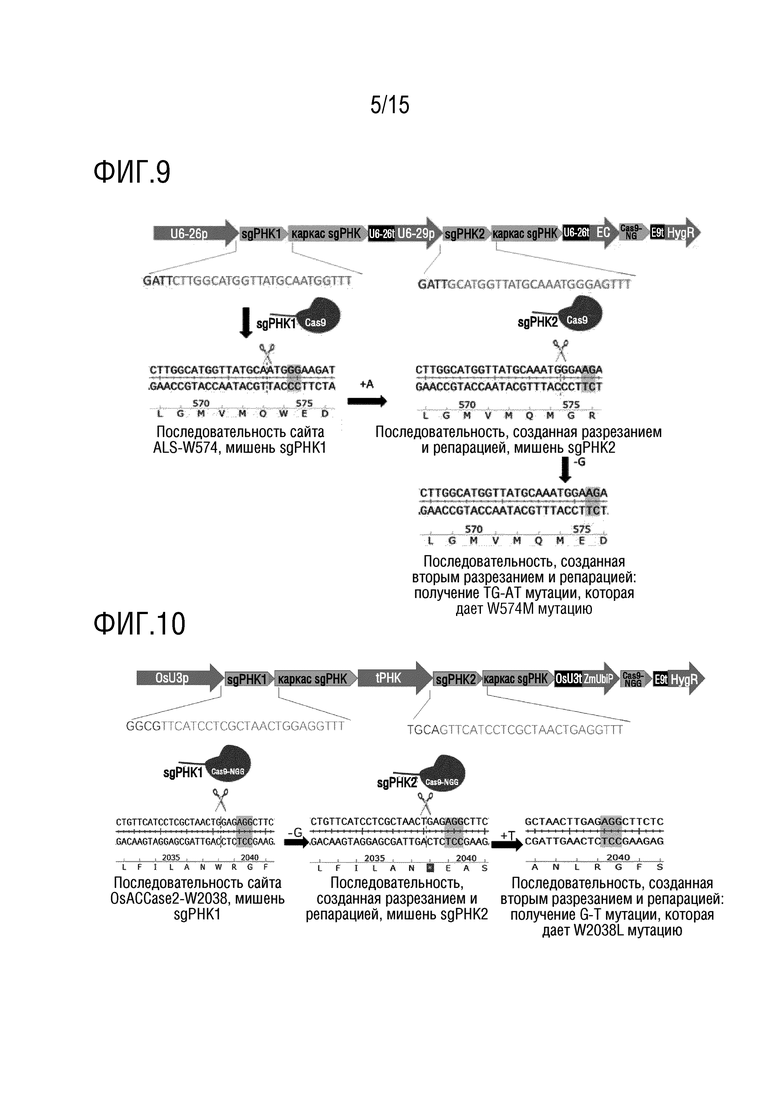
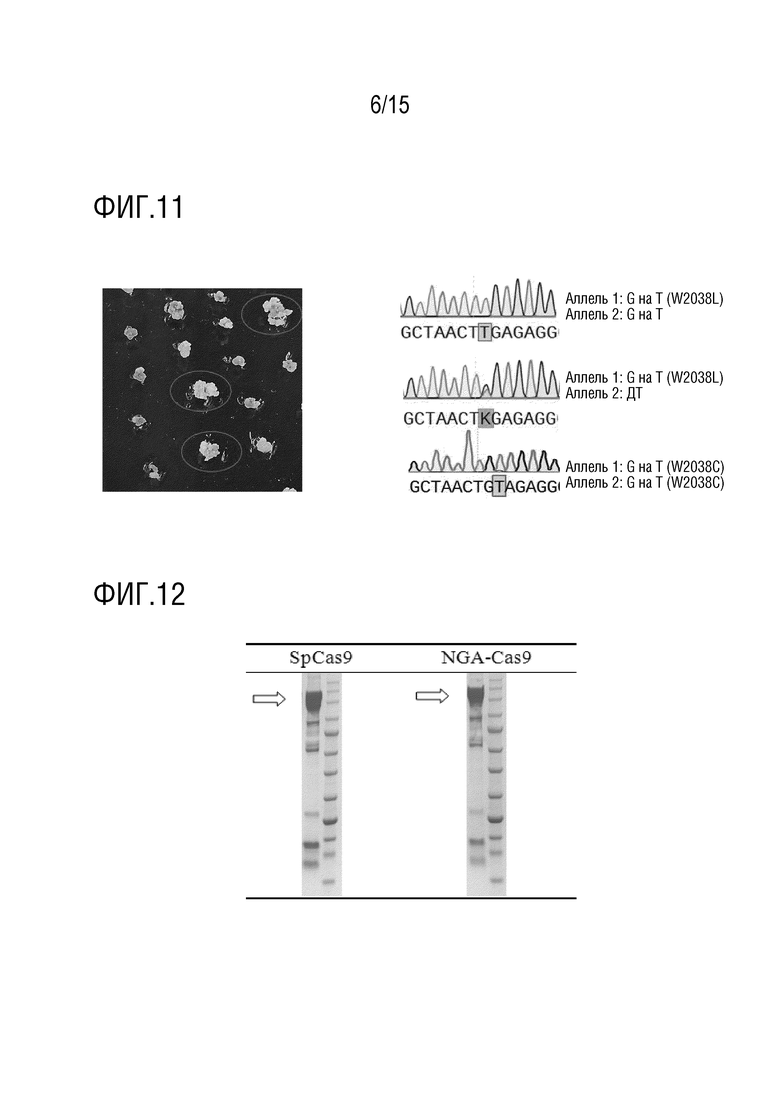
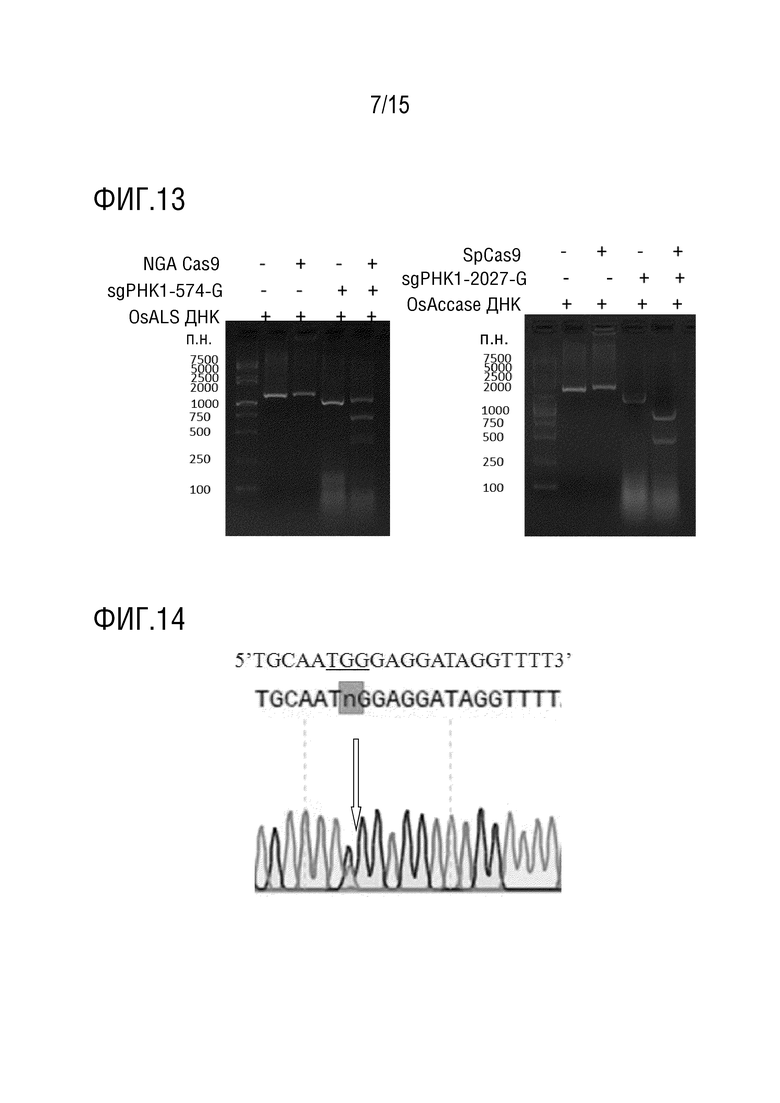
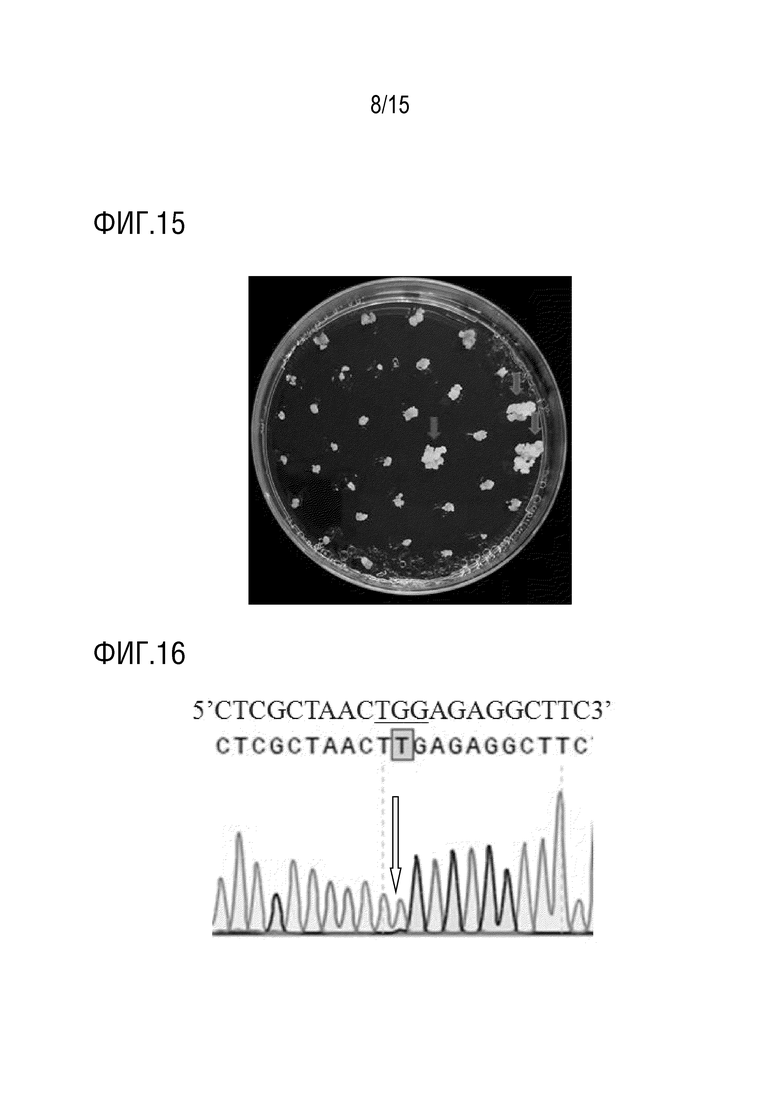

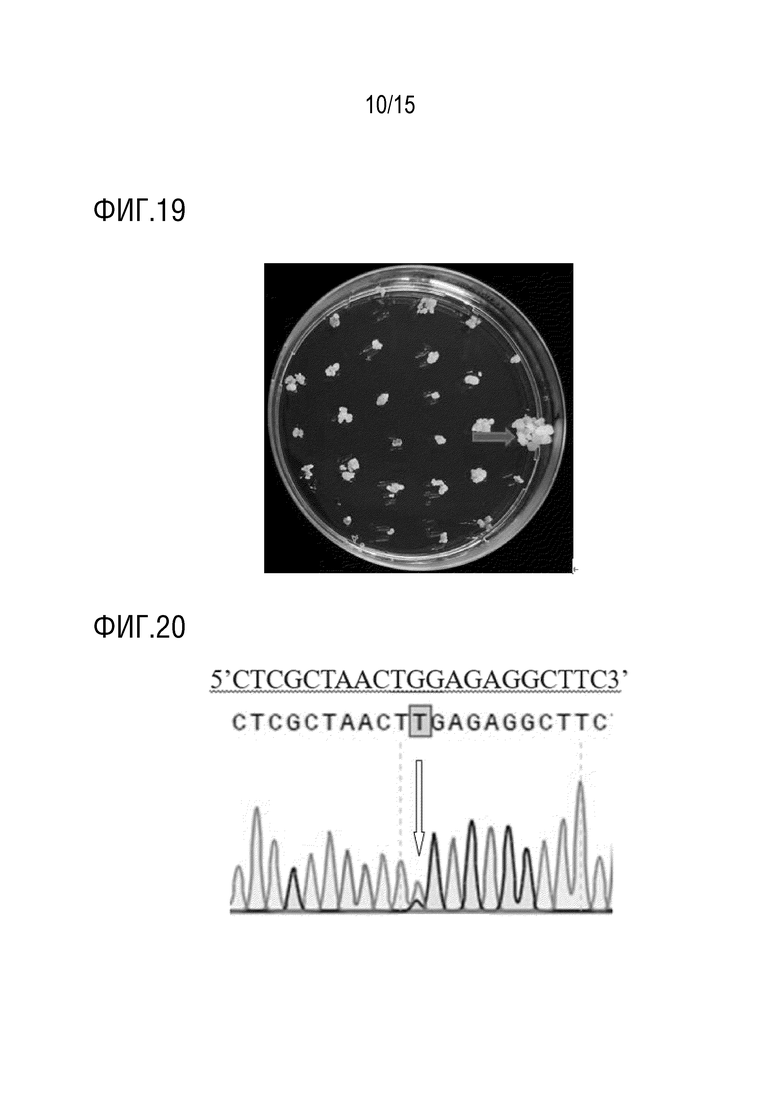
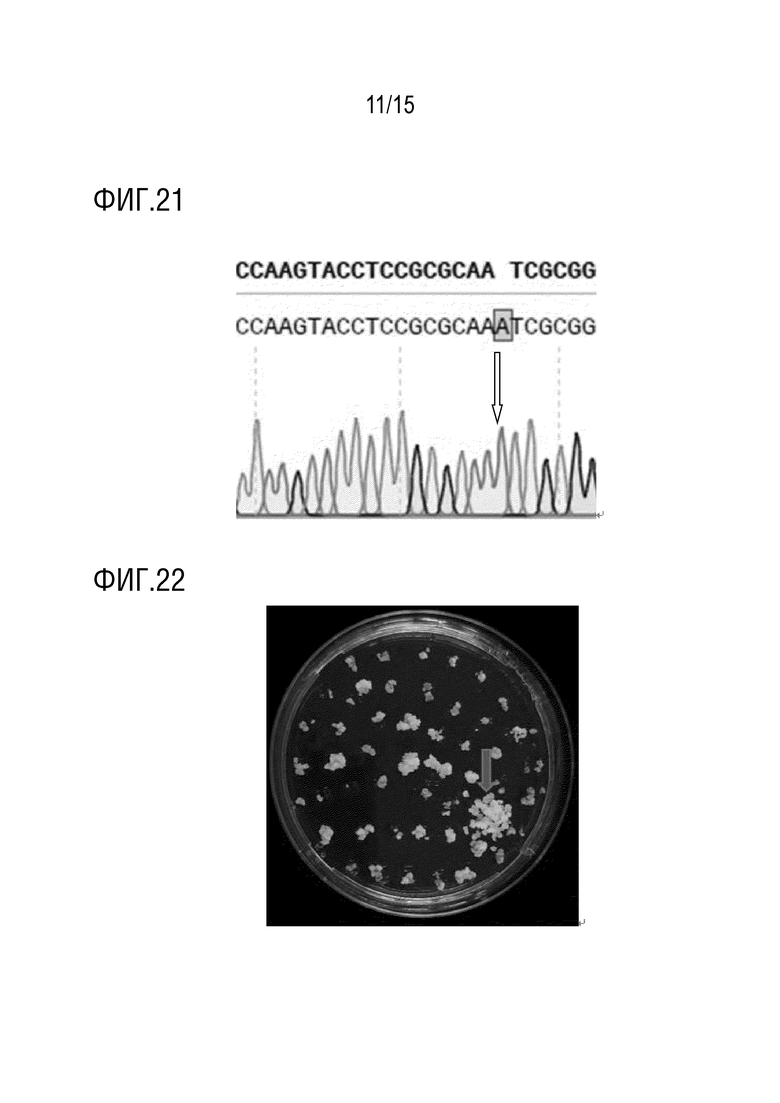
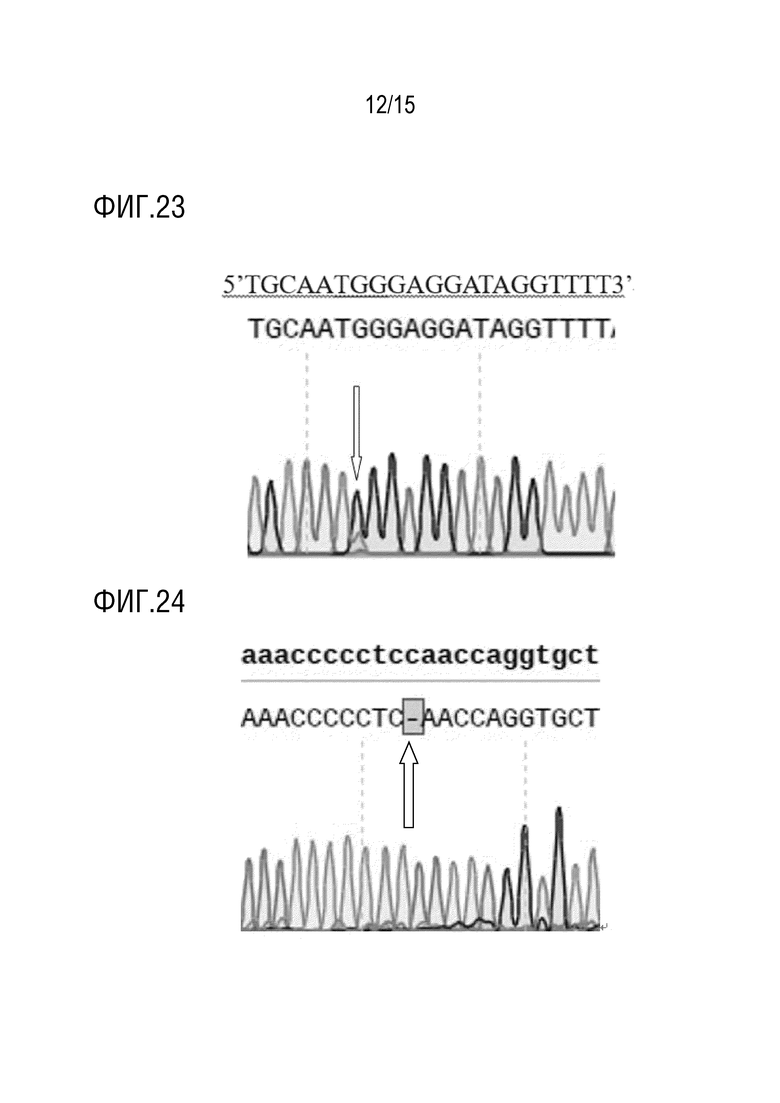
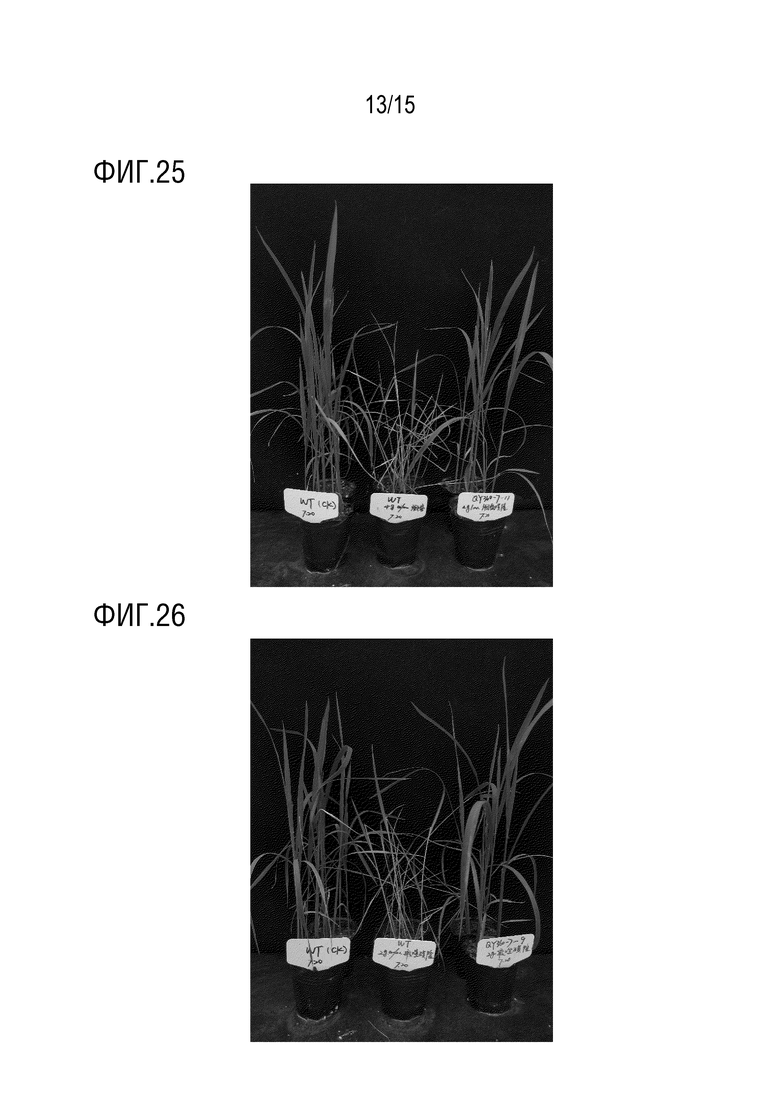
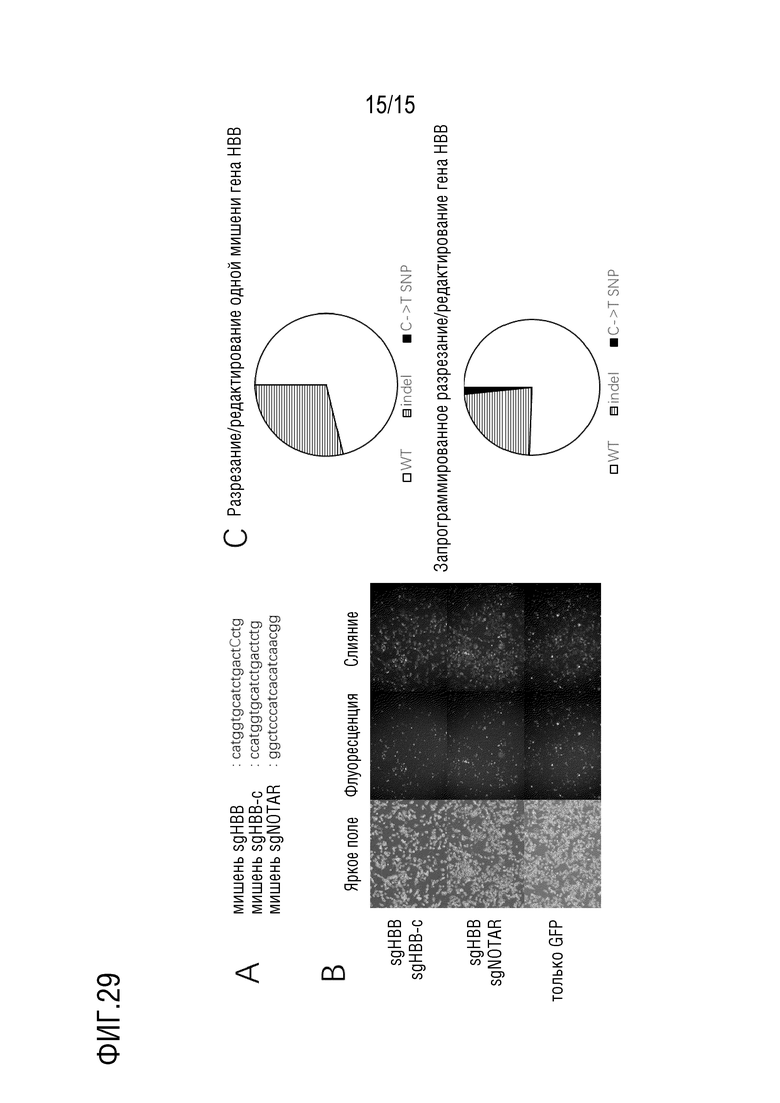
Изобретение относится к области биотехнологии, а именно к способу создания новой мутации в организме, который включает следующие стадии: последовательное создание двух или нескольких разрывов ДНК в специфичном сайте генома организма и их спонтанная репарация, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, созданной в результате предыдущей репарации разрыва ДНК, к способу лизиса ДНК-мишени, включающему контакт ДНК-мишени с комплексом, включающему полипептид Cas и, по меньшей мере, две таргетирующие РНК, а также к композиции и набору для их выполнения. Также раскрыты способ скрининга событий редактирования независимо от экзогенного трансгенного маркера, включающий последовательное создание двух или несколько разрывов ДНК в специфичном сайте первого гена-мишени клетки-реципиента и спонтанную репарацию, соответственно, где более поздний разрыв ДНК создают на основе новой последовательности, полученной в результате репарации предыдущего разрыва ДНК, а также способ нетрансгенного временного редактирования генома организма. Изобретение эффективно для создания новой мутации в организме. 6 н. и 20 з.п. ф-лы, 29 ил., 7 табл., 13 пр.
1. Способ создания новой мутации в организме, который включает следующие стадии: последовательное создание двух или нескольких разрывов ДНК в специфичном сайте генома организма и их спонтанная репарация, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, созданной в результате предыдущей репарации разрыва ДНК,
где «разрыв ДНК» достигается путем доставки нуклеазы с таргетирующим свойством в клетку организма для контакта со специфичным сайтом геномной ДНК, и
где «нуклеазой с таргетным свойством» является система CRISPR/Cas, где «последовательное создание двух или нескольких разрывов ДНК в специфичном сайте» относится к такому, которое основано на новой последовательности, образованной предыдущим событием репарации разрыва ДНК, созданным CRISPR/Cas системой, конструируют новую таргетирующую РНК для направления нуклеазы для повторного разрезания указанного сайта, где указанная таргетирующая РНК представляет собой sgРНК или CRISPR-РНК (crРНК).
2. Способ по п.1, отличающийся тем, что «два или несколько разрывов ДНК» создаются путем последовательной доставки разных таргетных нуклеаз в клетки-реципиенты разных поколений, где мутантную клетку, завершившую предыдущее редактирование, используют в качестве реципиента для получения доставки таргетной нуклеазы для последующего редактирования, тем самым выполняя второе редактирование для создания сайт-специфичной мутации; или
«два или несколько разрывов ДНК» создаются путем доставки разных таргетных нуклеаз для разных мишеней в одну и ту же клетку-реципиент; или
«два или несколько разрывов ДНК» создаются, когда комплексы RNP, образованные одной и той же CRISPR/Cas нуклеазой, соответственно, с разными crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени; или
«два или несколько разрывов ДНК» создаются, когда комплексы RNP, соответственно, образованные каждой из двух или нескольких CRISPR/Cas нуклеаз, которые распознают разные последовательности PAM, с соответствующими crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
3. Способ по п.2, отличающийся тем, что таргетной нуклеазой является любая CRISPR/Cas нуклеаза, способная осуществлять редактирование генома.
4. Способ по п.2, отличающийся тем, что целевая нуклеаза находится в форме ДНК;
или в форме mРНК или белка вместо ДНК.
5. Способ по п.2, отличающийся тем, что способ доставки таргетной нуклеазы в клетку выбран из: 1) способа ПЭГ-опосредованной трансфекции клеток; 2) способа липосомно-опосредованной трансфекции клеток; 3) способа электропорационной трансформации; 4) микроинъекции; 5) бомбардировки генной пушкой; или 6) способа Agrobacterium-опосредованной трансформации.
6. Способ лизиса ДНК-мишени, включающий контакт ДНК-мишени с комплексом, где комплекс включает:
(а) полипептид Cas; и
(b) по меньшей мере, две таргетирующие РНК, в которых первая таргетирующая РНК таргетирует ДНК-мишень, чтобы вызвать разрыв в специфичном сайте ДНК-мишени, и последняя таргетирующая РНК таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и снова создает разрыв в указанном специфичном сайте, где указанная таргетирующая РНК представляет собой sgРНК или crРНК.
7. Способ по п.6, отличающийся тем, что контакт включает введение в клетку следующего: (а) Cas полипептида или полинуклеотида, кодирующего Cas полипептид, и (b) тергетирующей РНК или ДНК полинуклеотида, кодирующего таргетирующую РНК.
8. Композиция для выполнения способа по п. 6 или 7, содержащая:
(а) Cas полипептид или полинуклеотид, кодирующий Cas полипептид; и
(b) по меньшей мере, две таргетирующие РНК или ДНК полинуклеотида, кодирующих указанные таргетирующие РНК, где первая РНК таргетирует ДНК-мишень, чтобы вызвать разрыв в специфичном сайте ДНК-мишени, и последняя таргетирующая РНК таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и снова создает разрыв в указанном специфичном сайте, где указанная таргетирующая РНК представляет собой sgРНК или crРНК.
9. Набор для выполнения способа по п. 6 или 7, включающий:
(а) Cas полипептид или нуклеиновую кислоту, содержащую нуклеотидную последовательность, кодирующую Cas полипептид; и
(b) по меньшей мере, две таргетирующие РНК или нуклеиновые кислоты, содержащие нуклеотидные последовательности, кодирующие указанные таргетирующие РНК, где первая РНК таргетирует ДНК-мишень, чтобы вызвать разрыв в специфичном сайте ДНК-мишени, и последняя таргетирующая РНК таргетирует последовательность, созданную в результате предыдущего события репарации разрыва, и снова создает разрыв в указанном специфичном сайте, где указанная таргетирующая РНК представляет собой sgРНК или crРНК;
где (а) и (b) находятся в одном и том же или отдельных контейнерах.
10. Набор по п.9, где указанные таргетирующие РНК в (b) находятся в одном и том же или отдельных контейнерах.
11. Способ скрининга событий редактирования независимо от экзогенного трансгенного маркера, включающий следующие стадии:
1) два или несколько разрывов ДНК последовательно создают в специфичном сайте первого гена-мишени клетки-реципиента и спонтанно репарируют, соответственно, где более поздний разрыв ДНК создают на основе новой последовательности, полученной в результате репарации предыдущего разрыва ДНК;
где «разрыв ДНК» достигается путем доставки нуклеазы с таргетирующим свойством в клетку-реципиент для контакта со специфичным сайтом гена-мишени, и
где «нуклеазой с таргетным свойством» является система CRISPR/Cas,
где признак «два или несколько разрывов ДНК последовательно создаются в специфичном сайте» относится к тому, что основано на новой последовательности, образованной предыдущим событием репарации разрыва ДНК, созданным системой CRISPR/Cas, создают новую таргетирующую РНК для направления нуклеазы для повторного разрезания указанного сайта, где таргетирующая РНК представляет собой sgРНК или CRISPR-РНК (crРНК);
2) события редактирования создают после того, как конкретный сайт первого гена-мишени последовательно отрезают и репарируют на стадии 1), что может придать мутантной клетке резистентность к давлению отбора для продуцирования фенотипического селектируемого признака, соответствующее давление отбора применяют для селекции признака, и выделяют клетку, ткань, орган или целый организм, который содержит такие события редактирования;
где «давлением отбора» может быть давление окружающей среды или давление, возникающее в результате добавления соединения; и
3) необязательно, в дополнение к первому гену-мишени, таргетную нуклеазу для, по меньшей мере, одного второго гена-мишени используют для одновременного редактирования другого сайта-мишени, и событие редактирования второго гена-мишени синхронно обогащают и подвергают скринингу посредством скрининга селектируемого признака, созданного мутациями первого гена-мишени, и выделяют клетку, ткань, орган или целый организм, которые одновременно содержат события редактирования первого гена-мишени и, по меньшей мере, одного второго гена-мишени.
12. Способ по п.11, отличающийся тем, что «первым геном-мишенью» является локус гена, кодирующий, по меньшей мере, один фенотипический селектируемый признак, где, по меньшей мере, одним фенотипическим селектируемым признаком является признак резистенции/толерантности или признак преимущества роста.
13. Способ по п.11 или 12, отличающийся тем, что «специфичный сайт первого гена-мишени» относится к сайту, в котором возникает мутация после последовательных разрезов и репараций, который способен придать клетке-реципиенту резистентность к давлению отбора для получения, по меньшей мере, одного фенотипически селектируемого признака резистентности/толерантности или признака преимущества роста;
отличающийся тем, что мутация включает замену одного основания, замену множества оснований или вставку или делецию неопределенного числа оснований.
14. Способ по любому из пп.11-13, отличающийся тем, что «разрыв ДНК» достигается путем доставки нуклеазы с таргетным свойством в клетку организма для контакта со специфичным сайтом геномной ДНК.
15. Способ по любому из пп.11-14, отличающийся тем, что «два или несколько разрывов ДНК» образуются, когда комплексы RNP, образованные одной и той же CRISPR/Cas нуклеазой, соответственно, с различными crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени; или
«два или несколько разрывов ДНК» образуются, когда комплексы RNP, образованные двумя или несколькими CRISPR/Cas нуклеазами, которые распознают разные последовательности РАМ с соответствующими crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
16. Способ по любому из пп.11-15, отличающийся тем, что «таргетная нуклеаза для, по меньшей мере, одного второго гена-мишени» и CRISPR/Cas нуклеаза, используемая для создания разрыва ДНК в специфичном сайте первого гена-мишени, являются одинаковыми или разными.
17. Способ по любому из пп.11-16, отличающийся тем, что таргетная нуклеаза имеет форму ДНК;
или форму mРНК или белка вместо ДНК.
18. Способ нетрансгенного временного редактирования генома организма, включающий следующие стадии:
1) создания и синтеза комбинации, по меньшей мере, двух фрагментов crРНК или комбинации, по меньшей мере, двух фрагментов sgРНК для специфичного сайта первого гена-мишени клетки-реципиента, где комбинация crРНК в комбинации с tracrРНК или только комбинация sgРНК способны управлять соответствующим Cas белком для последовательного создания двух или нескольких разрывов ДНК в конкретном сайте в первом гене-мишени клетки-реципиента и их спонтанной репарации, соответственно, где более поздний разрыв ДНК создается на основе новой последовательности, создаваемой из предыдущей репарации разрыва ДНК;
2) смешивания соответствующего количества белка CRISPR/Cas или его соответствующей mРНК с комбинацией фрагментов crРНК и фрагмента tracrРНК или только с комбинацией фрагментов sgРНК, как указано выше, созданной и синтезированной заранее, которая способна направлять сайт-специфическое редактирование первого гена-мишени для создания эндогенных селективных маркеров, необязательно, последующего добавления, по меньшей мере, одного из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней, и проведения инкубации in vitro с образованием комплекса RNP;
3) доставки вышеуказанного комплекса RNP в клетку-реципиент посредством способа кратковременного введения и контакта со специфичным сайтом геномной ДНК для осуществления редактирования гена;
4) в соответствии с фенотипическим селектируемым признаком, создаваемым сайт-специфическим редактированием первого гена-мишени комплексом RNP, применения соответствующего давления отбора, чтобы провести селекцию признака и выделения клетки, ткани, органа или всего организма, который содержит событие редактирования, и, необязательно, выделения клетки, ткани, органа или полного организма, которые одновременно содержат события редактирования первого гена-мишени и, по меньшей мере, одного из второго, третьего или нескольких генов-мишеней,
где «давлением отбора» является давление окружающей среды или давление, возникающее в результате добавления соединения.
19. Способ по п.18, отличающийся тем, что «первым геном-мишенью» является локус гена, кодирующий, по меньшей мере, один фенотипически селектируемый признак, где, по меньшей мере, одним фенотипически селектируемым признаком является признак резистентности/толерантности или признак преимущества роста.
20. Способ по п.18 или 19, отличающийся тем, что «конкретный сайт первого гена-мишени» относится к сайту, в котором мутация образуется после последовательных разрезов и репараций, который способен придавать клетке-реципиенту резистентность к давлению отбора для получения, по меньшей мере, одного фенотипически селектируемого признака резистентности/толерантности или признака преимущества роста;
отличающийся тем, что мутация включает замену одного основания, замену множества оснований или вставку или делецию неопределенного числа оснований.
21. Способ по любому из пп.18-20, отличающийся тем, что признак «для последовательного создания двух или нескольких разрывов ДНК в конкретном сайте» относится к признаку, основанному на новой последовательности, созданной предыдущим событием репарации разрыва ДНК, созданным системой CRISPR/Cas, конструируют новую таргетирующую РНК для повторного разрезания указанного сайта, где указанная таргетирующая РНК представляет собой sgРНК или crРНК.
22. Способ по любому из пп.18-21, отличающийся тем, что «два или несколько разрывов ДНК», образованные, когда комплексы RNP формируются одной и той же CRISPR/Cas нуклеазой, соответственно, с различными crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени; или
«два или несколько разрывов ДНК» создаются, когда комплексы RNP, соответственно, образованные каждой из двух или нескольких нуклеаз CRISPR/Cas, которые распознают разные последовательности PAM с соответствующими crРНК или sgРНК, последовательно разрезают соответствующие последовательности-мишени.
23. Способ по любому из пп.18-22, отличающийся тем, что «по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней» имеет тот же самый Cas белок, что и crРНК или sgРНК, таргетирующие первый ген-мишень; или
«по меньшей мере, один из искусственно синтезированных фрагментов crРНК и tracrРНК или искусственно синтезированных фрагментов sgРНК, таргетирующих второй, третий или несколько генов-мишеней», и crРНК или sgРНК, таргетирующих первый ген-мишень, используют Cas белки, которые распознают разные последовательности PAM.
24. Способ по любому из пп.18-23, отличающийся тем, что если организмом является растение, клетку-реципиент заменяют на растительную реципиентную клетку или ткань; где «реципиентной растительной клеткой или тканью» является любая клетка или ткань, которые могут служить реципиентом для временной экспрессии и могут быть регенерированы в полноценное растение через культуру ткани; где клеткой предпочтительно является протопластная клетка или суспензионная клетка; и тканью предпочтительно является каллюс, незрелый зародыш, зрелый зародыш, лист, верхушка побега, молодой колос или гипокотиль.
25. Способ по п.24, отличающийся тем, что «первым геном-мишенью» является, по меньшей мере, один эндогенный ген, который кодирует, по меньшей мере, один фенотипический селектируемый признак, выбранный из резистентности/толерантности к гербицидам, где резистентность/толерантность к гербицидам выбрана из группы, состоящей из резистентности/толерантности к ингибитору EPSPS, резистентности/толерантности к ингибитору синтеза глутамина, резистентности/толерантности к ингибитору ALS или AHAS, резистентности/толерантности к ингибитору АССазы, резистентности/толерантности к ингибитору биосинтеза каротиноидов, резистентности/толерантности к ингибитору целлюлозы, резистентности/толерантности к ингибитору синтеза жиров, резистентности/толерантности к ингибитору длинноцепочечных жирных кислот, резистентности/толерантности к ингибитору сборки микротрубочек, резистентности/толерантности к электрон шунтирующим агентам фотосистемы I, резистентности/толерантности к ингибитору фотосистемы II или резистентности/толерантности к ингибитору PPO и резистентности/толерантности к синтетическому гормону роста.
26. Способ по п.25, отличающийся тем, что «первым геном-мишенью» является ALS, и «специфичный сайт гена» относится к сайту A122, P197, R198, D204, A205, D376, R377, W574, S653 или G654 в аминокислотной последовательности белка Arabidopsis AtALS, и аминокислотные сайты в белке ALS другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам при использовании аминокислотной последовательности AtALS в качестве эталонного стандарта; или
указанная crРНК или sgРНК таргетирует последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности белка AtALS, выбранный из группы, состоящей из A122, P197, R198, D204, A205, D376, R377, W574, S653, G654 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке ALS другого растения, который соответствует указанному выше аминокислотному сайту, и любую их комбинацию, с использованием аминокислотной последовательности AtALS в качестве эталонного стандарта; или
«первым геном-мишенью» является АССаза, и «специфичный сайт гена» относится к сайту I1781, E1874, N1878, W1999, W2027, I2041, D2078, C2088 или G2096 в аминокислотной последовательности белка AmACCase Alopecurus myosuroides, и аминокислотным сайтам в белке ACCазе другого однодольного растения, которые соответствуют вышеупомянутым аминокислотным сайтам, при использовании аминокислотной последовательности AmACCase в качестве эталонного стандарта; или
crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности AmACCase, выбранный из группы, состоящей из I1781, E1874, N1878, W1999, W2027, I2041, D2078, C2088, G2096 или любую их комбинацию, и последовательность-мишень содержащую последовательность, кодирующую аминокислотный сайт в белке АССазы другого однодольного растения, который соответствует вышеупомянутым аминокислотным сайтам и любой их комбинации, при использовании аминокислотной последовательности AmACCase в качестве эталонного стандарта; или
«первым геном-мишенью» является HPPD, и «специфичный сайт гена» относится к сайту H141, L276, P277, N338, G342, R346, D370, P386, K418 или G419 в аминокислотной последовательности белка Oryza sativa OsHPPD, и аминокислотным сайтам в белке HPPD другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам, при использовании аминокислотной последовательности OsHPPD в качестве эталонного стандарта; или
указанная crРНК или sgРНК таргетирует последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsHPPD, выбранный из группы, состоящей из H141, L276, P277, N338, G342, R346, D370, P386, K418, G419 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке HPPD другого растения, который соответствует вышеупомянутому аминокислотному сайту и любой их комбинации, при использовании аминокислотной последовательности OsHPPD в качестве эталонного стандарта; или
«первым геном-мишенью» является РРО, и «специфичный сайт гена» относится к сайту S128, V217, S223, V364, K373, L423, Y425 или W470 в аминокислотной последовательности белка Oryza sativa OsPPO1, и аминокислотным сайтам в белке РРО другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам, при использовании аминокислотной последовательности OsPPO1 в качестве эталонного стандарта; или
crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsPPO1, выбранный из группы, состоящей из S128, V217, S223, V364, K373, L423, Y425, W470, или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке РРО другого растения, который соответствует указанному выше аминокислотному сайту и любой их комбинации, при использовании аминокислотной последовательности OsPPO1 в качестве эталонного стандарта; или
«первым геном-мишенью» является TIR1, и «специфичный сайт гена» относится к сайт F93, F357, C413 или S448 в аминокислотной последовательности белка Oryza sativa OsTIR1, и аминокислотным сайтам в белке TIR1 другого растения, которые соответствуют вышеупомянутым аминокислотным сайтам при использовании аминокислотной последовательности OsTIR1 в качестве эталонного стандарта; или
crРНК или sgРНК таргетируют последовательность-мишень, содержащую последовательность, кодирующую сайт аминокислотной последовательности OsTIR1, выбранный из группы, состоящей из F93, F357, C413, S448 или любой их комбинации, и последовательность-мишень, содержащую последовательность, кодирующую аминокислотный сайт в белке TIR1 другого растения, который соответствует указанному выше аминокислотному сайту, и любую их комбинацию, при использовании аминокислотной последовательности OsTIR1 в качестве эталонного стандарта.
| WO 2018202199 A1, 08.11.2018 | |||
| WO 2018140899 A1, 02.08.2018 | |||
| WO 2018205995 A1, 15.11.2018 | |||
| А.А | |||
| НЕМУДРЫЙ и др., Системы редактирования геномов TALEN и CRISPR/Cas - инструменты открытий, Acta naturae, 2014, т | |||
| Приспособление для точного наложения листов бумаги при снятии оттисков | 1922 |
|
SU6A1 |
Авторы
Даты
2025-01-30—Публикация
2020-10-13—Подача