Область техники, к которой относится изобретение
[0001] Настоящее изобретение относится к биспецифичному слитому белку с новой структурой и его применению. Настоящее изобретение также относится к полинуклеотиду, конструкции нуклеиновой кислоты и клетке-хозяину, кодирующей или продуцирующей слитый белок.
Уровень техники
[0002] С развитием биотехнологии антитела как зрелые терапевтические лекарственные средства играют важную роль в лечении опухолей и иммунных заболеваний. В настоящее время большинство препаратов на основе антител, представленных на рынке, нацелены на один антиген и могут действовать только на одну молекулу-мишень. Однако сложные заболевания часто имеют многофакторную природу, включая сверхэкспрессию или синергию медиаторов заболевания или активацию различных рецепторов, включая молекулярные взаимодействия в их сигнальных сетях. Следовательно, регулирование множественных сигнальных путей может повысить эффективность терапевтических антител. Терапия биспецифичными антителами (BsAb) с использованием стратегий двойного нацеливания стала одним из эффективных методов лечения опухолей и иммунных заболеваний (Kontermann R. Dual targeting strategies with bispecific antibodies [C]//MAbs. Taylor & Francis, 2012, 4(2): 182-197.).
[0003] Биспецифичные антитела могут специфично связываться с двумя разными антигенами или эпитопами (Carter P. Bispecific human IgG by design [J]. Journal of immunological methods, 2001, 248(1-2): 7-15.). Еще в 1980-х годах Morrison et al. первыми получили первое по-настоящему четырехвалентное биспецифичное антитело против декстрана и дансильной группы путем связывания одноцепочечных антител с различной специфичностью с использованием гибких пептидов для экспрессии слитого белка (Coloma MJ, Morrison SL. Design and production of novel tetravalent bispecific antibodies [J]. Nature biotechnology, 1997, 15(2): 159.). Однако из-за узких мест в технологии получения антител и отсутствия фундаментальных исследований сигнальных путей разработка биспецифичных антител была затруднена. Благодаря глубокому пониманию патогенеза заболевания и быстрому развитию технологий, связанных с терапевтическими моноклональными антителами (McAbs), технологии конструирования, экспрессии и очистки антител значительно улучшились, что позволяет разработчикам биспецифичных антител иметь технологии и мотивацию для преодоления ограничивающих факторов. В настоящее время существуют в основном следующие механизмы получения биспецифиченых или мультиспецифичных антител: (1) соединение клеток (в транс); (2) ингибирование рецептора (в цис) или активация рецептора (в цис); (3) имитация кофактора; (4) подходы PIgGyback (Aran F. Labrijn, et al. Bispecific antibodies: a mechanistic review of the pipeline, Nature Reviews Drug Discovery том 18, стр. 585-608(2019)).
[0004] При разработке биспецифичных антител очень важен выбор молекулярной структуры. Различные конструкции биспецифичных антител имеют свои преимущества и недостатки, но дизайн биспецифичных антител для целей клинического лечения должен решать следующие проблемы: во-первых, обеспечение правильного связывания или спаривания двух пар (или более) различных легких цепей и тяжелых цепей; во-вторых, поддержание независимости каждого связывающего домена каждого моноклонального антитела, чтобы при одновременном связывании разных эпитопов между ними не было пространственное влияние друг на друга; в-третьих, молекулы антител должны легко экспрессироваться в клетках млекопитающих без сложного процесса модификации белка; в-четвертых, требуется хорошая лекарственная устойчивость, такая как высокая термическая стабильность, высокая химическая стабильность, высокая растворимость, невысокая склонность к полимеризации, низкая вязкость, высокая экспрессия, подходящее время полужизни и тому подобное (Spiess C, Zhai Q, Carter P J. Alternative molecular formats and therapeutic applications for bispecific antibodies [J]. Molecular immunology, 2015, 67(2): 95-106).
[0005] В соответствии с различными формами молекулярной структуры (т.е. компонентами и способами конструирования) биспецифичные антитела можно разделить на множество типов. Например, по симметрии конструкции влево-вправо они делятся на симметричные и асимметричные структуры; в зависимости от наличия или отсутствия Fc-области их можно разделить на биспецифичные антитела, содержащие Fc-область, и биспецифические антитела, лишенные Fc-области; и в зависимости от количества антигенсвязывающих областей они делятся на двухвалентные, трехвалентные, четырехвалентные или поливалентные конфигурации и т.д. В настоящее время биспецифичные антитела насчитываются десятками (Spiess C, Zhai Q, Carter P J. Alternative molecular formats and therapeutic applications for bispecific antibodies [J]. Molecular immunology, 2015, 67(2): 95-106), эти десятки биспецифичных антител имеют различные структуры, примерно разделенные на 5 категорий, включая IgG-подобные биспецифичные антитела, IgG-подобные биспецифичные антитела с дополнительными функциональными областями, биспецифичные антитела с различными антигенсвязывающими фрагментами, соединенными последовательно, биспецифичные антитела типа слитого белка и химически связанные биспецифичные антитела.
Из всех этих типов для создания IgG-подобных биспецифичных антител в основном применяют технологию рекомбинантных ДНК для структурной модификации. Сконструированные IgG-подобные биспецифичные антитела имеют полные кристаллизующиеся фрагменты (т.е. Fc-фрагменты), и, таким образом, они могут связываться с различными антигенами, сохраняя при этом опосредованную Fc-фрагментом ADCC (антителозависимая клеточно-опосредованная цитотоксичность) и CDC (опосредованная комплементом цитотоксичность, комплемент-зависимая цитотоксичность) и т.п., и в то же время этот тип антител также сохраняет свойство увеличенного времени полужизни антитела in vivo за счет связывания с неонатальным рецептором Fc (FcRn) (Ridgway J B B, Presta L G, Carter P. «Knobs-into-holes» engineering of antibody CH3 domains for heavy chain heterodimerization [J]. Protein Engineering, Design and Selection, 1996, 9(7): 617-621.); IgG-подобные биспецифичные антитела с дополнительными функциональными областями обычно основаны на традиционных антителах IgG и получаются путем добавления других специфичных антигенсвязывающих фрагментов (то есть дополнительных функциональных областей, таких как однодоменные антитела, одноцепочечные антитела и т.д.) к тяжелой цепи и/или легкой цепи с помощью технологии слитых белков (LaFleur D, Abramyan D, Kanakaraj P, et al. Monoclonal antibody therapeutics with up to five specificities: functional enhancement through fusion of target-specific peptides [C]//MAbs. Taylor & Francis, 2013, 5(2): 208-218.); биспецифичные антитела с разными антигенсвязывающими фрагментами, соединенными в серию, получают путем связывания разных Fab, одноцепочечных антител, однодоменных антител или антигенсвязывающих фрагментов в определенной последовательности через связывающий пептид (Stork R, Müller D, Kontermann R E. A novel tri-functional antibody fusion protein with improved pharmacokinetic properties generated by fusing a bispecific single-chain diabody with an albumin-binding domain from streptococcal protein G [J]. Protein Engineering, Design & Selection, 2007, 20(11): 569-576.); биспецифичные антитела типа слитого белка представляют собой белковые молекулы, которые могут одновременно связывать два или более антигена, образованные путем связывания антител/фрагментов антител (IgG, Fab, scFv и т.д.) с различной специфичностью через линкер, такой как полипептидный фрагмент; и химически связанные биспецифичные антитела аналогичны биспецифичным антителам типа слитого белка, но для химически связанных биспецифичных антител необходимо сначала получить два антитела/фрагмента антитела по отдельности, а затем эти два антитела/фрагмента антитела соединяют посредством химических связей или их соединяют с белком-носителем.
[0006] Кроме того, с развитием фундаментальных исследований сигнальных путей и расширением механизмов действия би-антител появляется все больше и больше мишеней и комбинаций, доступных для отбора биспецифичных антител, таких как PD-1, PD-L1, CTLA- 4, LAG-3, FGL1, TIM-3, галектин-9, TIGIT, CD155 и рецептор TGF-β (TGF-βR), CD80, CD86, VEGFR, ловушка VEGF, FGL1, CD70, 4-1BBL, OX40L, и SIRPα; и опухолевые антигены, включая клаудин 18.2, HER-2, мезотелин, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, VEGF-A, нектин-4, FGFR, C-met, RANKL, PDGF, PDGFR, PDGFRα, DLL4, Ang-1 и Ang-2 и т.д. Комбинации мишеней иммунных контрольных точек или опухолевых мишеней с другими мишенями, относящимися к сопротивлению развитию раковых клеток или активации Т-клеток, или комбинация множественных мишеней иммунных контрольных значительно улучшают возможность лечения злокачественных заболеваний, таких как опухоли. На ранней стадии комбинация антител для двух отдельных контрольных точек использовалась клинически для повышения эффективности. Например, по сравнению с лечением одним ипилимумабом, лечение комбинацией ипилимумаба (анти-CTLA4) и ниволумаба (анти-PD1) может улучшить выживаемость пациентов с меланомой. В настоящее время безопасность и ранняя эффективность четырех bsAb PD-1×CTLA4 оцениваются в ранних клинических испытаниях. Концепция ингибиторов, блокирующих две иммунные контрольные точки, также используется клинически для других целевых комбинаций, таких как PD-1×LAG3, PD-1×TIM3 и PD-L1×CTLA4 (Wolchok, J. D. et al. Overall Survival with Combined Nivolumab and Ipilimumab in Advanced Melanoma, N.Engl. J. Med. 377(14): 1345-1356, (2017); Dovedi, S. J. et al. MEDI5752: A novel bispecific antibody that preferentially targets CTLA-4 on PD-1 expressing T-cells. Cancer Res.78(13), Supplement. аннотация 2776: (2018). Hedvat, M. et al. Simultaneous checkpoint - checkpoint or checkpoint - costimulatory receptortargeting with bispecific antibodies promotes enhanced human T cell activation [аннотация P664]. Представлено на конференции Общества иммунотерапии рака (SITC) в 2018 (2018); Aran F. Labrijn, et al. Bispecific antibodies: a mechanistic review of the pipeline, Nature Reviews Drug Discovery том 18, страницы 585-608 (2019).
[0007] В настоящее время большинство биспецифичных антител все еще находятся на стадии клинических или доклинических исследований, и только три биспецифичных антитела одобрены для выхода на рынок. Это катумаксомаб компании Trion Pharma (исключен из списка по коммерческим причинам в 2017 году), блинатумомаб компании Amgen и эмицизумаб компании Roche. Катумаксомаб представляет собой IgG-подобное биспецифичное антитело в форме Триомаб. Он нацелен на Т-клетки и опухолевые клетки, на которых происходит избыточная экспрессия молекул адгезии эпителиальных клеток (EpCAM), и ингибирует опухолевые клетки посредством активации Т-клеток, антителозависимой клеточно-опосредованной цитотоксичности (ADCC) и комплемент-зависимой цитотоксичности (CDC). Он был одобрен Европейским агентством по лекарственным средствам (EMA) в 2009 году для лечения злокачественного асцита, вызванного EpCAM-положительными опухолями, для которого было неэффективно или невозможно стандартное лечение, и это также первое биспецифичное антитело, одобренное для выхода на рынок. Блинатумомаб представляет собой биспецифичное антитело без Fc-области и принимает структурную форму BiTE (биспецифичный активатор Т-клеток). Блинатумомаб связывается с Т-клетками через анти-CD3 часть и со злокачественными и нормальными В-клетками через анти-CD19 часть, тем самым вызывая опосредованный Т-клетками эффект уничтожения опухолевых клеток. В 2014 году он был одобрен Управлением по контролю качества пищевых продуктов и лекарственных препаратов (FDA США) для лечения отрицательного по филадельфийской хромосоме острого лимфобластного лейкоза предшественников В-клеток. Эмицизумаб представляет собой модифицированное гуманизированное биспецифичное моноклональное антитело IgG4, которое может связываться с фактором IXa и фактором X. В этом антителе используется принцип укладки «выступы-во-впадины» (KiH) для дизайна Fc, «Общая легкая цепь-IgG (CLC-IgG)» для дизайна LC и «многомерная оптимизация» для оптимизации вариабельной области, и это антитело было одобрено FDA в 2017 году для рутинной профилактики гемофилии A в присутствии ингибиторов фактора VIII.
Таблица 1. Биспецифичные антитела, одобренные для выхода на рынок (в том числе исключенные из списка)
(исключено из списка в 2017)
[0008] Несмотря на то что в настоящее время существуют десятки биспецифичных антител/платформ, а также опыт исследований и разработок трех имеющихся на рынке лекарств, существует больше проблем и трудностей при разработке биспецифичных антител по сравнению с общими антителами из-за их сложной структуры и различных функциональных характеристик. Например, IgG-подобные биспецифичные антитела сталкиваются с проблемой несоответствия тяжелой цепи и тяжелой цепи или тяжелой цепи и легкой цепи во время процесса получения, наиболее типичным из таких антител является катумаксомаб. Хотя катумаксомаб является первым биспецифичным антителом, одобренным для выхода на рынок, существуют некоторые очень явные ограничения, которые в основном отражаются в сложном процессе производства антитела Triomab и проблеме иммуногенности, связанной с относительной склонностью к появлению гетерологичных антител, и в 2017 году этот препарат был исключен из списка по коммерческим причинам; в качестве другого примера, биспецифичные антитела с различными антигенсвязывающими фрагментами, соединенными последовательно, имеют короткое время полужизни из-за отсутствия константных областей, особенно отсутствия Fc-фрагментов, а их уровень экспрессии и внутриклеточная стабильность после экспрессии обычно ниже, чем у традиционных IgG (Stork R, Müller D, Kontermann R E. A novel tri-functional antibody fusion protein with improved pharmacokinetic properties generated by fusing a bispecific single-chain diabody with an albumin-binding domain from streptococcal protein G [J]. Protein Engineering, Design & Selection, 2007, 20(11): 569-576.). Кроме того, для большинства антител этого типа трудно использовать средства аффинной очистки в процессе очистки, поэтому процесс очистки и анализ примесей в готовом продукте также сложнее, чем в случае традиционных IgG (Tan P H, Sandmaier B M, Stayton P S. Contributions of a highly conserved VH/VL hydrogen bonding interaction to scFv folding stability and refolding efficiency [J]. Biophysical journal, 1998, 75(3): 1473-1482), наиболее типичным из таких антител является Блинатумомаб. Блинатумомаб имеет небольшую молекулярную массу из-за отсутствия Fc-фрагментов, а его время полужизни в крови составляет всего 1,25±0,63 часа, что намного короче, чем время полужизни интактного антитела, поэтому этот препарат требует непрерывных внутривенных инъекций в течение 4 недель для достижения эффективной дозы, и его необходимо поставлять с дополнительным устройством для непрерывной инфузии при использовании. Кроме того, он имеет плохую стабильность и легко образует агрегаты в процессе производства, что также влияет на качество продукта и увеличивает производственные затраты. Современные биспецифичные антитела имеют множество недостатков, таких как низкая экспрессия, плохая стабильность, сложные производственные процессы и значительно более высокие затраты на исследования и разработки, чем в случае моноклональных антител, все эти недостатки ограничивают прогресс в разработке биспецифичных антител. Следовательно, существует острая необходимость в разработке биспецифичных антител с новыми структурами, чтобы предоставить больше клинических возможностей.
Сущность изобретения
[0009] В ответ на вышеупомянутые проблемы в настоящем изобретении предложен биспецифичный слитый белок с новой структурой, который принадлежит к IgG-подобному биспецифичному слитому белку с дополнительными функциональными областями. В частности, слитый белок имеет структуру, в которой второй связывающий домен вставлен в шарнирную область полноразмерного иммуноглобулина G (IgG) через необязательный пептидный линкер, а Fab-область IgG представляет собой первый связывающий домен слитого белка; после вставки второго связывающего домена тяжелая цепь IgG представляет собой тяжелую цепь слитого белка, а легкая цепь IgG представляет собой легкую цепью слитого белка; второй связывающий домен выбран из человеческого рецептора или лиганда, фрагмента внеклеточной области рецептора или лиганда, фрагмента связывающего домена и комбинации фрагментов рецептора или лиганда; где рецептор или лиганд образует структуру димеризации или мультимеризации в естественном сигнальном пути для активации или ингибирования сигнального пути, а второй связывающий домен и первый связывающий домен нацелены на разные мишени. Биспецифичный слитый белок с вышеуказанной структурой был назван слитым белком со структурой Hibody (биспецифичное антитело с шарнирной вставкой) (в дальнейшем именуемым слитым белком со структурой Hibody или Hibody).
[0010] Кроме того, первый связывающий домен нацелен и связывается с молекулой иммунной контрольной точки или опухолевым антигеном.
[0011] Кроме того, молекула иммунной контрольной точки включает в себя PD-1 (белок 1 запрограммированной гибели клеток), PD-L1 (лиганд 1 белка 1 запрограммированной гибели клеток), CTLA-4 (антиген-4, связанный с цитотоксическими Т-лимфоцитами), LAG-3 (ген активации лимфоцитов-3), FGL1 (фибриноген-подобный белок 1), TIM-3 (Т-клеточный иммуноглобулин-3), галектин-9, TIGIT (Т-клеточный иммунорецептор с доменами Ig и ITIM), CD155, CD47; опухолевый антиген включает в себя клаудин 18.2, Her-2 (рецептор-2 эпидермального фактора роста человека), мезотелин, BCMA (антиген созревания B-клеток), SSTR2 (рецептор соматостатина 2), GPRC5D (член D группы 5 семейства C рецепторов, сопряженных с G-белком), PSMA (простат-специфический мембранный антиген), FcRH5 (Fc-рецептор-подобный протеин 5), CD33, CD123, CD20, A33, CEA (карцино-эмбриональный антиген), CD28, DLL3 (дельта-подобный протеин 3), EGFR (рецептор эпидермального фактора роста), VEGFR (рецептор фактора роста эндотелия сосудов), VEGFR2 (рецептор фактора роста эндотелия сосудов 2), VEGF-A (фактор роста эндотелия сосудов А), нектин-4, FGFR (рецепторы фактора роста фибробластов), C-met, RANKL (активатор рецептора ядерного фактора лиганда каппа-B), PDGF (фактор роста тромбоцитов), PDGFR (рецептор фактора роста тромбоцитов), PDGFRα (рецептор фактора роста тромбоцитов α), DLL4 (дельта-подобный лиганд 4), Ang-1 (Ангиопоэтин-1) и Ang-2 (Ангиопоэтин-2).
[0012] Кроме того, второй связывающий домен нацелен и связывается с человеческим TGF-β (трансформирующий фактор роста-β), CTLA-4, VEGF, LAG3, CD27, 4-1BB (CD137), OX40 (CD134), CD47, FGL1 (Фибриноген-подобный протеин 1), TLT-2 (Trem-подобный транскрипт 2), CD28, HGF (фактор роста гепатоцитов), CSF1 (колониестимулирующий фактор 1), CXCL1 (лиганд 1 хемокина CXC 1), CXCL2 (лиганд 2 хемокина CXC), CXCL3 (лиганд 3 хемокина CXC), CXCL5 (лиганд 5 хемокина CXC), CXCL6 (лиганд 6 хемокина CXC), CXCL7 (лиганд 7 хемокина CXC), CXCL8 (лиганд 8 хемокина CXC), CXCL9 (лиганд 9 хемокина CXC), CXCL10 (лиганд 10 хемокина CXC), CXCL12 (лиганд 12 хемокина CXC), GITR (рецептор фактора некроза опухоли, индуцируемого глюкокортикоидами), EGF (эпидермальный фактор роста) и ICOSL (лиганд индуцируемого костимулятора).
[0013] Кроме того, рецептор или лиганд представляет собой человеческий рецептор TGF-β (TGF-βR), CD80, CD86, VEGFR, ловушку VEGF, FGL1, CD70, 4-1BBL, OX40L, SIRPα (сигнальный регуляторный белок α), B7-H3 (CD276), C-met, CSF1R (рецептор колониестимулирующего фактора 1), CXCR2 (рецептор 2 хемокина CXC), CXCR3 (рецептор 3 хемокина CXC), CXCR4 (рецептор 4 хемокина CXC), GITRL (лиганд рецептора TNF, индуцируемого глюкокортикоидами), EGFR и ICOS (индуцируемый костимулятор).
[0014] Среди них CD80 и CD86 представляют собой трансмембранные гликопротеины, которые являются членами суперсемейства иммуноглобулинов (IgSF). Зрелая молекула CD80 состоит из 254 аминокислот, включая 208 аминокислот во внеклеточном домене (ECD), 25 аминокислот в трансмембранном домене и 21 аминокислоту во внутриклеточном домене. Аналогично зрелая молекула CD86 состоит из 303 аминокислот, включая 222 аминокислоты во внеклеточном домене, 20 аминокислот в трансмембранном домене и 61 аминокислоту во внутриклеточном домене. Внеклеточные домены CD80 и CD86 включают в себя V-область иммуноглобулина (IgV) и C-область иммуноглобулина (IgC). CD80 и CD86 связываются со своими лигандами CD28 и CTLA-4 через V-область иммуноглобулина (IgV). В случае связывания CD80 и CD86 с CD28, CD80 и CD86 оказывают важное регулирующее действие на индуцированную антигеном активацию, пролиферацию и выработку эффекторной функции Т-клеток, действуя как положительные регуляторы; тогда как в случае связывания CD80 и CD86 с CTLA-4, CD80 и CD86 подавляют иммунный ответ, действуя как негативные регуляторы. Следовательно, CD80 и CD86 являются костимулирующими факторами при активации Т-лимфоцитов, и они играют важную роль в аутоиммунном мониторинге, гуморальном иммунном ответе и ответе на трансплантацию. В отличие от использования агонистических анти-CD28 антител для активации Т-клеток, использование лиганда CD28 CD80 в качестве активирующего фактора для активации Т-клеток не вызывает серьезного цитокинового шторма, тем самым значительно снижая возможность подвергнуть опасности жизнь пациента.
[0015] В некоторых вариантах осуществления изобретения ECD CD80 выбран из CD80 человека (такого как CD80 человека с SEQ ID NO: 134) или ECD CD80 человека, полученного из изотипа 2 или изотипа 3 CD80 (SEQ ID NO: 135 и 136). ECD CD80 содержит V-область иммуноглобулина (IgV) CD80 (CD80-IgV, SEQ ID NO: 133). В одном варианте осуществления изобретения ECD CD80 содержит V-область и C-область иммуноглобулина CD80 человека (CD80-IgVIgC, SEQ ID NO: 32). В одном варианте осуществления изобретения ECD CD80 представляет собой ECD CD80 человека. В одном варианте осуществления изобретения ECD CD80 включает в себя IgV CD80 человека.
[0016] CD80-IgV (SEQ ID NO: 133):

[0017] CD80-IgVIgC (SEQ ID NO: 32):

[0018] CD80 изотип 1 (SEQ ID NO: 134):

[0019] CD80 изотип 2 (SEQ ID NO: 135):

[0020] CD80 изотип 3 (SEQ ID NO: 136):

[0021] Кроме того, первый связывающий домен и второй связывающий домен выбраны из следующих комбинаций:
[0022] (1) Первый связывающий домен нацелен и связывается с PD-L1, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, FGL1, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
[0023] (2) Первый связывающий домен нацелен и связывается с PD-1, а второй связывающий домен нацелен и связывается с человеческим CTLA-4, VEGF, HGF, EGF, CD28, LAG3, CD27, 4-1BB или OX40; или
[0024] (3) Первый связывающий домен нацелен и связывается с CTLA-4, а второй связывающий домен нацелен и связывается с человеческим CD28, VEGF, HGF, EGF, LAG3, CD27, 4-1BB или OX40; или
[0025] (4) Первый связывающий домен нацелен и связывается с LAG-3, а второй связывающий домен нацелен и связывается с человеческим CD28, VEGF, HGF, EGF, CTLA-4, CD27, 4-1BB или OX40; или
[0026] (5) Первый связывающий домен нацелен и связывается с FGL1, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
[0027] (6) Первый связывающий домен нацелен и связывается с TIM-3, а второй связывающий домен нацелен и связывается с человеческим VEGF, HGF, EGF, LAG3, CD27, 4-1BB или OX40; или
[0028] (7) Первый связывающий домен нацелен и связывается с галектином-9, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
[0029] (8) Первый связывающий домен нацелен и связывается с TIGIT, а второй связывающий домен нацелен и связывается с человеческим TGF-β, HGF, EGF или VEGF; или
[0030] (9) Первый связывающий домен нацелен и связывается с CD155, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, HGF, EGF, CD47 или CD155; или
[0031] (10) Первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2, мезотелином, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, VEGF-A, нектином-4, FGFR, C-met, RANKL, PDGF, PDGFR, PDGFRα, DLL-4, Ang-1 или Ang-2, а второй связывающий домен нацелен и связывается с человеческим CTLA-4, TGF-β, VEGF, FGL1, LAG3, 4-1BB, OX40, CD27, CD28, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, EGF или ICOSL.
[0032] Кроме того, первый связывающий домен и рецептор или лиганд выбраны из следующих комбинаций:
[0033] (1) Первый связывающий домен нацелен и связывается с PD-L1, а рецептор или лиганд выбран из TGF-βRII, VEGFR, LAG-3, SIRPα, TIGIT, C-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
[0034] (2) Первый связывающий домен нацелен и связывается с PD-1, а рецептор или лиганд выбран из человеческого CD80, CD86, VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
[0035] (3) Первый связывающий домен нацелен и связывается с CTLA-4, а рецептор или лиганд выбран из человеческого CD80, CD86, VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
[0036] (4) Первый связывающий домен нацелен на LAG-3 и связывается с ним, и рецептор или лиганд выбран из человеческого VEGFR, c-MET, EGFR, CD80, CD86, CD70, 4-1BBL или OX40L; или
[0037] (5) Первый связывающий домен нацелен и связывается с FGL1, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, SIRPα, TIGIT, c-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
[0038] (6) Первый связывающий домен нацелен и связывается с TIM-3, а рецептор или лиганд выбран из человеческого VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
[0039] (7) Первый связывающий домен нацелен и связывается с галектином-9, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, SIRPα, TIGIT, c-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
[0040] (8) Первый связывающий домен нацелен и связывается с TIGIT, а рецептор или лиганд выбран из человеческого TGF-βRII, c-MET, EGFR или VEGFR; или
[0041] (9) Первый связывающий домен нацелен и связывается с CD155, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, c-MET, EGFR, SIRPα или TIGIT; или
[0042] (10) Первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2, мезотелином, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, нектином-4, FGFR, c-MET, RANKL, PDGF, PDGFR, PDGFRα, DLL4, Ang-1 или Ang-2, а рецептор или лиганд выбран из человеческого CTLA-4, CD80, CD86, TGF-βRII, VEGFR, FGL1, LAG3, 4-1BB, OX40, CD27, CD28, CD70, c-MET, SIRPα, TIGIT, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS.
[0043] Кроме того, фрагмент рецептора или лиганда включает в себя фрагмент внеклеточной области и фрагмент связывающего домена рецептора или лиганда.
[0044] Кроме того, первый связывающий домен нацелен и связывается с PD-L1; второй связывающий домен представляет собой фрагмент человеческого TGF-βRII; или первый связывающий домен нацелен и связывается с PD-1, а второй связывающий домен включает в себя человеческий ECD CD80, V-область Ig CD80, человеческий IgVIgC CD80, ECD VEGFR1, ECD VEGFR2 или комбинацию второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2; или первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2 или EGFR, а второй связывающий домен выбран из человеческого ECD CD80, V-области Ig CD80, человеческого IgVIgC CD80, ECD VEGFR1, ECD VEGFR2 или комбинации второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2.
[0045] Кроме того, второй связывающий домен выбран из:
[0046] (1) Человеческого TGF-βRII, содержащего последовательность, показанную в SEQ ID NO: 31, 131 или 132, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 31, 131 или 132, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 31, 131 или 132; или
[0047] (2) ECD CD80, содержащего последовательность, показанную в SEQ ID NO: 32 или 133, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 32 или 133, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 32 или 133; или
[0048] (3) Комбинации второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2, содержащей последовательность, показанную в SEQ ID NO: 33, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 33, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 33.
[0049] Кроме того, С-конец или/и N-конец второго связывающего домена имеет пептидный линкер, и этот пептидный линкер состоит из 2-30 аминокислот.
[0050] Кроме того, пептидный линкер может представлять собой:
(1) (GGGGS)n; или
(2) AKTTPKLEEGEFSEAR (SEQ ID NO: 80); или
(3) AKTTPKLEEGEFSEARV (SEQ ID NO: 81); или
(4) AKTTPKLGG (SEQ ID NO: 82); или
(5) SAKTTPKLGG (SEQ ID NO: 83); или
(6) SAKTTP (SEQ ID NO: 84); или
(7) RADAAP (SEQ ID NO: 85); или
(8) RADAAPTVS (SEQ ID NO: 86); или
(9) RADAAAAGGPGS (SEQ ID NO: 87); или
(10) RADAAAA (SEQ ID NO: 88); или
(11) SAKTTPKLEEGEFSEARV (SEQ ID NO: 89); или
(12) ADAAP (SEQ ID NO: 90); или
(13) DAAPTVSIFPP (SEQ ID NO: 91); или
(14) TVAAP (SEQ ID NO: 92); или
(15) TVAAPSVFIFPP (SEQ ID NO: 93); или
(16) QPKAAP (SEQ ID NO: 94); или
(17) QPKAAPSVTLFPP (SEQ ID NO: 95); или
(18) AKTTPP (SEQ ID NO: 96); или
(19) AKTTPPSVTPLAP (SEQ ID NO: 97); или
(20) AKTTAP (SEQ ID NO: 98); или
(21) AKTTAPSVYPLAP (SEQ ID NO: 99); или
(22) ASTKGP (SEQ ID NO: 100); или
(23) ASTKGPSVFPLAP (SEQ ID NO: 101); или
(24) GENKVEYAPALMALS (SEQ ID NO: 102); или
(25) GPAKELTPLKEAKVS (SEQ ID NO: 103); или
(26) GHEAAAVMQVQYPAS (SEQ ID NO: 104); или
(27) GGGGSGGGGSGGGGSA (SEQ ID NO: 105),
где n равно 1, 2, 3 или 4;
когда n=1, (GGGGS)n представляет собой GGGGS (SEQ ID NO: 120);
когда n=2, (GGGGS)n представляет собой GGGGSGGGGS (SEQ ID NO: 121) или (GGGGS)2;
когда n=3, (GGGGS)n представляет собой GGGGSGGGGSGGGGS (SEQ ID NO: 122) или (GGGGS)3;
когда n=4, (GGGGS)n представляет собой GGGGSGGGGSGGGGSGGGGS (SEQ ID NO: 123) или (GGGGS)4.
[0051] Кроме того, размер вставленного второго связывающего домена не превышает 235, 240, 250, 260, 270, 280, 290 или 300 аминокислот.
[0052] Кроме того, сайт вставки второго связывающего домена расположен в средней передней части шарнирной области, и сайт вставки не влияет на образование дисульфидных связей иммуноглобулина, при этом средняя передняя часть шарнирной области в основном относится к части до 231А. Кроме того, часть аминокислот в шарнирной области до и после сайта вставки заменяется или удаляется. Например, шарнирная область содержит мутации D221G и/или C220V.
[0053] Кроме того, IgG выбран из IgG млекопитающих, гуманизированного IgG и человеческого IgG, и млекопитающее включает в себя мышь и крысу; предпочтительно, IgG представляет собой IgG1, IgG2, IgG3 или IgG4.
[0054] Кроме того, Fc-область слитого белка агликозилирована или дегликозилирована, или имеет пониженное фукозилирование, или афукозилирована.
[0055] Кроме того, IgG представляет собой атезолизумаб, авелумаб, дурвалумаб, ниволумаб, пембролизумаб, цемиплимаб или ипилимумаб. Предпочтительно, IgG представляет собой атезолизумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 106, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 107; или IgG представляет собой авелумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 108, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 109; или IgG представляет собой дурвалумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 110, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 111; или IgG представляет собой ниволумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 112, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 113; или IgG представляет собой пембролизумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 114, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 115; или IgG представляет собой ипилимумаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 116, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 117; или IgG представляет собой цемиплимаб, аминокислотная последовательность тяжелой цепи IgG показана в SEQ ID NO: 118, а аминокислотная последовательность легкой цепи IgG показана в SEQ ID NO: 119.
[0056] В частности, настоящее изобретение относится к слитому белку структуры Hibody с первым связывающим доменом, нацеленным на человеческий PD-L1, CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG имеют ту же последовательность CDR, что и антитело, определяемое следующей последовательностью, или имеющее 1-2 аминокислотные замены в CDR антитела, определяемого следующей последовательностью, антитело определяется следующим образом:
[0057] (1) Аминокислотная последовательность вариабельной области тяжелой цепи представлена в SEQ ID NO: 66; и/или
[0058] (2) Аминокислотная последовательность вариабельной области легкой цепи представлена в SEQ ID NO: 67.
[0059] Кроме того, CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG являются следующими:
[0060] (1) Для вариабельной области тяжелой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 1-5 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 1-5; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 6-10 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 6-10; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 11-15 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 11-15; и/или
[0061] (2) Для вариабельной области легкой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 16-20 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 16-20; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 21-25 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 21-25; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 26-30 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 26-30.
[0062] В конкретном варианте осуществления изобретения, в соответствии с используемыми различными методами измерения или системами идентификации, определяющие комплементарность области CDR 1-3 соответствующих вариабельных областей тяжелой цепи и легкой цепи показаны в Таблице 2.
Таблица 2. Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи и легкой цепи анти-PD-L1 антитела
GFSLSRY
WGVGT
NWGTADYFDY
GFSLSRYSVH
MIWGVGTTD
NWGTADYFDY
RYSVH
MIWGVGTTDYNSALKS
NWGTADYFDY
SRYSVH
WLGMIWGVGTTD
ARNWGTADYFD
GFSLSRYS
IWGVGTT
ARNWGTADYFDY
RASKSVHTSGYSYMH
LASNLES
QHSGELPYT
RASKSVHTSGYSYMH
LASNLES
QHSGELPYT
RASKSVHTSGYSYMH
LASNLES
QHSGELPYT
HTSGYSYMHWY
LLIYLASNLE
QHSGELPY
KSVHTSGYSY
LAS
QHSGELPYT
[0063] Кроме того, слитый белок содержит следующие последовательности CDR,
[0064] (1) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 1, 6, 11 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 1, 6, 11; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 16, 21, 26 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 16, 21, 26; или
[0065] (2) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 2, 7, 12 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 2, 7, 12; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 17, 22, 27 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 17, 22, 27; или
[0066] (3) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 3, 8, 13 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 3, 8, 13; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 18, 23, 28 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 18, 23, 28; или
[0067] (4) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 4, 9, 14 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 4, 9, 14; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 19, 24, 29 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 19, 24, 29; или
[0068] (5) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 5, 10, 15 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 5, 10, 15; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 20, 25, 30 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 20, 25, 30.
[0069] Кроме того, в тяжелой цепи слитого белка аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 3, 8, 13; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 18, 23, 28.
[0070] Кроме того, последовательности вариабельной области тяжелой цепи и вариабельной области легкой цепи слитого белка являются следующими:
[0071] (1) Вариабельная область тяжелой цепи имеет последовательность, показанную в SEQ ID NO: 66, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 66, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 66; и/или
[0072] (2) Вариабельная область легкой цепи имеет последовательность, показанную в SEQ ID NO: 67, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 67, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 67.
[0073] Более конкретно, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка являются следующими:
[0074] (1) Тяжелая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 72, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%. 97%, 98%, 99% по сравнению с SEQ ID NO: 72;
[0075] (2) Легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 73, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 73.
[0076] Кроме того, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка являются следующими:
[0077] (1) Аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 72;
[0078] (2) Аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 73.
[0079] В частности, настоящее изобретение относится к слитому белку структуры Hibody с первым связывающим доменом, нацеленным на человеческий PD-1, где CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab имеют ту же последовательность CDR, что и антитело, определяемое следующей последовательностью, или имеющее 1-2 аминокислотные замены в CDR антитела, определяемого следующей последовательностью, антитело определяется следующим образом:
[0080] (1) Аминокислотная последовательность вариабельной области тяжелой цепи представлена в SEQ ID NO: 64; и/или
[0081] (2) Аминокислотная последовательность вариабельной области легкой цепи представлена в SEQ ID NO: 65.
[0082] Кроме того, CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab в слитом белке являются следующими:
[0083] (1) Для вариабельной области тяжелой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 34-38 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 34-38; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 39-43 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 39-43; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 44-48 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 44-48; и/или
[0084] (2) Для вариабельной области легкой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 49-53 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 49-53; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 54-58 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 54-58; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 59-63 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 59-63.
[0085] В конкретном варианте осуществления изобретения, в соответствии с используемыми различными методами измерения или системами идентификации, определяющие комплементарность области CDR 1-3 соответствующих вариабельных областей тяжелой цепи и легкой цепи показаны в Таблице 3.
Таблица 3. Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи и легкой цепи анти-PD-1 антитела
GITFSNS
WYDGSK
NDDY
GITFSNSGMH
VIWYDGSKRY
NDDY
NSGMH
VIWYDGSKRYYADSVKG
NDDY
SNSGMH
WVAVIWYDGSKRY
ATNDD
GITFSNSG
IWYDGSKR
ATNDDY
RASQSVSSYLA
DASNRAT
QQSSNWPRT
RASQSVSSYLA
DASNRAT
QQSSNWPRT
RASQSVSSYLA
DASNRAT
QQSSNWPRT
SSYLAWY
LLIYDASNRA
QQSSNWPR
QSVSSY
DAS
QQSSNWPRT
[0086] Кроме того, CDR 1-3 вариабельной области слитого белка определены следующим образом:
[0087] (1) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 34, 39, 44 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 34, 39, 44; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 49, 54, 59 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 49, 54, 59; или
[0088] (2) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 35, 40, 45 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 35, 40, 45; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 50, 55, 60 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 50, 55, 60; или
[0089] (3) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 36, 41, 46 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 36, 41, 46; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 51, 56, 61 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 51, 56, 61; или
[0090] (4) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 37, 42, 47 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 37, 42, 47; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 52, 57, 62 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 52, 57, 62; или
[0091] (5) Аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 38, 43, 48 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 38, 43, 48; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 53, 58, 63 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 53, 58, 63.
[0092] Кроме того, CDR 1-3 вариабельной области слитого белка определены следующим образом: аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 36, 41, 46; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 51, 56, 61.
[0093] Кроме того, аминокислотные последовательности вариабельной области тяжелой цепи и вариабельной области легкой цепи слитого белка являются следующими:
[0094] (1) Вариабельная область тяжелой цепи имеет последовательность, показанную в SEQ ID NO: 64, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 64, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 64; и/или
[0095] (2) Вариабельная область легкой цепи имеет последовательность, показанную в SEQ ID NO: 65, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 65, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 65.
[0096] Более конкретно, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка определены следующим образом:
[0097] (1) Тяжелая цепь гибридного белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 68, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 68;
[0098] (2) Легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 69, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 69.
[0099] Кроме того, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка определены следующим образом:
[00100] (1) Аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 68;
[00101] (2) Аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 69.
[00102] Более конкретно, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка определены следующим образом:
[00103] (1) Тяжелая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 70, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 70;
[00104] (2) Легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 71, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 71.
[00105] Кроме того, аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка определены следующим образом:
[00106] (1) Аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 70;
[00107] (2) Аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 71.
[00108] Настоящее изобретение также относится к выделенному полинуклеотиду, кодирующему вышеуказанный слитый белок.
[00109] Настоящее изобретение также относится к конструкции нуклеиновой кислоты, содержащей указанный выше полинуклеотид, предпочтительно конструкция нуклеиновой кислоты представляет собой вектор.
[00110] Настоящее изобретение также относится к клетке-хозяину, содержащей указанный выше полинуклеотид или указанную выше конструкцию нуклеиновой кислоты или вектор, предпочтительно клетка является прокариотической клеткой, эукариотической клеткой, дрожжевой клеткой, клеткой млекопитающего, клеткой E. coli или клеткой СНО, клеткой NS0, клеткой Sp2/0 или клеткой BHK.
[00111] Настоящее изобретение также относится к фармацевтической композиции, содержащей указанный выше слитый белок и фармацевтически приемлемый носитель.
[00112] Настоящее изобретение также относится к способу лечения опухоли или рака, включающему в себя этап введения указанного выше слитого белка или фармацевтической композиции субъекту, нуждающемуся в лечении или облегчении.
[00113] Настоящее изобретение также относится к применению указанного выше слитого белка, полинуклеотида, конструкции нуклеиновой кислоты или вектора, или фармацевтической композиции в получении лекарственного средства для лечения или предотвращения опухоли или рака. Опухоль или рак включает в себя солидную опухоль или несолидную опухоль.
[00114] Настоящее изобретение также относится к диагностическому набору, содержащему указанный выше слитый белок.
[00115] Настоящее изобретение также относится к способу получения слитого белка, включающему в себя культивирование клетки-хозяина в условиях, позволяющих экспрессию вышеуказанной конструкции нуклеиновой кислоты, и выделение продуцированного слитого белка из культуры.
[00116] Слитый белок со структурой Hibody, раскрытый в настоящем изобретении, имеет те же преимущества экспрессии и продукции, что и IgG, поскольку второй связывающий домен вставлен в шарнирную область и может быть легко применим к существующей платформе экспрессии IgG и платформе очистки, что значительно снижает последующую стоимость разработки; по сравнению с биспецифичным антителом, созданным с помощью существующей платформы биспецифичных антител, биспецифичное антитело, созданное с помощью платформы Hibody, имеет более высокий уровень экспрессии и не имеет проблемы несоответствия легкой и тяжелой цепей, что также значительно снижает стоимость получения антител. Кроме того, неожиданно было обнаружено, что вставка второго связывающего домена определенного размера в две шарнирные области не только не влияет на связывающую активность Fab-области слитого белка и стабильность белка, но также дополнительно улучшает стабильность и обеспечивает более длительное время полужизни. В частности, активность связывания второго связывающего домена с целевым сайтом связывания значительно улучшена по сравнению с активностью соответствующего растворимого мономера природного связывающего фрагмента с соответствующей мишенью, что может объясняться тем, что поскольку рецептор или лиганд выбранного второго связывающего домена формирует структуру димеризации или мультимеризации в естественном сигнальном пути для активации или ингибирования сигнального пути, то когда второй связывающий домен вставлен в шарнирную область, его пространственное позиционное соотношение в шарнирной области способствует образованию димеризации соответствующего рецептора или лиганда или их фрагмента, так что путем имитации естественного сигнального пути генерируется значительно улучшенная связывающая активность, что позволяет слитому белку иметь значительно повышенную способность к активации или ингибированию по сравнению с растворимым рецептором, лигандом или их фрагментом. Кроме того, с помощью разумной комбинации двух связывающих доменов мишень ингибирования рецептора или активации рецептора может быть гибко выбрана для достижения эффективного синергетического эффекта. Биспецифичный слитый белок настоящего изобретения, обладает значительно большим лечебным действием для эффективного лечения или контроля процесса заболевания, особенно при различных опухолях и видах рака, по сравнению с раздельным введением двух целевых лекарственных средств или биспецифичного антитела предшествующего уровня техники.
[00117]
Краткое описание чертежей
[00118] На Фигуре 1 показано схематическое изображение слитого белка структуры Hibody.
[00119] На Фигуре 2 показана кривая ингибирования Hib-PDV и его контрольной группы у мышиной модели раковой опухоли толстой кишки.
[00120] На Фигуре 3 показана кривая ингибирования Hib-PDC и его контрольной группы в отношении раковой опухоли толстой кишки мыши.
[00121] На Фигуре 4 показана сравнительная диаграмма Hib-PLT и его контрольной группы по стимулированию клеточной секреции фактора IFN-γ при применении в дозе 10-100 нМ.
Подробное описание
Определения
[00122] Если не указано иное, то все используемые здесь технические и научные термины имеют то же значение, которое обычно понимается специалистами в данной области техники, имеющей отношение к настоящему изобретению. Специалисты в данной области техники могут обратиться к следующим источникам: Dictionary of Cell and Molecular Biology, third edition, 1999, Academic Press; the Concise Dictionary of Biomedicine and Molecular Biology, Juo, Pei-Show, второе издание, 2002, CRC Press; и the Oxford Dictionary of Biochemistry and Molecular Biology, переработанное издание, 2000 , Oxford University Press.
[00123] Аминокислоты, включенные в настоящее изобретение, могут быть представлены их общеизвестными трехбуквенными символами или однобуквенными символами, рекомендованными Комиссией по биохимической номенклатуре IUPAC-IUB. Точно так же нуклеотиды могут быть представлены их общепризнанными однобуквенными кодами.
[00124] В настоящем изобретении способ определения или нумерации определяющий комплементарность области (CDR) вариабельного домена антитела включает в себя IMGT, способ Кэбота (такой как указано в Kabat et al., Sequences of Proteins of Immunological Interest, 5th edition, Public Health Service, National Institutes of Health, Bethesda MD. (1991)), способ Чотиа, способ AbM и контактный способ.
[00125] Для целей настоящего изобретения термины «идентичность», «совпадение» или «сходство» между двумя нуклеотидными или аминокислотными последовательностями относится к проценту одинаковых нуклеотидов или аминокислотных остатков у двух сравниваемых последовательностей после оптимального выравнивания. Этот процент является чисто статистическим, и различия между двумя последовательностями распределены случайным образом и охватывают всю их длину. Сравнение между двумя нуклеотидными или аминокислотными последовательностями обычно выполняется путем сравнения этих последовательностей после их оптимального выравнивания, и может выполняться по сегменту или по «окну сравнения». В дополнение к «ручному» выравниванию, оптимальное выравнивание для сравнения последовательностей также может быть выполнено с помощью алгоритма локальной гомологии Смита и Уотермана (1981) [Ad. App. Math. 2: 482] с помощью алгоритма локальной гомологии Неддлмана и Вунша (1970) [J. MoI. Biol. 48: 443] с помощью метода поиска сходства Пирсона и Липмана (1988) [Proc. Natl. Acad. Sci. USA 85: 2444), а также с помощью компьютерного программного обеспечения с использованием этих алгоритмов (GAP, BESTFIT, FASTA и TFASTA в пакете программного обеспечения Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Dr., Madison, WI, или программы для сравнения BLAST N или BLAST P).
[00126] В широком смысле термин «иммуноглобулин», включенный в настоящее изобретение, относится к белку животного происхождения с активностью антител, состоящему из двух идентичных легких цепей и двух идентичных тяжелых цепей. Это белок, относящийся к важному классу иммунных эффекторных молекул и продуцируемый лимфоцитами иммунной системы высших животных, и он может быть преобразован в антитела под воздействием антигенов. Из-за разной структуры их можно разделить на 5 типов: IgG, IgA, IgM, IgD и IgE. Предпочтительно иммуноглобулин, включенный в настоящее изобретение, представляет собой IgG.
[00127] В широком смысле рецептор, включенный в настоящее изобретение, представляет собой тип особого белка, который существует в клеточной мембране или в клетке и может связываться со специфичной сигнальной молекулой вне клетки, чтобы активировать серию биохимических реакций в клетке, чтобы заставить клетку производить соответствующий эффект при действии внешних раздражителей. Биологически активные вещества, которые связываются с рецепторами, вместе называются лигандами. Связывание рецептора и лиганда приводит к изменению конформации молекулы, которое вызывает клеточные ответы, такие как опосредование межклеточной передачи сигнала, межклеточная адгезия, эндоцитоз и тому подобное.
[00128] Биспецифичный слитый белок настоящего изобретения основан на структуре иммуноглобулина со вторым связывающим доменом, вставленным в его шарнирную область. Таким образом, биспецифичный слитый белок настоящего изобретения имеет два связывающих домена.
Примеры
[00129] Настоящее изобретение будет дополнительно проиллюстрировано посредством следующих примеров. Следует отметить, что следующие примеры предназначены для дальнейшей разработки и объяснения настоящего изобретения, и их не следует рассматривать как ограничение настоящего изобретения.
[00130] Пример 1. Конструирование белков
[00131] В этом примере следующие белковые молекулы были сконструированы с использованием обычных для данной области техники методов, и их временную или стабильную экспрессию осуществляли при помощи обычных для данной области техники методов:
(1) Три биспецифиченых слитых белка Hibody: Hib-PLT, Hib-PDC и Hib-PDV;
(2) Контрольные белки 1-3: сконструированы в соответствии с бифункциональной молекулярной структурой, описанной в Международной патентной заявке WO2015/118175;
(3) Другие контрольные белки: TGF-β-R-His, VEGF-R-His, CD80-His, контрольные антитела к PD-L1.
Таблица 4. Целевые комбинации слитого белка
Нуклеотидные последовательности и аминокислотные последовательности тяжелой цепи и легкой цепи слитого белка Hib-PLT являются следующими:
[00132] Нуклеотидная последовательность тяжелой цепи слитого белка Hib-PLT (SEQ ID NO: 78):
CAGGTGCAGCTGCAGGAGTCCGGACCAGGACTGGTGAAGCCATCCGAGACCCTGAGCCTGACCTGTACAGTGTCCGGCTTCAGCCTGTCTAGGTACAGCGTGCACTGGATCAGACAGCCACCTGGCAAGGGACTGGAGTGGCTGGGCATGATCTGGGGCGTGGGCACCACAGACTACAACTCTGCTCTGAAGTCCAGACTGACCATCAGCAAGGATACATCTAAGAATCAGTTCAGCCTGAAGCTGTCCAGCGTGACCGCCGCTGACACAGCCGTGTACTATTGCGCTCGCAACTGGGGCACCGCCGACTACTTCGACTATTGGGGCCAGGGCACCACAGTGACAGTGTCTTCCGCTAGCACCAAGGGCCCATCGGTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAAAGTTGAGCCCAAATCTTGTGACAAAGGCGGGGGAGGCAGCGGCGGCGGAGGAAGCGGGGGCGGAGGTAGCACCATCCCTCCACACGTGCAGAAGAGCGTGAACAATGACATGATCGTCACCGACAACAACGGCGCCGTCAAGTTCCCCCAGCTGTGCAAATTCTGCGACGTGAGGTTCTCCACGTGCGACAACCAGAAGAGCTGTATGAGCAACTGCAGCATCACATCCATCTGCGAAAAACCCCAGGAAGTGTGCGTCGCCGTCTGGCGGAAGAACGACGAGAACATCACACTGGAGACCGTGTGCCACGACCCCAAACTGCCCTACCACGACTTCATCCTGGAGGACGCCGCCAGCCCAAAGTGCATCATGAAAGAGAAGAAGAAGCCGGGCGAGACTTTCTTCATGTGCTCCTGCAGCTCCGACGAGTGCAACGATAATATCATCTTCAGCGAAGAATACAACACATCTAACCCAGACGGAGGGGGCGGATCCGGGGGCGGCGGAAGCGGCGGGGGGGGCAGCACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTAAATGA
[00133] Примечание: Подчеркнутая и выделенная жирным шрифтом последовательность представляет собой нуклеотидную последовательность TGF-βRII.
[00134] Аминокислотная последовательность тяжелой цепи слитого белка Hib-PLT (SEQ ID NO: 72):

[00135] Примечание: Выделенный жирным шрифтом фрагмент в рамке представляет собой последовательность шарнирной области, пептидный линкер выделен курсивом, а подчеркнутая жирным шрифтом последовательность представляет собой аминокислотную последовательность TGF-βRII.
[00136] Нуклеотидная последовательность легкой цепи слитого белка Hib-PLT (SEQ ID NO: 79):
GATATCGTGCTGACCCAGTCTCCAGCTTCCCTGGCCGTGTCCCCAGGACAGAGGGCCACCATCACATGTCGGGCTTCCAAGAGCGTGCACACAAGCGGCTACTCTTATATGCATTGGTACCAGCAGAAGCCCGGCCAGCCCCCTAAGCTGCTGATCTATCTGGCTTCCAACCTGGAGAGCGGAGTGCCAGCTAGGTTCTCTGGCTCCGGCAGCGGCACCGACTTTACCCTGACAATCAATCCTGTGGAGGCCAACGATACAGCTAATTACTATTGCCAGCACTCCGGAGAGCTGCCATACACCTTCGGCGGAGGCACAAAGGTGGAGATCAAGCGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGTTAG
[00137] Аминокислотная последовательность легкой цепи слитого белка Hib-PLT (SEQ ID NO: 73):

Нуклеотидные последовательности и аминокислотные последовательности тяжелой и легкой цепей слитого белка Hib-PDC являются следующими:
[00138] Нуклеотидная последовательность тяжелой цепи слитого белка Hib-PDC (SEQ ID NO: 74):
CAGGTGCAGCTCGTGGAGAGCGGGGGAGGCGTCGTCCAGCCTGGCCGTAGCCTGCGGCTGGACTGTAAGGCTAGCGGAATCACGTTCAGCAACAGCGGAATGCACTGGGTCAGACAGGCTCCTGGCAAAGGCCTCGAATGGGTCGCCGTGATCTGGTACGACGGGAGCAAGAGATACTACGCAGATTCAGTGAAGGGCCGTTTCACAATCAGCCGTGACAACTCGAAGAACACACTGTTCCTGCAGATGAACAGCCTGAGGGCAGAAGACACAGCAGTCTACTACTGCGCTACAAATGACGACTACTGGGGCCAGGGAACACTGGTCACCGTGAGCTCAGCTTCCACCAAGGGCCCATCCGTCTTCCCCCTGGCGCCCTGCTCCAGGAGCACCTCCGAGAGCACAGCCGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACGAAGACCTACACCTGCAACGTAGATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGTCCGGTGGCGGCGGAAGCGGCGGCGGAGGCAGCGGCGGAGGCGGCAGCGTGATCCATGTCACGAAGGAGGTCAAAGAAGTGGCCACCCTCAGCTGCGGTCACAACGTCAGCGTGGAAGAGTTGGCCCAGACCAGAATCTACTGGCAGAAGGAGAAGAAGATGGTCTTGACCATGATGAGCGGCGACATGAACATCTGGCCAGAGTACAAGAATAGAACTATCTTCGACATCACCAATAACCTGAGCATCGTGATCCTCGCTTTGCGTCCGAGCGACGAAGGCACCTACGAGTGCGTAGTGCTAAAGTACGAGAAAGACGCCTTCAAGCGGGAGCACCTGGCAGAAGTAACCCTGAGCGTGAAGGCAGACTTCCCAACGCCTAGCATCTCCGACTTCGAGATCCCCACAAGCAACATCCGGCGGATCATCTGTAGCACCTCCGGGGGTTTCCCCGAGCCTCACCTGTCCTGGCTCGAGAACGGCGAGGAGCTGAACGCCATCAACACCACCGTGAGCCAGGACCCCGAGACAGAACTGTACGCCGTGAGCTCCAAGCTCGACTTCAACATGACCACAAATCACAGCTTCATGTGCCTCATCAAGTACGGACACCTGCGGGTGAATCAGACGTTCAACTGGAACGGCGGGGGGGGTAGCGGCGGCGGGGGCTCAGGTGGGGGGGGCTCAAAATATGGTCCCCCATGCCCACCATGCCCAGCACCTGAGTTCCTGGGGGGACCATCAGTCTTCCTGTTCCCCCCAAAACCCAAGGACACTCTCATGATCTCCCGGACCCCTGAGGTCACGTGCGTGGTGGTGGACGTGAGCCAGGAAGACCCCGAGGTCCAGTTCAACTGGTACGTGGATGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTTCAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAACGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGGCCTCCCGTCCTCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAGCCACAGGTGTACACCCTGCCCCCATCCCAGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAGGCTAACCGTGGACAAGAGCAGGTGGCAGGAGGGGAATGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACACAGAAGAGCCTCTCCCTGTCTCTGGGTAAATAG
[00139] Примечание: Подчеркнутая и выделенная жирным шрифтом последовательность представляет собой нуклеотидную последовательность CD80.
[00140] Аминокислотная последовательность тяжелой цепи слитого белка Hib-PDC (SEQ ID NO: 68):

[00141] Примечание: Пептидный линкер выделен курсивом, а подчеркнутая и выделенная жирным шрифтом последовательность представляет собой аминокислотную последовательность CD80.
[00142] Нуклеотидная последовательность легкой цепи слитого белка Hib-PDC (SEQ ID NO: 75):
GAGATCGTGCTGACACAGAGCCCAGCCACTCTGTCACTGTCCCCAGGAGAAAGGGCTACTCTGTCTTGCCGGGCAAGCCAGTCTGTCTCCAGCTACCTGGCCTGGTATCAGCAGAAGCCCGGACAGGCTCCTAGACTGCTGATCTACGACGCAAGTAACAGAGCCACCGGCATCCCCGCACGCTTCAGTGGCTCAGGCTCCGGAACAGACTTTACTCTGACCATCTCTAGTCTGGAGCCTGAAGATTTCGCCGTGTACTATTGTCAGCAGAGCTCTAATTGGCCTAGAACCTTCGGCCAGGGCACCAAAGTCGAGATCAAGCGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGTTAG
[00143] Аминокислотная последовательность легкой цепи слитого белка Hib-PDC (SEQ ID NO: 69):

Нуклеотидные последовательности и аминокислотные последовательности тяжелой и легкой цепей слитого белка Hib-PDV являются следующими:
[00144] Нуклеотидная последовательность тяжелой цепи слитого белка Hib-PDV (SEQ ID NO: 76):
CAGGTGCAGCTCGTGGAGAGCGGGGGAGGCGTCGTCCAGCCTGGCCGTAGCCTGCGGCTGGACTGTAAGGCTAGCGGAATCACGTTCAGCAACAGCGGAATGCACTGGGTCAGACAGGCTCCTGGCAAAGGCCTCGAATGGGTCGCCGTGATCTGGTACGACGGGAGCAAGAGATACTACGCAGATTCAGTGAAGGGCCGTTTCACAATCAGCCGTGACAACTCGAAGAACACACTGTTCCTGCAGATGAACAGCCTGAGGGCAGAAGACACAGCAGTCTACTACTGCGCTACAAATGACGACTACTGGGGCCAGGGAACACTGGTCACCGTGAGCTCAGCTTCCACCAAGGGCCCATCCGTCTTCCCCCTGGCGCCCTGCTCCAGGAGCACCTCCGAGAGCACAGCCGCCCTGGGCTGCCTGGTCAAGGACTACTTCCCCGAACCGGTGACGGTGTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCCGGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGCACGAAGACCTACACCTGCAACGTAGATCACAAGCCCAGCAACACCAAGGTGGACAAGAGAGTTGAGTCCGGTGGCGGCGGAAGCGGCGGCGGAGGCAGCGGCGGAGGCGGCAGCGGTAGACCATTCGTAGAGATGTACAGCGAAATCCCCGAAATCATCCACATGACTGAAGGAAGGGAGCTCGTCATCCCCTGCCGGGTTACGTCACCTAACATCACTGTTACTCTGAAGAAGTTCCCACTCGACACTTTGATCCCTGATGGAAAACGCATCATCTGGGACAGCAGAAAGGGCTTCATCATCTCAAATGCAACGTACAAAGAAATCGGACTCCTGACCTGTGAAGCAACAGTCAATGGACATTTGTATAAGACAAACTATCTCACACATCGACAGACCAATACAATCATCGATGTGGTTCTGAGCCCGTCTCATGGAATCGAACTATCTGTTGGAGAAAAGCTCGTCCTGAATTGTACAGCAAGAACTGAACTGAATGTGGGAATCGACTTCAACTGGGAATACCCTTCTTCGAAGCATCAGCATAAGAAACTCGTAAACCGAGACCTAAAGACCCAGTCTGGGAGCGAGATGAAGAAATTCTTGAGCACCCTGACTATCGATGGTGTAACCCGGAGCGACCAGGGATTGTACACCTGTGCAGCATCCAGCGGGCTGATGACCAAGAAGAACAGCACATTCGTCAGGGTCCATGAACCCGGCGGGGGGGGTAGCGGCGGCGGGGGCTCAGGTGGGGGGGGCTCAAAATATGGTCCCCCATGCCCACCATGCCCAGCACCTGAGTTCCTGGGGGGACCATCAGTCTTCCTGTTCCCCCCAAAACCCAAGGACACTCTCATGATCTCCCGGACCCCTGAGGTCACGTGCGTGGTGGTGGACGTGAGCCAGGAAGACCCCGAGGTCCAGTTCAACTGGTACGTGGATGGCGTGGAGGTGCATAATGCCAAGACAAAGCCGCGGGAGGAGCAGTTCAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAACGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGGCCTCCCGTCCTCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAGCCACAGGTGTACACCCTGCCCCCATCCCAGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTACCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAGGCTAACCGTGGACAAGAGCAGGTGGCAGGAGGGGAATGTCTTCTCATGCTCCGTGATGCATGAGGCTCTGCACAACCACTACACACAGAAGAGCCTCTCCCTGTCTCTGGGTAAATAG
[00145] Примечание: Подчеркнутая и выделенная жирным шрифтом последовательность представляет собой нуклеотидную последовательность VEGFR.
[00146] Аминокислотная последовательность тяжелой цепи слитого белка Hib-PDV (SEQ ID NO: 70):


[00147] Примечание: Пептидный линкер выделен курсивом, а подчеркнутая и выделенная жирным шрифтом последовательность представляет собой аминокислотную последовательность VEGF.
[00148] Нуклеотидная последовательность легкой цепи слитого белка Hib-PDV (SEQ ID NO: 77):
GAGATCGTGCTGACACAGAGCCCAGCCACTCTGTCACTGTCCCCAGGAGAAAGGGCTACTCTGTCTTGCCGGGCAAGCCAGTCTGTCTCCAGCTACCTGGCCTGGTATCAGCAGAAGCCCGGACAGGCTCCTAGACTGCTGATCTACGACGCAAGTAACAGAGCCACCGGCATCCCCGCACGCTTCAGTGGCTCAGGCTCCGGAACAGACTTTACTCTGACCATCTCTAGTCTGGAGCCTGAAGATTTCGCCGTGTACTATTGTCAGCAGAGCTCTAATTGGCCTAGAACCTTCGGCCAGGGCACCAAAGTCGAGATCAAGCGTACGGTGGCTGCACCATCTGTCTTCATCTTCCCGCCATCTGATGAGCAGTTGAAATCTGGAACTGCCTCTGTTGTGTGCCTGCTGAATAACTTCTATCCCAGAGAGGCCAAAGTACAGTGGAAGGTGGATAACGCCCTCCAATCGGGTAACTCCCAGGAGAGTGTCACAGAGCAGGACAGCAAGGACAGCACCTACAGCCTCAGCAGCACCCTGACGCTGAGCAAAGCAGACTACGAGAAACACAAAGTCTACGCCTGCGAAGTCACCCATCAGGGCCTGAGCTCGCCCGTCACAAAGAGCTTCAACAGGGGAGAGTGTTAG
[00149] Аминокислотная последовательность легкой цепи слитого белка Hib-PDV (SEQ ID NO: 71):

Аминокислотные последовательности тяжелой и легкой цепи Контрольного белка 1
[00150] Аминокислотная последовательность тяжелой цепи Контрольного белка 1 (SEQ ID NO: 124):

[00151] Примечание: Пептидный линкер выделен курсивом, а подчеркнутая и выделенная жирным шрифтом последовательность представляет собой аминокислотную последовательность TGF-βRII.
[00152] Аминокислотная последовательность легкой цепи Контрольного белка 1 (SEQ ID NO: 125):

Аминокислотные последовательности тяжелой и легкой цепи Контрольного белка 2
[00153] Аминокислотная последовательность тяжелой цепи Контрольного белка 2 (SEQ ID NO: 126):


[00154] Примечание: Пептидный линкер выделен курсивом, а подчеркнутая и выделенная жирным шрифтом последовательность представляет собой аминокислотную последовательность CD80.
[00155] Аминокислотная последовательность легкой цепи Контрольного белка 2 (SEQ ID NO: 127):

Аминокислотные последовательности тяжелой и легкой цепи Контрольного белка 3
[00156] Аминокислотная последовательность тяжелой цепи Контрольного белка 3 (SEQ ID NO: 128):

[00157] Примечание: Пептидный линкер выделен курсивом, а подчеркнутая и выделенная жирным шрифтом последовательность представляет собой аминокислотную последовательность VEGFR.
[00158] Аминокислотная последовательность легкой цепи Контрольного белка 3 (SEQ ID NO: 129):

[00159] TGF-β-R-His представляет собой His-меченный фрагмент внеклеточной области TGF-βRII, где аминокислотная последовательность фрагмента внеклеточной области TGF-βRII (SEQ ID NO: 137) является следующей:

Аминокислотная последовательность VEGF-R-His
[00160] VEGF-R-His представляет собой меченный His комбинированный фрагмент второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2, где аминокислотная последовательность комбинированного фрагмента второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2 (SEQ ID NO: 130) является следующей:

Аминокислотная последовательность CD80-His
[00161] CD80-His представляет собой His-меченный фрагмент CD80, где аминокислотная последовательность фрагмента CD80 (SEQ ID NO: 32) является следующей:

[00162] Пример 2. Экспрессия в клетках CHO
[00163] A. Размораживание и размножение клеток:
[00164] Клетки-хозяева СНО размораживали в объеме культивирования 10 мл и условиями культивирования при температуре 37,0°C, 200 об/мин, 8% CO2 и 80% влажности. После оттаивания плотность клеток составляла 2,82×105 клеток/мл, а жизнеспособность составляла 95,41%.
[00165] После 72 часов размножения и культивирования клеток плотность жизнеспособных клеток составляла 2,22×106 клеток/мл, а жизнеспособность составляла 98,32%.
[00166] Клетки повторно высевали при концентрации 5×105 клеток/мл для операции повторного посева клеток. После 48 часов непрерывного культивирования плотность жизнеспособных клеток составляла 2×106 клеток/мл, а жизнеспособность составляла 98,68%.
[00167] Клетки высевали в 6-луночный планшет из расчета 5×105 клеток на лунку в объеме культивирования 2 мл на лунку и помещали в инкубатор с диоксидом углерода (условия культивирования: 37,0°C, 8% CO2) на ночь для прикрепления и трансфекции на следующий день.
[00168] B. Трансфекция
[00169] В качестве реагента для трансфекции использовали реагент Lipofectamine®LTX (Invitrogen, каталожный № 15338-100), содержащий реагенты LTX и Plus.
[00170] Процедура трансфекции: Замена среды в 6-луночном планшете и приготовление комплекса для трансфекции: LTX: 9 мкл на лунку+плазмида: 2,5 мкг на лунку+плюс: 2,5 мкл на лунку. Комплекс для трансфекции добавляли в 6-луночный планшет в количестве 250 мкл на лунку. Среду в каждой лунке 6-луночного планшета заменяли через 5 часов после трансфекции клеток. После 48 часов трансфекции супернатант отбирали для определения содержания антител в супернатанте методом твердофазного ИФА с двойным антигеном.
[00171] C. Периодическое культивирование и периодическое культивирование с подпиткой
[00172] После 48 часов трансфекции клетки в 6-планшете переваривали, собирали в пробирку для культивирования клеток и адаптировали для роста в суспензии при плотности клеток 3×105 клеток/мл. Когда жизнеспособность клеток возвращалась примерно к 90%, пул клеток, экспрессирующих Hib-PLT, и пул клеток, экспрессирующих Контрольный белок 1, культивировали в периодической культуре, а клетки, экспрессирующие Hib-PDC, Контрольный белок 2, Hib-PDV и Контрольный белок 3 культивировали в периодической культуре с подпиткой.
[00173] Периодическое культивирование: Клетки культивировали в пробирке для культивирования клеток TPP при плотности клеток 3×105 клеток/мл, в объеме объемом 30 мл и условиях культивирования при температуре 37,0°C, 200 об/мин, 8% CO2 и 80% влажности, и жизнеспособность и метаболизм клеток контролировали каждый день. Культивирование прекращали, когда жизнеспособность клеток становилась <60%, и клеточный супернатант после завершения культивирования отбирали для определения концентрации.
[00174] Периодическое культивирование с подпиткой: Клетки культивировали в пробирке для культивирования клеток TPP при плотности клеток 3×105 клеток/мл, в объеме 30 мл и условиях культивирования при температуре 37,0°C, 200 об/мин, 8% CO2, и 80% влажности. Концентрированную среду для клеток подавали в соответствии с определенной стратегией культивирования с подпиткой (см. Таблицу 5), при которой содержание глюкозы поддерживали на уровне 2-6 г/л. Культивирование прекращали, когда жизнеспособность становилась <60%, и клеточный супернатант после завершения культивирования отбирали для определения концентрации.
[00175]
[00176] В Таблице 6 показаны результаты определения уровня экспрессии биспецифичных слитых белков Hibody Hib-PLT, Hib-PDC и Hib-PDV и их соответствующих контрольных белков. Результаты показывают, что относительное значение увеличения уровня экспрессии Hib-PLT увеличилось на 52,85% по сравнению с его соответствующим контрольным белком; относительное значение увеличения уровня экспрессии Hib-PDC увеличилось на 33,43% по сравнению с его соответствующим контрольным белком; и относительное значение увеличения уровня экспрессии Hib-PDV увеличилось на 25,6% по сравнению с его соответствующим контрольным белком, что указывает на то, что уровень экспрессии биспецифичного слитого белка Hibody настоящего изобретения значительно превосходит уровень экспрессии соответствующего контрольного белка, и биспецифичное антитело настоящего изобретения имеет неожиданные технические эффекты.
Таблица 6. Уровень экспрессии биспецифичных антител
Примечание. Относительное значение увеличения=(уровень экспрессии биспецифичного слитого белка, настоящего изобретения - уровень экспрессии соответствующего контрольного белка)/уровень экспрессии соответствующего контрольного белка.
[00177] Пример 3. Определение связывающей активности
[00178] 1. Определение связывающей активности Hib-PLT:
(1) Покрывающий антиген: TGF-β1 разбавляли до 2 мкг/мл покрывающим буфером, добавляли в 96-луночный планшет в количестве 100 мкл на лунку и оставляли на ночь при температуре 2-8°C;
(2) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(3) Блокирование планшета: Планшет блокировали используемым в качестве блокирующего раствора 3% БСА-ФСБТ в количестве 300 мкл на лунку, и инкубировали при температуре 37°C в течение 2 часов;
(4) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(5) Добавление образцов: Каждая точка концентрации образцов является следующей. Образец с каждой точкой концентрации последовательно добавляли в 96-луночный планшет в количестве 100 мкл на лунку, и каждая точка концентрации была параллельна 2-м повторным лункам, и планшет инкубировали при температуре 37°C в течение 2 часов;
(6) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(7) Добавление вторичного антитела:
Лунка с образцом Hib-PLT: Козье антитело против человеческого Fc-HRP разводили до 1:5000 и добавляли по 100 мкл на лунку. Затем планшет инкубировали при температуре 37°C в течение 2 часов;
Лунка с TGF-β-R-His: анти-His-HRP антитела разводили до 1:5000 и добавляли по 100 мкл на лунку. Затем планшет инкубировали при температуре 37°C в течение 2 часов;
(8) Промывание планшета: Планшет автоматически промывали 5 раз используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(9) Проявление цвета: Планшет подвергали проявлению цвета в течение 2 минут инкубации с используемым в качестве раствора для проявления цвета ТМБ в количестве 100 мкл на лунку;
(10) Остановка проявления цвета: Проявление цвета в планшете останавливали добавлением 2M H2SO4 в качестве стоп-раствора в количестве 50 мкл на лунку;
(11) Считывание: Значения оптической плотности считывали при длине волны 450/655 нм, соответственно, с использованием считывающего устройства для микропланшетов MD. Для построения кривой 4-PL концентрацию исследуемого образца наносили на ось абсцисс, а значение оптической плотности наносили на ось ординаты, и при помощи программного обеспечения автоматически вычисляли значение EC50.
[00179] 2. Определение связывающей активности Hib-PDV:
(1) Покрывающий антиген: VEGF разбавляли до 1 мкг/мл покрывающим буфером, добавляли в 96-луночный планшет в количестве 100 мкл на лунку и оставляли на ночь при температуре 2-8°C;
(2) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(3) Блокирование планшета: Планшет блокировали используемым в качестве блокирующего раствора 3% БСА-ФСБТ в количестве 300 мкл на лунку, и инкубировали при температуре 37°C в течение 2 часов;
(4) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(5) Добавление образцов: Каждая точка концентрации образцов является следующей. Образец с каждой точкой концентрации последовательно добавляли в 96-луночный планшет в количестве 100 мкл на лунку, и каждая точка концентрации была параллельна 2-м повторным лункам, и планшет инкубировали при температуре 37°C в течение 2 часов;
(6) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(7) Добавление вторичного антитела:
Лунка с образцом Hib-PVD: Козье антитело против человеческого Fc-HRP разводили до 1:5000 и добавляли по 100 мкл на лунку. Затем планшет инкубировали при температуре 37°C в течение 2 часов;
Лунка с VEGFR-His: анти-His-HRP антитела разводили до 1:5000 и добавляли по 100 мкл на лунку. Затем планшет инкубировали при температуре 37°C в течение 2 часов;
(8) Промывание планшета: Планшет автоматически промывали 5 раз используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(9) Проявление цвета: Планшет подвергали проявлению цвета в течение 2 минут инкубации с используемым в качестве раствора для проявления цвета ТМБ в количестве 100 мкл на лунку;
(10) Остановка проявления цвета: Проявление цвета в планшете останавливали добавлением 2M H2SO4 в качестве стоп-раствора в количестве 50 мкл на лунку;
(11) Считывание: Значения оптической плотности считывали при длине волны 450/655 нм, соответственно, с использованием считывающего устройства для микропланшетов MD. Для построения кривой 4-PL концентрацию исследуемого образца наносили на ось абсцисс, а значение оптической плотности наносили на ось ординаты, и при помощи программного обеспечения автоматически вычисляли значение EC50.
[00180] 3. Определение связывающей активности Hib-PDC:
(1) Покрывающий антиген: Белок CTLA-4 повторно растворяли в 1 мл стерилизованной воды до концентрации 200 мкг/мл, разбавляли до 20 мкг/мл покрывающим буфером, добавляли в 96-луночный планшет в количестве 100 мкл на лунку и оставляли на ночь при температуре 2-8°C;
(2) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(3) Блокирование планшета: Планшет блокировали используемым в качестве блокирующего раствора 3% БСА-ФСБТ в количестве 300 мкл на лунку, и инкубировали при температуре 37°C в течение 2 часов±20 минут;
(4) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(5) Добавление образцов: Биотинилированный образец разбавляли ФСБТ. Каждая точка концентрации образцов является следующей. Образец с каждой точкой концентрации последовательно добавляли в 96-луночный планшет в количестве 100 мкл на лунку, и каждая точка концентрации была параллельна 2-м повторным лункам, при этом в качестве отрицательного контроля использовали лунку с ФСБТ, и планшет инкубировали при температуре 37°C в течение 2 часов±20 минут;
(6) Промывание планшета: Планшет автоматически промывали 3 раза используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(7) Добавление вторичного антитела:
Лунка с образцом Hib-PDC и лунка с CD80-His: Стрептавидин-HRP разводили до 1:2000 и добавляли по 100 мкл на лунку. Затем планшет инкубировали при температуре 37°C в течение 1 часа±10 минут;
(8) Промывание планшета: Планшет автоматически промывали 5 раз используемым в качестве элюента ФСБТ в количестве 350 мкл на лунку, и стряхивали остатки жидкости, чтобы осушить досуха после промывания;
(9) Проявление цвета: добавляли субстрат ТМБ Double Slow в количестве 100 мкл на лунку, и защищенный от света планшет подвергали проявлению цвета в течение 6 минут при комнатной температуре;
(10) Остановка проявления цвета: Проявление цвета в планшете останавливали добавлением 2M H2SO4 в качестве стоп-раствора в количестве 50 мкл на лунку;
(11) Считывание: Значения оптической плотности считывали при длине волны 450/655 нм, соответственно, с использованием считывающего устройства для микропланшетов MD. Для построения кривой 4-PL концентрацию исследуемого образца наносили на ось абсцисс, а значение оптической плотности наносили на ось ординаты, и при помощи программного обеспечения автоматически вычисляли значение EC50 (См. Таблицу 7).
Таблица 7. Значение ЕС50 для каждой тестовой группы
[00181] Результаты определения связывающей активности показали, что при сравнении между Hib-PLT и мономером рецептора TGF-β Hib-PLT имеет значение EC50 0,164 нМ, мономер рецептора TGF-β имеет значение EC50 11,60 нМ и связывающая активность Hib-PLT примерно в 70 раз лучше, чем у мономера рецептора TGF-β; при сравнении между Hib-PDV и мономером рецептора VEGF Hib-PDV имеет значение EC50 0,061 нМ, мономер рецептора VEGF имеет значение EC50 2154 нМ, а связывающая активность Hib-PDV примерно в 35,311 раз лучше, чем у мономера рецептора VEGF; при сравнении между Hib-PDC и мономером CD80 Hib-PDC имеет значение EC50 24,36 нМ, мономер CD80 имеет значение EC50 50,69 нМ, а связывающая активность Hib-PDC примерно в 2 раза лучше, чем у мономера CD80. Таким образом, связывающие активности Hib-PLT, Hib-PDV и Hib-PDC, сконструированных с использованием модели би-антитела настоящего изобретения, намного лучше, чем у их соответствующих мономеров.
[00182] Пример 4. Эффективность Hib-PDV в отношении подкожно трансплантированной опухоли клеток рака толстой кишки MC38 у мышей линии C57BL/6J-hPD-1
[00183] В эксперименте участвовало 32 7-недельных гуманизированных трансгенных мыши линии PD-1 C57BL/6J (из Shanghai Model Organisms Center, Inc.). В правый бок мышей подкожно инокулировали 0,1 мл суспензии клеток рака толстой кишки MC38 (1×106 клеток на мышь). Когда размер опухоли достигал примерно 100 мм3, мышей случайным образом разделяли на группы в соответствии с размером опухоли, а именно группу, получавшую носитель (ФСБ), группу, получавшую Hib-PDV (1,25 мг/кг), группу, получавшую Hib-PDV (2,5 мг/кг) и группу, получавшую Hib-PDV (5 мг/кг), соответственно, всего 4 группы, по 8 мышей с опухолями в каждой группе. Препараты вводили мышам внутрибрюшинно один раз в неделю, всего 3 введения, в течение периода эксперимента, длинный и короткий диаметр опухоли и массу тела измеряли дважды в неделю. На 21-й день после введения для оценки эффективности лекарственного средства рассчитывали степень ингибирования роста опухоли (TGIRTV), как показано в Таблице 8.
Таблица 8. Схема лечения Hib-PDV при определении его активности ингибирования роста раковой опухоли толстой кишки у мышей
[00184] Формула расчета:
[00185] Объем опухоли: TV=D1×D22/2, где D1 и D2 представляют собой длинный диаметр и короткий диаметр опухоли соответственно;
[00186] Относительный объем опухоли: RTV=TVT/TV1, где TV1 - это объем опухоли до введения, а TVT - это объем опухоли при каждом измерении;
[00187] Относительная степень ингибирования опухоли: TGIRTV(%)=(1-TRTV/CRTV)×100%, где TRTV представляет собой RTV группы, получавшей препарат, или группы положительного контроля, а CRTV представляет собой RTV группы отрицательного контроля.
[00188] Результаты и выводы. Действие Hib-PDV по ингибированию опухоли у мышиной модели рака толстой кишки показано в Таблице 9 и на Фигуре 2, где в Таблице 9 показаны объем опухоли и степень ингибирования роста опухоли Hib-PDV у мышиной модели рака толстой кишки, а Фигура 2 представляет собой график роста опухоли после введения. Экспериментальные результаты показывают, что Hib-PDV оказывает значительное ингибирующее действие на опухоль у мышиной модели рака толстой кишки.
Таблица 9. Действие Hib-PDV на объем опухоли и степень ингибирования опухоли у мышиной модели рака толстой кишки (среднее+СКО)
[00189] Пример 5. Эффективность Hib-PDC в отношении подкожно трансплантированных hPD-L1-трансгенных клеток опухоли MC38 у мышей линии C57BL/6J-hPD-1
[00190] В эксперименте участвовало тридцать две 7-9 недельные гуманизированные PD-1-трансгенные самки гуманизированной линии мышей C57BL/6J (из Shanghai Model Organisms Center, Inc.). 0,1 мл суспензии трансгенных клеток hPD-L1 MC38 (1×106 клеток на мышь) подкожно имплантировали в заднюю часть правой передней конечности мыши. Когда размер опухоли достигал примерно 100 мм3, мышей случайным образом разделили на группы в соответствии с размером опухоли, а именно группу, получавшую носитель (ФСБ), группу, получавшую Hib-PDC (1,25 мг/кг), группу, получавшую Hib-PDC (2,5 мг/кг) и группу, получавшую Hib-PDC (5 мг/кг), соответственно, всего 4 группы, по 8 мышей с опухолями в каждой группе. Препараты вводили мышам внутрибрюшинно один раз в неделю, всего 3 введения, в течение периода эксперимента, длинный и короткий диаметр опухоли и массу тела измеряли дважды в неделю. На 21-й день после введения для оценки эффективности лекарственного средства рассчитывали степень ингибирования роста опухоли (TGIRTV), как показано в Таблице 10.
Таблица 10. Схема лечения Hib-PDC при определении его активности ингибирования роста раковой опухоли толстой кишки у мышей
[00191] Формула расчета:
[00192] Объем опухоли: TV=D1×D22/2, где D1 и D2 представляют собой длинный диаметр и короткий диаметр опухоли соответственно;
[00193] Относительный объем опухоли: RTV=TVT/TV1, где TV1 - это объем опухоли до введения, а TVT - это объем опухоли при каждом измерении;
[00194] Относительная степень ингибирования опухоли: TGIRTV(%)=(1-TRTV/CRTV)×100%, где TRTV представляет собой RTV группы, получавшей препарат, или группы положительного контроля, а CRTV представляет собой RTV группы отрицательного контроля.
[00195] Результаты и выводы. Действие Hib-PDC по ингибированию раковой опухоли толстой кишки у мышей показано в Таблице 11 и на Фигуре 3, где в Таблице 11 показано влияние Hib-PDC на объем опухоли и степень ингибирования опухоли у мышиной модели рака толстой кишки, а Фигура 3 представляет собой график роста опухоли после введения. Экспериментальные результаты показывают, что Hib-PDC оказывает значительное ингибирующее действие на подкожно трансплантированные опухоли колоректального рака у мышей.
Таблица 11. Действие Hib-PDC на объем опухоли и степень ингибирования опухоли у мышиной модели рака толстой кишки (среднее+СКО)
[00196] Пример 6. Влияние Hib-PLT на секрецию цитокинов в смешанной реакции аллогенных лимфоцитов
[00197] Метод твердофазного ИФА использовали для определения концентрации цитокина IFN-γ в клеточном супернатанте для оценки роли Hib-PLT в активации CD4+ Т-клеток. Мононуклеарные лимфоциты от донора 1 индуцировали в зрелые дендритные клетки in vitro с помощью набора для быстрого созревания ДК клеток in vitro (каталожный номер: CT-004, StemEry), клетки собирали через 48 ч и разбавляли до концентрации 2×105 клеток/мл. CD4+ Т-клетки от донора 2 обогащали при помощи магнитных гранул (Набор реагентов для обогащения CD4+ Т-клеток человека EasySep™, StemCell, каталожный номер 19052) и разбавляли до концентрации 1×106 клеток/мл. Зрелые дендритные клетки и CD4+Т-клетки смешивали в объемном соотношении 1:1, и смешанную суспензию клеток добавляли в 96-луночный планшет в объеме 100 мкл на лунку. Hib-PLT, контрольное антитело к PD-L1+ловушка TGF-β (комбинированный препарат), контрольное антитело к PD-L1, ловушку TGF-β и человеческий IgG1 (Biolegend) добавляли в 96-луночный планшет, содержащий суспензию смешанных клеток, с конечной концентрацией 0,01-100 нМ. Затем планшет помещали в инкубатор при температуре 37°C, 5% CO2 на 120 часов, центрифугировали при 2000 об/мин в течение 5 минут и собирали 150-200 мкл клеточного супернатанта. Наборы реагентов для твердофазного ИФА (Набор реагентов для твердофазного ИФА IL-2 человека Quantikine, R&D, каталожный номер: S2050; Набор реагентов для твердофазного ИФА IFN-γ человека Quantikine, R&D, каталожный номер: SIF50C) использовали для обнаружения фактора IFN-γ в супернатанте смешанной реакции аллогенных лимфоцитов. Для анализа различий между группами использовали однофакторный дисперсионный анализ.
[00198] Результаты и выводы: Как показано на Фигуре 4, уровень Hib-PLT в концентрации 10-100 нМ, способствующей секреции клетками фактора IFN-γ, был значимо выше, чем у группы применения комбинированного препарата контрольного антитела PD-L1 и ловушки TGF-β (p<0,05). Результаты показывают, что при уровне концентрации 10-100 нМ Hib-PLT оказывает лучшее действие, чем группа комбинированных препаратов, в стимулировании активации CD4+Т-клеток в реакции смешанных лимфоцитов.
[00199] Настоящее изобретение было проиллюстрировано различными конкретными примерами. Однако специалист в данной области техники может понять, что настоящее изобретение не ограничивается каждым конкретным вариантом осуществления, и специалист с обычной квалификацией может вносить различные изменения или модификации в пределах объема настоящего изобретения, и каждый технический признак, упомянутый в различных местах в настоящем описании может быть объединен с другим таким признаком без отступления от сущности и объема настоящего изобретения. Такие изменения и модификации находятся в пределах объема настоящего изобретения.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> RemeGen Co., Ltd.
<120> БИСПЕЦИФИЧНЫЙ СЛИТЫЙ БЕЛОК И ЕГО ПРИМЕНЕНИЕ
<130> OP0021-AU/CA/RU/US-0084
<160> 137
<170> Патентная версия 3.3
<210> 1
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 1
Gly Phe Ser Leu Ser Arg Tyr
1 5
<210> 2
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 2
Gly Phe Ser Leu Ser Arg Tyr Ser Val His
1 5 10
<210> 3
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 3
Arg Tyr Ser Val His
1 5
<210> 4
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 4
Ser Arg Tyr Ser Val His
1 5
<210> 5
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 5
Gly Phe Ser Leu Ser Arg Tyr Ser
1 5
<210> 6
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 6
Trp Gly Val Gly Thr
1 5
<210> 7
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 7
Met Ile Trp Gly Val Gly Thr Thr Asp
1 5
<210> 8
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 8
Met Ile Trp Gly Val Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys Ser
1 5 10 15
<210> 9
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 9
Trp Leu Gly Met Ile Trp Gly Val Gly Thr Thr Asp
1 5 10
<210> 10
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 10
Ile Trp Gly Val Gly Thr Thr
1 5
<210> 11
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 11
Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr
1 5 10
<210> 12
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 12
Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr
1 5 10
<210> 13
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 13
Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr
1 5 10
<210> 14
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 14
Ala Arg Asn Trp Gly Thr Ala Asp Tyr Phe Asp
1 5 10
<210> 15
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 15
Ala Arg Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr
1 5 10
<210> 16
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 16
Arg Ala Ser Lys Ser Val His Thr Ser Gly Tyr Ser Tyr Met His
1 5 10 15
<210> 17
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 17
Arg Ala Ser Lys Ser Val His Thr Ser Gly Tyr Ser Tyr Met His
1 5 10 15
<210> 18
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 18
Arg Ala Ser Lys Ser Val His Thr Ser Gly Tyr Ser Tyr Met His
1 5 10 15
<210> 19
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 19
His Thr Ser Gly Tyr Ser Tyr Met His Trp Tyr
1 5 10
<210> 20
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 20
Lys Ser Val His Thr Ser Gly Tyr Ser Tyr
1 5 10
<210> 21
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 21
Leu Ala Ser Asn Leu Glu Ser
1 5
<210> 22
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 22
Leu Ala Ser Asn Leu Glu Ser
1 5
<210> 23
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 23
Leu Ala Ser Asn Leu Glu Ser
1 5
<210> 24
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 24
Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu
1 5 10
<210> 25
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 25
Leu Ala Ser
1
<210> 26
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 26
Gln His Ser Gly Glu Leu Pro Tyr Thr
1 5
<210> 27
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 27
Gln His Ser Gly Glu Leu Pro Tyr Thr
1 5
<210> 28
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 28
Gln His Ser Gly Glu Leu Pro Tyr Thr
1 5
<210> 29
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 29
Gln His Ser Gly Glu Leu Pro Tyr
1 5
<210> 30
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 30
Gln His Ser Gly Glu Leu Pro Tyr Thr
1 5
<210> 31
<211> 137
<212> Белок
<213> Искусственная последовательность
<220>
<223> TGF-βRII человека
<400> 31
Thr Ile Pro Pro His Val Gln Lys Ser Val Asn Asn Asp Met Ile Val
1 5 10 15
Thr Asp Asn Asn Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys
20 25 30
Asp Val Arg Phe Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn
35 40 45
Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala
50 55 60
Val Trp Arg Lys Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His
65 70 75 80
Asp Pro Lys Leu Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser
85 90 95
Pro Lys Cys Ile Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe
100 105 110
Met Cys Ser Cys Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser
115 120 125
Glu Glu Tyr Asn Thr Ser Asn Pro Asp
130 135
<210> 32
<211> 198
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD80 ECD
<400> 32
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn
195
<210> 33
<211> 202
<212> Белок
<213> Искусственная последовательность
<220>
<223> VEGFR
<400> 33
Gly Arg Pro Phe Val Glu Met Tyr Ser Glu Ile Pro Glu Ile Ile His
1 5 10 15
Met Thr Glu Gly Arg Glu Leu Val Ile Pro Cys Arg Val Thr Ser Pro
20 25 30
Asn Ile Thr Val Thr Leu Lys Lys Phe Pro Leu Asp Thr Leu Ile Pro
35 40 45
Asp Gly Lys Arg Ile Ile Trp Asp Ser Arg Lys Gly Phe Ile Ile Ser
50 55 60
Asn Ala Thr Tyr Lys Glu Ile Gly Leu Leu Thr Cys Glu Ala Thr Val
65 70 75 80
Asn Gly His Leu Tyr Lys Thr Asn Tyr Leu Thr His Arg Gln Thr Asn
85 90 95
Thr Ile Ile Asp Val Val Leu Ser Pro Ser His Gly Ile Glu Leu Ser
100 105 110
Val Gly Glu Lys Leu Val Leu Asn Cys Thr Ala Arg Thr Glu Leu Asn
115 120 125
Val Gly Ile Asp Phe Asn Trp Glu Tyr Pro Ser Ser Lys His Gln His
130 135 140
Lys Lys Leu Val Asn Arg Asp Leu Lys Thr Gln Ser Gly Ser Glu Met
145 150 155 160
Lys Lys Phe Leu Ser Thr Leu Thr Ile Asp Gly Val Thr Arg Ser Asp
165 170 175
Gln Gly Leu Tyr Thr Cys Ala Ala Ser Ser Gly Leu Met Thr Lys Lys
180 185 190
Asn Ser Thr Phe Val Arg Val His Glu Pro
195 200
<210> 34
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 34
Gly Ile Thr Phe Ser Asn Ser
1 5
<210> 35
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 35
Gly Ile Thr Phe Ser Asn Ser Gly Met His
1 5 10
<210> 36
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 36
Asn Ser Gly Met His
1 5
<210> 37
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 37
Ser Asn Ser Gly Met His
1 5
<210> 38
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области тяжелой цепи
<400> 38
Gly Ile Thr Phe Ser Asn Ser Gly
1 5
<210> 39
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 39
Trp Tyr Asp Gly Ser Lys
1 5
<210> 40
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 40
Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr
1 5 10
<210> 41
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 41
Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 42
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 42
Trp Val Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr
1 5 10
<210> 43
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области тяжелой цепи
<400> 43
Ile Trp Tyr Asp Gly Ser Lys Arg
1 5
<210> 44
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 44
Asn Asp Asp Tyr
1
<210> 45
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 45
Asn Asp Asp Tyr
1
<210> 46
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 46
Asn Asp Asp Tyr
1
<210> 47
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 47
Ala Thr Asn Asp Asp
1 5
<210> 48
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области тяжелой цепи
<400> 48
Ala Thr Asn Asp Asp Tyr
1 5
<210> 49
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 49
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 50
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 50
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 51
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 51
Arg Ala Ser Gln Ser Val Ser Ser Tyr Leu Ala
1 5 10
<210> 52
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 52
Ser Ser Tyr Leu Ala Trp Tyr
1 5
<210> 53
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR1 вариабельной области легкой цепи
<400> 53
Gln Ser Val Ser Ser Tyr
1 5
<210> 54
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 54
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 55
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 55
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 56
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 56
Asp Ala Ser Asn Arg Ala Thr
1 5
<210> 57
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 57
Leu Leu Ile Tyr Asp Ala Ser Asn Arg Ala
1 5 10
<210> 58
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR2 вариабельной области легкой цепи
<400> 58
Asp Ala Ser
1
<210> 59
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 59
Gln Gln Ser Ser Asn Trp Pro Arg Thr
1 5
<210> 60
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 60
Gln Gln Ser Ser Asn Trp Pro Arg Thr
1 5
<210> 61
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 61
Gln Gln Ser Ser Asn Trp Pro Arg Thr
1 5
<210> 62
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 62
Gln Gln Ser Ser Asn Trp Pro Arg
1 5
<210> 63
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR3 вариабельной области легкой цепи
<400> 63
Gln Gln Ser Ser Asn Trp Pro Arg Thr
1 5
<210> 64
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
<400> 64
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser
<210> 65
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
<400> 65
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 66
<211> 120
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность вариабельной области тяжелой цепи
<400> 66
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Arg Tyr
20 25 30
Ser Val His Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Met Ile Trp Gly Val Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser
115 120
<210> 67
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность вариабельной области легкой цепи
<400> 67
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Ile Thr Cys Arg Ala Ser Lys Ser Val His Thr Ser
20 25 30
Gly Tyr Ser Tyr Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn
65 70 75 80
Pro Val Glu Ala Asn Asp Thr Ala Asn Tyr Tyr Cys Gln His Ser Gly
85 90 95
Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr
<210> 68
<211> 668
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Hib-PDC
<400> 68
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
210 215 220
Gly Gly Gly Ser Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala
225 230 235 240
Thr Leu Ser Cys Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr
245 250 255
Arg Ile Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser
260 265 270
Gly Asp Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp
275 280 285
Ile Thr Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp
290 295 300
Glu Gly Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe
305 310 315 320
Lys Arg Glu His Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe
325 330 335
Pro Thr Pro Ser Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg
340 345 350
Arg Ile Ile Cys Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser
355 360 365
Trp Leu Glu Asn Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser
370 375 380
Gln Asp Pro Glu Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe
385 390 395 400
Asn Met Thr Thr Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His
405 410 415
Leu Arg Val Asn Gln Thr Phe Asn Trp Asn Gly Gly Gly Gly Ser Gly
420 425 430
Gly Gly Gly Ser Gly Gly Gly Gly Ser Lys Tyr Gly Pro Pro Cys Pro
435 440 445
Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe
450 455 460
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
465 470 475 480
Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe
485 490 495
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
500 505 510
Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
515 520 525
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
530 535 540
Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala
545 550 555 560
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln
565 570 575
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
580 585 590
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
595 600 605
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
610 615 620
Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu
625 630 635 640
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
645 650 655
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
660 665
<210> 69
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Hib-PDC
<400> 69
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 70
<211> 672
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Hib-PDV
<400> 70
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
210 215 220
Gly Gly Gly Ser Gly Arg Pro Phe Val Glu Met Tyr Ser Glu Ile Pro
225 230 235 240
Glu Ile Ile His Met Thr Glu Gly Arg Glu Leu Val Ile Pro Cys Arg
245 250 255
Val Thr Ser Pro Asn Ile Thr Val Thr Leu Lys Lys Phe Pro Leu Asp
260 265 270
Thr Leu Ile Pro Asp Gly Lys Arg Ile Ile Trp Asp Ser Arg Lys Gly
275 280 285
Phe Ile Ile Ser Asn Ala Thr Tyr Lys Glu Ile Gly Leu Leu Thr Cys
290 295 300
Glu Ala Thr Val Asn Gly His Leu Tyr Lys Thr Asn Tyr Leu Thr His
305 310 315 320
Arg Gln Thr Asn Thr Ile Ile Asp Val Val Leu Ser Pro Ser His Gly
325 330 335
Ile Glu Leu Ser Val Gly Glu Lys Leu Val Leu Asn Cys Thr Ala Arg
340 345 350
Thr Glu Leu Asn Val Gly Ile Asp Phe Asn Trp Glu Tyr Pro Ser Ser
355 360 365
Lys His Gln His Lys Lys Leu Val Asn Arg Asp Leu Lys Thr Gln Ser
370 375 380
Gly Ser Glu Met Lys Lys Phe Leu Ser Thr Leu Thr Ile Asp Gly Val
385 390 395 400
Thr Arg Ser Asp Gln Gly Leu Tyr Thr Cys Ala Ala Ser Ser Gly Leu
405 410 415
Met Thr Lys Lys Asn Ser Thr Phe Val Arg Val His Glu Pro Gly Gly
420 425 430
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Lys Tyr Gly
435 440 445
Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser
450 455 460
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
465 470 475 480
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
485 490 495
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
500 505 510
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
515 520 525
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
530 535 540
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
545 550 555 560
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
565 570 575
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
580 585 590
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
595 600 605
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
610 615 620
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
625 630 635 640
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
645 650 655
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
660 665 670
<210> 71
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Hib-PDV
<400> 71
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 72
<211> 615
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Hib-PLT
<400> 72
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Arg Tyr
20 25 30
Ser Val His Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Met Ile Trp Gly Val Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Gly
210 215 220
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ile
225 230 235 240
Pro Pro His Val Gln Lys Ser Val Asn Asn Asp Met Ile Val Thr Asp
245 250 255
Asn Asn Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys Asp Val
260 265 270
Arg Phe Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn Cys Ser
275 280 285
Ile Thr Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala Val Trp
290 295 300
Arg Lys Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His Asp Pro
305 310 315 320
Lys Leu Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser Pro Lys
325 330 335
Cys Ile Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe Met Cys
340 345 350
Ser Cys Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser Glu Glu
355 360 365
Tyr Asn Thr Ser Asn Pro Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly
370 375 380
Ser Gly Gly Gly Gly Ser Thr His Thr Cys Pro Pro Cys Pro Ala Pro
385 390 395 400
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
405 410 415
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
420 425 430
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
435 440 445
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
450 455 460
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
465 470 475 480
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
485 490 495
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
500 505 510
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
515 520 525
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
530 535 540
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
545 550 555 560
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
565 570 575
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
580 585 590
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
595 600 605
Leu Ser Leu Ser Pro Gly Lys
610 615
<210> 73
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Hib-PLT
<400> 73
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Ile Thr Cys Arg Ala Ser Lys Ser Val His Thr Ser
20 25 30
Gly Tyr Ser Tyr Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn
65 70 75 80
Pro Val Glu Ala Asn Asp Thr Ala Asn Tyr Tyr Cys Gln His Ser Gly
85 90 95
Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 74
<211> 2007
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность тяжелой цепи Hib-PDC
<400> 74
caggtgcagc tcgtggagag cgggggaggc gtcgtccagc ctggccgtag cctgcggctg 60
gactgtaagg ctagcggaat cacgttcagc aacagcggaa tgcactgggt cagacaggct 120
cctggcaaag gcctcgaatg ggtcgccgtg atctggtacg acgggagcaa gagatactac 180
gcagattcag tgaagggccg tttcacaatc agccgtgaca actcgaagaa cacactgttc 240
ctgcagatga acagcctgag ggcagaagac acagcagtct actactgcgc tacaaatgac 300
gactactggg gccagggaac actggtcacc gtgagctcag cttccaccaa gggcccatcc 360
gtcttccccc tggcgccctg ctccaggagc acctccgaga gcacagccgc cctgggctgc 420
ctggtcaagg actacttccc cgaaccggtg acggtgtcgt ggaactcagg cgccctgacc 480
agcggcgtgc acaccttccc ggctgtccta cagtcctcag gactctactc cctcagcagc 540
gtggtgaccg tgccctccag cagcttgggc acgaagacct acacctgcaa cgtagatcac 600
aagcccagca acaccaaggt ggacaagaga gttgagtccg gtggcggcgg aagcggcggc 660
ggaggcagcg gcggaggcgg cagcgtgatc catgtcacga aggaggtcaa agaagtggcc 720
accctcagct gcggtcacaa cgtcagcgtg gaagagttgg cccagaccag aatctactgg 780
cagaaggaga agaagatggt cttgaccatg atgagcggcg acatgaacat ctggccagag 840
tacaagaata gaactatctt cgacatcacc aataacctga gcatcgtgat cctcgctttg 900
cgtccgagcg acgaaggcac ctacgagtgc gtagtgctaa agtacgagaa agacgccttc 960
aagcgggagc acctggcaga agtaaccctg agcgtgaagg cagacttccc aacgcctagc 1020
atctccgact tcgagatccc cacaagcaac atccggcgga tcatctgtag cacctccggg 1080
ggtttccccg agcctcacct gtcctggctc gagaacggcg aggagctgaa cgccatcaac 1140
accaccgtga gccaggaccc cgagacagaa ctgtacgccg tgagctccaa gctcgacttc 1200
aacatgacca caaatcacag cttcatgtgc ctcatcaagt acggacacct gcgggtgaat 1260
cagacgttca actggaacgg cggggggggt agcggcggcg ggggctcagg tggggggggc 1320
tcaaaatatg gtcccccatg cccaccatgc ccagcacctg agttcctggg gggaccatca 1380
gtcttcctgt tccccccaaa acccaaggac actctcatga tctcccggac ccctgaggtc 1440
acgtgcgtgg tggtggacgt gagccaggaa gaccccgagg tccagttcaa ctggtacgtg 1500
gatggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagtt caacagcacg 1560
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaacgg caaggagtac 1620
aagtgcaagg tctccaacaa aggcctcccg tcctccatcg agaaaaccat ctccaaagcc 1680
aaagggcagc cccgagagcc acaggtgtac accctgcccc catcccagga ggagatgacc 1740
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct accccagcga catcgccgtg 1800
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1860
tccgacggct ccttcttcct ctacagcagg ctaaccgtgg acaagagcag gtggcaggag 1920
gggaatgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacacagaag 1980
agcctctccc tgtctctggg taaatag 2007
<210> 75
<211> 645
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность легкой цепи Hib-PDC
<400> 75
gagatcgtgc tgacacagag cccagccact ctgtcactgt ccccaggaga aagggctact 60
ctgtcttgcc gggcaagcca gtctgtctcc agctacctgg cctggtatca gcagaagccc 120
ggacaggctc ctagactgct gatctacgac gcaagtaaca gagccaccgg catccccgca 180
cgcttcagtg gctcaggctc cggaacagac tttactctga ccatctctag tctggagcct 240
gaagatttcg ccgtgtacta ttgtcagcag agctctaatt ggcctagaac cttcggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 76
<211> 2019
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность тяжелой цепи Hib-PDV
<400> 76
caggtgcagc tcgtggagag cgggggaggc gtcgtccagc ctggccgtag cctgcggctg 60
gactgtaagg ctagcggaat cacgttcagc aacagcggaa tgcactgggt cagacaggct 120
cctggcaaag gcctcgaatg ggtcgccgtg atctggtacg acgggagcaa gagatactac 180
gcagattcag tgaagggccg tttcacaatc agccgtgaca actcgaagaa cacactgttc 240
ctgcagatga acagcctgag ggcagaagac acagcagtct actactgcgc tacaaatgac 300
gactactggg gccagggaac actggtcacc gtgagctcag cttccaccaa gggcccatcc 360
gtcttccccc tggcgccctg ctccaggagc acctccgaga gcacagccgc cctgggctgc 420
ctggtcaagg actacttccc cgaaccggtg acggtgtcgt ggaactcagg cgccctgacc 480
agcggcgtgc acaccttccc ggctgtccta cagtcctcag gactctactc cctcagcagc 540
gtggtgaccg tgccctccag cagcttgggc acgaagacct acacctgcaa cgtagatcac 600
aagcccagca acaccaaggt ggacaagaga gttgagtccg gtggcggcgg aagcggcggc 660
ggaggcagcg gcggaggcgg cagcggtaga ccattcgtag agatgtacag cgaaatcccc 720
gaaatcatcc acatgactga aggaagggag ctcgtcatcc cctgccgggt tacgtcacct 780
aacatcactg ttactctgaa gaagttccca ctcgacactt tgatccctga tggaaaacgc 840
atcatctggg acagcagaaa gggcttcatc atctcaaatg caacgtacaa agaaatcgga 900
ctcctgacct gtgaagcaac agtcaatgga catttgtata agacaaacta tctcacacat 960
cgacagacca atacaatcat cgatgtggtt ctgagcccgt ctcatggaat cgaactatct 1020
gttggagaaa agctcgtcct gaattgtaca gcaagaactg aactgaatgt gggaatcgac 1080
ttcaactggg aatacccttc ttcgaagcat cagcataaga aactcgtaaa ccgagaccta 1140
aagacccagt ctgggagcga gatgaagaaa ttcttgagca ccctgactat cgatggtgta 1200
acccggagcg accagggatt gtacacctgt gcagcatcca gcgggctgat gaccaagaag 1260
aacagcacat tcgtcagggt ccatgaaccc ggcggggggg gtagcggcgg cgggggctca 1320
ggtggggggg gctcaaaata tggtccccca tgcccaccat gcccagcacc tgagttcctg 1380
gggggaccat cagtcttcct gttcccccca aaacccaagg acactctcat gatctcccgg 1440
acccctgagg tcacgtgcgt ggtggtggac gtgagccagg aagaccccga ggtccagttc 1500
aactggtacg tggatggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 1560
ttcaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaac 1620
ggcaaggagt acaagtgcaa ggtctccaac aaaggcctcc cgtcctccat cgagaaaacc 1680
atctccaaag ccaaagggca gccccgagag ccacaggtgt acaccctgcc cccatcccag 1740
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctaccccagc 1800
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1860
cccgtgctgg actccgacgg ctccttcttc ctctacagca ggctaaccgt ggacaagagc 1920
aggtggcagg aggggaatgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1980
tacacacaga agagcctctc cctgtctctg ggtaaatag 2019
<210> 77
<211> 645
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность легкой цепи Hib-PDV
<400> 77
gagatcgtgc tgacacagag cccagccact ctgtcactgt ccccaggaga aagggctact 60
ctgtcttgcc gggcaagcca gtctgtctcc agctacctgg cctggtatca gcagaagccc 120
ggacaggctc ctagactgct gatctacgac gcaagtaaca gagccaccgg catccccgca 180
cgcttcagtg gctcaggctc cggaacagac tttactctga ccatctctag tctggagcct 240
gaagatttcg ccgtgtacta ttgtcagcag agctctaatt ggcctagaac cttcggccag 300
ggcaccaaag tcgagatcaa gcgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 78
<211> 1848
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность тяжелой цепи Hib-PLT
<400> 78
caggtgcagc tgcaggagtc cggaccagga ctggtgaagc catccgagac cctgagcctg 60
acctgtacag tgtccggctt cagcctgtct aggtacagcg tgcactggat cagacagcca 120
cctggcaagg gactggagtg gctgggcatg atctggggcg tgggcaccac agactacaac 180
tctgctctga agtccagact gaccatcagc aaggatacat ctaagaatca gttcagcctg 240
aagctgtcca gcgtgaccgc cgctgacaca gccgtgtact attgcgctcg caactggggc 300
accgccgact acttcgacta ttggggccag ggcaccacag tgacagtgtc ttccgctagc 360
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 600
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agaaagttga gcccaaatct 660
tgtgacaaag gcgggggagg cagcggcggc ggaggaagcg ggggcggagg tagcaccatc 720
cctccacacg tgcagaagag cgtgaacaat gacatgatcg tcaccgacaa caacggcgcc 780
gtcaagttcc cccagctgtg caaattctgc gacgtgaggt tctccacgtg cgacaaccag 840
aagagctgta tgagcaactg cagcatcaca tccatctgcg aaaaacccca ggaagtgtgc 900
gtcgccgtct ggcggaagaa cgacgagaac atcacactgg agaccgtgtg ccacgacccc 960
aaactgccct accacgactt catcctggag gacgccgcca gcccaaagtg catcatgaaa 1020
gagaagaaga agccgggcga gactttcttc atgtgctcct gcagctccga cgagtgcaac 1080
gataatatca tcttcagcga agaatacaac acatctaacc cagacggagg gggcggatcc 1140
gggggcggcg gaagcggcgg ggggggcagc actcacacat gcccaccgtg cccagcacct 1200
gaactcctgg ggggaccgtc agtcttcctc ttccccccaa aacccaagga caccctcatg 1260
atctcccgga cccctgaggt cacatgcgtg gtggtggacg tgagccacga agaccctgag 1320
gtcaagttca actggtacgt ggacggcgtg gaggtgcata atgccaagac aaagccgcgg 1380
gaggagcagt acaacagcac gtaccgtgtg gtcagcgtcc tcaccgtcct gcaccaggac 1440
tggctgaatg gcaaggagta caagtgcaag gtctccaaca aagccctccc agcccccatc 1500
gagaaaacca tctccaaagc caaagggcag ccccgagaac cacaggtgta caccctgccc 1560
ccatcccggg aggagatgac caagaaccag gtcagcctga cctgcctggt caaaggcttc 1620
tatcccagcg acatcgccgt ggagtgggag agcaatgggc agccggagaa caactacaag 1680
accacgcctc ccgtgctgga ctccgacggc tccttcttcc tctacagcaa gctcaccgtg 1740
gacaagagca ggtggcagca ggggaacgtc ttctcatgct ccgtgatgca tgaggctctg 1800
cacaaccact acacgcagaa gagcctctcc ctgtctccgg gtaaatga 1848
<210> 79
<211> 657
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Нуклеотидная последовательность легкой цепи Hib-PLT
<400> 79
gatatcgtgc tgacccagtc tccagcttcc ctggccgtgt ccccaggaca gagggccacc 60
atcacatgtc gggcttccaa gagcgtgcac acaagcggct actcttatat gcattggtac 120
cagcagaagc ccggccagcc ccctaagctg ctgatctatc tggcttccaa cctggagagc 180
ggagtgccag ctaggttctc tggctccggc agcggcaccg actttaccct gacaatcaat 240
cctgtggagg ccaacgatac agctaattac tattgccagc actccggaga gctgccatac 300
accttcggcg gaggcacaaa ggtggagatc aagcgtacgg tggctgcacc atctgtcttc 360
atcttcccgc catctgatga gcagttgaaa tctggaactg cctctgttgt gtgcctgctg 420
aataacttct atcccagaga ggccaaagta cagtggaagg tggataacgc cctccaatcg 480
ggtaactccc aggagagtgt cacagagcag gacagcaagg acagcaccta cagcctcagc 540
agcaccctga cgctgagcaa agcagactac gagaaacaca aagtctacgc ctgcgaagtc 600
acccatcagg gcctgagctc gcccgtcaca aagagcttca acaggggaga gtgttag 657
<210> 80
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 80
Ala Lys Thr Thr Pro Lys Leu Glu Glu Gly Glu Phe Ser Glu Ala Arg
1 5 10 15
<210> 81
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 81
Ala Lys Thr Thr Pro Lys Leu Glu Glu Gly Glu Phe Ser Glu Ala Arg
1 5 10 15
Val
<210> 82
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 82
Ala Lys Thr Thr Pro Lys Leu Gly Gly
1 5
<210> 83
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 83
Ser Ala Lys Thr Thr Pro Lys Leu Gly Gly
1 5 10
<210> 84
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 84
Ser Ala Lys Thr Thr Pro
1 5
<210> 85
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 85
Arg Ala Asp Ala Ala Pro
1 5
<210> 86
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 86
Arg Ala Asp Ala Ala Pro Thr Val Ser
1 5
<210> 87
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 87
Arg Ala Asp Ala Ala Ala Ala Gly Gly Pro Gly Ser
1 5 10
<210> 88
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 88
Arg Ala Asp Ala Ala Ala Ala
1 5
<210> 89
<211> 18
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 89
Ser Ala Lys Thr Thr Pro Lys Leu Glu Glu Gly Glu Phe Ser Glu Ala
1 5 10 15
Arg Val
<210> 90
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 90
Ala Asp Ala Ala Pro
1 5
<210> 91
<211> 11
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 91
Asp Ala Ala Pro Thr Val Ser Ile Phe Pro Pro
1 5 10
<210> 92
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 92
Thr Val Ala Ala Pro
1 5
<210> 93
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 93
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro
1 5 10
<210> 94
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 94
Gln Pro Lys Ala Ala Pro
1 5
<210> 95
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 95
Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro
1 5 10
<210> 96
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 96
Ala Lys Thr Thr Pro Pro
1 5
<210> 97
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 97
Ala Lys Thr Thr Pro Pro Ser Val Thr Pro Leu Ala Pro
1 5 10
<210> 98
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 98
Ala Lys Thr Thr Ala Pro
1 5
<210> 99
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 99
Ala Lys Thr Thr Ala Pro Ser Val Tyr Pro Leu Ala Pro
1 5 10
<210> 100
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 100
Ala Ser Thr Lys Gly Pro
1 5
<210> 101
<211> 13
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 101
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro
1 5 10
<210> 102
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 102
Gly Glu Asn Lys Val Glu Tyr Ala Pro Ala Leu Met Ala Leu Ser
1 5 10 15
<210> 103
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 103
Gly Pro Ala Lys Glu Leu Thr Pro Leu Lys Glu Ala Lys Val Ser
1 5 10 15
<210> 104
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 104
Gly His Glu Ala Ala Ala Val Met Gln Val Gln Tyr Pro Ala Ser
1 5 10 15
<210> 105
<211> 16
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 105
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala
1 5 10 15
<210> 106
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи атезолизумаба
<400> 106
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Ser
20 25 30
Trp Ile His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Trp Ile Ser Pro Tyr Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Ala Asp Thr Ser Lys Asn Thr Ala Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg His Trp Pro Gly Gly Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Ala Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 107
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи атезолизумаба
<400> 107
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Asp Val Ser Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Phe Leu Tyr Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Leu Tyr His Pro Ala
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 108
<211> 450
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи авелумаба
<400> 108
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ile Met Met Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Tyr Pro Ser Gly Gly Ile Thr Phe Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ile Lys Leu Gly Thr Val Thr Thr Val Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 109
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи авелумаба
<400> 109
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Thr Gly Thr Ser Ser Asp Val Gly Gly Tyr
20 25 30
Asn Tyr Val Ser Trp Tyr Gln Gln His Pro Gly Lys Ala Pro Lys Leu
35 40 45
Met Ile Tyr Asp Val Ser Asn Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Tyr Thr Ser Ser
85 90 95
Ser Thr Arg Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Asn Pro Thr Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Gly Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Lys Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 110
<211> 451
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи дурвалумаба
<400> 110
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Arg Tyr
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Asn Ile Lys Gln Asp Gly Ser Glu Lys Tyr Tyr Val Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Gly Trp Phe Gly Glu Leu Ala Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Phe Glu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Ser Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 111
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи дурвалумаба
<400> 111
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Arg Val Ser Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Asp Ala Ser Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Leu Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 112
<211> 440
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи ниволумаба
<400> 112
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala
210 215 220
Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
225 230 235 240
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
245 250 255
Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
260 265 270
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
275 280 285
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
290 295 300
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
305 310 315 320
Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
325 330 335
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr
340 345 350
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
355 360 365
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
370 375 380
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
385 390 395 400
Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe
405 410 415
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
420 425 430
Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210> 113
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи ниволумаба
<400> 113
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 114
<211> 447
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи пембролизумаба
<400> 114
Gln Val Gln Leu Val Gln Ser Gly Val Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Tyr Met Tyr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Asn Pro Ser Asn Gly Gly Thr Asn Phe Asn Glu Lys Phe
50 55 60
Lys Asn Arg Val Thr Leu Thr Thr Asp Ser Ser Thr Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Lys Ser Leu Gln Phe Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Asp Tyr Arg Phe Asp Met Gly Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro
210 215 220
Pro Cys Pro Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
260 265 270
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 115
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи пембролизумаба
<400> 115
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Lys Gly Val Ser Thr Ser
20 25 30
Gly Tyr Ser Tyr Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
35 40 45
Arg Leu Leu Ile Tyr Leu Ala Ser Tyr Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
65 70 75 80
Ser Leu Glu Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln His Ser Arg
85 90 95
Asp Leu Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 116
<211> 448
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи ипилимумаба
<400> 116
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Thr Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Thr Phe Ile Ser Tyr Asp Gly Asn Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Thr Gly Trp Leu Gly Pro Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 117
<211> 215
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи ипилимумаба
<400> 117
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Ser Ser
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Phe Ser Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Gly Ser Ser Pro
85 90 95
Trp Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 118
<211> 444
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи цемиплимаба
<400> 118
Glu Val Gln Leu Leu Glu Ser Gly Gly Val Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Phe
20 25 30
Gly Met Thr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Gly Gly Gly Arg Asp Thr Tyr Phe Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Gly Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Trp Gly Asn Ile Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro
210 215 220
Pro Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe
225 230 235 240
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
245 250 255
Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe
260 265 270
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
275 280 285
Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
290 295 300
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
305 310 315 320
Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala
325 330 335
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln
340 345 350
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
355 360 365
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
370 375 380
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
385 390 395 400
Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu
405 410 415
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
420 425 430
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440
<210> 119
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи цемиплимаба
<400> 119
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Ser Ile Thr Ile Thr Cys Arg Ala Ser Leu Ser Ile Asn Thr Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu His Gly Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Arg Thr Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Ser Asn Thr Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Val Val Asp Phe Arg Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 120
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 120
Gly Gly Gly Gly Ser
1 5
<210> 121
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 121
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 122
<211> 15
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 122
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 123
<211> 20
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный линкер
<400> 123
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 124
<211> 600
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Контрольного белка 1
<400> 124
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Arg Tyr
20 25 30
Ser Val His Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Met Ile Trp Gly Val Gly Thr Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Thr Ile Ser Lys Asp Thr Ser Lys Asn Gln Phe Ser Leu
65 70 75 80
Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Asn Trp Gly Thr Ala Asp Tyr Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr
210 215 220
His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser
225 230 235 240
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg
245 250 255
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro
260 265 270
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
275 280 285
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val
290 295 300
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
305 310 315 320
Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr
325 330 335
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
340 345 350
Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
355 360 365
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
370 375 380
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
385 390 395 400
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser
405 410 415
Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala
420 425 430
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr
450 455 460
Ile Pro Pro His Val Gln Lys Ser Val Asn Asn Asp Met Ile Val Thr
465 470 475 480
Asp Asn Asn Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys Asp
485 490 495
Val Arg Phe Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn Cys
500 505 510
Ser Ile Thr Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala Val
515 520 525
Trp Arg Lys Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His Asp
530 535 540
Pro Lys Leu Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser Pro
545 550 555 560
Lys Cys Ile Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe Met
565 570 575
Cys Ser Cys Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser Glu
580 585 590
Glu Tyr Asn Thr Ser Asn Pro Asp
595 600
<210> 125
<211> 218
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Контрольного белка 1
<400> 125
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Pro Gly
1 5 10 15
Gln Arg Ala Thr Ile Thr Cys Arg Ala Ser Lys Ser Val His Thr Ser
20 25 30
Gly Tyr Ser Tyr Met His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Tyr Leu Ala Ser Asn Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asn
65 70 75 80
Pro Val Glu Ala Asn Asp Thr Ala Asn Tyr Tyr Cys Gln His Ser Gly
85 90 95
Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
115 120 125
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
130 135 140
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
145 150 155 160
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
165 170 175
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
180 185 190
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
195 200 205
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 126
<211> 653
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Контрольного белка 2
<400> 126
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala
210 215 220
Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
225 230 235 240
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
245 250 255
Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
260 265 270
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
275 280 285
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
290 295 300
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
305 310 315 320
Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
325 330 335
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr
340 345 350
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
355 360 365
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
370 375 380
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
385 390 395 400
Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe
405 410 415
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
420 425 430
Ser Leu Ser Leu Ser Leu Gly Lys Gly Gly Gly Gly Ser Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Val Ile His Val Thr Lys Glu Val Lys
450 455 460
Glu Val Ala Thr Leu Ser Cys Gly His Asn Val Ser Val Glu Glu Leu
465 470 475 480
Ala Gln Thr Arg Ile Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr
485 490 495
Met Met Ser Gly Asp Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr
500 505 510
Ile Phe Asp Ile Thr Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg
515 520 525
Pro Ser Asp Glu Gly Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys
530 535 540
Asp Ala Phe Lys Arg Glu His Leu Ala Glu Val Thr Leu Ser Val Lys
545 550 555 560
Ala Asp Phe Pro Thr Pro Ser Ile Ser Asp Phe Glu Ile Pro Thr Ser
565 570 575
Asn Ile Arg Arg Ile Ile Cys Ser Thr Ser Gly Gly Phe Pro Glu Pro
580 585 590
His Leu Ser Trp Leu Glu Asn Gly Glu Glu Leu Asn Ala Ile Asn Thr
595 600 605
Thr Val Ser Gln Asp Pro Glu Thr Glu Leu Tyr Ala Val Ser Ser Lys
610 615 620
Leu Asp Phe Asn Met Thr Thr Asn His Ser Phe Met Cys Leu Ile Lys
625 630 635 640
Tyr Gly His Leu Arg Val Asn Gln Thr Phe Asn Trp Asn
645 650
<210> 127
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Контрольного белка 2
<400> 127
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 128
<211> 657
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность тяжелой цепи Контрольного белка 3
<400> 128
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Asp Cys Lys Ala Ser Gly Ile Thr Phe Ser Asn Ser
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Trp Tyr Asp Gly Ser Lys Arg Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asn Asp Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser
115 120 125
Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys
180 185 190
Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala
210 215 220
Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
225 230 235 240
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
245 250 255
Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val
260 265 270
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
275 280 285
Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
290 295 300
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly
305 310 315 320
Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
325 330 335
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr
340 345 350
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
355 360 365
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
370 375 380
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
385 390 395 400
Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe
405 410 415
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
420 425 430
Ser Leu Ser Leu Ser Leu Gly Lys Gly Gly Gly Gly Ser Gly Gly Gly
435 440 445
Gly Ser Gly Gly Gly Gly Ser Gly Arg Pro Phe Val Glu Met Tyr Ser
450 455 460
Glu Ile Pro Glu Ile Ile His Met Thr Glu Gly Arg Glu Leu Val Ile
465 470 475 480
Pro Cys Arg Val Thr Ser Pro Asn Ile Thr Val Thr Leu Lys Lys Phe
485 490 495
Pro Leu Asp Thr Leu Ile Pro Asp Gly Lys Arg Ile Ile Trp Asp Ser
500 505 510
Arg Lys Gly Phe Ile Ile Ser Asn Ala Thr Tyr Lys Glu Ile Gly Leu
515 520 525
Leu Thr Cys Glu Ala Thr Val Asn Gly His Leu Tyr Lys Thr Asn Tyr
530 535 540
Leu Thr His Arg Gln Thr Asn Thr Ile Ile Asp Val Val Leu Ser Pro
545 550 555 560
Ser His Gly Ile Glu Leu Ser Val Gly Glu Lys Leu Val Leu Asn Cys
565 570 575
Thr Ala Arg Thr Glu Leu Asn Val Gly Ile Asp Phe Asn Trp Glu Tyr
580 585 590
Pro Ser Ser Lys His Gln His Lys Lys Leu Val Asn Arg Asp Leu Lys
595 600 605
Thr Gln Ser Gly Ser Glu Met Lys Lys Phe Leu Ser Thr Leu Thr Ile
610 615 620
Asp Gly Val Thr Arg Ser Asp Gln Gly Leu Tyr Thr Cys Ala Ala Ser
625 630 635 640
Ser Gly Leu Met Thr Lys Lys Asn Ser Thr Phe Val Arg Val His Glu
645 650 655
Pro
<210> 129
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> Аминокислотная последовательность легкой цепи Контрольного белка 3
<400> 129
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Ser Ser Asn Trp Pro Arg
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 130
<211> 202
<212> Белок
<213> Искусственная последовательность
<220>
<223> фрагмент VEGF-R
<400> 130
Gly Arg Pro Phe Val Glu Met Tyr Ser Glu Ile Pro Glu Ile Ile His
1 5 10 15
Met Thr Glu Gly Arg Glu Leu Val Ile Pro Cys Arg Val Thr Ser Pro
20 25 30
Asn Ile Thr Val Thr Leu Lys Lys Phe Pro Leu Asp Thr Leu Ile Pro
35 40 45
Asp Gly Lys Arg Ile Ile Trp Asp Ser Arg Lys Gly Phe Ile Ile Ser
50 55 60
Asn Ala Thr Tyr Lys Glu Ile Gly Leu Leu Thr Cys Glu Ala Thr Val
65 70 75 80
Asn Gly His Leu Tyr Lys Thr Asn Tyr Leu Thr His Arg Gln Thr Asn
85 90 95
Thr Ile Ile Asp Val Val Leu Ser Pro Ser His Gly Ile Glu Leu Ser
100 105 110
Val Gly Glu Lys Leu Val Leu Asn Cys Thr Ala Arg Thr Glu Leu Asn
115 120 125
Val Gly Ile Asp Phe Asn Trp Glu Tyr Pro Ser Ser Lys His Gln His
130 135 140
Lys Lys Leu Val Asn Arg Asp Leu Lys Thr Gln Ser Gly Ser Glu Met
145 150 155 160
Lys Lys Phe Leu Ser Thr Leu Thr Ile Asp Gly Val Thr Arg Ser Asp
165 170 175
Gln Gly Leu Tyr Thr Cys Ala Ala Ser Ser Gly Leu Met Thr Lys Lys
180 185 190
Asn Ser Thr Phe Val Arg Val His Glu Pro
195 200
<210> 131
<211> 117
<212> Белок
<213> Искусственная последовательность
<220>
<223> TGF-βRII человека
<400> 131
Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys Asp Val Arg Phe
1 5 10 15
Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn Cys Ser Ile Thr
20 25 30
Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala Val Trp Arg Lys
35 40 45
Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His Asp Pro Lys Leu
50 55 60
Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile
65 70 75 80
Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe Met Cys Ser Cys
85 90 95
Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser Glu Glu Tyr Asn
100 105 110
Thr Ser Asn Pro Asp
115
<210> 132
<211> 115
<212> Белок
<213> Искусственная последовательность
<220>
<223> TGF-βRII человека
<400> 132
Val Lys Phe Pro Gln Leu Cys Lys Phe Cys Asp Val Arg Phe Ser Thr
1 5 10 15
Cys Asp Asn Gln Lys Ser Cys Met Ser Asn Cys Ser Ile Thr Ser Ile
20 25 30
Cys Glu Lys Pro Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp
35 40 45
Glu Asn Ile Thr Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr
50 55 60
His Asp Phe Ile Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys
65 70 75 80
Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe Met Cys Ser Cys Ser Ser
85 90 95
Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser Glu Glu Tyr Asn Thr Ser
100 105 110
Asn Pro Asp
115
<210> 133
<211> 112
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD80 ECD
<400> 133
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
<210> 134
<211> 288
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD80
<400> 134
Met Gly His Thr Arg Arg Gln Gly Thr Ser Pro Ser Lys Cys Pro Tyr
1 5 10 15
Leu Asn Phe Phe Gln Leu Leu Val Leu Ala Gly Leu Ser His Phe Cys
20 25 30
Ser Gly Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu
35 40 45
Ser Cys Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile
50 55 60
Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp
65 70 75 80
Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr
85 90 95
Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly
100 105 110
Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg
115 120 125
Glu His Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr
130 135 140
Pro Ser Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile
145 150 155 160
Ile Cys Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu
165 170 175
Glu Asn Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp
180 185 190
Pro Glu Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met
195 200 205
Thr Thr Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg
210 215 220
Val Asn Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro
225 230 235 240
Asp Asn Leu Leu Pro Ser Trp Ala Ile Thr Leu Ile Ser Val Asn Gly
245 250 255
Ile Phe Val Ile Cys Cys Leu Thr Tyr Cys Phe Ala Pro Arg Cys Arg
260 265 270
Glu Arg Arg Arg Asn Glu Arg Leu Arg Arg Glu Ser Val Arg Pro Val
275 280 285
<210> 135
<211> 256
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD80
<400> 135
Met Gly His Thr Arg Arg Gln Gly Thr Ser Pro Ser Lys Cys Pro Tyr
1 5 10 15
Leu Asn Phe Phe Gln Leu Leu Val Leu Ala Gly Leu Ser His Phe Cys
20 25 30
Ser Gly Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu
35 40 45
Ser Cys Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile
50 55 60
Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp
65 70 75 80
Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr
85 90 95
Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly
100 105 110
Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg
115 120 125
Glu His Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr
130 135 140
Pro Ser Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile
145 150 155 160
Ile Cys Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu
165 170 175
Glu Asn Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp
180 185 190
Pro Glu Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met
195 200 205
Thr Thr Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg
210 215 220
Val Asn Gln Thr Phe Asn Trp Asn Thr Ser Phe Ala Pro Arg Cys Arg
225 230 235 240
Glu Arg Arg Arg Asn Glu Arg Leu Arg Arg Glu Ser Val Arg Pro Val
245 250 255
<210> 136
<211> 162
<212> Белок
<213> Искусственная последовательность
<220>
<223> CD80
<400> 136
Met Gly His Thr Arg Arg Gln Gly Thr Ser Pro Ser Lys Cys Pro Tyr
1 5 10 15
Leu Asn Phe Phe Gln Leu Leu Val Leu Ala Gly Leu Ser His Phe Cys
20 25 30
Ser Gly Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu
35 40 45
Ser Cys Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile
50 55 60
Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp
65 70 75 80
Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr
85 90 95
Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly
100 105 110
Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg
115 120 125
Glu His Leu Ala Glu Val Thr Leu Ser Val Lys Gly Phe Ala Pro Arg
130 135 140
Cys Arg Glu Arg Arg Arg Asn Glu Arg Leu Arg Arg Glu Ser Val Arg
145 150 155 160
Pro Val
<210> 137
<211> 137
<212> Белок
<213> Искусственная последовательность
<220>
<223> фрагмент внеклеточной области TGF-βRⅡ
<400> 137
Thr Ile Pro Pro His Val Gln Lys Ser Val Asn Asn Asp Met Ile Val
1 5 10 15
Thr Asp Asn Asn Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys
20 25 30
Asp Val Arg Phe Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn
35 40 45
Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala
50 55 60
Val Trp Arg Lys Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His
65 70 75 80
Asp Pro Lys Leu Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser
85 90 95
Pro Lys Cys Ile Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe
100 105 110
Met Cys Ser Cys Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser
115 120 125
Glu Glu Tyr Asn Thr Ser Asn Pro Asp
130 135
| название | год | авторы | номер документа |
|---|---|---|---|
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| АНТАГОНИСТЫ NGF ДЛЯ МЕДИЦИНСКОГО ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2829812C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
| Слитые молекулы, происходящие от Cholix-токсина, для пероральной доставки биологически активных нагрузок | 2015 |
|
RU2723178C2 |
| ВАРИАНТ Fc И ЕГО ПОЛУЧЕНИЕ | 2021 |
|
RU2828852C1 |
| СЛИТЫЙ БЕЛОК GDF15 ДЛИТЕЛЬНОГО ДЕЙСТВИЯ И СОДЕРЖАЩАЯ ЕГО ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 2020 |
|
RU2836280C1 |
| НАЦЕЛЕННЫЕ НА ОПУХОЛЬ АГОНИСТИЧЕСКИЕ CD28-АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2019 |
|
RU2837821C1 |
| ВАРИАНТЫ IgG-FC ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2018 |
|
RU2814952C2 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
| ПОЛИВАЛЕТНЫЕ И ПОЛИСПЕЦИФИЧНЫЕ GITR-СВЯЗЫВАЮЩИЕ СЛИТЫЕ БЕЛКИ | 2016 |
|
RU2753439C2 |
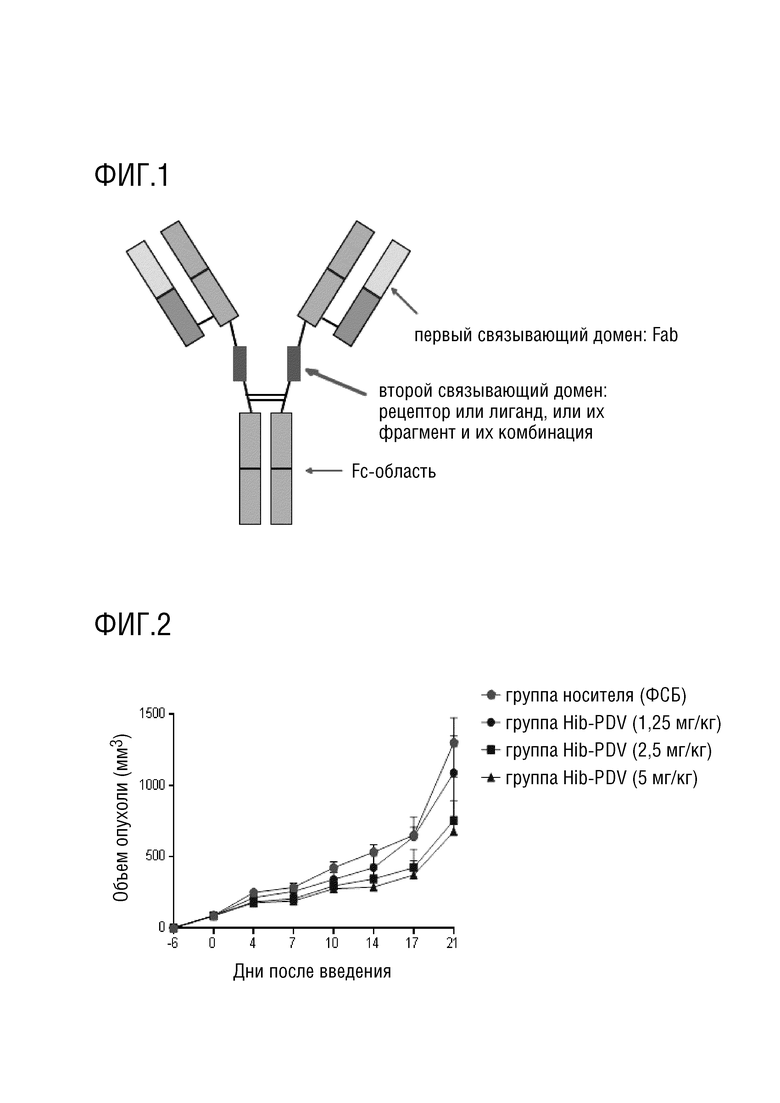
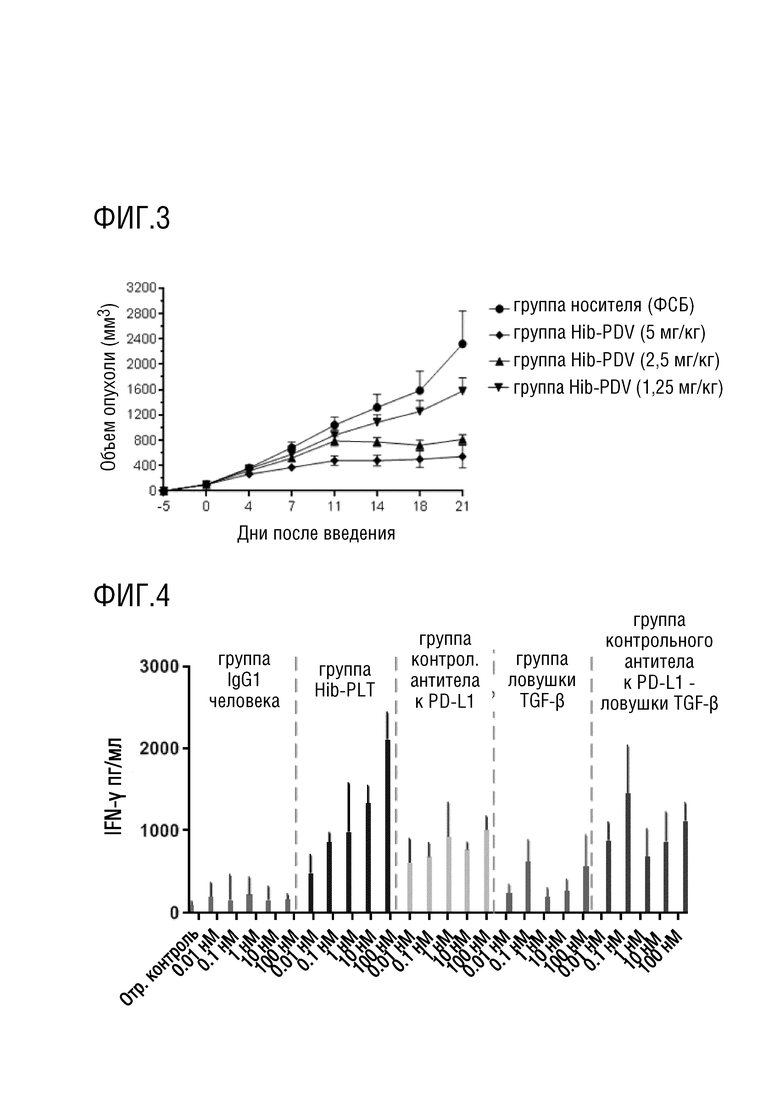
Настоящее изобретение относится к области биотехнологии, в частности к новым слитым терапевтическим белкам, и может быть использовано в медицине. Изобретение раскрывает биспецифичный слитый белок, в структуре которого второй связывающий структурный домен вставлен в шарнирную область полноразмерного иммуноглобулина G (IgG). Такой слитый белок имеет те же преимущества в экспрессии и продуцировании, что и исходный IgG, не влияет на связывающую активность Fab-области слитого белка и дополнительно улучшает стабильность и обеспечивает более продолжительное время полужизни. Изобретение может быть применимо при лечении различных злокачественных новообразований. 9 н. и 32 з.п. ф-лы, 6 пр., 11 табл., 4 ил.
1. Биспецифичный слитый белок для лечения или предотвращения злокачественной опухоли или рака, где слитый белок имеет структуру, в которой второй связывающий домен вставлен в шарнирную область полноразмерного иммуноглобулина G (IgG) через необязательный пептидный линкер, а Fab-область IgG представляет собой первый связывающий домен; после вставки второго связывающего домена тяжелая цепь IgG представляет собой тяжелую цепь слитого белка, а легкая цепь IgG представляет собой легкую цепь слитого белка; второй связывающий домен выбран из человеческого рецептора или лиганда, фрагмента рецептора или лиганда и комбинации фрагментов рецептора или лиганда; где рецептор или лиганд образует структуру димеризации или мультимеризации в естественном сигнальном пути для активации или ингибирования сигнального пути, а второй связывающий домен и первый связывающий домен нацелены на разные мишени;
где первый связывающий домен нацелен и связывается с молекулой иммунной контрольной точки или опухолевым антигеном;
где молекула иммунной контрольной точки включает в себя PD-1, PD-L1, CTLA-4, LAG-3, FGL1, TIM-3, галектин-9, TIGIT, CD155 и CD47; опухолевый антиген включает в себя клаудин 18.2, HER-2, мезотелин, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, VEGF-A, нектин-4, FGFR, C-met, RANKL, PDGF, PDGFR, PDGFRα, DLL4, Ang-1 и Ang-2;
где второй связывающий домен нацелен и связывается с человеческим TGF-β, CTLA-4, VEGF, LAG3, CD27, 4-1BB, OX40, CD47, FGL1, TLT-2, CD28, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, GITR, EGF и ICOSL.
2. Слитый белок по п.1, где первый связывающий домен и второй связывающий домен выбраны из следующих комбинаций:
(1) первый связывающий домен нацелен и связывается с PD-L1, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, FGL1, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
(2) первый связывающий домен нацелен и связывается с PD-1, а второй связывающий домен нацелен и связывается с человеческим CTLA-4, VEGF, HGF, EGF, CD28, LAG3, CD27, 4-1BB или OX40; или
(3) первый связывающий домен нацелен и связывается с CTLA-4, а второй связывающий домен нацелен и связывается с человеческим CD28, VEGF, HGF, EGF, LAG3, CD27, 4-1BB или OX40; или
(4) первый связывающий домен нацелен и связывается с LAG-3, а второй связывающий домен нацелен и связывается с человеческим CD28, VEGF, HGF, EGF, CTLA-4, CD27, 4-1BB или OX40; или
(5) первый связывающий домен нацелен и связывается с FGL1, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
(6) первый связывающий домен нацелен и связывается с TIM-3, а второй связывающий домен нацелен и связывается с человеческим VEGF, HGF, EGF, LAG3, CD27, 4-1BB или OX40; или
(7) первый связывающий домен нацелен и связывается с галектином-9, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, CXCL12, EGF или ICOSL; или
(8) первый связывающий домен нацелен и связывается с TIGIT, а второй связывающий домен нацелен и связывается с человеческим TGF-β, HGF, EGF или VEGF; или
(9) первый связывающий домен нацелен и связывается с CD155, а второй связывающий домен нацелен и связывается с человеческим TGF-β, VEGF, HGF, EGF, CD47 или CD155; или
(10) первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2, мезотелином, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, VEGF-A, нектином-4, FGFR, C-met, RANKL, PDGF, PDGFR, PDGFRα, DLL-4, Ang-1 или Ang-2, а второй связывающий домен нацелен и связывается с человеческим CTLA-4, TGF-β, VEGF, FGL1, LAG3, 4-1BB, OX40, CD27, CD28, CD47, CD155, HGF, CSF1, CXCL1, CXCL2, CXCL3, CXCL5, CXCL6, CXCL7, CXCL8, CXCL9, CXCL10, EGF или ICOSL.
3. Слитый белок по п.2, где первый связывающий домен и рецептор или лиганд выбраны из следующих комбинаций:
(1) первый связывающий домен нацелен и связывается с PD-L1, а рецептор или лиганд выбран из TGF-βRII, VEGFR, LAG-3, SIRPα, TIGIT, C-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
(2) первый связывающий домен нацелен и связывается с PD-1, а рецептор или лиганд выбран из человеческого CD80, CD86, VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
(3) первый связывающий домен нацелен и связывается с CTLA-4, а рецептор или лиганд выбран из человеческого CD80, CD86, VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
(4) первый связывающий домен нацелен на LAG-3 и связывается с ним, и рецептор или лиганд выбран из человеческого VEGFR, c-MET, EGFR, CD80, CD86, CD70, 4-1BBL или OX40L; или
(5) первый связывающий домен нацелен и связывается с FGL1, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, SIRPα, TIGIT, c-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
(6) первый связывающий домен нацелен и связывается с TIM-3, а рецептор или лиганд выбран из человеческого VEGFR, c-MET, EGFR, FGL1, CD70, 4-1BBL или OX40L; или
(7) первый связывающий домен нацелен и связывается с галектином-9, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, SIRPα, TIGIT, c-MET, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS; или
(8) первый связывающий домен нацелен и связывается с TIGIT, а рецептор или лиганд выбран из человеческого TGF-βRII, c-MET, EGFR или VEGFR; или
(9) первый связывающий домен нацелен и связывается с CD155, а рецептор или лиганд выбран из человеческого TGF-βRII, VEGFR, c-MET, EGFR, SIRPα или TIGIT; или
(10) первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2, мезотелином, BCMA, SSTR2, GPRC5D, PSMA, FCRH5, CD33, CD123, CD20, A33, CEA, CD28, DLL3, EGFR, VEGFR, VEGFR2, нектином-4, FGFR, c-MET, RANKL, PDGF, PDGFR, PDGFRα, DLL4, Ang-1 или Ang-2, а рецептор или лиганд выбран из человеческого CTLA-4, CD80, CD86, TGF-βRII, VEGFR, FGL1, LAG3, 4-1BB, OX40, CD27, CD28, CD70, c-MET, SIRPα, TIGIT, CSF1R, CXCR2, CXCR3, CXCR4, EGFR или ICOS.
4. Слитый белок по любому из пп.1-3, где фрагмент рецептора или лиганда включает в себя фрагмент внеклеточной области и фрагмент связывающего домена рецептора или лиганда.
5. Слитый белок по п.3, где первый связывающий домен нацелен и связывается с PD-L1; второй связывающий домен представляет собой фрагмент человеческого TGF-βRII; или первый связывающий домен нацелен и связывается с PD-1, а второй связывающий домен включает в себя человеческий ECD CD80, V-область Ig CD80, человеческий IgVIgC CD80, ECD VEGFR1, ECD VEGFR2 или комбинацию второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2; или первый связывающий домен нацелен и связывается с клаудином 18.2, HER-2 или EGFR, а второй связывающий домен выбран из человеческого ECD CD80, V-области Ig CD80, человеческого IgVIgC CD80, ECD VEGFR1, ECD VEGFR2 или комбинации второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2.
6. Слитый белок по п.5, где второй связывающий домен выбран из:
(1) человеческого TGF-βRII, содержащего последовательность, показанную в SEQ ID NO: 31, 131 или 132, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 31, 131 или 132, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 31, 131 или 132; или
(2) ECD CD80, содержащего последовательность, показанную в SEQ ID NO: 32 или 133, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 32 или 133, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 32 или 133; или
(3) комбинации второй внеклеточной области VEGFR1 и третьей внеклеточной области VEGFR2, содержащей последовательность, показанную в SEQ ID NO: 33, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с последовательностью, показанной в SEQ ID NO: 33, или аминокислотной последовательностью, имеющей 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 аминокислотных замен или делеций в последовательности, показанной в SEQ ID NO: 33.
7. Слитый белок по п.5 или 6, где первый связывающий домен нацелен и связывается с человеческим PD-L1, и CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG имеют ту же последовательность CDR, что и антитело, определяемое следующей последовательностью, или имеющее 1-2 аминокислотные замены в CDR антитела, определяемого следующей последовательностью, где антитело определяется следующим образом:
(1) аминокислотная последовательность вариабельной области тяжелой цепи представлена в SEQ ID NO: 66; и/или
(2) аминокислотная последовательность вариабельной области легкой цепи представлена в SEQ ID NO: 67.
8. Слитый белок по п.7, где CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG являются следующими:
(1) для вариабельной области тяжелой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 1-5 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 1-5; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 6-10 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 6-10; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 11-15 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 11-15; и/или
(2) для вариабельной области легкой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 16-20 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 16-20; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 21-25 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 21-25; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 26-30 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 26-30.
9. Слитый белок по п.8, где:
(1) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 1, 6, 11 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 1, 6, 11; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 16, 21, 26 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 16, 21, 26; или
(2) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 2, 7, 12 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 2, 7, 12; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 17, 22, 27 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 17, 22, 27; или
(3) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 3, 8, 13 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 3, 8, 13; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 18, 23, 28 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 18, 23, 28; или
(4) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 4, 9, 14 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 4, 9, 14; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 19, 24, 29 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 19, 24, 29; или
(5) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 5, 10, 15 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 5, 10, 15; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 20, 25, 30 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 20, 25, 30.
10. Слитый белок по п.9, где аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 3, 8, 13; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 18, 23, 28.
11. Слитый белок по п.10, где:
(1) вариабельная область тяжелой цепи имеет последовательность, показанную в SEQ ID NO: 66, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 66, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 66; и/или
(2) вариабельная область легкой цепи имеет последовательность, показанную в SEQ ID NO: 67, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 67, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 67.
12. Слитый белок по п.11, где:
(1) тяжелая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 72, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%. 97%, 98%, 99% по сравнению с SEQ ID NO: 72;
(2) легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 73, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 73.
13. Слитый белок по п.12, где:
(1) аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 72;
(2) аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 73.
14. Слитый белок по п.5, где первый связывающий домен нацелен и связывается с PD-1, и CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG имеют ту же последовательность CDR, что и антитело, определяемое следующей последовательностью, или имеющее 1-2 аминокислотные замены в CDR антитела, определяемого следующей последовательностью, где антитело определяется следующим образом:
(1) аминокислотная последовательность вариабельной области тяжелой цепи представлена в SEQ ID NO: 64; и/или
(2) аминокислотная последовательность вариабельной области легкой цепи представлена в SEQ ID NO: 65.
15. Слитый белок по п.14, где CDR вариабельной области тяжелой цепи и/или CDR вариабельной области легкой цепи в Fab IgG являются следующими:
(1) для вариабельной области тяжелой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 34-38 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 34-38; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 39-43 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 39-43; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 44-48 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 44-48; и/или
(2) для вариабельной области легкой цепи аминокислотная последовательность CDR1 выбрана из SEQ ID NO: 49-53 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 49-53; аминокислотная последовательность CDR2 выбрана из SEQ ID NO: 54-58 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 54-58; аминокислотная последовательность CDR3 выбрана из SEQ ID NO: 59-63 и аминокислотных последовательностей, содержащих 1 или 2 аминокислотные замены в SEQ ID NO: 59-63.
16. Слитый белок по п.15, где:
(1) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 34, 39, 44 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 34, 39, 44; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 49, 54, 59 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 49, 54, 59; или
(2) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 35, 40, 45 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 35, 40, 45; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 50, 55, 60 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 50, 55, 60; или
(3) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 36, 41, 46 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 36, 41, 46; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 51, 56, 61 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 51, 56, 61; или
(4) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 37, 42, 47 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 37, 42, 47; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 52, 57, 62 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 52, 57, 62; или
(5) аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 38, 43, 48 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 38, 43, 48; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 53, 58, 63 или аминокислотную последовательность, имеющую 1 или 2 аминокислотные замены в SEQ ID NO: 53, 58, 63.
17. Слитый белок по п.16, где аминокислотные последовательности CDR 1-3 вариабельной области тяжелой цепи представляют собой SEQ ID NO: 36, 41, 46; и/или аминокислотная последовательность CDR 1-3 вариабельной области легкой цепи представляет собой SEQ ID NO: 51, 56, 61.
18. Слитый белок по п.17, где:
(1) вариабельная область тяжелой цепи имеет последовательность, показанную в SEQ ID NO: 64, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 64, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 64; и/или
(2) вариабельная область легкой цепи имеет последовательность, показанную в SEQ ID NO: 65, или последовательность, которая имеет те же CDR 1-3, что и SEQ ID NO: 65, и имеет идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 65.
19. Слитый белок по п.18, где:
(1) тяжелая цепь гибридного белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 68, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 68;
(2) легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 69, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 69.
20. Слитый белок по п.19, где:
(1) аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 68;
(2) аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 69.
21. Слитый белок по п.18, где:
(1) тяжелая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 70, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 70;
(2) легкая цепь слитого белка имеет аминокислотную последовательность, показанную в SEQ ID NO: 71, или последовательность, имеющую идентичность более 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99% по сравнению с SEQ ID NO: 71.
22. Слитый белок по п.21, где:
(1) аминокислотная последовательность тяжелой цепи слитого белка имеет 1-15 мутаций аминокислотного сайта или имеет альтернативный пептидный линкер по сравнению с SEQ ID NO: 70;
(2) аминокислотная последовательность легкой цепи слитого белка имеет 1-10 мутаций аминокислотного сайта по сравнению с SEQ ID NO: 71.
23. Слитый белок по любому из пп.1-6, где IgG представляет собой атезолизумаб, авелумаб, дурвалумаб, ниволумаб, пембролизумаб, цемиплимаб или ипилимумаб.
24. Слитый белок по любому из пп.1-23, где С-конец или/и N-конец второго связывающего домена имеет пептидный линкер, и этот пептидный линкер состоит из 2-30 аминокислот.
25. Слитый белок по п.24, где пептидный линкер представляет собой:
(1) (GGGGS)n; или
(2) AKTTPKLEEGEFSEAR (SEQ ID NO: 80); или
(3) AKTTPKLEEGEFSEARV (SEQ ID NO: 81); или
(4) AKTTPKLGG (SEQ ID NO: 82); или
(5) SAKTTPKLGG (SEQ ID NO: 83); или
(6) SAKTTP (SEQ ID NO: 84); или
(7) RADAAP (SEQ ID NO: 85); или
(8) RADAAPTVS (SEQ ID NO: 86); или
(9) RADAAAAGGPGS (SEQ ID NO: 87); или
(10) RADAAAA (SEQ ID NO: 88); или
(11) SAKTTPKLEEGEFSEARV (SEQ ID NO: 89); или
(12) ADAAP (SEQ ID NO: 90); или
(13) DAAPTVSIFPP (SEQ ID NO: 91); или
(14) TVAAP (SEQ ID NO: 92); или
(15) TVAAPSVFIFPP (SEQ ID NO: 93); или
(16) QPKAAP (SEQ ID NO: 94); или
(17) QPKAAPSVTLFPP (SEQ ID NO: 95); или
(18) AKTTPP (SEQ ID NO: 96); или
(19) AKTTPPSVTPLAP (SEQ ID NO: 97); или
(20) AKTTAP (SEQ ID NO: 98); или
(21) AKTTAPSVYPLAP (SEQ ID NO: 99); или
(22) ASTKGP (SEQ ID NO: 100); или
(23) ASTKGPSVFPLAP (SEQ ID NO: 101); или
(24) GENKVEYAPALMALS (SEQ ID NO: 102); или
(25) GPAKELTPLKEAKVS (SEQ ID NO: 103); или
(26) GHEAAAVMQVQYPAS (SEQ ID NO: 104); или
(27) GGGGSGGGGSGGGGSA (SEQ ID NO: 105),
где n равно 1, 2, 3 или 4.
26. Слитый белок по любому из пп. 1-25, где размер встроенного второго связывающего домена не превышает 300 аминокислот.
27. Слитый белок по любому из пп.1-26, где сайт вставки второго связывающего домена расположен в средней передней части шарнирной области, и сайт вставки не влияет на образование дисульфидных связей иммуноглобулина.
28. Слитый белок по п.27, где средняя передняя часть шарнирной области относится к части до 231A.
29. Слитый белок по любому из пп. 1-28, где часть аминокислот в шарнирной области до и после сайта вставки заменена или удалена.
30. Слитый белок по п.29, где шарнирная область содержит мутации D221G и/или C220V.
31. Слитый белок по любому из пп. 1-30, где IgG выбран из IgG млекопитающих, гуманизированного IgG и человеческого IgG, и млекопитающее включает мышь, крысу и кролика; предпочтительно, IgG представляет собой IgG1, IgG2, IgG3 или IgG4.
32. Слитый белок по любому из пп. 1-31, где Fc-область слитого белка агликозилирована или дегликозилирована, или имеет пониженное фукозилирование, или афукозилирована.
33. Выделенный полинуклеотид, кодирующий слитый белок по любому из пп. 1-32.
34. Вектор экспрессии для лечения или предотвращения злокачественной опухоли или рака, содержащий полинуклеотид по п.33.
35. Клетка-хозяин для лечения или предотвращения злокачественной опухоли или рака, содержащая полинуклеотид по п.33 или вектор экспрессии по п.34.
36. Фармацевтическая композиция для лечения или предотвращения злокачественной опухоли или рака, содержащая слитый белок по любому из пп. 1-32 и фармацевтически приемлемый носитель.
37. Способ лечения злокачественной опухоли или рака, включающий в себя этап введения слитого белка по любому из пп. 1-32 или фармацевтической композиции по п.36 субъекту, нуждающемуся в лечении или облегчении.
38. Применение слитого белка по любому из пп. 1-32, полинуклеотида по п.33, вектора экспрессии по п.35 или фармацевтической композиции по п.36 в получении лекарственного средства для лечения или предотвращения злокачественной опухоли или рака.
39. Применение по п.38, где опухоль или рак включает в себя солидную опухоль или несолидную опухоль.
40. Применение слитого белка по любому из пп. 1-32, в диагностике злокачественной опухоли или рака.
41. Способ получения слитого белка по любому из пп. 1-32, включающий культивирование клетки-хозяина по п.35 в условиях, обеспечивающих экспрессию вектора экспрессии по п.34, и выделение полученного слитого белка из культуры.
| RU 2016135062 A3, 12.12.2018 | |||
| CN 109721657 A, 07.05.2019 | |||
| US 2020002439 A1, 02.01.2020 | |||
| BEZABEH B | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
Авторы
Даты
2023-08-10—Публикация
2021-03-19—Подача